用于预测乳腺癌的生物标记物组合、试剂盒及使用方法与流程
本发明涉及生物检测
技术领域:
,尤其涉及用于预测乳腺癌的生物标记物组合、试剂盒及使用方法。
背景技术:
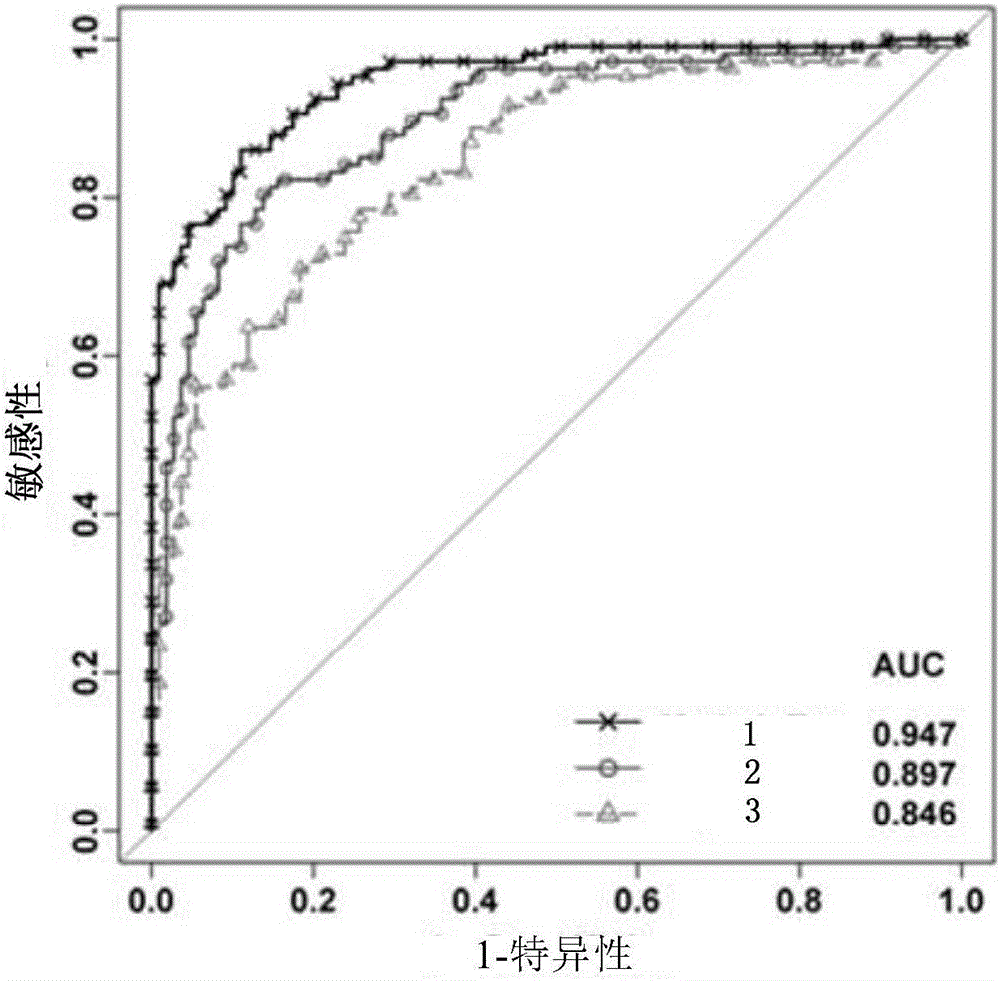
:随着当今社会的发展,恶性肿瘤已成为目前人类的主要杀手之一。而对于女性恶性肿瘤,由于乳腺癌是世界范围内造成妇女发病和死亡的重要原因,因而乳腺癌在女性恶性肿瘤中居于首位。大量研究表明,乳腺癌发病的原因大多是环境因素和遗传因素共同作用的结果,而遗传性乳腺癌在所有发病的乳腺癌中仅占有5-10%,因此,大多数乳腺癌无法通过乳腺癌易感基因的突变检测来确定是否发生乳腺癌病变。最近研究发现,DNA(deoxyribonucleicacid,脱氧核糖核酸)甲基化状态与乳腺癌的发生发展密切相关。DNA甲基化主要出现在DNA链鸟嘌呤前的胞嘧啶(即CpG位点),CpG位点在甲基转移酶的催化作用下添加甲基,CpG位点被甲基化,即DNA甲基化。当DNA甲基化达到一定程度时,常规的右手双螺旋型B-DNA向左手双螺旋型Z-DNA发生过渡。相对于B-DNA,Z-DNA的结构收缩、螺旋加深,这使得许多能够与蛋白质结合的原件缩入螺旋沟内,导致DNA转录起始困难,从而使得基因无法正常表达,即基因失活。人体存在的抑癌基因、致癌基因、DNA修复基因以及细胞周期调节基因等众多基因中都含有CpG位点,当抑癌基因中的CpG位点高度甲基化时,抑癌基因的基因表达被抑制,抑癌基因失活,进而导致抑癌基因丧失正常的抑癌功能;当致癌基因中的CpG位点甲基化比率较低时,致癌基因表达活跃,癌细胞的表达量增加,当癌细胞的表达量增加到一定程度时则发生癌变。同时,人体中还存在与甲基转移酶的作用相反的去甲基酶,通过去甲基酶和甲基转移酶的作用能够使DNA甲基化状态呈现可逆状态,因此,基因中DNA的甲基化水平或甲基化率能够作为衡量癌变的指标。另外,去甲基酶和甲基转移酶的含量会因为生活环境、饮食、药物等方式的不同而不同,因而能够通过改变生活环境、饮食、药物等来避免和减缓乳腺癌的发生发展。生物标记物是一种能够反映细胞生长增殖特性的标志物,能够在生物体受到严重损害之前,从分子、细胞等生物学水平上反应人体的异常化,这种异常化主要表现在生化代谢过程中的变化、异常代谢产物含量以及生理活性物质的异常表现等,因此,通过检测生物标记物能够得知人体当前所处的状态。生物标记物能够标记特定的基因,如与乳腺癌相关的抑癌基因、致癌基因以及DNA修复基因等,以反映出当前机体是否癌变。目前,DNA甲基化与乳腺癌的研究大多集中在单一基因的研究,因而生物标记物也为单一生物标记物。由于乳腺癌肿瘤在分裂生长的过程中,子细胞会出现基因方面的改变,使得乳腺癌肿瘤的生长速度、侵袭能力、治疗以及预后方面产生差异,即乳腺癌肿瘤存在异质性和复杂性,因而基因的突变以及复杂性使得单一生物标记物不能准确指示DNA的甲基化状态和甲基化率,进而单一基因的研究也就不能准确地表明DNA甲基化与乳腺癌的关系。另外,单一生物标记物在应用时存在较为严重的假阳性和假阴性问题,且检测灵敏度低,这使得检测结果的准确度降低。技术实现要素:本发明提供一种用于预测乳腺癌的生物标记物组合、试剂盒及使用方法,以解决现有生物标记物在指示DNA的甲基化状态和甲基化率时灵敏度低、准确度低的问题。第一方面,本发明提供一种用于预测乳腺癌的生物标记物组合,所述组合为甲基化引物组合,所述甲基化引物组合包括:根据端粒基因RAD50设计的序列为TTATATGTTATGTGATTATTGGAAATTAT的正向引物和序列为TTCCCAAAACTTAATCCTAAAACTC的反向引物;根据端粒基因RTEL设计的序列为GGGTTGGGTGTTAGTGAGTGT的正向引物和序列为CAACTCCCATACCCCAAAAA的反向引物;根据端粒基因TERC设计的序列为AAAATTTGTAGAGTAGGAATTAAGTTG的正向引物和序列为CCCCAAACCTAACTAACTAAAC的反向引物;和/或,根据端粒基因TRF1设计的序列为TAGTAGAATAGGAATTTTGGGAGT的正向引物和序列为AATACAACCTTAACTAAAAC的反向引物。第二方面,所述试剂盒包括甲基化引物组合。第三方面,本发明提供一种用于检测DNA样品中DNA片段的目标基因位置的甲基化率的方法,所述方法包括:DNA样本经亚硫酸盐处理后采用甲基化引物组合分别靶向PCR扩增,得到包含测序Tag序列的靶向PCR扩增产物;向所述包含测序Tag序列的靶向PCR扩增产物中插入上游通用测序引物和下游带条形码的测序引物,以区别不同样本的靶向PCR扩增产物;其中,相同样本的所述靶向PCR扩增产物插入上游通用测序引物和条形码相同的所述下游带条形码的测序引物,不同样本的所述靶向PCR扩增产物插入上游通用测序引物和条形码不同的所述带条形码的下游测序引物;所述反应体系分别PCR扩增,得到不同样本的PCR扩增产物;将不同样本的所述PCR扩增产物等比例混合构成扩增子文库;所述扩增子文库经纯化、定量和质控后得到甲基化测序文库;对所述甲基化测序文库双端测序、样本拆分和甲基化信息分析,得到各样本DNA中目的基因各CpG位点的甲基化状态和甲基化率。结合第三方面,所述靶向PCR扩增的扩增条件为:95℃保持10分钟,95℃变性保持15秒,58℃退火保持30秒,72℃延伸保持60秒,共40个循环;所述PCR扩增的扩增条件为:95℃保持10分钟,95℃变性保持15秒,60℃退火保持30秒,72℃延伸保持60秒,共15个循环,循环结束后在72℃下延伸反应3分钟。第四方面,本发明提供一种用于预测乳腺癌的生物标记物组合,所述组合为qPCR引物组合,所述qPCR引物组合包括:根据端粒基因RAD50设计的序列为TGGATATGCGAGGACGATG的正向引物和序列为TGTTGGCTCATCCAAGGCA的反向引物;根据端粒基因RTEL设计的序列为CATCGATGCTGTTGAGCTGC的正向引物和序列为GGATGATCTGGTCCAGCGAG的反向引物;根据端粒基因TERC设计的序列为CATGTGTGAGCCGAGTCCTG的正向引物和序列为GAAGAGGAACGGAGCGAGTC的反向引物;根据端粒基因TRF1设计的序列为GTCTGCGGTAACTGAATCCTCA的正向引物和序列为TTGTTGCTGGGTTCCATGTT的反向引物;和/或,根据参考基因GAPDH设计的序列为CCTCTCCCCAGCCAAAGAAG的正向引物和序列为TGACCCTTTTTGGACTTCAG的反向引物。第五方面,本发明提供一种用于预测乳腺癌的试剂盒,所述试剂盒包括qPCR引物组合。第六方面,本发明提供一种用于预测乳腺癌的生物标记物组合,所述组合包括甲基化引物组合和qPCR引物组合。第七方面,本发明提供一种用于预测乳腺癌的试剂盒,所述试剂盒包括包含有甲基化引物组合和qPCR引物组合的生物标记物组合。第八方面,本发明提供一种用于检测核酸样品中目标基因位置的目标基因甲基化率和目标基因表达量的方法,采用包含有甲基化引物组合和qPCR引物组合的生物标记物组合,其中,甲基化引物组合用于检测目的基因的甲基化率,qPCR引物组合用于检测目的基因的表达量。第九方面,本发明提供的生物标记物组合在制备预测乳腺癌试剂、乳腺癌复发检测试剂或乳腺癌治疗用药评估试剂中的应用。本发明的实施例提供的技术方案可以包括以下有益效果:在细胞分裂过程中,随着DNA分子的不断复制,端粒的长度不断缩小,当端粒缩小至几近没有时,细胞会立即激活凋亡机制,细胞走向凋亡;而仅存在于造血细胞、干细胞、生殖细胞以及癌细胞中的端粒酶能够延长端粒的长度,使得细胞不会因端粒的缩短而凋亡,因此端粒与细胞增殖有关。癌细胞中端粒酶的活性比造血细胞等正常细胞中端粒酶的活性大,因而癌细胞中端粒的延长长度大于造血细胞等正常细胞中端粒的延长长度,继而癌细胞的细胞增殖速度较快。在本发明提供的用于预测乳腺癌的生物标记物组合、试剂盒中,引物设计基因选自与癌细胞增殖相关的端粒基因,尤其选自在乳腺癌组织和正常组织中甲基化率差异最为显著的四种端粒基因RAD50、RTEL、TERC和TRF1。分别根据四种端粒基因的启动子区设计目标基因的甲基化引物,形成甲基化引物组合,用以检测目的基因启动子区的甲基化状态和甲基化率;分别根据四种端粒基因的编码区设计目标基因的qPCR引物,形成qPCR引物组合,检测目的基因的表达量水平。由于本发明提供的生物标记物组合是根据在乳腺癌组织和正常组织中甲基化率差异最为显著的端粒基因而设计,因而在检测样本是否为乳腺癌时,生物标记物组合能够特异性的捕获和扩增检测样本中的特定DNA片段,使得检测灵敏度提高,而且还具有较好的抗噪性、灵活性和准确度,且重复性好,能够应用于乳腺癌的早期预测、治疗用药评估检测和预后复发检测等。本发明提供的用于预测乳腺癌的方法操作快速、方法简便,具有较好的可行性与预测效果。应当理解的是,以上的一般描述和后文的细节描述仅是示例性和解释性的,并不能限制本发明。附图说明此处的附图被并入说明书中并构成本说明书的一部分,示出了符合本发明的实施例,并与说明书一起用于解释本发明的原理。为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,对于本领域普通技术人员而言,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。图1为本发明实施例提供的预测乳腺癌肿瘤组织的ROC曲线(receiveroperatingcharacteristiccurve,受试者工作特征曲线)图。具体实施方式这里将详细地对示例性实施例进行说明,其示例表示在附图中。下面的描述涉及附图时,除非另有表示,不同附图中的相同数字表示相同或相似的要素。以下示例性实施例中所描述的实施方式并不代表与本发明相一致的所有实施方式。相反,它们仅是与如所附权利要求书中所详述的、本发明的一些方面相一致的装置和方法的例子。端粒是存在于真核细胞线状染色体末端具有高度重复的TTAGGG序列的一小段DNA-蛋白质复合体,具有保持染色体完整性和控制细胞分离周期的特性。端粒存在于任何能够提取出的人类DNA样本中,每次细胞分裂时,由于端粒基因在细胞分裂时不能完全复制,因而每条染色体的端粒长度会逐渐变短。当端粒缩小至几近没有时,细胞会立即激活凋亡机制,细胞走向凋亡。而仅存在于造血细胞、干细胞、生殖细胞以及癌细胞中的端粒酶能够延长端粒的长度,使得细胞不会因端粒的缩短而凋亡,因此端粒与细胞增殖有关。研究表明,端粒酶的激活是肿瘤发生的基本步骤,端粒酶等相关蛋白的持续表达能够使得肿瘤细胞获得无限繁殖的能力,且癌细胞中端粒酶的活性比造血细胞等正常细胞中端粒酶的活性大,因而癌细胞中端粒的延长长度大于造血细胞等正常细胞中端粒的延长长度,继而癌细胞的细胞增殖速度较快,因此,端粒在癌症的发生发展中具有重要的作用。DNA甲基化检测技术具有损伤小、敏感度高的特性,且DNA的甲基化状态的改变通常出现在癌变之前,因而可通过基因中DNA的甲基化水平或甲基化率衡量正常细胞是否发生癌变。在本发明实施例提供的生物标记物、试剂盒及甲基化测试方法中所涉及的序列如表1所示。表1:序列在本发明实施例提供的生物标记物中,选取与乳腺癌相关的端粒基因设计引物。在选择端粒基因时,选取在乳腺癌组织和正常组织中甲基化率差异最为显著的四种端粒基因RAD50、RTEL、TERC和TRF1作为引物设计的基础基因。具体地,分别根据端粒基因RAD50、RTEL、TERC和TRF1的启动子区设计甲基化引物,形成甲基化引物组合,用以检测目的基因启动子区的甲基化状态和甲基化率。根据四种端粒基因RAD50、RTEL、TERC和TRF1设计的甲基化引物序列请参考表2。表2:甲基化引物序列编号基因名称正向引物(5'-3')反向引物(5'-3')1RAD50SEQ1SEQ22RTELSEQ3SEQ43TERCSEQ5SEQ64TRF1SEQ7SEQ8本发明实施例提供的四组甲基化引物以及四组甲基化引物组成的甲基化引物组合中的各组特异性扩增引物序列均能够特异性的地捕获待测样本中的特定DNA片段,即目的基因,并通过PCR反应(PolymeraseChainReaction,聚合酶链反应)扩增目的基因,进而得出目的基因的甲基化水平。本发明实施例还提供一种用于预测乳腺癌的试剂盒,该试剂盒包括第一轮PCR扩增试剂和第二轮PCR扩增试剂。第一轮PCR扩增试剂包括缓冲溶液、亚硫酸盐、dNTP(deoxy-ribonucleosidetriphosphate,脱氧核糖核苷三磷酸)、酶混合液和第一轮靶向PCR扩增引物。第二轮PCR扩增试剂包括缓冲溶液、dNTP、酶混合液和第二轮PCR扩增引物。基于同本发明实施例提供的生物标记物组合相同的原因,试剂盒能够检测出目的基因的甲基化水平。进一步,第一轮靶向PCR扩增引物包括如表2所示的甲基化引物或甲基化引物组合。每个正向引物的5'端添加序列SEQ19,每个反向引物的5'端添加序列SEQ20。第二轮PCR扩增引物包括上游通用测序引物序列P1和下游带条形码的测序引物序列Index1-6。具体地,亚硫酸盐用于处理待测样本中提取出的基因组DNA,以使基因组DNA中的胞嘧啶(C)转变为尿嘧啶(U),而已甲基化的胞嘧啶不受影响,进而便于得知基因组DNA中的甲基化状态和甲基化率。在本发明实施例中,缓冲溶液为TE缓冲液用于溶解核酸,能够稳定和储存DNA和RNA。反应预混液为PCR反应体系的基础液,酶混合液包括DNA聚合酶和MgCl2(氯化镁)等试剂。每对甲基化引物中的正向引物和反向引物均包括特异性扩增序列和与特异性扩增序列5'端连接的测序接头序列,而特异性扩增序列如表2所示,测序接头序列则为正向测序Tag序列和反向测序Tag序列,其中,正向测序Tag序列和反向测序Tag序列是测序的必要前置步骤。正向测序Tag序列的具体序列为表1中的SEQ19所示序列,反向测序Tag序列的具体序列为表1中的SEQ20所示序列。上游通用测序引物序列和下游带条形码的测序引物序列用于扩增并标记不同样本中提取的DNA。上游通用测序引物序列如表1中P1所示,下游带条形码的测序引物序列如表1中Index1-6所示。本发明实施例还提供一种用于检测DNA样品中DNA片段的目标基因位置的甲基化率的方法,该方法包括:S101:DNA样本经亚硫酸盐处理后采用甲基化引物组合进行靶向PCR扩增,得到包含测序Tag序列的靶向PCR扩增产物。采用DNA提取试剂从待测人体的乳腺组织样本中提取DNA样本,将DNA样本通过亚硫酸盐处理,以使DNA中的未甲基化的胞嘧啶转变为尿嘧啶。亚硫酸盐处理后的DNA样本使用分光光度计定量,以保证有足够的DNA样本参与到下一步反应中。在本发明实施例中,DNA提取试剂可以为TIANampGenomicDNAKit。定量后的DNA样本在甲基化引物组合的作用下发生靶向PCR扩增,即发生第一轮PCR扩增反应,以通过特异性的甲基化引物捕获DNA样本中的特定DNA片段,即捕获目的基因,完成第一轮PCR扩增。第一轮PCR扩增在捕获的目的基因的同时,扩增产物的两端连接有如表1中SEQ19和SEQ20所示的测序Tag序列。靶向PCR扩增的扩增条件为:95℃保持10分钟,95℃变性保持15秒,58℃退火保持30秒,72℃延伸保持60秒,共40个循环。第一轮PCR扩增后得到的靶向PCR扩增产物采用TE缓冲液稀释100倍,进而便于稳定和储存DNA。S102:向所述包含测序Tag序列的靶向PCR扩增产物中插入上游通用测序引物和下游带条形码的测序引物,以区别不同样本的靶向PCR扩增产物;其中,相同样本的所述靶向PCR扩增产物插入上游通用测序引物和条形码相同的所述下游带条形码的测序引物,不同样本的所述靶向PCR扩增产物插入上游通用测序引物和条形码不同的所述下游带条形码的测序引物。根据不同样本的靶向PCR扩增产物添加上游通用测序引物和条形码不同的下游带条形码的测序引物。由于下游条形码不同的带条形码的测序引物不同,所有的引物形成引物混合物。上游通用测序引物的序列如表1中P1所示,下游带条形码的测序引物的序列如表1中Index1-6所示。在本发明实施例中,上游通用测序引物和下游带条形码的测序引物的浓度均为2μM/L。7μl的预混液、1μl已插入测序Tag序列的靶向PCR扩增产物以及2μl第二轮PCR引物混合液构成PCR扩增的反应体系。S103:所述反应体系分别PCR扩增,得到不同样本的PCR扩增产物。不同样本的反应体系分别PCR扩增,得到不同样本的PCR扩增产物。PCR扩增的扩增条件为:95℃保持10分钟,95℃变性保持15秒,60℃退火保持30秒,72℃延伸保持60秒,共15个循环,循环结束后在72℃下延伸反应3分钟。S104:将不同的所述PCR扩增产物等比例混合构成扩增子文库;将不同样本的PCR扩增产物按照1:1:…:1的等比例混合,以保证各样本的数据是均一的。等比例混合后的PCR扩增产物构成扩增子文库。S105:所述扩增子文库经纯化、定量和质控后得到甲基化测序文库。扩增子文库通过AgencourtAMPureXPsystem纯化处理,以去除多余的引物和非特异性扩增产物。通过芯片生物分析仪检测纯化处理后的扩增子文库中的扩增产物片段的大小,以避免影响最后的测序数据的质量。进一步,扩增子文库通过dsDNAHSAssayKit进行精确定量和质控,形成甲基化测序文库。S106:对所述甲基化测序文库双端测序、样本拆分和甲基化信息分析,得到各样本DNA中目的基因各CpG位点的甲基化状态和甲基化率。甲基化测序文库采用MiSeqReagentKitv2500cycles试剂盒在MiSeq测序仪上完成双端2*250bp的测序,以获得更多的测序数据,减少后续的分析错误,其中,测序数据包括甲基化数据。双端测序结束后,将测序数据按照样本条形码的不同拆分为不同的样本数据,并使用Bismark软件将多个不同样本的测序数据与人类参考基因组序列对比,即可得知各样本每个基因上单个CpG位点的甲基化率。计算每个基因上CpG位点的甲基化率的算术平均值,得到各样本DNA中目的基因的甲基化率。Logistic回归分析法是一种研究二分类观察结果和一些影响因素之间关系的多变量分析方法。根据Logistic回归分析法建立Logistic回归模型,Logistic回归模型的预测值在0-1之间,Logistic回归模型默认的判别值为0.5,即≥0.5或<0.5。在疾病研究过程中,通过Logistic回归分析法能够分析出疾病和各危险因素之间的定量关系。在本发明实施例中,通过Logistic回归分析法建立Logistic回归模型,以从各样本DNA中目的基因的甲基化率分析出样本是否为乳腺癌样本。同样的,在本发明实施例中,预测值的判别为Logistic回归模型默认的判别值。当预测值大于或等于0.5时,样本判定为乳腺癌样本。当预测值小于0.5时,样本判定为正常乳腺组织。本发明实施例还提供了另一组生物标记物组合,该生物标记物组合中的分别根据端粒基因RAD50、RTEL、TERC和/或TRF1的编码区设计为qPCR(QuantitativePolymeraseChainReaction,定量聚合酶链反应)引物,四组qPCR引物单独或组合形成qPCR引物组合。qPCR引物组合以RNA(RibonucleicAcid,核糖核酸)为基础,能够检测出目的基因的表达量水平。GAPDH基因被称为管家基因,几乎在所有组织中都高水平表达,且在同种细胞或者组织中的蛋白质表达量一般是恒定的,因而被广泛用作基因表达量试验的标准化内参。在本发明实施例中,同样采用GAPDH基因作为标准化内参。根据端粒基因RAD50、RTEL、TERC和TRF1设计的qPCR引物的序列以及GAPDH基因的序列请参考表3。表3:qPCR引物序列及GAPDH基因序列编号基因名称正向引物(5'-3')反向引物(5'-3')1RAD50SEQ9SEQ102RTELSEQ11SEQ123TERCSEQ13SEQ144TRF1SEQ15SEQ165GAPDHSEQ17SEQ18本发明实施例还提供一种用于预测乳腺癌的试剂盒,该试剂盒包括如表3所示的qPCR引物组合。基于同本发明实施例提供的生物标记物组合相同的原因,该试剂盒能够检测出目的基因的表达量水平。进一步,试剂盒还包括逆转录酶、去核酸酶水和PCR预混液。其中,逆转录酶用于将所有的RNA逆转录为cDNA(complementarydeoxyribonucleicacid,互补脱氧核糖核酸),以便于qPCR扩增反应。去核酸酶水(Nuclease-freeWater)能够避免其他核酸污染本发明实施例中cDNA,影响测试结果。PCR预混液为PCR反应体系的基础液,在本发明实施例中,PCR预混液选用5μl2xSYBRGreenPCRmastermix。本发明实施例还提供一种用于检测RNA样品中RNA片段的目标基因位置的表达量水平的方法,该方法包括:S201:样本的总RNA逆转录为cDNA。提取组织样本中的总RNA,将总RNA使用逆转录酶逆转录为cDNA。S202:所述cDNA、参考基因、qPCR引物组合、去核酸酶水和PCR预混液构成反应体系。2μl的cDNA、1μl参考基因、qPCR引物组合、2μl去核酸酶水和5μlPCR预混液构成反应体系,其中,qPCR引物组合中每组qPCR引物的用量为1μl,浓度为2μΜ/L。S203:所述反应体系qPCR扩增,得到qPCR扩增产物,所述qPCR扩增产物通过测序系统检测得到所述参考基因的循环阈值及所述cDNA中目的基因的循环阈值;反应体系进行qPCR扩增,得到qPCR扩增产物。其中,qPCR扩增的扩增条件为:95℃保持10分钟,95℃变性保持15秒,60℃退火保持30秒,72℃延伸保持60秒,共40个循环。反应体系在qPCR扩增反应的过程中能够得出熔解曲线(MeltingCurveAnalysis),通过熔解曲线分析qPCR扩增产物的纯度。另外,通过测序系统检测能够得知参考基因的循环阈值(Ct,Thresholdcycle)和cDNA中目的基因的循环阈值。S204:根据所述参考基因的循环阈值及所述cDNA中目的基因的循环阈值确定所述目的基因表达量的平均循环阈值。同一组织样本平行测试三次,计算目的基因三次Ct值的算术平均值以及参考基因三次Ct值的算术平均值。将目的基因的Ct值算术平均值减去参考基因的Ct值算术平均值能够确定目的基因表达量的平均循环阈值(dCt,deltavalueofCyclethreshold)。在本发明实施例中,确定目的基因表达量后,同样通过Logistic回归分析法判定样本是否为乳腺癌样本。预测值的判别为Logistic回归模型默认的判别值。本发明实施例还提供一组生物标记物组合,该生物标记物包括甲基化引物组合和qPCR引物组合,甲基化引物组合和qPCR引物组合的具体引物序列,此处不再赘述。由于本发明实施例提供的生物标记物组合是根据在乳腺癌组织和正常组织中甲基化率差异最为显著的端粒基因而设计,因而在检测样本是否为乳腺癌时,生物标记物组合能够特异性的捕获和扩增检测样本中的特定DNA片段,使得检测灵敏度提高,而且还具有较好的抗噪性、灵活性和准确度,且重复性好,能够应用于乳腺癌的早期预测、治疗用药评估检测和预后复发检测等。本发明实施例还提供一种用于预测乳腺癌的试剂盒,该试剂盒包括甲基化引物组合和qPCR引物组合。基于同本发明实施例提供的生物标记物组合相同的原因,试剂盒能够同时检测出目的基因的甲基化水平和目的基因表达量水平。进一步,根据本发明实施例提供的包含甲基化引物组合的用于预测乳腺癌的试剂盒和包含qPCR引物组合的用于预测乳腺癌的试剂盒,本发明实施例提供的试剂盒还包括亚硫酸盐、TE缓冲液、反应预混液、第一轮PCR引物、第二轮PCR引物、逆转录酶、去核酸酶水和PCR预混液。基于本发明实施例提供的用于检测DNA样品中DNA片段的目标基因位置的甲基化率的方法以及用于检测RNA样品中RNA片段的目标基因位置的表达量水平的方法,本发明实施例提供一种用于检测核酸样品中目标基因位置的目标基因甲基化率和目标基因表达量的方法。在本发明实施例提供的方法中,包括用于检测DNA样品中DNA片段的目标基因位置的甲基化率的方法以及用于检测RNA样品中RNA片段的目标基因位置的表达量水平的方法,以能够同时检测目标基因的甲基化率以及目标基因的表达量水平,进而便于分析样本是否为乳腺癌样本。本发明实施例提供的包括甲基化引物组合的生物标记物组合、包括qPCR引物组合的生物标记物组合以及同时包括甲基化引物组合和qPCR引物组合的生物标记物组合均能够用于制备预测乳腺癌的试剂,以便于预测是组织样本否为乳腺癌样本。进一步,上述三种生物标记物组合还能够用于制备乳腺癌治疗试剂,以便于在乳腺癌治疗的过程中检测分析乳腺癌的治疗疗效以及治疗进程。更进一步,上述三种生物标记物组合还能够用于制备乳腺癌复发检测试剂,以便于在乳腺癌治疗后期监测乳腺癌是否复发。本发明实施例提供的包括甲基化引物组合的试剂盒、包括qPCR引物组合的试剂盒以及同时包括甲基化引物组合和qPCR引物组合的试剂盒均能够用于制备预测乳腺癌的试剂,以便于预测是组织样本否为乳腺癌样本。进一步,上述三种试剂盒还能够用于制备乳腺癌治疗试剂,以便于在乳腺癌治疗的过程中检测分析乳腺癌的治疗疗效以及治疗进程。更进一步,上述三种试剂盒还能够用于制备乳腺癌复发检测试剂,以便于在乳腺癌治疗后期监测乳腺癌是否复发。为评估本发明实施例提供的三种生物标记物组合在制备预测乳腺癌的试剂中所起的预测效果,结合Logistic回归分析法确定的回归模型,通过敏感性(Sensitivity)、特异性(Specificity)、准确性(Accuracy)和ROC(ReceiverOperatingCharacteristicCurve,受试者工作特征曲线)曲线的AUC(AreaUnderCurve)值四种评估指标评定三种生物标记物组合的预测效果。敏感性的计算公式为Sensitivity=TP/(TP+FN),特异性的计算公式为Specificity=TN/(TN+FP),准确性的计算公式为Accuracy=(TP+TN)/(TP+FP+TN+FN),其中,TP-truepositive;FP-falsepositive;TN-truenegative;FN-falsenegative。ROC曲线由横轴的1-特异性和纵轴的敏感性组成,能够反映两个模型数值在取不同的分类阈值时的相对关系。ROC曲线能够用于评估不同生物标记物组的预测性能,用ROC曲线下的面积AUC作为测量值来评估基因集合预测性能的优劣。在评定三种生物标记物组合的预测效果时,四种评估指标的指标值均在[0,1]之间,指标值越高时,说明根据生物标记物组合确定的回归模型的模型预测效果越好。下面结合具体实施例说明本发明实施例提供的三种生物标记物组合的性能评估以及预测乳腺癌的预测效果。选取114例未接受化疗或放疗的乳腺癌病人的肿瘤组织以及乳腺正常组织,其中,肿瘤组织和正常组织各占一半。对114例组织样本同时进行目的基因的甲基化检测和表达量检测。同时包括甲基化引物组合和qPCR引物组合的生物标记物组合所建立的模型标记为1,包含甲基化引物组合的生物标记物组合所建立的模型标记为2,包括qPCR引物组合的生物标记物组合所建立的模型标记为3。在甲基化检测和表达量检测过程中,通过留一交叉验证法(Leaveoneoutcrossvalidation,LOOCV)计算出回归模型的CVerror(CrossValidationPredictionError,交叉验证预测误差)。CVerror值越低,则表示回归模型的模拟越精确,且能够排除过拟合现象。目的基因Logistic回归模型的性能评估的结果值请参考表4,性能评估结果请参考附图1。在甲基化检测阶段,各样本之间具有较好的均一性,且目的基因的测序深度满足分析所需要的319X的平均测序深度。经过甲基化测序,87%的测序数据对比到PCR扩增产物的序列位置。大于97%的样本测序数据落在平均测序读数的两倍范围以内。根据4组甲基化引物得出的4个目的基因在肿瘤组织中的平均甲基化率显著高于正常组织,P值为3.54E-35。在表达量检测阶段,目的基因在肿瘤组织中的dCt值显著高于正常组织,即肿瘤组织中的目的基因表达低于正常组织中的目的基因表达。相对于端粒基因RAD50、RTEL、TERC和TRF1设计的甲基化引物,P值经Holm法矫正后的数值分别为1.07E-13,1.03E-5,2.38E-14,3.14E-13。上述结果表明,根据端粒基因RAD50、RTEL、TERC和TRF1设计的甲基化引物具有很好的肿瘤特异性。表4:目的基因Logistic回归模型的性能评估的结果值模型CVerror敏感性特异性准确性AUC10.1100.8320.890.8610.94720.1370.7940.8620.8290.89730.1700.7380.7610.7500.846由表4可知,回归模型1-3的敏感性、特异性、准确性和AUC四个指标的数值均高于0.7,而特异性越高假阳性越低,敏感性越高假阴性越低,由此说明,单独采用甲基化引物组合、qPCR引物组合以及组合使用甲基化引物组合和qPCR引物组合均能够很好的鉴别出乳腺癌样本。由附图1可知,曲线的随机波动程度较低,从而体现出重复性和抗噪性较好。在回归模型1-3中,回归模型1在敏感性、特异性、准确性和AUC四个指标的数值均最高,而CVerror值最低,仅为0.110。由此说明同时包括甲基化引物组合和qPCR引物组合的生物标记物组合更加能够准确的预测样本是否为乳腺癌样本。本发明实施例提供的生物标记组合由于根据在乳腺癌组织和正常组织中甲基化率差异最为显著的端粒基因而设计,因而在检测样本是否为乳腺癌时,生物标记物组合能够特异性的捕获和扩增检测样本中的特定DNA片段,使得检测灵敏度提高,而且还具有较好的抗噪性、灵活性和准确度,且重复性好,能够应用于乳腺癌的早期预测、治疗用药评估检测和预后复发检测等。本发明提供的用于预测乳腺癌的方法操作快速、方法简便,具有较好的可行性与预测效果,有潜力运用于乳腺癌风险评估等领域,具有较大的临床价值和潜在意义。本领域技术人员在考虑说明书及实践这里发明的公开后,将容易想到本发明的其它实施方案。本申请旨在涵盖本发明的任何变型、用途或者适应性变化,这些变型、用途或者适应性变化遵循本发明的一般性原理并包括本发明未公开的本
技术领域:
中的公知常识或惯用技术手段。说明书和实施例仅被视为示例性的,本发明的真正范围和精神由下面的权利要求指出。应当理解的是,诸如“第一”和“第二”等之类的关系术语仅仅用来将一个实体或者操作与另一个实体或操作区分开来,而不一定要求或者暗示这些实体或操作之间存在任何这种实际的关系或者顺序。本发明并不局限于上面已经描述并在附图中示出的精确结构,并且可以在不脱离其范围进行各种修改和改变。本发明的范围仅由所附的权利要求来限制。SEQUENCELISTING<110>湖南圣维基因科技有限公司<120>用于预测乳腺癌的生物标记物组合、试剂盒及使用方法<130>2016<160>18<170>PatentInversion3.3<210>1<211>29<212>DNA<213>人工序列<400>1ttatatgttatgtgattattggaaattat29<210>2<211>25<212>DNA<213>人工序列<400>2ttcccaaaacttaatcctaaaactc25<210>3<211>21<212>DNA<213>人工序列<400>3gggttgggtgttagtgagtgt21<210>4<211>20<212>DNA<213>人工序列<400>4caactcccataccccaaaaa20<210>5<211>27<212>DNA<213>人工序列<400>5aaaatttgtagagtaggaattaagttg27<210>6<211>22<212>DNA<213>人工序列<400>6ccccaaacctaactaactaaac22<210>7<211>24<212>DNA<213>人工序列<400>7tagtagaataggaattttgggagt24<210>8<211>20<212>DNA<213>人工序列<400>8aatacaaccttaactaaaac20<210>9<211>19<212>DNA<213>人工序列<400>9tggatatgcgaggacgatg19<210>10<211>19<212>DNA<213>人工序列<400>10tgttggctcatccaaggca19<210>11<211>20<212>DNA<213>人工序列<400>11catcgatgctgttgagctgc20<210>12<211>20<212>DNA<213>人工序列<400>12ggatgatctggtccagcgag20<210>13<211>20<212>DNA<213>人工序列<400>13catgtgtgagccgagtcctg20<210>14<211>20<212>DNA<213>人工序列<400>14gaagaggaacggagcgagtc20<210>15<211>22<212>DNA<213>人工序列<400>15gtctgcggtaactgaatcctca22<210>16<211>20<212>DNA<213>人工序列<400>16ttgttgctgggttccatgtt20<210>17<211>20<212>DNA<213>人工序列<400>17cctctccccagccaaagaag20<210>18<211>20<212>DNA<213>人工序列<400>18tgaccctttttggacttcag20<210>19<211>33<212>DNA<213>人工序列<400>19tcgtcggcagcgtcagatgtgtataagagacag33<210>20<211>34<212>DNA<213>人工序列<400>20gtctcgtgggctcggagatgtgtataagagacag34<210>21<211>62<212>DNA<213>人工序列<400>21aatgatacggcgaccaccgagatctacactcgtcggcagcgtcagatgtgtataagagac60ag62<210>22<211>68<212>DNA<213>人工序列<400>22caagcagaagacggcatacgagatgtatcgtcgtgtctcgtgggctcggagatgtgtata60agagacag68<210>23<211>68<212>DNA<213>人工序列<400>23caagcagaagacggcatacgagatgtgtatgcgtgtctcgtgggctcggagatgtgtata60agagacag68<210>24<211>68<212>DNA<213>人工序列<400>24caagcagaagacggcatacgagattgctcgtagtgtctcgtgggctcggagatgtgtata60agagacag68<210>25<211>68<212>DNA<213>人工序列<400>25caagcagaagacggcatacgagatgtcgtcgtctgtctcgtgggctcggagatgtgtata60agagacag68<210>26<211>68<212>DNA<213>人工序列<400>26caagcagaagacggcatacgagatgtgcgtgtgtgtctcgtgggctcggagatgtgtata60agagacag68<210>27<211>68<212>DNA<213>人工序列<400>27caagcagaagacggcatacgagatgcgtcgtgtagtctcgtgggctcggagatgtgtata60agagacag68当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1