一种用于流感病毒的有限复制型黏膜免疫疫苗的制作方法
本发明涉及一种用于流感病毒的有限复制型黏膜免疫疫苗。
背景技术:
流行性感冒病毒,是正粘病毒科(orthomyxoviridae)流行性感冒病毒(influenzavirus)属的代表种,简称流感病毒,包括人流感病毒和动物流感病毒。人流感病毒分为甲(a)、乙(b)、丙(c)三型,是流行性感冒(流感)的病原体。甲型流感病毒抗原性易发生变异,多次引起世界性大流行,例如1918~1919年的大流行中,全世界至少有2000万~4000万人死于流感。乙型流感病毒对人类致病性较低。丙型流感病毒只引起人类不明显的或轻微的上呼吸道感染,很少造成流行。甲型流感病毒于1933年分离成功,乙型流感病毒于1940年获得,丙型流感病毒直到1949年才成功分离。
甲型流感病毒的rna由8个节段组成,第1、2、3个节段编码的是rna多聚集酶,第4个节段负责编码血凝素;第5个节段负责编码核蛋白,第6个节段编码的是神经氨酸酶;第7个节段编码基质蛋白,第8个节段编码的是一种未知功能的能起到拼接rna功能的非结构蛋白。
技术实现要素:
本发明的目的是提供一种用于流感病毒的有限复制型黏膜免疫疫苗。
本发明首先保护一种突变蛋白,命名为ns1r38a/k41a蛋白,是将流感病毒的ns1蛋白进行如下两个氨基酸残基的突变得到的:第38位氨基酸残基由精氨酸突变为丙氨酸,第41位氨基酸残基由赖氨酸突变为丙氨酸。
编码所述突变蛋白的基因(命名为ns1r38a/k41a基因)也属于本发明的保护范围。
ns1r38a/k41a基因具体可为将编码ns1蛋白的基因进行如下两组突变得到的:第112-114位核苷酸由“cga”突变为“gca”,第121-123位核苷酸由“aag”突变为“gca”。
编码ns1蛋白的基因具体如序列表的序列1所示。
含有ns1r38a/k41a基因的质粒也属于本发明的保护范围。所述质粒具体可为质粒phh21-ns1r38a/k41a。质粒phh21-ns1r38a/k41a为将ns1r38a/k41a基因插入载体phh21的多克隆位点(例如bsmbi酶切位点)得到的重组质粒。
本发明还保护一种重组病毒,是将流感病毒基因组中编码ns1蛋白的两个氨基酸残基的密码子进行如下突变得到的重组病毒:自n末端第38位氨基酸残基的密码子由精氨酸密码子突变为丙氨酸密码子,自n末端第41位氨基酸残基的密码子由赖氨酸密码子突变为丙氨酸密码子。
所述重组病毒具体可为:是将流感病毒基因组中编码ns1蛋白的两个氨基酸残基的密码子进行如下突变得到的重组病毒:自n末端第38位氨基酸残基的密码子由精氨酸密码子“cga”突变为丙氨酸密码子“gca”,自n末端第41位氨基酸残基的密码子由赖氨酸密码子“aag”突变为丙氨酸密码子“gca”。
所述重组病毒具体可为:是将流感病毒基因组中序列表的序列1所示的编码ns1蛋白的基因进行如下两组突变得到的:第112-114位核苷酸由“cga”突变为“gca”,第121-123位核苷酸由“aag”突变为“gca”。
以上任一所述ns1蛋白为如下(a)或(b):
(a)由序列表中序列2所示的氨基酸序列组成的蛋白质;
(b)将(a1)经过一个或几个氨基酸残基的取代和/或缺失和/或添加且与具有相同功能的由其衍生的蛋白质。
本发明还保护一种重组病毒,其制备方法包括如下步骤:
将质粒phh21-pa、质粒phh21-pb1、质粒phh21-pb2、质粒phh21-ha、质粒phh21-np、质粒phh21-na、质粒phh21-m、质粒phh21-ns1r38a/k41a、质粒pcdna3.0-pa、质粒pcdna3.0-pb1、质粒pcdna3.0-pb2和质粒pcdna3.0-np共转染离体哺乳动物细胞后进行培养得到的重组病毒。
phh21-pa为在载体phh21的多克隆位点(例如bsmbi酶切位点)插入序列表的序列3所示的双链dna分子得到的重组质粒;质粒phh21-pb1为在载体phh21的多克隆位点(例如bsmbi酶切位点)插入序列表的序列4所示的双链dna分子得到的重组质粒;质粒phh21-pb2为在载体phh21的多克隆位点(例如bsmbi酶切位点)插入序列表的序列5所示的双链dna分子得到的重组质粒;质粒phh21-ha为在载体phh21的多克隆位点(例如bsmbi酶切位点)插入序列表的序列6所示的双链dna分子得到的重组质粒;质粒phh21-np为在载体phh21的多克隆位点(例如bsmbi酶切位点)插入序列表的序列7所示的双链dna分子得到的重组质粒;质粒phh21-na为在载体phh21的多克隆位点(例如bsmbi酶切位点)插入序列表的序列8所示的双链dna分子得到的重组质粒;质粒phh21-m为在载体phh21的多克隆位点(例如bsmbi酶切位点)插入序列表的序列9所示的双链dna分子得到的重组质粒;质粒phh21-ns1r38a/k41a为将ns1r38a/k41a基因插入载体phh21的多克隆位点(例如bsmbi酶切位点)得到的重组质粒;质粒pcdna3.0-pa为在载体pcdna3.0的多克隆位点(例如kpni和xhoi酶切位点之间)插入序列表的序列3所示的双链dna分子得到的重组质粒;质粒pcdna3.0-pb1为在载体pcdna3.0的多克隆位点(例如kpni和xhoi酶切位点之间)插入序列表的序列4所示的双链dna分子得到的重组质粒;质粒pcdna3.0-pb2为在载体pcdna3.0的多克隆位点(例如kpni和xhoi酶切位点之间)插入序列表的序列5所示的双链dna分子得到的重组质粒;质粒pcdna3.0-np为在载体pcdna3.0的多克隆位点(例如kpni和xhoi酶切位点之间)插入序列表的序列7所示的双链dna分子得到的重组质粒。
所述哺乳动物细胞具体可为293trig-iko细胞。
所述培养条件具体可为37℃培养6-72小时。
本发明还保护一种制备流感病毒疫苗的方法,包括如下步骤:
第1次传代的方法如下:将所述重组病毒感染哺乳动物细胞,培养后收集上清,即为含有f1代病毒的病毒液;
第2次传代的方法如下:采用上一次传代得到的病毒液感染哺乳动物细胞,培养后收集的上清为含有f2代病毒的病毒液;
重复按照第2次传代的方法进行连续传代;
第n-1次传代中,收集的上清具有病毒复制能力;
第n次传代中,收集的上清不具有病毒复制能力;
n为3以上的自然数;
将第n-1次传代收集的上清作为流感病毒疫苗的活性成分。
本发明还保护一种制备流感病毒疫苗的方法(方法甲),包括如下步骤:
(1)将所述重组病毒感染mdck细胞,培养后收集上清,即f1代病毒液;
(2)f1代病毒液感染mdck细胞,培养后收集上清,即f2代病毒液;
(3)f2代病毒液感染mdck细胞,培养后收集上清,即f3代病毒液;
(4)将f3代病毒液作为流感病毒疫苗的活性成分;
本发明还保护一种制备流感病毒疫苗的方法(方法乙),包括如下步骤:
(1)将所述重组病毒感染mdck细胞,培养后收集上清,即f1代病毒液;
(2)f1代病毒液感染a549细胞,培养后收集上清,即f2代病毒液;
(3)将f2代病毒液作为流感病毒疫苗的活性成分。
以上任一所述方法制备得到的流感病毒疫苗也属于本发明的保护范围。
本发明还保护一种用于制备流感病毒疫苗的试剂盒,包括以上任一所述重组病毒。所述试剂盒还包括293trig-iko细胞和/或mdck细胞和/或a549细胞和/或vero细胞。
本发明还保护以上任一所述重组病毒在制备流感病毒疫苗中的应用。
以上任一所述流感病毒具体可为a型流感病毒。
以上任一所述流感病毒具体可为动物感染的流感病毒。
以上任一所述流感病毒具体可为家禽感染的流感病毒或家畜感染的流感病毒。
所述a型流感病毒具体可为wsn病毒a/wsn/1933(h1n1)毒株。
本发明提供的流感病毒,采用vero细胞传代可以多代保持复制能力,采用mdck细胞传至4代失去复制能力,采用a549细胞传至3代失去复制能力。因此,可以采用vero细胞传代,采用mdck细胞或a549细胞制备疫苗(将失去复制能力的前一代病毒作为疫苗),并且可以采用黏膜免疫。本发明对于流感病毒的防治具有重大应用价值。
附图说明
图1为实施例1中westernblot结果。
图2为实施例2中f2细胞westernblot结果。
图3为实施例2中f3细胞westernblot结果。
图4为实施例2中f4细胞westernblot结果。
图5为实施例3中f2细胞westernblot结果。
图6为实施例3中f3细胞westernblot结果。
图7为实施例3中f4细胞westernblot结果。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。实施例中所用的pbs缓冲液,如无特殊说明,均为ph7.2-7.4、0.01m的pbs缓冲液。
载体phh21:neumann,g.etal.,generationofinfluenzaavirusesentirelyfromclonedcdnas.pnatlacadsciusa96(16),9345(1999)。
载体pcdna3.0:上海希匹吉生物技术有限公司,产品目录号cpc030。脂质体2000(lipofectamine2000):invitrogen。293trig-iko细胞:武汉普诺赛生命科技有限公司,货号cl-0001。mdck细胞:atcc,ccl-34。a549细胞:atcc,ccl-185。vero细胞:北京中科质检生物技术有限公司,货号v0180000。
病毒感染液:含2μg/mltpck处理的胰酶(胰酶是以胰酶母液的方式加入的,胰酶母液为用pbs缓冲液配制的胰酶浓度为0.25g/100ml的溶液)、100u/ml青霉素和100u/ml链霉素的无血清dmem培养基。
噬斑鉴定的方法:(1)将mdck细胞接种于12孔板中,每孔约1×105细胞,置于37℃、5%co2培养箱中培养12小时,吸弃上清并用pbs缓冲液清洗细胞;(2)将待测上清用病毒感染液梯度稀释后分别加入完成步骤(1)的各孔中,每个稀释度设置三个重复孔,37℃孵育1小时,吸弃上清并用pbs缓冲液清洗细胞;(3)完成步骤(2)后,每孔加入1毫升混合溶液(混合溶液的制备方法:将1体积份融化后降温至37℃左右的3g/100ml低熔点琼脂糖水溶液与1体积份预热到37℃的无酚红dmem培养基等体积混合,且混合物中加入tpck处理的胰酶、青霉素和链霉素,使得胰酶浓度为2μg/ml,青霉素和链霉素的浓度均为100u/ml);(4)完成步骤(3)后,将12孔板4℃放置15分钟以上,待琼脂凝固后,将孔板翻转过来倒置在37℃培养箱中培养,在显微镜下观察细胞病变情况,培养3天后,将12孔板从培养箱中取出,计数空斑数。
病毒-血清中和抗体效价的检测方法:(1)将mdck细胞接种于96孔板中,每孔约4×104细胞,培养12小时,吸弃上清并用pbs缓冲液清洗细胞;(2)1体积份血清与4体积份rde酶(受体破坏酶)混合,37℃孵育16小时,然后置于56℃水浴锅中灭活30min,然后加入1体积份20%鸡红细胞并混匀,4℃孵育12小时,然后1000g离心并收集上清,即为预处理后的血清;(3)用dmem培养液梯度稀释步骤(2)得到的预处理后的血清,得到各个抗体稀释液;(4)用dmem培养液稀释实施例1制备的wt-f1代上清,得到病毒稀释液;(5)将抗体稀释液与病毒稀释液(病毒含量为100tcid50)等体积混匀,37℃孵育1h,然后加入完成步骤(1)的96孔板中,100μl/孔,静置孵育1h;(6)完成步骤(5)后,吸弃上清,用pbs缓冲液清洗细胞,加入含2μg/μltpck-胰酶的dmem培养液,静置培养72h;(7)观察阳性感染细胞孔的数量,采用reed-muench法计算血清的中和效价。
ns1-r38a-f:5’-gacttctgatctgcgcgaagccgat-3’;
ns1-r38a-r:5’-attccttgatcggcttcgcgcagat-3’。
ns1-k41a-f:5’-tcctcttagggatgcctgatctcgg-3’;
ns1-k41a-r:5’-ttcgccgagatcaggcatccctaag-3’。
ns1-r38a/k41a-f:5’-ctcttagggatgcctgatctgcgcg-3’;
ns1-r38a/k41a-r:5’-ttcgcgcagatcaggcatccctaag-3’。
实施例1、突变体病毒的制备
一、构建重组质粒
ns1蛋白如序列表的序列2所示。编码ns1蛋白的基因命名为ns1基因。ns1基因cdna中的开放阅读框如序列表的序列1所示。在载体phh21的bsmbi酶切位点插入序列表的序列1所示的双链dna分子,得到质粒phh21-ns1。
以质粒phh21-ns1为模板,采用ns1-r38a-f和ns1-r38a-r组成的引物对引入单点突变,得到质粒phh21-ns1r38a。经测序,与质粒phh21-ns1相比,质粒phh21-ns1r38a的差异仅在于:将编码ns1蛋白自n末端第38位氨基酸残基的密码子由精氨酸密码子“cga”突变为了丙氨酸密码子“gca”。
以质粒phh21-ns1为模板,采用ns1-k41a-f和ns1-k41a-r组成的引物对引入单点突变,得到质粒phh21-ns1k41a。经测序,与质粒phh21-ns1相比,质粒phh21-ns1k41a的差异仅在于:将编码ns1蛋白自n末端第41位氨基酸残基的密码子由赖氨酸密码子“aag”突变为了丙氨酸密码子“gca”。
以质粒phh21-ns1为模板,采用ns1-r38a/k41a-f和ns1-r38a/k41a-r引入双点突变,得到质粒phh21-ns1r38a/k41a。经测序,与质粒phh21-ns1相比,质粒phh21-ns1r38a/k41a的差异仅在于:将编码ns1蛋白的两个氨基酸残基的密码子进行了突变,分别为:自n末端第38位氨基酸残基的密码子由精氨酸密码子“cga”突变为了丙氨酸密码子“gca”,自n末端第41位氨基酸残基的密码子由赖氨酸密码子“aag”突变为了丙氨酸密码子“gca”。
在载体phh21的bsmbi酶切位点插入序列表的序列3所示的双链dna分子,得到质粒phh21-pa。
在载体phh21的bsmbi酶切位点插入序列表的序列4所示的双链dna分子,得到质粒phh21-pb1。
在载体phh21的bsmbi酶切位点插入序列表的序列5所示的双链dna分子,得到质粒phh21-pb2。
在载体phh21的bsmbi酶切位点插入序列表的序列6所示的双链dna分子,得到质粒phh21-ha。
在载体phh21的bsmbi酶切位点插入序列表的序列7所示的双链dna分子,得到质粒phh21-np。
在载体phh21的bsmbi酶切位点插入序列表的序列8所示的双链dna分子,得到质粒phh21-na。
在载体phh21的bsmbi酶切位点插入序列表的序列9所示的双链dna分子,得到质粒phh21-m。
在载体pcdna3.0的kpni和xhoi酶切位点之间插入序列表的序列3所示的双链dna分子,得到质粒pcdna3.0-pa。
在载体pcdna3.0的kpni和xhoi酶切位点之间插入序列表的序列4所示的双链dna分子,得到质粒pcdna3.0-pb1。
在载体pcdna3.0的kpni和xhoi酶切位点之间插入序列表的序列5所示的双链dna分子,得到质粒pcdna3.0-pb2。
在载体pcdna3.0的kpni和xhoi酶切位点之间插入序列表的序列7所示的双链dna分子,得到质粒pcdna3.0-np。
二、病毒拯救
1、将293trig-iko细胞接种于60mm平皿,每皿1×106个细胞,培养12小时。
2、分组处理如下:
第一组:将质粒phh21-pa、质粒phh21-pb1、质粒phh21-pb2、质粒phh21-ha、质粒phh21-np、质粒phh21-na、质粒phh21-m、质粒phh21-ns1r38a、质粒pcdna3.0-pa、质粒pcdna3.0-pb1、质粒pcdna3.0-pb2和质粒pcdna3.0-np(每种质粒1μg)混合后通过脂质体2000共转染完成步骤1的细胞,37℃培养6小时,然后更换培养体系为病毒感染液,然后37℃培养72小时,分别收获上清和细胞。
第二组:与第一组的差异仅在于用质粒phh21-ns1k41a代替质粒phh21-ns1r38a。
第三组:与第一组的差异仅在于用质粒phh21-ns1r38a/k41a代替质粒phh21-ns1r38a。
第四组:与第一组的差异仅在于用质粒phh21-ns1代替质粒phh21-ns1r38a。
第五组:与第一组的差异仅在于不加入质粒phh21-ns1r38a。
将质粒phh21-pa、质粒phh21-pb1、质粒phh21-pb2、质粒phh21-ha、质粒phh21-np、质粒phh21-na、质粒phh21-m、质粒phh21-ns1、质粒pcdna3.0-pa、质粒pcdna3.0-pb1、质粒pcdna3.0-pb2和质粒pcdna3.0-np共转染293trig-iko细胞(即上述第四组),培养后得到wsn病毒a/wsn/1933(h1n1)毒株。wsn病毒a/wsn/1933(h1n1)毒株又称wsn病毒野生型。相应的,将质粒phh21-ns1r38a代替质粒phh21-ns1后,得到的病毒为r38a单突变病毒。相应的,将质粒phh21-ns1k41a代替质粒phh21-ns1后,得到的病毒为k41a单突变病毒。相应的,将质粒phh21-ns1r38a/k41a代替质粒phh21-ns1后,得到的病毒为r38a/k41a双突变病毒。
第一组得到的上清命名为r38a-f0代上清。
第二组得到的上清命名为k41a-f0代上清。
第三组得到的上清命名为r38a/k41a-f0代上清。
第四组得到的上清命名为wt-f0代上清。
第五组得到的上清命名为nc-f0代上清。
3、盲传一代后检测上清滴度
①将mdck细胞接种于直径为100mm的平皿,每皿7×106个细胞,培养12小时。
②完成步骤①后,每皿加入500μl步骤2收获的上清,然后培养72小时,然后收获上清。将收获的上清定义为f1代上清(其中含有初始拯救出的f1代重组病毒)。f1代上清进行噬斑鉴定,每毫升上清造成的噬斑数量以10为底的对数值即为滴度。
r38a-f0代上清进行步骤3得到的f1代上清命名为r38a-f1代上清。
k41a-f0代上清进行步骤3得到的f1代上清命名为k41a-f1代上清。
r38a/k41a-f0代上清进行步骤3得到的f1代上清命名为r38a/k41a-f1代上清。
wt-f0代上清进行步骤3得到的f1代上清命名为wt-f1代上清。
nc-f0代上清进行步骤3得到的f1代上清命名为nc-f1代上清。
r38a-f1代上清的滴度为4.1±0.1。k41a-f1代上清的滴度为4.5±0.2。r38a/k41a-f1代上清的滴度为3.3±0.1。wt-f1代上清的滴度为5.6±0.1。nc-f1代上清的滴度为0,即不能使mdck产生噬斑。
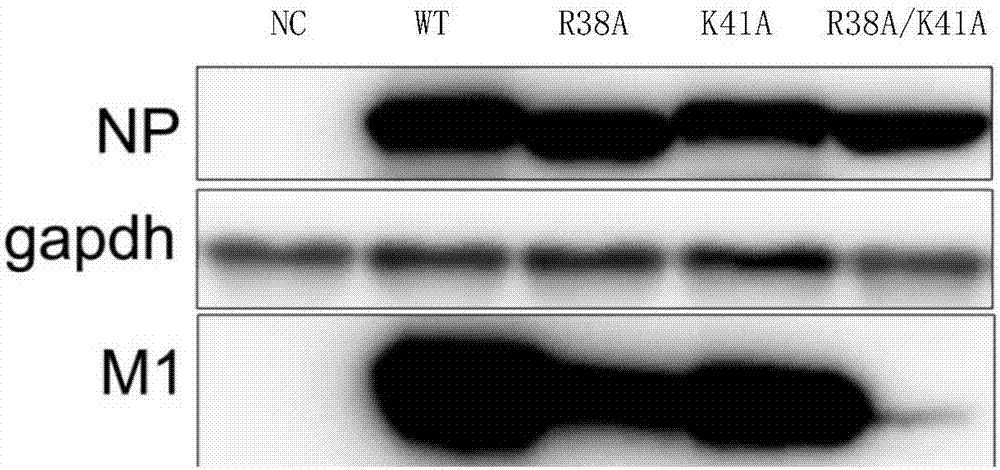
4、取步骤2收获的细胞,先进行破碎再进行westernblot(检测各主要病毒蛋白的表达)。westernblot中,用于检测np蛋白的一抗为抗np蛋白的多抗;用于检测m1蛋白的一抗为抗m1蛋白的单抗。westernblot结果见图1。第一组至第四组收获的细胞中流感病毒的两种重要蛋白(np蛋白、m1蛋白)均能够正常表达。
实施例2、重组病毒的有限复制特性鉴定(在mdck细胞上盲传)
分别将实施例1中制备得到的各个f1代上清(r38a-f1代上清、k41a-f1代上清、r38a/k41a-f1代上清、wt-f1代上清、nc-f1代上清)进行如下操作:
一、盲传
1、将200微升f1代上清加至60mm平皿中(每皿中已铺1×106个mdck细胞),然后培养72小时,然后分别收获上清和细胞。将收获的上清定义为f2代上清。为便于描述,将收获的细胞命名为f2细胞。
2、将200微升f2代上清加至60mm平皿中(每皿中已铺1×106个mdck细胞),然后培养72小时,然后分别收获上清和细胞。将收获的上清定义为f3代上清。为便于描述,将收获的细胞命名为f3细胞。
3、将200微升f3代上清加至60mm平皿中(每皿中已铺1×106个mdck细胞),然后培养72小时,然后分别收获上清和细胞。将收获的上清定义为f4代上清。为便于描述,将收获的细胞命名为f4细胞。
r38a-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为r38a-f2代上清、r38a-f3代上清、r38a-f4代上清。
k41a-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为k41a-f2代上清、k41a-f3代上清、k41a-f4代上清。
r38a/k41a-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为r38a/k41a-f2代上清、r38a/k41a-f3代上清、r38a/k41a-f4代上清。
wt-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为wt-f2代上清、wt-f3代上清、wt-f4代上清。
nc-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为nc-f2代上清、nc-f3代上清、nc-f4代上清。
二、f2代上清、f3代上清、f4代上清分别进行噬斑鉴定,每毫升上清造成的噬斑数量以10为底的对数值即为滴度。
r38a-f2代上清、r38a-f3代上清、r38a-f4代上清的滴度依次为4.0、3.8、3.6。
k41a-f2代上清、k41a-f3代上清、k41a-f4代上清的滴度依次为4.5、4.3、4.3。
r38a/k41a-f2代上清、r38a/k41a-f3代上清、r38a/k41a-f4代上清的滴度依次为2.5、2.0、0。
wt-f2代上清、wt-f3代上清、wt-f4代上清的滴度依次为6.0、6.2、6.1。
nc-f2代上清、nc-f3代上清、nc-f4代上清的滴度依次为0、0、0。
三、f2细胞、f3细胞、f4细胞分别进行如下步骤:先进行破碎再进行westernblot(检测各主要病毒蛋白的表达)。westernblot中,用于检测np蛋白的一抗为抗np蛋白的多抗;用于检测m1蛋白的一抗为抗m1蛋白的单抗。f2细胞westernblot结果见图2。f3细胞westernblot结果见图3。f4细胞westernblot结果见图4。
以上结果表明,r38a/k41a双突变病毒在盲传至第4代时失去复制能力。
实施例3、双突变重组病毒的有限复制特性鉴定(在a549细胞上传代)
分别将实施例1中制备得到的各个f1代上清(r38a-f1代上清、k41a-f1代上清、r38a/k41a-f1代上清、wt-f1代上清、nc-f1代上清)进行如下操作:
1、将f1代上清加至60mm平皿中(平皿中已铺a549细胞,moi=0.001),然后培养72小时,然后分别收获上清和细胞。将收获的上清定义为f2代上清。为便于描述,将收获的细胞命名为f2细胞。
2、将200微升f2代上清加至60mm平皿中(每皿中已铺1×106个a549细胞),然后培养72小时,然后分别收获上清和细胞。将收获的上清定义为f3代上清。为便于描述,将收获的细胞命名为f3细胞。
3、将200微升f3代上清加至60mm平皿中(每皿中已铺1×106个a549细胞),然后培养72小时,然后分别收获上清和细胞。将收获的上清定义为f4代上清。为便于描述,将收获的细胞命名为f4细胞。
r38a-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为r38a-f2代上清、r38a-f3代上清、r38a-f4代上清。
k41a-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为k41a-f2代上清、k41a-f3代上清、k41a-f4代上清。
r38a/k41a-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为r38a/k41a-f2代上清、r38a/k41a-f3代上清、r38a/k41a-f4代上清。
wt-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为wt-f2代上清、wt-f3代上清、wt-f4代上清。
nc-f1代上清进行上述步骤得到的f2代上清、f3代上清和f4代上清依次命名为nc-f2代上清、nc-f3代上清、nc-f4代上清。
二、f2代上清、f3代上清、f4代上清分别进行噬斑鉴定,每毫升上清造成的噬斑数量以10为底的对数值即为滴度。
r38a-f2代上清、r38a-f3代上清、r38a-f4代上清的滴度依次为3.7、3.4、3.3。
k41a-f2代上清、k41a-f3代上清、k41a-f4代上清的滴度依次为4.3、4.2、4.0。
r38a/k41a-f2代上清、r38a/k41a-f3代上清、r38a/k41a-f4代上清的滴度依次为2.2、0、0。
wt-f2代上清、wt-f3代上清、wt-f4代上清的滴度依次为5.8、5.5、5.3。
nc-f2代上清、nc-f3代上清、nc-f4代上清的滴度依次为0、0、0。
三、f2细胞、f3细胞、f4细胞分别进行如下步骤:先进行破碎再进行westernblot(检测各主要病毒蛋白的表达)。westernblot中,用于检测np蛋白的一抗为抗np蛋白的多抗;用于检测m1蛋白的一抗为抗m1蛋白的单抗。f2细胞westernblot结果见图5。f3细胞westernblot结果见图6。f4细胞westernblot结果见图7。
以上结果表明,r38a/k41a双突变病毒在盲传至第3代时失去复制能力。
实施例4、双突变重组病毒感染小鼠
一、体重和死亡率
体重约16g的5周龄balb/c小鼠经过乙醚麻醉后,分成5组,每组10只,分别处理如下:
第一组:试验第0天,利用鼻吸法吸入50μl实施例2制备的wt-f3代上清(病毒含量为103pfu),然后每日观察小鼠体重和死亡率。
第二组:试验第0天,利用鼻吸法吸入50μl实施例2制备的r38a/k41a-f3代上清(病毒含量为103pfu),然后每日观察小鼠体重和死亡率。
第三组:试验第0天,利用鼻吸法吸入50μl实施例3制备的wt-f2代上清(病毒含量为103pfu),然后每日观察小鼠体重和死亡率。
第四组:试验第0天,利用鼻吸法吸入50μl实施例3制备的r38a/k41a-f2代上清(病毒含量为103pfu),然后每日观察小鼠体重和死亡率。
第五组:试验第0天,利用鼻吸法吸入50微升灭菌后的pbs缓冲液。
相对体重=某一天的体重÷试验第0天的体重×100%。
相同体重和死亡率的结果见表1。
表1
二、病毒-血清中和抗体效价和肺脏病毒滴度
体重约16g的5周龄balb/c小鼠经过乙醚麻醉后,分成5组,每组9只,分别处理如下:
第一组:试验第0天,利用鼻吸法吸入50μl实施例2制备的wt-f3代上清(病毒含量为103pfu);
第二组:试验第0天,利用鼻吸法吸入50μl实施例2制备的r38a/k41a-f3代上清(病毒含量为103pfu),然后每日观察小鼠体重和死亡率。
第三组:试验第0天,利用鼻吸法吸入50μl实施例3制备的wt-f2代上清(病毒含量为103pfu),然后每日观察小鼠体重和死亡率。
第四组:试验第0天,利用鼻吸法吸入50μl实施例3制备的r38a/k41a-f2代上清(病毒含量为103pfu),然后每日观察小鼠体重和死亡率。
第五组:试验第0天,利用鼻吸法吸入50μl灭菌后的pbs缓冲液。
分别于试验第3天、试验第7天和试验第14天每组处死3只小鼠。试验第3天、第7天和第14天处死的小鼠取血清检测病毒-血清中和抗体效价(简称效价)。试验第3天和第7天处死的小鼠取肺脏,用研磨器将1g肺脏进行组织匀浆于1毫升冰浴pbs缓冲液中,匀浆液离心5000g×10min,收集上清,进行噬斑鉴定(每毫升上清造成的噬斑数量以10为底的对数值即为滴度)。
结果见表2。
表2
三、攻毒试验
体重约16g的5周龄balb/c小鼠经过乙醚麻醉后,分成3组,每组6只,分别处理如下:
第一组:试验第0天,利用鼻吸法吸入50μl实施例2制备的r38a/k41a-f3代上清(病毒含量为103pfu);试验第17天,利用鼻吸法吸入50μl实施例1制备的wt-f1代上清(病毒含量为103.88pfu,用dmem培养液调整体积),试验第32天统计存活率。
第二组:试验第0天,利用鼻吸法吸入50μl实施例3制备的r38a/k41a-f2代上清(病毒含量为103pfu);试验第17天,利用鼻吸法吸入50μl实施例1制备的wt-f1代上清(病毒含量为103.88pfu,用dmem培养液调整体积),试验第32天统计存活率。
第三组:试验第0天,利用鼻吸法吸入50μl灭菌后的pbs缓冲液;试验第17天,利用鼻吸法吸入50μl实施例1制备的wt-f1代上清(病毒含量为103.88pfu,用dmem培养液调整体积),试验第32天统计存活率。
第一组和第二组的存活率均为100%。
第三组的存活率为0%。
实施例5、大批量制备
1、将200微升实施例1中制备得到的r38a/k41a-f1代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f2代上清。
2、将200微升f2代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f3代上清。
3、将200微升f3代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f4代上清。
4、将200微升f4代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f5代上清。
5、将200微升f5代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f6代上清。
6、将200微升f6代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f7代上清。
7、将200微升f7代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f8代上清。
8、将200微升f8代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f9代上清。
9、将200微升f9代上清加至60mm平皿中(每皿中已铺1×106个vero细胞),然后培养72小时,然后收获上清。将收获的上清定义为f10代上清。
将f2代上清、f3代上清、f4代上清、f5代上清和f10代上清分别作为待测上清,进行噬斑鉴定,每毫升上清造成的噬斑数量以10为底的对数值即为滴度。
f2代上清的滴度为3.34。
f3代上清的滴度为4.84。
f4代上清的滴度为4.34。
f5代上清的滴度为5.18。
f10代上清的滴度为4.51。
结果表明,采用vero细胞,可以传代并制备病毒液。
sequencelisting
<110>中国科学院微生物研究所
<120>一种用于流感病毒的有限复制型黏膜免疫疫苗
<130>gncyx172149
<160>9
<170>patentinversion3.5
<210>1
<211>693
<212>dna
<213>artificialsequence
<400>1
atggatccaaacactgtgtcaagctttcaggtagattgctttctttggcatgtccgcaaa60
agagttgcagaccaagaactaggtgatgccccattccttgatcggcttcgccgagatcag120
aagtccctaagaggaagaggcagcactctcggtctggacatcgaaacagccacccgtgct180
ggaaagcaaatagtggagcggattctgaaggaagaatctgatgaggcactcaaaatgacc240
atggcctctgtacctgcatcgcgctacctaactgacatgactcttgaggaaatgtcaagg300
cactggttcatgctcatgcccaagcagaaagtggcaggccctctttgtatcagaatggac360
caggcgatcatggataagaacatcatactgaaagcgaacttcagtgtgatttttgaccgg420
ctggagactctaatattactaagggccttcaccgaagaggggacaattgttggcgaaatt480
tcaccactgccctctcttccaggacatactgatgaggatgtcaaaaatgcagttggggtc540
ctcatcggaggacttgaatggaataataacacagttcgagtctctgaaactctacagaga600
ttcgcttggagaagcagtaatgagaatgggagacctccactcactccaaaacagaaacgg660
aaaatggcgggaacaattaggtcagaagtttga693
<210>2
<211>230
<212>prt
<213>artificialsequence
<400>2
metaspproasnthrvalserserpheglnvalaspcyspheleutrp
151015
hisvalarglysargvalalaaspglngluleuglyaspalaprophe
202530
leuaspargleuargargaspglnlysserleuargglyargglyser
354045
thrleuglyleuaspilegluthralathrargalaglylysglnile
505560
valgluargileleulysglugluseraspglualaleulysmetthr
65707580
metalaservalproalaserargtyrleuthraspmetthrleuglu
859095
glumetserarghistrpphemetleumetprolysglnlysvalala
100105110
glyproleucysileargmetaspglnalailemetasplysasnile
115120125
ileleulysalaasnpheservalilepheaspargleugluthrleu
130135140
ileleuleuargalaphethrglugluglythrilevalglygluile
145150155160
serproleuproserleuproglyhisthraspgluaspvallysasn
165170175
alavalglyvalleuileglyglyleuglutrpasnasnasnthrval
180185190
argvalsergluthrleuglnargphealatrpargserserasnglu
195200205
asnglyargproproleuthrprolysglnlysarglysmetalagly
210215220
thrileargsergluval
225230
<210>3
<211>2202
<212>dna
<213>artificialsequence
<400>3
ctgattcaaaatggaagattttgtgcgacaatgcttcaatccgatgattgtcgagcttgc60
ggaaaaggcaatgaaagagtatggagaggacctgaaaatcgaaacaaacaaatttgcagc120
aatatgcactcacttggaagtgtgcttcatgtattcagattttcacttcatcgatgagca180
aggcgagtcaatagtcgtagaacttggcgatccaaatgcacttttgaagcacagatttga240
aataatcgagggaagagatcgcacaatagcctggacagtaataaacagtatttgcaacac300
tacaggggctgagaaaccaaagtttctaccagatttgtatgattacaagaagaatagatt360
catcgaaattggagtaacaaggagagaagttcacatatactatctggaaaaggccaataa420
aattaaatctgagaagacacacatccacattttctcattcactggggaggaaatggccac480
aaaggccgactacactctcgatgaagaaagcagggctaggatcaaaaccaggctattcac540
cataagacaagaaatggctagcagaggcctctgggattcctttcgtcagtccgagagagg600
cgaagagacaattgaagaaagatttgaaatcacaggaacaatgcgcaagcttgccgacca660
aagtctcccgccaaacttctccagccttgaaaattttagagcctatgtggatggattcga720
accgaacggctacattgagggcaagctttctcaaatgtccaaagaagtaaatgctagaat780
tgaaccttttttgaaatcaacaccacgaccacttagacttccggatgggcctccctgttc840
tcagcggtccaaattcctgctgatggatgccttaaaattaagcattgaggacccaagtca900
tgagggagaggggataccgctatatgatgcaatcaaatgcatgagaacattctttggatg960
gaaggaacccaatgttgttaaaccacacgaaaagggaataaatccaaattatcttctgtc1020
atggaagcaagtactggcagaactgcaggacattgagaatgaggagaaaattccaaggac1080
taaaaatatgaagaaaacgagtcagttaaagtgggcacttggtgagaacatggcaccaga1140
aaaggtagactttgacgattgtaaagatgtaggcgatttgaagcaatatgatagtgatga1200
accagaattgaggtcgcttgcaagttggattcagaatgagttcaacaaggcatgtgaact1260
gaccgattcaagctggatagagctcgatgagattggagaagatgcggctccaattgaaca1320
cattgcaagcatgagaaggaattatttcacagcagaggtgtctcattgcagagccacaga1380
atacataatgaagggggtgtacatcaatactgccttgcttaatgcatcctgtgcagcaat1440
ggatgatttccaattgattccaatgataagcaagtgtagaactaaggagggaaggcgaaa1500
gaccaatttgtacggtttcatcataaaaggaagatcccacttaaggaatgacaccgatgt1560
ggtaaactttgtgagcatggagttttccctcactgacccaagacttgaaccacacaaatg1620
ggagaagtactgtgttcttgaggtaggagatatgcttctaagaagtgccataggccatgt1680
gtcaaggcctatgttcttgtatgtgaggacaaatggaacctcaaaaattaaaatgaaatg1740
ggggatggaaatgaggcgttgcctccttcagtcacttcaacaaatcgagagtatgattga1800
agctgagtcctctgtcaaggagaaagacatgaccaaagagttctttgaaaacaaatcaga1860
aacatggcccgttggagagtcccccaaaggagtggaggaaggttccattgggaaggtctg1920
cagaactttattggcaaagtcggtattcaacagcttgtatgcatctccacaactggaagg1980
attttcagctgaatcaagaaaactgcttcttatcgttcaggctcttagggacaacctgga2040
acctgggacctttgatcttggggggctatatgaagcaattgaggagtgcctgattaatga2100
tccctgggttttgcttaatgcttcttggttcaactccttcctcacacatgcattgagata2160
gttgtggcaatgctactatttgctatccatactgtccaaaaa2202
<210>4
<211>2303
<212>dna
<213>artificialsequence
<400>4
atttgaatggatgtcaatccgactttacttttcttaaaagtgccagcacaaaatgctata60
agcacaactttcccttatactggagaccctccttacagccatgggacaggaacaggatac120
accatggatactgtcaacaggacacatcagtactcagaaaggggaagatggacaacaaac180
accgaaactggagcaccgcaactcaacccgattgatgggccactgccagaagacaatgaa240
ccaagtggttatgcccaaacagattgtgtattggaagcaatggccttccttgaggaatcc300
catcctggtatctttgagacctcgtgtcttgaaacgatggaggttgttcagcaaacacga360
gtggacaagctgacacaaggccgacagacctatgactggactctaaataggaaccagcct420
gctgcaacagcattggccaacacaatagaagtgttcagatcaaatggcctcacggccaat480
gaatctggaaggctcatagacttccttaaggatgtaatggagtcaatgaacaaagaagaa540
atggagatcacaactcattttcagagaaagagacgagtgagagacaatatgactaagaaa600
atggtgacacagagaacaataggtaaaaggaagcagagattgaacaaaaggagttatcta660
attagggcattaaccctgaacacaatgaccaaagatgctgagagagggaagctaaaacgg720
agagcaattgcaaccccagggatgcaaataagggggtttgtatactttgttgagacacta780
gcaaggagtatatgtgagaaacttgaacaatcaggattgccagttggaggcaatgagaag840
aaagcaaagttggcaaatgttgtaaggaagatgatgaccaattctcaggacactgaaatt900
tctttcaccatcactggagataacaccaaatggaacgaaaatcagaaccctcggatgttt960
ttggccatgatcacatatataaccagaaatcagcccgaatggttcagaaatgttctaagt1020
attgctccaataatgttctcaaacaaaatggcgagactgggaaaggggtacatgtttgag1080
agcaagagtatgaaacttagaactcaaatacctgcagaaatgctagcaagcatcgatttg1140
aaatacttcaatgattcaactagaaagaagattgaaaaaatccggccgctcttaatagat1200
gggactgcatcattgagccctggaatgatgatgggcatgttcaatatgttaagtactgta1260
ttaggcgtctccatcctgaatcttggacaaaagagacacaccaagactacttactggtgg1320
gatggtcttcaatcttctgatgattttgctctgattgtgaatgcacccaatcatgaaggg1380
attcaagccggagtcaacaggttttatcgaacctgtaagctacttggaattaatatgagc1440
aagaaaaagtcttacataaacagaacaggtacatttgaattcacaagttttttctatcgt1500
tatgggtttgttgccaatttcagcatggagcttcccagctttggggtgtctgggatcaac1560
gagtctgcggacatgagtattggagttactgtcatcaaaaacaatatgataaacaatgat1620
cttggtccagcaaccgctcaaatggcccttcagctgttcatcaaagattacaggtacacg1680
taccggtgccatagaggtgacacacaaatacaaacccgaagatcatttgaaataaagaaa1740
ctgtgggagcaaacccattccaaagctggactgctggtctccgacggaggcccaaattta1800
tacaacattagaaatctccacattcctgaagtctgcttgaaatgggaattaatggatgag1860
gattaccaggggcgtttatgcaacccactgaacccatttgtcaaccataaagacattgaa1920
tcagtgaacaatgcagtgataatgccagcacatggtccagccaaaaacatggagtatgat1980
gctgttgcaacaacacactcctggatccccaaaagaaatcgatccatcttgaatacaagc2040
caaagaggaatacttgaagatgaacaaatgtaccaaaagtgctgcaacttatttgaaaaa2100
ttcttccccagcagttcatacagaagaccagtcgggatatccagtatggtggaggctatg2160
gtttccagagcccgaattgatgcacgaattgatttcgaatctggaaggataaagaaagag2220
gagttcactgagatcatgaagatctgttccaccattgaagagctcagacggcaaaaatag2280
tgaatttagcttgtccttcatga2303
<210>5
<211>2290
<212>dna
<213>artificialsequence
<400>5
atggaaagaataaaagaactaaggaatctaatgtcgcagtctcgcactcgcgagatactc60
acaaaaaccaccgtggaccatatggccataatcaagaagtacacatcaggaagacaggag120
aagaacccagcacttaggatgaaatggatgatggcaatgaaatatccaattacagcagac180
aagaggataacggaaatgattcctgagagaaatgagcagggacaaactttatggagtaaa240
atgaatgacgccggatcagaccgagtgatggtatcacctctggctgtgacatggtggaat300
aggaatggaccagtgacaagtacagttcattatccaaaaatctacaaaacttattttgaa360
aaagtcgaaaggttaaaacatggaacctttggccctgtccattttagaaaccaagtcaaa420
atacgtcgaagagttgacataaatcctggtcatgcagatctcagtgccaaagaggcacag480
gatgtaatcatggaagttgttttccctaacgaagtgggagccaggatactaacatcggaa540
tcgcaactaacgacaaccaaagagaagaaagaagaactccagggttgcaaaatttctcct600
ctgatggtggcatacatgttggagagagaactggtccgcaaaacgagattcctcccagtg660
gctggtggaacaagcagtgtgtacattgaagtgttgcatttgacccaaggaacatgctgg720
gaacagatgtacactccaggaggggaggcgaggaatgatgatgttgatcaaagcttaatt780
attgctgctagaaacatagtaagaagagccacagtatcagcagatccactagcatcttta840
ttggagatgtgccacagcacgcagattggtggagtaaggatggtaaacatccttaggcag900
aacccaacagaagagcaagccgtggatatttgcaaggctgcaatgggactgagaattagc960
tcatccttcagttttggtggattcacatttaagagaacaagcggatcatcagtcaagaga1020
gaggaagaggtgcttacgggcaatcttcagacattgaagataagagtgcatgagggatat1080
gaagagttcacaatggttgggagaagagcaacagctatactcagaaaagcaaccaggaga1140
ttgattcagctgatagtgagtgggagagacgaacagtcgattgccgaagcaataattgtg1200
gccatggtattttcacaagaggattgtatgataaaagcagttagaggtgacctgaatttc1260
gtcaatagggcgaatcagcgattgaatcccatgcaccaacttttgagacattttcagaag1320
gatgcaaaggtgctctttcaaaattggggaattgaatccatcgacaatgtgatgggaatg1380
atcgggatattgcccgacatgactccaagcaccgagatgtcaatgagaggagtgagaatc1440
agcaaaatgggggtagatgagtattccagcgcggagaagatagtggtgagcattgaccgt1500
tttttgagagttagggaccaacgtgggaatgtactactgtctcccgaggaggtcagtgaa1560
acacagggaacagagaaactgacaataacttactcatcgtcaatgatgtgggagattaat1620
ggtcctgaatcagtgttggtcaatacctatcagtggatcatcagaaactgggaaactgtt1680
aaaattcagtggtcccagaatcctacaatgctgtacaataaaatggaatttgagccattt1740
cagtctttagttccaaaggccgttagaggccaatacagtgggtttgtgagaactctgttc1800
caacaaatgagggatgtgcttgggacatttgataccgctcagataataaaacttcttccc1860
ttcgcagccgctccaccaaagcaaagtggaatgcagttctcctcattgactataaatgtg1920
aggggatcaggaatgagaatacttgtaaggggcaattctccagtattcaactacaacaag1980
accactaaaagactcacagttctcggaaaggatgctggccctttaactgaagacccagat2040
gaaggcacagctggagttgagtccgcagttctgagaggattcctcattctgggcaaagaa2100
gacaggagatatggaccagcattaagcataaatgaactgagcaaccttgcgaaaggagag2160
aaggctaatgtgctaattgggcaaggagacgtggtgttggtaatgaaacggaaacggaac2220
tctagcatacttactgacagccagacagcgaccaaaagaattcggatggccatcaattag2280
tgtcgaatag2290
<210>6
<211>1725
<212>dna
<213>artificialsequence
<400>6
ccaaaatgaaggcaaaactactggtcctgttatatgcatttgtagctacagatgcagaca60
caatatgtataggctaccatgcgaacaactcaaccgacactgttgacacaatattcgaga120
agaatgtggcagtgacacattctgttaacctgctcgaagacagacacaacgggaaactat180
gtaaattaaaaggaatagccccactacaattggggaaatgtaacatcatcggatggctct240
tgggaaatccagaatgcgactcactgcttccagcgagatcatggtcctacattgtagaaa300
caccaaactctgagaatggagcatgttatccaggagatttcatcgactatgaggaactga360
gggagcaattgagctcagtatcatcattagaaagattcgaaatatttcccaaggaaagtt420
catggcccaaccacacattcaacggagtaacagcatcatgctcccataggggaaaaagca480
gtttttacagaaatttgctatggctgacgaagaagggggattcatacccaaagctgacca540
attcctatgtgaacaataaagggaaagaagtccttgtactatggggtgttcatcacccgt600
ctagcagtgatgagcaacagagtctctatagtaatggaaatgcttatgtctctgtagcgt660
cttcaaattataacaggagattcaccccggaaatagctgcaaggcccaaagtaaaagatc720
aacatgggaggatgaactattactggaccttgctagaacccggagacacaataatatttg780
aggcaactggtaatctaatagcaccatggtatgctttcgcactgagtagagggtttgagt840
ccggcatcatcacctcaaacgcgtcaatgcatgagtgtaacacgaagtgtcaaacacccc900
agggatctataaacagcaatctccctttccagaatatacacccagtcacaataggagagt960
gcccaaaatatgtcaggagtaccaaattgaggatggttacaggactaagaaacatcccat1020
ccattcaatacagaggtctatttggagccattgctggttttattgaggggggatggactg1080
gaatgatagatggatggtatggttatcatcatcagaatgaacagggatcaggctatgcag1140
cggatcaaaaaagcacacagaatgccattaacgggattacaaacaaggtgaactctatta1200
tcgagaaaatgaacactcaattcacagctgtgggtaaagaattcaacaacttagaaaaaa1260
ggatggaaaatttaaataaaaaagttgatgatgggtttctggacatttggacatataatg1320
cagaattgttagttctactggaaaatggaagaactttggatttccatgacttaaatgtga1380
agaatctgtacgagaaagtaaaaagccaattaaagaataatgccaaagaaatcggaaatg1440
ggtgttttgagttctaccacaagtgtgacaatgaatgcatggaaagtgtaagaaatggga1500
cttatgattatccaaaatattcagaagaatcaaagttgaacagggaaaagatagatggag1560
tgaaattggaatcaatgggggtgtatcagattctggcgatctactcaactgtcgccagtt1620
cactggtgcttttggtctccctgggggcaatcagtttctggatgtgttctaatgggtctt1680
tgcagtgcagaatatgcatctgagattaggatttcagaaatataa1725
<210>7
<211>1526
<212>dna
<213>artificialsequence
<400>7
tcactcacagagtgacatcgaaatcatggcgaccaaaggcaccaaacgatcttacgaaca60
gatggagactgatggagaacgccagaatgccactgaaatcagagcatctgtcggaaaaat120
gattgatggaattggacgattctacatccaaatgtgcaccgaacttaaactcagtgatta180
tgagggacggctgattcagaacagcttaacaatagagagaatggtgctctctgcttttga240
cgagaggaggaataaatatctagaagaacatcccagtgcggggaaagatcctaagaaaac300
tggaggacctatatacaggagagtagatggaaagtggaggagagaactcatcctttatga360
caaagaagaaataagacgaatctggcgccaagctaataatggtgacgatgcaacggctgg420
tctgactcacatgatgatctggcactccaatttgaatgatgcaacttaccagaggacaag480
agctcttgttcgcacaggaatggatcccaggatgtgctcactgatgcagggttcaaccct540
ccctaggaggtctggggccgcaggtgctgcagtcaaaggagttggaacaatggtgatgga600
attgatcagaatgatcaaacgtgggatcaatgatcggaacttctggaggggtgagaatgg660
acggagaacaaggattgcttatgaaagaatgtgcaacattctcaaagggaaatttcaaac720
agctgcacaaagaacaatggtggatcaagtgagagagagccggaatccaggaaatgctga780
gttcgaagatctcatctttttagcacggtctgcactcatattgagagggtcagttgctca840
caagtcctgcctgcctgcctgtgtgtatggatctgccgtagccagtggatacgactttga900
aagagagggatactctctagtcggaatagaccctttcagactgcttcaaaacagccaagt960
atacagcctaatcagaccaaatgagaatccagcacacaagagtcaactggtgtggatggc1020
atgccattctgctgcatttgaagatctaagagtatcaagcttcatcagagggacgaaagt1080
ggtcccaagagggaagctttccactagaggagttcaaattgcttccaatgaaaacatgga1140
gactatggaatcaagtacccttgaactgagaagcagatactgggccataaggaccagaag1200
tggagggaacaccaatcaacagagggcttcctcgggccaaatcagcatacaacctacgtt1260
ctcagtacagagaaatctcccttttgacagaccaaccattatggcagcattcactgggaa1320
tacagaggggagaacatctgacatgagaaccgaaatcataaggctgatggaaagtgcaag1380
accagaagatgtgtctttccaggggcggggagtcttcgagctctcggacgaaaaggcaac1440
gagcccgatcgtgccctcctttgacatgagtaatgaaggatcttatttcttcggagacaa1500
tgcagaggagtacgacaattaaagaa1526
<210>8
<211>1368
<212>dna
<213>artificialsequence
<400>8
atgaatccaaaccagaaaataataaccattgggtcaatctgtatggtagtcggaataatt60
agcctaatattgcaaataggaaatataatctcaatatggattagccattcaattcaaacc120
ggaaatcaaaaccatactggaatatgcaaccaaggcagcattacctataaagttgttgct180
gggcaggactcaacttcagtgatattaaccggcaattcatctctttgtcccatccgtggg240
tgggctatacacagcaaagacaatggcataagaattggttccaaaggagacgtttttgtc300
ataagagagccttttatttcatgttctcacttggaatgcaggaccttttttctgactcaa360
ggcgccttactgaatgacaagcattcaagggggacctttaaggacagaagcccttatagg420
gccttaatgagctgccctgtcggtgaagctccgtccccgtacaattcaaggtttgaatcg480
gttgcttggtcagcaagtgcatgtcatgatggaatgggctggctaacaatcggaatttct540
ggtccagatgatggagcagtggctgtattaaaatacaacggcataataactgaaaccata600
aaaagttggaggaagaatatattgagaacacaagagtctgaatgtacctgtgtaaatggt660
tcatgttttaccataatgaccgatggcccaagtgatgggctggcctcgtacaaaattttc720
aagatcgagaaggggaaggttactaaatcaatagagttgaatgcacctaattctcactac780
gaggaatgttcctgttaccctgataccggcaaagtgatgtgtgtgtgcagagacaattgg840
cacggttcgaaccgaccatgggtgtccttcgaccaaaacctagattataaaataggatac900
atctgcagtggggttttcggtgacaacccgcgtcccaaagatggaacaggcagctgtggc960
ccagtgtctgctgatggagcaaacggagtaaagggattttcatataagtatggtaatggt1020
gtttggataggaaggactaaaagtgacagttccagacatgggtttgagatgatttgggat1080
cctaatggatggacagagactgatagtaggttctctatgagacaagatgttgtggcaatg1140
actgatcggtcagggtacagcggaagtttcgttcaacatcctgagctaacagggctagac1200
tgtatgaggccttgcttctgggttgaattaatcagggggctacctgaggagaacgcaatc1260
tggactagtgggagcatcatttctttttgtggtgtgaatagtgatactgtagattggtct1320
tggccagacggtgctgagttgccgttcaccattgacaagtagtttgtt1368
<210>9
<211>984
<212>dna
<213>artificialsequence
<400>9
atgagtcttctaaccgaggtcgaaacgtacgttctctctatcgtcccgtcaggccccctc60
aaagccgagatcgcacagagacttgaagatgtctttgcagggaagaacaccgatcttgag120
gttctcatggaatggctaaagacaagaccaatcctgtcacctctgactaaggggatttta180
ggatttgtgttcacgctcaccgtgcccagtgagcggggactgcagcgtagacgctttgtc240
caaaatgctcttaatgggaacggagatccaaataacatggacaaagcagttaaactgtat300
aggaagcttaagagggagataacattccatggggccaaagaaatagcactcagttattct360
gctggtgcacttgccagttgtatgggcctcatatacaacaggatgggggctgtgaccact420
gaagtggcatttggcctggtatgcgcaacctgtgaacagattgctgactcccagcatcgg480
tctcataggcaaatggtgacaacaaccaatccactaatcagacatgagaacagaatggtt540
ctagccagcactacagctaaggctatggagcaaatggctggatcgagtgagcaagcagca600
gaggccatggatattgctagtcaggccaggcaaatggtgcaggcgatgagaaccgttggg660
actcatcctagctccagtgctggtctaaaagatgatcttcttgaaaatttgcaggcctat720
cagaaacgaatgggggtgcagatgcaacgattcaagtgatcctctcgtcattgcagcaaa780
tatcattggaatcttgcacttgatattgtggattcttgatcgtctttttttcaaatgcat840
ttatcgtcgctttaaatacggtttgaaaagagggccttctacggaaggagtgccagagtc900
tatgagggaagaatatcgaaaggaacagcagaatgctgtggatgttgacgatggtcattt960
tgtcaacatagagctggagtaaaa984
- 还没有人留言评论。精彩留言会获得点赞!