选择由活性化合物诱导的特定效应的基于RNA编辑的算法和体外方法与流程
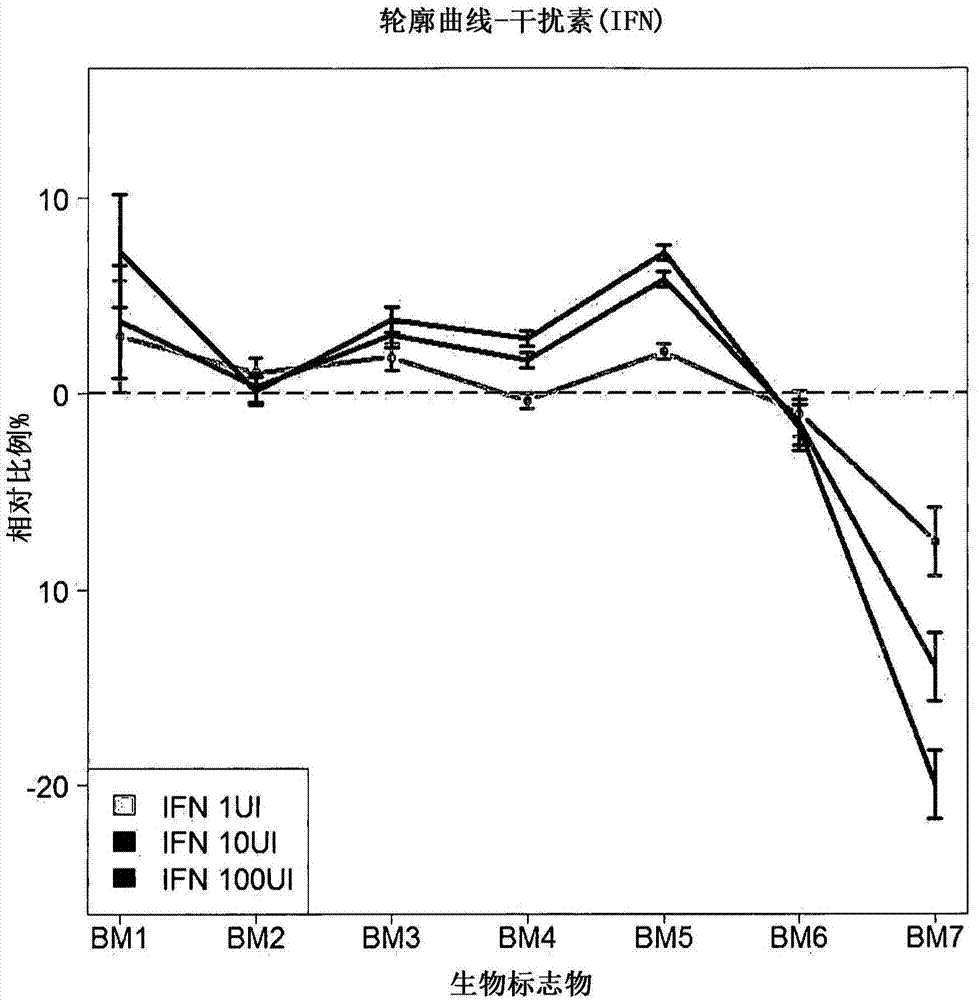
精神病症在全球健康系统中的权重越来越大(1)。其是西方社会的常见病症,且在1/5个体的一生中至少影响其1次。精神科病症由影响大脑回路、神经传递和神经可塑性的分子途径受扰所致。近期研究表明,诸如甲基化、乙酰化和脱氨等dna和rna上的表观遗传修饰的改变与例如重度抑郁症、双相情感障碍和精神分裂症相关(2,3)。近期研究还启发了催化rna上的腺苷脱氨(rna的a至i编辑)的编辑酶的重要性。已显示该具体机理会直接调节基本上编码高度保守的神经递质和突触相关因子的基因的功能(4-7)。重要的是,该rna编辑机制和同源adad酶(作用于rna的腺苷脱氨酶)在健康和疾病中的作用近来已通过其在罹患精神科病症的患者的大脑中的失调的累积证据而得到更深入的支持(8,9)。adar作用于双链前体mrna茎环以特异性地使腺苷残基优先脱氨。位于编码序列中的残基的脱氨会导致产生具有不同药理学性质的受体变体(例如,5-羟色胺2c受体、谷氨酸受体)的氨基酸取代(10)。已提出在大脑中的5-羟色胺生物学的异常是潜藏于抑郁和/或自杀行为的特征性状(11-13)。通过分析自杀牺牲者的死后脑组织,发明人等已观察到针对已知会极大地损害5-ht2cr药理学性质的5-羟色胺受体2c(5ht2cr)前体mrna的rna编辑活性的明显改变(10,14)。令人感兴趣的是,自杀牺牲者的人皮质中的5-ht2crmrna编辑谱的这些改变与在sh-sy5y细胞中观察到的经干扰素诱导的变化部分地重叠。发明人精确定位了特定的生物标志物来表征与抑郁/自杀患者相关联的5-ht2cr的“rna编辑特征”。已报道属于不同治疗类别的几种药物会潜在诱导严重的精神科副作用,尤其是抑郁和自杀倾向(15-18)。今天,尚未有鉴定这类分子的许可测试,而美国食品药品管理局(fda)仅发出了关于整个治疗类别的一般性警告。因此,存在对提供能以高精度和高区分力确定药物或候选药物诱导负面副作用的风险的体外测试的需求。发明人验证了此前设计的利用仔细选出的细胞系(sh-sy5y)预测药物诱导的精神科副作用的新型体外检测。发明人筛选了260种市场许可的化合物以检验药物诱导的5-ht2cr编辑的改变。化合物选自宽范围的已知会(带有fda警示标签和/或许多病例报告)或不会(没有经报道的精神科副作用)潜在诱导自杀倾向的治疗类别(抗抑郁剂、抗精神病药、抗肥胖剂、抗病毒剂、抗炎剂、抗真菌剂、抗癫痫药、情绪稳定剂等等)。数据被用于以高特异性和灵敏度来鉴定“有风险”的化合物。在第一方面,本发明涉及一种用于体外预测化合物、特别是药物在患者中诱导特定效应的概率的算法,其中,所述算法通过包括以下步骤的方法获得:a)-选择展示出rna的a至i编辑的至少一个靶标,其前体mrna是adar酶(作用于rna的腺苷脱氨酶)的底物,所述adar的作用导致产生不同的亚型或位点,-选择内源性表达所述至少一个靶标和至少所述adar酶的至少一个细胞系,-选择阳性对照化合物,在所述细胞系的细胞经所述阳性对照处理时所述阳性对照化合物能够以剂量依赖性方式改变所述靶亚型/或编辑位点的相对比例;-选择由一定比例的注释有诱导所述特定效应的风险评分的化合物的组成的分子集合,b)采用所述分子集合的每一种分子以及阴性对照和所述阳性对照来处理所述细胞系的细胞,c)分析已用所述集合的分子处理的每个样品中的所述至少一个靶标rna编辑谱,从而获得针对所述集合的每种分子且针对所述靶标的每种编辑亚型/或位点而言的所述靶标的rna编辑水平的比例,d)-i)通过单变量分析统计法,针对每种亚型/或编辑位点评估其精度及其区分分子诱导所述特定效应的风险的能力;和/或-ii)通过多变量分析统计法,针对亚型和/或编辑位点的每种组合来评估其精度及其区分分子诱导所述特定效应的风险的能力;和-iii)选择展示出最佳区分性能的组合,e)利用所选择的亚型/或编辑位点的组合来建立算法,和使用由此获得的算法来预测药物、化合物或分子在患者中诱导所述特定效应的概率。本说明书中的化合物意指矿物、化学或生物学化合物,特别是他们能对人、动物患者或者在植物中有活性。在本说明书中,用语“患者”也包括植物。术语“算法”也包括统计模型(例如cart模型)。在优选实施方式中,在根据本发明的所述算法中,所述特定效应是副作用,优选选自负面或希望的副作用、优选负面副作用。在优选实施方式中,展示出rna的a至i编辑的所述靶标选自由5-ht2cr、pde8a(磷酸二酯酶8a)、gria2(谷氨酸受体2)、gria3、gria4、grik1、grik2、grin2c、grm4、grm6、flnb(细丝蛋白b)、5-ht2a、gabra3(gabaα3)、flna、cyfip2组成的组。在优选实施方式中,所述特定效应、优选副作用、更优选希望的或负面的副作用选自包括以下的组:心血管、变态反应学、cns、特别是精神科、皮肤病学、内分泌学、胃肠病学、血液学、感染学(infectiology)、代谢、神经肌肉、肿瘤学、炎性和肥胖、负面副作用。更优选的是精神科负面副作用。在优选实施方式中,根据本发明的算法的所述细胞系的细胞来自内源性表达所述靶标和adar的细胞系。更优选地,所述细胞系选自由以下组成的组:-能够内源性表达所述靶标并展示与人皮质中所观察到的相似的adar酶表达稳态的人或动物细胞系,-成神经细胞瘤细胞系,优选人细胞系,-成神经细胞瘤细胞系,对其而言在相对于阴性对照或空载剂对照归一化时阳性对照以至少4倍、优选至少5或6倍的倍数诱导来诱导adar1a表达,和-人sh-sy5y细胞系。在优选实施方式中,在步骤b)中,所述细胞系的细胞在12h至72h、优选48h±4h的时段中用待测试的分子或对照进行处理,最优选48h。在优选实施方式中,在根据本发明的算法中,所述阳性对照是干扰素α,或能够以100iu/ml再现干扰素rna编辑谱的化合物(如例如图6中所示)。使用sh-sy5y人成神经细胞瘤细胞系,这是因为其内源性表达5-ht2crmrna并且展示出与人皮质干扰素α中所观察到的类似的adar酶表达稳态。在优选实施方式中,在根据本发明的算法或模型中,步骤c)包括以下步骤:相对于经空载剂处理的对照细胞,针对所述细胞系中的每种亚型或位点确定rna编辑的基础水平,从而针对每种分子和每种编辑亚型或编辑位点获得所述靶标的rna编辑水平的平均/中位数相对比例。优选地,所述经空载剂处理的对照细胞是经dmso处理的对照细胞。在优选实施方式中,在根据本发明的算法或模型中,所述方法是用于体外预测化合物、特别是药物无风险地或者以低风险或高风险(优选无风险或高风险)诱导特定效应(优选副作用,优选地选自负面或希望的副作用,优选负面副作用)的概率的方法。在特定的优选实施方式中,在根据本发明的算法或模型中,所述分子集合由平衡比例的分子组成,每种分子注释有诱导所述特定效应(其为副作用,优选地选自负面或希望的副作用,优选负面副作用)的高风险和极低风险(优选无风险)评分。“平衡比例的分子”意指对于所述希望的负面副作用的经良好注释的分子的集合,所述经良好注释的分子已知对于诱导所述负面副作用无风险或低风险或高风险,并且呈现至少3种、优选至少4种或5种不同治疗类别,特别是选自以下的组:心血管、变态反应学、cns、特别是精神科、皮肤病学、内分泌学、胃肠病学、血液学、感染学、代谢、神经肌肉、肿瘤学、炎性和肥胖治疗类别。优选地,所述至少3、4、5、6、7或8种不同治疗类别的每一种中所包括的分子数目代表了集合的分子总数的至少10%。在更优选的实施方式中,代表希望的特定效应(优选副作用,优选地选自负面或希望的副作用,优选负面副作用)的类别的治疗类别包括集合的分子总数的多于20%、优选25%、30%或35%。在优选实施方式中,在根据本发明的算法中,在步骤c)中,所述分子集合同时地、优选针对集合的每种分子以不同浓度进行分析。在优选实施方式中,在根据本发明的算法中,步骤1)d)i)包括对于每种亚型或其组合计算以下的步骤:-对于所述特定效应(优选副作用,优选地选自负面或希望的副作用,优选负面副作用)为至少60%、优选70%且优选高于80%的灵敏度(se%)和至少60%、优选70%且优选高于80%的特异性(sp%)的最佳阈值;-阳性(ppv,%)和阴性(npv,%)预测值,以评估真实存在[真阳性/(真阳性+假阳性]和真实缺失[真阴性/(真阴性+假阴性)],所述方法允许确定所述亚型/或位点或其组合的选择的全局性能。在优选实施方式中,在根据本发明的算法或模型中,在步骤c)中,rna编辑谱通过包括以下的方法进行:-包括ngs文库制备的ngs法(下一代测序),优选使用两步pcr法来选择性地对靶标的受关注序列片段(包括编辑位点)进行测序;-对所得到的所有ngs文库测序;以及可选地-对所述测序数据进行生物信息学分析,所述生物信息学分析优选包括以下步骤:-序列的预比对处理和品质控制-相对于参比序列的比对;和-编辑水平调用(calling),以获得所述靶标的编辑谱。在优选实施方式中,在根据本发明的算法中,在步骤d)i)和d)ii)中,以及在步骤e)中,允许获得所述算法的所述统计方法通过包括以下的方法来进行:-mroc程序,特别是用于鉴定线性组合,其使auc(曲线下面积)roc最大化,且其中各组合的公式提供如下且可用作新的虚拟标志物z:z=a1.(亚型1)+a2.(亚型2)+…ai.(亚型i)+….an.(亚型n)其中,a1是计算系数且(亚型i)是亚型的靶标的各自rna编辑水平的相对比例;和/或-应用于单变量和多变量分析的logistic回归模型,其用于估计在不同的亚型/或编辑位点值的分子的相对风险;和/或-应用于评估亚型/或编辑位点组合的cart(分类和回归树)法;和/或-应用于评估亚型/或编辑位点组合的随机森林(rt)法,其特别是用于对编辑亚型/或位点的重要性评级并将最佳亚型/或编辑位点组合以将分子的“相对风险”归类,和/或可选地-应用于针对分子的“相对风险”评估亚型或编辑位点组合的多变量分析,其选自由以下组成的组:-支持向量机(svm)法;-人工神经网络(ann)法;-贝叶斯网络法;-wknn(加权k最近邻)法;-偏最小二乘判别分析(pls-da);和-线性与二次判别分析(lda/qda)。在优选实施方式中,在根据本发明的算法中,-所述至少一种靶标为5-ht2cr,且-所述负面副作用为精神科负面副作用,且-所述细胞系为人sh-sy5y成神经细胞瘤细胞系,且-所述阳性对照为干扰素α,且且其中:-能够区分测试药物是否以低风险或高风险诱导所述精神科负面副作用的位点组合至少包括至少2、3、4或5个单一位点的组合,所述单一位点选自由以下5-ht2cr位点组成的组:a、b、c、d和e,优选至少3、4或5个所述位点的组合,-或能够区分测试药物是否以低风险或高风险诱导所述精神科负面副作用的亚型组合至少包括至少2、3、4、5、6、7、8、9、10、11、12或13个单一亚型的组合,所述单一亚型选自由以下5-ht2cr亚型组成的组:a、b、ab、abc、ac、c、d、ad、ae、acd、aec、abcd和ne,优选至少5、6或7个所述亚型的组合,并且可选的是其中:允许获得所述算法或模型的所述统计方法通过包括以下的方法来进行:-mroc程序、随机森林法和/或cart算法。在第二方面,本发明涉及一种体外预测药物、化合物或分子在患者中诱导特定效应(优选副作用、更优选负面或希望的副作用)的概率或风险的方法,所述方法使用展示出rna的a至i编辑的靶标,其前体mrna是adar酶的底物,所述adar的作用导致产生不同的亚型或编辑位点,其中所述方法包括以下步骤:a)分析已用所述药物或化合物或分子处理过的样品中的靶标rna编辑谱,从而获得针对每种所述靶标的编辑亚型的所述靶标的rna编辑水平的比例,且其中所述靶标rna编辑谱以对于所述特定效应获得的权利要求1至15中任一项所述的算法或模型中的分子集合的分子所获得那样获得;b)利用权利要求1至15中任一项所述的针对所述靶标和所述特定效应获得的算法或模型来计算端值或应用对于所述药物或化合物获得的算法或模型;和c)根据步骤b)所获得结果确定所述药物或化合物是否有风险、特别是低风险或高风险在患者中诱导所述特定效应。在另一个实施方式中,根据本发明的预测药物、化合物或分子在患者中诱导特定效应的概率或风险的所述体外方法使用了至少2个、3个或4个展示出rna的a至i编辑的靶标的组合,其前体mrna是adar酶的底物,所述adar的作用导致产生不同的亚型或位点,其中所述方法包括以下步骤:a)分析已用所述药物或化合物或分子处理过的样品中的所述靶标组合的每个靶标rna编辑谱,从而获得针对每种所述靶标的每种编辑亚型或位点的rna编辑水平的比例,且其中所述靶标rna编辑谱的每一个以对于所述特定效应获得的权利要求1至15中任一项所述的算法或模型中的分子集合的分子所获得那样获得;b)利用权利要求1至15中任一项所述的针对所述靶标和所述特定效应获得的算法或模型来计算端值或应用对于所述药物或化合物获得的算法或模型;和c)根据步骤b)所获得结果确定所述药物或化合物是否有风险、特别是低风险或高风险在患者中诱导所述特定效应。在另一个优选实施方式中,至少2个、3个或4个展示出rna的a至i编辑的靶标(其前体mrna是展示出rna的a至i编辑的adar酶靶标的底物)的所述组合选自以下靶标组合:所述靶标组合选自由5-ht2cr、pde8a(磷酸二酯酶8a)、gria2(谷氨酸受体2)、gria3、gria4、grik1、grik2、grin2c、grm4、grm6、flnb(细丝蛋白b)、5-ht2a、gabra3(gabaα3)、flna、cyfip2组成的组。在第三方面,本发明涉及用于确定化合物、优选药物是否有风险、特别是低风险或无风险或高风险在患者中诱导所述特定效应(优选副作用,优选地选自负面或希望的副作用,优选负面副作用)的试剂盒,其包括:1)使用根据本发明的算法或者应用用于预测根据本发明的预测化合物、优选药物在患者中诱导所述特定效应(优选副作用,优选地选自负面或希望的副作用,优选负面副作用)的概率或风险的方法的说明书,从而获得端值,对该端值的分析确定了所述测试药物在患者中诱导所述负面副作用的风险,所述说明书包括可选的roc曲线或cart决策树;和2)用于确定编辑rna谱的试剂,所述编辑rna谱根据用于获得分子集合的每种分子的编辑rna谱的试剂需求而针对所述测试药物获得,所述分子集合用于确定1)中所述说明书的所述算法或所述模型。在优选实施方式中,在本发明的算法或模型中使用该方法时所述试剂包括对于用于ngs文库制备的2步pcr所必须的引物组。在更优选的实施方式中,所述试剂包括用于针对所述靶标中的至少一个或对于至少2、3或4个靶标的组合获得根据权利要求1至17所述的rna编辑谱的寡核苷酸序列。在更优选的实施方式中,所述试剂包括对于用于ngs文库制备的2步pcr所必须的一个引物组或引物组的组合,并且其中所述至少一个靶标或所述靶标组合选自如下的靶标:所述靶标选自由5-ht2cr、pde8a(磷酸二酯酶8a)、gria2(谷氨酸受体2)、gria3、gria4、grik1、grik2、grin2c、grm4、grm6、flnb(细丝蛋白b)、5-ht2a、gabra3(gabaα3)、flna、cyfip2组成的组。在另一个更优选的实施方式中,所述试剂包括选自由以下组成的组的一个引物组或引物组的组合:-对于pde8a靶标pde8a_左:5’-caacccacttatttctgcctag-3’(seqidno.1)pde8a_右:5’-ttctgaaaacaatgggcacc-3’(seqidno.2);-对于fnlb靶标flnb_左:5’-aaatgggtcgtgcggtgtat-3’(seqidno.3)flnb_右:5’-cctgctcgggtggtgttaat-3’(seqidno.4);-对于gria2靶标gria2_左:5’-ctctttagtggagccagagtct-3’(seqidno.5)gria2_右:5’-tcctcagcactttcgatggg-3’(seqidno.6);-对于grik2靶标grik2_左:5’-cctgaatcctctctcccctg-3’(seqidno.7)grik2_右:5’-ccaaatgcctcccactatcc-3’(seqidno.8);和-对于gabra3靶标gabra3_左:5’-ccaccttgagtatcagtgcc-3’(seqidno.9)gabra3_右:5’-cgatgttgaaggtagtgctgg-3’(seqidno.10)。选择了以下实施例和附图及下文图例来为本领域技术人员提供完整的描述以便能够实施和应用本发明。这些实例并非旨在限制发明人所认为的发明的范围,也非旨在表明仅进行了下文的实验。本发明的其它特征和优点将在对实施例和附图的描述的其余部分中出现,其图例在下文中给出。附图说明:图1:干扰素α-诱导的rna标记(剂量响应)(ifnα)sh-sy5y人成神经细胞瘤细胞系中的5-ht2crmrna编辑‘谱’。在用fnα处理48小时后的干扰素α(ifnα)的效应的剂量响应分析。5-ht2crmrna的相对比例通过基于ngs的测序进行分析。所述谱通过对经ifnα处理的细胞中测定的5-ht2crmrna编辑的相对比例扣除在经空载剂处理的对照细胞中的5-ht2crmrna编辑的相对比例而获得。图2a-2b:体外检测中所测试的所有260种化合物的治疗分类的饼图。中枢神经系统(cns)作用性化合物的进一步亚分类显示于图的b部分。图3:在所选分子的测试期间应用的实验设定和方法的示意性呈现。所有260种化合物以5份生物学独立复制品(replicate)进行测试。各个细胞培养板用10种分子、空载剂对照(dmso)以及100iu/ml干扰素α进行处理。测试5份独立生物学复制品产生了恰好1620个样品,其通过基于ngs的rna编辑定量方法以相同方式进行处理。图4a-4i:各个孔中的adar1amrna表达对用所述分子处理48小时的sh-sy5y细胞中的adar1a表达的定量pcr(qpcr)分析。在用所述分子、空载剂(dmso)或ifnα处理48小时后对每个样品中的adar1amrna表达水平进行了定量。显示了单个生物学复制品(n=1)。如同预期,用ifnα处理的每个板孔展示出增加的adar1a表达(a至j)。应注意,分子165也展示出在暴露于该分子后的adar1amrna表达水平的显著增加。图5a-5b:所有空载剂对照和ifnα处理的(100ui/ml)sh-sy5y细胞的原始数据(a)对所有150种空载剂对照(dmso)和ifn处理的孔的全局分析。表格显示出所有5-ht2crmrna编辑亚型的所有基本统计特征。在分析中汇集了在整个实验期间获得的空对照和ifnα处理的条件(n=150)以生成对5-ht2cr上的ifn-诱导的rna编辑变化的标准测定。显示受ifn处理最显著影响的5ht2cr编辑亚型的柱形图。对于所有5-ht2crmrna编辑亚型对经空载剂处理(dmso)和ifnα处理的孔给出了平均值、中位数、标准偏差和变异系数(cv以百分比表达)。图6:轮廓曲线-rna编辑曲线ifn100通过对经ifnα处理的细胞中测定的5-ht2crmrna编辑的相对比例扣除在经空载剂处理的对照细胞中的5-ht2crmrna编辑的相对比例而获得的5ht2crmrna编辑谱。给出了平均值和中位数值,误差条表示平均值的标准误差(sem,n=150)。图7a-7b:在用各分子处理48小时后的获得的5ht2crmrna编辑谱的说明性实例。在各图中实例以4种“有风险”化合物(阿立哌唑、舍曲林、异维甲酸和泰伦那班)(a)和4种“低风险”分子(锂、氯胺酮、奥丹西隆和利巴韦林)(b)的组给出。各图中给出了ifn参照(以黑色示)。给出了平均值,误差条表示平均值的标准误差(sem,n=5)。图8:最具代表性的5ht2crmrna编辑亚型对于区分低风险分子与高风险分子的诊断潜力的说明性实例。箱线图是通过5-数摘要(最小观察值、下四分位数(q1)、中位数(q2)、上四分位数(q3)和最大观察值)以图示方式描述数字数据组的便利方式。箱线图可用于在不对基础统计分布进行任何假设的情况下展示群体间差异。对于p值使用wilcoxon秩和检验。符号*表示p值≤0.05,**表示p值≤0.01而***表示p值≤0.001。图9:使用从关于分子数据集(n=143,低风险相对于高风险分子)的图15的13种亚型的组中选出的2种亚型的组合的接收-运行-特征(roc)曲线的说明性实例。决策规则:z=0.121xacd-0.142xne图10:使用从关于分子数据集(n=143,低风险相对于高风险分子)的图15的13种亚型的组中选出的3种亚型的组合的接收-运行-特征(roc)曲线的说明性实例。决策规则:z=-0.1449xc+0.569xae-0.1548xne图11:使用从关于分子数据集(n=143,低风险相对于高风险分子)的图15的13种亚型的组中选出的4种亚型的组合的接收-运行-特征(roc)曲线的说明性实例。决策规则:z=0.0235xab+0.1567xacd+0.3880xaec-0.1355xne图12:使用从关于分子数据集(n=143,低风险相对于高风险分子)的图15的13种亚型的组中选出的5种亚型的组合的接收-运行-特征(roc)曲线的说明性实例。决策规则:z=0.016xab-0.0563xabc+0.183xacd+0.386xaec-0.1428xne图13:使用从关于分子数据集(n=143,低风险相对于高风险分子)的图15的13种亚型的组中选出的6种亚型的组合的接收-运行-特征(roc)曲线的说明性实例。决策规则:z=0.0157xab-0.0557xabc+0.0187xd+0.1817xacd+0.3883xaec-0.1426xne图14:使用从关于分子数据集(n=143,低风险相对于高风险分子)的图15的13种亚型的组中选出的7种亚型的组合的接收-运行-特征(roc)曲线的说明性实例。决策规则:z=-0.0505xb+0.0224xab+0.001xd+0.163xacd+0.389xaec-0.1402xabcd-0.1385xne图15:使用从关于分子数据集(n=143,低风险相对于高风险分子)的13种亚型的组合的接收-运行-特征(roc)曲线的说明性实例。决策规则:z=0.2035xa+0.1283xb+0.1979xab+0.1147xabc+0.1860xac+0.04331xc+0.1884xd+0.1259xad+0.7739xae+0.4295xacd+0.4775xaec-0.0415xabcd+0.0245xne图16a-16c:使用从关于分子数据集(n=143,低风险相对于高风险分子)的图14的7种亚型的组合的随机森林(rf)算法的接收-运行-特征(roc)曲线的说明性实例。所有数据集的roc曲线以黑线显示而测试数据集的roc曲线以虚线显示(a)。rf模型中的亚型的重要性(权重)(b)(c)。图17a-17c:采用rf法的诊断性能的实例。使用从关于分子数据集(n=143,低风险相对于高风险分子)的图15的13种亚型的组合的随机森林(rf)算法的接收-运行-特征(roc)曲线的说明性实例。所有数据集的roc曲线以黑线显示而测试数据集的roc曲线以虚线显示(a)。rf模型中的亚型的重要性(权重)(b)(c)。图18a-18c:通过额外靶标:gria2(a)、flnb(b)和pde8a(c)测定的rna编辑活性的定量。在所有情形中,ifn处理诱导了经编辑的亚型的相对比例的增加,如未经编辑(ne)的mrna的减少所示。图19a-19b:ln18(a)和ln229(b)成神经细胞瘤细胞系(htr2c)在ln18细胞(a)和ln229细胞(b)中通过对经ifnα处理的细胞中测定的5-ht2crmrna编辑的相对比例扣除在经空载剂处理的对照细胞中的5-ht2crmrna编辑的相对比例而获得的5ht2crmrna编辑谱。给出了5ht2crmrna的平均mrna编辑谱。图20:cart算法预测使用关于分子数据集(n=143,低风险相对于高风险分子)的6种亚型的cart算法的代表性决策树和诊断性能的说明性实例。图21a-21d:对于在患者中具有诱导特定效应的低风险或无风险的两种化合物获得的rna编辑谱。作为实例提供了与空载剂对照处理的细胞相比采用利多卡因(a)和奥丹西隆(b)获得的rna编辑谱。对于有高风险在患者中诱导特定效应的两种化合物(如利血平(c)和氟西汀(d))获得的rna编辑谱。图22a-22c:在htr2c上通过阿立哌唑(a)、干扰素(ifn)(b)和利血平(c)观察到的的rna编辑变化的时间进程分析。图23a-23c:在用三种不同化合物:氯氮平(a)、舍曲林(b)和氯胺酮(c)对sh-sy5y细胞处理后的rna编辑谱的剂量依赖性改变。实施例1:材料和方法药物诱导的精神科负面副作用的数据库的创建含有以精确10mm溶解于dmso中的1280种小分子的集合的化学文库购自prestwickchemicals。文库中所含的所有小分子是100%经许可药物(fda、ema和其它机构),表现了最大可能程度的药物相似性,且已针对其高度化学和药理学多样性以及在人中的已知生物利用度进行了筛选。在购买该化学文库(prestwickchemicals)时,提供了包含关于每一种分子的靶标、治疗类别/效果、专利和admet的详细信息的高度注释的数据库。发明人通过查询定期更新安全信息和案例报告的数据库(例如,fdamedwatch、emea等)检索了在开处方给人后发出自杀和抑郁相关的负面副作用的报告。其后,发明人汇总了查询结果并对化学文库中所含的每种药物分配了风险评分。考虑到各种参数,例如报告自杀和/或抑郁相关的负面副作用的案例数、药物的开处方程度、列于根据who的重要药物列表等等,建立了评分体系以便对潜在诱导负面精神科副作用(抑郁和/或自杀相关的负面副作用)的药物的风险进行定量。发明人获得了关于有风险诱导负面精神科副作用的具体信息的综合数据库。细胞培养使用了sh-sy5y人成神经细胞瘤细胞系,因其内源性表达5-ht2crmrna并且展示出与在人皮质中观察到的相似的adar1酶表达稳态(cavarec等,2013,weissmann等,2016translationalpsychiatry,patenttoxadar)。sh-sy5y人成神经细胞瘤细胞系购自sigmaaldrich。细胞在37℃在5%co2的湿润气氛中于标准条件下常规培养。由于血清中常常存在5-ht2crmrna表达被5-羟色胺去敏化和下调,因此相对于未透析的胎牛血清,优选经透析的胎牛血清(fbssciencetech参考数字fb-1280d/500)(saucier等,1998)。在实验进程期间,细胞在p8至p22之间的传代数进行培养。在将细胞接种至12孔细胞培养板之前,通过将经胰蛋白酶化的细胞悬浮物独立地加载至kovaslide(kovainternational)腔室、由具有血细胞计技术网格的光学透明塑料制成的一次性显微镜载玻片,进行对细胞数目的估算。两个腔室均由两名实验室技术人员进行计数,并将4次独立计数结果进一步用于计算细胞数和12孔细胞培养板的平板接种。药理学处理和细胞裂解在收到后,将整个prestwick化学文库转移至个体试管、编码、等分并储存于-80℃以待进一步使用。发明人由其自主生成的药物诱导的精神科负面副作用数据库选择了260种分子,其由平衡比例的注释为具有高风险和极低风险评分的药物组成。将药物编码并仔细地在整个实验设定下对分子进行随机处理。所有260种分子在每个实验中与阴性对照(空载剂dmso)和阳性对照(干扰素α)同时进行分析。在每个12孔细胞培养板上,添加阴性对照和阳性对照,剩余10个空缺位置留给测试分子。继而,每一个复制品由12孔的27个培养板组成(ref)。以完全相同的方式重复进行5次试验,如此对每个受测分子产生5个独立的生物学复制品(n=5)。在实验进程中,产生了总共1620个样品,即27(孔板数)x12(每板的板孔数)x5(复制品数)。初步实验使得可以鉴定出在10μm对sh-sy5y细胞致命的7种分子。对于这些分子将浓度进行调整并降低直到可以检测到减小的毒性。在实验前,成列地制备并安置分子和对照的所有稀释液。在处理前通过显微镜控制所有324个孔(27x12个孔)的细胞密度、形态、生活力和污染情况。另外,使用canoneos700数字相机对每个板孔拍照。在用分子处理细胞正好48小时后,使用该数字相机以所限定的参数对每个板孔拍照。在仔细地除去生长培养基后,添加350μl含1%β-巯基乙醇的rlt裂解缓冲液(qiagen)以使细胞完全地化学裂解。将12孔板堆叠并在rna提取前储存于冰箱中。总rna提取、品质控制和逆转录按照制造商的指南(qiagen)进行总rna提取。rneasymini试剂盒提供了利用硅膜rneasy旋转柱对来自细胞的高品质rna快速纯化。使用全自动化样品制备qiacube提取所有细胞裂解物。采用适当规程,使用标准程序以每轮12个样品(一个完整12孔板)分批进行提取。在样品制备和rna提取期间,采取标准预防措施以避免rna被rna酶降解。所有提取的rna样品通过labchipgx(perkinelmer)分析以对总rna进行定性和定量。还进行了通过qubit的基于荧光的定量,以验证labchipgx数据。对于每个个体样品确定rna品质评分(rqs评分)(1620个样品的平均rqs评分=9.6/10)。其后,对样品归一化并使用takara试剂盒(primescriptrttakararef#rr037a)从1μgrna材料起始以20μl最终反应体积进行纯化rna的逆转录。在peqstar96x热循环仪上于42℃进行15分钟cdna合成并将反应混合物保持在4℃待进一步使用。通过定量pcr(qpcr)的相对mrna表达在cdna合成后将样品储存于4℃,之后在lc480系统(roche)上通过qpcr对adar1amrna表达进行分析。使用标准曲线法对qpcr数据定量。已知通过干扰素α处理(ifnα)会诱导adar1a的mrna表达。如所预期的那样,已采用ifnα处理48小时的所有样品都显示出基因表达的倍数诱导为6至7的adar1a表达的增加。另外,利血平处理也一致地增加adar1amrna水平。ngs文库制备对于ngs文库制备采用了2步pcr法以便对将要进行rna编辑的5-ht2cr(如前文所述且由发明人等所证实)的外显子v进行选择性测序。使用了经验证的pcr引物来通过pcr扩增所关注的区域。对于pcr扩增,根据制造商指南(ref#m0494s)使用q5hotstarthighfidelity酶(newenglandbiolabs)。使用优化的pcr规程在peqstar96x热循环仪上进行pcr反应。在pcr之后,通过labchipgx(perkinelmer)对所有样品进行分析,并评估pcr产物的数量和品质。利用基于荧光的qubit方法确定扩增子的纯度并进行定量。在品质控制后,使用磁珠(来自mokascience的highpreppcrmagbio系统)对96个pcr反应物(微孔板)纯化。在纯化后,使用qubit系统将dna定量并计算纯化产率。其后,使用q5hotstarthighfidelitypcr酶(newenglandbiolabs)和illumina96索引试剂盒(nexteraxt索引试剂盒;illumina)通过pcr扩增对样品进行单独索引。在pcr后,将样品汇集至文库中并使用magbiopcr清理系统进行纯化。根据illumina指南使文库变性并加载至测序盒上以仅在miseq平台上对fastq测序。在每个文库中包括有含有确定量的5ht2cr亚型的质粒的库,以控制测序品质和每轮测序的误差。另外,在文库中引入入了标准rna库以确定在实验进程期间的不同测序流动池之间的可变性。为了对所有1620个样品测序,要求18个18miseq试剂盒v3(illumina)。所有ngs文库在14pm测序,并以10%phix(phixcontrolv3)加标以引入文库多样性。实施例2:测序数据的生物信息学分析1.fastq序列的预比对处理和品质控制测序数据从miseq测序仪(illumina)以fastq文件下载。为评估测序品质,使用fastqc软件0.11.5版执行了每个原始fastq文件的初始品质。进行了预处理步骤,包括除去接头序列(adaptersequence)并将序列根据其尺寸和品质分数进行过滤(除去所有短读出值(<50nts)和平均qc<30的读出值)。其后,为利于并改善序列比对的品质,使用了针对illuminangs数据的灵活性读出值修剪工具(trimmomatic程序0.35版)。在执行预处理步骤后,对每个经清理的fastq文件进行了额外品质控制然后进行进一步序列处理。2.针对参比序列的比对使用bowtie22.2.5版以端对端灵敏模式进行了经处理的读出值的比对。对于人基因组序列的最新注释(ucschg38)进行比对,并将具有多比对区的读出值、具有较差比对品质的读出系列(q<40)或含有插入/缺失的读出系列(indel)从进一步分析中剔除。文件比对的过滤采用samtools软件1.2版进行,其提供了以sam格式操作比对的各种功用,包括以按位置(per-position)格式分选、合并、索引和生成比对。3.标记水平调用接下来,使用samtoolsmpileup将从多个样品获得的比对结果数据同时堆集。运行自制脚本来对每个基因组位置处的不同atgc核苷酸的数目进行计数(“基础计数”)。因此,对于每个基因组位置,自制脚本计算出具有‘g’的读出值的百分比[‘g’读出值的数目/(‘g’读出值的数目+‘a’读出值的数目)*100]。具有‘g’读出值的百分比>0.5的基因组位置‘a’参考值被脚本自动检测到并被视为“a至i编辑位点”。最后一阶段是计算前述“a至i编辑位点”的所有可能的组合的百分比,以获得靶标的编辑谱。4.靶标的基线与分子编辑谱之间的比较发明人分析了一大组分子(n=260)的5ht2crrna编辑谱。为将分子共同比较,发明人在第一步确定了与经空载剂处理(dmso)的对照细胞相比在sh-sy5y人成神经细胞瘤细胞系中对于每种亚型/或位点而言的靶标的rna编辑的基础水平。为此,从大于150次空载剂独立实验(复制品)计算了rna编辑水平的平均值(实例已给出)。另外,自制脚本自动计算了每种分子复制品(n=5)与对照参比物(ctrl)的偏差。最后,对于每种分子和每个编辑亚型/或位点,获得了靶标的rna编辑水平的平均/中位数相对比例。实施例3:统计分析所有统计和图用“r/bioconductor”统计开源软件计算(19,20)。rna编辑值通常以平均值±平均值的标准误差(sem)来表示。采用非参数wilcoxon秩和检验和welcht检验来进行差异分析。采用多种检验方法时,重要的是调节每个编辑亚型的p值(例如:来自5个编辑位点(a、b、c、e、d)的包括5ht2cr的未经编辑亚型(ne)在内的32种rna编辑亚型)以控制错误发现率(fdr)。采用“多重检验包”对所有统计检验应用benjamini&hochberg(bh)程序,并认为低于0.05的经调节p值是统计显著的。编辑水平的相对比例正常分布,因此未应用归一化。对于每种显著亚型,所有数据分布以中位数和条形图(barplot)或箱线图(boxplot)示出。对于每种分子还示出了来自显著亚型且代表在sh-sy5y人成神经细胞瘤细胞系中的5ht2cr的rna编辑水平的编辑谱曲线。应用pearson检验关联来鉴定所有分子组的亚型相关性。5ht2cr编辑亚型的诊断性能可以由以下表征:代表其检测“高风险分子”组的能力的灵敏度,和代表其检测“无风险或低风险分子”组的特异性。对诊断检验的评估结果可以总结在比较这两个明确限定组的2×2相依表中。通过固定截止值(cut-off),可以根据检验结果将两组分为归类为阳性或阴性的分类。在给出具体亚型时,可以在“高风险”组中鉴定出具有阳性检验结果的a分子(“真阳性”:tp)和在“低风险”组中具有阳性检验结果的b分子(“真阴性”:tn)。以同样方式,观察到了在“高风险”组中具有阴性检验结果的c分子(“假阳性”:fp)和在“低风险”组中具有阴性检验结果的d分子(“假阴性”:fn)。灵敏度定义为tp/(tp+fn);其在本文称作“真阳性率”。特异性定义为tn/(tn+fp);其在本文称作“真阴性率”。使用接收运行特征(roc)分析评估了每种5ht2cr编辑亚型的精度及其区分能力。roc曲线是对于各种值的检验的灵敏度(se)和特异性(sp)的相互关系的图形可视化。另外,将所有5ht2cr编辑亚型各自组合以便使用如mroc程序[comput.methodsprogramsbiomed.2001;66:199-207]、logistic回归(22)以及两种监督学习算法cart(23)和随机森林(24)等几种方法来评估灵敏度和特异性的潜在增加。mroc是鉴定线性组合的专用程序(25,26),其使auc(曲线下面积)roc最大化(27)。如下提供了用于对应组合的方程,且其可以用作新虚拟标志物z:z=a×亚型1+b×亚型2+c×亚型3,其中,a、b、c是计算系数,而亚型1、2、3是亚型的靶标的个体rna编辑水平的相对比例。可将2、3或4个靶标的组合进行彼此组合以使用诸如mroc程序或logistic回归等多变量法来评估灵敏度和特异性的潜在增加。可如下计算各组合的方程,且其可用作新的虚拟标志物zn:zn=n1×靶标1+n2×靶标2+n3×靶标3,其中,n1,n2,n3…是计算系数,而靶标1、2、3是例如与靶标水平相关的值。logistic回归模型也被用于单变量或多变量分析,以评估在不同亚型或位点值的分子的相对风险。发明人将亚型作为连续变量(数据未示出)和分类变量(使用三分变量值(tertile)为截止点)进行分析。在后一种情形中,计算让步比(or)及其95%置信区间。此外对连续变量应用了logistic回归的惩罚版本(lasso、ridge或elastic-net法)。对于这些方法,采用软件包:r软件3.2.3的glmnet2.0-3版。cart(分类和回归树)法也被应用来评估亚型组合。该决策树法允许生成一组分类规则,其以使用者易于理解的层次图示出。在每个树节点处做出决定。传统上,左侧分支对应于对所关注问题的阳性响应,而右侧分支对应于对所关注问题的阴性响应。然后可将分类程序翻译为一组‘if-then’规则(例如参见图20)。随机森林(rf)法如前应用于评估亚型组合。该方法将breiman的“bagging”理念与特征的随机选择结合,从而构建具有受控方差的决策树集合。因此,可以使用随机森林来对编辑亚型的重要性评级,并将最佳亚型组合以对分子的“相对风险”分类(参见图16和17)。cart和随机森林是监督学习方法。这些方法要求使用用于构建模型的训练集和检验集来对其验证。因此,发明人将其数据集共享:2/3的数据集用于学习期,而1/3用于验证期。这种共享经随机化,且遵循每个样品中的各种规则的初始比例。为了估计这些分类器的误差预测,使用10倍交叉验证法,重复10次以避免过度拟合问题。对于这些方法,采用r软件3.2.3版的“rpart软件包4.1-10”和“randomforest软件包4.6-12”。对于分子的“相对风险”,可以采用另一多变量分析来评估5ht2cr编辑亚型组合,如:-支持向量机(svm)法(28);-人工神经网络(ann)法(29);-贝叶斯网络法(30);-wknn(加权k最近邻)法(31);-偏最小二乘判别分析(pls-da)(32);-线性与二次判别分析(lda/qda)(33);-及其它。实施例4:结果sh-sy5y细胞系的验证在实验前,用递增剂量的干扰素处理人成神经细胞瘤细胞系(sh-sy5y),并使用基于ngs的方法测定5ht2cr的rna编辑。如所预期的那样,5ht2cr亚型的相对比例发生变化且特别是能够剂量依赖性地增加(图1),这证实了此前所述的在该特定培养细胞系中的ifn诱导响应。ifn谱与此前采用截然不同的分析方法所获得的数据紧密匹配(34,35)。实验程序一旦细胞系显示出稳定生长特性并因此对于ifn处理产生相应的响应后,准备筛选制备260种分子。基于内部定义标准,对化学文库中的1280种分子的每一种分配风险评分。出于实践原因,选择了260种分子以在专有体外检测中进行进一步测试。在分子选择程序期间,谨慎地涵盖化学文库中所含的图2中所认定的所有主要治疗类别的一部分(优选至少3、4、5、6或7类)(图2)。在260种分子中,112种是作为抗惊厥剂和抗抑郁剂等其用于中枢神经系统病症的处方药(图2b)。在处理前将所有分子转移并等分在适当的试管中。选择用于260种分子的筛选的实验设定由用10种分子、空载剂对照(dmso)和100iu/ml干扰素α(其进而为每个细胞培养板生成阳性和阴性对照)分别处理的26个孔板(12孔板)组成。使用另外的细胞培养板来添加额外的对照孔。每个分子以3周间隔以5个生物学复制品进行测试(图3)。处理恰好48小时后,将细胞在适当的裂解缓冲液中裂解并在-20℃储存待进一步处理。所有rna提取采用qiacube自动化提取进行且对板单独处理(分批,每次提取12个样品)。相对adar1amrna表达在rna提取后,合成cdna并在384微孔板中于lc480lightcycler(roche)上评估adar1a表达。以这种方式,可以在一次qpcr运行中对相同批次的所有样品进行分析。在每个12孔板上观察到对于所有经ifn处理细胞而言干扰素依赖性的adar1a诱导,从而反映出响应的稳健性。令人感兴趣的是,165号分子也诱导adar1amrna表达(图4a-4i,板17)。该响应可以在所有生物学复制品(n=5)中看到。如此前在sh-sy5y细胞中所观察的,在相对于空载剂对照归一化时ifn以6.6的倍数诱导()来诱导adar1a表达。9.31%的变异系数清楚地说明了该生物学现象的再现性。表1:在sh-sy5y细胞中ifn处理后的adar1amrna表达的基本统计特征。平均倍数诱导(与dmso处理的对照细胞相比)标准偏差、中位数和cv(以百分比表达)。平均值(倍数诱导)6,61标准偏差0,62中位数6,62cv(%)9,31a)5ht2cr编辑亚型的单变量分析sh-sy5y细胞上的ifnrna编辑亚型与对照的比较在cdna合成步骤后,应用靶向5ht2cr的外显子v的2步pcr法建立ngs文库并对所有样品中的每一单独的5ht2crmrna的相对比例进行精确定量。所有空载剂对照和经ifn处理孔(n=150)的平均值显示在图5a中并以柱形图示出。可以观察到空载剂对照和ifn处理条件之间的亚型相对比例的明显差异(图5b)。这些数据以rna编辑谱表达,从而生成与此前所述的谱(参见图1和cavarec等)非常紧密匹配的前述rna编辑谱(图7a-7b)。举例而言,当在sh-sy5y细胞系上比较在ifn(n=150)与空载剂对照(空载剂,n=150)存在时的5-ht2crrna编辑亚型的水平,5-ht2cr的ac、abc、ab、a、ae、ace、d、abcd、abe、c、b、bc和abcerna编辑水平得到显著改变(图5a-5b和图6)。对于ifn分子与空载剂对照(basal0)的比较而言,5-ht2cr的未经编辑亚型(ne)的水平最为显著。而且,发明人观察到ac、abc、ab、a、ae、aec、abcd、abe、c、b、bc和abec的5-ht2crrna编辑水平的增加以及5-ht2crrna编辑的d和未经编辑(ne)亚型的水平的减少。这些结果表明,在ifn存在下在sh-sy5y细胞中,5-ht2cr的rna编辑活性全局增加。表2:在比较ifn分子(n=150)与对照(n=150)时的5-ht2crrna编辑水平的差异分析对照(n=150)sh-sy5y细胞上的高风险分子与低风险分子的rna编辑亚型水平的比较例如,在比较具有低风险的分子(n=82)和具有高风险的分子(n=61)时,5-ht2cr亚型的单编辑或未经编辑(ne)水平能被显著改变(图7a-7b)。基于对5-ht2cr亚型的rna编辑水平的接收-运行-特征(roc)分析,各个亚型的曲线下面积(auc)允许区分具有低风险或高风险的分子(表3)。表3:在比较低风险分子(n=82)和高风险分子(n=61)时单编辑亚型的区分性能每种亚型的精度及其区分能力利用接收运行特征(roc)分析进行评估。roc曲线是对各种值的检测的灵敏度(se)和特异性(sp)之间的相互关系的图形可视化。auc是指带有其置信区间(ci)的曲线下面积。roc曲线基于通过计算针对单标志物的灵敏度(se%)和特异性(sp%)的最佳阈值来预测分子相对风险的模型。计算针对单个rna编辑亚型的阳性(ppv,%)和阴性(npv,%)预测值来评估高风险分子在“自杀副作用组”中的真实存在[真阳性/(真阳性+假阳性]和真实缺失[真阴性/(真阴性+假阴性)]。b)5-ht2cr编辑亚型的多变量分析在比较低风险与高风险分子时采用mroc(多重接收-运行-特征)法的多重标志物分析显著改善了auc。亚型组合结合了选自以下组合的13种亚型的组的2种、3种、4种、5种、6种、7种或13中亚型:a+b+ab+abc+ac+c+d+ad+ae+acd+aec+abcd+ne,与cavarec等(2013)所得到的相比,通过本发明的的方法获得的组合具有如通过更高的灵敏度和特异性所报道的在高风险分子中更高的自杀副作用风险的预测值。组合了如本发明的组合中所鉴定的2种、3种、4种、5种、6种、7种、8种、9种、0种、11种、12种和13种亚型的统计分析生产了一系列决策规则;对于每种组合计算新的虚拟标志物(z),如图9至15以及以下相应表4至9所示(低风险分子相对高风险分子)。多亚型组的精度及其区分能力利用接收运行特征(roc)分析进行评估。roc曲线是对各种值的检测的灵敏度(se)和特异性(sp)之间的相互关系的图形可视化。auc是指带有其置信区间(ci)的曲线下面积。roc曲线基于通过计算针对多亚型组的灵敏度(se%)和特异性(sp%)的最佳阈值来预测毒性高风险的模型。计算针对组合标志物的阳性(ppv,%)和阴性(npv,%)预测值来评估高风险分子的自杀/抑郁诱导性负面副作用的真实存在[真阳性/(真阳性+假阳性]和真实缺失[真阴性/(真阴性+假阴性)]。表4:利用采用2种亚型的多变量分析的5-ht2cr编辑亚型性能(低风险分子相对于高风险分子)c2(2种亚型的组合):前10rd组合c2aucrocci95%阈值sp(%)se(%)vpp(%)vpn(%)精度1acd+ne0,845[0,776;0,914]0,125287,875,482,182,882,52aec+ne0,838[0,768;0,908]0,092182,978,777,484,081,13a+ne0,839[0,771;0,908]0,082278,182,073,585,379,74abc+ne0,84[0,771;0,909]0,170390,265,683,377,979,75b+ne0,842[0,773;0,911]0,059176,882,072,585,179,06ac+ne0,841[0,771;0,91]0,054276,882,072,585,179,07c+ne0,841[0,774;0,909]0,051776,878,771,682,977,68ae+ne0,839[0,771;0,907]0,023376,878,771,682,977,69ab+ac0,739[0,653;0,824]-0,025370,770,564,276,370,610a+acd0,715[0,625;0,804]0,136972,067,264,174,769,9决策规则:rd1:z=0,121xacd-0,142xne表5:利用采用3种亚型的多变量分析的5-ht2cr编辑亚型性能(低风险分子相对于高风险分子)c3:前25决策规则:rd1:z=-0,1449xc+0,569xae-0,1548xne表6:利用采用4种亚型的多变量分析的5-ht2cr编辑亚型性能(低风险分子相对于高风险分子)c4:前25决策规则:rd1:z=0,0235xab+0,1567xacd+0,3880xaec-0,1355xne表7:利用采用5种亚型的多变量分析的5-ht2cr编辑亚型性能(低风险分子相对于高风险分子)c5:前25决策规则:rd1:z=0,016xab-0,0563xabc+0,183xacd+0,386xaec-0,1428xne表8:利用采用6种亚型的多变量分析的5-ht2cr编辑亚型性能(低风险分子相对于高风险分子)c6:前25决策规则:rd1:z=0,0157xab-0,0557xabc+0,0187xd+0,1817xacd+0,3883xaec-0,1426xne表9:利用采用7种亚型的多变量分析的5-ht2cr编辑亚型性能(低风险分子相对于高风险分子)c7:前25决策规则:rd1:z=-0,0505xb+0,0224xab+0,001xd+0,163xacd+0,389xaec-0,1402xabcd-0,1385xne表10:利用采用13种亚型的多变量分析的5-ht2cr编辑亚型性能(低风险分子相对于高风险分子)c13z=0,2035xa+0,1283xb+0,1979xab+0,1147xabc+0,1860xac+0,04331xc+0,1884xd+0,1259xad+0,7739xae+0,4295xacd+0,4775xaec-0,0415xabcd+0,0245xnec)决策树法:多变量分析代表“分类和回归树”的cart算法是一种决策树方法。这些树将有助于建立分类规则组,其以使用者易于理解的等级图表示。所述树由内部节点(决策节点)、边界和末端叶组成。这些节点通过检验来标记并可以响应于与从该节点起的边界的标记匹配的检验。如果决策树是二元的,通过转换,传统上左边界对应于对检验的阳性响应而右边界对应于阴性响应。所获得的分类程序就决策规则而言可以得到即刻翻译。决策树是流行且有效的监督分类方法。该方法要求使用训练组来构建模型并使用检验组对其验证。因此,为了建立数据集,发明人将其“非模糊”分子(n=143)的列表共享:数据集的90%用于学习期(n=93种药物),而10%用于检验期(50种药物)。这种共享已经随机化并遵循每种分子的各种规则的初始比例。而且,例如发明人已将“ifn谱”中的6种rna编辑亚型与cart方法进行组合来构建决策模型(图20)。表11:在分子数据集上使用cart算法与选自亚型的5种亚型x1、x2、x3、x4和x5的组合或c13组合的5-ht2cr编辑亚型诊断性能(低风险分子相对于高风险分子)在数据集上使用5rna编辑亚型的cart模型的诊断性能对于区分低风险分子与高风险分子也可能极具吸引力。d)随机森林法:多变量分析随机森林是流行且有效的监督分类方法。该方法要求使用训练组来构建模型并使用检验组对其验证。因此,为了建立数据集,发明人将其“非模糊”分子(n=143)的列表共享:数据集的65%用于学习期(n=93种药物),而35%用于检验期(50种药物)。这种共享已经随机化并遵循每种分子的各种规则的初始比例。而且,发明人通过ifn权衡了学习数据集以改善具有“ifn谱”的药物和具有“basal0谱”的药物的区分能力。因此,发明人添加了12种ifn分子和从学习组(n=113)随机取出的8种对照。例如,发明人,在“ifn谱”中组合了7种和13种代表性rna编辑亚型(参见表9c7和表10c13的rd1),使用随机森林(rf)算法来构建决策模型(rf模型参数:mtrystart=1,stepfactor=2,ntree=500,improve=0.01;rf模型的袋外(oob)估计值=0.21)(图16a-c和图17a-c))。表12:在分子数据集上采用7种亚型的随机森林算法的相依表(低风险分子相对于高风险分子)学习检验所有数据特异性1007692灵敏度1009096精度1008494表13:在分子数据集上采用13种亚型的随机森林算法的5-ht2cr编辑亚型诊断性能(低风险分子相对于高风险分子)学习检验所有数据特异性1009096灵敏度1008695精度1008896利用5-ht2cr的7种或13种rna编辑亚型的诊断性能对于区分低风险分子与高风险分子极具吸引力,且具有优于90%(对于c7)和优于95%(对于c13)的灵敏度、特异性和精度,其显著优于cavarec等(2013)所公开的那些。实施例5:靶标多样化为了进一步补充sh-sy5y细胞中的5ht2crmrna编辑,发明人分析了额外的adar底物(gria2、flnb、pde8a、grik2和gabra3)。令人感兴趣的是,ifn处理对于所研究的所有三种靶标都改变了rna编辑亚型的相对比例(图11)。因此可预见添加额外生物标志物会进一步增加检验的诊断性能。实施例6:通过对各种靶标的基于ngs的分析获得的化合物特异性rna编辑谱已通过对gabra3、gria2、grik2和htr2c靶标的基于ngs的分析获得了化合物特异性rna编辑谱(参见图21a-21b)。在图21a和21b中,柱形图展示了与经空载剂对照处理的细胞相比,在用所指定化合物处理的人sh-sy5y成神经细胞瘤细胞系中的各个特定位点定量的rna编辑水平的相对比例。正值(%)表示与经空载剂处理的细胞相比由化合物诱导的特定位点处的rna编辑的增加。相反,负值(%)表示与经空载剂处理的细胞相比由化合物诱导的特定位点处的rna编辑的减少。已获得了对在患者中诱导特定效应具有低风险或无风险的两种化合物的rna编辑谱(参见图21a、21b)。作为实例,提供了与经空载剂对照处理的细胞相比,用利多卡因(a)和奥丹西隆(b)获得的rna编辑谱。对于在患者中诱导特定效应具有高风险的两种化合物如利血平(参见图21c)和氟西汀(参见图21d)已获得了rna编辑谱。实施例7:rna编辑的时间进程分析在htr2c观察到了通过阿立哌唑、干扰素(ifn)和利血平的rna编辑变化的时间进程分析(参见图22a-22c)。采用所有三种化合物对sh-sy5y细胞的处理都导致rna编辑谱的时间依赖性改变。这由显示出随时间减少的未经编辑的htr2c的对应相对比例所明确说明。令人感兴趣的是,通过所述处理诱导的变化的特异性由在阿立哌唑(参见图22a)和干扰素(参见图22b)或利血平(参见图22c)之间获得的差异谱说明。应用最优选的算法来确定在每个研究的时间点的每种化合物的风险评分(prob(算法))。虽然干扰素和利血平风险评分在所有时间点都高,但是阿立哌唑处理经鉴定在从24小时开始以后具有正面的风险(参见下表14)。表14:阿立哌唑、干扰素和利血平处理后在各时间点的风险评分水平分子prob(算法)预测阿立哌唑12h0.528nd阿立哌唑24h0.632pos阿立哌唑48h0.760posifn100ui12h0.720posifn100ui24h0.968posifn100ui48h0.970pos利血平12h0.878pos利血平24h0.986pos利血平48h0.976pos实施例8:在用不同化合物处理sh-sy5y细胞后的rna编辑谱的剂量依赖性改变在用3种不同化合物氯氮平、舍曲林和氯胺酮处理sh-sy5y细胞后已获得了rna编辑谱的剂量依赖性改变(参见图23a-23c)。rna编辑谱表示了与经空载剂处理的sh-sy5y细胞相比的htr2crna编辑的相对比例。参考文献1.(who)who.preventingsuicide:aglobalimperative.2014.2.labonteb,tureckig.theepigeneticsofsuicide:explainingthebiologicaleffectsofearlylifeenvironmentaladversity.archivesofsuicideresearch:officialjournaloftheinternationalacademyforsuicideresearch.2010;14(4):291-310.pubmedpmid:21082447.3.gurevichi,englandermt,adlersbergm,siegalnb,schmaussc.modulationofserotonin2creceptoreditingbysustainedchangesinserotonergicneurotransmission.thejournalofneuroscience:theofficialjournalofthesocietyforneuroscience.2002dec15;22(24):10529-32.pubmedpmid:12486144.4.sodhims,burnetpw,makoffaj,kerwinrw,harrisonpj.rnaeditingofthe5-ht(2c)receptorisreducedinschizophrenia.molecularpsychiatry.2001jul;6(4):373-9.pubmedpmid:11443520.5.alons,garrettsc,levanoney,olsons,graveleybr,rosenthaljj等,themajorityoftranscriptsinthesquidnervoussystemareextensivelyrecodedbya-to-irnaediting.elife.2015;4.pubmedpmid:25569156.pubmedcentralpmcid:4384741.6.khermeshk,d'erchiaam,barakm,annesea,wachtelc,levanoney等,reducedlevelsofproteinrecodingbya-to-irnaeditinginalzheimer'sdisease.rna.2016feb;22(2):290-302.pubmedpmid:26655226.pubmedcentralpmcid:4712678.7.porathht,carmis,levanoney.agenome-widemapofhyper-editedrnarevealsnumerousnewsites.naturecommunications.2014;5:4726.pubmedpmid:25158696.pubmedcentralpmcid:4365171.8.seeburgph,higuchim,sprengelr.rnaeditingofbrainglutamatereceptorchannels:mechanismandphysiology.brainresearchbrainresearchreviews.1998may;26(2-3):217-29.pubmedpmid:9651532.9.yangw,wangq,kanessj,murrayjm,nishikurak.alteredrnaeditingofserotonin5-ht2creceptorinducedbyinterferon:implicationsfordepressionassociatedwithcytokinetherapy.brainresearchmolecularbrainresearch.2004apr29;124(1):70-8.pubmedpmid:15093687.10.drachevas,pateln,wooda,marcussm,sieverlj,haroutunianv.increasedserotonin2creceptormrnaediting:apossibleriskfactorforsuicide.molecularpsychiatry.2008nov;13(11):1001-10.pubmedpmid:17848916.11.mannjj,brentda,arangov.theneurobiologyandgeneticsofsuicideandattemptedsuicide:afocusontheserotonergicsystem.neuropsychopharmacology:officialpublicationoftheamericancollegeofneuropsychopharmacology.2001may;24(5):467-77.pubmedpmid:11282247.12.mannjj,currierdm.stress,geneticsandepigeneticeffectsontheneurobiologyofsuicidalbehavioranddepression.europeanpsychiatry:thejournaloftheassociationofeuropeanpsychiatrists.2010jun;25(5):268-71.pubmedpmid:20451357.pubmedcentralpmcid:2896004.13.mannjj,huangyy,underwoodmd,kassirsa,oppenheims,kellytm等,aserotonintransportergenepromoterpolymorphism(5-httlpr)andprefrontalcorticalbindinginmajordepressionandsuicide.archivesofgeneralpsychiatry.2000aug;57(8):729-38.pubmedpmid:10920459.14.dinahweissmann,laurentvincent,markd.underwood,laurentcavarec,nicolassalvetat,siemvanderlaan等,regionspecificalterationsofrnaeditingofserotonin2creceptorincortexofsuicideswithmajordepression.translationalpsychiatry.2016;minorrevision.15.christensenr,kristensenpk,bartelsem,bliddalh,astrupa.efficacyandsafetyoftheweight-lossdrugrimonabant:ameta-analysisofrandomisedtrials.lancet.2007nov17;370(9600):1706-13.pubmedpmid:18022033.16.mihanovicm,restek-petrovicb,bodord,molnars,oreskovica,preseckip.suicidalityandsideeffectsofantidepressantsandantipsychotics.psychiatriadanubina.2010mar;22(1):79-84.pubmedpmid:20305596.17.moreirafa,crippaja.thepsychiatricside-effectsofrimonabant.revistabrasileiradepsiquiatria.2009jun;31(2):145-53.pubmedpmid:19578688.18.sundstroma,alfredssonl,sjolin-forsbergg,gerdenb,bergmanu,jokinenj.associationofsuicideattemptswithacneandtreatmentwithisotretinoin:retrospectiveswedishcohortstudy.bmj.2010;341:c5812.pubmedpmid:21071484.pubmedcentralpmcid:2978759.19.gey,sd,speedtp.resampling-basedmultipletestingformicroarraydatahypothesis.sociedaddeestadisticaeinvestigacionoperativatest.2003;12:1-77.20.gentlemanrc,careyvj,batesdm,bolstadb,dettlingm,dudoits等,bioconductor:opensoftwaredevelopmentforcomputationalbiologyandbioinformatics.genomebiology.2004;5(10):r80.pubmedpmid:15461798.pubmedcentralpmcid:545600.21.yb,yh.controllingthefalsediscoveryrate:apracticalandpowerfulapproachtomultipletesting.jroystatistsocser.1995;b57,1:289–300.22.d.g.k,l.l.k,e.m.m.appliedregressionanalysisandothermultivariatemethods.pws-kentpublishingcompany,boston.1988.23.l.breiman,j.friedman,olshenr,stone.c.cart:classificationandregressiontrees.wadsworthinternational.1984.24.breimanl.randomforests.machinelearning.2001;45((1)):5-32.25.sujq,liujs.linearcombinationsofmultiplediagnosticmarkers.journaloftheamericanstatisticalassociation;1993(88):1350-5.26.wangh.anoteoniterativemarginaloptimization:asimplealgorithmformaximumrankcorrelationestimation.computationalstatisticsanddataanalysis2007(51):2803-12.27.staacka,badendiecks,schnorrd,loeningsa,jungk.combineddeterminationofplasmammp2,mmp9,andtimp1improvesthenon-invasivedetectionoftransitionalcellcarcinomaofthebladder.bmcurology.2006;6:19.pubmedpmid:16901349.pubmedcentralpmcid:1560390.28.cortesc,vapnik.v.support-vectornetworks.machinelearning.1995;20.29.baxtwg.applicationofartificialneuralnetworkstoclinicalmedicine.lancet.1995oct28;346(8983):1135-8.pubmedpmid:7475607.30.n.f,al.e.usingbayesiannetworkstoanalyzeexpressiondata.jcomputbiol.2000;7((3-4)):601-20.31.k.h,k.p.s.weightedk-nearest-neighbortechniquesandordinalclassification.discussionpaper399,sfb386,ludwig-maximiliansuniversitymunich2004.32.wolds.pls-regression:abasictoolofchemometrics.chemometricsandintelligentlaboratorysystems.2001;58(108-130).33.fisherra.theuseofmultiplemeasurementsintaxonomicproblems.annalsofeugenics.1936;(2)(179-188).34.dw.evaluationofthepotentialriskofdruginducedmooddisturbanceandsuicide:useofadedicatedplatform.patentwo2008/152146.2008.35.cavarecl,vincentl,leborgnec,plusquellecc,olliviern,normandie-levip等,invitroscreeningfordrug-induceddepressionand/orsuicidaladverseeffects:anewtoxicogenomicassaybasedonce-sscpanalysisofhtr2cmrnaeditinginsh-sy5ycells.neurotoxicityresearch.2013jan;23(1):49-62.pubmedpmid:22528247.序列表<110>阿利瑟迪亚格公司国家科研中心<120>选择由活性化合物诱导的特定效应的基于rna编辑的算法和体外方法<130>alcb001pct<150>ep16000600.3<151>2016-03-11<160>10<170>patentinversion3.5<210>1<211>22<212>dna<213>人工(artificial)<220><223>pde8a靶标:正向引物<400>1caacccacttatttctgcctag22<210>2<211>20<212>dna<213>人工(artificial)<220><223>pde8a靶标:反向引物<400>2ttctgaaaacaatgggcacc20<210>3<211>20<212>dna<213>人工(artificial)<220><223>fnlb靶标:正向引物<400>3aaatgggtcgtgcggtgtat20<210>4<211>21<212>dna<213>人工(artificial)<220><223>fnlb靶标:反向引物<400>4cctgctcggggtggtgttaat21<210>5<211>22<212>dna<213>人工(artificial)<220><223>gria2靶标:正向引物<400>5ctctttagtggagccagagtct22<210>6<211>20<212>dna<213>人工(artificial)<220><223>gria2靶标:反向引物<400>6tcctcagcactttcgatggg<210>7<211>20<212>dna<213>人工(artificial)<220><223>grik2靶标:正向引物<400>7cctgaatcctctctcccctg<210>8<211>20<212>dna<213>人工(artificial)<220><223>grik2靶标:反向引物<400>8ccaaatgcctcccactatcc<210>9<211>20<212>dna<213>人工(artificial)<220><223>gabra3靶标:正向引物<400>9ccaccttgagtatcagtgcc<210>10<211>21<212>dna<213>人工(artificial)<220><223>gabra3靶标:反向引物<400>10cgatgttgaaggtagtgctgg20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1