LEMD3及其突变在BAVM诊治中的应用的制作方法
本发明属于生物医药领域,涉及lemd3及其突变在bavm诊治中的应用。
背景技术
脑动静脉畸形(brainarteriovenousmalformations,bavm)是一种先天性局部脑血管发育异常、后天不断发展所形成的血管团,是颅内最常见的血管畸形之一。其组织学特点是:紊乱的血管团内有动静脉的直接沟通,其内部可有脑组织,其周围为畸形团周围扩张的毛细血管网。虽然bavm的患病率仅为0.14%,但年轻人中与创伤无关的中风和颅内出血有2%的病因为bavm。研究中发现,未经治疗的bavm出血的可能性以每年3%~5%递增,而未经治疗的bavm破裂出血的死亡率是0.7%~1%(m.a.weber,etal,(2014)cerebralarteriovenousmalformations:resultsofthearubastudy.derradiologe243,6d7.)。
临床上常应用spetzler-martin评分表对bavm进行分级,该分级是基于畸形团的大小、静脉引流的模式(浅静脉或深静脉引流)及畸形团所在的位置(是否临近重要脑功能区)进行,共分为i-v级,另有独立的第vi级(指无法手术的病变,切除将不可避免地造成残疾性损害或死亡)。该分级越高,手术切除的难度及风险越大。随着影像学技术的不断发展,bavm的诊断率大大提高。而手术切除、介入及放射治疗手段的发展也日新月异,但高spetzler-martin级别的bavm患者在治疗过程中仍然面临着较高的致死、致残率。为此,bavm一直是神经科学领域的研究热点和难点。近年来,bavm的分子生物学研究逐渐得到重视,并为揭示bavm的发生、发展机制提供越来越多的证据。
目前的研究表明,bavm的形成可能是在先天性异常血管或基因缺陷存在的基础上,通过后天的环境因素(创伤、缺血、炎症、局部静脉高压等)引发获得性血管新生及重塑,最终造成紊乱的局灶性血流动力学改变及紊乱的血管结构(a.s.achroletal.,(2006)tumornecrosisfactor-alpha-238g>apromoterpolymorphismisassociatedwithincreasedriskofnewhemorrhageinthenaturalcourseofpatientswithbrainarteriovenousmalformations.stroke;ajournalofcerebralcirculation37,231.)。这种机制被认为是bavm病理生理学改变的关键步骤。对于先天因素即基因水平的研究,目前进展缓慢,研究与bavm发生发展相关的基因具有对于bavm的早期诊断和治疗具有重要的意义。
技术实现要素:
为了弥补现有技术的不足,本发明的目的之一在于提供一种与bavm相关的基因、基因突变以及蛋白突变体。
本发明的目的之二在于提供与bavm相关的基因、基因突变以及突变体在bavm的早期诊断和治疗中的应用。
本发明的目的之三在于提供一种bavm的斑马鱼模型及其构建方法,以及在该模型在筛选治疗bavm药物中的应用。
为了实现上述目的,本发明采用如下技术方案:
本发明的第一方面提供了突变的lemd3蛋白,与seqidno.1相比,所述蛋白的氨基酸序列具有突变p.thr879ser。
具体的,所述突变的lemd3蛋白的氨基酸序列如seqidno.2所示。
本发明的第二方面提供了一种编码本发明第一方面所述突变的lemd3蛋白的核苷酸序列。
本发明的第三方面提供了一种编码lemd3基因突变体的核苷酸序列,与seqidno.3相比,所述核苷酸序列具有突变c.2636c>g。
具体的,所述核苷酸序列如seqidno.4所示。
本发明的第四方面提供了一种试剂,所述试剂选自:
检测lemd3表达水平的试剂;或
检测lemd3蛋白第879位氨基酸位点的试剂;或
检测lemd3基因第2636位核苷酸位点的试剂。
进一步,所述试剂选自:
特异性识别lemd3基因的探针,或
特异性扩增lemd3基因的引物,或
特异性结合lemd3蛋白的抗体或配体。
进一步,特异性扩增lemd3基因的引物序列如seqidno.8和9所示。
本发明的第五方面提供了一种药物组合物,所述药物组合物包括lemd3的促进剂,所述促进剂包括提高lemd3基因或其表达产物稳定性、上调lemd3基因或其表达产物的表达水平、增加lemd3基因或其表达产物有效作用时间的物质。
进一步,所述促进剂为含有野生型lemd3mrna的表达载体。
进一步,所述药物组合物还包括药学上可接受的载体。所述载体包括(但不限于)稀释剂、赋形剂、粘合剂、湿润剂、吸收促进剂、表面活性剂、致湿剂、吸附载体、润滑剂、缓冲剂、稳定剂、抑菌剂、等渗剂、螯合剂、ph控制剂。
本发明的第六方面提供了一种构建bavm斑马鱼模型的方法,将lemd3抑制剂注射入斑马鱼胚胎中
进一步,所述lemd3抑制剂为吗啉反义寡核苷酸。
进一步,所述吗啉反义寡核苷酸的序列如seqidno.5所示。
进一步,所述斑马鱼胚胎为单细胞期至二细胞期的胚胎。
本发明的第七方面提供了一种bavm斑马鱼模型,所述模型通过本发明第六方面所述的方法构建而成。
本发明的第八方面提供了如下任一项所述的应用:
a.本发明第一方面所述的蛋白在制备诊断bavm的产品中的应用;
b.本发明第二方面或第三方面所述的核苷酸序列在制备诊断bavm的产品中的应用;
c.lemd3在制备诊断bavm的产品中的应用;
d.本发明第四方面所述的试剂在制备诊断bavm的产品中的应用;
e.lemd3在制备治疗bavm的药物中的应用;
f.本发明第五方面所述的药物组合物在制备治疗bavm的产品中的应用;
g.本发明第七方面所述的bavm斑马鱼模型在筛选治疗bavm的候选药物中的应用。
进一步,a中所述的产品包括检测蛋白突变位点的试剂,所述突变为p.thr879ser,当检测的879位的氨基酸为ser时,患者患有bavm;
b中所述的产品包括检测核苷酸突变位点的试剂,所述突变为c.2636c>g,当检测的2636位的核苷酸为g时,患者患有bavm;
c中所述的产品包括检测lemd3的试剂,所述试剂是检测lemd3表达水平的试剂。
g中,若施用待筛选物质后,斑马鱼的bavm的症状缓解,则该待筛选物质是治疗bavm的候选药物。
附图说明
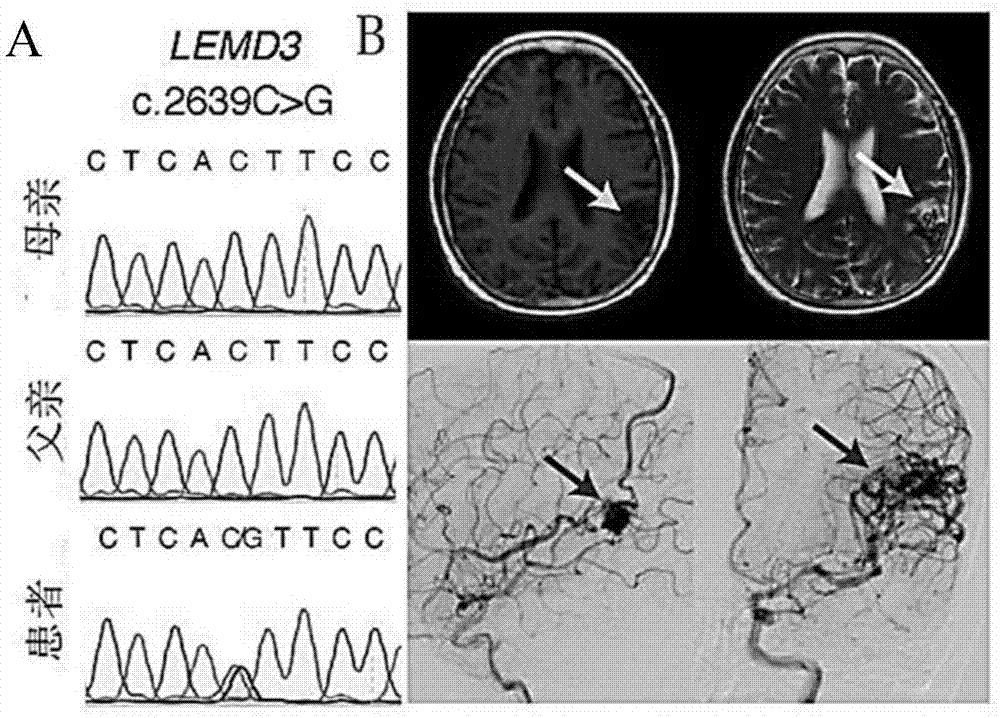
图1是携带lemd3突变的患者的sanger测序验证以及临床影像学诊断;其中,图asanger测序图,图b临床影像学诊断图。
图2是斑马鱼脑血管的共聚焦显微镜荧光图。
图3是斑马鱼突变体的bavm样表型模式图。
图4是斑马鱼突变体的生长和rt-pcr验证图;其中图a是生长图,图b是吗啉反义寡核苷酸的作用位点和上下游引物的结合位点图;图c是突变体的rt-pcr验证图。
图5是斑马鱼bavm表型数量统计图。
具体的实施方式
本发明经过广泛而深入的研究,利用全外显子组测序,通过核心家系与散发病例以及健康人群的对比研究,从病因学和基因学角度对bavm的出血风险、发展转归进行预测,有助于实现早期精准、个体化治疗。同时,为复杂多基因脑血管先天性疾病的病因学研究提供了新方法和经验。
在本发明中,“外显子”一词是指在成熟mrna中被保留下的部分,即成熟mrna对应于基因中的部分。内含子是在mrna加工过程中被剪切掉的部分,在成熟mrna中不存在。外显子和内含子都是对于基因而言的,编码的部分为外显子,不编码的为内含子,内含子没有遗传效应。
本发明可以利用本领域内已知的任何方法对基因及其编码的蛋白进行检测。本领域技术人员应当理解,检测基因的手段不是本发明的重要方面。本发明中的基因使用本领域普通技术人员已知的多种检测技术进行检测,这些技术包括但不限于:核酸测序、核酸杂交、核酸扩增技术、免疫检测技术。
检测基因表达水平的方法包括(但不限于):聚合酶链式反应(pcr)、逆转录聚合酶链式反应(rt-pcr)、转录介导的扩增(tma)、连接酶链式反应(lcr)、链置换扩增(sda)和基于核酸序列的扩增(nasba)、原位杂交(ish)、微阵列、southern或northern印迹、夹心elisa、放射免疫测定(ria)、直接、间接或对比酶联免疫吸附测定(elisa)、酶免疫测定(eia)、荧光免疫测定(fia)、蛋白质印迹法、免疫沉淀法和基于任何颗粒的免疫测定(如使用金颗粒、银颗粒或乳胶颗粒、磁性颗粒或量子点)。其中,pcr需要在扩增前将rna逆转录成dna(rt-pcr),tma和nasba直接扩增rna。
检测基因突变或蛋白突变的方法包括(但不限于):taqman法、质谱法、dna微阵列法、测序法、微测序、杂交、限制性片段分析、寡核苷酸连接检测、等位基因特异性pcr-hrm或联合应用上述方法。
在本发明中,示例性探针包括pcr引物以及基因特异性dna寡核苷酸探针,例如固定于微阵列基底上的微阵列探针、定量核酸酶保护检验探针、与分子条形码连接的探针、以及固定于珠上的探针。
本发明中术语“探针”指能与另一分子的特定序列或亚序列或其它部分结合的分子。除非另有指出,“探针”通常指能通过互补碱基配对与另一多核苷酸(往往称为“靶多核苷酸”)结合的多核苷酸探针。根据杂交条件的严谨性,探针能和与该探针缺乏完全序列互补性的靶多核苷酸结合。探针可作直接或间接的标记,其范围包括引物。杂交方式,包括,但不限于:溶液相、固相、混合相或原位杂交测定法。
作为探针,可以使用荧光标记、放射标记、生物素标记等对癌检测用多核苷酸进行了标记的标记探针。多核苷酸的标记方法本身是公知的。可通过如下方法检查试样中是否存在受试核酸:固定受试核酸或者其扩增物,与标记探针进行杂交,洗涤,以及然后测定与固相结合的标记。备选地,还可固定癌检测用多核苷酸,使受试核酸与其杂交,然后应用标记探针等检测结合于固相上的受试核酸。在这种情况下,结合于固相上的癌检测用多核苷酸也称为探针。使用多核苷酸探针测定受试核酸的方法在本领域也是公知的。可以如下进行该方法:在缓冲液中使多核苷酸探针与受试核酸在tm或者其附近(优选在±4℃以内)接触用于杂交,洗涤,然后测定杂交的标记探针或者与固相探针结合的模板核酸。
在作为探针使用的多核苷酸的大小优选为18个或更多个核苷酸、更优选为20个或更多个核苷酸,以及编码区域的全长或更少。作为引物使用时,该多核苷酸大小优选为18个或更多个核苷酸,以及50个或更少核苷酸。这些探针具有与靶点基因的特定的碱基序列互补的碱基序列。这里,所谓“互补”,只要是杂交即可,可以不是完全互补。这些多核苷酸通常相对于该特定的碱基序列具有80%以上、优选90%以上、更优选95%以上、特别优选100%的同源性。这些探针可以是dna,也可以是rna,另外,可以为在其一部分或全部中核苷酸通过pn、lna、ena、gna、tna等人工核酸置换得到的多核苷酸。
本发明的引物或探针可以使用磷酰亚胺固相支持法或其他熟知方法化学合成。也可以使用本领域已知的许多手段修饰所述核酸序列。这些修饰的非限制性实例是甲基化、加帽、用天然核苷酸的一种或多种类似物进行的置换和在核苷酸之间的修饰,例如,修饰不带电荷的连接体(例如,磷酸甲酯、磷酸三酯、磷酰亚胺、氨基甲酸酯等),或修饰带电荷的连接体(例如,硫代磷酸酯、二硫代磷酸酯等)。
在本发明中作为一种可选择的实施方式,诊断bavm的产品可以是试剂盒,所述试剂盒包含与一种或多种生物标志物的mrna特异性结合的一种或多种探针。在某些实施方案中,所述试剂盒还包含洗涤溶液。在某些实施方案中,所述试剂盒还包含进行杂交试验的试剂、mrna分离或纯化工具、检测工具以及阳性和阴性对照。在某些实施方案中,所述试剂盒还包含使用该试剂盒的说明书。所述试剂盒可以定制供在家使用、临床使用或研究使用。
在某些实施方案中,本文提供的是用于检测一种或多种生物标志物的蛋白质水平的试剂盒。在某些实施方案中,所述试剂盒包含用识别蛋白质生物标志物的抗体包被的试纸条、洗涤溶液、进行该试验的试剂、蛋白质分离或纯化工具、检测工具以及阳性和阴性对照。在某些实施方案中,所述试剂盒还包含使用该试剂盒的说明书。所述试剂盒可以定制供在家使用、临床使用或研究使用。
这样一种试剂盒可以采用例如试纸条、膜、芯片、盘、测试条、滤器、微球、载片、多孔板或光纤。所述试剂盒的固相支持物可以是例如塑料、硅片、金属、树脂、玻璃、膜、颗粒、沉淀物、凝胶、聚合物、薄片、球、多糖、毛细管、胶片、板或载片。
在本发明中,lemd3的抑制剂包括核酸抑制物,蛋白抑制剂,蛋白水解酶,蛋白结合分子。其中核酸抑制物选自:以lemd3或其转录本为靶序列、且能够抑制lemd3基因表达或基因转录的干扰分子,包括:shrna(小发夹rna)、小干扰rna(sirna)、dsrna、微小rna、反义核酸,或能表达或形成所述shrna、小干扰rna、dsrna、微小rna、反义核酸的构建物。蛋白结合分子选自:与siglec10蛋白特异性结合的物质,如能够抑制lemd3蛋白活性的抗体或配体。
作为本发明的一种优选的实施方式,所述lemd3的抑制剂为一种反义寡核苷酸,反义寡核苷酸是指与体内某rna序列具有互补顺序,并能通过碱基配对与互补链杂交,从而影响转录或翻译过程的rna或dna片段。更为优选的,所述的抑制剂为吗啉反义寡核苷酸,吗啉反义寡核苷酸(morpholinooligomers,即mo)因其核苷酸骨架上的吗啉环而得名,吗啉环取代了rna中的核糖核苷酸环或者dna中的脱氧核糖核苷酸环。吗啉反义寡核苷酸是一种新型反义寡核苷酸,能抑制细胞内mrna的剪接过程,从而抑制基因的表达;同时具有良好的稳定性、溶解度和细胞渗透性。
药物组合物
基于发明人的发现,本发明提供了一种药物组合物,所述药物组合物包括lemd3的促进剂。所述促进剂包括提高lemd3基因或其表达产物稳定性、上调lemd3基因或其表达产物的表达水平、增加lemd3基因或其表达产物有效作用时间的物质。
作为本发明的一种可选择的实施方式,所述的lemd3的促进剂是一种含有lemd3的表达载体。所述的表达载体通常还含有启动子、复制起点和/或标记基因等。
在本发明中,所述药物组合物还包括药学上可接受的载体,包括(但并不限于):稀释剂、赋形剂如乳糖、氯化钠、葡萄糖、尿素、淀粉、水等、填充剂如淀粉、蔗糖等;粘合剂如单糖浆、葡萄糖溶液、淀粉溶液、纤维素衍生物、藻酸盐、明胶和聚乙烯吡咯烷酮;湿润剂如甘油;崩解剂如干淀粉、海藻酸钠、海带多糖粉末、琼脂粉末、碳酸钙和碳酸氢钠;吸收促进剂季铵化合物、十二烷基硫酸钠等;表面活性剂如聚氧化乙烯山梨聚糖脂肪酸酯、十二烷基硫酸钠、硬脂酸单甘油酯、十六烷醇等;致湿剂如甘油、淀粉等;吸附载体如淀粉、乳糖、斑脱土、硅胶、高岭土和皂粘土等;润滑剂如滑石粉、硬脂酸钙和镁、聚乙二醇、硼酸粉末等。
在本发明中,药物组合物可以使用不同的添加剂进行制备,例如缓冲剂、稳定剂、抑菌剂、等渗剂、螯合剂、ph控制剂及表面活性剂。
本发明所述的药物组合物可口服给药、非胃肠道给药、通过吸入喷雾给药、局部给药、直肠给药、鼻给药、颊给药、阴道给药或通过植入的贮药装置给药。优选口服给药或注射给药。本发明的药物组合物可含有任何常用的无毒可药用载体、辅料或赋形剂。在某些情况下,药用酸、碱或缓冲剂可用来调节制剂的ph以提高所配制的化合物或其给药剂型的稳定性。本文所用术语非胃肠道包括皮下、皮内、静脉内、肌内、关节内、动脉内、滑膜内、胸骨内、鞠内、损伤部位内、和颅内注射或输注技术。只要能达到目标组织,本发明所述药物组合物可以通过任何途径给予受体。
本发明药物组合物可以以任何口服剂型的形式口服给药,包括但不限于胶囊、片剂、乳剂和水悬浮液、分散剂和溶液。对于口服片剂,常用载体包括乳糖和玉米淀粉。一般还加入润滑剂例如硬脂酸镁。为了以胶囊形式口服给药,适用的稀释剂包括乳糖和无水玉米淀粉。当口服施用水悬浮液和/或乳液时,可将活性组分悬浮或溶解在油相中,并与乳化剂和/或悬浮剂合并。如果需要的话,可加入一些甜味剂和/或矫味剂和/或着色剂。适当时,可将用于口服给药的剂量单位制剂包微囊。例如,通过在聚合物、蜡等中将颗粒物质包衣或包埋,也可制备所述制剂已延长或维持释放。
在本发明中,可将促进lemd3表达的化合物作为裸rna与递送试剂一起作为核酸(如重组质粒或病毒载体)给受试者施用,所述核酸包含增加lemd3表达的序列。递送试剂可以是亲脂试剂、聚阳离子、脂质体等。
本领域的技术人员熟知的方法能用于构建本发明所需的表达载体。这些方法包括体外重组dna技术、dna合成技术、体内重组技术等。所述的表达载体优选地包含一个或多个选择性标记基因,以提供用于选择转化的宿主细胞的表型性状,如卡拉霉素、庆大霉素、潮霉素、氨苄青霉素抗性。
在本发明中,是本领域已知的各种载体,如市售的载体、包括质粒、粘粒、噬菌体、病毒等。表达载体向宿主细胞中的导入可以使用电穿孔法、磷酸钙法、脂质体法、deae葡聚糖法、显微注射、病毒感染、脂质体转染、与细胞膜透过性肽的结合等周知的方法。
本发明所述药物组合物可口服给药、非胃肠道给药、通过吸入喷雾给药、局部给药、直肠给药、鼻给药、颊给药、阴道给药或通过植入的贮药装置给药。优选口服给药或注射给药。本发明药物组合物可含有任何常用的无毒可药用载体、辅料或赋形剂。
下面结合附图和实施例对本发明作进一步详细的说明。以下实施例仅用于说明本发明而不用于限制本发明的范围。实施例中未注明具体条件的实验方法,通常按照常规条件,例如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。
实施例1筛选与bavm相关的基因
1、样品收集
收集了2015年11月至2016年11月期间在北京天坛医院被确诊为bavm的患者123名(患者及其生物学父母血液被留取,患者+生物学父母=核心家系),脑动静脉畸形(bavm)的诊断通过数字减影血管造影(dsa)、磁共振成像/血管摄影或计算机断层摄影证实,所有的父母都进行了体格检查以排除严重的血管问题。
排除标准为:(1)已知的遗传性出血性毛细血管扩张症(hereditaryhemorrhagictelangiectasis,hht)、毛细血管性畸形-avm(capillarymalformation-arteriovenousmalformationsyndrome,cm-avm)、sturge-weber综合征或其他孟德尔遗传血管疾病;或(2)不完整的临床数据。
经排除之后,符合标准的有100名,患者及其临床表型正常的生物学父母均进行了wes。
本研究经北京天坛医院伦理委员会批准。患者或其父母签署知情同意。斑马鱼实验经北京大学动物保护和应用委员会(iacuc)批准。
2、全外显子测序及突变注释
1)提取dna
使用axygen的血基因组dna提取试剂盒提取患者及父母外周血全基因组dna,用1%琼脂糖凝胶电泳和nanodrop2000分光光度计进行dna定量检测。
2)构建dna文库
采用超声打断仪(covariss2,massachusetts,usa)将基因组dna随机打断成主带200-300bp的片段,取500ng提纯的dna片段进行末端修复并连接上标记性接头,构建dna测序文库。按照illumina标准建库方法进行3步酶促反应:通过末端修复,加“a”和连接illumina测序接头(通过pcr反应,将一个8bp的barcode连接至dna片段上),形成样本文库。
3)外显子捕获
使用sureselecthumanallexonv6+utrr2coredesign(91mb,agilent)完成外显子捕获,具体操作参见使用说明。
4)测序及分析
使用illuminahiseq4000平台进行测序,使用genomeanalysistoolkit3.4.0以及haplotypecaller检测snp位点和插入缺失标记,采用pumch进行注释,新发突变、复合杂合突变和隐形遗传突变运用gemini(version0.19.1)来计算。利用生物信息学预测工具预测候选变异的保守性和致病性。所有变异均对比公用数据库1000genomesproject(http://www.internationalgenome.org/),exomevariantserver,nhlbigoexomesequencingproject(esp)(http://evs.gs.washington.edu/evs/),和exomeaggregationconsortium(exac)(http://exac.broadinstitute.org/),筛选致病性突变。
3、结果
结果显示,与参考转录本(nm_001167614.1)相比,患者的lemd3基因出现了突变c.2636c>g(p.thr879ser),突变reads/总reads为61/114。
实施例2使用sanger测序对突变位点进行验证
1、将从患者和父母获得的基因组dna,并使用axygen-ap-gx-50套件试剂盒进行纯化,在abi3730测序仪上进行sanger测序。
2、结果
结果如图1所示,测序结果显示,患者中的lemd3第2636位的核苷酸由c变成了g。
实施例3斑马鱼验证
1、斑马鱼品系
利用tg(kdrl.4:mcherry)pku6转基因斑马鱼进行实验其体内mcherry红色荧光的表达是由kdrl.4(一种内皮细胞特异性基因)调控。斑马鱼和胚胎按照标准进行培育。为了避免真菌感染,胚胎在0.5g/l亚甲基蓝中进行孵化。1-苯基2-硫脲(0.003%)被用来抑制色素生成。
2、体外转录和胚胎注射
为了体内验证针对斑马鱼同源的lemd3的设计了反义吗啉反义寡核苷酸,mo1(针对外显子2-内含子2的剪切区),mo2(针对aug位点),具体序列如下:
mo1:5'-acaggacagatacttacatactgct-3'(seqidno.5)
mo2:5'-acgccatctttgcacgaaaaacgca-3'(seqidno.6)
对照mo:5'-cctcttacctcagttacaatttata-3'(seqidno.7)
将基因敲低试剂morpholino5ng注射入斑马鱼胚胎单细胞期至二细胞期的细胞。
将lemd3野生型和突变型的cdna序列克隆进pcs2+质粒,使用限制性内切酶消化,并使用ambionmmessagemmachine试剂盒转录出mrna,以200pg的剂量与morpholino(吗啉反义寡核苷酸)进行共同注射。
3、荧光观察
将48h的斑马鱼使用镊子剥去卵膜,使用tricane麻醉。将胚胎在低熔点胶中固定,使用共聚焦显微镜进行拍照,由两位研究者使用盲法评估斑马鱼脑血管表型。
4、rna提取
使用trizol法提取24h胚胎的总rna,将组织或细胞匀浆后,加入1.5ml离心管。加入0.2倍相同体积的氯仿,振荡混匀30s。室温离心15000g3min。将上清液转移到另一干净离心管中。在上清液中加入等体积的异丙醇,振荡器上振荡混均30s。室温离心15000g5min,rna沉淀将在离心底的侧面形成,小心吸弃上清液。清洗:在含rna沉淀的离心管中加入1ml75%乙醇,振荡器上振荡混均30s。室温15000g离心1min。小心吸弃上清液。重复清洗步骤。将离心管倒置于滤纸上以干燥rna。
5、逆转录
将rna产物使用onescriptreversetranscriptasekit试剂盒进行逆转录,将逆转录产物进行pcr扩增。引物序列如下:
lemd3的引物序列如下:
正向引物:5’-ttcagtgcccactacttgtcc-3’(seqidno.8)
反向引物:5’-cagggtcaccaccaatcaac-3’(seqidno.9)
pcr反应条件:94℃2min,(94℃30s,60℃60s,72℃1.5min)×20~30个循环
6、统计分析
利用spss15.0软件进行统计分析,所有的实验进行了至少三次重复,有统计学意义的定义为p<0.05。利用t检验分析斑马鱼表型比例数据。
7、结果
斑马鱼脑血管的免疫荧光分析显示一系列bavm特征:(1)异常的后循环连接血管节段(posteriorconnectingsegments,pcs)延伸并与靠近基底交通动脉(basalcommunicatingartery,bca)的原始后脑通道(theprimordialhidbrainchannel,phbc)融合,形成了半月形畸形表现;(2)bca和pcs扩张;(3)大脑前动脉的发育不良(图2)。这些结果与之前报道的avm斑马鱼模型相一致。
进一步验证lemd3基因及其突变的功能,lemd3突变体(lemd3敲低的斑马鱼)的头部、心周出现了不同程度的水肿,表现出体节发育阻滞及弯曲等畸形(图4a),而rt-pcr图,可见被截断了约41bp的lemd3转录本(图4c)。
对bavm表型的斑马鱼进行统计,发现lemd3敲除相比对照组显著提高了avm表型,说明lemd3与bavm的发生发展相关,而注射人类野生型lemd3基因mrna可以缓解lemd3突变体的bavm样表型,而注射人类lemd3突变体(c.971t>c)mrna却无作用(图5),说明lemd3基因的c.2636c>g(p.thr879ser)突变为致病突变。
上述实施例的说明只是用于理解本发明的方法及其核心思想。应当指出,对于本领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以对本发明进行若干改进和修饰,这些改进和修饰也将落入本发明权利要求的保护范围内。
序列表
<110>北京市神经外科研究所
中国医学科学院北京协和医院
<120>lemd3及其突变在bavm诊治中的应用
<160>9
<170>siposequencelisting1.0
<210>1
<211>910
<212>prt
<213>homosapiens
<400>1
metalaalaalaalaalaseralaproglnglnleuseraspgluglu
151015
leupheserglnleuargargtyrglyleuserproglyprovalthr
202530
gluserthrargprovaltyrleulyslysleulyslysleuargglu
354045
glugluglnglnglnhisargserglyglyargglyasnlysthrarg
505560
asnserasnasnasnasnthralaalaalathrvalalaalaalagly
65707580
proalaalaalaalaalaalaglymetglyvalargprovalsergly
859095
aspleusertyrleuargthrproglyglyleucysargileserala
100105110
serglyprogluserleuleuglyglyproglyglyalaseralaala
115120125
proalaalaglyserlysvalleuleuglypheserseraspgluser
130135140
aspvalglualaserproargaspglnalaglyglyglyglyarglys
145150155160
aspargalaserleuglntyrargglyleulysalaproproalapro
165170175
leualaalasergluvalthrasnserasnseralagluargarglys
180185190
prohissertrptrpglyalaargargproalaglyprogluleugln
195200205
thrproproglylysaspglyalavalgluaspglugluglyglugly
210215220
gluaspglyglugluargaspprogluthrglugluproleutrpala
225230235240
serargthrvalasnglyserargleuvalprotyrsercysargglu
245250255
asntyrseraspsergluglugluaspaspaspaspvalalaserser
260265270
argglnvalleulysaspaspserleuserarghisargproargarg
275280285
thrhisserlysproleuproproleuthralalysseralaglygly
290295300
argleugluthrservalglnglyglyglyglyleualametasnasp
305310315320
argalaalaalaalaglyserleuaspargserargasnleugluglu
325330335
alaalaalaalagluglnglyglyglycysaspglnvalaspserser
340345350
provalproargtyrargvalasnalalyslysleuthrproleuleu
355360365
proproproleuthraspmetaspserthrleuaspserserthrgly
370375380
serleuleulysthrasnasnhisileglyglyglyalapheserval
385390395400
aspserproargiletyrserasnserleuproproseralaalaval
405410415
alaalaserserserleuargileasnhisalaasnhisthrglyser
420425430
asnhisthrtyrleulysasnthrtyrasnlysprolysleuserglu
435440445
progluglugluleuleuglnglnphelysargglugluvalserpro
450455460
thrglyserpheseralahistyrleusermetpheleuleuthrala
465470475480
alacysleuphepheleuileleuglyleuthrtyrleuglymetarg
485490495
glythrglyvalsergluaspglygluleuserlysasnprophegly
500505510
gluthrpheglylysileglngluserglulysthrleumetmetasn
515520525
thrleutyrlysleuhisaspargleualaglnleualaglyasphis
530535540
glucysglyserserserglnargthrleuservalglnglualaala
545550555560
alatyrleulysaspleuglyproglutyrgluglyilepheasnthr
565570575
serleuglntrpileleugluasnglylysaspvalglyileargcys
580585590
valglypheglyprogluglugluleuthrasnilethraspvalgln
595600605
pheleuglnserthrargproleumetserphetrpcysargphearg
610615620
argalaphevalthrvalthrhisargleuleuleuleucysleugly
625630635640
valvalmetvalcysvalvalleuargtyrmetlystyrargtrpthr
645650655
lysglugluglugluthrargglnmettyraspmetvalvallysile
660665670
ileaspvalleuargserhisasnglualacysglngluasnlysasp
675680685
leuglnprotyrmetproileprohisvalargaspserleuilegln
690695700
prohisasparglyslysmetlyslysvaltrpaspargalavalasp
705710715720
pheleualaalaasngluserargvalargthrgluthrargargile
725730735
glyglyalaasppheleuvaltrpargtrpileglnproseralaser
740745750
cysasplysileleuvalileproserlysvaltrpglnglyglnala
755760765
phehisleuaspargargasnserproproasnserleuthrprocys
770775780
leulysileargasnmetpheaspprovalmetgluileglyaspgln
785790795800
trphisleualaileglnglualaileleuglulyscysseraspasn
805810815
aspglyilevalhisilealavalasplysasnserarggluglycys
820825830
valtyrvallyscysleuserproglutyralaglylysalaphelys
835840845
alaleuhisglysertrppheaspglylysleuvalthrvallystyr
850855860
leuargleuaspargtyrhishisargpheproglnalaleuthrser
865870875880
asnthrproleulysproserasnlyshismetasnsermetserhis
885890895
leuargleuargthrglyleuthrasnserglnglyserser
900905910
<210>2
<211>910
<212>prt
<213>homosapiens
<400>2
metalaalaalaalaalaseralaproglnglnleuseraspgluglu
151015
leupheserglnleuargargtyrglyleuserproglyprovalthr
202530
gluserthrargprovaltyrleulyslysleulyslysleuargglu
354045
glugluglnglnglnhisargserglyglyargglyasnlysthrarg
505560
asnserasnasnasnasnthralaalaalathrvalalaalaalagly
65707580
proalaalaalaalaalaalaglymetglyvalargprovalsergly
859095
aspleusertyrleuargthrproglyglyleucysargileserala
100105110
serglyprogluserleuleuglyglyproglyglyalaseralaala
115120125
proalaalaglyserlysvalleuleuglypheserseraspgluser
130135140
aspvalglualaserproargaspglnalaglyglyglyglyarglys
145150155160
aspargalaserleuglntyrargglyleulysalaproproalapro
165170175
leualaalasergluvalthrasnserasnseralagluargarglys
180185190
prohissertrptrpglyalaargargproalaglyprogluleugln
195200205
thrproproglylysaspglyalavalgluaspglugluglyglugly
210215220
gluaspglyglugluargaspprogluthrglugluproleutrpala
225230235240
serargthrvalasnglyserargleuvalprotyrsercysargglu
245250255
asntyrseraspsergluglugluaspaspaspaspvalalaserser
260265270
argglnvalleulysaspaspserleuserarghisargproargarg
275280285
thrhisserlysproleuproproleuthralalysseralaglygly
290295300
argleugluthrservalglnglyglyglyglyleualametasnasp
305310315320
argalaalaalaalaglyserleuaspargserargasnleugluglu
325330335
alaalaalaalagluglnglyglyglycysaspglnvalaspserser
340345350
provalproargtyrargvalasnalalyslysleuthrproleuleu
355360365
proproproleuthraspmetaspserthrleuaspserserthrgly
370375380
serleuleulysthrasnasnhisileglyglyglyalapheserval
385390395400
aspserproargiletyrserasnserleuproproseralaalaval
405410415
alaalaserserserleuargileasnhisalaasnhisthrglyser
420425430
asnhisthrtyrleulysasnthrtyrasnlysprolysleuserglu
435440445
progluglugluleuleuglnglnphelysargglugluvalserpro
450455460
thrglyserpheseralahistyrleusermetpheleuleuthrala
465470475480
alacysleuphepheleuileleuglyleuthrtyrleuglymetarg
485490495
glythrglyvalsergluaspglygluleuserlysasnprophegly
500505510
gluthrpheglylysileglngluserglulysthrleumetmetasn
515520525
thrleutyrlysleuhisaspargleualaglnleualaglyasphis
530535540
glucysglyserserserglnargthrleuservalglnglualaala
545550555560
alatyrleulysaspleuglyproglutyrgluglyilepheasnthr
565570575
serleuglntrpileleugluasnglylysaspvalglyileargcys
580585590
valglypheglyprogluglugluleuthrasnilethraspvalgln
595600605
pheleuglnserthrargproleumetserphetrpcysargphearg
610615620
argalaphevalthrvalthrhisargleuleuleuleucysleugly
625630635640
valvalmetvalcysvalvalleuargtyrmetlystyrargtrpthr
645650655
lysglugluglugluthrargglnmettyraspmetvalvallysile
660665670
ileaspvalleuargserhisasnglualacysglngluasnlysasp
675680685
leuglnprotyrmetproileprohisvalargaspserleuilegln
690695700
prohisasparglyslysmetlyslysvaltrpaspargalavalasp
705710715720
pheleualaalaasngluserargvalargthrgluthrargargile
725730735
glyglyalaasppheleuvaltrpargtrpileglnproseralaser
740745750
cysasplysileleuvalileproserlysvaltrpglnglyglnala
755760765
phehisleuaspargargasnserproproasnserleuthrprocys
770775780
leulysileargasnmetpheaspprovalmetgluileglyaspgln
785790795800
trphisleualaileglnglualaileleuglulyscysseraspasn
805810815
aspglyilevalhisilealavalasplysasnserarggluglycys
820825830
valtyrvallyscysleuserproglutyralaglylysalaphelys
835840845
alaleuhisglysertrppheaspglylysleuvalthrvallystyr
850855860
leuargleuaspargtyrhishisargpheproglnalaleuserser
865870875880
asnthrproleulysproserasnlyshismetasnsermetserhis
885890895
leuargleuargthrglyleuthrasnserglnglyserser
900905910
<210>3
<211>2733
<212>dna
<213>homosapiens
<400>3
atggcggcggcagcagcttcggcgcctcagcagctctcggatgaggagcttttctctcag60
ctccgccgttacggcctgtctcccggaccagtgacggagagcacccgcccggtctacctc120
aagaagctgaagaagcttcgagaggaagagcagcaacagcaccggtcagggggccgcggc180
aacaagacgcggaacagtaataacaataacacggcagccgccacggtcgcagccgcggga240
ccagcggcggcggcggccgcggggatgggggtccggccggtctcgggcgacctctcctac300
ttacggactcctgggggcctgtgccgaatctcggcctctggcccagagagcctcctggga360
gggcccgggggcgcctccgccgcccccgcggctggcagcaaagtgctgctgggcttcagc420
tcggacgagtcggacgtggaggccagtccccgggaccaggccggcggcggcgggaggaaa480
gaccgggcttcgctccagtaccgcgggctcaaagcgccgccggcgcccctggccgccagc540
gaggtgactaacagcaactctgcagagcgaaggaagccccactcgtggtggggggccagg600
aggccggcgggccccgagctgcagaccccgccggggaaagatggagcagtggaggacgag660
gaaggggagggagaggacggtgaggagagggacccggagaccgaggagccgctctgggcg720
agccggaccgtgaatggcagccggcttgtcccctacagctgccgggaaaactattcggac780
tcagaggaagaggacgacgacgacgtggcctccagcagacaggtattaaaggacgactcc840
ctttcccggcatcggcccagacgaacccatagtaagcctctccccccgctgactgctaaa900
tcggccggcggcaggctggagacttcagttcagggagggggaggactcgcgatgaatgac960
agggcggcggctgccgggagtctagacaggagccgaaacctcgaagaggcggcggccgcg1020
gagcagggaggagggtgtgatcaagtggactccagccccgttcctagataccgtgttaac1080
gctaagaaactgacccctctcctgcccccgccacttactgacatggactcaaccttggat1140
tcgtcaacaggctcccttctgaaaaccaataatcatattggcggtggggccttcagtgtg1200
gactcccccaggatttattctaacagtctccctcccagtgcggcggtggccgcctctagt1260
tcactcaggatcaatcacgccaatcatacgggctccaatcatacctacctgaaaaacaca1320
tacaacaaaccgaagctttccgaacccgaagaggaacttctccagcaatttaaacgggag1380
gaggtgtccccaacagggagtttcagtgcccactacttgtcgatgtttctcttaactgct1440
gcctgcttatttttcctaatactgggactgacttacctaggaatgagagggacaggagta1500
tctgaggatggagaactcagcaaaaacccctttggtgaaacatttggaaaaatacaagaa1560
agtgaaaaaactcttatgatgaacacattatataagcttcatgatcgattggcacagctt1620
gcaggagatcatgaatgtggcagttctagtcaaagaacgctttctgttcaagaggcagct1680
gcgtatttaaaagatttaggtcctgaatatgaaggtatatttaacacttcattgcagtgg1740
atcttagaaaatggaaaagatgttggaataaggtgtgttggttttggccctgaggaagaa1800
ttgacaaatataactgatgtgcagtttttacagtccacaagaccactgatgtctttttgg1860
tgtcgttttcgacgtgcttttgttactgtaactcacagattattgttgttatgcttaggt1920
gtagtgatggtttgtgtcgttctgcgttacatgaaatatcgatggacaaaagaagaggag1980
gaaacaaggcagatgtatgatatggtggtaaagattatagatgttttacgaagtcataat2040
gaagcctgccaggaaaacaaagatttacaaccttacatgcctattccacatgtacgcgat2100
tccttaatacagcctcatgacaggaaaaaaatgaagaaagtctgggatagagctgttgac2160
ttccttgctgctaatgagtctagagttcgcacggaaacacgaagaataggtggtgcagat2220
tttctggtttggcggtggatccagccttctgcatcctgtgacaaaatattagttatacct2280
tctaaagtatggcaaggtcaagcatttcatttagatagaagaaattcaccaccaaatagt2340
ttgacaccgtgtctaaagattcggaatatgtttgatcccgttatggaaataggggatcag2400
tggcatttggcaattcaagaagcaattttagaaaaatgcagtgataatgatggcattgtt2460
cacattgcagtagacaaaaattcacgtgagggttgtgtatatgttaaatgtctgtctcca2520
gaatatgctggaaaggcttttaaagcattgcatggctcttggtttgatgggaaattggtt2580
acagtaaaatatttacgactagatagataccaccatcgctttccccaggctctcacttcc2640
aacactccattgaagccatcaaataaacatatgaactccatgtctcatcttcgtcttcgg2700
actggcctaaccaattctcaaggaagttcctga2733
<210>4
<211>2733
<212>dna
<213>homosapiens
<400>4
atggcggcggcagcagcttcggcgcctcagcagctctcggatgaggagcttttctctcag60
ctccgccgttacggcctgtctcccggaccagtgacggagagcacccgcccggtctacctc120
aagaagctgaagaagcttcgagaggaagagcagcaacagcaccggtcagggggccgcggc180
aacaagacgcggaacagtaataacaataacacggcagccgccacggtcgcagccgcggga240
ccagcggcggcggcggccgcggggatgggggtccggccggtctcgggcgacctctcctac300
ttacggactcctgggggcctgtgccgaatctcggcctctggcccagagagcctcctggga360
gggcccgggggcgcctccgccgcccccgcggctggcagcaaagtgctgctgggcttcagc420
tcggacgagtcggacgtggaggccagtccccgggaccaggccggcggcggcgggaggaaa480
gaccgggcttcgctccagtaccgcgggctcaaagcgccgccggcgcccctggccgccagc540
gaggtgactaacagcaactctgcagagcgaaggaagccccactcgtggtggggggccagg600
aggccggcgggccccgagctgcagaccccgccggggaaagatggagcagtggaggacgag660
gaaggggagggagaggacggtgaggagagggacccggagaccgaggagccgctctgggcg720
agccggaccgtgaatggcagccggcttgtcccctacagctgccgggaaaactattcggac780
tcagaggaagaggacgacgacgacgtggcctccagcagacaggtattaaaggacgactcc840
ctttcccggcatcggcccagacgaacccatagtaagcctctccccccgctgactgctaaa900
tcggccggcggcaggctggagacttcagttcagggagggggaggactcgcgatgaatgac960
agggcggcggctgccgggagtctagacaggagccgaaacctcgaagaggcggcggccgcg1020
gagcagggaggagggtgtgatcaagtggactccagccccgttcctagataccgtgttaac1080
gctaagaaactgacccctctcctgcccccgccacttactgacatggactcaaccttggat1140
tcgtcaacaggctcccttctgaaaaccaataatcatattggcggtggggccttcagtgtg1200
gactcccccaggatttattctaacagtctccctcccagtgcggcggtggccgcctctagt1260
tcactcaggatcaatcacgccaatcatacgggctccaatcatacctacctgaaaaacaca1320
tacaacaaaccgaagctttccgaacccgaagaggaacttctccagcaatttaaacgggag1380
gaggtgtccccaacagggagtttcagtgcccactacttgtcgatgtttctcttaactgct1440
gcctgcttatttttcctaatactgggactgacttacctaggaatgagagggacaggagta1500
tctgaggatggagaactcagcaaaaacccctttggtgaaacatttggaaaaatacaagaa1560
agtgaaaaaactcttatgatgaacacattatataagcttcatgatcgattggcacagctt1620
gcaggagatcatgaatgtggcagttctagtcaaagaacgctttctgttcaagaggcagct1680
gcgtatttaaaagatttaggtcctgaatatgaaggtatatttaacacttcattgcagtgg1740
atcttagaaaatggaaaagatgttggaataaggtgtgttggttttggccctgaggaagaa1800
ttgacaaatataactgatgtgcagtttttacagtccacaagaccactgatgtctttttgg1860
tgtcgttttcgacgtgcttttgttactgtaactcacagattattgttgttatgcttaggt1920
gtagtgatggtttgtgtcgttctgcgttacatgaaatatcgatggacaaaagaagaggag1980
gaaacaaggcagatgtatgatatggtggtaaagattatagatgttttacgaagtcataat2040
gaagcctgccaggaaaacaaagatttacaaccttacatgcctattccacatgtacgcgat2100
tccttaatacagcctcatgacaggaaaaaaatgaagaaagtctgggatagagctgttgac2160
ttccttgctgctaatgagtctagagttcgcacggaaacacgaagaataggtggtgcagat2220
tttctggtttggcggtggatccagccttctgcatcctgtgacaaaatattagttatacct2280
tctaaagtatggcaaggtcaagcatttcatttagatagaagaaattcaccaccaaatagt2340
ttgacaccgtgtctaaagattcggaatatgtttgatcccgttatggaaataggggatcag2400
tggcatttggcaattcaagaagcaattttagaaaaatgcagtgataatgatggcattgtt2460
cacattgcagtagacaaaaattcacgtgagggttgtgtatatgttaaatgtctgtctcca2520
gaatatgctggaaaggcttttaaagcattgcatggctcttggtttgatgggaaattggtt2580
acagtaaaatatttacgactagatagataccaccatcgctttccccaggctctcagttcc2640
aacactccattgaagccatcaaataaacatatgaactccatgtctcatcttcgtcttcgg2700
actggcctaaccaattctcaaggaagttcctga2733
<210>5
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>5
acaggacagatacttacatactgct25
<210>6
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>6
acgccatctttgcacgaaaaacgca25
<210>7
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>7
cctcttacctcagttacaatttata25
<210>8
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>8
ttcagtgcccactacttgtcc21
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
cagggtcaccaccaatcaac20
- 还没有人留言评论。精彩留言会获得点赞!