高粱抗逆性相关基因SbERECTA及其编码蛋白与应用的制作方法
本发明涉及基因工程领域,特别是涉及一个高粱抗逆性相关基因sberecta、其编码蛋白及其在植物抗逆中的应用。
背景技术:
高粱(sorghumbiocolorl.moench)是畜牧业一种很好的饲草来源,既可做牧草放牧,又可刈割做青饲、青贮和干草。近年来,由于过度放牧,土壤风蚀、水蚀、盐碱化加重,以天然草地为主的草原畜牧业已没有优势可言。发展饲用高粱,对缓解由于畜多草少而造成的草原生态环境失衡具有重要意义。我国主栽高粱品种遗传资源丰富,主要性状(生物产量、籽粒产量等)变异幅度大,挖掘和鉴定高粱关键功能基因,对培育生物产量高、抗逆性强的饲用高粱新品种,解决土地资源贫瘠地区农业发展、畜牧业养殖、生物新能源开发等具有重要意义。
作物抗逆的机制是多基因参与的协同防御反应,包括外界逆境信号感知和传递机制、逆境胁迫的分子响应机制等。逆境会影响作物的各种生理生化过程,如抑制光合作用、改变细胞膜稳定性、改变激素和次级代谢物合成,甚至导致植株死亡。因此,研究高粱抗逆的功能基因,对于增加高粱的光合效率,提高高粱的生物学产量具有重要意义。
erecta基因可以调控拟南芥的气孔发育,影响植株的蒸腾效率和光合效率,增加植株的耐热性,是一个重要的功能基因。erecta蛋白是一个类受体激酶,主要参与调控植物的细胞分裂和组织发育。在拟南芥中过量表达erecta基因后,叶片气孔密度降低,叶面积增大,水分利用效率(wue)提高,株高增加,生物量显著增加,进一步研究发现erecta基因参与植物器官建成、细胞膨大和细胞间相关联系。另外,erecta基因能影响二磷酸核酮糖羧化酶(rubisco)的羧化速率和光电子转移能力,调节植物的光合作用,从杨树中克隆的pderecta基因转入拟南芥,拟南芥气孔密度降低,蒸腾效率增加,光合速率提高,生物量增加。erecta基因还参与了调控植物的抗病性,增强植株耐热性,如对稻瘟病产生非寄主性的抗性,过量表达显著提高番茄和水稻的耐热性和生物量。因此,erecta基因不仅参与调控气孔密度,影响植物的光合作用和生物量,又参与调控植物的抗病性和耐热性,是一个多功能基因。然而,迄今为止,尚未见到饲草作物,如高粱、高丹草等有关该基因的报道。为了深入剖析高粱sberecta(sber)基因功能,本研究对sber基因进行分离和初步的功能验证。
技术实现要素:
本发明提供一种高粱抗逆性相关sberecta蛋白。
其为:1)由seqidno.1所示的氨基酸序列组成的蛋白质(由982个aa组成);或2)在seqidno.1所示的氨基酸序列中经取代、缺失或添加一个或几个氨基酸且具有同等活性的由1)衍生的蛋白质。
本发明的另一目的是提供编码上述高粱抗逆性相关sberecta蛋白的sberecta基因。
sberecta基因具有seqidno.2所示的核苷酸序列(由3210个核苷酸组成)。本发明的高粱抗逆性相关sberecta基因是从高粱中克隆到的一个基因。
应当理解,本领域技术人员可根据本发明公开的氨基酸序列,在不影响其活性的前提下,取代、缺失和/或增加一个或几个氨基酸,得到所述蛋白的突变序列。
因此,本发明的高粱抗逆性相关sberecta蛋白还包括seqidno.1所示氨基酸序列经取代、替换和/或增加一个或几个氨基酸,具有高粱抗逆性相关sberecta蛋白同等活性的由高粱抗逆性相关sberecta蛋白衍生得到的蛋白质。本发明基因包括编码所述蛋白的核酸序列。此外,应理解,考虑到密码子的简并性以及不同物种密码子的偏爱性,本领域技术人员可以根据需要使用适合特定物种表达的密码子。
本发明的另一个目的是提供携带sberecta基因的植物表达载体,所述的植物表达载体为prres14-gsber。
本发明的sberecta基因,它编码的蛋白序列与玉米、水稻、高粱、小麦、大麦、二穗短柄草、拟南芥等的氨基酸序列之间的相似性相似度在80%以上,lrr区氨基酸序列非常保守;中部约570位到600位的氨基酸位点是该蛋白的跨膜区域,与其他物种的氨基酸序列差异较大,推测不同物种rlks的跨膜区功能差异性较大;3’末端约268个氨基酸是亮氨酸/苏氨酸的激酶区域,序列非常保守,包括典型的甘氨酸(g)、赖氨酸(k)和天冬氨酸(d),这些结构域与细胞分裂和增值、细胞凋亡和分化都有重要关系。这些结果暗示sberecta对于调控高粱生长发育具有关键作用。在高粱不同组织器官中,sberecta基因的表达量差异很大:在花梗组织中,sberecta基因表达量非常高;在幼穗中,sberecta基因表达量较高,但在幼茎、叶鞘和子房中,sberecta基因表达量明显降低,而在幼叶、花药和种子中,目的基因表达量较低,在根中几乎检测不到sberecta基因的表达。这些结果说明sberecta基因在未成熟的组织器官中具有较大的转录量,且在地上快速分化发育的幼嫩组织中,转录量极为丰富,暗示sberecta在高粱幼苗发育的早期就参与了植株的发育和形态构建成。在aba、brs、ga3和iaa处理下,高粱内源基因sbactin的相对表达量很稳定,变化不明显,而目标基因sberecta相对表达量较大,在不同处理时间段内表达差异水平达到极显著,且随着激素处理时间的延长,sberecta基因表达量呈现先增加后降低的趋势。
本发明将sberecta基因构建到表达载体pmd18-t上,获得到prres14-gsber过表达载体,并在大肠杆菌中扩繁。通过农杆菌介导转化方法,将prres14-gsber携带的sberecta基因转入模式植物拟南芥中,获得拟南芥转化植株。结果表明,sberecta早期就参与了植株的发育和形态构建成,具有提高转基因拟南芥的耐高温特性的作用。
本发明还提供含有sberecta核苷酸序列或其片段的克隆载体或各类表达载体、含有所述载体的宿主细胞、含有所述核苷酸序列或其特异片段的转化植物细胞和转基因植物。
本发明的另一个目的是提供sberecta基因及其编码蛋白sberecta在提高植物抗逆性,尤其是耐高温特性中的应用。
本发明的有益效果在于:
(1)提供了高粱sberecta基因及其编码蛋白sberecta;
(2)通过在植物中过表达sberecta基因提高植物抗逆性,尤其耐高温特性。
因此,sberecta基因及其编码的蛋白可以提高植物抗逆性,尤其耐高温特性。
附图说明
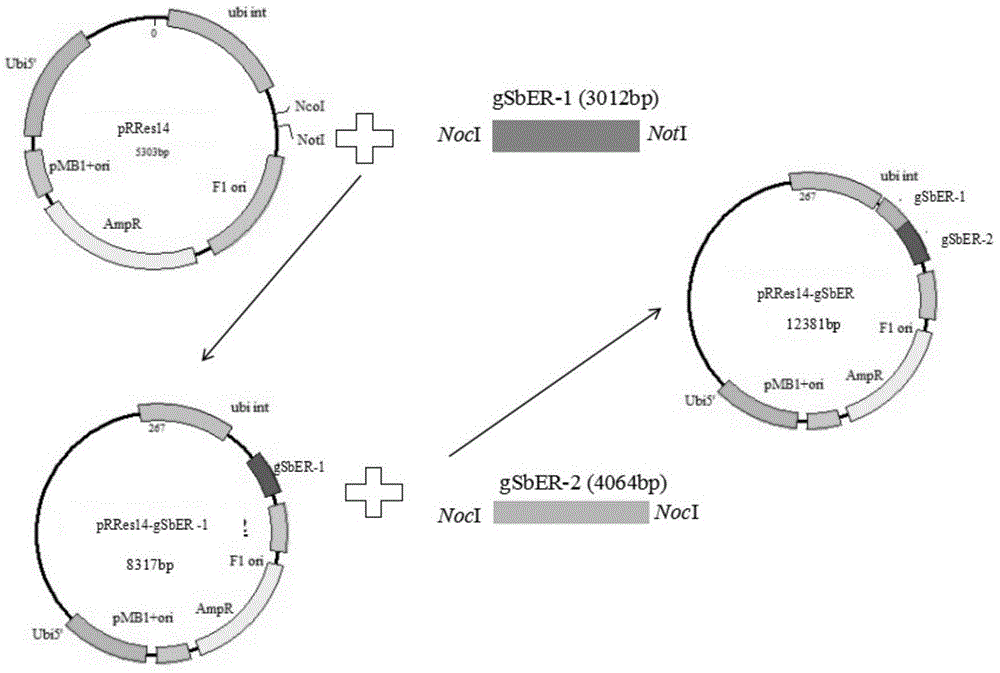
图1为本发明实施例1中高粱抗逆性相关基因sberecta的prres14-gsber重组载体结构示意图。
图2为本发明sberecta基因cdna和氨基酸序列分析示意图。
图3为高粱抗逆性相关基因sberecta保守区的结构域示意图。其中,lrrnt为亮氨酸富集区的n端;lrrdomain为亮氨酸串联区;transmembranedomain为跨膜区;s_tkc为丝氨酸/苏氨酸蛋白激酶区,催化区域;np_bind为atp结合位点(富含赖氨酸残基);act_site为质子接受位点(富含天冬氨酸残基)。
图4为sberecta基因的cdna和gdna序列分析示意图。
图5为sberecta氨基酸序列比对。其中,黑色直线将sberecta亮氨酸串联区和丝氨酸/苏氨酸激酶区分割开来。十字信号表示sberecta与其他物种rlks家族高度保守的氨基酸序列;箭头表示sberecta激酶区典型的氨基酸。每一个比对物种缩写号如下:taer=小麦;hv=大麦;bd=二穗短柄草;sber=高粱;zm=玉米;os=水稻;rc=蓖麻;vv=葡萄;gm=大豆;at=拟南芥;mt=苜蓿;cs=黄瓜;pt=杨树;sm=卷柏;pp=苔藓。
图6为高粱抗逆性相关基因sberecta组织特异性表达柱状图。
图7为高粱抗逆性相关基因sberecta在外源激素下的表达模式线条图。
图8为转sberecta基因的拟南芥幼苗高温处理后4个时间段的植株照片。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。
实施例1:高粱抗逆性相关基因sberecta的提取
1、实验材料
2016年4月,在安徽科技学院种植园播种高粱品种tx623b(已知高粱品种,可从育种单位购买)。在苗期取幼根、幼茎、幼叶和叶鞘样品,在开花初期取幼穗、花梗、花药和子房样品,在成熟期取种子样品,均在液氮中速冻,-80℃冰箱中保存备用,用以分析sberecta基因在高粱不同组织中的表达特性,并分离sberecta基因。
同时,高粱tx623b种子播种土壤中(营养土:蛭石=2:1),在光照培养箱培养。培养箱环境温度26℃,湿度50%-70%,白天光照16h,夜间光照8h,光强525μmol.s-1.m-2。10d以后,将幼苗从土壤中轻轻带土取出,清水冲洗干净,不要破坏根系,用滤纸清理水分后,立刻分别置于aba(100μm/l),brs(0.75μm/l),ga3(30mm/l)和iaa(10μm/l)溶液中培养,对照组利用清水处理。处理时间段为0h、1h、2h、4h、6h、12h、24h、48h和60h后,对照组和处理组分别取样,取样时将植株的根、茎和叶混合,每个样品取三株,作为生物学重复,液氮速冻,-80℃保存备用,分析sberecta基因在四种外源激素下的表达模式。
2、实验方法
2.1高粱rna的提取和cdna的合成
(1)rna提取(酚-氯仿-异戊醇法)
①液氮研磨植物组织,加入rna提取液,振荡器充分震荡;
②加入0.5ml的氯仿/异戊醇(24:1),继续涡旋30sec,4℃离心5min;
③上层水相转移到另一个新的2.0ml的离心管中,加入同等体积(约1ml)的氯仿/异戊醇(24:1),涡旋30s,4℃离心5min;
④上层水相转移到一个新的2.0ml的离心管中,微加入同等体积的的licl(4m),混匀后在冰箱里4℃温育过夜;
⑤4℃离心20min,弃掉上清,加入1ml70%的酒精,漂洗沉淀,在通风橱充分晾干后,去除样品中dna杂质;
⑥加入一样体积的苯酚/氯仿/异戊醇(25:24:1)涡旋混匀,离心5min;上清转移至新的离心管中;
⑦加入一样体积的氯仿/异戊醇(24:1),涡旋混匀,离心5min;上清转移至新的离心管中;
⑧加入2.5体积的无水乙醇和1/10体积的naoac溶液(3mph=5.2),-80℃下1小时;
⑨4℃离心20min,弃掉上清,1ml70%的酒精漂洗沉淀,弃上清,在通风橱充分晾干,加入20μlh2odepc溶解;
⑩nanodrop测定rna浓度,取1μgrna样品,琼脂糖电泳检测,保存rna样品在-80℃冰箱中备用。
(2)cdna的合成和检测
取5μg的总rna,利用(oligodt)12-18引物,合成的cdna原样,合成步骤参照takara公司的primescripttmii1ststrandcdnasynthesiskit试剂盒(6120a)说明书。合成的cdna样品用depc水稀释5倍后分装,-20℃冰箱保存。
2.2sberecta分离和序列分析
(1)sberecta序列分析
参照ncbi公布的小麦的taerecta序列(genebankaccessionjq599261.2),在phytozome(https://phytozome.jgi.doe.gov/pz/portal.html#)数据库中比对分析,获得高粱的sberecta基因的cdna序列和gdna序列。sberecta家族氨基酸翻译位点采用geneious6.0.5软件推测,sberecta家族的功能结构域分析采用conserveddomainsearch(http://www.ncbi.nlm.nih.gov/structure/cdd/wrpsb.cgi)和smart(http://smart.embl-heidelberg.de)数据库等进行。
(2)sberecta基因组织特异性表达和外源激素下的表达模式
利用采用实施定量(qrt-pcr)检测sberecta基因组织特异性表达和外源激素下的表达模式,目标基因sberecta的引物为sberectaq-f:gccagccgtgccaagctacatc(seqidno.3)和sberectaq-r:ctccctgcatccttcaactccc(seqidno.4)。内参基因sbactin的引物为sbactin-f:acggcctggatggcgacgtacatg(seqidno.5)和sbactin-r:gcagaaggacgcctacgttggtgac(seqidno.6)。利用abi7300(appliedbiosystems,usa)进行qrt-pcr分析,每个样品三次技术重复,反应体系为20μl,包括10μl2×sybrmix;0.3μl上游引物(10μm);0.3μl下游游引物(10μm);2μl(50ng)稀释的cdna模板,ddh2o补足20μl。反应程序为:95℃20s;95℃5s,61℃30s,40个循环。qrt-pcr数据分析参见以下公式(1):
公式中ex是目的基因引物的扩增效率,er看家基因引物的扩增效率,ct,x和ct,r分别为目的基因和看家基因在pcr反应过程中收集的ct值。如果有多个看家基因,则公式分母为多个看家基因计算数值的几何平均数(ramakersetal.2003;olsenetal.2012;rieuandpowers2009)。sberecta基因表达量的差异显著性分析利用spss19.0软件(ibmspssstatistics,usa)。
(3)sberecta基因全长cdna和gdna序列的分离
1)基因扩增
参照phytozome数据库中sberecta的序列设计引物,cdna序列分离引物分别为sbercdna-f:gcgagaacccacgaaaccaacc(seqidno.7)和sbercdna-rf:cccatgattccaacacggcaaa(seqidno.8);
gdna序列较大,采用分段扩增,中间设有一段重叠的区域,然后酶切连接在一起,引物分别为sbergdna-1f:atgcctgtccgcagctcagtggcc(seqidno.9)和sbergdna-1r:ggtggtatcgacccagttaacttattg(seqidno.10);sbergdna-2f:caataagttaactgggtcgataccacc(seqidno.11)和sbergdna-2r:ccccagacctctctctcttccctac(seqidno.12);利用高保真酶(kodfx-101,toyobo),以高粱tx623b的花梗组织cdna和dna为模板,进行pcr扩增,cdna序列长度为3376bp,gdna序列长度为7276bp,反应程序:94℃2min,35cycles(98℃10s,55℃30s,68℃1mim/kb),72℃10min,4℃保存。目的片段pcr产物的纯化按照百泰克试剂盒(dp1711)的说明进行。
2)连接转化
①如果纯化后的pcr产物为平末端,则需要在产物末端加上ploya尾巴,方便后续ta克隆连接,反应体系10μl:pcr产物6μl,taqbuffer2μl,taqpolymerase1μl,datp(10mm)0.2μl,ddh2o0.8μl,室温或者37℃反应30min后,70℃继续30min终止反应;
②将上述的pcr产物连接到克隆载体pmd18-t(6011,takara)上,反应体系为12μl,pcr产物6μl(适当调整),pmd18-tcloningvector1μl,solutioni溶液5μl;
③轻轻混匀,16℃连接30min,反应结束,将离心管置于冰上;
④加连接产物转入50μltop10感受态细胞中(提前在冰上慢慢解冻);
⑤42℃热激40s,立即置于冰上2min;
⑥加入150μl液体lb(无抗生素),200rpm,37℃孵育1h;
⑦将转化的菌液均匀的涂在lb平板上(含有100μg/mlamp+,并附加有40μl20mg/ml的x-gal和60μl200mg/ml的iptg);
⑧挑取白色克隆,摇菌备用。
3)酶切鉴定,测序
分别吸取50μl菌液,于5mllb液体培养基(含有终浓度100μg/mlamp+)中培养12-16h,按照质粒提取说明书(9760,takara)提取质粒,双酶切鉴定,片段大小正确,送样测序分析,测序正确,sberecta基因全长cdna和gdna序列片段分离成功。
4)超表达载体构建和拟南芥的转化
分段将sberecta基因gdna序列插入过表达载体(含有ubi组成型启动子),获得到prres14-gsber过表达载体(具体结构见图1)。结合载体的多克隆位点,利用限制酶ncoi和noti酶切第一片段gsber-1的质粒,限制酶ncoi酶切第二片段gsber-2的质粒,均回收酶切产物。酶切反应体系20μl:noti1μl,10×bufferh2μl,0.1%bsa2μl,0.1%
利用限制酶ncoi和noti酶切过表达载体prres14,然后将上面回收的gsber-1片段插入线性载体prres14,连接反应体系15μl:5×ligasereactionbuffer3μl,载体dna1.5μl,片段dna6μl,t4dnaligase(1units)1.5μl,ddh2o3μl,4℃过夜连接。测序分析,获得prres14-gsber-1重组载体。
利用限制酶ncoi酶切prres14-gsber-1重组载体,然后将上面回收的gsber-2片段插入线性载体prres14.101-gsber-1,连接体系如上。测序分析,ncoi和noti双酶切鉴定,获得prres14-gsber过表达重组载体。
提取prres14-gsber质粒,转化农杆菌,然后利用农杆菌侵染拟南芥(哥伦比亚0型)的花絮,获得sberecta过表达转基因拟南芥种子,连续筛选种植三代,获得t3带转基因拟南芥种子,备用。
5)转基因拟南芥高温处理
将t3代的转基因拟南芥和野生型拟南芥种子分别播种在空ms培养基上,组培室生长10d后,选取根长等长的转基因拟南芥和野生型拟南芥幼苗分别移栽到土壤(营养土:蛭石=2:1)中,在光照培养箱培养,培养箱环境温度26℃,湿度50%-70%,白天光照16h,夜间光照8h,光强525μmol.s-1.m-2。5天后,42℃高温处理不同的时间段,后放置培养箱继续培养10d,观察统计幼苗的复活情况。
实施例2:高粱抗逆性相关基因sberecta的cdna序列分析及其编码蛋白的氨基酸序列分析
1、cdna序列和氨基氨酸分析
本发明sberecta基因cdna和氨基酸序列分析示意图见图2,sberecta基因cdna序列含有2949bp开放的阅读框,编码983氨基酸,预测蛋白分子量106kda,等电点(pi)为6.21。进一步分析其编码的蛋白发现,sberecta属于典型的类受体激酶家族(rlks),n端是亮氨酸串联的富集区域,接受胞外信号转导,中间是疏水结构,是跨膜区,c端是丝氨酸/苏氨酸蛋白激酶区。高粱抗逆性相关基因sberecta保守区的结构域示意图见图3,其中,lrrnt为亮氨酸富集区的n端;lrrdomain为亮氨酸串联区;transmembranedomain为跨膜区;s_tkc为丝氨酸/苏氨酸蛋白激酶区,催化区域;np_bind为atp结合位点(富含赖氨酸残基);act_site为质子接受位点(富含天冬氨酸残基。sberecta基因gdna序列含有7076bp。
2、选择性剪切的特点分析
sberecta基因的cdna和gdna序列分析示意图见图4,比对sberecta基因cdna序列和gdna序列之后,发现sberecta的gdna序列具有27个外显子,26个内含子。然而,sberecta的cdna序列存在不同的内含子和外显子剪切体形式。深入分析表明,sberecta在gdna序列4127-4497bp区间内存在三种cdna序列的剪切体形式:①sb-cdna1,长度为3110,27个外显子,含有两个内含子;②sb-cdna2,长度为2880bp,26个外显子,无内含子;③sb-cdna3,长度2949,27个外显子,无内含子。
3、sberecta家族氨基酸序列结构分析
在phytozomev9.0数据库中,以高粱sberecta氨基酸序列为探针进行比对,下载玉米、水稻、高粱、小麦、大麦、二穗短柄草、拟南芥等相似度在80%以上的rlks家族氨基酸,具体氨基酸序列比对图见图5,其中,黑色直线将sberecta亮氨酸串联区和丝氨酸/苏氨酸激酶区分割开来。十字信号表示sberecta与其他物种rlks家族高度保守的氨基酸序列;箭头表示sberecta激酶区典型的氨基酸。每一个比对物种缩写号如下:taer=小麦;hv=大麦;bd=二穗短柄草;sber=高粱;zm=玉米;os=水稻;rc=蓖麻;vv=葡萄;gm=大豆;at=拟南芥;mt=苜蓿;cs=黄瓜;pt=杨树;sm=卷柏;pp=苔藓。多序列比对预测:sberecta与其他物种一样,lrr区氨基酸序列非常保守;中部约570位到600位的氨基酸位点是该蛋白的跨膜区域,与其他物种的氨基酸序列差异较大,推测不同物种rlks的跨膜区功能差异性较大;3’末端约268个氨基酸是亮氨酸/苏氨酸的激酶区域,序列非常保守,包括典型的甘氨酸(g)、赖氨酸(k)和天冬氨酸(d),这些结构域与细胞分裂和增值、细胞凋亡和分化都有重要关系。这些结果暗示sberecta对于调控高粱生长发育具有关键作用。
实施例3:sberecta基因组织特异性表达
在各个发育时期,收集高粱不同组织器官材料,利用sbactin内参基因作为参照,研究sberecta基因在不同高粱组织中相对表达情况,特异性表达柱状图见图6。结果发现,在高粱不同组织器官中,sberecta基因的表达量差异很大:在花梗组织中,sberecta基因表达量非常高;在幼穗中,sberecta基因表达量较高,但在幼茎、叶鞘和子房中,sberecta基因表达量明显降低,而在幼叶、花药和种子中,目的基因表达量较低,在根中几乎检测不到sberecta基因的表达。这些结果说明sberecta基因在未成熟的组织器官中具有较大的转录量,且在地上快速分化发育的幼嫩组织中,转录量极为丰富,暗示sberecta在高粱幼苗发育的早期就参与了植株的发育和形态构建成。
实施例4:sberecta在外源激素下的表达模式
在aba、brs、ga3和iaa处理下,高粱内源基因sbactin的相对表达量很稳定,变化不明显,而目标基因sberecta相对表达量较大,在不同处理时间段内表达差异水平达到极显著,且随着激素处理时间的延长,高粱抗逆性相关基因sberecta在外源激素下的表达模式线条图见图7,sberecta基因表达量呈现先增加后降低的趋势。
实施例5:sberecta超表达拟南芥株系耐高温特性鉴定
转sberecta基因的拟南芥幼苗分别在42℃高温下处理4h、8h、12h和15h四个时间段。结果显示(图8),在高温4h后,转基因株系和野生型株系存活率都为60%;在高温8h和12h后,转基因株系和野生型株系的存活率分别都为40%和20%;在高温15h后,转基因株系和野生型株系存活率也分别为40%和20%,说明在高温胁迫下,超表达sberecta基因的拟南芥幼苗具有很强的生命力,在一定程度上可以抵抗高温带来的伤害,而野生型拟南芥不具有这种能力,初步证明sberecta基因的过表达可以提高转基因拟南芥的耐高温特性。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>安徽科技学院
<120>高粱抗逆性相关基因sberecta及其编码蛋白与应用
<160>12
<170>siposequencelisting1.0
<210>1
<211>982
<212>prt
<213>高粱(sorghumbicolor)
<400>1
metprovalargserservalalametthrthrthralaalaargala
151015
leuvalalaleuleuleuvalalavalalavalalaaspaspglyala
202530
thrleuvalgluilelyslysserpheargasnvalglyasnvalleu
354045
tyrasptrpalaglyaspasptyrcyssertrpargglyvalleucys
505560
aspasnvalthrphealavalalaalaleuasnleuserglyleuasn
65707580
leugluglygluileserproalavalglyserleulysserleuval
859095
serileaspleulysserasnglyleuserglyglnileproaspglu
100105110
ileglyaspcysserserleuargthrleuasppheserpheasnasn
115120125
leuaspglyaspilepropheserileserlysleulyshisleuglu
130135140
asnleuileleulysasnasnglnleuileglyalaileproserthr
145150155160
leuserglnleuproasnleulysileleuaspleualaglnasnlys
165170175
leuthrglygluileproargleuiletyrtrpasngluvalleugln
180185190
tyrleuglyleuargglyasnhisleugluglyserleuserproasp
195200205
metcysglnleuthrglyleutrptyrpheaspvallysasnasnser
210215220
leuthrglyvalileproaspthrileglyasncysthrserphegln
225230235240
valleuaspleusertyrasnargphethrglyproilepropheasn
245250255
ileglypheleuglnvalalathrleuserleuglnglyasnlysphe
260265270
thrglyproileproservalileglyleumetglnalaleualaval
275280285
leuaspleusertyrasnglnleuserglyproileproserileleu
290295300
glyasnleuthrtyrthrglulysleutyrileglnglyasnlysleu
305310315320
thrglyserileproprogluleuglyasnmetserthrleuhistyr
325330335
leugluleuasnaspasnglnleuthrglyserileproprogluleu
340345350
glyargleuthrglyleupheaspleuasnleualaasnasnhisleu
355360365
gluglyproileproaspasnleusersercysvalasnleuasnser
370375380
pheasnalatyrglyasnlysleuasnglythrileproargserleu
385390395400
arglysleuglusermetthrtyrleuasnleuserserasnpheile
405410415
serglyserileproilegluleuserargileasnasnleuaspthr
420425430
leuaspleusercysasnmetmetthrglyproileproserserile
435440445
glyserleugluhisleuleuargleuasnleuserlysasnglyleu
450455460
valglypheileproalaglupheglyasnleuargservalmetglu
465470475480
ileaspleusertyrasnhisleuglyglyleuileproglngluleu
485490495
glumetleuglnasnleumetleuleulysleugluasnasnasnile
500505510
thrglyaspleuserserleumetasncyspheserleuasnileleu
515520525
asnvalsertyrasnasnleualaglyvalvalproalaaspasnasn
530535540
phethrargpheserproaspserpheleuglyasnproglyleucys
545550555560
glytyrtrpleuglysersercysargserthrglyhishisglulys
565570575
proproileserlysalaalaileileglyvalalavalglyglyleu
580585590
valileleuleumetileleuvalalavalcysargprohisargpro
595600605
proalaphelysaspvalthrvalserlysprovalargasnalapro
610615620
prolysleuvalileleuhismetasnmetalaleuhisvaltyrasp
625630635640
aspilemetargmetthrgluasnleuserglulystyrileilegly
645650655
tyrglyalaserserthrvaltyrlyscysvalleulysasncyslys
660665670
provalalailelyslysleutyralahistyrproglnserleulys
675680685
gluphegluthrgluleugluthrvalglyserilelyshisargasn
690695700
leuvalserleuglnglytyrserleuserprovalglyasnleuleu
705710715720
phetyrasptyrmetglucysglyserleutrpaspvalleuhisglu
725730735
glyserserlyslyslyslysleuasptrpgluthrargleuargile
740745750
alaleuglyalaalaglnglyleualatyrleuhishisaspcysser
755760765
proargileilehisargaspvallysserlysasnileleuleuasp
770775780
lysasptyrglualahisleuthrasppheglyilealalysserleu
785790795800
cysvalserlysthrhisthrserthrtyrvalmetglythrilegly
805810815
tyrileaspproglutyralaargthrserargleuasnglulysser
820825830
aspvaltyrsertyrglyilevalleuleugluleuleuthrglylys
835840845
lysprovalaspasnglucysasnleuhishisleuileleuserlys
850855860
thralaserasngluvalmetaspthrvalaspproaspileglyasp
865870875880
thrcyslysaspleuglygluvallyslysleupheglnleualaleu
885890895
leucysthrlysargglnproseraspargprothrmethisgluval
900905910
valargvalleuaspcysleuvalasnproaspproproprolyspro
915920925
seralahisglnleuproglnproserproalavalprosertyrile
930935940
asnglutyrvalserleuargglythrglyalaleusercysalaasn
945950955960
serthrserthrseraspalagluleupheleulyspheglygluala
965970975
ileserglnasnmetglu
980
<210>2
<211>3210
<212>dna
<213>高粱(sorghumbicolor)
<400>2
gctctgcccccattaaggcagggcaggcagaggaggcaggctcgctgcagcacgcttcgc60
gctccatcgtcgctgtcctcttctccctgtaatgtcactcccccgatgcctgtccgcagc120
tcagtggccatgacgacgacggccgcccgtgctctcgtcgccctcctcctcgtcgccgtc180
gccgtcgccgacgatggggcgacgctggtggagatcaagaagtccttccgcaacgtcggc240
aacgtactgtacgattgggccggcgacgactactgctcctggcgcggcgtcctgtgcgac300
aacgtcacattcgccgtcgctgcgctcaacctctctggcctcaaccttgagggcgagatc360
tctccagccgtcggcagcctcaagagcctcgtctccatcgatctgaagtcaaatgggcta420
tccgggcagatccctgatgagattggtgattgttcatcacttaggacgctggacttttct480
ttcaacaacttggatggcgacataccattttctatatcaaagctgaagcacctggagaac540
ttgatattgaagaacaaccagctgattggtgcgatcccatcaacattgtcacagctccca600
aatttgaagattttggatttggcacaaaacaaactgactggggagataccaaggcttatc660
tactggaatgaggttcttcaatatctgggcttacggggcaatcatttagaaggaagcctc720
tctcctgatatgtgccagctgactggcctttggtactttgatgtgaagaacaatagcttg780
accggggtgataccagacaccattgggaactgtacaagttttcaagtcttggatttgtct840
tacaaccgctttactggaccaatcccattcaacattggtttcctacaagtggctacacta900
tccttgcaagggaacaagttcaccggtccaattccttcagtaattggtcttatgcaggct960
ctcgctgttctagatctgagttacaaccaattatctggtcctataccatcaatactaggc1020
aacttgacatacactgagaagctgtacatccaaggcaataagttaactgggtcgatacca1080
ccagagttaggaaatatgtcaacacttcattacctagaactgaacgataatcaacttact1140
gggtcaattccaccagagcttggaaggctaacaggcttgtttgacctgaaccttgcgaat1200
aaccacctggaaggaccaattcctgacaacctaagttcatgtgtgaatctcaatagcttc1260
aatgcttatggcaacaagttaaatgggaccattcctcgttcgttgcggaaacttgaaagc1320
atgacctatttaaatctgtcatcaaacttcataagtggctctattcctattgagttatca1380
aggatcaacaatttggacacgctggatttatcctgtaacatgatgactggtccaattcca1440
tcatcaattggcagcctagagcatctattgagacttaacttgagcaagaatggtctagtt1500
ggattcatccccgcggagtttggtaatttgaggagtgtcatggagattgatttatcctat1560
aatcaccttggtggcctgattcctcaagaacttgaaatgctgcaaaacctgatgttgcta1620
aaactggaaaataacaatataactggtgatctgtcgtctctgatgaactgcttcagcctc1680
aatatcttaaatgtgtcgtacaataatttggctggtgttgtccctgctgacaacaacttc1740
acacggttttcacctgacagctttttaggtaatcctggactctgtggatactggcttggt1800
tcgtcgtgtcgttccactggccaccacgagaaaccgcctatctcaaaggctgccataatt1860
ggtgttgctgtgggtggacttgttatcctcttgatgatcttagtagctgtttgcaggcca1920
catcgtccacctgcttttaaagatgtcactgtaagcaagccagtgagaaatgctcccccc1980
aagctggtgatccttcatatgaacatggcccttcatgtatacgatgacataatgaggatg2040
actgagaacttgagtgagaaatacatcattggatacggggcgtcaagtacagtttataaa2100
tgtgtcctaaagaattgcaaaccggtggcaataaaaaagctgtatgcccactacccacag2160
agccttaaggaatttgaaactgagcttgagactgttggtagcatcaagcaccggaatcta2220
gtcagccttcaagggtactcattatcacctgttgggaacctcctcttttatgattatatg2280
gaatgtggcagcttatgggatgttttacatgaaggttcatccaagaagaaaaaacttgac2340
tgggagactcgcctacggattgctcttggtgcagctcaaggccttgcttaccttcaccat2400
gactgcagtccacggataattcatcgggatgtaaaatcaaagaatatactccttgacaaa2460
gattatgaggcccatcttacagactttggaattgctaagagcttatgtgtctcaaaaact2520
cacacatcaacctatgtcatgggaactattggctacattgatcctgagtacgcccgcact2580
tcccgtctcaacgaaaagtctgatgtctacagctatggcattgttctgctggagctgctg2640
actggcaagaagccagtggacaacgagtgcaatctccatcacttgatcctatcgaagacg2700
gcaagcaacgaggtcatggataccgtggaccctgacatcggggacacctgcaaggacctc2760
ggcgaggtgaagaagctgttccagctggcgctcctttgcaccaagcggcaaccctcggac2820
cgaccgacgatgcacgaggtggtgcgcgtcctggactgcctggtgaacccggacccgccg2880
ccaaagccgtcggcgcaccagctgccgcagccgtcgccagccgtgccaagctacatcaac2940
gagtacgtcagcctgcggggcaccggcgctctctcctgcgccaactcgaccagcacctcg3000
gacgccgagctgttcctcaagttcggcgaggccatctcgcagaacatggagtagggaaga3060
gagagaggtctggggagttgaaggatgcagggagtagtgggagtagctgactgacatttt3120
gcgggatgcaggaggagattaacatggggaactcagtagggtgttggttaactgtaaaaa3180
aagtcatgtgcctctagagcagcagtagag3210
<210>3
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>3
gccagccgtgccaagctacatc22
<210>4
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>4
ctccctgcatccttcaactccc22
<210>5
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>5
acggcctggatggcgacgtacatg24
<210>6
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>6
gcagaaggacgcctacgttggtgac25
<210>7
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>7
gcgagaacccacgaaaccaacc22
<210>8
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>8
cccatgattccaacacggcaaa22
<210>9
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>9
atgcctgtccgcagctcagtggcc24
<210>10
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>10
ggtggtatcgacccagttaacttattg27
<210>11
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>11
caataagttaactgggtcgataccacc27
<210>12
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>12
ccccagacctctctctcttccctac25
- 还没有人留言评论。精彩留言会获得点赞!