一种优化后的猪圆环病毒3型衣壳蛋白基因及其在制备病毒样颗粒中的应用的制作方法
本发明涉及一种优化后的猪圆环病毒3型衣壳蛋白基因,还涉及该基因在制备病毒样颗粒中的应用。本发明属于动物基因工程与动物病毒学技术领域。
背景技术:
2015年6月,美国北卡罗来纳州一个商品猪场母猪群暴发皮炎肾病综合征(pdns),发病期间,该场母猪死亡率较历史平均水平增加了10.2%,受胎率减少了0.6%,窝均木乃伊胎相比历史平均水平增加1.19%,流产出的木乃伊胎大小不一。临床上,感染母猪厌食,皮肤多处出现丘疹性皮炎;组织结构上观察,皮肤损伤以急性坏死性皮炎为特点;肾皮质区肾小管扩张,管状黏膜上皮细胞退化,肾皮质间质及肾小球大量淋巴细胞及巨噬细胞弥漫性浸润。死亡母猪表现出皮炎肾病综合征(pdns)。以上关于母猪眼观病变以及组织学病变,包括流产症状,都与以前报道的圆环病毒相关疾病基本一致。然而所有母猪组织样本,包括肾脏、淋巴结、肺脏以及皮肤组织样本,经免疫组化(ihc)检测以及定量pcr(qpcr)检测发现pcv2,prrsv,iav均为阴性,此外,木乃伊胎组织定量pcr检测显示pcv2,prrsv,ppv同样都为阴性。在针对样本采用宏基因组测序后,发现一种新型,基因型不同以往的圆环病毒,命名为pcv3。与pcv1和pcv2一样,pcv3病毒包含2个主要开放阅读框(orf)。其中,orf1编码由297个氨基酸(aa)组成复制酶蛋白rep,orf2编码由214个aa组成衣壳蛋白cap,两者复制方向相反。pcv3阳性地区已经分布于世界各地,我国的34个省份有21个省份有pcv3阳性报道。可以预想,pcv3会对养猪业造成很大的损失。因此,提出对于pcv3有效的防控措施极其重要。然而pcv3具有极难在体外分离培养的特点,这对疫苗的研究造成极大的困难。
病毒样颗粒(virus-likeparticles,vlp)是利用重组表达的方法,在体外将重组病毒衣壳蛋白自组装成与天然病毒有相似结构的空心颗粒。vlp是制备病毒疫苗较为理想的重组蛋白抗原,它具备如下优点:①不含病毒核酸,无减毒病毒疫苗(liveattenuatedvirusvaccine)或灭活病毒疫苗(inactivatedvirusvaccine)的感染风险;②与天然病毒的外部结构高度相似因而具有与病毒颗粒相近的免疫原性;③具有病原体相关模式分子(pathogen-associatedmolecularpatterns,panp)的活性,可激活固有免疫应答;④其形态和大小有利于抗原提呈细胞特别是树突细胞的摄取;⑤可经mhci类和ii类分子提呈,激活cd4+辅助t细胞和cd8+细胞毒性t淋巴细胞;⑥能激活b细胞产生高滴度的抗体,也能激发t细胞非依赖的igm反应;⑦可用于递送toll样受体(toll-likereceptor,tlr)激动剂和t细胞表位;⑧本身可有佐剂功能;⑨作为vlp呈现平台(vlpdisplayplatforms)呈现其他抗原。因此,构建猪圆环病毒3型的病毒样颗粒可能为解决该病毒的广泛流行提供一个新的方向。
目前有很多表达重组蛋白的系统,包括大肠杆菌表达系统,酵母表达系统和杆状病毒表达系统等。其中,相比其他表达系统,大肠杆菌表达系统具有成本低,表达量大的优势,更适合大规模工业生产。在本发明申请之前,已经有4个以大肠杆菌表达pcv3的cap基因的相关发明专利申请,分别是,发明专利申请1(申请号201710184557.9,一种猪圆环病毒基因工程亚单位疫苗及其制备方法与应用),其方法是以大肠杆菌表达了包含表达载体上32个氨基酸+pcv3cap蛋白的重组融合蛋白,未提及形成了vlp;发明专利申请2(申请号201711420604.1,一种猪圆环病毒3型cap蛋白疫苗及其制备方法和应用),其方法是以大肠杆菌表达gst融合的去除了1-30氨基酸(核定位信号肽,nls)的pcv3cap蛋白,未提及形成了vlp;发明专利申请3(申请号201810420766.3,猪圆环病毒3型病毒样颗粒的制备方法及其应用),其方法是以大肠杆菌表达sumo标签融合的去除1-33氨基酸(nls)的pcv3cap蛋白,在纯化了sumo标签融合截短的cap蛋白后,经蛋白酶酶切去除sumo蛋白后自组装形成了vlp;发明专利申请4(申请号201810743651.8,一种猪圆环病毒3型基因工程亚单位疫苗及其制备方法),其方法是以大肠杆菌表达经过截短剪接改造的pcv3cap蛋白,纯化后的蛋白形成了vlp。
本发明设计了6套pcv3cap重组蛋白的基因优化表达方案,包括5套不同基因优化方案的pcv3cap蛋白完整全长基因与1套去除核定位信号的pcv3cap蛋白,从中最终筛选到1套pcv3cap蛋白完整全长基因经过部分基因优化后的方案为最优方案。与已经公开的4个专利申请的加标签、截短和截短后剪接改造的设计思路相比,本发明的pcv3cap蛋白为完整全长基因的优化序列(部分基因优化),不引入外源标签和进行截短及截短后剪接改造,能够在大肠杆菌中高效表达并能在大肠杆菌细胞中自组装形成vlp,与以上4个相关发明专利申请相比具有明显不同。其中,与发明专利申请1相比,我们保持了完整的pcv3cap蛋白,没有引入额外的表达载体上的多余其他氨基酸,更接近于天然的pcv3cap蛋白,而且形成了vlp;与发明专利申请2相比,我们保持了完整的pcv3cap蛋白,没有截短和融合gst标签,更接近于天然的pcv3cap蛋白,而且形成了vlp;与发明专利申请3相比,我们保持了完整的pcv3cap蛋白,没有截短,也不需要纯化后用蛋白酶酶切去除sumo标签蛋白后再进行体外组装成vlp,本发明专利形成的vlp更接近天然构象且组装效率更高;与发明专利申请4相比,我们保持了完整的pcv3cap蛋白,没有截短和剪接改造,更接近于天然的pcv3cap蛋白,形成的vlp更接近天然构象。在基因的同源性方面,只有发明专利申请3和4表明形成了vlp,因此我们将本发明的优化基因序列与发明专利申请3和4所公开的序列进行了比对,结果显示本发明的优化基因序列与发明专利申请3序列的同源性为90.3%,与发明专利申请4序列的同源性为72.6%。
总而言之,本发明从6套不同的pcv3cap重组蛋白的基因优化表达方案中优选出一套高效表达并且形成了vlp的方案,本发明和以上相关专利申请相比设计思路明显不同,使用的表达载体也不同,基因的序列也差异显著,纯化的方法也不一样,而且,以上4个专利申请均未用其表达的pcv3cap蛋白或形成的vlp建立相应的pcv3血清学抗体检测方法。建立相应的低交叉背景的血清学抗体检测方法需要较高纯度的纯化抗原,而本发明通过系列的层析纯化方法获得了较高纯度的pcv3vlp,建立了交叉背景极低的pcv3血清学抗体检测方法,具有广阔的应用前景。
技术实现要素:
本发明的目的是提供一种人工合成的猪圆环病毒3型衣壳蛋白全长基因,该基因与其他优化方法相比能够高水平表达重组蛋白并形成猪圆环病毒3型病毒样颗粒;
本发明的另一目的是提供一种制备猪圆环病毒3型重组衣壳蛋白及其形成病毒样颗粒的方法;
本发明的再一目的是提供一种猪圆环病毒3型病毒样颗粒在抗体检测以及亚单位疫苗制备中的应用。
为了达到上述目的,本发明采用了以下技术手段:
本发明设计了6套pcv3cap重组蛋白的基因优化方案,包括5套pcv3cap蛋白完整全长基因与1套去除核定位信号的pcv3cap蛋白截短基因。具体而言,方案1:在考虑到密码子偏爱性和gc含量等因素的基础上对pcv3cap蛋白完整全长基因进行部分优化;方案2:在考虑到密码子偏爱性和gc含量等因素的基础上对pcv3cap蛋白完整全长基因进行不同于方案1的部分优化,方案3:在考虑到密码子偏爱性和gc含量等因素的基础上对pcv3cap蛋白完整全长基因进行不同于方案1和2的部分优化;方案4:将pcv3cap蛋白完整全长基因中的非大肠杆菌偏好密码子全部替换为偏好密码子;方案5:对pcv3cap蛋白完整全长基因不进行优化;方案6:在考虑到密码子偏爱性和gc含量等因素的基础上对去除信号肽的pcv3cap蛋白截短基因进行部分优化。以上6套不同优化表达方案的结果是:方案5的表达量极低;方案4的表达量也未达到期望的高效表达水平;方案1、2、3和4的表达量比方案5都要好,但其中方案1表达量最高,而且可以自组装形成vlp;另外,方案6的表达量也很高,但方案6的纯化产物未见形成vlp,根据我们多次重复的结果,去除信号肽后的pcv3cap蛋白截短的重组蛋白,在电子显微镜下始终没有看到病毒样颗粒。综上可见,方案1是我们设计的6套方案中的最优方案。
因此,在上述研究的基础上,本发明提出了一种优化后的猪圆环病毒3型衣壳蛋白全长基因,所述基因的核苷酸序列如seqidno.1所示。
含有所述的优化后的猪圆环病毒3型衣壳蛋白全长基因表达载体,以及含有所述的表达载体的宿主细胞也在本发明的保护范围之内。
进一步的,本发明还提出了所述的优化后的猪圆环病毒3型衣壳蛋白全长基因在制备猪圆环病毒3型病毒样颗粒中的应用。
一种制备猪圆环病毒3型病毒样颗粒的方法,包括以下步骤:
1)合成本发明所述的优化后的猪圆环病毒3型衣壳蛋白全长基因;
2)将合成的基因连接至pet30a载体,产物转化到e.colidh5α感受态细胞,挑菌鉴定后抽提质粒,重组质粒命名为pet30a-rcap;
3)将pet30a-rcap质粒转入大肠杆菌表达菌株bl21(de3),培养后,得到含有pet30a-rcap质粒的大肠杆菌菌液;
4)将菌液接种到含卡那霉素的液体tb培养基中,37℃,220rpm摇床上培养2h。然后加入终浓度为0.4mmol/l的iptg,25℃,220rpm进行诱导表达20h,随后离心收集菌液;
5)破碎步骤4)中收集的菌液上清,经两步层析纯化,包括第一步阴离子交换层析和第二步凝胶过滤层析,得到猪圆环病毒3型病毒样颗粒。
按照所述的方法制备得到的猪圆环病毒3型病毒样颗粒也在本发明的保护范围之内。
进一步的,本发明还提出了所述的猪圆环病毒3型病毒样颗粒在制备猪圆环病毒3型抗体检测试剂中的应用。以及
所述的猪圆环病毒3型病毒样颗粒在制备猪圆环病毒3型亚单位疫苗中的应用。
相较于现有技术,本发明的有益效果是:
1)本发明是国内外有关猪圆环病毒3型完整衣壳蛋白优化的基因在大肠杆菌中高效表达并在细胞内自组装成为病毒样颗粒的首次报道。
2)本发明表达的猪圆环病毒3型重组衣壳蛋白及其形成的病毒样颗粒将为猪圆环病毒3型的抗体检测及亚单位疫苗的研制打下良好的基础。
3)本发明方法简单易行,成本较低。
附图说明
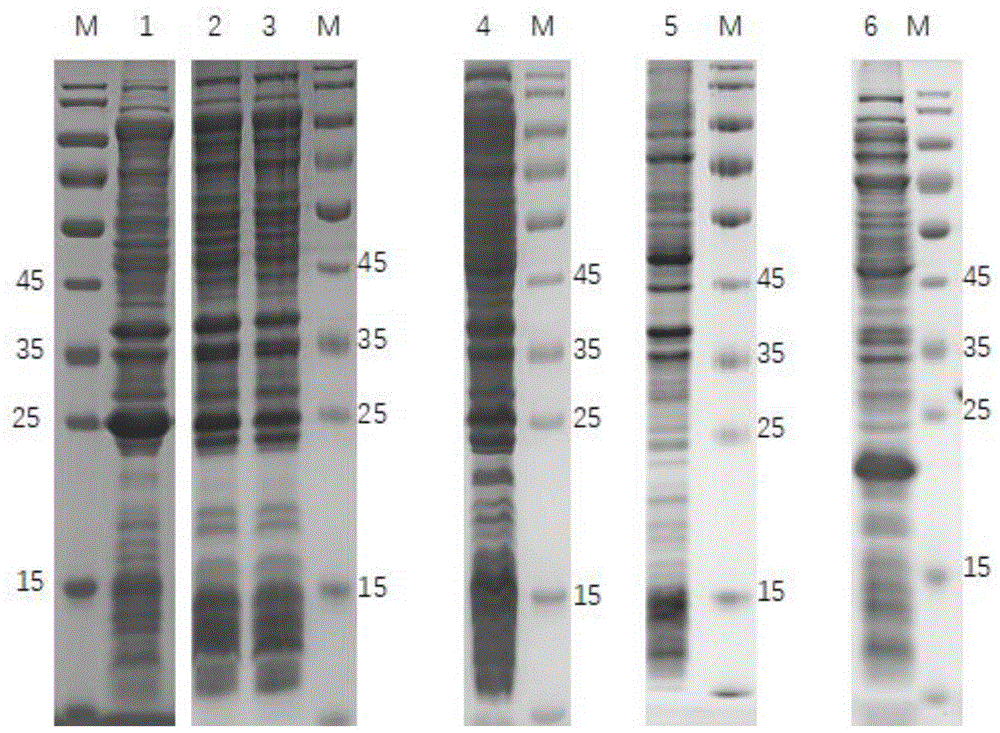
图1为大肠杆菌表达的本发明猪圆环病毒3型重组衣壳蛋白的sds-page结果;
其中:m为预染蛋白marker;1为seqidno.1所示序列表达结果;2为seqidno.2所示序列表达结果;3为seqidno.3所示序列表达结果;4为seqidno.4所示序列表达结果;5为seqidno.5所示序列表达结果;6为seqidno.6所示序列表达结果;
图2为seqidno.1所示序列高效表达重组蛋白并形成病毒样颗粒的电镜结果;
其中:a为100nm视野结果,b为200nm视野结果;
图3为seqidno.6所示序列高效表达重组蛋白但未形成病毒样颗粒的电镜结果;
图4为大肠杆菌表达的本发明猪圆环病毒3型重组衣壳蛋白在第一步离子交换层析纯化结果;
图5为大肠杆菌表达的本发明猪圆环病毒3型重组衣壳蛋白在第二步凝胶过滤层析纯化结果;
图6为基于本发明最终筛选的猪圆环病毒3型病毒样颗粒的间接elisa方法棋盘法优化结果;
图7为本发明基于猪圆环病毒3型病毒样颗粒的间接elisa方法特异性(a)和敏感性(b)实验结果;
其中:图7b中a-f为不同强度的pcv3阳性血清;
图8为基于本发明猪圆环病毒3型病毒样颗粒的间接elisa方法对373份临床猪血清抗体检测结果。
具体实施方式
下面通过实验并结合实施例对本发明做进一步说明,应该理解的是,这些实施例仅用于例证的目的,绝不限制本发明的保护范围。本领域普通技术人员理解,在本发明权利要求所设定的精神和范围内可对其进行许多改变,修改,甚至等效变更,但都将落入本发明的保护范围内。
实施例1经过优化的猪圆环病毒3型衣壳蛋白基因的合成
本发明设计了6套pcv3cap重组蛋白的基因优化方案,包括5套pcv3cap蛋白完整全长基因与1套去除核定位信号的pcv3cap蛋白截短基因。具体而言,方案1:在考虑到密码子偏爱性和gc含量等因素的基础上对pcv3cap蛋白完整全长基因进行部分优化,优化后的基因的核苷酸序列如seqidno.1所示;方案2:在考虑到密码子偏爱性和gc含量等因素的基础上对pcv3cap蛋白完整全长基因进行不同于方案1的部分优化,优化后的基因的核苷酸序列如seqidno.2所示;方案3:在考虑到密码子偏爱性和gc含量等因素的基础上对pcv3cap蛋白完整全长基因进行不同于方案1和2的部分优化,优化后的基因的核苷酸序列如seqidno.3所示;方案4:将pcv3cap蛋白完整全长基因中的非大肠杆菌偏好密码子全部替换为偏好密码子,优化后的基因的核苷酸序列如seqidno.4所示;方案5:对pcv3cap蛋白完整全长基因不进行优化,优化后的基因的核苷酸序列如seqidno.5所示;方案6:在考虑到密码子偏爱性和gc含量等因素的基础上对去除信号肽的pcv3cap蛋白截短基因进行部分优化,优化后的基因的核苷酸序列如seqidno.6所示。
实施例2对比各种优化的猪圆环病毒3型衣壳蛋白基因的重组蛋白表达水平及是否形成病毒样颗粒
1)cap蛋白表达载体的构建
分别将人工合成的优化后的cap蛋白基因(seqidno.1-6所示)与pet30a载体的ndei和xhoi酶切位点连接并在16℃过夜。之后将连接产物转化到e.colidh5α感受态细胞,涂至含卡那霉素的lb平板上,37℃培养12h。挑取平板上的单菌落,进行菌液pcr鉴定并测序。鉴定正确后抽提重组质粒。
2)重组cap蛋白的诱导表达
将重组质粒转入大肠杆菌表达菌株bl21(de3),以1:100的比例接入含卡那霉素的tb培养基中37℃,220rpm过夜培养。将活化的菌液以1:50比例接种到含卡那霉素的的液体tb培养基中,37℃,220rpm摇床上培养2h。然后加入终浓度为0.4mmol/l的iptg,25℃,220rpm进行诱导表达10h。随后离心收集菌液,按照100ml菌体加入10ml缓冲液(50mmtris-hcl,ph8.0)重悬菌体。对重悬的菌体进行超声破碎,冰上操作。工作3s,停顿6s,amp设置为39%,重复300个循环。随后将破碎完成的菌液离心,12000rpm,30min,4℃,收集上清。
样品处理过后进行sds-page分析(图1),目的蛋白大小约25kd。对比多套方案的表达水平,seqidno.1密码子部分优化的全长基因重组蛋白的表达水平远高于全部优化和不优化的序列,也高于另两种部分优化方法。seqidno.6对衣壳蛋白核定位信号截短表达也可以表达较高水平的重组蛋白。因此选取seqidno.1和seqidno.6进行后续电镜观察,判断是否形成猪圆环病毒3型病毒样颗粒。
3)电镜观察
分别将2)中获得的高表达的重组蛋白送至透射电镜观察。seqidno.1所示序列方案可以观察到明显的圆形空心颗粒(图2),直径大约为10nm,说明该套方案成功形成了猪圆环病毒3型病毒样颗粒。而seqidno.6所示序列方案未发现明显的病毒样颗粒(图3),说明该套方案虽然成功的高效表达重组衣壳蛋白,但并未自组装成为病毒样颗粒。所以最终确定seqidno.1所示的优化后的猪圆环病毒3型衣壳蛋白全长基因可以高效表达重组衣壳蛋白并形成猪圆环病毒3型病毒样颗粒。
实施例3猪圆环病毒3型衣壳蛋白的表达、纯化以及猪圆环病毒3型病毒样颗粒的制备
1)合成seqidno.1所示的优化后的猪圆环病毒3型衣壳蛋白全长基因;
2)将合成的基因连接至pet30a载体的ndei和xhoi酶切位点,产物转化到e.colidh5α感受态细胞,挑菌鉴定后抽提质粒,重组质粒命名为pet30a-rcap;
3)将pet30a-rcap质粒转入大肠杆菌表达菌株bl21(de3),培养后,得到含有pet30a-rcap质粒的大肠杆菌菌液;
4)将菌液接种到含卡那霉素的液体tb培养基中,37℃,220rpm摇床上培养2h。然后加入终浓度为0.4mmol/l的iptg,25℃,220rpm进行诱导表达20h,随后离心收集菌液;
5)破碎步骤4)中收集的菌液上清,经两步层析纯化,包括第一步阴离子交换层析和第二步凝胶过滤层析,得到猪圆环病毒3型病毒样颗粒。具体操作为:
重组cap蛋白的初步纯化:
用缓冲液(50mmtris-hcl,ph8.0)平衡deae阴离子交换柱,随后将裂解的菌液上清缓慢载入平衡后的柱子。选用含有一定盐浓度的缓冲液(50mmtris-hcl,150mmnacl,ph8.0)对柱子进行洗脱,可得一定纯度的目的蛋白。即初步纯化的目的蛋白,结果如图4所示。
重组cap蛋白的精细纯化:
选用含有一定盐浓度的缓冲液(50mmtris-hcl,150mmnacl,ph8.0)平衡琼脂糖6ff凝胶过滤层析柱,5%柱体积载入初步纯化后的目的蛋白溶液。收集在od280nm监测下第一个可吸收峰洗脱的样品,即精细纯化的目的蛋白,结果如图5所示。可以看到经过第二步纯化后,目的蛋白的纯度可以达到极高的水平,并可以自组装形成猪圆环病毒3型病毒样颗粒。
实施例4猪圆环病毒3型病毒样颗粒在抗体检测中的应用
1)间接elisa方法的建立
将实施例3制备得到的高纯度猪圆环病毒3型病毒样颗粒包被在elisa板上,用于检测针对猪圆环病毒3型的特异性抗体。使用特异性阳性血清和阴性血清对包被条件进行棋盘法优化。od450nm吸光值阳性接近1.0,阴性接近0.1,p/n值最大时为最佳条件。棋盘法结果如图6所示,包被浓度为5μg/ml,血清稀释度为1:200时p/n值最大为13.118。随后选取40份阴性血清确定阈值并测试本发明猪圆环病毒3型病毒样颗粒的间接elisa方法的特异性和敏感性(图7)。
2)利用本发明所建立的间接elisa方法对临床猪血清进行检测
基于1)中所确立elisa条件,对373份来自中国的临床猪血清进行抗体检测,这其中包括60份野猪血清。结果如图8所示,373份血清的抗体阳性率为52.82%,值得注意的是,野猪的阳性率(18.33%)远低于家猪59.11的阳性率。
序列表
<110>中国农业科学院哈尔滨兽医研究所(中国动物卫生与流行病学中心哈尔滨分中心)
哈尔滨维科生物技术有限公司
<120>一种优化后的猪圆环病毒3型衣壳蛋白基因及其在制备病毒样颗粒中应用
<130>klpi190678
<160>6
<170>patentin3.5
<210>1
<211>645
<212>dna
<213>artificialsequence
<400>1
atgcgtcatcgtgcgatctttcgtcgccgtccgcgtccgcgtcgccgtcgccgtcatcgt60
cgccgttatgcacgccgtcgcctgttcattcgccgtccgaccgcgggtacctactacacc120
aaaaagtacagcaccatgaatgtgattagcgttggtaccccgcagaataacaaaccgtgg180
catgcgaatcatttcattacccgcctgaacgaatgggaaaccgcgattacctttgaatac240
tacaaaattctgaaaatgaaagtgaccctgagcccggtgattagcccggcgcagcagacc300
aaaaccatgtttggtcataccgcgattgatctggatggtgcgtggaccaccaatacctgg360
ctgcaggatgatccgtatgcggaaagcagcacccgcaaagtgatgaccagcaaaaagaaa420
catagccgctatttcaccccgaaaccgctgctggcgggtaccaccagcgcgcatccgggt480
cagagcctgttcttctttagccgcccgaccccgtggctgaatacctatgatccgaccgtt540
cagtggggtgcgctgctgtggagcatctatgttccggagaaaaccggtatgaccgatttc600
tatggcaccaaagaagtgtggattcgctacaaaagcgtgctgtaa645
<210>2
<211>645
<212>dna
<213>artificialsequence
<400>2
atgcgccatcgcgcgatctttcgccgtcgcccgcgtccgcgccgtcgccgtcgccatcgt60
cgccgttatgcacgccgtcgtctgttcattcgccgtccgaccgcgggcacctactacacc120
aagaaatacagcaccatgaacgtgattagcgtgggcaccccgcagaacaacaaaccgtgg180
catgcgaaccatttcattacccgcctgaacgaatgggaaaccgcgattacctttgaatac240
tacaaaattctgaaaatgaaagtgaccctgagcccggtgattagcccggcgcagcagacc300
aaaaccatgtttggccataccgcgattgatctggatggcgcgtggaccaccaacacctgg360
ctgcaggatgatccgtatgcggaaagcagcacccgcaaagtgatgaccagcaagaagaaa420
catagccgctatttcaccccgaaaccgctgctggcgggcaccaccagcgcgcatccgggc480
cagagcctgttcttctttagccgcccgaccccgtggctgaacacctatgatccgaccgtg540
cagtggggcgcgctgctgtggagcatctatgtgccggagaaaaccggcatgaccgatttc600
tatggcaccaaagaagtgtggattcgctacaaaagcgtgctgtaa645
<210>3
<211>645
<212>dna
<213>artificialsequence
<400>3
atgcgccatcgcgcgatttttcgccgccgcccgcgcccgcgccgccgccgccgccatcgc60
cgccgctatgcacgccgccgtctgtttattcgccgcccgaccgcgggcacctattatacc120
aaaaaatatagcaccatgaacgtgattagcgtgggcaccccgcagaacaacaaaccgtgg180
catgcgaaccattttattacccgcctgaacgaatgggaaaccgcgattacctttgaatat240
tataaaattctgaaaatgaaagtgaccctgagcccggtgattagcccggcgcagcagacc300
aaaaccatgtttggccataccgcgattgatctggatggcgcgtggaccaccaacacctgg360
ctgcaggatgatccgtatgcggaaagcagcacccgcaaagtgatgaccagcaaaaaaaaa420
catagccgctattttaccccgaaaccgctgctggcgggcaccaccagcgcgcatccgggc480
cagagcctgtttttttttagccgcccgaccccgtggctgaacacctatgatccgaccgtg540
cagtggggcgcgctgctgtggagcatttatgtgccggaaaaaaccggcatgaccgatttt600
tatggcaccaaagaagtgtggattcgctataaaagcgtgctgtaa645
<210>4
<211>642
<212>dna
<213>artificialsequence
<400>4
atgcgtcatcgtgcaatttttcgtcgtcgtccgcgtccgcgtcgtcgtcgtcgtcatcgt60
cgtcgttatgcacgtcgtcgtctgtttattcgtcgtccgaccgcaggtacctattatacc120
aaaaaatatagcaccatgaatgttattagcgttggtaccccgcagaataataaaccgtgg180
catgcaaatcattttattacccgtctgaatgaatgggaaaccgcaattacctttgaatat240
tataaaattctgaaaatgaaagttaccctgagcccggttattagcccggcacagcagacc300
aaaaccatgtttggtcataccgcaattgatctggatggtgcatggaccaccaatacctgg360
ctgcaggatgatccgtatgcagaaagcagcacccgtaaagttatgaccagcaaaaaaaaa420
catagccgttattttaccccgaaaccgctgctggcaggtaccaccagcgcacatccgggt480
cagagcctgtttttttttagccgtccgaccccgtggctgaatacctatgatccgaccgtt540
cagtggggtgcactgctgtggagcatttatgttccggaaaaaaccggtatgaccgatttt600
tatggtaccaaagaagtttggattcgttataaaagcgttctg642
<210>5
<211>645
<212>dna
<213>artificialsequence
<400>5
atgagacacagagctatattcagaagaagaccccgcccaaggagacgacgacgccacaga60
aggcgctatgccagaagaagactattcattaggaggcccacagctggcacatactacaca120
aaaaaatactccaccatgaacgtcatatccgttggaacccctcagaataacaagccctgg180
cacgccaaccacttcattacccgcctaaacgaatgggaaactgcaattacctttgaatac240
tataaaatactaaagatgaaagttacactcagccctgtaatttctccggctcagcaaacc300
aaaactatgttcgggcacacagccatagatctagacggcgcctggaccacaaacacttgg360
ctccaagacgacccttacgcggaaagttccactcgtaaagttatgacttctaaaaaaaaa420
cacagccgttacttcacccccaaaccacttctggcgggaactaccagcgctcacccagga480
caaagcctcttctttttctccagacccaccccatggctcaacacatatgaccccaccgtt540
caatggggagcactgctttggagcatttatgtcccagaaaaaactggaatgacagacttc600
tacggcaccaaagaagtttggattcgttacaagtccgttctctag645
<210>6
<211>552
<212>dna
<213>artificialsequence
<400>6
atgccgaccgcgggtacctactacaccaaaaagtacagcaccatgaatgtgattagcgtt60
ggtaccccgcagaataacaaaccgtggcatgcgaatcatttcattacccgcctgaacgaa120
tgggaaaccgcgattacctttgaatactacaaaattctgaaaatgaaagtgaccctgagc180
ccggtgattagcccggcgcagcagaccaaaaccatgtttggtcataccgcgattgatctg240
gatggtgcgtggaccaccaatacctggctgcaggatgatccgtatgcggaaagcagcacc300
cgcaaagtgatgaccagcaaaaagaaacatagccgctatttcaccccgaaaccgctgctg360
gcgggtaccaccagcgcgcatccgggtcagagcctgttcttctttagccgcccgaccccg420
tggctgaatacctatgatccgaccgttcagtggggtgcgctgctgtggagcatctatgtt480
ccggagaaaaccggtatgaccgatttctatggcaccaaagaagtgtggattcgctacaaa540
agcgtgctgtaa552
- 还没有人留言评论。精彩留言会获得点赞!