用于一次性鉴别大肠杆菌五种菌毛基因的引物及方法与流程
本发明涉及大肠杆菌菌毛的检测,尤其涉及产肠毒素大肠杆菌五种菌毛的一次性鉴别的引物及所用的检测方法。
背景技术:
大肠埃希氏菌简称大肠杆菌,是革兰氏阴性菌,能够产生内毒素,大肠杆菌病属于国家三类传染病。其中产肠毒素大肠杆菌(enterotoxigenicescherichiacoli,etec)在临床上引起仔猪、犊牛、羔羊以及其它经济动物的水样腹泻,同时也是发展中国家婴儿腹泻和发达国家旅游者腹泻的病因之一。据报道,发展中国家平均每年将近40万儿童死于etec引起的腹泻,到目前为止尚无有效疫苗进行预防。etec感染对新生仔猪和断奶仔猪的危害最为严重,可引起仔猪白痢、黄痢以及水肿病,对养猪业造成了巨大的经济损失。临床症状表现为仔猪呕吐腹泻、排黄白色水样稀粪、眼睑水肿、消瘦脱水,并伴随其他疾病继发感染最终导致死亡。etec具有多种菌毛类型,其中f4(k88)、f5(k99)、f18ab(f107,2134p)、f18ac(8813)、f41在我国乃至世界范围都较为流行。etec的菌毛能够黏附到动物小肠绒毛特异性受体上,释放多种肠道毒素,继而引起水和电解质失衡,导致腹泻,同时这些菌毛可保护大肠杆菌免受肠道消化酶的影响,不利于肠道对大肠杆菌的清除。从防治角度而言,准确鉴定病畜所携带的大肠杆菌菌毛类型,有助于更好研究菌毛特性并进行有针对性的预防和治疗。目前,可以利用聚合酶链式反应(polymerasechainreaction,pcr)技术对菌毛进行鉴定。首先,uida基因编码大肠杆菌特异性表达的β-葡萄糖糖醛酸酶,通过pcr扩增uida能够对大肠杆菌进行菌株鉴别。在此基础上,可进一步鉴定菌毛类型,但是不同的菌毛基因序列不同,需要分别进行pcr反应,操作繁琐,耗时长,检测效率低。如果直接混合多种引物同时进行pcr扩增,首先引物之间会相互干扰,由于不同菌毛序列具有一定的保守性,引物与其他菌毛模板可发生非特异性结合或彼此互补配对,导致条带混乱;其次,一个退火温度通常不能适用于多种引物,导致无法进行有效扩增,不能达到鉴定目的。因此亟需一种快速准确鉴定多种流行的etec菌毛类型的多重pcr方法。
技术实现要素:
本发明的目的是提供一种用于一次性鉴别大肠杆菌五种菌毛基因的引物,该五种引物在多重pcr反应过程中能够分别扩增k88、k99、f18ab、f18ac、f41五种菌毛基因,并且不会相互干扰,鉴定结果准确,检测效率高。
为了解决上述问题,本发明采取如下技术方案:
用于一次性鉴别大肠杆菌五种菌毛基因的引物,具有如下核苷酸序列:
k88引物:
k88-f:5'-tggtagtatcactgcagat-3'
k88-r:5'-cactttcactgaaccaact-3';
k99引物:
k99-f:5'-gctcgtattgactggtct-3'
k99-r:5'-cagccgtagtgaatgaag-3';
f18ab引物:
f18ab-f:5'-ataacttggagcgggcagtta-3'
f18ab-r:5'-ttgtaagtaaccgcgtaagc-3';
f18ac引物:
f18ac-f:5'-tgcgagtagtgctcaagt-3'
f18ac-r:5'-attcacggtatctggtag-3';
f41引物:
f41-f:5'-tgctgattggacggaaggtc-3'
f41-r:5'-ggttgaagcactttgccctg-3'。
本发明还提供利用上述五种引物一次性鉴别大肠杆菌五种菌毛基因的方法,包括如下步骤:
(a)从动物内脏上采集菌株并提取dna模板;
(b)鉴定菌株是否属于大肠杆菌种属;
(c)如果所述菌株属于大肠杆菌种属,再利用权利要求1中的五种引物对步骤(a)中提取的dna进行多重pcr扩增,反应程序:94℃预变性3min,94℃变性30s,58℃退火30s,72℃延伸1min,变性至延伸重复30个循环,72℃延伸5min,4℃保存;
(d)琼脂糖凝胶电泳检测。
优选的,所用pcr反应体系为:
所述五种引物混合液4μl,所述dna模板1μl,taqpcrmix12.5μl,灭菌水补至25μl,所述五种引物等物质的量混合,总浓度为20μmol/l,所述dna模板浓度为800ng/μl。
优选的,步骤(b)中,通过鉴定uida基因来鉴定所述菌株是否属于大肠杆菌均属,并且所用uida引物为:
uida-f:5'-catgaagatgcggacttgcg-3'
uida-r:5'-gctaacgtatccacgccgta-3',
pcr反应程序:94℃预变性3min,94℃变性30s,72℃退火30s,72℃延伸1min,变性至延伸重复30个循环,72℃延伸5min,4℃保存。
优选的,pcr反应体系为:所述uida引物4μl,浓度为20μmol/l,所述dna模板1μl,浓度为800ng/μl,taqpcrmix12.5μl,灭菌水补至25μl。
本发明的有益效果在于,本方法及所用引物能够在同一pcr反应体系中同时检测k88、k99、f18ab、f18ac、f41五种菌毛基因,反应特异性强、灵敏性高,不会相互干扰,极大提高了菌毛的检测效率,针对大肠杆菌病提供了可靠性的检验手段。
附图说明
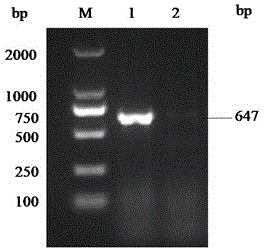
图1是猪大肠杆菌uida基因鉴定的电泳图(m.dnamarker;1.大肠杆菌escherichiacoli;2.沙门氏菌salmonellacholeraesuis);
图2是五种引物特异性试验的电泳图(m.dnamarker;1.k88;2.k99;3.f18ab;4.f18ac;5.f41);
图3是多重pcr退火温度筛选的电泳图(m.dnamarker;1.55℃;2.56.3℃;3.57.7℃;4.59.7℃;5.61.6℃);
图4是多重pcr敏感性试验电泳图(m.dnamarker;1.原液;2.10倍稀释;3.100倍稀释;4.1000倍稀释)
图5是临床分离的猪大肠杆菌uida基因鉴定的电泳图(m.dnamarker;1.石家庄;2.衡水;3.沧州)
图6是衡水地区猪大肠杆菌菌毛基因鉴定的电泳图(m.dnamarker;1、2、3:样品)。
具体实施方式
下面结合附图,通过具体实施例对本发明做进一步说明:
针对uida,k88,k99,f18ab,f18ac,f41保守区域设计6对特异性引物,使用网站上blast功能进行引物比对,引物由上海生物工程股份有限公司合成。引物序列及产物长度见(表1)。
实施例1细菌菌株的收集和处理
采用猪源大肠杆菌进行相关试验。标准阳性菌株猪源大肠杆菌、猪源沙门氏菌和五种菌毛类型的猪源大肠杆菌均购自中国兽医微生物菌种保藏管理中心。待检菌株分离于河北省石家庄、沧州、保定、张家口,衡水等地的养殖场腹泻仔猪的肝脏、肺脏、脾脏,将上述内脏器官选取病健交界处涂布到麦康凯培养基上,37℃培养24h左右,挑取边缘光滑红色单菌落,接种于伊红美兰培养基,待长出符合大肠杆菌的深紫色带金属光泽的菌落,同时使用革兰氏染色液对单菌落染色油镜观察其显微结构,再次挑取单菌落接种到普通营养琼脂培养基上待单菌落长出,最后接种到lb液体培养基扩大培养37℃,200r/min,振荡培养12h,使用紫外分光光度计测定od600为0.8时,取出菌液与甘油1:1混合,液氮速冻于-80℃长期保存。
实施例2细菌基因组dna提取
将实施例1中的阳性菌株接种到lb液体培养基中扩大培养,37℃,200r/min,振荡培养过夜。取出1ml菌液4℃,12000×g/min,离心5min,弃去培养上清,加100μl双蒸水将菌体沉淀重新悬浮,102℃金属浴加热10min,迅速放置冰上6min,使细菌菌体充分裂解。4℃,12000×g/min,离心5min,取上清作为细菌dna模板。nanodrop测定dna浓度。
实施例3对uida基因的pcr扩增
使用北京天根生化科技有限公司taqpcr试剂盒对uida基因进行pcr扩增。pcr反应体系为:表1中uida上下游引物各2μl,浓度为20μmol/l,所述dna模板1μl,浓度为800ng/μl,taqpcrmix12.5μl,灭菌水补至25μl。阳性对照为猪大肠埃希氏菌,阴性对照为猪霍乱沙门氏菌。pcr反应程序:94℃预变性3min,94℃变性30s,72℃退火30s,72℃延伸1min,变性至延伸重复30个循环,72℃延伸5min,4℃保存。试验结果如图1所示。
由图1可见,uida引物可以uida基因进行有效扩增,产物长度经为647bp,该引物不会扩增猪霍乱沙门氏菌,因此uida引物能够用于鉴别菌株是否属于大肠杆菌种属。
实施例4菌毛基因单一pcr反应
应用北京天根生化科技有限公司taqpcr预混剂分别对五种阳性菌株的菌毛基因k88、k99、f18ab、f18ac、f41进行扩增,所用引物分别为表1中第2-6组引物。pcr反应体系为25μl:上下游引物各2μl,浓度为20μmol/l,所述dna模板1μl,浓度为800ng/μl,taqpcrmix12.5μl,灭菌水补至25μl。反应程序:94℃预变性3min,94℃变性30s,57℃退火30s,72℃延伸1min,从变性至延伸重复30个循环,72℃延伸5min,4℃保存。1.5%的琼脂糖凝胶电泳检测。
试验结果:表1中各引物可分别对k88、k99、f18ab、f18ac、f41五种菌毛基因进行扩增,产物长度分别343bp、157bp、252bp,494bp和580bp,各引物单独扩增效果如图2所示。
实施例5菌毛基因多重pcr反应
将实施例4中五种引物和五种阳性菌株dna混合,并对引物退火温度进行多次优化。遵循生物实验方法原则,其他实验条件保持一致,针对某一条件进行优化。退火温度设定在55~61.6℃,五种引物等物质的量混合,混合引物总浓度为20μmol/l。所述五种引物混合液4μl,所述dna模板1μl,浓度为800ng/μl,taqpcrmix12.5μl,灭菌水补至25μl。反应程序:94℃预变性3min,94℃变性30s,在55~61.6℃选取五个温度点(55℃、56.3℃、57.7℃、59.7℃、61.6℃)分别进行30s退火,72℃延伸1min,变性至延伸重复30个循环,72℃延伸5min,4℃保存。1.5%的琼脂糖凝胶电泳。试验结果如图3所示。从图3可以看出,第3个泳道的退火温度较为合适,并最终确定最佳退火温度为58℃。由此可见,五种引物与五种菌毛dna模板进行多重pcr扩增,条带为五条单独条带,条带大小与预期结果符合,无非特异性结合,说明引物之间无交叉性。因此,上述五种引物可以进行多重pcr反应,效果良好。
实施例6多重pcr引物敏感性分析
根据实施例5中多重pcr反应条件,使用紫外分光光度计测定dna浓度及纯度,严格遵守生物实验原则,采用10倍倍比稀释法稀释模板dna,其他试验条件保持不变。pcr扩增后进行1.5%的琼脂糖凝胶电泳,结果如图4所示。由图4可见,多重pcr最低检测量为模板dna800pg/μl,最高检测稀释倍数为104,可见该方法在较低dna浓度存在的情况下扩增效果良好,具有较好的敏感性。
实施例7多重pcr反应对待检菌株的应用实例
对实施例1中分离的疑似大肠杆菌菌株进行初步种属鉴定,取3株按照实施例3所描述的方法pcr扩增uida基因后进行琼脂糖凝胶电泳,然后将pcr产物纯化后测序。结果如图5所示。鉴定结果显示,待检菌株与阳性菌株条带大小符合,同时测序结果显示与大肠杆菌uida基因组序列吻合,由此证明uida可以用来鉴定待检菌株是否属于大肠杆菌种属。
采用实施例5中描述的多重pcr反应体系及反应程序,并选取所述最佳退火温度,对实施例1中分离的河北省内101例大肠杆菌5种菌毛基因k88、k99、f18ab、f18ac、f41进行鉴定。其中衡水地区的三株菌株pcr扩增结果如图6所示,该结果显示衡水地区etec菌毛类型为f18ab,f18ac,k88,k99。对所有101株进行多重pcr分析,结果如表2所示,鉴定结果显示河北省101株etec菌毛类型均为多菌毛混合感染类型,分布情况呈散发流行趋势。
通过上述实施例可见,表1中的第1种引物能够鉴别菌株是否属于大肠杆菌种属,其余五种引物能够一次性鉴别大肠杆菌的菌毛是否为k88、k99、f18ab、f18ac、f41中的一种或几种。一次多重pcr扩增能够实现普通方法多次操作的效果,提升了检测便捷性,提高了工作效率。此外,本方法适用大批量鉴定样本,检出灵敏度高,为疫苗研究提供依据,同时适用于其他物种产肠毒素大肠杆菌感染的菌毛类型诊断。在探索上述实验过程中,还为每种菌毛分别设计了数十种引物,这些引物有的对该种菌毛的特异性差,扩增结果有杂带;有的虽然有特异性,但进行多重pcr反应中与其他引物相互结合干扰,扩增条带复杂混乱,无法用于鉴定分析。
上述实施例只是对本发明构思和实现的若干说明,并非对其进行限制,在本发明构思下,未经实质变换的技术方案仍然在保护范围内。
序列表
<110>河北农业大学
<120>用于一次性鉴别大肠杆菌五种菌毛基因的引物及方法
<160>12
<170>siposequencelisting1.0
<210>1
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>1
tggtagtatcactgcagat19
<210>2
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>2
cactttcactgaaccaact19
<210>3
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>3
gctcgtattgactggtct18
<210>4
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>4
cagccgtagtgaatgaag18
<210>5
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>5
ataacttggagcgggcagtta21
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
ttgtaagtaaccgcgtaagc20
<210>7
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>7
tgcgagtagtgctcaagt18
<210>8
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>8
attcacggtatctggtag18
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
tgctgattggacggaaggtc20
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
ggttgaagcactttgccctg20
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
catgaagatgcggacttgcg20
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
gctaacgtatccacgccgta20
- 还没有人留言评论。精彩留言会获得点赞!