一种用于新型冠状病毒感染患者无创诊断的肠道微生物基因标志物及其应用
本发明属于生物医药技术领域,具体涉及一种用于新型冠状病毒感染患者无创诊断的肠道微生物基因标志物及其应用。
背景技术:
新型冠状病毒肺炎(coronavirusdisease2019,covid-19)在世界范围内迅速传播,截止到2020年12月,已有超过5900万例感染病例和100万例死亡病例。新冠肺炎的广泛传播给全世界的卫生健康事业带来了极大的负担。通过逆转录聚合酶链反应(reversetranscription-polymerasechainreaction,rt-pcr)进行核酸检测来判断是否感染新型冠状病毒(severeacuterespiratorysyndromecoronavirus2,sars-cov-2),是目前公认的诊断“金标准”。但是,由于病毒窗口期等方面的原因,感染新冠病毒后,部分患者经rt-pcr检测出现假阴性的结果,且假阴性率高达20%,这极大的增加了疾病的传染源和传播范围。因此,建立一种新的区别健康人和新型冠状病毒感染者的诊断模型,以弥补现有检测方法的不足,对于新冠肺炎防治工作具有重大意义。
人体肠道微生态系统与健康和疾病密切相关。人体肠道内定植着数目庞大(1014)、结构复杂(超过1000种细菌)的微生物群落(约1.5公斤)。其细胞总量几乎是人体自身细胞数量的10倍,基因总量是人类自身基因的150倍。肠道菌群和宿主共生并共进化过程中,在调节宿主的消化吸收、免疫反应、代谢、炎症等方面发挥重要作用。肠道微生态失调促进慢性疾病的发生发展,包括肝硬化、心血管疾病、糖尿病、结肠癌、慢性肾脏病等。
肠道微生物中关健功能菌可成为人类疾病的新型生物标志物。肠道微生态的特征或者基于肠道微生物建立的区别模型作为特定疾病或肿瘤的区分工具正越来越多的被广泛报道和认可。肠道菌群的“肠型(enterotypes)”可反映人对疾病的易感性,提示肠道菌群具有潜在的预警和诊断作用。qinj等率先使用宏基因组学报道了肠道微生态与2型糖尿病的相关性,指出23个菌种可能成为区分2型糖尿病的肠道微生物标志物。qinn,lia等(2014年nature杂志)通过宏基因组测序技术解析了中国人群肝硬化患者肠道微生态结构,在微生物基因和功能水平上,鉴定了肝硬化特异性的15个标记物,创建了一个高准确度的肝硬化患者区分指数。renz等(advancedscience,2020)指出,肠道微生态对慢性肾脏疾病更为敏感,5个菌种可成为区分慢性肾脏疾病的肠道微生物标志物,并且肠道微生物也可反应慢性肾脏疾病进展的不同阶段。wangz等(nature,2011)指出依赖于肠道微生态的卵磷脂代谢促进了心血管疾病的进展,这一发现为心血管疾病新型的诊断方法和治疗策略提供了理论基础。ohpl等(ajt,2012)发现肠道微生态的变化与小肠移植排斥紧密相关,可成为一个潜在的移植排斥的诊断标记物。yuj等在不同民族患者中揭示并验证了结直肠癌的微生物标志物,指出微生物标志物作为一种可支付的、无创的结直肠癌的早期诊断标志物。因而,肠道微生物可能是不同疾病诊断的有力工具。然而,用于区别新型冠状病毒感染和非感染者的肠道微生物模型还未曾报道过。
技术实现要素:
本发明提供了一种用于区别新型冠状病毒感染和非感染者的肠道微生物基因标志物,由seqidno:1-7所示的7种微生物基因组成,所述微生物基因在人体肠道中富集。
seqidno:1-7所示的7种微生物基因的序列如下:
>otu470
cctacggggggctgcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgccgcgtgagtgaagaagtatctcggtatgtaaagctctatcagcagggaagaaaatgacggtacctgactaagaagccccggctaactacgtgccagcagccgcggtaatacgtagggggcaagcgttatccggatttactgggtgtaaagggagcgtagacggtgtggcaagtctgatgtgaaaggcatgggctcaacctgtggactgcattggaaactgtcatacttgagtgccggaggggtaagcggaattcctagtgtagcggtgaaatgcgtagatattaggaggaacaccagtggcgaaggcggcttactggacagtaactgacgttgaggctcgaaagcgtggggagcaaacaggattagatacccttgtagtc
>otu1000
cctacgggtggctgcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgccgcgtgaaggaagaagtatctcggtatgtaaacttctatcagcagggaagatagtgacggtacctgactaagaagccccggctaactacgtgccagcagccgcggtaatacgtagggggcaagcgttatccggatttactgggtgtaaagggagcgtagacggtgtggcaagtctgatgtgaaaggcatgggctcaacctgtggactgcattggaaactgtcatacttgagtgccggaggggtaagcggaattcctagtgtagcggtgaaatgcgtagatattaggaggaacaccagtggcgaaggcggcttactggacggtaactgacgttgaggctcgaaagcgtggggagcaaacaggattagataccctagtagtc
>otu1092
cctacgggtggctgcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgccgcgtggaggaagaaggtcttcggattgtaaactcctgttgttgaggaagataatgacggtactcaacaaggaagtgacggctaactacgtgccagcagccgcggtaaaacgtaggtcacaagcgttgtccggaattactgggtgtaaagggagcgtagacggtgtggcaagtctgatgtgaaaggcatgggctcaacctgtggactgcattggaaactgtcatacttgagtgccggaggggtaagcggaattcctagtgtagcggtgaaatgcgtagatattaggaggaacaccagtggcgaaggcggcttactggacggtaactgacgttgaggctcgaaagcgtggggagcaaacaggattagatacccgagtagtc
>otu1741
cctacgggtggcagcagtggggaatattggacaatggggggaaccctgatccagccatgccgcgtgtgtgaagaaggccctcgggttgtaaagcactttcagtgaggaagaacgcctggtggttaatacccatcaggaaagacatcactcacagaagaagcaccggctaactccgtgccagcagccgcggtaatacggagggtgcgagcgttaatcggaattactgggcgtaaagcgcgcgtaggtggcttgataagccggttgtgaaagccccgggctcaacctgggaacggcatccggaactgtcaagctagagtgcaggagaggaaggtagaattcccggtgtagcggtgaaatgcgtagagatcgggaggaataccagtggcgaaggcggccttctggactgacactgacactgaggtgcgaaagcgtgggtagcaaacaggattagataccctagtagtc
>otu1314
cctacgggtggcagcagtggggaatattggacaatgggcgcaagcctgatccagccatgccgcgtgagtgatgaaggccttagggttgtaaagctctttcaccggagaagataatgacggtatccggagaagaagccccggctaacttcgtgccagcagccgcggtaatacgaagggggctagcgttgttcggaattactgggcgtaaagcgcacgtaggcggactattaagtcaggggtgaaatcccggagctcaactccggaactgcctttgatactggtagtctagagaccgagagaggtaagtggaactcctagtgtagaggtggaattcgtagatattaggaagaacaccagtggcgaaggcggcttactggctcggaactgacgctgaggtgcgaaagcgtggggagcaaacaggattagatacccctgtagtc
>otu1473
cctacgggtggcagcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgccgcgtgagcgaagaagtatttcggtatgtaaagctctatcagcagggaagaagaaatgacggtacctgactaagaagcaccggctaaatacgtgccagcagccgcggtaaaacgtaggtcacaagcgttgtccggaattactgggtgtaaagggagcgcaggcgggagaacaagttggaagtgaaatccatgggctcaacccatgaactgctttcaaaactgtttttcttgagtagtgcagaggtaggcggaattcccggtgtagcggtggaatgcgtagatatcgggaggaacaccagtggcgaaggcggcctactgggcaccaactgacgctgaggctcgaaagtgtgggtagcaaacaggattagatacccctgtagtc
>otu239
cctacgggcggcagcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgccgcgtgagcgaagaagtatttcggtatgtaaagctctatcagcagggaagaaaatgacggtacctgactaagaagcaccggctaaatacgtgccagcagccgcggtaatacgtatggtgcaagcgttatccggatttactgggtgtaaagggagcgtagacggcaaggcaagtctgatgtgaaaacccagggcttaaccctgggactgcattggaaactgtctggctcgagtgccggagaggtaagcggaattcctagtgtagcggtgaaatgcgtagatattaggaggaacaccagtggcgaaggcggcttactggacggtaactgacgttgaggctcgaaagcgtggggagcaaacaggattagataccctagtagtc
另外本发明还提供了一种用于检测试剂,包括用于检测seqidno:1-7所示的7种微生物基因的引物。
所述引物序列为seqidno:8-9。
引物primers
测序区域v3+v4:338f-805r
上游引物:338factcctacgggaggcagca
下游引物:805rggactachvgggtwtctaat
检测试剂在制备新型冠状病毒感染和非感染的区分检测试剂盒中的应用,所述检测试剂适用于检测seqidno:1-7所示的7种微生物基因。
适用于区别新型冠状病毒感染和非感染者,新型冠状病毒感染者包括rt-pcr检测阳性的确诊患者及rt-pcr检测阴性但igg抗体阳性的临床诊断患者(疑似患者)。
对所述对象的粪便进行检测,以便确定该样本是否包含所述的微生物基因,是否可以建立区别新型冠状病毒感染和非感染者的微生物基因模型。
通过收集入组对象的粪便样本,抽提微生物总dna,完成微生物dna的16srdnamiseq测序,检测是否存在权利要求1所述的7种微生物基因。
进一步的,通过收集入组对象的粪便样本,抽提微生物总dna,进行肠道菌群的16srdnamiseq测序。基于高通量测序数据,在训练集中建立新型冠状病毒感染和非感染者的微生物区别模型,建立新冠肺炎患病率(probabilityofdisease,pod)指数;pod指数在验证集中计算其区别能力,进行验证;进一步在新冠肺炎疑似患者中进行验证,证实微生物基因区别模型弥补rt-pcr检测不足的可能性。
本发明还提供了一种区别新型冠状病毒感染和非感染者的肠道微生物基因区别模型,包括seqidno:1-7所示的7种微生物基因。
肠道微生物基因区别模型的建立方法具体包括:
(1)收集入组对象(新型冠状病毒感染和非感染者)的粪便样本,按照dna的标准抽提方法完成粪便样本中微生物总dna的抽提,在illuminamiseq平台完成肠道菌群的16srdna高通量测序工作;
(2)基于高通量测序数据,在微生物区别模型的训练集中,在24例新冠病毒感染患者和48例健康对照之间,基于一个随机森林模型,通过一个五倍交叉验证的算法,鉴定了用于该模型的最佳的7个微生物基因标志物。
(3)基于7个微生物基因标志物,通过使用随机生成的决策树的比率来计算新冠肺炎患病率的患病率(probabilityofdisease,pod)指数。
(4)该微生物区别模型在24例新冠肺炎确诊患者和48例健康对照之间的区别能力达到99.74%,pod指数在新冠病毒感染确诊患者中明显升高,两组之间有显著性差异(p=1.2x10-13)。
(5)在验证集中,该微生物区别模型在12例新冠肺炎确诊患者和24例健康对照之间的区别能力达到99.31%;pod指数在确诊患者中明显升高,两组之间有显著性差异(p=2.0x10-6)。
(6)在验证集中,该微生物区别模型在23例新冠肺炎疑似患者和24例健康对照之间的区别能力达到98.01%;pod指数在疑似患者中明显升高,两组之间有显著性差异(p=1.8x10-8)。
因此,本发明的微生物基因区别模型在新冠病毒感染和非感染患者中实现了良好的区别能力,也验证了该区别模型能够将核酸检测阴性而igg抗体阳性的疑似患者从健康人群中鉴别出来。
另外,还提供了一种用于区别新冠病毒感染和非感染的肠道微生物模型的试剂盒,包括用于检测权利要求1所述的seqidno:1-7所示的7种微生物基因的引物。
本发明的具体操作步骤如下:
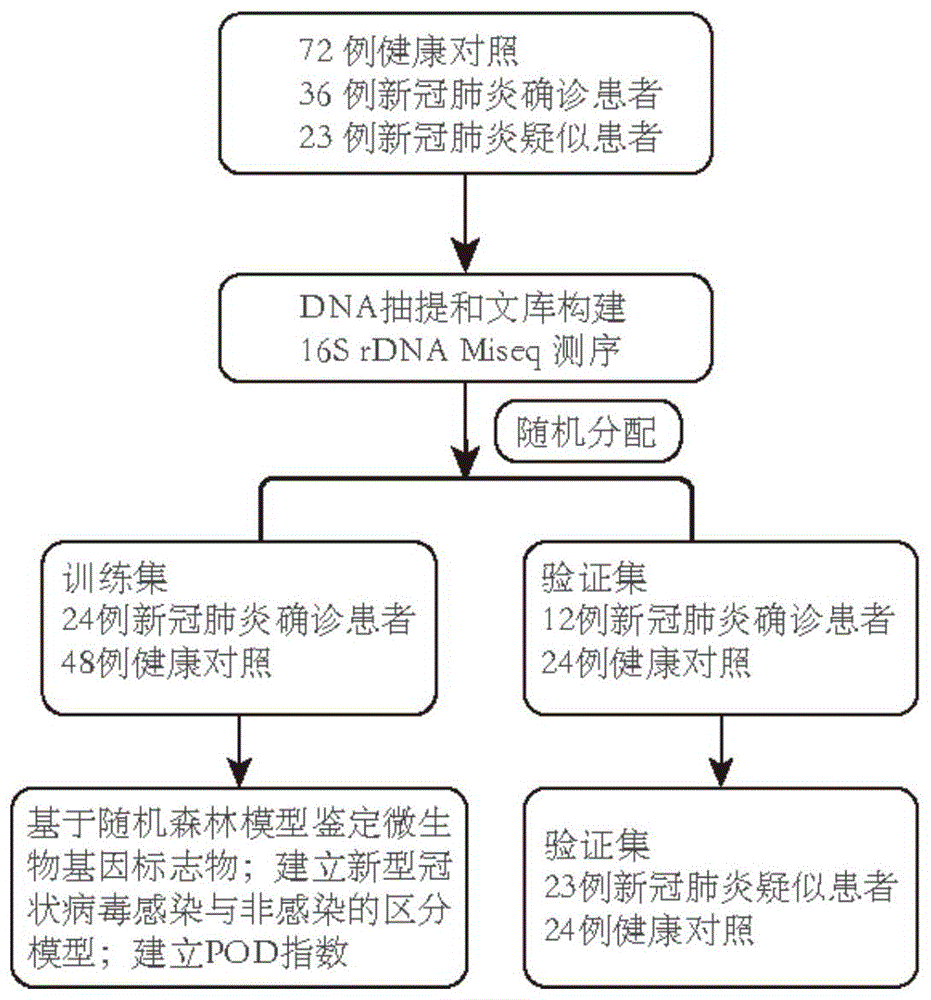
(1)按照前瞻性临床试验的设计原则,本发明的研究设计如图1所示。该研究方案得到了郑州大学第一附属医院和光山县人民医院伦理委员会的批准。所有入组的患者签署研究方案知情同意书和临床样本收集知情同意书。
(2)每一个入组的新冠病毒感染和非感染患者提供一份新鲜的粪便样本,研究实验人员将样本在70℃灭活1小时,然后按照每份200mg的重量分装成10份,并立即冻存于-80℃冰箱。粪便细菌总dna的抽提方法按照试剂盒的说明书进行。
(3)完成粪便细菌总dna样本的扩增和dna文库构建,在illuminamiseq测序平台完成16srdna测序。所有的输出序列完成基本的预处理和基本的生物信息学分析。
(4)从所有样本中随机抽选等量的序列数,按照uparse传递途径拼接成对应的16srdna基因序列分类单元(operationaltaxonomyunits,otus)。按照微生物基因标志物训练集和微生物基因标志物验证集,将产生的所有样本的otus基因序列收集整理。基于微生物基因序列,使用rdp分类器2.6版本注释。
(5)基于高通量测序数据产生的代表性序列,计算出微生物基因标志物发现集的otus频率文件和验证集的otus频率文件。这些otus用于一个相关性研究来鉴定在新冠病毒感染和非感染患者之间差异明显的otus丰度。使用wilcoxon检验方法统计分析新冠病毒感染和非感染患者之间差异的微生物基因标志物。选择出的7个otus微生物基因标志物做进一步的分析。
(6)在微生物区别模型的训练集中,包括24例新冠病毒感染确诊患者和48例健康对照,使用筛选出的otus丰度文件,在一个随机森林模型(r软件3.4.1和随机森林软件包4.6–12)中采用五倍交叉验证的算法(除了设置“importance=true”之外,软件参数默认)进行微生物基因标志物的筛选。采用五倍交叉验证的5次试验,获得了交叉验证错误曲线,其中最小的交叉验证错误点作为cut-off值使用。最小的交叉验证错误值加上对应值的标准差为cut-off值。筛选出小于cut-off值的错误率的7个以下的otus标志物的集合,选择最小数目otus的集合作为最佳的微生物基因标志物的集合,最终鉴定了用于该模型的最佳的7个微生物基因标志物(图2)。选择出的7个微生物otus标志物的基因序列见seqidno:1-7。
(7)通过使用随机生成的决策树的比率来计算患病率(probabilityofdisease,pod)指数。决策树预测样本为“cp”,设置的参数预测为:proximity=t,norm.votes=t,predict.all=true。在loo模式中构建的随机森林模型用于预测验证集中每一个样本的pod指数,最终计算每一个样本的平均预测的pod指数。
(8)使用r3.3.0程序包中的proc工具计算受试者工作曲线(roc),用来评估微生物区别模型,曲线下面积(auc)用于指定roc的效应值。
(9)该微生物区别模型在24例新冠病毒确诊患者和48例健康对照之间的区别能力达到99.74%(图4),pod指数在新冠病毒确诊患者中明显升高,两组之间有显著性差异(p=1.2x10-13)(图3)。
(10)在验证集中,pod指数在12例新冠肺炎确诊患者中明显升高,两组之间有显著性差异(p=2.0x10-6)(图5),该微生物区别模型在12例新冠肺炎确诊患者和24例健康对照之间的区别能力达到99.31%(图6)。
(11)在验证集中,pod指数在23例中新冠疑似患者中明显升高,两组之间有显著性差异(p=1.8x10-8)(图7),该微生物区别模型在在23例新冠疑似患者和24例健康对照之间的区别能力达到98.01%(图8)。
因此,本发明的微生物基因区别模型在新冠肺炎感染患者和非感染者中实现了良好的区别能力,也验证了该区别模型能够弥补rt-pcr检测的不足,将感染新冠病毒的疑似患者从健康人群的鉴定出来。
附图说明
图1.一种用于区别新冠肺炎感染和非感染的肠道微生物模型的研究设计和临床应用。
图2.基于随机森林模型采用五倍交叉验证法鉴定的最佳的肠道微生物基因标志物。
图3.在24例新冠肺炎确诊患者和48例健康对照的训练集中,患病率(pod)指数在两组之间的表达差异;
图4.在24例新冠肺炎确诊患者和48例健康对照的训练集中,微生物基因区别模型实现的区别效能;
图5.在验证集中,与24例健康对照人群相比,患病率(pod)指数在12例新冠肺炎确诊患者的表达差异;
图6.在验证集中,患病率(pod)指数在24例健康对照和12例新冠肺炎确诊患者之间的区别能力;
图7.在验证集中,与24例健康对照人群相比,患病率(pod)指数在23例新冠肺炎疑似患者的表达差异;
图8.在验证集中,患病率(pod)指数在24例健康对照和23例新冠肺炎疑似患者之间的区别能力;
具体实施方式
下面结合实施例对本发明作进一步的阐述,但本发明的保护内容不仅限于这些实施例。
下列实施例中所用方法如无特别说明,均为常规方法。下列实施例中所需要的材料或试剂,如无特殊说明均为公开商业途径获得。
本发明通过收集入组对象的粪便样本,抽提微生物总dna,进行肠道菌群的16srdnamiseq测序。基于高通量测序数据,在训练集中建立新型冠状病毒感染和非感染者的微生物区别模型,建立新冠肺炎患病率(probabilityofdisease,pod)指数;pod指数在验证集中计算其区别能力,进行验证;进一步在新冠肺炎疑似患者中进行验证,以证明微生物基因区别模型弥补rt-pcr的可能性。
其操作步骤如下:
(1)按照前瞻性临床试验的设计原则,本发明的研究设计如图1所示。该研究方案得到了郑州大学第一附属医院和光山县人民医院伦理委员会的批准。所有入组的患者签署研究方案知情同意书和临床样本收集知情同意书。
(2)每一个入组的新型冠状病毒感染患者和非感染患者提供一份新鲜的粪便样本,研究实验人员将样本在70℃灭活1小时,然后按照每份200mg的重量分装成10份,并立即冻存于-80℃冰箱。粪便细菌总dna的抽提方法按照试剂盒的说明书进行。
(3)完成粪便细菌总dna样本的扩增和dna文库构建,在illuminamiseq测序平台完成16srdna测序。所有的输出序列完成基本的预处理和基本的生物信息学分析。
(4)从所有样本中随机抽选等量的序列数,按照uparse传递途径拼接成对应的16srdna基因序列分类单元(operationaltaxonomyunits,otus)。按照微生物基因标志物训练集和微生物基因标志物验证集,将产生的所有样本的otus基因序列收集整理。基于微生物基因序列,使用rdp分类器2.6版本注释。
(5)基于高通量测序数据产生的代表性序列,计算出微生物基因标志物发现集的otus频率文件和验证集的otus频率文件。这些otus用于一个相关性研究来鉴定在新型冠状病毒感染和非感染者之间差异明显的otus丰度。使用wilcoxon检验方法统计分析新型冠状病毒感染和非感染者之间差异的微生物基因标志物。选择出的otus微生物基因标志物做进一步的分析。
(6)在微生物区别模型的训练集中,包括24例新冠肺炎确诊患者和48例健康对照,使用筛选出的otus丰度文件,在一个随机森林模型(r软件3.4.1和随机森林软件包4.6–12)中采用五倍交叉验证的算法(除了设置“importance=true”之外,软件参数默认)进行微生物基因标志物的筛选。采用五倍交叉验证的5次试验,获得了交叉验证错误曲线,其中最小的交叉验证错误点作为cut-off值使用。最小的交叉验证错误值加上对应值的标准差为cut-off值。筛选出小于cut-off值的错误率的7个以下的otus标志物的集合,选择最小数目otus的集合作为最佳的微生物基因标志物的集合,最终鉴定了用于该模型的最佳的7个微生物基因标志物(图2)。选择出的7个微生物otus标志物的基因序列见seqidno:1-7。
(7)通过使用随机生成的决策树的比率来计算患病率(probabilityofdisease,pod)指数。决策树预测样本为“cp”,设置的参数预测为:proximity=t,norm.votes=t,predict.all=true。在loo模式中构建的随机森林模型用于预测验证集中每一个样本的pod指数,最终计算每一个样本的平均预测的pod指数。
(8)使用r3.3.0程序包中的proc工具计算受试者工作曲线(roc),用来评估微生物区别模型,曲线下面积(auc)用于指定roc的效应值。
(9)该微生物区别模型在24例新冠肺炎确诊患者和48例健康对照之间的区别能力达到99.74%(图4),pod指数在新冠肺炎确诊患者中明显升高,两组之间有显著性差异(p=1.2x10-13)(图3)。
(10)在验证集中,pod指数在12例新冠肺炎确诊患者中明显升高,两组之间有显著性差异(p=2.0x10-6)(图5),该微生物区别模型在12例新冠肺炎确诊患者和24例健康对照之间的区别能力达到99.31%(图6)。
(11)在验证集中,pod指数在23例中新冠疑似患者中明显升高,两组之间有显著性差异(p=1.8x10-8)(图7),该微生物区别模型在在23例新冠疑似患者和24例健康对照之间的区别能力达到98.01%(图8)。
因此,本发明的微生物基因区别模型在新冠肺炎感染患者和非感染者中实现了良好的区别能力,也验证了该区别模型能够弥补rt-pcr检测的不足,将核酸检测阴性的新冠肺炎感染者从健康人群中鉴别出来。
序列表
<110>郑州大学第一附属医院
<120>一种用于新型冠状病毒感染患者无创诊断的肠道微生物基因标志物及其应用
<160>9
<170>siposequencelisting1.0
<210>1
<211>440
<212>dna
<213>肠道细菌(microorganism)
<400>1
cctacggggggctgcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgc60
cgcgtgagtgaagaagtatctcggtatgtaaagctctatcagcagggaagaaaatgacgg120
tacctgactaagaagccccggctaactacgtgccagcagccgcggtaatacgtagggggc180
aagcgttatccggatttactgggtgtaaagggagcgtagacggtgtggcaagtctgatgt240
gaaaggcatgggctcaacctgtggactgcattggaaactgtcatacttgagtgccggagg300
ggtaagcggaattcctagtgtagcggtgaaatgcgtagatattaggaggaacaccagtgg360
cgaaggcggcttactggacagtaactgacgttgaggctcgaaagcgtggggagcaaacag420
gattagatacccttgtagtc440
<210>2
<211>440
<212>dna
<213>肠道细菌(microorganism)
<400>2
cctacgggtggctgcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgc60
cgcgtgaaggaagaagtatctcggtatgtaaacttctatcagcagggaagatagtgacgg120
tacctgactaagaagccccggctaactacgtgccagcagccgcggtaatacgtagggggc180
aagcgttatccggatttactgggtgtaaagggagcgtagacggtgtggcaagtctgatgt240
gaaaggcatgggctcaacctgtggactgcattggaaactgtcatacttgagtgccggagg300
ggtaagcggaattcctagtgtagcggtgaaatgcgtagatattaggaggaacaccagtgg360
cgaaggcggcttactggacggtaactgacgttgaggctcgaaagcgtggggagcaaacag420
gattagataccctagtagtc440
<210>3
<211>440
<212>dna
<213>肠道细菌(microorganism)
<400>3
cctacgggtggctgcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgc60
cgcgtggaggaagaaggtcttcggattgtaaactcctgttgttgaggaagataatgacgg120
tactcaacaaggaagtgacggctaactacgtgccagcagccgcggtaaaacgtaggtcac180
aagcgttgtccggaattactgggtgtaaagggagcgtagacggtgtggcaagtctgatgt240
gaaaggcatgggctcaacctgtggactgcattggaaactgtcatacttgagtgccggagg300
ggtaagcggaattcctagtgtagcggtgaaatgcgtagatattaggaggaacaccagtgg360
cgaaggcggcttactggacggtaactgacgttgaggctcgaaagcgtggggagcaaacag420
gattagatacccgagtagtc440
<210>4
<211>465
<212>dna
<213>肠道细菌(microorganism)
<400>4
cctacgggtggcagcagtggggaatattggacaatggggggaaccctgatccagccatgc60
cgcgtgtgtgaagaaggccctcgggttgtaaagcactttcagtgaggaagaacgcctggt120
ggttaatacccatcaggaaagacatcactcacagaagaagcaccggctaactccgtgcca180
gcagccgcggtaatacggagggtgcgagcgttaatcggaattactgggcgtaaagcgcgc240
gtaggtggcttgataagccggttgtgaaagccccgggctcaacctgggaacggcatccgg300
aactgtcaagctagagtgcaggagaggaaggtagaattcccggtgtagcggtgaaatgcg360
tagagatcgggaggaataccagtggcgaaggcggccttctggactgacactgacactgag420
gtgcgaaagcgtgggtagcaaacaggattagataccctagtagtc465
<210>5
<211>440
<212>dna
<213>肠道细菌(microorganism)
<400>5
cctacgggtggcagcagtggggaatattggacaatgggcgcaagcctgatccagccatgc60
cgcgtgagtgatgaaggccttagggttgtaaagctctttcaccggagaagataatgacgg120
tatccggagaagaagccccggctaacttcgtgccagcagccgcggtaatacgaagggggc180
tagcgttgttcggaattactgggcgtaaagcgcacgtaggcggactattaagtcaggggt240
gaaatcccggagctcaactccggaactgcctttgatactggtagtctagagaccgagaga300
ggtaagtggaactcctagtgtagaggtggaattcgtagatattaggaagaacaccagtgg360
cgaaggcggcttactggctcggaactgacgctgaggtgcgaaagcgtggggagcaaacag420
gattagatacccctgtagtc440
<210>6
<211>442
<212>dna
<213>肠道细菌(microorganism)
<400>6
cctacgggtggcagcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgc60
cgcgtgagcgaagaagtatttcggtatgtaaagctctatcagcagggaagaagaaatgac120
ggtacctgactaagaagcaccggctaaatacgtgccagcagccgcggtaaaacgtaggtc180
acaagcgttgtccggaattactgggtgtaaagggagcgcaggcgggagaacaagttggaa240
gtgaaatccatgggctcaacccatgaactgctttcaaaactgtttttcttgagtagtgca300
gaggtaggcggaattcccggtgtagcggtggaatgcgtagatatcgggaggaacaccagt360
ggcgaaggcggcctactgggcaccaactgacgctgaggctcgaaagtgtgggtagcaaac420
aggattagatacccctgtagtc442
<210>7
<211>440
<212>dna
<213>肠道细菌(microorganism)
<400>7
cctacgggcggcagcagtggggaatattgcacaatgggggaaaccctgatgcagcgacgc60
cgcgtgagcgaagaagtatttcggtatgtaaagctctatcagcagggaagaaaatgacgg120
tacctgactaagaagcaccggctaaatacgtgccagcagccgcggtaatacgtatggtgc180
aagcgttatccggatttactgggtgtaaagggagcgtagacggcaaggcaagtctgatgt240
gaaaacccagggcttaaccctgggactgcattggaaactgtctggctcgagtgccggaga300
ggtaagcggaattcctagtgtagcggtgaaatgcgtagatattaggaggaacaccagtgg360
cgaaggcggcttactggacggtaactgacgttgaggctcgaaagcgtggggagcaaacag420
gattagataccctagtagtc440
<210>8
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>8
actcctacgggaggcagca19
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
ggactachvgggtwtctaat20
- 还没有人留言评论。精彩留言会获得点赞!