一种微滴数字PCR分析耐除草剂转基因大豆ZH10-6拷贝数的方法
本发明属于农业转基因生物安全评价技术领域,特别是涉及一种基于微滴数字pcr技术分析耐除草剂基因大豆新品种拷贝数的方法构建及其应用。具体说是一种基于数字pcr的转基因大豆zh10-6拷贝数分析方法,通过将体系溶液生成微滴,将pcr体系分配到足够小的反应单元中,实现每个反应单元只有单个模板分子,经pcr反应扩增后,由微滴读取器读取微滴并计算内外基因拷贝数浓度比值,实现精准检测转基因大豆zh10-6外源插入基因拷贝数的方法构建及其应用。
背景技术:
转基因作物是指将非本身的外源目的片段转化到目标植株得到的新作物。以获得更好的性状或质量。例如利用转基因技术培育的耐除草剂等具有抗性、产量多、质量高的作物,给人们带来了巨大的经济效益。转基因技术在农业和食品行业中发挥的作用尤为重要。然而,许多人认为转基因食品是非自然的和人工的,因此不能对它们做出积极的反应。确保食品安全和质量的食品分析与所有国家都有密切关系,对转基因食品进行食品安全评价变得极其重要。表达载体、目的基因整合情况、外源插入序列表达情况等分子特征是转基因生物安全评价的重要内容,其中转基因作物中插入外源基因的拷贝数鉴定是关键参数。
关于转基因作物对人类健康和环境具有潜在威胁的辩论日益激烈,许多国家和地区已经发布了与转基因作物有关的法规和法律,以严格限制其种植和应用。对于转基因作物、转基因食品我国要求转基因产品需要有明确合理的标识,生产和经营转基因食品要严格申请许可证。国家对转基因技术以及转基因产品的重视,建立对转基因作物科学严谨的检测方法迫切需要,并可为转基因管理制度的执行提供一定的支持。
数字pcr(digitalpolymerasechainreaction,dpcr)是近年来在实时荧光pcr基础上发展起来的微量dna分子定量检测新技术。dpcr是将pcr体系分配到足够小的反应单元中,实现每个反应单元只有单个模板分子进行pcr扩增,再采用泊松分布原理,根据阳性微滴与阴性微滴数的比例计算目标分子拷贝数,实现绝对定量。该方法降低了标准曲线对测量结果产生影响等问题,降低了基体效应,实现了pcr扩增的样品分离,消除了本底信号的影响,提高了低拷贝dna的扩增灵敏度。相比实时荧光pcr,数字pcr具有更好的测量独立性,且无需任何校准物,具有更高的特异性、灵敏度、精确性和稳定性。
转基因耐除草剂大豆zh10-6是由中国农业科学院作物科学研究所研发的转g2epsps基因和gat基因耐除草剂大豆新品系。研发人采用农杆菌介导转化法,将整合有g2epsps和gat两个基因的独立表达载体dna导入大豆受体中,通过多代筛选获得对草甘膦具有抗性的zh10-6转化体,在我国具有重要产业化应用前景。本研究以zh10-6外源插入基因和转化体特异性序列为靶标,建立了微滴式数字pcr精准分析外源插入基因拷贝数的方法,旨在为该转化体的安全评价、行政监管和知识产权保护提供重要的技术支撑。
技术实现要素:
本发明克服了southernblot和实时荧光定量pcr两种常用的外源基因拷贝数分析技术工作量大、周期长、操作要求高等缺陷,提供了一种快速、简便、精准分析转基因大豆新品系zh10-6的外源目的基因在基因组插入拷贝数的方法。本发明的目的是建立转基因大豆新品系zh10-6微滴数字pcr拷贝数分析方法,该方法较其他方法具有周期短、准确性高、重复性好等优势。
本发明的技术内容如下:
一种用于检测转基因大豆zh10-6的特异性引物及探针,其特征在于包括g2epsps外源插入目的基因序列:
正向引物序列:5’-tcgagattgatggtggtttgtc-3’
反向引物序列:5’-tcagcgccacttcaatcg-3’
探针序列:fam-ccagtacgtatcggccctgctgatg-bhq1;
gat外源插入目的基因序列:
正向引物序列:5’-gggcaaactgatttccatagctt-3’
反向引物序列:5’-cctcggagctggtactgtttct-3’
探针序列:fam-attccaccaggccgagcactcaga-bhq1;
3’端转化体特异性序列:
正向引物序列:5’-accttcggttccacccttgt-3’
反向引物序列:5’-aggaacaaaaggaaacaaaaaataat-3’
探针序列:fam-tagtttccgaggcagacacatcagtt-bhq1;
内源基因lectin基因序列:
正向引物序列:5’-gccctctactccaccccca-3’
反向引物序列:5’-gcccatctgcaagccttttt-3’
探针序列:fam-agcttcgccgcttccttcaacttcac-bhq1;
本发明进一步公开了用于分析耐除草剂转基因大豆zh10-6拷贝数的方法,其特征在于如下步骤:
(1)g2epsps外源插入目的基因序列:
正向引物序列:5’-tcgagattgatggtggtttgtc-3’
反向引物序列:5’-tcagcgccacttcaatcg-3’
探针序列:fam-ccagtacgtatcggccctgctgatg-bhq1;
gat外源插入目的基因序列:
正向引物序列:5’-gggcaaactgatttccatagctt-3’
反向引物序列:5’-cctcggagctggtactgtttct-3’
探针序列:fam-attccaccaggccgagcactcaga-bhq1;
3’端转化体特异性序列:
正向引物序列:5’-accttcggttccacccttgt-3’
反向引物序列:5’-aggaacaaaaggaaacaaaaaataat-3’
探针序列:fam-tagtttccgaggcagacacatcagtt-bhq1;
内参基因lectin基因序列:
正向引物序列:5’-gccctctactccaccccca-3’,
反向引物序列:5’-gcccatctgcaagccttttt-3’,
探针序列:fam-agcttcgccgcttccttcaacttcac-bhq1;
(2)pcr反应体系:总体积为20μl,包括bio-radddpcrsupermixforprobes10μl,10µmol/l上游引物1μl,10µmol/l下游引物1μl,10µmol/l探针1μl,25ng/μldna模板1μl,最终用无菌水补充至20μl;
(3)微滴生成:将20μl反应体系小心转移至微卡发生器,避免产生气泡,同时与微卡发生器相应位置加入微滴生成油70μl,盖上垫片,放入微滴生成器中生成微滴,微滴生成后用移液器倾斜30-45度吸取40μl反应产物,小心转移至数字96孔反应板中,170℃热封膜;
(4)pcr反应程序:94℃预变性热激活10min,1个循环;94℃变性30s,60℃退火1min,40个循环;98℃酶失活10min,1个循环;
(5)结果分析:微滴数字pcr仪自动计算每个反应的拷贝数浓度、总微滴数和阳性微滴数,总微滴数大于10000以上视为有效结果,按照公式a=b/c,计算出试样中耐除草剂转基因大豆zh10-6的外源目的基因在基因组插入的拷贝数;公式中a为耐除草剂转基因大豆zh10-6的外源目的基因在基因组插入的拷贝数,b为耐除草剂转基因大豆zh10-6外源基因序列的拷贝数浓度,c为内参基因lectin基因的拷贝数浓度。
本发明更进一步公开了用于分析转基因大豆zh10-6拷贝数的特异性引物及探针在用于微滴数字pcr技术快速高效分析转基因大豆zh10-6拷贝数方面的应用。实验结果显示该方法大大提高了工作效率,具有快速、灵活、高效、所需样本量少等优点,可快捷简便高效的用于对转基因大豆zh10-6品系拷贝数分析,并可为其他转基因大豆品系及其他转基因作物的微滴式数字pcr拷贝数分析方法建立提供参考。
本发明主要考察了微滴数字pcr技术分析转基因大豆zh10-6拷贝数的引物探针的微滴扩增效果,利用3’端转化体特异性序列的拷贝数验证转基因大豆zh10-6新品系的纯合性,重点是解决微滴数字pcr技术分析转基因大豆zh10-6拷贝数方法的建立,本发明的创新点在于首次建立了微滴数字pcr方法分析转基因大豆新品系zh10-6的外源目的基因在基因组插入拷贝数的方法,为转基因大豆新品系分子特征检测提供了可靠的技术手段。
本发明试验效果如下:
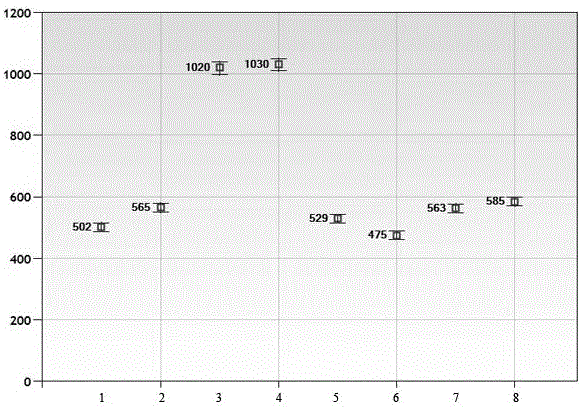
(1)以耐除草剂转基因大豆zh10-6的基因组dna为模板,以大豆内标准基因lectin为参照基因,应用构建的微滴数字pcr检测体系,对其进行微滴数字pcr分析,测定每个基因的拷贝数浓度。结果见图1,大豆内标准lectin基因、g2epsps外源插入目的基因、gat外源插入目的基因、3’端转化体特异性序列的拷贝数浓度分别为533copies/μl、1025copies/μl、502copies/μl、574copies/μl。
(2)计算试样中耐除草剂转基因大豆zh10-6拷贝数
将耐除草剂转基因大豆zh10-6目的基因和转化体特异性序列的拷贝数浓度和内源基因lectin的拷贝数浓度带入公式,计算出试样中耐除草剂转基因大豆zh10-6的外源目的基因在基因组插入的拷贝数。计算试样中耐除草剂转基因大豆zh10-6拷贝数的公式:
a=b/c,
式中:
a—试样中耐除草剂转基因大豆zh10-6的外源目的基因在基因组插入的拷贝数;
b—耐除草剂转基因大豆zh10-6的外源目的基因拷贝数浓度;
c—lectin内源基因拷贝数浓度;
试验结论:
本研究建立了微滴式数字pcr分析转基因大豆zh10-6拷贝数的方法。本研究材料转基因耐除草剂大豆zh10-6是由中国农业科学院作物科学研究所研究的转一个gat基因和2个g2epsps基因的耐除草剂大豆新品系。结果显示,以转基因大豆zh10-6基因组dna,进行外源目的基因g2epsps和gat、3’端转化体特异性序列的微滴数字pcr扩增,转基因大豆zh10-6的外源目的基因g2epsps和gat在基因组上的插入拷贝数分别为1.92、0.94,3’端转化体特异性序列的拷贝数为1.08,验证转基因大豆zh10-6为纯合子,因此鉴定转基因大豆zh10-6为单拷贝插入。结果表明本研究建立的转基因大豆zh10-6拷贝数分析方法周期短、准确性高、重复性好,可快捷简便高效的用于对转基因大豆zh10-6新品系的拷贝数分析,同时验证转基因大豆材料的纯合性,并可为其他转基因大豆品系及其他转基因作物的微滴式数字pcr拷贝数分析方法建立提供参考。
附图说明
图1:微滴数字pcr扩增结果;如图标注:1-2,lectin基因;3-4,g2epsps基因;5-6,gat基因;7-8,3’端转化体特异性序列;
图2:微滴数字pcr拷贝数浓度;如图标注:1-2,lectin基因;3-4,g2epsps基因;5-6,gat基因;7-8,3’端转化体特异性序列;
具体实施方式
下面通过具体的实施方案叙述本发明。除非特别说明,本发明中所用的技术手段均为本领域技术人员所公知的方法。另外,实施方案应理解为说明性的,而非限制本发明的范围,本发明的实质和范围仅由权利要求书所限定。对于本领域技术人员而言,在不背离本发明实质和范围的前提下,对这些实施方案中的物料成分和用量进行的各种改变或改动也属于本发明的保护范围。本发明所用原料及试剂均有市售。
实施例1
(1)样品1:转基因耐除草剂大豆zh10-6随机单株样品。
(2)试剂:bio-radddpcrsupermixforprobes预混液;上海生工合成的大豆内源lectin基因、外源目的基因和转化体特异性序列扩增引物和探针。
g2epsps外源插入目的基因序列:
正向引物序列:5’-tcgagattgatggtggtttgtc-3’
反向引物序列:5’-tcagcgccacttcaatcg-3’
探针序列:fam-ccagtacgtatcggccctgctgatg-bhq1;
gat外源插入目的基因序列:
正向引物序列:5’-gggcaaactgatttccatagctt-3’
反向引物序列:5’-cctcggagctggtactgtttct-3’
探针序列:fam-attccaccaggccgagcactcaga-bhq1;
3’端转化体特异性序列:
正向引物序列:5’-accttcggttccacccttgt-3’
反向引物序列:5’-aggaacaaaaggaaacaaaaaataat-3’
探针序列:fam-tagtttccgaggcagacacatcagtt-bhq1;
内源基因lectin基因序列:
正向引物序列:5’-gccctctactccaccccca-3’
反向引物序列:5’-gcccatctgcaagccttttt-3’
探针序列:fam-agcttcgccgcttccttcaacttcac-bhq1;
(3)pcr反应体系:总体积为20μl,包括bio-radddpcrsupermixforprobes10μl,上游引物(10µmol/l)1μl,下游引物(10µmol/l)1μl,探针(10µmol/l)1μl,dna模板(25ng/μl)1μl,最终用无菌水补充至20μl。
(4)微滴生成:将20μl反应体系小心转移至微卡发生器,避免产生气泡,同时与微卡发生器相应位置加入微滴生成油70μl,盖上垫片,放入微滴生成器中生成微滴,微滴生成后用移液器倾斜30-45度吸取40μl反应产物,小心转移至数字96孔反应板中,170℃热封膜;
(5)pcr反应程序:94℃预变性热激活10min,1个循环;94℃变性30s,60℃退火1min,40个循环;98℃酶失活10min,1个循环。
(6)微滴数字pcr仪自动计算每个反应的拷贝数浓度、总微滴数和阳性微滴数,总微滴数大于10000以上视为有效结果。按照公式a=b/c,计算出试样中耐除草剂转基因大豆zh10-6的外源目的基因在基因组上插入的拷贝数。将耐除草剂转基因大豆zh10-6外源基因序列的拷贝数浓度和内源基因lectin的拷贝数浓度带入公式。
结果见表1,计算出试样中耐除草剂转基因大豆zh10-6的外源目的基因在基因组上插入的拷贝数分别为1.85、0.98,鉴定为单拷贝插入,且通过转化体特异性序列的拷贝数鉴定单株大豆为纯合子。
式中:
a—试样中耐除草剂转基因大豆zh10-6的外源目的基因在基因组插入的拷贝数;
b—耐除草剂转基因大豆zh10-6外源基因序列的拷贝数浓度;
c—lectin内源基因的拷贝数浓度;
表1随机单株样品测试结果
试验结论:
应用本研究建立的微滴式数字pcr分析转基因大豆zh10-6拷贝数的方法,用单株转基因大豆zh10-6基因组dna,进行外源目的基因g2epsps和gat、3’端转化体特异性序列的微滴数字pcr扩增,转基因大豆zh10-6的外源目的基因g2epsps和gat在基因组上的插入拷贝数分别为1.85、0.98,同时3’端转化体特异性序列的拷贝数为1.08,验证单株转基因大豆zh10-6为纯合子,因此鉴定该单株转基因大豆zh10-6为单拷贝插入。结果表明本研究建立的转基因大豆zh10-6拷贝数分析方法周期短、准确性高、重复性好,可快捷简便高效的用于对转基因大豆zh10-6新品系的拷贝数分析,同时验证转基因大豆材料的纯合性,并可为其他转基因大豆品系及其他转基因作物的微滴式数字pcr拷贝数分析方法建立提供参考。
在详细说明的较佳实施例之后,熟悉该项技术人士可清楚地了解,在不脱离上述申请专利范围与精神下可进行各种变化与修改,凡依据本发明的技术实质对以上实施例所作任何简单修改、等同变化与修饰,均属于本发明技术方案的范围。且本发明亦不受说明书中所举实例实施方式的限制。
序列表
<110>天津市农业科学院
<120>一种微滴数字pcr分析耐除草剂转基因大豆zh10-6拷贝数的方法
<160>9
<170>siposequencelisting1.0
<210>1
<211>22
<212>dna
<213>人工序列1
<400>1
tcgagattgatggtggtttgtc22
<210>2
<211>18
<212>dna
<213>人工序列2
<400>2
tcagcgccacttcaatcg18
<210>3
<211>25
<212>dna
<213>人工序列3
<400>3
ccagtacgtatcggccctgctgatg25
<210>4
<211>23
<212>dna
<213>人工序列4
<400>4
gggcaaactgatttccatagctt23
<210>5
<211>22
<212>dna
<213>人工序列5
<400>5
cctcggagctggtactgtttct22
<210>6
<211>24
<212>dna
<213>人工序列6
<400>6
attccaccaggccgagcactcaga24
<210>7
<211>20
<212>dna
<213>人工序列7
<400>7
accttcggttccacccttgt20
<210>8
<211>26
<212>dna
<213>人工序列8
<400>8
aggaacaaaaggaaacaaaaaataat26
<210>9
<211>26
<212>dna
<213>人工序列9
<400>9
tagtttccgaggcagacacatcagtt26
- 还没有人留言评论。精彩留言会获得点赞!