用于病毒相关肿瘤的系统、方法和组合物与流程
技术领域
本发明的领域是来自组织样本和病毒基因组(特别是病毒相关的肿瘤和肿瘤相关的病毒)的遗传信息的计算机分析。
背景技术:
背景描述包括可以用于理解本发明的信息。其并非承认本文提供的任何信息是目前所主张发明的现有技术或与其相关,或者并非承认明确引用的或隐含引用的任何出版公开是现有技术。
在全世界的妇女中,宫颈癌是第三常见的癌症,2008年大约530,000例被诊断为宫颈癌,并且275,000例死亡。由于大部分宫颈癌的起源,普遍认为其是可预防的疾病。单就2004-2008的美国而言,平均11,967例宫颈癌,其中11,500例可归因于HPV感染(Gillison ML,et al.Cancer.2008;113:3036-3046)。本文确认的所有公开均以引用方式并入本文,如同表示每份单独的公开或专利申请明确地且单独地以引用方式并入本文。在所引用参考文献中术语的定义或用途与本文所提供术语的定义不一致或矛盾的情况下,采用本文所提供术语的定义,而不采用参考文献中的术语定义。除了Pap涂片,针对大部分HPV致癌病毒株的免疫(Gardasil和Cervarix)将是更加可用的,其提供早期诊断以及由此提供更好的治疗结果。
目前,已知超过120种人类乳头瘤病毒(HPV)亚型;这些亚型进一步分成低风险和高风险类型。目前有15种高风险类型,其中HPV 16、18、45、31、33、52和58是最普遍的。超过70%的高度上皮内瘤样变和浸润 性宫颈癌为HPV 16+或HPV 18+(Schiller JT,et al.Gynecol Oncol.2010 Jun;118(1Suppl):S12-17)。在超过12个月的一项研究中,75%的女性能够自发地清除其高风险HPV感染,并且在额外的6个月中,另外的22%女性能够自身清除感染(Steben,M.et al.Gynecologic Oncology 107(2):S2–S5)。但是,自身不能清除感染的那些人(例如由于免疫受损状态或未知原因)通常发展成宫颈癌。尽管新的感染可以通过疫苗来预防(例如Gardasil和Cervarix),但是对于已经感染的患者,疫苗是无效的。用于已经发展成宫颈癌的患者,目前的标准治疗方法是基于顺铂的化疗,其不能治愈至少15%至45%的巨块型(bulky)IB至IIIB期患者(A,et al.Cancer Treat Rev 2003,29:389-399)。不幸地,在市场上不具有新的且更有效的针对HPV的治疗。
值得注意地,已知HPV将其DNA整合至宿主基因组中,并且已经报道了HPV整合的优选位点(例如Popescu et al.Cancer Genet Cytogenet 42:157-171.1989)。如别处所报道,HPV已经显示整合至N-Myc基因(2p24)和c-Myc基因(8q24.1)的附近(Couturier et al.,J Virol 65:4534-4538.1991),这可以导致Myc蛋白的表达下调。在新建立的宫颈癌细胞系中,还观察到HPV与c-Myc的共同扩增(Gotoh et al.,Jpn.J.Cancer Res.82:1252-1257.1991)。在进一步的研究中,通过定量荧光免疫组织化学来测定宫颈活检中c-Myc蛋白的水平,并且数据表明c-Myc蛋白的水平在早期和晚期宫颈病变中发挥作用。认为早期病变中c-Myc延长的过表达允许DHFR稳定扩增,这在一定的时间内显示增加的基因扩增,并且持续指示肿瘤进展。在其他的报道中,认为高危型HPV中肿瘤蛋白E6和E7的表达分别使TP53和RB1失活,并因此控制细胞周期和细胞凋亡,同时鳞状上皮内病变中MDM2被上调(Arvanitis et al.,Oncol Rep.2008Oct;20(4):751-60)。
宫颈癌中不普遍但是与治疗更加相关的是ERBB2(红细胞白血病病毒癌基因同源物2)(更常称为HER2/neu)的扩增或过表达。ERBB2是定位于染色体17q12上的原癌基因。ERBB2的过表达最通常地与侵入性乳腺癌相关,但是近年来,作为用于其他癌症的生物标志物而开始引起注意。在大部分情况下,ERBB2在宫颈癌中具有相对低的表达,并且根据研究位点 和癌症严重性,大幅改变的结果使得宫颈癌中ERBB2的表达范围为1%-42%(Rosty C,et al.Int J Gynecol Pathol 2004;23:13–7)。无论普遍性如何,ERBB2的表达与通常导致较低生存率的大部分侵入性癌症类型中的一些相关(Berchuck A.et al.Cancer Res.1990Jul 1;50(13):4087-91),并且其他处描述了稀少的过表达ERBB2的宫颈腺癌(Fadare,et al.International Journal of Gynecological Pathology Volume 23,Issue 4Oct 2004)。使用人源化单克隆抗体曲妥单抗(trastuzumab)治疗宫颈癌患者目前在ERBB2+宫颈癌患者中进行审核。
因此,尽管用于多种癌症的许多治疗选择是可利用的,并且尽管患者和病原体基因组的分子特征变得越来越常规,但是仍需要更好地了解病毒相关的疾病中,特别是病毒相关的宫颈癌中病原体和患者基因组中发生的分子事件,从而有助于对个体疾病的定制治疗。
发明概述
本发明的主题涉及用于病毒相关肿瘤的系统、方法和装置,其有助于更好地了解病原体和患者基因组之间的相互作用和/或变化。除了其他之外,特别涉及指示基因组交换的相互作用的鉴定和表征,其可以基于基因组交换的特定类型而提供独特的治疗选择。
例如在本发明的主题的一个方面中,涉及一种方法,其包括将分析仪与序列数据库进行信息偶联的步骤,其中所述序列数据库储存了来自病毒相关肿瘤的核酸序列和嵌合参照核酸序列。最通常地,嵌合参照核酸序列包含一种或多种病毒核酸序列和哺乳动物核酸序列。在所涉及方法的另一个步骤中,分析仪用于鉴定嵌合参照核酸序列中至少一些病毒核酸序列与来自病毒相关肿瘤的核酸序列中的等位基因(例如致癌基因)的整合。在另一个步骤中,分析仪用于鉴定等位基因和病毒核酸序列的共同扩增,在另一个步骤中,一旦共同扩增达到阈值(例如高于10倍的扩增),则更新病毒相关肿瘤的治疗建议记录。
对于来自病毒相关肿瘤的核酸序列而言,涉及此类序列包含病毒相关肿瘤的至少一条或多条染色体的核酸序列、病毒相关肿瘤外显子组的至少 50%和/或病毒相关肿瘤完整基因组的至少50%。
在另一些所涉及的方面中,病毒相关肿瘤与HTLV-1(人类T细胞白血病病毒)、HPV病毒(人类乳头状瘤病毒)、HHV-8(人类疱疹病毒8)、EBV(EB病毒)、HBV(乙型肝炎病毒)、HCV(丙型肝炎病毒)、SV40(猿猴疱疹病毒40)、BKV(BK病毒)、JCV(JC病毒)、HERV(人类内源性逆转录病毒)、HMTV(人类乳腺肿瘤病毒)、KSHV(卡波氏肉瘤相关疱疹病毒)和/或TTV(细环病毒)相关。当然,在此类病毒具有不同的风险类型、基因型和/或血清型的情况下,还考虑了其他的亚类。例如病毒相关肿瘤可以与16、18、31、33、35、39、45、51、52、56、58、59、68、69、73或82型高风险HPV病毒相关。因此,涉及嵌合参照核酸序列可以包含来自多种不同病毒的多种病毒核酸序列。
最通常的但不是必须的,嵌合参照核酸序列中的哺乳动物核酸序列为匹配的正常的哺乳动物核酸序列。进一步优选来自病毒相关肿瘤的核酸序列和嵌合参照核酸序列为BAM/SAM/FASTA或FASTA索引格式。
在本发明的主题的其他方面中,涉及鉴定整合的步骤包括整合位点的鉴定,并且所涉及方法可以进一步包括确定用于包含共同扩增序列(的基因组排布的解决方法(例如环状或串联重复)的步骤。
因此,从不同的观点来看,本发明人还涉及一种方法,其中分析仪与序列数据库是信息偶联的,其中所述序列数据库储存了来自哺乳动物组织的核酸序列和嵌合参照核酸序列的核酸序列。最通常地,嵌合参照核酸序列包含至少一种病毒核酸序列和可任选地哺乳动物核酸序列。在另一个步骤中,分析仪鉴定来自哺乳动物组织的至少一些核酸序列在病毒核酸序列中的整合,以及来自哺乳动物组织的核酸序列的等位基因(例如致癌基因)与至少一些病毒核酸序列的共同扩增。在另一个步骤中,一旦共同扩增达到阈值(例如高于10倍的扩增),则更新哺乳动物的治疗建议记录。
最通常地,嵌合组织为疾病组织,并且最通常地为肿瘤的癌组织(例如病毒相关肿瘤的肿瘤组织)。还涉及来自哺乳动物组织的核酸序列包含哺乳动物组织的至少一条染色体核酸序列,哺乳动物组织外显子组的至少50%和/或哺乳动物组织完整基因组的至少50%。
类似地,通常优选嵌合参照核酸序列包含多种不同病毒的核酸序列, 并且最通常地包含一种或多种肿瘤相关病毒的核酸序列。例如所涉及的肿瘤相关病毒包括HTLV-1(人类T细胞白血病病毒)、HPV病毒(人类乳头状瘤病毒)、HHV-8(人类疱疹病毒8)、EBV(EB病毒)、HBV(乙型肝炎病毒)、HCV(丙型肝炎病毒)、SV40(猿猴疱疹病毒40)、BKV(BK病毒)、JCV(JC病毒)、HERV(人类内源性逆转录病毒)、HMTV(人类乳腺肿瘤病毒)、KSHV(卡波氏肉瘤相关疱疹病毒)和/或TTV(细环病毒)。此类病毒可以如上文所述进一步分成亚类。例如肿瘤相关病毒可以为16、18、31、33、35、39、45、51、52、56、58、59、68、69、73或82型高风险HPV病毒。尽管不限于此类方法的本发明的主题,通常优选嵌合参照核酸序列包含哺乳动物核酸序列。
在本发明的主题的其他方面中,哺乳动物核酸序列为匹配的正常的哺乳动物核酸序列,并且通常涉及本文提出的方法可以包括确定用于包含共同扩增序列的基因组排布的解决方法(例如环状方法或串联重复)的步骤。
因此,本发明人还涉及一种方法,其包括将分析仪与序列数据库进行信息偶联的步骤,其中所述序列数据库储存了来自患者宫颈肿瘤的核酸序列和嵌合参照核酸序列。如上文所述,在此类方法中通常优选嵌合参照核酸序列包含来自患者的匹配的正常核酸序列和HPV病毒的病毒核酸序列。在所涉及方法的另一个步骤中,分析仪用于鉴定嵌合参照核酸序列中至少一些病毒核酸序列与宫颈肿瘤核酸序列中致癌基因的等位基因的整合,所述分析仪进一步用于鉴定等位基因和病毒核酸序列(多种)的共同扩增。一旦达到预定的阈值,则更新或生成病毒相关肿瘤的治疗建议记录(例如施用靶向致癌基因的药品)。
在所涉及方法的一个典型方面中,来自宫颈肿瘤的核酸序列包含宫颈肿瘤的至少一条染色体核酸序列、宫颈肿瘤外显子组的至少50%和/或宫颈肿瘤完整基因组的至少50%。此外,对于嵌合参照核酸序列而言,涉及所述序列包含16、18、31、33、35、39、45、51、52、56、58、59、68、69、73和82型HPV病毒的至少2种核酸序列。此类序列优选地涵盖HPV病毒的完整基因组。
在其他所涉及的方面中,致癌基因可以为编码生长因子受体的基因,特别是ERBB2,并且进一步涉及所述的等位基因为纯合等位基因。如上文 所述,还涉及所述方法进一步包括确定用于包含共同扩增序列的基因组排布的解决方法(例如环状方法或串联重复)的步骤。
在本发明的主题的其他方面中,涉及一种方法,其包括将编辑仪与序列数据库进行信息偶联的步骤,其中所述序列数据库储存了来自哺乳动物组织的核酸序列(例如与哺乳动物匹配的正常序列)和来自多种不同病毒的多种病毒核酸序列。编辑仪则用于将来自哺乳动物组织的核酸序列与多种病毒核酸序列合并成单个嵌合核酸序列文件,其中来自哺乳动物组织的核酸序列根据染色体结构在单个嵌合核酸序列文件中被组织,并且其中多种病毒核酸序列的每一种作为单个染色体在单个嵌合核酸序列中被组织。在所涉及方法的其他步骤中,接着用单个嵌合基因组文件对序列数据库进行更新。
如上所述,涉及来自哺乳动物组织的核酸序列包含哺乳动物组织至少一条染色体的核酸序列、哺乳动物组织外显子组的至少50%,和/或哺乳动物组织完整基因组的至少50%。此外,涉及不同的病毒为肿瘤相关病毒,其可以包括HTLV-1(人类T细胞白血病病毒)、HPV病毒(人类乳头状瘤病毒)、HHV-8(人类疱疹病毒8)、EBV(EB病毒)、HBV(乙型肝炎病毒)、HCV(丙型肝炎病毒)、SV40(猿猴疱疹病毒40)、BKV(BK病毒)、JCV(JC病毒)、HERV(人类内源性逆转录病毒)、HMTV(人类乳腺肿瘤病毒)、KSHV(卡波氏肉瘤相关疱疹病毒)和/或TTV(细环病毒)。在所述病毒为HPV病毒的情况下,特别涉及的病毒包括16、18、31、33、35、39、45、51、52、56、58、59、68、69、73或82型HPV病毒。
尽管不限于本发明的主题,通常优选合并步骤包括将多种病毒核酸序列附加至来自哺乳动物组织的核酸序列上,和/或来自哺乳动物组织的至少一种(和最通常地所有的)核酸序列、多种病毒核酸序列和单个嵌合核酸序列文件为BAM/SAM/FASTA或FASTA索引格式。
通过下文中优选实施方案的详述以及附图(其中相同的数字代表相同的元件),本发明主题的多种目的、特征、方面和优点将更加显而易见。
附图简述
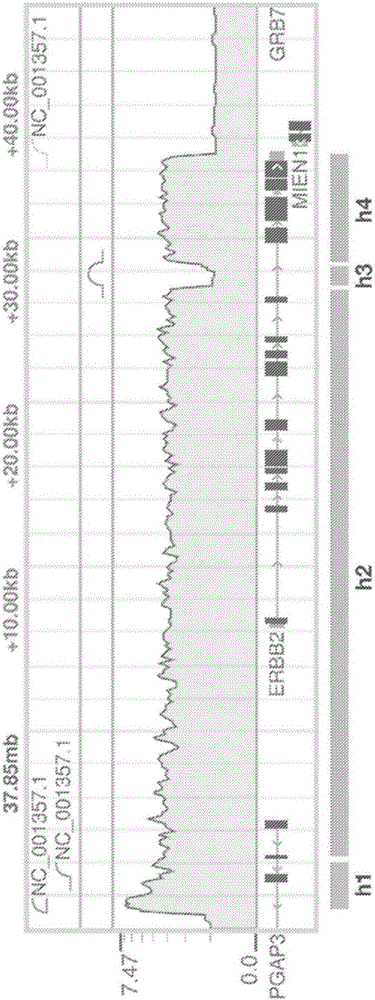
图1为肿瘤DNA分析的示例性结果的详细视图,其中描绘了肿瘤相关HPV-18DNA(NC_001357.1)在人类染色体17中的整合和共同扩增。仅描绘了染色体17的一部分。
图2为嵌合参照DNA分析的示例性结果的详细视图,其中描绘了人类DNA(17)在HPV-18的基因组中的整合和共同扩增。仅描绘了HPV-18病毒的病毒基因组。
图3为图1和2中所示共同扩增的HPV-18序列(v1、v2、v4)与人类序列(h1、h2、h4)之间基因组排布的环状解决方法的示例性图示。
图4为图3环状解决方法的示例性图表,其显示为HPV-18的嵌合重组病毒基因组。
发明详述
本发明人发现某些肿瘤可以是病毒核酸的宿主,特别是在已知肿瘤与现有的病毒感染有关(即病毒相关肿瘤)的情况下。相反地,本发明人还发现病毒可以获得宿主基因组的一部分。一旦获得,本发明人进一步发现肿瘤相关病毒核酸序列和宿主核酸序列在病毒复制的过程中共同扩增。在复制的病毒核酸包含来自宿主的致癌基因的情况下,共同扩增可以呈现不利的结果,还可以展示用于宿主的新的治疗选择。因此,本发明人考虑可以通过对患者和/或来自患者的病毒样品进行基因组分析而提供诊断、疗法和其他治疗选择,其中分析包括鉴定基因组内容物的(通常为相互的)交换和/或共同扩增。
例如并且如下文中更详细讨论,本发明人通过低分化宫颈腺癌患者样品的基因组分析发现患者基因组与HPV-18病毒的病毒基因组之间的基因组交换/融合。尽管之前报道过HPV-18整合入宿主基因组中,并且尽管之前表明宿主中的癌症归因于宿主基因组上病毒E6/E7基因的调节影响,本发明人现在发现完全不同的潜在作用模式。更具体而言,本发明人发现HPV与宿主基因组的病毒整合伴随着宿主致癌基因(在此为:ERBB2基因)与大部分病毒基因组的的基本上共同扩增。如本文所用,第一基因和第二基因的术语“共同扩增”是指第一基因和第二基因以相同的拷贝数扩 增。在大部分情况下以及如下文中更详细的进一步讨论,观察到共同扩增仅与单个等位基因相关。此类基因组融合可以以病毒序列与患者基因组的整合(通常为串联重复)和/或以染色体外结构(例如环状的病毒基因组)或者甚至病毒颗粒的形式存在。但是,例如在第一等位基因的独立扩增之后发生共同扩增的情况下,就第一等位基因和第二等位基因的拷贝数而言,共同扩增的结果还可以是不对称的。
因此,在共同扩增的宿主核酸包含致癌基因、肿瘤抑制基因、参与细胞周期调节的基因和/或参与细胞分裂的基因的情况下,检测此类宿主核酸(例如ERBB2)的病毒相关扩增可以影响/指导使用靶向宿主核酸的药品对病毒相关肿瘤的药品治疗。从另一种观点来看,应该领会的是,靶向扩增的宿主核酸的功能可以作为病毒相关肿瘤的新的治疗模式。
应该特别领会的是,所涉及的系统和方法在患者被诊断或疑似患有病毒相关癌症的情况下是特别有利的。由于在此类患者群体中,对病毒与宿主基因组之间潜在相互作用的了解可以重要地指示治疗选择,本文所涉及的系统和方法将基于对病毒与宿主基因组序列的基因组整合和/或病毒与宿主基因组序列的共同扩增进行快速鉴定和表征。本发明人现在发现这种快速分析可以通过以计算机模拟(in silico)方式对参照基因组(其可以得自健康宿主组织或得自非宿主组织)进行修饰来实现,其中一种或多种病毒基因组序列(以及最优选完整的病毒基因组)与参照基因组合并,从而形成嵌合参照核酸序列。
用于嵌合参照核酸序列中的合适的参照基因组包括相同患者的完整基因组核酸序列,并且通常得自未患病的组织。例如参照基因组核酸可以得自全血,得自与癌组织相邻的组织,或者得自颊粘膜拭子或活检。备选地,参照基因组还可以得自早期从患者采集的样品,或得自之前完整基因组测序尝试。在其他备选的方面中,参照基因组还可以为来自相同物种(例如人类或其他哺乳动物)的基因组序列,优选通过性别或者平均的或一致的序列对相同物种进行分层。最通常地,参照基因组为或涵盖完整的基因组。然而,还涉及更小部分的基因组,其包括至少一条染色体、或2-5条染色体、或5-10条染色体、或者多于10条染色体。备选地,参照基因组还可以仅代表完整外显子组或完整转录子组的一部分(例如1-10%、 10-30%、30-60%或60-90%)。因此并且从另一种观点来看,参照基因组通常包含人类(或其他物种)完整基因组的至少10%、至少30%、至少50%或至少70%。
用于嵌合参照核酸序列中的合适的病毒基因组包括至少一种病毒的完整基因组核酸序列,并且更通常地为已知与疾病相关的一组病毒,特别是肿瘤相关病毒(即已知与癌症疾病相关的病毒)。例如认为适用于本文的病毒基因组序列包括得自以下的那些:HTLV-1(与成人T细胞白血病相关)、HPV病毒(与宫颈癌、皮肤癌、头部和颈部癌症以及肛门生殖器癌症相关)、HHV-8(与Kaposi肉瘤、原发性渗出性淋巴瘤、Castleman疾病相关)、EBV(与Burkitt淋巴瘤、鼻咽癌、移植后淋巴瘤和Hodgkin疾病相关)、HBV和HCV(与肝细胞癌相关的)、SV40(与脑部癌症、骨癌、间皮瘤相关)、BKV(与前列腺癌相关的)、JCV(与脑部癌症相关)、HERV(与生殖细胞瘤、乳腺癌、卵巢癌和黑素瘤相关)、HMTV(与乳腺癌相关)、KSHV(与Kaposi肉瘤相关)和/或TTV(与胃肠癌、肺癌、乳腺癌和骨髓瘤相关)。但是,应该领会的是,合适的病毒还包括对于特定疾病的关联目前未知的那些。
另一方面,适用于在本文的病毒序列还可以通过一种或多种普通分类器进行分层,其可以包括一组病毒中的器官特异性(例如HBV、HCV)、癌症类型特异性或风险类型。例如在病毒为HPV病毒的情况下,合适的病毒基因组序列可以包括与高风险宫颈癌或其他泌尿生殖系统癌症相关的那些,包括16、18、31、33、35、39、45、51、52、56、58、59、68、69、73和/或82型HPV。最通常地,病毒基因组为或涵盖完整的基因组。然而,还涉及更小部分的基因组,其包括病毒基因组部分,例如一个或多个单一的病毒基因或转录单元,或者病毒的完整基因组的至少10%、至少30%、至少50%或至少70%。
特别优选嵌合参照核酸序列将包括病毒核酸序列作为附加于参照基因组核酸序列上的一个或多个个体单元。最通常地,用于各个病毒核酸序列的个体单元被组织/标记为个体染色体。在其他的优点中,应该注意使用这种排布(特别是在序列比较是使用增量同步比对(incremental synchronized alignment)来进行的情况下)允许对基因组整合位点、拷贝 数测定和受影响的等位基因进行快速鉴定。因此,还涉及病毒核酸序列以与参照基因组核酸序列相同的格式(例如BAM、SAM、FASTA或FASTA索引)被组织,然而不特意排除备选格式。就上文的观点而言,因此应该认为用于哺乳动物的嵌合参照核酸序列的染色体总数可以显著地超过来自病毒相关肿瘤的核酸序列的染色体总数。例如嵌合参照核酸序列的染色体总数可以超过来自病毒相关肿瘤的核酸序列的染色体总数至少1条、至少5条、至少10条、至少20条、至少50条以及甚至更多。事实上,确切的染色体总数将由所包含病毒基因组序列的数量而确定。
就此而言,本发明人涉及一种方法,其中通过将编辑仪与序列数据库进行信息偶联,对用于基因组分析的参照序列进行修饰,其中所述序列数据库储存了来自哺乳动物组织的一种或多种核酸序列,以及来自各种不同病毒的一种或多种病毒核酸序列。编辑仪接着用于将来自哺乳动物组织的核酸序列与多种病毒核酸序列合并成单个嵌合核酸序列文件。当然,应该领会的是,此类编辑可以使用相对少量的所选病毒基因组序列以人工方式实施,或者以自动化方式实施,其中病毒集合相对较大。此外,应该领会的是,编辑仪可以将任何格式的病毒序列合并至(例如哺乳动物/人类)参照序列中,并且病毒序列可以转化为所需最终格式(例如BAM、SAM、FASTA或FASTA索引格式)。然而,通常优选病毒序列已经为所需的最终格式(例如BAM、SAM、FASTA或FASTA索引格式)。例如,参照序列或多个参照序列可以以具有相关FASTA索引的FASTA文件储存,并且所述文件接着可以如上文所述与一种或多种病毒基因组序列合并。如果需要/必须,可以进一步转化为BAM格式。此外,来自患者肿瘤的测序数据(包含病毒序列)还可以以BAM文件储存。
此外,对于嵌合核酸序列的结构而言,特别优选来自哺乳动物组织的核酸序列根据染色体结构(例如在BAM格式的情况下)在单个嵌合核酸序列文件被组织,而病毒核酸序列作为各个单个染色体在单个嵌合核酸序列文件中被组织。一旦嵌合核酸序列文件被组装,优选接着用如此产生的嵌合核酸序列文件对序列数据库进行更新。当然,还应该认识到编辑仪还可以用于使来自哺乳动物组织的核酸序列在传输过程中与来自病毒基因组序列文库的一种或多种病毒核酸序列快速(on-the-fly)合并,这样可以 进行增量同步比对,如下文进一步讨论的那样。对于合适的序列及其部分,采取上文已经提供的相同考虑。
对于用于与嵌合核酸序列比较的合适的患者样品而言,通常优选患者样品得自被诊断或疑似患有癌症(特别是病毒相关癌症)的患者。具有多种获得此类样品的方式,并且通常优选样品包含癌症组织。接着以公知的方式对样品进行处理,从而由患病组织(例如病毒相关肿瘤)获得核酸序列。在本发明主题的特别优选的方面中,整个基因组的覆盖度为至少20倍,更多为至少30倍。因此,通常优选的是来自病毒相关肿瘤的核酸序列覆盖肿瘤的完整基因组。然而,还应该领会的是,病毒相关肿瘤的较小核酸部分也被认为是合适的,特别是在已经猜测整合事件的位点的情况下。因此,涉及来自病毒相关肿瘤的核酸序列包含病毒相关肿瘤至少1条染色体、3条染色体、5条染色体或10条染色体的核酸序列。从另一种观点来看,还涉及来自病毒相关肿瘤的核酸序列包含病毒相关肿瘤外显子组的至少一部分(例如至少10%或者10-30%之间,至少13%或者30-50%之间,至少50%或者50-80%之间)。因此,来自病毒相关肿瘤的核酸序列可以包含病毒相关肿瘤完整基因组的至少10%(例如10-30%之间或更高)、至少30%(例如30-50%之间或更高)、至少50%(例如50-80%之间或更高)。
就此而言,应该注意的是,由患病组织得到的所有样品均被认为是适用于本文的,无论此类组织是癌症或者甚至癌前组织与否。事实上,认为所有患病组织和疑似患病组织均适于本文。然而,特别优选组织包括由病毒相关肿瘤得到的那些。例如用于病毒相关肿瘤的示例性病毒包括与以下相关的那些:HTLV-1(人类T细胞白血病病毒)、HPV病毒(人类乳头状瘤病毒,特别是16、18、31、33、35、39、45、51、52、56、58、59、68、69、73和/或82高风险型)、HHV-8(人类疱疹病毒8)、EBV(EB病毒)、HBV(乙型肝炎病毒)、HCV(丙型肝炎病毒)、SV40(猿猴疱疹病毒40)、BKV(BK病毒)、JCV(JC病毒)、HERV(人类内源性逆转录病毒)、HMTV(人类乳腺肿瘤病毒)、KSHV(卡波氏肉瘤相关疱疹病毒)和/或TTV(细环病毒)。
如上文已经叙述的那样,多种数据格式适用于本文,然而,用于来自 病毒相关肿瘤的核酸序列的特别优选的数据格式包括BAM、SAM、FASTA或FASTA索引格式。此外,应该注意的是,用于来自病毒相关肿瘤的核酸序列和嵌合参照核酸序列的格式是相同的,从而允许快速进行增量同步比对。
在本发明主题的其他特别优选的方面中,使用增量同步比对处理嵌合参照核酸序列和来自病毒相关肿瘤的核酸序列,以快速鉴定整合、共同扩增和基因组交换的位点。例如并且并非限定本发明的主题,通常优选使用软件工具进行基因组分析,其中嵌合参照核酸序列(其包含来自健康或参照组织的基因组核酸序列)被同步化,并且与来自病毒相关肿瘤(或者其他患病组织)的核酸序列进行增量比较。一种特别优选的工具包括之前描述于WO2013/074058A1(其以引用方式并入本文)中的BAMBAM。
使用此类方法,应该特别领会的是,不仅可以在各种样品(例如病毒和患者)中发现存在种间交叉整合的序列,还可以发现位点、拷贝数、突变等,所有这些均可以对疾病的存在、进展和/或结果产生显著的影响。因此,本发明人不仅涉及检测一种或多种种间交叉整合事件的方法,还涉及此类事件的特征,其随后用作治疗和预后评估的基础。
还应该注意的是,所涉及的分析不必仅限于对患者肿瘤样品进行基因组分析的那些,还涉及多种检验和方法,其中使用原样的或者经过处理以富集病毒核酸内容物中的样品的患者样品(例如静脉抽血或其他生物体液(例如尿液、泪液、唾液、脑脊液等)的获取)。然后可以分析如此获得的样品,从而鉴定至少一部分病毒基因组的存在情况,其中所述部分还包含至少一部分宿主基因组。因此,所涉及的系统和方法还扩展至利用由样品获得的病毒基因组核酸序列与人类(或其他哺乳动物)参照基因组之间的增量同步比对来进行分析,其中所述人类(或其他哺乳动物)参照基因组可以是或者不是上文已经描述的嵌合参照核酸序列。
因此,所涉及的系统和方法还包括诊断检验,从而证明或排除目前或之前患者中病毒特别是肿瘤相关病毒的病毒感染。因此,所涉及的诊断方法将还包括查明肿瘤与之前(或目前)的病毒感染相关的步骤。可以通过检测病毒抗原、抗病毒抗原的抗体存在情况以及检测病毒核酸、表位和/或酶的分子生物学方法等,对病毒存在情况进行审核。一旦证明或疑似存 在,可以基于所检测病毒选择合适的病毒基因组序列(例如选择病毒的种类或类型,包括肝炎病毒或HPV分型)。
无论特定序列的性质如何,通常涉及核酸序列储存于数据库中,以由分析仪和/或编辑仪进行检索,并且此类数据库可以是单个式或分布式数据库。因此,术语“数据库”应该理解为不限于单个物理装置,而是包括彼此信息偶联的多个不同的储存装置。应该进一步注意的是,应该读取针对计算机的任何语言,从而包括计算装置的任何合适的组合,包括服务器、界面、系统、数据库、代理、对等机、发动机、控制器或者单独或共同运行的其他类型的计算装置。人们应该领会,计算装置包括处理器,其被配置为执行储存在有形非暂时性计算机可读储存介质(例如硬盘驱动器、固态硬盘、RAM、闪存、ROM等)上的软件指令。优选软件指令将计算装置配置为提供下文讨论的关于所公开仪器的作用、职责或其他功能性。在特别优选的实施方案中,多种服务器、系统、数据库或界面交换数据使用了标准的协议或算法(其可能基于HTTP、HTTPS、AES、公钥-私钥交换、网络服务API、已知的金融交易协议或其他电子信息交换方法)。优选数据交换在分组交换网络、因特网、LAN、WAN、VPN或其他类型的分组交换网络之间进行。
因此,本发明人涉及一种方法,其中分析仪与序列数据库信息偶联,其中所述序列数据库储存了来自病毒相关肿瘤的核酸序列和嵌合参照核酸序列,其中所述嵌合参照核酸序列包含至少一种病毒核酸序列和哺乳动物核酸序列。分析仪接着用于(a)鉴定嵌合参照核酸序列中的至少一些病毒核酸序列与来自病毒相关肿瘤的核酸序列中的等位基因的整合;和(b)鉴定等位基因与至少一些病毒核酸序列的共同扩增。在分析结束时,一旦共同扩增达到阈值(例如阈值为至少2倍扩增、至少3倍扩增、至少5倍扩增、至少10倍扩增或更高),则更新病毒相关肿瘤的治疗建议记录。最通常地,此类记录为电子形式,并且可以储存在与分析仪信息偶联的任何装置(例如患者或医护人员的移动装置、医学实践或医疗保健提供者的本地服务器等)上。
从不同的观点来看,本发明人还涉及一种方法,其中分析仪与序列数据库信息偶联,其中所述数据库储存了来自哺乳动物组织的核酸序列和嵌 合参照核酸序列。在此类方法中,嵌合参照核酸序列将包含至少一种病毒核酸序列(更通常地,至少2种、至少5种、至少10种和更多),并且还可以进一步包含哺乳动物(例如人类)核酸序列。分析仪接着用于鉴定来自哺乳动物组织的至少一些核酸序列在病毒核酸序列中的整合,并且进一步用于鉴定来自哺乳动物组织的核酸序列的等位基因与至少一些病毒核酸序列的共同扩增。如之前所述,一旦共同扩增达到阈值,则更新哺乳动物的治疗建议记录,此类记录将为电子形式,并且可以储存在与分析仪信息偶联的任何装置上。
同样地并且进一步针对下文所述的实施例,本发明人涉及一种方法,其中分析仪与序列数据库信息偶联,其中所述序列数据库储存了来自患者宫颈肿瘤的核酸序列和嵌合参照核酸序列,其中所述嵌合参照核酸序列包含来自患者的参照序列(优选地为匹配的正常的核酸序列)以及HPV病毒的一种或多种病毒核酸序列。分析仪接着用于鉴定嵌合参照核酸序列中至少一些病毒核酸序列与来自宫颈肿瘤的核酸序列中致癌基因(例如编码生长因子受体的基因(包括ERBB2)、肿瘤抑制基因、参与细胞周期调节的基因和/或参与细胞分裂的基因)的至少一个等位基因的整合。在另一个步骤中,分析仪还用于识别等位基因与至少一些病毒核酸序列的共同扩增,并且一旦共同扩增达到阈值,则更新病毒相关的肿瘤的治疗建议记录。
应该认识到同步增量分析和庞大的序列文件使得所述的此类方法完全不适用于人类实践,这是因为即使人们每天分析10000个碱基,此类文件分析也容易超过人类的生命周期。此外,用于基因组排布的解决方法的计算将进一步增加人类行为的不可能性。此外,应该指出的是,嵌合参照核酸(即合并的病毒核酸序列和哺乳动物核酸序列,其中病毒序列作为个体染色体被组织/索引)的特定文件结构将具有极大改善分析时间的技术作用,这是因为这种文件结构(a)与将整个序列加载至存储器中相比,可以快速地处理而无需太多的存储要求;和(b)允许基因组整合的快速分析和共同扩增的鉴定,这是由于此类方法仅需要分析2个序列文件,而不是分析3个或多个序列文件(其另外由病毒数量决定)。
此外,除非在本文中另外说明,或者上下文另外清楚地反驳,本文所述的所有方法均可以以任何合适的顺序实施。针对本文某些实施方案而提 供的任何和所有实例或示例性语言(如“例如”)的使用仅仅意在更好地说明本发明,并非对另外主张的本发明的范围进行限定。说明书中的语言均不应该解释为表明实践本发明所必需的任何未主张的要素。
在其他所涉及的方法中,应该认识到一旦鉴定出来自病毒宿主的基因(例如致癌基因、肿瘤抑制基因、参与细胞周期调节的基因和/或参与细胞分裂的基因)的共同扩增,所获得的结果可以以计算机模拟的方式建立宿主基因组被扩增部分的潜在影响的模型,由此鉴定其他的治疗选择和靶标。在其他建模系统中,特别优选的建模系统包括途径分析模型,特别是PARADIGM,其在WO2011/139345和WO/2013/062505中有所描述,这两份文献均以引用方式并入本文。例如在发现ERBB2被扩增的情况下,此类信息可以用于途径识别算法中,从而研究不同于或除了抑制ERBB2的基因产物以外的治疗方法。
实施例
患者病史:2012年3月,发现1名44岁女性患有宫颈息肉。对息肉进行活检,发现其为低分化的宫颈腺癌。该女性之前的帕氏涂片均是正常的,最后一次涂片是在2011年9月进行的。2012年4月,该女性进行广泛性子宫切除术,包括双侧输卵管切除术和淋巴结切除术。该患者选择保留其卵巢。外科病理学证明壁浸润至宫颈外部三分之一处。水平扩散至2.7cm。未见淋巴、血管或子宫浸润。淋巴结为阴性。患者未接受任何术后化疗或放射治疗。该患者的术后期并发盆腔脓肿和全身性感染,随后恢复。患者在8周内返回工作,并且其他是正常的。
2012年7月,患者发展成盆腔不适和排尿困难。病情检查(包括超声、CT扫描和PET扫描)显示多个盆腔肿块,其中最大SUV为43.9。针吸活检证实为低分化腺癌,与最初的活检一致。患者开始进行卡波铂和吉西他滨治疗,但是由于在别处实施的体外检验结果,2012年8月改为长春瑞滨、他莫昔芬和吉非替尼。在4周剂量后,CT/PET复查中发现肿瘤发生进展,患者恢复为卡波铂和吉西他滨治疗,并接受1个剂量。随后,患者选择进行“替代性治疗”。疾病持续进展,并且在2012年9月末发展成阻 塞性肾衰竭,使用输尿管支架进行治疗。
2012年11月末,患者进行其转移性肿瘤的腹腔镜活检,用于多种检验,包括全基因组测序、癌症基因窗(cancer gene panel)测序、RNA表达谱、免疫组织学和FISH,并异种移植至免疫缺陷型小鼠中。这些检验均证实HER2(ERBB2)基因扩增并且蛋白过表达。2012年12月,使用曲妥单抗和拉帕替尼对患者进行治疗,应答较佳。其腹部疼痛消除,减少了30磅腹水,并且其左下肢体淋巴水肿在3周内均消除。2013年2月的CT/PET扫描证实其肿瘤产生明显应答。
患者表现良好,直至2013年4月,其发展出运动性呼吸困难和疼痛。CT/PET扫描显示具有胸腔积液和重度肾积水的疾病进展。放置肾盂引流管。终止曲妥单抗,并且开始用曲妥珠单抗(ado-trastuzumab emtansine)治疗q3wks,以及帕妥珠单抗和拉帕替尼。2013年6月的CT/PET扫描复查显示持续的进展。实施针吸活检从而检测肿瘤是否保持HER阳性。基因分析和对FFPE切片的FISH检验证实HER2扩增和过表达,与之前于2012年11月进行的活检具有最小的基因变化。在4周的过程中,使用曲妥单抗、拉帕替尼、长春瑞滨和高剂量他莫昔芬治疗患者,但是骨盆疼痛、恶心和腹部气胀的症状加重,并且根据小鼠异种移植物研究的结果,用卡波铂替代长春瑞滨和他莫昔芬。目前,患者继续使用这种卡波铂、曲妥单抗和拉帕替尼的方案。9月3日的CT/PET扫描显示疾病的进展。
在使用卡波铂/吉西他滨、长春瑞滨/他莫昔芬/吉非替尼治疗后复发,并且对另外几轮卡波铂/吉西他滨无应答之后,在患者中实施全基因组测序和定量蛋白质组学分析。全基因组序列分析显示患者的肿瘤样品除了包含大量的体细胞突变、几种体细胞重排和染色体22的杂合性丢失(数据未示出)以外,还包含大量的映射18型HPV的读值。注意到了ERBB2、CDH1、CLTCL1和PTPRK中的突变,所有这些突变在乳腺癌中常见。患者的肿瘤基因组分析揭示了患者肿瘤样品中存在HPV-18的证据,包括导致ERBB2基因显著扩增的基因组整合。通过质谱对福尔马林固定的组织的蛋白质组学分析确认并定量了由于ERBB2扩增所导致的患者肿瘤细胞中HER2蛋白的过表达。在该患者中所见的过表达水平与FISH阳性扩增和曲妥珠单抗的临床效力有关。
根据这些综合的omic(基因组学加蛋白质组学)发现,在先前经过四线化疗后复发之后施用曲妥单抗(针对乳腺和胃癌的批准的治疗),几个月内患者产生明显应答,使疾病稳定。随后的CT/PET扫描证实作为对曲妥珠单抗的应答,患者的肿瘤明显收缩。因此并且从不同的观点来看,应该领会的是,癌症患者的分子标志与结构上的肿瘤类型无关,其需要使用全基因组测序来测定可以与生存结果直接相关的染色体重排。
方法:由Illumina Clinical Services Laboratory对活检组织进行全基因组测序,并且与正常产生的大约25亿读值进行匹配,使得测序深度分别为45.85×和30.69×。使用BWA(Burrows-Wheeler Aligner,例如参见http://bio-bwa.sourceforge.net/)将所有读值与经修正的UCSC人类参照HG19比对,其中所述经修正的UCSC人类参照HG19包含2013年3月5日NCBI中可利用的所有已知的病毒序列,其中每个病毒序列在参照文件中作为分开的染色体处理。使用Five3Genomics序列处理平台,从而产生拷贝数估值、体细胞变体和重排,如WO2013/074058A1、WO2011/149534A2和WO2013/086424A1中所述,所有上述文献以引用方式并入本文。
结果:基因组序列分析显示患者肿瘤样品除了包含48个体细胞突变、4个体细胞重排、染色体22的杂合性(LOH)丢失以外,还包含大量映射18型HPV的读值,这些改变中的2种(ERBB2和CDH1)以及HPV-18+状态均已知与癌症有关(如在the Sanger Cancer Gene Census中所述),由此被认为与患者的宫颈癌诊断有关。在文献综述揭示了XAB2在基因组稳定性和对PARP抑制剂的敏感性中的作用之后,XAB2中的错义突变还被鉴定为临床上相关。此外,染色体22经历了杂合性)LOH)丢失事件,其包括单个拷贝的已知癌症基因CLTCL1、SMARCB1、EP300和NF2的缺失。
ERBB2为原癌基因受体酪氨酸激酶,其编码蛋白质ErbB2生长因子受体(HER-2/neu);HER2与其他EGF受体结合,从而形成异源二聚体,该异源二聚体使配体的结合稳定,并促进激酶介导的下游途径,其中一些途径促进细胞生长并抑制细胞凋亡。已知HER2在乳腺、卵巢、胃和侵入性子宫癌中过表达,并且通常与较低的生存率有关。在该患者中,ERBB2表现为被扩增并且以可以直接映射HPV18序列的重排作为侧翼,如图1 中所示。更具体而言,图1描绘了患者染色体17的一部分的图示,其位于大约位置37.85mb和下游区域,如图表上方所示。在图表底部附近的箭头和箭头内的方框示意性地表示基因,其中ERBB2基因完全显示,方框表示各个基因的外显子。基因上方的方框以线性图的形式显示了特定区域序列的拷贝数。由线性图易于显而易见的是,染色体17上的大部分人类序列以基本相同的拷贝数扩增,之间具有一些小的缺口。缺口上方的半圆形图标表示基因组重排,其连接彼此面对的扩增序列的各个末端,而上方区段中的四分之一环形图标表示chr.17的人类序列与chr.17外部另一个序列的连接。在这种情况下,其他序列为NC_001357.1,其为HPV-18。因此,应该容易显而易见的是发生了chr.17和HPV-18序列的基因组重排。为了便于参考,来自chr.17的人类相邻的序列示意性地描绘成标记为h1,h2,h3和h3的方块。
图2示出了当查看嵌合参照核酸序列中的HPV-18病毒基因组序列时,由相同的患者得到的结果(此外仅示出针对HPV-18的“染色体”)。在此,再次以线形图示出扩增,而相应的病毒基因E1、E2,、E4、E5-7、L1和L2在线性图的下方以方框示出。由线性图上方的伸出的半环可见,存在连接病毒基因组末端的读值,其表示环状基因组。由半环上方的四分之一环形还易于显而易见的是,存在连接HPV-18序列和人类chr.17中的序列的读值。值得注意的是,病毒L1基因的一部分在HPV-18中缺失。再次,为了便于参考,相邻的病毒序列标记为v1、v2、v3和v4。
综上,由此应该易于显而易见的是,病毒HPV-18和人类chr.17的一部分共享了相邻的读值,代表发生了相互的基因组重排,其中HPV-18基因组的一部分整合至人类基因组中,而且其中人类基因组的一部分整合至病毒基因组中。图3示出了与图1和2中可见信息一致的图表式排布。由图3可见,扩增的人类区段h1的左端与扩增的病毒区段v4的左端连接(还参见图1中左侧的第一个四分之一环形和图2中右侧的最后一个四分之一环形),扩增的病毒区段v4的右端则连接扩增的病毒区段v1的左端(还参见图2中伸出的半环的末端)。可以对重排点进行分析,从而获得图4中示意性示出的环形方法,其中示出了包含大部分ERBB2的环形HVP-18病毒基因组(注意缺少h3不影响ERBB2中的任何外显子)。
扩增显示得到大约总共18个拷贝,其中最小的等位基因数量为1。这表明扩增事件仅涉及ERBB2的2个等位基因中的1个。在小部分的读值中,存在有效地删除外显子16的重排,尽管该重排的深度表明其可能是晚期事件。这种晚期重排的功能含义尚不清楚。来自该患者的活检组织的免疫组织化学证明其肿瘤细胞中HER2的过表达。
通过在患者基因组中发现HPV-18标志基因组以及通过活检样品的荧光原位杂交(FISH),发现该患者为HPV-18+。数据(参见图1-4,其描绘了具有明显拷贝数改变的患者染色体17q12,以及L1缺失的18型HPV)明确地显示了由HPV-18基因组至染色体17上HER2侧翼区域的清楚的重排,其中拷贝数直接支持HER2和HPV-18具有相等的拷贝数。这表明HPV基因组在L1(衣壳蛋白)位置处将HER2直接整合至其序列中,或者HPV-18整合至患者基因组中使得包含HER2的序列串联重复被多次拷贝入染色体17中。无论整合方式如何,应该理解的是,ERBB2的扩增强烈地表明患者中发现过表达的Her2是由于基因组整合和ERBB2基因的共同扩增,这本身提出了通过给予Her2抑制剂而观察到有效的新的治疗靶标。
患者呈现宫颈癌,但是基于测序的检验指向独特的显示—更通常地在乳腺癌中发现的突变;在该患者中,我们观察到ERBB2、CDH1、CLTCL1,所有这些均可以在乳腺癌中发现。患者的HPV-18+状态也值得注意,其中其L1衣壳蛋白基因缺失,这表明肿瘤的进展。
认为HPV-16和18为高风险HPV病毒株。由图4还可见,称为L1的HPV衣壳蛋白(发现其在该患者中缺失)通常在最初感染和生产阶段中存在,但是在癌前病变过程中缺失或不表达。L1表达的减少与宫颈上皮内瘤样病变进展成浸润性宫颈癌有关。癌症中值得注意的两个其他的HPV蛋白为E6和E7,其分别结合并使肿瘤抑制因子P53和pRb失活(关于人类乳头瘤病毒(HPV)和宫颈癌的WHO/ICO信息中心,www.who.int/hpvcentre)。
缺少L1衣壳蛋白基因的HPV-18基因组的存在以及CLTCL1笼形蛋白基因的缺失是潜在引人关注的巧合发现。HPV L1通过结合上皮细胞表面上的硫酸乙酰肝素蛋白多糖而介导笼形蛋白依赖性细胞进入。笼形蛋白质非依赖性内吞作用可以由小型衣壳蛋白L2通过模联蛋白(annexin)A2来 介导。CLTCL1是笼形蛋白介导的登革热病毒和人类肠道病毒71感染性的必需成分。
对于本领域技术人员应该显而易见的是,在不脱离本文所述的本发明理念的情况下,除了已经描述的那些以外,更多的修改是可行的。因此,除了所附的权利要求书的精神以外,本发明的主题不能被限定。此外,在理解说明书和权利要求书的过程中,所有的术语均应该以与上下文一致的最广泛可行的方式来理解。具体而言,术语“包含”(动词)和“包含”(动名词)应该解释为以非排除方式用于要素、组分或步骤,从而表明所涉及的要素、组分或步骤可以与未明确涉及的其他要素、组分或步骤一起存在、使用或结合。在本说明书的权利要求书涉及选自A、B、C……和N中的至少一种事物的情况下,该内容应该解释为仅需要选自上述的一种要素,既不是A加N,也不是B加N,等等。此外,如在本说明书及其后的整篇权利要求书中所用,“a”、“an”和“the”的含义包括复数指代,除非上下文另外清楚地说明。此外,如本说明书中所用,“在……里”的含义包括“在……里”和“在……上”的含义,除非上下文另外清楚地说明。
- 还没有人留言评论。精彩留言会获得点赞!