将非天然氨基酸掺入蛋白质中的制作方法
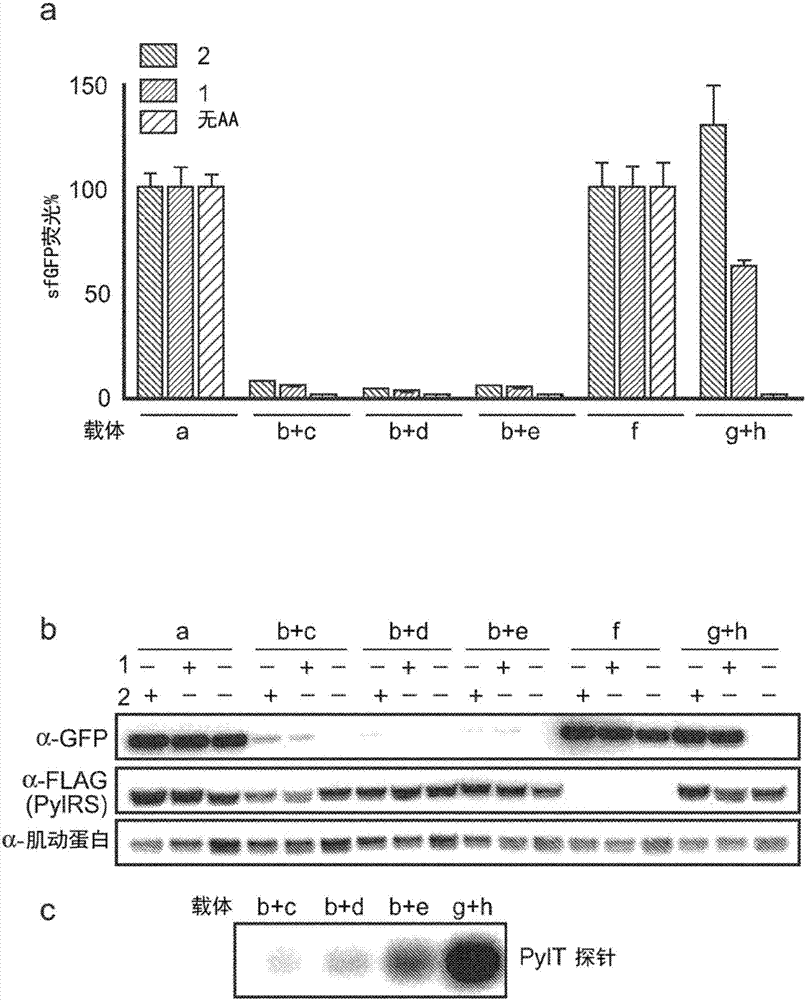
本发明属于蛋白质表达的一般领域,特别是将非天然氨基酸掺入目的蛋白质中的领域。
背景技术:
:遗传密码扩增允许将超过一百个非天然氨基酸位点特异性掺入蛋白质中。然而,这些方法的用途可能受限于非天然氨基酸掺入蛋白质中的效率。将非天然氨基酸有效、共翻译、位点特异性掺入蛋白质中将使得能够出现用于产生位点特异性修饰的重组蛋白(1,2)的方法,以及体内精确控制和成像蛋白质功能的策略(3,4)以及使用设计者非天然氨基酸来控制或报告蛋白质功能的许多其它方法。正交trna合成酶/trna对引导非天然氨基酸的掺入,最通常是响应于琥珀终止密码子(uag)。非天然氨基酸掺入的效率通过以下二者来定义:i)正交合成酶/trna对能够响应于核糖体的a位点中的uag密码子进行翻译延伸的内在效率,以及ii)释放因子与氨酰化正交trnacua竞争以终止蛋白质合成的效率。吡咯赖氨酰(pyrolysyl)-trna合成酶(pylrs)/trnacua对可以说是针对遗传密码扩增所开发的最有用的一对,因为i)它在包括大肠杆菌(e.coli)、酵母、哺乳动物细胞、线虫(c.elegans)和黑腹果蝇(d.melanogaster)在内的一系列宿主中是正交的(orthogonal),ii)pylrs不识别常见的20个氨基酸,iii)pylrs不识别同系物trnacua的反密码子,iv)pylrs的活性位点适应一系列荷载有用功能团的非天然氨基酸而不需要定向进化,v)pylrs的活性位点可以进化以识别荷载一系列在大肠杆菌中有用的功能团的结构多样的非天然氨基酸,和vi)在大肠杆菌中发现的合成酶变体可用于多种真核宿主,在此合成酶的定向进化的实施面临挑战(5)。目前非天然氨基酸的掺入在真核细胞中的效率比其在大肠杆菌中的效率更低。在实现遗传密码扩增方法的潜力的过程中,在真核细胞中向蛋白质中有效、位点特异性地掺入非天然氨基酸面临重大挑战。解决这一挑战将允许在真核细胞中合成修饰的重组蛋白质,并增加将新的化学功能引入蛋白质中以在真核细胞中以高的空间和时间精度控制和成像其功能的新兴策略。本发明寻求解决这个需求。发明概述本发明人已经开发了一种基于正交合成酶/trna对的表达系统,用于将一种或多种非天然氨基酸有效地掺入到在真核细胞(例如哺乳动物细胞或昆虫细胞)中表达的目的蛋白质中。有利地,本文所述的表达系统增加了在这些细胞中掺入非天然氨基酸的效率。此外,本发明人已经工程改造了erf1(其通常在哺乳动物细胞中的所有三个终止密码子上终止翻译)以提供响应于tag密码子的非天然氨基酸掺入的显著增加而不增加其他终止密码子的通读(read-through)。本文提供的数据首次证明,尽管天然erf1识别所有三个终止密码子,仍然可以工程改造erf1以选择性地提高真核细胞中响应于琥珀终止密码子的非天然氨基酸的掺入效率而不增加乳白或赭石终止密码子的通读。释放因子存在于原核系统中,如大肠杆菌(e.coli)表达系统。已经研究了温度敏感的释放因子(如tsrf1)用于原核表达系统中琥珀抑制的瞬时增加。细菌rf1与rrna的相互作用已被精确定位于原核系统中rrna的530环。例如,wo2008/065398在第4页第31至35行提到了这种相互作用。然而,没有从大肠杆菌系统到作为本发明主题的真核系统的跨越(crossover)。除了名称之外,原核和真核蛋白之间没有直接类推(analogy)。将突变体从非常不同的细菌蛋白转移到作为本发明主题的真核蛋白是不可能的。在原核系统中,不同的rf蛋白发挥不同的生物学功能,与之相比,真核系统有一种生物学上具有非常不同的“分裂功能”安排(arrangement)。相比之下,在真核系统中,单个erf1蛋白提供多种终止功能。因此,哺乳动物erf1蛋白质可能被认为在技术上与原核系统中的释放因子蛋白质非常不同。因此,通过选择性破坏rf1功能而在大肠杆菌中开发的用于增强响应于琥珀密码子的非天然氨基酸掺入的策略(12-16)不能扩展到真核系统。某些erf1突变体是本领域已知的。已经在尝试研究erf1功能的过程中纯粹描述了这些突变体。这些公开内容集中于erf1生物学的学术研究。相比之下,本发明人首次教导使用某些erf1突变体将非天然氨基酸掺入蛋白质中。实际上,已知没有报道工程改造真核翻译机制以提高使用正交trna合成酶/trna对在真核细胞中将非天然氨基酸位点特异性掺入蛋白质中的效率。本发明人首次在琥珀密码子表达系统的环境中实现erf1突变体的利用。因此,本发明的优点是本发明人首次教导通过使用erf1突变体增强了对琥珀密码子的抑制。因此,本发明的优点是本发明人首次教导使用erf1突变体来增强抑制琥珀密码子。有利地,通过将改良的表达系统与工程改造的erf1组合,携带单个非天然氨基酸的蛋白质的产量增加了17至20倍。含有非天然氨基酸的蛋白质的产率可以与不含终止密码子的基因产生蛋白质的产率相当。此外,改良的系统增加了蛋白质的产量,在多个位点(例如,3个或更多个位点)掺入非天然氨基酸从不可测量的低水平提高至无琥珀终止对照的43%。该方法可以有效地产生位点特异性修饰的治疗性蛋白质,并且能够使靶向细胞蛋白质用含有非天然氨基酸的形式定量替代,这使蛋白质功能的成像或合成调节成为可能。有利地,本公开内容可以使得能够在真核细胞中有效地产生位点特异性修饰的治疗性蛋白质,并能够使靶向细胞蛋白质用含有非天然氨基酸的形式定量替代,这使蛋白质功能成像或合成调节成为可能。因此,一方面,本发明提供了一种将非天然氨基酸掺入真核细胞的目的蛋白质中的方法,所述方法包括以下步骤:i)提供表达正交trna合成酶-trna对的真核细胞、编码所述目的蛋白质的目的核酸序列和突变体erf1,所述突变体erf1具有与seqidno:4的人野生型erf1序列具有至少67%序列同一性的氨基酸序列,所述目的核酸序列在用于掺入非天然氨基酸的位置上包含由trna识别的密码子;ii)在待掺入由所述目的核酸序列编码的蛋白质中的非天然氨基酸存在的情况下培养真核细胞,其中所述非天然氨基酸是正交trna合成酶的底物;和iii)培养真核细胞以允许通过正交trna合成酶-trna对将所述非天然氨基酸掺入目的蛋白质中。一方面,本发明涉及突变体erf1用于将非天然氨基酸掺入真核生物细胞的目的蛋白质中的用途,所述突变体erf1具有与seqidno:4的人野生型erf1序列具有至少60%、更适当地67%序列同一性的氨基酸序列。一方面,本发明涉及突变体erf1多肽或编码其的核酸,所述突变体erf1具有与seqidno:4的人野生型erf1序列具有至少60%、更适当地67%序列同一性的氨基酸序列,用于帮助通过翻译编码目的多肽的核酸将非天然氨基酸掺入所述目的多肽中,所述核酸包含引导将所述非天然氨基酸掺入所述目的多肽中的正交密码子。一方面,本发明涉及包含如上所述的突变体erf1多肽或核酸的真核宿主细胞。一方面,本发明涉及真核宿主细胞,其包含(i)正交trna合成酶-trna对,和(ii)突变体erf1,所述突变体erf1具有与seqidno:4的人野生型erf1序列具有至少60%、更适当地67%序列同一性的氨基酸序列,以及任选地(iii)编码目的蛋白质的目的核酸序列,所述目的核酸序列在用于掺入非天然氨基酸的位置上包含由trna识别的密码子。一方面,本发明涉及包含核酸的组合或试剂盒,所述核酸编码:(i)正交trna合成酶-trna对,和(ii)突变体erf1,所述突变体erf1具有与seqidno:4的人野生型erf1序列具有至少60%、更适当地67%序列同一性的氨基酸序列,以及任选地(iii)编码目的蛋白质的目的核酸序列,所述目的核酸序列在用于掺入非天然氨基酸的位置上包含由trna识别的密码子。一方面,本发明涉及如上所述的真核宿主细胞或如上所述的组合或试剂盒,还包含非天然氨基酸,例如bock或cypk或bcnk;更适当地是bock或cypk。适当地,所述突变体erf1相对于野生型erf1对照提供了增加的非天然氨基酸掺入效率。适当地,所述突变体erf1相对于seqidno:4包含选自以下的突变或突变的组合:(i)e55(ii)n129,k130(iii)t122,s123(iv)y125(v)t58,s60,s64,l125,n129(vi)s123,l124,y125(vii)s123,l124,y125(viii)s123,l124,y125(ix)m51,k130(x)s123,l124,y125(xi)s123,l124,y125(xii)s123,l124,y125(xiii)s123,l124,y125适当地,所述突变体erf1相对于seqidno:4包含选自以下的突变或突变的组合:(i)e55d(ii)n129p,k130q(iii)t122q,s123f(iv)e55a(v)y125f(vi)t58k,s60t,s64d,l125f,n129s(vii)s123a,l124i,y125l(viii)s123r,l124w,y125r(ix)s123h,l124a,y125g(x)m51a,k130m(xi)s123a,l124l,y125v(xii)s123l,l124c,y125s(xiii)s123l,l124s,y125s(xiv)s123v,l124t,y125p适当地,所述真核细胞是哺乳动物或昆虫细胞。适当地,所述密码子是终止密码子。更适当地,所述终止密码子是uag。适当地,正交trna合成酶-trna对包含吡咯赖氨酰-trna合成酶(pylrs)/pylttrnacua对。适当地,所述trna是:(i)pylt的u25c变体,或(ii)pylt的opt变体,或(iii)pylt的u25c-opt变体。本发明的进一步的方面和实施方案在第一方面,本发明提供了用于在真核细胞(例如哺乳动物细胞或昆虫细胞)中表达trna合成酶和trna对的核酸构建体,其包含:(i)编码trna合成酶的核酸序列,其可操作地连接到能够表达trna合成酶的第一启动子;和(ii)编码trna的核酸序列,其可操作地连接到能够表达trna的第二启动子,其中第一和第二启动子彼此方向相反,或者其中trna以核酸构建体上多个拷贝的形式存在。编码该构建体的示例性核苷酸序列如seqidno:1所示。适当地,核酸构建体还可以包含编码目的核酸序列的核酸序列,其可操作地连接到能够在真核细胞中表达目的核酸序列的另外的启动子。适当地,能够表达目的核酸序列的启动子在与上述第一方面的第一启动子相同的方向上取向。适当地,能够表达目的核酸序列的启动子与第一启动子相同或与第一启动子不同。在一个实施方案中,该启动子是或者衍生自本文所述的ef-1启动子,或者是或者衍生自cmv启动子。适当地,核酸构建体还包含编码如本文所述的突变体erf1的核酸序列。在一个实施方案中,erf1突变体由表达trna合成酶的第一polii开放阅读框下游(3')的cmv启动子表达。适当地,编码突变体erf1的核酸序列和编码trna合成酶的核酸序列通过相同开放阅读框中的自切割肽连接。适当地,编码trna合成酶的核酸序列和编码突变体erf1的核酸序列通过相同开放阅读框架中的自切割肽连接。在plosone6(4)(2011)中描述了示例性的t2a自切割肽。在第二方面,本发明提供了用于在真核细胞如哺乳动物细胞或昆虫细胞中表达trna和目的核酸序列的核酸构建体,所述目的核酸序列在用于掺入非天然氨基酸的位置上包含由trna识别的密码子,所述核酸构建体包含:(i)包含目的核酸序列的核酸序列,其可操作地连接到能够在真核细胞中表达目的核酸序列的第一启动子;和(ii)编码trna的核酸序列,其可操作地连接到能够表达trna的第二启动子,其中第一和第二启动子彼此方向相反,或者其中trna以核酸构建体上多个拷贝的形式存在。编码该构建体的示例性核苷酸序列如seqidno:2所示。适当地,构建体包含编码突变体erf1、适当地是突变型哺乳动物erf1、适当地是突变型智人(homosapiens)erf1的核酸序列,其中突变体erf1选自e55d、e55a、n129p/k130q和y125f,或其两种或更多种的组合。适当地,第一和第二启动子彼此方向相反,并且trna以核酸构建体上多个拷贝的形式存在。适当地,核酸构建体包含与启动子直接连接的trna。根据该实施方案,trna直接与启动子连接,而没有位于trna和启动子之间的任何中间序列。适当地,核酸构建体包含与启动子直接连接的trna。根据该实施方案,trna直接与启动子连接,没有位于trna和启动子之间的中间序列。trna的3'末端可以间接连接到终止子序列。举例来说,终止子序列(例如,ttttt)可以通过接头连接到trna,所述接头任选地包含序列gacaagtgcgg。适当地,编码trna的核酸序列的每个拷贝都由独立的(其本身的)启动子控制。适当地,启动子排列(arrangement)包括沿第一方向取向的延伸因子启动子和在第二方向上取向的poliii启动子。适当地,第一启动子是或者衍生自ef-1启动子。适当地,第二启动子是或者衍生自u6启动子。适当地,trna存在于核酸构建体上的4、5、6、7或8个或更多个拷贝中。适当地,trna是野生型或变体trna,适当地是pylt的u25c变体。适当地,目的核酸序列包含至少1、2、3或4个或更多个终止密码子,适当地至少1、2个或3个密码子。适当地,目的核酸序列编码抗体或抗体片段。适当地,所述trna合成酶与真核细胞中的内源性trna正交,和/或所述trna与真核细胞中的内源性trna合成酶正交,和/或所述trna合成酶与真核细胞中的内源性trna正交且所述trna与内源性trna合成酶正交。在另一方面,本发明提供了包含第一方面的核酸构建体和第二方面的核酸构建体的核酸构建体的组合。在另一方面,本发明提供了包含本发明第一方面的核酸构建体和本发明第二方面的核酸构建体的核酸构建体的组合。适当地,编码突变体erf1的核酸序列在另一个独立的构建体上。在另一方面,本发明提供了包含本发明第一方面的核酸构建体或本发明第二方面的核酸构建体的载体。在另一方面,本发明提供了包含含有本发明第一方面的核酸构建体的载体和本发明第二方面的核酸构建体的载体的载体的组合。适当地,编码突变体erf1的核酸序列位于另一个独立的载体上。在另一方面,本发明提供了包含本发明第一方面的核酸构建体或本发明第二方面的核酸构建体、核酸构建体的组合、载体或载体的组合的细胞。适当地,细胞还包含编码突变体erf1、合适地突变体智人erf1的核酸构建体。适当地,编码突变体erf1的核酸序列在独立的构建体或载体上。适当地,突变体erf1选自e55d、e55a、n129p/k130q和y125f,或其两种或更多种的组合,适当地,其中在如genbank登录号af095901.1中所述的智人erf1基因序列中进行突变。在一个实施方案中,突变在密码子优化的智人erf1基因序列中进行。密码子优化的智人erf1基因序列的实例如seqidno:3所示。适当地,细胞是哺乳动物细胞或昆虫细胞。适当地,用核酸瞬时或稳定转染细胞。在另一方面,本发明提供了一种用于将非天然氨基酸掺入真核细胞如哺乳动物细胞或昆虫细胞的蛋白质中的试剂盒,其包含:(i)本发明第一或第二方面的核酸构建体;或(ii)核酸构建体的组合;或(iii)载体;或(iv)载体的组合;或(v)真核细胞;和(vi)任选的非天然氨基酸。适当地,试剂盒还包含编码突变体erf1的核酸构建体或载体或包含其的细胞。在另一方面,本发明提供了一种将非天然氨基酸掺入到真核细胞如哺乳动物细胞或昆虫细胞的目的蛋白中的方法,包括以下步骤:i)提供细胞,其中所述细胞包含如本文所述的核酸构建体的组合或载体的组合;ii)在存在待被掺入由目的核酸序列编码的目的蛋白质中的非天然氨基酸的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和iii)培养细胞以允许通过正交trna-trna合成酶对将所述非天然氨基酸掺入目的蛋白质中。适当地,将至少1个、2个、3个、4个或5个非天然氨基酸掺入目的蛋白质中。在另一方面,本发明提供了制备抗体-药物缀合物的方法,其包括以下步骤:i)提供真核细胞例如哺乳动物细胞或昆虫细胞,其中目的核酸序列编码抗体或抗体片段,并且其中所述细胞包含本文所述核酸构建体的组合或载体的组合,和ii)在存在待掺入抗体或抗体片段中的非天然氨基酸的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;iii)获得其中已经被掺入了非天然氨基酸的抗体或抗体片段;和vi)通过非天然氨基酸将抗体或抗体片段与药物部分缀合。在另一方面,本发明提供了(i)本发明的第一或第二方面的核酸构建体;和/或(ii)核酸构建体的组合;和/或(iii)载体;和/或(iv)载体的组合;和/或(v)真核细胞例如哺乳动物细胞或昆虫细胞,用于在真核细胞中将非天然氨基酸掺入到目的蛋白质中。在另一方面,本发明提供了一种将非天然氨基酸掺入到真核细胞如哺乳动物细胞或昆虫细胞的目的蛋白质中的方法,包括以下步骤:i)提供表达trna合成酶和trna对、目的核酸序列和突变体erf1的真核细胞;ii)在存在待被掺入由目的核酸序列编码的目的蛋白质中的非天然氨基酸的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和iii)培养细胞以允许通过正交trna-trna合成酶对将所述非天然氨基酸掺入目的蛋白质中。在另一方面,本发明提供突变体erf1用于将非天然氨基酸掺入真核细胞如哺乳动物细胞或昆虫细胞的目的蛋白质中的用途。在另一方面,本发明提供了鉴定erf1的突变体的方法,其增加了目的蛋白中非天然氨基酸的掺入,所述方法包括以下步骤:(i)提供能够在其中将非天然氨基酸掺入目的蛋白质中的细胞,适当地,其中所述细胞包含本文所述核酸构建体的组合或载体的组合;(ii)在存在待被掺入目的蛋白质中的非天然氨基酸的情况下以及在存在和不存在erf1的突变体的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和(iii)在存在和不存在erf1的突变体的情况下测定掺入目的蛋白质中非天然氨基酸的水平,其中在存在erf1的突变体的情况下掺入目的蛋白质中非天然氨基酸的水平增加表示erf1的所述突变体增加了在目的蛋白质中非天然氨基酸的掺入。在另一方面,本发明提供了基本上如本文参照随附的说明书和附图所述的构建体、载体、细胞、试剂盒、方法或用途。附图说明现在将参考附图进一步描述本发明的实施方案,其中:图表1示出了所使用的质粒构建体和非天然氨基酸。(a)所使用的载体示意图。pylt是编码pyltrnacua的基因和pylt*编码u25c变体。u6表示u6启动子,cmv是cmv启动子,cmvenh是cmv启动子的5'增强子片段,ef1prom是ef-1α启动子。红色条表示琥珀终止密码子的位置。(b)1(nε-[(叔丁氧基)羰基]-1-赖氨酸)和2(nε-[((2-甲基环丙-2-烯-1-基)甲氧基)羰基]-1-赖氨酸)的化学结构。图1示出了优化用于在哺乳动物细胞中响应于tag密码子的非天然氨基酸的掺入的pylrs/trnacua表达载体。(a)在荧光测定中测量的1(2mm)和2(0.5mm)掺入sfgfp中的定量。指定的构建体在hek293t细胞中瞬时表达,并且在裂解物中通过485nm下激发、然后在520nm处的荧光定量sfgfp。每个载体组合包括无氨基酸对照。将数据绘制为具有亮氨酸密码子代替终止密码子的等同sfgfp对照质粒所显示的荧光的百分比(图表1a中构建体a)。数据表示一式三份的平均值±se表示。(b)通过蛋白质印迹显示的裂解物中的sfgfp产量。等量的用指定的载体转染的和在存在所示氨基酸的情况下生长的细胞的细胞裂解物或无氨基酸的细胞裂解物用α-gfp、α-肌动蛋白和α-flag抗体进行免疫印迹。(c)在不存在氨基酸的情况下,来自构建体b+c、b+d、b+e、g+h的相对pylt/pylt*表达的northern印迹分析。有关加载对照,请参见补充图1。图2示出了erf1突变和使用pylrs/trnacua对掺入1(2mm)对终止密码子通读的影响。(a)本研究中突变的erf1位置。来自erf1(pdbid:3e1y)的n末端结构域的结构29,本研究中突变的残基为红色。(b)在用perf1(x)和cmv-pylrs/cmv-dlr(tag)瞬时转染hek293t细胞后表达人erf1变体,其中x表示引入的突变。阴性对照(-)检测内源性erf1,shrna是内源性erf1的敲低。(c)通过在不存在抑制子trna的情况下在hek293t细胞中renilla-tag萤火虫萤光素酶报告基因和erf1变体的表达来确定所有三个终止密码子的通读。将cmv-pylrs/cmv-dlr(tag)(或相应的taa、tga或丝氨酸密码子变体)瞬时转染到细胞中,并在20小时后测定表达水平。tag、taa或tga通读相对于来自含有丝氨酸密码子(tcc)的构建体的数据进行标准化。数据以一式四份测量的平均值±se表示。阴性对照(-)检测内源性erf1,shrna是内源性erf1的敲低。wt是使用黑腹果蝇(d.melanogaster)密码子重新编码的人erf1。δ100突变体的数据超出指示范围(offscale),值为:1.6%(taa)、2%(tag)和15%(tga)。(d)用perf1(x)、表达来自u6启动子的pylt的质粒c(图表1a)和其中sfgfp被renilla-tag-萤火虫代替的cmv-pylrs/cmv-renilla-tag-萤火虫形式的质粒a(图表1a)瞬时转染hek293t细胞,其中x表示引入的突变。阴性对照(-)检测内源性erf1,shrna是内源性erf1的敲低。用α-erf1和α-肌动蛋白抗体对等量的细胞裂解物进行免疫印迹。(e)erf1(x)变体增加了使用吡咯赖氨酰trna/合成酶对响应于琥珀终止密码子的非天然氨基酸掺入。按照针对小图d所述转染hek293t细胞,并在存在1mm氨基酸1的情况下生长,并在20小时后进行测量。测量了相对于携带丝氨酸密码子代替琥珀终止密码子的renilla-tcc萤火虫报告子的通读%。图3示出了将erf1e55d与优化的pylrs/trnacua对表达系统组合使得能够在哺乳动物细胞中将多种非天然氨基酸有效地掺入重组蛋白中。(a)将质粒g、h(或i,图表1a)和erf1e55d瞬时转染到hek293t细胞中,并在存在或不存在2mm氨基酸1的情况下生长48小时。在细胞裂解物中485nm下激发、然后在520nm处定量全长sfgfp。数据以四次独立测量的平均值±se表示。(b)裂解物的蛋白质印迹。(c)除了使用0.5mm氨基酸2外,与小图a相同。(d)裂解物的蛋白质印迹。图4示出了掺入一个或三个非天然氨基酸的重组sfgfp的表达、纯化和表征。(a)将质粒g、h(或i,图表1a)和erf1e55d瞬时转染hek293t细胞中,并在存在或不存在2mm氨基酸1或0.5mm氨基酸2的情况下生长48小时。全长sfgfp通过ni-nta色谱纯化。(b)电喷雾离子化质谱证实在sfgfp中的一个或三个位点上定量掺入了非天然氨基酸1和2(另见补充附图4)。图5示出了基因组整合的erf1e55d的稳定表达增强了t-rex293flp-in细胞中的琥珀抑制。通过使用t-rex293flp-in系统(lifetechnologies)产生稳定的erf1系,由于插入到限定的靶基因座,插入的转基因得到均匀的表达。a将质粒g和h或i(图1,a)瞬时转染到含有使用黑腹果蝇密码子的基因组整合型erf1e55d的t-rex293细胞中。在存在或不存在0.5mm氨基酸2的情况下培养细胞48小时。在转染前16小时加入1μg/ml四环素诱导erf1e55d的表达。在所有实验中都使用经认证的无四环素生长培养基。在细胞裂解物中485nm下激发后在520nm处定量全长sfgfp。数据以4次独立测量的平均值±se表示。b如图a所示裂解物的蛋白质印迹。加载等量的细胞裂解物。通过用具有插入erf1wt或e55d与慢病毒shrna(erf1)构建体(santacruz,如图3所示)转化t-rex293flp-in系和对原始细胞的嘌呤霉素选择,产生了组成型表达针对内源性erf1的shrna的稳定系。由于在黑腹果蝇密码子优化后缺乏与shrna互补的序列,erf1wt和e55d对于shrna(erf1)是不反应的(refractory)。c在存在或不存在2的情况下瞬时转染构建体g和h(图1)并且在生长培养基中加入1μg/ml四环素,表达sfgfp(tag)48小时,以诱导释放因子变体的表达。在细胞裂解物中485nm下激发后在520nm处定量全长sfgfp。数据以四次独立测量的平均值±se表示。d.与c组相同,除了表达sfgfp(tag)3。图6示出了erf1突变和使用pylrs/trnacua对掺入1(1mm)对dmel细胞终止密码子通读的影响。a.使用perf1(x)和uas-pylrs/uas-gfp-(tag)-mcherry/(u6-pylt)4瞬时转染dmel细胞后,表达人erf1变体(使用黑腹果蝇密码子重新编码),其中x表示引入的突变体。无终止子对照作为全长蛋白质的大小标记,阴性对照(-)在存在内源性erf1的情况下建立基线抑制效率,而没有2的阴性对照表明在不存在1的情况下通读的水平。细胞转染并生长48小时。通过计算表示全长产物与截短产物的带的强度的比,在非饱和暴露条件下根据gfp免疫染色的印迹定量通读。每个条以三次独立转染和定量实验的平均值±se表示。b.通过蛋白质印迹显示裂解物中蛋白质的表达水平。对于用指定载体转染并在1mm氨基酸1存在下或不含氨基酸的情况下生长的细胞的等量细胞裂解物,用α-gfp、α-erf1和α-ha抗体进行免疫染色,如图a所述。将所示的α-gfp印迹曝光30秒,以显示印迹中(inprint)全长产品的特有条带。将用于定量的印迹的相应曝光曝光5秒,并且不显示饱和,额外的重复也如此。c.用于瞬时转染的构建体。图7示出了在存在erf1e55d突变体的情况下将四种不同的非天然氨基酸掺入sfgfp(tag)中。图8(补充图1)示出了使用通用pylt/pyltu25c探针的northern印迹。与图1a、b类似,在质粒b+c、b+d、b+e、g+h(图表1)瞬时转染后24小时提取总rna。图9(补充图2)示出了用perf1(x)和cmv-pylrs/cmv-dlr(tag)瞬时转染hek293t细胞后表达的人erf1变体。阴性对照(-)检测内源性erf1。双重萤光素酶测定中产生的蛋白质的绝对表达水平由α-renilla蛋白印迹显示。无tfx是未转染的细胞。图10(补充图3)示出了裂解缓冲液中sfgfp荧光定量的校准曲线。纯化和连续稀释的sfgfp的荧光强度相对于样品中sfgfp的浓度作图。蛋白质在细菌表达后进行纯化,通过280nm处的吸光度测定进行定量,并在加入蛋白酶抑制剂的ripa缓冲液中进行稀释。图11(补充图4)示出了电喷雾电离质谱法,其证实了在sfgfp中的三个位点上非天然氨基酸1和2的定量掺入。较小组分表示检测期间一个1或2的氨基甲酸酯基团的自发切割,产生天然赖氨酸。相应的质量损失分别计算为100或110da。我们以前已经观察到电喷雾电离过程中发生1的氨基甲酸酯切割7。图12在双重萤光素酶报告基因测定中筛选转基因黑腹果蝇萤火虫系的卵巢中erf1e55d或δ100表达对终止密码子通读的影响。图13显示了fac图。图14显示了一张照片。图15显示了条形图表。图16显示了pyltu25c和pyltu25copt变体。图17显示了pyltu25c和pyltu25copt变体的序列。图18显示了照片和条形图表。图19示出了图。图20显示了一个图。图21显示了(a)一个图、(b)化学结构和(c)两个图。发明详述构建体和载体如本文所用,术语“构建体”或“载体”通常是指能够转运其所连接的目的核酸序列的核酸。一种类型的载体是“质粒”,其是指可以连接另外的dna区段的环状双链dna环。另一种类型的载体是病毒载体,其中另外的dna区段可以连接到病毒基因组中。某些载体能够在其被引入其中的宿主细胞中自主复制(例如,具有细菌复制起点的细菌载体和附加型哺乳动物载体)。将其它载体(例如非附加型哺乳动物载体)导入宿主细胞后整合到宿主细胞的基因组中,从而与宿主基因组一起复制。目的核酸序列可以被掺入构建体或载体(例如表达载体)中。载体可用于在兼容的宿主细胞中复制核酸。载体可以从宿主细胞中回收。载体可以是用于在相容的宿主细胞(例如真核细胞(例如哺乳动物细胞或昆虫细胞))中表达目的核酸序列的表达载体。适当地,目的核酸序列可操作地连接到能够提供目的核酸序列在宿主细胞中的表达的控制序列(例如启动子或增强子)。术语“可操作地连接”是指所描述的组分处于允许它们以其预期方式起作用的关系中。可操作地连接到目的核酸序列的调节序列以这样的方式连接,使得在与控制序列相容的条件下实现目的核酸序列的表达。载体可以被转化或转染到合适的宿主细胞中以提供蛋白质的表达。该过程可以包括在提供编码蛋白质的目的核酸序列的载体的表达的条件下培养用表达载体转化的宿主细胞,和任选地回收表达的蛋白质。载体可以是例如提供有复制起点、任选地用于表达目标核酸序列的启动子和任选的启动子的调节子的质粒或病毒载体。在细菌质粒的情况下,载体可以含有一个或多个选择标记基因,例如氨苄青霉素抗性基因。一方面,本发明提供了用于在真核细胞中表达trna合成酶和trna对的核酸构建体,其包含:(i)编码trna合成酶的核酸序列,其与可表达trna合成酶的第一启动子可操作地连接;和(ii)编码trna的核酸序列,其与可表达trna的第二启动子可操作地连接,其中第一和第二启动子彼此方向相反,或者其中trna以核酸构建体上多个拷贝的形式存在。另一方面,本发明提供了用于在真核细胞中表达trna和目的核酸序列的核酸构建体,所述目的核酸序列在用于掺入非天然氨基酸的位置上包含由trna识别的密码子,所述核酸构建体包含:(i)包含目的核酸序列的核酸序列,其可操作地连接到能够在该细胞中表达目的核酸序列的第一启动子;和(ii)编码trna的核酸序列,其可操作地连接到能够表达trna的第二启动子,其中第一和第二启动子彼此方向相反,或者其中trna以核酸构建体上多个拷贝的形式存在。另一方面,本发明还提供了包含上述每个核酸构建体的核酸构建体的组合。另一方面,本发明还提供了包含或分别包含上述每个核酸构建体的载体。另一方面,本发明还提供了包含分别包含上述每种核酸构建体的载体的载体的组合。在某些实施方案中,核酸构建体中的第一和第二启动子是独立的启动子,其被放置在相反的方向上。根据该实施方案,第一和第二启动子可以说是双向启动子,其中每个启动子均被编码在相对的链上,其5'末端朝向彼此。与启动子可操作地连接的每个核酸将具有相应的取向。因此,例如,可以在反向链上编码启动子和待表达的trna序列。可以在正链上编码启动子和待表达的trna合成酶基因。作为进一步的实例,可以在反向链上编码u6启动子和待表达的trna序列。作为进一步的实例,可以在正链上编码ef-1a启动子和待表达的trna合成酶基因。除了第一和第二启动子之外,还可以包括一个或多个另外的启动子,其可以是与第一和/或第二启动子相同的启动子,或者可以与第一和/或第二启动子不同。进一步的启动子可以在与第一或第二启动子相同的方向上取向。适当地,进一步的启动子在与第一启动子相同的方向上取向。适当地,本文所述的构建体还包含编码如本文所述的突变体erf1的核酸序列。表达突变体erf1的启动子可以在与第一或第二启动子相同的方向上取向。适当地,启动子在与第一启动子相同的方向上取向。适当地,编码突变体erf1的核酸序列和编码trna合成酶的核酸序列在同一开放阅读框中通过自切割肽连接。适当地,编码trna合成酶的核酸序列和编码突变体erf1的核酸序列在相同开放阅读框架中通过自切割肽连接。在plosone6(4)(2011)中描述了示例性的t2a自切割肽。适当地,构建体提供了多拷贝trna排列。适当地,在本文所述的构建体中提供trna基因的至少2个、3个、4个、5个、6个、7个、8个、9个、10个、11个或12个拷贝。适当地,在构建体中提供至少4个拷贝的trna基因。适当地,在构建体中提供至少8个拷贝的trna基因。可以在相同或不同构建体上提供trna基因的多个拷贝。在一个实施方案中,在第一构建体上提供至少4个拷贝的trna基因,并且在第二构建体上提供至少4个拷贝的trna基因。在一个实施方案中,trna基因的多个拷贝处于单个启动子的控制之下。在另一个实施方案中,trna基因的多个拷贝处于多个不同启动子的控制之下。在另一个实施方案中,trna基因的每个拷贝都在独立的启动子的控制下,其可以是相同的启动子或两个或更多个不同的启动子。在另一个实施方案中,trna基因的每个拷贝处于多个启动子的控制之下,其可以是相同的启动子或两个或更多个不同的启动子。适当地,每个trna基因都在独立的启动子的控制下,每个trna基因是相同的启动子。适当地,所提供的控制每个trna基因的一个或多个启动子是相同的。在这种情况下,“相同”是指在其序列方面相同,而不是暗示单个启动子序列控制多个trna序列。显然,可以存在如本文所述的相同的启动子的多个拷贝。在一个实施方案中,提供多拷贝trna排列,其中提供至少4个拷贝的trna,其中每个拷贝可操作地连接至启动子。在另一个实施方案中,提供多拷贝trna排列,其中提供至少4个拷贝的trna,其中每个拷贝可操作地连接至启动子,每个启动子是相同的启动子-例如rnapoliii启动子,例如u6启动子。在一个实施方案中,trna的5'末端直接由用于表达trna的启动子的转录起始位点定义。应当注意,本文提供的核苷酸构建体具有广泛的应用,并且可以用于乳白(opal)和/或赭石(ochre)抑制以及琥珀抑制。当将本发明的核酸构建体应用于琥珀/乳白/赭石抑制时,本领域技术人员将与适当的琥珀/乳白/赭石密码子一起选择合适的trna和/或trna合成酶。构建体和载体的组合本文所述的构建体和载体的组合涉及用于将一种或多种非天然氨基酸掺入细胞中。作为实例,构建体的组合包括:(1)核酸构建体,其包含:(i)编码trna合成酶的核酸序列,可操作地连接到能够表达trna合成酶的第一启动子;和(ii)编码trna的核酸序列,其可操作地连接到能够表达trna的第二启动子,其中第一和第二启动子彼此方向相反,或者其中trna以核酸构建体上多个拷贝的形式存在;和(2)核酸构建体,其包含:(i)包含目的核酸序列的核酸序列,其可操作地连接到能够在真核细胞中表达目的核酸序列的第一启动子;和(ii)编码trna的核酸序列,其可操作地连接到能够表达trna的第二启动子,其中第一和第二启动子彼此方向相反,或者其中trna以核酸构建体上多个拷贝的形式存在。在一个实施方案中,上述构建体(1)的目的核酸序列还包含编码的目的核酸序列的核酸序列,其可操作地连接到能够在真核细胞中表达目的核酸序列的另外的启动子。根据该实施方案,构建体(1)不一定必须与构建体(2)一起使用,因为构建体(2)的目的核酸序列和trna被掺入构建体(1)中。根据该实施方案,可能需要在另一载体上包含一个或多个trna的另外的拷贝。该另一载体可以仅包含一个或多个启动子控制下的一个或多个trna。任选地,根据需要可以将其它元件掺入该另一载体。包含如上所述的(1)和(2)的构建体的组合可以与编码本文所述的突变体erf1的另外的构建体一起使用。或者,编码突变体erf1的核酸序列可以掺入构建体(1)和/或(2)中。适当地,将编码突变体erf1的核酸序列掺入构建体(1)中。根据该实施方案,本发明公开了核酸构建体,其包含:(i)编码trna合成酶的核酸序列,其可操作地连接到能够表达trna合成酶的第一启动子;和(ii)编码trna的核酸序列,其可操作地连接到能够表达trna的第二启动子,其中第一和第二启动子彼此方向相反,或者其中trna以核酸构建体上多个拷贝的形式存在;任选地(iii)编码目的核酸序列的核酸序列,其可操作地连接到能够在真核细胞中表达目的核酸序列的另外的启动子;和任选地(iv)编码如本文所述的突变体erf1的目的核酸序列。本发明还公开了包含这些构建体的各种组合的载体和细胞。trna合成酶本文使用的trna合成酶(适当地,氨酰-trna合成酶)可以变化。尽管在实施例中可以已经使用了具体的trna合成酶序列,但本发明并不仅限于这些实施例。原则上,可以使用提供trna装载(氨基酰化)功能的任何trna合成酶。例如,trna合成酶可以来自任何合适的物种,例如来自古细菌,例如来自methanosarcina——如巴氏甲烷八叠球菌ms;巴氏甲烷八叠球菌str.fusaro;马氏甲烷八叠球菌(methanosarcinamazei)go1;噬乙酸甲烷八叠球菌c2a;噬热甲烷八叠球菌;或者布氏拟甲烷球菌——例如布氏拟甲烷球菌。或者,trna合成酶可以来自细菌,例如来自desulfitobacterium——例如desulfitobacteriumhafniensedcb-2;desulfitobacteriumhafniensey51;desulfitobacteriumhafniensepcp1;或者desulfotomaculumacetoxidansdsm771。在一个实施方案中,所述trna合成酶是吡咯赖氨酰trna合成酶(pylrs),其是具有吡咯赖氨酰trna合成酶生物活性的蛋白质。pylrs能够用非天然氨基酸酰化trna。pylrs可以是野生型或遗传工程改造的pylrs。遗传工程改造的pylrs已经描述于例如neumann等人(natchembiol4:232,2008)和yanagisawa等人(chembiol2008,15:1187)和ep2192185a1中。适当地,选择提高非天然氨基酸的掺入效率的遗传工程改造的trna合成酶基因。根据一个实施方案,pylrs来自巴氏甲烷八叠球菌(mbpylrs),任选地包含或由以下所示的密码子优化的序列组成:atggactacaaggacgacgacgacaaggacaagaaacccctggacgtgctgatcagcgccaccggcctgtggatgagccggaccggcaccctgcacaagatcaagcaccacgaggtgtcaagaagcaaaatctacatcgagatggcctgcggcgaccacctggtggtgaacaacagcagaagctgccggaccgccagagccttccggcaccacaagtacagaaagacctgcaagcggtgccgggtgtccgacgaggacatcaacaactttctgaccagaagcaccgagagcaagaacagcgtgaaagtgcgggtggtgtccgcccccaaagtgaagaaagccatgcccaagagcgtgtccagagcccccaagcccctggaaaacagcgtgtccgccaaggccagcaccaacaccagccgcagcgtgcccagccccgccaagagcacccccaacagctccgtgcccgcctctgctcctgctcccagcctgacacggtcccagctggacagagtggaggccctgctgtcccccgaggacaagatcagcctgaacatggccaagcccttccgggagctggaacccgagctggtgacccggcggaagaacgacttccagcggctgtacaccaacgaccgggaggactacctgggcaagctggaacgggacatcaccaagttcttcgtggaccggggcttcctggaaatcaagagccccatcctgatccccgccgagtacgtggagcggatgggcatcaacaacgacaccgagctgtccaagcagattttccgggtggacaagaacctgtgcctgcggcctatgctggcccccaccctgtacaactacctgcggaaactggacagaatcctgcctggccccatcaagattttcgaagtgggaccctgctaccggaaagagagcgacggcaaagagcacctggaagagtttacaatggtgaatttttgccagatgggcagcggctgcacccgggagaacctggaagccctgatcaaagagttcctggattacctggaaatcgacttcgagatcgtgggcgacagctgcatggtgtacggcgacaccctggacatcatgcacggcgacctggaactgagcagcgccgtggtgggacccgtgtccctggaccgggagtggggcatcgacaagccctggatcggagccggcttcggcctggaacggctgctgaaagtgatgcacggcttcaagaacatcaagcgggccagcagaagcgagagctactacaacggcatcagcaccaacctgtgatgataa根据具体的实施方案,pylrs来自马氏甲烷八叠球菌(methanosarcinamazei)(mmpylrs),任选地包含或由以下所示的密码子优化的序列组成:atggactacaaggacgacgacgacaagggacaagaagcccctgaacaccctgatcagcgccacaggactgtggatgtccagaaccggcaccatccacaagatcaagcaccacgaggtgtcccggtccaaaatctacatcgagatggcctgcggcgatcacctggtcgtcaacaacagcagaagcagccggacagccagagccctgcggcaccacaagtacagaaagacctgcaagcggtgcagagtgtccgacgaggacctgaacaagttcctgaccaaggccaacgaggaccagaccagcgtgaaagtgaaggtggtgtccgcccccacccggaccaagaaagccatgcccaagagcgtggccagagcccccaagcccctggaaaacaccgaagccgctcaggcccagcccagcggcagcaagttcagccccgccatccccgtgtctacccaggaaagcgtcagcgtccccgccagcgtgtccaccagcatctctagcatctcaaccggcgccacagcttctgccctggtcaagggcaacaccaaccccatcaccagcatgtctgcccctgtgcaggcctctgccccagccctgaccaagtcccagaccgaccggctggaagtgctcctgaaccccaaggacgagatcagcctgaacagcggcaagcccttccgggagctggaaagcgagctgctgagccggcggaagaaggacctccagcaaatctacgccgaggaacgggagaactacctgggcaagctggaaagagagatcacccggttcttcgtggaccggggcttcctggaaatcaagagccccatcctgatccccctggagtacatcgagcggatgggcatcgacaacgacaccgagctgagcaagcagattttccgggtggacaagaacttctgcctgcggcccatgctggcccccaacctgtacaactacctgcggaaactggatcgcgctctgcccgaccccatcaagattttcgagatcggcccctgctaccggaaagagagcgacggcaaagagcacctggaagagtttacaatgctgaacttttgccagatgggcagcggctgcaccagagagaacctggaatccatcatcaccgactttctgaaccacctggggatcgacttcaagatcgtgggcgacagctgcatggtgtacggcgacaccctggacgtgatgcacggcgacctggaactgtctagcgccgtcgtgggacccatccctctggaccgggagtggggcatcgataagccctggatcggagccggcttcggcctggaacggctgctgaaagtcaagcacgactttaagaacatcaagcgggctgccagaagcgagagctactacaacggcatcagcaccaacctgtgatgataa适当地,编码trna合成酶的核苷酸序列是密码子优化的。trna本文使用的trna可以变化。虽然在实施例中可能已经使用了具体的trna,但本发明并不仅限于这些实施例。原则上,可以使用任何trna,条件是其与选定的trna合成酶相容。trna可以来自任何合适的物种,例如来自古细菌,例如来自甲烷八叠球菌(methanosarcina)——例如巴氏甲烷八叠球菌(methanosarcinabarkeri)ms;巴氏甲烷八叠球菌str.fusaro;马氏甲烷八叠球菌go1;噬乙酸甲烷八叠球菌(methanosarcinaacetivorans)c2a;嗜热甲烷八叠球菌(methanosarcinathermophila);或者拟甲烷球菌(methanococcoides)——例如布氏拟甲烷球菌(methanococcoidesburtonii)。或者,trna可以来自细菌,例如来自脱亚硫酸菌(desulfitobacterium)——例如desulfitobacteriumhafniensedcb-2;desulfitobacteriumhafniensey51;desulfitobacteriumhafniensepcp1;或者desulfotomaculumacetoxidansdsm771。trna基因可以是野生型trna基因,也可以是突变的trna基因。适当地,选择增加非天然氨基酸的掺入效率的突变的trna基因。在一个实施方案中,突变的trna基因(例如突变的trnacua基因)是如biochemistry(2013)52,10所述的pylt的u25c变体。在一个实施方案中,突变的trna基因(例如突变的trnacua基因)是如fan等人2015(nucleicacidsresearchdoi:10.1093/nar/gkv800)中所述的pylt的opt变体。在一个实施方案中,突变的trna基因(例如突变的trnacua基因)具有pylt的u25c和opt二者的变体,即在该实施方案中,trna如pylttrnacua基因包括u25c和opt二者的突变。在一个实施方案中,编码trna的序列是编码trnapyl、更合适的trnapylcua的来自马氏甲烷八叠球菌吡咯赖氨酸的吡咯赖氨酸trna(pylt)基因。这通过琥珀抑制、即通过识别琥珀密码子掺入非天然氨基酸。编码来自马氏甲烷八叠球菌的pylt的核酸序列的实例是:ggaaacctgatcatgtagatcgaatggactctaaatccgttcagccgggttagattcccgg在另一个实施方案中,来自马氏甲烷八叠球菌的pylt表达自具有接头的u6启动子,所述接头之后为pylt的3’端的终止子。示例性的序列是(u6启动子小写并加粗;plyt加下划线;接头大写并加粗;终止子大写并加下划线):trna合成酶/trna对适合地,trna-trna合成酶对是不识别20种天然存在的氨基酸中的任一种的对。应当理解,相应的或同系的trna或trna合成酶可以从不同物种组合,例如methanococcus细菌的不同物种。例如,可能一起使用来自马氏甲烷八叠球菌的吡咯赖氨酸trna和来自巴氏甲烷八叠球菌的吡咯赖氨酰trna合成酶。使用本领域已知的方法容易地测试此类配对的功能,例如通过将宿主细胞中的不同组分组合在一起并分析产生的完整目的蛋白质。在一个实施方案中,适当地,trna-trna合成酶对是来自methanococcus的吡咯赖氨酰-trna合成酶(pylrs)/trnacua对。在一个实施方案中,所述trna合成酶是或者衍生自来自巴氏甲烷八叠球菌(mbpylrs)的pylrs,并且trna是或者衍生自来自巴氏甲烷八叠球菌吡咯赖氨酸的吡咯赖氨酸trna(pylt)。在一个实施方案中,所述trna合成酶是或者衍生自来自马氏甲烷八叠球菌(mmpylrs)的pylrs,并且trna是或者衍生自来自马氏甲烷八叠球菌吡咯赖氨酸的吡咯赖氨酸trna(pylt)。适当地,所述trna合成酶与真核细胞中的内源性trna正交,和/或所述trna与真核细胞中的内源性trna合成酶正交,和/或所述trna合成酶与真核细胞中的内源性trna正交并且所述trna与内源性trna合成酶正交。控制序列可操作地连接到核酸序列的控制序列包括启动子、增强子和其他表达调控信号。这些控制序列可以被选择为与构建体或载体设计用于其的宿主细胞相容。术语启动子是本领域众所周知的,并且包括大小和复杂性从最小启动子到包括上游元件和增强子的启动子的范围内的核酸区域。适当地,启动子序列之一是rnapoliii启动子——例如u6启动子。适当地,rnapoliii启动子可操作地连接到trna基因。适当地,这样的排列在本发明的构建体中重复至少4、5、6、7或8次或更多次。适当地,启动子序列之一是真核延伸因子启动子——例如ef-1启动子(例如,ef-1α)。适当地,该启动子可操作地连接到trna合成酶基因和/或目的核酸序列。适当地,这样的排列在本发明的构建体中重复至少一次。rnapoliii启动子适当地,任何能够在真核细胞(例如哺乳动物或昆虫细胞)中指导rnapoliii转录的启动子可用于本文所述的构建体中。rnapoliii启动子包括内源和外源(内部和外部)启动子。适当地,所述启动子是或者衍生自真核u6启动子,适当地,智人u6启动子。一种示例性的u6启动子描述于thejournalofbiologicalchemistry(1987)262(3),1187–1193中。用于人和/或小鼠系统的示例性u6启动子描述于journaloftheamericanchemicalsociety(2010)132(12),4086–4088中。另一个示例性的u6启动子包含或由以下所示的序列组成:tgggcaggaagagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattagaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccg如本文所述,适当地,启动子是或者衍生自能够在哺乳动物细胞(例如小鼠或人类细胞)中指导rnapoliii转录的、可操作地连接到一个或多个trna基因的u6启动子。延伸因子启动子适当地,能够在真核细胞(例如哺乳动物或昆虫细胞)中指导表达的任何真核延伸因子启动子可用于本文所述的构建体中。适当地,所述启动子是或者衍生自真核延伸因子1(ef-1)启动子。适当地,所述启动子是或者衍生自ef-1α启动子。示例性的ef-1α启动子描述于anticancerres.(2002),22(6a),3325-30中。另一个示例性的ef-1α启动子包含或由以下所示的序列组成:ctagtaaggatctgcgatcgctccggtgcccgtcagtgggcagagcgcacatcgcccacagtccccgagaagttggggggaggggtcggcaattgaacgggtgcctagagaaggtggcgcggggtaaactgggaaagtgatgtcgtgtactggctccgcctttttcccgagggtgggggagaaccgtatataagtgcagtagtcgccgtgaacgttctttttcgcaacgggtttgccgccagaacacagctgaagcttcgaggggctcgcatctctccttcacgcgcccgccgccctacctgaggccgccatccacgccggttgagtcgcgttctgccgcctcccgcctgtggtgcctcctgaactgcgtccgccgtctaggtaagtttaaagctcaggtcgagaccgggcctttgtccggcgctcccttggagcctacctagactcagccggctctccacgctttgcctgaccctgcttgctcaactctacgtctttgtttcgttttctgttctgcgccgttacagatccaagctgtgaccggcgcctactctag适当地,启动子是或者衍生自能够在哺乳动物细胞(例如小鼠或人类细胞)中指导转录的、可操作地连接到如本文所述的trna合成酶和/或目的核酸序列的ef-1α启动子。宿主细胞合适的宿主细胞可以包括细菌细胞(例如大肠杆菌),但是最适当的宿主细胞是真核细胞,例如昆虫细胞(例如果蝇(drosophila)如黑腹果蝇)、酵母细胞、线虫(例如c.elegans)、小鼠(例如musmusculus)或者哺乳动物细胞(例如中国仓鼠卵巢细胞(cho)或cos细胞、人293t细胞、hela细胞、nih3t3细胞和小鼠红白血病(mel)细胞)或人细胞或其他真核细胞。其他合适的宿主细胞是本领域技术人员已知的。适当地,宿主细胞是哺乳动物细胞——例如人细胞或昆虫细胞。可以在本发明的实施方案中通常使用的其它合适的宿主细胞是实施例部分中提及的那些。可以通过常规转化或转染技术将载体dna引入宿主细胞。如本文所用,术语“转化”和“转染”旨在表示将外源核酸分子(例如dna)引入宿主细胞的各种公认的技术,包括磷酸钙或氯化钙共沉淀、deae-葡聚糖介导的转染、脂质体转染或电穿孔。用于转化或转染宿主细胞的合适方法是本领域众所周知的。当产生细胞系时,通常优选制备稳定的细胞系。例如,为了稳定转染哺乳动物细胞,已知根据所用的表达载体和转染技术,只有少部分细胞可以将外源dna整合到其基因组中。为了鉴定和选择这些整合体,通常将编码选择性标记(例如抗生素抗性)的基因与目的基因一起引入宿主细胞。优选的选择性标记包括赋予药物抗性的那些,例如g418、潮霉素或氨甲蝶呤。编码选择性标记的核酸分子可以在相同载体上引入宿主细胞中,或者可以在独立的载体上引入。用引入的核酸分子稳定转染的细胞可以通过药物选择来鉴定(例如,已经掺入选择性标记基因的细胞将存活,而其他细胞则死亡)。在一个实施方案中,本文所述的构建体整合到宿主细胞的基因组中。稳定整合的优点是实现了个体细胞或克隆之间的均匀性。另一个优点是可以选择最好的生产者。因此,期望产生稳定的细胞系。在另一个实施方案中,将本文所述的构建体转染入宿主细胞。将构建体转染到宿主细胞中的优点是可以使蛋白质产量最大化。在一个方面,本发明描述了包含本文所述的核酸构建体或载体的细胞。erf1除非从上下文中另外显而易见,否则本文中对“erf1”的引用是指真核erf1。当在本文中使用时,特别是在讨论erf1时,“突变体”具有“不是野生型”的天然含义。显然,野生型残基可以根据所使用的特定物种的erf1而变化。参考特定残基应参照genbank登录号af095901.1的erf1的智人野生型参考序列来解释。依赖于本发明的申请日的数据库版本。为避免疑问,这意味着2015年8月15日的geneticsequencedatabankncbi-genbankflatfilerelease209.0。为了避免疑问,野生型人erf1多肽序列被认为是:特别地,本申请中给出的氨基酸地址对应于上述erf1参考序列的编号。如果使用erf1的截短或延伸形式(例如,如果添加了6his标签或其中多肽的一部分缺失),那么氨基酸编号应该被视为对应于全长参考序列的等同部分,而不是一个“绝对”或极其不灵活的数字地址。作为说明,如果描述提及e55的置换,则这意味着上述erf1参考序列的氨基酸55。如果另一物种位置55在野生型中不是e,则例如通过将所述另一物种的erf1的序列与上述参考序列比对并选择相应的氨基酸来鉴定对应于如本领域众所周知的人野生型序列的e55的氨基酸酸。类似地,如果使用的多肽通过缺失前10个氨基酸被截短,则给出的地址仍将是e55(而不是例如e45)——如本领域常规知晓的,本领域技术人员将容易地理解参考上面的全长erf1序列而查阅相应上下文的氨基酸。本发明人教导了去除erf1的n末端结构域的其他截短物将具有相似的作用。erf1的n-末端结构域(大致氨基酸1-130)与信使rna和终止密码子相互作用。如果该结构域的全部或部分被删除,在使用中它应形成无活性erf1-erf3复合物(以δ100变体为例),并增加终止密码子通读(和毒性)。适当地,本发明中使用的erf1包含对应于seqidno:4的至少氨基酸131以前的氨基酸序列;适当地,包含对应于seqidno:4的至少氨基酸101以前的氨基酸序列。适当地,本发明中使用的erf1包含对应于seqidno:4的至少氨基酸101至末端的氨基酸序列。适当地,本发明中使用的erf1包含对应于seqidno:4的至少氨基酸131至末端的氨基酸序列。适当地,erf1的c末端不被截短或者仅相对于seqidno:4被最少截短。最适当地,相对于seqidno:4,erf1的c末端不被截短。所需的任何比对应通过眼睛或使用本领域已知的任何广泛可用的序列比对程序进行,例如gcg程序套件(gcggeneticscomputergroupuniversityresearchpark575sciencedrivemadison,wi53711)。最适合的比对方式是使用具有默认设置的clustalw。有利地,根据本公开可以使用erf1的某些突变体,以响应于一个或多个终止密码子而提供非天然氨基酸掺入的显著增加,而基本上不增加其它终止密码子的通读。因此,与某些erf1突变体一起在细胞中表达本文所述的核酸构建体可能是有利的。可以使用各种启动子(例如ef1启动子或cmv启动子)来表达erf1。最适当地,本发明的erf1突变体增加了非天然氨基酸掺入的效率。适当地,相对于自然翻译控制,本发明的erf1突变体增加了非天然氨基酸掺入的效率。适当地,相对于野生型erf1对照,本发明的erf1突变体增加了非天然氨基酸掺入效率。这可以如本文所教导的,例如通过参考实施例部分容易地确定。在某些实施方案中,将突变体erf1整合到宿主细胞中,适当地稳定整合入宿主细胞。在某些实施方案中,突变体erf1由宿主细胞中本文所述的一种或多种核酸构建体表达。在某些实施方案中,编码突变体erf1的核酸序列在独立的构建体或独立的载体上。真核翻译终止因子1(erf1),也称为tb3-1,是在人中由etf1基因编码的蛋白质。在真核生物中,这是识别所有三个终止密码子的唯一释放因子。蛋白质生物合成的终止和新生多肽链的释放是通过在核糖体的氨酰基位点存在框内终止密码子来传递信号的。翻译终止过程是通用的并且由蛋白质释放因子(rfs)和gtp介导。示例性erf1基因序列是genbank登录号af095901.1中所述的野生型智人erf1基因序列。适当地,erf1基因可以是密码子优化的,例如针对黑腹果蝇。使用shrna敲低哺乳动物细胞中的erf1表达在抑制瞬时增加后也可能是有害的。高水平的终止密码子通读也可能发生。显然,应当避免这些有害作用,并且在本公开内容中适当地不使用shrna。可用于本发明的erf1突变体在下表中公开:*在选择使用哪种erf1突变体时,本领域技术人员将考虑细胞活力与增加抑制之间的相互作用。例如,δ100erf1突变体可以导致高水平的终止密码子通读和病态细胞。因此,δ100erf1突变体不适用于本公开中。**作为掺入信号对于uag无效,但可能会发现用于uga。偶尔会提到未突变的位点,例如“l124l”。显然这不是一个突变,因为没有改变野生型“l”。这被理解为表明l124在该具体efr1中没有突变,即位置l124在突变的具体组合/具体示例性erf1中作为野生型(作为l)保留。对于给定位置处的每个突变,据信是许多相互密切关联的氨基酸将产生类似的效果。“示例性突变体”不是详尽无遗的。还考虑了与上述列i中鉴定的残基对“不是野生型”的突变。可以对列ii中提到的那些残基进行适当的保守置换,例如根据下表。第二列中相同组(block)中以及优选在下表第三列中的相同行内的氨基酸可以彼此置换:例如,列i中的e55可能被突变为“不是e”。更适当地,e55可以突变为对列ii中具体突变保守的氨基酸——例如e55d或e55a或e55g或e55p。最适当地,e55可以突变为列ii中特别提及的氨基酸,例如e55a或e55d,最适当地,e55d。这同样适用于列i中列出的其他残基。所有s123、l124、y125突变体在dlr测定中与e55d相比表现良好(比e55d好1-2倍),但在蛋白质表达测试中表现不如e55d。它们在本发明中仍然有用,但最优选的突变体是e55d。(sfgfp(3tag)。虽然一些突变以组合形式呈现,但是这些组合是本发明的特别优选的实例。本发明公开了使用个体erf1突变体;适当地,所述突变体erf1包含相对于seqidno:4选自e55、n129、k130、t122、s123、y125、t58、s60、s64、l125、s123、l124、m51和k130的突变。适当地,所述突变体erf1包含相对于seqidno:4选自e55d、n129p、k130q、t122q、s123f、e55a、y125f、t58k、s60t、s64d、l125f、n129s、s123a、l124i、y125l、s123r、l124w、y125r、s123h、l124a、y125g、m51a、k130m、y125v、s123l、l124c、y125s、s123l、l124s、y125s、s123v、l124t和y125p。可以通过瞬时表达或通过基因组整合在宿主细胞中提供erf1。例如,使用trexflip-in系统(人hek293-衍生的)在稳定的遗传背景中获得erf1突变体的诱导型表达。在一个实施方案中,通过瞬时转染引入相关核酸。在一个实施方案中,通过稳定的细胞系产生引入相关核酸。本文描述了各种特别合适的突变体和组合,包括m51a/k130m、t122q/s123f、s70a/g73s、e55d、e55a、n129p/k130q和y125f。适合地,本公开中使用的erf1突变体在e55处包含突变。适当地,本公开中使用的erf1突变体选自e55d、e55a、n129p/k130q和y125f或其两种或更多种的组合。这些突变相对于衍生自genbank登录号af095901.1的野生型智人erf1氨基酸序列或其密码子优化的变体进行。erf1蛋白在大多数真核生物体内表现出非常强的同源性。我们使用人erf1作为例子来引入我们的突变,但来自其他物种的erf1也可以携带相同的突变(例如人或昆虫erf1蛋白中的e55d)。我们教导这些替代物种突变体erf1蛋白质应具有与本文示例性erf所示相似的技术效果。我们使用优选的erf1突变体(工程改造人(智人)erf1变体)成功地增强了多种真核宿主细胞(包括cho细胞(中国仓鼠(c.griseus))、hek细胞(智人)和dmel细胞(黑腹果蝇))中非天然氨基酸的掺入。这些生物体中的erf1蛋白是高度保守的(表1)。表1:erf1在各种物种中的成对相似性。智人中国仓鼠黑腹果蝇酿酒酵母智人---中国仓鼠92%---黑腹果蝇84%78%----酿酒酵母67%62%67%---给出的百分比表示通过clustalw比对测定的跨蛋白质的保守氨基酸同一性。鉴于各种单细胞(酵母)和多细胞(哺乳动物、昆虫)真核生物之间的保守程度,支持来自各种真核生物的erf1变体将在多种其他真核物种宿主细胞中发挥作用。例如,宿主细胞可以是人、小鼠、线虫、驴、酵母或其他真核宿主细胞。适当地,突变体erf1具有与seqidno:4的人野生型erf1序列具有至少60%序列同一性的氨基酸序列;适当地,突变体erf1具有与seqidno:4的人野生型erf1序列具有至少67%序列同一性的氨基酸序列;适当地,突变体erf1具有与seqidno:4的人野生型erf1序列具有至少84%序列同一性的氨基酸序列;适当地,突变体erf1具有与seqidno:4的人野生型erf1序列具有至少92%序列同一性的氨基酸序列,适当地,突变体erf1具有与人野生型erf1具有至少95%序列同一性的氨基酸序列seqidno:4的序列,适当地,突变体erf1具有与seqidno:4的人野生型erf1序列具有至少98%序列同一性的氨基酸序列,或者甚至更多。在一个实施方案中,适当地,在引入针对突变体efr1的特异性突变之前,计算同一性百分比水平。优选地计算同一性百分比水平,包括突变体efr1所述的具体突变。适当地,宿主细胞是体外的。适当地,当宿主细胞处于生物体中时,宿主是非人。我们已经将示例的人erf1引入三种真核生物(人、仓鼠、蝇)的细胞系以及活的真核动物(蝇)中。工程改造的人erf1增强了天然erf1仅有84%保守性的物种(dmel昆虫细胞)中的非天然氨基酸掺入。我们已经使用构建体以用我们的示例性人erf1变体(衍生自nucleicacidsres。2010sep;38(16):5479-92的质粒)代替天然酵母erf1。这表明可以使用不同的erf1蛋白质,仅具有67%的保守性(序列同一性),并且覆盖大量真核生物。当然,erf1(或变体)的核酸序列不是重要的——一个示例性的人erf1变体不仅已被密码子优化以在昆虫细胞中表达良好,而且在人细胞系中也起作用。因此,核酸的密码子优化(如果需要)是本领域技术人员的事情。本发明的优点是琥珀抑制得到增加。适当地,本文所述的erf1突变体用于琥珀抑制。在另一方面,本发明提供了一种将非天然氨基酸掺入到真核细胞如哺乳动物细胞或昆虫细胞的目的蛋白质中的方法,包括以下步骤:i)提供表达trna合成酶和trna对、目的核酸序列和突变体erf1的真核细胞;ii)在存在待掺入由目的核酸序列编码的蛋白质中的非天然氨基酸的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和iii)培养细胞以允许通过正交trna-trna合成酶对将所述非天然氨基酸掺入目的蛋白质。本发明还公开了将突变体erf1用于将非天然氨基酸掺入到真核细胞的目的蛋白质中的用途。本发明还公开了鉴定erf1的突变体的方法,其还提供增加目的蛋白质中非天然氨基酸掺入的突变体。该方法包括以下步骤:(i)提供能够将非天然氨基酸掺入到目的蛋白质中的细胞,合适地其中所述细胞是如本文所述的真核细胞;(ii)在存在待掺入目的蛋白质的非天然氨基酸以及存在和不存在erf1的突变体的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和(iii)测定在存在和不存在erf1突变体的情况下非天然氨基酸掺入目的蛋白质的水平,其中在存在erf1突变体的情况下非天然氨基酸掺入目标蛋白中的水平增加表示,erf1的所述突变体增加了目的蛋白质中非天然氨基酸的掺入。将一个或多个突变掺入erf1中的方法包括本领域众所周知的定点诱变等。适当地,所选择的突变可以基于erf1中对琥珀密码子的终止具有影响的氨基酸的突变,如参考文献25-30中所述。适当地,所选择的突变可以位于与核糖体内的mrna上的终止密码子相互作用的erf1的n末端结构域中(参见图2a)。期望地,erf1突变体响应于所选择的密码子(例如tag)而导致有效的非天然氨基酸掺入,而不增加其他终止密码子的通读。在一个实施方案中,在本公开中不使用释放因子突变体,例如erf1。在一个实施方案中,内源性释放因子(例如erf1)的表达从宿主细胞降低或缺失。这可以通过例如破坏编码erf1的一个或多个基因组基因座,或通过rna介导的erf1基因沉默来实现。制备包含一个或多个非天然氨基酸的蛋白质在本公开内容中可以使用正交或扩展的遗传密码,其中存在于目的核酸序列中的一个或多个特定密码子被分配以编码非天然氨基酸,使得其可以通过使用正交trna合成酶/trna对在遗传上被掺入目的蛋白质中。正交trna合成酶/trna对能够用非天然氨基酸装载trna,并且能够响应于密码子将该非天然氨基酸掺入多肽链中。密码子可能是琥珀密码子、赭石密码子、乳白密码子或四联密码子。密码子需简单地对应于将用于携带非天然氨基酸的正交trna。适当地,密码是琥珀密码子。适当地,密码子是正交密码子。引入非天然氨基酸在很大程度上是在琥珀uag密码子上进行的。适当地,密码子是uag或uga,最合适的是uag(琥珀)。示例性突变最小化琥珀(uag)终止密码子(例如e55d)上的释放因子的活性。所描述的其它突变可能不影响琥珀终止密码子的识别,但是降低uga或uaa终止密码子(乳白/赭石)上的终止活性。这由s70a、g73s举例说明。本领域技术人员使用除uag(琥珀)之外的密码子时,会选择erf1突变体来满足他们的需要。应当注意,本文所示的具体实例已经使用琥珀密码子和相应的trna/trna合成酶。如上所述,这些可以是变化的。或者,为了使用其他密码子而不会出现使用或选择能够与非天然氨基酸一起使用的替代性trna/trna合成酶对的麻烦,trna的反密码子区域可以简单地被交换用于针对密码子选择的所需反密码子区域。反密码子区域不参与trna的装载或掺入功能,也不涉及trna合成酶的识别,所以这种互换完全在本领域技术人员的范围内。因此,如果需要,可以使用替代的正交trna合成酶/trna对。宿主细胞可用于产生(例如,表达)包含一种或多种非天然氨基酸的蛋白质。引入本文公开的构建体或载体的宿主细胞被培养或维持在合适的培养基中,使得产生trna、trna合成酶和目的蛋白质。培养基还包含非天然氨基酸,使得目的蛋白质掺入非天然氨基酸。这样的蛋白质由编码序列中包含本文所述的一个或多个密码子的核酸编码。正交trna合成酶/trna对用非天然氨基酸装载trna,并且响应于密码子将非天然氨基酸掺入多肽链中。在另一方面,本发明提供了一种将非天然氨基酸掺入真核细胞的目的蛋白质中的方法,包括以下步骤:i)提供包含本文所述构建体或载体的真核细胞;ii)在存在一种或多种待掺入目的核酸序列编码的目的蛋白质中的非天然氨基酸的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和iii)培养细胞以允许通过正交trna-trna合成酶对将所述非天然氨基酸掺入目的蛋白质。包含非天然氨基酸的蛋白质通过在宿主细胞中引入包含trna和trna合成酶与包含具有一个或多个框内正交(终止)密码子的目的核酸序列的本文所述的核酸构建体来制备。将宿主细胞暴露于包含非天然氨基酸的生理溶液中,然后将宿主细胞保持在允许目的蛋白质编码序列表达的条件下。响应于密码子,将非天然氨基酸掺入多肽链中。有利的是,将多于一种非天然氨基酸掺入目的蛋白质中。或者,可以在蛋白质的两个或更多个位点将两种或更多种非天然氨基酸掺入目的蛋白质中。适当地,可以在蛋白质的三个或更多个位点将至少三种非天然氨基酸掺入目的蛋白质中。适当地,可以在蛋白质中的四个或更多该位点将至少四种非天然氨基酸掺入目的蛋白质中。适当地,可以在蛋白质的五个或更多个位点将至少五种非天然氨基酸掺入目的蛋白质中。适当地,可以在蛋白质中六个或更多个位点将至少六种非天然氨基酸掺入目的蛋白质中。适当地,可以在蛋白质中的七个或更多个位点将至少七种非天然氨基酸掺入目的蛋白质中。适当地,可以在蛋白质的八个或更多个位点将至少八种非天然氨基酸掺入目的蛋白质中。当将多个非天然氨基酸掺入目的蛋白质中时,应当理解,需要在所需位置将多个密码子掺入编码核酸序列,使得trna合成酶/trna对可以响应于密码子指导非天然氨基酸的掺入。至少1、2、3、4、5、6、7或8个或更多个密码子编码核酸可以掺入目的核酸序列中。当希望将多于一种类型的非天然氨基酸掺入到单个蛋白质的目的蛋白质中时,可以使用第二个或另外的正交trna-trna合成酶对来掺入第二个或另外的非天然氨基酸;适当地,所述第二个或另外的正交trna-trna合成酶对识别编码目的蛋白质的核酸中的不同密码子,使得两种或更多种非天然氨基酸可以在单个制造步骤中特异性地掺入蛋白质的不同限定位点。在某些实施方案中,因此可以使用两个或更多个正交trna-trna合成酶对。一旦在宿主细胞中产生了掺入非天然氨基酸的目的蛋白质,就可以通过本领域已知的各种技术,包括酶促、化学和/或渗透裂解和物理破坏来提取它们。目的蛋白质可以通过本领域已知的标准技术纯化,例如制备色谱法、亲和纯化或任何其它合适的技术。非天然氨基酸如本文所用,术语“非天然氨基酸”是指除蛋白质中天然存在的20个氨基酸之外的氨基酸。非天然氨基酸的非限制性实例包括:对乙酰基-l-苯丙氨酸、对碘-l-苯丙氨酸、o-甲基-l-酪氨酸、对炔丙酰氧基苯丙氨酸、对炔丙基-苯丙氨酸、l-3-(2-萘基)丙氨酸、3-甲基-苯丙氨酸、o-4-烯丙基-l-酪氨酸、4-丙基-l-酪氨酸、三-o-乙酰基-glcnacβ-丝氨酸、l-多巴、氟化苯丙氨酸、异丙基-l-苯丙氨酸、对叠氮基-l-苯丙氨酸、对酰基-l-苯丙氨酸、对苯甲酰基-l-苯丙氨酸、l-磷脂酰丝氨酸、丝氨酸磷酸酯、酪氨酸磷酸酯、对溴苯丙氨酸、对氨基-l-苯丙氨酸、异丙基-l-苯丙氨酸、酪氨酸氨基酸的非天然类似物;谷氨酰胺氨基酸的非天然类似物;苯丙氨酸氨基酸的非天然类似物;丝氨酸氨基酸的非天然类似物;苏氨酸氨基酸的非天然类似物;烷基、芳基、酰基、叠氮基、氰基、卤素、肼、酰肼、羟基、烯基、炔基、醚、硫醇、磺酰基、硒基、酯、硫代酸、硼酸酯(borate)、硼化物(boronate)、二氧磷基(phospho)、磷酰基(phosphono)、膦、杂环、烯酮、亚胺、醛、羟胺、酮或氨基置换的氨基酸,或其组合;具有可光活化交联剂的氨基酸;自旋标记(spin-labeled)的氨基酸;荧光氨基酸;金属结合氨基酸;含金属氨基酸;放射性氨基酸;光笼化和/或可光异构化氨基酸;含有生物素或生物素类似物的氨基酸;含酮的氨基酸;包含聚乙二醇或聚醚的氨基酸;重原子取代的氨基酸;可化学切割或光可切割的氨基酸;具有延长侧链的氨基酸;含有毒性基团的氨基酸;糖取代的氨基酸;碳连接的含糖氨基酸;氧化还原活性氨基酸;含有α-羟基的酸;氨基硫代酸;α,α二取代氨基酸;β-氨基酸;除脯氨酸或组氨酸以外的环状氨基酸,以及除苯丙氨酸、酪氨酸或色氨酸以外的芳香族氨基酸。为了将一种或多种选择的非天然氨基酸掺入本文所述的蛋白质中,本领域技术人员简单地选择能够装载识别密码子的正交trna的正确合成酶。本文提供了引入不同非天然氨基酸的具体实例。在实施例2中,证明了(nε-[(叔丁氧基)羰基]-1-赖氨酸)和(nε-[((2-甲基环丙-2-烯-1-基)甲氧基)羰基]-1-赖氨酸)的掺入。这些化合物的结构如图表1b所示。这两种底物都是pylrs/trnacua对的已知有效底物(natbiotechnol2014,32,465和jamchemsoc2009,131,8720)。在实施例9中,显示了boc-k(nε-[(叔丁氧基)羰基]-l-赖氨酸、降冰片烯-k(nε-降冰片烯-2-基氧基羰基-l-赖氨酸)、环丙烯-k(nε-[((2-甲基环丙-2-烯-1-基)甲氧基)羰基]-l-赖氨酸)和双环炔-n(nε-([(1r,8s)-双环[6.1.0]壬-4-基-9-基甲氧基]羰基)-赖氨酸)的掺入。wo2010/139948描述了使用正交trna-trna合成酶对将能够支持炔基-叠氮基键合的脂肪族或直链碳主链氨基酸掺入目的蛋白质中。wo2013/10844描述了使用正交trna-trna合成酶对将降冰片烯氨基酸掺入目的蛋白质。抗体适当地,目的核酸序列编码抗体或抗体片段。可以将一种或多种非天然氨基酸——适当地,2、3、4、5、6、7或8种或更多种非天然氨基酸引入抗体或抗体片段中。如本文所用,术语“抗体”是指能够非共价、可逆地和以特异性的方式结合相应抗原的免疫球蛋白家族的蛋白质。该术语包括但不限于单克隆抗体、人抗体、人源化抗体、骆驼化抗体、嵌合抗体和抗独特型(抗-id)抗体。抗体可以是任何同种型/类,因此可以包括igg、ige、igm、igd、iga和igy,或抗体亚类如igg1、igg2、igg3、igg4、iga1和iga2。如本文所用,术语“抗体片段”是指抗体的一个或多个部分,其保留与抗原的表位(例如通过结合、空间位阻、稳定化/去稳定化、空间分布)特异性相互作用的能力。结合片段的实例包括但不限于单链fv(scfv)、二硫键连接的fv(sdfv)、fab片段、f(ab')片段、由vl、vh、cl和ch1结构域组成的单价片段;f(ab)2片段,包含在铰链区通过二硫桥连接的两个fab片段的二价片段;由vh和ch1结构域组成的fd片段;由抗体单臂的vl和vh结构域组成的fv片段;dab片段,其由vh结构域组成;和分离的互补决定区(cdr)或抗体的其他表位结合片段。此外,尽管fv片段的两个结构域vl和vh被独立的基因编码,但是它们可以通过合成接头使用重组方法连接,使得它们能够被制成单个的蛋白质链,其中vl和vh区对形成单价分子(称为单链fv(“scfv”)。此类单链抗体包括在术语“抗原结合片段”内。这些抗原结合片段使用本领域技术人员已知的常规技术获得,可以以与完整抗体相同的方式筛选活性。抗体可以是单特异性的、双特异性的或多特异性的。多特异性抗体可以对一种靶蛋白的不同表位是特异性的,或者可以含有对多于一种靶蛋白特异的抗原结合结构域。抗体可以与另一种功能性分子如另一种肽或蛋白质连接或共表达。例如,抗体或其片段可以与一个或多个其它分子实体(例如另一抗体或抗体片段)功能性连接以产生具有第二结合特异性的双特异性或多特异性抗体。功能性连接可以使用例如化学偶联、遗传融合或非共价缔合来实现。其他示例性双特异性形式包括基于scfv或双抗体的双特异性形式、igg-scfv融合体、双重可变结构域(dvd)-ig、杂交瘤(quadroma)、旋钮入孔(knobs-into-holes)、普通轻链(例如,具有旋钮入孔的普通轻链等)、crossmab、crossfab、(seed)体、亮氨酸拉链、duobody、igg1/igg2和双作用fab(daf)-igg双特异性形式(参见例如mabs(2012)4:6,1-11)。适当地,使用的抗体是人抗体。术语“人抗体”包括具有可变区的抗体,其中框架区和cdr区二者均衍生自人类起源的序列。此外,如果抗体含有恒定区,恒定区也衍生自此类人序列,例如人种系序列,或人种系序列的突变形式或衍生自人框架序列分析的含有共有框架序列的抗体。人抗体可以包括不由人序列编码的氨基酸残基(例如,通过体外随机或位点特异性诱变或通过体内体细胞突变引入的突变,或保守置换,以促进稳定性或制造)。抗体药物缀合物可以使用其中掺入一种或多种非天然氨基酸的抗体或抗体片段来制备抗体药物偶联物(adc)。adc包含与药物部分缀合的抗体或抗体片段。药物部分可以是对adc存在于其中的细胞具有期望的影响的任何药物部分。作为例子,药物部分可以是抗癌剂、抗血液学障碍的药剂、自身免疫治疗剂、抗炎剂、抗真菌剂、抗细菌剂、抗寄生虫剂、抗病毒剂、或麻醉剂、或放射性同位素等。抗体或抗体片段可以根据需要缀合至一个或多个相同或不同的药物部分。抗体或抗体片段可以与修饰给定生物学反应的药物部分缀合。因此,例如,药物部分可以是具有所需生物学活性的蛋白质、肽或多肽。这样的蛋白质可以包括例如毒素、细胞毒素、蛋白质——例如肿瘤坏死因子、细胞因子、凋亡剂、抗血管生成剂或生物学反应调节剂——例如淋巴因子。各种使药物部分与抗体或抗体片段缀合的方法是本领域众所周知的。例如,可以参考mabs(2014)6(1):46–53,其综述了当前使位点特异性药物与抗体缀合的方法。各种蛋白质化学修饰技术在本领域中也是已知的(参见例如natchembiol.(2011)7,876–84;bioconjugatetechniques,elsevier(2008)和chembiol.(2010)17,213–27)。药物部分可以通过接头与抗体或抗体片段连接。如本文所用,“接头”是指能够将抗体或抗体片段与药物抗性连接的任何化学部分。接头可以是易于切割的(可切割接头),如酸诱导的切割、光诱导的切割、肽酶诱导的切割、酯酶诱导的切割和二硫键切割。或者,接头可以基本上是耐受切割的(例如,稳定的接头或不可切除的接头)。可以通过本领域已知的各种测定法针对adc的物理/化学性质和/或生物学活性对adc进行表征和选择。例如,可以通过已知方法(例如elisa、facs、biacore或蛋白质印迹)测试抗体的抗原结合活性。转基因动物和细胞系特别可用于筛选具有作为预防性治疗或治疗性治疗潜力的adc。筛选有用的adc可以包括向转基因动物施用剂量范围上的候选adc,以及在不同时间点测定adc对所评估的疾病或病症的影响。或者或另外,如果适用的话,药物可以在暴露于疾病的诱导剂之前施用或同时施用。候选adc可以以中等或高通量筛选格式串行、单独或并行进行筛选。因此,在另一方面,本发明提供了制备抗体-药物缀合物的方法,其包括以下步骤:i)提供本文所述的真核细胞,其中目的核酸序列编码抗体或抗体片段;ii)在待掺入入抗体或抗体片段的非天然氨基酸存在下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;iii)获得其中已经掺入非天然氨基酸的抗体或抗体片段;和vi)经由非天然氨基酸将抗体或抗体片段与药物部分缀合。可以使用非天然氨基酸和药物部分之间的接头。在一个实施方案中,包含一个或多个(例如2、3、4、5、6、7或8个或更多个)非天然氨基酸的抗体或抗体片段通过非天然氨基与药物部分之间的连接缀合至药物部分。传统上,药物部分与抗体或抗体片段中的半胱氨酸或赖氨酸残基非选择性缀合。然而,这种策略通常会导致异质产物,从而使得优化adc的生物学、物理学和药理学性质面临挑战。使用非天然氨基酸作为缀合点来合成精确控制缀合位点和化学计量的均相adc具有许多优点,其可以包括改善的药代动力学和改善的效力。因此,位点特异性缀合方法是非常需要的。迄今为止,许多adc已经平均靶向4种药物/抗体。已经选择了该比例作为细胞毒性和药代动力学稳定性的最佳组合(参见acc.chem.res(2008)41(1)98-107和clin.cancer.res(2004)10(20):7063-7070)。因此,具体实施方案涉及包含约4种非天然氨基酸的抗体或抗体片段,所述非天然氨基酸可以是或者通过非天然氨基酸和药物部分之间的连接与药物部分缀合。在一个示例性实施方案中,抗体是抗her2/neuigg1人源化抗体或其变体或衍生物——例如曲妥珠单抗。将掺入不同的非天然存在的氨基酸(例如nε-[((2-甲基环丙-2-烯-1-基)甲氧基)羰基]-l-赖氨酸),用于缀合一种或多种小分子。使用四嗪化学,所述分子可以与含有一个或多个非天然氨基酸的mab缀合。该分子可以包括诱导免疫应答的细胞毒性分子或化学部分。有利地,nε-[((2-甲基环丙-2-烯-1-基)甲氧基)羰基]-l-赖氨酸)表现出在琥珀密码子非常高的掺入率(以及随后更高的蛋白质表达)。与在公共领域中用于蛋白质缀合的许多uaa相比,环丙烯具有更长的侧链和更多的溶剂暴露的缀合手柄。这种与高比例的琥珀密码子掺入组合的长侧链,应该允许将uaa掺入mab支架内的更多位点。这种灵活性对于定制不同的抗体-药物缀合物是重要的。试剂盒本发明还提供了用于产生包含一种或多种非天然氨基酸的目的蛋白质的试剂盒。一方面,本发明提供了用于将非天然氨基酸掺入真核细胞的蛋白质中的试剂盒,其包含:(i)本文所述的核酸构建体;或(ii)本文所述的核酸构建体的组合;或(iii)本文描述的载体;或(iv)本文所述载体的组合;或(v)本文所述的细胞;和(vi)任选的非天然氨基酸。适当地,试剂盒还包含编码突变体erf1的核酸构建体或载体或包含其的细胞。试剂盒还可以包括描述使用试剂生产此类蛋白质的方法的印刷说明材料。一般重组dna技术除非另有说明,否则本发明使用在本领域普通技术人员的能力范围内的化学、分子生物学、微生物学和重组dna技术的常规技术。此类技术在文献中有解释。参见例如m.green&j.sambrook,2012,molecularcloning:alaboratorymanual,coldspringharborlaboratorypress。该文通过引用并入本文。现在通过编号段落描述本发明:段落1.在真核细胞、适当地哺乳动物或昆虫细胞中表达trna合成酶和trna对的核酸构建体,其包含:(i)编码trna合成酶的核酸序列,其可操作地连接到能够表达trna合成酶的第一启动子;和(ii)编码trna的核酸序列,其可操作地连接到能够表达trna的第二启动子,其中第一和第二启动子彼此方向相反,或其中trna以核酸构建体上多个拷贝的形式存在。段落2.根据段落1的核酸构建体,其中所述核酸构建体还包含编码目的核酸序列的核酸序列,其可操作地连接到能够在真核细胞中表达目的核酸序列的另外的启动子。段落3.根据段落2的核酸构建体,其中所述启动子在与第一启动子相同的方向上取向,任选地,其中能够在真核细胞中表达目的核酸序列的启动子与第一启动子相同或不同于第一启动子。段落4.根据段落1至3中任一段落的核酸构建体,还包含编码突变体erf1的核酸序列,适当地,突变体智人erf1,适当地,其中突变体erf1选自e55d、e55a、n129p/k130q和y125f,或其两个或更多个的组合。段落5.根据段落4的核酸构建体,其中编码突变体erf1的核酸序列和编码trna合成酶的核酸序列通过自切割肽在相同开放阅读框架中连接。段落6.用于在真核细胞中表达trna和目的核酸序列的核酸构建体,所述目的核酸序列在用于掺入非天然氨基酸的位置上包含由trna识别的密码子,所述核酸构建体包含:(i)包含目的核酸序列的核酸序列,其可操作地连接到能够在真核细胞中表达目的核酸序列的第一启动子;和(ii)编码trna的核酸序列,其可操作地连接到能够表达trna的第二启动子,其中第一和第二启动子彼此方向相反,或其中trna以核酸构建体上多个拷贝的形式存在。段落7.根据段落6的核酸构建体,还包含编码突变体erf1、适当地是突变体智人erf1的核酸序列,其中突变体erf1选自e55d、e55a、n129p/k130q和y125f,或其两个或更多个的组合。段落8.根据前述段落中任一段落的核酸构建体,其中所述第一和第二启动子彼此方向相反,并且其中trna以核酸构建体上多个拷贝的形式存在。段落9.根据前述段落中任一段落的核酸构建体,其中trna直接连接至启动子或间接连接至启动子,适当地,其中核酸构建体包含与trna用接头连接的终止子序列。段落10.根据前述段落中任一段落的核酸构建体,其中编码trna的核酸序列的每个拷贝在独立的启动子的控制下。段落11.根据前述段落中任一段落的核酸构建体,其中启动子排列包括在第一方向上取向的延伸因子启动子和在第二方向上取向的poliii启动子。段落12.根据前述段落中任一段落的核酸构建体,其中第一启动子是或者衍生自ef-1启动子。段落13.根据前述段落中任一段落的核酸构建体,其中第二启动子是或者衍生自u6启动子。段落14.根据前述段落中任一段落的核酸构建体,其中trna在核酸构建体上存在4、5、6、7或8个或更多个拷贝。段落15.根据前述段落中任一段落的核酸构建体,其中trna是野生型或变体trna,合适地是pylt的u25c变体。段落16.根据前述段落中任一段落的核酸构建体,其中目的核酸序列包含至少1、2、3或4个终止密码子。段落17.根据段落2-16中任一段落的核酸构建体,其中目的核酸序列编码抗体或抗体片段。段落18.根据前述段落中任一段落的核酸构建体,其中所述trna合成酶与真核细胞中的内源性trna正交,和/或所述trna与真核细胞中的内源性trna合成酶正交,和/或所述trna合成酶与真核细胞中的内源性trna正交并且所述trna与内源性trna合成酶正交。段落19.包含根据段落1至5和8至18中任一段落的核酸构建体和根据段落6至18中任一项的核酸构建体的核酸构建体的组合。段落20.根据段落19的核酸构建体的组合,其中编码突变体erf1的核酸序列在独立的构建体上。段落21.包含根据段落1至18中任一段落的核酸构建体的载体。段落22.载体的组合,其包含含有段落1至5和8至18中任一段落的核酸构建体的载体和包含根据段落6至18中任一段落的核酸构建体的载体。段落23.根据段落22的载体的组合,其中编码突变体erf1的核酸序列在单独的载体上。段落24.细胞,其包含根据段落1至18中任一段落的核酸构建体,根据段落19或段落20的核酸构建体的组合,根据段落21的载体或根据段落22或段落23的载体的组合。段落25.根据段落24的细胞,还包含编码突变体erf1、合适地突变型智人erf1的核酸构建体。段落26.根据段落25的细胞,其中突变体erf1选自e55d、e55a、n129p/k130q和y125f,或其两种或更多种的组合,其中在如genbank登录号af095901.1中所述的智人erf1基因序列中进行突变。段落27.根据段落24-26中任一段落的细胞,其中细胞是昆虫细胞或哺乳动物细胞。段落28.根据段落24-27中任一段落的细胞,其中用核酸瞬时或稳定转染细胞。段落29.将非天然氨基酸掺入真核细胞(合适地哺乳动物或昆虫细胞)的蛋白质中的试剂盒,其包含:(i)根据段落1至5和8至18中任一段落的核酸构建体和根据段落6至18中任一段落的核酸构建体;或(ii)根据段落19或段落20的核酸构建体的组合;或(iii)根据段落21的载体;或(iv)根据段落22或段落23的载体的组合;或(v)根据段落27或段落28的昆虫或哺乳动物细胞;和(vi)任选的非天然氨基酸。段落30.根据段落29的试剂盒,还包含编码突变体erf1的核酸构建体或载体或包含其的细胞。段落31.将非天然氨基酸掺入真核细胞(合适地哺乳动物或昆虫细胞)中的目的蛋白质的方法,包括以下步骤:i)提供段落27或段落28的细胞,其中所述细胞包含根据段落19或段落20的核酸构建体的组合或根据段落22或段落23的载体的组合;和ii)在存在待被掺入由目的核酸序列编码的目的蛋白质中的非天然氨基酸的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和iii)培养细胞以允许通过正交trna-trna合成酶对将所述非天然氨基酸掺入目的蛋白质中。段落32.根据段落31的方法,其中将至少3、4或5个非天然氨基酸掺入目的蛋白质中。段落33.制备抗体-药物缀合物的方法,包括以下步骤:i)根据段落27或段落28提供细胞,其中所述细胞包含根据段落19或段落20的核酸构建体的组合或根据段落22或段落23的载体的组合,并且其中所述目的核酸序列编码抗体或抗体片段;ii)在存在待掺入抗体或抗体片段中的非天然氨基酸的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;iii)获得其中已经掺入了非天然氨基酸的抗体或抗体片段;和iv)通过非天然氨基酸将抗体或抗体片段与药物部分缀合。段落34.(i)根据段落1至5和8至18中任一段落的核酸构建体和根据段落6至18中任一段落的核酸构建体;或(ii)根据段落19或段落20的核酸构建体的组合;或(iii)根据段落21的载体;或(iv)根据段落22或段落23的载体组合;或(v)根据段落27或段落28的昆虫或哺乳动物细胞,用于在真核细胞、适当地哺乳动物或昆虫细胞中将非天然氨基酸掺入目的蛋白质中的用途。段落35.将非天然氨基酸掺入真核细胞(合适地哺乳动物或昆虫细胞)的目的蛋白质中的方法,包括以下步骤:i)提供表达正交trna合成酶和trna对、目的核酸序列和突变体erf1的真核细胞,所述目的核酸序列在用于掺入非天然氨基酸的位置上包含由trna识别的密码子;ii)在存在待被掺入由目的核酸序列编码的目的蛋白质中的非天然氨基酸的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和iii)培养真核细胞以允许通过正交trna-trna合成酶对将所述非天然氨基酸掺入目的蛋白质。段落36.突变体erf1用于将非天然氨基酸掺入真核细胞如哺乳动物细胞或昆虫细胞的目的蛋白质中的用途。段落37.鉴定增加了目的蛋白中非天然氨基酸的掺入的erf1的突变体的方法,所述方法包括以下步骤:(i)提供能够在其中将非天然氨基酸掺入目的蛋白质中的细胞,适当地,其中所述细胞表达正交trna合成酶和trna对、目的核酸序列和任选的突变体erf1,所述目的核酸序列在用于掺入非天然氨基酸的位置上包含由trna识别的密码子;在存在待被掺入目的蛋白质中的非天然氨基酸以及在存在和不存在erf1的突变体的情况下培养细胞,其中所述非天然氨基酸是trna合成酶的底物;和测定在存在和不存在erf1的突变体的情况下非天然氨基酸掺入目的蛋白质中的水平,其中在存在erf1的突变体的情况下掺入目的蛋白质中非天然氨基酸的水平增加表示,erf1的所述突变体增加在目的蛋白质中非天然氨基酸的掺入。段落38.基本上如本文参照所附描述和附图所述的构建体、载体、细胞、试剂盒、方法或用途。实施方案的描述虽然已经参考附图在此详细公开本发明的说明性实施方案,但是应当理解,本发明不限于精确的实施方案,并且本领域技术人员可以在其中实现各种改变和修改,而不脱离由所附权利要求及其等同物限定的本发明的范围。另外特别和优选的方面在附带的独立和从属权利要求中阐述。从属权利要求的特征可以适当地与独立权利要求的特征组合,并且除了权利要求中明确规定的那些之外的特征组合。在将设备特征描述为可操作以提供功能的情况下,应当理解,这包括提供该功能或适配或配置为提供该功能的装置特征。实施例实施例1——材料与方法dna构建体报告构建体衍生自先前描述的质粒pmbpylrs-mcherry-tag-egfp-ha1,其限制性位点用sfgfp150tag替代并进行密码子优化以用于在人细胞系(lifetechnologies)中表达(补充信息表s1),以产生质粒cmv-pylrs/cmv-sfgfp(tag)。用renilla-tag-萤火虫萤光素酶盒替换相同的区域以产生质粒cmv-pylrs/cmv-dlr(tag)。使用kod热启动聚合酶(novagen),将终止或有义密码子通过定点诱变引入pjet1.2(thermoscientific)中的sfgfp150tag或renilla-tag-萤火虫萤光素酶盒中。将得到的突变体亚克隆到pmbpylrs-mcherry-tag-egfp-ha中,得到sfgfp150leu(cmv-pylrs/cmv-sfgfp)和sfgfp101、133、150tag(cmv-pylrs/cmv–sfgfp(tag)3)以及含有taa(cmv-pylrs/cmv-dlr(taa))、tga(cmv-pylrs/cmv-dlr(tga))和ser(cmv-pylrs/cmv-dlr)的双重萤光素酶报道质粒。对于4xu6pylt质粒系列,使用pb220pa-1骨架(systembiosciences)。用来自cd532a-2(systembiosciences)的ef-1α盒替换cmv盒,将spei/sali片段亚克隆到pb220pa-1的spei/sali中。合成了具有spei/avrii侧翼位点的优化的u6启动子/马氏甲烷八叠球菌吡咯赖氨酸trna插入物(补充信息表s1)(lifetechnologies)。通过将spei/avrii片段重复插入到ef1启动子的独特spei位点5'中构建4xu6pylt串联盒。(u6-pylt*)4/ef-1α-pylrs、(u6-pylt*)4/ef-1α-sfgfp(tag)和(u6-pylt*)4/ef-1α-sfgfp(tag)3质粒通过亚克隆来自ag28、cmv-pylrs/cmv-sfgfp和cmv-pylrs/cmv-s2的相关基因构建。mcherry-tag-egfp盒在mfei和nheisfgfp(tag)3之间。将智人erf1基因(genbank:af095901.1)针对黑腹果蝇进行密码子优化(补充信息表s1),并通过n末端his6标签、c末端三个终止密码子(uaaugauag)扩增。密码子优化使用helixweb工具包2进行。通过定点诱变引入额外的突变(补充信息表2),并使用限制性位点hindiii/noti将所得构建体克隆到哺乳动物表达载体pcdnatm5/frt/to中,以产生perf1(x)载体,其中x表示突变,erf1由cmv启动子表达。细胞培养、抗体和测定将贴壁hek293t细胞在37℃在5%co2气氛中保持在补充有10%fbs的dulbecco改良的eagle's培养基(dmem)-glutamax(gibco)上。使用(mirus)转染试剂或以聚乙烯亚胺(maxpei,polysciences)按照制造商的操作方案按3:1的pei:dna比例进行瞬时转染。通过用针对erf1(ab30928,abcam)、肌动蛋白(#4967,cellsignalingtechnology)、flag(a8592,sigma-aldrich)、his(27e8,cellsignalingtechnology)、ha(c29f4,cellsignalingtechnology)和相应的hrp连接的二抗(#7074、#7076、#7077,cellsignalingtechnology)确认蛋白质的表达和erf1的耗尽。通过与其它转染的质粒等量的市售erf1shrna(sc-37871-sh,santacruzbiotechnology)转染来实现erf1的耗尽。氨基酸1可商购(e1610.0025,bachem),氨基酸2如前所述合成。对于蛋白质表达,将氨基酸溶液在培养基中制备为中和的储备溶液,并将其加入到培养的细胞中,或者与预温育的转染混合物(transit-293,96孔板)一起或在转染后四小时更换培养基时(pei,24孔板,10cm培养皿,t75瓶)。northern印迹使用qiazol裂解试剂(qiagen)从hek293t细胞中分离总rna,并用异丙醇沉淀。northern印迹用northernmax-gly试剂盒(ambion)进行;在乙二醛上样染料中将rna变性,在2%琼脂糖凝胶上分离,转移到brightstar-plus带正电的尼龙膜(ambion)上,并通过stratalinker2400uv交联剂(strategene)通过uv交联。膜在37℃下与5'-生物素化的dna探针(5’-ggaaaccccgggaatctaacccggctgaacggatttagag-3’)杂交过夜。使用化学发光核酸检测模块(thermoscientific)检测杂交探针。双重萤光素酶测定在96孔板(costar#3595,corning)中转染hek293t细胞,每孔用两倍建议量的dna(总计0.2μg)和试剂(0.6μl)。根据简化的制造商操作方案(promega)进行双重萤光素酶测定。培养16小时后,除去培养基,并将细胞在室温下在20μl被动裂解缓冲液中裂解15分钟。然后将10μl裂解液加入到在白色96孔板(nunc236105,thermoscientific)中的50μl萤光素酶测定缓冲液ii中。随后,进行萤火虫萤光素酶发光测量(pherastarfs,bmglabtech),淬灭反应,加入50μlstop&glo缓冲液后进行renilla发光测量。实验一式四份进行,三个重复用于双重萤光素酶测定,剩余的重复用于在50μl具有完全蛋白酶抑制剂(roche)的ripa缓冲液(sigma)中裂解后的免疫印迹。sfgfp变体的瞬时表达用于荧光测定的sfgfp(tag)和sfgfp(tag)3的表达在24孔板中进行。常规地,每孔接种100000个细胞,生长过夜,并使用160ngdna/质粒、1.5μl的pei(1mg/ml)和50μl还原血清培养基(opti-mem,gibco)瞬时转染。转染后4小时,交换培养基,将氨基酸1或2加入到新鲜生长培养基中。48小时生长后,小心取出上清液,摇动的同时在加入100μl具有完全蛋白酶抑制剂(roche)的ripa缓冲液(sigma)中裂解细胞。将50μl裂解物转移到96孔板(costar#3595,corning)中,并在485/520nm测定荧光强度(pherastarfs,bmglabtech)。使用校准曲线在裂解物中定量sfgfp(补充图s1)。为了校准目的,在pbad启动子控制下,在来自基于p15a的质粒的大肠杆菌dh10b细胞中表达sfgfp,如前所述通过ni亲和层析纯化4。通过sds-page后的考马斯染色证实蛋白质纯度。通过测量补充有完全蛋白酶抑制剂(roche)的ripa缓冲液(sigma)中连续稀释(每步1:10)的280nm处的吸光度(消光系数0.6845),来测定参考样品中的绝对蛋白质浓度。在96孔板(costar,corning)中一式三份测量给定浓度的sfgfp的荧光强度(激发485nm,发射520nm)。对于给定的得数设置,酶标仪(pherastarfs,bmglabtech)的线性范围之外的测量被丢弃,剩余的数据点拟合到线性曲线。在相同条件下进行细胞裂解物中sfgfp荧光的随后测量,并参照标准曲线测定sfgfp浓度(prism6,graphpad)。在10cm培养皿中进行用于质谱法的蛋白质表达。用pei将15μg的dna在10cm组织培养皿中转染hek293t。细胞培养基在转染后4小时进行交换,并用氨基酸培养72小时。细胞用pbs洗涤两次,并在1mlripa缓冲液中裂解。将澄清的裂解物加入到50μlm(chromotek)中,并在4℃下孵育4小时。将珠子磁分离并用1mlripa、1mlpbs、1mlpbs+500mmnacl、1mlddh2o洗涤并在1%乙酸/ddh2o中洗脱。质谱使用配有6130四极杆光谱仪的agilent1200lc-ms系统进行电喷雾质谱。溶剂系统由作为缓冲液a的含0.1%甲酸的h2o和作为缓冲液b的含0.1%甲酸的乙腈(mecn)组成。在214和280nm监测蛋白质的uv吸光度。以正离子模式进行蛋白质ms采集,并通过mschemstation软件(agilenttechnologies)中的反卷积计算总蛋白质量6。实施例2——增加的trna水平提高了非天然氨基酸的掺入效率我们优化了trnacua的表达水平,以提高哺乳动物细胞中非天然氨基酸掺入的效率。研究人员使用不同的pylrs和trna质粒,其改变了pylrs的拷贝数、trnacua和启动子17-20的选择。然而,没有报道可以量化哺乳动物细胞中带有用pylrs/trnacua配对掺入的非天然氨基酸的蛋白质的产量,也没有报道相对于不含琥珀终止密码子的基因表达的对照蛋白质来量化非天然氨基酸的掺入效率。这些实验对于了解哺乳动物细胞中非天然氨基酸掺入与天然蛋白质合成相比是至关重要的。我们首先使用质粒b和c检测非天然氨基酸的效率,所述质粒携带cmv启动子上的单拷贝pylrs和每个均由具有cmv增强子的u6启动子驱动的四拷贝的trnacua17,19(构建体示意图示于图表1a中)。该系统指导1(nε-[(叔丁氧基)羰基]-l-赖氨酸或2(nε-[((2-甲基环丙-2-烯-1-基)甲氧基)羰基]-l-赖氨酸(图表1b)的掺入,两者是pylrs/trnacua对的已知和有效底物21,22),其响应于sfgfp23(cmv-sfgfp(tag))中位置150处的琥珀密码子,效率为5%和7%(图1a、b);所有的掺入效率都报告为以携带代替150位琥珀密码子的亮氨酸密码子的相同对照构建体(质粒a,图表1)产生的sfgfp水平的百分比。接下来我们用在优化的u6启动子上用单拷贝的trnacua替换四拷贝的trnacua,导致非天然氨基酸掺入效率的小幅度降低(质粒b和d)。与原始的四拷贝盒(c)不同,新的u6trnacua盒(d)不含有cmv增强子,并且对于trna产生精确的5'末端,其不需要核酸酶处理。northern印迹(图1c)表明从d产生的pyltrnacua的水平与c产生的水平相当。这表明改变的trna表达构建体提供了更多的trna拷贝/trna基因拷贝。用u25c变体代替trnacua24将掺入效率从2.7%-3.5%略微提高到4.7-5.1%(质粒b和e,图表1),对trnacua水平具有很小的影响。创建串联阵列,每个包含u6pyltrnacua(带有u25c)的四个拷贝,并将来自cmv的蛋白质编码基因的启动子切换到ef-1α(质粒g和h,图表1),这导致该系统中携带1或2的sfgfp的显著增加。在该系统中,响应于sfgfp中的琥珀密码子而以62%的效率掺入氨基酸1,其中2的掺入效率为约129%。蛋白质印迹表明,将蛋白质编码基因的启动子改变为ef-1α不会改变pylrs(抗flagb+e对g+h,图1b)或wtsfgfp(avsf,图1b)的水平,证明通过改变为ef1α启动子,pylrs水平和sfgfp表达的最大水平都不会被显著改变。然而,northern印迹显示,该系统中的trna水平比所有其他所测试的系统都高得多,这表明我们观察到的非天然氨基酸掺入效率的大幅度增加是由trna水平的增加引起的。实施例3——选择的erf1变体的异位表达不会增加终止密码子的通读。接下来,我们询问是否可以通过工程改造erf1进一步增强非天然氨基酸的掺入效率,而不增加其他终止密码子的通读。虽然使用优化的合成酶和trna系统,非天然氨基酸的掺入效率已经很好,但我们设想,erf1工程改造可能进一步提高这种效率,并使我们有效地在蛋白质的多个位点掺入非天然氨基酸。我们首先鉴定erf1中在遗传或生物化学研究中报道对琥珀密码子终止有影响25-30的氨基酸位置。这些突变位于erf1的n末端结构域(图2a),与核糖体内的mrna的终止密码子相互作用。为了评估erf1突变体对哺乳动物细胞中的翻译终止的影响,我们定量了hek293t细胞中以及携带添加的过表达人erf1和erf1突变体的hek293t细胞中,琥珀、乳白和赭石终止密码子处的抑制trna非依赖性通读(图2b)。erf1与erf3主要通过erf137上的c末端结构域介导形成复合物,介导翻译终止。erf3以与内源性erf1相当的水平存在于细胞中,因此erf3限制了可能形成的终止复合物的数目35。erf1的n端结构域突变体的过表达可能会使这些复合物(通过质量作用)偏向含有erf1突变体,从而揭示erf1突变的表型。我们将每个erf1变体引入细胞(图2b),并使用三个双重萤光素酶报告基因(图2c)测量了终止密码子的基础通读。每个报告子都包含一个n-末端海肾萤光素酶,随后是一个终止密码子(琥珀、乳白或赭石)和一个c末端萤火虫萤光素酶31-34。终止密码子的通读在0.08和0.12%之间(tag0.09%、tga0.12%、taa0.08%),为进一步的实验提供了基准。erf1的异位过表达导致所有三个终止密码子的通读降低(tag0.03%、tga0.07%、taa0.04%),与细胞中erf1水平的增加一致35。通读的这种减少很小,和与内源erf1水平相当的erf3的水平一致,erf3水平限制了可以形成的功能终止复合物的数量35。引入erf1变体增加了对野生型erf1引入的终止密码子的通读。然而,对于所有测试的erf1突变体,除了两个外,所有三个终止密码子的阅读都没有增加到高于不存在异位表达的erf1时的水平。我们得出结论,测试的大多数erf1变体的异位表达不会增加终止密码子的通读高于基础水平。将终止密码子的通读增加高于通常在细胞中发现的水平的两个erf1突变体是erf1δ100(将通读增加到1.6%(taa)、2%(tag)和15%(tga)的突变体)和t122q、s123f突变体29,其选择性地增加tga密码子的通读2倍)。通过shrna降低内源性erf1水平增加了所有三个终止密码子的基础通读2-3倍。预期erf1δ100突变体对所有终止密码子通读的影响,原因是缺失残基的n末端结构域介导了mrna中所有三个终止密码子的识别,但不介导与erf3相互作用36-39。因此,预期该突变体与erf3形成无活性复合物,减少了可以介导终止的功能性erf1/erf3复合物的数量。同样地,预期针对erf1的shrna对所有终止密码子的作用40,因为erf1的减少应导致终止密码子上的终止减少。实施例4——erf1突变体增加非天然氨基酸掺入效率。为了研究erf1、erf1突变体和shrna对非天然氨基酸掺入的影响,我们用相关erf1突变体转染细胞(图2d)。每个样品还提供了琥珀抑制的双重萤光素酶报告基因、单拷贝的正交吡咯赖氨酰trna合成酶(pylrs)/trnacua对(图表1中显示为b+d的排列,但用双重萤光素酶报告基因替代sfgfp)。我们使用这个系统来最大化动态范围,我们可以用它来测量erf1变体提供的增强。将氨基酸1加入到所有细胞中。在一种情况下,添加shrna构建体到内源性erf140,使得我们可以比较异位表达的erf1突变体对敲低内源性erf1的影响。双重萤光素酶测定用于确定erf1对非天然氨基酸掺入效率的影响(图2e)。在没有异位表达释放因子的情况下,在该测定中非天然氨基酸掺入的效率为约5.3%。野生型释放因子异位表达后掺入效率略有下降,而内源性erf1的shrna敲低后则上调至13%。除了s70a、g73s突变体外,在所有突变体释放因子存在下,1的掺入效率增加。此突变体以前描述为双功能uar特异性erf1。28,41两个erf1突变体导致最有效的非天然氨基酸掺入:erf1(e55d),27%;和erf1(δ100),36%。δ100突变体和e55d突变体的掺入效率比不含异位表达释放因子的细胞中的掺入效率高5至7倍。有趣的是,虽然在存在和不存在pylrs/trnacua对时强烈增强琥珀通读,erf1δ100突变体显著降低了在两种情况下产生的萤光素酶的总量,这与对翻译效率具有全局影响的所有三个终止密码子处终止的剧烈破坏一致(支持性信息图2)。此外,δ100突变体导致在不存在抑制性trna的情况下对所有三个终止密码子的通读;因此,我们没有进一步调查这个释放因子突变体。我们将重点放在erf1(e55d)上。鉴于使用兔网织红细胞核糖体的体外测定,经鉴定这种释放因子突变体比响应于琥珀密码子更有效地响应于赭石和乳白密码子而从起始trna中除去甲酰-甲硫氨酸。25实施例5——用于掺入多个非天然氨基酸的优化系统。接下来,我们结合优化的合成酶和trna系统与erf1的e55d突变体(图3)。我们发现,在1存在下生长的含有pylrs/trnacua对的细胞中添加erf1(e55d)将1向sfgfp(tag)的掺入从62%增加到85%(图3a)。类似地,erf1(e55d)的添加将sfgfp(tag)3(其在gfp的k101、d133和v15014的位置含有琥珀终止密码子)中掺入1的效率提高了从5%到12%的(图3a、b)。来自sfgfp(tag)的sfgfp-1的产量在105个细胞中为0.65μg,而来自sfgfp-(tag)3的sfgfp-(1)3的产量为每105个细胞0.1μg(支持性信息图3;所有引用的产量为每接种细胞数,并在转染后48小时测量)。我们发现,在存在2的情况下生长的含有pylrs/trnacua对的细胞中添加erf1(e55d)使sfgfp(tag)中2的掺入从129%增加到157%,并且erf1(e55d)的添加将自sfgfp(tag)3产生sfgfp-(2)3的生产效率自11%提高至43%,提高了四倍(图3c、d)。来自sfgfp(tag)的sfgfp-2的产量为1.76μg/105个细胞,而来自sfgfp(tag)3的sfgfp-(2)3的产量为0.49μg/105个细胞(支持性信息图3)。从含有优化系统的细胞裂解物中纯化全长sfgfp(图4a)。电喷雾离子化质谱法证明了1和2的1个分子和3个分子分别从sfgfp(tag)和sfgfp(tag)3掺入到sfgfp中的位点特异性掺入(图4b,支持性信息图4)。该数据与图4a中的无氨基酸对照组合显示出在erf1(e55d)存在下非天然氨基酸的高保真掺入。实施例6——诱导型erf1突变体的基因组整合增强了琥珀抑制迄今为止描述的erf1增强表达系统依赖于三个质粒的平行转染,以便引入用于将非天然氨基酸掺入细胞中的所有必需组分。通过erf1e55d突变体的基因组整合,只需要转染两种质粒(图1,与h或i组合的构建体g),这增加了递送到细胞中的这些质粒的量。通过使用四环素诱导型启动子用于erf1,产生细胞系,其通过在生长培养基中加入四环素,可以在需要时从标准细胞培养维持模式切换到高效琥珀抑制模式。将erf1变体导入flp-intmt-rextm293细胞系(lifetechnologies)。该细胞系含有单个转录活性基因组frt靶位点,从而消除由于基因组插入位点引起的表达水平的变化。因此,它是比较不同稳定细胞系中多种遗传构建体的作用的理想工具。我们创建了基于pcdnatm5/frt/to(lifetechnologies)的必需frt供体质粒。黑腹果蝇重编码的人erf1wt和erf1e55d变体被成功整合到基因组中。所得到的trex293wt和e55d细胞系可以在诱导和未诱导条件下维持数月,这表明毒性作用可忽略不计。通过以1:1的比例瞬时转染含有pylrs/trnacua和sfgfp报告物的构建体(构建体g+h/i,图1)测定琥珀抑制效率。在0.5mm氨基酸2的存在下,通过加入1μg/ml四环素在诱导erf1表达后,与sfgfp(wt)相比较,琥珀抑制在sfgfp(tag)中从75%增加到99%,在sfgfp(tag)3中从8%增加至20%(图5a)。与通过瞬时转染引入erf1相比,观察到的效果不太明显(图4)。免疫染色erf1的细胞裂解物显示,erf1的水平在诱导后成功升高(图5d),但与瞬时转染后erf1表达相比不太明显(图4,b/d)。与通过每个细胞的瞬时转染递送质粒的多个拷贝相比,这与单个基因组整合位点的存在相一致。在内源性野生型erf1的存在下,仅当存在足够的erf1e55d以防止形成能够在uag密码子终止的野生型erf1/erf3复合物时,才能增强琥珀抑制。实施例7——shrna(erf1)的组成型表达在erf1e55d稳定整合的细胞系中增加了琥珀通读由于erf1变体的强表达能够增强琥珀抑制(图3),所以在转基因erf1存在下减少内源性erf1也是如此(carnes等人2003;ilegems等人2004)。通过shrna降低内源性erf1水平与基础终止密码子通读的非特异性增加相关(图3b,c)。根据具有增强的琥珀抑制潜力的稳定细胞系的概念,早期使用的erf1特异性shrnas设定通过随机慢病毒融合到基因组中而被引入细胞系(santacruzbiotechnology)。基于获得性嘌呤霉素抗性,选择成功整合事件的细胞。产生含有erf1e55d和erf1野生型的两种细胞系,并且可以在存在和不存在四环素的情况下维持数周。在瞬时转染pylrs/trnacua对和sfgfp(tag)或sfgfp(tag)3报告构建体(质粒g+h/i,图1)后测定琥珀抑制效率。琥珀抑制效率显示为高于背景的总sfgfp荧光,并且在同一细胞系中对sfgfp(wt)进行归一化。与“空白”亲本细胞系trex293flp-in相比,erf1e55d的整合和诱导在0.5mm的2的存在下导致琥珀抑制从87%增加到99%。相比之下,erf1wt的诱导平均降低琥珀抑制至81%。具有erf1特异性shrna的组成型表达的细胞系在erf1e55d背景中将相对荧光增加至114%,但在erf1wt背景下保持79%的抑制(图5c)。在trex293flp-in亲本细胞系中,sfgfp(tag)3报告物的琥珀抑制效率为7.6%。表达erf1e55d的稳定系显示琥珀抑制增加至19%,表达erf1e55d和shrna(erf1)二者的细胞系中进一步增加至31%。相比之下,表达erf1wt的细胞系与7.6%的亲代细胞系相比,琥珀抑制没有变化。表达erf1wt和shrna(erf1)的衍生细胞系显示抑制效率轻微降低至5%(图5d)。总的来说,通过单拷贝erf1e55d的稳定整合可以提高相对的琥珀抑制效率,进一步增强需要降低内源性erf1水平,或者增加erf1e55d的表达。在任一种情况下,可以实现sfgfp-2的定量表达,并且可以以sfgfp的30-40%的效率生产sfgfp-23。实施例8——erf1突变体增加了dmel细胞中非天然氨基酸的掺入效率我们还评估了选择的erf1突变体在基于昆虫细胞的表达系统中的作用。衍生自果蝇的细胞系,如schneider2细胞,常规用于蛋白质的大规模表达。已经使用m.mazeipylrs/trnacua对和合适的表达系统证明了在这些系统中向蛋白质中掺入非天然氨基酸(bianco等人,2012;elliott等人,2014)。针对每个erf1变体,四种构建体用于驱动非天然氨基酸掺入:pylrs/trnacua表达构建体,含有gfp-mcherry或gfp-tag-mcherry(uas驱动)的报道构建体,和表达构建体。总体而言,dmel细胞的转染效率低于哺乳动物系统,所得到的细胞裂解物和印迹代表转染和未转染细胞的混合物,降低了表观效应(图6a)。视觉指示剂是δ100突变体,显示在内源性erf1条带以下的对应于异位的截短erf1的弱带(图6b)。图6a中所示的琥珀抑制的定量是基于三次独立的转染实验,相应的印迹被单独定量。野生型释放因子的表达导致通读的非常微小的减少,而erf1δ100突变体的表达使通读水平增加了五倍,从4.8%增加到23%。这种效应伴随着截短的gfp总量的大幅度减少,这表明整体翻译过程的显著破坏(图6b)。e55d突变体显示了所测试变体的通读水平的第二大增长,增长了四倍,达到了15%。类似地,突变e55a和nk129pq使通读增加了大约三倍,达到12%。据报道增强uga通读的突变型s70ag73s对琥珀抑制没有影响。尽管在细胞背景和使用的报告系统中均存在差异,但是这些结果密切地反映了先前在哺乳动物细胞中观察到的相对效应大小。唯一的例外是突变y125f,其在dmel细胞中似乎具有可忽略的作用,但在哺乳动物系统中改善了uag通读。实施例9——erf1e55d突变体提高了广泛的非天然氨基酸和trna合成酶变体的掺入效率。如图7所示,在erf1e55d突变体的存在下向sfgfp(tag)掺入四种不同的非天然氨基酸-boc-k(nε-[(叔丁氧羰基)羰基]-l-赖氨酸、降冰片烯-k(nε-降冰片烯-2-基氧基羰基-l-赖氨酸)、环丙烯-k-(nε-[((2-甲基环丙-2-烯-1-基)甲氧基)羰基]-l-赖氨酸)和双环炔-n(nε-([(1r,8s)-双环[6.1.0]壬-4-基-9-基甲氧基]羰基)-赖氨酸)得到改善。将实施例6中生成的t-rex293erf1e55d用于诱导表达erf1e55d。通过以1:1的比例瞬时转染含有pylrs/trnacua和sfgfp150tag报告子的构建体(构建体g+h/i,图1)来确定琥珀抑制效率。琥珀抑制效率是基于利用蛋白质印迹法的sfgfp的表达测定的。在所有情况下,erf1e55d突变体的存在增加了sfgfp的表达。实施例10——讨论我们已经定义了相对于天然翻译控制的非天然氨基酸掺入的效率,这使我们能够定量评估非天然氨基酸掺入效率的改进。我们所创建的优化系统提供了氨基酸1和2的非天然氨基酸掺入效率的17至20倍的改善。对于氨基酸1,掺入效率从5%提高到85%,而对于氨基酸2掺入效率从无终止密码子对照的7%增加到157%。此外,优化的系统分别将在三个位置掺入1和2的蛋白质的产量从不可测量的低水平提高到无终止对照的12%和43%。各种因素有助于非天然氨基酸掺入的显著改善,其中包括:优化trnacua水平以优化pylrs/trnacua表达;以及开发和使用工程改造的erf1变体。尽管掺入非天然氨基酸对于单独使用优化的pylrs/trnacua系统的单个琥珀密码子的响应是非常有效的,但是通过加入erf1(例如e55d)进一步提高了效率。当在多个位点掺入非天然氨基酸时,erf1突变体对非天然氨基酸掺入的影响更显著,在三个位点上含有氨基酸1的蛋白质的产量增加了2-3倍;三个位点上含有2的蛋白质的产量增加了4倍。实施例11——替代erf1突变体的证明我们最初通过在hek293t细胞系中瞬时转染来筛选erf1变体。下表列出了通过其对非天然氨基酸掺入(bock)的影响进行排列的所筛选的突变。相关操作方案和支持材料如上述实施例。实施例12:erf1突变体在各种真核生物物种中的功能本发明适用于多种真核生物。本发明可以应用于昆虫细胞如蝇细胞(如果蝇)、真菌细胞如酵母细胞和其他真核生物。我们使用瞬时转染将突变体erf1引入昆虫(dmel)细胞系。方法:d.mel2细胞的瞬时转染(s2)按照标准细胞培养实践,将果蝇s2细胞(d.mel;invitrogen)保持在富含2mml谷氨酰胺和青/链霉素(50iu/ml青霉素,50μg/ml链霉素)的完全express5sfm培养基(lifetechnologiesltd.)的烧瓶t75中。在转染前24小时,通过刮擦或摇动将细胞从表面脱离,并将5ml悬浮的细胞用8ml预热培养基稀释。对于24孔板,每孔接种0.5ml悬浮细胞并生长过夜。使用fugenehd(promega)瞬时转染细胞。对于每个孔,将1.75μlfugenehd与15μl无菌水混合,并在室温下温育5分钟。类似地,在15μl无菌水中稀释0.75μgdna(各报告基因pylrs/pylt和erf1构建体0.3μg,对gal4为0.15μg),并加入到稀释的fugenehd试剂中,然后在室温下温育15分钟。在含有靶细胞的每个孔中,取出用过的生长培养基,并用500μl无菌pbs小心清洗细胞。然后,如果需要,每孔用500μlexpress5培养基、0.2mmcuso4(sigma)、2mm/ml谷氨酰胺和非天然氨基酸填充。1溶解于无菌水/naoh(20mm1)中,并加入生长培养基中。随后使用4mhcl将ph调节至6.5。除非另有说明,否则生长培养基中最终浓度为2mm。将30μl转染混合物的25μl滴加到每个孔中。轻轻摇动板,并在25℃下孵育过夜。转染后16小时,每孔用pbs洗涤,细胞在100μl具有完全蛋白酶抑制剂(roche)的ripa缓冲液(sigma)中裂解,同时在室温下振荡15分钟。通过gfptagmcherry构建体的表达测定非天然氨基酸的掺入。通过对gfp免疫染色的印迹进行定量测定全长蛋白质与截短蛋白质的比例,如针对hek293t细胞所述。果蝇黑腹果蝇基因构建体使用bamhi/noti限制性位点(引物erf1_sg105),将含有末端his6标签和三重终止终止信号的针对黑腹果蝇密码子使用优化的人erf1基因克隆到质粒sg105(bianco等人,2012)中。sg105衍生自uasp,并含有白色基因座下游的第二个多克隆位点(1998)。ab51包含六个拷贝的u6pylt盒和uaspylrs(ambrabianco,未发表的数据)。确定黑腹果蝇中非天然氨基酸的掺入效率使用果蝇胚胎注射服务(bestgeneinc.),通过p元件注射产生含有erf1变体的转基因蝇系。克隆到质粒sg105中的erf1δ100和e55d构建体被成功地显微注射,分别返回了九个和七个独特的系。在16℃的苹果盘上保持蝇系储备。对于遗传杂交和非天然氨基掺入实验,将蝇保持在25℃。含有erf1δ100或e55d变体的片段蝇系与来自ft74-s2-nos蝇系的未交配雌性杂交。根据每个erf1系筛选得到的后代的遗传标记的正确组合,维持用于进一步实验。ft74s2nos系支持使用pylrs/pyltcua对的非天然氨基酸掺入,并表达双重萤光素酶报告子。在基因组上整合的构建体是uas双重萤光素酶(tag)报告子、uaspylrs、四个拷贝的u6pylt盒(triple-rep-l)和nos-gal4-vp16盒(bloomington4937)。讨论了这些构建体和系的创建(bianco等人,2012)。将化合物2混合到酵母糊中加入蝇中。2溶于水/naoh至终浓度为10mm,加入干燥的酵母,直到达到糊状稠度。加入1mhcl以中和糊状物。为了测量蝇系中掺入非天然氨基酸的含量,在5只雄性的管中,表达用于非天然氨基掺入的必需组分以及erf1变体的每个系有15只雌性蝇被与5只雄性一起装在试管中,并饲以少量10mm2-酵母糊。在24和48小时后将蝇转移到具有新鲜的2-酵母糊的新管中。第三天早上将其转入具有2-酵母糊的新鲜管中,下午解剖雌性蝇。为了解剖,将蝇收集在co2垫上。雌性蝇转移到显微镜下的解剖玻璃上,通过仔细去除后尖部从胸部分离出卵巢。将10-12只蝇的卵巢收集在pbs缓冲液中,并转移到1.5ml微量离心管中。将管在最低设置的台式离心机中离心30秒,除去上清液。加入100μl1x被动裂解缓冲液(promega)后,使用塑料杵将卵巢研磨,直到溶液接近乳状外观。通过全速离心3分钟除去碎片。仔细取出上清液,全速再沉淀1分钟。每个双重萤光素酶测定法以96孔格式使用10μl的该裂解物,每个样品重复三次。如对哺乳动物细胞裂解物所述进行测定(promega)。参见图12。a.遗传背景的插图。表达吡咯赖氨酰aars/trnacua对、双重萤光素酶报告基因和卵巢特异性启动子的来自蝇系ft74-s2-nos/tm6的未交配过的雌性与显微注射后erf1e55d或δ100的随机基因组插入产生的平衡蝇系杂交(bestgene)。b、c.在双重萤光素酶测定中测量终止密码子通读。收集来自每个遗传杂交的十二只具有正确遗传标记的雌性蝇,并加入10mmcyp酵母糊72小时。通过解剖收集卵巢、汇集并测试uag通读。小图b列出了从erf1e55d整合事件生成的所有蝇系的结果;小图c列出了erf1δ100的结果。实施例13:pyltu25c和pyltu25copt变体均有效地在hek细胞中进行琥珀抑制用gfp150uag和4xpyltu25c或4xpyltu25copt转染的hek细胞通过facs分析gfp荧光,以粉色圆圈圈出gfp阳性群体,标记以百分比,用于图的重复mfi。标准转染和流式细胞术方案,在2mmbock中表达。参见图13。利用4xpyltu25c或4xpyltu25copt的曲妥珠单抗重链的蛋白质印迹,位置a114uag,lc和hc在独立的质粒上。测量图形重复印迹强度。标准转染和western印迹操作方案,在2mmbock中表达。参见图14和图15。用于比较的亲本m.mazeipyltrna(cua反密码子)的trna三叶草图、u25c衍生物、u25c和opt衍生物。以绿色显示u25c突变,以粉红色显示六核苷酸opt突变设置。参见图16。trna变体的dna序列的序列比对(黑色差异),注意到cca尾部没有在哺乳动物质粒中明确编码,而是由细胞转录后添加。参见图17。m.mazeipyltu25coptggaaacgtgatcatgtagatcgaacggactctaaatccgttcagtggggttagattccccacgtttccg(1x)人u6启动子::m.mazeipyltu25copt::3’utr和终止子cctagttgggcaggaagagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattagaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggaaacgtgatcatgtagatcgaacggactctaaatccgttcagtggggttagattccccacgtttccggacaagtgcggttttt(4x)人u6启动子::m.mazeipyltu25copt::3’utr和终止子,如所克隆的tgggcgatgtgcgctctgcccactgacgggcaccggagcgatcgcagatcccctagttgggcaggaagagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattagaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggaaacgtgatcatgtagatcgaacggactctaaatccgttcagtggggttagattccccacgtttccggacaagtgcggtttttagaattacaacttatatcgtatgggctagactcgagcctagttgggcaggaagagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattagaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggaaacgtgatcatgtagatcgaacggactctaaatccgttcagtggggttagattccccacgtttccggacaagtgcggtttttgcggccgcgatatctgcagaattcacactggactaggatccgagctccctagttgggcaggaagagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattagaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggaaacgtgatcatgtagatcgaacggactctaaatccgttcagtggggttagattccccacgtttccggacaagtgcggtttttggtaccaagcttaagcagactcgtcgtgactacattagcctagttgggcaggaagagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattagaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggaaacgtgatcatgtagatcgaacggactctaaatccgttcagtggggttagattccccacgtttccggacaagtgcggtttttcctagtggccttggaggccttttccccgtatccccccaggtgtctgcaggctcaaagagcagcgagaagc该4x盒可以代替以上述各种载体中的4xpyltu25c盒,例如具有gfp(150uag)的pkym1、曲妥珠单抗lc和hc(114uag))。实施例14:erf1e55d提高了抗体表达所有实验均用2mmbock进行,除非另有说明。列为“cypk”的片段用0.5mm环丙烯进行。两者都是吡罗立定模拟物。hc和lc分别在独立的质粒上,并使用标准pei方案以1:1的比例共转染。对于gfp研究,仅转染单个质粒。由于非天然机制的trna/合成酶与gfp/mab基因在同一质粒上,因此这些研究不需要额外的质粒。抗体表达以lc-ires-hc形式根据含有两个抗体基因以及吡咯赖氨酸非天然氨基酸trna和合成酶的单一载体进行。该4xpylt(u25c)/pyls构建体如上所述。erf1由pcdna5(标准表达载体)表达。将erf1相对抗体质粒以1:5的比例转染。在expi293细胞(一种悬浮hek系统)中进行实验,并使用标准方案用pei转染。培养物各30ml,60ug的dna,180μg的pei,表达7天。使用与hrp缀合的抗-hc和抗lc抗体,直接从培养物上清中进行蛋白质印迹。在重链位点ala114处具有uag琥珀密码子的曲妥珠单抗在hek细胞中以我们的表达系统显示具有erf1e55d的表达增加。参见图18。构建体如图19所示。质粒图如图20所示。序列如下。pkym1lc-ires-hc序列gttaggcgttttgcgctgcttcgcgatgtacgggccagatatacgcgttgacattgattattgactagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacggggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacgggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgtacggtgggaggtctatataagcagagctcgtttagtgaaccgtcagatcgcctggagacgccatccacgctgttttgacctccatagaagacaccgggaccgatccagcctccggactctagaggatcgaattaagcttggtaccgccgccaccatgggatggtcatgtatcatcctttttctagtagcaactgcaaccggtgtacattctgacatccagatgacccagtccccgagctctctgtctgcgtctgttggtgaccgcgttaccatcacctgccgtgcgtcccaggacgttaacaccgccgtggcgtggtatcaacagaaaccgggtaaagcgccaaaactgctgatctactccgcgtctttcctgtactctggtgttccgtctcgtttcagcggttctcgttctggtactgacttcaccctcaccatctcttctctgcagccggaagacttcgcgacctactactgccagcagcactacaccaccccaccgaccttcggtcagggcaccaaagttgaaatcaaacgtacggtggctgcaccatctgtcttcatcttcccgccatctgatgagcagttgaaatctggaactgcctctgttgtgtgcctgctgaataacttctaccccagagaagccaaagtgcagtggaaggtggacaacgccctgcagagcggaaacagccaggaaagcgtgacagagcaggattccaaggattccacatacagcctgagcagcacactgacactgtccaaggccgactacgagaagcacaaggtgtacgcctgcgaagtgacacaccagggactgtcctcccctgtgacaaagagcttcaacagaggagaatgctgaggccgcgcccctctccctcccccccccctaacgttactggccgaagccgcttggaataaggccggtgtgcgtttgtctatatgttattttccaccatattgccgtcttttggcaatgtgagggcccggaaacctggccctgtcttcttgacgagcattcctaggggtctttcccctctcgccaaaggaatgcaaggtctgttgaatgtcgtgaaggaagcagttcctctggaagcttcttgaagacaaacaacgtctgtagcgaccctttgcaggcagcggaaccccccacctggcgacaggtgcctctgcggccaaaagccacgtgtataagatacacctgcaaaggcggcacaaccccagtgccacgttgtgagttggatagttgtggaaagagtcaaatggctctcctcaagcgtattcaacaaggggctgaaggatgcccagaaggtaccccattgtatgggatctgatctggggcctcggtgcacatgctttacatgtgtttagtcgaggttaaaaaaacgtctaggccccccgaaccacggggacgtggttttcctttgaaaaacacgataatacctccggaatgggatggtcatgtatcatcctttttctagtagcaactgcaaccggtgtacattctgaagttcagctggttgaatctggtggtggtctggttcaaccgggtggctccctgcgtctgtcttgtgcggcctctggttttaacatcaaagatacctatatccactgggttcgtcaggcgccaggcaaaggtctggaatgggttgcgcgtatctacccgaccaacggttacacccgctacgcggactctgttaaaggtcgtttcaccatctctgcggacacctctaaaaacaccgcgtacctgcagatgaactctctgcgtgcggaagacaccgccgtttactactgctctcgttggggtggtgacggtttctacgcgatggactactggggtcagggtacgctggttaccgtttcttcttagtcgaccaagggcccatcggtcttccccctggcaccctcctccaagagcacctctgggggcacagcggccctgggctgcctggtcaaggactacttccccgaacctgtgacggtctcgtggaactcaggcgccctgaccagcggcgtgcacaccttcccggctgtcctacagtcctcaggactctactccctcagcagcgtggtgaccgtgccctccagcagcttgggcacccagacctacatctgcaacgtgaatcacaagcccagcaacaccaaggtggacaagaaagttgagcccaaatcttgtgacaaaactcacacatgcccaccgtgcccagcacctgaactcctggggggaccgtcagtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacccctgaggtcacatgcgtggtggtggacgtgagccacgaagaccctgaggtcaagttcaactggtacgtggacggcgtggaggtgcataatgccaagacaaagccgcgggaggagcagtacaacagcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaatggcaaggagtacaagtgcaaggtctccaacaaagccctcccagcccccatcgagaaaaccatctccaaagccaaagggcagccccgagaaccacaggtgtacaccctgcccccatcccgggaggagatgaccaagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagcgacatcgccgtggagtgggagagcaatgggcagccggagaacaactacaagaccacgcctcccgtgctggactccgacggctccttcttcctctatagcaagctcaccgtggacaagagcaggtggcagcaggggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactacacgcagaagagcctctccctgtccccgggttgagctcgagtctagagggttcgatccctaccggttagtaatgagtttaaactcgacaatcaacctctggattacaaaatttgtgaaagattgactggtattcttaactatgttgctccttttacgctatgtggatacgctgctttaatgcctttgtatcatgctattgcttcccgtatggctttcattttctcctccttgtataaatcctggttgctgtctctttatgaggagttgtggcccgttgtcaggcaacgtggcgtggtgtgcactgtgtttgctgacgcaacccccactggttggggcattgccaccacctgtcagctcctttccgggactttcgctttccccctccctattgccacggcggaactcatcgccgcctgccttgcccgctgctggacaggggctcggctgttgggcactgacaattccgtggtgttgtcggggaagctgacgtcctttccatggctgctcgcctgtgttgccacctggattctgcgcgggacgtccttctgctacgtcccttcggccctcaatccagcggaccttccttcccgcggcctgctgccggctctgcggcctcttccgcgtcttcgccttcgccctcagacgagtcggatctccctttgggccgcctccccgcctggaaacgggggaggctaactgaaacacggaaggagacaataccggaaggaacccgcgctatgacggcaataaaaagacagaataaaacgcacgggtgttgggtcgtttgttcataaacgcggggttcggtcccagggctggcactctgtcgataccccaccgagaccccattggggccaatacgcccgcgtttcttccttttccccaccccaccccccaagttcgggtgaaggcccagggctcgcagccaacgtcggggcggcaggccctgccatagcagatctgcgcagctctataaagtaacaaaacttttatgagggacagcccccccccaaagcccccagggatgtaattacgtccctcccccgctagggggcagcagcgagccgcccggggctccgctccggtccggcgctccccccgcatccccgagccggcagcgtgcggggacagcccgggcacggggaaggtggcacgggatcgctttcctctgaacgcttctcgctgctctttgagcctgcagacacctggggggatacggggaaaaggcctccaaggccactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactagtaaggatctgcgatcgctccggtgcccgtcagtgggcagagcgcacatcgcccacagtccccgagaagttggggggaggggtcggcaattgaacgggtgcctagagaaggtggcgcggggtaaactgggaaagtgatgtcgtgtactggctccgcctttttcccgagggtgggggagaaccgtatataagtgcagtagtcgccgtgaacgttctttttcgcaacgggtttgccgccagaacacagctgaagcttcgaggggctcgcatctctccttcacgcgcccgccgccctacctgaggccgccatccacgccggttgagtcgcgttctgccgcctcccgcctgtggtgcctcctgaactgcgtccgccgtctaggtaagtttaaagctcaggtcgagaccgggcctttgtccggcgctcccttggagcctacctagactcagccggctctccacgctttgcctgaccctgcttgctcaactctacgtctttgtttcgttttctgttctgcgccgttacagatccaagctgtgaccggcgcctactctagagctagcgtttaaacttaagcttgccaccatggactacaaggacgacgacgacaaggacaagaagcccctgaacaccctgatcagcgccacaggactgtggatgtccagaaccggcaccatccacaagatcaagcaccacgaggtgtcccggtccaaaatctacatcgagatggcctgcggcgatcacctggtcgtcaacaacagcagaagcagccggacagccagagccctgcggcaccacaagtacagaaagacctgcaagcggtgcagagtgtccgacgaggacctgaacaagttcctgaccaaggccaacgaggaccagaccagcgtgaaagtgaaggtggtgtccgcccccacccggaccaagaaagccatgcccaagagcgtggccagagcccccaagcccctggaaaacaccgaagccgctcaggcccagcccagcggcagcaagttcagccccgccatccccgtgtctacccaggaaagcgtcagcgtccccgccagcgtgtccaccagcatctctagcatctcaaccggcgccacagcttctgccctggtcaagggcaacaccaaccccatcaccagcatgtctgcccctgtgcaggcctctgccccagccctgaccaagtcccagaccgaccggctggaagtgctcctgaaccccaaggacgagatcagcctgaacagcggcaagcccttccgggagctggaaagcgagctgctgagccggcggaagaaggacctccagcaaatctacgccgaggaacgggagaactacctgggcaagctggaaagagagatcacccggttcttcgtggaccggggcttcctggaaatcaagagccccatcctgatccccctggagtacatcgagcggatgggcatcgacaacgacaccgagctgagcaagcagattttccgggtggacaagaacttctgcctgcggcccatgctggcccccaacctgtacaactacctgcggaaactggatcgcgctctgcccgaccccatcaagattttcgagatcggcccctgctaccggaaagagagcgacggcaaagagcacctggaagagtttacaatgctgaacttttgccagatgggcagcggctgcaccagagagaacctggaatccatcatcaccgactttctgaaccacctggggatcgacttcaagatcgtgggcgacagctgcatggtgtacggcgacaccctggacgtgatgcacggcgacctggaactgtctagcgccgtcgtgggacccatccctctggaccgggagtggggcatcgataagccctggatcggagccggcttcggcctggaacggctgctgaaagtcaagcacgactttaagaacatcaagcgggctgccagaagcgagagctactacaacggcatcagcaccaacctgtgatgaggatccgcggccgcgcccctctccctcccccccccctaacgttactggccgaagccggagttcttcgcccaccccaacttgtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgtatcttatcatgtctgtataccgtcgacctctagctagagcttggcgtaatcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatacgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcaatgctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatccagtctattaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcgccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatactcttcctttttcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctgacgtcgacggatcgggagatctcccgatcccctatggtcgactctcagtacaatctgctctgatgccgcatagttaagccagtatctgctccctgcttgtgtgttggaggtcgctgagtagtgcgcgagcaaaatttaagctacaacaaggcaaggcttgaccgacaattgcatgaagaatctgcttagg具有前导肽的hca114xdna序列atgggatggtcatgtatcatcctttttctagtagcaactgcaaccggtgtacattctgaagttcagctggttgaatctggtggtggtctggttcaaccgggtggctccctgcgtctgtcttgtgcggcctctggttttaacatcaaagatacctatatccactgggttcgtcaggcgccaggcaaaggtctggaatgggttgcgcgtatctacccgaccaacggttacacccgctacgcggactctgttaaaggtcgtttcaccatctctgcggacacctctaaaaacaccgcgtacctgcagatgaactctctgcgtgcggaagacaccgccgtttactactgctctcgttggggtggtgacggtttctacgcgatggactactggggtcagggtacgctggttaccgtttcttcttagtcgaccaagggcccatcggtcttccccctggcaccctcctccaagagcacctctgggggcacagcggccctgggctgcctggtcaaggactacttccccgaacctgtgacggtctcgtggaactcaggcgccctgaccagcggcgtgcacaccttcccggctgtcctacagtcctcaggactctactccctcagcagcgtggtgaccgtgccctccagcagcttgggcacccagacctacatctgcaacgtgaatcacaagcccagcaacaccaaggtggacaagaaagttgagcccaaatcttgtgacaaaactcacacatgcccaccgtgcccagcacctgaactcctggggggaccgtcagtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacccctgaggtcacatgcgtggtggtggacgtgagccacgaagaccctgaggtcaagttcaactggtacgtggacggcgtggaggtgcataatgccaagacaaagccgcgggaggagcagtacaacagcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaatggcaaggagtacaagtgcaaggtctccaacaaagccctcccagcccccatcgagaaaaccatctccaaagccaaagggcagccccgagaaccacaggtgtacaccctgcccccatcccgggaggagatgaccaagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagcgacatcgccgtggagtgggagagcaatgggcagccggagaacaactacaagaccacgcctcccgtgctggactccgacggctccttcttcctctatagcaagctcaccgtggacaagagcaggtggcagcaggggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactacacgcagaagagcctctccctgtccccgggt具有前导肽的lcdna序列atgggatggtcatgtatcatcctttttctagtagcaactgcaaccggtgtacattctgacatccagatgacccagtccccgagctctctgtctgcgtctgttggtgaccgcgttaccatcacctgccgtgcgtcccaggacgttaacaccgccgtggcgtggtatcaacagaaaccgggtaaagcgccaaaactgctgatctactccgcgtctttcctgtactctggtgttccgtctcgtttcagcggttctcgttctggtactgacttcaccctcaccatctcttctctgcagccggaagacttcgcgacctactactgccagcagcactacaccaccccaccgaccttcggtcagggcaccaaagttgaaatcaaacgtacggtggctgcaccatctgtcttcatcttcccgccatctgatgagcagttgaaatctggaactgcctctgttgtgtgcctgctgaataacttctaccccagagaagccaaagtgcagtggaaggtggacaacgccctgcagagcggaaacagccaggaaagcgtgacagagcaggattccaaggattccacatacagcctgagcagcacactgacactgtccaaggccgactacgagaagcacaaggtgtacgcctgcgaagtgacacaccagggactgtcctcccctgtgacaaagagcttcaacagaggagaatgctga具有a114x突变的hc蛋白序列,*代表uag非天然位点evqlvesggglvqpggslrlscaasgfnikdtyihwvrqapgkglewvariyptngytryadsvkgrftisadtskntaylqmnslraedtavyycsrwggdgfyamdywgqgtlvtvss*stkgpsvfplapsskstsggtaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtqtyicnvnhkpsntkvdkkvepkscdkthtcppcpapellggpsvflfppkpkdtlmisrtpevtcvvvdvshedpevkfnwyvdgvevhnaktkpreeqynstyrvvsvltvlhqdwlngkeykckvsnkalpapiektiskakgqprepqvytlppsreemtknqvsltclvkgfypsdiavewesngqpennykttppvldsdgsfflyskltvdksrwqqgnvfscsvmhealhnhytqkslslspglc蛋白序列diqmtqspsslsasvgdrvtitcrasqdvntavawyqqkpgkapklliysasflysgvpsrfsgsrsgtdftltisslqpedfatyycqqhyttpptfgqgtkveikrtvaapsvfifppsdeqlksgtasvvcllnnfypreakvqwkvdnalqsgnsqesvteqdskdstyslsstltlskadyekhkvyacevthqglsspvtksfnrgec实施例15:erf1e55d增强hek细胞中的bcn掺入hek2933天表达,1mmbcn(nepsilon-(双环[6.1.0]壬-4-炔-9-基甲氧基)羰基-l-赖氨酸)),1∶1比例的质粒,如上所述的标准方案。参见图21。geneticencodingofbicyclononynesandtrans-cyclooctenesforrapidsite-specificproteinlabelinginvitroandinlivemammaliancellsviafluorogenicdiels-alderreactionsj.am.chem.soc.2012134:10317-10320k.lang,l.davis,s.wallace,m.mahesh,d.j.cox,m.l.blackman,j.m.fox&j.w.chin.缩写pylrs,吡咯赖氨酰-trna合成酶;pylt,吡咯赖氨酰-trna合成酶;erf1,真核释放因子1;erf3,真核释放因子3;sfgfp,超级折叠绿色荧光蛋白。参考文献(1)lang,k.;chin,j.w.chemrev2014,114,4764.(2)kim,c.h.;axup,j.y.;schultz,p.g.curropinchembiol2013,17,412.(3)baker,a.s.;deiters,a.acschembiol2014,9,7(4)davis,l.;chin,j.w.natrevmolcellbiol2012,13,168.(5)chin,j.w.annurevbiochem2014,83:379(6)bertram,g.;innes,s.;minella,o.;richardson,j.;stansfield,i.microbiology2001,147,255.(7)jackson,r.j.;hellen,c.u.;pestova,t.v.advproteinchemstructbiol2012,86,45.(8)dever,t.e.;green,r.coldspringharbperspectbiol2012,4,a013706.(9)scolnick,e.;tompkins,r.;caskey,t.;nirenberg,m.procnatlacadsciusa1968,61,768.(10)petry,s.;brodersen,d.e.;murphy,f.v.t.;dunham,c.m.;selmer,m.;tarry,m.j.;kelley,a.c.;ramakrishnan,v.cell2005,123,1255.(11)konecki,d.s.;aune,k.c.;tate,w.;caskey,c.t.jbiolchem1977,252,4514.(12)wang,k.;neumann,h.;peak-chew,s.y.;chin,j.w.natbiotechnol2007,25,770.(13)mukai,t.;hayashi,a.;iraha,f.;sato,a.;ohtake,k.;yokoyama,s.;sakamoto,k.nucleicacidsres2010,38,8188.(14)johnson,d.b.;xu,j.;shen,z.;takimoto,j.k.;schultz,m.d.;schmitz,r.j.;xiang,z.;ecker,j.r.;briggs,s.p.;wang,l.natchembiol2011,7,779.(15)lajoie,m.j.;rovner,a.j.;goodman,d.b.;aerni,h.r.;haimovich,a.d.;kuznetsov,g.;mercer,j.a.;wang,h.h.;carr,p.a.;mosberg,j.a.;rohland,n.;schultz,p.g.;jacobson,j.m.;rinehart,j.;church,g.m.;isaacs,f.j.science2013,342,357.(16)wu,i.l.;patterson,m.a.;carpenterdesai,h.e.;mehl,r.a.;giorgi,g.;conticello,v.p.chembiochem2013,14,968.(17)mukai,t.;kobayashi,t.;hino,n.;yanagisawa,t.;sakamoto,k.;yokoyama,s.biochemicalandbiophysicalresearchcommunications2008,371,818.(18)chen,p.r.;groff,d.;guo,j.;ou,w.;cellitti,s.;geierstanger,b.h.;schultz,p.g.angewandtechemieinternationaledition2009,48,4052.(19)gautier,a.;nguyen,d.p.;lusic,h.;an,w.;deiters,a.;chin,j.w.jamchemsoc2010,132,4086.(20)xiao,h.;chatterjee,a.;choi,s.h.;bajjuri,k.m.;sinha,s.c.;schultz,p.g.angewchemintedengl2013,52,14080.(21)elliott,t.s.;townsley,f.m.;bianco,a.;ernst,r.j.;sachdeva,a.;elsasser,s.j.;davis,l.;lang,k.;pisa,r.;greiss,s.;lilley,k.s.;chin,j.w.natbiotechnol2014,32,465.(22)nguyen,d.p.;lusic,h.;neumann,h.;kapadnis,p.b.;deiters,a.;chin,j.w.jamchemsoc2009,131,8720.(23)miyake-stoner,s.j.;refakis,c.a.;hammill,j.t.;lusic,h.;hazen,j.l.;deiters,a.;mehl,r.a.biochemistry2010,49,1667.(24)chatterjee,a.;sun,s.b.;furman,j.l.;xiao,h.;schultz,p.g.biochemistry2013,52,10(25)kolosov,p.;frolova,l.;seit-nebi,a.;dubovaya,v.;kononenko,a.;oparina,n.;justesen,j.;efimov,a.;kisselev,l.nucleicacidsres2005,33,6418.(26)bulygin,k.n.;khairulina,y.s.;kolosov,p.m.;ven'yaminova,a.g.;graifer,d.m.;vorobjev,y.n.;frolova,l.y.;kisselev,l.l.;karpova,g.g.rna2010,16,1902.(27)seit-nebi,a.;frolova,l.;kisselev,l.emborep2002,3,881.(28)eliseev,b.;kryuchkova,p.;alkalaeva,e.;frolova,l.nucleicacidsres2011,39,599.(29)lekomtsev,s.;kolosov,p.;bidou,l.;frolova,l.;rousset,j.p.;kisselev,l.procnatlacadsciusa2007,104,10824.(30)kryuchkova,p.;grishin,a.;eliseev,b.;karyagina,a.;frolova,l.;alkalaeva,e.nucleicacidsres2013,41,4573.(31)grentzmann,g.;ingram,j.a.;kelly,p.j.;gesteland,r.f.;atkins,j.f.rna1998,4,479.(32)keeling,k.m.;lanier,j.;du,m.;salas-marco,j.;gao,l.;kaenjak-angeletti,a.;bedwell,d.m.rna2004,10,691.(33)salas-marco,j.;fan-minogue,h.;kallmeyer,a.k.;klobutcher,l.a.;farabaugh,p.j.;bedwell,d.m.molcellbiol2006,26,438.(34)conard,s.e.;buckley,j.;dang,m.;bedwell,g.j.;carter,r.l.;khass,m.;bedwell,d.m.rna2012,18,1210.(35)legoff,x.;philippe,m.;jean-jean,o.molcellbiol1997,17,3164.(36)merkulova,t.i.;frolova,l.y.;lazar,m.;camonis,j.;kisselev,l.l.febslett1999,443,41.(37)cheng,z.;saito,k.;pisarev,a.v.;wada,m.;pisareva,v.p.;pestova,t.v.;gajda,m.;round,a.;kong,c.;lim,m.;nakamura,y.;svergun,d.i.;ito,k.;song,h.genesdev2009,23,1106.(38)kononenko,a.v.;mitkevich,v.a.;dubovaya,v.i.;kolosov,p.m.;makarov,a.a.;kisselev,l.l.proteins2008,70,388.(39)desgeorges,a.;hashem,y.;unbehaun,a.;grassucci,r.a.;taylor,d.;hellen,c.u.;pestova,t.v.;frank,j.nucleicacidsres2014,42,3409.(40)janzen,d.m.;geballe,a.p.nucleicacidsres2004,32,4491.(41)liang,h.;wong,j.y.;bao,q.;cavalcanti,a.r.;landweber,l.f.jmolevol2005,60,337.补充参考文献(1)gautier,a.;nguyen,d.p.;lusic,h.;an,w.;deiters,a.;chin,j.w.jamchemsoc2010,132,4086.(2)hoover,d.m.;lubkowski,j.nucleicacidsres2002,30,e43.s12(3)elliott,t.s.;townsley,f.m.;bianco,a.;ernst,r.j.;sachdeva,a.;elsasser,s.j.;davis,l.;lang,k.;pisa,r.;greiss,s.;lilley,k.s.;chin,j.w.natbiotechnol2014,32,465.(4)arbely,e.;torres-kolbus,j.;deiters,a.;chin,j.w.jamchemsoc2012,134,11912.(5)wilkins,m.r.;gasteiger,e.;bairoch,a.;sanchez,j.c.;williams,k.l.;appel,r.d.;hochstrasser,d.f.methodsmolbiol1999,112,531.(6)nguyen,d.p.;garciam.m.;kapadnisp.b.;neumann,n.;chin,j.w.jamchemsoc2009,131,40.(7)nguyen,d.p.;mahesh,m.;elsasser,s.j.;hancock,s.m.;uttamapinant,c.;chin,j.w.jamchemsoc2014,136,2240.本文引用或描述的任何出版物提供了在本申请的申请日之前公开的相关信息。本文中的声明不应被解释为承认发明人无权先于此类披露。上述说明书中提及的所有出版物通过引用并入本文。本发明的各种修改和变化对本领域技术人员是显而易见的,其不脱离本发明的范围和精神。虽然已经结合具体的优选实施例描述了本发明,但是应当理解,所要求保护的本发明不应被不适当地限于这些具体实施例。实际上,对分子生物学和化学或相关领域的技术人员显而易见的用于实施本发明的所述模式的各种修改在所附权利要求的范围内。补充表s1本研究中使用的合成基因的核苷酸序列a.修饰的u6-pylt*表达盒的序列。pylt被加下划线,反密码子标有红色。b.sfgfp(tag)的序列,具有智人密码子优化和c末端多组氨酸标签。位置150处的琥珀密码子被标记为红色。c显示了具有n-末端聚组氨酸标签的黑腹果蝇密码子优化后的人erf1序列。a.u6-pyltu25cb.sfgfp(tag)c.h.sapienserf1补充表s2用于定点诱变的寡核苷酸序列seqidno:1actcttcctttttcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctaaattgtaagcgttaatattttgttaaaattcgcgttaaatttttgttaaatcagctcattttttaaccaataggccgaaatcggcaaaatcccttataaatcaaaagaatagaccgagatagggttgagtgttgttccagtttggaacaagagtccactattaaagaacgtggactccaacgtcaaagggcgaaaaaccgtctatcagggcgatggcccactacgtgaaccatcaccctaatcaagttttttggggtcgaggtgccgtaaagcactaaatcggaaccctaaagggagcccccgatttagagcttgacggggaaagccggcgaacgtggcgagaaaggaagggaagaaagcgaaaggagcgggcgctagggcgctggcaagtgtagcggtcacgctgcgcgtaaccaccacacccgccgcgcttaatgcgccgctacagggcgcgtcccattcgccattcaggctgcgcaactgttgggaagggcgatcggtgcgggcctcttcgctattacgccagctggcgaaagggggatgtgctgcaaggcgattaagttgggtaacgccagggttttcccagtcacgacgttgtaaaacgacggccagtgagcgcgcctcgttcattcacgtttttgaacccgtggaggacgggcagactcgcggtgcaaatgtgttttacagcgtgatggagcagatgaagatgctcgacacgctgcagaacacgcagctagattaaccctagaaagataatcatattgtgacgtacgttaaagataatcatgcgtaaaattgacgcatgtgttttatcggtctgtatatcgaggtttatttattaatttgaatagatattaagttttattatatttacacttacatactaataataaattcaacaaacaatttatttatgtttatttatttattaaaaaaaaacaaaaactcaaaatttcttctataaagtaacaaaacttttatgagggacagcccccccccaaagcccccagggatgtaattacgtccctcccccgctagggggcagcagcgagccgcccggggctccgctccggtccggcgctccccccgcatccccgagccggcagcgtgcggggacagcccgggcacggggaaggtggcacgggatcgctttcctctgaacgcttctcgctgctctttgagcctgcagacacctggggggatacggggaaaaggcctccaaggccactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactagtaaggatctgcgatcgctccggtgcccgtcagtgggcagagcgcacatcgcccacagtccccgagaagttggggggaggggtcggcaattgaacgggtgcctagagaaggtggcgcggggtaaactgggaaagtgatgtcgtgtactggctccgcctttttcccgagggtgggggagaaccgtatataagtgcagtagtcgccgtgaacgttctttttcgcaacgggtttgccgccagaacacagctgaagcttcgaggggctcgcatctctccttcacgcgcccgccgccctacctgaggccgccatccacgccggttgagtcgcgttctgccgcctcccgcctgtggtgcctcctgaactgcgtccgccgtctaggtaagtttaaagctcaggtcgagaccgggcctttgtccggcgctcccttggagcctacctagactcagccggctctccacgctttgcctgaccctgcttgctcaactctacgtctttgtttcgttttctgttctgcgccgttacagatccaagctgtgaccggcgcctactctagagctagcgtttaaacttaagcttgccaccatggactacaaggacgacgacgacaaggacaagaagcccctgaacaccctgatcagcgccacaggactgtggatgtccagaaccggcaccatccacaagatcaagcaccacgaggtgtcccggtccaaaatctacatcgagatggcctgcggcgatcacctggtcgtcaacaacagcagaagcagccggacagccagagccctgcggcaccacaagtacagaaagacctgcaagcggtgcagagtgtccgacgaggacctgaacaagttcctgaccaaggccaacgaggaccagaccagcgtgaaagtgaaggtggtgtccgcccccacccggaccaagaaagccatgcccaagagcgtggccagagcccccaagcccctggaaaacaccgaagccgctcaggcccagcccagcggcagcaagttcagccccgccatccccgtgtctacccaggaaagcgtcagcgtccccgccagcgtgtccaccagcatctctagcatctcaaccggcgccacagcttctgccctggtcaagggcaacaccaaccccatcaccagcatgtctgcccctgtgcaggcctctgccccagccctgaccaagtcccagaccgaccggctggaagtgctcctgaaccccaaggacgagatcagcctgaacagcggcaagcccttccgggagctggaaagcgagctgctgagccggcggaagaaggacctccagcaaatctacgccgaggaacgggagaactacctgggcaagctggaaagagagatcacccggttcttcgtggaccggggcttcctggaaatcaagagccccatcctgatccccctggagtacatcgagcggatgggcatcgacaacgacaccgagctgagcaagcagattttccgggtggacaagaacttctgcctgcggcccatgctggcccccaacctgtacaactacctgcggaaactggatcgcgctctgcccgaccccatcaagattttcgagatcggcccctgctaccggaaagagagcgacggcaaagagcacctggaagagtttacaatgctgaacttttgccagatgggcagcggctgcaccagagagaacctggaatccatcatcaccgactttctgaaccacctggggatcgacttcaagatcgtgggcgacagctgcatggtgtacggcgacaccctggacgtgatgcacggcgacctggaactgtctagcgccgtcgtgggacccatccctctggaccgggagtggggcatcgataagccctggatcggagccggcttcggcctggaacggctgctgaaagtcaagcacgactttaagaacatcaagcgggctgccagaagcgagagctactacaacggcatcagcaccaacctgtgatgataaggatccaaaatgtacagcggccgcgcccctctccctcccccccccctaacgttactggccgaagccgcttggaataaggccggtgtgcgtttgtctatatgttattttccaccatattgccgtcttttggcaatgtgagggcccggaaacctggccctgtcttcttgacgagcattcctaggggtctttcccctctcgccaaaggaatgcaaggtctgttgaatgtcgtgaaggaagcagttcctctggaagcttcttgaagacaaacaacgtctgtagcgaccctttgcaggcagcggaaccccccacctggcgacaggtgcctctgcggccaaaagccacgtgtataagatacacctgcaaaggcggcacaaccccagtgccacgttgtgagttggatagttgtggaaagagtcaaatggctctcctcaagcgtattcaacaaggggctgaaggatgcccagaaggtaccccattgtatgggatctgatctggggcctcggtgcacatgctttacatgtgtttagtcgaggttaaaaaaacgtctaggccccccgaaccacggggacgtggttttcctttgaaaaacacgataatacctccggaatgattgaacaagatggattgcacgcaggttctccggccgcttgggtggagaggctattcggctatgactgggcacaacagacaatcggctgctctgatgccgccgtgttccggctgtcagcgcaggggcgcccggttctttttgtcaagaccgacctgtccggtgccctgaatgaactgcaggacgaggcagcgcggctatcgtggctggccacgacgggcgttccttgcgcagctgtgctcgacgttgtcactgaagcgggaagggactggctgctattgggcgaagtgccggggcaggatctcctgtcatctcaccttgctcctgccgagaaagtatccatcatggctgatgcaatgcggcggctgcatacgcttgatccggctacctgcccattcgaccaccaagcgaaacatcgcatcgagcgagcacgtactcggatggaagccggtcttgtcgatcaggatgatctggacgaagagcatcaggggctcgcgccagccgaactgttcgccaggctcaaggcgcgcatgcccgacggcgaggatctcgtcgtgacccatggcgatgcctgcttgccgaatatcatggtggaaaatggccgcttttctggattcatcgactgtggccggctgggtgtggcggaccgctatcaggacatagcgttggctacccgtgatattgctgaagagcttggcggcgaatgggctgaccgcttcctcgtgctttacggtatcgccgctcccgattcgcagcgcatcgccttctatcgccttcttgacgagttcttctgagtcgacaatcaacctctggattacaaaatttgtgaaagattgactggtattcttaactatgttgctccttttacgctatgtggatacgctgctttaatgcctttgtatcatgttaacttgtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgtatcttatcatgtctggaattgactcaaatgatgtcaattagtctatcagaagctatctggtctcccttccgggggacaagacatccctgtttaatatttaaacagcagtgttcccaaactgggttcttatatcccttgctctggtcaaccaggttgcagggtttcctgtcctcacaggaacgaagtccctaaagaaacagtggcagccaggtttagccccggaattgactggattccttttttagggcccattggtatggctttttccccgtatccccccaggtgtctgcaggctcaaagagcagcgagaagcgttcagaggaaagcgatcccgtgccaccttccccgtgcccgggctgtccccgcacgctgccggctcggggatgcggggggagcgccggaccggagcggagccccgggcggctcgctgctgccccctagcgggggagggacgtaattacatccctgggggctttgggggggggctgtccctgatatctataacaagaaaatatatatataataagttatcacgtaagtagaacatgaaataacaatataattatcgtatgagttaaatcttaaaagtcacgtaaaagataatcatgcgtcattttgactcacgcggtcgttatagttcaaaatcagtgacacttaccgcattgacaagcacgcctcacgggagctccaagcggcgactgagatgtcctaaatgcacagcgacggattcgcgctatttagaaagagagagcaatatttcaagaatgcatgcgtcaattttacgcagactatctttctagggttaatctagctgcatcaggatcatatcgtcgggtcttttttccggctcagtcatcgcccaagctggcgctatctgggcatcggggaggaagaagcccgtgccttttcccgcgaggttgaagcggcatggaaagagtttgccgaggatgactgctgctgcattgacgttgagcgaaaacgcacgtttaccatgatgattcgggaaggtgtggccatgcacgcctttaacggtgaactgttcgttcaggccacctgggataccagttcgtcgcggcttttccggacacagttccggatggtcagcccgaagcgcatcagcaacccgaacaataccggcgacagccggaactgccgtgccggtgtgcagattaatgacagcggtgcggcgctgggatattacgtcagcgaggacgggtatcctggctggatgccgcagaaatggacatggataccccgtgagttacccggcgggcgcgcttggcgtaatcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatacgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatccagtctattaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcgccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatseqidno:2actcttcctttttcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctaaattgtaagcgttaatattttgttaaaattcgcgttaaatttttgttaaatcagctcattttttaaccaataggccgaaatcggcaaaatcccttataaatcaaaagaatagaccgagatagggttgagtgttgttccagtttggaacaagagtccactattaaagaacgtggactccaacgtcaaagggcgaaaaaccgtctatcagggcgatggcccactacgtgaaccatcaccctaatcaagttttttggggtcgaggtgccgtaaagcactaaatcggaaccctaaagggagcccccgatttagagcttgacggggaaagccggcgaacgtggcgagaaaggaagggaagaaagcgaaaggagcgggcgctagggcgctggcaagtgtagcggtcacgctgcgcgtaaccaccacacccgccgcgcttaatgcgccgctacagggcgcgtcccattcgccattcaggctgcgcaactgttgggaagggcgatcggtgcgggcctcttcgctattacgccagctggcgaaagggggatgtgctgcaaggcgattaagttgggtaacgccagggttttcccagtcacgacgttgtaaaacgacggccagtgagcgcgcctcgttcattcacgtttttgaacccgtggaggacgggcagactcgcggtgcaaatgtgttttacagcgtgatggagcagatgaagatgctcgacacgctgcagaacacgcagctagattaaccctagaaagataatcatattgtgacgtacgttaaagataatcatgcgtaaaattgacgcatgtgttttatcggtctgtatatcgaggtttatttattaatttgaatagatattaagttttattatatttacacttacatactaataataaattcaacaaacaatttatttatgtttatttatttattaaaaaaaaacaaaaactcaaaatttcttctataaagtaacaaaacttttatgagggacagcccccccccaaagcccccagggatgtaattacgtccctcccccgctagggggcagcagcgagccgcccggggctccgctccggtccggcgctccccccgcatccccgagccggcagcgtgcggggacagcccgggcacggggaaggtggcacgggatcgctttcctctgaacgcttctcgctgctctttgagcctgcagacacctggggggatacggggaaaaggcctccaaggccactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactaggaaaaaccgcacttgtccggaaaccccgggaatctaacccggctgaacggatttagagtccattcgatctacatgatcaggtttccggtgtttcgtcctttccacaagatatataaagccaagaaatcgaaatactttcaagttacggtaagcatatgatagtccattttaaaacataattttaaaactgcaaactacccaagaaattattactttctacgtcacgtattttgtactaatatctttgtgtttacagtcaaattaattctaattatctctctaacagccttgtatcgtatatgcaaatatgaaggaatcatgggaaataggccctcttcctgcccaactagtaaggatctgcgatcgctccggtgcccgtcagtgggcagagcgcacatcgcccacagtccccgagaagttggggggaggggtcggcaattgaacgggtgcctagagaaggtggcgcggggtaaactgggaaagtgatgtcgtgtactggctccgcctttttcccgagggtgggggagaaccgtatataagtgcagtagtcgccgtgaacgttctttttcgcaacgggtttgccgccagaacacagctgaagcttcgaggggctcgcatctctccttcacgcgcccgccgccctacctgaggccgccatccacgccggttgagtcgcgttctgccgcctcccgcctgtggtgcctcctgaactgcgtccgccgtctaggtaagtttaaagctcaggtcgagaccgggcctttgtccggcgctcccttggagcctacctagactcagccggctctccacgctttgcctgaccctgcttgctcaactctacgtctttgtttcgttttctgttctgcgccgttacagatccaagctgtgaccggcgcctactctagagctagcgaattcgaatttaaatcggatcaccaccatggtgtccaagggcgaggaactgttcaccggcgtggtgcccatcctggtggaactggatggcgacgtgaacggccacaagttctctgtgcggggagagggcgaaggcgacgccacaaatggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccttggcctaccctcgtgaccacactgacctacggcgtgcagtgcttcagcagataccccgaccatatgaagcggcacgacttcttcaagagcgccatgcccgagggctacgtgcaggaacggaccatcagcttcaaggacgacggcacctacaagaccagagccgaagtgaagttcgagggcgacaccctcgtgaaccggatcgagctgaagggcattgatttcaaagaggacggcaacatcctgggccacaagctggagtacaacttcaacagccactaggtgtacatcaccgccgacaagcagaagaacggcatcaaggccaacttcaagatccggcacaacgtggaagatggcagcgtgcagctggccgaccactaccagcagaacacccccatcggagatggccccgtgctgctgcccgacaaccactacctgagcacccagagcgtgctgagcaaggaccccaacgagaagcgggaccacatggtgctgctggaattcgtgaccgccgctggcatcacccacggcatggacgagctgtacaagggcagccaccatcaccatcaccattgataaggatcccagttaccatcgatgatccgcggccgcgcccctctccctcccccccccctaacgttactggccgaagccgcttggaataaggccggtgtgcgtttgtctatatgttattttccaccatattgccgtcttttggcaatgtgagggcccggaaacctggccctgtcttcttgacgagcattcctaggggtctttcccctctcgccaaaggaatgcaaggtctgttgaatgtcgtgaaggaagcagttcctctggaagcttcttgaagacaaacaacgtctgtagcgaccctttgcaggcagcggaaccccccacctggcgacaggtgcctctgcggccaaaagccacgtgtataagatacacctgcaaaggcggcacaaccccagtgccacgttgtgagttggatagttgtggaaagagtcaaatggctctcctcaagcgtattcaacaaggggctgaaggatgcccagaaggtaccccattgtatgggatctgatctggggcctcggtgcacatgctttacatgtgtttagtcgaggttaaaaaaacgtctaggccccccgaaccacggggacgtggttttcctttgaaaaacacgataatacctccggaatgattgaacaagatggattgcacgcaggttctccggccgcttgggtggagaggctattcggctatgactgggcacaacagacaatcggctgctctgatgccgccgtgttccggctgtcagcgcaggggcgcccggttctttttgtcaagaccgacctgtccggtgccctgaatgaactgcaggacgaggcagcgcggctatcgtggctggccacgacgggcgttccttgcgcagctgtgctcgacgttgtcactgaagcgggaagggactggctgctattgggcgaagtgccggggcaggatctcctgtcatctcaccttgctcctgccgagaaagtatccatcatggctgatgcaatgcggcggctgcatacgcttgatccggctacctgcccattcgaccaccaagcgaaacatcgcatcgagcgagcacgtactcggatggaagccggtcttgtcgatcaggatgatctggacgaagagcatcaggggctcgcgccagccgaactgttcgccaggctcaaggcgcgcatgcccgacggcgaggatctcgtcgtgacccatggcgatgcctgcttgccgaatatcatggtggaaaatggccgcttttctggattcatcgactgtggccggctgggtgtggcggaccgctatcaggacatagcgttggctacccgtgatattgctgaagagcttggcggcgaatgggctgaccgcttcctcgtgctttacggtatcgccgctcccgattcgcagcgcatcgccttctatcgccttcttgacgagttcttctgagtcgacaatcaacctctggattacaaaatttgtgaaagattgactggtattcttaactatgttgctccttttacgctatgtggatacgctgctttaatgcctttgtatcatgttaacttgtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgtatcttatcatgtctggaattgactcaaatgatgtcaattagtctatcagaagctatctggtctcccttccgggggacaagacatccctgtttaatatttaaacagcagtgttcccaaactgggttcttatatcccttgctctggtcaaccaggttgcagggtttcctgtcctcacaggaacgaagtccctaaagaaacagtggcagccaggtttagccccggaattgactggattccttttttagggcccattggtatggctttttccccgtatccccccaggtgtctgcaggctcaaagagcagcgagaagcgttcagaggaaagcgatcccgtgccaccttccccgtgcccgggctgtccccgcacgctgccggctcggggatgcggggggagcgccggaccggagcggagccccgggcggctcgctgctgccccctagcgggggagggacgtaattacatccctgggggctttgggggggggctgtccctgatatctataacaagaaaatatatatataataagttatcacgtaagtagaacatgaaataacaatataattatcgtatgagttaaatcttaaaagtcacgtaaaagataatcatgcgtcattttgactcacgcggtcgttatagttcaaaatcagtgacacttaccgcattgacaagcacgcctcacgggagctccaagcggcgactgagatgtcctaaatgcacagcgacggattcgcgctatttagaaagagagagcaatatttcaagaatgcatgcgtcaattttacgcagactatctttctagggttaatctagctgcatcaggatcatatcgtcgggtcttttttccggctcagtcatcgcccaagctggcgctatctgggcatcggggaggaagaagcccgtgccttttcccgcgaggttgaagcggcatggaaagagtttgccgaggatgactgctgctgcattgacgttgagcgaaaacgcacgtttaccatgatgattcgggaaggtgtggccatgcacgcctttaacggtgaactgttcgttcaggccacctgggataccagttcgtcgcggcttttccggacacagttccggatggtcagcccgaagcgcatcagcaacccgaacaataccggcgacagccggaactgccgtgccggtgtgcagattaatgacagcggtgcggcgctgggatattacgtcagcgaggacgggtatcctggctggatgccgcagaaatggacatggataccccgtgagttacccggcgggcgcgcttggcgtaatcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatacgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatccagtctattaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcgccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatseqidno:3atggccgatgatccaagcgccgcagaccgcaatgttgagatctggaagataaaaaagctgataaagagcttggaggccgcacggggcaacggcacgtccatgatatccctcattatcccaccaaaggatcagatcagccgtgtggcgaagatgttggcggatgagttcggaacggccagtaacattaaaagtcgcgtgaaccggctgtcggtcttgggcgccatcacttccgtgcagcaacgtctgaaactgtacaacaaagtcccacccaacggtttggtcgtctactgcggtacgatagttaccgaggaaggaaaggagaagaaagtgaatattgatttcgaaccatttaaaccgataaacactagcttgtacttgtgcgacaataagtttcatacagaggcactcacggccctgctgagcgacgactcgaaattcggattcattgtcattgatggaagtggagcgctgttcggcacgctgcagggtaacacgcgcgaggtcttgcacaaattcaccgtggacttgcccaaaaagcatggccgtggtggccagagcgccctcaggtttgcgcggctgcgcatggagaagcgccataactacgtgcgcaaggtcgcagagacggctgtgcagctgttcatctcgggtgataaggtaaatgtcgcgggactggtgctcgccggcagcgcggacttcaaaaccgagctgagtcagtccgacatgttcgatcagcgtctgcagtcgaaggtactgaagctcgtcgacattagctacggcggcgagaacggcttcaatcaggccatcgaactgagtaccgaagtcctcagtaacgtaaagtttattcaggaaaaaaagttgattggacgctactttgatgaaataagccaagatacgggcaaatactgttttggcgtcgaggatactctgaaagcgctcgagatgggagcagtggaaatactcatcgtatatgaaaatctcgatataatgcgctatgtactgcattgccaaggaacagaagaggagaaaattctctacctcaccccggagcaagagaaggacaagagccattttacagacaaggagacgggccaagagcacgagctcattgagtcgatgcccttgctcgaatggtttgccaacaactacaagaagttcggcgcgaccctggaaattgtcacggataaatcgcaggagggcagccagtttgtgaagggcttcggtggcatcggcggcatcctccgctaccgggtggatttccaaggcatggaatatcaaggtggagatgatgaattcttcgatttggatgattactaatgatag当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1