一种用于筛选弱MdmX抑制剂或测试弱MdmX抑制剂的抑制活性的融合蛋白的制作方法
本发明属于生物技术领域,涉及一种用于筛选MdmX抑制剂或测试Mdmx抑制剂的抑制活性的重组蛋白的制备和应用,特别是种用于筛选弱MdmX抑制剂或测试弱Mdmx抑制剂的抑制活性的重组蛋白的制备和应用,更具体涉及一种p53多肽和MdmX氨基端融合蛋白及其类似物的制备及其在筛选MdmX抑制剂或测试MdmX抑制剂的抑制活性中的应用。
背景技术:
p53是一种抑癌蛋白,在细胞周期阻滞、DNA损伤修复及细胞凋亡等生物过程中发挥作用,并已成为重要的肿瘤治疗靶点。p53的抑制癌变活性受到细胞内过表达MdmX和Mdm2的抑制,与过半数的癌症相关。解除MdmX和Mdm2对p53的抑制是开发新型癌症治疗药物的靶点。
MdmX和Mdm2是p53下游的重要调控基因蛋白,MdmX和Mdm2为同源蛋白,两者结构高度相似,但抑制p53的机理有所不同。目前Mdm2抑制剂的筛选和设计较为成熟,许多已经进入临床研究阶段,例如nutlin-3a改进型分子RG7112即将进入II期临床研究(Ray-Coquard I,Blay J Y,Italiano A,et al.Effect of the MDM2antagonist RG7112 on the P53 pathway in patients with MDM2-amplified,well-differentiated or dedifferentiated liposarcoma:an exploratory proof-of-mechanism study[J].The lancet oncology,2012,13(11):1133-1140)。但是,MdmX抑制剂的有效筛选和设计进展缓慢。由于Mdm2抑制剂的抗药性问题(Lane D P,Cheok C F,Lain S.p53-based cancer therapy[J].Cold Spring Harbor perspectives in biology,2010,2(9):a001222.),癌细胞中的MdmX会进一步过表达,加剧癌细胞的恶化。因此,寻找MdmX抑制剂,及以MdmX为基础而设计的MdmX/Mdm2双抑制剂是治疗近半数癌症的有效药物。
MdmX的N末端存在p53的结合域,MdmX的p53结合域可以和p53蛋白的转录激活结构域结合,形成p53/MdmX复合体,该复合体能够抑制p53的转录活性,但并不介导p53的降解,使p53蛋白失活而诱发肿瘤。
如若一种化合物,能和p53-MdmX复合物竞争性结合,释放p53的转录活性,或以竞争性或不可逆性结合游离的MdmX,则这种化合物可以解除或部分解除MdmX对p53的抑制,使得p53发挥抑癌的作用,因此,这种化合物可视为癌症的治疗或缓解用的药物。
现有技术中公开了一些通过与p53竞争性结合MdmX而测试MdmX抑制剂的技术方案。
中国专利文献CN105936646(公开日:2016.09.14)公开了一类迷你蛋白及其应用。所述迷你蛋白以白蛋白结合域的3α螺旋束为骨架蛋白,并且在所述骨架蛋白的三维结构表面、N端、C端中的至少一处引入有阳离子性氨基酸,并经所述阳离子性氨基酸至少连接p53与MDM2/MDMX结合的关键氨基酸位点。该迷你蛋白兼具入胞以及与白蛋白、MDM2/MDMX结合的能力,并可良好抑制p53和Mdm2/MdmX之间的相互作用,可用于制备抗癌药物等,可用于规模化生产和应用。小蛋白与Mdm2/MdmX的结合用的荧光偏振检测。
中国专利文献CN103923067(公开日:2014.07.16)公开了一种MdmX/Mdm2的小分子抑制剂,同时还涉及一种MdmX/Mdm2的小分子抑制剂的制备方法,该小分子抑制剂化合物能抑制MdmX蛋白质和p53蛋白的相互作用,也能抑制Mdm2蛋白质和p53蛋白的相互作用,该方法中MdmX抑制剂筛选采用荧光偏振法测定。
在上述检测MdmX的抑制剂的模型中存在一些不足:
1.p53片段与MdmX片段分别加入测试体系,二者的摩尔比就很难精确地确定,就会带来较大的系统误差或随机误差。
2.方案中采用荧光素标记p53片段,这就会使每次标记操作中,荧光素分子与p53片段分子的摩尔比不完全一致,就会带来较大的随机误差,就更难于进行精确地定量研究。
3.方案中采用荧光素标记p53片段,而荧光素在可见光下容易分解变性而失去荧光功能,这就要求相应操作避光,光致分解会降低检测方法的准确性,要求避光会增加操作的难度和实验成本。
4.由于荧光素的疏水性,降低检测方法和数据的准确性。
5.p53片段和MdmX需要分别制备,会增加操作的复杂度。
6.尽管MdmX与Mdm2为同源蛋白,两者三维结构高度相似,但现有Mdm2小分子抑制剂对MdmX的抑制效果甚弱。
7.对于作用力大小明显不同的MdmX抑制剂均用同一个模型来测试,无法区分强MdmX抑制剂于弱MdmX抑制剂,更重要的是,作用力较弱的MdmX抑制剂可能由于不能有效地竞争性结合p53结构域而没有检测到。
技术实现要素:
蛋白质在270~300nm吸收波长范围内有光吸收现象,这种光吸收性质主要是来自蛋白质中的色氨酸、酪氨酸和苯丙氨酸,所以含有该类氨基酸的蛋白质具有天然荧光性质。当蛋白质中没有色氨酸时,其最大发射波长约在304nm处,而且其荧光光谱的位置不受大分子构象的影响;当蛋白质中存在色氨酸时,它的荧光发射最大波长与色氨酸所处的疏水环境程度有明显关系,其荧光最大发射波长在308~355nm范围内(Vivian,J.T.Callis,P.R.,Mechanisms of tryptophan fluorescence shifts in proteins.HYPERLINK"https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1301402/"Biophys J.2001,80(5):2093–2109),当色氨酸完全埋藏在疏水腔中时,荧光发射波长在321nm左右,完全暴露于水相时,荧光发射波长在350nm左右。
已经解析的MdmX(23~111序列)与p53(15~29序列)所形成的复合物晶体结构(PDB ID:3DAB,参见文献:Popowicz G,Czarna A,Holak T.Structure of the human Mdmx protein bound to the p53tumor suppressor transactivation domain[J].Cell Cycle,2008,7(15):2441-3.)表明,p53的转录激活区(15~29序列)以α-螺旋形式通过3个关键氨基酸(Phe19、Trp23及Leu26)与MdmX的N-端疏水区域相互作用。MdmX上的3个关键作用位点连同其邻近的氨基酸一起组成三个结合口袋,即F19口袋、W23口袋及L26口袋。
在本文将人源MdmX的N-端结构域(23~111序列)对应的结构域称为p53结合结构域(定义为N-MdmX),将人源p53的15~29序列称对应的结构域为MdmX结合结构域(定义为p53p)。
为解决至少上述技术问题,本发明提供一种用于筛选MdmX抑制剂或测试MdmX抑制剂的抑制活性的融合蛋白。其核心构思如下文所述。
将p53p与N-MdmX通过一段连接臂氨基酸序列(定义为XX)连接,形成融合蛋白,该融合蛋白中p53p片段的色氨酸Trp23(此处的23代表该色氨酸来源于野生型p53蛋白的第23位,而不代表在融合蛋白中的位置,下同),既是与N-MdmX结合的关键氨基酸,又是内源性荧光探针。(,其荧光发射最大波长将反映p53p片段与N-MdmX的结合能力。由于该色氨酸深埋于N-MdmX结合口袋疏水腔中(Popowicz G,Czarna A,Holak T.Structure of the human Mdmx protein bound to the p53tumor suppressor transactivation domain[J].Cell Cycle,2008,7(15):2441-3.),其荧光发射最大波长在321nm。当MdmX抑制剂竞争性地与该融合蛋白中的MdmX部分的p53结合结构域相结合后,部分或全部p53p将与N-MdmX分离开,此时色氨酸Trp23完全暴露于水相,在321nm处的荧光信号强度将随抑制剂浓度增大而发生显著变化,并存在一定的数学关联性,同时荧光发射波长也向350nm方向红移。
因此,在添加待测MdmX抑制剂前后测量融合蛋白体系中321nm发射光的强度变化,就可计算出MdmX抑制剂对MdmX的抑制活性/抑制程度的大小,进而,该融合蛋白起到了筛选MdmX抑制剂的作用或测试MdmX抑制剂的抑制活性的作用。
对照现有技术,该融合蛋白至少具有如下一些优点:
MdmX的p53结合结构域与p53中的MdmX结合结构域由于存在于同一个肽链上,它们的摩尔数是相等的,相当于与MdmX抑制剂竞争的p53部分与MdmX的相对含量是固定的,这将减少批次操作间的随机误差。这种1:1的模式也能够更准确的反映筛选的配体与p53p部分竞争性结合MdmX蛋白表面疏水腔的能力。
该融合蛋白不需要标记荧光探针,就不会因为荧光标记的程度不同带来的误差。融合蛋白中的色氨酸也不会见光分解,而且色氨酸与融合蛋白的摩尔比是固定不变的,且p53p与MdmX结合的色氨酸处于结合或游离状态时相对应色氨酸的发射光谱不同。因此,不需要避光设备,而且上述融合蛋白中,游离色氨酸Trp23与游离的p53部分和与MdmX部分结合的MdmX抑制剂(首先假设MdmX抑制剂与MdmX结合的摩尔比是1:1)是等摩尔的,结合的色氨酸Trp23与p53部分与MdmX部分的复合物是等摩尔的,对应的发射光是321nm左右,也就是说加入MdmX抑制剂前后321nm发射光的强度的变化情况与MdmX抑制剂竞争性结合的程度是对应的,相对现有技术,有更小的系统误差或随机误差。
为了实现上述目的,该用于筛选MdmX抑制剂或测试MdmX抑制剂的抑制活性的融合蛋白的核心技术方案为:
所述融合蛋白包括测试序列、结合序列和连接臂序列;
所述测试序列为MdmX序列或包含MdmX序列中的p53结合结构域的MdmX片段;
所述结合序列为p53p序列、包含p53序列中的MdmX结合结构域的p53片段或保持与MdmX有结合活性的包含p53序列中的MdmX结合结构域的p53片段的突变体;
所述连接臂序列为一个肽段,所述连接臂的上下游分别与所述测试序列和所述结合序列之一连接;所述连接臂的氨基酸序列不参与所述融合蛋白的二级结构的形成或维持;所述连接臂的长度和柔性足以使所述测试序列的空间结构的任一表面部分与所述结合序列的空间结构的任一表面部分在空间上有机会相接触。
可选地,所述融合蛋白的上游或下游具有用于蛋白分离和/或纯化的标签序列。
进一步地,由于人源p53(GenBank:AB082923.1)中有四个色氨酸,其中仅有一个色氨酸落入其MdmX结合结构域;人源MdmX基因(GeneBank:AF007111.1)中有七个色氨酸,均未落入p53结合结构域。进一步的技术方案是,将p53的MdmX结构域与MdmX的p53结构域通过连接臂连接,且连接臂中不含有色氨酸。这样,整个融合蛋白中仅有唯一色氨酸残基,该色氨酸的荧光光谱随着色氨酸的环境而变化。具体地就是当p53结合结构域与MdmX结合结构域相结合的时候,融合蛋白中的唯一色氨酸残基藏在疏水腔中,荧光发射波长在321nm左右。当MdmX抑制剂与MdmX中的p53结合结构域结合后,就将部分或全部融合蛋白中的唯一色氨酸残基暴露于水相,荧光发射波长在350nm左右。此种情况下,在添加待测MdmX抑制剂前后测量融合蛋白体系中321nm发射光的强度,就避免了不随MdmX抑制剂结合而变化发光特性的色氨酸对色氨酸发光光谱的影响,也就可以使实验测试结果更加灵敏。
具体地,为了上述目的,该技术方案为:
该融合蛋白用式p53p-XX-N-MdmX表示,其特征在于,
p53p代表所述结合序列SEQ ID NO.1,具体为:SQETFSDLWKLLPEN;
XX代表所述连接臂序列SEQ ID NO.2,具体为:GSGSSENLYFQ;
N-MdmX代表所述测试序列SEQ ID NO.3,具体为:GSQINQVRPKLPLLKILHAAGAQGEMFTVKEVMHYLGQYIMVKQLYDQQEQHMVYCGGDLLGELLGRQSFSVKDPSPLYDMLRKNLVTLAT;
p53p-XX-N-MdmX融合蛋白中,氨基酸的一级结构表明,其序列中只存在一个色氨酸,且它还是对应于p53多肽和N-MdmX相互作用的关键氨基酸,可以根据色氨酸的内源荧光特征,来筛选和p53p-XX-N-MdmX融合蛋白中与p53多肽竞争性结合MdmX的小分子化合物。
为了融合蛋白表达后纯化的方便,可在融合蛋白上游融合组氨酸标签序列,融合了组氨酸标签的融合蛋白序列可为:
GSSHHHHHHGSSQETFSDLWKLLPENGSGSSENLYFQGSQINQVRPKLPLLKILHAAGAQGEMFTVKEVMHYLGQYIMVKQLYDQQEQHMVYCGGDLLGELLGRQSFSVKDPSPLYDMLRKNLVTLAT
不同的MdmX抑制剂与MdmX的结合能力会有不同,对于结合能力较强的MdmX抑制剂,其可以充分地与p53部分竞争结合MdmX部分。而对于结合能力较弱的MdmX抑制剂,其可能不足以发生可检测的竞争性结合的程度。因此上述融合蛋白就不能有效地检测该类MdmX抑制剂。
为了进一步使上述融合蛋白能够检测弱结合力的MdmX抑制剂,一种方案是通过突变MdmX结合结构域,使MdmX结合结构域与p53结合结构域之间的结合力变弱,例如将与MdmX结合的p53p关键氨基酸之一Phe19或Leu26(该19或26代表相应p53p中氨基酸在野生型中的位置,下同)突变,使MdmX结合结构域与p53结合结构域的结合变弱,以便使结合力更弱的MdmX抑制剂得以与MdmX结合结构域竞争性结合p53结合结构域,使融合蛋白能够用于筛选结合力较弱的MdmX抑制剂或测试结合力较弱的MdmX抑制剂的抑制活性。利用这两个弱化的模型可进一步筛选亲和力在微摩尔浓度范围内的小分子化合物。
具体地,该技术方案为:
p53p代表的所述结合序列突变为:SQETASDLWKLLPEN。
p53p代表的所述结合序列突变为:SQETFSDLWKLAPEN。
本发明还提供了一种核苷酸序列,所述核苷酸序列能够表达如上各个方案所述的用于筛选MdmX抑制剂或测试MdmX抑制剂的抑制活性的融合蛋白。
可选地,上述核苷酸序列为SEQ ID NO.04(用于表达p53p-XX-N-MdmX的碱基序列)、SEQ ID NO.05(用于表达p53pF19A-XX-N-MdmX的碱基序列)或SEQ ID NO.06(用于表达p53pL26A-XX-N-MdmX的碱基序列)所示的序列。
本发明还提供了一种用于筛选MdmX抑制剂或测试MdmX抑制剂的抑制活性的融合蛋白的表达载体,所述表达载体能够借助宿主表达如上各个方案所述的用于筛选MdmX抑制剂或测试MdmX抑制剂的抑制活性的融合蛋白。
可选地,所述载体是pET28a载体,所述宿主是大肠杆菌BL21(DE3)。
本发明还提供了一种如上各个方案所述的的用于筛选MdmX抑制剂或测试Mdmx抑制剂的抑制活性的融合蛋白在筛选MdmX抑制剂或测试Mdmx抑制剂的抑制活性的用途,其中,所述MdmX抑制剂是能够与P53竞争性结合MdmX的抑制剂。
更为具体地,nutlin-3a作为一种公认的较强的Mdm2抑制剂,对MdmX也具有一定的抑制作用,nutlin-3a与MdmX和Mdm2的Kd值分别为28μM、0.7μM(Laurie N A,Donovan S L,Shih C S,et al.Inactivation of the p53pathway in retinoblastoma.[J].Nature,2006,444(7115):61-6)。将nutlin-3a作为鉴定p53p-XX-N-MdmX融合蛋白模型有效性的代表性小分子是合理的。
除此之外,用商业可以获取的32个Mdm2小分子抑制剂对这一模型进行进一步鉴定。
为了实现上述的目的,本发明采用了以下技术措施:
本发明利用分子克隆构建了p53p-XX-N-MdmX、p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX融合蛋白表达质粒,并利用大肠杆菌工程菌BL21(DE3)表达、纯化制备。利用F-7000荧光分光光度计分析nutlin-3a及32个Mdm2抑制剂与p53p-XX-N-MdmX融合蛋白的竞争性结合能力以此来验证以此模型来筛选抑制剂的可行性与有效性。之后将关键氨基酸Phe19和Leu26分别突变为Ala构建了p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX两个融合蛋白筛选模型,并利用蛋白质变性剂盐酸胍验证了突变后融合蛋白的稳定性,结果也证明将关键氨基酸突变后p53p与MdmX氨基端的结合能力减弱,之后,通过PerkinElmer的LS45/55型荧光/磷光/发光分光光度计运用三种模型同时筛选nutlin-3a和32个Mdm2抑制剂,筛选结果显示,改进过的模型对筛选亲和力低的抑制剂是更加有效的。
附图说明
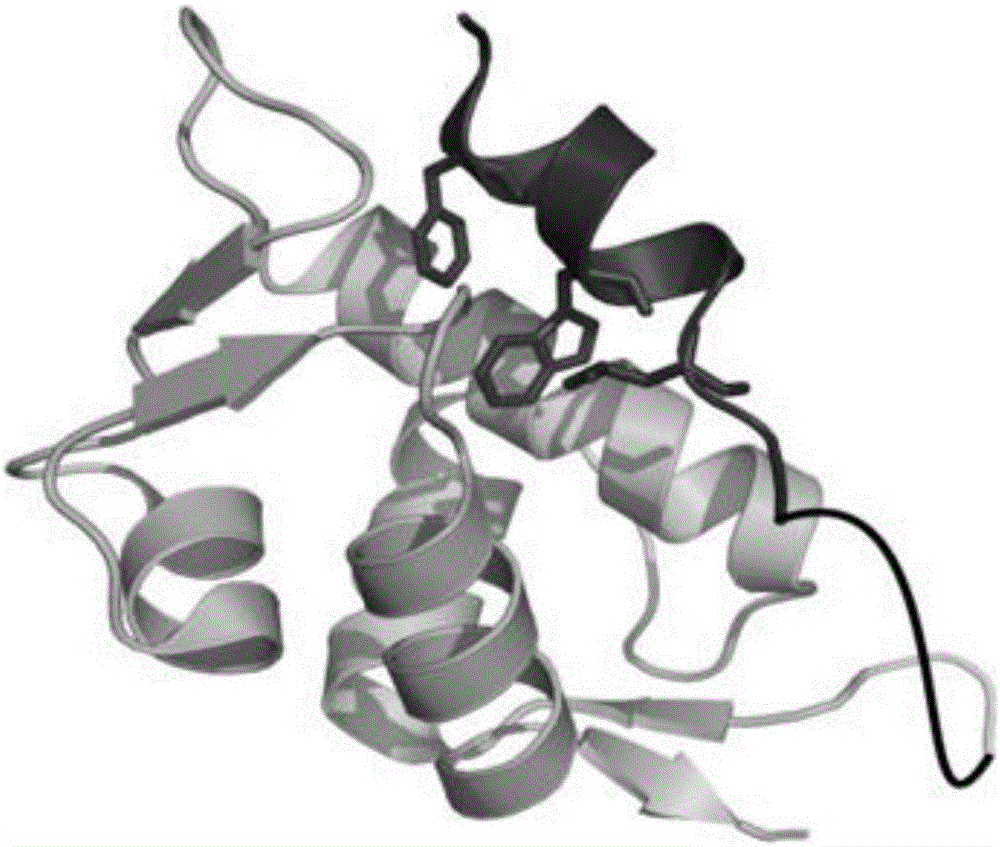
图1为p53p-XX-N-MdmX融合蛋白三级结构图。
图2为p53p-XX-N-MdmX融合蛋白表达质粒构建三步PCR反应条件。
图3为p53p-XX-N-MdmX融合蛋白纯化过程中样品通过Ni-NTA色谱柱纯化后SDS-PAGE胶图(a),通过凝胶色谱柱(Supredex 16/60075pg)进一步纯化后AKTA图谱(b)和SDS-PAGE胶图(c)。
图4为p53p-XX-N-MdmX融合蛋白在λEX为278nm时,λEM在290nm~500nm范围内的荧光光谱曲线(a)和在λEM为321nm时,λEX在245nm~300nm的激发光光谱曲线(b)。
图5为nutlin-3a滴定浓度为OD280nm=0.1的p53p-XX-N-MdmX融合蛋白时,λEX在278nm,随着nutlin-3a浓度增加,λEM为290nm~500nm范围内的荧光强度变化曲线(a)和λEM在321nm处荧光强度峰值趋势的拟合曲线(b)。
图6为nutlin-3a对p53p-XX-N-MdmX融合蛋白的竞争性结合模式图。
图7为λEX在278nm时,nutlin-3a(a)和p53p(b)在λEM在290nm~500nm范围内的荧光发射光谱曲线。
图8为DMSO(无nutlin-3a)滴定浓度为OD280nm=0.1的p53p-XX-N-MdmX融合蛋白时,λEX=278nm时,随着DMSO浓度增加,λEM为290nm~500nm范围内的荧光强度变化曲线(a)和λEM在321nm处荧光强度峰值的拟合曲线(b)。
图9为以nutlin-3a作为对照,对比nutlin-3a和32个Mdm2小分子抑制剂对p53p-XX-N-MdmX融合蛋白的竞争抑制常数Ki值。
图10为p53pF19A-XX-N-MdmX(a)和p53pL26A-XX-N-MdmX(b)融合蛋白三级结构模拟图。
图11为p53p-XX-N-MdmX融合蛋白突变体表达质粒构建三步PCR反应条件。
图12为p53pF19A-XX-N-MdmX融合蛋白分子克隆过程中DNA胶图。
图13为λEX在278nm时,随着盐酸胍浓度的增加,p53p-XX-N-MdmX(a)、p53pF19A-XX-N-MdmX(b)和p53pL26A-XX-N-MdmX(c)融合蛋白的荧光发射光谱曲线在λEM在290nm~500nm的变化趋势。
图14为随着盐酸胍浓度增加,三种融合蛋白分别在λmax为321nm、331nm和330nm处荧光强度峰值变化趋势图。
图15为以p53p-XX-N-MdmX融合蛋白为对照,用p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX融合蛋白模型筛选32个Mdm2小分子抑制剂和nutlin-3a来表明突变后融合蛋白对小分子结合能力的变化。
具体实施方式
为了更好的解释本发明的技术方案,下面结合附图详细介绍本发明的各个实施例。以下实施例用于进一步说明本发明,但不应理解为对本发明的固定或限制。若未特别指明,实施例中所用的技术特征可以替换为具有在不背离发明构思前提下等同或相似功能或效果的其他本领域已知的技术特征,本发明的不同模块的任意组合均落入本发明的保护范围。
实施例1:p53p-XX-N-MdmX融合蛋白表达质粒构建
以已经公布的p53的MdmX结合结构域p53p与MdmX氨基端的p53结合结构域的晶体结构(PDB ID:3DAB)为基础(参考Structure of the human Mdmx protein bound to the p53tumor suppressor transactivation domain,Popowicz,G.M.,Czarna,A.,Holak,T.A.(2008)CellCycle 7:2441-2443),我们建立了以下p53p-XX-N-MdmX融合蛋白模型。
p53p-XX-N-MdmX融合蛋白的氨基酸序列如下所示(除去标签GSSHHHHHHGS后蛋白质的三级结构运用PymolWin模拟得模拟图如图1所示):
GSSHHHHHHGSSQETFSDLWKLLPENGSGSSENLYFQGSQINQVRPKLPLLKILHAAGAQGEMFTVKEVMHYLGQYIMVKQLYDQQEQHMVYCGGDLLGELLGRQSFSVKDPSPLYDMLRKNLVTLAT
其中,SQETFSDLWKLLPEN来自p53的MdmX结合结构域序列,GSQINQVRPKLPLLKILHAAGAQGEMFTVKEVMHYLGQYIMVKQLYDQQEQHMVYCGGDLLGELLGRQSFSVKDPSPLYDMLRKNLVTLAT来自MdmX的p53结合结构域序列,GSGSSENLYFQ是连接臂氨基酸序列。GSSHHHHHHGS是有助于融合蛋白分离纯化的标签序列,其中两端的GSS或GS是柔性的氨基酸片段。
为了能够通过分子克隆构建以上氨基酸序列,根据大肠杆菌BL21(DE3)密码子偏好性合成了N-MdmX的DNA序列,其核苷酸编码序列如SEQ ID NO.4所示。
将该核苷酸通过Nco I和EcoR I双酶切,连接到pET28a质粒(Nco I和EcoR I双酶切)上,通过分子克隆流程,得到了含有N-MdmX的编码序列的重组质粒,命名为pET28a-N-MdmX。
为了构建和表达以上融合蛋白,根据已构建的pET28a-N-MdmX质粒,设计了以下引物序列:
Primer1:5'-CAT GCC ATG GGC AGC AGC CAT CAC CAT CAT CAC CAC GGC AGC-3'
Primer2:5'-GTT TCC ACA GAT CGC TAA AGG TTT CCT GGC TGC TGC CGT GGT GAT GAT G-3'
Primer3:5'-TTA GCG ATC TGT GGA AAC TGC TGC CGG AAA ATG GCA GCG GCA GCA GCG A-3'
Primer4:5'-CGG GAT CCC TGA AAA TAC AGG TTT TCG CTG CTG CCG CTG CCA-3'
以设计好的引物按照以下体系进行片段扩增。
PCR体系1:
PCR1反应条件如图2-a所示;
PCR体系2:
PCR2反应条件如图2-b所示;
PCR体系3:
PCR3反应条件如图2-c所示;
PCR3产物试剂盒回收,回收产物用Nco Ⅰ和BamH Ⅰ双酶切,作为插入片段。
PCR回收产物酶切体系:
37℃,3h,酶切产物试剂盒(OMEGA,Plasmid Mini Kit I)回收。
上述三个PCR反应中,PCR1和PCR2反应体系中不需要模版,两个引物碱基互补配对延伸,形成两段核苷酸序列,不用回收产物。以PCR1和PCR2产物为模版,进行融合PCR实验,为PCR3实验。进而得到了含有组氨酸标签、p53p和XX融合核苷酸序列,通过酶切后,连接到pET28a-N-MdmX质粒上。
将pET28a-N-MdmX质粒用Nco Ⅰ和BamH Ⅰ双酶切,作为载体。
质粒载体酶切体系:
37℃,3h,酶切产物切胶试剂盒(康为世纪,Gel Extraction Kit)回收。
连接反应按载体DNA与插入片段DNA摩尔比为1:3比例混合,加入与体系相同的高效DNA连接酶(TOYOBO,Ligation high Ver.2),16℃,连接3h。
将全部连接产物转入50μl DH5α(top10)大肠杆菌中,冰浴30min,42℃热激90s后立即放回冰浴,2min,加入200μl LB液体培养基活化,37℃,200rpm振荡培养1h复苏后再涂含有卡那霉素的LB固体培养基平板,37℃倒置培养12~16h,观察菌落生长情况。
有单菌落存在,进行菌液PCR鉴定,条带正确,提质粒pET28a-p53p-XX-N-MdmX,质粒酶切鉴定,条带正确,送质粒测序,测序结果正确,保藏菌种待用。
pET28a-p53p-XX-N-MdmX测序结果核苷酸序列如SEQ ID NO.4所示。
实施例2:p53p-XX-N-MdmX融合蛋白表达与纯化
将pET28a-p53p-XX-N-MdmX质粒化转入大肠杆菌BL21(DE3);挑单菌落到2ml LB K+(Kanamycin)培养基、37℃、200rpm过夜培养;取500μl菌液转入50ml LB K+培养基、37℃、200rpm培养3h;将50ml菌液全转入1L LB K+培养基、37℃、200rpm培养,直到菌体浓度达到OD280nm=0.8左右,加入IPTG(终浓度0.4mM)诱导,约20h收集菌体(收集菌体时离心机参数设置为3500rpm、30min、4℃)。
超声波细胞破碎机破碎细胞。按照菌体体积:缓冲液体积=1:5的比例,加入buffeA缓冲液(50mM Na2HPO4、200mM NaCl、10mM咪唑、1mM BME(β-巯基乙醇)、pH8.0),参数设置为:总时间2min;超声时间2s;间隙时间4s;温度报警24℃;超声功率40%(宁波新芝生物科技股份有限公司的JY92-IIN超声波细胞粉碎机)。重复2~3次,直到菌体破碎完全。将细胞破碎液用50ml离心管分装平衡,离心,30min、4℃、18000rpm,将上清、沉淀分别收集于试剂瓶(管)中。
将破碎上清通过AKTA pure过Ni-NTA色谱柱(图3a)和凝胶色谱柱(Supredex 16/60075pg或Superdex 26/60075pg,图3b,c),收集纯化的蛋白,浓缩,分装,每支200μl,液氮速冻,冻存于-80℃冰箱中。
实施例3:p53p-XX-N-MdmX融合蛋白模型荧光光谱分析
取一支冻存的200μl的p53p-XX-N-MdmX蛋白,快速解冻,用磷酸盐缓冲液稀释蛋白浓度至OD280nm=0.1。取400μl至石英比色皿中,在F-7000荧光分光光度计上进行荧光扫描。根据色氨酸的内源荧光特征,取λEX为278nm,扫描λEM在290nm~500nm范围内的荧光强度,发现最大荧光强度在321nm(图4a)。故定λEM为321nm,扫描λEX为245nm~300nm的荧光激发光谱曲线,结果如图4b所示,也可知在λEX在278nm时,荧光强度最大,故定激发光波长为278nm。
实施例4:利用p53p-XX-N-MdmX融合蛋白荧光法测定nutlin-3a与MdmX亲和力
取冻存于-80℃的p53p-XX-N-MdmX蛋白,快速解冻,用磷酸盐缓冲液稀释至蛋白浓度为OD280nm=0.1,取400μl至石英比色皿中,在F-7000荧光分光光度计上取λEX为278nm,λEM在290nm~500nm范围进行荧光扫描。按照如下表1中nutlin-3a(母液2.5mM,溶于DMSO)浓度梯度进行滴定(每加入一次nutlin-3a都要混合均匀)。
表1 nutlin-3a滴定p53p-XX-N-MdmX融合蛋白浓度梯度
Nutlin-3a对p53p-XX-N-MdmX融合蛋白的竞争性结合模式图,如图6所示。
如图5a所示为不同nutlin-3a浓度对融合蛋白荧光光谱的影响,随着nutlin-3a滴定浓度的增加,在最大发射波长321nm处的荧光强度发生显著变化。在图5b中以nutlin-3a为0μM的荧光强度峰值为标准,随着nutlin-3a浓度变化,在321nm的荧光强度值变化趋势图,对趋势曲线按照如下公式进行拟合,拟合结果可获取nutlin-3a对N-MdmX的抑制常数(Ki值,竞争结合解离常数),拟合结果如图5b所示。
拟合公式为:fi=I/[I+Ki(1+LT/Kd)](参考Chen Y,Prusoff W H.Relationship between the inhibition constant and the concentration of an inhibitor that cause a 50%inhibition of an enzyme reaction[J].Biochem Pharmacol,1973,22:3099-3108.);其中,fi为抑制剂对N-MdmX竞争的部分抑制程度(为荧光变化相对强度,具体单位为百分比,见下具体说明)。I为竞争性抑制中增加的抑制剂浓度(μM);Ki为竞争性解离常数,即抑制剂与p53p竞争性结合N-MdmX的亲和解离常数;LT为融合蛋白中p53p的浓度(μM);Kd为解离常数,即p53p与N-MdmX的亲和解离常数。
而抑制剂对N-MdmX竞争抑制程度fi可以通过荧光的衰减值来反映,即fi=(A1-y)/(A1-A2),其中A1为拟合曲线上荧光强度最大值,A2为拟合曲线上荧光强度最小值,y为拟合曲线上的任一点,整合之后的公式为:Y=A1-X(A1-A2)/[X+Ki(1+LT/Kd)]。
从图5a可以明显的判断随着nutlin-3a的浓度增大,图中存在两个明显的峰形波动,λEM为321nm和λEM为350nm~450nm,其中λEM为321nm处的是荧光强度逐渐降低的峰,以上已经清晰地分析,该峰的变化是色氨酸的发射荧光的强度变化。而在350nm~450nm范围内存在是一个荧光强度逐渐增高的侧峰,通过单独分别对小分子nutlin-3a和p53p(合成的含十五个氨基酸多肽:SQETFSDLWKLLPEN)在磷酸盐体系(无p53p-XX-N-MdmX,其它条件一致)中荧光强度峰值的验证,判断350nm~450nm范围内处的侧峰出现原因是由nutlin-3a本身的荧光引起还是与p53p竞争性结合后p53p游离引起。
与nutlin-3a滴定p53p-XX-N-MdmX相同的测试条件,取λEX为278nm,λEM为290~500nm,结果如图7a(nutlin-3a)和7b(p53p)所示。图7a所示,λmax为375nm,图7b所示,λmax为350nm,可以确定,在荧光发射波长为350nm~450nm处的侧峰是随着nutlin-3a的滴定,nutlin-3a引入的荧光强度,故在本实验测试和计算结果中是可以忽略不考虑的。
取400μl用磷酸盐缓冲液稀释到OD280nm=0.1的p53p-XX-N-MdmX蛋白做无nutlin-3a的空白对照实验,除了无nutlin-3a,其他实验条件与上述完全一致。实验结果如图8a(荧光强度变化)和图8b(荧光强度峰值随着浓度变化)所示。
Nutlin-3a滴定结果说明,nutlin-3a和p53p对MdmX的结合位点存在竞争性结合关系,才使得色氨酸在321nm处的发射荧光强度峰值降低明显。本发明将进一步用32个已知结合能力的小分子化合物作进一步验证。
实施例5:利用p53p-XX-N-MdmX融合蛋白荧光法和极化荧光法(FP)筛选小分子化合物库
(1)测试所用抑制剂:
小分子化合物库包含32个商业可以获取的Mdm2小分子抑制剂。其结构可参见表2。
表2小分子化合物库中的化合物结构及其两种Ki测量结果
(2)p53p-XX-N-MdmX融合蛋白荧光筛选法:
参照nutlin-3a滴定方案(细节参见实施例4),用磷酸盐缓冲液将p53p-XX-N-MdmX蛋白稀释至OD280nm=0.1,取400μl置于石英比色皿。将32个小分子每个按照如下表3中浓度梯度滴定(32个小分子母液浓度均是2.5mM,溶于DMSO)。
表3 32个小分子滴定p53p-XX-N-MdmX融合蛋白浓度梯度
滴定后得到的随着小分子浓度增加,在λEM为321nm处荧光强度峰值随着浓度变化的Ki值(小分子解离常数)按照公式进行线性拟合。具体各个分子的Ki值参见表2第三列。
将32个小分子的拟合Ki值和nutlin-3a的拟合Ki值进行汇总比较,然后比较它们的竞争结合能力和nutlin-3a的对比。对比结果如图9所示。
(2)p53p和N-MdmX极化荧光(FP)筛选法:
将p53p多肽(15~29位氨基酸)通过荧光素标记(fluorescein-GSGSSQETFSDLWKLLPEN,Flu-p53p)。
将pET28a-N-MdmX质粒化转入BL21(DE3),0.4mM IPTG诱导表达并纯化(表达纯化方法参照实施例2),液氮速冻,冻存于-80℃冰箱。
FP实验的缓冲液为PBS(137mM NaCl;2.7mM KCl;10mM Na2HPO4;2mM KH2PO4;pH7.4)、0.025%Tween-20;0.5%BSA;pH7.5。
将32个小分子化合物和nutlin-3a以DMSO稀释为终浓度为2mM。
取3μl以DMSO稀释的小分子化合物和nutlin-3a,加入47μl的缓冲液稀释小分子的终浓度为0.12mM。用移液枪取20μl小分子化合物按照标记好的顺序置于96孔板,每个小分子做一次重复,共66孔。以未加小分子化合物的同样条件的DMSO作为对照,准备4孔。
取N-MdmX蛋白快速解冻,14000rpm,10min,4℃,离心。
准备溶液1包含15nM Flu-p53p多肽和OD280nm=0.1的N-MdmX蛋白溶液,溶液2包含15nM的Flu-p53p多肽溶液。
将溶液1加入有小分子化合物的66孔和未加小分子化合物的对照孔中其中两孔(作为阴性对照),每孔40μl;将溶液2加入对照孔中的2孔中(作为阳性对照),每孔40μl。
将96孔板200g离心2min,在暗处室温混合30分钟。
使用PerkinElmer的EnVision多标记微孔板检测仪检测,取激发光为555nm,荧光为632nm检测。检测结果分析公式为:抑制率%=(阴性对照-样品值)/(阴性对照-阳性对照)(Zhang Q,Lu H.Identification of small molecules affecting p53-MDM2/MDMX interaction by fluorescence polarization[J].p53Protocols,2013:95-111.),结果如表2第四列所示。
以下是对图9和表2所反映的结果的分析。以Cpd20、Cpd23、Cpd28、Cpd31为例,这四个化合物是以1,4-苯二氮-2,5-(1H,4H)-二酮为骨架设计的一类化合物(以该骨架设计了一类Mdm2抑制剂,Novel 1,4-benzodiazepine-2,5-diones as Hdm2antagonists with improved cellular activity),而通过FP测得的它们与MdmX的Ki值分别为6.32μM、10.33μM、3.58μM、10.63μM,其结果与以p53p-XX-N-MdmX融合蛋白来筛选这四个化合物拟合的Ki值分别为5.90μM、9.79μM、2.07μM、8.29μM基本一致。
以Cpd4和CPd6为例,从图上可以看出Cpd4和Cpd6的Ki值差别较大,通过FP测得的它们与MdmX的Ki值分别为1.85μM和67.3μM,而通过p53p-XX-N-MdmX融合蛋白的荧光测定法Ki分别为1.49μM和51.73μM,这说明本发明的荧光方法与现有技术中的FP方法所测得的Ki值也是相符的。
表2所示结果的比较说明,以nutlin-3a和32个小分子的滴定实验结果虽然存在着一定误差,但用所建立的这种p53p-XX-N-MdmX融合蛋白模型来筛选合适的小分子抑制剂与现有技术中的FP方法所测结果也是基本符合的,说明该融合蛋白筛选系统是有效的。
实施例6:p53p-XX-N-MdmX融合蛋白突变体的质粒构建
p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX是p53p-XX-N-MdmX融合蛋白的突变体模型,它们是在p53p-XX-N-MdmX融合蛋白基础上分别把p53p肽段上的Phe19突变为Ala19、Leu26突变为Ala26(19和26指相应氨基酸在野生型p53多肽中的位置)。将结合在三个结合口袋的三个关键氨基酸残基中的两个氨基酸分别进行突变,来分别减弱p53p多肽段和MdmX N端结构域的结合能力,为筛选出结合能力较弱的新型小分子药物骨架提供基础。
两种突变体模型依据p53p-XX-N-MdmX融合蛋白模型建立如下:
p53pF19A-XX-N-MdmX氨基酸序列如下所示(三级结构运用PymolWin模拟得模拟图如图10a所示,未包括GSSHHHHHHGS):
GSSHHHHHHGSSQETASDLWKLLPENGSGSSENLYFQGSQINQVRPKLPLLKILHAAGAQGEMFTVKEVMHYLGQYIMVKQLYDQQEQHMVYCGGDLLGELLGRQSFSVKDPSPLYDMLRKNLVTLAT
p53pL26A-XX-N-MdmX氨基酸序列如下所示(三级结构运用PymolWin模拟得模拟图如图10b所示,未包括GSSHHHHHHGS):
GSSHHHHHHGSSQETFSDLWKLAPENGSGSSENLYFQGSQINQVRPKLPLLKILHAAGAQGEMFTVKEVMHYLGQYIMVKQLYDQQEQHMVYCGGDLLGELLGRQSFSVKDPSPLYDMLRKNLVTLAT
为了构建以上融合蛋白模型,根据已有质粒pET28a-p53p-XX-N-MdmX,设计了以下引物序列:
F19A-forward:5’-GGC AGC AGC CAG GAA ACC GCT AGC GAT CTG TGG AAA-3’
F19A-reverse:5’-TTT CCA CAG ATC GCT AGC GGT TTC CTG GCT GCT GCC-3’
L26A-forward:5’-AGC GAT CTG TGG AAA CTG GCG CCG GAA AAT GGC AGC-3’
L26A-reverse:5’-GCT GCC ATT TTC CGG CGC CAG TTT CCA CAG ATC GCT-3’
p53pF19A-XX-N-MdmX融合蛋白构建:
PCR体系1:
PCR1反应条件如图11a所示;
DNA胶图如图12a所示;
PCR体系2:
PCR2反应条件如图11b所示;
DNA胶图如图12b所示;
PCR体系3:
PCR3反应条件如图11c所示;
DNA胶图如图12c所示;
PCR产物回收,回收产物用Nco I和EcoR I双酶切,作为插入片段。
这里是融合PCR。PCR1和PCR2获得两段核苷酸序列,PCR3是融合PCR,只对单个氨基酸突变。
PCR回收产物酶切体系:
37℃,3h,酶切产物试剂盒回收。
将pET28a-N-MdmX质粒用Nco Ⅰ和EcoR Ⅰ双酶切,切胶回收,作为载体。
连接反应按载体DNA与插入片段DNA摩尔比为1:3比例混合,加入与体系相同的高效连接酶(TOYOBO,Ligation high Ver.2),16℃,连接3h。
将全部连接产物转入50μl DH5α(top10)大肠杆菌中,冰浴30min,42℃热激90s后立即放回冰浴,2min,加入200μl LB液体培养基活化,37℃,200rpm振荡培养1h复苏后再涂含有卡那霉素的LB固体培养基平板,37℃倒置培养12~16h,观察菌落生长情况。抽提pET28a-p53pF19A-XX-N-MdmX质粒,测序,测序结果正确,保藏菌种。
p53pF19A-XX--N-MdmX与p53pL26A-XX-N-MdmX的分子克隆过程除PCR过程中引物序列外,其他条件完全一致。
p53pF19A-XX-N-MdmX的核酸编码序列如SEQ ID NO.5所示,p53pL26A-XX-N-MdmX的核酸编码序列如SEQ ID NO.6所示。
实施例7:p53pF19A-XX-N-MdmX融合蛋白和p53pL26A-XX-N-MdmX融合蛋白的表达与纯化
将pET28a-p53pF19A-XX-N-MdmX和pET28a-p53pL26A-XX-N-MdmX质粒分别化学转化入大肠杆菌BL21(DE3);挑单菌落到2ml LB K+培养基,37℃、200rpm过夜培养;取500μl菌液转入50ml LB K+培养基,37℃、200rpm培养3h;将50ml菌液全转入1L LB K+培养基,37℃、200rpm培养,直到菌体浓度达到OD280=0.8左右,加入IPTG(终浓度0.4mM)诱导,约20h收菌(菌体收集时离心机参数设置为3500rpm、30min、4℃)。
超声波细胞破碎机破碎细胞。按照菌体体积:缓冲液体积=1:5的比例,加入buffeA缓冲液(50mM Na2HPO4、200mM NaCl、10mM咪唑、1mM BME、PH 8.0),参数设置为:总时间2min;超声时间2s;间隙时间4s;温度报警24℃;超声功率40%。重复2~3次(观察菌体的破碎后的粘稠程度来确定破碎次数),直到菌体破碎完全。将细胞破碎液用50ml离心管分装平衡,离心,30min、4℃、18000rpm,将上清、沉淀分别收集于试剂瓶(管)中。
将上清通过AKTA pure过Ni-NTA色谱柱和Gel-filtration(sSupredex 16/60075pg或Superdex 26/60075pg),收集纯化的蛋白,浓缩,分装,每支200μl,液氮速冻,冻存于-80℃冰箱。
实施例8:验证p53p-XX-N-MdmX、p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX三者的稳定性差异性
在室温下,3~4mol/L盐酸胍可使可使蛋白质从天然状态转变至变性状态,通常增加变性剂浓度可以提高变性程度,通常6mol/L盐酸胍可以使蛋白质完全转变为变性状态。
相较于p53p-XX-N-MdmX融合蛋白,p53pF19A-XX-N-MdmX融合蛋白和p53pL26A-XX-N-MdmX融合蛋白是在p53p-XX-N-MdmX的基础上分别把p53p多肽段上的Phe19改变为Ala19、Leu26改变为Ala26。将结合在三个结合口袋的三个关键氨基酸残基中的两个氨基酸分别进行突变,来分别减弱p53p和MdmX N端结构域的结合能力,使融合蛋白的稳定性减弱,以此来筛选竞争结合能力弱的新型骨架结构的小分子抑制剂。
用超纯水配制浓度为8M的盐酸胍溶液100ml。
取4支200μl/支的p53p-XX-N-MdmX(OD280=1.0),3支200μl/支的p53pF19A-XX-N-MdmX(OD280=1.38),2支200μl/支的p53pL26A-XX-N-MdmX(OD280=2.0)融合蛋白快速解冻,混合,14000rpm,10min,离心。
按照以下表4、表5和表6比例,分别配置不同盐酸胍浓度下的p53p-XX-N-MdmX、p53pF19A-XX-N-MdmX和p53pL26A–XX-N-MdmX融合蛋白溶液。
表4 p53p-XX-N-MdmX融合蛋白在0~4M盐酸胍浓度下的溶液
表5 p53pF19A-N-MdmX融合蛋白在0~4M盐酸胍浓度下的溶液
表6 p53pL26A–N-MdmX融合蛋白在0~4M盐酸胍浓度下的溶液
按照盐酸胍浓度梯度,取400μl样品到石英比色皿中,将比色皿放入F-7000荧光分光光度计,取λEX为278nm,λEM取290nm~500nm,根据盐酸胍浓度变化,p53p-XX-N-MdmX、p53pF19A-XX-N-MdmX和p53pL26A–XX-N-MdmX得到荧光强度变化图如图13a,b和c所示。
根据荧光强度峰值的变化,将三种融合蛋白的荧光强度峰值随着盐酸胍浓度变化的结果整理如图14所示:
根据图13a,13b和13c所示,荧光发射波长的峰值分别为p53p-XX-N-MdmX:λEM=321nm、p53pF19A-XX-N-MdmX:λEM=331nm和p53pL26A-XX-N-MdmX:λEM=330nm,根据荧光发射波长的峰值可以判断p53p上的色氨酸结合在N-MdmX的疏水腔中的深浅顺序依次为p53p-XX-N-MdmX、p53pL26A-XX-N-MdmX、p53pF19A-XX-N-MdmX,即可以初步判断融合蛋白的稳定性的强弱依次为:p53p-XX-N-MdmX、p53pL26A-XX-N-MdmX和p53pF19A-XX-N-MdmX。
图14所示,三种融合蛋白的荧光强度峰值随着蛋白质变性剂盐酸胍的浓度变化图可以看出,分析荧光强度峰值IC50发现,荧光强度峰值降低一半时,所需盐酸胍的浓度大小依次为:p53p-XX-N-MdmX、p53pL26A-XX-N-MdmX、p53pF19A-XX-N-MdmX,即可以更进一步的判断融合蛋白的稳定性的强弱依次为:p53p-XX-N-MdmX、p53pL26A-XX-N-MdmX和p53pF19A-XX-N-MdmX。
结果说明p53pL26A-XX-N-MdmX和p53pF19A-XX-N-MdmX比p53p-XX-N-MdmX结构更不稳定,说明前者两个结构域之间的亲和力更弱,进而前者更容易被亲和力较弱的MdmX抑制剂所竞争性结合。
这表明用p53pL26A-XX-N-MdmX和p53pF19A-XX-N-MdmX作为筛选模型筛选亲和力弱的小分子理论上是可行的。
实施例9:用上述32个小分子和nutlin-3a对p53p-XX-N-MdmX、p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX进行滴定,来验证结合能力的变化。
融合蛋白快速解冻,14000rpm,10min,离心,弃去因为变性而沉淀的蛋白。
小分子母液浓度10mM,取1μl小分子母液加9μl DMSO稀释为1mM。
按照小分子的终浓度为10μM,蛋白浓度为OD280nm=0.5,将溶液按照这个浓度加入96孔黑色微孔板(以加入相同体积的DMSO作为空白对照),在4℃冰箱混合2h。用PerkinElmer LS-45/55荧光/磷光/发光分光光度计读取96孔板荧光数据,先取λEX为278nm,p53p-XX-N-MdmXλEM为321nm,p53pF19A-XX-N-MdmXλEM为331nm,p53pL26A-XX-N-MdmXλEM为330nm,激发光栅15nm,荧光光栅20nm,阈值(cut off)290nm,可以读取到激发光为278nm时,三种融合蛋白的荧光强度值。再以λEX为278nm,p53p-XX-N-MdmX、p53pF19A-XXN-MdmX、p53pL26A-N-MdmXλEM为375nm,阈值(cut off)350nm可以分别读取到激发光为278nm时,三种融合蛋白的荧光强度值。每隔4h测一次,每次重复两遍。
96孔板最多一次可以同时测试32个抑制剂和nutlin-3a对三种融合蛋白的抑制效果(可做一组平行实验)。
以p53p-XX-N-MdmX融合蛋白为例,在λEM为321nm,截断波长为290nm时读取的荧光强度为加入小分子后的荧光强度值,在λEM为375nm,截断波长为350nm时读取的荧光强度为nutlin-3a(或者其他化合物)所引入的荧光强度值,两次读取荧光强度值的差值即为小分子加入后的色氨酸的荧光强度变化值,以加入相同体积的DMSO后的荧光强度值作为空白对照,加入小分子后的色氨酸的荧光强度变化值与空白对照之间的差值即为加入小分子化合物后与p53p竞争性结合引起的色氨酸的游离程度,负值越大,说明色氨酸游离程度越大,即该小分子的结合能力更强,定此差值为纵坐标,如若为正值,则说明色氨酸结合增强,即可能存在着相互作用使N-MdmX结构域更加稳定,p53p和N-MdmX的亲和力增强。以小分子编号为横坐标,本实验顺次为1#-32#以及nutlin-3a,整理结果如图15所示。
图15结果表明,对于单个小分子来说p53p-XX-N-MdmX的结果负值小于p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX的负值,这说明同一种小分子在p53p-XX-N-MdmX中竞争p53部分的能力弱于p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX,这说明p53p-XX-N-MdmX中p53结合结构域与MdmX结合结构域的结合强度更大,结果符合本发明的设计目的。
同时,同一种抑制剂分子对p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX中N-MdmX部分的竞争性结合能力更强,意味着p53pF19A-XX-N-MdmX和p53pL26A-XX-N-MdmX可以适用于与N-MdmX部分结合力更弱的小分子抑制剂的筛选或测试。这进一步说明结果符合本发明的设计目的。而且图15中p53p-XX-N-MdmX融合蛋白筛选模型所表现的与32个小分子抑制剂的亲和力与图8所反映的也是基本相吻合的,更进一步的说明了该模型的有效性。
以上各个实施例只是用于进一步说明本发明,并不是用来限制本发明的保护范围,凡是基于本发明的构思所作出的等同变换及对本发明的各个技术方案显而易见的改动,均落入本发明的保护范围。
SEQUENCE LISTING
<110> 湖北工业大学
<120> 一种用于筛选弱MdmX抑制剂或测试弱MdmX抑制剂的抑制活性的融合蛋白
<130> 2016-12-07
<160> 6
<170> PatentIn version 3.5
<210> 1
<211> 15
<212> PRT
<213> 人工序列
<400> 1
Ser Gln Glu Thr Phe Ser Asp Leu Trp Lys Leu Leu Pro Glu Asn
1 5 10 15
<210> 2
<211> 11
<212> PRT
<213> 人工序列
<400> 2
Gly Ser Gly Ser Ser Glu Asn Leu Tyr Phe Gln
1 5 10
<210> 3
<211> 91
<212> PRT
<213> 人工序列
<400> 3
Gly Ser Gln Ile Asn Gln Val Arg Pro Lys Leu Pro Leu Leu Lys Ile
1 5 10 15
Leu His Ala Ala Gly Ala Gln Gly Glu Met Phe Thr Val Lys Glu Val
20 25 30
Met His Tyr Leu Gly Gln Tyr Ile Met Val Lys Gln Leu Tyr Asp Gln
35 40 45
Gln Glu Gln His Met Val Tyr Cys Gly Gly Asp Leu Leu Gly Glu Leu
50 55 60
Leu Gly Arg Gln Ser Phe Ser Val Lys Asp Pro Ser Pro Leu Tyr Asp
65 70 75 80
Met Leu Arg Lys Asn Leu Val Thr Leu Ala Thr
85 90
<210> 4
<211> 384
<212> DNA
<213> 人工序列
<400> 4
ggcagcagcc atcaccatca tcaccacggc agcagccagg aaacctttag cgatctgtgg 60
aaactgctgc cggaaaatgg cagcggcagc agcgaaaacc tgtattttca gggatcccag 120
attaaccagg tgcgtccgaa actgccgctg ctgaaaattc tgcatgcggc gggcgcgcag 180
ggcgaaatgt ttaccgtgaa agaagtgatg cattatctgg gccagtatat tatggtgaaa 240
cagctgtatg atcagcagga acagcacatg gtgtattgcg gcggcgatct gctgggcgaa 300
ctgctgggcc gtcagagctt tagcgtgaaa gatccgagcc cgctgtatga tatgctgcgt 360
aaaaacctgg tgaccctggc gacc 384
<210> 5
<211> 384
<212> DNA
<213> 人工序列
<400> 5
ggcagcagcc atcaccatca tcaccacggc agcagccagg aaaccgctag cgatctgtgg 60
aaactgctgc cggaaaatgg cagcggcagc agcgaaaacc tgtattttca gggatcccag 120
attaaccagg tgcgtccgaa actgccgctg ctgaaaattc tgcatgcggc gggcgcgcag 180
ggcgaaatgt ttaccgtgaa agaagtgatg cattatctgg gccagtatat tatggtgaaa 240
cagctgtatg atcagcagga acagcacatg gtgtattgcg gcggcgatct gctgggcgaa 300
ctgctgggcc gtcagagctt tagcgtgaaa gatccgagcc cgctgtatga tatgctgcgt 360
aaaaacctgg tgaccctggc gacc 384
<210> 6
<211> 384
<212> DNA
<213> 人工序列
<400> 6
ggcagcagcc atcaccatca tcaccacggc agcagccagg aaacctttag cgatctgtgg 60
aaactggcgc cggaaaatgg cagcggcagc agcgaaaacc tgtattttca gggatcccag 120
attaaccagg tgcgtccgaa actgccgctg ctgaaaattc tgcatgcggc gggcgcgcag 180
ggcgaaatgt ttaccgtgaa agaagtgatg cattatctgg gccagtatat tatggtgaaa 240
cagctgtatg atcagcagga acagcacatg gtgtattgcg gcggcgatct gctgggcgaa 300
ctgctgggcc gtcagagctt tagcgtgaaa gatccgagcc cgctgtatga tatgctgcgt 360
aaaaacctgg tgaccctggc gacc 384
- 还没有人留言评论。精彩留言会获得点赞!