一种AsCpf1突变体蛋白、编码基因、重组表达载体及其制备方法与应用与流程
本发明属于生物化学与分子生物学技术领域,具体涉及一种ascpf1突变体蛋白、编码基因、重组表达载体及其制备方法与应用。
背景技术:
基因编辑是在基因组水平上进行精确修饰的一种技术,可完成基因定点删除突变、基因定点插入突变、多位点同时突变和小片段的删失等。基因编辑技术可用于基因功能及疾病发病机理研究、构建疾病动物模型、生物治疗、遗传和肿瘤相关疾病研究、整合病毒疾病研究和改良农畜牧物种。crispr-cas9是继“锌指核酸内切酶(zfn)”、“类转录激活因子效应物核酸酶(talen)”之后出现的第三代基因组定点编辑技术。
ascpf1(acidaminococcμssp.cpf1)是一种rna介导的v型crispr-cas9系统内切酶,在单链crrna(crisprrna)存在的情况下,ascpf1识别双链dna的5’-tttn-3’pam(protospaceradjacentmotif),并在双链dna的与crrna互补链的第18个碱基和非互补链的第23个碱基处对dna进行切割。与crispr-cas9基因编辑系统相比,crispr-ascpf1系统具有不需要tracrrna(trans-activatingcrrna)、切割靶向dna效率高、切割产生粘性末端等特点,是潜在的、可用于真核生物基因组编辑的工具。
pd-1(programmeddeath1),程序性死亡受体,是一种重要的免疫抑制分子,为cd28超家族成员,最初是从凋亡的小鼠t细胞杂交瘤2b4.11克隆出来。作为癌症免疫治疗的重要靶点,以基因编辑t细胞的pd-1基因为靶点的免疫调节对抗肿瘤、抗感染、抗自身免疫性疾病及器官移植存活等均有重要的意义。
技术实现要素:
本发明的目的之一是提供一种ascpf1突变体蛋白,可用于编辑t细胞的pd-1基因。
为实现上述目的,本发明提供的ascpf1突变体蛋白,其氨基酸序列如seqidno:1所示。
本发明的目的之二是提供一种重组ascpf1基因突变体,对ascpf1基因进行结构改造,可用于编码上述ascpf1突变体蛋白。
为实现上述目的,本发明提供的一种重组ascpf1基因突变体ascpf1(s589q/p711t),其核苷酸序列如seqidno:2所示。ascpf1(s589q/p711t)是对ascpf1基因进行密码子优化,并将野生型ascpf1中的两个氨基酸位点(s598q和p711t)突变得到。
本发明的目的之三是提供一种含重组ascpf1基因突变体的重组表达载体。
为实现上述目的,本发明提供的重组表达载体pet15b-ascpf1(s589q/p711t),是以seqidno:2所示的ascpf1(s589q/p711t)为模板设计引物进行pcr扩增,再将纯化的pcr产物用无缝克隆方法插入pet15b载体的多克隆位点ncoi和xhoi之间得到。
本发明的目的之四是提供ascpf1突变体蛋白的制备方法,得到高纯度的ascpf1突变体蛋白。
为实现上述目的,本发明提供的ascpf1突变体蛋白的制备方法,包括如下步骤:
①编码ascpf1突变体蛋白原核表达载体的构建:以合成的ascpf1(s589q/p711t)基因组dna为模板,用pcr的方法扩增ascpf1(s589q/p711t)编码序列,将得到的pcr产物进行纯化,用无缝克隆方法插入pet15b载体的多克隆位点ncoi和xhoi之间,得到重组表达载体,经测序验证阅读框及dna序列全部正确,即成功构建了含重组ascpf1基因突变体的原核表达载体,命名为pet15b-ascpf1(s589q/p711t);
②ascpf1突变体蛋白的表达:将原核表达载体pet15b-ascpf(s589q/p711t)转入表达宿主菌escherichiacolibl21(de3),挑取单克隆接种至新鲜的含50mg/l氨苄青霉素的lb培养基,经37℃摇床培养至od600为0.6,加入终浓度为0.6mm的iptg诱导ascpf1(s589q/p711t)的表达;
③ascpf1突变体蛋白的纯化:收集一升发酵液的细胞,并用40ml冰浴的buffera重悬,冰浴超声裂解细胞,离心取上清,上清通过ni-ida柱;上样完毕用50mlbuffere洗涤ni-ida柱;50mlbufferg洗脱ni-ida柱,分管收集;将洗脱到的目的蛋白过cm离子柱;100mlbufferd洗涤cm离子柱;10mlbufferb洗脱cm离子柱,分管收集;使用专业分析软件grab-it2.5分析tat-as-cpf1蛋白纯度;洗脱的ascpf1(s589q/p711t)合并后将蛋白透析至20mmtris,50mmnaclph7.5,20%glycerol中;其中,buffera的组成为:20mmtris-hcl,50mmnacl,1%triton-100,20%glycerolph7.5;buffere的组成为:20mmtris-hclph7.5,2mnacl,0.1%tritonx-100,20%glycerol;bufferg的组成为:20mmtris-hclph7.5,50mmnacl,0.1%tritonx-100,500mm咪唑,20%glycerol;bufferd的组成为:20mmtris-hclph7.5,50mmnacl,0.1%tritonx-100,20%glycerol;bufferb的组成为:20mmtris-hclph7.5,50mmnacl,0.1%tritonx-100,20%glycerol。
本发明通过载体构建将密码子优化的ascpf1(s589q/p711t)基因克隆到pet15b载体中,在低温条件下ascpf1(s589q/p711t)在大肠杆菌中蛋白表达量可以达到18%,可溶性达到95%。
本发明的目的之五是提供ascpf1突变体蛋白、重组ascpf1基因突变体和重组表达载体在基因编辑原代t细胞pd-1方面的应用。
本发明的目的之六是提供ascpf1突变体蛋白编辑原代t细胞pd-1的方法,能更有效、便捷地编辑原代t细胞pd-1的基因。
具体包括以下步骤:
(1)原代t细胞pd-1基因部分序列扩增
以提取的原代t细胞pd-1基因组dna为模板,pcr扩增并回收扩增片段,pcr扩增所用正向引物为:5’-actccccagacaggccctg-3’(seqidno:6),反向引物为:5’-caggggctggccggtgcg-3’(seqidno:7);pcr反应体系为:基因组dna模板100ng,20μm正向引物1μl,20μm反向引物1μl,2xultrapfumix25μl,加ddh2o至总体积50μl;pcr反应条件为:94℃热变性5分钟,56℃退火30秒,72℃延伸1分钟,共进行30个循环;
(2)ascpf1突变体蛋白体外切割pd-1
取1μg步骤(1)回收的pd-1产物、1μl纯化的ascpf1突变体蛋白、1μg合成的crrna、2μl10*reactionbuffer,配制成20μl体外切割反应体系,37℃水浴30min后,2%琼脂糖凝胶电泳检测切割;其中10*reactionbμffer的组成为:500mmkac、200mmtris-醋酸、100mmmg(ac)2、1mg/mlbsaph7.9@25℃;
(3)westernblotting检测ascpf1突变体蛋白处理原代t细胞后,pd-1表达水平变化
取1×106个原代t细胞pd-1,加入20μg纯化的ascpf1突变体蛋白,10μg合成的crrna混匀冰浴,电转化仪点击转化后铺6孔板,72h后提取蛋白,westernblotting检测pd-1蛋白表达水平变化。
本发明纯化得到的ascpf1突变体蛋白切割效率高,切割不需要复杂的tracrrna,操作方便,节约成本;发现了两个新的pd-1基因编辑位点;利用ascpf1突变体蛋白能够实现原代t细胞pd-1的敲降,为基因编辑t细胞pd-1基因及癌症免疫治疗打下基础。
附图说明
图1是重组表达载体pet15b-ascpf1(s589q/p711t)的质粒图谱;
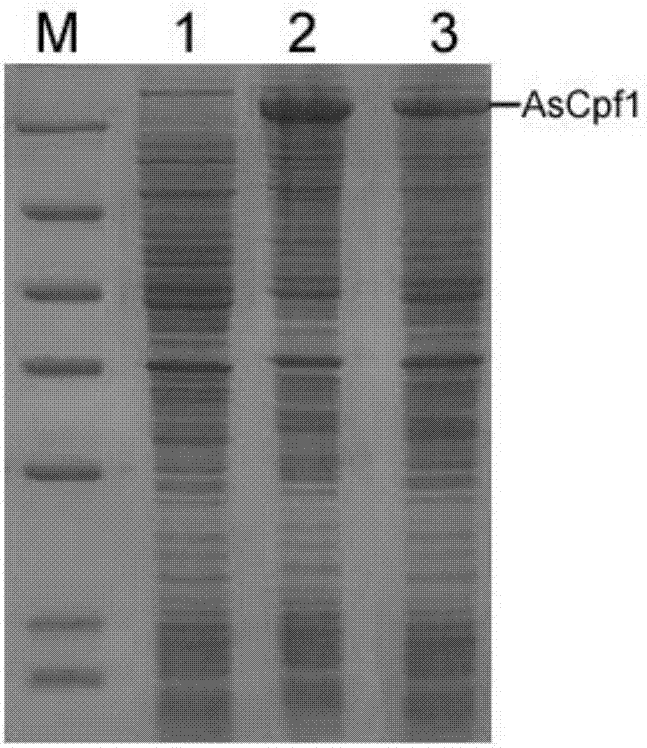
图2是重组ascpf1(s589q/p711t)蛋白表达的sds-page分析:泳道m为蛋白marker(kd),泳道1为iptg诱导前,泳道2和3为iptg诱导后ascpf1(s589q/p711t)的全菌蛋白表达;
图3是ni-ida柱纯化后的ascpf1(s589q/p711t)的sds-page(13%)分析:泳道1:超声上清;泳道2:流出;泳道3:50mm咪唑洗脱;泳道4:500mm咪唑洗脱1;泳道5:500mm咪唑洗脱2;泳道6:残留;泳道m为蛋白marker(kd);
图4是cm柱纯化后的ascpf1(s589q/p711t)的sds-page(13%)分析:泳道1:ni-ida纯化ascpf1(s589q/p711t);泳道2:cm流出;泳道3:cm洗脱1;泳道4:cm洗脱2;泳道5:cm洗脱3;泳道6:残留;泳道m为蛋白marker(kd);
图5是纯化的ascpf1(s589q/p711t)与野生型ascpf1(wt)体外切割活性对比(体外切割贪铜菌copr基因片段):
泳道m:dl2000;泳道1:copr+ascpf1(wt);泳道2:copr+crrna;泳道3:copr+ascpf1(wt)+crrna;泳道4:copr+ascpf1(s589q)+crrna;泳道5:copr+ascpf1(p117t)+crrna;泳道6:copr+ascpf1(s589q/p711t)+crrna;
图6是纯化的ascpf1(s589q/p711t)体外切割pd-1:泳道m为dnamarker(dl2000);泳道1:pd-1+ascpf1(s589q/p711t);泳道2:pd-1+ascpf1(s589q/p711t)+crrna1;泳道3:pd-1+ascpf1(s589q/p711t)+crrna2;泳道4:pd-1+ascpf1(s589q/p711t)+crrna3;
图7是ascpf1(s589q/p711t)处理原代t细胞后,westernblotting检测pd-1表达水平:
泳道1:con组1(原代t细胞不经任何处理);泳道2:con组2(原代t细胞+电击处理);泳道3:con组3(原代t细胞+ascpf1(s589q/p711t)+电击处理);泳道4:crrna1(原代t细胞+ascpf1(s589q/p711t)+crrna1+电击处理);泳道5:crrna2(原代t细胞+ascpf1(s589q/p711t)+crrna2+电击处理);泳道6:crrna3(原代t细胞+ascpf1(s589q/p711t)+crrna3+电击处理)。
具体实施方式
下面结合附图及具体实施例对发明作进一步详细描述。
下述实例中所用的实验方法如无特殊说明,均为常规方法。
下述实例中所用的材料、试剂等,如无特殊说明,均可从商业途径获得。
实施例1:ascpf1的优化设计和合成
首先对野生型ascpf1的序列进行以下优化:
5’端加上穿膜肽tat编码序列,编码序列为tacggtcgtaaaaaacgtcgtcagcgtcgtcgt(seqidno:3)。
3’端加上核定位序列(nls),编码序列为:aaaaggccggcggccacgaaaaaggccggccaggcaaaaaagaaaaagggatcc(seqidno:4)。
3’端加上6*his标签序列,编码序列为:catcatcatcatcatcac(seqidno:5)。
再将野生型ascpf1中的两个氨基酸位点突变:s598q和p711t。
密码子优化后ascpf1基因进行全基因合成,委托上海吉玛制药技术有限公司合成,命名为ascpf1(s589q/p711t)。合成的ascpf1(s589q/p711t)的核苷酸序列如seqidno:2所示。
实施例2:原核表达载体pet15b-ascpf1(s589q/p711t)的构建
以合成的ascpf1(s589q/p711t)基因组dna为模板,用pcr的方法扩增ascpf1(s589q/p711t)编码序列,设计的正向引物为:
5’-gtttaactttaagaaggagatataccatgtacggtcgtaaaaaacgtcgtcagcgtcgtcgtacacagttcgagggctttac-3’(seqidno:6)
反向引物为:
5’-tcgggctttgttagcagccggatcctcgagttagtgatgatgatgatgatgg-3’(seqidno:7)
pcr反应体系为:合成的ascpf1(s589q/p711t)模板50ng,正向引物(20μm)1μl,反向引物(20μm)1μl,2xultrapfumix25μl,加ddh2o至总体积50μl;
pcr扩增条件为:94℃热变性5分钟,60℃退火30秒,72℃延伸5分钟,共进行30个循环。
将得到的pcr产物进行纯化,用无缝克隆方法插入pet15b载体(购自novagen公司)的多克隆位点ncoi和xhoi之间,得到重组表达载体。对重组表达载体进行dna测序,分析其阅读框和编码序列的正确性。
将密码子优化的ascpf1(s589q/p711t)基因克隆至pet15b载体中,重组质粒经生工生物工程(上海)股份有限公司测序验证,阅读框及dna序列全部正确,即成功构建了含重组ascpf1基因突变体的原核表达载体,命名为pet15b-ascpf1(s589q/p711t),其简图参见图1。
实施例3:ascpf1突变体蛋白的表达及纯化
(1)ascpf1突变体蛋白的表达
分别将原核表达载体pet15b-ascpf(s589q/p711t)和对照载体pet15b转入表达宿主菌escherichiacolibl21(de3)(购自newenglandbiolabs公司),挑取单克隆接种至新鲜的lb培养基(含50mg/l氨苄青霉素)。待细菌长至od600为0.6左右,分别加入终浓度为0.6mm的iptg(异丙基-β-d-硫代半乳糖苷)诱导ascpf1(s589q/p711t)的表达。诱导两个小时后收集菌体,处理后的菌体蛋白样品经13%sds-page电泳,使用凝胶成像系统uvpwhite/ultraviolettransilluminator(购自美国upland公司)对考马斯亮蓝染色的凝胶进行扫描记录,采用专业分析软件grab-it2.5和gelwork分析目的蛋白占菌体总蛋白的比例。
本申请中,可溶性定义为:可溶性的目的蛋白/总的目的蛋白×100%。在iptg诱导后,出现了一条约为155kd的蛋白条带,和理论分子量大小吻合,经凝胶扫描灰度分析,ascpf1(s589q/p711t)蛋白约占菌体总蛋白的18%(图2),可溶性达到95%。
(2)ascpf1突变体蛋白的纯化
收集一升发酵液的细胞,并用40ml冰浴的buffera(20mmtris-hcl,50mmnacl,1%triton-100,20%glycerolph7.5)重悬,冰浴超声裂解细胞,离心取上清,上清通过ni-ida柱;上样完毕用50mlbuffere(20mmtris-hclph7.5,2mnacl,0.1%tritonx-100,20%glycerol)洗涤ni-ida柱;50mlbufferg(20mmtris-hclph7.5,50mmnacl,0.1%tritonx-100,500mm咪唑,20%glycerol)洗脱ni-ida柱,分管收集;将洗脱到的目的蛋白过cm离子柱;100mlbufferd(20mmtris-hclph7.5,50mmnacl,0.1%tritonx-100,20%glycerol)洗涤cm离子柱;10mlbufferb(20mmtris-hclph7.5,50mmnacl,0.1%tritonx-100,20%glycerol)洗脱cm离子柱,分管收集。使用专业分析软件grab-it2.5分析tat-as-cpf1蛋白纯度为90%。洗脱的ascpf1(s589q/p711t)合并后将蛋白透析至20mmtris,50mmnaclph7.5,20%glycerol中。
ni-ida柱纯化后的ascpf1(s589q/p711t)的sds-page(13%)分析如图3所示;cm柱纯化后的ascpf1(s589q/p711t)的sds-page(13%)分析如图4所示。
实施例4:纯化的ascpf1突变体蛋白与野生型ascpf1(wt)切割活性对比
(1)copr基因序列扩增
采用常规方法从天然沥青中提取贪铜菌,并以提取的贪铜菌基因组dna为模板,pcr扩增并回收copr基因片段,pcr扩增所用正向引物为:
5’-atgaaattgctggtagtcgaaga-3’(seqidno:8),反向引物为:5’-tcagtcgccttcctccggat-3’(seqidno:9)。pcr扩增体系为:基因组dna模板100ng,正向引物(20μm)1μl,反向引物(20μm)1μl,2xultrapfumix25μl,加ddh2o至总体积50μl;pcr反应条件为:94℃热变性10分钟,58℃退火30秒,72℃延伸1分钟,共进行35个循环。pcr扩增产物回收,用于ascpf1(s589q/p711t)和野生型ascpf1(wt)体外切割实验。
(2)切割活性对比
取1μg回收的copr产物、1μl纯化的ascpf1突变体蛋白或者ascpf1(wt)、1μg合成的crrna、2μl10*reactionbuffer,配制成20μl体外切割反应体系,37℃水浴30min后,2%琼脂糖凝胶电泳检测切割;其中10*reactionbuffer的组成为:500mmkac、200mmtris-醋酸、100mmmg(ac)2、1mg/mlbsaph7.9@25℃。
其中crrna委托上海吉玛制药技术有限公司合成,其序列为:
5’-cgcgggccagcagttcggcaaaggatctacaacagtagaaattccctatagtgagtcgtattaatttc-3’(seqidno:10)
体外切割实验结果如图5所示,结果表明ascpf1突变体蛋白相较于野生型ascpf1(wt),切割活性明显提高。
实施例5:ascpf1突变体蛋白编辑原代t细胞pd-1
(1)pd-1基因部分序列扩增
采用常规方法从人外周血中分离原代t细胞,并以提取的原代t细胞基因组dna为模板,pcr扩增并回收扩增片段,pcr扩增所用正向引物为:5’-actccccagacaggccctg-3’(seqidno:11),反向引物为:5’-caggggctggccggtgcg-3’(seqidno:12)。pcr扩增体系:基因组dna模板100ng,正向引物(20μm)1μl,反向引物(20μm)1μl,2xultrapfumix25μl,加ddh2o至总体积50μl;pcr反应条件为:94℃热变性5分钟,56℃退火30秒,72℃延伸1分钟,共进行30个循环。pcr扩增产物回收,用于ascpf1(s589q/p711t)体外切割实验。
(2)ascpf1(s589q/p711t)体外切割pd-1
取1μg回收的pd-1产物、1μl纯化的ascpf1(s589q/p711t)、1μg合成的crrna、2μl10*reactionbμffer,配制成20μl体外切割反应体系,37℃水浴30min后,2%琼脂糖凝胶电泳检测切割。其中10*reactionbμffer的组成为:500mmkac、200mmtris-醋酸、100mmmg(ac)2、1mg/mlbsaph7.9@25℃。
根据pd-1基因序列设计了3条靶向不同位点的crrna,委托上海吉玛制药技术有限公司合成:
crrna1:aauuucuacucuuguagaugcacgaagcucuccgauguguugg(seqidno:13);
crrna2:aauuucuacucuuguagauaucugcgccuugggggccagggag(seqidno:14);
crrna3:aauuucuacucuuguagaugaacuggccggcuggccuggguga(seqidno:15);
使用纯化的ascpf1突变体蛋白对pd-1进行体外切割实验,实验结果如图6所示,结果表明3条crrna均可介导ascpf1(s589q/p711t)对pd-1进行切割,说明ascpf1(s589q/p711t)识别并切割pd-1效率高。
(3)westernblotting检测ascpf1突变体蛋白处理原代t细胞后,pd-1表达水平变化
取1×106个原代t细胞pd-1,加入20μg纯化的ascpf1突变体蛋白,10μg合成的crrna混匀冰浴,电转化仪(bio-radmicropilsertm)点击转化后铺6孔板,72h后提取蛋白westernblotting检测pd-1蛋白表达水平变化。
用设计合成的3条crrna分别对原代t细胞进行处理,westernblotting检测结果如图7所示,crrna2和crrna3处理原代t细胞后,pd-1蛋白表达水平明显降低,表明ascpf1突变体蛋白及设计的两条针对pd-1基因的靶向crrna能有效敲降原代t细胞pd-1的表达。
<110>江苏溥博生物科技有限公司
<120>一种ascpf1突变体蛋白、编码基因、重组表达载体及其制备方法与应用
<160>15
<210>1
<211>1342
<212>prt
<213>人工序列
<220>
<223>
<400>1
mettyrglyarglyslysargargglnargargargthrglnpheglu
151015
glyphethrasnleutyrglnvalserlysthrleuargphegluleu
202530
ileproglnglylysthrleulyshisileglngluglnglypheile
354045
glugluasplysalaargasnasphistyrlysgluleulysproile
505560
ileaspargiletyrlysthrtyralaaspglncysleuglnleuval
65707580
glnleuasptrpgluasnleuseralaalaileaspsertyrarglys
859095
glulysthrglugluthrargasnalaleuileglugluglnalathr
100105110
tyrargasnalailehisasptyrpheileglyargthraspasnleu
115120125
thraspalaileasnlysarghisalagluiletyrlysglyleuphe
130135140
lysalagluleupheasnglylysvalleulysglnleuglythrval
145150155160
thrthrthrgluhisgluasnalaleuleuargserpheasplysphe
165170175
thrthrtyrpheserglyphetyrgluasnarglysasnvalpheser
180185190
alagluaspileserthralaileprohisargilevalglnaspasn
195200205
pheprolysphelysgluasncyshisilephethrargleuilethr
210215220
alavalproserleuarggluhisphegluasnvallyslysalaile
225230235240
glyilephevalserthrserileglugluvalpheserpheprophe
245250255
tyrasnglnleuleuthrglnthrglnileaspleutyrasnglnleu
260265270
leuglyglyileserargglualaglythrglulysilelysglyleu
275280285
asngluvalleuasnleualaileglnlysasnaspgluthralahis
290295300
ileilealaserleuprohisargpheileproleuphelysglnile
305310315320
leuseraspargasnthrleuserpheileleuglugluphelysser
325330335
aspglugluvalileglnserphecyslystyrlysthrleuleuarg
340345350
asngluasnvalleugluthralaglualaleupheasngluleuasn
355360365
serileaspleuthrhisilepheileserhislyslysleugluthr
370375380
ileserseralaleucysasphistrpaspthrleuargasnalaleu
385390395400
tyrgluargargilesergluleuthrglylysilethrlysserala
405410415
lysglulysvalglnargserleulyshisgluaspileasnleugln
420425430
gluileileseralaalaglylysgluleuserglualaphelysgln
435440445
lysthrsergluileleuserhisalahisalaalaleuaspglnpro
450455460
leuprothrthrleulyslysglngluglulysgluileleulysser
465470475480
glnleuaspserleuleuglyleutyrhisleuleuasptrppheala
485490495
valaspgluserasngluvalaspproglupheseralaargleuthr
500505510
glyilelysleuglumetgluproserleuserphetyrasnlysala
515520525
argasntyralathrlyslysprotyrservalglulysphelysleu
530535540
asnpheglnmetprothrleualaserglytrpaspvalasnlysglu
545550555560
lysasnasnglyalaileleuphevallysasnglyleutyrtyrleu
565570575
glyilemetprolysglnlysglyargtyrlysalaleuserpheglu
580585590
prothrglulysthrglngluglypheasplysmettyrtyrasptyr
595600605
pheproaspalaalalysmetileprolyscysserthrglnleulys
610615620
alavalthralahispheglnthrhisthrthrproileleuleuser
625630635640
asnasnpheilegluproleugluilethrlysgluiletyraspleu
645650655
asnasnproglulysgluprolyslyspheglnthralatyralalys
660665670
lysthrglyaspglnlysglytyrargglualaleucyslystrpile
675680685
aspphethrargasppheleuserlystyrthrlysthrthrserile
690695700
aspleuserserleuargthrserserglntyrlysaspleuglyglu
705710715720
tyrtyralagluleuasnproleuleutyrhisileserpheglnarg
725730735
ilealaglulysgluilemetaspalavalgluthrglylysleutyr
740745750
leupheglniletyrasnlysaspphealalysglyhishisglylys
755760765
proasnleuhisthrleutyrtrpthrglyleupheserprogluasn
770775780
leualalysthrserilelysleuasnglyglnalagluleuphetyr
785790795800
argprolysserargmetlysargmetalahisargleuglyglulys
805810815
metleuasnlyslysleulysaspglnlysthrproileproaspthr
820825830
leutyrglngluleutyrasptyrvalasnhisargleuserhisasp
835840845
leuseraspglualaargalaleuleuproasnvalilethrlysglu
850855860
valserhisgluileilelysaspargargphethrserasplysphe
865870875880
phephehisvalproilethrleuasntyrglnalaalaasnserpro
885890895
serlyspheasnglnargvalasnalatyrleulysgluhisproglu
900905910
thrproileileglyileaspargglygluargasnleuiletyrile
915920925
thrvalileaspserthrglylysileleugluglnargserleuasn
930935940
thrileglnglnpheasptyrglnlyslysleuaspasnargglulys
945950955960
gluargvalalaalaargglnalatrpservalvalglythrilelys
965970975
aspleulysglnglytyrleuserglnvalilehisgluilevalasp
980985990
leumetilehistyrglnalavalvalvalleugluasnleuasnphe
99510001005
glyphelysserlysargthrglyilealaglulysalavaltyrgln
111010151020
glnpheglulysmetleuileasplysleuasncysleuvalleulys
1125103010351040
asptyrproalaglulysvalglyglyvalleuasnprotyrglnleu
104510501055
thraspglnphethrserphealalysmetglythrglnserglyphe
106010651070
leuphetyrvalproalaprotyrthrserlysileaspproleuthr
107510801085
glyphevalaspprophevaltrplysthrilelysasnhisgluser
109010951100
arglyshispheleugluglypheasppheleuhistyraspvallys
1105111011151120
thrglyasppheileleuhisphelysmetasnargasnleuserphe
112511301135
glnargglyleuproglyphemetproalatrpaspilevalpheglu
114011451150
lysasngluthrglnpheaspalalysglythrpropheilealagly
115511601165
lysargilevalprovalilegluasnhisargphethrglyargtyr
117011751180
argaspleutyrproalaasngluleuilealaleuleugluglulys
1185119011951200
glyilevalpheargaspglyserasnileleuprolysleuleuglu
120512101215
asnaspaspserhisalaileaspthrmetvalalaleuileargser
122012251230
valleuglnmetargasnserasnalaalathrglygluasptyrile
123512401245
asnserprovalargaspleuasnglyvalcyspheaspserargphe
125012551260
glnasnproglutrpprometaspalaaspalaasnglyalatyrhis
1265127012751280
ilealaleulysglyglnleuleuleuasnhisleulysgluserlys
128512901295
aspleulysleuglnasnglyileserasnglnasptrpleualatyr
130013051310
ileglngluleuargasnlysargproalaalathrlyslysalagly
131513201325
glnalalyslyslyslysglyserhishishishishishis***
133013351340
<210>2
<211>4029
<212>dna
<213>人工序列
<220>
<223>
<400>2
atgtacggtcgtaaaaaacgtcgtcagcgtcgtcgtacacagttcgagggctttaccaac60
ctgtatcaggtgagcaagacactgcggtttgagctgatcccacagggcaagaccctgaag120
cacatccaggagcagggcttcatcgaggaggacaaggcccgcaatgatcactacaaggag180
ctgaagcccatcatcgatcggatctacaagacctatgccgaccagtgcctgcagctggtg240
cagctggattgggagaacctgagcgccgccatcgactcctatagaaaggagaaaaccgag300
gagacaaggaacgccctgatcgaggagcaggccacatatcgcaatgccatccacgactac360
ttcatcggccggacagacaacctgaccgatgccatcaataagagacacgccgagatctac420
aagggcctgttcaaggccgagctgtttaatggcaaggtgctgaagcagctgggcaccgtg480
accacaaccgagcacgagaacgccctgctgcggagcttcgacaagtttacaacctacttc540
tccggcttttatgagaacaggaagaacgtgttcagcgccgaggatatcagcacagccatc600
ccacaccgcatcgtgcaggacaacttccccaagtttaaggagaattgtcacatcttcaca660
cgcctgatcaccgccgtgcccagcctgcgggagcactttgagaacgtgaagaaggccatc720
ggcatcttcgtgagcacctccatcgaggaggtgttttccttccctttttataaccagctg780
ctgacacagacccagatcgacctgtataaccagctgctgggaggaatctctcgggaggca840
ggcaccgagaagatcaagggcctgaacgaggtgctgaatctggccatccagaagaatgat900
gagacagcccacatcatcgcctccctgccacacagattcatccccctgtttaagcagatc960
ctgtccgataggaacaccctgtctttcatcctggaggagtttaagagcgacgaggaagtg1020
atccagtccttctgcaagtacaagacactgctgagaaacgagaacgtgctggagacagcc1080
gaggccctgtttaacgagctgaacagcatcgacctgacacacatcttcatcagccacaag1140
aagctggagacaatcagcagcgccctgtgcgaccactgggatacactgaggaatgccctg1200
tatgagcggagaatctccgagctgacaggcaagatcaccaagtctgccaaggagaaggtg1260
cagcgcagcctgaagcacgaggatatcaacctgcaggagatcatctctgccgcaggcaag1320
gagctgagcgaggccttcaagcagaaaaccagcgagatcctgtcccacgcacacgccgcc1380
ctggatcagccactgcctacaaccctgaagaagcaggaggagaaggagatcctgaagtct1440
cagctggacagcctgctgggcctgtaccacctgctggactggtttgccgtggatgagtcc1500
aacgaggtggaccccgagttctctgcccggctgaccggcatcaagctggagatggagcct1560
tctctgagcttctacaacaaggccagaaattatgccaccaagaagccctactccgtggag1620
aagttcaagctgaactttcagatgcctacactggcctctggctgggacgtgaataaggag1680
aagaacaatggcgccatcctgtttgtgaagaacggcctgtactatctgggcatcatgcca1740
aagcagaagggcaggtataaggccctgagcttcgagcccacagagaaaacccaggagggc1800
tttgataagatgtactatgactacttccctgatgccgccaagatgatcccaaagtgcagc1860
acccagctgaaggccgtgacagcccactttcagacccacacaacccccatcctgctgtcc1920
aacaatttcatcgagcctctggagatcacaaaggagatctacgacctgaacaatcctgag1980
aaggagccaaagaagtttcagacagcctacgccaagaaaaccggcgaccagaagggctac2040
agagaggccctgtgcaagtggatcgacttcacaagggattttctgtccaagtataccaag2100
acaacctctatcgatctgtctagcctgcggacttcctctcagtataaggacctgggcgag2160
tactatgccgagctgaatcccctgctgtaccacatcagcttccagagaatcgccgagaag2220
gagatcatggatgccgtggagacaggcaagctgtacctgttccagatctataacaaggac2280
tttgccaagggccaccacggcaagcctaatctgcacacactgtattggaccggcctgttt2340
tctccagagaacctggccaagacaagcatcaagctgaatggccaggccgagctgttctac2400
cgccctaagtccaggatgaagaggatggcacaccggctgggagagaagatgctgaacaag2460
aagctgaaggatcagaaaaccccaatccccgacaccctgtaccaggagctgtacgactat2520
gtgaatcacagactgtcccacgacctgtctgatgaggccagggccctgctgcccaacgtg2580
atcaccaaggaggtgtctcacgagatcatcaaggataggcgctttaccagcgacaagttc2640
tttttccacgtgcctatcacactgaactatcaggccgccaattccccatctaagttcaac2700
cagagggtgaatgcctacctgaaggagcaccccgagacacctatcatcggcatcgatcgg2760
ggcgagagaaacctgatctatatcacagtgatcgactccaccggcaagatcctggagcag2820
cggagcctgaacaccatccagcagtttgattaccagaagaagctggacaacagggagaag2880
gagagggtggcagcaaggcaggcctggtctgtggtgggcacaatcaaggatctgaagcag2940
ggctatctgagccaggtcatccacgagatcgtggacctgatgatccactaccaggccgtg3000
gtggtgctggagaacctgaatttcggctttaagagcaagaggaccggcatcgccgagaag3060
gccgtgtaccagcagttcgagaagatgctgatcgataagctgaattgcctggtgctgaag3120
gactatccagcagagaaagtgggaggcgtgctgaacccataccagctgacagaccagttc3180
acctcctttgccaagatgggcacccagtctggcttcctgttttacgtgcctgccccatat3240
acatctaagatcgatcccctgaccggcttcgtggaccccttcgtgtggaaaaccatcaag3300
aatcacgagagccgcaagcacttcctggagggcttcgactttctgcactacgacgtgaaa3360
accggcgacttcatcctgcactttaagatgaacagaaatctgtccttccagaggggcctg3420
cccggctttatgcctgcatgggatatcgtgttcgagaagaacgagacacagtttgacgcc3480
aagggcacccctttcatcgccggcaagagaatcgtgccagtgatcgagaatcacagattc3540
accggcagataccgggacctgtatcctgccaacgagctgatcgccctgctggaggagaag3600
ggcatcgtgttcagggatggctccaacatcctgccaaagctgctggagaatgacgattct3660
cacgccatcgacaccatggtggccctgatccgcagcgtgctgcagatgcggaactccaat3720
gccgccacaggcgaggactatatcaacagccccgtgcgcgatctgaatggcgtgtgcttc3780
gactcccggtttcagaacccagagtggcccatggacgccgatgccaatggcgcctaccac3840
atcgccctgaagggccagctgctgctgaatcacctgaaggagagcaaggatctgaagctg3900
cagaacggcatctccaatcaggactggctggcctacatccaggagctgcgcaacaaaagg3960
ccggcggccacgaaaaaggccggccaggcaaaaaagaaaaagggatcccatcatcatcat4020
catcactaa4029
<210>3
<211>33
<212>dna
<213>人工序列
<220>
<223>
<400>3
tacggtcgtaaaaaacgtcgtcagcgtcgtcgt33
<210>4
<211>54
<212>dna
<213>人工序列
<220>
<223>
<400>4
aaaaggccggcggccacgaaaaaggccggccaggcaaaaaagaaaaagggatcc54
<210>5
<211>18
<212>dna
<213>人工序列
<220>
<223>
<400>5
catcatcatcatcatcac18
<210>6
<211>82
<212>dna
<213>人工序列
<220>
<223>
<400>6
gtttaactttaagaaggagatataccatgtacggtcgtaaaaaacgtcgtcagcgtcgtc60
gtacacagttcgagggctttac82
<210>7
<211>52
<212>dna
<213>人工序列
<220>
<223>
<400>7
tcgggctttgttagcagccggatcctcgagttagtgatgatgatgatgatgg52
<210>8
<211>23
<212>dna
<213>人工序列
<220>
<223>
<400>8
atgaaattgctggtagtcgaaga23
<210>9
<211>23
<212>dna
<213>人工序列
<220>
<223>
<400>9
atgaaattgctggtagtcgaaga23
<210>10
<211>68
<212>dna
<213>人工序列
<220>
<223>
<400>10
cgcgggccagcagttcggcaaaggatctacaacagtagaaattccctatagtgagtcgta60
ttaatttc68
<210>11
<211>19
<212>dna
<213>人工序列
<220>
<223>
<400>11
actccccagacaggccctg19
<210>12
<211>18
<212>dna
<213>人工序列
<220>
<223>
<400>12
caggggctggccggtgcg18
<210>13
<211>43
<212>dna
<213>人工序列
<220>
<223>
<400>13
aauuucuacucuuguagaugcacgaagcucuccgauguguugg43
<210>14
<211>43
<212>dna
<213>人工序列
<220>
<223>
<400>14
aauuucuacucuuguagauaucugcgccuugggggccagggag43
<210>15
<211>43
<212>dna
<213>人工序列
<220>
<223>
<400>15
aauuucuacucuuguagaugaacuggccggcuggccuggguga43
- 还没有人留言评论。精彩留言会获得点赞!
- 蝎神经毒素AaIT及其编码基因与应用的制造方法与工艺
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种花狭口蛙的基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种泽陆蛙基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 降低种子重量的白桦AP2基因及其编码蛋白的制造方法与工艺
- 可编码ArtRD蛋白基因及其蛋白和应用的制造方法与工艺
- 蝎神经毒素AaIT及其编码基因与应用的制造方法与工艺
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种花狭口蛙的基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种泽陆蛙基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 蛋白StTrxF、编码基因及其在提高植物淀粉含量中的应用的制造方法与工艺
- 蛋白质及其编码基因在调控植物抗黄萎病性中的应用的制造方法与工艺
- 甘薯耐逆相关蛋白IbFBA2、编码基因、重组载体与应用的制造方法与工艺
- 植物抗旱相关蛋白ZmNAC111及其编码基因与应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 蛋白StTrxF、编码基因及其在提高植物淀粉含量中的应用的制造方法与工艺
- 植物抗旱相关蛋白ZmNAC111及其编码基因与应用的制造方法与工艺
- 核盘菌凝集素SSL‑6蛋白及其编码基因与应用的制造方法与工艺
- 植物抗病毒相关蛋白GmSN1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 蛋白StTrxF、编码基因及其在提高植物淀粉含量中的应用的制造方法与工艺
- 蛋白质及其编码基因在调控植物抗黄萎病性中的应用的制造方法与工艺
- 植物抗旱相关蛋白ZmNAC111及其编码基因与应用的制造方法与工艺
- 核盘菌凝集素SSL‑6蛋白及其编码基因与应用的制造方法与工艺