用于商业化学品生产的生物平台的制作方法
本申请要求于2014年4月28日提交的美国临时专利申请第61/985,379号的权益,以其整体通过引用并入本文。
以ASCII文本上传序列表
将如下以ASCII文本形式上传的内容通过引用并入本文:序列表的计算机可读形式(CRF),文件名:514112007840SEQLIST.TXT,数据记录日期:2015年4月23日,大小:97KB。
关于政府资助研究的声明
本发明受美国农业部国家食品与农业研究所(USDA-NIFA)支持(基金编号:2011-67009-20060)。政府对本发明保有一定权利。本发明同样受加州能源委员会支持,基金编号55779A/08-03。能源委员会对本发明保有一定权利。
技术领域
本公开大致涉及将纤维素生物质转化成燃料和化学品的生物平台。更具体地,本公开涉及将纤维素生物质转化成将可用于生产商业化学品的糖酸或其盐。
技术背景
价廉并广泛存在的纤维素生物质是仅有的用于有机燃料、化学品和材料的可预见及可持续的资源(Lynd等人,2005;Lynd等人,2002;Lynd等人,1999)。特别地,纤维素生物质来源的燃料乙醇生产具有近乎零的温室气体排放并提供了许多其它的环境效益(Lynd等人,2005;Lynd等人,1999;Lynd等人,1991)。影响纤维素生物质来源的乙醇及其它化学品的生产的主要障碍是缺少低成本生产技术。
用于燃料和化学品生产的传统的生物化学平台或方法从纤维素原料中产生作为反应中间体的糖。然后可以将这些糖发酵以产生燃料和化学品。目前的生物化学平台涉及五个关键步骤:(1)预处理,(2)纤维素酶生产,(3)酶水解,(4)发酵,和(5)产品回收。前三步:预处理、纤维素酶生产和酶水解是生产过程中最昂贵的三个步骤,其占总生产成本的约65%。
第一步预处理是去除半纤维素和木质素以增加纤维素对随后酶水解的敏感性,因此允许暴露的纤维素通过纤维素酶水解成糖的过程。预处理过程倾向于热化学。此过程中所使用的技术包括用酸或碱处理,或者通过蒸汽爆破或氨爆破。大多数技术是能源密集型且昂贵的,并常常造成污染。此外,预处理反应器的资金成本是非常高的,这是因为在高温下对酸或碱的耐受有特别的材料要求。
预处理步骤后,在第二步骤中添加纤维素酶以水解纤维素,使得产生糖。尽管由于酶的工业化生产,其生产成本已大幅下降,但这一步骤的成本依然很高。降低前述两个步骤的成本对于实现有效生产生物质来源的乙醇和化学品是至关重要的。因此,需要用于将纤维素生物质转化成商业化学品的改善的组合物和方法。
技术实现要素:
在一个方面中,本公开涉及重组的宿主细胞,其包括:
a)与相应的野生型细胞相比,活性降低的具有β-葡萄糖苷酶活性的一种或多种多肽,其中所述一种或多种多肽中的每一种由与选自NCU00130、NCU04952、NCU05577、NCU07487、NCU08755和NCU03641基因的基因具有至少80%序列同一性的基因编码;b)与相应的野生型细胞相比,活性降低的具有纤维二糖酸盐磷酸化酶(cellobionate phosphorylase)活性的多肽,其中所述多肽由与NCU09425(NdvB)具有至少80%序列同一性的基因编码;c)与相应的野生型细胞相比,活性降低的由与NCU08807(CRE-1)具有至少80%序列同一性的基因编码的多肽;和d)与相应的野生型宿主细胞相比,活性降低的由与NCU09333(ACE-1)具有至少80%序列同一性的基因编码的多肽。在一些实施方案中,宿主细胞是真菌细胞。在一些实施方案中,真菌细胞是粗糙脉孢菌(Neurospora crassa)。在一些可以与任一前述实施方案组合的实施方案中,具有β-葡萄糖苷酶活性的一种或多种多肽的活性降低是由于基因突变。在一些实施方案中,基因突变存在于两个或更多个、三个或更多个、四个或更多个、五个或更多个或六个或更多个编码具有β-葡萄糖苷酶活性的多肽的基因中。在一些实施方案中,至少一个基因突变是敲除突变。在一些可以与任一前述实施方案组合的实施方案中,具有纤维二糖酸盐磷酸化酶活性的多肽的活性降低是由于基因突变。在一些实施方案中,基因突变是敲除突变。在一些可以与任一前述实施方案组合的实施方案中,由与NCU08807(CRE-1)具有至少80%序列同一性的基因编码的多肽的活性降低是由于基因突变。在一些实施方案中,基因突变是敲除突变。在一些可以与任一前述实施方案组合的实施方案中,由与NCU09333(ACE-1)具有至少80%序列同一性的基因编码的多肽的活性降低是由于基因突变。在一些实施方案中,基因突变是敲除突变。在一些可以与任一前述实施方案组合的实施方案中,宿主细胞还包含与相应的野生型细胞相比,活性降低的由与NCU08290(MUS51)具有至少80%序列同一性的基因编码的多肽。在一些实施方案中,由与NCU08290(MUS51)具有至少80%序列同一性的基因编码的多肽的活性降低是由于基因突变。在一些实施方案中,基因突变是敲除突变。在一些可以与任一前述实施方案组合的实施方案中,宿主细胞还包含与相应的野生型细胞相比,具有增加的漆酶表达或活性的多肽。在一些实施方案中,多肽由与NCU04528具有至少80%序列同一性的基因编码。在一些实施方案中,多肽的表达受组成型启动子的控制。在一些可以与任一前述实施方案组合的实施方案中,宿主细胞还包含与相应的野生型细胞相比,具有增加的纤维二糖脱氢酶表达或活性的多肽。在一些实施方案中,具有增加的纤维二糖脱氢酶表达或活性的多肽的表达受组成型启动子的控制。在一些可以与任一前述实施方案组合的实施方案中,宿主细胞从纤维素中产生糖酸。在一些可以与任一前述实施方案组合的实施方案中,宿主细胞产生纤维二糖。在一些可以与任一前述实施方案组合的实施方案中,糖酸是纤维二糖酸盐(cellobionate)。在一些实施方案中,与相应的野生型细胞相比,宿主细胞的纤维二糖酸盐的消耗降低了至少80%。
在另一方面中,本公开涉及重组的粗糙脉孢菌细胞,其包括:a)在NCU00130、NCU04952、NCU05577、NCU07487、NCU08755和NCU03641基因中的各个基因中的突变,其中所述突变降低了由所述基因编码的多肽的β-葡萄糖苷酶活性;b)在NCU09425(NdvB)基因中的突变,其中所述突变降低了由所述基因编码的多肽的纤维二糖酸盐磷酸化酶活性;c)在NCU08807(CRE-1)和NCU09333(ACE-1)基因中的各个基因中的突变,其中所述突变降低了由所述基因编码的多肽的活性;d)与野生型粗糙脉孢菌细胞相比,具有增加的表达或活性的漆酶,并且其中重组的粗糙脉孢菌细胞从纤维素中产生纤维二糖酸盐。
在另一方面中,本公开涉及产生糖酸的方法,所述方法包括:a)提供宿主细胞,所述宿主细胞具有i)与相应的野生型细胞相比,具有β-葡萄糖苷酶活性的一种或多种多肽的活性降低,其中每个所述一种或多种多肽由与选自NCU00130、NCU04952、NCU05577、NCU07487、NCU08755和NCU03641基因的基因具有至少80%序列同一性的基因编码;ii)与相应的野生型细胞相比,具有纤维二糖酸盐磷酸化酶活性的多肽的活性降低,其中所述多肽由与NCU09425(NdvB)具有至少80%序列同一性的基因编码;iii)与相应的野生型细胞相比,由与NCU08807(CRE-1)具有至少80%序列同一性的基因编码的多肽的活性降低;和iv)与相应的野生型宿主细胞相比,由与NCU09333(ACE-1)具有至少80%序列同一性的基因编码的多肽的活性降低;b)在包含纤维素的培养基中培养宿主细胞,其中所述宿主细胞从纤维素中产生糖酸。
在另一方面中,本公开涉及产生糖酸的方法,所述方法包括a)提供任一前述实施方案中的宿主细胞,以及b)在包含纤维素的培养基中培养所述宿主细胞,其中所述宿主细胞从纤维素中产生糖酸。在一些实施方案中,该方法还包括从培养基中充分纯化糖酸的步骤。在一些可以与任一前述实施方案组合的实施方案中,宿主细胞产生纤维二糖。在一些可以与任一前述实施方案组合的实施方案中,糖酸是纤维二糖酸盐。在一些实施方案中,与相应的野生型细胞相比,宿主细胞的纤维二糖酸盐的消耗降低了至少80%。在一些可以与任一前述实施方案组合的实施方案中,可以在存在外源漆酶的情况下培养宿主细胞。在一些可以与任一前述实施方案组合的实施方案中,在存在外源纤维二糖脱氢酶的情况下培养宿主细胞。在一些可以与任一前述实施方案组合的实施方案中,培养基还包括氧化还原介体。
附图说明
专利或申请文件包含至少一个彩色附图。附有彩色附图的本专利或专利申请公开的副本将由办公室根据请求并支付必要的费用提供。
图1A示出了在从纤维素生物质中生产商业化学品的方法中所使用的常规步骤的流程图。图1B示出了直接酶水解纤维素的方法的流程图。
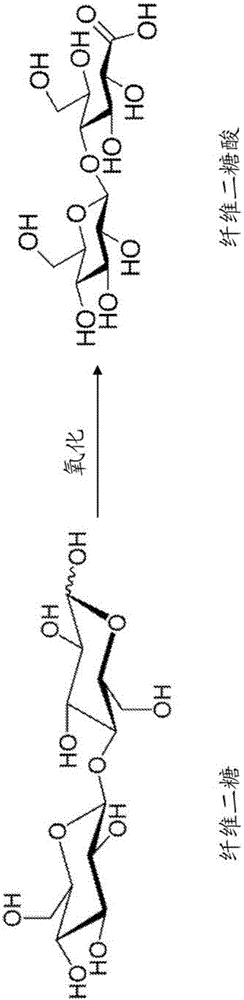
图2示出了纤维二糖向纤维二糖酸的化学转化。
图3示出了一些分解纤维素的微生物(如粗糙脉孢菌)的纤维素降解机制。
图4A示出了在添加或不添加外源纤维二糖脱氢酶的情况下由F5菌株生产纤维二糖。图4B示出了在添加或不添加外源纤维二糖脱氢酶的情况下由F5菌株生产纤维二糖酸盐。
图5A示出了由大肠杆菌(Escherichia coli)菌株KO 11将葡萄糖酸盐转化成乙醇和乙酸盐。图5B示出了由大肠杆菌菌株KO 11将葡萄糖和葡萄糖酸盐的混合物转化成乙醇和乙酸盐。
图6示出了纤维素转化和菌丝团产生的概述。
图7示出了漆酶和ABTS对各种菌株中纤维二糖和纤维二糖酸盐生产的影响。
图8A示出了不同菌株中的纤维二糖和纤维二糖酸盐的水平。图8B示出了纤维二糖酸盐的理论收率和残留收率。
图9A示出了在转化反应之前过滤的含细胞和纤维素的各种菌株中的纤维二糖和纤维二糖酸盐的水平。图9B示出了纤维二糖酸盐的理论收率和残留收率。
图10A示出了理论的纤维二糖和纤维二糖酸盐的浓度表。图10B示出了基于测量的或预测的纤维二糖水平的纤维二糖酸盐的理论转化。
图11A示出了不同菌株中的纤维二糖酸盐的水平。图11B示出了各种菌株中的纤维二糖的水平。图11C示出了各种菌株中的纤维二糖脱氢酶(CDH)的活性。
图12示出了F5Δcre-1Δace菌株和F5Δcre-1ΔaceΔndvB菌株的纤维二糖酸盐的利用。
图13示出了F5Δcre-1Δace菌株和F5Δcre-1ΔaceΔndvB+lac菌株的纤维二糖酸盐的生产。
图14A示出了各种菌株中的纤维二糖的生产。图14B示出了各种菌株中的纤维二糖酸盐的生产。所有菌株都生长于20g/L的微晶纤维素(Avicel)中。
图15示出了纤维二糖酸转化成α-D-葡萄糖-l-磷酸和D-葡萄糖酸。
图16示出了CDH对底物(纤维二糖/乳糖)的酶氧化。其中还原的CDH被氧化还原介体(ABTS)氧化,其又被漆酶氧化。还原的漆酶被氧氧化,而水是唯一的副产物。
图17A和17B示出了pH对由CDH-ABTS-漆酶介导的纤维二糖向CBA转化的影响。酸性条件(pH 4和pH 6)使用50mM柠檬酸钠缓冲液;pH 7.2和pH 8使用50mM磷酸钠缓冲液。Vogels pH 6条件使用1×Vogel's盐培养基。所示结果是生物学一式两份重复的平均值,误差条表示标准偏差。
图18A和18B示出了在1×Vogel's培养基和20g/L的微晶纤维素中,磷酸钾缓冲液浓度和纤维二糖向CBA转化的影响,所述转化是用F5Δace-1Δcre-1ΔndvB菌株由CDH-ABTS-漆酶介导的转化。所示的数值是生物学一式两份重复的平均值,误差条表示标准偏差。
图19A和19B示出了用在1×Vogel's培养基和20g/L微晶纤维素中生长的F5Δace-1Δcre-1ΔndvB菌株的漆酶(0.05U/mL)和ABTS(0.01mM)添加时间的优化。所示的数值是生物学一式三份重复的平均值,误差条表示标准偏差。
图20示出了粗糙脉孢菌在通过3μM放线菌酮在48小时诱导进入发酵中诱导的漆酶生产。所示结果是生物学一式三份重复的平均值,误差条表示标准偏差。
图21A和21B示出了在使用CDH-ABTS-漆酶转化体系的纤维二糖向CBA转化的过程中,粗糙脉孢菌漆酶与糙皮侧耳菌(P.ostreatus)漆酶的比较。所示数据是生物学一式两份重复的平均值,误差条表示标准偏差。
图22示出了利用重组的粗糙脉孢菌菌株的漆酶生产。
图23示出了在添加ABTS(在0小时添加ABTS)的指定菌株中的纤维二糖酸盐的生产。
图24示出了在添加或不添加ABTS的指定菌株中的纤维二糖酸盐的生产。
详细说明
进行如下描述使本领域技术人员能够制造和使用各种实施方案。具体设备、技术和应用的描述仅作为示例提供。对本文描述的实施例的各种修改对于本领域技术人员而言是显而易见的,并且本文所定义的一般原理可以应用于其它实施例和应用且不脱离各种实施方案的范畴和精神。因此,各种实施方案并不意图限制本文所述和所示的实施例,而与权利要求的保护范围一致。
概述
本公开大体涉及用于将纤维素生物质转化为燃料和化学品的生物平台。更具体地,本公开涉及将纤维素材料转化为糖酸或其盐,然后其可用于生产商业化学品。
图1A示出了在从纤维素生物质生产葡萄糖酸和异丁醇中使用的常规步骤,其涉及纤维素酶的生产和需氧发酵的步骤。申请人提出了一种可替代的路线,示于图1B中。其规避了纤维素酶生产的需求,而替代为基于直接酶水解纤维素以产生糖酸,如纤维二糖酸(参见图2)和随后的下游产物。
分解纤维素的微生物能够水解纤维素,因此此类微生物可以用作生产商业化学品。图3示出了一些分解纤维素的微生物(包括粗糙脉孢菌)的纤维素降解机制。纤维素被内切葡聚糖酶和外切葡聚糖酶水解成纤维寡糖,其中纤维二糖为主要组分。所获得的主要组分纤维二糖被纤维二糖脱氢酶氧化以产生纤维二糖-1,5-内酯(cellobiono-1,5-lactone),其与水自发地反应以形成纤维二糖酸。此外,纤维二糖酸通过溶解性多糖单氧化酶和纤维二糖脱氢酶的组合作用从纤维素中被释放。将纤维二糖酸转运到细胞质中,然后通过纤维二糖酸磷酸化酶(NCU09425)进行磷酸化以产生αGlc1P和D-葡萄糖酸。所释放的αGlc1P通过葡萄糖磷酸变位酶(EC2.7.5.1)转化为D-葡萄糖-6-磷酸以进入糖酵解途径。通过葡萄糖激酶(EC2.7.1.12)和6-磷酸葡萄糖酸盐脱氢酶(EC 1.1.1.44)的顺序反应,将释放的D-葡萄糖酸通过6-磷酸葡萄糖酸盐转化为核酮糖5-磷酸以进入戊糖磷酸途径(Nihira等人,2013)。本文所述的纤维素代谢途径的显著特征之一是真菌有效地使用ATP衍生的能量,这是因为可以直接磷酸化纤维二糖酸的D-葡萄糖残基而不通过ATP依赖的碳水化合物激酶消耗ATP。纤维二糖酸似乎是β-葡萄糖苷酶的较不理想的底物,这是因为该反应的水解产物D-葡萄糖酸是β-葡萄糖苷酶的非竞争性抑制剂(Nihira等人,2013)。此外,应当注意,子囊菌门的各种分解纤维素的真菌也具有纤维二糖酸磷酸化酶的同源蛋白。
在纤维寡糖产生之后,β-葡萄糖苷酶可以将纤维寡糖水解成葡萄糖,葡萄糖可以被真菌代谢。如果β-葡萄糖苷酶的酶活性降低并且纤维二糖脱氢酶(CDH)活性增加,则碳流可以指向糖醛酸生产。这些有机酸可以从液体培养基中不断地分离出去,例如通过氢氧化钙沉淀,以避免任何的可能的抑制作用。在实践中,一些β-葡萄糖苷酶活性必须在细胞中保持完整以支持细胞生长和酶生产。可以通过降低葡萄糖激酶的活性来防止葡萄糖酸利用,从而使该化合物可用于随后的乙醇发酵。葡萄糖酸被发现是黑曲霉(A.niger)的β-葡萄糖苷酶的非竞争性产物抑制剂,并且其通过残留的β-葡萄糖苷酶活性可以抑制纤维二糖酸盐的任何明显的水解。此外,水解实验表明,纤维二糖酸以比纤维二糖低约10倍的速率被β-葡萄糖苷酶水解,并且所形成的葡萄糖酸是β-葡萄糖苷酶的抑制剂(Cannella,2012)。
在本文中,申请人公开了将纤维素转化为下游产物的组合物和方法。本公开至少部分基于申请人的发现,即将粗糙脉孢菌工程化以包括在六种β-葡萄糖苷酶基因中的突变、在CRE-1基因中的突变、在ACE-1基因中的突变、在NdvB纤维二糖酸盐磷酸化酶基因中的突变以及工程化以过表达漆酶基因或在存在外源漆酶的情况下培养,产生出奇高量的纤维二糖酸(纤维二糖酸盐)。这一工程化菌株同时不能代谢所产生的纤维二糖酸盐,使得纤维二糖酸盐积累。如上所述,纤维二糖酸盐可用于生产下游产物,如葡萄糖酸和异丁醇。
具有修饰的活性的多肽
本公开涉及与相应的对照细胞(例如野生型细胞)相比,具有各种多肽的修饰的活性的重组宿主细胞。如本文所用,“多肽”是包括多个连续聚合的氨基酸残基(例如,至少约15个连续聚合的氨基酸残基)的氨基酸序列。如本文所用,“多肽”是指氨基酸序列、寡肽、肽、蛋白质或其部分,并且术语“多肽”和“蛋白质”可互换使用。
β-葡萄糖苷酶多肽
与相应的野生型细胞相比,本公开的宿主细胞的一种或多种β-葡萄糖苷酶多肽的活性降低。
本公开的β-葡萄糖苷酶(bgl)基因编码β-葡萄糖苷酶。如本文所用,“β-葡萄糖苷酶”是指β-D-葡萄糖苷葡萄糖水解酶(E.C.3.2.1.21),其催化末端非还原性β-D-葡萄糖残基的水解并释放β-D-葡萄糖。β-葡萄糖苷酶是任意的催化β-D-葡萄糖苷(如纤维糊精)中的末端非还原性残基水解并释放葡萄糖的酶。
本公开的β-葡萄糖苷酶可以是胞内β-葡萄糖苷酶或胞外β-葡萄糖苷酶。如本文所用,“胞内β-葡萄糖苷酶”在木质纤维素分解细胞内表达并水解转运入细胞的纤维糊精。如本文所用,“胞外β-葡萄糖苷酶”由木质纤维素分解细胞表达和分泌或在木质纤维素分解细胞的表面上表达。
在某些实施方案中,β-葡萄糖苷酶是糖基水解酶家族1成员。该基团的成员可以通过基序[LIVMFSTC]-[LIVFYS]-[LIV]-[LIVMST]-E-N-G-[LIVMFAR]-[CSAGN](SEQ ID NO:23)来鉴定。本文中,E是催化性谷氨酸。在一些实施方案中,β-葡萄糖苷酶来自粗糙脉孢菌。其它β-葡萄糖苷酶可包括来自糖基水解酶家族3的那些酶。这些β-葡萄糖苷酶可根据PROSITE数据库通过以下基序鉴定:[LIVM](2)-[KR]-x-[EQKRD]-x(4)-G-[LIVMFTC]-[LIVT]-[LIVMF]-[ST]-D-x(2)-[SGADNIT](SEQ ID NO:24)。本文D是催化性天冬氨酸。通常,包含在NCBI序列COG2723中发现的β-葡萄糖苷酶/6-磷酸-β-葡萄糖苷酶/β-半乳糖苷酶的保守结构域的任何β-葡萄糖苷酶都可以使用。
在某些实施方案中,本公开的β-葡萄糖苷酶包括例如来自粗糙脉孢菌由NCU00130、NCU04952、NCU05577、NCU07487、NCU08054、NCU08755和NCU03641编码的β-葡萄糖苷酶。本公开合适的β-葡萄糖苷酶还包括NCU00130、NCU04952、NCU05577、NCU07487、NCU08054、NCU08755和NCU03641的同源物、直系同源物和旁系同源物。
本公开的胞内β-葡萄糖苷酶包括例如由NCU00130、NCU05577、NCU07487、NCU08054、其同源物、其直系同源物和其旁系同源物编码的酶。本公开的胞外β-葡萄糖苷酶包括例如由NCU04952、NCU08755、NCU03641、其同源物、其直系同源物和其旁系同源物编码的酶。
在一些实施方案中,具有降低的β-葡萄糖苷酶活性的多肽是由NCU00130、NCU04952、NCU05577、NCU07487、NCU08755和/或NCU03641编码的多肽。由NCU00130编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:1。由NCU04952编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:3。由NCU05577编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:5。由NCU07487编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:7。由NCU08755编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:9。由NCU03641编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:11。在一些实施方案中,具有降低的β-葡萄糖苷酶活性的多肽是由与NCU00130、NCU04952、NCU05577、NCU07487、NCU08755或NCU03641基因具有至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少98%、至少99%或100%的同一性的基因编码。NCU00130的核苷酸序列如本文所示为SEQ ID NO:2。NCU04952的核苷酸序列在本文所示为SEQ ID NO:4。NCU05577的核苷酸序列在本文中所示为SEQ ID NO:6。NCU07487的核苷酸序列在本文中所示为SEQ ID NO:8。NCU08755的氨基酸序列在本文中所示为SEQ ID NO:10。NCU03641的核苷酸序列在本文中所示为SEQ ID NO:12。
其它各种的β-葡萄糖苷酶是本领域公知的,并且可以用于本公开的方法和组合物中。
在本公开的宿主细胞中一种或多种β-葡萄糖苷酶可具有降低的活性。本公开的宿主细胞可以具有活性降低的两种或更多种、三种或更多种、四种或更多种、五种或更多种或六种或更多种的具有β-葡萄糖苷酶活性的多肽。
纤维二糖酸盐磷酸化酶多肽
与相应的野生型细胞相比,本公开的宿主细胞具有活性降低的一种或多种纤维二糖酸盐磷酸化酶蛋白。
纤维二糖酸盐磷酸化酶在本领域中是公知的并且描述于本文中。纤维二糖酸盐磷酸化酶催化如图15所示的反应并具有与EC 2.4.1.321相关的活性。在该反应中,纤维二糖酸盐磷酸化酶催化纤维二糖酸(4-O-β-D-吡喃葡萄糖基-D-葡萄糖酸盐)的磷酸化以产生α-D-葡萄糖-1-磷酸和D-葡萄糖酸。在一些实施方案中,本公开的纤维二糖酸盐磷酸化酶包含一个或多个糖基转移酶家族36蛋白结构域。
在一些实施方案中,具有降低的纤维二糖酸盐磷酸化酶活性的多肽是由NCU09425编码的多肽,NCU09425编码来自粗糙脉孢菌的NdvB蛋白。由NCU09425编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:13。在一些实施方案中,具有降低的纤维二糖酸盐磷酸化酶活性的多肽是由与NCU09425具有至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少98%、至少99%或100%的同一性的基因编码。NCU09425的核苷酸序列在本文中示为SEQ ID NO:14。其它各种纤维二糖酸盐磷酸化酶是本领域公知的,并且可以用于本公开的方法和组合物中。其它示例性的纤维二糖酸盐磷酸化酶包括例如来自野油菜黄单胞菌(Xanthomonas campestris)的糖苷水解酶家族94纤维二糖酸盐磷酸化酶。
CRE-1多肽
与相应的野生型细胞相比,本公开的宿主细胞具有降低的CRE-1蛋白活性。CRE-1蛋白是本领域公知的并在本文中描述。CRE-1是转录因子蛋白。一般来说,转录因子多肽是参与细胞中基因表达调节的多肽。转录因子多肽可具有与核酸(例如DNA)的表达控制相关的活性。转录因子活性是本领域公知的。例如,转录因子多肽活性可以包括DNA结合活性或与基因表达的调节相关的活性。
普遍认为CRE-1涉及分解代谢产物的抑制。已示出cre的缺失增加纤维素酶的表达。cre-菌株在优选碳源中生长速率降低;然而,在微晶纤维素中,其产生了多30-50%的纤维素酶并且更快地消耗微晶纤维素(Sun等人,2011),并且cre的表达与纤维素酶的表达相关。
在一些实施方案中,具有CRE-1活性降低的多肽是由NCU08807编码的多肽,NCU08807编码来自粗糙脉孢菌的CRE-1蛋白。由NCU08807编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:15。在一些实施方案中,具有降低的CRE-1活性的多肽是由与NCU08807基因具有至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少70%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少98%、至少99%或100%的同一性的基因编码。NCU08807的核苷酸序列在本文中示为SEQ ID NO:16。其它各种CRE-1蛋白在本领域是公知的,并且可以用于本公开的方法和组合物中。
ACE-1多肽
与相应的野生型细胞相比,本公开的宿主细胞的ACE-1蛋白活性降低。ACE-1蛋白是本领域公知的并且在本文中描述。ACE-1是锌指转录因子蛋白。ace-1基因的突变(ace-)导致里氏木霉(T.reesei)中纤维素酶的表达更高(Aro等人,2003)。
在一些实施方案中,具有降低的ACE-1活性的多肽是由NCU09333编码的多肽,NCU09333编码来自粗糙脉孢菌的ACE-1蛋白。由NCU09333编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:17。在一些实施方案中,具有降低的ACE-1活性的多肽是由与NCU09333具有至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少98%、至少99%或100%的同一性的基因编码。NCU09333的核苷酸序列在本文中示为SEQ ID NO:18。各种其它ACE-1蛋白是本领域公知的,并且可以用于本公开的方法和组合物中。
MUS51多肽
与相应的野生型细胞相比,本公开的宿主细胞可具有降低的MUS51蛋白活性。MUS51蛋白是本领域公知的并且在本文中描述。MUS51是ATP依赖的DNA解旋酶II亚基1蛋白(E.C.3.6.4.12)。MUS51蛋白参与非同源末端连接(NHEJ)DNA双链断裂修复。不希望受理论束缚,一般认为在宿主细胞中降低MUS51蛋白活性将增加宿主细胞中同源重组的机会和/或频率,这可使宿主细胞的基因修饰更容易。
在一些实施方案中,具有降低的MUS51活性的多肽是由NCU08290编码的多肽,NCU08290编码来自粗糙脉孢菌的MUS51蛋白。在一些实施方案中,具有降低的MUS51活性的多肽是由与NCU08290具有至少10%、至少15%、至少20%、至少25%、至少30%、至少35%至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少98%、至少99%或100%的同一性的基因编码。各种其它MUS51蛋白是本领域公知的,并且可以用于本公开的方法和组合物中。
漆酶多肽
与相应的野生型细胞相比,本公开的宿主细胞可具有增加的漆酶蛋白的表达或活性。在本公开的方法中,漆酶蛋白也可以外源添加到培养基中。漆酶蛋白是本领域公知的并且在本文中描述。漆酶是“蓝色”含铜氧化酶(E.C.1.10.3.2)。漆酶催化以下反应:4苯二酚+O2=4苯半醌+2H2O。这些多铜酶对邻-(o-)和对(p-)醌醇具有低特异性作用,并且通常还作用于氨基苯酚和苯二胺。漆酶还可以作用于酚和类似分子,进行单电子氧化。由于漆酶蛋白属于氧化酶家族,这些酶需要氧作为酶促作用的第二底物。
在一些实施方案中,具有增加的漆酶表达或活性的多肽是由NCU04528编码的多肽,NCU04528编码来自粗糙脉孢菌的漆酶蛋白。由NCU04528编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:19。在一些实施方案中,具有增加的漆酶表达或活性的多肽是由与NCU04528具有至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少98%、至少99%或100%同一性的基因编码的多肽。NCU04528的核苷酸序列在本文中示为SEQ ID NO:20。各种其它漆酶蛋白是本领域公知的,并且可以用于本公开的方法和组合物中。
纤维二糖脱氢酶多肽
与相应的野生型细胞相比,本公开的宿主细胞具有增加的纤维二糖脱氢酶蛋白的表达或活性。在本公开的方法中,纤维二糖脱氢酶蛋白也可以外源添加到培养基中。纤维二糖脱氢酶(CDH)蛋白是本领域公知的并且在本文中描述。CDH酶催化以下反应:纤维二糖+受体=纤维二糖-1,5-内酯+还原的受体(E.C.1.1.99.18)。CDH蛋白含有N-末端血红素结构域和C-末端脱氢酶结构域。一些CDH蛋白在蛋白质的C末端还含有纤维素结合模块(CBM)。CDH血红素结构域的直系同源物仅在真菌蛋白中发现,而脱氢酶结构域的直系同源物存在于生命的所有结构域的蛋白质中;脱氢酶结构域是较大的GMC氧化还原酶超家族的一部分。
在一些实施方案中,具有增加的纤维二糖脱氢酶表达或活性的多肽是由NCU00206编码的多肽,NCU00206编码来自粗糙脉孢菌的纤维二糖脱氢酶蛋白。由NCU00206编码的蛋白质的氨基酸序列在本文中示为SEQ ID NO:21。在一些实施方案中,具有增加的纤维二糖脱氢酶表达或活性的多肽是由与基因NCU00206具有至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少98%、至少99%或100%的同一性的基因编码的多肽。NCU00206的核苷酸序列在本文中示为SEQ ID NO:22。各种其它纤维二糖脱氢酶蛋白是本领域公知的,并且可以用于本公开的方法和组合物中。其它纤维二糖脱氢酶包括例如编号:XM_411367、BAD32781、BAC20641、XM_389621、AF257654、AB187223、XM_360402、U46081、AF081574、AY187232、AF074951和AF029668的多肽。
编码多肽的多核苷酸
本公开还涉及编码具有本公开的修饰活性的多肽的多核苷酸。编码多肽的多核苷酸在本文中也称为“基因”。例如,本文提供了编码已知或假定的β-葡萄糖苷酶、纤维二糖酸盐磷酸化酶、CRE-1蛋白、ACE-1蛋白、MUS51蛋白、漆酶或纤维二糖脱氢酶多肽的多核苷酸。用于测定多肽与编码多肽的多核苷酸之间的关系的方法是本领域技术人员公知的。类似地,确定由多核苷酸序列编码的多肽序列的方法是本领域技术人员公知的。
如本文所用,术语“多核苷酸”、“核酸序列”、“核酸”及其变体应属于多脱氧核糖核苷酸(含2-脱氧-D-核糖)、多核糖核苷酸(含D-核糖)、为嘌呤或嘧啶碱基的N-糖苷的任何其它类型的多核苷酸以及含有非核苷酸主链的其它聚合物,只要此聚合物含有允许碱基配对和碱基堆积(如DNA和RNA中所发现的)的构型的核碱基。因此,这些术语包括已知类型的核酸序列修饰,例如,用类似物取代一个或多个天然存在的核苷酸以及核苷酸间的修饰。如本文所使用,核苷酸和多核苷酸的符号是由IUPAC-IUB生物化学命名委员会推荐。
本公开的多核苷酸的序列可以通过本领域已知的各种合适的方法制备,包括例如直接化学合成或克隆。对于直接化学合成,核酸的聚合物的形成通常涉及将3'-阻断和5'-阻断的核苷酸单体依次添加至延长的核苷酸链的5'-羟基末端,其中每次添加都是通过延长的链的5'-羟基末端亲核攻击所添加的单体的3'-位置,其通常是磷衍生物(例如磷酸三酯、亚磷酰胺等)。这种方法是本领域技术人员公知的,并且描述于相关文本和文献中(例如,在Matteucci等人,(1980)Tetrahedron Lett 21:719-722;美国专利4,500,707、5,436,327和5,700,637中)。此外,通过本领域技术人员已知的技术,例如利用聚合酶链式反应(PCR;例如美国专利4,683,195),通过使用合适的限制性酶分割DNA以从天然来源分离所需的序列,使用凝胶电泳分隔片段,然后从凝胶中回收所需的多核苷酸序列。
鉴定序列相似性的方法
本领域技术人员已知晓各种用于鉴定相似(例如同源物、直系同源物、旁系同源物等)多肽和/或多核苷酸序列的方法,包括系统发育法、序列相似性分析和杂交法。
系统发育树可以通过使用程序诸如CLUSTAL(Thompson等人Nucleic Acids Res.22:4673-4680(1994);Higgins等人,Methods Enzymol266:383-402(1996))或MEGA(Tamura等人,Mol.Biol.&Evo.24:1596-1599(2007))建立基因家族。一旦来自一个物种的基因的初始树被建立,可以将潜在的直系同源序列置于系统发育树中,并且可以确定它们与来自目的物种的基因的关系。进化关系也可以使用邻接法(Neighbor-Joining method)来推断(Saitou和Nei,Mol.Biol.&Evo.4:406-425(1987))。同源序列也可以通过倒数BLAST策略来确定。进化距离则可以使用泊松校正方法来计算(Zuckerkandl和Pauling,pp.97-166,Evolving Genes and Proteins,V.Bryson和H.J.Vogel编辑,Academic Press,New York(1965))。
此外,进化信息可用于预测基因功能。通过关注基因如何在基因序列变得相似(即通过进化过程)而不是序列本身相似(Eisen,Genome Res.8:163-167(1998)),可以极大地改善基因的功能预测。存在许多具体实例说明基因功能与基因系统发育密切相关(Eisen,Genome Res.8:163-167(1998))。通过使用系统发育分析,本领域技术人员将认识到由紧密相关的多肽推断相似功能是可预测的。
当使用系统发育程序如CLUSTAL分析一组相关序列时,紧密相关的序列通常聚集在一起或在相同的进化枝(一组相似的基因)中。也可以用成对BLAST分析鉴定相似基因的组(Feng和Doolittle,J.Mol.Evol.25:351-360(1987))。对落入一个进化枝内的具有相似功能的相似基因的组进行分析可以产生进化枝的特有的子序列。这些亚序列,称为共有序列,不仅可用于限定各个进化枝内的序列,而且可用于限定这些基因的功能;进化枝内的基因可以包含共有相同功能的旁系同源序列或直系同源序列(也参见例如Mount,Bioinformatics:Sequence andGenome Analysis,Cold Spring Harbor Laboratory Press,Cold Spring Harbor,N.Y.,543页(2001))。
为了找到与参考序列同源的序列,可以用BLASTN程序(分数=100,字长=12)进行BLAST核苷酸搜索以获得与编码本公开的蛋白质的核苷酸序列同源的核苷酸序列。可以用BLASTX程序(分数=50,字长=3)进行BLAST蛋白质搜索以获得与本公开的蛋白质或多肽同源的氨基酸序列。为了获得用于比较目的的缺口比对,可以使用如在Altschul等人(1997)Nucleic Acids Res.25:3389中所述的缺口BLAST(在BLAST 2.0中)。或者,可以使用PSI-BLAST(在BLAST 2.0中)进行检测分子间远距离关系的迭代搜索。参见Altschul等人(1997)(同上)。当使用BLAST、缺口BLAST或PSI-BLAST时,可以使用各自程序的默认参数(例如,用于核苷酸序列的BLASTN,用于蛋白质的BLASTX)。
用于序列比对和用于分析多肽和多核苷酸序列的相似性和同一性的方法是本领域公知的。
如本文所使用,“序列同一性”是指在待分析的序列中的相同位置的相同残基的百分比。如本文所用,“序列相似性”是指在待分析的序列中的相同位置的具有相似生物物理/生物化学特性(例如电荷、大小、疏水性)的残基的百分比。
用于比较的序列比对方法是本领域公知的,包括手动比对和计算机辅助序列比对和分析。由于计算机辅助方法提供的增加的吞吐量,后一种方法是本公开中的优选方法。如下所述,多种用于进行序列比对的计算机程序都是可用的,或由技术人员开发。
任何两个序列之间的序列同一性和/或相似性的百分比的确定可以使用数学算法来完成。此类数学算法的实例是Myers和Miller,CABIOS 4:11-17(1988)中的算法;Smith等人,Adv.Appl.Math.2:482(1981)中的局部同源算法;Needleman和Wunsch,J.Mol.Biol.48:443-453(1970)中的同源比对算法;Pearson和Lipman,Proc.Natl.Acad.Sci.85:2444-2448(1988)中的相似性搜索方法;Karlin和Altschul,Proc.Natl.Acad.Sci.USA 87:2264-2268(1990)中的算法;Karlin和Altschul,Proc.Natl.Acad.Sci.USA90:5873-5877(1993)中的改良版。
可以利用计算机执行这些数学算法用于比较序列以确定序列同一性和/或相似性。此类执行程序包括例如:PC/基因程序中的CLUSTAL(可从Intelligenetics,Mountain View,Calif获得);AlignX程序,10.3.0版本(Invitrogen,Carlsbad,CA)以及在Wisconsin遗传软件包(版本8)中的GAP、BESTFIT、BLAST、FASTA和TFASTA(可从Genetics Computer Group(GCG),575Science Drive,Madison,Wis.,USA获得)。使用这些程序进行比对时可采用默认参数。CLUSTAL程序详细描述于Higgins等人,Gene73:237-244(1988);Higgins等人,CABIOS 5:151-153(1989);Corpet等人,Nucleic Acids Res.16:10881-90(1988);Huang等人,CABIOS 8:155-65(1992)和Pearson等人,Meth.Mol.Biol.24:307-331(1994)。Altschul等人,J.Mol.Biol.215:403-410(1990)中的BLAST程序是基于Karlin和Altschul(1990)(如上)中的算法。
通过在严格或在高严格条件下相互杂交以鉴定与参考序列同源的多聚核苷酸。当单链多聚核苷酸基于各种良好表征的物理-化学力如氢键、溶剂排斥、碱基堆积等而相关联时,单链多聚核苷酸进行杂交。杂交的严格性反映了相关的核酸的序列同一性的程度,即严格性越高,两条多核苷酸链越相似。严格性受多种因素影响,包括温度、盐浓度和组成、有机和无机添加剂、溶剂等,这些因素存在于杂交和洗脱液和孵育(及其次数)中,在如下所引用的参考文献中进行了更详细地描述(例如,Sambrook等人,Molecular Cloning:A Laboratory Manual,第2版,Vol.1-3,Cold Spring Harbor Laboratory,Cold Spring Harbor,N.Y.("Sambrook")(1989);Berger和Kimmel,Guide to Molecular Cloning Techniques,Methods in Enzymology,vol.152Academic Press,Inc.,San Diego,Calif.("Berger和Kimmel")(1987);以及Anderson和Young,"Quantitative Filter Hybridisation.",Hames和Higgins编辑,Nucleic Acid Hybridisation,A Practical Approach.Oxford,TRL Press,73-111(1985))。
本公开所包括的是在各种严格性条件下能与所公开的多核苷酸序列和其片段杂交的多核苷酸序列(参见,例如,Wahl和Berger,Methods Enzymol.152:399-407(1987);以及Kimmel,Methods Enzymo.152:507-511,(1987))。本公开的多核苷酸的全长cDNA、同源物、直系同源物和旁系同源物可以使用公知的聚核苷酸杂交方法进行鉴定和分离。
表达多核苷酸的载体
本公开的各个多核苷酸可以并入表达载体中。“表达载体”或“载体”是指一种化合物和/或组合物,所述化合物和/或组合物转导、转换或感染宿主细胞,从而导致细胞可以表达原生细胞以外的或以非原生细胞的方式表达多核苷酸和/或蛋白质。“表达载体”包含由宿主细胞表达的多核苷酸序列(普通RNA或DNA)。任选地,表达载体还包含辅助多核苷酸进入宿主细胞的物质,如病毒、脂质体、蛋白包被等。预期用于本公开的表达载体包括那些能插入多核苷酸序列,连同任何优选或所需的操作元件的载体。此外,表达载体必须能被转移到宿主细胞中并在其中复制。优选的表达载体是质粒,尤其是具有已记录的限制性位点并包含优选或所需的用于多核苷酸序列转录的操作元件的质粒。此类质粒以及其它的表达载体在本领域中是公知的。
单个多核苷酸的并入是通过已知方法来完成,例如,使用限制性酶(例如BamHI、EcoRI、HhaI、XhoI、XmaI等)以裂解表达载体(例如质粒)中的特定位点。限制性酶产生单链末端,其可以使多核苷酸退火或合成以具有与所裂解的表达载体的末端序列互补的终端。使用合适的酶(如DNA连接酶)进行退火。如本领域技术人员所意识到的,常常用相同的限制性酶裂解表达载体和所需的多核苷酸,从而确保表达载体的末端和多核苷酸的末端是相互互补的。此外,可以用DNA连接子更容易地将多核苷酸序列连接到表达载体中。
也可以利用本领域已知的方法(例如美国专利4,683,195)组合一系列单个的多核苷酸。例如,在单独的PCR中,可以首先生成所需的每一种多核苷酸。然后,设计特异引物,使PCR产物的末端包含互补序列。当将PCR产物混合、变性和再退火时,具有匹配序列的链在其3’末端处重叠并充当相互的引物。由DNA聚合酶产生的这一重叠的延伸产生了一种分子,其中在所述分子中,原始序列被“拼接”在一起。用这一方式,可以使一系列单独的多核苷酸“拼接”在一起,并随后同时转导至宿主细胞中。因此,可以影响多个多核苷酸中的每一个的表达。
然后将单个多核苷酸或“拼接的”多核苷酸并入表达载体中。本公开并不限制多核苷酸被并入表达载体的过程。本领域技术人员很熟悉将多核苷酸并入表达载体的必要步骤。典型的表达载体包含所需的多核苷酸,在此之前有一个或多个调节区域,连同核蛋白体结合位点,例如:长度为3-9个核苷酸的核苷酸序列,位于大肠杆菌起始密码子上游的3-11核苷酸。参见Shine和Dalgarno(1975)Nature 254(5495):34-38以及Steitz(1979)Biological Regulation and Development(Goldberger,R.F.编辑),1:349-399(Plenum,New York)。
本文使用的术语“操作性连接”指的是将控制序列置于相对于DNA序列或多核苷酸的编码序列的合适位置上,使得控制序列引导多肽的表达。
调节区域包括,例如那些包含启动子和操作子的区域。启动子可以操作性连接到所需的多核苷酸上,从而通过RNA聚合酶启动多核苷酸的转录。操作子是毗邻启动子的多核苷酸序列,包含蛋白结合结构域(可以结合阻遏蛋白)。在缺少阻遏蛋白时,通过启动子启动转录。当存在阻遏蛋白时,特异于操纵子的蛋白结合结构域的阻遏蛋白与操纵子结合,从而抑制转录。通过这种方式,根据所用的特定调节区域和相应阻遏蛋白的存在与否,实现控制转录。实例包括乳糖启动子(当与乳糖接触时,Lad阻遏蛋白改变构象,从而阻止Lad阻遏蛋白与操纵子相结合)和色氨酸启动子(当与色氨酸结合时,TrpR阻遏蛋白具有与操纵子结合的构象;当缺少色氨酸时,TrpR阻遏蛋白具有未与操纵子结合的构象)。另一个实例是tac启动子(参见de Boer等人,(1983)Proc Natl Acad Sci USA 80(1):21-25)。
本公开制备宿主细胞的方法可包括将含有重组核酸的表达载体导入或转移至宿主细胞。对于本领域技术人员而言,此类将表达载体转移至宿主细胞中的方法是公知的。例如,一种用于转化有表达载体的细胞的方法包括氯化钙处理,通过钙沉淀导入表达载体。其它的盐,例如磷酸氢钙,也可以按照类似过程使用。此外,也可以用电穿孔转染法(即用电流提高细胞对核酸序列的渗透性)转染宿主细胞。也可以使用原生质球转化细胞(Schweizer,M,Proc.Natl.Acad.Sci.,78:5086-5090(1981))。微注射核酸序列也提供了转染宿主细胞的能力。也可以使用其它方法,例如脂质复合体、脂质体和树枝状分子。本领域技术人员能使用这些或其它方法用所需序列转染宿主细胞。
在某些情况下,在转化前,将细胞制备成原生质体或原生质球。可以通过例如用酶处理具有细胞壁的细胞以降解细胞壁。也可以例如用几丁质酶处理真菌细胞来制备原生质体或原生质球。
载体可以是自主复制的载体,即载体,作为染色体外实体存在,其复制过程独立于染色体的复制,例如质粒、染色体外元件、微型染色体或人造染色体。载体可包含确保自主复制的任何手段。或者,当把载体导入宿主细胞时,载体可被整合进基因组,并与将其整合的染色体一起复制。此外,也可以将一起包含全部DNA的单个载体或质粒或者两个或更多个载体或质粒导入宿主的基因组中,或使用转座子。
载体优选包含一个或多个选择标记,便于筛选转化的宿主细胞。选择标记是一种基因,其产物提供了例如杀生物剂或病毒的抗性、重金属抗性、使原养型向营养缺陷型转变等。细菌细胞的选择可以基于已知抗微生物抗性的基因,如amp、gpt、neo和hyg基因。
用于真菌宿主细胞的选择标记包括,例如amdS(乙酰胺酶)、argB(鸟氨酸氨甲酰基转移酶)、bar(草丁膦乙酰转移酶)、hph(潮霉素磷酸转移酶)、niaD(硝酸还原酶)、pyrU(乳清酸核苷-5’-磷酸基脱羧酶)、sC(硫酸腺苷酰基转移酶)和trpC(邻氨基苯甲酸合酶),及其等同物。
载体可包含允许载体整合至宿主的基因组或允许载体独立于基因组在细胞中自主复制的元件。
为整合进宿主基因组中,载体可依赖基因序列或任何其它的载体元件,其用于通过同源或非同源重组将载体整合进基因组。或者,载体可包含另外的核苷酸序列,其用于通过同源重组将整合导入进宿主的基因组中。所述另外的核苷酸序列能把载体整合至宿主基因组里的精确位置上。为了提高整合至精确位置的概率,整合元件应包含足够数量的核酸,如:100至10000个碱基对、400至10000个碱基对或800至10000个碱基对,其与对应的靶序列高度同源以提高同源重组的概率。整合元件可以是与宿主基因组中的靶序列同源的任何序列。此外,整合元件可以是非编码或编码的核苷酸序列。另一方面,载体可通过非同源重组整合到宿主的基因组中。
对于自主复制,载体还可以包含复制起点,使载体能够在宿主内自主复制。复制起点可以是细胞内介导自主复制功能的任何质粒复制因子。术语“复制起点”或“质粒复制因子”在本文中定义为使质粒或载体在体内复制的序列。
在载体中,各种调节本公开的重组核酸的表达的启动子是本领域公知的,并且包括例如组成型启动子和诱导型启动子。启动子描述于,例如Sambrook等人,Molecular Cloning:A Laboratory Manual,第3版,Cold Spring Harbor Laboratory Press,(2001)。启动子可以是病毒、细菌、真菌、哺乳动物或植物启动子。此外,启动子可以是组成型启动子、诱导型启动子、环境调控型启动子或发育调控型启动子。用于调节本公开的重组核酸的启动子的合适的实例包括,例如粗糙脉孢菌ccg-1组成型启动子,其响应昼夜节律和养分条件;粗糙脉孢菌gpd-1(甘油醛-3-磷酸脱氢酶)强组成型启动子;粗糙脉孢菌vvd(光)诱导型启动子;粗糙脉孢菌qa-2(奎尼酸)诱导型启动子;构巢曲霉(Aspergillus nidulans)gpdA启动子;构巢曲霉trpC组成型启动子;粗糙脉孢菌tef-1(转录延伸因子)高组成型启动子;以及粗糙脉孢菌xlr-1(XlnR同源)启动子,其在曲霉菌中经常使用。在一些实施方案中,本公开的重组多肽的表达受异源启动子控制。在一些优选的实施方案中,启动子是TEF1启动子以实现多肽的过表达。
可以将一个以上的基因拷贝插入到宿主中以增加基因产物的生成。可以通过以下来获得基因拷贝数的增加:将至少一个额外的基因拷贝整合至宿主基因组中,或通过包含伴随核苷酸序列的可扩增的选择标记基因,其中可以通过在存在合适的选择试剂的情况下培养细胞来选择含有扩增的选择标记基因拷贝以及由此含有额外的基因拷贝的细胞。
用以连接上述元件以构建本公开的重组表达载体的过程,对于本领域技术人员而言是公知的(参见,例如Sambrook等人,1989,如上)。当仅用单个表达载体时(没有添加中间体),载体将包含所有必需的核酸序列。
具有修饰的多肽活性的宿主细胞
具有修饰的多肽活性的本公开的重组宿主细胞能产生糖酸(如纤维二糖酸盐)以及来自纤维素或纤维素生物质的通用化学品。代谢纤维素的能力是木质纤维素降解细胞所表现出的特性,因此,本公开的宿主细胞优选是木质纤维素降解细胞。如本文所公开,木质纤维素降解细胞可以是需氧细胞或厌氧细胞。在某些优选的实施方案中,木质纤维素降解细胞是需氧细胞。
本公开的木质纤维素降解细胞产生降解木质纤维素或其组分的纤维素酶。在需氧(即富氧)或厌氧(即氧气不足)的条件下,木质纤维素降解细胞可以降解木质纤维素或其组分。在某些实施方案中,本公开的木质纤维素降解细胞能预处理木质纤维素生物质。此类木质纤维素降解细胞能同时降解木质素、溶解木质素,或将木质素变成修饰形式如脱甲基化木质素。木质素是高能化合物,其可用于产生能量(例如电能)。在其它实施方案中,本公开的木质纤维素降解细胞产生一种或多种纤维素酶、半纤维酶、木质素溶解酶或其组合。在某些实施方案中,一种或多种半纤维素酶和/或木质素溶解酶在木质纤维素细胞中重组表达。因此,本公开的木质纤维素降解细胞能从木质纤维素生物质中产生单糖(如葡萄糖)和纤维糊精(如纤维二糖、纤维三塘、纤维四糖、纤维五糖等)。此外,本公开的木质纤维素降解细胞也能从木质纤维素生物质中产生半纤维素寡糖,如木二糖。
本公开的木质纤维素降解细胞可包括,例如真菌和细菌。本公开的适合的木质纤维素真菌可包括,例如白腐菌、褐腐菌、软腐菌和子囊菌纲真菌。本公开的适合的木质纤维素细菌可包括,例如梭状芽胞杆菌(Clostridium sp.)和Thermanaerobacterium sp.。适合的宿主细胞的其它实例可包括,例如里氏木霉(Trichoderma reesei)、热纤维梭菌(Clostridium thermocellum)、巴氏梭状芽胞杆菌C7(Clostridium papyrosolvens C7)、Podospera anserine、球毛壳菌(Chaetomium globosum)、构巢曲霉(Aspergillus nidulans)、黑曲霉(Aspergillus niger)、米曲霉(Aspergillus oryzae)、酿酒酵母(Saccharomyces cerevisiae)、裂殖酵母菌(Schizosaccharomyces pombe)、黄孢原毛平革菌(Phanerochaete chrysosporium)、嗜热侧孢霉(Sporotrichum thermophile)(嗜热毁丝霉(Myceliophthora thermophila))、玉米赤霉(Gibberella zeae)、核盘菌(Sclerotinia sclerotiorum)、孢盘菌(Botryotinia fuckelian)、黑曲霉(Aspergillus niger)、太瑞斯梭孢壳霉(Thielavia terrestris)、镰孢菌(Fusarium spp.)、根霉(Rhizopus spp.)、新美鞭菌(Neocallimastix frontalis)、根囊鞭菌(Orpinomyces sp.)、梨囊鞭菌(Piromyces sp.)、青霉菌(Penicillium chrysogenum)细胞、裂褶菌(Schizophyllum commune)、褐腐菌(Postia placenta)、纤维素枝顶抱霉(Acremonium cellulolyticus)、解脂耶氏酵母(Yarrowia lipolytica)、多形汉逊酵母(Hansenula polymorpha)、乳酸克鲁维酵母(Kluyveromyces lactis)、毕赤酵母(Pichia pastoris)、金孢子菌(Chrysosporium lucknowense)、曲霉菌(Aspergillus sp.)、木霉菌(Trichoderma sp.)、Caldocellulosiruptor sp.、丁酸弧菌(Butyrivibrio sp.)、丁酸弧菌(Butyrivibrio sp.)、真杆菌(Eubacterium sp.)、梭状芽胞杆菌(Clostridium sp.)、拟杆菌(Bacteroides sp.)、醋孤菌(Acetivibrio sp.)、高温放线菌(Thermoactinomyces sp.)、Caldibacillus sp.、芽孢杆菌(Bacillus sp.)、热酸菌(Acidothermus sp.)、纤维菌(Cellulomonas sp.)、小单孢菌(Micromonospora sp.)、游动放线菌(Actinoplanes sp.)、链霉菌(Streptomyces sp.)、温双岐菌(Thermobifida sp.)、高温单孢菌(Thermomonospora sp.)、小双孢菌(Microbispora sp.)、纤维杆菌(Fibrobacter sp.)、生孢噬胞菌(Sporocytophaga sp.)、噬细胞菌(Cytophaga sp.)、黄杆菌(Flavobacterium sp.)、无色菌(Achromobacter sp.)、黄单胞菌(Xanthomonas sp.)、纤维孤菌(Cellvibrio sp.)、假单胞菌(Pseudomonas sp.)、黏球菌(Myxobacter sp.)、Clostridium phytofermentans、Clostridium japonicas和厌氧嗜热杆菌(Thermoanaerobacterium saccharolyticum)细胞。
在某些实施方案中,本公开的宿主细胞是丝状真菌细胞,其包括例如脉孢菌(Neurospora)细胞、木霉菌(Trichoderma)细胞和曲霉菌(Aspergillus)细胞。在某些优选的实施方案中,丝状真菌细胞是粗糙脉孢菌细胞。
本公开的宿主细胞是活的生物细胞,其被操作以改变例如细胞内一种或多种多肽的活性。比如,通过插入重组DNA或RNA可转化宿主细胞。此类重组DNA或RNA可以在表达载体中。此外,宿主细胞可受到突变以诱发编码多肽的多核苷酸中的突变。被基因修饰的宿主细胞是重组宿主细胞。
本公开的宿主细胞可以是基因修饰的。例如,可以将重组核酸引入宿主细胞中或宿主细胞可以具有引入内源和/或外源多核苷酸的突变,因此基因修饰的宿主细胞不会在自然界中产生。合适的宿主细胞可以是,例如能够表达用于不同功能(例如重组蛋白表达和/或靶向基因沉默)的一种或多种核酸构建体的宿主细胞。
本文使用的“重组核酸”或“异源核酸”或“重组多核苷酸”,“重组核苷酸”或“重组DNA”指的是一种核酸的聚合物,其至少是下列之一:(a)核酸序列对于给定的宿主细胞是外源的(即,不是自然存在的);(b)序列可能在给定的宿主细胞中自然存在,但以非正常的数量表达(例如比预期高);或(c)核酸序列包含两种或更多种在天然相互的相同关系中不存在的子序列。例如,对于例子(c)而言,重组的核酸序列将有两种或更多种来自不相关基因的序列,排列形成新功能的核酸。例如,本公开描述了将表达载体导入宿主细胞中,表达载体包含编码在宿主细胞中不是天然存在的蛋白质的核酸序列,或包含编码在细胞中天然存在但受不同调节序列控制的蛋白质的核酸。然后,参照宿主细胞的基因组,重组编码蛋白质的核酸序列。本文使用的术语“重组的多肽”指的是由如上所述的“重组核酸”或“异源核酸”或“重组多核苷酸”、“重组核苷”或“重组DNA”生成的多肽。
在一些实施方案中,宿主细胞天然产生本公开的任一多肽。在一些实施方案中,编码所需多肽的基因可以对于宿主细胞是异源的,或者这些基因可以对于宿主细胞是内源的但可操作地连接到异源启动子和/或控制区,导致例如在宿主细胞中基因表达更高或在宿主细胞中基因表达降低。
在一些实施方案中,本公开的宿主细胞可以被修饰以促进纤维素的代谢。例如,本公开的宿主细胞可以被修饰以含有一种或多种纤维糊精转运体。纤维糊精是不同长度的葡萄糖聚合物,包括例如纤维二糖(2个葡萄糖单体)、纤维三糖(3个葡萄糖单体)、纤维四糖(4个葡萄糖单体)、纤维五糖(5个葡萄糖单体)和纤维六糖(6个葡萄糖单体)。纤维糊精转运体是将纤维糊精分子从细胞外部运输到细胞内部和/或从细胞内部运输到细胞外部的任何跨膜蛋白。合适的纤维糊精转运体的实例可包括例如NCU00801、NCU00809、NCU8114、XP_001268541.1和LAC2。
修饰多肽活性的方法
与相应的对照细胞,例如相应的野生型细胞相比,本公开的宿主细胞具有修饰的多肽活性,以促进和/或增加糖酸的产生。多肽的活性可以被修饰,使得本公开的一种或多种多肽具有增加的活性或降低的活性。在一些实施方案中,宿主细胞可具有一种或多种具有增加的活性的多肽以及一种或多种具有降低的活性的多肽。修饰(例如增加和/或降低)本公开的一种或多种多肽的活性的方法是本领域公知的并且在本文中描述。降低的多肽活性
与相应的对照细胞例如野生型细胞相比,本公开的宿主细胞可以含有具有降低的活性的一种或多种多肽。在一些实施方案中,与相应的对照细胞相比,一种或多种的β-葡萄糖苷酶、纤维二糖酸盐磷酸化酶、CRE-1蛋白、ACE-1蛋白和/或MUS51蛋白在宿主细胞中具有降低的活性。降低多肽的表达、丰度和/或活性的方法是本领域公知的并且在本文中描述。
在一些实施方案中,降低多肽的活性涉及过表达作为多肽抑制剂的多肽。宿主细胞可过表达抑制本公开的一种或多种的β-葡萄糖苷酶、纤维二糖酸盐磷酸化酶、CRE-1蛋白、ACE-1蛋白和/或MUS51蛋白的表达和/或活性的抑制剂。在一些实施方案中,可以在宿主细胞中表达重组的β-葡萄糖苷酶、纤维二糖酸盐磷酸化酶、CRE-1蛋白、ACE-1蛋白和/或MUS51蛋白,使得重组的多肽干扰并降低内源多肽的活性。
在一些实施方案中,降低多肽(例如一种或多种的β-葡萄糖苷酶、纤维二糖酸盐磷酸化酶、CRE-1蛋白、ACE-1蛋白和/或MUS51蛋白)的活性包括降低编码多肽的核酸的表达。
通过将基因突变导入靶核酸中,可以实现核酸表达的降低。可以通过产生突变使用诱变的方法来破坏或“敲除”靶基因的表达。在一些实施方案中,诱变导致靶基因的部分缺失。在其它实施方案中,诱变导致靶基因的完全缺失。诱变微生物的方法是本领域公知的并且包括例如随机诱变和定点诱变以诱导突变。随机诱变的方法的实例包括,例如化学诱变(例如使用乙磺酸甲酯)、插入诱变和照射。
减少或抑制靶基因表达的一种方法是通过基因修饰靶基因并将其导入宿主细胞的基因组以通过同源重组取代野生型版本的基因(例如,如美国专利6,924,146中所述)。
另一种减少或抑制靶基因表达的方法是使用农杆菌(Agrobacterium tumefaciens)的T-DNA或转座子的插入诱变(参见Winkler等人,Methods Mol.Biol.82:129-136,1989和Martienssen Proc.Nati.Acad.Sci.95:2021-2026,1998)。产生插入突变后,可以筛选突变来鉴定那些在靶基因中含有插入的突变。通过插入诱变破坏靶基因的方法描述于例如美国专利5,792,633。通过转座子诱变破坏靶基因的方法描述于美国专利6,207,384。
另一种破坏靶基因的方法是使用cre-lox体系(例如,如美国专利4,959,317中所述)。
另一种破坏靶基因的方法是使用PCR诱变(例如,如美国专利7,501,275中所述)。
通过RNA干扰(RNAi)的方式也可减弱或抑制内源性基因表达,其利用具有与靶基因序列相同或相似序列的双链RNA。RNAi可包括使用微RNA(如人工miRNA)以抑制基因表达。
RNAi是当具有与靶基因序列相同或相似的序列的双链RNA被导入细胞时的一种现象,抑制插入的外源基因和内源靶基因的表达。双链RNA可以由两条独立的互补RNA形成,或由具有内部互补序列的单个RNA形成的双链RNA。
因此,在一些实施方案中,使用RNAi技术实现基因表达的降低或抑制。例如,为了使用RNAi实现编码蛋白质的DNA的表达的降低或抑制,将具有编码蛋白质的DNA的序列或具有基本上与其相似的序列的双链RNA(包括被设计为不翻译蛋白质的那些)或其片段引入感兴趣的宿主细胞。如本文所用,RNAi和dsRNA均指通过引入双链RNA分子(包括提及的具有双链区的分子,例如短发夹RNA分子)诱导的基因特异性沉默,参见例如,美国专利6,506,559和6,573,099。然后可以对所获得的细胞筛选与靶基因表达降低相关的表型,例如降低的纤维素酶表达,和/或通过监测靶基因转录物的稳态RNA水平。尽管用于RNAi的序列不需要与靶基因完全相同,其可以是与靶基因序列具有至少70%、80%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更多的同一性。参见,例如美国专利申请公开2004/0029283。编码具有与靶基因无关的茎-环结构并将其置于特异于感兴趣的基因的序列的远端的RNA分子的构建体也可用于抑制靶基因的表达。参见例如美国专利申请公开2003/0221211。
RNAi核酸可以包括全长靶RNA或对应于靶RNA的片段。在一些情况下,片段将具有少于对应于靶序列的100、200、300、400或500个核苷酸。此外,在一些方面,这些片段的长度为至少例如50、100、150、200或更多个核苷酸。干扰RNA可基于短双链体(即,双链序列的短区)进行设计。通常,短双链体的长度为至少约15、20或25-50个核苷酸(例如,双链RNA的每个互补序列的长度为15-50个核苷酸),通常约20-30个核苷酸,例如20、21、22、23、24、25、26、27、28、29或30个核苷酸。在一些情况下,用于RNAi的片段将对应于不存在于生物体中的其它蛋白质中或与生物体中的其它转录物几乎没有相似性的靶蛋白质区域,例如分析公开可用的序列数据库,通过比较序列进行选择。类似地,可以选择与感兴趣的基因家族的保守序列(例如本文所述的那些)有相似性或同一性的RNAi片段,使得RNAi靶向于含有保守序列的多种不同的基因转录物。
可以将RNAi作为较大DNA构建体的一部分引入宿主细胞。通常,此类构建体允许RNAi在引入后在细胞中稳定表达,例如通过将构建体整合到宿主基因组中。因此,在转染有载体的细胞中连续表达RNAi的表达载体可用于本公开。例如,可以使用表达小发夹或茎-环结构RNA或微小RNA前体的载体,其在体内加工成能够进行基因特异性沉默的小RNAi分子(Brummelkamp等人,Science 296:550-553,(2002);和Paddison等人,Genes&Dev.16:948-958,(2002))。通过双链RNA进行的转录后基因沉默进一步描述于Hammond等人,Nature Rev Gen 2:110-119,(2001);Fire等人,Nature 391:806-811,(1998)以及Timmons和Fire,Nature 395:854,(1998)。
用于选择和设计产生RNAi的序列的方法是本领域公知的(例如美国专利6,506,559、6,511,824和6,489,127)。
还可以通过将基于靶基因核酸序列的反义构建体引入宿主细胞中来获得靶基因在宿主细胞中的基因表达的减少或抑制。对于反义抑制,靶序列相对于表达载体中的启动子序列以相反方向排列。引入的序列不需要是全长cDNA或基因,也不需要与靶cDNA或待转化细胞中发现的基因相同。然而,通常,当引入的序列具有较短的长度时,使用与天然靶序列更高程度的同源性以实现有效的反义抑制。在一些方面,载体中引入的反义序列的长度为至少30个核苷酸,并且通常在反义序列的长度增加时观察到改善的反义抑制。在一些方面,载体中反义序列的长度将大于100个核苷酸。所述的反义构建体的转录导致RNA分子的产生,所述RNA分子是从内源靶基因转录的mRNA分子的反向互补序列。靶基因表达的抑制也可以使用核酶来实现。核酶的制备和使用公开于美国专利4,987,071和5,543,508中。
可使用本领域公知的方法构建表达框,所述表达框包含编码靶基因表达抑制剂的核酸,例如反义RNA或siRNA。构建体包含调节元件,调节元件包含启动子和其它用于表达和选择表达有构建体的细胞的序列。通常,真菌和/或细菌的转换载体包括受5’和3’调节序列转录控制的一个或多个克隆的编码序列(基因组或cDNA)以及显性选择标记。此类转换载体通常也包含启动子(例如控制诱导型或组成型、环境调节型或发育调节型表达的调控区)、转录起始位点、RNA处理信号(例如内含子剪接位点)、转录终止位点和/或聚腺苷酸化信号。
在某些实施方案中,可以修饰靶核酸的一部分,例如编码催化结构域的区域、编码区或需要表达编码区的控制序列。此类基因的控制序列可以是启动子序列或其功能部分,即足以影响基因表达的部分序列。例如,启动子序列可以是被灭活的,导致没有表达,或者较弱的启动子可以替代天然启动子序列以降低编码序列的表达。其它用于可能的修饰的控制序列包括,例如前导序列、前肽序列、信号序列、转录终止子和转录激活子。
增加的多肽活性
与相应的对照细胞,例如相应的野生型细胞相比,本公开的宿主细胞可以含有一种或多种具有增加的表达和/或活性的多肽。在一些实施方案中,本公开的宿主细胞含有编码本公开的重组多肽(例如重组漆酶和/或重组纤维二糖脱氢酶)的重组核酸。在某些实施方案中,重组核酸在宿主细胞中错误表达(例如组成型表达、诱导型表达等),使得与相应的对照细胞相比,错误表达导致增加的多肽活性。在一些实施方案中,含有编码重组多肽的重组核酸的宿主细胞含有比不含相应的重组核酸的相应对照细胞更多数量的多肽。当蛋白质或核酸以高于正常量的量在宿主细胞中产生或维持时,蛋白质或核酸是“过表达的”。在一些实施方案中,本公开的宿主细胞过表达多肽,例如漆酶和/或纤维二糖脱氢酶。宿主细胞可过表达一种或多种的漆酶和/或纤维二糖脱氢酶多肽,使得与相应的对照细胞相比,宿主细胞中这些蛋白质中的一种或多种的活性增加。相应的对照细胞可以是,例如未过表达在宿主细胞中过表达的一种或多种多肽的细胞,例如野生型细胞。各种对照细胞对于本领域技术人员而言是显而易见的。
各种增强多肽表达的方法是本领域公知的。例如,可以修饰与控制编码多肽的核酸的表达相关的其它基因区(例如增强子序列),使得增强核酸的表达。可以通过检测由基因编码的mRNA的水平和/或通过检测由核酸编码的多肽的水平或活性来评估核酸的表达水平。
在一些实施方案中,宿主细胞过表达多肽,所述多肽是漆酶和/或纤维二糖脱氢酶多肽中的一种或多种的激活剂。激活剂多肽的过表达可导致由激活剂激活的多肽的丰度和活性增加。激活剂可以增强漆酶和/或纤维二糖脱氢酶多肽中的一种或多种的表达。激活剂可以增强漆酶和/或纤维二糖脱氢酶多肽中的一种或多种的活性。
可以通过过表达多肽来实现本公开的多肽(例如漆酶和/或纤维二糖脱氢酶多肽)的丰度的增加以增加多肽活性。其它增加多肽丰度的方法是本领域已知的。例如,通过细胞降解机制(如蛋白酶体)降低多肽的降解可增加多肽的稳定性和丰度。可基因修饰多肽,使得其对细胞蛋白酶解的抗性增加,但在分子活性上没有变化。多肽是与漆酶和/或纤维二糖脱氢酶多肽中的一种或多种的降解相关的细胞因子的抑制剂,可以将所述多肽导入宿主细胞以增加一种或多种多肽的丰度。此外,可以用蛋白酶体的化学抑制剂(如环己酰亚胺)处理宿主细胞以增加本公开的一种或多种多肽的丰度。
生产糖酸的方法
本公开的具有修饰的多肽活性的宿主细胞能从纤维素或纤维素材料中产生糖酸,例如纤维寡糖醛酸盐(cellooligosaccharide aldonate)。本公开的宿主细胞能代谢纤维素(木质纤维素降解细胞),也能根据所存在的促纤维素代谢的内源基因机制来代谢纤维素,或被基因工程化以代谢纤维素。因此,本公开的宿主细胞将表达一种或多种的下列酶:纤维素酶、木聚糖酶、木质酶、氧化酶、脱氢酶和漆酶。一旦分解纤维素产生了产物(如纤维二糖),本公开的宿主细胞可以利用这些分解产物产生糖酸。
本公开的方法涉及使用具有修饰的多肽活性的宿主细胞以产生糖酸(如纤维二糖酸盐)以及来自纤维素或纤维素生物质的通用化学品。糖酸,也称为糖醛酸(SAA),是指分子内糖的CHO醛基官能团被羧酸基官能团(COOH)取代。可以将糖酸划分为四大类:(1)寡糖醛酸(OAA)、(2)二糖醛酸(DAA)、(3)单糖醛酸(MAA)和(4)杂多糖醛酸(HSAA)。寡糖醛酸(OAA)的实例包括:纤维三糖酸、纤维四糖酸、纤维庚糖酸、木糖三糖酸和木糖五糖酸。二糖醛酸(DAA)的实例包括:纤维二糖酸(CBA)、木糖二糖酸、半乳糖酸、4-O-β-D-吡喃半乳糖基葡萄糖酸和6-O-β-D-吡喃半乳糖基葡萄糖酸。单糖醛酸(MAA)的实例包括葡萄糖酸、木糖酸、半乳糖酸、阿糖酸和甘露糖酸。HSAA的实例是4-O-甲基-α-D-葡萄糖醛酸吡喃糖酸。
在DAA、OAA和HSAA的情况下,糖单元之间以及糖与醛酸末端之间的连接可以是直链或支链。例如,葡萄糖酸可以糖基连接在糖单元的1-、3-、4-或6-位的氧原子上。可以是糖和末端醛酸的任意组合。
糖酸通常以盐形式存在于水溶液中。无机盐和有机盐的实例是胺盐、锂盐、钠盐、镁盐、钙盐和铝盐,以及乙醇胺盐、三乙醇胺盐、吗啉盐、吡啶盐和哌啶盐。
本公开的方法包括在含有纤维素的培养基中,培养本公开的宿主细胞,其中宿主细胞产生糖酸,糖酸来自纤维素。在适合于通过宿主细胞从纤维素中产生糖酸的条件下,在生长培养基中培养本公开的宿主细胞,例如具有降低活性的一种或多种的β葡萄糖苷酶、纤维二糖酸盐磷酸化酶、CRE-1蛋白、ACE-1蛋白和/或MUS51蛋白的宿主细胞,和/或具有一种或多种的漆酶和/或纤维二糖脱氢酶的活性或表达增加的宿主细胞。
在生长/培养基中,本公开的宿主细胞能利用纤维素作为碳源。根据本公开的一些方面,培养基包含作为宿主细胞碳源的纤维素或纤维素源。“碳源”通常是指适合用作细胞生长碳源的底物或化合物,例如纤维素或纤维素源。在一些实施方案中,培养基仅包含作为唯一碳源的纤维素或纤维素源。在其它实施方案中,培养基可包含除纤维素以外的其它碳源。碳源可以以不同形式存在,例如聚合物、碳水化合物、酸、醇、醛、酮、氨基酸、多肽等。这包括例如各种单糖、寡糖、多糖、生物质聚合物如纤维素或半纤维素、木糖、阿拉伯糖、二糖如蔗糖、饱和或不饱和脂肪酸、琥珀酸盐、乳酸盐、乙酸盐、乙醇等或其混合物。除了合适的碳源之外,培养基可含有合适的矿物质、盐、辅因子、缓冲液和本领域技术人员已知的适合于培养物生长和促进参与糖酸生产的途径的其它组分。如本公开的方法中使用的微生物标准培养环境是本领域公知的并且在本文中描述。
纤维素源是本领域公知的并且在本文中描述。例如,可以商业购买纤维素,并作为碳源将其加入培养液中。纤维素源可以是包含纤维素的物质或分解释放纤维素的物质。如本文所使用,“包含纤维素的物质”是包含或能产生纤维素(包括生物质,如包含植物物质的生物质)的任何原料。适用于目前公开方法使用的生物质包括各种包含纤维素的物质,例如芒草、柳枝稷、大米草、黑麦草、利甘草、象草、普通芦苇、小麦秸秆、大麦秸秆、油菜秸秆、燕麦秸秆、玉米秆、大豆秆、燕麦壳、高粱、稻皮、黑麦皮、小麦皮、甘蔗渣、椰子核粉、椰子核球、棕榈仁粉、玉米纤维、干谷物酒糟及其可溶物(DDGS)、须芒草、玉米穗轴、松木、桦木、柳木、山杨木、白杨木、能源型甘蔗、废纸、锯末、林业废弃物、城市固体垃圾、废纸、农作物残留物、其它草和其它树木。纤维素源可能需要预处理以生成和/或释放纤维素或纤维素分解产物,例如用高温或高压处理。此类处理对本领域技术人员而言是公知的。
在一些实施方案中,在外源添加漆酶和/或纤维二糖脱氢酶的情况下培养本公开的宿主细胞。可以从商业供应商购买这些酶,或通过本领域技术人员易于理解的方法,将其重组表达并纯化。在一些实施方案中,例如将外源漆酶加入到培养基中的那些实施方案中,培养基可以进一步含有作为氧化还原介体的化合物,例如ABTS(2,2'-连氮基-双(3-乙基苯并噻唑啉-6-磺酸))。
在一些实施方案中,在本公开的宿主细胞产生糖酸之后,将糖酸沉淀、移除或从培养基中高度纯化。从培养基中高度纯化糖酸的方法是本领域已知的。
在一些实施方案中,本公开的方法涉及与相应的对照细胞相比具有增加的糖酸产率的宿主细胞。具有增加的糖酸产率的宿主细胞可以是那些降低了一种或多种的β-葡萄糖苷酶、纤维二糖酸盐磷酸化酶、CRE-1蛋白、ACE-1蛋白和/或MUS51蛋白的活性的宿主细胞,和/或增加一种或多种的漆酶和/或纤维二糖脱氢酶的表达或活性的宿主细胞。本公开的宿主细胞中,糖酸的产率可以是比相应的对照细胞的糖酸产率高至少0.1倍、至少0.2倍、至少0.3倍、至少0.4倍、至少0.5倍、至少0.6倍、至少0.7倍、至少0.8倍、至少0.9倍、至少1倍、至少1.25倍、至少1.5倍、至少1.75倍、至少2倍、至少2.25倍、至少2.5倍、至少2.75倍、至少3倍、至少3.25倍、至少3.5倍、至少3.75倍、至少4倍、至少4.25倍、至少4.5倍、至少4.75倍、至少5倍、至少5.25倍、至少5.5倍、至少5.75倍、至少6倍、至少7倍、至少8倍、至少9倍、至少10倍、至少20倍或至少30倍或更高,对照细胞是例如野生型细胞或不具有修饰的多肽活性的细胞。在一些实施方案中,相应的对照细胞不积累任何糖酸。在一些实施方案中,由宿主细胞产生的糖酸是纤维二糖酸盐。
在一些实施方案中,本公开的方法涉及与相应的对照细胞相比具有降低的糖酸消耗率的宿主细胞。具有降低的糖酸消耗率的宿主细胞可能是那些具有降低的一种或多种的β-葡萄糖苷酶、纤维二糖酸盐磷酸化酶、CRE-1蛋白、ACE-1蛋白和/或MUS51蛋白的活性的宿主细胞,和/或增加的一种或多种的漆酶和/或纤维二糖脱氢酶的表达和活性的宿主细胞。本公开的宿主细胞的糖酸消耗与相应的对照细胞相比可以降低约5%、约10%、约15%、约20%、约25%、约30%、约35%、约40%、约45%、约50%、约55%、约60%、约65%、约70%、约75%、约80%、约85%、约90%、约95%、约99%或约100%,对照细胞是例如野生型细胞或不具有修饰的多肽活性的细胞。在一些实施方案中,与相应的对照细胞相比,本公开的宿主细胞的纤维二糖酸盐的消耗降低约100%。
测定宿主细胞或细胞群的糖酸累积、生产和/或消耗或降解率的方法是本领域已知的并在本文进行描述。例如,确定培养基中的细胞或细胞群的糖酸消耗或降解率可包括允许细胞在含有糖酸的培养基中生长,并随时间在特定时间点测量生长培养基中的糖酸消耗并确定结果线图的斜率以确定糖的消耗/降解率(例如纤维二糖酸盐/L/h)。类似地,确定糖酸累积或生产可包括在含有纤维素的培养基中培养细胞或细胞群,并随时间在特定时间点测量纤维素中的糖酸生产。
用本公开的方法产生的糖酸可用作通用化学品(如燃料或燃料添加剂)的生产原料。示例性的通用化学品包括例如酒精、乙醇、丙醇、异丙醇、丙酮、丁醇、异丁醇、2-甲基-1-丁醇、3-甲基-1-丁醇、苯乙醇、脂肪醇、异戊醇、醛、乙醛、丙醛、丁醛、异丁醛、2-甲基-1-丁醛、3-甲基-1-丁醛、苯乙醛、脂肪醛、碳水化合物、烷烃、烯烃、类异戊二烯、脂肪酸、蜡酯、乙酯、氢及其组合。
实施例
提供以下实施例以说明所提供的实施方案,并不意图限制本公开的范围。
实施例1:从纤维素直接生产糖酸和糖寡聚物的代谢途径
本实施例示出了直接从纤维素生产糖酸的途径。通过缺失β-葡萄糖苷酶基因的各种拷贝、过表达漆酶基因并缺失纤维二糖酸盐磷酸化酶基因来构建真菌菌株。工程化的真菌能直接从纤维素直接产生糖酸(例如纤维二糖酸盐)而不需要添加任何外源性纤维素酶。所生产的糖酸可用作生产各种燃料和化学品的起始原料。
材料与方法
材料
野生型粗糙脉孢菌(2489)获得自真菌遗传保藏中心(FGSC)。本研究中使用的F5菌株是菌株2489,其七个中的六个β-葡萄糖苷酶(bgl)基因被敲除。来自糙皮侧耳菌(Pleurotus ostreatus)(一种有效的漆酶生产者)的漆酶获得自Sigma Aldrich,并用于需要外源添加漆酶的任何研究中。
2,2'-连氮基-双(3-乙基苯并噻唑啉-6-磺酸)(ABTS)获得自Sigma Aldrich。对于需要外源性CDH的实验,由申请人开发的重组毕赤酵母(Pichia pastoris)菌株用于产生CDH(Zhang等人,2011)。
菌株构建
如前所述,使用靶基因缺失与标记回收的方法,构建菌株F5Δcre-1和F5Δacre 1(Szewczyk等人,2013)。由遗传杂交构建F5Δcre-1和F5Δacre1(Fan等人,2012)。
如前所述,使用靶基因缺失与标记回收所述的方法,构建纤维二糖酸盐磷酸化酶(NdvB)敲除(F5Δcre-1Δace1ΔndvB)(Szewczyk等人,2013)。
漆酶的过表达
合成包含侧翼为来自构巢曲霉的gpdA启动子和trpC终止子的天然漆酶基因(NCU04528;Genbank登记号#J03505)的敲入盒,并由Life Technologies验证序列。gpdA启动子的上游区域含有与粗糙脉孢菌csr-1基因的3'末端同源的1020bp序列,并且trpC终止子的下游区域包含与csr-1基因的5'末端同源的1900bp序列。csr-1基因赋予了对环孢菌素A的敏感性。通过同源重组用敲入盒替换csr-1基因使得菌株对环孢菌素A具有抗性,为同核突变体提供了选择方法。收获菌株F5Δcre-1Δace1ΔndvB的10日分生孢子,通过干纱布过滤,并用50mL的1M冰冷的山梨醇洗涤并离心三次。将分生孢子浓缩成2.5×109分生孢子/mL的浓度。将90μL的细胞与1000μg的DNA混合,并在电击(0.2cm电击杯;1.5kV/cm;电容:25uFD;阻力:600ohm)前冰浴5分钟。电击后,立即将750mL的1M的冰冷山梨醇加入杯中,并将悬浮液转移至2mL含有1×Vogel’s盐和2%酵母提取物的恢复培养基中。将细胞在30℃以及200rpm的旋转振荡器中再生6小时。将350μL的再生溶液铺在含有l×Vogel’s盐、2%酵母提取物、1M山梨醇、20g L-1山梨糖、0.5g L-1果糖、0.5g L-1葡萄糖、5μg mL-1CsA和2%琼脂的烧瓶中。在30℃生长4-7天后,将菌落转移至斜面培养基(5μg mL-1CsA、1×Vogel’s、2%琼脂)并在30℃生长4天。通过监测生长于具有20g/L葡萄糖的1×Vogel’s盐上的转化株的漆酶的胞外表达,选择阳性转化株。在此条件下,亲本菌株不产生任何可检测的漆酶。根据以前公布的方法(Baminger等人,2001),用ABTS吸光度的增加来测量漆酶的表达。将一个漆酶活性单位定义为每分钟氧化1mmol的ABTS的酶量。菌株被命名为标签F5Δcre-1Δace1ΔndvB+lac。
结果
申请人先前构建了七个粗糙脉孢菌菌株,其中七个β-葡萄糖苷酶(bgl)基因中的六个被敲除(六倍体bgl敲除菌株),以及构建了一个七倍体bgl敲除菌株(Fan等人,2012)(Wu等人,2013)。申请人示出了可以通过最好的菌株之一从纤维素中生产纤维二糖酸盐,这一菌株被命名为F5(Fan等人,2012)。F5在每个如下β-葡萄糖苷酶基因中含有突变:NCU00130、NCU04952、NCU05577、NCU07487、NCU08755和NCU03641。这些结果能在图6中重复。例如,图4A示出F5菌株(其是六倍体bgl突变菌株)能积累纤维二糖。图4B示出F5菌株还可积累纤维二糖酸盐,并且外源添加纤维二糖脱氢酶(CDH)可增加该菌株中所存在的纤维二糖酸盐的浓度。图5A和图5B示出仅糖酸以及糖加糖酸(葡萄糖和葡萄糖酸盐)可被转化为燃料和化学品(Fan等人,2012)。
此外,图6提供了纤维素转化和菌丝团生产的总结。野生型细胞(其保留完整的β-葡萄糖苷酶活性)不累积纤维二糖或纤维二糖酸盐。相比之下,具有或不具有CDH的F5菌株积累纤维二糖和纤维二糖酸盐,并以比野生型低的收率产生菌丝团。
用ABTS和漆酶将纤维二糖转化成纤维二糖酸盐
为了进一步研究纤维素分解微生物的糖酸生产,申请人生成了另外的基因修饰的粗糙脉孢菌菌株。选择F5菌株(如上所述)进行进一步修饰,将cre-1基因和ace-1基因中的突变导入F5菌株。cre基因是已知的分解代谢物抑制子,并且该基因的缺失已示出增加纤维素酶的表达。cre-菌株在优选的碳源上具有降低的生长速率;然而,在微晶纤维素中,其产生30-50%更多的纤维素酶且消耗微晶纤维素更快(Sun等人,2011),cre表达与纤维素酶表达相关。ace-1基因的突变(ace-)导致里氏木霉中纤维素酶的更高表达(Aro等人,2003)。
为了分析F5菌株和F5Δcre-lΔace菌株中的纤维二糖酸盐生产,使这些菌株在Vogel’s培养基+20g/L微晶纤维素中生长。在96小时,将0.8U/mL漆酶和0.1mM的ABTS添加至培养基中。如图7所示,纤维二糖在F5和F5Δcre-lΔace菌株中积累。在96小时添加外源性漆酶和ABTS时,纤维二糖酸盐开始累积,然纤维二糖浓度迅速下降。结果表明,在添加ABTS和漆酶的条件下,所有纤维二糖都被转化为纤维二糖酸盐。
如上所述,在F5和F5Δcre-1Δace菌株的生长培养基中添加ABTS和漆酶诱导培养基中的纤维二糖酸盐的积累,即培养基中的纤维二糖被转化成纤维二糖酸盐。不希望受理论束缚,可以认为漆酶参与了存在于这些菌株中的纤维二糖脱氢酶(CDH)的活性的再生。当黄素酶(例如CDH)氧化底物(例如纤维二糖)时,其从底物中提取两个电子,将底物转化成其相应的内酯,其自发水解成醛酸。为了使CDH恢复功能,其必须被氧化。氧是CDH的热力学上有利的电子受体;然而,这是极端速率限制的。漆酶是氧化还原酶,与CDH相反,氧化还原酶被氧有效地氧化,唯一的副产物是水。含FAD的酶纤维二糖脱氢酶(CDH;EC1.1.99.18)将还原糖部分的C-1位上的乳糖氧化成乳糖醛酸内酯,其自发水解成乳糖酸。在该反应中使用2,20-连氮基-双(3-乙基苯并噻唑啉-6-磺酸)(ABTS)的阳离子基团作为一种可能的电子受体(氧化还原介体),并且被漆酶(LAC;EC1.10.3.2)、多铜氧化酶连续再氧化(van Hecke等人,2009)。
申请人进一步在F5和F5Δcre-1Δace菌株中研究纤维二糖酸盐的积累。图8A和图8B示出了这些菌株的其它实验以及在烧瓶发酵中使用漆酶/ABTS。细胞在具有20g/L的微晶纤维素的Vogel培养基中生长,96小时进入发酵,加入漆酶和ABTS。类似的实验示于图9A和图9B,除了在添加漆酶和ABTS之前,在开始转化反应之前,过滤细胞和纤维素。图10A和图10B提供了进一步的计算。转化率的计算是基于1摩尔的纤维二糖至1摩尔的纤维二糖酸盐的理论转化率。例如,如果从20mM的纤维二糖获得15mM的纤维二糖酸盐,则转化百分比是75%。如果从20mM的纤维二糖获得22mM的纤维二糖酸盐,则转化百分比是110%。可以看出,观察到多于100%的纤维二糖转化成纤维二糖酸盐的理论最大值。这可能是因为液体培养基中的一些其它纤维寡糖经CDH-漆酶-ABTS氧化还原介导的体系转化成纤维二糖酸盐。残留百分比是在给定时间检测的纤维二糖酸盐的量与在先前时间点检测的纤维二糖酸盐的最大量相比。例如,如果检测的纤维二糖酸盐的最大量为15mM,并且24小时后检测的纤维二糖酸盐的量是10mM,则纤维二糖酸盐的残留百分比是66%。不希望受理论束缚,可以认为,当纤维二糖被转化时,纤维素被连续转化(解除抑制),或者纤维寡糖也被转化为纤维二糖酸盐。
NdvB敲除菌株
申请人进一步修饰F5Δcre-1Δace菌株以包含编码纤维二糖酸盐磷酸化酶的ndvB基因中的突变。与亲本菌株(F5Δcre-1Δace)相比,选择两种ndvB敲除菌株(8.3.4.6&8.3.8.8)用于评价CDH-ABTS-漆酶氧化还原体系。进一步修饰两个ndvB敲除菌株以包含mus51突变。不希望受理论束缚,可以认为mus51突变增加了该菌株的基因修饰的容易性。这些菌株的发酵条件如上所述。在92.5小时进入发酵时,将漆酶(0.8U/mL和ABTS(0.1mM)添加到发酵液中以引发纤维二糖向纤维二糖酸盐的转化。通过HPLC监测纤维二糖至纤维二糖酸盐的浓度。如图11A和图11B所示,在向培养基中加入ABTS和漆酶时,所有的纤维二糖被转化为纤维二糖酸盐。与F5菌株相比,F5Δcre-1ΔaceΔmus51ΔndvB菌株产生了更高浓度的纤维二糖酸盐。F5Δcre-1ΔaceΔmus51ΔndvB菌株也展现出更高的CDH活性(图11C)。不希望受理论束缚,可以认为F5Δcre-1ΔaceΔmus51ΔndvB突变株的更高的CDH活性是由于这些菌株中CRE-1和ACE-1的活性丧失。
F5Δcre-1ΔaceΔndvB菌株中的纤维二糖酸盐消耗的分析
不希望受理论束缚,可以认为纤维二糖酸盐磷酸化酶的缺失导致纤维二糖酸盐消耗的阻断。为了进一步探索这一代谢,构建了四个单独的F5Δcre-1ΔaceΔndvB敲除菌株并与F5ΔaceΔcre-1菌株就纤维二糖酸盐的消耗进行比较。在具有0.5g/L葡萄糖(以起始细胞生长)和20g/L纤维二糖酸盐的1×Vogel’s盐中评估所有的菌株。通过HPLC使用具有4mM CaCl2流动相的Aminex 87C柱以0.6mL/分钟测定液体培养基上清液中的葡萄糖和纤维二糖酸盐的浓度。如图12所示,分析所有菌株在刚超过8小时时所消耗的葡萄糖。F5ΔaceΔcre-1菌株在刚超过27小时时消耗了5g/L的纤维二糖酸盐并且仅是在葡萄糖耗尽之后。相比之下,F5Δcre-1ΔaceΔndvB敲除菌株不消耗任何纤维二糖酸盐。
F5Δcre-1ΔaceΔndvB中的漆酶过表达
申请人分析了在基因工程化以过表达漆酶的F5Δcre-1ΔaceΔndvB菌株中的纤维二糖酸盐的积累。为了测试漆酶过表达的效果,收集菌株F5Δcre-1ΔaceΔndvB+lac和对照菌株F5Δcre-1Δace的十天分生孢子并接种到含有20g/L微晶纤维素和0.6g/L葡萄糖的烧瓶中。在27℃以200rpm在光照下进行发酵。在培养开始时加入0.1mM的ABTS。在不同时间间隔取样。通过HPLC测量纤维二糖和纤维二糖酸盐的浓度。如图13所示,F5Δcre-1Δace菌株产生少量的纤维二糖酸盐。然而,所产生的纤维二糖酸盐随后被真菌消耗。然而,F5Δcre-1ΔaceΔndvB+lac菌株产生约7mM的纤维二糖酸盐,并且没有观察到通过菌株消耗纤维二糖酸盐。
构建另外的漆酶过表达菌株以确认图13所获得的结果。如图14A和图14B所示,与F5Δcre-1Δace菌株相比,在F5Δcre-1Δace突变体背景中的纤维二糖酸盐磷酸化酶(ndvB)上的突变和漆酶的过表达都导致显著增加的纤维二糖酸盐生产。
总结
总之,本文呈现的数据证实了工程化的粗糙脉孢菌提高了来自纤维素的纤维二糖酸盐的产生。多个bgl基因的缺失将碳流重新导向纤维二糖/纤维二糖酸盐生产。cre/ace基因缺失促进了纤维素酶的表达。NdvB基因缺失消除了纤维二糖酸盐的消耗。最后,在存在CDH和氧化还原介体的情况下,漆酶的过表达提高了纤维二糖酸盐生产。
实施例2:利用工程化的粗糙脉孢菌菌株并添加漆酶和氧化还原介体从纤维素中生产纤维二糖酸盐。
本实施例示出了使用工程化真菌菌株并外源添加漆酶和氧化还原介体从纤维素中生产纤维二糖酸的生产方法。实施例1示出了工程化菌株粗糙脉孢菌(F5Δace-1Δcre-1ΔndvB)直接从纤维素生产纤维二糖酸盐而不添加外源纤维素酶。粗糙脉孢菌产生纤维素酶(其将纤维素水解成纤维二糖)和纤维二糖脱氢酶(CDH)(其将纤维二糖氧化成纤维二糖酸盐)。然而,纤维二糖向纤维二糖酸盐的转化受分子氧对CDH的缓慢再氧化的限制。通过向发酵中添加低浓度的漆酶和氧化还原介体,可以通过氧化还原介体有效地氧化CDH,并通过漆酶对氧化还原介体进行原位再氧化。在本实施例中,通过评估pH、缓冲液和漆酶以及氧化还原介体添加时间对纤维二糖酸盐收率的影响来优化纤维素到纤维二糖酸盐的转化。进行物料衡算,并将天然的粗糙脉孢菌漆酶在此体系中与外源的糙皮侧耳菌漆酶进行评估。
引言
用于生产有机酸的微生物发酵平台的发展在过去十年愈发受到关注(Jang等人,2012;Sauer等人,2008),这是由于该方法具有可靠性和高效性(Demain等人,2007)。近年来,由于羧酸的独特的物化性质,羧酸例如乳糖酸(LBA)已作为特殊的酸出现。LBA是高附加值的有机酸,在制药、食品和化妆品行业存在许多应用(Alonso等人,2013)。为了与基于石化过程的羧酸生产相竞争,发展利用低成本底物的微生物方法是必要的(Alonso等人,2013)。目前通过化学合成在能量密集型方法中生产LBA需要昂贵的金属催化剂。或者可以通过各种细菌和真菌菌株使用精炼的乳糖作为底物来生物生产LBA(Miyamoto等人,2000;Meiberg J等人,1990;Murakami H等人,2006;Murakami H等人,2003;Pedruzzi I等人,2011;Malvessi E等人,2013;Kiryu T等人,2012;Alonso S等人,2011;Alonso S等人,2012;Alonso S等人,2013)。还研究了在环境友好的发酵过程中通过腐臭假单胞菌(Pseudomonas taetrolens)将廉价的干酪乳清底物作为用于LBA生产的底物(Alonso S等人,2013;Alonso S等人,2011;Alonso S等人,2012)。催化生物转化的酶是乳糖脱氢酶(Miyamoto等人,2000;Meiberg J等人,1990;Murakami H等人,2006;Murakami H等人,2003;Pedruzzi I等人,2011;Malvessi E等人,2013;Kiryu T等人,2012;Alonso S等人,2011;Alonso S等人,2012;Alonso S等人,2013)。
也可以通过CDH的酶促反应从乳糖中产生LBA。CDH是由多种纤维素分解真菌产生的血黄素酶(hemoflavoenzyme)。该蛋白含有负责氧化乳糖或纤维二糖的C-末端黄素腺嘌呤二核苷酸(FAD)结构域,分别导致乳糖酸盐或纤维二糖酸盐的形成。两个电子随后从FAD结构域转移到N-末端的血红素结构域(Henriksson G等人,2000)。为了使CDH恢复功能,还原的血红素结构域必须在电子受体的帮助下被氧化。如果没有其它电子受体存在,则氧是该体系的电子受体。虽然总体反应在热力学上是有利的,但是分子氧对CDH的再氧化的速率非常慢,并且是将乳糖转化为LBA中的限速步骤(Roy BP等人,1996;Bao W等人,1993)。
除了氧以外,许多底物(例如金属离子、醌和有机染料)可以是CDH血红素结构域的可替代的电子受体(Baminger U等人,2001)。二氯酚靛酚(DCPIP)和2,2'-连氮基-双[3-乙基苯并噻唑啉-6-磺酸](ABTS)是两种能够非常有效地接受来自CDH电子的氧化还原介体(Baminger U等人,2001)。然而,通过添加氧化还原介体来促进CDH中的电子转移以提高乳糖到LBA的转化率是昂贵的,除非氧化还原介体可以原位再生。
Baminger等人报道了一种将乳糖快速氧化成LBA的新型CDH-ABTS-漆酶双酶体系(Baminger U等人,2001)。漆酶是重要的多铜氧化酶,其广泛分布于木材降解真菌中(Madhavi V等人,2009)。它们在白腐和褐腐真菌中尤其普遍,推测涉及木质素降解(Arora DS等人,2010)。与CDH相比,漆酶氧化大量的还原底物并且非常有效地使用氧作为最终的电子受体。一种增加通过CDH与作为最终电子受体的氧来氧化乳糖的速率的策略是使用催化量的DCPIP或ABTS以及经漆酶的氧化还原介体的原位再生。如图16所示,CDH被还原。当乳糖被氧化成乳糖酸时,还原的CDH在氧化还原介体的帮助下被再氧化,然后通过漆酶的氧化再生氧化还原介体。最后,当电子传至氧(最终的电子受体),漆酶被再生。发现此类双酶级联体系使用ABTS作为氧化还原介体,能显著促进乳糖转化为LBA的速率(Baminger U等人,2001;Van Hecke W等人,2009)。
纤维素生物质,其成本低且分布广泛且丰富,是潜在的生物生产羧酸的可替代底物。在实施例1中,申请人研究了使用工程化粗糙脉孢菌菌株并添加外源漆酶和氧化还原介体直接从纤维素中生产纤维二糖酸(CBA)的方法,其中纤维二糖酸是LBA的姐妹羧酸(立体异构体)。
通过将七个β-葡萄糖苷酶(BGL)基因中的六个缺失,将粗糙脉孢菌预先工程化以从纤维素中生产纤维二糖,获得菌株F5(Fan Z等人,2012;Wu W等人,2013)。该菌株还产生CDH,其可将纤维二糖氧化成CBA。申请人通过敲除纤维二糖酸盐磷酸化酶(ndvB)基因并且通过缺失碳分解代谢物抑制基因cre-1和ace-1改善纤维素酶的表达,进一步将该菌株工程化以防止CBA消耗,获得菌株F5Δace-1Δcre-1ΔndvB(Hildebrand A等人,20l4)(参见实施例1)。从20g/L微晶纤维素中,F5Δace-1Δcre-1ΔndvB菌株产生20mM(7g/L)的纤维二糖和10mM的CBA(3.5g/L)。在此体系中,氧是最终的电子受体。通过氧再氧化CDH是限速步骤,其导致大多数的纤维二糖不能完全转化为CBA。
在本实施例中,申请人探索了使用工程化的粗糙脉孢菌F5Δace-1Δcre-1ΔndvB菌株将纤维素分解成纤维二糖并产生CDH的可能性。外源添加漆酶和ABTS将补充CDH以形成双酶级联体系以将纤维二糖转化成CBA。优化发酵条件(包括pH、缓冲液以及漆酶和ABTS浓度及添加时间)以最大化纤维素到CBA的收率。此外,包括整个发酵的物料平衡。最后,研究了在CDH-ABTS-漆酶体系中使用天然粗糙脉孢菌漆酶的可能性。
材料和方法
菌株和试剂
野生型粗糙脉孢菌(2489)获得自真菌遗传保藏中心(FGSC)(McCluskey K等人,2010)。本实施例中使用的F5菌株是七个bgl基因中的六个被敲除的菌株2489(Fan Z等人,2012;Wu W等人,2013)。如Hildebrand A等人,2014中所述,通过敲除ace-1、cre-1和ndvB基因将F5菌株工程化用以增加纤维素酶的表达和纤维素的水解。本实施例中使用的菌株及其来源列于表1。
表1:本实施例中使用的示例性菌株
为了构建过表达天然粗糙脉孢菌漆酶的粗糙脉孢菌菌株,使用如前所述的基于环孢菌素A抗性的基因位置系统(gene placement system)过表达天然漆酶基因。gpdA启动子(来自黑曲霉)、漆酶基因(NCU04528.7)和trpC终止子由Life Technologies合成并连接到载体,其具与csr-1同源的侧翼。质粒在大肠杆菌中表达并通过质粒的限制性切割获得插入盒的线性DNA。将1200μg线性DNA与2.5×109分生孢子/mL混合并电穿孔(0.1cm电击杯-7.5kV/cm;电容:25uFD;电阻:600ohm)。电穿孔后,将细胞在750mL的1M冰冷山梨醇中再生,然后将其添加至2mL的恢复培养基(1×Vogel’s培养基、2%酵母提取物)。在30℃以及200rpm振摇6小时后,将350μL恢复的细胞涂布于琼脂板(1×Vogel’s、2%酵母提取物、1M山梨醇、20g L-1山梨糖、0.5g L-1果糖、0.5g L-1葡萄糖、5μg mL-1CsA和2%琼脂)。30℃生长5天后,将菌落置于斜面培养基(斜面培养基(5μg mL-1CsA、1×Vogel’s、2%琼脂斜面))并再生长4天。通过PCR验证转化株。
来自糙皮侧耳菌(P.ostreatus)漆酶获得自Sigma Aldrich并用于某些涉及外源添加漆酶的实验。ABTS获得自Sigma Aldrich。对于体外实验,所使用的CDH是由工程化的毕赤酵母菌株产生的重组粗糙脉孢菌CDH。根据如Zhang等人,2011中所述的方法生产该菌株。
使用CDH-ABTS-漆酶体系的纤维二糖向CBA的转化
为了测试CDH-漆酶-ABTS氧化还原体系对将纤维二糖转化为纤维二糖酸盐的适用性,将浓度约30mM的纤维二糖加入到含漆酶、ABTS、CDH以及指定浓度和pH的缓冲液的50mL离心管中。当研究pH的影响时,使用50mM浓度的柠檬酸钠缓冲液用于酸性条件,使用50mM浓度的磷酸钠缓冲液用于碱性条件,以及使用无pH调整的1×Vogel’s盐培养基用于pH6的条件。
发酵实验
粗糙脉孢菌菌株在30℃光照培养箱中生长于具有1×Vogel’s盐和1.5%蔗糖的琼脂上。3天后,从培养箱中取出烧瓶并在室温下生长7天。在总计10天的生长后,在DI水中收集分生孢子并用八层纱布过滤。在具有50mL工作体积、1×Vogel’s盐培养基、0.5g/L葡萄糖(以起始生长)和20g/L微晶纤维素的250mL无挡板烧瓶(unbaffled flask)中进行发酵实验。以一定体积接种分生孢子以使最终的OD420为0.15。在28℃的光照的200rpm的摇床中孵育烧瓶。如本文所示,将外源漆酶和ABTS加入到烧瓶中。在多个时间间隔取样以测量纤维二糖或纤维二糖酸盐浓度。为了研究起始pH对纤维二糖酸盐收率的影响,加入不同浓度的柠檬酸钠、磷酸钠、磷酸钾以及磷酸钠-钾缓冲液以达到所示pH。为了研究漆酶和ABTS添加时间对纤维二糖酸盐产生的影响,将漆酶和ABTS分别以0.05U/mL和0.01mM的终浓度加入到发酵液体培养基中。
用粗糙脉孢菌漆酶过表达菌株进行发酵实验
粗糙脉孢菌在30℃光照培养箱中生长于具有1×Vogels盐和1.5%蔗糖的琼脂上。3天后,从培养箱中取出烧瓶并在室温下生长7天。在总计10天的生长后,在DI水中收集分生孢子并用八层纱布过滤。在具有50mL工作体积的250mL无挡板烧瓶中进行发酵实验。生长培养基包括1×Vogel’s盐培养基和20g/L葡萄糖(以起始漆酶过表达),或者包括1×Vogel’s盐培养基、0.5g/L葡萄糖(以起始生长)和20g/L微晶纤维素。以一定体积接种分生孢子以使最终的OD420为0.15。在28℃的光照的200rpm的摇床中孵育烧瓶。加入初始浓度为0.1mM的ABTS。
粗糙脉孢菌生产漆酶
野生型菌株生长于含1.5%蔗糖的Vogel’s盐培养基中。根据文献(Huber M等人,1987;Linden RM等人,1991;Froehner SC等人,1974),在48小时加入终浓度为3μM的环己酰亚胺(用于漆酶生产的诱导剂)。根据下文在“酶浓度”部分中所描述的试验来监测漆酶的表达。在继续发酵142小时后,过滤液体培养基以去除残留的微晶纤维素和细胞,以获得在无细胞实验中的CDH-ABTS-漆酶氧化还原体系中用于评价的天然漆酶。
样品分析
使用装备有CARBOSep COREGEL-87C(Transgenomic,San Jose,CA,USA)柱的Shimadzu HPLC分析无细胞实验和发酵液体培养基中的纤维二糖和纤维二糖酸盐的浓度。使用流速为0.6mL/min的4mM氯化钙(CaCl2)作为流动相。
酶浓度
如前所述并进行少许修改,通过监测ABTS吸光度的增加来测量漆酶的活性(Van Hecke W等人,2009;Baminger U等人,1999)。反应混合物包括100mM的乙酸钠缓冲液中的5mM的ABTS,pH4.5。一个单位的漆酶活性被定义为在上述反应条件下每分钟氧化1μmol的ABTS的酶量。
根据先前建立的方法并进行轻微修改,通过在分光光度计中监测520nm处的DCPIP的吸光度的减少来确定CDH的浓度(Van Hecke W等人,2009;Baminger U等人,1999)。反应包括在100mM,pH4.5的乙酸钠缓冲液中的0.1mM的DCPIP、3mM纤维二糖和4mM氟化钠。一个单位的酶活性被定义为在上述反应条件下每分钟还原1μmol的DCIP的酶量。
菌丝体生物质的测定
通过从菌丝体中提取麦角固醇并通过HPLC测定数量来测定发酵样品中所含的菌丝体的干重(Gessner MO等人,1991)。通过0.8μm膜过滤以收集发酵残留物。收集包括菌丝体的所有残留物,在液氮中冷冻1小时,并向冷冻样品中添加乙醇(6mL)并在37℃振荡孵育2小时。向混合物中添加KOH溶液(60%w/v,0.8mL)的等分试样,然后加热至97℃持续20min。将样品冷却并用HCl(36.5%,约0.7mL)中和。用己烷(3×5mL)萃取溶液,合并己烷相,并使用空气以蒸发溶剂。将残留物溶解于乙醇(1mL)中,通过0.22μm膜过滤器过滤并采用HPLC与PDA检测器在反相柱(ZORBAX Eclipse Plus C18,4.6×250mm,5μm粒径,Agilent)上分析,并以1.0mL/min的甲醇-水(97:3v/v)洗脱。使用已知的粗糙脉孢菌干燥生物质制备的标准曲线定量生物质的量。通过从发酵残留物的干重中减去菌丝体生物质来计算残留的微晶纤维素的量。
结果
pH在使用CDH-ABTS-漆酶体系的纤维二糖向CBA的转化过程中的作用
测试酸性、中性和碱性条件以确定pH对纤维二糖向CBA转化的影响。纤维二糖向CBA的转化的时间过程示于图17A和图17B。对于pH6的条件,转化在24小时内完成,平均27.9mM的纤维二糖被转化为28.6mM的CBA,如预期,获得约1:1摩尔的转化。所获得的数据支持了CDH-ABTS-漆酶体系将纤维二糖转化为纤维二糖酸盐的功效。然而,对于在此实验中使用特定的CDH和漆酶将纤维二糖有效转化成CBA的过程而言,酸性条件的存在是重要的。
起始pH对CBA生产的影响
当F5Δace-1Δcre-1ΔndvB在20g/L的微晶纤维素上生长时,产生约20mM的纤维二糖和10mM的CBA。如果在纤维二糖向CBA的转化中添加漆酶和ABTS,则产生30mM的CBA(Hildebrand A等人,2014)(参见实施例1)。由于产生高浓度的CBA,pH迅速从pH6降至pH4。评估缓冲的发酵培养基的影响以确定此pH下降对纤维二糖向CBA的转化的效果。评估了三种不同浓度的磷酸钾缓冲液以控制pH下降的速率并与无缓冲的Vogel’s培养基相比较(图18A和图18B)。在无缓冲的Vogel’s培养基的情况下,在发酵过程中pH降至4。磷酸钠缓冲液的添加导致pH更缓慢地降低,并且随缓冲液浓度的增加,最终pH更高。200mM磷酸钾保持pH为6,直至发酵的最后一天pH降至5.5。虽然pH在所测试的条件下发生了变化,但对纤维二糖和CBA产生没有显著的影响。因此,在本实施例中,将无缓冲的Vogel’s培养基用于发酵实验的剩余部分。
于不同时间在CBA生产中添加漆酶和ABTS
将向使用20g/L微晶纤维素上的F5Δace-1Δcre-1ΔndvB菌株的发酵体系中添加漆酶和ABTS的过程优化。在不同时间点加入0.05U/mL的漆酶和0.01mM的ABTS。如图19A和图19B所示,纤维二糖在所有添加时间后的48小时内都完全转化为CBA,其中在120小时添加到发酵的添加时间是最佳的。在所有情况下,最大CBA浓度发生在168小时,之后略有降低。当没有加入漆酶和ABTS时,发酵中的最大纤维二糖浓度也发生在168小时,并且在该时间点纤维二糖酸盐产生也达到最高点。
在120小时将漆酶和ABTS加入发酵的平行实验中,在168小时,收集烧瓶以定量生物质和微晶纤维素利用。如表2所示,结果表明67%的微晶纤维素被水解,其中91%流向CBA生产,4%流向细胞质生产。在对照条件中,当不添加漆酶和ABTS,62%的微晶纤维素被水解,并且消耗的微晶纤维素的较小部分流向CBA生产和细胞质生产(分别为29%和3.3%)。不希望受理论束缚,在添加漆酶和ABTS的情况下,较高的纤维素转化表明纤维二糖向CBA的转化可以减少纤维二糖对纤维素酶的抑制,使得纤维寡糖水解并随后转化成CBA。
表2:在20g/L微晶纤维素上生长的F5Δace-1Δcre-1ΔndvB菌株中的微晶纤维素水解百分比和产生发酵产物的百分比。
基于标准偏差和误差理论分析误差。
粗糙脉孢菌天然漆酶的体外分析
在将外源漆酶加入发酵的实验中,漆酶宿主是糙皮侧耳菌(Pleureotus ostreatus),其是表达高水平的漆酶的担子菌(Okamoto K等人,2000;More S等人,2011)。虽然粗糙脉孢菌有天然的漆酶,但其不天然表达,仅在不适合有效发酵的压力条件下表达。然而,本文研究了使用天然的粗糙脉孢菌漆酶在CDH-ABTS-漆酶体系中使纤维二糖向CBA转化的可行性。
根据之前的研究,环己酰亚胺、D-苯丙氨酸和硫酸铜都是粗糙脉孢菌漆酶的可能的诱导剂(Huber M等人,1987;Linden RM等人,1991;Froehner SC等人,1974;Luke AK等人,2001)。为了在粗糙脉孢菌F5菌株中诱导漆酶,在培养48小时后,加入3μM环己酰亚胺。然后再发酵142小时后,获得0.15U/mL的漆酶,如图20所示,在纤维二糖转化体系中测试合适的浓度。
在离心管实验(无细胞)中,相较于糙皮侧耳菌,测试所产生的漆酶,如图21A和图21B所示,加入纤维二糖、CDH和ABTS并监测纤维二糖向CBA的转化。结果表明,两种漆酶具有相当的活性,都有效地在CDH-ABTS-漆酶转化体系中使纤维二糖转化成CBA。
纤维素分解真菌(例如粗糙脉孢菌)可潜在地产生将纤维素转化成CBA所需的所有酶。粗糙脉孢菌能产生纤维素酶和CDH。通过添加外源添加的漆酶和ABTS,粗糙脉孢菌能以非常高的收率将纤维素转化为CBA。该实施例示出,粗糙脉孢菌产生的天然漆酶与糙皮侧耳菌的漆酶一样有效。这为工程改造粗糙脉孢菌以产生将纤维素转化成CBA所需的所有酶提供了可能。在粗糙脉孢菌中漆酶的同源或异源表达可通过工程化天然或异源漆酶在组成型或诱导型启动子下表达而实现,从而允许在所测试的发酵条件下生产漆酶。
文献中的报道表明,在粗糙脉孢菌中过表达的天然漆酶的浓度为55mg/L(Schilling B等人,1992)。此浓度足以在存在CDH和ABTS时有效地将纤维二糖转化为CBA,并且因此可以在本文所述的菌株中进行天然粗糙脉孢菌漆酶的过表达。此外,一项研究表明,漆酶去抑制的突变体lah-1产生漆酶的水平甚至高于用环己酰亚胺诱导的野生型产生漆酶的水平(Tamaru H等人,1989)。去抑制是定位于连锁群I中的nit-2和leu-3之间的未知基因中的单突变所导致的。类似地,去抑制F5Δace-1Δcre-1ΔndvB菌株或在该菌中过表达漆酶都可以构建一种仅需添加低浓度的ABTS即可从纤维素中产生CBA的菌株。
粗糙脉孢菌的天然漆酶的体内分析
如上所述的使用内源粗糙脉孢菌漆酶的体外实验表明了在生产纤维二糖酸盐分析中用过表达这一内源基因代替添加外源漆酶的可能性。为了探索这一点,在F5Δace-1Δcre-1ΔndvB菌株背景下如上所述构建过表达天然粗糙脉孢菌漆酶基因的粗糙脉孢菌菌株。当菌株在葡萄糖上生长时,天然漆酶被成功表达。三株不同的菌株产生约0.03U/mL的漆酶(图22)。选择最好的生产转化体(2-2)用来进行另外的实验。
然后,探索了通过重组的菌株从微晶纤维素中生产纤维二糖酸盐。如图23所示,过表达漆酶的菌株(F5Δace-1Δcre-1ΔndvB(Lac+))比不产生漆酶的对照菌株(F5Δace-1Δcre-1ΔndvBΔmus51)产生更多的纤维二糖酸盐。这两种菌株都比F5Δace-1Δcre-1Δmus51产生更多的纤维二糖酸盐。在发酵开始时加入0.1mM的ABTS。
还检测了在过表达天然粗糙脉孢菌漆酶的重组菌株中加入ABTS对纤维二糖酸盐生产的影响。如图24所示,添加ABTS增加在这一菌株(F5Δace-1Δcre-1ΔndvB(Lac+))中产生的纤维二糖酸盐的量。然而,还观察到,即使不添加ABTS也可产生纤维二糖酸盐,这表明培养基中的一些其它试剂(如FeSO4)也可充当电子介质。
讨论
在测试的体系中,使用ABTS作为氧化还原介体。尽管其仅需要催化量,但其对于工业应用而言是非常昂贵的,可替代的低成本的氧化还原介体可能会更好。多种无机金属和有机染料可作为氧化还原介体(Kunamneni,A等人,2008)。值得注意的是,在纸和纸浆工业中作为废物所产生的木质素降解产物如香草醛、阿魏酸或对香豆酸被证明是非常有效的天然存在的漆酶氧化还原介体(Camarero S等人,2005;Canas A等人,2007;Camarero S等人,2007;Gutierrez A等人,2007)。这些化合物也可用作廉价的氧化还原介体源(Kunamneni,A等人,2008)。此外,如果使用预处理的木质纤维素生物质作为底物代替纯的纤维素,这些化合物可天然存在于原料流中,并且可以潜在地减少或避免外源添加氧化还原介体。
总结
该实施例证明,可以通过添加外源漆酶或过表达内源漆酶基因的工程化的粗糙脉孢菌菌株,从纤维素中生产CBA。由粗糙脉孢菌产生的天然漆酶与外源添加的漆酶具有同样的功能。测试的菌株可用于将纤维素以微生物法转化为CBA。这种转化理念也适用于其它工业相关的纤维素分解真菌用于从廉价的原料(例如纤维素生物质)中生产CBA。
参考文献
Lynd,L.R.,Zyl,W.H.,McBride,J.E.,and Laser,M.(2005)Consolidated bioprocessing of cellulosic biomass:an update,Curr Opin Biotechnol.
Lynd,L.R.,Weimer,P.J.,van Zyl,W.H.,and Pretorius,I.S.(2002)Microbial cellulose utilization:fundamentals and biotechnology,Microbiol Mol Biol Rev 66,506-577,目录。
Lynd,L.R.,Wyman,C.E.,and Gerngross,T.U.(1999)Biocommodity Engineering,Biotechnol Prog 15,777-793.
Lynd,L.R.,Cushman,J.H.,Nichols,R.J.,and Wyman,C.E.(1991)Fuel Ethanol from Cellulosic Biomass,Science 251,1318-1323.
Fan,Z.,Wu,W.,Hildebrand,A.,Kasuga,T.,Zhang,R.,and Xiong,X.(2012)A novel biochemical route for fuels and chemicals production from cellulosic biomass.PLoS One 7,e31693.
Wu,W.,Hildebrand,A.,Kasuga,T.,Xiong,X.,and Fan,Z.(2013)Direct cellobiose production from cellulose using sextuple beta-glucosidase gene deletion Neurospora crassa mutants.Enzyme and Microbial Technology 52,184-189.
Zhang,R.,Fan,Z.,and Kasuga,T.(2011)Expression of cellobiose dehydrogenase from Neurospora crassa in Pichia pastoris and its purification and characterization.Protein Expression and Purification75,63-69.
Szewczyk,E.,Kasuga,T.,and Fan,Z.(2013)Efficient sequential repetitive gene deletions in Neurospora crassa,employing self-excising beta-recombinase/six cassette.Journal of Microbiological Methods 92,236-243.
Baminger,U.,Ludwig,R.,Galhaup,C.,Leitner,C.,Kulbe,K.D.,and Haltrich,D.(2001)Continuous enzymatic regeneration of redox mediators used in biotransformation reactions employing flavoproteins.Journal of Molecular Catalysis B-Enzymatic 11,541-550.
Jang Y-S,Kim B,Shin JH,Choi YJ,Choi S,et al.(2012)Bio-based production of C2-C6 platform chemicals.Biotechnol Bioeng 109:2437-2459.
Sauer M,Porro D,Mattanovich D,Branduardi P(2008)Microbial production of organic acids:expanding the markets.Trends Biotechnol 26:100-108.
Demain AL(2007)The business of biotechnology.Ind Biotechnol 3:269-283.
Alonso S,Rendueles M,Díaz M(2013)Bio-production of lactobionic acid:current status,applications and future prospects.Biotechnol Adv 31:1275-1291.
Miyamoto Y,Ooi T,Kinoshita S(2000)Production of lactobionic acid from whey by Pseudomonas sp.LS13-1.Biotechnol Lett 22:427-430.
Meiberg J,PM B,Sloots B(1990)A process for the fermentative oxidation of reducing disaccharides.EP 0384534 A1
Murakami H,Kiryu T,Kiso T,Nakano H(2006)Production of Aldonic Acids from Monosaccharides by Washed Cells of Burkholderia cepacia and their Calcium Binding Capability.J Appl glycosci 279:277-279.
Murakami H,Seko A,Azumi M,Ueshima N,Yoshizumi H,et al.(2003)Fermentative Production of Lactobionic Acid by Burkholderia cepacia.J Appl glycosci 120:117-120.
Pedruzzi I,da Silva EAB,Rodrigues AE(2011)Production of lactobionic acid and sorbitol from lactose/fructose substrate using GFOR/GL enzymes from Zymomonas mobilis cells:a kinetic study.Enzyme Microb Technol 49:183-191.
Malvessi E,Carra S,Pasquali FC,Kern DB,da Silveira MM,et al.(2013)Production of organic acids by periplasmic enzymes present in free and immobilized cells of Zymomonas mobilis.J Ind Microbiol Biotechnol40:1-10.
Kiryu T,Yamauchi K,Masuyama A,Ooe K,Kimura T,et al.(2012)Optimization of Lactobionic Acid Production by Acetobacter orientalis Isolated from Caucasian Fermented Milk,“Caspian Sea Yogurt.”Biosci Biotechnol Biochem 76:361-363.
Alonso S,Rendueles M,Díaz M(2011)Efficient lactobionic acid production from whey by Pseudomonas taetrolens under pH-shift conditions.Bioresour Technol 102:9730-9736.
Alonso S,Rendueles M,Díaz M(2012)Role of dissolved oxygen availability on lactobionic acid production from whey by Pseudomonas taetrolens.Bioresour Technol 109:140-147.
Alonso S,Rendueles M,Díaz M(2013)Feeding strategies for enhanced lactobionic acid production from whey by Pseudomonas taetrolens.Bioresour Technol 134:134-142.
Henriksson G,Johansson G,Pettersson G(2000)A critical review of cellobiose dehydrogenases.J Biotechnol 78:93-113.
Roy BP,Dumonceaux T,Koukoulas AA,Archibald FS(1996)Purification and Characterization of Cellobiose Dehydrogenases from the White Rot Fungus Trametes versicolor.Appl Environ Microbiol 62:4417-4427.
Bao W,Usha SN,Renganathan V(1993)Purification and characterization of cellobiose dehydrogenase,a novel extracellular hemoflavoenzyme from the white-rot fungus Phanerochaete chrysosporium.Arch Biochem Biophys 300:705-713.
Baminger U,Ludwig R,Galhaup C,Leitner C,Kulbe KD,et al.(2001)Continuous enzymatic regeneration of redox mediators used in biotransformation reactions employing flavoproteins.J Mol Catal B Enzym 11:541-550.
Madhavi V,Lele SS(2009)Laccase:properties and applications.4:1694-1717.
Arora DS,Sharma RK,Singh Arora D,Kumar Sharma R(2010)Ligninolytic fungal laccases and their biotechnological applications.Appl Biochem Biotechnol 160:1760-1788.
Van Hecke W,Bhagwat A,Ludwig RR,Dewulf J,Haltrich D,et al.(2009)Kinetic Modeling of a Bi-Enzymatic System for Efficient Conversion of Lactose to Lactobionic Acid.Biotechnol Bioeng 102:1475-1482.
Lynd LR,Cushman J,Nichols R,Wyman C(1991)Fuel ethanol from cellulosic biomass.Science 251:1318-1323.
Fan Z,Wu W,Hildebrand A,Kasuga T,Zhang R,et al.(2012)Novel Biochemical Route for Fuels and Chemicals production from Cellulosic Biomass.PLoS One 7:8.
Wu W,Hildebrand A,Kasuga T,Xiong X,Fan Z(2013)Direct cellobiose production from cellulose using sextuple beta-glucosidase gene deletion Neurospora crassa mutants.Enzyme Microb Technol 52:184-189.
Hildebrand A,Szewczyk E,Lin H,Kasuga T,Fan J(2014)Engineering Neurospora crassa for improved cellobiose and cellobionate production.Appl Environ Microbiol:In press.
McCluskey K,Wiest A,Plamann M(2010)The fungal genetics stock center:A repository for 50 years of fungal genetics research.J Biosci 35:119-126.
Zhang R,Fan Z,Kasuga T(2011)Expression of cellobiose dehydrogenase from Neurospora crassa in Pichia pastoris and its purification and characterization.Protein Expr Purif 75:63-69.
Huber M,Lerch K(1987)The influence of copper on the induction of tyrosinase and laccase in Neurospora crassa.219:335-338.
Linden RM,Schilling BC,Germann U a,Lerch K(1991)Regulation of laccase synthesis in induced Neurospora crassa cultures.Curr Genet 19:375-381.
Froehner SC,Eriksson KE(1974)Induction of Neurospora crassa laccase with protein synthesis inhibitors.J Bacteriol 120:450-457.
Baminger U,Nidetzky B,Kulbe KD,Haltrich D(1999)A simple assay for measuring cellobiose dehydrogenase activity in the presence of laccase.35:253-259.
Gessner MO,Bauchrowitz MA,Escautier M,Renouvelables R,Marvig J (1991)Extraction and quantification of ergosterol as a measure of fungal biomass in leaf litter.Microb Ecol:285-291.
Okamoto K,Yanagi SO,Sakai T(2000)Purification and characterization of extracellular laccase from Pleurotus ostreatus.Mycoscience 41:7-13.
More S,S.RP,K.P,M.S,S.Malini,et al.(2011)Isolation,Purification,and Characterization of Fungal Laccase fromPleurotus sp.Enzyme Res2011:7.
Luke AK,Burton SG(2001)A novel application for Neurospora crassa:Progress from batch culture to a membrane bioreactor for the bioremediation of phenols.Enzyme Microb Technol 29:348-356.
Schilling B,Linden RM,Kupper U,Lerch K(1992)Expression of Neurospora crassa laccase under the control of the copper-inducible metallothionein-promoter.Curr Genet 22:197-203.
Tamaru H,Inoue H(1989)Isolation and characterization of a laccase-derepressed mutant of Neurospora crassa.J Bacteriol 171:6288-6293.
Kunamneni,A.,Camarero,S.,Garcia-Burgos,C.,Plou,F.J.,Ballesteros,A.,and Alcalde,M.(2008)Engineering and Applications of fungal laccases for organic synthesis,Microbial cell factories 7,17.
Camarero S,Ibarra D,Martínez MJ,Martínez AT.Lignin-derived compounds as efficient laccase mediators for decolorization of different types of recalcitrant dyes.Appl Environ Microbiol.2005;71:1775-1784.
A,Alcalde M,Plou FJ,Martínez MJ,Martínez AT,Camarero S.Transformation of polycyclic aromatic hydrocarbons by laccase is strongly enhanced by phenolic compounds present in soil.Environ Sci Technol.2007;41:2964-2971.
Camarero S,Ibarra D,Martínez AT,Romero J,Gutiérrez A,del Río JC.Paper pulp delignification using laccase and natural mediators.Enzyme Microb Technol.2007;40:1264-1271.
Gutiérrez A,Rencores J,Ibarra D,Molina S,Camarero S,Romero J,del Río JC,Martínez AT.Removal of lipophilic extractives from paper pulp by laccase and lignin-derived phenols as natural mediators.Environ Sci Technol.2007;41:4124-4129.
序列表
<110> 加利福尼亚大学董事会
范质良
阿曼达·希尔德布兰德
<120> 用于商业化学品生产的生物平台
<130> 514112007840
<140> 未分配
<141> 同时递交
<150> 61/985,379
<151> 2014-04-28
<160> 24
<170> FastSEQ for Windows Version 4.0
<210> 1
<211> 476
<212> PRT
<213> 粗糙脉孢菌
<400> 1
Met Ser Leu Pro Lys Asp Phe Leu Trp Gly Phe Ala Thr Ala Ala Tyr
1 5 10 15
Gln Ile Glu Gly Ala Ile His Ala Asp Gly Arg Gly Pro Ser Ile Trp
20 25 30
Asp Thr Phe Cys Asn Ile Pro Gly Lys Ile Ala Asp Gly Ser Ser Gly
35 40 45
Ala Val Ala Cys Asp Ser Tyr Asn Arg Thr Lys Glu Asp Ile Asp Leu
50 55 60
Leu Lys Ser Leu Gly Ala Thr Ala Tyr Arg Phe Ser Ile Ser Trp Ser
65 70 75 80
Arg Ile Ile Pro Val Gly Gly Arg Asn Asp Pro Ile Asn Gln Lys Gly
85 90 95
Ile Asp His Tyr Val Lys Phe Val Asp Asp Leu Leu Glu Ala Gly Ile
100 105 110
Thr Pro Phe Ile Thr Leu Phe His Trp Asp Leu Pro Asp Gly Leu Asp
115 120 125
Lys Arg Tyr Gly Gly Leu Leu Asn Arg Glu Glu Phe Pro Leu Asp Phe
130 135 140
Glu His Tyr Ala Arg Thr Met Phe Lys Ala Ile Pro Lys Cys Lys His
145 150 155 160
Trp Ile Thr Phe Asn Glu Pro Trp Cys Ser Ser Ile Leu Gly Tyr Asn
165 170 175
Ser Gly Tyr Phe Ala Pro Gly His Thr Ser Asp Arg Thr Lys Ser Pro
180 185 190
Val Gly Asp Ser Ala Arg Glu Pro Trp Ile Val Gly His Asn Leu Leu
195 200 205
Ile Ala His Gly Arg Ala Val Lys Val Tyr Arg Glu Asp Phe Lys Pro
210 215 220
Thr Gln Gly Gly Glu Ile Gly Ile Thr Leu Asn Gly Asp Ala Thr Leu
225 230 235 240
Pro Trp Asp Pro Glu Asp Pro Leu Asp Val Glu Ala Cys Asp Arg Lys
245 250 255
Ile Glu Phe Ala Ile Ser Trp Phe Ala Asp Pro Ile Tyr Phe Gly Lys
260 265 270
Tyr Pro Asp Ser Met Arg Lys Gln Leu Gly Asp Arg Leu Pro Glu Phe
275 280 285
Thr Pro Glu Glu Val Ala Leu Val Lys Gly Ser Asn Asp Phe Tyr Gly
290 295 300
Met Asn His Tyr Thr Ala Asn Tyr Ile Lys His Lys Lys Gly Val Pro
305 310 315 320
Pro Glu Asp Asp Phe Leu Gly Asn Leu Glu Thr Leu Phe Tyr Asn Lys
325 330 335
Lys Gly Asn Cys Ile Gly Pro Glu Thr Gln Ser Phe Trp Leu Arg Pro
340 345 350
His Ala Gln Gly Phe Arg Asp Leu Leu Asn Trp Leu Ser Lys Arg Tyr
355 360 365
Gly Tyr Pro Lys Ile Tyr Val Thr Glu Asn Gly Thr Ser Leu Lys Gly
370 375 380
Glu Asn Ala Met Pro Leu Lys Gln Ile Val Glu Asp Asp Phe Arg Val
385 390 395 400
Lys Tyr Phe Asn Asp Tyr Val Asn Ala Met Ala Lys Ala His Ser Glu
405 410 415
Asp Gly Val Asn Val Lys Gly Tyr Leu Ala Trp Ser Leu Met Asp Asn
420 425 430
Phe Glu Trp Ala Glu Gly Tyr Glu Thr Arg Phe Gly Val Thr Tyr Val
435 440 445
Asp Tyr Glu Asn Asp Gln Lys Arg Tyr Pro Lys Lys Ser Ala Lys Ser
450 455 460
Leu Lys Pro Leu Phe Asp Ser Leu Ile Lys Lys Asp
465 470 475
<210> 2
<211> 1431
<212> DNA
<213> 粗糙脉孢菌
<400> 2
atgtctcttc ctaaggattt cctctggggc ttcgctactg cggcctatca gattgagggt 60
gctatccacg ccgacggccg tggcccctct atctgggata ctttctgcaa cattcccggt 120
aaaatcgccg acggcagctc tggtgccgtc gcctgcgact cttacaaccg caccaaggag 180
gacattgacc tcctcaagtc tctcggcgcc accgcctacc gcttctccat ctcctggtct 240
cgcatcatcc ccgttggtgg tcgcaacgac cccatcaacc agaagggcat cgaccactat 300
gtcaagtttg tcgatgacct gctcgaggct ggtattaccc cctttatcac cctcttccac 360
tgggatcttc ccgatggtct cgacaagcgc tacggcggtc ttctgaaccg tgaagagttc 420
cccctcgact ttgagcacta cgcccgcact atgttcaagg ccattcccaa gtgcaagcac 480
tggatcacct tcaacgagcc ctggtgcagc tccatcctcg gctacaactc gggctacttt 540
gcccctggcc acacctccga ccgtaccaag tcacccgttg gtgacagcgc tcgcgagccc 600
tggatcgtcg gccataacct gctcatcgct cacgggcgtg ccgtcaaggt gtaccgagaa 660
gacttcaagc ccacgcaggg cggcgagatc ggtatcacct tgaacggcga cgccactctt 720
ccctgggatc cagaggaccc cttggacgtc gaggcgtgcg accgcaagat tgagttcgcc 780
atcagctggt tcgcagaccc catctacttt ggaaagtacc ccgactcgat gcgcaaacag 840
ctcggtgacc ggctgcccga gtttacgccc gaggaggtgg cgcttgtcaa gggttccaac 900
gacttctacg gcatgaacca ctacacagcc aactacatca agcacaagaa gggcgtccct 960
cccgaggacg acttcctcgg caacctcgag acgctcttct acaacaagaa gggtaactgc 1020
atcgggcccg agacccagtc gttctggctc cggccgcacg cccagggctt ccgcgacctg 1080
ctcaactggc tcagcaagcg ctacggatac cccaagatct acgtgaccga gaacgggacc 1140
agtctcaagg gcgagaacgc catgccgctc aagcaaattg tcgaggacga cttccgcgtc 1200
aagtacttca acgactacgt caacgccatg gccaaggcgc atagcgagga cggcgtcaac 1260
gtcaagggat atcttgcctg gagcttgatg gacaactttg agtgggccga gggctatgag 1320
acgcggttcg gcgttaccta tgtcgactat gagaacgacc agaagaggta ccccaagaag 1380
agcgccaaga gcttgaagcc gctctttgac tctttgatca agaaggacta a 1431
<210> 3
<211> 735
<212> PRT
<213> 粗糙脉孢菌
<400> 3
Met His Leu Arg Ile Phe Ala Val Leu Ala Ala Thr Ser Leu Ala Trp
1 5 10 15
Ala Glu Thr Ser Glu Lys Gln Ala Arg Gln Ala Gly Ser Gly Phe Ala
20 25 30
Ala Trp Asp Ala Ala Tyr Ser Gln Ala Ser Thr Ala Leu Ser Lys Leu
35 40 45
Ser Gln Gln Asp Lys Val Asn Ile Val Thr Gly Val Gly Trp Asn Lys
50 55 60
Gly Pro Cys Val Gly Asn Thr Pro Ala Ile Ala Ser Ile Gly Tyr Pro
65 70 75 80
Gln Leu Cys Leu Gln Asp Gly Pro Leu Gly Ile Arg Phe Gly Gly Ser
85 90 95
Val Thr Ala Phe Thr Pro Gly Ile Gln Ala Ala Ser Thr Trp Asp Val
100 105 110
Glu Leu Ile Arg Gln Arg Gly Val Tyr Leu Gly Ala Glu Ala Arg Gly
115 120 125
Val Gly Val His Val Leu Leu Gly Pro Val Ala Gly Ala Leu Gly Lys
130 135 140
Ile Pro Asn Gly Gly Arg Asn Trp Glu Gly Phe Gly Pro Asp Pro Tyr
145 150 155 160
Leu Thr Gly Ile Ala Met Ser Glu Thr Ile Glu Gly Ile Gln Ser Asn
165 170 175
Gly Val Gln Ala Cys Ala Lys His Phe Ile Leu Asn Glu Gln Glu Thr
180 185 190
Asn Arg Asp Thr Ile Ser Ser Val Val Asp Asp Arg Thr Met His Glu
195 200 205
Leu Tyr Leu Phe Pro Phe Ala Asp Ala Val His Ser Asn Val Ala Ser
210 215 220
Val Met Cys Ser Tyr Asn Lys Val Asn Gly Thr Trp Ala Cys Glu Asn
225 230 235 240
Asp Lys Ile Gln Asn Gly Leu Leu Lys Lys Glu Leu Gly Phe Lys Gly
245 250 255
Tyr Val Met Ser Asp Trp Asn Ala Gln His Thr Thr Asn Gly Ala Ala
260 265 270
Asn Ser Gly Met Asp Met Thr Met Pro Gly Ser Asp Phe Asn Gly Lys
275 280 285
Thr Ile Leu Trp Gly Pro Gln Leu Asn Thr Ala Val Asn Asn Gly Gln
290 295 300
Val Ser Lys Ala Arg Leu Asp Asp Met Ala Lys Arg Ile Leu Ala Ser
305 310 315 320
Trp Tyr Leu Leu Glu Gln Asn Ser Gly Tyr Pro Ala Thr Asn Leu Lys
325 330 335
Ala Asn Val Gln Gly Asn His Lys Glu Asn Val Arg Ala Val Ala Arg
340 345 350
Asp Gly Ile Val Leu Leu Lys Asn Asp Asp Asn Ile Leu Pro Leu Lys
355 360 365
Lys Pro Ser Lys Leu Ala Ile Ile Gly Ser Ser Ser Val Val Asn Pro
370 375 380
Ala Gly Arg Asn Ala Cys Thr Asp Arg Gly Cys Asn Thr Gly Ala Leu
385 390 395 400
Gly Met Gly Trp Gly Ser Gly Thr Ala Asp Tyr Pro Tyr Phe Val Ala
405 410 415
Pro Tyr Asp Ala Leu Lys Thr Arg Ala Gln Ser Asp Gly Thr Thr Val
420 425 430
Asn Leu Leu Ser Ser Asp Ser Thr Ser Gly Val Ala Asn Ala Ala Ser
435 440 445
Gly Ala Asp Ala Ala Leu Val Phe Ile Thr Ala Asp Ser Gly Glu Gly
450 455 460
Tyr Ile Thr Val Glu Gly Val Thr Gly Asp Arg Pro Asn Leu Asp Pro
465 470 475 480
Trp His Asn Gly Asn Gln Leu Val Gln Ala Val Ala Gln Ala Asn Lys
485 490 495
Asn Thr Ile Val Val Val His Ser Thr Gly Pro Ile Ile Leu Glu Thr
500 505 510
Ile Leu Ala Gln Pro Gly Val Lys Ala Val Val Trp Ala Gly Leu Pro
515 520 525
Ser Gln Glu Asn Gly Asn Ala Leu Val Asp Val Leu Tyr Gly Leu Val
530 535 540
Ser Pro Ser Gly Lys Leu Pro Tyr Thr Ile Ala Lys Ser Glu Ser Asp
545 550 555 560
Tyr Gly Thr Ala Val Gln Arg Gly Gly Thr Asp Leu Phe Thr Glu Gly
565 570 575
Leu Phe Ile Asp Tyr Arg His Phe Asp Lys Asn Gly Ile Ala Pro Arg
580 585 590
Tyr Glu Phe Gly Phe Gly Leu Ser Tyr Thr Asn Phe Thr Tyr Ser Ser
595 600 605
Leu Ser Ile Thr Ser Thr Ala Ser Ser Gly Pro Ala Ser Gly Asp Thr
610 615 620
Ile Pro Gly Gly Arg Ala Asp Leu Trp Glu Thr Val Ala Thr Val Thr
625 630 635 640
Ala Val Val Lys Asn Thr Gly Gly Val Gln Gly Ala Glu Ala Pro Gln
645 650 655
Leu Tyr Ile Thr Leu Pro Ser Ser Ala Pro Ser Ser Pro Pro Lys Gln
660 665 670
Leu Arg Gly Phe Ala Lys Leu Lys Leu Ala Pro Gly Glu Ser Lys Thr
675 680 685
Ala Thr Phe Ile Leu Arg Arg Arg Asp Leu Ser Tyr Trp Asp Thr Gly
690 695 700
Ser Gln Asn Trp Val Val Pro Ser Gly Ser Phe Gly Val Val Val Gly
705 710 715 720
Ala Ser Ser Arg Asp Leu Arg Leu Asn Gly Lys Phe Asp Val Tyr
725 730 735
<210> 4
<211> 2208
<212> DNA
<213> 粗糙脉孢菌
<400> 4
atgcaccttc gaatatttgc ggtgttggcc gcgacttccc tcgcctgggc cgagactagc 60
gagaaacaag ctcgtcaagc tggctcaggt tttgcggcgt gggacgcagc ctattctcag 120
gcaagcactg ctctctccaa gctttcacag caagacaagg tcaacatcgt caccggagtc 180
ggctggaata agggcccatg tgttggcaac accccagcta ttgcatcaat cggttatccc 240
cagctctgtt tacaagacgg ccctctcggc attcggtttg gaggaagtgt caccgcgttc 300
acgcctggta tccaggcggc ttcaacatgg gacgtcgaac tgattcgaca gcgcggcgtc 360
tacctcggtg cagaagccag aggggttggc gtacatgtcc ttcttggacc cgtggccgga 420
gcgcttggca agatccccaa tggtggacgt aactgggagg gctttggtcc ggatccctac 480
ctcacaggta ttgccatgag cgaaacaatt gaagggatcc agagcaatgg tgtacaagct 540
tgcgccaagc acttcattct caacgaacag gagacaaacc gcgatactat cagcagtgtc 600
gtcgacgacc gcaccatgca tgaactatac ctcttccctt ttgccgatgc cgtacactca 660
aatgttgcaa gtgtgatgtg cagctacaac aaggtcaacg gtacgtgggc atgtgagaat 720
gacaaaatcc agaatggcct tctcaagaaa gagctaggct tcaaaggata tgtcatgagt 780
gattggaacg cccagcacac cacgaacggc gctgcaaaca gtggtatgga tatgacgatg 840
ccaggcagtg actttaatgg caagacgatc ctgtggggac cacagctcaa caccgccgtc 900
aacaatggcc aggtctccaa agcaagactg gacgacatgg ccaagcgcat tctcgcatcg 960
tggtatttac tcgagcaaaa ctcaggctac cctgcgacta acctcaaggc caatgttcaa 1020
ggaaaccaca aggagaacgt tcgcgcagtg gcaagagacg gcattgttct gctgaagaac 1080
gacgataaca tcctcccgct caagaagcct agcaagctgg caatcattgg gtcatcgtcc 1140
gttgtcaacc ctgcgggaag gaacgcctgc accgatcgag gatgcaacac cggtgcgctc 1200
ggcatgggtt ggggctccgg cacggccgat tacccctact tcgtagcacc ctatgatgct 1260
ctcaagacgc gggctcagtc cgacggaaca actgtcaacc tactcagctc tgacagcacc 1320
agcggcgtag ccaacgctgc ctccggagcc gacgcggcac tagtcttcat cacagccgat 1380
tccggcgaag gctacatcac ggtcgagggc gtgaccggcg accgtcccaa cctcgatccc 1440
tggcacaacg gcaaccagct agtccaagcc gtggctcaag ccaacaagaa caccattgtc 1500
gtcgtccaca gtaccggccc catcattctg gagactatcc tcgcgcagcc gggcgtcaag 1560
gcggtcgtgt gggccggtct ccccagccaa gagaacggca acgcccttgt cgatgtccta 1620
tacggcttgg tctctccctc gggtaagctg ccgtatacta tcgccaagag cgaaagcgac 1680
tacggcactg ccgtgcaaag gggagggacg gatctgttca ctgagggtct gttcatcgat 1740
taccgccact ttgacaagaa cggtatcgct ccccggtatg agttcggttt cggtctttcc 1800
tacacgaact tcacctactc ctccctctcc atcacctcca ccgcctcctc cggtcccgcc 1860
tcgggtgaca ccatccctgg cggccgcgcc gacctctggg aaaccgtggc aaccgtcact 1920
gccgtcgtca aaaacacggg tggtgtgcag ggcgccgagg caccccagct atacatcacc 1980
ttgccctctt ccgcgccgtc gagcccgccg aaacagctca gagggtttgc aaagctgaag 2040
ctggcgcccg gggagagcaa gacagctacg ttcattttgc ggaggaggga tttgagttat 2100
tgggatacgg gcagccagaa ttgggtggtg cctagtggca gctttggggt ggtagtgggt 2160
gctagttcga gggatttgag gttgaatggg aagtttgatg tttattga 2208
<210> 5
<211> 849
<212> PRT
<213> 粗糙脉孢菌
<400> 5
Met Asn His Leu Ser Ser Leu Tyr Glu Gln Leu Leu Arg Leu Pro Thr
1 5 10 15
Ser Leu Pro Gly Pro Gly Tyr Phe Ala Asn Gln Ala Lys Glu Val Val
20 25 30
Phe Ser Met Lys Gly Ala Val Ala Leu Ile Cys Thr Ala Val Ser Leu
35 40 45
Gln Ser Cys Ser Ser Leu Pro Ser Ser Ser Ser Ser Ser Ser Ser Thr
50 55 60
Ser Ser Ser Gln His His Asp Gly Gln His Pro Ser Glu Leu Pro Tyr
65 70 75 80
Tyr Gly Leu Ser Pro Pro Phe Tyr Pro Thr Pro Val Ala Asn Gly Thr
85 90 95
Ser Ser Ser Arg Trp Ser Ser Ala Tyr Gln His Ala Leu Ala Leu Thr
100 105 110
Ser Gln Met Thr Leu Leu Glu Leu Gln Asn Leu Thr Arg Gly Phe Pro
115 120 125
Gly Pro Cys Val Gly Asn Thr Gly Ser Ile Asp Arg Leu Ser Ile Pro
130 135 140
Pro Leu Cys Phe Tyr Asp Gly Pro Ser Gly Val Arg Gly Gln Glu Phe
145 150 155 160
Ala Ser Ala Phe Pro Ala Gly Ile His Leu Ala Ala Thr Trp Asp Ala
165 170 175
Asp Leu Met Tyr Arg Tyr Gly Arg Ala Val Gly Ala Glu Tyr Arg Gly
180 185 190
Lys Gly Val Asn Ile Ala Leu Gly Pro Leu Ala Gly Pro Leu Gly Arg
195 200 205
Val Ala Lys Gly Gly Arg Asn Trp Glu Gly Leu Gly Ser Asp Pro Tyr
210 215 220
Leu Ala Gly Val Gly Met Gly Arg Ile Val Glu Gly Val Gln Gly Glu
225 230 235 240
Gly Val Ile Ala Thr Ala Lys His Phe Leu Leu Asn Glu Gln Glu Tyr
245 250 255
Arg Arg Arg Trp Gly Gln Asp Ala Pro Asp Gly Glu Gly His Ala Ile
260 265 270
Ser Ala Asn Val Gly Asp Arg Ala Leu Arg Glu Tyr Val Trp Pro Phe
275 280 285
Met Asp Ala Leu Arg Ala Gly Ala Gly Ala Val Met Cys Gly Tyr Asn
290 295 300
Arg Ala Asn His Ser Tyr Val Cys Gln Asn Ser Lys Leu Leu Asn Gly
305 310 315 320
Ile Leu Lys Thr Glu Leu Gly Phe Glu Gly Phe Val Val Thr Asp Trp
325 330 335
Asp Ala Gln Met Ser Gly Val Ala Ser Ala Asn Ala Gly Ala Asp Met
340 345 350
Val Met Pro Arg Asp Gly Phe Trp Gly Glu Lys Leu Ile Glu Ala Val
355 360 365
Lys Asn Gly Ser Val Ala Glu Glu Arg Leu Asn Asp Met Ala Thr Arg
370 375 380
Val Leu Ala Ala Trp Leu Tyr Ala Gly Gln Asp Asp Gly Thr Tyr Pro
385 390 395 400
Pro Val Gly Val Ala Pro Gly Gly Glu Leu Pro Gly Pro Val Asp Val
405 410 415
Gln Ala Asp His Ala Asp Leu Ile Arg Glu Ile Gly Ala Ala Gly Thr
420 425 430
Val Leu Val Lys Asn Ile Asn Gly Thr Leu Pro Leu Thr Arg Pro Lys
435 440 445
Phe Leu Cys Val Tyr Gly Tyr Asp Ala Ile Val Lys Ser Thr Pro Trp
450 455 460
Glu Asn Pro Asp Arg Tyr Gly Gly Gly Tyr Asp Val Asn Arg Gly Trp
465 470 475 480
Thr Thr Phe Asn Gly Thr Leu Ile Thr Gly Gly Gly Ser Gly Ser Ser
485 490 495
Thr Pro Pro Tyr Val Val Ser Pro Phe Glu Ala Leu Ser His Arg Ile
500 505 510
Arg Lys Asp Arg Gly Met Leu Arg Trp Asp Phe His Ser Ala Asn Pro
515 520 525
Ser Gln Gln Tyr Leu Asn Ala Asp Ala Cys Leu Val Phe Ile Asn Ala
530 535 540
Tyr Ala Ser Glu Met Ser Asp Arg Pro Ala Leu Ser Asp Thr Phe Ser
545 550 555 560
Asp Asp Leu Val Leu Asn Ile Ala Ser Trp Cys Ser Gln Thr Ile Val
565 570 575
Ile Val His Ser Ala Gly Ile Arg Leu Val Asp Pro Trp Ile Ser His
580 585 590
Pro Asn Val Thr Ala Val Leu Met Ala Gly Leu Pro Gly Gln Glu Ser
595 600 605
Gly Asn Ser Leu Val Asp Ile Leu Tyr Gly Asp Val Asn Pro Ser Gly
610 615 620
Arg Leu Pro Tyr Thr Ile Ala Lys Lys Glu Ser Asp Tyr Gly His Leu
625 630 635 640
Leu Asn Pro Ser Thr Gly Gln Glu Glu Glu Gly Gly Asp Pro Phe Phe
645 650 655
Pro Glu Asp Asn Phe Val Glu Gly Leu His Ile Asp Tyr Arg Phe Phe
660 665 670
Asp Arg His Gly Ile Ala Pro Arg Phe Glu Phe Gly Phe Gly Leu Ser
675 680 685
Tyr Thr Thr Phe Ser Tyr Ser Asp Leu Gln Ile Tyr Val Ser Ile Ala
690 695 700
Pro Gly Val Val Gln Glu Thr Thr Met Pro Ser Leu Ala Ala Thr Thr
705 710 715 720
Arg Phe Ala Glu Phe Pro Asp Pro Asp Thr Pro Val Ile Gln Gly Gly
725 730 735
His Pro Asp Leu Trp Glu Thr Leu Phe Val Val Arg Cys Arg Val Glu
740 745 750
Asn Ser Gly Gly Lys Tyr Glu Gly Arg Glu Val Val Gln Leu Tyr Val
755 760 765
Gly Ile Pro Ala Leu Asn Glu Gly Gly Glu Asp Lys Glu Thr Pro Val
770 775 780
Arg Gln Leu Arg Gly Phe Lys Arg Val Gly Pro Leu Ala Pro Gly Glu
785 790 795 800
Asp Gly Glu Ala Glu Phe Glu Leu Thr Arg Arg Asp Leu Ser Val Trp
805 810 815
Asp Val Glu Met Gln Gln Trp Arg Leu Arg Arg Gly Lys Tyr Lys Ile
820 825 830
Trp Val Gly Ala Ser Ser Arg Asp Leu Arg Leu Ser Gly Thr Phe Asp
835 840 845
Ile
<210> 6
<211> 2550
<212> DNA
<213> 粗糙脉孢菌
<400> 6
atgaaccatc tcagcagtct ctacgagcaa ctgctacggc tgccaacgag cctcccaggc 60
ccaggctact ttgccaacca agccaaagaa gtcgtcttct ccatgaaggg cgccgtagcc 120
ctcatctgca ccgccgtctc tctgcaatca tgcagtagtc taccatcatc atcatcatca 180
tcatcatcaa catcgtcctc acaacatcat gacgggcaac acccctctga actcccctac 240
tacggtctca gtccgccgtt ttaccccacc cccgtcgcca acggcacctc ctcctctcgc 300
tggtcatccg cctaccagca cgccctcgct ctcacctcgc aaatgacgct cttggaactc 360
caaaacctca ctcgcggttt tcctggtcct tgcgtgggaa acacgggctc aatcgaccgc 420
ctatccatac caccgctctg cttctacgat ggcccctccg gtgtgcgcgg ccaggaattc 480
gcctccgctt ttcccgcggg cattcacctc gccgcgacat gggacgcgga cctcatgtac 540
cgctacggcc gggctgtggg cgctgagtat cgcggtaaag gtgtgaacat tgctctaggg 600
ccgctggccg ggccgttggg acgggtggca aagggaggga ggaattggga aggcttgggg 660
agtgatccgt acctggctgg tgttgggatg gggaggatcg tggagggcgt gcaaggggag 720
ggggtcattg ccactgcgaa gcatttcttg ctcaacgaac aggagtatag acgaagatgg 780
ggacaggatg cgccagacgg agaaggacat gccatcagcg cgaatgttgg ggacagagcg 840
ctgagggagt acgtctggcc gtttatggac gcactgcgcg cgggtgccgg agccgtgatg 900
tgtggatata atagggcgaa tcacagctat gtgtgccaga attccaagtt attgaatgga 960
atactcaaga cggagctagg gtttgaagga tttgtggtta ctgattggga tgcgcagatg 1020
agcggggtcg cgtcagccaa cgccggagcg gatatggtga tgccacggga cgggttctgg 1080
ggcgagaagt tgattgaagc tgtaaagaac ggatctgtgg ccgaggagcg cttgaacgac 1140
atggcgaccc gcgtgctggc ggcatggcta tacgcaggcc aggacgatgg gacctaccca 1200
cccgttggtg tcgcgccggg cggtgagctg cccggcccgg ttgacgtaca agcggaccac 1260
gcggatctca tccgcgaaat cggagcggcg ggcacagtcc tggtcaagaa tatcaacggc 1320
accctccccc taacccgacc caaattcctc tgcgtttacg gctacgacgc catcgtcaaa 1380
tccactccct gggagaaccc cgaccgctac ggcggcggct acgacgtcaa ccgcggctgg 1440
accaccttca acggcacctt gatcaccggc ggcggctccg gcagctccac gcccccttac 1500
gtcgtttccc ccttcgaagc cttgtcccac cgtatccgca aagaccgcgg catgctccgc 1560
tgggacttcc actcggccaa cccctcccag caatacctca acgcggacgc ctgcctcgtc 1620
ttcatcaacg cctacgcctc cgagatgtcc gaccgtcctg ccctctccga caccttcagc 1680
gacgacctcg tgctcaatat agcctcctgg tgctcccaaa ccatcgtcat cgtccactcg 1740
gccggcatcc ggctcgtcga cccgtggatc tcccacccca acgtcaccgc cgtgctcatg 1800
gccggcttac ccggccaaga atccggtaac agtctggtcg acatcttata cggcgacgtc 1860
aacccttccg gccgtttacc ttataccatt gccaaaaaag agtccgatta cgggcatcta 1920
ctcaaccctt ccaccggcca agaagaagag ggcggcgacc cgtttttccc cgaagataac 1980
tttgtggagg gactacacat cgattaccgc ttcttcgacc gtcatggcat cgctccccgc 2040
ttcgaattcg gcttcggttt gtcttatacc actttctctt attcggactt gcaaatttac 2100
gtctcaatcg ctcccggtgt cgtgcaagag acgacgatgc cctccctcgc tgccaccacc 2160
cggttcgccg agtttcccga tccggacaca cctgttatcc agggcggaca tccggacttg 2220
tgggagactt tgttcgtggt aaggtgtcgc gtggagaatt ccggggggaa atatgaggga 2280
agagaagttg tgcagttata cgttggtatt cccgctttga atgagggggg agaggataag 2340
gaaacgccgg ttcgtcagct gcgcgggttt aaacgcgtgg gaccgctggc gccgggggag 2400
gatggggagg cggagtttga gttgacgaga agggatttga gcgtttggga tgtggagatg 2460
cagcagtggc ggttgaggag agggaagtat aagatttggg tgggggctag tagtagggat 2520
ttgaggttga gtgggacatt tgatatttga 2550
<210> 7
<211> 871
<212> PRT
<213> 粗糙脉孢菌
<400> 7
Met Thr Ser Leu Ser Ser Ile Ala Val Gly Gly Gly Pro Phe Glu Arg
1 5 10 15
Asp Thr Asp Pro Asn Pro Ser Pro Ser Ser Ser Ile Asp Ser Asn Thr
20 25 30
Thr Pro Asp Thr Asp Phe Thr Pro Met Gly Ser Pro Phe Gln Glu Val
35 40 45
Ala Val Pro Lys Asn Lys Arg Phe Leu Ala Lys Lys Lys Leu Ala Asn
50 55 60
Leu Thr Gln Glu Glu Lys Ile Ser Leu Leu Thr Ala Ala Asp Phe Trp
65 70 75 80
Arg Thr Lys Ser Ile Pro Glu Lys Gly Ile Pro Ala Val Lys Thr Ser
85 90 95
Asp Gly Pro Asn Gly Ala Arg Gly Gly Ile Phe Val Gly Gly Thr Lys
100 105 110
Ala Ala Leu Phe Pro Cys Gly Ile Ser Leu Ala Ala Thr Trp Asn Lys
115 120 125
Ala Leu Leu Arg Glu Val Gly Arg His Leu Ala Glu Glu Ala Lys Ala
130 135 140
Arg Gln Ala Asn Ile Leu Leu Ala Pro Thr Val Cys Met His Arg His
145 150 155 160
Pro Leu Gly Gly Arg Asn Phe Glu Ser Phe Ser Glu Asp Pro Leu Leu
165 170 175
Thr Gly Lys Leu Ala Ala Gln Tyr Ile Lys Gly Leu Gln Glu Arg Gly
180 185 190
Val Ala Ala Thr Ile Lys His Phe Val Gly Asn Glu Gln Glu Thr His
195 200 205
Arg Leu Thr Val Asn Ser Val Ile Ala Glu Arg Pro Leu Arg Glu Ile
210 215 220
Tyr Leu Arg Pro Phe Glu Ile Ala Val Arg Glu Ala Lys Pro Trp Ala
225 230 235 240
Val Met Ser Ser Tyr Asn Leu Glu Val Leu Arg Asp Glu Trp Lys Phe
245 250 255
Asp Gly Ala Val Met Ser Asp Trp Gly Gly Val Asn Ser Thr Ala Glu
260 265 270
Ser Ile Lys Ala Gly Cys Asp Ile Glu Phe Pro His Ser Lys Lys Trp
275 280 285
Arg Tyr Glu Lys Val Met Glu Ala Leu Asn Lys Gly Glu Leu Ser Gln
290 295 300
Ala Asp Ile Asp Arg Ala Ala Glu Asn Val Leu Thr Leu Val Glu Arg
305 310 315 320
Thr Lys Gly Ser Asp Leu Thr Pro Glu Ala Ala Glu Arg Glu Asp Asp
325 330 335
Arg Glu Glu Thr Arg Glu Leu Ile Arg Glu Ala Gly Ile Gln Gly Leu
340 345 350
Thr Leu Leu Lys Asn Glu Gly Ser Ile Leu Pro Ile Asn Pro Lys Thr
355 360 365
Thr Lys Val Ala Val Ile Gly Pro Asn Ala Asn Arg Ala Ile Ala Gly
370 375 380
Gly Gly Gly Ser Ala Ser Leu Asn Pro Tyr Tyr Thr Thr Leu Pro Leu
385 390 395 400
Asp Ser Ile Arg Lys Val Ala Glu Lys Pro Val Thr Tyr Ala Gln Gly
405 410 415
Cys His Ile Tyr Lys Trp Leu Pro Val Ala Ser Pro Tyr Cys Ser Asp
420 425 430
Lys Thr Gly Lys Pro Gly Val Thr Ile Glu Trp Phe Lys Gly Asp Lys
435 440 445
Phe Lys Gly Glu Pro Val Val Ile Gln Arg Arg Thr Asn Thr Asp Leu
450 455 460
Phe Leu Trp Asp Ser Ala Pro Leu Ala Gln Thr Gly Pro Glu Trp Ser
465 470 475 480
Ala Ile Ala Thr Thr Tyr Leu Thr Pro Lys His Ser Gly Lys His Thr
485 490 495
Ile Ser Tyr Met Ser Val Gly Pro Gly Lys Leu Tyr Ile Asn Gly Lys
500 505 510
Leu Ser Leu Asp Leu Trp Asp Trp Thr Glu Glu Gly Glu Ala Met Phe
515 520 525
Asp Gly Ser Val Asp Tyr Leu Val Glu Leu Glu Met Val Ala Asn Arg
530 535 540
Pro Val Glu Leu Arg Val Glu Met Thr Asn Glu Leu Arg Pro Leu Ser
545 550 555 560
Lys Gln Lys Gln Met Gly Met Thr His Arg Tyr Gly Gly Cys Arg Ile
565 570 575
Gly Tyr Lys Glu Ala Asp Gln Ile Asp Tyr Leu Gln Gln Ala Ile Ala
580 585 590
Ala Ala Ser Ser Ala Asp Val Ala Ile Val Ile Val Gly Leu Asp Ala
595 600 605
Glu Trp Glu Ser Glu Gly Tyr Asp Arg Gln Thr Met Asp Leu Pro Tyr
610 615 620
Asn Gly Ser Gln Asp Arg Leu Ile Glu Ala Val Val Ala Ala Asn Pro
625 630 635 640
Asn Thr Val Val Val Asn Gln Ser Gly Ser Pro Val Thr Met Pro Trp
645 650 655
Ala Asp Arg Val Pro Ala Ile Leu Gln Ala Trp Tyr Gln Gly Gln Glu
660 665 670
Ala Gly Asn Ala Leu Ala Asp Val Leu Phe Gly Leu Arg Asn Pro Ser
675 680 685
Gly Lys Leu Pro Cys Thr Phe Pro Lys Arg Leu Glu Asp Thr Pro Ala
690 695 700
Tyr His Asn Trp Pro Gly Glu Asn Leu Glu Val Ile Tyr Gly Glu Gly
705 710 715 720
Ile Tyr Ile Gly Tyr Arg His Tyr Asp Arg Thr Lys Ile Ala Pro Leu
725 730 735
Phe Pro Phe Gly His Gly Leu Ser Tyr Thr Lys Phe Glu Tyr Gly Arg
740 745 750
Pro Ser Leu Ser Ser Arg Val Leu Arg Glu Asn Gly Val Ile Glu Leu
755 760 765
Cys Val Ala Ile Ser Asn Val Gly Glu Tyr Asp Gly Ala Glu Thr Val
770 775 780
Gln Val Tyr Val Arg Asp Glu Lys Ser Lys Leu Pro Arg Pro Glu Lys
785 790 795 800
Glu Leu Val Ala Phe Glu Lys Val Ala Leu Glu Arg Gly Glu Thr Lys
805 810 815
His Leu Arg Met Glu Leu Asp Lys Tyr Ala Val Gly Tyr Tyr Asp Thr
820 825 830
Asp Lys Lys Gly Trp Val Ala Glu Glu Gly Arg Phe Val Val Leu Val
835 840 845
Gly Ser Ser Ala Gly Asp Ile Lys Tyr Ser Val Pro Phe Glu Val Lys
850 855 860
Gln Thr Phe Thr Trp Val Phe
865 870
<210> 8
<211> 2616
<212> DNA
<213> 粗糙脉孢菌
<400> 8
atgacttcac tttcctcgat agccgttggg ggaggcccct tcgagcggga tactgatccc 60
aatccctccc cttccagcag tatcgacagc aacaccacgc ccgataccga cttcacgccc 120
atgggaagcc cgttccagga agtggccgtc cccaagaaca agaggttcct ggcgaagaag 180
aagttggcaa atctgacaca ggaggagaag atttccctcc taactgccgc agacttctgg 240
aggacaaagt ccattccgga gaagggtatt cctgcagtga aaacaagtga tggacccaat 300
ggcgcccgag gtggtatctt tgttggcggt accaaggccg cattattccc ctgcggcatt 360
tcattggccg ccacatggaa caaggccctc ctccgcgaag taggccgtca tttggccgag 420
gaagcaaagg ctcgtcaggc caacattctc ctcgcaccca cggtctgcat gcacaggcat 480
cccttgggag gaaggaactt tgagtctttc tctgaggatc cgcttcttac aggcaagctt 540
gccgctcagt atatcaaggg tcttcaggag agaggtgtgg ctgccaccat caagcacttt 600
gttggcaatg agcaagagac acacagattg acggtcaact ccgtgattgc cgaaaggcca 660
ctccgtgaga tttacctacg gccatttgag attgccgtgc gcgaggccaa gccgtgggct 720
gtcatgtctt cgtacaacct tgaggttctt cgagatgagt ggaaatttga cggtgctgtc 780
atgtccgatt ggggtggcgt caactctacc gcggagtcca tcaaggccgg ctgtgacatc 840
gaattccccc actccaaaaa atggcgttat gaaaaggtca tggaggccct gaacaagggc 900
gaactctccc aggctgacat tgacagagcg gctgaaaacg tcctgactct tgtcgagcga 960
acaaagggaa gcgatctgac acccgaagct gcggaaagag aagatgacag agaggaaacc 1020
cgggagctga tccgcgaggc tggcatccag ggtctcaccc ttttgaaaaa cgaaggatca 1080
attctgccca tcaatcccaa gaccaccaag gttgccgtca tcggccccaa cgccaaccga 1140
gccatcgctg gaggtggtgg gagtgccagc ttgaacccct attacaccac cctgcctctt 1200
gatagcatcc gcaaggttgc cgaaaagcct gttacctatg cccaaggctg ccacatctac 1260
aagtggctcc ctgttgcctc tccttactgc tccgacaaga ctggcaagcc cggtgttacc 1320
atagagtggt tcaagggtga caagttcaag ggagagcccg tagtcatcca acgacggacc 1380
aacaccgatc tcttcctctg ggactctgct cccctcgccc aaaccggccc cgaatggtcc 1440
gccattgcca ccacctacct cacccccaag cactccggca aacacaccat ctcctacatg 1500
tccgtcggcc ccggcaagct ctacatcaac ggcaagctct ccctcgacct ctgggactgg 1560
accgaagaag gcgaagccat gttcgacggc tccgtcgact acctcgtcga gctcgaaatg 1620
gtcgccaacc gccccgtcga gctccgcgtc gaaatgacca acgagctccg ccccctttcc 1680
aagcaaaagc aaatgggcat gacccaccgc tacggcggct gccgcatcgg ctacaaggaa 1740
gccgaccaaa tcgactacct ccaacaagcc atcgccgccg cctcctccgc cgacgtggcc 1800
atcgtcatcg tcggcctcga cgccgagtgg gaatccgagg ggtacgatcg ccaaaccatg 1860
gacctgccct acaacggctc gcaagaccgg ctcatcgaag ccgtcgtggc cgccaaccct 1920
aacaccgtcg tcgtcaacca gtccggttca cccgtcacca tgccctgggc tgatcgcgtc 1980
cctgccatct tgcaagcgtg gtaccaaggc caagaagcag gtaacgccct ggccgacgtg 2040
ctctttgggc tgcgcaaccc cagcggaaaa ctcccctgca cgttccccaa gcgtctcgaa 2100
gatacgccgg cgtaccataa ttggccgggc gagaacctcg aggttattta cggcgagggg 2160
atctacattg gctatcgaca ctatgaccgg accaaaatcg cgccgctctt cccctttggt 2220
catggtcttt cgtatacgaa gtttgagtat gggcggccgt cgctgtctag tcgcgtgttg 2280
agggagaatg gagttatcga gctgtgtgtg gcgattagta atgtgggcga gtatgatggg 2340
gcggagacgg tgcaggtgta tgtgcgggat gagaaatcca aactgccgag gccggagaag 2400
gagctggtgg cgtttgagaa ggtggcgctg gaaagggggg agacgaagca tttgaggatg 2460
gaactggata agtatgcggt ggggtattat gatacagaca aaaaggggtg ggtggcggag 2520
gaagggaggt ttgtggtttt ggtggggagt tcggcggggg atatcaagta ctctgtgcca 2580
tttgaggtta agcagacttt cacttgggtc ttttag 2616
<210> 9
<211> 875
<212> PRT
<213> 粗糙脉孢菌
<400> 9
Met Lys Phe Ala Ile Pro Leu Ala Leu Leu Ala Ser Gly Asn Leu Ala
1 5 10 15
Leu Ala Ala Pro Glu Pro Ile His Pro Ser His Gln Gln Leu Asn Lys
20 25 30
Arg Ser Leu Ala Tyr Ser Glu Pro His Tyr Pro Ser Pro Trp Met Asp
35 40 45
Pro Lys Ala Ile Gly Trp Glu Glu Ala Tyr Glu Lys Ala Lys Ala Phe
50 55 60
Val Ser Gln Leu Thr Leu Leu Glu Lys Val Asn Leu Thr Thr Gly Ile
65 70 75 80
Gly Trp Gly Ala Glu Gln Cys Val Gly Gln Thr Gly Ala Ile Pro Arg
85 90 95
Leu Gly Leu Lys Ser Met Cys Met Gln Asp Ala Pro Leu Ala Ile Arg
100 105 110
Gly Thr Asp Tyr Asn Ser Val Phe Pro Ala Gly Val Thr Thr Ala Ala
115 120 125
Thr Phe Asp Arg Gly Leu Met Tyr Lys Arg Gly Tyr Ala Leu Gly Gln
130 135 140
Glu Ala Lys Gly Lys Gly Val Thr Val Leu Leu Gly Pro Val Ala Gly
145 150 155 160
Pro Leu Gly Arg Ala Pro Glu Gly Gly Arg Asn Trp Glu Gly Phe Ser
165 170 175
Thr Asp Pro Val Leu Thr Gly Ile Ala Met Ala Glu Thr Ile Lys Gly
180 185 190
Thr Gln Asp Ala Gly Val Val Ala Cys Ala Lys His Phe Ile Gly Asn
195 200 205
Glu Gln Glu His Phe Arg Gln Val Gly Glu Ser Gln Asp Tyr Gly Tyr
210 215 220
Asn Ile Ser Glu Thr Leu Ser Ser Asn Ile Asp Asp Lys Thr Met His
225 230 235 240
Glu Met Tyr Leu Trp Pro Phe Val Asp Ala Ile Arg Ala Gly Val Gly
245 250 255
Ser Phe Met Cys Ala Tyr Thr Gln Ala Asn Asn Ser Tyr Ser Cys Gln
260 265 270
Asn Ser Lys Leu Leu Asn Asn Leu Leu Lys Gln Glu Asn Gly Phe Gln
275 280 285
Gly Phe Val Met Ser Asp Trp Gln Ala His His Ser Gly Val Ala Ser
290 295 300
Ala Ala Ala Gly Leu Asp Met Ser Met Pro Gly Asp Thr Met Phe Asn
305 310 315 320
Ser Gly Arg Ser Tyr Trp Gly Thr Asn Leu Thr Leu Ala Val Leu Asn
325 330 335
Gly Thr Val Pro Gln Trp Arg Ile Asp Asp Met Ala Met Arg Ile Met
340 345 350
Ala Ala Phe Phe Lys Val Gly Gln Thr Val Glu Asp Gln Glu Pro Ile
355 360 365
Asn Phe Ser Phe Trp Thr Leu Asp Thr Tyr Gly Pro Leu His Trp Ala
370 375 380
Ala Arg Lys Asp Tyr Gln Gln Ile Asn Trp His Val Asn Val Gln Gly
385 390 395 400
Asp His Gly Ser Leu Ile Arg Glu Ile Ala Ala Arg Gly Thr Val Leu
405 410 415
Leu Lys Asn Thr Gly Ser Leu Pro Leu Lys Lys Pro Lys Phe Leu Ala
420 425 430
Val Ile Gly Glu Asp Ala Gly Pro Asn Pro Leu Gly Pro Asn Gly Cys
435 440 445
Ala Asp Asn Arg Cys Asn Asn Gly Thr Leu Gly Ile Gly Trp Gly Ser
450 455 460
Gly Thr Gly Asn Phe Pro Tyr Leu Val Thr Pro Asp Gln Ala Leu Gln
465 470 475 480
Ala Arg Ala Val Gln Asp Gly Ser Arg Tyr Glu Ser Val Leu Arg Asn
485 490 495
His Ala Pro Thr Glu Ile Lys Ala Leu Val Ser Gln Gln Asp Ala Thr
500 505 510
Ala Ile Val Phe Val Asn Ala Asn Ser Gly Glu Gly Phe Ile Glu Ile
515 520 525
Asp Gly Asn Lys Gly Asp Arg Leu Asn Leu Thr Leu Trp Asn Glu Gly
530 535 540
Asp Ala Leu Val Lys Asn Val Ser Ser Trp Cys Asn Asn Thr Ile Val
545 550 555 560
Val Leu His Thr Pro Gly Pro Val Leu Leu Thr Glu Trp Tyr Asp Asn
565 570 575
Pro Asn Ile Thr Ala Ile Leu Trp Ala Gly Met Pro Gly Gln Glu Ser
580 585 590
Gly Asn Ser Ile Thr Asp Val Leu Tyr Gly Arg Val Asn Pro Ser Gly
595 600 605
Arg Thr Pro Phe Thr Trp Gly Ala Thr Arg Glu Ser Tyr Gly Thr Asp
610 615 620
Val Leu Tyr Glu Pro Asn Asn Gly Asn Glu Ala Pro Gln Leu Asp Tyr
625 630 635 640
Thr Glu Gly Val Phe Ile Asp Tyr Arg His Phe Asp Lys Ala Asn Ala
645 650 655
Ser Val Leu Tyr Glu Phe Gly Phe Gly Leu Ser Tyr Thr Thr Phe Glu
660 665 670
Tyr Ser Asn Leu Lys Ile Glu Lys His Gln Val Gly Glu Tyr Thr Pro
675 680 685
Thr Thr Gly Gln Thr Glu Ala Ala Pro Thr Phe Gly Asn Phe Ser Glu
690 695 700
Ser Val Glu Asp Tyr Val Phe Pro Ala Ala Glu Phe Pro Tyr Val Tyr
705 710 715 720
Gln Phe Ile Tyr Pro Tyr Leu Asn Ser Thr Asp Met Ser Ala Ser Ser
725 730 735
Gly Asp Ala Gln Tyr Gly Gln Thr Ala Glu Glu Phe Leu Pro Pro Lys
740 745 750
Ala Asn Asp Gly Ser Ala Gln Pro Leu Leu Arg Ser Ser Gly Leu His
755 760 765
His Pro Gly Gly Asn Pro Ala Leu Tyr Asp Ile Met Tyr Thr Val Thr
770 775 780
Ala Asp Ile Thr Asn Thr Gly Lys Val Ala Gly Asp Glu Val Pro Gln
785 790 795 800
Leu Tyr Val Ser Leu Gly Gly Pro Glu Asp Pro Lys Val Val Leu Arg
805 810 815
Gly Phe Asp Arg Leu Arg Val Glu Pro Gly Glu Lys Val Gln Phe Lys
820 825 830
Ala Val Leu Thr Arg Arg Asp Val Ser Ser Trp Asp Thr Val Lys Gln
835 840 845
Asp Trp Val Ile Thr Glu Tyr Ala Lys Lys Val Tyr Val Gly Pro Ser
850 855 860
Ser Arg Lys Leu Asp Leu Glu Glu Val Leu Pro
865 870 875
<210> 10
<211> 2628
<212> DNA
<213> 粗糙脉孢菌
<400> 10
atgaagttcg ccattccgct tgccttgctg gcgagcggca atctcgccct ggctgcgccc 60
gagccgatcc atccttctca tcaacagctc aataagaggt cgcttgccta ctcagaaccc 120
cattatccat caccatggat ggaccccaag gccatcggct gggaagaagc ctacgaaaag 180
gcaaaggcct ttgtgtccca gttgaccctg ctcgaaaaag tcaaccttac aaccggcatc 240
ggctggggcg ctgaacaatg cgttggccaa acaggtgcca tccctcggct tggcttgaag 300
agcatgtgta tgcaagatgc ccccttggcc atccgtggca ccgactacaa ctcagtcttc 360
cccgccgggg tcacaaccgc cgctacgttt gatcgaggtc tcatgtataa acgcggatat 420
gctttaggcc aggaagccaa gggcaaggga gtcacggttc tcctcggacc tgttgccggt 480
cccctcggcc gggcacccga aggcggccgc aactgggaag gcttctcgac cgaccctgtt 540
ctcaccggta tcgccatggc ggagaccatc aagggaacac aggacgctgg cgtcgtcgct 600
tgcgccaagc actttatcgg caacgagcaa gaacacttca gacaggttgg agagtcacag 660
gactacggat acaacatctc cgaaacgcta tcctccaaca ttgacgacaa gaccatgcac 720
gaaatgtatt tgtggccgtt tgttgatgcc atcagagctg gtgtaggctc cttcatgtgc 780
gcctacactc aggccaacaa ctcgtacagc tgccagaact cgaagctcct caacaacctg 840
ctcaagcaag aaaacggatt ccagggcttt gtgatgagcg attggcaagc ccatcactct 900
ggtgtcgcga gcgctgctgc cggtctcgac atgagcatgc ccggtgatac tatgtttaac 960
agcggccgca gctactgggg aaccaacctc acactcgccg tcctcaatgg aactgtcccg 1020
cagtggcgca ttgacgacat ggccatgcgc atcatggctg ctttcttcaa ggttggccaa 1080
accgtcgagg accaagagcc catcaacttc tccttctgga ccctcgacac gtacggcccg 1140
ctgcactggg ccgctcgcaa agattatcag cagatcaact ggcatgttaa cgttcagggc 1200
gatcacggaa gccttattag ggagattgca gctcgcggaa cagttctgtt gaaaaacacc 1260
ggctcccttc ccctgaaaaa gcccaagttt ctggccgtca ttggtgagga tgctgggccg 1320
aaccctcttg ggccgaacgg atgcgccgac aatagatgta acaatggtac cttgggtatt 1380
ggctggggtt ccggcaccgg taacttccct tatcttgtga cgcccgacca agccctgcaa 1440
gcccgcgctg ttcaggatgg atctcgctac gagagtgtct tgaggaacca tgcgcccacg 1500
gaaatcaagg ccctggtttc ccagcaggac gcaactgcca ttgtctttgt caacgccaac 1560
tctggtgaag gcttcatcga gattgatggc aacaaaggtg atcggctgaa cctcaccctt 1620
tggaatgagg gcgatgctct ggtcaagaat gtttccagct ggtgtaacaa caccattgtc 1680
gtcctccaca cacctggtcc cgttttgctc acggaatggt acgacaaccc caatattacc 1740
gccattctct gggccggtat gcccggtcag gagagcggca actccatcac ggacgtcttg 1800
tacggtagag tcaacccatc tgggcgcact cccttcacct ggggtgccac gcgcgaaagc 1860
tacggcaccg atgttctcta tgagccgaac aacgggaacg aagctcctca gctggactac 1920
accgaaggag tctttatcga ttaccgtcac tttgacaagg ccaacgcctc ggtcctttac 1980
gagtttggct ttggattgag ctataccacc tttgagtaca gcaacttgaa gatcgagaag 2040
caccaagtcg gcgagtacac tcccaccaca gggcagacgg aagctgcgcc cacttttgga 2100
aacttttctg aaagcgtgga agactacgta ttccctgccg cagagttccc ctacgtctac 2160
cagtttattt acccctacct caacagcacc gacatgtctg cttccagcgg cgatgcgcag 2220
tacggccaga ctgccgagga gttcttgccg cccaaggcca acgatggatc ggcgcagcct 2280
ttgcttcgct catccggcct tcatcacccc ggtggaaacc ctgctcttta cgacatcatg 2340
tacactgtta cggcggatat caccaacacg ggcaaggtgg cgggtgatga agtaccgcag 2400
ctgtacgtga gcctcggtgg gccagaggat cccaaggtgg tgctgagagg gtttgacaga 2460
cttagggttg agcctgggga gaaggtgcag ttcaaggcgg tgctgacgag gagggatgtg 2520
agttcatggg atacggtgaa gcaggattgg gtgattactg agtacgcgaa gaaggtgtac 2580
gtcgggccaa gctcgaggaa gttggatctc gaggaggttc ttccctga 2628
<210> 11
<211> 827
<212> PRT
<213> 粗糙脉孢菌
<400> 11
Met Ala Val Arg Ser Asn Phe Val Trp Leu Thr Leu Ala Leu Ser Phe
1 5 10 15
Phe Asp Leu Thr Cys Asn Ala Ser Pro Val Arg Gln Arg Ala Ala Val
20 25 30
Pro Asp Gly Phe Tyr Ala Ala Pro Tyr Tyr Pro Thr Pro Tyr Gly Gly
35 40 45
Trp Glu Asp Ser Trp Lys Asp Ser Tyr Ala Lys Ala Gln Ala Leu Val
50 55 60
Gly Lys Met Thr Leu Ala Glu Lys Thr Asn Ile Thr Gly Gly Ser Gly
65 70 75 80
Met Phe Met Gly Pro Cys Val Gly Asn Ser Gly Ser Ala Tyr Arg Val
85 90 95
Gly Phe Pro Gln Leu Cys Leu Gln Asp Gly Ala Leu Gly Val Gly Asn
100 105 110
Thr Asp His Asn Thr Ala Phe Pro Ala Gly Ile Thr Thr Gly Ala Thr
115 120 125
Phe Asp Lys Asp Leu Ile Tyr Ala Arg Ala Val Ala Ile Gly Lys Glu
130 135 140
Phe Arg Gly Lys Gly Ala His Val Phe Leu Gly Pro Met Val Gly Pro
145 150 155 160
Leu Gly Arg Lys Pro Leu Gly Gly Arg Asn Trp Glu Gly Phe Gly Ala
165 170 175
Asp Pro Val Leu Gln Gly Ile Ala Gly Ala Leu Thr Ile Lys Gly Val
180 185 190
Gln Glu Gln Gly Val Ile Ala Thr Ile Lys His Leu Ile Gly Asn Glu
195 200 205
Gln Glu Met Tyr Arg Met Tyr Asn Pro Phe Gln Ala Gly Tyr Ser Ser
210 215 220
Asn Ile Val Ala Ser Ala Leu Ala Gly Leu Asp Met Ser Met Pro Gly
225 230 235 240
Asp Thr Gln Ile Pro Leu Phe Gly Asn Ser Pro Phe Lys Phe His Leu
245 250 255
Thr Glu Ala Val Leu Asn Gly Ser Val Pro Val Asp Arg Leu Asn Asp
260 265 270
Met Ala Thr Arg Ile Val Ala Ala Trp Tyr Gln Phe Gly Gln Asp Lys
275 280 285
Asn Phe Pro Ala Val Asn Phe His Ser Tyr Val Ser Ser Glu Lys Gly
290 295 300
Leu Leu Tyr Pro Gly Ala Leu Pro Val Ser Pro Ile Gly Lys Val Asn
305 310 315 320
Trp Phe Val Asp Val Gln Ala Asp His Gly Ser Val Ala Arg Gln Val
325 330 335
Ala Gln Asp Ala Ile Thr Leu Leu Lys Asn Asp Asp Asn Phe Leu Pro
340 345 350
Leu Ser Thr Lys Ser Ser Leu Arg Ile Phe Gly Ser Asp Ala Arg Val
355 360 365
Asp Pro Asp Gly Pro Asn Ala Cys Gly Asn Arg Ala Cys Asn Lys Gly
370 375 380
Thr Leu Gly Met Gly Trp Gly Ser Gly Val Ala Asn Tyr Pro Tyr Phe
385 390 395 400
Asp Asp Pro Ile Thr Ala Ile Lys Lys Arg Val Glu Asn Val Lys Leu
405 410 415
Tyr Asp Ser Asp Asp Phe Pro His Thr Leu Thr Pro Ser Pro Thr Asp
420 425 430
Asp Asp Ile Ala Ile Val Phe Ile Asn Ser Ala Ala Gly Glu Asn Ser
435 440 445
Leu Thr Val Glu Gly Asn His Gly Asp Arg Asp Asn Asp Lys Phe Ser
450 455 460
Ala Trp His Asn Gly Asp Asn Leu Val Gln Lys Ala Ala Glu Asn Tyr
465 470 475 480
Lys Asn Val Ile Val Val Val His Thr Val Gly Pro Leu Ile Leu Glu
485 490 495
Pro Trp Ile Asp Leu Pro Ser Val Lys Ala Val Leu Phe Ala His Leu
500 505 510
Pro Gly Gln Glu Ala Gly Glu Ser Leu Ala Asn Val Leu Phe Gly Asp
515 520 525
Val Ser Pro Ser Gly His Leu Pro Tyr Ser Ile Thr Lys Lys Glu Asn
530 535 540
Asp Leu Pro Asp Ser Val Thr Lys Leu Val Lys Glu Ile Ile Gly Gln
545 550 555 560
Pro Gln Asp Thr Tyr Ser Glu Gly Leu Tyr Ile Asp Tyr Arg Trp Leu
565 570 575
Asn Lys Gln Gly Ile Lys Pro Arg Tyr Ala Phe Gly His Gly Leu Ser
580 585 590
Tyr Thr Thr Phe Asn Tyr Thr Asp Ala His Ile Lys Ile Val Asn Ala
595 600 605
Leu Ser Ser Ala Leu Pro Pro Ala Arg Gln Pro Lys Pro Ser Val Ala
610 615 620
Ile Leu Ser Thr Glu Ile Pro Pro Ala Ser Asp Ala Tyr Glu Pro Ala
625 630 635 640
Gly Phe Ser Lys Val Trp Arg Tyr Ile Tyr Ser Trp Leu Ser Lys Ser
645 650 655
Asp Ala Asp Lys Ala Tyr Ala Val Gly Thr Ser Ser Ser Ser Lys Ser
660 665 670
Gly Ser Gln Thr Tyr Pro Tyr Pro Glu Gly Tyr Ser Ser Val Gln Lys
675 680 685
Pro Gly Val Pro Ala Gly Gly Gly Gln Gly Gly Asn Pro Ala Leu Phe
690 695 700
Asp Thr Ile Leu Glu Leu Asp Val Thr Val Gln Asn Thr Gly Ser Arg
705 710 715 720
His Lys Gly Lys Ala Ser Val Gln Ala Tyr Ile Gln Phe Pro Thr Asp
725 730 735
Ser Gly Tyr Asp Thr Pro Ile Ile Gln Leu Arg Asp Phe Ala Lys Thr
740 745 750
Lys Glu Leu Gly Thr Gly Glu Ser Glu Thr Val Thr Leu Arg Leu Arg
755 760 765
Arg Lys Asp Leu Ser Val Trp Asp Thr Gln Lys Gln Asn Trp Val Ala
770 775 780
Pro Gly Ala Leu Gly Ala Asn Gly Lys Ser Lys Arg Tyr Ile Val Trp
785 790 795 800
Leu Gly Glu Gly Ser Asp Lys Leu Phe Thr Arg Cys Phe Ser Asp Thr
805 810 815
Leu Val Cys Glu Arg Gly Val Glu Pro Pro Val
820 825
<210> 12
<211> 2484
<212> DNA
<213> 粗糙脉孢菌
<400> 12
atggctgtcc gatcgaactt tgtgtggctc actctcgcct tgagcttttt cgaccttacc 60
tgcaatgcaa gtccagtgcg acaacgagcg gccgtcccag atggtttcta tgcggctcct 120
tactacccaa cgccatacgg tggctgggaa gattcatgga aagacagcta tgcaaaggca 180
caggcactgg tgggcaagat gacactagca gagaagacga acatcacggg tggaagtggt 240
atgtttatgg gcccttgtgt aggaaacagc ggcagtgcat accgagtcgg ctttcctcag 300
ctttgtttgc aggatggtgc tctcggtgtg ggcaacacgg accataacac tgcatttcca 360
gctggtatca ccacgggagc gacgttcgac aaggacctta tctatgctcg cgcagtggct 420
attggtaaag aatttcgcgg caaaggagcc cacgtttttc ttggtcccat ggtaggtccc 480
ctaggtcgca agccactcgg tggccgcaac tgggaaggct tcggtgccga tcccgttctc 540
caaggtattg ccggtgcctt gactatcaag ggtgtccagg aacagggtgt tattgctact 600
atcaagcatc tcattggaaa cgagcaggag atgtatcgca tgtacaaccc atttcaagca 660
ggatacagtt cgaatattgt cgcatcagca ctggccggcc ttgacatgag catgcctgga 720
gacactcaaa tacctctctt tggtaatagc cccttcaagt tccatctcac ggaggctgtc 780
ttgaacggct ccgtgccagt ggacaggcta aacgacatgg caaccaggat tgtggccgct 840
tggtatcagt tcggccaaga caagaacttc ccagctgtta actttcactc atacgtatcg 900
agtgagaagg gacttctata tccaggtgct cttccggtct cgcccattgg taaggttaac 960
tggtttgtgg atgttcaagc ggaccacggc tcggtcgcac gccaggtcgc ccaagatgcc 1020
attactcttc tgaagaatga tgataatttc ctgccccttt ctaccaagtc atccctcagg 1080
atcttcggct ctgatgctcg ggttgaccct gatggcccaa atgcttgcgg aaaccgcgcc 1140
tgcaacaagg gcactctggg aatgggctgg ggctccggcg ttgcgaacta cccctatttt 1200
gatgacccca tcactgccat caagaagcgg gtcgaaaatg tcaagctcta cgacagcgat 1260
gacttccccc acacactcac tccctcgccc actgatgacg acattgcaat tgtctttatc 1320
aactccgccg cgggcgaaaa ctcgctcacc gttgaaggca accatggtga tcgcgacaat 1380
gacaagttct ccgcttggca caatggagat aatctcgtcc agaaggccgc ggagaactac 1440
aaaaacgtca tagtcgttgt ccacacagtc ggccccctta tactcgagcc ctggattgac 1500
ctgccctccg tgaaggccgt cctcttcgcc cacctccccg gccaagaagc cggcgagtcg 1560
ctcgccaacg ttctcttcgg cgacgtctca ccctccggcc acctccccta ctccatcacc 1620
aagaaggaga acgacctccc tgacagtgta accaagctcg tcaaggaaat catcggccag 1680
ccccaagata cctacagtga aggcctctat atcgattacc gctggctcaa taagcaaggc 1740
atcaagcccc gctatgcctt cggccacggt ctgagttaca ccaccttcaa ctacaccgac 1800
gctcacatca agatcgtcaa cgccctctcc tccgccctgc cgcccgctcg ccagcccaag 1860
ccctccgtcg ccatcctctc caccgagata ccgcccgctt cagatgccta tgagcccgcc 1920
ggcttctcca aggtctggcg ctacatttac tcctggctat ccaagtccga cgccgacaag 1980
gcctatgccg tcggtacctc ttcttcttcc aagtccggct cccaaactta cccctacccc 2040
gagggctact cgagcgtcca aaagcctggt gtccctgccg gcggcggaca gggtggtaac 2100
ccggccctct ttgacaccat cctcgagctg gacgtgactg tgcaaaacac tggttctcga 2160
cacaagggta aagcctctgt gcaggcgtat atccagtttc ccacggacag cgggtacgac 2220
acgcctatta tccagttgcg tgactttgcc aagacgaagg agttgggcac cggggagagc 2280
gagacggtta cgttaaggct gaggaggaag gatttgagcg tgtgggatac gcaaaagcag 2340
aattgggtgg ctcctggtgc cttgggggct aatgggaaga gtaagcggta cattgtgtgg 2400
ttgggtgagg ggagtgataa gcttttcact aggtgtttca gcgatacttt ggtttgtgaa 2460
aggggggttg agccacctgt ttga 2484
<210> 13
<211> 791
<212> PRT
<213> 粗糙脉孢菌
<400> 13
Met Thr Arg Lys Met Pro Thr Leu Val Arg Pro Thr His Asn Gly Glu
1 5 10 15
Arg Tyr Glu Ile Thr Asn Pro Thr Ala Met Pro Lys Ala Ala Gly Phe
20 25 30
Leu Trp Asn Gln Lys Met Met Ile Gln Ile Thr Cys Arg Gly Phe Ala
35 40 45
Thr Ala Gln Phe Met Gln Pro Glu Pro Ala Lys Tyr Ala Tyr Ala Pro
50 55 60
Asn Ile Glu Ala Lys Thr Phe Met Gln Pro Glu Pro Asn Tyr Tyr Ala
65 70 75 80
His His Pro Gly Arg Phe Val Tyr Ile Lys Asp Glu Glu Thr Gly Arg
85 90 95
Leu Phe Ser Ala Pro Tyr Glu Pro Val Arg Ala Pro His Asp Arg Phe
100 105 110
Val Phe Ser Ala Gly Lys Thr Asp Val Phe Trp Val Ile Glu Ser Met
115 120 125
Gly Ile Arg Val Glu Met Thr Met Gly Leu Pro Thr His His Val Ala
130 135 140
Glu Leu Trp Thr Ile Lys Val Lys Asn Leu Ser Ser Arg Pro Arg Lys
145 150 155 160
Leu Ser Val Thr Pro Tyr Phe Pro Ile Gly Tyr Met Ser Trp Met Asn
165 170 175
Gln Ser Ala Glu Trp Asn His Asn Leu Asn Gly Ile Val Ala Ser Cys
180 185 190
Val Thr Pro Tyr Gln Lys Ala Ala Asp Tyr Phe Lys Asn Lys Tyr Leu
195 200 205
Lys Asp Lys Thr Tyr Phe Leu Cys Asp Val Pro Pro Asp Ser Trp Glu
210 215 220
Ala Ser Gln Gln Ala Phe Glu Gly Glu Gly Gly Leu His Asn Pro Ser
225 230 235 240
Ala Leu Gln Glu Arg Asn Leu Ser Gly Ser Asp Ala Arg Tyr Glu Thr
245 250 255
Pro Thr Ala Ala Val Gln Tyr Lys Ile Ala Leu Gly Thr Gly Glu Gln
260 265 270
Gln Glu Tyr Arg Phe Leu Phe Gly Pro Ala His Asp Glu Ala Glu Ile
275 280 285
Gly Ala Met Arg Ser Lys Tyr Leu Ser Lys Glu Gly Phe Glu Gln Thr
290 295 300
Ala Ala Asp Tyr Ala Ala Tyr Met Ala Arg Gly Arg Gly Cys Leu His
305 310 315 320
Val Glu Thr Pro Asp Lys Asp Leu Asp Asn Phe Ile Asn Asn Trp Leu
325 330 335
Pro Arg Gln Val Tyr Tyr His Gly Asp Val Asn Arg Leu Thr Thr Asp
340 345 350
Pro Gln Thr Arg Asn Tyr Leu Gln Asp Asn Met Gly Met Asn Tyr Ile
355 360 365
Lys Pro Glu Val Ser Arg Arg Ala Phe Leu Thr Ala Ile Ala Gln Gln
370 375 380
Glu Ala Thr Gly Ala Met Pro Asp Gly Ile Leu Leu Val Glu Gly Ala
385 390 395 400
Glu Leu Lys Tyr Ile Asn Gln Val Pro His Thr Asp His Cys Val Trp
405 410 415
Leu Pro Val Thr Leu Glu Ala Tyr Leu Asn Glu Thr Gly Asp Tyr Ser
420 425 430
Leu Leu Lys Glu Lys Val Pro Ser Ala Asn Gly Asp Lys Leu Thr Val
435 440 445
Phe Glu Arg Phe Cys Arg Ala Met Asp Trp Leu Leu Lys Ser Arg Asp
450 455 460
His Arg Gly Leu Ser Tyr Ile Ala Gln Gly Asp Trp Cys Asp Pro Met
465 470 475 480
Asn Met Val Gly Tyr Lys Gly Lys Gly Val Ser Gly Trp Leu Thr Leu
485 490 495
Ala Thr Ala Phe Ser Leu Asn Ile Trp Ala Lys Val Cys Asp His Glu
500 505 510
Gly Glu Thr Asp Leu Ala Lys Arg Phe Arg Glu Gly Ala Asp Ala Cys
515 520 525
Asn Ala Ala Ala Asn Glu His Leu Trp Asp Gly Glu Trp Phe Ala Arg
530 535 540
Gly Ile Thr Asp Asp Asn Val Val Phe Gly Ile Lys Glu Asp Lys Glu
545 550 555 560
Gly Arg Ile Trp Leu Asn Pro Gln Ser Trp Ser Ile Leu Ser Gly Ala
565 570 575
Ala Ser Pro Glu Gln Ile Asp Lys Met Leu Pro Gln Ile Asp Ser His
580 585 590
Leu Asn Thr Pro Tyr Gly Ile Gln Met Phe Gly Pro Pro Tyr Thr Lys
595 600 605
Met Arg Glu Asp Val Gly Arg Val Thr Gln Lys Ala Ile Gly Ser Ala
610 615 620
Glu Asn Ala Ala Val Tyr Asn His Ala Gly Ile Phe Phe Ile His Ser
625 630 635 640
Leu Tyr Glu Leu Gly Ala Gln Gln Asp Arg Ala Phe Thr Leu Leu Arg
645 650 655
Gln Met Leu Pro Gly Pro Thr Asp Thr Asp Tyr Ile Gln Arg Gly Gln
660 665 670
Leu Pro Ile Tyr Ile Pro Asn Tyr Tyr Arg Gly Ala Trp Lys Glu Cys
675 680 685
Pro Arg Thr Ala Gly Arg Ser Ser Gln Leu Phe Asn Thr Gly Thr Val
690 695 700
Ser Trp Val Tyr Arg Cys Ile Ile Glu Gly Leu Cys Gly Leu Arg Gly
705 710 715 720
Asp Gly Glu Gly Leu Leu Ile Arg Pro Gln Leu Pro Ser Ser Trp Asn
725 730 735
Ser Met Lys Val Thr Arg Glu Phe Arg Gly Ala Thr Phe Asn Val Asp
740 745 750
Ile Arg Arg Gly Asn Val Lys Glu Val Thr Val Arg Asn Gly Asp Lys
755 760 765
Val Leu Pro Ala Pro His Val Lys Asp Ile Glu Pro Gly Gln Thr Tyr
770 775 780
Asn Leu Thr Val Thr Ile Pro
785 790
<210> 14
<211> 2376
<212> DNA
<213> 粗糙脉孢菌
<400> 14
atgaccagga aaatgccgac tctggtccgc ccgacccata acggcgagcg ctacgaaatc 60
accaacccca cagccatgcc caaggccgct ggcttcctgt ggaaccaaaa gatgatgatt 120
cagatcacct gccgtggttt tgcgactgcc cagttcatgc agcccgagcc tgcaaagtac 180
gcctatgcgc ccaatatcga ggccaagacc ttcatgcaac cggagccgaa ctactacgcc 240
caccatcccg gccgctttgt gtacatcaag gatgaggaga ccggccggct tttctctgct 300
ccctacgaac ccgtccgcgc cccacacgat cgcttcgtct tctccgccgg caagactgac 360
gtcttctggg tcatcgagtc gatgggaatt cgcgtggaga tgaccatggg cctccccacc 420
caccatgttg ccgaactctg gaccatcaaa gtcaaaaacc tttcaagccg tcctcggaaa 480
ctcagtgtga ccccctactt ccccattgga tacatgtcgt ggatgaacca gtcggccgag 540
tggaatcata acctcaacgg aatcgttgcc agctgtgtca ccccttacca gaaggccgcc 600
gattacttca agaacaagta tctcaaggac aagacctact tcctttgtga tgtccctcct 660
gactcctggg aagccagtca gcaagccttt gaaggtgagg gtggtcttca caaccccagt 720
gccctacaag agaggaatct cagcggcagt gatgcgcggt acgaaactcc cacggcagct 780
gtgcaataca agattgctct tggcacgggc gagcaacaag agtaccggtt cctctttggc 840
cccgcccatg atgaagctga gatcggagcc atgcggtcca agtacctaag caaggaaggt 900
ttcgaacaga ctgcagctga ttacgccgct tacatggccc gtgggcgcgg atgtctccac 960
gtggaaaccc cagataagga tctcgacaac ttcatcaaca actggctgcc ccgacaggtc 1020
tactaccacg gcgatgtcaa ccgcttgacc actgatccac aaacacgcaa ctacctacag 1080
gacaacatgg gcatgaacta catcaaaccc gaagtatccc gcagggcttt cctcacagcc 1140
attgcccagc aagaagctac cggcgccatg cccgacggca tccttttggt tgagggtgcc 1200
gaactcaaat acatcaacca ggtacctcac actgatcact gtgtgtggtt gccagttaca 1260
ctggaggcat acctcaacga gaccggtgat tacagcttgc taaaggagaa agtgccgagt 1320
gccaacggcg acaagctcac cgtcttcgag cgcttctgcc gtgctatgga ctggctgctc 1380
aagtctcgtg accatcgtgg tctgagttat attgcccagg gtgactggtg tgatcccatg 1440
aacatggtgg gctacaaggg caagggtgtt tctggctggc tcactctggc cacggccttt 1500
tccctcaaca tttgggccaa ggtttgcgat catgaaggcg agaccgatct cgccaagcgc 1560
ttccgtgagg gtgccgacgc atgcaacgct gctgcaaacg agcatctttg ggacggtgaa 1620
tggtttgctc gcggcatcac tgacgacaat gttgtctttg gcatcaagga ggacaaggaa 1680
ggccgcatct ggttgaaccc ccagtcctgg tccatcctca gcggtgctgc cagccctgaa 1740
cagattgaca agatgttgcc ccagattgac tcgcatctca acacaccata tggtatccag 1800
atgttcggtc ccccttacac caagatgcgc gaggatgtcg gtcgcgtaac gcaaaaagcc 1860
attggctcgg ccgagaacgc cgctgtgtac aaccacgctg gaatcttctt catccatagc 1920
ctgtacgagc ttggagcaca gcaagaccga gcatttaccc tattacggca aatgctccct 1980
ggtcctactg acaccgacta tatccaacga ggccagctgc ccatctatat tcccaactac 2040
taccgaggcg cctggaagga gtgtcctcgc acagctggcc ggtccagtca gttgtttaac 2100
acgggtaccg tttcttgggt ttaccgctgc atcattgaag ggctctgtgg cttgcgcggc 2160
gatggcgaag gtctactcat ccggccacag ctaccgagct cctggaacag catgaaggtc 2220
actcgcgagt ttcggggcgc caccttcaat gtcgacattc gtcgtggcaa tgtcaaggag 2280
gtcactgtca ggaatggcga caaggtcctg cctgcgcctc acgtcaagga catcgagcca 2340
ggccagacgt acaaccttac agtgactatt ccgtaa 2376
<210> 15
<211> 430
<212> PRT
<213> 粗糙脉孢菌
<400> 15
Met Gln Arg Val Gln Ser Ala Val Asp Phe Ser Asn Leu Leu Asn Pro
1 5 10 15
Ser Glu Ser Thr Ala Glu Lys Arg Asp His Ser Gly Ser Pro Arg Gln
20 25 30
Gln Thr Ala Gln Pro Gln Gln Gln Gln Gln Gln Pro Gln Pro Glu Ala
35 40 45
Asp Met Ala Thr Val Gly Leu Leu Arg Pro Asn Gly Pro Leu Pro Gly
50 55 60
Ala Gln Ala Thr Glu Pro Ala Asn Glu Leu Pro Arg Pro Tyr Lys Cys
65 70 75 80
Pro Leu Cys Asp Lys Ala Phe His Arg Leu Glu His Gln Thr Arg His
85 90 95
Ile Arg Thr His Thr Gly Glu Lys Pro His Ala Cys Gln Phe Pro Gly
100 105 110
Cys Ser Lys Lys Phe Ser Arg Ser Asp Glu Leu Thr Arg His Ser Arg
115 120 125
Ile His Ser Asn Pro Asn Ser Arg Arg Gly Asn Lys Gly Gln Gln Gln
130 135 140
Gln Gln His Pro Leu Val His Asn His Gly Leu Gln Pro Asp Met Met
145 150 155 160
Pro Pro Pro Gly Pro Lys Ala Ile Arg Ser Ala Pro Pro Thr Ala Met
165 170 175
Ser Ser Pro Asn Val Ser Pro Pro His Ser Tyr Ser Pro Tyr Asn Phe
180 185 190
Ala Pro Ser Gly Leu Asn Pro Tyr Ser His Ser Arg Ser Ser Ala Gly
195 200 205
Ser Gln Ser Gly Pro Asp Ile Ser Leu Leu Ala Arg Ala Ala Gly Gln
210 215 220
Val Glu Arg Asp Gly Ala Ala His His His Phe Gln Pro Arg Phe Gln
225 230 235 240
Phe Tyr Gly Asn Thr Leu His Ala Ala Thr Ala Ser Arg Asn Gln Leu
245 250 255
Pro Gly Leu Gln Ala Tyr His Met Ser Arg Ser His Ser His Glu Asp
260 265 270
His Asp Asp His Tyr Gly Gln Ser Tyr Arg His Ala Lys Arg Ser Arg
275 280 285
Pro Asn Ser Pro Asn Ser Thr Ala Pro Ser Ser Pro Thr Phe Ser His
290 295 300
Asp Ser Leu Ser Pro Thr Pro Asp His Thr Pro Leu Ala Thr Pro Ala
305 310 315 320
His Ser Pro Arg Leu Arg Pro His Pro Gly Leu Glu Leu Pro Pro Phe
325 330 335
Arg Asn Leu Ser Leu Gly Gln Gln His Thr Thr Pro Ala Leu Ala Pro
340 345 350
Leu Glu Pro Ala Leu Asp Gly Gln Phe Ser Leu Pro Gln Thr Pro Pro
355 360 365
Ala Ala Pro Arg Ser Ser Gly Met Ser Leu Thr Asp Ile Ile Ser Arg
370 375 380
Pro Asp Gly Thr Gln Arg Lys Leu Pro Val Pro Lys Val Ala Val Gln
385 390 395 400
Asp Leu Leu Gly Pro Ala Asp Gly Phe Asn Pro Ser Val Arg Asn Ser
405 410 415
Ser Ser Thr Ser Leu Ser Gly Ala Glu Met Met Asp Arg Leu
420 425 430
<210> 16
<211> 1293
<212> DNA
<213> 粗糙脉孢菌
<400> 16
atgcaacgcg tacagtcagc agtagatttc tccaatctgc tgaatccatc cgaatccacg 60
gccgagaaga gagatcacag cggctcccca agacaacaaa ccgcacaacc acaacagcag 120
cagcaacaac cacagcccga agcagacatg gcgacagtcg gcttgttgcg ccccaacggg 180
cccctgcccg gtgcccaagc tactgagccc gccaacgagc tcccacggcc ttacaagtgc 240
cctctgtgcg acaaggcttt ccaccgcctg gagcatcaga caagacacat tcgcacccac 300
acaggagaga agccgcacgc gtgccagttc ccggggtgta gcaagaagtt ctcgcgttcg 360
gacgagctga ctcggcactc gaggatacac agcaatccta actcgcggag gggaaacaag 420
ggccagcagc agcagcagca tccccttgtc cacaaccacg ggctgcagcc cgacatgatg 480
cctcctcctg gccccaaggc tatccgctct gctccgccta ctgccatgtc ctctcccaac 540
gtgtcacccc ctcactctta cagcccttat aactttgctc cctcgggcct caacccctat 600
agccattccc gtagctccgc gggaagccaa agtggtcctg atatctcgct cctcgcgagg 660
gccgccggac aagttgagcg ggacggcgcc gctcatcatc actttcagcc acgattccag 720
ttctacggaa acactctgca tgccgccacg gcttccagga atcagctccc tgggctccag 780
gcctatcaca tgtccaggtc gcactcccac gaggatcacg atgaccacta tggccagtct 840
tacaggcatg ccaagaggtc gaggcccaac tcgcccaact cgacagcacc ttcttccccg 900
acattctctc acgactcgct atcgcctact cccgatcaca ctccacttgc tactcctgct 960
cactcgcctc gtctgcgccc tcatcctggc cttgagctgc cccctttcag gaacctttcc 1020
ttgggccaac agcacacaac gcctgcgctg gctcctctcg agcccgctct tgatgggcag 1080
ttctctcttc cccagactcc tcccgctgcg cccaggagca gtggcatgtc cttgaccgac 1140
atcatcagcc gacctgatgg cactcaaagg aaacttcctg tgcccaaggt tgctgtccag 1200
gatctgcttg gaccagccga tgggttcaac ccaagtgtca ggaactcttc ttctaccagc 1260
ctctctggag cggagatgat ggaccggttg taa 1293
<210> 17
<211> 714
<212> PRT
<213> 粗糙脉孢菌
<400> 17
Met Ser Asn Pro Arg Arg Arg Pro Ala Gly Gly Leu Thr Leu Lys Thr
1 5 10 15
Asn Met Leu Leu Gln Lys Gly Ala Thr Phe His Ser Pro Thr Thr Pro
20 25 30
Ala Ser Gly Asp Ser Ser Glu Arg Val Phe Val Pro Pro Ser Leu Pro
35 40 45
Arg Arg Ser His Thr Asn Leu Asp Asp Val Ile Asp Ser Arg Cys Arg
50 55 60
Arg Val Ala Leu Ala Leu Asp Ala Ile Glu Arg Gln Leu Ala Ser Ser
65 70 75 80
Asn Asp Thr Phe Ala Ser Ala Ser Arg Ser Asp Lys Cys Ile Pro Pro
85 90 95
Pro Arg Gly Leu Leu Glu Arg Asn Leu Asp Ser Pro Ile Met Pro Lys
100 105 110
Glu Val Glu Pro Glu Arg Arg Met Leu Arg Pro Arg Thr Arg Arg Ser
115 120 125
Ser Arg His His Asp Ser Asp Ser Gly Leu Gly Ser Ser Ile Ala Ser
130 135 140
Thr Ser Glu Lys Asp Ala Ser Ser Lys Ala Lys Thr Thr Arg Thr Ser
145 150 155 160
Ala Val Ala Arg Ser Ala Thr Ala Arg Ala Ala Ser Thr Pro Asp Leu
165 170 175
Pro Gly Leu Gly Asp Arg Ala Thr Asn Arg Ile Val Glu Tyr Ile Leu
180 185 190
Lys Pro Leu Leu Ala Lys Pro Asn Leu Lys Glu Phe His Ser Leu Val
195 200 205
Leu Glu Cys Pro Lys Lys Ile Gln Glu Lys Glu Ile Leu Cys Leu Arg
210 215 220
Asp Leu Glu Lys Thr Leu Ile Leu Val Ala Pro Ala Thr Val Gln His
225 230 235 240
Leu Gly Asp Arg Glu Leu Thr Arg Pro Arg Asp Leu Pro Tyr Thr Ser
245 250 255
Gly Tyr Phe Val Asp Leu Val Asp Gln Phe Tyr Asn Tyr Ala Arg Gln
260 265 270
Ile Ala Glu Ser Asn Lys Thr Lys Glu Gly Ala Asn Asp Met Asp Ile
275 280 285
Asp Pro Thr Asp Gln Ile Lys Ile His Gly Gly Pro His Ile Asn Gly
290 295 300
Arg Leu Ser Glu Leu Val Arg Val Lys Lys Asn Gly Gln Ala Ile Ser
305 310 315 320
Leu Ala Thr Gly Leu Pro Val Asp Leu Cys Asp Lys Ala Pro Glu Thr
325 330 335
Pro Val Asn Phe Lys Arg Ser Gln Ser Glu Glu Ala Leu Asp Glu Glu
340 345 350
Glu Val Met Arg Ser Met Ala Arg Arg Lys Lys Asn Ala Ser Pro Glu
355 360 365
Glu Leu Ala Pro Lys Arg Cys Arg Glu Pro Gly Cys Asn Lys Glu Phe
370 375 380
Lys Arg Pro Cys Asp Leu Thr Lys His Glu Lys Thr His Ser Arg Pro
385 390 395 400
Trp Lys Cys Pro Val Lys Thr Cys Lys Tyr His Glu Tyr Gly Trp Pro
405 410 415
Thr Glu Lys Glu Met Asp Arg His His Asn Asp Lys His Ser Ser Ala
420 425 430
Pro Pro Met Tyr Glu Cys Leu Phe Lys Pro Cys Pro Tyr Lys Ser Lys
435 440 445
Arg Glu Ser Asn Cys Lys Gln His Met Glu Lys Ala His Gly Trp Thr
450 455 460
Tyr Val Arg Thr Lys Thr Asn Gly Lys Lys Pro Ser Thr Leu Pro Ser
465 470 475 480
Leu Gly Pro Asp Ser Gly His Pro Thr Pro Gln Leu Gln Asn Ile Gly
485 490 495
Thr Pro Ser Ser Asp Arg Ser Met Ser Ile Ala Thr Pro Ser Asp Asp
500 505 510
Trp Asn Ala Gly Leu Tyr Gln Thr Asn Ile Glu Phe Pro Ala Tyr Ala
515 520 525
Pro Glu Phe Asn Phe Asn Thr Ile Pro Gln Gln Leu Glu Leu Asp Tyr
530 535 540
Ser Pro Ile Asp Asn Gly Thr Pro Ser Pro Asp Ser Gly Met Asp His
545 550 555 560
Asn Ser Ala Tyr Gln Asp Leu Asn Glu Phe Thr Leu Ile Asp Asp Ile
565 570 575
Tyr Gly Ala Thr Val Gln Leu Pro Asn Gln Val Ile Ser Pro Phe Tyr
580 585 590
Leu Lys Asp Met Gly Gln His Leu Gly Ala Tyr Thr Ala Pro Asp Leu
595 600 605
Cys Gln Pro His Pro Ala His Ile Ser Pro Ile Gly Gln Gly Asn Thr
610 615 620
Met Leu Phe Thr Pro Pro Thr Ser Leu Gly Glu Val Asp Glu Gly Phe
625 630 635 640
Glu Asp His Asp Phe Ala Met Ser Asn Cys Asn Asn Val Pro Gly Asp
645 650 655
Phe Ile Leu Tyr Pro Pro Thr Thr Asp Ala Tyr Ser Lys Pro Thr Phe
660 665 670
Thr Glu Ser Leu Phe Ala Asn Val Asp Ile Pro Ser Met Ala Ala Gly
675 680 685
Tyr Ser Gln Pro Ser Ser Gln Asp Ile Leu His Ala Tyr Gln Gly Asp
690 695 700
Trp Thr Ser His Asp Met Asn Ala Tyr Phe
705 710
<210> 18
<211> 2145
<212> DNA
<213> 粗糙脉孢菌
<400> 18
atgtcgaacc ctcgcaggag accggcgggt ggattgaccc tcaagacgaa catgctccta 60
caaaagggag ctaccttcca ctctcccact actccagcct cgggggattc ttctgagcgc 120
gtcttcgtcc cgccgtcgct ccctcgccgc tcccacacca acttggacga tgttatcgac 180
tcccgctgcc gccgcgttgc cttagctttg gatgcgatcg agaggcaatt ggcctcttca 240
aatgacacct ttgcctcggc tagtcgcagt gacaagtgca ttcccccgcc acgtgggctg 300
ctggaaagaa acttggattc gcctatcatg cccaaggagg ttgagcctga gcgccggatg 360
ctccgtccac ggacacgccg ttcatctcgg caccacgatt cagacagcgg tctaggaagc 420
tcgattgctt ctacttccga gaaggatgcc tcttccaagg ccaagacgac gaggacttcg 480
gctgtcgcca gatccgctac cgcccgcgca gcatcgacac ctgatcttcc gggccttggg 540
gaccgtgcta ccaaccgcat cgttgaatac attttgaagc ctctgcttgc aaagccgaat 600
ctgaaagagt ttcactcgct tgtgcttgag tgtcccaaaa agatccagga gaaggagatc 660
ttgtgcctaa gagatcttga gaagactttg atcctcgtgg ctccggcaac tgtccagcac 720
cttggcgacc gcgagctgac tcgccctcgc gatctccctt acactagcgg ttattttgtt 780
gatctcgttg atcaattcta taactatgcc cggcagatcg cggaaagcaa caagaccaag 840
gaaggcgcca atgacatgga tattgatccc actgatcaaa tcaagataca cggtggccct 900
cacatcaacg gcaggctttc ggaactggtc cgcgtgaaga agaacggaca ggccatctcc 960
ttggctactg gattgcctgt ggacctttgt gataaggcgc cagaaacccc ggtaaacttc 1020
aagcgctccc agagcgagga ggccctggat gaagaggagg ttatgcgctc catggctcgc 1080
cggaagaaga acgccagccc tgaggagctg gcgccgaaga gatgccgcga gcctggttgc 1140
aacaaggagt tcaagcgccc ttgtgacctg accaagcacg agaagacaca ttctagacca 1200
tggaagtgcc cggtcaagac atgcaagtac cacgagtacg gctggcccac cgagaaggaa 1260
atggatcgtc atcacaacga caagcactcc tcagccccac ctatgtacga gtgcctcttc 1320
aagccttgcc cctacaagtc caagcgcgag tccaactgca agcagcatat ggaaaaggcc 1380
catggctgga cgtacgttag gactaaaacc aatggcaaga agcccagtac gcttcccagc 1440
ttggggcccg attccggcca tcccactccc cagctgcaga acattggtac cccttcgagc 1500
gaccgcagta tgagcatcgc cactccctcc gatgattgga acgctgggct gtaccagacc 1560
aacattgagt tccctgcata tgctcccgag ttcaacttca acaccatccc gcagcagctt 1620
gagcttgact attcacccat tgacaacggc actccatcac cggattctgg catggaccac 1680
aattctgcgt atcaagatct caacgaattc acgctcattg atgacatcta cggtgcgact 1740
gtgcaactgc ccaaccaagt catttctccc ttctacctca aagacatggg acagcatctc 1800
ggcgcgtaca ccgcgcccga cctttgccag ccacatcccg cccatatttc ccccattggg 1860
caaggaaaca cgatgctttt cacgccccct acttcattgg gggaagttga tgagggcttt 1920
gaggatcacg attttgccat gagtaactgc aataatgtcc ctggagactt cattttgtat 1980
cctccaacca ctgacgccta ctcaaaaccc acgttcactg aatcactttt cgcgaacgtg 2040
gacatcccga gcatggctgc cggctattcc cagccatcat ctcaggacat cttgcacgcc 2100
tatcaaggtg attggacttc gcatgatatg aatgcttact tctaa 2145
<210> 19
<211> 619
<212> PRT
<213> 粗糙脉孢菌
<400> 19
Met Lys Phe Leu Gly Ile Ala Ala Leu Val Ala Gly Leu Leu Ala Pro
1 5 10 15
Ser Leu Val Leu Gly Ala Pro Ala Pro Gly Thr Glu Gly Val Asn Leu
20 25 30
Leu Thr Pro Val Asp Lys Arg Gln Asp Ser Gln Ala Glu Arg Tyr Gly
35 40 45
Gly Gly Gly Gly Gly Gly Cys Asn Ser Pro Thr Asn Arg Gln Cys Trp
50 55 60
Ser Pro Gly Phe Asn Ile Asn Thr Asp Tyr Glu Leu Gly Thr Pro Asn
65 70 75 80
Thr Gly Lys Thr Arg Arg Tyr Lys Leu Thr Leu Thr Glu Thr Asp Asn
85 90 95
Trp Ile Gly Pro Asp Gly Val Ile Lys Asp Lys Val Met Met Val Asn
100 105 110
Asp Lys Ile Ile Gly Pro Thr Ile Gln Ala Asp Trp Gly Asp Tyr Ile
115 120 125
Glu Ile Thr Val Ile Asn Lys Leu Lys Ser Asn Gly Thr Ser Ile His
130 135 140
Trp His Gly Met His Gln Arg Asn Ser Asn Ile Gln Asp Gly Val Asn
145 150 155 160
Gly Val Thr Glu Cys Pro Ile Pro Pro Arg Gly Gly Ser Lys Val Tyr
165 170 175
Arg Trp Arg Ala Thr Gln Tyr Gly Thr Ser Trp Tyr His Ser His Phe
180 185 190
Ser Ala Gln Tyr Gly Asn Gly Ile Val Gly Pro Ile Val Ile Asn Gly
195 200 205
Pro Ala Ser Ala Asn Tyr Asp Val Asp Leu Gly Pro Phe Pro Leu Thr
210 215 220
Asp Tyr Tyr Tyr Asp Thr Ala Asp Arg Leu Val Leu Leu Thr Gln His
225 230 235 240
Ala Gly Pro Pro Pro Ser Asn Asn Val Leu Phe Asn Gly Phe Ala Lys
245 250 255
His Pro Thr Thr Gly Ala Gly Gln Tyr Ala Thr Val Ser Leu Thr Lys
260 265 270
Gly Lys Lys His Arg Leu Arg Leu Ile Asn Thr Ser Val Glu Asn His
275 280 285
Phe Gln Leu Ser Leu Val Asn His Ser Met Thr Ile Ile Ser Ala Asp
290 295 300
Leu Val Pro Val Gln Pro Tyr Lys Val Asp Ser Leu Phe Leu Gly Val
305 310 315 320
Gly Gln Arg Tyr Asp Val Ile Ile Asp Ala Asn Gln Ala Val Gly Asn
325 330 335
Tyr Trp Phe Asn Val Thr Phe Gly Gly Ser Lys Leu Cys Gly Asp Ser
340 345 350
Asp Asn His Tyr Pro Ala Ala Ile Phe Arg Tyr Gln Gly Ala Pro Lys
355 360 365
Ala Leu Pro Thr Asn Gln Gly Val Ala Pro Val Asp His Gln Cys Leu
370 375 380
Asp Leu Asn Asp Leu Lys Pro Val Leu Gln Arg Ser Leu Asn Thr Asn
385 390 395 400
Ser Ile Ala Leu Asn Thr Gly Asn Thr Ile Pro Ile Thr Leu Asp Gly
405 410 415
Phe Val Trp Arg Val Asn Gly Thr Ala Ile Asn Ile Asn Trp Asn Lys
420 425 430
Pro Val Leu Glu Tyr Val Leu Thr Gly Asn Thr Asn Tyr Ser Gln Ser
435 440 445
Asp Asn Ile Val Gln Val Glu Gly Val Asn Gln Trp Lys Tyr Trp Leu
450 455 460
Ile Glu Asn Asp Pro Asp Gly Ala Phe Ser Leu Pro His Pro Ile His
465 470 475 480
Leu His Gly His Asp Phe Leu Ile Leu Gly Arg Ser Pro Asp Val Thr
485 490 495
Ala Ile Ser Gln Thr Arg Tyr Val Phe Asp Pro Ala Val Asp Met Ala
500 505 510
Arg Leu Asn Gly Asn Asn Pro Thr Arg Arg Asp Thr Ala Met Leu Pro
515 520 525
Ala Lys Gly Trp Leu Leu Ile Ala Phe Arg Thr Asp Asn Pro Gly Ser
530 535 540
Trp Leu Met His Cys His Ile Ala Trp His Val Ser Gly Gly Leu Ser
545 550 555 560
Asn Gln Phe Leu Glu Arg Ala Gln Asp Leu Arg Asn Ser Ile Ser Pro
565 570 575
Ala Asp Lys Lys Ala Phe Asn Asp Asn Cys Asp Ala Trp Arg Ala Tyr
580 585 590
Phe Pro Asp Asn Ala Pro Phe Pro Lys Asp Asp Ser Gly Leu Arg Ser
595 600 605
Gly Val Lys Ala Arg Glu Val Lys Met Lys Trp
610 615
<210> 20
<211> 1860
<212> DNA
<213> 粗糙脉孢菌
<400> 20
atgaaattct tgggcattgc cgccctcgtt gcgggccttc ttgccccttc acttgttctc 60
ggtgctcctg cacctggcac cgagggagtg aatctcctca ctcccgttga taagagacag 120
gactcccaag ccgaacgtta tggaggtggc ggagggggtg gttgcaactc cccgaccaac 180
cggcagtgtt ggtctccagg attcaacatc aacaccgact atgaactcgg aactcctaat 240
accggcaaga ccagacggta caagctcacc ctcaccgaga ctgataactg gattggccct 300
gatggtgtca taaaggacaa ggtcatgatg gtcaacgaca agatcattgg acctaccatt 360
caggctgatt ggggcgacta tattgagatc accgtcatca acaaactgaa gtcgaatggc 420
acctcgatcc actggcacgg catgcaccag cgcaactcta acatccagga cggcgtcaac 480
ggcgtgaccg agtgccccat cccacccaga ggcggcagca aagtgtaccg ctggcgggcc 540
acccagtacg ggacgtcgtg gtaccactcg catttctcag cgcagtacgg caatggcatt 600
gttgggccca ttgtcatcaa cggaccggcc tcggccaact acgacgtcga cctcggccct 660
ttcccgctca cggactacta ttacgacacc gctgaccgtc tcgtgctgct cacacagcat 720
gccggccctc cgccgagcaa taatgtgctc ttcaacggct tcgccaagca tccgaccaca 780
ggcgctggcc agtatgccac cgtgtcgctc accaagggta agaagcatcg gctgcggctc 840
atcaacacct cggtcgagaa tcacttccaa ctgtcgctgg tgaatcactc catgaccatc 900
atctcggcgg atctggttcc cgtacagccc tacaaggtcg acagcctgtt cctcggcgtc 960
ggtcagcgct acgatgtcat catcgacgcc aaccaggccg ttggaaacta ctggttcaat 1020
gtgacgtttg gtgggagcaa gctgtgcggt gattcggaca accactaccc ggccgccatt 1080
ttccggtacc agggcgcgcc gaaggcactg ccgaccaacc agggcgttgc gccggtcgac 1140
caccaatgcc tggacctcaa cgacctgaag cccgtcctgc agcgcagcct gaacaccaac 1200
agcatcgcac tcaacactgg caacacgatc cccatcacgc tcgacgggtt cgtctggcgg 1260
gtcaatggaa cggccatcaa catcaactgg aacaagccgg tgctggagta cgtgctgacg 1320
ggcaacacca actactcgca gtccgacaac attgtgcagg tcgagggcgt caaccaatgg 1380
aagtactggc tcatcgagaa cgaccccgac ggtgccttta gcctgccgca tcccatccac 1440
ctgcacggcc acgactttct catcctcggt cggtcgcccg acgtgaccgc catcagccag 1500
acgcgctacg tgtttgaccc ggccgtggac atggctcggt tgaacggcaa caacccgacg 1560
cggcgcgaca cggccatgct cccggccaag ggctggctgc tcatcgcgtt ccgcaccgac 1620
aatcccgggt cgtggctgat gcactgccac attgcctggc acgtgtcggg aggtctgtcg 1680
aaccagttcc tggagcgggc gcaggacctc agaaacagta tcagccctgc ggataagaag 1740
gcctttaacg acaactgtga cgcgtggagg gcgtacttcc ctgacaatgc gccgttcccc 1800
aaggacgact ctggcctgag gagcggtgtc aaggcgaggg aggtgaagat gaagtggtag 1860
<210> 21
<211> 829
<212> PRT
<213> 粗糙脉孢菌
<400> 21
Met Arg Thr Thr Ser Ala Phe Leu Ser Gly Leu Ala Ala Val Ala Ser
1 5 10 15
Leu Leu Ser Pro Ala Phe Ala Gln Thr Ala Pro Lys Thr Phe Thr His
20 25 30
Pro Asp Thr Gly Ile Val Phe Asn Thr Trp Ser Ala Ser Asp Ser Gln
35 40 45
Thr Lys Gly Gly Phe Thr Val Gly Met Ala Leu Pro Ser Asn Ala Leu
50 55 60
Thr Thr Asp Ala Thr Glu Phe Ile Gly Tyr Leu Glu Cys Ser Ser Ala
65 70 75 80
Lys Asn Gly Ala Asn Ser Gly Trp Cys Gly Val Ser Leu Arg Gly Ala
85 90 95
Met Thr Asn Asn Leu Leu Ile Thr Ala Trp Pro Ser Asp Gly Glu Val
100 105 110
Tyr Thr Asn Leu Met Phe Ala Thr Gly Tyr Ala Met Pro Lys Asn Tyr
115 120 125
Ala Gly Asp Ala Lys Ile Thr Gln Ile Ala Ser Ser Val Asn Ala Thr
130 135 140
His Phe Thr Leu Val Phe Arg Cys Gln Asn Cys Leu Ser Trp Asp Gln
145 150 155 160
Asp Gly Val Thr Gly Gly Ile Ser Thr Ser Asn Lys Gly Ala Gln Leu
165 170 175
Gly Trp Val Gln Ala Phe Pro Ser Pro Gly Asn Pro Thr Cys Pro Thr
180 185 190
Gln Ile Thr Leu Ser Gln His Asp Asn Gly Met Gly Gln Trp Gly Ala
195 200 205
Ala Phe Asp Ser Asn Ile Ala Asn Pro Ser Tyr Thr Ala Trp Ala Ala
210 215 220
Lys Ala Thr Lys Thr Val Thr Gly Thr Cys Ser Gly Pro Val Thr Thr
225 230 235 240
Ser Ile Ala Ala Thr Pro Val Pro Thr Gly Val Ser Phe Asp Tyr Ile
245 250 255
Val Val Gly Gly Gly Ala Gly Gly Ile Pro Val Ala Asp Lys Leu Ser
260 265 270
Glu Ser Gly Lys Ser Val Leu Leu Ile Glu Lys Gly Phe Ala Ser Thr
275 280 285
Gly Glu His Gly Gly Thr Leu Lys Pro Glu Trp Leu Asn Asn Thr Ser
290 295 300
Leu Thr Arg Phe Asp Val Pro Gly Leu Cys Asn Gln Ile Trp Lys Asp
305 310 315 320
Ser Asp Gly Ile Ala Cys Ser Asp Thr Asp Gln Met Ala Gly Cys Val
325 330 335
Leu Gly Gly Gly Thr Ala Ile Asn Ala Gly Leu Trp Tyr Lys Pro Tyr
340 345 350
Thr Lys Asp Trp Asp Tyr Leu Phe Pro Ser Gly Trp Lys Gly Ser Asp
355 360 365
Ile Ala Gly Ala Thr Ser Arg Ala Leu Ser Arg Ile Pro Gly Thr Thr
370 375 380
Thr Pro Ser Gln Asp Gly Lys Arg Tyr Leu Gln Gln Gly Phe Glu Val
385 390 395 400
Leu Ala Asn Gly Leu Lys Ala Ser Gly Trp Lys Glu Val Asp Ser Leu
405 410 415
Lys Asp Ser Glu Gln Lys Asn Arg Thr Phe Ser His Thr Ser Tyr Met
420 425 430
Tyr Ile Asn Gly Glu Arg Gly Gly Pro Leu Ala Thr Tyr Leu Val Ser
435 440 445
Ala Lys Lys Arg Ser Asn Phe Lys Leu Trp Leu Asn Thr Ala Val Lys
450 455 460
Arg Val Ile Arg Glu Gly Gly His Ile Thr Gly Val Glu Val Glu Ala
465 470 475 480
Phe Arg Asn Gly Gly Tyr Ser Gly Ile Ile Pro Val Thr Asn Thr Thr
485 490 495
Gly Arg Val Val Leu Ser Ala Gly Thr Phe Gly Ser Ala Lys Ile Leu
500 505 510
Leu Arg Ser Gly Ile Gly Pro Lys Asp Gln Leu Glu Val Val Lys Ala
515 520 525
Ser Ala Asp Gly Pro Thr Met Val Ser Asn Ser Ser Trp Ile Asp Leu
530 535 540
Pro Val Gly His Asn Leu Val Asp His Thr Asn Thr Asp Thr Val Ile
545 550 555 560
Gln His Asn Asn Val Thr Phe Tyr Asp Phe Tyr Lys Ala Trp Asp Asn
565 570 575
Pro Asn Thr Thr Asp Met Asn Leu Tyr Leu Asn Gly Arg Ser Gly Ile
580 585 590
Phe Ala Gln Ala Ala Pro Asn Ile Gly Pro Leu Phe Trp Glu Glu Ile
595 600 605
Thr Gly Ala Asp Gly Ile Val Arg Gln Leu His Trp Thr Ala Arg Val
610 615 620
Glu Gly Ser Phe Glu Thr Pro Asp Gly Tyr Ala Met Thr Met Ser Gln
625 630 635 640
Tyr Leu Gly Arg Gly Ala Thr Ser Arg Gly Arg Met Thr Leu Ser Pro
645 650 655
Thr Leu Asn Thr Val Val Ser Asp Leu Pro Tyr Leu Lys Asp Pro Asn
660 665 670
Asp Lys Ala Ala Val Val Gln Gly Ile Val Asn Leu Gln Lys Ala Leu
675 680 685
Ala Asn Val Lys Gly Leu Thr Trp Ala Tyr Pro Ser Ala Asn Gln Thr
690 695 700
Ala Ala Asp Phe Val Asp Lys Gln Pro Val Thr Tyr Gln Ser Arg Arg
705 710 715 720
Ser Asn His Trp Met Gly Thr Asn Lys Met Gly Thr Asp Asp Gly Arg
725 730 735
Ser Gly Gly Thr Ala Val Val Asp Thr Asn Thr Arg Val Tyr Gly Thr
740 745 750
Asp Asn Leu Tyr Val Val Asp Ala Ser Ile Phe Pro Gly Val Pro Thr
755 760 765
Thr Asn Pro Thr Ala Tyr Ile Val Val Ala Ala Glu His Ala Ala Ala
770 775 780
Lys Ile Leu Ala Gln Pro Ala Asn Glu Ala Val Pro Lys Trp Gly Trp
785 790 795 800
Cys Gly Gly Pro Thr Tyr Thr Gly Ser Gln Thr Cys Gln Ala Pro Tyr
805 810 815
Lys Cys Glu Lys Gln Asn Asp Trp Tyr Trp Gln Cys Val
820 825
<210> 22
<211> 2490
<212> DNA
<213> 粗糙脉孢菌
<400> 22
atgaggacca cctcggcctt tctcagcggc ctggcggcgg tggcttcatt gctgtcgccc 60
gccttcgccc aaaccgctcc caagaccttc actcatcctg ataccggcat tgtcttcaac 120
acatggagtg cttccgattc ccagaccaaa ggtggcttca ctgttggtat ggctctgccg 180
tcaaatgctc ttactaccga cgcgactgaa ttcatcggtt atctggaatg ctcctccgcc 240
aagaatggtg ccaatagcgg ttggtgcggt gtttctctca gaggcgccat gaccaacaat 300
ctactcatta ccgcctggcc ttctgacgga gaagtctaca ccaatctcat gttcgccacg 360
ggttacgcca tgcccaagaa ctacgctggt gacgccaaga tcacccagat cgcgtccagc 420
gtgaacgcta cccacttcac ccttgtcttt aggtgccaga actgtttgtc atgggaccaa 480
gacggtgtca ccggcggcat ttctaccagc aataaggggg cccagctcgg ttgggtccag 540
gcgttcccct ctcccggcaa cccgacttgc cctacccaga tcactctcag tcagcatgac 600
aacggtatgg gccagtgggg agctgccttt gacagcaaca ttgccaatcc ctcttatact 660
gcatgggctg ccaaggccac caagaccgtt accggtactt gcagtggtcc agtcacgacc 720
agtattgccg ccactcctgt tcccactggc gtttcttttg actacattgt cgttggtggt 780
ggtgccggtg gtattcccgt cgctgacaag ctcagcgagt ccggtaagag cgtgctgctc 840
atcgagaagg gtttcgcttc cactggtgag catggtggta ctctgaagcc cgagtggctg 900
aataatacat cccttactcg cttcgatgtt cccggtcttt gcaaccagat ctggaaagac 960
tcggatggca ttgcctgctc cgataccgat cagatggccg gctgcgtgct cggcggtggt 1020
accgccatca acgccggtct ctggtacaag ccctacacca aggactggga ctacctcttc 1080
ccctctggct ggaagggcag cgatatcgcc ggtgctacca gcagagccct ctcccgcatt 1140
ccgggtacca ccactccttc tcaggatgga aagcgctacc ttcagcaggg tttcgaggtt 1200
cttgccaacg gcctcaaggc gagcggctgg aaggaggtcg attccctcaa ggacagcgag 1260
cagaagaacc gcactttctc ccacacctca tacatgtaca tcaatggcga gcgtggcggt 1320
cctctagcga cttacctcgt cagcgccaag aagcgcagca acttcaagct gtggctcaac 1380
accgctgtca agcgcgtcat ccgtgagggc ggccacatta ccggtgtgga ggttgaggcc 1440
ttccgcaacg gcggctactc cggaatcatc cccgtcacca acaccaccgg ccgcgtcgtt 1500
ctttccgccg gcaccttcgg cagcgccaag atccttctcc gttccggcat tggccccaag 1560
gaccagctcg aggtggtcaa ggcctccgcc gacggcccta ccatggtcag caactcgtcc 1620
tggattgacc tccccgtcgg ccacaacctg gttgaccaca ccaacaccga caccgtcatc 1680
cagcacaaca acgtgacctt ctacgacttt tacaaggctt gggacaaccc caacacgacc 1740
gacatgaacc tgtacctcaa tgggcgctcc ggcatcttcg cccaggccgc gcccaacatt 1800
ggccccttgt tctgggagga gatcacgggc gccgacggca tcgtccgtca gctgcactgg 1860
accgcccgcg tcgagggcag cttcgagacc cccgacggct acgccatgac catgagccag 1920
taccttggcc gtggcgccac ctcgcgcggc cgcatgaccc tcagccctac cctcaacacc 1980
gtcgtgtctg acctcccgta cctcaaggac cccaacgaca aggccgctgt cgttcagggt 2040
atcgtcaacc tccagaaggc tctcgccaac gtcaagggtc tcacctgggc ttaccctagc 2100
gccaaccaga cggctgctga ttttgttgac aagcaacccg taacctacca atcccgccgc 2160
tccaaccact ggatgggcac caacaagatg ggcaccgacg acggccgcag cggcggcacc 2220
gcagtcgtcg acaccaacac gcgcgtctat ggcaccgaca acctgtacgt ggtggacgcc 2280
tcgattttcc ccggtgtgcc gaccaccaac cctaccgcct acattgtcgt cgccgctgag 2340
catgccgcgg ccaaaatcct ggcgcaaccc gccaacgagg ccgttcccaa gtggggctgg 2400
tgcggcgggc cgacgtatac tggcagccag acgtgccagg cgccatataa gtgcgagaag 2460
cagaatgatt ggtattggca gtgtgtgtag 2490
<210> 23
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成的构建体
<220>
<221> 变体
<222> 1
<223> Xaa = Leu, Ile,Val, Met, Phe, Ser, Thr或Cys
<220>
<221> 变体
<222> 2
<223> Xaa = Leu, Ile, Val, Phe, Tyr或Ser
<220>
<221> 变体
<222> 3
<223> Xaa = Leu, Ile或Val
<220>
<221> 变体
<222> 4
<223> Xaa = Leu, Ile, Val, Met, Ser或Thr
<220>
<221> 变体
<222> 8
<223> Xaa = Leu, Ile, Val, Met, Phe, Ala或Arg
<220>
<221> 变体
<222> 9
<223> Xaa = Cys, Ser, Ala, Gly或Asn
<400> 23
Xaa Xaa Xaa Xaa Glu Asn Gly Xaa Xaa
1 5
<210> 24
<211> 18
<212> PRT
<213> 人工序列
<220>
<223> 合成的构建体
<220>
<221> 变体
<222> 1, 2
<223> Xaa = Leu, Ile, Val或Met
<220>
<221> 变体
<222> 3
<223> Xaa = Lys或Arg
<220>
<221> 变体
<222> 4
<223> Xaa = 任意氨基酸
<220>
<221> 变体
<222> 5
<223> Xaa = Glu, Gln, Lys, Arg或Asp
<220>
<221> 变体
<222> 6, 7, 8, 9
<223> Xaa = 任意氨基酸
<220>
<221> 变体
<222> 11
<223> Xaa = Leu, Ile, Val, Met, Phe, Thr或Cys
<220>
<221> 变体
<222> 12
<223> Xaa = Leu, Ile, Val或Thr
<220>
<221> 变体
<222> 13
<223> Xaa = Leu, Ile, Val, Met或Phe
<220>
<221> 变体
<222> 14
<223> Xaa = Ser或Thr
<220>
<221> 变体
<222> 16, 17
<223> Xaa = 任意氨基酸
<220>
<221> 变体
<222> 18
<223> Xaa = Ser, Gly, Ala, Asp, Asn, Ile或Thr
<400> 24
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Gly Xaa Xaa Xaa Xaa Asp Xaa
1 5 10 15
Xaa Xaa
- 还没有人留言评论。精彩留言会获得点赞!