突变CSGG孔的制作方法
本发明涉及一种新型蛋白孔及其用途。特别地,其涉及在核酸测序应用及分子感测中的生物纳米孔。
本发明涉及csgg的突变形式。本发明还涉及采用csgg的分析物检测和表征。
背景技术:
蛋白孔是形成膜中通道的跨膜多肽和复合物,离子和某些分子可以通过该通道。所述通道的最小直径典型的处在纳米范围内(10-9米),因此将这些多肽中的某些命名为“纳米孔”。
纳米孔具有作为生物传感器的巨大潜力。当结合在膜上的纳米孔被施加电势时,离子从所述通道中流过。该离子流可以作为电流被测量。例如,wo2000/28312和d.stoddartetal.,proc.natl.acad.sci.,2010,106,7702-7中描述了使用单通道记录设备的合适的电学检测技术。例如,wo2009/077734中描述了多通道记录技术。
通过所述孔,或结合到所述孔中或接近所述孔的分子转录用于阻碍并从而降低通过所述通道的离子流。离子流的降低程度指示孔内或其附近的阻塞的尺寸,该离子流降低程度通过电流的下降进行测量。因此被测量的电流可以用于测量通道阻塞的大小或程度。电流的变化可以用于识别分子或分子的一部分已经在所述孔(分子传感)或接近所述孔或在特定体系中结合,其可以用于基于分子大小测定位于孔内分子的身份(核酸测序)。
“链测序”方法是已知的采用生物纳米孔对核酸进行测序的方法。将单链多核苷酸通过纳米孔,当它们瞬时穿过纳米孔通道时,每个核苷酸上的碱基通过检测电流的变化进行测定。该方法相对于核酸测序的传统方法节省了大量时间和成本。
之前报道的蛋白纳米孔,例如突变体mspa(manraoetal.,naturebiotechnology,2012,30(4),349-353)以及α-溶血素纳米孔(nat.nanotechnol.,2009,4(4),265-70)已经用于使用“链测序”方法的核酸测序。相似地,对于蛋白传感其他孔例如α-溶血素(jamchemsoc,2012,134(5),2781-7)和clya(am.chem.soc.nano.2014,8(12),12826-35)(j.am.chem.soc,2013,135(36),13456-63)也已经被采用。
对克服现有技术缺陷的新型纳米孔的需求依然存在,尤其是优化用于分子传感应用和例如核酸测序应用的孔的维度和特征。
纳米孔传感是一种依靠观察个体结合或分析物分子和受体间相互作用的传感方法。纳米孔传感器可以通过在绝缘膜中放置纳米维度的单孔并测量在分析物分子存在下通过该孔的电压驱动的离子传输而产生。分析物的识别通过其独特的电流信号、显著的持续时间和电流块的范围以及电流水平的变化显示出来。
现在存在对快速且便宜、应用范围广泛的核酸(例如dna或rna)测序技术的需求。现有技术速度慢并且价格昂贵主要是因为其依赖扩增技术产生大量核酸并需要大量的用于信号检测的专用荧光化学物质。纳米孔传感有潜力通过减少核苷酸和所需试剂用量而提供一种快速且便宜的核酸测序方法。
采用纳米孔传感进行核酸测序的两个必要组分是(1)控制核酸移动通过该孔;和(2)核酸聚合物移动通过孔时对核苷酸的识别。在过去,为实现核苷酸的识别,使核酸穿过溶血素的突变体。这会提供电流信号,该电流信号显示是由序列决定的。这也表明当使用溶血素孔时,大量的核苷酸有助于观测电流,使得观测电流和多核苷酸之间的直接关系令人深思。
虽然通过溶血素孔的突变已经改善了用于识别核苷酸的电流范围,但是如果核苷酸之间的电流差异进一步提高,则测序系统将具有更好的性能。另外,观察到,当核酸移动通过孔时,某些电流状态表现出高变化性。还显示,某些突变溶血素孔比其他的展现出更高的变化性。虽然这些状态的变化可以包含序列的特异性信息,但是希望产生具有低变化性的孔来简化该系统。还期望减少有助于观测到的电流的核苷酸的数量。
技术实现要素:
发明人已经鉴定出细菌淀粉样蛋白分泌通道csgg的结构。所述csgg通道是一种跨膜寡聚蛋白,其形成最小直径约为0.9nm的通道。csgg纳米孔的结构使其适合用于蛋白质传感应用中,特别是用于核酸测序。csgg多肽的修饰变体可以用于进一步增强所述通道对上述特定应用的适用性。
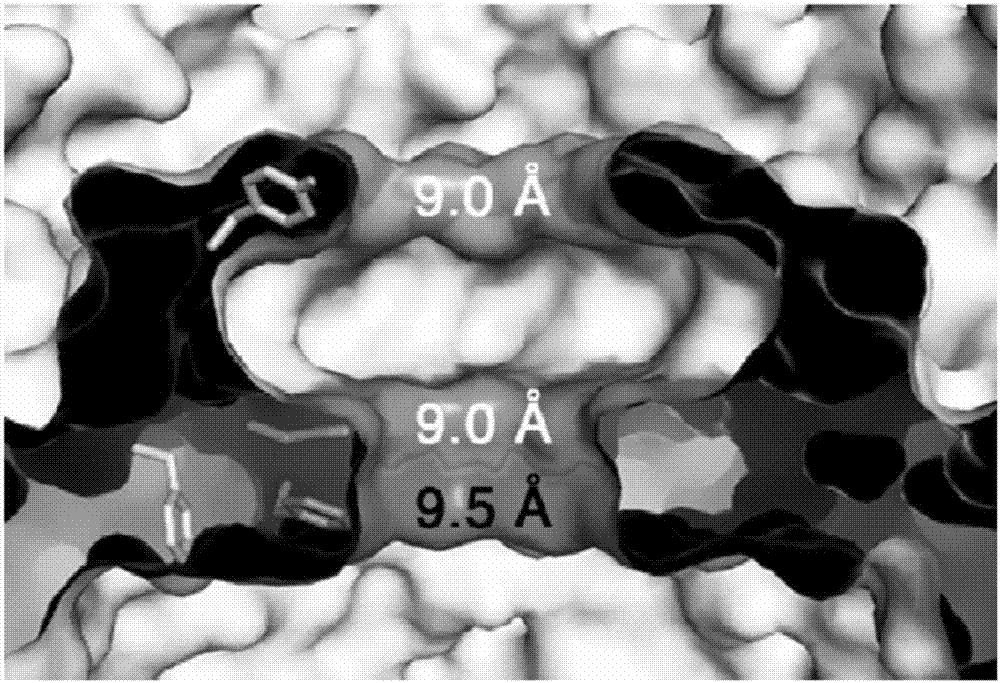
因为其结构更适合于dna测序应用,csgg孔较现有蛋白质孔例如clya或α-溶血素具有优势。csgg孔具有更有利的纵横比,包括比clya具有更短的跨膜通道。csgg孔与α-溶血素孔相比具有更宽的通道开口。对于某些应用,这可以促进酶的附着,例如对于核酸测序应用。在这些实施方式中,其也可以使位于酶和读取头(定义为孔的最窄部分)之间的核酸链段的长度最小化,从而导致改善的读出信号。在涉及核酸测序的本发明的实施方式中,csgg孔的通道中狭窄的内部收缩结构(constriction)还促进单链dna的易位。所述收缩结构由两个环形环组成,这两个环形环通过相邻蛋白质单体中位置51处的酪氨酸残基(tyr51)以及分别在位置56和55处的苯丙氨酸残基和天冬酰胺残基(phe56和asn55)并置而形成。所述收缩结构的尺寸可以被修改。clya具有更宽的内部收缩结构,使得目前没有用于测序的双链dna通过。所述α-溶血素孔不仅具有一个1.3nm宽的内部收缩结构,同时还具有一个2nm宽的具有额外读取头特征的β-桶结构。
在第一方面,本发明涉及一种用于分子传感的方法,其包括:
a)提供一种由绝缘层内的至少一种csgg单体形成的csgg生物孔;
b)在所述绝缘层上施加电势,从而建立通过所述生物孔的电流;
c)使所述csgg生物孔与测试底物接触;以及
d)测量流过所述生物孔的电流。
典型地,所述绝缘层为一种膜,例如磷脂双分子层。在一种实施方式中,通过所述孔的电流是由从所述绝缘层的第一侧流到所述绝缘层的第二侧的可溶性离子流携带。
在本发明的一种实施方式中,所述分子传感为分析物检测。在特定实施方式中,分析物检测的方法包括在步骤(d)之后的另外的步骤:通过与测试底物不存在时通过所述生物孔的电流相比,通过所述生物孔的电流减少,确定出测试底物的存在。
在本发明一种替代的实施方式中,所述分子传感是核酸测序。典型地,通过所述方法测序的核酸种类为dna或rna。在本发明特定的实施方式中,所述csgg生物孔适合于容纳额外的辅助蛋白。典型地,所述额外的辅助蛋白为选自以下的核酸加工酶:dna或rna聚合酶;异构酶;拓扑异构酶;促旋酶;端粒酶;核酸外切酶;和解旋酶。
在本发明的实施方式中,所述csgg生物孔为一种修饰的csgg孔,其中所述修饰的csgg孔对所述csgg孔的至少一个csgg单体中的单体野生型大肠杆菌csgg多肽序列至少具有一种修饰。典型地,对形成所述csgg孔的所有csgg单体进行相同的修饰。在本发明具体的实施方式中,所述修饰的csgg单体具有根据seqidno:4至388的从位置38至63的多肽序列。
在第二方面,本发明涉及修饰的csgg生物孔,包括至少一个csgg单体,其中所述修饰的csgg生物孔具有不多于一个的通道收缩结构,所述收缩结构的直径范围为0.5nm至1.5nm。典型地,所述修饰在所述csgg单体多肽序列的位置38至63之间。合适的,所述修饰处于选自以下的位置:tyr51;asn55;和phe56。在具体的实施方式中,所述修饰为在tyr51位置,或在asn55和phe56两个位置。
在本发明的实施方式中,对所述csgg单体的修饰选自以下:自然存在的氨基酸的取代;自然存在的氨基酸的缺失;和自然存在的氨基酸侧链的修饰。合适地,所述修饰降低或除去未修饰氨基酸的空间阻碍。在具体的实施方式中,所述孔的至少一个csgg单体具有根据seqidno4至388的从位置38至63的多肽序列。
在第三方面,本发明涉及分离的多肽,所述多肽编码本发明第二方面的修饰的csgg生物孔的至少一种csgg单体。
在第四方面,本发明涉及分离的核酸,所述核酸编码本发明第三方面的分离的多肽。
在第五方面,本发明涉及一种生物传感器,包括:
a)绝缘层;
b)在所述绝缘层中的csgg生物孔;以及
c)用于测量通过所述生物孔电流的设备。
在具体实施方式中,在所述生物传感器中的所述csgg生物孔为根据本发明第二方面所述的修饰的csgg生物孔。
在第六方面,本发明涉及用于生物传感应用的csgg生物孔的用途,其中所述生物感应应用为分析物检测或核酸测序。
本发明第六方面的一种实施方式中,所述核酸测序为dna测序或rna测序。
发明人意外地证明,csgg及其新突变体可用于表征分析物,例如多核苷酸。本发明涉及突变csgg单体,其中进行一个或多个修饰以提高所述单体与分析物,例如多核苷酸,相互作用的能力。发明人还意外的证明,包含新型突变单体的孔具有增强的与分析物,例如多核苷酸,相互作用的能力,并因此表现出用于评估分析物特性(例如多核苷酸序列)的改进的性能。所述突变孔意外的显示出改善的对核苷酸的鉴别。特别地,突变孔意外的显示出增加的电流范围,这使其更容易区分不同的核苷酸,并减少会增加信噪比的状态变化。另外,多核苷酸移动通过所述孔时促成电流的核苷酸数量降低。这使得确定多核苷酸移动通过孔时观察到的电流与多核苷酸之间的直接关系更加容易。另外,所述突变孔可以显示出增加的通量,例如,更可能与分析物,例如多核苷酸,相互作用。这使得采用所述孔表征分析物更加容易。所述突变孔可以更容易地插入膜。
因此,本发明提供一种包含seqidno:390所示序列变体的突变型csgg单体,其中所述变体包含在y51,n55和f56中的一个或多个位置处的突变。
因此,本发明提供一种包含seqidno:390所示序列变体的突变型csgg单体,其中所述变体包括以下的一种或多种:(i)在以下位置的一个或多个突变(即在一个或多个以下位置的突变)n40,d43,e44,s54,s57,q62,r97,e101,e124,e131,r142,t150和r192;(ii)在y51/n55,y51/f56,n55/f56或y51/n55/f56的突变;(iii)q42r或q42k;(iv)k49r;(v)n102r,n102f,n102y或n102w;(vi)d149n,d149q或d149r;(vii)e185n,e185q或e185r;(viii)d195n,d195q或d195r;(ix)e201n,e201q或e201r;(x)e203n,e203q或e203r;和(xi)一个或多个以下位置的缺失:f48,k49,p50,y51,p52,a53,s54,n55,f56和s57。
本发明还提供:
-包含两个或多个共价连接的csgg单体的构建体,其中至少一个单体是本发明所述的突变型单体;
-编码本发明突变型单体或本发明构建体的多核苷酸;
-一种衍生自包含本发明的相同突变型单体或本发明的相同构建体的csgg的同源寡聚孔;
-一种衍生自包含至少一个本发明突变型单体或至少一个本发明构建体的csgg的异源寡聚孔;
-用于确定目标分析物的存在、不存在或一种或多种特性的方法,包括:
a)将目标分析物与csgg孔或其突变体接触,以使所述目标分析物相对于所述孔移动;和
b)所述分析物相对于所述孔移动时获取一个或多个测量值并因此确定所述分析物的存在、不存在或其一种或多种特性;
-一种形成用于表征目标多核苷酸的传感器的方法,包括形成csgg孔或其突变体和多核苷酸结合蛋白之间的复合物,并从而形成用于表征目标多核苷酸的传感器;
-用于表征目标多核苷酸的传感器,包括csgg孔或其突变体和多核苷酸结合蛋白之间的复合物;
-csgg孔或其突变体在确定目标分析物存在、不存在或一个或多个特性中的应用;
-一种用于表征目标分析物的试剂盒,包括(a)csg孔或其突变体,以及
(b)膜的组分;
-一种用于表征样品中目标分析物的设备,包括(a)多个csgg孔或其突变体和(b)多个膜;
-表征目标多核苷酸的方法,包括:
a)使所述多核苷酸与csgg孔或其突变体、聚合酶和标记的核苷接触,以使磷酸标记物(phosphatelabelledspecie)通过聚合酶顺序添加到目标多核苷酸,其中所述磷酸标记物含有对每个核苷酸特异性的标记;以及
b)采用所述孔检测所述磷酸标记物并从而表征所述多核苷酸;和
生产本发明所述突变单体或本发明所述构建体的方法,包括在合适的宿主细胞中表达本发明所述的多核苷酸并从而产生本发明所述的突变单体或构建体。
附图说明
图1表示带面表示法(ribbonandsurfacerepresentation)中csgg九聚物在其通道构象中的侧面剖视图。
图2表示csgg通道收缩结构(即在纳米孔感测应用环境下的孔读取头)的截面图以及相关的直径测量。
图3表示形成孔收缩结构的结构基序,包括三个堆叠的同心侧链层:tyr51,asn55和phe56。
图4表示csgg同源物中的序列同源性,包括csgg样蛋白的多重序列比对(seqidno:442至seqidno:448)。所选序列选自跨csgg样序列系统发生树的单系分支(monophyleticclade)(未显示),以给出序列多样性的代表性视图。二级结构元素显示为,对于β-链和α-螺旋分别用箭头或棒状物表示,并且所述二级结构元素基于大肠杆菌csgg晶体结构。重要地,相当于大肠杆菌tyr51,asn55和phe56的残基由箭头突出显示。这些残基形成孔的内部收缩结构,即在前述纳米孔传感应用中的所述孔读取头。
图5表示在平面磷脂双分子层中重组并在+50mv(n=33)或-50mv(n=13)的电场下测量的csgg的代表性单通道电流记录(a)和电导直方图(b)。
图6表示ppb重组的csgg的单通道电流记录,该电流记录在+50mv或-50mv,并辅以csge浓度增量的条件下进行。水平刻度条位于0pa。
图7表示a.c8e4/ldao溶解的csgg原始负染色em照片。箭头表示在图g所示尺寸洗脱曲线中标记的不同颗粒群,其为(i)csgg九聚体的聚集体,(ii)csgg十八聚体和(iii)csgg九聚体。比例尺(scalebar),20nm。b,所指示低聚态顶视图和侧视图的类平均示意图,c,顶视图中ldao溶解的csgg的旋转自相关函数图,显示出九重对称性,d,csggc1s的原始负染色em照片-箭头表示图g中通过尺寸排阻色谱观察到的十六聚体(iv)和八聚体(v)颗粒。e,csggc1s低聚物侧视图的类平均示意图。没有观察该构建体的俯视图,f,由图g中表示的尺寸排阻色谱观察到的csggc1s和csgg颗粒的洗脱体积(ev)、计算的分子质量(mwcalc)、对应于csgg低聚状态(csggn)的预期分子质量(mwcsgg)以及通过负染色电镜(em)和x射线晶体观察到的颗粒的对称性的表格。g,csggc1s(黑色)和c8e4/ldao-溶解的csgg(灰色)在superdex20010/300gl(gehealthcare)上进行的尺寸排阻色谱图,h,i,结晶低聚物顶视图和侧视图的带状示意图,其显示csggc1s(h)的d8十六聚体和膜提取的csgg(i)中的d9十八聚体。一个原聚体从n端(蓝色)到c端(红色)用彩虹色进行着色。在晶体中捕获的形成尾接尾二聚体的两个c8八聚体(csggc1s)或c9九聚体(csgg)被着色成蓝色和棕褐色。r和θ分别表示半径和原聚体间的转角(interprotomerrotation)。
图8表示csggc1s在
图9表示采用以c端到n端方向显示的伸展构象的聚丙氨酸链螺旋穿过所述通道模型化的csgg收缩结构的顶视图(图9a)和侧视图(图9b)。聚丙氨基酸链的模拟溶剂化(modelledsolvation),即图9b中的位置,如图9c所示,其中为清楚起见,c环结构被除去(显示的溶剂分子是距整个聚丙氨酸链
图10:表示大肠杆菌中的csgg。
图11:表示csgg的尺寸。
图12:说明在
图13:说明在
图14:说明在
图15-17:突变孔与野生型(wt)相比,表现出增加的范围。
图18-19:突变孔与野生型(wt)相比,表现出增加的通量。
图20-21:突变孔与野生型(wt)相比,表现出增强的插入。
图22:表示用于实施例18中的dna构建体x。标记为1的区域对应于30个spc3间隔区。标记为2的区域对应于seqidno:415。标记为3的区域对应于4个isp18间隔区。标记为4的区域对应于seqidno:416。标记为5的区段对应于4个5-硝基吲哚。标记为6的区域对应于seqidno:417。标记为7的区域对应于seqidno:418。标记为8的区域对应于seqidno:419,其具有4个isp18间隔区(标记为9的区域)连接在seqidno:419的3'端。在isp18间隔区的另一端是3'胆固醇系链(标记为10)。标记为11的区域对应于4个spc3间隔区。
图23:表示csgg蛋白的链球菌性陷阱(streptrap)(gehealthcare)纯化的示例性色谱踪迹(x轴标记=洗脱体积(ml),y轴标记=吸光度(mau))。将样品加样在25mmtris,150mmnacl,2mmedta,0.01%ddm中,并用10mm脱硫生物素洗脱。将洗脱的csgg蛋白中的洗脱峰标记为e1。
图24:表示在初始strep纯化后,csgg蛋白的典型sds-page可视化的示例。4-20%tgx凝胶(biorad)在1xtgs缓冲液中300v下运行22分钟。所述凝胶用syproruby染色剂染色。泳道1-3表示如箭头所示的含有csgg蛋白的主要洗脱峰(图23中标记为e1)。泳道4-6对应于含有污染物的主要洗脱峰(图23中标记为e1)的尾部的洗脱馏分。m表示所使用的分子量标记物,其为未染色的novexsharp(单位=kd)。
图25:表示csgg蛋白尺寸排阻色谱(sec)的示例(120mls200gehealthcare,x轴标记=洗脱体积(ml),y轴标记=吸光度(mau))。所述sec在strep纯化和加热蛋白质样品后进行。sec的电泳缓冲液为25mm三羟甲基氨基甲烷(tris),150mmnacl,2mmedta,0.01%ddm,0.1%sds,ph8.0,并且色谱柱以1ml/分钟的速率运行。标记为x的踪迹在220nm处有吸光度,标记为y的踪迹在280nm处有吸光度。收集标记为星号的峰。
图26:表示sec后,csgg蛋白的典型sds-page可视化的示例。4-20%tgx凝胶(biorad)在1xtgs缓冲液中300v下运行22分钟,并且所述凝胶用syproruby染色剂染色。泳道1表示strep纯化后并加热但在sec之前的csgg蛋白样品。泳道2-8表示图25中运行大约48ml-60ml(中间峰=55ml)的峰上收集的馏分,并在图25中用星号标记。m表示所使用的分子量标记,其为未染色的novexsharp(单位=kd)。对应于大肠杆菌csgg孔的条带由箭头表示。
图27至33:突变孔与野生型(wt)相比,表现出增加的范围。
图34-39:突变孔与野生型(wt)相比,表现出增加的范围。
图40表示模拟中在0和20ns取得的位于孔(csgg-eco-(y51t/f56q)-strepll(c))9(具有突变y51t/f56q的seqidno:390,其中stepll(c)为seqidno:435并且在c端被连接到孔突变体no.20))顶部的酶(t4dda-(e94c/c109a/c136a/a360c)(具有突变e94c/c109a/c136a/a360c以及(△m1)g1g2)的seqidno:412)的快照(运行图1至3)。
图41表示模拟中在30和40ns取得的位于孔(csgg-eco-(y51t/f56q)-strepll(c)9(具有突变y51t/f56q的seqidno:390,其中stepll(c)为seqidno:435并且在c端被连接到孔突变体no.20))顶部的酶(t4dda-(e94c/c109a/c136a/a360c)(具有突变e94c/c109a/c136a/a360c以及(△m1)g1g2)的seqidno:412)的快照(运行图1至3)。
图42表示处于前孔构象的csggc1s的x射线结构。a,csggc1s单体的带状图,其从n端到c端着色为蓝色到红色的彩虹。二级结构元素根据abd样折叠进行标记,额外的n-端和c-端的α-螺旋和连接β1和α1的延伸环分别标记为αn,αc和c环(cl)。b,csggc1sc8八聚体的侧视图,其具有由颜色区分的亚单元和一个具有如图a中进行取向和着色的亚单元。
图43表示处于其通道构象的csgg的结构。a,csggc1s(蓝色)和膜抽提的csgg(红色)的atr-ftir光谱的酰胺i区域(1,700-1,600cm-1)。b,tm1和tm2序列(seqidno:449和seqidno:450)(面向双分子层的残基,为蓝色)以及与野生型csgg(wt)、空载体或缺乏tm1或tm2的下划线片段的csgg互补的大肠杆菌bw25141δcsgg的刚果红结合区。数据采用3组生物学重复试验表示。c,处于前孔构象(浅蓝色;tm1粉色,tm2紫色)和通道构象(褐色;tm1绿色;tm2橙色)的csgg单体的覆盖图。cl,c环。d,e,带面表示法中的csgg九聚体的侧视图(d)和截面图(e);螺旋2,核心结构域和tm发夹结构分别用蓝色,浅蓝色和褐色表示。单个原聚体如图42a进行着色。品红色球表示leu2的位置。om,外膜。
图44表示csgg通道收缩结构。a,csgg通道收缩结构的截面图及其排除溶剂的直径。b,收缩结构由三个堆叠的同心侧链层组成:tyr51,asn55和phe56,其前面是来自周质侧的phe48。c,csgg通道的拓扑结构。d,与csgg(wt)、空载体或带有显示收缩结构突变的csgg互补的大肠杆菌bw25141δcsgg的刚果红结合区。数据采用6组生物学重复试验表示。e,f,表示在平面磷脂双分子层中重组并在+50mv(n=33)或-50mv(n=13)的电场下测量的csgg代表性单通道电流记录(e)和电导直方图(f)。
图45表示csgg转运机制的模型。a,csge(e),csgg(g)和补充过量csgg(e+g)的csgg的非变性凝胶电泳图(nativepage),显示了csgg-csge复合物(e-g*)的形成。数据采用包括4组不同批次蛋白的7组实验表示。b,从非变性凝胶电泳(nativepage)回收的csge(e),csgg(g)和e-g*复合物的sds-page。数据采用2组重复试验表示。m,分子量标记。c,csgg-csge颗粒的选取种类的平均图。从左至右:通过冷冻电子显微镜(cryo-em)看到的顶视图和侧视图,以及负染色侧视图与csgg九聚体的比较图。d,顶视和斜侧视的csge颗粒冷冻电子显微镜(cryo-em)平均值。旋转自相关显示为九重对称。e,csgg-csge的三维重构图(24a°分辨率,1221个单颗粒)显示了包括csgg(蓝色)的九聚体颗粒以及另外表示为csge九聚体(橙色)的密度。f,ppb重组csgg的单通道电流记录,该电流记录在+50mv或-50mv,并补充增加csge浓度的条件下进行。水平刻度条位于0pa处。g,csgg介导的蛋白质分泌的试验模型(tentativemodel)。csgg和cage被提出用于形成捕获csga的分泌复合物(在图54中讨论),在通道上产生熵势。在通道收缩结构中捕获csga后,ds-整流的布朗扩散(ds-rectifiedbrowniandiffusion)促使多肽逐渐移位穿过外膜。
图46表示大肠杆菌中的curli生物合成途径。较大的curli亚单元csga(浅绿色)作为可溶性单体蛋白从细胞中分泌出来。较小的curli亚单元csgb(深绿色)与外膜(om)相连并且作为将csga从可溶性蛋白转化为淀粉样蛋白沉淀的成核剂。csgg(橙色)组装成外膜中的寡聚curli-特异性转运通道。csge(紫色)和csgf(浅蓝色)形成多产csga以及csgb的转运和沉积所需的可溶性辅助蛋白。csgc形成功能未知的假定的氧化还原酶。所有curli蛋白具有假定的用于跨细胞质(内)膜(im)转运的sec信号序列。
图47表示通过尺寸排阻色谱和负染色电子显微镜分析的csgg和csggc1s在溶液中的低聚态。a,c8e4/ldao溶解的csgg原始负染色em照片。箭头表示如g中所示的尺寸洗脱曲线中标记的不同颗粒群,其为(i)csgg九聚体的聚集体,(ii)csgg十八聚体和(iii)csgg九聚体。比例尺,20nm。b,所指示的低聚态的顶视图和侧视图的类平均示意图。c,顶视图中ldao溶解的csgg的旋转自相关函数图,显示为九重对称性。d,csggc1s的原始负染色em照片,箭头表示图g中通过尺寸排阻色谱观察到的十六聚体(iv)和八聚体(v)颗粒。e,csggc1s低聚物侧视图的类平均示意图。未观察该构建体的顶视图。f,由图g中表示的尺寸排阻色谱观察到的csggc1s和csgg颗粒的洗脱体积(ev)、计算的分子质量(mwcalc)、对应csgg低聚态(csggn)的预期分子质量(mwcsgg)以及通过负染色em和x射线晶体衍射观察的颗粒的对称性的表格。g,csggc1s(黑色)和c8e4/ldao-溶解的csgg(灰色)在superdex20010/300gl(gehealthcare)上进行的尺寸排阻色谱图。h,i,顶视图和侧视图中结晶低聚物的带状示意图,显示了csggc1s(h)的d8十六聚体和膜提取的csgg(i)的d9十八聚体。一个原聚体从n端(蓝色)到c端(红色)以彩虹色进行着色。在晶体中捕获的形成尾到尾相接(tail-to-tail)的二聚体的两个c8八聚体(csggc1s)或c9九聚体(csgg)被染成蓝色和棕褐色。r和h分别表示半径和原聚体间的转角。
图48表示csgg与结构同源物的比较和与csgg接触的原聚体间的接触。a,b,csggc1s单体(例如为前孔构象的csgg)(a)和tolb的核苷酸结合结构域样结构域(b)(pdb2hqs)的条带图,二者均从n端(蓝色)到c端(红色)着色为彩虹色。普通的二级结构元素以同样的方式进行标记。c,csggc1s(灰色),从左到右,与油菜黄单胞菌属罕见脂蛋白(xanthomonascampestrisrarelipoprotein)b(pdb2r76,着色为粉红色)、希瓦氏菌假定脂蛋白(shewanellaoneidensishypotheticallipoprotein)duf330(pdb2iqi,着色为粉红色)和大肠杆菌tolb(pdb2hqs,其n-端和b-螺旋桨(b-propeller)结构域分别着色为粉红色和黄色)层叠在一起。csgg特异性结构元素被标记和上色,如左上图所示。d,e,在csgg结构中发现的两个相邻的原聚体的条带图,该图沿双分子层的平面从低聚物的外部(c)或内部(d)观察。第一个原聚体从n端到c端用彩虹色(深蓝色至红色)表示;第二个原聚体用浅蓝色(核心结构域)、蓝色(螺旋2)和褐色(tm结构域)表示。四个主要的低聚界面是明显的:b6-b39主链在b-桶结构内的相互作用,收缩环(cl),与b1-b3-b4-b5抵接的螺旋1(α1)的侧链堆叠以及螺旋2(α2)的螺旋-螺旋堆叠。还观察到,连接脂质锚定物(品红色球表示leu2中ca位置)和n端螺旋(αn)的18个残基的n-端环包裹相邻的两个原聚体。脂质锚定物的投影位置预期与+2原聚体的tm1和tm2发夹结构相抵接(为清楚起见未示出)。
图49表示csgg寡聚体中选取的表面残基的cys可接近性(accessibility)测试。a-c,周质(a),侧面(b)和胞外(c)视图中显示的csgg九聚体的条带示意图。一个原聚体从n端(蓝色)到c端(红色)着色为彩虹色。半胱氨酸取代基被标记,并且s原子的等效位置显示为球体,根据对大肠杆菌外膜中mal-peg(5,000da)标记的可接近性进行上色。d,在sds-page上分析的与mal-peg反应的样品的蛋白质印迹(westernblot),显示了所引入的半胱氨酸在mal-peg上的结合增加了5kda。可接近的(11和111)、中等可接近的(1)和不可接近的(2)位点在a-e中分别着色为绿色、橙色和红色。对于arg97和arg110,存在44kda处的第二物质,对应于引入的和天然的半胱氨酸均被标记的蛋白质的一部分。数据采用4组独立生物学重复性试验表示。e,存在于x射线结构中的d9十八聚体中的二聚界面的侧视图。在二聚体界面或d9颗粒的内腔内引入的半胱氨酸被标记。在与膜结合的csgg中,这些残基对于mal-peg是可接近的,证明d9颗粒是膜提取的csgg浓缩溶液的产物并且c9复合物形成生理上相关的物质。发现,c端螺旋中的残基(ac;lys242,asp248和his255)是不可接近至难以接近的,表明ac可与大肠杆菌细胞膜,可能是肽聚糖层,形成额外的接触。
图50表示用聚丙氨酸链模型对csgg收缩结构进行的分子动力学模拟。a,b,以伸展构象螺旋穿过通道的聚丙氨酸链模拟的csgg收缩结构的顶视图(a)和侧视图(b),此处采用c-端至n-端的方向表示。通过csgg转运体的底物通道本身不是序列特异性的参考文献16,23。为了清楚,将聚丙氨酸链用于模拟一正经过的多肽链的假定的相互作用。所述模拟的区域由9个同心的csggc环结构组成,每个c环结构均包含残基47-58。连接所述收缩结构的侧链用棒状示意图表示,其中phe51着色为板岩色(slateblue),asn55(酰胺夹)着色为青色,phe48和phe56(φ夹))分别着色为浅橙色和深橙色。n,o和h原子(只示出了羟基或侧链上的酰胺h原子)分别着色为蓝色、红色和白色。所述聚丙氨酸链中的c,n,o和h原子分别着色为绿色、蓝色、红色和白色。在所述收缩结构中所述聚丙氨酸残基
图51表示csgg同源物中的序列保守性。a,csgg九聚体的表面示意图,依据序列相似性着色(对着色的黄色到蓝色进行从低到高的保守性打分),并从周质(最左侧)、侧面(左中)、细胞外环境(右中)观察或作为横截面视图(最右侧)。所述附图显示了,周质前庭的入口、收缩环的前庭侧和tm结构域的腔表面的最高序列保守性映射图的区域。b,csgg样脂蛋白的多重序列比对。所选序列选自跨系统发育的三个csgg样序列的单系分支(monophyleticclades)(未显示),以给出序列多样性的代表性视图。二级结构元素的β-链和α-螺旋分别用箭头或棒状物表示,并且所述二级结构元素基于大肠杆菌csgg晶体结构。c,d,csgg原聚体的二级结构示意图(c)和csgg九聚体表面示意图的横截面视图(d),其均为灰色,并且三个连续的高序列保守性的区域着色为红色(hcr1)、蓝色(hcr2)和黄色(hcr3)。hcr1和hcr2形成收缩环的前庭侧;hcr3对应于螺旋2,位于周质前庭的入口处。在所述收缩结构内部,phe56为100%保守,而asn55可以被ser或thr保守性取代,例如被可作为氢键供体/受体的小极性侧链取代。所述收缩结构出口处的同心侧链环(tyr51)不是保守的。在所述收缩结构入口处的phe-环的存在于拓扑结构上类似于炭疽保护性抗原pa63中的phe427环(称为φ夹)),其中显示其催化多肽的捕获和通过过程参考文献20。toxb超家族蛋白的mst显示在phe环高度处具有保守的基序d(d/q)(f)(s/n)s。这类似于curli样转运体中所见的基序s(q/nt)(f)st。尽管所述孔构象中原子级分辨率的pa63结构尚无法得到,但已得到的结构表明,所述phe-环可以类似地随后由保守性氢键供体/受体(ser/asn428),作为随后的移位通道中的同心环(需说明的是,在两个转运体中所述元素的取向都是反转的)。
图52表示csgg和csgg:csge孔的单通道电流分析.a,在负场电位下,csgg孔显示出两种电导状态。左上图和右上图分别显示了正常(在+50,0和-50mv下测量)的和低电导形式(在0,+50和-50mv下测量)的代表性单通道电流轨迹。在总观察时间(n=22)内,没有观察到两种状态之间的转换,表明所述电导状态具有长寿命(时间尺度为秒到分钟)。左下图显示在+50和-50mv(n=33)下获得的csgg孔的正常的和低电导形式的电流直方图。csgg孔在常规和低电导下的i-v曲线用右下图表示。所述数据表示在至少四个独立记录下的平均值和标准方差。所述低电导形式的自然或生理存在是未知的。b,采用辅助因子csge滴定的csgg通道的电生理学性质。该曲线显示了作为csge浓度的函数的处于开放状态、中间态和闭合状态的通道的比例。csgg的开放和关闭状态如图45f所示。将csge浓度提高到多于10nm导致csgg孔闭合。所述效应发生在+50mv(左图)和-50mv(右图)下,排除了csge(计算的pi4.7)电泳到csgg孔中导致所述孔堵塞的可能性。低概率(,5%)的中间状态大约具有开放通道电导的一半。其可能表示csge引起的所述csgg通道的不完全闭合;或者,其可能表示由来自电解质溶液的残留csgg单体结合到嵌入膜的孔引起的csgg二聚体的暂时形成。从单通道电流迹线的全点直方图分析可获得上述三种状态的比例。上述直方图产生的最多三个状态的峰值区域,以及某一给定状态的比例可通过将相应的峰面积除以记录中所有其他状态的总和获得。在负场电位下,两个开放电导状态被识别,类似于对csgg的观察结果(参见图a)。因为两种开放通道的变化都被较高的cage浓度所阻碍,所以b中的“开放”迹线结合了两种电导形式。曲线图中的数据表示从三个独立记录得到的平均值和标准方差。c,晶体结构,尺寸排阻色谱和em显示,洗涤剂提取的csgg孔在较高蛋白质浓度下形成非天然尾-尾堆叠的二聚体(例如,两个如d9颗粒的九聚体;图47)。这些二聚体也可以在单通道记录中观察到。上图显示在+50,0和-50mv(从左到右)条件下堆叠的csgg孔的单通道电流迹线。左下图显示在+50和-50mv条件下记录的二聚csgg孔的电流直方图。分别在+50和-50mv条件下的实验电导值+16.2±1.8和-16.0±3.0pa(n=15)均接近理论计算值23pa。右下图显示堆叠的csgg孔的i-v曲线。数据表示六个独立记录的平均值和标准方差。d,通过电生理学测试csge结合和阻断堆叠的csgg孔的能力。其显示的为在+50mv(上图)和-50mv(下图)条件下,10或100nmcsge存在下,堆叠的csgg孔的单通道电流迹线。所述电流迹线表明,csge的原本饱和浓度并不会导致堆叠的csgg二聚体的孔闭合。这些观察结果与csgg-cage接触区至螺旋2和csgg周质腔入口的映射图很一致,如由em和定点突变所识别(图45和图52)。
图53表示csge寡聚体和csgg-csge复合物,a,csge的尺寸排阻色谱(superose6,16/600;电泳缓冲液20mmtris-hclph8,100mmnacl,2.5%甘油)显示两种寡聚状态,1和2,的一种平衡,表观分子质量比为9.16:1。峰1的负染色电子显微镜(negative-stainem)观察显示,离散的csge颗粒(插图为五个代表性种类平均图像,按增加的倾斜角度排序)与9个csge复本的尺寸相当。b,由冷冻电子显微镜(cryo-em)从顶部视角观察到的csge低聚物的所选种类平均图像并且其旋转自相关图显示c9对称性的存在。c,csgg-csge冷冻电子显微镜(cryo-em)模型的fsc分析。采用对应1221个颗粒的125类,在相关性为0.5的阈值下,通过fsc分析确定了,三维重构实现了
图54表示csgg分泌复合物的组装和底物征集。所述curli转运体csgg与可溶性分泌辅助因子csge形成化学计量比为9:9的分泌复合物,其围成一个
图55表示csggc1s和csgg的数据采集统计图和电子密度映射图。a,csggc1s和csggx射线结构的数据采集统计图。b,在
图56表示与携带单链dna突出端的dna发夹结构相互作用的csggwt蛋白质的单通道电流迹线(左)和其放大区域(右)。所述迹线显示,响应于在+50mv或-50mv间隔下测量的电位而改变的电流(如箭头所示)。最后的+50mv区段的下行电流的阻滞表明发夹双链同时进入孔腔内,并将单链发夹末端螺旋入孔的内部结构,导致几乎完全的电流阻滞。将电场反转至-50mv导致孔的电泳解阻塞(electrophoreticunblocking)。新的+50mv阶段再次导致dna发夹结构的进入/螺旋化以及孔的堵塞。在所述+50mv区段上,发夹结构的解旋可以导致所述电流阻滞的停止,如所述电流阻滞的反转所示。所述发夹结构——其具有序列:3'gcggggagcgtattagagttggatcggatgcagctggctactgacgtcatgacgtcagtagccagcatgcatccgatc-5'——被加入到所述腔室的顺式侧,终浓度为10nm。
图57表示csgg-δρyρα的纯化和通道性质,所述csgg-δρyρα是突变的csgg孔,其中,在所述收缩残基y51处pypa序列(残基50-53)突变为gg。
序列表说明
seqidno:1表示包含信号序列的野生型大肠杆菌csgg的氨基酸序列(uniprot访问号p0aea2)。
seqidno:2表示包含信号序列的野生型大肠杆菌csgg的多核苷酸序列(基因id:12932538)。
seqidno:3表示seqidno:2中位置53至77的野生型大肠杆菌csgg的氨基酸序列。这对应于成熟野生型大肠杆菌csgg单体的位置38至63的氨基酸序列(即缺少所述信号序列)。
seqidno:4至388表示缺失信号序列的csgg的修饰单体的第38至63位的氨基酸序列。
seqidno:389表示编码来自大肠杆菌str.k-12substr.mc4100的野生型csgg单体的密码子优化的多核苷酸序列。该单体缺少所述信号序列。
seqidno:390表示来自大肠杆菌str.k-12substr.mc4100的成熟的野生型csgg单体的氨基酸序列。该单体缺少所述信号序列。用于此处的缩写csgg=csgg-eco.
seqidno:391表示假设蛋白cko_02032的yp_001453594.1:1-248的氨基酸序列[克氏柠檬酸杆菌(citrobacterkoseri)atccbaa-895],其与seqidno:390序列99%相同。
seqidno:392表示curli生产组装/运输组件csgg的wp_001787128.1:16-238的氨基酸序列,部分[沙门氏菌(salmonellaenterica)],其与seqidno:390序列98%相同。
seqidno:393表示curli生产组装/运输蛋白csgg的key44978.1|:16-277的氨基酸序列[无丙二酸柠檬酸杆菌(citrobacteramalonaticus)],其与seqidno:390序列98%相同。
seqidno:394表示curli生产组装/运输组件的yp_003364699.1:16-277的氨基酸序列[鼠柠檬酸杆菌(citrobacterrodentium)icc168],其与seqidno:390序列97%相同。
seqidno:395表示curli生产组装/运输组件csgg的yp_004828099.1:16-277的氨基酸序列[阿氏肠杆菌(enterobacterasburiae)lf7a],其与seqidno:390序列94%相同。
seqidno:396表示编码phi29dna聚合酶的多核苷酸序列。
seqidno:397表示phi29dna聚合酶的氨基酸序列。
seqidno:398表示源自大肠杆菌sbcb基因的密码子优化的多核苷酸序列。它编码来自大肠杆菌的核酸外切酶i酶(ecoexoi)。
seqidno:399表示来自大肠杆菌的核酸外切酶i酶(ecoexoi)的氨基酸序列。
seqidno:400表示源自大肠杆菌的xtha基因的密码子优化的多核苷酸序列。它编码来自大肠杆菌的核酸外切酶iii酶。
seqidno:401表示来自大肠杆菌的核酸外切酶iii酶的氨基酸序列。该酶从3'至5'方向对双链dna(dsdna)的其中一条链进行5'单磷酸核苷的分布式消解。所述酶在链上的引发需要约4个核苷酸的5'突出结构。
seqidno:402表示源自极端嗜热链球菌(t.thermophilus)的recj基因的密码子优化的多核苷酸序列。它编码来极端嗜热链球菌的recj酶(tthrecj-cd)。
seqidno:403表示来自极端嗜热链球菌的recj酶的氨基酸序列(tthrecj-cd)。该酶从5'至3'方向对单链dna(ssdna)的5'单磷酸核苷进行进行性消解(processivedigestion)。所述酶在链上的引发需要至少4个核苷酸。
seqidno:404表示源自细菌噬菌体λ外切(redx)基因的密码子优化的多核苷酸序列。它编码所述细菌噬菌体λ核酸外切酶。
seqidno:405表示细菌噬菌体λ核酸外切酶的氨基酸序列。该序列是组装成三聚体的三个相同的亚基之一。该酶从5'至3'方向对双链dna(dsdna)的其中一条链进行核苷酸的高度进行性消解(http://www.neb.com/nebecomm/products/productm0262.asp)。所述酶在链上的引发优选需要约4个带有5'磷酸的核苷酸的突出结构。
seqidno:406表示hel308mbu的氨基酸序列。
seqidno:407表示hel308csy的氨基酸序列。
seqidno:408表示hel308tga的氨基酸序列。
seqidno:409表示hel308mhu的氨基酸序列。
seqidno:410表示traleco的氨基酸序列。
seqidno:411表示xpdmbu的氨基酸序列。
seqidno:412表示dda1993的氨基酸序列。
seqidno:413表示trwccba的氨基酸序列。
seqidno:414表示转运体的wp_006819418.1:19-280[雷金斯堡约克氏菌(yokenellaregensburgei)]的氨基酸序列,其与seqidno:390序列91%相同.
seqidno:415表示curli生产组装/运输蛋白csgg的wp_024556654.1:16-277[粉尘克洛诺菌(cronobacterpulveris)]的氨基酸序列,其与seqidno:390序列89%相同。
seqidno:416表示curli生产组装/运输蛋白csgg的yp_005400916.1:16-277[水生拉恩氏菌(rahnellaaquatilis)hx2]的氨基酸序列,其与seqidno:390序列84%相同。
seqidno:417表示csgg家族curli生产组装/运输组件的kfc99297.1:20-278的氨基酸序列[抗坏血酸克吕沃尔菌(kluyveraascorbata)atcc33433],其与seqidno:390序列82%相同。
seqidno:418表示csgg家族curli生产组装/运输组件的kfc86716.11:16-274的氨基酸序列[蜂房哈夫尼菌(hafniaalvei)atcc13337],其与seqidno:390序列81%相同。
seqidno:419表示参与形成curli聚合物的未表征蛋白的yp_007340845.1|:16-270的氨基酸序列[肠杆菌科细菌菌株(enterobacteriaceaebacteriumstrain)fgi57],其与seqidno:390序列76%相同。
seqidno:420表示curli生产组装/运输蛋白csgg的wp_010861740.1:17-274的氨基酸序列[邻单胞痢疾杆菌(plesiomonasshigelloides)],其与seqidno:390序列70%相同。
seqidno:421表示curli生产组装/运输外膜脂蛋白组分csgg的yp_205788.1:23-270的氨基酸序列[费氏弧菌(vibriofischeri)es114],其与seqidno:390序列60%相同。
seqidno:422表示curli生产组装蛋白csgg的wp_017023479.1:23-270的氨基酸序列[变形菌(aliivibriologei)],其与seqidno:390序列59%相同。
seqidno:423表示curli生产组装/运输组分csgg的wp_007470398.1:22-275的氨基酸序列[发光杆菌属(photobacteriumsp.)ak15],其与seqidno:390序列57%相同。
seqidno:424表示curli生产组装蛋白csgg的wp_021231638.1:17-277的氨基酸序列[维氏气单胞菌(aeromonasveronii)],其与seqidno:390序列56%相同。
seqidno:425表示curli生产组装/运输蛋白csgg的wp_033538267.1:27-265的氨基酸序列[希瓦氏菌属(shewanellasp.)ecsmb14101],其与seqidno:390序列56%相同。
seqidno:426表示curli生产组装蛋白csgg的wp_003247972.1:30-262的氨基酸序列[恶臭假单胞菌(pseudomonasputida)],其与seqidno:390序列54%相同。
seqidno:427表示curli生产组装/运输组分csgg的yp_003557438.1:1-234的氨基酸序列[紫色希瓦氏菌(shewanellaviolacea)dss12],其与seqidno:390序列53%相同。
seqidno:428表示curli生产组装/运输蛋白csgg的wp_027859066.1:36-280的氨基酸序列[詹氏海杆菌(marinobacteriumjannaschii)],其与seqidno:390序列53%相同。
seqidno:429表示curli生产组装/运输组分csgg的cej70222.1:29-262的氨基酸序列[金黄杆菌(chryseobacteriumoranimense)g311],其与seqidno:390序列50%相同。
seqidno:430表示实施例18中使用的多核苷酸序列。
seqidno:431表示实施例18中使用的多核苷酸序列。
seqidno:432表示实施例18中使用的多核苷酸序列。
seqidno:433表示实施例18中使用的多核苷酸序列。
seqidno:434表示实施例18中使用的多核苷酸序列。连接到seqidno:434的3'端的是6个isp18间隔区,其另一端连接到两个胸腺嘧啶和3'胆固醇teg上。
seqidno:435表示stepii(c)的氨基酸序列。
seqidno:436表示pro的氨基酸序列。
seqidno:437至440表示来自实施例1的引物。
seqidno:441表示来自实施例21的发夹结构。
seqidno:442至448表示来自图4的序列。
seqidno:449至450表示来自图43的序列。
具体实施方式
除非特别说明,本发明的实施采用化学,分子生物学,微生物学,重组dna技术以及化学方法中的常规技术,其在本领域普通技术人员的能力范围之内。这些技术也均在文献中具有解释,例如,m.r.green,j.sambrook,2012,分子克隆:实验室手册,第四版,1-3册,coldspringharborlaboratory出版社,coldspringharbor,ny;ausubel,f.m.efal.(1995和定期补充;分子生物学电流协议,第9,13和16章,johnwiley&sons,newyork,n.y.);b.roe,j.crabtree,anda.kahn,1996,dna分离和测序:基本技术,johnwiley&sons;j.m.polakandjameso'd.mcgee,1990,原位杂交:原理与实践,牛津大学出版社;m.j.gait(编辑),1984,寡核苷酸的合成:实用方法,irl出版社;以及d.m.j.lilley和j.e.dahlberg,1992,酶学方法:dna结构a部分:酶学中dna合成和物理分析方法,academic出版社。上述这些文献的全部内容均通过引用纳入本文。
在阐述本发明之前,提供许多定义将有助于本发明的理解。本文引用的所有参考文献通过援引整体并入本文。除非特别说明,本文使用的所有技术和科学术语与本发明所属领域的普通技术人员通常的理解具有相同的含义。
如本文所使用的,术语“包括”是指必须包括任何所列举的要素,并且也可以任选地包括其他元素。“基本上由...组成”意味着必须包括任何列举的要素,排除对列举的要素的基本和新特性具有重大影响的元素,并且可以可选地包括其它元素。“由...组成”是指不包括所有未列举的元素。由这些术语中的每一个定义的实施例在本发明的范围内。
本文所用的术语“核酸”是指单链或双链共价连接的核苷酸序列,其中每个核苷酸上的3'和5'末端通过磷酸二酯键连接。所述多核苷酸可以由脱氧核糖核苷酸碱基或核糖核苷酸碱基组成。核酸可以包括dna和rna,并可以在体外合成制备或从自然资源中分离。核酸可以进一步包括修饰的dna或rna,例如甲基化的dna或rna,或经过翻译后修饰的rna,例如用7-甲基鸟苷进行的5'-盖帽,3'-端加工,例如裂解和多腺苷化,以及拼接。核酸还可以包括合成核酸(xna),例如己糖醇核酸(hna),环己烯核酸(cena),苏糖核酸(tna),甘油核酸(gna),锁核酸(lna)和肽核酸(pna)。核酸(本文也称为“多核苷酸”)的大小通常用双链多核苷酸的碱基对(bp)数目表示,或在单链多核苷酸的情况下用核苷酸的数目(nt)表示。1千个bp或nt等于一个千碱基对(kb)。长度小于约40个核苷酸的多核苷酸通常称为“寡核苷酸”,并且可以包含用于dna操作(例如通过聚合酶链式反应(pcr))中的引物。
本发明中的术语“氨基酸”使用其最广泛的含义,其含义包括天然存在的lα-氨基酸或残基。本文使用的天然存在氨基酸通常使用单字母或三字母缩写为:a=ala;c=cys;d=asp;e=glu;f=phe;g=gly;h=his;i=iie;k=lys;l=leu;m=met;n=asn;p=pro;q=gln;r=arg;s=ser;t=thr;v=val;w=trp;andy=tyr(lehninger,a.l,(1975)生物化学,第2版,71-92页,worthpublishers,newyork)。一般术语“氨基酸”还包括d-氨基酸,复古反转氨基酸(retro-inversoaminoacid)以及化学修饰的氨基酸,例如氨基酸类似物,通常不包含在蛋白质中的天然存在的氨基酸,例如正亮氨酸,以及化学合成的具有本领域已知的氨基酸特征的化合物,如β-氨基酸。例如,苯丙氨酸或脯氨酸的类似物或模拟物——其允许所述肽化合物与天然phe或pro具有相同的构象限制——包括在氨基酸的定义中。这种类似物和模拟物在本文中称为相应氨基酸的“功能等价物”。氨基酸的其它实例列举在:roberts和vellaccio,肽:分析,合成,生物学,grossandmeiehofer,eds.,第5卷,341页,academicpress,inc.,n.y.1983,其通过引用并入本文。
“多肽”是指通过肽键连接的氨基酸残基的聚合物,无论是天然产生的还是通过合成方法体外产生的。长度小于约12个氨基酸残基的多肽通常称为“肽”,长度在约12至约30个氨基酸残基之间的多肽可称为“寡肽”。本文所用的术语“多肽”表示天然存在的多肽,前体形式或前蛋白的产物。多肽也可以经过熟化或翻译后修饰过程,其可以包括但不限于:糖基化,蛋白水解断裂,脂化,信号肽裂解,前肽裂解,磷酸化等。本文所用术语“蛋白质”是指含有一个或多个多肽链的大分子。
“生物孔”是指限定通道或孔的跨膜蛋白质结构,其允许分子和离子从膜的一侧移位到另一侧。离子类物质穿过孔的移位可以通过施加到所述孔的任一侧的电势差来驱动。纳米孔是一种生物孔,其中分子或离子通过的通道的最小直径是纳米级的(10-9米)。
对本发明所有实施方式或方面,多核苷酸可以包含与野生型大肠杆菌csgg相比具有至少50%,60%,70%,80%,90%,95%或99%完整序列同一性的多核苷酸,如seqidno:2所示。同样,所述多肽可以包含与野生型大肠杆菌csgg相比具有至少50%,60%,70%,80%,90%,95%或99%完整序列同一性的多肽,如seqidno:1所示。多肽可以包含含有pfam结构域pf03783的多肽,pfam结构域pf03783为csgg样蛋白的特征。目前已知的csgg同源物和csgg体系结构的列表可以在http://pfam.xfam.org//family/pf03783找到。序列同一性也因而可以是全长多核苷酸或多肽的片段或部分。因此,序列可以与本发明的序列具有仅50%的总体序列同一性,但是特定区域、结构域或亚基可以与本发明的序列共享80%,90%或至多99%的序列同一性。根据本发明,对seqidno:2的核酸序列的同源性并不仅限于序列同一性。许多核酸序列彼此能够表现出显著的生物学同源性,尽管其具有明显很低的序列同一性。在本发明中,同源核酸序列被认为是在低严格条件下彼此杂合的序列(m.r.green,j.sambrook,2012,分子克隆:实验室手册,第四版,1-3册,coldspringharborlaboratory出版社,coldspringharbor,ny)。
术语“载体”用于表示线性或圆形的dna分子,可以将适当大小的另一核酸(通常为dna)序列片段整合到其中。上述dna片段可以包括为由dna序列片段编码的基因的转录准备的额外片段。所述额外片段可以包括但不限于启动子,转录终止子,增强子,内部核糖体进入位点,非翻译区,多腺苷化信号,可选择标记,复制起点等等。可以使用各种适用于原核(例如,β-内酰胺酶和乳糖启动子系统,碱性磷酸酶,色氨酸(trp)启动子系统,大肠杆菌的lac,tac,t3,t7启动子)宿主和真核(例如,猿猴病毒40早期或晚期启动子,劳斯氏肉瘤病毒(roussarcomavirus)长末端重复启动子,巨细胞病毒启动子,腺病毒晚期启动子,eg-1a启动子)宿主的启动子。表达载体通常来自质粒,粘粒,病毒载体和酵母人工染色体;载体通常是含有来自几种来源的dna序列的重组分子。本发明的具体实施方式提供编码如本文所述的野生型或修饰的csgg多肽的表达载体。术语“可操作地连接”,当应用于dna序列时,例如在如上所述的核酸载体中,表示所述序列进行排列使得它们协同地发挥作用以达到其预定目的,即启动子序列允许转录的启始通过相关的编码序列进行到终止序列。
生物孔的跨膜蛋白结构本质上可以是单体或寡聚体。通常,所述孔包括围绕中心轴布置的多个多肽亚基,从而形成蛋白质内衬的通道,该通道的延伸基本上垂直于纳米孔残基所在的膜。多肽亚基的数量不受限制。通常,亚基的数量从5至最多30,合适的亚基数量为6至10。或者,在裂解素(perfringolysin)或相关的大膜孔的情况下,亚基的数量未限定。在形成蛋白内衬通道的纳米孔内的蛋白亚基部分通常包括二级结构基序,其可以包括一种或多种跨膜β-桶状结构,和/或α-螺旋部分。
应当理解,所公开产品和方法的不同应用可以根据本领域的具体需要进行调整。还应当理解,本文使用的术语仅出于描述本发明具体实施方式的目的,而不是限制性的。
另外如本说明书和所附权利要求书所使用的,除非另有内容明确规定,单数形式“一”,“一个”以及“所述”包括复数指代。因此,例如,提及“一个多核苷酸”时包括两个或多个多核苷酸,提及“一个多核苷酸结合蛋白”时包括两个或多个这种蛋白,提及“一个解旋酶”时包括两个或多个解旋酶,提及“一个单体”时是指两种或多种单体,提及“一个孔”时包括两个或多个孔等。
本文引用的所有出版物,专利和专利申请,无论是在上文还是下文,它们的全部内容均通过援引包含在本文中。
本发明一部分涉及细菌淀粉样蛋白分泌通道(csgg),其制备方法及其在核酸测序应用和分子传感中的用途。
csgg是存在于大肠杆菌外膜中的膜脂蛋白(uniprot访问号p0aea2;基因id:12932538)。在所述外脂质膜中,csgg形成纳米孔,其包含九个csgg单体亚基的寡聚复合物。通过ii型(脂蛋白)信号序列,csgg前蛋白移位穿过sec易位子,随后在成熟csgg(即带有裂解的ii型信号序列的csgg)的n末端cys残基上进行三酰化。三酰化或“脂质化”的csgg被输送到革兰氏阴性宿主的外膜,其中它插入双分子层中作为九聚体孔。非脂质化形式的csgg,例如,csggc1s作为处于前孔构象的可溶性蛋白存在于细胞周质中(图42)。
野生型csgg纳米孔的x射线结构(goyaletal.,自然,2014,516(7530),250-3)显示其具有
该收缩结构的作用是限制离子和其他分子通过csgg通道的路径。使用标准电解质条件和+50mv或-50mv的电位,在平面磷脂双分子层中重组的csgg的单通道电流记录分别导致43.1±4.5pa(n=33)或-45.1±4.0pa(n=13)的稳定电流(图5)。
通过添加化学计量的周质因子csge(uniprot访问号poae95;实施例10至12)可以有效地阻止电流流过所述csgg通道。不希望受理论束缚,电流的证据指向一种机制,即cage与csgg孔形成复合物,用于封闭通道的一端。可以使用标准单通道记录技术检测到通过csgg通道的离子流量的显著减少(实施例12和13,图6)。发明人已经发现,根据本发明的一个实施方案,电流的测量参数(最大电流和监测电流变化的能力)使得纳米孔适合用于核酸测序和分子传感的应用中。
因此,本发明的一部分涉及基于流过纳米孔的电流的电学测量值的变化,csgg纳米孔蛋白复合物在核酸测序中的方法和用途。
核酸特别适用于纳米孔测序。dna和rna中的天然存在的核酸碱基可以通过其物理尺寸来区分。当核酸分子或单个碱基通过纳米孔的通道时,碱基之间的尺寸差异导致通过通道的离子流的直接相关的降低。离子流中的所述变化可以被记录。用于记录离子流变化的合适的电学测量技术描述于例如wo2000/28312和d.stoddartetal.,proc.natl.acad.sci.,2010,106,pp7702-7中(单通道记录设备);以及例如wo2009/077734(多通道记录技术)中。通过适当的校准,离子流的特征的减少可用于识别实时穿过通道的特定核苷酸和相关碱基。
跨膜通道中最窄收缩结构的尺寸通常是决定纳米孔用于核酸测序应用的适宜性的关键因素。如果收缩结构太小,待测序的分子将无法通过。然而,为了实现对通过通道的离子流的最大影响,在其最窄点处(即在收缩结构处),通道不应该太大。理想地,任何收缩结构在直径上应尽可能地与通过的碱基的尺寸相近。对于核酸和核酸碱基的测序,合适的收缩结构直径在纳米范围内(10-9米范围)。合适地,直径应在0.5至1.5nm范围内,通常直径在0.7至1.2nm范围内。野生型大肠杆菌csgg中的收缩结构具有约
对于与核酸测序相关的应用,所述csgg纳米孔可以以野生型形式使用,或者可以进一步修饰,例如通过特定氨基酸残基的定向突变,以进一步增强所使用的纳米孔所需要的性质。例如,在本发明的实施方式中,考虑采用突变改变通道内收缩结构的数量,大小,形状,位置或取向。修饰的突变csgg纳米孔复合物可以通过已知的基因工程技术制备,其导致多肽序列中特异性靶向氨基酸残基的插入,取代和/或缺失。在寡聚csgg纳米孔的情况下,可以在每个单体多肽亚基,或任何一个单体或所有单体上进行突变。合适地,在本发明的一个实施方式中,对在寡聚蛋白结构内的所有单体多肽进行所述的突变。
根据本发明的一个实施方式,提供了一种修饰的突变csgg纳米孔,其中所述孔内通道收缩结构的数量减少。
野生型大肠杆菌csgg孔包括两个通道收缩结构(参见图1)。这些收缩结构由(i)氨基酸残基phe56和asn55,和(ii)氨基酸残基tyr51形成,作为更宽结构的一部分,其包括从54位至53位的另外的氨基酸以及c-环基序(图2和图3)。
在通常的纳米孔核酸测序中,当感兴趣的核酸序列的各个核苷酸顺序地通过所述纳米孔的通道时,由于所述核苷酸对所述通道的部分阻塞,所述开放通道的离子流被减少。使用上述合适的记录技术测量上述这种离子流的减少。离子流的减少可以校准为已知核苷酸通过所述通道时所测得的离子流的减少,得到用于确定哪个核苷酸通过所述通道的方法,并且从而得到当核酸顺序通过所述通道时,确定通过所述纳米孔的核酸的核苷酸序列的方法。为了精确测定各个核苷酸,通常要求通过所述通道的离子流的减少与通过所述单个收缩结构(或“读取头”)的各个核苷酸的大小直接相关。应当理解,可以通过例如相关的聚合酶的作用,使所述测序是针对"螺旋"穿过(“threaded”through)所述孔的完整核酸聚合物进行的。或者,序列可以由已经从靠近孔的目标核酸上顺序除去的核苷酸三磷酸碱基的通过进行测定(参见例如wo2014/187924)。
当存在两个或更多个收缩结构并且它们相互间隔开时,每个收缩结构可以同时在核酸链中读取分离的核苷酸或与其相互作用。在这种情况下,通过所述通道的离子流的减少将是所有含有核苷酸的收缩结构的流的合并限制的结果。因此,在一些情况下,双重收缩结构可能导致复合电流信号。在某些情况下,当存在两个这样的读取头时,对一个收缩结构或“读取头”的电流读数可能不能被单独确定。
野生型csgg孔结构可以通过重组基因技术重新设计,以扩大,改变或去除两个收缩结构中的一个,使得在通道内留下单个收缩结构,从而限定单个读取头。csgg寡聚孔中的收缩结构基序位于野生型大肠杆菌csgg多肽中38至63位置的氨基酸残基处。该区域的野生型氨基酸序列如seqidno:3所示。在考虑该区域时,在位置50至53,54至56和58至59的任何氨基酸残基的突变均被视为在本发明的范围内。基于与csgg同源物的序列相似性(图4),位置38至49,53,57以及61至63的氨基酸残基被认为是高度保守的,因此可能不太适合用于取代或其他修饰。由于tyr51,asn55和phe56侧链在野生型csgg结构的通道内的关键定位,这些位置的突变可能是有利的,以便修饰或改变读取头的特性。
在单体csgg蛋白指定位置的突变可以导致处于该位置的野生型氨基酸被任意其它天然或非天然氨基酸取代。在本发明的一个实施方式中,希望扩大或除去一个收缩结构;合适地,选择修饰的csgg蛋白中的氨基酸侧链,使得与其所取代的野生型结构中的氨基酸侧链相比具有较小的空间位阻。指定位置上替换的氨基酸残基可以具有相似的静电性质,或者其也可以具有不同的静电性质。合适地,替换的氨基酸侧链将具有与其所取代的野生型结构中的氨基酸侧链相似的静电电荷,以最小化对二级结构或通道性质的破坏。
替换的氨基酸的选择可以基于blosum62矩阵,其提供了基于大量多重序列比对计算氨基酸被另一个取代的可能性的标准方法。blosum62矩阵的实例对技术人员而言可在互联网上免费获得;参见例如国家生物技术信息中心的网站(http://www.ncbi.nlm.nih.gov/class/structure/aa/aa_explorer.cgi)。
对于野生型csgg结构中的tyr51,其在csgg通道中形成第一收缩结构,提供任意氨基酸的取代。特别地,在本发明的某些实施方式中,tyr51可被丙氨酸,甘氨酸,缬氨酸,亮氨酸,异亮氨酸,天冬酰胺,谷氨酰胺和苯丙氨酸取代(seqid:39-318)。在本发明的实施方式中,用丙氨酸或甘氨酸对tyr51的取代是特别合适的(seqid:39-108)。在实施方式中,残基50至53(野生型序列中的pypa)可以用甘氨酸-甘氨酸(gg)取代(seqid:354-388)。
对于asn55——其有助于所述csgg通道中的第二收缩结构,提供任意氨基酸的取代。特别地,在本发明的某些实施方式中,asn55可以被丙氨酸,甘氨酸,缬氨酸,丝氨酸或苏氨酸取代(seqid:9-33,44-68,79-103,14-14,138,149-173,184-208,219-243,254-278,289-313和324-348)。
对于phe56——其形成所述csgg通道中的第二收缩结构的一部分,提供任意氨基酸的取代。特别地,在本发明的某些实施方式中,phe56可以被丙氨酸,甘氨酸,缬氨酸,亮氨酸,异亮氨酸,天冬酰胺和谷氨酰胺取代(seqid:5-13,15-18,20-23,25-28,30-33,40-43,45-48,50-53,55-58,60-63,65-68,75-78,80-83,85-88,90-93,95-98,100-103,110-113,115-118,120-123,125-128,130-133,135-138,145-148,150-153,155-158,160-163,165-168,170-173,180-183,185-188,190-193,195-198,200-203,205-208,215-218,220-223,225-228,230-233,235-238,240-243,250-253,255-258,260-263,265-268,270-273,275-578,285-288,290-293,295-298,300-303,305-308,310-313,320-323,325-328,330-333,335-338,340-343和345-348)。在本发明的实施方式中,丙氨酸和甘氨酸对phe56的取代是特别合适的(seqid:5,10,15,20,25,30,35,40,45,50,55,60,65,70,75,80,85,90,95,100,105,110,115,120,125,130,135,140,145,150,155,160,165,170,175,180,185,190,195,200,205,210,215,220,225,230,235,240,245,250,255,260,265,270,275,280,285,290,295,300,305,310,315,320,325,330,335,340,345,350,355,360,365,370,375,380,385,6,11,16,21,26,31,36,41,46,51,56,61,66,71,76,81,86,91,96,101,106,111,116,121,126,131,136,141,146,151,156,161,166,171,176,181,186,191,196,201,206,211,216,221,226,231,236,241,246,251,256,261,266,271,276,281,286,291,296,301,306,311,316,321,326,331,336,341346,351,356,361,366,371,376,381和386)。
在给定的突变csgg蛋白中,tyr51的取代可以与位置55和56中的任一个同时进行(seqid:44,49,54,59,64,79,84,89,94,99,114,119,124,129,134,149,154,159,164,169,184,189,194,199,204,219,224,229,234,239,254,259,264,269,274,289,294,299,304,309,359,364,369,374,379,40,41,42,75,7677,110,111,112,145,146,147,180,181,182,215,216,217,250,251,252,285,286和287),其中所述通道内至少一个合适尺寸的收缩结构被保持。或者,tyr51的取代与位置asn55和phe56处的取代相互排斥(seqid:39,74,109,144,179,214,249,284,354,10,11,12,15,16,17,20,21,22,25,26,27,30,31和32)。
或者,野生型csgg蛋白中tyr51,asn55或phe56中的一个或多个可以缺失(seqid:319-353,34-38,69-73,104-108,139-143,174-178,209-213,244-248,279-283,314-318,384-388,8,13,18,23,28,33,43,48,53,58,63,68,78,83,88,93,98,103,113,118,123,128,133,138,148,153,158,163,168,173,183,188,193,198,203,208,218,223,228,233,238,243,253,258,263,268,273,278,288,293,298,303,308和313)。为保持通道中至少一个收缩结构,在给定的实施方式中,氨基酸残基tyr51的缺失与氨基酸残基asn55和phe56的缺失相互排斥(seqid:319-322,324-327,329-332,334-337,339-342,344-347,38,73,108,143,178,213,248,283和318)。位置53和54以及48和49的某些相邻的氨基酸残基也可以缺失。
应当理解,本发明提供的实施方式,其中上述修饰可以单独或以任何组合方式进行。
除去在tyr51的收缩结构或在asn55/tyr56的收缩结构,导致csgg通道内的单个收缩结构。不希望被理论束缚,假设asn55/tyr56的收缩结构与可能是期望的在tyr51的收缩结构相比具有更高的构象稳定性。然而,与核苷酸相比,asn55/tyr56收缩结构可能太高(沿中心孔的轴测量)。这可能导致dna链移位中各个碱基对的分辨率差。
对于tyr51收缩结构,则可能是相反的。除去asn55/tyr56收缩结构后,寡聚物中剩下的tyr51残基的环可能与天然结构相比在构象上不太稳定。然而,tyr51收缩结构较短(当沿着中心孔的轴测量时),并且可能更能够在通道内提供可区分各个碱基的收缩结构。
在任一实施方式中,当在核酸测序应用中使用所述孔时,单个狭窄收缩结构的存在(当沿中心孔轴测量时)可能降低电流读取的复杂性。核酸通过所述孔移位时产生的观测电流的调节将因此仅反映通过单个收缩结构或读数头的分离的核苷酸的通过。
一个收缩结构的有效去除也可以增加孔突变体的开放通道电流。增加的开放通道电流将是有利的,因为较高的背景电导导致针对不同核酸碱基对信号的更高分辨率的电流阻滞水平。采用这种方式,对读取头的修饰可以改善生物孔对核酸测序和其他分子传感应用的适用性。
作为替代的实施方式,或除了上述序列修饰之外,还提供了,asn55/phe56收缩结构可进一步适于调整其高度(沿中心孔轴测量)。asn55/phe56收缩结构的这种进一步调整可能伴随着或未伴随着tyr51或csgg通道内其他位置的突变。合适地,asn55/phe56收缩结构的进一步调整考虑作为扩大或去除由tyr51残基形成的收缩结构的突变的一部分。
在野生型形式中,asn55/phe56通道收缩结构由两个彼此垂直相邻而设的氨基酸环组成。因此,所述收缩结构具有大于1nm的长度。1nm长的收缩结构可能不允许分辨对核酸链移位中分离碱基的离子流产生的电信号。通常地,用于核酸测序的已知纳米孔的所述收缩结构一般长度小于1nm。例如,用于dna测序的mspa纳米孔具有0.6nm的收缩结构高度(沿中心孔轴测量;manraoetal.,naturebiotechnology,2012,30(4),349-353)。
为降低asn55/phe56收缩结构的高度(沿中心孔轴测量),两个残基中的任一个可以被取代或缺失,导致收缩结构顶部或底部的扩大。
对于形成csgg通道中第二收缩结构一部分的asn55,可以考虑用任意氨基酸取代。特别地,用丙氨酸,甘氨酸,缬氨酸,丝氨酸或苏氨酸取代(seqid:9-33,44-68,79-103,14-14,138,149-173,184-208,219-243,254-278,289-313和324-348)。
对于形成csgg通道中第二收缩结构一部分的phe56,可以考虑用任意氨基酸取代。特别地,用丙氨酸,甘氨酸,缬氨酸,亮氨酸,异亮氨酸,天冬酰胺和谷氨酰胺取代(seqid:5-13,15-18,20-23,25-28,30-33,40-43,45-48,50-53,55-58,60-63,65-68,75-78,80-83,85-88,90-93,95-98,100-103,110-113,115-118,120-123,125-128,130-133,135-138,145-148,150-153,155-158,160-163,165-168,170-173,180-183,185-188,190-193,195-198,200-203,205-208,215-218,220-223,225-228,230-233,235-238,240-243,250-253,255-258,260-263,265-268,270-273,275-578,285-288,290-293,295-298,300-303,305-308,310-313,320-323,325-328,330-333,335-338,340-343和345-348)。在本发明的实施方式中,丙氨酸和甘氨酸对phe56的取代是特别合适的(seqid:5,10,15,20,25,30,35,40,45,50,55,60,65,70,75,80,85,90,95,100,105,110,115,120,125,130,135,140,145,150,155,160,165,170,175,180,185,190,195,200,205,210,215,220,225,230,235,240,245,250,255,260,265,270,275,280,285,290,295,300,305,310,315,320,325,330,335,340,345,350,355,360,365,370,375,380,385,6,11,16,21,26,31,36,41,46,51,56,61,66,71,76,81,86,91,96,101,106,111,116,121,126,131,136,141,146,151,156,161,166,171,176,181,186,191,196,201,206,211,216,221,226,231,236,241,246,251,256,261,266,271,276,281,286,291,296,301,306,311,316,321,326,331,336,341346,351,356,361,366,371,376,381和386)。
用于调整野生型csgg孔的收缩结构的最小直径的修饰也是可以想到的。csgg孔中两个收缩结构的最小直径约为0.9nm
上述列出的任意修饰可以有利地改变收缩结构处的氨基酸的亲水性和电荷分布,以改善移位核酸链的通过和与移位核酸链的非共价相互作用,从而改善电流读出。上述列出的任意突变也可以有利地改变靠近通道收缩结构的亲水性和电荷分布,以优化通过所述收缩结构的电解质离子的流动,并且实现更好的区别移位核酸链中通过的核苷酸。
csgg蛋白的进一步修饰被认为可能导致改变通道腔内的表面电荷分布。在本发明的一个实施方式中,进行上述这些修饰可以避免移位核酸不希望地静电吸附到通道壁上。由于核酸带有负电荷,并且csgg通道内腔含有一些正电荷(图1),假定静电相互作用可能干扰核酸测序时的螺旋或移位。合适地带正电荷的氨基酸残基例如赖氨酸,组氨酸和精氨酸,可以被中性或带负电荷的侧链取代,以进一步提高核酸移位通过孔的效率,从而提高电流读数的清楚度。
本发明的实施方式还提供了对野生型大肠杆菌csgg或突变csgg的通道内腔进行修饰以促进核酸链(或各个核苷酸)移位或螺旋进入所述孔收缩结构。具有内部收缩结构的csgg跨膜部分类似于带有以中心孔为特征的盖状结构的桶状结构。通过在最靠近所述桶状结构和盖状结构的孔内腔中增加额外的环,可以促进螺旋进入过程。
本发明具体实施方式提供了,所述csgg孔可以由共价连接的一种或多种单体,二聚体或寡聚物组成。作为非限制性实施例,单体可以以任意构象进行基因融合,例如通过它们的末端氨基酸。在这种情况下,一个单体的氨基末端可以与另一个单体的羧基末端融合。
根据本发明的一个实施方式,还提供了,所述csgg孔可以适应于容纳额外的辅助蛋白,该辅助蛋白可以在分子从所述孔通过时具有有利的性质。对所述孔的适应性可能有助于核酸加工酶的锚定。核酸加工酶可以包括dna或rna聚合酶;异构酶;拓扑异构酶;促旋酶;端粒酶;和解旋酶。将这些酶中的一种或多种与纳米孔相关联可以在增强核酸螺旋进入所述孔以及控制核酸链移位通过孔的速度方面具有益处(manraoetal.,naturebiotechnology,2012,30(4),349-353)。控制核酸链通过孔的移位速度具有以下优点:在更适合读取且更一致的离子流的电流测量方面提供改进的响应。
在本发明的实施方式中,设想对纳米孔膜外区域的修饰可能有助于通过提供一个或多个结合/锚定位点促进合适的核酸加工酶(例如dna-聚合酶)在通道的内腔中或邻近通道内腔位置的对接(docking)。合适的锚定位点可以包括用于酶静电结合的静电区块(electrostaticpatches);允许共价偶联的一个或多个半胱氨酸残基;和/或用于提供空间锚的跨膜通道部分的改变的内部宽度。
本发明在下面更详细列出的具体实施方式中提供用于核酸测序的csgg野生型孔的进一步适应性变化。
在本发明的实施方式中,所述csgg孔的膜外区域(如图1所示的底部)可以被截短或除去,以促进核酸链在所述通道内腔的另一侧上的离去。膜外区域的截短或除去也可以改善来自通道中离子流的电信号的电流分辨率。后者的益处是通过由膜外区域降低对离子流的阻力而带来的。在本发明的csgg孔中,跨膜通道、内部收缩结构和盖帽区域代表连续的三个阻力区域。去除或降低上述区域之一的贡献会增加开放通道电流,从而提高电流分辨率。这种改变可以包括缺失α-螺旋2(α2),c-末端α-螺旋(αc)(图1),和/或它们的组合。
在本发明的进一步实施方式中,面向在野生型大肠杆菌csgg孔的外表面上的氨基酸的膜可以被修饰以促进所述孔插入到所述膜中。在某些实施方式中,提供了,单个氨基酸取代可以用合适的更疏水的类似物替代野生型残基。例如,残基ser136,gly138,gly140,ala148,ala188或gly202中的一个或多个可以改变为ala,val,leu或iie。此外,芳香类残基如酪氨酸或色氨酸可以取代适当的野生型氨基酸,使得它们位于疏水膜和亲水溶剂的界面处。例如,残基leu154或leu182中的一个或多个可被tyr,phe或trp取代。
在本发明的实施方式中,可以增加所述蛋白孔的热稳定性。这导致测序装置中纳米孔的保质期的有利增加。在实施方式中,蛋白孔的热稳定性的增加通过修饰β-折叠序列(beta-turnsequences)或改善蛋白表面的静电相互作用实现。在一种实施方式中,跨膜区域中的β-发夹结构可以通过在相邻β-发夹结构中或相邻β-发夹结构之间跨两个相邻β-链形成共价二硫化物进行稳定。这种交叉链半胱氨酸对的实例可以是val139-asp203;gly139-gly205;lys135-thr207;glu201-ala141;gly147-gly189;asp149-gln187;gln151-glu185;thr207-glu185;gly205-gln187;asp203-gly189;ala153-lys135;gly137-gln151;val139-asp149或ala141-gly147。
在本发明的进一步实施方式中,根据biotechnol.j.,2011,6(6),650-659中描述的方法,用于多肽序列的密码子可以被修饰以允许以高水平和低错误率表达csgg蛋白。所述修饰也可以靶向mrna的任何二级结构。
本发明还提供蛋白序列的改变以改善其对蛋白酶的稳定性。这可以通过去除柔性环区域实现,例如缺失α-螺旋2(α2),c-末端α-螺旋(αc)(图1),和/或其组合。
在本发明的实施方式中,改变所述csgg多肽序列/表达系统以避免蛋白质可能的聚集。
本发明还提供多肽序列的改变以提高蛋白质的折叠效率。合适的技术提供在biotechnol.j.,2011,6(6),650-659中。
本发明的一种实施方式进一步提供生物亲和性标签的替换或增加以便于所述csgg蛋白的纯化。公开的所述csgg孔的结构包含strepll标签(goyaletal.,nature,2014,516(7530),250-3)。本发明的实施方式包括其它生物亲和性标签,例如用于促进通过金属螯合亲和色谱纯化的组氨酸标签。在本发明的替代实施方式中,所述标签可以包括flag标签或表位标签,例如myc-标签或ha-标签。在本发明的进一步实施方式中,所述纳米孔可以通过采用生物素或其类似物(例如脱硫生物素)的生物素化进行修饰,从而有助于通过与链霉亲和素的相互作用进行纯化。
在本发明的实施方式中,可以添加在蛋白质末端的负电荷以增加所述多肽的净电荷并促进蛋白质在聚丙烯酰胺凝胶电泳中的迁移。这可能导致在对这些种类感兴趣的情况下对csgg异源寡聚体的改进的分离(参见howorkaetal.proc.nat.acad.sci.,2001,98(23),12996-13001)。异源寡聚体可用于对每孔引入一个单半胱氨酸残基,这可以有利于促进合适的核酸加工酶的连接,例如如上所述的dna聚合酶。
本发明的一部分涉及野生型或修饰的csgg纳米孔基于流过纳米孔电流的电学测量值的变化在分子感测应用中的用途。
分子在所述csgg孔的通道中或在所述通道任一开口附近的结合对通过所述孔的开放通道离子流有影响。以与上述核酸测序应用相似的方式,可以使用合适的测量技术通过电流变化来测量开放通道离子流的变化(例如,wo2000/28312和d.stoddartetal,proc.natl.acad.sci.,2010,106,7702-7或wo2009/077734)。离子流的减少程度(通过电流的减少测量)与孔内或孔附近的阻塞尺寸有关。因此感兴趣的分子,也称为分析物,在孔内或孔附近的结合提供了可检测和可测量的事件,从而形成生物传感器的基础。用于纳米孔感应的合适分子包括核酸;蛋白质;肽类和小分子如药物,毒素或细胞因子。
对生物分子存在的检测可以应用于个性化药物开发,医药,诊断,生命科学研究,环境监测以及安全和/或国防工业。
在本发明的实施方式中,本申请公开的野生型或修饰的大肠杆菌csgg纳米孔或其同源物可用作分子传感器。分析物检测过程描述于howorkaetal.naturebiotechnology(2012)jun7;30(6)506-7。待检测的所述分析物分子可以与通道的任意一面结合,或者结合在通道本身的内腔内。结合位置可以通过待感测分子的大小确定。野生型csgg孔可以作为传感器,或本发明的实施方式,通过重组或化学方法来修饰csgg孔,以增加结合强度,结合位置或待感测分子的结合特异性。通常的修饰包括加入与待感测分子的结构互补的特异性结合部分。当分析物分子包含核酸时,该结合部分可以包含环糊精或寡核苷酸;对于小分子,这可以是已知的互补结合区域,例如抗体或非抗体分子的抗原结合部分,包括来自t细胞受体(tcr)的单链可变片段(scfv)区域或抗原识别结构域;或对于蛋白质,其可以是目标蛋白质的已知配体。以这种方式,野生型或修饰的大肠杆菌csgg纳米孔或其同源物可以表现出能够用作分子传感器,用于检测样品中可以包括细胞表面抗原的合适抗原(包括表位)的存在,包括受体,实体肿瘤或血液癌细胞标志物(如淋巴瘤或白血病),病毒抗原,细菌抗原,原生动物抗原,过敏源,过敏相关分子,白蛋白(如人,啮齿动物或牛),荧光分子(包括荧光素),血液组抗原,小分子,药物,酶,酶或酶底物的催化位点,以及酶底物的过渡态类似物。
可以使用已知的基因工程和重组dna技术来实现修饰。任何适应的定位将取决于待感测分子的性质,例如尺寸,三维结构及其生物化学性质。适应性结构的选择可以利用计算机结构设计。一系列定制的csgg纳米孔被设想出来,每个都特异性适用于其注定的感测应用。蛋白-蛋白相互作用或蛋白质-小分子相互作用的测定和优化可以使用例如
在一个实施方式中,所述csgg孔可以是水溶性的八聚体形式,其中n端cys残基被另一氨基酸替代,以防止蛋白质n端的脂化。在替代实施方式中,蛋白质可以通过除去n-端前导序列在细胞质中表达,以避免通过细菌脂质化途径的加工。
goyal等人(nature,2014;516(7530)250-3)描述了csgg单体可溶性蛋白,八聚体可溶性蛋白和寡聚脂质化csgg孔的制备方法,该文献通过援引加入本申请以及实施例1和2中。
突变csgg单体
本发明提供突变型csgg单体。突变csgg单体可用于形成本发明所述的孔。突变csgg单体是其序列从野生型csgg单体的序列变化而言且保留形成孔的能力的单体。用于确认突变单体形成孔的能力的方法在本领域中是公知的,下面将更详细地讨论。
所述突变单体具有提高的多核苷酸读取性质,即显示改进的多核苷酸捕获和核苷酸识别。特别地,由突变单体构建的孔比野生型更容易捕获核苷酸和多核苷酸。此外,由突变单体构建的孔显示增加的电流范围(这使得更容易区分不同的核苷酸)和减少的状态变化(这增加了信噪比)。此外,随着多核苷酸移动通过由突变体构建的孔,贡献于电流的核苷酸数目减少。这使得更容易鉴定当多核苷酸移动通过所述孔时观察到的电流与多核苷酸序列之间的直接关系。此外,由突变单体构建的孔,可以显示增加的通量,即更可能与分析物例如多核苷酸相互作用。这使得使用所述孔表征分析物更容易。由突变单体构建的孔可以更容易地插入膜中。
本发明所述突变单体包括seqidno:390所示序列的变体。seqidno:390是来自大肠杆菌str.k-12亚株mc4100的野生型csgg单体。seqidno:390的变体是具有与seqidno:390的序列不同的氨基酸序列并保留其形成孔的能力的多肽。可以使用本领域已知的任何方法测定变体形成孔的能力。例如,可以将变体与其他合适的亚基一起插入两亲性分子层,并且可以确定变体寡聚而形成孔的能力。将亚基插入膜,例如双亲性分子层,中的方法是本领域已知的。例如,亚基可以在含有三嵌段共聚物膜的溶液中以纯化的形式悬浮,使得其扩散到所述膜并通过结合到膜上并组装成功能状态而插入。
在本申请的所有讨论中,对氨基酸使用标准的单字母代码。这些代码如下:丙氨酸(a),精氨酸(r),天冬酰胺(n),天冬氨酸(d),半胱氨酸(c),谷氨酸(e),谷氨酰胺(q),甘氨酸(g),亮氨酸(l),赖氨酸(k),甲硫氨酸(m),苯丙氨酸(f),脯氨酸(p),丝氨酸(s),苏氨酸(t),色氨酸(w)和缬氨酸(ⅴ)。还使用标准取代符号,即q42r表示42位置的q被r替换。
在一种实施方式中,本发明的突变单体包括seqidno:390的突变体,其包含以下中的一个或多个:(i)在以下位置的一个或多个突变(即在一个或多个以下位置的突变):n40,d43,e44,s54,s57,q62,r97,e101,e124,e131,r142,t150和r192,例如以下位置的一个或多个突变(即在一个或多个以下位置的突变):n40,d43,e44,s54,s57,q62,e101,e131和t150或n40,d43,e44,e101和e131;(ii)在y51n55,y51f56,n55f56或y51n55f56的突变;(iii)q42r或q42k;(iv)k49r;(v)n102r,n102f,n102y或n102w;(vi)d149n,d149q或d149r;(vii)e185n,e185q或e185r;(viii)d195n,d195q或d195r;(ix)e201n,e201q或e201r;(x)e203n,e203q或e203r;和(xi)缺失以下一个或多个位置:f48,k49,p50,y51,p52,a53,s54,n55,f56和s57。所述变体可以包含(i)至(xi)的任意组合。特别地,所述变体可以包括{i}{ii}{iii}{iv}{v}{vi}{vii}{viii}{ix}{x}{xi}{i,ii}{i,iii}{i,iv}{i,v}{i,vi}{i,vii}{i,viii}{i,ix}{i,x}{i,xi}{ii,iii}{ii,iv}{ii,v}{ii,vi}{ii,vii}{ii,viii}{ii,ix}{ii,x}{ii,xi}{iii,iv}{iii,v}{iii,vi}{iii,vii}{iii,viii}{iii,ix}{iii,x}{iii,xi}{iv,v}{iv,vi}{iv,vii}{iv,viii}{iv,ix}{iv,x}{iv,xi}{v,vi}{v,vii}{v,viii}{v,ix}{v,x}{v,xi}{vi,vii}{vi,viii}{vi,ix}{vi,x}{vi,xi}{vii,viii}{vii,ix}{vii,x}{vii,xi}{viii,ix}{viii,x}{viii,xi}{ix,x}{ix,xi}{x,xi}{i,ii,iii}{i,ii,iv}{i,ii,v}{i,ii,vi}{i,ii,vii}{i,ii,viii}{i,ii,ix}{i,ii,x}{i,ii,xi}{i,iii,iv}{i,iii,v}{i,iii,vi}{i,iii,vii}{i,iii,viii}{i,iii,ix}{i,iii,x}{i,iii,xi}{i,iv,v}{i,iv,vi}{i,iv,vii}{i,iv,viii}{i,iv,ix}{i,iv,x}{i,iv,xi}{i,v,vi}{i,v,vii}{i,v,viii}{i,v,ix}{i,v,x}{i,v,xi}{i,vi,vii}{i,vi,viii}{i,vi,ix}{i,vi,x]{i,vi,xi}{i,vii,viii}{i,vii,ix}{i,vii,x}{i,vii,xi}{i,viii,ix}{i,viii,x}{i,viii,xi}{i,ix,x}{i,ix,xi}{i,x,xi}{ii,iii,iv}{ii,iii,v}{ii,iii,vi]{ii,iii,vii}{ii,iii,viii}{ii,iii,ix}{ii,iii,x}{ii,iii,xi}{ii,iv,v}{ii,iv,vi}{ii,iv,vii}{ii,iv,viii}{ii,iv,ix}{ii,iv,x}{ii,iv,xi}{ii,v,vi]{ii,v,vii}{ii,v,viii}{ii,v,ix}{ii,v,x}{ii,v,xi}{ii,vi,vii}{ii,vi,viii}{ii,vi,ix}{ii,vi,x}{ii,vi,xi}{ii,vii,viii}{ii,vii,ix}{ii,vii,x]{ii,vii,xi}{ii,viii,ix}{ii,viii,x}{ii,viii,xi}{ii,ix,x}{ii,ix,xi}{ii,x,xi}{iii,iv,v}{iii,iv,vi}{iii,iv,vii}{iii,iv,viii}{iii,iv,ix}{iii,iv,x}{iii,iv,xi}{iii,v,vi}{iii,v,vii}{iii,v,viii}{iii,v,ix}{iii,v,x}{iii,v,xi}{iii,vi,vii}{iii,vi,viii}{iii,vi,ix}{iii,vi,x}{iii,vi,xi}{iii,vii,viii}{iii,vii,ix}{iii,vii,x}{iii,vii,xi}{iii,viii,ix}{iii,viii,x}{i,ii,iii,iv}{i,ii,iii,v}{i,ii,iii,vi}{i,ii,iii,vii}{i,ii,iii,viii]{i,ii,iii,ix}{i,ii,iii,x}{i,ii,iii,xi}{i,ii,iv,v}{i,ii,iv,vi}{i,ii,iv,vii}{i,ii,iv,viii}{i,ii,iv,ix}{i,ii,iv,x}{i,ii,iv,xi}{i,ii,v,vi}{i,ii,v,vii}{i,ii,v,viii}{i,ii,v,ix}{i,ii,v,x}{i,ii,v,xi}{i,ii,vi,vii}{i,ii,vi,viii}{i,ii,vi,ix}{i,ii,vi,x}{i,ii,vi,xi}{i,ii,vii,viii}{i,ii,vii,ix}{i,ii,vii,x}{i,ii,vii,xi}{i,ii,viii,ix}{i,ii,viii,x}{i,ii,viii,xi}{i,ii,ix,x}{i,ii,ix,xi}{i,ii,x,xi}{i,iii,iv,v}{i,iii,iv,vi}{i,iii,iv,vii]{i,iii,iv,viii}{i,iii,iv,ix}{i,iii,iv,x}{i,iii,iv,xi}{i,iii,v,vi}{i,iii,v,vii}{i,iii,v,viii}{i,iii,v,ix}{i,iii,v,x}{i,iii,v,xi}{i,iii,vi,vii]{i,iii,vi,viii}{i,iii,vi,ix}{i,iii,vi,x}{i,iii,vi,xi}{i,iii,vii,viii}{i,iii,vii,ix}{i,iii,vii,x}{i,iii,vii,xi}{i,iii,viii,ix}{i,iii,viii,x}{i,iii,viii,xi}{i,iii,ix,x}{i,iii,ix,xi}{i,iii,x,xi}{i,iv,v,vi}{i,iv,v,vii}{i,iv,v,viii}{i,iv,v,ix}{i,iv,v,x}{i,iv,v,xi}{i,iv,vi,vii}{i,iv,vi,viii}{i,iv,vi,ix}{i,iv,vi,x}{i,iv,vi,xi}{i,iv,vii,viii}{i,iv,vii,ix}{i,iv,vii,x}{i,iv,vii,xi}{i,iv,viii,ix}{i,iv,viii,x}{i,iv,viii,xi}{i,iv,ix,x}{i,iv,ix,xi}{i,iv,x,xi}{i,v,vi,vii}{i,v,vi,viii}{i,v,vi,ix}{i,v,vi,x}{i,v,vi,xi}{i,v,vii,viii}{i,v,vii,ix}{i,v,vii,x}{i,v,vii,xi}{i,v,viii,ix}{i,v,viii,x}{i,v,viii,xi}{i,v,ix,x}{i,v,ix,xi}{i,v,x,xi}{i,vi,vii,viii}{i,vi,vii,ix}{i,vi,vii,x]{i,vi,vii,xi}{i,vi,viii,ix}{i,vi,viii,x}{i,vi,viii,xi}{i,vi,ix,x}{i,vi,ix,xi}{i,vi,x,xi}{i,vii,viii,ix}{i,vii,viii,x}{i,vii,viii,xi]{i,vii,ix,x}{i,vii,ix,xi}{i,vii,x,xi}{i,viii,ix,x}{i,viii,ix,xi}{i,viii,x,xi}{i,ix,x,xi}{ii,iii,iv,v}{ii,iii,iv,vi}{ii,iii,iv,vii]{ii,iii,iv,viii}{ii,iii,iv,ix}{ii,iii,iv,x}{ii,iii,iv,xi}{ii,iii,v,vi}{ii,iii,v,vii}{ii,iii,v,viii}{ii,iii,v,ix}{ii,iii,v,x}{ii,iii,v,xi}{ii,iii,vi,vii}{ii,iii,vi,viii}{ii,iii,vi,ix}{ii,iii,vi,x}{ii,iii,vi,xi}{ii,iii,vii,viii}{ii,iii,vii,ix}{ii,iii,vii,x}{ii,iii,vii,xi}{ii,iii,viii,ix]{ii,iii,viii,x}{ii,iii,viii,xi}{ii,iii,ix,x}{ii,iii,ix,xi}{ii,iii,x,xi}{ii,iv,v,vi}{ii,iv,v,vii}{ii,iv,v,viii}{ii,iv,v,ix}{ii,iv,v,x}{ii,iv,v,xi}{ii,iv,vi,vii}{ii,iv,vi,viii}{ii,iv,vi,ix}{ii,iv,vi,x}{ii,iv,vi,xi}{ii,iv,vii,viii}{ii,iv,vii,ix}{ii,iv,vii,x}{ii,iv,vii,xi}{ii,iv,viii,ix}{ii,iv,viii,x}{ii,iv,viii,xi}{ii,iv,ix,x}{ii,iv,ix,xi}{ii,iv,x,xi}{ii,v,vi,vii}{ii,v,vi,viii}{ii,v,vi,ix}{ii,v,vi,x}{ii,v,vi,xi}{ii,v,vii,viii}{ii,v,vii,ix}{ii,v,vii,x}{ii,v,vii,xi}{ii,v,viii,ix}{ii,v,viii,x}{ii,v,viii,xi}{ii,v,ix,x}{ii,v,ix,xi}{ii,v,x,xi}{ii,vi,vii,viii}{ii,vi,vii,ix}{ii,vi,vii,x}{ii,vi,vii,xi}{ii,vi,viii,ix}{ii,vi,viii,x}{ii,vi,viii,xi}{ii,vi,ix,x}{ii,vi,ix,xi}{ii,vi,x,xi}{ii,vii,viii,ix}{ii,vii,viii,x}{ii,vii,viii,xi}{ii,vii,ix,x}{ii,vii,ix,xi}{ii,vii,x,xi}{ii,viii,ix,x}{ii,viii,ix,xi}{ii,viii,x,xi}{ii,ix,x,xi}{iii,iv,v,vi}{iii,iv,v,vii}{iii,iv,v,viii}{iii,iv,v,ix}{iii,iv,v,x}{iii,iv,v,xi}{iii,iv,vi,vii}{iii,iv,vi,viii]{iii,iv,vi,ix}{iii,iv,vi,x}{iii,iv,vi,xi}{iii,iv,vii,viii}{iii,iv,vii,ix}{iii,iv,vii,x}{iii,iv,vii,xi}{iii,iv,viii,ix}{iii,iv,viii,x}{iii,iv,viii,xi}{iii,iv,ix,x}{iii,iv,ix,xi}{iii,iv,x,xi}{iii,v,vi,vii}{iii,v,vi,viii}{iii,v,vi,ix}{iii,v,vi,x}{iii,v,vi,xi}{iii,v,vii,viii]{iii,v,vii,ix}{iii,v,vii,x}{iii,v,vii,xi}{iii,v,viii,ix}{iii,v,vii,x}{iii,v,viii,xi}{iii,v,ix,x}{iii,v,ix,xi}{iii,v,x,xi}{iii,vi,vii,viii}{iii,vi,vii,ix}{iii,vi,vii,x}{iii,vi,vii,xi}{iii,vi,viii,ix}{iii,vi,viii,x}{iii,vi,viii,xi}{iii,vi,ix,x}{iii,vi,ix,xi}{iii,vi,x,xi]{iii,vii,viii,ix}{iii,vii,viii,x}{iii,vii,viii,xi}{iii,vii,ix,x}{iii,vii,ix,xi}{iii,vii,x,xi}{iii,viii,ix,x}{iii,viii,ix,xi}{iii,viii,x,xi]{iii,ix,x,xi}{iv,v,vi,vii}{iv,v,vi,viii}{iv,v,vi,ix}{iv,v,vi,x}{iv,v,vi,xi}{iv,v,vii,viii}{iv,v,vii,ix}{iv,v,vii,x}{iv,v,vii,xi}{iv,v,viii,ix}{iv,v,viii,x}{iv,v,viii,xi}{iv,v,ix,x}{iv,v,ix,xi}{iv,v,x,xi}{iv,vi,vii,viii}{iv,vi,vii,ix}{iv,vi,vii,x]{iv,vi,vii,xi}{iv,vi,viii,ix}{iv,vi,viii,x}{iv,vi,viii,xi}{iv,vi,ix,x}{iv,vi,ix,xi}{iv,vi,x,xi}{iv,vii,viii,ix}{iv,vii,viii,x]{iv,vii,viii,xi}{iv,vii,ix,x}{iv,vii,ix,xi}{iv,vii,x,xi}{iv,viii,ix,x}{iv,viii,ix,xi}{iv,viii,x,xi}{iv,ix,x,xi}{v,vi,vii,viii]{v,vi,vii,ix}{v,vi,vii,x}{v,vi,vii,xi}{v,vi,viii,ix}{v,vi,viii,x}{v,vi,viii,xi}{v,vi,ix,x}{v,vi,ix,xi}{v,vi,x,xi}{v,vii,viii,ix]{v,vii,viii,x}{v,vii,viii,xi}{v,vii,ix,x}{v,vii,ix,xi}{v,vii,x,xi}{v,viii,ix,x}{v,viii,ix,xi}{v,viii,x,xi}{v,ix,x,xi}{vi,vii,viii,ix}{vi,vii,viii,x}{vi,vii,viii,xi}{vi,vii,ix,x}{vi,vii,ix,xi}{vi,vii,x,xi}{vi,viii,ix,x}{vi,viii,ix,xi}{vi,viii,x,xi}{vi,ix,x,xi}{vii,viii,ix,x}{vii,viii,ix,xi}{vii,viii,x,xi}{vii,ix,x,xi}{viii,ix,x,xi}{i,ii,iii,iv,v}{i,ii,iii,iv,vi}{i,ii,iii,iv,vii}{i,ii,iii,iv,viii}{i,ii,iii,iv,ix}{i,ii,iii,iv,x}{i,ii,iii,iv,xi}{i,ii,iii,v,vi}{i,ii,iii,v,vii}{i,ii,iii,v,viii}{i,ii,iii,v,ix}{i,ii,iii,v,x]{i,ii,iii,v,xi}{i,ii,iii,vi,vii}{i,ii,iii,vi,viii}{i,ii,iii,vi,ix}{i,ii,iii,vi,x}{i,ii,iii,vi,xi}{i,ii,iii,vii,viii}{i,ii,iii,vii,ix}{i,ii,iii,vii,x}{i,ii,iii,vii,xi}{i,ii,iii,viii,ix}{i,ii,iii,viii,x}{i,ii,iii,viii,xi}{i,ii,iii,ix,x}{i,ii,iii,ix,xi}{i,ii,iii,x,xi}{i,ii,iv,v,vi}{i,ii,iv,v,vii}{i,ii,iv,v,viii}{i,ii,iv,v,ix}{i,ii,iv,v,x}{i,ii,iv,v,xi}{i,ii,iv,vi,vii}{i,ii,iv,vi,viii}{i,ii,iv,vi,ix}{i,ii,iv,vi,x}{i,ii,iv,vi,xi}{i,ii,iv,vii,viii}{i,ii,iv,vii,ix}{i,ii,iv,vii,x}{i,ii,iv,vii,xi}{i,ii,iv,viii,ix}{i,ii,iv,viii,x}{i,ii,iv,viii,xi}{i,ii,iv,ix,x}{i,ii,iv,ix,xi]{i,ii,iv,x,xi}{i,ii,v,vi,vii}{i,ii,v,vi,viii}{i,ii,v,vi,ix}{i,ii,v,vi,x}{i,ii,v,vi,xi}{i,ii,v,vii,viii}{i,ii,v,vii,ix}{i,ii,v,vii,x}{i,ii,v,vii,xi}{i,ii,v,viii,ix}{i,ii,v,viii,x}{i,ii,v,viii,xi}{i,ii,v,ix,x}{i,ii,v,ix,xi}{i,ii,v,x,xi}{i,ii,vi,vii,viii}{i,ii,vi,vii,ix}{i,ii,vi,vii,x}{i,ii,vi,vii,xi}{i,ii,vi,viii,ix}{i,ii,vi,viii,x}{i,ii,vi,viii,xi}{i,ii,vi,ix,x}{i,ii,vi,ix,xi}{i,ii,vi,x,xi}{i,ii,vii,viii,ix]{i,ii,vii,viii,x}{i,ii,vii,viii,xi}{i,ii,vii,ix,x}{i,ii,vii,ix,xi}{i,ii,vii,x,xi}{i,ii,viii,ix,x}{i,ii,viii,ix,xi}{i,ii,viii,x,xi}{i,ii,ix,x,xi}{i,iii,iv,v,vi}{i,iii,iv,v,vii}{i,iii,iv,v,viii}{i,iii,iv,v,ix}{i,iii,iv,v,x}{i,iii,iv,v,xi}{i,iii,iv,vi,vii}{i,iii,iv,vi,viii}{i,iii,iv,vi,ix}{i,iii,iv,vi,x}{i,iii,iv,vi,xi}{i,iii,iv,vii,viii}{i,iii,iv,vii,ix}{i,iii,iv,vii,x}{i,iii,iv,vii,xi}{i,iii,iv,viii,ix}{i,iii,iv,viii,x}{i,iii,iv,viii,xi}{i,iii,iv,ix,x}{i,iii,iv,ix,xi}{i,iii,iv,x,xi}{i,iii,v,vi,vii}{i,iii,v,vi,viii}{i,iii,v,vi,ix}{i,iii,v,vi,x}{i,iii,v,vi,xi]{i,iii,v,vii,viii}{i,iii,v,vii,ix}{i,iii,v,vii,x}{i,iii,v,vii,xi}{i,iii,v,viii,ix}{i,iii,v,viii,x}{i,iii,v,viii,xi}{i,iii,v,ix,x}{i,iii,v,ix,xi}{i,iii,v,x,xi}{i,iii,vi,vii,viii}{i,iii,vi,vii,ix}{i,iii,vi,vii,x}{i,iii,vi,vii,xi}{i,iii,vi,viii,ix}{i,iii,vi,viii,x}{i,iii,vi,viii,xi}{i,iii,vi,ix,x}{i,iii,vi,ix,xi}{i,iii,vi,x,xi}{i,iii,vii,viii,ix}{i,iii,vii,viii,x}{i,iii,vii,viii,xi}{i,iii,vii,ix,x}{i,iii,vii,ix,xi}{i,iii,vii,x,xi}{i,iii,viii,ix,x}{i,iii,viii,ix,xi}{i,iii,viii,x,xi}{i,iii,ix,x,xi}{i,iv,v,vi,vii}{i,iv,v,vi,viii}{i,iv,v,vi,ix}{i,iv,v,vi,x}{i,iv,v,vi,xi}{i,iv,v,vii,viii}{i,iv,v,vii,ix}{i,iv,v,vii,x}{i,iv,v,vii,xi}{i,iv,v,viii,ix}{i,iv,v,viii,x}{i,iv,v,viii,xi]{i,iv,v,ix,x}{i,iv,v,ix,xi}{i,iv,v,x,xi}{i,iv,vi,vii,viii}{i,iv,vi,vii,ix}{i,iv,vi,vii,x}{i,iv,vi,vii,xi}{i,iv,vi,viii,ix}{i,iv,vi,viii,x}{i,iv,vi,viii,xi}{i,iv,vi,ix,x}{i,iv,vi,ix,xi}{i,iv,vi,x,xi}{i,iv,vii,viii,ix}{i,iv,vii,viii,x}{i,iv,vii,viii,xi}{i,iv,vii,ix,x}{i,iv,vii,ix,xi}{i,iv,vii,x,xi}{i,iv,viii,ix,x}{i,iv,viii,ix,xi}{i,iv,viii,x,xi}{i,iv,ix,x,xi}{i,v,vi,vii,viii}{i,v,vi,vii,ix}{i,v,vi,vii,x}{i,v,vi,vii,xi}{i,v,vi,viii,ix}{i,v,vi,viii,x}{i,v,vi,viii,xi}{i,v,vi,ix,x}{i,v,vi,ix,xi}{i,v,vi,x,xi}{i,v,vii,viii,ix}{i,v,vii,viii,x}{i,v,vii,viii,xi}{i,v,vii,ix,x}{i,v,vii,ix,xi}{i,v,vii,x,xi}{i,v,viii,ix,x}{i,v,viii,ix,xi}{i,v,viii,x,xi}{i,v,ix,x,xi}{i,vi,vii,viii,ix}{i,vi,vii,viii,x}{i,vi,vii,viii,xi}{i,vi,vii,ix,x}{i,vi,vii,ix,xi}{i,vi,vii,x,xi}{i,vi,viii,ix,x}{i,vi,viii,ix,xi}{i,vi,viii,x,xi}{i,vi,ix,x,xi}{i,vii,viii,ix,x}{i,vii,viii,ix,xi}{i,vii,viii,x,xi}{i,vii,ix,x,xi}{i,viii,ix,x,xi}{ii,iii,iv,v,vi}{ii,iii,iv,v,vii}{ii,iii,iv,v,viii}{ii,iii,iv,v,ix}{ii,iii,iv,v,x}{ii,iii,iv,v,xi}{ii,iii,iv,vi,vii}{ii,iii,iv,vi,viii}{ii,iii,iv,vi,ix}{ii,iii,iv,vi,x}{ii,iii,iv,vi,xi}{ii,iii,iv,vii,viii}{ii,iii,iv,vii,ix}{ii,iii,iv,vii,x}{ii,iii,iv,vii,xi}{ii,iii,iv,viii,ix}{ii,iii,iv,viii,x}{ii,iii,iv,viii,xi}{ii,iii,iv,ix,x}{ii,iii,iv,ix,xi}{ii,iii,iv,x,xi}{ii,iii,v,vi,vii}{ii,iii,v,vi,viii}{ii,iii,v,vi,ix}{ii,iii,v,vi,x}{ii,iii,v,vi,xi}{ii,iii,v,vii,viii}{ii,iii,v,vii,ix}{ii,iii,v,vii,x}{ii,iii,v,vii,xi}{ii,iii,v,viii,ix}{ii,iii,v,viii,x}{ii,iii,v,viii,xi}{ii,iii,v,ix,x}{ii,iii,v,ix,xi}{ii,iii,v,x,xi}{ii,iii,vi,vii,viii}{ii,iii,vi,vii,ix}{ii,iii,vi,vii,x}{ii,iii,vi,vii,xi}{ii,iii,vi,viii,ix}{ii,iii,vi,viii,x}{ii,iii,vi,viii,xi}{ii,iii,vi,ix,x}{ii,iii,vi,ix,xi}{ii,iii,vi,x,xi}{ii,iii,vii,viii,ix}{ii,iii,vii,viii,x}{ii,iii,vii,viii,xi}{ii,iii,vii,ix,x}{ii,iii,vii,ix,xi}{ii,iii,vii,x,xi}{ii,iii,viii,ix,x}{ii,iii,viii,ix,xi}{ii,iii,viii,x,xi}{ii,iii,ix,x,xi}{ii,iv,v,vi,vii}{ii,iv,v,vi,viii}{ii,iv,v,vi,ix}{ii,iv,v,vi,x}{ii,iv,v,vi,xi}{ii,iv,v,vii,viii}{ii,iv,v,vii,ix}{ii,iv,v,vii,x}{ii,iv,v,vii,xi}{ii,iv,v,viii,ix}{ii,iv,v,viii,x}{ii,iv,v,viii,xi}{ii,iv,v,ix,x}{ii,iv,v,ix,xi}{ii,iv,v,x,xi}{ii,iv,vi,vii,viii}{ii,iv,vi,vii,ix}{ii,iv,vi,vii,x}{ii,iv,vi,vii,xi}{ii,iv,vi,viii,ix}{ii,iv,vi,viii,x}{ii,iv,vi,viii,xi}{ii,iv,vi,ix,x}{ii,iv,vi,ix,xi}{ii,iv,vi,x,xi}{ii,iv,vii,viii,ix}{ii,iv,vii,viii,x}{ii,iv,vii,viii,xi}{ii,iv,vii,ix,x}{ii,iv,vii,ix,xi}{ii,iv,vii,x,xi}{ii,iv,viii,ix,x}{ii,iv,viii,ix,xi}{ii,iv,viii,x,xi}{ii,iv,ix,x,xi}{ii,v,vi,vii,viii}{ii,v,vi,vii,ix}{ii,v,vi,vii,x}{ii,v,vi,vii,xi}{ii,v,vi,viii,ix}{ii,v,vi,viii,x}{ii,v,vi,viii,xi}{ii,v,vi,ix,x}{ii,v,vi,ix,xi}{ii,v,vi,x,xi}{ii,v,vii,viii,ix}{ii,v,vii,viii,x}{ii,v,vii,viii,xi}{ii,v,vii,ix,x}{ii,v,vii,ix,xi}{ii,v,vii,x,xi}{ii,v,viii,ix,x}{ii,v,viii,ix,xi}{ii,v,viii,x,xi}{ii,v,ix,x,xi}{ii,vi,vii,viii,ix}{ii,vi,vii,viii,x}{ii,vi,vii,viii,xi}{ii,vi,vii,ix,x}{ii,vi,vii,ix,xi}{ii,vi,vii,x,xi}{ii,vi,viii,ix,x}{ii,vi,viii,ix,xi}{ii,vi,viii,x,xi}{ii,vi,ix,x,xi}{ii,vii,viii,ix,x}{ii,vii,viii,ix,xi}{ii,vii,viii,x,xi}{ii,vii,ix,x,xi}{ii,viii,ix,x,xi}{iii,iv,v,vi,vii}{iii,iv,v,vi,viii}{iii,iv,v,vi,ix}{iii,iv,v,vi,x}{iii,iv,v,vi,xi}{iii,iv,v,vii,viii}{iii,iv,v,vii,ix}{iii,iv,v,vii,x}{iii,iv,v,vii,xi}{iii,iv,v,viii,ix}{iii,iv,v,viii,x}{iii,iv,v,viii,xi}{iii,iv,v,ix,x}{iii,iv,v,ix,xi}{iii,iv,v,x,xi}{iii,iv,vi,vii,viii}{iii,iv,vi,vii,ix}{iii,iv,vi,vii,x}{iii,iv,vi,vii,xi}{iii,iv,vi,viii,ix}{iii,iv,vi,viii,x}{iii,iv,vi,viii,xi}{iii,iv,vi,ix,x}{iii,iv,vi,ix,xi}{iii,iv,vi,x,xi}{iii,iv,vii,viii,ix}{iii,iv,vii,viii,x}{iii,iv,vii,viii,xi}{iii,iv,vii,ix,x}{iii,iv,vii,ix,xi}{iii,iv,vii,x,xi}{iii,iv,viii,ix,x}{iii,iv,viii,ix,xi}{iii,iv,viii,x,xi}{iii,iv,ix,x,xi}{iii,v,vi,vii,viii}{iii,v,vi,vii,ix}{iii,v,vi,vii,x}{iii,v,vi,vii,xi}{iii,v,vi,viii,ix}{iii,v,vi,viii,x}{iii,v,vi,viii,xi}{iii,v,vi,ix,x}{iii,v,vi,ix,xi}{iii,v,vi,x,xi}{iii,v,vii,viii,ix}{iii,v,vii,viii,x}{iii,v,vii,viii,xi}{iii,v,vii,ix,x}{iii,v,vii,ix,xi}{iii,v,vii,x,xi}{iii,v,viii,ix,x}{iii,v,viii,ix,xi}{iii,v,viii,x,xi}{iii,v,ix,x,xi}{iii,vi,vii,viii,ix}{iii,vi,vii,viii,x}{iii,vi,vii,viii,xi}{iii,vi,vii,ix,x}{iii,vi,vii,ix,xi}{iii,vi,vii,x,xi}{iii,vi,viii,ix,x}{iii,vi,viii,ix,xi}{iii,vi,viii,x,xi}{iii,vi,ix,x,xi}{iii,vii,viii,ix,x}{iii,vii,viii,ix,xi}{iii,vii,viii,x,xi}{iii,vii,ix,x,xi}{iii,viii,ix,x,xi}{iv,v,vi,vii,viii}{iv,v,vi,vii,ix}{iv,v,vi,vii,x}{iv,v,vi,vii,xi}{iv,v,vi,viii,ix}{iv,v,vi,viii,x}{iv,v,vi,viii,xi}{iv,v,vi,ix,x}{iv,v,vi,ix,xi}{iv,v,vi,x,xi}{iv,v,vii,viii,ix}{iv,v,vii,viii,x}{iv,v,vii,viii,xi}{iv,v,vii,ix,x}{iv,v,vii,ix,xi}{iv,v,vii,x,xi}{iv,v,viii,ix,x}{iv,v,viii,ix,xi}{iv,v,viii,x,xi}{iv,v,ix,x,xi}{iv,vi,vii,viii,ix}{iv,vi,vii,viii,x}{iv,vi,vii,viii,x{iv,vi,vii,ix,x}{iv,vi,vii,ix,xi}{iv,vi,vii,x,xi}{iv,vi,viii,ix,x}{iv,vi,viii,ix,xi}{iv,vi,viii,x,xi}{iv,vi,ix,x,xi}{iv,vii,viii,ix,x}{iv,vii,viii,ix,xi}{iv,vii,viii,x,xi}{iv,vii,ix,x,xi}{iv,viii,ix,x,xi}{v,vi,vii,viii,ix}{v,vi,vii,viii,x}{v,vi,vii,viii,xi}{v,vi,vii,ix,x}{v,vi,vii,ix,xi}{v,vi,vii,x,xi}{v,vi,viii,ix,x}{v,vi,viii,ix,xi}{v,vi,viii,x,xi}{v,vi,ix,x,xi}{v,vii,viii,ix,x}{v,vii,viii,ix,xi}{v,vii,viii,x,xi}{v,vii,ix,x,xi}{v,viii,ix,x,xi}{vi,vii,viii,ix,x}{vi,vii,viii,ix,xi}{vi,vii,viii,x,xi}{vi,vii,ix,x,xi}{vi,viii,ix,x,xi}{vii,viii,ix,x,xi}{i,ii,iii,iv,v,vi}{i,ii,iii,iv,v,vii}{i,ii,iii,iv,v,viii}{i,ii,iii,iv,v,ix}{i,ii,iii,iv,v,x}{i,ii,iii,iv,v,xi}{i,ii,iii,iv,vi,vii}{i,ii,iii,iv,vi,viii}{i,ii,iii,iv,vi,ix}{i,ii,iii,iv,vi,x}{i,ii,iii,iv,vi,xi}{i,ii,iii,iv,vii,viii}{i,ii,iii,iv,vii,ix}{i,ii,iii,iv,vii,x}{i,ii,iii,iv,vii,xi}{i,ii,iii,iv,viii,ix}{i,ii,iii,iv,viii,x}{i,ii,iii,iv,viii,xi}{i,ii,iii,iv,ix,x}{i,ii,iii,iv,ix,xi}{i,ii,iii,iv,x,xi}{i,ii,iii,v,vi,vii}{i,ii,iii,v,vi,viii}{i,ii,iii,v,vi,ix}{i,ii,iii,v,vi,x}{i,ii,iii,v,vi,xi}{i,ii,iii,v,vii,viii}{i,ii,iii,v,vii,ix}{i,ii,iii,v,vii,x}{i,ii,iii,v,vii,xi}{i,ii,iii,v,viii,ix}{i,ii,iii,v,viii,x}{i,ii,iii,v,viii,xi}{i,ii,iii,v,ix,x}{i,ii,iii,v,ix,xi}{i,ii,iii,v,x,xi}{i,ii,iii,vi,vii,viii}{i,ii,iii,vi,vii,ix}{i,ii,iii,vi,vii,x}{i,ii,iii,vi,vii,xi}{i,ii,iii,vi,viii,ix}{i,ii,iii,vi,viii,x}{i,ii,iii,vi,viii,xi}{i,ii,iii,vi,ix,x}{i,ii,iii,vi,ix,xi}{i,ii,iii,vi,x,xi}{i,ii,iii,vii,viii,ix}{i,ii,iii,vii,viii,x}{i,ii,iii,vii,viii,xi}{i,ii,iii,vii,ix,x}{i,ii,iii,vii,ix,xi}{i,ii,iii,vii,x,xi}{i,ii,iii,viii,ix,x}{i,ii,iii,viii,ix,xi}{i,ii,iii,viii,x,xi}{i,ii,iii,ix,x,xi}{i,ii,iv,v,vi,vii}{i,ii,iv,v,vi,viii}{i,ii,iv,v,vi,ix}{i,ii,iv,v,vi,x}{i,ii,iv,v,vi,xi}{i,ii,iv,v,vii,viii}{i,ii,iv,v,vii,ix}{i,ii,iv,v,vii,x}{i,ii,iv,v,vii,xi}{i,ii,iv,v,viii,ix}{i,ii,iv,v,viii,x}{i,ii,iv,v,viii,xi}{i,ii,iv,v,ix,x}{i,ii,iv,v,ix,xi}{i,ii,iv,v,x,xi}{i,ii,iv,vi,vii,viii}{i,ii,iv,vi,vii,ix}{i,ii,iv,vi,vii,x}{i,ii,iv,vi,vii,xi}{i,ii,iv,vi,viii,ix}{i,ii,iv,vi,viii,x}{i,ii,iv,vi,viii,xi}{i,ii,iv,vi,ix,x}{i,ii,iv,vi,ix,xi}{i,ii,iv,vi,x,xi}{i,ii,iv,vii,viii,ix}{i,ii,iv,vii,viii,x}{i,ii,iv,vii,viii,xi}{i,ii,iv,vii,ix,x}{i,ii,iv,vii,ix,xi}{i,ii,iv,vii,x,xi}{i,ii,iv,viii,ix,x}{i,ii,iv,viii,ix,xi}{i,ii,iv,viii,x,xi}{i,ii,iv,ix,x,xi}{i,ii,v,vi,vii,viii}{i,ii,v,vi,vii,ix}{i,ii,v,vi,vii,x}{i,ii,v,vi,vii,xi}{i,ii,v,vi,viii,ix}{i,ii,v,vi,viii,x}{i,ii,v,vi,viii,xi}{i,ii,v,vi,ix,x}{i,ii,v,vi,ix,xi}{i,ii,v,vi,x,xi}{i,ii,v,vii,viii,ix}{i,ii,v,vii,viii,x}{i,ii,v,vii,viii,xi}{i,ii,v,vii,ix,x}{i,ii,v,vii,ix,xi}{i,ii,v,vii,x,xi}{i,ii,v,viii,ix,x}{i,ii,v,viii,ix,xi}{i,ii,v,viii,x,xi}{i,ii,v,ix,x,xi}{i,ii,vi,vii,viii,ix}{i,ii,vi,vii,viii,x}{i,ii,vi,vii,viii,xi}{i,ii,vi,vii,ix,x}{i,ii,vi,vii,ix,xi}{i,ii,vi,vii,x,xi}{i,ii,vi,viii,ix,x}{i,ii,vi,viii,ix,xi}{i,ii,vi,viii,x,xi}{i,ii,vi,ix,x,xi}{i,ii,vii,viii,ix,x}{i,ii,vii,viii,ix,xi}{i,ii,vii,viii,x,xi}{i,ii,vii,ix,x,xi}{i,ii,viii,ix,x,xi}{i,iii,iv,v,vi,vii}{i,iii,iv,v,vi,viii}{i,iii,iv,v,vi,ix}{i,iii,iv,v,vi,x}{i,iii,iv,v,vi,xi}{i,iii,iv,v,vii,viii}{i,iii,iv,v,vii,ix}{i,iii,iv,v,vii,x}{i,iii,iv,v,vii,xi}{i,iii,iv,v,viii,ix}{i,iii,iv,v,viii,x}{i,iii,iv,v,viii,xi}{i,iii,iv,v,ix,x}{i,iii,iv,v,ix,xi}{i,iii,iv,v,x,xi}{i,iii,iv,vi,vii,viii}{i,iii,iv,vi,vii,ix}{i,iii,iv,vi,vii,x}{i,iii,iv,vi,vii,xi}{i,iii,iv,vi,viii,ix}{i,iii,iv,vi,viii,x}{i,iii,iv,vi,viii,xi}{i,iii,iv,vi,ix,x}{i,iii,iv,vi,ix,xi}{i,iii,iv,vi,x,xi}{i,iii,iv,vii,viii,ix}{i,iii,iv,vii,viii,x}{i,iii,iv,vii,viii,xi}{i,iii,iv,vii,ix,x}{i,iii,iv,vii,ix,xi}{i,iii,iv,vii,x,xi}{i,iii,iv,viii,ix,x}{i,iii,iv,viii,ix,xi}{i,iii,iv,viii,x,xi}{i,iii,iv,ix,x,xi}{i,iii,v,vi,vii,viii}{i,iii,v,vi,vii,ix}{i,iii,v,vi,vii,x}{i,iii,v,vi,vii,xi}{i,iii,v,vi,viii,ix}{i,iii,v,vi,viii,x}{i,iii,v,vi,viii,xi}{i,iii,v,vi,ix,x}{i,iii,v,vi,ix,xi}{i,iii,v,vi,x,xi}{i,iii,v,vii,viii,ix}{i,iii,v,vii,viii,x}{i,iii,v,vii,viii,xi}{i,iii,v,vii,ix,x}{i,iii,v,vii,ix,xi}{i,iii,v,vii,x,xi}{i,iii,v,viii,ix,x}{i,iii,v,viii,ix,xi}{i,iii,v,viii,x,xi}{i,iii,v,ix,x,xi}{i,iii,vi,vii,viii,ix}{i,iii,vi,vii,viii,x}{i,iii,vi,vii,viii,xi}{i,iii,vi,vii,ix,x}{i,iii,vi,vii,ix,xi}{i,iii,vi,vii,x,xi}{i,iii,vi,viii,ix,x}{i,iii,vi,viii,ix,xi}{i,iii,vi,viii,x,xi}{i,iii,vi,ix,x,xi}{i,iii,vii,viii,ix,x}{i,iii,vii,viii,ix,xi}{i,iii,vii,viii,x,xi}{i,iii,vii,ix,x,xi}{i,iii,viii,ix,x,xi}{i,iv,v,vi,vii,viii}{i,iv,v,vi,vii,ix}{i,iv,v,vi,vii,x}{i,iv,v,vi,vii,xi}{i,iv,v,vi,viii,ix}{i,iv,v,vi,viii,x}{i,iv,v,vi,viii,xi}{i,iv,v,vi,ix,x}{i,iv,v,vi,ix,xi}{i,iv,v,vi,x,xi}{i,iv,v,vii,viii,ix}{i,iv,v,vii,viii,x}{i,iv,v,vii,viii,xi}{i,iv,v,vii,ix,x}{i,iv,v,vii,ix,xi}{i,iv,v,vii,x,xi}{i,iv,v,viii,ix,x}{i,iv,v,viii,ix,xi}{i,iv,v,viii,x,xi}{i,iv,v,ix,x,xi}{i,iv,vi,vii,viii,ix}{i,iv,vi,vii,viii,x}{i,iv,vi,vii,viii,xi}{i,iv,vi,vii,ix,x}{i,iv,vi,vii,ix,xi}{i,iv,vi,vii,x,xi}{i,iv,vi,viii,ix,x}{i,iv,vi,viii,ix,xi}{i,iv,vi,viii,x,xi}{i,iv,vi,ix,x,xi}{i,iv,vii,viii,ix,x}{i,iv,vii,viii,ix,xi}{i,iv,vii,viii,x,xi}{i,iv,vii,ix,x,xi}{i,iv,viii,ix,x,xi}{i,v,vi,vii,viii,ix}{i,v,vi,vii,viii,x}{i,v,vi,vii,viii,xi}{i,v,vi,vii,ix,x}{i,v,vi,vii,ix,xi}{i,v,vi,vii,x,xi}{i,v,vi,viii,ix,x}{i,v,vi,viii,ix,xi}{i,v,vi,viii,x,xi}{i,v,vi,ix,x,xi}{i,v,vii,viii,ix,x}{i,v,vii,viii,ix,xr{i,v,vii,viii,x,xi}{i,v,vii,ix,x,xi}{i,v,viii,ix,x,xi}{i,vi,vii,viii,ix,x}{i,vi,vii,viii,ix,xi}{i,vi,vii,viii,x,xi}{i,vi,vii,ix,x,xi}{i,vi,viii,ix,x,xi}{i,vii,viii,ix,x,xi}{ii,iii,iv,v,vi,vii}{ii,iii,iv,v,vi,viii}{ii,iii,iv,v,vi,ix}{ii,iii,iv,v,vi,x}{ii,iii,iv,v,vi,xi}{ii,iii,iv,v,vii,viii}{ii,iii,iv,v,vii,ix}{ii,iii,iv,v,vii,x}{ii,iii,iv,v,vii,xi}{ii,iii,iv,v,viii,ix}{ii,iii,iv,v,viii,x}{ii,iii,iv,v,viii,xi}{ii,iii,iv,v,ix,x}{ii,iii,iv,v,ix,xi}{ii,iii,iv,v,x,xi}{ii,iii,iv,vi,vii,viii}{ii,iii,iv,vi,vii,ix}{ii,iii,iv,vi,vii,x}{ii,iii,iv,vi,vii,xi}{ii,iii,iv,vi,viii,ix}{ii,iii,iv,vi,viii,x}{ii,iii,iv,vi,viii,xi}{ii,iii,iv,vi,ix,x}{ii,iii,iv,vi,ix,xi}{ii,iii,iv,vi,x,xi}{ii,iii,iv,vii,viii,ix}{ii,iii,iv,vii,viii,x}{ii,iii,iv,vii,viii,xi}{ii,iii,iv,vii,ix,x}{ii,iii,iv,vii,ix,xi}{ii,iii,iv,vii,x,xi}{ii,iii,iv,viii,ix,x}{ii,iii,iv,viii,ix,xi}{ii,iii,iv,viii,x,xi}{ii,iii,iv,ix,x,xi}{ii,iii,v,vi,vii,viii}{ii,iii,v,vi,vii,ix}{ii,iii,v,vi,vii,x}{ii,iii,v,vi,vii,xi}{ii,iii,v,vi,viii,ix}{ii,iii,v,vi,viii,x}{ii,iii,v,vi,viii,xi}{ii,iii,v,vi,ix,x}{ii,iii,v,vi,ix,xi}{ii,iii,v,vi,x,xi}{ii,iii,v,vii,viii,ix}{ii,iii,v,vii,viii,x}{ii,iii,v,vii,viii,xi}{ii,iii,v,vii,ix,x}{ii,iii,v,vii,ix,xi}{ii,iii,v,vii,x,xi}{ii,iii,v,viii,ix,x}{ii,iii,v,viii,ix,xi}{ii,iii,v,viii,x,xi}{ii,iii,v,ix,x,xi}{ii,iii,vi,vii,viii,ix}{ii,iii,vi,vii,viii,x}{ii,iii,vi,vii,viii,xi}{ii,iii,vi,vii,ix,x}{ii,iii,vi,vii,ix,xi}{ii,iii,vi,vii,x,xi}{ii,iii,vi,viii,ix,x}{ii,iii,vi,viii,ix,xi}{ii,iii,vi,viii,x,xi}{ii,iii,vi,ix,x,xi}{ii,iii,vii,viii,ix,x}{ii,iii,vii,viii,ix,xi}{ii,iii,vii,viii,x,xi}{ii,iii,vii,ix,x,xi}{ii,iii,viii,ix,x,xi}{ii,iv,v,vi,vii,viii}{ii,iv,v,vi,vii,ix}{ii,iv,v,vi,vii,x}{ii,iv,v,vi,vii,xi}{ii,iv,v,vi,viii,ix}{ii,iv,v,vi,viii,x}{ii,iv,v,vi,viii,xi}{ii,iv,v,vi,ix,x}{ii,iv,v,vi,ix,xi}{ii,iv,v,vi,x,xi}{ii,iv,v,vii,viii,ix}{ii,iv,v,vii,viii,x}{ii,iv,v,vii,viii,xi}{ii,iv,v,vii,ix,x}{ii,iv,v,vii,ix,xi}{ii,iv,v,vii,x,xi}{ii,iv,v,viii,ix,x}{ii,iv,v,viii,ix,xi}{ii,iv,v,viii,x,xi}{ii,iv,v,ix,x,xi}{ii,iv,vi,vii,viii,ix}{ii,iv,vi,vii,viii,x}{ii,iv,vi,vii,viii,xi}{ii,iv,vi,vii,ix,x}{ii,iv,vi,vii,ix,xi}{ii,iv,vi,vii,x,xi}{ii,iv,vi,viii,ix,x}{ii,iv,vi,viii,ix,xi}{ii,iv,vi,viii,x,xi}{ii,iv,vi,ix,x,xi}{ii,iv,vii,viii,ix,x}{ii,iv,vii,viii,ix,xi}{ii,iv,vii,viii,x,xi}{ii,iv,vii,ix,x,xi}{ii,iv,viii,ix,x,xi}{ii,v,vi,vii,viii,ix}{ii,v,vi,vii,viii,x}{ii,v,vi,vii,viii,xi}{ii,v,vi,vii,ix,x}{ii,v,vi,vii,ix,xi}{ii,v,vi,vii,x,xi}{ii,v,vi,viii,ix,x}{ii,v,vi,viii,ix,xi}{ii,v,vi,viii,x,xi}{ii,v,vi,ix,x,xi}{ii,v,vii,viii,ix,x}{ii,v,vii,viii,ix,xi}{ii,v,vii,viii,x,xi}{ii,v,vii,ix,x,xi}{ii,v,viii,ix,x,xi}{ii,vi,vii,viii,ix,x}{ii,vi,vii,viii,ix,xi}{ii,vi,vii,viii,x,xi}{ii,vi,vii,ix,x,xi}{ii,vi,viii,ix,x,xi}{ii,vii,viii,ix,x,xi}{iii,iv,v,vi,vii,viii}{iii,iv,v,vi,vii,ix}{iii,iv,v,vi,vii,x}{iii,iv,v,vi,vii,xi}{iii,iv,v,vi,viii,ix}{iii,iv,v,vi,viii,x}{iii,iv,v,vi,viii,xi}{iii,iv,v,vi,ix,x}{iii,iv,v,vi,ix,xi}{iii,iv,v,vi,x,xi}{iii,iv,v,vii,viii,ix}{iii,iv,v,vii,viii,x}{iii,iv,v,vii,viii,xi}{iii,iv,v,vii,ix,x}{iii,iv,v,vii,ix,xi}{iii,iv,v,vii,x,xi}{iii,iv,v,viii,ix,x}{iii,iv,v,viii,ix,xi}{iii,iv,v,viii,x,xi}{iii,iv,v,ix,x,xi}{iii,iv,vi,vii,viii,ix}{iii,iv,vi,vii,viii,x}{iii,iv,vi,vii,viii,xi}{iii,iv,vi,vii,ix,x}{iii,iv,vi,vii,ix,xi}{iii,iv,vi,vii,x,xi}{iii,iv,vi,viii,ix,x}{iii,iv,vi,viii,ix,xi}{iii,iv,vi,viii,x,xi}{iii,iv,vi,ix,x,xi}{iii,iv,vii,viii,ix,x}{iii,iv,vii,viii,ix,xi}{iii,iv,vii,viii,x,xi}{iii,iv,vii,ix,x,xi}{iii,iv,viii,ix,x,xi}{iii,v,vi,vii,viii,ix}{iii,v,vi,vii,viii,x}{iii,v,vi,vii,viii,xi}{iii,v,vi,vii,ix,x}{iii,v,vi,vii,ix,xi}{iii,v,vi,vii,x,xi}{iii,v,vi,viii,ix,x}{iii,v,vi,viii,ix,xi}{iii,v,vi,viii,x,xi}{iii,v,vi,ix,x,xi}{iii,v,vii,viii,ix,x}{iii,v,vii,viii,ix,xi}{iii,v,vii,viii,x,xi}{iii,v,vii,ix,x,xi}{iii,v,viii,ix,x,xi}{iii,vi,vii,viii,ix,x}{iii,vi,vii,viii,ix,xi}{iii,vi,vii,viii,x,xi}{iii,vi,vii,ix,x,xi}{iii,vi,viii,ix,x,xi}{iii,vii,viii,ix,x,xi}{iv,v,vi,vii,viii,ix}{iv,v,vi,vii,viii,x}{iv,v,vi,vii,viii,xi}{iv,v,vi,vii,ix,x}{iv,v,vi,vii,ix,xi}{iv,v,vi,vii,x,xi}{iv,v,vi,viii,ix,x^iv,v,vi,viii,ix,xi}{iv,v,vi,viii,x,xi}{iv,v,vi,ix,x,xi}{iv,v,vii,viii,ix,x}{iv,v,vii,viii,ix,xi}{iv,v,vii,viii,x,xiiv,v,vii,ix,x,xi}{iv,v,viii,ix,x,xi}{iv,vi,vii,viii,ix,x}{iv,vi,vii,viii,ix,xi}{iv,vi,vii,viii,x,xi}{iv,vi,vii,ix,x,xiiv,vi,viii,ix,x,xi}{iv,vii,viii,ix,x,xi}{v,vi,vii,viii,ix,x}{v,vi,vii,viii,ix,xi}{v,vi,vii,viii,x,xi}{v,vi,vii,ix,x,xiv,vi,viii,ix,x,xi}{v,vii,viii,ix,x,xi}{vi,vii,viii,ix,x,xi}{i,ii,iii,iv,v,vi,vii}{i,ii,iii,iv,v,vi,viii}{i,ii,iii,iv,v,vi,ixi,iv,v,vi,x}{i,ii,iii,iv,v,vi,xi}{i,ii,iii,iv,v,vii,viii}{i,ii,iii,iv,v,vii,ix}{i,ii,iii,iv,v,vii,x}{i,ii,iii,iv,v,vii,xii,iv,v,viii,ix}{i,ii,iii,iv,v,viii,x}{i,ii,iii,iv,v,viii,xi}{i,ii,iii,iv,v,ix,x}{i,ii,iii,iv,v,ix,xi}{i,ii,iii,iv,v,x,xii,iv,vi,vii,viii}{i,ii,iii,iv,vi,vii,ix}{i,ii,iii,iv,vi,vii,x}{i,ii,iii,iv,vi,vii,xi}{i,ii,iii,iv,vi,viii,ix}{i,ii,iii,iv,vi,viii,xi,iv,vi,viii,xi}{i,ii,iii,iv,vi,ix,x}{i,ii,iii,iv,vi,ix,xi}{i,ii,iii,iv,vi,x,xi}{i,ii,iii,iv,vii,viii,ix}{i,ii,iii,iv,vii,viii,x^i,iv,vii,viii,xi}{i,ii,iii,iv,vii,ix,x}{i,ii,iii,iv,vii,ix,xi}{i,ii,iii,iv,vii,x,xi}{i,ii,iii,iv,viii,ix,x}{i,ii,iii,iv,viii,ix,xii,iv,viii,x,xi}{i,ii,iii,iv,ix,x,xi}{i,ii,iii,v,vi,vii,viii}{i,ii,iii,v,vi,vii,ix}{i,ii,iii,v,vi,vii,x}{i,ii,iii,v,vi,vii,xii,v,vi,viii,ix}{i,ii,iii,v,vi,viii,x}{i,ii,iii,v,vi,viii,xi}{i,ii,iii,v,vi,ix,x}{i,ii,iii,v,vi,ix,xi}{i,ii,iii,v,vi,x,xi}{i,ii,iii,v,vii,viii,ix}{i,ii,iii,v,vii,viii,x}{i,ii,iii,v,vii,viii,xi}{i,ii,iii,v,vii,ix,x}{i,ii,iii,v,vii,ix,xi}{i,ii,iii,v,vii,x,xii,v,viii,ix,x}{i,ii,iii,v,viii,ix,xi}{i,ii,iii,v,viii,x,xi}{i,ii,iii,v,ix,x,xi}{i,ii,iii,vi,vii,viii,ix}{i,ii,iii,vi,vii,viii,xi,vi,vii,viii,xi}{i,ii,iii,vi,vii,ix,x}{i,ii,iii,vi,vii,ix,xi}{i,ii,iii,vi,vii,x,xi}{i,ii,iii,vi,viii,ix,x}{i,ii,iii,vi,viii,ix,xii,vi,viii,x,xi}{i,ii,iii,vi,ix,x,xi}{i,ii,iii,vii,viii,ix,x}{i,ii,iii,vii,viii,ix,xi}{i,ii,iii,vii,viii,x,xi}{i,ii,iii,vii,ix,x,xii,viii,ix,x,xi}{i,ii,iv,v,vi,vii,viii}{i,ii,iv,v,vi,vii,ix}{i,ii,iv,v,vi,vii,x}{i,ii,iv,v,vi,vii,xi}{i,ii,iv,v,vi,viii,ixiv,v,vi,viii,x}{i,ii,iv,v,vi,viii,xi}{i,ii,iv,v,vi,ix,x}{i,ii,iv,v,vi,ix,xi}{i,ii,iv,v,vi,x,xi}{i,ii,iv,v,vii,viii,ix}{i,ii,iv,v,vii,viii,x}{i,ii,iv,v,vii,viii,xi}{i,ii,iv,v,vii,ix,x}{i,ii,iv,v,vii,ix,xi}{i,ii,iv,v,vii,x,xi}{i,ii,iv,v,viii,ix,xiv,v,viii,ix,xi}{i,ii,iv,v,viii,x,xi}{i,ii,iv,v,ix,x,xi}{i,ii,iv,vi,vii,viii,ix}{i,ii,iv,vi,vii,viii,x}{i,ii,iv,vi,vii,viii,xiiv,vi,vii,ix,x}{i,ii,iv,vi,vii,ix,xi}{i,ii,iv,vi,vii,x,xi}{i,ii,iv,vi,viii,ix,x}{i,ii,iv,vi,viii,ix,xi}{i,ii,iv,vi,viii,x,xiiv,vi,ix,x,xi}{i,ii,iv,vii,viii,ix,x}{i,ii,iv,vii,viii,ix,xi}{i,ii,iv,vii,viii,x,xi}{i,ii,iv,vii,ix,x,xi}{i,ii,iv,viii,ix,x,xi}{i,ii,v,vi,vii,viii,ix}{i,ii,v,vi,vii,viii,x}{i,ii,v,vi,vii,viii,xi}{i,ii,v,vi,vii,ix,x}{i,ii,v,vi,vii,ix,xi}{i,ii,v,vi,vii,x,xiv,vi,viii,ix,x}{i,ii,v,vi,viii,ix,xi}{i,ii,v,vi,viii,x,xi}{i,ii,v,vi,ix,x,xi}{i,ii,v,vii,viii,ix,x}{i,ii,v,vii,viii,ix,xiv,vii,viii,x,xi}{i,ii,v,vii,ix,x,xi}{i,ii,v,viii,ix,x,xi}{i,ii,vi,vii,viii,ix,x}{i,ii,vi,vii,viii,ix,xi}{i,ii,vi,vii,viii,x,xivi,vii,ix,x,xi}{i,ii,vi,viii,ix,x,xi}{i,ii,vii,viii,ix,x,xi}{i,iii,iv,v,vi,vii,viii}{i,iii,iv,v,vi,vii,ix}{i,iii,iv,v,vi,vii,x}{i,iii,iv,v,vi,vii,xi}{i,iii,iv,v,vi,viii,ix}{i,iii,iv,v,vi,viii,x}{i,iii,iv,v,vi,viii,xi}{i,iii,iv,v,vi,ix,x}{i,iii,iv,v,vi,ix,xi,iv,v,vi,x,xi}{i,iii,iv,v,vii,viii,ix}{i,iii,iv,v,vii,viii,x}{i,iii,iv,v,vii,viii,xi}{i,iii,iv,v,vii,ix,x}{i,iii,iv,v,vii,ix,xi,iv,v,vii,x,xi}{i,iii,iv,v,viii,ix,x}{i,iii,iv,v,viii,ix,xi}{i,iii,iv,v,viii,x,xi}{i,iii,iv,v,ix,x,xi}{i,iii,iv,vi,vii,viii,ix,iv,vi,vii,viii,x}{i,iii,iv,vi,vii,viii,xi}{i,iii,iv,vi,vii,ix,x}{i,iii,iv,vi,vii,ix,xi}{i,iii,iv,vi,vii,x,xi}{i,iii,iv,vi,viii,ix,x}{i,iii,iv,vi,viii,ix,xi}{i,iii,iv,vi,viii,x,xi}{i,iii,iv,vi,ix,x,xi}{i,iii,iv,vii,viii,ix,x}{i,iii,iv,vii,viii,ix,xi}{i,iii,iv,vii,viii,x,xi}{i,iii,iv,vii,ix,x,xi}{i,iii,iv,viii,ix,x,xi}{i,iii,v,vi,vii,viii,ix}{i,iii,v,vi,vii,viii,x}{i,iii,v,vi,vii,viii,xi}{i,iii,v,vi,vii,ix,x}{i,iii,v,vi,vii,ix,xi}{i,iii,v,vi,vii,x,xi}{i,iii,v,vi,viii,ix,x}{i,iii,v,vi,viii,ix,xi}{i,iii,v,vi,viii,x,xi}{i,iii,v,vi,ix,x,xi,v,vii,viii,ix,x}{i,iii,v,vii,viii,ix,xi}{i,iii,v,vii,viii,x,xi}{i,iii,v,vii,ix,x,xi}{i,iii,v,viii,ix,x,xi}{i,iii,vi,vii,viii,ix,x}{i,iii,vi,vii,viii,ix,xi}{i,iii,vi,vii,viii,x,xi}{i,iii,vi,vii,ix,x,xi}{i,iii,vi,viii,ix,x,xi}{i,iii,vii,viii,ix,x,xi}{i,iv,v,vi,vii,viii,ix}{i,iv,v,vi,vii,viii,x}{i,iv,v,vi,vii,viii,xi}{i,iv,v,vi,vii,ix,x}{i,iv,v,vi,vii,ix,xi}{i,iv,v,vi,vii,x,xi}{i,iv,v,vi,viii,ix,x}{i,iv,v,vi,viii,ix,xi}{i,iv,v,vi,viii,x,xi}{i,iv,v,vi,ix,x,xi}{i,iv,v,vii,viii,ix,x}{i,iv,v,vii,viii,ix,xi}{i,iv,v,vii,viii,x,xi}{i,iv,v,vii,ix,x,xi}{i,iv,v,viii,ix,x,xi}{i,iv,vi,vii,viii,ix,x}{i,iv,vi,vii,viii,ix,xi}{i,iv,vi,vii,viii,x,xi}{i,iv,vi,vii,ix,x,xiiv,vi,viii,ix,x,xi}{i,iv,vii,viii,ix,x,xi}{i,v,vi,vii,viii,ix,x}{i,v,vi,vii,viii,ix,xi}{i,v,vi,vii,viii,x,xi}{i,v,vi,vii,ix,x,xi}{i,v,vi,viii,ix,x,xi}{i,v,vii,viii,ix,x,xi}{i,vi,vii,viii,ix,x,xi}{ii,iii,iv,v,vi,vii,viii}{ii,iii,iv,v,vi,vii,ix}{ii,iii,iv,v,vi,vii,x}{ii,iii,iv,v,vi,vii,xi}{ii,iii,iv,v,vi,viii,ix}{ii,iii,iv,v,vi,viii,x}{ii,iii,iv,v,vi,viii,xi}{ii,iii,iv,v,vi,ix,x}{ii,iii,iv,v,vi,ix,x}{ii,iii,iv,v,vi,x,xi}{ii,iii,iv,v,vii,viii,ix}{ii,iii,iv,v,vii,viii,x}{ii,iii,iv,v,vii,viii,xi}{ii,iii,iv,v,vii,ix,x}{ii,iii,iv,v,vii,ix,x{ii,iii,iv,v,vii,x,xi}{ii,iii,iv,v,viii,ix,x}{ii,iii,iv,v,viii,ix,xi}{ii,iii,iv,v,viii,x,xi}{ii,iii,iv,v,ix,x,xi}{ii,iii,iv,vi,vii,viii,ix}{ii,iii,iv,vi,vii,viii,x}{ii,iii,iv,vi,vii,viii,xi}{ii,iii,iv,vi,vii,ix,x}{ii,iii,iv,vi,vii,ix,xi}{ii,iii,iv,vi,vii,x,xi}{ii,iii,iv,vi,viii,ix,x}{ii,iii,iv,vi,viii,ix,xi}{ii,iii,iv,vi,viii,x,xi}{ii,iii,iv,vi,ix,x,xi}{ii,iii,iv,vii,viii,ix,x}{ii,iii,iv,vii,viii,ix,xi}{ii,iii,iv,vii,viii,x,x{ii,iii,iv,vii,ix,x,xi}{ii,iii,iv,viii,ix,x,xi}{ii,iii,v,vi,vii,viii,ix}{ii,iii,v,vi,vii,viii,x}{ii,iii,v,vi,vii,viii,xi}{ii,iii,v,vi,vii,ix,x}{ii,iii,v,vi,vii,ix,xi}{ii,iii,v,vi,vii,x,xi}{ii,iii,v,vi,viii,ix,x}{ii,iii,v,vi,viii,ix,xi}{ii,iii,v,vi,viii,x,xi}{ii,iii,v,vi,ix,x,x}{ii,iii,v,vii,viii,ix,x}{ii,iii,v,vii,viii,ix,xi}{ii,iii,v,vii,viii,x,xi}{ii,iii,v,vii,ix,x,xi}{ii,iii,v,viii,ix,x,xi}{ii,iii,vi,vii,viii,ix,x}{ii,iii,vi,vii,viii,ix,xi}{ii,iii,vi,vii,viii,x,xi}{ii,iii,vi,vii,ix,x,xi}{ii,iii,vi,viii,ix,x,xi}{ii,iii,vii,viii,ix,x,xi}{ii,iv,v,vi,vii,viii,ix}{ii,iv,v,vi,vii,viii,x}{ii,iv,v,vi,vii,viii,xi}{ii,iv,v,vi,vii,ix,x}{ii,iv,v,vi,vii,ix,xi}{ii,iv,v,vi,vii,x,xi}{ii,iv,v,vi,viii,ix,x}{ii,iv,v,vi,viii,ix,xi}{ii,iv,v,vi,viii,x,xi}{ii,iv,v,vi,ix,x,xi}{ii,iv,v,vii,viii,ix,x}{ii,iv,v,vii,viii,ix,xi}{ii,iv,v,vii,viii,x,x}{ii,iv,v,vii,ix,x,xi}{ii,iv,v,viii,ix,x,xi}{ii,iv,vi,vii,viii,ix,x}{ii,iv,vi,vii,viii,ix,xi}{ii,iv,vi,vii,viii,x,xi}{ii,iv,vi,vii,ix,x,x}{ii,iv,vi,viii,ix,x,xi}{ii,iv,vii,viii,ix,x,xi}{ii,v,vi,vii,viii,ix,x}{ii,v,vi,vii,viii,ix,xi}{ii,v,vi,vii,viii,x,xi}{ii,v,vi,vii,ix,x,x{ii,v,vi,viii,ix,x,xi}{ii,v,vii,viii,ix,x,xi}{ii,vi,vii,viii,ix,x,xi}{iii,iv,v,vi,vii,viii,ix}{iii,iv,v,vi,vii,viii,x}{iii,iv,v,vi,vii,viii,xi}{iii,iv,v,vi,vii,ix,x}{iii,iv,v,vi,vii,ix,xi}{iii,iv,v,vi,vii,x,xi}{iii,iv,v,vi,viii,ix,x}{iii,iv,v,vi,viii,ix,x}{iii,iv,v,vi,viii,x,xi}{iii,iv,v,vi,ix,x,xi}{iii,iv,v,vii,viii,ix,x}{iii,iv,v,vii,viii,ix,xi}{iii,iv,v,vii,viii,x,xi}{iii,iv,v,vii,ix,x,xiii,iv,v,viii,ix,x,xi}{iii,iv,vi,vii,viii,ix,x}{iii,iv,vi,vii,viii,ix,xi}{iii,iv,vi,vii,viii,x,xi}{iii,iv,vi,vii,ix,x,xriii,iv,vi,viii,ix,x,xi}{iii,iv,vii,viii,ix,x,xi}{iii,v,vi,vii,viii,ix,x}{iii,v,vi,vii,viii,ix,xi}{iii,v,vi,vii,viii,x,xi}{iii,v,vi,vii,ix,x,xi}{iii,v,vi,viii,ix,x,xi}{iii,v,vii,viii,ix,x,xi}{iii,vi,vii,viii,ix,x,xi}{iv,v,vi,vii,viii,ix,x}{iv,v,vi,vii,viii,ix,xi}{iv,v,vi,vii,viii,x,xi}{iv,v,vi,vii,ix,x,xi}{iv,v,vi,viii,ix,x,xi}{iv,v,vii,viii,ix,x,xi}{iv,vi,vii,viii,ix,x,xi}{v,vi,vii,viii,ix,x,xi}{i,ii,iii,iv,v,vi,vii,viii}{i,ii,iii,iv,v,vi,vii,ix}{i,ii,iii,iv,v,vi,vii,x}{i,ii,iii,iv,v,vi,vii,xi}{i,ii,iii,iv,v,vi,viii,ix}{i,ii,iii,iv,v,vi,viii,x}{i,ii,iii,iv,v,vi,viii,xi}{i,ii,iii,iv,v,vi,ix,x}{i,ii,iii,iv,v,vi,ix,xi}{i,ii,iii,iv,v,vi,x,xi}{i,ii,iii,iv,v,vii,viii,ix}{i,ii,iii,iv,v,vii,viii,x}{i,ii,iii,iv,v,vii,viii,x}{i,ii,iii,iv,v,vii,ix,x}{i,ii,iii,iv,v,vii,ix,xi}{i,ii,iii,iv,v,vii,x,xi}{i,ii,iii,iv,v,viii,ix,x}{i,ii,iii,iv,v,viii,ix,xi,ii,iii,iv,v,viii,x,xi}{i,ii,iii,iv,v,ix,x,xi}{i,ii,iii,iv,vi,vii,viii,ix}{i,ii,iii,iv,vi,vii,viii,x}{i,ii,iii,iv,vi,vii,viii,x}{i,ii,iii,iv,vi,vii,ix,x}{i,ii,iii,iv,vi,vii,ix,xi}{i,ii,iii,iv,vi,vii,x,xi}{i,ii,iii,iv,vi,viii,ix,x}{i,ii,iii,iv,vi,viii,ix,x}{i,ii,iii,iv,vi,viii,x,xi}{i,ii,iii,iv,vi,ix,x,xi}{i,ii,iii,iv,vii,viii,ix,x}{i,ii,iii,iv,vii,viii,ix,xi}{i,ii,iii,iv,vii,viii,x,x}{i,ii,iii,iv,vii,ix,x,xi}{i,ii,iii,iv,viii,ix,x,xi}{i,ii,iii,v,vi,vii,viii,ix}{i,ii,iii,v,vi,vii,viii,x}{i,ii,iii,v,vi,vii,viii,x}{i,ii,iii,v,vi,vii,ix,x}{i,ii,iii,v,vi,vii,ix,xi}{i,ii,iii,v,vi,vii,x,xi}{i,ii,iii,v,vi,viii,ix,x}{i,ii,iii,v,vi,viii,ix,x}{i,ii,iii,v,vi,viii,x,xi}{i,ii,iii,v,vi,ix,x,xi}{i,ii,iii,v,vii,viii,ix,x}{i,ii,iii,v,vii,viii,ix,xi}{i,ii,iii,v,vii,viii,x,x}{i,ii,iii,v,vii,ix,x,xi}{i,ii,iii,v,viii,ix,x,xi}{i,ii,iii,vi,vii,viii,ix,x}{i,ii,iii,vi,vii,viii,ix,xi}{i,ii,iii,vi,vii,viii,x,x}{i,ii,iii,vi,vii,ix,x,xi}{i,ii,iii,vi,viii,ix,x,xi}{i,ii,iii,vii,viii,ix,x,xi}{i,ii,iv,v,vi,vii,viii,ix}{i,ii,iv,v,vi,vii,viii,x}{i,ii,iv,v,vi,vii,viii,xi}{i,ii,iv,v,vi,vii,ix,x}{i,ii,iv,v,vi,vii,ix,xi}{i,ii,iv,v,vi,vii,x,xi}{i,ii,iv,v,vi,viii,ix,x}{i,ii,iv,v,vi,viii,ix,xi}{i,ii,iv,v,vi,viii,x,xi}{i,ii,iv,v,vi,ix,x,xi}{i,ii,iv,v,vii,viii,ix,x}{i,ii,iv,v,vii,viii,ix,x}{i,ii,iv,v,vii,viii,x,xi}{i,ii,iv,v,vii,ix,x,xi}{i,ii,iv,v,viii,ix,x,xi}{i,ii,iv,vi,vii,viii,ix,x}{i,ii,iv,vi,vii,viii,ix,x}{i,ii,iv,vi,vii,viii,x,xi}{i,ii,iv,vi,vii,ix,x,xi}{i,ii,iv,vi,viii,ix,x,xi}{i,ii,iv,vii,viii,ix,x,xi}{i,ii,v,vi,vii,viii,ix,x}{i,ii,v,vi,vii,viii,ix,xi}{i,ii,v,vi,vii,viii,x,xi}{i,ii,v,vi,vii,ix,x,xi}{i,ii,v,vi,viii,ix,x,xi}{i,ii,v,vii,viii,ix,x,xi}{i,ii,vi,vii,viii,ix,x,xi}{i,iii,iv,v,vi,vii,viii,ix}{i,iii,iv,v,vi,vii,viii,x}{i,iii,iv,v,vi,vii,viii,xi}{i,iii,iv,v,vi,vii,ix,x}{i,iii,iv,v,vi,vii,ix,xi}{i,iii,iv,v,vi,vii,x,xi}{i,iii,iv,v,vi,viii,ix,x}{i,iii,iv,v,vi,viii,ix,xi}{i,iii,iv,v,vi,viii,x,xi}{i,iii,iv,v,vi,ix,x,xi}{i,iii,iv,v,vii,viii,ix,x}{i,iii,iv,v,vii,viii,ix,xi}{i,iii,iv,v,vii,viii,x,xi}{i,iii,iv,v,vii,ix,x,xi}{i,iii,iv,v,viii,ix,x,xi}{i,iii,iv,vi,vii,viii,ix,x}{i,iii,iv,vi,vii,viii,ix,xi}{i,iii,iv,vi,vii,viii,x,xi}{i,iii,iv,vi,vii,ix,x,xi}{i,iii,iv,vi,viii,ix,x,xi}{i,iii,iv,vii,viii,ix,x,xi}{i,iii,v,vi,vii,viii,ix,x}{i,iii,v,vi,vii,viii,ix,xi}{i,iii,v,vi,vii,viii,x,xi}{i,iii,v,vi,vii,ix,x,xi}{i,iii,v,vi,viii,ix,x,xi}{i,iii,v,vii,viii,ix,x,xi}{i,iii,vi,vii,viii,ix,x,xi}{i,iv,v,vi,vii,viii,ix,x}{i,iv,v,vi,vii,viii,ix,xi}{i,iv,v,vi,vii,viii,x,xi}{i,iv,v,vi,vii,ix,x,xi}{i,iv,v,vi,viii,ix,x,xi}{i,iv,v,vii,viii,ix,x,xi}{i,iv,vi,vii,viii,ix,x,xi}{i,v,vi,vii,viii,ix,x,xi}{ii,iii,iv,v,vi,vii,viii,ix}{ii,iii,iv,v,vi,vii,viii,x}{ii,iii,iv,v,vi,vii,viii,xi}{ii,iii,iv,v,vi,vii,ix,x}{ii,iii,iv,v,vi,vii,ix,xi}{ii,iii,iv,v,vi,vii,x,xi}{ii,iii,iv,v,vi,viii,ix,x}{ii,iii,iv,v,vi,viii,ix,xi}{ii,iii,iv,v,vi,viii,x,xi}{ii,iii,iv,v,vi,ix,x,xi}{ii,iii,iv,v,vii,viii,ix,x}{ii,iii,iv,v,vii,viii,ix,xi}{ii,iii,iv,v,vii,viii,x,xi}{ii,iii,iv,v,vii,ix,x,xi}{ii,iii,iv,v,viii,ix,x,xi}{ii,iii,iv,vi,vii,viii,ix,x}{ii,iii,iv,vi,vii,viii,ix,xi}{ii,iii,iv,vi,vii,viii,x,xi}{ii,iii,iv,vi,vii,ix,x,xi}{ii,iii,iv,vi,viii,ix,x,xi}{ii,iii,iv,vii,viii,ix,x,xi}{ii,iii,v,vi,vii,viii,ix,x}{ii,iii,v,vi,vii,viii,ix,xii,v,vi,vii,viii,x,xi}{ii,iii,v,vi,vii,ix,x,xi}{ii,iii,v,vi,viii,ix,x,xi}{ii,iii,v,vii,viii,ix,x,xi}{ii,iii,vi,vii,viii,ix,x,xiv,v,vi,vii,viii,ix,x}{ii,iv,v,vi,vii,viii,ix,xi}{ii,iv,v,vi,vii,viii,x,xi}{ii,iv,v,vi,vii,ix,x,xi}{ii,iv,v,vi,viii,ix,x,xiv,v,vii,viii,ix,x,xi}{ii,iv,vi,vii,viii,ix,x,xi}{ii,v,vi,vii,viii,ix,x,xi}{iii,iv,v,vi,vii,viii,ix,x}{iii,iv,v,vi,vii,viii,ix,xi}{iii,iv,v,vi,vii,viii,x,xi}{iii,iv,v,vi,vii,ix,x,xi}{iii,iv,v,vi,viii,ix,x,xi}{iii,iv,v,vii,viii,ix,x,xi}{iii,iv,vi,vii,viii,ix,x,xi}{iii,v,vi,vii,viii,ix,x,xi}{iv,v,vi,vii,viii,ix,x,xi}{i,ii,iii,iv,v,vi,vii,viii,ix}{i,ii,iii,iv,v,vi,vii,viii,x}{i,ii,iii,iv,v,vi,vii,viii,xi}{i,ii,iii,iv,v,vi,vii,ix,x}{i,ii,iii,iv,v,vi,vii,ix,xi}{i,ii,iii,iv,v,vi,vii,x,xi}{i,ii,iii,iv,v,vi,viii,ix,x}{i,ii,iii,iv,v,vi,viii,ix,xi}{i,ii,iii,iv,v,vi,viii,x,xi}{i,ii,iii,iv,v,vi,ix,x,xi}{i,ii,iii,iv,v,vii,viii,ix,x}{i,ii,iii,iv,v,vii,viii,ix,xi}{i,ii,iii,iv,v,vii,viii,x,xi}{i,ii,iii,iv,v,vii,ix,x,xi}{i,ii,iii,iv,v,viii,ix,x,xi}{i,ii,iii,iv,vi,vii,viii,ix,x}{i,ii,iii,iv,vi,vii,viii,ix,xi}{i,ii,iii,iv,vi,vii,viii,x,xi}{i,ii,iii,iv,vi,vii,ix,x,xi}{i,ii,iii,iv,vi,viii,ix,x,xi}{i,ii,iii,iv,vii,viii,ix,x,xi}{i,ii,iii,v,vi,vii,viii,ix,x}{i,ii,iii,v,vi,vii,viii,ix,xi}{i,ii,iii,v,vi,vii,viii,x,xi}{i,ii,iii,v,vi,vii,ix,x,xi}{i,ii,iii,v,vi,viii,ix,x,xi}{i,ii,iii,v,vii,viii,ix,x,xi}{i,ii,iii,vi,vii,viii,ix,x,xi}{i,ii,iv,v,vi,vii,viii,ix,x}{i,ii,iv,v,vi,vii,viii,ix,xi}{i,ii,iv,v,vi,vii,viii,x,xi}{i,ii,iv,v,vi,vii,ix,x,xi}{i,ii,iv,v,vi,viii,ix,x,xi}{i,ii,iv,v,vii,viii,ix,x,xi}{i,ii,iv,vi,vii,viii,ix,x,xi}{i,ii,iv,vi,vii,viii,ix,x,xi}{i,iii,iv,v,vi,vii,viii,ix,x}{i,iii,iv,v,vi,vii,viii,ix,xi}{i,iii,iv,v,vi,vii,viii,x,xi}{i,iii,iv,v,vi,vii,ix,x,xi}{i,iii,iv,v,vi,viii,ix,x,xi}{i,iii,iv,v,vii,viii,ix,x,xi}{i,iii,iv,vi,vii,viii,ix,x,xi}{i,iii,v,vi,vii,viii,ix,x,xi}{i,iv,v,vi,vii,viii,ix,x,xi}{ii,iii,iv,v,vi,vii,viii,ix,x}{ii,iii,iv,v,vi,vii,viii,ix,xi}{ii,iii,iv,v,vi,vii,viii,x,xi}{ii,iii,iv,v,vi,vii,ix,x,xi}{ii,iii,iv,v,vi,viii,ix,x,xi}{ii,iii,iv,v,vii,viii,ix,x,xi}{ii,iii,iv,vi,vii,viii,ix,x,xi}{ii,iii,v,vi,vii,viii,ix,x,xi}{ii,iv,v,vi,vii,viii,ix,x,xi}{iii,iv,v,vi,vii,viii,ix,x,xi}{i,ii,iii,iv,v,vi,vii,viii,ix,x}{i,ii,iii,iv,v,vi,vii,viii,ix,xi}{i,ii,iii,iv,v,vi,vii,viii,x,xi}{i,ii,iii,iv,v,vi,vii,ix,x,xi}{i,ii,iii,iv,v,vi,viii,ix,x,xi}{i,ii,iii,iv,v,vii,viii,ix,x,xi}{i,ii,iii,iv,vi,vii,viii,ix,x,xi}{i,ii,iii,v,vi,vii,viii,ix,x,xi}{i,ii,iv,v,vi,vii,viii,ix,x,xi}{i,iii,iv,v,vi,vii,viii,ix,x,xi}{ii,iii,iv,v,vi,vii,viii,ix,x,xi}或{i,ii,iii,iv,v,vi,viiviii,ix,x,xi}。
如果所述变体包括(i)和(iii)至(xi)中的任意一种,其可以进一步包括位于y51,n55和f56中的一个或多个位置的突变,例如位于y51,n55,f56,y51n55,y51/f56,n55/f56或y51/n55/f56。
在(i)中,所述变体可以包括n40,d43,e44,s54,s57,q62,r97,e101,e124,e131,r142,t150和r192中任意数量和组合的突变。在(i)中,所述变优选包括位于以下位置的一个或多个突变(即在以下一个或多个位置进行突变):n40,d43,e44,s54,s57,q62,e101,e131和t150。在(i)中,所述变优选包括位于以下位置的一个或多个突变(即在一个或多个以下位置的突变):n40,d43,e44,e101和e131。在(i)中,所述变体优选包括位于s54和/或s57的突变。在(i)中,所述变体更优选包括位于(a)s54和/或s57和(b)y51,n55和f56中的一个或多个,例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56的突变。如果在(xi)中缺失s54和/或s57,则它/它们不能在(i)中突变,反之亦然。在(i)中,所述变体优选包括位于t150,例如t150i的突变。或者所述变体优选包括位于(a)t150和(b)y51,n55和f56中的一个或多个,例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56的突变。在(i)中,所述变体优选包括位于q62,例如q62r或q62k的突变。或者所述变体优选包括位于(a)q62和(b)y51,n55和f56中的一个或多个,例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56的突变。所述变体可以包括位于d43,e44,q62或其任意组合的突变,例如d43,e44,q62,d43/e44,d43/q62,e44/q62或d43/e44/q62。或者所述变体优选包括位于(a)d43,e44,q62,d43/e44,d43/q62,e44/q62或d43/e44/q62和(b)y51,n55和f56中的一个或多个,例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56的突变。
在本申请的(ii)和其他地方中,符号/表示“和”,例如y51/n55是指y51和n55。在(ii)中,所述变体优选包括位于y51/n55的突变。已经提出,csgg中的所述收缩结构由三个由残基y51,n55和f56的侧链形成的堆叠的同心环组成(goyaletal,2014,nature,516,250-253)。在(ii)中这些残基的突变因而可以减少当多核苷酸移动穿过所述孔时对电流有贡献的核苷酸的数量,从而使得更容易识别观察到的电流(当多核苷酸移动通过所述孔时)与多核苷酸之间的直接关系。y56可以以下文关于本发明方法中可用变体和孔而论述的任何方式进行突变。
在(v)中,所述变体可以包括n102r,n102f,n102y或n102w。所述变体优选包括(a)n102r,n102f,n102y或n102w和(b)位于y51,n55和f56中的一个或多个位置例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56的突变。
在(xi)中,任意数量和组合的以下位置:k49,p50,y51,p52,a53,s54,n55,f56和s57可以缺失。优选地,k49,p50,y51,p52,a53,s54,n55和s57中的一个或多个可以缺失。如果y51,n55和f56中的任意一个在(xi)中缺失,则它/它们不能在(ii)中突变,反之亦然。
在(i)中,所述变体优选包括以下取代中的一个或多个:n40r,n40k,d43n,d43q,d43r,d43k,e44n,e44q,e44r,e44k,s54p,s57p,q62r,q62k,r97n,r97g,r97l,e101n,e101q,e101r,e101k,e101f,e101y,e101w,e124n,e124q,e124r,e124k,e124f,e124y,e124w,e131d,r142e,r142n,t150i,r192e和r192n,例如n40r,n40k,d43n,d43q,d43r,d43k,e44n,e44q,e44r,e44k,s54p,s57p,q62r,q62k,e101n,e101q,e101r,e101k,e101f,e101y,e101w,e131d和t150i中的一个或多个,或n40r,n40k,d43n,d43q,d43r,d43k,e44n,e44q,e44r,e44k,e101n,e101q,e101r,e101k,e101f,e101y,e101w和e131d中的一个或多个。所述变体可以包括任意数量的这些取代和这些取代的任意组合。在(i)中,所述变体优选包括s54p和/或s57p。在(i)中,所述突变体优选包括(a)s54p和/或s57p和(b)位于y51,n55和f56中的一个或多个位置的突变,例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56。位于y51,n55和f56中一个或多个位置的所述突变可以是如下所述突变中的任意一种。在(i)中,所述变体优选包括f56a/s57p或者s54p/f56a。所述变体优选包括t150i。或者所述变体优选包括位于(a)t150i和(b)y51,n55和f56中的一个或多个,例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56的突变。
在(i)中,所述变体优选包括q62r或q62k。或者所述变体优选包括(a)q62r或q62k和(b)位于y51,n55和f56中的一个或多个位置的突变,例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56。所述变体可以包括d43n,e44n,q62r或q62k或其任意的组合,例如d43n,e44n,q62r,q62k,d43n/e44n,d43n/q62r,d43n/q62k,e44n/q62r,e44n/q62k,d43n/e44n/q62r或d43n/e44n/q62k。或者所述变体优选包括(a)d43n,e44n,q62r,q62k,d43n/e44n,d43n/q62r,d43n/q62k,e44n/q62r,e44n/q62k,d43n/e44n/q62r或d43n/e44n/q62k和(b)位于y51,n55和f56中的一个或多个位置例如位于y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56的突变。
在(i)中,所述变体优选包括d43n。
在(i)中,所述变体优选包括e101r,e101s,e101f或e101n。
在(i)中,所述变体优选包括e124n,e124q,e124r,e124k,e124f,e124y,e124w或e124d,例如e124n。
在(i)中,所述变体优选包括r142e和r142n。
在(i)中,所述变体优选包括r97n,r97g或者r97l。
在(i)中,所述变体优选包括r192e和r192n。
在(ii)中,所述变体优选包括f56n/n55q,f56n/n55r,f56n/n55k,f56n/n55sf56n/n55g,f56n/n55a,f56n/n55t,f56q/n55q,f56q/n55r,f56q/n55k,f56q/n55sf56q/n55g,f56q/n55a,f56q/n55t,f56r/n55q,f56r/n55r,f56r/n55k,f56r/n55sf56r/n55g,f56r/n55a,f56r/n55t,f56s/n55q,f56s/n55r,f56s/n55k,f56s/n55s,f56s/n55gf56s/n55a,f56s/n55t,f56g/n55q,f56g/n55r,f56g/n55k,f56g/n55s,f56g/n55g,f56g/n55a,f56g/n55t,f56a/n55q,f56a/n55r,f56a/n55k,f56a/n55s,f56a/n55g,f56a/n55af56a/n55t,f56k/n55q,f56k/n55r,f56k/n55k,f56k/n55s,f56k/n55g,f56k/n55a,f56k/n55tf56n/y51l,f56n/y51v,f56n/y51a,f56n/y51n,f56n/y51q,f56n/y51s,f56n/y51g,f56q/y51lf56q/y51v,f56q/y51a,f56q/y51n,f56q/y51q,f56q/y51s,f56q/y51g,f56r/y51l,f56r/y51v,f56r/y51a,f56r/y51n,f56r/y51q,f56r/y51s,f56r/y51g,f56s/y51l,f56s/y51v,f56s/y51af56s/y51n,f56s/y51q,f56s/y51s,f56s/y51g,f56g/y51l,f56g/y51v,f56g/y51a,f56g/y51nf56g/y51q,f56g/y51s,f56g/y51g,f56a/y51l,f56a/y51v,f56a/y51a,f56a/y51n,f56a/y51qf56a/y51s,f56a/y51g,f56k/y51l,f56k/y51v,f56k/y51a,f56k/y51n,f56k/y51q,f56k/y51s/f56k/y51g,n55q/y51l,n55q/y51v,n55q/y51a,n55q/y51n,n55q/y51q,n55q/y51s/n55q/y51g,n55r/y51l,n55r/y51v,n55r/y51a,n55r/y51n,n55r/y51q,n55r/y51s/n55r/y51g,n55k/y51l,n55k/y51v,n55k/y51a,n55k/y51n,n55k/y51q,n55k/y51s/n55k/y51g,n55s/y51l,n55s/y51v,n55s/y51a,n55s/y51n,n55s/y51q,n55s/y51s/n55s/y51g,n55g/y51l,n55g/y51v,n55g/y51a,n55g/y51n,n55g/y51q,n55g/y51sn55g/y51g,n55a/y51l,n55a/y51v,n55a/t51a,n55a/t51n,n55a/y51q,n55ay51sn55a/y51g,n55t/y51l,n55t/y51v,n55t/t51a,n55t/t51n,n55t/y51q,n55t/y51s,n55t/y51g,f56n/n55q/y51l,f56n/n55q/y51v,f56n/n55q/y51a,f56n/n55q/y51n,f56n/n55q/y51q,f56n/n55q/y51s,f56n/n55q/y51g,f56n/n55r/y51l,f56n/n55r/y51v,f56n/n55r/y51a,f56n/n55r/y51n,f56n/n55r/y51q,f56n/n55r/y51s,f56n/n55r/y51g,f56n/n55k/y51lf56n/n55k/y51v,f56n/n55k/y51a,f56n/n55k/y51n,f56n/n55k/y51q,f56n/n55k/y51s,f56n/n55k/y51g,f56n/n55s/y51l,f56n/n55s/y51v,f56n/n55s/y51a,f56n/n55s/y51n,f56n/n55s/y51q,f56n/n55s/y51s,f56n/n55s/y51g,f56n/n55g/y51l,f56n/n55g/y51v,f56n/n55g/y51a,f56n/n55g/y51n,f56n/n55g/y51q,f56n/n55g/y51s,f56n/n55g/y51g,f56n/n55a/y51l,f56n/n55a/y51v,f56n/n55a/y51a,f56n/n55a/y51n,f56n/n55a/y51q,f56n/n55a/y51s,f56n/n55a/y51g,f56n/n55t/y51l,f56n/n55t/y51v,f56n/n55t/y51a,f56n/n55t/y51n,f56n/n55t/y51q,f56n/n55t/y51s,f56n/n55t/y51g,f56q/n55q/y51l,f56q/n55q/y51v,f56q/n55q/y51a,f56q/n55q/y51n,f56q/n55q/y51q,f56q/n55q/y51s,f56q/n55q/y51g,f56q/n55r/y51l,f56q/n55r/y51v,f56q/n55r/y51a,f56q/n55r/y51n,f56q/n55r/y51q,f56q/n55r/y51s,f56q/n55r/y51g,f56q/n55k/y51l,f56q/n55k/y51v,f56q/n55k/y51a,f56q/n55k/t51n,f56q/n55k/y51q,f56q/n55k/y51s,f56q/n55k/y51g,f56q/n55s/y51l,f56q/n55s/y51v,f56q/n55s/y51a,f56q/n55s/y51n,f56q/n55s/y51q,f56q/n55s/y51s,f56q/n55s/y51g,f56q/n55g/y51l,f56q/n55g/y51v,f56q/n55g/y51a,f56q/n55g/y51n,f56q/n55g/y51q,f56q/n55g/y51s,f56q/n55g/y51g,f56q/n55a/y51l,f56q/n55a/y51v,f56q/n55a/y51a,f56q/n55a/y51n,f56q/n55a/y51q,f56q/n55a/y51s,f56q/n55a/y51g,f56q/n55t/y51l,f56q/n55t/y51v,f56q/n55t/y51a,f56q/n55t/y51n,f56q/n55t/y51q,f56q/n55t/y51s,f56q/n55t/y51g,f56r/n55q/y51l,f56r/n55q/y51v,f56r/n55q/y51a,f56r/n55q/y51n,f56r/n55q/y51q,f56r/n55q/y51s,f56r/n55q/y51g,f56r/n55r/y51l,f56r/n55r/y51v,f56r/n55r/y51a,f56r/n55r/y51n,f56r/n55r/y51q,f56r/n55r/y51s,f56r/n55r/y51g,f56r/n55k/y51l,f56r/n55k/y51v,f56r/n55k/y51a,f56r/n55k/y51n,f56r/n55k/y51q,f56r/n55k/y51s,f56r/n55k/y51g,f56r/n55s/y51l,f56r/n55s/y51v,f56r/n55s/y51a,f56r/n55s/y51n,f56r/n55s/y51q,f56r/n55s/y51s,f56r/n55s/y51g,f56r/n55g/y51l,f56r/n55g/y51v,f56r/n55g/y51a,f56r/n55g/y51n,f56r/n55g/y51q,f56r/n55g/y51s,f56r/n55g/y51g;f56r/n55a/y51l,f56r/n55a/y51v,f56r/n55a/y51a,f56r/n55a/y51n,f56r/n55a/y51q,f56r/n55a/y51s,f56r/n55a/y51gf56r/n55t/y51l,f56r/n55t/y51v,f56r/n55t/y51a,f56r/n55t/y51n,f56r/n55t/y51qf56r/n55t/y51s,f56r/n55t/y51g,f56s/n55q/y51l,f56s/n55q/y51v,f56s/n55q/y51af56s/n55q/y51n,f56s/n55q/y51q,f56s/n55q/y51s,f56s/n55q/y51g,f56s/n55r/y51lf56s/n55r/y51v,f56s/n55r/y51a,f56s/n55r/y51n,f56s/n55r/y51q,f56s/n55r/y51sf56s/n55r/y51g,f56s/n55k/y51l,f56s/n55k/y51v,f56s/n55k/y51a,f56s/n55k/y51nf56s/n55k/y51q,f56s/n55k/y51s,f56s/n55k/y51g,f56s/n55s/y51l,f56s/n55s/y51vf56s/n55s/y51a,f56s/n55s/y51n,f56s/n55s/y51q,f56s/n55s/y51s,f56s/n55s/y51gf56s/n55g/y51l,f56s/n55g/y51v,f56s/n55g/y51a,f56s/n55g/y51n,f56s/n55g/y51qf56s/n55g/y51s,f56s/n55g/y51g,f56s/n55a/y51l,f56s/n55a/y51v,f56s/n55a/y51af56s/n55a/y51n,f56s/n55a/y51q,f56s/n55a/y51s,f56s/n55a/y51g,f56s/n55t/y51lf56s/n55t/y51v,f56s/n55t/y51a,f56s/n55t/y51n,f56s/n55t/y51q,f56s/n55t/y51sf56s/n55t/y51g,f56g/n55q/y51l,f56g/n55q/y51v,f56g/n55q/y51a,f56g/n55q/y51nf56g/n55q/y51q,f56g/n55q/y51s,f56g/n55q/y51g;f56g/n55r/y51l,f56g/n55r/y51vf56g/n55r/y51a,f56g/n55r/y51n,f56g/n55r/y51q,f56g/n55r/y51s,f56g/n55r/y51gf56g/n55k/y51l,f56g/n55k/y51v,f56g/n55k/y51a,f56g/n55k/y51n,f56g/n55k/y51qf56g/n55k/y51s,f56g/n55k/y51g,f56g/n55s/y51l,f56g/n55s/y51v,f56g/n55s/y51af56g/n55s/y51n,f56g/n55s/y51q,f56g/n55s/y51s,f56g/n55s/y51g,f56g/n55g/y51lf56g/n55g/y51v,f56g/n55g/y51a,f56g/n55g/y51n,f56g/n55g/y51q,f56g/n55g/y51sf56g/n55g/y51g,f56g/n55a/y51l,f56g/n55a/y51v,f56g/n55a/y51a,f56g/n55a/y51nf56g/n55a/y51q,f56g/n55a/y51s,f56g/n55a/y51g,f56g/n55t/y51l,f56g/n55t/y51v,f56g/n55t/y51a,f56g/n55t/y51n,f56g/n55t/y51q,f56g/n55t/y51s,f56g/n55t/y51g,f56a/n55q/y51l,f56a/n55q/y51v,f56a/n55q/y51a,f56a/n55q/y51n,f56a/n55q/y51q,f56a/n55q/y51s,f56a/n55q/y51g,f56a/n55r/y51l,f56a/n55r/y51v,f56a/n55r/y51a,f56a/n55r/y51n,f56a/n55r/y51q,f56a/n55r/y51s,f56a/n55r/y51g,f56a/n55k/y51l,f56a/n55k/y51v,f56a/n55k/y51a,f56a/n55k/y51n,f56a/n55k/y51q,f56a/n55k/y51s,f56a/n55k/y51g,f56a/n55s/y51l,f56a/n55s/y51v,f56a/n55s/y51a,f56a/n55s/y51n,f56a/n55s/y51q,f56a/n55s/y51s,f56a/n55s/y51g,f56a/n55g/y51l,f56a/n55g/y51v,f56a/n55g/y51a,f56a/n55g/y51n,f56a/n55g/y51q,f56a/n55g/y51s,f56a/n55g/y51g,f56a/n55a/y51l,f56a/n55a/y51v,f56a/n55a/y51a,f56a/n55a/y51n,f56a/n55a/y51q,f56a/n55a/y51s,f56a/n55a/y51g,f56a/n55t/y51l,f56a/n55t/y51v,f56a/n55t/y51a,f56a/n55t/y51n,f56a/n55t/y51q,f56a/n55t/y51s,f56a/n55t/y51g,f56k/n55q/y51l,f56k/n55q/y51v,f56k/n55q/y51a,f56k/n55q/y51n,f56k/n55q/y51q,f56k/n55q/y51s,f56k/n55q/y51g,f56k/n55r/y51l,f56k/n55r/y51v,f56k/n55r/y51a,f56k/n55r/y51n,f56k/n55r/y51q,f56k/n55r/y51s,f56k/n55r/y51g,f56k/n55k/y51l,f56k/n55k/y51v,f56k/n55k/y51a,f56k/n55k/y51n,f56k/n55k/y51q,f56k/n55k/y51s,f56k/n55k/y51g,f56k/n55s/y51l,f56k/n55s/y51v,f56k/n55s/y51a,f56k/n55s/y51n,f56k/n55s/y51q,f56k/n55s/y51s,f56k/n55s/y51g,f56k/n55g/y51l,f56k/n55g/y51v,f56k/n55g/y51a,f56k/n55g/y51n,f56k/n55g/y51q,f56k/n55g/y51s,f56k/n55g/y51g,f56k/n55a/y51l,f56k/n55a/y51v,f56k/n55a/y51a,f56k/n55a/y51n,f56k/n55a/y51q,f56k/n55a/y51s,f56k/n55a/y51g,f56k/n55t/y51l,f56k/n55t/y51v,f56k/n55t/y51a,f56k/n55t/y51n,f56k/n55t/y51q,f56k/n55t/y51s,f56k/n55t/y51g,f56e/n55r,f56e/n55k,f56d/n55r,f56d/n55k,f56r/n55e,f56r/n55d,f56k/n55e或f56k/n55d。
在(ii)中,所述变体优选包括y51r/f56q,y51n/f56n,y51m/f56q,y51l/f56q,y51i/f56q,y51v/f56q,y51a/f56q,y51p/f56q,y51g/f56q,y51c/f56q,y51q/f56q,y51n/f56q,y51s/f56q,y51e/f56q,y51d/f56q,y51k/f56q或y51h/f56q。
在(ii)中,所述变体优选包括y51t/f56q,y51q/f56q或者y51a/f56q。
在(ii)中,所述变体优选包括y51t/f56f,y51t/f56m,y51t/f56l,y51t/f56i,y51t/f56v,y51t/f56a,y51t/f56p,y51t/f56g,y51t/f56c,y51t/f56q,y51t/f56n,y51t/f56t,y51t/f56s,y51t/f56e,y51t/f56d,y51t/f56k,y51t/f56hory51t/f56r。
在(ii)中,所述变体优选包括y51t/n55q,y51t/n55s或者y51t/n55a。
在(ii)中,所述变体优选包括y51a/f56f,y51a/f56l,y51a/f56i,y51a/f56v,y51a/f56a,y51a/f56p,y51a/f56g,y51a/f56c,y51a/f56q,y51a/f56n,y51a/f56t,y51a/f56s,y51a/f56e,y51a/f56d,y51a/f56k,y51a/f56h或y51a/f56r。
在(ii)中,所述变体优选包括y51c/f56a,y51e/f56a,y51d/f56a,y51k/f56a,y51h/f56a,y51q/f56a,y51n/f56a,y51s/f56a,y51p/f56a或y51v/f56a。
在(xi)中,所述变体优选包括y51/p52,y51/p52/a53,p50至p52,p50至a53,k49至y51,k49至a53的缺失和采用单脯氨酸(p),k49至s54进行的替换和采用单p,y51至a53,y51至s54,n55/f56,n55至s57,n55/f56进行的替换,和采用单p,n55/f56进行的替换和采用单甘氨酸(g),n55/f56进行的替换和采用单丙氨酸(a),n55/f56进行的替换和采用p和y51n,n55/f56进行的替换和采用单p和y51q,n55/f56进行的替换和采用单p和y51s,n55/f56进行的替换和采用单甘氨酸(g)和y51n,n55/f56进行的替换和采用单g和y51q,n55/f56进行的替换和采用单g和y51s,n55/f56进行的替换和采用单丙氨酸(a)和y51n,n55/f56进行的替换和采用单丙氨酸(a),y51q或n55/f56进行的替换和采用单丙氨酸(a)和y51s进行的替换。
所述变体更优选包括d195n/e203n,d195q/e203n,d195n/e203q,d195q/e203q,e201n/e203n,e201q/e203n,e201n/e203q,e201q/e203q,e185n/e203q,e185q/e203q,e185n/e203n,e185q/e203n,d195n/e201n/e203n,d195q/e201n/e203n,d195n/e201q/e203n,d195n/e201n/e203q,d195q/e201q/e203n,d195q/e201n/e203q,d195n/e201q/e203q,d195q/e201q/e203q,d149n/e201n,d149q/e201n,d149n/e201q,d149q/e201q,d149n/e201n/d195n,d149q/e201n/d195n,d149n/e201q/d195n,d149n/e201n/d195q,d149q/e201q/d195n,d149q/e201n/d195q,d149n/e201q/d195q,d149q/e201q/d195q,d149n/e203n,d149q/e203n,d149n/e203q,d149q/e203q,d149n/e185n/e201n,d149q/e185n/e201n,d149n/e185q/e201n,d149n/e185n/e201q,d149q/e185q/e201n,d149q/e185n/e201q,d149n/e185q/e201q,d149q/e185q/e201qd149n/e185n/e203n,d149q/e185n/e203n,d149n/e185q/e203n,d149n/e185n/e203q,d149q/e185q/e203n,d149q/e185n/e203q,d149n/e185q/e203q,d149q/e185q/e203qd149n/e185n/e201n/e203n,d149q/e185n/e201n/e203n,d149n/e185q/e201n/e203n,d149n/e185n/e201q/e203n,d149n/e185n/e201n/e203q,d149q/e185q/e201n/e203n,d149q/e185n/e201q/e203n,d149q/e185n/e201n/e203q,d149n/e185q/e201q/e203n,d149n/e185q/e201n/e203q,d149n/e185n/e201q/e203q,d149q/e185q/e201q/e203q,d149q/e185q/e201n/e203q,d149q/e185n/e201q/e203q,d149n/e185q/e201q/e203q,d149q/e185q/e201q/e203n,d149n/e185n/d195n/e201n/e203n,d149q/e185n/d195n/e201n/e203n,d149n/e185q/d195n/e201n/e203n,d149n/e185n/d195q/e201n/e203n,d149n/e185n/d195n/e201q/e203n,d149n/e185n/d195n/e201n/e203q,d149q/e185q/d195n/e201n/e203n,d149q/e185n/d195q/e201n/e203n,d149q/e185n/d195n/e201q/e203n,d149q/e185n/d195n/e201n/e203q,d149n/e185q/d195q/e201n/e203n,d149n/e185q/d195n/e201q/e203n,d149n/e185q/d195n/e201n/e203q,d149n/e185n/d195q/e201q/e203n,d149n/e185n/d195q/e201n/e203q,d149n/e185n/d195n/e201q/e203q,d149q/e185q/d195q/e201n/e203n,d149q/e185q/d195n/e201q/e203n,d149q/e185q/d195n/e201n/e203q,d149q/e185n/d195q/e201q/e203n,d149q/e185n/d195q/e201n/e203q,d149q/e185n/d195n/e201q/e203q,d149n/e185q/d195q/e201q/e203n,d149n/e185q/d195q/e201n/e203q,d149n/e185q/d195n/e201q/e203q,d149n/e185n/d195q/e201q/e203q,d149q/e185q/d195q/e201q/e203n,d149q/e185q/d195q/e201n/e203q,d149q/e185q/d195n/e201q/e203q,d149q/e185n/d195q/e201q/e203q,d149n/e185q/d195q/e201q/e203q,d149q/e185q/d195q/e201q/e203q,d149n/e185r/e201n/e203n,d149q/e185r/e201n/e203n,d149n/e185r/e201q/e203n,d149n/e185r/e201n/e203q,d149q/e185r/e201q/e203n,d149q/e185r/e201n/e203q,d149n/e185r/e201q/e203q,d149q/e185r/e201q/e203q,d149r/e185n/e201n/e203n,d149r/e185q/e201n/e203n,d149r/e185n/e201q/e203n,d149r/e185n/e201n/e203q,d149r/e185q/e201q/e203n,d149r/e185q/e201n/e203q,d149r/e185n/e201q/e203q,d149r/e185q/e201q/e203q,d149r/e185n/d195n/e201n/e203n,d149r/e185q/d195n/e201n/e203n,d149r/e185n/d195q/e201n/e203n,d149r/e185n/d195n/e201q/e203n,d149r/e185q/d195n/e201n/e203q,d149r/e185q/d195q/e201n/e203n,d149r/e185q/d195n/e201q/e203n,d149r/e185q/d195n/e201n/e203q,d149r/e185n/d195q/e201q/e203n,d149r/e185n/d195q/e201n/e203q,d149r/e185n/d195n/e201q/e203q,d149r/e185q/d195q/e201q/e203n,d149r/e185q/d195q/e201n/e203q,d149r/e185q/d195n/e201q/e203q,d149r/e185n/d195q/e201q/e203q,d149r/e185q/d195q/e201q/e203q,d149n/e185r/d195n/e201n/e203n,d149q/e185r/d195n/e201n/e203n,d149n/e185r/d195q/e201n/e203n,d149n/e185r/d195n/e201q/e203n,d149n/e185r/d195n/e201n/e203q,d149q/e185r/d195q/e201n/e203n,d149q/e185r/d195n/e201q/e203n,d149q/e185r/d195n/e201n/e203q,d149n/e185r/d195q/e201q/e203n,d149n/e185r/d195q/e201n/e203q,d149n/e185r/d195n/e201q/e203q,d149q/e185r/d195q/e201q/e203n,d149q/e185r/d195q/e201n/e203q,d149q/e185r/d195n/e201q/e203q,d149n/e185r/d195q/e201q/e203q,d149q/e185r/d195q/e201q/e203q,d149n/e185r/d195n/e201r/e203n,d149q/e185r/d195n/e201r/e203n,d149n/e185r/d195q/e201r/e203n,d149n/e185r/d195n/e201r/e203q,d149q/e185r/d195q/e201r/e203n,d149q/e185r/d195n/e201r/e203q,d149n/e185r/d195q/e201r/e203q,d149q/e185r/d195q/e201r/e203q,e131d/k49r,e101n/n102f,e101n/n102y,e101n/n102w,e101f/n102f,e101f/n102y,e101f/n102w,e101y/n102f,e101y/n102y,e101y/n102w,e101w/n102f,e101w/n102y,e101w/n102w,e101n/n102r,e101f/n102r,e101y/n102r或e101w/n102f。
本发明形成所述孔的优选变体——其中当多核苷酸移动通过孔时较少的核苷酸有助于电流——包括y51a/f56a,y51a/f56n,y51i/f56a,y51l/f56a,y51t/f56a,y51i/f56n,y51l/f56n或y51t/f56n或更优选y51i/f56a,y51l/f56a或y51t/f56a。如上所述,这使得更容易鉴定观察到的电流(当多核苷酸移动通过所述孔时)与多核苷酸之间的直接关系。
形成显示增加的范围的孔的优选变体包括以下位置的突变:
y51,f56,d149,e185,e201和e203;
n55和f56;
y51和f56;
y51,n55和f56;或
f56和n102。
形成显示增加的范围的孔的优选变体包括:
y51n,f56a,d149n,e185r,e201n和e203n;
n55s和f56q;
y51a和f56a;
y51a和f56n;
y51i和f56a;
y51l和f56a;
y51t和f56a;
y51i和f56n;
y51l和f56n;
y51t和f56n;
y51t和f56q;
y51a,n55s和f56a;
y51a,n55s和f56n;
y51t,n55s和f56q;或
f56q和n102r。
形成所述孔的优选变体——其中当多核苷酸移动通过所述孔时较少的核苷酸贡献于电流——包括在下列位置的突变:
n55和f56,例如n55x和f56q,其中x是任意氨基酸;或
y51和f56,例如y51x和f56q,其中x是任意氨基酸。
形成显示增加的通量的孔的优选变体包括以下位置的突变:
d149,e185和e203;
d149,e185,e201和e203;或者
d149,e185,d195,e201和e203。
形成显示增加的通量的孔的优选变体包括:
d149n,e185n和e203n;
d149n,e185n,e201n和e203n;
d149n,e185r,d195n,e201n和e203n;或
d149n,e185r,d195n,e201r和e203n。
形成所述孔的优选变体——其中多核苷酸的捕获增加——包括以下突变:
d43n/y51t/f56q;
e44n/y51t/f56q;
d43n/e44n/y51t/f56q;
y51t/f56q/q62r;
d43n/y51t/f56q/q62r;
e44n/y51t/f56q/q62r;或
d43n/e44n/y51t/f56q/q62r。
优选的变体包括以下突变:
d149r/e185r/e201r/e203r或y51t/f56q/d149r/e185r/e201r/e203r;
d149n/e185n/e201n/e203n或y51t/f56q/d149n/e185n/e201n/e203n;
e201r/e203r或y51t/f56q/e201r/e203r
e201n/e203r或y51t/f56q/e201n/e203r;
e203r或y51t/f56q/e203r;
e203n或y51t/f56q/e203n;
e201r或y51tf56q/e201r;
e201n或y51t/f56q/e201n;
e185r或y51t/f56q/e185r;
e185n或y51t/f56q/e185n;
d149r或y51t/f56q/d149r;
d149n或y51t/f56q/d149n;
r142e或y51t/f56q/r142e;
r142n或y51t/f56q/r142n;
r192e或y51t/f56q/r192e;或者
r192n或y51t/f56q/r192n。
优选的变体包括以下突变:
y51a/f56q/e101n/n102r;
y51a/f56q/r97n/n102g;
y51a/f56q/r97n/n102r;
y51a/f56q/r97n;
y51a/f56q/r97g;
y51a/f56q/r97l;
y51a/f56q/n102r;
y51a/f56q/n102f;
y51a/f56q/n102g;
y51a/f56q/e101r;
y51a/f56q/e101f;
y51a/f56q/e101n;或
y51a/f56q/e101g。
本发明还提供了包含seqidno:390所示序列的变体的突变型csgg单体,其中所述变体包括在t150位置的突变。形成表现出增加的插入的孔的优选变体包括t150i。在t150位置的突变,例如t150i,可以与上述任意突变或突变的组合进行结合。
本发明还提供包含seqidno:390所示序列的变体的突变型csgg单体,所述变体包含实施例中公开的变体中存在的突变的组合。
引入或取代天然存在氨基酸的方法是本领域公知的。例如,在编码突变单体的多核苷酸的相关位置,通过用精氨酸的密码子(cgt)替换甲硫氨酸的密码子(atg),可使甲硫氨酸(m)被精氨酸(r)取代。所述多核苷酸可以随后如下所述进行表达。
引入或取代非天然存在氨基酸的方法也是本领域公知的。例如,非天然存在的氨基酸可以通过在用于表达突变单体的ivtt系统中包含合成氨酰基-trna来引入。或者,它们可以通过在那些特定氨基酸的合成(即非天然存在的)类似物的存在下通过在对特定氨基酸为营养缺陷型的大肠杆菌中表达所述突变单体来引入。如果使用部分肽合成来产生突变单体,则它们也可以通过裸连接来制备。
本发明的其他单体
在另一个实施方式中,本发明提供一种突变csgg单体,其包括seqidno:390所示的序列的变体,其中所述变体包括在位置y51,n55和f56中的一个或多个的突变。所述变体可以包含在位置y51;n55;f56;y51/n55;y51/f56;n55/f56;或y51/n55/f56的突变。所述变体优选包括位置y51,n55或f56的突变。所述变体可以包含上述讨论的在y51,n55和f56中一个或多个位置的任意特异性突变和其任意组合。y51,n55和f56的一个或多个可以用任意氨基酸取代。y51可以被f,m,l,i,v,a,p,g,c,q,n,t,s,e,d,k,h或r取代,例如a,s,t,n或q。n55可以被f,m,l,i,v,a,p,g,c,q,,t,s,e,d,k,h或r取代,例如a,s,t,或q。f56可以被m,l,i,v,a,p,g,c,q,n,t,s,e,d,k,h或r取代,例如a,s,t,n或q。
所述变体可以进一步包括以下中的一个或多个:(i)在以下位置的一个或多个突变(即在一个或多个以下位置的突变):(i)n40,d43,e44,s54,s57,q62,r97,e101,e124,e131,r142,t150和r192,(iii)q42r或q42k;(iv)k49r;(v)n102r,n102f,n102y或n102w;(vi)d149n,d149q或d149r;(vii)e185n,e185q或e185r;(viii)d195n,d195q或d195r;(ix)e201n,e201q或e201r;(x)e203n,e203q或e203r;和(xi)缺失以下位置中的一个或多个:f48,k49,p50,y51,p52,a53,s54,n55,f56和s57。所述变体可以包含上述讨论的(i)和(iii)至(xi)的任意组合。所述变体可以包含上述讨论的对(i)和(iii)至(xi)的任意实施方式。
变体
除上述具体突变之外,变体可以包括其他突变。在seqidno:390的氨基酸序列的整个长度上,基于氨基酸同一性,变体优选与所述序列至少50%同源。更优选,基于氨基酸同一性,所述变体与seqidno:390的氨基酸序列在整个长度上可以至少55%,至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%以及更优选至少95%,97%或99%同源。在100个以上,例如125,150,175或200以上的连续氨基酸延伸链上,可以具有至少80%,例如至少85%,90%或95%的氨基酸同一性(“严格硬同源性”(hardhomology))。
现有技术的标准方法可以用于测定同源性。例如uwgcg软件包提供了可用于计算同源性的bestfit程序,例如使用其默认设置(devereuxetal.(1984)nucleicacidsresearch12,p387-395)。pileup和blast算法可用于计算同源性或序列排序(例如识别等效残基或相应序列(通常以其默认设置)),例如如altschuls.f.(1993)jmolevol36290-300;altschul,s.fetal(1990)jmolbiol215403-10中描述的。用于进行blast分析的软件可通过国家生物技术信息中心公开获得(http://www.ncbi.nlm.nih.gov)。
seqidno:390是来自大肠杆菌str.k-12substr.mc4100的野生型csgg单体。序列seqidno:390的变体可以包括存在于另一个csgg同源物中的任意取代。优选的csgg同源物在seqid:no:391至395和414至429中示出。所述变体可以包括与seqidno:390相比存在于seqid:no:391至395和414至429中的一个或多个取代的组合。
除上述讨论的氨基酸序列之外,还可以对seqidno:390的氨基酸序列进行氨基酸取代,例如至多1,2,3,4,5,10,20或30个取代。保守取代是将氨基酸用其他具有相似化学结构、相似化学性质或相似侧链体积的氨基酸替换。引入的所述氨基酸可与其替换的氨基酸相比,具有相似的极性,亲水性,疏水性,碱性,酸性,中性或电荷。或者,所述保守取代可以引入另一芳族的或脂肪族的氨基酸代替之前存在的芳族或脂肪族氨基酸。保守的氨基酸改变是本领域公知的,并且可以根据下表1中定义的20个主要氨基酸的性质进行选择。当氨基酸具有相似的极性时,这也可以参考表2中氨基酸侧链的亲水性水平来确定。
表1-氨基酸的化学性质
表2-亲水性水平
seqidno:390的氨基酸序列的一个或多个氨基酸残基可另外从上述多肽中缺失。多达1,2,3,4,5,10,20或30个以上残基可以缺失。
变体可以包含seqidno:390的片段。所述片段保留形成孔的活性。所述片段的长度可以为至少50,至少100,至少150,至少200或至少250个氨基酸。所述片段可以用于产生所述孔。片段优选包括seqidno:390的跨膜结构域,即k135-q153和s183-s208。
一个或多个氨基酸可以替换地或额外地加入到上述多肽中。可以在seqidno:390或其多肽变体或其片段的氨基酸序列的氨基末端或羧基末端提供延伸。所述延伸可以是相当短的,例如1至10个氨基酸长度。替代地,所述延伸可以更长,例如达到50或100个氨基酸。载体蛋白可以根据本发明与氨基酸序列融合。下面更详细地讨论其他融合蛋白。
如上所述,变体是具有由seqidno:390的序列变化而来并且保留其形成孔能力的氨基酸序列的多肽。变体通常包含seqidno:390中负责孔形成的区域。包含β-桶结构的csgg的孔形成能力由每个亚基中的β-片体(β-sheets)提供。seqidno:390的变体通常包括seqidno:390中形成β-变体的区域,即k135-q153和s183-s208。只要所得到的变体保持其形成孔的能力,就可以对seqidno:390中形成β-片体的区域进行一个或多个修饰。seqidno:390的变体优选包括在其α-螺旋和/或环结构区域内的一个或多个修饰,例如取代,增加或缺失。
衍生自csgg的单体可以进行修饰,以帮助对它们的鉴定或纯化,例如通过添加链霉亲和素标签或通过添加用以促进其从细胞中分泌的信号序列,其中所述单体不天然含有这种序列。下面更详细地讨论其他合适的标签。所述单体可以用显示标签标记。所述显示标签可以是允许所述单体被检测的任意合适的标签。合适的标签如下所述。
衍生自csgg的单体也可以使用d-氨基酸制备。例如,衍生自csgg的单体可以包括l-氨基酸和d-氨基酸的混合物。这在制备上述蛋白质或肽的技术领域中是常规的。
衍生自csgg的单体包含一个或多个特异性修饰以便于核苷酸的区分。衍生自csgg的单体也可以包含其他非特异性修饰,只要其不会干扰孔的形成。许多非特异性侧链修饰是本领域已知的,并且可以针对衍生自csgg的单体的侧链进行。所述修饰包括,例如通过下述进行的氨基酸还原性烷基化:与醛反应然后用nabh4还原,用亚氨酰乙酸甲酯(methylacetimidate)进行脒基化或用乙酸酐进行酰化来氨基酸。
所述衍生自csgg的单体可以采用本领域已知的方法产生。所述衍生自csgg的单体可以合成制备或通过重组方法制备。例如,所述单体可以通过体外翻译和转录(ivtt)来合成。生产所述孔和单体的合适方法在国际申请号pct/gb09/001690(公开为wo2010/004273),pct/gb09/001679(公开为wo2010/004265)或pct/gb10/000133(公开为wo2010/086603)中有所讨论。将孔插入膜的方法如下所述。
在一些实施方式中,所述突变单体经过化学修饰。所述突变单体可以通过任意方式和在任意位点进行化学修饰。所述突变单体优选通过将分子连接到一个或多个半胱氨酸(半胱氨酸连接),分子连接到一个或多个赖氨酸,将分子连接到一个或多个非天然氨基酸,抗原表位的酶修饰或末端修饰,来进行化学修饰。实施上述修饰的合适方法是本领域公知的。所述突变单体可以通过任意分子的连接进行化学修饰。例如,所述突变单体可以通过连接染料或荧光团进行化学修饰。
在一些实施方式中,所述突变单体用分子适配器(adaptor)进行化学修饰,分子适配器促进包含所述单体的孔和目标核苷酸或目标多核苷酸序列之间的相互作用。所述适配器的存在改善所述孔和所述核苷酸或多核苷酸的主-客体化学,因而提高了由所述突变单体形成的孔的测序能力。主-客体化学的原理是本领域公知的。所述适配器对所述孔的物理或化学性质有影响,能改善其与核苷酸或多核苷酸序列的相互作用。所述适配器可以改变孔的桶状结构或通道结构的电荷,或特异性地与所述核苷酸或多核苷酸序列相互作用或结合从而促进其与所述孔的相互作用。
所述分子适配器优选为环状分子,环糊精,能够进行杂合的物质,dna结合剂或螯合剂,肽或肽类似物,合成聚合物,芳族平面分子,带正电荷的小分子或能够进行氢键合的小分子。
所述适配器可以为环状的。环状适配器优选具有与所述孔相同的对称性。由于csgg通常围绕中心轴具有8个或9个亚基,因此所述适配器优选具有8重或9重的对称性。这将在下面更详细地讨论。
所述适配器通常通过主-客体化学与所述核苷酸或多核苷酸序列相互作用。所述适配器通常能够与所述核苷酸或多核苷酸序列相互作用。所述适配器包括一个或多个能够与所述核苷酸或多核苷酸序列相互作用的化学基团。所述一个或多个化学基团优选通过非共价相互作用与所述核苷酸或多核苷酸序列相互作用,例如,疏水性相互作用,氢键,范得华力,π-阳离子相互作用和/或静电力。能够与所述核苷酸或多核苷酸序列相互作用的一个或多个化学基团优选为带正电荷的。能够与所述核苷酸或多核苷酸序列相互作用的一个或多个化学基团更优选包括氨基基团。所述氨基基团可以连接在伯、仲或叔碳原子上。所述适配器甚至更优选包括氨基基团的环,例如6,7或8个氨基基团的环。所述适配器最优选包括8个氨基基团的环。质子化的氨基基团的环可以与在所述核苷酸或多核苷酸序列中带负电荷的磷酸基团相互作用。
通过所述适配器与包括所述突变单体的孔之间的主-客体化学可以促进所述适配器在所述孔中的正确定位。所述适配器优选包括一个或多个能够与一个或多个所述孔中的氨基酸相互作用的化学基团。所述适配器更优选包括一个或多个能够通过非共价相互作用与所述孔中一个或多个氨基酸相互作用的化学基团,例如,疏水性相互作用,氢键,范得华力,π-阳离子相互作用和/或静电力。所述能够与所述孔中一个或多个氨基酸相互作用的化学基团通常为羟基或胺。所述羟基基团可以连接在伯、仲或叔碳原子上。所述羟基基团可以与所述孔中未带电荷的氨基酸形成氢键。可以使用任意促进所述孔和所述核苷酸或多核苷酸序列的适配器。
合适的适配器包括,但不限于,环糊精、环肽和葫芦脲。所述适配器优选为环糊精或其衍生物。所述环糊精或其衍生物可以为在eliseev,a.v.,和schneider,h-j.(1994)j.am.chem.soc.116,6081-6088中公开的任意一种。所述适配器更优选为七-6-氨基-β-环糊精(am7-βcd),6-单脱氧-6-单氨基-β-环糊精(am1-βcd)或七-(6-脱氧-6-胍基)-环糊精(gu7-βcd)。所述gu7-βcd中的胍基基团比am7-βcd中的伯胺具有更高的pka值,因此其更容易带正电荷。所述gu7-βcd适配器可用于增加所述孔中核苷酸的停留时间,以提高测量的残留电流的准确度,以及增加高温或低数据采集率下的基础检出率。
如果如下更详细所述的使用3-(2-吡啶基二硫代)丙酸琥珀酰亚胺酯(spdp)交联剂,则适配器优选为七(6-脱氧-6-氨基)-6-n-单(2-吡啶基)二硫丙酰基-β-环糊精(am6ampdp1-βcd)。
更合适的适配器包括包含9个糖单元的γ-环糊精(并因此具有九重对称)。γ-环糊精可以包含连接体分子,或者可以被修饰成包含上述β-环糊精实施例中使用的所有或多个修饰的糖单元。
所述分子适配器优选为共价连接到所述突变单体上。所述适配器可以通过任意本领域已知的方法共价连接到所述孔上。所述适配器通常通过化学连接进行连接。如果通过半胱氨酸连接基团连接所述分子适配器,则优选将一个或多个半胱氨酸通过取代引入所述突变体,例如引入所述桶结构中。所述突变单体可以通过将分子适配器连接到所述突变单体中的一个或多个半胱氨酸上进行化学修饰。所述一个或多个半胱氨酸可以是天然存在的,即位于seqidno:390中1和/或215位置。替代地,所述突变单体可以通过将分子连接到引入到其他位置的一个或多个半胱氨酸上进行化学修饰。位于位置215的半胱氨酸可以被除去,例如通过取代除去,以保证所述分子适配器不会连接到除位于位置1的半胱氨酸以外的位置或连接到在另一位置引入的半胱氨酸上。
半胱氨酸残基的反应活性可以通过修饰其邻近残基进行增强。例如,侧链精氨酸,组氨酸或赖氨酸残基的碱性基团会将半胱氨酸硫醇基团的pka改变为更具反应性的s-基团的pka。所述半胱氨酸残基的反应活性可以被硫醇保护基如dtnb保护。在连接连接体之前,这些保护基团可以与所述突变单体的一个或多个半胱氨酸残基反应。
所述分子可以直接连接到所述突变单体上。所述分子优选通过连接体,例如化学交联剂或肽连接体,连接到所述突变单体上。
合适的化学交联剂在本领域是公知的。优选的交联剂包括2,5-二氧代吡咯烷-1-基3-(吡啶-2-基硫烷基)丙酸酯,2,5-二氧代吡咯烷-1-基4-(吡啶-2-基硫烷基)丁酸乙酯和2,5-二氧代吡咯烷-1-基8-(吡啶-2-基二硫烷基)辛酸酯。最优选的交联剂为3-(2-吡啶基二硫代)丙酸琥珀酰亚胺酯(spdp)通常地,在所述分子/交联剂复合物共价连接到所述突变单体之前,所述分子共价连接到所述双功能交联剂上,但也可能是在所述双功能交联剂/单体复合物连接到所述分子之前,将所述双功能交联剂共价连接到所述单体上。
所述连接体优选对二硫苏糖醇(dtt)具有抗性。合适的连接体包括,但不限于,碘乙酰胺系和马来酰亚胺系连接体。
在另一实施方式中,所述单体可以连接到多核苷酸结合蛋白上。这形成可用于本发明测序方法中的模块化测序系统。以下内容讨论多核苷酸结合蛋白。
所述多核苷酸结合蛋白优选共价连接到所述突变单体上。所述蛋白可以采用任意本领域已知方法共价连接到所述单体上。所述单体和蛋白可以进行化学融合或遗传融合。如果整个构建体由单个多核苷酸序列表达,则单体和蛋白质可以进行遗传融合。单体与多核苷酸结合蛋白的遗传融合在国际申请号pct/gb09/001679(公开为wo2010/004265)中有所讨论。
如果所述多核苷酸结合蛋白通过半胱氨酸连接基团连接,则所述一个或多个半胱氨酸优选通过取代引入到所述突变体中。所述一个或多个半胱氨酸优选引入到环状区域,所述环状区域在同源物中具有低保守性,这表明可以耐受突变或插入。它们因此适合用于连接多核苷酸结合蛋白。在这类实施方式中,251位置的天然存在的半胱氨酸可以被除去。半胱氨酸残基的反应活性可以通过上文描述的修饰进行增强。
所述多核苷酸结合蛋白可以被直接或通过一个或多个连接体连接到所述突变单体上。可以使用国际申请号pct/gb10/000132(公开为wo2010/086602)中描述的杂合连接体将所述分子连接到所述突变体单体上。替代地,可以使用肽连接体。肽连接体为氨基酸序列。所述肽连接体的长度,柔性和亲水性通常被设计成使其不破坏所述单体和分子的功能。优选的柔性肽连接体具有2至20个,例如4,6,8,10或16个丝氨酸和/或甘氨酸的链长。更优选的柔性连接体包括(sg)1,(sg)2,(sg)3,(sg)4,(sg)5and(sg)8,其中s为丝氨酸而g为甘氨酸。优选的刚性连接体具有2至30个,例如4,6,8,16或24个脯氨酸的链长。更优选的刚性连接体包括(p)12,其中p为脯氨酸。
所述突变单体可以采用分子适配器和多核苷酸结合蛋白进行化学修饰。
所述分子(用其对所述单体进行化学修饰)可以直接连接到所述单体或如在国际申请pct/gb09/001690(公布号wo2010/004273),pct/gb09/001679(公布号wo2010/004265)或pct/gb10/000133(公开号wo2010/086603)中公开的通过连接体连接。
本文所述的任意蛋白质,例如本发明所述的突变体单体和孔,可以被修饰以有助于对它们的鉴定或纯化,例如通过添加组氨酸残基(his标签),天冬氨酸残基(asp标签),链霉亲和素标签,旗号标签(flagtag),sumo标签,gst标签或mbp标签,或者通过添加促进它们从细胞分泌的信号序列,其中多肽不天然含有这类序列。引入遗传标签的替代方法是将标签化学反应到蛋白上的天然或改造过的位置。这样的一个例子可以是使凝胶移位试剂与在蛋白质外部改造的半胱氨酸反应。该方法已倍证明是一种分离溶血素异源寡聚体的方法(chembiol.1997jul;4(7)497-505)。
任意本文所述的蛋白质,例如本发明所述的突变单体和孔,可以用显示标记进行标记。所述显示标记可以是允许所述蛋白质被检测的任意合适标记。合适的标记包括,但不限于,荧光分子,放射性同位素例如125i,35s,酶,抗体,抗原,多核苷酸和配体例如生物素。
任意本文所述的蛋白质,例如本发明所述的单体和孔,可以合成制备或通过重组方法制备。例如,所述蛋白质可以通过体外翻译和转录(ivtt)来合成。所述蛋白质的氨基酸序列可以被修饰为,包括非天然存在的氨基酸或增加所述蛋白质的稳定性。当通过合成方法制备所述蛋白质时,可以在制备过程中引入这种氨基酸。在合成或重组制备之后,所述蛋白也可能被改变。
蛋白质也可以采用d-氨基酸制备。例如,所述蛋白质可以包括l-氨基酸和d-氨基酸的混合物。这在制备上述蛋白质或肽的技术领域中是常规的。
所述蛋白质也可以包含其他非特异性修饰,只要它们不干扰所述蛋白质的功能。许多非特异性侧链修饰是本领域已知的并可以对所述蛋白的侧链进行。所述修饰包括,例如通过下述进行的氨基酸还原性烷基化:与醛反应然后用nabh4还原,用亚氨酰乙酸甲酯进行脒基化或用乙酸酐进行酰化来氨基酸。
本文所述的任意蛋白,包括本发明所述单体和孔,可以采用本领域已知的标准方法制备。编码蛋白的多核苷酸序列可以采用本领域的标准方法进行衍生和复制。编码蛋白的多核苷酸序列采用本领域标准技术在细菌宿主细胞中表达。所述蛋白可以从重组表达载体通过在细胞中原位表达所述多肽进行制备。所述表达载体任选地带有诱导型启动子以控制所述多肽的表达。这些方法在sambrook,j.andrussell,d.(2001).molecularcloningalaboratorymanual,3rdedition.coldspringharborlaboratorypress,coldspringharbor,ny中描述。
从产生蛋白质的生物体中或重组表达后经任意蛋白质液相色谱系统纯化后,蛋白可以规模生产。典型的蛋白质液相色谱系统包括fplc,akta系统,bio-cad系统,bio-radbiologic系统和gilsonhplc系统。
构建体
本发明也提供一种包括两个以上共价连接的csgg单体的构建体,其中至少一个所述单体是本发明所述的突变单体。本发明所述的构建体保持其形成孔的能力。其可以如上所述进行确定。本发明的一个或多个构建体可以用于形成用于表征的孔,例如多核苷酸测序。所述构建体可以包括至少2,至少3,至少4,至少5,至少6,至少7,至少8,至少9或至少10个单体。所述构建体优选包括2个单体。所述两个或多个单体可以为相同的或不同的。
所述构建体中至少一个单体为本发明所述的突变单体。所述构建体中2个以上,3个以上,4个以上,5个以上,6个以上,7个以上,8个以上,9个以上,10个以上单体可以为本发明所述的突变单体。所述构建体中所有单体优选为本发明中所述突变单体。所述突变单体可以是相同的或不同的。在优选的实施方式中,所述构建体包括两个本发明所述的突变单体。
所述构建体中本发明所述突变单体优选为具有大约相同的长度或具有相同的长度。所述构建体中本发明突变单体的桶状结构优选为具有大约相同的长度或具有相同的长度。长度可以用氨基酸数量和/或单元长度进行测量。
所述构建体可以包括一个或多个不是本发明突变单体的单体。不为本发明突变单体的csgg突变单体包括包含seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的单体或seqidno:390,391,392,393,394,395,414,415,416,417,418,419,414,421,422,423,424,425,426,427,428或429的对照变体,其中上述氨基酸/位置均未进行突变。所述构建体中的至少一个单体可以包括seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429或者seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429中所示序列的对照变体。seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的对照变体与390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428or429,40或41在其整个序列上基于氨基酸的同一性至少50%同源。更优选,所述对照变体与seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的氨基酸序列在整个长度上基于氨基酸同一性可以至少55%,至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%以及更优选至少95%,97%或99%同源。
所述构建体中的单体优选为遗传融合的。如果整个构建体由单个多核苷酸序列表达,则单体可以进行遗传融合。所述单体的编码序列可以以任意方式重组以形成编码所述构建体的单个多核苷酸序列。
所述单体可以以任意构象进行遗传融合。所述单体可以通过其末端氨基酸进行融合。例如,一个所述单体的氨基末端可以与另一单体的羧基末端融合。构建体中第二和其后的单体(从氨基至羧基方向)可以在它们的氨基末端(各自与之前单体的羧基末端融合)包含一个甲硫氨酸。例如,如果m是一个单体(不带有氨基末端甲硫氨酸)且mm是带有氨基末端甲硫氨酸的单体,则所述构建体可以包括序列m-mm,m-mm-mm或m-mm-mm-mm。这些甲硫氨酸的存在通常来自编码整个构建体的多核苷酸中编码第二个或其后的单体的多核苷酸的5'末端的起始密码子(即atg)的表达。所述构建体中的第一单体(从氨基到羧基方向)也可以包括甲硫氨酸(例如mm-mm,mm-mm-mm或mm-mm-mm-mm)。
所述两个或更多个单体可以直接遗传融合在一起。所述单体优选采用连接体遗传融合。所述连接体可以设计为限制所述单体的移动。优选的连接体是氨基酸序列(即肽连接体)。上文所述的任意肽连接体均可以使用。
在另一优选的实施方式中,所述单体是化学融合的。如果两个单体是化学连接的,例如通过化学交联剂连接,则所述两个单体是化学融合的。下文所述的任意化学交联剂均可使用。所述连接体可以连接到一个或多个引入到本发明突变单体中的半胱氨酸残基上。或者,所述连接体可以连接到所述构建体中所述单体之一的末端。
如果构建体包含不同单体,可以通过在大量过量的所述单体中保持所述连接体的浓度防止单体彼此间的交联。替代地,可以在使用两个连接体时采用“锁和钥”设置。每个连接体的仅一端可以一起反应以形成更长的连接体,并且所述连接体的另一端各自与不同的单体反应。这种连接体在国际申请号pct/gb10/000132(公开号:wo2010/086602)中描述。
多核苷酸
本发明还提供编码本发明所述突变单体的多核苷酸序列。所述突变单体可以是如上所述的任意一种。所述多核苷酸序列优选包括基于核苷酸同一性与序列seqidno:389的序列在整个序列上至少50%,60%,70%,80%,90%或95%同源的序列。在300个以上,例如375,450,525或600个以上的连续核苷酸延伸链上,可以具有至少80%例如至少85%,90%或95%的核苷酸同一性(“严格同源性”)。同源性可以如上所述进行计算。所述多核苷酸序列可以包含基于遗传密码的简并性而与seqidno:389不同的序列。
本发明还提供编码任意本发明所述遗传融合构建体的多核苷酸序列。所述多核苷酸优选包括seqidno:389中所示序列的两个或更多个变体。所述多核苷酸序列优选包括两个或更多个与seqidno:389序列基于核苷酸同一性在整个序列上具有至少50%,60%,70%,80%,90%或95%同源性的序列。在600个以上,例如750,900,1050或1200个以上的连续核苷酸延伸链上,可以具有至少80%例如至少85%,90%或95%的核苷酸同一性(”严格同源性”)。同源性可以如上所述进行计算。
多核苷酸序列可以采用本领域的标准方法进行衍生和复制。编码野生型csgg的染色体dna可以从产生孔的生物体如大肠杆菌中提取。编码所述孔亚基的基因可以使用包括特异性引物的pcr进行扩增。所述扩增的序列随后可以进行定点突变。定点突变的合适方法是本领域已知的并且包括,例如,组合链式反应。编码本发明构建的多核苷酸可以采用本领域公知的技术制备,例如在sambrook,j.andrussell,d.(2001).molecularcloningalaboratorymanual,3rdedition.coldspringharborlaboratorypress,coldspringharbor,ny中描述的那些。
所得到的多核苷酸序列随后可以被整合到重组可复制载体上,例如克隆载体。所述载体可以用于在相容的宿主细胞中复制所述多核苷酸。因此多核苷酸序列可以通过将多核苷酸引入到可复制载体中,将载体引入相容的宿主细胞中,并在引起载体复制的条件下使宿主细胞生长而进行制备。所述载体可以从所述宿主细胞中回收。用于多核苷酸克隆的合适宿主细胞是本领域已知的并且在下文进行更具体的描述。
所述多核苷酸序列可以克隆进合适的表达载体中。在表达载体中,所述多核苷酸序列通常可操作地连接到能够提供宿主细胞对编码序列的表达的控制序列。所述表达载体可以用于表达孔亚基。
术语"可操作地连接"是指并置连接(juxtaposition),其中所描述的组分处于允许它们以其预期方式发挥作用的关系。控制序列“可操作地连接”到编码序列是指以使得编码序列的表达在与对照序列相容的条件下实现的方式连接。可以将相同或不同的多核苷酸序列的多个复本引入到所述载体中。
所述表达载体可以随后被引入到合适的宿主细胞中。因此,本发明的突变体单体或构建体可以通过将多核苷酸序列插入到表达载体中,将载体引入到相容的细菌宿主细胞中,并在能引起所述多核苷酸序列的表达的条件下使宿主细胞生长来制备。重组表达的单体或构建体可以自组装到在所述宿主细胞膜中的孔中。替代地,以这种方式制备的重组孔可以从所述宿主细胞中移出并插入到另一膜中。当产生包含至少两种不同单体或构建体的孔时,所述不同的单体或构建体可以如上所述在不同宿主细胞中分别表达,从宿主细胞中移出并组装到分离的膜中的孔中,例如兔细胞膜或合成膜。
所述载体可以为例如提供有复制起点的质粒,病毒或噬菌体载体,任选地用于表达所述多核苷酸序列的启动子,和任选地所述启动子的调控子。所述载体可以包含一个或多个可选择的标记基因,例如,四环素抗性基因。可以对启动子和其他表达调节信号进行选择,使其与设计表达载体所针对的宿主细胞相容。通常使用t7,trc,lac,ara或λl启动子。
所述宿主细胞通常以高水平表达所述单体或构建体。用多核苷酸序列转化的宿主细胞被选择为,与用于转化所述细胞的表达载体相容。所述宿主细胞通常为细菌并优选大肠杆菌。具有λde3溶源菌(lysogen)的任何细胞,例如c41(de3),bl21(de3),jm109(de3),b834(de3),tuner,origami和origamib,可以表达包含t7启动子的载体。除上述条件之外,在caoetal,2014,pnas,structureofthenonamericbacterialamyloidsecretionchannel,doi-1411942111和goyaletal,2014,nature,516,250-253structuralandmechanisticinsightsintothebacterialamyloidsecretionchannelcsgg中引用的任何方法也可以用于表达csgg蛋白。
本发明还包括产生本发明突变单体或本发明构建体的方法。所述方法包括在合适的宿主细胞中表达本发明的多核苷酸。所述多核苷酸优选为载体的一部分并且优选可操作地连接到启动子上。
孔
本发明还提供多种孔。本发明所述的孔理想的用于表征多核苷酸序列,例如测序多核苷酸序列,因为它们可以高灵敏度区分不同的核苷酸。所述孔可以出人预料的区分dna和rna中的四种核苷酸。本发明所述孔甚至可以区分甲基化和未甲基化的核苷酸。本发明所述孔的碱基分辨率出人预料的高。所述孔显示对全部四种dna核苷酸的几乎完全分离。所述孔基于在所述孔中的停留时间和流过所述孔的电流,进一步区分脱氧胞嘧啶单磷酸(dcmp)和甲基-dcmp。
本发明所述孔还可以在一系列条件下区分不同核苷酸。特别地,所述孔在有利于核酸表征如测序的条件下区分核苷酸。通过改变施加的电势,盐浓度,缓冲液,温度和添加剂如脲,甜菜碱和dtt的存在,可以控制本发明所述的孔区分不同核苷酸的程度。这允许孔的功能被精细调控,特别是在测序时。这将在下面更详细地讨论。本发明所述的孔也可以用于通过与一种或多种单体的相互作用而不是在以核苷酸为基础的核苷酸上,来鉴定多核苷酸聚合物。
本发明所述孔可以是分离的,基本上分离的,纯化的或基本纯化的。如果完全不含任何其它组分,例如脂质体或其它孔,则本发明所述的孔被分离或纯化。如果孔与不会干扰其预期用途的载体或稀释剂混合,则该孔被基本上被分离。例如,如果孔以包含小于10%,小于5%,小于2%或小于1%的其它组分如三嵌段共聚物,脂质体或其它孔的形式存在,则所述孔基本上被分离或基本上被纯化。替代地,本发明所述孔可以存在于膜中。合适的膜如下所述。
本发明的孔可以以单独的或单一的孔存在。替代地,本发明所述孔可以存在于两个或更多个孔的同源或异源群体中。
同源寡聚孔
本发明还提供一种衍生自包含相同本发明突变单体的csgg的同源寡聚孔。所述同源寡聚孔可以包括本发明的任意突变体。本发明所述的同源寡聚孔理想的用于多核苷酸表征,例如测序。本发明所述同源寡聚孔可以具有上述的任何优点。
所述同源寡聚孔可以包含任意数量的突变单体。所述孔一般包含至少7,至少8,至少9或至少10个相同的突变单体,例如,7,8,9或10个突变单体。所述孔优选包含8或9个相同的突变单体。所述突变单体的一个或多个,例如2,3,4,5,6,7,8,9或10个,优选如上述讨论的被化学修饰。
下面更详细的讨论用于制备所述孔的方法。
异源寡聚孔
本发明还提供衍生自包含至少一种本发明的突变单体的csgg的异源寡聚孔。本发明所述的异源寡聚孔理想的用于多核苷酸表征,例如测序。异源寡聚孔可以用本领域已知的方法制备(例如proteinsci.2002jul;11(7)1813-24)。
所述异源寡聚孔包含足够的单体以形成所述孔。所述单体可以为任何种类。所述孔一般包含至少7,至少8,至少9或至少10个单体,例如,7,8,9或10个单体。所述孔优选包含8或9个单体。
在优选的实施方式中,所有所述单体(例如10,9,8或7个单体)为本发明的突变单体,并且其中至少一个单体与其他单体不同。在更优选的实施方式中,所述孔包括八个或九个本发明所述的突变单体,并且其中至少一个单体与其他单体不同。它们也可以全部彼此不同。
在所述孔中本发明的突变单体优选为具有大约相同的长度或具有相同的长度。所述孔中本发明所述突变单体的桶状结构优选为具有大约相同的长度或具有相同的长度。长度可以用氨基酸数量和/或长度单元进行测量。
在另一优选的实施方式中,至少一个所述突变单体不是本发明的突变单体。在这个实施方式中,剩下的单体优选为本发明所述的突变单体。因此,所述孔可以包括9,8,7,6,5,4,3,2或1个本发明的突变单体。所述孔中任意数量的单体可以不是本发明的突变单体。所述孔优选包括七个或八个本发明的突变单体以及一个不是本发明的单体的单体。本发明所述的突变单体可以是相同或不同的。
所述构建体中本发明所述突变单体优选为具有大约相同的长度或具有相同的长度。所述构建体中本发明所述突变单体的桶状结构优选为具有大约相同的长度或具有相同的长度。长度可以用氨基酸数量和/或长度单元进行测量。
所述孔可以包括一个或多个不是本发明突变单体的单体。作为不是本发明突变单体的csgg单体包括包含seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的单体或者seqidno:390,391,392,393,394,395,414,415,416,417,418,419,414,421,422,423,424,425,426,427,428或429的对照变体,其中所有如上所述的与本发明相关的氨基酸/位置均没有被突变/取代。seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的对照变体通常为与seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429在其整个序列上基于氨基酸的同一性至少50%同源。更优选,所述对照变体可以为基于氨基酸同一性,与seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的氨基酸序列在整个序列上,至少55%,至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%以及更优选至少95%,97%或99%同源。
在如上所述的所有实施方式中,一个或更多,例如2,3,4,5,6,7,8,9或10个,所述突变单体优选如上所述进行化学修饰。
下面更详细的讨论用于制备所述孔的方法。
含有构建体的孔
本发明还提供一种包括至少一个本发明构建体的孔。本发明的构建体包括两个或更多个共价连接的衍生自csgg的单体,其中至少一个所述单体为本发明的突变单体。也就是说,构建体必须包含多于一个的单体。所述孔含有足够的构建体和,如果必要,足够的单体以形成所述孔。例如,八聚体孔可以包含(a)四个构建体,每个构建体包含两个单体,(b)两个构建体,每个构建体包含四个单体或(b)一个构建体,其包含两个单体和六个不构成构建体一部分的单体。例如,九聚体孔可以包括(a)各自包含两个单体的四个构建体以及一个不形成构建体一部分的单体,(b)各自包含四个单体的两个构建体以及一个不形成构建体一部分的单体或(b)一个包含两个单体的构建体和七个不构成构建体一部分的单体。本领域技术人员可以设想构建体和单体的其他组合。
至少两个在所述孔中的所述单体为本发明构建体的形式。所述构建体,以及因此所述孔,包括至少一个本发明的突变单体。所述孔一般共包含至少7,至少8,至少9或至少10个单体,例如,7,8,9或10个单体(其中的至少两个必须在构建体中)。所述孔优选包括八个或九个单体(其中的至少两个必须在构建体中)。
包含构建体的孔可以为同源寡聚体(即包括相同的构建体)或为异源寡聚体(即其中至少一个构建体与其他构建体不同)。
孔通常包含(a)包含两个单体的一个构建体和(b)5,6,7或8个单体。所述构建体可以是如上所述的任意一种。所述单体可以为如上所述任意一种,包括本发明的突变单体,包含seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的单体以及包含如上所述seqidno:390,391,392,393,394,395,414,415,416,417,418,419,414,421,422,423,424,425,426,427,428或429的对照变体的突变单体。
另一种典型的孔包括多于一个的本发明构建体,例如两个,三个或四个本发明的构建体。如果必要,所述孔进一步包含足够的额外单体或构建体以形成所述孔。所述额外单体可以为如上所述任意一种,包括本发明的突变单体,包含seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的单体以及包含如上所述seqidno:390,391,392,393,394,395,414,415,416,417,418,419,414,421,422,423,424,425,426,427,428或429的对照变体的突变单体。所述额外构建体可以为如上所述任意一种或者可以为包括两个或更多个共价连接的csgg单体的构建体,所述csgg单体各自包括包含seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429的单体以及包含如上所述seqidno:390,391,392,393,394,395,414,415,416,417,418,419,414,421,422,423,424,425,426,427,428或429的对照变体的单体。
本发明进一步的孔仅包括包含两个单体的构建体,例如孔可以包括4,5,6,7或8个包含两个单体的构建体。至少一个构建体为本发明的构建体,即,在至少一个构建体中的至少一个单体,并且优选在至少一个构建体中的每个单体,为本发明的突变单体。所有包含两个单体的构建体可以为本发明的构建体。
本发明的特定孔包括四个本发明的构建体,每个构建体包含两个单体,其中每个构建体中的至少一个单体,并且优选每个构建体中的每个单体,为本发明的突变单体。所述构建体可以寡聚成具有使得每个构建体中只有一个单体有助于孔通道的结构的孔。通常地,所述构建体中的其他单体将在所述孔的通道的外部。例如,本发明的孔可以包括7,8,9或10个包含两个单体的构建体,其中所述通道包括7,8,9或10个单体。
突变可以引入到如上所述的构建体中。所述突变可以是交替的,即所述突变对两个单体构建体中的每个单体均是不同的,并且所述构建体被组装为导致交替修饰的同源寡聚体。也就是说,包含muta和mutb的单体被融合并且被组装以形成a-b:a-b:a-b:a-b孔。替代地,所述突变可以是相邻的,即将相同的突变引入一个构建体中的两个单体中,然后与不同的突变单体或构建体进行寡聚。也就是说,包含muta的单体进行融合,然后与含mutb的单体进行寡聚以形成a-a:b:b:b:b:b:b。
在含有构建体的孔中一个或多个本发明的单体可以如上所述进行化学修饰。
分析物的表征
本发明提供一种确定目标分析物的存在、不存在或一种或多种特性的方法。该方法涉及将所述目标分析物与csgg孔或其突变体(例如本发明的孔)接触,使得所述目标分析物相对于,例如穿过,所述孔移动,并且当所述分析物相对于所述孔移动时获取一个或多个测量值,从而确定所述分析物的存在、不存在或一种或多种特性。所述目标分析物也可以被称为模板分析物或感兴趣的分析物。
所述方法包括将所述目标分析物与csgg孔或其突变体(例如本发明的孔)接触,使得所述目标分析物移动穿过所述孔。所述孔一般包含至少7,至少8,至少9或至少10个单体,例如,7,8,9或10个单体。所述孔优选包含8或9个相同的单体。所述单体中的一个或多个,例如2,3,4,5,6,7,8,9或10个,优选如上述讨论的被化学修饰。
所述csgg孔可以来源于任何生物体。所述csgg孔可以包含包括seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429中所示序列的单体。所述csgg孔可以包含至少7,至少8,至少9或至少10个单体,例如,7,8,9或10个单体,每个单体包括seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429中所示序列(即所述孔是包含来自相同生物体的相同单体的同源寡聚物)。所述csgg孔可以包含单体的任意组合,每个所述单体包括seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429中所示序列。例如,所述孔可以包含7个包括seqidno:390中所示序列的单体和2个包括seqidno:391中所示序列的单体。
所述csgg突变体可以包含任意数量的突变单体,例如至少7,至少8,至少9或至少10个单体,例如,7,8,9或10个单体。所述突变单体可以包括seqidno:390,391,392,393,394,395,414,415,416,417,418,419,420,421,422,423,424,425,426,427,428或429中所示序列的比较变体。比较变体已经在前文中讨论过。比较变体必须能够形成孔,并可以相比于本发明所述孔具有任意百分比的前文讨论的同源性。
所述csgg突变体优选包含9个单体并且所述单体中至少一个是seqidno:390中所示序列的变体,所述变体包括:(a)在一个或多个以下位置的突变:n40,q42,d43,e44,k49,y51,s54,n55,f56,s57,q62,e101,n102,e124,e131,r142,d149,t150,e185,r192,d195,e201和e203,例如在一个或多个以下位置的突变:n40,q42,d43,e44,k49,y51,s54,n55,f56,s57,q62,e101,n102,e131,d149,t150,e185,d195,e201和e203,或一个或多个在一个或多个以下位置的突变:n40,q42,d43,e44,k49,y51,n55,f56,e101,n102,e131,d149,t150,e185,d195,e201和e203;和/或(b)一个或多个以下位置的缺失:f48,k49,p50,y51,p52,a53,s54,n55,f56和s57。所述变体可以包含(a);(b);或(a)和(b)。在(a)中,任意数量和组合的以下位置:n40,q42,d43,e44,k49,y51,s54,n55,f56,s57,q62,e101,n102,r124,e131,r142,d149,e185,r192,d195,e201以及e203,可以发生突变。如前文所讨论的,y51,n55和f56中的一个或多个发生突变可以减少当多核苷酸移动穿过所述孔时对电流有贡献的核苷酸的数量,从而使得更容易识别当多核苷酸移动通过所述孔时观察到的电流与多核苷酸之间的直接关系。
在(b)中,任意数量和组合的以下位置:f48,k49,p50,y51,p52,a53,s54,n55,f56和s57可以缺失。所述变体可以包含前文针对本发明突变单体讨论的任意特定突变/取代或其组合。
用于本发明所述方法的优选变体包括一个或多个以下取代:(a)f56n,f56q,f56r,f56s,f56g,f56a或f56k或f56a,f56p,f56r,f56h,f56s,f56q,f56i,f56l,f56t或f56g;(b)n55q,n55r,n55k,n55s,n55g,n55a或n55t;(c)y51l,y51v,y51a,y51n,y51q,y51s或y51g;(d)t150i;(e)s54p;以及(f)s57p。所述变体可以包含(a)至(f)的任意数量和组合。
用于本发明所述方法的优选变体包含q62r或q62k。
用于本发明所述方法的优选变体包含在d43,e44,q62或其任意组合的突变,例如d43,e44,q62,d43/e44,d43/q62,e44/q62或d43/e44/q62。
所述变体可以包含以下位置的突变:
y51,f56,d149,e185,e201和e203,例如y51n,f56a,d149n,e185r,e201n和e203n;
n55,例如n55a或n55s;
y51,例如y51n或y51t;
s54,例如s54p;
s57,例如s57p;
f56,例如f56n,f56q,f56r,f56s,f56g,f56a或f56k或f56a,f56p,f56r,f56h,f56s,f56q,f56i,f56l,f56t或f56g;
y51和f56,例如y51a和f56a,y51a和f56n,y51i和f56a,y51l和f56a,y51t和f56a,y51t和f56q,y51i和f56n,y51l和f56n或y51t和f56n,优选,y51i和f56a,y51l和f56a或y51t和f56a,更优选y51t和f56q或进一步优选y51x和f56q,其中x是任意一种氨基酸;
n55和f56,例如n55x和f56q,其中x是任意一种氨基酸;
y51,n55和f56,例如y51a,n55s和f56a,y51a,n55s和f56n或y51t,n55s和f56q;
s54和f56,例如s54p和f56a或s54p和f56n;
f56和s57,例如f56a和s57p或f56n和s57p;
d149,e185和e203,例如d149n,e185n和e203n;
d149,e185,e201和e203,例如d149n,e185n,e201n和e203n;
d149,e185,d195,e201和e203,例如d149n,e185r,d195n,e201n和e203n或d149n,e185r,d195n,e201r和e203n;
f56和n102,例如f56q和n102r;
(a)q62,例如q62r或q62k,和(b)y51,n55和f56的一个或多个,例如在y51,n55,f56,y51n55,y51f56,n55f56或y51n55f56,例如y51tf56qq62r;
(i)d43,e44,q62或其任意组合,例如d43,e44,q62,d43/e44,d43/q62,e44/q62或d43/e44/q62和(ii)y51,n55和f56的一种或多种,例如在y51,n55,f56,y51/n55,y51/f56,n55/f56或y51/n55/f56,例如d43n/y51t/f56q,e44n/y51t/f56q,d43n/e44n/y51t/f56q,d43n/y51t/f56q/q62r,e44n/y51t/f56q/q62r或d43n/e44n/y51t/f56q/q62r;或
t150,例如t150i。
用于本发明方法的优选孔包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包括一个或多个以下取代:(a)f56n,f56q,f56r,f56s,f56g,f56a或f56k或f56a,f56p,f56r,f56h,f56s,f56q,f56i,f56l,f56t或f56g;(b)n55q,n55r,n55k,n55s,n55g,n55a或n55t;(c)y51l,y51v,y51a,y51n,y51q,y51s或y51g;(d)t150i;(e)s54p;以及(f)s57p。所述变体可以包含(a)至(f)中的任意数量和组合。所述单体优选在这些优选的孔中是相同的。
用于本发明所述方法的优选孔包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包括:
y51n,f56a,d149n,e185,e201n和e203n;
n55a;
n55s;
y51n;
s54p;
s57p;
f56n,f56q,f56r,f56s,f56g,f56a或f56k;
f56a,f56p,f56r,f56h,f56s,f56q,f56i,f56l,f56t或f56g;
y51a和f56a;
y51a和f56n;
y51i和f56a;
y51l和f56a;
y51t和f56a;
y51t和f56q;
y51i和f56n;
y51l和f56n;
y51t和f56n;
n55s和f56q;
y51a,n55s和f56a;
y51a,n55s和f56n;
y51t,n55s和f56q;
s54p和f56a;
s54p和f56n;
f56a和s57p;
f56n和s57p;
d149n,e185n和e203n;
d149n,e185n,e201n和e203n;
d149n,e185r,d195n,e201n和e203n;
d149n,e185r,d195n,e201r和e203n;
t150i;
f56q和n102r;
f56和n102,例如f56q和n102r;
y51t/f56q/q62r;
d43n/y51t/f56q;
e44n/y51t/f56q;
d43n/e44n/y51t/f56q;
d43n/y51t/f56q/q62r;
e44n/y51t/f56q/q62r;或
d43n/e44n/y51t/f56q/q62r。
用于本发明方法的所述csgg突变体优选包含9个单体并且所述单体中的至少一个是seqidno:390中所示序列的变体,所述变体包括在位置y51,n55和f56中的一个或多个位置的突变。用于本发明所述方法的优选孔包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含在位置y51,n55和f56中的一个或多个处的突变。所述单体优选在这些优选的孔中是相同的。所述变体可以包含在位置y51;n55;f56;y51/n55;y51/f56;n55/f56;或y51/n55/f56的突变。所述变体可以包含上述讨论的位置y51,n55和f56中的一个或多个位置处的任意特异性突变和其任意组合。y51,n55和f56中的一个或多个可以用任意氨基酸取代。y51可以被f,m,l,i,v,a,p,g,c,q,n,t,s,e,d,k,h或r取代,例如a,s,t,n或q。n55可以被f,m,l,i,v,a,p,g,c,q,,t,s,e,d,k,h或r取代,例如a,s,t,或q。f56可以被m,l,i,v,a,p,g,c,q,n,t,s,e,d,k,h或r取代,例如a,s,t,n或q。所述变体可以进一步包含以下中的一个或多个:(i)在以下位置的一个或多个突变(即在一个或多个以下位置的突变)(i)n40,d43,e44,s54,s57,q62,r97,e101,e124,e131,r142,t150和r192;(iii)q42rorq42k;(iv)k49r;(v)n102r,n102f,n102y或n102w;(vi)d149n,d149q或d149r;(vii)e185n,e185q或e185r;(viii)d195n,d195q或d195r;(ix)e201n,e201q或e201r;(x)e203n,e203q或e203r;以及(ix)缺失一个或多个以下位置:f48,k49,p50,y51,p52,a53,s54,n55,f56和s57。所述变体可以包含上述讨论的(i)和(iii)至(xi)的任意组合。所述变体可以包含上述针对(i)和(iii)至(xi)讨论的的任意实施方式。
(1)用于本发明所述方法的优选变体或(2)用于本发明所述方法的优选孔,包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含:y51r/f56q,y51n/f56n,y51m/f56q,y51l/f56q,y51i/f56q,y51v/f56q,y51a/f56q,y51p/f56q,y51g/f56q,y51c/f56q,y51q/f56q,y51n/f56q,y51s/f56q,y51e/f56q,y51d/f56q,y51k/f56q或y51h/f56q。
(1)用于本发明所述方法的优选突变体或(2)用于本发明所述方法的优选孔,包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含:y51tf56q,y51qf56q或y51a/f56q。
(1)用于本发明所述方法的优选突变体或(2)用于本发明所述方法的优选孔,包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含:y51t/f56f,y51t/f56m,y51t/f56l,y51t/f56i,y51t/f56v,y51t/f56a,y51t/f56p,y51t/f56g,y51t/f56c,y51t/f56q,y51t/f56n,y51t/f56t,y51t/f56s,y51t/f56e,y51t/f56d,y51t/f56k,y51t/f56h或y51t/f56r。
(1)用于本发明所述方法的优选变体或(2)用于本发明所述方法的优选孔,包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含:y51t/n55q,y51t/n55s或y51t/n55a。
(1)用于本发明所述方法的优选变体包含或(2)用于本发明所述方法的优选孔,包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含:y51a/f56f,y51a/f56l,y51a/f56i,y51a/f56v,y51a/f56a,y51a/f56p,y51a/f56g,y51a/f56c,y51a/f56q,y51a/f56n,y51a/f56t,y51a/f56s,y51a/f56e,y51a/f56d,y51a/f56k,y51a/f56h或y51a/f56r。
(1)用于本发明所述方法的优选变体或(2)用于本发明所述方法的优选孔,包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含:y51c/f56a,y51e/f56a,y51d/f56a,y51k/f56a,y51h/f56a,y51q/f56a,y51n/f56a,y51s/f56a,y51p/f56a或y51v/f56a。
(1)用于本发明所述方法的优选变体或(2)用于本发明所述方法的优选孔,包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含:
d149r/e185r/e201r/e203r或y51t/f56q/d149r/e185r/e201r/e203r;
d149n/e185n/e201n/e203n或y51t/f56q/d149n/e185n/e201n/e203n;
e201r/e203r或y51t/f56q/e201r/e203r
e201n/e203r或y51t/f56q/e201n/e203r;
e203r或y51t/f56q/e203r;
e203n或y51t/f56q/e203n;
e201r或y51t/f56q/e201r;
e201n或y51t/f56q/e201n;
e185r或y51t/f56q/e185r;
e185n或y51t/f56q/e185n;
d149r或y51t/f56q/d149r;
d149n或y51tf56q/d149n;
r142e或y51t/f56q/r142e;
r142n或y51t/f56q/r142n;
r192e或y51t/f56q/r192e;或者
r192n或y51t/f56q/r192n;
(1)用于本发明所述方法的优选变体或(2)用于本发明所述方法的优选孔,包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含:
y51a/f56q/e101n/n102r;
y51a/f56q/r97n/n102g;
y51a/f56q/r97n/n102r;
y51a/f56q/r97n;
y51a/f56q/r97g;
y51a/f56q/r97l;
y51a/f56q/n102r;
y51a/f56q/n102f;
y51a/f56q/n102g;
y51a/f56q/e101r;
y51a/f56q/e101f;
y51a/f56q/e101n;或
y51a/f56q/e101g。
所述单体优选在这些优选的孔中是相同的。
用于本发明所述方法的优选孔包含至少7,至少8,至少9或至少10个单体,例如7,8,9或10个单体,每个所述单体包括seqidno:390的变体,所述变体包含f56a,f56p,f56r,f56h,f56s,f56q,f56i,f56l,f56t或f56g。所述单体优选在这些优选的孔中是相同的。
所述csgg突变体可以包含实施例中公开的seqidno:390的任何变体或者可以是实施例中公开的任何孔。
所述csgg突变体最优选为本发明的孔。
步骤(a)和(b)优选在跨孔施加电势的情况下进行。如下更详细讨论的内容,所施加的电势一般导致在所述孔和多核苷酸结合蛋白之间形成复合物。所述施加的电势可以是电压电势。替代地,所施加的电势可以是化学电势。其一个例子是跨越双亲性分子层使用盐梯度。盐梯度被公开在holdenetal.,jamchemsoc.2007jul11;129(27)8650-5中。
所述方法用于确定目标分析物的存在、不存在或一种或多种特性。所述方法可以用于确定至少一种目标分析物的存在、不存在或一种或多种特性。所述方法可以涉及确定两种或更多种目标分析物的存在、不存在或一种或多种特性。所述方法可以包括确定任意数量目标分析物的存在、不存在或一种或多种特性,例如2,5,10,15,20,30,40,50,100或更多种目标分析物。一种或多种目标分析物的任意数量的特性可以被确定,例如1,2,3,4,5,10或更多种特性。
所述目标分析物优选为金属离子,无机盐,聚合物,氨基酸,肽,多肽,蛋白,核苷酸,寡核苷酸,多核苷酸,染料,漂白剂,药物,诊断剂,娱乐性毒品,爆炸物或环境污染物。所述方法可以涉及确定两种或更多种相同类别的目标分析物的存在、不存在或一种或多种特性,例如,两种以上蛋白,两种以上核苷酸或两种以上药物。或者,所述方法可以涉及确定两种或更多种不同类别的目标分析物的存在、不存在或一种或多种特性,例如,一种或多种蛋白,一种或多种核苷酸和一种或多种药物。
所述目标分析物可以通过细胞分泌。或者,所述目标分析物可以是存在细胞内的分析物使得在进行本发明前所述分析物必须被从细胞中抽提出来。
所述分析物优选为氨基酸,肽,多肽和/或蛋白。所述氨基酸,肽,多肽或蛋白可以是天然存在的或非天然存在的。所述多肽或蛋白可以在其中包含合成或修饰的氨基酸。对氨基酸的大量不同类型的修饰是本领域已知的。合适的氨基酸和对其的修饰如上所述。为了本发明的目的,可以理解的是,目标分析物可以通过任何本领域已知方法进行修饰。
所述蛋白可以是酶,抗体,激素,生长因子或生长调节蛋白,例如细胞因子。所述细胞因子可以选自白介素,优选ifn-1,il-1,il-2,il-4,il-5,il-6,il-10,il-12和il-13,干扰素,优选il-γ,以及其他细胞因子,例如tnf-a。所述蛋白可以是细菌蛋白,真菌蛋白,病毒蛋白或寄生虫源蛋白。
所述目标分析物优选为核苷酸,寡核苷酸或多核苷酸。以下讨论核苷酸和多核苷酸。寡核苷酸是短链核苷酸聚合物,一般具有50个或更少的核苷酸,例如40个或更少,30个或更少,20个或更少,10个或更少或5个个或更少的核苷酸。所述寡核苷酸可以包括以下讨论的任何核苷酸,包括无碱基和修饰的核苷酸。
所述目标分析物,例如目标多核苷酸,可以存在于下面讨论的任何合适的样品中。
如下所述,所述孔一般存在于膜中。可以采用如下讨论的方法将目标分析物偶联或传递至所述膜上。
如下讨论的任何测量方法可以用于确定目标分析物的存在、不存在或一种或多种特性。所述方法优选包括将所述目标分析物与所述孔接触使得所述分析物相对于(例如移动穿过)所述孔移动,并且测量当所述分析物相对于所述孔移动时穿过所述孔的电流,从而确定所述分析物的存在、不存在或一种种或多种特性。
如果所述电流以对分析物特异性的方式流过孔(即如果检测到与所述分析物相关的特异性电流流过所述孔),则目标分析物存在。如果电流没有以对核苷酸特异性的方式流过所述孔,则所述分析物不存在。对照实验可以在所述分析物存在下进行以确定是否影响电流流过所述孔的方式。
本发明可以用于区分相似结构的分析物——基于它们对穿过孔的电流的不同影响。各个分析物可以从它们与所述孔相互作用时它们的电流幅值在单个分子水平上进行识别。本发明也可以用于确定样品中是否存在特定分析物。本发明也可以用于测量样品中特定分析物的浓度。使用除csgg之外的孔进行分析物表征是本领域已知的。
多核苷酸的表征
本发明提供一种表征目标多核苷酸的方法,例如多核苷酸测序。存在两种使用纳米孔表征或测序多核苷酸的主要策略,即链表征/测序和外切核酸酶表征/测序。本发明所述方法可以涉及任何一种方法。
在链测序中,dna顺着或逆着所施加的电势位移通过纳米孔。以渐进方式或前进方式(processively)作用在双链dna上的外切核酸酶可以用在所述孔的顺式侧以在施加电势条件下将剩余单链送入并通过所述孔,或在反向电势条件下在所述孔的反式侧上将剩余单链送入并通过所述孔。相似地,解开双链dna的解旋酶也可以采用类似的方式使用。也可以使用聚合酶。还可能用于要求逆着施加电势进行链移位的测序应用,但是所述dna必须在反向电势或无电势下首先被酶“捕获”。然后随着电势在结合后切换回来,所述链将从顺式到反式穿过所述孔并且通过电流保持处于伸展构象。单链dna外切核酸酶或单链dna依赖性聚合酶可以作为分子马达,以受控的逐步方式,逆着施加的电势,从反式到顺式,将刚刚移位的单链拉回并通过所述孔。
在一种实施方式中,所述表征目标多核苷酸的方法包括使目标序列与孔和解旋酶接触。任何解旋酶均可以用于所述方法。以下讨论合适的解旋酶。解旋酶可以相对于所述孔以两种模式工作。第一,所述方法优选使用解旋酶进行,使其通过由施加电压产生的场控制目标序列通过所述孔的运动。在这种模式中,首先将dna的5'端捕获到所述孔中,并且所述酶控制dna移动进入所述孔中,使得所述目标序列在所述电场下通过所述孔直到其最终移位到双分子层的反式侧。或者,所述方法优选这样进行:使得解旋酶控制所述目标序列逆着由施加电压产生的场通过所述孔的运动。在这种模式中,dna的3'端首先被捕获到所述孔中,并且所述酶控制dna通过所述孔的运动,使得所述目标序列逆着所施加的场被拉出所述孔直到其最终弹回到双分子层的顺式侧。
在核酸外切酶测序中,核酸外切酶从目标多核苷酸的一端释放各单个核苷酸,并且如下所述鉴定这些单个核苷酸。在另一种实施方式中,表征目标多核苷酸的方法包括使目标序列与孔和核酸外切酶接触。如下所述的任意核酸外切酶均可以用于所述方法。所述酶可以如下所述共价连接到所述孔上。
核酸外切酶是通常锁定到多核苷酸的一端并从该端一次消化所述序列的一个核苷酸的酶。所述核酸外切酶可以在5'至3'方向或3'至5'方向消化所述多核苷酸。多核苷酸的结合有核酸外切酶的末端通常通过选择所使用的酶和/或采用本领域已知的方法决定。多核苷酸任意一端的羟基或盖帽结构通常可以用于预防或促进核酸外切酶与多核苷酸特定末端的结合。
所述方法包括将所述多核苷酸与所述核酸外切酶接触,使得所述核苷酸从所述多核苷酸的末端以允许表征或鉴别如上所述的一部分核苷酸的速率被消化。进行这些步骤的方法是本领域公知的。例如,埃德曼(edman)降解用于从多肽的末端顺序消化单个氨基酸,使得它们可以使用高效液相色谱(hplc)进行鉴定。同源性的方法可以用于本发明。
所述核酸外切酶作用的速率一般低于野生型核酸外切酶的最优速率。本发明所述方法中的所述核酸外切酶活性的合适速率包括每秒消化0.5到1000个核苷酸,每秒消化0.6到500个核苷酸,每秒消化0.7到200个核苷酸,每秒消化0.8到100个核苷酸,每秒消化0.9到50个核苷酸或每秒消化1到20或10个核苷酸。所述速率优选为每秒1,10,100,500或1000个核苷酸。核酸外切酶活性的合适速率可以通过多种方式实现。例如,根据本发明可以使用具有降低的最佳活性速率的不同的核酸外切酶。
在链表征的实施方式中,所述方法包括使所述多核苷酸与csgg孔或其突变体,例如本发明的孔,接触,使得所述多核苷酸相对于所述孔移动,例如通过所述孔,并且当所述多核苷酸相对于所述孔移动时,获取一个或多个测量值,其中所述测量值指示所述多核聚苷酸的一个或多个特征,从而表征所述目标多核苷酸。
在核酸外切酶表征的实施方式中,所述方法包括使所述多核苷酸与csgg孔或其突变体,例如本发明中的孔,以及核酸外切酶接触,使得所述核酸外切酶从所述目标多核苷酸的一端消化各个核苷酸并且所述各个核苷酸相对于所述孔移动,例如通过所述孔,并且当所述各个核苷酸相对于所述孔移动时获取一个或多个测量值,其中所述测量值指示所述各个核苷酸的一个或多个特征,从而表征所述目标多核苷酸。
各个核苷酸是单个的核苷酸。各个核苷酸是不通过核苷酸键与另一个核苷酸或多核苷酸结合的核苷酸。核苷酸键包括与另一核苷酸的糖基结合的核苷酸的磷酸基团之一。各个核苷酸通常不通过核苷酸键与另一个具有至少5个,至少10个,至少20个,至少50个,至少100个,至少200个,至少500个,至少1000个或至少5000个核苷酸的多核苷酸结合。例如,所述各个核苷酸已经从目标多核苷酸序列被消化,例如,dna或rna链。所述核苷酸可以是如下所述的任意一种。
所述各个核苷酸可以与所述孔以任意方式在任意位点相互作用。所述核苷酸优选通过适配器或与适配器连接而可逆地与所述孔结合,如上文所述。所述核苷酸最优选在它们跨膜穿过所述孔时通过适配器或与适配器连接而可逆地与所述孔结合。所述核苷酸也可以当它们跨膜穿过所述孔时通过适配器或与所述适配器接合而可逆地与所述孔的桶状结构或通道结合。
在所述各个核苷酸与所述孔的相互作用中,所述核苷酸通常以对核苷酸特异性的方式影响流过所述孔的电流。例如,一种特定的核苷酸将以特定的平均时间周期和特定的程度减少流过所述孔的电流。也就是说,流过所述孔的电流对特定核苷酸是特有的。可以进行对照实验来确定特定核苷酸对流过所述孔的电流的影响。然后将对测试样品实施本发明方法得到的结果与来自上述对照实验的结果进行比较,以便鉴定样品中的特定核苷酸或确定样品中是否存在特定核苷酸。以指明特定核苷酸的方式影响流过所述孔的电流的频率可用于确定样品中所述核苷酸的浓度。样品中不同核苷酸的比例也可以计算得出。例如,dcmp对甲基-dcmp的比例可以计算得出。
所述方法包括测量所述目标多核苷酸的一种或多种特性。所述目标多核苷酸也可以被称为模板多核苷酸或感兴趣的多核苷酸。
这个实施方式也可以使用csgg孔或其突变体,例如本发明的孔。上文针对目标分析物所述的任意孔和实施方式均可以使用。
多核苷酸
多核苷酸,例如核酸,是包含两个或多个核苷酸的大分子。所述多核苷酸或核酸可以包含任意核苷酸的任意组合。所述核苷酸可以是天然存在的或人工合成的。所述多核苷酸中的一个或多个核苷酸可以被氧化或甲基化。所述多核苷酸中的一个或多个核苷酸可以被损伤。例如,所述多核苷酸可以包含嘧啶二聚体。这种二聚体通常与由紫外线造成的损伤有关并且是皮肤黑色素瘤的主要成因。所述多核苷酸中的一个或多个核苷酸可以被修饰,例如用标记或标签。合适的标记如下所述。所述多核苷酸可以包含一个或多个间隔区。
核苷酸通常含有核碱基,糖和或至少一个磷酸基团。所述核碱基和糖形成核苷。
所述核碱基通常是杂环的。核碱基包括,但不限于,嘌呤和嘧啶,更具体的是腺嘌呤(a),鸟嘌呤(g),胸腺嘧啶(t),尿嘧啶(u)和胞嘧啶(c)。
所述糖通常是戊糖。核苷糖包括,但不限于,核糖和脱氧核糖。所述糖优选是脱氧核糖。
所述多核苷酸优选包括如下核苷:脱氧腺苷(da),脱氧尿苷(du)和/或脱氧胸苷(dt),脱氧鸟苷(dg)和脱氧胞苷(dc)。
所述核苷酸通常是核糖核苷酸或脱氧核糖核苷酸。所述核苷酸通常含有单磷酸,二磷酸或三磷酸。所述核苷酸可以包含多于三个磷酸,例如4或5个磷酸。磷酸可以连接在核苷酸的3’侧或5’侧。核苷酸包括,但不限于,单磷酸腺苷(amp),单磷酸鸟苷(gmp),单磷酸胸苷(tmp),单磷酸尿苷(ump),单磷酸5-甲基胞苷,单磷酸5-羟基甲基胞苷,单磷酸胞苷(cmp),环单磷酸腺苷(camp),环单磷酸鸟苷(cgmp),单磷酸脱氧腺苷(damp),单磷酸脱氧鸟苷(dgmp),单磷酸脱氧胸苷(dtmp),单磷酸脱氧尿苷(dump),单磷酸脱氧胞苷(dcmp)和单磷酸脱氧甲基胞苷。所述核苷酸优选选自amp,tmp,gmp,cmp,ump,damp,dtmp,dgmp,dcmp和dump。
核苷酸可以是无碱基的(即缺少核碱基)。核苷酸也可以缺少核碱基和糖(即为c3间隔区)。
所述多核苷酸中的核苷酸可以任意方式相互连接。所述核苷酸通常通过其糖基和磷酸基团连接,如在核酸中一样。所述核苷酸可以通过其核碱基连接,如在嘧啶二聚体中一样。
所述多核苷酸可以是单链或双链的。所述多核苷酸的至少一部分优选是双链的。
所述多核苷酸可以是核酸,例如脱氧核糖核酸(dna)或核糖核酸(rna)。所述多核苷酸可以包含一条rna链,所述rna链杂合到一条dna链。所述多核苷酸可以是任意现有技术已知的合成核酸,例如肽核酸(pna),甘油核酸(gna),苏糖核酸(tna),锁核酸(lna)或具有核苷酸侧链的其他合成聚合物。所述pna骨架是由通过肽键连接的重复的n-(2-氨基乙基)-甘氨酸单元组成。所述gna骨架是由通过磷酸二酯键连接的重复乙二醇单元组成。所述tna骨架是由通过磷酸二酯键连接在一起的重复苏糖基组成。lna由上述核糖核酸形成,具有连接核糖部分中2’氧和4’碳的额外桥连结构。桥连的核酸(bna)是修饰的rna核苷酸。它们也可以称为限制的或不可接近的rna。bna单体可以含有5元,6元或甚至7元桥连结构并带有“固定的”c3'-内糖折叠结构(c3'-endosugarpuckering)。所述桥连结构被合成引入核糖的2',4'-位,以产生2',4'-bna单体。
所述多核苷酸最优选核糖核酸(rna)或脱氧核糖核酸(dna)。
所述多核苷酸可以为任意长度。例如,所述多核苷酸的长度可以是至少10,至少50,至少100,至少150,至少200,至少250,至少300,至少400或至少500个核苷酸或核苷酸对。所述多核苷酸的长度可以为1000个或更多个核苷酸或核苷酸对,5000个或更多个核苷酸或核苷酸对或100000个或更多个核苷酸或核苷酸对。
任意数量的多核苷酸可以被研究。例如本发明方法可以涉及表征2,3,4,5,6,7,8,9,10,20,30,50,100个或更多个多核苷酸。如果两个或更多个多核苷酸被表征,它们可以是不同的多核苷酸或相同多核苷酸的两种情形。
所述多核苷酸可以是天然存在的或人工合成的。例如,所述方法可用于验证所制备的寡核苷酸的序列。所述方法通常在体外进行。
样品
所述多核苷酸通常存在于任意合适的样品中。本发明通常针对已知包含或怀疑含有所述多核苷酸的样品进行。或者,本发明可以针对某一样品进行以确认已知存在或预期存在于所述样品中的多核苷酸的身份。
所述样品可以是生物样品。本发明可以针对从任何生物体或微生物中获得或提取的样品在体外实施。所述生物体或微生物通常是古细菌的(archaeal),原核的或真核的,并且通常属于以下五界之一:植物界,动物界,真菌,原核生物和原生生物。本发明针对从任何病毒中获得或提取的样品在体外实施。所述样品优选是流体样品。样品通常包括患者的体液。所述样品可以是尿液,淋巴液,唾液,粘液或羊水,但优选血液,血浆或血清。
通常,所述样品来源于人,但替代地可以是来自其他哺乳动物,如自商业上养殖的动物如马,牛,绵羊,鱼,鸡或猪,或者替代地可以是宠物如猫或狗。或者,所述样品可以来源于植物,例如从商业作物获得的样品,如谷类,豆类,水果或蔬菜,例如小麦,大麦,燕麦,芸苔,玉米,大豆,水稻,大黄,香蕉,苹果,番茄,土豆,葡萄,烟草,菜豆,小扁豆,甘蔗,可可,棉花。
所述样品可以是非生物样品。所述非生物样品优选为流体样品。非生物样品的实例包括手术液,水如饮用水、海水或河水,以及用于实验室试验的试剂。
所述样品通常是在本发明中使用前处理,例如通过离心,或通过膜过滤掉不需要的分子或细胞,例如红细胞。所述样品可在采集后立即测量。样品也可通常在测定前被存储,优选低于-70℃存储。
表征
所述方法可以涉及测量多核苷酸的两个,三个,四个或五个或更多个特征。所述一个或多个特征优选选自(i)多核苷酸的长度,(ii)多核苷酸的身份,(iii)多核苷酸的序列,(iv)多核苷酸的二级结构,以及(v)多核苷酸是否被修饰。根据本发明可以测量(i)至(v)的任意组合,例如{i},{ii},{iii},{iv},{v},{i,ii},{i,iii},{i,iv},{i,v},{ii,iii},{ii,iv},{ii,v},{iii,iv},{iii,v},{iv,v},{i,ii,iii},{i,ii,iv},{i,ii,v},{i,iii,iv},{i,iii,v},{i,iv,v},{ii,iii,iv},{ii,iii,v},{ii,iv,v},{iii,iv,v},{i,ii,iii,iv},{i,ii,iii,v},{i,ii,iv,v},{i,iii,iv,v},{ii,iii,iv,v}或{i,ii,iii,iv,v}。与第二多核苷酸相比,(i)至(v)的不同组合可以测量第一多核苷酸,包括上述所列出的任意组合。
对于(i),可以例如通过确定多核苷酸和孔之间相互作用的数量或多核苷酸和孔之间相互作用的持续时间对多核苷酸的长度进行测量。
对于(ii),可以以多种方式测量多核苷酸的身份,多核苷酸的身份可以结合多核苷酸序列的测量或不结合多核苷酸序列的测量进行测量。前者较为简单;对多核苷酸进行测序进而进行识别。后者可以通过几种不同方式完成。例如,可以测量多核苷酸中特定基序的存在(无需测量多核苷酸的其余序列)。或者,所述方法中特定的电和/或光信号的测量可以识别出所述多核苷酸来自特定来源。
对于(iii),多核苷酸的序列可以如先前所述进行测定。合适的测序方法,特别是使用电学测量方法的测序方法,描述在stoddartdetal.,procnatlacadsci,12;106(19)7702-7,liebermankretal,jamchemsoc.2010;132(50)17961-72,以及国际申请wo2000/28312中。
对于(iv),二级结构可以采用多种方法测量。例如,如果所述方法涉及电学测量方法,则可以使用停留时间的变化或流过孔的电流的变化来测量所述二级结构。这允许区分单链和双链多核苷酸的区域。
对于(v),可以测量是否存在任何修饰。所述方法优选包括,测定多核苷酸是否通过甲基化,氧化,损伤,用一种或多种蛋白或用一种或多种标记,标签或间隔区进行修饰。特定的修饰将导致与所述孔的特异性相互作用,其可以使用下述方法进行测量。例如,甲基胞嘧啶可以基于其与每个核苷酸相互作用期间流过所述孔的电流而与胞嘧啶区分开来。
所述目标多核苷酸与csgg孔或其突变体接触,例如如本发明的孔。所述孔通常存在于膜中。合适的膜如下所述。所述方法可以使用适合于研究膜/孔系统——其中孔存在于膜中——的的任何装置进行。所述方法可以使用适合用于跨膜孔感侧的任何装置进行。例如,所述装置包括包含水性溶液的腔室和将腔室分成两个部分的屏障。所述屏障通常具有孔洞,在孔洞中形成包含孔的膜。或者所述屏障形成膜,所述膜中存在孔。
所述方法可以使用描述于国际申请号pct/gb08/000562(wo2008/102120)中的装置进行。
可以进行各种不同类型的测量。这包括但不限于电学测量和光学测量。可能的电学测量包括电流测量,阻抗测量,隧道测量(tunnellingmeasurement)(ivanovapetal.,nanolett.2011jan12;11(1):279-85)以及fet测量(国际申请wo2005/124888)。光学测量可以与电学测量结合(sonigvetal.,revsciinstrum.2010jan;81(1)014301)。所述测量可以是跨膜电流测量,例如流过所述孔的离子电流的测量。
电学测量可以使用描述在stoddartdetal.,procnatlacadsci,12;106(19)7702-7,liebermankretal,jamchemsoc.2010;132(50)17961-72和国际申请wo2000/28312中的标准单通道记录设备进行。替代地,电学测量可以使用多通道系统进行,例如如国际申请wo2009/077734和国际申请wo2011/067559中描述的。
所述方法优选采用跨膜施加的电势进行。所述施加的电势可以是电压电势。替代地,所施加的电势可以是化学电势。其一实例为采用跨膜,例如双亲性分子层,的盐梯度进行。盐梯度被公开在holdenetal.,jamchemsoc.2007jul11;129(27):8650-5中。在一些情况下,多核苷酸相对所述孔移动时流过所述孔的电流用于估算或确定所述多核苷酸的序列。这就是链测序。
所述方法可以包括测量多核苷酸相对所述孔移动时流过所述孔的电流。因此用于所述方法的设备也可以包括能够施加电势并测量穿过膜和孔的电信号的电路。所述方法可以采用膜片钳或电压钳进行。所述方法优选涉及使用电压钳。
本发明方法可以包括测量多核苷酸相对所述孔移动时流过所述孔的电流。测量通过跨膜蛋白孔的离子流的合适条件是本领域已知的并且在实施例中公开。所述方法通常通过施加在所述膜和所述孔上的电压进行。所使用的电压通常为从+5v至-5v,例如从从+4v至-4v,从+3v至-3v或从+2v至-2v。所使用的电压通常为从-600mv至+600v或-400mv至+400mv。所使用的电压优选在具有选自-400mv,-300mv,-200mv,-150mv,-100mv,-50mv,-20mv和0mv的下限和独立地选自+10mv,+20mv,+50mv,+100mv,+150mv,+200mv,+300mv和+400mv的上限的范围内。所使用的电压更优选在100mv至240mv的范围内并且最优选在120mv至220mv的范围内。通过使用增加的施加电势,可以增加孔对不同核苷酸的识别。
所述方法通常在存在任何电荷载体的情况下进行,例如金属盐例如碱金属盐,卤化物盐例如氯化物盐,例如碱金属氯化物盐。电荷载体可以包括离子液体或有机盐,例如四甲基氯化铵,三甲基苯基氯化铵,苯基三甲基氯化铵或1-乙基-3-甲基咪唑鎓氯化物。在上述示例性装置中,盐存在于所述腔室中的水性溶液中。通常使用氯化钾(kcl),氯化钠(nacl),氯化铯(cscl)或亚铁氰化钾和铁氰化钾的混合物。kcl,nacl和亚铁氰化钾和铁氰化钾的混合物是优选的。电荷载体在所述膜上可以是不对称的。例如,电荷载体的类型和/或浓度可以在所述膜的每一侧上不同。
所述盐的浓度可以是饱和的。所述盐的浓度可以为3m或更低,并且通常为0.1至2.5m,0.3至1.9m,0.5至1.8m,0.7至1.7m,0.9至1.6m或1m至1.4m。所述盐的浓度优选为150mm至1m。所述方法优选使用至少0.3m,例如至少0.4m,至少0.5m,至少0.6m,至少0.8m,至少1.0m,至少1.5m,至少2.0m,至少2.5m或至少3.0m的盐浓度进行。高盐浓度提供高的信噪比,并允许通过电流指示在正常电流波动背景下待识别的核苷酸的存在。
所述方法通常在存在缓冲液的情况下进行。在上述示例性装置中,所述缓冲液存在于所述腔室中的水性溶液中。任意缓冲液可以用于本发明的方法。通常地,所述缓冲液为磷酸缓冲液。其他合适的缓冲液为hepes或tris-hcl缓冲液。所述方法通常在ph为4.0至12.0,4.5至10.0,5.0至9.0,5.5至8.8,6.0至8.7或7.0至8.8或7.5至8.5下进行。使用的ph值优选约7.5。
所述方法可以在0℃至100℃,15℃至95℃,16℃至90℃,17℃至85℃,18℃至80℃,19℃至70℃或20℃至60℃温度下进行。所述方法通常在室温下进行。所述方法任选的在支持酶功能的温度下进行,例如约37℃。
多核苷酸结合蛋白
链表征方法优选包括使多核苷酸与多核苷酸结合蛋白接触,使得所述蛋白控制多核苷酸相对于孔的移动,例如,通过所述孔。
更优选地,所述方法包括(a)使多核苷酸与csgg孔或其突变体(例如,本发明的孔)和多核苷酸结合蛋白接触,使得所述蛋白控制多核苷酸相对于孔的移动,例如,通过孔,和(b)当多核苷酸相对于孔移动时获取一个或多个测量值,其中,所述测量值指示多核苷酸的一个或多个特征,从而表征多核苷酸。
更优选地,所述方法包括(a)使多核苷酸与csgg孔或其突变体(例如,本发明的孔)和多核苷酸结合蛋白接触,使得所述蛋白控制多核苷酸相对于孔的移动,例如,通过孔,和(b)当多核苷酸相对于孔移动时测量通过孔的电流,其中,所述电流指示多核苷酸的一个或多个特征,从而表征多核苷酸。
多核苷酸结合蛋白可以是能够结合多核苷酸并控制其移动通过孔的任何蛋白。本领域中判断蛋白是否与多核苷酸结合是比较简单的。蛋白通常与多核苷酸相互作用并改性多核苷酸的至少一种性质。蛋白可以通过裂解多核苷酸以形成各单个核苷酸或核苷酸的短链(例如,二核苷酸或三核苷酸)来对其进行改性。蛋白可以通过使多核苷酸定向或将其移动到特定位置来对其进行改性,即,控制它的移动。
多核苷酸结合蛋白优选衍生自多核苷酸处理酶。多核苷酸处理酶是能够与多核苷酸相互作用并改性多核苷酸的至少一种性质的多肽。所述酶可以通过裂解多核苷酸以形成各单个核苷酸或核苷酸的短链(例如,二核苷酸或三核苷酸)来对其进行改性。所述酶可以通过使多核苷酸定向或将其移动到特定位置来对其进行改性。多核苷酸处理酶不需要显示酶活性,只要其能够结合多核苷酸并控制其通过孔的移动即可。例如,可以对所述酶进行改性以去除其酶活性,或者可以在防止其用作酶的条件下进行使用。以下对这种条件进行更详细的讨论。
多核苷酸处理酶优选衍生自溶核酶(nucleolyticenzyme)。在酶的构建体中使用的多核苷酸处理酶更优选地衍生自酶分类(ec)基团3.1.11,3.1.13,3.1.14,3.1.15,3.1.16,3.1.21,3.1.22,3.1.25,3.1.26,3.1.27,3.1.30和3.1.31中的任一个。所述酶可以是申请号为pct/gb10/000133(公开为wo2010/086603)的国际申请中公开的那些酶中的任一个。
优选地,所述酶为聚合酶、外切核酸酶、解旋酶和拓扑异构酶,例如,促旋酶。合适的酶包括但不限于来自大肠杆菌的核酸外切酶i(seqidno:399)、来自大肠杆菌的核酸外切酶iii酶(seqidno401)、来自极端嗜热链球菌的recj(seqidno:403)和细菌噬菌体λ外切核酸酶(seqidno:405)、tatd外切核酸酶及其变体。三个包括seqidno:403或其变体所示序列的亚基相互作用形成三聚体外切核酸酶。这些外切核酸酶也可以用于本发明的外切核酸酶方法中。聚合酶可以是
所述酶最优选衍生自解旋酶,例如,hel308mbu(seqidno:406)、hel308csy(seqidno:407)、hel308tga(seqidno:408)、hel308mhu(seqidno:409)、traleco(seqidno:410)、xpdmbu(seqidno:411)或其变体。本发明中可以使用任何解旋酶。所述解旋酶可以为或衍生自hel308解旋酶、recd解旋酶,例如,tral解旋酶或trwc解旋酶、xpd解旋酶或dda解旋酶。所述解旋酶可以是以下国际申请中公开的解旋酶、改性解旋酶或解旋酶构建体中的任一个:pct/gb2012/052579(公开为wo2013/057495);pct/gb2012/053274(公开为wo2013/098562);pct/gb2012/053273(公开为wo2013098561);pct/gb2013/051925(公开为wo2013/057495);pct/gb2012/053274(公开为wo2013/098562);pct/gb2012/053273(公开为wo2013098561);pct/gb2013/051925(公开为wo2014/013260);pct/gb2013/051924(公开为wo2014/013259);pct/gb2013/051928(公开为wo2014/013262)和pct/gb2014/052736。
解旋酶优选地包括seqidno:413(trwccba)或其变体所示的序列、seqidno:406(hel308mbu)或其变体所示的序列,或者seqidno:412(dda)或其变体所示的序列。变体可能以下述针对跨膜孔所讨论的任何方式不同于天然序列。seqidno:412的优选变体包括(a)e94c和a360c或(b)e94c,a360c,c109a和c136a,然后,可选地(δm1)g1g2(即,缺失m1,然后加入g1和g2)。
根据本发明,可以使用任何数量的解旋酶。例如,可以使用1,2,3,4,5,6,7,8,9,10或更多个解旋酶。在一些实施例中,可以使用不同数目的解旋酶。
本发明的方法优选包括使多核苷酸与两个或更多个解旋酶接触。所述两个或更多个解旋酶通常是相同的解旋酶。所述两个或更多个解旋酶可以是不同的解旋酶。
所述两个或更多个解旋酶可以是上述解旋酶的任意组合。所述两个或更多个解旋酶可以是两个或更多个dda解旋酶。所述两个或更多个解旋酶可以是一种或多种dda解旋酶和一种或多种trwc解旋酶。所述两个或更多个解旋酶可以是相同解旋酶的不同变体。
所述两个或更多个解旋酶优选地彼此连接。所述两个或更多个解旋酶更优选地彼此共价连接。解旋酶可以以任何顺序并使用任何方法连接。本发明中采用的优选解旋酶构建体在以下国际申请中进行描述:pct/gb2013/051925(公开为wo2014/013260);pct/gb2013/051924(公开为wo2014/013259);pct/gb2013/051928(公开为wo2014/013262)和pct/gb2014/052736。
seqidno:397,399,401,403,405,406,407,408,409,410,411,412或413的变体为具有氨基酸序列的酶,所述氨基酸序列从seqidno:397,399,401,403,405,406,407,408,409,410,411,412或413的氨基酸序列变化而来并且保留多核苷酸结合能力。这可以使用本领域已知的任何方法来测量。例如,变体可以与多核苷酸接触,并且可以对其与多核苷酸结合及沿多核苷酸移动的能力进行测量。变体可以包括促进多核苷酸结合和/或促进其在高盐浓度和/或室温下活性的改性。可以对变体进行改性,使得它们结合多核苷酸(即,保留多核苷酸结合能力),但不起到解旋酶的作用(即,当提供有促进移动的所有必需组分时,不沿多核苷酸移动,例如atp和mg2+)。这种改性是本领域已知的。例如,解旋酶中mg2+结合结构域的改性通常导致变体不能用作解旋酶。这些类型的变体可以作为分子制动剂(见下文)。
在seqidno:397,339,401,403,405,406,407,408,409,410,411,412或413的氨基酸序列的整个长度上,基于氨基酸同一性,变体优选与该序列至少50%同源。更优选地,基于氨基酸同一性,变体多肽可以与seqidno:397,399,401,403,405,406,407,408,409,410,411,412或413的氨基酸序列在整个序列上至少55%,至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%,更优选地,至少95%,97%或99%同源。在200或更多个,例如,230,250,270,280,300,400,500,600,700,800,900或1000或更多个连续氨基酸的长度段上,可以存在至少80%,例如,至少85%,90%或95%的氨基酸同一性(”严格同源性”)。如上所述,确定同源性。变体可以按上述针对seqidno:390所讨论的任何方式,不同于野生型序列。酶可以与孔共价连接。可以采用任何方法将酶与孔共价连接。
优选的分子制动剂为trwccba-q594a(具有突变q594a的seqidno:413)。这种变体不用作解旋酶(即,当提供有促进移动的所有必需组分时,与多核苷酸结合,但不沿其移动,例如,atp和mg2+)。
在链测序时,多核苷酸随着或逆着所施加的电势移位通过孔。可以将对双链多核苷酸逐渐或渐进性起作用的外切核酸酶用在孔的顺式侧上以在施加电势的情况下将剩余的单链送入并通过所述孔,或者在反向电势的情况下,在反式侧上将剩余单链送入并通过所述孔。同样地,解开双链dna的解旋酶也可以以类似的方式进行使用。也可以使用聚合酶。还可能用于要求逆着施加电势进行链移位的测序应用,但dna必须在反向电势或不存在电势的情况下首先被酶“捕获”。然后随着电势在结合后切换回来,所述链将从顺式到反式穿过所述孔并且通过电流保持处于伸展构象。单链dna外切核酸酶或单链dna依赖性聚合酶可以作为分子马达,以受控的逐步方式,逆着施加的电势,从反式到顺式,将刚刚移位的单链拉回并通过所述孔。
所述方法中可以使用任何解旋酶。解旋酶相对于孔可以以两种模式工作。第一,所述方法优选使用解旋酶进行,使得解旋酶通过由施加电压产生的场移动所述多核苷酸通过所述孔。在这种模式中,首先将多核苷酸的5'端捕获到所述孔中,并且所述解旋酶将所述多核苷酸移动到所述孔中,使得其在所述电场下通过所述孔直到其最终移位到膜的反式侧。或者,所述方法优选这样进行:使得解旋酶逆着由施加电压产生的场使多核苷酸移动通过所述孔。在这种模式中,多核苷酸的3'端首先被捕获到所述孔中,并且所述解旋酶使多核苷酸移动通过所述孔,使得其逆着所施加的场被拉出所述孔直到其最终弹回到膜的顺式侧。
也可以沿相反方向执行所述方法。可以在孔中首先捕获多核苷酸的3’端,并且解旋酶可以移动多核苷酸到孔中,使得其利用场通过所述孔,直到其最终移位到膜的反式侧。
当解旋酶未提供有促进移动的必要组分或解旋酶被改性以阻碍或阻止其移动时,其可以结合多核苷酸并且在其被所施加的场牵引进孔中时用作减慢多核苷酸移动的制动剂。在非主动模式中,多核苷酸的3’或5’端是否被捕获是无关紧要的,是所施加的场通过用作制动剂的酶将多核苷酸朝向反式侧牵引到孔中。当处于非主动模式时,解旋酶对多核苷酸进行的移动控制可以以多种方式进行描述,包括棘轮(ratcheting)、滑动和制动。也可以以这种方式使用缺乏解旋酶活性的解旋酶变体。
可以以任何顺序使多核苷酸与多核苷酸结合蛋白和孔接触。优选地,当多核苷酸与多核苷酸结合蛋白(比如:解旋酶)和孔接触时,多核苷酸首先与蛋白形成复合物。当在孔上施加电压时,多核苷酸/蛋白复合物此时与孔形成复合物并控制多核苷酸移动通过孔。
采用多核苷酸结合蛋白的方法中的任何步骤通常在游离核苷酸或游离核苷酸类似物以及促进多核苷酸结合蛋白的作用的酶辅助因子存在的情况下进行。游离核苷酸可以是上面讨论的任何一个或多个单个核苷酸。游离核苷酸包括但不限于腺苷一磷酸(amp)、腺苷二磷酸(adp)、腺苷三磷酸(atp)、鸟苷一磷酸(gmp)、鸟苷二磷酸(gdp)、鸟苷三磷酸(gtp)、胸苷单磷酸(tmp)、胸苷二磷酸(tdp)、胸苷三磷酸(ttp)、尿苷单磷酸(ump)、尿苷二磷酸(udp)、尿苷三磷酸(utp)、胞苷单磷酸(cmp)、胞苷二磷酸(cdp)、胞苷三磷酸(ctp)、环腺苷酸(camp)、环磷酸鸟苷(cgmp)、脱氧腺苷一磷酸(damp)、脱氧腺苷二磷酸(dadp)、脱氧腺苷三磷酸(datp)、脱氧鸟苷一磷酸(dgmp)、脱氧鸟苷二磷酸(dgdp)、脱氧鸟苷三磷酸(dgtp)、脱氧胸苷单磷酸(dtmp)、脱氧胸苷二磷酸(dtdp)、脱氧胸苷三磷酸(dttp)、脱氧尿苷单磷酸(dump)、脱氧尿苷二磷酸(dudp)、脱氧尿苷三磷酸(dutp)、脱氧胞苷单磷酸(dcmp)、脱氧胞苷二磷酸(dcdp)和脱氧胞苷三磷酸(dctp)。游离核苷酸优选地选自amp、tmp、gmp、cmp、ump、damp、dtmp、dgmp或dcmp。游离核苷酸优选为腺苷三磷酸(atp)。酶辅助因子为使得构建体起作用的因子。酶辅助因子优选为二价金属阳离子。二价金属阳离子优选为mg2+、mn2+、ca2+、或co2+。酶辅助因子最优选为mg2+。
解旋酶和分子制动剂
在优选实施例中,所述方法包括:
(a)向多核苷酸提供一个或多个解旋酶以及与多核苷酸连接的一个或多个分子制动剂;
(b)使多核苷酸与csgg孔或其突变体,例如本发明的孔,接触,并在孔上施加电势使得一个或多个解旋酶以及一个或多个分子制动剂聚集在一起,二者均控制多核苷酸相对于孔的移动,例如,通过孔;
(c)在多核苷酸相对于孔移动时,获取一个或多个测量值,其中,所述测量值指示多核苷酸的一个或多个特征,从而表征多核苷酸。
这类方法在申请号为pct/gb2014/052737的国际申请中进行详细讨论。
所述一个或多个解旋酶可以是上述讨论的任何一种。所述一个或多个分子制动剂可以是与多核苷酸结合并减慢多核苷酸通过孔的移动的任何化合物或分子。所述一个或多个分子制动剂优选包括一个或多个与多核苷酸结合的化合物。所述一个或多个化合物优选为一个或多个大环化合物。合适的大环化合物包括但不限于环糊精、杯芳烃、环肽、冠醚、瓜环、柱芳烃、其衍生物或其组合。环糊精或其衍生物可以是eliseev,a.v.和schneider,h-j.(1994)j.am.chem.soc116,6081-6088中公开的任一种。该试剂更优选为七-6-氨基-β-环糊精(am7-βcd)、6-单脱氧-6-单氨基-β-环糊精(am1-βcd)或七-(6-脱氧-6-胍基)-环糊精(gu7-βcd)。
所述一个或多个分子制动剂优选为一个或多个单链结合蛋白(ssb)。所述一个或多个分子制动剂更优选地为包括不具有净负电荷的羧基末端(c-末端)区域的单链结合蛋白(ssb)或(ii)在其c-末端区域具有一处或多处修饰的改性ssb,所述修饰减少了c-末端区域的净负电荷。所述一个或多个分子制动剂最优选地为国际申请pct/gb2013/051924(公开为wo2014/013259)中公开的ssb之一。
所述一个或多个分子制动剂优选地为一个或多个多核苷酸结合蛋白。多核苷酸结合蛋白可以是任何能够与多核苷酸结合并控制其通过孔的移动的蛋白。本领域中可以容易地判断蛋白是否与多核苷酸结合。蛋白通常与多核苷酸相互作用并改性多核苷酸的至少一种性质。蛋白可以通过裂解多核苷酸形成各单个核苷酸或核苷酸的短链(例如,二核苷酸或三核苷酸)来对其进行改性。该部分可以通过使多核苷酸定向或将其移动到特定位置,即,控制其的移动,来对其进行改性。
多核苷酸结合蛋白优选衍生自多核苷酸处理酶。所述一个或多个分子制动剂可以衍生自上述任何一种多核苷酸处理酶。作为分子制动剂的phi29聚合酶(seqidno:396)的改性版本公开在美国专利5,576,204中。所述一个或多个分子制动剂优选衍生自解旋酶。
可以使用衍生自解旋酶的任意数量的分子制动剂。例如,可以将1,2,3,4,5,6,7,8,9,10或更多个解旋酶用作分子制动剂。如果两个或更多个解旋酶被用作分子制动剂,则所述两个或更多个解旋酶通常是相同的解旋酶。所述两个或更多个解旋酶可以是不同的解旋酶。
所述两个或更多个解旋酶可以是上述解旋酶的任意组合。所述两个或更多个解旋酶可以是两个或更多个dda解旋酶。所述两个或更多个解旋酶可以是一个或多个dda解旋酶和一个或多个trwc解旋酶。所述两个或更多个解旋酶可以是同一解旋酶的不同变体。
所述两个或更多个解旋酶优选地彼此连接。所述两个或更多个解旋酶更优选地彼此共价连接。解旋酶可以以任何顺序并使用任何方法进行连接。优选地,对衍生自解旋酶的所述一个或多个分子制动剂进行改性以减小多核苷酸结合结构域中开口的大小,其中,多核苷酸在至少一个构象状态下通过所述多核苷酸结合结构域从解旋酶上解开。这在wo2014/013260中进行了公开。
本发明中使用的优选解旋酶构建体在以下国际申请中进行描述:pct/gb2013/051925(公开为wo2014/013260);pct/gb2013/051924(公开为wo2014/013259);pct/gb2013/051928(公开为wo2014/013262)和pct/gb2014/052736。
如果所述一个或多个解旋酶在主动模式下使用(即,当所述一个或多个解旋酶提供有便于移动的所有必需组分时,例如,atp和mg2+),所述一个或多个分子制动剂优选地(a)在非主动模式下使用(即,在不存在促进移动的必要组件或不能主动移动的情况下使用);(b)在主动模式下使用,其中,所述一个或多个分子制动剂沿与所述一个或多个解旋酶相反的方向移动;或(c)在主动模式下使用,其中,所述一个或多个分子制动剂沿与所述一个或多个解旋酶相同的方向移动,并且比所述一个或多个解旋酶移动得更慢。
如果所述一个或多个解旋酶在非主动模式下(即,当所述一个或多个解旋酶未提供有促进移动的所有必需组分,例如,atp和mg2+时或不能主动移动时)使用,所述一个或多个分子制动剂优选地(a)在非主动模式下(即,用在不存在促进移动的必要组件或不能主动移动的情况下)使用或(b)在主动模式下使用,其中,所述一个或多个分子制动剂以与多核苷酸通过孔的方向相同的方向沿着多核苷酸移动。
所述一个或多个解旋酶和一个或多个分子制动剂可以在任何位置与多核苷酸连接,使得它们被聚集在一起,并且二者均控制多核苷酸通过孔的移动。所述一个或多个解旋酶和一个或多个分子制动剂是至少一个分开的核苷酸,例如,至少5个、至少10个、至少50个、至少100个、至少500个、至少1000个、至少5000个、至少10,000个、至少50,000个或更多个分离开的核苷酸。如果该方法涉及表征一端设置有y适配器且另一端设置有发夹环适配器的双链多核苷酸,则所述一个或多个解旋酶优选地与y适配器连接,且所述一个或多个分子制动剂优选地与发夹环适配器连接。在这一实施方式中,所述一个或多个分子制动剂优选地为一个或多个进行了改性使得它们与多核苷酸结合但不用作解旋酶的解旋酶。与y适配器连接的所述一个或多个解旋酶优选地停滞在以下进行更详细讨论的间隔区处。与发夹环适配器连接的所述一个或多个分子制动剂优选地不停滞在间隔区处。当所述一个或多个解旋酶到达发夹环时,所述一个或多个解旋酶和一个或多个分子制动剂优选地被聚集在一起。在y适配器与多核苷酸连接之前或在y适配器与多核苷酸连接之后,可以将所述一个或多个解旋酶与y适配器连接。在发夹环适配器与多核苷酸连接之前或者在发夹环适配器与多核苷酸连接之后,可以将所述一个或多个分子制动剂与发夹环适配器连接。
所述一个或多个解旋酶和所述一个或多个分子制动剂优选地不相互连接。所述一个或多个解旋酶和所述一个或多个分子制动剂更优选地彼此不共价连接。如国际申请pct/gb2013/051925(公开为wo2014/013260);pct/gb2013/051924(公开为wo2014/013259);pct/gb2013/051928(公开为wo2014/013262)和pct/gb2014/052736中所述,所述一个或多个解旋酶和一个或多个分子制动剂优选地不连接。
间隔区
所述一个或多个解旋酶可以停滞在国际申请pct/gb2014/050175中所述的一个或多个间隔区处。在本发明中可以使用该国际申请中公开的一个或多个解旋酶和一个或多个间隔区的任何构象。
当多核苷酸的一部分进入孔并沿着由所施加电势产生的场移动通过孔时,随着多核苷酸移动通过孔,所述一个或多个解旋酶通过孔移动通过间隔区。这是因为多核苷酸(包括所述一个或多个间隔区)移动通过孔,而所述一个或多个解旋酶留在孔的顶部。
所述一个或多个间隔区优选是多核苷酸的一部分,例如,它们打断多核苷酸序列。所述一个或多个间隔区优选地不是与多核苷酸杂交的一个或多个阻断分子例如减速块(speedbump)的一部分。
多核苷酸中可以存在任意数量的间隔区,例如1,2,3,4,5,6,7,8,9,10或更多个间隔区。多核苷酸中优选存在两个、四个或六个间隔区。多核苷酸的不同区域中可以存在一个或多个间隔区,例如,y适配器和/或发夹环适配器中的一个或多个间隔区。
所述一个或多个间隔区各自提供一个能垒,所述一个或多个解旋酶即使在主动模式下也无法克服该能垒。所述一个或多个间隔区可以通过减少解旋酶的牵引力(例如,通过去除多核苷酸中的核苷酸的碱基)或物理性阻断所述一个或多个解旋酶的移动(例如,采用庞大的化学基团)来阻滞所述一个或多个解旋酶。
所述一个或多个间隔区可以包括使所述一个或多个解旋酶停滞的任一分子或分子组合。所述一个或多个间隔区可以包括防止所述一个或多个解旋酶沿多核苷酸移动的任一分子或分子组合。在不存在跨膜孔和施加电势的情况下,直接确定所述一个或多个解旋酶是否停滞在一个或多个间隔区处。例如,可以通过page测量解旋酶移动穿过间隔区并替代dna互补链的能力。
所述一个或多个间隔区通常包括线性分子,例如,聚合物。所述一个或多个间隔区通常具有与多核苷酸不同的结构。例如,如果多核苷酸是dna,则所述一个或多个间隔区通常不是dna。特别地,如果多核苷酸是脱氧核糖核酸(dna)或核糖核酸(rna),则所述一个或多个间隔区优选包括肽核酸(pna)、甘油核酸(gna)、苏糖核酸(tna)、锁核酸(lna)或具有核苷酸侧链的合成聚合物。所述一个或多个间隔区可以包括与多核苷酸方向相反的一个或多个核苷酸。例如,当多核苷酸处于5’至3’方向时,所述一个或多个间隔区可以在3’至5’方向上包括一个或多个核苷酸。核苷酸可以是以上所讨论的任一种。
所述一个或多个间隔区优选包括一个或多个硝基吲哚,例如,一个或多个5-硝基吲哚、一个或多个肌苷、一个或多个吖啶、一个或多个2-氨基嘌呤、一个或多个2-6-二氨基嘌呤、一个或多个5-溴-脱氧尿苷、一个或多个反式胸苷(反式dt)、一个或多个反式二脱氧胸腺嘧啶(ddt)、一个或多个双脱氧胞苷(ddc)、一个或多个5-甲基胞苷、一个或多个5-羟甲基胞苷、一个或多个2’-o-甲基rna碱基、一个或多个异脱氧胞苷(iso-dc)、一个或多个异脱氧鸟苷(iso-dg)、一个或多个ispc3基团(即,缺乏糖和碱基的核苷酸)、一个或多个光可裂解(pc)基团、一个或多个己二醇基团、一个或多个间隔区9(isp9)基团、一个或多个间隔区18(isp18)基团、聚合物或一个或多个硫醇连接。所述一个或多个间隔区可以包括这些基团的任一组合。许多这些基团可以从
所述一个或多个间隔区可以包括任一数量的这些基团。例如,对于2-氨基嘌呤、2-6-二氨基嘌呤、5-溴-脱氧尿苷、反式dt、ddt、ddc、5-甲基胞苷、5-羟甲基胞苷、2’-o-甲基rna碱基、iso-dc、iso-dg、ispc3基团、pc基团、己二醇基团和硫醇连接,所述一个或多个间隔区优选包括2,3,4,5,6,7,8,9,10,11,12或更多个。所述一个或多个间隔区优选包括2,3,4,5,6,7,8或更多个isp9基团。所述一个或多个间隔区优选包括2,3,4,5或6或更多个isp18基团。最优选的间隔区为四个isp18基团。
聚合物优选为多肽或聚乙二醇(peg)。所述多肽优选包括2,3,4,5,6,7,8,9,10,11,12或更多个氨基酸。所述peg优选包括2,3,4,5,6,7,8,9,10,11,12或更多个单体单元。
所述一个或多个间隔区优选包括一个或多个无碱基核苷酸(即,缺少核碱基的核苷酸),例如,2,3,4,5,6,7,8,9,10,11,12或更多个无碱基核苷酸。所述核碱基可以由无碱基核苷酸中的-h(idsp)或-oh替代。通过从一个或多个相邻核苷酸中去除核碱基,可以将无碱基间隔区插入多核苷酸中。例如,可以将多核苷酸改性为包括3-甲基腺嘌呤、7-甲基鸟嘌呤、1,n6-亚乙烯基腺嘌呤肌苷(ethenoadenineinosine)或次黄嘌呤,并且可以使用人类烷基腺嘌呤dna糖基化酶(haag)从这些核苷酸中去除核碱基。替代地,可以将多核苷酸改性成包括尿嘧啶和用尿嘧啶-dna糖基化酶(udg)去除的核碱基。在一实施例中,所述一个或多个间隔区不包括任何无碱基核苷酸。
所述一个或多个解旋酶可以由每个线性分子间隔区阻滞(即,在间隔区之前)或在每个线性分子间隔区上阻滞。如果使用线性分子间隔区,则多核苷酸优选地设置有与每个间隔区末端相邻的多核苷酸双链区域,所述一个或多个解旋酶移动通过每个间隔区末端。所述双链区域通常有助于在相邻间隔区上阻滞所述一个或多个解旋酶。如果在约100mm或更低的盐浓度下执行该方法,则特别优选存在双链区域。每个双链区域通常是至少10个,例如,至少12个,长度的核苷酸。如果本发明中使用的多核苷酸为单链的,则可以通过将较短的多核苷酸与邻近间隔区的区域进行杂合来形成双链区域。所述较短的多核苷酸通常由与多核苷酸相同的核苷酸形成,但也可以由不同的核苷酸形成。例如,所述较短的多核苷酸可以由lna形成。
如果使用线性分子间隔区,则多核苷酸优选在每个间隔区的末端设置有阻断分子,所述末端与所述一个或多个解旋酶移动通过的末端相对。这可以有助于确保所述一个或多个解旋酶在每个间隔区上保持停滞。还可以有助于在所述一个或多个解旋酶在溶液中扩散的情况下,使它/它们保留在多核苷酸上。所述阻断分子可以是以下讨论的任一化学基团,这些化学基团物理上导致所述一个或多个解旋酶停滞。所述阻断分子可以是多核苷酸的双链区域。
所述一个或多个间隔区优选地包括一个或多个物理上导致所述一个或多个解旋酶停滞的化学基团。所述一个或多个化学基团优选为一个或多个侧链化学基团。所述一个或多个化学基团可以与多核苷酸中的一个或多个核碱基连接。所述一个或多个化学基团可以与多核苷酸骨架连接。可以存在任一数目的这些化学基团,例如,2,3,4,5,6,7,8,9,10,11,12或更多个。合适的基团包括但不限于荧光团、链霉亲和素和/或生物素、胆固醇、亚甲蓝、二硝基苯酚(dnp)、地高辛和/或抗地高辛和二苄基环辛炔基。
多核苷酸中的不同间隔区可以包括不同的阻滞分子。例如,一个间隔区可以包括上述线性分子之一,另一个间隔区可以包括物理上使所述一个或多个解旋酶停滞的一个或多个化学基团。间隔区可以包括上述线性分子中的任何一种和物理上导致所述一种或多种解旋酶停滞的一个或多个化学基团,例如,一个或多个脱碱基(abasics)和荧光团。
可以根据多核苷酸的类型和执行本发明所述方法的条件来设计合适的间隔区。大多数解旋酶结合dna并沿着dna移动,因此可能会被任何不是dna的物质停滞。上文对合适的分子进行了讨论。
优选在游离核苷酸和/或解旋酶辅助因子存在的情况下,执行本发明所述方法。这在以下进行更详细的讨论。在不存在跨膜孔和施加电势的情况下,所述一个或多个间隔区优选能够在存在游离核苷酸和/或存在解旋酶辅助因子的情况下阻滞所述一个或多个解旋酶。
如果在存在游离核苷酸和解旋酶辅助因子的情况下执行本发明所述方法(使得所述一个或多个解旋酶处于主动模式),如下所述,则通常使用一个或多个较长的间隔区来确保所述一个或多个解旋酶在与跨膜孔相接触且施加电势之前停滞在多核苷酸上。在不存在游离核苷酸和解旋酶辅助因子的情况下,可以使用一个或多个较短的间隔区(使得所述一个或多个解旋酶处于非主动模式)。
盐浓度也影响所述一个或多个间隔区阻滞所述一个或多个解旋酶的能力。在不存在跨膜孔和施加电势的情况下,所述一个或多个间隔区优选能够在约100mm或更低的盐浓度下使所述一个或多个解旋酶停滞。本发明所述方法中使用的盐浓度越高,通常使用的所述一个或多个间隔区越短,反之亦然。
特征的优选组合在下表3中示出。
表3
该方法可以涉及使两个或更多个解旋酶移动通过间隔区。在这种情况下,通常增加间隔区的长度,以防止尾部解旋酶在不存在孔和施加电势的情况下推动先导解旋酶经过间隔区。如果该方法涉及使两个或更多个解旋酶移动通过一个或多个间隔区,则上述间隔区长度可以增加至少1.5倍,例如,2倍、2.5倍或3倍。例如,如果该方法涉及使两个或更多个解旋酶移动通过一个或多个间隔区,则上述表3第三列中的间隔区长度可以增加1.5倍、2倍、2.5倍或3倍。
膜
本发明所述孔可以存在于膜中。在本发明所述方法中,多核苷酸通常在膜中与csgg孔或其突变体例如本发明所述孔接触。根据本发明,可以使用任何膜。合适的膜是本领域公知的。膜优选为两亲层。两亲层是由两亲分子形成的层,例如,磷脂,其具有亲水性和亲油性。两亲分子可以是合成的或天然存在的。非天然存在的两亲物和形成单层的两亲物是本领域已知的,包括:例如,嵌段共聚物(gonzalez-perez等人;langmuir;2009,25,10447-10450)。嵌段共聚物为聚合物材料,其中,两个或更多个单体子单元聚合在一起形成单一聚合物链。嵌段共聚物通常具有由每个单体子单元贡献的性质。然而,嵌段共聚物可以具有由各个子单元形成的聚合物所不具有的独特性质。可以对嵌段共聚物进行设计,使得其中一个单体子单元为疏水的(即亲油的),而另一个子单元在水性介质中为亲水的。在这种情况下,嵌段共聚物可以具有两亲性质并且可以形成模拟生物膜的结构。嵌段共聚物可以是二嵌段(由两个单体子单元组成),但也可以由多于两个的单体子单元构成,从而形成表现为两亲物的更加复杂的结构。共聚物可以是三嵌段、四嵌段或五嵌段共聚物。膜优选为三嵌段共聚物膜。
古细菌双极性四醚脂质是天然存在的脂质,其构建成使得脂质形成单层膜。通常在存活于恶劣生物环境、嗜热菌、嗜盐菌和嗜酸性细菌的极端微生物中发现这些脂质。其稳定性被认为是源于最终双层的融合性质。通过产生具有亲水-疏水-亲水这一通用基序的三嵌段聚合物来直接构建模拟这些生物实体的嵌段共聚物材料。该材料可以形成单体膜,所述单体膜行为与脂质双层类似,并且包括从囊泡到层式膜发一系列相行为。由这些三嵌段共聚物形成的膜相比生物脂质膜具有多个优点。由于三嵌段共聚物是合成的,因此可以仔细控制精确的结构,以提供形成膜以及与孔和其它蛋白相互作用所需的正确链长度和性质。
嵌段共聚物也可以由未分类为脂质子物质的子单元构成;例如,疏水性聚合物可以由硅氧烷或其它非烃系单体制成。嵌段共聚物的亲水性部分也可以具有低的蛋白结合性质,这使得膜暴露于原始生物样本时具有高度抗性。这种头部基团单元还可以衍生自非典型脂质头部基团。
与生物脂质膜相比,三嵌段共聚物膜也增加了机械和环境稳定性,例如,更高的操作温度或ph范围。嵌段共聚物的合成性质为定制基于聚合物的能广泛应用的膜提供了平台。
最优选地,膜为国际申请pct/gb2013/052766或pct/gb2013/052767中公开的膜之一。
可以对两亲性分子进行化学改性或功能化以促进多核苷酸的偶联。
两亲层可以是单层或双层。两亲层通常是平面的。两亲层可以是弯曲的。可以对两亲层进行支撑。
两亲性膜通常是天然可移动的,基本上作为二维流体,其脂质扩散速率约为10-8cms-1。这意味着孔和偶联的多核苷酸通常可以在两亲膜内移动。
膜可以为脂质双层。脂质双层为细胞膜的模型,并作为一系列实验研究的优秀平台。例如,脂质双层通过单通道记录可以用于膜蛋白的体外研究。替代地,脂质双层可用作用以检测一系列物质存在的生物传感器。脂质双层可以是任何脂质双层。合适的脂质双层包括但不限于平面脂质双层、支撑双层或脂质体。脂质双层优选为平面脂质双层。以下国际申请中公开了合适的脂质双层:pct/gb08/000563(公开为wo2008/102121)、申请号为pct/gb08/004127(公开为wo2009/077734)和申请号为pct/gb2006/001057(公开为wo2006/100484)。
脂质双层的形成方法是本领域已知的。脂质双层通常通过montal和mueller(proc.natl.acad.sci.usa.),1972;69:3561-3566)的方法形成,其中脂质单层承载在水溶液/空气界面上,经过与该界面垂直的孔的任一侧。通常,通过先将脂质溶解在有机溶剂中,然后在孔的任一侧上使所述溶剂滴在水溶液表面上蒸发,从而将脂质添加到水性电解质溶液的表面。一旦有机溶剂蒸发,孔的任一侧上的溶液/空气界面物理地上下移动通过孔,直到形成双层。可以在膜中的孔上或在凹陷内的开口上形成平面脂质双层。
montal和mueller的方法受大众所喜爱,因为它是一种用于形成适合蛋白孔插入的优质脂质双层的经济且较简单方法。其它形成双层的常见方法包括尖端浸渍、涂覆双层和脂质体双层的贴片钳。
尖部浸渍形成双层必需使孔表面(例如,移液管尖部)接触到承载脂质单层的测试溶液的表面。另外,首先通过将溶解在有机溶剂中的脂质的液滴在溶液表面蒸发而在溶液/空气界面处产生脂质单层。然后由langmuir-schaefer工艺形成所述双层,并且所述双层需要机械自动化以相对于溶液表面移动所述孔。
对于涂覆双层,将溶解在有机溶剂中的脂质的液滴直接施加到所述孔,该孔浸没在水性测试溶液中。使用画笔或等效物将脂质溶液薄薄地涂在孔上。溶剂的稀释(thinning)导致脂质双层的形成。然而,从双层完全去除溶剂是困难的,并因此由该方法形成的双层是较不稳定的且在电化学测量过程中更容易有噪声。
膜片钳通常在生物细胞膜的研究中使用。细胞膜通过抽吸被夹到移液管的端部,且膜的小块贴附到孔上。该方法已被改为用于通过夹住脂质体,然后脂质体爆裂而使脂质双层密封移液管的孔,而产生脂质双层。该方法需要稳定的、巨大的和单层的脂质体,以及需要在具有玻璃表面的材料中制造小孔。
脂质体可通过超声、挤出或mozafari法(colas等人(2007)micron38:841–847)来形成。
在优选的实施方案中,脂质双层如在国际申请no.pct/gb08/004127(公开为wo2009/077734)中描述的来形成。本方法中有利的是,脂质双层是由干燥的脂质形成的。在最优选的实施方案中,脂质双层跨越开口而形成,如在wo2009/077734(pct/gb08/004127)中所述。
脂质双层是由脂质的两个相对的层形成的。脂质的两层被排列为使得它们的疏水性尾部基团面向彼此以形成疏水性内部。脂质的亲水性头部基团面向外朝向该双层的每一侧上的含水环境。该双层可存在于许多脂质相中,所述脂质相包括但不限于,液态无序相(流体层状)、液态有序相、固态有序相(层状凝胶相、相互交叉的凝胶相)和平面双层晶体(层状子凝胶相、层状结晶相)。
可使用形成脂质双层的任何脂质组合物。脂质组合物被选择为,使得形成的脂双层具有所需的特性,例如表面电荷,具有支撑膜蛋白的能力、填充密度或机械特性。脂质组合物可包含一个或多个不同的脂质。例如,脂质组合物可含有多达100个脂质。脂质组合物优选含有1至10个脂质。脂质组合物可以包括天然存在的脂质和/或人工脂质。
脂质通常包括头部基团、界面部分和可以相同或不同的两个疏水尾部基团。合适的头部基团包括,但不限于,中性头部基团,如二酰基甘油酯(dg)和神经酰胺(cm);两性离子头部基团,如磷脂酰胆碱(pc)、磷脂酰乙醇胺(pe)和鞘磷脂(sm);带负电荷的头部基团,如磷脂酰甘油(pg);磷脂酰丝氨酸(ps)、磷脂酰肌醇(pi)、磷酸(pa)和心磷脂(ca);和带正电荷的头部基团,如三甲基铵-丙烷(tap)。合适的界面部分包括,但不限于,天然存在的界面部分,例如基于甘油或基于神经酰胺的部分。合适的疏水性尾部基团包括,但不限于,饱和烃链,如月桂酸(正十二烷酸)、肉豆蔻酸(正十四烷酸)、棕榈酸(正十六烷酸)、硬脂酸(正十八烷酸)和花生酸(正二十烷酸);不饱和烃链,如油酸(顺-9-十八烷酸);和支链烃链,如植烷酰(phytanoyl)。链的长度和不饱和烃链中双键的位置和数量可以改变。链的长度和支链烃链中支链(如甲基基团)的位置和数目可以改变。疏水性尾部基团可以被连接到作为醚或酯的界面部分。脂质可以是霉菌酸。
脂质也可以是被化学修饰的。脂质的头部基团或尾部基团可以是经化学修饰的。头部基团已被化学修饰的合适的脂质包括,但不限于,peg修饰的脂质,例如1,2-二酰基-sn-甘油基-3-磷酸乙醇胺-n-[甲氧基(聚乙二醇)-2000];官能化的peg脂质,如1,2-二硬脂酰-sn-甘油基-3-磷酸乙醇胺-n-[生物素基(聚乙二醇)2000];为共轭而修饰的脂质,如1,2-二油酰-sn-甘油基-3-磷酸乙醇胺-n-(琥珀酰基)和1,2-二棕榈酰基-sn-甘油基-3-磷酸乙醇胺-n-(生物素基)。尾部基团已被化学修饰的合适的脂质包括,但不限于,可聚合的脂质,如1,2-双(10,12-二十三二炔酰)-sn甘油基-3-磷酸胆碱;氟化脂质,例如1-棕榈酰基-2-(16-氟代棕榈酰基)-sn-甘油基-3-磷酸胆碱;氘化脂质,如1,2-二棕榈酰基-d62-sn-甘油基-3-磷酸胆碱;和醚连接的脂质,如1,2-二-o-植烷基-sn-甘油基-3-磷酸胆碱。脂质可以是化学修饰的或官能化的,以有助于多核苷酸的偶联。
两亲层例如,脂质组合物,通常包含将影响该层的性能的一个或多个添加剂。合适的添加剂包括,但不限于,脂肪酸,如棕榈酸、肉豆蔻酸和油酸;脂肪醇,如棕榈醇、肉豆蔻醇和油醇;甾醇,如胆固醇、麦角固醇、羊毛甾醇,谷甾醇和豆甾醇;溶血磷脂,如1-酰基-2-羟基-sn-甘油基-3-磷酸胆碱;和神经酰胺。
在另一个优选的实施方案中,所述膜包括固态层。固态层可以由有机和无机材料形成,所述有机和无机材料包括但不限于,微电子材料、绝缘材料,如si3n4、al2o3和sio,有机和无机聚合物,例如聚酰胺、塑料如
该方法通常使用(i)包含孔的人工两亲层,(ⅱ)分离的、包含孔的天然存在的脂质双层,或(iii)具有嵌入其中的孔的细胞,而进行。该方法通常使用人工两亲层进行,如人工三嵌段共聚物层。除了孔以外,所述层可包含其它跨膜和/或膜内蛋白以及其他分子。合适的设备和条件在下面讨论。本发明的方法通常在体外实施。
偶联
多核苷酸优选与包括孔的膜偶联。所述方法可以包括将多核苷酸与包括孔的膜偶联。优选采用一个或多个锚(anchor)将多核苷酸与膜偶联。可以使用任何已知方法将多核苷酸与膜偶联。
每个锚包括与多核苷酸偶联(或结合)的基团和与膜偶联(或结合)的基团。每个锚可与多核苷酸和/或膜共价偶联(或结合)。如果使用了y适配器和/或发夹环适配器,则优选使用该适配器将多核苷酸与膜偶联。
可以采用任一数量的锚将多核苷酸与膜偶联,例如,2,3,4或更多个锚。例如,可以采用两个锚,将多核苷酸与膜偶联,其中,每个锚均与多核苷酸和膜偶联(或结合)。
所述一个或多个锚可以包括所述一个或多个解旋酶和/或所述一个或多个分子制动剂。
如果膜为两亲层,例如,共聚物膜或脂质双层,则所述一个或多个锚优选包括存在于膜中的多肽锚和/或存在于膜中的疏水锚。疏水锚优选为脂质、脂肪酸、固醇、碳纳米管、多肽、蛋白或氨基酸,例如,胆固醇、棕榈酸酯或生育酚。在优选实施例中,所述一个或多个锚不是孔。
可以对膜的组分,例如,两亲分子、共聚物或脂质,进行化学改性或官能化以形成所述一个或多个锚。以下对合适的化学改性以及膜组分的合适官能化方式的示例进行更详细的讨论。可以对任一比例的膜组分进行官能化,例如,至少0.01%、至少0.1%、至少1%、至少10%、至少25%、至少50%或100%。
可以直接将多核苷酸与膜偶联。用于将多核苷酸与膜偶联的一个或多个锚优选包括连接体。所述一个或多个锚可以包括一个或多个,例如,2,3,4或更多个连接体。可以使用一个连接体将多于一个,例如,2,3,4或更多个多核苷酸与膜偶联。
优选的连接体包括但不限于诸如多核苷酸、聚乙二醇(peg)、多糖和多肽等聚合物。这些连接体可以是直链、支链或环状的。例如,连接体可以是环状多核苷酸。多核苷酸可以杂合到所述环状多核苷酸连接体上的互补序列。
所述一个或多个锚或一个或多个连接体可以包括可以切割分解(cuttobrokendown)的组分,例如,限制位点或光不稳定基团。
官能化连接体及其可以偶联分子的方式是本领域已知的。例如,用马来酰亚胺基团官能化的连接体将与蛋白中的半胱氨酸残基反应并连接。在本发明的上下文中,蛋白可以存在于膜中或可以用于与多核苷酸偶联(或结合)。这将在以下进行更详细的讨论。
可以采用“锁和钥匙”结构来避免多核苷酸交联。各连接体仅一端可以一起反应以形成更长的连接体,各连接体的另一端分别与多核苷酸或膜反应。这样的连接体在国际申请pct/gb10/000132(公开为wo2010/086602)中进行描述。
优选在以下所讨论的测序实施例中使用连接体。多核苷酸在与孔相互作用时不偶联(即,在步骤(b)或(e)中不偶联),就这一点而言,如果多核苷酸直接与膜持久偶联,则由于膜和孔之间的距离,当测序运行不能继续到多核苷酸的末端时,某些序列数据将丢失。如果使用连接体,则多核苷酸可以进行到完成为止。
偶联可以是持久的或稳定的。换言之,偶联可以为以下这样:使得当与孔相互作用时,多核苷酸仍与膜偶联。
偶联可以是瞬态的。换言之,偶联可以为以下这样:使得当与孔相互作用时,多核苷酸可以从膜上解偶联。
对于某些应用,如适体(aptamer)检测,优选偶联为瞬态性质的。如果持久或稳定的连接体直接与多核苷酸的5’或3’末端连接,并且连接体比膜与跨膜孔通道之间的距离短,则当测序运行不能继续到多核苷酸的末端时,某些序列数据将丢失。如果偶联是瞬态的,则当偶联端随机摆脱膜时,则可以完全处理多核苷酸。以下对形成持久性/稳定性或瞬态链接的化学基团进行更详细的讨论。多核苷酸可以利用胆固醇或脂肪酰基链与两亲层或三嵌段共聚物膜瞬时偶联。可以使用长度为6至30个碳原子的任一脂肪酰基链,例如,十六烷酸。
在优选实施例中,将多核苷酸(例如,核酸)与诸如三嵌段共聚物膜或脂质双层等两亲层偶联。之前已使用各种不同的系链策略来将核酸与合成脂质双层偶联。这些总结在下表4中。
表4
可以在合成反应中使用经改性的亚磷酰胺对合成多核苷酸和/或连接体进行官能化,这易于直接加入合适的锚定基团,例如,胆固醇、生育酚、棕榈酸酯、硫醇、脂质和生物素基团。这些不同的连接用化学物质提供了一套用于与多核苷酸连接的选择。每个不同的改性基团以稍微不同的方式偶联多核苷酸,并且偶联并不总是永久的,因此为多核苷酸与膜的连接提供了不同的停留时间。上文中讨论了瞬态偶联的优点。
如果可以将互补的反应基团或锚定基团加入到多核苷酸中,则多核苷酸与连接体或官能化膜的偶联也可以通过许多其它方式实现。向多核苷酸的任一末端添加反应基团从前已经被报道。可以采用t4多核苷酸激酶和atpγs向ssdna或dsdna的5’末端添加硫醇基团(grant,g.p.和p.z.qin(2007);“afacilemethodforattachingnitroxidespinlabelsatthe5’terminusofnucleicacids.”nucleicacidsres35(10):e77)。可以采用t4多核苷酸激酶和γ-[2-叠氮基乙基]-atp或γ-[6-叠氮基己基]-atp向ssdna或dsdna的5’-磷酸酯添加叠氮化物基团。采用硫醇或点击化学,将系链(tether)与多核苷酸共价连接,所述系链含有硫醇、碘乙酰胺opss或马来酰亚胺基团(对硫醇具有反应性)或dibo(二苯并环辛炔(dibenzocyclooxtyne))或炔基(对叠氮化物具有反应性)。可以采用末端转移酶使化学基团(例如,生物素、硫醇和荧光团)的选择更多样化,从而将改性的寡核苷酸结合到ssdna的3’端(kumar,a.,p.tchen等人(1988);“nonradioactivelabelingofsyntheticoligonucleotideprobeswithterminaldeoxynucleotidyltransferase.”analbiochem169(2):376-82)。链霉亲和素/生物素和/或链霉亲和素/脱硫生物素偶联可用于任何其它多核苷酸。以下实施例描述了如何使用链霉亲和素/生物素和链霉亲和素/脱硫生物素将多核苷酸与膜偶联。也可以采用具有适当改性的核苷酸的末端转移酶(例如胆固醇或棕榈酸酯)将锚直接加入到多核苷酸中。
所述一个或多个锚优选通过杂合将多核苷酸与膜偶联。所述一个或多个锚中的杂合允许以如上所述的瞬时方式偶联。杂合可以存在于所述一个或多个锚的任何部分中,例如,在所述一个或多个锚与多核苷酸之间、在所述一个或多个锚内或者在所述一个或多个锚与膜之间。例如,连接体可以包括两个或更多个杂合在一起的多核苷酸,例如,3,4或5个多核苷酸。所述一个或多个锚可以与多核苷酸杂合。所述一个或多个锚可以直接与多核苷酸杂合,或直接与连接到多核苷酸的y适配器和/或前导序列杂合,或直接与连接到多核苷酸的发夹环适配器杂合(如下所述)。替代地,所述一个或多个锚可以与一个或多个(例如,2或3个)杂合到多核苷酸的中间多核苷酸(或“夹板(splint)”)杂合,与连接到多核苷酸的y适配器和/或前导序列杂合,或与连接到多核苷酸的发夹环适配器杂合(如下所述)。
所述一个或多个锚可以包括单链或双链多核苷酸。锚的一部分可以与单链或双链多核苷酸连接。采用t4rna连接酶i对ssdna的短片段的连接已被报道(troutt,a.b.,m.g.mcheyzer-williams等人(1992);“ligation-anchoredpcr:asimpleamplificationtechniquewithsingle-sidedspecificity.”procnatlacadsciusa89(20):9823-5)。替代地,单链或双链多核苷酸可以与双链多核苷酸连接,然后通过热或化学变性分离这两条链。对于双链多核苷酸,可以将一段单链多核苷酸添加到所述双链体的一端或两端,或将双链多核苷酸添加到所述双链体的一端或两端。对于将单链多核苷酸添加到双链多核苷酸,这可以同连接到单链多核苷酸其它区域一样,使用t4rna连接酶i来实现。对于将双链多核苷酸添加到双链多核苷酸,此时连接可以用分别在多核苷酸和添加的多核苷酸上的互补的3'da/dt尾部(这是常规针对许多样品制备应用进行的,以防止串多联体(concatemer)或者二聚体形成)或使用由多核苷酸的限制消化以及相容适配器的连接而产生的“粘性末端”,进行“平端终止(blunt-ended)”。然后,当双链体熔化时,每条单链将具有5'或3'修饰,如果使用单链多核苷酸进行连接或修饰,则在5’末端或3'末端进行修饰,或如果使用双链多核苷酸进行连接,则在这两端进行修饰。
如果多核苷酸是合成链,则在化学合成多核苷酸期间可以掺入所述一个或多个锚。例如,可以采用具有与其连接的反应基团的引物合成多核苷酸。
腺苷酸化多核苷酸是连接反应中的中间体,其中,腺苷单磷酸与多核苷酸的5’-磷酸连接。可以使用各种试剂盒来产生该中间体,例如,来自neb的5’dna腺苷酸化试剂盒。通过在反应中用atp替代修饰的核苷酸三磷酸酯,然后可以向多核苷酸的5’添加反应基团(如硫醇、胺、生物素、叠氮化物等)。也可以采用带有适当修饰的核苷酸(例如,胆固醇或棕榈酸酯)的5’dna腺苷酸化试剂盒将锚直接添加到多核苷酸。
扩增基因组dna片段的常见技术是使用聚合酶链反应(pcr)。这里,采用两个合成的寡核苷酸引物,可以产生同一dna片段的多个复本,其中,对于每个复本,双链体中每个链的5’将是合成的多核苷酸。可以通过采用聚合酶将单个或多个核苷酸添加到单链或双链dna的3’末端。可以使用的聚合酶的实例包括但不限于末端转移酶、克列诺酶(klenow)和大肠杆菌poly(a)聚合酶。通过在反应中用atp替代修饰的核苷酸三磷酸酯,就可将诸如胆固醇、硫醇、胺、叠氮化物、生物素或脂质等锚掺入双链多核苷酸中。因此,扩增的多核苷酸的每个复本都含有锚。
理想地,多核苷酸与膜偶联而不必使多核苷酸官能化。这可以通过将所述一个或多个锚(例如,多核苷酸结合蛋白或化学基团)与膜偶联并使所述一个或多个锚与所述多核苷酸相互作用或通过使膜官能化来实现。所述一个或多个锚可以通过本文所述的任何方法与膜偶联。特别地,所述一个或多个锚可以包括一个或多个连接体,例如,马来酰亚胺官能化连接体。
在本实施例中,多核苷酸通常是rna、dna、pna、tna或lna,并且可以是双链或单链的。本实施例特别适用于基因组dna多核苷酸。
所述一个或多个锚可以包括与单链或双链多核苷酸、多核苷酸内的特定核苷酸序列或多核苷酸内改性核苷酸的模式或存在于多核苷酸上的任何其它配体进行偶联、结合或相互作用的任何基团。
合适的用于锚中的结合蛋白包括但不限于:大肠杆菌单链结合蛋白、p5单链结合蛋白、t4gp32单链结合蛋白、topovdsdna结合区、人组蛋白、大肠杆菌hudna结合蛋白和其它古细菌、原核或真核单链或双链多核苷酸(或核酸)结合蛋白,包括下述所列的那些。
特定核苷酸序列可以是转录因子、核糖体、内切核酸酶、拓扑异构酶或复制起始因子所识别的序列。改性核苷酸的模式可以是甲基化或损伤的模式。
所述一个或多个锚可以包括与多核苷酸偶联、结合、嵌入或相互作用的任一基团。所述基团可以通过静电、氢键或范德华相互作用而插入多核苷酸或与多核苷酸相互作用。这种基团包括赖氨酸单体、聚赖氨酸(其与ssdna或dsdna相互作用)、溴化乙锭(其插入dsdna)、通用碱基或通用核苷酸(其可以与任何多核苷酸杂合)和锇复合物(其可以与甲基化碱基反应)。因此,可以采用与膜连接的一个或多个通用核苷酸将多核苷酸与膜偶联。每个通用核苷酸可以采用一个或多个连接体与膜偶联。通用核苷酸优选包括以下核碱基之一:次黄嘌呤、4-硝基吲哚、5-硝基吲哚、6-硝基吲哚、福尔马林、3-硝基吡咯、硝基咪唑、4-硝基吡唑、4-硝基苯并咪唑、5-硝基吲唑、4-氨基苯并咪唑或苯基(c6-芳环)。通用核苷酸更优选地包括以下核苷之一:2’-脱氧肌苷、肌苷、7-脱氮杂-2’-脱氧肌苷、7-脱脱氮肌苷、2-氮杂-脱氧肌苷、2-氮杂-肌苷、2-o’-甲基肌苷、4-硝基吲哚2’-脱氧核糖核苷、4-硝基吲哚核糖核苷、5-硝基吲哚2’-脱氧核糖核苷、5-硝基吲哚核糖核苷、6-硝基吲哚2’-脱氧核糖核苷、6-硝基吲哚核糖核苷、3-硝基吡咯2’-脱氧核糖核苷、3-硝基吡咯核糖核苷、次黄嘌呤的非环状糖类似物、硝基咪唑2’-脱氧核糖核苷、硝基咪唑核糖核苷、4-硝基吡唑2’-脱氧核糖核苷、4-硝基吡唑核糖核苷、4-硝基苯并咪唑2’-脱氧核糖核苷、4-硝基苯并咪唑核糖核苷、5-硝基吲唑2’-脱氧核糖核苷、5-硝基吲唑核糖核苷、4-氨基苯并咪唑2’-脱氧核糖核苷、4-氨基苯并咪唑核糖核苷、苯基c-核糖核苷、苯基c-2’-脱氧核糖基核苷、2'-脱氧水粉蕈素、2'-脱氧异鸟苷、k-2'-脱氧核糖、p-2'脱氧核糖和吡咯烷。通用核苷酸更优选包含2'-脱氧肌苷。通用核苷酸更优选为imp或dimp。通用核苷酸最优选dpmp(2'-脱氧-p-核苷单磷酸)或dkmp(n6-甲氧基-2,6-二氨基嘌呤单磷酸)。
所述一个或多个锚可以通过hoogsteen氢键(其中,两个核碱基通过氢键保持在一起)或反向hoogsteen氢键(其中,一个核碱基相对于另一个核碱基旋转180°)与多核苷酸偶联(或结合)。例如,所述一个或多个锚可以包括一个或多个核苷酸、一个或多个寡核苷酸或一个或多个与多核苷酸形成hoogsteen氢键或反向hoogsteen氢键的多核苷酸。这些类型的氢键允许第三个多核苷酸链缠绕在双链螺旋上,形成三链体。所述一个或多个锚可以通过与双链体形成三链体而与双链多核苷酸偶联(或结合)。
在本实施例中,至少1%、至少10%、至少25%、至少50%或100%的膜组分可以被官能化。
当所述一个或多个锚包括蛋白时,它们能够无需进行进一步官能化而直接锚定到膜中,例如,如果它已经具有与膜相容的外部疏水区域。这种蛋白的实例包括但不限于跨膜蛋白、膜内蛋白和膜蛋白。替代地,蛋白可以用与膜相容的基因融合疏水区域表达。这种疏水蛋白区域是本领域已知的。
所述一个或多个锚在与膜接触之前优选与多核苷酸混合,但是所述一个或多个锚可以与膜接触并随后与多核苷酸接触。
另一方面,可以使用上述方法对多核苷酸进行官能化,使得其可以被特定的结合基团识别。具体地,可以用诸如生物素(用于与链霉亲和素结合)、直链淀粉(用于与麦芽糖结合蛋白或融合蛋白结合)、ni-nta(用于与多组氨酸或多组氨酸标记蛋白结合)或肽(例如,抗原)等配体对多核苷酸进行官能化。
根据优选实施例,当多核苷酸连接到优先旋入孔的前导序列时,所述一个或多个锚可以用于将多核苷酸与膜偶联。以下将对前导序列进行更详细的讨论。优选地,将多核苷酸连接(例如,接合)到优先旋入孔的前导序列。这种前导序列可以包括均聚多核苷酸或无碱区域。前导序列通常设计为直接地或经由一个或多个中间多核苷酸(或夹板)与所述一个或多个锚杂合。在这种情况下,所述一个或多个锚通常包括与前导序列中的序列或一个或多个中间多核苷酸(或夹板)中的序列互补的多核苷酸序列。在这种情况下,所述一个或多个夹板通常包括与前导序列中的序列互补的多核苷酸序列。
化学连接中使用的分子的实例是edc(1-乙基-3-[3-二甲基氨基丙基]碳二亚胺盐酸盐)。也可以采用市售的试剂盒(thermopierce,货号:22980)向多核苷酸的5’末端添加反应基团。合适的方法包括但不限于使用组氨酸残基和ni-nta进行瞬时亲和连接以及通过反应性半胱氨酸、赖氨酸或非天然氨基酸进行更稳定的共价连接。
双链多核苷酸
多核苷酸可以是双链的。如果多核苷酸是双链的,则所述方法优选还包括:在接触步骤之前将诸如发夹环等桥接部分与多核苷酸的一端连接。然后,根据本发明,多核苷酸的两条链可以在多核苷酸与孔接触之时或接触之前分离开来。当多核苷酸移动通过孔是由诸如解旋酶或分子制动剂等多核苷酸结合蛋白控制时,两条链可以分离开来。
以这种方式在双链构建体上连接和询问两条链增加了表征的效率和准确性。
桥接部分能够连接目标多核苷酸的两条链。桥接部分通常共价连接目标多核苷酸的两条链。假如桥接部分不干扰单链多核苷酸移动通过跨膜孔,则桥接部分可以是任何能够连接目标多核苷酸的两条链的物质。
桥接部分可以通过本领域已知的任一合适方法与目标多核苷酸连接。桥接部分可以单独合成并且化学连接或酶连接到目标多核苷酸。替代地,桥接部分可以在处理目标多核苷酸时产生。
桥接部分在目标多核苷酸的一端或靠近目标多核苷酸的一端与目标多核苷酸连接。桥接部分优选在目标多核苷酸末端的10个核苷酸内与目标多核苷酸连接。
合适的桥接部分包括但不限于聚合物连接体、化学连接体、多核苷酸或多肽。优选地,桥接部分包括dna、rna、改性dna(例如,脱碱基dna)、rna、pna、lna或peg。桥接部分更优选为dna或rna。
桥接部分最优选是发夹环或发夹环适配器。可以使用本领域已知的方法来设计合适的发夹适配器。发夹环可以是任何长度。发夹环长度通常为110个或更少的核苷酸,例如,100个或更少的核苷酸、90个或更少的核苷酸、80个或更少的核苷酸、70个或更少的核苷酸、60个或更少的核苷酸、50个或更少的核苷酸、40个或更少的核苷酸、30个或更少的核苷酸、20个或更少的核苷酸或10个或更少的核苷酸。发夹环的长度优选为约1至10、2至100、5至80或6至50个核苷酸。如果所述环涉及适配器的差分选择性,则优选长度较长的发夹环,例如,50至110个核苷酸。类似地,如下所述,如果所述环不涉及如下所述的选择性结合,则优选长度较短的发夹环,例如,1至5个核苷酸。
发夹适配器可以与第一和/或第二多核苷酸的任一端,即,5’或3’末端,连接。可以使用本领域已知的任何方法将发夹适配器与第一和/或第二多核苷酸连接。可以使用连接酶连接发夹适配器,例如,t4dna连接酶、大肠杆菌dna连接酶、taqdna连接酶、tmadna连接酶和9°ndna连接酶。
可以使用本领域已知的任何方法将多核苷酸的两条链分离开来。例如,它们可以被多核苷酸结合蛋白或者利用有利于脱杂合的条件(有利于脱杂合的条件的实例包括但不限于高温、高ph以及添加可以破坏氢键合或碱基配对的试剂,如甲酰胺和脲)分离开来。
发夹适配器优选包括选择性结合部分。这使得第一和/或第二多核苷酸能够被纯化或分离。选择性结合部分为可以基于其结合性质进行选择的部分。因此,选择性结合部分优选为特异性与表面结合的部分。如果选择性结合部分与表面结合的程度比本发明中使用的任何其它部分更大,则所述选择性结合部分与表面特异性结合。在优选实施例中,所述部分与本发明中使用的任何其它部分所未结合的表面结合。
合适的选择性结合部分是本领域已知的。优选的选择性结合部分包括但不限于:生物素、多核苷酸序列、抗体、抗体片段,例如,fab和scsv,抗原、多核苷酸结合蛋白、多组氨酸尾部和gst标签。最优选的选择性结合部分为生物素和选择性多核苷酸序列。生物素特特异性地与涂覆有亲和素的表面结合。选择性多核苷酸序列特异性地与涂覆有同源序列的表面结合(即,杂合)。替代地,选择性多核苷酸序列特异性地与涂覆有多核苷酸结合蛋白的表面结合。
发夹适配器和/或选择性结合部分可以包括能够切割、切开(nicked)、裂解或水解的区域。这种区域可以设计为,使第一和/或第二多核苷酸在纯化或分离后从其所结合的表面去除。合适的区域是本领域已知的。合适的区域包括但不限于rna区域、包括脱硫生物素和链霉亲和素的区域、二硫键和光可裂解区域。
双链目标多核苷酸优选地在桥接部分的相对端包括前导序列,例如,发夹环或发夹环适配器。前导序列将在以下进行更详细的讨论。
拐角测序(roundthecornersequencing)
在优选实施例中,目标双链多核苷酸的一端设置有桥接部分,例如,发夹环或发夹环适配器,并且所述方法包括使多核苷酸与孔接触,使得多核苷酸的两条链移动通过孔,以及在多核苷酸的两条链相对于孔移动时获取一个或多个测量值,其中,所述测量值指示多核苷酸的两条链的一个或多个特征,从而表征目标双链多核苷酸。在另一个优选实施例中,目标双链多核苷酸的一端设置有桥接部分,例如,发夹环或发夹环适配器,并且所述方法包括使多核苷酸与孔和外切核酸酶接触,使得多核苷酸的两条链均被消化从而形成单个核苷酸。上述任一实施方式同样适用于本实施方式。
前导序列
链表征/测序方法中的接触步骤之前,所述方法优选包括使多核苷酸与优先旋入孔的前导序列连接。前导序列有利于本发明方法。前导序列被设计为优先旋入孔中,从而促进多核苷酸通过孔的移动。前导序列也可以用于将多核苷酸与如上所述的一个或多个锚连接。
前导序列通常包括聚合物。所述聚合物优选是带负电荷的。所述聚合物优选为多核苷酸(例如dna或rna)、改性多核苷酸(例如,脱碱基dna)、pna、lna、聚乙二醇(peg)或多肽。前导序列优选包括多核苷酸,更优选地包括单链多核苷酸。前导序列可以包括上述任一多核苷酸。单链前导序列最优选包括单链dna,例如,聚dt区段。前导序列优选地包括所述一个或多个间隔区。
前导序列可以是任何长度,但是长度通常为10至150个核苷酸,例如,长度为20至150个核苷酸。前导序列的长度通常取决于所述方法中使用的跨膜孔。
前导序列优选为下文限定的y适配器的一部分。
双偶联
本发明的方法可以涉及双链多核苷酸的双偶联。在优选实施方式中,本发明的方法包括:
(a)在双链多核苷酸的一端设置y适配器,另一端设置诸如发夹环适配器等桥接部分适配器,其中,所述y适配器包括一个或多个用于将多核苷酸与膜偶联第一锚,所述桥接部分适配器包括一个或多个用于将多核苷酸与膜偶联的第二锚,其中,桥接部分适配器与膜偶联的强度大于y适配器与膜偶联的强度;
(b)使步骤(a)中提供的多核苷酸与csgg孔或其突变体,比如本发明的孔,相接触,使得多核苷酸相对于孔移动,例如,通过孔;以及
(c)在多核苷酸相对于孔移动时获取一个或多个测量值,其中,所述测量值指示多核苷酸的一个或多个特征,从而表征目标多核苷酸。
这种方法在申请号为1406147.7的英国申请中进行了详细讨论。
双链多核苷酸的一端设置有y适配器,另一端设置有桥接部分适配器。所述y适配器和/或桥接部分适配器通常是多核苷酸适配器。它们可以由上述任一多核苷酸形成。
y适配器通常包括(a)双链区域和(b)单链区域或在另一端不互补的区域。如果y适配器包括单链区域,则可以将其描述为具有突出端(overhang)。在y适配器中存在非互补区域使得适配器具有y形状,因为与双链部分不同,这两条链通常彼此不杂合。y适配器包括所述一个或多个第一锚。上文对锚进行了更详细的讨论。
y适配器优选地包括优先旋入孔的前导序列。这在上文中讨论过。
如上所述,桥接部分适配器优选包括选择性结合部分。如上所述,桥接部分适配器和/或选择性结合部分可以包括能够切割、切开、裂解或水解的区域。
如上所述,如果使用一个或多个解旋酶和一个或多个分子制动剂,则y适配器优选地包括所述一个或多个解旋酶,并且桥接部分适配器优选包括所述一个或多个分子制动剂。
可以使用本领域已知的任何方法将y适配器和/或桥接部分适配器与多核苷酸连接。可以使用诸如t4dna连接酶、大肠杆菌dna连接酶、taqdna连接酶、tmadna连接酶和9°ndna连接酶等连接酶连接所述适配器中的一者或两者。替代地,可以使用以下所讨论的本发明的方法将适配器添加到多核苷酸。
在优选实施例中,所述方法的步骤a)包括对双链多核苷酸进行改性,使得其一端包括y适配器,另一端包括桥接部分适配器。可以采用任何改性方式。所述方法优选包括根据本发明对双链多核苷酸进行改性。这将在以下进行更详细的讨论。改性和表征方法可以以任何方式进行组合。
桥接部分适配器与膜的偶联(或结合)强度大于y适配器与膜的偶联(或结合)强度。这可以以任何方式进行测量。申请号为1406147.7的英国申请的实施例中公开了一种用于测量偶联(或结合)强度的合适方法。
桥接部分适配器的偶联(或结合)强度优选为y适配器偶联(或结合)强度的至少1.5倍,例如,为锚适配器的偶联(或结合)强度的至少两倍、至少三倍、至少四倍、至少五倍或至少十倍。用于膜的桥接部分适配器的亲和常数(kd)优选为y适配器的亲和常数的至少1.5倍,例如为y适配器偶联强度的至少两倍,至少三次,至少四次,至少五次或至少十倍。
桥接部分适配器比y适配器更强地与膜偶联(或结合)的方式有多种。例如,桥接部分适配器可以包括比y适配器更多的锚。例如,桥接部分适配器可以包括2,3或更多个第二锚,而y适配器可以包括一个第一锚。
所述一个或多个第二锚与膜偶联(或结合)的强度可以大于所述一个或多个第一锚与膜偶联(或结合)的强度。所述一个或多个第二锚与桥接部分适配器偶联(或结合)的强度可以大于所述一个或多个第一锚与y适配器偶联(或结合)的强度。所述一个或多个第一锚和所述一个或多个第二锚可以通过杂合与其各自的适配器连接,并且所述一个或多个第二锚中的杂合强度比所述一个或多个第一锚中的杂合强度大。这些实施例的任一组合也可用于本发明。可以使用本领域的已知技术来测量偶联(或结合)强度。
所述一个或多个第二锚优选地包括一个或多个与膜偶联(或结合)的基团,其强度大于所述一个或多个与膜偶联(或结合)的第一锚中的一个或多个基团。在优选实施例中,采用胆固醇将桥接部分适配器/一个或多个第二锚与膜偶联(或结合),采用棕榈酸酯将y适配器/一个或多个第一锚与膜偶联(或结合)。胆固醇比棕榈酸酯能更强地与三嵌段共聚物膜和脂质膜结合。在替代实施例中,桥接部分适配器/一个或多个第二锚采用诸如棕榈酸酯等单酰基物质与膜偶联(或结合),而y适配器/一个或多个第一锚采用诸如二棕榈酰卵磷脂等二酰基物质与膜偶联(或结合)。
添加发夹环和前导序列
在提供之前,双链多核苷酸可以与mua转座酶和一组双链mua底物接触,其中,所述组中的一部分底物为包括前导序列的y适配器,并且所述组中的一部分底物为发夹环适配器。转座酶将双链多核苷酸分析物分裂成片段,并将mua底物与片段的一端或两端连接。这产生了多个改性双链多核苷酸,其一端包括前导序列,另一端包括发夹环。然后,可以使用本发明的方法对改性双链多核苷酸进行研究。
所述组中的每个底物优选包括通用核苷酸的至少一个突出端,使得转座酶将模板多核苷酸分裂成片段,并将底物与双链片段的一端或两端连接,从而产生多个片段/底物构建体,其中,所述方法还包括将突出端与构建体中的片段连接,从而产生多个经改性的双链多核苷酸。以上对合适的通用核苷酸进行了讨论。突出端的长度优选为5个核苷酸。
替代地,组中的每个底物优选包括(i)至少一个突出端和(ii)在与所述至少一个突出端相同的链中的至少一个核苷酸,其包括不存在于模板多核苷酸中的核苷,使得转座酶将模板多核苷酸分裂成片段并将底物与双链片段的一端或两端连接,从而产生多个片段/底物构建体,所述方法还包括(a)通过选择性地去除所述至少一个核苷酸来从构建体中去除突出端,从而产生多个包括单链间隙的双链构建体;和(b)修复构建体中的单链间隙,从而产生多个经改性的双链多核苷酸。多核苷酸通常包括核苷脱氧腺苷(da)、脱氧尿苷(du)和/或胸苷(dt)、脱氧鸟苷(dg)和脱氧胞苷(dc)。不存在于多核苷酸中的核苷优选为脱碱基的腺苷(a)、尿苷(u)、5-甲基尿苷(m5u)、胞苷(c)或鸟苷(g),或包括脲、5,6-二羟基胸腺嘧啶、胸腺嘧啶、5-羟基-5-甲基乙内酰脲(5-hydroxy-5methylhydanton)、尿嘧啶二醇、6-羟基-5,6-二氢硫胺、甲基丙醇二酰脲(tartronylurea)、7,8-二氢-8-氧鸟嘌呤(8-氧鸟嘌呤)、8-氧代腺嘌呤、fapy-鸟嘌呤、甲基-fapy-鸟嘌呤、fapy-腺嘌呤、黄曲霉毒素b1-fapy-鸟嘌呤、5-羟基胞嘧啶、5-羟基尿嘧啶、3-甲基腺嘌呤、7-甲基鸟嘌呤、1,n6-亚乙烯基腺嘌呤、次黄嘌呤、5-羟基尿嘧啶、5-羟基甲基尿嘧啶、5-甲酰基尿嘧啶或顺式-环丁烷嘧啶二聚体。所述至少一个核苷酸优选为所述突出端中的10个或更少个核苷酸。所述至少一个核苷酸为突出端中的第一个核苷酸。突出端中的所有核苷酸优选包括不存在于模板多核苷酸中的核苷。
这些基于mua的方法在申请号为pct/gb2014/052505的国际申请中进行了公开。这些方法也在申请号为1406147.7的英国申请中进行了详细讨论。
一个或多个解旋酶可以在其与双链多核苷酸和mua转座酶接触之前与mua底物y适配器连接。替代地,一个或多个解旋酶可以在其与双链多核苷酸和mua转座酶接触之前与mua底物y适配器连接。
一个或多个分子制动剂可以在其与双链多核苷酸和mua转座酶接触之前与mua底物发夹环适配器连接。替代地,一个或多个分子制动剂可以在其与双链多核苷酸和mua转座酶接触之前与mua底物发夹环适配器连接。
解偶联
本发明所述方法可以涉及表征多个目标多核苷酸和解偶联至少第一目标多核苷酸。
在优选实施例中,本发明涉及表征两个或更多个目标多核苷酸。该方法包括:
(a)在第一样本中提供第一多核苷酸;
(b)在第二样本中提供第二多核苷酸;
(c)采用一个或多个锚将所述第一样本中的所述第一多核苷酸与膜偶联;
(d)使所述第一多核苷酸与csgg孔或其突变体例如本发明的孔接触,使得多核苷酸相对于孔移动,例如,通过孔;
(e)当所述第一多核苷酸相对于孔移动时获取一个或多个测量值,其中,所述测量值指示所述第一多核苷酸的一个或多个特征,从而表征所述第一多核苷酸;
(f)将所述第一多核苷酸从膜中解偶联;
(g)采用一个或多个锚将所述第二样本中的所述第二多核苷酸与膜偶联;
(h)使所述第二多核苷酸与csgg孔或其突变体例如本发明的孔接触,使得所述第二多核苷酸相对于孔移动,例如,通过孔;以及
(i)当所述第二多核苷酸相对于孔移动时获取一个或多个测量值,其中,所述测量值指示所述第二多核苷酸的一个或多个特征,从而表征所述第二个多核苷酸。
这种方法在申请号为1406155.0的英国申请中进行详细讨论。
步骤(f)(即,第一多核苷酸的解偶联)可以在步骤(g)之前(即,在将第二多核苷酸与膜偶联之前)执行。步骤(g)可以在步骤(f)之前执行。如果第二多核苷酸在第一多核苷酸解偶联之前与膜偶联,则步骤(f)优选包括选择性地将第一多核苷酸与膜解偶联(即,将第一多核苷酸而不是第二多核苷酸与膜解偶联)。技术人员可以设计一种实现选择性解偶联的系统。步骤(f)和(g)可以同时执行。这将在以下进行更详细的讨论。
在步骤(f)中,优选至少10%的第一多核苷酸从膜中解偶联。例如,至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%或至少95%的第一多核苷酸可以从膜中解偶联。优选地,所有的第一多核苷酸从膜中解偶联。可以使用孔来确定从膜中解偶联的第一多核苷酸的数量。这在各实施例中进行公开。
第一多核苷酸和第二多核苷酸可以彼此不同。替代地,第一和第二多核苷酸可以是不同的多核苷酸。在这种情况下,在添加第二多核苷酸之前可以无需去除至少部分所述第一样本。这将在以下进行更详细的讨论。如果该方法涉及研究三个或更多个多核苷酸,则它们可以彼此不同,或者它们中的一些可能彼此不同。
第一多核苷酸和第二多核苷酸可以是同一多核苷酸的两个实例。第一多核苷酸可以与第二多核苷酸相同。这使得可以校阅(proofreading)。如果该方法涉及研究三个或更多个多核苷酸,则它们可以全部为同一多核苷酸的三个或更多个实例,或者它们中的一些可以是同一多核苷酸的不同实例。
第一样本和第二样本可以彼此不同。例如,第一样本可以来源于人,第二样本可以来源于病毒。如果第一和第二样本彼此不同,则它们可以含有或被怀疑含有相同的第一和第二多核苷酸。如果所述方法涉及研究三个或更多个样本,则它们可以全部彼此不同,或者它们中的一些可以彼此不同。
第一样本和第二样本优选为同一样本的两个实例。第一样本优选与第二样本相同。这使得可以校阅。如果所述方法涉及研究三个或更多个样本,则它们可以全部是同一样本的三个或更多个实例,或者它们中的一些可以是同一样本的不同实例。
可以研究任一数目的多核苷酸。例如,本发明方法可以涉及表征3,4,5,6,7,8,9,10,20,30,50,100或更多个多核苷酸。如果使用本发明所述方法研究三个或更多个多核苷酸,则第二多核苷酸也从膜中解偶联,并针对第三多核苷酸添加必需的步骤数。对于四个或更多个多核苷酸也是如此。
本发明所述方法涉及将第一多核苷酸从膜中解偶联。如果正在研究三个或更多个多核苷酸,则本发明所述方法可以涉及将第二多核苷酸从膜中解偶联。
可以采用任何已知的方法将第一多核苷酸从膜中解偶联。优选地,不采用跨膜孔将第一多核苷酸从步骤(f)中的膜解偶联。优选地,不采用电压或施加电势将第一多核苷酸从膜中解偶联。
步骤(f)优选包括通过从膜中去除一个或多个锚来使第一多核苷酸从膜中解偶联。如果锚被去除,则采用其它(或单独的)锚将第二多核苷酸与膜偶联。用于偶联第二多核苷酸的锚可以与用于偶联第一多核苷酸的锚类型相同或可以为不同类型的锚。
步骤(f)更优选地包括使一个或多个锚与一种试剂接触,所述试剂对于一个或多个锚的亲和性比锚对于膜的亲和性高。各种用于竞争性结合或免疫放射测定法以确定分子的特定结合能力的方案是本领域公知的(例如,参见maddox等人,j.exp.med.158,1211-1226,1993)。该试剂将锚从膜中去除,从而使第一多核苷酸解偶联。该试剂优选为糖。可以采用任何这样的糖:其与一个或多个锚结合的亲和性要比一个或多个锚对于膜的亲和性高。所述糖可以是如下所述的环糊精或其衍生物。
如果一个或多个锚包括疏水锚,例如,胆固醇,则所述试剂优选为环糊精或其衍生物或脂质。环糊精或其衍生物可以是eliseev,a.v.和schneider,h-j(1994)j.am.chem.soc.116,6081-6088中公开的任一种。所述试剂更优选为七-6-氨基-β-环糊精(am7-βcd)、6-单脱氧-6-单氨基-β-环糊精(am1-βcd)或七-(6-脱氧-6-胍基)-环糊精(gu7-βcd)。可以使用本文中公开的任何脂质。
如果锚包括链霉亲和素、生物素或脱硫生物素,则该试剂优选为生物素、脱硫生物素或链霉亲和素。生物素和脱硫生物素与链霉亲和素结合的亲和性均高于链霉亲和素与膜结合的亲和性,反之亦然。生物素比脱硫生物素具有更强的对链霉亲和素的亲和性。因此,可以使用生物素或链霉亲和素将包括链霉亲和素的锚从膜中去除,反之亦然。
如果锚包括蛋白,则所述试剂优选为与蛋白特异性结合的抗体或其片段。如果抗体优先或以高亲和性与蛋白结合而不是仅以低亲和性与其它或不同蛋白结合,则所述抗体与蛋白特异性结合。如果抗体以1×10-6m或更小的,更优选地1×10-7m或更小的,5×10-8m或更小的,更优选地1×10-8m或更小的或更优选地5×10-9m或更小的kd结合,则其优先或以高亲和性结合。如果抗体以1×10-6m或更多的,更优选地1×10-5m或更多的,更优选地1×10-4m或更多的,更优选地1×10-3m或更多的,进一步优选地1×10-2m或更多的kd结合,则其是以低亲和性结合。任何方法可用于检测结合或特异性结合。定量测量抗体与蛋白结合的方法是本领域已知的。抗体可以是单克隆抗体或多克隆抗体。抗体的合适片段包括但不限于fv、f(ab’)和f(ab’)2片段以及单链抗体。此外,抗体或其片段可以是嵌合抗体或其片段、cdr移植抗体或其片段或人源化抗体(humanisedantibody)或其片段。
步骤(f)优选包括使所述一个或多个锚与一种能降低所述一个或多个锚与膜偶联的能力的试剂接触。例如,所述试剂可能干扰一个或多个锚的结构和/或疏水性,并从而降低它们与膜偶联的能力。如果锚包括胆固醇,则所述试剂优选为胆固醇脱氢酶。如果锚包括脂质,则所述试剂优选为磷脂酶。如果锚包括蛋白,则所述试剂优选为蛋白酶或脲。合适的锚和试剂的其它组合对于本领域技术人员而言是清楚的。
步骤(f)更优选地包括通过使第一多核苷酸与一个或多个锚分离来将第一多核苷酸从膜中解偶联。这可以以任何方式完成。例如,连接体可以在包括连接体的锚中被切割。本实施例特别适用于涉及通过杂合而连接的锚。这种锚在上文中进行了讨论。
步骤(f)更优选地包括通过使第一多核苷酸及一个或多个锚与试剂接触来将第一多核苷酸从膜中解偶联,其中,所述试剂与第一多核苷酸竞争以便与一个或多个锚结合。竞争性结合的确定和测量方法是本领域已知的。所述试剂优选为与第一多核苷酸竞争以便与所述一个或多个锚杂合的多核苷酸。例如,如果采用一个或多个涉及杂合的锚将第一多核苷酸与膜偶联,则可以通过使所述一个或多个锚与同样杂合到杂合位点的多核苷酸接触来对多核苷酸进行解偶联。通常,以高于第一多核苷酸和一个或多个锚浓度的浓度添加多核苷酸试剂。替代地,与第一多核苷酸相比,多核苷酸试剂可以更强地与一个或多个锚杂合。
步骤(f)更优选地包括(i)使第一多核苷酸及一个或多个锚与脲、三(2-羧基乙基)膦(tcep)、二硫苏糖醇(dtt)、链霉亲和素或生物素、uv光、酶或结合剂接触;(ii)加热第一多核苷酸和一个或多个锚;或(iii)改变ph值。脲、三(2-羧基乙基)膦(tcep)或二硫苏糖醇(dtt)能够破坏锚并将第一多核苷酸与膜分离。如果锚包括链霉亲和素-生物素连接,则链霉亲和素试剂争取与生物素结合。如果锚包括链霉亲和素-脱硫生物素连接,则生物素试剂争取与链霉亲和素结合。uv光可用于降解光不稳定基团。酶和结合剂可用于切割、分解或解开锚。优选的酶包括但不限于外切核酸酶、内切核酸酶或解旋酶。优选的结合剂包括但不限于酶、抗体或其片段或单链结合蛋白(ssb)。可以使用任何以下讨论的酶或上述讨论的抗体。热量和ph可用于破坏杂合和其它连接。
如果通过将第一多核苷酸与一个或多个锚分离来将第一多核苷酸从膜中解偶联,则一个或多个锚将保留在膜中。步骤(g)优选地包括采用与第一多核苷酸分离的一个或多个锚将第二多核苷酸与膜偶联。例如,第二多核苷酸还可以具有一个或多个与保留在膜中的一个或多个锚杂合的多核苷酸。替代地,步骤(g)优选地包括采用与第一多核苷酸(即一个或多个其它锚)分离的锚中的一个或多个单独的锚将第二多核苷酸与膜偶联。所述一个或多个单独的锚可以与将第一多核苷酸与膜偶联的锚是同一类型,或者可以是不同类型的锚。步骤(g)优选地包括采用一个或多个锚将第二多核苷酸与膜偶联,其中所述一个或多个锚不同于与第一多核苷酸分离的一个或多个锚。
在优选实施例中,步骤(f)和(g)包括通过使膜与第二多核苷酸接触使得第二多核苷酸与第一多核苷酸竞争以便与一个或多个锚结合并取代第一多核苷酸来将第一多核苷酸从膜中解偶联。例如,如果采用一个或多个涉及杂合的锚来将第一多核苷酸与膜偶联,则可以通过使锚与连接到多核苷酸(其也与一个或多个锚中的杂合位点杂合)的第二多核苷酸接触来对第一多核苷酸进行解偶联。通常以高于第一多核苷酸和一个或多个锚浓度的浓度添加第二多核苷酸。替代地,相比第一多核苷酸,第二多核苷酸可以更强地与一个或多个锚杂合。
去除或洗涤
虽然在步骤(f)中将第一多核苷酸从膜中解偶联,但其并不一定被去除或洗去。如果可以容易地将第二多核苷酸与第一多核苷酸区分开来,则不需要去除第一多核苷酸。
在步骤(f)和(g)之间,所述方法优选地还包括从膜中去除至少一些第一样本。可以去除至少10%的第一样本,例如,可以去除至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%或至少90%的第一样本。
所述方法更优选地还包括从膜中去除所有第一样本。这可以以任何方式完成。例如,在第一多核苷酸进行解偶联后,可以用缓冲液洗涤膜。以下对合适的缓冲液进行讨论。
改性的多核苷酸
在表征之前,在使用目标多核苷酸作为模板将聚合酶形成改性多核苷酸的条件下,可以通过使多核苷酸与聚合酶和一组游离核苷酸接触来对目标多核苷酸进行改性,其中,当形成改性多核苷酸时,所述聚合酶用不同核苷酸种类替代目标多核苷酸中的一个或多个核苷酸种类。然后,可以向改性多核苷酸提供与多核苷酸连接的一个或多个解旋酶以及与多核苷酸连接的一个或多个分子制动剂。这种类型的改性在申请号为1403096.9的英国申请中进行了描述。可以采用上述任一聚合酶。聚合酶优选为克列诺酶(klenow)或9°north。
在使用模板多核苷酸作为模板将聚合酶形成改性多核苷酸的条件下,使模板多核苷酸与聚合酶接触。这些条件是本领域已知的。例如,多核苷酸通常与市售的聚合酶缓冲液中的聚合酶接触,例如,来自newengland
使用跨膜孔进行的多核苷酸表征,如测序,通常包括对由k个核苷酸组成的聚合物单元进行分析,其中,k是正整数(即,‘k-聚体(k-mers)’)。这在申请号为pct/gb2012/052343(公开为wo2013/041878)的国际申请中进行了讨论。尽管希望在不同k-聚体的电流测量值之间有明确的分离,但是这些测量值中的一些发生重叠是很常见的。特别是k-聚体中具有高数量的聚合物单元,即,高k值,分辨不同k-聚体产生的测量值变得比较困难,从而不利于得到有关多核苷酸的信息,例如,估计多核苷酸的潜在序列。
通过用改性多核苷酸中的不同核苷酸种类替代目标多核苷酸中的一个或多个核苷酸种类,所述改性多核苷酸包含与目标多核苷酸不同的k-聚体。改性多核苷酸中的不同k-聚体能够产生与目标多核苷酸中的k-聚体不同的电流测量值,因此改性多核苷酸提供与目标多核苷酸不同的信息。来自改性多核苷酸的附加信息可以使得更容易表征目标多核苷酸。在一些情况下,改性多核苷酸本身可能更易于表征。例如,改性多核苷酸可以设计为包括电流测量值之间具有增加的分离或清楚的分离的k-聚体,或具有降低的噪声的k-聚体。
当形成改性多核苷酸时,聚合酶优选用不同核苷酸种类替代目标多核苷酸中的两个或更多个核苷酸。聚合酶可以用不同的核苷酸种类替代目标多核苷酸中的所述两个或更多个核苷酸种类的每一个。聚合酶可以用同一核苷酸种类替代目标多核苷酸中的所述两个或更多个核苷酸种类的每一个。
如果目标多核苷酸为dna,则改性多核苷酸中的不同核苷酸种类通常包括不同于腺嘌呤、鸟嘌呤、胸腺嘧啶、胞嘧啶或甲基胞嘧啶的核碱基和/或包括不同于脱氧腺苷、脱氧鸟苷、胸苷、脱氧胞苷或脱氧甲基胞苷的核苷。如果目标多核苷酸为rna,则改性多核苷酸中的不同核苷酸种类通常包括不同于腺嘌呤、鸟嘌呤、尿嘧啶、胞嘧啶或甲基胞嘧啶的核碱基和/或包括不同于腺苷、鸟苷、尿苷、胞苷或甲基胞苷的核苷。不同的核苷酸种类可以是上述任一通用核苷酸。
聚合酶可以用不同的核苷酸种类替代一个或多个核苷酸种类,其中,所述不同的核苷酸种类包括所述一个或多个核苷酸种类中不存在的化学基团或原子。化学基团可以是丙炔基、硫基、氧代基、甲基、羟甲基、甲酰基、羧基、羰基、苄基、炔丙基或炔丙胺基。
聚合酶可以用不同核苷酸种类替代一个或多个核苷酸种类,其中,所述不同核苷酸种类缺少所述一个或多个核苷酸种类中存在的化学基团或原子。聚合酶可以用具有改变的电负性的不同核苷酸种类替代一个或多个核苷酸种类。具有改变的电负性的不同核苷酸种类优选地包括卤素原子。
所述方法优选地还包括选择性地从改性多核苷酸的一个或多个不同核苷酸种类中去除核碱基。
分析物输送
目标分析物优选地与将分析物向膜输送的微粒连接。这种类型的输送在申请号为1418469.1的英国申请中进行了公开。
可以使用任何类型的微粒和连接方法。
其它表征方法
在另一实施例中,通过检测聚合酶添加给目标多核苷酸的标记种类然后进行释放来对多核苷酸进行表征。聚合酶使用多核苷酸作为模板。每个标记种类对每个核苷酸具有特异性。多核苷酸与csgg孔或其突变体例如本发明所述孔、聚合酶以及标记的核苷酸接触,使得磷酸酯标记物质由聚合酶顺序地加入到多核苷酸,其中,磷酸酯物质包含对每个核苷酸特异性的标记。标记的物质可以在其从核苷酸释放之前(即,当其加入到目标多核苷酸中时)或在其从核苷酸释放之后采用孔进行检测。
聚合酶可以是上述任一种。采用孔检测磷酸酯标记物质,从而表征多核苷酸。这种方法在申请号为13187149.3(公开为ep2682460)的欧洲申请中进行了公开。上述任一实施例同样适用于该方法。
标记的物质的实例包括但不限于聚合物、聚乙二醇、糖、环糊精、荧光团、药物、代谢物、肽。这种标签的非限制性实例可以在kumar等人scirep.2012;2:684电子出版2012年9月21日的著作中发现。
传感器的形成方法
本发明还提供了形成传感器以用于表征目标多核苷酸的方法。所述方法包括在诸如本发明所述孔等csgg孔或其突变体与诸如解旋酶或外切核酸酶等多核苷酸结合蛋白之间形成复合物。可以通过在存在目标多核苷酸的情况下使孔和蛋白接触,然后在孔上施加电势来形成复合物。施加的电势可以是如上所述的化学电势或电压电势。替代地,可以通过使孔与蛋白共价连接来形成复合物。用于共价连接的方法是本领域已知的并在以下国际申请中进行了公开:例如,申请号为pct/gb09/001679(公开为wo2010/004265)和pct/gb10/000133(公开为wo2010/086603)的国际申请。复合物是一种用于表征目标多核苷酸的传感器。所述方法优选地包括在诸如本发明所述孔等csgg孔或其突变体和解旋酶之间形成复合物。上述任一实施例同样适用于该方法。
本发明还提供了一种用于表征目标多核苷酸的传感器。传感器包括在诸如本发明所述孔等csgg孔或其突变体与多核苷酸结合蛋白之间的复合物。上述任一实施例同样适用于本发明所述传感器。
试剂盒
本发明还提供了一种用于表征目标多核苷酸的试剂盒。试剂盒包括csgg孔或其突变体,例如,本发明所述孔,以及膜的组分。膜优选由各组分形成。孔优选存在于膜中。试剂盒可以包括上述任一膜的组分,例如,两亲层或三嵌段共聚物膜。
试剂盒还可以包括多核苷酸结合蛋白。可以使用上述任一多核苷酸结合蛋白。
试剂盒还可以包括一个或多个用于将多核苷酸与膜偶联的锚。
试剂盒优选用于表征双链多核苷酸,并且优选包括y适配器和发夹环适配器。y适配器优选具有一个或多个连接的解旋酶,发夹环适配器优选地具有一个或多个连接的分子制动剂。y适配器优选包括一个或多个用于将多核苷酸与膜偶联的第一锚,发夹环适配器优选地包括一个或多个用于将多核苷酸与膜偶联的第二锚,并且发夹环适配器与膜的偶联强度优选地大于y适配器与膜的偶联强度。
本发明所述试剂盒可以另外包括一个或多个能够使上述任一实施例得以执行的其它试剂或仪器。这种试剂或仪器包括以下一种或多种:合适的缓冲液(水性溶液)、从受体(例如,血管或包括针头的仪器)获得样本的装置、用于扩增和/或表达多核苷酸的装置或电压或膜片钳装置。试剂可以以干燥状态存在于试剂盒中,使得流体样本使试剂重新悬浮。试剂盒还可以可选地包括能够使试剂盒用于本发明所述方法的说明或有关所述方法可以用于哪种生物体的细节。
设备
本发明还提供了一种用于表征目标分析物(例如,目标多核苷酸)的设备。所述设备包括多个csgg孔或其突变体和多个膜。所述多个孔优选存在于所述多个膜中。孔和膜的数量优选相等。优选地,每个膜中存在单个孔。
所述设备优选地还包括用于实施本发明所述方法的指令。所述设备可以是任一用于分析物分析的常规设备,例如,阵列或芯片。结合本发明所述方法所讨论的任一实施例同样适用于本发明所述设备。所述设备还可以包括本发明所述试剂盒中存在的任何特征。
优选地,对所述设备进行设置以便执行本发明所述方法。
所述设备优选地包括:
传感器装置,其能够支撑多个孔和膜并且可操作地采用孔和膜进行分析物表征;和
至少一个端口,用于传送用以进行表征材料。
替代地,所述设备优选地包括:
传感器装置,其能够支撑多个孔和膜并可操作地采用孔和膜进行分析物表征;和
至少一个储存器,用于容纳用以进行表征的材料。
所述设备更优选地包括:
传感器装置,其能够支撑膜和多个孔,并且可操作地采用孔和膜进行分析物表征;
至少一个储存器,用于容纳用以进行表征的材料;
流体系统,用于可控地将材料从所述至少一个储存器向所述传感器装置供应;和
一个或多个容器,用于接收相应的样本,所述流体系统用于选择性地将样本从所述一个或多个容器向所述传感器装置供应。
所述设备可以是以下国际申请中描述的任一种:申请号为pct/gb08/004127(公开为wo2009/077734),pct/gb10/000789(公开为wo2010/122293),申请号为pct/gb10/002206(公开wo2011/067559)或申请号为pct/us99/25679(公开为wo00/28312)的国际申请。
下列实施例对本发明进行说明。
实施例
实施例1:克隆和csgg菌株
已经描述了用于生产外膜局部c-末端strepii标记的csgg(ppg1)和周质c-末端strepii标记的csggc1s(ppg2)的表达构建体(goyal,p.etal.,actacrystallogr.f.struct.biol.cryst.commun.2013,69,1349-1353)。对于硒代甲硫氨酸(selenomethionine)标记,strepii标记的csggc1s由于产率增加而在细胞质中进行表达。因此,对ppg2进行改变以便采用带有引物5’-tctttaaccgccccgcctaaag-3’(正向)(seqidno:437)和5’-cattttttgccctcgttatc-3’(反向)(ppg3)(seqidno:438)的反向pcr去除n末端信号肽。对于表型测定,通过所描述的方法[datsenko,k.a.etal.,proc.natlacad.sci.usa97,6640-6645(2000)]构建大肠杆菌bw25141(大肠杆菌nvg2)的csgg缺失突变体(用引物5’-aataactcaaccgatttttaagccccagcttcataaggaaaataatcgtgtaggctggagctgcttc-3’(seqidno:439)和5’-cgcttaaacagtaaaatgccggatgataattccggcttttttatctgcatatgaatatcctccttag-3’(seqidno:440))。在trc启动子控制下,通过始于pmc2(含有csgg的ptrc99a载体)的定点诱变(quikchange方案;stratagene)来构建用于cys可接近性试验并用于通道收缩结构的表型探测的各种csgg取代型突变体(robinson,l.s.etal.,mol.microbiol.2006,59,870-881)。
实施例2:蛋白的表达和纯化
如所述(robinson,l.s.etal.,mol.microbiol.2006,59,870-881)对csgg和csggc1s进行表达和纯化。简言之,在大肠杆菌bl21(de3)中重组产生csgg,其中,所述大肠杆菌bl21(de3)采用ppg1转化并使用缓冲液a(50mmtris-hclph8.0,500mmnacl,1mmedta;1mm二硫苏糖醇(dtt))中1%的正十二烷基-β-d-麦芽糖苷(ddm)从分离的外膜中进行提取。将strepii标记的csgg装载到5毫升strep-tactin琼脂糖柱(ibagmbh)上,并通过用20个柱体积的补充有0.5%的四甘醇单辛基醚(c8e4;affymetrix)和4mm月桂基二甲基胺-n-氧化物(ldao;affymetrix)的缓冲液a洗涤来进行洗涤剂交换。将蛋白通过添加2.5mmd-脱硫生物素进行洗脱,并浓缩至5mgml-1用于结晶实验。对于硒代甲硫氨酸标记,在用ppg3转化并在补充有40mgl-1l-硒代甲硫氨酸的m9基本培养基上生长的met营养缺陷型菌株b834(de3)中产生csggc1s。使细胞颗粒(cellpellet)重悬于补充有完整的蛋白酶抑制剂混合物(proteaseinhibitorcocktail(roche))的50mmtris-hclph8.0,150mmnacl,1mmedta,5mmdtt中,并通过穿过以20×103lbin-2操作的ts系列细胞破碎剂(constantsystemsltd)而破碎。如所述(robinson,l.s.etal.,mol.microbiol.2006,59,870-881),对标记的csggc1s进行纯化。在整个纯化过程中加入dtt(5mm)以避免硒代甲硫氨酸的氧化。
在携带有pnh27的大肠杆菌nebc2566细胞中产生csge(nenninger,a.a.etal.,mol.microbiol.2011,81,468-499)。将25mmtris-hclph8.0,150mmnacl,25mm咪唑,5%(v/v)glycerol甘油中的细胞溶解产物装载在histrapff上(gehealthcare)。用在20mmtris-hciph8.0,150mmnacl,5%(v/v)甘油缓冲液中的500mm咪唑采用线性梯度对csge-组氨酸(csge-his)进行洗脱。对含有csge的馏分补充250mm(nh4)2so4,并施加到用20mmtris-hclph8.0,100mmnacl,250mm(nh4)2so4、5%(v/v)甘油平衡的5mlhitrap苯基hp柱(gehealthcare)上。用20mmtris-hclph8.0,10mmnacl,5%(v/v)甘油以线性梯度进行洗脱。将含有馏分的csge装载到superose6prepgrade10/600柱(gehealthcare)上,所述柱在20mmtris-hclph8.0,100mmnacl,5%(v/v)甘油中平衡。
实施例3:溶液中寡聚状态评估
将各约0.5mg的洗涤剂溶解的csgg(0.5%c8e4,4mmldao)和csggc1s施用到用25mmtris-hclph8.0,500mmnacl,1mmdtt,4mmldao和0.5%c8e4(csgg)平衡或用25mmtris-hclph8.0,200mmnacl(csggc1s)平衡的superdex20010/300gl分析凝胶过滤柱(gehealthcare)上,并以0.7mlmin-1运行。用牛甲状腺球蛋白、牛γ-球蛋白、鸡卵清蛋白、马肌球蛋白和维生素b12(bio-rad)对柱洗脱体积进行校准(图7)。采用swamy,m.,etal.,sci.stke2006,pl4[http://dx.doi.org/10.1126/stke.3452006pl4(2006)]中所描述的程序,将膜提取的csgg,即20μg洗涤剂溶解的蛋白,也在3-10%蓝色天然page上运行(图7)。nativemark(lifetechnologies)未染色蛋白标准物(7μl)用于分子质量估计。主要发现成熟的csgg是具有c9对称性的离散型九聚体成孔颗粒以及九聚体孔的尾对尾二聚体(即,具有d9对称性的十八聚体)。出于纳米孔感测应用的目的,蛋白的优选状态为单个九聚体孔。可以通过在体积排阻色谱法和/或插入用于纳米孔感测应用的脂质双层之前加热样本来增加九聚体孔对d9十八聚体孔的数量。
实施例4:结晶、数据收集和结构测定
通过对包含有100mmph值为4.2的乙酸钠、8%peg4000和100mmph值为7.0的丙二酸钠的溶液进行坐滴式(sitting-drop)蒸气扩散,将硒代甲硫氨酸标记的csggc1s浓缩至3.8mgml-1并进行结晶。在补充有15%甘油的晶体缓冲液中对晶体进行培养,并在液氮中快速冷冻。通过对包含有100mmtris-hclph8.0、8%peg4000、100mmnacl和500mmmgcl2的溶液进行悬滴式(hanging-drop)蒸气扩散,将洗涤剂溶解的csgg浓缩至5mgml-1并进行结晶。将晶体在液氮中快速冷冻并通过存在于晶体溶液中的洗涤剂进行冷冻保护。为了优化结晶条件并筛选具有良好衍射质量的晶体,在光束线proxima-1和proxima-2a(soleil,法国)、px-l(瑞士光源,瑞士)、102、103、104和124(钻石光源,英国)以及id14eh2、id23eh1和id23eh2(esrf,法国)上对晶体进行分析。分别在光束线104和103收集用于csggc1s和csgg结构测定的最终衍射数据(表5)。
csggc1s和膜提取的csgg(均从单个晶体收集)的数据统计;
*最高分辨率壳在括号中示出。
↑沿互逆方向a*,b*和c*上的各向异性截断的纠正值。
表5
采用xia2和xds封装(winter,g.,j.appl.cryst.,2010,43,186-190;kabsch,w.,actacrystallogr.dbiol.crystallogr.2010,66,125-132)处理csggc1s的衍射数据。csggc1s的晶体属于空间群p1,晶胞大小为
在空间群c2中,采用xds封装(winter,g.,j.appl.cryst.2010,43,186-190;kabsch,w.,actacrystallogr.dbiol.crystallogr.2010,66,125-132)从
实施例5:刚果红试验
为了分析刚果红结合,在lb(lysogenybroth)培养基中对lb中在37℃生长的细菌过夜培养物进行稀释,直至达到0.5的d600。在补充有氨苄青霉素(100mgl-1)、刚果红(100mgl-1)和0.1%(w/v)异丙基β-d-硫代半乳糖苷(iptg)的lb琼脂板上点样5μl样本。在室温(20-22℃)下将平板培养48小时以诱导卷曲菌毛表达。观察菌落形态发展和染色结合情况48h。
实施例6:半胱氨酸可接近性试验
采用定点突变在pmc2中产生半胱氨酸突变体,并在大肠杆菌lsr12(chapman,m.r.etal.,science,2002,295,851-855)中进行表达。将生长过夜的细菌培养物点样到含有1mmiptg和100mgl-1氨苄青霉素的lb琼脂板上。将平板在室温下培养,并在48小时后刮下细胞,重悬于1ml的pbs中并用d600归一化。通过超声降解法裂解细胞,并在4℃温度下以3,000g离心20秒,以便从细胞裂解液和悬浮膜中去除未破裂的细胞。将上清液中的蛋白在室温下用15mm甲氧基聚乙二醇-马来酰亚胺(mal-peg5kda)标记1小时。用100mmdtt终止反应,并在4℃温度下在50.4ti转子中以40,000r.p.m(~100,000g)离心20分钟以便将总的膜制成小球。用1%月桂酰肌氨酸钠洗涤所述小球以溶解细胞质膜并再次离心。将所得的外膜用含有1%ddm的pbs重新悬浮并溶解。使用镍珠的金属亲和性下拉(pulldowns)用于sds-page和anti-his蛋白质印迹。具有空载pmc2载体的大肠杆菌lsr12细胞用作阴性对照。
实施例7:atr-ftir光谱法
在equinox55红外光谱仪(bruker)上进行atr-ftir测量,连续用干燥空气吹扫,配备有液氮制冷型汞镉碲化物探测器和goldengate反射附件(specac)。内反射元件为金刚石晶体(2mm×2mm),光束入射角为45°。将每个纯化的蛋白样本(1μl)在晶体表面铺展并在气态氮气流下干燥以形成膜。以2cm-1分辨率记录的每个光谱为128个累计值的平均值用于提高的信噪比。所有光谱采用水蒸汽贡献减法来处理,通过衍射控象法(apodization)在最终分辨率为4cm-1情况下变平滑,并在amidei谱带(1,700-1,600cm-1)的区域上进行归一化以使其进行比较(goormaghtigh,e.;ruysschaert,j.m.,spectrochim.acta,1994,50a,2137-2144)。
实施例8:阴性染色em和对称性测定
阴性染色em用于监测csgg、csggc1s和csge的溶液中(in-solution)寡聚状态。将csge、csggc1s和两亲性结合(amphipol-bound)的csgg调节成0.05mgml-1的浓度,并施加到辉光放电的碳涂覆铜网格上(cf-400;electronmicroscopysciences)。培养1分钟后,将样本染上印记,然后在2%的乙酸双氧铀中洗涤并染色。在tecnait12biotwinlab6显微镜上收集图像,操作电压为120kv,放大倍数为×49,000,散焦为800至2,000nm之间。如对于cryo-em所述,执行对比传递函数(ctf)、相位翻转和颗粒选择。对于膜提取的(membrane-extracted)csgg,分别从九聚体和顶视图分析十八聚体颗粒(共1,780个)。对于纯化的csge,总共分析了2,452个颗粒。正如以下csgg-csgecryo-em分析所述,获得三维模型,并通过几轮多参考对准(multi-referencealignment,mra)、多统计分析(msa)和锚设置细化进行细化。在所有情况下,归一化和中心化后,采用如cryo-em部分中所述的imagic-4d对图像进行分类。对于每组颗粒,选择与特征视图对应的最佳分类。采用最佳分类平均值进行csgg顶视图的对称性测定,每类大约有20张图像。使用imagic计算旋转自相关函数并进行绘制。
实施例9:阴性染色em和对称性测定
阴性染色em用于监测csgg、csggc1s和csge的溶液中(in-solution)寡聚状态。将csge、csggc1s和两亲性聚合物结合(amphipol-bound)的csgg调节成0.05mgml-1的浓度,并施加到辉光放电的碳涂覆铜网上(cf-400;electronmicroscopysciences)。培养1分钟后,将样本染上印迹,然后在2%的乙酸双氧铀中洗涤并染色。在tecnait12biotwinlab6显微镜上收集图像,操作电压为120kv,放大倍数为×49,000,散焦为800至2,000nm之间。如对于cryo-em所述,执行对比传递函数(ctf)、相位翻转和颗粒选择。对于膜提取的csgg,分别通过九聚体和顶视图分析十八聚体颗粒(共1,780个)。对于纯化的csge,总共分析了2,452个颗粒。针对以下csgg-csgecryo-em分析所述,获得三维模型,并通过几轮多参考对准(multi-referencealignment,mra)、多统计分析(msa)和锚设置细化进行细化。在所有情况下,归一化和中心化后,采用如cryo-em部分中所述的imagic-4d对图像进行分类。对于每组颗粒,选择与特征视图对应的最佳分类。采用最佳分类平均值进行csgg顶视图的对称性测定,每类大约有20张图像。使用imagic计算旋转自相关函数并进行绘制。
实施例10:csgg-csge复合物形成
对于csgg-csge复合物形成,通过向300ml洗涤剂去稳定的脂质体(1mgml-11,2-二肉豆蔻酰-sn-甘油-3-磷酸胆碱(dmpc)和0.4%的ldao)添加120mlcsgg(0.5%c8e4中的24mgml-1蛋白、4mmldao、25mmph值为8.0的tris-hcl、500mmnacl、1mmdtt)并在加入90ml(在100mgml-1原料(stock)下)的a8-35两亲聚合物(a8-35amphipols)前在冰上培养5分钟将纯化的csgg中的增溶洗涤剂替换为两亲聚合物a8-35(anatrace)。在冰上培养另外15分钟后,将样本装载到superose610/300gl(gehealthcare)柱上,并在200mmnacl、2.5%木糖醇、25mmph为8的tris-hcl、0.2mmdtt中进行凝胶过滤。将200mmnacl、2.5%木糖醇、25mmph值为8的tris-hcl、0.2mmdtt中的等体积纯化单体csge以对于csge和csgg分别为15和5μm的最终蛋白浓度加入到两亲聚合物溶解的csgg,样本在温度为18℃、为、125v电压下在0.5×tbe缓冲液中4.5%的天然page上运行。对于第二个变性维度,将对应于csgg-csge复合物的条带从在同一凝胶上平行运行的未染色的泳道(lanes)中切除,在laemmli缓冲液(60mmph值为6.8的tris-hcl、2%的sds、10%甘油、5%的2-巯基乙醇、0.01%溴酚蓝)中煮沸5分钟,并在4-20%的sds-page上运行。纯化的csge和csgg作为对照样本与复合物一起运行。用考马斯亮蓝(instantbluecoomassie)对凝胶进行染色,以便进行目视检查,或用sypro橙染色,以便通过对sds-page上的csge和csgg条带进行荧光检测(typhoonfla9000)来对csgg-csge复合物进行化学计量评估,得到0.97的csgg/csge比。
实施例11:csgg-csgecryo-em
cryo电子显微镜用于测定c9csgg-csge复合物的溶液中(in-solution)结构。将如上所述制备的csgg-csge复合物与histrapff(gehealthcare)结合并洗脱以去除未结合的csgg,洗脱后立即将其应用于quantifoilr2/2碳涂覆网格(quantifoilmicrotoolsgmbh),所述quantifoilr2/2碳涂覆网格已在20ma下辉光放电30秒。使用自动化系统(leica)将样本冷冻浸入液氮中,并在feif20显微镜下观察,其操作电压为200kv,并且在低剂量条件下标称放大倍数为×50,000,散焦范围为1.4-3mm。在falconii探测器上记录图像帧。标本级别的像素大小为每像素
实施例12:csgg和csgg+csge孔的单通道电流分析
在负场电势下,csgg孔显示出两种电导状态。图5分别显示正常(在+50,0和-50mv下测量)和低电导形式(在0,+50和-50mv下测量)的代表性单通道电流迹线。在总观察时间(n=22)期间,没有观察到两种状态之间的转换,表明电导状态具有长的寿命(秒到分的时间量程)。左下图显示了在+50和-50mv(n=33)情况下获得的csgg孔的正常和低电导形式的电流直方图。右下图显示了具有规则和低电导率的csgg孔的i-v曲线。数据表示至少四个独立记录值的平均值和标准偏差。低电导形式的性质或生理存在是未知的。
图6显示了用辅助因子csge滴定的csgg通道的电生理学结果。该图将开放、中间和闭合通道的馏分显示为csge浓度的函数。csgg的开放和闭合状态如图6所示(分别为0nm和100nm的csge)。将csge的浓度增加到10nm以上导致csgg孔闭合。该效应发生在+50mv(左)和-50mv(右),排除了将csge(计算的pl为4.7)电泳到csgg孔中导致孔堵塞的可能性。不频繁(<5%)的中间状态的电导大约是开放通道的一半。这可能表示csge引起csgg通道不完全闭合;可选地,这可能表示电解质溶液中的残留csgg单体与膜嵌入孔结合而引起csgg二聚体的暂时形成。通过对单通道电流迹线进行全点直方图分析获得三种状态的馏分。直方图中产生的多达三种状态的峰值区域、给定状态的馏分通过将相应的峰值区域除以记录中所有其它状态的总和来获得。在负场电势下,可以看到两个开放电导状态,类似于csgg的观察结果(见a)。由于两个开放通道的变化都被较高的csge浓度阻断,图6(0nm)中的“开放”迹线结合了两种电导形式。图中的数据表示三个独立记录值的平均值和标准偏差。
晶体结构、尺寸排阻层析和em显示了洗涤剂提取的csgg孔在较高的蛋白浓度下形成非天然的尾对尾层叠式(stacked)二聚体(例如,两个九聚体作为d9颗粒,图7)。这些二聚体也可以在单通道记录中观察到。上图显示了+50,0和-50mv(从左到右)情况下的层叠式csgg孔的单通道电流迹线。左下图显示了+50和-50mv情况下记录的二聚csgg孔的电流直方图。在+50和-50mv情况下,+16.2±1.8和-16.0±3.0pa(n=15)的实验电导值分别接近理论计算值23pa。右下图显示了层叠式csgg孔的i-v曲线。数据表示六个独立记录值的平均值和标准偏差。
通过电生理学对csge结合和阻断层叠式csgg孔的能力进行测试。示出了在+50mv(上位)和-50mv(下位)情况下,在10或100nmcsge存在下,层叠式csgg孔的单通道电流迹线。电流痕迹表明,csge的其它饱和浓度不会导致层叠式csgg二聚体的孔闭合。由em和定点诱变可以看出,这些观察结果与csgg-csge接触区映射到螺旋物2以及csgg周质腔体的开口很好地吻合(图5和图6)。
实施例13:胆汁盐毒性试验
通过在含有胆汁盐的琼脂板上减慢生长来研究外膜渗透性。将大肠杆菌lsr12(chapman,m.r.etal.,science,2002,295,851-855)细胞的十倍连续稀释液(5ml)点样在mcconkey琼脂板上,其中,所述稀释液同时含有plr42(nenninger,a.a.etal.,mol.microbiol.,2011,81,486-499)和pmc2(robinson,l.s.etal.,mol.microbiol.2006,59,870-881)(或衍生的螺旋物2突变体),所述琼脂板含有100mgl-1氨苄青霉素、25mgl-1氯霉素、1mm含有或不含0.2%(w/v)l-阿拉伯糖的iptg。在37℃下培养过夜后,检查菌落生长。
实施例14:单通道电流记录
采用带有来自nanion的orbit16试剂盒的并行高分辨率电记录进行单通道电流记录。简言之,在16通道多电极空腔阵列(meca)芯片(lonera)(delriomartinez,j.m.,acsnanosmall,2014,5,8080-8088)中的(亚皮升体积的)微腔上形成1,2-二植烷酰基-sn-甘油基-3-磷酸胆碱的水平双层(avantipolarlipids)。双层上方和下方的顺式和反式腔均含有1.0mkcl、25mmph值为8.0的tris-hcl。为了将通道插入膜中,将溶解在25mmph值为8.0的tris-hcl、500mmnacl、1mmdtt、0.5%c8e4、5mmldao中的csgg加入到顺式腔中直至最终浓度为90-300nm。为了测试csgg通道与csge的相互作用,将溶解在25mmph值为8.0的tris-hcl、150mmnacl中的后一种蛋白的溶液加入到顺式腔中直至最终浓度为0.1、1、10和100nm。采用tecellatriton16通道放大器,在保持电势为+50mv和-50mv(顺式侧接地)情况下,记录跨膜电流,其中,所述16通道放大器的低通滤波频率为3khz且采样频率为10khz。使用pclamp套件(分子装置)的clampfit来分析电流迹线。使用origin8.6(microcal)(movileanu,l.,naturebiotechnol.,2000,18,1091-1095)生成图。
基于csggx射线结构的孔尺寸,将测量电流与计算出的电流进行比较,模拟由三段组成:跨膜段、周质前庭和连接两者的内通道收缩。将前两个段建模为圆锥形,而收缩表示为圆柱体。相应的电阻r1,r2和r3分别计算为
r1=l1/(πd1d1k)
r2=l2/(πd2d2k)
r3=l3/(πd1d2k)
其中,l1、l2和l3为各段的轴向长度,分别测量为3.5、4.0和2.0nm,并且d1、d1、d2和d2为段1和段2的最大和最小直径,分别测量为4.0、0.8、3.4和0.8nm。电导率k的值为10.6sm-1。通过将r1、r2和r3以及电压v=50mv插入到下式来计算电流:
i=v/(r1+r2+r3)
未发现接入电阻显著改变预测电流。
可以在存在分析物的情况下,进行如上所述的单通道电流记录,从而使得通道起到生物传感器的作用。
实施例15:采用模型聚丙氨酸链对csgg收缩结构进行分子动力学模拟
利用聚丙氨酸链穿过扩展构象的通道对csgg收缩结构进行建模,如图9所示,在c端至n端方向。穿过csgg转运体的底物通道本身不是序列特异性的(nenninger,a.a.etal.,mol.microbiol.,2011,81,486-499;vangerven,n.etal.,mol.microbiol.2014,91,1022-10352014)。为了清楚起见,使用聚丙氨酸链来对穿过多肽链的推定相互作用进行建模。建模区域由9个同心式csggc-环组成,每个包括残基47-58。沿收缩结构排列成行的侧链在图中示为棒状,其中标记出asn55和phe56。收缩结构内的
实施例16:csgg纳米孔用于核酸测序的用途
phi29dna聚合酶(dnap)可以与位于膜内的野生型或突变型csgg纳米孔一起用作分子马达,以允许寡聚探针dna链通过所述孔的可控运动。可以跨所述孔施加电压,并且由所述纳米孔任一侧的盐溶液中的离子的运动产生电流。随着探针dna移动通过所述孔,流经所述孔的离子流相对于所述dna变化。该信息已显示为序列依赖性的,并允许从电流测量值,例如上述实施例14中所述的测量值,精确地读取探针的序列。
实施例17
本实施例描述了为研究csgg中的dna行为而进行的模拟。
材料与方法
实施拉伸分子动力学模拟以研究csgg-eco和各种突变体对dna移位的能垒大小。所述模拟使用4.0.5版gromacs软件包,并采用gromos53a6力场和spc水模型实施。从蛋白质数据库中获取csgg-eco(seqidno:390)的结构,登录号为4uv3。为了制作csgg-eco突变体的模型,其野生型蛋白质结构使用pymol进行突变。研究的突变体为csgg-eco-(f56a)(带有突变f56a的seqidno:390),csgg-eco-(f56a-n55s)(带有突变f56a/n55s的seqidno:390)以及csgg-eco-(f56a-n55s-y51a)(带有突变f56a/n55s/y51a的seqidno:390)。
然后dna被放入所述孔中。建立两种不同的系统:
i.将单个鸟嘌呤核苷酸放置在所述孔内,正好在收缩结构区域的上方。
(所述残基56环上方大约5-10埃)
ii.单链dna(ssdna)沿所述孔的轴线放置,并且其5'端朝向所述孔的β-桶结构侧。在上述建立中,所述ssdna预穿过所述孔的整个长度。
然后模拟盒被溶剂化,并使用最速下降算法将能量最小化。
每个系统都在npt系统中模拟,使用berendsen恒温算法和berendsen恒压算法至300k。在整个模拟过程中,所述孔的骨架受到限制。
为了将dna拉过所述孔,在单个鸟嘌呤模拟中对其磷原子施加拉力。在所述ssdna模拟中,所述拉力施加到所述链5'端的磷原子上。通过连接上述dna磷原子和平行于孔轴线以恒定速率行进的虚拟点之间的弹性元件,以恒定速率施加所述拉力。需注意的是,所述弹性元件没有任何形状,也不会发生任何流体动力学阻力。弹性常数等于
实验结果
单个g的移位
如图12所示,所述拉力与时间的图表明,核苷酸进入所述野生型csgg-eco孔中的苯丙氨酸残基f56的环的能垒较大。对于所研究的csgg-eco突变体,未观察到鸟嘌呤移位存在显著能垒。
ssdna移位
对于ssdna移位,每个孔进行两次模拟,每次模拟施加不同的拉力速度(
实施例18
本实施例描述对多种csgg突变体的表征。
材料与方法
在建立实验之前,dna构建体x(终浓度为0.1nm,参见用于图像显示构建体x的图22和说明书)与t4dda-e94c/c109a/c136a/a360c(带有突变e94c/c109a/c136a/a360c的seqidno:412,加入到纳米孔体系的终浓度为10nm,其在缓冲液(151.5mmkcl,25mm磷酸钾,5%甘油,ph7.0,1mmedta)中提供)一起在室温下预培养5分钟。5分钟后,将tmad(100μm)加入到所述预混合物中,并将所述混合物再培养5分钟。最后,将mgcl2(加入到所述纳米孔体系中的终浓度为1.5mm),atp(加入到所述纳米孔体系中的终浓度为1.5mm),kcl(加入到所述纳米孔体系中的终浓度为500mm)和磷酸钾缓冲液(加入到所述纳米孔体系中的终浓度为25mm)加入到所述预混合物中。
从插入到缓冲液(25mmk磷酸盐缓冲液,150mm亚铁(ii)氰化钾,150mm铁(iii)氰化钾,ph8.0)的嵌段共聚物中的各种单个csgg纳米孔获得电学测量结果。在实现将单个孔插入所述嵌段共聚物中后,缓冲液(2ml,25mmk磷酸盐缓冲液,150mm亚铁(ii)氰化钾,150mm铁(iii)氰化钾,ph8.0)流过所述系统以除去任何过量csgg纳米孔。150ul的500mmkcl,25mmk磷酸盐,1.5mmmgcl2,1.5mmatp,ph8.0的溶液随后流过所述系统。10分钟后,150ul的500mmkcl,25mmk磷酸钾,1.5mmmgcl2,1.5mmatp,ph8.0的溶液流过所述系统,并且然后酶(t4dda-e94c/c109a/c136a/a360c,终浓度为10nm),dna构建体x(终浓度为0.1nm),养料(mgcl2终浓度1.5mm,atp终浓度1.5mm)的预混液(共150ul)流入所述单个纳米孔实验系统。所述实验在-120mv和监测解旋酶控制的dna移动下进行。
实验结果
孔显示增加的范围(图15至17以及图27至39)
csgg-eco-(strepll(c))(seqidno:390,其中stepll(c)为seqidno:435并且连接在其c末端)具有~10pa的范围(参见图15(a)),而以下csgg-eco孔突变体显示增加的电流范围-
1-csgg-eco-(y51n-f56a-d149n-e185r-e201n-e203n-strepll(c))9(具有突变y51n/f56a/d149n/e185r/e201n/e203n的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~30pa的范围(参见图15(b))。
2-csgg-eco-(n55a-strepll(c))9(具有突变n55a的seqidno:390,其中stepll(c)具有seqidno:435并且连接在c末端)显示为~35pa的范围(参见图15(c))。
3-csgg-eco-(n55s-strepll(c))9(具有突变n55s的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~40pa的范围(参见图16(a))。
4-csgg-eco-(y51n-strepll(c))9(具有突变y51n的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~40pa的范围(参见图16(b))。
5-csgg-eco-(y51a-f56a-strepll(c))9(具有突变y51a/f56a的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~30pa的范围(参见图16(c))。
6-csgg-eco-(y51a-f56n-strepll(c))9(具有突变y51a/f56n的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~20pa的范围(参见图17(a))。
7-csgg-eco-(y51a-n55s-f56a-strepll(c))9(具有突变y51a/n55s/f56a的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~30pa的范围(参见图17(b))。
8-csgg-eco-(y51a-n55s-f56n-strepll(c))9(具有突变y51a/n55s/f56n的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~30pa的范围(参见图17(c))。
13-csgg-eco-(f56h-strepll(c))9(具有突变f56h的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~35pa的范围(参见图27)。
14-csgg-eco-(f56q-strepll(c))9(具有突变f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在其c末端)显示为~40pa的范围(参见图28)。
15-csgg-eco-(f56t-strepll(c))9(具有突变f56t的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~35pa的范围(参见图29)。
16-csgg-eco-(s54p/f56a-strepll(c))9(具有突变s54p/f56a的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~35pa的范围(参见图30)。
17-csgg-eco-(y51t/f56a-strepll(c))9(具有突变y51t/f56a的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~30pa的范围(参见图31)。
18-csgg-eco-(f56p-strepll(c))9(具有突变f56p的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~30pa的范围(参见图32)。
19-csgg-eco-(f56a-strepll(c))9(具有突变f56a的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~40pa的范围(参见图33)。
20-csgg-eco-(y51t/f56q-strepll(c))9(具有突变y51t/f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~30pa的范围(参见图34)。
21-csgg-eco-(n55s/f56q-strepll(c))9(具有突变n55s/f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~35pa的范围(参见图35)。
22-csgg-eco-(y51t/n55s/f56q-strepll(c))9(具有突变y51t/n55s/f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~35pa的范围(参见图36)。
23-csgg-eco-(f56q/n102r-strepll(c))9(具有突变f56q/n102r的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~30pa的范围(参见图37)。
24-csgg-eco-(y51q/f56q-strepll(c))9(具有突变y51q/f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~40pa的范围(参见图38)。
25-csgg-eco-(y51a/f56q-strepll(c))9(具有突变y51a/f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)显示为~35pa的范围(参见图39)。
显示增加的通量的孔(图18和19)
从图18和19可以看出,以下突变孔(以下9-12)在4小时内显示出每个通道多个解旋酶控制的dna移动(图18和19中标记为x),而如图18(a)所示的csgg-eco-(strepll(c))(seqidno:390,其中stepii(c)为seqidno:435并且连接在c末端),通常在4小时内每个通道仅显示1或2个解旋酶控制的dna运动(在图18(a)中标记为x),并相反地,显示延长的阻挡区域(在图18(a)中标记为y)。
9-csgg-eco-(d149n-e185n-e203n-strepll(c))9(具有突变d149n/e185n/e203n的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)(参见图18(b))。
10-csgg-eco-(d149n-e185n-e201n-e203n-strepll(c))9(具有突变d149n/e185n/e201n/e203n的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)(参见图18(c))。
11-csgg-eco-(d149n-e185r-d195n-e201n-e203n)-strepll(c))9(具有突变d149n/e185r/d195n/e201n/e203n的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)(参见图19(a))。
11-csgg-eco-(d149n-e185r-d195n-e201r-e203n)-strepll(c))9(具有突变d149n/e185r/d195n/e201r/e203n的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)(参见图19(b))。
显示增加的插入的孔(图20和21)
通过对比图20和21可以看出,与csgg-eco-(strepll(c))(seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端)孔相比(如图20所示),如图21中所示的突变孔csgg-eco-(t1501-strepll(c))9(具有突变t150l的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端),以增加的孔数(~4-5倍)存在于膜中。图20和21中的箭头表示在4小时的实验中插入到嵌段共聚物中的csgg-eco纳米孔的数目(图20中的130-140以及图21中的1-11,各自对应于单独的纳米孔实验)。对于csgg-eco-(strepll(c))(seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端),三次实验显示插入一个纳米孔,而对于突变孔(csgg-eco-(t150l-strepll(c))9),每次实验显示插入至少一个纳米孔并且多次实验显示多个孔的插入。
实施例19
本实施例描述发展用于纯化csgg孔的大肠杆菌纯化方法。
材料与方法
编码多肽pro-csgg-eco-(strepll(c)(seqidno:390,其中stepll(c)为seqidno:435并连接在c末端,并且其中pro为seqidno:436并连接在n末端)的dna在genscriptusainc.中合成并被克隆到含有氨苄青霉素抗性基因的pt7载体中。所述pt7载体的蛋白质表达通过异丙基β-d-1-硫代吡喃半乳糖苷(lptg)诱导。所述dna溶液的浓度调节至400ng/ul。使用dna(1μl)来转化lemo21(de3)感受态(competent)大肠杆菌细胞(50μl,neb,目录号c2528h)。在转化前,csgg基因从lemo21(de3)细胞(genebridgesgmbh,德国)中敲除。然后所述细胞接种在含有氨苄青霉素(0.1mg/ml)的lb琼脂上,并在37℃下培养约16小时。
在含有氨苄青霉素的lb平板上生长的菌落含有csgg质粒。使用一个这样的菌落来接种含有氨苄青霉素(0.1mg/ml)的lb培养基(100ml)上的起始培养物。所述起始培养物在37℃,搅拌下生长直到od600达到1.0-1.2。所述起始培养物用于接种到新鲜的500ml的含有氨苄青霉素(0.1mg/ml)和鼠李糖(500μm)的lb培养基上,以达到0.1的o.d.600。所述起始培养物在37℃,搅拌下生长直到od600达到0.6。然后将所述培养物的温度调至18℃,并通过添加lptg(终浓度为0.2mm)引发诱导。诱导在18℃搅拌下进行约18小时。
诱导后,所述培养物通过在6000g下离心30分钟制成小球。所述小球在含有蛋白酶抑制剂(merckmillipore539138),苯甲酸酶核酸酶(sigmae1014)和1x抽提液(merckmillipore70921)ph8.0的50mmtris,300mmnacl溶液(每克小球约10ml缓冲液)中重悬。所述悬液充分混合直到其完全均匀,然后样品在4℃下转移到滚筒混合器中约5小时。通过在20000g下离心45分钟使细胞裂解物制成小球,上清液通过0.22μmpes注射过滤器进行过滤。含有csgg的上清液(称为样品1),进一步通过柱层析提纯。
样品1上样到5mlstreptrap柱(gehealthcare)。所述柱用25mmtris,150mmnacl,2mmedta,0.01%ddmph8的溶液洗涤,直到维持10个柱体积的稳定基线。然后所述柱在回到150mm缓冲液之前,用25mmtris,2mnacl,2mmedta,0.01%ddmph8的溶液洗涤。洗脱采用10mm脱硫生物素进行。图23中显示csgg蛋白的streptrap(gehealthcare)纯化的色谱痕迹的例子。洗脱峰用e1标记。图24示出在初始strep纯化后,csgg-eco蛋白的典型sds-page可视化的示例。泳道1-3显示了如箭头所示的含有csgg蛋白的主要洗脱峰(图23中标记为e1)。泳道4-6对应于含有污染物的主要洗脱峰(图23中标记为e1)的尾部的洗脱馏分。
合并所述洗脱峰,并加热至65℃15分钟以除去热不稳定的杂质蛋白。所述加热的溶液在20000g下离心10分钟,并弃去所述小球。上清液在120mlsephadexs200柱(gehealthcare)上通过25mmtris,150mmnacl,2mmedta,0.01%ddm,0.1%sdsph8溶液进行凝胶过滤。由于蛋白质的色氨酸组分低,监测在220nm下进行。样品在约55ml体积处洗脱出来(图25显示尺寸排阻柱的痕迹并用星号标记在55ml处的样品峰)。洗脱峰在4-20%tgx(参见图26,biorad)上进行电泳,以证实感兴趣的csgg-eco-strepll(c))(seqidno:390,其中stepll为seqidno:435并且连接在c末端)的孔的存在。合并鉴定出的馏分,并用50kd的amicon自旋柱进行浓缩。
实施例20
本实施例描述用来研究csgg-eco-(y51t/f56q-strepll(c))9(具有突变y51t/f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端孔突变体no.20)与t4dda-(e94c/c109a/c136a/a360c)(具有突变e94c/c109a/c136a/a360c的seqidno:412并且然后(δm1)g1g2)之间相互作用的模拟。
模拟方法
所述模拟使用4.0.5版gromacs软件包,并采用gromos53a6力场和spc水模型进行。
csgg-eco-(y51t/f56q-strepll(c))9(具有突变y51t/f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端孔突变体no.20)的模型是基于蛋白质数据库中发现的csgg的晶体结构,登录号为4uv3和4q79。相关的突变采用pymol制作。然后所得到的孔模型使用最速下降算法将能量最小化。t4dda-(e94c/c109a/c136a/a360c)(具有突变e94c/c109a/c136a/a360c的seqidno:412以及然后的(δm1)g1g2)的模型是基于蛋白质数据库中发现的dda1993结构,登录号为3upu。再次,相关的突变采用pymol制作,并使用最速下降算法将能量最小化。
t4dda-(e94c/c109a/c136a/a360c)(具有突变e94c/c109a/c136a/a360c的seqidno:412以及然后的(δm1)g1g2)的模型然后放置于csgg-eco-(y51t/f56q-strepll(c))9(具有突变y51t/f56q的seqidno:390,其中stepll(c)为seqidno:435并且连接在c末端孔突变体no.20)的上方。使用不同的初始酶构象进行三次模拟(试验1至3(0ns),参见图40):
在所有酶构象中,酶被取向使得dna的5'末端指向孔,并且在整个模拟过程中酶是无限定的。孔骨架受到限制并且模拟盒被溶剂化。所述系统在npt系统中模拟40ns,使用berendsen恒温算法和berendsen恒压算法至300k。
酶与孔之间的接触使用gromacs分析软件以及本地书写的代码进行分析。下面的表格显示了观察到的孔和酶氨基酸的接触次数。表6-8显示与酶上的氨基酸接触位点相互作用的孔上的氨基酸接触位点。在三次模拟中的两次模拟中,酶在孔的顶部倾斜(参见试验2和3(20,30和40ns),图40和41)。试验1显示酶没有倾斜,因此可以优化表6中显示具有高相互作用的位点,以增加孔帽上的酶稳定性。
表6=试验1酶和孔接触相互作用
表7=试验2酶和孔接触相互作用
表8=试验3酶和孔接触相互作用
实施例21:csgg纳米孔在通道收缩结构中对捕获核酸的能力
纳米孔用于核酸测序的用途要求通过纳米孔捕获和穿过单链dna。在本实施例中,csggwt蛋白的单通道电流迹线伴随着携带单链dna突出结构的dna发夹的存在。在图56中表示的所述实施例的迹线显示对应于在+50mv或-50mv间隔(如箭头所示)测量的电势而改变的电流。在最后+50mv段的下行电流的阻滞表明将单链发夹结构末端穿入内部的孔结构,所述内部的孔结构导致几乎完全的电流阻滞。将电场反转至-50mv导致孔的电泳解除阻塞。新的+50mv时段再次导致dna发夹结构的结合以及孔的堵塞。在所述+50mv段上,发夹结构的展开可以导致所述电流阻滞的停止,如所述电流阻滞的反转所示。具有序列为:3'gcggggagcgtattagagttggatcggatgcagctggctactgacgtcatgacgtcagtagccagcatgcatccgatc-5'(seqidno:441)的发夹结构在10nm的终浓度下被加入到所述腔室的顺式侧。
实施例22本实施例描述具有改变的内部收缩结构的突变csgg孔的产生。
在本实施例中,证明了csgg-δρυρα的稳定性和通道性质,所述csgg-δρυρα为一种突变csgg孔,其中收缩结构环中的序列pypa(残基50-53)被gg取代。对于该突变体,位置38至63的收缩结构基序对应于seqidno:354。这种突变预见到从所述孔的收缩结构中除去y51以及收缩结构环的缩短,以便降低在所述孔读取头中的复杂性,即所述孔的最窄部分,在感测应用中在此处测量电导率,对结合或穿过所述孔的底物的性质最为敏感。所述pypa序列的替换保留所述csgg九聚体的稳定性,并导致具有如图57所示增加的电导率的孔。
利用来自nanion(德国慕尼黑)的orbit16试剂盒,使用平行高分辨率电子记录,所述csgg-δpυρa孔突变体通过单通道电流记录进行分析。简而言之,在16-通道多电极空腔阵列(meca)芯片(lonera,弗莱堡,德国)51中的(亚皮秒体积的)微腔上形成1,2-二植烷酰基-sn-甘油-3-磷酸胆碱(avantipolarlipids)的水平双分子层。所述双分子层上方和下方的顺式和反式腔室均含有1.0mkcl,25mmtris-hcl,ph8.0。为了将通道插入所述膜中,将溶解在25mmtrisph8.0,500mmnacl,1mmdtt,0.5%c8e4,5mmldao中的csgg加入到顺式腔室中至终浓度为100nm。使用tecellatriton16通道放大器,在3khz的低通滤波频率和10khz的采样频率下,在+50mv和-50mv(顺式侧接地)的保持电势记录h跨膜电流。电流迹线使用pclamp组件的clampfit(moleculardevices,usa)进行分析。
实施例23:
本实施例描述对细菌淀粉样蛋白分泌通道csgg结构和机理的深入观察。
curli是功能性淀粉样蛋白纤维,其构成由拟杆菌和变形杆菌形成的防护生物膜中的细胞外基质的主要蛋白质组分(主要是a类和c类)1-3。它们提供致病菌株中的适应性优势,并在菌血症期间诱导强烈的促炎症反应1,4,5。curli的形成需要专门的蛋白质分泌机制,所述机制包括外膜脂蛋白csgg和两种可溶性辅助蛋白,csge和csgf6,7。我们在这里报道了大肠杆菌csgg非脂化,可溶形式以及天然膜提取构象的x射线结构。csgg形成由9个反密码子-结合-结构域样单元组成的寡聚转运复合体,其产生横贯所述双分子层的β-桶结构并连接到周质中的笼状前庭(vestibule)。其跨膜和周质结构域被由三个堆叠的同心苯丙氨酸,天冬酰胺和酪氨酸环组成的0.9nm通道收缩结构分开,所述环可以引导伸展的多肽底物通过所述分泌孔。特异性因子cage形成结合和封闭分泌通道的周质面的九聚体接合体,产生一个
curli是细菌表面附属物,其具有淀粉样蛋白纤维的结构和生理特征,该蛋白纤维在人类退行性疾病中最为人所知7-9。然而,细菌淀粉样蛋白例如curli的作用是促进生物膜的形成4,10。与作为蛋白错误折叠产物的致病性淀粉样蛋白不同,curli的形成由在两个专用操纵子,csgbac(curli特异性基因bac)和大肠杆菌中的csgdefg,中编码的蛋白调控(图46)6,7。分泌后,csgb使csga亚基成核形成curli纤维7,11,12。csga和csgb的分泌和细胞外沉积依赖于两种可溶性辅助因子,分别为csge和csgf,以及位于外膜中的262-残基的脂蛋白csgg13-16。由于在外膜上缺少水解能源或离子梯度,因此csgg属于一种特殊类型的蛋白质移位体,其必须通过交替供能的运输机制进行操作。在没有结构模型的情况下,csgg如何以跨生物膜的调节方式促进高度稳定的淀粉样蛋白纤维的分泌和组装的动力学工作方式迄今仍然是未知的。
在插入所述外膜之前,脂蛋白通过脂蛋白定位(lol)途径穿过周质17。我们观察到,非脂化csgg(csggc1s)可以分离作为可溶性周质中间体,类似于在成孔蛋白和毒素中观察到的前孔形态18。除少量的离散低聚复合物之外,csggc1s主要作为单体被发现(图47)19。所述可溶的csggc1s寡聚体进行结晶,并且其结构确定至
在csggc1s结构中,将α1连接到脂质锚蛋白n-末端的残基1-17是无序的,并且没有明显的跨膜(tm)结构域可以被辨别(图42)。csggc1s和天然的膜提取csgg的衰减全反射傅里叶变换红外光谱(atr-ftir)显示后者在β折叠区域(1625-1630cm-1)具有更高的吸收,而无规则卷曲和α-螺旋区域(分别为1645-1650cm-1和1656cm-1;图43a)则伴随吸收减少,表明膜相关的csgg含有β-桶结构域。在连接β3-β4(残基134-154)和β5-α3(残基184-204)的无序的延伸环中发现延伸用于β-链形成的候选序列;这些序列的敲除导致无法形成curli(图43b)。洗涤剂提取的csgg的晶体结构证实两个区域的构象重排为两个相邻的β-发夹结构,延伸通过β3-β4(tm1)和β5-α3(tm2)形成的β-折叠(图43c)。它们在csgg寡聚体中的并排放置产生复合36-链的β-桶结构(图43d)。而结晶的csggc1s寡聚物显示d8对称性,csgg结构显示出d9对称性,并且csgg原聚体保留了相当的原聚体间的接触,除相对于中轴的5°旋转以及沿径向轴
因此,csgg形成宽度为
在磷脂双分子层平面中重组的csgg的单通道电流记录使用标准电解质条件和分别为+50mv或-50mv的电势,导致43.1±4.5pa(n=33)或-45.1±4.0pa(n=13)的稳定电流(图44e,f以及图52)。所述观察到的电流与基于简单三-片段孔模型计算的46.6pa的预测值和x射线结构中观察到的中心收缩结构的尺寸非常一致(图44c)。在负电场电势(-26.2±3.6pa(n=13);图52)条件下还可以观察到第二个低电导构象(图52).然而,这种构象是否存在于生理条件下并不清楚。
我们的结构数据和单通道记录表明csgg形成了非门控的肽扩散通道。在pa63,即模型肽扩散通道中,多肽的通过取决于δρh驱动的布朗棘轮模型,其对移位通道中的扩散步骤进行校正20-22。然而,这种质子梯度并不存在于外膜上,需要替代的驱动力。而在升高的浓度下,csgg促进周质多肽的非选择性扩散性渗出,在天然条件下对csga而言分泌是特异性的,并且需要周质因子csge16,23。在过量的cage存在下,纯化的csgg在天然性page上形成更缓慢迁移物种(图45a)。sds-page分析显示,该新物种由以1:1化学计量比存在的csgg-cage复合物组成(图45b)。通过下拉亲和纯化分离的csgg-csge的冷冻电子显微镜(cryo-em)可视化发现了对应于csgg九聚体的九倍对称颗粒,和在周质门庭的入口处的另外的加盖密度,其尺寸和形状类似于同样通过单粒子em和尺寸排阻色谱观察到的c9csge寡聚物(图45c-e以及图53)。观察到的csgg-cage接触界面的位置被csgg螺旋2中的阻塞点突变所证实。与加帽功能一致,单通道记录显示cage与所述移位体结合导致其离子电导的特异性沉默(图45f以及图52)。通道中csge的加帽似乎在csge九聚体结合的功能中是一个全有-或-全无的反应。在饱和条件下,csge的结合诱导通道的完全阻断,而在约10nm条件下,csge结合与解离事件之间的平衡导致间歇性阻断的或完全开放的移位体。在1nm或以下的条件下,瞬时(<1ms)局部阻塞事件可能源于短期与单体csge的遭遇。
因此,csgg和csge似乎形成一个包含~
curli诱导的生物膜在致病性肠杆菌中形成适合因子和毒力因子4,5。其独特的分泌和组装性能也迅速获得对(生物)技术应用的兴趣23,28,29。我们对两个关键分泌成分的结构表征和生物化学研究提供了一种在不存在可水解能源,膜电势或离子梯度的情况下,展开的蛋白底物的膜移位的重复机制的试验模型(图45e和图54)。所提出的分泌模型中贡献因子的完全验证和解构将需要移位子的体外重组,以允许在单分子水平下精确地遵循转运动力学。
方法
克隆和菌株。已在参考文献19中描述了用于制备局部外膜的c-末端strepll标记的csgg(ppg1)和周质c-末端strepll标记的csggc1s(ppg2)的表达构建体。对于硒代甲硫氨酸标记,strepll标记的csggc1s由于增加的产量而在细胞质中进行表达。因此,ppg2改变为采用带有引物5’-tctttaaccgccccgcctaaag-3’(正向)和5’-cattttttgccctcgttatc-3’(反向)(ppg3)的反向pcr去除n末端信号肽。对于表型测定,通过参考文献30中所描述的方法(带有引物5’-aataactcaaccgatttttaagccccagcttcataaggaaaataatcgtgtaggctggagctgcttc-3’和5’-cgcttaaacagtaaaatgccggatgataattccggcttttttatctgcatatgaatatcctccttag-3’)构建大肠杆菌bw25141(大肠杆菌nvg2)的csgg缺失突变体。在trc启动子控制下,通过始于pmc2,即含有csgg的ptrc99a载体,的定点诱变(quikchange方案;stratagene)来构建各种csgg替代型突变体,所述csgg替代型突变体用于cys可接近性试验并用于通道收缩的表型探测14。
蛋白表达和纯化。如上所述,对csgg和csggc1s进行表达和纯化19。简言之,在大肠杆菌bl21(de3)中重组地产生csgg,其中,所述大肠杆菌采用ppg1转化,并使用缓冲液a(50mmtris-hclph8.0;500mmnacl;1mmedta;1mm二硫苏糖醇(dtt))中的1%正十二烷基-b-d-麦芽糖苷(ddm)从分离的外膜中进行提取。将strepll标记的csgg装载到5毫升strep-tactin琼脂糖凝胶柱(lbagmbh)上,并通过用20个柱体积的补充有0.5%的四甘醇单辛基醚(c8e4;affymetrix)和4mm月桂基二甲基氧化胺(ldao:lauryldimethylamine-n-oxide;affymetrix)的缓冲液a洗涤来进行清洁剂交换。通过添加2.5mmd-脱硫生物素将蛋白洗脱,并浓缩至5mgml-1用于结晶实验。对于硒代甲硫氨酸标记,在用ppg3转化的met营养缺陷型菌株b834(de3)中制备csggc1s,并在补充有40mgl-1l-硒代甲硫氨酸的m9基本培养基上生长。细胞颗粒重悬于补充有完整的罗氏蛋白酶抑制剂混合物(proteaseinhibitorcocktail(roche))的50mmtris-hclph8.0、150mmnacl、1mmedta、5mmdtt中,并通过穿过以20×103lbin-2操作的ts系列细胞破碎剂(constantsystemsltd)而分裂。如上所述,对标记的csggc1s进行纯化19。在整个纯化过程中添加dtt(5mm)以避免硒代甲硫氨酸氧化。
在携带有pnh27的大肠杆菌nebc2566细胞中产生csge(参考文献16)。将25mmph值为8.0的tris-hcl、150mmnacl、25mm咪唑、5%(v/v)甘油中的细胞溶解产物装载在histrapff上(gehealthcare)。csge-his用20mmtris-hclph8.0,150mmnacl,5%(v/v)甘油缓冲液中的500mm咪唑进行线性梯度洗脱。含有csge的馏分用250mm(nh4)2so4增补,并施加到用20mmtris-hclph8.0、100mmnacl、250mm(nh4)2so4、5%(v/v)甘油平衡的5mlhitrapphenylhp柱上(gehealthcare)。向20mmtris-hclph8.0、10mmnacl、5%(v/v)甘油施加线性梯度以便进行洗脱。将含有馏分的csge装载到superose6prepgrade10/600(gehealthcare)柱上,所述柱在20mmtris-hclph8.0、100mmnacl、5%(v/v)甘油中平衡。
溶液中寡聚状态评估。将各约0.5mg的洗涤剂溶解的csgg(0.5%c8e4,4mmldao)和csggc1s施加到用25mmtris-hclph8.0、500mmnacl、1mmdtt、4mmldao和0.5%c8e4(csgg)或25mmtris-hclph8.0、200mmnacl(csggc1s)平衡的superdex20010/300gl分析凝胶过滤柱(gehealthcare)上,并以0.7mlmin-1运行。用牛甲状腺球蛋白、牛γ-球蛋白、鸡卵清蛋白、马肌球蛋白和维生素b12(bio-rad)对柱洗脱体积进行校准(图47)。采用参考文献31中描述的程序(图47),将膜提取的csgg,即20mg洗涤剂溶解的蛋白也在3-10%蓝色天然page上运行。nativemark(lifetechnologies)未染色蛋白标准物(7ml)用于分子量估计。
结晶、数据收集和结构测定。通过对包含有100mmph值为4.2的乙酸钠、8%peg4000和100mm丙二酸钠ph7.0的溶液进行坐滴式(sitting-drop)蒸气扩散,将硒代甲硫氨酸标记的csggc1s浓缩至3.8mgml-1并进行结晶。在补充有15%甘油的晶体缓冲液中对晶体进行培养,并在液氮中快速冷冻。通过对包含有100mmtris-hclph8.0、8%peg4000、100mmnacl和500mmmgcl2的溶液进行悬滴式(hanging-drop)蒸气扩散,将洗涤剂溶解的csgg浓缩至5mgml-1并进行结晶。将晶体在液氮中快速冷冻并通过存在于晶体溶液中的洗涤剂进行冷冻保护。为了优化结晶条件并筛选具有良好衍射质量的晶体,在光束线proxima-1和proxima-2a(soleil,法国)、px-l(瑞士光源,瑞士)、102、103、104和124(钻石光源,英国)以及id14eh2、id23eh1和id23eh2(esrf,法国)上对晶体进行分析。分别在光束线104和103上收集到用于csggc1s和csgg结构测定的最终衍射数据(数据收集和细化统计参见图55a)。采用xia2和xds32,33封装处理csggc1s的衍射数据。csggc1s的晶体属于空间群p1,晶胞大小为
在空间群c2中,采用xds封装32,33从
刚果红试验。为了分析刚果红结合,在lb(lysogenybroth)培养基中对lb中在37℃生长的细菌过夜培养物进行稀释,直至达到0.5的d600。在补充有氨苄青霉素(100mgl-1)、刚果红(100mgl-1)和0.1%(w/v)异丙基β-d-硫代半乳糖苷(iptg)的lb琼脂板上点样5μl样本。在室温(20-22℃)下将平板培养48小时以诱导卷曲菌毛表达。观察菌落形态发展和染色结合情况。
半胱氨酸可接近性试验。采用定点突变在pmc2中产生半胱氨酸突变体,并在大肠杆菌lsr12中进行表达(参考文献7)。将生长过夜的细菌培养物点样到含有1mmiptg和100mgl-1氨苄青霉素的lb琼脂板上。将板在室温下培育,并在48小时后刮下细胞,重悬于1ml的pbs中并用d600归一化。通过超声处理裂解细胞,并在4℃温度下以3,000g离心20秒,以便从细胞裂解液和悬浮膜中去除未破裂的细胞。将上清液中的蛋白在室温下用15mm甲氧基聚乙二醇-马来酰亚胺(mal-peg5kda)标记1小时。用100mmdtt终止反应,并在4℃温度下在50.4ti转子中以40,000r.p.m(~100,000g)离心20分钟以将整个膜制成小球。将小球用1%月桂酰肌氨酸钠洗涤以溶解细胞质膜并再次离心。将所得的外膜用含有1%ddm的pbs重新悬浮并溶解。用镍珠进行的金属亲和性下拉(metal-affinitypulldownswithnickelbeads)用于sds-page和抗-his蛋白质免疫印迹。带有空pmc2载体的大肠杆菌lsr12细胞用作阴性对照。
atr-ftir光谱法。在equinox55红外光谱仪(bruker)上进行atr-ftir测量,所述红外光谱仪连续用干燥空气吹扫,并配备有液氮制冷型碲汞镉探测器(mercurycadmiumtelluridedetector)和goldengate反射附件(specac)。内反射元件为金刚石晶体(2mm×2mm),光束入射角为45°。将每个纯化的蛋白样本(1μl)在晶体表面铺展并在气态氮气流下干燥以形成膜。以2cm-1分辨率记录的每个光谱为128个累计值的平均值,用以提高信噪比。所有光谱采用水蒸汽贡献减法(watervapourcontributionsubtraction)来处理,通过变迹法(apodization)在4cm-1最终分辨率下变平滑,并在酰胺i谱带(1,700-1,600cm-1)区域上归一化以对它们进行比较46。
阴性染色em和对称性测定。阴性染色em用于监测csgg、csggc1s和csge的溶液中(in-solution)寡聚状态。将csge、csggc1s和两亲聚合物结合的csgg调节成0.05mgml-1的浓度,并施加到辉光放电的碳涂覆铜网格上(cf-400;electronmicroscopysciences)。培育1分钟后,将样本染上印记,然后在2%的乙酸双氧铀中洗涤并染色。在tecnait12biotwinlab6显微镜上收集图像,操作电压为120kv,放大倍数为×49,000,散焦为800至2,000nm。如针对cryo-em所述,执行对比传递函数(ctf)、相位翻转和颗粒选择。对于膜提取的csgg,分别通过九聚体和顶视图分析十八聚体颗粒(共1,780个)。对于纯化的csge,总共分析了2,452个颗粒。按以下针对csgg-csgecryo-em分析所述,获得三维模型,并通过几轮多参考比对(multi-referencealignment,mra)、多统计分析(msa)和锚设置细化(anchorsetrefinement)来细化三维模型。在所有情况下,归一化和中心化后,如cryo-em部分中所述采用imagic-4d对图像进行分类。对于每组颗粒,选择与特征视图对应的最佳分类。采用最佳分类平均值进行csgg顶视图的对称性测定,每类大约有20张图像。使用imagic计算旋转自相关函数并进行绘制。
csgg-csge复合物形成。对于csgg-csge复合物形成,通过向300μl洗涤剂去稳定的脂质体(1mgml-11,2-二肉豆蔻酰-sn-甘油基-3-磷酸胆碱(dmpc)和0.4%的ldao)中加入120μlcsgg(0.5%c8e4中的24mgml-1蛋白、4mmldao,25mmtris-hclph8.0,500mmnacl,1mmdtt)并在加入90ml(在100mgml-1原料下)的a8-35两亲聚合物前在冰上培育5分钟,从而将纯化的csgg中的增溶洗涤剂交换为两亲聚合物a8-35(anatrace)。在冰上培育另外15分钟后,将样本装载到superose610/300gl(gehealthcare)柱上,并在200mmnacl,2.5%木糖醇,25mmtris-hclph为8,0.2mmdtt中进行凝胶过滤。,将200mmnacl、2.5%木糖醇、25mmph值为8的tris-hcl、0.2mmdtt中的等体积纯化单体csge以对于csge和csgg分别为15和5μm的最终蛋白浓度加入到两亲聚合物溶解的csgg,样本在温度为18℃、电压为125v情况下在0.5×tbe缓冲液中在4.5%的天然page上运行。对于第二个变性维度,将对应于csgg-csge复合物的条带从在同一凝胶上平行运行的未染色通道(lane)中切除,在laemmli缓冲液(60mmph值为6.8的tris-hcl、2%的sds、10%甘油、5%的2-巯基乙醇、0.01%溴酚蓝)中煮沸5分钟,并在4-20%的sds-page上运行。纯化的csge和csgg作为对照样本与复合物一起运行。用考马斯亮蓝(instantbluecoomassie)对凝胶进行染色,以便进行目视检查,或用sypro橙染色,以便通过对sds-page上的csge和csgg条带进行荧光检测(typhoonfla9000)来对csgg-csge复合物进行化学计量评估,得到0.97的csgg/csge比。
csgg-csgecryo-em。cryo电子显微镜用于测定c9csgg-csge复合物的溶液内(in-solution)结构。将如上所述制备的csgg-csge复合物与histrapff(gehealthcare)结合并从其上洗脱以去除未结合的csgg,洗脱后立即将其应用于在20ma下辉光放电30秒的quantifoilr2/2碳涂覆网格(quantifoilmicrotoolsgmbh)。使用自动化系统(leica)将样本冷冻浸入液氮中,并在feif20显微镜下观察,其中操作电压为200kv,并且在低剂量条件下标称放大倍数为×50,000,散焦范围为1.4-3μm。在falconii探测器上记录图像帧。标本级别的像素大小为每像素
胆汁盐毒性试验。通过在含有胆汁盐的琼脂板上减慢的生长来研究外膜渗透性。将大肠杆菌lsr12(参考文献7)细胞的十倍连续稀释液(5μl)点样在mcconkey琼脂板上,其中,所述稀释液含有plr42(参考文献16)和pmc2(参考文献14)(或衍生的螺旋物2突变体),所述琼脂板含有100μgl-1氨苄青霉素、25μgl-1氯霉素、1mm含有或不含0.2%(w/v)l-阿拉伯糖的iptg。在37℃下培育过夜后,检查菌落生长。
单通道电流记录。采用带有来自nanion的orbit16试剂盒的并行高分辨率电记录进行单通道电流记录。简言之,在16通道多电极空腔阵列(meca)芯片(lonera)51中的微腔(亚皮升体积(subpicolitrevolume)的)上形成1,2-二植烷酰基-sn-甘油基-3-磷酸胆碱(avantipolarlipids)的水平双层。双层上方和下方的顺式和反式腔均含有1.0mkcl、25mmph值为8.0的tris-hcl。为了将通道插入膜中,将溶解在25mmph值为8.0的tris-hcl、500mmnacl、1mmdtt、0.5%c8e4、5mmldao中的csgg加入到顺式腔室中直至最终浓度为90-300nm。为了测试csgg通道与csge的相互作用,将溶解在25mmph值为8.0的tris-hcl、150mmnacl中的csge的溶液加入到顺式腔室中直至最终浓度为0.1、1、10和100nm。采用tecellatriton16通道放大器,在保持电势为+50mv和-50mv(顺式侧接地)情况下,记录跨膜电流,其中,所述16通道放大器的低通滤波频率为3khz且采样频率为10khz。使用pclamp套件(moleculardevices)的clampfit分析电流迹线。使用origin8.6(microcal)52生成图。
基于csggx射线结构的孔尺寸,将测量的电流与计算出的电流进行比较,建模为由三段组成:跨膜段、周质前庭和连接两者的内通道收缩结构。将前两个段建模为圆锥形,而收缩结构表示为圆柱体。相应的电阻r1,r2和r3分别计算为
r1=l1/(πd1d1k)
r2=l2/(πd2d2k)
r3=l3/(πd1d2k)
其中,l1、l2和l3为各段的轴向长度,经测量分别为3.5、4.0和2.0nm,并且d1、d1、d2和d2为段1和段2的最大和最小直径,经测量分别为4.0、0.8、3.4和0.8nm。电导率k的值为10.6sm-1。通过将r1、r2和r3以及电压v=50mv代入到下式来计算电流:
i=v/(r1+r2+r3)
未发现接入电阻显著改变预测电流。
参考文献:
1.olsen,a.,jonsson,a.&normark,s.fibronectinbindingmediatedbyanovelclassofsurfaceorganellesonescherichiacoli.nature338,652-655(1989).
2.collinson,s.k.etal.thin,aggregativefimbriaemediatebindingofsalmonellaenteritidistofibronectin.j.bacteriol.175,12-18(1993).
3.dueholm,m.s.,albertsen,m.,otzen,d.&nielsen,p.h.curlifunctionalamyloidsystemsarephylogeneticallywidespreadanddisplaylargediversityinoperonandproteinstructure.plosone7,e51274(2012).
4.cegelski,l.etal.small-moleculeinhibitorstargetescherichiacoliamyloidbiogenesisandbiofilmformation.naturechem.biol.5,913-919(2009).
5.herwald,h.etal.activationofthecontact-phasesystemonbacterialsurfaces-acluetoseriouscomplicationsininfectiousdiseases.naturemed.4,298-302(1998).
6.hammar,m.,amqvist,a.,bian,z.,olsen,a.&normark,s.expressionoftwocsgoperonsisrequiredforproductionoffibronectin-andcongored-bindingcurlipolymersinescherichiacolik-12.mol.microbiol.18,661-670(1995).
7.chapman,m.r.etal.roleofescherichiacolicurlioperonsindirectingamyloidfiberformation.science295,851-855(2002).
8.wang,x.,smith,d.r.,jones,j.w.&chapman,m.r.invitropolymerizationofafunctionalescherichiacoliamyloidprotein.j.biol.chem.282,3713-3719(2007).
9.dueholm,m.s.etal.fibrillationofthemajorcurlisubunitcsgaunderawiderangeofconditionsimpliesarobustdesignofaggregation.biochemistry50,8281-8290(2011).
10.hung,c.etal.escherichiacolibiofilmshaveanorganizedandcomplexextracellularmatrixstructure.mbio4,e00645-13(2013).
11.hammar,m.,bian,z.&normark,s.nucleator-dependentintercellularassemblyofadhesivecurliorganellesinescherichiacoli.proc.natlacad.sci.usa93,6562-6566(1996).
12.bian,z.&normark,s.nucleatorfunctionofcsgbfortheassemblyofadhesivesurfaceorganellesinescherichiacoli.emboj.16,5827-5836(1997).
13.loferer,h.,hammar,m.&normark,s.availabilityofthefibresubunitcsgaandthenucleatorproteincsgbduringassemblyoffibronectin-bindingcurliislimitedbytheintracellularconcentrationofthenovellipoproteincsgg.mol.microbiol.26,11-23(1997).
14.robinson,l.s.,ashman,e.m.,hultgren,s.j.&chapman,m.r.secretionofcurlifibresubunitsismediatedbytheoutermembrane-localizedcsggprotein.mol.microbiol.59,870-881(2006).
15.nenninger,a.a.,robinson,l.s.&hultgren,s.j.localizedandefficientcurlinucleationrequiresthechaperone-likeamyloidassemblyproteincsgf.proc.natlacad.sci.usa106,900-905(2009).
16.nenninger,a.a.etal.csgeisacurlisecretionspecificityfactorthatpreventsamyloidfibreaggregation.mol.microbiol.81,486-499(2011).
17.okuda,s.&tokuda,h.lipoproteinsortinginbacteria.annu.rev.microbiol.65,239-259(2011).
18.lacovache,i.,bischofberger,m.&vandergoot,f.g.structureandassemblyofpore-formingproteins.curr.opin.struct.biol.20,241-246(2010).
19.goyal,p.,vangerven,n.,jonckheere,w.&remaut,h.crystallizationandpreliminaryx-raycrystallographicanalysisofthecurlitransportercsgg.actacrystallogr.fstruct.biol.cryst.commun.69,1349-1353(2013).
20.krantz,b.a.etal.aphenylalanineclampcatalyzesproteintranslocationthroughtheanthraxtoxinpore.science309,777-781(2005).
21.janowiak,b.e.,fischer,a.&collier,r.j.effectsofintroducingasinglechargedresidueintothephenylalanineclampofmultimericanthraxprotectiveantigen.j.biol.chem.285,8130-8137(2010).
22.feld,g.k.,brown,m.j.&krantz,b.a.ratchetingupproteintranslocationwithanthraxtoxin.proteinsci.21,606-624(2012).
23.vangerven,n.etal.secretionandfunctionaldisplayoffusionproteinsthroughthecurlibiogenesispathway.mol.microbiol.91,1022-1035(2014).
24.brinker,a.etal.dualfunctionofproteinconfinementinchaperonin-assistedproteinfolding.cell107,223-233(2001).
25.busby,j.n.,panjikar,s.,landsberg,m.j.,hurst,m.r.&lott,j.s.thebccomponentofabctoxinsisanrhs-repeat-containingproteinencapsulationdevice.nature501,547-550(2013).
26.takagi,f.,koga,n.&takada,s.howproteinthermodynamicsandfoldingmechanismsarealteredbythechaperonincage:molecularsimulations.proc.natlacad.sci.usa100,11367-11372(2003).
27.zhou,h.x.proteinfoldinginconfinedandcrowdedenvironments.arch.biochem.biophys.469,76-82(2008).
28.chen,a.y.etal.synthesisandpatterningoftunablemultiscalematerialswithengineeredcells.naturemater.13,515-523(2014).
29.sivanathan,v.&hochschild,a.abacterialexportsystemforgeneratingextracellularamyloidaggregates.natureprotocols8,1381-1390(2013).
30.datsenko,k.a.&wanner,b.l.one-stepinactivationofchromosomalgenesinescherichiacolik-12usingpcrproducts.proc.natlacad.sci.usa97,6640-6645(2000).
31.swamy,m.,siegers,g.m.,minguet,s.,wollscheid,b.&schamel,w.w.a.bluenativepolyacrylamidegelelectrophoresis(bn-page)fortheidentificationandanalysisofmultiproteincomplexes.sci.stke2006,pl4,http://dx.doi.org/10.1126/stke.3452006p14(2006).
32.winter,g.xia2:anexpertsystemformacromolecularcrystallographydatareduction.j.appl.cryst.43,186-190(2010).
33.kabsch,w.xds.actacrystallogr.dbiol.crystallogr.66,125-132(2010).
34.sheldrick,g.m.experimentalphasingwithshelxc/d/e:combiningchaintracingwithdensitymodification.actacrystallogr.dbiol.crystallogr.66,479-485(2010).
35.bricogne,g.,vonrhein,c.,flensburg,c.,schiltz,m.&paciorek,w.generation,representationandflowofphaseinformationinstructuredetermination:recentdevelopmentsinandaroundsharp2.0.actacrystallogr.dbiol.crystallogr.59,2023-2030(2003).
36.cowtan,k.recentdevelopmentsinclassicaldensitymodification.actacrystallogr.dbiol.crystallogr.66,470-478(2010).
37.cowtan,k.thebuccaneersoftwareforautomatedmodelbuilding.1.tracingproteinchains.actacrystallogr.dbiol.crystallogr.62,1002-1011(2006).
38.adams,p.d.etal.phenix:acomprehensivepython-basedsystemformacromolecularstructuresolution.actacrystallogr.dbiol.crystallogr.66,213-221(2010).
39.emsley,p.,lohkamp,b.,scott,w.g.&cowtan,k.featuresanddevelopmentofcoot.actacrystallogr.dbiol.crystallogr.66,486-501(2010).
40.davis,i.w.etal.molprobity:all-atomcontactsandstructurevalidationforproteinsandnucleicacids.nucleicacidsres.35(suppl2),w375-w383(2007).
41.strong,m.etal.towardthestructuralgenomicsofcomplexes:crystalstructureofape/ppeproteincomplexfrommycobacteriumtuberculosis.proc.natlacad.sci.usa103,8060-8065(2006).
42.mccoy,a.j.etal.phasercrystallographicsoftware.j.appl.cryst.40,658-674(2007).
43.smart,o.s.etal.exploitingstructuresimilarityinrefinement:automatedncsandtarget-structurerestraintsinbuster.actacrystallogr.dbiol.crystallogr.68,368-380(2012).
44.murshudov,g.n.etal.refmac5fortherefinementofmacromolecularcrystalstructures.actacrystallogr.dbiol.crystallogr.67,355-367(2011).
45.nicholls,r.a.,long,f.&murshudov,g.n.low-resolutionrefinementtoolsinrefmac5.actacrystallogr.dbiol.crystallogr.68,404-417(2012).
46.goormaghtigh,e.&ruysschaert,j.m.subtractionofatmosphericwatercontributioninfouriertransforminfraredspectroscopyofbiologicalmembranesandproteins.spectrochim.acta50a,2137-2144(1994).
47.mindell,j.a.&grigorieff,n.accuratedeterminationoflocaldefocusandspecimentiltinelectronmicroscopy.j.struct.biol.142,334-347(2003).
48.shaikh,t.r.etal.spiderimageprocessingforsingle-particlereconstructionofbiologicalmacromoleculesfromelectronmicrographs.natureprotocols3,1941-1974(2008).
49.tang,g.etal.eman2:anextensibleimageprocessingsuiteforelectronmicroscopy.j.struct.biol.157,38-46(2007).
50.pettersen,e.f.etal.ucsfchimera-avisualizationsystemforexploratoryresearchandanalysis.j.comput.chem.25,1605-1612(2004).
51.delriomartinez,j.m.,zaitseva,e.,petersen,s.,baaken,g.&behrends,j.c.automatedformationoflipidmembranemicroarraysforionicsinglemoleculesensingwithproteinnanopores.smallhttp://dx.doi.org/10.1002/smll.201402016(13august2014).
52.movileanu,l.,howorka,s.,braha,o.&bayley,h.detectingproteinanalytesthatmodulatetransmembranemovementofapolymerchainwithinasingleproteinpore.naturebiotechnol.18,1091-1095(2000).
53.pronk,s.etal.gromacs4.5:ahigh-throughputandhighlyparallelopensourcemolecularsimulationtoolkit.bioinformatics29,845-854(2013).
54.lindorff-larsen,k.etal.improvedside-chaintorsionpotentialsfortheamberff99sbproteinforcefield.proteins78,1950-1958(2010).
55.capra,j.a.&singh,m.predictingfunctionallyimportantresiduesfromsequenceconservation.bioinformatics23,1875-1882(2007).
序列表
<110>vibvzw
布鲁塞尔自由大学
stefanhoworka
牛津纳米孔技术公司
<120>突变孔
<130>hr/npore/517
<150>gb1415455.3
<151>2014-09-01
<150>gb1422079.2
<151>2014-12-11
<150>gb1506489.2
<151>2015-04-16
<150>gb1506754.9
<151>2015-04-21
<150>gb1508287.8
<151>2015-05-14
<150>gb1511203.0
<151>2015-06-25
<150>gb1515240.8
<151>2015-08-27
<160>450
<170>patentinversion3.5
<210>1
<211>277
<212>prt
<213>大肠杆菌(escherichiacoli)
<400>1
metglnargleupheleuleuvalalavalmetleuleuserglycys
151015
leuthralaproprolysglualaalaargprothrleumetproarg
202530
alaglnsertyrlysaspleuthrhisleuproalaprothrglylys
354045
ilephevalservaltyrasnileglnaspgluthrglyglnphelys
505560
protyrproalaserasnpheserthralavalproglnseralathr
65707580
alametleuvalthralaleulysaspserargtrppheileproleu
859095
gluargglnglyleuglnasnleuleuasngluarglysileilearg
100105110
alaalaglngluasnglythrvalalaileasnasnargileproleu
115120125
glnserleuthralaalaasnilemetvalgluglyserileilegly
130135140
tyrgluserasnvallysserglyglyvalglyalaargtyrphegly
145150155160
ileglyalaaspthrglntyrglnleuaspglnilealavalasnleu
165170175
argvalvalasnvalserthrglygluileleuserservalasnthr
180185190
serlysthrileleusertyrgluvalglnalaglyvalpheargphe
195200205
ileasptyrglnargleuleugluglygluvalglytyrthrserasn
210215220
gluprovalmetleucysleumetseralailegluthrglyvalile
225230235240
pheleuileasnaspglyileaspargglyleutrpaspleuglnasn
245250255
lysalagluargglnasnaspileleuvallystyrarghismetser
260265270
valproprogluser
275
<210>2
<211>834
<212>dna
<213>大肠杆菌
<400>2
atgcagcgcttatttcttttggttgccgtcatgttactgagcggatgcttaaccgccccg60
cctaaagaagccgccagaccgacattaatgcctcgtgctcagagctacaaagatttgacc120
catctgccagcgccgacgggtaaaatctttgtttcggtatacaacattcaggacgaaacc180
gggcaatttaaaccctacccggcaagtaacttctccactgctgttccgcaaagcgccacg240
gcaatgctggtcacggcactgaaagattctcgctggtttataccgctggagcgccagggc300
ttacaaaacctgcttaacgagcgcaagattattcgtgcggcacaagaaaacggcacggtt360
gccattaataaccgaatcccgctgcaatctttaacggcggcaaatatcatggttgaaggt420
tcgattatcggttatgaaagcaacgtcaaatctggcggggttggggcaagatattttggc480
atcggtgccgacacgcaataccagctcgatcagattgccgtgaacctgcgcgtcgtcaat540
gtgagtaccggcgagatcctttcttcggtgaacaccagtaagacgatactttcctatgaa600
gttcaggccggggttttccgctttattgactaccagcgcttgcttgaaggggaagtgggt660
tacacctcgaacgaacctgttatgctgtgcctgatgtcggctatcgaaacaggggtcatt720
ttcctgattaatgatggtatcgaccgtggtctgtgggatttgcaaaataaagcagaacgg780
cagaatgacattctggtgaaataccgccatatgtcggttccaccggaatcctga834
<210>3
<211>25
<212>prt
<213>大肠杆菌
<400>3
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serasnpheserthralavalprogln
2025
<210>4
<211>25
<212>prt
<213>大肠杆菌
<400>4
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serasnpheserthralavalprogln
2025
<210>5
<211>25
<212>prt
<213>大肠杆菌
<400>5
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serasnalaserthralavalprogln
2025
<210>6
<211>25
<212>prt
<213>大肠杆菌
<400>6
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serasnglyserthralavalprogln
2025
<210>7
<211>25
<212>prt
<213>大肠杆菌
<400>7
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serasnvalserthralavalprogln
2025
<210>8
<211>24
<212>prt
<213>大肠杆菌
<400>8
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serasnserthralavalprogln
20
<210>9
<211>25
<212>prt
<213>大肠杆菌
<400>9
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
seralapheserthralavalprogln
2025
<210>10
<211>25
<212>prt
<213>大肠杆菌
<400>10
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
seralaalaserthralavalprogln
2025
<210>11
<211>25
<212>prt
<213>大肠杆菌
<400>11
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
seralaglyserthralavalprogln
2025
<210>12
<211>25
<212>prt
<213>大肠杆菌
<400>12
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
seralavalserthralavalprogln
2025
<210>13
<211>24
<212>prt
<213>大肠杆菌
<400>13
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
seralaserthralavalprogln
20
<210>14
<211>25
<212>prt
<213>大肠杆菌
<400>14
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serglypheserthralavalprogln
2025
<210>15
<211>25
<212>prt
<213>大肠杆菌
<400>15
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serglyalaserthralavalprogln
2025
<210>16
<211>25
<212>prt
<213>大肠杆菌
<400>16
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serglyglyserthralavalprogln
2025
<210>17
<211>25
<212>prt
<213>大肠杆菌
<400>17
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serglyvalserthralavalprogln
2025
<210>18
<211>24
<212>prt
<213>大肠杆菌
<400>18
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serglyserthralavalprogln
20
<210>19
<211>25
<212>prt
<213>大肠杆菌
<400>19
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
servalpheserthralavalprogln
2025
<210>20
<211>25
<212>prt
<213>大肠杆菌
<400>20
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
servalalaserthralavalprogln
2025
<210>21
<211>25
<212>prt
<213>大肠杆菌
<400>21
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
servalglyserthralavalprogln
2025
<210>22
<211>25
<212>prt
<213>大肠杆菌
<400>22
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
servalvalserthralavalprogln
2025
<210>23
<211>24
<212>prt
<213>大肠杆菌
<400>23
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
servalserthralavalprogln
20
<210>24
<211>25
<212>prt
<213>大肠杆菌
<400>24
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serserpheserthralavalprogln
2025
<210>25
<211>25
<212>prt
<213>大肠杆菌
<400>25
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serseralaserthralavalprogln
2025
<210>26
<211>25
<212>prt
<213>大肠杆菌
<400>26
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serserglyserthralavalprogln
2025
<210>27
<211>25
<212>prt
<213>大肠杆菌
<400>27
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serservalserthralavalprogln
2025
<210>28
<211>24
<212>prt
<213>大肠杆菌
<400>28
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serserserthralavalprogln
20
<210>29
<211>25
<212>prt
<213>大肠杆菌
<400>29
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serthrpheserthralavalprogln
2025
<210>30
<211>25
<212>prt
<213>大肠杆菌
<400>30
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serthralaserthralavalprogln
2025
<210>31
<211>25
<212>prt
<213>大肠杆菌
<400>31
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serthrglyserthralavalprogln
2025
<210>32
<211>25
<212>prt
<213>大肠杆菌
<400>32
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serthrvalserthralavalprogln
2025
<210>33
<211>24
<212>prt
<213>大肠杆菌
<400>33
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serthrserthralavalprogln
20
<210>34
<211>24
<212>prt
<213>大肠杆菌
<400>34
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serpheserthralavalprogln
20
<210>35
<211>24
<212>prt
<213>大肠杆菌
<400>35
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
seralaserthralavalprogln
20
<210>36
<211>24
<212>prt
<213>大肠杆菌
<400>36
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serglyserthralavalprogln
20
<210>37
<211>24
<212>prt
<213>大肠杆菌
<400>37
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
servalserthralavalprogln
20
<210>38
<211>23
<212>prt
<213>大肠杆菌
<400>38
valtyrasnileglnaspgluthrglyglnphelysprotyrproala
151015
serserthralavalprogln
20
<210>39
<211>25
<212>prt
<213>大肠杆菌
<400>39
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serasnpheserthralavalprogln
2025
<210>40
<211>25
<212>prt
<213>大肠杆菌
<400>40
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serasnalaserthralavalprogln
2025
<210>41
<211>25
<212>prt
<213>大肠杆菌
<400>41
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serasnglyserthralavalprogln
2025
<210>42
<211>25
<212>prt
<213>大肠杆菌
<400>42
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serasnvalserthralavalprogln
2025
<210>43
<211>24
<212>prt
<213>大肠杆菌
<400>43
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serasnserthralavalprogln
20
<210>44
<211>25
<212>prt
<213>大肠杆菌
<400>44
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
seralapheserthralavalprogln
2025
<210>45
<211>25
<212>prt
<213>大肠杆菌
<400>45
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
seralaalaserthralavalprogln
2025
<210>46
<211>25
<212>prt
<213>大肠杆菌
<400>46
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
seralaglyserthralavalprogln
2025
<210>47
<211>25
<212>prt
<213>大肠杆菌
<400>47
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
seralavalserthralavalprogln
2025
<210>48
<211>24
<212>prt
<213>大肠杆菌
<400>48
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
seralaserthralavalprogln
20
<210>49
<211>25
<212>prt
<213>大肠杆菌
<400>49
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serglypheserthralavalprogln
2025
<210>50
<211>25
<212>prt
<213>大肠杆菌
<400>50
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serglyalaserthralavalprogln
2025
<210>51
<211>25
<212>prt
<213>大肠杆菌
<400>51
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serglyglyserthralavalprogln
2025
<210>52
<211>25
<212>prt
<213>大肠杆菌
<400>52
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serglyvalserthralavalprogln
2025
<210>53
<211>24
<212>prt
<213>大肠杆菌
<400>53
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serglyserthralavalprogln
20
<210>54
<211>25
<212>prt
<213>大肠杆菌
<400>54
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
servalpheserthralavalprogln
2025
<210>55
<211>25
<212>prt
<213>大肠杆菌
<400>55
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
servalalaserthralavalprogln
2025
<210>56
<211>25
<212>prt
<213>大肠杆菌
<400>56
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
servalglyserthralavalprogln
2025
<210>57
<211>25
<212>prt
<213>大肠杆菌
<400>57
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
servalvalserthralavalprogln
2025
<210>58
<211>24
<212>prt
<213>大肠杆菌
<400>58
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
servalserthralavalprogln
20
<210>59
<211>25
<212>prt
<213>大肠杆菌
<400>59
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serserpheserthralavalprogln
2025
<210>60
<211>25
<212>prt
<213>大肠杆菌
<400>60
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serseralaserthralavalprogln
2025
<210>61
<211>25
<212>prt
<213>大肠杆菌
<400>61
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serserglyserthralavalprogln
2025
<210>62
<211>25
<212>prt
<213>大肠杆菌
<400>62
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serservalserthralavalprogln
2025
<210>63
<211>24
<212>prt
<213>大肠杆菌
<400>63
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serserserthralavalprogln
20
<210>64
<211>25
<212>prt
<213>大肠杆菌
<400>64
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serthrpheserthralavalprogln
2025
<210>65
<211>25
<212>prt
<213>大肠杆菌
<400>65
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serthralaserthralavalprogln
2025
<210>66
<211>25
<212>prt
<213>大肠杆菌
<400>66
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serthrglyserthralavalprogln
2025
<210>67
<211>25
<212>prt
<213>大肠杆菌
<400>67
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serthrvalserthralavalprogln
2025
<210>68
<211>24
<212>prt
<213>大肠杆菌
<400>68
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serthrserthralavalprogln
20
<210>69
<211>24
<212>prt
<213>大肠杆菌
<400>69
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serpheserthralavalprogln
20
<210>70
<211>24
<212>prt
<213>大肠杆菌
<400>70
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
seralaserthralavalprogln
20
<210>71
<211>24
<212>prt
<213>大肠杆菌
<400>71
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serglyserthralavalprogln
20
<210>72
<211>24
<212>prt
<213>大肠杆菌
<400>72
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
servalserthralavalprogln
20
<210>73
<211>23
<212>prt
<213>大肠杆菌
<400>73
valtyrasnileglnaspgluthrglyglnphelysproalaproala
151015
serserthralavalprogln
20
<210>74
<211>25
<212>prt
<213>大肠杆菌
<400>74
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serasnpheserthralavalprogln
2025
<210>75
<211>25
<212>prt
<213>大肠杆菌
<400>75
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serasnalaserthralavalprogln
2025
<210>76
<211>25
<212>prt
<213>大肠杆菌
<400>76
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serasnglyserthralavalprogln
2025
<210>77
<211>25
<212>prt
<213>大肠杆菌
<400>77
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serasnvalserthralavalprogln
2025
<210>78
<211>24
<212>prt
<213>大肠杆菌
<400>78
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serasnserthralavalprogln
20
<210>79
<211>25
<212>prt
<213>大肠杆菌
<400>79
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
seralapheserthralavalprogln
2025
<210>80
<211>25
<212>prt
<213>大肠杆菌
<400>80
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
seralaalaserthralavalprogln
2025
<210>81
<211>25
<212>prt
<213>大肠杆菌
<400>81
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
seralaglyserthralavalprogln
2025
<210>82
<211>25
<212>prt
<213>大肠杆菌
<400>82
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
seralavalserthralavalprogln
2025
<210>83
<211>24
<212>prt
<213>大肠杆菌
<400>83
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
seralaserthralavalprogln
20
<210>84
<211>25
<212>prt
<213>大肠杆菌
<400>84
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serglypheserthralavalprogln
2025
<210>85
<211>25
<212>prt
<213>大肠杆菌
<400>85
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serglyalaserthralavalprogln
2025
<210>86
<211>25
<212>prt
<213>大肠杆菌
<400>86
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serglyglyserthralavalprogln
2025
<210>87
<211>25
<212>prt
<213>大肠杆菌
<400>87
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serglyvalserthralavalprogln
2025
<210>88
<211>24
<212>prt
<213>大肠杆菌
<400>88
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serglyserthralavalprogln
20
<210>89
<211>25
<212>prt
<213>大肠杆菌
<400>89
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
servalpheserthralavalprogln
2025
<210>90
<211>25
<212>prt
<213>大肠杆菌
<400>90
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
servalalaserthralavalprogln
2025
<210>91
<211>25
<212>prt
<213>大肠杆菌
<400>91
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
servalglyserthralavalprogln
2025
<210>92
<211>25
<212>prt
<213>大肠杆菌
<400>92
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
servalvalserthralavalprogln
2025
<210>93
<211>24
<212>prt
<213>大肠杆菌
<400>93
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
servalserthralavalprogln
20
<210>94
<211>25
<212>prt
<213>大肠杆菌
<400>94
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serserpheserthralavalprogln
2025
<210>95
<211>25
<212>prt
<213>大肠杆菌
<400>95
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serseralaserthralavalprogln
2025
<210>96
<211>25
<212>prt
<213>大肠杆菌
<400>96
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serserglyserthralavalprogln
2025
<210>97
<211>25
<212>prt
<213>大肠杆菌
<400>97
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serservalserthralavalprogln
2025
<210>98
<211>24
<212>prt
<213>大肠杆菌
<400>98
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serserserthralavalprogln
20
<210>99
<211>25
<212>prt
<213>大肠杆菌
<400>99
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serthrpheserthralavalprogln
2025
<210>100
<211>25
<212>prt
<213>大肠杆菌
<400>100
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serthralaserthralavalprogln
2025
<210>101
<211>25
<212>prt
<213>大肠杆菌
<400>101
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serthrglyserthralavalprogln
2025
<210>102
<211>25
<212>prt
<213>大肠杆菌
<400>102
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serthrvalserthralavalprogln
2025
<210>103
<211>24
<212>prt
<213>大肠杆菌
<400>103
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serthrserthralavalprogln
20
<210>104
<211>24
<212>prt
<213>大肠杆菌
<400>104
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serpheserthralavalprogln
20
<210>105
<211>24
<212>prt
<213>大肠杆菌
<400>105
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
seralaserthralavalprogln
20
<210>106
<211>24
<212>prt
<213>大肠杆菌
<400>106
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serglyserthralavalprogln
20
<210>107
<211>24
<212>prt
<213>大肠杆菌
<400>107
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
servalserthralavalprogln
20
<210>108
<211>23
<212>prt
<213>大肠杆菌
<400>108
valtyrasnileglnaspgluthrglyglnphelysproglyproala
151015
serserthralavalprogln
20
<210>109
<211>25
<212>prt
<213>大肠杆菌
<400>109
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serasnpheserthralavalprogln
2025
<210>110
<211>25
<212>prt
<213>大肠杆菌
<400>110
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serasnalaserthralavalprogln
2025
<210>111
<211>25
<212>prt
<213>大肠杆菌
<400>111
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serasnglyserthralavalprogln
2025
<210>112
<211>25
<212>prt
<213>大肠杆菌
<400>112
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serasnvalserthralavalprogln
2025
<210>113
<211>24
<212>prt
<213>大肠杆菌
<400>113
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serasnserthralavalprogln
20
<210>114
<211>25
<212>prt
<213>大肠杆菌
<400>114
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
seralapheserthralavalprogln
2025
<210>115
<211>25
<212>prt
<213>大肠杆菌
<400>115
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
seralaalaserthralavalprogln
2025
<210>116
<211>25
<212>prt
<213>大肠杆菌
<400>116
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
seralaglyserthralavalprogln
2025
<210>117
<211>25
<212>prt
<213>大肠杆菌
<400>117
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
seralavalserthralavalprogln
2025
<210>118
<211>24
<212>prt
<213>大肠杆菌
<400>118
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
seralaserthralavalprogln
20
<210>119
<211>25
<212>prt
<213>大肠杆菌
<400>119
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serglypheserthralavalprogln
2025
<210>120
<211>25
<212>prt
<213>大肠杆菌
<400>120
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serglyalaserthralavalprogln
2025
<210>121
<211>25
<212>prt
<213>大肠杆菌
<400>121
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serglyglyserthralavalprogln
2025
<210>122
<211>25
<212>prt
<213>大肠杆菌
<400>122
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serglyvalserthralavalprogln
2025
<210>123
<211>24
<212>prt
<213>大肠杆菌
<400>123
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serglyserthralavalprogln
20
<210>124
<211>25
<212>prt
<213>大肠杆菌
<400>124
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
servalpheserthralavalprogln
2025
<210>125
<211>25
<212>prt
<213>大肠杆菌
<400>125
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
servalalaserthralavalprogln
2025
<210>126
<211>25
<212>prt
<213>大肠杆菌
<400>126
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
servalglyserthralavalprogln
2025
<210>127
<211>25
<212>prt
<213>大肠杆菌
<400>127
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
servalvalserthralavalprogln
2025
<210>128
<211>24
<212>prt
<213>大肠杆菌
<400>128
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
servalserthralavalprogln
20
<210>129
<211>25
<212>prt
<213>大肠杆菌
<400>129
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serserpheserthralavalprogln
2025
<210>130
<211>25
<212>prt
<213>大肠杆菌
<400>130
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serseralaserthralavalprogln
2025
<210>131
<211>25
<212>prt
<213>大肠杆菌
<400>131
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serserglyserthralavalprogln
2025
<210>132
<211>25
<212>prt
<213>大肠杆菌
<400>132
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serservalserthralavalprogln
2025
<210>133
<211>24
<212>prt
<213>大肠杆菌
<400>133
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serserserthralavalprogln
20
<210>134
<211>25
<212>prt
<213>大肠杆菌
<400>134
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serthrpheserthralavalprogln
2025
<210>135
<211>25
<212>prt
<213>大肠杆菌
<400>135
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serthralaserthralavalprogln
2025
<210>136
<211>25
<212>prt
<213>大肠杆菌
<400>136
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serthrglyserthralavalprogln
2025
<210>137
<211>25
<212>prt
<213>大肠杆菌
<400>137
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serthrvalserthralavalprogln
2025
<210>138
<211>24
<212>prt
<213>大肠杆菌
<400>138
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serthrserthralavalprogln
20
<210>139
<211>24
<212>prt
<213>大肠杆菌
<400>139
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serpheserthralavalprogln
20
<210>140
<211>24
<212>prt
<213>大肠杆菌
<400>140
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
seralaserthralavalprogln
20
<210>141
<211>24
<212>prt
<213>大肠杆菌
<400>141
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serglyserthralavalprogln
20
<210>142
<211>24
<212>prt
<213>大肠杆菌
<400>142
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
servalserthralavalprogln
20
<210>143
<211>23
<212>prt
<213>大肠杆菌
<400>143
valtyrasnileglnaspgluthrglyglnphelysprovalproala
151015
serserthralavalprogln
20
<210>144
<211>25
<212>prt
<213>大肠杆菌
<400>144
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serasnpheserthralavalprogln
2025
<210>145
<211>25
<212>prt
<213>大肠杆菌
<400>145
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serasnalaserthralavalprogln
2025
<210>146
<211>25
<212>prt
<213>大肠杆菌
<400>146
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serasnglyserthralavalprogln
2025
<210>147
<211>25
<212>prt
<213>大肠杆菌
<400>147
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serasnvalserthralavalprogln
2025
<210>148
<211>24
<212>prt
<213>大肠杆菌
<400>148
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serasnserthralavalprogln
20
<210>149
<211>25
<212>prt
<213>大肠杆菌
<400>149
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
seralapheserthralavalprogln
2025
<210>150
<211>25
<212>prt
<213>大肠杆菌
<400>150
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
seralaalaserthralavalprogln
2025
<210>151
<211>25
<212>prt
<213>大肠杆菌
<400>151
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
seralaglyserthralavalprogln
2025
<210>152
<211>25
<212>prt
<213>大肠杆菌
<400>152
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
seralavalserthralavalprogln
2025
<210>153
<211>24
<212>prt
<213>大肠杆菌
<400>153
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
seralaserthralavalprogln
20
<210>154
<211>25
<212>prt
<213>大肠杆菌
<400>154
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serglypheserthralavalprogln
2025
<210>155
<211>25
<212>prt
<213>大肠杆菌
<400>155
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serglyalaserthralavalprogln
2025
<210>156
<211>25
<212>prt
<213>大肠杆菌
<400>156
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serglyglyserthralavalprogln
2025
<210>157
<211>25
<212>prt
<213>大肠杆菌
<400>157
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serglyvalserthralavalprogln
2025
<210>158
<211>24
<212>prt
<213>大肠杆菌
<400>158
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serglyserthralavalprogln
20
<210>159
<211>25
<212>prt
<213>大肠杆菌
<400>159
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
servalpheserthralavalprogln
2025
<210>160
<211>25
<212>prt
<213>大肠杆菌
<400>160
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
servalalaserthralavalprogln
2025
<210>161
<211>25
<212>prt
<213>大肠杆菌
<400>161
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
servalglyserthralavalprogln
2025
<210>162
<211>25
<212>prt
<213>大肠杆菌
<400>162
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
servalvalserthralavalprogln
2025
<210>163
<211>24
<212>prt
<213>大肠杆菌
<400>163
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
servalserthralavalprogln
20
<210>164
<211>25
<212>prt
<213>大肠杆菌
<400>164
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serserpheserthralavalprogln
2025
<210>165
<211>25
<212>prt
<213>大肠杆菌
<400>165
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serseralaserthralavalprogln
2025
<210>166
<211>25
<212>prt
<213>大肠杆菌
<400>166
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serserglyserthralavalprogln
2025
<210>167
<211>25
<212>prt
<213>大肠杆菌
<400>167
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serservalserthralavalprogln
2025
<210>168
<211>24
<212>prt
<213>大肠杆菌
<400>168
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serserserthralavalprogln
20
<210>169
<211>25
<212>prt
<213>大肠杆菌
<400>169
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serthrpheserthralavalprogln
2025
<210>170
<211>25
<212>prt
<213>大肠杆菌
<400>170
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serthralaserthralavalprogln
2025
<210>171
<211>25
<212>prt
<213>大肠杆菌
<400>171
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serthrglyserthralavalprogln
2025
<210>172
<211>25
<212>prt
<213>大肠杆菌
<400>172
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serthrvalserthralavalprogln
2025
<210>173
<211>24
<212>prt
<213>大肠杆菌
<400>173
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serthrserthralavalprogln
20
<210>174
<211>24
<212>prt
<213>大肠杆菌
<400>174
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serpheserthralavalprogln
20
<210>175
<211>24
<212>prt
<213>大肠杆菌
<400>175
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
seralaserthralavalprogln
20
<210>176
<211>24
<212>prt
<213>大肠杆菌
<400>176
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serglyserthralavalprogln
20
<210>177
<211>24
<212>prt
<213>大肠杆菌
<400>177
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
servalserthralavalprogln
20
<210>178
<211>23
<212>prt
<213>大肠杆菌
<400>178
valtyrasnileglnaspgluthrglyglnphelysproleuproala
151015
serserthralavalprogln
20
<210>179
<211>25
<212>prt
<213>大肠杆菌
<400>179
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serasnpheserthralavalprogln
2025
<210>180
<211>25
<212>prt
<213>大肠杆菌
<400>180
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serasnalaserthralavalprogln
2025
<210>181
<211>25
<212>prt
<213>大肠杆菌
<400>181
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serasnglyserthralavalprogln
2025
<210>182
<211>25
<212>prt
<213>大肠杆菌
<400>182
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serasnvalserthralavalprogln
2025
<210>183
<211>24
<212>prt
<213>大肠杆菌
<400>183
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serasnserthralavalprogln
20
<210>184
<211>25
<212>prt
<213>大肠杆菌
<400>184
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
seralapheserthralavalprogln
2025
<210>185
<211>25
<212>prt
<213>大肠杆菌
<400>185
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
seralaalaserthralavalprogln
2025
<210>186
<211>25
<212>prt
<213>大肠杆菌
<400>186
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
seralaglyserthralavalprogln
2025
<210>187
<211>25
<212>prt
<213>大肠杆菌
<400>187
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
seralavalserthralavalprogln
2025
<210>188
<211>24
<212>prt
<213>大肠杆菌
<400>188
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
seralaserthralavalprogln
20
<210>189
<211>25
<212>prt
<213>大肠杆菌
<400>189
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serglypheserthralavalprogln
2025
<210>190
<211>25
<212>prt
<213>大肠杆菌
<400>190
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serglyalaserthralavalprogln
2025
<210>191
<211>25
<212>prt
<213>大肠杆菌
<400>191
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serglyglyserthralavalprogln
2025
<210>192
<211>25
<212>prt
<213>大肠杆菌
<400>192
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serglyvalserthralavalprogln
2025
<210>193
<211>24
<212>prt
<213>大肠杆菌
<400>193
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serglyserthralavalprogln
20
<210>194
<211>25
<212>prt
<213>大肠杆菌
<400>194
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
servalpheserthralavalprogln
2025
<210>195
<211>25
<212>prt
<213>大肠杆菌
<400>195
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
servalalaserthralavalprogln
2025
<210>196
<211>25
<212>prt
<213>大肠杆菌
<400>196
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
servalglyserthralavalprogln
2025
<210>197
<211>25
<212>prt
<213>大肠杆菌
<400>197
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
servalvalserthralavalprogln
2025
<210>198
<211>24
<212>prt
<213>大肠杆菌
<400>198
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
servalserthralavalprogln
20
<210>199
<211>25
<212>prt
<213>大肠杆菌
<400>199
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serserpheserthralavalprogln
2025
<210>200
<211>25
<212>prt
<213>大肠杆菌
<400>200
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serseralaserthralavalprogln
2025
<210>201
<211>25
<212>prt
<213>大肠杆菌
<400>201
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serserglyserthralavalprogln
2025
<210>202
<211>25
<212>prt
<213>大肠杆菌
<400>202
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serservalserthralavalprogln
2025
<210>203
<211>24
<212>prt
<213>大肠杆菌
<400>203
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serserserthralavalprogln
20
<210>204
<211>25
<212>prt
<213>大肠杆菌
<400>204
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serthrpheserthralavalprogln
2025
<210>205
<211>25
<212>prt
<213>大肠杆菌
<400>205
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serthralaserthralavalprogln
2025
<210>206
<211>25
<212>prt
<213>大肠杆菌
<400>206
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serthrglyserthralavalprogln
2025
<210>207
<211>25
<212>prt
<213>大肠杆菌
<400>207
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serthrvalserthralavalprogln
2025
<210>208
<211>24
<212>prt
<213>大肠杆菌
<400>208
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serthrserthralavalprogln
20
<210>209
<211>24
<212>prt
<213>大肠杆菌
<400>209
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serpheserthralavalprogln
20
<210>210
<211>24
<212>prt
<213>大肠杆菌
<400>210
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
seralaserthralavalprogln
20
<210>211
<211>24
<212>prt
<213>大肠杆菌
<400>211
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serglyserthralavalprogln
20
<210>212
<211>24
<212>prt
<213>大肠杆菌
<400>212
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
servalserthralavalprogln
20
<210>213
<211>23
<212>prt
<213>大肠杆菌
<400>213
valtyrasnileglnaspgluthrglyglnphelysproileproala
151015
serserthralavalprogln
20
<210>214
<211>25
<212>prt
<213>大肠杆菌
<400>214
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serasnpheserthralavalprogln
2025
<210>215
<211>25
<212>prt
<213>大肠杆菌
<400>215
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serasnalaserthralavalprogln
2025
<210>216
<211>25
<212>prt
<213>大肠杆菌
<400>216
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serasnglyserthralavalprogln
2025
<210>217
<211>25
<212>prt
<213>大肠杆菌
<400>217
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serasnvalserthralavalprogln
2025
<210>218
<211>24
<212>prt
<213>大肠杆菌
<400>218
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serasnserthralavalprogln
20
<210>219
<211>25
<212>prt
<213>大肠杆菌
<400>219
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
seralapheserthralavalprogln
2025
<210>220
<211>25
<212>prt
<213>大肠杆菌
<400>220
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
seralaalaserthralavalprogln
2025
<210>221
<211>25
<212>prt
<213>大肠杆菌
<400>221
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
seralaglyserthralavalprogln
2025
<210>222
<211>25
<212>prt
<213>大肠杆菌
<400>222
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
seralavalserthralavalprogln
2025
<210>223
<211>24
<212>prt
<213>大肠杆菌
<400>223
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
seralaserthralavalprogln
20
<210>224
<211>25
<212>prt
<213>大肠杆菌
<400>224
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serglypheserthralavalprogln
2025
<210>225
<211>25
<212>prt
<213>大肠杆菌
<400>225
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serglyalaserthralavalprogln
2025
<210>226
<211>25
<212>prt
<213>大肠杆菌
<400>226
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serglyglyserthralavalprogln
2025
<210>227
<211>25
<212>prt
<213>大肠杆菌
<400>227
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serglyvalserthralavalprogln
2025
<210>228
<211>24
<212>prt
<213>大肠杆菌
<400>228
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serglyserthralavalprogln
20
<210>229
<211>25
<212>prt
<213>大肠杆菌
<400>229
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
servalpheserthralavalprogln
2025
<210>230
<211>25
<212>prt
<213>大肠杆菌
<400>230
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
servalalaserthralavalprogln
2025
<210>231
<211>25
<212>prt
<213>大肠杆菌
<400>231
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
servalglyserthralavalprogln
2025
<210>232
<211>25
<212>prt
<213>大肠杆菌
<400>232
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
servalvalserthralavalprogln
2025
<210>233
<211>24
<212>prt
<213>大肠杆菌
<400>233
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
servalserthralavalprogln
20
<210>234
<211>25
<212>prt
<213>大肠杆菌
<400>234
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serserpheserthralavalprogln
2025
<210>235
<211>25
<212>prt
<213>大肠杆菌
<400>235
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serseralaserthralavalprogln
2025
<210>236
<211>25
<212>prt
<213>大肠杆菌
<400>236
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serserglyserthralavalprogln
2025
<210>237
<211>25
<212>prt
<213>大肠杆菌
<400>237
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serservalserthralavalprogln
2025
<210>238
<211>24
<212>prt
<213>大肠杆菌
<400>238
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serserserthralavalprogln
20
<210>239
<211>25
<212>prt
<213>大肠杆菌
<400>239
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serthrpheserthralavalprogln
2025
<210>240
<211>25
<212>prt
<213>大肠杆菌
<400>240
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serthralaserthralavalprogln
2025
<210>241
<211>25
<212>prt
<213>大肠杆菌
<400>241
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serthrglyserthralavalprogln
2025
<210>242
<211>25
<212>prt
<213>大肠杆菌
<400>242
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serthrvalserthralavalprogln
2025
<210>243
<211>24
<212>prt
<213>大肠杆菌
<400>243
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serthrserthralavalprogln
20
<210>244
<211>24
<212>prt
<213>大肠杆菌
<400>244
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serpheserthralavalprogln
20
<210>245
<211>24
<212>prt
<213>大肠杆菌
<400>245
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
seralaserthralavalprogln
20
<210>246
<211>24
<212>prt
<213>大肠杆菌
<400>246
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serglyserthralavalprogln
20
<210>247
<211>24
<212>prt
<213>大肠杆菌
<400>247
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
servalserthralavalprogln
20
<210>248
<211>23
<212>prt
<213>大肠杆菌
<400>248
valtyrasnileglnaspgluthrglyglnphelysproasnproala
151015
serserthralavalprogln
20
<210>249
<211>25
<212>prt
<213>大肠杆菌
<400>249
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serasnpheserthralavalprogln
2025
<210>250
<211>25
<212>prt
<213>大肠杆菌
<400>250
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serasnalaserthralavalprogln
2025
<210>251
<211>25
<212>prt
<213>大肠杆菌
<400>251
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serasnglyserthralavalprogln
2025
<210>252
<211>25
<212>prt
<213>大肠杆菌
<400>252
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serasnvalserthralavalprogln
2025
<210>253
<211>24
<212>prt
<213>大肠杆菌
<400>253
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serasnserthralavalprogln
20
<210>254
<211>25
<212>prt
<213>大肠杆菌
<400>254
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
seralapheserthralavalprogln
2025
<210>255
<211>25
<212>prt
<213>大肠杆菌
<400>255
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
seralaalaserthralavalprogln
2025
<210>256
<211>25
<212>prt
<213>大肠杆菌
<400>256
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
seralaglyserthralavalprogln
2025
<210>257
<211>25
<212>prt
<213>大肠杆菌
<400>257
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
seralavalserthralavalprogln
2025
<210>258
<211>24
<212>prt
<213>大肠杆菌
<400>258
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
seralaserthralavalprogln
20
<210>259
<211>25
<212>prt
<213>大肠杆菌
<400>259
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serglypheserthralavalprogln
2025
<210>260
<211>25
<212>prt
<213>大肠杆菌
<400>260
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serglyalaserthralavalprogln
2025
<210>261
<211>25
<212>prt
<213>大肠杆菌
<400>261
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serglyglyserthralavalprogln
2025
<210>262
<211>25
<212>prt
<213>大肠杆菌
<400>262
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serglyvalserthralavalprogln
2025
<210>263
<211>24
<212>prt
<213>大肠杆菌
<400>263
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serglyserthralavalprogln
20
<210>264
<211>25
<212>prt
<213>大肠杆菌
<400>264
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
servalpheserthralavalprogln
2025
<210>265
<211>25
<212>prt
<213>大肠杆菌
<400>265
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
servalalaserthralavalprogln
2025
<210>266
<211>25
<212>prt
<213>大肠杆菌
<400>266
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
servalglyserthralavalprogln
2025
<210>267
<211>25
<212>prt
<213>大肠杆菌
<400>267
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
servalvalserthralavalprogln
2025
<210>268
<211>24
<212>prt
<213>大肠杆菌
<400>268
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
servalserthralavalprogln
20
<210>269
<211>25
<212>prt
<213>大肠杆菌
<400>269
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serserpheserthralavalprogln
2025
<210>270
<211>25
<212>prt
<213>大肠杆菌
<400>270
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serseralaserthralavalprogln
2025
<210>271
<211>25
<212>prt
<213>大肠杆菌
<400>271
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serserglyserthralavalprogln
2025
<210>272
<211>25
<212>prt
<213>大肠杆菌
<400>272
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serservalserthralavalprogln
2025
<210>273
<211>24
<212>prt
<213>大肠杆菌
<400>273
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serserserthralavalprogln
20
<210>274
<211>25
<212>prt
<213>大肠杆菌
<400>274
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serthrpheserthralavalprogln
2025
<210>275
<211>25
<212>prt
<213>大肠杆菌
<400>275
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serthralaserthralavalprogln
2025
<210>276
<211>25
<212>prt
<213>大肠杆菌
<400>276
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serthrglyserthralavalprogln
2025
<210>277
<211>25
<212>prt
<213>大肠杆菌
<400>277
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serthrvalserthralavalprogln
2025
<210>278
<211>24
<212>prt
<213>大肠杆菌
<400>278
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serthrserthralavalprogln
20
<210>279
<211>24
<212>prt
<213>大肠杆菌
<400>279
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serpheserthralavalprogln
20
<210>280
<211>24
<212>prt
<213>大肠杆菌
<400>280
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
seralaserthralavalprogln
20
<210>281
<211>24
<212>prt
<213>大肠杆菌
<400>281
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serglyserthralavalprogln
20
<210>282
<211>24
<212>prt
<213>大肠杆菌
<400>282
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
servalserthralavalprogln
20
<210>283
<211>23
<212>prt
<213>大肠杆菌
<400>283
valtyrasnileglnaspgluthrglyglnphelysproglnproala
151015
serserthralavalprogln
20
<210>284
<211>25
<212>prt
<213>大肠杆菌
<400>284
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serasnpheserthralavalprogln
2025
<210>285
<211>25
<212>prt
<213>大肠杆菌
<400>285
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serasnalaserthralavalprogln
2025
<210>286
<211>25
<212>prt
<213>大肠杆菌
<400>286
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serasnglyserthralavalprogln
2025
<210>287
<211>25
<212>prt
<213>大肠杆菌
<400>287
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serasnvalserthralavalprogln
2025
<210>288
<211>24
<212>prt
<213>大肠杆菌
<400>288
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serasnserthralavalprogln
20
<210>289
<211>25
<212>prt
<213>大肠杆菌
<400>289
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
seralapheserthralavalprogln
2025
<210>290
<211>25
<212>prt
<213>大肠杆菌
<400>290
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
seralaalaserthralavalprogln
2025
<210>291
<211>25
<212>prt
<213>大肠杆菌
<400>291
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
seralaglyserthralavalprogln
2025
<210>292
<211>25
<212>prt
<213>大肠杆菌
<400>292
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
seralavalserthralavalprogln
2025
<210>293
<211>24
<212>prt
<213>大肠杆菌
<400>293
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
seralaserthralavalprogln
20
<210>294
<211>25
<212>prt
<213>大肠杆菌
<400>294
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serglypheserthralavalprogln
2025
<210>295
<211>25
<212>prt
<213>大肠杆菌
<400>295
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serglyalaserthralavalprogln
2025
<210>296
<211>25
<212>prt
<213>大肠杆菌
<400>296
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serglyglyserthralavalprogln
2025
<210>297
<211>25
<212>prt
<213>大肠杆菌
<400>297
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serglyvalserthralavalprogln
2025
<210>298
<211>24
<212>prt
<213>大肠杆菌
<400>298
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serglyserthralavalprogln
20
<210>299
<211>25
<212>prt
<213>大肠杆菌
<400>299
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
servalpheserthralavalprogln
2025
<210>300
<211>25
<212>prt
<213>大肠杆菌
<400>300
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
servalalaserthralavalprogln
2025
<210>301
<211>25
<212>prt
<213>大肠杆菌
<400>301
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
servalglyserthralavalprogln
2025
<210>302
<211>25
<212>prt
<213>大肠杆菌
<400>302
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
servalvalserthralavalprogln
2025
<210>303
<211>24
<212>prt
<213>大肠杆菌
<400>303
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
servalserthralavalprogln
20
<210>304
<211>25
<212>prt
<213>大肠杆菌
<400>304
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serserpheserthralavalprogln
2025
<210>305
<211>25
<212>prt
<213>大肠杆菌
<400>305
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serseralaserthralavalprogln
2025
<210>306
<211>25
<212>prt
<213>大肠杆菌
<400>306
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serserglyserthralavalprogln
2025
<210>307
<211>25
<212>prt
<213>大肠杆菌
<400>307
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serservalserthralavalprogln
2025
<210>308
<211>24
<212>prt
<213>大肠杆菌
<400>308
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serserserthralavalprogln
20
<210>309
<211>25
<212>prt
<213>大肠杆菌
<400>309
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serthrpheserthralavalprogln
2025
<210>310
<211>25
<212>prt
<213>大肠杆菌
<400>310
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serthralaserthralavalprogln
2025
<210>311
<211>25
<212>prt
<213>大肠杆菌
<400>311
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serthrglyserthralavalprogln
2025
<210>312
<211>25
<212>prt
<213>大肠杆菌
<400>312
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serthrvalserthralavalprogln
2025
<210>313
<211>24
<212>prt
<213>大肠杆菌
<400>313
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serthrserthralavalprogln
20
<210>314
<211>24
<212>prt
<213>大肠杆菌
<400>314
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serpheserthralavalprogln
20
<210>315
<211>24
<212>prt
<213>大肠杆菌
<400>315
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
seralaserthralavalprogln
20
<210>316
<211>24
<212>prt
<213>大肠杆菌
<400>316
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serglyserthralavalprogln
20
<210>317
<211>24
<212>prt
<213>大肠杆菌
<400>317
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
servalserthralavalprogln
20
<210>318
<211>23
<212>prt
<213>大肠杆菌
<400>318
valtyrasnileglnaspgluthrglyglnphelyspropheproala
151015
serserthralavalprogln
20
<210>319
<211>24
<212>prt
<213>大肠杆菌
<400>319
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
asnpheserthralavalprogln
20
<210>320
<211>24
<212>prt
<213>大肠杆菌
<400>320
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
asnalaserthralavalprogln
20
<210>321
<211>24
<212>prt
<213>大肠杆菌
<400>321
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
asnglyserthralavalprogln
20
<210>322
<211>24
<212>prt
<213>大肠杆菌
<400>322
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
asnvalserthralavalprogln
20
<210>323
<211>23
<212>prt
<213>大肠杆菌
<400>323
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
asnserthralavalprogln
20
<210>324
<211>24
<212>prt
<213>大肠杆菌
<400>324
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
alapheserthralavalprogln
20
<210>325
<211>24
<212>prt
<213>大肠杆菌
<400>325
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
alaalaserthralavalprogln
20
<210>326
<211>24
<212>prt
<213>大肠杆菌
<400>326
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
alaglyserthralavalprogln
20
<210>327
<211>24
<212>prt
<213>大肠杆菌
<400>327
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
alavalserthralavalprogln
20
<210>328
<211>23
<212>prt
<213>大肠杆菌
<400>328
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
alaserthralavalprogln
20
<210>329
<211>24
<212>prt
<213>大肠杆菌
<400>329
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
glypheserthralavalprogln
20
<210>330
<211>24
<212>prt
<213>大肠杆菌
<400>330
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
glyalaserthralavalprogln
20
<210>331
<211>24
<212>prt
<213>大肠杆菌
<400>331
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
glyglyserthralavalprogln
20
<210>332
<211>24
<212>prt
<213>大肠杆菌
<400>332
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
glyvalserthralavalprogln
20
<210>333
<211>23
<212>prt
<213>大肠杆菌
<400>333
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
glyserthralavalprogln
20
<210>334
<211>24
<212>prt
<213>大肠杆菌
<400>334
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
valpheserthralavalprogln
20
<210>335
<211>24
<212>prt
<213>大肠杆菌
<400>335
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
valalaserthralavalprogln
20
<210>336
<211>24
<212>prt
<213>大肠杆菌
<400>336
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
valglyserthralavalprogln
20
<210>337
<211>24
<212>prt
<213>大肠杆菌
<400>337
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
valvalserthralavalprogln
20
<210>338
<211>23
<212>prt
<213>大肠杆菌
<400>338
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
valserthralavalprogln
20
<210>339
<211>24
<212>prt
<213>大肠杆菌
<400>339
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
serpheserthralavalprogln
20
<210>340
<211>24
<212>prt
<213>大肠杆菌
<400>340
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
seralaserthralavalprogln
20
<210>341
<211>24
<212>prt
<213>大肠杆菌
<400>341
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
serglyserthralavalprogln
20
<210>342
<211>24
<212>prt
<213>大肠杆菌
<400>342
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
servalserthralavalprogln
20
<210>343
<211>23
<212>prt
<213>大肠杆菌
<400>343
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
serserthralavalprogln
20
<210>344
<211>24
<212>prt
<213>大肠杆菌
<400>344
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
thrpheserthralavalprogln
20
<210>345
<211>24
<212>prt
<213>大肠杆菌
<400>345
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
thralaserthralavalprogln
20
<210>346
<211>24
<212>prt
<213>大肠杆菌
<400>346
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
thrglyserthralavalprogln
20
<210>347
<211>24
<212>prt
<213>大肠杆菌
<400>347
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
thrvalserthralavalprogln
20
<210>348
<211>23
<212>prt
<213>大肠杆菌
<400>348
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
thrserthralavalprogln
20
<210>349
<211>23
<212>prt
<213>大肠杆菌
<400>349
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
pheserthralavalprogln
20
<210>350
<211>23
<212>prt
<213>大肠杆菌
<400>350
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
alaserthralavalprogln
20
<210>351
<211>23
<212>prt
<213>大肠杆菌
<400>351
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
glyserthralavalprogln
20
<210>352
<211>23
<212>prt
<213>大肠杆菌
<400>352
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
valserthralavalprogln
20
<210>353
<211>22
<212>prt
<213>大肠杆菌
<400>353
valtyrasnileglnaspgluthrglyglnphelysproproalaser
151015
serthralavalprogln
20
<210>354
<211>23
<212>prt
<213>大肠杆菌
<400>354
valtyrasnileglnaspgluthrglyglnphelysglyglyserasn
151015
pheserthralavalprogln
20
<210>355
<211>23
<212>prt
<213>大肠杆菌
<400>355
valtyrasnileglnaspgluthrglyglnphelysglyglyserasn
151015
alaserthralavalprogln
20
<210>356
<211>23
<212>prt
<213>大肠杆菌
<400>356
valtyrasnileglnaspgluthrglyglnphelysglyglyserasn
151015
glyserthralavalprogln
20
<210>357
<211>23
<212>prt
<213>大肠杆菌
<400>357
valtyrasnileglnaspgluthrglyglnphelysglyglyserasn
151015
valserthralavalprogln
20
<210>358
<211>22
<212>prt
<213>大肠杆菌
<400>358
valtyrasnileglnaspgluthrglyglnphelysglyglyserasn
151015
serthralavalprogln
20
<210>359
<211>23
<212>prt
<213>大肠杆菌
<400>359
valtyrasnileglnaspgluthrglyglnphelysglyglyserala
151015
pheserthralavalprogln
20
<210>360
<211>23
<212>prt
<213>大肠杆菌
<400>360
valtyrasnileglnaspgluthrglyglnphelysglyglyserala
151015
alaserthralavalprogln
20
<210>361
<211>23
<212>prt
<213>大肠杆菌
<400>361
valtyrasnileglnaspgluthrglyglnphelysglyglyserala
151015
glyserthralavalprogln
20
<210>362
<211>23
<212>prt
<213>大肠杆菌
<400>362
valtyrasnileglnaspgluthrglyglnphelysglyglyserala
151015
valserthralavalprogln
20
<210>363
<211>22
<212>prt
<213>大肠杆菌
<400>363
valtyrasnileglnaspgluthrglyglnphelysglyglyserala
151015
serthralavalprogln
20
<210>364
<211>23
<212>prt
<213>大肠杆菌
<400>364
valtyrasnileglnaspgluthrglyglnphelysglyglysergly
151015
pheserthralavalprogln
20
<210>365
<211>23
<212>prt
<213>大肠杆菌
<400>365
valtyrasnileglnaspgluthrglyglnphelysglyglysergly
151015
alaserthralavalprogln
20
<210>366
<211>23
<212>prt
<213>大肠杆菌
<400>366
valtyrasnileglnaspgluthrglyglnphelysglyglysergly
151015
glyserthralavalprogln
20
<210>367
<211>23
<212>prt
<213>大肠杆菌
<400>367
valtyrasnileglnaspgluthrglyglnphelysglyglysergly
151015
valserthralavalprogln
20
<210>368
<211>22
<212>prt
<213>大肠杆菌
<400>368
valtyrasnileglnaspgluthrglyglnphelysglyglysergly
151015
serthralavalprogln
20
<210>369
<211>23
<212>prt
<213>大肠杆菌
<400>369
valtyrasnileglnaspgluthrglyglnphelysglyglyserval
151015
pheserthralavalprogln
20
<210>370
<211>23
<212>prt
<213>大肠杆菌
<400>370
valtyrasnileglnaspgluthrglyglnphelysglyglyserval
151015
alaserthralavalprogln
20
<210>371
<211>23
<212>prt
<213>大肠杆菌
<400>371
valtyrasnileglnaspgluthrglyglnphelysglyglyserval
151015
glyserthralavalprogln
20
<210>372
<211>23
<212>prt
<213>大肠杆菌
<400>372
valtyrasnileglnaspgluthrglyglnphelysglyglyserval
151015
valserthralavalprogln
20
<210>373
<211>22
<212>prt
<213>大肠杆菌
<400>373
valtyrasnileglnaspgluthrglyglnphelysglyglyserval
151015
serthralavalprogln
20
<210>374
<211>23
<212>prt
<213>大肠杆菌
<400>374
valtyrasnileglnaspgluthrglyglnphelysglyglyserser
151015
pheserthralavalprogln
20
<210>375
<211>23
<212>prt
<213>大肠杆菌
<400>375
valtyrasnileglnaspgluthrglyglnphelysglyglyserser
151015
alaserthralavalprogln
20
<210>376
<211>23
<212>prt
<213>大肠杆菌
<400>376
valtyrasnileglnaspgluthrglyglnphelysglyglyserser
151015
glyserthralavalprogln
20
<210>377
<211>23
<212>prt
<213>大肠杆菌
<400>377
valtyrasnileglnaspgluthrglyglnphelysglyglyserser
151015
valserthralavalprogln
20
<210>378
<211>22
<212>prt
<213>大肠杆菌
<400>378
valtyrasnileglnaspgluthrglyglnphelysglyglyserser
151015
serthralavalprogln
20
<210>379
<211>23
<212>prt
<213>大肠杆菌
<400>379
valtyrasnileglnaspgluthrglyglnphelysglyglyserthr
151015
pheserthralavalprogln
20
<210>380
<211>23
<212>prt
<213>大肠杆菌
<400>380
valtyrasnileglnaspgluthrglyglnphelysglyglyserthr
151015
alaserthralavalprogln
20
<210>381
<211>23
<212>prt
<213>大肠杆菌
<400>381
valtyrasnileglnaspgluthrglyglnphelysglyglyserthr
151015
glyserthralavalprogln
20
<210>382
<211>23
<212>prt
<213>大肠杆菌
<400>382
valtyrasnileglnaspgluthrglyglnphelysglyglyserthr
151015
valserthralavalprogln
20
<210>383
<211>22
<212>prt
<213>大肠杆菌
<400>383
valtyrasnileglnaspgluthrglyglnphelysglyglyserthr
151015
serthralavalprogln
20
<210>384
<211>22
<212>prt
<213>大肠杆菌
<400>384
valtyrasnileglnaspgluthrglyglnphelysglyglyserphe
151015
serthralavalprogln
20
<210>385
<211>22
<212>prt
<213>大肠杆菌
<400>385
valtyrasnileglnaspgluthrglyglnphelysglyglyserala
151015
serthralavalprogln
20
<210>386
<211>22
<212>prt
<213>大肠杆菌
<400>386
valtyrasnileglnaspgluthrglyglnphelysglyglysergly
151015
serthralavalprogln
20
<210>387
<211>22
<212>prt
<213>大肠杆菌
<400>387
valtyrasnileglnaspgluthrglyglnphelysglyglyserval
151015
serthralavalprogln
20
<210>388
<211>21
<212>prt
<213>大肠杆菌
<400>388
valtyrasnileglnaspgluthrglyglnphelysglyglyserser
151015
thralavalprogln
20
<210>389
<211>786
<212>dna
<213>大肠杆菌
<400>389
tgtctgaccgcaccgccgaaagaagcggcacgtccgaccctgatgccgcgtgcacagtct60
tataaagatctgacccatctgccggctccgacgggcaaaatttttgttagcgtctataac120
atccaggacgaaaccggtcaatttaaaccgtacccggcgagtaatttctccacggccgtt180
ccgcagagtgcaaccgctatgctggtcacggcactgaaagattcccgttggttcattccg240
ctggaacgccagggcctgcaaaacctgctgaatgaacgtaaaattatccgcgcagctcag300
gaaaacggtaccgtggccattaacaatcgtattccgctgcaaagcctgaccgccgcaaac360
atcatggttgaaggctctatcatcggttacgaatcaaacgtcaaatcgggcggtgtgggc420
gcacgttattttggcattggtgctgatacccagtaccaactggaccagatcgcagttaac480
ctgcgcgtggttaatgtcagcaccggcgaaattctgagctctgtgaataccagcaaaacg540
atcctgtcttacgaagtgcaggctggtgtttttcgtttcattgattatcaacgcctgctg600
gaaggcgaagtcggttacacctcaaacgaaccggtgatgctgtgtctgatgtcggcgatt660
gaaacgggtgttattttcctgatcaatgatggcatcgaccgtggtctgtgggatctgcag720
aacaaagccgaacgtcaaaatgacattctggtgaaataccgccacatgagtgttccgccg780
gaatcc786
<210>390
<211>262
<212>prt
<213>大肠杆菌
<400>390
cysleuthralaproprolysglualaalaargprothrleumetpro
151015
argalaglnsertyrlysaspleuthrhisleuproalaprothrgly
202530
lysilephevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thralametleuvalthralaleulysaspserargtrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalaileasnasnargilepro
100105110
leuglnserleuthralaalaasnilemetvalgluglyserileile
115120125
glytyrgluserasnvallysserglyglyvalglyalaargtyrphe
130135140
glyileglyalaaspthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluileleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglygluvalglytyrthrser
195200205
asngluprovalmetleucysleumetseralailegluthrglyval
210215220
ilepheleuileasnaspglyileaspargglyleutrpaspleugln
225230235240
asnlysalagluargglnasnaspileleuvallystyrarghismet
245250255
servalproprogluser
260
<210>391
<211>248
<212>prt
<213>克氏柠檬酸杆菌(citrobacterkoseri)
<400>391
metproargalaglnsertyrlysaspleuthrhisleuprometpro
151015
thrglylysilephevalservaltyrasnileglnaspgluthrgly
202530
glnphelysprotyrproalaserasnpheserthralavalprogln
354045
seralathralametleuvalthralaleulysaspserargtrpphe
505560
ileproleugluargglnglyleuglnasnleuleuasngluarglys
65707580
ileileargalaalaglngluasnglythrvalalaileasnasnarg
859095
ileproleuglnserleuthralaalaasnilemetvalgluglyser
100105110
ileileglytyrgluserasnvallysserglyglyvalglyalaarg
115120125
tyrpheglyileglyalaaspthrglntyrglnleuaspglnileala
130135140
valasnleuargvalvalasnvalserthrglygluileleuserser
145150155160
valasnthrserlysthrileleusertyrgluvalglnalaglyval
165170175
pheargpheileasptyrglnargleuleugluglygluileglytyr
180185190
thrserasngluprovalmetleucysleumetseralailegluthr
195200205
glyvalilepheleuileasnaspglyileaspargglyleutrpasp
210215220
leuglnasnlysalagluargglnasnaspileleuvallystyrarg
225230235240
hismetservalproprogluser
245
<210>392
<211>223
<212>prt
<213>沙门氏菌(salmonellaenterica)
<400>392
cysleuthralaproprolysglnalaalalysprothrleumetpro
151015
argalaglnsertyrlysaspleuthrhisleuproalaprothrgly
202530
lysilephevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thralametleuvalthralaleulysaspserargtrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalametasnasnargilepro
100105110
leuglnserleuthralaalaasnilemetvalgluglyserileile
115120125
glytyrgluserasnvallysserglyglyvalglyalaargtyrphe
130135140
glyileglyalaaspthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluileleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglygluileglytyrthrser
195200205
asngluprovalmetleucysleumetseralailegluthrgly
210215220
<210>393
<211>262
<212>prt
<213>无丙二酸柠檬酸杆菌(citrobacteramalonaticus)
<400>393
cysleuthralaproprolysglualaalalysprothrleumetpro
151015
argalaglnsertyrlysaspleuthrhisleuproileprothrgly
202530
lysilephevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thralametleuvalthralaleulysaspserargtrpphevalpro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalaileasnasnargilepro
100105110
leuglnserleuthralaalaasnilemetvalgluglyserileile
115120125
glytyrgluserasnvallysserglyglyvalglyalaargtyrphe
130135140
glyileglyalaaspthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluileleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglygluileglytyrthrser
195200205
asngluprovalmetleucysleumetseralailegluthrglyval
210215220
ilepheleuileasnaspglyileaspargglyleutrpaspleugln
225230235240
asnlysalaaspargglnasnaspileleuvallystyrarghismet
245250255
servalproprogluser
260
<210>394
<211>262
<212>prt
<213>鼠柠檬酸杆菌(citrobacterrodentium)
<400>394
cysleuthrthrproprolysglualaalalysprothrleumetpro
151015
argalaglnsertyrlysaspleuthrhisleuprovalprothrgly
202530
lysilephevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thralametleuvalthralaleulysaspserargtrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalaileasnasnargilepro
100105110
leuproserleuthralaalaasnilemetvalgluglyserileile
115120125
glytyrgluserasnvallysserglyglyalaglyalaargtyrphe
130135140
glyileglyalaaspthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluileleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglygluileglytyrthrser
195200205
asngluprovalmetleucysleumetseralailegluthrglyval
210215220
ilepheleuileasnaspglyileaspargglyleutrpaspleugln
225230235240
asnlysalaaspargglnasnaspileleuvallystyrargglnmet
245250255
servalproprogluser
260
<210>395
<211>262
<212>prt
<213>阿氏肠杆菌(enterobacterasburiae)
<400>395
cysleuthralaproprolysglualaalalysprothrleumetpro
151015
argalaglnsertyrargaspleuthrhisleuproalaprothrgly
202530
lysilephevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thralametleuvalthralaleulysaspserhistrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalaasnasnasnargmetpro
100105110
leuglnserleualaalaalaasnvalmetilegluglyserileile
115120125
glytyrgluserasnvallysserglyglyvalglyalaargtyrphe
130135140
glyileglyalaaspthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluvalleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglygluileglytyrthrser
195200205
asngluprovalmetmetcysleumetseralailegluthrglyval
210215220
ilepheleuileasnaspglyileaspargglyleutrpaspleugln
225230235240
asnlysalaaspalaglnasnprovalleuvallystyrargaspmet
245250255
servalproprogluser
260
<210>396
<211>1830
<212>dna
<213>枯草芽孢杆菌噬菌体phi29(bacillussubtilisphagephi29)
<400>396
atgaaacacatgccgcgtaaaatgtatagctgcgcgtttgaaaccacgaccaaagtggaa60
gattgtcgcgtttgggcctatggctacatgaacatcgaagatcattctgaatacaaaatc120
ggtaacagtctggatgaatttatggcatgggtgctgaaagttcaggcggatctgtacttc180
cacaacctgaaatttgatggcgcattcattatcaactggctggaacgtaatggctttaaa240
tggagcgcggatggtctgccgaacacgtataataccattatctctcgtatgggccagtgg300
tatatgattgatatctgcctgggctacaaaggtaaacgcaaaattcataccgtgatctat360
gatagcctgaaaaaactgccgtttccggtgaagaaaattgcgaaagatttcaaactgacg420
gttctgaaaggcgatattgattatcacaaagaacgtccggttggttacaaaatcaccccg480
gaagaatacgcatacatcaaaaacgatatccagatcatcgcagaagcgctgctgattcag540
tttaaacagggcctggatcgcatgaccgcgggcagtgatagcctgaaaggtttcaaagat600
atcatcacgaccaaaaaattcaaaaaagtgttcccgacgctgagcctgggtctggataaa660
gaagttcgttatgcctaccgcggcggttttacctggctgaacgatcgtttcaaagaaaaa720
gaaattggcgagggtatggtgtttgatgttaatagtctgtatccggcacagatgtacagc780
cgcctgctgccgtatggcgaaccgatcgtgttcgagggtaaatatgtttgggatgaagat840
tacccgctgcatattcagcacatccgttgtgaatttgaactgaaagaaggctatattccg900
accattcagatcaaacgtagtcgcttctataagggtaacgaatacctgaaaagctctggc960
ggtgaaatcgcggatctgtggctgagtaacgtggatctggaactgatgaaagaacactac1020
gatctgtacaacgttgaatacatcagcggcctgaaatttaaagccacgaccggtctgttc1080
aaagatttcatcgataaatggacctacatcaaaacgacctctgaaggcgcgattaaacag1140
ctggccaaactgatgctgaacagcctgtatggcaaattcgcctctaatccggatgtgacc1200
ggtaaagttccgtacctgaaagaaaatggcgcactgggttttcgcctgggcgaagaagaa1260
acgaaagatccggtgtataccccgatgggtgttttcattacggcctgggcacgttacacg1320
accatcaccgcggcccaggcatgctatgatcgcattatctactgtgataccgattctatt1380
catctgacgggcaccgaaatcccggatgtgattaaagatatcgttgatccgaaaaaactg1440
ggttattgggcccacgaaagtacgtttaaacgtgcaaaatacctgcgccagaaaacctac1500
atccaggatatctacatgaaagaagtggatggcaaactggttgaaggttctccggatgat1560
tacaccgatatcaaattcagtgtgaaatgcgccggcatgacggataaaatcaaaaaagaa1620
gtgaccttcgaaaacttcaaagttggtttcagccgcaaaatgaaaccgaaaccggtgcag1680
gttccgggcggtgtggttctggtggatgatacgtttaccattaaatctggcggtagtgcg1740
tggagccatccgcagttcgaaaaaggcggtggctctggtggcggttctggcggtagtgcc1800
tggagccacccgcagtttgaaaaataataa1830
<210>397
<211>608
<212>prt
<213>枯草芽孢杆菌噬菌体phi29
<400>397
metlyshismetproarglysmettyrsercysalaphegluthrthr
151015
thrlysvalgluaspcysargvaltrpalatyrglytyrmetasnile
202530
gluasphisserglutyrlysileglyasnserleuaspgluphemet
354045
alatrpvalleulysvalglnalaaspleutyrphehisasnleulys
505560
pheaspglyalapheileileasntrpleugluargasnglyphelys
65707580
trpseralaaspglyleuproasnthrtyrasnthrileileserarg
859095
metglyglntrptyrmetileaspilecysleuglytyrlysglylys
100105110
arglysilehisthrvaliletyraspserleulyslysleuprophe
115120125
provallyslysilealalysaspphelysleuthrvalleulysgly
130135140
aspileasptyrhislysgluargprovalglytyrlysilethrpro
145150155160
gluglutyralatyrilelysasnaspileglnileilealagluala
165170175
leuleuileglnphelysglnglyleuaspargmetthralaglyser
180185190
aspserleulysglyphelysaspileilethrthrlyslysphelys
195200205
lysvalpheprothrleuserleuglyleuasplysgluvalargtyr
210215220
alatyrargglyglyphethrtrpleuasnaspargphelysglulys
225230235240
gluileglygluglymetvalpheaspvalasnserleutyrproala
245250255
glnmettyrserargleuleuprotyrglygluproilevalpheglu
260265270
glylystyrvaltrpaspgluasptyrproleuhisileglnhisile
275280285
argcysgluphegluleulysgluglytyrileprothrileglnile
290295300
lysargserargphetyrlysglyasnglutyrleulyssersergly
305310315320
glygluilealaaspleutrpleuserasnvalaspleugluleumet
325330335
lysgluhistyraspleutyrasnvalglutyrileserglyleulys
340345350
phelysalathrthrglyleuphelysasppheileasplystrpthr
355360365
tyrilelysthrthrsergluglyalailelysglnleualalysleu
370375380
metleuasnserleutyrglylysphealaserasnproaspvalthr
385390395400
glylysvalprotyrleulysgluasnglyalaleuglypheargleu
405410415
glygluglugluthrlysaspprovaltyrthrprometglyvalphe
420425430
ilethralatrpalaargtyrthrthrilethralaalaglnalacys
435440445
tyraspargileiletyrcysaspthraspserilehisleuthrgly
450455460
thrgluileproaspvalilelysaspilevalaspprolyslysleu
465470475480
glytyrtrpalahisgluserthrphelysargalalystyrleuarg
485490495
glnlysthrtyrileglnaspiletyrmetlysgluvalaspglylys
500505510
leuvalgluglyserproaspasptyrthraspilelyspheserval
515520525
lyscysalaglymetthrasplysilelyslysgluvalthrpheglu
530535540
asnphelysvalglypheserarglysmetlysprolysprovalgln
545550555560
valproglyglyvalvalleuvalaspaspthrphethrilelysser
565570575
glyglyseralatrpserhisproglnpheglulysglyglyglyser
580585590
glyglyglyserglyglyseralatrpserhisproglnpheglulys
595600605
<210>398
<211>1390
<212>dna
<213>大肠杆菌
<400>398
atgatgaacgatggcaaacagcagagcaccttcctgtttcatgattatgaaaccttcggt60
acccatccggccctggatcgtccggcgcagtttgcggccattcgcaccgatagcgaattc120
aatgtgattggcgaaccggaagtgttttattgcaaaccggccgatgattatctgccgcag180
ccgggtgcggtgctgattaccggtattaccccgcaggaagcgcgcgcgaaaggtgaaaac240
gaagcggcgtttgccgcgcgcattcatagcctgtttaccgtgccgaaaacctgcattctg300
ggctataacaatgtgcgcttcgatgatgaagttacccgtaatatcttttatcgtaacttt360
tatgatccgtatgcgtggagctggcagcatgataacagccgttgggatctgctggatgtg420
atgcgcgcgtgctatgcgctgcgcccggaaggcattaattggccggaaaacgatgatggc480
ctgccgagctttcgtctggaacatctgaccaaagccaacggcattgaacatagcaatgcc540
catgatgcgatggccgatgtttatgcgaccattgcgatggcgaaactggttaaaacccgt600
cagccgcgcctgtttgattatctgtttacccaccgtaacaaacacaaactgatggcgctg660
attgatgttccgcagatgaaaccgctggtgcatgtgagcggcatgtttggcgcctggcgc720
ggcaacaccagctgggtggccccgctggcctggcacccggaaaatcgtaacgccgtgatt780
atggttgatctggccggtgatattagcccgctgctggaactggatagcgataccctgcgt840
gaacgcctgtataccgccaaaaccgatctgggcgataatgccgccgtgccggtgaaactg900
gttcacattaacaaatgcccggtgctggcccaggcgaacaccctgcgcccggaagatgcg960
gatcgtctgggtattaatcgccagcattgtctggataatctgaaaatcctgcgtgaaaac1020
ccgcaggtgcgtgaaaaagtggtggcgatcttcgcggaagcggaaccgttcaccccgagc1080
gataacgtggatgcgcagctgtataacggcttctttagcgatgccgatcgcgcggcgatg1140
aaaatcgttctggaaaccgaaccgcgcaatctgccggcgctggatattacctttgttgat1200
aaacgtattgaaaaactgctgtttaattatcgtgcgcgcaattttccgggtaccctggat1260
tatgccgaacagcagcgttggctggaacatcgtcgtcaggttttcaccccggaatttctg1320
cagggttatgcggatgaactgcagatgctggttcagcagtatgccgatgataaagaaaaa1380
gtggcgctgc1390
<210>399
<211>485
<212>prt
<213>大肠杆菌
<400>399
metmetasnaspglylysglnglnserthrpheleuphehisasptyr
151015
gluthrpheglythrhisproalaleuaspargproalaglnpheala
202530
alaileargthraspserglupheasnvalileglygluprogluval
354045
phetyrcyslysproalaaspasptyrleuproglnproglyalaval
505560
leuilethrglyilethrproglnglualaargalalysglygluasn
65707580
glualaalaphealaalaargilehisserleuphethrvalprolys
859095
thrcysileleuglytyrasnasnvalargpheaspaspgluvalthr
100105110
argasnilephetyrargasnphetyraspprotyralatrpsertrp
115120125
glnhisaspasnserargtrpaspleuleuaspvalmetargalacys
130135140
tyralaleuargprogluglyileasntrpprogluasnaspaspgly
145150155160
leuproserpheargleugluhisleuthrlysalaasnglyileglu
165170175
hisserasnalahisaspalametalaaspvaltyralathrileala
180185190
metalalysleuvallysthrargglnproargleupheasptyrleu
195200205
phethrhisargasnlyshislysleumetalaleuileaspvalpro
210215220
glnmetlysproleuvalhisvalserglymetpheglyalatrparg
225230235240
glyasnthrsertrpvalalaproleualatrphisprogluasnarg
245250255
asnalavalilemetvalaspleualaglyaspileserproleuleu
260265270
gluleuaspseraspthrleuarggluargleutyrthralalysthr
275280285
aspleuglyaspasnalaalavalprovallysleuvalhisileasn
290295300
lyscysprovalleualaglnalaasnthrleuargprogluaspala
305310315320
aspargleuglyileasnargglnhiscysleuaspasnleulysile
325330335
leuarggluasnproglnvalargglulysvalvalalailepheala
340345350
glualagluprophethrproseraspasnvalaspalaglnleutyr
355360365
asnglyphepheseraspalaaspargalaalametlysilevalleu
370375380
gluthrgluproargasnleuproalaleuaspilethrphevalasp
385390395400
lysargileglulysleuleupheasntyrargalaargasnphepro
405410415
glythrleuasptyralagluglnglnargtrpleugluhisargarg
420425430
glnvalphethrproglupheleuglnglytyralaaspgluleugln
435440445
metleuvalglnglntyralaaspasplysglulysvalalaleuleu
450455460
lysalaleutrpglntyralaglugluilevalserglyserglyhis
465470475480
hishishishishis
485
<210>400
<211>804
<212>dna
<213>大肠杆菌
<400>400
atgaaatttgtctcttttaatatcaacggcctgcgcgccagacctcaccagcttgaagcc60
atcgtcgaaaagcaccaaccggatgtgattggcctgcaggagacaaaagttcatgacgat120
atgtttccgctcgaagaggtggcgaagctcggctacaacgtgttttatcacgggcagaaa180
ggccattatggcgtggcgctgctgaccaaagagacgccgattgccgtgcgtcgcggcttt240
cccggtgacgacgaagaggcgcagcggcggattattatggcggaaatcccctcactgctg300
ggtaatgtcaccgtgatcaacggttacttcccgcagggtgaaagccgcgaccatccgata360
aaattcccggcaaaagcgcagttttatcagaatctgcaaaactacctggaaaccgaactc420
aaacgtgataatccggtactgattatgggcgatatgaatatcagccctacagatctggat480
atcggcattggcgaagaaaaccgtaagcgctggctgcgtaccggtaaatgctctttcctg540
ccggaagagcgcgaatggatggacaggctgatgagctgggggttggtcgataccttccgc600
catgcgaatccgcaaacagcagatcgtttctcatggtttgattaccgctcaaaaggtttt660
gacgataaccgtggtctgcgcatcgacctgctgctcgccagccaaccgctggcagaatgt720
tgcgtagaaaccggcatcgactatgaaatccgcagcatggaaaaaccgtccgatcacgcc780
cccgtctgggcgaccttccgccgc804
<210>401
<211>268
<212>prt
<213>大肠杆菌
<400>401
metlysphevalserpheasnileasnglyleuargalaargprohis
151015
glnleuglualailevalglulyshisglnproaspvalileglyleu
202530
glngluthrlysvalhisaspaspmetpheproleuglugluvalala
354045
lysleuglytyrasnvalphetyrhisglyglnlysglyhistyrgly
505560
valalaleuleuthrlysgluthrproilealavalargargglyphe
65707580
proglyaspaspgluglualaglnargargileilemetalagluile
859095
proserleuleuglyasnvalthrvalileasnglytyrpheprogln
100105110
glygluserargasphisproilelyspheproalalysalaglnphe
115120125
tyrglnasnleuglnasntyrleugluthrgluleulysargaspasn
130135140
provalleuilemetglyaspmetasnileserprothraspleuasp
145150155160
ileglyileglyglugluasnarglysargtrpleuargthrglylys
165170175
cysserpheleuproglugluargglutrpmetaspargleumetser
180185190
trpglyleuvalaspthrphearghisalaasnproglnthralaasp
195200205
argphesertrppheasptyrargserlysglypheaspaspasnarg
210215220
glyleuargileaspleuleuleualaserglnproleualaglucys
225230235240
cysvalgluthrglyileasptyrgluileargsermetglulyspro
245250255
serasphisalaprovaltrpalathrpheargarg
260265
<210>402
<211>1275
<212>dna
<213>嗜热细菌(thermusthermophilus)
<400>402
atgtttcgtcgtaaagaagatctggatccgccgctggcactgctgccgctgaaaggcctg60
cgcgaagccgccgcactgctggaagaagcgctgcgtcaaggtaaacgcattcgtgttcac120
ggcgactatgatgcggatggcctgaccggcaccgcgatcctggttcgtggtctggccgcc180
ctgggtgcggatgttcatccgtttatcccgcaccgcctggaagaaggctatggtgtcctg240
atggaacgcgtcccggaacatctggaagcctcggacctgtttctgaccgttgactgcggc300
attaccaaccatgcggaactgcgcgaactgctggaaaatggcgtggaagtcattgttacc360
gatcatcatacgccgggcaaaacgccgccgccgggtctggtcgtgcatccggcgctgacg420
ccggatctgaaagaaaaaccgaccggcgcaggcgtggcgtttctgctgctgtgggcactg480
catgaacgcctgggcctgccgccgccgctggaatacgcggacctggcagccgttggcacc540
attgccgacgttgccccgctgtggggttggaatcgtgcactggtgaaagaaggtctggca600
cgcatcccggcttcatcttgggtgggcctgcgtctgctggctgaagccgtgggctatacc660
ggcaaagcggtcgaagtcgctttccgcatcgcgccgcgcatcaatgcggcttcccgcctg720
ggcgaagcggaaaaagccctgcgcctgctgctgacggatgatgcggcagaagctcaggcg780
ctggtcggcgaactgcaccgtctgaacgcccgtcgtcagaccctggaagaagcgatgctg840
cgcaaactgctgccgcaggccgacccggaagcgaaagccatcgttctgctggacccggaa900
ggccatccgggtgttatgggtattgtggcctctcgcatcctggaagcgaccctgcgcccg960
gtctttctggtggcccagggcaaaggcaccgtgcgttcgctggctccgatttccgccgtc1020
gaagcactgcgcagcgcggaagatctgctgctgcgttatggtggtcataaagaagcggcg1080
ggtttcgcaatggatgaagcgctgtttccggcgttcaaagcacgcgttgaagcgtatgcc1140
gcacgtttcccggatccggttcgtgaagtggcactgctggatctgctgccggaaccgggc1200
ctgctgccgcaggtgttccgtgaactggcactgctggaaccgtatggtgaaggtaacccg1260
gaaccgctgttcctg1275
<210>403
<211>425
<212>prt
<213>嗜热细菌
<400>403
metpheargarglysgluaspleuaspproproleualaleuleupro
151015
leulysglyleuargglualaalaalaleuleugluglualaleuarg
202530
glnglylysargileargvalhisglyasptyraspalaaspglyleu
354045
thrglythralaileleuvalargglyleualaalaleuglyalaasp
505560
valhispropheileprohisargleuglugluglytyrglyvalleu
65707580
metgluargvalprogluhisleuglualaseraspleupheleuthr
859095
valaspcysglyilethrasnhisalagluleuarggluleuleuglu
100105110
asnglyvalgluvalilevalthrasphishisthrproglylysthr
115120125
proproproglyleuvalvalhisproalaleuthrproaspleulys
130135140
glulysprothrglyalaglyvalalapheleuleuleutrpalaleu
145150155160
hisgluargleuglyleuproproproleuglutyralaaspleuala
165170175
alavalglythrilealaaspvalalaproleutrpglytrpasnarg
180185190
alaleuvallysgluglyleualaargileproalasersertrpval
195200205
glyleuargleuleualaglualavalglytyrthrglylysalaval
210215220
gluvalalapheargilealaproargileasnalaalaserargleu
225230235240
glyglualaglulysalaleuargleuleuleuthraspaspalaala
245250255
glualaglnalaleuvalglygluleuhisargleuasnalaargarg
260265270
glnthrleugluglualametleuarglysleuleuproglnalaasp
275280285
proglualalysalailevalleuleuaspprogluglyhisprogly
290295300
valmetglyilevalalaserargileleuglualathrleuargpro
305310315320
valpheleuvalalaglnglylysglythrvalargserleualapro
325330335
ileseralavalglualaleuargseralagluaspleuleuleuarg
340345350
tyrglyglyhislysglualaalaglyphealametaspglualaleu
355360365
pheproalaphelysalaargvalglualatyralaalaargphepro
370375380
aspprovalarggluvalalaleuleuaspleuleuprogluprogly
385390395400
leuleuproglnvalphearggluleualaleuleugluprotyrgly
405410415
gluglyasnprogluproleupheleu
420425
<210>404
<211>738
<212>dna
<213>细菌噬菌体λ(bacteriophagelambda)
<400>404
tccggaagcggctctggtagtggttctggcatgacaccggacattatcctgcagcgtacc60
gggatcgatgtgagagctgtcgaacagggggatgatgcgtggcacaaattacggctcggc120
gtcatcaccgcttcagaagttcacaacgtgatagcaaaaccccgctccggaaagaagtgg180
cctgacatgaaaatgtcctacttccacaccctgcttgctgaggtttgcaccggtgtggct240
ccggaagttaacgctaaagcactggcctggggaaaacagtacgagaacgacgccagaacc300
ctgtttgaattcacttccggcgtgaatgttactgaatccccgatcatctatcgcgacgaa360
agtatgcgtaccgcctgctctcccgatggtttatgcagtgacggcaacggccttgaactg420
aaatgcccgtttacctcccgggatttcatgaagttccggctcggtggtttcgaggccata480
aagtcagcttacatggcccaggtgcagtacagcatgtgggtgacgcgaaaaaatgcctgg540
tactttgccaactatgacccgcgtatgaagcgtgaaggcctgcattatgtcgtgattgag600
cgggatgaaaagtacatggcgagttttgacgagatcgtgccggagttcatcgaaaaaatg660
gacgaggcactggctgaaattggttttgtatttggggagcaatggcgatctggctctggt720
tccggcagcggttccgga738
<210>405
<211>226
<212>prt
<213>细菌噬菌体λ
<400>405
metthrproaspileileleuglnargthrglyileaspvalargala
151015
valgluglnglyaspaspalatrphislysleuargleuglyvalile
202530
thralasergluvalhisasnvalilealalysproargserglylys
354045
lystrpproaspmetlysmetsertyrphehisthrleuleualaglu
505560
valcysthrglyvalalaprogluvalasnalalysalaleualatrp
65707580
glylysglntyrgluasnaspalaargthrleuphegluphethrser
859095
glyvalasnvalthrgluserproileiletyrargaspglusermet
100105110
argthralacysserproaspglyleucysseraspglyasnglyleu
115120125
gluleulyscysprophethrserargaspphemetlyspheargleu
130135140
glyglypheglualailelysseralatyrmetalaglnvalglntyr
145150155160
sermettrpvalthrarglysasnalatrptyrphealaasntyrasp
165170175
proargmetlysarggluglyleuhistyrvalvalilegluargasp
180185190
glulystyrmetalaserpheaspgluilevalproglupheileglu
195200205
lysmetaspglualaleualagluileglyphevalpheglyglugln
210215220
trparg
225
<210>406
<211>760
<212>prt
<213>伯顿拟甲烷球菌(methanococcoidesburtonii)
<400>406
metmetilearggluleuaspileproargaspileileglyphetyr
151015
gluaspserglyilelysgluleutyrproproglnalaglualaile
202530
glumetglyleuleuglulyslysasnleuleualaalaileprothr
354045
alaserglylysthrleuleualagluleualametilelysalaile
505560
arggluglyglylysalaleutyrilevalproleuargalaleuala
65707580
serglulysphegluargphelysgluleualapropheglyilelys
859095
valglyileserthrglyaspleuaspserargalaasptrpleugly
100105110
valasnaspileilevalalathrserglulysthraspserleuleu
115120125
argasnglythrsertrpmetaspgluilethrthrvalvalvalasp
130135140
gluilehisleuleuaspserlysasnargglyprothrleugluval
145150155160
thrilethrlysleumetargleuasnproaspvalglnvalvalala
165170175
leuseralathrvalglyasnalaargglumetalaasptrpleugly
180185190
alaalaleuvalleuserglutrpargprothraspleuhisglugly
195200205
valleupheglyaspalaileasnpheproglyserglnlyslysile
210215220
aspargleuglulysaspaspalavalasnleuvalleuaspthrile
225230235240
lysalagluglyglncysleuvalphegluserserargargasncys
245250255
alaglyphealalysthralaserserlysvalalalysileleuasp
260265270
asnaspilemetilelysleualaglyilealaglugluvalgluser
275280285
thrglygluthraspthralailevalleualaasncysilearglys
290295300
glyvalalaphehishisalaglyleuasnserasnhisarglysleu
305310315320
valgluasnglypheargglnasnleuilelysvalileserserthr
325330335
prothrleualaalaglyleuasnleuproalaargargvalileile
340345350
argsertyrargargpheaspserasnpheglymetglnproilepro
355360365
valleuglutyrlysglnmetalaglyargalaglyargprohisleu
370375380
aspprotyrglygluservalleuleualalysthrtyraspgluphe
385390395400
alaglnleumetgluasntyrvalglualaaspalagluaspiletrp
405410415
serlysleuglythrgluasnalaleuargthrhisvalleuserthr
420425430
ilevalasnglyphealaserthrargglngluleupheaspphephe
435440445
glyalathrphephealatyrglnglnasplystrpmetleugluglu
450455460
valileasnaspcysleuglupheleuileasplysalametvalser
465470475480
gluthrgluaspilegluaspalaserlysleupheleuargglythr
485490495
argleuglyserleuvalsermetleutyrileaspproleusergly
500505510
serlysilevalaspglyphelysaspileglylysserthrglygly
515520525
asnmetglyserleugluaspasplysglyaspaspilethrvalthr
530535540
aspmetthrleuleuhisleuvalcysserthrproaspmetarggln
545550555560
leutyrleuargasnthrasptyrthrilevalasnglutyrileval
565570575
alahisseraspgluphehisgluileproasplysleulysgluthr
580585590
asptyrglutrpphemetglygluvallysthralametleuleuglu
595600605
glutrpvalthrgluvalseralagluaspilethrarghispheasn
610615620
valglygluglyaspilehisalaleualaaspthrserglutrpleu
625630635640
methisalaalaalalysleualagluleuleuglyvalglutyrser
645650655
serhisalatyrserleuglulysargileargtyrglyserglyleu
660665670
aspleumetgluleuvalglyileargglyvalglyargvalargala
675680685
arglysleutyrasnalaglyphevalservalalalysleulysgly
690695700
alaaspileservalleuserlysleuvalglyprolysvalalatyr
705710715720
asnileleuserglyileglyvalargvalasnasplyshispheasn
725730735
seralaproileserserasnthrleuaspthrleuleuasplysasn
740745750
glnlysthrpheasnaspphegln
755760
<210>407
<211>707
<212>prt
<213>共生餐古菌(cenarchaeumsymbiosum)
<400>407
metargilesergluleuaspileproargproalaileglupheleu
151015
gluglygluglytyrlyslysleutyrproproglnalaalaalaala
202530
lysalaglyleuthraspglylysservalleuvalseralaprothr
354045
alaserglylysthrleuilealaalailealametileserhisleu
505560
serargasnargglylysalavaltyrleuserproleuargalaleu
65707580
alaalaglulysphealaglupheglylysileglyglyileproleu
859095
glyargprovalargvalglyvalserthrglyasppheglulysala
100105110
glyargserleuglyasnasnaspileleuvalleuthrasngluarg
115120125
metaspserleuileargargargproasptrpmetaspgluvalgly
130135140
leuvalilealaaspgluilehisleuileglyaspargserarggly
145150155160
prothrleuglumetvalleuthrlysleuargglyleuargserser
165170175
proglnvalvalalaleuseralathrileserasnalaaspgluile
180185190
alaglytrpleuaspcysthrleuvalhisserthrtrpargproval
195200205
proleusergluglyvaltyrglnaspglygluvalalametglyasp
210215220
glyserarghisgluvalalaalathrglyglyglyproalavalasp
225230235240
leualaalagluservalalagluglyglyglnserleuilepheala
245250255
aspthrargalaargseralaserleualaalalysalaseralaval
260265270
ileproglualalysglyalaaspalaalalysleualaalaalaala
275280285
lyslysileileserserglyglygluthrlysleualalysthrleu
290295300
alagluleuvalglulysglyalaalaphehishisalaglyleuasn
305310315320
glnaspcysargservalvalglugluglupheargserglyargile
325330335
argleuleualaserthrprothrleualaalaglyvalasnleupro
340345350
alaargargvalvalileserservalmetargtyrasnserserser
355360365
glymetsergluproileserileleuglutyrlysglnleucysgly
370375380
argalaglyargproglntyrasplysserglyglualailevalval
385390395400
glyglyvalasnalaaspgluilepheaspargtyrileglyglyglu
405410415
progluproileargseralametvalaspaspargalaleuargile
420425430
hisvalleuserleuvalthrthrserproglyilelysgluaspasp
435440445
valthrgluphepheleuglythrleuglyglyglnglnserglyglu
450455460
serthrvallyspheservalalavalalaleuargpheleuglnglu
465470475480
gluglymetleuglyargargglyglyargleualaalathrlysmet
485490495
glyargleuvalserargleutyrmetaspprometthralavalthr
500505510
leuargaspalavalglyglualaserproglyargmethisthrleu
515520525
glypheleuhisleuvalserglucyssergluphemetproargphe
530535540
alaleuargglnlysasphisgluvalalaglumetmetleugluala
545550555560
glyargglygluleuleuargprovaltyrsertyrglucysglyarg
565570575
glyleuleualaleuhisargtrpileglygluserproglualalys
580585590
leualagluaspleulysphegluserglyaspvalhisargmetval
595600605
gluserserglytrpleuleuargcysiletrpgluileserlyshis
610615620
glngluargproaspleuleuglygluleuaspvalleuargserarg
625630635640
valalatyrglyilelysalagluleuvalproleuvalserilelys
645650655
glyileglyargvalargserargargleupheargglyglyilelys
660665670
glyproglyaspleualaalavalprovalgluargleuserargval
675680685
gluglyileglyalathrleualaasnasnilelysserglnleuarg
690695700
lysglygly
705
<210>408
<211>720
<212>prt
<213>抗辐射热球菌(thermococcusgammatolerans)
<400>408
metlysvalaspgluleuprovalaspgluargleulysalavalleu
151015
lysgluargglyileglugluleutyrproproglnalaglualaleu
202530
lysserglyalaleugluglyargasnleuvalleualaileprothr
354045
alaserglylysthrleuvalsergluilevalmetvalasnlysleu
505560
ileglngluglyglylysalavaltyrleuvalproleulysalaleu
65707580
alagluglulystyrarggluphelysglutrpglulysleuglyleu
859095
lysvalalaalathrthrglyasptyraspserthraspasptrpleu
100105110
glyargtyraspileilevalalathralaglulyspheaspserleu
115120125
leuarghisglyalaargtrpileasnaspvallysleuvalvalala
130135140
aspgluvalhisleuileglysertyraspargglyalathrleuglu
145150155160
metileleuthrhismetleuglyargalaglnileleualaleuser
165170175
alathrvalglyasnalaglugluleualaglutrpleuaspalaser
180185190
leuvalvalserasptrpargprovalglnleuargargglyvalphe
195200205
hisleuglythrleuiletrpgluaspglylysvalglusertyrpro
210215220
gluasntrptyrserleuvalvalaspalavallysargglylysgly
225230235240
alaleuvalphevalasnthrargargseralaglulysglualaleu
245250255
alaleuserlysleuvalserserhisleuthrlysproglulysarg
260265270
alaleugluserleualaserglnleugluaspasnprothrserglu
275280285
lysleulysargalaleuargglyglyvalalaphehishisalagly
290295300
leuserargvalgluargthrleuilegluaspalapheargglugly
305310315320
leuilelysvalilethralathrprothrleuseralaglyvalasn
325330335
leuproserpheargvalileileargaspthrlysargtyralagly
340345350
pheglytrpthraspileprovalleugluileglnglnmetmetgly
355360365
argalaglyargproargtyrasplystyrglyglualaileileval
370375380
alaargthraspgluproglylysleumetgluargtyrilearggly
385390395400
lysproglulysleuphesermetleualaasngluglnalaphearg
405410415
serglnvalleualaleuilethrasnpheglyileargserphepro
420425430
gluleuvalargpheleugluargthrphetyralahisglnarglys
435440445
aspleuserserleuglutyrlysalalysgluvalvaltyrpheleu
450455460
ilegluasnglupheileaspleuaspleugluaspargpheilepro
465470475480
leupropheglylysargthrserglnleutyrileaspproleuthr
485490495
alalyslysphelysaspalapheproalailegluargasnproasn
500505510
propheglyilepheglnleuilealaserthrproaspmetalathr
515520525
leuthralaargargargglumetgluasptyrleuaspleualatyr
530535540
gluleugluasplysleutyralaserileprotyrtyrgluaspser
545550555560
argpheglnglypheleuglyglnvallysthralalysvalleuleu
565570575
asptrpileasngluvalproglualaargiletyrgluthrtyrser
580585590
ileaspproglyaspleutyrargleuleugluleualaasptrpleu
595600605
mettyrserleuilegluleutyrlysleuphegluprolysgluglu
610615620
ileleuasntyrleuargaspleuhisleuargleuarghisglyval
625630635640
argglugluleuleugluleuvalargleuproasnileglyarglys
645650655
argalaargalaleutyrasnalaglypheargservalglualaile
660665670
alaasnalalysproalagluleuleualavalgluglyileglyala
675680685
lysileleuaspglyiletyrarghisleuglyileglulysargval
690695700
thrgluglulysprolysarglysglythrleugluasppheleuarg
705710715720
<210>409
<211>799
<212>prt
<213>亨氏甲烷螺菌(methanospirillumhungatei)
<400>409
metgluilealaserleuproleuproaspserpheileargalacys
151015
hisalalysglyileargserleutyrproproglnalaglucysile
202530
glulysglyleuleugluglylysasnleuleuileserileprothr
354045
alaserglylysthrleuleualaglumetalamettrpserargile
505560
alaalaglyglylyscysleutyrilevalproleuargalaleuala
65707580
serglulystyraspglupheserlyslysglyvalileargvalgly
859095
ilealathrglyaspleuaspargthraspalatyrleuglygluasn
100105110
aspileilevalalathrserglulysthraspserleuleuargasn
115120125
argthrprotrpleuserglnilethrcysilevalleuaspgluval
130135140
hisleuileglysergluasnargglyalathrleuglumetvalile
145150155160
thrlysleuargtyrthrasnprovalmetglnileileglyleuser
165170175
alathrileglyasnproalaglnleualaglutrpleuaspalathr
180185190
leuilethrserthrtrpargprovalaspleuargglnglyvaltyr
195200205
tyrasnglylysileargpheseraspsergluargproileglngly
210215220
lysthrlyshisaspaspleuasnleucysleuaspthrilegluglu
225230235240
glyglyglncysleuvalphevalserserargargasnalaglugly
245250255
phealalyslysalaalaglyalaleulysalaglyserproaspser
260265270
lysalaleualaglngluleuargargleuargaspargaspglugly
275280285
asnvalleualaaspcysvalgluargglyalaalaphehishisala
290295300
glyleuileargglngluargthrileileglugluglypheargasn
305310315320
glytyrilegluvalilealaalathrprothrleualaalaglyleu
325330335
asnleuproalaargargvalileileargasptyrasnargpheala
340345350
serglyleuglymetvalproileprovalglyglutyrhisglnmet
355360365
alaglyargalaglyargprohisleuaspprotyrglyglualaval
370375380
leuleualalysaspalaproservalgluargleuphegluthrphe
385390395400
ileaspalaglualagluargvalaspserglncysvalaspaspala
405410415
serleucysalahisileleuserleuilealathrglyphealahis
420425430
aspglnglualaleuserserphemetgluargthrphetyrphephe
435440445
glnhisprolysthrargserleuproargleuvalalaaspalaile
450455460
argpheleuthrthralaglymetvalglugluarggluasnthrleu
465470475480
seralathrargleuglyserleuvalserargleutyrleuasnpro
485490495
cysthralaargleuileleuaspserleulyssercyslysthrpro
500505510
thrleuileglyleuleuhisvalilecysvalserproaspmetgln
515520525
argleutyrleulysalaalaaspthrglnleuleuargthrpheleu
530535540
phelyshislysaspaspleuileleuproleuprophegluglnglu
545550555560
gluglugluleutrpleuserglyleulysthralaleuvalleuthr
565570575
asptrpalaaspgluphesergluglymetileglugluargtyrgly
580585590
ileglyalaglyaspleutyrasnilevalaspserglylystrpleu
595600605
leuhisglythrgluargleuvalservalglumetproglumetser
610615620
glnvalvallysthrleuservalargvalhishisglyvallysser
625630635640
gluleuleuproleuvalalaleuargasnileglyargvalargala
645650655
argthrleutyrasnalaglytyrproaspproglualavalalaarg
660665670
alaglyleuserthrilealaargileileglygluglyilealaarg
675680685
glnvalileaspgluilethrglyvallysargserglyilehisser
690695700
seraspaspasptyrglnglnlysthrprogluleuleuthraspile
705710715720
proglyileglylyslysmetalaglulysleuglnasnalaglyile
725730735
ilethrvalseraspleuleuthralaaspgluvalleuleuserasp
740745750
valleuglyalaalaargalaarglysvalleualapheleuserasn
755760765
serglulysgluasnserserserasplysthrglugluileproasp
770775780
thrglnlysileargglyglnsersertrpgluasppheglycys
785790795
<210>410
<211>1756
<212>prt
<213>大肠杆菌
<400>410
metmetserilealaglnvalargseralaglyseralaglyasntyr
151015
tyrthrasplysaspasntyrtyrvalleuglysermetglygluarg
202530
trpalaglylysglyalagluglnleuglyleuglnglyservalasp
354045
lysaspvalphethrargleuleugluglyargleuproaspglyala
505560
aspleuserargmetglnaspglyserasnlyshisargproglytyr
65707580
aspleuthrpheseralaprolysservalsermetmetalametleu
859095
glyglyasplysargleuileaspalahisasnglnalavalaspphe
100105110
alavalargglnvalglualaleualaserthrargvalmetthrasp
115120125
glyglnsergluthrvalleuthrglyasnleuvalmetalaleuphe
130135140
asnhisaspthrserargaspglngluproglnleuhisthrhisala
145150155160
valvalalaasnvalthrglnhisasnglyglutrplysthrleuser
165170175
serasplysvalglylysthrglypheilegluasnvaltyralaasn
180185190
glnilealapheglyargleutyrargglulysleulysgluglnval
195200205
glualaleuglytyrgluthrgluvalvalglylyshisglymettrp
210215220
glumetproglyvalprovalglualapheserglyargserglnala
225230235240
ileargglualavalglygluaspalaserleulysserargaspval
245250255
alaalaleuaspthrarglysserlysglnhisvalaspprogluile
260265270
argmetalaglutrpmetglnthrleulysgluthrglypheaspile
275280285
argalatyrargaspalaalaaspglnargthrgluileargthrgln
290295300
alaproglyproalaserglnaspglyproaspvalglnglnalaval
305310315320
thrglnalailealaglyleusergluarglysvalglnphethrtyr
325330335
thraspvalleualaargthrvalglyileleuproprogluasngly
340345350
valilegluargalaargalaglyileaspglualaileserargglu
355360365
glnleuileproleuaspargglulysglyleuphethrserglyile
370375380
hisvalleuaspgluleuservalargalaleuserargaspilemet
385390395400
lysglnasnargvalthrvalhisproglulysservalproargthr
405410415
alaglytyrseraspalavalservalleualaglnaspargproser
420425430
leualailevalserglyglnglyglyalaalaglyglnarggluarg
435440445
valalagluleuvalmetmetalaarggluglnglyarggluvalgln
450455460
ileilealaalaaspargargserglnmetasnleulysglnaspglu
465470475480
argleuserglygluleuilethrglyargargglnleuleuglugly
485490495
metalaphethrproglyserthrvalilevalaspglnglyglulys
500505510
leuserleulysgluthrleuthrleuleuaspglyalaalaarghis
515520525
asnvalglnvalleuilethraspserglyglnargthrglythrgly
530535540
seralaleumetalametlysaspalaglyvalasnthrtyrargtrp
545550555560
glnglyglygluglnargproalathrileilesergluproasparg
565570575
asnvalargtyralaargleualaglyaspphealaalaservallys
580585590
alaglyglugluservalalaglnvalserglyvalarggluglnala
595600605
ileleuthrglnalaileargsergluleulysthrglnglyvalleu
610615620
glyhisprogluvalthrmetthralaleuserprovaltrpleuasp
625630635640
serargserargtyrleuargaspmettyrargproglymetvalmet
645650655
gluglntrpasnprogluthrargserhisaspargtyrvalileasp
660665670
argvalthralaglnserhisserleuthrleuargaspalaglngly
675680685
gluthrglnvalvalargileserserleuaspsersertrpserleu
690695700
pheargproglulysmetprovalalaaspglygluargleuargval
705710715720
thrglylysileproglyleuargvalserglyglyaspargleugln
725730735
valalaservalsergluaspalametthrvalvalvalproglyarg
740745750
alagluproalaserleuprovalseraspserprophethralaleu
755760765
lysleugluasnglytrpvalgluthrproglyhisservalserasp
770775780
seralathrvalphealaservalthrglnmetalametaspasnala
785790795800
thrleuasnglyleualaargserglyargaspvalargleutyrser
805810815
serleuaspgluthrargthralaglulysleualaarghisproser
820825830
phethrvalvalsergluglnilelysalaargalaglygluthrleu
835840845
leugluthralaileserleuglnlysalaglyleuhisthrproala
850855860
glnglnalailehisleualaleuprovalleugluserlysasnleu
865870875880
alaphesermetvalaspleuleuthrglualalysserphealaala
885890895
gluglythrglyphethrgluleuglyglygluileasnalaglnile
900905910
lysargglyaspleuleutyrvalaspvalalalysglytyrglythr
915920925
glyleuleuvalserargalasertyrglualaglulysserileleu
930935940
arghisileleugluglylysglualavalthrproleumetgluarg
945950955960
valproglygluleumetgluthrleuthrserglyglnargalaala
965970975
thrargmetileleugluthrseraspargphethrvalvalglngly
980985990
tyralaglyvalglylysthrthrglnpheargalavalmetserala
99510001005
valasnmetleuproalasergluargproargvalvalglyleu
101010151020
glyprothrhisargalavalglyglumetargseralaglyval
102510301035
aspalaglnthrleualaserpheleuhisaspthrglnleugln
104010451050
glnargserglygluthrproasppheserasnthrleupheleu
105510601065
leuaspglusersermetvalglyasnthrglumetalaargala
107010751080
tyralaleuilealaalaglyglyglyargalavalalasergly
108510901095
aspthraspglnleuglnalailealaproglyglnserphearg
110011051110
leuglnglnthrargseralaalaaspvalvalilemetlysglu
111511201125
ilevalargglnthrprogluleuargglualavaltyrserleu
113011351140
ileasnargaspvalgluargalaleuserglyleugluserval
114511501155
lysproserglnvalproargleugluglyalatrpalaproglu
116011651170
hisservalthrglupheserhisserglnglualalysleuala
117511801185
glualaglnglnlysalametleulysglyglualapheproasp
119011951200
ileprometthrleutyrglualailevalargasptyrthrgly
120512101215
argthrproglualaarggluglnthrleuilevalthrhisleu
122012251230
asngluaspargargvalleuasnsermetilehisaspalaarg
123512401245
glulysalaglygluleuglylysgluglnvalmetvalproval
125012551260
leuasnthralaasnileargaspglygluleuargargleuser
126512701275
thrtrpglulysasnproaspalaleualaleuvalaspasnval
128012851290
tyrhisargilealaglyileserlysaspaspglyleuilethr
129513001305
leuglnaspalagluglyasnthrargleuileserproargglu
131013151320
alavalalagluglyvalthrleutyrthrproasplysilearg
132513301335
valglythrglyaspargmetargphethrlysseraspargglu
134013451350
argglytyrvalalaasnservaltrpthrvalthralavalser
135513601365
glyaspservalthrleuseraspglyglnglnthrargvalile
137013751380
argproglyglngluargalagluglnhisileaspleualatyr
138513901395
alailethralahisglyalaglnglyalasergluthrpheala
140014051410
ilealaleugluglythrgluglyasnarglysleumetalagly
141514201425
phegluseralatyrvalalaleuserargmetlysglnhisval
143014351440
glnvaltyrthraspasnargglnglytrpthraspalaileasn
144514501455
asnalavalglnlysglythralahisaspvalleugluprolys
146014651470
proasparggluvalmetasnalaglnargleupheserthrala
147514801485
arggluleuargaspvalalaalaglyargalavalleuarggln
149014951500
alaglyleualaglyglyaspserproalaargpheilealapro
150515101515
glyarglystyrproglnprotyrvalalaleuproalapheasp
152015251530
argasnglylysseralaglyiletrpleuasnproleuthrthr
153515401545
aspaspglyasnglyleuargglypheserglygluglyargval
155015551560
lysglyserglyaspalaglnphevalalaleuglnglyserarg
156515701575
asnglygluserleuleualaaspasnmetglnaspglyvalarg
158015851590
ilealaargaspasnproaspserglyvalvalvalargileala
159516001605
glygluglyargprotrpasnproglyalailethrglyglyarg
161016151620
valtrpglyaspileproaspasnservalglnproglyalagly
162516301635
asnglygluprovalthralagluvalleualaglnargglnala
164016451650
gluglualaileargarggluthrgluargargalaaspgluile
165516601665
valarglysmetalagluasnlysproaspleuproaspglylys
167016751680
thrgluleualavalargaspilealaglyglngluargasparg
168516901695
seralailesergluarggluthralaleuprogluservalleu
170017051710
arggluserglnarggluargglualavalarggluvalalaarg
171517201725
gluasnleuleuglngluargleuglnglnmetgluargaspmet
173017351740
valargaspleuglnlysglulysthrleuglyglyasp
174517501755
<210>411
<211>726
<212>prt
<213>伯顿拟甲烷球菌
<400>411
metserasplysproalaphemetlystyrphethrglnsersercys
151015
tyrproasnglnglnglualametaspargilehisseralaleumet
202530
glnglnglnleuvalleuphegluglyalacysglythrglylysthr
354045
leuseralaleuvalproalaleuhisvalglylysmetleuglylys
505560
thrvalileilealathrasnvalhisglnglnmetvalglnpheile
65707580
asnglualaargaspilelyslysvalglnaspvallysvalalaval
859095
ilelysglylysthralametcysproglnglualaasptyrgluglu
100105110
cysservallysarggluasnthrphegluleumetgluthrgluarg
115120125
gluiletyrleulysargglngluleuasnseralaargaspsertyr
130135140
lyslysserhisaspproalaphevalthrleuargaspgluleuser
145150155160
lysgluileaspalavalgluglulysalaargglyleuargasparg
165170175
alacysasnaspleutyrgluvalleuargseraspserglulysphe
180185190
argglutrpleutyrlysgluvalargserproglugluileasnasp
195200205
hisalailelysaspglymetcysglytyrgluleuvallysargglu
210215220
leulyshisalaaspleuleuilecysasntyrhishisvalleuasn
225230235240
proaspilepheserthrvalleuglytrpileglulysgluprogln
245250255
gluthrilevalilepheaspglualahisasnleugluseralaala
260265270
argserhisserserleuserleuthrgluhisserileglulysala
275280285
ilethrgluleuglualaasnleuaspleuleualaaspaspasnile
290295300
hisasnleupheasnilepheleugluvalileseraspthrtyrasn
305310315320
serargphelyspheglygluarggluargvalarglysasntrptyr
325330335
aspileargileseraspprotyrgluargasnaspilevalarggly
340345350
lyspheleuargglnalalysglyasppheglyglulysaspaspile
355360365
glnileleuleuserglualasergluleuglyalalysleuaspglu
370375380
thrtyrargaspglntyrlyslysglyleuserservalmetlysarg
385390395400
serhisileargtyrvalalaaspphemetseralatyrilegluleu
405410415
serhisasnleuasntyrtyrproileleuasnvalargargaspmet
420425430
asnaspgluiletyrglyargvalgluleuphethrcysileprolys
435440445
asnvalthrgluproleupheasnserleupheservalileleumet
450455460
seralathrleuhispropheglumetvallyslysthrleuglyile
465470475480
thrargaspthrcysglumetsertyrglythrserpheprogluglu
485490495
lysargleuserilealavalserileproproleuphealalysasn
500505510
argaspasparghisvalthrgluleuleugluglnvalleuleuasp
515520525
serilegluasnserlysglyasnvalileleuphepheglnserala
530535540
pheglualalysargtyrtyrserlysilegluproleuvalasnval
545550555560
provalpheleuaspgluvalglyileserserglnaspvalargglu
565570575
gluphepheserileglyglugluasnglylysalavalleuleuser
580585590
tyrleutrpglythrleusergluglyileasptyrargaspglyarg
595600605
glyargthrvalileileileglyvalglytyrproalaleuasnasp
610615620
argmetasnalavalgluseralatyrasphisvalpheglytyrgly
625630635640
alaglytrpgluphealaileglnvalprothrilearglysilearg
645650655
glnalametglyargvalvalargserprothrasptyrglyalaarg
660665670
ileleuleuaspglyargpheleuthraspserlyslysargphegly
675680685
lyspheservalphegluvalpheproproalagluargsergluphe
690695700
valaspvalaspproglulysvallystyrserleumetasnphephe
705710715720
metaspasnaspglugln
725
<210>412
<211>439
<212>prt
<213>t4噬菌体(bacteriophaget4)
<400>412
metthrpheaspaspleuthrgluglyglnlysasnalapheasnile
151015
valmetlysalailelysglulyslyshishisvalthrileasngly
202530
proalaglythrglylysthrthrleuthrlyspheileilegluala
354045
leuileserthrglygluthrglyileileleualaalaprothrhis
505560
alaalalyslysileleuserlysleuserglylysglualaserthr
65707580
ilehisserileleulysileasnprovalthrtyrglugluasnval
859095
leuphegluglnlysgluvalproaspleualalyscysargvalleu
100105110
ilecysaspgluvalsermettyrasparglysleuphelysileleu
115120125
leuserthrileproprotrpcysthrileileglyileglyaspasn
130135140
lysglnileargprovalaspproglygluasnthralatyrileser
145150155160
prophephethrhislysaspphetyrglncysgluleuthrgluval
165170175
lysargserasnalaproileileaspvalalathraspvalargasn
180185190
glylystrpiletyrasplysvalvalaspglyhisglyvalarggly
195200205
phethrglyaspthralaleuargaspphemetvalasntyrpheser
210215220
ilevallysserleuaspaspleuphegluasnargvalmetalaphe
225230235240
thrasnlysservalasplysleuasnserileilearglyslysile
245250255
phegluthrasplysasppheilevalglygluileilevalmetgln
260265270
gluproleuphelysthrtyrlysileaspglylysprovalserglu
275280285
ileilepheasnasnglyglnleuvalargileileglualaglutyr
290295300
thrserthrphevallysalaargglyvalproglyglutyrleuile
305310315320
arghistrpaspleuthrvalgluthrtyrglyaspaspglutyrtyr
325330335
argglulysilelysileileserseraspglugluleutyrlysphe
340345350
asnleupheleuglylysthralagluthrtyrlysasntrpasnlys
355360365
glyglylysalaprotrpseraspphetrpaspalalysserglnphe
370375380
serlysvallysalaleuproalaserthrphehislysalaglngly
385390395400
metservalaspargalapheiletyrthrprocysilehistyrala
405410415
aspvalgluleualaglnglnleuleutyrvalglyvalthrarggly
420425430
argtyraspvalphetyrval
435
<210>413
<211>970
<212>prt
<213>深海海洋柠檬酸微菌(citromicrobiumbathyomarinum)
<400>413
metleuservalalaasnvalargserproseralaalaalasertyr
151015
phealaseraspasntyrtyralaseralaaspalaaspargsergly
202530
glntrpileglyaspglyalalysargleuglyleugluglylysval
354045
glualaargalapheaspalaleuleuargglygluleuproaspgly
505560
serservalglyasnproglyglnalahisargproglythraspleu
65707580
thrpheservalprolyssertrpserleuleualaleuvalglylys
859095
aspgluargileilealaalatyrargglualavalvalglualaleu
100105110
histrpalaglulysasnalaalagluthrargvalvalglulysgly
115120125
metvalvalthrglnalathrglyasnleualaileglyleuphegln
130135140
hisaspthrasnargasnglngluproasnleuhisphehisalaval
145150155160
ilealaasnvalthrglnglylysaspglylystrpargthrleulys
165170175
asnaspargleutrpglnleuasnthrthrleuasnserilealamet
180185190
alaargpheargvalalavalglulysleuglytyrgluproglypro
195200205
valleulyshisglyasnpheglualaargglyileserargglugln
210215220
valmetalapheserthrargarglysgluvalleuglualaargarg
225230235240
glyproglyleuaspalaglyargilealaalaleuaspthrargala
245250255
serlysgluglyilegluaspargalathrleuserlysglntrpser
260265270
glualaalaglnserileglyleuaspleulysproleuvalasparg
275280285
alaargthrlysalaleuglyglnglymetglualathrargilegly
290295300
serleuvalgluargglyargalatrpleuserargphealaalahis
305310315320
valargglyaspproalaaspproleuvalproproservalleulys
325330335
glnaspargglnthrilealaalaalaglnalavalalaseralaval
340345350
arghisleuserglnargglualaalaphegluargthralaleutyr
355360365
lysalaalaleuasppheglyleuprothrthrilealaaspvalglu
370375380
lysargthrargalaleuvalargserglyaspleuilealaglylys
385390395400
glygluhislysglytrpleualaserargaspalavalvalthrglu
405410415
glnargileleusergluvalalaalaglylysglyaspserserpro
420425430
alailethrproglnlysalaalaalaservalglnalaalaalaleu
435440445
thrglyglnglypheargleuasngluglyglnleualaalaalaarg
450455460
leuileleuileserlysaspargthrilealavalglnglyileala
465470475480
glyalaglylysserservalleulysprovalalagluvalleuarg
485490495
aspgluglyhisprovalileglyleualaileglnasnthrleuval
500505510
glnmetleugluargaspthrglyileglyserglnthrleualaarg
515520525
pheleuglyglytrpasnlysleuleuaspaspproglyasnvalala
530535540
leuargalaglualaglnalaserleulysasphisvalleuvalleu
545550555560
aspglualasermetvalserasngluasplysglulysleuvalarg
565570575
leualaasnleualaglyvalhisargleuvalleuileglyasparg
580585590
lysglnleuglyalavalaspalaglylysprophealaleuleugln
595600605
argalaglyilealaargalaglumetalathrasnleuargalaarg
610615620
aspprovalvalargglualaglnalaalaalaglnalaglyaspval
625630635640
arglysalaleuarghisleulysserhisthrvalglualaarggly
645650655
aspglyalaglnvalalaalagluthrtrpleualaleuasplysglu
660665670
thrargalaargthrseriletyralaserglyargalaileargser
675680685
alavalasnalaalavalglnglnglyleuleualaserarggluile
690695700
glyproalalysmetlysleugluvalleuaspargvalasnthrthr
705710715720
argglugluleuarghisleuproalatyrargalaglyargvalleu
725730735
gluvalserarglysglnglnalaleuglyleupheileglyglutyr
740745750
argvalileglyglnasparglysglylysleuvalgluvalgluasp
755760765
lysargglylysargpheargpheaspproalaargileargalagly
770775780
lysglyaspaspasnleuthrleuleugluproarglysleugluile
785790795800
hisgluglyaspargileargtrpthrargasnasphisargarggly
805810815
leupheasnalaaspglnalaargvalvalgluilealaasnglylys
820825830
valthrphegluthrserlysglyaspleuvalgluleulyslysasp
835840845
aspprometleulysargileaspleualatyralaleuasnvalhis
850855860
metalaglnglyleuthrseraspargglyilealavalmetaspser
865870875880
arggluargasnleuserasnglnlysthrpheleuvalthrvalthr
885890895
argleuargasphisleuthrleuvalvalaspseralaasplysleu
900905910
glyalaalavalalaargasnlysglyglulysalaseralaileglu
915920925
valthrglyservallysprothralathrlysglyserglyvalasp
930935940
glnprolysservalglualaasnlysalaglulysgluleuthrarg
945950955960
serlysserlysthrleuasppheglyile
965970
<210>414
<211>262
<212>prt
<213>雷金斯堡约克氏菌(yokenellaregensburgei)
<400>414
cysleuthralaproprolysglualaalalysprothrleumetpro
151015
argalaglnsertyrargaspleuthrhisleuproleuprosergly
202530
lysvalphevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thralametleuvalthralaleulysaspserargtrpphevalpro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalaaspasnasnargilepro
100105110
leuglnserleuthralaalaasnvalmetilegluglyserileile
115120125
glytyrgluserasnvallysserglyglyvalglyalaargtyrphe
130135140
glyileglyalaaspthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluvalleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
phevalasptyrglnargleuleugluglygluileglytyrthrser
195200205
asngluprovalmetleucysleumetseralailegluthrglyval
210215220
iletyrleuileasnaspglyilegluargglyleutrpaspleugln
225230235240
glnlysalaaspvalaspasnproileleualaargtyrargasnmet
245250255
seralaproprogluser
260
<210>415
<211>262
<212>prt
<213>粉尘克洛诺菌(cronobacterpulveris)
<400>415
cysleuthralaproprolysglualaalalysprothrleumetpro
151015
argalaglnsertyrargaspleuthrasnleuproaspprolysgly
202530
lysleuphevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thrsermetleuvalthralaleulysaspserargtrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalagluasnasnargmetpro
100105110
leuglnserleuvalalaalaasnvalmetilegluglyserileile
115120125
glytyrgluserasnvallysserglyglyvalglyalaargtyrphe
130135140
glyileglyglyaspthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluvalleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglygluileglytyrthrala
195200205
asngluprovalmetleucysleumetseralailegluthrglyval
210215220
ilehisleuileasnaspglyileasnargglyleutrpgluleulys
225230235240
asnlysglyaspalalysasnthrileleualalystyrargsermet
245250255
alavalproprogluser
260
<210>416
<211>262
<212>prt
<213>水生拉恩氏菌(rahnellaaquatilis)
<400>416
cysleuthralaalaprolysglualaalaargprothrleuleupro
151015
argalaprosertyrthraspleuthrhisleuproserproglngly
202530
argilephevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalacysasnpheserthralavalproglnserala
505560
thralametleuvalseralaleulysaspserlystrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglyservalalaileasnasnglnargpro
100105110
leuserserleuvalalaalaasnileleuilegluglyserileile
115120125
glytyrgluserasnvallysserglyglyvalglyalaargtyrphe
130135140
glyileglyalaserthrglntyrglnleuaspglnilealavalasn
145150155160
leuargalavalaspvalasnthrglygluvalleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglygluleuglytyrthrthr
195200205
asngluprovalmetleucysleumetseralailegluserglyval
210215220
iletyrleuvalasnaspglyilegluargasnleutrpglnleugln
225230235240
asnprosergluileasnserproileleuglnargtyrlysasnasn
245250255
ilevalproalagluser
260
<210>417
<211>259
<212>prt
<213>抗坏血酸克吕沃尔菌(kluyveraascorbata)
<400>417
cysilethrserproprolysglnalaalalysprothrleuleupro
151015
argserglnsertyrglnaspleuthrhisleuprogluproglngly
202530
argleuphevalservaltyrasnileseraspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthrservalproglnserala
505560
thralametleuvalseralaleulysaspserasntrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalavalasnasnargthrgln
100105110
leuproserleuvalalaalaasnileleuilegluglyserileile
115120125
glytyrgluserasnvallysserglyglyalaglyalaargtyrphe
130135140
glyileglyalaserthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluvalleuserservalasn
165170175
thrserlysthrileleusertyrglupheglnalaglyvalphearg
180185190
tyrileasptyrglnargleuleugluglygluvalglytyrthrval
195200205
asngluprovalmetleucysleumetseralailegluthrglyval
210215220
iletyrleuvalasnaspglyileserargasnleutrpglnleulys
225230235240
asnalaseraspileasnserprovalleuglulystyrlysserile
245250255
ilevalpro
<210>418
<211>259
<212>prt
<213>蜂房哈夫尼菌(hafniaalvei)
<400>418
cysleuthralaproprolysglnalaalalysprothrleumetpro
151015
argalaglnsertyrglnaspleuthrhisleuprogluproalagly
202530
lysleuphevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thralametleuvalseralaleulysaspserglytrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythralaalavalasnasnglnhisgln
100105110
leuserserleuvalalaalaasnvalleuvalgluglyserileile
115120125
glytyrgluserasnvallysserglyglyalaglyalaargphephe
130135140
glyileglyalaserthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalaspvalasnthrglyglnvalleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
tyrileasptyrglnargleuleugluglygluileglytyrthrthr
195200205
asngluprovalmetleucysvalmetseralailegluthrglyval
210215220
iletyrleuvalasnaspglyileasnargasnleutrpthrleulys
225230235240
asnproglnaspalalysserservalleugluargtyrlysserthr
245250255
ilevalpro
<210>419
<211>255
<212>prt
<213>肠杆菌科细菌菌株fgi57(enterobacteriaceaebacteriumstrainfgi57)
<400>419
cysilethrthrproproglnglualaalalysprothrleuleupro
151015
argaspalathrtyrlysaspleuvalserleuproglnproarggly
202530
lysiletyrvalalavaltyrasnileglnaspgluthrglyglnphe
354045
glnprotyrproalaserasnpheserthrservalproglnserala
505560
thralametleuvalserserleulysaspserargtrpphevalpro
65707580
leugluargglnglyleuasnasnleuleuasngluarglysileile
859095
argalaalaglnglnasnglythrvalglyaspasnasnalaserpro
100105110
leuproserleutyrseralaasnvalilevalgluglyserileile
115120125
glytyralaserasnvallysthrglyglypheglyalaargtyrphe
130135140
glyileglyglyserthrglntyrglnleuaspglnvalalavalasn
145150155160
leuargilevalasnvalhisthrglygluvalleuserservalasn
165170175
thrserlysthrileleusertyrgluileglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglyglualaglyphethrthr
195200205
asngluprovalmetthrcysleumetseralaileglugluglyval
210215220
ilehisleuileasnaspglyileasnlyslysleutrpalaleuser
225230235240
asnalaalaaspileasnsergluvalleuthrargtyrarglys
245250255
<210>420
<211>258
<212>prt
<213>邻单胞痢疾杆菌(plesiomonasshigelloides)
<400>420
ilethrgluvalprolysglualaalalysprothrleumetproarg
151015
alaserthrtyrlysaspleuvalalaleuprolysproasnglylys
202530
ileilevalservaltyrservalglnaspgluthrglyglnphelys
354045
proleuproalaserasnpheserthralavalproglnserglyasn
505560
alametleuthrseralaleulysaspserglytrpphevalproleu
65707580
gluarggluglyleuglnasnleuleuasngluarglysileilearg
859095
alaalaglngluasnglythrvalalaalaasnasnglnglnproleu
100105110
proserleuleuseralaasnvalvalilegluglyalaileilegly
115120125
tyraspseraspilelysthrglyglyalaglyalaargtyrphegly
130135140
ileglyalaaspglylystyrargvalaspglnvalalavalasnleu
145150155160
argalavalaspvalargthrglygluvalleuleuservalasnthr
165170175
serlysthrileleusersergluleuseralaglyvalpheargphe
180185190
ileglutyrglnargleuleugluleuglualaglytyrthrthrasn
195200205
gluprovalmetmetcysmetmetseralaleuglualaglyvalala
210215220
hisleuilevalgluglyileargglnasnleutrpserleuglnasn
225230235240
proseraspileasnasnproileileglnargtyrmetlysgluasp
245250255
valpro
<210>421
<211>248
<212>prt
<213>费氏弧菌(vibriofischeri)
<400>421
progluthrsergluserprothrleumetglnargglyalaasntyr
151015
ileaspleuileserleuprolysproglnglylysilephevalser
202530
valtyrasppheargaspglnthrglyglntyrlysproglnproasn
354045
serasnpheserthralavalproglnglyglythralaleuleuthr
505560
metalaleuleuaspserglutrpphetyrproleugluargglngly
65707580
leuglnasnleuleuthrgluarglysileileargalaalaglnlys
859095
lysglngluserileserasnhisglyserthrleuproserleuleu
100105110
seralaasnvalmetilegluglyglyilevalalatyraspserasn
115120125
ilelysthrglyglyalaglyalaargtyrleuglyileglyglyser
130135140
glyglntyrargalaaspglnvalthrvalasnileargalavalasp
145150155160
valargserglylysileleuthrservalthrthrserlysthrile
165170175
leusertyrgluvalseralaglyalapheargphevalasptyrlys
180185190
gluleuleugluvalgluleuglytyrthrasnasngluprovalasn
195200205
ilealaleumetseralaileaspseralavalilehisleuileval
210215220
lysglyvalglnglnglyleutrpargproalaasnleuaspthrarg
225230235240
asnasnproilephelyslystyr
245
<210>422
<211>248
<212>prt
<213>变形菌(aliivibriologei)
<400>422
proaspalasergluserprothrleumetglnargglyalathrtyr
151015
leuaspleuileserleuprolysproglnglylysiletyrvalser
202530
valtyrasppheargaspglnthrglyglntyrlysproglnproasn
354045
serasnpheserthralavalproglnglyglythralaleuleuthr
505560
metalaleuleuaspserglutrpphetyrproleugluargglngly
65707580
leuglnasnleuleuthrgluarglysileileargalaalaglnlys
859095
lysglngluserileserasnhisglyserthrleuproserleuleu
100105110
seralaasnvalmetilegluglyglyilevalalatyraspserasn
115120125
ilelysthrglyglyalaglyalaargtyrleuglyileglyglyser
130135140
glyglntyrargalaaspglnvalthrvalasnileargalavalasp
145150155160
valargserglylysileleuthrservalthrthrserlysthrile
165170175
leusertyrgluleuseralaglyalapheargphevalasptyrlys
180185190
gluleuleugluvalgluleuglytyrthrasnasngluprovalasn
195200205
ilealaleumetseralaileaspseralavalilehisleuileval
210215220
lysglyileglugluglyleutrpargprogluasnglnasnglylys
225230235240
gluasnproilephearglystyr
245
<210>423
<211>254
<212>prt
<213>发光杆菌属ak15(photobacteriumsp.ak15)
<400>423
progluthrserlysgluprothrleumetalaargglythralatyr
151015
glnaspleuvalserleuproleuprolysglylysvaltyrvalser
202530
valtyrasppheargaspglnthrglyglntyrlysproglnproasn
354045
serasnpheserthralavalproglnglyglyalaalaleuleuthr
505560
thralaleuleuaspserargtrpphemetproleugluargglugly
65707580
leuglnasnleuleuthrgluarglysileileargalaalaglnlys
859095
lysaspgluileprothrasnhisglyvalhisleuproserleuala
100105110
seralaasnilemetvalgluglyglyilevalalatyraspthrasn
115120125
ileglnthrglyglyalaglyalaargtyrleuglyvalglyalaser
130135140
glyglntyrargthraspglnvalthrvalasnileargalavalasp
145150155160
valargthrglyargileleuleuservalthrthrserlysthrile
165170175
leuserlysgluleuglnthrglyvalphelysphevalasptyrlys
180185190
aspleuleuglualagluleuglytyrthrthrasngluprovalasn
195200205
leualavalmetseralaileaspalaalavalvalhisvalileval
210215220
aspglyilelysthrglyleutrpgluproleuargglygluaspleu
225230235240
glnhisproileileglnglutyrmetasnargserlyspro
245250
<210>424
<211>261
<212>prt
<213>维氏气单胞菌(aeromonasveronii)
<400>424
cysalathrhisileglyserprovalalaaspglulysalathrleu
151015
metproargservalsertyrlysgluleuileserleuprolyspro
202530
lysglylysilevalalaalavaltyrasppheargaspglnthrgly
354045
glntyrleuproalaproalaserasnpheserthralavalthrgln
505560
glyglyvalalametleuserthralaleutrpaspserglntrpphe
65707580
valproleugluarggluglyleuglnasnleuleuthrgluarglys
859095
ilevalargalaalaglnasnlysproasnvalproglyasnasnala
100105110
asnglnleuproserleuvalalaalaasnileleuilegluglygly
115120125
ilevalalatyraspserasnvalargthrglyglyalaglyalalys
130135140
tyrpheglyileglyalaserglyglutyrargvalaspglnvalthr
145150155160
valasnleuargalavalaspileargserglyargileleuasnser
165170175
valthrthrserlysthrvalmetserglnglnvalglnalaglyval
180185190
pheargphevalglutyrlysargleuleuglualaglualaglyphe
195200205
serthrasngluprovalglnmetcysvalmetseralailegluser
210215220
glyvalileargleuilealaasnglyvalargaspasnleutrpgln
225230235240
leualaaspglnargaspileaspasnproileleuglnglutyrleu
245250255
glnaspasnalapro
260
<210>425
<211>239
<212>prt
<213>希瓦氏菌属ecsmb14101(shewanellasp.ecsmb14101)
<400>425
alaserserserleumetprolysglyglusertyrtyraspleuile
151015
asnleuproalaproglnglyvalmetleualaalavaltyraspphe
202530
argaspglnthrglyglntyrlysproileproserserasnpheser
354045
thralavalproglnserglythralapheleualaglnalaleuasn
505560
aspsersertrppheileprovalgluarggluglyleuglnasnleu
65707580
leuthrgluarglysilevalargalaglyleulysglyaspalaasn
859095
lysleuproglnleuasnseralaglnileleumetgluglyglyile
100105110
valalatyraspthrasnvalargthrglyglyalaglyalaargtyr
115120125
leuglyileglyalaalathrglnpheargvalaspthrvalthrval
130135140
asnleuargalavalaspileargthrglyargleuleuserserval
145150155160
thrthrthrlysserileleuserlysgluilethralaglyvalphe
165170175
lyspheileaspalaglngluleuleuglusergluleuglytyrthr
180185190
serasngluprovalserleucysvalalaseralailegluserala
195200205
valvalhismetilealaaspglyiletrplysglyalatrpasnleu
210215220
alaaspglnalaserglyleuargserprovalleuglnlystyr
225230235
<210>426
<211>233
<212>prt
<213>恶臭假单胞菌(pseudomonasputida)
<400>426
glnaspsergluthrprothrleuthrproargalaserthrtyrtyr
151015
aspleuileasnmetproargprolysglyargleumetalavalval
202530
tyrglypheargaspglnthrglyglntyrlysprothrproalaser
354045
serpheserthrservalthrglnglyalaalasermetleumetasp
505560
alaleuseralaserglytrpphevalvalleugluarggluglyleu
65707580
glnasnleuleuthrgluarglysileileargalaserglnlyslys
859095
proaspvalalagluasnilemetglygluleuproproleuglnala
100105110
alaasnleumetleugluglyglyileilealatyraspthrasnval
115120125
argserglyglygluglyalaargtyrleuglyileaspileserarg
130135140
glutyrargvalaspglnvalthrvalasnleuargalavalaspval
145150155160
argthrglyglnvalleualaasnvalmetthrserlysthriletyr
165170175
servalglyargseralaglyvalphelyspheilegluphelyslys
180185190
leuleuglualagluvalglytyrthrthrasngluproalaglnleu
195200205
cysvalleuseralailegluseralavalglyhisleuleualagln
210215220
glyilegluglnargleutrpglnval
225230
<210>427
<211>234
<212>prt
<213>紫色希瓦氏菌dss12(shewanellaviolaceadss12)
<400>427
metprolysseraspthrtyrtyraspleuileglyleuprohispro
151015
glnglysermetleualaalavaltyrasppheargaspglnthrgly
202530
glntyrlysalaileproserserasnpheserthralavalprogln
354045
serglythralapheleualaglnalaleuasnaspsersertrpphe
505560
valprovalgluarggluglyleuglnasnleuleuthrgluarglys
65707580
ilevalargalaglyleulysglyglualaasnglnleuproglnleu
859095
serseralaglnileleumetgluglyglyilevalalatyraspthr
100105110
asnilelysthrglyglyalaglyalaargtyrleuglyileglyval
115120125
asnserlyspheargvalaspthrvalthrvalasnleuargalaval
130135140
aspileargthrglyargleuleuserservalthrthrthrlysser
145150155160
ileleuserlysgluvalseralaglyvalphelyspheileaspala
165170175
glnaspleuleuglusergluleuglytyrthrserasngluproval
180185190
serleucysvalalaglnalailegluseralavalvalhismetile
195200205
alaaspglyiletrplysargalatrpasnleualaaspthralaser
210215220
glyleuasnasnprovalleuglnlystyr
225230
<210>428
<211>245
<212>prt
<213>詹氏海杆菌(marinobacteriumjannaschii)
<400>428
leuthrargargmetserthrtyrglnaspleuileaspmetproala
151015
proargglylysilevalthralavaltyrserpheargaspglnser
202530
glyglntyrlysproalaproserserserpheserthralavalthr
354045
glnglyalaalaalametleuvalasnvalleuasnaspserglytrp
505560
pheileproleugluarggluglyleuglnasnileleuthrgluarg
65707580
lysileileargalaalaleulyslysaspasnvalprovalasnasn
859095
seralaglyleuproserleuleualaalaasnilemetleuglugly
100105110
glyilevalglytyraspserasnilehisthrglyglyalaglyala
115120125
argtyrpheglyileglyalaserglulystyrargvalaspgluval
130135140
thrvalasnleuargalaileaspileargthrglyargileleuhis
145150155160
servalleuthrserlyslysileleuserarggluileargserasp
165170175
valtyrargpheilegluphelyshisleuleuglumetglualagly
180185190
ilethrthrasnaspproalaglnleucysvalleuseralaileglu
195200205
seralavalalahisleuilevalaspglyvalilelyslyssertrp
210215220
serleualaaspproasngluleuasnserprovalileglnalatyr
225230235240
glnglnglnargile
245
<210>429
<211>234
<212>prt
<213>金黄杆菌g311(chryseobacteriumoranimenseg311)
<400>429
proseraspprogluargserthrmetglygluleuthrproserthr
151015
alagluleuargasnleuproleuproasnglulysilevalilegly
202530
valtyrlyspheargaspglnthrglyglntyrlysprosergluasn
354045
glyasnasntrpserthralavalproglnglythrthrthrileleu
505560
ilelysalaleugluaspserargtrppheileproilegluargglu
65707580
asnilealaasnleuleuasngluargglnileileargserthrarg
859095
glnglutyrmetlysaspalaasplysasnserglnserleupropro
100105110
leuleutyralaglyileleuleugluglyglyvalilesertyrasp
115120125
serasnthrmetthrglyglypheglyalaargtyrpheglyilegly
130135140
alaserthrglntyrargglnaspargilethriletyrleuargala
145150155160
valserthrleuasnglygluileleulysthrvaltyrthrserlys
165170175
thrileleuserthrservalasnglyserphepheargtyrileasp
180185190
thrgluargleuleuglualagluvalglyleuthrglnasnglupro
195200205
valglnleualavalthrglualaileglulysalavalargserleu
210215220
ileilegluglythrargasplysiletrp
225230
<210>430
<211>12
<212>dna
<213>人工序列
<220>
<223>实施例18中使用的多核苷酸序列
<400>430
tttttttttttt12
<210>431
<211>27
<212>dna
<213>人工序列
<220>
<223>实施例18中使用的多核苷酸序列
<400>431
ggttgtttctgttggtgctgatattgc27
<210>432
<211>3635
<212>dna
<213>人工序列
<220>
<223>实施例18中使用的多核苷酸序列
<400>432
gccatcagattgtgtttgttagtcgctgccatcagattgtgtttgttagtcgcttttttt60
ttttggaattttttttttggaattttttttttgcgctaacaacctcctgccgttttgccc120
gtgcatatcggtcacgaacaaatctgattactaaacacagtagcctggatttgttctatc180
agtaatcgaccttattcctaattaaatagagcaaatccccttattgggggtaagacatga240
agatgccagaaaaacatgacctgttggccgccattctcgcggcaaaggaacaaggcatcg300
gggcaatccttgcgtttgcaatggcgtaccttcgcggcagatataatggcggtgcgttta360
caaaaacagtaatcgacgcaacgatgtgcgccattatcgcctagttcattcgtgaccttc420
tcgacttcgccggactaagtagcaatctcgcttatataacgagcgtgtttatcggctaca480
tcggtactgactcgattggttcgcttatcaaacgcttcgctgctaaaaaagccggagtag540
aagatggtagaaatcaataatcaacgtaaggcgttcctcgatatgctggcgtggtcggag600
ggaactgataacggacgtcagaaaaccagaaatcatggttatgacgtcattgtaggcgga660
gagctatttactgattactccgatcaccctcgcaaacttgtcacgctaaacccaaaactc720
aaatcaacaggcgccggacgctaccagcttctttcccgttggtgggatgcctaccgcaag780
cagcttggcctgaaagacttctctccgaaaagtcaggacgctgtggcattgcagcagatt840
aaggagcgtggcgctttacctatgattgatcgtggtgatatccgtcaggcaatcgaccgt900
tgcagcaatatctgggcttcactgccgggcgctggttatggtcagttcgagcataaggct960
gacagcctgattgcaaaattcaaagaagcgggcggaacggtcagagagattgatgtatga1020
gcagagtcaccgcgattatctccgctctggttatctgcatcatcgtctgcctgtcatggg1080
ctgttaatcattaccgtgataacgccattacctacaaagcccagcgcgacaaaaatgcca1140
gagaactgaagctggcgaacgcggcaattactgacatgcagatgcgtcagcgtgatgttg1200
ctgcgctcgatgcaaaatacacgaaggagttagctgatgctaaagctgaaaatgatgctc1260
tgcgtgatgatgttgccgctggtcgtcgtcggttgcacatcaaagcagtctgtcagtcag1320
tgcgtgaagccaccaccgcctccggcgtggataatgcagcctccccccgactggcagaca1380
ccgctgaacgggattatttcaccctcagagagaggctgatcactatgcaaaaacaactgg1440
aaggaacccagaagtatattaatgagcagtgcagatagagttgcccatatcgatgggcaa1500
ctcatgcaattattgtgagcaatacacacgcgcttccagcggagtataaatgcctaaagt1560
aataaaaccgagcaatccatttacgaatgtttgctgggtttctgttttaacaacattttc1620
tgcgccgccacaaattttggctgcatcgacagttttcttctgcccaattccagaaacgaa1680
gaaatgatgggtgatggtttcctttggtgctactgctgccggtttgttttgaacagtaaa1740
cgtctgttgagcacatcctgtaataagcagggccagcgcagtagcgagtagcattttttt1800
catggtgttattcccgatgctttttgaagttcgcagaatcgtatgtgtagaaaattaaac1860
aaaccctaaacaatgagttgaaatttcatattgttaatatttattaatgtatgtcaggtg1920
cgatgaatcgtcattgtattcccggattaactatgtccacagccctgacggggaacttct1980
ctgcgggagtgtccgggaataattaaaacgatgcacacagggtttagcgcgtacacgtat2040
tgcattatgccaacgccccggtgctgacacggaagaaaccggacgttatgatttagcgtg2100
gaaagatttgtgtagtgttctgaatgctctcagtaaatagtaatgaattatcaaaggtat2160
agtaatatcttttatgttcatggatatttgtaacccatcggaaaactcctgctttagcaa2220
gattttccctgtattgctgaaatgtgatttctcttgatttcaacctatcataggacgttt2280
ctataagatgcgtgtttcttgagaatttaacatttacaacctttttaagtccttttatta2340
acacggtgttatcgttttctaacacgatgtgaatattatctgtggctagatagtaaatat2400
aatgtgagacgttgtgacgttttagttcagaataaaacaattcacagtctaaatcttttc2460
gcacttgatcgaatatttctttaaaaatggcaacctgagccattggtaaaaccttccatg2520
tgatacgagggcgcgtagtttgcattatcgtttttatcgtttcaatctggtctgacctcc2580
ttgtgttttgttgatgatttatgtcaaatattaggaatgttttcacttaatagtattggt2640
tgcgtaacaaagtgcggtcctgctggcattctggagggaaatacaaccgacagatgtatg2700
taaggccaacgtgctcaaatcttcatacagaaagatttgaagtaatattttaaccgctag2760
atgaagagcaagcgcatggagcgacaaaatgaataaagaacaatctgctgatgatccctc2820
cgtggatctgattcgtgtaaaaaatatgcttaatagcaccatttctatgagttaccctga2880
tgttgtaattgcatgtatagaacataaggtgtctctggaagcattcagagcaattgaggc2940
agcgttggtgaagcacgataataatatgaaggattattccctggtggttgactgatcacc3000
ataactgctaatcattcaaactatttagtctgtgacagagccaacacgcagtctgtcact3060
gtcaggaaagtggtaaaactgcaactcaattactgcaatgccctcgtaattaagtgaatt3120
tacaatatcgtcctgttcggagggaagaacgcgggatgttcattcttcatcacttttaat3180
tgatgtatatgctctcttttctgacgttagtctccgacggcaggcttcaatgacccaggc3240
tgagaaattcccggaccctttttgctcaagagcgatgttaatttgttcaatcatttggtt3300
aggaaagcggatgttgcgggttgttgttctgcgggttctgttcttcgttgacatgaggtt3360
gccccgtattcagtgtcgctgatttgtattgtctgaagttgtttttacgttaagttgatg3420
cagatcaattaatacgatacctgcgtcataattgattatttgacgtggtttgatggcctc3480
cacgcacgttgtgatatgtagatgataatcattatcactttacgggtcctttccggtgaa3540
aaaaaaggtaccaaaaaaaacatcgtcgtgagtagtgaaccgtaagccgttctgtttatg3600
tttcttggacactgattgacacggtttagtagaac3635
<210>433
<211>3636
<212>dna
<213>人工序列
<220>
<223>实施例18中使用的多核苷酸序列
<400>433
ttttttttttttttttttttttttttttcaagaaacataaacagaacgtgcttacggttc60
actactcacgacgatgttttttttggtaccttttttttcaccggaaaggacccgtaaagt120
gataatgattatcatctacatatcacaacgtgcgtggaggccatcaaaccacgtcaaata180
atcaattatgacgcaggtatcgtattaattgatctgcatcaacttaacgtaaaaacaact240
tcagacaatacaaatcagcgacactgaatacggggcaacctcatgtcaacgaagaacaga300
acccgcagaacaacaacccgcaacatccgctttcctaaccaaatgattgaacaaattaac360
atcgctcttgagcaaaaagggtccgggaatttctcagcctgggtcattgaagcctgccgt420
cggagactaacgtcagaaaagagagcatatacatcaattaaaagtgatgaagaatgaaca480
tcccgcgttcttccctccgaacaggacgatattgtaaattcacttaattacgagggcatt540
gcagtaattgagttgcagttttaccactttcctgacagtgacagactgcgtgttggctct600
gtcacagactaaatagtttgaatgattagcagttatggtgatcagtcaaccaccagggaa660
taatccttcatattattatcgtgcttcaccaacgctgcctcaattgctctgaatgcttcc720
agagacaccttatgttctatacatgcaattacaacatcagggtaactcatagaaatggtg780
ctattaagcatattttttacacgaatcagatccacggagggatcatcagcagattgttct840
ttattcattttgtcgctccatgcgcttgctcttcatctagcggttaaaatattacttcaa900
atctttctgtatgaagatttgagcacgttggccttacatacatctgtcggttgtatttcc960
ctccagaatgccagcaggaccgcactttgttacgcaaccaatactattaagtgaaaacat1020
tcctaatatttgacataaatcatcaacaaaacacaaggaggtcagaccagattgaaacga1080
taaaaacgataatgcaaactacgcgccctcgtatcacatggaaggttttaccaatggctc1140
aggttgccatttttaaagaaatattcgatcaagtgcgaaaagatttagactgtgaattgt1200
tttattctgaactaaaacgtcacaacgtctcacattatatttactatctagccacagata1260
atattcacatcgtgttagaaaacgataacaccgtgttaataaaaggacttaaaaaggttg1320
taaatgttaaattctcaagaaacacgcatcttatagaaacgtcctatgataggttgaaat1380
caagagaaatcacatttcagcaatacagggaaaatcttgctaaagcaggagttttccgat1440
gggttacaaatatccatgaacataaaagatattactatacctttgataattcattactat1500
ttactgagagcattcagaacactacacaaatctttccacgctaaatcataacgtccggtt1560
tcttccgtgtcagcaccggggcgttggcataatgcaatacgtgtacgcgctaaaccctgt1620
gtgcatcgttttaattattcccggacactcccgcagagaagttccccgtcagggctgtgg1680
acatagttaatccgggaatacaatgacgattcatcgcacctgacatacattaataaatat1740
taacaatatgaaatttcaactcattgtttagggtttgtttaattttctacacatacgatt1800
ctgcgaacttcaaaaagcatcgggaataacaccatgaaaaaaatgctactcgctactgcg1860
ctggccctgcttattacaggatgtgctcaacagacgtttactgttcaaaacaaaccggca1920
gcagtagcaccaaaggaaaccatcacccatcatttcttcgtttctggaattgggcagaag1980
aaaactgtcgatgcagccaaaatttgtggcggcgcagaaaatgttgttaaaacagaaacc2040
cagcaaacattcgtaaatggattgctcggttttattactttaggcatttatactccgctg2100
gaagcgcgtgtgtattgctcacaataattgcatgagttgcccatcgatatgggcaactct2160
atctgcactgctcattaatatacttctgggttccttccagttgtttttgcatagtgatca2220
gcctctctctgagggtgaaataatcccgttcagcggtgtctgccagtcggggggaggctg2280
cattatccacgccggaggcggtggtggcttcacgcactgactgacagactgctttgatgt2340
gcaaccgacgacgaccagcggcaacatcatcacgcagagcatcattttcagctttagcat2400
cagctaactccttcgtgtattttgcatcgagcgcagcaacatcacgctgacgcatctgca2460
tgtcagtaattgccgcgttcgccagcttcagttctctggcatttttgtcgcgctgggctt2520
tgtaggtaatggcgttatcacggtaatgattaacagcccatgacaggcagacgatgatgc2580
agataaccagagcggagataatcgcggtgactctgctcatacatcaatctctctgaccgt2640
tccgcccgcttctttgaattttgcaatcaggctgtcagccttatgctcgaactgaccata2700
accagcgcccggcagtgaagcccagatattgctgcaacggtcgattgcctgacggatatc2760
accacgatcaatcataggtaaagcgccacgctccttaatctgctgcaatgccacagcgtc2820
ctgacttttcggagagaagtctttcaggccaagctgcttgcggtaggcatcccaccaacg2880
ggaaagaagctggtagcgtccggcgcctgttgatttgagttttgggtttagcgtgacaag2940
tttgcgagggtgatcggagtaatcagtaaatagctctccgcctacaatgacgtcataacc3000
atgatttctggttttctgacgtccgttatcagttccctccgaccacgccagcatatcgag3060
gaacgccttacgttgattattgatttctaccatcttctactccggcttttttagcagcga3120
agcgtttgataagcgaaccaatcgagtcagtaccgatgtagccgataaacacgctcgtta3180
tataagcgagattgctacttagtccggcgaagtcgagaaggtcacgaatgaactaggcga3240
taatggcgcacatcgttgcgtcgattactgtttttgtaaacgcaccgccattatatctgc3300
cgcgaaggtacgccattgcaaacgcaaggattgccccgatgccttgttcctttgccgcga3360
gaatggcggccaacaggtcatgtttttctggcatcttcatgtcttacccccaataagggg3420
atttgctctatttaattaggaataaggtcgattactgatagaacaaatccaggctactgt3480
gtttagtaatcagatttgttcgtgaccgatatgcacgggcaaaacggcaggaggttgtta3540
gcgcaaaaaaaaaattccaaaaaaaaaattccaaaaaaaaaaagcgactaacaaacacaa3600
tctgatggcagcgactaacaaacacaatctgatggc3636
<210>434
<211>28
<212>dna
<213>人工序列
<220>
<223>实施例18中使用的多核苷酸序列
<400>434
gcaatatcagcaccaacagaaacaacct28
<210>435
<211>10
<212>prt
<213>人工序列
<220>
<223>stepll(c)的氨基酸序列
<400>435
seralatrpserhisproglnpheglulys
1510
<210>436
<211>15
<212>prt
<213>人工序列
<220>
<223>pro的氨基酸序列
<400>436
metglnargleupheleuleuvalalavalmetleuleusergly
151015
<210>437
<211>22
<212>dna
<213>人工序列
<220>
<223>引物
<400>437
tctttaaccgccccgcctaaag22
<210>438
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>438
cattttttgccctcgttatc20
<210>439
<211>67
<212>dna
<213>人工序列
<220>
<223>引物
<400>439
aataactcaaccgatttttaagccccagcttcataaggaaaataatcgtgtaggctggag60
ctgcttc67
<210>440
<211>66
<212>dna
<213>人工序列
<220>
<223>引物
<400>440
cgcttaaacagtaaaatgccggatgataattccggcttttttatctgcatatgaatatcc60
tcctta66
<210>441
<211>78
<212>dna
<213>人工序列
<220>
<223>发夹
<400>441
gcggggagcgtattagagttggatcggatgcagctggctactgacgtcatgacgtcagta60
gccagcatgcatccgatc78
<210>442
<211>239
<212>prt
<213>假交替单胞菌属(种类sm9913)(pseudoalteromonassp.(strainsm9913))
<400>442
metglyasnleuileproproserlysglnseralavalthrleuglu
151015
prothraspvalpheseraspleulysserleuprolysproalagly
202530
serileprovalservaltyrserpheargaspglnthrglyglntyr
354045
lysproglnthrasnvalserserpheserthralavalthrglngly
505560
alaasnserileleuvalglnalaleuhisgluserasptrpphethr
65707580
provalgluarggluglyleuglnasnileleuthrgluarglysile
859095
ileargalaalaglnalathrasnproasnglnglygluleupropro
100105110
leuthrthralalysileileleugluglyglyileilesertyrasp
115120125
thrasnvallysthrglyglyleuglymetglutyrpheglyilegly
130135140
alasergluleutyrarggluaspalaileseriletyrleuargala
145150155160
valaspvalargthrglyglnvalleuleuservalalathrserlys
165170175
lysvalleuserglnglumetargalaglyphepheargtyrvalser
180185190
tyrlysargleualaglualaglualaglypheseraspasnglupro
195200205
metserilecysvalthrglnalaileglulysalaleuthraspleu
210215220
ilethrlysglyileasplysglyleutrpalalysglnalathr
225230235
<210>443
<211>252
<212>prt
<213>希瓦氏菌(shewanellabaltica)
<400>443
cysserleuileprolysproaspleuasnilethrproalagluval
151015
asnproleusergluvalmetargglyleuglnthrglnproglypro
202530
lyspheproileprovalalavaltyrserpheargaspglnthrgly
354045
glntyrlysproglnalaasnvalserserpheserthralavalthr
505560
glnglyalathrsermetleumetglnthrleuleuaspserlystrp
65707580
phethrprovalgluarggluglyleuglnasnleuleuthrgluarg
859095
lysileserasnlysglnserglythrlysglyaspaspvalproval
100105110
leuserthralaargleuleuleugluglyglyvalilesertyrglu
115120125
thrasnthrserthrglyglyserglyvalglutyrtyrglyilegly
130135140
alaserglumettyrarggluaspglnvalthriletyrleuargala
145150155160
valaspvalhisthrglylysvalmetmetservalserthrserlys
165170175
argvalleuserglnglumetargalaglyleupheargtyrthrser
180185190
leuasnargleualaglualagluileglyphethrthrasnglupro
195200205
valglnphecysvalleuglnalailegluleualavalalagluleu
210215220
ileasplysglyilelysglnglytyrtrpserprothrglnvalthr
225230235240
glnprovalglulysalaglngluleuserglnser
245250
<210>444
<211>264
<212>prt
<213>假单胞菌(pseudomonasentomophila)
<400>444
cysglyleuarggluprometproalagluglnaspalagluthrpro
151015
thrleuthrproargalaserthrtyrtyraspleuileasnmetpro
202530
argproargglyargleumetalavalvaltyrglypheargaspgln
354045
thrglyglntyrlysprothrproalaserserpheserthrserval
505560
thrglnglyalaalasermetleumetaspalaleuasnalasergly
65707580
trpphevalvalleugluarggluglyleuglnasnleuleuthrglu
859095
arglysileileargalaserglnlyslysproaspvalalagluasn
100105110
ileglnglygluleuproproleuglnalaalaasnleumetleuglu
115120125
glyglyileilealatyraspthrasnvalargserglyglyglugly
130135140
alaargtyrleuglyileaspileserargglutyrargvalaspgln
145150155160
valthrvalasnleuargalavalaspvalargthrglyglnvalleu
165170175
alaasnvalmetthrserlysthriletyrservalglyargserala
180185190
glyvalphelyspheilegluphelyslysleuleuglualagluval
195200205
glytyrthrthrasngluproalaglnleucysvalleuseralaile
210215220
glualaalavalglyhisleuleualaglnglyilegluargargleu
225230235240
trpglnvalalaalaaspglyalaglyasplysalaleuleuasplys
245250255
tyrleuserglntyrglnglnpro
260
<210>445
<211>261
<212>prt
<213>蜂房哈夫尼菌
<400>445
cysalaglymetvalalathrsergluasnleugluglyalagluala
151015
thrleuthrproargglyalathrtyrglnaspleuvalserleupro
202530
proproserglylysilephevalservaltyrasppheargaspgln
354045
thrglyglntyrargproalaproalaserthrpheserthralaval
505560
thrglnglyalaalaalametleuthrglyserleuseraspsergly
65707580
trppheileproleugluargvalglyleuglnasnleuleuthrglu
859095
argargileileargalagluphegluargpheglyglnproaspthr
100105110
leuproserleuargalaalaservalmetleugluglyglyileile
115120125
alatyrgluserasnileargthrglyglyalaglyalaglutyrphe
130135140
glyileglyalaserglyglntyrglnvalaspglnvalthrvalasn
145150155160
leuargalavalgluileserthrglygluileleualaasnvalthr
165170175
thrthrlysthriletyrserlysgluleuargalaglyvaltyrarg
180185190
pheileasppheserargleuleuglualaglualaglyilethrala
195200205
asngluprovalglnleualavalmetseralailegluseralaval
210215220
ilehisleuilealaargglyilegluasnargleutrpasnleuala
225230235240
proglyvalserleuglngluserileleuasnasptyrleuasnala
245250255
proileprometleu
260
<210>446
<211>262
<212>prt
<213>大肠杆菌
<400>446
cysleuthralaproprolysglualaalaargprothrleumetpro
151015
argalaglnsertyrlysaspleuthrhisleuproalaprothrgly
202530
lysilephevalservaltyrasnileglnaspgluthrglyglnphe
354045
lysprotyrproalaserasnpheserthralavalproglnserala
505560
thralametleuvalthralaleulysaspserargtrppheilepro
65707580
leugluargglnglyleuglnasnleuleuasngluarglysileile
859095
argalaalaglngluasnglythrvalalaileasnasnargilepro
100105110
leuglnserleuthralaalaasnilemetvalgluglyserileile
115120125
glytyrgluserasnvallysserglyglyvalglyalaargtyrphe
130135140
glyileglyalaaspthrglntyrglnleuaspglnilealavalasn
145150155160
leuargvalvalasnvalserthrglygluileleuserservalasn
165170175
thrserlysthrileleusertyrgluvalglnalaglyvalphearg
180185190
pheileasptyrglnargleuleugluglygluvalglytyrthrser
195200205
asngluprovalmetleucysleumetseralailegluthrglyval
210215220
ilepheleuileasnaspglyileaspargglyleutrpaspleugln
225230235240
asnlysalagluargglnasnaspileleuvallystyrarghismet
245250255
servalproprogluser
260
<210>447
<211>263
<212>prt
<213>东方弧菌(vibrioorientalis)
<400>447
cysserasnsermetthrileproaspalaaspglualaprothrleu
151015
metproargglyalathrtyrglnaspleuvalglnleuproglupro
202530
lysglyargileleuvalservaltyrasppheargaspglnthrgly
354045
glntyrlysproglnproasnserasnpheserthralavalprogln
505560
glyglythralaleuleuthralaserleuleuaspserargtrpphe
65707580
ileproleugluarggluglyleuglnasnleuleuthrgluarglys
859095
ileileargalaalaglnlyslysglyglnalaalaserasnhisgly
100105110
aspaspleuseralaleuserseralaasnvalvalilegluglygly
115120125
ilevalalatyraspserasnilelysthrglyglyleuglyalaarg
130135140
tyrleuglyileglyserserglylystyrargthraspglnvalthr
145150155160
valasnleuargalavalasnvalargthrglyglnileleuleuser
165170175
valthrthrserlysthrilepheserhisgluleuthralaglyala
180185190
pheargpheileasptyrlysaspleuleugluvalglumetglytyr
195200205
thrasnasngluprovalasnvalalavalmetseralaileaspala
210215220
alavalilehisleuilevallysglyileglyargglyleutrpgln
225230235240
progluasnlysgluaspleuglnaspgluthrileglnargtyrala
245250255
lysglnthrhisglnileleu
260
<210>448
<211>267
<212>prt
<213>shewanellapealeana
<400>448
cysserthrmetsergluilegluglnilethrproserserserleu
151015
metprolyssergluthrtyrtyraspleuileglyleuproalapro
202530
glnglysermetvalalaalavaltyrasppheargaspglnthrgly
354045
glntyrlysproileproserserasnpheserthralavalprogln
505560
serglythralapheleualaglnalaleuasnaspsersertrpphe
65707580
thrprovalgluarggluglyleuglnasnleuleuthrgluarglys
859095
ilevalargalaglyleulysglyaspalaalaserleuserglnleu
100105110
asnseralaglnileleumetgluglyglyilevalalatyraspthr
115120125
asnileargthrglyglyalaglyalaargtyrleuglyileglyala
130135140
serglyglnpheargvalaspserilethrvalasnleuargalaval
145150155160
aspileargthrglyargleuleuserservalthrthrthrlysser
165170175
valleuserlysgluleuthralaglyvalphelyspheileaspala
180185190
glngluleuleuglusergluileglytyrthrserasngluproval
195200205
serleucysvalalaglnalailegluseralavalvalhismetile
210215220
alaaspglyiletrplysargalatrpasnleualaaspserglnthr
225230235240
glyleugluasnproileleulyslystyrtrpleuglualahisser
245250255
valgluargvalglnalaargleugluglngly
260265
<210>449
<211>21
<212>prt
<213>人工序列
<220>
<223>tm1发夹(图43)
<400>449
vallysserglyglyvalglyalaargtyrpheglyileglyalaasp
151015
thrglntyrglnleu
20
<210>450
<211>27
<212>prt
<213>人工序列
<220>
<223>tm2发夹(图43)
<400>450
leusertyrgluvalglnalaglyvalpheargpheileasptyrgln
151015
argleuleugluglygluvalglytyrthrser
2025
- 还没有人留言评论。精彩留言会获得点赞!