纤维素可及度测量的融合蛋白及其应用的制作方法
本发明涉及生物化工领域,特别涉及用于纤维素可及度测量的融合蛋白及其应用。
背景技术:
由于化石资源短缺日趋严峻和环境污染日益严重,以来源广泛且可再生的木质纤维素为原料生产生物能源和工业化学品受到了世界各国的广泛关注。其中以生物技术为核心的木质纤维素的生物转化已经成为生物能源开发、生物质资源加工和绿色化工过程的关键技术之一,相关研究也已成为当前工业微生物技术的一个热点和难点。然而,植物在长期进化过程中形成了复杂的木质纤维素结构以防御动物和微生物的攻击,这种木质纤维素抵抗微生物和酶降解的各种特性统称为“抗生物降解屏障”。因此在木质纤维素生物转化过程中,首先必须要对木质纤维素进行预处理以提高纤维素的可及度,这是纤维素生物转化和利用过程的关键步骤之一。
纤维素的可及度是指纤维素对纤维素酶的可接触程度,是表征木质纤维素生物降解和预处理效果的重要标准。纤维素的可及度与基质的孔结构、比表面积及颗粒尺寸密切相关。而传统化学和材料科学中用于分析固体材料孔结构特性的方法亦可用于分析木质纤维素基质的孔结构。目前可用于表征木质纤维素基质孔结构特性的方法主要有 BET 氮气分子吸附法、溶质排斥法、差示扫描量热法和核磁共振法。但用这些方法测定纤维素可及度时,不同程度的存在某些问题。例如,BET 氮气分子吸附法、差示扫描量热法和核磁共振法测量纤维素可及度时,被测量原料需要进行干燥,而干燥会导致底物孔隙特性改变。另外,由于探针分子大小与纤维素酶分子大小相差较大,因而所测定的可及度与实际可及度偏差较大。溶质排斥法虽然可以测定含水状态下木质纤维素基质的纤维素可及度,但该方法费时费力,且重复性很差,难以测定纤维素表面贡献的可及表面积。
因此,要准确测定木质纤维素原料及预处理后基质中纤维素的可及度,所用探针分子需要满足如下要求:(1)探针分子需要与纤维素酶分子具有相当的尺寸大小;(2)探针分子需要可特异性识别纤维素,就像纤维素酶分子的纤维素结合元件(CBM)那样,可在热力学上自发地识别纤维素;(3)探针分子可定量分析或者能够给出可定量测量的信号。因此,本发明提供了一种新的纤维素可及度测量方法,通过构建与真菌纤维素酶具有类似纤维素识别功能且分子大小相当的融合蛋白探针分子,且该融合蛋白可发出定量分析的信号,从而实现准确定量测量纤维素对真菌纤维素酶的可及度。
技术实现要素:
现有技术测量纤维素可及度,存在被测量材料需要干燥、测量偏差较大、测量过程费时费力和可重复性差的问题。针对以上问题,本发明提供一种可特异性识别纤维素且具有与真菌纤维素酶关键组分外切葡聚糖酶相当分子量大小的融合蛋白分子,以及将其用作探针分子来准确测量纤维素可及度的新方法。
本发明提供了一种纤维素可及度测量的融合蛋白,其包含纤维素识别多肽、连接肽和荧光蛋白,或者由纤维素识别多肽、连接肽和荧光蛋白组成,其中所述连接肽位于纤维素识别多肽和荧光蛋白之间,并且所述融合蛋白的分子量为30-80 kD。
优选地,所述融合蛋白中的纤维素识别多肽为选自曲霉属、木霉属和青霉属中任一种真菌的纤维素酶的纤维素结合元件(CBM),优选为外切葡聚糖酶的纤维素结合元件;进一步优选地,其中所述纤维素识别多肽包含SEQ ID No:1所示的氨基酸序列,或由SEQ ID No:1所示的氨基酸序列组成。
优选地,所述融合蛋白的连接肽为选自曲霉属、木霉属和青霉属中任一种真菌的外切葡聚糖酶的连接肽,优选为外切葡聚糖酶中的CBM与催化域之间的连接肽;进一步优选地,其中所述连接肽包含如SEQ ID No:2所示的氨基酸序列,或者由SEQ ID No:2所示的氨基酸序列组成。
优选地,所述融合蛋白的荧光蛋白选自绿色荧光蛋白或其变体,包括蓝色荧光蛋白、蓝绿色荧光蛋白、原绿色荧光蛋白、增强型绿色荧光蛋白、增强型黄色荧光蛋白、光活性绿色荧光蛋白等;进一步优选地,其中所述荧光蛋白为分子量为20-60kD的绿色荧光蛋白;更进一步优选地,所述融合蛋白中包含1- 3个绿色荧光蛋白;再进一步优选地,其中所述荧光蛋白包含SEQ ID No:3 所示的氨基酸序列,或者由SEQ ID No:3 所示的氨基酸序列组成。
本发明提供了一种核酸分子,其编码所述的融合蛋白。
本发明提供了一种重组质粒,其包含所述的核酸分子;进一步优选地,所述重组质粒的表达载体选自大肠杆菌pET28a载体。
本发明还提供了一种构建所述重组质粒的方法,其包括以下步骤:
(1)设计并合成所述的核酸分子序列,使得其所表达的融合蛋白的分子量为 30-80kD;
(2)PCR扩增所合成的核酸分子序列,优选地,所使用的扩增引物如SEQ ID No.4和SEQ ID No.5所示;以及
(3)将步骤(2)中所扩增的产物回收并连接到大肠杆菌表达载体上,挑选出阳性克隆。
本发明还提供了一种表达所述融合蛋白的方法,其包括以下步骤:
(1)构建前述重组质粒;以及
(2)将步骤(1)中所构建的重组质粒转化到大肠杆菌表达菌株中,挑选出阳性克隆,并且诱导培养所述的阳性克隆表达融合蛋白;优选地,所述诱导培养条件如下:诱导剂,终浓度为1 mM IPTG;诱导温度,30℃;诱导时间,6 h。
本发明还提供了利用所述融合蛋白来测量纤维素可及度的方法;优选地,所述方法包括以下步骤:将1-100 mg/ml 的底物悬浮在pH 4至9的缓冲溶液中,加入1至20 μg/ml所述融合蛋白,于10至40℃下反应10至180分钟,测定吸附前后液相荧光强度变化,由此计算出底物的纤维素可及度。
优选地,本发明提供的方法可用于测定纯的纤维素或者经预处理后的木质纤维素中纤维素的可及度。
经过实验,用本发明提供的融合蛋白对木质纤维素的可及度进行测量,与传统方法相比,可以更准确地测定纤维素的可及度。
此外,为避免融合蛋白表达不完全,以及提高融合蛋白的表达量和稳定性,所述融合蛋白可以以CBM-GFP或者GFP-CBM的形式表达。
本发明的优点在于:
1、本发明提供的融合蛋白含有真菌纤维素酶的纤维素识别组件(CBM),可特异性地识别纤维素底物。
2、本发明提供的融合蛋白探含有荧光蛋白,可以获得可视化分析和定量测量的信号。
3、本发明提供的融合蛋白模拟了真菌纤维素酶的吸附特性。
4、本发明提供的融合蛋白的大小与纤维素酶中的关键组分外切葡聚糖酶(CBH)类似,因而可以提高测量的精确度。
5、本发明提供的融合蛋白可以用于含水状态下底物可及度的测定,避免了干燥带来的底物孔结构的改变。
附图说明:
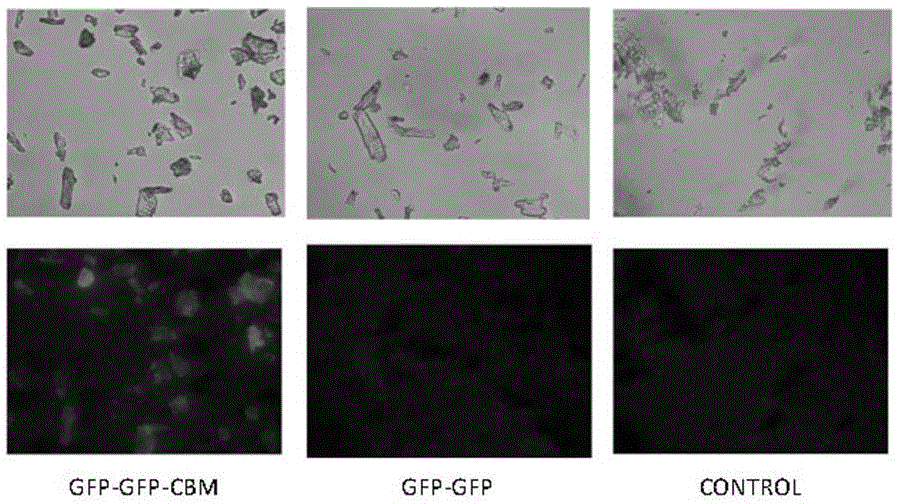
附图1:荧光显微镜下GFP-GFP-CBM和GFP-GFP蛋白以滤纸为底物的吸附结果
附图2:荧光显微镜下GFP-GFP-CBM和GFP-GFP蛋白以微晶纤维素为底物吸附结果
附图3:融合蛋白分子吸附量与预处理后麦秆纤维素酶解性能的关系
具体实施方式
为了更详细地说明本发明,给出下述制备实例。但本发明的范围并不局限于此。
实施例1
大肠杆菌重组质粒pET28a-GFP2-CBM的构建
根据Genebank中GFP基因、CBM基因的序列,设计两个GFP与CBM融合蛋白的基因序列并合成于pUC57-simple载体上。根据融合蛋白GFP2-CBM的基因序列及大肠杆菌表达载体pET28a上多克隆位点的特征,设计合成以下两条引物:
GFP_Fe: 5’-GACgaattcATGCGTAAAGGCGAAGAGCTGTTC-3’
CBM_Rh:5’- GACaagcttTTACAAGCACTGAGAGTAGTAAGG-3’
GFP_Fe、CBM_Rh两端分别设计了EcoRI和HindIII酶切位点,以小写字体表示。采用GFP_Fe、CBM_Rh引物,以本实验室构建的重组质粒pUC57-simple-GFP2-CBM为模板, 通过PCR方法扩增出GFP2-CBM基因。PCR条件:预变性95℃ 5分钟;再进行30个循环的95℃ 变性30秒, 60℃退火1分钟,72℃延伸 1 分30秒;最后72℃延伸 10分钟。扩增产物(大小约1.7kb)切胶回收后,将回收后的GFP2-CBM基因和载体pET28a分别进行EcoRI和HindIII双酶切,用T4连接酶连接,构建重组质粒pET28a-GFP2-CBM。将连接产物进行EcoRI和HindIII双酶切验证和测序验证。并对序列进行测序分析。
结果分析:重组质粒上的GFP2-CBM基因序列与设计的基因序列一致,说明大肠杆菌重组质粒pET28a-GFP2-CBM的基因构建正确。
实施例2
融合蛋白GFP2-CBM的表达
将重组质粒pET28a-GFP2-CBM转化到大肠杆菌表达菌种BL21,将菌液涂布于含50 μg/ml Kanamycin的LB平板上进行抗性筛选,挑取单克隆PCR检测,选择阳性克隆。
挑取重组大肠杆菌表达菌株BL21-pET28a-GFP2-CBM,接种于5 ml LB培养基中,37℃,200rpm摇床培养过夜。再取1 ml菌液转接100 ml LB培养基中,37℃,200rpm摇床培养至OD600为0.6左右,添加IPTG至终浓度为1 mM,30℃,200rpm摇床培养6 h。离心收集菌体,超声破碎取上清液,经Ni-NTA介质柱纯化,透析除盐,最终蛋白产量为100 mg/L菌液。经SDS-PAGE电泳检测,得到分子量70 Kda的目的蛋白条带,与外切葡聚糖酶的分子量相当。
结果分析:本实施例表达的融合蛋白,经SDS-PAGE电泳检测,得到分子量70 KDa的蛋白条带,外切葡聚糖酶的分子量为66-70KDa,说明本实施例表达的融合蛋白与外切葡聚糖酶的分子量相当,所以本实施例表达的融合蛋白与真菌纤维素酶关键组分外切葡聚糖酶具有类似的分子大小。
实施例3 融合蛋白对纯纤维素的识别
按照实施例1和2所述步骤获得融合蛋白GFP-GFP-CBM。并采用类似的步骤获得不含CBM的荧光蛋白GFP-GFP。利用GFP-GFP-CBM和GFP-GFP两种蛋白分别以滤纸及微晶纤维素为底物在37 ℃,200 rpm条件下吸附2 h,并在荧光显微镜下观察吸附后的滤纸纤维素(附图1)及微晶纤维素(附图2),可以发现,由于CBM对纤维素的识别作用,使GFP-GFP-CBM融合蛋白可以有效地吸附在纤维素上,而GFP-GFP融合蛋白则不具备此功能,因此在荧光显微镜下无明显的荧光现象。
结果分析:本实施例结果显示,本发明提供的融合蛋白以获得可视化分析信号,并且可以特异性地识别纤维素。
实施例4融合蛋白用于分析预处理后底物的可及度
通过用不同浓度的稀硫酸(0.4%, 1%,3%,5% ,8%)处理甘蔗渣样品,获得一系列不同半纤维素含量的预处理样品。在5%固液比,15 FPU/g固体的酶浓度条件下,对获得的样品在50℃,150 rpm条件下酶解120 h。同时,在经过1 h的过量牛血清蛋白吸附后,向样品中分别添加相同浓度的融合蛋白溶液在37 ℃,200 rpm条件下吸附2 h,将吸附的蛋白量与酶解120 h时的转化率关联可得随着稀酸浓度增加,半纤维素脱除增加,纤维素可及度增加,如附图3。结果表明融合蛋白分子可用于分析木质纤维素底物的可及度。
结果分析:本实施例结果表明,本发明提供的融合蛋白分子对预处理后木质纤维素底物中的纤维素亦具有特异性识别,可用于预处理后底物的纤维素可及度分析。
以上实施例说明本发明大肠杆菌重组质粒pET28a-GFP2-CBM的基因构建正确,表达的融合蛋白与外切葡聚糖酶的分子量相当,可以比较精确地测量出纤维素的可及度。本发明提供的融合蛋白含有荧光蛋白,可以获得可视化分析信号,具有真菌纤维素酶的吸附特性。通过本发明制备的融合蛋白分子可用于准确测量木质纤维素底物的可及度,与现有技术相比效果突出。
以上所述,仅为本发明较佳的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本领域的技术人员在本发明揭露的技术范围内,可轻易想到的变化或替换,都应涵盖在本发明的保护范围之内。因此,本发明的保护范围应该以权利要求的保护范围为准。
- 还没有人留言评论。精彩留言会获得点赞!
- 一种抗CD24人源化抗体融合蛋白的制造方法与工艺
- 特异性人EWS‑FLI1融合蛋白抗体的制备方法及在制备尤文肉瘤诊断抗体试剂的应用与制造工艺
- 一种融合蛋白表达纯化方法与制造工艺
- 抗USP2a蛋白单克隆抗体杂交瘤细胞及其产生的抗USP2a单克隆抗体和应用的制造方法与工艺
- 与癌症相关的基因融合体和基因变异体的制造方法与工艺
- 包含邻近氨基酸端的志贺毒素A亚基效应子区和细胞靶向性免疫球蛋白型结合区的蛋白的制造方法与工艺
- 杀虫融合蛋白改进的制造方法与工艺
- vin‑cdtb在大肠杆菌中的表达和制备的制造方法与工艺
- 一种抗肿瘤侵袭转移的多靶点融合蛋白的制备及用途的制造方法与工艺
- 重组人成纤维细胞生长因子21融合蛋白及其在制备治疗代谢疾病药物中的应用的制造方法与工艺