用于单细胞全基因组重亚硫酸氢盐测序的文库构建方法与流程
本发明属于基因检测领域,具体地说,涉及一种用于单细胞全基因组重亚硫酸氢盐测序的文库构建方法。
背景技术:
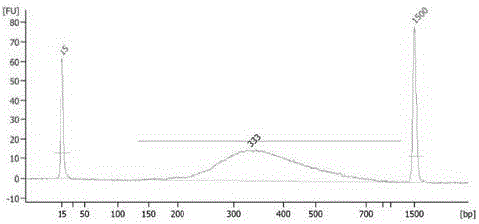
:DNA甲基化(DNAmethylation)是真核生物基因组DNA的一种重要的表观遗传学修饰,即在DNA甲基化转移酶(DNAmethyltransferase,DNMT)的作用下,将S-腺苷甲硫氨酸(SAM)的甲基基团共价结合到DNA分子的胞嘧啶上形成5-甲基胞嘧啶(5-mC)的过程。DNA甲基化在维持高等生物正常细胞功能、遗传印记、胚胎发育、衰老以及人类肿瘤的发生等生物学过程中起着重要作用。在无脊椎动物中,基因组DNA甲基化通过调控基因的表达模式,参与调节机体对环境的适应过程。因此,获得全基因组范围内所有胞嘧啶位点的甲基化数据,对于表观遗传学的时空特异性研究具有重要意义。现有的BisulfiteSequencing(重亚硫酸氢盐测序)将基因组中未发生甲基化的C碱基转换成U,经PCR扩增后变成T,与原本具有甲基化修饰的C碱基区分开来。Bisulfite处理与高通量测序技术相结合,可精确分析每一个C碱基的甲基化状态,从而来绘制单碱基分辨率的DNA甲基化图谱,用于研究特定DNA区域甲基化与特定表型之间的关联,为细胞分化、组织发育等基础机制研究,以及动植物育种、人类健康与疾病研究奠定基础。当前普遍采用的方法是从大量的组织样品中获取基因组DNA进行测序。但此传统方法依赖于一次性总体分析数百万个细胞的基因表达,往往会掩盖特异组织中某些特化细胞具有生物学意义的甲基化修饰差异。并且,在健康组织或肿瘤等病变组织中,某些细胞的数量稀少,除了单细胞方法没有任何其他的技术能够对它们进行分析。Sebastien等提出了单细胞全基因组重亚硫酸氢盐测序技术,流程包括:(1)采集单细胞;(2)将采集的单细胞进行裂解,得到裂解后的单细胞样本;(3)将所述裂解后的单细胞样本进行重亚硫酸氢盐处理,得到重亚硫酸氢盐处理产物;(4)以上述的重亚硫酸氢盐处理产物为模板,使用含生物素标记的oligo1引物合成第一条链;(5)使用链霉亲和素磁珠调取生物素标记的第一条链;(6)以上述生物素标记的第一条链为模板,使用oligo2引物合成第二条链;以及(7)以所述合成的第二条链作为模板进行PCR扩增,从而构建二代测序用DNA文库。但是,该方法做出的实验分析结果中,数据的比对率(UniqueMappedRatio)较低。参考文献[1]SébastienSmallwoodSA,LeeHJetal.2014.Single-cellgenome-widebisulfitesequencingforassessingepigeneticheterogeneity.Naturemethods,8,11(8):817-820.技术实现要素:本发明的主要目的在于提供一种单细胞全基因组重亚硫酸氢盐测序的文库构建方法,以及用于测定单细胞基因组DNA样本的甲基化位点的试剂盒。以提高数据比对率(UniqueMappedRatio)。1.一种单细胞全基因组重亚硫酸氢盐测序用文库的构建方法,该方法包括:步骤A:将单细胞进行裂解,得到裂解后的单细胞样本;步骤B:将所述裂解后的单细胞样本进行重亚硫酸氢盐处理,加入tRNA进行纯化,得到纯化后的重亚硫酸氢盐处理产物;步骤C:以步骤B所述重亚硫酸氢盐处理产物为模板,使用含生物素标记的oligo1引物合成第一条链;步骤D:使用链霉亲和素磁珠调取所述步骤C合成的生物素标记的第一条链;步骤E:以步骤D所述调取的第一条链为模板,使用oligo2引物合成第二条链,获得合成第二条链的产物;以及步骤F:以所述合成第二条链的产物为模板进行PCR扩增,从而构建二代测序用DNA文库。2.根据项1所述的文库构建方法,其中步骤B中tRNA的使用量为10ng。3.根据项1所述的文库构建方法,其中所述单细胞来源于哺乳动物、植物和/或微生物。4.根据项1所述的文库构建方法,其中步骤C所述SEQIDNO:1引物序列:5'-(Biotin)TACACTCTTTCCCTACACGACGCTCTTCCGATCTNNNNNNNNN-3'。5.根据项1所述的文库构建方法,其中在步骤D之前包括步骤E-1:消化单链DNA。6.根据项1所述的文库构建方法,其中步骤E使用所述SEQIDNO:2引物:5'-GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTNNNNNNNNN-3'。7.根据项1所述的文库构建方法,其中,在所述步骤C中对消化处理后的体系进行磁珠纯化,得到纯化的生物素标记的第一条链;和/或在所述步骤D中,对所述PCR扩增的产物进行磁珠纯化。8.根据项7所述的文库构建方法,其中磁珠纯化使用量为0.8倍以上。9.根据项7所述的文库构建方法,其中磁珠纯化使用0.9倍AmpureXP磁珠进行。10.一种用于单细胞全基因组重亚硫酸氢盐测序的文库构建的试剂盒,其包括选自下组中的至少一种或全部:用于对所述单细胞进行裂解的试剂;用于对所述单细胞裂解样本进行重亚硫酸氢盐处理试剂;用于对所述重亚硫酸氢盐处理后产物第一条链合成的试剂;用于对所述合成第一条链进行调取的试剂;用于对所述第一条链为模板合成第二条链的试剂;用于对所述第二条链为模板进行PCR扩增的试剂。有益效果:本发明能够从单细胞量起始量中,扩增得到可供后续重亚硫酸氢盐测序使用的高通量文库,结果提高了数据比对率,扩展了表观遗传学甲基化检测的使用范围,具有重要的生物学意义。附图说明图1为样本Ser1安捷伦2100检测结果图2为样本Ser2安捷伦2100检测结果发明的具体实施方式需要说明的是,在不冲突的情况下,本申请中的实施例及实施例中的特征可以相互组合。下面将参考附图并结合实施例来详细说明本发明。本发明方法包括:步骤A:采集单细胞;步骤B:将采集的单细胞进行裂解,得到裂解后的单细胞样本;步骤C:将所述裂解后的单细胞样本进行重亚硫酸氢盐处理,加入tRNA进行纯化,得到重亚硫酸氢盐处理产物;步骤D:以上述重亚硫酸氢盐处理产物为模板,使用含生物素标记的oligo1引物合成第一条链;步骤E:使用链霉亲和素磁珠调取生物素标记的第一条链;步骤F:以步骤D所述调取的第一条链为模板,使用oligo2引物合成第二条链,获得合成第二条链的产物;以及步骤G:以所述合成的第二条链作为模板进行PCR扩增,从而构建二代测序用DNA文库。在本发明所使用的单细胞可以源于哺乳动物、植物或/和微生物。根据本发明的一些实施例,哺乳动物可以为人和小鼠的至少一种,根据本发明的一个实施例,样本为一个来自于小鼠MII期的卵母细胞。本发明人经研究发现,在所述步骤C中,对单细胞样本进行重亚硫酸氢盐处理加入tRNA,可提高最后测序结果的比对率。因此为了更好地显现本发明的技术效果,优选使用5~20ng的tRNA,更优选10ng的tRNA。在本实施例中使用10ngtRNA,对于所述步骤C中采用的重亚硫酸氢盐处理体系及反应条件没有特殊限制,本领域技术人员可以适宜选择能够在重亚硫酸氢盐处理过程中使用的那些体系及反应条件。在第一条链合成反应完毕后,体系中还会有一些DNA以单链形式存在,例如未用完的含生物素标记的oligo1引物。在所述步骤E中,使用核酸外切酶消化这些单链DNA,将其降解。所述消化的反应条件可以由本领域技术人员适当选择能够在scBS方法中使用的那些消化的反应条件。然后,在所述步骤E中,使用链霉亲和素磁珠调取生物素标记的第一条链。这里,对于所述链霉亲和素磁珠的种类没有特殊限制,例如可以是M280磁珠(Invitrogen公司)。对于用于调取的条件没有特殊限制,本领域技术人员可以适当选择能够在scBS方法中使用的那些用于调取的条件。接下来,在所述步骤F中,以调取到的生物素标记的第一条链为模板,使用oligo2引物合成第二条链。对于所述步骤F中采用的第二条链的合成体系及反应条件没有特殊限制,本领域技术人员可以适宜选择能够在scBS方法中使用的那些第二条链的合成体系及反应条件。例如,可以采用如下体系:再接下来,在所述步骤G中,以所述第二条链作为模板进行PCR扩增,从而构建二代测序用DNA文库。对于所述PCR扩增的反应条件没有特殊限制,本领域技术人员可以适宜选择能够在scBS方法中使用的那些PCR扩增的反应条件。在另一方面中,本发明还提供一种用于单细胞全基因组重亚硫酸氢盐测序的文库构建的试剂盒(本发明的试剂盒),其可用于实施本发明的方法。本发明的试剂盒必须包含用于对重亚硫酸氢盐处理的试剂,该试剂包括tRNA,其使用量为5~20ng,优选10ng。用于对所述单细胞进行裂解的试剂,如无钙镁PBS缓冲液、LysisBuffer、2%SDS、蛋白酶K等;用于对所述重亚硫酸氢盐处理后产物第一条链合成的试剂,如Oligo1、10×BlueBuffer、Klenowexo-、dNTPs、核酸外切酶I、AmpureXP磁珠等;用于对所述合成第一条链进行调取的试剂,如M-280磁珠、BeadBindingBuffer、BeadWashBuffer等;用于对所述第一条链为模板合成第二条链的试剂,如dNTPs、Oligo2、10×BlueBuffer等;用于对所述第二条链为模板进行PCR扩增的试剂,如公共引物、Index、HiFiUracilMix等。除了采用所述tRNA、AmpureXP磁珠、Oligo1引物、Oligo2引物、公共引物、Index引物以外,本发明的试剂盒中所包含的试剂装置等可以与现有的scBS建库方法的试剂相同。实施例1以下通过实施例对本发明进行更具体的说明。应当理解,此处所描述的实施例是用于解释本发明,而非用于限定本发明。1.采集单细胞单细胞以0.5μL以内体积收集到0.2mL薄壁PCR管中,其中包含1μL无钙镁PBS缓冲液。采集的单细胞按照下面的形式进行命名。2.裂解细胞配制LysisBuffer10mMTris-Cl(pH7.4),2%SDS。0.2mL离心管中的单细胞样品,体积约为1μL,无需转管,直接将11.5μLLysisBuffer及0.5μL蛋白酶K加入管中,轻微震荡混匀后稍加离心。在PCR仪上37℃反应1小时,反应结束后补ddH2O至总体积20μL。3.重亚硫酸氢盐处理使用EZDNAMethylation-GoldKit对2所得样本进行Bisulfite处理。向步骤2样本所在的PCR管中加入130μLCTConversionReagent混匀,在PCR仪上进行反应,反应程序如下:98℃10分钟,64℃2.5小时,4℃保存。添加600μLM-BindingBuffer到Zymo-SpinICColumn中,并将该吸附柱放入试剂盒所提供的CollectionTube中,将带有样本的所有反应液共150μL加入吸附柱内,加入10ngtRNA混匀,12000rpm离心30秒,弃去流出液。添加200μLM-WashBuffer到柱中,并将该吸附柱放入原CollectionTube中,12000rpm离心30秒,弃去流出液。添加200μLM-DesulphonationBuffer到柱中,并将该吸附柱放入原CollectionTube中,室温20℃-30℃放置15-20分钟,12000rpm离心30秒,弃去流出液。添加200μLM-WashBuffer到柱中,并将该吸附柱放入原CollectionTube中,12000rpm离心30秒,弃去流出液。重复进行一次。添加18.5μLM-ElutionBuffer到柱中,并将该吸附柱放入1.5mL离心管中,12000rpm离心30秒,收集DNA溶液。4.一链合成4.1按照下表中前4种试剂混匀后,在PCR仪上置于65℃反应3分钟,降至4℃后,加入56UVazymeKlenowexo-,执行程序:4℃5分钟,每15秒升温1℃至37℃,37℃30分钟。一链合成所需引物oligo1序列:5'-(Biotin)TACACTCTTTCCCTACACGACGCTCTTCCGATCTNNNNNNNNN-3'4.2一链合成第2轮,将上步骤4.1中反应液立即置于95℃反应1分钟后,立即置于冰上;加入下表反应液共2.5μL,执行程序4℃5分钟,每15秒升温1℃至37℃,37℃30分钟。4.3重复步骤4.2共3次,至此总体积变为35μL(一链合成共5轮)。4.4消化单链DNA。加入2μL(40U)核酸外切酶I(NEBExonucleaseI)、4.11μLNEBExoIBuffer,37℃反应1小时。使用0.9倍AmpureXP磁珠纯化,样品溶解于30μL洗脱缓冲液EB中。5.M-280磁珠钓取5.1M-280磁珠准备:磁珠置于室温30分钟,每个样品使用20μL磁珠进行清洗。将每管20μLM-280磁珠置于磁力架上,吸弃上清,取50μLBeadBindingBuffer重悬磁珠,振荡混匀,轻微离心,清洗2次,磁珠溶于30μLBeadBindingBuffer中。5.2M-280磁珠钓取:将30μL样品与上步30μL磁珠混匀,20℃反应15分钟,恒温振荡混匀仪,每2分钟以600rpm转速震荡15秒。5.3钓取样品的清洗:将步骤5.2反应液置于磁力架上,吸弃上清后,使用200μLBeadWashBuffer重悬振荡清洗2次,200μLBufferEB重悬振荡清洗2次,最终置于磁力架上,弃上清。6.二链合成配制二链合成反应Mix:二链合成所需引物oligo2序列:5'-GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTNNNNNNNNN-3'使用二链合成反应Mix溶解步骤5.3中的磁珠,将带磁珠的反应液转入0.2mLPCR管中,置于PCR仪上95℃,反应45秒后,立即转移至冰上,加入3.7μLKlenowexo-(100U),总体积50μL。执行程序4℃5分钟;每15秒升温1℃升温至37℃,37℃90分钟。7.PCR扩增将6反应液用BufferEB重悬清洗后,弃上清,样本GT1使用公共引物及GT1-Index;样本GT2使用公共引物及GT1-Index;样本N使用公共引物及N-Index,使用下表PCR混合液50μL重悬磁珠,其中将6反应液用BufferEB重悬清洗后,弃上清,使用下表PCR混合液50μL重悬磁珠;执行程序:95℃2分钟;(94℃80秒,65℃30秒,72℃30秒)×13循环;72℃3分钟;4℃保存。使用0.9倍AmpureXP磁珠进行纯化,溶解于25μL洗脱缓冲液EB中,即为最终文库。使用引物序列如下:8.文库质控文库构建完成后,使用安捷伦2100生物分析仪对所构建的文库进行质检。如图1,2所示,所构建的文库的片段主要分布在200~500bp之间,且峰值出现在300bp左右。文库质检合格。对比例1按照Sebastien等提出的构建单细胞全基因组重亚硫酸氢盐测序文库方法进行,不同地方在于(1)在第3步重亚硫酸氢盐处理中不加入tRNA;(2)在磁珠纯化步骤中使用的磁珠规格为0.8倍AmpureXP。最终得到的文库命名为Ser1、Ser2。表1展示了GT1、GT2、Ser1、Ser2、N、这五个文库的数据分析结果。可知对于传统的单细胞中重亚硫酸氢盐测序方法,采用本发明的方法构建的文库在比对率(UniqueMappedRatio)方面的优势。表1样品的比对率统计SampleGT1GT2Ser1Ser2NCleanReads15,423,79219,375,36015,447,64115,544,73412,726,610UniqueMappedReads4,212,8146,209,5342,825,0973,980,61775,022UniqueMappedRatio(%)27.33218.325.60.6InsertSize(bp)163.19158.65——158.48ErrorRate(%)5.821.69——1.94DupRatio(%)23.099.7828.832.634.68CleanReads:过滤后的Reads数;UniqueMappedReads:唯一比对到基因组上的Reads数;UniqueMappedRatio(%):唯一比对到基因组上的Reads数占CleanReads的百分比;InsertSize(bp):插入片段的大小;ErrorRate(%):Lambda基因组上被测成C的次数/C位点总测序次数(在得到全基因组所有碱基比对信息后计算得到的),其中转化率=100(%)-ErrorRate(%)。DupRatio(%):Reads1Reads2有重复的Reads比例。还需要说明的是,在可实施且不明显违背本发明的主旨的前提下,在本说明书中作为某一技术方案的构成部分所描述的任一技术特征或技术特征的组合同样也可以适用于其它技术方案;并且,在可实施且不明显违背本发明的主旨的前提下,作为不同技术方案的构成部分所描述的技术特征之间也可以以任意方式进行组合,来构成其它技术方案。本发明也包含在上述情况下通过组合而得到的技术方案,并且这些技术方案相当于记载在本说明书中。上述说明示出并描述了本发明的优选实施例,如前所述,应当理解本发明并非局限于本文所披露的形式,不应看作是对其他实施例的排除,而可用于各种其他组合、修改和环境,并能够在本文所述发明构想范围内,通过上述教导或相关领域的技术或知识进行改动。而本领域技术人员所进行的改动和变化不脱离本发明的精神和范围,则都应在本发明所附权利要求的保护范围内。工业实用性根据本发明,能够适用于单细胞样本的重亚硫酸氢盐测序用文库的构建方法及建库试剂盒。序列表<110>安诺优达基因科技(北京)有限公司<120>用于单细胞全基因组重亚硫酸氢盐测序的文库构建方法<130>30CN<160>6<170>PatentInversion3.3<210>1<211>34<212>DNA<213>人工序列<400>Oligo1引物tacactctttccctacacgacgctcttccgatctnnnnnnnnn43<210>2<211>34<212>DNA<213>人工序列<400>Oligo2引物gtgactggagttcagacgtgtgctcttccgatctnnnnnnnnn43<210>3<211>45<212>DNA<213>人工序列<400>公共引物aatgatacggcgaccaccgagatctacactctttccctacacgac45<210>4<211>45<212>DNA<213>人工序列<400>GT1-Indexcaagcagaagacggcatacgagatgttaacctgtgactggagttc45<210>5<211>45<212>DNA<213>人工序列<400>GT2-Indexcaagcagaagacggcatacgagatgttgcaacgtgactggagttc45<210>6<211>45<212>DNA<213>人工序列<400>N-Indexcaagcagaagacggcatacgagattaattgaggtgactggagttc45当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1