htrA基因缺失的减毒沙门氏菌突变株及其构建方法以及在提高黑色素瘤靶向性中的应用与流程
本发明涉及微生物领域,具体涉及一种htrA基因缺失的减毒沙门氏菌突变株的构建与靶向小鼠黑色素瘤的应用。
背景技术:
恶性肿瘤是一类严重威胁人类生命健康的疾病,世界卫生组织报告显示2012年约有1400万新发癌症病例和820万例癌症相关死亡,并且预计今后二十年新发病例数将增加约70%。当前肿瘤治疗的主要方式包括外科手术、化疗和放射治疗,但是它们在治疗肿瘤时均存在严重不足。外科手术的适用范围较窄,若肿瘤组织超越局部或区域淋巴结时,直接切除不能很好的治愈;化疗和放射治疗靶向性低,组织内渗透性不足,并且对肿瘤细胞毒性有限,因此会对正常组织产生毒性,并且不能彻底清除所有肿瘤细胞。针对上述传统肿瘤治疗方式的缺陷和不足,迫切需要进一步研究和发展肿瘤治疗的新途径。
200多年前,一位骨外科医生——William B.Coley首先发现细菌感染能够抑制肿瘤生长。近年来,随着对细菌基因组学的深入了解和实验技术的不断进步,利用细菌介导的肿瘤治疗途径获得迅速的发展。越来越多研究发现一些专性或兼性厌氧细菌能够特异性靶向肿瘤组织,在其乏氧或坏死区中生长和繁殖,并且表现出抑制肿瘤细胞生长的作用。沙门氏菌就是这样一类肿瘤靶向性细菌。然而野生沙门氏菌是一种致病菌,为避免细菌引起的人和动物的感染,在野生沙门氏菌14028基础上对参与脂多糖合成基因(msbB)和嘌呤合成基因(purI)进行改造,获得了减毒沙门氏菌VNP20009。VNP20009能够倾向性地在肿瘤组织中生长繁殖,达到肿瘤组织中细菌滴度与正常组织中细菌滴度比例1000-10000:1。因此VNP20009不仅可以直接治疗肿瘤,并且可以作为基因治疗载体,携带更多的有效基因进行肿瘤治疗,以获得更好的肿瘤抑制效果。尽管VNP20009介导的基因治疗取得了一定的治疗效果,并且在I期临床实验中,其安全性得到了验证,但是临床实验结果显示,VNP20009不能有效地靶向肿瘤组织,抑制肿瘤生长,因此需要对VNP20009细菌治疗载体进行优化。
细菌的靶向性依赖于巨噬细胞在正常组织中对细菌的清除,以及细菌在肿瘤组织中的复制,因此肿瘤的靶向性与细菌在巨噬细胞内的存活密切相关。因此,要建立一个安全的、高肿瘤靶向性的减毒沙门氏菌,以实现细菌在肿瘤组织中高滴度,在正常组织中低滴度,细菌肿瘤靶向性提高,这是本发明要解决的问题。
技术实现要素:
为解决上述问题,本发明的目的是提供一株减毒沙门氏菌突变株及其构建方法,同时也提供其靶向到小鼠肿瘤中的应用。
本发明提供了一种减毒沙门氏菌突变株,将减毒沙门氏菌VNP20009的htrA基因敲除后获得。
进一步地,减毒沙门氏菌VNP20009的htrA基因的阅读框序列为SEQ ID NO:1。
进一步地,减毒沙门氏菌VNP20009的序列为SEQ ID NO:1的多核苷酸片段被缺失,替换成序列为SEQ ID NO:2的多核苷酸片段。
进一步地,本发明的减毒沙门氏菌突变株,其特征是与减毒沙门氏菌VNP20009相比,其形态(包括长度和宽度)、体外生长速率、在B16F10小鼠黑色素瘤细胞中的生长、及对RAW264.7巨噬细胞的侵染能力均没有显著性差异。
进一步地,本发明的减毒沙门氏菌突变株,其特征是与减毒沙门氏菌VNP20009相比,其在RAW264.7巨噬细胞内的增殖能力明显下降。
本发明提供了所述的htrA基因缺失的减毒沙门氏菌突变株的构建方法,包括下列步骤:1)以减毒沙门氏菌VNP20009为出发菌株,将质粒pKD46转化到减毒沙门氏菌VNP20009的感受态细胞,并诱导其表达λred同源重组蛋白系统;2)根据减毒沙门氏菌VNP20009基因组htrA基因的上下游DNA序列,设计基因敲除鉴定引物,其序列如SEQ ID N0:5-6所示;根据质粒PKD4分子序列,设计线性打靶片段的引物,其序列如SEQ ID N0:3-4所示;3)将线性打靶片段转化到步骤(1)所得减毒沙门氏菌VNP20009的感受态细胞,筛选具有卡纳霉素抗性的减毒沙门氏菌菌落,PCR测序鉴定,获得卡纳霉素抗性基因取代htrA基因的重组菌株;4)将步骤(3)所得重组菌株经筛选,得到消除质粒pKD46的重组菌株;5)将表达FLP重组酶的质粒pCP20电转化入步骤(4)所得的重组菌株,经筛选、培养,诱导卡纳霉素抗性基因和温敏型质粒pCP20的消除;6)对步骤(5)所得的重组菌株进行氨苄青霉素和卡纳霉素敏感性检测,筛选对氨苄青霉素和卡纳霉素均敏感的重组菌株菌落,经PCR产物电泳和测序鉴定证实基因htrA被彻底敲除,即得到减毒沙门氏菌VNP20009的htrA基因敲除的突变株△htrA。
本发明所述的减毒沙门氏菌突变株在靶向肿瘤中的应用,其特征是其在肿瘤组织中的细菌滴度与减毒沙门氏菌VNP20009相比,两者无显著性差异。
进一步地,本发明所述的减毒沙门氏菌突变株在靶向肿瘤中的应用,其特征是与减毒沙门氏菌VNP20009相比,其在脾脏中的细菌滴度明显降低。
进一步地,本发明所述的减毒沙门氏菌突变株在靶向肿瘤中的应用,其特征是与减毒沙门氏菌VNP20009相比,其在肿瘤中细菌滴度与脾脏中的细菌滴度比例提高,即靶向性提高。
进一步地,所述减毒沙门氏菌突变株为htrA基因缺失的减毒沙门氏菌突变株。
有益效果:本发明提供了一种高肿瘤靶向性的、全新的、安全的、低廉的、方便的减毒沙门氏菌突变株及其靶向到小鼠黑色素瘤中的应用。该减毒沙门氏菌与VNP20009相比,其在肿瘤组织中滴度保持不变,而在脾脏中细菌滴度明显降低,因此表现出良好的肿瘤治疗应用前景。
本发明的特点在于:
(1)选择htrA作为敲除靶点,利用Red同源重组方法敲除减毒沙门氏菌VNP20009上的htrA基因。htrA基因敲除后有利于巨噬细胞在正常组织中对沙门氏菌的清除,而不影响其在肿瘤组织内的生存和繁殖。
(2)本发明选择在减毒沙门氏菌VNP20009的遗传背景上敲除htrA基因,VNP20009是一株经过临床试验验证安全的减毒沙门氏菌菌株,我们在减毒沙门氏菌VNP20009的基础上继续敲除htrA基因,可以进一步提高菌株的安全性。
(3)由于利用方便培养的沙门氏菌作为靶向肿瘤的工具,成本低廉,具有大规模推广的应用前景。
(4)htrA基因缺失的减毒沙门氏菌突变株与VNP20009相比,其在细菌主要富集的正常器官如脾脏内,数量明显降低,而在肿瘤组织中细菌数量保持不变,因此减毒沙门氏菌突变株的肿瘤靶向性提高,显示出潜在的治疗前景。
附图说明
图1为本发明减毒沙门氏菌突变株的生物学分析
(1A)减毒沙门氏菌突变株电镜示意图;其中:1、减毒沙门氏菌VNP20009;2、减毒沙门氏菌突变株△htrA;
(1B)减毒沙门氏菌突变株体外生长曲线示意图;其中:1、减毒沙门氏菌VNP20009;2、减毒沙门氏菌突变株△htrA;
(1C)减毒沙门氏菌突变株体外侵染巨噬细胞示意图;其中:1、减毒沙门氏菌VNP20009;2、减毒沙门氏菌突变株△htrA;
(1D)减毒沙门氏菌突变株在黑色素瘤细胞存活能力分析;1、减毒沙门氏菌VNP20009;2、减毒沙门氏菌突变株△htrA;
(1E)减毒沙门氏菌突变株在巨噬细胞存活能力分析;1、减毒沙门氏菌VNP20009;2、减毒沙门氏菌突变株△htrA;
图2为本发明减毒沙门氏菌突变株在荷瘤小鼠中的生物学分布
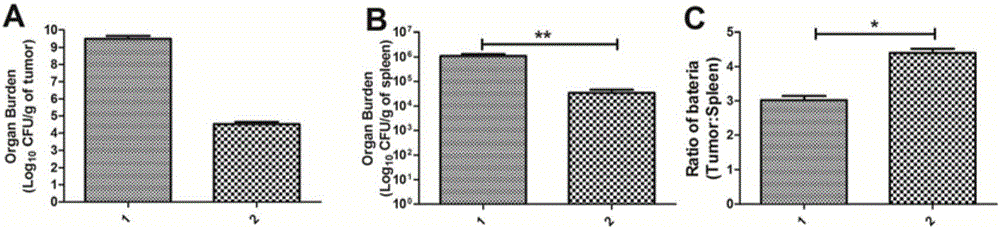
(2A)减毒沙门氏菌突变株在肿瘤中的分布;1、减毒沙门氏菌VNP20009;2、减毒沙门氏菌突变株△htrA;
(2B)减毒沙门氏菌突变株在脾脏中的分布;1、减毒沙门氏菌VNP20009;2、减毒沙门氏菌突变株△htrA;
(2C)减毒沙门氏菌突变株在荷瘤小鼠肿瘤/脾脏的细菌数量比例分析;1、减毒沙门氏菌VNP20009;2、减毒沙门氏菌突变株△htrA;
具体实施方式
以下通过实施例进一步说明本发明。应该理解的是,这些实施例是本发明的阐释和举例,并不以任何形式限制本发明的范围。
实施例1减毒沙门氏菌htrA基因敲除突变株的构建
(1)电转化法制备减毒沙门氏菌VNP20009感受态细胞
减毒沙门氏菌接种于LB培养基中,在37℃摇床上剧烈振荡培养至OD600=0.5左右;将菌液迅速置于冰上冷却10分钟,4℃下5000rpm/min冷冻离心4分钟;弃上清,加入1ml冰冷的10%甘油,用移液枪轻轻混匀,使细胞重新悬浮,4℃下5000rpm/min冷冻离心4分钟;弃上清,加入1ml冰冷的10%甘油,用移液枪轻轻混匀,使细胞重新悬浮,4℃下5000rpm/min冷冻离心4分钟;加入80μl冰冷10%的甘油,用移液器轻轻混匀,使细胞重新悬浮,立即使用或迅速置于-70℃超低温保存。
(2)将pKD46质粒电转化到减毒沙门氏菌VNP20009感受态细胞中
将20ng pKD46质粒加入到制备好的减毒沙门氏菌感受态细胞中,轻轻摇勻,冰上放置30~60s;调节电击仪,使电脉冲为25μF,电压2.5KV,电阻400Ω;将细菌与质粒DNA的混合物加至冷的电击杯内,电击;电击完成后,室温下加入1ml LB培养基,重悬细胞后,转移到1.5ml的离心管中,37℃轻柔振荡复苏1h;取200μl菌液涂布平板,37℃倒置培养16小时左右。
(3)Red重组酶的诱导表达和打靶序列转化的感受态细胞的制备
将含pKD46的减毒沙门氏菌在含有氨苄青霉素(浓度为50μg/ml)的LB培养基中于30℃振荡培养过夜;次日按1:100接种至100ml LB培养基(含氨苄青霉素和L-阿拉伯糖),30℃培养至OD600为0.4~0.6,立刻冰上预冷15~20min;然后4℃下5000rpm/min离心4min,弃培养基收集菌体;用预冷的10%甘油离心洗涤3次,浓缩100倍成1ml的感受态细胞,分装每管约100μl用于电击转化。
(4)线性基因打靶序列的转化
取约400ng打靶引物扩增的外源打靶片段与100μl感受态细胞混勻,转入预冷的0.2cm的Bio-Rad电击杯中,然后进行电击,电击条件为电阻400Ω,频率25μF,电压2.5kV;电击完成后迅速加入1ml LB培养基,30℃震荡培养1小时后,涂布卡纳霉素平板,37℃培养过夜,挑选转化子。
(5)质粒pKD46的消除:
质粒pKD46具有氨苄抗性,是温敏型质粒,在温度高时无法复制。
(6)卡纳霉素抗性基因的去除
将以上获得卡纳霉素抗性的减毒沙门氏菌突变株制成电转感受态细胞,导入编码FLP位点专一性重组酶的质粒pCP20;在含有氨苄青霉素的平板上30℃培养,筛选阳性转化子,然后转接至无抗性的液体LB培养基中,42℃培养4-6个小时,在无抗性的LB平板上37℃划线分离单菌落,对单菌落进行氨苄青霉素和卡纳霉素敏感性检测,获得对两种抗生素均敏感的克隆,即为去除了pCP20质粒和卡纳霉素抗性基因的htrA基因缺失的减毒沙门氏菌突变株。
(7)htrA基因敲除后的PCR鉴定和测序分析
实施例2减毒沙门氏菌突变株△htrA与VNP20009的生物学性状比较
通过扫描电子显微镜对△htrA和VNP20009的形态特征进行观察。通过电子显微镜图片对细菌的长度和宽度进行统计学分析(图1A),△htrA的形态特征与VNP20009相比,没有显著性差异。
以LB培养基中分别培养减毒沙门氏菌突变株△htrA与VNP20009,绘制生长曲线,比较两者在培养基中的生长曲线。如图1B,△htrA与VNP20009在体外最适培养条件下都呈指数生长状态,且两者生长曲线基本重合,这说明△htrA的体外生长速率与VNP20009相比没有差异。
在体外侵染巨噬细胞实验中,△htrA与VNP20009相比,侵染率没有显著性差异(图1C)。并且△htrA与VNP20009在小鼠黑色瘤B16F10细胞内增殖率没有显著性差异(图1D)。然而在侵染巨噬细胞5小时后,△htrA在巨噬细胞的增殖率明显低于VNP20009(图1E)。
实验1菌株的生长曲线的测定
沙门氏菌接种到LB液体培养基中在37℃细菌培养箱中复苏培养过夜,次日复苏菌株转接到新鲜LB中培养至OD为0.6-0.8;取等量的△htrA和VNP20009菌株分别转接到等量的新鲜LB中,并置于37℃细菌摇床中连续培养8h;自细菌转接到LB 2小时后,每隔一个小时将吸取细菌稀释涂布平板,最终利用所测定的数据进行沙门氏菌生长曲线的绘制。
实验2扫描电子显微镜观察菌株的形态特征
沙门氏菌接种到LB液体培养基中在37℃细菌培养箱中复苏培养过夜,次日转接到新鲜LB中培养至OD为0.6-0.8;随后吸取菌液3000rpm/min离心3min去除上清收集细菌,细菌加入500μl PBS洗涤三遍;接着细菌加入4%戊二醛充分混匀并置于4℃冰箱中过夜;次日3000rpm/min离心3min去除上清收集细菌,细菌加入500μl PBS洗涤三遍;随后细菌加入2%戊二醛充分混匀置于4℃冰箱中1h,再次3000rpm/min离心3min去除上清收集细菌,细菌加入500μl PBS洗涤三遍;然后进行乙醇梯度脱水,乙醇梯度为30%,50%,70%,80%,90%和100%,每次脱水10min。最后使用扫描电子显微镜观察细菌的形态特征。
实验3细菌侵染及在细胞内复制实验
细菌侵染前一天,将处于指数生长期的小鼠黑色素瘤细胞B16F10或巨噬细胞Raw 264.7采用0.25%的胰酶消化,接种于12孔板,细胞浓度调整为1×106个/孔,37℃5%CO2培养箱培养12h后,按照细菌/细胞比10:1进行侵染;为让细菌充分与细胞接触以起始侵染,12孔板被置于水平离心机中250g离心5min;然后将12孔板置于37℃5%CO2培养箱静置培养。1小时后,每孔加入庆大霉素(100μg/ml)以杀死细胞外细菌。2小时后,庆大霉素浓度降至10μg/ml。在细菌侵染2、5小时后,细胞用PBS清洗,并用0.1%triton X-100裂解细胞以释放细胞内细菌。释放的细菌稀释涂平板,平板在37℃培养箱中倒置培养过夜后计数。
实施例3减毒沙门氏菌突变株在荷黑色素瘤小鼠的肿瘤和脾脏内细菌滴度分析
为了确定减毒沙门氏菌△htrA与VNP20009在荷瘤小鼠中的生物学分布,当荷瘤小鼠的肿瘤体积达到150mm3时,每只小鼠腹腔注射5×105CFU的减毒沙门氏菌,在细菌感染3天后。在无菌条件下取出小鼠的肿瘤和脾脏,并分析其中的减毒沙门氏菌的数量。结果如图所示:在肿瘤组织中,减毒沙门氏菌△htrA与VNP20009菌株菌落数相近,结果没有统计学差异,这说明它们具有相近的靶向肿瘤能力(图2A)。同时△htrA在脾脏中的数量明显低于VNP20009菌株(P<0.05)(图2B)。除此之外,细菌肿瘤靶向性分析(细菌在肿瘤组织中的数量与在脾脏中的数量相比)显示:减毒沙门氏菌突变株△htrA的细菌比率明显高于VNP20009菌株(P<0.05)(图2C)。减毒沙门氏菌突变株△htrA所表现出的在正常器官中数量减少,而在肿瘤组织中大量分布的特性表明其具有潜在的治疗用途。
实验1
B16F10小鼠黑色素瘤模型构建
B16F10小鼠黑色素瘤细胞采用DMEM培养基培养至指数生长期,用0.5%胰酶消化,1000rpm/min离心3min,PBS洗涤2次后,细胞计数,用PBS重悬细胞,将终浓度调整为1×106个/ml。每只小鼠接种100μl于C57BL/6小鼠腋下,即1×105个/只。接种小鼠饲养于清洁级动物房,待肿瘤生长至体积约150mm3时进行后续实验。
实验2
减毒沙门氏菌在荷B16F10黑色素瘤小鼠中的生物学分布
△htrA和VNP20009菌株分别接种到LB中过夜复苏培养,次日转接至新鲜LB中培养至OD值为0.6-0.8,随后5000rpm/min离心4min收集细菌并用无菌PBS洗涤两遍,用PBS重悬细菌将其终浓度调整为5×106CFU/ml。取荷B16F10黑色素瘤小鼠4只,随机分成2组,并分别注射沙门氏菌重悬液100μl,即5×105CFU。注射细菌后小鼠正常进食并在3天后处死,在无菌环境下取出小鼠的部分肿瘤和脾脏,分别称重后置于2ml PBS中使用组织匀浆机进行匀浆(频率:40Hz;时间:180s)。接着梯度稀释后涂布在LB固体培养基中,并倒置于37℃细菌培养箱中培养12h,然后对培养基中的菌落数进行计数统计,最后根据组织的重量得出相应细菌在荷瘤小鼠中的生物学分布,单位为CFU/g。
综上所述,本发明以减毒沙门氏菌VNP20009为遗传背景,通过RED同源重组的方法敲除了其htrA基因,使其在荷瘤小鼠的脾脏内细菌数量下降,而在肿瘤组织中保持不变,因而提高了其肿瘤靶向性。为以细菌作为基因治疗载体,携带更多的有效基因进行肿瘤治疗打下了良好的基础。
以上显示和描述了本发明的基本原理、主要特征和本发明的优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下,本发明还会有各种变化和改进,本发明要求保护范围由所附的权利要求书、说明书及其等效物界定。
SEQUENCE LISTING
<110> 南京大学
<120> htrA基因缺失的减毒沙门氏菌突变株及其构建方法以及在提高黑色素瘤靶向
性中的应用
<130> zxx110201
<160> 6
<170> PatentIn version 3.3
<210> 1
<211> 1428
<212> DNA
<213> 人工序列
<400> 1
atgaaaaaaa ccacattagc aatgagtgca ctggctctga gtttaggttt ggcattgtcg 60
cctctgtctg ccacggcggc tgaaacgtcc tcttcagcaa tgactgccca gcagatgcca 120
agcctggcac cgatgctcga aaaagtgatg ccatcggtgg tcagtattaa tgttgaaggt 180
agcaccacgg tgaatacgcc gcgtatgccg cgtaatttcc agcagttctt tggcgatgac 240
tccccgttct gccaggacgg ttctccgttc cagaattctc cgttctgcca gggcggcggt 300
aacggcggca acggcggtca acaacagaaa ttcatggcgc tgggctccgg cgtaattatt 360
gacgccgcga agggctacgt cgtcaccaac aaccacgtgg ttgataacgc cagcgtgatt 420
aaagtacagc ttagcgatgg gcgtaaattc gatgctaaag tggtgggcaa agatccgcgt 480
tctgatatcg cgctgattca aattcagaat ccgaagaacc tgacggcgat taagctggcg 540
gactccgacg cgctgcgcgt gggggattat accgtcgcta ttggtaaccc gtttggtctg 600
ggcgaaacgg tgacgtcagg tatcgtttcg gcgctggggc gtagcggcct gaacgtagaa 660
aattacgaga actttattca gaccgacgcc gcgattaacc gtggtaactc cggcggcgcg 720
ctggtgaacc tgaacggtga gctgatcggt attaacaccg cgattctggc gccggacggc 780
ggcaacatcg gtatcggctt cgctatcccc agtaacatgg tgaaaaacct gacgtcgcag 840
atggtggaat acggccaggt gaaacgcggc gaactgggga tcatggggac tgagctgaat 900
tccgaattgg cgaaagcgat gaaagtcgac gcccagcgag gcgcgttcgt cagccaggtg 960
atgccgaatt cgtccgcggc gaaagcgggt atcaaagccg gggatgtcat tacctcgctg 1020
aacggtaaac cgatcagcag ctttgcggcg ctgcgcgctc aggtcggcac tatgccggtc 1080
ggcagcaaaa tcagcctcgg tctgctgcgt gaaggtaaag cgattacggt gaatctggaa 1140
ctgcagcaga gcagccagag tcaggttgat tccagcacca tcttcagcgg gattgaaggc 1200
gctgaaatga gcaataaagg ccaggataaa ggcgttgtgg tgagcagcgt gaaagcgaac 1260
tcacccgccg cgcaaattgg cctcaaaaaa ggcgatgtga ttatcggcgc taaccagcag 1320
ccggtgaaaa atatcgccga gctgcgtaag attctcgaca gcaagccgtc ggttctggcg 1380
ctgaatattc agcgtggtga tagttctatt tatttgctga tgcagtaa 1428
<210> 2
<211> 81
<212> DNA
<213> 人工序列
<400> 2
gtaggctgga gctgcttcga agttcctata ctttctagag aataggaact tcggaatagg 60
aactaaggag gatattcata t 81
<210> 3
<211> 60
<212> DNA
<213> 人工序列
<400> 3
acagcaattt tgcgttacct gttaatcgag attgaaacac gtgtaggctg gagctgcttc 60
<210> 4
<211> 60
<212> DNA
<213> 人工序列
<400> 4
agtgttgccc ccttccatgg cggaaggggg acaaaggtga catatgaata tcctccttag 60
<210> 5
<211> 20
<212> DNA
<213> 人工序列
<400> 5
ggcggtcgaa cagtaaatgg 20
<210> 6
<211> 20
<212> DNA
<213> 人工序列
<400> 6
ggaagatgac aggtctcgac 20
- 还没有人留言评论。精彩留言会获得点赞!