藻类培养中臂尾轮虫的快速定量检测qPCR方法与流程
本发明涉及微生物分子检测
技术领域:
,尤其是涉及在藻类大规模培养中对臂尾轮虫进行早期监测的qPCR方法。
背景技术:
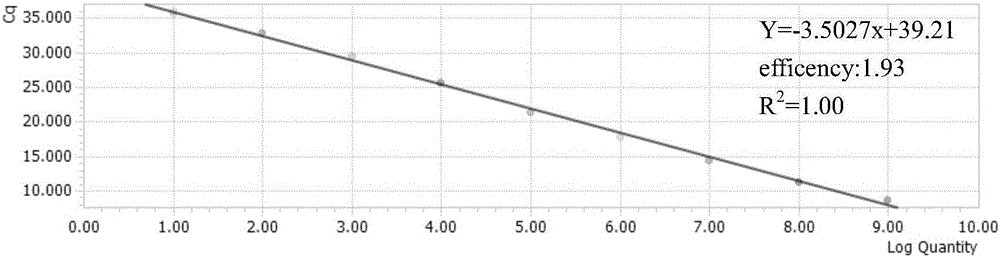
:臂尾轮虫是在藻类大规模培养中存在的一种重要污染原生动物,它们是一类广泛分布于内陆和沿岸咸水生境的世界性种类轮虫。目前已经在小球藻、栅藻以及血球藻的大规模培养中发现并爆发,一经感染,可使藻类大规模培养在2至4天内完全溃败,导致“泡汤”,从而给藻类的大规模培养带来极大危害。因此对于藻类培养中存在的臂尾轮虫的早期监测和分子鉴定方法的确定至关重要。目前对臂尾轮虫的研究尤其是褶皱臂尾轮虫的研究较为透彻,研究领域涉及到其分布、种群动态、生理、有性生殖、休眠卵的诱导、生化组分、超微结构、组织学与组织化学等诸多方面。但是对于其对大规模藻类培养造成的危害研究比较局限,在环境条件不利于生产繁殖的时候便形成休眠孢囊存在,对于在大规模藻类培养很难在显微镜中观察到它们的存在,并且传统的形态学观察灵敏度低,在原生动物存在数量很大的前提下才可以完成。而DNA直接测序及聚合酶链反应(PCR)存在检测效率低,当DNA含量很低的情况下不能被检出,检测结果出现假阳性的现象,也经常会影响对实际情况的判断。并且采用以上方法对臂尾轮虫进行检测的时候,就说明存在数量已经很大,而这种存在量已足以使藻类大规模培养发生完全溃败。因此,对臂尾轮虫的早期监测和分子鉴定的方法尤为重要。随着分子生物学检测技术的发展,在原生动物的检测手段方面已发展迅速,实时荧光定量PCR(QuantitativeReal-timePCR,qPCR)以其检测速度快,灵敏度高,特异性强的特点在原生动物的分子检测中和贝类病原检测中已得到广泛的应用。在贝类上的原生动物检测灵敏度以达到30拷贝。因此,对于臂尾轮虫的检测急需要荧光定量PCR这种灵敏度高、特异性强、检测速度快的检测手段。技术实现要素:本发明的目的是为了在藻类培养早期快速监测到污染物的存在,提供一种臂尾轮虫的荧光定量PCR检测方法。该方法较传统的PCR方法具有更高的灵敏度,且操作简便,可进行大量的样品检测,并对臂尾轮虫进行定量,从而为藻类培养早期臂尾轮虫污染的检测和监控提供有效方法。本发明的第一个技术目的是提供臂尾轮虫CoI基因在作为用于藻类培养中危害臂尾轮虫的快速定量检测qPCR方法的分子标记中的应用。由于原生动物基因序列相似度较大,如直接采用DNA直接测序及聚合酶链反应(PCR),经常出现检测效率低,检测结果出现假阳性的现象。而本发明经过反复研究后,发现采用臂尾轮虫CoI基因的特异性区域片段作为分子标记进行qPCR,可以有效的将臂尾轮虫在藻类培养过程中有其他污染原生动物存在的条件下鉴定出来。同时,本发明以CoI基因作为分子标记,还具有以下特点:(1)CoI基因作为分子标记可以使用很短的序列就可以进行区分,而其他的分子标记所涉序列比较长,比较复杂且容易误判;(2)CoI基因能够准确区分出物种差异,具有特异性;(3)CoI基因存在于线粒体,每个生物细胞内有若干个线粒体,检测时可以放大,具有很高灵敏度。本发明的第二个技术目的是提供一种DNA分子标记,包含臂尾轮虫CoI基因的三段特异性区域序列,其序列分别如SEQIDNo.1-3所示。接着提供一组引物组,包含三对引物,分别用于特异性扩增序列SEQIDNo.1-3,各自的上下游引物序列如下(如SEQIDNo.4-9所示):(1)上游引物CoI-1F:5’-CAGCAATTTTATTAATTACTAGG-3’;下游引物CoI-1R:5’-ATAATACAGGATTGCCTCCAC-3’;(2)上游引物CoI-2F:5’-CGGCTTAATTGGTCTTAGCATA-3’;下游引物CoI-2R:5’-CCTAACATAAGTGGAATAAGTCA-3’;(3)上游引物CoI-3F:5’-TCTTCCGCTATTGATGCTG-3’;下游引物CoI-3R:5’-ACGGTCTAACGAGATTCTTT-3’。上述分子标记或引物可应用在藻类培养中危害臂尾轮虫的快速定量检测qPCR方法中。进一步地,本发明提供一种藻类培养中危害臂尾轮虫的快速定量检测qPCR方法,采用上述的特异性引物组,对待测样品的基因组DNA进行荧光定量PCR检测。该方法较传统的PCR方法具有更高的灵敏度,且操作简便,可进行大量的样品检测,并对臂尾轮虫进行定量,从而为藻类培养早期臂尾轮虫污染的检测和监控提供有效方法。所述的快速定量检测qPCR方法,还包括步骤:采用所述的特异性引物组,将包含目的扩增片段的基因克隆到质粒PGEM-T中,构建的重组质粒PGEMT-CoI作为检测臂尾轮虫浓度的定量标准品。所述的快速定量检测qPCR方法,其中特异性引物:上游引物和下游引物的浓度均为10μmol/L,反应终浓度为0.25μmol/L。荧光定量PCR采用的荧光染料优选为SYBRGreen染料,荧光检测波长为470-514nm。优选的,所述荧光定量PCR的反应体系为:20μL反应体系中包含:2×的LightCycler480SYBRGreenⅠMaster10μL,10μmol/L的上游引物0.5μL,10μmol/L的下游引物0.5μL,DNA模板1μL,加水至总体积20μL。所述荧光定量PCR的反应程序为:95℃5min,1个循环;95℃10s、56℃20s、72℃30s,40个循环。所述荧光定量PCR,以结果的判断方法如下:(1)阳性对照的Ct值应小于33.0,阴性对照的Ct值应大于36.0。阳性模板为质粒定量标准品PGEMT-CoI-1、PGEMT-CoI-2和PGEMT-CoI-3,阴性模板为无菌双蒸水;(2)若Ct值小于33.0,且呈现典型的扩增曲线,则判定为阳性结果,表明检测样品中含有臂尾轮虫;(3)若Ct值大于36.0或无扩增信号,则判定为阴性结果,表明检测样品中无臂尾轮虫;(4)若Ct值在33.0~36.0之间,则视为可疑结果,样品需重复检验一次;若结果仍在此范围内,则判定为阴性结果;结果呈阳性的样品,根据其Ct值以及建立的定量标准曲线计算样品内的臂尾轮虫含量;计算公式如下:样品含臂尾轮虫的只数(cells/ml)=conc/N,其中N代表每只褶皱臂尾轮虫中CoI基因的拷贝数。本发明最主要的技术效果是找到一种分子标记并设计出特异性引物,可以有效的将臂尾轮虫在藻类培养过程中有其他污染原生动物存在的条件下鉴定出来。所采用的荧光定量PCR技术巧妙的利用了PCR技术的DNA高效扩增、特异性引物的高特异性以及光谱技术的敏感性和实时定量的优点,克服了常规PCR定性检测的一些不足,极大地提高了检测的敏感性和特异性,缩短了试验时间,简化了实验操作,而且荧光检测的结果由电脑软件分析,避免了产物后处理过程所致的污染,较常规PCR更为客观、灵敏、准确。本发明的方法也可设计为臂尾轮虫早期快速鉴定试剂盒。其具有对样本低要求,高效率,精确检测,省时省力省费用,效果显著等特点。所述试剂盒中包含所述的特异性引物组。具体的,所述试剂盒可以包括检测试剂和基因组DNA提取试剂。所述检测试剂包括荧光染料、特异性引物、阳性标准品。所述基因组DNA提取试剂优选包括Roche的HighPureTemplatePreparationKit试剂盒。本发明提供的检测方法对臂尾轮虫的检测具有特异性强、灵敏度高的特点,利用该方法确定了单只褶皱臂尾轮虫线粒体CoI基因的拷贝数为6332±109copies/只。该方法可检测出的最低浓度分别可达到95.49copies/μL、8.11copies/μL、27.71copies/μL臂尾轮虫线粒体CoI基因的存在,对藻类培养污染物的早期监测具有重要意义。对本发明方法的灵敏度、稳定性、特异性以及对环境样品的有效性进行综合评价如下:(1)灵敏度:用灭菌双蒸水分别将质粒定量标准品PGEMT-CoI-1、PGEMT-CoI-2和PGEMT-CoI-3稀释1010、109、108、107、106、105、104、103、102、101、100copies/μL,采用经优化后的体系进行检测,验证所建立的方法的灵敏度。本检测方法在分别在103~109copies/reaction、101~109copies/reaction、101~109copies/reaction数量级范围内具有良好的线性关系,相关系数分别为R1=1.00、R2=1.00、R3=0.99,其检测范围分别可达到7、9、9个数量级,其灵敏度分别可到1000、10、10拷贝以下。(2)稳定性:我们从批间重复和批内重复两个方面进行评价。批内重复性:选取同一份标本,设5个平行反应管同时进行反应,检验其Ct值的变异系数约为1.32%。批间重复性:选取2个不同浓度的重组质粒标准品,一周内重复检验3次,检验其Ct值的变异系数为1.97%和1.65%。表明本发明的检测方法具有良好的稳定性和重复性。(3)特异性:本研究选取了22份样品进行检测:3份轮虫样品,8份纤毛虫样品,3份变形虫样品,3份鞭毛虫样品以及5份藻类样品。采用PCR方法进行检测,检测结果显示除了臂尾轮虫外,其余样品均为阴性结果。附图说明图1是臂尾轮虫CoI基因绝对定量的标准曲线,y1=-3.6664×log(conc)+43.26(注:conc表示反应体系中的标准质粒拷贝数)。图2是臂尾轮虫CoI基因绝对定量的标准曲线,y2=-3.5027×log(conc)+39.21(注:conc表示反应体系中的标准质粒拷贝数)。图3是臂尾轮虫CoI基因绝对定量的标准曲线,y3=-3.5421×log(conc)+41.11(注:conc表示反应体系中的标准质粒拷贝数)。图4是第一对引物荧光定量PCR敏感度检测的荧光信号图,从左至右的标准品浓度分别为109~103copies/μL。图5是第二对引物荧光定量PCR敏感度检测的荧光信号图,从左至右的标准品浓度分别为109~101copies/μL。图6是第三对引物荧光定量PCR敏感度检测的荧光信号图,从左至右的标准品浓度分别为109~101copies/μL。图7是第一对引物特异性验证的PCR产物琼脂糖凝胶电泳图,M:100bpDNALadder;1:Branchionusplicatilis;2:Lecanesp.;3:Philodinasp.;4:Tetrahymenasp.;5:Euplotessp.;6:Gastrostylasp.;7:Sterkiellasp.;8:Parameciumsp.;9:Vorticellasp.;10:Cyclidiummarinum;11:Colpodasp.;12:Nucleariasimplex;13:Hartmannellasp.;14:Mayorellasp.;15:Poterioochromonasmalhamensis;16:Eeuglenasp.;17:Bodonidaesp.;18:Haematoculcuspluvialis;19:Chlamydomonas;20:Chlorella;21:Scenedesmus;22:Paraphysodermasedebokerensis;23:Water。图8是第二对引物特异性验证的PCR产物琼脂糖凝胶电泳图,M:100bpDNALadder;1:Branchionusplicatilis;2:Lecanesp.;3:Philodinasp.;4:Tetrahymenasp.;5:Euplotessp.;6:Gastrostylasp.;7:Sterkiellasp.;8:Parameciumsp.;9:Vorticellasp.;10:Cyclidiummarinum;11:Colpodasp.;12:Nucleariasimplex;13:Hartmannellasp.;14:Mayorellasp.;15:Poterioochromonasmalhamensis;16:Eeuglenasp.;17:Bodonidaesp.;18:Haematoculcuspluvialis;19:Chlamydomonas;20:Chlorella;21:Scenedesmus;22:Paraphysodermasedebokerensis;23:Water。图9是第三对引物特异性验证的PCR产物琼脂糖凝胶电泳图,M:100bpDNALadder;1:Branchionusplicatilis;2:Lecanesp.;3:Philodinasp.;4:Tetrahymenasp.;5:Euplotessp.;6:Gastrostylasp.;7:Sterkiellasp.;8:Parameciumsp.;9:Vorticellasp.;10:Cyclidiummarinum;11:Colpodasp.;12:Nucleariasimplex;13:Hartmannellasp.;14:Mayorellasp.;15:Poterioochromonasmalhamensis;16:Eeuglenasp.;17:Bodonidaesp.;18:Haematoculcuspluvialis;19:Chlamydomonas;20:Chlorella;21:Scenedesmus;22:Paraphysodermasedebokerensis;23:Water。图10是第一对引物1-7和10只褶皱臂尾轮虫检测的荧光信号图,从左至右分别为10和7-1只褶皱臂尾轮虫。图11是第二对引物1-7和10只褶皱臂尾轮虫检测的荧光信号图,从左至右分别为10和7-1只褶皱臂尾轮虫。图12是第三对引物1-7和10只褶皱臂尾轮虫检测的荧光信号图,从左至右分别为10和7-1只褶皱臂尾轮虫。具体实施方式以下实施例进一步说明本发明的内容,但不应理解为对本发明的限制。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本发明的范围。若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。实施例1.设计引物,步骤:分子条形码方法常被用于鉴定微生物污染,目前主要研究在所有真核生物当中都存在的一个基因:细胞色素氧化酶I(CoIcytochromecoxidaseI)基因的一部分。这部分DNA序列能够准确区分出物种差异,具有每个物种的特异性,就像物种的“身份证”编号。在藻类培养过程中,会出现不同种微生物的污染,为了明确污染物的信息,我们首次将臂尾轮虫CoI基因作为监测的优先选择。利用积累获得的褶皱臂尾轮虫和在GenBank获取的1059条萼花臂尾轮虫以及413条褶皱臂尾轮虫CoI基因序列,以及在GenBank获取的43条原生动物的CoI基因序列,通过生物信息学比对分析,选取序列特异性区域,应用PrimerPrimer5.0软件,设计三对PCR引物,目的扩增片段为分别为120bp、184bp和202bp,扩增序列分别如SEQIDNo.1-3所示:SEQIDNo.1:ATAATACAGGATTGCCTCCACCCGCAGGGTCGAAGAAGGAAGTGTTAAAATTACGATCAGTAAGAAGCATAGTAATAGCACCTGCTAGTACGGGTAGCCTAGTAATTAATAAAATTGCTG;SEQIDNo.2:GGCTTAATTGGTCTTAGCATAAGGTTTCTTATTCGTCTAGAGTTAGGGGTCGTAGGGTCCTACTTAGGTGATGAGCATCTTTACAATGTTCTAGTTACTGCCCACGCTTTCGTAATGATTTTCTTTATAGTAATGCCCGTCTCTATAGGAGGTTTTGGTAATTGACTTATTCCACTTATGTTAG;SEQIDNo.3:CGGTCTAACGAGATTCTTTTTGTAGTACGAGAGCAAATAATAGTAGTTAAGAAGTTGATTCTCCCTAAAATAGAAGAAATACCTGATAGATGTAAACTAAAAATAGCTAAGTCAACTGAAACACCTGCATGATAAGTTGAGTCGGATAAAGGAGGATAAACAGTTCAACCAGTACCAGCACCAGCATCAATAGCGGAAGAAA;引物序列分别如SEQIDNo.4-9所示:1.上游引物CoI-1F:5’-CAGCAATTTTATTAATTACTAGG-3’;下游引物CoI-1R:5’-ATAATACAGGATTGCCTCCAC-3’;2.上游引物CoI-2F:5’-CGGCTTAATTGGTCTTAGCATA-3’;下游引物CoI-2R:5’-CCTAACATAAGTGGAATAAGTCA-3’;3.上游引物CoI-3F:5’-TCTTCCGCTATTGATGCTG-3’;下游引物CoI-3R:5’-ACGGTCTAACGAGATTCTTT-3’;从GenBank获取的原生动物CoI序列的物种信息见表1:表1:从GenBank获取的原生动物CoI序列的物种信息实施例2.质粒定量标准品的构建和制备实验步骤:1.定量标准品的构建利用PCR方法克隆得到含有靶基因序列的CoI基因片段,重组到载体PGEM-T中,并进行DNA序列测定。构建的重组质粒作为定量标准品,命名为PGEMT-CoI-1、PGEMT-CoI-2和PGEMT-CoI-3。2.定量标准品的制备质粒PGEMT-CoI用Omega质粒提取试剂盒进行提取纯化,根据Nanodrop8000测定的质粒的浓度及质量以及阿伏伽德罗常数换算成拷贝数。计算公式如下:一个碱基对的平均分子量为660g/mol;质粒的总长度为载体的总长度加上插入片段的长度;N:代表阿伏伽德罗常数(6.02×1023copies/mol)。实施例3.基因组DNA的提取实验步骤:1.臂尾轮虫的收集在藻类培养体系中获得的臂尾轮虫经清洗、饥饿72小时后,在解剖镜下分离单只个体及多只个体,分装入1.5mlEP(eppendorf)管中,后在解剖镜下确认分离的个体数。后经液氮速冻5min后置于95℃温浴5min,重复3次后备用。2.基因组DNA的提取比较不同的DNA提取试剂盒提取臂尾轮虫DNA的提取效率,分别采用了DNeasyBlood&TissueKit(QIAGEN)、DNeasyPlantMiniKit(QIAGEN)以及HighPureTemplatePreparationKit(Roche)三种试剂盒、CTAB法、珠打法和发明名称为“一种单只轮虫基因组DNA的快速提取方法”的专利申请所描述的方法,结果显示HighPureTemplatePreparationKit(Roche)的提取效率最高。对收集到的臂尾轮虫采用HighPureTemplatePreparationKit(Roche)提取试剂盒提取DNA,利用Nanodrop8000对DNA的浓度及质量进行测定。选取浓度和质量最佳的DNA保存备用。实施例4.荧光定量PCR检测方法的建立和优化实验步骤:1.绝对定量标准曲线的建立1)质粒PGEMT-CoI用Omega质粒提取试剂盒进行提取纯化,根据Nanodrop8000测定的质粒的浓度及质量以及阿伏伽德罗常数换算成拷贝数。2)定量PCR反应条件优化通过对反应体系中不同浓度的定量PCR引物和不同公司的supermix以及反应程序中不同的退火温度和退火时间等的实验结果进行比较,选择反应灵敏度高、本底荧光信号低、具有典型S型扩增荧光信号曲线、反应效率接近于1的反应体系和反应条件。本发明使用的荧光定量PCR仪为Roche公司的LightCycle@96。荧光检测的程序设置在每个循环第三步结束时采集荧光信号,检测波长为470-514nm。定量PCR循环采用三步法,退火和延伸分开进行。反应体系的筛选:反应条件的筛选:条件筛选条件退火温度52℃、54℃、56℃、58℃退火时间10s、20s、30s经优化的定量PCR程序为:经优化的定量PCR反应体系(20μL反应体系)为:3)绝对定量的扩增标准曲线建立用灭菌双蒸水10倍梯度稀释定量标准品PGEMT-CoI-1、PGEMT-CoI-2和PGEMT-CoI-3。稀释后质粒浓度分别为1010、109、108、107、106、105、104、103、102、101、100copies/μL,采用经优化后的体系和条件进行反应。检测结束后利用荧光PCR自带的软件(LightCycler96SW1.1)绘制标准曲线(图1、图2、图3)。本检测方法的三对引物分别在103~109copies/reaction、101~109copies/reaction、101~109copies/reaction数量级范围内具有良好的线性关系,相关系分别为R1=1.00、R2=1.00、R3=0.99,其检测范围分别可达到7、9、9个数量级,其灵敏度分别可到1000、10、10拷贝以下(图4、图5、图6)。检测反应中单只臂尾轮虫CoI基因的拷贝数的计算公式:(a、b取于标准曲线y=ax+b)样品中臂尾轮虫浓度的计算公式:样品含臂尾轮虫的只数(cells/ml)=conc/N(注:N代表每只褶皱臂尾轮虫中CoI基因的拷贝数)2.稳定性分析从批间重复性和批内重复性两个方面进行评价建立的荧光定量PCR反应体系的稳定性。1)批内重复性检验:选取同一份标本,按照实施例2和3所述的方法对其提取核酸。采用经优化后的体系和条件设置5个平行反应管同时进行反应,利用统计学方法检验其Ct值的变异系数。批内重复性检测的Ct值的变异系数(CV)分别为1.32%。2)批间重复性检验:选取2个不同浓度的重组质粒标准品,采用实施例4所述的反应体系和条件,一周内重复检测3次,利用统计学方法检验其Ct值的变异系数。批间重复性检测的Ct值的变异系数(CV)分别为1.97%和1.65%。表明本发明建立的检测方法具有良好的稳定性和重复性。3.特异性分析1)为检验建立的荧光定量PCR方法的适用性,本研究选取了22份样品进行检测:3份轮虫样品,8份纤毛虫样品,3份变形虫样品,3份鞭毛虫样品以及5份藻类样品。采用PCR方法进行检测,检测结果显示除了臂尾轮虫外,其余样品均为阴性结果(图7、图8、图9)。4.检测结果的判定及臂尾轮虫的定量1)阳性对照的Ct值应小于33.0,阴性对照的Ct值应大于36.0。阳性模板为质粒定量标准品PGEMT-CoI-1、PGEMT-CoI-2和PGEMT-CoI-3,阴性模板为无菌双蒸水。2)若Ct值小于33.0,且呈现典型的扩增曲线,则判定为阳性结果,表明检测样品中含有臂尾轮虫。3)若Ct值大于36.0或无扩增信号,则判定为阴性结果,表明检测样品中无臂尾轮虫。4)若Ct值在33.0~36.0之间,则视为可疑结果,样品需重复检验一次。若结果仍在此范围内,则判定为阴性结果。结果呈阳性的样品,根据其Ct值以及建立的定量标准曲线计算样品内的臂尾轮虫含量。计算公式如下:样品含臂尾轮虫的只数(cells/ml)=conc/N(注:N代表每只褶皱臂尾轮虫中CoI基因的拷贝数)实施例5.线粒体CoI基因拷贝数的确定实验步骤:分别取1-7以及10只褶皱臂尾轮虫收集于1.5mlEP管内。按照实施例2和3所述的方法对其提取核酸。采用经优化后的体系和条件进行反应。检测结束后按照实施例4所述的方法结合标准曲线分别计算出数量不同的褶皱臂尾轮虫的CoI基因拷贝数,再计算单只褶皱臂尾轮虫的线粒体CoI基因拷贝数。检测结果见表2,不同引物检测的荧光信号见图10、图11、图12。计算公式如下:单只褶皱臂尾轮虫的线粒体CoI基因拷贝数(copies/cell)=conc×V/M(注:V代表提取DNA时的溶解体积与实验中所用DNA体积的比值,M代表褶皱臂尾轮虫只数)表2.数量不同的褶皱臂尾轮虫的荧光定量PCR结果利用该方法确定了单只褶皱臂尾轮虫线粒体CoI基因的拷贝数为6332±109copies/只。该方法对臂尾轮虫的检测具有特异性强、灵敏度高的特点,该方法可检测出的最低浓度分别可达到95.49copies/μL、8.11copies/μL、27.71copies/μL臂尾轮虫线粒体CoI基因的存在,对藻类培养污染物的早期监测具有重要意义。以上所述仅是本发明的优选实施方式,应当指出,对于本
技术领域:
的普通技术人员来说,在不脱离本发明技术原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。序列表<110>国家开发投资公司中国科学院水生生物研究所<120>藻类培养中臂尾轮虫的快速定量检测qPCR方法<130>P1610236<160>9<170>PatentInversion3.5<210>1<211>120<212>DNA<213>臂尾轮虫CoI基因序列片段<400>1ataatacaggattgcctccacccgcagggtcgaagaaggaagtgttaaaattacgatcag60taagaagcatagtaatagcacctgctagtacgggtagcctagtaattaataaaattgctg120<210>2<211>184<212>DNA<213>臂尾轮虫CoI基因序列片段<400>2ggcttaattggtcttagcataaggtttcttattcgtctagagttaggggtcgtagggtcc60tacttaggtgatgagcatctttacaatgttctagttactgcccacgctttcgtaatgatt120ttctttatagtaatgcccgtctctataggaggttttggtaattgacttattccacttatg180ttag184<210>3<211>202<212>DNA<213>臂尾轮虫CoI基因序列片段<400>3cggtctaacgagattctttttgtagtacgagagcaaataatagtagttaagaagttgatt60ctccctaaaatagaagaaatacctgatagatgtaaactaaaaatagctaagtcaactgaa120acacctgcatgataagttgagtcggataaaggaggataaacagttcaaccagtaccagca180ccagcatcaatagcggaagaaa202<210>4<211>23<212>DNA<213>人工序列<400>4cagcaattttattaattactagg23<210>5<211>21<212>DNA<213>人工序列<400>5ataatacaggattgcctccac21<210>6<211>22<212>DNA<213>人工序列<400>6cggcttaattggtcttagcata22<210>7<211>23<212>DNA<213>人工序列<400>7cctaacataagtggaataagtca23<210>8<211>19<212>DNA<213>人工序列<400>8tcttccgctattgatgctg19<210>9<211>20<212>DNA<213>人工序列<400>9acggtctaacgagattcttt20当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1