扩增兜兰属B类MADS‑box基因的引物的制作方法
本发明涉及生物技术领域,尤其涉及用于扩增兜兰属B类MADS-box基因,包括PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的引物。具体而言,本发明涉及上述引物以及包括所述引物的试剂盒。本发明还涉及开发上述引物的方法以及利用上述引物分离B类MADS-box基因的方法。本发明另外涉及利用上述引物扩增得到的基因序列进行遗传关系分析的应用。
背景技术:
MADS-box基因的命名源于最初发现的四个家族成员的首字母:MCM1(MiniChromosom Maintenancel,酵母微染色体维持基因)、AGAMOUS(拟南芥轮生器官确定性基因)、DEFICIENS(金鱼草轮生器官确定性基因)和SRF(Serum Response Factor,人血清应答因子)(Norman C等,“Isolation and properties of cDNA clones encoding SRF,a transcription factor that binds to the c-Fos serum response element,”Cell,1988,55:989-1003;Passmore等,“Saccharomyces cerevisiae protein involved in plasmid maintenance is necessary for mating of MAT a cells,”Journal of Biochemistry and Molecular Biology,1988,204:593-606;Sommer等,“Genetic control of flower development by homeotic genes in Antirrhinum majus,”Science,1990,250(4983):931-936;Yanofsky等,“The protein encoded by the Arabidopsis homeotic gene agamous resembles transcription factors,”Nature,1990,346(6279):35-39)。这4种基因编码的蛋白质都有一段由56-58个氨基酸组成的位于N端的高度保守区域,将编码这类蛋白因子的基因称为MADS-box基因(Riechmann等,“MADS domain proteins in plant development,”Biological Chemistry,1997,378:1079-1101)。MADS-box基因所编码的MADS-box蛋白质是一类重要的转录调控因子,其功能贯穿于整个植物发育过程,包括确定花分生组织,调节植物开花时间,调控花器官,影响果实、根、叶等器官的发育等。
根据基因结构、蛋白结构和系统进化关系的不同,MADS-box基因分为Type I和Type II两大类型(Becker等,“The major clades of MADS-box genes and their role in the development and evolution of flowering plants,”Molecular Phylogenetics and Evolution,2003,29(2003):464-489)。在陆生植物中,这两大类型分别被称为M型MADS-box基因和MIKC型MADS-box基因。目前从植物中分离得到的A类、B类、C类、D类、E类等同源异型基因,大部分均属于MIKCC型MADS-box家族,它们在植物花器官进化、形态多样性等方面起到关键作用。
兰科植物的花具有独特的结构与形态,花器官结构呈两侧对称排布。与双子叶植物典型的花器官形态不同,兰花的三枚萼片瓣化,其中一枚花瓣高度特化形成唇瓣,雄蕊和雌蕊合生成合蕊柱。故兰花的花器官结构由外向内分为3轮:第一轮由3个萼片组成;第二轮由2个侧瓣和1个唇瓣组成;第三轮由合蕊柱组成。部分兰花的萼片和侧瓣性状极为相似,合称为花被。兰花作为重要的观赏植物,这些奇异的花型特征使其成为研究单子叶植物花发育分子机制,尤其是相关MADS-box基因表达调控机制的理想材料。
在被子植物中,B类MADS-box基因分为两种类型:PI类基因和AP3类基因。这两类基因在花发育过程中共同行使B类基因的功能。现有的研究揭示了MADS-box基因在兰花成花转换及花器官发育过程中的重要作用(田敏,龚茂江,徐小雅,王彩霞,“兰科植物花发育的基因调控研究进展,”浙江农林大学学报,2011,28(3):494-499),尤其是B类MADS-box基因亚家族在兰科植物花被片的发育中发挥重要的作用。大部分兰科植物中具有6个B类MADS-box基因,它们与其他家族基因共同决定了兰科植物复杂的花器官发育。PI类基因在兰科中可以分为两个亚类:PI-1和PI-2。而AP3类基因可以分为四个亚类:AP3B1、AP3B2、AP3A1和AP3A2(Mondragon-Palomino M等,“MADS about the evolution of orchid flowers.”Trends in Plant Science,2008,13(2):51-59)。
兜兰属(Paphiopedilum Pfitz)植物是兰科植物最具特色的物种之一,具有重要的观赏价值。该属包括杏黄兜兰(P.armeniacum)、同色兜兰(P.concolor)、亨利兜兰(P.henryanum)等重要的物种。在以往的研究中从蝴蝶兰、石斛兰、文心兰等兰科植物中分离得到了B类MADS-box基因。在兜兰中,只在同色兜兰等兜兰中扩增到PI-2基因,在多个兜兰中扩增到AP3A1和AP3B1基因,在一个兜兰杂交种(P.‘Macabre’)中也克隆到AP3A2,但在所有兜兰中都没有克隆到PI-1、AP3B2。我们利用文献中公布的现有引物没有克隆到兜兰的PI-1,AP3B2基因,而且也没有克隆到亨利兜兰任何B类MADS-box基因。
因此,为了解析兜兰花瓣发育过程,有必要分离兜兰属B类MADS-box基因,从而可以对该类基因的序列结构、表达模式和功能进行分析,为兰科植物的遗传多样性分析以及为分子育种的实践提供理论指导。
技术实现要素:
为了解决上述问题,本发明提供了用于分离兜兰属B类MADS-box基因PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的引物组、包含所述引物的试剂盒、开发和筛选用于分离兜兰属B类MADS-box基因的引物的方法、利用所述引物分离兜兰属B类MADS-box基因的方法、以及由上述方法分离得到的兜兰属B类MADS-box基因。
本发明的第一方面提供了用于分离兜兰属B类MADS-box基因PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的引物组,其包括下列5’→3’引物的组合:
本发明的第二方面提供了一种试剂盒,其中所述试剂盒包括本发明如上所述的引物组。
本发明的第三方面提供了本发明所述的引物组在制备用于通过巣式PCR方法分离兜兰属B类MADS-box基因PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的试剂盒中的用途。
本发明的第四方面提供了一种通过巣式PCR方法分离兜兰属B类MADS-box基因PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的方法。
在一个实施方式中,所述方法使用本发明如上所述的引物组。
在一个优选的实施方式中,所述方法包括如下步骤:
1).提取兜兰属植物的总RNA;
2).采用3’RACE技术扩增得到目的基因完整的3’序列,包括:
2-a).利用总RNA为模板进行第一链cDNA的合成,其中初始反应体系包括:Oligo(dT)17(100pmol/μl)1μl、dNTP(10mM)1μl和mRNA 11μl;
2-b).利用步骤2-a)所得的cDNA为模板,采用巣式PCR的方法进行3'RACE第一轮PCR反应和3'RACE第二轮PCR反应,以扩增目的基因片段,其中涉及的引物如下表1-1所示:
其中,3'RACE第一轮PCR反应体系如下表1-2所示:
其中F1分别是上述AP3A1_F1、AP3A2_F1、AP3B1_F1、AP3B2_F1或PI_F1,
其中,3'RACE第二轮PCR反应体系如下表1-3所示:
其中F2分别是上述AP3A1_F2、AP3A2_F2、AP3B1_F2、AP3B2_F2或PI_F2;
3).采用5’RACE技术扩增得到目的基因完整的5’序列,包括:
3-a).使步骤1)的RNA脱磷酸、去帽、并进行连接反应:
3-b).利用步骤3-a)得到的已进行连接反应的RNA为模板,进行第一链cDNA的合成,其中初始反应体系包括:GeneRacer Oligo dT引物(50pmol/μl)1μl、dNTP Mix(10mM)1μl和DEPC H2O 1μl;
3-c).利用步骤3-b)所得的cDNA为模板,采用巣式PCR的方法进行5'RACE第一轮PCR反应和5'RACE第二轮PCR反应,以扩增目的基因片段,其中涉及的引物如下表2-1所示:
其中,5'RACE第一轮PCR反应体系如下表2-2所示:
其中R1分别是上述AP3A1_R1、AP3A2_R1、AP3B1_R1、AP3B2_R1或PI_R1,
其中,5'RACE第二轮PCR反应体系如下表2-3所示:
其中R2分别是上述AP3A1_R2、AP3A2_R2、AP3B1_R2、AP3B2_R2或PI_R2;和
4).回收上述步骤2)和3)所得的PCR扩增产物、测序后进行序列分析并将同一基因的相应5’序列和3’序列拼接,得到完整的基因序列,分别命名为PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2。
在一个优选的实施方式中,所述方法还包括将所得的完整的基因序列与已知的兰科其他的相应B类基因的序列进行序列比对的步骤,以确定所得完整的基因序列是否是目的基因序列。
本发明的第五方面提供了一种开发兜兰属B类MADS-box基因PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的引物的方法,其中所述引物选自如上所述的引物组。
在一个实施方式中,所述方法包括:
获得其他兰科植物的B类基因序列并进行比对,以得到相对保守但在兜兰近缘种中又相对特异的序列区域;
针对上述步骤得到的序列区域进行引物设计;
合成设计得到的引物并进行筛选,从而得到如上所述的引物组。
在一个优选的实施方式中,筛选引物的步骤包括:
进行3'RACE第一轮PCR反应,经1%琼脂糖凝胶电泳检测PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA,从而筛选合适的F1引物;
进行3'RACE第二轮PCR反应,经1%琼脂糖凝胶电泳检测PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA进行测序,从而筛选合适的F2引物;
进行5'RACE第一轮PCR反应,经1%琼脂糖凝胶电泳检测PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA,从而筛选合适的R1引物;
进行5'RACE第二轮PCR反应,经1%琼脂糖凝胶电泳检测PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA进行测序,从而筛选合适的R2引物;
利用每一种基因的F1和F2引物的不同组合进行两轮3'RACE PCR反应,经1%琼脂糖凝胶电泳检测最终的PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA进行测序,从而筛选合适的F1和F2引物的组合;以及
利用每一种基因的R1和R2引物的不同组合进行两轮5'RACE PCR反应,经1%琼脂糖凝胶电泳检测最终的PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA进行测序,从而筛选合适的R1和R2引物的组合。
本发明的第六方面提供了兜兰属(优选地,亨利兜兰)B类MADS-box基因PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的核苷酸序列。在一个优选的实施方式中,所述PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2基因的CDS序列分别如序列表中SEQ ID NOs:1-5所示。
本发明的第七方面还提供了兰科植物的B类MADS-box基因的多序列比对分析以及系统进化分析的结果,如附图4-6所示,从而有助于了解兰科物种间的亲缘关系,帮助解析兰花花器官决定的分子调控机理,为兰科植物分子育种的实践提供理论依据。
本发明的有益技术效果
本发明提供的引物有效、准确地分离得到了兜兰属B类MADS-box基因PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的序列,为进一步研究兰科,尤其是兜兰属植物的B类MADS-box基因及其在花器官发育过程中的功能奠定了基础。而且,通过上述引物分离得到的B类MADS-box基因序列还可进一步用于遗传多样性和系统进化分析,从而有助于了解兰科植物的遗传多样性分布以及物种间的亲缘关系,为分子育种和品种改良提供理论指导,也为利用分子遗传学的方法操纵和改变控制花性状的某些基因的表达来改良兰花花型提供理论参考,为其他观赏植物的花器官发育等相关研究提供新思路。
前述内容仅仅是示意性的并且决不旨在是限制性的。除了上述示意性方面、实施方式和特征,通过参考下述详细说明,进一步的方面、实施方式和特征将更容易被理解。
附图简述
通过参考下述附图,本发明的进一步的方面、特征将更容易被理解。本领域技术人员应该理解,这些附图仅示意性地阐述了根据本发明的一些实施方式,而不应将其视为是对本发明范围的限制。
图1示出了亨利兜兰花芽总RNA琼脂糖凝胶电泳图,其中泳道1-3所示为提取的RNA的三个重复。
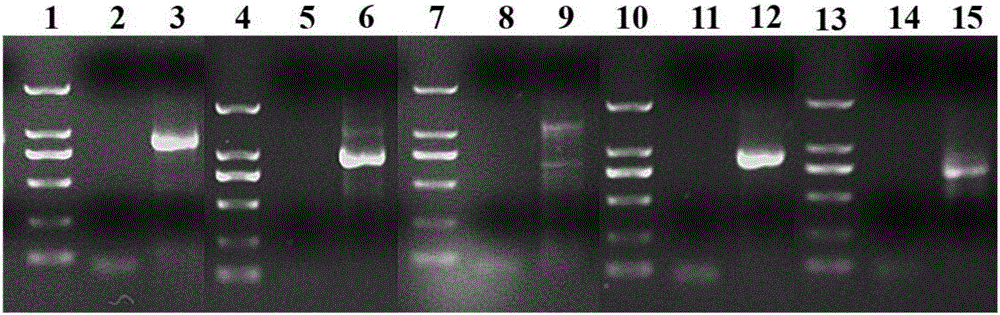
图2示出了B类MADS-box基因3'RACE第二轮PCR产物的琼脂糖凝胶电泳图,其中泳道1、4、7、10、13代表2K的Marker;2、5、8、11、14分别为不同基因的空白对照;3代表PahePI基因的条带;6代表PaheAP3A1基因的条带;9代表PaheAP3A2基因的条带;12代表PaheAP3B1基因的条带;15代表PaheAP3B2基因的条带。
图3示出了B类MADS-box基因5'RACE第二轮PCR产物的琼脂糖凝胶电泳图,其中泳道1-5代表基因PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的条带。
图4A-C示出了亨利兜兰与部分兰科植物PI类基因多序列比对结果,其中“**”表示亨利兜兰的B类MADS-box基因,基因的M、I、K、C区已用黑色箭头标出,红框区域为PI基序。
图5A-C示出了亨利兜兰与部分兰科植物AP3类基因多序列比对结果,其中“**”表示克隆到的亨利兜兰的B类MADS-box基因,黑色箭头标出基因的M、I、K、C区,红框区域为PI衍生基序和paleoAP3基序。
图6示出了基于DNA序列通过邻接法建立的部分兰科植物B类MADS-box基因的谱系进化树,其中“★”代表克隆得到的基因。
图7A示出了亨利兜兰生殖生长过程中3个发育阶段的花芽B1、B2、B3。
具体实施方式
在下文中,仅简单地描述了某些示例性实施例。正如本领域技术人员可认识到的那样,在不脱离本发明的精神或范围的情况下,可通过各种不同方式修改所描述的实施例。因此,附图和描述被认为本质上是示例性的而非限制性的。
实施例1.设计用于分离兜兰属B类MADS-box基因的引物
从Genbank中下载其他兰科植物的B类基因序列,用MEGA 6.0将序列进行比对,选择基因保守的区域但在兜兰近缘种中又相对特异的序列区域设计引物。引物的长度在18-24bp,Tm值一般控制在55-60℃,避免在引物的3’端使用碱基A;单个引物尽量不要出现发夹结构,引物之间尽量不要出现二聚体;引物的GC含量一般为40-60%,且上下游引物的GC含量不能相差太大;引物中四种碱基的分布最好是随机的,不存在聚嘌呤和聚嘧啶,尤其在引物的3’端不应超过3个连续的G或C。尽可能地设计出所有符合条件的引物。
从设计的引物当中挑选15对,进行引物序列合成(上海生工生物技术有限公司),用于后续试验筛选。
实施例2.筛选用于分离兜兰属B类MADS-box基因的引物
参考下面实施例3中所示的实验方法以及材料,执行下述实验,以筛选合适的引物组:
利用现有Oligo(dT)17引物与表1-1中不同的F1引物(AP3A1_F1、AP3A2_F1、AP3B1_F1、AP3B2_F1或PI_F1)组合,进行3'RACE第一轮PCR反应,经1%琼脂糖凝胶电泳检测PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA,从而筛选合适的F1引物;
利用Oligo(dT)17与表1-1中不同的F2引物(AP3A1_F2、AP3A2_F2、AP3B1_F2、AP3B2_F2或PI_F2)组合,进行3'RACE第二轮PCR反应,经1%琼脂糖凝胶电泳检测PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA进行测序,从而筛选合适的F2引物;
利用现有的GeneRacer 5’引物与表2-1中不同的R1引物(AP3A1_R1、AP3A2_R1、AP3B1_R1、AP3B2_R1或PI_R1)组合,进行5'RACE第一轮PCR反应,经1%琼脂糖凝胶电泳检测PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA,从而筛选合适的R1引物;
利用现有的GeneRacer 5’巣式引物与表2-1中不同的R2引物(AP3A1_R2、AP3A2_R2、AP3B1_R2、AP3B2_R2或PI_R2)组合,进行5'RACE第二轮PCR反应,经1%琼脂糖凝胶电泳检测PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA进行测序,从而筛选合适的R2引物;
利用每一种基因的F1和F2引物的不同组合进行两轮3'RACE PCR反应,经1%琼脂糖凝胶电泳检测最终的PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA进行测序,从而筛选合适的F1和F2引物的组合;以及
利用每一种基因的R1和R2引物的不同组合进行两轮5'RACE PCR反应,经1%琼脂糖凝胶电泳检测最终的PCR产物,挑选并回收电泳条带清晰、单一且长度在预期范围内的凝胶DNA进行测序,从而筛选合适的R1和R2引物的组合。
利用引物B.1作为对照,经过上述实验,筛选得到的3'RACE引物序列如表1-1所示,筛选得到的5'RACE引物序列如表2-1所示。
实施例3.分离兜兰属B类MADS-box基因
1、试验材料
(1)植物材料:亨利兜兰。
(2)克隆载体:pEASY-T1和pEASY-T3(北京全式金生物技术有限公司)。
(3)试剂:RNA提取采用RNA prep Pure多糖多酚植物总RNA提取试剂盒(离心柱型,DP441,北京天根生化科技有限公司);反转录合成第一链cDNA采用Invitrogen的Superscript III反转录酶和相关体系(18080-044),购自北京华中海威科技有限公司;5’RACE使用Invitrogen的Kit(L150202),购自北京巨华泰克科技有限公司;胶回收采用普通琼脂糖凝胶DNA回收试剂盒(DP209-02),购自北京天根生化科技有限公司;Rnase Inhibitor(2313A)、Takara Taq酶、dNTP均购自Takara公司;KAPA Hifi HotStart ReadyMix PCR Kit(KK2601)购自北京巨华泰克科技有限公司;DNA Marker(BM111-01)购自北京全式金生物技术有限公司。
2、总RNA的提取与检测
取50-100mg新鲜的亨利兜兰花芽(长约1.5cm)在液氮中迅速研磨成粉末,具体操作步骤详见说明书(http://www.tiangen.com/asset/imsupload/up0828128001368061652.pdf)。重复说明书中最后一步,可多得10%-20%的RNA。用Nanodrop 2000检测总RNA浓度,并用电泳检测RNA质量。
凝胶电泳显示,所提取的总RNA电泳条带清晰,18S和28S条带的亮度比接近1:2,Nanodrop 2000检测总RNA的浓度为151.6ng/μl,A260/A280=2.09,A260/A230=1.93:说明提取的RNA质量较好,可用于后续试验,如图1所示。
3、3’RACE
(1)第一链cDNA的合成
a.按如下体系建立初始反应体系:
Oligo(dT)17(100pmol/μl) 1μl
dNTP(10mM) 1μl
mRNA 11μl
b.70℃变性2min,取出后置于冰上,向初始反应体系中加入下列试剂:
c.42℃水浴60min,进行第一链cDNA的合成。
d.70℃变性15min,使Superscript III反转录酶失活。
e.将反转录产物于-20℃冰箱保存。
(2)cDNA序列的扩增
采用巢式PCR的方法进行3'RACE第一轮PCR反应和3'RACE第二轮PCR反应,引物序列见表1-1。将每轮PCR扩增的产物用1%的琼脂糖凝胶电泳进行检测。PCR反应体系见表1-2和表1-3。
凝胶电泳结果显示,得到了5个长度在900-1100bp的单一目的片段(如图2所示)。
4、5’RACE
(1)mRNA的去磷酸化
冰上建立初始反应体系,见表3,轻轻混合,简短离心。50℃孵育1h,简短离心,置冰上。
(2)沉淀RNA
a.向上述混合液中加入90μl DEPC水和100μl苯酚:氯仿,剧烈涡旋30s。
b.室温下,最大速度离心5min,将上层水相转移至新的离心管中。
c.加入2μl 10mg/ml的肌糖原与10μl 3M的醋酸钠混匀后,加入220μl 95%乙醇,简短涡旋。
d.置干冰上10min,4℃下最大速度离心20min,除去上清。
e.加入500μl 70%乙醇,颠倒几次,简短涡旋,4℃下离心2min。
f.除去乙醇留下沉淀,再离心,除去乙醇,室温放置1-2min晾干沉淀。
g.7μl DEPC水溶解沉淀。
表3 mRNA脱磷酸的初始反应体系
(3)去mRNA帽子结构
a.冰上建立初始反应体系,见表4,轻轻混合,简短离心。
表4去mRNA帽子结构的初始反应体系
b.37℃孵育1h,简短离心,置冰上。
c.按照步骤(2)的操作沉淀RNA,用7μl DEPC水溶解沉淀。
(4)连接反应
a.向0.25μg冻干的GeneRacer RNA Oligo中加入7μl已脱磷酸、去帽的RNA,吸打几次混合,简短离心。
b.65℃孵育5min,置于冰上2min,简短离心。
c.建立4μl DE连接反应初始反应体系,见表5,与步骤b的混合液轻轻混合,简短离心。
d.37℃孵育1h,简短离心,置冰上。
e.按照步骤(2)的操作沉淀RNA,加入10μl DEPC水溶解沉淀。
表5连接反应的初始反应体系
(5)反转录合成第一链cDNA
a.向10μl已进行连接反应的RNA中加入:1μl(50pmol/μl)GeneRacer Oligo dT引物;1μl(10mM)dNTP Mix;1μl DEPC H2O。
b.65℃孵育5min,冰上放置2min,简短离心。
c.向13μl的上述混合物中加入:4μl 5×First Strand缓冲液;1μl 0.1M DTT;1μl RNaseOut(40U/μl);1μl Superscript III RT(200U/μl)。吸打混合,简短离心,50℃孵育30-60min。
d.70℃孵育15min,冰上放置2min,简短离心。
e.加入1μl RNase H(2U),37℃孵育20min,简短离心,立即用于PCR或存于-20℃。
(6)cDNA序列的扩增
采用巢式PCR的方法进行5'RACE第一轮PCR反应和5'RACE第二轮PCR反应,引物序列见表2-1。将每轮PCR扩增的产物用1%的琼脂糖凝胶电泳进行检测。PCR反应体系见表2-2和表2-3。
凝胶电泳结果显示,得到了5个长度在760左右的单一目的片段(如图3所示)。
5、电泳及凝胶回收
PCR结束后,产物用1.5%琼脂糖凝胶分离。
6、目的片段与载体连接、转化大肠杆菌及测序
使用TransGen的pEASY-T3Cloning Kit(3’RACE黏性末端)和pEASY-Blunt Simple Cloning Kit(5’RACE平末端)连接转化体系进行目的片段与载体的连接。
将PCR扩增的产物用1%的琼脂糖凝胶电泳进行检测。选择有单一目的条带的菌液送北京六合华大基因科技股份有限公司进行测序。
将序列拼接,得到亨利兜兰中B类MADS-box基因的全长,分别命名为PahePI、PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2,长度都在1000bp左右,其CDS序列分别如SEQ ID NOs:1-5所示。
PahePI基因全长986bp,包含一段633bp的CDS序列,编码210个氨基酸。BLAST结果表明,PahePI与兜兰属植物PI基因的一致性最高。例如,PahePI与同色兜兰PcGLO基因(JN565030)的序列一致性达到97%,与兜兰杂交种P.‘Macabre’的PaphPI基因(EU444049)的序列一致性达到99%,与其他兰科植物PI基因的序列一致性也达到近90%。
PaheAP3A1基因全长962bp,包含一段675bp的CDS序列,编码224个氨基酸。BLAST分析表明,PaheAP3A1与兜兰属植物AP3类基因的一致性非常高。例如,PaheAP3A1与同色兜兰PcDEF(JN565029)、P.‘Macabre’的AP3-1(EU444046)的序列一致性均达到97%。
PaheAP3A2基因全长1034bp,包含一段678bp的CDS序列,编码225个氨基酸。BLAST分析表明,PaheAP3A2与兜兰属植物P.‘Macabre’的AP3-2(EU444047)和长叶南美兜兰DEF4(FJ804108)的序列一致性分别达到99%和94%。
PaheAP3B1基因全长948bp,包含一段687bp的CDS序列,编码228个氨基酸。BLAST分析表明,PaheAP3A1与兜兰属植物P.‘Macabre’的AP3-3(EU444048)和长叶南美兜兰DEF2(FJ804106)的序列一致性分别达到97%和90%。
PaheAP3B2基因全长1007bp,包含一段636bp的CDS序列,编码211个氨基酸,是4个AP3类基因拷贝中最短的。BLAST分析表明,与PaheAP3B2基因序列一致性最高的是长叶南美兜兰DEF1(FJ804105)基因,达到93%,而未见到其他兜兰属植物中克隆到该类拷贝。与之相似度次之的是意大利红门兰(Orchis italica)的OitaDEF2基因(AB857727),一致性达80%。
以上分析结果以及下面实施例4-5的分析结果均表明上述5个分离得到的基因属于B类MADS-box基因家族。
上述基因编码的蛋白质序列的基本性质分析结果见下表6。
表6亨利兜兰AP3类基因编码的蛋白质序列的基本性质分析
实施例4.亨利兜兰B类MADS-box基因多序列比对分析
收集了NCBI数据库中已公开的49个具有代表性的兰科植物及水稻(Oryza sativa)、玉米(Zea mays)2个重要的单子叶植物的B类基因CDS序列,进行序列比对,结果如图4-5所示。
氨基酸序列比对结果表明,从亨利兜兰中克隆得到的5个B类基因均含有高度保守的MADS盒、较为保守的K盒、中度保守的I结构域。而C末端的保守性较低。PahePI基因的C末端具有典型的PI基序;与PI基因不同,4个拷贝的AP3基因PaheAP3A1、PaheAP3A2、PaheAP3B1、PaheAP3B2的氨基酸序列C末端含有两个保守的基序,即PI衍生基序和paeloAP3基序。
实施例5.亨利兜兰B类MADS-box基因系统进化分析
根据实施例4的结果,以3个水稻B类基因和4个玉米的B类基因作为外类群,采用邻接法构建了基于DNA序列的部分兰科植物B类MADS-box基因系统进化树,如图6所示。
根据谱系进化树可知,在所选择的兰科植物中,亨利兜兰与兜兰杂交种P.(‘Macabre’已公布的4个B类MADS-box基因PaphPI、PaphAP3-1、PaphAP3-2、PaphAP3-3的遗传距离最近;与长叶美洲兜兰(P.longifolium)中的同源基因遗传距离次之;与小兰屿蝴蝶兰(P.equestris)、文心兰(O.Gower Ramsey)等兰花中的同源基因遗传距离较远;与香荚兰(V.planifolia)中同源基因的遗传距离最远。
实施例6.亨利兜兰花发育相关B类MADS-box基因的表达模式分析
分别提取亨利兜兰生殖生长过程中3个发育阶段的花芽B1、B2、B3(如图7所示);花器官合萼片、中萼片、花瓣、唇瓣、合蕊柱、子房;以及生长期的根、花茎、叶中的总RNA。
参考实施例3,利用不同样品的总RNA分别反转录合成第一链cDNA。
按如下表7所示的体系配制反应混合液,进行实时荧光定量PCR。所有反应进行三次生物学重复,每个生物学重复进行三次技术重复。
表7实时荧光定量PCR反应体系
利用标准曲线计算引物的扩增效率,保证引物的扩增效率为90%-110%,对于扩增效率不高的引物进行更换。引物序列见表8,其中ACT-F和ACT-R是现有引物。
选用肌动蛋白(β-Actin)作为内参基因,运用2-(ΔΔCt)法计算目的基因的相对表达量。
表8实时荧光定量PCR所用引物序列
凝胶电泳检测结果显示为目的片段大小的单一的条带,扩增效率均在95.2%-103.1%,表明引物的特异性很好。以试验中不同组织的cDNA模板的混合样进行梯度稀释(1×、10×、100×、1000×、10000×)制作标准曲线,Slope≈-3.3,相关系数:R2>0.99。
实验结果表明,PahePI、PaheAP3A1、PaheAP3B1基因在根、花茎、叶中几乎不表达;PaheAP3A2和PaheAP3B2在根、花茎、叶中有微弱的表达,并且相对表达量相似。PahePI在花器官各组织中均有表达,且表达量差异不大。PaheAP3A1基因在萼片中几乎不表达,花瓣中微弱表达,合蕊柱中的表达量较高,在唇瓣中高度表达,其表达量达到合蕊柱中的2倍以上。PaheAP3A2在各轮花器官中由外向内表达量有逐渐上升的趋势,在子房中的表达量最高。PaheAP3B1只在合蕊柱中有较高的表达量,在其他部位的表达量极微弱,几乎不表达。PaheAP3B2在唇瓣、子房中微弱表达,在合蕊柱较高表达,在萼片和花瓣中高度表达,其平均表达量是合蕊柱中的近3倍。
以上所述,仅为本发明的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,可轻易想到其各种变化或替换,这些都应涵盖在本发明的保护范围之内。因此,本发明的保护范围应以所述权利要求的保护范围为准。
序列表
<110> 中国农业科学院蔬菜花卉研究所
<120> 扩增兜兰属B类MADS-box基因的引物
<130>
<160> 31
<170> PatentIn 版本3.5
<210> 1
<211> 633
<212> DNA
<213> 亨利兜兰(Paphiopedium henryanum)
<220>
<223> PahePI基因的CDS序列
<400> 1
atggggcggg gaaagataga gatcaagcga atcgagaact caacgaatcg gcaagtgacc 60
ttctcgaagc ggaggaatgg aatcatgaag aaggcgaagg agatcagcgt tctatgcgat 120
gctcaggtct cgctagtcat cttctccagc ctaggaaaga tgtccgagta ctgcagccct 180
gccaccacgt tgtcgaagat gctggagaag taccagcaaa actcgggaaa gaagctgtgg 240
gacgcgaagc acgagaactt gagcgcggag atagatcgga taaagaagga aaacgataac 300
atgcagatcg agctcaggca tttgaagggg gaagatctga actctctcaa cccaaaagag 360
cttatcccaa tcgaggatgc gcttcagaat ggtttgacta gtgttcgtga taagcagatg 420
aatttcttga agatgctcaa aaagaatgaa agaatgctgg aagatgaaaa taaaaggctt 480
acgtatctgt tgcaccacca ggaactggca atggaaggta gtatgagaga actggacatc 540
agttatcacc agaaagatcg agagtatgga aaccagatgc caatggcctt tcgtgtgcag 600
cctatccagc ccaacttgca aggaaataaa tga 633
<210> 2
<211> 675
<212> DNA
<213> 亨利兜兰
<220>
<223> PaheAP3A1基因的CDS序列
<400> 2
atggggagag ggaagataga gattaagagg atagagaacc caacgagcag gcaggttacg 60
tattccaagc ggagggctgg gatcatgaag aaggcaagcg agctctccgt tctctgtgat 120
gccgagctct ccctcgtcat gttctccagc accggcaggt tctccgagta ctgcagcccc 180
tccaccgacg ccaagagtat gtatgatcga taccagcagg ccaccggcat cgacctctgg 240
agcacgcagt acgagaagat gcagaatatg ttgagccatc tgaaggaggt gaaccgcagt 300
ctgaggaggg agataagcca aaggatggga gaggatctgg atgggatgga catcaaggaa 360
ctgcgtggtc ttgagcaaaa catagatgaa gcacttaatc ttgttcgcag ccgaaaatac 420
catgtcatca gcacacaaac agacacgtac aagaaaaagt tgaagaactc tcaagaaacc 480
cacaagaacc tcatacgtga gctggaaatg gaggagcatg cagtcttcgg ttacgtggat 540
gatgatcaat gcaacaacga tggtgcactt gctctagtaa atgaggcttc gtacaatttc 600
tcattccgtg cacagccgag cgcagcaaac tttctcggaa tgggctatgg ctcgcatgat 660
ctgcgtcttg catga 675
<210> 3
<211> 678
<212> DNA
<213> 亨利兜兰
<220>
<223> PaheAP3A2基因的CDS序列
<400> 3
atggggaggg gaaagataga gataaagaag atagaaaacc ccacgaatcg gcaggttaca 60
tactcgaaga ggagggcagg gatcatgaag aaggcgaggg agctcactgt cctgtgcgac 120
gctgaggtat ctctcataat gttctcaagc accggcaagt tctccgagta ctgcagcccc 180
tccaccgatg cgaagaaggt gtttgatcgt taccagcaag tatccgggat caacttgtgg 240
agcgtgcagt acgagaagat gcagactacg ctgaaccatt tgaaggagat caaccacagc 300
ttgagaaggg agataaggca gaggatgggc gaggatctgg agggactgga catcaaggaa 360
ctgcgcggtc ttgagcaaaa catggatgag gctctgaagc ttgtgaggaa taggaaatat 420
cacgtaatca gtactcagac ggacacctac aagaagaagt tgaagaactc tcaagaaaca 480
catcgcaatt tgctgcgtga actggaaatt atagaggatc acccggtctt cgggtatgtt 540
gatgatgatc cgagcaacta cgagggtacg cttgctcttg caaatgggag ctctcacatg 600
tttgccttcc gtgttcagcc cagccagcca aaccttcacg gaatgggatt tggatctcat 660
gacctgcgtc ttgcctga 678
<210> 4
<211> 687
<212> DNA
<213> 亨利兜兰
<220>
<223> PaheAP3B1基因的CDS序列
<400> 4
atggggaggg gaaaggttga gataaagaag attgaaaatc ccacgaacag gcaagtcaca 60
tactcaaaga ggaggctcgg catcatgaag aaggccaagg agctctctgt cctctgcgac 120
gcacagctct ctctcatcat gttctccaac accggcaagt tagccgacta ctgcagcccc 180
tccactgacg tcaagaggat atttgagagg taccaggttg tcacaggcat caatctctgg 240
aatgcacaat acgagaggat gcagagaaca ctgaagcatc tcgaggaaat caaccaaagc 300
cttcgcaagg agattatgca gaggtccgga gagggattgg aggggatgaa catggaggaa 360
ctgcgcggtc ttgagcaaac attggatgac tcgctgagaa ttgttcggca gaggaagtat 420
catgtgattg ccacacaaac tgacacttac aagaaaaagc ttaaaagcac cagagaatca 480
tacaggcttc taatgcaaga actggagatg aaggatgagc attcgcatta tgattttgtc 540
gccgaagaac acctcagaat ttgcgagaat tcgatcccaa tggtgaatgg cgcccctccc 600
atgtatgcct tcaaggttgt gcagcaaatc cagccaaatc tgatgggcat gagctatgat 660
cattcacatg atctgagcct tgcttga 687
<210> 5
<211> 636
<212> DNA
<213> 亨利兜兰
<220>
<223> PaheAP3B2基因的CDS序列
<400> 5
atggggagag ggaagataga gatgaagaag atagagaata ccacaagcag gcaagtgact 60
tactcgaaga gaaggatggg tatcatgaag aaggcaaagg agctcactgt gctctgcgac 120
gccaaagtct ccctcatcat attctcaagc tccggaaagt taacagaata ctgcagcccc 180
tccaccaacg ccaagcagat atttcagagt tacagccgag tgaccggcat cgatatatgg 240
aatgccgaat acgagagaat gcaaaaaact ctgaataatc tcaaagaggt caaccaaaat 300
cttcgcatgg agataaggca gaggaggggg gagggtctgg aagttctgga catggaggaa 360
ctgcgcggtc ttgagcaaaa tttggaagag tcgattaaaa ttgttcgcca cagaaagtat 420
catgtgatct ccacgcaaat cgacacttac aagaaaaagg taaaaagctc ccaggaagca 480
tacaaagccc tgatgcatga actggttagt aagccactca tcgacgttta tgagaatctc 540
agctcagtgg ttaatgggag acgttacagt gaagtccagc caagagagct tagcctgcag 600
ggcattgatt atgagtggca tgagctgaga ttttga 636
<210> 6
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> Oligo(dT)17引物
<400> 6
ccggatcctc tagagcggcc gct 23
<210> 7
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> PI_F1引物
<400> 7
cgagaactca acgaatcg 18
<210> 8
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> PI_F2引物
<400> 8
gcaagtgacc ttctcgaag 19
<210> 9
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> AP3A1_F1引物
<400> 9
gatttaggga tacaaggatg 20
<210> 10
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> AP3A1_F1引物
<400> 10
atagagaacc caacgagcag 20
<210> 11
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> AP3A2_F1引物
<400> 11
gatagaaaac cccacgaatc 20
<210> 12
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> AP3A2_F2引物
<400> 12
atgaagaagg cgagggag 18
<210> 13
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> AP3B1_F1引物
<400> 13
tgacatactc aaagaggagg 20
<210> 14
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> AP3B1_F2引物
<400> 14
gcttggcatc atgaagaag 19
<210> 15
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> AP3B2_F1引物
<400> 15
ggagagggaa gatagagat 19
<210> 16
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> AP3B2_F2引物
<400> 16
atcatattct caagctccgg 20
<210> 17
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> GeneRacer 5’引物
<400> 17
cgactggagc acgaggacac tga 23
<210> 18
<211> 25
<212> DNA
<213> 人工序列
<220>
<223> GeneRacer 5’巣式引物
<400> 18
gacactgaca tggactgaag gagta 25
<210> 19
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> PI_R1引物
<400> 19
gcaagttggg ctggataggc tgc 23
<210> 20
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> PI_R2引物
<400> 20
accattctga agcgcatcct cgattg 26
<210> 21
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> AP3A1_R1引物
<400> 21
ccgagaaagt ttgctgcgct cgg 23
<210> 22
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> AP3A1_R2引物
<400> 22
taaccgaaga ctgcatgctc ctccat 26
<210> 23
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> AP3A2_R1引物
<400> 23
tccgtgaagg tttggctggc tgg 23
<210> 24
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> AP3A2_R2引物
<400> 24
cgaagaccgg gtgatcctct ataatt 26
<210> 25
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> AP3B1_R1引物
<400> 25
tgaaggcata catgggaggg gcg 23
<210> 26
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> AP3B1_R2引物
<400> 26
gaatgctcat ccttcatctc cagttc 26
<210> 27
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> AP3B2_R1引物
<400> 27
ctgcaggcta agctctcttg gct 23
<210> 28
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> AP3B2_R2引物
<400> 28
ggcttactaa ccagttcatg catcag 26
<210> 29
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> 对照引物B.1
<400> 29
gacctggccg tcaggcagct cg 22
<210> 30
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> ACT-F引物
<400> 30
aggctggatt tgctggtga 19
<210> 31
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> ACT-R引物
<400> 31
cagttgctta caattccgtg c 21
- 还没有人留言评论。精彩留言会获得点赞!