一种检测进行性家族性肝胆汁淤积症基因突变的试剂盒的制作方法
本实用新型涉及检测基因突变试剂盒,特别是涉及一种检测进行性家族性肝胆汁淤积症基因突变的试剂盒。
背景技术:
进行性家族性肝内胆汁淤积症是一种严重的胆汁淤积性肝病,为常染色体隐性遗传疾病。主要是由于基因突变而造成肝细胞和胆管上皮细胞上各功能蛋白的生成、修饰、调控缺陷导致肝细胞性胆汁淤积,通常在婴儿期或儿童期起病,最终发展为肝衰竭。根据其致病基因不同,PFIC主要分为3型:①PFIC1(Byler病)由ATP8B1基因突变引起,导致该基因编码的P型ATP酶-FIC1缺陷,PFIC1患者肝细胞胆汁酸过负荷,不过FIC1缺陷如何进展成胆汁淤积尚不清楚;②PFIC2源于编码胆盐排泄泵(bile salt export pump,BSEP)蛋白的基因ABCB11突变,影响了毛细胆管膜胆盐转运蛋白(BSEP)的表达,导致胆盐分泌降低,从而使得肝细胞内胆盐聚积而造成严重损伤;③PFIC3为编码多药耐药糖蛋白(MDR3)的ABCB4基因的突变,影响了毛细胆管磷脂转运器,导致磷脂输出障碍。
ATP8B1基因位于18q21-22,含有28个外显子。据文献报道,PFIC1的ATP8B1基因突变多是无义突变和缺失突变,严重影响了FIC1蛋白功能,而且许多FIC1病患者是复合杂合子,更难以明辨基因型-表型相关性。
ABCB11(BSEP)位于2q24-31,含有28个外显子。PFIC2基因型-表型相关性亦不明了。但大多数BSEP突变儿童,不论其突变类型,肝细胞毛细胆管膜均无BSEP蛋白表达;现已报道PFIC2患者BSEP的突变可达100余种。严重表型常与蛋白截断或蛋白生成衰竭的基因突变有关。其中大多为无效突变,表现为终止密码子提前出现及碱基的插入或缺失。而发生移码突变,余多为复合错义突变,这些突变均严重影响了BSEP蛋白的表达和(或)功能。
已报道与PFIC3相关的ABCB4突变达30余种,ABCB4位于7q21.1,含有28个外显子,全长74kb。已有的研究显示ABCB4基因突变热点涉及外显子6、9、12、14、18、23、26,均位于编码蛋白的主体区,并且突变的类型与胆淤的严重性相关。近1/3病例突变致截断蛋白生成,肝脏免疫染色检测不出MDR3糖蛋白。另2/3病例为错义突变,多发生在高度保守的涉及ATP结合的Walker A和B基序。这些氨基酸变化并不影响ATP酶活性与转运过程,而是造成细胞内MDR3糖蛋白组装错误,功能缺陷。
PFIC1型及PFIC2型临床表型相似,通过对ATP8B1和ABCB11基因的检测,有助于进行确诊。PFIC为常染色体隐性遗传病,患儿兄弟姐妹有25%的发病、50%的携带致病突变的可能性。对无症状兄弟姐妹进行致病基因检测不但有助于早期发现疾病、早期治疗。
技术实现要素:
本实用新型所要解决的技术问题是提供一种检测进行性家族性肝胆汁淤积症基因突变的试剂盒。本实用新型将样本保存、进行性家族性胆汁淤积症致病基因突变检测整合到一个试剂盒中,通过直接测序技术,可以直接进行DNA序列分析。直接测序技术是现有基因突变检测方法中最直接、最准确的方法,可以实现机械化自动化。因此,本实用新型携带方便、结果更加直观、准确。
本实用新型解决其技术问题所采用的技术方案是:提供一种检测进行性家族性肝胆汁淤积症基因突变的试剂盒。
一种检测进行性家族性肝胆汁淤积症基因突变的试剂盒,包括盒体1、盒盖2和设置在盒体内的第一衬垫3,第一衬垫3上设有小孔,样本保存管4、装有PCR扩增试剂的试剂管5、装有PCR产物纯化试剂的试剂管6、含DNA测序试剂的试剂管7被设置于衬垫3的小孔内;试剂盒内还放置有与第一衬垫3平行的第二衬垫10,盒盖2内侧设有条码区9。
所述小孔在衬垫3上分成两排平行排列。
所述第二衬垫10上有凹槽,凹槽内设有装有正反向引物的2块96孔板8,该引物既为PCR扩增引物,又为DNA测序引物。
所述96孔板8均用封口膜封口。
试剂盒两侧板底部相对各设有一条凹槽,凹槽内滑动插入第二衬垫10。
试剂盒两侧板底部设置有安装柱,第二衬垫10放置在安装柱上。
所述条码区9内含试剂盒中试剂保存、配制信息。
PCR扩增试剂包括:0.05U/μLTaq DNA聚合酶、400μM dNTPs、4mM MgCl2、20mM Tris-HCL(PH8.0)和50mM KCL。
所述引物试剂包括ATP8B1基因、ABCB4基因、ABCB11基因共154条引物。
有益效果
由于采用了上述的技术方案,本实用新型与现有技术相比,具有以下的优点和积极效果:本实用新型将样本保存、进行性家族性胆汁淤积症致病基因突变检测整合到一个试剂盒中,通过直接测序技术,可以直接进行DNA序列分析;直接测序技术是现有基因突变检测方法中最直接、最准确的方法,可以实现机械化自动化。因此,本实用新型携带方便、结果更加直观、准确。
本实用新型将样本保存、以及ATP8B1基因、ABCB4基因、ABCB11基因的突变筛查整合在一个试剂盒内,设计合理,方便携带、保存,使用方法简单,通过筛查可降低进行性家族性肝胆汁淤积症的发病率。
附图说明
图1是本实用新型的一种用于检测检测进行性家族性肝胆汁淤积症基因突变的试剂盒结构示意图;
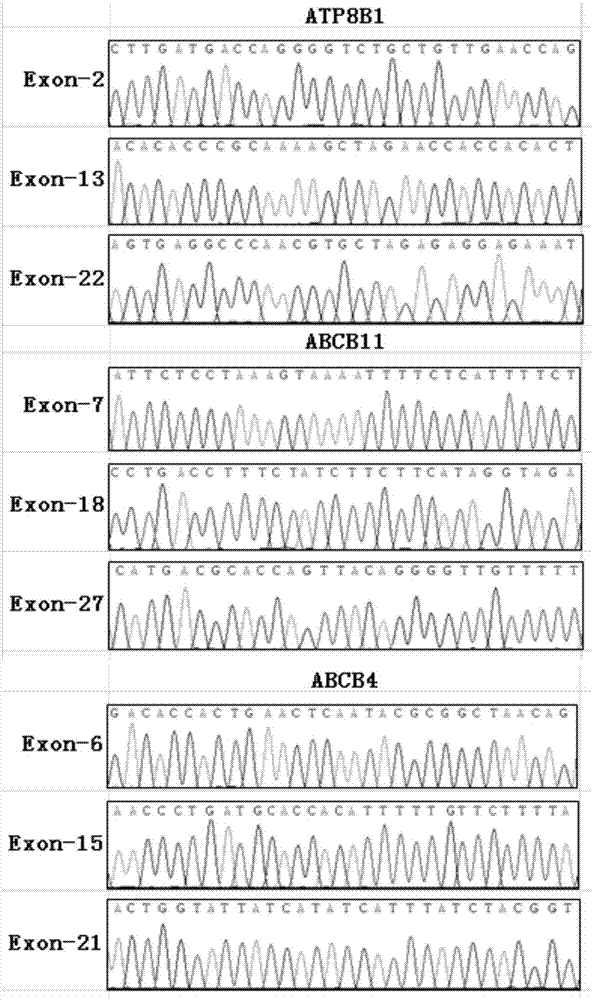
图2为ATP8B1、ABCB11、ABCB4基因外显子实验无突变的结果示意图;
图3为ATP8B1、ABCB11、ABCB4基因外显子实验有突变的结果示意图。
具体实施方式
下面结合具体实施例,进一步阐述本实用新型。应理解,这些实施例仅用于说明本实用新型而不用于限制本实用新型的范围。此外应理解,在阅读了本实用新型讲授的内容之后,本领域技术人员可以对本实用新型作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所使用的试剂、耗材等,如无特殊说明,均可从商业途径获得。
本实用新型的实施方式涉及一种检测进行性家族性肝胆汁淤积症基因突变的试剂盒。
如图1所示,为本实用新型提供的一种检测检测进行性家族性肝胆汁淤积症基因突变的试剂盒结构示意图,包括盒体1、盒盖2和设置在盒体内的衬垫3,第一衬垫3将盒体分为上下两层,第一衬垫3上设有4个小孔,小孔平行排列成两排,样本保存管4、装有PCR扩增试剂的试剂管5、装有PCR产物纯化试剂的试剂管6、含DNA测序试剂的试剂管7被设置于衬垫3小孔内;试剂盒内还放置有与第一衬垫3平行的第二衬垫10,试剂盒两侧板底部相对各设有一条凹槽,凹槽内滑动插入第二衬垫10,其中第二衬垫10上有凹槽,凹槽内设有装有正反向引物的2块96孔板8,盒盖2内侧设有条码区9,包含试剂盒中试剂保存、配制信息。
测试步骤如下:
步骤1:采集被测者外周血标本,样本保存于样本保存管4中;
步骤2:采用柱式提取法对样本中的基因组DNA进行提取;
步骤3:PCR扩增
PCR扩增试剂(总体积为1mL)成分包括:0.05U/μLTaqDNA聚合酶、400μMdNTPs、4mMMgCl2、20mMTris-HCL(PH8.0)和50mMKCL。
引物序列如下:
ATP8B1基因:
ATP8B1-1-F:CGCCGCCAACTAGACACGC;
ATP8B1-1-R:ACTCCCGAGCCCGCACAGGT;
ATP8B1-2-F:GCAAGAGTCATAATTCACTCTGGTTTCT;
ATP8B1-2-R:CGTGCCAAAGATGATCGGTCTAT;
ATP8B1-3-F:CTGAGGGATGGAGGTTTAGGAC;
ATP8B1-3-R:CCTTCCACGTAACCACCTGC;
ATP8B1-4-F:CATAAGCCACCGCACCCAGACA;
ATP8B1-4-R:TCCATTTGCTCCCTTTCCCT;
ATP8B1-5/6-F:TGATGACGGTGATGAGACTTGG;
ATP8B1-5/6-R:GTAATCCCATCTACTCGGAGGCTGA;
ATP8B1-7/8-F:CTTTAATATGTTTACATCCATTAAAGAGC;
ATP8B1-7/8-R:CAGTGGAATGAATGTGCCTT;
ATP8B1-9/10-F:TTTAATTCATTTTTTCCCCATTC;
ATP8B1-9/10-R:TCTTCTTTTGGTTTTGATGGAC;
ATP8B1-11-F:CTGTCTCCTGGGTTCAAGTGATTCT;
ATP8B1-11-R:TCCAGTTCCCGTCATCAAAGCAC;
ATP8B1-12-F:CTAGGAAATGCAAGAGGTTGGAAATC;
ATP8B1-12-R:AAATGAAGGCACTATGTTGGGAGAA;
ATP8B1-13-F:GGGATTCTAACATTCAGACCATAGC;
ATP8B1-13-R:CCAGCAATGCCAGGAGAC;
ATP8B1-14-F:TAGGGCACTGAGGGGCTGAGGA;
ATP8B1-14-R:TGGGCAACCACCAGGAGCAG;
ATP8B1-15-F:ATAATTGAAACCTTGCCTTTGAA;
ATP8B1-15-R:TGTTTCCTACTTTGCTCTTCCAT;
ATP8B1-16-F:TGGCATAACCCTTCCAAGTCAAT;
ATP8B1-16-R:CCCAGCCAACAATACCAGTTTCA;
ATP8B1-17-F:AGCCTGGACAACAGAGCAAGAC;
ATP8B1-17-R:CAGTTAACATACAGCTTTCATGCAA;
ATP8B1-18-F:AAGTGTATTTGTACAGAATCTGTCATGTT;
ATP8B1-18-R:ACCTTCTTCCATTGTGCCA;
ATP8B1-19-F:GGAGAGCAGCAACCAGGATGT;
ATP8B1-19-R:AAGGAAGAAACTGCTACTGAGGG;
ATP8B1-20-F:TGAATCCTGGAGGTTGAGGCTGT;
ATP8B1-20-R:GCTGAGAAGGAATGGAGAAAAATGG;
ATP8B1-21-F:AAGTCAAAGTTATCTCAGAGTCAAG;
ATP8B1-21-R:CTTTCATGTATAGGCTAAGAGACTT;
ATP8B1-22-F:CAGAATGAAATTCTTCTCGAGA;
ATP8B1-22-R:AAACATCTGCTCTATGAAAACCTA;
ATP8B1-23-F:TGGAACCATTTTTATCAACTGAT;
ATP8B1-23-R:GCACCAGAAACATTTAGAGAAGTC;
ATP8B1-24-F:AGCAAGACCCCCATCTCTATTAAAA;
ATP8B1-24-R:TATTACAAAACAAGAAGCACAGCCA;
ATP8B1-25-F:GCCTTCCTAAAATTTAACAGCAG;
ATP8B1-25-R:TCAGGCAAGAAATAGAAAATGTG;
ATP8B1-26-F:TTGCCTGAGTCAAGGATGGTTTA;
ATP8B1-26-R:AGCCACTGTGCCCAGCCATT;
ATP8B1-27-F:CACCACACCTGGCGAAATATTAA;
ATP8B1-27-R:ATCATTCTTACCAAACTTCCCATGAG;
ATP8B1-28-F:GGGGAGTTCATCAGTGTTCATTTC;
ATP8B1-28-R:ATAGTCCAACCCAAGGAGTTTGTTAT;
ABCB4基因:
ABCB4-2-F:CGCGAGGTTCGAGGTGAG
ABCB4-2-F:TTTGTTTTGGAGGCTTCTTT
ABCB4-3-F:TAAGGCAGGCGACCATTT;
ABCB4-3-R:CTCAAGCAACCCTCCCAC;
ABCB4-4-F:CTCCTTTTCTAAGACATTCATTT;
ABCB4-4-R:AAGATGGTAATGAATAGCAAAAT;
ABCB4-5-F:TAAAGAGAAACTTAACAATAGCATC;
ABCB4-5-R:ATAGGTGAATCTGGGTAAAGAGTAC;
ABCB4-6-F:GGTATTTAATAGAGCCTTTCTC;
ABCB4-6-R:CCTTGACATATTTTCACACAG;
ABCB4-7-F:GTGTTTGTTGGATGTCTACTTCA;
ABCB4-7-R:GTGCTGGGATACAGGAATGA;
ABCB4-8-F:GAACACTGGCATTTGCTACAT;
ABCB4-8-R:GCTTTAGAGTCTGACATTCAACTAT;
ABCB4-9-F:GGTCTCACCATGGGTTCATT;
ABCB4-9-R:TCATCTTTCAAAAAGGAGCGA;
ABCB4-10-F:TAATGAATGCCAGAATGTGAC;
ABCB4-10-R:TCAAAAATATGCAAACTAAAGCCA;
ABCB4-11/12-F:ACTTGTTTGTGCTATGATGGAAT;
ABCB4-11/12-R:AAGGGTGTGAAGGCATTATC;
ABCB4-13-F:GGATGTTTTTCATGAATGGTCC;
ABCB4-13-R:TGGACAATCTTGCATCTCAAA;
ABCB4-14-F:CTCAGTTAGGGGTTAAAGGATTA;
ABCB4-14-R:CATTTGCTATGTTTCTGTTTCTCA;
ABCB4-15-F:TTTGCCATAATCACGCAGAG;
ABCB4-15-R:TGCTCAGTATAGCATTCACTGGA;
ABCB4-16-F:TTGATTGAGAAGCAGTTAGGAAA;
ABCB4-16-R:GTATGGCTCATAGTAGCAGTCATCT;
ABCB4-17-F:CGAACAACCCATACTCAGCTTATG;
ABCB4-17-R:GAGGTTGGGAGAAGCAGCAGC;
ABCB4-18-F:ATGTGACACTCAAGCCACTATT;
ABCB4-18-R:TGAGGGCAAACTTGTAATGT;
ABCB4-19-F:GGCAACTGTAAAACAACTCATAACT;
ABCB4-19-R:CATCCTTGCGTGAATACCCT;
ABCB4-20-F:AGGCTTTGTCTGGTTTTCTTT;
ABCB4-20-R:TGGGTATGCTACATGCTTATCTA;
ABCB4-21-F:AGCCAATGTAGCTCAGGCAT;
ABCB4-21-R:AGTTGTAGTGGGCACAAACATT;
ABCB4-22-F:TAGTCTTGAACAGATTATGCCTT;
ABCB4-22-R:CTTGGAGACCTATTCTTGTTGTT;
ABCB4-23-F:AAGGAGGCTGAAGAGATGGT;
ABCB4-23-R:AGGATGGAAACTGTGGTAAAT;
ABCB4-24-F:TATGAAAATGTATGTCGGGG;
ABCB4-24-R:TTAAATGTCAGTCAAGTTGCC;
ABCB4-25-F:TTATGTTTCTCTACTACAGTCTTTG;
ABCB4-25-R:CAGTTGGGGTTTATAGAATGT;
ABCB4-26-F:GCTTAATCACCATAAAAATCATCA;
ABCB4-26-R:AGGTAGGCATAATGTATTCCCA;
ABCB4-27-F:TACCATTGAGAATTAAAGTCACTAG;
ABCB4-27-R:TTGAGGCAAGAGAATCGCT;
ABCB4-28-F:ATACAATTTTTGGGATAAGGTGTC;
ABCB4-28-R:TGATGACAAACCAGAAACTTATT;
ABCB11基因:
ABCB11-2-F:CTCTTTCAGGGAGTTATTAACCGAT;
ABCB11-2-R:CAGCAACAGTAGGTAGAAGTATCAT;
ABCB11-3-F:GAGAGACAATATGAGCAGGAAGA;
ABCB11-3-R:TCAAAGGCACAAAGCAGAGAATACT;
ABCB11-4-F:AAATCACCACCTAGGGAGAAT;
ABCB11-4-R:GAAAGTCATCACAACAATCAC;
ABCB11-5F:ATTCCCATGAAATTTGGTGTTGAGTACTA;
ABCB11-5-R:AGCTCAGCCAGTAAAATCCCCTCTATA;
ABCB11-6-F:GTAATCTCTGGTGGCTTGATCCTA;
ABCB11-6-R:TTGTATCCTCCTAAGTTTCCATCTG;
ABCB11-7-F:AATTACTTTCCCCCTTTTCTCAA;
ABCB11-7-R:AACATAGGAAAACTGAAAATATTTTTAAAT;
ABCB11-8-F:TAGGGATAGAGAGATGGGAATGT;
ABCB11-8-R:GGGAGTAATCTAACAAACTACATGA;
ABCB11-9-F:CAGACTGACTTACCTAATTTCTTGGACT;
ABCB11-9-R:AAACATACTGCTAAAGGCTTGGGACT;
ABCB11-10-F:TCCCTGAAGCTGCTCTGTGTTTG;
ABCB11-10-R:GAAGGAAATGCTATGTCTCGGTC;
ABCB11-11-F:GCTACCTTGCTTAGACTTCTTACT;
ABCB11-11-R:TTCTTCAGGAGTTCATTCTGTGCC;
ABCB11-12-F:CTAAGAGGCACAATAAATGTATACATCTTCA;
ABCB11-12-R:GAAACAGAGTCAGGCTTCAGAAAAT;
ABCB11-13-F:CTCATCCTTGCCAATGTTTCCT;
ABCB11-13-R:AAGCGTGTCCCATCAATTCAGTA;
ABCB11-14-F:AGAATCTTATTGGCCTCTATTTTTTCTGC;
ABCB11-14-R:TTGGGAATCATACGAGAAGAAATGTGTA;
ABCB11-15-F:GACAGAAAGGACATTATAGTGG;
ABCB11-15-R:AATCAGACTAGATGCATGAACCC;
ABCB11-16-F:GATGCAAAGGTCAGTGTCAGCT;
ABCB11-16-R:TGCCAGAGTTGTTGGGAGAA;
ABCB11-17-F:GGTTTAAATGACTCAGTCTTGC;
ABCB11-17-R:TGAGGATTAGGACTACAGAGGA;
ABCB11-18-F:TACCATGTGACCTACCAAACATTTCTA;
ABCB11-18-R:CCTAGAAAACTCATATTCTCAGGCTTAA;
ABCB11-19-F:TGTGAATGCCAAAGGATCTGC;
ABCB11-19-R:TTGCCTTCTTACCCTCTGTGTG;
ABCB11-20/21-F:CCACAGCTTACATTAGGGTTCACTC;
ABCB11-20/21-R:GAATGCTCTAATGAAAGAATGCC;
ABCB11-22-F:ATATTTTATCACAACTTTATAAAAGGTCTGA;
ABCB11-22-R:GCTTCCTTCAGTCTCTTCGTACTACT;
ABCB11-23-F:GCAGCCACTGAAATGTCACGAA;
ABCB11-23-R:ACCAGGCTATTCCTTCCTTGTGT;
ABCB11-24-F:ATATTTGGTCCTTTCCTGGCAGAAC;
ABCB11-24-R:TACCCCACACCATCCCCTGA;
ABCB11-25-F:TTTGGCAGCATGGTTTGAAGG;
ABCB11-25-R:GAGTCTGGCAAAGCAAAAACTTGAAG;
ABCB11-26-F:ATGGATGCCACTCCTGATAGACA;
ABCB11-26-R:GGATTTTCAGATTAGGGATGCTC;
ABCB11-27-F:TCACTCACTGTTCCCTAGTTCAA;
ABCB11-27-R:GCTCTAGATAATTGTCTTTTGG;
ABCB11-28-F:CATCAACTTTCCATCTTCTCTTTGC;
ABCB11-28-R:TTGGGTTTTCCCTCATATGGAC。
在ABI Veriti型PCR扩增仪上进行,分为154个反应孔,每孔分别加入1对引物,每对正反引物各1μL(引物浓度为5μmol/L),每孔加入检测样本2μL,PCR反应液10μL,去离子水6μL,反应总体积20μL。
反应条件为:95℃预变性5min;95℃30秒、62℃30秒、72℃40秒,15个循环;95℃30秒,55℃30秒,72℃40秒,25个循环;最后72℃10分钟后,降温至4℃。
步骤4:PCR产物纯化
每个反应孔反应体系总体积为11μL,包括:PCR产物纯化试剂2μL和PCR产物9μL组成。反应条件为:37℃50分钟,95℃5分钟。
步骤5:DNA测序反应
每个反应孔反应体系总体积为5μL,包含:PCR纯化产物1μL、DNA测序试剂1μL、测序引物1μL和去离子水2μL组成。反应条件为:95℃预变性4分钟;95℃15秒、56℃20秒,25个循环、60℃2分钟后,降温至4℃。
反应结束后,每管加入2μL 125mM乙二胺四乙酸二钠EDTA-2Na和无水乙醇15μL,涡旋混匀后3700转/分,4℃离心30分钟,弃上清;每孔加入50μL 75%乙醇,3700转/分,4℃离心15分钟,弃上清;室温放置15分钟后加入HIDI溶液5μL。置于PCR扩增仪上,95℃预变性5分钟,至ABI3500测序仪上机测序。
步骤6:结果分析
在测序仪上进行结果读取,用软件Chromas查看测序检测结果。
1.未检出突变:未发现ATP8B1、ABCB11、ABCB4基因外显子上存在突变;
2.检出突变:发现ATP8B1、ABCB11、ABCB4基因外显子上存在突变。
在测序仪上进行结果读取,用软件Chromas查阅测序检测结果。若基因无突变,则分析结果如图2所示;若基因存在突变,则分析结果如图3所示。
- 还没有人留言评论。精彩留言会获得点赞!