一种水稻胚乳粉质相关基因OsmtSSB及其编码蛋白质和应用的制作方法
本发明属于基因工程领域,具体涉及一种水稻胚乳粉质相关基因osmtssb及其编码蛋白质和应用。
背景技术:
随着生活水平的提高,人们对稻米品质也提出了越来越高的要求,而改善品质也是提高我国稻米国际竞争力的必要手段。但是目前在我国水稻生产中,稻米品质不高,因此提高品质是当前一项十分重要的工作。
水稻种子在成熟过程中积累大量的淀粉,为种子萌发和幼苗生长发育提供主要的能量,同时这些数量可观的淀粉也成为人类重要的食物来源。水稻种子中淀粉积累的多少往往决定了种子的大小和重量,因此和水稻的产量直接相关,同时种子中直链淀粉和支链淀粉的比例直接影响着稻米的食味品质,因此研究稻米中淀粉的合成过程具有重要的应用价值,开展其发育和遗传控制机理的深入研究,对改良稻米品质和提高产量有重要的意义。
尽管许多参与淀粉合成过程的酶和调控因子已经被鉴定出来,但是人类尚不能利用体外系统合成淀粉,这说明植物中淀粉的合成是一个复杂而精细的过程,新的参与淀粉合成的关键因子仍然需要进一步鉴定。
水稻淀粉突变体包括糯性(waxy,wx)、糖质(sugary,su)、粉质(floury,flo)、皱缩(shrunken,sh)、暗色(dull,du)、心白(white-core,wc)等类型。在水稻中,筛选胚乳异常突变体(粉质胚乳)是寻找新的淀粉合成关键因子的重要方法。随着分子生物学和分子遗传学方法与技术的快速发展,多位研究者对水稻粉质胚乳性状进行了基因定位研究。迄今为止,已经有一些水稻粉质基因被精细定位和克隆,但是水稻淀粉合成的调控机制还不清楚,这就需要我们定位和克隆更多的淀粉合成基因来进一步揭示水稻淀粉合成的机制。
技术实现要素:
本发明的第一个目的还提供一种水稻胚乳淀粉合成相关基因osmtssb编码的蛋白序列,其氨基酸序列如seqidno.3所示,含有206氨基酸。
本发明的另一目的是提供一种水稻胚乳淀粉合成相关基因osmtssb的核苷酸序列。
所述基因osmtssb优选为如下1)或2)或3)所述的dna分子:
1)seqidno.1所示的dna分子;
2)seqidno.2所示的dna分子;
3)在严格条件下与1)或2)限定的dna序列杂交且编码所述蛋白的dna分子;
4)与1)或2)或3)限定的dna序列具有90%以上同源性,且编码植物淀粉合成相关蛋白的dna分子。
含有以上任一所述基因的重组表达载体也属于本发明的保护范围。
可用现有的植物表达载体构建含有所述基因的重组表达载体。
所述植物表达载体包括双元农杆菌载体和可用于植物微弹轰击的载体等。所述植物表达载体还可包含外源基因的3’端非翻译区域,即包含聚腺苷酸信号和任何其它参与mrna加工或基因表达的dna片段。所述聚腺苷酸信号可引导聚腺苷酸加入到mrna前体的3’端,如农杆菌冠瘿瘤诱导(ti)质粒基因(如胭脂合成酶nos基因)、植物基因(如大豆贮存蛋白基因)3’端转录的非翻译区均具有类似功能。
使用所述基因构建重组植物表达载体时,在其转录起始核苷酸前可加上任何一种增强型启动子或组成型启动子,如花椰菜花叶病毒(camv)35s启动子、玉米的泛素启动子(ubiquitin),它们可单独使用或与其它的植物启动子结合使用;此外,使用本发明的基因构建植物表达载体时,还可使用增强子,包括翻译增强子或转录增强子,这些增强子区域可以是atg起始密码子或邻接区域起始密码子等,但必需与编码序列的阅读框相同,以保证整个序列的正确翻译。所述翻译控制信号和起始密码子的来源是广泛的,可以是天然的,也可以是合成的。翻译起始区域可以来自转录起始区域或结构基因。
为了便于对转基因植物细胞或植物进行鉴定及筛选,可对所用植物表达载体进行加工,如加入可在植物中表达的编码可产生颜色变化的酶或发光化合物的报告基因(gus基因、萤光素酶luc基因等)、具有抗性的抗生素标记物(庆大霉素标记物、卡那霉素标记物等)或是抗化学试剂标记基因(如抗除莠剂基因)等。从转基因植物的安全性考虑,可不加任何选择性标记基因,直接以逆境筛选转化植株。
所述重组过表达载体可为在用限制性内切酶kpni和bamhi双酶切载体pcambia1390的重组位点插入所述基因(osmtssb)得到的重组质粒。将含有osmtssb的pcambia1390命名为pcambia1390-osmtssb。
含有以上任一所述基因(osmtssb)的表达盒及重组菌均属于本发明的保护范围。
扩增所述基因(osmtssb)全长或任一片段的引物对也属于本发明的保护范围,所述的引物对优选primer1/primer2、primer3/primer4;其中primer1的核苷酸序列如seqidno.4所示,primer2的核苷酸序列如seqidno.5所示,primer3的核苷酸序列如seqidno.6所示,primer4的核苷酸序列如seqidno.7所示。
在精细定位此基因过程中涉及到的定位引物(见表1),indel引物为本实验需要而自行设计的引物,这些自行设计的引物也属于本发明的保护范围。
有益效果
本发明首次发现、定位并克隆得到一个新的植物胚乳粉质相关蛋白的基因osmtssb。本发明的植物胚乳粉质相关蛋白影响植物的淀粉合成过程。抑制该蛋白编码基因的表达可导致植物种子中淀粉合成的障碍,从而可以培育胚乳变异的转基因植物和植物淀粉含量降低的转基因植物。将所述蛋白的编码基因导入淀粉含量降低的植物中,可以培育淀粉含量正常的植物。所述蛋白及其编码基因可以应用于植物遗传改良。
附图说明
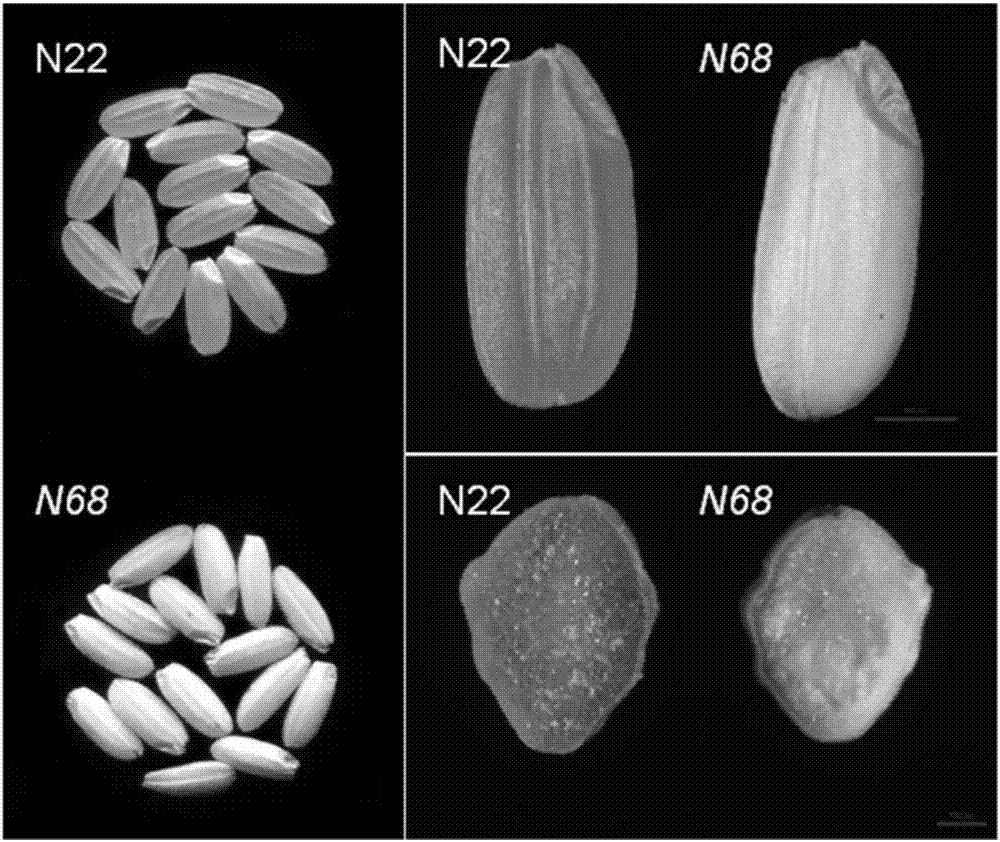
图1为野生型n22与突变体n68的淀粉含量测定。
图2为野生型n22与突变体n68的籽粒外型。
图3为野生型n22与突变体n68籽粒扫描电镜观察。
图4为突变基因在第5染色体上的精细定位。
图5为转pcambia1390-osmtssb的t0代植株的t1籽粒表型。
图6为转pcambia1390-osmtssb的t0代植株的t1籽粒扫描电镜观察。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。
实施例1、水稻淀粉合成相关位点及其编码基因的发现
一、水稻胚乳粉质突变体n68的淀粉含量分布分析及遗传分析
在利用mnu化学诱变产生的n22突变体中,筛选出一个胚乳粉质突变体,命名为n68。与n22比较,n68的主要特征是:种子中淀粉含量下降(图1),同时具有种子不透明的表型(图2)。图2显示n22具有半透明的胚乳,n68胚乳是不透明的。这说明基因突变影响了淀粉的合成,导致淀粉粒的结构和性质发生了变化,使胚乳在表观上表现出巨大的差异。
对野生型和突变体n68种子的横截面进行了扫描电镜观察(见图3),从放大200倍的照片上,野生型种子的横截面的淀粉片层结构比较规则整齐,而突变体比较松散。由此可以推断,这些淀粉粒结构和排布的改变可能导致中胚乳呈现粉质的表型。
二、突变基因位点的图位克隆
1、突变基因的定位
首先,配制了突变体和正常粳稻品种nipponbare的杂交组合,f1自交一代后获得了f2种子。将f2种子去壳,以表型透明和不透明为标准进行统计分析,调查分离比例,结果显示,在f2分离群体中(共计1235个f2个体),具有野生型表型个体(933个)和突变体表型个体(302个),符合3:1的分离比例(χ2=0.1687<χ20.05,1=3.84)。由此可见,该突变由一对隐性基因控制。
然后用10株极端个体进行连锁,将目的基因确定在水稻第5染色体上,利用实验室的公用引物,增加极端个体数量至130株,缩小定位区段至570kb内,完成初定位。
挑选与突变体表型一致的f2种子发苗,获得1013株隐性极端个体的叶片,利用公用引物与自己设计的引物,最终将目的基因确定在标记199-2和h199-1之间,区段大小86kb(图4)。
上述ssr标记分析的方法如下所述:
(1)提取上述选取单株的总dna作为模板,具体方法如下:
①取0.2g左右的水稻幼嫩叶片,置于2.0mleppendorf管中,管中放置一粒钢珠,把装好样品的eppendorf管在液氮中冷冻5min,置于2000型geno/grinder仪器上粉碎样品1min。
②加入660μl提取液(含100mmtris-hcl(ph8.0),20mmedta(ph8.0),1.4mnacl,0.2g/mlctab的溶液),漩涡器上剧烈涡旋混匀,冰浴30min。
③加入40μl20%sds,65℃温浴10min,每隔两分钟轻轻上下颠倒混匀。
④加入100μl5mnacl,温和混匀。
⑤加入100μl10×ctab,65℃温浴10min,间断轻轻上下颠倒混匀。
⑥加入900μl氯仿,充分混匀,12000rpm离心3min。
⑦转移上清液至1.5mleppendorf管中,加入600μl异丙醇,混匀,12000rpm离心5min。
⑧弃上清液,沉淀用70%(v/v)乙醇漂洗一次,室温凉干。
⑨加入100μl1×te(121gtris溶于1升水中,用盐酸调ph值至8.0)溶解dna。
⑩取2μl电泳检测dna质量,并用du800分光光度仪测定浓度(beckmaninstrumentinc.u.s.a)。
(2)将上述提取的dna稀释成约20ng/μl,作为模板进行pcr扩增;
pcr反应体系(10μl):dna(20ng/μl)1μl,上游引物(2pmol/μl)1μl,下游引物(2pmol/μl)1μl,10×buffer(mgcl2free)1μl,dntp(10mm)0.2μl,mgcl2(25mm)0.6μl,rtaq(5u/μl)0.1μl,ddh2o5.1μl,共10μl。
pcr反应程序:94.0℃变性5min;94.0℃变性30s、55℃退火30s、72℃延伸1min,共循环35次;72℃延伸7min;10℃保存。pcr反应在mjresearchptc-225热循环仪中进行。
(3)ssr标记的pcr产物检测
扩增产物用8%非变性聚丙烯酰胺凝胶电泳分析。以50bp的dnaladder为对照比较扩增产物的分子量大小,银染显色。
上述引物开发过程如下:
(1)ssr标记开发
将公共图谱的ssr标记与水稻基因组序列进行整合,下载突变位点附近的bac/pac克隆序列。用ssrhunter(李强等,遗传,2005,27(5):808-810)或ssrit在线软件(http://archive.gramene.org/db/markers/ssrtool/)搜索克隆中潜在的ssr序列(重复次数≥6);将这些ssr及其邻近400~500bp的序列在ncbi通过blast程序在线与相应的籼稻序列进行比较,如果两者的ssr重复次数有差异,初步推断该ssr引物的pcr产物在籼、粳间存在多态性;再利用primerpremier5.0软件设计ssr引物,并由上海英骏生物技术有限公司合成。将自行设计的ssr成对引物等比例混合,检测其在n22和nipponbare之间的多态性,表现多态者用作精细定位的分子标记。用于精细定位的分子标记见表1。
表1用于精细定位的分子标记
2、粉质基因的获得
经过对86kb区间内的测序,发现了mtssb基因存在一个单碱基的突变,
根据网上公布的序列设计引物,序列如下所述:
primer1:5'aacgccgccagccgcctc3'(seqidno.4);
primer2:5'tttgagaggccagcatcat3'(seqidno.5)。
以primer1和primer2为引物,以n22的发育中胚乳cdna为模板,进行pcr扩增获得目的基因。扩增反应在ptc-200(mjresearchinc.)pcr仪上进行:94℃3min;94℃30sec,60℃45sec,72℃10min,35个循环;72℃5min。将pcr产物回收纯化后连接到pmd18-t(takara),转化大肠杆菌dh5α感受态细胞(北京tiangen公司cb101),挑选阳性克隆后,进行测序。
序列测定结果表明,pcr反应获得的片段具有seqidno.2所示的核苷酸序列,编码206个氨基酸残基组成的蛋白质(见序列表的seqidno.3)。将seqidno.3所示的蛋白命名为osmtssb,将seqidno.3所示的蛋白的编码基因命名osmtssb。
实施例2、转基因植物的获得和鉴定
一、重组表达载体构建
以n22(来自南京农业大学水稻所种质资源库)的基因组dna为模板,进行pcr扩增获得osmtssb基因,pcr引物序列如下:
primer3:
5'tgggcccggcgcgccaagcttaccatgttgccaaatgcc3'(seqidno.6);
primer4:
5'gaattcccggggatccctagaacaatccttctcct3'(seqidno.7)。
上述引物分别位于seqidno.2所示基因的上游2kb和下游1.5kb,扩增产物包含了该基因的启动子部分,将pcr产物回收纯化。采用infusion重组试剂盒(takara)将pcr产物克隆到载体pcambia1390中。infusion重组反应体系(10μl):pcr产物1.0μl,pcambia13056.0μl,5×infusionbuffer2.0μl,infusionenzymemix1μl。短暂离心后将混合体系37℃水浴15分钟,而后50℃水浴15分钟,取2.5μl反应体系用热激法转化大肠杆菌dh5α感受态细胞(北京tiangen公司;cb101)。将全部转化细胞均匀涂布在含50mg/l卡那霉素的lb固体培养基上。37℃培养16h后,挑取克隆阳性克隆,进行测序。测序结果表明,得到了含有seqidno.1所示基因的重组表达载体,将含有osmtssb的pcambia1390命名为pcambia1390-osmtssb,osmtssb基因片段利用infusion重组试剂盒(takara)插入到该载体的hindiii和bamhi酶切位点之间。。
二、重组农杆菌的获得
用电击法将pcambia1390-osmtssb转化农杆菌eha105菌株(购自美国英俊公司),得到重组菌株,提取质粒进行pcr及酶切鉴定。将pcr及酶切鉴定正确的重组菌株命名为eh-pcambia1305-osmtssb。
用pcambia1390作为对照载体转化农杆菌eha105菌株,方法同上,得到转空载体对照菌株。
三、转基因植物的获得
分别将eh-pcambia1390-osmtssb和转空载体对照菌株转化水稻成熟谷蛋白含量降低突变体n68,具体方法为:
(1)28℃培养eh-pcambia1390-osmtssb(或转空载体对照菌株)16小时,收集菌体,并稀释到n6液体培养基(sigma公司,c1416)中至浓度为od600≈0.5,获得菌液;
(2)将培养至一个月的n68水稻成熟胚胚性愈伤组织与步骤(1)的菌液混合侵染30min,滤纸吸干菌液后转入共培养培养基(n6固体共培养培养基,sigma公司)中,24℃共培养3天;
(3)将步骤(2)的愈伤接种在含有100mg/l潮霉素的n6固体筛选培养基上第一次筛选(16天);
(4)挑取健康愈伤转入含有100mg/l潮霉素的n6固体筛选培养基上第二次筛选,每15天继代一次;
(5)挑取健康愈伤转入含有50mg/l潮霉素的n6固体筛选培养基上第三次筛选,每15天继代一次;
(6)挑取抗性愈伤转入分化培养基上分化,得到分化成苗的t0代阳性植株。
四、转基因植株的鉴定
1、pcr分子鉴定
本研究中利用潮霉素标记鉴定转基因植株。
标记分析的pcr反应体系:dna(20ng/μl)2μl,primer3(10pmol/μl)2μl,primer4(10pmol/μl)2μl,10×buffer(mgcl2free)2μl,dntp(10mm)0.4μl,mgcl2(25mm)1.2μl,rtaq(5u/μl)0.4μl,ddh2o10μl,总体积20μl。
扩增反应在ptc-200(mjresearchinc.)pcr仪上进行:94℃3min;94℃30sec,55℃(引物不同,有所调整)45sec,72℃2.5min,35个循环;72℃5min。
pcr产物纯化回收,按试剂盒(北京tiangen公司)步骤进行。用8%的非变性page胶分离,银染。确定转基因阳性植株。
2、表型鉴定
分别将t0代转pcambia1390-osmtssb阳性植株、t0代转空载体对照植株、突变体n68和n22种植在南京农业大学土桥水稻育种基地转基因田内。种子成熟后,收取各材料种子,观察到pcambia1390-osmtssb植株的种子中出现了透明的种子(图5),进一步的扫描电镜分析显示,转入pcambia1390-osmtssb的n68种子的淀粉颗粒形态恢复至正常水平(图6)。因此证明n68中的突变表型是由osmtssb的突变造成的。pcambia1390-osmtssb可以使n68株系的淀粉合成恢复至正常水平。
<110>南京农业大学
<120>一种水稻胚乳粉质基因osmtssb及其编码蛋白质和应用
<160>7
<210>1
<211>4758
<212>dna
<213>稻属水稻(oryzasativavar.n22)
<220>
<223>胚乳粉质基因osmtssb的基因序列
<400>1
ataactcaaccttttgatctcgctacgaagctcctccgctccacccaggcacgagcgccg60
ccgcggccgactccaccggacaccacccaacgccgccagccgcctcctcccatcctccct120
cgatccgtacggatagcctccccgctcgctcggtctgcgcccgtcgtcgtcgtcccgccg180
tcttatccctcccgcgactcggcgacgcgcgaaggcctccggcgcggggctgaaggtacg240
tccttttcccgagctcgttctactgttccaactctgccatggctgcagttttccattttg300
ggctgggtctgccactgttctatggggtatgcaactgttgtcgacgtcgcgtgaagcccc360
tgaatggccagtgtgcgatgcttgcaaggtgtttgttaaaatgtctcggtagaatatctt420
ggaccgtttgcatattttatccatgcaaagtttagtccagtaggctgttaatttttctgt480
gattgcctatttagattttaggtggtgttaattcagcctcctgaagagggtggttattgg540
atactacgatcctagaatgccgctgcagcttagctaggcggatcagccgcttaggcaggt600
agagcagcttccttgtcttggccggatgtcgtccctgaggttccaacggaatggagctgc660
tgtagctgaataagaccataagttgtttttgtgcaggaaagaataagcagaactgtcatg720
gaactcctctgcactgttgagacaaacctgggatgcctcatcataaagaataagtgtctc780
ccacgtgataggccatcctgatgcaccttatttcctcatgtagttttttccttgtagaag840
gtcacactgaacaaatgcttttgagaatagaactgctaccaggaaaatgttgaatgaagt900
tgacgactgtgatgtttatgttgttttgaaatccattaaaggaataggaaaaaaatatat960
gttctgatgattatttttatgattttattatttaacgtttttcctcaatgtgcgtagatt1020
taccatgaattctctcagcaatggattactgaagggcttgaggagggtcttagaacagca1080
gagaaaaccaattggtgggttgtttttcatggttgtttcaagatttctactcagttttta1140
ctgagtaagttttgattaaatatttgtctgtgtagatttctacagaaaatctcaagcttg1200
gagttctactgtttccttttctgatattgatgagaagagtgaaatgggaggtgatgatga1260
ttatacagattcaagacgagagttagaaccccaaagcgtagaccccaagaagggctgggg1320
attccgtggtgttcacagggtatttaagtataatatgaaaaggatacttcatgttttggt1380
tttcaagcttaacttggtattaattgattgtttcaactttctaggctataatatgcggaa1440
aagtcggacaggttcctgtgcagaaaattttaaggaatggtcgtacagtgactgttttta1500
cagttggaactggtggcatgtttgaccagagggtagtaggggatgcagatctgccaaagc1560
cagctcagtggcatcggatagccattcataatgaccagctaggtgcttttgctgtccaga1620
agctggtgaagaagtacgtggtttcagtgagaagatcaccctttttcttttctccataca1680
agtatacaactatataccatttcctagttctatggcttgaaattcttttttactgtggtt1740
ggtgctgtctttgttcttgccttatttcagttctgcagtttatgttgagggtgatattga1800
aaccagagtatacaatgacagcattaatgatcaagtgaaaaatataccagagatttgtct1860
tcgacgcgatggtatgttgatctgccattccactagaaactttgtggtagtaatatttta1920
tagcaagaataaataacgtttgaagtttgaacaattttctcggggtaactccataactct1980
gttaccaaactggacacagaagcctgctgctttgtttaattatactgcatttgagtaatg2040
ttgatagctattcttttgtacttatttattttgattgtctttgttgttgtaaaagcttat2100
ccttggaaaaatatttatatgtacaggtaagattcggctgattaaatctggcgagagtgc2160
tgctagcatttcattagatgagctaagtaagttcactgaaaaccttaatattctgattta2220
tgagaatcttgcattacttgtatttgttgtgattatgcaactatgattggtctccacaat2280
tatctgcatgcagatgtccagtgttttagattttagtatttaccatttaatcatgacaac2340
ctttgtatgcaggagaaggattgttctagtttgaaagatctcagcacaagcaggccagga2400
gtacaggcatgtttcaactcttttgttgattgttttaacagtattaggattttgacttct2460
aagaagtgtccatgcaccctgttagctgtgggtcatgtgttttttattaatttgaacttg2520
aaatgtgcatattgtgaattatctgtagaacaccttcagctttcaatcaagcatttccct2580
gccaatacttatctgtcattgattggtgcctcgtgctatctttcagtagaaagtgatatt2640
ttgatgcctttaccaggctcatttcttcataagtttctcatagcgtggtattttacttgt2700
atcgcgtcttcatcttaatgaaatatgttgttacaacttcagcttgttacataagggtga2760
ttacaaattataagaaatgtcatagattcacggatcttgcgaatcaaatattacaaacct2820
ggaatggtagagtacctaattctatttatatttcttcactctatttgtacatgttgctga2880
tagattctccctggggtatttgtacatgaatagttgcacagtgacaatcttaggccaatt2940
tgttcaatgattttaagaatgtaagacactgaaccataactagtctctagttggtggaat3000
aaaagtgatcacttgtcattattaattttcatcatagatcatcaaaatcaattaggtgag3060
aaacaaacctctgagtaaatattctcgagtggcgaagaagaacacaggataacctgcatg3120
ttcacacaaaagcatagtatctcttgtcaacgaagtttgtagatcttcatgctgaaatgt3180
tgtgatcttgaaacttattcatatgaccaaattgtatttacacttgacatgttgccaggg3240
aatataaaaggtaatactagtaaattaactgttgagtaggttgagagaaatcttacatca3300
gtcacacccttatctaaaagcataattctgatgttcttcgctttggaaccttgtcaattg3360
tcaccaagtaagaatgggtgtgcttttgatggtatttcaggctttaccttatcctttgac3420
ctctttctgagcatctccttcatgccatccgccaccttgactgcagggcttgatagtgcc3480
atcctgcagaatgccatatgagctttgatagcatcttcgatgccatcctgttctcttctg3540
tctttactgagctcatcccctaccgcctctgagcataagccacaaagccatttccctccg3600
aagtttgccttgacattctctatgtattcctgggtgcaatcctcttccaggccacagcac3660
gcacacctcactgattcaatgtccatcttcaatgctgatgcctgatggaaagaggactaa3720
aagaacaatagtagatgcatgggagtagagaaggagaaagaaggtgggagaaggcaaata3780
gaaggaaacagtatggagttcaggactaggttgctgcttgtgtggataatttttgtagac3840
ctctccttccttttggattggcttgtttagcttggtatgttattgaaattttgagatttg3900
cccagacccatagtatcttcaacagtgtactacctcgtgaaagaataagaaagataatat3960
gatttttttgatgcctaccagtttcctaggtaaaaatataggaaaccgtagttcacatga4040
ctgcaactgtgttgaaggtaaaggcaggagcaggttcccatagcctgaattagctagtac4100
agttttgtttttcctgtgtgaagacgcacgatggatgacaaataaacatgctataagatc4160
caaatgaaaatgcaaaagaaaagttattcccatatctaaaaattgtatacttgatcacat4220
gcggtcaattagccatctacatatgcacattacaaagaacacaacacaagcttatctacc4280
tttagtgaaggagtgggggcatatcaccatcacattagcttatccttttacgacagtata4320
aatttcaatgattttgttttatataagtctttacatgatgatgtctttaaaggttgttac4380
atgcctaatatgtgtgatatatactagaaatgtcagacacctggtttctaacgcaacaac4440
cttgtttaggttgattactgaaatgtcgcactcctaatattagtcgcatcgccttcgttt4500
gtacgtatatacttttctgggcaggtcgtaacctgtttatctgtgcattattaggttcaa4560
aagataaagtggatacgtgcaggctcaacgatgatgctggcctctcaaagaaaagaaaaa4620
cgacgatgctgcttcaccgaagcgaaatttgttgtagcaggcatgtgaaccttgtcctgg4680
ccgcccttccagtcgtattttcattatcaacatcctttaatgtactcttgattaacatct4740
tagcagtcaaatttacag4758
<210>2
<211>621
<212>dna
<213>稻属水稻(oryzasativavar.n22)
<220>
<223>胚乳粉质基因osmtssb的cds序列
<400>2
atgaattctctcagcaatggattactgaagggcttgaggagggtcttagaacagcagaga60
aaaccaattgatttctacagaaaatctcaagcttggagttctactgtttccttttctgat120
attgatgagaagagtgaaatgggaggtgatgatgattatacagattcaagacgagagtta180
gaaccccaaagcgtagaccccaagaagggctggggattccgtggtgttcacagggctata240
atatgcggaaaagtcggacaggttcctgtgcagaaaattttaaggaatggtcgtacagtg300
actgtttttacagttggaactggtggcatgtttgaccagagggtagtaggggatgcagat360
ctgccaaagccagctcagtggcatcggatagccattcataatgaccagctaggtgctttt420
gctgtccagaagctggtgaagaattctgcagtttatgttgagggtgatattgaaaccaga480
gtatacaatgacagcattaatgatcaagtgaaaaatataccagagatttgtcttcgacgc540
gatggtaagattcggctgattaaatctggcgagagtgctgctagcatttcattagatgag600
ctaagagaaggattgttctag621
<210>3
<211>513
<212>prt
<213>稻属水稻(oryzasativavar.n22)
<220>
<223>胚乳粉质基因osmtssb氨基酸序列
<400>3
metasnserleuserasnglyleuleulysglyleuargargvalleu
151015
gluglnglnarglysproileaspphetyrarglysserglnalatrp
202530
serserthrvalserpheseraspileaspglulysserglumetgly
354045
glyaspaspasptyrthraspserargarggluleugluproglnser
505560
valaspprolyslysglytrpglypheargglyvalhisargalaile
65707580
ilecysglylysvalglyglnvalprovalglnlysileleuargasn
859095
glyargthrvalthrvalphethrvalglythrglyglymetpheasp
100105110
glnargvalvalglyaspalaaspleuprolysproalaglntrphis
115120125
argilealailehisasnaspglnleuglyalaphealavalglnlys
130135140
leuvallysasnseralavaltyrvalgluglyaspilegluthrarg
145150155160
valtyrasnaspserileasnaspglnvallysasnileprogluile
165170175
cysleuargargaspglylysileargleuilelysserglygluser
180185190
alaalaserileserleuaspgluleuarggluglyleuphe
195200205
<210>4
<211>18
<212>dna
<213>人工序列
<220>
<223>primer1
<400>4
aacgccgccagccgcctc18
<210>5
<211>18
<212>dna
<213>人工序列
<220>
<223>primer2
<400>5
tttgagaggccagcatcat18
<210>6
<211>39
<212>dna
<213>人工序列
<220>
<223>primer3
<400>6
tgggcccggcgcgccaagcttaccatgttgccaaatgcc39
<210>7
<211>35
<212>dna
<213>人工序列
<220>
<223>primer4
<400>7
gaattcccggggatccctagaacaatccttctcct35
- 还没有人留言评论。精彩留言会获得点赞!