一种基于CLCF1基因的胶质母细胞瘤辅助诊断、预后评价试剂盒及其使用方法与流程
本发明属于检测试剂盒
技术领域:
,具体涉及一种基于clcf1基因的胶质母细胞瘤辅助诊断、预后评价试剂盒及其使用方法。
背景技术:
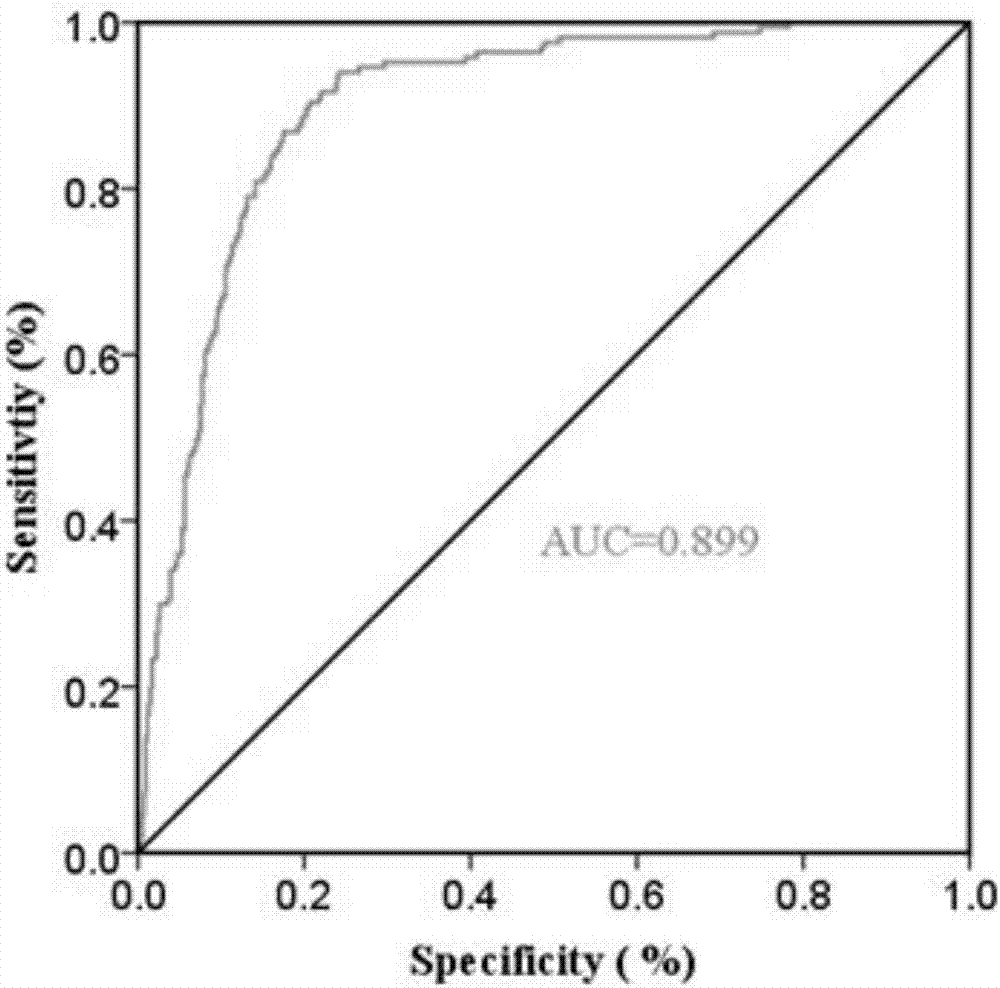
:胶质母细胞瘤是恶性程度最高、最常见的原发性脑肿瘤,约占中枢神经系统肿瘤的50%。尽管以手术为主联合放射及化学治疗的综合方案已成为治疗指南,但胶质母细胞瘤病人的预后并没有得到明显改善,平均生存时间只有12~15个月。低级别胶质瘤(lowgradeglioma,lgg)患者经过或长或短的时间发展,一般都会发展成为高级别胶质瘤(glioblastoma,gbm)。目前常用的鉴别低级别胶质瘤与高级别胶质瘤主要依靠组织病理诊断,但病理诊断的结果存在一定的人为因素,有一定的不可靠性。因此,发现新的特异的分子诊断及预后评价标记物势在必行。心肌营养素样细胞因子1(cardiotrophinlikecytokinefactor1,clcf1,alsoknownasnnt-1orbsf-3),分布于胞外区,属于细胞因子或生长因子,是il6家族的一个重要成员。clcf1是一种神经营养因子和b细胞刺激剂,与神经内分泌及免疫调节密切相关。clcf1在肿瘤中的表达及其临床意义研究很少,尤其在胶质瘤中的表达情况及预后指示情况尚不为所知。技术实现要素:本发明主要提供了一种基于clcf1基因的胶质母细胞瘤辅助诊断、预后评价试剂盒及其使用方法,发现clcf1基因在lgg和gbm中的转录表达水平存在显著差异。与lgg组织相比,clcf1基因在gbm组织中表达显著上调,通过检测clcf1基因转录水平的表达情况,可以诊断胶质瘤患者的恶性程度。同时,依据clcf1的表达情况,可以评估患者预后情况。其技术方案如下:一种基于clcf1基因的胶质母细胞瘤辅助诊断、预后评价试剂盒,包括扩增clcf1基因的pcr引物对,所述引物对为:正向引物序列为5’-cctcctgcttcttgcctacc-3’,如seqidno:1所示;反向引物序列为5’-tgccaccatgtttgtttcct-3’,如seqidno:2所示。优选的,所述试剂盒还包括扩增管家基因gapdh的pcr引物对,所述引物对为:正向引物序列为5’-gcaccgtcaaggctgagaac-3’,如seqidno:3所示;反向引物序列为5’-tggtgaagacgccagtgga-3’,如seqidno:4所示。优选的,所述试剂盒还包括sybrgreen聚合酶链式反应体系,所述sybrgreen聚合酶链式反应体系包括pcr缓冲液、dntps、sybrgreen荧光染料、无酶水和荧光定量加样板。优选的,所述试剂盒还包括rna提取试剂,所述rna提取试剂包含trizol、氯仿、异丙醇、75%乙醇和无酶水。优选的,所述试剂盒还包括将mrna反转录为cdna的体系,该反转录体系包含gdnaeraser、gdnaeraserbuffer、t重复寡核苷酸oligo(dt)、反转录反应液、反转录酶和dntps。一种基于clcf1基因的胶质母细胞瘤辅助诊断、预后评价试剂盒及其使用方法,包括以下步骤:(1)将获取的新鲜组织经过液氮处理研磨后进行rna抽提;(2)将抽提出的rna反转录成对应的cdna;(3)将反转录后的cdna进行对clcf1和gapdh基因的荧光定量pcr扩增;(4)以gapdh作为内参,记录每个反应的ct值,检测结果以δct表示,其中δct=ctgene-ctgapdh。clcf1基因在制备胶质母细胞瘤辅助诊断、预后评价试剂盒中的应用。采用上述方案,本发明具有以下优点:(1)本发明首次公开了clcf1基因与胶质瘤疾病进展相关,clcf1基因有望成为诊断gbm的分子标志物,并为研究胶质瘤疾病进展的分子机理提供新的思路;(2)clcf1基因在lgg和gbm中的转录表达水平存在显著差异,与lgg组织相比,clcf1基因在gbm组织中表达显著上调,通过检测clcf1基因转录水平的表达情况,可以诊断胶质瘤患者的恶性程度,从而可以制备含有clcf1基因的胶质母细胞瘤辅助诊断试剂盒,利用检测基因转录水平表达的方式诊断胶质母细胞瘤的方法更加灵敏更加特异,有利于疾病早期的诊断;(3)本发明首次证实了clcf1基因在gbm中表达的高低与患者预后直接相关,高表达clcf1的gbm患者与低表达clcf1的患者相比,生存时间显著减少。通过检测clcf1转录水平的表达情况,可以预估gbm患者的预后情况,从而制备含有clcf1基因的胶质母细胞瘤预后评价试剂盒,可以更加精准、更加客观的评判患者预后;(4)本发明还提示可以通过干预clcf1的表达阻断胶质瘤疾病进展,其有可能作为胶质瘤未来靶向治疗的特定靶点,为胶质瘤疾病的治疗提供新思路。附图说明图1为tcga数据库中clcf1基因mrna在530例lgg患者、167例gbm患者组织中的表达情况;图2为基于tcga数据库中clcf1基因mrna表达情况,绘制的clcf1对于诊断gbm的roc曲线;图3为cgga数据库中clcf1基因mrna在181例lgg患者、144例gbm患者组织中的表达情况;图4为基于cgga数据库中clcf1基因mrna表达情况,绘制的clcf1对于诊断gbm的roc曲线;图5为临床标本来源的clcf1基因mrna在41例lgg患者、41例gbm患者组织中的表达情况;图6为基于临床标本来源clcf1基因mrna表达情况,绘制的clcf1对于诊断gbm的roc曲线;图7为tcga的gbm(illuminahiseq)数据库中,基于clcf1基因mrna高低绘制的生存曲线;图8为tcga的gbm(affyu133a)数据库中,基于clcf1基因mrna高低绘制的生存曲线;图9为基于41例临床标本来源gbm的clcf1基因mrna高低绘制的生存曲线。具体实施方式具体实施例以下实施例中的实验方法如无特殊规定,均为常规方法,所涉及的实验试剂及材料如无特殊规定均为常规生化试剂和材料。一、tcga数据库对比lgg与gbm中clcf1差异表达肿瘤基因图谱计划(thecancergenomeatlas,tcga)项目组最初由美国国家肿瘤研究所(thenationalcancerinstitute,nci)和美国国家人类基因组研究所(thenationalhumangenomeresearchinstitute,nhgri)组成,并发展成一个非常大的数据研究平台。低级别胶质瘤(lgg)及胶质母细胞瘤(gbm)作为其最早研究的肿瘤之一,具有大量全面的数据结果。tcga中lgg和gbm数据来源于(https://genome-cancer.ucsc.edu),其中lgg收录了530例肿瘤样本,gbm收录了167例样本。clcf1基因转录水平结果来自illuminahiseq测序平台的rna-seq结果。图1为tcga数据库中clcf1基因mrna在530例lgg患者、167例gbm患者组织中的表达情况。如图1所示,clcf1基因在gbm患者组织中表达水平较lgg患者组织中表达水平显著上升(p<0.0001)。二、基于clcf1在tcga中差异表达绘制gbm诊断roc曲线基于上述tcga胶质瘤组织样本lgg和gbm的clcf1表达水平,绘制用于诊断gbm的roc曲线。在roc曲线评价方法中,roc曲线下的面积值auc在大于0.5的情况下,越接近于1,说明诊断效果越好。auc在0.5~0.7时有较低准确性,auc在0.7~0.9时有一定准确性,auc在0.9以上时有较高准确性。结果如图2所示,clcf1诊断胶质母细胞瘤的roc曲线下面积(auc)为0.899,即诊断具有一定准确性。三、cgga数据库获取lgg与gbm中clcf1差异表达中国胶质瘤基因图谱(chinesegliomagenomeatlas,cgga)是2012年由北京天坛医院发起并建成的中国胶质瘤患者样本解析的数据库,是我国特有的能够揭示中国人胶质瘤发生发展分子机制的大型数据库,为我国胶质瘤研究提供了大量基础和临床支持。在cgga数据库中(http://www.cgga.org.cn),共计包含lgg181例,gbm144例样本。clcf1基因转录水平结果来自illunimahiseq2000平台rna-seq结果。图2为cgga数据库中clcf1基因mrna在181例lgg患者、144例gbm患者组织中的表达情况。如图3所示,clcf1基因在gbm患者组织中表达水平较lgg患者组织中表达水平显著上升(p<0.0001)。四、基于clcf1在cgga中差异表达绘制gbm诊断roc曲线基于上述cgga胶质瘤组织样本lgg和gbm的clcf1表达水平,绘制用于诊断gbm的roc曲线。结果如图4所示,clcf1诊断胶质母细胞瘤的roc曲线下面积(auc)为0.861,即诊断具有一定准确性。五、临床组织样本检测lgg与gbm中clcf1差异表达收集郑州大学第一附属医院经病理诊断为低级别胶质瘤(lgg)41例和高级别胶质瘤(gbm)组织样本41例,共计82例组织样本。对这些样本进行rna提取和反转录,对反转录得到的cdna进行clcf1和gapdh基因的荧光定量pcr扩增。结果如图5所示。由图5可知,clcf1基因在gbm患者组织中表达水平较lgg患者组织中表达水平显著上升(p<0.0001)。六、基于clcf1在临床组织中差异表达绘制gbm诊断roc曲线基于上述收集的82例胶质瘤组织样本clcf1表达水平,绘制用于诊断gbm的roc曲线。结果如图6所示,clcf1诊断胶质母细胞瘤的roc曲线下面积(auc)为0.911,即诊断具有较高准确性。七、tcga的gbm(illuminahiseq)数据库中clcf1表达高低绘制生存曲线在tcga数据库中,gbm(illuminahiseq)包含167例肿瘤样本,带有预后信息的为165例。将这165例样本根据clcf1的mrna表达水平排序,高表达组为前87例,低表达组为后78例,绘制生存曲线,图7为基于clcf1基因mrna高低绘制的生存曲线。通过longranktest分析得p=0.0180,即预后有统计学意义。说明在gbm中,高表达clcf1的患者预后显著差于低表达clcf1患者。八、tcga的gbm(affyu133a)数据库中clcf1表达高低绘制生存曲线在tcga数据库中,gbm(affyu133a)包含539例肿瘤样本,带有预后信息的为524例。将这524例样本根据clcf1的mrna表达水平排序,高表达组为前248例,低表达组为后276例,绘制生存曲线,图8为基于clcf1基因mrna高低绘制的生存曲线。通过longranktest分析得p=0.0453,即预后有统计学意义。进一步说明在gbm中,高表达clcf1的患者预后显著差于低表达clcf1患者。九、临床标本来源的gbm中clcf1表达高低绘制生存曲线临床收集的胶质母细胞瘤41例患者均带有随访信息。根据41例样本clcf1的mrna表达水平排序,高表达组为前21例,低表达组为后20例,绘制生存曲线,图9为基于clcf1基因mrna高低绘制的生存曲线。通过longranktest分析得p=0.0051,即预后有统计学意义。进一步在gbm临床样本中证实了高表达clcf1的患者预后显著差于低表达clcf1患者。十、基于41例临床标本来源gbm的cox多因素回归分析性别、年龄、肿瘤大小及clcf1的表达水平被纳入cox多因素回归分析模型,分析这些因素与gbm预后的关系。分析结果如表1。表1基于41例临床标本来源gbm的cox多因素回归分析结果风险率95%clp值年龄1.0250.973-1.0810.353性别1.5180.621-3.7140.360kps1.0040.976-1.0330.781fuca11.5361.100-2.1430.012该结果说明了clcf1表达水平对gbm具有独立判断预后的价值(p=0.012)。十一、基于clcf1基因的胶质母细胞瘤辅助诊断试剂盒的制备及使用1.胶质母细胞瘤辅助诊断试剂盒包括扩增clcf1基因的pcr引物对、扩增管家基因gapdh的pcr引物对、sybrgreen聚合酶链式反应体系、rna提取试剂、mrna反转录为cdna的体系。其中扩增clcf1基因的pcr引物对为:正向引物序列为5’-cctcctgcttcttgcctacc-3’,如seqidno:1所示;反向引物序列为5’-tgccaccatgtttgtttcct-3’,如seqidno:2所示。扩增管家基因gapdh的pcr引物对为:正向引物序列为5’-gcaccgtcaaggctgagaac-3’,如seqidno:3所示;反向引物序列为5’-tggtgaagacgccagtgga-3’,如seqidno:4所示。所述sybrgreen聚合酶链式反应体系包括pcr缓冲液、dntps、sybrgreen荧光染料、无酶水和荧光定量加样板。所述rna提取试剂包含trizol、氯仿、异丙醇、75%乙醇和无酶水。所述反转录为cdna的体系包含gdnaeraser、gdnaeraserbuffer、t重复寡核苷酸oligo(dt)、反转录反应液、反转录酶和dntps。2.诊断试剂盒的检测过程如下:1)将待测的新鲜组织在液氮作用下研碎,在碎裂后的组织中加入1mltrizol,使用1ml的移液枪反复吹打,室温孵育5min,充分分离核蛋白复合物;2)每管加入500μl氯仿,上下颠倒混匀10次。待溶液充分乳化后,室温静置5min。4℃离心,12000rpm,15min;3)从离心机中小心取出离心管,此时匀浆液分为三层,即:无色的水相、中间的白色蛋白层及带有颜色的下层有机相。吸取水相转移至新的离心管中;4)向水相中加入等体积的异丙醇,上下颠倒离心管充分混匀后,室温静置10min;4℃,12000rpm离心10min,离心后,ep管底部会出现沉淀;5)小心弃去上清,缓慢地沿离心管壁加入lml预冷75%乙醇(切勿触及沉淀),12000rpm,4℃离心5min,弃去上清;6)室温干燥沉淀2-5min,加入适量的rnase-free水溶解沉淀,必要时可用移液枪轻轻吹打沉淀;7)利用nanodrop检测rna的浓度及纯度。od260/od280比值在1.80-2.0之间,说明rna纯度符合实验要求;8)反转录:首先配置5×gdnaeraserbuffer(2μl)、gdnaeraser(1μl)、rna(1μ)和rnase-free水混合体系,共10μl,42℃,2min;将5×primescriptbuffer2(4μl)、primescriptrtenzymemix(1μl)、rtprimermix(1μl)、rnase-free水(4μl)及上述混合液(10μl)进行混合,短暂离心后放入pcr仪,反应条件:37℃反应15min,85℃反应5秒,获得cdna;9)加样:将荧光定量板置于冰上,每个样本设2个复孔,将cdna(稀释4~5倍,稀释后加2μl)、sybergreen和rcr缓冲液混合一起每孔12μl,引物每孔8μl,配置好总体系后进行分装,减少误差;10)加样结束后,使用pe手套小心将荧光定量板膜盖上密封板,离心,避免气泡产生;11)设置程序:95℃10min;95℃10s,60℃10s,72℃10s,40个循环+溶解曲线;12)实验数据分析:以gapdh作为内参,记录每个反应的ct值,ct值为每个反应管内的荧光信号到达设定的阈值时所经历的循环数。δct=ctgene-ctgapdh,δct越小表示其起始拷贝数越多,表达越高。对本领域的技术人员来说,可根据以上描述的技术方案以及构思,做出其它各种相应的改变以及形变,而所有的这些改变以及形变都应该属于本发明权利要求的保护范围之内。序列表<110>郑州大学第一附属医院<120>一种基于clcf1基因的胶质母细胞瘤辅助诊断、预后评价试剂盒及其使用方法<160>4<170>siposequencelisting1.0<210>1<211>20<212>dna<213>人工序列(artificialsequence)<400>1cctcctgcttcttgcctacc20<210>2<211>20<212>dna<213>人工序列(artificialsequence)<400>2tgccaccatgtttgtttcct20<210>3<211>20<212>dna<213>人工序列(artificialsequence)<400>3gcaccgtcaaggctgagaac20<210>4<211>19<212>dna<213>人工序列(artificialsequence)<400>4tggtgaagacgccagtgga19当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1