一种基于CRISPR-Cas10核酸蛋白复合物的快捷核酸检测定量法的制作方法
本发明属于核酸检测领域,具体涉及到一种基于CRISPR-Cas10核酸蛋白复合物的快捷的核酸检测及定量方法。
背景技术:
核酸检测是应用分子生物学的手段检测受检样品的遗传物质或携带的病毒、病原体的基因,是体外诊断的重要分支。核酸检测技术目前主要分为核酸扩增技术(PCR)、原位杂交技术(ISH)、基因芯片以及二代高通量基因测序技术四大类。目前应用最多的为实时定量PCR,恒温扩增(RPA),其次是原位杂交和基因芯片技术,而高速发展的二代测序技术也为更精准的核酸检测提供技术支撑。目前,核酸检测产品应用在临床疾病如病原菌感染、肿瘤、遗传等的诊断占比达到70%以上。面对体外诊断行业的高速增长和极为广阔的市场前景,现有的核酸检测技术还面临着工序复杂、成本高等问题,因此还需开发更方便高效、成本更低廉的核酸检测新技术。
CRISPR-Cas(clustered regularly interspaced short palindromic repeats-CRISPR-associat ed)是一种原核生物所特有的获得性免疫系统,主要用于抵御病毒、质粒等外源遗传物质的入侵。CRISPR-Cas系统目前已被分为六个不同的类型,即I型、II型、III型、IV型、V型和VI型。目前,基于II型CRISPR-Cas9和V型CRISPR-Cpf1系统的基因组编辑工具广泛地应用于生命三域的科学研究中。基于VI型CRISPR-Cas13a系统的核酸检测应用也为体外分子诊断提供了新的技术手段,但该技术同样具有原料生产困难复杂、使用成本高、难于对核酸进行定量等问题。
针对上述问题,申请人发现,由多蛋白组成的III型CRISPR-Cas系统核酸蛋白复合物 (CRISPR-Cas10)识别配对的靶标RNA长度达到32-47个核苷酸,其不仅能切割靶标RN A,结合靶标RNA之后还能对序列非特异的单链DNA进行切割(冰岛硫化叶菌Cmr-β复合物的单链DNA切割活性还能被ATP进一步激活提高);研究还发现,对CRISPR-Cas10 核酸蛋白复合物的一个亚基进行突变改造能使其丧失对靶标RNA的切割活性。因此,CRI SPR-Cas10核酸蛋白复合物与只能切割单链RNA的单蛋白CRISPR系统Cas13a相比,在核酸检测应用上具有诸多明显优势。此外,由于靶标RNA能被CRISPR-Cas10核酸蛋白复合物切割后解离从而使得复合物丧失单链DNA切割活性,所以单链DNA探针被切割后所释放的信号强弱可以准确反映受检样品中靶标RNA的含量,基于此原理,我们可以利用该核酸蛋白复合物开发一种全新的核酸定量技术。
技术实现要素:
鉴于核酸检测的市场需求巨大,而现有技术和方法在检测工序、检测时间周期、检测灵敏度和检测成本方面都有待优化和提高。本发明的目的在于提供一种基于CRISPR-Cas10核酸蛋白复合物的快捷核酸检测定量法。
一种基于CRISPR-Cas10核酸蛋白复合物的快捷核酸检测定量法,包括以下步骤:
(a)确定待检样品靶标核酸序列;
(b)如果待检核酸样品中的靶标核酸为单链RNA,即可直接进行步骤(c),如果受检核酸样品中的靶标核酸为双链或者单链DNA,则需要利用RNA聚合酶将其转录成单链 RNA,再进行步骤(c);
(c)生产携带与步骤(b)中靶标核酸RNA序列配对的crRNA的CRISPR-Cas10核酸蛋白复合物,所述的CRISPR-Cas10核酸蛋白复合物通过原核生物细胞内源表达共纯化方式获得;
(d)在检测反应体系中加入适量待检测样品、步骤(C)生产的CRISPR-Cas10核酸蛋白复合物以及单链DNA探针;
所述的单链DNA探针的5’端和3’端分别标记荧光基团和淬灭基团;
优选的,所述的单链DNA探针为5-HEX/TTTTT/3Dabcyl;
(e)将检测反应体系放置在20-75℃下反应30-90分钟,待检测反应结束后,通过荧光酶标仪或荧光分光光度计,检测反应的荧光值,进行定性分析。
(d)对靶标RNA进行定量:配制不同浓度的靶标核酸标准品,绘制荧光值与浓度对应的标准曲线,再根据受检样品的荧光值计算其靶标核酸的浓度。
以上所述的方案中,优选的,当做定性分析时,CRISPR-Cas10核酸蛋白复合物为突变型的结构蛋白或核酸酶亚基(Cmr/Csm蛋白)与crRNA的复合体。
以上所述的方案中,优选的,使用CRISPR-Cas10核酸蛋白复合物通过冰岛硫化叶菌表达制备得到。
以上所述的方案中,优选的,所述的CRISPR-Cas10核酸蛋白复合物为携带与靶标核酸RN A序列配对的crRNA的Cmr-α核酸蛋白复合物或Cmr-β核酸蛋白复合物。
以上所述的步骤中,所述的待检测样品为含有核糖核酸RNA、脱氧核糖核酸DNA或两种核酸混合的样品,进一步包括含有核酸的动植物病和人体病原样品、人类肿瘤和遗传疾病样品以及环境样品如土壤和水等。
与现有技术相比,本发明具有以下优点:
本发明所采用的CRISPR-Cas10核酸蛋白复合物是从原核生物宿主细胞中制备的,含有 40-55个核苷酸的crRNA。该复合物能特异性地识别并结合与其crRNA序列相匹配的靶标R NA(32-47个核苷酸),形成具有双重核酸酶活性的三元CRISPR-Cas复合物(Cas蛋白、c rRNA和靶标RNA),它既能特异性切割靶标RNA,又能高效地、非特异性地切割单链DN A。此外,一旦靶标RNA被切割,CRISPR-Cas10复合物又恢复其无活性构型。因此,我们可以荧光标记的单链DNA作为报告探针,测定其DNA切割活性,同时,单链DNA探针被 CRISPR-Cas10核酸蛋白复合物切割后所释放的信号强弱可以准确反映受检样品中靶标RN A的含量,即本发明方法的基本原理(图1)。
本发明方法具有如下特点:操作流程和仪器需求简单,检测时间周期短;CRISPR-Cas1 0核酸蛋白复合物识别的靶标RNA较长,既保证了核酸检测特异性,又能区分不同的靶标核酸突变体;所使用的核酸蛋白复合物由原核生物内源表达生产,更方便更稳定;使用荧光报告探针为单链DNA,合成成本低,且不易降解;具有较高的灵敏度,对靶标核酸的直接检测限为10pM;更为重要的是,本方法能对6.4pM-2nM浓度范围内的靶标核酸进行精准定量,是一种全新的更为简便的核酸定量技术,将核酸蛋白复合物进行突变,可提高核酸检测灵敏度到100fM。该方法应用范围广,受检核酸样品中靶标核酸为RNA时可以直接进行检测,受检核酸样品中靶标核酸为DNA时可以将其转录成RNA之后再进行检测,可用于人类肿瘤标志物(microRNAs、lncRNAs)、遗传疾病、动植物和人体病原微生物的检测和诊断,还可用于环境样品如土壤和水中特定核酸的检测和定量。本发明方法有望替代目前广泛应用的qPCR技术,使核酸检测和定量技术提高到一个新水平。
附图说明
图1为本发明原核生物细胞内源表达生产CRISPR-Cas10核酸蛋白复合物并利用其进行核酸检测定量的原理示意图。
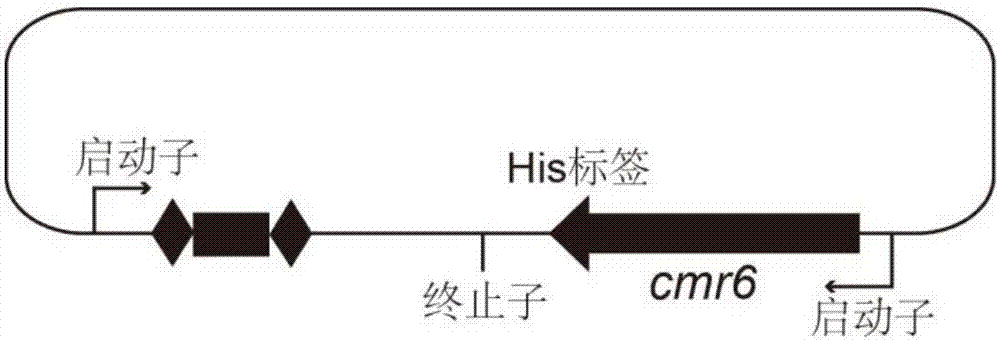
图2为本发明用于共纯化CRISPR-Cas10核酸蛋白复合物的表达质粒pSS1-Cmr6α/β结构图。
图3为冰岛硫化叶菌CRISPR-Cas10系统Cmr-α核酸蛋白复合物对浓度为100nM的靶标R NA的检测结果示意图。
图4为冰岛硫化叶菌CRISPR-Cas10系统Cmr-α核酸蛋白复合物对浓度分别为1nM和10 pM的靶标RNA的检测结果示意图。
图5为冰岛硫化叶菌CRISPR-Cas10系统Cmr-β核酸蛋白复合物对浓度分别为1nM,100 pM和10pM的靶标RNA的检测结果示意图。
图6为冰岛硫化叶菌CRISPR-Cas10系统Cmr-β核酸蛋白复合物检测浓度为1nM的靶标R NA,在反应体系中添加和不添加ATP的检测结果示意图。
图7为冰岛硫化叶菌CRISPR-Cas10系统Cmr-α核酸蛋白复合物对不同浓度靶标RNA进行检测,反应80分钟时的各检测反应的荧光值与靶标RNA浓度的线性关系示意图。
图8为冰岛硫化叶菌CRISPR-Cas10系统Cmr-α核酸蛋白复合物对不同浓度靶标RNA进行检测,各靶标RNA样品的浓度(2000pM,800pM,400pM,300pM,160pM,32pM, 6.4pM)与其荧光值之间的线性方程示意图。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别说明,实施例中的实验操作均按常规实验条件或按照材料、试剂制造厂商说明书建议的条件。
实施例1:
本实施例利用冰岛硫化叶菌S.islandicus REY15A CRISPR-Cas10系统Cmr-α核酸蛋白复合物对人工合成的一段小RNA进行检测为例来说明:
1.本实施例选择人工合成的一段小RNA为靶标核酸,,因此受检核酸样品为人工配制但其不影响该方法的在其它天然受检核酸样品的应用推广,根据该RNA序列设计两条引物(SS1-SpF/SS1-SpR)(表1),以上两条引物通过95℃处理5分钟后自然冷却退火生成一段具有粘性末端的spacer片段;
表1.本发明所用到引物序列
上表中形成spacer粘性末端的四个碱基被黑色加粗显示。
2.将上面得到的spacer片段与经BspMI酶切处理的载体pSe-Rp(Peng et al.,2015) 酶连并转化大肠杆菌DH5α菌株,得到人工mini-CRISPR质粒pAC-SS1;
3.用表1中的引物对Cmr6α-F/Cmr6α-R从冰岛硫化叶菌基因组DNA上扩增出cmr6 α基因片段,随后用NdeI/SalI克隆到冰岛硫化叶菌表达载体pSeSD1(Deng et al.,2009) 上得到Cmr-α系统亚基之一Cmr6α的表达质粒pSeSD-Cmr6α;
4.用表1中的引物对Cmr6-Insert-F/Cmr6-Insert-R以步骤3得到的表达质粒pSeSD- Cmr6α为模板扩增整个Cmr6α的表达元件,用SalI/XhoI克隆到步骤2得到的人工mini-CRI SPR质粒pAC-SS1上,得到Cmr-α核酸蛋白复合物的共纯化表达质粒pSS1-Cmr6α(图2);
5.将上面得到的共纯化表达质粒pSS1-Cmr6α转化到冰岛硫化叶菌中,表达纯化得到 Cmr-α核酸蛋白复合物;
6.选择黑色酶标板做检测反应,在反应体系中分别加入10μL 10×cleavage buffer (500mM Tris-HCl,100mM MgCl2,100mM DTT),其它反应组分及终浓度分别为:20 0nM荧光核酸报告探针(/5-HEX/TTTTT/3Dabcyl/),50nM Cmr-α核酸蛋白复合物,不同浓度的靶标核酸(100nM,1nM,100pM,0nM),最后用无菌水补至100μL;
7.在室温下反应5min后用多功能酶标仪检测荧光值(Ex/Em=535/556nm),每5 分钟检测一次,连续检测1小时;
8.绘制反应时间和接收荧光值的曲线(图3,图4),结果显示检测灵敏度能达到10 pM。
实施例2:
本实施例利用冰岛硫化叶菌S.islandicus REY15A CRISPR-Cas10系统Cmr-β核酸蛋白复合物对人工合成的一段小RNA进行检测为例来说明:
1.本实施例同样选用实施例1中的靶标核酸,
2.用表1中的引物对Cmr6β-F/Cmr6β-R从冰岛硫化叶菌基因组DNA上扩增出cmr6 β基因片段,随后用NdeI/SalI克隆到冰岛硫化叶菌表达载体pSeSD1(Deng et al.,2009) 上得到Cmr-β系统亚基之一Cmr6β的表达质粒pSeSD-Cmr6β;
3.用表1中的引物对Cmr6-Insert-F/Cmr6-Insert-R以步骤3得到的表达质粒pSeSD- Cmr6β为模板扩增整个Cmr6β的表达元件,用SalI/XhoI克隆到实施例1步骤2得到的人工 mini-CRISPR质粒pAC-SS1上,得到Cmr-β核酸蛋白复合物的共纯化表达质粒pSS1-Cmr6β;
4.将上面得到的共纯化表达质粒pSS1-Cmr6β转化到冰岛硫化叶菌中,表达纯化得到Cmr-β核酸蛋白复合物;
5.选择黑色酶标板做检测反应,在反应体系中分别加入10μL 10×cleavage buffer (500mM Tris-HCl,100mM MgCl2,100mM MnCl2,100mM DTT),其它反应组分及终浓度分别为:200nM荧光核酸报告探针(/5-HEX/TTTTT/3Dabcyl/),50nM Cmr-β核酸蛋白复合物,不同浓度的靶标核酸(1nM,100pM,10pM,0nM),200nM ATP,最后加无菌水补至100μL;
6.在室温下反应5min后用多功能酶标仪检测荧光值(Ex/Em=535/556nm),每5 分钟检测一次,连续检测1小时;
7.绘制反应时间和接收荧光值的曲线(图5,图6),结果显示检测灵敏度能达到10 pM,同时ATP能提高Cmr-β核酸蛋白复合物切割核酸报告探针的活性,提高荧光检测信号。
实施例3:
本实施例利用冰岛硫化叶菌S.islandicus REY15A CRISPR-Cas10系统Cmr-α核酸蛋白复合物对人工配制的核酸溶液进行靶标RNA定量分析为例来说明:
1.人工配制不同浓度的靶标核酸溶液(4000pM,2000pM,800pM,400pM,300 pM,160pM,32pM,6.4pM,1.28pM,0.256pM),利用野生型Cmr-α核酸蛋白复合物做检测反应;
2.选择黑色酶标板做检测反应,在反应体系中分别加入10μL 10×cleavage buffer (500mM Tris-HCl,100mM MgCl2,100mM MnCl2,100mM DTT),其它反应组分及终浓度分别为:400nM荧光核酸报告探针(/5-HEX/TTTTT/3Dabcyl/),50nM Cmr-α核酸蛋白复合物,最后加水至100μL;
3.在室温下反应5min后用多功能酶标仪检测荧光值(Ex/Em=535/556nm),每5 分钟检测一次,连续检测90分钟,结果显示荧光值随靶标RNA浓度和反应时间的增加而增加;
4.选取反应时间80分钟为节点,绘制荧光值与靶标核酸浓度的曲线(图7),结果显示荧光值和靶标核酸浓度具有较高的线性相关性;进一步选取线性相关性强的靶标核酸浓度范围(2000pM,800pM,400pM,300pM,160pM,32pM,6.4pM)绘制标准曲线,线性方程为y=0.9267x+219.44,R2=0.9987;
5.同实施例1中所述,另取已知浓度为80pM的RNA溶液作为受检样品,得到反应, 80分钟时的荧光值为287;
6.根据步骤4绘制的标准曲线,计算对应的靶标核酸浓度为72.9pM,表明在本实施例采用的反应体系下,核酸浓度在6.4pM-2000pM范围内可以用该方法进行精确定量。
实施例4:
本实施例通过将冰岛硫化叶菌S.islandicus REY15A CRISPR-Cas10系统Cmr-α核酸蛋白复合物中Cmr4α亚基的第27位氨基酸天冬酰胺D突变成丙氨酸A,使得该突变型Cmr- α核酸蛋白复合物丧失对靶标RNA的切割活性,从而提高对人工合成的一段小RNA的检测灵敏度为例来说明:
1.我们采用基于内源CRISPR-Cas系统基因组编辑技术在冰岛硫化叶菌基因组上对c mr4α基因进行定点突变,获得携带Cmr4αD27A的突变株(基因组编辑方法详见公开专利C N201510639204.4),具体步骤如下:
(a)在Cmr4α第27位氨基酸即基因序列第79-81位碱基附近选取protospacer序列,即 5’-ATAGGTTATCCAATAGTCTATGGCTCTAGCTTTAAGGGAG-3’,依据此protospacer 序列合成一对引物Cmr4αD27-SpF/Cmr4αD27-SpR(表1),两条引物通过95℃处理5分钟后自然冷却退火生成一段具有粘性末端的spacer片段然后克隆到pSe-Rp上构建成干涉靶标cmr4α上protospacer的人工mini-CRISPR质粒pAC-4αD27;
(b)分别用表1中的引物对D27A-SalIF/D27A-SOE-R和D27A-SOE-F/D27A-SalIR从冰岛硫化叶菌基因组DNA上扩增出标记Cmr4αD27的左右臂(编码的核苷酸序列为GAT,我们将其变成GCT,使得D27突变成A27),然后采用SOE-PCR的方法将两条同源臂连接起来,进一步将同源臂克隆到步骤(a)中得到的人工mini-CRISPR质粒pAC-4αD27 上,获得基因编辑质粒pGE-Cmr4αD27A。
(c)将基因编辑质粒pGE-Cmr4αD27A电转化入冰岛硫化叶菌,得到突变株Cmr4αD27A,并采用添加5-FOA的培养基消除质粒。
2.将实施例1中的Cmr-α核酸蛋白复合物的共纯化表达质粒pSS1-Cmr6α电转化到本实施例步骤1得到的突变株Cmr4αD27A中,表达共纯化得到携带Cmr4αD27A的突变型Cmr-α核酸蛋白复合物;
3.该核酸蛋白复合物能有效结合靶标RNA,但其不能切割靶标RNA,因此其结合靶标RNA之后能持续具有单链DNA切割活性,有效提高核酸检测灵敏度到100fM。
本发明保护范围不限于上述实施例。
尽管上文已经用一般性说明及具体实施例对本发明做了详尽的描述,但在本发明基础上作一些修改或改进,这对本领域技术人员而言是一望而知的,毋庸赘述。因此,在未偏离本发明主旨的基础上做的或多或少的修改或改进,均属于本发明要求保护的范围。
- 还没有人留言评论。精彩留言会获得点赞!