基于PCR反应的多核苷酸扩增方法与流程
基于pcr反应的多核苷酸扩增方法
技术领域
[0001]
本发明属于多核苷酸扩增领域,具体地涉及基于pcr反应的多核苷酸扩增。
背景技术:
[0002]
单细胞全基因组测序是单细胞研究中最重要最常用的技术之一,能揭示肿瘤等稀有临床样品中的细胞异质性,发现基因组的基因拷贝数变异、小片段插入或缺失、单核苷酸突变及结构变异,评估基因组不稳定性。在癌症发展过程中能够更加精确地挖掘出病理组织样本中的隐藏信息,具有广泛的应用。
[0003]
单细胞甲基化组测序是读取基因组上dna甲基化修饰的分布信息。dna甲基化多发生于cpg二核苷酸序列上,而cpg富集区域多位于基因的启动子区域,一般会导致基因转录受到抑制。dna甲基化水平异常与众多疾病相关,在临床疾病的检测分析上应用广泛。
[0004]
以上两种单细胞测序技术的实施均有赖于有效的基因组扩增。然而,单细胞的基因组起始量低,许多变异信息都仅有一个拷贝,为了满足二代和三代测序仪的测序需要,增加变异检测重复性,需要先对基因组进行扩增。有效的全基因组扩增需要满足以下特点:一、扩增产率高,稳定性强;二、基因组覆盖率高;三、均匀无偏好性;四、扩增错误率低,避免引入假阳性变异。目前已有的单细胞基因组扩增技术包括单纯基于pcr扩增的dop-pcr技术,多重置换扩增(mda)技术,多重退火和基于环的扩增循环(malbac)技术,通过转座子插入进行线性扩增的lianti技术,以及过转座子插入进行长片段扩增匹配三代测序平台的smile-seq技术。其中,malbac技术是目前使用最广泛的单细胞全基因组扩增技术,smile-seq是最新的单细胞全基因组扩增技术。对于单细胞甲基化组测序常用的方法为pbat测序,该方法使用先对基因组dna进行亚硫酸盐转化之后再进行基因组扩增。
[0005]
现有的单细胞基因组扩增技术产率有限并具有一定程度的扩增偏好性,限制了测序得到的基因组覆盖度和相应甲基化修饰位点的数量,难以获得单细胞全面的基因组信息。对于稀有细胞的基因组测定,可能丢失一些重要的单基因位点变异、结构变异和修饰变异信息。目前尚无一种简单通用的方法可以改善这一问题。
技术实现要素:
[0006]
本发明提供了一种基于pcr反应的多核苷酸扩增方法,所述方法包括对待扩增样品进行冷冻处理的步骤。所述方法适于各种基于pcr反应进行多核苷酸扩增的方法,尤其是少量细胞或单细胞的全基因组或甲基化组测序的方法,可以提高多核苷酸扩增的产率,尤其是可以有效提高基于pcr反应的单细胞基因组或甲基化组扩增技术的产率,测序所得的单细胞全基因组序列的基因组覆盖度和单细胞甲基化组的甲基化修饰位点数量。
[0007]
第一方面,本发明提供了一种基于pcr反应的多核苷酸扩增方法,所述方法包括对待扩增样品进行冷冻处理的步骤。
[0008]
在一些实施方案中,所述方法包括在进行pcr扩增前将待扩增样品在-60℃至-90℃的温度下冷冻2天-3个月的时间的步骤。
[0009]
优选地,将待扩增样品在-70℃至-90℃的温度下进行冷冻。
[0010]
优选地,将待扩增样品在-75℃至-90℃的温度下进行冷冻。
[0011]
优选地,将待扩增样品在-80℃至-85℃的温度下进行冷冻。
[0012]
优选地,将待扩增样品在-80℃的温度下进行冷冻。
[0013]
优选地,将待扩增样品冷冻2天-3个月的时间。
[0014]
优选地,将待扩增样品冷冻2天-1个月的时间。
[0015]
优选地,将待扩增样品冷冻2天-20天的时间。
[0016]
优选地,将待扩增样品冷冻2天-10天的时间。
[0017]
优选地,将待扩增样品冷冻2天-7天的时间。
[0018]
优选地,将待扩增样品冷冻7天。
[0019]
冷冻时间的选择标准是保证待扩增样品的扩增质量不受冷冻期间降解的影响。
[0020]
在一些实施方案中,所述基于pcr反应的多核苷酸扩增方法是基于pcr反应的细胞多核苷酸扩增方法。
[0021]
优选地,所述基于pcr反应的细胞多核苷酸扩增方法是基于pcr反应的单细胞多核苷酸扩增方法。
[0022]
优选地,所述基于pcr反应的多核苷酸扩增方法是基于pcr反应的单细胞全基因组或甲基化组的扩增方法。
[0023]
优选地,所述基于pcr反应的多核苷酸扩增方法包括但不限于基于pcr反应的smile-seq单细胞全基因组测序方法、malbac单细胞全基因组测序方法和pbat单细胞甲基化组测序方法。
[0024]
优选地,所述方法可用于单细胞全基因组或甲基化组的扩增与测序中,从而提高多核苷酸扩增产率。
[0025]
优选地,所述方法可用于单细胞全基因组或甲基化组的扩增与测序中,从而获得单细胞基因组测序覆盖度的增加及单细胞甲基化组测序得到的甲基化修饰位点数量的增加。
[0026]
优选地,将单细胞裂解液在-70℃至-90℃的温度下进行冷冻。
[0027]
优选地,将单细胞裂解液在-75℃至-90℃的温度下进行冷冻。
[0028]
优选地,将单细胞裂解液在-80℃至-85℃的温度下进行冷冻。
[0029]
优选地,将单细胞裂解液在-80℃的温度下进行冷冻。
[0030]
优选地,将单细胞裂解液冷冻2天-3个月的时间。
[0031]
优选地,将单细胞裂解液冷冻2天-1个月的时间。
[0032]
优选地,将单细胞裂解液冷冻2天-20天的时间。
[0033]
优选地,将单细胞裂解液冷冻2天-10天的时间。
[0034]
优选地,将单细胞裂解液冷冻2天-7天的时间。
[0035]
优选地,将单细胞裂解液冷冻7天。
[0036]
冷冻时间的选择标准是保证待扩增样品的扩增质量不受冷冻期间降解的影响。
[0037]
在一些实施方案中,所述基于pcr反应的细胞多核苷酸扩增方法包括以下步骤:
[0038]
(1)用细胞裂解液对细胞进行处理获得细胞裂解物;
[0039]
(2)使细胞裂解物中的蛋白酶失活后,进行冷冻处理获得待扩增样品;和
[0040]
(3)对步骤(2)获得的待扩增样品进行pcr扩增。
[0041]
优选地,所述基于pcr反应的细胞多核苷酸扩增方法是基于pcr反应的单细胞多核苷酸扩增方法。
[0042]
优选地,所述基于pcr反应的细胞多核苷酸扩增方法应用于smile-seq单细胞全基因组测序方法。
[0043]
在多核苷酸扩增前,对蛋白酶失活的细胞裂解物进行冷冻处理可以显著提高smile-seq单细胞全基因组扩增产物的产率,而且测序得到基因组覆盖度显著提高。
[0044]
优选地,所述基于pcr反应的细胞多核苷酸扩增方法应用于malbac单细胞全基因组测序方法。
[0045]
在多核苷酸扩增前,对细胞蛋白酶失活的细胞裂解物进行冷冻处理可以显著提高malbac单细胞全基因组扩增产物的产率,而且测序得到的基因组覆盖度显著提高。
[0046]
优选地,所述基于pcr反应的细胞多核苷酸扩增方法应用于pbat单细胞甲基化组测序方法。
[0047]
在多核苷酸扩增前,对细胞蛋白酶失活的细胞裂解物进行冷冻处理可以显著提高pbat单细胞甲基化组扩增产物的产率,而且甲基化修饰位点检出数量显著增加。
[0048]
用细胞裂解液对细胞进行裂解的时间是将足以将细胞裂解的时间,例如约10分钟至约20分钟,约20分钟至约30分钟,约30分钟至约1小时,约1小时至约3小时,约3小时至约6小时,约6小时至约9小时,约9小时至约12小时,约12小时至约15小时,约15小时至约18小时,约18小时至约21小时,或约21小时至约24小时。用细胞裂解液对细胞进行裂解的温育温度也根据具体的细胞和细胞裂解液进行选择,例如在50-75℃的温度范围内,优选地在55-65℃的温度范围内进行温育。
[0049]
细胞裂解液中还可以含有洗涤剂,包括但不限于:十二烷基硫酸钠(sds),月桂基sds,sds c12,triton x-100,triton x-114,np-40,tween-20,tween-80,辛基葡糖苷,辛硫基葡糖苷,3-((3-胆酰胺基丙基)二甲氨基)-1-丙磺酸盐(chaps),十二烷基硫酸锂等。
[0050]
细胞裂解液中还可以含有蛋白酶以消化细胞蛋白质。适用于本发明方法的蛋白酶包括但不限于丝氨酸蛋白酶、枯草杆菌蛋白酶型蛋白酶,例如蛋白酶k、brofasin等。
[0051]
在一些实施方案中,在足以消化细胞蛋白质的条件下,例如在50℃下温育3小时或足以消化细胞蛋白质的任何其它合适的温度和时间,将细胞与蛋白酶k一起温育。
[0052]
在smile-seq单细胞全基因组测序方法中,基因组dna的片段化可以使用转座子和转座酶复合物将序列插入或连接到核酸中,例如,使用tn5复合物进行基因组dna片段化。
[0053]
片段化的基因组dna的大小可选择在25-800bp范围内,例如75-725bp范围内,100-700bp范围内,125-675bp范围内,150-650bp范围内,175-625bp范围内,200-600bp范围内,225-575bp范围内,250-550bp范围内,275-525bp范围内,300-500bp范围内,325-475bp范围内,350-450bp范围内,375-425bp范围内。此外,为了配合三代测序,片段化的基因组dna的大小可选择在3kb-15kb的范围内,例如5kb-13kb范围内,7kb-11kb范围内,例如9kb-10kb范围内。
[0054]
malbac反应中,简并随机引物组退火至基因组dna并延伸,在2个周期后,在延伸和变性后形成发夹环。这些发夹不参与后面的反应循环,这种“准线性”预扩增之后,使用采用单个引物的pcr以指数方式扩增环状产物。
[0055]
本发明的基于pcr反应的多核苷酸扩增方法的一个优点是可应用于单细胞基因组测序及单细胞甲基化组测序中,所述单细胞基因组测序包括但不限于smile-seq测序、malbac测序。单细胞甲基化组测序包括但不限于pbat测序,对多核苷酸扩增产率具有显著性提升,对smile-seq测序和malbac测序得到的基因组覆盖率有显著性提升,而且对pbat测序得到的甲基化修饰位点检出数量有显著性提高。
[0056]
本发明的基于pcr反应的多核苷酸扩增方法的另一个优点是简单易行,不需要采购其它试剂耗材,仅需要将单细胞裂解样品放入实验室常规配有的-80℃冰箱冷冻3天-3个月即可,具有很高的可操作性和可推广性。
[0057]
本发明的方法的其他优点还表现在扩展性强,可应用于不同种类的基于pcr反应的基因组扩增技术,尤其是单细胞基因组和甲基化组测序中,所述单细胞基因组测序包括但不限于smile-seq测序、malbac测序;单细胞甲基化组测序包括但不限于pbat测序,本发明的方法可以随着基于pcr反应的基因组扩增技术的改进进行相应的适配。
[0058]
扩增中所需的样本量为单细胞或微量的细胞样本。
[0059]
第二方面,本发明提供了第一方面的基于pcr反应的多核苷酸扩增方法在基因组和甲基化测序或分析中的应用。
[0060]
在一些实施方案中,所述的基于pcr反应的多核苷酸扩增方法可用于单细胞基因组测序。
[0061]
在一些实施方案中,所述的基于pcr反应的多核苷酸扩增方法可用于cgh,单核苷酸多态性分型(snp),str分型,单核苷酸变异分析(snv),拷贝数变异(cnv)定量或sscp分析。
[0062]
在一些实施方案中,所述的基于pcr反应的多核苷酸扩增方法可用于单细胞基因分型,产前筛查,肿瘤细胞基因组的异质性检测,刑侦现场痕量取证,和微生物单细胞变异检测。
[0063]
以肿瘤细胞为例,同一患者的原发肿瘤和转移肿瘤存在差异,又或者同一肿瘤组织内的个体细胞间也存在差异,本发明的基于pcr反应的多核苷酸扩增方法可以深入了解个体肿瘤间的遗传异质性,包括,单细胞分辨率下的基因组异质状态,表观遗传特性,细胞空间动态,以及肿瘤微环境。具体而言,本发明的基于pcr反应的多核苷酸扩增方法可以用于肿瘤单细胞测序,循环肿瘤细胞单细胞测序。
[0064]
以产前筛查为例,本发明的基于pcr反应的多核苷酸扩增方法可以用于体外受精的植入前遗传学诊断,单生殖细胞测序。
[0065]
本发明的基于pcr反应的多核苷酸扩增方法还可以用于追踪造血干细胞的分化。
[0066]
单细胞全基因组测序技术,是在单细胞水平对全基因组进行扩增与测序的一项新技术。其原理是将分离的单个细胞的微量全基因组dna进行扩增,获得高覆盖率的完整的基因组后进行高通量测序用于揭示细胞群体差异和细胞进化关系。
[0067]
基因组覆盖度,是指测序获得的序列占整个靶序列如基因组序列的比例。由于基因组中的高gc、重复序列等复杂结构的存在,测序最终拼接组装获得的序列往往无法覆盖所有的区域。例如,如果基因组测序的覆盖度是98%,就意味着2%的序列区域未通过测序获得。单细胞基因组覆盖度是指单细胞基因组扩增后测序得到的基因组序列占整个基因组(参考基因组)序列的百分比。
[0068]
术语“核酸”、“核酸分子”、“寡核苷酸”和“多核苷酸”可互换使用,并且是指任何长度的核苷酸的聚合形式,即脱氧核糖核苷酸或核糖核苷酸,或其类似物。术语涵盖例如dna,rna及其改性形式。多核苷酸可以具有任何三维结构,并且可进行任何已知或未知的功能。多核苷酸的非限制性实例包括基因、基因片段、外显子、内含子、信使rna(mrna)、转移rna、核糖体rna、核酶、cdna、重组多核苷酸、支链多核苷酸、质粒、载体、任何序列的分离的dna、控制区,任何序列的分离的rna、核酸探针和引物。核酸分子可以是线性的或环状的。
附图说明
[0069]
图1显示了实施例1中应用样品冷冻方法提高smile-seq单细胞全基因组测序的产率及基因组覆盖度的结果。
[0070]
图2显示了实施例2中应用样品冷冻方法提高malbac单细胞全基因组测序的产率及基因组覆盖度的结果。
[0071]
图3显示了实施例3中应用样品冷冻方法提高pbat单细胞甲基化组测序的产率及cpg位点即甲基化修饰位点检出数量的结果。
具体实施例
[0072]
以下所述的是本发明的优选实施方式,本发明所保护的不限于以下优选实施方式。应当指出,对于本领域的技术人员来说在此发明创造构思的基础上,做出的若干变形和改进,都属于本发明的保护范围。所用试剂未注明生产商者,均为可以通过市购获得的常规产品。
[0073]
实施例1样品冷冻提高smile-seq单细胞全基因组测序的产率及基因组覆盖度以k562细胞的smile-seq测序为例,具体包括如下步骤:
[0074]
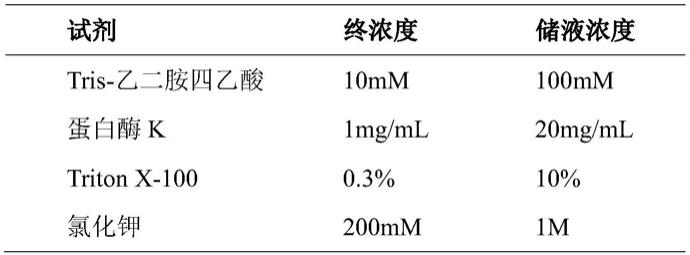
(1)单细胞裂解:细胞裂解液的配方如下表1所示:
[0075]
表1:
[0076][0077]
将单个细胞移入独立的反应管中,每管中加入2.5μl的上述细胞裂解液,震荡混合后,50℃孵育3小时,然后70℃孵育30分钟使蛋白酶充分失活,在进行下一步操作前,将实验组样品冻存于-80℃,3天后继续下一步,对照组直接进入下一步。
[0078]
(2)tn5转座酶接头包埋:将下表3所示的dna序列接头稀释到100μm,1:1退火反应得到接头混合液,在pcr仪中进行梯度退火反应:75℃,15分钟;60℃,10分钟;50℃,10分钟;40℃,10分钟;25℃,30分钟。包埋体系如下表2所示,充分吹打混匀,30℃孵育1小时,得到tn5转座酶复合物,于-20℃保存。
[0079]
表2:
[0080][0081]
表3:
[0082][0083]
(3)转座子介导的dna片段化:片段化反应液配方如下表4所示。向2.5μl的细胞裂解产物中加入7.5μl片段化反应液,温和吹打几次使混匀。在pcr仪上进行如下反应:55℃,10分钟,之后于4℃持温。加入2.5μl的0.2%sds终止反应,混匀后室温放置5分钟完成dna片段化。
[0084]
表4:
[0085][0086]
(4)pcr扩增反应:向上一步反应产物中加入37.5μl的pcr扩增反应液使最终反应体系为50μl,pcr扩增反应液的配方如下表5所示:
[0087]
表5:
[0088][0089]
pcr扩增程序如下表6所示:
[0090]
表6:
[0091][0092]
pcr引物i5-pb包含了16bp的随机条码序列和14bp与模板dna末端接头互补的锚定序列,具体序列参见下表7:
[0093]
表7:
[0094][0095]
(5)样品混合及纯化:将连接上不同细胞条码的单细胞基因组dna扩增样品混合在一起,用0.4
×
ampure pb磁珠纯化,先将0.4倍体积的磁珠与产物混合,室温孵育5分钟,然后置于磁力架上,待磁珠吸附到侧壁,去除上清。磁珠用80%乙醇洗涤2次,晾干,用适量体积的水溶液重悬磁珠,室温静置2分钟后,置于磁力架上,上清产物转移到新的样品管中。重复纯化步骤一次,纯化产物溶解在适量缓冲液中使终浓度大于50ng/μl以满足后续三代测序文库构建的起始要求。
[0096]
(6)文库构建和测序:使用trueprep dna library prep kit v2 for illumina(vazyme,cat:td502)对扩增产物进行文库构建。在illumina hiseq xten测序平台进行上机测序。
[0097]
实验结果如图1所示,经过3天-80℃冻存的实验组smile-seq单细胞全基因组扩增产物的产率相比于对照组更高。经过二代建库测序,实验组测序数据的全基因组覆盖度更高。实验组单细胞全基因测序所得序列与参考基因组相比所占比例显著提高,增加的部分为对照组扩增偏好性导致丢失的部分,降低了扩增偏好性。图1a对比了非冷冻处理和冷冻处理样品使用smile-seq测序方法进行扩增后的dna产率,发现应用样品冷冻方法处理的实验组产率显著高于非冷冻处理组;图1b对比了非冷冻处理和冷冻处理样品使用smile-seq测序方法进行建库测序结果的基因组覆盖率,发现应用样品冷冻方法处理的实验组基因组覆盖率显著高于非冷冻处理组。
[0098]
实施例2样品冷冻提高malbac单细胞全基因组测序的产率及基因组覆盖度
[0099]
以k562细胞的malbac测序为例,具体包括如下步骤:
[0100]
(1)单细胞裂解:细胞裂解液配方如下表8所示。将单个细胞移入独立的反应管中,
每管中加入5μl细胞裂解液,置于冰上,7000rpm离心1分钟。50℃孵育3小时,然后70℃孵育30分钟使蛋白酶充分失活,在进行下一步操作前,将实验组样品冻存于-80℃,7天后继续下一步,对照组直接进入下一步。
[0101]
表8:
[0102][0103]
gat3g引物序列如下表9所示:
[0104]
表9:
[0105][0106]
(2)准线性预扩增:在每个单细胞反应体系中加入30μl线性扩增混合液,扩增混合液配方如下表10所示。7000rpm离心1分钟后立刻置于冰上。之后进行pcr扩增,扩增后立刻置于冰上。
[0107]
表10:
[0108][0109]
gat3t引物的序列如下表11所示:
[0110]
表11:
[0111][0112]
pcr扩增程序如下表12所示:
[0113]
表12:
[0114][0115][0116]
(3)指数性扩增:在每个单细胞反应体系中加入30μl指数扩增混合液,震荡混匀,扩增混合液配方如下表13所示。7000rpm离心1分钟后立刻置于冰上,之后进行pcr扩增。
[0117]
表13:
[0118][0119]
gatcom引物的序列如下表14所示:
[0120]
表14:
[0121][0122]
pcr扩增程序如下表15所示:
[0123]
表15:
[0124][0125]
(4)扩增产物纯化:用pcr产物纯化试剂盒对上一步扩增产物进行纯化。
[0126]
(5)建库及测序:使用trueprep dna library prep kit v2 for illumina(vazyme,cat:td502)对扩增产物进行文库构建。在illunima hiseq xten测序平台进行上机测序。
[0127]
实验结果如图2所示,经过7天-80℃冻存的实验组malbac单细胞全基因组扩增产物的产率相比于对照组更高。经过二代建库测序,实验组测序数据的全基因组覆盖度更高。实验组单细胞全基因测序所得序列与参考基因组相比所占比例显著提高,增加的部分为对照组扩增偏好性导致丢失的部分,降低了扩增偏好性。具体而言,图2a对比了非冷冻处理和冷冻处理样品使用malbac测序方法进行扩增后的dna产率,发现应用样品冷冻方法处理的实验组产率显著高于非冷冻处理组;图2b对比了非冷冻处理和冷冻处理样品使用malbac测序方法进行建库测序结果的基因组覆盖率,发现应用样品冷冻方法处理的实验组基因组覆盖率显著高于非冷冻处理组。
[0128]
实施例3样品冷冻提高了pbat单细胞甲基化组测序的产率及cpg位点检出的数量
[0129]
以k562细胞的pbat测序为例,具体包括如下步骤:
[0130]
(1)单细胞裂解:细胞裂解液配方如下表16所示,将单个细胞移入独立的反应管中,每管中加入4.5μl细胞裂解液。50℃孵育3小时,然后75℃孵育30分钟使蛋白酶充分失活。9000g,4度离心1分钟,立刻置于冰上。下一步操作前,将实验组样品冻存于-80℃,7天后继续下一步,对照组直接进入下一步。
[0131]
表16:
[0132][0133]
(2)单细胞基因组dna的亚硫酸盐转化:使用ez-96dna methylation-direct
tm
magprep进行亚硫酸盐转化。在单细胞裂解体系中加入32.5μl转化试剂。将转化体系置于98℃孵育8分钟,然后64℃孵育3.5小时,最后4℃持温。最终基因组dna溶解于20μl浓度为10mm的tris-盐酸ph 8.5缓冲液中。
[0134]
(3)第一轮随机引物结合:取19.5μl亚硫酸盐转化的dna溶解液,加入4.5μl第一轮随机引物混合液,混合液配方如下表17所示。将混合体系置于65℃孵育3分钟,然后4℃持温。之后加入1μl浓度为50u/μl的恒温dna聚合酶(无外切酶活性)。将混合体系置于4℃孵育5分钟,之后每15秒升温1度直到达到25℃,维持5分钟,然后再每15秒升温1度直到37℃,在37℃孵育30分钟。
[0135]
表17:
[0136][0137]
scbs-p5-n6-oligo1引物序列如下表18所示:
[0138]
表18:
[0139][0140]
(4)第二轮随机引物结合:将上述反应液置于95℃孵育1分钟,之后立刻置于冰上。加入2.5μl第二轮随机引物反应混合液,混合液配方如下表19所示,其中scbs-p5-n6-oligo1和三磷酸脱氧核糖核苷酸储液均由1
×
neb缓冲液2号按1:1000的比例稀释得到。将混合体系置于4℃孵育5分钟,之后每15秒升温1度直到达到37℃,在37℃孵育30分钟。
[0141]
表19:
[0142][0143][0144]
(5)重复第二轮随机引物结合:对于单细胞需要重复直到完成四轮随机引物结合。
[0145]
(6)去除过量随机引物。在上述体系中加入2μl浓度为20u/μl的核酸外切酶,在37℃孵育1小时。用0.8
×
ampure pb磁珠纯化,先将0.8倍体积的磁珠与产物混合,室温孵育5分钟,然后置于磁力架上,待磁珠吸附到侧壁,去除上清。磁珠用80%乙醇洗涤2次,晾干,用39μl的浓度为10mm的tris-盐酸ph8.5缓冲液重悬磁珠,室温静置2分钟后,置于磁力架上,上清产物转移到新的样品管中。
[0146]
(7)第二链合成:在纯化后的dna中加入9μl第二链合成反应混合液,混合液配方如下表20所示。将上述反应液置于95℃孵育45秒,之后立刻置于冰上。加入2μl浓度为50u/μl的恒温dna聚合酶(无外切酶活性),将混合体系置于4℃孵育5分钟,之后每15秒升温1度直到达到37℃,在37℃孵育30分钟。用0.8
×
ampure pb磁珠纯化,先将0.8倍体积的磁珠与产物混合,室温孵育5分钟,然后置于磁力架上,待磁珠吸附到侧壁,去除上清。磁珠用80%乙醇洗涤2次,晾干,用23μl的浓度为10mm的tris-盐酸ph 8.5缓冲液重悬磁珠,室温静置2分钟后,置于磁力架上,上清产物转移到新的样品管中。
[0147]
表20:
[0148][0149]
scbs-p7-n6-oligo2引物序列如下表21所示:
[0150]
表21:
[0151][0152]
(8)pcr扩增:在上述23μl的纯化dna体系中加入50μlpcr反应体系,反应体系配方如下表22所示。
[0153]
表22:
[0154][0155]
pcr扩增程序如下表23所示:
[0156]
表23:
[0157][0158]
(9)扩增产物纯化:用0.8
×
ampure pb磁珠纯化,先将0.8倍体积的磁珠与产物混合,室温孵育5分钟,然后置于磁力架上,待磁珠吸附到侧壁,去除上清。磁珠用80%乙醇洗涤2次,晾干,用25μl的浓度为10mm的tris-盐酸ph8.5缓冲液重悬磁珠,室温静置2分钟后,置于磁力架上,上清产物转移到新的样品管中。
[0159]
(10)测序:在illumina novaseq测序平台进行上机测序。
[0160]
如图3所示,结果表明,经过7天-80℃冻存的实验组pbat单细胞甲基化组扩增产物的产率相比于对照组更高。经过二代测序,实验组测序得到的cpg位点检出数量更多。具体而言,图3a对比了非冷冻处理和冷冻处理样品使用pbat测序方法进行扩增后的dna产率,发现应用样品冷冻方法处理的实验组产率显著高于非冷冻处理组;图3b对比了非冷冻处理和冷冻处理样品使用pbat测序方法进行建库测序得到的cpg位点检出数量,发现应用样品冷冻方法处理的实验组cpg位点检出数量显著高于非冷冻处理组。cpg位点即甲基化修饰位点。
[0161]
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1