前列腺癌基因谱和使用所述基因谱的方法与流程
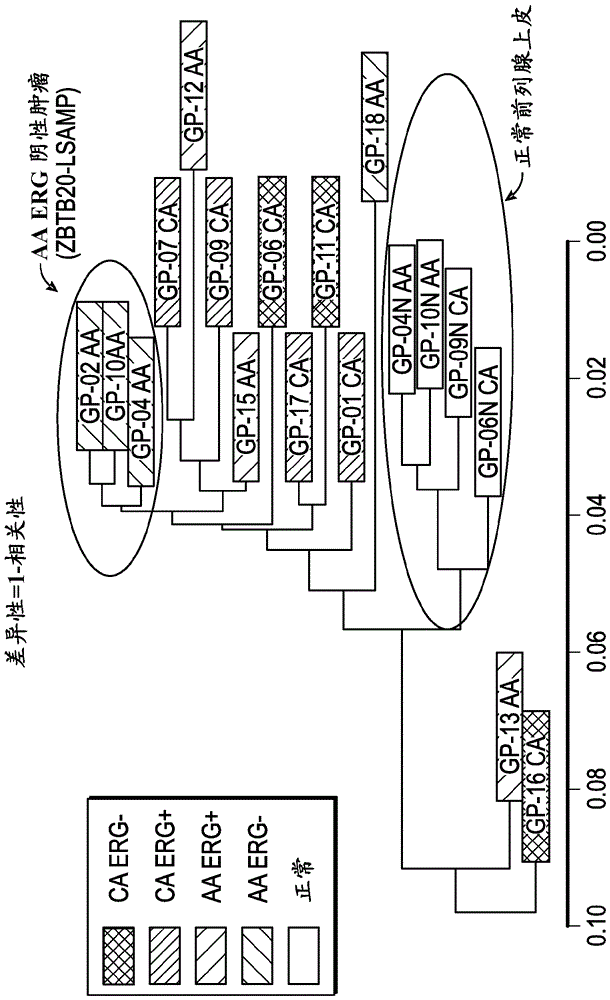
本申请要求2013年12月30日提交的美国临时专利申请号61/921,739的权益,并依赖于其提交日期,其整个内容通过引用结合到本申请中。政府利益本发明部分地在政府支持下完成。政府在本发明中享有一定权益。序列表本申请含有电子提交的ASCII格式的序列表,并通过引用以其整体结合到本文中。2014年12月29日创建的所述ASCII副本名为HMJ-145-PCT_SL.txt,大小为344,410字节。发明背景到2013年,估计238,590名男性将被诊断为前列腺癌(CaP),并估计29,720名男性将死于该疾病[1]。这种恶性肿瘤是美国男性癌症相关死亡的第二主要原因。另外,与其它人种相比,非裔美国(AA)男性具有因CaP所致的最高发病率和死亡率[1]。从表现和诊断到治疗、生存和生命质量,都存在人种差异[2]。研究人员提出社会经济地位(SES)对包括CaP特异性死亡率在内的这些差异产生显著影响[3]。同样,有证据显示,获得护理的机会减少与CaP结局差有关,所述结局差在AA男性中比在高加索美国(CA)男性中更普遍[4]。然而,存在其中AA男性具有与CA男性类似的结局的群体。Sridhar和同事[5]发表了元分析,其中他们得出结论,当说明SES时,在AA和CA男性之间在总体生存和CaP特异性生存方面没有差异。类似地,军人和退伍军人群体(同样的访问和筛选系统)在种族间在生存方面未观察到差异[6],且到2000年代早期在退伍军人组群中在诊断时病理期的差异缩小[7]。值得注意的是,这两项研究表明,与CA男性相比,AA男性更可能具有较高的Gleason评分和PSA水平[6,7]。虽然社会经济因素可能对CaP结局产生影响,但是它们似乎无法说明与诊断和疾病风险有关的所有变量。若干研究支持与CA男性相比AA男性具有较高的CaP发病率[1,8,9]。研究还显示AA男性具有诊断时显著较高的PSA、活组织检查中较高等级的疾病、各阶段较大的肿瘤体积和根治性前列腺切除术前较短的PSA倍增时间[10-12]。在有关应激和炎性反应的肿瘤微环境中注意到CA和AA男性的前列腺癌之间的生物学差异[13]。虽然在生物学差异的作用方面仍有争议,但是所表现的发病率和疾病侵袭性中观察到的差异表明AA和CA男性之间前列腺癌病变的不同途径的潜在作用。在过去十年内,大量研究集中在癌基因的改变及其在CaP中的作用[14-16]。在CaP中过量表达并且是CaP中最普遍已知的癌基因的TMPRSS2/ERG基因融合物中注意到种族性和人种间流行率的变化[17,18]。累计数据表明在种族性间ERG致癌变化有差异[17,19-21]。在描述ERG过量表达和ERG剪接变体的最初的论文中注意到与AA男性相比CA男性中显著较大的ERG表达[17,21]。该差异在有高Gleason等级(8-10)肿瘤的患者中在CA和AA之间甚至更明显(50%与16%)。因此,ERG是这些人种群(ethnicgroup)间的主要体细胞基因改变。但除TMPRSS2/ERG以外,对有关AA和CA男性间的CaP差异的遗传基础知之甚少,甚至未知[24]。因此,需要对不同种族人群有特异性并提供更准确的诊断和/或预后潜力的新的生物标志物和治疗标志物。因此,可利用非洲和高加索血统患者的单独基因表达谱以诊断或预测不同种族人群中的CaP,并根据这些种族特异性基因表达特性提供更有信息的治疗选择。发明概述本公开内容提供与前列腺癌有关的基因表达谱和使用所述基因表达谱的方法。可利用基因表达谱检测样品中的前列腺癌细胞或预测患者发生前列腺癌的可能性。基因表达谱也可用来评价前列腺癌的严重程度或病期或评估疗法的有效性或在治疗后监测前列腺癌的进程或消退(例如手术后的无病复发)。可测量核酸或蛋白质水平下的基因表达谱。一方面,基因表达谱是非洲血统患者特有的。另一方面,基因表达谱是高加索血统患者特有的。因此,一方面涉及与非洲血统患者中的前列腺癌有关的基因表达谱,其中基因表达谱包含下列基因的组合:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。另一方面涉及与高加索血统患者中的前列腺癌有关的基因表达谱,其中基因表达谱包含下列基因的组合:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN。又一方面涉及代表两个人种群中前列腺癌的最大差异表达基因的基因表达谱,并包括下列基因的组合:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4。在某些实施方案中,基因表达谱至少包含DLX1和NKX2-3。在一个实施方案中,该组合至少包括DLX1和NKX2-3。这些基因谱可用于收集用于诊断或预测前列腺癌的数据的方法,所述方法包括测量已公开的基因谱之一中代表性数目的基因的表达,其中在获自患者的样品中测量基因表达。所收集的基因表达数据可用来预测受试者是否患有前列腺癌或将发生前列腺癌或预测前列腺癌的病期或严重程度。所收集的基因表达数据还可用于知会有关治疗或监测患者的决定。鉴于这些独特基因表达谱的鉴定,本领域技术人员可确定哪一个已鉴定的基因包括在基因谱型分析中。基因的代表性数目可包括列于具体谱中的基因的全部或基因的某些较少数目,例如3或4或更多个。在某些实施方案中,该方法进一步包括检测与前列腺癌有关的一个或多个其它基因的表达,包括但不限于ERG、PSA和PCA3。另一方面涉及用于诊断或预测前列腺癌的试剂盒。在一个实施方案中,试剂盒被设计成用于诊断或预测非洲血统患者的前列腺癌,并包含用于检测下列基因(或由所述基因编码的多肽)的至少1个(优选至少3个)的多种探针:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。在某些实施方案中,多种探针选自多种寡核苷酸探针、多种抗体或多种多肽探针。在其它实施方案中,多种探针含有用于不超过500、100、50、25、15、10、9、8、7、6、5、4、3或2个基因(或多肽)的探针。在一个实施方案中,试剂盒进一步包含用于检测与前列腺癌有关的一个或多个其它基因的表达的探针,包括但不限于ERG、PSA和PCA3。在另一个实施方案中,试剂盒被设计成用于诊断或预测高加索血统患者的前列腺癌,并包含用于检测下列基因(或由所述基因编码的多肽)的至少1个(优选至少3个)的多种探针:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN。在某些实施方案中,多种探针选自多种寡核苷酸探针、多种抗体或多种多肽探针。在其它实施方案中,多种探针含有用于不超过500、100、50、25、15、10、9、8、7、6、5、4、3或2个基因(或多肽)的探针。在某些实施方案中,该方法进一步包括检测与前列腺癌有关的一个或多个其它基因的表达,包括但不限于ERG、PSA和PCA3。在另外又一个实施方案中,用于诊断或预测前列腺癌的试剂盒包含用于检测下列基因(或由所述基因编码的多肽)的至少1个(优选至少4个)的多种探针:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4。在一个实施方案中,所述基因包含DLX1和/或NKX2-3。在某些实施方案中,多种探针选自多种寡核苷酸探针、多种抗体或多种多肽探针。在其它实施方案中,多种探针含有用于不超过500、100、50、25、15、10、9、8、7、6、5、4、3或2个基因(或多肽)的探针。在某些实施方案中,该方法进一步包括检测与前列腺癌有关的一个或多个其它基因的表达,包括但不限于ERG、PSA和PCA3。在相关方面,本公开内容提供用于诊断和/或预测前列腺癌的阵列。在一个实施方案中,阵列包含(a)基质和(b)固定在基质上用于检测下列人基因的至少3个的表达的多种探针:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。在另一个实施方案中,阵列包含(a)基质和(b)固定在基质上用于检测下列人基因的至少3个的表达的多种探针:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN。在另外又一个实施方案中,阵列包含(a)基质和(b)固定在基质上用于检测下列人基因的至少4个的表达的多种探针:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4。在某些实施方案中,阵列进一步包含用于检测与前列腺癌有关的一个或多个其它基因的表达的探针,包括但不限于ERG、PSA和PCA3。探针优选排列在可寻址元件(addressableelement)内的基质上以利于检测。优选阵列包含有限数量的可寻址元件使得将阵列与较综合的阵列(例如基因组阵列等)区分开来。因此,在一个实施方案中,阵列包含500个或更少个可寻址元件。在另一个实施方案中,阵列包含不超过250、100、50或25个可寻址元件。在另一个实施方案中,将不超过1000种多核苷酸探针固定在阵列上。另一方面,本公开内容提供使用本文所述阵列检测生物样品中的基因表达的方法。使用检测基因表达的这些阵列也可以是用于检测或预测生物样品中的前列腺癌的方法的一部分。另一方面,本公开内容提供使用基因表达谱以鉴定需要前列腺癌治疗的患者的方法。在一个实施方案中,患者具有非洲血统,并且所述方法包括a)测试患者生物样品的多个基因的过量表达,其中因为患者具有非洲血统而选择所述多个基因,并且所述多个基因包含下列基因的至少3个:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1;和b)如果与对照样品或阈值相比,生物样品中COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1基因的一个或多个过量表达,则鉴定患者为需要前列腺癌治疗。在另一个实施方案中,患者具有高加索血统,且所述方法包括a)测试患者生物样品的多个基因的过量表达,其中因为患者具有高加索血统而选择所述多个基因,并且所述多个基因包含下列基因的至少4个:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3;和b)如果与对照样品或阈值相比,生物样品中PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3基因的一个或多个过量表达,则鉴定患者为需要前列腺癌治疗。在某些实施方案中,该方法进一步包括检测与前列腺癌有关的一个或多个其它基因的表达,包括但不限于ERG、PSA和PCA3。所述方法还进一步包括治疗患者的步骤。附图简述并入且构成本说明书的一部分的附图阐明某些实施方案,并且与书面描述一起,用来解释本文公开的抗体和方法的某些原理。图1显示采用平均连锁方法(average-linkagemethod)的14个肿瘤和4个正常样品的分层聚类分析(hierarchicalclusteringanalysis,HCA)。HCA揭示正常患者的不同聚类(GP-04、GP-10、GP-09和GP-06)和AA、ERG融合阴性患者的另一种不同的聚类(GP-02、GP-10和GP-04)。聚类基于基因的表达水平。所有组都被彩色编码。图2A是具有14个肿瘤和4个正常样品聚类的热图,其中非裔美籍患者在热图的左侧,CA在热图的右侧。热图中存在的基因是AA和CA患者的过量表达和表达不足基因(肿瘤与正常)的重叠。图2B提供在来自AA和CA患者的肿瘤和正常样品两者中最前3种过量表达基因的表达值(log2)。图3A是显示在AA患者中始终过量表达且在CA患者中同时表达不足或显示无变化的基因的热图。图3B是显示在CA患者中始终过量表达且在AA患者中同时表达不足或显示无变化的基因的热图。图4显示本发明一些实施方案的系统的示意图。具体地说,该图说明按照所公开的系统和方法可用于计算机系统106执行的各种硬件、软件和其它资源。在所示实施方案中,计算机系统106可包括与在操作系统控制下或与操作系统联合操作的随机存储器连接的一个或多个处理器110。实施方案中的处理器110可包括在一个或多个服务器、群集器或其它计算机或硬件资源中,或可采用基于云的资源执行。操作系统可为例如LinuxTM操作系统、UnixTM操作系统或其它开放源码或专有操作系统或平台的分配。处理器110可与数据存储112(例如保存在硬盘驱动器或驱动器阵列上的数据库)通信以访问或保存程序指令或其它数据。处理器110可进一步通过网络接口108通信,网络接口108进而可通过一个或多个网络104(例如互连网或其它公共或专用网络)通信,使得可从客户机102或其它装置或服务程序中接受查询或其它要求。另外,处理器110可利用网络接口108通过一个或多个网络104向用户发送信息、指令、工作流查询、部分工作流或其它数据。网络接口104可包括一个或多个服务器或与一个或多个服务器通信连接。客户机102可为例如与互连网连接的个人计算机。一般而言,可对处理器110进行编程或配制以执行实施本文公开的方法的控制逻辑和控制操作。处理器110可与协处理器114进一步通信连接(即通过通信信道连接)。协处理器114可为经配制以实施本文公开的方法的专用硬件和/或固件部件。因此,可通过处理器110和/或协处理器114实施本文公开的方法。计算机系统106的其它配置、相关网络连接和其它硬件、软件和服务程序资源是可能的。发明详述下面将详细参考不同的示例性实施方案,在附图中说明了其实例。要了解下面的详细描述为了使读者更全面地理解本发明方面的某些实施方案、特征和详细资料而提供,不应解释为限制本发明的范围。定义为了更容易地理解本发明,首先定义了某些术语。在整个详细描述中给出了其它定义。术语“非洲血统的”是指自认为具有非洲血统的个人,包括自认为是非洲裔美国人的个人和已确定具有与非洲祖先相关的遗传标记(亦称祖先信息标记(AIM))的个人,所述祖先信息标记例如JudithKidd等,Analysesofasetof128ancestryinformativesingle-nucleotidepolymorphismsinaglobalsetof119populationsamples(整套119群样品中一套128种提供祖先信息的单核苷酸多态性的分析),InvestigativeGenetics,(2):1,2011(所述参考资料通过引用以其整体予以结合)中鉴定的AIM。术语“高加索血统的”是指自认为具有高加索血统的个人,包括自认为是高加索美国人的个人和已确定具有与高加索(例如欧洲、北非或亚洲(西亚、中亚或南亚)祖先相关的遗传标记(亦称祖先信息标记(AIM))的个人,祖先信息标记例如JudithKidd等,Analysesofasetof128ancestryinformativesingle-nucleotidepolymorphismsinaglobalsetof119populationsamples(整套119群样品中一套128种提供祖先信息的单核苷酸多态性的分析),InvestigativeGenetics,(2):1,2011(所述参考资料通过引用以其整体予以结合)中鉴定的AIM。术语“抗体”是指免疫球蛋白或其抗原结合片段,并包括包含抗原结合片段或抗原结合结构域的任何多肽。该术语包括但不限于多克隆、单克隆、单特异性、多特异性、人源化、人、单链、嵌合、合成、重组、杂合、突变、移植和体外产生的抗体。除非之前有措词“完整”,否则术语“抗体”包括抗体片段例如Fab、F(ab')2、Fv、scFv、Fd、dAb和保留抗原结合功能的其它抗体片段。除非另有说明,否则抗体不必来自任何特殊来源,也不通过任何特殊方法产生。术语“检测”意指本领域已知的用于测定核酸或蛋白质的存在或量的各种方法的任一种。如本说明书中所用,术语“检测”包括定性或定量检测。术语“基因表达谱”是指样品中多个基因的表达水平。如本领域所理解的一样,可通过测量核酸(例如基因组DNA或mRNA)或由核酸编码的多肽的表达,分析基因的表达水平。术语“分离的”当在多肽或核酸的情况下使用时是指其天然环境基本上没有,因此可与可能天然存在的多肽或核酸区分开来的多肽或核酸。例如,分离的多肽或核酸基本上不含细胞物质或其来源于其中的细胞或组织源的其它多肽或核酸。术语“多肽”、“肽”和“蛋白质”在本文可互换使用,是指氨基酸的聚合物。本文所用术语“多肽探针”是指可用于蛋白质检测测定法(例如质谱法)以定量测定生物样品中的目标多肽的标记的(例如同位素标记的)多肽。术语“引物”意指能够与靶核酸的区域或其互补序列结合,并促进靶核酸的核酸扩增的多核苷酸。一般来讲,引物可具有可通过核酸聚合酶延伸的游离3’端。引物一般还包括能够通过直接与靶核酸的至少1条链或与该靶序列互补的链进行互补碱基相互使用而杂交的碱基序列。引物可包含靶标特异性序列和任选不与靶序列互补的其它序列。这些非互补序列可包含例如启动子序列或限制性内切核酸酶识别位点。“变异”或“变体”是指在小至单一碱基或较长间隔上不同于参比的等位基因序列。术语“ERG”或“ERG基因”是指Ets相关基因(ERG),其被指定唯一的HugoGeneNomenclatureCommittee(HGNC)标识码:HGNC:3446,并包括在前列腺癌中是普遍的ERG基因融合产物,包括TMPRSS2-ERG融合产物。分析ERG或ERG基因的表达包括分析与前列腺癌有关的ERG基因融合产物的表达,例如TMPRSS2-ERG。前列腺癌中的基因表达谱采用下一代测序技术以鉴定CaP的新的生物标志物和治疗靶标。对7名高加索血统患者和7名非洲血统患者(包括各患者的匹配对照的共28个样品)的组群间的获自组织学上确定的和精确解剖的原发CaP样本(80-95%肿瘤,主要Gleason模式3)的高质量基因组序列数据和覆盖度进行了比较,以评价所观察到的2个人种群之间CaP发病率和死亡率的差异。这些数据和分析提供来自针对临床病理特征匹配的非洲和高加索血统的CaP患者的前列腺癌基因组的第一手评价。两个人种群中CaP的最前面的差异表达基因包括:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4。因此,从生物样品中收集这些基因的至少2、3、4、5、6、7、8、9或10个的表达数据提供唯一的基因表达谱用于诊断或预测受试者的前列腺癌。某些实施方案涉及收集数据用于诊断或预测CaP的方法,所述方法包括检测生物样品中下列基因的至少2、3、4、5、6、7、8、9、10、11、12、13、14或15个的表达:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4。所述方法可任选包括从受试者中获得生物样品的额外步骤。所述方法可任选包括利用所收集的基因表达数据诊断或预测CaP的额外步骤。在一个实施方案中,与对照样品或阈值相比,下列基因的至少2、3、4、5、6、7、8、9、10、11、12、13、14或15个的过量表达:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4,表明生物样品中存在CaP或发生CaP的可能性增加。收集数据或诊断和/或预测CaP的方法可进一步包括检测与前列腺癌有关的其它基因的表达,包括但不限于ERG、PSA和PCA3。在某些实施方案中,所述方法包括检测不超过100、90、80、70、60、50、40、30、25、20、15、14、13、12、11、10、9、8、7、6、5、4、3或2个基因的表达。在一个实施方案中,所述方法包括检测DLX1和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测NKX2-3和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测DLX1和NKX2-3和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测PHGR1和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测THBS4和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测GAP43和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测FFAR2和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测GCNT1和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测SIM2和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测STX19和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测的表达KLB和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测APOF和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测LOC283177和表1所列一个或多个其它基因的表达。在另一个实施方案中,所述方法包括检测TRPM4和表1所列一个或多个其它基因的表达。已知人DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4的核酸和氨基酸序列。表1提供这些基因的由HugoGeneNomenclatureCommittee(HGNC)和EntrezGene指定的唯一标识码和代表性序列的登记号,所述序列通过引用以其整体结合到本文中。表1基因HGNCIDEntrezGeneID登记号DLX129141745NM_178120.4,GI:84043957NKX2-37836159296NM_145285.2 GI:148746210CRISP31690410321NM_006061.2 GI:300244559PHGR137226644844NM_001145643.1 GI:224548949THBS4117887060NM_003248.4 GI:291167798AMACR45123600NM_014324.5 GI:266456114GAP4341402596AK091466.1 GI:21749841FFAR245012867NM_005306.2 GI:227430361GCNT142032650NM_001097634.1 GI:148277030SIM2108336493NM_005069.3 GI:194239685STX1919300415117NM_001001850.2 GI:344313159KLB15527152831NM_175737.3 GI:198041706APOF615319BC026257.1 GI:20072209LOC283177N/A283177AK095081.1 GI:21754271TRPM41799354795NM_017636.3 GI:304766649下列基因被鉴定为与非洲血统患者相比在高加索血统患者的前列腺肿瘤中过量表达:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3。因此,获得这些基因的至少1、2、3、4、5、6、7或8个的表达数据提供唯一的基因表达谱用于诊断或预测高加索血统患者的前列腺癌。某些实施方案涉及收集数据用于诊断或预测高加索血统患者的CaP的方法,所述方法包括检测生物样品中下列基因的至少1、2、3、4、5、6、7或8个的表达:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3,其中生物样品获自高加索血统患者。所述方法可任选包括获取高加索血统患者的生物样品的额外步骤。所述方法可任选包括利用所收集的基因表达数据诊断或预测CaP的额外步骤。在诊断或预测CaP的方法中,与对照样品或阈值相比,下列基因的至少1、2、3、4、5、6、7或8个的过量表达:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3,表明生物样品中存在CaP或发生CaP的风险增加。收集数据或诊断和/或预测CaP的方法可进一步包括检测与前列腺癌有关的其它基因的表达,包括但不限于ERG、PSA和PCA3。在某些实施方案中,所述方法包括检测的表达不超过100、90、80、70、60、50、40、30、25、20、15、14、13、12、11、10、9、8、7、6、5、4、3或2个基因。在一个实施方案中,所述方法包括检测ALOX15以及PCA3、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3的一个或多个的表达。在另一个实施方案中,所述方法包括检测CDH19以及PCA3、AMACR、ALOX15、OR51E2/PSGR、F5、FZD8和CLDN3的一个或多个的表达。在另一个实施方案中,所述方法包括检测F5以及PCA3、AMACR、ALOX15、OR51E2/PSGR、CDH19、FZD8和CLDN3的一个或多个的表达。在另一个实施方案中,所述方法包括检测FZD8以及PCA3、AMACR、ALOX15、OR51E2/PSGR、CDH19、F5和CLDN3的一个或多个的表达。在另一个实施方案中,所述方法包括检测CLDN3以及PCA3、AMACR、ALOX15、OR51E2/PSGR、CDH19、F5和FZD8的一个或多个的表达。在另一个实施方案中,所述方法包括检测PCA3以及AMACR和ALOX15、CDH19、F5、FZD8和CLDN3的一个或多个的表达。表2提供在高加索血统患者中更频繁地过量表达的这些基因的HGNC和EntrezGene指定的唯一标识码和代表性序列的登记号,所述序列通过引用以其整体结合到本文中。表2基因HGNCIDEntrezGeneIDNCBI参考PCA3863750652AF103907.1 GI:6165973ALOX15433246NM_001140.3 GI:40316936AMACR45123600NM_014324.5 GI:266456114CDH19175828513NM_021153.3 GI:402534572OR51E2/PSGR1519581285AY033942.1 GI:16943640F535422153NM_000130.4 GI:119395710FZD840468325AB043703.1 GI:13623798CLDN320451365NM_001306.3 GI:171541813下列基因被鉴定为与高加索血统患者相比在非洲世系患者的前列腺肿瘤中过量表达:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。因此,获得这些基因的至少1、2、3、4、5、6或7个的表达数据提供唯一的基因表达谱用于诊断或预测非洲血统患者的前列腺癌。某些实施方案涉及收集数据用于诊断或预测非洲血统患者的CaP的方法,所述方法包括检测生物样品中下列基因的至少1、2、3、4、5、6或7个的表达:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1,其中生物样品获自非洲血统患者。所述方法可任选包括获得非洲血统患者的生物样品的额外步骤。所述方法可任选包括利用所收集的基因表达数据诊断或预测CaP的额外步骤。在诊断或预测CaP的方法中,与对照样品或阈值相比,下列基因的至少1、2、3、4、5、6或7个的过量表达:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1,表明生物样品中存在CaP或发生CaP的风险增加。收集数据或诊断和/或预测CaP的方法可进一步包括检测与前列腺癌有关的其它基因的表达,包括但不限于ERG、PSA和PCA3。在某些实施方案中,所述方法包括检测的表达不超过100、90、80、70、60、50、40、30、25、20、15、14、13、12、11、10、9、8、7、6、5、4、3或2个基因。在一个实施方案中,所述方法包括检测COL10A1以及HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1的一个或多个的表达。在另一个实施方案中,所述方法包括检测HOXC4以及COL10A1、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1的一个或多个的表达。在另一个实施方案中,所述方法包括检测ESPL1以及COL10A1、HOXC4、MMP9、ABCA13、PCDHGA1和AGSK1的一个或多个的表达。在另一个实施方案中,所述方法包括检测MMP9以及COL10A1、HOXC4、ESPL1、ABCA13、PCDHGA1和AGSK1的一个或多个的表达。在另一个实施方案中,所述方法包括检测ABCA13以及COL10A1、HOXC4、ESPL1、MMP9、PCDHGA1和AGSK1的一个或多个的表达。在另一个实施方案中,所述方法包括检测PCDHGA1以及COL10A1、HOXC4、ESPL1、MMP9、ABCA13和AGSK1的一个或多个的表达。在另一个实施方案中,所述方法包括检测AGSK1以及COL10A1、HOXC4、ESPL1、MMP9、ABCA13和PCDHGA1的一个或多个的表达。表3提供在非洲血统患者中更频繁地过量表达的这些基因的由HGNC和EntrezGene指定的唯一标识码和代表性序列的登记号,所述序列通过引用以其整体结合到本文中。表3另外,28个样品的全基因组序列分析鉴定出65个基因突变以较高置信度存在于所分析的14个前列腺肿瘤的至少1个中。在所分析的前列腺肿瘤中具有最高等位基因频率的65个基因突变发生在下列基因中:GLI1、IRX4、PAPPA、SPOP、TEX15、ZNF292、ANKRD11、FAT4、HECW2、KIAA1109、SHROOM3、SPOP、TTC36、ZNRF3、C17orf65、DEGS2、NEK3、KIAA0947、LSP1、NOX3、AKR1B1、ARHGAP12、ITGA4、PVRL4、RBM26、UCN3、CATSPERB、FCRL2、CACNA1E、CORO6、DMKN、EXT1、HEATR7B2、NDUFB5、GPR180、LRRC4、TPRA1、ZIM2、C12orf50、ELMO2、RBM26、SEC14L1、TNFSF11、C9orf125、CDC73、ITSN1、KCNK16、LRRC7、METTL6、MOSC1、RP11-50B3.2、STAB2、STARD13、PTPRT、RBPJ、UBA2、DIAPH3、IL18R1、LIPF、SLITRK5、TMEM132E、POT1、RB1CC1、TAOK1和UNC5A。这65个基因中,已知只有SPOP具有与前列腺癌有关的突变。因此,鉴定样品中这些基因突变的一个或多个可提供可用于诊断或预测前列腺癌的基因特征。某些实施方案涉及收集数据用于诊断或预测CaP的方法,所述方法包括检测生物样品中下列基因的至少2、3、4、5、6、7、8、9、10、15、20、25、30、35、40、45或50个的一个或多个突变的表达:GLI1、IRX4、PAPPA、SPOP、TEX15、ZNF292、ANKRD11、FAT4、HECW2、KIAA1109、SHROOM3、SPOP、TTC36、ZNRF3、C17orf65、DEGS2、NEK3、KIAA0947、LSP1、NOX3、AKR1B1、ARHGAP12、ITGA4、PVRL4、RBM26、UCN3、CATSPERB、FCRL2、CACNA1E、CORO6、DMKN、EXT1、HEATR7B2、NDUFB5、GPR180、LRRC4、TPRA1、ZIM2、C12orf50、ELMO2、RBM26、SEC14L1、TNFSF11、C9orf125、CDC73、ITSN1、KCNK16、LRRC7、METTL6、MOSC1、RP11-50B3.2、STAB2、STARD13、PTPRT、RBPJ、UBA2、DIAPH3、IL18R1、LIPF、SLITRK5、TMEM132E、POT1、RB1CC1、TAOK1和UNC5A。所述方法可任选包括从受试者中获得生物样品的额外步骤。所述方法可任选包括使用所收集的基因突变数据诊断或预测CaP的额外步骤。在诊断或预测CaP的方法中,检出下列基因的至少2、3、4、5、6、7、8、9、10、15、20、25、30、35、40、45或50个的一个或多个突变:GLI1、IRX4、PAPPA、SPOP、TEX15、ZNF292、ANKRD11、FAT4、HECW2、KIAA1109、SHROOM3、SPOP、TTC36、ZNRF3、C17orf65、DEGS2、NEK3、KIAA0947、LSP1、NOX3、AKR1B1、ARHGAP12、ITGA4、PVRL4、RBM26、UCN3、CATSPERB、FCRL2、CACNA1E、CORO6、DMKN、EXT1、HEATR7B2、NDUFB5、GPR180、LRRC4、TPRA1、ZIM2、C12orf50、ELMO2、RBM26、SEC14L1、TNFSF11、C9orf125、CDC73、ITSN1、KCNK16、LRRC7、METTL6、MOSC1、RP11-50B3.2、STAB2、STARD13、PTPRT、RBPJ、UBA2、DIAPH3、IL18R1、LIPF、SLITRK5、TMEM132E、POT1、RB1CC1、TAOK1和UNC5A,表明生物样品中存在CaP或发生CaP的风险增加。在某些实施方案中,所述方法包括检测不超过100、90、80、70、60、50、40、30、25、20、15、14、13、12、11、10、9、8、7、6、5、4、3或2个突变基因的表达。表4提供这些基因的HGNC指定的唯一标识码及其EntrezGeneID,所述序列通过引用以其整体结合到本文中。另外,表4提供在前列腺肿瘤和匹配的正常样品中鉴定的各个突变的频率。表4基因肿瘤频率正常频率HGNCIDEntrezGeneIDGLI1G:21/T:23(52%)G:37/T:0(0%)2050079820IRX4G:18/A:19(51%)G:42/A:0(0%)1487579368PAPPAC:21/T:20(48%)C:40/T:0(0%)1392777SPOPA:18/C:15(45%)A:39/C:0(0%)2135684940TEX15C:25/T:21(45%)C:32/T:0(0%)2506393099ZNF292T:14/A:11(44%)T:40/A:0(0%)35122131ANKRD11C:19/A:14(42%)C:41/A:0(0%)26857133558FAT4C:26/T:19(42%)C:30/T:0(0%)77004711HECW2C:22/T:16(42%)C:42/T:0(0%)28899160897KIAA1109A:19/G:14(42%)A:47/G:0(0%)1558664101SHROOM3G:23/A:16(41%)G:39/A:0(0%)30413131601SPOPG:20/C:14(41%)G:37/C:0(0%)1287523619TTC36G:21/A:15(41%)G:30/A:0(0%)26665160419ZNRF3C:15/T:10(40%)C:31/T:0(0%)1723363916C17orf65T:19/C:12(38%)T:41/C:0(0%)2032764062DEGS2C:19/G:12(38%)C:41/G:0(0%)106986397NEK3G:24/C:15(38%)G:32/C:0(0%)119268600KIAA0947G:28/A:17(37%)G:35/A:0(0%)2818084302LSP1G:18/T:11(37%)G:38/T:0(0%)1678379577NOX3C:22/T:13(37%)C:38/T:0(0%)61836453AKR1B1T:19/A:11(36%)T:47/A:0(0%)1446483795ARHGAP12G:24/A:14(36%)G:40/A:0(0%)1853157554ITGA4A:30/G:17(36%)A:35/G:0(0%)28343131965PVRL4C:26/T:15(36%)C:21/T:0(0%)2618964757RBM26C:30/G:17(36%)C:61/G:0(0%)1544626121UCN3T:24/G:14(36%)T:41/G:0(0%)1862955576CATSPERBT:36/G:20(35%)T:46/G:0(0%)1916490627FCRL2G:26/A:14(35%)G:32/A:0(0%)968211122CACNA1EC:25/T:13(34%)C:40/T:0(0%)57243516CORO6T:30/A:16(34%)T:24/A:0(0%)3066110054DMKNA:23/C:12(34%)A:37/C:0(0%)1548081624EXT1G:23/A:12(34%)G:31/A:0(0%)59888809HEATR7B2C:23/T:12(34%)C:41/T:0(0%)66228513NDUFB5A:32/C:17(34%)A:55/C:0(0%)2029526050GPR180A:32/G:16(33%)A:41/G:0(0%)26991124842LRRC4T:14/G:7(33%)T:40/G:0(0%)1728425913TPRA1A:18/C:9(33%)A:33/C:0(0%)155749821ZIM2C:28/T:14(33%)C:30/T:0(0%)2925957551C12orf50C:35/A:17(32%)C:38/A:0(0%)1256790249ELMO2T:35/C:17(32%)T:45/C:0(0%)2050079820RBM26C:34/T:16(32%)C:58/T:0(0%)1487579368SEC14L1T:31/G:15(32%)T:35/G:0(0%)1392777TNFSF11A:33/C:16(32%)A:44/C:0(0%)2135684940C9orf125T:26/G:12(31%)T:27/G:0(0%)2506393099CDC73G:31/T:14(31%)G:44/T:0(0%)35122131ITSN1T:31/C:14(31%)T:40/C:0(0%)26857133558KCNK16C:24/A:11(31%)C:39/A:0(0%)77004711LRRC7C:39/T:18(31%)C:32/T:0(0%)28899160897METTL6A:28/G:13(31%)A:35/G:0(0%)1558664101MOSC1A:20/G:9(31%)A:35/G:0(0%)30413131601RP11-50B3.2G:26/A:12(31%)G:44/A:0(0%)1287523619STAB2G:20/A:9(31%)G:38/A:0(0%)26665160419STARD13C:24/T:11(31%)C:35/T:0(0%)1723363916PTPRTC:30/T:13(30%)C:40/T:0(0%)2032764062RBPJC:23/T:9/G:1(30%)C:30/T:0(0%)106986397UBA2T:25/A:11(30%)T:46/A:0(0%)119268600DIAPH3C:39/A:16(29%)C:32/A:0(0%)2818084302IL18R1G:34/T:14(29%)G:42/T:0(0%)1678379577LIPFG:29/T:12(29%)G:43/T:0(0%)61836453SLITRK5G:22/A:9(29%)G:45/A:0(0%)1446483795TMEM132EC:34/T:14(29%)C:32/T:0(0%)1853157554POT1T:30/C:12(28%)T:46/C:0(0%)28343131965RB1CC1A:27/C:11(28%)A:42/C:0(0%)2618964757TAOK1A:25/C:10(28%)A:44/C:0(0%)1544626121UNC5AG:27/A:11(28%)G:38/A:0(0%)1862955576GLI1突变在所分析的肿瘤中显示最高的等位基因频率,并且还与SPOP(一种已知具有与前列腺癌有关的突变的基因)具有共同途径。因此,在一个实施方案中,本文所述方法包括单独或与表4所列基因突变的一个或多个组合检测GLI1突变。收集数据或诊断和/或预测CaP的方法可进一步包括检测与前列腺癌有关的其它基因的表达,包括但不限于ERG、PSA和PCA3。检测基因表达如本文所用,测量或检测前述基因或核酸任一个的表达包括测量或检测相当于目标基因的任何核酸转录物(例如mRNA、cDNA或基因组DNA)或由其编码的蛋白质。如果基因与一个以上的mRNA转录物或同种型有关,则可通过测量或检测基因的mRNA转录物的一个或多个或与基因有关的mRNA转录物的全部,来测量或检测基因的表达。通常,可根据mRNA或cDNA水平检测或测量基因表达,尽管适当时也可使用蛋白质水平。可采用用于测量mRNA水平、cDNA或蛋白质水平的任何定量或定性方法。检测或测量mRNA或cDNA水平的合适方法包括例如RNA印迹法、微阵列分析或核酸扩增程序,例如反转录PCR(RT-PCR)或实时RT-PCR,亦称定量RT-PCR(qRT-PCR)。所述方法是本领域众所周知的。参见例如Sambrook等,MolecularCloning:ALaboratoryManual,第4版,ColdSpringHarborPress,ColdSpringHarbor,N.Y.,2012。其它技术包括基因表达的数字式多路复用分析,例如nCounter®(NanoStringTechnologies,Seattle,WA)基因表达测定法,其进一步描述于[22]、[23]、US20100112710和US20100047924,以上所有通过引用以其整体结合到本文中。检测目标核酸通常包括靶标(例如mRNA、cDNA或基因组DNA)和探针之间的杂交。用于前列腺癌基因表达谱的基因的序列是已知的(见上文)。因此,本领域技术人员可容易地设计用于检测这些基因的杂交探针。参见例如Sambrook等,MolecularCloning:ALaboratoryManual,第4版,ColdSpringHarborPress,ColdSpringHarbor,N.Y.,2012。各个探针基本上对其靶标应有特异性,以避免任何交叉杂交和假阳性。在从转录物中衍生物质时,使用特异性探针的一个替代是使用特异性试剂(例如在cDNA产生期间,或在扩增期间使用靶标特异性引物)。在两种情况下,可通过与在所分析的基因组内基本上是唯一的靶标的部分杂交,实现特异性,例如与聚A尾杂交不会提供特异性。如果靶标具有多个剪接变体,则有可能设计识别对各变体是共同的区域的杂交试剂和/或使用一种以上试剂,所述试剂的每一种可识别一个或多个变体。优选采用微阵列分析或基于PCR的方法。在这方面,测量前列腺癌组织中的前述核酸的表达可包括例如使含有或疑似含有前列腺癌细胞的样品与对目标基因有特异性的多核苷酸探针或与经设计以扩增目标基因的一部分的引物接触,并分别检测探针与核酸靶标的结合或核酸的扩增。设计PCR引物的详细方案是本领域已知的。参见例如Sambrook等,MolecularCloning:ALaboratoryManual,第4版,ColdSpringHarborPress,ColdSpringHarbor,N.Y.,2012。类似地,制备和使用微阵列分析基因表达的详细方案是本领域已知和本文描述的。备选或此外,基因的表达水平可在蛋白质水平上测定,意味着测量由上述基因编码的蛋白质的水平。用于测定蛋白质的水平的若干方法和装置是众所周知的,包括例如描述于例如美国专利号6,143,576、6,113,855、6,019,944、5,985,579、5,947,124、5,939,272、5,922,615、5,885,527、5,851,776、5,824,799、5,679,526、5,525,524、5,458,852和5,480,792的免疫测定法,其每一个通过引用以其整体结合到本文中。这些测定法包括各种夹心、竞争性或非竞争性测定方式,以产生与目标蛋白质的存在或量有关的信号。可采用任何合适的免疫测定法,例如,侧向流动、酶联免疫测定法(ELISA)、放射免疫测定法(RIA)、竞争结合测定法等。描述了用于抗体阵列的多种形式。所述阵列通常包括对待检测的不同蛋白质有特异性的不同抗体。例如,可使用至少100种不同的抗体来检测100种不同的蛋白质靶标,每种抗体对每个靶标有特异性。还可使用对特定蛋白质靶标有特异性的其它配体,例如公开于WO/2008/048970的合成抗体,其通过引用以其整体结合到本文中。可从肽或小分子的随机文库中选择具有所需结合特异性的其它化合物。美国专利号5,922,615(其通过引用以其整体结合到本文中)描述了使用膜上的固定化抗体的多个分立区带以检测阵列中的多种靶抗原的装置。可使用微量滴定板或自动化以利于大量不同蛋白质的检测。免疫测定法的一个类型,称为核酸检测免疫测定法(NADIA),将免疫测定法的蛋白质抗原检测的特异性与聚合酶链式反应(PCR)的灵敏度和精确度结合。这种增强的DNA-免疫测定方法与酶免疫测定法的类似,包括抗体结合反应和中间洗涤步骤,只是酶标记被DNA链置换,且采用扩增技术(例如PCR)通过扩增反应检验。示例性NADIA技术描述于美国专利号5,665,539和公开的美国申请2008/0131883,其两个通过引用以其整体结合到本文中。简单地说,NADIA使用对目标蛋白质有特异性和用试验特异性核酸标记的第一(报道)抗体。核酸的存在不干扰抗体的结合,抗体也不干扰核酸扩增和检测。通常,把对目标蛋白质上的不同表位有特异性的第二(俘获)抗体包被在固相(例如顺磁粒子)上。使报道抗体/核酸缀合物在微量滴定板中与样品反应,以与靶抗原形成第一免疫复合物。然后将免疫复合物俘获在用俘获抗体包被的固相粒子上,形成不溶性夹心免疫复合物。洗涤微粒子以除去过量的未结合的报道抗体/核酸缀合物。然后通过使悬浮粒子进行扩增反应(例如PCR)并监测扩增的核酸产物,来检测结合的核酸标记。虽然免疫测定法通常已被用于蛋白质的鉴定和量化,但是质谱学(MS)技术的最新发展导致灵敏的高通量MS蛋白质分析的研发。MS方法可用来检测复杂生物样品中的少量蛋白质。例如,可在MS分析之前使生物样品分级来进行定向MS。用于进行MS分析前的这类分级的常见技术包括二维电泳、液相色谱法和毛细管电泳[25],所述参考文献通过引用以其整体结合到本文中。还出现选择反应监测(SRM),亦称多反应监测(MRM),作为有用的高通量基于MS的技术用于定量测定复杂生物样品中的靶定蛋白质,包括由基因融合(例如TMPRSS2/ERG)编码的前列腺癌生物标志物[26,27],所述参考文献通过引用以其整体结合到本文中。样品本申请所述方法包括前列腺细胞中基因表达谱的分析。这些前列腺细胞存在于生物样品中,例如前列腺组织、血液、血清、血浆、尿液、唾液或前列腺液。可在检测基因表达前从细胞中分离核酸或多肽。在一个实施方案中,生物样品包含前列腺组织并通过活组织检查(例如直肠或经会阴活检)获得。在另一个实施方案中,生物样品是尿液。尿液样品可在直肠指检(DRE)或前列腺活检后收集。在另一个实施方案中,样品是血液、血清或血浆,并含有从原发性肿瘤剥离的循环肿瘤细胞。样品还可含有肿瘤来源的外来体。外来体是小的(通常30-100nm)膜结合颗粒,它从正常细胞、患病细胞和肿瘤细胞中释放,并存在于血液和其它体液中。本申请公开的方法可与从多种哺乳动物收集的样品,但优选与从人类受试者获得的样品一起使用。对照对照可以是任何合适的参比,其允许评价与在包含非癌前列腺细胞(例如匹配受试者的正常前列腺上皮细胞)的样品或所述样品库中相同基因的表达相比的前列腺癌细胞中的基因表达水平。因此,例如,对照可以是与试验样品一起同时或相继分析的来自同一受试者的样品,或对照可以是已知是非癌的前列腺样品库中的上述目标基因的平均表达水平。或者,对照可通过样品中其它基因的mRNA拷贝数界定,其它基因例如可用来使基因表达水平归一化的持家基因(例如PBGD或GAPDH)。因此,对照可包括在例如用作标准物或参比的预制备微阵列中或在反映样品或非癌样品库中相关基因的表达谱的数据中,例如可以是电子数据库或计算机程序的一部分。基因过量表达和表达降低可通过任何合适的方法测定,例如通过将试验样品中基因的表达与对照(例如阳性或阴性对照)进行比较,或通过使用绝对表达的预定“截止值”或阈值。可如前所述提供对照。不论所用方法,过量表达和表达降低可定义为大于或小于非癌前列腺细胞或组织中相同基因或其它基因(例如持家基因)的表达水平的任何表达水平。进一步举例说明,过量表达可定义为是与非癌前列腺细胞或组织相比的至少约1.2倍、1.5倍、2倍、2.5倍、5倍、10倍、20倍、50倍、100倍或甚至更大表达的表达,表达降低可类似地定义为是与非癌前列腺细胞或组织相比的至少约1/1.2、1/1.5、1/2、1/2.5、1/5、1/10、1/20、1/50、1/100或甚至更低表达的表达。在一个实施方案中,本文所用的过量表达或表达降低被定义为分别是与对照样品或阈值相比的至少约2倍或1/2的表达。前列腺癌本公开内容提供与前列腺癌有关的基因表达谱。可利用基因表达谱检测样品中的前列腺癌细胞或测量前列腺癌的严重程度或侵袭性,例如区分高度分化的前列腺(WD)癌和分化差的(PD)前列腺癌。当在活检中发现前列腺癌时,通常进行分级以估计它将如何快地生长和扩散。最常用的前列腺癌分级系统,称为Gleason分级,根据其在显微镜下观察到的形式,以1-5的标度评价前列腺癌细胞。仍像健康前列腺细胞的癌细胞具有边界清晰可辨的均一形式,并被视为高度分化的(Gleason1和2级)。癌细胞越接近地类似于前列腺组织,细胞将越像正常前列腺组织运行,且癌越少侵袭性。Gleason3级,最常见的等级,显示中等分化的细胞,也就是说仍有些高度分化,但是边界非清晰可辨。分化差的癌细胞具有边界分辨不清的随机形式,并且不再像前列腺组织(Gleason4和5级),表明更大侵袭性的癌。前列腺癌常常具有不同等级的区域。通过将肿瘤内的2种最常见的癌细胞形式的等级相加求出综合Gleason评分。例如,如果最常见的形式是4级,次最常见的形式是3级,则综合Gleason评分为4+3=7。如果肿瘤内只有一种形式,则综合Gleason评分可低至1+1=2或高达5+5=10。2-4的综合评分被视为高度分化的,5-6的评分被视为中等分化的,7-10的评分被视为分化差。有高Gleason评分的癌症更可能在发现时早已扩散到前列腺以外。一般而言,Gleason评分越低,癌症侵袭性越小且预后越好(展望治愈或长期生存)。Gleason评分越高,癌症侵袭性越大且长期无转移生存的预后越差。阵列平行测量多个基因的RNA转录物水平的一种便利方法是使用阵列(在本领域亦称为微阵列)。使用阵列评价和比较基因表达水平的技术是本领域众所周知的,包括合适的杂交、检测和数据处理方案。有用的阵列包括固定在单独位置(例如可寻址元件)处的固体基质(例如玻璃支持物例如显微镜载玻片或膜)上的多种多核苷酸探针(通常为DNA),使得可检测杂交可发生在探针和转录物之间以表明存在的各种转录物的量。本申请公开的阵列可用于检测所需基因组合的表达的方法中,在整个本申请中论述了所述组合。在一个实施方案中,阵列包含(a)基质和(b)2、3、4、5、6、7、8、9或10个或更多个不同的可寻址元件,所述各可寻址元件包含至少1种多核苷酸探针用于检测下列人基因之一的mRNA转录物的表达(或从mRNA转录物合成的cDNA):DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4。在另一个实施方案中,阵列包含(a)基质和(b)2、3、4、5、6、7或8个或更多个不同的可寻址元件,所述各可寻址元件包含至少1种多核苷酸探针用于检测下列人基因之一的mRNA转录物的表达(或从mRNA转录物合成的cDNA):PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3。在另外又一个实施方案中,阵列包含(a)基质和(b)2、3、4、5、6或7个或更多个不同的可寻址元件,所述各可寻址元件包含至少1种多核苷酸探针用于检测下列人基因之一的mRNA转录物的表达(或从mRNA转录物合成的cDNA):COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。本文所用术语“可寻址元件”意指这样的元件,即在预定位置与基质连接并特异性结合已知靶分子,使得当检测出靶标结合时(例如通过荧光标记),根据基质上元件的位置,提供有关所结合的分子的特性的信息。如果可寻址元件不与相同的靶标基因结合,则对于本公开内容的目的是“不同的”。可寻址元件包含对指定基因的mRNA转录物有特异性一种或多种多核苷酸探针或从mRNA转录物合成的cDNA。可寻址元件可包含1拷贝以上的多核苷酸,可包含一种以上不同的多核苷酸,条件是所有的多核苷酸结合相同的靶分子。在已知基因表达一种以上的mRNA转录物时,基因的可寻址元件可包含针对不同转录物的不同探针,或被设计成检出对转录物的两种或更多种(或全部)是共同的核酸序列的探针。或者,阵列可包含不同转录物的可寻址元件。可寻址元件还可包含可检测标记,其合适的实例是本领域众所周知的。阵列可包含与除以下以外的mRNA或cDNA结合的可寻址元件:1)DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4;2)PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3;或3)COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。然而,能够检测多种靶标(例如mRNA或多肽靶标)的阵列,例如设计成用于细胞系、染色体、基因组等的全面表达概况的阵列,对于收集数据以用于诊断和/或预测前列腺癌是不经济和不方便的。因此,为了促进作为例如与本文所述方法联合的诊断工具或筛查的阵列的便利使用,阵列优选包含少数可寻址元件。在这一方面,在一个实施方案中,阵列包含不超过约1000个不同的可寻址元件,更优选不超过约500个不同的可寻址元件,不超过约250个不同的可寻址元件,或甚至不超过约100个不同的可寻址元件,例如约75个或更少个不同的可寻址元件,或甚至约50个或更少个不同的可寻址元件。当然,甚至较小的阵列可包含约25个或更少个不同的可寻址元件,例如约15个或更少个不同的可寻址元件或约12个或更少个不同的可寻址元件。甚至可将阵列限于约7个不同的可寻址元件而不干扰其功能性。还可通过限制阵列上多核苷酸探针的数目,将这些诊断阵列与更全面的基因组阵列等区分开来。因此,在一个实施方案中,阵列具有固定在基质上的针对不超过1000个基因的多核苷酸探针。在其它实施方案中,阵列具有固定在基质上的针对不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2个基因的寡核苷酸探针。基质可以是多核苷酸可与之共价或非共价连接的任何刚性或半刚性支持物。合适的基质包括膜、滤器、芯片、载玻片、圆片(wafer)、纤维、珠粒、凝胶、毛细管、板、聚合物、微粒子等。对基质是合适的材料包括例如尼龙、玻璃、陶瓷、塑料、二氧化硅、铝硅酸盐、硼硅酸盐、金属氧化物例如氧化铝和氧化镍、各种粘土、硝化纤维等。可寻址元件的多核苷酸(亦称为“探针”)可在预定的一维或二维排列上与基质连接,使得与探针杂交或结合的形式易与特定基因的表达相关联。因为探针位于基质的指定位置上(即元件是“可寻址的”),杂交或结合形式和强度产生特有的表达谱,这可根据特定基因的表达水平来解释,并且可按照本文所述方法与前列腺癌相关联。多核苷酸和多肽探针可通过本领域已知的任何合适方法产生(参见例如Sambrook等,MolecularCloning:ALaboratoryManual,第4版,ColdSpringHarborPress,ColdSpringHarbor,N.Y.,2012)。例如,可通过常规技术(例如PCR或合成),使用mRNA或cDNA靶标本身的核酸序列(例如表1-4中公开的核酸序列),产生与本文所述基因的mRNA转录物(或从其合成的cDNA)特异性结合的多核苷酸探针。本文所用术语“片段”意指包含约10个或更多个核苷酸、约15个或更多个核苷酸、约20个或更多个核苷酸、约30或更多个或甚至约50个或更多个核苷酸的多核苷酸序列的连续部分或一部分。进一步举例说明,可通过包含与mRNA转录物(例如SEQIDNO:2)或其片段互补或与SEQIDNO:2充分互补或其选择性与SEQIDNO:2结合的片段的核酸序列的多核苷酸,提供与DLX1的mRNA转录物(或其相应的cDNA)结合的多核苷酸探针。对于本文所述其它基因也同样如此。多核苷酸探针的确切性质对本发明不是关键的;可使用可选择性结合mRNA或cDNA靶标的任何探针。通常,多核苷酸探针可包含10个或更多个核酸、20个或更多个、50个或更多个或100个或更多个核酸。为了赋予足够的特异性,探针可与靶序列(例如表1-4中公开的核酸序列)的互补序列有约90%或更高、优选约95%或更高(例如约98%或更高或约99%或更高)的序列同一性,如通过例如应用众所周知的基础局部比对搜索工具(BasicLocalAlignmentSearchTool,BLAST)算法(可通过NationalCenterforBiotechnologyInformation(NCBI),Bethesda,Md.获得)测定。杂交反应的严格性可容易地通过本领域普通技术人员确定,一般根据探针长度、洗涤温度和盐浓度任经验计算。一般而言,对于适当退火,较长的探针需要较高的温度,而较短的探针需要较低的温度。当互补链存在于低于其解链温度的环境下时,杂交一般取决于变性核酸序列再退火的能力。探针和可杂交序列之间所需同源性的程度越高,可使用的相对温度越高。因此,因此得出结论,较高的相对温度往往可使反应条件较严格,而较低的相对温度则较不严格。有关杂交反应严格性的其它详情和解释参见Ausubel等,CurrentProtocolsinMolecularBiology,WileyIntersciencePublishers,(1995)。本文所用“严格条件”或“高严格性条件”根据以下识别,但不限于此:(1)对于洗涤使用低的离子强度和高的温度,例如0.015M氯化钠/0.0015M柠檬酸钠/0.1%十二烷基硫酸钠,在50℃下;(2)在杂交期间使用变性剂,例如甲酰胺,例如50%(v/v)甲酰胺与0.1%牛血清白蛋白/0.1%聚蔗糖(Ficoll)/0.1%聚乙烯吡咯烷酮/50mM磷酸钠缓冲液(pH6.5)与750mM氯化钠、75mM柠檬酸钠,在42℃下;或(3)使用50%甲酰胺、5XSSC(0.75MNaCl、0.075M柠檬酸钠)、50mM磷酸钠(pH6.8)、0.1%焦磷酸钠、5XDenhardt溶液、声波处理的鲑精DNA(50μg/ml)、0.1%SDS和10%硫酸葡聚糖,在42℃下,其中在42℃下在0.2XSSC(氯化钠/柠檬酸钠)中并在50%甲酰胺中在55℃下洗涤,接着包括含有0.1XSSC的EDTA、在55℃下的高严格性洗涤。Sambrook等,MolecularCloning:ALaboratoryManual,NewYork:ColdSpringHarborPress,1989描述了“中等严格条件”,但不限于此,包括使用不比上述那些严格的洗涤溶液和杂交条件(例如温度、离子强度和%SDS)。中等严格条件的实例是在包含以下的溶液中在37℃下温育过夜:20%甲酰胺、5XSSC(150mMNaCl、15mM柠檬酸三钠)、50mM磷酸钠(pH7.6)、5XDenhardt溶液、10%硫酸葡聚糖和20mg/mL变性已剪切鲑精DNA,接着在1XSSC中在约37-50℃下洗涤过滤器。技术人员应了解需要时如何调节温度、离子强度等以适应例如探针长度等因素。阵列可包含为多核苷酸阵列所共有的其它元件。例如,阵列还可包括用作对照、标准物或参比分子(例如持家基因或其部分(例如PBGD或GAPDH))的一个或多个元件,以助于表达水平的归一化或核酸品质和结合特征、试剂品质和有效性、杂交成功、分析阈值和成功等的测定。阵列或可寻址元件的这些其它共同方面,以及符合本发明的用于构建和使用阵列的方法,包括产生、标记和使合适探针与基质连接是本领域众所周知的。就本发明的方法而论,阵列的其它方面如本文之前描述的一样。在一个实施方案中,阵列包含(a)基质和(b)两个或更多个不同的可寻址元件,所述各可寻址元件包含至少1种多核苷酸探针用于检测下列人基因之一的mRNA转录物的表达:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4,其中阵列包含不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2个可寻址元件。在某些实施方案中,阵列包含至少3、4、5、6、7、10、12或15个不同的可寻址元件。在另一个实施方案中,阵列包含两个或更多个不同的可寻址元件,其每一个包含至少1种多核苷酸探针用于检测下列人基因之一的mRNA转录物的表达:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3,其中阵列包含不超过500、不超过250、不超过100、不超过50、不超过25或不超过15个可寻址元件。在一个实施方案中,阵列包含至少3、4、5、6、7、10、12或15个不同的可寻址元件。在另一个实施方案中,阵列包含两个或更多个不同的可寻址元件,其每一个包含至少1种多核苷酸探针用于检测下列人基因之一的mRNA转录物的表达:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1,其中阵列包含不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2个可寻址元件。在一个实施方案中,阵列包含至少3、4、5、6、7、10、12或15个不同的可寻址元件。阵列也可用来平行测量多种蛋白质的蛋白质水平。所述阵列包含携带与多种蛋白质特异性结合的多种配体的一个或多个支持物,其中多种蛋白质包含不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2种不同的蛋白质。配体任选与平面支持物或珠粒连接。在一个实施方案中,配体是抗体。待使用阵列检测的蛋白质相当于由本文所述目标核酸编码的蛋白质,包括公开的特定基因表达谱。因此,各配体(例如抗体)被设计成与靶标蛋白质之一(例如表1-4公开的多肽序列)结合。如同核酸阵列一样,各配体优选与不同的可寻址元件缔合以利于检测样品中不同的蛋白质。患者治疗本申请描述了诊断和预测获自受试者的样品中的前列腺癌的方法,其中分析了前列腺细胞和/或组织中的基因表达。如果样品显示过量表达某些基因或表达某些基因突变,则受试者患有前列腺癌或不同程度的晚期(例如WD或PD前列腺癌)前列腺癌的可能性加大。如果是这样的结果的话,检测或预测前列腺癌的方法可包括下列步骤的一个或多个:知会患者他们可能患有前列腺癌、WD前列腺癌或PD前列腺癌;前列腺组织的证实性组织学检查;和/或通过前列腺癌疗法治疗患者。因此,在某些方面,如果检测步骤表明受试者患有前列腺癌,则该方法进一步包括从受试者中采取前列腺活检和检查活检(例如组织学检查)中的前列腺组织的步骤以证实患者是否患有前列腺癌。或者,检测或预测前列腺癌的方法可用来评价对治疗的需要或监测对治疗的反应(例如手术或其它疗法后的无病复发),并因此可包括治疗患有前列腺癌的受试者的额外步骤。前列腺癌治疗选择包括手术、放射疗法、激素疗法、化学疗法、生物疗法或高强度聚焦超声。获准用于前列腺癌的药物包括:Enzalutamide(XTANDI)、醋酸阿比特龙、卡巴他赛(Cabazitaxel)、地加瑞克、Jevtana(卡巴他赛)、泼尼松、Provenge(西普鲁塞T)、西普鲁塞T或多西他赛。因此在阳性结果之后,本申请所述方法可包括手术、放射疗法、激素疗法、化学疗法、生物疗法或高强度聚焦超声的进一步步骤。药物筛选由本申请所述方法提供的与前列腺癌有关的基因表达谱或其缺乏也可用于在临床试验中或在前列腺癌动物模型中筛选药物。可按与监测各个患者类似的方式对药物进行临床试验,只是将平行给予前列腺癌患者人群,通常与给予安慰剂的对照人群进行比较。可分析各个患者及治疗人群或对照人群间基因表达水平的变化。在各个患者水平上的分析提供试验结束时患者总体状况的指示(即基因表达谱是否表明前列腺癌的存在或严重程度(例如WD或PD))和/或该表达谱在试验过程中是接近还是远离这类指示变化的指示。可汇集人群中各个患者的结果,以允许在治疗人群和对照人群之间进行比较。可在非人动物前列腺癌模型中进行类似试验。既然这样,在正对其进行试验的非人动物的任何物种中,所检测的基因的表达水平是上文引用的人基因的物种变体或同源物。虽然在人前列腺癌患者中测定的人基因的平均表达水平不必直接与动物模型中同源基因的表达水平相比较,但是人类值仍然可用来提供非人同源物表达水平的改变是处于朝向还是远离前列腺癌的诊断或WD或PD前列腺癌的预测的指示。试验中各动物的表达谱可提供试验结束时动物状况的指示(即,基因表达谱是否表明前列腺癌的存在或严重程度(例如WD或PD))和/或试验期间所述状况的变化。可在群体和所比较的治疗人群和对照人群间汇集各动物的结果。然后可比较2个群体间的基因表达水平的平均变化。计算机执行模型按照本发明的所有方面和实施方案,所提供的方法可为计算机执行的。可在数字计算机中,分析基因表达水平并与受试者的状况(例如前列腺癌的存在或疾病的严重程度(例如WD或PD前列腺癌))相关联。任选所述计算机与接收与基因表达水平相关的实验测定信号的扫描仪等直接连接。或者,可通过其它方法输入表达水平。可对计算机编程以将原始信号转化成表达水平(绝对或相对),将测量表达水平与一个或多个参比表达水平或这类值的标度进行比较。还可根据与一个或多个参比表达水平的比较对计算机编程以对表达水平赋值或指定其它标识,并汇集表达谱中多个基因的所述值或标识。还可对计算机编程以输出提供表示前列腺癌的存在或严重程度的值或其它标识以及用于确定所述值或标识的任何原始或中间数据。一台典型的计算机(参见美国专利号6,785,613;图4和5)包括使以下主要子系统互连的总线:例如中央处理器、系统存储器、输入/输出控制器、外部设备例如经由并行端口打印机、经由显示适配器的显示屏、串行端口、键盘、固定磁盘驱动器和有效接收外部存储设备的端口(例如USB端口)。可连接许多其它设置,例如经由I/O控制器的扫描仪、与串行端口或网络接口连接的鼠标。计算机含有保存允许计算机执行多种功能的代码的计算机可读介质。这些功能包括上述接收输入和发送输出的控制自动化设备。自动化设备可包括用于递送测定表达水平的试剂的机器臂以及小容器,例如用于进行表达分析的微量滴定板孔。一台典型的计算机系统106还可包括与在操作系统控制下或与操作系统联合操作的随机存储器连接的如图4给出和上文所述的一个或多个处理器110。在一个实施方案中,本发明的计算机执行方法的任一种可包括通过至少1个处理器获得反映生物样品中的下列人基因的至少2、3、4、5、6、7、8、9、10、11、12、13、14或15个的表达水平的信息的步骤:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4。在一个实施方案中,所述计算机执行方法包括至少1个处理器获得反映DLX1和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映NKX2-3和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映DLX1和NKX2-3和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映PHGR1和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映THBS4和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映GAP43和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映FFAR2和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映GCNT1和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映SIM2和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映STX19和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映KLB和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映APOF和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映LOC283177和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映TRPM4和表1所列一个或多个其它基因的表达水平的信息。在另一个实施方案中,本发明的计算机执行方法的任一种可包括获得至少1个处理器反映获自高加索血统患者的生物样品中下列人基因的至少2、3、4、5、6、7或8个的表达水平的信息的步骤:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3。在一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映ALOX15以及PCA3、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映CDH19以及PCA3、AMACR、ALOX15、OR51E2/PSGR、F5、FZD8和CLDN3的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括过至少1个处理器获得反映F5以及PCA3、AMACR、ALOX15、OR51E2/PSGR、CDH19、FZD8和CLDN3的一个或多个的表达水平的通信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映FZD8以及PCA3、AMACR、ALOX15、OR51E2/PSGR、CDH19、F5和CLDN3的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映CLDN3以及PCA3、AMACR、ALOX15、OR51E2/PSGR、CDH19、F5和FZD8的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映PCA3以及AMACR和ALOX15、CDH19、F5、FZD8和CLDN3的一个或多个的表达水平的信息。在另一个实施方案中,本发明的计算机执行方法的任一种可包括通过至少1个处理器获得反映获自非洲血统患者的生物样品中的下列人基因的至少2、3、4、5、6或7个的表达水平的信息的步骤:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。在一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映COL10A1以及HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映HOXC4以及COL10A1、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映ESPL1以及COL10A1、HOXC4、MMP9、ABCA13、PCDHGA1和AGSK1的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映MMP9以及COL10A1、HOXC4、ESPL1、ABCA13、PCDHGA1和AGSK1的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映ABCA13以及COL10A1、HOXC4、ESPL1、MMP9、PCDHGA1和AGSK1的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映PCDHGA1以及COL10A1、HOXC4、ESPL1、MMP9、ABCA13和AGSK1的一个或多个的表达水平的信息。在另一个实施方案中,所述计算机执行方法包括通过至少1个处理器获得反映AGSK1以及COL10A1、HOXC4、ESPL1、MMP9、ABCA13和PCDHGA1的一个或多个的表达水平的信息。在本发明的计算机执行方法的另一个实施方案中,所述方法可另外包括以下步骤:i)通过至少1个处理器测定一个或多个对照基因的表达水平和以下的表达水平之间的差异:1)生物样品中下列人基因的至少2、3、4、5、6、7、8、9、10、11、12、13、14或15个:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4;2)获自高加索血统患者的生物样品中下列人基因的至少2、3、4、5、6、7或8个:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3;或3)获自非洲血统患者的生物样品中下列人基因的至少2、3、4、5、6或7个:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1;和(ii)以用户可读格式输出测定步骤中得到的差异。在本发明的计算机执行方法的另一个实施方案中,所述方法可进一步包括根据输出步骤中得到的差异,以用户可读格式输出受试者患有前列腺癌、高度分化的前列腺癌或分化差的前列腺癌的测定结果。试剂盒可在试剂盒中安排用于本申请所述方法的多核苷酸探针和/或引物或抗体或多肽探针。因此,一个实施方案涉及用于诊断或预测前列腺癌的试剂盒,其包含用于检测下列人基因的至少2、3、4、5、6、7、8、8、9、10、11、12、13、14或15个的多种多核苷酸探针:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4,其中多种多核苷酸探针含有针对不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2个基因的多核苷酸探针。在一个实施方案中,多种多核苷酸探针包含用于检测上述基因的至少4或5个的多核苷酸探针,其中多种多核苷酸探针含有用于不超过10个基因的多核苷酸探针。多核苷酸探针可以是任选标记的。试剂盒可任选包括用于扩增下列人基因的至少2、3、4、5、6、7、8、8、9、10、11、12、13、14或15个的mRNA转录物的一部分的多核苷酸引物:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM4。另一个实施方案涉及用于诊断或预测高加索血统患者的前列腺癌的试剂盒,试剂盒包含用于检测下列人基因的至少3、4、5、6、7或8个的多种多核苷酸探针:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3,其中多种多核苷酸探针含有用于不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2个基因的多核苷酸探针。在一个实施方案中,多种多核苷酸探针包含用于检测上述基因的至少4或5个的多核苷酸探针,其中多种多核苷酸探针含有用于不超过10个基因的多核苷酸探针。多核苷酸探针可以是任选标记的。试剂盒可任选包括用于扩增下列人基因的至少3、4、5、6、7或8个的mRNA转录物的多核苷酸引物:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3。又一个实施方案涉及用于诊断或预测非洲血统患者的前列腺癌的试剂盒,试剂盒包含用于检测下列人基因的至少3、4、5、6或7个的多种多核苷酸探针:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1,其中多种多核苷酸探针含有用于不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2个基因的多核苷酸探针。在一个实施方案中,多种多核苷酸探针包含用于检测上述基因的至少4或5个的多核苷酸探针,其中多种多核苷酸探针含有用于不超过10个基因的多核苷酸探针。多核苷酸探针可以是任选标记的。试剂盒可任选包括用于扩增下列人基因的至少3、4、5、6或7个的mRNA转录物的多核苷酸引物:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。用于诊断或预测前列腺癌的试剂盒还可包含抗体。因此,在一个实施方案中,用于诊断或预测前列腺癌的试剂盒包含用于检测由下列人基因编码的多肽的至少3、4、5、6、7、8、9、10、11、12、13、14或15种的多种抗体:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM,其中多种抗体含有不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2种多肽的抗体。在一个实施方案中,多种抗体包含用于检测由上述基因编码的多肽的至少4或5种的抗体且其中多种抗体含有不超过10种多肽的抗体。抗体可以是任选标记的。在另一个实施方案中,用于诊断或预测高加索血统患者的前列腺癌的试剂盒包含用于检测由下列人基因编码的多肽的至少3、4、5、6或7种的多种抗体:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3,其中多种抗体含有不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2种多肽的抗体。在一个实施方案中,多种抗体包含用于检测由上述基因编码的多肽的至少4或5种的抗体且其中多种抗体含有不超过10种多肽的抗体。抗体可以是任选标记的。在另外又一个实施方案中,用于诊断或预测非洲血统患者的前列腺癌的试剂盒,其包含用于检测由下列人基因编码的多肽的至少3、4、5、6或7种的多种抗体:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1,其中多种抗体含有不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2种多肽的抗体。在一个实施方案中,多种抗体包含用于检测由上述基因编码的多肽的至少4或5种的抗体且其中多种抗体含有不超过10种多肽的抗体。抗体可以是任选标记的。另一方面,用于诊断或预测前列腺癌的试剂盒可包含可用于例如光谱测定方法(例如质谱法)的多肽探针。因此,在一个实施方案中,用于诊断或预测前列腺癌的试剂盒包含用于检测由下列人基因编码的多肽的至少3、4、5、6、7、8、9、10、11、12、13、14或15种的多种多肽探针:DLX1、NKX2-3、CRISP3、PHGR1、THBS4、AMACR、GAP43、FFAR2、GCNT1、SIM2、STX19、KLB、APOF、LOC283177和TRPM,其中多种多肽探针含有不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2种多肽的多肽探针。在一个实施方案中,多种多肽探针包含用于检测由上述基因编码的多肽的至少4或5种的多肽探针,且其中多种多肽探针含有不超过10种多肽的多肽探针。多肽探针可以是任选标记的。在另一个实施方案中,用于诊断或预测高加索血统患者的前列腺癌的试剂盒包含用于检测由下列人基因编码的多肽的至少3、4、5、6、7或8种的多种多肽探针:PCA3、ALOX15、AMACR、CDH19、OR51E2/PSGR、F5、FZD8和CLDN3,其中多种多肽探针含有不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2种多肽的多肽探针。在一个实施方案中,多种多肽探针包含用于检测由上述基因编码的多肽的至少4或5种的多肽探针,且其中多种多肽探针含有不超过10种多肽的多肽探针。多肽探针可以是任选标记的。在另外又一个实施方案中,用于诊断或预测非洲血统患者的前列腺癌的试剂盒包含用于检测由下列人基因编码的多肽的至少3、4、5、6或7种的多种多肽探针:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1,其中多种多肽探针含有不超过500、250、100、50、25、15、10、9、8、7、6、5、4、3或2种多肽的多肽探针。在一个实施方案中,多种多肽探针包含用于检测由上述基因编码的多肽的至少4或5种的多肽探针,且其中多种多肽探针含有不超过10种多肽的多肽探针。多肽探针可以是任选标记的。在一个实施方案中,试剂盒包括公开了在所公开的方法中使用试剂盒内容物的方法的说明资料。说明资料可以许多形式提供,包括但不限于书面形式(例如硬拷贝纸等)、以电子形式(例如计算机磁盘或光盘)提供或可以是可视的(例如可视文件)。试剂盒还可包括有利于试剂盒针对其而设计的特殊应用的部件。因此,例如,试剂盒可另外包括常规用于实施特定方法的其它试剂,包括但不限于缓冲液、酶、标记化合物等。所述试剂盒和合适的内容物为本领域技术人员所熟知。试剂盒还可包括参比或对照样品。参比或对照样品可以是生物样品或数据库。如上所述,本申请中描述的多核苷酸或多肽探针和抗体任选用可检测标记进行标记。可以使用与本领域普通技术人员已知的探针或抗体技术联用的任何可检测标记。在一个具体的实施方案中,探针用选自以下的可检测标记进行标记:荧光标记、化学发光标记、猝灭剂、放射性标记、生物素、质量标签和/或金。除非另有定义,否则本文所用的全部技术和科学术语都具有本领域普通技术人员通常理解的相同含义。虽然类似或等同于本文所述的方法和材料可用于实施或测试本发明,但下面描述了合适的方法和材料。本文提及的所有出版物、专利申请、专利和其它参考文献通过引用以其整体结合到本文中。万一有抵触,则以本说明书(包括定义)为准。另外,材料、方法和实施例只是说明性的,并无意是限制性的。实施例实施例1.比较基因组DNA分析在7名AA和7名CACaP患者(28个样本)的组群中,使用原发性前列腺肿瘤和相应的正常组织(血液)进行了比较全基因组分析。根据以下标准选择组群:基本治疗根治性前列腺切除术、无新辅助治疗、Gleason3+3和3+4级(代表在诊断/基本治疗时PSA筛查的CaP的大部分)、具有80%或更高肿瘤细胞内容物的冷冻肿瘤组织、得到超过2μg高分子量基因组DNA的解剖肿瘤组织、相应血液基因组DNA的可得性和患者临床病理数据。将28个样品送往IlluminaInc.(UK)以测序。应用Illumina的ELAND比对算法,对来自肿瘤样品的序列与参比基因组作图。测序报告良好的覆盖度(平均37)。应用Strelka算法,同时进行调用单核苷酸多态性(SNP)、小插入与小缺失(InDel)、拷贝数变异(CNV)和结构变异(SV)的变体。在该组群中,以预期频率鉴定出所有已建立的CaP突变(TMPRSS2/ERG、SPOP、CHD1和PTEN)。在14名患者的至少2名中鉴定出具有SNV、CNV或InDel体细胞突变的31个基因(包括已知突变):AC091435.2、APC、ASMTL、ASMTL-AS1、CDC73、CHD1、CSF2RA、EYS、FRG1、FRG1B、HK2、IL3RA、KLLN、LIPF、LOC100293744、MT-ATP6、MT-BD4、MT-CO1、MT-CYB、MT-ND2、MT-ND3、MUC16、MUC6、NOX3、PDHA2、PTEN、SLC25A6、SLC9B1、SPOP、TRAV20和USH2A。突变似乎不显示与任何特定组(AA-、AA+、CA-、CA+)有关。然而,未料到AA患者中不存在PTEN缺失。在2个CA病例中检测到明确的PTEN缺失,在另外3个CA病例中PTEN缺失不太明显,表明在CA病例中PTEN缺失的潜在排外性。实施例2.比较RNA分析为了补充基因组DNA分析,在相同的前列腺肿瘤样品组群中进行了RNA-Seq分析。RNA-Seq技术具有询问包括基因融合、基因和转录物表达在内的转录物组的多个方面的能力。从14名患者的4位中收集周围的正常组织。正常组织样品中的2个来自AA男性,2个来自CA男性。将RNA样品运送到ExpressionAnalysis(Durham,NC)用于转录物组测序。测序统计学的详细资料如下:测序类型:配对末端,各样品的平均读取长度:50nt,平均读取质量:37,读取数目:每个样品约3100万。以fastq格式获取各样品的表达分析的原始读取。这些文件含有通过Illumina的纯度滤器的所有序列和由Illuminaphred度量限定的全碱基(per-base)质量分数。在实际分析之前,针对低质量读取(质量分数<20)、伪像/复制序列和衔接序列,将这些原始读取过滤。在该分析中,从UCSC网站下载用于制图的人参比基因组(hg19)。应用TopHat软件2.0.8版(一种可用于制图的免费开放源码工具),使CleanPaired末端读取与hg19参比基因组进行比对,并且在比对中对于各读取允许不超过2个错配。采用Bowtie(一种超高通量短读取比对器(aligner)),将TopHat图读取至参比基因组。软件输出多份文件用于进一步分析:制图读取(mappedread)、融合连接处、剪接点、插入和缺失文件。应用Cufflinks,一种装配转录物、估计其丰度和测试RNA-Seq样品中的差异表达和调节的开放源码工具,将比对的读取装配成转录物。Cufflinks根据该基因的所有已知的剪接变体/同种型,计算出基因的表达水平。Cufflinks测量每百万制图读取每千碱基转录物的片段(FragmentsperKilobaseoftranscriptperMillionmappedreads,FPKM)中的转录物和基因丰度水平。FPKM方程式如下定义:C为特征(转录物、外显子)中可制图读取的数目,L为特征中的长度(单位:kb),N为特征中可制图读取的总数(单位:百万)。FPKM归一化方法消除用于比较样品间基因表达的差异的基因长度和测序偏差。Cufflinks包中包括的Cuffdiff程序计算出CaP肿瘤和正常样品间的差异表达,并包括样品间表达的各实测变化的p值。根据实验条件,Cuffdiff允许输入多个样品文件。基因和转录物表达水平以表格格式报告,并且这些文件含有基因信息、各基因的log2标度倍数变化、P值和错误发现率(FDR)。用GenomatixPathway分析软件进行有关统计显著性基因的途径分析和基因本体论生物进程(GeneOntologyBiologicalProcesses)。根据其表达水平,应用分层聚类(应用R包进行)将样品分类。图1.所有18个样品(14个肿瘤和4个正常)的聚类表明肿瘤和正常样品之间的清晰分界。然而,未按照AA和CA组使大多数肿瘤样品聚类。引人关注的是,4个融合阴性AA样品中的3个聚类在1组中,并且根据患者随访研究,3名患者中的2名发生转移(在该组群中仅2名转移),第3人有生化复发。从14个肿瘤(7个AA和7个CA)和4个正常(2个AA和2个CA)样品获得基因表达谱。肿瘤和正常样品之间的比较分析的限制是所有肿瘤样品的正常样品的可得性。因此,在当前的分析中,将各组内的2个正常样品合并在一起,将平均值与其相应组进行比较。在初步分析中,通过将各组内的肿瘤与正常样品进行比较产生各个患者的基因表达谱。使用在肿瘤中过量表达/表达不足至少2倍的倍数变化(肿瘤/正常比)和P值<0.05提取统计显著性基因。在非裔美国人和高加索美国人组中,101个基因和180个基因分别为统计显著性的(2倍且P值<0.05)。非裔美国人列表中几个前列腺癌文献相关基因包括CRISP3、SIM2、THBS4和MMP9;高加索美国人列表包括AMACR、APOF、CRISP3、OR51E2(PSGR)、SIM2和THBS4。AA基因列表与CA基因列表的比较显示在两个人种群中84个基因是共同的。共同基因列表包括前列腺癌中少数充分研究的基因像AMACR、CRIPS3和SIM2。DLX1、NKX3-2和CRISP3在该列表中是最前的过量表达的基因。图2.在两个人种群中最始终过量表达的基因是DLX1和NKX2-3。表5列出在AA和CA前列腺肿瘤两者中前15个过量表达的基因(通过倍数变化排序)。表5类似地,将CA组(180个基因)的肿瘤/正常比与AA组的肿瘤/正常比进行比较。该基因列表显示一些充分研究的前列腺癌基因,例如与AA组相比,PCA3(10倍)、PSGR(5倍)和AMACR(2倍)在CA组中过量表达(图3B)。另外,将TMPRSS2/ERG融合阳性样品的基因表达水平与融合阴性样品进行比较。CRISP3、GLDC和TDRD1是TMPRSS2/ERG融合阳性样品中的最前面的差异表达基因,而COL2A1和PLA2G7是TMPRSS2/ERG融合阴性样品中的最前面的差异表达基因。表6给出与AA组相比CA组前列腺肿瘤中最前面的差异表达基因。表6将AA组(101个基因)中差异表达基因列表的肿瘤/正常比与CA组中的肿瘤/正常比进行比较以评价AA人种特异性基因表达趋势。图3A的热图显示在AA组中始终上调且在CA组中同时下调(或表达无变化)的基因。在该列表中,MMP9是被发现在AA组中极强上调但在CA组中下调的首位基因。表7给出与CA组相比AA组前列腺肿瘤中最前面的差异表达基因。表7基因符号AACACOL10A1539.8616.81HOXC472.0613.13ESPL135.491.92MMP932.230.27ABCA1322.652.02PCDHGA115.151.82AGSK16.090.98本文引用的所有专利、专利申请和发表的参考文献均通过引用以其整体结合到本文中。虽然参照其优选的实施方案特别显示和描述了本发明,但本领域技术人员应了解,可在不偏离随附权利要求书所包括的本发明的范围的情况下在其中进行形式和详细资料的各种改变。参考文献下列参考文献被引用于本申请中,并且提供有关本发明领域的总体信息以及提供本申请中论述的测定法和其它详细资料。下列参考文献通过引用以其整体结合到本文中。1.Siegel,R.;Naishadham,D.;Jemal,A.Cancerstatistics(癌症统计学).CACancerJ.Clin.2013,63,11-30。2.Chornokur,G.;Dalton,K.;Borysova,M.E.;Kumar,N.B.Disparitiesatpresentation,diagnosis,treatment,andsurvivalinAfricanAmericanmenaffectedbyprostatecancer(患有前列腺癌的非裔美国男性的表现、诊断、治疗和生存期的差异).Prostate2011,71,985-997。3.Schwartz,K.;Powell,I.J.;Underwood,W.,3rd;George,J.;Yee,C.;Banerjee,M.Interplayofrace,socioeconomicstatus,andtreatmentonsurvivalofpatientswithprostatecancer(人种、社会经济地位和治疗对前列腺癌患者的生存的相互作用).Urology2009,74,1296-1302。4.Major,J.M.;Oliver,M.N.;Doubeni,C.A.;Hollenbeck,A.R.;Graubard,B.I.;Sinha,R.Socioeconomicstatus,healthcaredensity,andriskofprostatecanceramongAfricanAmericanandCaucasianmeninalargeprospectivestudy(大规模前瞻性研究中非裔美国男性和高加索美国男性间的社会经济地位、医疗保健密度和前列腺癌风险).CancerCausesControl2012,23,1185-1191。5.Sridhar,G.;Masho,S.W.;Adera,T.;Ramakrishnan,V.;Roberts,J.D.DoAfricanAmericanmenhavelowersurvivalfromprostatecancercomparedwithWhitemen(非裔美国男性因前列腺癌的生存比白种人男性的低吗)Ameta-analysis.Am.JMens.Health2010,4,189-206。6.Cullen,J.;Brassell,S.;Chen,Y.;Porter,C.;L’Esperance,J.;Brand,T.;McLeod,D.G.Racial/ethnicpatternsinprostatecanceroutcomesinanactivesurveillancecohort(积极监督组群中前列腺癌结局的种族/人种模式).ProstateCancer2011,2011,doi:10.1155/2011/234519。7.Berger,A.D.;Satagopan,J.;Lee,P.;Taneja,S.S.;Osman,I.Differencesinclinicopathologicfeaturesofprostatecancerbetweenblackandwhitepatientstreatedinthe1990sand2000s(在1990和2000年代治疗的黑人和白人患者之间前列腺癌的临床病理学特征的差异).Urology2006,67,120-124。8.Kheirandish,P.;Chinegwundoh,F.Ethnicdifferencesinprostatecancer(前列腺癌的种族差异).Br.J.Cancer2011,105,481-485。9.Odedina,F.T.;Akinremi,T.O.;Chinegwundoh,F.;Roberts,R.;Yu,D.;Reams,R.R.;Freedman,M.L.;Rivers,B.;Green,B.L.;Kumar,N.ProstatecancerdisparitiesinblackmenofAfricandescent:AcomparativeliteraturereviewofprostatecancerburdenamongblackmenintheUnitedStates,Caribbean,UnitedKingdom,andWestAfrica(非洲血统黑人男性的前列腺癌差异:美国、加勒比海、联合王国和西非的黑人男性间前列腺癌负荷的比较文献综述).Infect.AgentsCancer2009,4,doi:10.1186/1750-9378-4S1-S2。10.Heath,E.I.;Kattan,M.W.;Powell,I.J.;Sakr,W.;Brand,T.C.;Rybicki,B.A.;Thompson,I.M.;Aronson,W.J.;Terris,M.K.;Kane,C.J.;等,Theeffectofrace/ethnicityontheaccuracyofthe2001PartinTablesforpredictingpathologicstageoflocalizedprostatecancer(人种/种族性对用于预测局限性前列腺癌的病理分期的2001Partin表的准确性的影响).Urology2008,71,151-155。11.Moul,J.W.;Sesterhenn,I.A.;Connelly,R.R.;Douglas,T.;Srivastava,S.;Mostofi,F.K.;McLeod,D.G.Prostate-specificantigenvaluesatthetimeofprostatecancerdiagnosisinAfrican-Americanmen(非裔美国男性中在前列腺癌诊断时的前列腺特异性抗原值).JAMA1995,274,1277-1281。12.Tewari,A.;Horninger,W.;Badani,K.K.;Hasan,M.;Coon,S.;Crawford,E.D.;Gamito,E.J.;Wei,J.;Taub,D.;Montie,J.等,Racialdifferencesinserumprostate-specific(PSA)doublingtime,histopathologicalvariablesandlong-termPSArecurrencebetweenAfrican-AmericanandwhiteAmericanmenundergoingradicalprostatectomyforclinicallylocalizedprostatecancer(进行临床局限性前列腺癌的根治性前列腺切除术的非裔美国男性和美国白种男性间血清前列腺特异性(PSA)倍增时间、组织病理学变量和长期PSA复发的人种差异).BJUInt.2005,96,29-33。13.Wallace,T.A.;Prueitt,R.L.;Yi,M.;Howe,T.M.;Gillespie,J.W.;Yfantis,H.G.;Stephens,R.M.;Caporaso,N.E.;Loffredo,C.A.;Ambs,S.TumorimmunobiologicaldifferencesinprostatecancerbetweenAfrican-AmericanandCaucasian-Americanmen(非裔美国男性和高加索美国美国男性间前列腺癌的肿瘤免疫生物学差异).CancerRes.2008,68,927-936。14.Prensner,J.R.;Rubin,M.A.;Wei,J.T.;Chinnaiyan,A.M.BeyondPSA:Thenextgenerationofprostatecancerbiomarkers(超出PSA:下一代的前列腺癌生物标志物).Sci.Transl.Med.2012,4,doi:10.1126/scitranslmed.3003180。15.Rubin,M.A.;Maher,C.A.;Chinnaiyan,A.M.Commongenerearrangementsinprostatecancer(前列腺癌中的共同基因重排).J.Clin.Oncol.2011,29,3659-3668。16.Sreenath,T.L.;Dobi,A.;Petrovics,G.;Srivastava,S.OncogenicactivationofERG:Apredominantmechanisminprostatecancer(ERG的致癌基因激活:前列腺癌的主要机制).J.Carcinog.2011,11,10-21。17.Petrovics,G.;Liu,A.;Shaheduzzaman,S.;Furasato,B.;Sun,C.;Chen,Y.;Nau,M.Ravindranath,L.;Chen,Y.;Dobi,A.等,FrequentoverexpressionofETS-relatedgene-1(ERG1)inprostatecancertranscriptome(前列腺癌转录物组中ETS相关基因-1(ERG1)的频繁过量表达).Oncogene2005,24,3847-3852。18.Tomlins,S.A.;Rhodes,D.R.;Perner,S.;Dhanasekaran,S.M.;Mehra,R.;Sun,X.W.;Varambally,S.;Cao,X.;Tchinda,J.;Kuefer,R.等,RecurrentfusionofTMPRSS2andETStranscriptionfactorgenesinprostatecancer(前列腺癌中TMPRSS2和ETS转录因子基因的反复融合).Science2005,310,644-648。19.Magi-Galluzzi,C.;Tsusuki,T.;Elson,P.;Simmerman,K.;LaFarque,C.;Esqueva,R.;Klein,E.;Rubin,M.A.;Zhou,M.TMPRSS2-ERGgenefusionprevalenceandclassaresignificantlydifferentinprostatecancerofCaucasian,African-AmericanandJapanesepatients(TMPRSS2-ERG基因融合发生率和类别在高加索美国、非裔美国和日本患者的前列腺癌中显著不同).Prostate2011,71,489-497。20.Rosen,P.;Pfister,D.;Young,D.;Petrovics,G.;Chen,Y.;Cullen,J.;Bohm,D.;Perner,S.;Dobi,A.;McLeod,D.G.等,DifferencesinfrequencyofERGoncoproteinexpressionbetweenindextumorsofCaucasianandAfricanAmericanpatientswithprostatecancer(患有前列腺癌的高加索美国患者和非裔美国患者在指标肿瘤间ERG癌蛋白质表达的频率的差异).Urology2012,80,749-753。21.Hu,Y.;Dobi,A.;Sreenath,T.;Cook,C.;Tadase,A.Y.;Ravindranath,L.;Cullen,J.;Furusato,B.;Chen,Y.;Thanqapazham,R.L.等,DelineationofTMPRSS2-ERGsplicevariantsinprostatecancer(前列腺癌中TMPRSS2-ERG剪接变体的描述).Clin.CancerRes.2008,14,4719-4725。22.GaryKGeiss,等(2008)Directmultiplexedmeasurementofgeneexpressionwithcolor-codedprobepairs(用彩色编码探针对的基因表达的直接多路复用测量),NatureBiotechnology26:317-25。23.PaoloFortina和SaulSurrey,(2008)DigitalmRNAProfiling(数字式mRNA概况分析),NatureBiotechnology26:317-25。24.FarrellJ,PetrovicsG,McLeodDG,SrivastavaS.:GeneticandmoleculardifferencesinprostatecarcinogenesisbetweenAfricanAmericanandCaucasianAmericanmen(非裔美国男性和高加索美国美国男性间前列腺癌病变的遗传和分子差异).InternationalJournalofMolecularSciences.2013;14(8):15510-31。25.Rodriquez-Suarez等,Urineasasourceforclinicalproteomeanalysis:Fromdiscoverytoclinicalapplication(作为临床蛋白质组分析来源的尿液:从发现到临床应用),BiochimicaetBiophysicaActa(2013)。26.Shi等,Antibody-free,targetedmass-spectrometricapproachforquantificationofproteinsatlowpicogrampermilliliterlevelsinhumanplasma/serum(定量测定人血浆/血清中微微克/毫升的低水平蛋白质的无抗体定向质谱方法),PNAS,109(38):15395-15400(2012)。27.Elentiboba-Johnson和Lim,Fusionpeptidesfromoncogenicchimericproteinsasspecificbiomarkersofcancer(来自作为癌症的特异性生物标志物的致癌嵌合蛋白的融合肽),MolCellProteomics,12:2714(2013)。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1