一种基因敲除选育wnt16基因缺失型斑马鱼的方法与流程
本发明涉及基因敲除技术领域,尤其涉及一种基因敲除选育wnt16基因缺失型斑马鱼的方法。
背景技术:
Wnt基因位于人类12q13,是编码356个氨基酸的转录因子。一般认为Wnt信号转导途径分为经典Wnt信号途径和非经典的Wnt信号途径。经典W nt信号途径也称为Wnt/p-catenin信号途径。在不同物种中Wnt/p-catenin信号转导的分子机制具有极高的保守性。Wnt16是非经典的wnt配体,一般认为该基因介导β-catenin信号通路,可负调节RANK信号抑制破骨细胞的形成,参与骨形成和骨吸收等过程。通过基因差异表达谱分析和基因组关联分析等,发现Wnt16基因是骨骼的一个重要的调节因子,与骨质疏松密切相关。
斑马鱼与人类在骨骼发育过程中的基因、信号通路有高度同源性,且wnt16基因进化上较为保守,在人类中,存在可变剪切体,wnt16基因有两个亚型wnt16a和wnt16b。而且,与其他动物模型相比,斑马鱼个体小、幼鱼通体透明,利于骨骼发育的观察。通过CRISPR/Cas9基因打靶技术,在斑马鱼wnt16基因上设计合适的打靶位点,将体外合成的特异性sgRNA(终浓度30ng/μL)和Cas9-mRNA(终浓度500ng/μL)显微共注射进斑马鱼一细胞内,并通过活性检测证实了所选位点的有效性。基因打靶技术起源于20世纪80年代末,是一种通过对基因组进行定点修饰来研究基因功能的重要的方法手段,也可用于治疗人类的各种遗传性疾病。该技术主要是利用缺失突变、基因灭活、染色体大片段删除以及外源基因导入等方式来改变生物的遗传信息,并且在生殖系中稳定遗传后表达突变性状,从而研究生物体内特定基因在生长发育过程中的作用,所以这类技术手段已成为现代分子生物学研究热点。传统的基因打靶技术是建立在胚胎干细胞(ESC)和同源重组技术的基础之上,故打靶技术效率极低。2013年初,一种全新的人工核酸内切酶clustered regularly interspaced shortpalindromic repeats(CRISPR)/CRISPR-associated(Cas)9,能更高效且更精确地在生物体基因组中沉默特定基因,且制作简单、成本低,且可同时对靶基因上多个位点进行剪切,沉默任意数目的单个基因,但同时该技术存在一定的缺陷,其脱靶率相对较高。
技术实现要素:
本发明的目的就在于为了解决上述问题而提供一种基因敲除选育wnt16基因缺失型斑马鱼的方法。
本发明通过以下技术方案来实现上述目的:
本发明包括以下步骤:
步骤一:CRISPR/Cas9基因敲除靶位点设计
在GenBankTM、Ensembl或UCSC数据库上查询斑马鱼wnt16基因的基因组DNA序列及其功能结构域,根据CRISPR/Cas敲除原理,设计一对wnt16基因的靶位点,靶点的选择必须遵循5’-20bp-NGG-3’标准:其中5’端的GG二核苷酸是T7启动子的一部分,设计靶位点时不受此限制,但是必须保证靶位点的3’端是NGG,靶点的选择必须确保靶点位置碱基的插入或者缺失可以影响wnt16基因的整个结构域,从而改变基因的表达,
两对特异性PCR引物如下:
F1(靶位点a正向引物):
tgTAATACGACTCACTATAgacacaagcctgttgggcagGTTTTAGAGCTAGAAATAGC
F2(靶位点b正向引物):
tgTAATACGACTCACTATAggcctcctcaccacgggtcgGTTTTAGAGCTAGAAATAGC
R(共用的反向引物):AAGCACCGACTCGGTGCCACT
步骤二:构建gRNA表达载体以及gRNA体外合成:
首先将gRNA骨架克隆到p42250载体上,取1-2μL质粒进行琼脂糖凝胶电泳检测;
特异性gRNA体外合成:用BsaI限制性内切酶线性化此质粒,一般来说,酶切反应总体积为20μL,体系如下:
离心混匀后于37℃水浴,酶切4h以上,
以线性化的p42250载体为模板,通过下面特异性引物进行PCR,扩增出用于特异性gRNA合成的双链DNA,
>PCR引物
PCR引物上下游引物分别位于3号外显子两端的内含子上:
F(5’-AGAAATCTCTCAGCCAAACACG-3’)
R(5’-ATACTGCGAACAATTCCTTGCT-3’)
正向引物F:T7启动子+20bp靶序列+gRNA上游序列
反向引物R:gRNA下游序列
PCR反应体系(25μL)如下:
震荡混匀之后,4℃离心,于PCR仪上进行扩增反应,反应条件为:预变性95℃8min,(变性95℃30s,退火64℃30s,延伸72℃20s)30个循环,再72℃10min,待PCR扩增完成后,取1μL样品点样于2.0%琼脂糖凝胶上进行电泳,凝胶成像系统拍摄结果;
检测确定条带正确之后,进行琼脂糖凝胶DNA回收,纯化回收PCR产物,
测定纯化之后的DNA浓度(尽量达到1μg),再以此DNA为模板,用20μL体系进行体外转录,合成特异性gRNA,转录实验中所用Tip头,EP管均为DEPC处理过的RNase-Free产品,具体操作如下:
体外转录反应体系(20μL):
将反应物都加入1.5mL RNase-Free的EP管中,混匀之后,于37℃水浴1.5h;
水浴结束后,取1μL样品,用配制好的2.0%的琼脂糖凝胶进行电泳,以检测转录结果,若转录产物大小与预期的相符,则说明转录成功;
向转录体系中加入1μL DNA酶,放置于37℃水浴锅中反应20min,用以消化DNA模板,再取1μL转录终产物,即gRNA进行琼脂糖凝胶电泳,以检测转录效率,
特异性gRNA的纯化:
用RNeasy Mini kit试剂盒纯化转录成功的gRNA,保存于-20℃,吸取1μL溶液进行琼脂糖凝胶电泳,以检验纯化产物,并测定纯化之后的gRNA浓度,
步骤三:斑马鱼胚胎的显微注射:
尽量在受精后30min之内,用吸管吸取胚胎转移至用琼脂糖制作的显微注射专用培养皿中,
在进行显微注射之前,将Cas9mRNA和gRNA配成混合液,充分混匀,使Cas9mRNA的终浓度为500ng/μL,gRNA的终浓度为30ng/μL,注射约1.8nL Cas9mRNA和gRNA混合液于0-1个细胞水平的受精卵内,注射过的受精卵放置于E3水(5mmol/L NaCl,0.33mmol/L CaCl2,0.33mmol/L MgSO4,0.17mmol/L KCl,)中,28℃孵化,在体式显微镜下观察胚胎表型,筛选正常发育的胚胎用于靶位点突变分析,
显微注射体系如下:
步骤四:T7E1法和Sanger测序检测靶位点的有效性:
对斑马鱼胚胎进行显微注射之后,挑选部分发育正常的早期胚胎,检测其wnt16基因是否存在突变,可以提前确认此次选择的靶位点是否有效果,显微注射操作是否规范;
步骤五:注射两个月之后,进行剪尾鉴定,同上鉴定步骤,
步骤六:目的序列的TA克隆
T7E1酶切初步鉴定有突变可能的目的序列再进行Sanger测序,若测序峰图有双峰,并且测序结果显示有插入或缺失现象的目的序列,接下来进行TA克隆之后挑取单克隆作进一步检测;
步骤七:质粒的Sanger测序:
将双酶切检测结果显示条带大小符合预期结果的质粒送往测序,根据测序之后给出的峰图和序列,在NCBI上与标准目的序列进行对比,根据比对结果,分析出每个单克隆的突变类型,
步骤八:获得可遗传的斑马鱼突变体的F1代:
通过前面一系列筛选确定了斑马鱼突变体F0代,紧接着将F0代突变体分别与野生型斑马鱼杂交得到F1代胚胎,置于28℃培养,在初期观察F1代的存活率,受精三天后,每个突变体F1代分别取10个胚胎进行突变遗传性鉴定,将每个胚胎单独提取基因组,然后进行PCR扩增出700bp的靶位点附近区域,再进行T7E1酶切分析并送部分去测序,确定此突变是否可以遗传到F1代,
如果从F1代胚胎中检测到存在突变,则将斑马鱼突变体的F1代养大至2-3个月,再分别对每条F1代斑马鱼成鱼进行剪尾,筛选F1代突变体(具体方法如前面所述),
步骤九:获得斑马鱼突变体的F2代纯合子
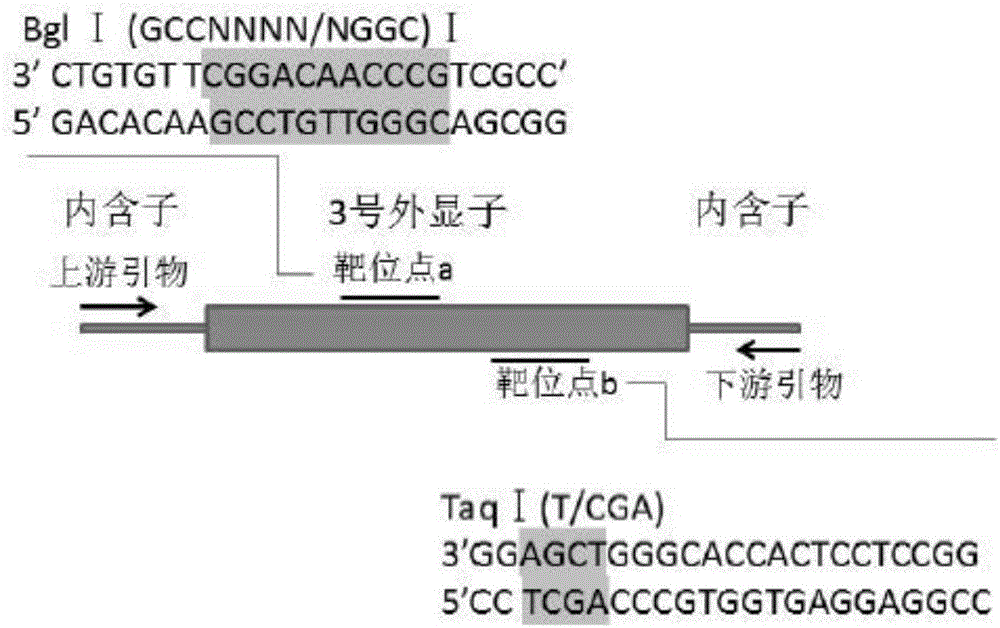
从F1代突变体中挑选相同突变的雌鱼和雄鱼,杂交得到F2代,放置于28℃培养,受精三天后取部分胚胎进行鉴定,将每个胚胎单独提取基因组,PCR扩增纯化后通过BglⅠ限制性酶切分析并测序,初步检验是否可以得到wnt16突变体纯合子,如检验结果证明存在纯合子,则养大后再单条剪尾鉴定。
所述步骤四中,检测其wnt16基因是否存在突变的方法如下:
(1)提取斑马鱼基因组
斑马鱼胚胎受精50小时后(50hpf),分别收集总数1/3野生型(做对照)和注射后胚胎于1.5mL Ep管中(每管5只胚胎),按照下述方法提取基因组DNA,具体步骤如下:
用移液枪将EP管中多余的水吸尽,向装有胚胎的Ep管中加入400μL细胞裂解液,2μL蛋白酶K,放置于55℃水浴锅中裂解4小时(期间每隔半小时,轻轻颠倒混匀,以保证胚胎被充分裂解完全);
裂解完成后,放在振荡器上充分震荡,加入等体积(400μL)异丙醇(预先冷却)于Ep管中,充分颠倒混匀,于4℃条件下,13000rpm离心10min,倒掉上清液;
加入75%乙醇500μL,于4℃条件下,13000rpm离心5min,弃上清液;
然后进行空甩,13000rpm离心1min,用移液枪小心吸取多余液体,室温干燥10-15min或烘箱干燥5min;
加入35μL去离子水,充分吹打混匀,琼脂糖凝胶电泳检测提取效率,放4℃冰箱保存,
(2)PCR扩增目的序列
提取基因组DNA之后,根据CRISPR靶位点上下游约150-200bp的基因组区域,利用Primer Premier 5.0软件设计引物序列以扩增出目的DNA片段,
PCR反应体系(50μL)如下:
震荡混匀之后,4℃离心,于PCR仪上进行扩增反应,反应条件为:预变性94℃2min,(变性94℃30s,退火55℃30s,延伸72℃23s)30个循环,再72℃2min,待反应结束后,离心PCR产物,取1μL样品点样于2.0%琼脂糖凝胶上进行电泳,检测PCR产物大小是否正确,
(3)若PCR产物正确,则用2.0%琼脂糖凝胶电泳分离PCR产物,在紫外下切下目的条带,进行纯化回收,
(4)T7核酸内切酶I(T7E1)法
用T7E1分析检测是否存在突变,首先,取纯化回收之后的DNA 15μL装于200μL的Ep管中,置于95℃热水中进行变性,然后自然冷却至室温(至少30min),再取变性之后的DNA进行T7E1酶切,体系如下:
混匀体系后,于37℃水浴锅中酶切反应30min,再用2%的凝胶进行电泳,以检测目的DNA片段是否被切开,如若目的DNA片段下面有被切开的条带,则使用ImageJ软件,通过酶切后条带的亮度来估计非同源末端连接的频率,
另外,送部分纯化之后的目的DNA片段进行Sanger测序,由测序的峰图来初步获得插入或缺失的信息。
本发明的有益效果在于:
本发明是一种基因敲除选育wnt16基因缺失型斑马鱼的方法,与现有技术相比,本发明能更高效且更精确地在生物体基因组中沉默特定基因,且制作简单、成本低,且可同时对靶基因上多个位点进行剪切,沉默任意数目的单个基因。用于进行基因与骨骼发育的相关研究,也可进项其他方面探索研究,检测wnt16基因的缺失与其他器官的发育是否具有相关性,例如心脏具有很好的医学研究价值,同时敲除wnt16基因的斑马鱼其成长周期明显缩短,也具有很好的商业价值。
附图说明
图1是CRISPR/Cas9打靶系统的原理图;
图2是wnt16基因上靶位点的结构图;
图3是缺失型和WT基因型系列反向对比图;
图4是靶位点附近缺失对比图;
图5是21号鱼F1代突变体二级结构改变的预测图。
具体实施方式
下面结合附图对本发明作进一步说明:
包括以下步骤:
步骤一:CRISPR/Cas9基因敲除靶位点设计
在GenBankTM、Ensembl或UCSC数据库上查询斑马鱼wnt16基因的基因组DNA序列及其功能结构域,根据CRISPR/Cas敲除原理,设计一对wnt16基因的靶位点,靶点的选择必须遵循5’-20bp-NGG-3’标准:其中5’端的GG二核苷酸是T7启动子的一部分,设计靶位点时不受此限制,但是必须保证靶位点的3’端是NGG,靶点的选择必须确保靶点位置碱基的插入或者缺失可以影响wnt16基因的整个结构域,从而改变基因的表达,
两对特异性PCR引物如下:
F1(靶位点a正向引物):
tgTAATACGACTCACTATAgacacaagcctgttgggcagGTTTTAGAGCTAGAAATAGC
F2(靶位点b正向引物):
tgTAATACGACTCACTATAggcctcctcaccacgggtcgGTTTTAGAGCTAGAAATAGC
R(共用的反向引物):AAGCACCGACTCGGTGCCACT
步骤二:构建gRNA表达载体以及gRNA体外合成:
首先将gRNA骨架克隆到p42250载体上,取1-2μL质粒进行琼脂糖凝胶电泳检测;
特异性gRNA体外合成:用BsaI限制性内切酶线性化此质粒,一般来说,酶切反应总体积为20μL,体系如下:
离心混匀后于37℃水浴,酶切4h以上,
以线性化的p42250载体为模板,通过下面特异性引物进行PCR,扩增出用于特异性gRNA合成的双链DNA,
>PCR引物
PCR引物上下游引物分别位于3号外显子两端的内含子上:
F(5’-AGAAATCTCTCAGCCAAACACG-3’)
R(5’-ATACTGCGAACAATTCCTTGCT-3’)
正向引物F:T7启动子+20bp靶序列+gRNA上游序列
反向引物R:gRNA下游序列
PCR反应体系(25μL)如下:
震荡混匀之后,4℃离心,于PCR仪上进行扩增反应,反应条件为:预变性95℃8min,(变性95℃30s,退火64℃30s,延伸72℃20s)30个循环,再72℃10min,待PCR扩增完成后,取1μL样品点样于2.0%琼脂糖凝胶上进行电泳,凝胶成像系统拍摄结果;
检测确定条带正确之后,进行琼脂糖凝胶DNA回收,纯化回收PCR产物,
测定纯化之后的DNA浓度(尽量达到1μg),再以此DNA为模板,用20μL体系进行体外转录,合成特异性gRNA,转录实验中所用Tip头,EP管均为DEPC处理过的RNase-Free产品,具体操作如下:
体外转录反应体系(20μL):
将反应物都加入1.5mL RNase-Free的EP管中,混匀之后,于37℃水浴1.5h;
水浴结束后,取1μL样品,用配制好的2.0%的琼脂糖凝胶进行电泳,以检测转录结果,若转录产物大小与预期的相符,则说明转录成功;
向转录体系中加入1μL DNA酶,放置于37℃水浴锅中反应20min,用以消化DNA模板,再取1μL转录终产物,即gRNA进行琼脂糖凝胶电泳,以检测转录效率,
特异性gRNA的纯化:
用RNeasy Mini kit试剂盒纯化转录成功的gRNA,保存于-20℃,吸取1μL溶液进行琼脂糖凝胶电泳,以检验纯化产物,并测定纯化之后的gRNA浓度,
步骤三:斑马鱼胚胎的显微注射:
尽量在受精后30min之内,用吸管吸取胚胎转移至用琼脂糖制作的显微注射专用培养皿中,
在进行显微注射之前,将Cas9mRNA和gRNA配成混合液,充分混匀,使Cas9mRNA的终浓度为500ng/μL,gRNA的终浓度为30ng/μL,注射约1.8nL Cas9mRNA和gRNA混合液于0-1个细胞水平的受精卵内,注射过的受精卵放置于E3水(5mmol/L NaCl,0.33mmol/L CaCl2,0.33mmol/L MgSO4,0.17mmol/L KCl,)中,28℃孵化,在体式显微镜下观察胚胎表型,筛选正常发育的胚胎用于靶位点突变分析,
显微注射体系如下:
步骤四:T7E1法和Sanger测序检测靶位点的有效性:
对斑马鱼胚胎进行显微注射之后,挑选部分发育正常的早期胚胎,检测其wnt16基因是否存在突变,可以提前确认此次选择的靶位点是否有效果,显微注射操作是否规范;
步骤五:注射两个月之后,进行剪尾鉴定,同上鉴定步骤,
步骤六:目的序列的TA克隆
T7E1酶切初步鉴定有突变可能的目的序列再进行Sanger测序,若测序峰图有双峰,并且测序结果显示有插入或缺失现象的目的序列,接下来进行TA克隆之后挑取单克隆作进一步检测;
步骤七:质粒的Sanger测序:
将双酶切检测结果显示条带大小符合预期结果的质粒送往测序,根据测序之后给出的峰图和序列,在NCBI上与标准目的序列进行对比,根据比对结果,分析出每个单克隆的突变类型,
步骤八:获得可遗传的斑马鱼突变体的F1代:
通过前面一系列筛选确定了斑马鱼突变体F0代,紧接着将F0代突变体分别与野生型斑马鱼杂交得到F1代胚胎,置于28℃培养,在初期观察F1代的存活率,受精三天后,每个突变体F1代分别取10个胚胎进行突变遗传性鉴定,将每个胚胎单独提取基因组,然后进行PCR扩增出700bp的靶位点附近区域,再进行T7E1酶切分析并送部分去测序,确定此突变是否可以遗传到F1代,
如果从F1代胚胎中检测到存在突变,则将斑马鱼突变体的F1代养大至2-3个月,再分别对每条F1代斑马鱼成鱼进行剪尾,筛选F1代突变体(具体方法如前面所述),
步骤九:获得斑马鱼突变体的F2代纯合子
从F1代突变体中挑选相同突变的雌鱼和雄鱼,杂交得到F2代,放置于28℃培养,受精三天后取部分胚胎进行鉴定,将每个胚胎单独提取基因组,PCR扩增纯化后通过BglⅠ限制性酶切分析并测序,初步检验是否可以得到wnt16突变体纯合子,如检验结果证明存在纯合子,则养大后再单条剪尾鉴定。
2、根据权利要求1所述的基因敲除选育wnt16基因缺失型斑马鱼的方法,其特征在于:所述步骤四中,检测其wnt16基因是否存在突变的方法如下:
提取斑马鱼基因组:
斑马鱼胚胎受精50小时后(50hpf),分别收集总数1/3野生型(做对照)和注射后胚胎于1.5mL Ep管中(每管5只胚胎),按照下述方法提取基因组DNA,具体步骤如下:
用移液枪将EP管中多余的水吸尽,向装有胚胎的Ep管中加入400μL细胞裂解液,2μL蛋白酶K,放置于55℃水浴锅中裂解4小时(期间每隔半小时,轻轻颠倒混匀,以保证胚胎被充分裂解完全);
裂解完成后,放在振荡器上充分震荡,加入等体积(400μL)异丙醇(预先冷却)于Ep管中,充分颠倒混匀,于4℃条件下,13000rpm离心10min,倒掉上清液;
加入75%乙醇500μL,于4℃条件下,13000rpm离心5min,弃上清液;
然后进行空甩,13000rpm离心1min,用移液枪小心吸取多余液体,室温干燥10-15min或烘箱干燥5min;
加入35μL去离子水,充分吹打混匀,琼脂糖凝胶电泳检测提取效率,放4℃冰箱保存,
PCR扩增目的序列:
提取基因组DNA之后,根据CRISPR靶位点上下游约150-200bp的基因组区域,利用Primer Premier 5.0软件设计引物序列以扩增出目的DNA片段,
PCR反应体系(50μL)如下:
震荡混匀之后,4℃离心,于PCR仪上进行扩增反应,反应条件为:预变性94℃2min,(变性94℃30s,退火55℃30s,延伸72℃23s)30个循环,再72℃2min,待反应结束后,离心PCR产物,取1μL样品点样于2.0%琼脂糖凝胶上进行电泳,检测PCR产物大小是否正确,
若PCR产物正确,则用2.0%琼脂糖凝胶电泳分离PCR产物,在紫外下切下目的条带,进行纯化回收,
T7核酸内切酶I(T7E1)法:
用T7E1分析检测是否存在突变,首先,取纯化回收之后的DNA 15μL装于200μL的Ep管中,置于95℃热水中进行变性,然后自然冷却至室温(至少30min),再取变性之后的DNA进行T7E1酶切,体系如下:
混匀体系后,于37℃水浴锅中酶切反应30min,再用2%的凝胶进行电泳,以检测目的DNA片段是否被切开,如若目的DNA片段下面有被切开的条带,则使用ImageJ软件,通过酶切后条带的亮度来估计非同源末端连接的频率,
另外,送部分纯化之后的目的DNA片段进行Sanger测序,由测序的峰图来初步获得插入或缺失的信息。
由于筛选到的21号突变体的F1代的wnt16基因部分碱基缺失没有造成整个基因的移码突变,不能完全改变斑马鱼wnt16基因的表达。接下来,利用蛋白质空间结构预测软件Phyre对筛选到的F1代突变体Wnt16的结构进行预测,结果如图1、图2和图3所示,是野生型斑马鱼和21号斑马鱼F1代突变体的wnt16蛋白的二级结构的对比结果,显示wnt16的某些保守结构域处有改变(图中用圆圈标识)。将碱基系列翻译成氨基酸序列,发现仅改变部分氨基酸没有造成移码,在整个WNT16蛋白上突变区域位于第146-182氨基酸,wnt16基因所编码的蛋白质的二级结构发生改变。结果说明所筛选到的21号斑马鱼的F1代整个wnt16结构域有可能改变,可能会影响斑马鱼的骨骼生长机制
以上显示和描述了本发明的基本原理和主要特征及本发明的优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下,本发明还会有各种变化和改进,这些变化和改进都落入要求保护的本发明范围内。本发明要求保护范围由所附的权利要求书及其等效物界定。
- 还没有人留言评论。精彩留言会获得点赞!