一种热稳定性提高的β‑淀粉酶突变体的制作方法
本发明涉及一种热稳定性提高的β-淀粉酶突变体,属于基因工程和酶工程技术领域。
背景技术:
β-淀粉酶又称为麦芽糖苷酶、糖原淀粉酶,其系统名为α-1,4-葡聚糖-4-麦芽糖水解酶(α-1,4-dglucanmaltohydrolase),e.c.编号为3.2.1.2.作用于淀粉时,从其非还原性末端顺次切下麦芽糖,水解产物是β-麦芽糖、β-极限糊精及非常少量的β-葡萄糖。β-淀粉酶具有相当大的工业应用价值,长期以来植物来源的大麦β-淀粉酶一直被广泛用于多个领域。例如,在啤酒发酵业中部分代替大麦芽用于啤酒的生产,在食品业中用于水解淀粉制造麦芽糖浆和饴糖等。自1974年higashihara等人首次确定巨大芽孢杆菌(bacillusmegaterium)胞外淀粉酶含有β-淀粉酶以来,相继发现了几种能够产生该酶的微生物,这些菌株大都属于细菌,如蜡状芽孢杆菌(bacilluscereus)、多粘芽孢杆菌(bacilluspolymyxa)、环状芽孢杆菌(bacilluscirculans)、高温放线菌(thermeoactinomycessp.)和热硫梭菌(clostridiumthermosulfurogenes)等。与植物β-淀粉酶相比,微生物来源的β-淀粉酶具有便于大规模工业化生产的优点,因此筛选寻找理想的微生物β-淀粉酶源成为淀粉酶研究应用领域的一个重要目标。
定向进化属于蛋白质的非理性设计,不需要事先了解蛋白质的结构、催化机制、保守序列等因素,而是通过模拟自然进化机制来为酶基因创造体外进化的条件,进而对目的蛋白进行分子改造,并定向筛选研究进化酶的酶学性质。易错pcr技术是目前应用最广泛的定向进化方法之一,具有操作方便、快速、高效的优点。对于微生物来源β-淀粉酶的研究,目前主要是围绕不同宿主的克隆表达、酶学定性,而针对其酶学性质的分子改造显得较少,在定向进化研究其热稳定性方面,尚未见相关文献报道。
发明人在前期的研究中从菜地土壤中成功的筛选到了一株高产β-淀粉酶的弯曲芽孢杆菌(bacillusflexus)cctcc2015368的菌株,并将该菌株的β-淀粉酶基因序列克隆出来,成功的在大肠杆菌(escherichiacoli)宿主中表达。
技术实现要素:
本发明的目的在于提供一种热稳定性提高的β-淀粉酶突变体。
所述热稳定性提高的β-淀粉酶突变体,是对亲本弯曲芽孢杆菌(bacillusflexus)cctcc2015368β-淀粉酶进行定向进化,通过分子进化的手段之一易错pcr技术,获得β-淀粉酶突变体。亲本β-淀粉酶氨基酸序列如seqidno:1所示。采用“原始氨基酸-替换位置-突变后氨基酸”来表示β-淀粉酶突变体,β-淀粉酶突变体为thr461tyr/asp506asn/asp519lys,氨基酸序列为seqidno:2。
用于表达所述的β-淀粉酶突变体的表达载体为pet24a(+);
用于所述的表达载体转化的微生物宿主为大肠杆菌(escherichiacoli)。
与亲本弯曲芽孢杆菌β-淀粉酶相比(55℃下的半衰期t50为18.0min),所述β-淀粉酶突变体在55℃下的半衰期t50提高了3倍,达到54min。本发明的有益效果:运用了分子生物学的方法对亲本弯曲芽孢杆菌β-淀粉酶进行了定向进化研究,通过易错pcr技术获得了β-淀粉酶突变体,与亲本弯曲芽孢杆菌β-淀粉酶相比(55℃下的半衰期t50为18.0min),β-淀粉酶突变体在55℃下的半衰期t50提高了3倍,达到54min。
附图说明
图1:野生型β-淀粉酶与突变体bea-10的最适温度比较;
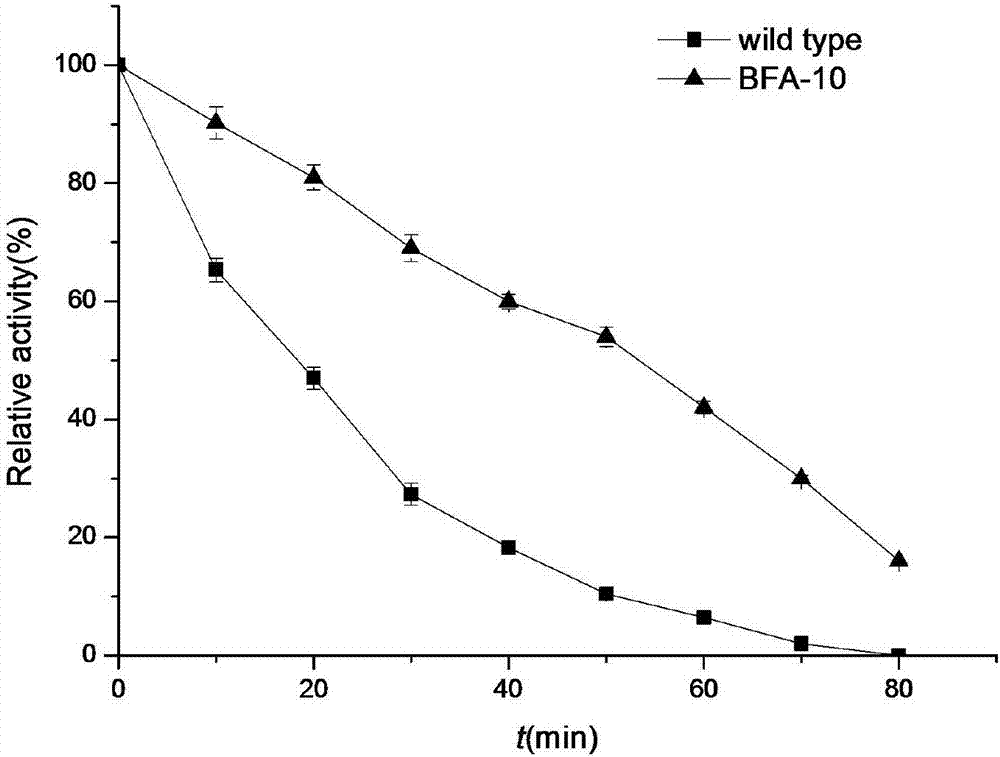
图2:野生型β-淀粉酶与突变体bea-10在55℃下的热稳定性比较。
具体实施方式
实例中涉及到的培养基及试剂配方如下:
lb液体培养基:酵母粉5.0g/l,胰蛋白胨10.0g/l,nacl10.0g/l。
lb固体培养基:在lb液体培养基的基础配方上,添加1.0%-2.0%(m/v)的琼脂。
tb培养基:酵母粉24.0g/l,甘油5.0g/l,胰蛋白胨12.0g/l,k2hpo4·3h2o16.43g/l,kh2po42.31g/l。
sob培养基:胰蛋白胨20.0g/l,酵母粉5.0g/l,mgcl20.952g/l,nacl0.5g/l,kcl0.186g/l。
lb琼脂淀粉板:在lb液体培养基的基础上添加可溶性淀粉1g/l,用于突变体的初步筛选。
实施例1:基于易错pcr技术构建β-淀粉酶突变体文库
运用易错pcr技术向弯曲芽孢杆菌β-淀粉酶基因中引入核苷酸突变。易错pcr的反应条件如下:
其中上游引物f和下游引物r的序列分别为:
f:(5′-aaccatggcggtaaatggacagtcgtt-3′);
r:(5′-aactcgagttaccaattattcgtatacgttg-3′)。
pcr扩增程序为:94℃4min;98℃10s,53℃10s,72℃1min40s,30个循环;72℃延伸10min,并于4℃下保温。
易错pcr产物经琼脂糖凝胶试剂盒胶回收,目的基因与载体pmd-18t连接转化jm109,克隆培养后,用限制酶ncoi和xhoi将目的基因片段回收与经过ncoi和xhoi双酶切消化后的表达载体pet24a(+)相连,转化至e.colijm109感受态细胞。涂布lb抗性平板(kan),37℃培养8-10个小时,将转化子全部转移至lb液体培养基中培养,并抽提重组突变体质粒。
将重组突变体质粒转化e.colibl21(de3)感受态细胞。在lb平板上(kan)培养获取β-淀粉酶突变体文库。
实施例2:热稳定性提高的β-淀粉酶突变体的筛选
以pet24a(+)表达亲本β-淀粉酶的大肠杆菌作为对照菌。
lb琼脂淀粉板初筛:重组菌e.colibl21(de3)/pet24a(+)-bfa于tb发酵培养基中诱导表达48h,离心获得胞外上清,利用高通量筛选系统将96空板中每一个突变体的发酵液一一转移到lb琼脂淀粉板上。37℃下过夜培养8h左右,用碘液(0.03%w/v)涂布琼脂淀粉板,观察透明圈,选取比对照明显大的透明圈所对应的突变菌株。
96孔板dns法定量筛选:挑选透明圈比对照高的突变菌株,用排枪快速将发酵上清转移到pcr管中(转移前用1:1的磷酸缓冲液与发酵液混合),在55℃的金属浴上保温40min,采用dns法测定保温前与保温后的酶活,在540nm处下测定吸光值并记录数据,通过计算残留酶活力从而选出热稳定性有提高的突变株。
实施例3:酶活分析方法
一个β-淀粉酶酶活单位(1u)定义(u/ml):1ml酶液在ph7.0,温度55℃,1h水解可溶性淀粉生产1mg麦芽糖所需的酶量称为一个酶活力单位。
测定方法:准确吸取0.5ml2%可溶性淀粉溶液,置于15ml比色管中,加0.4ml50mmph7.0磷酸盐缓冲溶液,摇匀,于55℃水浴预热5min,准确加入100μl酶液,立即计时,摇匀,于55℃水浴准确保温酶解反应10min,立即加入dns1ml,摇匀,并煮沸5min,冰水中冷却。以同样条件下用缓冲液代替酶液的反应体系作为对照,上述反应体系加10ml蒸馏水,混匀,1cm比色皿,540nm波长下测吸光度。
实施例3:β-淀粉酶突变体的纯化
将发酵上清液于4℃,12000rpm离心10min除去细胞。将上清液置于磁力搅拌器中并缓缓加入45%固体硫酸铵盐,于4℃过夜盐析。然后4℃,12000rpm离心20min。取沉淀物用适量ph7.0,50mm磷酸-柠檬酸缓冲液溶解,通过0.4μm透析膜过滤透析后得到浓缩酶样品。经过凝胶层析柱纯化后得到电泳纯的β-淀粉酶。
实施例4:β-淀粉酶突变体的最适温度和热稳定性
酶分子热稳性的提高可以用半衰期(t50)来体现。即酶分子的活力降低到初始活力一半时所需要的时间。酶的半衰期时间越长,则酶的热稳定性越高。反之,它的热稳定就比较差。
以ph7.0的柠檬酸-磷酸氢二钠(50mm)为缓冲液,在30-70℃温度范围测定野生型β-淀粉酶及突变体bfa-10(即thr461tyr/asp506asn/asp519lys)的最适温度。突变体和野生型β-淀粉酶的最适温度和热稳定性见图1和图2。
从图1可以得知,野生型和突变体bfa-10β-淀粉酶的最适温度均为55℃;在30-55℃的温度范围内,β-淀粉酶的相对活力没有明显的变化;值得注意的是,当温度升高到60℃和65℃时,突变体bfa-10能够保留最初酶活的75%和44%,而野生型仅保留其酶活的37%和23%。在最适温度55℃的条件下,测定不同时间点残留酶活来确定突变体bfa-10的热稳定性,并与野生型相比较(图2);从图2可知,突变体bfa-10在55℃下的半衰期为54min,而野生型的半衰期只有18min。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>江南大学
<120>一种热稳定性提高的β-淀粉酶突变体
<160>4
<170>patentinversion3.3
<210>1
<211>545
<212>prt
<213>弯曲芽孢杆菌
<400>1
mettyrlysproilelyslysphealaserleuilevalleuleuser
151015
phevalalaalapheileleuglyprothrasnserglnalaalaval
202530
asnglyglnserpheasnserasntyrlysthrtyrleumetalapro
354045
leulyslysvalthrgluphethrthrtrpglualaphegluasnasp
505560
leuarglysalalysglnasnglyphetyralavalthrvalaspphe
65707580
trptrpglyaspmetglulysasnglyaspglnglnpheasppheser
859095
tyralaglnargphealaglnalaalaargasnalaglyilelysmet
100105110
valproileileserthrhisglncysglyglyasnvalglyaspasp
115120125
cysasnthrproleuprosertrpiletrpasnthrlysthraspasp
130135140
serleutyrphelyssergluthrglythrvalasnlysgluthrval
145150155160
asnproleualathraspvalilethrlysglntyrglygluleutyr
165170175
thralaphealaglnalaleualaprotyrlysaspileileprolys
180185190
valtyrleuserglyglyproalaglygluleuargtyrprosertyr
195200205
thralaalaaspglythrglytyrproserargglylyspheglnala
210215220
tyrthraspphealalysserlyspheglnmettrpalavalasnlys
225230235240
tyrglyserleualaglyvalasnglnalatrpglyleuserleuthr
245250255
serthrserglnileleuproproseraspglyasnglnpheleulys
260265270
aspglytyrasnthrasntyrglylysasppheleuglutrptyrgln
275280285
glyvalleuglnasphisalalysargileglyalaleualahisgln
290295300
alapheaspprovalpheasnvalprovalglyalalysilealagly
305310315320
ilehistrpglntyrasnasnprothrmetprohisalaalaglulys
325330335
proalaglytyrasnasntyrserthrleuleuaspserphelysthr
340345350
alalysleuaspleuthrphethrcysleuglumetvalaspsergly
355360365
thrtyrproglutyrsermetprolysthrleuvallysgluvalala
370375380
serleualaasnalalysglyilevalleuasnglygluasnalaleu
385390395400
serileglyserglugluglntyrlysargalaalaglumetthrphe
405410415
asntyrasnphealaglyphethrleuleuargphetyraspvalile
420425430
asnasnserthrargmetserglnpheasnglnhisleuasnilelys
435440445
provalalaglnthrmetvalvallysasnalaprothrsersergly
450455460
gluservaltyrilevalglyaspargprogluleuglyglntrpasp
465470475480
thrilealatyrproilelysleusertyrasnserthrtyrglyasp
485490495
trpargglythrvalasnpheproalaaspargservalglnphelys
500505510
alaileilelysargseraspglyserleulyssertrpglnprothr
515520525
glnglntyrtrpasnvalproglythrprothrthrtyrthrasnasn
530535540
trp
545
<210>2
<211>545
<212>prt
<213>弯曲芽孢杆菌
<400>2
mettyrlysproilelyslysphealaserleuilevalleuleuser
151015
phevalalaalapheileleuglyprothrasnserglnalaalaval
202530
asnglyglnserpheasnserasntyrlysthrtyrleumetalapro
354045
leulyslysvalthrgluphethrthrtrpglualaphegluasnasp
505560
leuarglysalalysglnasnglyphetyralavalthrvalaspphe
65707580
trptrpglyaspmetglulysasnglyaspglnglnpheasppheser
859095
tyralaglnargphealaglnalaalaargasnalaglyilelysmet
100105110
valproileileserthrhisglncysglyglyasnvalglyaspasp
115120125
cysasnthrproleuprosertrpiletrpasnthrlysthraspasp
130135140
serleutyrphelyssergluthrglythrvalasnlysgluthrval
145150155160
asnproleualathraspvalilethrlysglntyrglygluleutyr
165170175
thralaphealaglnalaleualaprotyrlysaspileileprolys
180185190
valtyrleuserglyglyproalaglygluleuargtyrprosertyr
195200205
thralaalaaspglythrglytyrproserargglylyspheglnala
210215220
tyrthraspphealalysserlyspheglnmettrpalavalasnlys
225230235240
tyrglyserleualaglyvalasnglnalatrpglyleuserleuthr
245250255
serthrserglnileleuproproseraspglyasnglnpheleulys
260265270
aspglytyrasnthrasntyrglylysasppheleuglutrptyrgln
275280285
glyvalleuglnasphisalalysargileglyalaleualahisgln
290295300
alapheaspprovalpheasnvalprovalglyalalysilealagly
305310315320
ilehistrpglntyrasnasnprothrmetprohisalaalaglulys
325330335
proalaglytyrasnasntyrserthrleuleuaspserphelysthr
340345350
alalysleuaspleuthrphethrcysleuglumetvalaspsergly
355360365
thrtyrproglutyrsermetprolysthrleuvallysgluvalala
370375380
serleualaasnalalysglyilevalleuasnglygluasnalaleu
385390395400
serileglyserglugluglntyrlysargalaalaglumetthrphe
405410415
asntyrasnphealaglyphethrleuleuargphetyraspvalile
420425430
asnasnserthrargmetserglnpheasnglnhisleuasnilelys
435440445
provalalaglnthrmetvalvallysasnalaprotyrsersergly
450455460
gluservaltyrilevalglyaspargprogluleuglyglntrpasp
465470475480
thrilealatyrproilelysleusertyrasnserthrtyrglyasp
485490495
trpargglythrvalasnpheproalaasnargservalglnphelys
500505510
alaileilelysargserlysglyserleulyssertrpglnprothr
515520525
glnglntyrtrpasnvalproglythrprothrthrtyrthrasnasn
530535540
trp
545
<210>3
<211>27
<212>dna
<213>人工序列,用于pcr
<400>3
aaccatggcggtaaatggacagtcgtt27
<210>4
<211>31
<212>dna
<213>人工序列,用于pcr
<400>4
aactcgagttaccaattattcgtatacgttg31
- 还没有人留言评论。精彩留言会获得点赞!