一种碱性耐盐普鲁兰酶PulA及其基因和应用的制作方法
本发明涉及基因工程领域,具体地,本发明涉及一种碱性耐盐普鲁兰酶pula及其基因和应用。
背景技术:
淀粉是由α-d葡萄糖单体通过α-1,4-糖苷键或α-1,6-糖苷键聚合而成的高分子化合物。动植物中存在着大量的淀粉。各类植物中含有较高比例的淀粉,淀粉是植物体储存营养物质的一种方式,植物中的淀粉是以球状颗粒的形式贮藏,分布于种子、根、块茎等。
淀粉酶(amylase)是指能水解淀粉和糖原的一类酶的总称。淀粉酶作为一种催化剂,已经被广泛地应用于各个工业领域。大部分的植物、动物、微生物中都含有淀粉酶。根据淀粉酶水解底物产生的糖异构体类型的不同而将其分为α和β淀粉酶。根据淀粉酶的最适反应温度和热稳定性的不同,可将其可分为:高温淀粉酶、中温淀粉酶和低温淀粉酶。根据淀粉酶的最适ph和ph稳定性的不同可将其分为:酸性、中性和碱性淀粉酶。根据淀粉酶水解糖苷键的方式的不同,可将其主要分为:α-淀粉酶、β-淀粉酶、葡萄糖淀粉酶、异淀粉酶、支链淀粉酶等。
普鲁兰酶是一类淀粉脱支酶,能够专一性水解糖原和支链淀粉分支点中的α-1,6-糖苷键,切下支链淀粉的整个侧链将其变成直链淀粉。普鲁兰酶与异淀粉酶有所不同,普鲁兰酶能够水解由两到三个葡萄糖残基构成的侧链而异淀粉酶却不能。按水解位点的不同,普鲁兰酶可分为两种类型,一种是ⅰ型普鲁兰酶,专一性水解α-1,6-糖苷键;另一种是ⅱ型普鲁兰酶,也被称为淀粉普鲁兰酶,其同时具有水解α-1,6-糖苷键和内切低聚糖α-1,4-糖苷键的能力。普鲁兰酶作为一种重要的糖苷水解酶,在淀粉加工、葡萄糖浆生产、啤酒生产和医药化工等方面具有巨大的应用价值。
目前已报导的产普鲁兰酶的微生物很多,其中大部为细菌,如芽胞杆菌、微小杆菌和嗜冷希瓦氏菌属等。此外,还有部分普鲁兰酶从嗜热古菌和真菌中被发现。因为淀粉的糖化过程需要在较高的温度(55~60℃)、微酸性条件下(ph4.5~5.5)进行,酸性普鲁兰酶在糖浆工业中可提高利用率、降低粮耗和提高产品质量,从而目前报道较多的普鲁兰酶大都是酸性酶。碱性普鲁兰酶在洗涤剂工业中具有重要的应用前景。然而目前对于碱性普鲁兰酶的研究报导很少。此外,关于碱性普鲁兰酶的专利也鲜有报导。
本发明从大布苏盐碱湖底泥中分离了一株能够产碱性耐盐普鲁兰酶的嗜盐嗜碱菌alkalibacteriumsp.,利用分子生物学和基因工程的方法获得了该碱性耐盐普鲁兰酶的编码基因,并对该基因进行了重组表达,重组酶具有在碱性条件下高活性和高稳定性的特点。此外,该酶具有很好的盐耐受性和稳定性,在4mnacl存在的情况下还剩余45%以上的酶活。此外,该酶对普鲁兰糖的水解产物为三糖,表明该酶为i型普鲁兰酶。因此,该普鲁兰酶所具有的碱性耐盐的特点,在洗涤剂和食品工业领域具有很大的应用潜力。
技术实现要素:
本发明的目的是提供一种碱性耐盐普鲁兰酶pula及其基因和应用。
本发明的另一目的是提供包含上述基因的重组载体。
本发明的另一目的是提供包含上述基因的重组菌株。
本发明的另一目的是提供一种制备上述碱性耐盐普鲁兰酶的基因工程方法。
本发明的另一目的提供上述碱性耐盐普鲁兰酶的应用。
本发明首先获得一种碱性耐盐普鲁兰酶pula,在碱性及高盐条件下具有很高的酶活。这些性质符合洗涤工业中对普鲁兰酶的碱性的要求,增强去污能力。
本发明所述碱性耐盐普鲁兰酶pula,其氨基酸序列如seqidno.1所示。
本发明的普鲁兰酶pula总共含1184个氨基酸,经软件预测无信号肽,理论分子量为132.09kda,理论等电点为4.07。
本发明的普鲁兰酶pula的最适ph为9.0,在ph10.0时剩余77.4%的酶活。该普鲁兰酶在ph6.0-10.0之间均很稳定,在此ph范围内处理60min后剩余酶活性在75%以上,这说明此酶具有较好的ph稳定性。
本发明的普鲁兰酶pula的最适温度为50℃,在40-55℃时剩余50%以上的酶活。在不存在底物的情况下,该酶在45℃较稳定,处理一小时后剩余50%左右的酶活。该酶在50℃时的热稳定性不太好,处理一小时只剩余20%左右的酶活。
本发明的普鲁兰酶pula还具有很好的盐的耐受性和稳定性。在0.25–3.0mnacl存在的情况下剩余55%以上的酶活。而且在0.25mnacl存在的时候该酶的活性得到促进,活力为对照的105%。此外,该酶在3m和4mnacl的浓度下处理60min,均剩余95%以上的酶活,表明该酶具有很好的盐耐受性。
本发明还提供了编码上述碱性耐盐普鲁兰酶pula的基因,该基因的碱基序列如如seqidno.2所示。
本发明通过pcr的方法分离克隆了这一普鲁兰酶基因pula,dna全序列分析结果表明,普鲁兰酶pula结构基因pula全长3555bp,起始密码子为atg,终止密码子为taa,gc含量50%,编码1184个氨基酸组成的多肽(pula),无信号肽序列,n端为一个第41家族的碳水化合物结合结构域(cbm),c端为一个普鲁兰酶的催化结构域。在genbank中的比对结果表明它与alkalibacteriumpelagium(sek70797)的预测的普鲁兰酶最高一致性为93%,而且数据库中与它相似性大于50%以上的普鲁兰酶基因均未做过性质研究,表明pula是一个新的普鲁兰酶。
本发明还提供了包含上述碱性耐盐普鲁兰酶基因pula的重组载体,优选为pet22b-pula。将本发明的普鲁兰酶基因pula插入到表达载体合适的限制性酶切位点之间,使其核苷酸序列可操作的与表达调控序列相连接。作为本发明的一个最优选的实施方案,优选为将普鲁兰酶基因pula插入到质粒pet22b(+)上的ncoi和noti限制性酶切位点之间,得到重组表达质粒pet22b-pula。
本发明还提供了包含上述碱性耐盐普鲁兰酶基因pula的重组菌株,优选所述菌株为大肠杆菌、酵母菌、芽孢杆菌和丝状真菌,优选为重组菌株bl21(de3)/pula。
本发明还提供了一种制备碱性耐盐普鲁兰酶pula的方法,包括以下步骤:
1)用上述的重组载体转化宿主细胞,得重组菌株;
2)培养重组菌株,诱导重组普鲁兰酶的表达;
3)回收并纯化所表达的普鲁兰酶pula。
其中,优选所述宿主细胞为大肠杆菌细胞,优选将重组大肠杆菌表达质粒转化大肠杆菌细胞bl21(de3),得到重组菌株bl21/pula。
本发明还提供了上述碱性耐盐普鲁兰酶pula的应用。
本发明提供了一种性质优良的、适合于在洗涤和食品工业中应用的新普鲁兰酶pula。本发明所提供的普鲁兰酶pula最适反应温度为50℃,该酶的最适ph为9.0,在ph10.0的时候剩余77.4%的酶活。该普鲁兰酶还具有很好的ph耐受性,在ph6.0~11.0非常稳定。此外,该酶在0.25–3.0mnacl存在的情况下剩余50%以上的酶活,且在3m和4mnacl的浓度下非常稳定。本发明的普鲁兰酶pula具有碱性耐盐的特点,因此本发明的碱性耐盐普鲁兰酶pula可在洗涤剂工业中使用,增强去污能力。此外,该酶还可以应用于食品工业。
附图说明
图1:在大肠杆菌中表达的重组普鲁兰酶pula的sds-page分析。其中,1:低分子量蛋白质marker;2:未诱导的含有普鲁兰酶基因载体pet-pula的大肠杆菌培养上清液;3:诱导的含有普鲁兰酶基因载体pet-pula的大肠杆菌培养上清液浓缩液;4:通过镍柱纯化的达到电泳纯的pula蛋白。
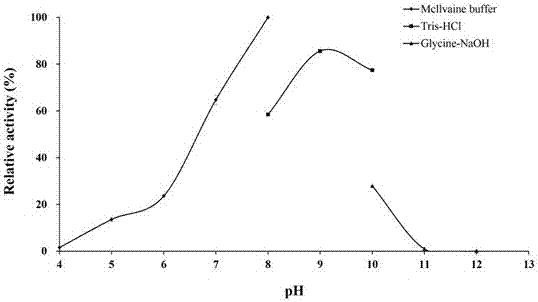
图2:重组普鲁兰酶pula的最适ph。
图3:重组普鲁兰酶pula的ph稳定性。
图4:重组普鲁兰酶pula的最适温度。
图5:重组普鲁兰酶pula的热稳定性。
图6:钙离子对重组普鲁兰酶pula活性的影响。
图7:重组普鲁兰酶pula的盐耐受性。
图8:重组普鲁兰酶pula的盐稳定性。
图9:重组普鲁兰酶pula的对普鲁兰糖的降解产物分析。
具体实施方式
试验材料和试剂
1、菌株及载体:大肠杆菌表达载体pet22b(+)及菌株escherichiacolibl21(de3)购自于novagen公司。
2、酶类及其它生化试剂:限制性内切酶、t4dna连接酶、dna聚合酶、dntps和pmd18-t载体购于日本takara公司。基因组提取试剂盒购于北京tiangen公司,纯化及质粒提取试剂盒购于美国omega公司。普鲁兰糖(pullulan)购于美国sigma公司;蛋白胨(tryptone)、酵母提取物(yeastextract)为英国oxoid公司产品,其余试剂为国产分析纯。
3、培养基:
(1)lb培养基(g/l):酵母粉5.0,蛋白胨10.0,nacl10.0,ph7.0。
(2)平板筛选培养基(g/l):酵母粉5.0,蛋白胨10.0,nacl10.0,琼脂15.0,ph7.0。
说明:以下实施例中未作具体说明的分子生物学实验方法,均参照《分子克隆实验指南》(第三版)j.萨姆布鲁克一书中所列的具体方法进行,或者按照试剂盒和产品说明书进行。
实施例1:普鲁兰酶基因pula的克隆
嗜盐嗜碱菌基因组dna的提取:利用淀粉为唯一碳源的筛选培养基从吉林乾安大布苏盐碱湖的底泥中分离得到一株产碱性普鲁兰酶的微生物,经16s鉴定为嗜盐嗜碱菌属。采用天根公司的细菌基因组提取试剂盒(dp302)提取该嗜盐嗜碱菌的基因组dna,具体操作完全按照该试剂盒的说明书进行。
根据普鲁兰酶基因的保守(hvrdftsdp和fdmmgdhd)序列设计合成了简并引物pul-f和pul-r:
pul-f:5'-gtncgngayttyacntcngayc-3';
pul-r:5'-tcrtgrtcncccatcatrtc-3'
以上述的嗜盐嗜碱菌基因组总dna为模板进行pcr扩增。采用降落pcr方法进行基因片段的扩增。反应参数为:94℃预变性5min;94℃变性30sec,50-45℃退火30sec,72℃延伸30sec,10个循环(每个循环降落0.5℃)。然后94℃变性30sec,45℃退火30sec,72℃延伸30sec,30个循环,72℃保温5min。得到一约570bp片段,将该片段回收后与pmd18-t载体相连送到英俊公司测序。
根据测序得到的普鲁兰酶基因的保守区的核甘酸序列,设计上游和下游各三条tail-pcr特异性引物:设计方向为需要扩增的未知区域方向,sp2的位置设计在sp1的内侧,sp3位于sp2的内侧。每两个引物之间的距离没有严格规定(为方便电泳识别结果,sp2和sp3一般间距100bp),引物长度一般22~30nt,退火温度在62℃。并将它们分别命名为sl4-usp1,sl4-usp2,sl4-usp3(上游特异性引物);sl4-dsp1,sl4-dsp2,sl4-dsp3(下游特异性引物)见表1。按照tail-pcr中的程序对两端侧翼序列进行扩增。
表1普鲁兰酶pulatail-pcr扩增和全长扩增所需的引物
通过tail-pcr得到已知基因序列的侧翼序列,扩增得到产物回收后送英俊公司测序。通过序列拼接获得该片段的上下游侧翼序列,全序列共长4.2kb,找到一个完整的开放阅读框(orf)。普鲁兰酶基因pula由3555个碱基组成,编码1184个氨基酸和一个终止密码子,在genbank中的比对结果表明它与来源于alkalibacteriumpelagium(sek70797)的预测的普鲁兰酶最高一致性为93%。而且数据库中与它相似性大于50%以上的普鲁兰酶基因均未做过性质研究。pula编码蛋白预计分子量为132kda,等电点为4.07。经预测,pula无信号肽序列,中间为一个第41家族的碳水化合物结构域,其c末端为一普鲁兰酶催化结构域。
实施例2重组普鲁兰酶的制备。
以在基因5’和3’端分别引入了ncoi和noti酶切位点pula-m-f和pula-m-r为引物对(见表1),嗜盐嗜碱菌基因组dna为模板,进行pcr扩增。pcr反应参数为:94℃预变性5min;94℃变性30sec,54℃退火30sec,72℃延伸4min,30个循环,72℃保温5min。将表达载体pet22b(+)进行双酶切(ncoi+noti),同时将编码普鲁兰酶的基因pula双酶切(ecori+noti),切好的编码普鲁兰酶的基因片段与表达载体pet22b(+)连接,获得含有普鲁兰酶基因pula的重组质粒pet22b-pula并转化大肠杆菌bl21(de3),获得重组大肠杆菌菌株bl21/pula。
取含有重组质粒pet22b-pula的bl21菌株,接种于5mllb(100µg/ml的氨苄青霉素)培养液中,37℃培养过夜。将培养好的菌液按1%接种于20mllb(加有100µg/ml的氨苄青霉素),37℃振荡培养约2~3h(od600达到0.5)后加入终浓度1mmol/l的诱导剂iptg,30℃180rpm震荡培养12h。12000rpm离心5min,收集培养基上清。dns法测定普鲁兰酶的活力。sds-page结果(图1)表明,重组普鲁兰酶在大肠杆菌中得到了表达。所表达的普鲁兰酶经过纯化之后,其蛋白质的含量达到总蛋白的95%以上。
实施例3重组普鲁兰酶pula的性质测定
1、重组普鲁兰酶的活性分析
重组普鲁兰酶的活性测定采取3,5-二硝基水杨酸(dns)法:具体方法如下:在ph9.0,50℃条件下,1ml的反应体系包括100μl适当的稀释酶液,900μl底物(1%榉木普鲁兰糖),反应10min,加入1.5mldns终止反应,沸水煮5min。冷却后540nm测定od值。1个酶活单位(u)定义为在给定的条件下每分钟释放出1μmol还原糖的所需的酶量。
2、重组普鲁兰酶pula的最适ph和ph稳定性的测定
最适ph测定:将纯化好的重组普鲁兰酶pula在37℃下和0.1mph4.0–11.0的缓冲液中进行酶促反应。酶的ph稳定性测定:将酶液置于0.1mph4.0–11.0的缓冲液中,在37℃下处理1h,然后在ph9.0及50℃下进行酶促反应,以未处理的酶液作为对照。缓冲液为:0.1mmcilvainebuffer(ph4.0–7.0),0.1mtris-hclbuffer(ph7.0–9.0)和0.1mglycine-naoh(ph9.0–11.0)。以1%普鲁兰糖为底物,反应10min,测定pula的活力。结果表明:pula的最适ph为9.0,在ph10.0剩余77.4%的酶活(图2);此普鲁兰酶在ph6.0-10.0之间均很稳定,在此ph范围内处理60min后剩余酶活性在75%以上,这说明此酶具有较好的ph稳定性(图3)。
3、重组普鲁兰酶pula的最适温度及热稳定性测定
酶的最适温度的测定:在ph9.0的缓冲液中,于10–60℃下进行酶促反应。酶的热稳定性测定:将同样酶量的酶液置于45℃、50℃和55℃中,处理0–60min后,在ph9.0及50℃下进行酶促反应,以未处理的酶液作为对照。以1%普鲁兰糖为底物,反应10min。酶反应最适温度测定结果(图4)表明,其最适温度为50℃。酶的热稳定性性试验表明(图5),重组酶在45℃时稳定性较好。55℃下保温60min,剩余酶活性为13%,。
4、重组普鲁兰酶pula的vmax和km的测定
以不同浓度的普鲁兰糖(1,0.8,0.4,0.2,0.15和0.1%)为底物,在最适条件下测定酶活性,计算相应的反应速度,利用米氏方程双倒数法求得km值及vmax。结果表明:该酶的vmax为38.02±2.55μmolmin–1mg–1,km为0.09±0.01mgml–1。
5、不同金属离子化学试剂对pula酶活的影响测定
在酶促反应体系中加入5mm的金属离子及化学试剂,研究其对酶活性的影响。在50℃及ph9.0条件下,以普鲁兰糖为底物测定酶活性。结果(表2)表明:ca2+,zn2+,mn2+,fe3+和co2+能够分别提高酶活力40%以上;cu2+和hg2+几乎完全抑制酶活性;ni2+,edta和sds对酶活有微弱的抑制作用。其它试剂对酶活力有微弱的促进作用。
表二金属离子及化学试剂对重组普鲁兰糖pula活性的影响
6、不同浓度ca2+对重组普鲁兰酶pula的活性的影响
普鲁兰酶pula中含有ca2+结合位点,因此研究了不同浓度ca2+对该酶活性的影响:在酶促反应体系中加入不同浓度的ca2+,研究其对酶活性的影响。在50℃及ph9.0条件下,以普鲁兰糖为底物测定酶活性。结果表明,浓度为30mm以下的ca2+对该酶活性都具有促进作用,其中浓度为5nm时活性最大,为不加钙离子的1.42倍(图6)。
7、不同浓度nacl对重组普鲁兰酶pula的活性和稳定的测定
nacl对重组普鲁兰酶pula的活性影响:在酶促反应体系中加入不同浓度的nacl,研究其对酶活性的影响。在50℃及ph9.0条件下,以普鲁兰糖为底物测定酶活性。nacl对重组普鲁兰酶pula的稳定性的影响:将酶液分别置于含有3m和4m的ph7.0的缓冲液中,在37℃下处理1h,然后在ph9.0及50℃下进行酶促反应,以未处理的酶液作为对照。结果表明:在0.25–4.0mnacl存在的情况下,该酶能够剩余45%以上的酶活(图7)。在3m和4mnacl的浓度下处理60min,剩余95%以上的酶活(图8)。
8、重组普鲁兰酶pula对普鲁兰糖的降解产物分析
样品处理过程如下:使用过量纯酶液与ph9.0buffer配置的1%普鲁兰糖底物混匀,放置在重组酶最稳定的温度中水浴反应,分别于10min,30min,1h,2h,4h和8h下取样,沸水浴5min灭活酶液,冷却至室温后13000r/min离心5min,取相同体积的样品点样在硅胶板上。薄层层析实验表明,该酶对普鲁兰糖的水解产物只有三塘,表明该酶为i型普鲁兰酶(图9)。
应当理解的是,对本领域普通技术人员来说,可以根据上述说明加以改进或变换,而所有这些改进和变换都应属于本发明所附权利要求的保护范围。
sequencelisting
<110>福州大学
<120>一种碱性耐盐普鲁兰酶pula及其基因和应用
<130>10
<160>10
<170>patentinversion3.3
<210>1
<211>1184
<212>prt
<213>氨基酸序列
<400>1
metalaaspglyglythralaleuleuthrvalaspasnglyserleu
151015
ilealaglugluilegluthrproaspaspseraspgluglnleuglu
202530
gluthrgluvalglugluglyphepheargilehisphealaserleu
354045
proseraspasparggluthrleuglyleutrpiletrpasnaspval
505560
lysgluprosergluasnargglyalatrpproasnglyalathrser
65707580
phethrglualavalglnthrasptyrglytrptyrmetaspileglu
859095
leuglugluasnproglnserileglypheleuileasnservalser
100105110
glyasnasnleuserglyaspilevalleuargleuleuthrserglu
115120125
metasnglnvaltrpleuasphisglutyralametthrprotyrglu
130135140
proleuleuglulysaspmetileargileasntyrlysargaspasn
145150155160
asnasptyraspasptrpglyleutrpthrtrpaspaspvalalaglu
165170175
prothrgluasntrpproalaglyalaglnaspseraspglyvalgly
180185190
proasnglythrtyrpheasnleuthrleualagluaspseraspgln
195200205
ileglypheleupheleuasnlysalaaspglyserglnthrargasp
210215220
tyrthrpheserasnleuseralahisserglnleuphemetargglu
225230235240
glyaspaspthriletyrthrasnprotyrtyrvalserglualagly
245250255
metileargalagluleuilesergluthrgluilegluvalphephe
260265270
hisserthrgluglyleugluglualaaspleuleugluleuilegln
275280285
leuthraspalagluglyargaspvalleupheaspalaalavalasp
290295300
hisaspargargvalvalserleuthrglyasppheservalgluhis
305310315320
alaprotyrthrvalthrpheaspgluthrglualaglualaargmet
325330335
glytrpargleulysaspalaleutyralatyraspglygluleugly
340345350
leuthrpheasngluaspglythralaaspleulysvaltrpserpro
355360365
seralaaspalavalthrvalvalleutyrasplysaspaspglnthr
370375380
valvalvalargaspaspileglumetthralaglugluserglyval
385390395400
trpargvalvalleuaspgluaspthrthrglyleuaspaspvalthr
405410415
glytyrphetyrhisphealailegluargasnglygluthrvalleu
420425430
alaleuaspprotyralaargsermetalaalatrpasnserserasp
435440445
proaspasntyrileglylysalaalailevalasnprosergluile
450455460
glyarggluleuasptyralaglnilegluglytyrasplysargglu
465470475480
aspalaileiletyrgluilehisvalargaspphethrserasppro
485490495
serilegluaspgluleuthrserglnpheglythrpheseralaphe
500505510
ileglulysleuasptyrilegluserleuglyvalthrhisvalgln
515520525
leuleuprovalmetsertyrphephealaasngluphegluasnala
530535540
gluargmetleuasptyrglyserthrglnthrasntyrasntrpgly
545550555560
tyraspproglnsertyrpheserleuthrglymettyrsergluasn
565570575
prolysaspproalaargargileglugluphelysasnleuileasp
580585590
alailehisserhisglymetglyvalileleuaspvalvaltyrasn
595600605
histhralaarggluhisilephegluaspleugluproasntyrtyr
610615620
hisphemetaspalaaspglythrserargthrserpheglyglygly
625630635640
argleuglythrthrhisglumetalaargargileleuvalaspser
645650655
ilethrtyrtrpvalgluglutyrlysvalaspglypheargpheasp
660665670
metmetglyasphisaspalagluserileglnmetalapheaspglu
675680685
alaglnlysleuasnproasnileleumetileglygluglytrparg
690695700
thrphevalglyaspgluglytyrgluaspvalmetproalaaspgln
705710715720
asptrpmetglnhisthrglnalavalglyserpheseraspaspphe
725730735
argasngluleulysserglypheglysergluglygluproargphe
740745750
ilethrglyglythrargserileglnargilepheaspasnleuthr
755760765
alaasnprohisasnphemetalathraspproglyaspvalvalpro
770775780
tyrilealaalahisaspasnleuthrleuhisaspvalilealagln
785790795800
serileglnlysaspproglutyrhisglnglugluilehisglnarg
805810815
ileargleuglyasnleumetvalleuthrserglnglythrprophe
820825830
valhisalaglyglnglutyrglyargthrlysglnpheargasppro
835840845
asppheilegluprovalalaasnaspglnvalprotyrlysserthr
850855860
phemetthraspgluaspglyasnpropheleutyrprotyrpheile
865870875880
hisaspsertyraspserthraspalavalasnargpheglutrpasp
885890895
lysvalthraspalaglualatyrproileasnthrglnthrglnser
900905910
tyrthrserglyleuilealaleuargargserthraspalapheser
915920925
lysglythrmetglugluilealaaspmetvalserleuvalaspala
930935940
progluilegluaspgluaspleuvalilevaltyrargalagluasp
945950955960
serasnglyaspargtyrtyrvalphevalasnalaaspaspserglu
965970975
argthrleuthrleuaspseraspleuthrgluglyhisvalleuval
980985990
aspserglnglnalaglythrargalailealaargprogluglyile
99510001005
thrvalgluglnalaglyvalthrleualaproleuthralaser
101010151020
valvalleuleuthrasparggluilegluprovalglugluser
102510301035
aspgluaspglyaspgluglythraspproglyasnglyglugln
104010451050
proglyglygluserglyproglythraspglnglyseraspgly
105510601065
aspaspprovalserglyglyglugluthralaaspprogluarg
107010751080
aspalagluglyaspasptyrprogluaspaspthraspleuser
108510901095
gluaspproglyalaglyglnaspserglyaspserilealaasp
110011051110
glyaspglnglyhisseraspglyproleuaspglyproaspgly
111511201125
aspgluthrglylysalagluglyaspsersergluserglnthr
113011351140
glygluglnasnglygluargleuproserthralathrleuleu
114511501155
trpthrvalglyalavalglyleumetserleuleuthrglyval
116011651170
valvalargglnilelyslyslysasnlysthr
11751180
<210>2
<211>3555
<212>dna
<213>基因序列
<400>2
atggcagacggcggaacagcactcctgacagtggataacgggtcactgattgcggaagaa60
attgagacaccggatgattcagatgaacagctagaagaaaccgaggtggaagaaggattt120
tttcgcatccattttgcgtcgcttccgtcagatgatcgcgagactcttgggctctggatc180
tggaacgatgtaaaagagccttctgaaaacagaggggcatggccgaatggggcgacctca240
tttacagaggctgttcagaccgactacggctggtatatggatattgaattggaagagaat300
ccacagtcgattggcttccttatcaattccgtctcggggaacaatctgtcaggagatatc360
gtcctgcggcttctgacttctgagatgaatcaggtatggcttgatcatgagtatgccatg420
acaccttatgagcctttacttgaaaaggacatgatccggatcaactacaaacgagacaac480
aatgactacgatgactggggcctctggacctgggacgatgtggcagaaccaacagaaaac540
tggcctgcaggcgctcaggacagcgatggcgtcgggcctaacggaacctattttaatctt600
acgctggcagaagattccgatcagatcggttttctgttcctgaataaagctgatggttcc660
cagacccgggattacacattctcgaacttgtccgctcacagccagctatttatgcgtgaa720
ggagacgatactatttataccaatccgtactatgtcagtgaagccggcatgatccgagcc780
gagctgatttctgaaactgaaatcgaagtgtttttccattcgacagaaggactggaggag840
gctgatctgcttgaattaattcagctgaccgatgcagaaggacgagacgtgctatttgat900
gcggctgtcgatcatgaccgacgagtcgttagtctgaccggtgacttctcagtggagcat960
gctccttataccgtgacttttgacgaaacggaagcagaagcgcgaatgggctggcgcctg1020
aaggatgcgctctatgcctatgacggagaacttggtctgacttttaacgaagacgggacc1080
gctgatctgaaagtctggtctccaagtgccgacgcagtgacggtcgttttatatgataaa1140
gatgatcagactgtcgttgtcagagatgatatcgaaatgactgccgaggagtcaggcgtg1200
tggcgtgttgttcttgatgaagacacgaccggactggacgatgtgacggggtacttctac1260
cattttgcgattgaaagaaatggcgaaaccgtcctggcactggatccttatgcccgctca1320
atggccgcatggaacagcagtgatccggataattacatcggcaaagctgccatcgtgaat1380
ccgagtgagatcggtcgggaactggattatgctcagatcgaaggctatgacaagcgcgaa1440
gatgcgatcatttatgaaattcatgtcagagacttcacatccgatccgtcaattgaagat1500
gaactgaccagtcagttcgggacgttcagtgctttcattgaaaagctcgattatatcgaa1560
agtctgggtgtgacccatgttcagctgcttccggtcatgagctatttctttgcaaatgaa1620
ttcgaaaacgccgaacggatgttggactacggatcgactcagaccaactataactggggt1680
tacgatccgcagagctacttctctctgacgggaatgtattcagaaaatccaaaagacccg1740
gcgagacgcattgaagaattcaaaaatctgatcgatgcgattcattcccacggcatgggt1800
gtcattcttgatgtggtctataatcacacggcacgggagcacatttttgaagatctggaa1860
ccgaactattaccatttcatggatgcggacggcacgtcacgaacaagtttcggaggcggc1920
cgactaggcaccactcatgagatggcccgccgtattctggtcgattcgatcacatactgg1980
gtggaagaatataaagttgacggcttccgcttcgatatgatgggcgatcacgatgccgaa2040
agcattcagatggcatttgacgaagcacagaaactgaatccgaatatcctgatgatcggt2100
gagggctggcggacctttgtcggtgacgaaggctatgaagacgtcatgccggctgaccag2160
gactggatgcagcacacacaagccgttggatcgttctcggatgacttcagaaatgaactg2220
aaatccggattcggcagtgagggagaaccgcgctttattaccggaggcacacggtccatc2280
caacgaatttttgacaatctgacagccaatccacataactttatggcgacagatccgggc2340
gatgtggtcccttatatcgcggcgcacgacaacctgacgcttcatgatgtcatcgctcaa2400
agcattcagaaagatccggaataccaccaggaagagatccatcagcgtatccgtctgggc2460
aatctgatggtgctgacttctcagggcacgccattcgtccatgccggtcaggaatacgga2520
cggaccaagcaattccgtgatcctgactttatcgaacctgtagcaaacgatcaggttcct2580
tacaagtcgacattcatgacagatgaagacgggaatccgttcctttacccgtatttcatt2640
catgactcatacgattcaacggatgcggtcaaccgttttgaatgggataaagtgacggat2700
gctgaagcttatccgattaatacgcagacccagtcttacacatctggtctgattgcattg2760
cggcgcagtacggatgccttcagtaaaggaacgatggaagagatcgcggacatggtgtcg2820
ctagtggatgcgccggaaatcgaggacgaagacctggttatcgtctatcgtgcagaagat2880
tccaatggcgatcgttactacgtctttgtgaatgcagatgattccgaacgaacgctgaca2940
cttgattctgatttgactgaagggcacgtcctggtcgatagtcagcaggccggcacacga3000
gcgatcgccagaccagaaggcatcacggtcgaacaggctggtgtcacgctggctcctttg3060
acggcatccgtggttttacttacggatagagaaattgagccggttgaagagagtgacgaa3120
gatggcgatgaaggaactgacccaggcaacggggaacagcctggtggagaatctggacca3180
ggaacagatcagggatccgatggcgacgatcctgtctcaggtggcgaagaaacggctgat3240
ccagaaagagatgccgagggtgatgattatccggaagacgacactgatttgtctgaggat3300
cctggtgctggtcaggatagtggagattcgatagctgatggcgaccaaggtcattcagat3360
ggtccactagatgggccggatggtgatgaaacaggcaaagcggaaggcgattcttctgaa3420
tcccagactggtgagcagaacggagaaaggctcccctctacagcaaccctactctggact3480
gtaggagcagtcggactgatgagcctcttgacaggggtagtggtcagacagatcaaaaag3540
aaaaataagacataa3555
<210>3
<211>26
<212>dna
<213>人工序列
<400>3
gtcagttcgggacgttcagtgctttc26
<210>4
<211>28
<212>dna
<213>人工序列
<400>4
cgaaagtctgggtgtgacccatgttcag28
<210>5
<211>30
<212>dna
<213>人工序列
<400>5
gtcattcttgatgtggtctataatcacacg30
<210>6
<211>30
<212>dna
<213>人工序列
<400>6
ccagtatgtgatcgaatcgaccagaatacg30
<210>7
<211>31
<212>dna
<213>人工序列
<400>7
catccatgaaatggtaatagttcggttccag31
<210>8
<211>28
<212>dna
<213>人工序列
<400>8
ggtctgagtcgatccgtagtccaacatc28
<210>9
<211>29
<212>dna
<213>人工序列
<400>9
gaaccatggatggcagacggcggaacagc29
<210>10
<211>41
<212>dna
<213>人工序列
<400>10
ggagcggccgctgtcttatttttctttttgatctgtctgac41
- 还没有人留言评论。精彩留言会获得点赞!