一种锌指蛋白转录因子基因RkMSN4及其应用的制作方法
本发明属于生物技术领域和遗传工程领域,涉及一种锌指蛋白转录因子基因rkmsn4及其重组表达载体,具体涉及从红冬孢酵母(rhodosporidiumkratochvilovae)ym25235中克隆的锌指蛋白转录因子基因rkmsn4及将该基因连接到载体转入到酵母菌中,并对其调节酵母菌生产多不饱和脂肪酸的能力进行研究,为多不饱和脂肪酸的大规模生产提供参考。
背景技术:
多不饱和脂肪酸(polyunsaturatedfattyacids,pufas)是指含有两个或两个以上c=c且碳链长度为18~22个碳原子的脂肪酸。根据第一个c=c出现在从甲基原子数起第3个或第6个碳原子上,通常分为n-3和n-6系列。多不饱和脂肪酸具有多种重要的生理功能,包括可降低冠心病、神经退行性疾病的风险。此外,pufas还可以作为很多具有生物活性物质的前体物质,如前列腺素、白三烯和血栓素,这些生物活性物质可以有效缓解炎症、疼痛和发烧。
长期以来人们都在研究如何从动植物油脂中提取pufas,但因为动植物的生长随着季节、地理位置等的影响而不断发生变化,所以pufas含量和构成也随之而变,况且从动植物中提取pufas的成本高,周期长,不能适应市场的需要。另外动植物油脂资源含油量及不饱和脂肪酸类型、比例均受到一定的限制。因此,近年来人们一直在探索利用pufas的新来源---即利用微生物技术来生产pufas。微生物具有发酵周期短、油脂含量高、培养成本低、不受原料控制、对环境的适应性强、生物转化率高等优点。近年来对于脂质积累的研究主要集中于单细胞微生物,如酵母和微藻。
锌指蛋白msn4是一类转录因子,很多研究表明,不同环境胁迫条件下其能促进宿主细胞中胁迫适应相关基因的转录,从而促进宿主对高盐、高渗透压等环境胁迫适应性,但是目前没有锌指蛋白msn4低温条件下促进多不饱和脂肪酸合成的研究报道。
技术实现要素:
本发明的目的是提供一种锌指蛋白转录因子基因rkmsn4,其参与调节低温环境下红冬孢酵母ym25235抗低温环境关键酶rkd12脂肪酸脱氢酶的活性,使酵母大量产生多不饱和脂肪酸抵御低温环境。该基因是从红冬孢酵母(rhodosporidiumkratochvilovae)ym25235中分离得到,该基因核苷酸序列如seqidno:1所示,该基因序列长为2625bp(碱基),该基因编码的氨基酸序列如seqidno:2所示的多肽或其片段,共编码874个氨基酸。
本发明另一目的是提供一种含有锌指转录因子rkmsn4的重组表达载体,是将seqidno:1所示基因直接与原核表达载体prh2034连接所构建的重组载体prh2034rkmsn4。
本发明另一目的是将锌指蛋白转录因子基因rkmsn4应用在低温下生产多不饱和脂肪酸中。
本发明从红冬孢酵母(rhodosporidiumkratochvilovae)ym25235的总rna基因中分离得到调控红冬孢酵母抗低温环境锌指蛋白转录因子基因rkmsn4,该基因全长2625bp。研究表明锌指转录因子rkmsn4在低温下能提高红冬孢酵母ym25235中催化油酸转化为亚油酸和亚麻酸的δ12/15-脂肪酸脱氢酶基因rkd12基因的mrna转录水平的提高,从而引起细胞脂中pufas的合成量显著增加;本研究结果有助于阐明产油酵母-红冬孢酵母ym25235中pufas低温合成调节机制,为揭示微生物低温适应性的机制提供参考,将有助于通过基因工程手段对其进行改造来提高多不饱和脂肪酸-亚油酸(la)和α-亚麻酸(ala)的含量,对多不饱和脂肪酸的工业化生产提供有良好的应用前景和经济效益,为大规模商业化生产多不饱和脂肪酸奠定基础。
附图说明
图1为本发明的红冬孢酵母ym25235锌指蛋白转录因子rkmsn4基因pcr扩增图;
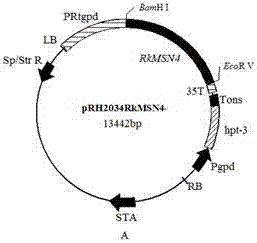
图2为利用本发明的红冬孢酵母ym25235锌指蛋白转录因子rkmsn4基因构建的红冬孢酵母表达载体prh2034rkmsn4;
图3为本发明所构建的重组表达质粒prh2034rkmsn4的酶切分析图;其中:1为dnamaker;2为空质粒prh2034bamhi、ecorv双酶切对照;3、4为重组质粒prh2034rkmsn4bamhi、ecorv双酶切;5为rkmsn4基因pcr产物;
图4为低温条件下rkmsn4基因转化的红冬孢酵母ym25235细胞脂肪酸气相色谱分析;a:30℃培养的ym25235/prh2304;b:30℃培养的ym25235/prh2034rkmsn4;
图5为低温条件下rkmsn4基因转化的红冬孢酵母ym25235细胞脂肪酸气相色谱分析;a:15℃培养的ym25235/prh2304;b:15℃培养的ym25235/prh2034rkmsn4。
具体实施方式
下面结合附图和实施例对本发明作进一步详细说明,但本发明保护范围不局限于所述内容,实施例中使用的试剂和方法,如无特殊说明,均采用常规试剂和使用常规方法。
实施例1:红冬孢酵母(rhodosporidiumkratochvilovae)ym25235锌指蛋白转录因子rkmsn4基因克隆
采用omega试剂盒e.z.n.afungalrnakit提取红冬孢酵母ym25235总rna,反转录试剂盒takarafirststrandcdnasynthesiskit合成cdna。根据红冬孢酵母ym25235的转录组序列设计特异性引物(引物1和引物2)进行pcr扩增;反应所用引物、组份和扩增条件如下:
引物1:rkmsn4-f:5’-aatggatccatggcgcctgctccccgt-3’(seqidno:3)
引物2:rkmsn4-r:5’-atcgatatctcacggcatcgcccacacct-3’(seqidno:4)
(ggatcc为bamhi酶切位点,gatatc为ecorv酶切位点)
pcr扩增体系如下(50μl):
pcr扩增条件为:95℃预变性5min,95℃变性30s、64℃退火30s、72℃延伸3min进行30个循环,最后72℃延伸10min。反应完后取产物1μl,然后在浓度为1%的琼脂糖凝胶中进行电泳分析。结果如图1所示,扩增获得大约2700bp大小的片段,然后用百泰克生物技术有限公司多动能dna纯化回收试剂盒回收目的片段,然后将pcr扩增得到的目的基因连接到pmd18-t上,连接产物转化至大肠杆菌dh5α感受态细胞中,用含有氨苄青霉素(amp+)的lb固体平板进行筛选,挑取平板上的转化子进行菌落pcr筛选阳性克隆,然后送去上海生工测序。测序结果表明,获得一段2625bp长的序列,编码874个氨基酸,命名为rkmsn4,序列组成如seqidno:1所示的核苷酸序列和氨基酸序列。
实施例2:重组表达质粒prh2034rkmsn4的构建
采用实施例1中测cdna为模板进行pcr扩增,反应所以引物组合、反应组分和扩增条件如下:
引物1:rkmsn4-f:5’-aatggatccatggcgcctgctccccgt-3’(seqidno:3)
引物2:rkmsn4-r:5’-atcgatatctcacggcatcgcccacacct-3’(seqidno:4)
(ggatcc为bamhi酶切位点,gatatc为ecorv酶切位点)
pcr扩增体系如下(50μl):
pcr扩增条件为:95℃预变性5min,95℃变性30s、64℃退火30s、72℃延伸3min进行30个循环,最后72℃延伸10min。将上述pcr获得的基因片段和表达载体prh2034用bamhi和ecorv核酸内切酶进行双酶切,用百泰克生物技术有限公司多动能dna纯化回收试剂盒回收酶切片段,并用t4连接酶在16℃连接过夜,将片段连接到表达载体prh2304上;连接产物转化大肠杆菌dh5α,用含有壮观霉素(spectinomycin)的lb固体平板进行筛选,挑取平板上的转化子进行菌落pcr,筛选阳性克隆,同时进一步对转化子进行双酶切验证分析,结果显示,如图3第3、4泳道所示,用ecorv和bamhi双酶切,重组质粒产生两条带,小分子条带与泳道5中该基因的pcr产物大小一致,大分子条带与泳道2中用相同酶切质粒prh2034后产生的条带大小一致,这表明所构建的重组质粒正确,进一步测序分析也证明这一点;将重组表达质粒命名为prh2034rkmsn4,该质粒图谱如图2所示。
实施例3:rkmsn4基因与低温环境中合成多不饱和脂肪酸实验
1、农杆菌介导转化红冬孢酵母ym25235
采用农杆菌介导转化法将重组质粒prh2034rkmsn4转化至红冬孢酵母ym25235菌株,以含潮霉素b(hygromycinb)终浓度为150µg/ml的ypd培养基筛选转化子;提取基因组dnapcr验证转化子。
2、rkmsn4基因与红冬孢酵母脂肪酸含量变化的分析
将转基因菌株ym25235/prh2034rkmsn4及对照菌株ym25235/prh2304分别先在30℃培养24h后,一份30℃继续培养24h,另一份立即转移至15℃冷处理培养24h;分别提取冷处理后4株菌的细胞中总脂肪酸,并进行甲酯化。将样品进行气相色谱分析,利用面积归一法计算菌株脂肪酸的含量;4株菌的脂肪酸气相色谱分析图谱如图4、5所示,在图中保留时间约为11min为饱和脂肪酸c18:1油酸oa,保留时间约为12.5min为不饱和脂肪酸c18:2亚油酸la,保留时间约为15min为不饱和脂肪酸c18:3亚麻酸ala。从图4、5,我们可以发现,当转基因菌株ym25235/prh2034rkmsn4和对照菌株ym25235/prh2304在30℃条件下,油酸(oa)、la及ala的峰面积大小基本一致;而当转基因菌株ym25235/prh2034rkmsn4和对照菌株ym25235/prh2304在15℃培养后,油酸(oa)的峰面积大小均呈下降趋势,la及ala的峰面积大小均呈上升趋势,且转基因菌株ym25235/prh2034rkmsn4的la及ala的峰面积均大于对照菌株ym25235/prh2304ala的峰面积(图4a:30℃培养的ym25235/prh2304,b:30℃培养的ym25235/prh2034rkmsn4;图5a:15℃培养的ym25235/prh2304,b:15℃培养的ym25235/prh2034rkmsn4)。
根据下表中脂肪酸含量数据,我们可以计算得出,在经15℃培养后,转基因菌株ym25235/prh2034rkmsn4中la和ala含量分别为31.56%和17.32%,相对于同一菌株30℃培养条件下la和ala的含量分别为16.81%和6.14%,变化不明显,但是相比较于15℃培养条件下对照菌株ym25235/prh2304中la和ala的含量(分别为24.18%和8.73%)分别提高了1.31倍和1.98倍,而且oa的含量减少了0.58倍。这些结果表明,在红冬孢酵母ym25235菌株中过表达rkmsn4基因会引起低温条件下la及ala两种多不饱和脂肪酸含量的增加。
表1:15℃条件下rkmsn4转基因红冬孢酵母ym25235细胞总脂肪酸含量
序列表
<110>昆明理工大学
<120>一种锌指蛋白转录因子基因rkmsn4及其应用
<160>4
<170>patentinversion3.3
<210>1
<211>2625
<212>dna
<213>rhodosporidiumkratochvilovae
<400>1
atggcgcctgctccccgtggttctgccagcgggcagaccttcccctgccccgactgcgac60
aaggtcttctcccgcaaggagtacatggcgcgacattaccggtcgaggcatagcaaggag120
aagcccttccaatgcgagtactgcgaacacgccttcagccgcagtgacctgctgaggcgt180
caccacaaaacgtgcagccaggctaaggcggcgaggggcgagggcgactcgccggatctt240
ccgcgcgagtcgcccaccgagcctgccgcgcccgtagcgtcgacctccgccatgcccgtc300
gacatactgtacccgccgtacccaccgcccgtcgcctcgtcctcgtccgccgttcctgcg360
ccgacctactcgccatatgacacaggcgcgcaggacctgcctgtccccccagccagcttc420
ctcgacctcgtctcttcgcaaccgtaccctgcgccctccccgtccctccccgccttctcg480
cccaacatggcgagcctgtatcccatcccattgaggaccggcagcacagcctcgcccgcc540
tcgagcggcatgtccggctcgccgttctcgcgcgccacgtacgcctcgacgaccggcaca600
tcgcccgacatggcgatgcagaaggagctgccgctcacgctccctgacgtgatggcttcc660
atctcggcgacgacgagcgcgccgcgccctcaaccgccgcaggagcagcaagcgatgccg720
acgctcgccgcgctcgacccgtacggcgccgcgccgcagccttcgtccgcattcaaggcc780
ggcgtcacgcagcacgatacgctcgcgcccggcctgtcgcgcacgggcagcttcaccaag840
gacgaagtgctcgcgtccgaggtcctgcaggacctgatgcgcacgccgttcggcgcgccg900
taccccgcgccgccagcgagcacgagcggcgtgccctggcacggcgcgaaggcggcgcag960
cgcgcggcggagcagcccgagtcggcgcaggcgaacgaagctgtcagcctggtcgactcg1020
tccggcggcgactgggctttcagcctcggcggcggcgcggttctgcccatcggcgggttc1080
ggcgggcccgtctcaaacaagctggaggagacgccagcggcgcagcagctcgccgagtac1140
ttcaataagggcggcgtcggcggcatcacggcgctcgacctcggcttcacggtcgagccg1200
agcctgtggcctgagtggatcacgatggagccgaagccggtgcacgacgacatgaagcgg1260
tggtggctgccagagcagaagttctgcctcggctatctctacccttggcatgtcccgccc1320
cttcccgttctctccggatacgcccgcaaggctacggagcagctcctcgcagctgtgccc1380
gtcctgcatggtccgtccgcggtcatgaccgagctaccgacgcacacggcgtttgcgctg1440
accgtcgccggcggggcgtacgagcccgagggccagagcttcagtaacgagatgctcgtc1500
gagaagagggtgttcctcgtgcgcggcttccaggagaaggacaagagctgggaggaccgc1560
ttcgcgtcactccagtcgctcctgctctaccagcttctcggcatcttccaccgcgacgag1620
cagcagcgcctgctctcccactccttccattcggcgctcgtctacatgtttcgcgcgctc1680
gacctcccgaaccgcgtaacgcagaccgcgctcgtcaagccgcgggcagacatgcatggt1740
ggggagctcgagaaggcgtggaaggagtggatcgaggtcgaaacccggcggcgagtcgcg1800
ttcatcgttttcctcatcgacctcgagcacgcggcggcgaccgagacgccgcagctcctc1860
gcgctcagcgacctcgacctcgaccttcccgcctccgagcgcgcgtggaaggcagagaac1920
gccgccgaatggctcgatcgcagtacctcgcccctctaccccgaaaccatctccttcctc1980
gccgccatccgcgccctcctctcgactaccccgcccgagcccttctccgccgcaggcgtt2040
ctcctcgccgaactcggccgcctctcgtccttcccgctcctcatcctctcgcggacgctc2100
tcgtacctcgagcgcaagacgcaggaggcgctcgcgcaaatcgacccgttcaagagcctc2160
ctcggcggcctcggcgtccttgagggccgcgagaccgagaatcgcgccgtcctcgagcgc2220
attcgtcgcggccgcgaagtcctgcgccgcctgcctggcgggattgcgcgcgggggtgga2280
gagggctggttcaacgagatcatcccctcggccaaggatttcgtcccagcgtcggactcg2340
cccgcagcgtcgagttcgagcgcgggcacctccccgcgggacgcgttctcctcctcctcc2400
gcaagcgggacggcatcgacatttccctcgccgcaagacgcgcccgcctcgctcgaggag2460
ctcttcgccgagttcgacgagcagccgtaccgcccgttccatggcgcgggcggcgcgacg2520
cccggcgagacgtacgagcaggcgcaggagcgcctgcgcaagcatgccgagcggcgcgtg2580
cgcgatgcgcagtccgcactgcccgaggtgtgggcgatgccgtga2625
<210>2
<211>874
<212>prt
<213>rhodosporidiumkratochvilovae
<400>2
metalaproalaproargglyseralaserglyglnthrphepro
110
cysproaspcysasplysvalpheserarglysglutyrmetala
2030
arghistyrargserarghisserlysglulyspropheglncys
40
glutyrcysgluhisalapheserargseraspleuleuargarg
5060
hishislysthrcysserglnalalysalaalaargglyglugly
70
aspserproaspleuproarggluserprothrgluproalaala
8090
provalalaserthrseralametprovalaspileleutyrpro
100
protyrproproprovalalaserserserseralavalproala
110120
prothrtyrserprotyraspthrglyalaglnaspleuproval
130
proproalaserpheleuaspleuvalserserglnprotyrpro
140150
alaproserproserleuproalapheserproasnmetalaser
160
leutyrproileproleuargthrglyserthralaserproala
170180
serserglymetserglyserpropheserargalathrtyrala
190
serthrthrglythrserproaspmetalametglnlysgluleu
200
proleuthrleuproaspvalmetalaserileseralathrthr
220
seralaproargproglnproproglngluglnglnalametpro
230
thrleualaalaleuaspprotyrglyalaalaproglnproser
250
seralaphelysalaglyvalthrglnhisaspthrleualapro
260
glyleuserargthrglyserphethrlysaspgluvalleuala
280
sergluvalleuglnaspleumetargthrpropheglyalapro
290
tyrproalaproproalaserthrserglyvalprotrphisgly
310
alalysalaalaglnargalaalagluglnprogluseralagln
320
alaasnglualavalserleuvalaspserserglyglyasptrp
340
alapheserleuglyglyglyalavalleuproileglyglyphe
350
glyglyprovalserasnlysleuglugluthrproalaalagln
370
glnleualaglutyrpheasnlysglyglyvalglyglyilethr
380
alaleuaspleuglyphethrvalgluproserleutrpproglu
400
trpilethrmetgluprolysprovalhisaspaspmetlysarg
410
trptrpleuprogluglnlysphecysleuglytyrleutyrpro
430
trphisvalproproleuprovalleuserglytyralaarglys
440
alathrgluglnleuleualaalavalprovalleuhisglypro
460
seralavalmetthrgluleuprothrhisthralaphealaleu
470
thrvalalaglyglyalatyrgluprogluglyglnserpheser
490
asnglumetleuvalglulysargvalpheleuvalargglyphe
500
glnglulysasplyssertrpgluaspargphealaserleugln
520
serleuleuleutyrglnleuleuglyilephehisargaspglu
530
glnglnargleuleuserhisserphehisseralaleuvaltyr
550
metpheargalaleuaspleuproasnargvalthrglnthrala
560
leuvallysproargalaaspmethisglyglygluleuglulys
580
alatrplysglutrpilegluvalgluthrargargargvalala
590
pheilevalpheleuileaspleugluhisalaalaalathrglu
610
thrproglnleuleualaleuseraspleuaspleuaspleupro
620
alasergluargalatrplysalagluasnalaalaglutrpleu
640
aspargserthrserproleutyrprogluthrileserpheleu
650
alaalaileargalaleuleuserthrthrproprogluprophe
670
seralaalaglyvalleuleualagluleuglyargleuserser
680
pheproleuleuileleuserargthrleusertyrleugluarg
700
lysthrglnglualaleualaglnileaspprophelysserleu
710
leuglyglyleuglyvalleugluglyarggluthrgluasnarg
730
alavalleugluargileargargglyarggluvalleuargarg
740
leuproglyglyilealaargglyglyglygluglytrppheasn
760
gluileileproseralalysaspphevalproalaseraspser
770
proalaalaserserserseralaglythrserproargaspala
790
pheserserserseralaserglythralaserthrpheproser
800
proglnaspalaproalaserleuglugluleuphealagluphe
820
aspgluglnprotyrargprophehisglyalaglyglyalathr
830
proglygluthrtyrgluglnalaglngluargleuarglyshis
850
alagluargargvalargaspalaglnseralaleuprogluval
860
trpalametpro***
874
<210>3
<211>27
<212>dna
<213>人工序列
<400>3
aatggatccatggcgcctgctccccgt27
<210>4
<211>29
<212>dna
<213>人工序列
<400>4
atcgatatctcacggcatcgcccacacct29
- 还没有人留言评论。精彩留言会获得点赞!
- 大豆威廉姆斯82中NAC膜结合转录因子基因GmNTL1及其应用的制作方法与工艺
- 一种锌指蛋白转录因子基因RkMSN4及其应用的制作方法与工艺
- 三七MYB转录因子基因PnMYB2及其应用的制作方法与工艺
- WRKY转录因子在制备抗逆转基因甜橙中的应用的制造方法与工艺
- 能够提供热耐受性的转录调节因子的表达的制造方法与工艺
- 具有用于舒适性、空间调节和个性化舒适性基因的辅助空气源的空气悬浮座椅的制造方法与工艺
- 一种大豆转录因子GmDISS1及其编码基因与应用的制造方法与工艺
- 一种与棉纤维发育相关的WRKY转录因子基因及其应用的制造方法与工艺
- 一个棉花产量性状关联的乙烯响应转录因子基因的制造方法与工艺
- 一种NF‑Y类核转录因子基因ZmNF‑YA13、其编码的蛋白及其应用的制造方法与工艺