一个棉花产量性状关联的乙烯响应转录因子基因的制作方法
本发明属于生物技术应用领域,涉及一个棉花产量性状关联的乙烯响应转录因子基因。
技术背景
棉花作为天然纤维的主要来源,是一种重要的经济作物。棉花生产不仅对我国农业乃至国民经济的发展有着重要的影响,而且在世界棉花贸易市场上也起着举足轻重的作用。随着纺织工业的发展和社会需求的提高,选育种植产量高、纤维品质优良的棉花品种尤为重要,因此深入挖掘和利用棉花产量的遗传变异就显得格外重要。由于棉花基因组的复杂性,先前对棉花产量性状的研究还处于qtl(quantitativetraitlocus)定位水平。
全基因组关联分析(genome-wideassociationstudy;gwas)是以基因组中数以百万计的单核苷酸多态性(singlenucleotideploymorphism,snp)为分子遗传标记,进行全基因组水平上的相关性分析,通过比较发现影响复杂性状的基因变异的一种新策略。利用全基因组关联分析挖掘和克隆农艺性状相关基因,无需事先假定候选基因,其检测能力强,精度高,已成为分子育种研究的热点。aranzana等(2005)采用gwas分析方法进行拟南芥开花和抗逆性方面的研究,成功的获得了拟南芥开花期(fri)和抗病性相关基因(rpm和rps)。belo等(2008)对553份优良自交系的8,950的snp进行了gwas分析,鉴定出于油酸含量相关的位点,这也是玉米首次真正意义上的全基因组关联分析。huang等(2011)利用第二代测序技术对517份水稻地方种进行了重测序并获得上百万的snp,然后对水稻的14个农艺性状进行gwas分析,并成功鉴定出80个性状关联的位点。此外,他们还对多达950份水稻群体进行重测序,对开花期和10个产量相关性状进行gwas分析,鉴定出了很多已知功能基因(huangetal.2012)。lin等(2014)对世界各地的360份番茄种质进行全基因组重测序,通过群体分化分析,首次发现决定粉果果皮颜色的关键变异位点,即slmyb12基因启动子区域的603bp缺失,进而抑制该基因的表达,从而使得成熟的粉果番茄果皮中不能积累类黄酮,导致鲜食番茄和加工番茄的差异。zhou等(2015)对302份大豆野生、地方品种以及改良品种进行重测序,结合gwas分析技术,发现96个gwas关联位点与之前报道的qtl有关联,并鉴别出含油量、株高和茸毛生成相关的新的关联位点。wang等(2016)利用全球不同地区的玉米自交系的自然变异群体进行gwas分析,找到83个遗传变异位点与玉米苗期抗旱性显著相关。并进一步证明抗旱性关联基因zmvpp1可以提高干旱敏感材料的抗旱性。以上充分说明,全基因组关联分析具有很高的定位精度,甚至可以达到单基因的水平,利用获得的与目标性状相关的功能标记,进行目标性状的筛选,加快育种进程和效率。
植物转录因子的数量和种类繁多,它们参与各种信号转导途径及生长发育的过程,是真核生物中最大的一个功能类别,大约占了全基因组的8%(weirauchandhughes,2011)。常见的植物转录因子有:myb、ap2/erebp、nac、bzip、homeobox、zincfinger、mads、wrky、yabby、arf、dof等。其中,乙烯响应转录因子家族ap2/erebp(apetala2/ethyleneresponsiveelementbandingprotein)是最大的三个基因家族之一(riechmann,2000),其家族成员广泛且保守地存在于植物中。其ap2保守结构域是包含约60个氨基酸残基的dna序列。β-转角中有两个保守的氨基酸残基,14位的丙氨酸(ala)和19位的天冬氨酸(asp)是识别ap2/erebp的重要特征(sakumaetal.,2002)。sakuma等(2002)根据结构域的数目和相似性把ap2/erebp转录因子分成了5个亚家族ap2(apetala2)、rav、dreb(dehydration-responsiveelementbindingprotein)、erf(ethyleneresponsivefactor)和其它。ap2/erebp转录因子一种植物调节蛋白,其生物学功能涉及植物初级代谢和次级代谢过程、植物生产发育调节(种子、花、果实等)和多种生物和非生物胁迫响应过程(francescoetal,2013)。
技术方案
本发明的目的是提供一个乙烯响应转录因子基因。全基因组关联分析结果表明该基因与棉花铃数、衣分和籽指这三个重要产量性状密切关联。
本发明的另一目的是提供该基因的应用。
本发明的目的可通过如下技术方案实现:
乙烯响应转录因子基因ghail6,在所述的乙烯响应转录因子基因ghail6在四倍体陆地棉tm-1中的cdna序列为:seqidno.1,基因组序列为:seqidno.2;所述的乙烯响应转录因子基因ghail6含有两个非同义突变的snp位点,分别在基因组序列的672bp和703bp位置上,这两个snp基因型紧密连锁;第一个snp位点碱基从a变为g,对应的氨基酸从asn变为asp;第二个snp位点碱基从g变为a,对应的氨基酸从gly变为asp;突变后的基因型衣分和产量显著高于野生型。
本发明所述的乙烯响应转录因子基因ghail6在鉴定高产量陆地棉品种中的应用。
本发明所述的乙烯响应转录因子基因ghail6在改良棉花产量性状中的应用。
本发明所述的乙烯响应转录因子基因ghail6在通过基因工程手段培育棉花高产新品种中的应用。
一种用于检测所述的snp位点的引物对,上游引物为:seqidno.3,下游引物为:seqidno.4。
所述的引物对在筛选高产棉花品种中的应用。
一种筛选高产棉花品种的方法,检测所述的两个snp位点,选择基因组序列的672bp和703bp位置上的碱基分别为g和a的棉花即为高产棉花品种。
有益效果
本发明的优点表现在:
本发明通过棉花品种群体重测序和全基因组关联分析挖掘了一个与棉花产量性状铃数、衣分和籽指同时关联的基因ghail6,属于ap2类乙烯响应转录因子基因(ap2/erebp)。本发明的乙烯响应因子家族基因ghail6在全基因组关联分析中与棉花产量性状密切相关。本发明提供的ghail6cdna和基因组序列由pcr技术获得,该技术具有起始模板量小,试验步骤简单易行而且灵敏度高的优点。
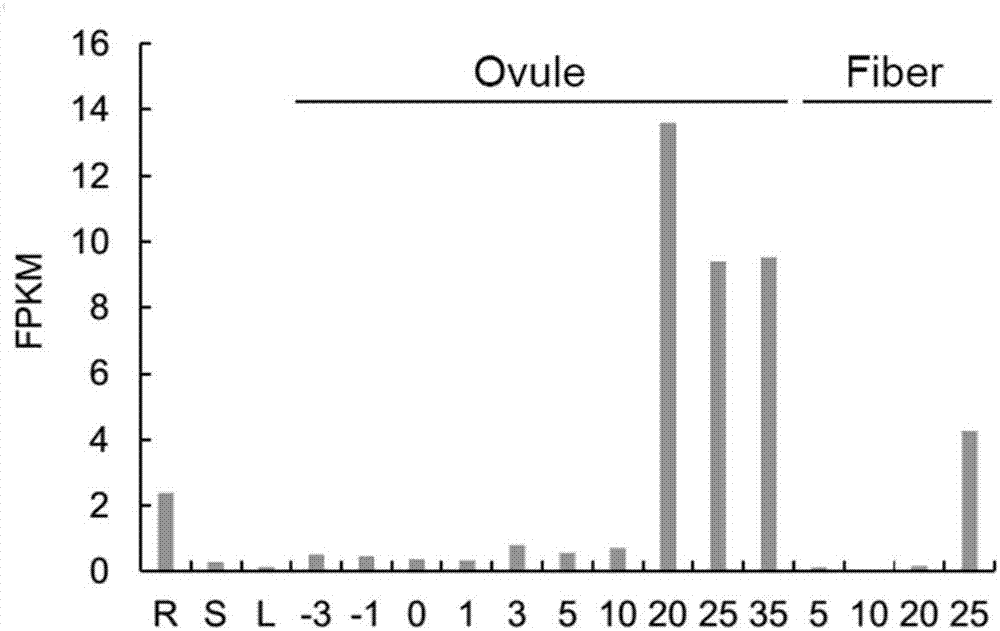
ghail6在棉花不同组织和发育时期的表达水平分析由转录组测序所得。该基因在棉花开花后20、25和35天的胚珠种子种优势表达,表明该基因与产量性状构成因子相关。
ghail6在相对高产和低产品种群体中的snp基因型通过pcr技术进行了验证,较易操作、灵敏度高和准确性好。
根据ghail6的不同snp基因型可以将品种群体分为两大类,统计学分析方法发现,这两类群体之间的铃数、衣分和籽指性状存在显著性差异(表1),进一步证明该基因与棉花产量性状之间的相关性。
附图说明
图1棉花不同产量性状gwas关联分析结果。
lp、bn和si分别代表产量性状衣分、铃数和籽指。红色箭头表示性状关联基因ghail6上的snp位点。横坐标表示染色体上的位置(mb),纵坐标表示snp位点关联的显著性,用-log10(pvalue)表示。
图2ghail6在棉花不同组织和发育时期的表达水平。
横坐标代表不同组织,包括根(r)、茎(s)、叶(l)、胚珠(ovule)和纤维(fiber)。胚珠组织包括了开花前3和1天、开花当天、开花后1到35天。纤维组织包括开花后5到25天。
图3ghail6的序列信息和不同单倍型的鉴定。
在品种群体中检测到ghail6序列存在两个非同义突变的snp位点,分别在基因组序列的672bp和703bp位置上,并且这两个snp基因型紧密连锁。第一个snp位点碱基从a变为g,对应的氨基酸从asn变为asp。第二个snp位点碱基从g变为a,对应的氨基酸从gly变为asp。根据两个snp位点碱基信息,将品种群体分为不同单倍型,标记为ghail6llb和ghail6hlb。llb表示低衣分和低铃数。hlb代表高衣分和高产量。
图4ghail6的不同单倍型之间产量性状的比较分析。
箱图代表品种群体的产量性状的分布情况。含有ghail6llb和ghail6hlb两种单倍型的品种分别是69和186个。蓝色代表单倍型ghail6llb的产量性状分布,橙色代表ghail6hlb的产量性状分布。方框内的横线代表性状分布的中值。**表示在0.05和0.01水平有差异。
具体实施方式
实施例1棉花产量性状关联的乙烯响应转录因子基因ghail6的挖掘:
针对258份现代品种或者品系,从2007到2009年,我们分别在河南安阳、江苏南京和新疆库车进行了产量性状(铃数、衣分和籽指)详细调查。同时,对这258份棉花品种进行了全基因组重测序,获得2.54tb测序数据,平均测序深度2.5×。我们将这些序列比对到棉花陆地棉基因组序列,利用samtools软件进行全基因组snp的鉴定,共挖掘了1,871,401个高质量的snp(最小基因频率>0.05)用于后续分析。利用emmax软件,我们进一步进行全基因组关联分析,再根据p<1×10-6筛选snp关联信号位点,并最终获得71个棉花产量性状关联位点。在这些关联位点中,我们发现了a02染色体上的一个snp信号关联位点(a02:79153947)可以同时关联铃数、衣分和籽指三个性状(图1)。这个snp位点正好处于基因外显子区,并造成了氨基酸序列的突变。该基因为乙烯响应转录因子基因aintegumenta-like6,ghail6(gh_a02g1392),属于ap2类基因。
实施例2乙烯响应转录因子基因ghail6的获得:
从陆地棉基因组序列中获取了ghail6(gh_a02g1392)的cdna序列和基因组序列,见seqidno.1和seqidno.2。根据cdna两端设计基因全长引物,进行pcr扩增,引物序列为f1:seqidno.3和r1:seqidno.4。pcr反应程序如下:94℃预变性5min;94℃变性30sec,60℃退火1min,72℃延伸1min,30个循环;最后72℃延伸7min。pcr扩增产物进行测序,与cdna进一步比对确定序列的准确性。
实施例3ghail6在棉花不同组织和发育时期的表达水平分析:
本实验采取了棉花不同组织和不同发育时期的rna样本进行转录组测序。样本材料包括了根、茎、叶、胚珠和纤维。胚珠组织包括了开花前3和1天、开花当天、开花后1到35天。纤维组织包括开花后5到25天。转录组测序采用illuminahiseq2500平台,每个样本平均测序深度达到6gb。基因表达水平的计算是利用tophat软件(verson2.0.8)将测序得到的reads与陆地棉基因组进行比对,计算出的表达水平以每百万测序碱基中每千个转录子测序碱基中所包含的测序片断数(fpkm)表示。实验结果中基因ghail6在棉花开花后20、25和35天的胚珠种子种优势表达,表明该基因与产量性状构成因子相关。
实施例4乙烯响应转录因子基因ghail6在鉴定高产量棉花品种和改良产量性状中的应用:
基于snp位点(a02:79153947)在染色体a02上的位置,在其两端设计基因组扩增引物,引物序列为f2:seqidno.5和r2:seqidno.6。利用这对引物,在258个品种的dna中进行pcr扩增并测序。pcr反应程序如下:94℃预变性5min;94℃变性30sec,58℃退火1min,72℃延伸45sec,30个循环;最后72℃延伸7min。根据测序结果分析该snp位点上每个品种群体的基因型。我们证实了ghail6序列中两个非同义突变的snp位点,分别在基因组序列的672bp和703bp位置上,并且这两个snp基因型紧密连锁(图3)。第一个snp位点碱基从a变为g,对应的氨基酸从asn变为asp。第二个snp位点碱基从g变为a,对应的氨基酸从gly变为asp。根据两个snp位点碱基信息,将低衣分和低铃数品种材料标记为ghail6llb,将高衣分和高产量品种材料标记为ghail6hlb(图3)。
根据ghail6基因组序列672bp和703bp位置上snp基因型,我们鉴定出单倍型ghail6llb材料69个,单倍型ghail6hlb材料186个(图3和表1)。利用t-test检测方法,我们计算了两组单倍型之间产量性状的相关性(图4)。结果显出,相比ghail6llb,单倍型ghail6hlb的铃数增产了11.33%,与铃数性状呈极显著正相关(p=0.0003);单倍型ghail6hlb的衣分增产了4.38%,与衣分性状呈极显著正相关(p=0.0004);单倍型ghail6hlb的籽指减产了6.01%,与籽指性状呈极显著负相关(p=0.003)。这与棉花产量性状衣分和籽指之间呈负相关相符合。
通过以上结果可以看出,基因ghail6在改良棉花产量性状和培育棉花高产新品种中具有重要的研究价值。一方面可以根据基因ghail6的两种单倍型设计分子标记,可以有效的鉴定棉花产量性状,在高产棉花品种选育研究中有很好的应用价值。另一方面,可以通过基因工程的手段将含有高产单倍型ghail6hlb的基因转入棉花品种中提高棉花产量,也可以将低产单倍型ghail6llb中的snp位点进行定点突变,改造成高产单倍型从而培育高产棉花新品种。
表1高产和低产单倍型在群体品种材料中的鉴定
<110>南京农业大学
<120>一个棉花产量性状关联的乙烯响应转录因子基因
<160>6
<210>1
<211>1509
<212>dna
<213>四倍体陆地棉tm-1
<220>
<223>乙烯响应转录因子基因ghail6cdna序列
<400>1
atggcaccagcaacagtgagtaactggctttcattttcactatccccgatggagatgttg60
aggtcttcatctgagcctcagtttgtgtcatatgaaggatcctcagctgctactgcttct120
gctgcttctcctcattacttgatcgataacttctatgccaatgactggacgaatccaaaa180
catcaaactcaacaaccggccatggcggccgatgagtcacccattctctcaagctttcac240
catcatcaagttccgaaactcgaagatttcctcggagattcctcgtcaatcgttagatat300
tcagataatagccaaaccgaaacccaggactcgtccttaactcatttaactcaaatctac360
gatcaccacaacgtcggtgctgctgcttacttcaacgaccaccaagatctcaaagccatt420
actgggtttcaagcgttttcgactaactcggggtctgaagttgatgactcagcttcaatg480
ggacgaactcagttagctgctgttgagtttcctggccactcaaattgtcccaccgccggt540
tccttatccctaggtgttaaccaagcctccgaaatcaacaccaccaccaccaataaagcc600
gtcgtttcagttgactccgattgttcaaagaaaatcgttgatacttttggtcagcgaact660
tcaatttacaggggtgtcaccagacaccgatggacgggtagatacgaagctcatttatgg720
gataacagctgtaggagagaaggtcaagctcggaaagggcgtcaaggtggatatgataaa780
gaagaaaaggcagctcgggcttacgatttagctgcgctgaaatactggggtcctactgca840
accaccaactttccgattacaaactattcgaaagagttggaggagatgaaacatgtgaca900
aagcaagaatttattgcttctttaaggaggaagagcagcggattttcgaggggagcatca960
atttacagaggtgtcacgaggcatcatcaacaaggtcgatggcaagcaaggattggtcgt1020
gttgctggaaacaaggacctttacctgggcacttttgctaccgaagaggaggcagcagag1080
gcctacgatatcgcggccatcaagtttaggggcataaatgcggtgaccaacttcgagatg1140
agccgctacgacgtcgaagcaattgccaagagttctctccccatcggtggagcagctaag1200
cggctaaagatctcactcgagtcggagcaaaaaccagtagtagtaaaccacgaacaacaa1260
ccccagtgcagctctaacagcaacataagttttgcccccatacagcagtcgatatctact1320
atcccttgcggaatcccgtttgatgcagcggcattttatcagcagaatctctaccaccac1380
cttcaggcttccaatatcagcatctcggatctccccggttcttcctcaacgatgaccaca1440
actccaacgaccttaatgtcgcaaccaacagcggatcagtttttcttatggcctcaccag1500
tcttactga1509
<210>2
<211>2927
<212>dna
<213>四倍体陆地棉tm-1
<220>
<223>乙烯响应转录因子基因ghail6基因组序列
<400>2
atggcaccagcaacagtgagtaactggctttcattttcactatccccgatggagatgttg60
aggtcttcatctgagcctcagtttgtgtcatatgaaggatcctcagctgctactgcttct120
gctgcttctcctcattacttgatcgataacttctatgccaatggtaatattttctttctt180
tcttttcttttcatggtttctgtttggttggcgagaaaatatagggtgttgtatactgtt240
acatataaaataaacccagaaagacatactgagttgactcgttttatgtgcatttttggt300
tttttttaatagactggacgaatccaaaacatcaaactcaacaaccggccatggcggccg360
atgagtcacccattctctcaagctttcaccatcatcaagttccgaaactcgaagatttcc420
tcggagattcctcgtcaatcgttagatattcagataatagccaaaccgaaacccaggact480
cgtccttaactcatttaactcaaatctacgatcaccacaacgtcggtgctgctgcttact540
tcaacgaccaccaagatctcaaagccattactgggtttcaagcgttttcgactaactcgg600
ggtctgaagttgatgactcagcttcaatgggacgaactcagttagctgctgttgagtttc660
ctggccactcaaattgtcccaccgccggttccttatccctaggtgttaaccaagcctccg720
aaatcaacaccaccaccaccaataaagccgtcgtttcagttgactccgattgttcaaaga780
aaatcgttgatacttttggtcagcgaacttcaatttacaggggtgtcaccaggtatacaa840
catcattttactattttttcccgagaaatttttaatttttttttaatggaggttacttac900
atggcaatggtaaatacagacaccgatggacgggtagatacgaagctcatttatgggata960
acagctgtaggagagaaggtcaagctcggaaagggcgtcaaggtacataaatacatacat1020
atatggttatatatacactgtttgacagtgaaaagaaacttgcatgcattcaaatgatag1080
ccatttgttgtcagcctttttttttgatgattgaatgactgatgttatatataatgtata1140
aatgttactttttctttacagccaaacaaaacacatttttattttttatttttttcttct1200
ttgattgatttgatcagatatgccgccattttagcaccctgtgtttgaaacgtcccacac1260
ccacacacccactggttcagtccccagggtctgtgagagagaaggtctgggtttgaccaa1320
accacccattgggtttattttatatgaaagataaaaaaaaaaaagtatgtgtgaaacccc1380
cacttttatagcctaaacttattgacagtgcctcttggctaaaaaagctgataatgtggg1440
ctatgttctggttcgcactcaagctttaataatctagccatagttgtaattttattttaa1500
tatttaacctaatgtaaattttgaagtttaattaaagggtaaaataatagtgaagtgcaa1560
ggattgaacagtataaaaaatgattgttatattttggctttgcattgctttgtatgaatg1620
attggaagcagaaaattttgactaaatttattgcctgatattgacaattttttgctactt1680
ttttgcatgaaaatgcgggatcgtggccgatgtttgttttcaacagtgtacttgggtaag1740
aaaactcaatacagtttcatgttttgttgtatttagttttaagtttgtaggctgataatg1800
tgagattttgtaattttcctcaggtggatatgataaagaagaaaaggcagctcgggctta1860
cgatttagctgcgctgaaatactggggtcctactgcaaccaccaactttccggtatatat1920
gatttcaaggtcctttttgttgctgaatgatattttgtgttaactcgtttaattgttcga1980
tcttatgcttggtttagattacaaactattcgaaagagttggaggagatgaaacatgtga2040
caaagcaagaatttattgcttctttaaggaggtaaaagcatagtattcttaatcagtttg2100
tgattccttattttcttggttaattgtggtgatttaatgtatcctttgtctcgattgaaa2160
tttaataggaagagcagcggattttcgaggggagcatcaatttacagaggtgtcacgagg2220
ttagattttgaattatgcgctccggattgttttcgattattacaaaccaagactaatgct2280
tgaattatcaacttaattttggcaggcatcatcaacaaggtcgatggcaagcaaggattg2340
gtcgtgttgctggaaacaaggacctttacctgggcacttttggtaaggctgcagatgcgt2400
gtgatgatgatccgatgccttattgtaagggtaactatagatacctaataataatcgtct2460
tatgtatgatgacagctaccgaagaggaggcagcagaggcctacgatatcgcggccatca2520
agtttaggggcataaatgcggtgaccaacttcgagatgagccgctacgacgtcgaagcaa2580
ttgccaagagttctctccccatcggtggagcagctaagcggctaaagatctcactcgagt2640
cggagcaaaaaccagtagtagtaaaccacgaacaacaaccccagtgcagctctaacagca2700
acataagttttgcccccatacagcagtcgatatctactatcccttgcggaatcccgtttg2760
atgcagcggcattttatcagcagaatctctaccaccaccttcaggcttccaatatcagca2820
tctcggatctccccggttcttcctcaacgatgaccacaactccaacgaccttaatgtcgc2880
aaccaacagcggatcagtttttcttatggcctcaccagtcttactga2927
<210>3
<211>23
<212>dna
<213>人工序列
<220>
<223>引物f1
<400>3
acagttttggtttttagtgttgg23
<210>4
<211>23
<212>dna
<213>人工序列
<220>
<223>引物r1
<400>4
tataagcctgatttaagggtcat23
<210>5
<211>20
<212>dna
<213>人工序列
<220>
<223>引物f2
<400>5
cgtcggtgctgctgcttact20
<210>6
<211>20
<212>dna
<213>人工序列
<220>
<223>引物r2
<400>6
tgaccttctctcctacagct20
- 还没有人留言评论。精彩留言会获得点赞!
- 大豆威廉姆斯82中NAC膜结合转录因子基因GmNTL1及其应用的制作方法与工艺
- 一种锌指蛋白转录因子基因RkMSN4及其应用的制作方法与工艺
- 三七MYB转录因子基因PnMYB2及其应用的制作方法与工艺
- WRKY转录因子在制备抗逆转基因甜橙中的应用的制造方法与工艺
- 能够提供热耐受性的转录调节因子的表达的制造方法与工艺
- 具有用于舒适性、空间调节和个性化舒适性基因的辅助空气源的空气悬浮座椅的制造方法与工艺
- 一种大豆转录因子GmDISS1及其编码基因与应用的制造方法与工艺
- 一种与棉纤维发育相关的WRKY转录因子基因及其应用的制造方法与工艺
- 一个棉花产量性状关联的乙烯响应转录因子基因的制造方法与工艺
- 一种NF‑Y类核转录因子基因ZmNF‑YA13、其编码的蛋白及其应用的制造方法与工艺