D-木糖酸脱水酶及其应用的制作方法
本发明属于微生物基因工程领域,具体涉及一种d-木糖酸脱水酶及其应用。
背景技术:
d-木糖酸脱水酶,系统命名是d-木糖酸氢-裂解酶,能催化木糖酸脱水反应。目前木糖酸脱水酶有不同的菌株来源,比如嗜盐的古细菌富盐菌、新月丙杆菌和假单胞菌。不同来源的d-木糖酸脱水酶可以利用不同的底物,比如d-葡萄糖酸、d-木糖酸和d-木糖。虽然已知源自大肠杆菌的木糖酸脱水酶yjhg具有催化d-木糖酸脱水的酶学活性,但关于其性质的研究报道较少。
目前,已经可以确定d-木糖酸脱水酶是许多代谢途径中的重要酶。在一些高附加值的化学品如乙二醇、三元醇和四元醇等生物合成途径中,其中来源于大肠杆菌的木糖酸脱水酶yjhg被用来催化d-木糖酸脱水反应。例如在利用d-木糖通过生物途径生产d-1,2,4-丁三醇的过程中,该合成途径包括:(1)在d-木糖脱氢酶的催化下将d-木糖转化为d-木糖酸;(2)d-木糖酸在d-木糖酸脱水酶的催化下生成d-3-脱氧甘油戊酮糖酸;(3)d-3-脱氧甘油戊酮糖酸在2-酮酸脱羧酶的催化下生成d-3,4-二羟基丁醛;(4)在脱氢酶催化下d-3,4-二羟基丁醛生成d-1,2,4-丁三醇。该酶对微生物发酵生产乙二醇、三元醇和四元醇的产量和质量有重要影响,但是,木糖酸脱水酶存在着反应速率缓慢,导致催化反应时间较长。因此,继续筛选一种具有高催化效率的木糖酸脱水酶,以提高木糖酸脱水酶的反应速率。
技术实现要素:
本发明要解决的技术问题是通过定向进化的技术手段提供一种具有高催化效率的木糖酸脱水酶yjhg。
本发明还要解决的技术问题是提供上述木糖酸脱水酶yjhg在制备多元醇中的应用。
为解决上述技术问题,本发明采用如下技术方案:
d-木糖酸脱水酶,所述d-木糖酸脱水酶的氨基酸序列如seqidno.:1~4所示。
编码上述d-木糖酸脱水酶的基因序列在本发明的保护范围之内,将seqidno.:1~4所示的核苷酸序列翻译为基因序列即可得到本发明中所述的编码d-木糖酸脱水酶的基因序列。
一种重组质粒,该重组质粒中包含上述编码d-木糖酸脱水酶的基因序列。
一种重组细胞,该重组细胞中包含上述编码d-木糖酸脱水酶的基因序列。
上述d-木糖酸脱水酶在制备多元醇中的应用在本发明的保护范围之内。
作为优选,所述的多元醇为1,2,4-丁三醇。
一株产1,2,4-丁三醇的基因工程菌,在大肠杆菌中导入了本发明中的编码d-木糖酸脱水酶的基因序列。
产1,2,4-丁三醇的基因工程菌,该大肠杆菌中导入了d-木糖酸脱水酶基因、d-木糖脱氢酶基因、苯甲酰甲酸脱羧酶基因、醇脱氢酶基因,所述的d-木糖脱氢酶基因的核苷酸序列如seqidno.:5所示、苯甲酰甲酸脱羧酶基因的核苷酸序列如seqidno.:6所示、醇脱氢酶基因的核苷酸序列如seqidno.:7所示。
上述产1,2,4-丁三醇的基因工程菌的构建方法,包括如下步骤:
(1)将d-木糖酸脱水酶基因、d-木糖脱氢酶基因、苯甲酰甲酸脱羧酶基因、醇脱氢酶基因分别克隆至表达质粒中,所述的表达质粒包括bt-62和bt-286,得到重组质粒,
(2)再将重组载体转化宿主菌,所述的宿主菌为trans1t1,得到产1,2,4-丁三醇的基因工程菌。
上述产1,2,4-丁三醇的基因工程菌在制备1,2,4-丁三醇中的应用在本发明的保护范围之内。
本发明通过易错pcr对所述来自大肠杆菌的d-木糖酸脱水酶yjhg的基因序列进行易错pcr,通过随机突变改变木糖酸脱水酶(yjhg)基因的密码子,易错pcr随机突变改变yjhg基因的密码子碱基的位置,最终筛选获得具有高木糖酸脱水酶活性的突变株91、96、62、286号。分别提取质粒,并送上海生工生物工程技术服务有限公司测序确定突变的yjhg基因序列。
突变株91号(氨基酸序列如seqidno.1所示)第143位碱基由t突变成c,相应的氨基酸由组氨酸突变成苏氨酸,第236位碱基由a突变成g,相应的氨基酸由赖氨酸突变成精氨酸;第520位碱基由g突变成a,相应的氨基酸由丙氨酸突变成苏氨酸;第938位碱基由t突变成c,相应的氨基酸由谷氨酰胺突变成脯氨酸。
突变株96号(氨基酸序列如seqidno.2所示)第967位碱基由g突变成a,相应的氨基酸由丙氨酸突变成苏氨酸。
突变株62号(氨基酸序列如seqidno.3所示)第967位碱基由g突变成a,相应的氨基酸由丙氨酸突变成苏氨酸,第1855位碱基由g突变成a,相应的氨基酸由天冬氨酸突变成天冬酰胺,第1898位碱基由c突变成t,相应的氨基酸由苏氨酸突变成异亮氨酸。
突变株286号(氨基酸序列如seqidno.4所示)第163位碱基由t突变成c,相应的氨基酸由丝氨酸突变成脯氨酸,第203位氨基酸由g突变成a,相应的氨基酸由丝氨酸突变为天冬酰胺,第967位碱基由g突变成a,相应的氨基酸由丙氨酸突变成苏氨酸。
有益效果:本发明相比于现有技术,具有如下优势:
本发明应用易错pcr技术对yjhg基因进行随机突变改造,获得几组活力较高的突变株91号、96号、62号和286号,这几个菌株在12h内所消耗木糖酸分别为4.2g/l、4.8g/l、6.22g/l、5.43g/l,而出发菌株(亲株a)在同等条件下消耗的木糖酸为2.15g/l,分别提高了2.1倍、2.4倍、2.89倍、2.5倍。应用易错pcr技术对yjhg基因进行随机突变改造,获得了在以木糖酸为底物时转化效率明显提高的突变菌株,育种目标明确、效率高。
附图说明
图1第一轮易错pcr复筛后的突变菌株与重组菌株的酶活对比
图2第二轮易错pcr初筛后的突变菌株与重组菌株的酶活对比。
图3第二轮易错pcr复筛后的突变菌株与重组菌株的酶活对比。
图4第二轮易错pcr复筛后的突变菌株产丁三醇的对比。
图5第一轮易错pcr复筛后的突变菌株产丁三醇的对比。
具体实施方式
根据下述实施例,可以更好地理解本发明。然而,本领域的技术人员容易理解,实施例所描述的内容仅用于说明本发明,而不应当也不会限制权利要求书中所详细描述的本发明。
实施例中所用的d-木糖酸脱水酶是来自大肠杆菌的基因,将该基因连接到pcwj表达载体上,转化至大肠杆菌trans1t1感受态细胞中,得到的阳性克隆培养至od600为0.6-1.0,加iptg终浓度为0.5mmol·l-1,33℃培养12~18h获得。
实施例1:含yjhg基因菌株的构建
(1)利用yjhg基因5’端和3’端引物引入酶切位点(ncoi和hindⅲ),对yjhg基因和pcwj质粒进行双酶切,然后将yjhg基因连接到pcwj载体上;
(2)将上述连接液转入大肠杆菌trans1t1的感受态细胞(全式金生物技术有限公司)里,涂布在带有50mg/l氯霉抗性的lb平板,37℃过夜培养。
(3)挑取平板上生长的单菌落,转接到含有50mg/l氯霉素抗性的lb培养基里,然后提取质粒,再经限制性内切酶spei和kpni进行酶切验证,最后得到了重组质粒pcwj-yjhg。
实施例2:yjhg基因突变体库的构建
(1)将实施例1得到的大肠杆菌trans1t1/pcwj-yjhg接种于lb液体培养基中,37℃摇床里培养,10h后,6000g离心5min,收集菌体细胞,用质粒小提试剂盒(购自上海生工生物工程技术服务有限公司)抽提质粒pcwj-yjhg;lb液体培养基组成为:蛋白胨10g/l,酵母粉5g/l,氯化钠5g/l,氯霉素50mg/l。
(2)易错pcr反应的建立:
(1a)设计含有限制性酶切位点的正向引物(yjhg-ncoi-f,catgccatggaaatgtctgttcgcaatatttttgctgac,下划线为ncoi酶切位点)和反向引物(yjhg-hindⅲ-r,cccaagctttcagtttttattcataaaatcgcgcaaagc,下划线为hindⅲ酶切位点);
(1b)配置50ul的易错pcr反应混合液:
易错pcr反应体系为:10×taqbuffer5ul、mn2+(50mmol/l)1ul、mg2+(50mmol/l)7ul、dna模板10ul、10×dntp5ul、上游引物yjhg-ncoi-f2ul、下游引物yjhg-hindⅲ-r2ul、taq酶0.5ul、ddh2o18ul;
(1c)将易错pcr反应混合液置于pcr仪上进行易错pcr扩增yjhg基因,扩增程序如下:
94℃变性3min,20个循环,每个循环包括94℃变性60s,57℃退火60s,72℃延伸60s,循环结束后,72℃无需延伸,然后保存在4℃下,扩增结束后,用浓度为1.5%琼脂糖凝胶电泳确认pcr产物大小。
(3)yjhg基因突变体库的构建:
(2a)用琼脂糖回收试剂盒(天根生化科技有限公司)回收pcr扩增产物;
(2b)配置50ul的酶切反应混合液,用限制性内切酶ncoi和hindⅲ对步骤(2b)的胶回收后的pcr产物和pcwj载体分别进行双酶切,双酶切体系为:片段dna模板:42ul,内切酶ncoi:1.5ul,内切酶hindⅲ:1.5ul,10×qcutbuffer:5ul,pcwj载体体系为:pcwj载体:42ul,内切酶ncoi:1.5ul,内切酶hindⅲ:1.5ul,10×qcutbuffer:5ul。分别将其于37℃水浴锅中酶切45min;
(2c)将酶切产物在浓度为1.5%琼脂糖凝胶上电泳确认大小,来验证酶切效果,并将目的片段用纯化试剂盒回收,再将pcwj载体切胶,用dna凝胶回收试剂盒(天根生化科技有限公司)回收。
(2d)配置10ul的连接反应混合液,体系为:t4ligationbuffer:1ul,步骤(2c)获得的双酶切后的pcwj载体:2.5ul,步骤(c)获得的双酶切后的dna片段:3.5ul,t4dnaligase:0.5ul,置于25℃水浴锅中反应30min。按《分子克隆实验指南》将连接产物转化至25ul大肠杆菌trans1t1感受态中。
(2f)最后将其涂布在lb固体培养基平板(蛋白胨10g/l,酵母粉5g/l,氯化钠5g/l,氯霉素50mg/ml),在37℃下正向放置以吸收过多的液体,然后倒置培养过夜;
(2g)将转化平板上长出的所有转化子单菌落挑至含50mg/l氯霉素的lb固体培养基平板上划线,于37℃恒温培养箱倒置培养过夜。将长出的所有转化子用引物对其进行菌落pcr。
实施例3:高产yjhg的基因工程菌的获得
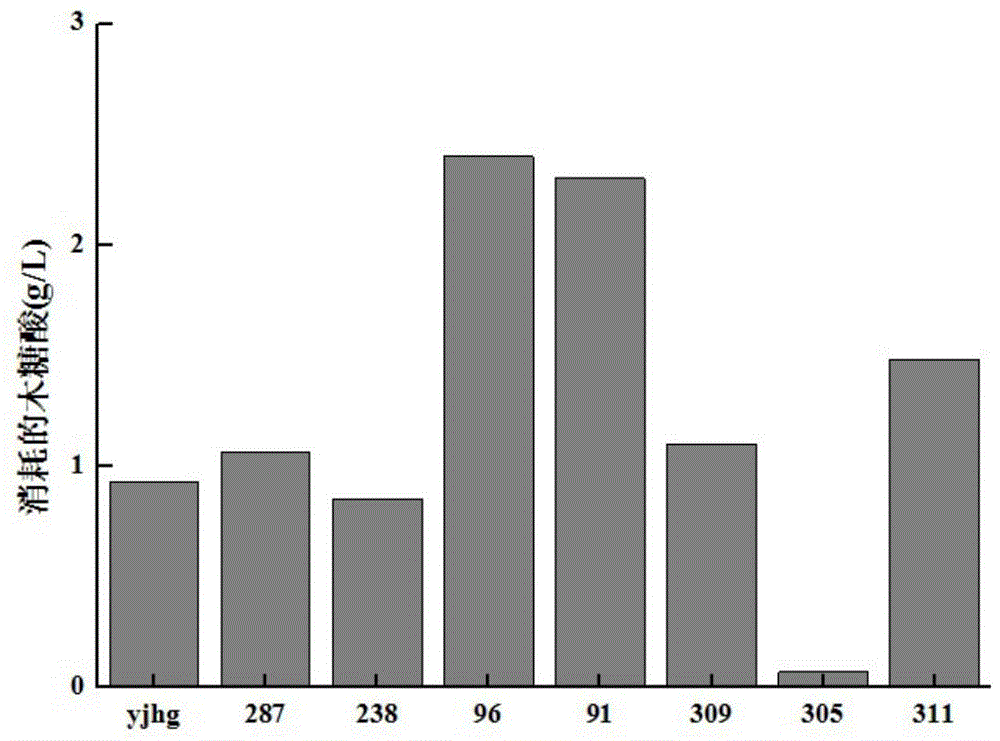
将实施例2中构建的突变体库中的转化子和pcwj-yjhg菌株分别接入96孔板中的1mllb液体培养基(蛋白胨10g/l,酵母粉5g/l,氯化钠5g/l,氯霉素50mg/ml)中,加入0.5mmol的iptg,于33℃、200rpm转速下摇床培养12h,培养结束后,8000r/min、4℃离心10min,收集细胞,加入500ul木糖酸溶液使其终浓度为100g/l,500ul水,混匀后,于33℃,200rpm摇床转化12h,加入30mm高碘酸试剂,与木糖酸反应可以生成甲醛,通过酶标仪在410nm处测定吸光度值进行初筛,再利用高效液相检测进行复筛,筛选出yjhg的酶活比亲株a明显提高的突变株91号、96号,如图1所示,其中,91号突变株在12h内消耗的木糖酸为2.3g/l,96号突变株在12h内消耗的木糖酸为2.5g/l,图中未突变菌株yjhg菌株在12h内消耗的木糖酸为0.93g/l。
将基因工程菌91号与96号先分别进行如下培养:
(1)从平板上分别挑取重组菌株pcwj-yjhg的单菌落接种到含有50mg/l氯霉素抗性的5mllb的摇管里,37℃培养6~8h后转接到含有50mg/l氯霉素抗性的100ml的lb培养基里,培养至od600=0.6~0.9,加入0.5mmol的iptg,诱导12h离心收菌,得到重组菌株pcwj-yjhg的细胞
(2)发酵培养:培养基配方:蛋白胨:10g/l,酵母粉:5g/l,氯化钠:5g/l,溶剂为水,将步骤(a)得到的细胞进行发酵培养,培养温度:33℃,培养时间:12h。
将预培养后的基因工程菌91号与96号分别加入木糖酸的水溶液,控制木糖酸溶液在初始反应体系中的浓度为20g/l,菌株的od600=60,于37℃反应12h,通过高效液相色谱检测木糖酸消耗了多少。如图2所示,91号和96号突变株分别在12h内消耗了木糖酸4.2g/l和4.8g/l左右,比亲株a在同样条件下分别提高了2.1与2.4倍。
将91号与96号突变菌株的木糖酸脱水酶基因(yjhg)利用实施例1中的酶切位点将突变的yjhg片段切下来,将突变的yjhg基因与d-木糖脱氢酶基因、苯甲酰甲酸脱羧酶基因、醇脱氢酶基因分别导入质粒中,得到重组质粒bt-91、bt-96,然后将重组质粒分别转化宿主菌trans1t1,得到重组菌bt-t1-91和bt-t1-96。控制反应体系为20ml,底物为20g/l,od=60,ph=7.0。如图5所示,突变菌株bt-t1-91和bt-t1-96在反应48h内消耗的木糖酸分别是11.13g/l和13.3g/l,与未突变菌株(pgp)相比,提高了约2倍和2.4倍,其中91号和96号突变菌株的1,2,4-丁三醇的产量分别达10.45g/l和12.55g/l,而未突变菌株的1,2,4-丁三醇的产量为5.75g/l,因此,与为突变菌株相比,提高了约1.86倍和2.1倍。
实施例4:对高产菌株96号再进行易错pcr
按照实施例1中的yjhg的突变库的构建再对突变株96号建立突变库,并构建到pcwj载体上,按照实施例3的方法筛选出酶活较高的突变菌株。
实施例5:高产突变株yjhg基因序列的分析
将实施例4复筛得到的突变株62号与286号所含质粒按实施例1中所述方法获得,并送上海生工生物工程技术服务有限公司测序确定突变的yjhg基因序列,并用dnaman软件进行序列比对;突变株62号基因的氨基酸序列为seqidno.3所示;突变株286号基因的氨基酸序列为seqidno.4所示。
突变株yjhg突变位点分析:第967位碱基由g突变成a,相应的氨基酸由丙氨酸突变成苏氨酸,第1855位碱基由g突变成a,相应的氨基酸由天冬氨酸突变成天冬酰胺,第1898位碱基由c突变成t,相应的氨基酸由苏氨酸突变成异亮氨酸。
突变株286号第163位碱基由t突变成c,相应的氨基酸由丝氨酸突变成脯氨酸,第203位氨基酸由g突变成a,相应的氨基酸由丝氨酸突变为天冬酰胺,第967位碱基由g突变成a,相应的氨基酸由丙氨酸突变成苏氨酸。
实施例5:
将基因工程菌62号与286号先分别进行如下培养:
(1)从平板上分别挑取重组菌株pcwj-yjhg的单菌落接种到含有50mg/l氯霉素抗性的5mllb的摇管里,37℃培养6~8h后转接到含有50mg/l氯霉素抗性的100ml的lb培养基里,培养至od600=0.6~0.9,加入0.5mmol的iptg,诱导12h离心收菌,得到重组菌株pcwj-yjhg的细胞
(2)发酵培养:培养基配方:蛋白胨:10g/l,酵母粉:5g/l,氯化钠:5g/l,溶剂为水,将步骤(a)得到的细胞进行发酵培养,培养温度:33℃,培养时间:12h。
将预培养后的基因工程菌62号与238号分别加入木糖酸的水溶液,控制木糖酸溶液在初始反应体系中的浓度为20g/l,菌株的od600=60,于37℃反应12h,通过高效液相色谱检测木糖酸消耗了多少。如图2所示,91号和96号突变株分别在12h内消耗了木糖酸4.2g/l和4.8g/l左右,比亲株a在同样条件下分别提高了2.1与2.4倍。
实施例6:
将62号与286号突变菌株的木糖酸脱水酶基因(yjhg)利用实施例1中的酶切位点将突变的yjhg片段切下来,将突变的yjhg基因与d-木糖脱氢酶基因、苯甲酰甲酸脱羧酶基因、醇脱氢酶基因分别导入质粒中,得到重组质粒bt-91、bt-96,然后将重组质粒分别转化宿主菌trans1t1,得到重组菌bt-t1-62和bt-t1-238。控制反应体系为20ml,底物为20g/l,od=60,ph=7.0。如图3所示,突变菌株bt-t1-62和bt-t1-286的1,2,4-丁三醇的产量分别达18.2g/l和15.69g/l,而未突变菌株的1,2,4-丁三醇的产量为5.43g/l,因此,与为突变菌株相比,提高了约2.89倍和2.5倍。
序列表
<110>南京工业大学
<120>d-木糖酸脱水酶及其应用
<160>7
<170>siposequencelisting1.0
<210>1
<211>655
<212>prt
<213>人工序列(artificialsequence)
<400>1
metservalargasnilephealaaspgluserhisaspiletyrthr
151015
valargthrhisalaaspglyproaspglygluleuproleuthrala
202530
glumetleuileasnargproserglyaspleupheglymetthrthr
354045
asnalaglymetglytrpserproaspgluleuaspargaspglyile
505560
leuleuleuserthrleuglyglyleuargglyalaaspglyargpro
65707580
valalaleualaleuhisglnglyhistyrgluleuaspileglnmet
859095
lysalaalaalagluvalilelysalaasnhisalaleuprotyrala
100105110
valtyrvalseraspprocysaspglyargthrglnglythrthrgly
115120125
metpheaspserleuprotyrargasnaspalasermetvalmetarg
130135140
argleuileargserleuproaspalalysalavalileglyvalala
145150155160
sercysasplysglyleuproalathrmetmetalaleuthralagln
165170175
hisasnilealathrvalleuvalproglyglyalathrleuproala
180185190
lysaspglygluaspasnglylysvalglnthrileglyalaargphe
195200205
alaasnglygluleuserleuglnaspalaargargalaglycyslys
210215220
alacysalaserserglyglyglycysglnpheleuglythralagly
225230235240
thrserglnvalvalalagluglyleuglyleualaileprohisser
245250255
alaleualaproserglygluprovaltrparggluilealaargala
260265270
seralaargalaalaleuasnleuserglnlysglyilethrthrarg
275280285
gluileleuthrasplysalailegluasnalametthrvalhisala
290295300
alapheglyglyserthrasnleuproleuhisileproalaileala
305310315320
hisglnalaglycyshisileprothrvalaspasptrpileargile
325330335
asnlysargvalproargleuvalservalleuproasnglyproval
340345350
tyrhisprothrvalasnalaphemetalaglyglyvalprogluval
355360365
metleuhisleuargserleuglyleuleuhisgluaspvalmetthr
370375380
valthrglyserthrleulysgluasnleuasptrptrpgluhisser
385390395400
gluargargglnargphelysglnleuleuleuaspglngluglnile
405410415
asnalaaspgluvalilemetserproglnglnalalysalaarggly
420425430
leuthrserthrilethrpheprovalglyasnilealaproglugly
435440445
servalilelysserthralaileaspprosermetileaspglugln
450455460
glyiletyrtyrhislysglyvalalalysvaltyrleuserglulys
465470475480
seralailetyraspilelyshisasplysilelysalaglyaspile
485490495
leuvalileileglyvalglyproserglythrglymetglugluthr
500505510
tyrglnvalthrseralaleulyshisleusertyrglylyshisval
515520525
serleuilethraspalaargpheserglyvalserthrglyalacys
530535540
ileglyhisvalglyproglualaleualaglyglyproileglylys
545550555560
leuargthrglyaspleuilegluilelysileaspcysarggluleu
565570575
hisglygluvalasnpheleuglythrargseraspgluglnleupro
580585590
serglngluglualathralaileleuasnalaargproserhisgln
595600605
aspleuleuproaspprogluleuproaspaspthrargleutrpala
610615620
metleuglnalavalserglyglythrtrpthrglycysiletyrasp
625630635640
valasnlysileglyalaalaleuargaspphemetasnlysasn
645650655
<210>2
<211>655
<212>prt
<213>人工序列(artificialsequence)
<400>2
metservalargasnilephealaaspgluserhisaspiletyrthr
151015
valargthrhisalaaspglyproaspglygluleuproleuthrala
202530
glumetleuileasnargproserglyaspleupheglymetthrmet
354045
asnalaglymetglytrpserproaspgluleuaspargaspglyile
505560
leuleuleuserthrleuglyglyleuargglyalaaspglylyspro
65707580
valalaleualaleuhisglnglyhistyrgluleuaspileglnmet
859095
lysalaalaalagluvalilelysalaasnhisalaleuprotyrala
100105110
valtyrvalseraspprocysaspglyargthrglnglythrthrgly
115120125
metpheaspserleuprotyrargasnaspalasermetvalmetarg
130135140
argleuileargserleuproaspalalysalavalileglyvalala
145150155160
sercysasplysglyleuproalathrmetmetalaleualaalagln
165170175
hisasnilealathrvalleuvalproglyglyalathrleuproala
180185190
lysaspglygluaspasnglylysvalglnthrileglyalaargphe
195200205
alaasnglygluleuserleuglnaspalaargargalaglycyslys
210215220
alacysalaserserglyglyglycysglnpheleuglythralagly
225230235240
thrserglnvalvalalagluglyleuglyleualaileprohisser
245250255
alaleualaproserglygluprovaltrparggluilealaargala
260265270
seralaargalaalaleuasnleuserglnlysglyilethrthrarg
275280285
gluileleuthrasplysalailegluasnalametthrvalhisala
290295300
alapheglyglyserthrasnleuleuleuhisileproalaileala
305310315320
hisglnthrglycyshisileprothrvalaspasptrpileargile
325330335
asnlysargvalproargleuvalservalleuproasnglyproval
340345350
tyrhisprothrvalasnalaphemetalaglyglyvalprogluval
355360365
metleuhisleuargserleuglyleuleuhisgluaspvalmetthr
370375380
valthrglyserthrleulysgluasnleuasptrptrpgluhisser
385390395400
gluargargglnargphelysglnleuleuleuaspglngluglnile
405410415
asnalaaspgluvalilemetserproglnglnalalysalaarggly
420425430
leuthrserthrilethrpheprovalglyasnilealaproglugly
435440445
servalilelysserthralaileaspprosermetileaspglugln
450455460
glyiletyrtyrhislysglyvalalalysvaltyrleuserglulys
465470475480
seralailetyraspilelyshisasplysilelysalaglyaspile
485490495
leuvalileileglyvalglyproserglythrglymetglugluthr
500505510
tyrglnvalthrseralaleulyshisleusertyrglylyshisval
515520525
serleuilethraspalaargpheserglyvalserthrglyalacys
530535540
ileglyhisvalglyproglualaleualaglyglyproileglylys
545550555560
leuargthrglyaspleuilegluilelysileaspcysarggluleu
565570575
hisglygluvalasnpheleuglythrargseraspgluglnleupro
580585590
serglngluglualathralaileleuasnalaargproserhisgln
595600605
aspleuleuproaspprogluleuproaspaspthrargleutrpala
610615620
metleuglnalavalserglyglythrtrpthrglycysiletyrasp
625630635640
valasnlysileglyalaalaleuargaspphemetasnlysasn
645650655
<210>3
<211>655
<212>prt
<213>人工序列(artificialsequence)
<400>3
metservalargasnilephealaaspgluserhisaspiletyrthr
151015
valargthrhisalaaspglyproaspglygluleuproleuthrala
202530
glumetleuileasnargproserglyaspleupheglymetthrmet
354045
asnalaglymetglytrpserproaspgluleuaspargaspglyile
505560
leuleuleuserthrleuglyglyleuargglyalaaspglylyspro
65707580
valalaleualaleuhisglnglyhistyrgluleuaspileglnmet
859095
lysalaalaalagluvalilelysalaasnhisalaleuprotyrala
100105110
valtyrvalseraspprocysaspglyargthrglnglythrthrgly
115120125
metpheaspserleuprotyrargasnaspalasermetvalmetarg
130135140
argleuileargserleuproaspalalysalavalileglyvalala
145150155160
sercysasplysglyleuproalathrmetmetalaleualaalagln
165170175
hisasnilealathrvalleuvalproglyglyalathrleuproala
180185190
lysaspglygluaspasnglylysvalglnthrileglyalaargphe
195200205
alaasnglygluleuserleuglnaspalaargargalaglycyslys
210215220
alacysalaserserglyglyglycysglnpheleuglythralagly
225230235240
thrserglnvalvalalagluglyleuglyleualaileprohisser
245250255
alaleualaproserglygluprovaltrparggluilealaargala
260265270
seralaargalaalaleuasnleuserglnlysglyilethrthrarg
275280285
gluileleuthrasplysalailegluasnalametthrvalhisala
290295300
alapheglyglyserthrasnleuleuleuhisileproalaileala
305310315320
hisglnthrglycyshisileprothrvalaspasptrpileargile
325330335
asnlysargvalproargleuvalservalleuproasnglyproval
340345350
tyrhisprothrvalasnalaphemetalaglyglyvalprogluval
355360365
metleuhisleuargserleuglyleuleuhisgluaspvalmetthr
370375380
valthrglyserthrleulysgluasnleuasptrptrpgluhisser
385390395400
gluargargglnargphelysglnleuleuleuaspglngluglnile
405410415
asnalaaspgluvalilemetserproglnglnalalysalaarggly
420425430
leuthrserthrilethrpheprovalglyasnilealaproglugly
435440445
servalilelysserthralaileaspprosermetileaspglugln
450455460
glyiletyrtyrhislysglyvalalalysvaltyrleuserglulys
465470475480
seralailetyraspilelyshisasplysilelysalaglyaspile
485490495
leuvalileileglyvalglyproserglythrglymetglugluthr
500505510
tyrglnvalthrseralaleulyshisleusertyrglylyshisval
515520525
serleuilethraspalaargpheserglyvalserthrglyalacys
530535540
ileglyhisvalglyproglualaleualaglyglyproileglylys
545550555560
leuargthrglyaspleuilegluilelysileaspcysarggluleu
565570575
hisglygluvalasnpheleuglythrargseraspgluglnleupro
580585590
serglngluglualathralaileleuasnalaargproserhisgln
595600605
aspleuleuproaspprogluleuproaspaspthrargleutrpala
610615620
metleuglnalavalserglyglythrtrpthrglycysiletyrasp
625630635640
valasnlysileglyalaalaleuargaspphemetasnlysasn
645650655
<210>4
<211>655
<212>prt
<213>人工序列(artificialsequence)
<400>4
metservalargasnilephealaaspgluserhisaspiletyrthr
151015
valargthrhisalaaspglyproaspglygluleuproleuthrala
202530
glumetleuileasnargproserglyaspleupheglymetthrmet
354045
asnalaglymetglytrpproproaspgluleuaspargaspglyile
505560
leuleuleuasnthrleuglyglyleuargglyalaaspglylyspro
65707580
valalaleualaleuhisglnglyhistyrgluleuaspileglnmet
859095
lysalaalaalagluvalilelysalaasnhisalaleuprotyrala
100105110
valtyrvalseraspprocysaspglyargthrglnglythrthrgly
115120125
metpheaspserleuprotyrargasnaspalasermetvalmetarg
130135140
argleuileargserleuproaspalalysalavalileglyvalala
145150155160
sercysasplysglyleuproalathrmetmetalaleualaalagln
165170175
hisasnilealathrvalleuvalproglyglyalathrleuproala
180185190
lysaspglygluaspasnglylysvalglnthrileglyalaargphe
195200205
alaasnglygluleuserleuglnaspalaargargalaglycyslys
210215220
alacysalaserserglyglyglycysglnpheleuglythralagly
225230235240
thrserglnvalvalalagluglyleuglyleualaileprohisser
245250255
alaleualaproserglygluprovaltrparggluilealaargala
260265270
seralaargalaalaleuasnleuserglnlysglyilethrthrarg
275280285
gluileleuthrasplysalailegluasnalametthrvalhisala
290295300
alapheglyglyserthrasnleuleuleuhisileproalaileala
305310315320
hisglnthrglycyshisileprothrvalaspasptrpileargile
325330335
asnlysargvalproargleuvalservalleuproasnglyproval
340345350
tyrhisprothrvalasnalaphemetalaglyglyvalprogluval
355360365
metleuhisleuargserleuglyleuleuhisgluaspvalmetthr
370375380
valthrglyserthrleulysgluasnleuasptrptrpgluhisser
385390395400
gluargargglnargphelysglnleuleuleuaspglngluglnile
405410415
asnalaaspgluvalilemetserproglnglnalalysalaarggly
420425430
leuthrserthrilethrpheprovalglyasnilealaproglugly
435440445
servalilelysserthralaileaspprosermetileaspglugln
450455460
glyiletyrtyrhislysglyvalalalysvaltyrleuserglulys
465470475480
seralailetyraspilelyshisasplysilelysalaglyaspile
485490495
leuvalileileglyvalglyproserglythrglymetglugluthr
500505510
tyrglnvalthrseralaleulyshisleusertyrglylyshisval
515520525
serleuilethraspalaargpheserglyvalserthrglyalacys
530535540
ileglyhisvalglyproglualaleualaglyglyproileglylys
545550555560
leuargthrglyaspleuilegluilelysileaspcysarggluleu
565570575
hisglygluvalasnpheleuglythrargseraspgluglnleupro
580585590
serglngluglualathralaileleuasnalaargproserhisgln
595600605
aspleuleuproaspprogluleuproaspaspthrargleutrpala
610615620
metleuglnalavalserglyglythrtrpthrglycysiletyrasp
625630635640
valasnlysileglyalaalaleuargaspphemetasnlysasn
645650655
<210>5
<211>744
<212>dna
<213>人工序列(artificialsequence)
<400>5
atgtcttctgctatctacccgtctctgaaaggtaaacgtgttgttatcaccggtggtggt60
tctggtatcggtgctggtctgaccgctggtttcgctcgtcagggtgctgaagttatcttc120
ctggacatcgctgacgaagactctcgtgctctggaagctgaactggctggttctccgatc180
ccgccggtttacaaacgttgcgacctgatgaacctggaagctatcaaagctgttttcgct240
gaaatcggtgacgttgacgttctggttaacaacgctggtaacgacgaccgtcacaaactg300
gctgacgttaccggtgcttactgggacgaacgtatcaacgttaacctgcgtcacatgctg360
ttctgcacccaggctgttgctccgggtatgaaaaaacgtggtggtggtgctgttatcaac420
ttcggttctatctcttggcacctgggtctggaagacctggttctgtacgaaaccgctaaa480
gctggtatcgaaggtatgacccgtgctctggctcgtgaactgggtccggacgacatccgt540
gttacctgcgttgttccgggtaacgttaaaaccaaacgtcaggaaaaatggtacaccccg600
gaaggtgaagctcagatcgttgctgctcagtgcctgaaaggtcgtatcgttccggaaaac660
gttgctgctctggttctgttcctggcttctgacgacgcttctctgtgcaccggtcacgaa720
tactggatcgacgctggttggcgt744
<210>6
<211>1644
<212>dna
<213>人工序列(artificialsequence)
<400>6
atgtatacagtaggagattacctgttagaccgattacacgagttgggaattgaagaaatt60
tttggagttcctggtgactataacttacaatttttagatcaaattatttcacgcgaagat120
atgaaatggattggaaatgctaatgaattaaatgcttcttatatggctgatggttatgct180
cgtactaaaaaagctgccgcatttctcaccacatttggagtcggcgaattgagtgcgatc240
aatggactggcaggaagttatgccgaaaatttaccagtagtagaaattgttggttcacca300
acttcaaaagtacaaaatgacggaaaatttgtccatcatacactagcagatggtgatttt360
aaacactttatgaagatgcatgaacctgttacagcagcgcggactttactgacagcagaa420
aatgccacatatgaaattgaccgagtactttctcaattactaaaagaaagaaaaccagtc480
tatattaacttaccagtcgatgttgctgcagcaaaagcagagaagcctgcattatcttta540
gaaaaagaaagctctacaacaaatacaactgaacaagtgattttgagtaagattgaagaa600
agtttgaaaaatgcccaaaaaccagtagtgattgcaggacacgaagtaattagttttggt660
ttagaaaaaacggtaactcagtttgtttcagaaacaaaactaccgattacgacactaaat720
tttggtaaaagtgctgttgatgaatctttgccctcatttttaggaatatataacgggaaa780
ctttcagaaatcagtcttaaaaattttgtggagtccgcagactttatcctaatgcttgga840
gtgaagcttacggaccgctcaacaggtgcattcacacatcatttagatgaaaataaaatg900
atttcactaaacatagatgaaggaataattttcaataaagtggtagaagattttgatttt960
agagcagtggtttcttctttatcagaattaaaaggaatagaatatgaaggacaatatatt1020
gataagcaatatgaagaatttattccatcaagtgctcccttatcacaagaccgtctatgg1080
caggcagttgaaagtttgactcaaagcaatgaaacaatcgttgctgaacaaggaacctca1140
ttttttggagcttcaacaattttcttaaaatcaaatagtcgttttattggacaaccttta1200
tggggttctattggatatacttttccagcggctttaggaagccaaattgcggataaagag1260
agcagacaccttttatttattggtgatggttcacttcaacttaccgtacaagaattagga1320
ctatcaatcagagaaaaactcaatccaatttgttttatcataaataatgatggttataca1380
gttgaaagagaaatccacggacctactcaaagttataacgacattccaatgtggaattac1440
tcgaaattaccagaaacatttggagcaacagaagatcgtgtagtatcaaaaattgttaga1500
acagagaatgaatttgtgtctgtcatgaaagaagcccaagcagatgtcaatagaatgtat1560
tggatagaactagttttggaaaaagaagatgcgccaaaattactgaaaaaaatgggtaaa1620
ttatttgctgagcaaaataaataa1644
<210>7
<211>1011
<212>dna
<213>人工序列(artificialsequence)
<400>7
atgaaggctgcagttgttacgaaggatcatcatgttgacgttacgtataaaacactgcgc60
tcactgaaacatggcgaagccctgctgaaaatggagtgttgtggtgtatgtcataccgat120
cttcatgttaagaatggcgattttggtgacaaaaccggcgtaattctgggccatgaaggc180
atcggtgtggtggcagaagtgggtccaggtgtcacctcattaaaaccaggcgatcgtgcc240
agcgtggcgtggttctacgaaggatgcggtcattgcgaatactgtaacagtggtaacgaa300
acgctctgccgttcagttaaaaatgccggatacagcgttgatggcgggatggcggaagag360
tgcatcgtggtcgccgattacgcggtaaaagtgccagatggtctggactcggcggcggcc420
agcagcattacctgtgcgggagtcaccacctacaaagccgttaagctgtcaaaaattcgt480
ccagggcagtggattgctatctacggtcttggcggtctgggtaacctcgccctgcaatac540
gcgaagaatgtctttaacgccaaagtgatcgccattgatgtcaatgatgagcagttaaaa600
ctggcaaccgaaatgggcgcagatttagcgattaactcacacaccgaagacgccgccaaa660
attgtgcaggagaaaactggtggcgctcacgctgcggtggtaacagcggtagctaaagct720
gcgtttaactcggcagttgatgctgtccgtgcaggcggtcgtgttgtggctgtcggtcta780
ccgccggagtctatgagcctggatatcccacgtcttgtgctggatggtattgaagtggtc840
ggttcgctggtcggcacgcgccaggatttaactgaagccttccagtttgccgccgaaggt900
aaagtggtgccgaaagtcgccctgcgtccgttagcggacatcaacaccatctttactgag960
atggaagaaggcaaaatccgtggccgcatggtgattgatttccgtcactaa1011
- 还没有人留言评论。精彩留言会获得点赞!