一种用于鉴定莴苣杂交种真实性的方法及其使用的KASP引物组合与流程
本发明属于生物
技术领域:
,具体涉及一种用于鉴定莴苣杂交种真实性的方法及其使用的kasp引物组合。
背景技术:
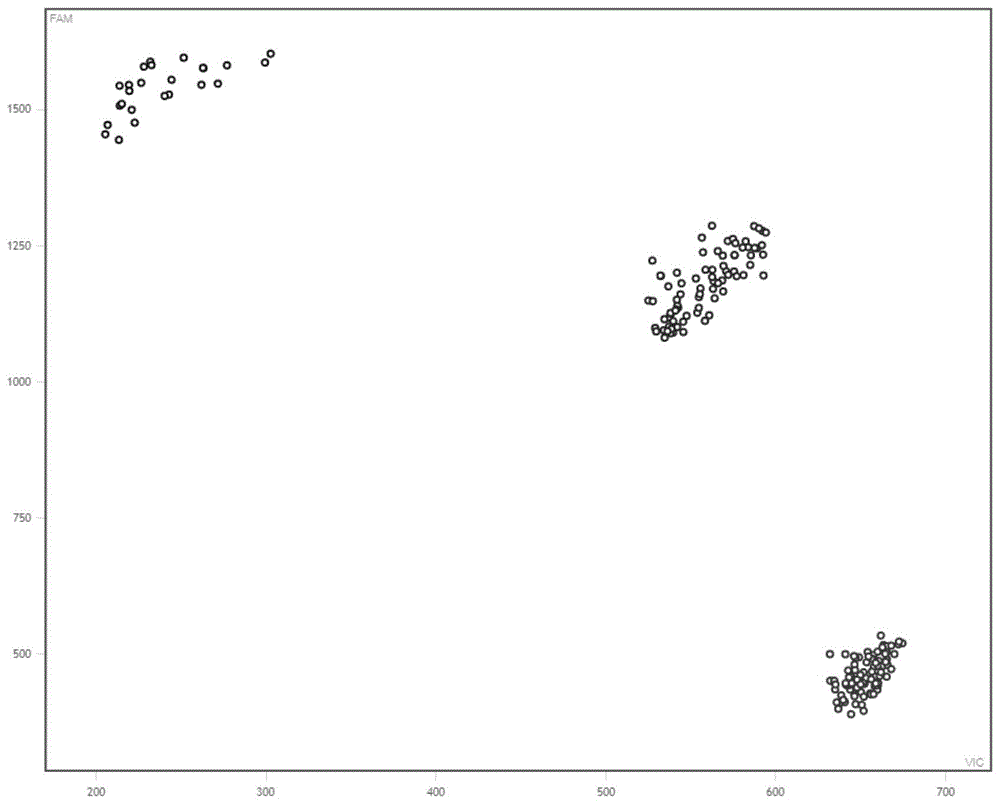
:莴苣(lactucasatival.)起源于欧洲地中海沿岸和西亚一带,由野生种驯化而来。叶用莴苣(生菜)是莴苣的栽培变种,是全球范围内种植面积最广泛的蔬菜之一,也是产量最大的蔬菜之一。莴苣花为舌状花、花小,柱头包裹在花药里面,1朵花由20多朵舌状小花组成。莴苣的花无法在蕾期去雄,只能在开花期进行去雄,但是开花时间很短,这就给真杂种的获得带来不便。目前国内莴苣主要是常规种,受利益的驱动,以常规种(或亲本)冒充杂交种或不同的自交系混杂冒充杂交种等掺杂使假现象时有发生。依据形态性状进行莴苣杂交种田间鉴定的传统手段时效性差,且容易受到环境与主观因素的影响。以dna为基础的分子标记具有准确可靠、简单快速、易于自动化的优点。目前主流的分子标记是ssr(simplesequencerepeats)和snp(singlenucleotidepolymorphism)标记。ssr是第二代分子标记,具有多态性高、呈共显性、易于操作、成本低等优点。基于ssr标记在品种指纹图谱构建、纯度鉴定与遗传多样性分析等方面的研究已有大量报道,但在实践中,ssr也暴露出检测位点较少、数据量少、结果代表性较差、不易实现数据共享等难以克服的缺陷。snp是指基因组核苷酸序列中由单个核苷酸水平上的变异而引起的基因组水平上的dna序列多态性,主要包括单个碱基的缺失、插入、转换及颠换等。基于snp位点设计的标记即为在此基础上发展起来的第三代分子标记,该标记类型具有突变频率低,遗传稳定性高;位点丰富,分布广泛;检测快速,筛选规模化等特点,在纯系鉴定及分子辅助单株选择等方面具有优势。snp标记作为第3代分子标记,与ssr标记相比具有以下优势:一是在基因组中密度更高分布更均匀;二是更适合数据库整合和数据共享;三是检测位点数量远远高于ssr;四是与功能基因甚至植物表型关联度高。snp标记目前被公认是极具应用前景的分子标记技术。snp位点有基于测序、芯片及pcr等各种平台的检测方法。其中,kasp(kompetitiveallelespecificpcr,即竞争性等位基因特异性pcr)技术是基于引物末端碱基的特异匹配对snp分型,可在广泛的基因组dna样品中对snp位点进行精准的双等位基因判断,具有高度稳定性与准确性。与第二代分子标记相比,kasp标记不需要根据dna片段大小进行分型,能够摆脱传统凝胶电泳这种步骤相对繁琐,低通量,价格又较贵的检测方法。因此,更加适用于现阶段飞速发展的高通量分子检测平台。开发适合于高通量分子检测平台的莴苣杂交种kasp分子标记对于推广普及分子标记技术的应用、提高我国莴苣的杂交育种效率和育种水平具有重要意义。技术实现要素:本发明的目的是鉴定莴苣品种(尤其是41个莴苣品种)和莴苣杂交种(尤其是32个莴苣杂交种)。本发明首先保护引物组合,可包括用于扩增sol_kasp01位点的引物组1、用于扩增sol_kasp02位点的引物组2、用于扩增sol_kasp03位点的引物组3、用于扩增sol_kasp04位点的引物组4、用于扩增sol_kasp05位点的引物组5、用于扩增sol_kasp06位点的引物组6、用于扩增sol_kasp07位点的引物组7、用于扩增sol_kasp08位点的引物组8、用于扩增sol_kasp09位点的引物组9、用于扩增sol_kasp10位点的引物组10、用于扩增sol_kasp11位点的引物组11、用于扩增sol_kasp12位点的引物组12、用于扩增sol_kasp13位点的引物组13、用于扩增sol_kasp14位点的引物组14、用于扩增sol_kasp15位点的引物组15、用于扩增sol_kasp16位点的引物组16、用于扩增sol_kasp17位点的引物组17和用于扩增sol_kasp18位点的引物组18。所述sol_kasp01位点—所述sol_kasp18位点可为莴苣基因组的18个snp位点。所述sol_kasp01位点为1号染色体上的第126697665位核苷酸。所述sol_kasp02位点为1号染色体上的第147985160位核苷酸。所述sol_kasp03位点为2号染色体上的第122143189位核苷酸。所述sol_kasp04位点为2号染色体上的第210259848位核苷酸。所述sol_kasp05位点为3号染色体上的第27187844位核苷酸。所述sol_kasp06位点为3号染色体上的第212222861位核苷酸。所述sol_kasp07位点为3号染色体上的第247666277位核苷酸。所述sol_kasp08位点为4号染色体上的第215625068位核苷酸。所述sol_kasp09位点为4号染色体上的第340636295位核苷酸。所述sol_kasp10位点为5号染色体上的第313574466位核苷酸。所述sol_kasp11位点为6号染色体上的第88254911位核苷酸。所述sol_kasp12位点为7号染色体上的第210730056位核苷酸。所述sol_kasp13位点为7号染色体上的第89707923位核苷酸。所述sol_kasp14位点为7号染色体上的第103614221位核苷酸。所述sol_kasp15位点为8号染色体上的第215217745位核苷酸。所述sol_kasp16位点为8号染色体上的第240162601位核苷酸。所述sol_kasp17位点为9号染色体上的第84172204位核苷酸。所述sol_kasp18位点为9号染色体上的第173010178位核苷酸。所述引物组合中,所述引物组1可由seqidno:1所示的正向引物01f1、seqidno:2所示的正向引物01f2和seqidno:3所示的反向引物01r组成。所述引物组2可由seqidno:4所示的正向引物02f1、seqidno:5所示的正向引物02f2和seqidno:6所示的反向引物02r组成。所述引物组3可由seqidno:7所示的正向引物03f1、seqidno:8所示的正向引物03f2和seqidno:9所示的反向引物03r组成。所述引物组4可由seqidno:10所示的正向引物04f1、seqidno:11所示的正向引物04f2和seqidno:12所示的反向引物04r组成。所述引物组5可由seqidno:13所示的正向引物05f1、seqidno:14所示的正向引物05f2和seqidno:15所示的反向引物05r组成。所述引物组6可由seqidno:16所示的正向引物06f1、seqidno:17所示的正向引物06f2和seqidno:18所示的反向引物06r组成。所述引物组7可由seqidno:19所示的正向引物07f1、seqidno:20所示的正向引物07f2和seqidno:21所示的反向引物07r组成。所述引物组8可由seqidno:22所示的正向引物08f1、seqidno:23所示的正向引物08f2和seqidno:24所示的反向引物08r组成。所述引物组9可由seqidno:25所示的正向引物09f1、seqidno:26所示的正向引物09f2和seqidno:27所示的反向引物09r组成。所述引物组10可由seqidno:28所示的正向引物10f1、seqidno:29所示的正向引物10f2和seqidno:30所示的反向引物10r组成。所述引物组11可由seqidno:31所示的正向引物11f1、seqidno:32所示的正向引物11f2和seqidno:33所示的反向引物11r组成。所述引物组12可由seqidno:34所示的正向引物12f1、seqidno:35所示的正向引物12f2和seqidno:36所示的反向引物12r组成。所述引物组13可由seqidno:37所示的正向引物13f1、seqidno:38所示的正向引物13f2和seqidno:39所示的反向引物13r组成。所述引物组14可由seqidno:40所示的正向引物14f1、seqidno:41所示的正向引物14f2和seqidno:42所示的反向引物14r组成。所述引物组15可由seqidno:43所示的正向引物15f1、seqidno:44所示的正向引物15f2和seqidno:45所示的反向引物15r组成。所述引物组16可由seqidno:46所示的正向引物16f1、seqidno:47所示的正向引物16f2和seqidno:32所示的反向引物16r组成。所述引物组17可由seqidno:49所示的正向引物17f1、seqidno:50所示的正向引物17f2和seqidno:51所示的反向引物17r组成。所述引物组18可由seqidno:52所示的正向引物18f1、seqidno:53所示的正向引物18f2和seqidno:54所示的反向引物18r组成。所述引物组合中,所述引物组1可由seqidno:1自5’末端起第22至45位所示的正向引物01f1、seqidno:2自5’末端起第22至45位所示的正向引物01f2和seqidno:3所示的反向引物01r组成。所述引物组2可由seqidno:4自5’末端起第22至45位所示的正向引物02f1、seqidno:5自5’末端起第22至44位所示的正向引物02f2和seqidno:6所示的反向引物02r组成。所述引物组3可由seqidno:7自5’末端起第22至46位所示的正向引物03f1、seqidno:8自5’末端起第22至47位所示的正向引物03f2和seqidno:9所示的反向引物03r组成。所述引物组4可由seqidno:10自5’末端起第22至48位所示的正向引物04f1、seqidno:11自5’末端起第22至48位所示的正向引物04f2和seqidno:12所示的反向引物04r组成。所述引物组5可由seqidno:13自5’末端起第22至43位所示的正向引物05f1、seqidno:14自5’末端起第22至44位所示的正向引物05f2和seqidno:15所示的反向引物05r组成。所述引物组6可由seqidno:16自5’末端起第22至48位所示的正向引物06f1、seqidno:17自5’末端起第22至48位所示的正向引物06f2和seqidno:18所示的反向引物06r组成。所述引物组7可由seqidno:19自5’末端起第22至47位所示的正向引物07f1、seqidno:20自5’末端起第22至47位所示的正向引物07f2和seqidno:21所示的反向引物07r组成。所述引物组8可由seqidno:22自5’末端起第22至44位所示的正向引物08f1、seqidno:23自5’末端起第22至46位所示的正向引物08f2和seqidno:24所示的反向引物08r组成。所述引物组9可由seqidno:25自5’末端起第22至43位所示的正向引物09f1、seqidno:26自5’末端起第22至44位所示的正向引物09f2和seqidno:27所示的反向引物09r组成。所述引物组10可由seqidno:28自5’末端起第22至46位所示的正向引物10f1、seqidno:29自5’末端起第22至46位所示的正向引物10f2和seqidno:30所示的反向引物10r组成。所述引物组11可由seqidno:31自5’末端起第22至45位所示的正向引物11f1、seqidno:32自5’末端起第22至45位所示的正向引物11f2和seqidno:33所示的反向引物11r组成。所述引物组12可由seqidno:34自5’末端起第22至44位所示的正向引物12f1、seqidno:35自5’末端起第22至44位所示的正向引物12f2和seqidno:36所示的反向引物12r组成。所述引物组13可由seqidno:37自5’末端起第22至41位所示的正向引物13f1、seqidno:38自5’末端起第22至41位所示的正向引物13f2和seqidno:39所示的反向引物13r组成。所述引物组14可由seqidno:40自5’末端起第22至41位所示的正向引物14f1、seqidno:41自5’末端起第22至41位所示的正向引物14f2和seqidno:42所示的反向引物14r组成。所述引物组15可由seqidno:43自5’末端起第22至42位所示的正向引物15f1、seqidno:44自5’末端起第22至42位所示的正向引物15f2和seqidno:45所示的反向引物15r组成。所述引物组16可由seqidno:46自5’末端起第22至46位所示的正向引物16f1、seqidno:47自5’末端起第22至46位所示的正向引物16f2和seqidno:32所示的反向引物16r组成。所述引物组17可由seqidno:49自5’末端起第22至47位所示的正向引物17f1、seqidno:50自5’末端起第22至47位所示的正向引物17f2和seqidno:51所示的反向引物17r组成。所述引物组18可由seqidno:52自5’末端起第22至45位所示的正向引物18f1、seqidno:53自5’末端起第22至45位所示的正向引物18f2和seqidno:54所示的反向引物18r组成。上述任一所述引物组中,名称中含有“f1”的引物、名称中含有“f2”的引物和名称中含有“r”的引物的摩尔比具体可为1:1:1。上述任一所述引物组合具体可由所述引物组1—所述引物组18组成。上文中,seqidno:2自5′末端起第1至21位所示的核苷酸序列为荧光标签序列(即fam荧光标签序列),荧光信号具体为蓝色。seqidno:1自5′末端起第1至21位所示的核苷酸序列也为荧光标签序列(即hex荧光标签序列),荧光信号具体为红色。含有上述任一所述引物组合的试剂盒也属于本发明的保护范围。所述试剂盒的制备方法也属于本发明的保护范围。所述试剂盒的制备方法包括将上述任一所述引物组中的各条引物分别单独包装或混合包装的步骤。所述试剂盒的应用也属于本发明的保护范围。所述试剂盒的应用可为x3)、x4)、x7)或x8):x3)鉴定41个莴苣品种;x4)鉴别41个莴苣品种真伪性;x7)鉴定32个莴苣杂交种;x8)鉴别32个莴苣杂交种真伪性。本发明还保护上述任一所述引物组合的应用,可为x1)至x8)中的任一种:x1)制备用于鉴定41个莴苣品种的试剂盒;x2)制备用于鉴别41个莴苣品种真伪性的试剂盒;x3)鉴定41个莴苣品种;x4)鉴别41个莴苣品种真伪性;x5)制备用于鉴定32个莴苣杂交种的试剂盒;x6)制备用于鉴别32个莴苣杂交种真伪性的试剂盒;x7)鉴定32个莴苣杂交种;x8)鉴别32个莴苣杂交种真伪性。本发明还保护一种鉴定待测莴苣属于41个莴苣品种和32个莴苣杂交种中哪个品种的方法,包括如下步骤:分别检测待测莴苣、41个莴苣品种和32个莴苣杂交种基于所述18个snp位点的基因型,然后进行如下判断:如果待测莴苣基于所述18个snp位点的基因型和41个莴苣品种或32个莴苣杂交种中某品种基于所述18个snp位点的基因型完全一致,则待测莴苣与该莴苣品种属于同一个品种;如果待测莴苣基于所述18个snp位点的基因型和41个莴苣品种或32个莴苣杂交种中各个品种基于所述18个snp位点的基因型均不一致,则待测莴苣与41个莴苣品种的品种均不相同。上述方法中,所述“检测待测莴苣、41个莴苣品种和32个莴苣杂交种基于所述18个snp位点的基因型”的步骤可如下:(1)分别以待测莴苣的基因组dna、41个莴苣品种和32个莴苣杂交种的基因组dna为模板,采用上述任一所述引物组合中引物组的进行pcr扩增,得到pcr扩增产物;(2)完成步骤(1)后,采用仪器检测pcr扩增产物的荧光信号,根据荧光信号的颜色获得待测莴苣、41个莴苣品种和32个莴苣杂交种基于所述18个snp位点的基因型。上述方法中,所述“检测待测莴苣、41个莴苣品种和32个莴苣杂交种基于所述18个snp位点的基因型”的步骤可如下:(1)分别以待测莴苣的基因组dna、41个莴苣品种和32个莴苣杂交种的基因组dna为模板,采用上述任一所述引物组合中引物组的进行pcr扩增,得到pcr扩增产物;(2)取步骤(1)得到的pcr扩增产物,测序;(3)根据步骤(2)得到的测序结果,获得待测莴苣、41个莴苣品种和32个莴苣杂交种基于所述18个snp位点的基因型。本发明还保护一种鉴定待测莴苣属于41个莴苣品种和32个莴苣杂交种中哪个品种的方法,包括如下步骤:(1)以待测莴苣的基因组dna为模板,采用上述任一所述引物组合中引物组的进行pcr扩增,得到pcr扩增产物;以标准莴苣品种群中的各个莴苣品种的基因组dna为模板,采用上述任一所述引物组合中引物组的进行pcr扩增,得到pcr扩增产物;所述标准莴苣品种群由41个莴苣品种组成;以标准莴苣杂交种群中的各个莴苣品种的基因组dna为模板,采用上述任一所述引物组合中引物组的进行pcr扩增,得到pcr扩增产物;所述标准莴苣杂交种群由32个莴苣杂交种组成;(2)将待测莴苣得到的各个pcr扩增产物与每个标准莴苣品种或每个标准莴苣杂交种对应的pcr扩增产物进行聚类分析,聚类分析中待测莴苣与哪个标准莴苣品种或哪个标准莴苣杂交种为同一类,该待测莴苣与该标准莴苣品种或该标准莴苣杂交种即属于同一个品种。上述任一所述的方法中,所述采用上述任一所述引物组合中引物组的进行pcr扩增可采用荧光定量pcr仪ab-q6flex(赛默飞世尔科技(中国)有限公司)或arraytape平台(douglasscientific公司)进行。采用荧光定量pcr仪ab-q6flex检测时的反应体系为5μl,包括1.0μl莴苣的基因组dna(浓度为50ng/μl)、0.07μl引物混合液(引物混合液中各条引物的浓度均为100pmol/l)、2.5μl2×kaspmix(艾吉析科技(上海)有限公司,货号为1536)和1.43μlddh20。反应程序为:95℃预变性10min;95℃变性20s,55℃退火60s,40个循环。按照荧光定量pcr仪ab-q6操作手册,编辑样品表,执行运行程序,保存数据。采用arraytape平台检测时的反应体系为1.6μl,包括0.8μl莴苣的基因组dna(浓度为50ng/μl)、0.03μl引物混合液(引物混合液中各条引物的浓度均为100pmol/l)和0.8μl2×kaspmix。反应程序为:95℃预变性10min;95℃变性20s,55℃退火60s,40个循环。按照arraytape平台仪器操作手册,运行程序,保存数据。数据记录:利用上述两种检测平台获得的数据中,纯合位点的基因型数据记录为allele1/allele1或allele2/allele2,杂合位点的基因型数据记录为allele1/allele2,其中allele1、allele2分别为该变异位点上两个等位碱基;缺失位点基因型数据记录为0;例如样品在某个位点上基因型为allele1/allele2,该位点的等位碱基为a/c,allele1代表碱基a,在该位点的基因型记录为a/c。上述方法中,所述聚类分析可为使用r(v3.5.1)软件的adegenet、poppr、ape、phangorn、hierfstat程序包对上述41种莴苣的dna指纹图谱进行聚类分析并导出tree格式文件,通过evolview软件进行聚类作图。上述任一所述41个莴苣品种可为c385、b117、b90、c345、r776、n1195、、l1056、m560、m546、m1070、l452、n1190、l1018、l467、m587、m1062、m1060、m1110、m1097、m540、s1253、、、r811、l399、l1015、l521、n703、n742、b902、c371、m1090、m1106、n1151、c306、sy-18、m586、l471、sy-19、n644、n1159、、n1144和n1158。上述任一所述32个莴苣杂交种可为n742×b902、l1056×s1253、l1056×m560、m546×m1070、m1090×m1106、m1070×m546、m1060×m1110、m1110×m1060、l452×n1190、m540×m1097、m1062×m587、l1018×l467、r811×l399、n1190×l452、n1190×l1015、l467×l1018、m1097×m540、m587×m1062、、l521×n1195、c345×r776、、n703×c371、n703×b90、n1151×n1159、n1151×n158、c385×b117、n1151×n1144、、n703×b117、c385×b90、、n1151×l471、sy-19×n644、sy-18×m586和n1151×c306。本发明提供的引物组合可用来鉴定待测莴苣样品,还可以用来鉴定莴苣杂交种的真实性,使得辨别莴苣杂交种真伪的操作变得准确便捷。本发明提供的方法具有高通量、准确、低成本、操作简单、节约人力、物力等优点,且不易受到环境影响,具有十分广阔的应用前景。本发明具有重要的应用价值。附图说明图1为采用表2所示的18个引物组鉴定表3所示的41种莴苣的结果。图2为采用人工合成的其它引物组鉴定表3所示的41种莴苣的结果。图3为41种莴苣在18个snp位点的基因型。图4为41种莴苣的dna指纹图谱的聚类分析。具体实施方式下面结合具体实施方式对本发明进行进一步的详细描述,给出的实施例仅为了阐明本发明,而不是为了限制本发明的范围。以下提供的实施例可作为本
技术领域:
普通技术人员进行进一步改进的指南,并不以任何方式构成对本发明的限制。下述实施例中的实验方法,如无特殊说明,均为常规方法,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。实施例1、用于鉴定莴苣的引物组合的获得一、18个snp位点的发现本发明通过对莴苣基因组进行大量序列分析和功能分析,获得多态性较好、在染色体上均匀分布、特异性高的200个snp位点。设计200个ksap引物组,之后进行大量筛选,选择出扩增效果稳定、分型清晰、能区分出待测样本的引物组18个,这18个引物组对应的snp位点的基本信息详见表1,其中snp位点在染色体上的位置是参考莴苣基因组v8版本(网址为:https://genomevolution.org/coge/genomeinfo.pl?gid=28333)确定的。表1.18个snp位点的基本信息snp位点的名称snp位点的碱基类型所在染色体snp位点在染色体上的位置sol_kasp01a/g1126697665sol_kasp02a/g1147985160sol_kasp03g/c2122143189sol_kasp04g/a2210259848sol_kasp05t/a327187844sol_kasp06a/t3212222861sol_kasp07g/c3247666277sol_kasp08c/t4215625068sol_kasp09g/a4340636295sol_kasp10c/g5313574466sol_kasp11g/a688254911sol_kasp12a/g7210730056sol_kasp13a/c789707923sol_kasp14g/a7103614221sol_kasp15t/c8215217745sol_kasp16t/a8240162601sol_kasp17g/t984172204sol_kasp18c/t9173010178二、用于鉴定莴苣的引物组合的获得引物组合由18个引物组组成。每个引物组的名称见表2中第2列。每个引物组由3条引物序列组成,用于扩增一个snp位点。18个引物组中各个引物的核苷酸序列如表2中第4列所示。采用上述18个引物组鉴定表3所示的41种莴苣的结果见图1。采用其它引物组鉴定表3所示的41种莴苣的结果见图2。表2.18个引物组及其引物的核苷酸序列注:粗体为hex荧光标签序列,斜体为fam荧光标签序列。表3实施例2、莴苣dna指纹库构建及应用一、莴苣dna指纹库的构建1、采用ctab法分别提取表3所示的41种莴苣幼苗叶片的基因组dna,得到相应的莴苣的基因组dna。2、分别以41种莴苣的基因组dna为模板,采用18个引物组进行pcr扩增(18个引物组在同一反应体系中),检测在18个snp位点的基因型。采用荧光定量pcr仪ab-q6flex(赛默飞世尔科技(中国)有限公司)或arraytape平台(douglasscientific公司)进行。采用荧光定量pcr仪ab-q6flex检测时的反应体系为5μl,包括1.0μl莴苣的基因组dna(浓度为50ng/μl)、0.07μl引物混合液(引物混合液中各条引物的浓度均为100pmol/l)、2.5μl2×kaspmix(艾吉析科技(上海)有限公司,货号为1536)和1.43μlddh20。反应程序为:95℃预变性10min;95℃变性20s,55℃退火60s,40个循环。按照荧光定量pcr仪ab-q6操作手册,编辑样品表,执行运行程序,保存数据。采用arraytape平台检测时的反应体系为1.6μl,包括0.8μl莴苣的基因组dna(浓度为50ng/μl)、0.03μl引物混合液(引物混合液中各条引物的浓度均为100pmol/l)和0.8μl2×kaspmix。反应程序为:95℃预变性10min;95℃变性20s,55℃退火60s,40个循环。按照arraytape平台仪器操作手册,运行程序,保存数据。数据记录:利用上述两种检测平台获得的数据中,纯合位点的基因型数据记录为allele1/allele1或allele2/allele2,杂合位点的基因型数据记录为allele1/allele2,其中allele1、allele2分别为该变异位点上两个等位碱基;缺失位点基因型数据记录为0;例如样品在某个位点上基因型为allele1/allele2,该位点的等位碱基为a/c,allele1代表碱基a,在该位点的基因型记录为a/c。41种莴苣在18个snp位点的基因型见图3,即41种莴苣的dna指纹图谱。3、使用r(v3.5.1)软件的adegenet、poppr、ape、phangorn、hierfstat程序包对上述41种莴苣的dna指纹图谱进行聚类分析并导出tree格式文件,通过evolview软件进行聚类作图。结果见图4。结果表明,实施例1用于鉴定莴苣的引物组合可以将表3所示的41种莴苣完全区分。二、莴苣dna指纹库的应用1、采用ctab法提取待测莴苣幼苗叶片的基因组dna,得到待测莴苣的基因组dna。2、以待测莴苣的基因组dna为模板,采用18个引物组进行pcr扩增(18个引物组在同一反应体系中),检测在18个snp位点的基因型。检测方法参考步骤一中2。3、完成步骤2后,使用r(v3.5.1)软件的adegenet、poppr、ape、phangorn、hierfstat程序包比对待测莴苣dna指纹图谱和41种莴苣的dna指纹图谱,如果待测莴苣dna指纹图谱与41种莴苣的dna指纹图谱中的某个莴苣的dna指纹图谱完全一致,则待测莴苣与该莴苣属于同一个品种。4、按照上述方法,获得2个待测莴苣的在18个snp位点的基因型,之后进行如下判断:如果差异位点数≥2,则两个待测莴苣为不同品种;如果差异位点数为1,则两个待测莴苣为近似品种;如果没有差异位点数,则两个待测莴苣为极近似或相同品种。实施例3、实施例1获得的用于鉴定莴苣的引物组合在鉴定莴苣杂交种真实性中的应用32个莴苣杂交种的杂交组合详见表4,所有莴苣杂交种均为杂交f1。表41、采用ctab法分别提取表4所示的32个莴苣杂交种叶片的基因组dna,得到相应的莴苣杂交种的基因组dna。2、分别以32个莴苣杂交种的基因组dna为模板,采用18个引物组进行pcr扩增(18个引物组在同一反应体系中),检测在18个snp位点的基因型。检测方法参考步骤一中2。3、获得32个莴苣杂交种在18个snp位点的基因型,即32个莴苣杂交种的dna指纹图谱。4、采用ctab法提取待测莴苣幼苗叶片的基因组dna,得到待测莴苣的基因组dna。5、以待测莴苣的基因组dna为模板,采用18个引物组进行pcr扩增(18个引物组在同一反应体系中),检测在18个snp位点的基因型。检测方法参考步骤一中2。6、将待测莴苣基于18个snp位点的基因型和32个莴苣杂交种基于18个snp位点的基因型进行比对,进行如下判断:如果待测莴苣基于18个snp位点的基因型和某一莴苣杂交种基于18个snp位点的基因型完全一致,则待测莴苣为该莴苣杂交种的真实的杂交种f1。例如c385×b90杂交组合,亲本c385是典型的绿色,亲本b90是典型的紫红色,杂交f1是淡紫红色。取二者杂交后得到的f1在田间是淡紫红色的植株,进行上述步骤1-6。结果表明,上述植株18个snp位点的基因型依次g/g、a/g、g/g、g/g、t/a、a/t、g/g、t/t、a/a、c/c、g/g、a/g、c/c、a/a、t/c、a/a、g/t和c/t,与c385×b90杂交种基于18个snp位点的基因型完全一致,证明该植株即是c385×b90真实的杂交种f1。以上对本发明进行了详述。对于本领域技术人员来说,在不脱离本发明的宗旨和范围,以及无需进行不必要的实验情况下,可在等同参数、浓度和条件下,在较宽范围内实施本发明。虽然本发明给出了特殊的实施例,应该理解为,可以对本发明作进一步的改进。总之,按本发明的原理,本申请欲包括任何变更、用途或对本发明的改进,包括脱离了本申请中已公开范围,而用本领域已知的常规技术进行的改变。按以下附带的权利要求的范围,可以进行一些基本特征的应用。<110>北京农业生物技术研究中心<120>一种用于鉴定莴苣杂交种真实性的方法及其使用的kasp引物组合<160>54<170>patentinversion3.5<210>1<211>45<212>dna<213>artificialsequence<400>1gaaggtcggagtcaacggattgcagcacctacaacagctataaca45<210>2<211>45<212>dna<213>artificialsequence<400>2gaaggtgaccaagttcatgctgcagcacctacaacagctataacg45<210>3<211>26<212>dna<213>artificialsequence<400>3cctctaatcatcagaatatttcccag26<210>4<211>45<212>dna<213>artificialsequence<400>4gaaggtcggagtcaacggattttgggtccacttgttaatgtgtta45<210>5<211>44<212>dna<213>artificialsequence<400>5gaaggtgaccaagttcatgcttgggtccacttgttaatgtgttg44<210>6<211>25<212>dna<213>artificialsequence<400>6aaatccataggagaagtgttgcttt25<210>7<211>46<212>dna<213>artificialsequence<400>7gaaggtcggagtcaacggattataaaggttgactagcatcaagtcg46<210>8<211>47<212>dna<213>artificialsequence<400>8gaaggtgaccaagttcatgcttataaaggttgactagcatcaagtcc47<210>9<211>24<212>dna<213>artificialsequence<400>9gaacttcttcctagtggtccttgc24<210>10<211>48<212>dna<213>artificialsequence<400>10gaaggtcggagtcaacggattaattaccaagaagtagtcacagaacag48<210>11<211>48<212>dna<213>artificialsequence<400>11gaaggtgaccaagttcatgctaattaccaagaagtagtcacagaacaa48<210>12<211>24<212>dna<213>artificialsequence<400>12ttaggttgatcggtggtttgtact24<210>13<211>43<212>dna<213>artificialsequence<400>13gaaggtcggagtcaacggatttggtgttcatgggtgatttctt43<210>14<211>44<212>dna<213>artificialsequence<400>14gaaggtgaccaagttcatgctttggtgttcatgggtgatttcta44<210>15<211>24<212>dna<213>artificialsequence<400>15tgtgaccaccttaggattgtatgc24<210>16<211>48<212>dna<213>artificialsequence<400>16gaaggtcggagtcaacggattcatcttttccattttatatgtttccta48<210>17<211>48<212>dna<213>artificialsequence<400>17gaaggtgaccaagttcatgctcatcttttccattttatatgtttcctt48<210>18<211>21<212>dna<213>artificialsequence<400>18ggagattgttgatcggttgcc21<210>19<211>47<212>dna<213>artificialsequence<400>19gaaggtcggagtcaacggattatgtttcaaaaaaagaaagagtcaag47<210>20<211>47<212>dna<213>artificialsequence<400>20gaaggtgaccaagttcatgctatgtttcaaaaaaagaaagagtcaac47<210>21<211>27<212>dna<213>artificialsequence<400>21tagtctctgcttactctctttgtctga27<210>22<211>44<212>dna<213>artificialsequence<400>22gaaggtcggagtcaacggattgataaccttttggattccagatc44<210>23<211>46<212>dna<213>artificialsequence<400>23gaaggtgaccaagttcatgctgagataaccttttggattccagatt46<210>24<211>20<212>dna<213>artificialsequence<400>24cggagtagccggatggtttc20<210>25<211>43<212>dna<213>artificialsequence<400>25gaaggtcggagtcaacggattactctatcgagcaagaaggctg43<210>26<211>44<212>dna<213>artificialsequence<400>26gaaggtgaccaagttcatgctgactctatcgagcaagaaggcta44<210>27<211>23<212>dna<213>artificialsequence<400>27aatccttgcatcattaaccaaag23<210>28<211>46<212>dna<213>artificialsequence<400>28gaaggtcggagtcaacggattgtgggtaatctctgaagagatgaac46<210>29<211>46<212>dna<213>artificialsequence<400>29gaaggtgaccaagttcatgctgtgggtaatctctgaagagatgaag46<210>30<211>25<212>dna<213>artificialsequence<400>30tgctaagtcgattagcaagtaaatg25<210>31<211>45<212>dna<213>artificialsequence<400>31gaaggtcggagtcaacggattagatgaatgttttgaaggattgtc45<210>32<211>45<212>dna<213>artificialsequence<400>32gaaggtgaccaagttcatgctagatgaatgttttgaaggattgtt45<210>33<211>23<212>dna<213>artificialsequence<400>33cactgaccaatccttgctaccta23<210>34<211>44<212>dna<213>artificialsequence<400>34gaaggtcggagtcaacggattggaagaacctctatatcccatca44<210>35<211>44<212>dna<213>artificialsequence<400>35gaaggtgaccaagttcatgctggaagaacctctatatcccatcg44<210>36<211>21<212>dna<213>artificialsequence<400>36attccccacttcataaatggc21<210>37<211>41<212>dna<213>artificialsequence<400>37gaaggtcggagtcaacggattcaacctgcctctccgatgga41<210>38<211>41<212>dna<213>artificialsequence<400>38gaaggtgaccaagttcatgctcaacctgcctctccgatggc41<210>39<211>22<212>dna<213>artificialsequence<400>39cccatatgtttttggaggactt22<210>40<211>41<212>dna<213>artificialsequence<400>40gaaggtcggagtcaacggattgtgccaaacgcttggaccag41<210>41<211>41<212>dna<213>artificialsequence<400>41gaaggtgaccaagttcatgctgtgccaaacgcttggaccaa41<210>42<211>21<212>dna<213>artificialsequence<400>42cttttgttggctgaggagtgg21<210>43<211>42<212>dna<213>artificialsequence<400>43gaaggtcggagtcaacggattgaggctcctccatccctattt42<210>44<211>42<212>dna<213>artificialsequence<400>44gaaggtgaccaagttcatgctgaggctcctccatccctattc42<210>45<211>23<212>dna<213>artificialsequence<400>45gttgtcctcattgactacatgca23<210>46<211>46<212>dna<213>artificialsequence<400>46gaaggtcggagtcaacggattgttttagagagctgtggtaactcat46<210>47<211>46<212>dna<213>artificialsequence<400>47gaaggtgaccaagttcatgctgttttagagagctgtggtaactcaa46<210>48<211>20<212>dna<213>artificialsequence<400>48ctgcacgtacatccctttca20<210>49<211>47<212>dna<213>artificialsequence<400>49gaaggtcggagtcaacggatttccttttataacttgccttatttttg47<210>50<211>47<212>dna<213>artificialsequence<400>50gaaggtgaccaagttcatgcttccttttataacttgccttatttttt47<210>51<211>26<212>dna<213>artificialsequence<400>51agttttatgacatggagatttttacg26<210>52<211>45<212>dna<213>artificialsequence<400>52gaaggtcggagtcaacggattcaagagagtggttacggttgtttc45<210>53<211>45<212>dna<213>artificialsequence<400>53gaaggtgaccaagttcatgctcaagagagtggttacggttgtttt45<210>54<211>24<212>dna<213>artificialsequence<400>54tgaagcaacactataagcccaaca24当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1