产生人抗体的转基因鸡的制作方法
交叉引用
本申请要求于2018年3月21日提交的美国临时申请系列号62/646,319的权益,该申请通过引用并入本文。
背景
与所有其它脊椎动物一样,鸡中b细胞发育的关键检查点是读框内v(d)j重排,从而导致功能性b细胞受体复合物在细胞表面表达(1-3)。鸡的重排过程利用与哺乳动物相同的重组信号序列和酶,将v、d和j基因重组为功能性v区(4-8)。鸡中的主要区别在于,在轻链和重链基因座中,只有一个种系v基因和一个种系j基因,而在重链中则具有多个高度相似的d区段(5、8),而不是在人中的大量的v、d和j基因。因此,重排过程在最初的b细胞库中几乎没有产生序列多样性。v-j和v-d-j连接处的不完美连接和核酸外切回切可以产生一些多样性,但是鸡b细胞不表达tdt(9),因此cdr-h3中没有n-添加,并且一般来说,由基因重排过程产生的免疫球蛋白多样性是最低程度的(5、10、11)。
为了产生多样化的库,鸡采用基因转换的过程,其中轻链和重链基因座中的上游假基因充当序列供体,以突变表达的功能性v(8、11-13)。这些假基因不包含启动子或重组信号序列,因此它们本身无法表达,但是它们的序列以可变长度的区段整合到单个功能性v中。来自库中不同假基因的多轮重叠基因转换导致高度多样化的幼稚库。除了基因转换外,非模板化的体细胞超突变也促成库的多样性(14-16)。尽管在鸡中存在v(d)j重排的局限性,但cdr-h3表现出与哺乳动物相当的长度和序列多样性(1、17)。鸡d在v(d)j重排中的功能可能与提供cdr-h3内二硫键桥用于稳定抗原结合环更相关(17),因为大多数d编码单个半胱氨酸残基,因此d-d连接会因此编码配对的半胱氨酸。鸡cdr-h3中的多样性来自基因转换/体细胞超突变,而不是重排过程本身。
本公开内容提供了产生人抗体的转基因鸡。
概述
本公开内容尤其提供了具有修饰的免疫球蛋白重链免疫球蛋白基因座的转基因鸡。修饰的基因座没有内源性的v-d-j区,而是具有人vh区段、人d簇、人j区段和基于人vh序列的多个上游假基因。修饰的igh基因座在鸡中经历v(d)j重组,发生上游假基因和人vh区段之间的基因转换,并且鸡产生具有多样化的免疫球蛋白重链(包括长度不同的cdr3)的抗体。
与设计用于产生重组抗体的其他转基因鸡相比,本发明的转基因鸡可以具有某些优点。例如,本发明的转基因鸡的igh基因座缺少内源性的v-d-j区,从而防止了人序列在v(d)j重组期间被去除(例如,通过鸡d和人j之间的重组)。此外,在小鼠中,v-d-j区包含对于生育能力必不可少的基因(即,adam6a和adam6b基因,参见例如,marcello等人,j.biol.chem.2011286:13060-70以及us8,697,940)。目前尚不清楚鸡基因组中内源性v-d-j区的完整序列。如此,尚不清楚鸡基因组中的内源性v-d-j区是否包含必需基因,或者是否可以制备包含缺少整个连续内源性鸡v-d-j区的igh基因座的鸡。此外,转基因鸡产生具有多样化的免疫球蛋白重链的抗体,其中v(d)j重组、基因转换和体细胞超突变的组合促成多样化。最后,因为(a)重链cdr3中的大量多样性是通过v(d)j重组实现的(与单独的基因转换相反),和(b)鸡中末端转移酶的缺乏将cdr的长度限制在d基因所编码的长度,所以鸡产生的抗体要短得多,并且可以认为是比鸡抗体(其由于d-d连接(其是在鸡中所选择的)而通常具有更长的重链cdr3)更加“人”的。
在野生型鸡中,较长的cdr-h3通过串联d-d连接产生,也可通过基因转换(其可用于插入序列、缺失序列以及仅改变序列)产生。因此,即使没有末端转移酶,cdr-h3仍然可以很长。在目前的鸡中,cdr-h3比野生型鸡和人的cdr-h3短,并且比通过在没有回切的情况下简单地连接v、d和j会出现的16-23个氨基酸的理论范围短,这表明目前的鸡中没有d-d连接或其他产生长cdr的机制。将从本发明的鸡制造的抗体与从人产生的抗体进行比较,cdr-h3在本发明的鸡中更短(平均长度=12,而在人中为15-16)。
本发明还提供了转基因鸡的产生方法和使用方法,以及由鸡产生的抗体组合物。
附图的简要说明
当结合附图阅读时,根据以下详细描述可以最好地理解本发明的一些方面。要强调的是,根据惯例,附图的各个特征未按比例绘制。实际上,为了清楚起见,各个特征的尺寸被任意地扩大或缩小。附图部分中包括了以下附图。
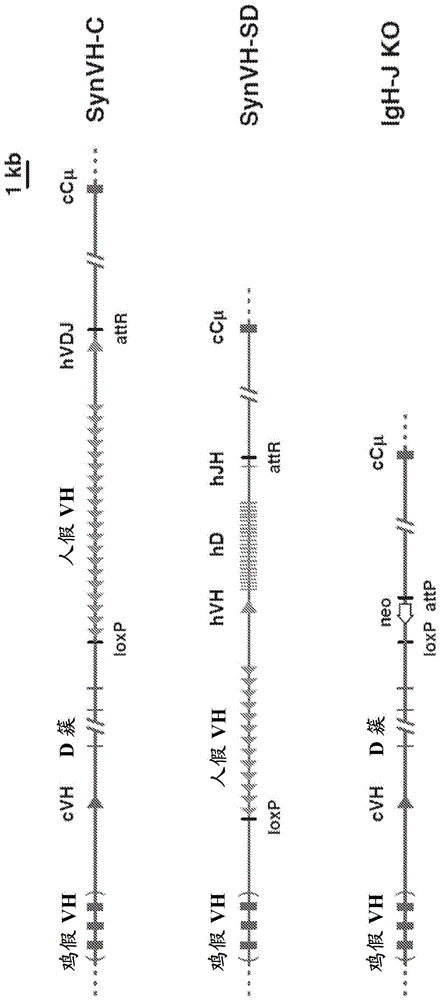
图1提供了鸡中预先重排的人vh区段和正在重排的人vh区段的图。synvh-c(预先重排的)和synvh-sd(正在重排的)转基因和重链敲除(igh-jko)的比例图。人序列以红色显示,鸡以蓝色显示。最上面一行,synvh-c转基因由与上游的人假基因阵列预先重组的人v区(hvdj)组成。人v区剪接至下游鸡恒定区(仅显示cmu)。鸡种系v和d基因以及假基因阵列位于人v基因的上游。未显示鸡假基因的精确作图(如括号所示),但到功能性鸡v的距离是准确的。显示了来自插入事件的剩余loxp和attr位点。中间一行,synvh-sd转基因包含单个人种系vh3-23基因、单个jh6基因和24个独特的人d。所有间插序列和重组位点均来自鸡重链基因座(以蓝色显示)。人种系v基因的上游是一系列基于人的假基因。上游鸡种系v和d基因被缺失,但是鸡假基因仍然存在。最下面的一行,鸡重链敲除的结构(21)。在这项研究中,转基因鸡的基因型是synvh-c或sd“敲入”/igh敲除。单个鸡jh基因被无启动子的neo基因替换。与neo基因相邻的attp位点是在其中插入synvh-c和synvh-sd构建体的位置,然后进行cre-lox重组以去除选择标记和质粒主链序列。
图2,a-b提供了在转基因鸡中观察到的信号肽变化的图以及种系信号肽序列的比对。synvh-sd信号肽的疏水性比synvh-c的疏水性小,并且经过改变以变得更加疏水。a.在转基因鸡中观察到的信号肽变化的图。通过将鸡信号肽外显子剪接至人v区外显子,或通过v区外显子中该部分的基因转换,可以将信号肽改变为鸡序列。人信号肽显示为黄色,鸡显示为橙色。显示了每种类型的信号肽的平均疏水性(kyte-doolittle,未进行标准化)。b.来自wt鸡vh区段、synvh-c和synvh-sd的种系信号肽序列的比对。v区外显子中编码的四个氨基酸为红色。将synvh-sd中的经常被突变为疏水残基的赖氨酸残基框示出来。平均疏水性标示在右侧。
图3提供了cdr-h3的长度。cdr-h3长度显示出在synvh-sd中更广泛的分布。基于imgt位置105-117计算来自所有序列(“未选择”,上方两个图)的cdr-h3长度,并将其基于具有清晰cdr-h3的所有序列(对于synvh-c,n=3,099,355,对于synvh-sd,n=1,050,398)中每个长度的频率绘制图。误差棒表示禽类之间的差异;synvh-c,n=6只禽类,synvh-sd,n=3只禽类。给出了平均值和sd。下方两个图显示了pgrn特异性mab的cdr-h3长度。
图4提供了synvh-c和synvh-sd氨基酸频率。对于大多数氨基酸,cdr-h3中的synvh-c和synvh-sd氨基酸频率相似。在不由v或j基因促成的位置(107-109或111)中,在cdr-h3的总氨基酸含量中每个氨基酸的频率。来自每只禽类的前1000个独特序列的12、13、14和15个残基的特定cdr-h3长度用于计算频率,然后将其平均。误差棒表示禽类之间的差异;synvh-c,n=6只禽类,synvh-sd,n=3只禽类。
图5描绘了synvh-c和synvh-sd中的cdr-h3中的序列多样性。cdr-h3中的序列多样性在synvh-c和synvh-sd中相似。对于synvh-c和synvh-sd的12个残基(顶部两个图)和15个残基(底部两个图)的特定长度,显示了cdr-h3imgt位置105-117的每个位置的香农熵和氨基酸分布。序列是从来自每只禽类的前1000个序列中得出的;synvh-c,6只禽类,synvh-sd,3只禽类。
图6描绘了synvh-c和synvh-sd中的cdr-h3的疏水性。cdr-h3的疏水性在synvh-c和synvh-sd中相似。对于来自每只禽类的前1000个序列,计算基于长度为12-15个残基的cdr-h3(imgt位置105-117)的标准化kyte-doolittle量表(34,35)的平均疏水性。图中显示了每个疏水性值的频率(分组到0.1的增量中)。
图7描绘了ngs数据中mab序列的频率。ngs数据中mab序列的频率显示提供gem筛选鉴定出的许多mab在脾脏群体中很少见。在来自每只禽的所有测序数据中,发现了在gem筛选中鉴定出的每种抗原特异性mab序列的次数。如所示的,mab按禽类和转基因synvh-c或synvh-sd分组。在曲线下方标示了来自每只禽类的mab数量。在整个数据集中被测序5次或更少次的mab以红色显示,表明在gem筛选中发现了罕见的mab。
图8描绘了人设计的假基因与每个构建体中的功能性v序列的比对。对于synvh-c,可从nihest数据库中查询人vh3-23基因获得不同的cdr-h1、2和3序列。种系vh3-23框架序列包含在一些假基因中,而其他的则与功能性vh序列(huvh,最上面的一行)(其是相对于种系具有9个变化的体细胞衍生的序列)匹配。对于synvh-sd,cdr-h1和2衍生自人vh3家族成员,并置于vh3-23框架支架上。不包括特定的cdr-h3序列;显示的序列是位于假基因之间的间隔区。(huvh示于seqidno:6;synvh48示于seqidno:7;synvh49示于seqidno:8;synvh38示于seqidno:9;synvh39示于seqidno:10;synvh40示于seqidno:11;synvh41示于seqidno:12;synvh42示于seqidno:13;synvh43示于seqidno:14;synvh44示于seqidno:15;synvh45示于seqidno:16;synvh46示于seqidno:17;synvh47示于seqidno:18;synvh50示于seqidno:19;synvh51示于seqidno:20;synvh52示于seqidno:21;synvh53示于seqidno:22;synvh54示于seqidno:23;synvh55示于seqidno:24;synvh56示于seqidno:25;synvh57示于seqidno:26;huvdj示于seqidno:27;synvh58示于seqidno:28;synvh59示于seqidno:29;synvh60示于seqidno:30;synvh61示于seqidno:31;synvh62示于seqidno:32;synvh63示于seqidno:33;synvh64示于seqidno:34;synvh65示于seqidno:35;synvh70示于seqidno:36;synvh71示于seqidno:37;synvh72示于seqidno:38;synvh73示于seqidno:39;synvh74示于seqidno:40)
图9描绘了来自synvh-c和synvh-sd的所有比对的v区序列的香农熵。包括了来自每只禽类的前1000个序列。标示了cdr名称(imgt)。
图10显示了crispr靶向的策略。a.用于crispr靶向的pgc第472-138行中存在的鸡igh基因座的图。igh基因座在d簇和恒定区(仅显示cμ)之间包含先前获得的jh基因区段的敲除(jh-ko),其被替换为选择标记盒。将grna1至4设计为靶向单个功能性vh区上游的区域(用箭头指示),将grna5设计为靶向egfp基因。b.用含有对egfp特异的cas9或cas9/grna5的构建体瞬时转染第472-138行的pgc。培养9天后,通过流式细胞术分析细胞的绿色荧光损失。
图11显示了pgc中crispr介导的ighko6b靶向。a.igh基因座的详细图。在上方显示了用于设计grna的ighko6b中5'和3'同源区之间的122bp序列。grna1-4的位置在序列上方用蓝线表示,前间隔区序列相邻基序(pam)用红线表示。修复载体ighko6b(下图)包含黄色的5'和3'同源区(hr)、单个loxp位点(蓝色箭头)和潮霉素选择盒(橙色)。以黑色箭头显示用于5'和3'靶向测定的引物结合位点的位置。jh-ko中的下游选择标记由flox化(floxed)的egfp(绿色框)和puro基因(蓝色框)以及方向相反的无启动子neo基因(粉红色框)组成。loxp位点是蓝色箭头。b.对通过将4种不同grna与cas9和ighko6b一起共转染到472-138细胞中获得的独立非克隆细胞群体进行5'靶向测定。对于每个grna转染,分析了3个潮霉素抗性群体。阳性对照(+)是dt40细胞系,其含有功能性v区的敲除[22],而阴性对照(-)是亲本ighko6b质粒。c.对用grna2获得的9个独立克隆(有12个克隆,但克隆4、7和12的生长较慢,目前未进行测试)进行5'和3'靶向测定。带强度的变化可能是由于模板gdna量的变化,因为收获的细胞数量未标准化。阴性对照(-)是来自jh-ko转基因禽类的基因组dna,阳性对照(+)是来自b中的grna2实验的细胞库(g2)。nt:无模板对照。d.对通过将1783–10嵌合细胞系培育到野生型获得的egfp+禽类进行相同的5'和3'靶向测定。
图12显示了crispr靶向的loxp位点的cre重组。a.cre重组前后的靶向的igh基因座的示意图。crispr靶向的loxp位点上游的正向引物与jh-ko盒中loxp位点下游的两种不同的反向引物一起使用。在非重组等位基因中,正向和反向引物在染色体上相隔约28kb。cre重组后,保留了单个loxp位点和无启动子的neo基因,并且引物相距1.6或2kb,其可轻松扩增。b.重组细胞的pcr。cre+:来自用cre转染的1783–9细胞的gdna模板;cre-,亲本1783–9细胞;jh-ko,来自杂合jh-ko禽类的gdna;ntc,无模板对照。
定义
短语“转基因鸡”是指包含含有外源核酸(即非鸡天然的重组核酸)的细胞的动物。外源核酸应存在于鸡的所有细胞中,某些单倍体生殖细胞可能除外。外源核酸分子被称为“转基因”,并且可以包含不是来自于鸡的一个或多个基因等。转基因鸡能够在其种系中稳定地传递外源核酸。
术语“内含子”是指在大多数真核生物的许多基因序列中间发现的dna序列。这些内含子序列被转录,但在mrna被翻译成蛋白质之前已从前体mrna转录物中去除。内含子去除的此过程是通过将内含子两侧的序列(外显子)剪接在一起而发生的。
术语“可操作地连接”是指单个核酸片段上的核酸序列的缔合,使得一个核酸序列的功能受到另一个核酸序列的影响。例如,当启动子能够影响编码序列的表达时(即,该编码序列在该启动子的转录控制下),该启动子与该编码序列可操作地连接。类似地,当内含子与编码序列可操作地连接时,该内含子从mrna中剪接出来以提供编码序列的表达。在基因转换的上下文下,如果一个序列可以通过基因转换将序列“施予”给另一序列,则两个核酸序列可操作地连接。如果两个序列未连接,因为一个序列可以通过基因转换将序列施予给另一个序列,则施予序列可以位于另一个序列的上游或下游,并且两个序列可以彼此相邻,即,没有其他介于中间的基因。“未连接”是指相关的遗传元件彼此之间不紧密关联,并且其一个的功能不影响另一个。
参照转录方向使用术语“上游”和“下游”。
术语“假基因”用于描述包含开放阅读框的未转录核酸区域,所述开放阅读框可以包含或可以不包含起始密码子和/或终止密码子。氨基酸序列可以在开放阅读框的核苷酸序列可以经由计算机模拟翻译以产生氨基酸序列的意义上由假基因“编码”。在重链和轻链免疫球蛋白基因座的情况下,假基因不包含启动子区、重组信号序列或前导序列。
术语“纯合的”表示相同的等位基因位于同源染色体上的相同基因座上。相反,“杂合的”表示不同的等位基因位于同源染色体的相同基因座上。转基因动物对于转基因可以是纯合的或杂合的。
关于基因,术语“内源性的”表示该基因是细胞天然的,即该基因存在于未修饰的细胞的基因组中的特定基因座上。内源性基因可以是野生型细胞中存在于该基因座上的野生型基因(自然界中发现的)。如果内源性基因存在于基因组中与野生型基因相同的基因座上,则它可以是修饰的内源性基因。这种修饰的内源性基因的例子是其中插入了外源核酸的基因。内源性基因可能存在于核基因组、线粒体基因组等中。
术语“构建体”是指重组核酸,通常是重组dna,其已出于表达特定核苷酸序列的目的而产生,或将用于构建其他重组核苷酸序列。构建体可以存在于载体或基因组中。
术语“重组”是指不在宿主细胞中天然存在的多核苷酸或多肽。重组分子可以包含以非天然存在的方式连接在一起的两个或更多个天然存在的序列。重组细胞包含重组多核苷酸或多肽。如果细胞接受重组核酸,则该核酸对细胞是“外源的”。
术语“选择标记”是指能够在宿主中表达的蛋白质,其允许容易地选择含有引入的核酸或载体的那些宿主。选择标记的例子包括但不限于赋予抗微生物剂抗性的蛋白质(例如潮霉素、博来霉素或氯霉素),赋予宿主细胞代谢优势例如营养优势的蛋白质,以及赋予细胞功能或表型优势(例如细胞分裂)的蛋白质。
如本文所用,术语“表达”是指基于基因的核酸序列产生多肽的过程。该过程包括转录和翻译。
在将核酸序列插入细胞中的上下文中的术语“引入”是指“转染”或“转化”或“转导”,并且包括提及将核酸序列掺入到真核或原核细胞中,其中核酸序列可以掺入到细胞的基因组(例如,染色体、质粒、质体或线粒体dna)中、转化为自主复制子或瞬时表达(例如,转染的mrna)。
在用一个遗传基因座替换另一个遗传基因座的上下文中,术语“替换”是指单步方案或多步方案。
术语“编码序列”是指当置于适当调节元件的控制下时编码一部分的蛋白质的核酸序列。本文所用的编码序列可以具有连续的orf,可以是orf的一部分,或者可以具有因内含子或非编码序列的存在而中断的orf。假基因可以包含未转录的编码序列。
术语“在相反的方向上”是指在不同链上的编码序列。例如,如果将转录区描述为在与假基因相反的方向上,则由转录区编码的氨基酸序列由上链或下链编码,而由假基因编码的氨基酸序列由相对于转录区的另一条链编码。
术语“抗体”和“免疫球蛋白”在本文可互换使用。这些术语是本领域技术人员众所周知的,并且是指由一种或多种特异性结合抗原的多肽组成的蛋白质。一种形式的抗体构成抗体的基本结构单元。这种形式是四聚体并由两对相同的抗体链组成,每对具有一条轻链和一条重链。在每对中,轻链和重链可变区共同负责与抗原的结合,恒定区负责抗体效应子功能。
公认的免疫球蛋白多肽包含κ和λ轻链以及α、γ(igg1、igg2、igg3、igg4)、δ、ε和μ重链或其他物种中的等同物。全长免疫球蛋白“轻链”(约25kda或约214个氨基酸)在nh2末端包含约110个氨基酸的可变区,而在cooh末端包含κ或λ恒定区。全长免疫球蛋白“重链”(约50kda或约446个氨基酸)类似地包含可变区(约116个氨基酸)和上述重链恒定区之一,例如γ(约330个氨基酸)。
术语“抗体”和“免疫球蛋白”包括任何同种型的抗体或免疫球蛋白、保留与抗原的特异性结合的抗体的片段包括但不限于fab、fv、scfv和fd片段、嵌合抗体、人源化抗体、单链抗体以及包含抗体的抗原结合部分和非抗体蛋白的融合蛋白。可以用例如放射性同位素、产生可检测产物的酶、荧光蛋白等可检测地标记抗体。抗体可以进一步与其他部分(例如特异性结合对的成员,例如生物素(生物素-亲和素特异性结合对的成员)等)缀合。抗体也可以结合到固体支持物上,固体支持物包括但不限于聚苯乙烯板或珠等。该术语还包括fab'、fv、f(ab’)2和/或保持与抗原的特异性结合的其他抗体片段,以及单克隆抗体和多克隆抗体。
抗体可以以多种其他形式存在,包括例如fv、fab和f(ab’)2,以及双功能(即双特异性)杂合抗体(例如,lanzavecchia等人,eur.j.immunol.17,105(1987))和单链(例如,huston等人,proc.natl.acad.sci.u.s.a.,85,5879-5883(1988)和bird等人,science,242,423-426(1988),其通过引用并入本文)。(一般参见hood等人,"immunology",benjamin,n.y.,2nded.(1984),和hunkapillerandhood,nature,323,15-16(1986))。
免疫球蛋白轻链或重链可变区由“框架”区(fr)间插三个高变区(也称为“互补决定区”或“cdr”)组成。已经精确地定义了框架区和cdr的范围(参见lefranc等人,imgt,theinternationalimmunogeneticsinformationsystem.nucleicacidsres.2009vol.37(databaseissue):d1006-12.epub2008oct31;参见全球网站imgt.org,下文称为“imgt系统”)。本文讨论的所有抗体氨基酸序列的编号均符合imgt系统。不同轻链或重链的框架区的序列在一个物种内是相对保守的。抗体的框架区(即轻链和重链组分的组合的框架区)用于定位和排列cdr。cdr主要负责与抗原表位的结合。
嵌合抗体是其中已通常通过遗传工程从属于不同物种的抗体可变区和恒定区基因构建轻链和重链基因的抗体。例如,可将来自鸡或兔单克隆抗体的基因的可变区段连接至人恒定区段,例如γ1和γ3。治疗性嵌合抗体的例子是包含来自鸡或兔抗体的可变或抗原结合结构域和来自人抗体的恒定或效应结构域的杂合蛋白(例如,由atcc保藏号crl9688的细胞制备的抗tac嵌合抗体),尽管可以使用其他哺乳动物物种。
如本文所用,术语“人框架”是指具有与人抗体的氨基酸序列(例如抗体的人种系序列的氨基酸序列)至少90%相同,例如至少95%,至少98%或至少99%相同的氨基酸序列的框架。在某些情况下,人框架可以是完整的人框架,在这种情况下,该框架的氨基酸序列与人抗体例如种系抗体的氨基酸序列相同。
如本文所用,术语“人源化抗体”或“人源化免疫球蛋白”是指包含已被来自人抗体的相应位置的氨基酸替换的一个或多个氨基酸(例如在框架区、恒定区或cdr中)的非人抗体。通常,与相同抗体的非人源化形式相比,预期人源化抗体在人宿主中产生降低的免疫反应。
应当理解,通过本发明的方法设计和生产的人源化抗体可以具有另外的保守氨基酸替换,其对抗原结合或其他抗体功能基本上没有影响。保守替换是指预期的组合,例如来自以下的那些组:gly、ala;val、ile、leu;asp、glu;asn、gln;ser、thr;lys、arg;和phe、tyr。不存在于同一组中的氨基酸是“实质上不同”的氨基酸。
术语“特异性结合”是指抗体优先结合存在于不同分析物的均匀混合物中的特定分析物的能力。在某些实施方案中,特异性结合相互作用将在样品中期望的和非期望的分析物之间进行区分,在一些实施方案中,区分超过约10至100倍或更多(例如,超过约1000倍或10,000倍)。
在某些实施方案中,当抗体和分析物在抗体/分析物复合物中特异性结合时,抗体和分析物之间的亲和力的特征在于小于10-6m、小于10-7m、小于10-8m、小于10-9m、小于10-9m、小于10-11m或小于约10-12m或更小的kd(解离常数)。
抗体重或轻链的“可变区”是包含cdr1、cdr2和cd3以及框架区的链的n端成熟结构域。抗体的重链和轻链均包含可变结构域。所有结构域、cdr和残基编号均基于序列比对和结构知识进行分配。框架和cdr残基的鉴定和编号由imgt系统定义。
vh是抗体重链的可变结构域。vl是抗体轻链的可变结构域。
如本文所用,当在分离的抗体的上下文中使用时,术语“分离的”是指不含至少60%、至少75%、至少90%、至少95%、至少98%和甚至至少99%的在纯化前与抗体缔合的其他成分。
本文所用的术语“治疗”、“医治”和类似术语是指对哺乳动物尤其是例如人或小鼠的任何疾病或病况的任何治疗,并且包括:a)防止疾病、病况或疾病或病况的症状在可能易患该疾病但尚未被诊断为患有该疾病的受试者中的发生;b)抑制疾病、病况或疾病或病况的症状,例如阻止其发展和/或延迟其在患者中的发作或表现;和/或c)减轻疾病、病况或疾病或病况的症状,例如引起病况或疾病和/或其症状的消退。
术语“受试者”、“宿主”、“患者”和“个体”在本文中可互换使用,是指希望对其进行诊断或治疗的任何哺乳动物受试者,特别是人。其他受试者可以包括牛、狗、猫、豚鼠、兔子、大鼠、小鼠、马等。
“天然”抗体是其中抗体的重和轻链免疫球蛋白已经由多细胞生物体的免疫系统天然选择的抗体,与例如由例如噬菌体展示产生的非天然配对的抗体相反。这样,某些抗体不包含任何病毒(例如噬菌体m13)衍生的序列。脾脏、淋巴结和骨髓是在动物体内产生天然抗体的组织的例子。
在将核酸序列插入细胞中的上下文中的术语“引入”是指“转染”或“转化”或“转导”,并且包括提及将核酸序列掺入到真核或原核细胞中,其中所述核酸序列可以瞬时存在于细胞中或可以掺入细胞的基因组(例如染色体、质粒、质体或线粒体dna)中,转化为自主复制子。
术语“多个”是指至少2个、至少5个、至少10个、至少20个、至少50个、至少100个、至少200个、至少500个、至少1000个、至少2000个、至少5000个或至少10,000个或至少50,000个或更多个。在某些情况下,多个包括至少10个至50个。在其他实施方案中,多个可以是至少50个至1,000个。
进一步的定义可以在本公开内容的其他地方。
示例实施方案的描述
本发明提供了具有修饰的免疫球蛋白重链免疫球蛋白基因座的转基因鸡。如上所述,并且如将在下面更详细地描述的,修饰的基因座没有内源性的v-d-j区,而是具有人vh区段、人d簇、人j区段和基于人vh序列的多个上游假基因序列。修饰的igh基因座在鸡中经历v(d)j重组,发生上游假基因和人vh区段之间的基因转换,并且鸡产生具有多样化免疫球蛋白重链的抗体。
在进一步描述本发明之前,应当理解,本发明不限于所描述的特定实施方案,并且因此当然可以变化。还应理解,本文中使用的术语仅出于描述特定实施方案的目的,而无意于限制本发明,因为本发明的范围仅由所附权利要求书限制。
在提供数值范围的情况下,应理解的是,介于该范围的上限和下限之间的每个中间值(达到下限单位的十分之一,除非上下文另有明确规定)以及所述范围中的任何其他陈述的值或中间值包括在本发明内。
除非另有定义,否则本文使用的所有技术和科学术语具有与本发明所属领域的普通技术人员通常所理解的相同含义。尽管类似于或等同于本文描述的那些方法和材料的任何方法和材料都可以用于本发明的实践或测试,但是现在描述优选的方法和材料。本文提及的所有出版物通过引用并入本文,以公开和描述与引用出版物有关的方法和/或材料。
必须注意的是,如本文和所附权利要求书中所使用的,单数形式的“一个(a)”、“一种(an)”和“该(the)”包括复数对象,除非上下文另外明确指出。因此,例如,对“一个细胞”的提及包括多个细胞,而对“候选试剂”的提及包括提及本领域技术人员已知的一种或多种候选试剂及其等同物,等等。还应注意,权利要求书可以被草拟为排除任何可选要素。这样,该陈述旨在作为与权利要求要素的叙述结合使用诸如“唯一”、“仅”等排他性术语或使用“否定”限制的先行基础。
提供本文讨论的出版物仅是针对其在本申请的提交日期之前的公开内容。本文中的任何内容均不得解释为承认本发明不是早于利用在先发明的此类出版物。此外,提供的公开日期可能与实际公开日期有所不同,实际公开日期可能需要独立确认。
本说明书中引用的所有出版物和专利均通过引用并入本文,就好像每个单独的出版物或专利均被具体地和单独地指出通过引用并入本文一样,并且其通过引用公开内容而并入本文并描述与引用的出版物相关的方法和/或材料。任何出版物的引用均是针对其在申请日之前的公开内容,并且不应被解释为承认本发明不是早于利用在先发明的此类出版物。此外,提供的公开日期可能与实际公开日期有所不同,实际公开日期可能需要独立确认。
对于本领域技术人员而言,在阅读本公开内容后将明显的是,本文描述和示出的每个单独的实施方案具有离散的组分和特征,其可以容易地与其他几个实施方案中的任何一个的特征分离或组合在一起,而不会脱离本发明的范围或精神。任何叙述的方法可以以所述的事件的顺序或以逻辑上可能的任何其他顺序执行。
转基因鸡及其制备方法
提供了包含基因组的转基因鸡,所述基因组包含修饰的内源性免疫球蛋白重链(igh)基因座。该修饰的基因座在图1中示意性地图示为“synvh-sd”。如所示的,该修饰的基因座缺少整个连续内源性鸡v-d-j区。该基因座的该序列目前未知,但认为其长度为10-12kb(例如约11kb)或至少15kb(例如长度为15kb至25kb或约20kb)。基于以下实施例部分中描述的结果,与其他动物(例如小鼠)不同,野生型基因座不含任何必需基因(除了内源性鸡v-d-j序列)。去除内源性鸡v-d-j区可防止v(d)j与下文所述的v-d-j人序列重组。这样的重组事件将去除很多基因座,从而使其失活。去除内源性鸡v-d-j区可防止这些事件的发生。
除了缺少整个连续内源性鸡v-d-j区外,基因座还以可操作的连接包含:(i)免疫球蛋白重链基因启动子,例如鸡免疫球蛋白重链基因启动子;(ii)人种系vh区段,其中所述人种系vh区段包含可变结构域的编码序列,所述可变结构域包含fr1、cdr1、fr2、cdr2和fr3。在本领域中,vh区段有时被称为“种系vh基因”,“功能性vh片段”或“种系vh序列”。除假基因外,据信在单倍体人基因组中有超过40个种系vh区段(参见例如kohsaka等人,j.clin.invest.199698:2794-800),其可以分为至少七个家族(参见例如,schroeder等人,internationalimmunology,2:41-501989)。在一些实施方案中,人种系vh区段可以例如来自vh3家族、vh1家族或vh4家族。这样的区段缺少重链cdr3和fr4编码序列。
如图1所示,在人种系vh区段的下游,修饰的基因座还包含(iii)人d簇(即,例如10-27个人多样性(d)基因区段的阵列,其可以由鸡基因组中的序列(即,间插序列)以及单个人连接(joining)(j)区段(例如,来自人种系的6个j区段之一,参见例如li等人,blood2004103:4602–4409)。在某些实施方案中,d区段中半胱氨酸的任何密码子可以已突变为编码另一个氨基酸,例如酪氨酸或色氨酸,以使由鸡产生的抗体中的二硫键(特别是在重链cdr3区中)最少化。可变(v)、多样性(d)和连接(j)基因区段两侧的重组信号序列(rss)可以来自鸡,但是鸡重组酶应该识别人rrs,因为这些序列几乎是相同的。
在j区段的下游,基因座包含多个序列,其编码可能对鸡是内源性的恒定区。该基因座包含将j区段的转录物的3'末端连接至一个拷贝的恒定区编码序列的5'末端的内含子。如所示的,修饰的基因座在人种系vh区段上游还包含多个假基因(例如,至少10个或10至30个假基因),其中假基因具有fr1-cdr1-fr2-cdr2-fr3的结构,其中:(i)假基因中的fr1、fr2和fr3区段(即“编码”抗体的重链fr1、fr2和fr3序列的序列)各自具有与人种系vh区段中的相应区段fr1、fr2和fr3区段基本上相同的(即具有与其至少95%相同或同一的序列)序列,以及(ii)cdr1和cdr2序列,其可以编码由与人种系vh区段处于同一家族的不同人vh区段编码的cdr1和cdr2。例如,如果人种系vh区段是vh3家族的成员,则假基因将包含来自该区段的fr1、fr2和fr3序列,以及来自vh3家族中其他人种系vh区段的cdr1/cdr2序列。类似地,如果人种系vh区段是vh1家族的成员,则假基因将包含来自该区段的fr1、fr2和fr3序列,以及来自vh1家族中其他人种系vh区段的cdr1/cdr2序列等。在一些实施方案中,假基因处于与人种系vh区段相反的方向。人种系vh区段可以选自以下序列:vh1-18、vh1-2、vh1-24、vh1-3、vh1-45、vh1-46、vh1-58、vh1-69、vh1-8、vh2-26、vh2-5、vh2-70、vh3-11、vh3-13、vh3-15、vh3-16、vh3-20、vh3-21、vh3-23、vh3-30、vh3-33、vh3-35、vh3-38、vh3-43、vh3-48、vh3-49、vh3-53、vh3-64、vh3-66、vh3-7、vh3-72、vh3-73、vh3-74、vh3-9、vh4-28、vh4-31、vh4-34、vh4-39、vh4-4、vh4-59、vh4-61、vh5-51、vh6-1和vh7-81。有关不同种系序列的描述,参见pctwo2005/005604。
尽管如上所述,人种系vh区段和假基因的fw区段可以彼此相同,而人种系vh区段和假基因的cdr区段可以不同,从而允许在假基因和种系序列的cdr区段之间进行基因转换。此外,cdr的长度可以变化。在某些实施方案中,重链cdr1的长度可以在6至12个氨基酸残基的范围内,重链cdr2的长度可以在4至12个氨基酸残基的范围内,重链cdr3的长度可以在3至25个氨基酸残基的范围内,尽管还预期了具有在这些范围之外的长度的cdr的抗体。
在上述和图1所示的构型中,修饰的igh基因座在鸡中经历v(d)j重组,多个人种系vh假基因在v(d)j重组后通过基因转换将核苷酸序列施予给人种系vh区段(至少在人种系vh区段中的cdr1和cdr2编码序列上);鸡产生包含多样化免疫球蛋白重链的抗体。鸡产生的抗体在重链cdr1、cdr2和cdr3区中多样化,并且重链cdr3的中位长度为11-13,例如约12个残基。cdr3区由通过v(d)j重组和体细胞超突变选择的d区段多样化。鸡中tdt的缺乏将重链cdr3的长度限制为由d基因编码的长度和/或可通过基因转换插入的长度,因此,相对于野生型鸡,重链cdr3相对较短并观察到人抗体和少量d-d连接。
鸡对于修饰的igh基因座可以是纯合的,或者对于修饰的igh基因座可以是杂合的。如果该鸡对于修饰的igh基因座是杂合的,则可以敲除同源染色体上的内源性igh基因座。例如,在一些实施方案中,同源染色体上的内源性igh基因座可缺少其j区或整个连续内源性鸡v-d-j区。鸡对于野生型igh基因座也可以是杂合的。
在某些实施方案中,转基因鸡还可表达与鸡轻链恒定区连接的免疫球蛋白轻链v区或与人轻链恒定区连接的人v区(例如人免疫球蛋白轻链),从而使由鸡产生的抗体是具有人v区和鸡恒定区的完全人或嵌合轻链。
在某些实施方案中,由主题转基因动物产生的抗体可以包含鸡恒定结构域和人可变结构域。由于在这些实施方案中可以使用内源性恒定区,所以抗体仍可以经历类别转换和亲和力成熟,这允许动物经历正常的免疫系统发育并发起正常的免疫应答。在特定的实施方案中,转基因鸡在重链基因座中具有三个内源性恒定区,其编码igm、igy和iga。在b细胞发育的早期,b细胞表达igm。随着亲和力成熟的进行,类别转换将恒定区转换为igy或iga。igy为成年和新生鸡提供体液免疫,它们通过沉积在蛋黄中的储备液接收约200mgigy。iga主要存在于淋巴组织(例如,脾脏、peyer淋巴集结和harderian腺)和输卵管中,尽管少量转移到蛋中并随后被发育中的胚胎吸收。
如上所述,本发明还提供了在其同源染色体中的一个或两个中都缺少其整个连续内源性鸡v-d-j区的转基因鸡。本发明还提供了来自转基因鸡的b细胞。
提供了一种制备转基因鸡的方法。在某些实施方案中,该方法包括:(a)从鸡的免疫球蛋白重链(igh)基因座中去除整个连续内源性鸡v-d-j区;和(b)将包含以下的构建体插入基因座:(i)免疫球蛋白重链基因启动子;(ii)人种系vh区段,其包含可变结构域的编码序列,该可变结构域包含fr1、cdr1、fr2、cdr2和fr3;(iii)人d簇;(iv)人j区段;(vi)多个假基因,每个假基因包含:与(b)(ii)的功能性人vh区段基本上相同的fr1、fr2和fr3编码序列;以及cdr1和cdr2编码序列。步骤(a)和(b)可以按任何顺序进行。但是,非编码序列(内含子)可以以内源性构型保留,以保留其中可以包含的内源性调控元件。
一旦制备了主题转基因动物,就可以通过用抗原免疫动物而容易地获得针对抗原的抗体。多种抗原可用于免疫转基因宿主动物。此类抗原包括微生物,例如活的、减毒的或死亡的病毒和单细胞生物(例如细菌和真菌),微生物的片段或分离自微生物的抗原分子。
在一些实施方案中,该方法可包括:(a)用抗原免疫如上所述的转基因鸡;(b)从转基因鸡中获得特异性结合抗原的抗体,例如多克隆或单克隆抗体。在一些实施方案中,该方法可包括(c)使用转基因鸡的b细胞制备杂交瘤;(d)筛选所述杂交瘤以鉴定产生特异性结合抗原的抗体的杂交瘤。该方法可以进一步包括使用pcr从转基因动物的b细胞中扩增编码重链和轻链可变区的核酸,并使用所述扩增的核酸表达重组抗体。也可以使用gem测定(美国专利8030095和8415173;izquierdo等人,2014)、深度测序或任何其他b细胞讯问技术从转基因鸡中回收单克隆抗体(例如,参见abcellera网站)。
在某些实施方案中,可以用以下免疫转基因鸡以产生治疗性抗体:gd2,egf-r,cea,cd52,cd20,lym-1,cd6,补体激活受体(car),egp40,vegf,肿瘤相关糖蛋白tag-72afp(甲胎蛋白),blys(tnf和apol相关配体),ca125(癌抗原125),cea(癌胚抗原),cd2(t细胞表面抗原),cd3(与tcr相关的异源多聚体),cd4,cd11a(整合素α-l),cd14(单核细胞分化抗原),cd20,cd22(b细胞受体),cd23(低亲和力ige受体),cd25(il-2受体α链),cd30(细胞因子受体),cd33(髓样细胞表面抗原),cd40(肿瘤坏死因子受体),cd44v6(介导白细胞粘附),cd52(campath-1),cd80(cd28和ctla-4的共刺激剂),补体成分c5,ctla,egfr,嗜酸性粒细胞趋化因子(细胞因子a11),her2/neu,her3,hla-dr,hla-dr10,hlaii类,ige,gpiib/iiia(整联蛋白),整联蛋白avβ3,整联蛋白a4β1和a4β7,整联蛋白β2,ifn-γ,il-1β,il-4,il-5,il-6r(il6受体),il-12,il-15,kdr(vegfr-2),lewisy,间皮素,muc1,muc18,ncam(神经细胞粘附分子),癌胚胎纤连蛋白,pdgfβr(β血小板衍生的生长因子受体),pmsa,肾癌抗原g250,rsv,e-选择蛋白,tgfbeta1,tgfbeta2,tnfα,dr4,dr5,dr6,vap-1(血管粘附蛋白1)或vegf等。
可以以任何方便的方式在有或没有佐剂的情况下将抗原施用至转基因鸡,并且可以按照预定的时间表施用抗原。
免疫后,可以对来自免疫的转基因动物的血清进行分级分离以纯化对抗原具有特异性的药用级多克隆抗体。对于转基因禽类,还可以通过分离蛋黄来制备抗体。浓缩的、纯化的免疫球蛋白级分可以通过色谱法(亲和力、离子交换、凝胶过滤等)、用例如硫酸铵的盐、例如乙醇的有机溶剂或例如聚乙二醇的聚合物选择性沉淀来获得。
为了制备单克隆抗体,可以从免疫的转基因动物中分离产生抗体的细胞,例如脾细胞或其他细胞,并用于与经转化的细胞系细胞融合以产生杂交瘤,或通过标准克隆分子生物学技术克隆编码抗体的cdna并在经转染的细胞中表达。制备单克隆抗体的方法在本领域中是良好建立的。参见例如,欧洲专利申请0583980a1,美国专利号4,977,081,wo97/16537和ep0491057b1,其公开内容通过引用并入本文。andris-widhopf等人,jimmunolmethods242:159(2000)和burton,immunotechnology1:87(1995)已描述了从克隆的cdna分子体外生产单克隆抗体的方法,其公开内容通过引用并入本文。
抗体组合物和筛选方法
提供了抗体组合物。如上所述,抗体的重链和轻链可变结构域与动物的免疫系统天然配对。在某些情况下,此类抗体可以被宿主细胞翻译后修饰(例如,糖基化),并且可以具有转基因鸡的物种的糖基化模式和组成特征。
可以筛选由主题转基因鸡产生的抗体以鉴定感兴趣的抗体。通常,此方法包括使用上述方法产生多种产生单克隆抗体的杂合细胞,并使用一种测定或多种测定的组合来筛选多种单克隆抗体。通常,这些测定是功能性测定,并且可以分为以下几类:检测抗体的结合亲和力或特异性的测定,以及检测抗体抑制某一过程能力的测定。
被鉴定为具有与抗原特异性结合活性或抑制活性的单克隆抗体被称为感兴趣的单克隆抗体。
结合测定
在这些测定中,测试抗体特异性结合底物的能力。在抗体结合的上下文中,术语“特异性”是指抗体与特异性抗原即多肽或表位的高亲合力和/或高亲和力结合。在许多实施方案中,特异性抗原是用于免疫从中分离产生抗体的细胞的动物宿主的抗原(或抗原的片段或子部分)。特异性结合抗原或其片段的抗体比相同抗体与其他抗原的结合更强。特异性结合多肽的抗体可以能够以较弱但可检测的水平(例如,与感兴趣的多肽显示的结合的10%或更少)结合其他多肽。这样的弱结合或背景结合很容易从特异性抗体与主题多肽的结合中辨别出来,例如,通过使用适当的对照。通常,特异性抗体以10-7m或更高,例如10-8m或更高(例如10-9m、10-10m、10-11m等)的结合亲和力与抗原结合。通常,具有10-6m或更小的结合亲和力的抗体是无用的,因为它不会以使用当前使用的常规方法可检测的水平结合抗原。
通常,在进行筛选测定时,将由产生抗体的宿主细胞的文库产生的抗体样品以可以鉴定每种抗体的方式(例如,使用板编号和在板上的位置,或使用其他允许鉴定产生抗体的宿主细胞培养物的标识符)沉积在固体支持物上。
可以通过本领域已知的任何方法筛选本发明的抗体的免疫特异性结合。可以使用的免疫测定包括但不限于使用诸如蛋白质印迹、放射免疫测定、elisa(酶联免疫吸附测定)、“夹心”免疫测定、免疫沉淀测定、沉淀素反应、凝胶扩散沉淀素反应、免疫扩散测定、凝集测定、补体固定测定、免疫放射测定、荧光免疫测定和蛋白a免疫测定的技术的竞争性和非竞争性测定系统。这样的测定是常规的并且在本领域中是众所周知的(参见例如,ausubel等人,eds,1994,currentprotocolsinmolecularbiology,vol.1,johnwiley&sons,inc.,newyork,其通过引用整体并入本文)。示例性的免疫测定在下文简要描述(但不旨在作为限制)。
免疫沉淀方案通常涉及在裂解缓冲液例如补充有蛋白质磷酸酶和/或蛋白酶抑制剂(例如edta、pmsf、抑肽酶、钒酸钠)的ripa缓冲液(1%np-40或tritonx-100、1%脱氧胆酸钠、0.1%sds、0.15mnacl、0.01m磷酸钠,ph7.2,1%trasylol)中裂解细胞群,将感兴趣的抗体添加到细胞裂解物中,在4℃孵育一段时间(例如1-4小时),将蛋白a和/或蛋白g琼脂糖珠加入细胞裂解液中,在4℃孵育约一小时或更长时间,将珠在裂解缓冲液中洗涤,然后将珠重悬于sds/样品缓冲液中。感兴趣的抗体免疫沉淀特定抗原的能力可以通过例如蛋白质印迹分析来评估。本领域技术人员将了解可以被修改以增加抗体与抗原的结合并降低背景的参数(例如,用琼脂糖凝胶珠预先清除细胞裂解物)。
蛋白质印迹分析通常涉及蛋白质样品的制备,然后在聚丙烯酰胺凝胶(例如,取决于抗原的分子量8%-20%的sds-page)中对蛋白质样品进行电泳,然后从聚丙烯酰胺凝胶将分离的蛋白质样品转移至膜例如硝化纤维素、pvdf或尼龙。转移后,将膜在封闭溶液(例如含有3%bsa或脱脂牛奶的pbs)中封闭,在洗涤缓冲液(例如pbs-tween20)中洗涤,并与在封闭缓冲区中稀释的第一抗体(感兴趣的抗体)一起孵育。孵育后,将膜在洗涤缓冲液中洗涤,与缀合至酶促底物(例如辣根过氧化物酶或碱性磷酸酶)或放射性分子(例如32p或125i)的第二抗体(其识别第一抗体,例如抗人抗体)孵育,然后在进一步的洗涤后,可以检测抗原的存在。本领域技术人员将了解可以被修改以增加检测到的信号并减少背景噪声的参数。
elisa包括制备抗原,用抗原包被96孔微量滴定板的孔,将与可检测化合物例如酶促底物(例如辣根过氧化物酶或碱性磷酸酶)缀合的感兴趣的抗体添加至孔中并孵育一段时间,并检测抗原的存在。在elisa中,感兴趣的抗体不是必须与可检测的化合物缀合;相反,可以将与可检测化合物缀合的第二抗体(识别感兴趣的抗体)添加到孔中。此外,代替用抗原包被孔,可以将抗体包被至孔。在这种情况下,在将感兴趣的抗原添加到包被的孔中之后,可以添加与可检测化合物缀合的第二抗体。本领域技术人员将了解可以被修改以增加检测到的信号的参数以及本领域已知的elisa的其他变型。
抗体对抗原的结合亲和力和抗体-抗原相互作用的解离速率可以通过竞争性结合测定来确定。竞争性结合测定的一个实例是放射免疫测定,其包括在递增量的未标记抗原的存在下将标记的抗原(例如3h或125i)与感兴趣的抗体一起孵育,以及检测与标记的抗原结合的抗体。感兴趣的抗体对特定抗原的亲和力和结合解离速率可以通过scatchard图分析从数据中确定。还可以使用放射免疫测定确定与第二抗体的竞争。在这种情况下,在递增量的未标记的第二抗体的存在下,将抗原与缀合至标记的化合物(例如3h或125i)的感兴趣的抗体一起孵育。
可以使用免疫细胞化学方法在用能够表达抗原的载体或者用单独的载体使用本领域公知的技术转染的细胞(例如哺乳动物细胞,例如cho细胞)上使用免疫细胞化学方法筛选本发明的抗体。结合抗原转染的细胞但不结合仅载体转染的细胞的抗体是抗原特异性的。
然而,在某些实施方案中,测定是抗原捕获测定,并且抗体的阵列或微阵列可用于该目的。制备和使用多肽微阵列的方法是本领域已知的(参见例如美国专利6,372,483、6,352,842、6,346,416和6,242,266)。
抑制剂测定
在某些实施方案中,该测定测量抗体对第一化合物与第二化合物(例如两种生物聚合化合物)之间相互作用的特异性抑制或特异性地抑制反应(例如酶促反应)。在相互作用抑制测定中,一种相互作用底物(通常是生物聚合化合物,例如蛋白质,例如受体)可以结合到反应容器中的固体支持物上。将抗体添加到反应容器中,然后加入用于底物的可检测的结合伴侣,通常是生物聚合化合物,例如蛋白质,例如受体的放射性标记配体。洗涤容器后,可以通过确定容器中存在的可检测的结合伴侣的量来测量相互作用抑制。当与不包含抗体的对照测定相比,结合伴侣的结合减少大于约20%、大于约50%、大于约70%、大于约80%或大于约90%或95%或更多时,发生相互作用抑制。
在反应抑制测定中,酶可以结合到反应容器中的固体支持物上。通常将抗体添加到反应容器中,然后添加酶的底物。在许多实施方案中,酶和底物之间的反应产物是可检测的,并且在一定时间后,通常停止反应。在反应停止后,可以通过确定容器中存在的可检测反应产物的水平来测量反应抑制。当与不包含抗体的对照测定相比,反应速率减少大于约20%、大于约50%、大于约70%、大于约80%或大于约90%或95%或更多时,发生反应抑制。
体内测定
在某些实施方案中,单克隆抗体在体内进行测试。通常,该方法包括向疾病或病况的动物模型施用主题单克隆抗体,并确定单克隆抗体对模型动物的疾病或病况的作用。本发明的体内测定包括对照,其中合适的对照包括不存在单克隆抗体的样品。通常,多种测定混合物与不同的抗体浓度平行运行,以获得对各种浓度的差异反应。通常,这些浓度之一用作阴性对照,即处于零浓度或低于检测水平。
感兴趣的单克隆抗体是这样抗体,其在与不存在抗体的对照相比时调节即减少或增加动物模型疾病或病况的症状至少约10%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约80%、至少约90%或更高。通常,感兴趣的单克隆抗体将使得主题动物与未患有该疾病或病况的相同动物更相似。使用本发明的方法和组合物鉴定出的具有治疗价值的单克隆抗体称为“治疗性”抗体。
由于表达感兴趣的抗体的杂合细胞包含编码免疫球蛋白重链和轻链的核酸,因此,如果鉴定出表达感兴趣的单克隆抗体的宿主细胞,则可以鉴定出编码感兴趣的单克隆抗体的核酸。这样,可以通过本领域技术人员已知的多种方法来鉴定主题核酸。使用类似的方法用于鉴定使用杂交瘤技术的单克隆抗体生产(harlow等人,antibodies:alaboratorymanual,firstedition(1988)coldspringharbor,n.y.)中的宿主细胞培养物。
例如,在鉴定了感兴趣的单克隆抗体后,可以使用“查找”表来鉴定表达感兴趣的抗体的宿主细胞,该表为每个抗体样品列出了相应的宿主细胞培养物。在某些其他实施方案中,包含抗体文库样品标识符、相应的表达盒文库样品标识符和/或宿主细胞标识符的查找表可用于鉴定主题核酸。
一旦鉴定好了,就可以使用本领域技术人员熟悉的技术恢复、表征和操作感兴趣的单克隆抗体(ausubel等人,shortprotocolsinmolecularbiology,3rded.,wiley&sons,(1995和sambrook,等人,molecularcloning:alaboratorymanual,thirdedition,(2001)coldspringharbor,n.y.)。
抗体表达
本发明还提供了几种产生感兴趣的单克隆抗体的方法。通常,这些方法包括在足以产生抗体的条件下孵育包含编码感兴趣的单克隆抗体的核酸的宿主细胞。
在一些实施方案中,产生感兴趣的单克隆抗体的方法包括将鉴定的用于感兴趣的单克隆抗体的表达盒转移到合适的载体中,并将重组载体转移到宿主细胞中以提供单克隆抗体的表达。在一些实施方案中,本发明的方法包括将来自鉴定的重链和轻链的至少可变结构域编码序列转移至适于它们在免疫球蛋白重链和轻链中的表达的载体。此时可以将合适的恒定结构域编码序列和/或其他抗体结构域编码序列添加至可变结构域编码序列。这些核酸修饰还可以允许主题抗体的人源化。
主题单克隆抗体可以通过本领域已知的用于抗体合成的任何方法特别是通过重组表达技术来产生。
主题单克隆抗体或其片段、衍生物或类似物的重组表达通常需要构建包含编码该抗体的多核苷酸的表达载体。可以使用本领域技术人员众所周知的方法来构建含有抗体编码序列以及适当的转录和翻译控制信号的表达载体。这些方法包括例如体外重组dna技术和合成技术。因此,本发明提供了包含编码本发明的抗体分子的核苷酸序列的载体。
通过常规技术将表达载体转移至宿主细胞,然后培养转染的细胞以产生主题抗体。在大多数实施方案中,编码重链和轻链的载体在宿主细胞中共表达以提供整个免疫球蛋白分子的表达。
可以使用多种宿主表达载体系统来表达主题单克隆抗体。这些包括但不限于用含有抗体编码序列的重组噬菌体dna、质粒dna或粘粒dna表达载体转化的微生物,例如细菌(例如,大肠杆菌、枯草芽孢杆菌);用含有抗体编码序列的重组酵母表达载体转化的酵母(例如,酵母属、毕赤酵母属);用含有抗体编码序列的重组病毒表达载体(例如杆状病毒)感染的昆虫细胞系统;用重组病毒表达载体(例如花椰菜花叶病毒,camv;烟草花叶病毒,tmv)感染的植物细胞系统,或用含有抗体编码序列的重组质粒表达载体(例如ti质粒)转化的植物细胞系统;或具有包含来源于哺乳动物细胞基因组(例如金属硫蛋白启动子)或哺乳动物病毒(例如腺病毒后期启动子;牛痘病毒7.5k启动子)的启动子的重组表达构建体的哺乳动物细胞系统(例如cos、cho、bhk、293、3t3细胞等)。在许多实施方案中,细菌细胞例如大肠杆菌和真核细胞用于表达整个重组抗体分子。例如,哺乳动物细胞(例如中国仓鼠卵巢细胞(cho))与载体(例如来自人巨细胞病毒的主要中间早期基因启动子元件)一起是抗体的有效表达系统(foecking等人,gene45:101(1986);cockett等人,bio/technology8:2(1990))。
在细菌系统中,可以基于表达的抗体分子的预期用途选择许多表达载体。例如,当要生产大量这样的蛋白质时,为了产生抗体分子的药物组合物,可能需要指导易于纯化的融合蛋白产物的高水平表达的载体。这样的载体包括但不限于大肠杆菌表达载体pur278(ruther等人,emboj.2:1791(1983)),其中抗体编码序列可以单独地与lacz编码区同框地连接到载体中,从而产生融合蛋白;pin载体(inouye&inouye,nucleicacidsres.13:3101-3109(1985);vanheeke&schuster,j.biol.chem.24:5503-5509(1989))等。pgex载体也可用于表达外源多肽作为与谷胱甘肽s-转移酶(gst)的融合蛋白。通常,这样的融合蛋白是可溶的,并且可以通过吸附并结合到谷胱甘肽-琼脂糖珠基质上,然后在游离谷胱甘肽的存在下洗脱而容易地从裂解的细胞中纯化。pgex载体设计为包含凝血酶或因子xa蛋白酶切割位点,因此可以从gst部分释放克隆的靶基因产物。
在昆虫系统中,autographacalifomica核多角体病毒(acnpv)被用作表达抗体的载体。该病毒在草地贪夜蛾细胞中生长。可以将抗体编码序列单独克隆到病毒的非必需区(例如多角体蛋白基因)中,并置于acnpv启动子(例如多角体蛋白启动子)的控制下。
在哺乳动物宿主细胞中,许多基于病毒的表达系统可用于表达主题抗体。在将腺病毒用作表达载体的情况下,可以将感兴趣的抗体编码序列连接至腺病毒转录/翻译控制复合体,例如晚期启动子和三联体前导序列。然后可以通过体外或体内重组将该嵌合基因插入腺病毒基因组。插入病毒基因组的非必需区(例如,e1或e3区)将产生重组病毒,该重组病毒是可存活的并且能够在被感染的宿主中表达抗体分子。(例如,参见logan&shenk,proc.natl.acad.sci.usa81:355-359(1984))。可以通过包含适当的转录增强子元件\转录终止子等来增强表达的效率(参见bittner等人,methodsinenzymol.153:51-544(1987))。
为了长期、高产量地生产重组抗体,可以使用稳定的表达。例如,可以工程化稳定表达抗体分子的细胞系。可以使用免疫球蛋白表达盒和选择标记来转化宿主细胞,而不是使用包含病毒复制起点的表达载体。引入外源dna后,可使工程化细胞在富集培养基中生长1-2天,然后切换至选择性培养基。重组质粒中的选择标记赋予对选择的抗性,并允许细胞将质粒稳定整合到染色体中并生长以形成中心点,其进而可以被克隆并扩增成细胞系。这样的工程化细胞系在筛选和评估与抗体分子直接或间接相互作用的化合物时可以特别有用。
可以使用许多选择系统,包括但不限于单纯疱疹病毒胸苷激酶(wigler等人,cell11:223(1977))、次黄嘌呤-鸟嘌呤磷酸核糖基转移酶(szybalska&szybalski,proc.natl.acad.sci.usa48:202(1992))以及腺嘌呤磷酸核糖基转移酶((lowy等人,cell22:817(1980))基因可以分别在tk-、hgprt-或aprt-细胞中使用。而且,抗代谢物抗性可以用作以下基因的选择基础:dhfr,其赋予对氨甲蝶呤的抗性(wigler等人,natl.acad.sci.usa77:357(1980);o'hare等人,proc.natl.acad.sci.usa78:1527(1981));gpt,其赋予对麦考酚酸的抗性(mulligan&berg,proc.natl.acad.sci.usa78:2072(1981));neo,其赋予对氨基糖苷g-418的抗性(clinicalpharmacy12:488-505;wuandwu,biotherapy3:87-95(1991);tolstoshev,ann.rev.pharnacol.toxicol.32:573-596(1993);mulligan,science260:926-932(1993);和morganandanderson,ann.rev.biochem.62:191-217(1993);tibtech11(5):155-215(1993));以及hygro,其赋予对潮霉素的抗性(santerre等人,gene30:147(1984))。重组dna技术领域中通常已知的方法可以常规地用于选择所需的重组克隆,并且这种方法例如在ausubel等人(eds.),currentprotocolsinmolecularbiology,johnwiley&sons,ny(1993);kriegler,genetransferandexpression,alaboratorymanual,stocktonpress,ny(1990);和dracopoli等人(eds),currentprotocolsinhumangenetics,johnwiley&sons,ny(1994)的第12和13章;colberre-garapin等人,j.mol.biol.150:1(1981)中描述。
宿主细胞可以用本发明的两种表达载体共转染,第一载体编码重链来源的多肽,第二载体编码轻链来源的多肽。这两种载体可以包含不同的选择标记和复制起点,这使得重链和轻链多肽能够平等表达。或者,可以使用单一载体,其编码并且能够表达重链和轻链多肽两者。
一旦产生了本发明的抗体分子,就可以通过本领域已知的用于纯化免疫球蛋白分子的任何方法进行纯化,例如,通过色谱法(例如,离子交换,亲和力,特别是通过在蛋白质a后对特定抗原的亲和力,和大小分级柱层析)、离心、差异溶解度或通过用于纯化蛋白质的任何其他标准技术。在许多实施方案中,抗体从细胞分泌到培养基中并从培养基中收获。
应用
本发明还提供了使用本发明的至少一种抗体调节或治疗如本领域已知或如本文所述的细胞、组织、器官、动物或患者中的至少一种与抗原相关的疾病的方法,例如将治疗有效量的抗体施用至或接触细胞、组织、器官、动物或患者。本发明还提供了调节或治疗细胞、组织、器官、动物或患者中的至少一种抗原相关疾病的方法,所述疾病包括但不限于肥胖症、免疫相关疾病、心血管疾病、传染病、恶性疾病或神经系统疾病中的至少一种。
通常,通过施用有效量或剂量的至少一种抗体组合物来实现病理病况的治疗,取决于组合物中所含活性剂的比活性,所述抗体组合物的有效量或剂量总计平均为每千克患者每剂量至少约0.01至500毫克的至少一种抗体,并且优选每单次或多次施用为至少约0.1至100毫克抗体/千克患者。或者,有效血清浓度可以包括每次单次或多次施用0.1-5000ng/ml的血清浓度。合适的剂量是医学从业者已知的,并且当然将取决于特定的疾病状态、所施用的组合物的比活性以及正在接受治疗的特定患者。在某些情况下,为了达到所需的治疗量,可以有必要提供重复施用,即重复单独施用以特定的监测或计量剂量,其中重复单独施用直至达到所需的日剂量或效果。
在某些实施方案中,主题抗体还可以用于诊断中,其中抗体与可检测标记物缀合或用作与缀合至可检测标记物的第二抗体一起使用的第一抗体。可检测标记包括放射性和非放射性标记,并且是本领域技术人员众所周知的。常见的非放射性标记包括可检测的酶例如辣根过氧化物酶、碱性磷酸酶和荧光分子。荧光分子吸收一个波长的光,然后发射另一波长的光,因此可以用例如荧光显微镜观察。分光光度计、荧光显微镜、荧光板读数器和流式分选仪是众所周知的,通常用于检测通过将其共价偶联至荧光染料而变成荧光的特定分子。有用的标记的实例包括荧光染料,例如绿色荧光蛋白、绿色荧光蛋白的红移突变体、氨基香豆素乙酸(amca)、异硫氰酸荧光素(fitc)、异硫氰酸四甲基氯丹明(tritc)、texasred、cy3.0和cy5.0。
如果使用荧光标记,则该分子可用于细胞分离策略,例如荧光激活细胞分选(facs)。在荧光激活细胞分选中,将标记有荧光分子的细胞在流式细胞仪例如becton-dickinson(sanjose,calif.)facsiv细胞仪或等效仪器上进行电子分选。荧光分子是识别特定细胞表面抗原的抗体。抗体缀合至荧光标记,例如异硫氰酸荧光素(fitc)或藻红蛋白(pe)。
实施方案
实施方案1.一种转基因鸡,其中所述转基因鸡包含基因组,所述基因组包含修饰的内源性免疫球蛋白重链(igh)基因座,所述基因座:
(a)缺少整个连续内源性鸡v-d-j区;和
(b)以可操作的连接包含:
(i)免疫球蛋白重链基因启动子;
(ii)人种系vh区段,其包含可变结构域的编码序列,所述可变结构域包含fr1、cdr1、fr2、cdr2和fr3序列;
(iii)人d簇;
(iv)单个人j区段;
(v)多个编码恒定区的序列;和在(b)(ii)的人种系vh区段的上游:
(vi)结构fr1-cdr1-fr2-cdr2-fr3的多个假基因,其各自包含:
与(b)(ii)的功能性人vh区段基本上相同的fr1、fr2和fr3序列;和
在假基因之间不同的cdr1和cdr2序列,
其中:
修饰的igh基因座在鸡中经历v(d)j重组;
v(d)j重组后,多个人种系vh假基因通过基因转换将核苷酸序列施予给人种系vh区段;和
所述鸡产生包含多样化的免疫球蛋白重链的抗体。
实施方案2.实施方案1的转基因鸡,其中(b)(vi)的cdr1和cdr2序列编码与(b)(ii)的功能性人vh区段属于同一家族的不同人vh区段的cdr1和cdr2。
实施方案3.前述实施方案中任一项的转基因鸡,其中(a)的整个连续内源性鸡v-d-j区的长度为至少15kb。
实施方案4.前述实施方案中任一项的转基因鸡,其中(b)(i)的启动子是鸡免疫球蛋白重链基因启动子。
实施方案5.前述实施方案中任一项的转基因鸡,其中(b)(ii)的人种系vh区段和(vi)的cdr1和cdr2序列来自vh3家族、vh1家族或vh4家族。
实施方案6.前述实施方案中任一项的转基因鸡,其中在d簇中,半胱氨酸的任何密码子已被突变以编码另一种氨基酸。
实施方案7.实施方案6的转基因鸡,其中在d簇中,半胱氨酸的任何密码子已被突变以编码酪氨酸或色氨酸。
实施方案8.前述实施方案中任一项的转基因鸡,其中d簇中的间插序列来自鸡。
实施方案9.前述实施方案中任一项的转基因鸡,其中(b)(v)的多个假基因包含至少10个假基因。
实施方案10.前述实施方案中任一项的转基因鸡,其中(b)(v)的假基因与(b)(ii)的人种系vh区段处于相反方向。
实施方案11.前述实施方案中任一项的转基因鸡,其中编码(b)(v)的恒定区的多个序列对于所述鸡是内源性的。
实施方案12.前述实施方案中任一项的转基因鸡,其中所述修饰的免疫球蛋白重链(igh)基因座的转录物包含将编码j的序列的一个拷贝的3'端与(b)(v)的恒定区编码序列的一个拷贝的5'端连接的内含子。
实施方案13.前述实施方案中任一项的转基因鸡,其中所述鸡对于修饰的igh基因座是纯合的。
实施方案14.前述实施方案中任一项的转基因鸡,其中所述鸡对于修饰的igh基因座是杂合的,并且同源染色体上的igh基因座被敲除。
实施方案15.前述实施方案中任一项的转基因鸡,其中所述鸡对于修饰的igh基因座是杂合的,并且同源染色体上的igh基因座是野生型的。
实施方案16.实施方案14的转基因鸡,其中所述鸡对于修饰的igh基因座是杂合的,并且同源染色体上的igh基因座缺少其整个连续内源性鸡v-d-j区。
实施方案17.实施方案14的转基因鸡,其中所述鸡对于修饰的igh基因座是杂合的,并且同源染色体上的igh基因座缺少j区。
实施方案18.前述实施方案中任一项的转基因鸡,其中所述鸡产生的抗体在重链cdr1、cdr2和cdr3区中是多样化的。
实施方案19.一种转基因鸡,其在其同源染色体中的一个或两个中缺少其整个连续内源性鸡v-d-j区。
实施方案20.一种来自任何前述实施方案的转基因鸡的b细胞。
实施方案21.一种方法,包括:
(a)用抗原免疫任何前述实施方案的转基因鸡;和
(b)从所述鸡中获得与所述抗原特异性结合的抗体。
实施方案22.实施方案21的方法,其中所述抗体是多克隆的。
实施方案23.实施方案21的方法,其中所述抗体是单克隆的。
实施方案24.实施方案21至23中任一项的方法,还包括:
(c)使用所述转基因鸡的b细胞制作杂交瘤;和
(d)筛选所述杂交瘤以鉴定产生特异性结合抗原的抗体的杂交瘤。
实施方案25.实施方案21至24中任一项的方法,其进一步包括使用pcr从转基因动物的b细胞扩增编码重链和轻链可变区的核酸,并使用所述扩增的核酸表达重组抗体。
实施方案26.一种由实施方案1-19中任一项的转基因鸡产生的抗体。
实施方案27.一种方法,包括:
(a)从鸡的免疫球蛋白重链(igh)基因座中去除整个连续内源性鸡v-d-j区;和
(b)将构建体插入基因座,所述构建体包含:
(i)免疫球蛋白重链基因启动子;
(ii)人种系vh区段,其包含可变结构域的编码序列,所述可变结构域包含fr1、cdr1、fr2、cdr2和fr3序列;
(iii)人d簇;
(iv)人j区段;和
(vi)结构fr1-cdr1-fr2-cdr2-fr3的多个假基因,其各自包含:
与(b)(ii)的功能性人vh区段基本上相同的fr1、fr2和fr3序列;和
在假基因之间不同的cdr1和cdr2编码序列,其中步骤(a)和(b)以任何顺序进行。
实施例
本发明提供以下实施例以证明和进一步举例说明本发明的某些实施方案和方面,并且不应解释为限制其范围。
实施例1
产生合成synvh-sd鸡
在这项研究中,转基因鸡被工程化以产生人可变区抗体库。从药物学的角度来看,在鸡中使用单个v框架具有优势,因为可以选择具有最佳制造和可开发性特征的优选框架。可以设计转基因,使得多样性集中于cdr,同时保持种系或近种系框架序列。该策略是为了保留鸡b细胞系统用于整合多样性的优势,其使用具有上游基于人的假基因的单个人框架,而不是插入可能在鸡种不能被正确调节的大型人基因组片段(携带多个人v、d和j基因)。为了使单个人框架能够为任何潜在靶标提供治疗候选物,转基因产生的多样性水平必须足够。产生了两种转基因鸡品系:一种携带预先重排的功能性vh区,其完全依赖于基因转换/体细胞超突变以产生多样性,另一种先经历v(d)j重排以产生功能性vh区,从而可以增加cdr-h3多样性的水平,然后进行基因转换/体细胞超突变。
材料和方法
先前描述了预先重排的vh区构建体synvh-c(18)。v(d)j重排构建体synvh-sd是通过几个部分的基因合成然后进行连接制成的。v、d和j区如下组装。人种系vh3-23*01基因是使用的单个v基因,而jh6基因是单个j基因。24个非冗余人d的两侧是重组信号序列和来自鸡d基因座的间插区(这些间隔区每个为约100-200bp)。人d2家族中的半胱氨酸密码子被突变为编码酪氨酸(7个实例)或色氨酸(2个实例)。重排元件与鸡vh启动子一起克隆以驱动重链表达,并包括了用于剪接至内源性恒定区的鸡j-c内含子的一小段。人假基因包含来自人种系vh3-23基因的fr和来自vh3种系基因家族的cdr1和2。设计了13个假基因。每个假基因之间的间隔区序列来自鸡假基因区,但不包括鸡v序列本身。包括了用于插入到靶向鸡重链基因座的attp位点中的attb位点(21,22),以及用于以后与靶基因组中的loxp位点重组的loxp位点。将synvh-sd构建体转染到含重链attp的细胞中。在这些细胞中,先前已通过crispr介导的靶向在鸡vh区段的上游插入了loxp位点(22)。插入synvh-sd转基因后,通过synvh-sd转基因以与第一个loxp位点相同的方向直接在人假基因阵列的上游引入第二个loxp位点。培育至cre鸡去除了loxp位点之间的所有dna,其中包括鸡的vh和d基因以及在转染过程中使用的选择标记,从而导致图1所示的结构。对于轻链,本研究中使用的所有转基因鸡从构建体synvk-ck表达人v-κ轻链(18)。这些禽类中的轻链和重链由人可变区和鸡恒定区组成。在重链基因座上,在所有情况下转基因都是杂合的,其中另一个等位基因被敲除,得到基因型ighsynvh/ighjh-ko。在轻链基因座上,基因型始终为iglsynvk-ck/iglvjc-ko。
如(18)所述,对含有synvh-c的鸡进行免疫。按照与synvh-c禽类相似的时间表,用pgrn蛋白或dna与pgrn蛋白质的组合对synvh-sd禽类进行免疫。
通过ficoll密度离心制备脾淋巴细胞。使用rneasy(qiagen)提取脾淋巴细胞(约107个)的总rna。将10-15ng总rna用于使用引物chvh-f9(5’-caccagtcggctccgcaaccatg-3’(seqidno:1))和cigy-ngs-r(5’-gggcgatgtggggctcgc-3’(seqidno:2))的onestepahead(qiagen)校对聚合酶反应中。获得了大约450bp的扩增子,并在abm(richmond,bc,canada)进行测序。abm执行配对末端合并、聚类分析、序列与先前鉴定的mab序列的匹配以及cdr-h3长度确定。
使用dnastar软件对序列进行比对和分析。excel宏是从annemariehonegger(universityofzurich)的网站下载的,并用于计算氨基酸频率。
根据ligandpharmaceuticalsiacuc批准的方案并在iacuc委员会的监督下进行动物实验。
结果
分析了来自具有两种不同人重链转基因的鸡的人vh序列。这些转基因之一(synvh-c)包含预先重排的功能性v区(18),另一个转基因synvh-sd包含在b细胞中经历重排的种系v、d和j区段(图1)。synvh-c中的v区是通过筛选人文库获得的,并且其包含重排的vh3-23/d1/jh4区,相对于种系vh3-23基因有9个框架(fr)变化。synvh-sd构建体包含单个种系v基因vh3-23、所有人d元件和单个jh6基因。通过插入来自鸡d基因座的序列(包括高度保守的重组信号序列)分离d元件。两种构建体中的所有其他非编码序列(启动子和内含子)均来源于鸡重链基因座用于最佳的转录和转录后调控。通过基因靶向和整合酶介导的插入的组合将转基因构建体插入内源性重链基因座(18、22)。两个转基因中的人v区剪接至内源性下游鸡恒定区。
这两个转基因均含有上游的基于人的假基因,这些假基因可以参与功能性人v的基因转换(8、11)。假基因被设计为具有主要在cdr中的多样性(图8),尽管有些synvh-c假基因在框架中也有变化,这些假基因是从头设计的,并且与人基因组中的v基因假基因没有任何关系。采用了两种不同的假基因cdr设计方法。在synvh-c中,cdr来源于表达序列的人序列数据库(est)中发现的天然存在的cdr序列。所有三个cdr都包含在假基因中,并且每个假基因的3'端均包含cdr3,但不延伸到fr4中或包含标记cdr3的边界的不变的trp-118残基。在一个假基因的cdr3和下一个假基因的起始位置(fr1)之间,放置了100bp的间隔区序列。在synvh-sd中,cdr来自人种系vh3家族成员cdr1和cdr2,在假基因中没有特定的cdr3序列,因为种系v基因不含cdr3。fr3的下游放置了间隔区序列,这些间隔区序列是可以用于基因转换的多样序列,即使它们不是来源于人cdr3(图8)。插入后,在两种情况下鸡假基因阵列均仍存在于上游。在synvh-c的情况下,仍然存在正常经历vdj重排的单个鸡种系vh和d簇,但由于没有种系jh区,它们不能重组为功能性v区,并且下游的人v区已完全重排。在synvh-sd转基因中,缺失了鸡v和d基因(22),以消除鸡基因直接与人jh基因重组并替换人v和d基因的可能性。两个禽类品系的基因组中存在的唯一jh区是synvh转基因中的人jh(图1)。
通过ngs扩增子测序,从9只经免疫的禽类的脾淋巴细胞群体中对重链v区进行整体测序(免疫的细节参见下文)。逆转录后,使用5'utr中的正向引物和igy恒定区ch1结构域中的反向引物,从淋巴细胞rna中扩增vh区,并通过miseq(appliedbiologicalmaterials,canada)进行测序。扩增vh区的引物被选择以使得它们会扩增人v区或鸡v区(假如它们被表达)。在人转基因中,唯一的人序列是v区编码序列。在两个synvh构建体中,所有非编码序列(5'utr,内含子等)和恒定区编码序列均为鸡序列。将末端配对读长组装并翻译以提供理论的蛋白质序列。表1给出了每只禽类的读长、独特的核苷酸和蛋白质序列的数量。每只禽类中最常见的序列的发现次数为约6700至89,000。对于某些分析,使用每个样品中最常见的前1000个独特序列,占每个样品的总序列数据的34-62%。
表1.测序数据概述。
该表按禽类编号列出了来自6个synvh-c和3个synvh-sd禽类的样品。给出了独特的核苷酸和蛋白质序列的数量,以及被测序到两次或更多次(2x深度)的独特肽序列的数量。在最后一列中给出了由前1000个序列代表的总序列读数的比例。
v区和信号肽的使用
来自9只禽类的vh区的分析首先是确定表达的序列是否如预期的那样是完全人的,或是否包含任何鸡序列(表2)。来自synvh-sd的所有v区都是人序列,这是预期的,因为内源性鸡v区在synvh-sd间插序列的上游被缺失了。出乎意料的是,来自synvh-c转基因的序列中的约5%为鸡vh区。这些序列包含与cdr3和人j序列融合的鸡cdr1-2和fr1-3,这是唯一的可以性,因为转基因鸡的基因组中存在的唯一jh序列是synvh转基因上的人j。cdr-h3似乎是人的,因为没有非典型半胱氨酸,其预期是在鸡cdr-h3中的(17)。synvh中表达的抗体库中有两个潜在的鸡vh序列来源:来自鸡假基因(其仍存在于人假基因的上游)的基因转换,或通过第二重排类型机制将人功能性v替换为鸡功能性v,其中鸡v基因通过v基因3'端的隐蔽重组信号序列与人v基因重排(23-26)。这些鸡v区似乎最可能来自第二重排,在转基因中的人fr3-cdr3连接处在读框内融合,从而缺失人v基因。支持基因替换而不是基因转换的主要证据是信号肽序列也是鸡的,而假基因不包含信号肽序列,因此不能将人信号肽突变为鸡信号肽序列。另外,如果基因转换可以用鸡v区替换表达的v区,那么可以期望在synvh-sd序列中会出现同样的情况,该序列也包含上游假基因。但是,在synvh-sd中未观察到v替换。已在synvh-sd转基因上游缺失了功能性鸡v和d簇,这排除了基因替换的可以性。
信号肽并不总是与转基因构建体中引入的序列匹配。尽管synvh-c和synvh-sd转基因中80-90%的序列包含与人v序列融合的人信号肽,但发现信号肽中的序列变异。观察到信号肽中变化的三种模式。在第一种模式中,将完整的鸡信号肽外显子直接在读框内剪接至人v区外显子,其在synvh-sd中约占20%,在synvh-c中约占5%(图2和表2)。synvh-sd中信号肽变化的更高频率表明存在此类变化的选择。synvh-c、synvh-sd和鸡vh中信号肽序列的比较显示,synvh-sd在中心的疏水结构域中包含lys残基,其中synvh-c包含ile,这使其疏水性低于synvh-c信号肽或鸡vh信号肽而成为较低效率的信号肽(27)(图2)。synvh-sd信号肽与人种系vh3-23基因相同,而synvh-c信号肽包含相对于种系的ile突变。在信号肽变化的第二种模式中,第一个外显子中编码的信号肽部分是人的,但v外显子中编码的信号肽的4个氨基酸是鸡的(图2)。在大约15%的synvh-sd序列中观察到此模式,但在synvh-c中未观察到。此区域存在于一些鸡假基因中(8),因此可能的是基因转换导致将信号肽的此区域突变为鸡。在这些情况下,此区域中的鸡来源的序列稍微更远地延伸到成熟的vh区的第三个氨基酸(见下文)。在信号肽变化的第三种模式中,synvh-sd序列保留了完整的人信号肽,但在lys残基上带有点突变(图2),从而将其变为疏水残基(val、ile或met)。这种模式出现在大约40%的synvh-sd序列中。所有这三种类型的变化均增加信号肽的疏水性。这些不同类型的变化在synvh-sd信号肽中的高出现率强烈表明针对这些变化的积极选择。
表2.synvh-c但非synvh-sd显示被鸡vh的基因替换,而两者均具有在信号肽中的变化。
该表显示了鸡和人vh区的数量(以及来自每个转基因的总序列百分比)。合并了来自六个synvh-c样品和三个synvh-sd样品的数据(总计为来自synvh-c禽类的6000个序列和来自synvh-sd禽类的3000个序列)。在第一栏中显示了包含鸡vh的序列,分类为具有鸡信号肽(chsigpep)或人信号肽(husigpep)的序列。在第二栏中是具有人vh的序列,类似地针对信号肽序列进行分类。所有序列均具有人jh(右列)。vh区的总数略少于序列的总数,因为一些序列具有vh缺失。
鸡假基因在synvh-c和synvh-sd中的基因转换极少见
鸡假基因阵列仍然存在于synvh-c和synvh-sd转基因的上游(图1)。在synvh-sd中,鸡假基因紧靠人功能性v,紧邻人假基因的上游,而在synvh-c中,鸡v和d基因位于两个假基因阵列之间。如上所述,与fr1相邻的信号肽序列在synvh-sd中经历基因转换,因此感兴趣的是知晓是否发生了鸡假基因对人框架的进一步基因转换。在dna水平上,鸡种系vh区段与转基因中的v基因总体上约65%相同。最长的同源性片段是11bp,并且1-6bp的错配分布在整个v区。尚不清楚这种水平和同源性模式是否足以实现基因转换。为了确定导致蛋白质序列改变的基因转换的功能性相关水平,在蛋白质水平上分析了fr1和3,以寻找长片段的鸡残基替换的证据(fr2在鸡和人之间过于保守而不能明确地检测基因转换事件)。观察到人fr1和3在synvh-c(在5652个序列中,在fr1中为0.07%,在fr3中为0.3%)和synvh-sd(在fr1中为2%,但在fr3中没有)中的非常低水平的基因转换(表3)。基因转换的最常见实例是synvh-sd中的信号肽变化,其发生在大约15%的序列中,这表明如果序列变化处于选择下,则可以以更高的频率观察到基因转换。鸡假基因在人fr中的基因转换事件的低频率表明它们是罕见的未经选择的事件。synvh-sdfr1中略微更高的频率(2%)可能是基因转换事件持续的结果,该事件始于基因5'端的信号肽区并持续到fr1中。另一个潜在的促成因素可能是人功能性v的染色体与synvh-sd中的鸡假基因的更近的物理邻近度,因为在该转基因中缺失了鸡v和d簇(图1)。在来源于这些禽类的抗原特异性mab序列中(见下文),在任何序列中均未观察到来源于鸡的fr残基,这支持了以下观点:这些稀有基因转换衍生的序列不处于选择下,并且它们也不是产生抗原结合剂所必需的。
表3.鸡假基因对人fr的基因转换极少见。
该表给出了在来自每只禽类的人序列中观察到的基因转换事件的数量。基因转换被定义为一长串为鸡序列的序列(即整个fr1)。分析了蛋白质序列,而不是核苷酸序列。右侧给出了分析的人序列的数量。
nd:未确定。鸡fr2与人转基因仅一个(synvh-sd)或两个(synvh-c)氨基酸不同,因此不可能明确评估基因转换。
*在v区外显子中编码的信号肽加上fr1的前三个氨基酸的部分,而不是在第一个外显子中编码的部分。
cdr3长度多样性
从imgt位置105-117分析了两种基因型中的cdr3长度多样性。在synvh-c序列中,范围是3-22个氨基酸,平均值是11.56±2.06(图3)。synvh-c的种系中的cdr-h3长度为11个密码子的固定长度,因为它是预先重排的,并且任何长度上的变化都只能是基因转换或缺失或插入序列的体细胞超突变的结果。这些机制更可能增加cdr长度,因为55%具有大于11个残基的长度,而只有27%的序列具有小于11的长度。在重排转基因synvh-sd中,平均cdr-h3长度稍微更长,并且分布更宽(11.89±2.68个氨基酸,范围为3-21;长度数据集都不符合正态分布)(图3)。更长的cdr-h3在synvh-sd中更常见,23%的cdr的长度为15个残基及以上,而对于synvh-c为3.6%。如果使用单个d,synvh-sd中假设的长度范围(没有编码序列的回切)将为16-23个密码子,这表明大多数序列经历由于v(d)j重组期间的回切或由于基因转换/体细胞超突变而引起的长度减少。尽管mab序列受到限制并且仅针对一种抗原,但cdr-h3长度对于synvh-sd偏向于更长的长度,而synvh-c频率似乎与整体测序相匹配(图3)。
鸡b细胞缺少tdt活性(9),因此在v(d)j重排过程中无法添加其他核苷酸。在野生型人和鸡的库中,cdr-h3倾向于比本文呈现的那些更长,其中平均cdr-h3长度为15-16个残基,且呈正态分布(17、28、29)。在野生型鸡中,cdr-h3的范围很广的长度是通过使用单个d(假设使用单个d,不具有回切的假定长度范围将是20-22个密码子)、串联d-d连接、核酸外切修剪和基因转换的组合来产生的(6、8、29)。在重排的synvh-sd转基因中,这些机制似乎没有在人v区中产生与wt鸡抗体中相同的长度。可能的是鸡d已经在演化中经历选择以偏爱串联d-d连接,这在鸡中很常见,并且是掺入成对的非典型半胱氨酸以形成cdr3内二硫键桥的机制(见下文)。相比之下,d-d连接通常未在人库中找到(29-31)。尽管没有针对d的使用进行任何分析,但在人序列中发现的更有限的cdr-h3长度表明d-d连接没有发生,或者如果存在,存在针对由鸡中人基因的d-d连接产生的更长cdr-h3的选择。
synvh-c和synvh-sd中的氨基酸含量
在synvh-c和synvh-sd数据集中分析了cdr的氨基酸含量(表4)。去除鸡v区序列,并仅分析人序列。这些数据集是基于经免疫的禽类,因此它们不代表幼稚库,但是可以将由两种转基因产生的库的特征相互比较,因为对于所有禽类免疫原、人颗粒蛋白前体(pgrn)是相同的。对于cdr-h1和h2(使用imgt定义),所有序列都包括在内,无论长度如何。
在cdr-h1中,大多数氨基酸在来自两个转基因的序列中以相似的频率呈现。一个例外是trp,它在synvh-c中不存在,但在synvh-sd中的发现频率为3.3%,全部位于imgt位置38处。可能的解释是trp存在于3个synvh-sd假基因中位置38处,但不存在于任何synvh-c假基因中。类似地,ala在synvh-c序列中更为普遍,并反映了ala在20个synvh-c假基因中的15个中在imgt位置38处存在。asp在synvh-sd中更常见,再次很可能是含有asp的假基因的基因转换的结果(16个中的7个在cdr1中具有至少一个asp)。尽管克隆选择可能改变相关氨基酸的频率,但通过增加参与抗原与pgrn的结合或影响属性诸如表达水平的残基的频率,至少一些频率差异应首先归因于变化的产生率的差异。
在cdr-h2中,两种基因型中的氨基酸分布也非常相似,同样,偏倚通常可追溯至假基因库中发现的残基。trp在synvh-sd中更常见,并且两个synvh-sd假基因在imgt位置58处包含trp,而synvh-c假基因均不包含trp。为了使trp存在于synvh-c序列中,这只能由体细胞超突变引起。
为了比较来自synvh-c和synvh-sd转基因的cdr-h3,有必要将分析集中在不是由jh区段促成的区域上,因为每个转基因具有不同的jh种系基因。synvh-sd中使用的jh6基因包含一串5个tyr残基,其使得数据中的氨基酸频率偏向tyr,并因此如果包括整个cdr则降低其他氨基酸的频率(无论边界是imgt位置105-117或107-114)。synvh-c中的j基因是jh4,其较短并且仅包含两个tyr残基。因此,焦点是特定长度的cdr-h3,并计算了通常由d区促成的位置(对于12-14个残基长度的位置为107-109,对于15个残基的位置为107-111)的氨基酸含量。这些长度的cdr-h3中的这些位置处的每个氨基酸的平均频率如图4和表4所示。在分析的cdr-h3部分中,synvh-c和synvh-sd序列之间的氨基酸含量显著不同(χ2值为2047,自由度为19,p<0.0001),并且与synvh-c相比,似乎在synvh-sd序列中的20个氨基酸中更均匀地分布(图4)。(synvh-c的标准偏差是synvh-sd的4倍)。尽管在两个转基因中以相似的频率发现许多残基,但也有一些值得注意的例外。最显著的差异是丝氨酸(synvh-c中为17.6%,相比之下synvh-sd中为2.9%)和谷氨酰胺(synvh-c中为0.5%,相比之下synvh-sd中为6.3%)。酪氨酸是抗原接触部位的重要组成部分(32、33),其在synvh-sd中(3.8%)比在synvh-c中(2.0%)略高。人d2家族成员中发现的cys密码子在synvh-sd中突变以主要编码tyr,其可以增加了tyr在库中的频率。synvh-sd中的半胱氨酸含量也较高(对于细节参见下文)。与synvh-c(0.26%)相比,synvh-sd(5.8%)中的组氨酸频率高得多,尽管带正电荷的氨基酸(k、r、h)的总体频率是相似的(synvh-sd13.0%,synvh-c11.7%)。在cdr-h3中,不可能将氨基酸频率的任何这些差异追溯到假基因库中可用残基的差异(图8)。ser、gln和his残基可在两个假基因阵列的cdr-h3区中找到。
为了测量两个转基因的cdr-h3中的氨基酸变异性,计算了长度为12-15个密码子的cdr3子集中每个位置的香农熵。图5中显示了12和15个残基的长度;13和14个残基给出了相似的结果。在cdr-h3部分中,环主要由d区段促成(对于12个残基的cdr的位置107-109和对于15个残基的cdr的位置107-111),这两个转基因的多样性是相似的。因此,单独的基因重排和基因转换可以在非由jh编码的区域中导致相似水平的多样性。在由jh编码的区域中,synvh-sdcdr-h3(尤其是来自trp-118的5-7个残基的位置(例如图5a中长度为12的cdr-h3中的位置110、112和113))中的多样性较少。这些位置在由jh6编码的酪氨酸的串联序列中,并且在测序数据中这些位置处的酪氨酸含量很高。但是,发现jh6基因中编码酪氨酸的其他位置(例如位置113和114)在synvh-sd禽类中发生了体细胞突变,并且可能是高度多样化的。位置112和112a处多样性的缺乏可能反映了这些位置在抗原结合中降低的参与性,导致体细胞突变的较小的选择压力。synvh-sd假基因中特定的cdr3序列的缺乏也可能是一个促成因素,但是位置108-111和113-114中的变异性与synvh-c相比没有降低(尽管这些位置也缺少假基因供体)。
为了确定fr区中的多样性水平,在v区的整个长度上计算了香农熵(图9)。fr几乎没有多样性,尤其是fr2。与synvh-c相比,synvh-sd中的fr变异性略低,这是可以预期的,因为synvh-c中的假基因包含一些fr变化,而synvh-sd中的假基因则没有。synvh-sd中fr的任何变化都必须来自非模板化的超突变,或者可能来自鸡假基因。在synvh-sd中观察到的fr1变异性部分来自鸡假基因(如上所述),但总体变异性水平仍低于synvh-c的fr1中的总体变异性水平。fr2基本没有变异。
synvh-c功能性v区在fr3中包含基序rlf(imgt位置90-92),其代表与在vh3-23基因中发现的种系残基qmn相比的体细胞突变。几个synvh-c假基因在这些位置处包含qmn残基,使得能够通过基因转换回复为种系fr3序列。在94%的序列中观察到这种回复,强烈表明rlf基序在结构上是不利的并且在库中选择了qmn残基。
表4.cdr1-3中的氨基酸分布。
该表使用imgtcdr命名。对于cdr-h3,计算并平均12-15个残基的cdr长度中的氨基酸频率,其中去除了由v和j基因编码的残基。仅分析了来自6只synvh-c禽类(n=5652)和3只synvh-sd禽类(n=3000)的人序列。
cdr3的疏水性
基于氨基酸疏水性的标准化的kyte-doolittle量表,计算了来自synvh-c和synvh-sd的每个cdr-h3(imgt位置105-117)的平均疏水性(34、、35)。合并了来自12-15个残基的cdr-h3长度的数据(图6)。这些值的平均值落在synvh-c和synvh-sd的量表的亲水性侧,并且范围似乎类似于先前对于人和小鼠库所报道的(36-38)。与synvh-c相比,synvh-sdcdr-h3的平均值向疏水性方向略有偏移(两组的疏水性值的平均值的差异是显著的(未配对的kolmogorov-smirnov检验,p<0.0001))。
fr和cdr中的半胱氨酸含量
发现低频率的非典型半胱氨酸残基分散在synvh-c和synvh-sd序列的整个v区中,作为单个未配对的半胱氨酸或可以潜在地形成二硫键桥的半胱氨酸对(表5)。在来自两个转基因的8652个vh序列中发现总共123个未配对的单个半胱氨酸(表5)。在synvh-c和synvh-sd的所有fr和cdr(包括cdr-h3)中都发现了这些残基,但synvh-c的fr1除外。未配对的半胱氨酸的频率低于野生型鸡中观察到的(来自synvh相对野生型的cdr1、fr2和cdr2中的0.08、0.13和0.17%相对2.1、0.8和2.4%(17))。
在cdr-h3中,synvh-c序列中的总半胱氨酸含量为0.01%,并且在synvh-sd中为2.05%(表4),相比之下在人中报道的为1.21%(28)。因此,在来源于鸡或人的人序列之间cdr-h3中半胱氨酸的出现是相似的。除了未配对的半胱氨酸外,在两个转基因的cdr-h3中(synvh-c中的3个序列,synvh-sd中的110个序列)还观察到少量可形成二硫键桥的配对的半胱氨酸(表4和表5)。synvh-c还在cdr-h2中具有配对的半胱氨酸的6个实例,但synvh-sd没有。这些配对的半胱氨酸形成潜在的二硫键稳定的环,但是环序列本身几乎没有多样性。仅发现各自来自synvh-c和synvh-sd的两个独特的序列,表明这些配对的半胱氨酸很仅少地发生,然后通过克隆扩充在其家族树中传播。令人惊讶的是,synvh-sd在cdr-h3中包含的潜在二硫键环的实例多于synvh-c(3.7%的synvh-sd序列包含潜在的cdr3内二硫键环,相比之下synvh-c序列为0.05%)。尽管synvh-sd转基因具有原则上可以提供更高的半胱氨酸含量的d簇,但该转基因被设计为使得通常在人种系类d2家族成员中发现的cys密码子全被突变为编码tyr或trp。在synvh-sd假基因的序列中,fr3下游的区域确实包含一些cys密码子,因此基因转换(以及体细胞超突变)可以已添加了半胱氨酸。synvh-sd假基因设计基于种系vh3家族cdr,并且由于种系v基因不包含cdr3,假基因中fr3下游的区域仅是假基因之间的间隔区序列。如果将这些间隔区用于基因转换,则其可以提供潜在的多样性来源。
来自鸡的人序列的半胱氨酸含量很低,与人来源的序列一样,这与正常鸡抗体的半胱氨酸含量形成鲜明对比(17)。在野生型鸡中,53%的未经选择的vh克隆在cdr-h3中包含两个非典型半胱氨酸,相比之下来自鸡的人序列为1.3%。这些配对的半胱氨酸可以形成稳定鸡cdr-h3中的抗原结合结构的小环。在本文呈现的人序列中半胱氨酸被2-4个氨基酸隔开,而在某些情况下,鸡抗体中的环可以更长(17)。鸡vh序列通常在cdr3中包含一个半胱氨酸,并在vh区中的其他位置包含第二个半胱氨酸,从而形成从cdr3到v区的另一部分的潜在二硫键桥(wu等人的3-6类型)。在来自鸡的人序列中未观察到这样的配对的半胱氨酸。来自两个转基因的8652个中只有一个序列在v区的不同部分中具有两个非典型半胱氨酸,即在fr2和fr3中具有半胱氨酸的单个synvh-c序列。在鸡中未见这种特殊模式(17)。
表5.synvh-c和synvh-sd禽类中非典型半胱氨酸含量较低。
数字是指来自每只禽类的前1000个独特序列中包含单个未配对的cys残基或两个潜在配对的cys残基的独特序列的数量(仅分析了人v区;总计为对于synvh-c的n=5652和对于synvh-sd的n=3000)。括号中显示了潜在环的序列。唯一的其他潜在二硫键环是在fr2和fr3中具有cys残基的单个synvh-c序列。在fr2和fr4中,非经典cys的所有实例都是trp->cys变化的情况,这可以通过单个核苷酸置换来发生。没有观察到超过2个非典型半胱氨酸的实例。
mab与ngs数据的比较
作为ngs数据的来源的脾淋巴细胞来自经免疫并用于产生抗原特异性抗体的禽类。六只synvh-c和三只synvh-sd禽类用测试免疫原人颗粒蛋白前体(pgrn)进行免疫。通过在gem测定中筛选脾细胞从每只禽类鉴定抗原特异性mab组(18、39)。通过elisa确认mab与pgrn的结合,并且获得了重链和轻链v区的序列。所有这些禽类中的轻链由人v-κ转基因提供(18)。禽类对于重链和轻链转基因是杂合的,在其他等位基因上具有轻链和重链敲除。因此,在禽类中仅产生人v区抗体。
将来自每只单个禽类的抗原特异性mab的独特vh序列与来自同一只禽类的ngs数据(总计177个mab)进行比较。ngs数据中每个mab的测序次数如图7所示。在ngs数据中发现所有177个mab序列,具有不同程度的匹配。在ngs数据中,79个mab(45%)被测序5次或更少,并且37个mab仅被测序一次,表明gem筛选可以容易地鉴定稀少的mab。如果仅基于序列数据选择mab,则可能会错过这些稀少的mab。其他的被发现了多次,多达约7000次。大约三分之一(63/177)的mab序列与ngs数据中的独特肽序列完全匹配,而其余的相对于最接近的匹配则有一些变化。这些变化可能反映了以下事实:ngs数据代表脾脏的简况,最多约106个b细胞(平均值,从每只禽类获得的序列总数),而mab来源于较大样品量(大约7×106个b细胞)的gem筛选。在gem筛选中选出的来自单个细胞的序列可能与ngs数据中的家族树中的序列相关,尽管在ngs数据中找不到确切的序列。通过反转录或文库制备引入的序列错误也可能是一个因素。
讨论
鸡基因转换/体细胞超突变系统已经演化以产生多样化的库,其能够对其宿主赋予保护性免疫。组合机制缺少多样性似乎并不限制cdr-h3库。当引入编码人可变区的两个转基因时(一个不能经历重排但仅通过基因转换和/或体细胞超突变而强迫产生其序列多样性,另一个经历重排并可以选择所有人d来产生组合多样性),氨基酸多样性水平是相似的。因为数据集来自经免疫的禽类,所以存在针对来自免疫的特定残基的一些偏倚,但两个转基因的行为相似,尽管其具有不同的多样性产生方式。在cdr1和2以及fr中,可能的是将氨基酸频率与两个转基因中人假基因中发现的残基相关联,但对于cdr3则并非如此。
cdr-h3长度的范围和均值在预先重排和正在重排的转基因中也非常相似。令人惊讶的是,鉴于正在重排的转基因原则上可以从d使用中的变化产生远更大的范围,但平均cdr-h3长度在两个转基因中非常相似。鸡中tdt的缺乏将长度限制到d基因编码的长度,或者可以通过基因转换插入的长度,这可以解释此处所见的与正常人库相比较短的长度。单独的基因转换可能无法产生与使用tdt活性的重排(如人中的)或使用d-d连接的重排(如wt鸡中的)一样宽的cdr-h3长度范围。synvh-sd没有产生通过d-d连接甚至单个d重排理论上可能实现的长度范围。对cdr-h3长度的限制可能来自与转基因鸡中特定v-κ轻链的配对、此类人d-d序列的潜在不利结构属性或促进鸡中未包括在人synvh-sd构建体中的d-d连接的未知顺式作用序列。即使cdr-h3长度范围和平均值相似,但synvh-sd的确含有相当一部分的较长cdr-h3(15个残基及以上),这可以代表抗体发现计划的优势,因为可能会有更多机会用于增加功能性,例如更广泛的表位覆盖、激动或拮抗功能以及动力学。最近显示,具有超长(约60个氨基酸)h3结构域的牛抗体可以是在许多hiv分化支中有效的中和剂,可能是由于它们具有结合病毒上保守但封闭的表位的能力(40)。
鸡中常见的结构(例如非典型二硫键桥)在人序列中极为罕见。在库发展期间可用的人种系序列和假基因不编码这种结构,因此没有明显的机制来产生它们。为了在鸡中获得高亲和力的人抗体,这些结构不是必需的,因为在本研究中比较的mab具有低至0.11nm的亲和力范围,和与野生型鸡中相似的表位覆盖率(18)。这些数据得到了以下观察结果的支持:来自wt鸡的识别新表位的mab不需要二硫键桥,尽管它们频繁存在(41)。
对于synvh-sd中的种系vh3-23信号肽和对于synvh-c的fr3中的qmn基序,针对功能序列的选择显然是有效的。种系vh3-23基因在信号肽中包含赖氨酸残基,其在人体细胞来源的synvh-c信号肽中被突变(为ile)。多种机制参与突变鸡中的synvh-sd信号肽序列,包括在v基因的5'端的基因转换。该结果表明,当鸡假基因的基因转换可以提供有用的目的时,它可以作用于人功能性v。尽管来自鸡假基因的基因转换作用于人v区的能力,但在整体测序中几乎观察不到人fr的基因转换,并且在mab序列中,根本没有发现鸡基因转换。从基因转换机制的角度来看,鸡假基因由于其与人功能性v的物理距离和其降低的同源性水平可能会处于不利地位(5、11、13)。这可以解释通过鸡假基因的基因转换的缺乏。从功能的角度来看,通过人假基因和体细胞超突变可实现的多样性足够广泛以使抗体能够结合抗原,因此通常没有驱动通过鸡假基因的进一步突变的选择压力。
实施例2
生成vdj敲除
指导rna设计:在mit服务器上分析了用于指导rna设计的鸡功能性重链v上游的159bp区域。选择四个指导rna,合成并分别克隆到含有野生型cas9核酸酶的ge6载体(horizon)中:grna1,aaatcattaatcaacccgac(seqidno:43);grna2,aacacgactccgggcctaga(seqidno:44);grna3,tgattaattgggcgcccgtc(seqidno:45);grna4,atttaatggccgtctaggcc(seqidno:46)。这些grna具有很少的预测的脱靶位点,其中没有一个在已知的编码序列中。还制备了含有对egfp具有特异性的grna5的对照构建体(aagttcgagggcgacaccc(seqidno:47))。
靶向载体ighko6b设计:从携带原始jh-ko的纯合敲除鸡中pcr扩增和克隆1133bp和1011bp的同源区(schusser等人,procnatlacadsciusa.2013110:20170–20175)。用引物5’-gcccctaataagtggtttaattatg-3’(seqidno:48)和5’-tctgcgctgagttctttgat-3’(seqidno:49)扩增5′hr;用引物5’-aagtcgaggctgacgagaaa-3’(seqidno:50)和5’-cttttccccaccaaatttca-3’(seqidno:51)扩增3′hr。纯合dna确保同源区对用于靶向的细胞(472-138)(其对于jh-ko是杂合的)中携带jh-ko的等位基因是同基因的。这两个等位基因可能是多态的,因为用于衍生这些pgc的鸡是远系杂交种。在igh基因座中,同源区被一段122bp的区段隔开,该区段包含由grna靶向的序列,确保当grna被共转染到细胞中时,它们将仅靶向基因组而非靶向载体本身。同源区位于用于在pgc中的选择的β-球蛋白hs4隔离的潮霉素抗性基因和设计用于与jh-ko选择标记盒中的下游loxp位点重组的loxp位点的侧翼。
用于靶向的细胞:pgc品系472-138含有先前靶向的重链基因座,其中jh区(schusser等人,procnatlacadsciusa.2013110:20170–20175)被替换为flox化的选择标记盒。通过将已注射jh-kopgc的种系嵌合雄性动物繁殖至野生型母鸡并培养来自4-8阶段(hamburger和hamilton)egfp阳性胚胎的生殖新月区的细胞来衍生该细胞系。如[6]所述培养pgc。简而言之,pgc在ko-dmem(lifetechnologies)中生长,其中的40%在水牛大鼠肝细胞(brl,atcc)上进行了预处理,并补充了7.5%的胎牛血清(hyclone)、2.5%的经辐照的鸡血清、1x非必需氨基酸、2mm谷氨酰胺、1mm丙酮酸钠、0.1mmβ-巯基乙醇(均来自lifetechnologies)、4ng/ml重组人成纤维细胞生长因子、6ng/ml重组小鼠干细胞因子(均来自r&dsystems)并在经辐射的brl细胞的饲养层上生长。每周3次将细胞传代到新鲜的饲养层上。
转染和注射:为了测试egfp基因座的失活,将15μg的egfp特异性grna5/cas9或单独的cas9添加到3×106个细胞中,并用v缓冲液(lonza,walkersville)制成100μl的体积。将细胞悬浮液转移到2mm的比色皿中,并经受350伏/100微秒的8个方波脉冲(btx830电穿孔仪)。电穿孔后,将细胞重悬于培养基中并培养9天,以允许稀释掉细胞中剩余的egfp。使用attune流式细胞仪(lifetechnologies)分析细胞的绿色荧光损失。对于靶向igh基因座的稳定转染子,将15μg环形grna1、2、3或4/cas9和2.5、5或15μg环形ighko6b添加到5×106个细胞中,并如上所述进行转染,然后与抗潮霉素的经辐照的brl一起铺板并以每孔105个细胞的密度接种在48孔板中。还进行了在不使用grna/cas9的情况下使用5μgighko6b的对照转染。3天后,加入40μg/ml潮霉素以选择具有ighko6b稳定整合的细胞。在鉴定出稳定的克隆后,扩增细胞并通过pcr确认ighko6b整合。将已确认的克隆注射到处于14-16阶段(h&h)的受体鸡胚中。将已注射的胚胎转移到替换壳中,并在37℃孵育直至孵化。在孵化后通过w染色体的pcr确定雏鸡的性别。
筛选ighko6b的靶向:通过pcr针对ighko6b的靶向分析潮霉素抗性克隆。对于5'测定,正向引物为chvh-f5:5’-tggtttggttgatggaagaatgta-3’(seqidno:52),反向引物为ha-r:5’-atacgatgttccagattacgctt-3’(seqidno:53)。对于3'测定,正向引物是ko6b-f:5’-gctgaactagaatgcatcaagc-3’(seqidno:54)和反向引物chvh-r33:5’-acaaacctttgccgcatcca-3’(seqidno:55)。
在igh基因座中插入的loxp位点的cre重组:如上所述用20μgβ-肌动蛋白-cre表达构建体瞬时转染来自1783–9品系的3×106个细胞(携带crispr靶向的loxp位点和jh-koloxp位点),并培养10天。然后在切除后重复cre转染以增加细胞百分比,四天后,收获细胞用于两个最外层loxp位点之间cre/lox重组的pcr分析。进行了两次pcr测定:均使用在上游vh侧翼区中的5'引物(chvh-f3ab:5’-gatggggggtggcaatggaatgat-3’(seqidno:56))。3'引物位于jh-ko中的neo基因中(neo-f1:5'-agctgtgctcgacgttgtcact-3'(seqidno:57))(产生1.6kb的扩增子),或位于选择标记下游的igh基因座中(chjc-r45:5’-gcccaaaatggccccaaaac-3’(seqidno:58))(产生2kb的扩增子)。
为了测试crispr/cas9是否可用于编辑pgc基因组,首先进行实验以灭活插入igh基因座的增强型绿色荧光蛋白(egfp)转基因。egfp基因是用于敲除jh-kopgc细胞系472-138中的jh基因片段的选择标记盒的一部分(图10,a)。将先前描述的针对egfp的grna(grna5)克隆到还带有野生型cas9核酸酶的u6表达载体(ge6)中。用编码cas9的构建体在有和没有grna5的情况下瞬时转染pgc。转染后第9天,通过流式细胞术分析细胞群体,与对照转染相比,cas9/grna5群体中约9%的细胞失去了egfp表达(图10,b)。
为了使用crispr/cas9产生遗传修饰的禽类,pgc中产生的修饰必须能够通过种系传给下一代。为此,其中每个细胞携带所需突变的克隆群体是优选的。与crispr/cas9结合设计药物选择策略以选择和生长携带修饰的克隆。jh-ko细胞中单个免疫球蛋白重链可变区(vh)上游的区域被靶向用于将loxp位点引入到igh基因座中。设计了四个引导rna以指导在vh的翻译起始位点上游约300bp的位点处通过cas9进行基因组双链切割(图11,a),将其各自使用cas9分别克隆到ge6载体中。cas9切割位点均距离供体靶向载体ighko6b中的同源区约50bp。使用侧翼有潮霉素选择盒的约1kb的短同源性区构建ighko6b(图11,a)。
尽管在基因组数据库中鸡igh基因座仅由短序列片段组成,没有组织成重叠群,但是有足够的独特序列可用于设计pcr引物并扩增这些短同源性区。同源区是从纯合的jh-ko基因组dna扩增的,因此对于472-138细胞系中包含jh-ko的等位基因是同基因的。这些细胞中的其他等位基因可能是多态的,因为用于衍生细胞系472-138的禽类是远系杂交种。
针对vh的4个grna分别与环状ighko6b一起共转染,并用潮霉素选择稳定的转化子。在第一组转染中,我们使用15μg的每种质粒(ighko6b和grna/cas9)用于5×106个细胞,此dna量在单独使用线性化靶向载体时通常将产生每48孔板大约1-10个集落。但是,当将ighko6b与grna/cas9结合使用时,转染效率很高,以使得所有4次转染的每个孔都含有潮霉素抗性细胞的多个克隆。尽管这些不是克隆群体,但收获来自每次转染的3个孔以测试通过ighko6b的靶向,并且所有均包含正确的靶向事件(图11,b)。对于grna1、3和4,大多数孔的每个孔具有约4-5个集落,表明靶向效率是20-25%(最差的)(如果在4-5中只有1个阳性克隆)。对于grna2,大多数孔仅具有约2-3个集落,表明约33%的较高潜在效率,因此对于后续转染,我们使用grna2,并使用较少量的供体ighko6b以减少抗性集落的数量。使用2.5μgighko6b+grna2/cas9的转染每个48孔板产生12个集落,针对靶向筛选其中的9个。所有9个克隆群体均具有正确的靶向(图11,c)。使用5μgighko6b+grna2/cas9的转染产生>50个集落,而使用5μgighko6b而没有grna/cas9的对照转染仅产生2个集落,其均未被正确靶向(数据未显示)。
利用位于靶向的等位基因中的潮霉素基因附近的loxp位点,独立确认了vh区的靶向。下游jh-ko选择标记的两侧是loxp位点(flox化),并且应与vhloxp位点位于同一染色体上。如果靶向正确,则cre重组应切除介于中间的dna,并留下单个loxp位点和无启动子的neo基因(图12,a)。crispr靶向的克隆1783–9的细胞用cre表达构建体连续瞬时转染两次(以增加具有切除的细胞的百分比),并且在第二次转染后将细胞生长四天以允许重组。位于loxp位点外部的pcr引物扩增预期大小的产物,而在未重组的细胞中,引物相距约28kb,未观察到产物(图12,b),表明靶向crispr的vhloxp位点位于正确的位置和方向。
将五种靶向的细胞系(1783–1、3、6、9和10)注射到胚胎中以产生种系嵌合体。使用jh-ko选择标记盒中的egfp转基因,将雄性嵌合体繁殖至野生型雌性动物,并通过绿色荧光筛选种系后代。5种细胞系中的4种以不同程度传播种系后代。一种细胞系1783-10表现出较高的种系传播率,包括一种嵌合体,其具有经注射的细胞的近100%的传播率。孵化了来自细胞系1783–10的传播的46个egfp阳性后代,并针对crispr靶向的ighko6b进行了分型。所有egfp阳性禽类均包含ighko6b插入序列,从而证实pgc克隆包含正确的稳定整合。
参考资料
1.ratcliffemjh.antibodies,immunoglobulingenesandthebursaoffabriciusinchickenbcelldevelopment.devcompimmunol(2006)30:101–118.doi:10.1016/j.dci.2005.06.018
2.pikek,ratcliffem.ligand-independentsignalingduringearlyavianbcelldevelopment.immunolres(2006)35:103–116.
3.aliahmadp,pikeka,ratcliffemjh.cellsurfaceimmunoglobulinregulatedcheckpointsinchickenbcelldevelopment.vetimmunolimmunopathol(2005)108:3–9.doi:10.1016/j.vetimm.2005.08.009
4.mccormackw,tjoelkerl,carlsonl,petryniakb,(null),humphriese,thompsonc.chickeniglgenerearrangementinvolvesdeletionofacircularepisomeandadditionofsinglenonrandomnucleotidestobothcodingsegments.cell(1989)56:785–91.
5.reynaudc,anquezv,dahana,weillj.asinglerearrangementeventgeneratesmostofthechickenimmunoglobulinlightchaindiversity.cell(1985)40:283.
6.reynaudc,anquezv,weillj.thechickendlocusanditscontributiontotheimmunoglobulinheavychainrepertoire.eurjimmunol(1991)21:2661–70.
7.benatart,tkalecl,ratcliffemj.stochasticrearrangementofimmunoglobulinvariable-regiongenesinchickenb-celldevelopment.procnatlacadsciusa(1992)89:7615–7619.
8.reynaudc,dahana,anquezv,weillj.somatichyperconversiondiversifiesthesinglevhsegmentofthechickenwithahighincidenceinthedregion.cell(1989)59:171–83.
9.yangb,gathykn,colemanms.t-cellspecificaviantdt:characterizationofthecdnaandrecombinantenzyme.nucleicacidsres(1995)23:2041–2048.
10.mansikkaa,sandbergm,lassilao,toivanenp.rearrangementofimmunoglobulinlightchaingenesinthechickenoccurspriortocolonizationoftheembryonicbursaoffabricius.procnatlacadsciusa(1990)87:9416–9420.
11.reynaudc-a,anquezv,grimalh,weillj-c.ahyperconversionmechanismgeneratesthechickenlightchainpreimmunerepertoire.cell(1987)48:379–388.doi:10.1016/0092-8674(87)90189-9
12.carlsonlm,mccormackwt,postemace,humphrieseh,thompsoncb.templatedinsertionsintherearrangedchickeniglvgenesegmentarisebyintrachromosomalgeneconversion.(1990)4:536–47.
13.mccormackwt,thompsoncb.chickeniglvariableregiongeneconversionsdisplaypseudogenedonorpreferenceand5“to3”polarity.genesdev(1990)4:548–558.
14.parvarir,zive,lantnerf,hellerd,schechteri.somaticdiversificationofchickenimmunoglobulinlightchainsbypointmutations.procnatlacadsciusa(1990)87:3072–3076.
15.arakawah,saribasakh,buersteddej.activation-inducedcytidinedeaminaseinitiatesimmunoglobulingeneconversionandhypermutationbyacommonintermediate.plosbiol(2004)2:e179.
16.salej,calandrinid,takatam,takedas,neubergerm.ablationofxrcc2/3transformsimmunoglobulinvgeneconversionintosomatichypermutation.nature(2001)412:921–926.
17.wul,oficjalskak,lambertm,fennellbj,darmanin-sheehana,níshúilleabháind,autinb,cumminse,tchistiakoval,blooml,etal.fundamentalcharacteristicsoftheimmunoglobulinvhrepertoireofchickensincomparisonwiththoseofhumans,mice,andcamelids.jimmunol(2012)188:322–333.doi:10.4049/jimmunol.1102466
18.chingkh,collariniej,abdicheyn,bedingerd,pedersend,izquierdos,harrimanr,zhul,etchesrj,vandelavoirm-c,etal.chickenswithhumanizedimmunoglobulingenesgenerateantibodieswithhighaffinityandbroadepitopecoveragetoconservedtargets.mabs(2017)doi:10.1080/19420862.2017.1386825
19.leightonpa,schusserb,yih,glanvillej,harrimanw.adiverserepertoireofhumanimmunoglobulinvariablegenesinachickenbcelllineisgeneratedbybothgeneconversionandsomatichypermutation.frontimmun(2015)6:126.doi:10.3389/fimmu.2015.00126
20.tillert,schusteri,depped,siegersk,strohnerr,herrmannt,berenguerm,poujold,stehlej,starky,etal.afullysynthetichumanfabantibodylibrarybasedonfixedvh/vlframeworkpairingswithfavorablebiophysicalproperties.mabs(2013)5:445–470.doi:10.4161/mabs.24218
21.schusserb,collariniej,yih,izquierdosm,feslerj,pedersend,klasingkc,kaspersb,harrimanwd,vandelavoirmc,etal.immunoglobulinknockoutchickensviaefficienthomologousrecombinationinprimordialgermcells.procnatlacadsciusa(2013)110:20170–20175.doi:10.1073/pnas.1317106110
22.dimitrovl,pedersend,chingkh,yih,collariniej,izquierdos,vandelavoirm-c,leightonpa.germlinegeneeditinginchickensbyefficientcrispr-mediatedhomologousrecombinationinprimordialgermcells.plosone(2016)11:e0154303.doi:10.1371/journal.pone.0154303
23.chenc,nagyz,prakel,weigertm.immunoglobulinheavychaingenereplacement:amechanismofreceptorediting.immunity(1995)3:747–755.
24.dewildtr,hoetr,vanvenrooijw,tomlinsoni,winterg.analysisofheavyandlightchainpairingsindicatesthatreceptoreditingshapesthehumanantibodyrepertoire.jmolbiol(1999)285:895–901.
25.cascalhom,maa,lees,masatl,wablm.aquasi-monoclonalmouse.science(1996)272:1649–1652.
26.brineybs,croweje.secondarymechanismsofdiversificationinthehumanantibodyrepertoire.frontimmun(2013)4:42.doi:10.3389/fimmu.2013.00042
27.haryadir,hos,kokyj,puhx,zhengl,pereirana,lib,bix,gohl-t,yangy,etal.optimizationofheavychainandlightchainsignalpeptidesforhighlevelexpressionoftherapeuticantibodiesinchocells.plosone(2015)10:e0116878.doi:10.1371/journal.pone.0116878
28.zemlinm,klingerm,linkj,zemlinc,bauerk,englerja,schroederhw,kirkhampm.expressedmurineandhumancdr-h3intervalsofequallengthexhibitdistinctrepertoiresthatdifferintheiraminoacidcompositionandpredictedrangeofstructures.jmolbiol(2003)334:733–749.
29.larimorek,mccormickmw,robinshs,greenbergpd.shapingofhumangermlineighrepertoiresrevealedbydeepsequencing.thejournalofimmunology(2012)189:3221–3230.doi:10.4049/jimmunol.1201303
30.ohm-laursenl,nielsenm,larsensr,baringtont.noevidencefortheuseofdir,d-dfusions,chromosome15openreadingframesorvhreplacementintheperipheralrepertoirewasfoundonapplicationofanimprovedalgorithm,jointml,to6329humanimmunoglobulinhrearrangements.immunology(2006)119:265–277.doi:10.1111/j.1365-2567.2006.02431.x
31.corbettsj,tomlinsonim,sonnhammerel,buckd,winterg.sequenceofthehumanimmunoglobulindiversity(d)segmentlocus:asystematicanalysisprovidesnoevidencefortheuseofdirsegments,inverteddsegments,“minor”dsegmentsord-drecombination.jmolbiol(1997)270:587–597.doi:10.1006/jmbi.1997.1141
32.collisavj,brouwerap,martinacr.analysisoftheantigencombiningsite:correlationsbetweenlengthandsequencecompositionofthehypervariableloopsandthenatureoftheantigen.jmolbiol(2003)325:337–354.
33.ofrany,schlessingera,rostb.automatedidentificationofcomplementaritydeterminingregions(cdrs)revealspeculiarcharacteristicsofcdrsandbcellepitopes.thejournalofimmunology(2008)181:6230–6235.
34.kytej,doolittler.asimplemethodfordisplayingthehydropathiccharacterofaprotein.jmolbiology(1982)157:105–132.
35.eisenbergd.three-dimensionalstructureofmembraneandsurfaceproteins.annurevbiochem(1984)53:595–623.doi:10.1146/annurev.bi.53.070184.003115
36.ivanovii,schelonkarl,zhuangy,gartlandgl,zemlinm,schroederhw.developmentoftheexpressedigcdr-h3repertoireismarkedbyfocusingofconstraintsinlength,aminoaciduse,andchargethatarefirstestablishedinearlybcellprogenitors.jimmunol(2005)174:7773–7780.
37.mroczekes,ippolitogc,rogoscht,hoikh,hwangpota,brandmg,zhuangy,liucr,schneiderda,zemlinm,etal.differencesinthecompositionofthehumanantibodyrepertoirebybcellsubsetsintheblood.frontimmun(2014)5:96.doi:10.3389/fimmu.2014.00096
38.rogoscht,kerzels,hoikh,zhangz,maierrf,ippolitogc,zemlinm.immunoglobulinanalysistool:anoveltoolfortheanalysisofhumanandmouseheavyandlightchaintranscripts.frontimmun(2012)3:176.doi:10.3389/fimmu.2012.00176
39.mettler-izquierdos,varelas,parkm,collariniej,lud,pramanicks,ruckerj,lopalcol,etchesr,harrimanw.high-efficiencyantibodydiscoveryachievedwithmultiplexedmicroscopy.microscopy(oxf)(2016)65:341–352.doi:10.1093/jmicro/dfw014
40.sokd,lekm,vadnaism,saye-franciscok,jardinejg,torresj,berndsenzt,kongl,stanfieldr,ruizj,etal.rapidelicitationofbroadlyneutralizingantibodiestohivbyimmunizationincows.nature(2017)doi:10.1038/nature23301
41.
本领域技术人员还将认识到,尽管以上已经根据优选实施方案描述了本发明,但是本发明不限于此。上述发明的各种特征和方面可以单独或联合使用。此外,尽管已经在特定环境中实现本发明的背景下并且对于特定应用描述了本发明,但本领域技术人员将认识到其实用性不限于此,并且本发明可以在需要检查其他样品的任何数量的环境和实施下有益地利用。因此,应鉴于本文所公开的本发明的全部范围和精神来解释所提出的权利要求。
序列表
<110>晶体生物科学股份有限公司
<120>产生人抗体的转基因鸡
<130>crys-009wo
<150>us62/646,319
<151>2018-03-21
<160>58
<170>patentinversion3.5
<210>1
<211>23
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>1
caccagtcggctccgcaaccatg23
<210>2
<211>18
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>2
gggcgatgtggggctcgc18
<210>3
<211>19
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>3
metserproleuvalserserleuleuleuleualaalaleuprogly
151015
leumetala
<210>4
<211>19
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>4
metglupheglyleusertrpilepheleuvalalaileleulysgly
151015
valglncys
<210>5
<211>19
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>5
metglupheglyleusertrpleupheleuvalalalysilelysgly
151015
valglncys
<210>6
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>6
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrphethrasntyr
202530
aspmetsertrpvalargglnalaproglygluglyleuglutrpval
354045
seralaileserglyserglyaspthrthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysglythrtrpasnthrphepheasptyrtrpglyleuglythr
100105110
leuvalthrvalserser
115
<210>7
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>7
gluvalglnleuvalgluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalavalserglypheprophesersertyr
202530
alametsertrpvalargglnalaproglygluglyleuglutrpval
354045
seralaserserglyserglyalailethrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysaspmetvalprotyrarggluglyglyasnserglytyrtyr
100105110
tyrglymetaspvalasp
115
<210>8
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>8
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheserserser
202530
alaargsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileserglyserglyglyserthrphetyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysalatrpgluleuleuhisileglnproaspalaalaprogly
100105110
pheserglyalahisser
115
<210>9
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>9
gluvalglnleuvalgluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglypheserphesersertyr
202530
alametsertrpvalargglnalaproglygluglyleuglutrpval
354045
serileileservalserglythrserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alaargaspargglyglyilealaalaglnaspthrhisglyvalhis
100105110
valserargalaalaleu
115
<210>10
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>10
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheserasntyr
202530
glymetthrtrpvalargglnproproglylysglyleuglutrpval
354045
serglyileargglyserglyglyglyalatyrtyralaaspserval
505560
argglyargphethrvalserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysaspserthrvalsertrptyrargalaleupheglnhisthr
100105110
proglyphemetgluser
115
<210>11
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>11
gluvalglnleuvalgluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrphethrsertyr
202530
alametsertrpvalargglnalaproglygluglyleuglutrpval
354045
serserileserglyasnglyglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysaspleuglytyrtyrtyraspserserglyasptyrglygly
100105110
serargglymetaspval
115
<210>12
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>12
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
glymetsertrpvalargglnalaproglygluglyleuglutrpval
354045
serglyileseralaserglyasnseralatyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alagluaspsertyrasnthrglytrpthrglutyrpheglnhisile
100105110
aspglylysglymetarg
115
<210>13
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>13
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
aspmetsertrpvalargglnalaproglygluglyleuglutrpval
354045
serglyileserglyargglyglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysprogluglyglyargglyalaalaalaglypropheaspala
100105110
pheaspileserprogly
115
<210>14
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>14
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheaspiletyr
202530
alametsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileserglyseralatyrserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspserserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysaspglyilealaalaglyhisthrargalaargglyserala
100105110
tyrproglyglumetasp
115
<210>15
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>15
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheserasnphe
202530
alametthrtrpvalargglnalaproglylysglyproglutrpval
354045
seralaileserhisserglyglyserthrtyrseralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysargaspargargserserilealaalaargleuleuglytyr
100105110
tyrtyrtyrtyrglymet
115
<210>16
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>16
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheserserhis
202530
alametsertrpvalargglnalaproglygluglyleuglutrpval
354045
seralaileserglyasnglyvalseralatyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysasppropheglytyrserasntyrglypheasptyrpheala
100105110
servalserproglyphe
115
<210>17
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>17
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheserthrtyr
202530
alametsertrpvalargglnalaproglygluglyleuglutrpval
354045
serargileserglyserglyglyserthrasptyralagluserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alaargserlysgluasptyraspileleuthrglytyrasnleugln
100105110
tyrtyrtyrtyrglymet
115
<210>18
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>18
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
alavalsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileserglyseralaaspserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysserthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysserglyargpheglyaspleutrpgluvaltyrtyrtyrgly
100105110
leuaspvalalatyrarg
115
<210>19
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>19
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglypheserphesersertyr
202530
alametasntrpvalargglnalaproglygluglyleuglutrpval
354045
serglyileserglyargglyglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alaargasppheargtyrtyraspalaileasnasntyrglyileleu
100105110
valthrtrpthralaarg
115
<210>20
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>20
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheargsertyr
202530
alametasntrpvalargglnalaproglygluglyleuglutrpval
354045
serleuileserglyserglygluseriletyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alaasnarggluglnleutrppheserserglygluthrtyrtyrtyr
100105110
tyrtyrglymetaspval
115
<210>21
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>21
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheserasntyr
202530
alametthrtrpvalargglnalaproglygluglyleuglutrpval
354045
servalileserserserglyaspthrthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysaspargtrptrpglythraspseraspalaglyvalglygln
100105110
hisserglyleuileval
115
<210>22
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>22
gluvalglnleuleuaspserglyglyglyleuvalglnproglygly
151015
serleuthrleusercysalaalaserglyphethrpheserasntyr
202530
alametglytrpvalargglnalaproglygluglyleuglutrpval
354045
serglyileseralaserglyglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysgluasnasnarghisglyglyvalserthrpheglyglyval
100105110
leuthrserprohisgly
115
<210>23
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>23
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
valmetsertrpvalargglnalaproglygluglyleuglutrpval
354045
seraspileargglyserglyaspserthrtyrtyrseraspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysalaalaalahistyrtyraspileleuthrglytyrphepro
100105110
sertyrtyrtyrtyrgly
115
<210>24
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>24
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheseriletyr
202530
alametsertrpvalargglnalaproglygluglyleuglutrpval
354045
servalileileglyserglyaspargilehistyralaaspserval
505560
lysglyargphethrileserargaspserserasnasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysileserglyaspilevalvalvalproalaalaphehisasp
100105110
valalaproglyleuasn
115
<210>25
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>25
gluvalglnleuvalgluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysthralaserglyphethrpheargasntyr
202530
alametprotrpvalargglnalaproglygluglyleuglutrpval
354045
serthrleuseraspasnglyglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysglyalaserargasnserleuleuprotyrasnproglyphe
100105110
glyglyvalilehisser
115
<210>26
<211>118
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>26
aspvalglnleuvalgluserglyglyglyvalvalargproglyglu
151015
serleuargleusercysalaalaserglyphethrpheserserasp
202530
alametsertrpvalargglnalaproglygluglyleuglutrpval
354045
serserileserglyserglyleuasnthrasptyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuargleupheserleuargalagluaspthralailetyrtyrcys
859095
alalysvalglytrpgluleuprotyrglyvalserproglyphegly
100105110
glyleualahisglypro
115
<210>27
<211>124
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>27
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
alametsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileserglyserglyglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysglutyrasntrpasnasptyrtyrtyrtyrtyrglymetasp
100105110
valtrpglyglnglythrthrvalthrvalserser
115120
<210>28
<211>115
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>28
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
trpmetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralailelysglnaspglyserglulystyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysasnproglypheglyglyvalilehisserprohisglyglu
100105110
ileargala
115
<210>29
<211>115
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>29
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrpheaspasptyr
202530
alametsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralailesertrpasnserglyseriletyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysaspvalalaproglyleuasnglyglyleuargaspproile
100105110
proglyglu
115
<210>30
<211>115
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>30
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrpheserasptyr
202530
tyrmetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileserserserglyserthriletyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysalaargthrglyleuthrglythrargvalhisleuhisleu
100105110
glyargleu
115
<210>31
<211>115
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>31
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
aspmetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileglythralaglyaspthrtyrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysglyglyserhisarghishisargvalalaserserserala
100105110
serglylys
115
<210>32
<211>116
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>32
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrpheserasnala
202530
trpmetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralailelysserlysthraspglyglythrthrtyrtyralaasp
505560
servallysglyargphethrileserargaspasnserlysasnthr
65707580
leutyrleuglnmetasnserleuargalagluaspthralavaltyr
859095
tyrcysalalysglymetgluarggluvalmetthrilelysproglu
100105110
cysprothrpro
115
<210>33
<211>115
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>33
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrpheaspasptyr
202530
glymetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileasntrpasnglyglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysproprothrprothrcysprothrproargproproleuthr
100105110
glupromet
115
<210>34
<211>115
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>34
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
sermetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralailesersersersersertyriletyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysileleuvalthrtrpthralaargglyhisasppheilecys
100105110
iletrpglu
115
<210>35
<211>115
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>35
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
alametsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralailesertyraspglyserasnlystyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysargalaproileserproargglyleutrpalaproleuasn
100105110
proglyala
115
<210>36
<211>114
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>36
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrvalserserasn
202530
tyrmetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralailetyrserglyglyserthrtyrtyralaaspservallys
505560
glyargphethrileserargaspasnserlysasnthrleutyrleu
65707580
glnmetasnserleuargalagluaspthralavaltyrtyrcysala
859095
lysargalaleuileserglnproglytrpservalthrproleuser
100105110
alaglu
<210>37
<211>113
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>37
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
alametsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileserserasnglyglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysargcysthrthrargvalproleuproserileserprogly
100105110
ala
<210>38
<211>117
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>38
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrpheserasphis
202530
tyrmetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralathrargasnlysalaasnsertyrthrthrtyrtyralaasp
505560
servallysglyargphethrileserargaspasnserlysasnthr
65707580
leutyrleuglnmetasnserleuargalagluaspthralavaltyr
859095
tyrcysalalysserproglyleuglyserleuserleuproargasn
100105110
proglyprothrleu
115
<210>39
<211>116
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>39
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrpheserglyser
202530
alametsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileargserlysalaasnsertyralathrtyrtyralaasp
505560
servallysglyargphethrileserargaspasnserlysasnthr
65707580
leutyrleuglnmetasnserleuargalagluaspthralavaltyr
859095
tyrcysalalysglnglyglyproserlysglyargthrargilepro
100105110
leuprosermet
115
<210>40
<211>114
<212>prt
<213>人工序列
<220>
<223>合成序列
<400>40
gluvalglnleuleugluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalaalaserglyphethrphesersertyr
202530
trpmetsertrpvalargglnalaproglylysglyleuglutrpval
354045
seralaileasnseraspglyserserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnserlysasnthrleutyr
65707580
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
859095
alalysglysermetthrglucysproprometproargtrpthrglu
100105110
glnarg
<210>41
<211>122
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>41
gtggataacgataattagaaggggacaaattaatgagaaaacacgactccgggcctagac60
ggccattaaatcattaatcaacccgacgggcgcccaattaatcataaagttattaagcaa120
tt122
<210>42
<211>122
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>42
aattgcttaataactttatgattaattgggcgcccgtcgggttgattaatgatttaatgg60
ccgtctaggcccggagtcgtgttttctcattaatttgtccccttctaattatcgttatcc120
ac122
<210>43
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>43
aaatcattaatcaacccgac20
<210>44
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>44
aacacgactccgggcctaga20
<210>45
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>45
tgattaattgggcgcccgtc20
<210>46
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>46
atttaatggccgtctaggcc20
<210>47
<211>19
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>47
aagttcgagggcgacaccc19
<210>48
<211>25
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>48
gcccctaataagtggtttaattatg25
<210>49
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>49
tctgcgctgagttctttgat20
<210>50
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>50
aagtcgaggctgacgagaaa20
<210>51
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>51
cttttccccaccaaatttca20
<210>52
<211>24
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>52
tggtttggttgatggaagaatgta24
<210>53
<211>23
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>53
atacgatgttccagattacgctt23
<210>54
<211>22
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>54
gctgaactagaatgcatcaagc22
<210>55
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>55
acaaacctttgccgcatcca20
<210>56
<211>24
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>56
gatggggggtggcaatggaatgat24
<210>57
<211>22
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>57
agctgtgctcgacgttgtcact22
<210>58
<211>20
<212>dna
<213>人工序列
<220>
<223>合成序列
<400>58
gcccaaaatggccccaaaac20
- 还没有人留言评论。精彩留言会获得点赞!