嵌合抗原受体(CAR)、组合物及其使用方法与流程
本申请是根据2016年6月24日提交的国际pct申请no.pct/us16/39306之权益,及2016年7月29日提交的美国临时申请no.62/369,004,皆以全文引用之方式併入本文中。
背景技术:
t细胞,淋巴细胞的一种,在细胞介导之免疫性中起重要作用。其与其他淋巴细胞(诸如b细胞及自然杀手细胞(nk细胞))之区别在于细胞表面上存在t细胞受体(tcr)。t辅助细胞,亦称为cd4+t或cd4t细胞,在其表面上表达cd4糖蛋白。辅助t细胞在暴露于由mhc(主要组织相容复合体)ii类分子呈递之肽抗原时活化。一旦活化,此等细胞快速增殖且分泌可调节免疫反应之细胞因子。细胞毒性t细胞,亦称为cd8+t细胞或cd8t细胞,在细胞表面上表达cd8糖蛋白。cd8+t细胞在暴露于由mhci类分子呈递之肽抗原时活化。记忆t细胞,一种t细胞亚群,长期留存并回应于其等同源抗原,因此向免疫系统提供针对过去感染及/或肿瘤细胞之“记忆”。
经基因改造后,t细胞可于其表面产生特殊受体,称为嵌合抗原受体(car)。car为允许t细胞识别肿瘤细胞上之特定蛋白(抗原)之蛋白。此等经工程改造之cart细胞接着在实验室中生长直至其数目达到数十亿。接着将扩增之cart细胞群输注至患者中。
迄今为止,临床试验已证实嵌合抗原受体(car)t细胞在对标准化学疗法具有抗性之血液科恶性疾病中具有巨大前景。最值得注意的是,特定之cd19car(cd19car)t细胞疗法具有显著效果,包括b细胞恶性肿瘤之长期缓解(kochenderfer,wilson等人2010,kalos,levine等人2011,porter,levine等人2011,davila,riviere等人2013,grupp,frey等人2013,grupp,kalos等人2013,kalos,nazimuddin等人2013,kochenderfer,dudley等人2013,kochenderfer,dudley等人2013,lee,shah等人2013,park,riviere等人2013,maude,frey等人2014)。
尽管car疗法在b细胞白血病及淋巴瘤中取得成功,但尚未有明确之car疗法应用于t细胞恶性肿瘤。鉴于t细胞恶性肿瘤之疗法比b细胞恶性肿瘤之疗法具有明显更差之效果(abramson,feldman等人2014),因此car疗法具有进一步解决巨大临床需求之潜力。
迄今为止,当前成果集中于在治疗多种b细胞恶性肿瘤中有显示功效之cart细胞。尽管在使用cd19car之b-all中约90%之初始缓解率为常见的,但大部分在一年内复发。至少部分复发原因归于抗原逃逸。因此,现时迫切地需要更有效的cart细胞治疗以防止复发。标靶探索及选择为初始步骤,因为不存在可确保或引导有效car设计之一般法则。
现时存在一些阻碍car治疗性方法之更广泛应用之障碍。其中最常见挑战为:(1)抗原标靶及嵌合抗原受体之选择;(2)car设计;(3)肿瘤异质性,尤其肿瘤抗原之表面表达之差异。靶向单一抗原具有免疫逃逸之风险,此可由靶向多个所需抗原来解决;(4)免疫抑制微环境。cart细胞可以在到达肿瘤部位时被抑制和失效。
大部分car嵌合抗原受体为来源于单株抗体之scfv且部分此等单株抗体已用于临床试验或疾病治疗。然而,单株抗体具有有限功效,表明需要替代的及更强效的靶向方法,诸如car。scfvs为car之最常用的嵌合抗原受体。然而,抗原上所识别之抗原决定基之car亲和结合及位置可影响功能。此外,t细胞或nk细胞之表面car表达量受适合的前导序列及启动子影响。此外,过表达之car蛋白可对细胞具有毒性。
因此,现时仍然需要经改良之基于嵌合抗原受体之疗法,其可慮及t细胞相关恶性肿瘤之更有效、安全及有效靶向。
技术实现要素:
在一个实施例中,本发明公开提供了具有第一嵌合抗原受体多肽的工程改造细胞,所述第一嵌合抗原受体多肽包括第一抗原识别结构域,第一信号肽,第一铰链区,第一跨膜结构域,第一共刺激结构域和第一信号域;第二嵌合抗原受体多肽,包括第二抗原识别结构域,第二信号肽,第二铰链区,第二跨膜结构域,第二共刺激结构域和第二信号结构域;其中第一抗原识别结构域不同于第二抗原识别结构域。
在另一个实施例中,本发明公开提供了包含嵌合抗原受体和增强子的工程改造多肽。
在另一个实施例中,本发明公开提供了包含嵌合抗原受体多肽和增强子的工程改造多肽。
在另一个实施例中,本发明公开提供了工程改造嵌合抗原受体多肽,所述多肽包括:信号肽,cd45抗原识别结构域,铰链区,跨膜结构域,至少一个共刺激结构域和信号传导结构域。在另一个实施例中,本发明公开提供了编码上述多肽的多核苷酸。
在另一个实施例中,本发明公开提供了具有上述工程改造多肽或多核苷酸的工程改造细胞。
在另一个实施例中,本发明公开内容提供了减少靶细胞数量的方法,包括以下步骤:i)使所述靶细胞与有效量的具有至少一种嵌合抗原受体多肽的工程改造细胞接触,具有多个嵌合抗原受体多肽的工程改造细胞,其每个嵌合抗原受体多肽是独立的;和ii)任选地,测定所述细胞数量的减少。靶细胞包括至少一种选自白细胞介素6受体,ny-eso-1,甲胎蛋白(afp),磷脂酰肌醇蛋白聚糖-3(gpc3),baff-r,baff,april,bcma,taci的细胞表面抗原,ley,cd5,cd13,cd14,cd15,cd19,cd20,cd22,cd33,cd41,cd45,cd61,cd64,cd68,cd117,cd123,cd138,cd267,cd269,cd38,flt3受体和cs1。
在另一个实施例中,本发明公开提供了通过施用任何工程改造的方法治疗b细胞淋巴瘤,t细胞淋巴瘤,多发性骨髓瘤,慢性髓性白血病,b细胞急性淋巴细胞白血病(b-all)和细胞增殖性疾病的方法。上述细胞给需要的患者。
【图式简单说明】
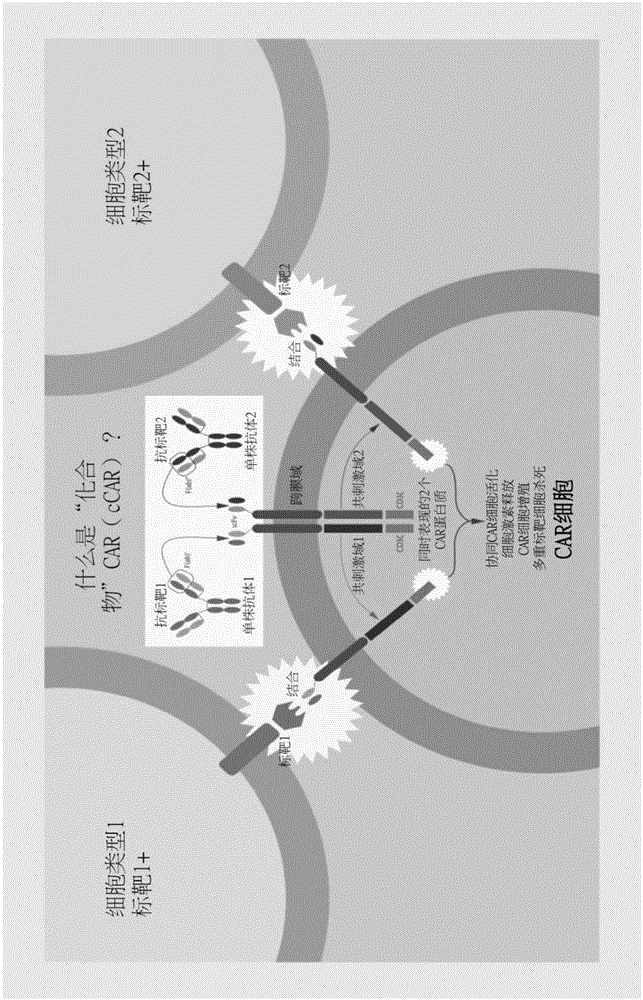
图1.ccar构筑体(下文中,“多个car或复合car”)之示意图。多样或复合的car靶向多种抗原(例如细胞类型1或细胞类型2或相同细胞类型)。多样的或ccart细胞免疫疗法包含个别组件car,其包含不同或相同的抗原识别域、铰链区、跨膜域、多种共刺激域及胞内信号传导域。
图2a.ccar-t构筑体之示意图。构筑体包含驱使由p2a肽连接之car之多个模组单元之表达的sffv启动子。在连接子裂解后,ccar裂解且在标靶表达cd33及/或cd123时接合。作为新颖的ccar构筑体,构筑体之活化域可包括(但不限于)cd33car区段上之4-1bb及cd123car上之cd28区域。
图2b.描绘经转导之cd33cd123ccar-t细胞之表达之蛋白印迹法。该图描绘两种不同car蛋白质,亦即cd33car及cd123car之表达。在连接子裂解时表达cd33及cd123car之ccar-t细胞产生两种不同且始终强烈的蛋白质谱带。包括绿色萤光蛋白质(gfp)作为阴性对照。
图2c.表达转导效率之流式细胞术。上图展示在用于ucb(脐带血)及pb(外周血)t细胞之前,在293fthek(人胚肾)细胞上测试以检验最大转导效率之cd33cd123ccar(亦称为cd33cd123-2g-car)之慢病毒效价。下图展示经包含cd33cd123ccar构筑体之慢病毒载体转导之cd33cd123ccar(亦称为cd33cd123-2g-car)t细胞及作为对照物之经gfp转导之细胞。由黄色环指示之百分比为转导效率之替代物。
图3.展示产生高效复合car(ccar)之方法的示意图。用复合car质粒dna和lipofectamine2000转染hek-293-ft细胞;在约36小时和约60小时收集病毒上清液;过滤并储存在-80摄氏度。用抗小鼠cd3抗体和il-2活化t细胞至少2天。在有经重组人纤维蛋白片段涂布的平板上用解冻之慢病毒将活化的t细胞转导至少一次;再以0.3×106个t细胞/ml进行至少一次过夜转导约2天,减少t细胞的数量以提高转导效率。转导后,洗涤并扩增细胞;进行流式分析(f(ab')2标记)以确认第3天的car效率;总共5-7天扩展。在体外将ccart细胞与靶细胞共培养,并在体内评估ccart细胞杀死癌细胞之功效(小鼠)。
图4.cd33cd123-2gcar-t细胞(ccar)与前髓细胞性白血病细胞系hl60一起培育之共培养分析。比较ccar-t细胞(下图)与对照性经gfp转导之t细胞(上图)。藉由在培育约24小时之后留存之cd33+细胞群体(黄色环中包围)来测量杀伤力。
图5.ccar-t细胞与骨髓性白血病细胞系kg-1a一起培育之共培养分析,其表达约100%cd33及约50-80%cd123。比较ccar-t细胞(下图)与对照性经gfp转导之t细胞(上图)。藉由在培育约24小时之后留存之cd33+细胞群体来测量杀伤力。
图6.cd33cd123ccar-t细胞与aml-9以5:1的比例共培养。ccar-t细胞与aml患者样品(此处称为aml-9)一起培育之共培养分析。患者细胞包括混合细胞群,诸如白血病细胞、单核细胞及其他类型之原始细胞。cd33充当car-t作用标记物以及cd34(白血病细胞之特异性标记物)之标记物。比较car-t图(右侧)与对照性经gfp转导之t细胞(中间)。藉由在培育至少24小时之后留存之cd33+/cd34+细胞群体来测量杀伤力。
图7.cd33-cd123ccar-t细胞与sp-bm-b6以5:1的比例共培养。ccar-t细胞与b-all患者样品(此处称为sp-bm-b6)一起培育之共培养分析。患者细胞包括混合细胞群,诸如白血病细胞、单核细胞及其他类型之原始细胞。cd34充当白血病细胞之特定标记物。比较car-t图(右侧)与对照性经gfp转导之t细胞(中间)。借由在培育至少24小时之后留存之cd34+细胞群体来测量杀伤力。
图8.nk-92细胞中之cd33cd123ccar表达。使用山羊抗小鼠抗体f(ab)2侦测cd33cd123ccar表达。
图9.cd33cd123ccarnk-92细胞与hl-60一起培育之共培养分析。比较ccarnk-92细胞与经gfp转导之nk-92细胞。藉由在培育约24小时之后留存之cd33+细胞群体来测量杀伤力。
图10.ccarnk-92细胞与kg1a一起培育之共培养分析。比较ccarnk细胞图与经gfp转导之nk-92细胞。藉由在培育约24小时之后留存之cd33+细胞群体来测量杀伤力。
图11.与hl-60或kg1a细胞共培养之cd33cd123ccar(car-cd33/123)nk-92细胞之剂量反应。藉由在培育约24小时之后留存之cd33+细胞群体来测量杀伤力。
图12.在两个kg11细胞群中,cd33cd123ccarnk-92细胞杀伤力与对照物杀伤力之比较。在car-cd33/123(cd33cd123ccarnk-92细胞)及标靶细胞kg1a之不同比率下共培养进行分析。藉由在培养约24小时之后留存之cd33+cd123+或cd33+cd123-细胞群体来测量杀伤力。
图13a.通过p2a和t2a的链接示意图显示单个构筑体中之ccar-t和4-1bbl。该构筑体由sffv启动子组成,该启动子驱动car的两个模块单元之表达,肽蛋白和增强子4-1bbl。当连接子裂解时,ccar和4-1bbl分裂并结合表达cd33和/或cd123及4-1bbl的靶标。复合car,cd33cd123cart细胞不仅通过cd28,而且还通过4-1bb配体(4-1bbl或cd137l)接受共刺激。由cd3-zeta信号传导结构域完整该car-t之组装。
图13b.在t细胞表面上表达cd33cd123-41bbl-2g构筑体。来自健康供体的外周血t细胞用cd33cd123-4-1bbl-2g构筑体在6孔板中转导,与2ml病毒上清液一起培养。用f(ab)’标记car蛋白的表面表达来测定car表达,随后进行facs分析。将转导之细胞与同时标记之对照t细胞进行比较。在转导期结束后1天测定表达并圈出转导之群体。
图14.通过p2a和t2a的链接示意图显示单个构筑体中之ccar-t和il-15/il-15sushi。该构筑体由sffv启动子组成,该启动子驱动两个模块单元之表达,car和增强子il-15/il-15sushi。当连接子裂解时,ccar和il-15/il-15sushi分裂并与表达cd33和/或cd123之靶标结合。有cd3-zeta信号传导结构域完整该car-t之组装。增强子包括,但不限于ccar上的il-15/il-15sushi。
图15.ccar之示意图。构筑体包含驱使由连接子连接之car之多个模组单元之表达的sffv启动子。当连接子裂解时,ccar裂解且在标靶表达多种标靶抗原(cd19及/或cd20,及/或cd22及/或138)之组合时接合。多种ccar使用相同或不同共刺激域,诸如(但不限于)4-1bb(亦标记为4-bb)及/或cd28。
图16.转导活化后之t细胞以制备cd19cd20-2g,cd19cd22-2gcart细胞(均为l8)。(16a)复合car的设计。(16b)蛋白印迹法。用对照载体(道1),cd19cd20-2g(道2)及cd19cd22-2g(道3)慢病毒质粒转染hek-293t细胞。转染48小时后,除去上清液,并收获细胞。裂解细胞以用于蛋白印迹法,并用小鼠抗人cd3z一抗和山羊抗小鼠hrp二抗探测。(16c)用抗cd3抗体活化pmbc白血球层t细胞3天。用对照载体(左),cd19cd20-2g(中)或cd19cd22-2g(右)慢病毒上清液转导细胞。培养3天后,收获细胞并与缀合生物素之山羊抗小鼠fab2或山羊igg抗体一起培养30分钟洗涤和悬浮细胞,并用链霉亲和素-pe和小鼠抗人cd3-percp染色30分钟。洗涤并将细胞悬浮与2%福尔马林中,然后通过流式细胞术分析以确定car效率。(n=2)
图17.使用不同之前导序列表达复合cd19cd22cart细胞。用抗cd3抗体将pmbc白血球层t细胞活化3天。用对照载体(左),l8-cd19cd22-2gcar(左中),l45-cd19cd22-2gcar(右中),或csf-cd19cd22-2gcar(右)慢病毒上清液转导细胞。3倍浓缩各个上清液。培养3天后,收获细胞并与缀合生物素之山羊抗小鼠fab2或山羊igg抗体一起培养30分钟。洗涤和悬浮细胞,并用链霉亲和素-pe和小鼠抗人cd3-percp染色30分钟。洗涤并将细胞悬浮与2%福尔马林中,然后通过流式细胞术分析以确定car效率。(n=2)
图18.使用浓缩的与未浓缩的l8-cd19cd22-2g或l8-cd19cd20-2g慢病毒上清液比较转导效率。(18a)pmbc白血球层t细胞用抗cd3抗体活化3天。用对照载体(左),未浓缩l8-cd19cd22-2gcar(中)或3倍浓缩l8-cd19cd22-2gcar(右)慢病毒上清液转导细胞。培养3天后,收获细胞并与缀合生物素之山羊抗小鼠fab2或山羊igg抗体一起培养30分钟。洗涤和悬浮细胞,并用链霉亲和素-pe和小鼠抗人cd3-percp染色30分钟。洗涤并将细胞悬浮与2%福尔马林中,然后通过流式细胞术分析以确定car效率。(n=2)(18b)相同之实验用于测试含有l8-cd19cd20-2g未浓缩或2.5倍浓缩慢病毒载体之构筑体。
图19.l8-cd19cd22-2gcart细胞在过夜培养中溶解sp53肿瘤细胞。将对照(上),或l8-cd19cd22-2g(下)慢病毒上清液转导活化pmbct细胞与sp53细胞以1:1(左),2:1(中)和5:1(右)的效应细胞:靶细胞比例进行共培养。在37摄氏度下培养24小时后,洗涤样品并用抗人cd3-percp和抗人cd19-apc染色,洗涤,并通过流式细胞术分析。单独sp53细胞样品显示在最右上方,每个比例之溶解百分比概述在右下方。(n=2)
图20.l8-cd19cd22-2gcart细胞在过夜培养中溶解jeko-1肿瘤细胞。用对照(左)或l8-cd19cd22-2g(中)之3倍浓缩慢病毒上清液转导的活化pmbct细胞与jeko-1细胞以2:1(上)和5:1(下)的效应细胞:靶细胞比例进行共培养。在37摄氏度下培养24小时后,洗涤样品并用抗人cd3-percp和cd19-apc染色,洗涤,并通过流式细胞术分析。单独jeko-1细胞样品和细胞溶解之总结显示在右侧。(n=2)
图21.l8-cd19cd22-2gcart细胞在过夜培养中溶解aml患者细胞。用对照(左)或l8-cd19cd22-2g(中)之3倍浓缩慢病毒上清液转导的活化pmbct细胞与来自aml患者(pt1)之以cmtmr染色之细胞以2:1(上)和5:1(中)的效应细胞:靶细胞比例进行共培养。在37摄氏度下培养24小时后,洗涤样品并用抗人cd3-percp和cd19-apc染色,洗涤,并通过流式细胞术分析。单独患者细胞样品和细胞溶解之总结显示在右侧。(n=2)
图22a.l8-cd19cd22-2gcart细胞耗尽cd19+b-all患者细胞。用对照(左)或l8-cd19cd22-2g(中)慢病毒上清液转导之活化pmbct细胞与来自b-all患者(pt2)之以cmtmr染色之细胞以1:1的效应细胞:靶细胞比例在含有2.5%fbs和il-2的培养基中进行共培养4天。在37摄氏度下培养后,洗涤样品并用抗人cd3-percp和抗人cd19-apc染色,洗涤,并通过流式细胞术分析。单独患者细胞样品和细胞溶解之总结显示在右侧。
图22b.l8-cd19cd22-2gccart细胞显示对cd22+k562之作用。通过转导入野生型k562细胞生产表达cd22之人造k562细胞系(k562xp22)。之后,吾人通过靶向次要的k562细胞cd22+群体以测试cd19cd22ccar之抗肿瘤特性。与对照相比,1:1(效应细胞:靶细胞)比例之共培养显示出适度且显著的细胞毒性作用。通过流式细胞术将共培养物用cd3,cd19和cd22染色以分离效应细胞及靶细胞群。其结果绘制成了图表。细胞毒性结果与其他报道之针对人工抗原呈递细胞系的抗肿瘤活性之数据一致。
图23.各种bc1ccar慢病毒之转导方案。(23a)方法一是基本转导方案,是将细胞分2次转导,每次24小时。方案根据该图进行。(23b)方法二具有与方法一相同之方法,只是将第二次转导替换成继续培养。(23c)修订后之方法二是将细胞直接与病毒上清液培养48小时。
图24a-24c.car构筑方案及转导方法之比较。(24a)bc1ccar之模块设计包括通过自切割p2a肽,cd8衍生之铰链域(h)及跨膜域(tm)与抗cd319(cs1)scfv融合的抗cd269(bcma)单链可变片段(scfv)区域,和与cd3ζ信号传导结构域连接之串联cd28和4-1bb共刺激结构域。形成病毒启动子(sffv)和cd8签到序列之强脾脏焦点用于在t细胞表面上有效表达cd3car分子。(24b)通过针对同型对照之流式细胞术测量bc1ccar之表达。圈出之群体代表转导后之car细胞。(24c)通过最佳方法改善转导效率。
图24d.在hek-293ft细胞上之bc1ccar和bcma-cs1-2g之蛋白表达。用gfp(道1)及bc1ccar(道2)慢病毒质粒转染hek-293ft细胞。转染48小时后,除去上清液,并除去细胞。将细胞溶解用于蛋白印迹法,并用小鼠抗人cd3z抗体探测。(c)通过最佳方法改善转导效率。
图25a-25c.体外评估bc1ccart细胞对骨髓瘤细胞系之作用。(25a)将bc1ccar和对照t细胞与高度bcma阳性的mm1s和rpmi-8226细胞以2:1和5:1效应细胞:靶细胞之比例一起培养24小时。用cytotracker染料(cmtmr)将mm1s和rpmi-8226靶细胞染色以区分效应t细胞和靶细胞。圈出的群体是肿瘤细胞。(25b)u266靶标耗尽。bc1ccar和对照t细胞同时也与表达bcma及cs1亚组之u266细胞一起培养。圈出的群体是肿瘤细胞。(25c)bc1ccart细胞抗人类骨髓瘤细胞系之活性之总结。2:1和5:1效应细胞:靶细胞比例之bc1ccart细胞在体外对各种骨髓瘤细胞系之细胞毒性之图形总结。
图26a-26d.bc1ccart细胞对原发性骨髓瘤肿瘤细胞之抗肿瘤活性之表征。(26a)剂量依赖性针对mm7-g原发性双表型肿瘤之影响。将bc1ccar和对照t细胞与bcma+cs1+原发性骨髓瘤细胞mm7-g共培养24小时。用cmtmr对靶细胞进行预染色,并以2:1,5:1和10:1之效应细胞:靶细胞比例进行共培养。通过bcma,cs1及cmtmr对群体进行闸控。在流式细胞术图表中圈出的群体为靶肿瘤细胞群体(左)。为了清楚起见,显示了总结体外细胞毒性之条形图(右)。(26b)mm10-g中之群体针对性消耗。在类似条件下进行与mm10-g原发性肿瘤细胞之共培养。当用抗cs1和抗bcma抗体染色时,mm10-g显示出不同之群体。bcma+cs1+双阳性群体呈紫色,而仅cs1阳性群体呈深蓝色。针对每个群体之bc1ccart细胞之细胞毒性总结在下面之条形图中。(26c)剂量依赖性针对cs1dimbcmaneg之影响。mm11-g原发肿瘤。使用bcmadimcs1dim原代细胞(mm11-g)之第三个实验进一步显示了在一系列的效应细胞:靶细胞(e:t)剂量下之bc1ccar细胞毒性作用。(26d)概述小图显示了具有多种bcma和cs1组合之针对骨髓瘤细胞系和原发肿瘤细胞之bc1ccart细胞细胞毒性。
图27a-27d.bc1ccar抗原针对性之功能验证。(27a)吾人设计了独立表达bcma或cs1之cml细胞系k562。野生型k562显示为阴性峰值,而表达bcma之k562(bcmaxpk562)和表达cs1之k562(cs1xp562)显示其各自抗原表达范围之群体移位。(27b)与bcmaxpk562及cs1xpk562细胞一起短期(4小时-12小时)培养之bc1ccart细胞显示与e:t剂量增加相关之抗原特异性细胞毒性。野生型k562细胞之实验作为阴性对照。吾人生产了针对cs1之单一car以比较bc1ccar对cs1xpk562细胞之功效,并在相应的图中用红线描绘。在培养24小时后,吾人还观察到针对cs1dimnk-92细胞之抗cs1特异性活性。(27c)混合细胞实验中单个抗原car与bc1ccart细胞之比较。用bcmaxpk562细胞及cs1xpk562细胞之5:1混合物进行超过48小时之长期培养。bc1ccar,cs1-car,bcma-car及对照t细胞以5:1之e:t比例加入每个处理孔中并通过流式细胞术分析。所示之柱状图显示了每种处理条件下所残留的bcma或cs1细胞群体,其中红线划分t细胞或靶细胞群。柱状图中的数值代表残留之靶肿瘤细胞闸控后之群体。(27d)bc1ccar对原发性骨髓抽吸物中cs1亚群之活性。使用骨髓抽吸样品作为表达cs1之少数亚群体进行进一步之共培养实验。以2:1(左图),5:1(中图)或10:1(右图)e:t比例添加bc1ccar或对照t细胞,圈出的群体代表表达cs1靶细胞群体。通过流式细胞术分析结果(上)。针对骨髓亚群之抗cs1活性之总结图(下)。
图28a-28c.长期连续杀上实验和再次挑战肿瘤实验。(28a)构建长期连续杀伤实验之方案。在没有外源细胞因子的情况下,进行168小时初始培养物为1:1e:t比例的car细胞或对照细胞与mm1s肿瘤细胞之共培养。48小时后,获取小样品进行流式细胞术分析,并将mm1s细胞重新引入每个处理孔中。一直重复此步骤直到实验进行到168小时。(26b)48小时后的t细胞增殖及反应。在进行流式细胞分析术的当天拍摄图像,并用抗bcma,抗cs1和抗cd3抗体染色。mm1s细胞高度表达bcma抗原及cs1抗抗原。圈出的蓝色群体代表mm1s肿瘤群体。(28c)108小时后car细胞的增殖与抗原的消耗。在108小时时间点进行类似之图像采集和流式细胞术分析。
图29a-29c.bc1ccar细胞在体内表现出抗白血病之作用。(29a)注射了mm1sluc+细胞之小鼠模型之ivis成像。对nsg小鼠进行亚致死照射并在静脉内注射表达荧光素酶之mm1s多发性骨髓瘤细胞以诱导可测量之肿瘤形成。3天后在小鼠静脉内注射5x106个bc1ccart细胞或对照gfpt细胞。在第3天,第6天,第8天和第11天,在小鼠皮下注射redijectd-luciferin并进行ivis成像。(29b)bc1ccart细胞控制mm1s肿瘤成长。将注射bc1ccart细胞之小鼠的平均光强度与注射gfp对照t细胞之小鼠的平均光强度进行比较。(29c)bc1ccart细胞改善小鼠之存活前景。测量小鼠的存活百分比并在两组之间进行比较,用log-rankmantel-cox测试以计算改善的存货前景之显著性。
图29d.bcma-car和bc1ccart细胞在异种移植小鼠模型中对表达bcma和cs1之k562细胞混合物表现出显著之抗白血病作用。将表达bcma之荧光素酶阳性k562细胞与表达cs1之荧光素酶阳性k562细胞以4:1的bcma:cs1k562细胞比例混合。然后在对小鼠进行亚致死照射后24小时在静脉内注射混合的k562细胞(0.5x106个细胞)。第3天后,在小鼠静脉内注射一定量之bcmacart细胞,bc1ccart细胞或对照t细胞(每组n=5)。在第3天,第7天,第10天和第12天使用ivis成像系统测量从背侧观察的肿瘤负荷。在第7天bcma小鼠#3有大肿瘤。在第10天,从背侧观察,与对照相比,bcma小鼠的肿瘤减少47.7%,而ccar小鼠的肿瘤减少53.8%;在第12天(仅腹侧视图),从背侧观察,与对照相比,bcma小鼠的肿瘤减少43.8%,而ccar小鼠的肿瘤减少60.7%。
图29e.bcma和bc1ccart细胞在体内显著减轻肿瘤负荷。相对于对照,随着时间的变化,用bcmacart细胞或ccar(bc1ccar)处理之小鼠的肿瘤减少百分比。
图30a-30b.bc1ccar转导入nk-92细胞。(30a)bc1ccar之模块设计如前所示。(30b)nk-92细胞表面的car表达。通过与病毒上清液一起培养48小时将构筑体转导入nk-92细胞,并用f(ab)’抗体检测标记car蛋白表面之表达。将已转导之群体圈出并与对照nk-92细胞进行比较。
图31a-31b.bc1ccarnk-92抗肿瘤特性之表征。(31a)bc1ccarnk细胞溶解骨髓瘤细胞系和原代细胞。除了原代mm7-g肿瘤细胞外,吾人还将bc1ccarnk-92细胞与u266,rpmi-8226和mm1s骨髓瘤细胞系一起培养。以5:1的e:t比例进行2小时共培养,并用抗cs1和抗bcma抗体标记以区分不同群体。圈出的群体为肿瘤群体。用细胞cytotracker染料(cmtmr)染色mm7-g原发性肿瘤细胞以区分nk-92细胞,并圈出肿瘤细胞。bc1ccarnk-92细胞毒活性总结在条形图中(31b)。(31c)使用人工的表达bcma之k562(bcmaxpk562)和表达cs1之k562(cs1xpk562)细胞测试bc1ccarnk-92细胞的抗原特异性活性。以5:1的e:t比例进行4小时共培养。k562群体预先用cmtmr染色并在流式细胞分析图中圈出。条形图总结了抗肿瘤活性。
图32a-32c.不同baff-car构筑体的产生和表征。(32a)l45-baff-28car表达在t细胞表面。将l45-baff-28car转导到t细胞中并使用f(ab)’抗体评估表面表达。将闸控后之群体与对照比较。(32b)car表达取决于前导序列。测试使用不同前导序列之baff-car构筑体以确定转导效率是否可以改善。使用f(ab)’抗体以评估car表达,圈出之群体为转导后群体。(32c)car表达取决于构筑体设计。包含不同前导序列和构筑体设计(附加单位)的baff-car构筑体被验证并用于确认是否可以改善car转导效率。与对照t细胞相比,闸控后圈出的群体为转导后的群体。csf-baff-2841bbl是具有csf前导序列之共表达4-1bbl(41bbl)之baffcar。csf-baff-28il-15ra是与csf前导序列共表达il-15/il-15sushi(il-15ra)的baffcar。
图33.l45-baff-28cart抗肿瘤特性之表征。l45-baff-28cart细胞具有针对mm1s肿瘤细胞之抗肿瘤活性。将l45-baff28cart细胞以3:1的e:t比例针对mm1s骨髓瘤细胞培养48小时。显示复制实验之样品。条形图总结了细胞毒活性。
图34a-34b.使用不同之baff-car构筑体及增强子之抗肿瘤活性之表征。(34a)baff-car构筑体针对mm1s细胞。将l8-baff-28il-15/il-15sushi和l8-baff-2841bblcar以5:1的e:t比例针对mm1s肿瘤细胞培养24小时。圈出之群体为肿瘤细胞。(34b)baff-car构筑体针对sp53细胞。以上两种car和l45-baff-28car均以5:1的e:t比例针对sp53肿瘤细胞(b谱系)培养24小时。(34c)细胞毒活性之总结条形图。
图35.显示ccar构筑体之示意图。该构筑体由sffv启动子组成,该启动子驱动由p2a肽连接之两个模块car单元之表达。在切割该p2a肽后,ccar分裂并与表达bcma和/或cd19的靶标结合。两个单元car使用不同或相同之共刺激结构域。共刺激结构域可以是,但不限于4-1bb或cd28。
图36a-36b.bcmacar单元之表征。(36a)bcmacar有效地消耗bcma+mm1s细胞。将bcmacar转导入t细胞并与mm1s肿瘤细胞一起培养。生成cs1car以用于稳健性。mm1s细胞具有bcma和cs1双阳性。用bcma和cs1抗体进行共培养48小时,以鉴定肿瘤中心位置。圈出的群体代表培养后残留的mm1s肿瘤细胞。(36b)评估bcmacar和cs1car对原发性mm7-g骨髓瘤患者细胞之抗肿瘤特性。mm7-g群体是多数是bcma+cs1+群体,还具有少数但显著的cs1+群体。bcmacar和cs1car一起用于评估细胞毒性,celltracker(cmtmr)用于区分肿瘤细胞群和car细胞。
图37a-37c.cd19car之表征。(37a)cdcar单元之设计。(37b)蛋白印迹法。用对照载体(道1)和cd19-2g(道2)慢病毒质粒转染hek-293t细胞。转染48小时后,除去上清液并收获细胞。溶解细胞以用于蛋白印迹法,并用小鼠抗人cd3z一抗和山羊抗小鼠hrp二抗探测。用抗cd3抗体将pmbc白血球层t细胞活化3天。用对照载体(左)和l8-cd19-2g(右)慢病毒上清液转导细胞。培养3天后,收获细胞并与缀合生物素的山羊抗小鼠fab2或山羊igg抗体一起培养30分钟。洗涤及悬浮细胞,并用链霉亲和素-pe和小鼠抗人cd3-percp染色30分钟。洗涤细胞后将细胞悬浮在2%福尔马林中,并通过流式细胞术分析确定car效率。(n=2)
图38a-38b.使用不同前导序列之复合cd19cart细胞之表达。(38a)设计car构筑体以表达具有不同前导序列之融合蛋白。(38b)用抗cd3抗体将pmbc白血球层t细胞活化3天。用对照载体(左),ha-cd19-2g(上中),il2-cd19-2g(右上),l8-cd19-2g(左中下)l45-cd19-2g(下中右)或csf-cd19-2gcar(右下)慢病毒上清液转导细胞。培养3天后,收获细胞并与缀合生物素之山羊抗小鼠fab2或山羊igg抗体一起培养30分钟。洗涤及悬浮细胞,并用链霉亲和素-pe和小鼠抗人cd3-percp染色30分钟。洗涤细胞后将细胞悬浮在2%福尔马林中,并通过流式细胞术分析确定car效率。(n=2)
图39a-39b.使用不同之cd19scfv序列在t细胞上表达cd19car。(39a)设计car构筑体以表达具有不同scfv序列的融合蛋白。(39b)用cd3抗体将pmbc白血球层t细胞活化3天。用对照载体(左),l8-cd19-2g(右上)或l8-cd19b-bb-2g(右)慢病毒上清液转导细胞。培养3天后,收获细胞并与缀合生物素之山羊抗小鼠fab2或山羊igg抗体一起培养30分钟。洗涤及悬浮细胞,并用链霉亲和素-pe和小鼠抗人cd3-perco染色30分钟。洗涤细胞后将细胞悬浮在2%福尔马林中,并通过流式细胞术分析以确定car效率。(n=2)
图40.l8-cd19-2g和cd19b-bbcart细胞在过夜共培养中溶解sp53肿瘤细胞。用对照(左),l8-cd19-2g(中)或l8-cd19b-bb-2g(右)慢病毒上清液转导之活化pmbct细胞与sp53细胞以2:1(上)和5:1(下)的效应细胞:靶细胞比例一起培养。在37摄氏度下培养24小时后,洗涤样品并用抗人cd3-percp和抗人cd19-apc染色,洗涤,并通过流式细胞术分析。单独sp53细胞样品和细胞溶解之总结在最右侧。(n=2)
图41.l8-cd19-2g和cd19b-bbcart细胞在过夜共培养中溶解jeko-1肿瘤细胞。用对照(左),l8-cd19-2g(中)或l8-cd19b-bb-2g(右)慢病毒上清液转导之活化pmbct细胞与jeko-1细胞以2:1(上)和5:1(下)的效应细胞:靶细胞比例一起培养。在37摄氏度下培养24小时后,洗涤样品并用抗人cd3-percp和抗人cd19-apc染色,洗涤,并通过流式细胞术分析。单独jeko-1细胞样品和细胞溶解之总结在最右侧。(n=2)
图42.l8-cd19-2g和l8-cd19b-bb-2gcart细胞在过夜共培养中溶解aml患者细胞。用对照(左),l8-cd19-2g(中)或l8-cd19b-bb-2g(右)慢病毒上清液转导之活化pmbct细胞与来自aml患者之已用cmtmr染色之细胞以2:1(上)和5:1(下)的效应细胞:靶细胞比例一起培养。在37摄氏度下培养24小时后,洗涤样品并用抗人cd3-percp和抗人cd19-apc染色,洗涤,并通过流式细胞术分析。单独患者细胞样品和细胞溶解之总结在最右侧。(n=2)
图43.l8-cd19-2g和l8-cd19b-bb-2gcart细胞耗尽cd19+患者细胞。用对照(左),l8-cd19-2g(中)或l8-cd19b-bb-2g(右)慢病毒上清液转导之活化pmbct细胞与来自b-all患者之已用cmtmr染色之细胞一起培养。l8-cd19-2gt细胞与患者细胞以1:1的比例过夜培养(上),而l8-cd19b-bb-2gt细胞与患者细胞以5:1的比例培养40小时(下)。在37摄氏度下培养后,洗涤样品并用抗人cd3-percp和抗人cd19-apc染色,洗涤,并通过流式细胞术分析。单独患者细胞样品和细胞溶解之总结在最右侧。(n=2)
图44.显示ccar构筑体之示意图。该构筑体由sffv启动子组成,该启动子驱动由p2a肽连接之两个模块car单元之表达。在切割该p2a肽后,ccar分裂并与表达bcma和/或cd19b的靶标结合。两个单元car使用不同或相同之共刺激结构域。共刺激结构域可以是,但不限于4-1bb或cd28。
图45a-45c.不同baff-car构筑体之产生和表征。(45a)将l45-baff-28car转导至t细胞中,并使用f(ab)’抗体评估表面表达。将闸控后的群体与对照进行比较。(45b)测试使用不同前导序列之baff-car构筑体以确定转导效率是否可以改善。使用f(ab)’抗体以评估car表达,圈出之群体为转导后群体。(45c)包含不同前导序列和构筑体设计(附加单位)的baff-car构筑体被验证并用于确认是否可以改善car转导效率。与对照t细胞相比,闸控后圈出的群体为转导后的群体。csf-baff-2841bbl是具有csf前导序列之共表达4-1bbl(41bbl)之baffcar。csf-baff-28il-15ra是与csf前导序列共表达il-15/il-15sushi(il-15ra)的baffcar。
图46a-46b.l45-baff-28cart细胞具有针对mm1s肿瘤细胞的抗肿瘤活性。l45-baff-28cart抗肿瘤特性之表征。(45a)体外baffcar细胞毒活性总结自(46b)。(46b)l45-baff-28cart细胞具有针对mm1s肿瘤细胞的抗肿瘤活性。将l45-baff-28cart细胞以3:1的e:t的比例针对mm1s骨髓瘤细胞培养48小时。显示复制实验之样品。
图47a-47b.使用不同构筑体和增强子的baff-car抗肿瘤活性之表征。(47a)l8-baff-28il-15/il-15sushi和l8-baff-284-1bblcar以5:1的e:t比例针对mm1s肿瘤细胞培养24小时。圈出的群体为肿瘤群体。(47b)以上两种car和l45-baff-28car以5:1的e:t比例针对sp53肿瘤细胞(b谱系)培养24小时。
图48.crispr/cas9干扰系统。分别藉由u6及sffv促进剂驱使sgrna及cas9嘌呤霉素之表达。cas9藉由e2a自裂解序列与嘌呤霉素抗性基因连接。
图49a.产生靶向血液恶性肿瘤的cart或nk细胞的步骤。
图49b.使用crispr/cas9慢病毒系统之表达稳定cd45阻断基因nk-92细胞之产生及分选。流式细胞术分析指示nk-92细胞表面上之cd45表达量(左图)。在sgcd45bcrispr转导至nk-92细胞中之后,经转导之细胞在含有嘌呤霉素之培养基中培养数周。使用cd45抗体测定cd45阴性nk-92细胞且进行分选。藉由流式细胞术分析测定稳定nk45i-92(表达cd45阻断基因)nk-92细胞之纯度(右图)。此资料证实成功产生及获得nk45i-92细胞。
图50.野生型、经gfp转导之nk-92或nk45i-92nk细胞之细胞生长曲线。为了评估nk-92细胞中由cd45阻断基因表达(kd)引起之细胞增殖作用,在接种至24孔板中之后第48小时及第96小时对nk-92(●)、经gfp转导之-92(■)及nk45i-92(▲)细胞之数目进行计数。在第48小时时间点时添加il-2(一式两份地进行n=3独立实验)。资料为平均值±s.d.。此等资料指示nk-92上cd45受体之阻断基因表达与未经转导之nk-92或经gfp转导之nk-92细胞相比展示类似的细胞生长曲线。重复在48小时时间点加il-2入24孔,n=3。
图51a-51b.用ccrf-cem(标靶:t)及gfpnk-92或gfpnk45i-92细胞(效应子:e),5:1(e:t)比率进行之共培养分析。16小时培育。(a)对仅ccrf-cem(左图中之蓝点)、在与ccrf-cem及对照性经gfp转导之nk-92细胞(中间图)或gfpnk45i-92细胞(右图)之共培养之流式细胞术分析。所有图中之蓝点指示残余标靶ccrf-cem细胞且红点展示共培养分析法之效应细胞。每个实验大多数之蓝点位于图的左上方。培养时间为6小时,效应t细胞:靶细胞之比例为5:1。所有实验均一式两份进行。(51b)此等资料表明与gfp对照性nk-92细胞活体外共培养分析法相比,nk-92细胞中cd45之阻断基因表现在针对ccrf-cem细胞之杀伤活性方面没有显著差异。
图52a-52b.将ccrf-cem(标靶:t)及gfpnk-92、cd5carnk-92或cd5carnk45i-92细胞(效应子:e)以5:1(e:t)比率进行16小时培育之共培养分析法。(a)自右向左,对仅ccrf-cem(左图)、在与ccrf-cem及对照性gfpnk-92细胞(中间左图)、cd5carnk-92细胞(中间右图)、cd5carnk45i-92细胞(右图)之共培养之流式细胞术分析。所有图中之蓝点指示残余标靶ccrf-cem细胞且红点展示共培养分析法之效应细胞。总培育时间为16小时且效应子t细胞:标靶细胞之比率为5:1。一式两份地进行所有实验。(b)柱状图指示在用ccrf-cem进行之共培养分析法中,cd5carnk-92细胞或cd5carnk45i-92细胞与对照性gfpnk92细胞相比之细胞溶解百分比。资料为平均值±s.d.。与对照性gfpnk-92细胞相比,cd5carnk细胞及cd5carnk45i-92细胞皆展示针对cd5阳性ccrf-cem之约100%细胞杀伤活性。此等资料表明与gfp对照性nk-92细胞活体外共培养分析法相比,cd5carnk细胞及cd5carnk45i-92细胞可有效地溶解表达cd5之ccrf-cem细胞,且提供cd45之阻断基因表达不影响nk-92细胞中关于杀伤活性之细胞功能的证据。
图53a-53b.cd45car构筑体之组织及其表达。(a)cd45car慢病毒载体(lentiviralvector)之示意图。cd45car构筑体为模组化信号传导域,其含有:前导序列、抗cd45scfv、铰链域(h)、跨膜域(tm)、两个定义构筑体为第3代car之共刺激域(cd28及4-1bb)及胞内信号传导域cd3ζ。(b)hek-293ft细胞用gfp(道1)及cd45car(道2)之慢病毒质体转染。在转染之后48小时,移出上清液,且亦移出细胞。细胞与小鼠抗人类cd3z抗体一起溶解以用于蛋白印迹法及调查。
图54.将cd45car转导至nk45i-92细胞中且将经cd45car转导之细胞进行细胞分选。在cd45car慢病毒转导至nk45i-92细胞中之后,与nk45i-92细胞(左图)相比,藉由流式细胞术分析测定之nk45i-92上cd45car之表达量(中间图中之蓝圈)。分选表达cd45car之nk45i-92细胞且藉由流式细胞术分析测定细胞表面上之cd45表达量(右图)。藉由流式细胞术分析侦测到细胞表面上约87%cd45car表达。
图55a-55b.用ccrf-cem(标靶:t)及gfpnk-92或cd45carnk45i-92细胞(效应子:e)进行共培养分析。5:1(e:t)比率。16小时培育。(a)对与ccrf-cem及对照性经gfp转导之nk-92细胞(左图)或cd45carnk45i-92细胞(右图)之共培养之流式细胞术分析。所有图中之蓝点指示残余标靶ccrf-cem细胞且红点展示共培养分析法之效应子nk-92细胞。总培育时间为16小时且效应子t细胞:标靶细胞之比率为5:1。一式两份地进行所有实验。(b)柱状图指示在用ccrf-cem进行之共培养分析法中,与对照性gfpnk92细胞相比,cd45carnk45i-92细胞之细胞溶解百分比。资料为平均值±s.d.。与对照性gfpnk-92细胞相比,cd45carnk45i-92细胞展示针对ccrf-cem细胞之约70%细胞溶解。此等资料表明与gfp对照性nk-92细胞活体外共培养分析法相比,cd45carnk45i-92细胞有效溶解表达cd45之ccrf-cem细胞。
图56a-56c.与jurkat细胞(标靶:t)及gfp对照组或cd45carnk45i-92细胞(效应子:e)之共培养分析。5:1或2:1(e:t)比率。6小时培育。(a)在用cmtmr细胞追踪剂染料将jurkat细胞染色之后,进行流式细胞术分析。此等资料证实jurkat细胞为cd45阳性(左图)且大部分为cd56阴性细胞(右图)。(56b和56c)对与jurkat细胞(标靶:t)及对照物或cd45carnk45i-92细胞(效应子:e)之共培养之流式细胞术分析。以5:1(56b)或2:1(56c)(e:t)之比例进行共培养分析。左图展示以5:1(e:t)比率进行之与对照性gfp或cd45car/cd45kdnk-92细胞之共培养且右图指示以2:1(e:t)比率进行之与对照性gfp或cd45carnk45i-92细胞之共培养。图中之蓝点指示残余标靶jurkat细胞且红点表示共培养分析法之效应细胞。总培育时间为6小时。一式两份地进行所有实验。(c)柱状图展示在5:1或2:1(e:t)比率下,与对照性gfpnk92细胞相比,cd45carnk45i-92细胞之细胞溶解百分比。资料为平均值±s.d.。在两种条件下,与对照性gfpnk-92细胞相比,cd45carnk45i-92细胞展示针对jurkat细胞之约60%细胞溶解。此资料表明与gfp对照性nk-92细胞活体外共培养分析法相比,cd45carnk45i-92细胞有效溶解在细胞表面上表达cd45之jurkat细胞。
图57a-57c.用gfp-nk-92细胞(标靶:t)及未经转导之nk-92细胞或cd45carnk45i-92细胞(效应子:e)进行之共培养分析法。5:1或2:1(e:t)比率。6小时培育(57a)使用gfp对照性nk-92细胞进行流式细胞术分析。此等资料证实gfp对照性nk-92细胞为约99%gfp阳性细胞(绿点)。(57b)对与gfp对照性nk-92细胞(标靶:t)及未经转导或cd45carnk45i-92细胞(效应子:e)之共培养分析法之流式细胞术分析。以5:1(57a)或2:1(e:t)进行共培养分析法之比率。左图展示以5:1(e:t)比率进行之与未经转导或cd45carnk45i-92细胞之共培养且右图指示以2:1(e:t)比率进行之与未经转导或cd45carnk45i-92细胞之共培养。图中之绿点指示残余标靶gfpnk-92细胞且红点表示共培养分析法之效应细胞。培育时间为6小时。一式两份地进行所有实验。(57c)柱状图展示在5:1或2:1(e:t)比率下,与未经转导之nk-92细胞相比,由cd45carnk45i-92细胞进行之gfpnk-92细胞之细胞溶解百分比。资料为平均值±s.d.。与未经转导之nk-92细胞相比,针对gfpnk-92细胞,cd45carnk45i-92细胞在2:1(e:t)比率下展示约20%细胞溶解且在5:1(e:t)比率下展示约55%细胞溶解。此资料表明与未经转导之nk-92细胞活体外共培养分析法相比,cd45carnk45i-92细胞有效溶解在细胞表面上表达cd45之gfpnk-92细胞。
图57d.将cd45b-bb或cd45b-28转导至nk45i-92细胞中且进行经cd45b-bb或cd45b-28转导之nk45i-92细胞之细胞分选。cd45b-bb之共刺激结构域是4-1bb,而cd45b-28之共刺激结构域是cd28。在cd45b-bb或cd45b-28慢病毒转导至nk45i-92细胞中之后,与nk45i-92细胞(左图)相比,藉由流式细胞术分析来测定nk45i-92上cd45b-bbcar或cd45b-28car之表面表达量(中间图中之蓝圈)。藉由流式细胞术分析分选表达cd45b-bb或cd45b-28car之nk45i-92细胞及表达水平。藉由流式细胞术分析侦测到细胞表面上约74%cd45b-bbcar或82%cd45b-28car表达。
图57e.与reh细胞(标靶:t)及gfpnk-92细胞或cd45carnk45i-92细胞或cd45b-bbnk45i-92细胞或cd45b-28nk45i-92细胞(效应子:e)之共培养分析法。5:1(e:t)比率。20小时培育。对仅reh细胞(左图)、与reh细胞及对照性经gfp转导之nk-92细胞(左侧第2个图)、cd45carnk45i-92细胞(中间图)、cd45b-bbnk45i-92细胞(左侧第4个图)或cd45b-28nk45i-92细胞(右图)之共培养之流式细胞术分析。所有图中之蓝点指示残余标靶reh细胞且红点展示共培养分析法之效应子gfp或car-nk-92细胞。reh为b急性淋巴细胞白血病细胞系。总培育时间为20小时且效应子nk-细胞:标靶细胞之比率为5:1。一式两份地进行所有实验。柱状图指示在用reh细胞进行之共培养分析法中,与对照性gfpnk92细胞相比,cd45carnk45i-92细胞、cd45b-bbnk45i-92细胞或cd45b-28nk45i-92细胞之细胞溶解百分比。资料为平均值±s.d.。与对照性gfpnk-92细胞相比,针对reh细胞,cd45carnk45i-92细胞展示约76%细胞溶解,cd45b-bbnk45i-92细胞展示约79%细胞溶解且cd45b-28nk45i-92展示100%细胞溶解。这些数据表明与gfp对照nk-92细胞在体外共培养测定相比,这三种cd45carnk45i-92细胞有效溶解reh细胞,其表征为表达cd45之b细胞。
图57fa-57fi.将u937细胞(标靶:t)与gfpnk-92细胞或cd45-28nk45i-92细胞以2:1(e:t)比率进行20小时共培养测定。(57fa)对单独u937细胞(单核细胞白血病细胞系)进行流式细胞术分析(左图),将u937细胞与gfp转导之对照nk-92细胞(中图)或cd45b-28nk45i-92细胞(右图)共培养。所有图中之蓝点代表剩余之u937靶细胞,红点代表效应gfp细胞或cd45b-28nk45i-92细胞。培养时间为6小时,效应nk细胞:靶细胞之比例为2:1。(57fb)条形图显示与u937共培养测定中的对照gfpnk-92细胞相比,cd45b-28nk45i-92细胞之细胞溶解百分比。与对照gfpnk-92细胞相比,cd45b-28nk45i-92细胞对u937细胞展示约81%细胞溶解。
图57ga-57gb.将molm-13细胞(靶标:t)与gfpnk-92细胞或cd45b-28nk45i-92细胞以5:1(e:t)比例进行20小时共培养测定。(57ga)对单独molm13细胞(单核细胞白血病细胞系)进行流式细胞术分析(左图),将molm13细胞与gfp转导之对照nk-92细胞(中图)或cd45b-28nk45i-92细胞(右图)共培养。所有图中之蓝点代表剩余的molm13靶细胞,红点代表效应gfp细胞或cd45b-28nk45i-92细胞。培养时间为20小时,效应nk细胞:靶细胞之比例为5:1。(57gb)条形图显示与molm13共培养测定中的对照gfpnk92细胞相比,cd45b-28nk45i-92细胞之细胞溶解百分比。与对照gfpnk-92细胞相比,cd45b-28nk45i-92细胞对molm13细胞展示约91.6%细胞溶解。
图57ha-57hb.将jeko-1细胞(靶标:t)及gfpnk-92细胞或cd45b-28nk45i-92细胞以2:1(e:t)比例进行6小时共培养测定。(57ha)对单独jeko-1细胞(套细胞淋巴瘤)进行流式细胞术分析(左图),将jeko-1细胞与gfp转导之对照nk-92细胞(中图)或cd45b-28nk45i-92细胞(右图)共培养。所有图中之蓝点代表剩余的jeko-1细胞,红点代表效应gfp细胞或cd45b-28nk45i-92细胞。培养时间为6小时,效应nk细胞:靶细胞比例为2:1。(57hb)条形图显示与jeko-1细胞共培养测定中的对照gfpnk92细胞相比,cd45b-28nk45i-92细胞之细胞溶解百分比。与对照gfpnk-92细胞相比,cd45b-28nk45i-92细胞对jeko-1细胞展示约44.6%细胞溶解。
图57ia-57ib.将sp53细胞(靶标:t)以及gfpnk-92细胞或cd45b-28nk45i-92以2:1(e:t)比例进行6小时共培养测定。(57ia)对单独sp53细胞(套细胞淋巴瘤细胞系)进行流式细胞术分析(左图),将sp53细胞与gfp转导之对照nk-92细胞(中图)或cd45b-28nk45i-92细胞(右图)共培养。所有图中之蓝点代表剩余的sp53靶细胞,红点代表效应gfp细胞或cd45b-28nk45i-92细胞。培养时间为6小时,效应nk细胞:靶细胞比例为2:1。(57ib)条形图显示与sp53细胞共培养测定中的对照gfpnk92细胞相比,cd45b-28nk45i-92细胞之细胞溶解百分比。与对照gfpnk-92细胞相比,cd45b-28nk45i-92细胞对sp53细胞展示约45%细胞溶解。
图57j.在48小时共培养中消除cd34(+)脐带血干细胞。将源自人脐带血的cd34(+)干细胞与对照或cd45b-28carnk细胞以2:1之低比例(效应细胞:靶标细胞)进行共培养48小时,然后标记细胞。与对照相比,cd45b-28carnk细胞消除了约96%的cd34(+)细胞。
图58a.通过p2a链接之单个构筑体中之ccar-t和4-1bbl或il-15/il-15sushi之示意图。该构筑体由驱动car表达之sffv启动子和增强子4-1bbl组成。当连接子裂解时,cd45car(或cd45bcar)与4-1bbl或il-15/il-15sushi分裂并与表达cd45之靶标结合。cd45cart细胞不仅通过cd28接受共刺激,还通过4-1bb配体(4-1bbl或cd137l)或il-15/il-15sushi。由cd3ζ信号传导结构域完整该car-t之组装。
图58b.使用流式细胞术分析测定cd45b-28-2g-4-1bblcar转导之nk45i-92细胞表面cd45bcar表达水平。左图(nk92细胞)和中图(gfp-nk92)展示阴性对照,右图展示cd45bcar之表面表达。其使用针对scfv区域之山羊抗小鼠f(ab’)2-pe标记(蓝色,圈出之群体)。转导之细胞在细胞表面表达86.99%之cd45b-car。
图58c.使用流式细胞术分析测定cd45b-28-2g-il-15/il-15sushicar转导之nk45i-92细胞表面cd45bcar表达水平。左图(nk92细胞)和中图(gfp-nk92)展示阴性对照,右图展示cd45bcar之表面表达。其使用针对scfv区域之山羊抗小鼠f(ab’)2-pe标记(蓝色,圈出之群体)。与阴性对照细胞相比,cd45b-28-2gil15ra病毒转导之细胞表面上表达55.96%的cd45b-car。
图59a-59b.用于阐明构筑体及其在t或nk细胞表达之示意图。(59a)将car(第三代)和il-15α受体之il-15/sushi结构域的组合装配在表达载体上,且其表达由sffv启动子驱动。用p2a自切割序列连接包含il-15/sushi之car。il-15/sushi部分由与il-15融合之il-2信号肽组成,并通过26个氨基酸的聚脯氨酸连接子与sushi结构域连接。(59b)car和il-15/sushi在t或nk细胞上。
图59c.使用流式细胞术分析测定cd45b-28-2g-il-15/il-15sushicar转导之nk45i-92细胞表面cd45bcar表达水平。左图(nk92细胞)和中图(gfp-nk92)展示阴性对照,右图展示cd45bcar之表面表达。其使用针对scfv区域之山羊抗小鼠f(ab’)2-pe标记(蓝色,圈出之群体)。与阴性对照细胞相比,cd45b-28-2g-il-15/il-15sushi病毒转导之细胞表面上表达55.96%的cd45b-car。cd45b-28-2g-il-15/il-15sushink细胞展示出强大之功能活性。
图60a-60b.cd4il-15/il-15sushi之表达。(60a)用gfp(道1)和cd4il-15/il-15sushicar(道2)的慢病毒质粒转染hek-293ft细胞。转染48小时后,除去上清液及细胞,并用小鼠抗人cd3z抗体进行蛋白印迹法。(60b)用源自转染hek-293ft细胞之gfp(左)或cd4il-15/il-15sushi(右)病毒上清液转导hek-293细胞。培养3天后,收获细胞,并用山羊抗小鼠f(ab’)2染色后通过流式细胞术分析。
图61.用cd4il-15/il-15sushicar转导nk细胞。用源自转染hek-293ft细胞之gfp(左)或cd4il-15/il-15sushicar(右)病毒上清液转导nk-92细胞。第一次转导后24小时进行第二次转导。在第二次转导后24小时收获细胞,洗涤并移至具有新鲜培养基和il-2的组织培养板。培养3天后,收获细胞并用山羊抗小鼠f(ab’)2抗体或山羊igg(对照)以1:250比例染色30分钟。洗涤细胞并用链霉亲和素-pe缀合物以1:500染色,洗涤,悬浮于2%福尔马林中,并通过流式细胞术分析。
图62.用cd4il15racar转导t细胞。左边为蛋白印迹法结果。用gfp(道1)和cd4il15ra-car(道2)之慢病毒质粒转染hek-293ft细胞。转染48小时后,除去上清液,收集细胞,用小鼠抗人cd3ζ抗体进行蛋白印迹法。右边为cd4il15racar之表达。用gfp(左)或浓缩之cd4il14racar(右)病毒上清液转导来自脐带血白血球层之已活化之t细胞。第一次转导后24小时进行第二次转导。在第二次转导后24小时,收获细胞,洗涤并移至具有新鲜培养基和il-2的组织培养板。培养3天后,收获细胞并用山羊抗小鼠f(ab’)2染色。洗涤细胞并用链霉亲和素-pe缀合物以1:500染色,洗涤,悬浮于2%福尔马林中,并通过流式细胞术分析。
图63a-63b.cd4carnk-92细胞和cd4il-15/il-15sushicarnk细胞在共培养中消除karpas299t白血病细胞。(63a)将gfp对照(右上),cd4car(左下)或cd4il-15/il-15sushi(右下)慢病毒上清液转导之nk-92细胞与karpas299细胞以5:1比例一起培养。共培养4小时后,用小鼠抗人cd(apc)和cd3(percp)抗体将细胞染色,并通过流式细胞术分析(n=2)。左上图显示单独标记之karpas299细胞。溶解之靶细胞百分比显示在图(63b)中。
图64.cd4carnk-92细胞和cd4il-15/il-15sushicarnk-92细胞在共培养中消除molt4t白血病细胞。将gfp对照(左),cd4car(中)或cd4il-15/il-15sushi(右二)慢病毒上清液转导之nk-92细胞与molt4细胞一起培养,效应细胞:靶细胞比例为1:1或2:1。在过夜共培养后,用小鼠抗人cd4(apc)和cd56(percp)抗体将细胞染色,并通过流式细胞术分析(n=2)。右上图显示单独标记之molt4细胞。溶解之靶细胞百分比显示在图中。
图65a-65b.cd4car和cd4il-15/il-15sushicart细胞在体内表现出抗白血病作用。对nsg小鼠进行亚致死照射并在静脉(尾静脉)内注射表达荧光素酶之molm13细胞以诱导可测量之肿瘤形成(65a)。molm-13细胞几乎是100%cd4阳性。3天后,在小鼠静脉内注射一个疗程的8x106个cd4car,cd4il-15/il-15sushicart细胞,或对照载体t细胞。在第3,6,9和11天,给小鼠皮下注射redijectd-luciferin并进行ivis成像(65b)。
图65c-65d.(65c)将测量之cd4car和cd4il-15/il-15sushicart小鼠之平均光强度与对照载体t小鼠之平均光强度进行比较,以剩余之肿瘤负荷确定溶解百分比。(65d)测量并比较三组小鼠之存活百分比。
图66a-66b.cd4il-15/il-15sushicarnk细胞在体内应激条件下表现出强大之抗白血病活性。第二天对nsg小鼠进行亚致死照射并在静脉(尾静脉)注射表达荧光素酶之jurkat细胞以诱导可测量之肿瘤形成(66a)。jurkat细胞具有低于60%之cd4+。3天后,给小鼠静脉内注射一个疗程之8x106cd4car,cd4il-15/il-15sushicarnk细胞,或载体对照nk细胞。在第3,7,10和14天,给小鼠皮下注射redijectd-luciferin并进行ivis成像(66b)。
图66c.将测量之注射cd4car和cd4il-15/il-15sushink细胞之小鼠之平均光强度与注射载体对照nk细胞之小鼠之平均光强度进行比较,以剩余之肿瘤负荷确定溶解百分比。
图67.重复体内实验,证明cd4il-15/il-15sushicarnk细胞对jurkat肿瘤细胞之溶解之稳健性,展示了与图66类似之结果。
图68a-68b.分泌之il-15/il-15sushi对car和未转导的相邻细胞之影响。将稳定表达cd4car或cd4il15ra的nk-92细胞以50:50之比例与稳定表达gfp之nk-92细胞混合。将这些细胞在添加了il-2或不添加il-2之培养基内共培养。(68a)在第0天(共培养开始)和第7天在20x荧光显微镜下拍摄之照片,没有添加il-2。(68b)在整个实验(直至第14天)中,与il-2共培养或不与il-2共培养的nk-92细胞之总细胞计数。
图69.比较分泌之il-15及il-15/il-15sushi对nk-92细胞生长之影响。在没有il-2的情况下,cd4il-15/il-15sushi,cdil-15和对照转导之nk-92细胞在常规nk细胞培养基下从250,000个细胞开始培养6天。两种转导之细胞均具有10%表面car表达,而cd4il-15/il-15sushi转导之nk-92细胞能够在第6天比cd4il-15转导之nk-92细胞高约3倍之速度扩增。在第4天,cd4il-15转导之nk-92细胞之生长速率略高于对照,但显著低于cd4il-15/il-15sushi转导之nk-92细胞。该研究指出了il-15/il-15sushi之共表达功能性复合物在促进nk-92细胞生长中之重要性。
图70.显示靶向tregs之tregcart构筑体示意图。该构筑体由sffv启动子组成,该启动子驱动由p2a肽连接之两个嵌合抗原受体之表达。每个单元含有cd45前导肽序列(信号肽)。在连接子裂解后,两个肽单元分开并与表达cd4和cd25之靶标结合。cd4嵌合抗原受体多肽单元包括信号肽,cd4抗原识别结构域,铰链区,跨膜结构域和cd3ζ链;cd25嵌合抗原受体多肽单元包括信号肽,cd25抗原识别结构域,铰链区,跨膜结构域,及共刺激结构域。tregcar可以增强共表达cd4和cd25细胞之溶解活性,同时使携带cd4或cd25抗原之细胞最少化。
图71a-71b.cd4zetacd25car之表征。(71a)通过病毒培养将cd4zetacd25car转导入t细胞48小时,并用f(ab)’抗体染色以测定car表面表达。圈出之群体代表转导后之细胞。(71b)使用cd4和cd25抗体表征c4-25zcar以验证构筑体功能。圈出之两个最相关之群体:cd4+cd25+和cd4-cd25+。双阳性群体和其他表型组之消耗总结在条形图中。
图72.cd4zetacd25cart细胞主要靶向共表达cd4和cd25之细胞。活化3天后,收获用对照载体(左),cd4car(中)或cd4zetacd25(右)慢病毒上清液转导之pmbc白血球层t细胞,并与小鼠抗人cd25-pe和小鼠抗人cd4-apc一起培养30分钟。洗涤细胞并将细胞悬浮于2%福尔巴林后,通过流式细胞术分析。
图73a.显示cd5car-52构筑体之示意图。该构筑体由驱动cd5car和cd52表面抗原共表达之sffv启动子组成。cd5嵌合抗原受体多肽单元包括信号肽,cd5抗原识别结构域,铰链区,跨膜结构域,cd28之共刺激结构域和cd3ζ链;cd52肽包括信号肽,cd52抗原识别结构域(抗cd52scfv),铰链区,跨膜结构域(衍生自cd28)。
图73b.确定血液中cd5car-52t细胞耗竭之实验设计。在亚致死照射后,将cd5car-52t细胞(5x106个细胞)静脉内注射刀妹纸nsg小鼠中。约24小时后,通过i.p.(腹腔注射)注射pbs或0.1mg/kgcampath。n=3.6小时和24小时后,从每只小鼠收集外周血并用cd3和cd45抗体标记,以确定通过campath处理作为急性期反应之car-t细胞之消耗。5天后,从每只小鼠收集全血并用cd3和cd45抗体标记以确定car-t细胞之持久性。使用流式细胞术分析以测定car-t细胞。
图73c.有或没有campath治疗后在6小时和24小时内外周血中cd5car-52t之消耗。流式细胞术分析显示在有或没有campath处理之小鼠外周血中cd5car-52t细胞(蓝点)之持久性。用cd3和cd45抗体标记血液样品以检测cd5car-52t细胞。未输注之car-t细胞(左图)之血液样品未显示cd3和cd45阳性细胞(阴性对照)。与未注射campath小鼠之6小时(左侧第二张图)和24小时(右侧第二张图)血液样品相比,0.1mg/kg注射campath小鼠显示在6小时(中图)和24小时(右图)时消除cd5car-52t细胞。n=3.这些结果表明campath治疗可以在短时间内从血液中消除car-t细胞。
图73d.在有或没有campath处理之情况下,5天后全血中cd5car-52t之消耗。流式细胞术分析显示来自具有或不具有campath处理之小鼠之全血样品中cd5car-52t细胞(蓝点)之持久性。用cd3和cd45抗体标记血液样品以检测cd5car-52t细胞持久性。未输注之car-t细胞之血液样品(左图)未显示cd3和cd45阳性细胞(阴性对照)。与未注射campath之小鼠之血液样品(中图)相比0.1mg/kgcampath处理之小鼠(右图)消除了cd5car-52t细胞。这些结果表明campath治疗可以从血液中消除car-t细胞。
图74.在6孔组织培养盘中,在具有10%fbs之dmem中,使用所指示之体积,hek293细胞用ef1-gfp或sffv-gfp病毒上清液转导。在第二天早晨更换培养基。四十八小时之后,在10×下使用gfp在evos萤光显微镜上观测经转导之细胞。
图75.使用来自前述图之体积,使经ef1-gfp或sffv-gfp病毒上清液转导之hek293细胞胰蛋白酶化,悬浮于福马林中且用流式细胞术分析,使用fitc通道以测定gfp+细胞之百分比。
图76a-76b.在转导之后第7、14、21及28天,在少量或大量病毒上清液情况下,使经ef1-gfp或sffv-gfp病毒上清液转导之经活化之脐带血白血球层t细胞胰蛋白酶化,悬浮于福马林中且用流式细胞术分析,使用fitc通道以测定gfp+细胞之百分比。(76a)在少量或大量上清液情况下经转导之细胞之gfp+t细胞百分比。(76b)相对于在少量sffv-gfp上清液情况下经转导之t细胞中之gfp+细胞百分比,在大量ef1-gfp上清液情况下经转导之gfp+t细胞之百分比。(使用50μlsffv-gfp及1mlef1-gfp上清液)。(n=2)。
图77.恶性浆细胞中之配位体受体相互作用。april配位体结合tac1或bcma。baff配位体结合tac1、bcma或baff-r。
图78.通过共表达分泌型il-15/il-15sushi之car消除肿瘤之步骤。(i)肿瘤及其微环境。巨噬细胞,t细胞,树突细胞和nk细胞是肿瘤微环境中针对肿瘤之免疫应答细胞,他们分泌低水平之不稳定的内源性il-15,其与il-15ra的可溶性细胞外结构域合成。该复合物形成更稳定的分子,极大增强了免疫细胞之存活和扩增。在肿瘤微环境中,癌细胞表达程序性死亡配体-1(pd-1)作为跨膜蛋白,其被认为在包括癌症内之特定事件期间在抑制免疫系统中起主要作用。pd-l1与其t细胞,b细胞和骨髓细胞上发现之受体pd-1结合,以抑制这些细胞免疫活性。(ii)靶向肿瘤细胞之cart或nk细胞可以是向肿瘤微环境递送增强子之载体。将cart或nk细胞工程改造以共表达分泌型融合蛋白,il-15/il-15sushi融合体。(iii)工程改造之cart或nk细胞与肿瘤靶细胞(亚组或所有细胞)结合。(iv)肿瘤微环境中之工程改造之cart或nk细胞靶向肿瘤细胞,与car靶向抗原结合,并引发肿瘤细胞溶解和通过扩增cart或nk细胞以大量分泌可溶性il-15/il-15sushi融合体。可溶性il-15/il-15sushi融合体是稳定的,且具有cart/nk细胞及其相邻肿瘤免疫应答细胞之意外且强大之免疫调节作用。分泌之il-15/il-15sushi蛋白将参与其他t细胞,树突细胞,巨噬细胞和nk细胞向肿瘤微环境之运输,其还:1)通过补充无法消除非靶向癌细胞之cart或nk细胞之缺陷溶解肿瘤细胞;2)增强cart/nk细胞之持久性和抗肿瘤活性。il-15/il-15sushi之过量表达压倒了pd-l1抑制免疫应答之能力。该car疗法可与检查点阻断之使用协同使用,包括但不限于pd-l1,ctla-4抑制剂,以获得更好之功效。
图79.显示了b细胞和浆细胞发育过程中之表面标记。baff和april均与受体,bcma和taci结合。baff还与baff-r受体结合。
图80.不同物种间il-2信号肽之蛋白序列之比对。
图81.不同物种间baff细胞外结构域之蛋白序列之比对。
【实施方式】
本发明提供嵌合抗原受体(car)组合物,其制造及使用方法。
嵌合抗原受体(car)多肽包括信号肽、抗原识别域、铰链区、跨膜域、至少一个共刺激域及信号传导域。
第一代car包括cd3z作为胞内信号传导域,而第二代car包括来源于多种蛋白质之至少一个单一共刺激域。共刺激域之实例包括(但不限于)cd28、cd2、4-1bb(cd137,亦称为「4-bb」)及ox-40(cd124)。第三代car包括两个共刺激域,诸如(但不限于)cd28、4-1bb、cd134(ox-40)、cd2及/或cd137(4-1bb)。
如本文中所使用,术语「肽」、「多肽」及「蛋白质」可互换使用,且是指具有由肽键共价连接之胺基酸残基之化合物。蛋白质或肽必须含有至少两个胺基酸,且对可包括蛋白质或肽序列之胺基酸最大数目无限制。多肽包括任何具有两个或更多个藉由肽键彼此接合之胺基酸之肽或蛋白质。如本文所使用之术语是指短链(其在此项技术中亦通常称为例如肽、寡肽及寡聚物)及长链(其在此项技术中通常称为蛋白质,其存在多种类型)。「多肽」尤其包括例如生物活性片段、实质上同源多肽、寡肽、均二聚体、杂二聚体、多肽变异体、经修饰之多肽、衍生物、类似物、融合蛋白。多肽包括天然肽、重组型肽、合成肽或其组合。
「信号肽」包括肽序列,其引导细胞内肽及任何连接之多肽之传递及定位,例如传递及定位至某一细胞器(诸如内质网)及/或细胞表面。如本文中所使用,「信号肽」及「前导序列」可互换使用。
信号肽为任何分泌或跨膜蛋白之肽,其引导所揭示之多肽传递至细胞膜及细胞表面,且提供本发明之多肽之正确定位。特定言之,本发明之信号肽将本发明之多肽引导至细胞膜,其中多肽之细胞外部分在细胞表面上显示,跨膜部分横跨质膜,且活性域位于细胞质部分或细胞之内部中。
在一个实施例中,信号肽在通过内质网(er)之后裂解,亦即为可裂解之信号肽。在一个实施例中,信号肽为i、ii、iii或iv型人类蛋白质。在一个实施例中,信号肽包括免疫球蛋白重链信号肽。
在一个实施例中,信号肽包括来自的人类cd45之信号肽。(uniprotkb/swiss-prot寄存编号p08575)。cd45信号肽长度为23个氨基酸(mylwlkllafgfafldtevfvtg)。在一些实施例中,信号肽可以是cd45信号肽之功能性片段。功能性片段包括cd45信号肽的至少10个氨基酸片段,其中将所附多肽指向细胞膜和细胞表面。人类cd45信号肽片段的实例包括:mylwlkllafg,fafldtevfvtg和lkllafgfafldte。
我们还考虑了人类cd45信号肽之功能同等物。如本文中所使用,「功能同等物」应该理解为突变体,其在至少一个上述序列位置中显示除了特别提及的氨基酸取代物之外的氨基酸取代物,但仍导致显示与野生型cd45肽相同或相似特性之突变体。功能等同物包括与人类cd45信号肽,其功能性片段或其功能同等物具有至少80%,至少85%,至少90%或至少95%相同之多肽。
功能同等物还包括来自其他物种之同源蛋白之cd45信号肽。这些信号肽的实例包括来自小鼠cd45之信号肽(mglwlkllafgfalldtevfvtg);来自大鼠cd45之信号肽(mylwlkllafslallgpevfvtg);来自绵羊cd45之信号肽(mtmylwlkllafgfafldtavsvag);来自黑猩猩cd45之信号肽(mylwlkllafgfafldtevfvtg);来自猴子cd45之信号肽(mtmylwlkllafgfafldtevfvag)。
在另一个实施例中,信号肽包括一下序列:mx1lwlkllafx2x3ax4lx5x6x7vx8vx9g;其中x1,x2,x3,x4,x5,x6,x7,x8,和x9为独立的y,g,s,f,l,d,p,t,e,或a。在一个实施例中,x1为y或g;x2为g或s;x3和x4为独立的f或l;x5为d或g;x6为p或t;x7为e或a;x8为f或s;和x9为a或t。
在一个实施例中,信号肽包括来自人类cd8a(malpvtalllplalllhaarp)。在一些实施例中,信号肽可以是cd8a信号肽之功能性片段。功能性片段包括至少10个氨基酸之cd8a信号肽片段,其将所附多肽指向细胞膜和细胞表面。人类cd8a信号肽片段实例包括:malpvtalllplalllhaa,malpvtalllp,pvtalllplall和lllplalllhaarp。
在另一个实施例中,信号肽包括来自人类cd8b之信号肽(mrprlwlllaaqltvlhgnsv)。在一些实施例中,信号肽可以是cd8b信号肽之功能性片段。功能性片段包括至少10个氨基酸之cd8b信号肽片段,其将所附多肽只想细胞膜和细胞表面。人类cd8b信号肽片段实例包括:mrprlwlllaaq,rlwlllaaqltvlhg和lwlllaaqltvlhgnsv。
我们还考虑了人类cd8a或cd8b信号肽之功能等同物。如本文中所使用,「功能同等物」应该理解为突变体,其在至少一个上述序列位置中显示除了特别提及的氨基酸取代物之外的氨基酸取代物,但仍导致显示与野生型cd8a或cd8b肽相同或相似特性之突变体。功能等同物包括与人类cd8信号肽,其功能片段或其功能等同物具有至少80%,至少85%,至少90%或至少95%相同之多肽。
功能等同物还包括来自其他物种之同源蛋白之cd8a和cd8b信号肽。
在一个实施例中,信号肽包括来自人类il-2之信号肽。il-2信号肽之长度为23个氨基酸(myrmqllscialslalvtns)。在一些实施例中,信号肽可以是il-2信号肽之功能性片段。功能性片段包括至少10个氨基酸之il-2信号肽片段,其将所附多肽指向细胞膜和细胞表面。人类il-2信号肽片段之实例包括:myrmqllscial,qllscialslal和scialslalvtns。
我们还考虑了il-2信号肽之功能等同物。如本文中所使用,「功能同等物」应该理解为突变体,其在至少一个上述序列位置中显示除了特别提及的氨基酸取代物之外的氨基酸取代物,但仍导致显示与野生型il-2信号肽相同或相似特性之突变体。功能等同物包括与人类il-2信号肽,其功能片段或其功能等同物具有至少80%,至少85%,至少90%或至少95%相同之多肽。
功能等同物还包括来自其他物种之同源蛋白之il-2信号肽,参见图80。
在另一个实施例中,信号肽包括一下序列:myx1x2qlx3scx4x5lx6lx7lx8x9x10x11;其中x1,x2,x3,x4,x5,x6,x7,x8,x9,x10,和x11为独立的r,k,s,m,i,v,l,a,i,t,n,s,或g。在一个实施例中,x1为r,k,或s;x2为m,i,或v;x3为l或a;x4和x5为独立的i,a,v,或t;x6为s或t;x7为a或v;x8,x9,x10,和x11为独立的v,l,t,a,n,s,或g.
「抗原识别域」包括对以下各者具有选择性或靶向以下各者之多肽:标靶之抗原、受体、肽配位体或蛋白质配位体;或标靶之多肽。
抗原识别域可自与配位体结合及/或信号转导相关之广泛多种细胞外域或分泌蛋白质中之任一者获得。抗原识别域可包括与ig轻链之一部分连接之ig重链之一部分,其组成特异性结合于标靶抗原之可变单链片段(scfv)。抗体可为单株或多株抗体或可具有任何特异性结合于标靶抗原之类型。在另一实施例中,抗原识别域可为受体或配位体。在特定实施例中,标靶抗原对特定疾病病状具有特异性且该疾病病状可为任何类型,只要其具有细胞表面抗原即可,该细胞表面抗原可由复合car架构中存在之嵌合受体构筑体中之至少一者识别。在特定实施例中,嵌合受体可用于任何其中存在特异性单株或多株抗体或能够产生特异性单株或多株抗体之癌症。特定言之,诸如神经母细胞瘤、小细胞肺癌、黑素瘤、卵巢癌、肾细胞癌、结肠癌、霍奇金氏淋巴瘤(hodgkin'slymphoma)及儿童急性淋巴细胞白血病之癌症具有对嵌合受体具有之抗原。
在一些实施例中,抗原识别结构域可以是非抗体蛋白质支架,例如但不限于,可以被设计为以高亲和力结合多种特定靶标之非抗体蛋白质支架,centyrins。centyrins是基于人类共有之肌腱蛋白fn3结构域之支架蛋白,通常小于在car分子上之scfv分子。
标靶特异性抗原识别域较佳包括来源于针对标靶之抗原之抗体的抗原结合域,或结合标靶之抗原之肽,或结合抗体(其结合标靶之抗原)之肽或蛋白质,或结合标靶上之受体之肽或蛋白质配位体(包括(但不限于)生长因子、细胞激素或荷尔蒙),或来源于结合标靶上之肽或蛋白质配位体之受体(包括(但不限于)生长因子受体、细胞激素受体或荷尔蒙受体)之域。
在一个实施例中,抗原识别域包括针对标靶(对标靶具有选择性)之单株或多株抗体之结合部分或可变区。
在另一实施例中,抗原识别域包括骆驼科(camelid)单域抗体,或其部分。在一个实施例中,骆驼科单域抗体包括在骆驼中发现之重链抗体,或vhh抗体。骆驼科(例如骆驼、单峰驼、大羊驼及羊驼)之vhh抗体系指骆驼单链抗体之可变片段(参见nguyen等人,2001;muyldermans,2001),且亦包括经分离之骆驼vhh抗体、重组型骆驼
vhh抗体或合成骆驼vhh抗体。
在另一实施例中,抗原识别域包括接合其同源受体之配位体。作为实例,april为结合tac1受体或bcma受体之配位体。根据本文中所揭示之发明,抗原识别域包括april,或其片段。作为另一实例,baff为结合baff-r受体或bcma受体之配位体。根据本发明,抗原识别域包括baff,或其片段。在另一实施例中,抗原识别域经人类化。
抗原识别域可在其序列内包括一些可变性且仍对本文中所揭示之标靶具有选择性。因此,预期抗原识别域之多肽可与本文中所揭示之抗原识别域多肽具有至少95%、至少90%、至少80%或至少70%一致性,且仍对本文中所描述之标靶具有选择性且属于本发明之范畴内。
标靶包括介白素6受体、ny-eso-1、α胎蛋白(afp)、磷脂醯肌醇蛋白聚糖-3(gpc3)、bcma、baff-r、taci、ley、cd13、cd14、cd15、cd19、cd20、cd22、cd33、cd41、cd61、cd64、cd68、cd117、cd123、cd138、cd267、cd269、cd38、flt3受体、cs1、cd45、ror1、psma、magea3、糖脂、磷脂醯肌醇蛋白聚糖3、f77、gd-2、wt1、cea、her-2/neu、mage-3、mage-4、mage-5、mage-6、α-胎蛋白、ca19-9、ca72-4、ny-eso、fap、erbb、c-met、mart-1、cd30、egfrviii、免疫球蛋白κ及λ、cd38、cd52、cd3、cd4、cd8、cd5、cd7、cd2及cd138。
在另一实施例中,标靶包括任何部分介白素6受体、ny-eso-1、α胎蛋白(afp)、磷脂醯肌醇蛋白聚糖-3(gpc3)、bcma、baff-r、taci、ley、cd13、cd14、cd15cd19、cd20、cd22、cd33、cd41、cd61、cd64、cd68、cd117、cd123、cd138、cd267、cd269、cd38、flt3受体、cs1、cd45、taci、ror1、psma、magea3、糖脂、磷脂醯肌醇蛋白聚糖3、f77、gd-2、wt1、cea、her-2/neu、mage-3、mage-4、mage-5、mage-6、α-胎蛋白、ca19-9、ca72-4、ny-eso、fap、erbb、c-met、mart-1、cd30、egfrviii、免疫球蛋白κ及λ、cd38、cd52、cd3、cd4、cd8、cd5、cd7、cd2及cd138。
在一个实施例中,标靶包括以下各者之表面暴露部分:介白素6受体、ny-eso-1、α胎蛋白(afp)、磷脂醯肌醇蛋白聚糖-3(gpc3)、bcma、baff-r、taci、ley、cd5、cd13、cd14、cd15cd19、cd20、cd22、cd33、cd41、cd61、cd64、cd68、cd117、cd123、cd138、cd267、cd269、cd38、flt3受体、cs1、cd45、taci、ror1、psma、magea3、糖脂、磷脂醯肌醇蛋白聚糖3、f77、gd-2、wt1、cea、her-2/neu、mage-3、mage-4、mage-5、mage-6、α-胎蛋白、ca19-9、ca72-4、ny-eso、fap、erbb、c-met、mart-1、cd30、egfrviii、免疫球蛋白κ及λ、cd38、cd52、cd3、cd4、cd8、cd5、cd7、cd2及cd138多肽。
例如靶标包括baff之表面暴露区域,如图81所示。靶标可以包括baff之表面暴露区域的一部分。例如,baff之部分包括人类baff之残留1-200,1-100,50-150或100-200。
在另一个实施例中,把抗原包括病毒或真菌抗原,例如来自人乳头瘤病毒(hpv)或ebv(epsteinbarr病毒)抗原之e6和e7;其部分;或表面暴露之区域。
在一个实施例中,taci抗原识别域包括seqidno.24。
在一个实施例中,bcma抗原识别域包括seqidno.25。
在一个实施例中,cs1抗原识别域包括seqidno.26。
在一个实施例中,baff-r抗原识别域包括seqidno.27。
在一个实施例中,cd33抗原识别域包括seqidno.28。
在一个实施例中,cd123抗原识别域包括seqidno.29。
在一个实施例中,cd19抗原识别域包括seqidno.30。
在一个实施例中,cd20抗原识别域包括seqidno.31。
在另一实施例中,cd20抗原识别域包括seqidno.32。
在一个实施例中,cd22抗原识别域包括seqidno.33。
在一个实施例中,cd45抗原识别域包括seqidno.34。
在一个实施例中,cd4抗原识别域包括seqidno.35。
在一个实施例中,cd25抗原识别域包括seqidno.36。
铰链区为安置于例如包括(但不限于)嵌合抗原受体与至少一个共刺激域及信号传导域之间的序列。可获得铰链序列,包括例如来自任何属之任何适合的序列,包括人类或其部分。此项技术中已知此类铰链区。在一个实施例中,铰链区包括人类蛋白质之铰链区,包括cd-8α、cd28、4-1bb、ox40、cd3-ζ、t细胞受体α或β链、cd3ζ链、cd28、cd3ε、cd45、cd4、cd5、cd8、cd8a、cd9、cd16、cd22、cd33、cd37、cd64、cd80、cd86、cd134、cd137、icos、cd154、其功能衍生物及其组合。
在一个实施例中,铰链区包括cd8a铰链区。
在一些实施例中,铰链区包括选自(但不限于)免疫球蛋白(例如igg1、igg2、igg3、igg4及igd)之一者。
跨膜域包括横跨细胞膜之疏水性多肽。特定言之,跨膜域自细胞膜之一侧(细胞外)横跨细胞膜之另一侧(细胞内或细胞质)。
跨膜域可呈α螺旋或β筒或其组合形式。跨膜域可包括异地同型蛋白质,其具有多个跨膜区段,各呈α-螺旋、β片或其组合形式。
在一个实施例中,使用与car中之一个域天然相关之跨膜域。在另一实施例中,选择跨膜域或藉由胺基酸取代修饰以避免此类域与相同或不同表面膜蛋白质之跨膜域之结合,从而最小化与受体复合物之其他成员之相互作用。
举例而言,跨膜域包括以下各者之跨膜域:t细胞受体α或β链、cd3ζ链、cd28、cd3ε、cd45、cd4、cd5、cd7、cd8、cd9、cd16、cd22、cd33、cd37、cd64、cd80、cd86、cd68、cd134、cd137、icos、cd41、cd154、其功能衍生物及其组合。
在一个实施例中,跨膜域经人工设计使得域中超过25%、超过50%或超过75%胺基酸残基为疏水性残基,诸如白胺酸及缬胺酸。在一个实施例中,在合成跨膜域之各端发现苯丙胺酸、色胺酸及缬胺酸之三联体。
在一个实施例中,跨膜域为cd8跨膜域。在另一实施例中,跨膜域为cd28跨膜域。此类跨膜域为此项技术中已知的。
信号传导域及共刺激域包括可活化免疫细胞以刺激或活化免疫细胞信号传导路径之至少一些态样之多肽。
在一个实施例中,信号传导域包括以下各者之功能性信号传导域之多肽:cd3ζ、共同fcrγ(fcer1g)、fcγrlla、fcrβ(fcεrib)、cd3γ、cd3δ、cd3ε、cd79a、cd79b、dnax活化蛋白质10(dap10)、dnax活化蛋白质12(dap12)、其活性片段、其功能衍生物及其组合。此类信号传导域为此项技术中已知的。
在一个实施例中,car多肽还包含一个或多个共刺激结构域。在一个实施例中,共刺激结构域是来自蛋白的功能性信号传导结构域,包括以下一种或多种:ox40;cd27;cd28;cd30;cd40;pd-1;cd2;cd7;cd258;自然杀手第2组成员c(nkg2c);自然杀手第2组成员d(nkg2d),b7-h3;与cd83,icam-1,lfa-1(cd11a/cd18),icos和4-1bb(cd137)中的至少一种结合的配体;cds;icam-1;lfa-1(cd1a/cd18);cd40;cd27;cd7;b7-h3;nkg2c;pd-1;icos;其活性片段;功能衍生物;及其组合。
如本文中所使用,至少一个共刺激域及信号传导域可共同称为胞内域。如本文中所使用,铰链区及抗原识别可共同称为细胞外域。
本发明亦提供编码上述嵌合抗原受体多肽之聚核苷酸。
如本文所使用之术语「聚核苷酸」定义为核苷酸链。聚核苷酸包括dna及rna。此外,核酸为核苷酸之聚合物。因此,如本文所使用之核酸及聚核苷酸为可互换的。熟习此项技术者具有核酸为聚核苷酸之常识,聚核苷酸其可水解成单体「核苷酸」。单体核苷酸可水解成核苷。如本文所使用,聚核苷酸包括(但不限于)藉由此项技术中可
使用的任何手段获得的所有核酸序列,包括(但不限于)重组手段,亦即使用一般选殖技术自重组型文库或细胞基因体选殖核酸序列,及聚合酶链反应(pcr),及其类似手段,及藉由合成成段。
编码car之聚核苷酸可藉由任何习知方法自指定car之胺基酸序列容易地制备。关于各域之胺基酸序列,编码胺基酸序列之基底序列可自前述ncbirefseqid或genbenk之寄存编号获得,且本发明之核酸可使用标准分子生物及/或化学程序制备。举例而言,基于基底序列,可合成聚核苷酸,且可藉由组合dna片段来制备本发明之聚核苷酸,该等dna片段系使用聚合酶链反应(pcr)自cdna文库获得。
在一个实施例中,本文中所揭示之聚核苷酸为基因之一部分,或表现或选殖卡匣。
上述聚核苷酸可选殖至载体中。「载体」为物质之组合物,其包括经分离之聚核苷酸,且其可用于将经分离之聚核苷酸传递至细胞内部。此项技术中已知的大量载体包括(但不限于)线性聚核苷酸、与离子性或两性化合物相关之聚核苷酸、质体、噬菌粒、黏质体及病毒。病毒包括噬菌体、噬菌体衍生物。因此,术语「载体」包括自主复制质体或病毒。该术语亦应视为包括非质体及非病毒化合物,其促进核酸转移至细胞中,诸如聚离胺酸化合物、脂质体及其类似物。病毒载体之实例包括(但不限于)腺病毒载体、腺相关病毒载体、反转录病毒载体、慢病毒载体及其类似物。在一个实施例中,载体包括选殖载体、表现载体、复制载体、探针产生载体、整合载体及测序载体。
在一实施例中,载体为病毒载体。在一个实施例中,病毒载体为反转录病毒载体或慢病毒载体。在一个实施例中,经工程改造之细胞以病毒方式转导以表现聚核苷酸序列。
已针对基因转移至哺乳动物细胞中开发出许多基于病毒之系统。举例而言,反转录病毒提供基因传递系统之适宜平台。所选基因可插入载体中且使用此项技术中已知之技术封装于反转录病毒粒子中。接着可分离重组型病毒且活体内或离体传递至患者之细胞中。此项技术中已知许多反转录病毒系统。在一些实施例中,使用腺病毒载体。此项技术中已知许多腺病毒载体。在一个实施例中,使用慢病毒载体。
病毒载体技术为此项技术中熟知的且描述于例如sambrook等人(2001,molecularcloning:alaboratorymanual,coldspringharborlaboratory,newyork)及其他病毒学及分子生物学手册中。适用作载体之病毒包括(但不限于)反转录病毒、腺病毒、腺相关病毒、疱疹病毒及慢病毒。通常,适合的载体含有在至少一种生物体、启动子序列、适宜的限制内切酶位点及一或多种可选标记物中具有功能性的复制起点(例如wo01/96584;wo01/29058;及美国专利第6,326,193号)。
已熟知慢病毒载体将基因高效转移至人类t细胞中之能力,但载体编码之基因之表达取决于驱使其表达之内部启动子。对于具有其他编码增生性细胞激素之共刺激域或基因之第三代或第四代car,强启动子尤其重要,因为增加之car主体尺寸并不确保等效表达量。存在多种具有不同强度及细胞类型特异性之启动子。使用cart细胞之基因疗法依赖于t细胞表达充分的car主体及长时间段维持表达之能力。通常选择ef-1α启动子用于car表达。
本发明是关于含有用于t细胞或nk细胞中高基因表达量之强启动子之表现载体。在其他实施例中,本发明者揭示适用于t细胞或nk细胞中之高car表达量之强启动子。在特定实施例中,强启动子与sffv启动子有关,其被选择性引入表现载体中,以在t细胞或nk细胞中获得高表达量及长时间段维持表达。所表达之基因偏好car、t细胞共刺激因子及用于免疫疗法之细胞激素。
适合的启动子之一个实例为前早期巨细胞病毒(cmv)启动子序列。此启动子序列为能够驱使任何与其以操作方式连接之聚核苷酸序列之高表达量的强组成性启动子序列。适合的启动子之另一实例为延长生长因子-1a(ef-1a)。然而,亦可使用其他组成性启动子序列,包括(但不限于)猴病毒40(sv40)早期启动子、小鼠乳房肿瘤病毒(mmtv)、人类免疫缺陷病毒(hiv)长末端重复序列(ltr)启动子、momulv启动子、禽类白血病病毒启动子、埃-巴二氏病毒前早期启动子、劳斯肉瘤病毒启动子(roussarcomaviruspromoter),以及人类基因启动子,诸如(但不限于)肌动蛋白启动子、肌球蛋白启动子、血红蛋白启动子及肌酸激酶启动子。此外,本发明不应限于使用组成性启动子,亦涵盖诱导性启动子作为本发明之一部分。诱导性启动子之使用提供分子开关,该分子开关能够在需要此类表达时打开其以操作方式连接之聚核苷酸序列表现,或在不需要时关闭表现。诱导性启动子之实例包括(但不限于)金属硫蛋白启动子、糖皮质激素启动子、孕酮启动子及四环素启动子。
可使用例如表现载体获得嵌合抗原受体聚核苷酸之表达,包括(但不限于)sffv(脾病灶形成病毒)(例如seqidno.23)或人类延长因子11α(ef)启动子、cag(具有cmv强化子之鸡β-肌动蛋白启动子)启动子人类延长因子1α(ef)启动子中之至少一者。所使用之强度较弱/表达量较低之启动子之实例可包括(但不限于)猴病毒40(sv40)早期启动子、巨细胞病毒(cmv)前早期启动子、泛素c(ubc)启动子及磷酸甘油酸激酶1(pgk)启动子或其部分。嵌合抗原受体之诱导性表达可使用例如四环素反应启动子获得,包括(但不限于)tre3gv(tet反应元件,包括所有世代且较佳为第3代)、诱导性启动子(clontechlaboratories,mountainview,ca)或其部分或组合。
在一个较佳实施例中,启动子为sffv启动子或其衍生物。且意外发现sffv启动子在根据本发明之经转导之细胞中提供更强表达及更大程度的持久性。
「表现载体」是指包括重组性聚核苷酸之载体,该重组性聚核苷酸包含与待表现之核苷酸序列以操作方式连接之表现控制序列。表现载体包括足够的顺式作用元素用于表现;用于表现之其他元素可由宿主细胞供应或在活体外表现系统中。表现载体包括此项技术中已知之所有表现载体,诸如并入重组性聚核苷酸之黏质体、质体(例如裸露或含于脂质体中)及病毒(例如慢病毒、反转录病毒、腺病毒及腺相关病毒)。表现载体可为双顺反子或多顺反子表现载体。双顺反子或多顺反子表现载体,可包括(1)与每个开放阅读框架融合之多个启动子;(2)在基因之间插入编接信号;表达由单个启动子驱使之基因之融合;(3)在基因(自裂解肽)之间插入蛋白水解分裂位点;及(iv)在基因之间插入内部核糖体进入位点(ires)。
在一个实施例中,本发明提供具有至少一个嵌合抗原受体多肽或聚核苷酸之经工程改造之细胞。
「经工程改造之细胞」意谓任何生物体之经修饰、经转型或借由基因、dna或rna序列或蛋白质或多肽之添加或修饰而操作之任何细胞。本发明之经分离之细胞、宿主细胞及经基因工程改造之细胞包括经分离之免疫细胞,诸如含有编码嵌合抗原受体或嵌合抗原受体复合物之dna或rna序列且在细胞表面上表达嵌合受体之nk细胞及t细胞。经分离之宿主细胞及经工程改造之细胞可用于例如增强nk细胞活性或t淋巴细胞活性、治疗癌症及治疗感染性疾病。
在一个实施例中,经工程改造之细胞包括免疫调节细胞。免疫调节细胞包括t细胞,诸如cd4t细胞(辅助t细胞)、cd8t细胞(细胞毒性t细胞、ctl)及记忆体t细胞或记忆体干细胞t细胞。在另一实施例中,t细胞包括自然杀手t细胞(nkt细胞)。
在一个实施例中,经工程改造之细胞包括自然杀手细胞。自然杀手细胞为此项技术中所熟知的。在一个实施例中,自然杀手细胞包括细胞系,诸如nk-92细胞。nk细胞系之其他实例包括nkg、yt、nk-ys、hank-1、yts细胞及nkl细胞。
nk细胞在无gvhd风险情况下介导抗肿瘤作用且相对于t细胞为短生命期。因此,nk细胞将在毁坏癌细胞之后不久耗竭,减少对car构筑体上将消融经修饰之细胞之诱导性自杀基因之需要。
根据本发明,吾人意外发现nk细胞提供易于获得的经工程改造以含有及表达本文中所揭示之嵌合抗原受体多肽之细胞。
同种异体或自体nk细胞诱导快速免疫反应,但归因于其有限使用寿命而自循环相对快速地消失。因此,申请人意外发现使用基于car细胞之疗法可减少持续性副作用问题。
根据本发明中的一个方面,可以用根据本发明的car多核苷酸扩增和转染nk细胞。nk细胞可以源自脐带血,外周血,ips细胞和胚胎干细胞。根据本发明内容中的一个方面,可以扩增nk-92细胞并用car转染。nk-92是一种不断增长的细胞系,具有自然杀伤(nk)细胞的特征和特性(arai,meagher等人,2008)。nk-92细胞系依赖il-2,并且已被证明是安全的(arai,meagher等人,2008)并且是可行的。表达nk-92细胞的car可以在无血清培养基中扩增,有或没有与饲养细胞共培养。可以通过分选获得携带感兴趣的car之纯nk-92群体。
在一个实施例中,经工程改造之细胞包括从供体获得的同种异体t细胞,其被修饰以灭活参与mhc识别的tcr(t细胞受体)组分。结果,缺少tcr的t细胞不会引起植物抗宿主病(gvhd)。
在一些实施例中,可以修饰经工程改造之细胞以防止细胞表面抗原之表达。例如,可以对经工程改造细胞进行遗传修饰以删除天然cd45基因以防止其表达和细胞表面展示。
在一些实施例中,经工程改造之细胞包括诱导性自杀基因(「安全开关」)或安全开关之组合,其可在载体上组装,诸如(但不限于)反转录病毒载体、慢病毒载体、腺病毒载体或质体。引入「安全开关」可极大地增加安全概况及限制复合car之命中标靶或偏离肿瘤毒性。「安全开关」可为诱导性自杀基因,诸如(但不限于)半胱天冬酶9基因、胸苷激酶、胞嘧啶脱胺酶(cd)或细胞色素p450。其他用于消除不合需要之经修饰之t细胞之安全开关涉及t细胞中cd20或cd52或cd19或经截短之表皮生长因子受体之表现。已涵盖所有有可能的安全开关且在本发明中实施。
在一些实施例中,将自杀基因整合至经工程改造之细胞基因体中。
在一个实施例中,本发明提供具有cd45嵌合抗原受体聚核苷酸之经工程改造之细胞。在一个实施例中,cd45car多肽包括seqidno.13及相应的聚核苷酸序列seqidno.14。在另一实施例中,cd45car多肽包括seqidno.15,及相应的聚核苷酸序列seqidno.16。在另一实施例中,cd45car多肽包括seqidno.17,及相应的聚核苷酸序列seqidno.18。
在特定的实施例中,经工程改造之细胞包括通过p2a裂解序列与il-15/il-15sushi连接的cd45car。提供该实施例之多肽包括seqidno.43和相应之多核苷酸序列seqidno.44。
在特定的实施例中,经工程改造之细胞包括通过p2a裂解系列与4-1bbl(cd137l)连接的cd45car。提供该实施例之多肽包括seqidno.42和相应之多核苷酸序列seqidno.41。
多个car单元
在一个实施例中,本发明提供了具有至少两个不同或分开的car单元之经工程改造的细胞。两个car单元可以是完整car单元或不完整car单元。如本文所用,“不同之car多肽”和“不同之car多肽单元”可互换使用。
本发明提供了嵌合抗原受体多肽,其具有信号肽,抗原识别结构域,铰链区,跨膜结构域,信号传导结构域和至少一个共刺激结构域,其定义为car单元或完整car单元。如本文所用,不完整car单元包括信号肽,抗原识别结构域,铰链区,跨膜结构域和信号传导结构域或至少一个共刺激结构域之多肽。不完整car单元不同时包含信号结构域和至少一个共刺激域,而是两者中的一个。
在一个实施例中,本发明提供了具有第一嵌合抗原受体多肽之经工程改造细胞,其多肽包含第一抗原识别结构域和共刺激结构域(第一不完整car单元);第二嵌合抗原受体多肽具有第二抗原识别结构域和信号结构域(第二不完整car单元);其中第一抗原识别结构域不同于第二抗原识别结构域。
因此,当两种靶抗原同时与抗原识别结构域组合时,具有两个不完整car单元的经工程改造细胞恻才能被完全激活。该策略提供了额外之特异性,直到靶标都与不完整car单元的抗原识别结构域结合后,经工程改造之细胞才能被完全激活。
此外,经工程改造细胞包括两个不完整car单元的实施例中,抗原识别结构域可以针对和与链霉亲和素,生物素,his,myc,ha,琼脂糖,v5,麦芽糖,gst,gfp,cd52,4-1bb,或cd28中的其中之一个结合。
如本文中所使用,复合car(ccar)或多个car是指具有至少两种不同且完整的嵌合抗原受体多肽之经工程改造之细胞。如本文中所使用,「不同的嵌合抗原受体多肽」具有独特抗原识别域、信号肽、铰链区、跨膜域、至少一个共刺激域及信号传导域。因此,两个独特的嵌合抗原受体多肽将具有不同的抗原识别域。在两个不同的嵌合抗原受体多肽之间,信号肽、铰链区、跨膜域、至少一个共刺激域及信号传导域可相同或不同。如本文中所使用,嵌合抗原受体(car)单元是指不同的嵌合抗原受体多肽,或编码不同的嵌合抗原受体多肽之聚核苷酸。
如本文中所使用,独特抗原识别域为对单个标靶或标靶之单个抗原决定基具有特异性或靶向其之抗原识别域。
在一些实施例中,一个复合car靶向两种不同抗原。
产生携带不同car单元之复合car可极具挑战性:(1)car与car之间的相互作用可具有不利影响且适合的car设计为抵消此作用之关键;(2)单一构筑体中之复合car可增加表达盒之长度,其可引起病毒效价及蛋白质表达量降低;(3)需要适合的设计以包括多种car主体元素,尤其选择策略以在单一载体中表达多个car;(4)强启动子对于携带其他car单元之复合car尤其重要;(5)car中之铰链区需要经设计使得较佳避免各car单元之间铰链区之相互相用;(6)表达于细胞中之两个或更多个car单元可引起毒性作用(car-car相互相用)。本文中之申请人提供可解决此等障碍之新颖及出人意料的car组合物及方法。
在一个实施例中,本发明提供具有多个car单元之经工程改造之细胞。此使得单个经工程改造之细胞靶向多个抗原。一个多个car单元同时靶向多个表面标记物或抗原可防止抗性纯系之选择及减少肿瘤复发。尚未研发用于任何恶性肿瘤之多个cart细胞免疫疗法,其中每个个别组分car包含多个域及活化位点。
在本发明之一个态样中,ccar包括多个car单元。在一些实施例中,ccar包括至少两个car单元。在另一实施例中,ccar包括至少三个car单元。在另一实施例中,ccar包括至少四个单元。
在一个实施例中,本发明提供经工程改造之细胞,其具有至少两种不同的嵌合抗原受体多肽,各自具有不同的抗原识别域。
在一个实施例中,具有至少两个不同嵌合抗原受体多肽之经工程改造细胞为t细胞。t细胞可以被改造使其不表达细胞表面抗原。例如,可以改造t细胞使其不表达cd45细胞表面抗原。
在一个较佳实施例中,具有至少两种不同嵌合抗原受体多肽之经工程改造之细胞为自外周血或脐带血及nk-92细胞分离之原生nk细胞,使得其「现成地」投与任何患有疾病或癌症之哺乳动物。
在一个实施例中,经工程改造之细胞包括(i.)第一嵌合抗原受体多肽,其包含第一抗原识别域、第一信号肽、第一铰链区、第一跨膜域、第一共刺激域及第一信号传导域;及(ii.)第二嵌合抗原受体多肽,其包含第二抗原识别域、第二信号肽、第二铰链区、第二跨膜域、第二共刺激域及第二信号传导域。第一抗原识别域与第二抗原识别域不同。
在一个较佳实施例中,每个经工程改造之car单元聚核苷酸具有不同核苷酸序列以避免同源重组。
在一个实施例中,第一抗原识别域之表吧选自由以下组成之群:介白素6受体、ny-eso-1、α胎蛋白(afp)、磷脂醯肌醇蛋白聚糖-3(gpc3)、baff-r、bcma、taci、ley、cd4、cd5、cd13、cd14、cd15、cd19、cd20、cd22、cd33、cd41、cd61、cd64、cd68、cd117、cd123、cd138、cd267、cd269、cd38、flt3受体、cll-1及cs1;且第二识别域之标靶选自由以下组成之群:介白素6受体、ny-eso-1、α胎蛋白(afp)、磷脂醯肌醇蛋白聚糖-3(gpc3)、baff-r、bcma、taci、ley、cd4、cd5、cd13、cd14、cd15、cd19、cd20、cd22、cd33、cd41、cd61、cd64、cd68、cd117、cd123、cd138、cd267、cd269、cd38、flt3受体、cll-1及cs1。
在一个实施例中,经工程改造之细胞包括具有cd19抗原识别域之第一嵌合抗原受体多肽及具有cd20识别域之第二嵌合抗原受体多肽。在一个实施例中,此经工程改造之细胞包括seqidno.3之多肽及相应的seqidno.4之聚核苷酸。
在一个实施例中,经工程改造之细胞包括具有cd19抗原识别域之第一嵌合抗原受体多肽及具有cd22识别域之第二嵌合抗原受体多肽。在一个实施例中,此经工程改造之细胞包括seqidno.5之多肽及相应的seqidno.6之聚核苷酸。
在一个实施例中,经工程改造之细胞包括具有cd19抗原识别域之第一嵌合抗原受体多肽及具有cd123识别域之第二嵌合抗原受体多肽。在一个实施例中,此经工程改造之细胞包括seqidno.7之多肽及相应的seqidno.8之聚核苷酸。
在一个实施例中,经工程改造之细胞包括具有cd33抗原识别域之第一嵌合抗原受体多肽及具有cd123抗原识别域之第二嵌合抗原受体多肽。在一个实施例中,此经工程改造之细胞包括seqidno.9之多肽及相应的seqidno.10之聚核苷酸。在另一实施例中,此经工程改造之细胞包括seqidno.11之多肽及相应的seqidno.12之聚核苷酸。
在一个实施例中,经工程改造之细胞包括具有baff-r抗原识别域之第一嵌合抗原受体多肽及具有cs1抗原识别域之第二嵌合抗原受体多肽。
在一个实施例中,经工程改造之细胞包括具有cd269抗原识别域之第一嵌合抗原受体多肽及具有cs1识别域之第二嵌合抗原受体多肽。在一个实施例中,经工程改造之细胞包括有包括seqidno.19之多肽及相应的聚核苷酸seqidno.20。在一个实施例中,经工程改造之细胞包括有包括seqidno.21之多肽及相应的聚核苷酸seqidno.22。
在一个实施例中,经工程改造之细胞包括具有cd33抗原识别域之第一嵌合抗原受体多肽及具有cd123识别域之第二嵌合抗原受体多肽。
在一个实施例中,各car单元包括相同或不同铰链区。在另一实施例中,各car单元包括相同或不同跨膜区。在另一实施例中,各car单元包括相同或不同胞内域。
在一个实施例中,各car单元包括cd3ζ链信号传导域。
在一个实施例中,每个不同的car单元包括不同的共刺激域以避免相互相用。举例而言,第一嵌合抗原受体多肽包括4-bb共刺激域;且第二嵌合抗原受体多肽包括cd28共刺激域。
在另一实施例中,铰链区经设计以排除可能引起不合需要的分子内或分子间相互作用之胺基酸。举例而言,铰链区可经设计以排除或最小化半胱胺酸残基以防止形成二硫键。在另一实施例中,铰链区可经设计以排除或最小化疏水性残基以防止不合需要之疏水相互作用。
复合car可独立地或组合杀死细胞。多重或复合car包含相同或不同铰链区、相同或不同跨膜、相同或不同共刺激及相同或不同细胞内域。较佳选择铰链区以避免相互相用位点。
本发明之复合car可靶向t或nk细胞中之相同或不同肿瘤群。第一个car例如可靶向大型肿瘤群且随后或第二个car例如可根除癌症或白血病干细胞,以避免癌症复发。
根据本发明,意外发现t或nk细胞中靶向不同或相同肿瘤群之复合car对抗可引起对car杀死活性之抗性之癌细胞的肿瘤因子,借此产生自癌细胞表面之标靶抗原之下调。亦意外发现此能够引起癌细胞「躲避」car疗法,称为「抗原逃逸」,及肿瘤非均质性,由此不同肿瘤细胞可呈现不同表面抗原表达概况。
具有car多肽及强化子之经工程改造之细胞
在另一实施例中,本发明提供具有至少一个嵌合抗原受体多肽及强化子之经工程改造之细胞。
在一个实施例中,本发明提供具有至少两种不同嵌合抗原受体多肽及强化子之经工程改造之细胞。
如本文中所使用,强化子包括生物分子,其促进或增强具有嵌合抗原受体多肽之经工程改造之细胞之活性。强化子包括细胞激素。在另一实施例中,强化子包括il-2、il-7、il-10、il-12、il-15、il-18、il-21、pd-1、pd-l1、csf1r、ctal-4、tim-3及tgfrβ、其受体及其功能片段。
强化子可由本文中所描述之经工程改造之细胞表达且在经工程改造之细胞之表面上呈现,或强化子可由经工程改造之细胞分泌至周围细胞外空间中。表面呈现及分泌之方法为此项技术中所熟知的。举例而言,强化子可为融合蛋白,其具有提供表面呈现或对细胞外空间之分泌的肽。
强化子之作用可由其他因子补充,诸如强化子受体及其功能片段。其他因子可与强化子一起以融合蛋白形式共表达,或表达为单独的肽且分泌至细胞外空间中。
增强子可以是从经工程改造之car细胞分泌的细胞因子,并且被设计为与car多肽共表达。在car与同源抗原结合时大量释放。肿瘤细胞周围的炎性细胞与癌细胞的进展及转移显著相关。炎症细胞可包括t细胞和先天免疫应答细胞,例如nk细胞、巨噬细胞和树突细胞,并且它们的增殖和抗肿瘤活性受细胞因子调节。诸如cart或nk细胞之car细胞与标靶癌症细胞结合并引发cart/nk细胞扩增的增强子大量分泌。分泌之增强子有效地促进针对爱细胞之免疫应答细胞的存活,分化和活化。增强子与car之共表达可以补充cart或nk细胞不能完全消除靶向癌细胞的缺陷(图78)。
car细胞可以是细胞因子之载体,并且细胞因子可以通过car细胞递送至标靶癌症位置,以降低高剂量外源细胞因子的系统毒性(图78)。
为了改善car细胞的持续存活或持久性,结合膜增强子可与car共表达以改善car持久性。
在一个实施例中,增强子为il-15。在这种情况下,上述另外之因子是il-15受体及其功能片段。功能性片段包括il-15受体,il-15ra和il-15ra(il-15sushi)之sushi结构域。可溶性il-15ra或il15sushi通过预防il-15降解显著增强il-15之功能活性。可溶性il-15/il-15ra或il-15/il-15sushi复合物在体内比单独之il-15稳定且更具刺激作用。
在一个实施例中,il-15与il-15受体,il-15ra和il-15ra(il-15sushi)的sushi结构域中之至少一种共表达为融合蛋白。在一个实施例中,il-15受体,il-15ra或il-15ra(il-15sushi)的sushi结构域位于il-15的n末端。在另一个实施例中,il-15受体,il-15ra或il-15ra(il-15sushi)的sushi结构域位于il-15的c末端。如本文所用,il-15/il-15sushi表示il-15sushi在融合蛋白中位于il-15的c末端;il-15sushi/il-15表示il-15sushi位于融合蛋白中il-15的n末端。
在一些实施例中,il-15和il-15受体或其功能片段多肽在单个多肽分子上通过连接子隔开,肽连接子长度可为1-25个氨基酸残基,25-100个氨基酸残基,或50-200个氨基酸残基。该连接子可包括本文所述的高效切割位点。
合适的sushi结构域之实例包括car构筑体,seqidno.1。根据本发明,本文公开之任何嵌合抗原受体多肽可以与人类白细胞介素2信号肽seqidno.2一起与人类白细胞介素15共表达。
白细胞介素(il)-15及其特异性受体链il-15rα(il-15-ra)在各种效应细胞中起关键作用,包括nk和cd8t细胞。可以修饰cd8+t细胞以表达自分泌生长因子,包括但不限于il-2/il-7,il-21或il-15,以维持在体内转移后的存活。不希望受理论束缚,据信il-15克服了cd4的缺乏以诱导原发性及召回记忆t细胞。在cd8t细胞上过度表达il-15-ra或il-15-ra融合体在体内和体外显著增强其存活和增殖。在一些实施例中,cd4car或任何car与il-15、il-15ra、il-15/il-15ra、il-15ra/il-15或其一部分或其组合中的至少一种共表达,以增强cart或nk的存活或增殖,并改善记忆carcd8+t细胞的扩增。
本发明公开内容提供了具有如本文所述之car多肽和il-15、il-15ra、il-15sushi、il-15/il-15ra、il-15ra/il-15、il-15/il-15sushi、il-15sushi/il-15或其片段及其组合中至少一种的经工程改造的细胞,以增强cart或nk的存活、续航力及增值,用于治疗患者的癌症。
在另一个实施例中,本发明公开提供了具有il-15、il-15ra、il-15sushi、il-15/il-15ra、il-15ra/il-15、il-15/il-15sushi、il-15sushi/il-15或其功能片段及其组合中至少一种重组组合的经工程改造的细胞;和至少一种不同的car多肽,其中抗原识别结构域包括ny-eso-1,甲胎蛋白(afp)磷脂酰肌醇蛋白聚糖-3(gpc3),bcma,baff-r,bcma,taci,ley,cd5,cd7,cd2,cd3,cd4,cd45,cd13,cd14,cd15,cd19,cd20,cd22,cd33,cd41,cd61,cd64,cd68,cd117,cd123,cd138,cd267,cd269,cd38,flt3受体,ror1,psma,magea3,糖脂、f77,gd-2,wt1,cea,her-2/neu,mage-3,mage-4,mage-5,mage-6,ca19-9,ca72-4,ny-eso,fap,erbb,c-met,mart-1,cd30,egfrviii,免疫球蛋白和,cd38和cs1。靶抗原还可以包括病毒或真菌抗原,例如来自人乳头瘤病毒(hpv)或ebv(epsteinbarr病毒)抗原的e6和e7。在进一步的实施例中,抗原识别多肽(scfv)和相应的多核苷酸cd2,cd3,cd5,cd7和cd52描述在pct申请no.pct/us2016/39306内,其内容通过引用并入本文。
不希望受理论束缚,据信当与检查点抑制剂或调节剂组合时,il-15/il-15sushi和其它类型的il-15或il-15ra蛋白或其蛋白片段提供car多肽的协同功效(例如抗-pd-1)。
在一个实施例中,本发明公开提供了cd4car工程改造之细胞,其包括il-15/il-15sushi(seqidno.1)和相应的多核苷酸(seqidno.2)。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15sushi的cd4car工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd4car多肽的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,本发明公开内容提供了具有cd45嵌合抗原受体多肽和il-15/il-15sushi(seqidno.44)的经工程改造之细胞,以及相应的核苷酸(seqidno.43)。
在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15sushi的cd45car之经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd4car多肽的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括cd45嵌合抗原受体多肽和4-1bbl(seqidno.74),以及相应的核苷酸(seqidno.73)。
在一个实施例中,本发明公开内容提供了通过向有需要的患者施用共表达4-1bbl的cd45car经工程改造之细胞,向患有癌症的患者提供长期持久缓解的方法。不希望受理论束缚,据信4-1bbl与cd45car的共表达通过增加car经工程改造之细胞的持久性以向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括cd19嵌合抗原受体多肽和il-15/il-15sushi(seqidno.59),和相应的多核苷酸(seqidno.60)。在一个实施例中,本发明公开提供了通过向有需要的患者施用包含il-15/il-15sushi的cd19car经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd19car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括cd20嵌合抗原受体多肽和il-15/il-15sushi(seqidno.58),和相应的多核苷酸(seqidno.57)。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15sushi的cd20car经工程改造之细胞,向癌症患者中提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd20car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括cd22嵌合抗原受体多肽和il-15/il-15sushi(seqidno.62),和相应的多核苷酸(seqidno.61)。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15sushi的cd22car经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd22car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括cd269嵌合抗原受体多肽和il-15/il-15sushi(seqidno.44),和相应的多核苷酸(seqidno.45)。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15sushi的cd269car经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd269car的共表达通过募集先天性免疫细胞到癌细胞,如浆细胞或骨髓瘤细胞(通常是昏暗的cd269(bcma)阳性),或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括car,cd4多肽seqidno.90,和相应多核苷酸seqidno.89。
在一个实施例中,经工程改造之细胞包括cd4嵌合抗原受体多肽和il-15/il-15sushi(seqidno.96),和相应的多核苷酸(seqidno.95)。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15sushi的cd4car经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd4car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括cd4嵌合抗原受体多肽和il-15/il-15ra(膜结合的)(seqidno.98),和相应的多核苷酸(seqidno.97)。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15ra的cd4car经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15ra(膜结合)与cd4car的共表达通过增加car经工程改造之细胞的持久性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括复合car,cd33cd123多肽和il-15/il-15sushi(seqidno.40),和相应的多核苷酸(seqidno.39)。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15sushi的cd33cd123复合car经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd33cd123car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括复合car,cd33cd123多肽和4-1bbl(seqidno.38),和相应的多核苷酸(seqidno.37)。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用共表达4-1bbl的cd33cd123复合car经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信4-1bbl与cd33cd123ccar的共表达通过增加ccar经工程改造之细胞的持久性在患者中提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括具有cd45前导序列(seqidno.78)的baffcar多肽和相应的多核苷酸序列(seqidno.77)。
在一个实施例中,经工程改造之细胞包括baffcar多肽,其具有cd8a前导序列(包括seqidno.80)和相应的多核苷酸序列(seqidno.79)。
在一个实施例中,经工程改造细胞包括baffcar多肽和il-15/il-15sushi(seqidno.84),和相应的多核苷酸(seqidno.83)。
在一个实施例中,本发明公开内容提供了通过向有需要的患者施用包含il-15/il-15sushi的baffcar经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与baffcar的共表达通过募集先天性免疫细胞到癌细胞(作为baff受体的癌细胞,cd269(bcma)在浆细胞和骨髓瘤细胞中微弱表达)或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括baffcar多肽和4-1bbl(seqidno.82),和相应的多核苷酸(seqidno.81)。在一个实施例中,本发明公开提供了通过向有需要的患者施用共表达4-1bbl的baffcar经工程改造之细胞,向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信4-1bbl与baffcar的共表达通过增加car经工程改造之细胞的持久性向患者提供长期持久的缓解。
在一个实施例中,经工程改造之细胞包括复合car,baffcd19b多肽seqidno.86和相应多核苷酸seqidno.85。
在一个实施例中,本发明公开内容提供了通过向有需要的患者施用baffcd19b复合car经工程改造之细胞来治疗患者的自身免疫病症之方法。不希望受理论束缚,据信baffcd19b复合car经工程改造之细胞为与自身免疫疾病相关的b细胞和浆细胞的耗尽提供了更好的治疗结果。
在一个实施例中,经工程改造之细胞包括aprilcd19b复合car多肽seqidno.88和相应多核苷酸seqidno.77。
在一个实施例中,本发明公开内容提供了通过向有需要的患者施用aprilcd19b复合car工程改造细胞来消耗有此需要的患者中的b细胞和浆细胞之方法。不希望受理论束缚,据信aprilcd19b复合car经工程改造之细胞可以为与自身免疫疾病相关的b细胞和浆细胞的耗尽提供更好的治疗结果。
在一个实施例中,经工程改造之细胞包括复合car,cd269cs1多肽seqidno.48和相应多核苷酸seqidno.47。在一个实施例中,本发明公开提供了通过向有需要的患者施用cd269cs1复合car经工程改造之细胞来治疗患者的骨髓瘤之方法。
不希望受理论束缚,据信cd269cs1复合car经工程改造之细胞为骨髓瘤患者提供更好的治疗结果,并防止抗原逃逸或疾病复发。
在一个实施例中,经工程改造细胞包括复合car,cd269cd19b多肽seqidno.50,和相应多核苷酸seqidno.49。
在一个实施例中,本发明公开内容提供了通过向有需要的患者施用cd269cd19b复合car经工程改造之细胞来耗尽患者中的b细胞和浆细胞之方法。不希望受理论束缚,据信cd269cd19b复合car经工程改造之细胞通过耗尽与自身免疫疾病相关的b细胞和浆细胞而为患有自身免疫疾病的患者提供更好的治疗结果。
在一个实施例中,经工程改造之细胞包括另一种复合car,cd269cd19多肽seqidno.52和相应多核苷酸seqidno.51。在一个实施例中,本发明公开内容提供了通过向有需要的患者施用cd269cd19复合car经工程改造之细胞来消耗患者中的b细胞和浆细胞之方法。不希望受理论束缚,据信cd269cd19复合car经工程改造细胞通过耗尽与自身免疫疾病相关的b细胞和浆细胞向患有自身免疫疾病的患者提供更好的治疗结果。
在一个实施例中,本发明公开提供了具有cd19嵌合抗原受体多核苷酸的经工程改造之细胞。在一个实施例中,cd19car多肽包括seqidno.54和相应的多核苷酸序列seqidno.53。在另一个实施例中,cd19car多肽包括seqidno.56,和相应的多核苷酸序列seqidno.55。
在一个实施例中,经工程改造细胞包括cd30car多肽和il-15/il-15sushi多肽(seqidno.100),和相应的多核苷酸(seqidno.99)。靶向疾病是恶性霍奇金淋巴瘤,癌细胞表达cd30。
在一个实施例中,本发明公开提供了通过向有需要的患者施用分泌il-15/il-15复合物的cd30car经工程改造之细胞来重新激活患者肿瘤微环境中的t细胞和先天免疫细胞之方法。不希望受理论束缚,据信从经工程改造细胞分泌的il-15/il-15复合物(例如il-15/il-15sushi复合物)可以重新激活肿瘤微环境中的t细胞和先天免疫细胞,然后恢复或增强他们对霍奇金淋巴瘤或间变性大细胞淋巴瘤的抗肿瘤免疫反应。
在一个实施例中,本发明公开内容提供了恢复或增强t细胞或先天免疫细胞活化或扩增之方法,包括il-15/il-15sushi与本文公开的car多肽的共表达。
在另一个实施例中,本发明公开内容提供了嵌合抗原受体多肽,其具有对cd30抗原特异的抗原识别结构域。
在一个实施例中,cd30car包括至少一个共刺激结构域。在另一个实施例中,cd30car包含至少两个共刺激结构域。
在一些实施例中,本发明公开内容包括共表达il-15/il-15sushi与cd30car之方法。在进一步的实施例中,在car与靶细胞结合时,稳定的功能性il-15/il-15sushi复合物大量分泌。
在另一个实施例中,本发明公开内容提供了通过向有需要的患者施用cd30car经工程改造之细胞来治疗患有霍奇金淋巴瘤或与表达cd30抗原的恶性细胞相关的癌症的患者之方法。表达cd30的恶性细胞的实例包括间变性大细胞淋巴瘤。
恶性霍奇金淋巴瘤携带cd30+reed-sternberg或reed-sternberg样细胞,其被大量的t细胞和先天免疫细胞包围。这些t或先天免疫细胞具有免疫耐受性,因为它们不能消除癌细胞。因此,治疗霍奇金淋巴瘤的关键方面之一是在肿瘤微环境中重新激活t细胞和先天免疫细胞,然后恢复或增强其抗肿瘤免疫应答。
在一些实施例中,本发明公开包括il-15/il-15sushi与cd30car共表达之方法。经工程改造之cd30cart或nk细胞与靶向癌细胞结合,从cd30cart或nk细胞的扩增中引发il-15/il-15sushi的大量分泌,从而令分泌性il-15/il-15sushi有效恢复或增强t或先天免疫细胞对癌细胞克服免疫抑制肿瘤微环境。
在一个实施例中,本发明公开内容提供了通过向有需要的患者施用共表达il-15/il-15sushi的cd30car经工程改造细胞向癌症患者提供长期持久缓解的方法。不希望受理论束缚,据信il-15/il-15sushi与cd30car的共表达通过增加car识别靶癌细胞的敏感性或募集先天免疫细胞针对靶癌细胞克服免疫抑制性肿瘤微环境,以向患者提供长期持久的缓解。
在一些实施例中,本发明公开内容提供了共表达il-15/il-15sushi和cd30car多肽的经工程改造之细胞。不希望受理论束缚,据信当与检查点抑制剂或调节剂(例如抗pd-1)组合时,cd30car经工程改造之后细胞与il-15/il-15sushi的共表达的组合提供协同功效。
在一些实施例中,本发明公开提供了通过向有需要的患者施用共表达il-15/il-15sushi的cd30car经工程改造之细胞来治疗患者的霍奇金淋巴瘤之方法。不希望受理论束缚,cd30car多肽和il-15/il-15sushi的共表达比单独cd30car表达有更好的霍奇金淋巴瘤或间变性大细胞治疗效果,因为cd30不在所有癌细胞中表达。
共表达分泌型il-15/il-15sushi的car消除肿瘤之步骤(图78)
在一些实施例中,本发明公开内容提供了通过向有需要的患者施用共表达il-15/il-15sushi的aprilcar经工程改造之细胞向癌症患者提供长期持久缓解之方法。不希望受理论束缚,据信il-15/il-15sushi与aprilcar多肽的共表达通过募集先天性免疫细胞到癌细胞(april受体cd269(bcma)在浆细胞和骨髓瘤细胞中弱表达)或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在具体的实施例中,本发明公开内容提供了用于消除肿瘤细胞之方法,包括使所述肿瘤细胞与共表达il-2以破坏所述肿瘤细胞的经工程改造的car细胞接触。
il-15最初被认为是t细胞和nk细胞的白细胞介素-2(il-2)样因子。与il-2不同,il-15是记忆t细胞的存活因子。
在特定实施例中,可以通过组合以下步骤中的至少一个或多个来实现肿瘤的消除:
(1)通过靶向car或nk抗原将本文公开的car工程改造t细胞或nk细胞与一部分肿瘤细胞结合;
(2)从共表达该分子的cart/nk细胞的扩增引发il-15/il-15sushi或il-2的大量分泌,延长半衰期;
(3)招募和刺激各种先天性和适应性免疫细胞对抗肿瘤;
(4)通过施用检查点阻断例如pd-l1和ctla-4抑制剂来减少肿瘤中存在的肿瘤抑制。
不希望受理论束缚,据信上述步骤的组合通过协同的先天和适应性免疫应答提供有效的抗肿瘤作用。
本文所述的工程改造细胞和方法适用于治疗任何癌症,其中存在特定的单克隆或多克隆抗体或能够根据现有技术产生。特别地,已经考虑了以下癌症并且被认为是在本发明公开的范围内,神经母细胞瘤,肺癌,黑素瘤,卵巢癌,肾细胞癌,结肠癌,脑癌,霍奇金淋巴瘤,b细胞淋巴瘤/白血病和t细胞淋巴瘤/白血病。所有这些都具有可被本文公开的嵌合抗原受体多肽和方法靶向的细胞表面抗原。
由于靶向抗原的丧失或cart/nk的耗竭,许多肿瘤逃脱了特定的cart/nk杀伤。本发明公开提供了克服这种逃避的方法。不希望受理论束缚,本发明公开内容提供了通过将具有本文公开的增强子或细胞因子的car工程改造细胞,特别是il-15或il-2,施用于肿瘤部位来预防肿瘤逃逸的方法。据信这直接刺激了先天性和适应性免疫应答。此外,据信来自car工程改造细胞的il-15和/或il-2分泌促进输注的cart细胞或carnk细胞的扩增和免疫细胞向肿瘤部位的入侵,这被认为能导致肿瘤破坏。
在实施例中,可以在fc结构域(例如il-15fc或il-2fc)的存在下建立半衰期延长和延长的治疗活性。对于il-15细胞因子,优选il-15/il-15sushi或il-15/il-15sushifc。fc结构域被称为与各种效应分子(所谓的fc融合蛋白)融合的iggfc结构域。
单抗原定向car免疫疗法,例如但不限于cd19,cd20,cd22,cd2,cd3,cd4,cd5,cd7,cd33,cd30,cd123,cd45,bcma,cs1,baff,taci和aprilcar由于靶抗原的完全丧失或靶抗原表达的改变,在患者中具有缓解风险。在此基础上,本发明公开内容提供了通过施用具有本文公开的car多肽的工程改造细胞和il-15/il-15sushi的共表达来提高car识别靶向癌细胞的敏感性或将先天免疫细胞募集到癌细胞的方法,为患者提供长期持久的缓解。
cart细胞可能难以根除一些大量实体瘤或淋巴瘤。免疫抑制微环境需要被克服,因为当接触肿瘤时cart细胞可能最终被简单地灭活或抑制。
在一些实施例中,本发明公开内容提供了在工程改造细胞中共表达分泌型il-15/il-15sushi和嵌合抗原受体多肽之方法。
在一些实施例中,本发明公开内容提供了通过在所述工程改造细胞中共表达分泌型il-15/il-15sushi来增加car工程改造细胞在体内半衰期的方法。不希望受理论束缚,据信il-15/il-15sushi的分泌复合物在功能上是稳定的并且有效地促进含car的工程改造细胞的存活。
在一些实施例中,本发明公开内容提供了使用car作为载体将il-15/il-15sushi递送至靶向癌症部位的方法,以促进先天免疫应答细胞针对癌细胞的增殖,防止肿瘤微环境的抑制,并减少高剂量外源性细胞因子的系统毒性。
在一些实施例中,本发明公开内容提供了使用car作为载体将il-15/il-15sushi递送至靶向癌症部位的方法,以将其他效应免疫细胞募集至该位点并帮助它们杀死癌细胞。
在一些实施例中,本发明公开提供了使用car作为载体将il-15/il-15sushi递送至靶向癌症部位以激活附近的免疫细胞以根除失去cart/nk细胞抗原的癌细胞之方法。
产生工程改造细胞之方法
可以通过本领域已知的任何方法将本文公开的任何多核苷酸引入工程改造细胞中。
在一个实施例中,通过本文公开的任何病毒载体将car多核苷酸递送至工程改造细胞。
在一个实施例中,为了实现增强的安全性特征或治疗指数,本文公开的任何工程改造细胞可构建为瞬时rna修饰的“可生物降解的”形式或衍生物,或其组合。可以将本发明公开中rna修饰的car电穿孔到t细胞或nk细胞中。复合car的表达可在几天内逐渐减少。
在本发明公开的一些实施例中,本文公开的任何工程改造细胞可以在转录系统(也称为“睡美人”)中构建,没有用病毒载体将cardna整合到宿主基因组中。
产生具有多个car单元的工程改造细胞的方法
在另一个实施例中,本发明公开提供了制备具有至少两个car单元的工程改造细胞之方法。
在一些实施例中,使用双顺反子或多顺反子表达载体在t或nk细胞中表达多个car单位。有几种策略可用于构建双顺反子或多顺反子载体,包括但不限于(1)与car的开放阅读框融合的多个启动子;(2)在car单元之间插入剪接信号;car的融合,其表达由单个启动子驱动;(3)在car单元之间插入蛋白水解切割位点(自切割肽);(iv)插入内部核糖体进入位点(ires)。
在优选的实施例中,多个car单元在单个开放阅读框(orf)中表达,从而产生具有多个car单元的单个多肽。在该实施例中,在每个car单元之间设置含有高效切割位点的氨基酸序列或接头。
如本文所用,高切割效率定义为超过50%,超过70%,超过80%或超过90%的转化蛋白质被切割。如kim2011所述,可通过蛋白印迹分析测量切割效率。
此外,在优选的实施例中,存在等量的裂解产物,如蛋白质印迹分析所示。
高效切割位点的实例包括猪teschovirus-21a(p2a),fmdv2a(本文缩写为f2a);马鼻炎a病毒(erav)2a(e2a);和那些甲病毒2a(t2a),细胞质多角体病毒2a(bmcpv2a)和flacherie病毒2a(bmifv2a),或其组合。在优选的实施例中,高效切割位点是p2a,kimjh,lees-r,lil-h,parkh-j,parkj-h,leeky等人(2011)描述了高效切割位点。在人细胞系,斑马鱼和小鼠中源自猪teschovirus-1的2a肽的高切割效率。plosone6(4):e18556之内容通过引用并入本文。
在其中多个car单元在单个开放阅读框(orf)中表达的实施例中,表达受强启动子的控制。强启动子的实例包括sffv启动子及其衍生物。
具有car多肽和增强子的工程改造细胞
在另一个实施例中,本发明公开提供了制备表达至少一种car单元和增强子的工程改造细胞的方法。
在一些实施例中,使用双顺反子或多顺反子表达载体在t或nk细胞中表达至少一种car单元和增强子。有几种策略可用于构建双顺反子或多顺反子载体,包括但不限于(1)与car的开放阅读框融合的多个启动子;(2)在car单元之间插入剪接信号;car的融合,其表达由单个启动子驱动;(3)在car单元之间插入蛋白水解切割位点(自切割肽);(4)插入内部核糖体进入位点(ires)。
在优选的实施例中,至少一个car单元和增强子在单个开放阅读框(orf)中表达,从而产生具有至少一个car单元和增强子的单一多肽。在该实施例中,含有高效切割位点的氨基酸序列或接头位于每个car单元之间和car单元及增强子之间。在该实施例中,orf处于强启动子的控制下。强启动子的实例包括sffv启动子及其衍生物。
此外,在优选的实施例中,存在等量的裂解产物,如蛋白质印迹分析所示。
使用本文中公开的组合物之治疗方法
在另一个实施例中,本发明公开提供了一种靶向cd45的方法,用于在癌症治疗中的同种异体移植之前进行调节。cd45也称为白细胞共同抗原(lca),并且是除了红细胞和血小板之外几乎在所有造血细胞来源的细胞上表达的酪氨酸磷酸酶。大多数血液系统恶性肿瘤表达cd45。例如,85%至90%的急性淋巴和髓样白血病表达cd45。但是并没有在非造血起源中发现cd45。另外,cd45在恶性细胞和白细胞上以每个细胞约200,000个分子的高密度表达。cd45是各种血液系统恶性肿瘤的理想靶点。然而,cart和nk细胞也表达cd45。没有内源性cd45的灭活,携带靶向cd45的cart或nk细胞可导致自杀。
cd45与tcr复合物的结合对于响应抗原的t细胞活化的调节是必需的。cd45缺陷型t细胞不能呈递抗原是由于通过t细胞受体(tcr)的信号传导减少。tcr是细胞表面受体,其响应于抗原的呈递,且在t细胞的活化中起重要作用。tcr通常由α和β两条链组成,其与转导亚基cd3相关,以形成细胞表面上存在的t细胞受体复合物。
令人惊讶地发现,本发明公开的多种car(复合car,ccar)是对抗癌细胞抵抗car活性的关键机制,即靶抗原从癌细胞表面的下调或异质表达。该机制允许癌细胞“隐藏”car治疗,这种现象被称为“抗原逃逸”。本发明公开通过识别两种或更多种抗原的组合来快速消除肿瘤来预防癌症抗原逃逸。
本发明公开内容提供了使用ccar同时靶向多抗原的方法,其通过最小化基于靶抗原丢失或下调的肿瘤选择的可能性而改善的肿瘤控制。
所公开的公开内容包括靶向肿瘤细胞中存在的不同或相同表面抗原的t或nk细胞中的复合(多种或化合物)ccar。本发明公开的复合嵌合抗原受体包含至少多个嵌合受体构筑体,其通过相同或不同抗原的接头和靶标连接。例如,存在于复合car(ccar)构筑体中的每种car构筑体包括抗原识别结构域,细胞外结构域,跨膜结构域和/或细胞质结构域。细胞外结构域和跨膜结构域可以源自这些结构域的任何所需来源。多个car构筑体通过链接子链接。复合car构筑体的表达由启动子驱动。接头可以是肽或蛋白质的一部分,其在产生蛋白质或肽(也称为自切割肽)后自切割。
在一个实施例中,本发明公开的复合car靶向骨髓增生异常综合征和急性髓性白血病(aml)群体。骨髓增生异常综合征(mds)仍然是一种无法治愈的造血干细胞恶性肿瘤,在老年人中最常发生,在美国每年约有14,000例新病例。大约30-40%的mds病例发展为aml。随着人口老龄化,mds的发病率继续增加。尽管已经对mds和aml进行了认真研究,但尚未开发出令人满意的治疗方法。
本发明的组合物和方法可用于产生t淋巴细胞或nk细胞群,其递送初级和共刺激信号,用于治疗癌症的免疫疗法,特别是肺癌,黑素瘤,乳腺癌,前列腺癌,结肠癌,肾细胞癌,卵巢癌,脑癌,肉瘤,白血病和淋巴瘤。
免疫疗法通常依赖于使用免疫效应细胞和分子来靶向及破坏癌细胞。效应子可以是携带表面分子的淋巴细胞,所述表面分子直接或间接地与肿瘤细胞靶标相互作用。各种效应细胞包括细胞毒性t细胞,nk细胞和nk-92细胞。本发明公开中描述的组合物和方法可以与其他类型的癌症治疗结合使用,例如化疗,手术,放射,基因治疗等。本发明公开中描述的组合物和方法可以用于治疗依赖免疫应答的其他疾病状况,例如炎症,免疫疾病和传染病。
在一些实施例中,本发明公开的复合car可通过实现具有最小残留疾病且不再对化疗有反应的患者的完全缓解而充当骨髓移植的桥梁。在其他实施例中,复合car消除白血病细胞,然后进行骨髓干细胞拯救以支持白细胞减少症。
在一些实施例中,本发明公开的复合car是抗癌细胞通过下调靶抗原来抵抗car活性的关键对抗机制。在另一个实施例中,本发明的复合car还可以对抗癌细胞的异质性,这在常规cart/nk细胞疗法中引起了重大挑战。在另一个实施例中,所公开的复合car设计成第一个car靶向庞大的肿瘤群体和癌症或白血病干细胞,以避免癌症复发。
在一个实施例中,本发明公开内容提供了通过使所述细胞与具有至少一种具有cd33抗原识别结构域的嵌合抗原受体多肽和嵌合抗原的工程改造细胞接触来破坏具有cd33抗原或cd123抗原或两者的细胞之方法。受体多肽具有cd23抗原识别结构域。工程改造细胞可以是t或nk细胞。
具有cd33抗原和cd123抗原中的至少一种的细胞包括急性髓性白血病,前体急性淋巴细胞白血病,慢性骨髓增生性肿瘤,慢性髓性白血病,脊髓发育不良综合征,原发性浆细胞样树突状肿瘤(bpdcn),霍奇金淋巴瘤,肥大细胞增多症和毛细胞白血病细胞。
在另一个实施例中,本发明公开内容提供了一种用于造血干细胞移植的骨髓清除预处理方案之方法。在该实施例中,将具有cd33单元和cd123单元的t或nk工程改造细胞施用于有需要的患者。
在进一步的实施例中,本发明公开内容提供了根除或杀死表达cd123或cd33或两者的白血病干细胞(lsc)或大量白血病细胞之方法。在该实施例中,将具有cd33单元和cd123单元的t或nk工程改造细胞施用于需要的患者。
在进一步的实施例中,t或nk细胞中的复合car可用于根除或杀死表达cd123或cd33或两者皆有的大量白血病细胞或cd34+cd38-的白血病干细胞。
在一些实施例中,复合car靶向表达cd19或cd20抗原或两者皆有的细胞。在另一个实施例中,复合car靶向表达cd19或cd22抗原或两者皆有的细胞。靶细胞可以是癌细胞,例如但不限于b细胞淋巴瘤或白血病。在进一步的实施例中,靶抗原可包括该组中的至少一种,但不限于ror1,psma,magea3,糖脂,磷脂酰肌醇蛋白聚糖3,f77,gd-2,wt1,cea,her-2/neu,mage-3,mage-4,mage-5,mage-6,甲胎蛋白,ca19-9,ca72-4,ny-eso,fap,erbb,c-met,mart-1,cd30,egfrviii,免疫球蛋白κ和λ,cd38,cd52,cd3,cd4,cd8,cd5,cd7,cd2,和cd138。靶抗原还可以包括病毒或真菌抗原,例如来自人乳头瘤病毒(hpv)或ebv(epsteinbarr病毒)抗原的e6和e7。
在一些实施例中,复合car工程改造细胞靶向具有细胞表面cd19抗原或细胞表面cd123抗原或两者皆有的细胞。靶细胞是癌细胞,例如但不限于b细胞淋巴瘤或白血病。
cd19cart细胞治疗的临床试验表明,80-94%的b-all患者完全缓解,但相当大比例的患者最终复发。b-all中cd123表达的普遍性很高,并且可以用作b-all的car靶标。
在一些实施例中,复合car靶向表达cd19或cd123抗原或两者皆有的细胞。不希望受理论束缚,据信cd19和/或cd123复合car工程改造细胞可抵消由于cd19或cd123抗原的丧失或阻止b-all或其他类型b细胞淋巴瘤/白血病复发而引发的肿瘤逃逸。
在进一步的实施例中,cd19和/或cd20复合car工程改造细胞靶向具有细胞表面cd19抗原和/或cd20细胞表面抗原的细胞。在另一个实施例中,靶细胞是恶性b细胞淋巴瘤/白血病,例如但不限于b-all,高级b细胞淋巴瘤,低级b细胞淋巴瘤,弥漫性大b细胞淋巴瘤,burkett淋巴瘤,套细胞淋巴瘤,cll,边缘区b细胞淋巴瘤和滤泡性淋巴瘤。
不希望受理论束缚,据信cd19和/或cd20car工程改造细胞提供了针对b细胞恶性肿瘤的过继性t/nk细胞疗法中抗原逃逸和预防疾病复发的有效保护。
具有至少一种抗原cd19,cd20,cd22和cd123的car靶细胞包括前体急性淋巴细胞白血病,b细胞淋巴瘤/白血病,慢性淋巴细胞白血病/淋巴瘤,地幔淋巴瘤,滤泡性淋巴瘤,边缘区b细胞淋巴瘤,弥漫性大b细胞淋巴瘤,burkett淋巴瘤,急性浆细胞样树突状肿瘤(bpdcn),霍金淋巴瘤和毛细胞白血病细胞。
在一个实施例中,工程改造细胞包括具有cd19抗原识别结构域的第一嵌合抗原受体多肽和具有cd22抗原识别结构域的第二嵌合抗原受体多肽。在一个实施例中,该工程改造细胞包括多肽seqidno.64和相应多核苷酸seqidno.63。
在一个实施例中,工程改造细胞包括具有cd19抗原识别结构域的第一嵌合抗原受体多肽和具有cd20抗原识别结构域的第二嵌合抗原受体多肽。在一个实施例中,该工程改造细胞包括多肽seqidno.66和相应多核苷酸seqidno.65。
在一个实施例中,工程改造细胞包括具有cd19抗原识别结构域的第一嵌合抗原受体多肽和具有cd123抗原识别结构域的第二嵌合抗原受体多肽。在一个实施例中,该工程改造细胞包括多肽seqidno.68和相应多核苷酸seqidno.67。
多发性骨髓瘤是一种无法治愈的疾病,表现出骨髓中浆细胞的不可控的增殖。cs1和bcma在骨髓瘤细胞上广泛表达,但不在造血干/祖细胞中表达。因此,bcma和cs1是cart/nk细胞疗法的理想靶标。
在进一步的实施例中,本发明公开内容提供了具有cs1(slam7)抗原识别结构域和/或靶向b细胞成熟抗原(bcma)的抗原识别结构域的复合car工程改造细胞。在另一个实施例中,靶细胞是恶性浆细胞,例如但不限于多发性骨髓瘤。
不希望受理论束缚,据信具有cs1和bcma抗原识别结构域中的至少一个的复合car工程改造细胞增强了针对多发性骨髓瘤的功能并抵消了抗原逃逸。
在一些实施例中,car靶向表达多种抗原的细胞,包括但不限于cs1,bcma,cd267,baff-r,cd38,cd138,cd52,cd19,taci,cd20,白细胞介素6受体和ny-eso-1抗原。在另一个实施例中,靶细胞是浆细胞,b细胞,恶性浆细胞,例如但不限于多发性骨髓瘤。
在一些实施例中,复合car靶向表达多种抗原的细胞,包括但不限于cs1,bcma,cd267,baff-r,cd38,cd138,cd52,cd19,taci,cd20,白细胞介素6受体和ny-eso-1抗原。在另一个实施例中,靶细胞是恶性浆细胞,例如但不限于多发性骨髓瘤。
在一些实施例中,复合car靶向表达多种抗原的细胞,包括但不限于甲胎蛋白(afp)和磷脂酰肌醇蛋白聚糖-3(gpc3)。在另一个实施例中,靶向细胞是肝细胞癌,纤维板层癌,肝母细胞瘤,未分化的胚胎肉瘤和肝,肺鳞状细胞癌,睾丸非精原细胞生殖细胞肿瘤,脂肪肉瘤,卵巢和性腺外卵黄囊肿瘤,卵巢绒毛膜癌,畸胎瘤,卵巢透明细胞癌,和胎盘部位滋养细胞肿瘤。
不希望受理论束缚,本发明公开提供了靶向不同或相同抗原的复合car工程改造t细胞或nk细胞抵消肿瘤逃逸并提供肿瘤细胞的同步靶向。
包含本文公开的复合car的t或nk宿主细胞体现在本发明公开内容中。复合car的核苷酸和多肽构筑体,序列,宿主细胞和载体被认为是本发明公开内容的一部分并且在本文中体现。
在一些实施例中,复合car工程改造细胞与目前正在开发或市场上可获得的任何化学治疗剂组合施用。在一些实施例中,复合car工程改造细胞被施用作疾病的一线治疗,所述疾病包括但不限于血液恶性肿瘤,癌症,非血液恶性肿瘤,炎性疾病,感染性疾病例如hiv和htlv等。在一个实施例中,将表达复合car工程改造t细胞与表达相同或不同复合car的nk细胞共同施用作为适应性免疫疗法。复合carnk细胞提供靶向细胞的具有快速及先天活性的细胞而复合t细胞提供相对持久的适应性免疫活性。
在一个实施例中,复合car工程改造细胞被施用作为哺乳动物的骨髓干细胞移植的桥梁,例如,对化疗有抗药性且不具备骨髓干细胞移植资格的患者。
在一些实施例中,复合car共表达转基因并在靶向肿瘤病变中释放转基因产物,例如il-12,并进一步调节肿瘤微环境。
在一个实施例中,将复合car工程改造细胞施用于哺乳动物以用于骨髓髓细胞消融,作为疾病治疗的一部分。
在一个具体实施例中,表达复合car的细胞可以是t细胞或nk细胞,给予哺乳动物,例如人类。本发明公开内容包括通过施用复合car治疗患有病症或疾病的哺乳动物之方法。靶细胞可以是癌细胞,或受任何其他疾病影响的细胞,例如传染病,炎症和自身免疫疾病。
本发明公开内容旨在包括复合car或抗原的片段,突变体或变体(例如,修饰形式)的用途,其保留诱导刺激和t/nk细胞的增殖之能力。“蛋白质形式”旨在表示与至少一种car或抗原具有显着同源性并且能够实现t/nk细胞的刺激和增殖的蛋白质。如本文所用,术语“生物活性”或“蛋白质的生物活性形式”意指包括能够影响细胞的抗肿瘤活性的蛋白质或变体的形式。
本发明的组合物和方法可用于产生t/nk细胞群,其递送初级和共刺激信号,用于治疗癌症的免疫疗法,特别是肺癌,黑素瘤,乳腺癌,前列腺癌,结肠癌,肾细胞癌,卵巢癌,神经母细胞瘤,横纹肌肉瘤,白血病和淋巴瘤的治疗。本发明公开中描述的组合物和方法可以与其他类型的癌症治疗结合使用,例如化疗,手术,放射,基因治疗等。
1)在一些实施例中,本发明公开提供了通过向患者施用car或复合cart细胞或nk细胞来消耗患有自身免疫疾病的患者中的b细胞,未成熟b细胞,记忆b细胞,浆母细胞,长寿浆细胞或浆细胞的方法。car靶向细胞是表达一种或两种或所有抗原的b细胞或浆细胞,bcma,taci和baff-r。自身免疫性疾病包括系统性硬皮病,多发性硬化症,牛皮癣,皮炎,炎症性肠病(如克罗恩病和溃疡性结肠炎),系统性红斑狼疮,血管炎,类风湿性关节炎,sjorgen综合征,多发性肌炎,肺泡蛋白沉积症,肉芽肿病和血管炎,addison's疾病,抗原-抗体复合物介导的疾病和抗肾小球基底膜疾病。
现在正在研究多种细胞外细胞标记物作为肿瘤相关抗原的价值,以作为cart/nk细胞疗法的潜在靶标。然而,除了脱靶毒性之外,这些抗原在健康组织上的表达导致靶向的肿瘤外不良事件仍然是主要的安全问题。此外,cart/nk细胞疗法的主要限制在于当靶向对肿瘤发生不必要的分子时选择抗原逃逸变体的可能性。因此,尽管具有高亲和力作用,但在很少或不表达靶抗原的情况下持续存在的恶性细胞可以逃避cart/nk细胞。
根据本发明公开,天然杀伤(nk)细胞代表用于car驱动杀灭的替代细胞毒性效应物。与t细胞不同,nk细胞不需要预激活并且能够有结构性地表现出细胞溶解功能。在nk细胞中进一步表达ccar允许nk细胞有效杀死癌症,特别是对nk细胞治疗具有抗性的癌细胞。
此外,已知nk细胞在没有诱导移植物抗宿主病(gvhd)的风险下能介导抗癌作用。
研究显示cd34+cd38-aml细胞上cd123的异常过表达,而正常骨髓对应cd34+cd38-不表达cd123(jordan,upchurch等人,2000)。这种cd123+,cd34+cd38-群体被认为是lscs,因为这些细胞能够在免疫缺陷小鼠中启动并维持白血病。
cd34+/cd38-/cd123+lscs的数量可用于预测aml患者的临床结果。aml患者中cd34+/cd38-/cd123+细胞(大于15%)与缺乏完全缓解和不利的细胞遗传学特征相关。此外,超过1%的cd34+/cd38-/cd123+细胞的存在也可能对无病生存和总体存活产生负面影响。
目前,mds和aml的治疗集中在白血病胚细胞上,因为它们非常丰富并且清楚地代表了患者最直接的问题。重要的是,白血病干细胞(lscs)与大多数其他白血病细胞(“胚”细胞)完全不同,它们构成罕见的亚群。虽然杀死胚细胞可以提供短期缓解,但lscs如果不被破坏,将永远重新生长,导致患者复发。为了实现mds疾病的持久治疗,必须销毁lscs。不幸的是,标准药物方案对mds或amllscs无效。因此,开发能够特异性针对白血病干细胞群和体积庞大的白血病群的新疗法至关重要。本发明公开中公开的复合car靶向这两种群体并且在本文中体现。
根据本发明,令人惊讶地发现nk细胞提供现成的产品,其可用作同种异体产品用于治疗。因此,根据本发明公开所示,根据现有技术的需要在患者特异性的基础上进行ccar细胞疗法。本发明公开的申请人已经发现了一种新的免疫疗法,其中不需要分离患者的淋巴细胞或肿瘤浸润的淋巴细胞用于有效且基于car细胞的疗法。
预期同种异体或自体nk细胞诱导快速免疫应答,但由于其有限的寿命而从循环中相对快速地消失。因此,申请人惊奇地发现使用基于ccar细胞的疗法减少了持续副作用的担忧。
根据本发明公开内容的一个方面,可以用根据本发明公开的ccar扩增和转染nk细胞。nk细胞可以源自脐带血,外周血,ips细胞和胚胎干细胞。根据本发明公开内容的一个方面,可以扩增nk-92细胞并用ccar转染。nk-92是一种不断增长的细胞系,具有自然杀伤(nk)细胞的特点和特征。nk-92细胞系依赖il-2,已被证明是安全可行的。表达nk-92细胞的ccar可以在有或没有饲养细胞共培养情况下在无血清培养基中扩增。可以通过分选获得纯携带ccar的nk-92群体。
鉴定合适的表面靶抗原是在适应性免疫疗法中开发cart/nk细胞的先决条件。
在本发明公开的一个方面,cd123抗原是ccar治疗的靶标之一。cd123是白细胞介素3受体的α链,在各种血液系统恶性肿瘤中过表达,包括急性髓性白血病(aml),b细胞急性淋巴细胞白血病(b-all),毛细胞白血病和原发性浆细胞样树突状肿瘤。cd123在正常造血干细胞上不存在或极少表达。更重要的是,cd123在与白血病干细胞(lscs)相关的白血病细胞子集上表达,其消融对于预防疾病的难治性和复发是必不可少的。
在本发明公开的一个方面,cd33抗原是ccar治疗的靶标之一。cd33是在急性髓性白血病中90%的恶性细胞上表达的跨膜受体。因此,根据本发明公开内容,从安全性的观点来看,cd123和cd33靶抗原特别有吸引力。
根据本发明,复合cd33cd123car可以非常有效地用于治疗慢性粒细胞白血病(cml)群体。在慢性粒细胞白血病(cml)中,存在罕见的cd34+cd38-细胞亚群。该群体被视为由lsc组成。增加的lscs数量与疾病的进展相关。小分子bcr-abl酪氨酸激酶抑制剂(tki)可显着改善cp-cml患者的总体存活率。然而,lscs被认为对tki疗法具有抗性。迫切需要一种靶向cml抗性lscs的新疗法来治疗cml,并且该新疗法体现在本发明公开中公开的复合cd33cd123car中。cd123在cd34+cd38-群体中有高表达。根据本发明,复合cd33cd123car对该群体的治疗非常有效。
在本发明公开的一个实施例中,在ccar中表达cd123和cd33的白血病细胞用作治疗性治疗。cd33在骨髓谱系,髓样白血病母细胞和成熟单核细胞的细胞上表达,但不是正常的多能造血干细胞上表达。cd33在cml,骨髓增生性肿瘤和mds中的白血病细胞中广泛表达。
由于大量急性髓性白血病(aml)患者难以接受标准化疗方案或治疗后出现疾病复发(burnett2012),因此amlcart细胞免疫治疗的发展有可能满足临床需求。在大多数这些患者中,白血病细胞表达cd123和cd33,赋予本文公开的复合cd33cd123car广泛的临床适用性。因此,本发明公开内容公开了一种新的多重ccart/nk细胞构筑体,其包含靶向多种白血病相关抗原的多种car,从而通过共刺激结构域激活的协同作用抵消靶向白血病细胞(包括白血病干细胞)的抗原逃逸机制,从而提供更有效和安全的治疗。
本发明公开进一步公开了复合car构筑体,其具有增强的针对共表达靶抗原的细胞的抗肿瘤活性的效力,并且仍然保持对仅表达一种抗原的肿瘤细胞的敏感性。此外,复合car的每个car包括一个或两个共刺激结构域,并且在特定靶标存在下表现出有效的杀伤能力。
在关于双重特异性的临床前研究中,针对包括乳腺癌和上皮卵巢癌在内的实体瘤的转录信号传导car,cd3ζ细胞内信号传导结构域,与来自第二代car的共刺激结构域分离。换句话说,一种car含有第一代car而没有任何共刺激结构域,另一种car缺乏cd3zeta细胞内结构域。因此,t细胞活化和有效杀伤需要两种靶抗原的存在。因此,提出它们作为降低由两种靶抗原之一的健康组织表达引起的肿瘤外毒性的方法,增加靶特异性,但是以灵敏度为代价。在一个实施例中,复合car是复合cd123cd19car。已经显示超过90%的b-all在一部分群体中表达cd123。与aml和mds一样,人们认为b-all中存在罕见的lsc群体。因此,根据本发明公开内容靶向白血病干细胞和大体积白血病群体可以应用于b-all。根据本发明公开内容,在b-all中表达的cd123和cd19表面抗原可以是靶标,因为cd19在b细胞淋巴样群的不同阶段中广泛表达。
多发性骨髓瘤(mm)是美国第二常见的血液系统恶性肿瘤,来源于骨髓或髓外部位积累的克隆浆细胞。mm是一种无法治愈的疾病,中位生存期约为4.5年。已经开发了临床前的抗骨髓瘤car,且car靶标包括cd38,cs1和b细胞成熟抗原(bcma)。然而,表面抗原表达的异质性通常发生在恶性浆细胞中,这使其成为car的困难靶标。恶性浆细胞也表达低水平的cd19。以前已经表明,骨髓瘤干细胞也表达一些b细胞标志物,包括cd19。与标准和其他骨髓瘤car疗法相结合,针对该群体可有效治疗骨髓瘤。
多发性骨髓瘤(mm)是血浆恶性肿瘤,具有浆细胞的克隆扩增。尽管治疗取得了重大进展,但骨髓瘤仍然是一种无法治愈的疾病;因此迫切需要新的治疗方法。
cs1(也称为cd319或slamf7)是由slamf7基因编码的蛋白质。表面抗原cs1是正常浆细胞和骨髓瘤细胞(恶性浆细胞)的强力标记物。
肿瘤坏死因子受体超家族成员17(tnfrsf17),也称为b细胞成熟抗原(bcma)或cd269几乎仅在浆细胞和恶性浆细胞的末期表达。其表达缺乏其他组织,表明作为cart或nk细胞的靶标的潜力。
恶性浆细胞显示cd269和cs1的不同程度的抗原异质性。靶向cd269或cs1的单个car单位产物可靶向肿瘤中的大多数细胞,导致最初强烈的抗肿瘤反应。随后,残留的稀有非靶向细胞扩增并引起疾病复发。虽然多发性骨髓瘤特别异质,但这种现象可以确定地适用于其他白血病或肿瘤。美国国立卫生研究院最近一项使用bcmacart细胞的临床试验结果显示,一些患有多发性骨髓瘤的患者完全恢复。然而,这些患者在17周后复发,这可能是由于抗原逃逸。在cd19car和ny-eso1cart细胞处理中也观察到抗原逃逸。因此,迫切需要更有效的cart细胞治疗以防止复发。
在本发明公开的一个方面,bcma和cs1是bcmacs1car治疗的靶标。
在一些实施例中,复合car靶向表达bcma或cs1抗原或两者皆有的细胞。靶细胞可以是癌细胞,例如但不限于淋巴瘤,或白血病或浆细胞肿瘤。在进一步的实施例中,浆细胞肿瘤选自浆细胞白血病,多发性骨髓瘤,浆细胞瘤,重链疾病,淀粉样变性,waldestrom巨球胶质细胞,重链疾病,孤立性骨浆细胞瘤,意义不明的单克隆丙种球蛋白病(mgus)和阴燃多发性骨髓瘤。
在一些实施例中,本发明公开内容提供了符合car多肽工程改造细胞,其靶向表达bcma或cd19抗原或两者皆有的细胞。靶细胞可以是癌细胞,例如但不限于淋巴瘤,或白血病或浆细胞肿瘤。在进一步的实施例中,浆细胞肿瘤选自浆细胞白血病,多发性骨髓瘤,浆细胞瘤,重链疾病,淀粉样变性,华氏巨球蛋白血症,重链疾病,孤立性骨浆细胞瘤,意义不明的单克隆丙种球蛋白病(mgus)和冒烟型多发性骨髓瘤。
baff(b细胞活化因子)和april(增殖诱导的配体)是特异性结合taci(也称为tnfrsf13b或cd267)和bcma的两种tnf同源物,具有高亲和力。baff(也称为blys)结合baff-r并且在功能上涉及增强b细胞后期的存活和增殖。baff已被证明涉及一些自身免疫性疾病。april在抗体类别转换的增强中起重要作用。baff和april都被认为是恶性浆细胞的生长和存活因子。
恶性浆细胞或正常浆细胞中的配体-受体相互作用描述于图77和79中。
在一些实施例中,本发明公开内容提供了复合car工程改造细胞,其靶向表达taci或cs1抗原或两者皆有的细胞。在另一个实施例中,复合car工程改造细胞靶向表达taci或cs1抗原或两者皆有的细胞。靶细胞可以是癌细胞,例如但不限于淋巴瘤,或白血病或浆细胞肿瘤。在进一步的实施例中,浆细胞肿瘤选自浆细胞白血病,多发性骨髓瘤,浆细胞瘤,重链疾病,淀粉样变性,华氏巨球蛋白血症,重链疾病,孤立性骨浆细胞瘤,意义不明的单克隆丙种球蛋白病(mgus)和冒烟型多发性骨髓瘤。靶细胞还可以是b细胞,未成熟b细胞,初始b细胞,中心母细胞,中心细胞,记忆b细胞,浆母细胞,长寿浆细胞,浆细胞中的一种或两种或多种不同细胞类型。这些细胞涉及自身免疫性疾病,包括系统性硬皮病,多发性硬化症,牛皮癣,皮炎,炎症性肠病(如克罗恩病和溃疡性结肠炎),系统性红斑狼疮,血管炎,类风湿性关节炎,sjorgen综合征,多发性肌炎,肉芽肿病和血管炎,艾迪生病,抗原-抗体复合物介导的疾病和抗肾小球基底膜疾病。
在一些实施例中,本发明公开内容提供了复合car工程改造细胞,其靶向表达baff-r或cs1抗原或两者皆有的细胞。在另一个实施例中,复合car工程改造细胞靶向表达baff-r或cs1抗原或两者的细胞。靶细胞可以是癌细胞,例如但不限于淋巴瘤,或白血病或浆细胞肿瘤。在进一步的实施例中,浆细胞肿瘤选自浆细胞白血病,多发性骨髓瘤,浆细胞瘤,重链疾病,淀粉样变性,华氏巨球蛋白血症,重链疾病,孤立性骨浆细胞瘤,意义不明的单克隆丙种球蛋白病(mgus)和冒烟型多发性骨髓瘤。
自身免疫性疾病如红斑狼疮(sle),寻常型天疱疮和多发性硬化症(ms)引起显着的发病率和残疾。这些疾病对目前的治疗反应不佳,并且在疾病过程中经常复发。b和浆细胞在自身免疫疾病的发病机理中起关键作用,因为它们是自身抗体产生的来源。b和浆细胞可通过抗原呈递,细胞因子分泌或抗体产生促进疾病进展和复发。使用cart/nk细胞方法删除b细胞或浆细胞或两者可能是有益的疗法。
器官移植代表了人的新生命,可移植的器官可包括肾脏,心脏,肺,胰腺和肠。然而,许多患者由于预先存在或正在发展针对供体抗原如人白细胞抗原(hla)的供体特异性抗体而不能接受可能挽救生命的器官。因此,患者可能失去捐赠的器官。目前,可用于抗体介导的排斥的治疗选择很少,并且在该领域中对于抗体介导的排斥的有效治疗存在巨大的未满足需求。使用cart/nk细胞删除b细胞或浆细胞或两者提供抗体介导的排斥的治疗。
所公开的公开内容提供了与car工程改造细胞有关的组合物和方法,所述car工程改造细胞靶向表达cd19或cd20的细胞,其导致b细胞的缺失但在具有抗体介导的器官排斥或自身免疫病症的患者中备用长寿浆细胞,包括但不限于,系统性红斑狼疮(sle),类风湿性关节炎(ra)和寻常型天疱疮和多发性硬化症(ms)。car对b细胞的缺失有利于降低疾病活动。
本发明公开内容还提供了与car工程改造细胞相关的组合物和方法,所述car工程改造细胞靶向表达bcma或baff-r,taci的细胞,其导致患有抗体介导的器官排斥或自身免疫病症的患者中浆细胞的缺失,所述自身免疫病包括但不限于全身性红斑狼疮(sle),类风湿性关节炎(ra)和寻常型天疱疮和多发性硬化症(ms)。浆细胞的缺失可有助于降低疾病活动。
在一些实施例中,本发明公开内容提供了与car工程改造细胞相关的组合物和方法,所述car工程改造细胞用于消耗成熟的记忆b细胞和短的长寿浆细胞,用于治疗自身免疫疾病和器官抗体介导的器官排斥。在一个实施例中,本发明公开内容提供了使用以下策略中的一种或多种消耗成熟的记忆b细胞和短寿命的浆细胞的方法:
1)通过使所述细胞与具有针对cd19或cd20或cd22的scfv的car工程改造细胞接触,消耗成熟的记忆b细胞和短寿命的浆细胞;
2)通过使所述细胞与具有针对bcma或taci或baff-r的scfv的car工程改造细胞接触来消耗短寿命和长寿浆细胞;要么
3)通过使所述细胞与具有包含bcma或taci或baff-r结合结构域(baff或april)的抗原识别结构域的car工程改造细胞接触来消耗短寿命和长寿浆细胞;
4)缺失成熟的记忆b细胞和接触所述细胞的短寿命的浆细胞,其中靶向多于一种不同抗原的复合car工程改造细胞为具有抗体介导的器官排斥或自身免疫疾病的患者提供疾病活性的降低。
5)通过接触靶向选自cd19,cd20,cd22,bcma,taci,april和baff的多于一种不同抗原的car工程改造细胞,删除成熟的记忆b细胞和短寿命的浆细胞。
在一些实施例中,复合car(ccar)靶向表达baff-r,bcma,taci和cs1抗原中的一种或两种或全部的细胞。
在一些实施例中,ccar中car的单位可包含:1)针对baff-r,bcma,taci和cs1的scfv;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
在一些实施例中,baffcar可以是ccar中car的单位,其包含:1)bcma或taci或baff-r结合结构域;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
在一些实施例中,aprilcar可以是ccar中car的单位,其包含:1)bcma或taci结合结构域;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
进一步的实施例中,bcma或tac1或baff-r结合结构域可以是april和baff分子的一部分或全部。
在一些实施例中,ccar中car的单位可包含:1)针对bcma或cs1的scfv;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
在一些实施例中,ccar中car的单位可包含:1)针对bcma或cd19的scfv;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
在一些实施例中,ccar中car的单位可包含:1)针对bcma或cd20的scfv;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
在一些实施例中,ccar中car的单位可包含:1)baff-r结合结构域或针对bcma的scfv;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
在一些实施例中,ccar中car的单位可包含:1)baff-r结合结构域或针对cd19的scfv;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
在一些实施例中,ccar中car的单位可包含:1)baff-r结合结构域或针对cd20的scfv;2)铰链区域;3)共刺激结构域和细胞内信号传导结构域。
出乎意料的是,一些骨髓瘤细胞对于bcma是暗淡的(弱的)或阴性的。为了增加car抗原识别在骨髓瘤细胞中的敏感性,靶向多种抗原来治愈这种疾病至关重要。
taci,bcma和baff-r是baff的受体。还出乎意料的是,一些骨髓瘤细胞在bcma上表达taci或baff-r。
在一些实施例中,本发明公开提供了包含baffcar的方法,所述baffcar靶向表达受体,baff-r,taci和bcma中的至少一种的细胞,以提高治疗功效并降低抗原疾病逃逸的风险。
baff与bcma结合的亲和力在微摩尔范围内,其远低于纳摩尔范围内的baff-r或taci。
在一些实施例中,本发明公开内容提供了产生包含baff和bcmacar的复合ccar的方法,以进一步消除一些不能被baffcar消除的骨髓瘤细胞。
在进一步的实施例中,ccar可包含一个或两个或多个car单元。每个单元car可以具有相同或不同的铰链区和共刺激结构域。
在进一步的实施例中,ccar可包含两个或更多个car多肽单元的多肽。每个单元car可以携带不同的多核苷酸序列以避免同源重组。
在一些实施例中,通过合并的car工程改造细胞可以实现靶向一种以上不同的抗原,所述car工程改造细胞由至少两种单独的cart或nk细胞产生。如本文所用,合并的car工程改造细胞包括具有一个以上不同的car多肽单元的工程改造细胞群。举例来说,合并的工程改造细胞包括具有不同car多肽的工程改造细胞群和具有不同的car多肽的工程改造细胞群。此外,合并的car工程改造细胞包括具有ccar多肽的工程改造细胞。
合并的cart或nk细胞可通过以下步骤完成:
1)产生靶向不同抗原的至少两种单独的car构筑体;
2)将单个构筑体转导至t或nk细胞并在标准培养基中离体扩增;
3)以适当的比例汇集不同的扩增的t或nk细胞;和
4)将合并的cart或nk细胞给予受试者。
或者,不同的工程改造细胞可以单独扩增并独立或顺序施用。
在进一步的实施例中,靶抗原可包括该组中的至少一种,但不限于ror1,psma,magea3,糖脂,磷脂酰肌醇蛋白聚糖3,f77,gd-2,wt1,cea,her-2/neu,mage-3,mage-4,mage-5,mage-6,甲胎蛋白,ca19-9,ca72-4,ny-eso,fap,erbb,c-met,mart-1,cd30,egfrviii,免疫球蛋白κ和λ,cd38,cd52,cd3,cd4,cd8,cd5,cd7,cd2和cd138。靶抗原还可以包括病毒或真菌抗原,例如来自人乳头瘤病毒(hpv)或ebv(epsteinbarr病毒)抗原的e6和e7。
在一些实施例中,ccar工程改造细胞靶向表达cd19或cd20抗原或其两者皆有的细胞。在另一个实施例中,ccar工程改造细胞靶向表达cd19或cd22抗原或两者皆有的细胞。靶细胞是与自身免疫疾病或癌细胞如b细胞淋巴瘤或白血病相关的正常b细胞。
急性移植物抗宿主病(gvhd)仍然是异基因造血干细胞移植后发病率和死亡率的最重要原因。在gvhd的效应阶段,t细胞受体(tcr),α和β链的异二聚体,在t细胞表面表达,tcr识别宿主细胞上hla分子上的一些抗原,增强t细胞增殖,并释放导致宿主细胞损伤的细胞毒性剂。tcr基因失活在防止潜在的移植物抗宿主反应方面是有效的。tcr的失活可导致tcr识别同种异体抗原并因此防止gvhd。
cd45在nk细胞中的作用与t细胞的作用完全不同。来自cd45的小鼠的nk细胞对原型肿瘤细胞系yac-1具有正常的细胞毒活性。此外,cd45缺陷型nk细胞正常增殖并对il-15和il-21起反应。因此,cd45破坏或缺失不会影响nk细胞的杀伤和增殖。
本发明公开内容包括在t或nk细胞中永久性缺失cd45并随后稳定引入cd45特异性car的方法。结果,工程改造的t细胞显示出cd45重定向特异性的所需特性,而不会引起自杀和对抗原呈递的反应。在另一个实施例中,工程改造t细胞可以作为用于治疗恶性肿瘤或其他疾病的现成疗法具有功效。
本发明公开涉及一种方法,其中t细胞被工程改造以通过内源性cd45的失活或缺失,减少或丧失tcr信号传导时允许增殖。tcr信号传导的减少或丧失可导致预防gvhd。
在另一个实施例中,通过cd45的失活或减少或丧失tcr信号传导的t细胞可以用作“现成的”治疗产品。
本发明公开包括修饰的t或nk细胞之方法,其包括:(a)通过灭活cd45修饰t或nk细胞;(b)扩展这些修饰细胞;(c)分选不表达cd45的修饰的t或nk细胞;(d)引入cd45car。
在实施例中,cd45car基因编码嵌合抗原受体(car),其中car包含抗原识别结构域,铰链区,跨膜结构域和t细胞刺激结构域中的至少一种,并且抗原识别结构域被重定向。cd45表面抗原存在于细胞上。抗原识别结构域包括单克隆抗体或针对cd45抗原的多克隆抗体。抗原识别结构域包括单克隆抗体或多克隆抗体的结合部分或可变区。
本发明公开包括通过共表达分泌型il-15/il-15sushi复合物来改善cd45cart/nk细胞扩增,持久性和抗肿瘤活性的方法。在另一个实施例中,工程改造的cd45cart/nk细胞包含分泌型il-15/il-15sushi复合物,其可以促进特定cart/nk细胞的扩增,并促进先天免疫细胞向肿瘤位点的浸润,导致肿瘤破坏。
本发明公开内容提供了另一种策略,其中工程改造cd45cart细胞不仅通过cd28途径接受共刺激,而且还共表达4-1bb配体(cd137l),其提供高治疗功效。
在一些实施例中,修饰的t细胞获自同种异体供体并用作“现成产品”。
使用cart或nk细胞靶向cd45可能导致自杀,因为t和nk细胞表达该表面抗原。为了克服该缺点,本发明公开内容提供了缺乏cd45的工程改造细胞。如本文所用,当基因表达减少或消除时,工程改造细胞缺乏特定基因。表达的减少或消除可以通过本领域已知的任何遗传方法完成。在一个实例中,cd45基因可以通过使用工程改造的crispr/cas9系统,锌指核酸酶(zfn)和tale核酸酶(talen)和大范围核酸酶来灭活。用靶向表达cd45的肿瘤的car进一步转导t或nk细胞中cd45的丧失。
本发明公开包括用于消除或减少骨髓,血液和器官中的异常或恶性细胞的方法。在一些实施例中,表达cd45的恶性细胞存在于患有急性白血病,慢性白血病t细胞淋巴瘤,骨髓性白血病,急性淋巴细胞性淋巴瘤或白血病,原发性积液淋巴瘤,网状组织细胞瘤,唐氏综合征的短暂性骨髓增生性疾病,淋巴细胞为主的霍奇金淋巴瘤,骨髓性白血病或肉瘤,树细胞瘤,组织细胞肉瘤,腱鞘巨细胞瘤,交叉树突状细胞肉瘤,移植后淋巴组织增生性疾病等的患者中。
造血干细胞移植(hsct)已被广泛用于治疗血液系统恶性肿瘤或非血液系统疾病。非血液学疾病包括遗传疾病和免疫缺陷和自身免疫疾病。遗传疾病包括但不限于镰状细胞病,地中海贫血和溶酶体贮积病。在干细胞移植之前,患者需要接受条件疗法以消耗骨髓壁龛中的造血干/祖细胞,以促进供体干细胞植入和增殖。高剂量的化学疗法和全身照射用于条件疗法,其引起严重的毒性和长期并发症,特别是在非造血器官例如严重的肠粘膜炎中。此外,传统的条件疗法可能破坏骨髓壁龛,从而导致造血细胞恢复。安全问题是将hsct扩展到各种非血液疾病的主要障碍。cd45仅在造血细胞上表达,靶向cd45应尽量减少对非造血器官的毒性。
在一些实施例中,cd45car细胞用于通过去除造血细胞在骨髓中产生用于骨髓干细胞移植的空间,同时去除白血病/淋巴瘤细胞或能够移植排斥的免疫细胞。
在一些实施例中,cd45car工程改造细胞用于消耗造血干/祖细胞,同时保留骨髓的结构和脉管系统,与全身照射对这些组织的破坏作用形成对比。保留骨髓的结构和脉管系统允许在瞬时cd45car治疗后更快的造血恢复。
在另一个实施例中,cd45car细胞可以在进行骨髓移植以接受干细胞之前用于患者的预处理。在另一个实施例中,cd45car可用作造血干细胞移植的髓源性预处理方案。
在优选的实施例中,cd45car工程改造细胞疗法是短暂的,以允许更快地恢复骨髓和外周造血细胞。可以通过使用短寿命工程改造细胞或提供具有如本文所述的自杀系统的工程改造细胞来完成瞬时治疗。
在一些实施例中,本发明公开内容包括选择性消除或消融内源性造血干/祖细胞群的方法,其中所述内源性造血干/祖细胞表达cd45,通过使所述细胞与特异性靶向表达cd45的cd45car工程改造细胞接触造血干/祖细胞。
在一些实施例中,cd45car细胞用于在干细胞移植和/或化学疗法后治疗或预防残留疾病。
在一些实施例中,cd45car是表达基因或盒的一部分。在优选的实施例中,除cd45car外,表达基因或盒还包括辅助基因或标签或其部分。辅助基因可以是诱导型自杀基因或其部分,包括但不限于半胱天冬酶9基因,胸苷激酶,胞嘧啶脱氨酶(cd)或细胞色素p450。“自杀基因”消融方法提高了基因治疗的安全性,并且仅在被特定化合物或分子激活时杀死细胞。在一些实施例中,自杀基因是可诱导的并且使用特定的二聚化学诱导剂(cid)活化。
在一些实施例中,安全开关可以包括辅助标签是c-myc标签,cd20,cd52(campath),截短的egfr基因(egfrt)或其部分或组合。附件标签可以用作非免疫原性选择工具或用于跟踪标记。在一些实施例中,安全开关可包括对应于rsvf糖蛋白a2菌株(nsellslindmpitndqkklmsnn)的残基254-277的24残基肽。在一些实施例中,安全开关可包括由单克隆抗tnfα药物结合的tnfα的氨基酸序列。
il-15及其受体在增强cart和nk细胞功能中的作用
最近的研究已经证明t细胞持久性与cart细胞治疗功效很好地相关。最近的试验证明,注入少量cart细胞可以产生有效和持久的抗肿瘤活性,这表明输注产品的质量而不是数量对于促进抗肿瘤活性更重要。
白细胞介素(il)-15是促进淋巴细胞发育和止血的细胞因子。增加的il-15水平促进t细胞增殖并增强t细胞效应子反应。最近研究的数据表明,il-15对于记忆cd8t细胞的产生和维持至关重要,这是与抗肿瘤活性相关的关键因素之一。il-15结合il-15受体α链(也称为il-15ra或ra),有助于il-15介导的作用,例如t细胞存活,增殖和记忆t细胞的产生。
il-15ra通过与t细胞和其他类型细胞的细胞表面上的il-15ra/βγ复合物结合,结合t细胞和il-15信号表面中的βγ复合物。
单独转染il-15不会显着影响t细胞功能,il-15/1il-15ra的转染允许t细胞自主存活和增殖。
单独施用il-15的功效可能受到游离il-15ra的可用性及其短半衰期的限制。可溶性il-15/ra复合物的施用极大地增强了体内ii-15半衰期和生物利用度。因此,用这种复合物处理小鼠,但不是单独的il-15处理,导致记忆cd8t细胞和nk细胞的强烈增殖和维持。il-15ra的细胞外区域的一部分称为sushi结构域(il-15sushi),其与il-15的结合是必需的(wei等人,j.immunol.,vol.167(1),p:277-282,2001)。il-15/sushi融合蛋白也称为il-15/il-15sushi,其含有的接头比il-15和单独的可溶性il-15ra(il-15sushi)更有效。il-15/ra(膜结合形式)或il-15/sushi(可溶形式)的组合可以使il-15活性最大化。通过保持il-15与转导细胞表面上的il-15ra结合,膜结合形式il-15/ra不会释放游离il-15。
在本发明公开中,出乎意料地发现从转导细胞释放的可溶性il-15/il-15sushi能够促进转导细胞及其相邻细胞的扩增,并进一步刺激它们抵抗肿瘤。
本发明公开内容提供了在单一构筑体中具有car和il-15/il-15sushi或il-15/ra的工程改造细胞。在一些实施例中,本发明公开内容包括产生更高病毒滴度并使用更强启动子来驱动car和il-15/ra或il-15/il-15sushi的方法。
在一些实施例中,本发明公开内容提供了工程改造细胞,其具有:(1)靶向抗原的car,包括但不限于cd4,cd2,cd3,cd7,cd5,cd45,cd20,cd19,cd33,cd123,cs1和b细胞成熟抗原(bcma);(2)il-15;(3)il-15ra(膜结合)或sushi(il-15/il-15sushi)。在进一步的实施例中,car包含嵌合抗原受体,一种或多种共刺激内部结构域,包括但不限于cd28,cd2,4-1bb,4-1bbl(cd137l),b7-2/cd86,ctla-4,b7-h1/pd-l1,icos,b7-h2,pd-1,b7-h3,pd-l2,b7-h4,cd40配体/tnfsf5,dppiv/cd26,dap12和ox40,以及cd3ζ链的细胞内结构域。在进一步的实施例中,强启动子可以是但不限于sffv。car,il-15/ra或sushi和诱导型自杀基因(“安全开关”)或组合可以组装在载体上,例如慢病毒载体,腺病毒载体和逆转录病毒载体或质粒。“安全开关”的引入可以显着提高安全性,并限制car的靶向或非肿瘤毒性。
在一个实施例中,工程改造细胞包括cd2嵌合抗原受体多肽和il-15/il-15sushi(seqidno.102),和相应的多核苷酸(seqidno.101)。不希望受理论束缚,据信il-15/il-15sushi与cd2car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,工程改造细胞包括cd3嵌合抗原受体多肽和il-15/il-15sushi(seqidno.104),和相应的多核苷酸(seqidno.103)。不希望受理论束缚,据信il-15/il-15sushi与cd3car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,工程改造细胞包括cd7嵌合抗原受体多肽和il-15/il-15sushi(seqidno.106),和相应的多核苷酸(seqidno.105)。在一些实施例中,本发明公开内容提供了通过向有需要的患者施用共表达il-15/il-15sushi的cd7car工程改造细胞向癌症患者提供长期持久缓解之方法。不希望受理论束缚,据信il-15/il-15sushi与cd7car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
在一个实施例中,工程改造细胞包括cd5嵌合抗原受体多肽和il-15/il-15sushi(seqidno.107),和相应的多核苷酸(seqidno.108)。在一些实施例中,本发明公开内容提供了通过向有需要的患者施用共表达il-15/il-15sushi的cd5car工程改造细胞向癌症患者提供长期持久缓解之方法。不希望受理论束缚,据信il-15/il-15sushi与cd5car的共表达通过募集先天性免疫细胞到癌细胞或增加car识别靶向的敏感性向患者提供长期持久的缓解。
car靶向cd4+cd25+调节性t细胞
调节性t细胞(tregs)又称抑制性t细胞,是调节免疫系统和维持自身抗原耐受的t细胞亚群。treg约占血液中cd4+t细胞总量的1-5%,在移植、过敏、哮喘、传染病、移植物抗宿主病(gvhd)、自身免疫等方面的临床应用多种多样。treg的生物标志物有cd4、foxp3和cd25。treg被认为是来源于原始cd4细胞。
在癌症中,treg在抑制肿瘤免疫和阻碍机体控制癌细胞生长的先天能力方面发挥着重要作用。
treg在癌症患者中扩展,通常在肿瘤微环境中富集。tregs浸润肿瘤,抑制抗肿瘤免疫。treg的耗竭能使小鼠对正常生长的肿瘤产生排斥反应。
使用针对cd25的抗体耗尽treg会导致部分treg降低,但抗肿瘤活性有限。treg的高水平消耗是一个深刻的抗肿瘤效果所必需的。此外,tregs与活化的cd4+和cd8+淋巴细胞以及活化的nk细胞共享cd25表达,在特异性方面存在显著问题。一般来说,treg很难从效应子t细胞和nk细胞中有效的识别出来,这使得treg的研究非常困难。
在一个实施例中,该工程改造细胞包括具有cd4抗原识别区域的第一嵌合抗原受体多肽和具有cd25抗原识别区域的第二嵌合抗原受体多肽。在一个实施例中,该工程改造细胞包括具有cd45前导序列的多肽seqidno.92和相应的多核苷酸seqidno.91。
在一个实施例中,该工程改造细胞包括具有cd4抗原识别区域的第一嵌合抗原受体多肽和具有cd25抗原识别区域的第二嵌合抗原受体多肽。在一个实施例中,该工程改造细胞包括具有cd8a前导序列的多肽seqidno.94和相应的多核苷酸seqidno.93。
用于t调节细胞的具体实施例
t淋巴细胞(t细胞)是白细胞的一种亚型,在细胞介导免疫中起着关键作用。t细胞分为cd4细胞和cd8细胞。自然杀伤细胞(nk细胞)是一种对先天免疫至关重要的细胞毒性细胞。
调节性t细胞(tregs)是介导免疫耐受和抑制的cd4+细胞,与辅助t细胞不同。tregs表达cd4、cd25和foxp3(cd4cd25+调节性t细胞)。
treg在肿瘤微环境中富集,被认为是阻碍抗肿瘤免疫反应和促进癌细胞耐受的重要物质。肿瘤中浸润性treg的增加与包括血液病恶性肿瘤和实体肿瘤在内的多种癌症的生存率低有关。
treg似乎优先转运到肿瘤微环境中,并在免疫抑制中发挥关键作用(ethanm.shevach等人,annualreviewofimmunology,vol.18:423-449,2000)。
通过靶向cd25,许多不同的方法被用来删除用于癌症治疗的treg,从而导致treg的部分减少。然而,这可能是有问题的,因为:(1)cd25也在活化的cd4、cd8和nk细胞中表达。cd25在活化的b细胞、巨噬细胞、成骨细胞、胸腺细胞、髓系前体和寡齿细胞中表达。(2)treg的消耗对疗效的要求非常高(xingruili等人,eur.j.immunol.2010.40:3325–3335)。
car的设计是将患者或供体的免疫细胞以一种主要组织相容性复合体(mhc)独立的方式重新定向到特定的“目标”抗原上。car蛋白结构通常包括许多模块组件或对功能积分的区域。“目标”识别模块是细胞外单链可变片段(scfv)。该成分来源于一种单克隆抗体,该单克隆抗体对精心选择的目标抗原具有特定的方向。与scfv串联的合适长度的铰链区域传递scfv区域的可移动性,以便与目标蛋白进行最佳结合。跨膜区域是细胞外结合区域和共刺激域(如cd28和/或4-1bb)之间的通道。最后一个模块包括cd3zeta细胞内信号区域。
本发明提供了一种针对细胞共表达cd4和cd25的新型tregcar(也称为cd4zetacd25car或c4-25zcar)构建的方法。tregcar特别消耗treg,同时保留了大多数不能共同表达cd4和cd25的细胞。
在一些实施例中,t细胞激活可以在tregcar中两个scfv分子同时作用于cd4和cd25时实现。在进一步实施例中,t细胞激活和共刺激信号均使用两个不同/分离的嵌合抗原受体多肽提供。
在某些实施例中,tregcar包括(1)第一嵌合抗原受体多肽单元,该多肽单元包括第一信号肽、第一抗原识别域、第一铰链区、第一跨膜域和细胞内信号域;和(2)第二个嵌合抗原受体多肽单位组成第二个信号肽,第二个抗原识别领域,第二个铰链区,第二个跨膜域和共刺激域(s)。第一个嵌合抗原受体多肽单位和第二个嵌合基因工程改造多肽单位表表达单一多肽分子上,其中,在第一嵌合抗原受体多肽单元与第二嵌合抗原受体多肽单元之间设有包含高效裂解位点的氨基酸序列。
在某些实施例中,tregcar增强了共表达cd4和cd25的细胞的裂解活性,同时最小化了仅携带cd4或cd25抗原的细胞。
在某些实施例中,为了避免同源重组,第一个嵌合抗原受体多肽单元的核苷酸序列与第二个嵌合工程改造多肽单元不同。
在某些实施例中,tregcar中的高效裂解位点为p2a。
在一些实施例中,第一个抗原识别域的目标为cd4或cd25,第二个抗原识别域的目标为cd4或cd25;其中,第一个抗原识别域与第二个抗原识别域不同。
在一个实施例中,抗原识别区域包括针对(选择性地)目标的单克隆或多克隆抗体的结合部分或可变区域。在进一步实施例中,目标抗原为cd4或cd25。
在一些实施例中,包含针对不同或相同抗原的tregcar的t或nk细胞。
在一些实施例中,t或nk细胞包括tregcar,这些tregcar以表达cd4和cd25的treg为靶标,同时保留了大多数细胞,这些细胞不能共同表达cd4和cd25。
在一些实施例中,t或nk细胞包括耗尽treg的tregcar。
在一些实施例中,本发明提供了一种tregcar的生成方法,该方法可用于治疗或预防受试者中与cd4+cd25+foxp3+t调节细胞(treg)相关的疾病。在另一个实施例中,使用tregcar治疗的疾病包括但不限于癌症。
在一些实施例中,目前披露代tregcar提供一种方法用于治疗或预防cd4+cd25+foxp3+t调控细胞(treg)相关的癌症包括,但不局限,肝细胞癌,纤维板层癌,肝母细胞瘤,未分化胚胎肉瘤和肝,肺鳞状细胞癌,睾丸非精原细胞生殖细胞肿瘤,脂肪肉瘤,卵巢和性腺外卵黄囊瘤,卵巢绒毛膜癌,畸胎瘤,卵巢透明细胞癌,胎盘部位的间充质错构瘤滋养细胞肿瘤,淋巴瘤和白血病。
在一些实施例中,本发明公开提供了一种用于抑制受试者肿瘤生长的tregcar生成方法。
在某些实施例中,tregcar与目前正在开发或市场上可用的任何化疗药物联合使用。在某些实施例中,tregcar被用作包括但不限于血液病恶性肿瘤和癌症在内的疾病的一线治疗。
在一些实施例中,表达tregcar的细胞与免疫调节药物(如,但不限于,ctla-4和pd-1/pd-l1阻滞)或细胞因子(如il-2、il-15或il-15/il-15sushi或il-15/ra和il-12或集落刺激因子-1受体抑制剂(csf1r)共同施用,以获得更好的治疗效果。
在某些实施例中,表达tregcar的细胞与其他免疫调节药物或car表达细胞共同施用,以在受试者中提供协同效应。
在具体实施例中,表达tregcar的细胞可以是t细胞或nk细胞,给哺乳动物(如人类)喂食。
在一些实施例中,tregcar被用于癌症的免疫治疗。癌症可从肺癌、黑色素瘤、乳腺癌、前列腺癌、结肠癌、肾癌、卵巢癌、宫颈癌、头颈癌、胃癌、肝癌、神经母细胞瘤、横纹肌肉瘤、白血病和淋巴瘤组中选择。本发明中描述的组合物和方法可以与其他癌症治疗类型一起使用,如化疗、手术、放疗、基因治疗等。
为了提高安全性或治疗指标,本发明公开的tregcar可以构建为短暂的rna修饰的“生物降解”版本或衍生物,或其组合。本发明的rna修饰car可以电穿孔进t细胞或nk细胞。tregcar的表达可能会在几天内逐渐减弱。
使用tregcar在主题中治疗癌症的方法体现在本发明公开中。该方法包括:
(1)从受试者或供体获得t/nk细胞。
(2)培养淋巴细胞/t细胞或nk细胞。
(3)将tregcar结构引入培养的t细胞或nk细胞中。
(4)tregcart细胞或nk细胞扩增。
(5)用治疗有效的剂量治疗受试者。
肿瘤浸润淋巴细胞(tils)的体外扩增在目前的过继细胞治疗中得到了成功的应用。在一个实施例中,获取tils并成功地在体外扩展。
在一些实施例中,可以从肿瘤组织样本中获取tils并扩大tils的数量。tregcar-t或nk细胞被用于与tils共培养,以消耗treg细胞,增强til对癌症的反应,这对疾病治疗是有价值的。
在一些实施例中,cd4car携带一组car构筑体,包括抗原识别域、铰链区、共刺激域和细胞内域(cd3zeta链)。在另一个实施例中,cd4car消耗treg,然后增强t细胞对癌细胞的反应,提高抗肿瘤活性的治疗效果。
在一些实施例中,cd25car携带一组car构筑体,包括抗原识别域、铰链区、共刺激域和细胞内域(cd3zeta链)。在另一个实施例中,cd25car消耗treg,然后增强t细胞对癌细胞的反应,提高抗肿瘤活性的治疗效果。
参考下面列出的实施例可以更好地理解本发明公开。提出以下实施例以向本领域普通技术人员提供如何制备和评价本文要求保护的化合物,组合物,制品,装置和/或方法的完整公开和描述,并且旨在纯粹地示例性的并且不旨在限制本发明公开。
可以通过共同施用car增强自来补充本文所述的任何工程改造细胞的施用。car增强自的实例包括增强car活性的免疫调节药物,例如但不限于靶向免疫检查点途径的药剂,集落刺激因子-1受体(csf1r)的抑制剂以获得更好的治疗结果。靶向免疫检查点途径的药剂包括结合抑制性免疫受体ctla-4,pd-1和pd-l1的小分子,蛋白质或抗体,并导致ctla-4和pd-1/pd-l1阻断。如本文所用,增强子包括如上所述的增强子。
这里使用的“病人”包括哺乳动物。此处所指的哺乳动物可以是任何哺乳动物。本法所称哺乳动物,是指任何哺乳动物,包括但不限于啮齿目哺乳动物,如小鼠、仓鼠等,以及兔等啮齿目哺乳动物。哺乳动物可能来自食肉目,包括猫科和犬科。哺乳动物可能属于偶蹄目,包括牛(牛)和猪(猪),也可能属于蹄目,包括马(马)。哺乳动物可能属于灵长目、头鲸目或类人猿目(猴子),也可能属于类人猿目(人类和类人猿)。最好,哺乳动物是人类。病人包括受试者。
在特定的化身,病人是一种人类0到6个月大的时候,6到12个月大的时候,1-5岁,5到10岁,5到12岁,10到15岁,15到20岁,13至19岁,20到25岁,25-30岁,20到65岁,30到35岁,35至40岁,40到45岁,45至50岁,50到55岁,55到60岁,60到65岁,65年到70岁,70-75岁,75年到80岁,80年到85年的历史,85到90岁,90到95岁或者95到100岁。
本文所用的术语“有效量”和“治疗有效量”是指足以提供所需治疗或生理或效果或结果的工程改造细胞。这样的效果或结果包括减少或改善细胞疾病的症状。不良影响,例如副作用,有时表现出所需的治疗效果;因此,从业者在确定适当的“有效金额”时,将潜在利益与潜在风险进行平衡。所需的确切量将因患者而异,取决于患者的种类,年龄和一般状况,给药方式等。因此,可能无法指定确切的“有效量”。然而,本领域普通技术人员仅使用常规实验可确定任何个别情况下的适当“有效量”。通常,工程改造细胞以足以减少靶细胞增殖的量和条件给予。
在施用用于治疗,抑制或预防癌症的递送系统后,可以以本领域技术人员熟知的各种方式评估治疗性工程改造细胞的功效。例如,本领域普通技术人员将理解,通过观察治疗性工程改造细胞减少癌细胞负荷或预防,与化学佐剂联合递送的治疗性工程改造细胞在治疗或抑制患者的癌症方面是有效的。癌细胞负荷进一步增加。癌细胞负荷可以通过本领域已知的方法测量,例如,使用聚合酶链反应测定来检测某些癌细胞核酸的存在或使用例如抗体鉴定血液中的某些癌细胞标记物。用于检测来自受试者或患者的样品(例如但不限于血液)中标志物的存在的测定法,或通过测量患者体内循环癌细胞抗体水平的水平。
在整个说明书中,数量由范围以及范围的下边界和上边界定义。每个下边界可以与每个上边界组合以定义范围。下边界和上边界应分别作为单独的元素。
在整个说明书中对“一个实施例”,“实施例”,“一个示例”或“示例”的引用意味着结合该实施例或示例描述的特定特征,结构或特性包括在至少一个实施例中。本实施例。因此,贯穿本说明书在各个地方出现的短语“在一个实施例中”,“在实施例中”,“一个示例”或“示例”不一定都指代相同的实施例或示例。此外,特定特征,结构或特性可以在一个或多个实施例或示例中以任何合适的组合和/或子组合进行组合。另外,应理解,此处提供的附图仅用于解释本领域普通技术人员的目的,并且附图不一定按比例绘制。
如本文所使用的,术语“包括”,“包含”,“包括”,“包括”,“具有”,“具有”或其任何其他变型旨在涵盖非排他性的包含。例如,包括元素列表的过程,物品或装置不一定仅限于那些元素,而是可以包括未明确列出的或该过程,物品或装置固有的其他元素。
此外,除非有相反的明确说明,否则“或”是指包含性的“或”而不是排他性的“或”。例如,条件a或b由以下任何一个满足:a为真(或存在)且b为假(或不存在),a为假(或不存在)且b为真(或存在),a和b都是真的(或存在)。
另外,本文给出的任何示例或说明不以任何方式被视为对使用它们的任何术语的限制,限制或表达定义。相反,这些示例或说明应被视为关于一个特定实施例进行描述并且仅是说明性的。本领域普通技术人员将理解,利用这些示例或说明的任何术语或术语将包括可以或可以不在其中或在说明书中的其他地方给出的其他实施例,并且所有这些实施例旨在包括在该术语或术语的范围。指定这种非限制性示例和说明的语言包括但不限于:“例如”,“例如”,“例如”和“在一个实施例中”。
在本说明书中,描述了包含多个成员的各种参数的组。在一组参数内,每个成员可以与任何一个或多个其他成员组合以产生另外的子组。例如,如果组的成员是a,b,c,d和e,则特别考虑的其他子组包括任何一个,两个,三个或四个成员,例如a和c;a,d和e;b,c,d和e;等等
如本文所用,xxxx抗原识别结构域是对xxxx具有选择性的多肽。“xxxx”表示如本文和上文所讨论的目标。例如,cd38抗原识别结构域是对cd38特异的多肽。
如本文所用,cdxcar是指具有cdx抗原识别结构域的嵌合抗原受体。
如本文所用,car工程改造细胞是如本文所述的工程改造细胞,其包括嵌合抗原受体多肽。举例来说,cd45工程改造细胞是具有本文公开的cd45嵌合抗原受体多肽的工程改造细胞。
如本文所用,复合car(ccar)工程改造细胞是如本文所述的工程改造细胞,其包括至少两种不同的嵌合抗原受体多肽。举例来说,cd19cd22复合car工程改造细胞是如本文所述的工程改造细胞,其包括具有cd19抗原识别结构域的第一嵌合抗原受体多肽和具有cd22抗原识别结构域的第二嵌合抗原受体多肽。
参照下面列出的例子,本披露可能更容易被理解。下面的例子提出提供普通的技能在艺术与一个完整的披露和描述的化合物,成分、文章、设备和/或声称此方法和评估,目的是纯粹的模范,并不是为了限制披露。
实例
ccar靶向工程改造细胞表达cd33或cd123或两者兼有
cd33cd123ccar之建筑遵循图1及图2a中之示意图。其包括驱动具有两个不同car单元之功能性复合car(ccar)之表达的sffv(脾病灶形成病毒)启动子。抗原受体朝向,抗cd33及抗cd123之scfv(单链可变片段)核苷酸序列。由于对于双顺反子基因构筑体之自裂解动力学之高效机制而使用来源于小rna病毒之p2a肽。自裂解p2a肽用于在表达期间将car、cd33car及cd123car中之两个独立单元连接在一起。此方法相比于文献中通常使用之内部核糖体进入位点(ires)之优势包括其尺寸较小及在2a肽之两个上游及下游单元蛋白质之间的高裂解功效。此外,当使用ires时,使用自裂解p2a肽可避免ires之前及之后基因之间的表达量差异问题。
模组单元,cd33car包括cd33scfv域、cd8a铰链区、cd8a跨膜域、4-bb共刺激域及cd3ζ链之胞内域。第二模组car,cd123car具有与cd33car相同的铰链、跨膜及细胞内信号传导域,但具有不同的scfv及共刺激域。cd33car识别其相应抗原且cd123car结合于其相应抗原。铰链区经设计使得避免其中二硫化物相互作用之序列。使用不同的共刺激域4-bb及cd28。将cd33cd123复合car次选殖至慢病毒质体中。
产生高效复合car(ccar)
借由根据制造商说明用脂染胺2000(lipofectamine2000)转染hek-293ft细胞来产生复合car慢病毒,但由于插入物之尺寸较大而使用2倍载体dna以增加效价,如图2中所示。在约12-16小时培育之后,移出含有脂染胺之培养基且置换为含有10%fbs、20mmhepes、1mm丙酮酸钠及1mm丁酸钠之dmem。在约24小时之后,收集且冷藏上清液,且置换为新鲜培养基。在约24小时之后,收集上清液且与前述上清液合并,且经由0.45μm滤片过滤。将上清液分成等分试样,用液氮急骤冷冻且在-80℃下储存。收集hek-293ft细胞,冷冻储存,且溶解用于后续电泳及蛋白印迹法(图2b)。
pb(外周血)或cb(人类脐带血)白血球层细胞用抗cd3抗体及il-2活化2天。将ccar慢病毒上清液旋涂在经重组人纤维蛋白片段涂布之多孔培养盘上。在多个孔中用慢病毒上清液以约0.3×106个细胞/毫升之低浓度转导经活化之t细胞,以增加转导功效(图3)。
在第一次隔夜转导之后,除非细胞看起来不健康,否则细胞在不洗涤情况下直接添加至第二个经病毒涂布之盘中以用于第二次转导。在第二次隔夜转导之后,洗涤细胞,合并且在经组织培养物处理之培养盘中培育。使cart细胞扩增至多约5天,随后进行共培养杀死分析。在培育约3天之后,细胞与结合有生物素之山羊抗小鼠f(ab')2或山羊igg(同型)抗体一起培育,洗涤且接着与抗生蛋白链菌素-pe及结合之抗人类cd3一起培育。在洗涤且悬浮于2%福马林中之后,接着藉由流式细胞术分析细胞以测定转导功效百分比。
cd33cd123ccar之表征
经转染之cd33cd123ccarhek293t细胞经历蛋白印迹分析以确认符合构筑体。用抗cd3ζ单株抗体进行之免疫印迹法展示复合carcd3ζ融合蛋白之预测尺寸之谱带(图1b)。重要的是,如所预期,在信号传导p2a肽之成功高裂解作用之墨点上观测到具有类似强度之两个不同谱带。如所预期,未发现gfp对照载体之cd3ζ表达。亦在hek293细胞(图2c)及原生t细胞(图2c)上测试scfv之表面表达。
在hek293细胞系中测试复合cd33cd23car慢病毒之转导功效且藉由流式细胞术分析(beckmancoulter)(图2c)。流式细胞术表明约67%hek细胞表达cd33cd123car。人类外周血(pb)通常用于自体t细胞疗法。人类pb白血球层细胞用抗cd3抗体及il-2活化,且用cd4car或对照性(gfp)慢病毒转导。在转导之后,流式细胞术分析表明约22%t细胞表达cd33cd123car(图2c)。
来源于脐带血(ucb)及外周血(pb)之cd33cd123ccart细胞特异性杀死表达cd33之肿瘤细胞
分别在约100,000、200,000、500,000、约1百万或2百万个效应细胞至约50,000、100,000、200,000个标靶细胞下,cd33cd123ccart细胞或gfpt细胞(对照物)以0.5:1、50:1范围内之比率,较佳以约2:1、5:1、10:1、20:1、50:1之比率与标靶细胞一起在约1-2mlt细胞培养基中,在无il-2之情况下培育约24小时。标靶细胞为来自白血病患者之白血病细胞系及白血病细胞。在共培养约24小时之后,细胞用小鼠抗人类cd33、cd123、cd34及cd3抗体染色。
产生表达cd33car及cd123car之cd33cd123ccart细胞且使用hl60及kg-1a细胞系测试抗白血病功能。hl60细胞系为高度富集cd33之前髓细胞性白血病细胞系。约100%其细胞群体为cd33+,其中其小型子集(<10%)为dimcd123+。在培养物中,测试此细胞系以测定cd33cd123car之效用,其中强调靶向表达cd33之白血病细胞。此外,归因于hl60中之强cd33表达,cd33cd123ccar作用可为显著的。实际上,在具有多种效应子:标靶细胞之比率之24小时共培养条件期间,cd33cd123ccar呈现显著白血病细胞杀死特性(图4)。首先测试cb衍生之cd33cd123cart细胞杀死hl60细胞之能力。在约24小时培育及在约0.5:1至50:1,较佳1:1至约5:1,更佳约2:1至4:1范围内之低效应子:标靶(e:t)比率下,当与gfp对照物比较时,cd33cd123car细胞消除约55%表达cd33之hl60细胞。在约5:1之比率下,杀伤作用提升至约82%。
来源于外周血单核细胞(pbmc)之cd33cd123car与骨髓性白血病细胞系kg1a共培养,与hl60及50-80%cd123相比,kg1a亦以适度表达约100%cd33。因此,kg1a为相对双重标靶细胞群体,其对由cd33cd123car靶向之抗原具有双重阳性。使用约24小时培育及约0.5:1至50:1范围内之低效应子:标靶(e:t)比率。尽管在约2:1之低e:t比率下,cd33cd123car仍呈现适度的约26%之抗白血病活性,与gfp对照物相比,e:t比率增加至10:1可引起杀死约62%kg1a(图5),表明cd33标记物之强度可为杀伤力之指示物,其中强烈呈现hl60且与kg1a相比利用更多的car作用。此等实验提供完全cd33cd123car针对其相关抗原呈现细胞群体之功能的证据。
已产生其他复合car,cd33cd123-bbccar。此复合car包含两个独立car单元,cd33及cd123。第一car包含结合于cd33之scfv且第二car具有不同的识别cd123之scfv。两个car皆含有相同铰链区、跨膜、共刺激及细胞内域。产生cd33cd123-bbccar慢病毒且在kg-1a细胞中测试其杀伤力。如图5中所示,在约10:1之比率下存在实质性杀死,但其与cd33cd123ccar相比强度较弱。
cd33cd123ccar具有针对表达cd33及/或cd123之患者样品之活性
除细胞系实验以外,亦对患者样品进行研究以测试每个个别car单元之功能。使用侵袭性急性骨髓白血病(aml),aml-9测试cd33cd123ccar之功效。归因于患者细胞群体之非均质性,该细胞群体包括aml-9样品中之多种细胞类型,用cd34及cd33闸控白血病母细胞,因为其对此等两种标记物呈阳性。在cart细胞:标靶细胞之某一比率下,观测到白血病细胞之此cd33+cd34+群体之消耗比gfp对照物高48%(图6)。
亦测试cd123阳性且cd33阴性之白血病细胞。为此目的,选择人类b细胞急性淋巴细胞白血病(b-all)样品,sp-bm-b6。此样品中之所有白血病母细胞为cd34+cd33-,且超过约50%对cd123呈阳性。与gfp对照物相比,cd33cd123ccart细胞之cd34+白血病细胞群体消耗率为约86%(图7)。基于细胞系及人类样品研究,吾人之资料明确表明复合cd33cd123car能够靶向表达cd33或cd123或其两者之白血病细胞。
cd33cd123ccarnk细胞靶向表达cd33或cd23或其两者之白血病细胞
自然杀手(nk)细胞为cd56+cd3-且可有效杀死受感染及肿瘤细胞类cd8+t细胞。与cd8+t细胞不同,nk细胞发起针对肿瘤之细胞毒性而无需活化以杀死细胞。nk细胞比效应细胞更安全,因为其可避免细胞激素风暴(cytokinestorms)之潜在致死性并发症。然而,完全未探索使用cd33或cd123或两种carnk细胞杀死白血病。
产生cd33cd123ccarnk细胞
nk-92细胞用cd33cd123car慢病毒上清液转导两个连续隔夜,转导之间的变化在于经重组人纤维蛋白片段涂布之培养盘及经病毒涂布之培养盘。经转导之细胞扩增3或4天且接着藉由流式细胞术分析car表达。收集细胞且以约1:250与山羊抗小鼠f(ab')2一起培育约30分钟。洗涤细胞,悬浮且用抗生蛋白链菌素-pe染色约30分钟。洗涤细胞且悬浮于2%福马林中,且藉由流式细胞术分析。接着如上文所述标记表达cd33cd123ccar之nk-92细胞且在facsaria上分选,其中收集且培养顶部0.2%表达f(ab')2之细胞。经分选、扩增之细胞之后续标记展示约89%nk-92细胞对抗小鼠f(ab')2呈阳性(图8)。
cd33cd123ccarnk细胞有效溶解或消除白血病细胞
首先,吾人藉由评估cd33cd123ccarnk-92细胞杀死共培养物中hl-60癌细胞系之能力来测试其功能。几乎所有hl-60细胞皆高度表达cd33,但此细胞系中之cd123表达仅小于10%(弱)。因此,cd33cd123ccar之杀伤力可能取决于ccar正确靶向cd33之能力。
cd33cd123ccarnk-92细胞与hl-60细胞一起在无il-2之nk细胞培养基中共培养约24小时。在培育之后,标记cd33cd123ccarnk-92细胞且与非car之对照物gfpnk-92细胞比较。与对照物gfpnk-92细胞相比,观测到cd33cd123ccarnk-92细胞显著杀死hl-60细胞。此外,cd33cd123ccarnk-92细胞之杀伤力具有剂量依赖性,其中与对照物比较,约10:1比率为约100%(图9及11)。
使用kg1a、使用骨髓白血病细胞系进行第二共培养实验,kg1a在所有细胞中表达cd33,但与hl-60相比以中等量表达。cd123抗原表达于约50-80%kg1a细胞中。实验设计与上述hl-60杀死分析法之第一实验类似,其具有相同培育时间、效应子:癌细胞比率及gfpnk-92细胞对照物。结果展示与gfpnk-92细胞对照物相比,cd33cd123ccarnk-92细胞以剂量依赖性方式显著杀死kg1a细胞。在10:1之效应子:标靶之比率下,与gfp对照物相比,cd33cd123ccarnk-92细胞杀死约85%kg1a细胞(图10及11)。
kg1a细胞之分析展示两种不同群体cd33+cd123-及cd33+cd123-。图11展示在两个群体中发现之细胞杀死之剂量依赖性增加。惊人的是,双重阳性群体在各增加之比率下展示较高有效杀死,表明cd33及cd123之两个模组car之可能的协同作用(图12)。
我们还产生了复合的cd33cd123cart细胞,其不仅通过cd28共刺激而且还在单个构筑体中共表达4-1bb配体(4-1bbl或cd137l),其提供更好的治疗功效(图13a)。来自健康供体的外周血的t细胞用cd33cd123-4-1bbl-2g构筑体在6孔板中转导,与2ml病毒上清液一起培养。用f(ab)'标记car蛋白的表面表达来测定car表达,随后进行facs分析。将转导的细胞与同时标记的对照t细胞进行比较。在转导期结束后1天测定表达并将转导的群体包围在图中。t细胞上的表面cd33cd123-41bbl-2gcar表达约为60%(图13b)。当构筑体中包含4-1bbl时,cd33cd123car改善功能活化。
增强子il-15/il-15sushi也包括在cd33cd123car构筑体中作为替代方法。复合car,cd33cd123和il-15/il-15sushi都在单一构筑体中(图14)。il-15/il-15sushi能够促进cart/nk细胞的扩增,并将先天免疫细胞浸润到肿瘤部位,这可能导致更好的肿瘤破坏。
工程改造ccar靶向细胞表达:1)cd19或cd20或两者兼有;2)cd19或cd20或两者兼容;3)cd19或cd138或两者都有产生cd19cd20、cd19cd22、cd19cd138ccar
已使用与上述cd33cd123ccar类似之策略产生三种ccar(图15)。
第二代复合car(cd19cd20、cd19cd22)
复合car(ccar)的结构如图16a所示。它由sffv(脾病灶形成病毒)启动子组成,该启动子驱动两种不同单元的功能性ccar的表达。第一种car是完整的l8-cd19-2g车(使用人类cd8a前导序列,称为l-8),通过从小核糖核酸病毒中提取的高效p2a自裂肽与完整的第二种car(cd20-2g或cd22-2g)连接。整个序列在框架中,最初形成一个大的融合蛋白,在细胞表面表达之前被分裂成两半。该方法保证了两种car的表达水平相等。ccardna分子随后被亚克隆到与上述相同的慢病毒质粒中。
转导的t细胞有效表达ccar
从用cd19cd20-2g或cd19cd22-2g载体构筑体或对照载体转染的hek293t细胞产生慢病毒载体上清液。收集慢病毒上清液后,收集细胞,裂解并电泳,然后进行蛋白印迹转移。用抗cd3ζ单株抗体进行之免疫印迹法展示carcd3ζ融合蛋白之预测尺寸之谱带,cd19car略大于cd20或cd22car单元(图16b)。接下来,将外周血白血球层细胞活化3天,在经重组人纤维蛋白片段涂布之培养盘上用浓缩的cd19cd20-2g、cd19cd22-2g或控制载体慢病毒上清转转导。第一次转导后24小时重复转导过程。转导后3天,用山羊抗小鼠f(ab')2抗体和小鼠抗人cd3抗体对转导的t细胞进行染色,证实car在t细胞表面的表达。
与对照转导相比,图16显示用浓缩的l8-cd19cd20-2g慢病毒上清液转导的26.9%的细胞和用浓缩的cd19cd22-2g慢病毒上清液转导的35.6%的t细胞通过流式细胞术测定为f(ab')2和cd3阳性。
基于前导序列,转导的t细胞以不同水平表达cd19cd22-2g
然后,我们确定了导致ccar细胞表面表达水平最高的前导序列,制备了三个构筑体,其中包含人cd8a(l8),cd45(l45)和集落刺激因子(csf)的前导序列(图17)。在用这些载体中的每一种产生的慢病毒上清液转导人外周血t细胞后,使用如上的f(ab')2抗体测定t细胞的转导效率。l8前导序列再次导致最高的转导效率(43.8%),其次是l45(9.8%)和csf(1.3%)。(图17)这表明复合car的最佳设计,如单个car,部分取决于表面car表达的前导序列。
慢病毒上清液的浓度可以导致ccar的更高转导效率
为了提高转导的t细胞中的car效率,将cd19cd20-2g和cd19cd22-2g的慢病毒上清液以3,880xg离心24小时。将得到的病毒颗粒以其原始体积的三分之一悬浮在培养基中,使其浓缩3倍。该浓缩物用于转导与非浓缩病毒相同体积的活化t细胞。图18显示用3x浓缩的cd19cd22-2g慢病毒上清液转导的t细胞的car效率几乎增加三倍,而用2.5x浓缩的cd19cd20-2g慢病毒上清液转导的t细胞的car效率增加近10倍(图18)。这说明了将慢病毒载体浓缩用于较长的ccar构筑体的重要性。
ccarcart细胞特异性靶向表达cd19的肿瘤细胞系
进行t细胞共培养杀伤测定以确定l8-cd19cd22-2g和l8-cd19cd20-2gcart细胞有效裂解cd19+细胞系sp53和jeko-1(两种套细胞淋巴瘤细胞系)的能力。简言之,每种靶细胞系用cmtmr膜染料预标记,然后与载体对照,l8-cd19cd22-2g或l8-cd19cd20-2gcart细胞以2:1和5:1效应物的比例共培养。:靶细胞(200,000或500,000个效应细胞至100,000个靶细胞,在1ml无血清或il-2的t细胞培养基中)。培养过夜后,用抗人cd3-percp和cd19-apc标记细胞30分钟,洗涤,并悬浮在2%福尔马林中用于通过流式细胞术分析。l8-cd19cd22-2gcart细胞显示出对肿瘤细胞的强烈裂解(图19),分别以2:1和5:1的比例裂解53.4%和93%的sp53细胞。在相同的比例下,l8-cd19cd22-2gcart细胞能够溶解69%和97.3%的jeko-1细胞(图20)。
ccarcart细胞从aml和b-all患者样品中消除cd19+细胞
使用患者样品再次进行研究。用cmtmr预标记这些原代细胞的白血球层级分,并以与肿瘤细胞系相同的方式与载体对照或l8-cd19cd22-2gcart细胞共培养。l8-cd19cd22-2gcart细胞在过夜共培养物中分别裂解了54.3%和77%的aml患者细胞,其中cd19以2:1和5:1的比例异常表达,并且裂解了84.3%的b在与培养基中加入2.5%fbs和il-2的4天共培养物中,1:1比例的-all肿瘤细胞(图21,22a)。由于这些aml患者细胞仅包含65%的胚细胞并且其中75%表达cd19,因此l8-cd19cd22-2gcart细胞可能能够消除整个cd19阳性胚细胞群。
ccarcart细胞裂解表达cd22的k562细胞
产生通过转导入野生型k562细胞的表达人k562的cd22细胞系(k562xp22)。随后,我们测试了cd19cd22ccar的抗肿瘤特性以靶向k562细胞的次要cd22+群体。与对照相比,1:1比例的共培养实验(有效:靶标)对表达k562的cd22群体显示出适度的显着细胞毒性作用。细胞毒性结果与报道的针对人工抗原呈递细胞系的抗肿瘤活性的其他数据保持一致(图22b)。
工程改造之ccar靶向表达bcma和cs1的细胞
产生包括bcmacs1ccar及bcmacd19ccar之用于治疗多发性骨髓瘤及自身免疫性疾病之ccar
已进行ccar之临床前研究以靶向表面抗原,包括cd38、cs1、cd138、b细胞成熟抗原(bcma)及cd38。cd19car亦在i期临床试验中显示一些用于治疗多发性骨髓瘤之功效。然而,鉴于恶性浆细胞中通常存在表面抗原表达之非均质性(ruiz-arguelles及sanmiguel1994),单个标靶不大可能足以消除此疾病。产生bcmacs1ccar、bcmacd19ccar、bcmacd38ccar及bcmacd138ccar且实验设计与如上文所描述之cd33cd123ccar类似。
产生包括bcmacs1ccar(bc1ccar)之用于治疗多发性骨髓瘤及自身免疫性疾病之ccar。
bcma-cs1ccar(bc1ccar)构筑体的产生和表征
我们已经观察到,与单一抗原car相比,复合car构筑体的转导通常缺乏高效基因转移率。无论是由于构筑体大小还是效应细胞的代谢应激或其他因素,复合car的转导方案的优化仍然是必要的。我们比较了3种不同的转导方案,主要差异包括是否在病毒上清液中发生培养,转导程序频率和每次处理的最终细胞密度数。方法1每次2小时转导24小时,并使用经重组人纤维蛋白片段涂布之培养盘,首先与病毒一起培养,吸出,然后与t细胞一起培养至终浓度为0.5×106个细胞/ml。方法2使用相同的病毒重组人纤维蛋白片段程序,然而,它交换至第二转导期以继续培养至总共48小时,最终细胞密度为0.3×106个细胞/ml。方法3是修订的方法2使用培养方案,将病毒上清液与细胞一起在经重组人纤维蛋白片段涂布之培养盘上以0.3×106个细胞/ml的细胞密度直接培养48小时(图23)。
bc1ccar之模组设计由藉由自裂解p2a肽与抗cd319(cs1)融合之抗cd269(bcma,b细胞成熟抗原)单链可变片段(scfv)区域、cd8衍生之铰链(h)及跨膜(tm)区域以及连接至cd3ζ信号传导域之串联4-1bb共活化域组成(图14a)。强脾病灶形成病毒启动子(sffv)及cd8前导序列用于t细胞表面上bc1ccar)car分子之有效表达。
在活化2天后,用bc1ccar慢病毒转导从人外周血白血球层分离的t细胞。根据上述不同的转导方案,报道了每种技术的各种转导效率(图24b)。我们发现,通常,以降低的细胞密度(0.3×106个细胞/ml)与病毒上清液一起培养48小时的细胞支持最高的基因转移效率(图24b)。因此,当我们改进我们的转导方案时,我们观察到相应更高的基因转移率(图24c)。
经转染之bc1ccarhek293t细胞经历蛋白印迹分析以确认复合构筑体。用抗cd3ζ单株抗体进行之免疫印迹法展示复合carcd3ζ融合蛋白之预测尺寸之谱带(图24d)。重要的是,如所预期,在信号传导p2a肽之成功高裂解作用之墨点上观测到具有类似强度之两个不同谱带。如所预期,未发现gfp对照载体之cd3ζ表达。
bc1ccart细胞特异性溶解bcma+及cs1+骨髓瘤细胞系
为了评估bc1ccart细胞之细胞毒性能力,吾人用骨髓瘤细胞系:mm1s(bmca+cs1+)、rpmi-8226(bcma+cs1-)及u266(bcma+cs1dim)进行共培养分析。藉由流式细胞术分析定量bc1ccart细胞溶解标靶细胞之能力,且标靶细胞用cytotracker染料(cmtmr)染色。在24小时共培养中,bc1ccar呈现mm1s细胞之几乎完全溶解,其中在2:1之e:t比率下消耗超过90%标靶细胞且在5:1之e:t下消耗超过95%(图25a和25c)。在rpmi-8226细胞中,bc1ccar在2:1之e:t比率下溶解超过70%bcma+标靶细胞,且在5:1之e:t下溶解超过75%标靶细胞(图16)。在与u266标靶细胞之24小时共培养中,bc1ccar在2:1之e:t比率下溶解80%bcma+u266细胞,达到饱和(图25b和25c)。由于骨髓瘤细胞系大部分都是bcma+,这些结果表明bc1ccart细胞的bcma靶向主要促进有效的细胞溶解。
原生患者骨髓瘤样品中bc1ccart细胞特异性靶向bcma+及cs1+群体我们用bc1ccart细胞对原发肿瘤细胞进行共培养,以评估其杀死多种原发性骨髓瘤细胞类型的能力。mm10-g初级样品的流式细胞术分析揭示了不同且一致的bcma+和cs1+群体亚群。mm7-g样品显示完整的bcma+cs1+表型,而mm11-g表现出嘈杂的bcmadimcs1dim表型,可能归因于其作为骨髓抽吸物的性质。bc1ccart细胞显示mm7-g原发患者样品的强烈剂量依赖性消融,在e:t比例为5:1时超过75%裂解,在10:1时增加至超过85%(图26a)。
bc1ccar还通过显着消除mm10-g共培养物中的bcma+cs1+和bcma-cs1+群体亚群,显示出靶向和特异性裂解能力。在e:t比例为2:1时,bc1ccart细胞消融超过60%的bcma+cs1+群体和70%的仅cs1+群体(图26b)。在e:t比为5:1时,仅cs1+群体的消融增加至80%(图26b)。针对mm11-g(图26c),bc1ccart细胞也能够以剂量依赖性方式证明细胞毒活性(图26c)。总之,bc1ccart细胞对骨髓瘤细胞系和呈现不同bcma和cs1组合的原发肿瘤细胞表现出强大的抗肿瘤活性(图26d)。
bc1ccar抗原特异性活性的功能评估
为了评估和表征bc1ccar在其抗原靶向方面的生物学特性,我们建立了一个模型,允许我们独立测试bc1ccarscfv功能。使用cml细胞系阴性的骨髓瘤标志物(k562),我们通过将cs1cdna转导入k562细胞并随后通过嘌呤霉素选择促进稳定表达,建立了稳定的表达cs5的k562细胞系(cs1xpk562)(图27a)。为了测试bcmascfv功能,我们从nih(kochenderferlab)获得了表达bcma的k562细胞系(bcmaxpk562)。在我们确认每种抗原表达细胞系的每种抗原的独立表达后(图27a),我们在共培养实验中使用它们来确定bc1ccart靶向功能。
在短期培养物(<24小时)中,bc1ccart细胞显示出针对bcmaxpk562细胞的细胞毒活性,而对野生型k562细胞没有显示出作用(图27b)。接下来,针对cs1xpk562细胞的短期培养物显示出针对表达cs1的靶细胞的类似反应。此外,bc1ccart细胞似乎比针对cs1xpk562细胞的cs1特异性car具有更强的细胞毒性作用(图27b)。在表达nk-92细胞的cs1dim上进行抗cs1活性的进一步验证,其中细胞毒性表现为剂量依赖性效应(图27b)。
为了在潜在的临床情景中模拟抗原逃逸,我们进行了组合的共培养实验。我们以1:1的比例混合bcmaxpk562和cs1xpk562,并寻找可能导致在现实世界情景中复发的抗原残留群体的证据。共培养超过48小时以确保抗原耗尽。接下来,构建直方图,其代表t细胞群和靶肿瘤细胞群。每个直方图中的数字表示残留的门控目标肿瘤群。我们发现,与对照t细胞相比,bcma特异性car和cs1特异性car能够消耗或对其各自的群体具有显着的细胞毒性作用。然而,cs1特异性car留下了显着的残留bcma+群体,而bcma特异性car实现了高度的细胞毒性,但仍然留下了一个小的但确定的cs1+群体(图27c)。相反,bc1ccart细胞有效地耗尽了两个靶群(图27c)。我们推测,拥有1种抗原的残留肿瘤群可能导致仅使用单一抗原特异性car进行治疗的患者复发。
由于正常骨髓表达可以表达cs1的一小部分浆细胞,因此担心cs1定向的car可能具有不利的细胞毒性。虽然骨髓中的cs1群体确实以剂量依赖性方式受bc1ccar影响(图27d),但cs1子集本身很小。
bc1ccart细胞即使在肿瘤再激发时也表现出持久性和连续杀伤能力
我们接下来研究了bc1ccart细胞在由细胞裂解,碎片和肿瘤再激发引起的不利微环境中以顺序方式杀死肿瘤细胞的能力。使用图28a中的方案,我们使用mm1s细胞作为模型骨髓瘤进行长期共培养,并且用新鲜mm1s定期再次攻击bc1ccart细胞和其他car构筑体以模拟肿瘤扩增或复发。初始共培养条件以1:1的e:t比进行。在没有外源性细胞因子的情况下,我们发现48小时后所有car细胞都完成靶抗原的消耗,具有显着的聚集和t细胞增殖(图28b)。相比之下,对照t细胞没有显示出反应和增殖,产生的肿瘤群体现在已经扩大了两倍于其初始数量。在用新鲜mm1s细胞重新挑战所有处理孔后,我们发现即使没有外源细胞因子,所有car仍然保持高度细胞毒性。到108小时,新输入的mm1s细胞几乎已被bcma-car和bc1ccar耗尽,仍然从cs1-car观察到显着的细胞毒性。然而,在该阶段,流式细胞术显示cs1-car群体减少并且肿瘤抗原群体的相对生长至约17%(图28c),表明cs1-cart细胞可能摇摆不定。此时,对照t细胞已被肿瘤细胞完全过度生长。所有car肿瘤裂解和细胞毒性在168小时后停止,然而,bcma-car和bc1ccar仍显示可检测的少数t细胞群,而对照t细胞和cs1-cart细胞几乎全部消失(数据未显示)。
bc1ccart细胞在体内显示出显着的控制和肿瘤减少
为了评估bc1ccart细胞的体内抗肿瘤活性,我们开发了一种异种小鼠模型,其使用nsg小鼠亚致射线照射并静脉内注射表达荧光素酶的mm1s细胞(多发性骨髓瘤细胞系),以诱导可测量的肿瘤形成。肿瘤细胞注射后3天,给小鼠静脉注射单剂量的5×106bc1ccart细胞或对照gfp细胞。在第3天,第6天,第8天和第11天,给小鼠皮下注射redijectd-荧光素(perkinelmer)并进行ivis成像以测量肿瘤负荷(图29a)。将测量的bc1ccart细胞小鼠的平均光强度与gfp对照小鼠的平均光强度进行比较,以确定通过bc1ccar处理控制肿瘤生长(图29b)。非配对t检验分析显示,与对照相比,在第6天两组之间具有极小的光强度并因此在bc1ccart细胞注射组中较少的肿瘤负荷之间存在极显着差异(p<0.01)(图29b)。接下来,我们比较了两组的小鼠存活率(图29c)。所有注射bc1ccart细胞的小鼠在第50天存活并且超过四分之一保持在第65天。基于long-rankmantel-cox测试,对照和处理小鼠之间的p值为0.0011。注射对照t细胞的小鼠的存活百分比在第50天开始减少,并在第55天死亡。总之,与对照细胞相比,这些体内数据表明bc1ccart细胞显着降低肿瘤负荷并延长注射mm1s的nsg小鼠的存活率。
bc1ccart细胞在混合抗原异种小鼠模型中表现出改善的细胞毒性作用为了评估可能阻止抗原逃逸的复合car的双重靶向性质,我们使用nsg小鼠设计了异种小鼠模型,所述nsg小鼠通过亚致射线照射并静脉内注射表达荧光素酶的k562细胞,其表达稳定转导的bcma或cs1。在嘌呤霉素选择后进一步分选表达bcma和cs1的k562细胞以表达,并建立稳定的同源单抗原群。然后在注射前分别以4:1的比例混合表达bcma和cs1的k562细胞以模拟潜在的抗原逃逸。肿瘤细胞注射后3天,给小鼠静脉内注射15×106个对照t细胞,bcma特异性car或bc1ccart细胞。由于t细胞输注和细胞聚集期间的技术问题,两只对照小鼠由于注射程序而死亡。在第3,7,10和12天,给小鼠皮下注射redijectd-荧光素(perkinelmer)并进行ivis成像以显现肿瘤负荷(图29d)。将注射bc1ccart细胞的小鼠的平均光强度(表示肿瘤负荷)与注射bcma特异性car和gfp对照的小鼠的平均光强度进行比较,以确定通过治疗控制肿瘤生长(图29d)。到第10天,与对照相比,bcma特异性car和bc1ccart细胞均显示出超过47%的肿瘤减少。然而,早在小鼠背侧第10天,肿瘤负荷减少有利于bc1ccar的差异为6%。到第12天,肿瘤减少有17%的差异,有利于背侧的bc1ccar(图29d和e)。该数字接近注射的cs1-k562细胞(20%)与bcma-k562(80%)的百分比。可能是表达k562细胞的cs1存活和增殖作为抗原逃逸模型的结果。总之,这些体内数据表明bc1ccart细胞似乎显示出对多种抗原群体的改善的肿瘤负荷控制。
bc1ccar转导和验证nk细胞中的抗肿瘤特性
为了进一步评估bc1ccar在不同环境中的稳健性,我们将bc1ccar构筑体转导到模型nk细胞系nk-92中。该构筑体成功地能够通过慢病毒培养转导到nk-92细胞中48小时,并在基因转移后产生62.1%的表面表达谱(图30a和30b)。以0.3-0.5×106个细胞/ml的密度维持nk-92细胞导致稳定的群体。为了在体外测试bc1ccar抗肿瘤活性,我们对骨髓瘤细胞系和主要患者样品进行了共培养。bc1ccar在培养物中以e:t比例为5:1接近针对mm1s,u266和rpmi-8226细胞系的80%裂解。它还成功地裂解了超过60%的原发性mm7-g肿瘤(图31a和31b)。这些结果在与bc1ccart细胞的可比性方面相似。接下来,我们测定了bc1ccar对其独立裂解bcma+或cs1+细胞的能力的抗原特异性。对bc1ccart细胞进行类似的测定(图27)。在用bcma表达k562(bcmaxpk562)或cs1表达k562(cs1xpk562细胞)的4小时培养物中,我们发现bc1ccarnk细胞能够对任一群体具有细胞毒性作用(图31c)。
产生ccar,包括bcmacd19或bcmacd19b,用于治疗浆细胞骨髓瘤或自身免疫性疾病
bcma-cd19ccar或bcma-cd19bccar构筑体的产生和表征
bc1ccar的模块化设计包括通过自切割p2a肽,cd8衍生的铰链(h)和跨膜(tm)区与抗cd319(cs1)scfv融合的抗cd269(bcma)单链可变片段(scfv)区,和cd1ζ信号传导结构域连接的4-1bb共刺激结构域(图35)。使用形成病毒启动子(sffv)和cd8前导序列的强脾脏焦点用于在t细胞表面上有效表达bcmacd19ccar分子和使用上述类似方法在体外和体内抗肿瘤活性。
测试bcmacd19car中car的每个单位的抗浆细胞或抗b细胞活性。我们发现bcmacar单元能够有效地裂解任何bcma+群体。我们首先针对双bcmacs1阳性浆细胞系mm1s进行共培养,并使用cs1car作为稳健性的次要测量。我们观察到bcma和cs1特异性car均能够高效裂解mm1s靶标(图36a)。接下来,我们将bcmacar和cs1car培养成对多数bcma+原发性骨髓瘤样品mm7-g。我们发现,就bcma表达而言,bcmacar能够几乎耗尽所有bcma+细胞。相反,cs1car留下残留的bcma+群体(图36b)。这些结果表明bcmacar在其细胞毒性作用中实现高效力和特异性。
我们接下来测试了cd19car单元的抗b细胞活性。抗cd19分子的单链可变片段(scfv)核苷酸序列用于两种不同的构筑体,cd19-2g和cd19b-bbcar。为了改善信号转导,cd19car设计有与cd3zeta信号传导结构域融合的4-1bb共刺激结构域,使其成为第二代car(图37a)。靶向cd19的第二代cart细胞先前已用于临床试验。为了在t细胞表面上有效表达cd19car分子,使用强脾脏形成焦点的病毒启动子(sffv),并将cd8a的前导序列掺入构筑体中。为了比较,还制备了使用cd45,csf,人白蛋白(ha)或il-2的前导序列的cd19car构筑体。通过cd-8衍生的铰链(h)和跨膜(tm)区域将抗cd19scfv与细胞内信号传导结构域分离(图37)。随后将具有不同前导序列或不同scfv序列的cd19cardna分子亚克隆到慢病毒质粒中。
转导的t细胞有效表达cd19car-从用cd19-2g载体构筑体和对照载体转染的hek293t细胞产生慢病毒载体上清液。收集慢病毒上清液后,收集细胞,裂解并电泳,然后进行蛋白印迹转移。用抗人cd3zeta抗体培养印迹膜,在含有来自用cd19-2g转染的细胞的裂解物的泳道中产生~56kda条带,所述细胞是表达的融合蛋白的预测大小(图37b)。接下来,将外周血白血球层细胞活化3天,并用经重组人纤维蛋白片段涂布之培养盘上的l8-cd19-2g或对照载体慢病毒上清液转导。在第一次转导后24小时重复转导程序。通过用山羊抗小鼠fab抗体和小鼠抗人cd3染色转导的t细胞,在转导后3天证明t细胞表面上的car表达。图37c显示,与对照转导相比,通过流式细胞术测定,用l8-cd19-2g病毒转导的19.8%的细胞对f(ab')2和cd3都是阳性的。
基于前导序列,转导的t细胞在不同水平表达cd19-2g-为了确定导致cd19-2gcar的最高水平的细胞表面表达的前导序列,制备了几种构筑体,其掺入了人cd8a(l8),cd45(l45),集落刺激因子(csf)的前导序列,人白蛋白(ha)和il2(图38a)。在用这些载体中的每一种产生的慢病毒上清液转导人外周血t细胞后,使用如上的f(ab')2抗体测定t细胞的转导效率。仅结合l8前导序列的cd19-2g构筑体导致任何明显的car细胞表面表达(32.5%),而l45前导序列仅导致3.3%的转导效率,并且csf,ha和il2低于1%(图38b)。这表明cd19-2gcar的最佳设计部分取决于所用的前导序列。
基于scfv序列,转导的t细胞以不同水平表达cd19-2g
为了确定cd19的scfv序列,其将导致cd19-2gcar的最高水平的细胞表面表达,在cd19-2gcar(图39a),cd19和cd19b的设计中使用两种不同的序列。两者都使用了l8领导者序列。在相同条件下用这些载体中的每一种产生的慢病毒上清液转导人外周血t细胞后,使用如上的f(ab')2抗体测定t细胞的转导效率。cd19-2g构筑体产生18.2%的car细胞,但cd19b-bb-2g构筑体导致54.7%的car效率(图39b)。这表明cd19-2gcar的最佳设计还部分取决于所用scfv的序列。
cd19-2g和cd19b-bb-2gcart细胞特异性靶向表达cd19的细胞系
进行t细胞共培养杀伤测定以确定cd19-2g和cd19b-bb-2gcart细胞有效裂解cd19+细胞系sp53和jeko-1(两种套细胞淋巴瘤细胞系)的能力。简而言之,每种靶细胞系用cmtmr膜染料预标记,然后与载体对照,l8-cd19-2g或l8-cd19b-bb-2gcart细胞以2:1和5的比例共培养:1效应物:靶细胞(200,000或500,000个效应细胞至100,000个靶细胞,在1ml无血清或il-2的t细胞培养基中)。培养过夜后,用抗人cd3-percp和cd19-apc标记细胞30分钟,洗涤,并悬浮在2%福尔马林中用于通过流式细胞术分析。cd19-2g和cd19b2gcart细胞均显示b细胞系sp53和jeko-1的强烈裂解(图40和41)。
cd19-2g和cd19b-bb-2gcart细胞消除aml和b-all患者样本中的cd19+细胞
还使用患者样品进行了研究。使用两名患有cd19+细胞的患者:一名诊断为aml(cd19的异常表达),一名患有b-all,用于该研究。患者的血液分别含有26.4%和90%的cd19+细胞(图41a,42a)。用cmtmr预标记这些原代细胞的血沉棕黄层级分,并以与肿瘤细胞系相同的方式和比例与载体对照,l8-cd19-2g或l8-cd19b-bb-2gt细胞共培养。l-8-cd19-2gcar和l-8-cd19b-2g细胞均能够完全消除表达cd19的靶细胞(图42和43)。
随着插入物大小的增加,病毒滴度通常降低,cd19bscfv的序列为cd19bcar提供更高的滴度(图39)。因此,cd19bscfv用于产生复合bcmacd19bcar(图44)。
另一种用于骨髓瘤和浆细胞的car设计
我们设计了一种表达car的配体,其与各种b细胞活化因子受体结合。虽然为可能与肿瘤群体结合的任何潜在抗原或配体因子设计cars似乎是一个合乎逻辑的飞跃,但car技术中的技术故障排除仍然是一个高且持久的障碍。由于未定义的分子相互作用或设计问题,并非所有car构筑好体都能够实现一致或足够的表面表达。我们能够实现cd45前导序列baff-car的表面表达,其具有约21%的cd28细胞内信号传导结构域(图45a)。然而,具有来自cd8或csf的替代前导序列的baff-car未实现任何有意义的表达(图45b)。考虑car设计时还观察到另一个因素。在一种情况下,我们使用4-1bbl配体结合结构域作为支持性刺激途径设计了baff-car构筑好体。另一方面,我们在构筑体中添加了表达臂的il-15/il-15sushi装甲。在两种情况下,与4-1bbl或il-15/il-15sushi配对的cd8前导序列均获得比csf前导序列更高的表面表达(图45c)。
baff-car的抗浆细胞特性
我们通过用浆细胞骨髓瘤细胞(mm1s)或地幔(mcl)细胞(sp53)与baff-car共培养来表征各种baff-car的生物学特性,所述细胞均表达浆细胞标记物cd138的组分,其中baff是配体结合复合物。l45-baff-28car能够在e:t比例为3:1接近60%后48小时后溶解mm1s肿瘤细胞(图46)。此外,l8-baff-28il-15/il-15sushi和l8-baff-284-1bblcar也能够达到相当程度的细胞毒性(图47a,47b)。与b细胞套细胞系sp53的共培养显示出有限的效果,仅对l8-baff-28il-15/il-15car观察到约25%的细胞毒性(图47)。
cd45car疗法
用chopchop设计三对sgrna以靶向相关基因。接着将基因特异性sgrnas选殖至与e2a自裂解连接子连接之表现人类cas9及嘌呤霉素抗性基因之慢病毒载体(lentiu6-sgrna-sffv-cas9-puro-wpre)中。u6-sgrna卡匣位于cas9元件之前。sgrna及cas9puro之表现分别由u6启动子及sffv驱使(图48)。
使用及构筑以下基因特异性sgrna序列。
在本发明之非限制性实施例中,已如下文所阐述设计及构筑例示性基因特异性sgrna:
cd45sgrna构筑体:
lenti-u6-sgcd45a-sffv-cas9-purogtggtgtgagtaggtaa
lenti-u6-sgcd45b-sffv-cas9-purogagttttgcattggcgg
lenti-u6-sgcd45c-sffv-cas9-purogagggtggttgtcaatg
图49a展示产生靶向血液科恶性疾病之cd45cart或nk细胞之步骤。
crispr/cas核酸酶靶向nk细胞上之cd45
使用具有基因特异性sgrna之慢病毒转导nk-92细胞。藉由流式细胞术分析测定nk-92细胞上cd45表达之减少。分选及扩增nk-92细胞之cd45阴性群体(图25)。使用经分选及扩增之cd45阴性nk-92细胞产生cd45carnk细胞。使用所得cd45carnk细胞测试其杀死cd45+细胞之能力。
在crispr/cas核酸酶标靶之后,经cd45不活化之nk-92细胞(nk45i-92)之功能性表征
吾人证明,在cd45之crispr/cas核酸酶不活化之后,nk45i-92细胞之生长与野生型nk-92细胞类似(图50)。cd45之不活化不显著影响nk-92之细胞增殖。此外,吾人证实当细胞与白血病细胞ccrf共培养时,nk45i-92细胞之溶解能力与野生型nk-92相容(图51)。
为了说明经cd45不活化之nk-92与car溶解相容,nk45i-92细胞及其野生型nk-92用表达cd5car或gfp之慢病毒转导。藉由facs分选所得cd5carnk45i-92细胞及gfpnk45i-92且用于比较其杀死标靶细胞之能力。cd5carnk45i-92细胞在其与ccrf-cem细胞共培养时,在2:1及5:1之比率(e:t)下显示稳定杀死cd5标靶白血病细胞之能力。吾人cd5carnk45i-92与cd5carnk-92细胞之间存在类似的活体外ccrf-cem细胞消除功效(图52)。此表明cd45表达之减少不会降低carnk细胞之抗肿瘤活性。
产生cd45car构筑体
吾人随后研究白血病细胞中nk45i-92细胞中cd45car对cd45抗原之反应吾人产生cd45car。cd45car由抗cd45单链可变片段(scfv)区域,cd8衍生之铰链(h)及跨膜tm)区域以及连接至cd3ζ信号传导域之串联cd28及4-1bb共活化域组成(图53a)。使用强脾病灶形成病毒启动子(sffv)及cd8前导序列。使用适合的载体对照经cd45car慢病毒质体转染之hek293-ft细胞之蛋白印迹法表征cd45car蛋白质。此外,抗cd3ζ单株抗体免疫印迹法显示cd45car蛋白质之预测尺寸之谱带,其中在载体对照物中未观测到谱带(图53b)。
cd45carnk45i-92nk细胞
在经萤光活化之细胞分选(facs)以富集nk45i-92细胞之后,测定cd45carnk-92转导功效为87%,如在分选之后藉由流式细胞术测定(图30)。在nk45i-92细胞之facs收集之后,cd45car表达量在至少10次继代中保持始终稳定。
cd45carnk45i-92细胞特异性溶解cd45+白血病细胞
为了评估cd45carnk45i-92抗白血病活性,吾人使用t-all细胞系,ccrf-cem及jurkat以及nk细胞系及nk-92细胞进行共培养分析,因为其皆表达cd45(图55,56及57)。吾人证明cd45carnk45i-92细胞持续显示白血病细胞之稳定溶解。在针对标靶细胞之中低有效性下培育6小时(e:t比率5:1)之后,cd45carnk45i-92细胞有效溶解超过60%ccrf-cem细胞(图55)。在6小时共培养之后,cd45carnk45i-92细胞在2:1或5:1之间e:t比率下亦能够消除约60%jurkat细胞(图56)。在6小时共培养之后,cd45carnk45i-92细胞在2:1之间e:t比率下有效溶解20%cd45阳性nk-92细胞,其中5:1之e:t下将近60%溶解(图57a-57c)。
为了进一步分析血液科恶性疾病之cd45标靶,吾人亦产生另外两种car:cd45-28及cd45-bb,且使用表cd45-28或cd45-bbcar之间慢病毒转导nk45i-92细胞.cd45-28和cd45-bbcar含有新型抗cd45scfv,其与上述cd45car不同,cd45-28car使用cd28共刺激域,而cd45-bb具有4-bb共刺激域。cars使用cd8衍生之铰链(h)跨膜(tm)区域和cd3ζ信号传导域。cd45car显示b急性淋巴细胞性细胞系reh之稳定溶解。cd45carnk45i-92细胞溶解约76%reh细胞与对照性gfpnk-92细胞相比,cd45b-bbcarnk45i-92细胞和cd45b-28carnk45i-92细胞分解展示约79%及100%reh细胞溶解(图57d-e)。cd45b-28carnk45i-92细胞呈现最高的reh细胞溶解能力(b-all细胞)。
我们还研究了cd45b-28carnk45i-92细胞是否可以溶解其他类型的白血病细胞。如图57f所示,用u937细胞(靶标:t)和gfpnk-92细胞或cd45b-28nk45i-92细胞(效应子:e)以2:1(e:t)比例进行共培养测定。与对照gfpnk-92细胞相比,cd45b-28nk45i-92细胞在20小时后表现出强大的抗白血病活性,对u937细胞有约81%的细胞裂解。u937是一种急性髓性白血病细胞系。当用molm-13细胞(靶标:t)和gfpnk-92细胞或cd45b-28nk45i-92细胞(效应子:e)以5:1(e:t)进行共培养测定时,观察到类似的发现。比例为20小时(图57g)。molm-13细胞来源于患有侵袭性急性单核细胞白血病的患者。还在两个套细胞系sp53和jeko中检查了抗白血病活性(图57h和i)。与相对较短的共培养的对照gfpnk-92细胞相比,cd45b-28nk45i-92具有2:1的低比例(e:t),能够裂解超过40%的sp53细胞或jeko白血病细胞。一段时间,6个小时。这些研究表明cd45b-28nk45i-92对不同类型的恶性白血病具有显着的抗白血病特性。
我们进一步研究了cd45b-28nk45i-92细胞是否可以裂解cd34+造血干/祖细胞。将源自人脐带血的cd34(+)干细胞与对照或cd45b-28carnk细胞以2:1的低比例(有效:靶标)共培养48小时。与对照相比,cd45b-28nk45i-92细胞几乎消除了cd34+造血前体细胞(图57j)。
另一种增强cd45car活性的car设计
我们还产生了工程改造cd45car细胞,不仅通过cd28进行共刺激,还在单一构筑体中共表达4-1bb配体(4-1bbl或cd137l),提供更好的治疗效果(图58a),他们的例子是如下面所描述的:
实施例:产生cd45b-28-2g-4-1bbl,并且产生的cd45bcar细胞可以接受共刺激途径,cd28和4-1bb。将cd45b-28-2g-4-1bbl病毒浓缩4倍并用于转导nk45i-92细胞。其car表面表达约为87%(图58b)。将cd45b-28-2g-4-1bbl病毒浓缩4倍并用于转导。当构筑体中包含4-1bbl时,cd45b-2gcar细胞的抗肿瘤活性显着改善。
增强子il-15/il-15sushi也包括在cd45car构筑体中,作为增强cd45car抗肿瘤活性的替代方法。cd45car和il-15/il-15sushi都在单一构筑体中(图58)。当il-15/il-15sushi包含在构筑体中时,cd45b-2gcar细胞的抗肿瘤活性显着提高。
实施例:产生cd45b-28-2g-il-15/il-15sushink细胞。表面cd45bcar表达约为60%。(图58c)。当il-15/il-15sushi包含在构筑体中时,cd45b-2gcar细胞的抗肿瘤活性显着提高。
表征cd4il-15/il-15sushicar
已生成cd4il-15/il-15sushi-car,其含有与il-15/il-15sushi连接的第三代cd4car(图59)。将car(第三代),sushi/il-15的组合装配在表达载体上,并且它们的表达由sffv启动子驱动(图59)。具有il-15/il-15sushi的car与p2a切割序列连锁。il-15/il-15sushi部分由与il-15融合的il-2信号肽通过26-氨基酸聚脯氨酸接头与il-15susi连接组成(图59)。il-2信号肽提供更好的分泌信号。il-15/il-15sushi的稳定的功能性复合物可以从转导的细胞分泌,并且分泌由il-2信号肽指导。
为了验证cd4il-15/il-15sushi构筑体,hek293ft细胞用gfp(对照物)或cd4il-15/il-15sushi之前的病毒质体转染。在转染之后约60小时,收集hek-293ft细胞及上清液。细胞溶解于含有蛋白酶抑制剂混合物之ripa缓冲液中和进行电泳。凝胶转移至immobilonfl墨点膜,阻断且在1:500下用小鼠抗人类cd3z抗体探测。在洗涤之后,薄膜用山羊抗小鼠hrp结合物探测,洗涤,且在用hyglohrp受质处理之后暴露于薄膜。cd4il15ra-car在hek293细胞中成功表达(道2,图60a)。藉由新鲜hek-293细胞之转导进一步检验cd4il-15/il-15sushi-car慢病毒上清液(图35a)。hek-293细胞来源经转染之hek-293ft细胞之gfp或cd4il-15/il-15sushi-car病毒上清液转导添加凝聚胺达到4μl/ml。在16小时之后更换培养基和用不含病毒上清液或凝聚胺之培养基置换。在转导之后三天,收集细胞和用山羊抗小鼠f(ab')2抗体以1:250染色30分钟洗涤细胞且使用抗生蛋白链菌素-pe结合物以1:500染色,洗涤,悬浮于2%福马林中而通过流式细胞术分析图60b展示经cd4il-15/il-15sushi-car慢病毒转导之hek-293细胞对f(ab)2-pe呈现80%阳性(圆圈,图60b),而经gfp对照慢病毒转导之细胞对f(ab)2-pe最小(图60b)。
产生cd4il-15/il-15sushi-carnk细胞
nk-92细胞用cd4il-15/il-15sushi-car慢病毒上清液转导。在培育5天之后,收集细胞且以1:250与山羊抗小鼠f(ab')2一起培育30分钟。洗涤细胞,悬浮且用抗生蛋白链菌素-pe染色30分钟。洗涤细胞且悬浮于2%福马林中,且藉由流式细胞术分析,引起约70%经转导之细胞表达cd4il15ra-car(圆圈,图61)。cd4il-15/il-15sushi-car之其他实验测试将包括活体外及活体内白血病/淋巴瘤杀死分析,及经cd4car转导之细胞之标靶杀死及增殖率之对比。本发明者亦使用与上文所述相同的策略产生cd19il-15/il-15sushi-car,cd20il-15/il-15sushi-car及cd22il-15/il-15sushi-car。
产生cd4il-15/il-15sushi-cart细胞
人类脐带白血球层细胞用cd4il-15/il-15sushi-car慢病毒上清液转导。在培育5天之后,收集细胞且以1:250与山羊抗小鼠f(ab')2一起培育30分钟。洗涤细胞,悬浮且用抗生蛋白链菌素-pe染色30分钟。洗涤细胞且悬浮于2%福马林中,且藉由流式细胞术分析,引起63%经转导之细胞表达cd4il-15/il-15sushi-car(圆圈,图62)。cd4il-15/il-15sushi-car之其他实验测试将包括活体外及活体内白血病/淋巴瘤杀死分析,及经cd4car转导之细胞之标靶杀死及增殖率之对比。
藉由将cd4il-15/il-15sushicarnk细胞与以下cd4阳性细胞系共培养来测试其相对于cd4carnk细胞之活体外抗白血病活性:karpas299及molt4
karpas299细胞系来源于具有多形性大型t细胞淋巴瘤之患者。自19周岁急性淋巴母细胞白血病(t-all)患者之外周血产生表达cd4之molt4细胞系。在4小时共培养实验期间,cd4il-15/il-15sushicarnk细胞在5:1之效应子:标靶比率下,在甚至高于cd4carnk细胞之比率下展示karpas299细胞之显著杀死(95%)(82%,图63)。类似地,当以1:1与molt4细胞共培养时,在隔夜分析法中,cd4il-15/il-15sushicarnk细胞以高于cd4carnk细胞之比率(84%至65%)溶解标靶细胞(图64)。此等结果证实cd4il-15/il-15sushicarnk细胞至少可与cd4carnk细胞相同地消除肿瘤细胞。
cd4car及cd4il-15/il-15sushicart细胞呈现比cd4car更强效的活体内抗肿瘤活性
为了评估cd4car及cd4il-15/il-15sushicart细胞之活体内抗肿瘤活性,及测定cd4il15racart细胞相对于cd4cart细胞之持久性之可能增加,吾人研究使用经亚致死剂量辐射及静脉内注射表现萤光素酶之molm13细胞(一种急性骨髓白血病细胞系(m5),其为100%cd4)以诱导可测量之肿瘤形成之nsg小鼠的异种小鼠模型。在肿瘤细胞注射之后三天,向6只小鼠各自静脉内注射8×106个cd4car、cd4il-15/il-15sushicart细胞或载体对照性t细胞之疗程。在第3、6、9及11天,向小鼠皮下注射redijectd-luciferin(perkinelmer)且让小鼠经历ivis成像以测量肿瘤负荷(图65b)。在第6天,经cd4cart细胞处理之小鼠相对于对照物具有52%肿瘤负荷降低,而经cd4il-15/il-15sushicart细胞处理之小鼠具有74%肿瘤负荷降低(图65c)。在第11天,在此等组中之两者中,几乎所有肿瘤细胞溶解。在第9天,不成对t测试分析显示对照物与两个组之间的极显著差异(p=0.0045),其中与对照物相比经cd4car及cd4il-15/il-15sushicart细胞处理之组中存在较低光强度且因此存在较低肿瘤负荷。
接下来,我们比较了两组的小鼠存活率(图65d)。注射cd4il-15/il-15sushicart细胞的所有白血病小鼠比cd4cart细胞存活的时间更长。总之,这些体内数据表明,与对照细胞相比,cd4il-15/il-15sushicart细胞显着降低肿瘤负荷并延长cd4il-15/il-15sushcart注射的nsg小鼠的存活。
cd4il-15/il-15sushicarnk细胞在体内表现出强大且持久的抗肿瘤活性
为了进一步评估cd4il-15/il-15sushicar功能,我们利用nkcar细胞和jurkat肿瘤细胞创造了一种压力条件。nk细胞具有短的半衰期特性,白血病jurkat细胞显示低于60%的cd4+表型(图66a)。在这种情况下,它允许我们研究分泌性可溶性il-15sushi如何影响car功能的持久性和杀伤能力。然后,我们使用我们的异种nsg小鼠模型,使用nsg小鼠进行亚致死照射并静脉内注射表达荧光素酶的jurkat细胞以诱导可测量的肿瘤形成。与molm-13细胞相反,jurkat细胞显示低于60%的cd4+表型(图66a)。jurkat细胞注射后3天,给小鼠静脉内注射10×106个cd4car,cd4il-15/il-15sushi或载体对照nk细胞。在第3天(治疗前一天),第7,10和14天,对小鼠进行ivis成像以测量肿瘤负荷(图66b)。将测量的cd4car和cd4il-15/il-15sushink注射小鼠的平均光强度与载体对照nk注射小鼠的平均光强度进行比较,以确定jurkat细胞的裂解百分比(图66c)。尽管两种条件在第7天显示出显着的肿瘤细胞裂解,但cd4carnk细胞的裂解百分比与第14天保持相同,而cd4il-15/il-15sushink细胞增加至超过97%。(图66d)。非配对t检验分析显示,到第14天两组之间存在极显着差异(p<0.0001)。这些结果表明jurkat肿瘤细胞的cd4carnk细胞裂解无法跟上cd4-jurkat细胞的扩增,而分泌il-15/il-15sushi的nkcar细胞的持续扩增有效地裂解。分泌型il-15/il-15sushi与car的共表达可以补充cart或nk细胞不能消除暗淡表达的癌细胞或非靶向癌细胞的缺陷。重复实验(图67)显示与图66中描述的结果类似的结果。
分泌的il-15/il-15sushi替代nk细胞存活和扩增中的il-2
测定分泌il-15/il-15sushi的nk细胞对细胞存活的影响。在存在或不存在il-2的情况下培养用cd4car或cd4il-15/il-15sushi稳定转导的nk-92细胞,以确定单独的il-15/il-15sushi分泌是否可以导致存活和扩增。在没有il-2的情况下培养的表达cd4car的nk细胞在第7天死亡,而在没有il-2的情况下培养的表达cd4il-15/il-15sushi的nk细胞以与cd4car或cd4il-15/il-15sushi细胞大致相同的速率扩增。与il-2一起培养(图68b),显示分泌的il-15/il-15sushi可以替代il-2。此外,我们能够证明分泌il-15/il-15sushi的nk细胞可以帮助共培养中未转导的nk-92细胞的存活和扩增。在该实验中,在存在或不存在il-2的情况下,将相同比例的表达nkgfp的细胞与表达cd4car或cd4il-15/il-15sushi的nk细胞一起培养。每2-3天计数细胞(图68a)。到第7天,未给予il-2的cd4carnk细胞死亡,但没有il-2的cd4il-15/il-15sushink细胞以与和il-2一起培养cd4car或cd4il-15/il-15sushi细胞大致相同的速率存活和扩增。表达gfp的细胞的数量与cd4il-15/il-15sushink细胞一起上升(图68b),表明分泌的il-15/il-15sushi对gfpnk细胞存活具有正面影响。在实验过程中,gfp阳性细胞的百分比从50%上升到超过70%(数据未显示)。在第二个实验中(图69),gfpnk和对照载体nk或cd4il-15/il-15sushink细胞以10比1的比例混合,没有il-2。到第6天,与对照细胞共培养的细胞全部死亡,但用分泌il-15/il-15sushi的nk细胞培养的细胞的存活存活至第10天。
为了进一步确定这种效应是由于单独的分泌蛋白质还是共培养细胞之间的相互作用,我们设计了一个实验,其中gfpnk细胞在培养的cd4car或cd4il-15/il-15sushink上方的室中培养。细胞或未转导的nk-92细胞。在这种情况下,只有蛋白质而非细胞才能在分离两种培养物的膜之间通过。将细胞在没有il-2的情况下培养,计数并每隔一天以1:1分裂。虽然nk-92细胞上方上腔室中的gfpnk细胞在第6天死亡,但是cd4il-15/il-15sushink细胞上方的gfpnk细胞在第12天存活并扩增(图70),从而表明它是il-15/il-15sushi蛋白由cd4il-15/il-15sushink细胞分泌,使其保持活力,而不是直接的细胞与细胞接触。在该模型中,上室代表肿瘤微环境,其中通过从cd4il-15/il-15sushink细胞分泌il-15/il-15sushi来改善t细胞或nk细胞的存活。
分泌的il-15/il-15sushi对cart和未转导的邻近细胞的影响
我们还比较了存在或不存在il-2的cd4car和cd4il-15/il-15sushi转导的t细胞的细胞生长。对于具有或不具有il-2的转导细胞,在整个实验(直至第17天)中计算的总细胞计数。与cd4car转导的t细胞相比,cd4il-15/il-15sushi转导的t细胞似乎对il-2的缺乏更耐受。
实例
生成tregcar靶treg细胞
tregcar(也称为cd4zetacd25car或c4-25z)遵循图70中的示意图。它包含sffv(脾脏焦点形成病毒)启动子,其驱动由p2a切割肽连接的两个不同单元的不完整car的表达。cd4嵌合抗原受体多肽单元包含cd45信号肽,cd4抗原识别结构域,铰链区(衍生的cd8a),跨膜结构域(cd8a)和cd3ζ链;cd25嵌合抗原受体多肽单元包含cd45信号肽,cd25抗原识别结构域,铰链区(cd8a),跨膜结构域(cd8a),共刺激结构域,cd28。tregcar可以增强共表达cd4和cd25的细胞的裂解活性,同时使单独携带cd4或cd25抗原的细胞最小化。
在测定中转导cd4zetacd25car(c4-25z)(tregcar)。与对照t细胞相比,cd4zetacd25car细胞显示约15%的表面表达,这足以观察构筑体功能的以下表型验证(图70a)。用cd4和cd25抗体测定cd4zetacd25car细胞和对照t细胞,以使用流式细胞术分析寻找逻辑门控行为。由于构筑体设计,cd4zetacd25car细胞将增强共表达cd4和cd25抗原的细胞的裂解活性。在这里,我们显示cd4+cd25+双阳性群体的耗尽(~95%),其他表型病例中的非逻辑事件几乎没有影响。条形图摘要显示逻辑门控car构造设计仅显着影响双阳性群体(图70b)。
我们通过将cd4zetacd25car与cd4car进行比较进一步表征了cd4zetacd25car。如所预期的,cd4cart细胞仅具有表达cd4的细胞的显着裂解能力,而cd4zetacd25cart细胞对该群体具有有限的杀伤能力(图71)。cd4zetacd25cart细胞也显示出几乎完全耗尽表达cd4和cd25抗原的细胞(图71)。这些研究表明,已经建立了共表达cd4和cd25的稳健的cd4zetacd25car靶向细胞。由于构筑体中的人特异性cd4或cd25scfv,cd4zetacd25car的功能特性难以在动物中测试。
在一些实施例中,所公开的公开内容还包括改善cd4zetacd25car治疗活性的方法。该示例如下所述。
实例
根据本发明制备工程改造的cd4zetacd25car细胞。
进行细胞杀伤测定
当与4-1bbl或il-15/il-15sushi或il-15/il-15ra共表达时,cd4zetacd25car杀伤的靶向细胞得到改善。
安全开关
引入“安全开关”极大地增加了安全性,“安全开关”可以是诱导型自杀基因,例如但不限于半胱天冬酶9基因,胸苷激酶,胞嘧啶脱氨酶(cd)或细胞色素p450。用于消除不需要的修饰t细胞的其他安全开关涉及t细胞中cd20或cd52或cd19或截短的表皮生长因子受体的共表达。
实施例:与cd52共表达的使用靶向t细胞恶性肿瘤的cd5car作为实例。
对于使用cart细胞对抗t细胞恶性肿瘤的临床治疗,在肿瘤耗尽后或由于car治疗引起的意外副作用的紧急情况下,可能需要建立消除患者cart细胞的安全方法。t细胞和b细胞在细胞表面表达cd52,cd52特异性抗体campath(alemtuzumab)可以从循环中消除cd52+细胞。因此,我们将人cd52序列掺入cd5car载体构筑体中(图73a)。该额外的cd52构筑体机械地将信号传导与天然cd52分开。目的是在campath处理后优先考虑天然cd52抗原在cart细胞表面逃逸的可能性。cd5car-cd52慢病毒蛋白和表达通过蛋白印迹和流式细胞术分析使用cd52抗体在转导的hek293细胞上进行确认。我们还发现共表达cd52会影响cart细胞功能。
用campath处理后体内耗尽输注的cd5car-cd52t细胞
为了评估通过campath(阿仑单抗)治疗消除car的效果,我们进行了如所述的体内方法(图72b和72c)。我们将静脉内注射5×106个cd5car-52t细胞注射到受照射的小鼠中。第二天,我们通过ip注射添加0.1mg/kg的campath或pbs,每组3只小鼠。在campath处理后6和24小时后,我们从小鼠尾部收集外周血并通过facs分析确定cd5car-52t细胞的存在。campath注射在6小时和24小时几乎完全耗尽血液中的cd5car-cd52t细胞(图72c)。campath给药后5天,cd5car-cd52细胞在骨髓和脾中也完全耗尽(图72d)。这些发现支持使用campath是一种有用的策略,作为从循环和淋巴器官中消耗car-t细胞的安全触发器。
在一个实施例中,工程改造细胞包括cd5嵌合抗原受体多肽和锚cd52(seqidno.70),和相应的多核苷酸(seqidno.69)。在一些实施例中,cd52掺入cd5car工程改造细胞或任何car工程改造细胞中,并可用作car治疗的“安全开关”。
使用gfp报告子进行启动子测试
在sffv,ef1或cag启动子下用表达gfp的慢病毒质粒转染hek293ft细胞。转染后约60小时,从各自收集上清液。通过首先用来自3种启动子中的每一种的上清液转导hek293细胞来测定相对病毒滴度。用来自3个转染的hek-293ft细胞中的每一个的gfp病毒上清液转导hek-293细胞。加入polybrene至4μl/ml。16小时后更换培养基,更换为不含病毒上清液或聚凝胺的培养基。转导三天后,收获细胞并洗涤,悬浮于2%福尔马林中,并通过流式细胞术分析gfp表达(fitc)。在每个样品中观察到gfp表达,但是用使用sffv启动子制备的病毒转导的细胞最高。
用来自每个启动子的gfp慢病毒上清液(基于hek293转导效率的结果的量)转导活化的人脐带白血球层细胞。培养5天后,收获细胞,洗涤并悬浮于2%福尔马林中,并通过流式细胞术分析gfp表达。43%的细胞以高水平(>103)表达gfp,而使用启动子ef1(15%)和cag(3%)用病毒转导的细胞的gfp表达相当低。五天后,以相同方式分析的细胞显示出几乎相同的百分比(分别为46%,15%和3%)。这些结果表明sffv启动子导致比ef1或cag启动子更强的表达,并且表达在转导后至少10天保持高水平。进一步的实验测试将包括超过10天窗口的转导细胞的更长培养时间。
上清液中病毒载体颗粒的功能滴度(通过流式细胞术测定的%gfp细胞允许代理病毒滴度调节,因为更高滴度的病毒渗入更多细胞,导致更高的%gfp细胞群)。
为了确定每个上清液中病毒载体颗粒的功能滴度,用ef1-gfp或sffv-gfp病毒上清液转导hek293细胞,30μl(低),125μl(中等)或500μl(高)每孔12孔组织培养处理的板。第二天早上将培养基更换为dmem加10%fbs(图73)。
然后将转导的细胞用胰蛋白酶消化,洗涤并悬浮在福尔马林中并进行流动分析。使用fitc通道通过流式细胞术测定每种条件下gfp+细胞的百分比(图74)。在每种情况下,用sffv-gfp转导的细胞中gfp+的百分比高于用相应体积的ef1-gfp病毒上清液转导的细胞(低50%至18%,培养基80%至40%,和82%)。高%到70%。由此,我们确定使用最高体积的ef1-启动子病毒与使用最低体积的sffv-启动子病毒的滴度相当,并且可以比较以下转导实验的相对启动子强度。
还在evos荧光显微镜下使用gfp以20x在相同的暴露条件下对每个孔显示转导的细胞(图73)。用sffv-gfp病毒上清液转导的细胞比用ef1-gfp转导的细胞显着更亮。此外,将高病毒体积负荷下的ef1-启动子的图像与使用低病毒体积负荷的sffv-启动子的图像进行比较显示出相似的荧光强度。这表明sffv启动子是基因表达的更强驱动因子。
原代t细胞中不同启动子的表面表达和持久性的比较(通过流式细胞术测定的t细胞转导的%gfp细胞显示gfp细胞群的预期差异,如先前在hek293细胞上的实验所预期的)
为了确定原代t细胞中启动子转导效率和表面表达的持久性,在经重组人纤维蛋白片段涂布之12孔培养盘上用50μlsffv-gfp或1mlef1-gfpef1-gfp病毒上清液转导活化的脐带血白血球层t细胞。在两次过夜转导后,将细胞在具有300iu/mlil-2(peprotech)的t细胞培养基上培养并维持在1.0-4.0×106/ml。洗涤细胞,悬浮在福尔马林中,并使用fitc通道进行流式细胞术分析,以在转导后7,14,21和28天确定gfp+细胞的百分比。与ef1-gfp转导的t细胞相比,用sffv-gfp转导的t细胞的gfp+细胞百分比始终更高(图75a),即使在此期间总gfp+细胞的百分比降低。进一步的比较显示,用较高(1ml)量的ef1-gfp上清液转导的t细胞实际上相对于用较低量(50μl,或少20倍)的sffv-转导的gfp+细胞的百分比降低百分比。在第7天和第28天之间,gfp从超过60%到低于40%(图75b)。这表明使用sffv启动子的转导导致转导细胞的更持久性。
产生car基因的方法包括t抗原识别部分(cd4,cd8,cd3,cd5,cd7和cd2中的至少一种,或其部分或组合中的至少一种),铰链区和t细胞。提供刺激域。
产生靶向抗原的多个car单元(ccar)的方法,所述抗原包括cd33,cd123,cd19,cd20,cd22,cd269,cs1,cd38,cd52,ror1,psma,baff,taci,cd138和gpc3中的至少一种。或者,提供铰链区和t细胞刺激域的一部分或组合。
所提供的方法还包括:1)产生靶向表达cd45的白血病和淋巴瘤的cart或nk细胞并避免自杀;2)产生“装甲”cart或nk细胞,其设计用于克服抑制性肿瘤微环境并表现出增强的抗肿瘤活性和长期持久性。
虽然已经描述了目前被认为是本发明公开的优选实施例的内容,但是本领域技术人员将认识到,在不脱离本发明公开的精神的情况下,可以对其进行其他和进一步的改变和修改,并且意图是预期的。要求所有这些修改和变化都在本发明公开的真实范围内。
在整个说明书和权利要求中使用与本发明公开的方面有关的各种术语。除非另有说明,否则这些术语将在本领域中给出它们的普通含义。其他具体定义的术语应以与本文提供的定义一致的方式解释。
序列表
<110>美商生物细胞基因治疗有限公司
<120>嵌合抗原受体(car)、组合物及其使用方法
<130>2541-3pctii
<140>
<141>
<150>62/369,004
<151>2016-07-29
<150>pct/us16/39306
<151>2016-06-24
<160>125
<170>patentinversion3.5
<210>1
<211>830
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>1
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalmetthrglnserproaspserleu
202530
alavalserleuglygluargvalthrmetasncyslyssersergln
354045
serleuleutyrserthrasnglnlysasntyrleualatrptyrgln
505560
glnlysproglyglnserprolysleuleuiletyrtrpalaserthr
65707580
arggluserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleuthrileserservalglnalagluaspvalalaval
100105110
tyrtyrcysglnglntyrtyrsertyrargthrpheglyglyglythr
115120125
lysleugluilelysglyglyglyglyserglyglyglyglysergly
130135140
glyglyglyserglnvalglnleuglnglnserglyprogluvalval
145150155160
lysproglyalaservallysmetsercyslysalaserglytyrthr
165170175
phethrsertyrvalilehistrpvalargglnlysproglyglngly
180185190
leuasptrpileglytyrileasnprotyrasnaspglythrasptyr
195200205
aspglulysphelysglylysalathrleuthrseraspthrserthr
210215220
serthralatyrmetgluleuserserleuargsergluaspthrala
225230235240
valtyrtyrcysalaargglulysaspasntyralathrglyalatrp
245250255
phealatyrtrpglyglnglythrleuvalthrvalserserthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcysargserlysargserargleuleuhisserasptyrmet
340345350
asnmetthrproargargproglyprothrarglyshistyrglnpro
355360365
tyralaproproargaspphealaalatyrargserlysargglyarg
370375380
lyslysleuleutyrilephelysglnprophemetargprovalgln
385390395400
thrthrglnglugluaspglycyssercysargpheproglugluglu
405410415
gluglyglycysgluleuargvallyspheserargseralaaspala
420425430
proalatyrglnglnglyglnasnglnleutyrasngluleuasnleu
435440445
glyargarggluglutyraspvalleuasplysargargglyargasp
450455460
proglumetglyglylysproglnargarglysasnproglnglugly
465470475480
leutyrasngluleuglnlysasplysmetalaglualatyrserglu
485490495
ileglymetlysglygluargargargglylysglyhisaspglyleu
500505510
tyrglnglyleuserthralathrlysaspthrtyraspalaleuhis
515520525
metglnalaleuproproargglyserglyalathrasnpheserleu
530535540
leulysglnalaglyaspvalglugluasnproglypromettyrarg
545550555560
metglnleuleusercysilealaleuserleualaleuvalthrasn
565570575
serglyilehisvalpheileleuglycyspheseralaglyleupro
580585590
lysthrglualaasntrpvalasnvalileseraspleulyslysile
595600605
gluaspleuileglnsermethisileaspalathrleutyrthrglu
610615620
seraspvalhisprosercyslysvalthralametlyscyspheleu
625630635640
leugluleuglnvalileserleugluserglyaspalaserilehis
645650655
aspthrvalgluasnleuileileleualaasnasnserleuserser
660665670
asnglyasnvalthrgluserglycyslysglucysglugluleuglu
675680685
glulysasnilelysglupheleuglnserphevalhisilevalgln
690695700
metpheileasnthrserserglyglyglyserglyglyglyglyser
705710715720
glyglyglyglyserglyglyglyglyserglyglyglyserleugln
725730735
alaproargargalaargglycysargthrleuglyleuproalaleu
740745750
leuleuleuleuleuleuargproproalathrargglyilethrcys
755760765
proproprometservalgluhisalaaspiletrpvallyssertyr
770775780
serleutyrserarggluargtyrilecysasnserglyphelysarg
785790795800
lysalaglythrserserleuthrglucysvalleuasnlysalathr
805810815
asnvalalahistrpthrthrproserleulyscysilearg
820825830
<210>2
<211>2509
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>2
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgatgacccaaagccccgacagcctggccgtgagcctgggcg120
agagggtgaccatgaactgcaaaagcagccagtccctgctgtactccaccaaccagaaga180
actacctggcttggtatcaacagaagcccggacagagccccaagctgctgatctattggg240
ccagcactagggaaagcggcgtgcccgataggttcagcggcagcgggagcggcacagact300
tcactctgaccattagcagcgtgcaggctgaggatgtggccgtctactactgccagcagt360
actacagctacaggacctttgggggcggaactaagctggagatcaagggaggggggggat420
ccgggggaggaggctccggcggaggcggaagccaagtgcaactgcagcagagcggcccag480
aggtggtcaaacctggggcaagcgtgaagatgagctgcaaggctagcggctataccttca540
ccagctatgtgatccactgggtgaggcagaaaccaggacagggcctggactggatcggct600
acatcaacccctacaatgacggcaccgattatgacgaaaaattcaaggggaaggccaccc660
tgaccagcgacaccagcacaagcaccgcctacatggagctgtccagcctgaggtccgagg720
acaccgccgtgtattactgtgccagggagaaggacaattacgccaccggcgcttggttcg780
cctactggggccagggcacactggtgacagtgagcagcaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcaggagtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgcc1080
gccccgggcccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcct1140
atcgctccaaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagac1200
cagtacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaag1260
gaggatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagg1320
gccagaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttgg1380
acaagagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctc1440
aggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattg1500
ggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagta1560
cagccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcg1620
gagccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggcccca1680
tgtacagaatgcagctgctgagctgcatcgccctgagcctggccctggtgaccaacagcg1740
gcatccacgtgttcatcctgggctgcttcagcgccggcctgcccaagaccgaggccaact1800
gggtgaacgtgatcagcgacctgaagaagatcgaggacctgatccagagcatgcacatcg1860
acgccaccctgtacaccgagagcgacgtgcaccccagctgcaaggtgaccgccatgaagt1920
gcttcctgctggagctgcaggtgatcagcctggagagcggcgacgccagcatccacgaca1980
ccgtggagaacctgatcatcctggccaacaacagcctgagcagcaacggcaacgtgaccg2040
agagcggctgcaaggagtgcgaggagctggaggagaagaacatcaaggagttcctgcaga2100
gcttcgtgcacatcgtgcagatgttcatcaacaccagctccggcggcggctccggcggcg2160
gcggctccggcggcggcggctccggcggcggcggctccggcggcggctccctgcaggccc2220
ccagaagagccagaggctgcagaaccctgggcctgcccgccctgctgctgctgctgctgc2280
tgagaccccccgccaccagaggcatcacctgccccccccccatgagcgtggagcacgccg2340
acatctgggtgaagagctacagcctgtacagcagagagagatacatctgcaacagcggct2400
tcaagagaaaggccggcaccagcagcctgaccgagtgcgtgctgaacaaggccaccaacg2460
tggcccactggaccacccccagcctgaagtgcatcagataagtttaaac2509
<210>3
<211>995
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>3
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspileglnmetthrglnthrthrserserleu
202530
seralaserleuglyaspargvalthrilesercysargalasergln
354045
aspileserlystyrleuasntrptyrglnglnlysproaspglythr
505560
vallysleuleuiletyrhisthrserargleuhisserglyvalpro
65707580
serargpheserglyserglyserglythrasptyrserleuthrile
859095
serasnleugluglngluaspilealathrtyrphecysglnglngly
100105110
asnthrleuprotyrthrpheglyglyglythrlysleugluilethr
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglyserglu
130135140
vallysleuglngluserglyproglyleuvalalaproserglnser
145150155160
leuservalthrcysthrvalserglyvalserleuproasptyrgly
165170175
valsertrpileargglnproproarglysglyleuglutrpleugly
180185190
valiletrpglysergluthrthrtyrtyrasnseralaleulysser
195200205
argleuthrileilelysaspasnserlysserglnvalpheleulys
210215220
metasnserleuglnthraspaspthralailetyrtyrcysalalys
225230235240
histyrtyrtyrglyglysertyralametasptyrtrpglyglngly
245250255
thrservalthrvalserserthrthrthrproalaproargpropro
260265270
thrproalaprothrilealaserglnproleuserleuargproglu
275280285
alacysargproalaalaglyglyalavalhisthrargglyleuasp
290295300
phealacysaspiletyriletrpalaproleualaglythrcysgly
305310315320
valleuleuleuserleuvalilethrleutyrcyslysargglyarg
325330335
lyslysleuleutyrilephelysglnprophemetargprovalgln
340345350
thrthrglnglugluaspglycyssercysargpheproglugluglu
355360365
gluglyglycysgluleuargvallyspheserargseralaaspala
370375380
proalatyrglnglnglyglnasnglnleutyrasngluleuasnleu
385390395400
glyargarggluglutyraspvalleuasplysargargglyargasp
405410415
proglumetglyglylysproglnargarglysasnproglnglugly
420425430
leutyrasngluleuglnlysasplysmetalaglualatyrserglu
435440445
ileglymetlysglygluargargargglylysglyhisaspglyleu
450455460
tyrglnglyleuserthralathrlysaspthrtyraspalaleuhis
465470475480
metglnalaleuproproargglyserglyalathrasnpheserleu
485490495
leulysglnalaglyaspvalglugluasnproglyprometalaleu
500505510
provalthralaleuleuleuproleualaleuleuleuhisalaala
515520525
argproglnilevalleuserglnserproalaileleuseralaser
530535540
proglyglulysvalthrmetthrcysargalaserserservalser
545550555560
tyrilehistrppheglnglnlysproglyserserprolysprotrp
565570575
iletyralathrserasnleualaserglyvalprovalargpheser
580585590
glyserglyserglythrsertyrserleuthrileserargvalglu
595600605
alagluaspalaalathrtyrtyrcysglnglntrpthrserasnpro
610615620
prothrpheglyglyglythrlysleugluilelysglyglyglygly
625630635640
serglyglyglyglyserglyglyglyglyserglnvalglnleugln
645650655
glnproglyalagluleuvallysproglyalaservallysmetser
660665670
cyslysalaserglytyrthrphethrsertyrasnmethistrpval
675680685
lysglnthrproglyargglyleuglutrpileglyalailetyrpro
690695700
glyasnglyaspthrsertyrasnglnlysphelysglylysalathr
705710715720
leuthralaasplysserserserthralatyrmetglnleuserser
725730735
leuthrsergluaspseralavaltyrtyrcysalaargserthrtyr
740745750
tyrglyglyasptrptyrpheasnvaltrpglyalaglythrthrval
755760765
thrvalseralathrthrthrproalaproargproprothrproala
770775780
prothrilealaserglnproleuserleuargproglualacysarg
785790795800
proalaalaglyglyalavalhisthrargglyleuaspphealacys
805810815
aspiletyriletrpalaproleualaglythrcysglyvalleuleu
820825830
leuserleuvalilethrleutyrcysargserlysargserargleu
835840845
leuhisserasptyrmetasnmetthrproargargproglyprothr
850855860
arglyshistyrglnprotyralaproproargaspphealaalatyr
865870875880
argserargvallyspheserargseralaaspalaproalatyrgln
885890895
glnglyglnasnglnleutyrasngluleuasnleuglyargargglu
900905910
glutyraspvalleuasplysargargglyargaspproglumetgly
915920925
glylysproglnargarglysasnproglngluglyleutyrasnglu
930935940
leuglnlysasplysmetalaglualatyrsergluileglymetlys
945950955960
glygluargargargglylysglyhisaspglyleutyrglnglyleu
965970975
serthralathrlysaspthrtyraspalaleuhismetglnalaleu
980985990
proproarg
995
<210>4
<211>3004
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>4
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatccagatgacacagactacatcctccctgtctgcctctctgggag120
acagagtcaccatcagttgcagggcaagtcaggacattagtaaatatttaaattggtatc180
agcagaaaccagatggaactgttaaactcctgatctaccatacatcaagattacactcag240
gagtcccatcaaggttcagtggcagtgggtctggaacagattattctctcaccattagca300
acctggagcaagaagatattgccacttacttttgccaacagggtaatacgcttccgtaca360
cgttcggaggggggaccaagctggagatcacaggtggcggtggctcgggcggtggtgggt420
cgggtggcggcggatctgaggtgaaactgcaggagtcaggacctggcctggtggcgccct480
cacagagcctgtccgtcacatgcactgtctcaggggtctcattacccgactatggtgtaa540
gctggattcgccagcctccacgaaagggtctggagtggctgggagtaatatggggtagtg600
aaaccacatactataattcagctctcaaatccagactgaccatcatcaaggacaactcca660
agagccaagttttcttaaaaatgaacagtctgcaaactgatgacacagccatttactact720
gtgccaaacattattactacggtggtagctatgctatggactactggggccaaggaacct780
cagtcaccgtctcctcaaccacgacgccagcgccgcgaccaccaacaccggcgcccacca840
tcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcag900
tgcacacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccggga960
cttgtggggtccttctcctgtcactggttatcaccctttactgcaaacggggcagaaaga1020
aactcctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagaggaag1080
atggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtgaagt1140
tcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagc1200
tcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctg1260
agatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgc1320
agaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggg1380
gcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacg1440
cccttcacatgcaggccctgccccctcgcggaagcggagccaccaacttcagcctgctga1500
agcaggccggcgacgtggaggagaaccccggccccatggccttaccagtgaccgccttgc1560
tcctgccgctggccttgctgctccacgccgccaggccgcagatcgtgctgagccagagcc1620
ctgccatcctgtccgcaagcccaggcgagaaggtgaccatgacctgtagggccagcagct1680
ccgtgagctacatccactggtttcagcagaagcctggaagcagccctaagccctggatct1740
acgccacaagcaatctggctagcggcgtgcccgtgaggttcagcggcagcgggagcggga1800
ccagctacagcctgactatcagcagggtggaggccgaggacgccgccacatactactgcc1860
aacagtggacctccaacccacccacctttggaggagggacaaaactggagatcaaagggg1920
gcggagggtccggaggcggcggaagcgggggagggggaagccaggtccaactgcaacagc1980
ccggagcagaactggtcaaaccaggcgccagcgtgaagatgagctgcaaggccagcgggt2040
acaccttcacttcctataacatgcactgggtgaagcagaccccaggaaggggcctggagt2100
ggatcggggcaatctatcccggcaacggcgacacaagctacaaccagaagttcaagggga2160
aagccactctgaccgccgacaagtccagctccaccgcctacatgcagctgagctccctga2220
ccagcgaggacagcgccgtgtactattgcgccagaagcacttattacggaggggactggt2280
acttcaacgtgtggggggcagggaccaccgtgaccgtgtccgccaccacgacgccagcgc2340
cgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagagg2400
cgtgccggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgata2460
tctacatctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatca2520
ccctttactgcaggagtaagaggagcaggctcctgcacagtgactacatgaacatgactc2580
cccgccgccccgggcccacccgcaagcattaccagccctatgccccaccacgcgacttcg2640
cagcctatcgctccagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagg2700
gccagaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttgg2760
acaagagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctc2820
aggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattg2880
ggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagta2940
cagccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgctaagttt3000
aaac3004
<210>5
<211>1001
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>5
aspargmetalaleuprovalthralaleuleuleuproleualaleu
151015
leuleuhisalaalaargproaspileglnmetthrglnthrthrser
202530
serleuseralaserleuglyaspargvalthrilesercysargala
354045
serglnaspileserlystyrleuasntrptyrglnglnlysproasp
505560
glythrvallysleuleuiletyrhisthrserargleuhissergly
65707580
valproserargpheserglyserglyserglythrasptyrserleu
859095
thrileserasnleugluglngluaspilealathrtyrphecysgln
100105110
glnglyasnthrleuprotyrthrpheglyglyglythrlysleuglu
115120125
ilethrglyglyglyglyserglyglyglyglyserglyglyglygly
130135140
sergluvallysleuglngluserglyproglyleuvalalaproser
145150155160
glnserleuservalthrcysthrvalserglyvalserleuproasp
165170175
tyrglyvalsertrpileargglnproproarglysglyleuglutrp
180185190
leuglyvaliletrpglysergluthrthrtyrtyrasnseralaleu
195200205
lysserargleuthrileilelysaspasnserlysserglnvalphe
210215220
leulysmetasnserleuglnthraspaspthralailetyrtyrcys
225230235240
alalyshistyrtyrtyrglyglysertyralametasptyrtrpgly
245250255
glnglythrservalthrvalserserthrthrthrproalaproarg
260265270
proprothrproalaprothrilealaserglnproleuserleuarg
275280285
proglualacysargproalaalaglyglyalavalhisthrarggly
290295300
leuaspphealacysaspiletyriletrpalaproleualaglythr
305310315320
cysglyvalleuleuleuserleuvalilethrleutyrcyslysarg
325330335
glyarglyslysleuleutyrilephelysglnprophemetargpro
340345350
valglnthrthrglnglugluaspglycyssercysargpheproglu
355360365
gluglugluglyglycysgluleuargvallyspheserargserala
370375380
aspalaproalatyrglnglnglyglnasnglnleutyrasngluleu
385390395400
asnleuglyargarggluglutyraspvalleuasplysargarggly
405410415
argaspproglumetglyglylysproargarglysasnproglnglu
420425430
glyleutyrasngluleuglnlysasplysmetalaglualatyrser
435440445
gluileglymetlysglygluargargargglylysglyhisaspgly
450455460
leutyrglnglyleuserthralathrlysaspthrtyraspalaleu
465470475480
hismetglnalaleuproproargglyserglyalathrasnpheser
485490495
leuleulysglnalaglyaspvalglugluasnproglyprometala
500505510
leuprovalthralaleuleuleuproleualaleuleuleuhisala
515520525
alaargproaspileglnmetthrglnserproserserleuserala
530535540
servalglyaspargvalthrilethrcysargserserglnserile
545550555560
valhisservalglyasnthrpheleuglutrptyrglnglnlyspro
565570575
glylysalaprolysleuleuiletyrlysvalserasnargpheser
580585590
glyvalproserargpheserglyserglyserglythraspphethr
595600605
leuthrileserserleuglnprogluaspphealathrtyrtyrcys
610615620
pheglnglyserglnpheprotyrthrpheglyglnglythrlysval
625630635640
gluilelysglyglyglyglyserglyglyglyglyserglyglygly
645650655
glysergluvalglnleuvalgluserglyglyglyleuvalglnpro
660665670
glyglyserleuargleusercysalaalaserglytyrglupheser
675680685
argsertrpmetasntrpvalargglnalaproglylysglyleuglu
690695700
trpvalglyargiletyrproglyaspglyaspthrasntyrsergly
705710715720
lysphelysglyargphethrileseralaaspthrserlysasnthr
725730735
alatyrleuglnmetasnserleuargalagluaspthralavaltyr
740745750
tyrcysalaargaspglysersertrpasptrptyrpheaspvaltrp
755760765
glyglnglythrleuvalthrvalserserthrthrthrproalapro
770775780
argproprothrproalaprothrilealaserglnproleuserleu
785790795800
argproglualacysargproalaalaglyglyalavalhisthrarg
805810815
glyleuaspphealacysaspiletyriletrpalaproleualagly
820825830
thrcysglyvalleuleuleuserleuvalilethrleutyrcyslys
835840845
argglyarglyslysleuleutyrilephelysglnprophemetarg
850855860
provalglnthrthrglnglugluaspglycyssercysargphepro
865870875880
glugluglugluglyglycysgluleuargvallyspheserargser
885890895
alaaspalaproalatyrglnglnglyglnasnglnleutyrasnglu
900905910
leuasnleuglyargarggluglutyraspvalleuasplysargarg
915920925
glyargaspproglumetglyglylysproargarglysasnprogln
930935940
gluglyleutyrasngluleuglnlysasplysmetalaglualatyr
945950955960
sergluileglymetlysglygluargargargglylysglyhisasp
965970975
glyleutyrglnglyleuserthralathrlysaspthrtyraspala
980985990
leuhismetglnalaleuproproarg
9951000
<210>6
<211>3016
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>6
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatccagatgacacagactacatcctccctgtctgcctctctgggag120
acagagtcaccatcagttgcagggcaagtcaggacattagtaaatatttaaattggtatc180
agcagaaaccagatggaactgttaaactcctgatctaccatacatcaagattacactcag240
gagtcccatcaaggttcagtggcagtgggtctggaacagattattctctcaccattagca300
acctggagcaagaagatattgccacttacttttgccaacagggtaatacgcttccgtaca360
cgttcggaggggggaccaagctggagatcacaggtggcggtggctcgggcggtggtgggt420
cgggtggcggcggatctgaggtgaaactgcaggagtcaggacctggcctggtggcgccct480
cacagagcctgtccgtcacatgcactgtctcaggggtctcattacccgactatggtgtaa540
gctggattcgccagcctccacgaaagggtctggagtggctgggagtaatatggggtagtg600
aaaccacatactataattcagctctcaaatccagactgaccatcatcaaggacaactcca660
agagccaagttttcttaaaaatgaacagtctgcaaactgatgacacagccatttactact720
gtgccaaacattattactacggtggtagctatgctatggactactggggccaaggaacct780
cagtcaccgtctcctcaaccacgacgccagcgccgcgaccaccaacaccggcgcccacca840
tcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcag900
tgcacacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccggga960
cttgtggggtccttctcctgtcactggttatcaccctttactgcaaacggggcagaaaga1020
aactcctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagaggaag1080
atggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtgaagt1140
tcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagc1200
tcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctg1260
agatggggggaaagccgagaaggaagaaccctcaggaaggcctgtacaatgaactgcaga1320
aagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggca1380
aggggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgccc1440
ttcacatgcaggccctgccccctcgcggaagcggagccaccaacttcagcctgctgaagc1500
aggccggcgacgtggaggagaaccccggccccatggccttaccagtgaccgccttgctcc1560
tgccgctggccttgctgctccacgccgccaggccggatatccagatgacccagagcccca1620
gctccctgtccgcatccgtgggcgacagagtgacaattacctgtagaagcagccaaagca1680
tcgtgcatagcgtcggcaacacttttctggagtggtatcaacagaagcccgggaaggccc1740
ccaaactgctgatctacaaggtgagcaacagattcagcggggtcccaagcagattctccg1800
gcagcggctccgggactgacttcaccctgaccattagcagcctgcagccagaggacttcg1860
ccacatactactgcttccaagggagccagttcccctacaccttcggccaaggcactaagg1920
tggagatcaaaggggggggaggaagcggcggaggagggagcggaggcgggggatccgaag1980
tgcaactggtcgaatccggaggggggctggtccagcctggagggtccctgagactgagct2040
gcgccgcaagcggctacgagttctccaggtcctggatgaactgggtgaggcaggccccag2100
gaaaagggctggaatgggtgggcaggatctaccctggcgacggcgataccaactactccg2160
gaaagttcaagggcaggttcactatcagcgccgacactagcaagaataccgcctacctgc2220
agatgaatagcctgagggccgaggacaccgccgtgtattactgcgctagagacggcagca2280
gctgggattggtacttcgacgtgtggggccagggcactctggtgactgtgagcagcacca2340
cgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccc2400
tgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggact2460
tcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttctcctgt2520
cactggttatcaccctttactgcaaacggggcagaaagaaactcctgtatatattcaaac2580
aaccatttatgagaccagtacaaactactcaagaggaagatggctgtagctgccgatttc2640
cagaagaagaagaaggaggatgtgaactgagagtgaagttcagcaggagcgcagacgccc2700
ccgcgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaagagagg2760
agtacgatgttttggacaagagacgtggccgggaccctgagatggggggaaagccgagaa2820
ggaagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcct2880
acagtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttacc2940
agggtctcagtacagccaccaaggacacctacgacgcccttcacatgcaggccctgcccc3000
ctcgctaagtttaaac3016
<210>7
<211>990
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>7
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspileglnmetthrglnthrthrserserleu
202530
seralaserleuglyaspargvalthrilesercysargalasergln
354045
aspileserlystyrleuasntrptyrglnglnlysproaspglythr
505560
vallysleuleuiletyrhisthrserargleuhisserglyvalpro
65707580
serargpheserglyserglyserglythrasptyrserleuthrile
859095
serasnleugluglngluaspilealathrtyrphecysglnglngly
100105110
asnthrleuprotyrthrpheglyglyglythrlysleugluilethr
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglyserglu
130135140
vallysleuglngluserglyproglyleuvalalaproserglnser
145150155160
leuservalthrcysthrvalserglyvalserleuproasptyrgly
165170175
valsertrpileargglnproproarglysglyleuglutrpleugly
180185190
valiletrpglysergluthrthrtyrtyrasnseralaleulysser
195200205
argleuthrileilelysaspasnserlysserglnvalpheleulys
210215220
metasnserleuglnthraspaspthralailetyrtyrcysalalys
225230235240
histyrtyrtyrglyglysertyralametasptyrtrpglyglngly
245250255
thrservalthrvalserserthrthrthrproalaproargpropro
260265270
thrproalaprothrilealaserglnproleuserleuargproglu
275280285
alacysargproalaalaglyglyalavalhisthrargglyleuasp
290295300
phealacysaspiletyriletrpalaproleualaglythrcysgly
305310315320
valleuleuleuserleuvalilethrleutyrcyslysargglyarg
325330335
lyslysleuleutyrilephelysglnprophemetargprovalgln
340345350
thrthrglnglugluaspglycyssercysargpheproglugluglu
355360365
gluglyglycysgluleuargvallyspheserargseralaaspala
370375380
proalatyrglnglnglyglnasnglnleutyrasngluleuasnleu
385390395400
glyargarggluglutyraspvalleuasplysargargglyargasp
405410415
proglumetglyglylysproglnargarglysasnproglnglugly
420425430
leutyrasngluleuglnlysasplysmetalaglualatyrserglu
435440445
ileglymetlysglygluargargargglylysglyhisaspglyleu
450455460
tyrglnglyleuserthralathrlysaspthrtyraspalaleuhis
465470475480
metglnalaleuproproargglyserglyalathrasnpheserleu
485490495
leulysglnalaglyaspvalglugluasnproglyprometalaleu
500505510
provalthralaleuleuleuproleualaleuleuleuhisalaala
515520525
argproaspvalglnilethrglnserprosertyrleualaalaser
530535540
proglygluthrilethrileasncysargalaserlysserileser
545550555560
lysaspleualatrptyrglnglulysproglylysthrasnlysleu
565570575
leuiletyrserglyserthrleuglnserglyileproserargphe
580585590
serglyserglyserglythraspphethrleuthrileserserleu
595600605
gluprogluaspphealamettyrtyrcysglnglnhisasnlystyr
610615620
protyrthrpheglyglyglythrlysleugluilelysglyglygly
625630635640
glyserglyglyglyglyserglyglyglyglyserglnvalglnleu
645650655
glnglnproglyalagluleuvalargproglyalaservallysleu
660665670
sercyslysalaserglytyrthrphethrsertyrtrpmetasntrp
675680685
vallysglnargproaspglnglyleuglutrpileglyargileasp
690695700
protyraspsergluthrhistyrasnglnlysphelysasplysala
705710715720
ileleuthrvalasplysserserserthralatyrmetglnleuser
725730735
serleuthrsergluaspseralavaltyrtyrcysalaargglyasn
740745750
trpaspasptyrtrpglyglnglythrthrleuthrvalserserthr
755760765
thrthrproalaproargproprothrproalaprothrilealaser
770775780
glnproleuserleuargproglualacysargproalaalaglygly
785790795800
alavalhisthrargglyleuaspphealacysaspiletyriletrp
805810815
alaproleualaglythrcysglyvalleuleuleuserleuvalile
820825830
thrleutyrcysargserlysargserargleuleuhisserasptyr
835840845
metasnmetthrproargargproglyprothrarglyshistyrgln
850855860
protyralaproproargaspphealaalatyrargserargvallys
865870875880
pheserargseralaaspalaproalatyrglnglnglyglnasngln
885890895
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
900905910
asplysargargglyargaspproglumetglyglylysproglnarg
915920925
arglysasnproglngluglyleutyrasngluleuglnlysasplys
930935940
metalaglualatyrsergluileglymetlysglygluargargarg
945950955960
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
965970975
aspthrtyraspalaleuhismetglnalaleuproproarg
980985990
<210>8
<211>2989
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>8
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatccagatgacacagactacatcctccctgtctgcctctctgggag120
acagagtcaccatcagttgcagggcaagtcaggacattagtaaatatttaaattggtatc180
agcagaaaccagatggaactgttaaactcctgatctaccatacatcaagattacactcag240
gagtcccatcaaggttcagtggcagtgggtctggaacagattattctctcaccattagca300
acctggagcaagaagatattgccacttacttttgccaacagggtaatacgcttccgtaca360
cgttcggaggggggaccaagctggagatcacaggtggcggtggctcgggcggtggtgggt420
cgggtggcggcggatctgaggtgaaactgcaggagtcaggacctggcctggtggcgccct480
cacagagcctgtccgtcacatgcactgtctcaggggtctcattacccgactatggtgtaa540
gctggattcgccagcctccacgaaagggtctggagtggctgggagtaatatggggtagtg600
aaaccacatactataattcagctctcaaatccagactgaccatcatcaaggacaactcca660
agagccaagttttcttaaaaatgaacagtctgcaaactgatgacacagccatttactact720
gtgccaaacattattactacggtggtagctatgctatggactactggggccaaggaacct780
cagtcaccgtctcctcaaccacgacgccagcgccgcgaccaccaacaccggcgcccacca840
tcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcag900
tgcacacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccggga960
cttgtggggtccttctcctgtcactggttatcaccctttactgcaaacggggcagaaaga1020
aactcctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagaggaag1080
atggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtgaagt1140
tcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagc1200
tcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctg1260
agatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgc1320
agaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggg1380
gcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacg1440
cccttcacatgcaggccctgccccctcgcggaagcggagccaccaacttcagcctgctga1500
agcaggccggcgacgtggaggagaaccccggccccatggccttaccagtgaccgccttgc1560
tcctgccgctggccttgctgctccacgccgccaggccggacgtgcagatcacccagagcc1620
ccagctacctggccgccagccccggcgagaccatcaccatcaactgcagagccagcaaga1680
gcatcagcaaggacctggcctggtaccaggagaagcccggcaagaccaacaagctgctga1740
tctacagcggcagcaccctgcagagcggcatccccagcagattcagcggcagcggcagcg1800
gcaccgacttcaccctgaccatcagcagcctggagcccgaggacttcgccatgtactact1860
gccagcagcacaacaagtacccctacaccttcggcggcggcaccaagctggagatcaagg1920
gaggggggggatccgggggaggaggctccggcggaggcggaagccaggtgcagctgcagc1980
agcccggcgccgagctggtgagacccggcgccagcgtgaagctgagctgcaaggccagcg2040
gctacaccttcaccagctactggatgaactgggtgaagcagagacccgaccagggcctgg2100
agtggatcggcagaatcgacccctacgacagcgagacccactacaaccagaagttcaagg2160
acaaggccatcctgaccgtggacaagagcagcagcaccgcctacatgcagctgagcagcc2220
tgaccagcgaggacagcgccgtgtactactgcgccagaggcaactgggacgactactggg2280
gccagggcaccaccctgaccgtgagcagcaccacgacgccagcgccgcgaccaccaacac2340
cggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcgg2400
cggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgc2460
ccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcagga2520
gtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggc2580
ccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctcca2640
gagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctct2700
ataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggcc2760
gggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtaca2820
atgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagc2880
gccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggaca2940
cctacgacgcccttcacatgcaggccctgccccctcgctaagtttaaac2989
<210>9
<211>1066
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>9
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproglnilevalleuthrglnserproalailemet
202530
seralaserproglyglulysvalthrilethrcysseralaserser
354045
serilesertyrmethistrppheglnglnlysproglythrserpro
505560
lysleutrpiletyrthrthrserasnleualaserglyvalproala
65707580
argpheserglyserglyserglythrsertyrserleuthrileser
859095
argmetglualagluaspalaalathrtyrtyrcyshisglnargser
100105110
thrtyrproleuthrpheglyserglythrlysleugluleulysgly
115120125
glyglyglyserglyglyglyglyserglyglyglyglyserglnval
130135140
glnleuglnglnserglyalagluleualalysproglyalaserval
145150155160
lysmetsercyslysalaserglytyrthrphethrsertyrargmet
165170175
histrpvallysglnargproglyglnglyleuglutrpileglytyr
180185190
ileasnproserthrglytyrthrglutyrasnglnlysphelysasp
195200205
lysalathrleuthralaasplysserserserthralatyrmetgln
210215220
leuserserleuthrphegluaspseralavaltyrtyrcysalaarg
225230235240
glyglyglyvalpheasptyrtrpglyglnglythrthrleuthrval
245250255
serserthrthrthrproalaproargproprothrproalaprothr
260265270
ilealaserglnproleuserleuargproglualacysargproala
275280285
alaglyglyalavalhisthrargglyleuaspphealacysaspile
290295300
tyriletrpalaproleualaglythrcysglyvalleuleuleuser
305310315320
leuvalilethrleutyrcysargserlysargserargleuleuhis
325330335
serasptyrmetasnmetthrproargargproglyprothrarglys
340345350
histyrglnprotyralaproproargaspphealaalatyrargser
355360365
lysargglyarglyslysleuleutyrilephelysglnprophemet
370375380
argprovalglnthrthrglnglugluaspglycyssercysargphe
385390395400
proglugluglugluglyglycysgluleuargvallyspheserarg
405410415
seralaaspalaproalatyrlysglnglyglnasnglnleutyrasn
420425430
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
435440445
argglyargaspproglumetglyglylysproargarglysasnpro
450455460
glngluglyleutyrasngluleuglnlysasplysmetalagluala
465470475480
tyrsergluileglymetlysglygluargargargglylysglyhis
485490495
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
500505510
alaleuhismetglnalaleuproproargglyserglyalathrasn
515520525
pheserleuleulysglnalaglyaspvalglugluasnproglypro
530535540
metalaleuprovalthralaleuleuleuproleualaleuleuleu
545550555560
hisalaalaargproaspvalglnilethrglnserprosertyrleu
565570575
alaalaserproglygluthrilethrileasncysargalaserlys
580585590
serileserlysaspleualatrptyrglnglulysproglylysthr
595600605
asnlysleuleuiletyrserglyserthrleuglnserglyilepro
610615620
serargpheserglyserglyserglythraspphethrleuthrile
625630635640
serserleugluprogluaspphealamettyrtyrcysglnglnhis
645650655
asnlystyrprotyrthrpheglyglyglythrlysleugluilelys
660665670
glyglyglyglyserglyglyglyglyserglyglyglyglysergln
675680685
valglnleuglnglnproglyalagluleuvalargproglyalaser
690695700
vallysleusercyslysalaserglytyrthrphethrsertyrtrp
705710715720
metasntrpvallysglnargproaspglnglyleuglutrpilegly
725730735
argileaspprotyraspsergluthrhistyrasnglnlysphelys
740745750
asplysalaileleuthrvalasplysserserserthralatyrmet
755760765
glnleuserserleuthrsergluaspseralavaltyrtyrcysala
770775780
argglyasntrpaspasptyrtrpglyglnglythrthrleuthrval
785790795800
serserthrthrthrproalaproargproprothrproalaprothr
805810815
ilealaserglnproleuserleuargproglualacysargproala
820825830
alaglyglyalavalhisthrargglyleuaspphealacysaspile
835840845
tyriletrpalaproleualaglythrcysglyvalleuleuleuser
850855860
leuvalilethrleutyrcysargserlysargserargleuleuhis
865870875880
serasptyrmetasnmetthrproargargproglyprothrarglys
885890895
histyrglnprotyralaproproargaspphealaalatyrargser
900905910
lysargglyarglyslysleuleutyrilephelysglnprophemet
915920925
argprovalglnthrthrglnglugluaspglycyssercysargphe
930935940
proglugluglugluglyglycysgluleuargvallyspheserarg
945950955960
seralaaspalaproalatyrlysglnglyglnasnglnleutyrasn
965970975
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
980985990
argglyargaspproglumetglyglylysproargarglysasnpro
99510001005
glngluglyleutyrasngluleuglnlysasplysmetalaglu
101010151020
alatyrsergluileglymetlysglygluargargargglylys
102510301035
glyhisaspglyleutyrglnglyleuserthralathrlysasp
104010451050
thrtyraspalaleuhismetglnalaleuproproarg
105510601065
<210>10
<211>3217
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>10
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccgcagatcgtgctgacccagagccccgccatcatgagcgccagccccggcg120
agaaggtgaccatcacctgcagcgccagcagcagcatcagctacatgcactggttccagc180
agaagcccggcaccagccccaagctgtggatctacaccaccagcaacctggccagcggcg240
tgcccgccagattcagcggcagcggcagcggcaccagctacagcctgaccatcagcagaa300
tggaggccgaggacgccgccacctactactgccaccagagaagcacctaccccctgacct360
tcggcagcggcaccaagctggagctgaagggaggggggggatccgggggaggaggctccg420
gcggaggcggaagccaggtgcagctgcagcagagcggcgccgagctggccaagcccggcg480
ccagcgtgaagatgagctgcaaggccagcggctacaccttcaccagctacagaatgcact540
gggtgaagcagagacccggccagggcctggagtggatcggctacatcaaccccagcaccg600
gctacaccgagtacaaccagaagttcaaggacaaggccaccctgaccgccgacaagagca660
gcagcaccgcctacatgcagctgagcagcctgaccttcgaggacagcgccgtgtactact720
gcgccagaggcggcggcgtgttcgactactggggccagggcaccaccctgaccgtgagca780
gcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccc840
tgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggc900
tggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttc960
tcctgtcactggttatcaccctttactgcaggagtaagaggagcaggctcctgcacagtg1020
actacatgaacatgactccccgccgccccgggcccacccgcaagcattaccagccctatg1080
ccccaccacgcgacttcgcagcctatcgctccaaacggggcagaaagaaactcctgtata1140
tattcaaacaaccatttatgagaccagtacaaactactcaagaggaagatggctgtagct1200
gccgatttccagaagaagaagaaggaggatgtgaactgagagtgaagttcagcaggagcg1260
cagacgcccccgcgtacaagcagggccagaaccagctctataacgagctcaatctaggac1320
gaagagaggagtacgatgttttggacaagagacgtggccgggaccctgagatggggggaa1380
agccgagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatgg1440
cggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatg1500
gcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttcacatgcagg1560
ccctgccccctcgcggaagcggagctactaacttcagcctgctgaagcaggctggagacg1620
tggaggagaaccctggacctatggccttaccagtgaccgccttgctcctgccgctggcct1680
tgctgctccacgccgccaggccggacgtgcagatcacccagagccccagctacctggccg1740
ccagccccggcgagaccatcaccatcaactgcagagccagcaagagcatcagcaaggacc1800
tggcctggtaccaggagaagcccggcaagaccaacaagctgctgatctacagcggcagca1860
ccctgcagagcggcatccccagcagattcagcggcagcggcagcggcaccgacttcaccc1920
tgaccatcagcagcctggagcccgaggacttcgccatgtactactgccagcagcacaaca1980
agtacccctacaccttcggcggcggcaccaagctggagatcaagggaggggggggatccg2040
ggggaggaggctccggcggaggcggaagccaggtgcagctgcagcagcccggcgccgagc2100
tggtgagacccggcgccagcgtgaagctgagctgcaaggccagcggctacaccttcacca2160
gctactggatgaactgggtgaagcagagacccgaccagggcctggagtggatcggcagaa2220
tcgacccctacgacagcgagacccactacaaccagaagttcaaggacaaggccatcctga2280
ccgtggacaagagcagcagcaccgcctacatgcagctgagcagcctgaccagcgaggaca2340
gcgccgtgtactactgcgccagaggcaactgggacgactactggggccagggcaccaccc2400
tgaccgtgagcagcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcg2460
cgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgc2520
acacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggactt2580
gtggggtccttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggc2640
tcctgcacagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcatt2700
accagccctatgccccaccacgcgacttcgcagcctatcgctccaaacggggcagaaaga2760
aactcctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagaggaag2820
atggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtgaagt2880
tcagcaggagcgcagacgcccccgcgtacaagcagggccagaaccagctctataacgagc2940
tcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctg3000
agatggggggaaagccgagaaggaagaaccctcaggaaggcctgtacaatgaactgcaga3060
aagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggca3120
aggggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgccc3180
ttcacatgcaggccctgccccctcgctaagtttaaac3217
<210>11
<211>985
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>11
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproglnilevalleuthrglnserproalailemet
202530
seralaserproglyglulysvalthrilethrcysseralaserser
354045
serilesertyrmethistrppheglnglnlysproglythrserpro
505560
lysleutrpiletyrthrthrserasnleualaserglyvalproala
65707580
argpheserglyserglyserglythrsertyrserleuthrileser
859095
argmetglualagluaspalaalathrtyrtyrcyshisglnargser
100105110
thrtyrproleuthrpheglyserglythrlysleugluleulysgly
115120125
glyglyglyserglyglyglyglyserglyglyglyglyserglnval
130135140
glnleuglnglnserglyalagluleualalysproglyalaserval
145150155160
lysmetsercyslysalaserglytyrthrphethrsertyrargmet
165170175
histrpvallysglnargproglyglnglyleuglutrpileglytyr
180185190
ileasnproserthrglytyrthrglutyrasnglnlysphelysasp
195200205
lysalathrleuthralaasplysserserserthralatyrmetgln
210215220
leuserserleuthrphegluaspseralavaltyrtyrcysalaarg
225230235240
glyglyglyvalpheasptyrtrpglyglnglythrthrleuthrval
245250255
serserthrthrthrproalaproargproprothrproalaprothr
260265270
ilealaserglnproleuserleuargproglualacysargproala
275280285
alaglyglyalavalhisthrargglyleuaspphealacysaspile
290295300
tyriletrpalaproleualaglythrcysglyvalleuleuleuser
305310315320
leuvalilethrleutyrcyslysargglyarglyslysleuleutyr
325330335
ilephelysglnprophemetargprovalglnthrthrglngluglu
340345350
aspglycyssercysargpheproglugluglugluglyglycysglu
355360365
leuargvallyspheserargseralaaspalaproalatyrglngln
370375380
glyglnasnglnleutyrasngluleuasnleuglyargarggluglu
385390395400
tyraspvalleuasplysargargglyargaspproglumetglygly
405410415
lysproglnargarglysasnproglngluglyleutyrasngluleu
420425430
glnlysasplysmetalaglualatyrsergluileglymetlysgly
435440445
gluargargargglylysglyhisaspglyleutyrglnglyleuser
450455460
thralathrlysaspthrtyraspalaleuhismetglnalaleupro
465470475480
proargglyserglyalathrasnpheserleuleulysglnalagly
485490495
aspvalglugluasnproglyprometalaleuprovalthralaleu
500505510
leuleuproleualaleuleuleuhisalaalaargproaspvalgln
515520525
ilethrglnserprosertyrleualaalaserproglygluthrile
530535540
thrileasncysargalaserlysserileserlysaspleualatrp
545550555560
tyrglnglulysproglylysthrasnlysleuleuiletyrsergly
565570575
serthrleuglnserglyileproserargpheserglyserglyser
580585590
glythraspphethrleuthrileserserleugluprogluaspphe
595600605
alamettyrtyrcysglnglnhisasnlystyrprotyrthrphegly
610615620
glyglythrlysleugluilelysglyglyglyglyserglyglygly
625630635640
glyserglyglyglyglyserglnvalglnleuglnglnproglyala
645650655
gluleuvalargproglyalaservallysleusercyslysalaser
660665670
glytyrthrphethrsertyrtrpmetasntrpvallysglnargpro
675680685
aspglnglyleuglutrpileglyargileaspprotyraspserglu
690695700
thrhistyrasnglnlysphelysasplysalaileleuthrvalasp
705710715720
lysserserserthralatyrmetglnleuserserleuthrserglu
725730735
aspseralavaltyrtyrcysalaargglyasntrpaspasptyrtrp
740745750
glyglnglythrthrleuthrvalserserthrthrthrproalapro
755760765
argproprothrproalaprothrilealaserglnproleuserleu
770775780
argproglualacysargproalaalaglyglyalavalhisthrarg
785790795800
glyleuaspphealacysaspiletyriletrpalaproleualagly
805810815
thrcysglyvalleuleuleuserleuvalilethrleutyrcysarg
820825830
serlysargserargleuleuhisserasptyrmetasnmetthrpro
835840845
argargproglyprothrarglyshistyrglnprotyralapropro
850855860
argaspphealaalatyrargserargvallyspheserargserala
865870875880
aspalaproalatyrglnglnglyglnasnglnleutyrasngluleu
885890895
asnleuglyargarggluglutyraspvalleuasplysargarggly
900905910
argaspproglumetglyglylysproglnargarglysasnprogln
915920925
gluglyleutyrasngluleuglnlysasplysmetalaglualatyr
930935940
sergluileglymetlysglygluargargargglylysglyhisasp
945950955960
glyleutyrglnglyleuserthralathrlysaspthrtyraspala
965970975
leuhismetglnalaleuproproarg
980985
<210>12
<211>2974
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>12
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccgcagatcgtgctgacccagagccccgccatcatgagcgccagccccggcg120
agaaggtgaccatcacctgcagcgccagcagcagcatcagctacatgcactggttccagc180
agaagcccggcaccagccccaagctgtggatctacaccaccagcaacctggccagcggcg240
tgcccgccagattcagcggcagcggcagcggcaccagctacagcctgaccatcagcagaa300
tggaggccgaggacgccgccacctactactgccaccagagaagcacctaccccctgacct360
tcggcagcggcaccaagctggagctgaagggaggggggggatccgggggaggaggctccg420
gcggaggcggaagccaggtgcagctgcagcagagcggcgccgagctggccaagcccggcg480
ccagcgtgaagatgagctgcaaggccagcggctacaccttcaccagctacagaatgcact540
gggtgaagcagagacccggccagggcctggagtggatcggctacatcaaccccagcaccg600
gctacaccgagtacaaccagaagttcaaggacaaggccaccctgaccgccgacaagagca660
gcagcaccgcctacatgcagctgagcagcctgaccttcgaggacagcgccgtgtactact720
gcgccagaggcggcggcgtgttcgactactggggccagggcaccaccctgaccgtgagca780
gcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccc840
tgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggc900
tggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttc960
tcctgtcactggttatcaccctttactgcaaacggggcagaaagaaactcctgtatatat1020
tcaaacaaccatttatgagaccagtacaaactactcaagaggaagatggctgtagctgcc1080
gatttccagaagaagaagaaggaggatgtgaactgagagtgaagttcagcaggagcgcag1140
acgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaa1200
gagaggagtacgatgttttggacaagagacgtggccgggaccctgagatggggggaaagc1260
cgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatgg1320
cggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatg1380
gcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttcacatgcagg1440
ccctgccccctcgcggaagcggagctactaacttcagcctgctgaagcaggctggagacg1500
tggaggagaaccctggacctatggccttaccagtgaccgccttgctcctgccgctggcct1560
tgctgctccacgccgccaggccggacgtgcagatcacccagagccccagctacctggccg1620
ccagccccggcgagaccatcaccatcaactgcagagccagcaagagcatcagcaaggacc1680
tggcctggtaccaggagaagcccggcaagaccaacaagctgctgatctacagcggcagca1740
ccctgcagagcggcatccccagcagattcagcggcagcggcagcggcaccgacttcaccc1800
tgaccatcagcagcctggagcccgaggacttcgccatgtactactgccagcagcacaaca1860
agtacccctacaccttcggcggcggcaccaagctggagatcaagggaggggggggatccg1920
ggggaggaggctccggcggaggcggaagccaggtgcagctgcagcagcccggcgccgagc1980
tggtgagacccggcgccagcgtgaagctgagctgcaaggccagcggctacaccttcacca2040
gctactggatgaactgggtgaagcagagacccgaccagggcctggagtggatcggcagaa2100
tcgacccctacgacagcgagacccactacaaccagaagttcaaggacaaggccatcctga2160
ccgtggacaagagcagcagcaccgcctacatgcagctgagcagcctgaccagcgaggaca2220
gcgccgtgtactactgcgccagaggcaactgggacgactactggggccagggcaccaccc2280
tgaccgtgagcagcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcg2340
cgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgc2400
acacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggactt2460
gtggggtccttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggc2520
tcctgcacagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcatt2580
accagccctatgccccaccacgcgacttcgcagcctatcgctccagagtgaagttcagca2640
ggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatc2700
taggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctgagatgg2760
ggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaag2820
ataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaagg2880
ggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttc2940
acatgcaggccctgccccctcgctaagtttaaac2974
<210>13
<211>546
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>13
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproseraspilevalleuthrglnserproalaser
202530
leualavalserleuglyglnargalathrilesercysargalaser
354045
lysservalserthrserglytyrsertyrleuhistrptyrglngln
505560
lysproglyglnproprolysleuleuiletyrleualaserasnleu
65707580
gluserglyvalproalaargpheserglyserglyserglythrasp
859095
phethrleuasnilehisprovalgluglugluaspalaalathrtyr
100105110
tyrcysglnhisserarggluleuprophethrpheglyserglythr
115120125
lysleugluilelyslysileserglyglyglyglyserglyglygly
130135140
glyserglyglyglyglyserglyglyglyglyserglyglyglygly
145150155160
serglnvalglnleuvalgluserglyglyglyleuvalglnprogly
165170175
glyserleulysleusercysalaalaserglypheasppheserarg
180185190
tyrtrpmetsertrpvalargglnalaproglylysglyleuglutrp
195200205
ileglygluileasnprothrserserthrileasnphethrproser
210215220
leulysasplysvalpheileserargaspasnalalysasnthrleu
225230235240
tyrleuglnmetserlysvalargsergluaspthralaleutyrtyr
245250255
cysalaargglyasntyrtyrargtyrglyaspalametasptyrtrp
260265270
glyglnglythrservalthrvalserthrthrthrproalaproarg
275280285
proprothrproalaprothrilealaserglnproleuserleuarg
290295300
proglualacysargproalaalaglyglyalavalhisthrarggly
305310315320
leuaspphealacysaspiletyriletrpalaproleualaglythr
325330335
cysglyvalleuleuleuserleuvalilethrleutyrcysargser
340345350
lysargserargleuleuhisserasptyrmetasnmetthrproarg
355360365
argproglyprothrarglyshistyrglnprotyralaproproarg
370375380
aspphealaalatyrargserlysargglyarglyslysleuleutyr
385390395400
ilephelysglnprophemetargprovalglnthrthrglngluglu
405410415
aspglycyssercysargpheproglugluglugluglyglycysglu
420425430
leuargvallyspheserargseralaaspalaproalatyrglngln
435440445
glyglnasnglnleutyrasngluleuasnleuglyargarggluglu
450455460
tyraspvalleuasplysargargglyargaspproglumetglygly
465470475480
lysproglnargarglysasnproglngluglyleutyrasngluleu
485490495
glnlysasplysmetalaglualatyrsergluileglymetlysgly
500505510
gluargargargglylysglyhisaspglyleutyrglnglyleuser
515520525
thralathrlysaspthrtyraspalaleuhismetglnalaleupro
530535540
proarg
545
<210>14
<211>1657
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>14
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccgagcgacatcgtgctgacccagagccccgccagcctggccgtgagcctgg120
gccagagagccaccatcagctgcagagccagcaagagcgtgagcaccagcggctacagct180
acctgcactggtaccagcagaagcccggccagccccccaagctgctgatctacctggcca240
gcaacctggagagcggcgtgcccgccagattcagcggcagcggcagcggcaccgacttca300
ccctgaacatccaccccgtggaggaggaggacgccgccacctactactgccagcacagca360
gagagctgcccttcaccttcggcagcggcaccaagctggagatcaagaagatcagcggcg420
gcggcggcagcggcggcggcggcagcggcggcggcggcagcggcggcggcggcagcggcg480
gcggcggcagccaggtgcagctggtggagagcggcggcggcctggtgcagcccggcggca540
gcctgaagctgagctgcgccgccagcggcttcgacttcagcagatactggatgagctggg600
tgagacaggcccccggcaagggcctggagtggatcggcgagatcaaccccaccagcagca660
ccatcaacttcacccccagcctgaaggacaaggtgttcatcagcagagacaacgccaaga720
acaccctgtacctgcagatgagcaaggtgagaagcgaggacaccgccctgtactactgcg780
ccagaggcaactactacagatacggcgacgccatggactactggggccagggcaccagcg840
tgaccgtgagcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgt900
cgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcaca960
cgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtg1020
gggtccttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggctcc1080
tgcacagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcattacc1140
agccctatgccccaccacgcgacttcgcagcctatcgctccaaacggggcagaaagaaac1200
tcctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagaggaagatg1260
gctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtgaagttca1320
gcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagctca1380
atctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctgaga1440
tggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcaga1500
aagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggca1560
aggggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgccc1620
ttcacatgcaggccctgccccctcgctaagtttaaac1657
<210>15
<211>490
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>15
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalleuthrglnserproalaserleu
202530
alavalserleuglyglnargalathrilesercysargalaserlys
354045
servalserthrserglytyrsertyrleuhistrptyrglnglnlys
505560
proglyglnproprolysleuleuiletyrleualaserasnleuglu
65707580
serglyvalproalaargpheserglyserglyserglythraspphe
859095
thrleuasnilehisprovalgluglugluaspalaalathrtyrtyr
100105110
cysglnhisserarggluleuprophethrpheglyserglythrlys
115120125
leugluilelysglyglyglyglyserglyglyglyglyserglygly
130135140
glyglyserglnvalglnleuvalgluserglyglyglyleuvalgln
145150155160
proglyglyserleulysleusercysalaalaserglypheaspphe
165170175
serargtyrtrpmetsertrpvalargglnalaproglylysglyleu
180185190
glutrpileglygluileasnprothrserserthrileasnphethr
195200205
proserleulysasplysvalpheileserargaspasnalalysasn
210215220
thrleutyrleuglnmetserlysvalargsergluaspthralaleu
225230235240
tyrtyrcysalaargglyasntyrtyrargtyrglyaspalametasp
245250255
tyrtrpglyglnglythrservalthrvalserthrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
argserlysargserargleuleuhisserasptyrmetasnmetthr
340345350
proargargproglyprothrarglyshistyrglnprotyralapro
355360365
proargaspphealaalatyrargserargvallyspheserargser
370375380
alaaspalaproalatyrglnglnglyglnasnglnleutyrasnglu
385390395400
leuasnleuglyargarggluglutyraspvalleuasplysargarg
405410415
glyargaspproglumetglyglylysproglnargarglysasnpro
420425430
glngluglyleutyrasngluleuglnlysasplysmetalagluala
435440445
tyrsergluileglymetlysglygluargargargglylysglyhis
450455460
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
465470475480
alaleuhismetglnalaleuproproarg
485490
<210>16
<211>1489
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>16
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgctgacccagagccccgccagcctggccgtgagcctgggcc120
agagggccaccatcagctgcagggccagcaagagcgtgagcaccagcggctacagctacc180
tgcactggtaccagcagaagcccggccagccccccaagctgctgatctacctggccagca240
acctggagagcggcgtgcccgccaggttcagcggcagcggcagcggcaccgacttcaccc300
tgaacatccaccccgtggaggaggaggacgccgccacctactactgccagcacagcaggg360
agctgcccttcaccttcggcagcggcaccaagctggagatcaagggaggggggggatccg420
ggggaggaggctccggcggaggcggaagccaggtgcagctggtggagagcggcggcggcc480
tggtgcagcccggcggcagcctgaagctgagctgcgccgccagcggcttcgacttcagca540
ggtactggatgagctgggtgaggcaggcccccggcaagggcctggagtggatcggcgaga600
tcaaccccaccagcagcaccatcaacttcacccccagcctgaaggacaaggtgttcatca660
gcagggacaacgccaagaacaccctgtacctgcagatgagcaaggtgaggagcgaggaca720
ccgccctgtactactgcgccaggggcaactactacaggtacggcgacgccatggactact780
ggggccagggcaccagcgtgaccgtgagcaccacgacgccagcgccgcgaccaccaacac840
cggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcgg900
cggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgc960
ccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcagga1020
gtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggc1080
ccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctcca1140
gagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctct1200
ataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggcc1260
gggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtaca1320
atgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagc1380
gccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggaca1440
cctacgacgcccttcacatgcaggccctgccccctcgctaagtttaaac1489
<210>17
<211>491
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>17
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalleuthrglnserproalaserleu
202530
alavalserleuglyglnargalathrilesercysargalaserlys
354045
servalserthrserglytyrsertyrleuhistrptyrglnglnlys
505560
proglyglnproprolysleuleuiletyrleualaserasnleuglu
65707580
serglyvalproalaargpheserglyserglyserglythraspphe
859095
thrleuasnilehisprovalgluglugluaspalaalathrtyrtyr
100105110
cysglnhisserarggluleuprophethrpheglyserglythrlys
115120125
leugluilelysglyglyglyglyserglyglyglyglyserglygly
130135140
glyglyserglnvalglnleuvalgluserglyglyglyleuvalgln
145150155160
proglyglyserleulysleusercysalaalaserglypheaspphe
165170175
serargtyrtrpmetsertrpvalargglnalaproglylysglyleu
180185190
glutrpileglygluileasnprothrserserthrileasnphethr
195200205
proserleulysasplysvalpheileserargaspasnalalysasn
210215220
thrleutyrleuglnmetserlysvalargsergluaspthralaleu
225230235240
tyrtyrcysalaargglyasntyrtyrargtyrglyaspalametasp
245250255
tyrtrpglyglnglythrservalthrvalserthrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
lysargglyarglyslysleuleutyrilephelysglnprophemet
340345350
argprovalglnthrthrglnglugluaspglycyssercysargphe
355360365
proglugluglugluglyglycysgluleuargvallyspheserarg
370375380
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
385390395400
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
405410415
argglyargaspproglumetglyglylysproglnargarglysasn
420425430
proglngluglyleutyrasngluleuglnlysasplysmetalaglu
435440445
alatyrsergluileglymetlysglygluargargargglylysgly
450455460
hisaspglyleutyrglnglyleuserthralathrlysaspthrtyr
465470475480
aspalaleuhismetglnalaleuproproarg
485490
<210>18
<211>1492
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>18
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgctgacccagagccccgccagcctggccgtgagcctgggcc120
agagggccaccatcagctgcagggccagcaagagcgtgagcaccagcggctacagctacc180
tgcactggtaccagcagaagcccggccagccccccaagctgctgatctacctggccagca240
acctggagagcggcgtgcccgccaggttcagcggcagcggcagcggcaccgacttcaccc300
tgaacatccaccccgtggaggaggaggacgccgccacctactactgccagcacagcaggg360
agctgcccttcaccttcggcagcggcaccaagctggagatcaagggaggggggggatccg420
ggggaggaggctccggcggaggcggaagccaggtgcagctggtggagagcggcggcggcc480
tggtgcagcccggcggcagcctgaagctgagctgcgccgccagcggcttcgacttcagca540
ggtactggatgagctgggtgaggcaggcccccggcaagggcctggagtggatcggcgaga600
tcaaccccaccagcagcaccatcaacttcacccccagcctgaaggacaaggtgttcatca660
gcagggacaacgccaagaacaccctgtacctgcagatgagcaaggtgaggagcgaggaca720
ccgccctgtactactgcgccaggggcaactactacaggtacggcgacgccatggactact780
ggggccagggcaccagcgtgaccgtgagcaccacgacgccagcgccgcgaccaccaacac840
cggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcgg900
cggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgc960
ccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcaaac1020
ggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccagtacaaacta1080
ctcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtgaac1140
tgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagc1200
tctataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtg1260
gccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgt1320
acaatgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcg1380
agcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaagg1440
acacctacgacgcccttcacatgcaggccctgccccctcgctaagtttaaac1492
<210>19
<211>997
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>19
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspvalvalmetthrglnserhisargphemet
202530
serthrservalglyaspargvalserilethrcysargalasergln
354045
aspvalasnthralavalsertrptyrglnglnlysproglyglnser
505560
prolysleuleuilepheseralasertyrargtyrthrglyvalpro
65707580
aspargphethrglyserglyserglyalaaspphethrleuthrile
859095
serservalglnalagluaspleualavaltyrtyrcysglnglnhis
100105110
tyrserthrprotrpthrpheglyglyglythrlysleugluilelys
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglysergln
130135140
ileglnleuvalglnserglyproaspleulyslysproglygluthr
145150155160
vallysleusercyslysalaserglytyrthrphethrasnphegly
165170175
metasntrpvallysglnalaproglylysglyphelystrpmetala
180185190
trpileasnthrthrargtyrthrglyglusertyrphealaaspasp
195200205
phelysglyargphealapheservalgluthrseralathrthrala
210215220
tyrleuglnileasnasnleulysthrgluaspthralathrtyrphe
225230235240
cysalaargglygluiletyrtyrglytyraspglyglyphealatyr
245250255
trpglyglnglythrleuvalthrvalseralathrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
lysargglyarglyslysleuleutyrilephelysglnprophemet
340345350
argprovalglnthrthrglnglugluaspglycyssercysargphe
355360365
proglugluglugluglyglycysgluleuargvallyspheserarg
370375380
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
385390395400
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
405410415
argglyargaspproglumetglyglylysproglnargarglysasn
420425430
proglngluglyleutyrasngluleuglnlysasplysmetalaglu
435440445
alatyrsergluileglymetlysglygluargargargglylysgly
450455460
hisaspglyleutyrglnglyleuserthralathrlysaspthrtyr
465470475480
aspalaleuhismetglnalaleuproproargglyserglyalathr
485490495
asnpheserleuleulysglnalaglyaspvalglugluasnprogly
500505510
proalaleuprovalthralaleuleuleuproleualaleuleuleu
515520525
hisalaalaargproaspileglnmetthrglnserproserserleu
530535540
seralaservalglyaspargvalthrilethrcyslysalasergln
545550555560
aspvalglyilealavalalatrptyrglnglnlysproglylysval
565570575
prolysleuleuiletyrtrpalaserthrarghisthrglyvalpro
580585590
aspargpheserglyserglyserglythraspphethrleuthrile
595600605
serserleuglnprogluaspvalalathrtyrtyrcysglnglntyr
610615620
sersertyrprotyrthrpheglyglnglythrlysvalgluilelys
625630635640
glyglyglyglyserglyglyglyglyserglyglyglyglyserglu
645650655
valglnleuvalgluserglyglyglyleuvalglnproglyglyser
660665670
leuargleusercysalaalaserglypheasppheserargtyrtrp
675680685
metsertrpvalargglnalaproglylysglyleuglutrpilegly
690695700
gluileasnproaspserserthrileasntyralaproserleulys
705710715720
asplyspheileileserargaspasnalalysasnserleutyrleu
725730735
glnmetasnserleuargalagluaspthralavaltyrtyrcysala
740745750
argproaspglyasntyrtrptyrpheaspvaltrpglyglnglythr
755760765
leuvalthrvalserserthrthrthrproalaproargproprothr
770775780
proalaprothrilealaserglnproleuserleuargprogluala
785790795800
cysargproalaalaglyglyalavalhisthrargglyleuaspphe
805810815
alacysaspiletyriletrpalaproleualaglythrcysglyval
820825830
leuleuleuserleuvalilethrleutyrcysargserlysargser
835840845
argleuleuhisserasptyrmetasnmetthrproargargprogly
850855860
prothrarglyshistyrglnprotyralaproproargasppheala
865870875880
alatyrargserargvallyspheserargseralaaspalaproala
885890895
tyrglnglnglyglnasnglnleutyrasngluleuasnleuglyarg
900905910
arggluglutyraspvalleuasplysargargglyargaspproglu
915920925
metglyglylysproglnargarglysasnproglngluglyleutyr
930935940
asngluleuglnlysasplysmetalaglualatyrsergluilegly
945950955960
metlysglygluargargargglylysglyhisaspglyleutyrgln
965970975
glyleuserthralathrlysaspthrtyraspalaleuhismetgln
980985990
alaleuproproarg
995
<210>20
<211>2994
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>20
atggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacgccgccagg60
ccggatgtggtgatgacccagagccatcgctttatgagcaccagcgtgggcgatcgcgtg120
agcattacctgccgcgcgagccaggatgtgaacaccgcggtgagctggtatcagcagaaa180
ccgggccagagcccgaaactgctgatttttagcgcgagctatcgctataccggcgtgccg240
gatcgctttaccggcagcggcagcggcgcggattttaccctgaccattagcagcgtgcag300
gcggaagatctggcggtgtattattgccagcagcattatagcaccccgtggacctttggc360
ggcggcaccaaactggaaattaaaggaggggggggatccgggggaggaggctccggcgga420
ggcggaagccagattcagctggtgcagagcggcccggatctgaaaaaaccgggcgaaacc480
gtgaaactgagctgcaaagcgagcggctatacctttaccaactttggcatgaactgggtg540
aaacaggcgccgggcaaaggctttaaatggatggcgtggattaacaccacccgctatacc600
ggcgaaagctattttgcggatgattttaaaggccgctttgcgtttagcgtggaaaccagc660
gcgaccaccgcgtatctgcagattaacaacctgaaaaccgaagataccgcgacctatttt720
tgcgcgcgcggcgaaatttattatggctatgatggcggctttgcgtattggggccagggc780
accctggtgaccgtgagcgcgaccacgacgccagcgccgcgaccaccaacaccggcgccc840
accatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggc900
gcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggcc960
gggacttgtggggtccttctcctgtcactggttatcaccctttactgcaaacggggcaga1020
aagaaactcctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagag1080
gaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtg1140
aagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataac1200
gagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggac1260
cctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaa1320
ctgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccgg1380
aggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacacctac1440
gacgcccttcacatgcaggccctgccccctcgcggaagcggagccaccaacttcagcctg1500
ctgaagcaggccggcgacgtggaggagaaccccggccccgccttaccagtgaccgccttg1560
ctcctgccgctggccttgctgctccacgccgccaggccggatattcagatgacccagagc1620
ccgagcagcctgagcgcgagcgtgggcgatcgcgtgaccattacctgcaaagcgagccag1680
gatgtgggcattgcggtggcgtggtatcagcagaaaccgggcaaagtgccgaaactgctg1740
atttattgggcgagcacccgccataccggcgtgccggatcgctttagcggcagcggcagc1800
ggcaccgattttaccctgaccattagcagcctgcagccggaagatgtggcgacctattat1860
tgccagcagtatagcagctatccgtatacctttggccagggcaccaaagtggaaattaaa1920
ggaggggggggatccgggggaggaggctccggcggaggcggaagcgaagtgcagctggtg1980
gaaagcggcggcggcctggtgcagccgggcggcagcctgcgcctgagctgcgcggcgagc2040
ggctttgattttagccgctattggatgagctgggtgcgccaggcgccgggcaaaggcctg2100
gaatggattggcgaaattaacccggatagcagcaccattaactatgcgccgagcctgaaa2160
gataaatttattattagccgcgataacgcgaaaaacagcctgtatctgcagatgaacagc2220
ctgcgcgcggaagataccgcggtgtattattgcgcgcgcccggatggcaactattggtat2280
tttgatgtgtggggccagggcaccctggtgaccgtgagcagcaccacgacgccagcgccg2340
cgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcg2400
tgccggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatc2460
tacatctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacc2520
ctttactgcaggagtaagaggagcaggctcctgcacagtgactacatgaacatgactccc2580
cgccgccccgggcccacccgcaagcattaccagccctatgccccaccacgcgacttcgca2640
gcctatcgctccagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggc2700
cagaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttggac2760
aagagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcag2820
gaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattggg2880
atgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtaca2940
gccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgctaa2994
<210>21
<211>997
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>21
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspvalvalmetthrglnserhisargphemet
202530
serthrservalglyaspargvalserilethrcysargalasergln
354045
aspvalasnthralavalsertrptyrglnglnlysproglyglnser
505560
prolysleuleuilepheseralasertyrargtyrthrglyvalpro
65707580
aspargphethrglyserglyserglyalaaspphethrleuthrile
859095
serservalglnalagluaspleualavaltyrtyrcysglnglnhis
100105110
tyrserthrprotrpthrpheglyglyglythrlysleuaspilelys
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglysergln
130135140
ileglnleuvalglnserglyproaspleulyslysproglygluthr
145150155160
vallysleusercyslysalaserglytyrthrphethrasnphegly
165170175
metasntrpvallysglnalaproglylysglyphelystrpmetala
180185190
trpileasnthrtyrthrglyglusertyrphealaaspaspphelys
195200205
glyargphealapheservalgluthrseralathrthralatyrleu
210215220
glnileasnasnleulysthrgluaspthralathrtyrphecysala
225230235240
argglygluiletyrtyrglytyraspglyglyphealatyrtrpgly
245250255
glnglythrleuvalthrvalseralathrthrthrproalaproarg
260265270
proprothrproalaprothrilealaserglnproleuserleuarg
275280285
proglualacysargproalaalaglyglyalavalhisthrarggly
290295300
leuaspphealacysaspiletyriletrpalaproleualaglythr
305310315320
cysglyvalleuleuleuserleuvalilethrleutyrcyslysarg
325330335
glyarglyslysleuleutyrilephelysglnprophemetargpro
340345350
valglnthrthrglnglugluaspglycyssercysargpheproglu
355360365
gluglugluglyglycysgluleuargvallyspheserargserala
370375380
aspalaproalatyrglnglnglyglnasnglnleutyrasngluleu
385390395400
asnleuglyargarggluglutyraspvalleuasplysargarggly
405410415
argaspproglumetglyglylysproglnargarglysasnprogln
420425430
gluglyleutyrasngluleuglnlysasplysmetalaglualatyr
435440445
sergluileglymetlysglygluargargargglylysglyhisasp
450455460
glyleutyrglnglyleuserthralathrlysaspthrtyraspala
465470475480
leuhismetglnalaleuproproargglyserglyalathrasnphe
485490495
serleuleulysglnalaglyaspvalglugluasnproglypromet
500505510
alaleuprovalthralaleuleuleuproleualaleuleuleuhis
515520525
alaalaargproaspileglnmetthrglnserproserserleuser
530535540
alaservalglyaspargvalthrilethrcyslysalaserglnasp
545550555560
valglyilealavalalatrptyrglnglnlysproglylysvalpro
565570575
lysleuleuiletyrtrpalaserthrarghisthrglyvalproasp
580585590
argpheserglyserglyserglythraspphethrleuthrileser
595600605
serleuglnprogluaspvalalathrtyrtyrcysglnglntyrser
610615620
sertyrprotyrthrpheglyglnglythrlysvalgluilelysgly
625630635640
glyglyglyserglyglyglyglyserglyglyglyglysergluval
645650655
glnleuvalgluserglyglyglyleuvalglnproglyglyserleu
660665670
argleusercysalaalaserglypheasppheserargtyrtrpmet
675680685
sertrpvalargglnalaproglylysglyleuglutrpileglyglu
690695700
ileasnproaspserserthrileasntyralaproserleulysasp
705710715720
lyspheileileserargaspasnalalysasnserleutyrleugln
725730735
metasnserleuargalagluaspthralavaltyrtyrcysalaarg
740745750
proaspglyasntyrtrptyrpheaspvaltrpglyglnglythrleu
755760765
valthrvalserserthrthrthrproalaproargproprothrpro
770775780
alaprothrilealaserglnproleuserleuargproglualacys
785790795800
argproalaalaglyglyalavalhisthrargglyleuasppheala
805810815
cysaspiletyriletrpalaproleualaglythrcysglyvalleu
820825830
leuleuserleuvalilethrleutyrcyslysargglyarglyslys
835840845
leuleutyrilephelysglnprophemetargprovalglnthrthr
850855860
glnglugluaspglycyssercysargpheprogluglugluglugly
865870875880
glycysgluleuargvallyspheserargseralaaspalaproala
885890895
tyrglnglnglyglnasnglnleutyrasngluleuasnleuglyarg
900905910
arggluglutyraspvalleuasplysargargglyargaspproglu
915920925
metglyglylysproglnargarglysasnproglngluglyleutyr
930935940
asngluleuglnlysasplysmetalaglualatyrsergluilegly
945950955960
metlysglygluargargargglylysglyhisaspglyleutyrgln
965970975
glyleuserthralathrlysaspthrtyraspalaleuhismetgln
980985990
alaleuproproarg
995
<210>22
<211>3009
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>22
cgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacgc60
cgccaggccggatgtggtgatgacccagagccatcgctttatgagcaccagcgtgggaga120
tcgagtgagcattacctgccgcgcgagccaggatgtgaacaccgcggtgagctggtatca180
gcagaaaccgggccagagcccgaaactgctgatttttagcgcgagctatcgctataccgg240
cgtgccggatcgctttaccggcagcggcagcggcgcggattttaccctgaccattagcag300
cgtgcaggcggaagatctggcggtgtattattgccagcagcattatagcaccccgtggac360
ctttggcggcggcaccaaactggatattaaaggaggggggggatccgggggaggaggctc420
cggcggaggcggaagccagattcagctggtgcagagcggcccggatctgaaaaaaccggg480
cgaaaccgtgaaactgagctgcaaagcgagcggctatacctttaccaactttggcatgaa540
ctgggtgaaacaggcgccgggcaaaggctttaaatggatggcgtggattaacacctatac600
cggcgaaagctattttgcggatgattttaaaggccgctttgcgtttagcgtggaaaccag660
cgcgaccaccgcgtatctgcagattaacaacctgaaaaccgaagataccgcgacctattt720
ttgcgcgcgcggcgaaatttattatggctatgatggcggctttgcgtattggggccaggg780
caccctggtgaccgtgagcgcgaccacgacgccagcgccgcgaccaccaacaccggcgcc840
caccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcgggggg900
cgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggc960
cgggacttgtggggtccttctcctgtcactggttatcaccctttactgcaaacggggcag1020
aaagaaactcctgtatatattcaagcaaccatttatgagaccagtacaaactactcaaga1080
ggaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagt1140
gaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataa1200
cgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccggga1260
ccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatga1320
actgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccg1380
gaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacaccta1440
cgacgcccttcacatgcaggccctgccccctcgcggaagcggagccaccaacttcagcct1500
gctgaagcaggccggcgacgtggaggagaaccccggccccatggccttaccagtgaccgc1560
cttgctcctgccgctggccttgctgctccacgccgccaggccggatattcagatgaccca1620
gagcccgagcagcctgagcgcgagcgtgggcgaccgcgtgaccattacctgcaaagcgag1680
ccaggatgtgggcattgcggtggcgtggtatcagcagaaaccgggcaaagtgccgaaact1740
gctgatttattgggcgagcacccgccataccggcgtgccggatcgctttagcggcagcgg1800
cagcggcaccgattttaccctgaccattagcagcctgcagccggaagatgtggcgaccta1860
ttattgccagcagtatagcagctatccgtatacctttggccagggcaccaaagtggaaat1920
taaaggaggggggggatccgggggaggaggctccggcggaggcggaagcgaagtgcagct1980
ggtggaaagcggcggcggcctggtgcagccgggcggcagcctgcgcctgagctgcgcggc2040
gagcggctttgattttagccgctattggatgagctgggtgcgccaggcgccgggcaaagg2100
cctggaatggattggcgaaattaacccggatagcagcaccattaactatgcgccgagcct2160
gaaagataaatttattattagccgcgataacgcgaaaaacagcctgtatctgcagatgaa2220
cagcctgcgcgcggaagataccgcggtgtattattgcgcgcgcccggatggcaactattg2280
gtattttgatgtgtggggccagggcaccctggtgaccgtgagcagcaccacgacgccagc2340
gccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccaga2400
ggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtga2460
tatctacatctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttat2520
caccctttactgcaaacggggcagaaagaaactcctgtatatattcaagcaaccatttat2580
gagaccagtacaaactactcaagaggaagatggctgtagctgccgatttccagaagaaga2640
agaaggaggatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtacca2700
gcagggccagaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgt2760
tttggacaagagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaa2820
ccctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtga2880
gattgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtct2940
cagtacagccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcta3000
agtttaaac3009
<210>23
<211>423
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>23
agctagctgcagtaacgccattttgcaaggcatggaaaaataccaaaccaagaatagaga60
agttcagatcaagggcgggtacatgaaaatagctaacgttgggccaaacaggatatctgc120
ggtgagcagtttcggccccggcccggggccaagaacagatggtcaccgcagtttcggccc180
cggcccgaggccaagaacagatggtccccagatatggcccaaccctcagcagtttcttaa240
gacccatcagatgtttccaggctcccccaaggacctgaaatgaccctgcgccttatttga300
attaaccaatcagcctgcttctcgcttctgttcgcgcgcttctgcttcccgagctctata360
aaagagctcacaacccctcactcggcgcgccagtcctccgacagactgagtcgcccgggt420
acc423
<210>24
<211>165
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>24
metserglyleuglyargserargargglyglyargserargvalasp
151015
glnglugluargpheproglnglyleutrpthrglyvalalametarg
202530
sercysproglugluglntyrtrpaspproleuleuglythrcysmet
354045
sercyslysthrilecysasnhisglnserglnargthrcysalaala
505560
phecysargserleusercysarglysgluglnglylysphetyrasp
65707580
hisleuleuargaspcysilesercysalaserilecysglyglnhis
859095
prolysglncysalatyrphecysgluasnlysleuargserproval
100105110
asnleuproprogluleuargargglnargserglygluvalgluasn
115120125
asnseraspasnserglyargtyrglnglyleugluhisargglyser
130135140
glualaserproalaleuproglyleulysleuseralaaspglnval
145150155160
alaleuvaltyrser
165
<210>25
<211>54
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>25
metleuglnmetalaglyglncysserglnasnglutyrpheaspser
151015
leuleuhisalacysileprocysglnleuargcysserserasnthr
202530
proproleuthrcysglnargtyrcysasnalaservalthrasnser
354045
vallysglythrasnala
50
<210>26
<211>204
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>26
serglyprovallysgluleuvalglyservalglyglyalavalthr
151015
pheproleulysserlysvallysglnvalaspserilevaltrpthr
202530
pheasnthrthrproleuvalthrileglnprogluglyglythrile
354045
ilevalthrglnasnargasnarggluargvalasppheproaspgly
505560
glytyrserleulysleuserlysleulyslysasnaspserglyile
65707580
tyrtyrvalglyiletyrserserserleuglnglnproserthrgln
859095
glutyrvalleuhisvaltyrgluhisleuserlysprolysvalthr
100105110
metglyleuglnserasnlysasnglythrcysvalthrasnleuthr
115120125
cyscysmetgluhisglyglugluaspvaliletyrthrtrplysala
130135140
leuglyglnalaalaasngluserhisasnglyserileleuproile
145150155160
sertrpargtrpglygluseraspmetthrpheilecysvalalaarg
165170175
asnprovalserargasnpheserserproileleualaarglysleu
180185190
cysgluglyalaalaaspaspproaspsersermet
195200
<210>27
<211>46
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>27
metaspaspserthrgluarggluglnserargleuthrsercysleu
151015
lyslysarggluglumetlysleulysglucysvalserileleupro
202530
arglysgluserproservalargserserlysaspglylys
354045
<210>28
<211>242
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>28
aspproasnphetrpleuglnvalglngluservalthrvalglnglu
151015
glyleucysvalleuvalprocysthrphephehisproileprotyr
202530
tyrasplysasnserprovalhisglytyrtrpphearggluglyala
354045
ileileserargaspserprovalalathrasnlysleuaspglnglu
505560
valglnglugluthrglnglyargpheargleuleuglyaspproser
65707580
argasnasncysserleuserilevalaspalaargargargaspasn
859095
glysertyrphepheargmetgluargglyserthrlystyrsertyr
100105110
lysserproglnleuservalhisvalthraspleuthrhisargpro
115120125
lysileleuileproglythrleugluproglyhisserlysasnleu
130135140
thrcysservalsertrpalacysgluglnglythrproproilephe
145150155160
sertrpleuseralaalaprothrserleuglyproargthrthrhis
165170175
serservalleuileilethrproargproglnasphisglythrasn
180185190
leuthrcysglnvallysphealaglyalaglyvalthrthrgluarg
195200205
thrileglnleuasnvalthrtyrvalproglnasnprothrthrgly
210215220
ilepheproglyaspglyserglylysglngluthrargalaglyval
225230235240
valhis
<210>29
<211>287
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>29
thrlysgluaspproasnproproilethrasnleuargmetlysala
151015
lysalaglnglnleuthrtrpaspleuasnargasnvalthraspile
202530
glucysvallysaspalaasptyrsermetproalavalasnasnser
354045
tyrcysglnpheglyalaileserleucysgluvalthrasntyrthr
505560
valargvalalaasnpropropheserthrtrpileleupheproglu
65707580
asnserglylysprotrpalaglyalagluasnleuthrcystrpile
859095
hisaspvalasppheleusercyssertrpalavalglyproglyala
100105110
proalaaspvalglntyraspleutyrleuasnvalalaasnargarg
115120125
glnglntyrglucysleuhistyrlysthraspalaglnglythrarg
130135140
ileglycysargpheaspaspileserargleuserserglysergln
145150155160
serserhisileleuvalargglyargseralaalapheglyilepro
165170175
cysthrasplysphevalvalpheserglnilegluileleuthrpro
180185190
proasnmetthralalyscysasnlysthrhisserphemethistrp
195200205
lysmetargserhispheasnarglyspheargtyrgluleuglnile
210215220
glnlysargmetglnprovalilethrgluglnvalargaspargthr
225230235240
serpheglnleuleuasnproglythrtyrthrvalglnileargala
245250255
arggluargvaltyrglupheleuseralatrpserthrproglnarg
260265270
pheglucysaspglnglugluglyalaasnthrargalatrparg
275280285
<210>30
<211>272
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>30
proglugluproleuvalvallysvalglugluglyaspasnalaval
151015
leuglncysleulysglythrseraspglyprothrglnglnleuthr
202530
trpserarggluserproleulyspropheleulysleuserleugly
354045
leuproglyleuglyilehismetargproleualailetrpleuphe
505560
ilepheasnvalserglnglnmetglyglyphetyrleucysglnpro
65707580
glyproproserglulysalatrpglnproglytrpthrvalasnval
859095
gluglyserglygluleupheargtrpasnvalseraspleuglygly
100105110
leuglycysglyleulysasnargsersergluglyproserserpro
115120125
serglylysleumetserprolysleutyrvaltrpalalysasparg
130135140
progluiletrpgluglygluproprocysleuproproargaspser
145150155160
leuasnglnserleuserglnaspleuthrmetalaproglyserthr
165170175
leutrpleusercysglyvalproproaspservalserargglypro
180185190
leusertrpthrhisvalhisprolysglyprolysserleuleuser
195200205
leugluleulysaspaspargproalaargaspmettrpvalmetglu
210215220
thrglyleuleuleuproargalathralaglnaspalaglylystyr
225230235240
tyrcyshisargglyasnleuthrmetserphehisleugluilethr
245250255
alaargprovalleutrphistrpleuleuargthrglyglytrplys
260265270
<210>31
<211>6
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>31
proilecysvalthrval
15
<210>32
<211>47
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>32
lysileserhispheleulysmetgluserleuasnpheileargala
151015
histhrprotyrileasniletyrasncysgluproalaasnproser
202530
glulysasnserproserthrglntyrcystyrserileglnser
354045
<210>33
<211>480
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>33
aspserserlystrpvalphegluhisprogluthrleutyralatrp
151015
gluglyalacysvaltrpileprocysthrtyrargalaleuaspgly
202530
aspleugluserpheileleuphehisasnproglutyrasnlysasn
354045
thrserlyspheaspglythrargleutyrgluserthrlysaspgly
505560
lysvalprosergluglnlysargvalglnpheleuglyasplysasn
65707580
lysasncysthrleuserilehisprovalhisleuasnaspsergly
859095
glnleuglyleuargmetgluserlysthrglulystrpmetgluarg
100105110
ilehisleuasnvalsergluargpropheproprohisileglnleu
115120125
proprogluileglngluserglngluvalthrleuthrcysleuleu
130135140
asnphesercystyrglytyrproileglnleuglntrpleuleuglu
145150155160
glyvalprometargglnalaalavalthrserthrserleuthrile
165170175
lysservalphethrargsergluleulyspheserproglntrpser
180185190
hishisglylysilevalthrcysglnleuglnaspalaaspglylys
195200205
pheleuserasnaspthrvalglnleuasnvallyshisthrprolys
210215220
leugluilelysvalthrproseraspalailevalarggluglyasp
225230235240
servalthrmetthrcysgluvalserserserasnproglutyrthr
245250255
thrvalsertrpleulysaspglythrserleulyslysglnasnthr
260265270
phethrleuasnleuarggluvalthrlysaspglnserglylystyr
275280285
cyscysglnvalserasnaspvalglyproglyargserglugluval
290295300
pheleuglnvalglntyralaprogluproserthrvalglnileleu
305310315320
hisserproalavalgluglyserglnvalglupheleucysmetser
325330335
leualaasnproleuprothrasntyrthrtrptyrhisasnglylys
340345350
glumetglnglyargthrgluglulysvalhisileprolysileleu
355360365
protrphisalaglythrtyrsercysvalalagluasnileleugly
370375380
thrglyglnargglyproglyalagluleuaspvalglntyrpropro
385390395400
lyslysvalthrthrvalileglnasnprometproileargglugly
405410415
aspthrvalthrleusercysasntyrasnserserasnproserval
420425430
thrargtyrglutrplysprohisglyalatrpglugluproserleu
435440445
glyvalleulysileglnasnvalglytrpaspasnthrthrileala
450455460
cysalaalacysasnsertrpcyssertrpalaserprovalalaleu
465470475480
<210>34
<211>480
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>34
glnserprothrproserprothrglyleuthrthralalysmetpro
151015
servalproleuserseraspproleuprothrhisthrthralaphe
202530
serproalaserthrphegluarggluasnaspphesergluthrthr
354045
thrserleuserproaspasnthrserthrglnvalserproaspser
505560
leuaspasnalaseralapheasnthrthrglyvalserservalgln
65707580
thrprohisleuprothrhisalaaspserglnthrproseralagly
859095
thraspthrglnthrpheserglyseralaalaasnalalysleuasn
100105110
prothrproglyserasnalaileseraspvalproglygluargser
115120125
thralaserthrpheprothraspprovalserproleuthrthrthr
130135140
leuserleualahishisserseralaalaleuproalaargthrser
145150155160
asnthrthrilethralaasnthrseraspalatyrleuasnalaser
165170175
gluthrthrthrleuserproserglyseralavalileserthrthr
180185190
thrilealathrthrproserlysprothrcysaspglulystyrala
195200205
asnilethrvalasptyrleutyrasnlysgluthrlysleuphethr
210215220
alalysleuasnvalasngluasnvalglucysglyasnasnthrcys
225230235240
thrasnasngluvalhisasnleuthrglucyslysasnalaserval
245250255
serileserhisasnsercysthralaproasplysthrleuileleu
260265270
aspvalproproglyvalglulyspheglnleuhisaspcysthrgln
275280285
valglulysalaaspthrthrilecysleulystrplysasnileglu
290295300
thrphethrcysaspthrglnasnilethrtyrargpheglncysgly
305310315320
asnmetilepheaspasnlysgluilelysleugluasnleuglupro
325330335
gluhisglutyrlyscysaspsergluileleutyrasnasnhislys
340345350
phethrasnalaserlysileilelysthrasppheglyserprogly
355360365
gluproglnileilephecysargserglualaalahisglnglyval
370375380
ilethrtrpasnproproglnargserphehisasnphethrleucys
385390395400
tyrilelysgluthrglulysaspcysleuasnleuasplysasnleu
405410415
ilelystyraspleuglnasnleulysprotyrthrlystyrvalleu
420425430
serleuhisalatyrileilealalysvalglnargasnglyserala
435440445
alametcyshisphethrthrlysseralaproproserglnvaltrp
450455460
asnmetthrvalsermetthrseraspasnsermethisvallyscys
465470475480
<210>35
<211>371
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>35
lyslysvalvalleuglylyslysglyaspthrvalgluleuthrcys
151015
thralaserglnlyslysserileglnphehistrplysasnserasn
202530
glnilelysileleuglyasnglnglyserpheleuthrlysglypro
354045
serlysleuasnaspargalaaspserargargserleutrpaspgln
505560
glyasnpheproleuileilelysasnleulysilegluaspserasp
65707580
thrtyrilecysgluvalgluaspglnlysglugluvalglnleuleu
859095
valpheglyleuthralaasnseraspthrhisleuleuglnglygln
100105110
serleuthrleuthrleugluserproproglyserserproserval
115120125
glncysargserproargglylysasnileglnglyglylysthrleu
130135140
servalserglnleugluleuglnaspserglythrtrpthrcysthr
145150155160
valleuglnasnglnlyslysvalgluphelysileaspilevalval
165170175
leualapheglnlysalaserserilevaltyrlyslysgluglyglu
180185190
glnvalglupheserpheproleualaphethrvalglulysleuthr
195200205
glyserglygluleutrptrpglnalagluargalaserserserlys
210215220
sertrpilethrpheaspleulysasnlysgluvalservallysarg
225230235240
valthrglnaspprolysleuglnmetglylyslysleuproleuhis
245250255
leuthrleuproglnalaleuproglntyralaglyserglyasnleu
260265270
thrleualaleuglualalysthrglylysleuhisglngluvalasn
275280285
leuvalvalmetargalathrglnleuglnlysasnleuthrcysglu
290295300
valtrpglyprothrserprolysleumetleuserleulysleuglu
305310315320
asnlysglualalysvalserlysargglulysalavaltrpvalleu
325330335
asnproglualaglymettrpglncysleuleuseraspserglygln
340345350
valleuleugluserasnilelysvalleuprothrtrpserthrpro
355360365
valglnpro
370
<210>36
<211>219
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>36
gluleucysaspaspaspproprogluileprohisalathrphelys
151015
alametalatyrlysgluglythrmetleuasncysglucyslysarg
202530
glypheargargilelysserglyserleutyrmetleucysthrgly
354045
asnserserhissersertrpaspasnglncysglncysthrserser
505560
alathrargasnthrthrlysglnvalthrproglnprogluglugln
65707580
lysgluarglysthrthrglumetglnserprometglnprovalasp
859095
glnalaserleuproglyhiscysarggluproproprotrpgluasn
100105110
glualathrgluargiletyrhisphevalvalglyglnmetvaltyr
115120125
tyrglncysvalglnglytyrargalaleuhisargglyproalaglu
130135140
servalcyslysmetthrhisglylysthrargtrpthrglnprogln
145150155160
leuilecysthrglyglumetgluthrserglnpheproglygluglu
165170175
lysproglnalaserprogluglyargproglusergluthrsercys
180185190
leuvalthrthrthrasppheglnileglnthrglumetalaalathr
195200205
metgluthrserilephethrthrglutyrgln
210215
<210>37
<211>3799
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>37
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccgcagatcgtgctgacccagagccccgccatcatgagcgccagccccggcg120
agaaggtgaccatcacctgcagcgccagcagcagcatcagctacatgcactggttccagc180
agaagcccggcaccagccccaagctgtggatctacaccaccagcaacctggccagcggcg240
tgcccgccagattcagcggcagcggcagcggcaccagctacagcctgaccatcagcagaa300
tggaggccgaggacgccgccacctactactgccaccagagaagcacctaccccctgacct360
tcggcagcggcaccaagctggagctgaagggaggggggggatccgggggaggaggctccg420
gcggaggcggaagccaggtgcagctgcagcagagcggcgccgagctggccaagcccggcg480
ccagcgtgaagatgagctgcaaggccagcggctacaccttcaccagctacagaatgcact540
gggtgaagcagagacccggccagggcctggagtggatcggctacatcaaccccagcaccg600
gctacaccgagtacaaccagaagttcaaggacaaggccaccctgaccgccgacaagagca660
gcagcaccgcctacatgcagctgagcagcctgaccttcgaggacagcgccgtgtactact720
gcgccagaggcggcggcgtgttcgactactggggccagggcaccaccctgaccgtgagca780
gcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccc840
tgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggc900
tggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttc960
tcctgtcactggttatcaccctttactgcaaacggggcagaaagaaactcctgtatatat1020
tcaaacaaccatttatgagaccagtacaaactactcaagaggaagatggctgtagctgcc1080
gatttccagaagaagaagaaggaggatgtgaactgagagtgaagttcagcaggagcgcag1140
acgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaa1200
gagaggagtacgatgttttggacaagagacgtggccgggaccctgagatggggggaaagc1260
cgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatgg1320
cggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatg1380
gcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttcacatgcagg1440
ccctgccccctcgcggaagcggagctactaacttcagcctgctgaagcaggctggagacg1500
tggaggagaaccctggacctatggccttaccagtgaccgccttgctcctgccgctggcct1560
tgctgctccacgccgccaggccggacgtgcagatcacccagagccccagctacctggccg1620
ccagccccggcgagaccatcaccatcaactgcagagccagcaagagcatcagcaaggacc1680
tggcctggtaccaggagaagcccggcaagaccaacaagctgctgatctacagcggcagca1740
ccctgcagagcggcatccccagcagattcagcggcagcggcagcggcaccgacttcaccc1800
tgaccatcagcagcctggagcccgaggacttcgccatgtactactgccagcagcacaaca1860
agtacccctacaccttcggcggcggcaccaagctggagatcaagggaggggggggatccg1920
ggggaggaggctccggcggaggcggaagccaggtgcagctgcagcagcccggcgccgagc1980
tggtgagacccggcgccagcgtgaagctgagctgcaaggccagcggctacaccttcacca2040
gctactggatgaactgggtgaagcagagacccgaccagggcctggagtggatcggcagaa2100
tcgacccctacgacagcgagacccactacaaccagaagttcaaggacaaggccatcctga2160
ccgtggacaagagcagcagcaccgcctacatgcagctgagcagcctgaccagcgaggaca2220
gcgccgtgtactactgcgccagaggcaactgggacgactactggggccagggcaccaccc2280
tgaccgtgagcagcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcg2340
cgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgc2400
acacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggactt2460
gtggggtccttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggc2520
tcctgcacagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcatt2580
accagccctatgccccaccacgcgacttcgcagcctatcgctccagagtgaagttcagca2640
ggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatc2700
taggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctgagatgg2760
ggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaag2820
ataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaagg2880
ggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttc2940
acatgcaggccctgccccctcgcggcagcggcgaaggccgcggcagcctgctgacctgcg3000
gcgatgtggaagaaaacccgggccccatggaatacgcctctgacgcttcactggaccccg3060
aagccccgtggcctcccgcgccccgcgctcgcgcctgccgcgtactgccttgggccctgg3120
tcgcggggctgctgctgctgctgctgctcgctgccgcctgcgccgtcttcctcgcctgcc3180
cctgggccgtgtccggggctcgcgcctcgcccggctccgcggccagcccgagactccgcg3240
agggtcccgagctttcgcccgacgatcccgccggcctcttggacctgcggcagggcatgt3300
ttgcgcagctggtggcccaaaatgttctgctgatcgatgggcccctgagctggtacagtg3360
acccaggcctggcaggcgtgtccctgacggggggcctgagctacaaagaggacacgaagg3420
agctggtggtggccaaggctggagtctactatgtcttctttcaactagagctgcggcgcg3480
tggtggccggcgagggctcaggctccgtttcacttgcgctgcacctgcagccactgcgct3540
ctgctgctggggccgccgccctggctttgaccgtggacctgccacccgcctcctccgagg3600
ctcggaactcggccttcggtttccagggccgcttgctgcacctgagtgccggccagcgcc3660
tgggcgtccatcttcacactgaggccagggcacgccatgcctggcagcttacccagggcg3720
ccacagtcttgggactcttccgggtgacccccgaaatcccagccggactcccttcaccga3780
ggtcggaataagtttaaac3799
<210>38
<211>1260
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>38
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproglnilevalleuthrglnserproalailemet
202530
seralaserproglyglulysvalthrilethrcysseralaserser
354045
serilesertyrmethistrppheglnglnlysproglythrserpro
505560
lysleutrpiletyrthrthrserasnleualaserglyvalproala
65707580
argpheserglyserglyserglythrsertyrserleuthrileser
859095
argmetglualagluaspalaalathrtyrtyrcyshisglnargser
100105110
thrtyrproleuthrpheglyserglythrlysleugluleulysgly
115120125
glyglyglyserglyglyglyglyserglyglyglyglyserglnval
130135140
glnleuglnglnserglyalagluleualalysproglyalaserval
145150155160
lysmetsercyslysalaserglytyrthrphethrsertyrargmet
165170175
histrpvallysglnargproglyglnglyleuglutrpileglytyr
180185190
ileasnproserthrglytyrthrglutyrasnglnlysphelysasp
195200205
lysalathrleuthralaasplysserserserthralatyrmetgln
210215220
leuserserleuthrphegluaspseralavaltyrtyrcysalaarg
225230235240
glyglyglyvalpheasptyrtrpglyglnglythrthrleuthrval
245250255
serserthrthrthrproalaproargproprothrproalaprothr
260265270
ilealaserglnproleuserleuargproglualacysargproala
275280285
alaglyglyalavalhisthrargglyleuaspphealacysaspile
290295300
tyriletrpalaproleualaglythrcysglyvalleuleuleuser
305310315320
leuvalilethrleutyrcyslysargglyarglyslysleuleutyr
325330335
ilephelysglnprophemetargprovalglnthrthrglngluglu
340345350
aspglycyssercysargpheproglugluglugluglyglycysglu
355360365
leuargvallyspheserargseralaaspalaproalatyrglngln
370375380
glyglnasnglnleutyrasngluleuasnleuglyargarggluglu
385390395400
tyraspvalleuasplysargargglyargaspproglumetglygly
405410415
lysproglnargarglysasnproglngluglyleutyrasngluleu
420425430
glnlysasplysmetalaglualatyrsergluileglymetlysgly
435440445
gluargargargglylysglyhisaspglyleutyrglnglyleuser
450455460
thralathrlysaspthrtyraspalaleuhismetglnalaleupro
465470475480
proargglyserglyalathrasnpheserleuleulysglnalagly
485490495
aspvalglugluasnproglyprometalaleuprovalthralaleu
500505510
leuleuproleualaleuleuleuhisalaalaargproaspvalgln
515520525
ilethrglnserprosertyrleualaalaserproglygluthrile
530535540
thrileasncysargalaserlysserileserlysaspleualatrp
545550555560
tyrglnglulysproglylysthrasnlysleuleuiletyrsergly
565570575
serthrleuglnserglyileproserargpheserglyserglyser
580585590
glythraspphethrleuthrileserserleugluprogluaspphe
595600605
alamettyrtyrcysglnglnhisasnlystyrprotyrthrphegly
610615620
glyglythrlysleugluilelysglyglyglyglyserglyglygly
625630635640
glyserglyglyglyglyserglnvalglnleuglnglnproglyala
645650655
gluleuvalargproglyalaservallysleusercyslysalaser
660665670
glytyrthrphethrsertyrtrpmetasntrpvallysglnargpro
675680685
aspglnglyleuglutrpileglyargileaspprotyraspserglu
690695700
thrhistyrasnglnlysphelysasplysalaileleuthrvalasp
705710715720
lysserserserthralatyrmetglnleuserserleuthrserglu
725730735
aspseralavaltyrtyrcysalaargglyasntrpaspasptyrtrp
740745750
glyglnglythrthrleuthrvalserserthrthrthrproalapro
755760765
argproprothrproalaprothrilealaserglnproleuserleu
770775780
argproglualacysargproalaalaglyglyalavalhisthrarg
785790795800
glyleuaspphealacysaspiletyriletrpalaproleualagly
805810815
thrcysglyvalleuleuleuserleuvalilethrleutyrcysarg
820825830
serlysargserargleuleuhisserasptyrmetasnmetthrpro
835840845
argargproglyprothrarglyshistyrglnprotyralapropro
850855860
argaspphealaalatyrargserargvallyspheserargserala
865870875880
aspalaproalatyrglnglnglyglnasnglnleutyrasngluleu
885890895
asnleuglyargarggluglutyraspvalleuasplysargarggly
900905910
argaspproglumetglyglylysproglnargarglysasnprogln
915920925
gluglyleutyrasngluleuglnlysasplysmetalaglualatyr
930935940
sergluileglymetlysglygluargargargglylysglyhisasp
945950955960
glyleutyrglnglyleuserthralathrlysaspthrtyraspala
965970975
leuhismetglnalaleuproproargglyserglygluglyarggly
980985990
serleuleuthrcysglyaspvalglugluasnproglyprometglu
99510001005
tyralaseraspalaserleuaspproglualaprotrppropro
101010151020
alaproargalaargalacysargvalleuprotrpalaleuval
102510301035
alaglyleuleuleuleuleuleuleualaalaalacysalaval
104010451050
pheleualacysprotrpalavalserglyalaargalaserpro
105510601065
glyseralaalaserproargleuarggluglyprogluleuser
107010751080
proaspaspproalaglyleuleuaspleuargglnglymetphe
108510901095
alaglnleuvalalaglnasnvalleuleuileaspglyproleu
110011051110
sertrptyrseraspproglyleualaglyvalserleuthrgly
111511201125
glyleusertyrlysgluaspthrlysgluleuvalvalalalys
113011351140
alaglyvaltyrtyrvalphepheglnleugluleuargargval
114511501155
valalaglygluglyserglyservalserleualaleuhisleu
116011651170
glnproleuargseralaalaglyalaalaalaleualaleuthr
117511801185
valaspleuproproalaserserglualaargasnseralaphe
119011951200
glypheglnglyargleuleuhisleuseralaglyglnargleu
120512101215
glyvalhisleuhisthrglualaargalaarghisalatrpgln
122012251230
leuthrglnglyalathrvalleuglyleupheargvalthrpro
123512401245
gluileproalaglyleuproserproargserglu
125012551260
<210>39
<211>3856
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>39
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccgcagatcgtgctgacccagagccccgccatcatgagcgccagccccggcg120
agaaggtgaccatcacctgcagcgccagcagcagcatcagctacatgcactggttccagc180
agaagcccggcaccagccccaagctgtggatctacaccaccagcaacctggccagcggcg240
tgcccgccagattcagcggcagcggcagcggcaccagctacagcctgaccatcagcagaa300
tggaggccgaggacgccgccacctactactgccaccagagaagcacctaccccctgacct360
tcggcagcggcaccaagctggagctgaagggaggggggggatccgggggaggaggctccg420
gcggaggcggaagccaggtgcagctgcagcagagcggcgccgagctggccaagcccggcg480
ccagcgtgaagatgagctgcaaggccagcggctacaccttcaccagctacagaatgcact540
gggtgaagcagagacccggccagggcctggagtggatcggctacatcaaccccagcaccg600
gctacaccgagtacaaccagaagttcaaggacaaggccaccctgaccgccgacaagagca660
gcagcaccgcctacatgcagctgagcagcctgaccttcgaggacagcgccgtgtactact720
gcgccagaggcggcggcgtgttcgactactggggccagggcaccaccctgaccgtgagca780
gcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccc840
tgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggc900
tggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttc960
tcctgtcactggttatcaccctttactgcaaacggggcagaaagaaactcctgtatatat1020
tcaaacaaccatttatgagaccagtacaaactactcaagaggaagatggctgtagctgcc1080
gatttccagaagaagaagaaggaggatgtgaactgagagtgaagttcagcaggagcgcag1140
acgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaa1200
gagaggagtacgatgttttggacaagagacgtggccgggaccctgagatggggggaaagc1260
cgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatgg1320
cggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatg1380
gcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttcacatgcagg1440
ccctgccccctcgcggaagcggagctactaacttcagcctgctgaagcaggctggagacg1500
tggaggagaaccctggacctatggccttaccagtgaccgccttgctcctgccgctggcct1560
tgctgctccacgccgccaggccggacgtgcagatcacccagagccccagctacctggccg1620
ccagccccggcgagaccatcaccatcaactgcagagccagcaagagcatcagcaaggacc1680
tggcctggtaccaggagaagcccggcaagaccaacaagctgctgatctacagcggcagca1740
ccctgcagagcggcatccccagcagattcagcggcagcggcagcggcaccgacttcaccc1800
tgaccatcagcagcctggagcccgaggacttcgccatgtactactgccagcagcacaaca1860
agtacccctacaccttcggcggcggcaccaagctggagatcaagggaggggggggatccg1920
ggggaggaggctccggcggaggcggaagccaggtgcagctgcagcagcccggcgccgagc1980
tggtgagacccggcgccagcgtgaagctgagctgcaaggccagcggctacaccttcacca2040
gctactggatgaactgggtgaagcagagacccgaccagggcctggagtggatcggcagaa2100
tcgacccctacgacagcgagacccactacaaccagaagttcaaggacaaggccatcctga2160
ccgtggacaagagcagcagcaccgcctacatgcagctgagcagcctgaccagcgaggaca2220
gcgccgtgtactactgcgccagaggcaactgggacgactactggggccagggcaccaccc2280
tgaccgtgagcagcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcg2340
cgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgc2400
acacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggactt2460
gtggggtccttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggc2520
tcctgcacagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcatt2580
accagccctatgccccaccacgcgacttcgcagcctatcgctccagagtgaagttcagca2640
ggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatc2700
taggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctgagatgg2760
ggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaag2820
ataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaagg2880
ggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttc2940
acatgcaggccctgccccctcgcggcagcggcgaaggccgcggcagcctgctgacctgcg3000
gcgatgtggaagaaaacccgggccccatgtacagaatgcagctgctgagctgcatcgccc3060
tgagcctggccctggtgaccaacagcggcatccacgtgttcatcctgggctgcttcagcg3120
ccggcctgcccaagaccgaggccaactgggtgaacgtgatcagcgacctgaagaagatcg3180
aggacctgatccagagcatgcacatcgacgccaccctgtacaccgagagcgacgtgcacc3240
ccagctgcaaggtgaccgccatgaagtgcttcctgctggagctgcaggtgatcagcctgg3300
agagcggcgacgccagcatccacgacaccgtggagaacctgatcatcctggccaacaaca3360
gcctgagcagcaacggcaacgtgaccgagagcggctgcaaggagtgcgaggagctggagg3420
agaagaacatcaaggagttcctgcagagcttcgtgcacatcgtgcagatgttcatcaaca3480
ccagctccggcggcggctccggcggcggcggctccggcggcggcggctccggcggcggcg3540
gctccggcggcggctccctgcaggcccccagaagagccagaggctgcagaaccctgggcc3600
tgcccgccctgctgctgctgctgctgctgagaccccccgccaccagaggcatcacctgcc3660
ccccccccatgagcgtggagcacgccgacatctgggtgaagagctacagcctgtacagca3720
gagagagatacatctgcaacagcggcttcaagagaaaggccggcaccagcagcctgaccg3780
agtgcgtgctgaacaaggccaccaacgtggcccactggaccacccccagcctgaagtgca3840
tcagataagtttaaac3856
<210>40
<211>1279
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>40
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproglnilevalleuthrglnserproalailemet
202530
seralaserproglyglulysvalthrilethrcysseralaserser
354045
serilesertyrmethistrppheglnglnlysproglythrserpro
505560
lysleutrpiletyrthrthrserasnleualaserglyvalproala
65707580
argpheserglyserglyserglythrsertyrserleuthrileser
859095
argmetglualagluaspalaalathrtyrtyrcyshisglnargser
100105110
thrtyrproleuthrpheglyserglythrlysleugluleulysgly
115120125
glyglyglyserglyglyglyglyserglyglyglyglyserglnval
130135140
glnleuglnglnserglyalagluleualalysproglyalaserval
145150155160
lysmetsercyslysalaserglytyrthrphethrsertyrargmet
165170175
histrpvallysglnargproglyglnglyleuglutrpileglytyr
180185190
ileasnproserthrglytyrthrglutyrasnglnlysphelysasp
195200205
lysalathrleuthralaasplysserserserthralatyrmetgln
210215220
leuserserleuthrphegluaspseralavaltyrtyrcysalaarg
225230235240
glyglyglyvalpheasptyrtrpglyglnglythrthrleuthrval
245250255
serserthrthrthrproalaproargproprothrproalaprothr
260265270
ilealaserglnproleuserleuargproglualacysargproala
275280285
alaglyglyalavalhisthrargglyleuaspphealacysaspile
290295300
tyriletrpalaproleualaglythrcysglyvalleuleuleuser
305310315320
leuvalilethrleutyrcyslysargglyarglyslysleuleutyr
325330335
ilephelysglnprophemetargprovalglnthrthrglngluglu
340345350
aspglycyssercysargpheproglugluglugluglyglycysglu
355360365
leuargvallyspheserargseralaaspalaproalatyrglngln
370375380
glyglnasnglnleutyrasngluleuasnleuglyargarggluglu
385390395400
tyraspvalleuasplysargargglyargaspproglumetglygly
405410415
lysproglnargarglysasnproglngluglyleutyrasngluleu
420425430
glnlysasplysmetalaglualatyrsergluileglymetlysgly
435440445
gluargargargglylysglyhisaspglyleutyrglnglyleuser
450455460
thralathrlysaspthrtyraspalaleuhismetglnalaleupro
465470475480
proargglyserglyalathrasnpheserleuleulysglnalagly
485490495
aspvalglugluasnproglyprometalaleuprovalthralaleu
500505510
leuleuproleualaleuleuleuhisalaalaargproaspvalgln
515520525
ilethrglnserprosertyrleualaalaserproglygluthrile
530535540
thrileasncysargalaserlysserileserlysaspleualatrp
545550555560
tyrglnglulysproglylysthrasnlysleuleuiletyrsergly
565570575
serthrleuglnserglyileproserargpheserglyserglyser
580585590
glythraspphethrleuthrileserserleugluprogluaspphe
595600605
alamettyrtyrcysglnglnhisasnlystyrprotyrthrphegly
610615620
glyglythrlysleugluilelysglyglyglyglyserglyglygly
625630635640
glyserglyglyglyglyserglnvalglnleuglnglnproglyala
645650655
gluleuvalargproglyalaservallysleusercyslysalaser
660665670
glytyrthrphethrsertyrtrpmetasntrpvallysglnargpro
675680685
aspglnglyleuglutrpileglyargileaspprotyraspserglu
690695700
thrhistyrasnglnlysphelysasplysalaileleuthrvalasp
705710715720
lysserserserthralatyrmetglnleuserserleuthrserglu
725730735
aspseralavaltyrtyrcysalaargglyasntrpaspasptyrtrp
740745750
glyglnglythrthrleuthrvalserserthrthrthrproalapro
755760765
argproprothrproalaprothrilealaserglnproleuserleu
770775780
argproglualacysargproalaalaglyglyalavalhisthrarg
785790795800
glyleuaspphealacysaspiletyriletrpalaproleualagly
805810815
thrcysglyvalleuleuleuserleuvalilethrleutyrcysarg
820825830
serlysargserargleuleuhisserasptyrmetasnmetthrpro
835840845
argargproglyprothrarglyshistyrglnprotyralapropro
850855860
argaspphealaalatyrargserargvallyspheserargserala
865870875880
aspalaproalatyrglnglnglyglnasnglnleutyrasngluleu
885890895
asnleuglyargarggluglutyraspvalleuasplysargarggly
900905910
argaspproglumetglyglylysproglnargarglysasnprogln
915920925
gluglyleutyrasngluleuglnlysasplysmetalaglualatyr
930935940
sergluileglymetlysglygluargargargglylysglyhisasp
945950955960
glyleutyrglnglyleuserthralathrlysaspthrtyraspala
965970975
leuhismetglnalaleuproproargglyserglygluglyarggly
980985990
serleuleuthrcysglyaspvalglugluasnproglypromettyr
99510001005
argmetglnleuleusercysilealaleuserleualaleuval
101010151020
thrasnserglyilehisvalpheileleuglycyspheserala
102510301035
glyleuprolysthrglualaasntrpvalasnvalileserasp
104010451050
leulyslysilegluaspleuileglnsermethisileaspala
105510601065
thrleutyrthrgluseraspvalhisprosercyslysvalthr
107010751080
alametlyscyspheleuleugluleuglnvalileserleuglu
108510901095
serglyaspalaserilehisaspthrvalgluasnleuileile
110011051110
leualaasnasnserleuserserasnglyasnvalthrgluser
111511201125
glycyslysglucysglugluleugluglulysasnilelysglu
113011351140
pheleuglnserphevalhisilevalglnmetpheileasnthr
114511501155
serserglyglyglyserglyglyglyglyserglyglyglygly
116011651170
serglyglyglyglyserglyglyglyserleuglnalaproarg
117511801185
argalaargglycysargthrleuglyleuproalaleuleuleu
119011951200
leuleuleuleuargproproalathrargglyilethrcyspro
120512101215
proprometservalgluhisalaaspiletrpvallyssertyr
122012251230
serleutyrserarggluargtyrilecysasnserglyphelys
123512401245
arglysalaglythrserserleuthrglucysvalleuasnlys
125012551260
alathrasnvalalahistrpthrthrproserleulyscysile
126512701275
arg
<210>41
<211>2314
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>41
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgctgacccagagccccgccagcctggccgtgagcctgggcc120
agagggccaccatcagctgcagggccagcaagagcgtgagcaccagcggctacagctacc180
tgcactggtaccagcagaagcccggccagccccccaagctgctgatctacctggccagca240
acctggagagcggcgtgcccgccaggttcagcggcagcggcagcggcaccgacttcaccc300
tgaacatccaccccgtggaggaggaggacgccgccacctactactgccagcacagcaggg360
agctgcccttcaccttcggcagcggcaccaagctggagatcaagggaggggggggatccg420
ggggaggaggctccggcggaggcggaagccaggtgcagctggtggagagcggcggcggcc480
tggtgcagcccggcggcagcctgaagctgagctgcgccgccagcggcttcgacttcagca540
ggtactggatgagctgggtgaggcaggcccccggcaagggcctggagtggatcggcgaga600
tcaaccccaccagcagcaccatcaacttcacccccagcctgaaggacaaggtgttcatca660
gcagggacaacgccaagaacaccctgtacctgcagatgagcaaggtgaggagcgaggaca720
ccgccctgtactactgcgccaggggcaactactacaggtacggcgacgccatggactact780
ggggccagggcaccagcgtgaccgtgagcaccacgacgccagcgccgcgaccaccaacac840
cggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcgg900
cggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgc960
ccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcagga1020
gtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggc1080
ccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctcca1140
gagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctct1200
ataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggcc1260
gggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtaca1320
atgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagc1380
gccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggaca1440
cctacgacgcccttcacatgcaggccctgccccctcgcggcagcggcgaaggccgcggca1500
gcctgctgacctgcggcgatgtggaagaaaacccgggccccatggaatacgcctctgacg1560
cttcactggaccccgaagccccgtggcctcccgcgccccgcgctcgcgcctgccgcgtac1620
tgccttgggccctggtcgcggggctgctgctgctgctgctgctcgctgccgcctgcgccg1680
tcttcctcgcctgcccctgggccgtgtccggggctcgcgcctcgcccggctccgcggcca1740
gcccgagactccgcgagggtcccgagctttcgcccgacgatcccgccggcctcttggacc1800
tgcggcagggcatgtttgcgcagctggtggcccaaaatgttctgctgatcgatgggcccc1860
tgagctggtacagtgacccaggcctggcaggcgtgtccctgacggggggcctgagctaca1920
aagaggacacgaaggagctggtggtggccaaggctggagtctactatgtcttctttcaac1980
tagagctgcggcgcgtggtggccggcgagggctcaggctccgtttcacttgcgctgcacc2040
tgcagccactgcgctctgctgctggggccgccgccctggctttgaccgtggacctgccac2100
ccgcctcctccgaggctcggaactcggccttcggtttccagggccgcttgctgcacctga2160
gtgccggccagcgcctgggcgtccatcttcacactgaggccagggcacgccatgcctggc2220
agcttacccagggcgccacagtcttgggactcttccgggtgacccccgaaatcccagccg2280
gactcccttcaccgaggtcggaataagtttaaac2314
<210>42
<211>765
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>42
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalleuthrglnserproalaserleu
202530
alavalserleuglyglnargalathrilesercysargalaserlys
354045
servalserthrserglytyrsertyrleuhistrptyrglnglnlys
505560
proglyglnproprolysleuleuiletyrleualaserasnleuglu
65707580
serglyvalproalaargpheserglyserglyserglythraspphe
859095
thrleuasnilehisprovalgluglugluaspalaalathrtyrtyr
100105110
cysglnhisserarggluleuprophethrpheglyserglythrlys
115120125
leugluilelysglyglyglyglyserglyglyglyglyserglygly
130135140
glyglyserglnvalglnleuvalgluserglyglyglyleuvalgln
145150155160
proglyglyserleulysleusercysalaalaserglypheaspphe
165170175
serargtyrtrpmetsertrpvalargglnalaproglylysglyleu
180185190
glutrpileglygluileasnprothrserserthrileasnphethr
195200205
proserleulysasplysvalpheileserargaspasnalalysasn
210215220
thrleutyrleuglnmetserlysvalargsergluaspthralaleu
225230235240
tyrtyrcysalaargglyasntyrtyrargtyrglyaspalametasp
245250255
tyrtrpglyglnglythrservalthrvalserthrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
argserlysargserargleuleuhisserasptyrmetasnmetthr
340345350
proargargproglyprothrarglyshistyrglnprotyralapro
355360365
proargaspphealaalatyrargserargvallyspheserargser
370375380
alaaspalaproalatyrglnglnglyglnasnglnleutyrasnglu
385390395400
leuasnleuglyargarggluglutyraspvalleuasplysargarg
405410415
glyargaspproglumetglyglylysproglnargarglysasnpro
420425430
glngluglyleutyrasngluleuglnlysasplysmetalagluala
435440445
tyrsergluileglymetlysglygluargargargglylysglyhis
450455460
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
465470475480
alaleuhismetglnalaleuproproargglyserglygluglyarg
485490495
glyserleuleuthrcysglyaspvalglugluasnproglypromet
500505510
glutyralaseraspalaserleuaspproglualaprotrppropro
515520525
alaproargalaargalacysargvalleuprotrpalaleuvalala
530535540
glyleuleuleuleuleuleuleualaalaalacysalavalpheleu
545550555560
alacysprotrpalavalserglyalaargalaserproglyserala
565570575
alaserproargleuarggluglyprogluleuserproaspasppro
580585590
alaglyleuleuaspleuargglnglymetphealaglnleuvalala
595600605
glnasnvalleuleuileaspglyproleusertrptyrserasppro
610615620
glyleualaglyvalserleuthrglyglyleusertyrlysgluasp
625630635640
thrlysgluleuvalvalalalysalaglyvaltyrtyrvalphephe
645650655
glnleugluleuargargvalvalalaglygluglyserglyserval
660665670
serleualaleuhisleuglnproleuargseralaalaglyalaala
675680685
alaleualaleuthrvalaspleuproproalaserserglualaarg
690695700
asnseralapheglypheglnglyargleuleuhisleuseralagly
705710715720
glnargleuglyvalhisleuhisthrglualaargalaarghisala
725730735
trpglnleuthrglnglyalathrvalleuglyleupheargvalthr
740745750
progluileproalaglyleuproserproargserglu
755760765
<210>43
<211>2371
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>43
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgctgacccagagccccgccagcctggccgtgagcctgggcc120
agagggccaccatcagctgcagggccagcaagagcgtgagcaccagcggctacagctacc180
tgcactggtaccagcagaagcccggccagccccccaagctgctgatctacctggccagca240
acctggagagcggcgtgcccgccaggttcagcggcagcggcagcggcaccgacttcaccc300
tgaacatccaccccgtggaggaggaggacgccgccacctactactgccagcacagcaggg360
agctgcccttcaccttcggcagcggcaccaagctggagatcaagggaggggggggatccg420
ggggaggaggctccggcggaggcggaagccaggtgcagctggtggagagcggcggcggcc480
tggtgcagcccggcggcagcctgaagctgagctgcgccgccagcggcttcgacttcagca540
ggtactggatgagctgggtgaggcaggcccccggcaagggcctggagtggatcggcgaga600
tcaaccccaccagcagcaccatcaacttcacccccagcctgaaggacaaggtgttcatca660
gcagggacaacgccaagaacaccctgtacctgcagatgagcaaggtgaggagcgaggaca720
ccgccctgtactactgcgccaggggcaactactacaggtacggcgacgccatggactact780
ggggccagggcaccagcgtgaccgtgagcaccacgacgccagcgccgcgaccaccaacac840
cggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcgg900
cggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgc960
ccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcagga1020
gtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggc1080
ccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctcca1140
gagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctct1200
ataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggcc1260
gggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtaca1320
atgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagc1380
gccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggaca1440
cctacgacgcccttcacatgcaggccctgccccctcgcggcagcggcgaaggccgcggca1500
gcctgctgacctgcggcgatgtggaagaaaacccgggccccatgtacagaatgcagctgc1560
tgagctgcatcgccctgagcctggccctggtgaccaacagcggcatccacgtgttcatcc1620
tgggctgcttcagcgccggcctgcccaagaccgaggccaactgggtgaacgtgatcagcg1680
acctgaagaagatcgaggacctgatccagagcatgcacatcgacgccaccctgtacaccg1740
agagcgacgtgcaccccagctgcaaggtgaccgccatgaagtgcttcctgctggagctgc1800
aggtgatcagcctggagagcggcgacgccagcatccacgacaccgtggagaacctgatca1860
tcctggccaacaacagcctgagcagcaacggcaacgtgaccgagagcggctgcaaggagt1920
gcgaggagctggaggagaagaacatcaaggagttcctgcagagcttcgtgcacatcgtgc1980
agatgttcatcaacaccagctccggcggcggctccggcggcggcggctccggcggcggcg2040
gctccggcggcggcggctccggcggcggctccctgcaggcccccagaagagccagaggct2100
gcagaaccctgggcctgcccgccctgctgctgctgctgctgctgagaccccccgccacca2160
gaggcatcacctgccccccccccatgagcgtggagcacgccgacatctgggtgaagagct2220
acagcctgtacagcagagagagatacatctgcaacagcggcttcaagagaaaggccggca2280
ccagcagcctgaccgagtgcgtgctgaacaaggccaccaacgtggcccactggaccaccc2340
ccagcctgaagtgcatcagataagtttaaac2371
<210>44
<211>784
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>44
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalleuthrglnserproalaserleu
202530
alavalserleuglyglnargalathrilesercysargalaserlys
354045
servalserthrserglytyrsertyrleuhistrptyrglnglnlys
505560
proglyglnproprolysleuleuiletyrleualaserasnleuglu
65707580
serglyvalproalaargpheserglyserglyserglythraspphe
859095
thrleuasnilehisprovalgluglugluaspalaalathrtyrtyr
100105110
cysglnhisserarggluleuprophethrpheglyserglythrlys
115120125
leugluilelysglyglyglyglyserglyglyglyglyserglygly
130135140
glyglyserglnvalglnleuvalgluserglyglyglyleuvalgln
145150155160
proglyglyserleulysleusercysalaalaserglypheaspphe
165170175
serargtyrtrpmetsertrpvalargglnalaproglylysglyleu
180185190
glutrpileglygluileasnprothrserserthrileasnphethr
195200205
proserleulysasplysvalpheileserargaspasnalalysasn
210215220
thrleutyrleuglnmetserlysvalargsergluaspthralaleu
225230235240
tyrtyrcysalaargglyasntyrtyrargtyrglyaspalametasp
245250255
tyrtrpglyglnglythrservalthrvalserthrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
argserlysargserargleuleuhisserasptyrmetasnmetthr
340345350
proargargproglyprothrarglyshistyrglnprotyralapro
355360365
proargaspphealaalatyrargserargvallyspheserargser
370375380
alaaspalaproalatyrglnglnglyglnasnglnleutyrasnglu
385390395400
leuasnleuglyargarggluglutyraspvalleuasplysargarg
405410415
glyargaspproglumetglyglylysproglnargarglysasnpro
420425430
glngluglyleutyrasngluleuglnlysasplysmetalagluala
435440445
tyrsergluileglymetlysglygluargargargglylysglyhis
450455460
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
465470475480
alaleuhismetglnalaleuproproargglyserglygluglyarg
485490495
glyserleuleuthrcysglyaspvalglugluasnproglypromet
500505510
tyrargmetglnleuleusercysilealaleuserleualaleuval
515520525
thrasnserglyilehisvalpheileleuglycyspheseralagly
530535540
leuprolysthrglualaasntrpvalasnvalileseraspleulys
545550555560
lysilegluaspleuileglnsermethisileaspalathrleutyr
565570575
thrgluseraspvalhisprosercyslysvalthralametlyscys
580585590
pheleuleugluleuglnvalileserleugluserglyaspalaser
595600605
ilehisaspthrvalgluasnleuileileleualaasnasnserleu
610615620
serserasnglyasnvalthrgluserglycyslysglucysgluglu
625630635640
leugluglulysasnilelysglupheleuglnserphevalhisile
645650655
valglnmetpheileasnthrserserglyglyglyserglyglygly
660665670
glyserglyglyglyglyserglyglyglyglyserglyglyglyser
675680685
leuglnalaproargargalaargglycysargthrleuglyleupro
690695700
alaleuleuleuleuleuleuleuargproproalathrargglyile
705710715720
thrcysproproprometservalgluhisalaaspiletrpvallys
725730735
sertyrserleutyrserarggluargtyrilecysasnserglyphe
740745750
lysarglysalaglythrserserleuthrglucysvalleuasnlys
755760765
alathrasnvalalahistrpthrthrproserleulyscysilearg
770775780
<210>45
<211>2369
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>45
atggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacgccgccagg60
ccggatgtggtgatgacccagagccatcgctttatgagcaccagcgtgggcgaccgagtg120
agcattacctgccgcgcgagccaggatgtgaacaccgcggtgagctggtatcagcagaaa180
ccgggccagagcccgaaactgctgatttttagcgcgagctatcgctataccggcgtgccg240
gatcgctttaccggcagcggcagcggcgcggattttaccctgaccattagcagcgtgcag300
gcggaagatctggcggtgtattattgccagcagcattatagcaccccgtggacctttggc360
ggcggcaccaaactggacattaaaggaggggggggatccgggggaggaggctccggcgga420
ggcggaagccagattcagctggtgcagagcggcccggatctgaaaaaaccgggcgaaacc480
gtgaaactgagctgcaaagcgagcggctatacctttaccaactttggcatgaactgggtg540
aaacaggcgccgggcaaaggctttaaatggatggcgtggattaacaccacccgctatacc600
ggcgaaagctattttgcggatgattttaaaggccgctttgcgtttagcgtggaaaccagc660
gcgaccaccgcgtatctgcagattaacaacctgaaaaccgaagataccgcgacctatttt720
tgcgcgcgcggcgaaatttattatggctatgatggcggctttgcgtattggggccagggc780
accctggtgaccgtgagcgcgaccacgacgccagcgccgcgaccaccaacaccggcgccc840
accatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggc900
gcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggcc960
gggacttgtggggtccttctcctgtcactggttatcaccctttactgcaaacggggcaga1020
aagaaactcctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagag1080
gaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtg1140
aagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataac1200
gagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggac1260
cctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaa1320
ctgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccgg1380
aggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacacctac1440
gacgcccttcacatgcaggccctgccccctcgcggaagcggagccaccaacttcagcctg1500
ctgaagcaggccggcgacgtggaggagaaccccggccccatgtacagaatgcagctgctg1560
agctgcatcgccctgagcctggccctggtgaccaacagcggcatccacgtgttcatcctg1620
ggctgcttcagcgccggcctgcccaagaccgaggccaactgggtgaacgtgatcagcgac1680
ctgaagaagatcgaggacctgatccagagcatgcacatcgacgccaccctgtacaccgag1740
agcgacgtgcaccccagctgcaaggtgaccgccatgaagtgcttcctgctggagctgcag1800
gtgatcagcctggagagcggcgacgccagcatccacgacaccgtggagaacctgatcatc1860
ctggccaacaacagcctgagcagcaacggcaacgtgaccgagagcggctgcaaggagtgc1920
gaggagctggaggagaagaacatcaaggagttcctgcagagcttcgtgcacatcgtgcag1980
atgttcatcaacaccagctccggcggcggctccggcggcggcggctccggcggcggcggc2040
tccggcggcggcggctccggcggcggctccctgcaggcccccagaagagccagaggctgc2100
agaaccctgggcctgcccgccctgctgctgctgctgctgctgagaccccccgccaccaga2160
ggcatcacctgccccccccccatgagcgtggagcacgccgacatctgggtgaagagctac2220
agcctgtacagcagagagagatacatctgcaacagcggcttcaagagaaaggccggcacc2280
agcagcctgaccgagtgcgtgctgaacaaggccaccaacgtggcccactggaccaccccc2340
agcctgaagtgcatcagataagtttaaac2369
<210>46
<211>786
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>46
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspvalvalmetthrglnserhisargphemet
202530
serthrservalglyaspargvalserilethrcysargalasergln
354045
aspvalasnthralavalsertrptyrglnglnlysproglyglnser
505560
prolysleuleuilepheseralasertyrargtyrthrglyvalpro
65707580
aspargphethrglyserglyserglyalaaspphethrleuthrile
859095
serservalglnalagluaspleualavaltyrtyrcysglnglnhis
100105110
tyrserthrprotrpthrpheglyglyglythrlysleuaspilelys
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglysergln
130135140
ileglnleuvalglnserglyproaspleulyslysproglygluthr
145150155160
vallysleusercyslysalaserglytyrthrphethrasnphegly
165170175
metasntrpvallysglnalaproglylysglyphelystrpmetala
180185190
trpileasnthrthrargtyrthrglyglusertyrphealaaspasp
195200205
phelysglyargphealapheservalgluthrseralathrthrala
210215220
tyrleuglnileasnasnleulysthrgluaspthralathrtyrphe
225230235240
cysalaargglygluiletyrtyrglytyraspglyglyphealatyr
245250255
trpglyglnglythrleuvalthrvalseralathrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
lysargglyarglyslysleuleutyrilephelysglnprophemet
340345350
argprovalglnthrthrglnglugluaspglycyssercysargphe
355360365
proglugluglugluglyglycysgluleuargvallyspheserarg
370375380
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
385390395400
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
405410415
argglyargaspproglumetglyglylysproglnargarglysasn
420425430
proglngluglyleutyrasngluleuglnlysasplysmetalaglu
435440445
alatyrsergluileglymetlysglygluargargargglylysgly
450455460
hisaspglyleutyrglnglyleuserthralathrlysaspthrtyr
465470475480
aspalaleuhismetglnalaleuproproargglyserglyalathr
485490495
asnpheserleuleulysglnalaglyaspvalglugluasnprogly
500505510
promettyrargmetglnleuleusercysilealaleuserleuala
515520525
leuvalthrasnserglyilehisvalpheileleuglycyspheser
530535540
alaglyleuprolysthrglualaasntrpvalasnvalileserasp
545550555560
leulyslysilegluaspleuileglnsermethisileaspalathr
565570575
leutyrthrgluseraspvalhisprosercyslysvalthralamet
580585590
lyscyspheleuleugluleuglnvalileserleugluserglyasp
595600605
alaserilehisaspthrvalgluasnleuileileleualaasnasn
610615620
serleuserserasnglyasnvalthrgluserglycyslysglucys
625630635640
glugluleugluglulysasnilelysglupheleuglnserpheval
645650655
hisilevalglnmetpheileasnthrserserglyglyglysergly
660665670
glyglyglyserglyglyglyglyserglyglyglyglyserglygly
675680685
glyserleuglnalaproargargalaargglycysargthrleugly
690695700
leuproalaleuleuleuleuleuleuleuargproproalathrarg
705710715720
glyilethrcysproproprometservalgluhisalaaspiletrp
725730735
vallyssertyrserleutyrserarggluargtyrilecysasnser
740745750
glyphelysarglysalaglythrserserleuthrglucysvalleu
755760765
asnlysalathrasnvalalahistrpthrthrproserleulyscys
770775780
ilearg
785
<210>47
<211>3019
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>47
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggatgtggtgatgacccagagccatcgctttatgagcaccagcgtgg120
gcgaccgagtgagcattacctgccgcgcgagccaggatgtgaacaccgcggtgagctggt180
atcagcagaaaccgggccagagcccgaaactgctgatttttagcgcgagctatcgctata240
ccggcgtgccggatcgctttaccggcagcggcagcggcgcggattttaccctgaccatta300
gcagcgtgcaggcggaagatctggcggtgtattattgccagcagcattatagcaccccgt360
ggacctttggcggcggcaccaaactggacattaaaggaggggggggatccgggggaggag420
gctccggcggaggcggaagccagattcagctggtgcagagcggcccggatctgaaaaaac480
cgggcgaaaccgtgaaactgagctgcaaagcgagcggctatacctttaccaactttggca540
tgaactgggtgaaacaggcgccgggcaaaggctttaaatggatggcgtggattaacacca600
cccgctataccggcgaaagctattttgcggatgattttaaaggccgctttgcgtttagcg660
tggaaaccagcgcgaccaccgcgtatctgcagattaacaacctgaaaaccgaagataccg720
cgacctatttttgcgcgcgcggcgaaatttattatggctatgatggcggctttgcgtatt780
ggggccagggcaccctggtgaccgtgagcgcgaccacgacgccagcgccgcgaccaccaa840
caccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccag900
cggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctggg960
cgcccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgca1020
aacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccagtacaaa1080
ctactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtg1140
aactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaacc1200
agctctataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagac1260
gtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcc1320
tgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaag1380
gcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccacca1440
aggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcggagccacca1500
acttcagcctgctgaagcaggccggcgacgtggaggagaaccccggccccatggccctgc1560
ccgtgaccgccctgctgctgcccctggccctgctgctgcacgccgccaggcccgatattc1620
agatgacccagagcccgagcagcctgagcgcgagcgtgggcgaccgggtgaccattacct1680
gcaaagcgagccaggatgtgggcattgcggtggcgtggtatcagcagaaaccgggcaaag1740
tgccgaaactgctgatttattgggcgagcacccgccataccggcgtgccggatcgcttta1800
gcggcagcggcagcggcaccgattttaccctgaccattagcagcctgcagccggaagatg1860
tggcgacctattattgccagcagtatagcagctatccgtatacctttggccagggcacca1920
aagtggaaattaaaggaggggggggatccgggggaggaggctccggcggaggcggaagcg1980
aagtgcagctggtggaaagcggcggcggcctggtgcagccgggcggcagcctgcgcctga2040
gctgcgcggcgagcggctttgattttagccgctattggatgagctgggtgcgccaggcgc2100
cgggcaaaggcctggaatggattggcgaaattaacccggatagcagcaccattaactatg2160
cgccgagcctgaaagataaatttattattagccgcgataacgcgaaaaacagcctgtatc2220
tgcagatgaacagcctgcgcgcggaagataccgcggtgtattattgcgcgcgcccggatg2280
gcaactattggtattttgatgtgtggggccagggcaccctggtgaccgtgagcagcacca2340
ccacccccgcccccaggccccccacccccgcccccaccatcgccagccagcccctgagcc2400
tgaggcccgaggcctgcaggcccgccgccggcggcgccgtgcacaccaggggcctggact2460
tcgcctgcgacatctacatctgggcccccctggccggcacctgcggcgtgctgctgctga2520
gcctggtgatcaccctgtactgcaagaggggcaggaagaagctgctgtacatcttcaagc2580
agcccttcatgaggcccgtgcagaccacccaggaggaggacggctgcagctgcaggttcc2640
ccgaggaggaggagggcggctgcgagctgagggtgaagttcagcaggagcgccgacgccc2700
ccgcctaccagcagggccagaaccagctgtacaacgagctgaacctgggcaggagggagg2760
agtacgacgtgctggacaagaggaggggcagggaccccgagatgggcggcaagccccaga2820
ggaggaagaacccccaggagggcctgtacaacgagctgcagaaggacaagatggccgagg2880
cctacagcgagatcggcatgaagggcgagaggaggaggggcaagggccacgacggcctgt2940
accagggcctgagcaccgccaccaaggacacctacgacgccctgcacatgcaggccctgc3000
cccccaggtaagtttaaac3019
<210>48
<211>999
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>48
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspvalvalmetthrglnserhisargphemet
202530
serthrservalglyaspargvalserilethrcysargalasergln
354045
aspvalasnthralavalsertrptyrglnglnlysproglyglnser
505560
prolysleuleuilepheseralasertyrargtyrthrglyvalpro
65707580
aspargphethrglyserglyserglyalaaspphethrleuthrile
859095
serservalglnalagluaspleualavaltyrtyrcysglnglnhis
100105110
tyrserthrprotrpthrpheglyglyglythrlysleuaspilelys
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglysergln
130135140
ileglnleuvalglnserglyproaspleulyslysproglygluthr
145150155160
vallysleusercyslysalaserglytyrthrphethrasnphegly
165170175
metasntrpvallysglnalaproglylysglyphelystrpmetala
180185190
trpileasnthrthrargtyrthrglyglusertyrphealaaspasp
195200205
phelysglyargphealapheservalgluthrseralathrthrala
210215220
tyrleuglnileasnasnleulysthrgluaspthralathrtyrphe
225230235240
cysalaargglygluiletyrtyrglytyraspglyglyphealatyr
245250255
trpglyglnglythrleuvalthrvalseralathrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
lysargglyarglyslysleuleutyrilephelysglnprophemet
340345350
argprovalglnthrthrglnglugluaspglycyssercysargphe
355360365
proglugluglugluglyglycysgluleuargvallyspheserarg
370375380
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
385390395400
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
405410415
argglyargaspproglumetglyglylysproglnargarglysasn
420425430
proglngluglyleutyrasngluleuglnlysasplysmetalaglu
435440445
alatyrsergluileglymetlysglygluargargargglylysgly
450455460
hisaspglyleutyrglnglyleuserthralathrlysaspthrtyr
465470475480
aspalaleuhismetglnalaleuproproargglyserglyalathr
485490495
asnpheserleuleulysglnalaglyaspvalglugluasnprogly
500505510
prometalaleuprovalthralaleuleuleuproleualaleuleu
515520525
leuhisalaalaargproaspileglnmetthrglnserproserser
530535540
leuseralaservalglyaspargvalthrilethrcyslysalaser
545550555560
glnaspvalglyilealavalalatrptyrglnglnlysproglylys
565570575
valprolysleuleuiletyrtrpalaserthrarghisthrglyval
580585590
proaspargpheserglyserglyserglythraspphethrleuthr
595600605
ileserserleuglnprogluaspvalalathrtyrtyrcysglngln
610615620
tyrsersertyrprotyrthrpheglyglnglythrlysvalgluile
625630635640
lysglyglyglyglyserglyglyglyglyserglyglyglyglyser
645650655
gluvalglnleuvalgluserglyglyglyleuvalglnproglygly
660665670
serleuargleusercysalaalaserglypheasppheserargtyr
675680685
trpmetsertrpvalargglnalaproglylysglyleuglutrpile
690695700
glygluileasnproaspserserthrileasntyralaproserleu
705710715720
lysasplyspheileileserargaspasnalalysasnserleutyr
725730735
leuglnmetasnserleuargalagluaspthralavaltyrtyrcys
740745750
alaargproaspglyasntyrtrptyrpheaspvaltrpglyglngly
755760765
thrleuvalthrvalserserthrthrthrproalaproargpropro
770775780
thrproalaprothrilealaserglnproleuserleuargproglu
785790795800
alacysargproalaalaglyglyalavalhisthrargglyleuasp
805810815
phealacysaspiletyriletrpalaproleualaglythrcysgly
820825830
valleuleuleuserleuvalilethrleutyrcyslysargglyarg
835840845
lyslysleuleutyrilephelysglnprophemetargprovalgln
850855860
thrthrglnglugluaspglycyssercysargpheproglugluglu
865870875880
gluglyglycysgluleuargvallyspheserargseralaaspala
885890895
proalatyrglnglnglyglnasnglnleutyrasngluleuasnleu
900905910
glyargarggluglutyraspvalleuasplysargargglyargasp
915920925
proglumetglyglylysproglnargarglysasnproglnglugly
930935940
leutyrasngluleuglnlysasplysmetalaglualatyrserglu
945950955960
ileglymetlysglygluargargargglylysglyhisaspglyleu
965970975
tyrglnglyleuserthralathrlysaspthrtyraspalaleuhis
980985990
metglnalaleuproproarg
995
<210>49
<211>3040
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>49
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggatgtggtgatgacccagagccatcgctttatgagcaccagcgtgg120
gcgaccgagtgagcattacctgccgcgcgagccaggatgtgaacaccgcggtgagctggt180
atcagcagaaaccgggccagagcccgaaactgctgatttttagcgcgagctatcgctata240
ccggcgtgccggatcgctttaccggcagcggcagcggcgcggattttaccctgaccatta300
gcagcgtgcaggcggaagatctggcggtgtattattgccagcagcattatagcaccccgt360
ggacctttggcggcggcaccaaactggacattaaaggaggggggggatccgggggaggag420
gctccggcggaggcggaagccagattcagctggtgcagagcggcccggatctgaaaaaac480
cgggcgaaaccgtgaaactgagctgcaaagcgagcggctatacctttaccaactttggca540
tgaactgggtgaaacaggcgccgggcaaaggctttaaatggatggcgtggattaacacca600
cccgctataccggcgaaagctattttgcggatgattttaaaggccgctttgcgtttagcg660
tggaaaccagcgcgaccaccgcgtatctgcagattaacaacctgaaaaccgaagataccg720
cgacctatttttgcgcgcgcggcgaaatttattatggctatgatggcggctttgcgtatt780
ggggccagggcaccctggtgaccgtgagcgcgaccacgacgccagcgccgcgaccaccaa840
caccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccag900
cggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctggg960
cgcccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgca1020
aacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccagtacaaa1080
ctactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtg1140
aactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaacc1200
agctctataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagac1260
gtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcc1320
tgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaag1380
gcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccacca1440
aggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcggagccacca1500
acttcagcctgctgaagcaggccggcgacgtggaggagaaccccggccccatggccctgc1560
ccgtgaccgccctgctgctgcccctggccctgctgctgcacgccgccaggcccgaggtcc1620
agctgcagcagtctggacctgagctgataaagcctggggcttcagtgaagatgtcctgca1680
aggcttctggatacacattcactagctatgttatgcactgggtgaagcagaagcctgggc1740
agggccttgagtggattggatatattaatccttacaatgatggtactaagtacaatgaga1800
agttcaaaggcaaggccacactgacttcagacaaatcctccagcacagcctacatggagc1860
tcagcagcctgacctctgaggactctgcggtctattactgtgcaagagggacttattact1920
acggtagtagggtatttgactactggggccaaggcaccactctcacagtctcctcaggtg1980
gagggggctcaggcggaggtggctctgggggtggaggctcggacattgtgatgactcagg2040
ctgcaccctctatacctgtcactcctggagagtcagtatccatctcctgcaggtctagta2100
agagtctcctgaatagtaatggcaacacttacttgtattggttcctgcagaggccaggcc2160
agtctcctcagctcctgatatatcggatgtccaaccttgcctcaggagtcccagacaggt2220
tcagtggcagtgggtcaggaactgctttcacactgagaatcagtagagtggaggctgagg2280
atgtgggtgtttattactgtatgcaacatctagaatatccgttcacgttcggtgctggga2340
ccaagctggagctgaaacggaccaccacccccgcccccaggccccccacccccgccccca2400
ccatcgccagccagcccctgagcctgaggcccgaggcctgcaggcccgccgccggcggcg2460
ccgtgcacaccaggggcctggacttcgcctgcgacatctacatctgggcccccctggccg2520
gcacctgcggcgtgctgctgctgagcctggtgatcaccctgtactgcaggagtaagagga2580
gcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggcccacccgca2640
agcattaccagccctatgccccaccacgcgacttcgcagcctatcgctccagggtgaagt2700
tcagcaggagcgccgacgcccccgcctaccagcagggccagaaccagctgtacaacgagc2760
tgaacctgggcaggagggaggagtacgacgtgctggacaagaggaggggcagggaccccg2820
agatgggcggcaagccccagaggaggaagaacccccaggagggcctgtacaacgagctgc2880
agaaggacaagatggccgaggcctacagcgagatcggcatgaagggcgagaggaggaggg2940
gcaagggccacgacggcctgtaccagggcctgagcaccgccaccaaggacacctacgacg3000
ccctgcacatgcaggccctgccccccaggtaagtttaaac3040
<210>50
<211>1006
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>50
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspvalvalmetthrglnserhisargphemet
202530
serthrservalglyaspargvalserilethrcysargalasergln
354045
aspvalasnthralavalsertrptyrglnglnlysproglyglnser
505560
prolysleuleuilepheseralasertyrargtyrthrglyvalpro
65707580
aspargphethrglyserglyserglyalaaspphethrleuthrile
859095
serservalglnalagluaspleualavaltyrtyrcysglnglnhis
100105110
tyrserthrprotrpthrpheglyglyglythrlysleuaspilelys
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglysergln
130135140
ileglnleuvalglnserglyproaspleulyslysproglygluthr
145150155160
vallysleusercyslysalaserglytyrthrphethrasnphegly
165170175
metasntrpvallysglnalaproglylysglyphelystrpmetala
180185190
trpileasnthrthrargtyrthrglyglusertyrphealaaspasp
195200205
phelysglyargphealapheservalgluthrseralathrthrala
210215220
tyrleuglnileasnasnleulysthrgluaspthralathrtyrphe
225230235240
cysalaargglygluiletyrtyrglytyraspglyglyphealatyr
245250255
trpglyglnglythrleuvalthrvalseralathrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
lysargglyarglyslysleuleutyrilephelysglnprophemet
340345350
argprovalglnthrthrglnglugluaspglycyssercysargphe
355360365
proglugluglugluglyglycysgluleuargvallyspheserarg
370375380
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
385390395400
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
405410415
argglyargaspproglumetglyglylysproglnargarglysasn
420425430
proglngluglyleutyrasngluleuglnlysasplysmetalaglu
435440445
alatyrsergluileglymetlysglygluargargargglylysgly
450455460
hisaspglyleutyrglnglyleuserthralathrlysaspthrtyr
465470475480
aspalaleuhismetglnalaleuproproargglyserglyalathr
485490495
asnpheserleuleulysglnalaglyaspvalglugluasnprogly
500505510
prometalaleuprovalthralaleuleuleuproleualaleuleu
515520525
leuhisalaalaargprogluvalglnleuglnglnserglyproglu
530535540
leuilelysproglyalaservallysmetsercyslysalasergly
545550555560
tyrthrphethrsertyrvalmethistrpvallysglnlysprogly
565570575
glnglyleuglutrpileglytyrileasnprotyrasnaspglythr
580585590
lystyrasnglulysphelysglylysalathrleuthrserasplys
595600605
serserserthralatyrmetgluleuserserleuthrsergluasp
610615620
seralavaltyrtyrcysalaargglythrtyrtyrtyrglyserarg
625630635640
valpheasptyrtrpglyglnglythrthrleuthrvalsersergly
645650655
glyglyglyserglyglyglyglyserglyglyglyglyseraspile
660665670
valmetthrglnalaalaproserileprovalthrproglygluser
675680685
valserilesercysargserserlysserleuleuasnserasngly
690695700
asnthrtyrleutyrtrppheleuglnargproglyglnserprogln
705710715720
leuleuiletyrargmetserasnleualaserglyvalproasparg
725730735
pheserglyserglyserglythralaphethrleuargileserarg
740745750
valglualagluaspvalglyvaltyrtyrcysmetglnhisleuglu
755760765
tyrprophethrpheglyalaglythrlysleugluleulysargthr
770775780
thrthrproalaproargproprothrproalaprothrilealaser
785790795800
glnproleuserleuargproglualacysargproalaalaglygly
805810815
alavalhisthrargglyleuaspphealacysaspiletyriletrp
820825830
alaproleualaglythrcysglyvalleuleuleuserleuvalile
835840845
thrleutyrcysargserlysargserargleuleuhisserasptyr
850855860
metasnmetthrproargargproglyprothrarglyshistyrgln
865870875880
protyralaproproargaspphealaalatyrargserargvallys
885890895
pheserargseralaaspalaproalatyrglnglnglyglnasngln
900905910
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
915920925
asplysargargglyargaspproglumetglyglylysproglnarg
930935940
arglysasnproglngluglyleutyrasngluleuglnlysasplys
945950955960
metalaglualatyrsergluileglymetlysglygluargargarg
965970975
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
980985990
aspthrtyraspalaleuhismetglnalaleuproproarg
99510001005
<210>51
<211>3022
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>51
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggatgtggtgatgacccagagccatcgctttatgagcaccagcgtgg120
gcgaccgagtgagcattacctgccgcgcgagccaggatgtgaacaccgcggtgagctggt180
atcagcagaaaccgggccagagcccgaaactgctgatttttagcgcgagctatcgctata240
ccggcgtgccggatcgctttaccggcagcggcagcggcgcggattttaccctgaccatta300
gcagcgtgcaggcggaagatctggcggtgtattattgccagcagcattatagcaccccgt360
ggacctttggcggcggcaccaaactggacattaaaggaggggggggatccgggggaggag420
gctccggcggaggcggaagccagattcagctggtgcagagcggcccggatctgaaaaaac480
cgggcgaaaccgtgaaactgagctgcaaagcgagcggctatacctttaccaactttggca540
tgaactgggtgaaacaggcgccgggcaaaggctttaaatggatggcgtggattaacacca600
cccgctataccggcgaaagctattttgcggatgattttaaaggccgctttgcgtttagcg660
tggaaaccagcgcgaccaccgcgtatctgcagattaacaacctgaaaaccgaagataccg720
cgacctatttttgcgcgcgcggcgaaatttattatggctatgatggcggctttgcgtatt780
ggggccagggcaccctggtgaccgtgagcgcgaccacgacgccagcgccgcgaccaccaa840
caccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccag900
cggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctggg960
cgcccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgca1020
aacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccagtacaaa1080
ctactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtg1140
aactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaacc1200
agctctataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagac1260
gtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcc1320
tgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaag1380
gcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccacca1440
aggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcggagccacca1500
acttcagcctgctgaagcaggccggcgacgtggaggagaaccccggccccatggccctgc1560
ccgtgaccgccctgctgctgcccctggccctgctgctgcacgccgccaggcccgacatcc1620
agatgacacagactacatcctccctgtctgcctctctgggagacagagtcaccatcagtt1680
gcagggcaagtcaggacattagtaaatatttaaattggtatcagcagaaaccagatggaa1740
ctgttaaactcctgatctaccatacatcaagattacactcaggagtcccatcaaggttca1800
gtggcagtgggtctggaacagattattctctcaccattagcaacctggagcaagaagata1860
ttgccacttacttttgccaacagggtaatacgcttccgtacacgttcggaggggggacca1920
agctggagatcacaggtggcggtggctcgggcggtggtgggtcgggtggcggcggatctg1980
aggtgaaactgcaggagtcaggacctggcctggtggcgccctcacagagcctgtccgtca2040
catgcactgtctcaggggtctcattacccgactatggtgtaagctggattcgccagcctc2100
cacgaaagggtctggagtggctgggagtaatatggggtagtgaaaccacatactataatt2160
cagctctcaaatccagactgaccatcatcaaggacaactccaagagccaagttttcttaa2220
aaatgaacagtctgcaaactgatgacacagccatttactactgtgccaaacattattact2280
acggtggtagctatgctatggactactggggccaaggaacctcagtcaccgtctcctcaa2340
ccaccacccccgcccccaggccccccacccccgcccccaccatcgccagccagcccctga2400
gcctgaggcccgaggcctgcaggcccgccgccggcggcgccgtgcacaccaggggcctgg2460
acttcgcctgcgacatctacatctgggcccccctggccggcacctgcggcgtgctgctgc2520
tgagcctggtgatcaccctgtactgcaagaggggcaggaagaagctgctgtacatcttca2580
agcagcccttcatgaggcccgtgcagaccacccaggaggaggacggctgcagctgcaggt2640
tccccgaggaggaggagggcggctgcgagctgagggtgaagttcagcaggagcgccgacg2700
cccccgcctaccagcagggccagaaccagctgtacaacgagctgaacctgggcaggaggg2760
aggagtacgacgtgctggacaagaggaggggcagggaccccgagatgggcggcaagcccc2820
agaggaggaagaacccccaggagggcctgtacaacgagctgcagaaggacaagatggccg2880
aggcctacagcgagatcggcatgaagggcgagaggaggaggggcaagggccacgacggcc2940
tgtaccagggcctgagcaccgccaccaaggacacctacgacgccctgcacatgcaggccc3000
tgccccccaggtaagtttaaac3022
<210>52
<211>1000
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>52
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspvalvalmetthrglnserhisargphemet
202530
serthrservalglyaspargvalserilethrcysargalasergln
354045
aspvalasnthralavalsertrptyrglnglnlysproglyglnser
505560
prolysleuleuilepheseralasertyrargtyrthrglyvalpro
65707580
aspargphethrglyserglyserglyalaaspphethrleuthrile
859095
serservalglnalagluaspleualavaltyrtyrcysglnglnhis
100105110
tyrserthrprotrpthrpheglyglyglythrlysleuaspilelys
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglysergln
130135140
ileglnleuvalglnserglyproaspleulyslysproglygluthr
145150155160
vallysleusercyslysalaserglytyrthrphethrasnphegly
165170175
metasntrpvallysglnalaproglylysglyphelystrpmetala
180185190
trpileasnthrthrargtyrthrglyglusertyrphealaaspasp
195200205
phelysglyargphealapheservalgluthrseralathrthrala
210215220
tyrleuglnileasnasnleulysthrgluaspthralathrtyrphe
225230235240
cysalaargglygluiletyrtyrglytyraspglyglyphealatyr
245250255
trpglyglnglythrleuvalthrvalseralathrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
lysargglyarglyslysleuleutyrilephelysglnprophemet
340345350
argprovalglnthrthrglnglugluaspglycyssercysargphe
355360365
proglugluglugluglyglycysgluleuargvallyspheserarg
370375380
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
385390395400
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
405410415
argglyargaspproglumetglyglylysproglnargarglysasn
420425430
proglngluglyleutyrasngluleuglnlysasplysmetalaglu
435440445
alatyrsergluileglymetlysglygluargargargglylysgly
450455460
hisaspglyleutyrglnglyleuserthralathrlysaspthrtyr
465470475480
aspalaleuhismetglnalaleuproproargglyserglyalathr
485490495
asnpheserleuleulysglnalaglyaspvalglugluasnprogly
500505510
prometalaleuprovalthralaleuleuleuproleualaleuleu
515520525
leuhisalaalaargproaspileglnmetthrglnthrthrserser
530535540
leuseralaserleuglyaspargvalthrilesercysargalaser
545550555560
glnaspileserlystyrleuasntrptyrglnglnlysproaspgly
565570575
thrvallysleuleuiletyrhisthrserargleuhisserglyval
580585590
proserargpheserglyserglyserglythrasptyrserleuthr
595600605
ileserasnleugluglngluaspilealathrtyrphecysglngln
610615620
glyasnthrleuprotyrthrpheglyglyglythrlysleugluile
625630635640
thrglyglyglyglyserglyglyglyglyserglyglyglyglyser
645650655
gluvallysleuglngluserglyproglyleuvalalaprosergln
660665670
serleuservalthrcysthrvalserglyvalserleuproasptyr
675680685
glyvalsertrpileargglnproproarglysglyleuglutrpleu
690695700
glyvaliletrpglysergluthrthrtyrtyrasnseralaleulys
705710715720
serargleuthrileilelysaspasnserlysserglnvalpheleu
725730735
lysmetasnserleuglnthraspaspthralailetyrtyrcysala
740745750
lyshistyrtyrtyrglyglysertyralametasptyrtrpglygln
755760765
glythrservalthrvalserserthrthrthrproalaproargpro
770775780
prothrproalaprothrilealaserglnproleuserleuargpro
785790795800
glualacysargproalaalaglyglyalavalhisthrargglyleu
805810815
aspphealacysaspiletyriletrpalaproleualaglythrcys
820825830
glyvalleuleuleuserleuvalilethrleutyrcyslysarggly
835840845
arglyslysleuleutyrilephelysglnprophemetargproval
850855860
glnthrthrglnglugluaspglycyssercysargpheprogluglu
865870875880
glugluglyglycysgluleuargvallyspheserargseralaasp
885890895
alaproalatyrglnglnglyglnasnglnleutyrasngluleuasn
900905910
leuglyargarggluglutyraspvalleuasplysargargglyarg
915920925
aspproglumetglyglylysproglnargarglysasnproglnglu
930935940
glyleutyrasngluleuglnlysasplysmetalaglualatyrser
945950955960
gluileglymetlysglygluargargargglylysglyhisaspgly
965970975
leutyrglnglyleuserthralathrlysaspthrtyraspalaleu
980985990
hismetglnalaleuproproarg
9951000
<210>53
<211>1483
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>53
gcgatcgcaccatggccctgcccgtgaccgccctgctgctgcccctggccctgctgctgc60
acgccgccaggcccgacatccagatgacacagactacatcctccctgtctgcctctctgg120
gagacagagtcaccatcagttgcagggcaagtcaggacattagtaaatatttaaattggt180
atcagcagaaaccagatggaactgttaaactcctgatctaccatacatcaagattacact240
caggagtcccatcaaggttcagtggcagtgggtctggaacagattattctctcaccatta300
gcaacctggagcaagaagatattgccacttacttttgccaacagggtaatacgcttccgt360
acacgttcggaggggggaccaagctggagatcacaggtggcggtggctcgggcggtggtg420
ggtcgggtggcggcggatctgaggtgaaactgcaggagtcaggacctggcctggtggcgc480
cctcacagagcctgtccgtcacatgcactgtctcaggggtctcattacccgactatggtg540
taagctggattcgccagcctccacgaaagggtctggagtggctgggagtaatatggggta600
gtgaaaccacatactataattcagctctcaaatccagactgaccatcatcaaggacaact660
ccaagagccaagttttcttaaaaatgaacagtctgcaaactgatgacacagccatttact720
actgtgccaaacattattactacggtggtagctatgctatggactactggggccaaggaa780
cctcagtcaccgtctcctcaaccacgacgccagcgccgcgaccaccaacaccggcgccca840
ccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcg900
cagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccg960
ggacttgtggggtccttctcctgtcactggttatcaccctttactgcaaacggggcagaa1020
agaaactcctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagagg1080
aagatggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtga1140
agttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacg1200
agctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggacc1260
ctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaac1320
tgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccgga1380
ggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacg1440
acgcccttcacatgcaggccctgccccctcgctaagtttaaac1483
<210>54
<211>487
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>54
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspileglnmetthrglnthrthrserserleu
202530
seralaserleuglyaspargvalthrilesercysargalasergln
354045
aspileserlystyrleuasntrptyrglnglnlysproaspglythr
505560
vallysleuleuiletyrhisthrserargleuhisserglyvalpro
65707580
serargpheserglyserglyserglythrasptyrserleuthrile
859095
serasnleugluglngluaspilealathrtyrphecysglnglngly
100105110
asnthrleuprotyrthrpheglyglyglythrlysleugluilethr
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglyserglu
130135140
vallysleuglngluserglyproglyleuvalalaproserglnser
145150155160
leuservalthrcysthrvalserglyvalserleuproasptyrgly
165170175
valsertrpileargglnproproarglysglyleuglutrpleugly
180185190
valiletrpglysergluthrthrtyrtyrasnseralaleulysser
195200205
argleuthrileilelysaspasnserlysserglnvalpheleulys
210215220
metasnserleuglnthraspaspthralailetyrtyrcysalalys
225230235240
histyrtyrtyrglyglysertyralametasptyrtrpglyglngly
245250255
thrservalthrvalserserthrthrthrproalaproargpropro
260265270
thrproalaprothrilealaserglnproleuserleuargproglu
275280285
alacysargproalaalaglyglyalavalhisthrargglyleuasp
290295300
phealacysaspiletyriletrpalaproleualaglythrcysgly
305310315320
valleuleuleuserleuvalilethrleutyrcyslysargglyarg
325330335
lyslysleuleutyrilephelysglnprophemetargprovalgln
340345350
thrthrglnglugluaspglycyssercysargpheproglugluglu
355360365
gluglyglycysgluleuargvallyspheserargseralaaspala
370375380
proalatyrglnglnglyglnasnglnleutyrasngluleuasnleu
385390395400
glyargarggluglutyraspvalleuasplysargargglyargasp
405410415
proglumetglyglylysproglnargarglysasnproglnglugly
420425430
leutyrasngluleuglnlysasplysmetalaglualatyrserglu
435440445
ileglymetlysglygluargargargglylysglyhisaspglyleu
450455460
tyrglnglyleuserthralathrlysaspthrtyraspalaleuhis
465470475480
metglnalaleuproproarg
485
<210>55
<211>1501
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>55
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggaggtccagctgcagcagtctggacctgagctgataaagcctggggctt120
cagtgaagatgtcctgcaaggcttctggatacacattcactagctatgttatgcactggg180
tgaagcagaagcctgggcagggccttgagtggattggatatattaatccttacaatgatg240
gtactaagtacaatgagaagttcaaaggcaaggccacactgacttcagacaaatcctcca300
gcacagcctacatggagctcagcagcctgacctctgaggactctgcggtctattactgtg360
caagagggacttattactacggtagtagggtatttgactactggggccaaggcaccactc420
tcacagtctcctcaggtggagggggctcaggcggaggtggctctgggggtggaggctcgg480
acattgtgatgactcaggctgcaccctctatacctgtcactcctggagagtcagtatcca540
tctcctgcaggtctagtaagagtctcctgaatagtaatggcaacacttacttgtattggt600
tcctgcagaggccaggccagtctcctcagctcctgatatatcggatgtccaaccttgcct660
caggagtcccagacaggttcagtggcagtgggtcaggaactgctttcacactgagaatca720
gtagagtggaggctgaggatgtgggtgtttattactgtatgcaacatctagaatatccgt780
tcacgttcggtgctgggaccaagctggagctgaaacggaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcaaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccag1080
tacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggag1140
gatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggcc1200
agaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttggaca1260
agagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcagg1320
aaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattggga1380
tgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacag1440
ccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgctaagtttaaa1500
c1501
<210>56
<211>494
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>56
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprogluvalglnleuglnglnserglyprogluleu
202530
ilelysproglyalaservallysmetsercyslysalaserglytyr
354045
thrphethrsertyrvalmethistrpvallysglnlysproglygln
505560
glyleuglutrpileglytyrileasnprotyrasnaspglythrlys
65707580
tyrasnglulysphelysglylysalathrleuthrserasplysser
859095
serserthralatyrmetgluleuserserleuthrsergluaspser
100105110
alavaltyrtyrcysalaargglythrtyrtyrtyrglyserargval
115120125
pheasptyrtrpglyglnglythrthrleuthrvalserserglygly
130135140
glyglyserglyglyglyglyserglyglyglyglyseraspileval
145150155160
metthrglnalaalaproserileprovalthrproglygluserval
165170175
serilesercysargserserlysserleuleuasnserasnglyasn
180185190
thrtyrleutyrtrppheleuglnargproglyglnserproglnleu
195200205
leuiletyrargmetserasnleualaserglyvalproaspargphe
210215220
serglyserglyserglythralaphethrleuargileserargval
225230235240
glualagluaspvalglyvaltyrtyrcysmetglnhisleuglutyr
245250255
prophethrpheglyalaglythrlysleugluleulysargthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcyslysargglyarglyslysleuleutyrilephelysgln
340345350
prophemetargprovalglnthrthrglnglugluaspglycysser
355360365
cysargpheproglugluglugluglyglycysgluleuargvallys
370375380
pheserargseralaaspalaproalatyrglnglnglyglnasngln
385390395400
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
405410415
asplysargargglyargaspproglumetglyglylysproglnarg
420425430
arglysasnproglngluglyleutyrasngluleuglnlysasplys
435440445
metalaglualatyrsergluileglymetlysglygluargargarg
450455460
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
465470475480
aspthrtyraspalaleuhismetglnalaleuproproarg
485490
<210>57
<211>2293
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>57
gcgatcgcaccatggccctgcccgtgaccgccctgctgctgcccctggccctgctgctgc60
acgccgccaggcccgacattcagctgacccagtctccagcaatcctttctgcatctccag120
gggagaaggtcacaatgacttgcagggccagctcaagtttaagtttcatgcactggtacc180
agcagaagccaggatcctcccccaaaccctggatttatgccacatccaacctggcttctg240
gagtccctgctcgcttcagtggcagtgggtctgggacctcttactctctcacaatcagca300
cagtggaggctgaagatgctgcctcttatttctgccatcagtggagtagtaacccgctca360
cgttcggtgctgggaccaagctggagatcagctcgggcggcggcggctcgggcggcggcg420
gctccggagacgtcatgggggtggattctgggggaggcttagtgcagcctggagggtccc480
ggaaactctcctgtgcagcccctggattcactttcagtagctttgggatgcactgggttc540
gtcaggctccagagaaggggttggagtgggtcgcatacattagtagtcccagtagtaccc600
tccactatgcagacagagtgaagggccgattcaccatctccagagacaatcccaagaaca660
ccctgttcctgcaaatgaaactaccctcactatgctatggactactggggccaagggacc720
acgttcacaccaccacccccgcccccaggccccccacccccgcccccaccatcgccagcc780
agcccctgagcctgaggcccgaggcctgcaggcccgccgccggcggcgccgtgcacacca840
ggggcctggacttcgcctgcgacatctacatctgggcccccctggccggcacctgcggcg900
tgctgctgctgagcctggtgatcaccctgtactgcaggagtaagaggagcaggctcctgc960
acagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcattaccagc1020
cctatgccccaccacgcgacttcgcagcctatcgctccagggtgaagttcagcaggagcg1080
ccgacgcccccgcctaccagcagggccagaaccagctgtacaacgagctgaacctgggca1140
ggagggaggagtacgacgtgctggacaagaggaggggcagggaccccgagatgggcggca1200
agccccagaggaggaagaacccccaggagggcctgtacaacgagctgcagaaggacaaga1260
tggccgaggcctacagcgagatcggcatgaagggcgagaggaggaggggcaagggccacg1320
acggcctgtaccagggcctgagcaccgccaccaaggacacctacgacgccctgcacatgc1380
aggccctgccccccaggggaagcggagccaccaacttcagcctgctgaagcaggccggcg1440
acgtggaggagaaccccggccccatgtacagaatgcagctgctgagctgcatcgccctga1500
gcctggccctggtgaccaacagcggcatccacgtgttcatcctgggctgcttcagcgccg1560
gcctgcccaagaccgaggccaactgggtgaacgtgatcagcgacctgaagaagatcgagg1620
acctgatccagagcatgcacatcgacgccaccctgtacaccgagagcgacgtgcacccca1680
gctgcaaggtgaccgccatgaagtgcttcctgctggagctgcaggtgatcagcctggaga1740
gcggcgacgccagcatccacgacaccgtggagaacctgatcatcctggccaacaacagcc1800
tgagcagcaacggcaacgtgaccgagagcggctgcaaggagtgcgaggagctggaggaga1860
agaacatcaaggagttcctgcagagcttcgtgcacatcgtgcagatgttcatcaacacca1920
gctccggcggcggctccggcggcggcggctccggcggcggcggctccggcggcggcggct1980
ccggcggcggctccctgcaggcccccagaagagccagaggctgcagaaccctgggcctgc2040
ccgccctgctgctgctgctgctgctgagaccccccgccaccagaggcatcacctgccccc2100
cccccatgagcgtggagcacgccgacatctgggtgaagagctacagcctgtacagcagag2160
agagatacatctgcaacagcggcttcaagagaaaggccggcaccagcagcctgaccgagt2220
gcgtgctgaacaaggccaccaacgtggcccactggaccacccccagcctgaagtgcatca2280
gataagtttaaac2293
<210>58
<211>757
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>58
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspileglnleuthrglnserproalaileleu
202530
seralaserproglyglulysvalthrmetthrcysargalaserser
354045
serleuserphemethistrptyrglnglnlysproglyserserpro
505560
lysprotrpiletyralathrserasnleualaserglyvalproala
65707580
argpheserglyserglyserglythrsertyrserleuthrileser
859095
thrvalglualagluaspalaalasertyrphecyshisglntrpser
100105110
serasnproleuthrpheglyalaglythrlysleugluileserser
115120125
glyglyglyglyserglyglyglyglyserglyaspvalmetglyval
130135140
aspserglyglyglyleuvalglnproglyglyserarglysleuser
145150155160
cysalaalaproglyphethrpheserserpheglymethistrpval
165170175
argglnalaproglulysglyleuglutrpvalalatyrileserser
180185190
proserserthrleuhistyralaaspargvallysglyargphethr
195200205
ileserargaspasnprolysasnthrleupheleuglnmetlysleu
210215220
proserleucystyrglyleuleuglyproargasphisvalhisthr
225230235240
thrthrproalaproargproprothrproalaprothrilealaser
245250255
glnproleuserleuargproglualacysargproalaalaglygly
260265270
alavalhisthrargglyleuaspphealacysaspiletyriletrp
275280285
alaproleualaglythrcysglyvalleuleuleuserleuvalile
290295300
thrleutyrcysargserlysargserargleuleuhisserasptyr
305310315320
metasnmetthrproargargproglyprothrarglyshistyrgln
325330335
protyralaproproargaspphealaalatyrargserargvallys
340345350
pheserargseralaaspalaproalatyrglnglnglyglnasngln
355360365
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
370375380
asplysargargglyargaspproglumetglyglylysproglnarg
385390395400
arglysasnproglngluglyleutyrasngluleuglnlysasplys
405410415
metalaglualatyrsergluileglymetlysglygluargargarg
420425430
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
435440445
aspthrtyraspalaleuhismetglnalaleuproproargglyser
450455460
glyalathrasnpheserleuleulysglnalaglyaspvalgluglu
465470475480
asnproglypromettyrargmetglnleuleusercysilealaleu
485490495
serleualaleuvalthrasnserglyilehisvalpheileleugly
500505510
cyspheseralaglyleuprolysthrglualaasntrpvalasnval
515520525
ileseraspleulyslysilegluaspleuileglnsermethisile
530535540
aspalathrleutyrthrgluseraspvalhisprosercyslysval
545550555560
thralametlyscyspheleuleugluleuglnvalileserleuglu
565570575
serglyaspalaserilehisaspthrvalgluasnleuileileleu
580585590
alaasnasnserleuserserasnglyasnvalthrgluserglycys
595600605
lysglucysglugluleugluglulysasnilelysglupheleugln
610615620
serphevalhisilevalglnmetpheileasnthrserserglygly
625630635640
glyserglyglyglyglyserglyglyglyglyserglyglyglygly
645650655
serglyglyglyserleuglnalaproargargalaargglycysarg
660665670
thrleuglyleuproalaleuleuleuleuleuleuleuargpropro
675680685
alathrargglyilethrcysproproprometservalgluhisala
690695700
aspiletrpvallyssertyrserleutyrserarggluargtyrile
705710715720
cysasnserglyphelysarglysalaglythrserserleuthrglu
725730735
cysvalleuasnlysalathrasnvalalahistrpthrthrproser
740745750
leulyscysilearg
755
<210>59
<211>2389
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>59
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggaggtccagctgcagcagtctggacctgagctgataaagcctgggg120
cttcagtgaagatgtcctgcaaggcttctggatacacattcactagctatgttatgcact180
gggtgaagcagaagcctgggcagggccttgagtggattggatatattaatccttacaatg240
atggtactaagtacaatgagaagttcaaaggcaaggccacactgacttcagacaaatcct300
ccagcacagcctacatggagctcagcagcctgacctctgaggactctgcggtctattact360
gtgcaagagggacttattactacggtagtagggtatttgactactggggccaaggcacca420
ctctcacagtctcctcaggtggagggggctcaggcggaggtggctctgggggtggaggct480
cggacattgtgatgactcaggctgcaccctctatacctgtcactcctggagagtcagtat540
ccatctcctgcaggtctagtaagagtctcctgaatagtaatggcaacacttacttgtatt600
ggttcctgcagaggccaggccagtctcctcagctcctgatatatcggatgtccaaccttg660
cctcaggagtcccagacaggttcagtggcagtgggtcaggaactgctttcacactgagaa720
tcagtagagtggaggctgaggatgtgggtgtttattactgtatgcaacatctagaatatc780
cgttcacgttcggtgctgggaccaagctggagctgaaacggaccacgacgccagcgccgc840
gaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgt900
gccggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatct960
acatctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcaccc1020
tttactgcaaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagac1080
cagtacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaag1140
gaggatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagg1200
gccagaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttgg1260
acaagagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctc1320
aggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattg1380
ggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagta1440
cagccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcg1500
gagccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggcccca1560
tgtacagaatgcagctgctgagctgcatcgccctgagcctggccctggtgaccaacagcg1620
gcatccacgtgttcatcctgggctgcttcagcgccggcctgcccaagaccgaggccaact1680
gggtgaacgtgatcagcgacctgaagaagatcgaggacctgatccagagcatgcacatcg1740
acgccaccctgtacaccgagagcgacgtgcaccccagctgcaaggtgaccgccatgaagt1800
gcttcctgctggagctgcaggtgatcagcctggagagcggcgacgccagcatccacgaca1860
ccgtggagaacctgatcatcctggccaacaacagcctgagcagcaacggcaacgtgaccg1920
agagcggctgcaaggagtgcgaggagctggaggagaagaacatcaaggagttcctgcaga1980
gcttcgtgcacatcgtgcagatgttcatcaacaccagctccggcggcggctccggcggcg2040
gcggctccggcggcggcggctccggcggcggcggctccggcggcggctccctgcaggccc2100
ccagaagagccagaggctgcagaaccctgggcctgcccgccctgctgctgctgctgctgc2160
tgagaccccccgccaccagaggcatcacctgccccccccccatgagcgtggagcacgccg2220
acatctgggtgaagagctacagcctgtacagcagagagagatacatctgcaacagcggct2280
tcaagagaaaggccggcaccagcagcctgaccgagtgcgtgctgaacaaggccaccaacg2340
tggcccactggaccacccccagcctgaagtgcatcagataagtttaaac2389
<210>60
<211>789
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>60
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprogluvalglnleuglnglnserglyprogluleu
202530
ilelysproglyalaservallysmetsercyslysalaserglytyr
354045
thrphethrsertyrvalmethistrpvallysglnlysproglygln
505560
glyleuglutrpileglytyrileasnprotyrasnaspglythrlys
65707580
tyrasnglulysphelysglylysalathrleuthrserasplysser
859095
serserthralatyrmetgluleuserserleuthrsergluaspser
100105110
alavaltyrtyrcysalaargglythrtyrtyrtyrglyserargval
115120125
pheasptyrtrpglyglnglythrthrleuthrvalserserglygly
130135140
glyglyserglyglyglyglyserglyglyglyglyseraspileval
145150155160
metthrglnalaalaproserileprovalthrproglygluserval
165170175
serilesercysargserserlysserleuleuasnserasnglyasn
180185190
thrtyrleutyrtrppheleuglnargproglyglnserproglnleu
195200205
leuiletyrargmetserasnleualaserglyvalproaspargphe
210215220
serglyserglyserglythralaphethrleuargileserargval
225230235240
glualagluaspvalglyvaltyrtyrcysmetglnhisleuglutyr
245250255
prophethrpheglyalaglythrlysleugluleulysargthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcyslysargglyarglyslysleuleutyrilephelysgln
340345350
prophemetargprovalglnthrthrglnglugluaspglycysser
355360365
cysargpheproglugluglugluglyglycysgluleuargvallys
370375380
pheserargseralaaspalaproalatyrglnglnglyglnasngln
385390395400
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
405410415
asplysargargglyargaspproglumetglyglylysproglnarg
420425430
arglysasnproglngluglyleutyrasngluleuglnlysasplys
435440445
metalaglualatyrsergluileglymetlysglygluargargarg
450455460
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
465470475480
aspthrtyraspalaleuhismetglnalaleuproproargglyser
485490495
glyalathrasnpheserleuleulysglnalaglyaspvalgluglu
500505510
asnproglypromettyrargmetglnleuleusercysilealaleu
515520525
serleualaleuvalthrasnserglyilehisvalpheileleugly
530535540
cyspheseralaglyleuprolysthrglualaasntrpvalasnval
545550555560
ileseraspleulyslysilegluaspleuileglnsermethisile
565570575
aspalathrleutyrthrgluseraspvalhisprosercyslysval
580585590
thralametlyscyspheleuleugluleuglnvalileserleuglu
595600605
serglyaspalaserilehisaspthrvalgluasnleuileileleu
610615620
alaasnasnserleuserserasnglyasnvalthrgluserglycys
625630635640
lysglucysglugluleugluglulysasnilelysglupheleugln
645650655
serphevalhisilevalglnmetpheileasnthrserserglygly
660665670
glyserglyglyglyglyserglyglyglyglyserglyglyglygly
675680685
serglyglyglyserleuglnalaproargargalaargglycysarg
690695700
thrleuglyleuproalaleuleuleuleuleuleuleuargpropro
705710715720
alathrargglyilethrcysproproprometservalgluhisala
725730735
aspiletrpvallyssertyrserleutyrserarggluargtyrile
740745750
cysasnserglyphelysarglysalaglythrserserleuthrglu
755760765
cysvalleuasnlysalathrasnvalalahistrpthrthrproser
770775780
leulyscysilearg
785
<210>61
<211>2389
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>61
gcgatcgcaccatggccctgcccgtgaccgccctgctgctgcccctggccctgctgctgc60
acgccgccaggcccgatatccagatgacccagagccccagctccctgtccgcatccgtgg120
gcgacagagtgacaattacctgtagaagcagccaaagcatcgtgcatagcgtcggcaaca180
cttttctggagtggtatcaacagaagcccgggaaggcccccaaactgctgatctacaagg240
tgagcaacagattcagcggggtcccaagcagattctccggcagcggctccgggactgact300
tcaccctgaccattagcagcctgcagccagaggacttcgccacatactactgcttccaag360
ggagccagttcccctacaccttcggccaaggcactaaggtggagatcaaaggggggggag420
gaagcggcggaggagggagcggaggcgggggatccgaagtgcaactggtcgaatccggag480
gggggctggtccagcctggagggtccctgagactgagctgcgccgcaagcggctacgagt540
tctccaggtcctggatgaactgggtgaggcaggccccaggaaaagggctggaatgggtgg600
gcaggatctaccctggcgacggcgataccaactactccggaaagttcaagggcaggttca660
ctatcagcgccgacactagcaagaataccgcctacctgcagatgaatagcctgagggccg720
aggacaccgccgtgtattactgcgctagagacggcagcagctgggattggtacttcgacg780
tgtggggccagggcactctggtgactgtgagcagcaccaccacccccgcccccaggcccc840
ccacccccgcccccaccatcgccagccagcccctgagcctgaggcccgaggcctgcaggc900
ccgccgccggcggcgccgtgcacaccaggggcctggacttcgcctgcgacttttgggtgc960
tggtggtggttggtggagtcctggcttgctatagcttgctagtaacagtggcctttatta1020
ttttctgggtgaggagtaagaggagcaggctcctgcacagtgactacatgaacatgactc1080
cccgccgccccgggcccacccgcaagcattaccagccctatgccccaccacgcgacttcg1140
cagcctatcgctccagggtgaagttcagcaggagcgccgacgcccccgcctaccagcagg1200
gccagaaccagctgtacaacgagctgaacctgggcaggagggaggagtacgacgtgctgg1260
acaagaggaggggcagggaccccgagatgggcggcaagccccagaggaggaagaaccccc1320
aggagggcctgtacaacgagctgcagaaggacaagatggccgaggcctacagcgagatcg1380
gcatgaagggcgagaggaggaggggcaagggccacgacggcctgtaccagggcctgagca1440
ccgccaccaaggacacctacgacgccctgcacatgcaggccctgccccccaggggaagcg1500
gagccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggcccca1560
tgtacagaatgcagctgctgagctgcatcgccctgagcctggccctggtgaccaacagcg1620
gcatccacgtgttcatcctgggctgcttcagcgccggcctgcccaagaccgaggccaact1680
gggtgaacgtgatcagcgacctgaagaagatcgaggacctgatccagagcatgcacatcg1740
acgccaccctgtacaccgagagcgacgtgcaccccagctgcaaggtgaccgccatgaagt1800
gcttcctgctggagctgcaggtgatcagcctggagagcggcgacgccagcatccacgaca1860
ccgtggagaacctgatcatcctggccaacaacagcctgagcagcaacggcaacgtgaccg1920
agagcggctgcaaggagtgcgaggagctggaggagaagaacatcaaggagttcctgcaga1980
gcttcgtgcacatcgtgcagatgttcatcaacaccagctccggcggcggctccggcggcg2040
gcggctccggcggcggcggctccggcggcggcggctccggcggcggctccctgcaggccc2100
ccagaagagccagaggctgcagaaccctgggcctgcccgccctgctgctgctgctgctgc2160
tgagaccccccgccaccagaggcatcacctgccccccccccatgagcgtggagcacgccg2220
acatctgggtgaagagctacagcctgtacagcagagagagatacatctgcaacagcggct2280
tcaagagaaaggccggcaccagcagcctgaccgagtgcgtgctgaacaaggccaccaacg2340
tggcccactggaccacccccagcctgaagtgcatcagataagtttaaac2389
<210>62
<211>789
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>62
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspileglnmetthrglnserproserserleu
202530
seralaservalglyaspargvalthrilethrcysargsersergln
354045
serilevalhisservalglyasnthrpheleuglutrptyrglngln
505560
lysproglylysalaprolysleuleuiletyrlysvalserasnarg
65707580
pheserglyvalproserargpheserglyserglyserglythrasp
859095
phethrleuthrileserserleuglnprogluaspphealathrtyr
100105110
tyrcyspheglnglyserglnpheprotyrthrpheglyglnglythr
115120125
lysvalgluilelysglyglyglyglyserglyglyglyglysergly
130135140
glyglyglysergluvalglnleuvalgluserglyglyglyleuval
145150155160
glnproglyglyserleuargleusercysalaalaserglytyrglu
165170175
pheserargsertrpmetasntrpvalargglnalaproglylysgly
180185190
leuglutrpvalglyargiletyrproglyaspglyaspthrasntyr
195200205
serglylysphelysglyargphethrileseralaaspthrserlys
210215220
asnthralatyrleuglnmetasnserleuargalagluaspthrala
225230235240
valtyrtyrcysalaargaspglysersertrpasptrptyrpheasp
245250255
valtrpglyglnglythrleuvalthrvalserserthrthrthrpro
260265270
alaproargproprothrproalaprothrilealaserglnproleu
275280285
serleuargproglualacysargproalaalaglyglyalavalhis
290295300
thrargglyleuaspphealacysaspphetrpvalleuvalvalval
305310315320
glyglyvalleualacystyrserleuleuvalthrvalalapheile
325330335
ilephetrpvalargserlysargserargleuleuhisserasptyr
340345350
metasnmetthrproargargproglyprothrarglyshistyrgln
355360365
protyralaproproargaspphealaalatyrargserargvallys
370375380
pheserargseralaaspalaproalatyrglnglnglyglnasngln
385390395400
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
405410415
asplysargargglyargaspproglumetglyglylysproglnarg
420425430
arglysasnproglngluglyleutyrasngluleuglnlysasplys
435440445
metalaglualatyrsergluileglymetlysglygluargargarg
450455460
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
465470475480
aspthrtyraspalaleuhismetglnalaleuproproargglyser
485490495
glyalathrasnpheserleuleulysglnalaglyaspvalgluglu
500505510
asnproglypromettyrargmetglnleuleusercysilealaleu
515520525
serleualaleuvalthrasnserglyilehisvalpheileleugly
530535540
cyspheseralaglyleuprolysthrglualaasntrpvalasnval
545550555560
ileseraspleulyslysilegluaspleuileglnsermethisile
565570575
aspalathrleutyrthrgluseraspvalhisprosercyslysval
580585590
thralametlyscyspheleuleugluleuglnvalileserleuglu
595600605
serglyaspalaserilehisaspthrvalgluasnleuileileleu
610615620
alaasnasnserleuserserasnglyasnvalthrgluserglycys
625630635640
lysglucysglugluleugluglulysasnilelysglupheleugln
645650655
serphevalhisilevalglnmetpheileasnthrserserglygly
660665670
glyserglyglyglyglyserglyglyglyglyserglyglyglygly
675680685
serglyglyglyserleuglnalaproargargalaargglycysarg
690695700
thrleuglyleuproalaleuleuleuleuleuleuleuargpropro
705710715720
alathrargglyilethrcysproproprometservalgluhisala
725730735
aspiletrpvallyssertyrserleutyrserarggluargtyrile
740745750
cysasnserglyphelysarglysalaglythrserserleuthrglu
755760765
cysvalleuasnlysalathrasnvalalahistrpthrthrproser
770775780
leulyscysilearg
785
<210>63
<211>3049
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>63
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggaggtccagctgcagcagtctggacctgagctgataaagcctggggctt120
cagtgaagatgtcctgcaaggcttctggatacacattcactagctatgttatgcactggg180
tgaagcagaagcctgggcagggccttgagtggattggatatattaatccttacaatgatg240
gtactaagtacaatgagaagttcaaaggcaaggccacactgacttcagacaaatcctcca300
gcacagcctacatggagctcagcagcctgacctctgaggactctgcggtctattactgtg360
caagagggacttattactacggtagtagggtatttgactactggggccaaggcaccactc420
tcacagtctcctcaggtggagggggctcaggcggaggtggctctgggggtggaggctcgg480
acattgtgatgactcaggctgcaccctctatacctgtcactcctggagagtcagtatcca540
tctcctgcaggtctagtaagagtctcctgaatagtaatggcaacacttacttgtattggt600
tcctgcagaggccaggccagtctcctcagctcctgatatatcggatgtccaaccttgcct660
caggagtcccagacaggttcagtggcagtgggtcaggaactgctttcacactgagaatca720
gtagagtggaggctgaggatgtgggtgtttattactgtatgcaacatctagaatatccgt780
tcacgttcggtgctgggaccaagctggagctgaaacggaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcaaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccag1080
tacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggag1140
gatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggcc1200
agaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttggaca1260
agagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcagg1320
aaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattggga1380
tgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacag1440
ccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcggag1500
ccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggccccatgg1560
ccctgcccgtgaccgccctgctgctgcccctggccctgctgctgcacgccgccaggcccg1620
atatccagatgacccagagccccagctccctgtccgcatccgtgggcgacagagtgacaa1680
ttacctgtagaagcagccaaagcatcgtgcatagcgtcggcaacacttttctggagtggt1740
atcaacagaagcccgggaaggcccccaaactgctgatctacaaggtgagcaacagattca1800
gcggggtcccaagcagattctccggcagcggctccgggactgacttcaccctgaccatta1860
gcagcctgcagccagaggacttcgccacatactactgcttccaagggagccagttcccct1920
acaccttcggccaaggcactaaggtggagatcaaaggggggggaggaagcggcggaggag1980
ggagcggaggcgggggatccgaagtgcaactggtcgaatccggaggggggctggtccagc2040
ctggagggtccctgagactgagctgcgccgcaagcggctacgagttctccaggtcctgga2100
tgaactgggtgaggcaggccccaggaaaagggctggaatgggtgggcaggatctaccctg2160
gcgacggcgataccaactactccggaaagttcaagggcaggttcactatcagcgccgaca2220
ctagcaagaataccgcctacctgcagatgaatagcctgagggccgaggacaccgccgtgt2280
attactgcgctagagacggcagcagctgggattggtacttcgacgtgtggggccagggca2340
ctctggtgactgtgagcagcaccaccacccccgcccccaggccccccacccccgccccca2400
ccatcgccagccagcccctgagcctgaggcccgaggcctgcaggcccgccgccggcggcg2460
ccgtgcacaccaggggcctggacttcgcctgcgacttttgggtgctggtggtggttggtg2520
gagtcctggcttgctatagcttgctagtaacagtggcctttattattttctgggtgagga2580
gtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggc2640
ccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctcca2700
gggtgaagttcagcaggagcgccgacgcccccgcctaccagcagggccagaaccagctgt2760
acaacgagctgaacctgggcaggagggaggagtacgacgtgctggacaagaggaggggca2820
gggaccccgagatgggcggcaagccccagaggaggaagaacccccaggagggcctgtaca2880
acgagctgcagaaggacaagatggccgaggcctacagcgagatcggcatgaagggcgaga2940
ggaggaggggcaagggccacgacggcctgtaccagggcctgagcaccgccaccaaggaca3000
cctacgacgccctgcacatgcaggccctgccccccaggtaagtttaaac3049
<210>64
<211>1010
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>64
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprogluvalglnleuglnglnserglyprogluleu
202530
ilelysproglyalaservallysmetsercyslysalaserglytyr
354045
thrphethrsertyrvalmethistrpvallysglnlysproglygln
505560
glyleuglutrpileglytyrileasnprotyrasnaspglythrlys
65707580
tyrasnglulysphelysglylysalathrleuthrserasplysser
859095
serserthralatyrmetgluleuserserleuthrsergluaspser
100105110
alavaltyrtyrcysalaargglythrtyrtyrtyrglyserargval
115120125
pheasptyrtrpglyglnglythrthrleuthrvalserserglygly
130135140
glyglyserglyglyglyglyserglyglyglyglyseraspileval
145150155160
metthrglnalaalaproserileprovalthrproglygluserval
165170175
serilesercysargserserlysserleuleuasnserasnglyasn
180185190
thrtyrleutyrtrppheleuglnargproglyglnserproglnleu
195200205
leuiletyrargmetserasnleualaserglyvalproaspargphe
210215220
serglyserglyserglythralaphethrleuargileserargval
225230235240
glualagluaspvalglyvaltyrtyrcysmetglnhisleuglutyr
245250255
prophethrpheglyalaglythrlysleugluleulysargthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcyslysargglyarglyslysleuleutyrilephelysgln
340345350
prophemetargprovalglnthrthrglnglugluaspglycysser
355360365
cysargpheproglugluglugluglyglycysgluleuargvallys
370375380
pheserargseralaaspalaproalatyrglnglnglyglnasngln
385390395400
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
405410415
asplysargargglyargaspproglumetglyglylysproglnarg
420425430
arglysasnproglngluglyleutyrasngluleuglnlysasplys
435440445
metalaglualatyrsergluileglymetlysglygluargargarg
450455460
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
465470475480
aspthrtyraspalaleuhismetglnalaleuproproargglyser
485490495
glyalathrasnpheserleuleulysglnalaglyaspvalgluglu
500505510
asnproglyprometalaleuprovalthralaleuleuleuproleu
515520525
alaleuleuleuhisalaalaargproaspileglnmetthrglnser
530535540
proserserleuseralaservalglyaspargvalthrilethrcys
545550555560
argserserglnserilevalhisservalglyasnthrpheleuglu
565570575
trptyrglnglnlysproglylysalaprolysleuleuiletyrlys
580585590
valserasnargpheserglyvalproserargpheserglysergly
595600605
serglythraspphethrleuthrileserserleuglnprogluasp
610615620
phealathrtyrtyrcyspheglnglyserglnpheprotyrthrphe
625630635640
glyglnglythrlysvalgluilelysglyglyglyglyserglygly
645650655
glyglyserglyglyglyglysergluvalglnleuvalglusergly
660665670
glyglyleuvalglnproglyglyserleuargleusercysalaala
675680685
serglytyrglupheserargsertrpmetasntrpvalargglnala
690695700
proglylysglyleuglutrpvalglyargiletyrproglyaspgly
705710715720
aspthrasntyrserglylysphelysglyargphethrileserala
725730735
aspthrserlysasnthralatyrleuglnmetasnserleuargala
740745750
gluaspthralavaltyrtyrcysalaargaspglysersertrpasp
755760765
trptyrpheaspvaltrpglyglnglythrleuvalthrvalserser
770775780
thrthrthrproalaproargproprothrproalaprothrileala
785790795800
serglnproleuserleuargproglualacysargproalaalagly
805810815
glyalavalhisthrargglyleuaspphealacysaspphetrpval
820825830
leuvalvalvalglyglyvalleualacystyrserleuleuvalthr
835840845
valalapheileilephetrpvalargserlysargserargleuleu
850855860
hisserasptyrmetasnmetthrproargargproglyprothrarg
865870875880
lyshistyrglnprotyralaproproargaspphealaalatyrarg
885890895
serargvallyspheserargseralaaspalaproalatyrglngln
900905910
glyglnasnglnleutyrasngluleuasnleuglyargarggluglu
915920925
tyraspvalleuasplysargargglyargaspproglumetglygly
930935940
lysproglnargarglysasnproglngluglyleutyrasngluleu
945950955960
glnlysasplysmetalaglualatyrsergluileglymetlysgly
965970975
gluargargargglylysglyhisaspglyleutyrglnglyleuser
980985990
thralathrlysaspthrtyraspalaleuhismetglnalaleupro
99510001005
proarg
1010
<210>65
<211>2953
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>65
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggaggtccagctgcagcagtctggacctgagctgataaagcctggggctt120
cagtgaagatgtcctgcaaggcttctggatacacattcactagctatgttatgcactggg180
tgaagcagaagcctgggcagggccttgagtggattggatatattaatccttacaatgatg240
gtactaagtacaatgagaagttcaaaggcaaggccacactgacttcagacaaatcctcca300
gcacagcctacatggagctcagcagcctgacctctgaggactctgcggtctattactgtg360
caagagggacttattactacggtagtagggtatttgactactggggccaaggcaccactc420
tcacagtctcctcaggtggagggggctcaggcggaggtggctctgggggtggaggctcgg480
acattgtgatgactcaggctgcaccctctatacctgtcactcctggagagtcagtatcca540
tctcctgcaggtctagtaagagtctcctgaatagtaatggcaacacttacttgtattggt600
tcctgcagaggccaggccagtctcctcagctcctgatatatcggatgtccaaccttgcct660
caggagtcccagacaggttcagtggcagtgggtcaggaactgctttcacactgagaatca720
gtagagtggaggctgaggatgtgggtgtttattactgtatgcaacatctagaatatccgt780
tcacgttcggtgctgggaccaagctggagctgaaacggaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcaaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccag1080
tacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggag1140
gatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggcc1200
agaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttggaca1260
agagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcagg1320
aaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattggga1380
tgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacag1440
ccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcggag1500
ccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggccccatgg1560
ccctgcccgtgaccgccctgctgctgcccctggccctgctgctgcacgccgccaggcccg1620
acattcagctgacccagtctccagcaatcctttctgcatctccaggggagaaggtcacaa1680
tgacttgcagggccagctcaagtttaagtttcatgcactggtaccagcagaagccaggat1740
cctcccccaaaccctggatttatgccacatccaacctggcttctggagtccctgctcgct1800
tcagtggcagtgggtctgggacctcttactctctcacaatcagcacagtggaggctgaag1860
atgctgcctcttatttctgccatcagtggagtagtaacccgctcacgttcggtgctggga1920
ccaagctggagatcagctcgggcggcggcggctcgggcggcggcggctccggagacgtca1980
tgggggtggattctgggggaggcttagtgcagcctggagggtcccggaaactctcctgtg2040
cagcccctggattcactttcagtagctttgggatgcactgggttcgtcaggctccagaga2100
aggggttggagtgggtcgcatacattagtagtcccagtagtaccctccactatgcagaca2160
gagtgaagggccgattcaccatctccagagacaatcccaagaacaccctgttcctgcaaa2220
tgaaactaccctcactatgctatggactactggggccaagggaccacgttcacaccacca2280
cccccgcccccaggccccccacccccgcccccaccatcgccagccagcccctgagcctga2340
ggcccgaggcctgcaggcccgccgccggcggcgccgtgcacaccaggggcctggacttcg2400
cctgcgacatctacatctgggcccccctggccggcacctgcggcgtgctgctgctgagcc2460
tggtgatcaccctgtactgcaggagtaagaggagcaggctcctgcacagtgactacatga2520
acatgactccccgccgccccgggcccacccgcaagcattaccagccctatgccccaccac2580
gcgacttcgcagcctatcgctccagggtgaagttcagcaggagcgccgacgcccccgcct2640
accagcagggccagaaccagctgtacaacgagctgaacctgggcaggagggaggagtacg2700
acgtgctggacaagaggaggggcagggaccccgagatgggcggcaagccccagaggagga2760
agaacccccaggagggcctgtacaacgagctgcagaaggacaagatggccgaggcctaca2820
gcgagatcggcatgaagggcgagaggaggaggggcaagggccacgacggcctgtaccagg2880
gcctgagcaccgccaccaaggacacctacgacgccctgcacatgcaggccctgcccccca2940
ggtaagtttaaac2953
<210>66
<211>978
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>66
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprogluvalglnleuglnglnserglyprogluleu
202530
ilelysproglyalaservallysmetsercyslysalaserglytyr
354045
thrphethrsertyrvalmethistrpvallysglnlysproglygln
505560
glyleuglutrpileglytyrileasnprotyrasnaspglythrlys
65707580
tyrasnglulysphelysglylysalathrleuthrserasplysser
859095
serserthralatyrmetgluleuserserleuthrsergluaspser
100105110
alavaltyrtyrcysalaargglythrtyrtyrtyrglyserargval
115120125
pheasptyrtrpglyglnglythrthrleuthrvalserserglygly
130135140
glyglyserglyglyglyglyserglyglyglyglyseraspileval
145150155160
metthrglnalaalaproserileprovalthrproglygluserval
165170175
serilesercysargserserlysserleuleuasnserasnglyasn
180185190
thrtyrleutyrtrppheleuglnargproglyglnserproglnleu
195200205
leuiletyrargmetserasnleualaserglyvalproaspargphe
210215220
serglyserglyserglythralaphethrleuargileserargval
225230235240
glualagluaspvalglyvaltyrtyrcysmetglnhisleuglutyr
245250255
prophethrpheglyalaglythrlysleugluleulysargthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcyslysargglyarglyslysleuleutyrilephelysgln
340345350
prophemetargprovalglnthrthrglnglugluaspglycysser
355360365
cysargpheproglugluglugluglyglycysgluleuargvallys
370375380
pheserargseralaaspalaproalatyrglnglnglyglnasngln
385390395400
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
405410415
asplysargargglyargaspproglumetglyglylysproglnarg
420425430
arglysasnproglngluglyleutyrasngluleuglnlysasplys
435440445
metalaglualatyrsergluileglymetlysglygluargargarg
450455460
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
465470475480
aspthrtyraspalaleuhismetglnalaleuproproargglyser
485490495
glyalathrasnpheserleuleulysglnalaglyaspvalgluglu
500505510
asnproglyprometalaleuprovalthralaleuleuleuproleu
515520525
alaleuleuleuhisalaalaargproaspileglnleuthrglnser
530535540
proalaileleuseralaserproglyglulysvalthrmetthrcys
545550555560
argalaserserserleuserphemethistrptyrglnglnlyspro
565570575
glyserserprolysprotrpiletyralathrserasnleualaser
580585590
glyvalproalaargpheserglyserglyserglythrsertyrser
595600605
leuthrileserthrvalglualagluaspalaalasertyrphecys
610615620
hisglntrpserserasnproleuthrpheglyalaglythrlysleu
625630635640
gluileserserglyglyglyglyserglyglyglyglyserglyasp
645650655
valmetglyvalaspserglyglyglyleuvalglnproglyglyser
660665670
arglysleusercysalaalaproglyphethrpheserserphegly
675680685
methistrpvalargglnalaproglulysglyleuglutrpvalala
690695700
tyrileserserproserserthrleuhistyralaaspargvallys
705710715720
glyargphethrileserargaspasnprolysasnthrleupheleu
725730735
glnmetlysleuproserleucystyrglyleuleuglyproargasp
740745750
hisvalhisthrthrthrproalaproargproprothrproalapro
755760765
thrilealaserglnproleuserleuargproglualacysargpro
770775780
alaalaglyglyalavalhisthrargglyleuaspphealacysasp
785790795800
iletyriletrpalaproleualaglythrcysglyvalleuleuleu
805810815
serleuvalilethrleutyrcysargserlysargserargleuleu
820825830
hisserasptyrmetasnmetthrproargargproglyprothrarg
835840845
lyshistyrglnprotyralaproproargaspphealaalatyrarg
850855860
serargvallyspheserargseralaaspalaproalatyrglngln
865870875880
glyglnasnglnleutyrasngluleuasnleuglyargarggluglu
885890895
tyraspvalleuasplysargargglyargaspproglumetglygly
900905910
lysproglnargarglysasnproglngluglyleutyrasngluleu
915920925
glnlysasplysmetalaglualatyrsergluileglymetlysgly
930935940
gluargargargglylysglyhisaspglyleutyrglnglyleuser
945950955960
thralathrlysaspthrtyraspalaleuhismetglnalaleupro
965970975
proarg
<210>67
<211>3010
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>67
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggaggtccagctgcagcagtctggacctgagctgataaagcctggggctt120
cagtgaagatgtcctgcaaggcttctggatacacattcactagctatgttatgcactggg180
tgaagcagaagcctgggcagggccttgagtggattggatatattaatccttacaatgatg240
gtactaagtacaatgagaagttcaaaggcaaggccacactgacttcagacaaatcctcca300
gcacagcctacatggagctcagcagcctgacctctgaggactctgcggtctattactgtg360
caagagggacttattactacggtagtagggtatttgactactggggccaaggcaccactc420
tcacagtctcctcaggtggagggggctcaggcggaggtggctctgggggtggaggctcgg480
acattgtgatgactcaggctgcaccctctatacctgtcactcctggagagtcagtatcca540
tctcctgcaggtctagtaagagtctcctgaatagtaatggcaacacttacttgtattggt600
tcctgcagaggccaggccagtctcctcagctcctgatatatcggatgtccaaccttgcct660
caggagtcccagacaggttcagtggcagtgggtcaggaactgctttcacactgagaatca720
gtagagtggaggctgaggatgtgggtgtttattactgtatgcaacatctagaatatccgt780
tcacgttcggtgctgggaccaagctggagctgaaacggaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcaaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccag1080
tacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggag1140
gatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggcc1200
agaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttggaca1260
agagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcagg1320
aaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattggga1380
tgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacag1440
ccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcggag1500
ccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggccccatgg1560
ccctgcccgtgaccgccctgctgctgcccctggccctgctgctgcacgccgccaggcccg1620
acgtgcagatcacccagagccccagctacctggccgccagccccggcgagaccatcacca1680
tcaactgcagagccagcaagagcatcagcaaggacctggcctggtaccaggagaagcccg1740
gcaagaccaacaagctgctgatctacagcggcagcaccctgcagagcggcatccccagca1800
gattcagcggcagcggcagcggcaccgacttcaccctgaccatcagcagcctggagcccg1860
aggacttcgccatgtactactgccagcagcacaacaagtacccctacaccttcggcggcg1920
gcaccaagctggagatcaagggaggggggggatccgggggaggaggctccggcggaggcg1980
gaagccaggtgcagctgcagcagcccggcgccgagctggtgagacccggcgccagcgtga2040
agctgagctgcaaggccagcggctacaccttcaccagctactggatgaactgggtgaagc2100
agagacccgaccagggcctggagtggatcggcagaatcgacccctacgacagcgagaccc2160
actacaaccagaagttcaaggacaaggccatcctgaccgtggacaagagcagcagcaccg2220
cctacatgcagctgagcagcctgaccagcgaggacagcgccgtgtactactgcgccagag2280
gcaactgggacgactactggggccagggcaccaccctgaccgtgagcagcaccaccaccc2340
ccgcccccaggccccccacccccgcccccaccatcgccagccagcccctgagcctgaggc2400
ccgaggcctgcaggcccgccgccggcggcgccgtgcacaccaggggcctggacttcgcct2460
gcgacatctacatctgggcccccctggccggcacctgcggcgtgctgctgctgagcctgg2520
tgatcaccctgtactgcaggagtaagaggagcaggctcctgcacagtgactacatgaaca2580
tgactccccgccgccccgggcccacccgcaagcattaccagccctatgccccaccacgcg2640
acttcgcagcctatcgctccagggtgaagttcagcaggagcgccgacgcccccgcctacc2700
agcagggccagaaccagctgtacaacgagctgaacctgggcaggagggaggagtacgacg2760
tgctggacaagaggaggggcagggaccccgagatgggcggcaagccccagaggaggaaga2820
acccccaggagggcctgtacaacgagctgcagaaggacaagatggccgaggcctacagcg2880
agatcggcatgaagggcgagaggaggaggggcaagggccacgacggcctgtaccagggcc2940
tgagcaccgccaccaaggacacctacgacgccctgcacatgcaggccctgccccccaggt3000
aagtttaaac3010
<210>68
<211>997
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>68
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprogluvalglnleuglnglnserglyprogluleu
202530
ilelysproglyalaservallysmetsercyslysalaserglytyr
354045
thrphethrsertyrvalmethistrpvallysglnlysproglygln
505560
glyleuglutrpileglytyrileasnprotyrasnaspglythrlys
65707580
tyrasnglulysphelysglylysalathrleuthrserasplysser
859095
serserthralatyrmetgluleuserserleuthrsergluaspser
100105110
alavaltyrtyrcysalaargglythrtyrtyrtyrglyserargval
115120125
pheasptyrtrpglyglnglythrthrleuthrvalserserglygly
130135140
glyglyserglyglyglyglyserglyglyglyglyseraspileval
145150155160
metthrglnalaalaproserileprovalthrproglygluserval
165170175
serilesercysargserserlysserleuleuasnserasnglyasn
180185190
thrtyrleutyrtrppheleuglnargproglyglnserproglnleu
195200205
leuiletyrargmetserasnleualaserglyvalproaspargphe
210215220
serglyserglyserglythralaphethrleuargileserargval
225230235240
glualagluaspvalglyvaltyrtyrcysmetglnhisleuglutyr
245250255
prophethrpheglyalaglythrlysleugluleulysargthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcyslysargglyarglyslysleuleutyrilephelysgln
340345350
prophemetargprovalglnthrthrglnglugluaspglycysser
355360365
cysargpheproglugluglugluglyglycysgluleuargvallys
370375380
pheserargseralaaspalaproalatyrglnglnglyglnasngln
385390395400
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
405410415
asplysargargglyargaspproglumetglyglylysproglnarg
420425430
arglysasnproglngluglyleutyrasngluleuglnlysasplys
435440445
metalaglualatyrsergluileglymetlysglygluargargarg
450455460
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
465470475480
aspthrtyraspalaleuhismetglnalaleuproproargglyser
485490495
glyalathrasnpheserleuleulysglnalaglyaspvalgluglu
500505510
asnproglyprometalaleuprovalthralaleuleuleuproleu
515520525
alaleuleuleuhisalaalaargproaspvalglnilethrglnser
530535540
prosertyrleualaalaserproglygluthrilethrileasncys
545550555560
argalaserlysserileserlysaspleualatrptyrglnglulys
565570575
proglylysthrasnlysleuleuiletyrserglyserthrleugln
580585590
serglyileproserargpheserglyserglyserglythraspphe
595600605
thrleuthrileserserleugluprogluaspphealamettyrtyr
610615620
cysglnglnhisasnlystyrprotyrthrpheglyglyglythrlys
625630635640
leugluilelysglyglyglyglyserglyglyglyglyserglygly
645650655
glyglyserglnvalglnleuglnglnproglyalagluleuvalarg
660665670
proglyalaservallysleusercyslysalaserglytyrthrphe
675680685
thrsertyrtrpmetasntrpvallysglnargproaspglnglyleu
690695700
glutrpileglyargileaspprotyraspsergluthrhistyrasn
705710715720
glnlysphelysasplysalaileleuthrvalasplysserserser
725730735
thralatyrmetglnleuserserleuthrsergluaspseralaval
740745750
tyrtyrcysalaargglyasntrpaspasptyrtrpglyglnglythr
755760765
thrleuthrvalserserthrthrthrproalaproargproprothr
770775780
proalaprothrilealaserglnproleuserleuargprogluala
785790795800
cysargproalaalaglyglyalavalhisthrargglyleuaspphe
805810815
alacysaspiletyriletrpalaproleualaglythrcysglyval
820825830
leuleuleuserleuvalilethrleutyrcysargserlysargser
835840845
argleuleuhisserasptyrmetasnmetthrproargargprogly
850855860
prothrarglyshistyrglnprotyralaproproargasppheala
865870875880
alatyrargserargvallyspheserargseralaaspalaproala
885890895
tyrglnglnglyglnasnglnleutyrasngluleuasnleuglyarg
900905910
arggluglutyraspvalleuasplysargargglyargaspproglu
915920925
metglyglylysproglnargarglysasnproglngluglyleutyr
930935940
asngluleuglnlysasplysmetalaglualatyrsergluilegly
945950955960
metlysglygluargargargglylysglyhisaspglyleutyrgln
965970975
glyleuserthralathrlysaspthrtyraspalaleuhismetgln
980985990
alaleuproproarg
995
<210>69
<211>1767
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>69
gcgatcgctgacctctagaatgtatttgtggcttaaactcttggcatttggctttgcctt60
tctggacacagaagtatttgtgacaggggacatccaggtgacccagagccccagcagcct120
gagcgccagcctgggcgagagaatcagcctgacctgcagaaccagccaggacatcagcaa180
ctacctgaactggttccagcagaagcccgacggcaccttcaagagactgatctacgccac240
cagcagcctggacagcggcgtgcccaagagattcagcggcagcggcagcggcagcgacta300
cagcctgaccatcagcagcctggagagcgaggacttcgccgactactactgcctgcagta360
cgccagctaccccttcaccttcggcagcggcaccaagctggagatcaagggagggggggg420
atccgggggaggaggctccggcggaggcggaagcgaggtgcagctgcaggagagcggccc480
cggcctggtgaagcccagccagaccctgagcctgacctgcagcgtgaccggctacagcat540
caccagcggctactactggcactggatcagacagttccccggcaacaagctgcagtggat600
gggctacatcagctacagcggcttcaccaactacaagaccagcctgatcaacagaatcag660
catcacccacgacaccagcgagaaccagttcttcctgaacctgaacagcgtgaccaccga720
ggacaccgccacctactactgcgccggcgacagaaccggcagctggttcgcctactgggg780
ccagggcaccctggtgaccgtgagcgccaccacgacgccagcgccgcgaccaccaacacc840
ggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggc900
ggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgcc960
cttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcaggag1020
taagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggcc1080
cacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctccag1140
agtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctcta1200
taacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccg1260
ggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaa1320
tgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcg1380
ccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacac1440
ctacgacgcccttcacatgcaggccctgccccctcgcggcagcggcgaaggccgcggcag1500
cctgctgacctgcggcgatgtggaagaaaacccgggccccatgtacctgtggctgaagct1560
gctggccttcggcttcgccttcctggacaccgaggtgttcgtgaccggcggccagaacga1620
caccagccagaccagcagccccagcagcccgctgtttccgggcccgagcaaaccgttttg1680
ggtgctggtggtggtgggcggcgtgctggcgtgctatagcctgctggtgaccgtggcgtt1740
tattattttttgggtgtaagtttaaac1767
<210>70
<211>579
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>70
mettyrleutrpleulysleuleualapheglyphealapheleuasp
151015
thrgluvalphevalthrglyaspileglnvalthrglnserproser
202530
serleuseralaserleuglygluargileserleuthrcysargthr
354045
serglnaspileserasntyrleuasntrppheglnglnlysproasp
505560
glythrphelysargleuiletyralathrserserleuaspsergly
65707580
valprolysargpheserglyserglyserglyserasptyrserleu
859095
thrileserserleuglusergluaspphealaasptyrtyrcysleu
100105110
glntyralasertyrprophethrpheglyserglythrlysleuglu
115120125
ilelysglyglyglyglyserglyglyglyglyserglyglyglygly
130135140
sergluvalglnleuglngluserglyproglyleuvallysproser
145150155160
glnthrleuserleuthrcysservalthrglytyrserilethrser
165170175
glytyrtyrtrphistrpileargglnpheproglyasnlysleugln
180185190
trpmetglytyrilesertyrserglyphethrasntyrlysthrser
195200205
leuileasnargileserilethrhisaspthrsergluasnglnphe
210215220
pheleuasnleuasnservalthrthrgluaspthralathrtyrtyr
225230235240
cysalaglyaspargthrglysertrpphealatyrtrpglyglngly
245250255
thrleuvalthrvalseralathrthrthrproalaproargpropro
260265270
thrproalaprothrilealaserglnproleuserleuargproglu
275280285
alacysargproalaalaglyglyalavalhisthrargglyleuasp
290295300
phealacysaspiletyriletrpalaproleualaglythrcysgly
305310315320
valleuleuleuserleuvalilethrleutyrcysargserlysarg
325330335
serargleuleuhisserasptyrmetasnmetthrproargargpro
340345350
glyprothrarglyshistyrglnprotyralaproproargaspphe
355360365
alaalatyrargserargvallyspheserargseralaaspalapro
370375380
alatyrglnglnglyglnasnglnleutyrasngluleuasnleugly
385390395400
argarggluglutyraspvalleuasplysargargglyargasppro
405410415
glumetglyglylysproglnargarglysasnproglngluglyleu
420425430
tyrasngluleuglnlysasplysmetalaglualatyrsergluile
435440445
glymetlysglygluargargargglylysglyhisaspglyleutyr
450455460
glnglyleuserthralathrlysaspthrtyraspalaleuhismet
465470475480
glnalaleuproproargglyserglygluglyargglyserleuleu
485490495
thrcysglyaspvalglugluasnproglypromettyrleutrpleu
500505510
lysleuleualapheglyphealapheleuaspthrgluvalpheval
515520525
thrglyglyglnasnaspthrserglnthrserserproserserpro
530535540
leupheproglyproserlysprophetrpvalleuvalvalvalgly
545550555560
glyvalleualacystyrserleuleuvalthrvalalapheileile
565570575
phetrpval
<210>71
<211>1880
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>71
atgtacctgtggctgaagctgctggccttcggcttcgccttcctggacaccgaggtgttc60
gtgaccggcgacatccaggtgacccagagccccagcagcctgagcgccagcctgggcgag120
agaatcagcctgacctgcagaaccagccaggacatcagcaactacctgaactggttccag180
cagaagcccgacggcaccttcaagagactgatctacgccaccagcagcctggacagcggc240
gtgcccaagagattcagcggcagcggcagcggcagcgactacagcctgaccatcagcagc300
ctggagagcgaggacttcgccgactactactgcctgcagtacgccagctaccccttcacc360
ttcggcagcggcaccaagctggagatcaagggaggggggggatccgggggaggaggctcc420
ggcggaggcggaagcgaggtgcagctgcaggagagcggccccggcctggtgaagcccagc480
cagaccctgagcctgacctgcagcgtgaccggctacagcatcaccagcggctactactgg540
cactggatcagacagttccccggcaacaagctgcagtggatgggctacatcagctacagc600
ggcttcaccaactacaagaccagcctgatcaacagaatcagcatcacccacgacaccagc660
gagaaccagttcttcctgaacctgaacagcgtgaccaccgaggacaccgccacctactac720
tgcgccggcgacagaaccggcagctggttcgcctactggggccagggcaccctggtgacc780
gtgagcgccaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcg840
cagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacg900
agggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggg960
gtccttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggctcctg1020
cacagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcattaccag1080
ccctatgccccaccacgcgacttcgcagcctatcgctccaaacggggcagaaagaaactc1140
ctgtatatattcaaacaaccatttatgagaccagtacaaactactcaagaggaagatggc1200
tgtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtgaagttcagc1260
aggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagctcaat1320
ctaggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctgagatg1380
gggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaa1440
gataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaag1500
gggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgccctt1560
cacatgcaggccctgccccctcgcatcgatggcagcggcgagggcaggggcagcctgctg1620
acctgcggcgacgtggaggagaaccccggccccatggccttaccagtgaccgccttgctc1680
ctgccgctggccttgctgctccacgccgccaggccgggccagaacgacaccagccagacc1740
agcagccccagcagccccctgttccccggccccagcaagcccttctgggtgctggtggtg1800
gtgggcggcgtgctggcctgctacagcctgctggtgaccgtggccttcatcatcttctgg1860
gtgaggagctaagtttaaac1880
<210>72
<211>623
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>72
mettyrleutrpleulysleuleualapheglyphealapheleuasp
151015
thrgluvalphevalthrglyaspileglnvalthrglnserproser
202530
serleuseralaserleuglygluargileserleuthrcysargthr
354045
serglnaspileserasntyrleuasntrppheglnglnlysproasp
505560
glythrphelysargleuiletyralathrserserleuaspsergly
65707580
valprolysargpheserglyserglyserglyserasptyrserleu
859095
thrileserserleuglusergluaspphealaasptyrtyrcysleu
100105110
glntyralasertyrprophethrpheglyserglythrlysleuglu
115120125
ilelysglyglyglyglyserglyglyglyglyserglyglyglygly
130135140
sergluvalglnleuglngluserglyproglyleuvallysproser
145150155160
glnthrleuserleuthrcysservalthrglytyrserilethrser
165170175
glytyrtyrtrphistrpileargglnpheproglyasnlysleugln
180185190
trpmetglytyrilesertyrserglyphethrasntyrlysthrser
195200205
leuileasnargileserilethrhisaspthrsergluasnglnphe
210215220
pheleuasnleuasnservalthrthrgluaspthralathrtyrtyr
225230235240
cysalaglyaspargthrglysertrpphealatyrtrpglyglngly
245250255
thrleuvalthrvalseralathrthrthrproalaproargpropro
260265270
thrproalaprothrilealaserglnproleuserleuargproglu
275280285
alacysargproalaalaglyglyalavalhisthrargglyleuasp
290295300
phealacysaspiletyriletrpalaproleualaglythrcysgly
305310315320
valleuleuleuserleuvalilethrleutyrcysargserlysarg
325330335
serargleuleuhisserasptyrmetasnmetthrproargargpro
340345350
glyprothrarglyshistyrglnprotyralaproproargaspphe
355360365
alaalatyrargserlysargglyarglyslysleuleutyrilephe
370375380
lysglnprophemetargprovalglnthrthrglnglugluaspgly
385390395400
cyssercysargpheproglugluglugluglyglycysgluleuarg
405410415
vallyspheserargseralaaspalaproalatyrglnglnglygln
420425430
asnglnleutyrasngluleuasnleuglyargarggluglutyrasp
435440445
valleuasplysargargglyargaspproglumetglyglylyspro
450455460
glnargarglysasnproglngluglyleutyrasngluleuglnlys
465470475480
asplysmetalaglualatyrsergluileglymetlysglygluarg
485490495
argargglylysglyhisaspglyleutyrglnglyleuserthrala
500505510
thrlysaspthrtyraspalaleuhismetglnalaleuproproarg
515520525
ileaspglyserglygluglyargglyserleuleuthrcysglyasp
530535540
valglugluasnproglyprometalaleuprovalthralaleuleu
545550555560
leuproleualaleuleuleuhisalaalaargproglyglnasnasp
565570575
thrserglnthrserserproserserproleupheproglyproser
580585590
lysprophetrpvalleuvalvalvalglyglyvalleualacystyr
595600605
serleuleuvalthrvalalapheileilephetrpvalargser
610615620
<210>73
<211>2314
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>73
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgctgacccagagccccgccagcctggccgtgagcctgggcc120
agagggccaccatcagctgcagggccagcaagagcgtgagcaccagcggctacagctacc180
tgcactggtaccagcagaagcccggccagccccccaagctgctgatctacctggccagca240
acctggagagcggcgtgcccgccaggttcagcggcagcggcagcggcaccgacttcaccc300
tgaacatccaccccgtggaggaggaggacgccgccacctactactgccagcacagcaggg360
agctgcccttcaccttcggcagcggcaccaagctggagatcaagggaggggggggatccg420
ggggaggaggctccggcggaggcggaagccaggtgcagctggtggagagcggcggcggcc480
tggtgcagcccggcggcagcctgaagctgagctgcgccgccagcggcttcgacttcagca540
ggtactggatgagctgggtgaggcaggcccccggcaagggcctggagtggatcggcgaga600
tcaaccccaccagcagcaccatcaacttcacccccagcctgaaggacaaggtgttcatca660
gcagggacaacgccaagaacaccctgtacctgcagatgagcaaggtgaggagcgaggaca720
ccgccctgtactactgcgccaggggcaactactacaggtacggcgacgccatggactact780
ggggccagggcaccagcgtgaccgtgagcaccacgacgccagcgccgcgaccaccaacac840
cggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcgg900
cggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgc960
ccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcagga1020
gtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggc1080
ccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctcca1140
gagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctct1200
ataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggcc1260
gggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtaca1320
atgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagc1380
gccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggaca1440
cctacgacgcccttcacatgcaggccctgccccctcgcggcagcggcgaaggccgcggca1500
gcctgctgacctgcggcgatgtggaagaaaacccgggccccatggaatacgcctctgacg1560
cttcactggaccccgaagccccgtggcctcccgcgccccgcgctcgcgcctgccgcgtac1620
tgccttgggccctggtcgcggggctgctgctgctgctgctgctcgctgccgcctgcgccg1680
tcttcctcgcctgcccctgggccgtgtccggggctcgcgcctcgcccggctccgcggcca1740
gcccgagactccgcgagggtcccgagctttcgcccgacgatcccgccggcctcttggacc1800
tgcggcagggcatgtttgcgcagctggtggcccaaaatgttctgctgatcgatgggcccc1860
tgagctggtacagtgacccaggcctggcaggcgtgtccctgacggggggcctgagctaca1920
aagaggacacgaaggagctggtggtggccaaggctggagtctactatgtcttctttcaac1980
tagagctgcggcgcgtggtggccggcgagggctcaggctccgtttcacttgcgctgcacc2040
tgcagccactgcgctctgctgctggggccgccgccctggctttgaccgtggacctgccac2100
ccgcctcctccgaggctcggaactcggccttcggtttccagggccgcttgctgcacctga2160
gtgccggccagcgcctgggcgtccatcttcacactgaggccagggcacgccatgcctggc2220
agcttacccagggcgccacagtcttgggactcttccgggtgacccccgaaatcccagccg2280
gactcccttcaccgaggtcggaataagtttaaac2314
<210>74
<211>765
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>74
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalleuthrglnserproalaserleu
202530
alavalserleuglyglnargalathrilesercysargalaserlys
354045
servalserthrserglytyrsertyrleuhistrptyrglnglnlys
505560
proglyglnproprolysleuleuiletyrleualaserasnleuglu
65707580
serglyvalproalaargpheserglyserglyserglythraspphe
859095
thrleuasnilehisprovalgluglugluaspalaalathrtyrtyr
100105110
cysglnhisserarggluleuprophethrpheglyserglythrlys
115120125
leugluilelysglyglyglyglyserglyglyglyglyserglygly
130135140
glyglyserglnvalglnleuvalgluserglyglyglyleuvalgln
145150155160
proglyglyserleulysleusercysalaalaserglypheaspphe
165170175
serargtyrtrpmetsertrpvalargglnalaproglylysglyleu
180185190
glutrpileglygluileasnprothrserserthrileasnphethr
195200205
proserleulysasplysvalpheileserargaspasnalalysasn
210215220
thrleutyrleuglnmetserlysvalargsergluaspthralaleu
225230235240
tyrtyrcysalaargglyasntyrtyrargtyrglyaspalametasp
245250255
tyrtrpglyglnglythrservalthrvalserthrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
argserlysargserargleuleuhisserasptyrmetasnmetthr
340345350
proargargproglyprothrarglyshistyrglnprotyralapro
355360365
proargaspphealaalatyrargserargvallyspheserargser
370375380
alaaspalaproalatyrglnglnglyglnasnglnleutyrasnglu
385390395400
leuasnleuglyargarggluglutyraspvalleuasplysargarg
405410415
glyargaspproglumetglyglylysproglnargarglysasnpro
420425430
glngluglyleutyrasngluleuglnlysasplysmetalagluala
435440445
tyrsergluileglymetlysglygluargargargglylysglyhis
450455460
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
465470475480
alaleuhismetglnalaleuproproargglyserglygluglyarg
485490495
glyserleuleuthrcysglyaspvalglugluasnproglypromet
500505510
glutyralaseraspalaserleuaspproglualaprotrppropro
515520525
alaproargalaargalacysargvalleuprotrpalaleuvalala
530535540
glyleuleuleuleuleuleuleualaalaalacysalavalpheleu
545550555560
alacysprotrpalavalserglyalaargalaserproglyserala
565570575
alaserproargleuarggluglyprogluleuserproaspasppro
580585590
alaglyleuleuaspleuargglnglymetphealaglnleuvalala
595600605
glnasnvalleuleuileaspglyproleusertrptyrserasppro
610615620
glyleualaglyvalserleuthrglyglyleusertyrlysgluasp
625630635640
thrlysgluleuvalvalalalysalaglyvaltyrtyrvalphephe
645650655
glnleugluleuargargvalvalalaglygluglyserglyserval
660665670
serleualaleuhisleuglnproleuargseralaalaglyalaala
675680685
alaleualaleuthrvalaspleuproproalaserserglualaarg
690695700
asnseralapheglypheglnglyargleuleuhisleuseralagly
705710715720
glnargleuglyvalhisleuhisthrglualaargalaarghisala
725730735
trpglnleuthrglnglyalathrvalleuglyleupheargvalthr
740745750
progluileproalaglyleuproserproargserglu
755760765
<210>75
<211>2371
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>75
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgctgacccagagccccgccagcctggccgtgagcctgggcc120
agagggccaccatcagctgcagggccagcaagagcgtgagcaccagcggctacagctacc180
tgcactggtaccagcagaagcccggccagccccccaagctgctgatctacctggccagca240
acctggagagcggcgtgcccgccaggttcagcggcagcggcagcggcaccgacttcaccc300
tgaacatccaccccgtggaggaggaggacgccgccacctactactgccagcacagcaggg360
agctgcccttcaccttcggcagcggcaccaagctggagatcaagggaggggggggatccg420
ggggaggaggctccggcggaggcggaagccaggtgcagctggtggagagcggcggcggcc480
tggtgcagcccggcggcagcctgaagctgagctgcgccgccagcggcttcgacttcagca540
ggtactggatgagctgggtgaggcaggcccccggcaagggcctggagtggatcggcgaga600
tcaaccccaccagcagcaccatcaacttcacccccagcctgaaggacaaggtgttcatca660
gcagggacaacgccaagaacaccctgtacctgcagatgagcaaggtgaggagcgaggaca720
ccgccctgtactactgcgccaggggcaactactacaggtacggcgacgccatggactact780
ggggccagggcaccagcgtgaccgtgagcaccacgacgccagcgccgcgaccaccaacac840
cggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcgg900
cggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgc960
ccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcagga1020
gtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggc1080
ccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctcca1140
gagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctct1200
ataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggcc1260
gggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtaca1320
atgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagc1380
gccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggaca1440
cctacgacgcccttcacatgcaggccctgccccctcgcggcagcggcgaaggccgcggca1500
gcctgctgacctgcggcgatgtggaagaaaacccgggccccatgtacagaatgcagctgc1560
tgagctgcatcgccctgagcctggccctggtgaccaacagcggcatccacgtgttcatcc1620
tgggctgcttcagcgccggcctgcccaagaccgaggccaactgggtgaacgtgatcagcg1680
acctgaagaagatcgaggacctgatccagagcatgcacatcgacgccaccctgtacaccg1740
agagcgacgtgcaccccagctgcaaggtgaccgccatgaagtgcttcctgctggagctgc1800
aggtgatcagcctggagagcggcgacgccagcatccacgacaccgtggagaacctgatca1860
tcctggccaacaacagcctgagcagcaacggcaacgtgaccgagagcggctgcaaggagt1920
gcgaggagctggaggagaagaacatcaaggagttcctgcagagcttcgtgcacatcgtgc1980
agatgttcatcaacaccagctccggcggcggctccggcggcggcggctccggcggcggcg2040
gctccggcggcggcggctccggcggcggctccctgcaggcccccagaagagccagaggct2100
gcagaaccctgggcctgcccgccctgctgctgctgctgctgctgagaccccccgccacca2160
gaggcatcacctgccccccccccatgagcgtggagcacgccgacatctgggtgaagagct2220
acagcctgtacagcagagagagatacatctgcaacagcggcttcaagagaaaggccggca2280
ccagcagcctgaccgagtgcgtgctgaacaaggccaccaacgtggcccactggaccaccc2340
ccagcctgaagtgcatcagataagtttaaac2371
<210>76
<211>784
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>76
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalleuthrglnserproalaserleu
202530
alavalserleuglyglnargalathrilesercysargalaserlys
354045
servalserthrserglytyrsertyrleuhistrptyrglnglnlys
505560
proglyglnproprolysleuleuiletyrleualaserasnleuglu
65707580
serglyvalproalaargpheserglyserglyserglythraspphe
859095
thrleuasnilehisprovalgluglugluaspalaalathrtyrtyr
100105110
cysglnhisserarggluleuprophethrpheglyserglythrlys
115120125
leugluilelysglyglyglyglyserglyglyglyglyserglygly
130135140
glyglyserglnvalglnleuvalgluserglyglyglyleuvalgln
145150155160
proglyglyserleulysleusercysalaalaserglypheaspphe
165170175
serargtyrtrpmetsertrpvalargglnalaproglylysglyleu
180185190
glutrpileglygluileasnprothrserserthrileasnphethr
195200205
proserleulysasplysvalpheileserargaspasnalalysasn
210215220
thrleutyrleuglnmetserlysvalargsergluaspthralaleu
225230235240
tyrtyrcysalaargglyasntyrtyrargtyrglyaspalametasp
245250255
tyrtrpglyglnglythrservalthrvalserthrthrthrproala
260265270
proargproprothrproalaprothrilealaserglnproleuser
275280285
leuargproglualacysargproalaalaglyglyalavalhisthr
290295300
argglyleuaspphealacysaspiletyriletrpalaproleuala
305310315320
glythrcysglyvalleuleuleuserleuvalilethrleutyrcys
325330335
argserlysargserargleuleuhisserasptyrmetasnmetthr
340345350
proargargproglyprothrarglyshistyrglnprotyralapro
355360365
proargaspphealaalatyrargserargvallyspheserargser
370375380
alaaspalaproalatyrglnglnglyglnasnglnleutyrasnglu
385390395400
leuasnleuglyargarggluglutyraspvalleuasplysargarg
405410415
glyargaspproglumetglyglylysproglnargarglysasnpro
420425430
glngluglyleutyrasngluleuglnlysasplysmetalagluala
435440445
tyrsergluileglymetlysglygluargargargglylysglyhis
450455460
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
465470475480
alaleuhismetglnalaleuproproargglyserglygluglyarg
485490495
glyserleuleuthrcysglyaspvalglugluasnproglypromet
500505510
tyrargmetglnleuleusercysilealaleuserleualaleuval
515520525
thrasnserglyilehisvalpheileleuglycyspheseralagly
530535540
leuprolysthrglualaasntrpvalasnvalileseraspleulys
545550555560
lysilegluaspleuileglnsermethisileaspalathrleutyr
565570575
thrgluseraspvalhisprosercyslysvalthralametlyscys
580585590
pheleuleugluleuglnvalileserleugluserglyaspalaser
595600605
ilehisaspthrvalgluasnleuileileleualaasnasnserleu
610615620
serserasnglyasnvalthrgluserglycyslysglucysgluglu
625630635640
leugluglulysasnilelysglupheleuglnserphevalhisile
645650655
valglnmetpheileasnthrserserglyglyglyserglyglygly
660665670
glyserglyglyglyglyserglyglyglyglyserglyglyglyser
675680685
leuglnalaproargargalaargglycysargthrleuglyleupro
690695700
alaleuleuleuleuleuleuleuargproproalathrargglyile
705710715720
thrcysproproprometservalgluhisalaaspiletrpvallys
725730735
sertyrserleutyrserarggluargtyrilecysasnserglyphe
740745750
lysarglysalaglythrserserleuthrglucysvalleuasnlys
755760765
alathrasnvalalahistrpthrthrproserleulyscysilearg
770775780
<210>77
<211>1223
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>77
gcgatcgctgccatcgataccatgtatttgtggcttaaactcttggcatttggctttgcc60
tttctggacacagaagtatttgtgacaggggccgttcagggtccagaagaaacagtcact120
caagactgcttgcaactgattgcagacagtgaaacaccaactatacaaaaaggatcttac180
acatttgttccatggcttctcagctttaaaaggggaagtgccctagaagaaaaagagaat240
aaaatattggtcaaagaaactggttacttttttatatatggtcaggttttatatactgat300
aagacctacgccatgggacatctaattcagaggaagaaggtccatgtctttggggatgaa360
ttgagtctggtgactttgtttcgatgtattcaaaatatgcctgaaacactacccaataat420
tcctgctattcagctggcattgcaaaactggaagaaggagatgaactccaacttgcaata480
ccaagagaaaatgcacaaatatcactggatggagatgtcacattttttggtgcattgaaa540
ctgctgaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcag600
cccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagg660
gggctggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtc720
cttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggctcctgcac780
agtgactacatgaacatgactccccgccgccccgggcccacccgcaagcattaccagccc840
tatgccccaccacgcgacttcgcagcctatcgctccagagtgaagttcagcaggagcgca900
gacgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatctaggacga960
agagaggagtacgatgttttggacaagagacgtggccgggaccctgagatggggggaaag1020
ccgagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatggcg1080
gaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatggc1140
ctttaccagggtctcagtacagccaccaaggacacctacgacgcccttcacatgcaggcc1200
ctgccccctcgctaagtttaaac1223
<210>78
<211>397
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>78
mettyrleutrpleulysleuleualapheglyphealapheleuasp
151015
thrgluvalphevalthrglyalavalglnglyproglugluthrval
202530
thrglnaspcysleuglnleuilealaaspsergluthrprothrile
354045
glnlysglysertyrthrphevalprotrpleuleuserphelysarg
505560
glyseralaleugluglulysgluasnlysileleuvallysgluthr
65707580
glytyrphepheiletyrglyglnvalleutyrthrasplysthrtyr
859095
alametglyhisleuileglnarglyslysvalhisvalpheglyasp
100105110
gluleuserleuvalthrleupheargcysileglnasnmetproglu
115120125
thrleuproasnasnsercystyrseralaglyilealalysleuglu
130135140
gluglyaspgluleuglnleualaileproarggluasnalaglnile
145150155160
serleuaspglyaspvalthrphepheglyalaleulysleuleuthr
165170175
thrthrproalaproargproprothrproalaprothrilealaser
180185190
glnproleuserleuargproglualacysargproalaalaglygly
195200205
alavalhisthrargglyleuaspphealacysaspiletyriletrp
210215220
alaproleualaglythrcysglyvalleuleuleuserleuvalile
225230235240
thrleutyrcysargserlysargserargleuleuhisserasptyr
245250255
metasnmetthrproargargproglyprothrarglyshistyrgln
260265270
protyralaproproargaspphealaalatyrargserargvallys
275280285
pheserargseralaaspalaproalatyrglnglnglyglnasngln
290295300
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
305310315320
asplysargargglyargaspproglumetglyglylysproargarg
325330335
lysasnproglngluglyleutyrasngluleuglnlysasplysmet
340345350
alaglualatyrsergluileglymetlysglygluargargarggly
355360365
lysglyhisaspglyleutyrglnglyleuserthralathrlysasp
370375380
thrtyraspalaleuhismetglnalaleuproproarg
385390395
<210>79
<211>1214
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>79
gcgatcgctgccatcgataccatggccttaccagtgaccgccttgctcctgccgctggcc60
ttgctgctccacgccgccaggccggttcagggtccagaagaaacagtcactcaagactgc120
ttgcaactgattgcagacagtgaaacaccaactatacaaaaaggatcttacacatttgtt180
ccatggcttctcagctttaaaaggggaagtgccctagaagaaaaagagaataaaatattg240
gtcaaagaaactggttacttttttatatatggtcaggttttatatactgataagacctac300
gccatgggacatctaattcagaggaagaaggtccatgtctttggggatgaattgagtctg360
gtgactttgtttcgatgtattcaaaatatgcctgaaacactacccaataattcctgctat420
tcagctggcattgcaaaactggaagaaggagatgaactccaacttgcaataccaagagaa480
aatgcacaaatatcactggatggagatgtcacattttttggtgcattgaaactgctgacc540
acgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtcc600
ctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggac660
ttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttctcctg720
tcactggttatcaccctttactgcaggagtaagaggagcaggctcctgcacagtgactac780
atgaacatgactccccgccgccccgggcccacccgcaagcattaccagccctatgcccca840
ccacgcgacttcgcagcctatcgctccagagtgaagttcagcaggagcgcagacgccccc900
gcgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaagagaggag960
tacgatgttttggacaagagacgtggccgggaccctgagatggggggaaagccgagaagg1020
aagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctac1080
agtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccag1140
ggtctcagtacagccaccaaggacacctacgacgcccttcacatgcaggccctgccccct1200
cgctaagtttaaac1214
<210>80
<211>394
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>80
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprovalglnglyproglugluthrvalthrglnasp
202530
cysleuglnleuilealaaspsergluthrprothrileglnlysgly
354045
sertyrthrphevalprotrpleuleuserphelysargglyserala
505560
leugluglulysgluasnlysileleuvallysgluthrglytyrphe
65707580
pheiletyrglyglnvalleutyrthrasplysthrtyralametgly
859095
hisleuileglnarglyslysvalhisvalpheglyaspgluleuser
100105110
leuvalthrleupheargcysileglnasnmetprogluthrleupro
115120125
asnasnsercystyrseralaglyilealalysleuglugluglyasp
130135140
gluleuglnleualaileproarggluasnalaglnileserleuasp
145150155160
glyaspvalthrphepheglyalaleulysleuleuthrthrthrpro
165170175
alaproargproprothrproalaprothrilealaserglnproleu
180185190
serleuargproglualacysargproalaalaglyglyalavalhis
195200205
thrargglyleuaspphealacysaspiletyriletrpalaproleu
210215220
alaglythrcysglyvalleuleuleuserleuvalilethrleutyr
225230235240
cysargserlysargserargleuleuhisserasptyrmetasnmet
245250255
thrproargargproglyprothrarglyshistyrglnprotyrala
260265270
proproargaspphealaalatyrargserargvallyspheserarg
275280285
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
290295300
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
305310315320
argglyargaspproglumetglyglylysproargarglysasnpro
325330335
glngluglyleutyrasngluleuglnlysasplysmetalagluala
340345350
tyrsergluileglymetlysglygluargargargglylysglyhis
355360365
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
370375380
alaleuhismetglnalaleuproproarg
385390
<210>81
<211>2039
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>81
gcgatcgctgccatcgataccatggccttaccagtgaccgccttgctcctgccgctggcc60
ttgctgctccacgccgccaggccggttcagggtccagaagaaacagtcactcaagactgc120
ttgcaactgattgcagacagtgaaacaccaactatacaaaaaggatcttacacatttgtt180
ccatggcttctcagctttaaaaggggaagtgccctagaagaaaaagagaataaaatattg240
gtcaaagaaactggttacttttttatatatggtcaggttttatatactgataagacctac300
gccatgggacatctaattcagaggaagaaggtccatgtctttggggatgaattgagtctg360
gtgactttgtttcgatgtattcaaaatatgcctgaaacactacccaataattcctgctat420
tcagctggcattgcaaaactggaagaaggagatgaactccaacttgcaataccaagagaa480
aatgcacaaatatcactggatggagatgtcacattttttggtgcattgaaactgctgacc540
acgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtcc600
ctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggac660
ttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttctcctg720
tcactggttatcaccctttactgcaggagtaagaggagcaggctcctgcacagtgactac780
atgaacatgactccccgccgccccgggcccacccgcaagcattaccagccctatgcccca840
ccacgcgacttcgcagcctatcgctccagagtgaagttcagcaggagcgcagacgccccc900
gcgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaagagaggag960
tacgatgttttggacaagagacgtggccgggaccctgagatggggggaaagccgagaagg1020
aagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctac1080
agtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccag1140
ggtctcagtacagccaccaaggacacctacgacgcccttcacatgcaggccctgccccct1200
cgcggcagcggcgaaggccgcggcagcctgctgacctgcggcgatgtggaagaaaacccg1260
ggccccatggaatacgcctctgacgcttcactggaccccgaagccccgtggcctcccgcg1320
ccccgcgctcgcgcctgccgcgtactgccttgggccctggtcgcggggctgctgctgctg1380
ctgctgctcgctgccgcctgcgccgtcttcctcgcctgcccctgggccgtgtccggggct1440
cgcgcctcgcccggctccgcggccagcccgagactccgcgagggtcccgagctttcgccc1500
gacgatcccgccggcctcttggacctgcggcagggcatgtttgcgcagctggtggcccaa1560
aatgttctgctgatcgatgggcccctgagctggtacagtgacccaggcctggcaggcgtg1620
tccctgacggggggcctgagctacaaagaggacacgaaggagctggtggtggccaaggct1680
ggagtctactatgtcttctttcaactagagctgcggcgcgtggtggccggcgagggctca1740
ggctccgtttcacttgcgctgcacctgcagccactgcgctctgctgctggggccgccgcc1800
ctggctttgaccgtggacctgccacccgcctcctccgaggctcggaactcggccttcggt1860
ttccagggccgcttgctgcacctgagtgccggccagcgcctgggcgtccatcttcacact1920
gaggccagggcacgccatgcctggcagcttacccagggcgccacagtcttgggactcttc1980
cgggtgacccccgaaatcccagccggactcccttcaccgaggtcggaataagtttaaac2039
<210>82
<211>669
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>82
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprovalglnglyproglugluthrvalthrglnasp
202530
cysleuglnleuilealaaspsergluthrprothrileglnlysgly
354045
sertyrthrphevalprotrpleuleuserphelysargglyserala
505560
leugluglulysgluasnlysileleuvallysgluthrglytyrphe
65707580
pheiletyrglyglnvalleutyrthrasplysthrtyralametgly
859095
hisleuileglnarglyslysvalhisvalpheglyaspgluleuser
100105110
leuvalthrleupheargcysileglnasnmetprogluthrleupro
115120125
asnasnsercystyrseralaglyilealalysleuglugluglyasp
130135140
gluleuglnleualaileproarggluasnalaglnileserleuasp
145150155160
glyaspvalthrphepheglyalaleulysleuleuthrthrthrpro
165170175
alaproargproprothrproalaprothrilealaserglnproleu
180185190
serleuargproglualacysargproalaalaglyglyalavalhis
195200205
thrargglyleuaspphealacysaspiletyriletrpalaproleu
210215220
alaglythrcysglyvalleuleuleuserleuvalilethrleutyr
225230235240
cysargserlysargserargleuleuhisserasptyrmetasnmet
245250255
thrproargargproglyprothrarglyshistyrglnprotyrala
260265270
proproargaspphealaalatyrargserargvallyspheserarg
275280285
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
290295300
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
305310315320
argglyargaspproglumetglyglylysproargarglysasnpro
325330335
glngluglyleutyrasngluleuglnlysasplysmetalagluala
340345350
tyrsergluileglymetlysglygluargargargglylysglyhis
355360365
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
370375380
alaleuhismetglnalaleuproproargglyserglygluglyarg
385390395400
glyserleuleuthrcysglyaspvalglugluasnproglypromet
405410415
glutyralaseraspalaserleuaspproglualaprotrppropro
420425430
alaproargalaargalacysargvalleuprotrpalaleuvalala
435440445
glyleuleuleuleuleuleuleualaalaalacysalavalpheleu
450455460
alacysprotrpalavalserglyalaargalaserproglyserala
465470475480
alaserproargleuarggluglyprogluleuserproaspasppro
485490495
alaglyleuleuaspleuargglnglymetphealaglnleuvalala
500505510
glnasnvalleuleuileaspglyproleusertrptyrserasppro
515520525
glyleualaglyvalserleuthrglyglyleusertyrlysgluasp
530535540
thrlysgluleuvalvalalalysalaglyvaltyrtyrvalphephe
545550555560
glnleugluleuargargvalvalalaglygluglyserglyserval
565570575
serleualaleuhisleuglnproleuargseralaalaglyalaala
580585590
alaleualaleuthrvalaspleuproproalaserserglualaarg
595600605
asnseralapheglypheglnglyargleuleuhisleuseralagly
610615620
glnargleuglyvalhisleuhisthrglualaargalaarghisala
625630635640
trpglnleuthrglnglyalathrvalleuglyleupheargvalthr
645650655
progluileproalaglyleuproserproargserglu
660665
<210>83
<211>2096
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>83
gcgatcgctgccatcgataccatggccttaccagtgaccgccttgctcctgccgctggcc60
ttgctgctccacgccgccaggccggttcagggtccagaagaaacagtcactcaagactgc120
ttgcaactgattgcagacagtgaaacaccaactatacaaaaaggatcttacacatttgtt180
ccatggcttctcagctttaaaaggggaagtgccctagaagaaaaagagaataaaatattg240
gtcaaagaaactggttacttttttatatatggtcaggttttatatactgataagacctac300
gccatgggacatctaattcagaggaagaaggtccatgtctttggggatgaattgagtctg360
gtgactttgtttcgatgtattcaaaatatgcctgaaacactacccaataattcctgctat420
tcagctggcattgcaaaactggaagaaggagatgaactccaacttgcaataccaagagaa480
aatgcacaaatatcactggatggagatgtcacattttttggtgcattgaaactgctgacc540
acgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtcc600
ctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggac660
ttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttctcctg720
tcactggttatcaccctttactgcaggagtaagaggagcaggctcctgcacagtgactac780
atgaacatgactccccgccgccccgggcccacccgcaagcattaccagccctatgcccca840
ccacgcgacttcgcagcctatcgctccagagtgaagttcagcaggagcgcagacgccccc900
gcgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaagagaggag960
tacgatgttttggacaagagacgtggccgggaccctgagatggggggaaagccgagaagg1020
aagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctac1080
agtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccag1140
ggtctcagtacagccaccaaggacacctacgacgcccttcacatgcaggccctgccccct1200
cgcggcagcggcgaaggccgcggcagcctgctgacctgcggcgatgtggaagaaaacccg1260
ggccccatgtacagaatgcagctgctgagctgcatcgccctgagcctggccctggtgacc1320
aacagcggcatccacgtgttcatcctgggctgcttcagcgccggcctgcccaagaccgag1380
gccaactgggtgaacgtgatcagcgacctgaagaagatcgaggacctgatccagagcatg1440
cacatcgacgccaccctgtacaccgagagcgacgtgcaccccagctgcaaggtgaccgcc1500
atgaagtgcttcctgctggagctgcaggtgatcagcctggagagcggcgacgccagcatc1560
cacgacaccgtggagaacctgatcatcctggccaacaacagcctgagcagcaacggcaac1620
gtgaccgagagcggctgcaaggagtgcgaggagctggaggagaagaacatcaaggagttc1680
ctgcagagcttcgtgcacatcgtgcagatgttcatcaacaccagctccggcggcggctcc1740
ggcggcggcggctccggcggcggcggctccggcggcggcggctccggcggcggctccctg1800
caggcccccagaagagccagaggctgcagaaccctgggcctgcccgccctgctgctgctg1860
ctgctgctgagaccccccgccaccagaggcatcacctgccccccccccatgagcgtggag1920
cacgccgacatctgggtgaagagctacagcctgtacagcagagagagatacatctgcaac1980
agcggcttcaagagaaaggccggcaccagcagcctgaccgagtgcgtgctgaacaaggcc2040
accaacgtggcccactggaccacccccagcctgaagtgcatcagataagtttaaac2096
<210>84
<211>688
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>84
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprovalglnglyproglugluthrvalthrglnasp
202530
cysleuglnleuilealaaspsergluthrprothrileglnlysgly
354045
sertyrthrphevalprotrpleuleuserphelysargglyserala
505560
leugluglulysgluasnlysileleuvallysgluthrglytyrphe
65707580
pheiletyrglyglnvalleutyrthrasplysthrtyralametgly
859095
hisleuileglnarglyslysvalhisvalpheglyaspgluleuser
100105110
leuvalthrleupheargcysileglnasnmetprogluthrleupro
115120125
asnasnsercystyrseralaglyilealalysleuglugluglyasp
130135140
gluleuglnleualaileproarggluasnalaglnileserleuasp
145150155160
glyaspvalthrphepheglyalaleulysleuleuthrthrthrpro
165170175
alaproargproprothrproalaprothrilealaserglnproleu
180185190
serleuargproglualacysargproalaalaglyglyalavalhis
195200205
thrargglyleuaspphealacysaspiletyriletrpalaproleu
210215220
alaglythrcysglyvalleuleuleuserleuvalilethrleutyr
225230235240
cysargserlysargserargleuleuhisserasptyrmetasnmet
245250255
thrproargargproglyprothrarglyshistyrglnprotyrala
260265270
proproargaspphealaalatyrargserargvallyspheserarg
275280285
seralaaspalaproalatyrglnglnglyglnasnglnleutyrasn
290295300
gluleuasnleuglyargarggluglutyraspvalleuasplysarg
305310315320
argglyargaspproglumetglyglylysproargarglysasnpro
325330335
glngluglyleutyrasngluleuglnlysasplysmetalagluala
340345350
tyrsergluileglymetlysglygluargargargglylysglyhis
355360365
aspglyleutyrglnglyleuserthralathrlysaspthrtyrasp
370375380
alaleuhismetglnalaleuproproargglyserglygluglyarg
385390395400
glyserleuleuthrcysglyaspvalglugluasnproglypromet
405410415
tyrargmetglnleuleusercysilealaleuserleualaleuval
420425430
thrasnserglyilehisvalpheileleuglycyspheseralagly
435440445
leuprolysthrglualaasntrpvalasnvalileseraspleulys
450455460
lysilegluaspleuileglnsermethisileaspalathrleutyr
465470475480
thrgluseraspvalhisprosercyslysvalthralametlyscys
485490495
pheleuleugluleuglnvalileserleugluserglyaspalaser
500505510
ilehisaspthrvalgluasnleuileileleualaasnasnserleu
515520525
serserasnglyasnvalthrgluserglycyslysglucysgluglu
530535540
leugluglulysasnilelysglupheleuglnserphevalhisile
545550555560
valglnmetpheileasnthrserserglyglyglyserglyglygly
565570575
glyserglyglyglyglyserglyglyglyglyserglyglyglyser
580585590
leuglnalaproargargalaargglycysargthrleuglyleupro
595600605
alaleuleuleuleuleuleuleuargproproalathrargglyile
610615620
thrcysproproprometservalgluhisalaaspiletrpvallys
625630635640
sertyrserleutyrserarggluargtyrilecysasnserglyphe
645650655
lysarglysalaglythrserserleuthrglucysvalleuasnlys
660665670
alathrasnvalalahistrpthrthrproserleulyscysilearg
675680685
<210>85
<211>2755
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>85
gcgatcgcaccatggccctgcccgtgaccgccctgctgctgcccctggccctgctgctgc60
acgccgccaggcccgccgttcagggtccagaagaaacagtcactcaagactgcttgcaac120
tgattgcagacagtgaaacaccaactatacaaaaaggatcttacacatttgttccatggc180
ttctcagctttaaaaggggaagtgccctagaagaaaaagagaataaaatattggtcaaag240
aaactggttacttttttatatatggtcaggttttatatactgataagacctacgccatgg300
gacatctaattcagaggaagaaggtccatgtctttggggatgaattgagtctggtgactt360
tgtttcgatgtattcaaaatatgcctgaaacactacccaataattcctgctattcagctg420
gcattgcaaaactggaagaaggagatgaactccaacttgcaataccaagagaaaatgcac480
aaatatcactggatggagatgtcacattttttggtgcattgaaactgctgaccacgacgc540
cagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcc600
cagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggacttcgcct660
gtgatatctacatctgggcgcccttggccgggacttgtggggtccttctcctgtcactgg720
ttatcaccctttactgcaggagtaagaggagcaggctcctgcacagtgactacatgaaca780
tgactccccgccgccccgggcccacccgcaagcattaccagccctatgccccaccacgcg840
acttcgcagcctatcgctccagagtgaagttcagcaggagcgcagacgcccccgcgtacc900
agcagggccagaaccagctctataacgagctcaatctaggacgaagagaggagtacgatg960
ttttggacaagagacgtggccgggaccctgagatggggggaaagccgagaaggaagaacc1020
ctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgaga1080
ttgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctca1140
gtacagccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaa1200
gcggagccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggcc1260
ccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacgccgcca1320
ggccggaggtccagctgcagcagtctggacctgagctgataaagcctggggcttcagtga1380
agatgtcctgcaaggcttctggatacacattcactagctatgttatgcactgggtgaagc1440
agaagcctgggcagggccttgagtggattggatatattaatccttacaatgatggtacta1500
agtacaatgagaagttcaaaggcaaggccacactgacttcagacaaatcctccagcacag1560
cctacatggagctcagcagcctgacctctgaggactctgcggtctattactgtgcaagag1620
ggacttattactacggtagtagggtatttgactactggggccaaggcaccactctcacag1680
tctcctcaggtggagggggctcaggcggaggtggctctgggggtggaggctcggacattg1740
tgatgactcaggctgcaccctctatacctgtcactcctggagagtcagtatccatctcct1800
gcaggtctagtaagagtctcctgaatagtaatggcaacacttacttgtattggttcctgc1860
agaggccaggccagtctcctcagctcctgatatatcggatgtccaaccttgcctcaggag1920
tcccagacaggttcagtggcagtgggtcaggaactgctttcacactgagaatcagtagag1980
tggaggctgaggatgtgggtgtttattactgtatgcaacatctagaatatccgttcacgt2040
tcggtgctgggaccaagctggagctgaaacggaccacgacgccagcgccgcgaccaccaa2100
caccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccag2160
cggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctggg2220
cgcccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgca2280
aacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccagtacaaa2340
ctactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtg2400
aactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaacc2460
agctctataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagac2520
gtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcc2580
tgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaag2640
gcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccacca2700
aggacacctacgacgcccttcacatgcaggccctgccccctcgctaagtttaaac2755
<210>86
<211>911
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>86
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproalavalglnglyproglugluthrvalthrgln
202530
aspcysleuglnleuilealaaspsergluthrprothrileglnlys
354045
glysertyrthrphevalprotrpleuleuserphelysargglyser
505560
alaleugluglulysgluasnlysileleuvallysgluthrglytyr
65707580
phepheiletyrglyglnvalleutyrthrasplysthrtyralamet
859095
glyhisleuileglnarglyslysvalhisvalpheglyaspgluleu
100105110
serleuvalthrleupheargcysileglnasnmetprogluthrleu
115120125
proasnasnsercystyrseralaglyilealalysleugluglugly
130135140
aspgluleuglnleualaileproarggluasnalaglnileserleu
145150155160
aspglyaspvalthrphepheglyalaleulysleuleuthrthrthr
165170175
proalaproargproprothrproalaprothrilealaserglnpro
180185190
leuserleuargproglualacysargproalaalaglyglyalaval
195200205
histhrargglyleuaspphealacysaspiletyriletrpalapro
210215220
leualaglythrcysglyvalleuleuleuserleuvalilethrleu
225230235240
tyrcysargserlysargserargleuleuhisserasptyrmetasn
245250255
metthrproargargproglyprothrarglyshistyrglnprotyr
260265270
alaproproargaspphealaalatyrargserargvallyspheser
275280285
argseralaaspalaproalatyrglnglnglyglnasnglnleutyr
290295300
asngluleuasnleuglyargarggluglutyraspvalleuasplys
305310315320
argargglyargaspproglumetglyglylysproargarglysasn
325330335
proglngluglyleutyrasngluleuglnlysasplysmetalaglu
340345350
alatyrsergluileglymetlysglygluargargargglylysgly
355360365
hisaspglyleutyrglnglyleuserthralathrlysaspthrtyr
370375380
aspalaleuhismetglnalaleuproproargglyserglyalathr
385390395400
asnpheserleuleulysglnalaglyaspvalglugluasnprogly
405410415
prometalaleuprovalthralaleuleuleuproleualaleuleu
420425430
leuhisalaalaargprogluvalglnleuglnglnserglyproglu
435440445
leuilelysproglyalaservallysmetsercyslysalasergly
450455460
tyrthrphethrsertyrvalmethistrpvallysglnlysprogly
465470475480
glnglyleuglutrpileglytyrileasnprotyrasnaspglythr
485490495
lystyrasnglulysphelysglylysalathrleuthrserasplys
500505510
serserserthralatyrmetgluleuserserleuthrsergluasp
515520525
seralavaltyrtyrcysalaargglythrtyrtyrtyrglyserarg
530535540
valpheasptyrtrpglyglnglythrthrleuthrvalsersergly
545550555560
glyglyglyserglyglyglyglyserglyglyglyglyseraspile
565570575
valmetthrglnalaalaproserileprovalthrproglygluser
580585590
valserilesercysargserserlysserleuleuasnserasngly
595600605
asnthrtyrleutyrtrppheleuglnargproglyglnserprogln
610615620
leuleuiletyrargmetserasnleualaserglyvalproasparg
625630635640
pheserglyserglyserglythralaphethrleuargileserarg
645650655
valglualagluaspvalglyvaltyrtyrcysmetglnhisleuglu
660665670
tyrprophethrpheglyalaglythrlysleugluleulysargthr
675680685
thrthrproalaproargproprothrproalaprothrilealaser
690695700
glnproleuserleuargproglualacysargproalaalaglygly
705710715720
alavalhisthrargglyleuaspphealacysaspiletyriletrp
725730735
alaproleualaglythrcysglyvalleuleuleuserleuvalile
740745750
thrleutyrcyslysargglyarglyslysleuleutyrilephelys
755760765
glnprophemetargprovalglnthrthrglnglugluaspglycys
770775780
sercysargpheproglugluglugluglyglycysgluleuargval
785790795800
lyspheserargseralaaspalaproalatyrglnglnglyglnasn
805810815
glnleutyrasngluleuasnleuglyargarggluglutyraspval
820825830
leuasplysargargglyargaspproglumetglyglylysprogln
835840845
argarglysasnproglngluglyleutyrasngluleuglnlysasp
850855860
lysmetalaglualatyrsergluileglymetlysglygluargarg
865870875880
argglylysglyhisaspglyleutyrglnglyleuserthralathr
885890895
lysaspthrtyraspalaleuhismetglnalaleuproproarg
900905910
<210>87
<211>2734
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>87
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggtgctcacccaaaaacagaagaagcagcactctgtcctgcacctgg120
ttcccattaacgccacctccaaggatgactccgatgtgacagaggtgatgtggcaaccag180
ctcttaggcgtgggagaggcctacaggcccaaggatatggtgtccgaatccaggatgctg240
gagtttatctgctgtatagccaggtcctgtttcaagacgtgactttcaccatgggtcagg300
tggtgtctcgagaaggccaaggaaggcaggagactctattccgatgtataagaagtatgc360
cctcccacccggaccgggcctacaacagctgctatagcgcaggtgtcttccatttacacc420
aaggggatattctgagtgtcataattccccgggcaagggcgaaacttaacctctctccac480
atggaaccttcctggggtttgtgaaactgaccacgacgccagcgccgcgaccaccaacac540
cggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcgg600
cggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgc660
ccttggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcagga720
gtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggc780
ccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctcca840
gagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctct900
ataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggcc960
gggaccctgagatggggggaaagccgagaaggaagaaccctcaggaaggcctgtacaatg1020
aactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgcc1080
ggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacacct1140
acgacgcccttcacatgcaggccctgccccctcgcggaagcggagccaccaacttcagcc1200
tgctgaagcaggccggcgacgtggaggagaaccccggccccatggccttaccagtgaccg1260
ccttgctcctgccgctggccttgctgctccacgccgccaggccggaggtccagctgcagc1320
agtctggacctgagctgataaagcctggggcttcagtgaagatgtcctgcaaggcttctg1380
gatacacattcactagctatgttatgcactgggtgaagcagaagcctgggcagggccttg1440
agtggattggatatattaatccttacaatgatggtactaagtacaatgagaagttcaaag1500
gcaaggccacactgacttcagacaaatcctccagcacagcctacatggagctcagcagcc1560
tgacctctgaggactctgcggtctattactgtgcaagagggacttattactacggtagta1620
gggtatttgactactggggccaaggcaccactctcacagtctcctcaggtggagggggct1680
caggcggaggtggctctgggggtggaggctcggacattgtgatgactcaggctgcaccct1740
ctatacctgtcactcctggagagtcagtatccatctcctgcaggtctagtaagagtctcc1800
tgaatagtaatggcaacacttacttgtattggttcctgcagaggccaggccagtctcctc1860
agctcctgatatatcggatgtccaaccttgcctcaggagtcccagacaggttcagtggca1920
gtgggtcaggaactgctttcacactgagaatcagtagagtggaggctgaggatgtgggtg1980
tttattactgtatgcaacatctagaatatccgttcacgttcggtgctgggaccaagctgg2040
agctgaaacggaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgt2100
cgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcaca2160
cgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtg2220
gggtccttctcctgtcactggttatcaccctttactgcaaacggggcagaaagaaactcc2280
tgtatatattcaaacaaccatttatgagaccagtacaaactactcaagaggaagatggct2340
gtagctgccgatttccagaagaagaagaaggaggatgtgaactgagagtgaagttcagca2400
ggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatc2460
taggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctgagatgg2520
ggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaag2580
ataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaagg2640
ggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttc2700
acatgcaggccctgccccctcgctaagtttaaac2734
<210>88
<211>904
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>88
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprovalleuthrglnlysglnlyslysglnhisser
202530
valleuhisleuvalproileasnalathrserlysaspaspserasp
354045
valthrgluvalmettrpglnproalaleuargargglyargglyleu
505560
glnalaglnglytyrglyvalargileglnaspalaglyvaltyrleu
65707580
leutyrserglnvalleupheglnaspvalthrphethrmetglygln
859095
valvalserarggluglyglnglyargglngluthrleupheargcys
100105110
ileargsermetproserhisproaspargalatyrasnsercystyr
115120125
seralaglyvalphehisleuhisglnglyaspileleuservalile
130135140
ileproargalaargalalysleuasnleuserprohisglythrphe
145150155160
leuglyphevallysleuthrthrthrproalaproargproprothr
165170175
proalaprothrilealaserglnproleuserleuargprogluala
180185190
cysargproalaalaglyglyalavalhisthrargglyleuaspphe
195200205
alacysaspiletyriletrpalaproleualaglythrcysglyval
210215220
leuleuleuserleuvalilethrleutyrcysargserlysargser
225230235240
argleuleuhisserasptyrmetasnmetthrproargargprogly
245250255
prothrarglyshistyrglnprotyralaproproargasppheala
260265270
alatyrargserargvallyspheserargseralaaspalaproala
275280285
tyrglnglnglyglnasnglnleutyrasngluleuasnleuglyarg
290295300
arggluglutyraspvalleuasplysargargglyargaspproglu
305310315320
metglyglylysproargarglysasnproglngluglyleutyrasn
325330335
gluleuglnlysasplysmetalaglualatyrsergluileglymet
340345350
lysglygluargargargglylysglyhisaspglyleutyrglngly
355360365
leuserthralathrlysaspthrtyraspalaleuhismetglnala
370375380
leuproproargglyserglyalathrasnpheserleuleulysgln
385390395400
alaglyaspvalglugluasnproglyprometalaleuprovalthr
405410415
alaleuleuleuproleualaleuleuleuhisalaalaargproglu
420425430
valglnleuglnglnserglyprogluleuilelysproglyalaser
435440445
vallysmetsercyslysalaserglytyrthrphethrsertyrval
450455460
methistrpvallysglnlysproglyglnglyleuglutrpilegly
465470475480
tyrileasnprotyrasnaspglythrlystyrasnglulysphelys
485490495
glylysalathrleuthrserasplysserserserthralatyrmet
500505510
gluleuserserleuthrsergluaspseralavaltyrtyrcysala
515520525
argglythrtyrtyrtyrglyserargvalpheasptyrtrpglygln
530535540
glythrthrleuthrvalserserglyglyglyglyserglyglygly
545550555560
glyserglyglyglyglyseraspilevalmetthrglnalaalapro
565570575
serileprovalthrproglygluservalserilesercysargser
580585590
serlysserleuleuasnserasnglyasnthrtyrleutyrtrpphe
595600605
leuglnargproglyglnserproglnleuleuiletyrargmetser
610615620
asnleualaserglyvalproaspargpheserglyserglysergly
625630635640
thralaphethrleuargileserargvalglualagluaspvalgly
645650655
valtyrtyrcysmetglnhisleuglutyrprophethrpheglyala
660665670
glythrlysleugluleulysargthrthrthrproalaproargpro
675680685
prothrproalaprothrilealaserglnproleuserleuargpro
690695700
glualacysargproalaalaglyglyalavalhisthrargglyleu
705710715720
aspphealacysaspiletyriletrpalaproleualaglythrcys
725730735
glyvalleuleuleuserleuvalilethrleutyrcyslysarggly
740745750
arglyslysleuleutyrilephelysglnprophemetargproval
755760765
glnthrthrglnglugluaspglycyssercysargpheprogluglu
770775780
glugluglyglycysgluleuargvallyspheserargseralaasp
785790795800
alaproalatyrglnglnglyglnasnglnleutyrasngluleuasn
805810815
leuglyargarggluglutyraspvalleuasplysargargglyarg
820825830
aspproglumetglyglylysproglnargarglysasnproglnglu
835840845
glyleutyrasngluleuglnlysasplysmetalaglualatyrser
850855860
gluileglymetlysglygluargargargglylysglyhisaspgly
865870875880
leutyrglnglyleuserthralathrlysaspthrtyraspalaleu
885890895
hismetglnalaleuproproarg
900
<210>89
<211>1498
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>89
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgatgacccaaagccccgacagcctggccgtgagcctgggcg120
agagggtgaccatgaactgcaaaagcagccagtccctgctgtactccaccaaccagaaga180
actacctggcttggtatcaacagaagcccggacagagccccaagctgctgatctattggg240
ccagcactagggaaagcggcgtgcccgataggttcagcggcagcgggagcggcacagact300
tcactctgaccattagcagcgtgcaggctgaggatgtggccgtctactactgccagcagt360
actacagctacaggacctttgggggcggaactaagctggagatcaagggaggggggggat420
ccgggggaggaggctccggcggaggcggaagccaagtgcaactgcagcagagcggcccag480
aggtggtcaaacctggggcaagcgtgaagatgagctgcaaggctagcggctataccttca540
ccagctatgtgatccactgggtgaggcagaaaccaggacagggcctggactggatcggct600
acatcaacccctacaatgacggcaccgattatgacgaaaaattcaaggggaaggccaccc660
tgaccagcgacaccagcacaagcaccgcctacatggagctgtccagcctgaggtccgagg720
acaccgccgtgtattactgtgccagggagaaggacaattacgccaccggcgcttggttcg780
cctactggggccagggcacactggtgacagtgagcagcaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcaggagtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgcc1080
gccccgggcccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcct1140
atcgctccagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccaga1200
accagctctataacgagctcaatctaggacgaagagaggagtacgatgttttggacaaga1260
gacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctcaggaag1320
gcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattgggatga1380
aaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacagcca1440
ccaaggacacctacgacgcccttcacatgcaggccctgccccctcgctaagtttaaac1498
<210>90
<211>493
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>90
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalmetthrglnserproaspserleu
202530
alavalserleuglygluargvalthrmetasncyslyssersergln
354045
serleuleutyrserthrasnglnlysasntyrleualatrptyrgln
505560
glnlysproglyglnserprolysleuleuiletyrtrpalaserthr
65707580
arggluserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleuthrileserservalglnalagluaspvalalaval
100105110
tyrtyrcysglnglntyrtyrsertyrargthrpheglyglyglythr
115120125
lysleugluilelysglyglyglyglyserglyglyglyglysergly
130135140
glyglyglyserglnvalglnleuglnglnserglyprogluvalval
145150155160
lysproglyalaservallysmetsercyslysalaserglytyrthr
165170175
phethrsertyrvalilehistrpvalargglnlysproglyglngly
180185190
leuasptrpileglytyrileasnprotyrasnaspglythrasptyr
195200205
aspglulysphelysglylysalathrleuthrseraspthrserthr
210215220
serthralatyrmetgluleuserserleuargsergluaspthrala
225230235240
valtyrtyrcysalaargglulysaspasntyralathrglyalatrp
245250255
phealatyrtrpglyglnglythrleuvalthrvalserserthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcysargserlysargserargleuleuhisserasptyrmet
340345350
asnmetthrproargargproglyprothrarglyshistyrglnpro
355360365
tyralaproproargaspphealaalatyrargserargvallysphe
370375380
serargseralaaspalaproalatyrglnglnglyglnasnglnleu
385390395400
tyrasngluleuasnleuglyargarggluglutyraspvalleuasp
405410415
lysargargglyargaspproglumetglyglylysproglnargarg
420425430
lysasnproglngluglyleutyrasngluleuglnlysasplysmet
435440445
alaglualatyrsergluileglymetlysglygluargargarggly
450455460
lysglyhisaspglyleutyrglnglyleuserthralathrlysasp
465470475480
thrtyraspalaleuhismetglnalaleuproproarg
485490
<210>91
<211>2560
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>91
gcgatcgcatgtatttgtggcttaaactcttggcatttggctttgcctttctggacacag60
aagtatttgtgacaggggacatcgtgatgacccaaagccccgacagcctggccgtgagcc120
tgggcgagagggtgaccatgaactgcaaaagcagccagtccctgctgtactccaccaacc180
agaagaactacctggcttggtatcaacagaagcccggacagagccccaagctgctgatct240
attgggccagcactagggaaagcggcgtgcccgataggttcagcggcagcgggagcggca300
cagacttcactctgaccattagcagcgtgcaggctgaggatgtggccgtctactactgcc360
agcagtactacagctacaggacctttgggggcggaactaagctggagatcaagggagggg420
ggggatccgggggaggaggctccggcggaggcggaagccaagtgcaactgcagcagagcg480
gcccagaggtggtcaaacctggggcaagcgtgaagatgagctgcaaggctagcggctata540
ccttcaccagctatgtgatccactgggtgaggcagaaaccaggacagggcctggactgga600
tcggctacatcaacccctacaatgacggcaccgattatgacgaaaaattcaaggggaagg660
ccaccctgaccagcgacaccagcacaagcaccgcctacatggagctgtccagcctgaggt720
ccgaggacaccgccgtgtattactgtgccagggagaaggacaattacgccaccggcgctt780
ggttcgcctactggggccagggcacactggtgacagtgagcagcaccacgacgccagcgc840
cgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagagg900
cgtgccggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgata960
tctacatctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatca1020
ccctttactgcagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggcc1080
agaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttggaca1140
agagacgtggccgggaccctgagatggggggaaagccgagaaggaagaaccctcaggaag1200
gcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattgggatga1260
aaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacagcca1320
ccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcggagcca1380
ccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggcccctacctgt1440
ggctgaagctgctggccttcggcttcgccttcctggacaccgaggtgttcgtgaccggcg1500
acatccagatgacccagagccccagcaccctgagcgccagcgtgggcgacagggtgacca1560
tcacctgcagcgccagcagcagcatcagctacatgcactggtaccagcagaagcccggca1620
aggcccccaagctgctgatctacaccaccagcaacctggccagcggcgtgcccgccaggt1680
tcagcggcagcggcagcggcaccgagttcaccctgaccatcagcagcctgcagcccgacg1740
acttcgccacctactactgccaccagaggagcacctaccccctgaccttcggccagggca1800
ccaaggtggaggtgaagggcggcggcggcagcggcggcggcggcagcggcggcggcggca1860
gccaggtgcagctggtgcagagcggcgccgaggtgaagaagcccggcagcagcgtgaagg1920
tgagctgcaaggccagcggctacaccttcaccagctacaggatgcactgggtgaggcagg1980
cccccggccagggcctggagtggatcggctacatcaaccccagcaccggctacaccgagt2040
acaaccagaagttcaaggacaaggccaccatcaccgccgacgagagcaccaacaccgcct2100
acatggagctgagcagcctgaggagcgaggacaccgccgtgtactactgcgccaggggcg2160
gcggcgtgttcgactactggggccagggcaccctggtgaccgtgagcagcaccaccaccc2220
ccgcccccaggccccccacccccgcccccaccatcgccagccagcccctgagcctgaggc2280
ccgaggcctgcaggcccgccgccggcggcgccgtgcacaccaggggcctggacttcgcct2340
gcgacttttgggtgctggtggtggttggtggagtcctggcttgctatagcttgctagtaa2400
cagtggcctttattattttctgggtgaggagtaagaggagcaggctcctgcacagtgact2460
acatgaacatgactccccgccgccccgggcccacccgcaagcattaccagccctatgccc2520
caccacgcgacttcgcagcctatcgctcctaagtttaaac2560
<210>92
<211>847
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>92
mettyrleutrpleulysleuleualapheglyphealapheleuasp
151015
thrgluvalphevalthrglyaspilevalmetthrglnserproasp
202530
serleualavalserleuglygluargvalthrmetasncyslysser
354045
serglnserleuleutyrserthrasnglnlysasntyrleualatrp
505560
tyrglnglnlysproglyglnserprolysleuleuiletyrtrpala
65707580
serthrarggluserglyvalproaspargpheserglyserglyser
859095
glythraspphethrleuthrileserservalglnalagluaspval
100105110
alavaltyrtyrcysglnglntyrtyrsertyrargthrpheglygly
115120125
glythrlysleugluilelysglyglyglyglyserglyglyglygly
130135140
serglyglyglyglyserglnvalglnleuglnglnserglyproglu
145150155160
valvallysproglyalaservallysmetsercyslysalasergly
165170175
tyrthrphethrsertyrvalilehistrpvalargglnlysprogly
180185190
glnglyleuasptrpileglytyrileasnprotyrasnaspglythr
195200205
asptyraspglulysphelysglylysalathrleuthrseraspthr
210215220
serthrserthralatyrmetgluleuserserleuargsergluasp
225230235240
thralavaltyrtyrcysalaargglulysaspasntyralathrgly
245250255
alatrpphealatyrtrpglyglnglythrleuvalthrvalserser
260265270
thrthrthrproalaproargproprothrproalaprothrileala
275280285
serglnproleuserleuargproglualacysargproalaalagly
290295300
glyalavalhisthrargglyleuaspphealacysaspiletyrile
305310315320
trpalaproleualaglythrcysglyvalleuleuleuserleuval
325330335
ilethrleutyrcysargvallyspheserargseralaaspalapro
340345350
alatyrglnglnglyglnasnglnleutyrasngluleuasnleugly
355360365
argarggluglutyraspvalleuasplysargargglyargasppro
370375380
glumetglyglylysproargarglysasnproglngluglyleutyr
385390395400
asngluleuglnlysasplysmetalaglualatyrsergluilegly
405410415
metlysglygluargargargglylysglyhisaspglyleutyrgln
420425430
glyleuserthralathrlysaspthrtyraspalaleuhismetgln
435440445
alaleuproproargglyserglyalathrasnpheserleuleulys
450455460
glnalaglyaspvalglugluasnproglyprotyrleutrpleulys
465470475480
leuleualapheglyphealapheleuaspthrgluvalphevalthr
485490495
glyaspileglnmetthrglnserproserthrleuseralaserval
500505510
glyaspargvalthrilethrcysseralaserserserilesertyr
515520525
methistrptyrglnglnlysproglylysalaprolysleuleuile
530535540
tyrthrthrserasnleualaserglyvalproalaargphesergly
545550555560
serglyserglythrgluphethrleuthrileserserleuglnpro
565570575
aspaspphealathrtyrtyrcyshisglnargserthrtyrproleu
580585590
thrpheglyglnglythrlysvalgluvallysglyglyglyglyser
595600605
glyglyglyglyserglyglyglyglyserglnvalglnleuvalgln
610615620
serglyalagluvallyslysproglyserservallysvalsercys
625630635640
lysalaserglytyrthrphethrsertyrargmethistrpvalarg
645650655
glnalaproglyglnglyleuglutrpileglytyrileasnproser
660665670
thrglytyrthrglutyrasnglnlysphelysasplysalathrile
675680685
thralaaspgluserthrasnthralatyrmetgluleuserserleu
690695700
argsergluaspthralavaltyrtyrcysalaargglyglyglyval
705710715720
pheasptyrtrpglyglnglythrleuvalthrvalserserthrthr
725730735
thrproalaproargproprothrproalaprothrilealasergln
740745750
proleuserleuargproglualacysargproalaalaglyglyala
755760765
valhisthrargglyleuaspphealacysaspphetrpvalleuval
770775780
valvalglyglyvalleualacystyrserleuleuvalthrvalala
785790795800
pheileilephetrpvalargserlysargserargleuleuhisser
805810815
asptyrmetasnmetthrproargargproglyprothrarglyshis
820825830
tyrglnprotyralaproproargaspphealaalatyrargser
835840845
<210>93
<211>2551
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>93
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgatgacccaaagccccgacagcctggccgtgagcctgggcg120
agagggtgaccatgaactgcaaaagcagccagtccctgctgtactccaccaaccagaaga180
actacctggcttggtatcaacagaagcccggacagagccccaagctgctgatctattggg240
ccagcactagggaaagcggcgtgcccgataggttcagcggcagcgggagcggcacagact300
tcactctgaccattagcagcgtgcaggctgaggatgtggccgtctactactgccagcagt360
actacagctacaggacctttgggggcggaactaagctggagatcaagggaggggggggat420
ccgggggaggaggctccggcggaggcggaagccaagtgcaactgcagcagagcggcccag480
aggtggtcaaacctggggcaagcgtgaagatgagctgcaaggctagcggctataccttca540
ccagctatgtgatccactgggtgaggcagaaaccaggacagggcctggactggatcggct600
acatcaacccctacaatgacggcaccgattatgacgaaaaattcaaggggaaggccaccc660
tgaccagcgacaccagcacaagcaccgcctacatggagctgtccagcctgaggtccgagg720
acaccgccgtgtattactgtgccagggagaaggacaattacgccaccggcgcttggttcg780
cctactggggccagggcacactggtgacagtgagcagcaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaacc1080
agctctataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagac1140
gtggccgggaccctgagatggggggaaagccgagaaggaagaaccctcaggaaggcctgt1200
acaatgaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcg1260
agcgccggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaagg1320
acacctacgacgcccttcacatgcaggccctgccccctcgcggaagcggagccaccaact1380
tcagcctgctgaagcaggccggcgacgtggaggagaaccccggccccatggccctgcccg1440
tgaccgccctgctgctgcccctggccctgctgctgcacgccgccaggcccgacatccaga1500
tgacccagagccccagcaccctgagcgccagcgtgggcgacagggtgaccatcacctgca1560
gcgccagcagcagcatcagctacatgcactggtaccagcagaagcccggcaaggccccca1620
agctgctgatctacaccaccagcaacctggccagcggcgtgcccgccaggttcagcggca1680
gcggcagcggcaccgagttcaccctgaccatcagcagcctgcagcccgacgacttcgcca1740
cctactactgccaccagaggagcacctaccccctgaccttcggccagggcaccaaggtgg1800
aggtgaagggcggcggcggcagcggcggcggcggcagcggcggcggcggcagccaggtgc1860
agctggtgcagagcggcgccgaggtgaagaagcccggcagcagcgtgaaggtgagctgca1920
aggccagcggctacaccttcaccagctacaggatgcactgggtgaggcaggcccccggcc1980
agggcctggagtggatcggctacatcaaccccagcaccggctacaccgagtacaaccaga2040
agttcaaggacaaggccaccatcaccgccgacgagagcaccaacaccgcctacatggagc2100
tgagcagcctgaggagcgaggacaccgccgtgtactactgcgccaggggcggcggcgtgt2160
tcgactactggggccagggcaccctggtgaccgtgagcagcaccaccacccccgccccca2220
ggccccccacccccgcccccaccatcgccagccagcccctgagcctgaggcccgaggcct2280
gcaggcccgccgccggcggcgccgtgcacaccaggggcctggacttcgcctgcgactttt2340
gggtgctggtggtggttggtggagtcctggcttgctatagcttgctagtaacagtggcct2400
ttattattttctgggtgaggagtaagaggagcaggctcctgcacagtgactacatgaaca2460
tgactccccgccgccccgggcccacccgcaagcattaccagccctatgccccaccacgcg2520
acttcgcagcctatcgctcctaagtttaaac2551
<210>94
<211>844
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>94
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalmetthrglnserproaspserleu
202530
alavalserleuglygluargvalthrmetasncyslyssersergln
354045
serleuleutyrserthrasnglnlysasntyrleualatrptyrgln
505560
glnlysproglyglnserprolysleuleuiletyrtrpalaserthr
65707580
arggluserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleuthrileserservalglnalagluaspvalalaval
100105110
tyrtyrcysglnglntyrtyrsertyrargthrpheglyglyglythr
115120125
lysleugluilelysglyglyglyglyserglyglyglyglysergly
130135140
glyglyglyserglnvalglnleuglnglnserglyprogluvalval
145150155160
lysproglyalaservallysmetsercyslysalaserglytyrthr
165170175
phethrsertyrvalilehistrpvalargglnlysproglyglngly
180185190
leuasptrpileglytyrileasnprotyrasnaspglythrasptyr
195200205
aspglulysphelysglylysalathrleuthrseraspthrserthr
210215220
serthralatyrmetgluleuserserleuargsergluaspthrala
225230235240
valtyrtyrcysalaargglulysaspasntyralathrglyalatrp
245250255
phealatyrtrpglyglnglythrleuvalthrvalserserthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcysargvallyspheserargseralaaspalaproalatyr
340345350
glnglnglyglnasnglnleutyrasngluleuasnleuglyargarg
355360365
gluglutyraspvalleuasplysargargglyargaspproglumet
370375380
glyglylysproargarglysasnproglngluglyleutyrasnglu
385390395400
leuglnlysasplysmetalaglualatyrsergluileglymetlys
405410415
glygluargargargglylysglyhisaspglyleutyrglnglyleu
420425430
serthralathrlysaspthrtyraspalaleuhismetglnalaleu
435440445
proproargglyserglyalathrasnpheserleuleulysglnala
450455460
glyaspvalglugluasnproglyprometalaleuprovalthrala
465470475480
leuleuleuproleualaleuleuleuhisalaalaargproaspile
485490495
glnmetthrglnserproserthrleuseralaservalglyasparg
500505510
valthrilethrcysseralaserserserilesertyrmethistrp
515520525
tyrglnglnlysproglylysalaprolysleuleuiletyrthrthr
530535540
serasnleualaserglyvalproalaargpheserglyserglyser
545550555560
glythrgluphethrleuthrileserserleuglnproaspaspphe
565570575
alathrtyrtyrcyshisglnargserthrtyrproleuthrphegly
580585590
glnglythrlysvalgluvallysglyglyglyglyserglyglygly
595600605
glyserglyglyglyglyserglnvalglnleuvalglnserglyala
610615620
gluvallyslysproglyserservallysvalsercyslysalaser
625630635640
glytyrthrphethrsertyrargmethistrpvalargglnalapro
645650655
glyglnglyleuglutrpileglytyrileasnproserthrglytyr
660665670
thrglutyrasnglnlysphelysasplysalathrilethralaasp
675680685
gluserthrasnthralatyrmetgluleuserserleuargserglu
690695700
aspthralavaltyrtyrcysalaargglyglyglyvalpheasptyr
705710715720
trpglyglnglythrleuvalthrvalserserthrthrthrproala
725730735
proargproprothrproalaprothrilealaserglnproleuser
740745750
leuargproglualacysargproalaalaglyglyalavalhisthr
755760765
argglyleuaspphealacysaspphetrpvalleuvalvalvalgly
770775780
glyvalleualacystyrserleuleuvalthrvalalapheileile
785790795800
phetrpvalargserlysargserargleuleuhisserasptyrmet
805810815
asnmetthrproargargproglyprothrarglyshistyrglnpro
820825830
tyralaproproargaspphealaalatyrargser
835840
<210>95
<211>2149
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>95
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgatgacccaaagccccgacagcctggccgtgagcctgggcg120
agagggtgaccatgaactgcaaaagcagccagtccctgctgtactccaccaaccagaaga180
actacctggcttggtatcaacagaagcccggacagagccccaagctgctgatctattggg240
ccagcactagggaaagcggcgtgcccgataggttcagcggcagcgggagcggcacagact300
tcactctgaccattagcagcgtgcaggctgaggatgtggccgtctactactgccagcagt360
actacagctacaggacctttgggggcggaactaagctggagatcaagggaggggggggat420
ccgggggaggaggctccggcggaggcggaagccaagtgcaactgcagcagagcggcccag480
aggtggtcaaacctggggcaagcgtgaagatgagctgcaaggctagcggctataccttca540
ccagctatgtgatccactgggtgaggcagaaaccaggacagggcctggactggatcggct600
acatcaacccctacaatgacggcaccgattatgacgaaaaattcaaggggaaggccaccc660
tgaccagcgacaccagcacaagcaccgcctacatggagctgtccagcctgaggtccgagg720
acaccgccgtgtattactgtgccagggagaaggacaattacgccaccggcgcttggttcg780
cctactggggccagggcacactggtgacagtgagcagcaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcaggagtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgcc1080
gccccgggcccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcct1140
atcgctccaaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagac1200
cagtacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaag1260
gaggatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagg1320
gccagaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttgg1380
acaagagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctc1440
aggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattg1500
ggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagta1560
cagccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcg1620
gagccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggcccca1680
tgtacagaatgcagctgctgagctgcatcgccctgagcctggccctggtgaccaacagcg1740
gcatccacgtgttcatcctgggctgcttcagcgccggcctgcccaagaccgaggccaact1800
gggtgaacgtgatcagcgacctgaagaagatcgaggacctgatccagagcatgcacatcg1860
acgccaccctgtacaccgagagcgacgtgcaccccagctgcaaggtgaccgccatgaagt1920
gcttcctgctggagctgcaggtgatcagcctggagagcggcgacgccagcatccacgaca1980
ccgtggagaacctgatcatcctggccaacaacagcctgagcagcaacggcaacgtgaccg2040
agagcggctgcaaggagtgcgaggagctggaggagaagaacatcaaggagttcctgcaga2100
gcttcgtgcacatcgtgcagatgttcatcaacaccagctaagtttaaac2149
<210>96
<211>710
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>96
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalmetthrglnserproaspserleu
202530
alavalserleuglygluargvalthrmetasncyslyssersergln
354045
serleuleutyrserthrasnglnlysasntyrleualatrptyrgln
505560
glnlysproglyglnserprolysleuleuiletyrtrpalaserthr
65707580
arggluserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleuthrileserservalglnalagluaspvalalaval
100105110
tyrtyrcysglnglntyrtyrsertyrargthrpheglyglyglythr
115120125
lysleugluilelysglyglyglyglyserglyglyglyglysergly
130135140
glyglyglyserglnvalglnleuglnglnserglyprogluvalval
145150155160
lysproglyalaservallysmetsercyslysalaserglytyrthr
165170175
phethrsertyrvalilehistrpvalargglnlysproglyglngly
180185190
leuasptrpileglytyrileasnprotyrasnaspglythrasptyr
195200205
aspglulysphelysglylysalathrleuthrseraspthrserthr
210215220
serthralatyrmetgluleuserserleuargsergluaspthrala
225230235240
valtyrtyrcysalaargglulysaspasntyralathrglyalatrp
245250255
phealatyrtrpglyglnglythrleuvalthrvalserserthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcysargserlysargserargleuleuhisserasptyrmet
340345350
asnmetthrproargargproglyprothrarglyshistyrglnpro
355360365
tyralaproproargaspphealaalatyrargserlysargglyarg
370375380
lyslysleuleutyrilephelysglnprophemetargprovalgln
385390395400
thrthrglnglugluaspglycyssercysargpheproglugluglu
405410415
gluglyglycysgluleuargvallyspheserargseralaaspala
420425430
proalatyrglnglnglyglnasnglnleutyrasngluleuasnleu
435440445
glyargarggluglutyraspvalleuasplysargargglyargasp
450455460
proglumetglyglylysproglnargarglysasnproglnglugly
465470475480
leutyrasngluleuglnlysasplysmetalaglualatyrserglu
485490495
ileglymetlysglygluargargargglylysglyhisaspglyleu
500505510
tyrglnglyleuserthralathrlysaspthrtyraspalaleuhis
515520525
metglnalaleuproproargglyserglyalathrasnpheserleu
530535540
leulysglnalaglyaspvalglugluasnproglypromettyrarg
545550555560
metglnleuleusercysilealaleuserleualaleuvalthrasn
565570575
serglyilehisvalpheileleuglycyspheseralaglyleupro
580585590
lysthrglualaasntrpvalasnvalileseraspleulyslysile
595600605
gluaspleuileglnsermethisileaspalathrleutyrthrglu
610615620
seraspvalhisprosercyslysvalthralametlyscyspheleu
625630635640
leugluleuglnvalileserleugluserglyaspalaserilehis
645650655
aspthrvalgluasnleuileileleualaasnasnserleuserser
660665670
asnglyasnvalthrgluserglycyslysglucysglugluleuglu
675680685
glulysasnilelysglupheleuglnserphevalhisilevalgln
690695700
metpheileasnthrser
705710
<210>97
<211>2623
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>97
gcgatcgcatggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacg60
ccgccaggccggacatcgtgatgacccaaagccccgacagcctggccgtgagcctgggcg120
agagggtgaccatgaactgcaaaagcagccagtccctgctgtactccaccaaccagaaga180
actacctggcttggtatcaacagaagcccggacagagccccaagctgctgatctattggg240
ccagcactagggaaagcggcgtgcccgataggttcagcggcagcgggagcggcacagact300
tcactctgaccattagcagcgtgcaggctgaggatgtggccgtctactactgccagcagt360
actacagctacaggacctttgggggcggaactaagctggagatcaagggaggggggggat420
ccgggggaggaggctccggcggaggcggaagccaagtgcaactgcagcagagcggcccag480
aggtggtcaaacctggggcaagcgtgaagatgagctgcaaggctagcggctataccttca540
ccagctatgtgatccactgggtgaggcagaaaccaggacagggcctggactggatcggct600
acatcaacccctacaatgacggcaccgattatgacgaaaaattcaaggggaaggccaccc660
tgaccagcgacaccagcacaagcaccgcctacatggagctgtccagcctgaggtccgagg720
acaccgccgtgtattactgtgccagggagaaggacaattacgccaccggcgcttggttcg780
cctactggggccagggcacactggtgacagtgagcagcaccacgacgccagcgccgcgac840
caccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgcc900
ggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgatatctaca960
tctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatcacccttt1020
actgcaggagtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgcc1080
gccccgggcccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcct1140
atcgctccaaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagac1200
cagtacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaag1260
gaggatgtgaactgagagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagg1320
gccagaaccagctctataacgagctcaatctaggacgaagagaggagtacgatgttttgg1380
acaagagacgtggccgggaccctgagatggggggaaagccgcagagaaggaagaaccctc1440
aggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagtgagattg1500
ggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggtctcagta1560
cagccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgcggaagcg1620
gagccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggcccca1680
tgtacagaatgcagctgctgagctgcatcgccctgagcctggccctggtgaccaacagcg1740
gcatccacgtgttcatcctgggctgcttcagcgccggcctgcccaagaccgaggccaact1800
gggtgaacgtgatcagcgacctgaagaagatcgaggacctgatccagagcatgcacatcg1860
acgccaccctgtacaccgagagcgacgtgcaccccagctgcaaggtgaccgccatgaagt1920
gcttcctgctggagctgcaggtgatcagcctggagagcggcgacgccagcatccacgaca1980
ccgtggagaacctgatcatcctggccaacaacagcctgagcagcaacggcaacgtgaccg2040
agagcggctgcaaggagtgcgaggagctggaggagaagaacatcaaggagttcctgcaga2100
gcttcgtgcacatcgtgcagatgttcatcaacaccagctccggcggcggctccggcggcg2160
gcggctccggcggcggcggctccggcggcggcggctccggcggcggctccctgcagatca2220
cctgccccccccccatgagcgtggagcacgccgacatctgggtgaagagctacagcctgt2280
acagcagagagagatacatctgcaacagcggcttcaagagaaaggccggcaccagcagcc2340
tgaccgagtgcgtgctgaacaaggccaccaacgtggcccactggaccacccccagcctga2400
agtgcatcagaaccaccacccccgcccccaggccccccacccccgcccccaccatcgcca2460
gccagcccctgagcctgaggcccgaggcctgcaggcccgccgccggcggcgccgtgcaca2520
ccaggggcctggacttcgcctgcgacgtggctatctccacgtccactgtcctgctgtgtg2580
ggctgagcgctgtgtctctcctggcatgctactaagtttaaac2623
<210>98
<211>868
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>98
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalmetthrglnserproaspserleu
202530
alavalserleuglygluargvalthrmetasncyslyssersergln
354045
serleuleutyrserthrasnglnlysasntyrleualatrptyrgln
505560
glnlysproglyglnserprolysleuleuiletyrtrpalaserthr
65707580
arggluserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleuthrileserservalglnalagluaspvalalaval
100105110
tyrtyrcysglnglntyrtyrsertyrargthrpheglyglyglythr
115120125
lysleugluilelysglyglyglyglyserglyglyglyglysergly
130135140
glyglyglyserglnvalglnleuglnglnserglyprogluvalval
145150155160
lysproglyalaservallysmetsercyslysalaserglytyrthr
165170175
phethrsertyrvalilehistrpvalargglnlysproglyglngly
180185190
leuasptrpileglytyrileasnprotyrasnaspglythrasptyr
195200205
aspglulysphelysglylysalathrleuthrseraspthrserthr
210215220
serthralatyrmetgluleuserserleuargsergluaspthrala
225230235240
valtyrtyrcysalaargglulysaspasntyralathrglyalatrp
245250255
phealatyrtrpglyglnglythrleuvalthrvalserserthrthr
260265270
thrproalaproargproprothrproalaprothrilealasergln
275280285
proleuserleuargproglualacysargproalaalaglyglyala
290295300
valhisthrargglyleuaspphealacysaspiletyriletrpala
305310315320
proleualaglythrcysglyvalleuleuleuserleuvalilethr
325330335
leutyrcysargserlysargserargleuleuhisserasptyrmet
340345350
asnmetthrproargargproglyprothrarglyshistyrglnpro
355360365
tyralaproproargaspphealaalatyrargserlysargglyarg
370375380
lyslysleuleutyrilephelysglnprophemetargprovalgln
385390395400
thrthrglnglugluaspglycyssercysargpheproglugluglu
405410415
gluglyglycysgluleuargvallyspheserargseralaaspala
420425430
proalatyrglnglnglyglnasnglnleutyrasngluleuasnleu
435440445
glyargarggluglutyraspvalleuasplysargargglyargasp
450455460
proglumetglyglylysproglnargarglysasnproglnglugly
465470475480
leutyrasngluleuglnlysasplysmetalaglualatyrserglu
485490495
ileglymetlysglygluargargargglylysglyhisaspglyleu
500505510
tyrglnglyleuserthralathrlysaspthrtyraspalaleuhis
515520525
metglnalaleuproproargglyserglyalathrasnpheserleu
530535540
leulysglnalaglyaspvalglugluasnproglypromettyrarg
545550555560
metglnleuleusercysilealaleuserleualaleuvalthrasn
565570575
serglyilehisvalpheileleuglycyspheseralaglyleupro
580585590
lysthrglualaasntrpvalasnvalileseraspleulyslysile
595600605
gluaspleuileglnsermethisileaspalathrleutyrthrglu
610615620
seraspvalhisprosercyslysvalthralametlyscyspheleu
625630635640
leugluleuglnvalileserleugluserglyaspalaserilehis
645650655
aspthrvalgluasnleuileileleualaasnasnserleuserser
660665670
asnglyasnvalthrgluserglycyslysglucysglugluleuglu
675680685
glulysasnilelysglupheleuglnserphevalhisilevalgln
690695700
metpheileasnthrserserglyglyglyserglyglyglyglyser
705710715720
glyglyglyglyserglyglyglyglyserglyglyglyserleugln
725730735
ilethrcysproproprometservalgluhisalaaspiletrpval
740745750
lyssertyrserleutyrserarggluargtyrilecysasnsergly
755760765
phelysarglysalaglythrserserleuthrglucysvalleuasn
770775780
lysalathrasnvalalahistrpthrthrproserleulyscysile
785790795800
argthrthrthrproalaproargproprothrproalaprothrile
805810815
alaserglnproleuserleuargproglualacysargproalaala
820825830
glyglyalavalhisthrargglyleuaspphealacysaspvalala
835840845
ileserthrserthrvalleuleucysglyleuseralavalserleu
850855860
leualacystyr
865
<210>99
<211>2389
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>99
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggacatcgtgatgagccagagccccagcagcctggccgtgagcgtgg120
gcgagaagttcaccgtgaactgcaagagcagccagagcctgctgtacagcagcaaccaga180
agaacttcctggcctggtaccagcagaagcccggccagagccccaagctgctgatctact240
gggccagcaccagagagagcggcgtgcccgacagattcatcggcagcggcagcggcaccg300
acttcaccctgaccatcagcagcgtgaaggccgaggacctggccgtgtactactgccagc360
agcactacagatacccctggaccttcggcggcggcaccaagctggagatcaagagagcca420
gaggaggggggggatccgggggaggaggctccggcggaggcggaagccaggtgaccctga480
aggagagcggccccggcatcctgcagcccagccagaccctgagcctgacctgcagcttca540
gcggcttcagcctgaacaccagcggcgtgggcgtgggctggatcagacagcccagcggca600
agggcctggagtggctggcccacatctggtgggacgacgacgagagatacaaccccgtgc660
tgaagagcagactgaccatcagcaaggacaccagcagcaaccaggtgttcctgaagatcg720
ccaacgtggacaccgccgacagcgccacctactactgcgtgagaagcatggtggcctggt780
tcttcccctactggggcagaggcaccctggtgaccgtgagcgccaccacgacgccagcgc840
cgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgcccagagg900
cgtgccggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgtgata960
tctacatctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatca1020
ccctttactgcaggagtaagaggagcaggctcctgcacagtgactacatgaacatgactc1080
cccgccgccccgggcccacccgcaagcattaccagccctatgccccaccacgcgacttcg1140
cagcctatcgctccagggtgaagttcagcaggagcgccgacgcccccgcctaccagcagg1200
gccagaaccagctgtacaacgagctgaacctgggcaggagggaggagtacgacgtgctgg1260
acaagaggaggggcagggaccccgagatgggcggcaagccccagaggaggaagaaccccc1320
aggagggcctgtacaacgagctgcagaaggacaagatggccgaggcctacagcgagatcg1380
gcatgaagggcgagaggaggaggggcaagggccacgacggcctgtaccagggcctgagca1440
ccgccaccaaggacacctacgacgccctgcacatgcaggccctgccccccaggggaagcg1500
gagccaccaacttcagcctgctgaagcaggccggcgacgtggaggagaaccccggcccca1560
tgtacagaatgcagctgctgagctgcatcgccctgagcctggccctggtgaccaacagcg1620
gcatccacgtgttcatcctgggctgcttcagcgccggcctgcccaagaccgaggccaact1680
gggtgaacgtgatcagcgacctgaagaagatcgaggacctgatccagagcatgcacatcg1740
acgccaccctgtacaccgagagcgacgtgcaccccagctgcaaggtgaccgccatgaagt1800
gcttcctgctggagctgcaggtgatcagcctggagagcggcgacgccagcatccacgaca1860
ccgtggagaacctgatcatcctggccaacaacagcctgagcagcaacggcaacgtgaccg1920
agagcggctgcaaggagtgcgaggagctggaggagaagaacatcaaggagttcctgcaga1980
gcttcgtgcacatcgtgcagatgttcatcaacaccagctccggcggcggctccggcggcg2040
gcggctccggcggcggcggctccggcggcggcggctccggcggcggctccctgcaggccc2100
ccagaagagccagaggctgcagaaccctgggcctgcccgccctgctgctgctgctgctgc2160
tgagaccccccgccaccagaggcatcacctgccccccccccatgagcgtggagcacgccg2220
acatctgggtgaagagctacagcctgtacagcagagagagatacatctgcaacagcggct2280
tcaagagaaaggccggcaccagcagcctgaccgagtgcgtgctgaacaaggccaccaacg2340
tggcccactggaccacccccagcctgaagtgcatcagataagtttaaac2389
<210>100
<211>789
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>100
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilevalmetserglnserproserserleu
202530
alavalservalglyglulysphethrvalasncyslyssersergln
354045
serleuleutyrserserasnglnlysasnpheleualatrptyrgln
505560
glnlysproglyglnserprolysleuleuiletyrtrpalaserthr
65707580
arggluserglyvalproaspargpheileglyserglyserglythr
859095
aspphethrleuthrileserservallysalagluaspleualaval
100105110
tyrtyrcysglnglnhistyrargtyrprotrpthrpheglyglygly
115120125
thrlysleugluilelysargalaargglyglyglyglyserglygly
130135140
glyglyserglyglyglyglyserglnvalthrleulysglusergly
145150155160
proglyileleuglnproserglnthrleuserleuthrcysserphe
165170175
serglypheserleuasnthrserglyvalglyvalglytrpilearg
180185190
glnproserglylysglyleuglutrpleualahisiletrptrpasp
195200205
aspaspgluargtyrasnprovalleulysserargleuthrileser
210215220
lysaspthrserserasnglnvalpheleulysilealaasnvalasp
225230235240
thralaaspseralathrtyrtyrcysvalargsermetvalalatrp
245250255
phepheprotyrtrpglyargglythrleuvalthrvalseralathr
260265270
thrthrproalaproargproprothrproalaprothrilealaser
275280285
glnproleuserleuargproglualacysargproalaalaglygly
290295300
alavalhisthrargglyleuaspphealacysaspiletyriletrp
305310315320
alaproleualaglythrcysglyvalleuleuleuserleuvalile
325330335
thrleutyrcysargserlysargserargleuleuhisserasptyr
340345350
metasnmetthrproargargproglyprothrarglyshistyrgln
355360365
protyralaproproargaspphealaalatyrargserargvallys
370375380
pheserargseralaaspalaproalatyrglnglnglyglnasngln
385390395400
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
405410415
asplysargargglyargaspproglumetglyglylysproglnarg
420425430
arglysasnproglngluglyleutyrasngluleuglnlysasplys
435440445
metalaglualatyrsergluileglymetlysglygluargargarg
450455460
glylysglyhisaspglyleutyrglnglyleuserthralathrlys
465470475480
aspthrtyraspalaleuhismetglnalaleuproproargglyser
485490495
glyalathrasnpheserleuleulysglnalaglyaspvalgluglu
500505510
asnproglypromettyrargmetglnleuleusercysilealaleu
515520525
serleualaleuvalthrasnserglyilehisvalpheileleugly
530535540
cyspheseralaglyleuprolysthrglualaasntrpvalasnval
545550555560
ileseraspleulyslysilegluaspleuileglnsermethisile
565570575
aspalathrleutyrthrgluseraspvalhisprosercyslysval
580585590
thralametlyscyspheleuleugluleuglnvalileserleuglu
595600605
serglyaspalaserilehisaspthrvalgluasnleuileileleu
610615620
alaasnasnserleuserserasnglyasnvalthrgluserglycys
625630635640
lysglucysglugluleugluglulysasnilelysglupheleugln
645650655
serphevalhisilevalglnmetpheileasnthrserserglygly
660665670
glyserglyglyglyglyserglyglyglyglyserglyglyglygly
675680685
serglyglyglyserleuglnalaproargargalaargglycysarg
690695700
thrleuglyleuproalaleuleuleuleuleuleuleuargpropro
705710715720
alathrargglyilethrcysproproprometservalgluhisala
725730735
aspiletrpvallyssertyrserleutyrserarggluargtyrile
740745750
cysasnserglyphelysarglysalaglythrserserleuthrglu
755760765
cysvalleuasnlysalathrasnvalalahistrpthrthrproser
770775780
leulyscysilearg
785
<210>101
<211>2371
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>101
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggacattatgatgacacagtcgccatcatctctggctgtgtctgcag120
gagaaaaggtcactatgacctgtaagtccagtcaaagtgttttatacagttcaaatcaga180
agaactacttggcctggtaccagcagaaaccagggcagtctcctaaactactgatctact240
gggcatccactagggaatctggtgtccctgatcgcttcacaggcagtggatctgggacag300
attttactcttaccatcagcagtgtgcaacctgaagacctggcagtttattactgtcatc360
aatacctctcctcgcacacgttcggaggggggaccaagctggaaataaaacggggtggcg420
gtggctcgggcggtggtgggtcgggtggcggcggatctcaactgcagcagcctggggctg480
agctggtgaggcctgggtcttcagtgaagctgtcctgcaaggcttctggctacaccttca540
ccaggtactggatacattgggtgaagcagaggcctatacaaggccttgaatggattggta600
acattgatccttctgatagtgaaactcactacaatcaaaagttcaaggacaaggccacat660
tgactgtagacaaatcctccggcacagcctacatgcagctcagcagcctgacatctgagg720
actctgcggtctattactgtgcaacagaggatctttactatgctatggagtactggggtc780
aaggaacctcagtcaccgtctcctctaccacgacgccagcgccgcgaccaccaacaccgg840
cgcccaccatcgcgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcgg900
ggggcgcagtgcacacgagggggctggacttcgcctgtgatatctacatctgggcgccct960
tggccgggacttgtggggtccttctcctgtcactggttatcaccctttactgcaggagta1020
agaggagcaggctcctgcacagtgactacatgaacatgactccccgccgccccgggccca1080
cccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgctccagag1140
tgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctctata1200
acgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggccggg1260
accctgagatggggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatg1320
aactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgcc1380
ggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacacct1440
acgacgcccttcacatgcaggccctgccccctcgcggaagcggagccaccaacttcagcc1500
tgctgaagcaggccggcgacgtggaggagaaccccggccccatgtacagaatgcagctgc1560
tgagctgcatcgccctgagcctggccctggtgaccaacagcggcatccacgtgttcatcc1620
tgggctgcttcagcgccggcctgcccaagaccgaggccaactgggtgaacgtgatcagcg1680
acctgaagaagatcgaggacctgatccagagcatgcacatcgacgccaccctgtacaccg1740
agagcgacgtgcaccccagctgcaaggtgaccgccatgaagtgcttcctgctggagctgc1800
aggtgatcagcctggagagcggcgacgccagcatccacgacaccgtggagaacctgatca1860
tcctggccaacaacagcctgagcagcaacggcaacgtgaccgagagcggctgcaaggagt1920
gcgaggagctggaggagaagaacatcaaggagttcctgcagagcttcgtgcacatcgtgc1980
agatgttcatcaacaccagctccggcggcggctccggcggcggcggctccggcggcggcg2040
gctccggcggcggcggctccggcggcggctccctgcaggcccccagaagagccagaggct2100
gcagaaccctgggcctgcccgccctgctgctgctgctgctgctgagaccccccgccacca2160
gaggcatcacctgccccccccccatgagcgtggagcacgccgacatctgggtgaagagct2220
acagcctgtacagcagagagagatacatctgcaacagcggcttcaagagaaaggccggca2280
ccagcagcctgaccgagtgcgtgctgaacaaggccaccaacgtggcccactggaccaccc2340
ccagcctgaagtgcatcagataagtttaaac2371
<210>102
<211>783
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>102
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspilemetmetthrglnserproserserleu
202530
alavalseralaglyglulysvalthrmetthrcyslyssersergln
354045
servalleutyrserserasnglnlysasntyrleualatrptyrgln
505560
glnlysproglyglnserprolysleuleuiletyrtrpalaserthr
65707580
arggluserglyvalproaspargphethrglyserglyserglythr
859095
aspphethrleuthrileserservalglnprogluaspleualaval
100105110
tyrtyrcyshisglntyrleuserserhisthrpheglyglyglythr
115120125
lysleugluilelysargglyglyglyglyserglyglyglyglyser
130135140
glyglyglyglyserglnleuglnglnproglyalagluleuvalarg
145150155160
proglyserservallysleusercyslysalaserglytyrthrphe
165170175
thrargtyrtrpilehistrpvallysglnargproileglnglyleu
180185190
glutrpileglyasnileaspproseraspsergluthrhistyrasn
195200205
glnlysphelysasplysalathrleuthrvalasplyssersergly
210215220
thralatyrmetglnleuserserleuthrsergluaspseralaval
225230235240
tyrtyrcysalathrgluaspleutyrtyralametglutyrtrpgly
245250255
glnglythrservalthrvalserserthrthrthrproalaproarg
260265270
proprothrproalaprothrilealaserglnproleuserleuarg
275280285
proglualacysargproalaalaglyglyalavalhisthrarggly
290295300
leuaspphealacysaspiletyriletrpalaproleualaglythr
305310315320
cysglyvalleuleuleuserleuvalilethrleutyrcysargser
325330335
lysargserargleuleuhisserasptyrmetasnmetthrproarg
340345350
argproglyprothrarglyshistyrglnprotyralaproproarg
355360365
aspphealaalatyrargserargvallyspheserargseralaasp
370375380
alaproalatyrglnglnglyglnasnglnleutyrasngluleuasn
385390395400
leuglyargarggluglutyraspvalleuasplysargargglyarg
405410415
aspproglumetglyglylysproglnargarglysasnproglnglu
420425430
glyleutyrasngluleuglnlysasplysmetalaglualatyrser
435440445
gluileglymetlysglygluargargargglylysglyhisaspgly
450455460
leutyrglnglyleuserthralathrlysaspthrtyraspalaleu
465470475480
hismetglnalaleuproproargglyserglyalathrasnpheser
485490495
leuleulysglnalaglyaspvalglugluasnproglypromettyr
500505510
argmetglnleuleusercysilealaleuserleualaleuvalthr
515520525
asnserglyilehisvalpheileleuglycyspheseralaglyleu
530535540
prolysthrglualaasntrpvalasnvalileseraspleulyslys
545550555560
ilegluaspleuileglnsermethisileaspalathrleutyrthr
565570575
gluseraspvalhisprosercyslysvalthralametlyscysphe
580585590
leuleugluleuglnvalileserleugluserglyaspalaserile
595600605
hisaspthrvalgluasnleuileileleualaasnasnserleuser
610615620
serasnglyasnvalthrgluserglycyslysglucysglugluleu
625630635640
gluglulysasnilelysglupheleuglnserphevalhisileval
645650655
glnmetpheileasnthrserserglyglyglyserglyglyglygly
660665670
serglyglyglyglyserglyglyglyglyserglyglyglyserleu
675680685
glnalaproargargalaargglycysargthrleuglyleuproala
690695700
leuleuleuleuleuleuleuargproproalathrargglyilethr
705710715720
cysproproprometservalgluhisalaaspiletrpvallysser
725730735
tyrserleutyrserarggluargtyrilecysasnserglyphelys
740745750
arglysalaglythrserserleuthrglucysvalleuasnlysala
755760765
thrasnvalalahistrpthrthrproserleulyscysilearg
770775780
<210>103
<211>2356
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>103
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggacatccagatgacccagagccccagcagcctgagcgccagcgtgg120
gcgacagagtgaccatcacctgcagcgccagcagcagcgtgagctacatgaactggtacc180
agcagacccccggcaaggcccccaagagatggatctacgacaccagcaagctggccagcg240
gcgtgcccagcagattcagcggcagcggcagcggcaccgactacaccttcaccatcagca300
gcctgcagcccgaggacatcgccacctactactgccagcagtggagcagcaaccccttca360
ccttcggccagggcaccaagctgcagatcggcggcggcggcagcggcggcggcggcagcg420
gcggcggcggcagccaggtgcagctggtgcagagcggcggcggcgtggtgcagcccggca480
gaagcctgagactgagctgcaaggccagcggctacaccttcaccagatacaccatgcact540
gggtgagacaggcccccggcaagggcctggagtggatcggctacatcaaccccagcagag600
gctacaccaactacaaccagaaggtgaaggacagattcaccatcagcagagacaacagca660
agaacaccgccttcctgcagatggacagcctgagacccgaggacaccggcgtgtacttct720
gcgccagatactacgacgaccactactgcctggactactggggccagggcacccccgtga780
ccgtgagcagcaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgt840
cgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcaca900
cgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtg960
gggtccttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggctcc1020
tgcacagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcattacc1080
agccctatgccccaccacgcgacttcgcagcctatcgctccagagtgaagttcagcagga1140
gcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatctag1200
gacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctgagatggggg1260
gaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaagata1320
agatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaaggggc1380
acgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttcaca1440
tgcaggccctgccccctcgcggaagcggagccaccaacttcagcctgctgaagcaggccg1500
gcgacgtggaggagaaccccggccccatgtacagaatgcagctgctgagctgcatcgccc1560
tgagcctggccctggtgaccaacagcggcatccacgtgttcatcctgggctgcttcagcg1620
ccggcctgcccaagaccgaggccaactgggtgaacgtgatcagcgacctgaagaagatcg1680
aggacctgatccagagcatgcacatcgacgccaccctgtacaccgagagcgacgtgcacc1740
ccagctgcaaggtgaccgccatgaagtgcttcctgctggagctgcaggtgatcagcctgg1800
agagcggcgacgccagcatccacgacaccgtggagaacctgatcatcctggccaacaaca1860
gcctgagcagcaacggcaacgtgaccgagagcggctgcaaggagtgcgaggagctggagg1920
agaagaacatcaaggagttcctgcagagcttcgtgcacatcgtgcagatgttcatcaaca1980
ccagctccggcggcggctccggcggcggcggctccggcggcggcggctccggcggcggcg2040
gctccggcggcggctccctgcaggcccccagaagagccagaggctgcagaaccctgggcc2100
tgcccgccctgctgctgctgctgctgctgagaccccccgccaccagaggcatcacctgcc2160
ccccccccatgagcgtggagcacgccgacatctgggtgaagagctacagcctgtacagca2220
gagagagatacatctgcaacagcggcttcaagagaaaggccggcaccagcagcctgaccg2280
agtgcgtgctgaacaaggccaccaacgtggcccactggaccacccccagcctgaagtgca2340
tcagataagtttaaac2356
<210>104
<211>777
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>104
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspileglnmetthrglnserproserserleu
202530
seralaservalglyaspargvalthrilethrcysseralaserser
354045
servalsertyrmetasntrptyrglnglnthrproglylysalapro
505560
lysargtrpiletyraspthrserlysleualaserglyvalproser
65707580
argpheserglyserglyserglythrasptyrthrphethrileser
859095
serleuglnprogluaspilealathrtyrtyrcysglnglntrpser
100105110
serasnprophethrpheglyglnglythrlysleuglnileglygly
115120125
glyglyserglyglyglyglyserglyglyglyglyserglnvalgln
130135140
leuvalglnserglyglyglyvalvalglnproglyargserleuarg
145150155160
leusercyslysalaserglytyrthrphethrargtyrthrmethis
165170175
trpvalargglnalaproglylysglyleuglutrpileglytyrile
180185190
asnproserargglytyrthrasntyrasnglnlysvallysasparg
195200205
phethrileserargaspasnserlysasnthralapheleuglnmet
210215220
aspserleuargprogluaspthrglyvaltyrphecysalaargtyr
225230235240
tyraspasphistyrcysleuasptyrtrpglyglnglythrproval
245250255
thrvalserserthrthrthrproalaproargproprothrproala
260265270
prothrilealaserglnproleuserleuargproglualacysarg
275280285
proalaalaglyglyalavalhisthrargglyleuaspphealacys
290295300
aspiletyriletrpalaproleualaglythrcysglyvalleuleu
305310315320
leuserleuvalilethrleutyrcysargserlysargserargleu
325330335
leuhisserasptyrmetasnmetthrproargargproglyprothr
340345350
arglyshistyrglnprotyralaproproargaspphealaalatyr
355360365
argserargvallyspheserargseralaaspalaproalatyrgln
370375380
glnglyglnasnglnleutyrasngluleuasnleuglyargargglu
385390395400
glutyraspvalleuasplysargargglyargaspproglumetgly
405410415
glylysproglnargarglysasnproglngluglyleutyrasnglu
420425430
leuglnlysasplysmetalaglualatyrsergluileglymetlys
435440445
glygluargargargglylysglyhisaspglyleutyrglnglyleu
450455460
serthralathrlysaspthrtyraspalaleuhismetglnalaleu
465470475480
proproargglyserglyalathrasnpheserleuleulysglnala
485490495
glyaspvalglugluasnproglypromettyrargmetglnleuleu
500505510
sercysilealaleuserleualaleuvalthrasnserglyilehis
515520525
valpheileleuglycyspheseralaglyleuprolysthrgluala
530535540
asntrpvalasnvalileseraspleulyslysilegluaspleuile
545550555560
glnsermethisileaspalathrleutyrthrgluseraspvalhis
565570575
prosercyslysvalthralametlyscyspheleuleugluleugln
580585590
valileserleugluserglyaspalaserilehisaspthrvalglu
595600605
asnleuileileleualaasnasnserleuserserasnglyasnval
610615620
thrgluserglycyslysglucysglugluleugluglulysasnile
625630635640
lysglupheleuglnserphevalhisilevalglnmetpheileasn
645650655
thrserserglyglyglyserglyglyglyglyserglyglyglygly
660665670
serglyglyglyglyserglyglyglyserleuglnalaproargarg
675680685
alaargglycysargthrleuglyleuproalaleuleuleuleuleu
690695700
leuleuargproproalathrargglyilethrcyspropropromet
705710715720
servalgluhisalaaspiletrpvallyssertyrserleutyrser
725730735
arggluargtyrilecysasnserglyphelysarglysalaglythr
740745750
serserleuthrglucysvalleuasnlysalathrasnvalalahis
755760765
trpthrthrproserleulyscysile
770775
<210>105
<211>2401
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>105
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccgggcgcccagcccgccatggccgcctacaaggacatccagatgaccc120
agaccaccagcagcctgagcgccagcctgggcgacagagtgaccatcagctgcagcgcca180
gccagggcatcagcaactacctgaactggtaccagcagaagcccgacggcaccaacaagc240
tgctgatctactacaccagcagcctgcacagcggcgtgcccagcagattcagcggcagcg300
gcagcggcaccgactacagcctgcacagcaacctggagcccgaggacatcgccacctact360
actgccagcagtacagcaagctgccctacaccttcggcggcggcaccaagctggagatca420
agagaggcggcggcggcagcggcggcggcggcagcggcggcggcggcagcggcggcggcg480
gcagcgaggtgcagctggtggagagcggcggcggcctggtgaagcccggcggcagcctga540
agctgagctgcgccgccagcggcctgaccttcagcagctacgccatgagctggaacagac600
agacccccgagaagagactggagtgggtggccagcatcagcagcggcggcttcacctact660
accccgacagcaacaagggcagattcaccatcagcagagacaacgccagaaacatcctgt720
acctgcagatgagcagcctgagaagcgaggacaccgccatgtactactgcgccagagacg780
aggtgagaggctacctggacgtgtggggcgccggcaccaccgtgaccgtgagcagcacca840
cgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccc900
tgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggact960
tcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttctcctgt1020
cactggttatcaccctttactgcaggagtaagaggagcaggctcctgcacagtgactaca1080
tgaacatgactccccgccgccccgggcccacccgcaagcattaccagccctatgccccac1140
cacgcgacttcgcagcctatcgctccagagtgaagttcagcaggagcgcagacgcccccg1200
cgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaagagaggagt1260
acgatgttttggacaagagacgtggccgggaccctgagatggggggaaagccgcagagaa1320
ggaagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcct1380
acagtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttacc1440
agggtctcagtacagccaccaaggacacctacgacgcccttcacatgcaggccctgcccc1500
ctcgcggaagcggagccaccaacttcagcctgctgaagcaggccggcgacgtggaggaga1560
accccggccccatgtacagaatgcagctgctgagctgcatcgccctgagcctggccctgg1620
tgaccaacagcggcatccacgtgttcatcctgggctgcttcagcgccggcctgcccaaga1680
ccgaggccaactgggtgaacgtgatcagcgacctgaagaagatcgaggacctgatccaga1740
gcatgcacatcgacgccaccctgtacaccgagagcgacgtgcaccccagctgcaaggtga1800
ccgccatgaagtgcttcctgctggagctgcaggtgatcagcctggagagcggcgacgcca1860
gcatccacgacaccgtggagaacctgatcatcctggccaacaacagcctgagcagcaacg1920
gcaacgtgaccgagagcggctgcaaggagtgcgaggagctggaggagaagaacatcaagg1980
agttcctgcagagcttcgtgcacatcgtgcagatgttcatcaacaccagctccggcggcg2040
gctccggcggcggcggctccggcggcggcggctccggcggcggcggctccggcggcggct2100
ccctgcaggcccccagaagagccagaggctgcagaaccctgggcctgcccgccctgctgc2160
tgctgctgctgctgagaccccccgccaccagaggcatcacctgccccccccccatgagcg2220
tggagcacgccgacatctgggtgaagagctacagcctgtacagcagagagagatacatct2280
gcaacagcggcttcaagagaaaggccggcaccagcagcctgaccgagtgcgtgctgaaca2340
aggccaccaacgtggcccactggaccacccccagcctgaagtgcatcagataagtttaaa2400
c2401
<210>106
<211>793
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>106
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproglyalaglnproalametalaalatyrlysasp
202530
ileglnmetthrglnthrthrserserleuseralaserleuglyasp
354045
argvalthrilesercysseralaserglnglyileserasntyrleu
505560
asntrptyrglnglnlysproaspglythrasnlysleuleuiletyr
65707580
tyrthrserserleuhisserglyvalproserargpheserglyser
859095
glyserglythrasptyrserleuhisserasnleugluprogluasp
100105110
ilealathrtyrtyrcysglnglntyrserlysleuprotyrthrphe
115120125
glyglyglythrlysleugluilelysargglyglyglyglysergly
130135140
glyglyglyserglyglyglyglyserglyglyglyglysergluval
145150155160
glnleuvalgluserglyglyglyleuvallysproglyglyserleu
165170175
lysleusercysalaalaserglyleuthrphesersertyralamet
180185190
sertrpasnargglnthrproglulysargleuglutrpvalalaser
195200205
ileserserglyglyphethrtyrtyrproaspserasnlysglyarg
210215220
phethrileserargaspasnalaargasnileleutyrleuglnmet
225230235240
serserleuargsergluaspthralamettyrtyrcysalaargasp
245250255
gluvalargglytyrleuaspvaltrpglyalaglythrthrvalthr
260265270
valserserthrthrthrproalaproargproprothrproalapro
275280285
thrilealaserglnproleuserleuargproglualacysargpro
290295300
alaalaglyglyalavalhisthrargglyleuaspphealacysasp
305310315320
iletyriletrpalaproleualaglythrcysglyvalleuleuleu
325330335
serleuvalilethrleutyrcysargserlysargserargleuleu
340345350
hisserasptyrmetasnmetthrproargargproglyprothrarg
355360365
lyshistyrglnprotyralaproproargaspphealaalatyrarg
370375380
serargvallyspheserargseralaaspalaproalatyrglngln
385390395400
glyglnasnglnleutyrasngluleuasnleuglyargarggluglu
405410415
tyraspvalleuasplysargargglyargaspproglumetglygly
420425430
lysproglnargarglysasnproglngluglyleutyrasngluleu
435440445
glnlysasplysmetalaglualatyrsergluileglymetlysgly
450455460
gluargargargglylysglyhisaspglyleutyrglnglyleuser
465470475480
thralathrlysaspthrtyraspalaleuhismetglnalaleupro
485490495
proargglyserglyalathrasnpheserleuleulysglnalagly
500505510
aspvalglugluasnproglypromettyrargmetglnleuleuser
515520525
cysilealaleuserleualaleuvalthrasnserglyilehisval
530535540
pheileleuglycyspheseralaglyleuprolysthrglualaasn
545550555560
trpvalasnvalileseraspleulyslysilegluaspleuilegln
565570575
sermethisileaspalathrleutyrthrgluseraspvalhispro
580585590
sercyslysvalthralametlyscyspheleuleugluleuglnval
595600605
ileserleugluserglyaspalaserilehisaspthrvalgluasn
610615620
leuileileleualaasnasnserleuserserasnglyasnvalthr
625630635640
gluserglycyslysglucysglugluleugluglulysasnilelys
645650655
glupheleuglnserphevalhisilevalglnmetpheileasnthr
660665670
serserglyglyglyserglyglyglyglyserglyglyglyglyser
675680685
glyglyglyglyserglyglyglyserleuglnalaproargargala
690695700
argglycysargthrleuglyleuproalaleuleuleuleuleuleu
705710715720
leuargproproalathrargglyilethrcysproproprometser
725730735
valgluhisalaaspiletrpvallyssertyrserleutyrserarg
740745750
gluargtyrilecysasnserglyphelysarglysalaglythrser
755760765
serleuthrglucysvalleuasnlysalathrasnvalalahistrp
770775780
thrthrproserleulyscysilearg
785790
<210>107
<211>2359
<212>dna
<213>artificialsequence
<220>
<223>syntheticsequence
<400>107
gcgatcgcaccatggccttaccagtgaccgccttgctcctgccgctggccttgctgctcc60
acgccgccaggccggacatccaggtgacccagagccccagcagcctgagcgccagcctgg120
gcgagagaatcagcctgacctgcagaaccagccaggacatcagcaactacctgaactggt180
tccagcagaagcccgacggcaccttcaagagactgatctacgccaccagcagcctggaca240
gcggcgtgcccaagagattcagcggcagcggcagcggcagcgactacagcctgaccatca300
gcagcctggagagcgaggacttcgccgactactactgcctgcagtacgccagctacccct360
tcaccttcggcagcggcaccaagctggagatcaagggaggggggggatccgggggaggag420
gctccggcggaggcggaagcgaggtgcagctgcaggagagcggccccggcctggtgaagc480
ccagccagaccctgagcctgacctgcagcgtgaccggctacagcatcaccagcggctact540
actggcactggatcagacagttccccggcaacaagctgcagtggatgggctacatcagct600
acagcggcttcaccaactacaagaccagcctgatcaacagaatcagcatcacccacgaca660
ccagcgagaaccagttcttcctgaacctgaacagcgtgaccaccgaggacaccgccacct720
actactgcgccggcgacagaaccggcagctggttcgcctactggggccagggcaccctgg780
tgaccgtgagcgccaccacgacgccagcgccgcgaccaccaacaccggcgcccaccatcg840
cgtcgcagcccctgtccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgc900
acacgagggggctggacttcgcctgtgatatctacatctgggcgcccttggccgggactt960
gtggggtccttctcctgtcactggttatcaccctttactgcaggagtaagaggagcaggc1020
tcctgcacagtgactacatgaacatgactccccgccgccccgggcccacccgcaagcatt1080
accagccctatgccccaccacgcgacttcgcagcctatcgctccagagtgaagttcagca1140
ggagcgcagacgcccccgcgtaccagcagggccagaaccagctctataacgagctcaatc1200
taggacgaagagaggagtacgatgttttggacaagagacgtggccgggaccctgagatgg1260
ggggaaagccgcagagaaggaagaaccctcaggaaggcctgtacaatgaactgcagaaag1320
ataagatggcggaggcctacagtgagattgggatgaaaggcgagcgccggaggggcaagg1380
ggcacgatggcctttaccagggtctcagtacagccaccaaggacacctacgacgcccttc1440
acatgcaggccctgccccctcgcggaagcggagccaccaacttcagcctgctgaagcagg1500
ccggcgacgtggaggagaaccccggccccatgtacagaatgcagctgctgagctgcatcg1560
ccctgagcctggccctggtgaccaacagcggcatccacgtgttcatcctgggctgcttca1620
gcgccggcctgcccaagaccgaggccaactgggtgaacgtgatcagcgacctgaagaaga1680
tcgaggacctgatccagagcatgcacatcgacgccaccctgtacaccgagagcgacgtgc1740
accccagctgcaaggtgaccgccatgaagtgcttcctgctggagctgcaggtgatcagcc1800
tggagagcggcgacgccagcatccacgacaccgtggagaacctgatcatcctggccaaca1860
acagcctgagcagcaacggcaacgtgaccgagagcggctgcaaggagtgcgaggagctgg1920
aggagaagaacatcaaggagttcctgcagagcttcgtgcacatcgtgcagatgttcatca1980
acaccagctccggcggcggctccggcggcggcggctccggcggcggcggctccggcggcg2040
gcggctccggcggcggctccctgcaggcccccagaagagccagaggctgcagaaccctgg2100
gcctgcccgccctgctgctgctgctgctgctgagaccccccgccaccagaggcatcacct2160
gccccccccccatgagcgtggagcacgccgacatctgggtgaagagctacagcctgtaca2220
gcagagagagatacatctgcaacagcggcttcaagagaaaggccggcaccagcagcctga2280
ccgagtgcgtgctgaacaaggccaccaacgtggcccactggaccacccccagcctgaagt2340
gcatcagataagtttaaac2359
<210>108
<211>779
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>108
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproaspileglnvalthrglnserproserserleu
202530
seralaserleuglygluargileserleuthrcysargthrsergln
354045
aspileserasntyrleuasntrppheglnglnlysproaspglythr
505560
phelysargleuiletyralathrserserleuaspserglyvalpro
65707580
lysargpheserglyserglyserglyserasptyrserleuthrile
859095
serserleuglusergluaspphealaasptyrtyrcysleuglntyr
100105110
alasertyrprophethrpheglyserglythrlysleugluilelys
115120125
glyglyglyglyserglyglyglyglyserglyglyglyglyserglu
130135140
valglnleuglngluserglyproglyleuvallysproserglnthr
145150155160
leuserleuthrcysservalthrglytyrserilethrserglytyr
165170175
tyrtrphistrpileargglnpheproglyasnlysleuglntrpmet
180185190
glytyrilesertyrserglyphethrasntyrlysthrserleuile
195200205
asnargileserilethrhisaspthrsergluasnglnphepheleu
210215220
asnleuasnservalthrthrgluaspthralathrtyrtyrcysala
225230235240
glyaspargthrglysertrpphealatyrtrpglyglnglythrleu
245250255
valthrvalseralathrthrthrproalaproargproprothrpro
260265270
alaprothrilealaserglnproleuserleuargproglualacys
275280285
argproalaalaglyglyalavalhisthrargglyleuasppheala
290295300
cysaspiletyriletrpalaproleualaglythrcysglyvalleu
305310315320
leuleuserleuvalilethrleutyrcysargserlysargserarg
325330335
leuleuhisserasptyrmetasnmetthrproargargproglypro
340345350
thrarglyshistyrglnprotyralaproproargaspphealaala
355360365
tyrargserargvallyspheserargseralaaspalaproalatyr
370375380
glnglnglyglnasnglnleutyrasngluleuasnleuglyargarg
385390395400
gluglutyraspvalleuasplysargargglyargaspproglumet
405410415
glyglylysproglnargarglysasnproglngluglyleutyrasn
420425430
gluleuglnlysasplysmetalaglualatyrsergluileglymet
435440445
lysglygluargargargglylysglyhisaspglyleutyrglngly
450455460
leuserthralathrlysaspthrtyraspalaleuhismetglnala
465470475480
leuproproargglyserglyalathrasnpheserleuleulysgln
485490495
alaglyaspvalglugluasnproglypromettyrargmetglnleu
500505510
leusercysilealaleuserleualaleuvalthrasnserglyile
515520525
hisvalpheileleuglycyspheseralaglyleuprolysthrglu
530535540
alaasntrpvalasnvalileseraspleulyslysilegluaspleu
545550555560
ileglnsermethisileaspalathrleutyrthrgluseraspval
565570575
hisprosercyslysvalthralametlyscyspheleuleugluleu
580585590
glnvalileserleugluserglyaspalaserilehisaspthrval
595600605
gluasnleuileileleualaasnasnserleuserserasnglyasn
610615620
valthrgluserglycyslysglucysglugluleugluglulysasn
625630635640
ilelysglupheleuglnserphevalhisilevalglnmetpheile
645650655
asnthrserserglyglyglyserglyglyglyglyserglyglygly
660665670
glyserglyglyglyglyserglyglyglyserleuglnalaproarg
675680685
argalaargglycysargthrleuglyleuproalaleuleuleuleu
690695700
leuleuleuargproproalathrargglyilethrcyspropropro
705710715720
metservalgluhisalaaspiletrpvallyssertyrserleutyr
725730735
serarggluargtyrilecysasnserglyphelysarglysalagly
740745750
thrserserleuthrglucysvalleuasnlysalathrasnvalala
755760765
histrpthrthrproserleulyscysilearg
770775
<210>109
<211>21
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>109
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargpro
20
<210>110
<211>116
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>110
aspilevalmetserglnserproserserleualavalservalgly
151015
glulysphethrvalasncyslysserserglnserleuleutyrser
202530
serasnglnlysasnpheleualatrptyrglnglnlysproglygln
354045
serprolysleuleuiletyrtrpalaserthrarggluserglyval
505560
proaspargpheileglyserglyserglythraspphethrleuthr
65707580
ileserservallysalagluaspleualavaltyrtyrcysglngln
859095
histyrargtyrprotrpthrpheglyglyglythrlysleugluile
100105110
lysargalaarg
115
<210>111
<211>15
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>111
glyglyglyglyserglyglyglyglyserglyglyglyglyser
151015
<210>112
<211>119
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>112
glnvalthrleulysgluserglyproglyileleuglnprosergln
151015
thrleuserleuthrcysserpheserglypheserleuasnthrser
202530
glyvalglyvalglytrpileargglnproserglylysglyleuglu
354045
trpleualahisiletrptrpaspaspaspgluargtyrasnproval
505560
leulysserargleuthrileserlysaspthrserserasnglnval
65707580
pheleulysilealaasnvalaspthralaaspseralathrtyrtyr
859095
cysvalargsermetvalalatrpphepheprotyrtrpglyarggly
100105110
thrleuvalthrvalserala
115
<210>113
<211>45
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>113
thrthrthrproalaproargproprothrproalaprothrileala
151015
serglnproleuserleuargproglualacysargproalaalagly
202530
glyalavalhisthrargglyleuaspphealacysasp
354045
<210>114
<211>24
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>114
iletyriletrpalaproleualaglythrcysglyvalleuleuleu
151015
serleuvalilethrleutyrcys
20
<210>115
<211>41
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>115
argserlysargserargleuleuhisserasptyrmetasnmetthr
151015
proargargproglyprothrarglyshistyrglnprotyralapro
202530
proargaspphealaalatyrargser
3540
<210>116
<211>113
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>116
argvallyspheserargseralaaspalaproalatyrglnglngly
151015
glnasnglnleutyrasngluleuasnleuglyargarggluglutyr
202530
aspvalleuasplysargargglyargaspproglumetglyglylys
354045
proglnargarglysasnproglngluglyleutyrasngluleugln
505560
lysasplysmetalaglualatyrsergluileglymetlysglyglu
65707580
argargargglylysglyhisaspglyleutyrglnglyleuserthr
859095
alathrlysaspthrtyraspalaleuhismetglnalaleupropro
100105110
arg
<210>117
<211>22
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>117
glyserglyalathrasnpheserleuleulysglnalaglyaspval
151015
glugluasnproglypro
20
<210>118
<211>20
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>118
mettyrargmetglnleuleusercysilealaleuserleualaleu
151015
valthrasnser
20
<210>119
<211>21
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>119
glyilehisvalpheileleuglycyspheseralaglyleuprolys
151015
thrglualaasntrp
20
<210>120
<211>50
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>120
valasnvalileseraspleulyslysilegluaspleuileglnser
151015
methisileaspalathrleutyrthrgluseraspvalhisproser
202530
cyslysvalthralametlyscyspheleuleugluleuglnvalile
354045
serleu
50
<210>121
<211>50
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>121
gluserglyaspalaserilehisaspthrvalgluasnleuileile
151015
leualaasnasnserleuserserasnglyasnvalthrglusergly
202530
cyslysglucysglugluleugluglulysasnilelysglupheleu
354045
glnser
50
<210>122
<211>12
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>122
phevalhisilevalglnmetpheileasnthrser
1510
<210>123
<211>26
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>123
serglyglyglyserglyglyglyglyserglyglyglyglysergly
151015
glyglyglyserglyglyglyserleugln
2025
<210>124
<211>29
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>124
alaproargargalaargglycysargthrleuglyleuproalaleu
151015
leuleuleuleuleuleuargproproalathrarggly
2025
<210>125
<211>65
<212>prt
<213>artificialsequence
<220>
<223>syntheticsequence
<400>125
ilethrcysproproprometservalgluhisalaaspiletrpval
151015
lyssertyrserleutyrserarggluargtyrilecysasnsergly
202530
phelysarglysalaglythrserserleuthrglucysvalleuasn
354045
lysalathrasnvalalahistrpthrthrproserleulyscysile
505560
arg
65
- 还没有人留言评论。精彩留言会获得点赞!