用于检测RNA水平基因融合的方法、电子设备和计算机存储介质与流程
本公开总体上涉及生物信息检测处理,并且具体地,涉及用于检测基因融合的方法、电子设备和计算机存储介质。
背景技术:
基因融合(fusiongene)是两个或者两个以上的基因部分或者全部的序列构成新的杂合基因的过程,是导致癌症发生和发展的重要机制之一。例如,针对ntrk融合靶点的loxo-101的靶向药物对泛癌种具有广泛的药效,有数据表明,其可以达到整体70-80%的癌症控制率。因此,基因融合检测是对于肿瘤预测、临床肿瘤治疗的靶向用药设计具有非常重要意义。传统的基于rna层面的二代测序技术的融合检测方案例如包括:,利用全转录组测序或者利用预定义的panel来筛选想要的融合基因,目前有了多种方法软件。例如,star-fusion、fusionseq、tophat-fusion、defuse、fusionhunter、fusionmap,soapfus等等。
在传统的融合检测的方案中,就单个样本的产生结果文件里,会产生很多的候选(candicate)融合,并且无法直接判断这些候选融合的真假,因此存在着大量的假阳性,无法直接满足临床检验的精准需求。
技术实现要素:
本公开提供一种检测rna水平基因融合的方法、电子设备和计算机存储介质,有助于对假阳性的快速正确识别,能够明显降低基因融合检测结果的假阳性。
根据本公开的第一方面,提供了一种检测基因融合的方法。该方法包括:接收全基因组比对信息和全转录组比对信息,全基因组比对信息和全转录组比对信息是基于双末端测序数据分别与全基因组参考序列和全转录组参考序列的比对结果而分别生成的,双末端测序数据包括待测样本的多个成对读长;针对成对失调读长进行聚类,以生成多个大簇,成对失调读长是基于全基因组比对信息而获取的;针对符合第一预定条件的大簇进行全基因组层面的基因注释,以便基于对应基因生成用于标识大簇的第一基因名称组合;基于全转录组比对信息,针对多个成对读长进行全转录组层面的基因注释,以便基于对应基因生成用于标识多个成对读长的第二基因名称组合;以及识别第一基因名称组合和第二基因名称组合所关联的相同的对应基因,以便将相同的对应基因确定为潜在的融合基因。
检测基因融合根据本发明的第二方面,还提供了一种计算设备,该设备包括:存储器,被配置为存储一个或多个计算机程序;以及处理器,耦合至存储器并且被配置为执行一个或多个程序使装置执行本公开的第一方面的方法。
根据本公开的第三方面,还提供了一种非瞬态计算机可读存储介质。该非瞬态计算机可读存储介质上存储有机器可执行指令,该机器可执行指令在被执行时使机器执行本公开的第一方面的方法。
提供发明内容部分是为了以简化的形式来介绍对概念的选择,它们在下文的具体实施方式中将被进一步描述。发明内容部分无意标识本公开的关键特征或主要特征,也无意限制本公开的范围。
附图说明
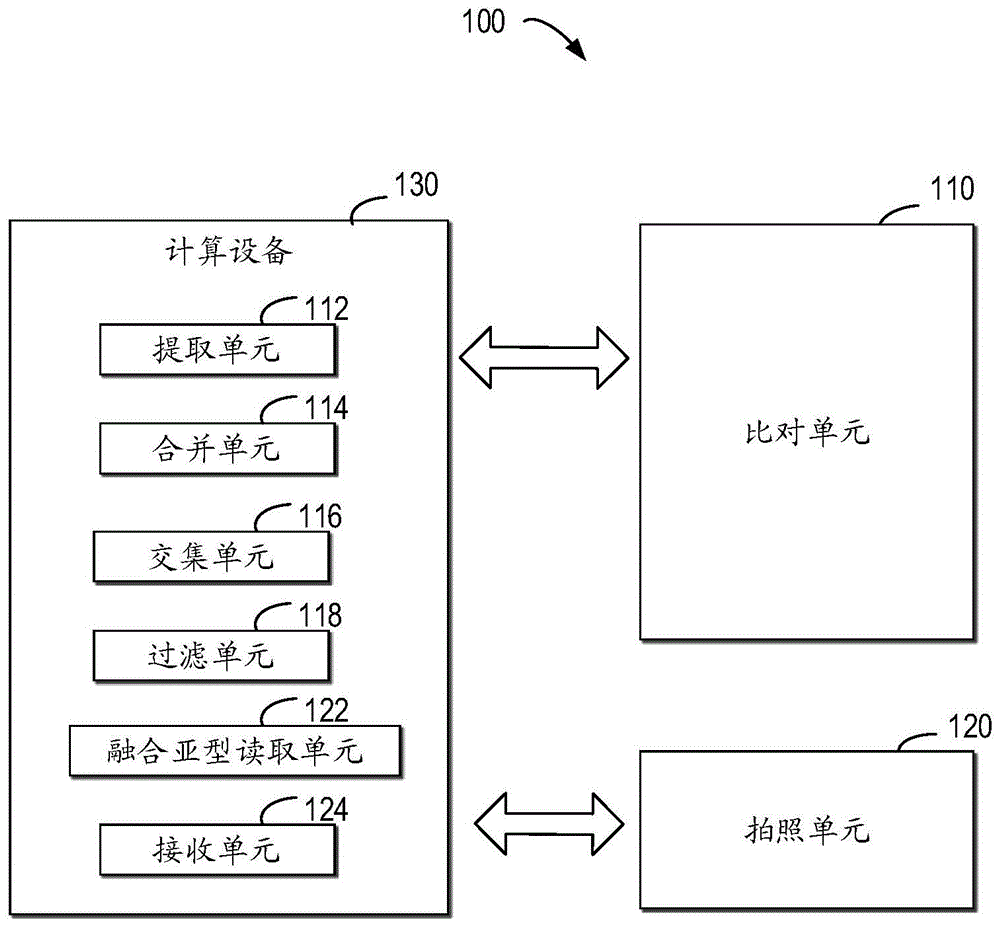
图1示出了根据本公开的实施例的用于检测rna水平基因融合的方法的系统100的示意图;
图2示出了根据本公开的实施例的用于检测rna水平基因融合的方法200的流程图;
图3示出了根据本公开的实施例的用于确定基因融合的可靠性的方法300的流程图;
图4示出了根据本公开的实施例的用于留下可靠的潜在的融合基因的方法400的流程图;
图5示出了根据本公开的实施例的用于留下可靠的潜在的融合基因的方法500的流程图;
图6示出了根据本公开的实施例的融合支持簇上断点的特征的示意图;
图7示出了根据本公开的实施例的用于留下可靠的潜在的融合基因的方法700的流程图;
图8示出了根据本公开实施例的融合支持簇中的成对的读长的类别的示意图;
图9示出了根据本公开的实施例的用于显示潜在的基因融合的方法900的流程图;
图10示出了根据本公开的实施例的基于所采集igv图像确定参与融合的exon号的示意图;
图11示出了根据本公开的实施例的验证例的检测结果示意图;
图12示出了根据本公开的实施例的用于检测可靠的rna水平基因融合的方法1200的流程图;以及
图13示意性示出了适于用来实现本公开实施例的电子设备1300的框图;以及。
在各个附图中,相同或对应的标号表示相同或对应的部分。
具体实施方式
下面将参照附图更详细地描述本公开的优选实施例。虽然附图中显示了本公开的优选实施例,然而应该理解,可以以各种形式实现本公开而不应被这里阐述的实施例所限制。相反,提供这些实施例是为了使本公开更加透彻和完整,并且能够将本公开的范围完整地传达给本领域的技术人员。
关于比对信息来源,以下实施例中,在进行之前,先对待测样本通过探针捕获得到的各个测序片段进行双末端测序得到双末端数据,该双末端数据包括一对对成对的读长;再将获得的双末端数据比对到参考基因组上得到比对信息。
在本文中使用的术语“包括”及其变形表示开放性包括,即“包括但不限于”。除非特别申明,术语“或”表示“和/或”。术语“基于”表示“至少部分地基于”。术语“一个示例实施例”和“一个实施例”表示“至少一个示例实施例”。术语“另一实施例”表示“至少一个另外的实施例”。术语“第一”、“第二”等等可以指代不同的或相同的对象。
另外,在本文中使用的术语“测序片段”,其通常是将来自目标个体的待测样本经过测序平台适配的文库构建流程构建出来的rna文库,其组成是一定长度的rna随机片段。
在本文中使用的术语“读长”表示对测序片段末端测序得到的测序序列。术语“成对的读长”表示:双末端测序得到的来自同一个测序片段两端的两个测序序列,根据成对的读长在全基因组参考序列上比对的结果可以将成对的读长分为不同类型的成对的读长。术语“失调读长对”或者“成对失调读长”表示:上述比对的结果显示成对的读长不能按正常的映射间距、方向比对到全基因组参考序列上,或其中的一个或两个读长都不能完整地在一个位置比对到全基因组参考序列上,或者两个成对的读长不能比对在同一个染色体上,通常指pediscordantreads和clippedreads对。术语“读长起点位置”表示:上述映射的起点在参考基因组上所处的位置。术语“读长终点位置”表示:上述映射的终点在参考基因组上所处的位置。
在本文中使用的术语“映射距离(intersize)”表示:上述成对的读长起点之间的距离,也即成对的读长的两个读长起点位置之间的距离。术语“映射方向”表示:指一个读长比对在参考序列上的方向。术语“映射位置”表示:上述读长起点在参考序列上所处的位置。
术语“断点”表示:一个读长上,与参考序列连续匹配和与参考序列连续不匹配的交界处碱基所在的位点称为该读长的断点。术语“同一断点”表示:断点在全基因组参考序列上映射到同一位置的所有断点统称为同一断点。术语“基因注释”表示:基因结构被精确注释到内含子(intron)、外显子(exon)具体组成结构的基因结构。术语“基因名称组合”表示:对特定区域的读长(reads)进行注释,能注释到的基因名称进行组合为一个基因名称组合,将该基因名称组合用于标识该特定区域。比如针对一个特定区域进行基因注释,注释到的对应基因的名称分别为a基因和b基因,则用于标识该特定区域的基因名称组合为a+b。由相同的基因组合名称所标识,表示都注释到相同的对应基因,这里相同的对应基因是指对应基因的名称相同,不是指具体的基因结构相同。
经研究发现,在上述传统的用于检测基因融合的方案中,针对单个样本所产生的检测结果文件中,会产生很多无法直接判断真假的候选融合。另外,由于在rna层面比对到参考基因组上时,因为rna不包含内含子(intron)区域,因此会面临“将短片段化比对至基因组所导致的专一性较为困难”的挑战,从而导致候选基因的判断真假的不确定性,进而导致假阳性很高。并且,传统的用于检测基因融合的方案中存在着大量例如是由相邻基因的可变剪接、基因家族、假基因等所导致的假阳性,因此在准确检测基因融合方面存在局限性。
为了至少部分地解决上述问题以及其他潜在问题中的一个或者多个,本公开的示例实施例提出了一种用于检测基因融合的方案。该方案包括:接收全基因组比对信息和全转录组比对信息,全基因组比对信息和全转录组比对信息是基于双末端测序数据分别与全基因组参考序列和全转录组参考序列的比对结果而分别生成的,双末端测序数据包括待测样本的多个成对读长;针对成对失调读长进行聚类,以生成多个大簇,成对失调读长是基于全基因组比对信息而获取的;针对符合第一预定条件的大簇进行全基因组层面的基因注释,以便基于对应基因生成用于标识大簇的第一基因名称组合;基于全转录组比对信息,针对多个成对读长进行全转录组层面的基因注释,以便基于对应基因生成用于标识多个成对读长的第二基因名称组合;以及识别第一基因名称组合和第二基因名称组合所关联的相同的对应基因,以便将相同的对应基因确定为潜在的融合基因。
在上述方案中,通过获得双末端测序数据分别与全基因组参考序列和全转录组参考序列的比对结果而分别生成的全基因组比对信息和全转录组比对信息;并且通过针对由基于全基因组比对信息而获取的成对失调读长所聚合的大簇进行全基因组层面的基因注释,基于全转录组比对信息、针对成对读长进行全转录组层面的基因注释,以及基于全基因组层面的基因注释和全转录组层面的基因注释来识别相同的对应基因,以便将其确定为潜在的融合基因;本公开能够将rna测序数据同时比对基因组和转录组,可以得到有效的候选基因融合组合对,以及通过抽取纯支持某种基因融合转录组的转录本信息对和区域,从而回避了传统方法intron的问题和短片段比对不专一问题,因此能够明显降低检测结果的假阳性,增加基因融合的检出率以及准确率。
图1示出了根据本公开的实施例的用于检测基因融合的方法的系统100的示意图。如图1所示,系统100包括比对单元110、提取单元112、合并单元114、交集单元116、过滤单元118、拍照单元120、融合亚型读取单元122、接收单元124。
在一些实施例中,提取单元112、合并单元114、交集单元116、过滤单元118、融合亚型读取单元122、接收单元124可以配置在一个或者多个计算设备130上。比对单元110和拍照单元可以独立于计算设备130之外。计算设备130可以获取比对单元110所生成的比对信息。比对单元110的比对信息例如包括:双末端测序数据、全基因组比对信息和全转录组比对信息。计算设备130可以通过有线或者无线的方式与比对单元110进行数据交互。
比对单元110用于针对待测样本通过探针捕获得到的各个测序片段进行双末端测序得到双末端数据;然后基于双末端数据与全基因组参考序列的比对结果而生成全基因组比对信息;以及基于双末端数据与全转录组参考序列的比对结果而生成全转录组比对信息。其中全基因组比对信息可以包括各个读长比对得到的在全基因组上的映射方向、映射位置、以及成对的读长比对得到的intersize。在一些实施例中,全基因组比对信息还可以包括:通过读长的匹配情况找出的各个与不同读长分别对应的断点、以及成对读长的类型。全转录组比对信息例如包括双末端测序数据中的成对的读长能比对到的相应转录本。
提取单元112用于提取比对异常的成对失调读长以便用于后继分析。提取单元112可以根据比对结果所显示的成对的读长不能按正常的映射间距、方向比对到全基因组参考序列上,或其中的一个或两个读长都不能完整地在一个位置比对到全基因组参考序列上,或者两个成对的读长不能比对在同一个染色体上,而提取比对异常的成对失调读长。
合并单元114用于针对读长进行聚类、基因注释以及数据合并。在一些实施例中,合并单元114可以直接针对成对失调读长进行聚类以生成多个大簇。在一些实施例中,合并单元114可以首先根据失调读长对所对应的映射位置之间的间距是否满足预定聚类距离来针对成对失调读长进行聚类以生成多个小簇;然后再将小簇所包括的成对失调读长的对应关系相同的小簇合并以生成大簇。合并单元114还可以针对符合第一预定条件的大簇进行全基因组层面的基因注释,以便基于对应基因生成用于标识大簇的第一基因组合名称。合并单元114还可以基于全转录组比对信息,针对多个成对读长进行全转录组层面的基因注释,以便基于对应基因生成用于标识多个成对读长的第二基因组合名称。
交集单元116用于识别潜在的融合基因。例如,交集单元116可以识别用于标识大簇的第一基因组合名称和用于标识多个成对读长的第二基因组合名称的交集,即第一基因组合名称和第二基因组合名称所关联的相同的对应基因,以便将相同的对应基因确定为潜在的融合基因。交集单元116还可以基于具有相同基因名称的支持簇,确定潜在的融合基因的潜在支持簇。
过滤单元118用于过滤rna水平得到的比对非特异性组合(arti)和转录组水平相邻基因的可变剪接,以便留下可靠的潜在融合基因,过滤掉不可靠候选融合基因。rna水平的融合是检测细胞转录水平的融合,相对于细胞的基因组水平(dna)水平对于融合蛋白的表达的反映更直接。例如,过滤单元118可以确定潜在融合基因是否满足以下去除条件。该去除条件为:属于基因家族、假基因、预定的转录本比对非特异性组合(arti,例如常见转录本比对非特异性组合)、ncrna、或者非5’-3’转录结构,或者属于相邻基因并且相邻基因中的两个基因所分别对应的经与参考转录组比对所获得的支持簇读长方向为相对的。如果判断满足以上去除条件时,则将相应的潜在融合基因去除。否则(即不满足去除条件),则留下潜在融合基因。
拍照单元120用于将基因组合的readssupport证据用拍照方式保存。
融合亚型读取单元122用于对rna水平形成的融合亚型进行预测。融合亚型读取单元122可以基于前文所确定潜在融合基因确定融合partner和融合亚型。
接收单元124用于接收成功拍照的基因组合的断点信息和基因结构注释信息,预测融合转录本的信息。
以下将结合图2描述根据本公开的实施例的用于检测rna水平基因融合的方法。图2示出了根据本公开的实施例的用于检测基因融合的方法200的流程图。应当理解,方法200例如可以在图13所描述的电子设备1300处执行。也可以在图1所描述的计算设备130处执行。应当理解,方法200还可以包括未示出的附加动作和/或可以省略所示出的动作,本公开的范围在此方面不受限制。
在框202处,计算设备130可以接收全基因组比对信息和全转录组比对信息,全基因组比对信息和全转录组比对信息是基于双末端测序数据分别与全基因组参考序列和全转录组参考序列的比对结果而分别生成的,双末端测序数据包括待测样本的多个成对读长。在一些实施例中,计算设备130经由所配置的通讯单元(未在图1中示出)从比对单元110接收全基因组比对信息和全转录组比对信息。
关于全基因组比对信息,在一些实施例中,全基因组比对信息包括:各个读长比对得到的在全基因组上的映射方向、映射位置。在一些实施例中,全基因组比对信息还包括:成对的读长比对得到的intersize、通过读长的匹配情况找出的各与不同读长分别对应的断点、成对读长的类型中的至少一项。关于全转录组比对信息,在一些实施例中,其包括:双末端测序数据中的成对的读长能比对到的相应转录本。在框204处,计算设备130可以针对成对失调读长进行聚类,以生成多个大簇,成对失调读长是基于全基因组比对信息而获取的。例如,计算设备130可以基于框202处所获取的rna转录组双末端测序数据与全基因组参考序列进行比对的具体比对信息,筛选出成对失调读长(pediscordantreads);然后针对成对失调读长进行聚类以便生成多个大簇。
关于生成多个大簇的方式包括多种。在一些实施例中,生成多个大簇的方式包括:计算设备130可以直接基于预定聚类规则(例如成对失调读长所对应的映射位置之间的间距)针对成对失调读长进行聚类以生成多个大簇。在一些实施例中,计算设备130也可以首先根据失调读长对所对应的映射位置之间的间距是否满足预定聚类距离来针对成对失调读长进行聚类以生成多个小簇;然后再将所包括的成对失调读长的对应关系相同的小簇合并以生成大簇。下文将对“先生成小簇、再将小簇合并成大簇”的方式进行说明。
首先,如果计算设备130确定成对失调读长对应的映射位置之间的间距满足预定聚类距离,针对成对失调读长进行聚类,以生成多个小簇。例如,如果计算设备130可以确定当前读长的终点位置和下一个相邻的读长的起点位置之间的间距,然后判断该间距是否满足预定聚类距离(预定聚类距离例如为小于或者等于500-1000bp)。例如,可以将位于第一个读长的终点位置与邻近读长的始点位置之间的间距与预定聚类距离进行比较,依此进行,直到出现一个读长的终点位置与邻近读长的始点位置之间的间距超过预定聚类距离,则该读长为该小簇中的最后一个读长。以此类推,可以生成多个小簇。
然后,计算设备130确定小簇所包括的成对失调读长的对应关系是否相同;如果确定小簇的间距满足预定距离,合并小簇,以生成大簇。计算设备130可以将所有上述得到的小簇中的间距满足预定距离(例如小于500bp)进行合并得到各个大簇。通过采用上述手段,即基于失调读长对对应的映射位置之间的间距与预定聚类距离先聚成小簇,以及将间距满足预定距离的小簇合并成大簇,有利于提高潜在基因融合的确定精度。
在框206处,计算设备130可以针对符合第一预定条件的大簇进行全基因组层面的基因注释,以便基于对应基因生成用于标识大簇的第一基因名称组合。其中第一预定条件例如包括:大簇所包括的成对失调读长的数量大于或者等于第一预定对数。
例如,计算设备130将大簇中的read按照开始位置(startlocation)进行全基因组层面的基因注释,以便标识各read的所在基因名称组合,以便生成标识大簇的第一基因名称组合。在一些实施例中,计算设备130还可以进一步将各标记好所在基因名称组合的read,按照nameid相同的pe关系(一对pereads中r1和r2的nameid前缀相同),记录pereadid及其对应所在基因名称组合。
在一些实施例中,计算设备130针对符合第一预定条件的大簇(例如包括的成对失调读长的对数满足大于等于第一预定对数)的各个大簇对应的区域分别进行全基因组层面的基因注释,以便得到与各个大簇分别对应的第一基因名称组合,并将第一基因名称组合相同的大簇进行合并得到与该第一基因名称组合相对应的支持簇。例如,计算设备130首先判断框204处所生成的大簇中包括的成对失调读长的对数是否满足大于等于第一预定对数(例如而不限于是5),对于满足第一预定对数的,计算设备130继续进行全基因组层面的基因注释以便得到相应的第一基因名称组合。例如,基因注释到基因a和基因b的名称,则基因组合名称就为a+b。
在框208处,计算设备130基于全转录组比对信息,针对多个成对读长进行全转录组层面的基因注释,以便基于对应基因生成用于标识多个成对读长的第二基因名称组合。例如,计算设备130可以基于框202处所获取的rna转录组双末端测序数据与全转录组参考序列进行比对的具体比对信息,将pe成对readsid比对到的转录本并就其对应的基因进行全转录组层面的基因注释,得到pereadid及其对应所在基因名称组合。
在框210处,计算设备130可以识别第一基因名称组合和第二基因名称组合所关联的相同的对应基因,以便基于相同的对应基因确定潜在的融合基因。例如,计算设备130可以针对框206处所生成基于全基因组层面的基因注释的第一基因名称组合,以及208处所生成的基于全转录组层面的基因注释的第二基因名称组合进行逻辑交集处理,得到全基因组和全转录组均比对到相同的对应基因上的readsids与其对应所在的基因名称组合。然后对不同的对应基因的reads进行数量统计,得到跨不同组合基因的pediscordantreads支持对数和readsids文件。
在上述方案中,由于基因rna融合检测方法将分别在基因组水平和转录组水平分别得到的注释为同一个基因名称组合的基因组合作为相应的潜在融合基因,并具有相同的基因名称的支持簇确定为该潜在的融合基因的潜在支持簇,继续通过可靠性判断就能找出可靠的融合基因,从而避免了将rna片段reads单独通过全基因组检测融合基因时,由于不包含intron区域而短片段化比对造成的假阳性很高的问题,同时又能避免单独直接比对到转录本水平由于多个转录本可变剪接问题或rna片段部分交联而检测融合基因时比对不特异导致的融合基因漏检的问题,也即本发明的基因rna融合检测方法既能降低假阳性概率,又能提高实际的融合基因的检出率。
在一些实施例中,方法200还还包括:计算设备130合并由相同的基因组合名称所标识的大簇,以生成与相同的基因组合名称相关联的支持簇;基于具有相同的基因名称的支持簇,确定潜在的融合基因的潜在支持簇。例如,当将各个满足第一预定对数的大簇注释完成后,计算设备130将第一基因名称组合相同的大簇进行合并,这样就得到该基因组合名称对应的支持簇。例如有两个大簇注释后的第一基因名称组合都为a+b,其中一个大簇中包括的成对的失调对数为5,另外一个大簇中包括的成对的失调对数为6,则该两个大簇进行合并,得到5+6=11对成对的失调对数。计算设备130可以将上述11对成对的失调对数确定为对应于基因组合名称a+b的基因组合的支持簇。
在一些实施例中,方法200还包括潜在的融合基因的可靠性判断方法。下文将结合图3至8说明融合基因的可靠性判断方法的相关方式,在此,不再赘述。
在一些实施例中,方法200还包括潜在的融合基因的可视化方法。下文将结合图9至10说明融合基因的可靠性判断方法的相关方式,在此,不再赘述。
图3示出了根据本公开的实施例的用于确定基因融合的可靠性的方法300的流程图。应当理解,方法300例如可以在图13所描述的电子设备1300处执行。也可以在图1所描述的计算设备130处执行。应当理解,方法300还可以包括未示出的附加动作和/或可以省略所示出的动作,本公开的范围在此方面不受限制。
在框302处,计算设备130可以确定潜在支持簇所包括的成对失调读长的数量是否大于或者等于第二预定对数。
在框304处,如果计算设备130确定潜在支持簇所包括的成对失调读长的数量大于或者等于第二预定对数,确认潜在融合基因是否满足以下去除条件:属于基因家族、假基因预定的转录本比对非特异性组合、非编码核糖核酸(ncrna)中的至少一种去除;属于相邻基因并且相邻基因中的两个基因所分别对应的经与参考转录组比对所获得的支持簇读长方向为相对的;或者不属于预定转录结构(预定转录结构例如为五三转录结构)。例如,计算设备130可以针对框210处所确定的潜在的基因组合所对应reads支持对数超过5条的,进行基因家族、假基因、相邻基因并且对应支持簇read方向相对、常见转录本比对arti组合、ncrna、非五三转录结构(非5’-3’转录结构)的识别去除,然后就留下的潜在的基因组合进行后续处理。
在一些实施例中,如果潜在融合基因中的两个基因为相邻基因,并且该两个基因所分别对应的经与参考转录组比对所获得的支持簇读长方向为相对的。例如,潜在的基因组合a和b被确定为相邻基因,并且基因a对应的经与参考转录组比对所获得的支持簇read方向,与基因组合b对应的经与参考转录组比对所获得的支持簇read方向是相对的。上述相对的情形例如是指基因组合a-b形成5’-3’的可变剪接转录本。通过采用上述手段,将满足相邻基因但是对应支持簇的读长方向不相对的潜在的基因组合留下,能够避免漏检。
在框306处,如果计算设备130确定潜在的融合基因不满足去除条件,留下潜在的融合基因。相反,如果计算设备130确定潜在的融合基因满足去除条件,则至框310处去除该融合基因。
在框308处,计算设备130确定所留下的潜在的融合基因为可靠的潜在的融合基因。
通过采用上述手段,本公开能够提高所确定的潜在的融合基因的可靠性。
在一些实施例中,方法300还进一步包括用于过滤潜在的融合基因以便留下可靠的潜在的rna融合的方法。下文将结合图4至图7具体说明留下可靠潜在的融合基因的方法。
图4示出了根据本公开的实施例的用于留下可靠的潜在的融合基因的方法400的流程图。应当理解,方法400例如可以在图13所描述的电子设备1300处执行。也可以在图1所描述的计算设备130处执行。应当理解,方法400还可以包括未示出的附加动作和/或可以省略所示出的动作,本公开的范围在此方面不受限制。
在框402处,计算设备130可以确定潜在融合基因是否不满足至少一项去除条件。
在框404处,如果计算设备130可以确定潜在融合基因不满足去除条件,将与对应基因所相应的成对失调读长和对应基因的预定转录本的转录参考序列进行单独比对,以便确定比对质量。相反,如果计算设备130确定潜在融合基因满足任一项去除条件,则至框412处去该除潜在融合基因。
例如,某个潜在融合基因并非属于基因家族、假基因、预定的转录本比对非特异性组合、ncrna、非五三转录结构中的至少一种,也并非属于相邻基因并且相邻基因中的两个基因所分别对应的经与参考转录组比对所获得的支持簇读长方向为相对的,则进一步确定比对质量。例如,潜在融合基因的第一基因名称组合为a+b,该第一基因名称组合中对应基因为a和b。则将潜在支持簇中与对应基因a相应的成对失调读长和该基因单常见转录本的转录参考序列进行单个比对。另外,将潜在支持簇中与对应基因b相应的成对失调读长和该基因单常见转录本的转录参考序列进行单个比对。基于上述两个单个对比分别生成比对质量。
在框406处,计算设备130确定比对质量是否大于或者等于预定比对质量。在一些实施例中,预定比对质量例如为10。例如,确定比对质量之后,看该比对质量大于或者等于10。
在框408处,如果计算设备130确定比对质量大于或者等于预定比对质量,确定潜在支持簇中满足预定过滤条件的成对失调读长的数量是否大于或者等于第三预定对数。如果确定比对质量小于预定比对质量,则至框412处执行相应去除。
例如,针对前文提及的对应基因a的上述单个比对的质量大于等于10,而针对对应基因b的单个比对质量小于10,则可以从潜在支持簇中将对应基因a所对应的所有成对失调读长提取出来,以便根据预定过滤条件进行过滤,并判断所有满足预定过滤条件的成对失调读长的对数是否满足大于等于第三预定对数。
关于预定过滤条件,在一些实施例中,其例如是:成对的读长的在全基因组参考序列上的比对为至少一端为完全比对或者其两端都为部分比对。在一些实施例中,第三预定对数例如为5。
在框410处,如果计算设备130确定满足预定过滤条件的成对失调读长的数量大于或者等于第三预定对数,留下潜在的融合基因。如果潜在支持簇中满足预定过滤条件的成对失调读长的数量较少,例如,小于第三预定对数,则至框412处执行相应去除。例如,当潜在支持簇中所有满足预定过滤条件的成对失调读长的对数满足大于或者等于第三预定对数时,将相应的潜在融合基因留下,以便作为可靠地潜在的基因;以及将相应的所有满足预定过滤条件的成对失调读长作为相应的潜在融合基因的融合支持簇。
通过采用上述手段,本公开能够进一步提高所确定的潜在的融合基因的可靠性。
图5示出了根据本公开的实施例的用于留下可靠的潜在的融合基因的方法500的流程图。应当理解,方法500例如可以在图13所描述的电子设备1300处执行。也可以在图1所描述的计算设备130处执行。应当理解,方法500还可以包括未示出的附加动作和/或可以省略所示出的动作,本公开的范围在此方面不受限制。
在框502处,计算设备130可以确定与比对质量大于或者等于预定比对质量的潜在融合基因相应的融合支持簇。在一些实施例中,预定比对质量例如为10。在一些实施例中,计算设备130可以将框404处获得的各基因组合readsid与单基因单常见转录本的reference序列进行单个比对,将比对质量(mapq)较好的比对到各组合基因对的一对转录本上的reads提取出来,过滤留下成对的读长中的两个读长至少有一端完全匹配(wholemapping)或者clippedreads对,形成各基因组合小sam文件,同时重新计算小sam文件中包含的reads支持对数作为该组合基因对的融合支持reads对数。
在框504处,计算设备130将融合支持簇对应在全转录组参考序列上的位置的覆盖区域按相邻预定间距范围进行合并,以便生成合并区域。在一些实施例中,预定间距范围为0-500bp。例如,框502处所确定的融合支持簇中的各个成对失调读长在全转录组参考序列上对应分布到不同的位置。从第一个失调读长开始,按比对位置顺序看相邻的失调读长之间的间距,如果确定一个失调读长与相邻的下一个失调读长之间的间距范围不属于0-500bp,则将该失调读长以及之前的失调读长各自覆盖区域进行合并,以便生成合并区域。以此类推,直到合并完融合支持簇中所有的失调读长的覆盖区域,进而生成多个不同的合并区域。
在框506处,计算设备130确定合并区域的数量是否小于或者等于预定个数。在一些实施例中,预定个数例如而不限于为6。例如,确定框504处所生成的合并区域的个数是否小于等于6。
在框508处,如果计算设备130确定合并区域的数量小于或者等于预定个数,留下潜在的融合基因。如果计算设备130确定合并区域的数量大于预定个数,则至框510处去除该潜在的融合基因。例如,计算设备130针对框502处所获得的各基因组合对的reads分布的区域(region),并对相邻500bp范围之内的进行区域合并,以形成合并区域,如果合并区域后的区域的个数少于6个,则留下潜在的融合基因。进一步的,计算设备130可以抽取出对应基因对的转录本-exon注释bed,load进igv进行后续拍照处理。
应当理解,合并区域后的基因组合所包含的区域个数所反映的是比对的特异性。前文提及的可靠性过滤方式主要基于预定的条件,可能存在意外条件所导致的比对不专一,该比对不专一可能出现未被很好地识别的情形,而通过采用多个合并区域所反映出比对的特异性,可以识别上述比对不专一的情形。因此,通过采用上述手段,本公开能够更为精确地确定潜在的融合基因可靠性。另外,有利于避免发生漏检的问题,同时又能保证处理效率,提高了处理速度。
在一些实施例中,如果框508处可以确定合并区域的数量小于或者等于预定个数,还可以进一步确定相应的融合支持簇是否满足以下至少一项条件:融合支持簇中存在的断点满足预定一致性条件;融合支持簇在转录本上的比对为连续比对。如果确定融合支持簇满足以上至少一项条件,即融合支持簇中存在的断点满足预定一致性条件或者融合支持簇在转录本上的比对为连续比对,则留下潜在的融合基因以作为可靠的潜在的融合基因。
关于预定一致性条件,在一些实施例中,其例如包括:融合支持簇中具有同一断点的所有失调读长之间以同一断点为起点存在相同的不能连续比对的序列,以及具有同一断点的所有失调读长的数量大于或者等于2。
关于连续比对,在一些实施例中,其例如包括:融合支持簇中的各个成对的读长在转录本上的比对覆盖到的区域,具有连续性,不是具有间隔的不连续区域。
以下结合图6说明融合支持簇中存在的断点满足预定一致性条件的情形。图6示出了根据本公开的实施例的融合支持簇上断点的特征的示意图。如图6所示,图中具有同一断点的各个失调读长呈纵向排列分别与参考基因组进行比对,610指示的灰色部分表示存在同一断点的各个映射能匹配的部分,620指示的黑色部分表示不能匹配的部分。在图6中,各个失调读长不能比对的部分存在连续相同的序列,这些融合支持簇的断点满足一致性条件。因此,潜在融合基因留下以确定为可靠的潜在基因。
在一些实施例中,方法500还包括用于过滤潜在的融合基因以便留下可靠的潜在的融合基因的方法700。图7示出了根据本公开的实施例的用于留下可靠的潜在的融合基因的方法700的流程图。应当理解,方法700例如可以在图13所描述的电子设备1300处执行。也可以在图1所描述的计算设备130处执行。应当理解,方法700还可以包括未示出的附加动作和/或可以省略所示出的动作,本公开的范围在此方面不受限制。
在框702处,计算设备130将相应的融合支持簇中的成对的读长在全基因组参考序列进行比对,以便基于两端是否存在断点来确定成对的读长的类别。
在一些实施例中,所确定的成对的读长的类别包括:两端都没有断点(例如为i类)、两端都有断点(例如为ii类)以及只有一端有断点(例如为iii类)。图8示出了根据本公开实施例的融合支持簇中的成对的读长的类别的示意图。在图8中,810所指示的为“两端都没有断点”的类别,其中成对的读长中的两个读长都没有断点,即两端都完全匹配(wholemapping),没有穿过图8中虚线840和842所示的断点。820所指示的为“两端都有断点”(twobp)的类型,即成对的读长中的两个读长都存在图8中虚线840和842所示的断点。830所指示的为“只有一端有断点”(onepartnerbp)的类型,即成对的读长中的两个读长中的一个存在图8中虚线840所示的断点,另一个不存在图8中虚线842所示的断点。
在框704处,计算设备130融合支持簇中至少包括两种不同类型的成对的读长。
在框706处,如果计算设备130确定融合支持簇中至少包括两种不同类型的成对的读长,确定不同类型的成对的读长在全基因组参考序列上对应的区域是否存在重合的融合区域。如果计算设备130确定融合支持簇中未包括至少两种不同类型的成对的读长,则至框710处去除该潜在的融合基因。
在一些实施例中,如果计算设备130确定融合支持簇中至少包括两种类型的成对的读长时,例如,存在810所示的i类和820所示的ii类两种成对的读长的类型时,进一步针对不同类型的成对的读长在全基因组参考序列上对应的区域确定是否存在重合的融合区域。以下表一示意性示出了成对的读长1至6的类别以及在全基因组参考序列上对应的区域。由表一可知,类型为i类的所有成对的读长所对应的所有区域为5-70。类型为ii类的所有成对的读长所对应的所有区域为18至85。然后确定上述两种区域之间是否存在相互覆盖区域,以便将其作为融合区域。
表一
在框708处,如果计算设备130确定不同类型的成对的读长在全基因组参考序列上对应的区域存在重合的融合区域,留下潜在的融合基因。如果计算设备130确定不存在重合的融合区域,则至框710处去除该潜在的融合基因。
例如,如表一所示,类型为i类的所有成对的读长所对应的所有区域为5-70。类型为ii类的所有成对的读长所对应的所有区域为18至85。上述两种区域之间存在相互重叠的融合区域18-70,则留下该潜在融合基因,即该确定的为可靠的融合基因的融合区域就为18-70。反之,如果一个可靠支持簇只包括一种类型的成对的读长时,舍弃该潜在融合基因。如果一个可靠支持簇包括两种类型及以上的成对的读长时,则继续确定是否存在重合的融合区域,如果存在重合的融合区域,则留下该潜在融合基因。对于有清晰一致断点和reads连续转录组上比对reads,即可认为是可靠的基因融合。进一步的计算设备130可以对其从头进行基因结构注释,得到一致的基因融合及其参与exon亚型。
通过采用上述手段,本公开能够更为精确地确定潜在融合基因的可靠性。另外,所确定的不同类型的成对的读长在全基因组参考序列上对应的区域存在重合的融合区域其实就是融合基因实际发生融合的区域,因此,本公开还能够实现基于该融合区域的注释分析而获得融合位点和原因。
图9示出了根据本公开的实施例的用于显示潜在的基因融合的方法900的流程图。应当理解,方法900例如可以在图13所描述的电子设备1300处执行。也可以在图1所描述的计算设备130处执行。应当理解,方法900还可以包括未示出的附加动作和/或可以省略所示出的动作,本公开的范围在此方面不受限制。
在框902处,计算设备130对融合支持簇中具有同一断点并且比对的箭头方向朝向右的所有失调读长,确定从exon号为1开始到断点位置之前能匹配上的位置包括的exon号为5’端参与融合的exon号。例如,计算设备130采集所留下潜在的基因融合的igv图像,以便基于igv图像确定参与融合的外显子(exon)号。图10示出了根据本公开的实施例的基于所采集igv图像确定参与融合的exon号的示意图。如图10所示,图10包括一组读长。该组读长的箭头方向整体朝右(未示出)。未完全匹配上的部分的断点位置的exon号例如为exonn,以该断点为分界线,分为能匹配上的区域(matchregion)和未匹配上的区域(softclipregion),能匹配上的区域指该组读长中所有能匹配到参考本上的部分覆盖的所有区域。未匹配上的区域指该组读长中所有不能匹配到参考本上的部分覆盖的所有区域,exon号从匹配到的区域的最左端的exon1向右直到最右端(最末端)的lastexonn。其中exon1开始直到能匹配上的位置(也即exonn-1),包括的所有exon号,即为前文提及的5’端参与融合的exon号。
在框904处,计算设备130对融合支持簇中具有同一断点并且比对的箭头方向朝向左的所有失调读长,确定断点位置向右的第一个匹配得上的位置的exon号到向右直到最末端的exon号为3’端参与融合的exon号。如图10所示,断点向右的第一个匹配上的位置为exonn+1,最末端的为lastexonn为3’端参与融合的exon号。
通过采用上述手段,能够全面且直观展示所得到的断点、基因名称组合中的不同基因组合、可靠支持簇中的各个成对的读长的比对情况进行呈现,以便对靶向用药起到指导作用。
在一些实施例中,计算设备130例如将有清晰一致断点和reads连续转录组上比对reads确认为是可靠的基因融合,对其从头进行基因结构注释,得到一致的基因融合及其参与exon亚型。计算设备130可以进行将所得到的断点、基因名称组合中的不同基因组合、可靠支持簇中的各个成对的读长的比对情况进行呈现,并将最后得到为可靠的融合基因以及找出的相应的融合区域部分,根据同一断点位置,可以重新进行详细的基因注释,以找出具体参与exon亚型。在一些实施例中,计算设备130还可以将断点、subsam、基因组合,基因功能影响等信息进行汇总呈现。
图11示出了根据本公开的实施例的验证例的检测结果示意图。图11所示的是针对验证例为软组织肿瘤ffpe样本(119c5124s1n1)的检测结果。已知该样本具有lmna-ntrk1融合基因,对其采用实施例的方法进行检测,得到的结果如图11所示的检测结果。如图11所示,从图中可以直观地看出,通过实施例的方法,找出的区域显示为lmna基因和ntrk1基因的融合,左边为lmna基因,右边ntrk1基因,且能找出两个基因分布具体参与融合的exon亚型,还能看出支持该融合基因的成对的读长的分布情况以及断点位置。以下表二具体示意出所确定的lmna基因和ntrk1基因的融合的相关信息。
表二
由于本公开的基因rna融合检测方法将分别在基因组层面和转录组层面分别得到的注释为同一个基因名称组合的基因组合作为相应的潜在融合基因,并把具有相同的基因名称的成对失调读长为该潜在的融合基因的潜在支持对,继续通过可靠性判断就能找出可靠的融合基因,从而避免了将rna片段单独通过全基因组检测融合基因时,由于不包含intron区域而短片段化比对造成的假阳性很高的问题,同时又能避免单独直接比对到转录本水平由于多个转录本可变剪接问题或rna片段部分交联而检测融合基因时比对不特异导致的融合基因漏检的问题,也即本发明的基因rna融合检测方法既能降低假阳性概率,又能提高实际的融合基因的检出率。
图12示出了根据本公开的实施例的用于检测可靠的rna水平基因融合的方法1200的流程图。应当理解,方法1200例如可以在图13所描述的电子设备1300处执行。也可以在图1所描述的计算设备130处执行。应当理解,方法1200还可以包括未示出的附加动作和/或可以省略所示出的动作,本公开的范围在此方面不受限制。
在框1202处,计算设备130可以接收全基因组比对信息和全转录组比对信息。例如,可以针对样本提取mrna,然后目标平台(targetpanel)捕获后上机测序(例如,pe150x2、20m目标测序数据量),以便获得fastq文件。然后针对所获得的fastq文件进行与参考基因组hg19进行比对(bwamem),以便获得bam格式的比对结果文件。另外,针对上述fastq文件进行与人类hg19参考转录组进行比对(例如,进行minimap2与gencodetanscript.fa),以便获得paf比对结果文件。
在框1204处,计算设备130针对符合预定条件的成对失调读长进行聚类,以生成多个大簇。例如,计算设备130可以根据成对失调读长(pereads)之间比对的intersize参数(是否大于2kb)和cigar(例如,[hs],butnot[di]),来从上述bam格式的比对结果文件中的dedup.bam中筛选提取出关于候选的嵌合读长(chimericreads)的subset.bam文件。然后计算设备130将所获得的subset.bam文件转换成bed并且进行合并(merge,-d10),以便进行聚类(cluster,-d10)。例如,将多对pereads聚类到相同的多处区域(region)的读长(reads)合并在一起。
在框1206处,计算设备130针对大簇进行全基因组层面的基因注释,以便基于对应基因生成用于标识大簇的第一基因名称组合。例如,计算设备130针对合并区域进行基因注释,获得基因组水平各基因组合所对应的读长标识(readsids)文件,并且针对读长进行计数(readscount)。该读长标识(readsids)文件可以指示第一基因名称组合。然后,针对所获得的多个support.sub.bam文件的支持区域(supportregion)进行基因注释,以便获得候选的基因组合列表。
在框1208处,计算设备130针对多个成对读长进行全转录组层面的基因注释,以便基于对应基因生成用于标识所述多个成对读长的第二基因名称组合。例如,计算设备130将框1206处获得的读长标识(readsids)文件按照成对关系(pair)所比对到的转录本进行注释和聚类,然后对转录本的基因名称进行注释,获得转录组水平各基因组合所对应的读长标识(readsids)文件,该读长标识(readsids)文件可以指示第二基因名称组合。
在框1210处,计算设备130基于第一基因名称组合和所述第二基因名称组合对应一致的基因,确定潜在的rna水平的基因融合。例如,计算设备130基于将框1206处所获得基因组水平各基因组合所对应的读长标识(readsids)文件和框1208处所获得的转录组水平各基因组合所对应的读长标识(readsids)文件比对一致的对应基因,生成潜在的基因融合列表文件。通过采用上述手端,本公开通过rna测序数据同时比对基因组和转录组,能够得到有效的rna潜在的(candicate)基因融合组合对。
在框1212处,如果计算设备130确定潜在的rna水平的基因融合属于基因家族、假基因、相邻基因并且相邻基因中的两个基因所分别对应的经与参考转录组比对所获得的支持簇读长方向为相对的,预定的转录本比对非特异性组合、非编码核糖核酸(ncrna)、非五三转录结构中的至少一种,去除该潜在的rna水平的基因融合,否则留下该潜在的rna水平的基因融合。例如,计算设备130对照一个关于过滤条件的配置(config)文件,如果框1210处所确定的潜在的rna水平的基因融合属于同一基因家族、假基因、rna比对常见arti、非五三转录结构中的任一种情形,或者属于相邻基因并且相邻基因中的两个基因所分别对应的经与参考转录组比对所获得的支持簇读长方向为相对的,则去除该基因组合。将经留下来的基因组合列表中的各个基因组合所对应的读长标识(readsid)提取成support.ids文件。
在框1214处,计算设备130将融合支持簇对应在全转录组参考序列上的位置的覆盖区域按相邻预定间距范围进行合并以便生成合并区域,以及对合并区域进行过滤。例如,计算设备130可以将框1212处获得的各个基因组合对的support.ids文件进行minimap与单基因对应最常见转录本的tanscript.fa(例如,hg19参考转录组)进行比对,以便获得sam文件,然后将sam文件中的读长(reads)的区域分布进行合并,以便形成合并区域(-d500)。然后,过滤合并区域的数量小于或者等于6的基因组合对。
在框1216处,计算设备130如果确定不同类型的所述成对的读长在全基因组参考序列上对应的区域存在重合的融合区域,留下所述潜在的融合基因。例如,将框1214处获得的(留下的)各基因组合对的转录本比对sam文件进行其各合并区域内read的比对开始位置多样性的计算,统计含有大于或者等于2个多样性的区域的个数,过滤这些合并区域总数里没有一个符合前面条件的基因组合对。
在框1218处,计算设备130采集所留下所述潜在的基因融合的igv图像,以便基于所述igv图像确定参与融合的外显子(exon)号读取融合基因的亚型。例如,计算设备130基于框1216处获得的过滤后的基因组合对的bam和对应的转录本exon结构信息,还有区间信息,load进igv,以便进行igv快照(snapshot)。然后,基于拍到的图片中读长的特征,最终根据基因融合的方式(pattern)判定出真融合的基因组合对。通过采用上述手段,本公开能够通过拍照直观有效地根据阳性rna融合的支持读长方式(supportreadspattern)来确认基因融合。
图13示意性示出了适于用来实现本公开实施例的电子设备1300的框图。设备1300可以是用于实现执行图2至6所示的方法200-500、700、900和1200的设备。如图7所示,设备1300包括中央处理单元(cpu)1301,其可以根据存储在只读存储器(rom)1302中的计算机程序指令或者从存储单元1308加载到随机访问存储器(ram)1303中的计算机程序指令,来执行各种适当的动作和处理。在ram1303中,还可存储设备1300操作所需的各种程序和数据。cpu1301、rom1302以及ram1303通过总线1304彼此相连。输入/输出(i/o)接口1305也连接至总线1304。
设备1300中的多个部件连接至i/o接口1305,包括:输入单元1306、输出单元1307、存储单元1308,处理单元1301执行上文所描述的各个方法和处理,例如执行方法200-500、700、900和1200。例如,在一些实施例中,方法200-500、700、900和1200可被实现为计算机软件程序,其被存储于机器可读介质,例如存储单元1308。在一些实施例中,计算机程序的部分或者全部可以经由rom1302和/或通信单元1309而被载入和/或安装到设备1300上。当计算机程序加载到ram1303并由cpu1301执行时,可以执行上文描述的方法200-500、700、900和1200的一个或多个操作。备选地,在其他实施例中,cpu1301可以通过其他任何适当的方式(例如,借助于固件)而被配置为执行方法200-500、700、900和1200的一个或多个动作。
需要进一步说明的是,本公开可以是方法、装置、系统和/或计算机程序产品。计算机程序产品可以包括计算机可读存储介质,其上载有用于执行本公开的各个方面的计算机可读程序指令。
计算机可读存储介质可以是可以保持和存储由指令执行设备使用的指令的有形设备。计算机可读存储介质例如可以是但不限于电存储设备、磁存储设备、光存储设备、电磁存储设备、半导体存储设备或者上述的任意合适的组合。计算机可读存储介质的更具体的例子(非穷举的列表)包括:便携式计算机盘、硬盘、随机存取存储器(ram)、只读存储器(rom)、可擦式可编程只读存储器(eprom或闪存)、静态随机存取存储器(sram)、便携式压缩盘只读存储器(cd-rom)、数字多功能盘(dvd)、记忆棒、软盘、机械编码设备、例如其上存储有指令的打孔卡或凹槽内凸起结构、以及上述的任意合适的组合。这里所使用的计算机可读存储介质不被解释为瞬时信号本身,诸如无线电波或者其他自由传播的电磁波、通过波导或其他传输媒介传播的电磁波(例如,通过光纤电缆的光脉冲)、或者通过电线传输的电信号。
这里所描述的计算机可读程序指令可以从计算机可读存储介质下载到各个计算/处理设备,或者通过网络、例如因特网、局域网、广域网和/或无线网下载到外部计算机或外部存储设备。网络可以包括铜传输电缆、光纤传输、无线传输、路由器、防火墙、交换机、网关计算机和/或边缘服务器。每个计算/处理设备中的网络适配卡或者网络接口从网络接收计算机可读程序指令,并转发该计算机可读程序指令,以供存储在各个计算/处理设备中的计算机可读存储介质中。
用于执行本公开操作的计算机程序指令可以是汇编指令、指令集架构(isa)指令、机器指令、机器相关指令、微代码、固件指令、状态设置数据、或者以一种或多种编程语言的任意组合编写的源代码或目标代码,该编程语言包括面向对象的编程语言—诸如smalltalk、c++等,以及常规的过程式编程语言—诸如“c”语言或类似的编程语言。计算机可读程序指令可以完全地在用户计算机上执行、部分地在用户计算机上执行、作为一个独立的软件包执行、部分在用户计算机上部分在远程计算机上执行、或者完全在远程计算机或服务器上执行。在涉及远程计算机的情形中,远程计算机可以通过任意种类的网络—包括局域网(lan)或广域网(wan)—连接到用户计算机,或者,可以连接到外部计算机(例如利用因特网服务提供商来通过因特网连接)。在一些实施例中,通过利用计算机可读程序指令的状态信息来个性化定制电子电路,例如可编程逻辑电路、现场可编程门阵列(fpga)或可编程逻辑阵列(pla),该电子电路可以执行计算机可读程序指令,从而实现本公开的各个方面。
这里参照根据本公开实施例的方法、设备(系统)、和计算机程序产品的流程图和/或框图描述了本公开的各个方面。应当理解,流程图和/或框图的每个方框以及流程图和/或框图中各方框的组合,都可以由计算机可读程序指令实现。
这些计算机可读程序指令可以提供给语音交互装置中的处理器、通用计算机、专用计算机或其它可编程数据处理装置的处理单元,从而生产出一种机器,使得这些指令在通过计算机或其它可编程数据处理装置的处理单元执行时,产生了实现流程图和/或框图中的一个或多个方框中规定的功能/动作的装置。也可以把这些计算机可读程序指令存储在计算机可读存储介质中,这些指令使得计算机、可编程数据处理装置和/或其他设备以特定方式工作,从而,存储有指令的计算机可读介质则包括一个制造品,其包括实现流程图和/或框图中的一个或多个方框中规定的功能/动作的各个方面的指令。
也可以把计算机可读程序指令加载到计算机、其它可编程数据处理装置、或其它设备上,使得在计算机、其它可编程数据处理装置或其它设备上执行一序列操作步骤,以产生计算机实现的过程,从而使得在计算机、其它可编程数据处理装置、或其它设备上执行的指令实现流程图和/或框图中的一个或多个方框中规定的功能/动作。
附图中的流程图和框图显示了根据本公开的多个实施例的设备、方法和计算机程序产品的可能实现的体系架构、功能和操作。在这点上,流程图或框图中的每个方框可以代表一个模块、程序段或指令的一部分,该模块、程序段或指令的一部分包含一个或多个用于实现规定的逻辑功能的可执行指令。在有些作为替换的实现中,方框中所标注的功能也可以以不同于附图中所标注的顺序发生。例如,两个连续的方框实际上可以基本并行地执行,它们有时也可以按相反的顺序执行,这依所涉及的功能而定。也要注意的是,框图和/或流程图中的每个方框、以及框图和/或流程图中的方框的组合,可以用执行规定的功能或动作的专用的基于硬件的系统来实现,或者可以用专用硬件与计算机指令的组合来实现。
以上已经描述了本公开的各实施例,上述说明是示例性的,并非穷尽性的,并且也不限于所披露的各实施例。在不偏离所说明的各实施例的范围和精神的情况下,对于本技术领域的普通技术人员来说许多修改和变更都是显而易见的。本文中所用术语的选择,旨在最好地解释各实施例的原理、实际应用或对市场中的技术改进,或者使本技术领域的其它普通技术人员能理解本文披露的各实施例。
以上该仅为本公开的可选实施例,并不用于限制本公开,对于本领域的技术人员来说,本公开可以有各种更改和变化。凡在本公开的精神和原则之内,所作的任何修改、等效替换、改进等,均应包含在本公开的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!