抗‑Siglec‑15抗体的制作方法
技术领域
本发明涉及可用作骨代谢异常的治疗剂和/或预防剂的物质以及治疗和/或预防骨代谢异常的方法。
背景技术:
已知骨骼为动态性器官,其通过重复形成和吸收,进行持续重建,从而改变其本身的形态并维持血钙水平。健康的骨骼维持由成骨细胞进行的骨形成和由破骨细胞进行的骨吸收之间的平衡关系,从而骨量保持恒定。然而,当骨形成和骨吸收之间的平衡被破坏时,则产生诸如骨质疏松症等骨代谢异常(WO 07/093042和Endocrinological Review, (1992) 13, 第66-80页)。
作为调节骨代谢的因子,报道了很多全身性激素以及局部性细胞因子,且这些因子互相合作以形成和维持骨(WO 07/093042和Endocrinological Review, (1996) 17, 第308-332页)。作为因老龄化导致的骨组织的变化,骨质疏松症的发病众所周知,但是其发病机理包含各种因素,如性激素的分泌降低以及该激素受体异常、骨局部细胞因子表达变动、老化基因的表达、破骨细胞或成骨细胞的分化衰退或功能不全,因此,难以将它理解为单纯的年龄相关的生理现象。原发性骨质疏松症主要分为由雌激素分泌降低导致的绝经后骨质疏松症和由老龄化导致的老年性骨质疏松症,为了阐明其发病机理和开发针对它的治疗剂,在骨形成和骨吸收的调节机制方面的基础研究的进展很重要。
破骨细胞是源自造血干细胞的多核细胞,通过在破骨细胞粘附的骨表面上释放氯离子和氢离子,破骨细胞使骨表面与破骨细胞之间的间隙成为酸性,并且还分泌作为酸性蛋白酶的组织蛋白酶K等(American Journal of Physiology, (1991) 260, C1315-C1324)。这引发磷酸钙的降解、酸性蛋白酶的活化以及骨基质蛋白的降解,导致骨吸收。
业已发现破骨细胞前体细胞通过存在于骨表面上的成骨细胞/基质细胞的细胞膜上表达的 RNAKL (NF-κB配体的受体活化因子)的刺激,分化为破骨细胞(Proceedings of the National Academy of Science of the United States of America,(1998)95, 第3597-3602页, 和Cell,(1998)93, 第165-176页)。已发现RNAKL 是成骨细胞/基质细胞所产生的膜蛋白,其表达受骨吸收因子的调节,RNAKL 诱导破骨细胞前体细胞向成熟的多核破骨细胞的分化等(Proceedings of the National Academy of Science of the United States of America,(1998)95, 第3597-3602页, 和Journal of Bone and Mineral Research,(1998)23,S222)。此外,已经发现敲除 RNAKL 的小鼠发生(develop)骨硬化病样疾病,因此,已经证明RNAKL是生理性的破骨细胞分化诱导因子(Nature,(1999)397, 第315-323页)。
使用二膦酸盐、活性维生素D3、降钙素及其衍生物、激素例如雌二醇、SERM(选择性雌激素受体调节剂)、依普黄酮、维生素K2(四烯甲萘醌)、PTH和钙制剂等作为治疗骨代谢疾病或缩短治疗时间的药物制剂。但是,这些药物不会总是表现出令人满意的治疗效果,已经需要开发具有更有效的治疗效果的药物。
免疫细胞的细胞膜上被多种聚糖(诸如唾液酸化的聚糖)高度覆盖,所述聚糖被各种聚糖结合蛋白识别。结合唾液酸的免疫球蛋白样凝集素(在下文中称为“Siglecs”)为识别并结合唾液酸化的聚糖的I型膜蛋白家族。许多siglecs在免疫细胞的细胞膜上表达,并识别类似地存在于免疫细胞的细胞膜上的唾液酸,调节细胞相互作用或细胞功能,并被认为参与免疫应答(Nature Reviews Immunology,(2007)7, 第255-266页)。然而,也有很多尚未阐明其生理功能的siglec分子。最近报道Siglec-15(结合唾液酸的免疫球蛋白样凝集素15)为属于Siglecs的分子(参见,例如,非专利文件10),其与被称为CD33L3(CD33分子样3)的分子相同。该分子从鱼类到人类是进化高度保守的,业已发现在人脾和淋巴结的树突细胞和/或巨噬细胞中强表达。此外,使用唾液酸探针的结合测试的结果是:已经发现人Siglec-15结合Neu5Acα2-6GalNAc,小鼠Siglec-15进一步结合Neu5Acα2-3Galβ1-4Glc等(参见,例如,Glycobiology, (2007) 17, 第838-846页)。直至最近,还未揭示Siglec-15的生理作用,但业已报道,随着破骨细胞的分化和成熟,Siglec-15的表达增加,通过用RNA干扰使Siglec-15的表达下降,可以抑制破骨细胞的分化(参见,例如,WO 07/093042)。此外,在WO 09/48072 (2009年4月16日公开)中已经首次披露了抗-Siglec-15抗体对破骨细胞分化的作用,但是,尚未阐明可以施用给人类的抗体序列。
技术实现要素:
发明所要解决的问题
本发明的目的在于提供:在各种形式的骨代谢异常(可见为骨质疏松症、类风湿性关节炎、癌的骨转移等)中特异表达的基因;抑制破骨细胞的分化和成熟及其活性的物质;以及骨代谢异常的治疗剂和/或预防剂。
解决问题的手段
为了找到对骨代谢异常具有治疗和/或预防效果的物质,本发明人以阐明破骨细胞的分化、成熟和活化机制为目标进行了研究。结果本发明人发现随着破骨细胞的分化和成熟,Siglec-15基因的表达增加,还发现特异性地结合Siglec-15的抗体可以抑制破骨细胞的分化。此外,发明人人源化了得到的大鼠抗-小鼠Siglec-15抗体从而完成了本发明。
即,本发明包括以下发明。
(1) 一种抗体或所述抗体的功能片段,所述抗体结合包含由SEQ ID NO: 2表示的氨基酸序列的氨基酸残基39-165的多肽,并抑制破骨细胞形成和/或破骨性骨吸收。
(2) 根据(1)所述的抗体或所述抗体的功能片段,其特征在于:
重链序列含有具有CDRH1、CDRH2和CDRH3的可变区,且所述CDRH1包含由SEQ ID NO: 44表示的氨基酸序列,所述CDRH2包含由SEQ ID NO: 45和SEQ ID NO: 97表示的任一个氨基酸序列,所述CDRH3包含由SEQ ID NO: 46表示的氨基酸序列;且
轻链序列含有具有CDRL1、CDRL2和CDRL3的可变区,且所述CDRL1包含由SEQ ID NO: 47表示的氨基酸序列,所述CDRL2包含由SEQ ID NO: 48表示的氨基酸序列,且所述CDRL3包含由SEQ ID NO: 49表示的氨基酸序列。
(3) 根据(2)所述的抗体或所述抗体的功能片段,其特征在于含有:包含由SEQ ID NO: 41表示的氨基酸序列的氨基酸残基20-140的重链可变区序列和包含由SEQ ID NO: 43表示的氨基酸序列的氨基酸残基21-132的轻链可变区序列。
(4) 根据(1)至(3)中任一项所述的抗体功能片段,其选自:Fab、F(ab’)2、Fab’和Fv。
(5) 根据(1)至(3)中任一项所述的抗体,其特征在于是scFv。
(6) 根据(1)至(4)中任一项所述的抗体或所述抗体的功能片段,其特征在于所述抗体是嵌合抗体。
(7) 根据(6)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 41表示的氨基酸序列的氨基酸残基20-470,且所述轻链序列包含由SEQ ID NO: 43表示的氨基酸序列的氨基酸残基21-237。
(8) 根据(1)至(4)和(6)中任一项所述的抗体或所述抗体的功能片段,其特征在于所述抗体是人源化的。
(9) 根据(7)或(8)所述的抗体,其中所述重链具有人免疫球蛋白G2重链的恒定区,且所述轻链具有人免疫球蛋白κ轻链的恒定区。
(10) 一种抑制破骨细胞形成和/或破骨性骨吸收的抗体或所述抗体的功能片段,其中所述抗体含有:
(a) 选自下述氨基酸序列的重链可变区:
a1) 一种氨基酸序列,其包含由SEQ ID NO: 51表示的氨基酸序列的氨基酸残基20-140;
a2) 一种氨基酸序列,其包含由SEQ ID NO: 53表示的氨基酸序列的氨基酸残基20-140;
a3) 一种氨基酸序列,其包含由SEQ ID NO: 55表示的氨基酸序列的氨基酸残基20-140;
a4) 一种氨基酸序列,其包含由SEQ ID NO: 57表示的氨基酸序列的氨基酸残基20-140;
a5) 一种氨基酸序列,其包含由SEQ ID NO: 59表示的氨基酸序列的氨基酸残基20-140;
a6) 一种氨基酸序列,其包含由SEQ ID NO: 99表示的氨基酸序列的氨基酸残基20-140;
a7) 一种氨基酸序列,其包含由SEQ ID NO: 101表示的氨基酸序列的氨基酸残基20-140;
a8) 一种氨基酸序列,其与选自a1)至a7)的任一个氨基酸序列具有至少95%同源性;
a9) 一种氨基酸序列,其与选自a1)至a7)的任一个氨基酸序列具有至少99%同源性;和
a10) 一种氨基酸序列,其包含在选自a1)至a7)的任一个氨基酸序列中的一个至几个氨基酸残基的置换、删除或添加;和
(b) 选自下述氨基酸序列的轻链可变区:
b1) 一种氨基酸序列,其包含由SEQ ID NO: 61表示的氨基酸序列的氨基酸残基21-133;
b2) 一种氨基酸序列,其包含由SEQ ID NO: 63表示的氨基酸序列的氨基酸残基21-133;
b3) 一种氨基酸序列,其包含由SEQ ID NO: 65表示的氨基酸序列的氨基酸残基21-133;
b4) 一种氨基酸序列,其包含由SEQ ID NO: 67表示的氨基酸序列的氨基酸残基21-132;
b5) 一种氨基酸序列,其包含由SEQ ID NO: 69表示的氨基酸序列的氨基酸残基21-133;
b6) 一种氨基酸序列,其包含由SEQ ID NO: 71表示的氨基酸序列的氨基酸残基21-133;
b7) 一种氨基酸序列,其包含由SEQ ID NO: 103表示的氨基酸序列的氨基酸残基21-133;
b8) 一种氨基酸序列,其包含由SEQ ID NO: 105表示的氨基酸序列的氨基酸残基21-133;
b9) 一种氨基酸序列,其与选自b1)至b8)的任一个氨基酸序列具有至少95%同源性;
b10) 一种氨基酸序列,其与选自b1)至b8)的任一个氨基酸序列具有至少99%同源性;和
b11) 一种氨基酸序列,其包含在选自b1)至b8)的任一个氨基酸序列中的一个至几个氨基酸残基的置换、删除或添加。
(11) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 51表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 61表示的氨基酸序列的氨基酸残基21-133。
(12) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 53表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 63表示的氨基酸序列的氨基酸残基21-133。
(13) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 55表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 65表示的氨基酸序列的氨基酸残基21-133。
(14) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 55表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 67表示的氨基酸序列的氨基酸残基21-132。
(15) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 57表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 69表示的氨基酸序列的氨基酸残基21-133。
(16) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 59表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 71表示的氨基酸序列的氨基酸残基21-133。
(17) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 99表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 69表示的氨基酸序列的氨基酸残基21-133。
(18) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 101表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 69表示的氨基酸序列的氨基酸残基21-133。
(19) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 99表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 103表示的氨基酸序列的氨基酸残基21-133。
(20) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列含有重链可变区,所述重链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 99表示的氨基酸序列的氨基酸残基20-140,且所述轻链序列含有轻链可变区,所述轻链可变区包含这样的氨基酸序列,其包含由SEQ ID NO: 105表示的氨基酸序列的氨基酸残基21-133。
(21) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 51表示的氨基酸序列的氨基酸残基20-470,且所述轻链序列包含由SEQ ID NO: 61表示的氨基酸序列的氨基酸残基21-238。
(22) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 53表示的氨基酸序列的氨基酸残基20-470,且所述轻链序列包含由SEQ ID NO: 63表示的氨基酸序列的氨基酸残基21-238。
(23) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 55表示的氨基酸序列的氨基酸残基20-470,且所述轻链序列包含由SEQ ID NO: 65表示的氨基酸序列的氨基酸残基21-238。
(24) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 55表示的氨基酸序列的氨基酸残基20-470,且所述轻链序列包含由SEQ ID NO: 67表示的氨基酸序列的氨基酸残基21-237。
(25) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 57表示的氨基酸序列的氨基酸残基20-470,且所述轻链序列包含由SEQ ID NO: 69表示的氨基酸序列的氨基酸残基21-238。
(26) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 59表示的氨基酸序列的氨基酸残基20-470,且所述轻链序列包含由SEQ ID NO: 71表示的氨基酸序列的氨基酸残基21-238。
(27) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 99表示的氨基酸序列的氨基酸残基20-466,且所述轻链序列包含由SEQ ID NO: 69表示的氨基酸序列的氨基酸残基21-238。
(28) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 101表示的氨基酸序列的氨基酸残基20-466,且所述轻链序列包含由SEQ ID NO: 69表示的氨基酸序列的氨基酸残基21-238。
(29) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 99表示的氨基酸序列的氨基酸残基20-466,且所述轻链序列包含由SEQ ID NO: 103表示的氨基酸序列的氨基酸残基21-238。
(30) 根据(10)所述的抗体或所述抗体的功能片段,其特征在于含有重链序列和轻链序列,所述重链序列包含由SEQ ID NO: 99表示的氨基酸序列的氨基酸残基20-466,且所述轻链序列包含由SEQ ID NO: 105表示的氨基酸序列的氨基酸残基21-238。
(31) 一种药物组合物,其特征在于包含至少一种根据(1)至(30)所述的抗体或所述抗体的功能片段。
(32) 根据(31)所述的药物组合物,其特征在于是骨代谢异常的治疗剂和/或预防剂。
(33) 一种用于治疗和/或预防骨代谢异常的药物组合物,其特征在于包含至少一种根据(1)至(30)所述的抗体或所述抗体的功能片段和至少一个选自下述的成员:二膦酸盐、活性维生素D3、降钙素及其衍生物、激素例如雌二醇、SERM (选择性雌激素受体调节剂)、依普黄酮、维生素K2 (四烯甲萘醌)、钙制剂、PTH (甲状旁腺激素)、非甾体类抗炎药、可溶性TNF受体、抗-TNF-α抗体或所述抗体的功能片段、抗-PTHrP (甲状旁腺激素相关蛋白)抗体或所述抗体的功能片段、IL-1 受体拮抗剂、抗-IL-6 受体抗体或所述抗体的功能片段、抗-RANKL抗体或所述抗体的功能片段以及OCIF (破骨细胞生成抑制因子)。
(34) 根据(32)或(33)所述的药物组合物,其中所述骨代谢异常选自:骨质疏松症、伴随类风湿性关节炎的骨破坏、癌性高钙血症、伴随多发性骨髓瘤或癌的骨转移的骨破坏、巨细胞瘤、骨质减少、牙周炎导致的牙缺失、人工关节周围的骨溶解、慢性骨髓炎中的骨破坏、骨佩吉特病、肾性骨营养不良和成骨不全。
(35) 根据(34)所述的药物组合物,其特征在于所述骨代谢异常是骨质疏松症、伴随类风湿性关节炎的骨破坏、或伴随癌的骨转移的骨破坏。
(36) 根据(35)所述的药物组合物,其特征在于所述骨代谢异常是骨质疏松症。
(37) 根据(36)所述的药物组合物,其特征在于所述骨质疏松症是绝经后骨质疏松症、老年性骨质疏松症、由使用诸如类固醇或免疫抑制剂等治疗剂导致的继发性骨质疏松症、或伴随类风湿性关节炎的骨质疏松症。
(38) 一种治疗和/或预防骨代谢异常的方法,其特征在于施用至少一种根据(1)至(30)所述的抗体或所述抗体的功能片段。
(39) 一种治疗和/或预防骨代谢异常的方法,其特征在于同时或相继施用至少一种根据(1)至(30)所述的抗体或所述抗体的功能片段和至少一个选自下述的成员:二膦酸盐、活性维生素D3、降钙素及其衍生物、激素例如雌二醇、SERM (选择性雌激素受体调节剂)、依普黄酮、维生素K2 (四烯甲萘醌)、钙制剂、PTH (甲状旁腺激素)、非甾体类抗炎药、可溶性TNF受体、抗-TNF-α抗体或所述抗体的功能片段、抗-PTHrP (甲状旁腺激素相关蛋白)抗体或所述抗体的功能片段、IL-1 受体拮抗剂、抗-IL-6 受体抗体或所述抗体的功能片段、抗-RANKL抗体或所述抗体的功能片段以及OCIF (破骨细胞生成抑制因子)。
(40) 根据(38)或(39)所述的治疗和/或预防方法,其特征在于所述骨代谢异常是骨质疏松症、伴随类风湿性关节炎的骨破坏、或伴随癌的骨转移的骨破坏。
(41) 根据(40)所述的治疗和/或预防方法,其特征在于所述骨代谢异常是骨质疏松症。
(42) 根据(41)所述的治疗和/或预防方法,其特征在于所述骨质疏松症是绝经后骨质疏松症、老年性骨质疏松症、由使用诸如类固醇或免疫抑制剂等治疗剂导致的继发性骨质疏松症、或伴随类风湿性关节炎的骨质疏松症。
(43) 一种多核苷酸,其编码根据(3)、(7)和(10)至(30)中任一项所述的抗体。
(44) 根据(43)所述的多核苷酸,其特征在于含有:包含由SEQ ID NO: 40表示的核苷酸序列的核苷酸58-420的核苷酸序列,和包含由SEQ ID NO: 42表示的核苷酸序列的核苷酸61-396的核苷酸序列。
(45) 根据(44)所述的多核苷酸,其特征在于含有:包含由SEQ ID NO: 40表示的核苷酸序列的核苷酸58-1410的核苷酸序列,和包含由SEQ ID NO: 42表示的核苷酸序列的核苷酸61-711的核苷酸序列。
(46) 根据(43)所述的多核苷酸,其特征在于含有:
(a) 一种多核苷酸,其选自下述的核苷酸序列:
a1) 一种核苷酸序列,其包含由SEQ ID NO: 50表示的核苷酸序列的核苷酸58-420;
a2) 一种核苷酸序列,其包含由SEQ ID NO: 52表示的核苷酸序列的核苷酸58-420;
a3) 一种核苷酸序列,其包含由SEQ ID NO: 54表示的核苷酸序列的核苷酸58-420;
a4) 一种核苷酸序列,其包含由SEQ ID NO: 56表示的核苷酸序列的核苷酸58-420;
a5) 一种核苷酸序列,其包含由SEQ ID NO: 58表示的核苷酸序列的核苷酸58-420;
a6) 一种核苷酸序列,其包含由SEQ ID NO: 98表示的核苷酸序列的核苷酸58-420;
a7) 一种核苷酸序列,其包含由SEQ ID NO: 100表示的核苷酸序列的核苷酸58-420;
a8) 多核苷酸的核苷酸序列,所述多核苷酸在严格条件下与包含选自a1)至a7)的任一个核苷酸序列的互补核苷酸序列的多核苷酸杂交;和
a9) 一种核苷酸序列,其包含在选自a1)至a7)的任一个核苷酸序列中的一个至几个核苷酸的置换、删除或添加;和
(b) 一种多核苷酸,其选自下述的核苷酸序列:
b1) 一种核苷酸序列,其包含由SEQ ID NO: 60表示的核苷酸序列的核苷酸61-399;
b2) 一种核苷酸序列,其包含由SEQ ID NO: 62表示的核苷酸序列的核苷酸61-399;
b3) 一种核苷酸序列,其包含由SEQ ID NO: 64表示的核苷酸序列的核苷酸61-399;
b4) 一种核苷酸序列,其包含由SEQ ID NO: 66表示的核苷酸序列的核苷酸61-396;
b5) 一种核苷酸序列,其包含由SEQ ID NO: 68表示的核苷酸序列的核苷酸61-399;
b6) 一种核苷酸序列,其包含由SEQ ID NO: 70表示的核苷酸序列的核苷酸61-399;
b7) 一种核苷酸序列,其包含由SEQ ID NO: 102表示的核苷酸序列的核苷酸61-399;
b8) 一种核苷酸序列,其包含由SEQ ID NO: 104表示的核苷酸序列的核苷酸61-399;
b9) 多核苷酸的核苷酸序列,所述多核苷酸在严格条件下与包含选自b1)至b8)的任一个核苷酸序列的互补核苷酸序列的多核苷酸杂交;和
b10) 一种核苷酸序列,其包含在选自b1)至b8)的任一个核苷酸序列中的一个至几个核苷酸的置换、删除或添加。
(47) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 50表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 60表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(48) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 52表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 62表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(49) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 54表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 64表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(50) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 54表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 66表示的核苷酸序列的核苷酸61-396的核苷酸序列。
(51) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 56表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 68表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(52) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 58表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 70表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(53) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 98表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 68表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(54) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 100表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 68表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(55) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 98表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 102表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(56) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 98表示的核苷酸序列的核苷酸58-420的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 104表示的核苷酸序列的核苷酸61-399的核苷酸序列。
(57) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 50表示的核苷酸序列的核苷酸58-1410的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 60表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(58) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 52表示的核苷酸序列的核苷酸58-1410的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 62表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(59) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 54表示的核苷酸序列的核苷酸58-1410的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 64表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(60) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 54表示的核苷酸序列的核苷酸58-1410的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 66表示的核苷酸序列的核苷酸61-711的核苷酸序列。
(61) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 56表示的核苷酸序列的核苷酸58-1410的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 68表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(62) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 58表示的核苷酸序列的核苷酸58-1410的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 70表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(63) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 98表示的核苷酸序列的核苷酸58-1398的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 68表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(64) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 100表示的核苷酸序列的核苷酸58-1398的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 68表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(65) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 98表示的核苷酸序列的核苷酸58-1398的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 102表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(66) 根据(46)所述的多核苷酸,其特征在于含有:这样的多核苷酸,其含有包含由SEQ ID NO: 98表示的核苷酸序列的核苷酸58-1398的核苷酸序列;和这样的多核苷酸,其含有包含由SEQ ID NO: 104表示的核苷酸序列的核苷酸61-714的核苷酸序列。
(67) 一种载体,其包含根据(43)至(66)所述的任一种多核苷酸。
(68) 一种转化的宿主细胞,其包含根据(43)至(66)所述的任一种多核苷酸。
(69) 一种转化的宿主细胞,其包含根据(67)所述的载体。
(70) 一种生产根据(3)、(7)和(10)至(30)中任一项所述的抗体的方法,所述方法包括:培养根据(68)或(69)所述的宿主细胞,并从得到的培养产物纯化抗体。
发明优点
根据本发明,可以得到骨代谢异常的治疗剂和/或预防剂,其作用机制是抑制破骨细胞的分化和成熟及其骨吸收活性。
附图说明
图1 显示了通过SDS-聚丙烯酰胺电泳和银染色评估用HisTrap HP柱色谱法和Resource Q柱色谱法纯化的小鼠Siglec-15-His纯度的结果。
图2 显示了通过SDS-聚丙烯酰胺电泳和使用抗-V5-HRP抗体的蛋白印迹法检测用HisTrap HP柱色谱法和Resource Q柱色谱法纯化的小鼠Siglec-15-His 特征的结果。
图3 显示了通过SDS-聚丙烯酰胺电泳和银染色评估用HiTrap蛋白A柱色谱法纯化的小鼠Siglec-15-Fc纯度的结果。
图4 显示了通过ELISA法测试大鼠抗-小鼠Siglec-15 单克隆抗体结合其上固定有小鼠Siglec-15-Fc的板的结果。符号(◆)表示#1A1抗体,符号(■)表示#3A1抗体,符号(▲)表示#8A1抗体,符号(×)表示#24A1抗体,符号(●)表示#32A1抗体,符号(○)表示#34A1抗体,符号(+)表示#39A1抗体,符号(-)表示#40A1抗体,符号(—)表示#41B1抗体,符号(◇)表示#61A1抗体,且符号(□)表示对照IgG。
图5 显示了测试添加大鼠抗-小鼠Siglec-15 单克隆抗体(#3A1、#8A1或#32A1) 对小鼠骨髓非贴壁细胞向破骨细胞分化(用RANKL刺激)的影响的结果。此外,图中大鼠对照IgG是图5和6共有的阴性对照。
图6 显示了测试添加大鼠抗-小鼠Siglec-15 单克隆抗体(#34A1、#39A1或#40A1)对小鼠骨髓非贴壁细胞向破骨细胞分化(用RANKL刺激)的影响的结果。此外,图中兔抗-小鼠Siglec-15 多克隆抗体No.3是图5和6共有的阳性对照。
图7 显示了通过SDS-聚丙烯酰胺电泳和银染色评价用HisTrap HP柱色谱法和Resource Q柱色谱法纯化的人Siglec-15-His纯度的结果。
图8 显示了通过SDS-聚丙烯酰胺电泳评价用蛋白A柱色谱法纯化的人Siglec-15-Fc纯度的结果。
图9 显示了通过ELISA法测试大鼠抗-小鼠Siglec-15 单克隆抗体结合其上固定有人Siglec-15-Fc的板的结果。符号(◆)表示#1A1抗体,符号(■)表示#3A1抗体,符号(▲)表示#8A1抗体,符号(×)表示#24A1抗体,符号(●)表示#32A1抗体,符号(○)表示#34A1抗体,符号(+)表示#39A1抗体,符号(-)表示#40A1抗体,符号(—)表示#41B1抗体,符号(◇)表示#61A1抗体,且符号(□)表示对照IgG。
图10 显示了通过TRAP染色描绘的通过添加大鼠抗-小鼠Siglec-15 单克隆抗体抑制从正常人破骨细胞前体细胞形成巨大破骨细胞的显微照片。
图11 显示了通过TRAP染色描绘的通过添加大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体)抑制从正常人破骨细胞前体细胞形成巨大破骨细胞的显微照片。
图12的图显示了通过添加大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体) 抑制正常人破骨细胞的骨吸收活性(N=6)。
图13A的图显示了当将大鼠抗-小鼠Siglec-15 单克隆抗体施用给卵巢切除的大鼠4周时,增加腰椎骨矿物质密度的效应;且图13B的图显示了当将大鼠抗-小鼠Siglec-15 单克隆抗体施用给卵巢切除的大鼠4周时,减少尿脱氧吡啶啉排泄的效应。
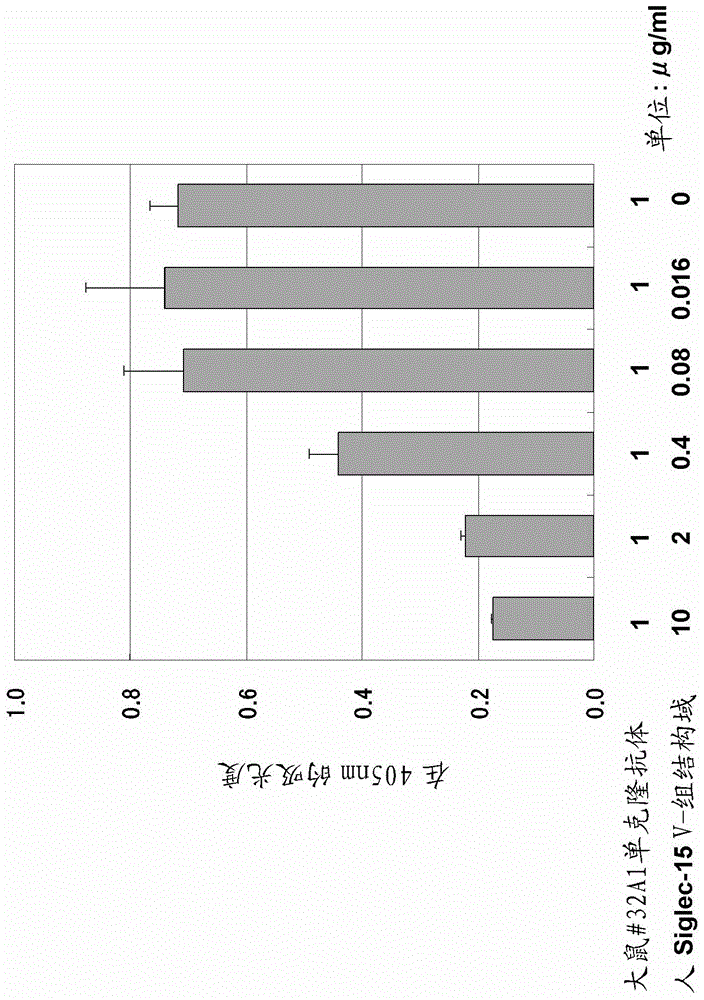
图14是通过竞争ELISA显示大鼠抗-小鼠Siglec-15 单克隆抗体#32A1结合人Siglec-15的V-组结构域的图。
图15是通过竞争ELISA显示大鼠#32A1抗体及其人嵌合抗体具有基本上相同的对小鼠Siglec-15-Fc的亲和力的图。
图16 显示了用TRAP酶活性描绘的通过添加大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体)及其嵌合抗体抑制小鼠破骨细胞形成的图(N=3)。
图17 显示了通过TRAP染色描绘的通过添加大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体抑制从正常人破骨细胞前体细胞形成巨大破骨细胞的显微照片。
图18是这样的图,其中使用其上面固定化有小鼠Siglec-15-Fc的板通过ELISA法证实了6类人源化的大鼠抗-小鼠Siglec-15抗体以抗体浓度依赖性的方式结合小鼠Siglec-15蛋白。
图19是这样的图,其中使用其上面固定化有人Siglec-15-Fc 的板通过ELISA法证实了6类人源化的大鼠抗-小鼠Siglec-15抗体以抗体浓度依赖性的方式结合人Siglec-15蛋白。
图20 显示了通过TRAP染色描绘的通过添加大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体)或其嵌合抗体抑制在TNFα刺激下形成小鼠巨大破骨细胞的显微照片。
图21 显示了用TRAP酶活性描绘的通过添加大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体)或其嵌合抗体抑制在TNFα刺激下形成小鼠破骨细胞的图(N=3)。
图22 显示了用TRAP酶活性描绘的通过添加大鼠抗-小鼠Siglec-15 单克隆抗体(#8A1或#32A1抗体)抑制大鼠破骨细胞形成的图(N=3)。
图23 显示了克隆的大鼠#32A1重链的核苷酸序列及其氨基酸序列。
图24 显示了克隆的大鼠#32A1轻链的核苷酸序列及其氨基酸序列。
图25 显示了#32A1人嵌合抗体重链的核苷酸序列及其氨基酸序列。
图26 显示了#32A1人嵌合抗体轻链的核苷酸序列及其氨基酸序列。
图27 显示了h#32A1-T1H的核苷酸序列及其氨基酸序列。
图28 显示了h#32A1-T2H的核苷酸序列及其氨基酸序列。
图29 显示了h#32A1-T3H的核苷酸序列及其氨基酸序列。
图30 显示了h#32A1-T5H的核苷酸序列及其氨基酸序列。
图31 显示了h#32A1-T6H的核苷酸序列及其氨基酸序列。
图32 显示了h#32A1-T1L的核苷酸序列及其氨基酸序列。
图33 显示了h#32A1-T2L的核苷酸序列及其氨基酸序列。
图34 显示了h#32A1-T3L的核苷酸序列及其氨基酸序列。
图35 显示了h#32A1-T4L的核苷酸序列及其氨基酸序列。
图36 显示了h#32A1-T5L的核苷酸序列及其氨基酸序列。
图37 显示了h#32A1-T6L的核苷酸序列及其氨基酸序列。
图38 显示了h#32A1-T7H、h#32A1-T8H和h#32A1-T9H的氨基酸序列。
图39 显示了h#32A1-T10H、h#32A1-T11H和h#32A1-T12H的氨基酸序列。
图40 显示了h#32A1-T7L、h#32A1-T8L、h#32A1-T9L、h#32A1-T10L和h#32A1-T11L的氨基酸序列。
图41 显示了h#32A1-T12L、h#32A1-T13L、h#32A1-T14L、h#32A1-T15L和h#32A1-T16L的氨基酸序列。
图42 显示了h#32A1-T17L、h#32A1-T18L、h#32A1-T19L、h#32A1-T20L和h#32A1-T21L的氨基酸序列。
图43 显示了h#32A1-T22L、h#32A1-T23L、h#32A1-T24L和h#32A1-T25L的氨基酸序列。
图44 显示了用TRAP酶活性描绘的通过添加人源化的大鼠抗-小鼠Siglec-15抗体(h#32A1-T1、h#32A1-T2或h#32A1-T3)抑制小鼠破骨细胞形成的图。此外,图中大鼠#32A1抗体是图44和45共有的阳性对照。
图45 显示了用TRAP酶活性描绘的通过添加人源化的大鼠抗-小鼠Siglec-15抗体(h#32A1-T4、h#32A1-T5或h#32A1-T6)抑制小鼠破骨细胞形成的图。
图46 显示了用TRAP酶活性描绘的通过添加人源化的大鼠抗-小鼠Siglec-15抗体(h#32A1-H1-1/L5或h#32A1-H5/L5)抑制小鼠破骨细胞形成的图。此外,图中大鼠#32A1抗体和#32A1人嵌合抗体是图46和47共有的阳性对照。
图47 显示了用TRAP酶活性描绘的通过添加人源化的大鼠抗-小鼠Siglec-15抗体(h#32A1-H1-1/L2-16或h#32A1-H-1/L2-15)抑制小鼠破骨细胞形成的图。
图48 显示的图描绘了通过添加人源化的大鼠抗-小鼠Siglec-15抗体(h#32A1-T1、h#32A1-T2、h#32A1-T3、h#32A1-T4、h#32A1-T5或h#32A1-T6)抑制正常人破骨细胞的骨吸收活性。此外,图中大鼠#32A1抗体和#32A1人嵌合抗体是图48和49共有的阳性对照。
图49 显示的图描绘了通过添加人源化的大鼠抗-小鼠Siglec-15抗体(h#32A1-H1-1/L5、h#32A1-H5/L5、h#32A1-H1-1/L2-16或h#32A1-H1-1/L2-15)抑制正常人破骨细胞的骨吸收活性。
图50是为了测定h#32A1-H5/L5抗体的热稳定性而得到的热分析图。
图51是为了测定h#32A1-H1-1/L5抗体的热稳定性而得到的热分析图。
图52是为了测定h#32A1-H1-1/L2-15抗体的热稳定性而得到的热分析图。
图53是为了测定h#32A1-H1-1/L2-16抗体的热稳定性而得到的热分析图。
图54 显示了h#32A1-H1-1的核苷酸序列及其氨基酸序列。
图55 显示了h#32A1-H5的核苷酸序列及其氨基酸序列。
图56 显示了h#32A1-L2-15的核苷酸序列及其氨基酸序列。
图57 显示了h#32A1-L2-16的核苷酸序列及其氨基酸序列。
具体实施方式
本文使用的术语“基因”不仅包括DNA,还包括mRNA、cDNA以及cRNA。
本文使用的术语“多核苷酸”用于与核酸相同的含义,并且还包括DNA、RNA、探针、寡核苷酸以及引物。
本文使用的术语“多肽”和“蛋白”无区别使用。
本文使用的术语“RNA级分”是指包含RNA的级分。
本文使用的术语“细胞”也包括动物个体内的细胞和培养的细胞。
本文使用的术语“Siglec-15”以与“Siglec-15蛋白”相同的含义使用。
本文使用的术语“破骨细胞形成”以与“破骨细胞分化”或“破骨细胞成熟”相同的含义使用。
本文使用的术语“抗体功能片段”是指具有抗原结合活性的抗体的一部分片段,包括Fab、F(ab’)2、Fv、scFv、双特异抗体、线性抗体、从抗体片段形成的多特异性抗体等。该术语也包括Fab’,其为在还原条件下处理F(ab’)2 得到的抗体的可变区的单价片段。但是,该术语不限于这些分子,只要所述片段具有与抗原的结合亲和力。此外,这些功能片段不仅包括用适当的酶处理抗体蛋白的全长分子得到的片段,还包括使用遗传修饰的抗体基因在适当宿主细胞中产生的蛋白。
本文使用的术语“表位”是指特定的抗-Siglec-15抗体结合的Siglec-15的部分肽或部分三级结构。通过诸如免疫测定法等本领域技术人员熟知的方法,可以确定作为Siglec-15的部分肽的上述表位。但是,例如,可以采用以下的方法。制备Siglec-15的各种部分结构。在制备部分结构时,可使用已知的寡肽合成技术。例如,使用本领域技术人员已知的基因重组技术,制备从Siglec-15的C末端或N末端顺次缩短所得到的具有适当的缩短长度的一系列多肽。此后,检测这些多肽的抗体的反应性,并粗略确定识别部位。然后,合成长度更短的肽,并检测与这些肽的反应性,由此可确定表位。此外,通过X-射线结构分析描述邻近抗体的Siglec-15的氨基酸残基,可以确定作为Siglec-15 的部分三级结构的表位(其结合特定的Siglec-15抗体)。如果第二抗-Siglec-15抗体结合第一抗-Siglec-15抗体所结合的部分肽或部分三级结构,可以确定第一抗体与第二抗体具有相同的表位。此外,通过证实第二抗-Siglec-15抗体与第一抗-Siglec-15抗体竞争结合Siglec-15(即第二抗体抑制Siglec-15和第一抗体之间的结合),可以确定第一抗体和第二抗体具有相同的表位,即使尚未确定特定表位的序列或结构。此外,当第一抗体和第二抗体结合相同的表位,且第一抗体具有诸如抗原中和活性等特殊效应时,可以预期第二抗体具有相同的活性。
已知抗体分子的每个重链和轻链具有3个互补性决定区 (CDR)。互补性决定区也称作超变结构域,且存在于抗体的每个重链和轻链的可变区中。它是在它的一级结构中具有非常高的可变性的位点,且在每个重和轻多肽链的一级结构中存在3个单独的CDR。在本说明书中,关于抗体的互补性决定区,重链的互补性决定区由来自重链氨基酸序列的氨基端侧的CDRH1、CDRH2和CDRH3表示,轻链的互补性决定区由来自轻链氨基酸序列的氨基端侧的CDRL1、CDRL2和CDRL3表示。这些位点在三级结构中彼此邻近,决定了抗体结合的抗原的特异性。
本文使用的短语“在严格条件下进行杂交”是指在可实现鉴定的条件下进行杂交,其中在市售的的杂交溶液ExpressHyb杂交溶液(由Clontech, Inc.生产)中,在68 ℃进行杂交,或者使用其上固定有DNA 的滤器,在0.7-1.0M NaCl存在下,在68℃进行杂交,然后使用0.1-2X的SSC溶液(1 x SSC溶液由150 mM NaCl和15 mM柠檬酸钠组成)在68℃进行洗涤,或在与其等同的条件下杂交。
1.Siglec-15
本发明人已经发现Siglec-15基因在巨细胞瘤中特异性表达,还已经发现当单核细胞-衍生的细胞系分化为破骨细胞时,Siglec-15基因的表达水平增加。
关于在本发明中所使用的Siglec-15,Siglec-15直接地从人、非人哺乳动物(例如豚鼠、大鼠、小鼠、兔、猪、绵羊、牛或猴)或鸡的单核细胞或骨髓细胞纯化和使用,或者制备上述细胞的细胞膜级分和使用。此外,通过体外合成Siglec-15或通过基因工程使其在宿主细胞内产生,可以得到Siglec-15。具体地,在基因工程生产中,将Siglec-15 cDNA整合到能表达Siglec-15 cDNA的载体中,然后在含有转录和翻译所需的酶、底物和能量物质的溶液中合成Siglec-15,或者转化另一种原核或真核宿主细胞,以表达Siglec-15,由此可得到该蛋白。
人Siglec-15 cDNA的核苷酸序列以登录号NM_213602登记在GenBank中,并表示为序列表的SEQ ID NO:1,其氨基酸序列以序列表的SEQ ID NO:2表示。小鼠Siglec-15 cDNA的核苷酸序列以登录号XM_884636登记在GenBank中,并表示为序列表的SEQ ID NO:3,其氨基酸序列以序列表的SEQ ID NO:4表示。已经去除了信号序列的成熟的人Siglec-15对应着包含由SEQ ID NO: 2表示的氨基酸序列的氨基酸残基21-328的氨基酸序列。此外,已经去除了信号序列的小鼠Siglec-15对应着包含由SEQ ID NO: 4表示的氨基酸序列的氨基酸残基21-341的氨基酸序列。此外,Siglec-15有时被称为CD33 抗原-样3、CD33分子-样3、CD33-样3或CD33L3,这些都表示相同的分子。
可以如下得到Siglec-15 cDNA:通过例如所谓的PCR 方法,其中使用表达Siglec-15 cDNA的cDNA文库作为模板,并使用特异性扩增Siglec-15 cDNA的引物,进行聚合酶链式反应(在下文中称作“PCR”) (Saiki, R. K., 等人, Science, (1988) 239, 487-49)。
此外,具有下述特征的多核苷酸也视作Siglec-15 cDNA:它在严格条件下与包含至少一个选自序列表中的SEQ ID NO: 1和3所表示的核苷酸序列的互补核苷酸序列的多核苷酸杂交,并且编码具有与Siglec-15相当的生物活性的蛋白。此外,具有下述特征的多核苷酸也视作Siglec-15 cDNA:它是从人或小鼠Siglec-15基因座转录的剪接变体,或与其互补序列在严格条件下杂交,且编码具有与Siglec-15相当的生物活性的蛋白。
此外,具有下述特征的蛋白也视作Siglec-15:它包含这样的氨基酸序列,所述序列包含在选自序列表的SEQ ID NO: 2和4表示的氨基酸序列的至少一个氨基酸序列中的一个或几个氨基酸的置换、删除或添加,或通过从这些序列中的任一个去除信号序列所得到的氨基酸序列,且具有与Siglec-15相当的生物活性。此外,具有下述特征的蛋白也视作Siglec-15:它包含由从人或小鼠Siglec-15基因座转录的剪接变体编码的氨基酸序列,或包含在其中的一个或几个氨基酸的置换、删除或添加的氨基酸序列,且具有与Siglec-15相当的生物活性。
2. 骨代谢异常的检测
对来自各种人骨组织的测试样品组中的Siglec-15基因表达水平的分析显示该基因在巨细胞瘤 (GCT) 中的表达水平显著增加,所述巨细胞瘤是破骨细胞样多核巨大细胞大量出现的骨肿瘤,且其特征是在临床上发现骨溶解性的骨破坏(Bullough 等,Atlas of Orthopedic Pathology 第2版, 第17.6-17.8页,Lippincott Williams & Wilkins Publishers(1992))。
还发现,当单核细胞-衍生的细胞系分化为破骨细胞时,Siglec-15基因的表达水平增加。
因此,认为Siglec-15与诸如GCT等其中骨吸收增加的人病理学有关。换而言之,通过测量Siglec-15基因和/或Siglec-15在各细胞和/或各组织中的表达水平,能够确定伴随Siglec-15的过表达的骨代谢异常状态。本文使用的骨代谢异常是指特征在于净骨流失的障碍,其具体实例包括但不限于:骨质疏松症 (绝经后骨质疏松症、老年性骨质疏松症、由使用诸如类固醇或免疫抑制剂等治疗剂导致的继发性骨质疏松症、或伴随类风湿性关节炎的骨质疏松症)、伴随类风湿性关节炎的骨破坏、癌性高钙血症、伴随多发性骨髓瘤或癌的骨转移的骨破坏、巨细胞瘤、骨质减少、牙周炎导致的牙缺失、人工关节周围的骨溶解、慢性骨髓炎中的骨破坏、骨佩吉特病、肾性骨营养不良和成骨不全。
在本发明中,用于检测Siglec-15基因和/或Siglec-15的表达水平的“测试样品”是指从测试受试者得到的下述组织的样品:骨髓、骨、前列腺、精巢、阴茎、膀胱、肾脏、口腔、咽、唇、舌、牙龈、鼻咽、食道、胃、小肠、大肠、结肠、肝脏、胆囊、胰脏、鼻、肺、软组织、皮肤、乳房、子宫、卵巢、脑、甲状腺、淋巴结、肌肉、脂肪组织等,或从测试受试者得到的血液、体液、排泄物等,临床样本等,但是,在本发明中,更优选血液或骨髓。
关于已知与破骨细胞分化有关的RANKL,已经生产了敲除的小鼠,并已经分析了当已经丧失RANKL功能时的表型 (Young-Yun Kong, 等人, Nature (1999) 397, 第315-323页)。通过以与上述相同的方式生产缺失Siglec-15的敲除的小鼠,可以分析当已经丧失Siglec-15功能时的表型。
3. 抗-Siglec-15抗体的制备
可以如下获得本发明的抗Siglec-15的抗体:根据常规的方法,用Siglec-15或选自Siglec-15的氨基酸序列的任意多肽免疫动物,并采集和纯化体内产生的抗体。用作抗原的Siglec-15的生物物种不限于人,也可以用从诸如小鼠或大鼠等除人以外的动物得到的Siglec-15免疫动物。在这种情况下,通过测试抗体与得到的异源 Siglec-15和人Siglec-15的结合之间的交叉反应性,可以选择可适用于人疾病的抗体。
此外,根据已知的方法(例如,Kohler和Milstein, Nature, (1975) 256, 第495-497页;Kennet,R.编,Monoclonal Antibody, 第365-367页, Prenum Press,N.Y.(1980)),通过使生产抗Siglec-15抗体的抗体生产细胞与骨髓瘤细胞融合,建立杂交瘤,可以得到单克隆抗体。
此外,可以如下得到要用作抗原的Siglec-15:通过基因工程,使宿主细胞生产Siglec-15基因。
具体而言,制备能表达Siglec-15基因的载体,将得到的载体转染进宿主细胞中,以表达所述基因,然后纯化表达的Siglec-15。在下文中具体描述了获得抗Siglec-15抗体的方法。
(1)抗原的制备
用于制备抗-Siglec-15抗体的抗原的实例包括Siglec-15,包含含有Siglec-15的至少6 个连续氨基酸的部分氨基酸序列的多肽,以及通过向其附加给定的氨基酸序列或载体而得到的衍生物。
Siglec-15可从人肿瘤组织或肿瘤细胞中直接纯化,并使用。此外,通过体外合成或通过基因工程造成宿主细胞生产Siglec-15,可以得到Siglec-15。
关于基因工程,具体地,将Siglec-15 cDNA整合到能表达Siglec-15 cDNA的载体中,然后在含有转录和翻译所需的酶、底物和能量物质的溶液中合成Siglec-15,或者转化另一种原核或真核宿主细胞,以表达Siglec-15,由此可得到该抗原。
此外,通过在适当的宿主-载体系统中表达融合蛋白,所述融合蛋白通过将作为膜蛋白的Siglec-15的胞外结构域连接到抗体的恒定区上而得到,也可以得到作为分泌蛋白的抗原。
可以如下得到Siglec-15 cDNA:通过例如所谓的PCR 方法,其中使用表达Siglec-15 cDNA的cDNA文库作为模板,并使用特异性扩增Siglec-15 cDNA的引物,进行聚合酶链式反应(在下文中称作“PCR”) (参见Saiki, R. K., 等人, Science, (1988) 239, 第487-489页)。
作为多肽的体外合成,例如可以例举由Roche Diagnostics, Inc. 生产的快速翻译系统(RTS),但不限于此。
原核宿主的实例包括大肠杆菌(Escherichia coli)和枯草芽孢杆菌(Bacillus subtilis)。为了用靶基因转化宿主细胞,使用含有复制子(即来自与所述宿主相容的物种的复制起点)和调节序列的质粒载体转化宿主细胞。此外,所述载体优选具有能赋予转化细胞表型选择性的序列。
真核宿主细胞的实例包括脊椎动物细胞、昆虫细胞和酵母细胞。作为脊椎动物细胞经常使用例如猿COS细胞(Gluzman, Y., Cell, (1981) 23, 第175-182页, ATCC CRL-1650)的二氢叶酸还原酶-缺陷株 (Urlaub, G.和Chasin, L. A., Proc. Natl. Acad. Sci. USA (1980) 77, 第4126-4220页)、鼠成纤维细胞NIH3T3 (ATCC No. CRL-1658)和中国仓鼠卵巢细胞(CHO细胞;ATCC: CCL-61)等,但是它们不限于此。
根据常规方法,可以培养这样得到的转化体,并通过培养该转化体,在细胞内或在细胞外生产靶多肽。
根据使用的宿主细胞,可以从各种常用的培养基中选择培养使用的合适培养基。如果使用大肠杆菌,则可以使用例如根据需要在其中添加诸如氨苄西林或IPMG等抗生素的LB培养基。
通过多种已知的分离方法中的任一种,利用蛋白的物理性质或化学性质,可以分离和纯化通过这种培养由转化体在细胞内或在细胞外生产的重组蛋白。
所述方法的具体实例包括:使用常规的蛋白沉淀剂进行处理、超滤、诸如分子筛色谱法(凝胶过滤)、吸附色谱法、离子交换色谱法和亲和色谱法等各类液相色谱法、透析法、以及它们的组合。
此外,通过将6组氨酸残基标签连接到要表达的重组蛋白上,可以使用镍亲和柱有效地纯化蛋白。或者,通过将IgG Fc区连接到要表达的重组蛋白上,可以使用蛋白A柱有效地纯化蛋白。
通过组合上述方法,可以高产率、高纯度地容易地制备大量靶多肽。
(2)抗-Siglec-15 单克隆抗体的制备
特异性地结合Siglec-15的抗体的实例包括特异性地结合Siglec-15的单克隆抗体,该抗体的获得方法如下所述。
单克隆抗体的制备一般需要下述操作步骤:
(a)纯化要用作抗原的生物聚合物;
(b)如下制备抗体生产细胞:通过注射抗原来免疫动物,采集血液,测定其抗体滴度,以确定脾摘除时间;
(c)制备骨髓瘤细胞(在下文中称作“骨髓瘤”);
(d)将抗体生产细胞和骨髓瘤融合;
(e)筛选生产靶抗体的杂交瘤群;
(f)将杂交瘤分成单细胞克隆(克隆化);
(g)任选地,培养杂交瘤,或培育植入了杂交瘤的动物,以制备大量单克隆抗体;
(h)检测如此制备的单克隆抗体的生物活性及其结合特异性,或者测定它们作为标记试剂的性质等。
在下文中,按照上述步骤对单克隆抗体的制备方法进行详述,但是所述方法不限于此,例如,可以使用除了脾细胞以外的抗体生产细胞和骨髓瘤。
(a)抗原的纯化
可以使用按照上述方法制备的Siglec-15或其部分肽作为抗原。
此外,由表达Siglec-15的重组细胞制备的膜组分、或表达Siglec-15的重组细胞本身、以及通过本领城技术人员已知的方法化学合成的本发明蛋白的部分肽,也可以用作抗原。
(b)抗体生产细胞的制备
将在步骤(a)中得到的抗原与佐剂(如弗氏完全或不完全佐剂、或硫酸钾铝)混合,将得到的混合物用作免疫原,免疫实验动物。在已知的杂交瘤制备方法中所使用的任何动物,可以无困难地用作实验动物。具体而言,例如,可以使用小鼠、大鼠、山羊、绵羊、牛、马等。但是,从容易获得与提取的抗体生产细胞融合的骨髓瘤细胞的角度考虑,优选使用小鼠或大鼠作为被免疫的动物。
此外,要使用的小鼠和大鼠的品系没有特别限制,在小鼠的情况下,可以使用诸如A、AKR、BALB/c、BDP、BA、CE、C3H、57BL、C57BL、C57L、DBA、FL、HTH、HT1、LP、NZB、NZW、RF、R III、SJL、SWR、WB和129 等不同的品系,在大鼠的情况下,可以使用例如Wistar、Low、Lewis、Sprague、Dawley、ACI、BN、Fischer等。
这些小鼠和大鼠可从实验动物培育商/分销商购得,例如,CLEA Japan, Inc.和Charles River Laboratories Japan, Inc。
在它们中,考虑到与下述的骨髓瘤细胞的融合相容性,特别优选地作为被免疫的动物的是:在小鼠的情况下,BALB/c品系;在小鼠的情况下,Wistar和Low品系。
此外,考虑到人和小鼠之间的抗原同源性,还优选使用具有降低的生物功能(以除去自身抗体)的小鼠,即具有自身免疫疾病的小鼠。
小鼠或大鼠在免疫时的年龄优选为5-12周龄,更优选6-8周龄。
为了使用Siglec-15或其重组体免疫动物,可以使用例如在下述文献中详述的已知方法:例如,Weir, D. M., Handbook of Experimental Immunology Vol. I. II. III., Blackwell Scientific Publications, Oxford (1987), Kabat, E. A.和Mayer, M. M., Experimental Immunochemistry, Charles C Thomas Publisher Springfield, Illinois (1964)等。
在这些免疫方法中,本发明优选的具体方法例如如下所述。
即,首先,将作为抗原的膜蛋白级分或被造成表达抗原的细胞真皮内地或腹膜内地施用给动物。
但是,为提高免疫效率,优选两种施用途径的组合,如果在前半阶段进行真皮内施用,则在后半阶段或只在最后一次给药时进行腹膜内施用,可以特别提高免疫效率。
抗原的施用计划随被免疫动物的种类、个体差异等而异。但是,一般而言,优选的施用计划是这样的,其中抗原的施用频率是3-6次,且给药间隔是2-6周;更优选的施用计划是这样的,其中抗原的施用频率是3-4次,且给药间隔是2-4周。
此外,抗原剂量随被免疫动物的种类、个体差异等而异。但是,剂量通常设定为0.05-5mg,优选约0.1-0.5mg。
在如上所述施用抗原后1-6 周、优选2-4 周、更优选2-3 周,进行加强免疫。
在进行加强免疫时抗原的剂量随动物的种类或大小等而异。但是,在小鼠的情况下,剂量通常设定为0.05-5mg,优选0.1-0.5mg,更优选约0.1-0.2 mg。
加强免疫后1-10天、优选2-5 天、更优选2-3天,从被免疫的动物无菌摘取包含抗体生产细胞的脾细胞或淋巴细胞。
此时,测定抗体滴度,如果将抗体滴度足够升高的动物作为抗体生产细胞供给源,可以更有效地进行后续操作。
这里采用的测定抗体滴度的方法的实例包括RIA法和ELISA法,但所述方法不限于此。
例如,如果使用ELISA法,则可以根据如下所述的方法测定本发明中的抗体滴度。
首先,将纯化的或部分纯化的抗原吸附于固相(诸如用于ELISA的96孔板)的表面,再用与抗原无关的蛋白(诸如牛血清白蛋白(在下文中称作“BSA”))覆盖未吸附抗原的固相表面。洗涤表面后,使表面接触作为第一抗体的系列稀释的样品(例如小鼠血清),使样品中的抗体结合抗原。
再加入作为第二抗体的用酶标记的针对小鼠抗体的抗体,使之结合小鼠抗体。洗涤后,加入该酶的底物,测量吸光度的变化(其因底物降解等诱导的显色而发生),并基于测量结果,计算抗体滴度。
根据已知的方法(例如,Kohler 等人, Nature (1975), 256, 第495页; Kohler 等人, Eur. J. Immunol. (1977), 6, 第511页; Milstein 等人, Nature (1977), 266, 第550页; Walsh, Nature (1977), 266, 第495页),可以从脾细胞或淋巴细胞分离抗体生产细胞。
例如,在脾细胞的情况下,可以采用常规方法,其中通过将脾匀浆化,用不锈钢网过滤,得到细胞,然后使细胞悬浮于Eagle氏最低基础培养基(MEM)中,从而分离抗体生产细胞。
(c)骨髓瘤细胞(在下文中称作“骨髓瘤”)的制备
细胞融合所使用的骨髓瘤细胞没有特别限制,可从已知的细胞系中选择适合的细胞。但是,基于从融合的细胞中选择杂交瘤时的便利性考虑,优选使用其选择方法已经得到确立的HGPRT (次黄嘌呤-鸟嘌呤磷酸核糖基转移酶) 缺陷品系。
更具体地,HGPRT-缺陷品系的实例包括:源自小鼠的X63-Ag8(X63)、NS1-ANS/1(NS1)、P3X63-Ag8.U1(P3U1)、X63-Ag8.653(X63.653)、SP2/0-Ag14(SP2/0)、MPC11-45.6TG1.7(45.6TG)、F0、S149/5XXO和BU.1;源自大鼠的210.RSY3.Ag.1.2.3(Y3);和源自人的U266AR(SKO-007)、GM1500·GTG-A12(GM1500)、UC729-6、LICR-LOW-HMy2(HMy2)和8226AR/NIP4-1(NP41)。
这些HGPRT-缺陷株可从例如美国典型培养物保藏中心 (ATCC)等得到。
在诸如8-氮杂鸟嘌呤培养基[通过将8-氮杂鸟嘌呤加入添加了谷氨酰胺、2-巯基乙醇、庆大霉素和胎牛血清 (在下文中称作“FCS”)的RPMI-1640 培养基中所得到的培养基]、Iscove氏改良的Dulbecco氏培养基(在下文中称作“IMDM”)、或Dulbecco氏改良的Eagle培养基(在下文中称作“DMEM”)等适当的培养基中,继代培养这些细胞品系。在该情况下,在进行细胞融合之前3-4天,在正常培养基[例如含有10 % FCS 的ASF104 培养基(Ajinomoto Co., Ltd.生产]中继代培养细胞,以在细胞融合当天得到不少于2×107的细胞。
(d)细胞融合
根据已知的方法 (Weir, D. M. Handbook of Experimental Immunology Vol. I. II. III., Blackwell Scientific Publications, Oxford (1987), Kabat, E. A. 和Mayer, M. M., Experimental Immunochemistry, Charles C Thomas Publisher, Springfield, Illinois (1964), 等),在不使细胞存活率过度降低的条件下,可以适当地进行抗体生产细胞与骨髓瘤细胞之间的融合。
作为这样的方法,可以使用:在含有高浓度的诸如聚乙二醇等聚合物的溶液中,将抗体生产细胞和骨髓瘤细胞混合的化学方法;利用电刺激的物理方法,等。
在这些方法中,化学方法的具体实例如下所述。
即,在将聚乙二醇用于含有高浓度聚合物的溶液中的情况下,在分子量为1500-6000、更优选2000-4000 的聚乙二醇溶液中,在30-40℃、优选35-38℃的温度,使抗体生产细胞和骨髓瘤细胞混合1-10分钟、优选5-8分钟。
(e)杂交瘤群的选择
选择通过上述细胞融合得到的杂交瘤的方法没有特别限制。通常,采用HAT (次黄嘌呤、氨基蝶呤、胸苷)选择方法(Kohler 等人, Nature (1975), 256, 第495页; Milstein 等人, Nature (1977), 266, 第550页)。
当使用在氨基蝶呤存在下无法存活的HGPRT-缺陷品系的骨髓瘤细胞获得杂交瘤时,该方法是有效的。
即,通过在HAT培养基中培养未融合的细胞和杂交瘤,选择性地仅容许对氨基蝶呤有抗性的杂交瘤存活并增殖。
(f)分成单细胞克隆(克隆化)
作为杂交瘤的克隆方法,可以使用已知的方法,例如甲基纤维素法、软琼脂糖法、或有限稀释法等(参见,例如,Barbara, B. M.和Stanley, M. S.: Selected Methods in Cellular Immunology, W. H. Freeman和Company, San Francisco (1980))。在这些方法中,特别地优选有限稀释法。
在该方法中,在微量培养板上接种来自胎大鼠的成纤维细胞品系,或诸如正常小鼠脾细胞、胸腺细胞或腹水细胞等饲养细胞。
同时,在培养基中稀释杂交瘤,以得到0.2-0.5个细胞/0.2ml的细胞密度。向每个孔中加入稀释的杂交瘤悬浮液的0.1 ml 等分试样,继续培养约2周,同时在预定的时间间隔(例如每3天),将约1/3的培养液更换为新鲜培养基,由此可使杂交瘤克隆增殖。
使已确定了抗体滴度的孔中的杂交瘤经受克隆化,例如通过有限稀释法,重复2-4次。选择已确定具有稳定抗体滴度的杂交瘤,作为生产抗-Siglec-15 单克隆抗体的杂交瘤品系。
这样克隆的杂交瘤品系的实例包括在 WO 09/48072中所述的杂交瘤#32A1。于2008年8月28日,将杂交瘤#32A1保藏在独立行政法人产业技术综合研究所特许生物寄托中心(International Patent Organism Depositary of the National Institute of Advanced Industrial Science and Technology,位于:Central 6, 1-1-1 Higashi, Tsukuba-shi, Ibaraki-ken, 305-8566, Japan),并已经在抗-Siglec-15 杂交瘤#32A1(anti--Siglec-15 Hybridoma #32A1)的名称下获得登录号FERM BP-10999。此外,在本说明书中,由杂交瘤#32A1生产的抗体称作“#32A1抗体”,或简称“#32A1”。此外,在本说明书的实施例中得到的除了#32A1抗体以外的抗体,也以相同的方式用抗体名称来表示。含有#32A1抗体的重链可变区的部分片段具有这样的氨基酸序列,其包含序列表中的SEQ ID NO: 28 的氨基酸残基20-167。此外,含有#32A1抗体的轻链可变区序列的部分片段具有这样的氨基酸序列,其包含序列表中的SEQ ID NO: 30的氨基酸残基21-139。
(g)通过培养杂交瘤制备单克隆抗体
通过培养这样选择的杂交瘤,可以有效地获得单克隆抗体。但是,在培养之前,优选地对生产目标单克隆抗体的杂交瘤进行筛选。
在这样的筛选中,可采用已知的方法。
通过例如在上述(b)项中解释的ELISA法,可以测量本发明的抗体滴度。
在液氮中或在-80℃或以下的冷冻箱中,可以以冷冻状态保存由上述方法得到的杂交瘤。
完成克隆化以后,将培养基由HT 培养基更换为正常培养基,并培养杂交瘤。
通过使用大型培养瓶的旋转培养或通过旋动培养,进行大规模培养。
从大规模培养得到的上清液中,通过使用诸如凝胶过滤等本领域技术人员已知的方法进行纯化,可以得到特异性地结合本发明的蛋白的单克隆抗体。
此外,向与杂交瘤同品系的小鼠(例如上述的BALB/c)或Nu/Nu小鼠的腹腔内注射杂交瘤,使杂交瘤增殖,由此可以获得含有大量本发明的单克隆抗体的腹水。
在腹腔内施用杂交瘤的情况下,如果提前3-7天施用诸如2,6,10,14-四甲基十五烷(姥鲛烷)等的矿物油,则可得到更大量的腹水。
例如,预先向与杂交瘤同品系的小鼠的腹腔内注射免疫抑制剂,使T细胞失活。20天后,使106-107个杂交瘤克隆细胞悬浮于不含血清的培养基中(0.5 ml),然后向小鼠腹腔内注射该悬浮液。一般而言,当腹部膨胀、充满腹水时,从小鼠采集腹水。
通过该方法,可得到比培养液中的浓度高约100 倍或更高浓度的单克隆抗体。
通过在例如Weir, D. M.: Handbook of Experimental Immunology Vol. I, II, III, Blackwell Scientific Publications, Oxford (1978)中描述的方法,可以纯化由上述方法得到的单克隆抗体。
也就是说,所述方法的实例包括硫酸胺沉淀法、凝胶过滤法、离子交换色谱法和亲和色谱法。
作为简单的纯化方法,也可以使用市售的单克隆抗体纯化试剂盒(例如,由Pharmacia, Inc.生产的MAbTrap GII试剂盒)等。
这样得到的单克隆抗体对于Siglec-15具有高抗原特异性。
(h)单克隆抗体的测定
可以如下确定这样得到的单克隆抗体的同种型和亚类。
首先,鉴定方法的实例包括奥脱洛尼(Ouchterlony)法、ELISA法以及RIA法。
奥脱洛尼法是简便的,但是当单克隆抗体浓度较低时,需要浓缩操作。
另一方面,当使用ELISA法或RIA法时,通过使培养上清液与吸附有抗原的固相直接反应,并使用与各种免疫球蛋白同种型和亚类对应的抗体作为第二抗体,可以鉴定单克隆抗体的同种型和亚类。
此外,作为更简便的方法,也可以使用市售的鉴定试剂盒(例如,由Bio-Rad Laboratories, Inc.生产的小鼠分型试剂盒)等。
此外,通过Folin Lowry法,以及基于在280nm的吸光度的计算方法 [1.4 (OD 280) = 1 mg免疫球蛋白/ml],可以进行蛋白的定量测定。
(3)其它抗体
本发明的抗体不仅包括上述抗Siglec-15的单克隆抗体,而且包括以降低对人的异种抗原性为目的而人工修饰得到的重组抗体,例如嵌合抗体、人源化抗体和人抗体。使用已知的方法,可以制备这些抗体。
作为嵌合抗体,可以例举这样的抗体,其中抗体可变区和恒定区源自不同的物种,例如,其中使小鼠-或大鼠-衍生的抗体可变区与人-衍生的恒定区相连的嵌合抗体(参见Proc. Natl. Acad. Sci. USA, 81, 6851-6855, (1984))。作为源自大鼠抗-小鼠抗体#32A1的嵌合抗体的一个实例,可以例举这样的抗体,其包含重链和轻链,所述重链具有包含序列表中的SEQ ID NO: 41的氨基酸残基20-470的氨基酸序列,且所述轻链具有包含序列表中的SEQ ID NO: 43的氨基酸残基21-237 的氨基酸序列。
作为人源化抗体,可以例举通过只将互补性决定区 (CDR)整合到人-衍生的抗体中所得到的抗体 (参见Nature (1986) 321, 第页522-525);以及通过CDR-移植法,将构架的氨基酸残基的一部分和CDR 序列移植到人抗体中所得到的抗体(WO 90/07861)。作为大鼠抗体#32A1的人源化抗体的一个实例,可以例举含有下述重链可变区的重链和含有下述轻链可变区的轻链的任意组合,所述重链可变区包含这样的氨基酸序列,其含有序列表中的 SEQ ID NO: 51的氨基酸残基20-140、SEQ ID NO: 53的氨基酸残基20-140、SEQ ID NO: 55的氨基酸残基20-140、SEQ ID NO: 57的氨基酸残基20-140、SEQ ID NO: 59的氨基酸残基20-140、SEQ ID NO: 99的氨基酸残基20-140、SEQ ID NO: 101的氨基酸残基20-140、或由SEQ ID NO: 72-77表示的任一个氨基酸序列的氨基酸残基1-121,所述轻链可变区包含这样的氨基酸序列,其含有序列表中的SEQ ID NO: 61的氨基酸残基21-133、SEQ ID NO: 63的氨基酸残基21-133、SEQ ID NO: 65的氨基酸残基21-133、SEQ ID NO: 67的氨基酸残基21-132、SEQ ID NO: 69的氨基酸残基21-133、SEQ ID NO: 71的氨基酸残基21-133、SEQ ID NO: 103的氨基酸残基21-133、SEQ ID NO: 105的氨基酸残基21-133、或由SEQ ID NO: 78-96表示的任一个氨基酸序列的氨基酸残基1-113。
作为一个优选组合,可以例举这样的抗体,其包含含有下述重链可变区的重链和含有下述轻链可变区的轻链,所述重链可变区包含含有SEQ ID NO: 51的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 61的氨基酸残基21-133的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 53的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 63的氨基酸残基21-133的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 55的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 65的氨基酸残基21-133的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 55的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 67的氨基酸残基21-132的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 57的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 69的氨基酸残基21-133的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 59的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 71的氨基酸残基21-133的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 99的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 69的氨基酸残基21-133的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 101的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 69的氨基酸残基21-133的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 99的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 103的氨基酸残基21-133的氨基酸序列,或者所述重链可变区包含含有SEQ ID NO: 99的氨基酸残基20-140的氨基酸序列,且所述轻链可变区包含含有SEQ ID NO: 105的氨基酸残基21-133的氨基酸序列。
作为一个更优选的组合,可以例举包含重链和轻链的抗体,所述重链具有包含SEQ ID NO: 51的氨基酸残基20-470的氨基酸序列,且所述轻链具有包含SEQ ID NO: 61的氨基酸残基21-238的氨基酸序列,或者所述重链具有包含SEQ ID NO: 53的氨基酸残基20-470的氨基酸序列,且所述轻链具有包含SEQ ID NO: 63的氨基酸残基21-238的氨基酸序列,或者所述重链具有包含SEQ ID NO: 55的氨基酸残基20-470的氨基酸序列,且所述轻链具有包含SEQ ID NO: 65的氨基酸残基21-238的氨基酸序列,或者所述重链具有包含SEQ ID NO: 55的氨基酸残基20-470的氨基酸序列,且所述轻链具有包含SEQ ID NO: 67的氨基酸残基21-237的氨基酸序列,或者所述重链具有包含SEQ ID NO: 57的氨基酸残基20-470的氨基酸序列,且所述轻链具有包含SEQ ID NO: 69的氨基酸残基21-238的氨基酸序列,或者所述重链具有包含SEQ ID NO: 59的氨基酸残基20-470的氨基酸序列,且所述轻链具有包含SEQ ID NO: 71的氨基酸残基21-238的氨基酸序列,或者所述重链具有包含SEQ ID NO: 99的氨基酸残基20-466 的氨基酸序列,且所述轻链具有包含SEQ ID NO: 69的氨基酸残基21-238的氨基酸序列,或者所述重链具有包含SEQ ID NO: 101的氨基酸残基20-466 的氨基酸序列,且所述轻链具有包含SEQ ID NO: 69的氨基酸残基21-238的氨基酸序列,或者所述重链具有包含SEQ ID NO: 99的氨基酸残基20-466 的氨基酸序列,且所述轻链具有包含SEQ ID NO: 103的氨基酸残基21-238的氨基酸序列,或者所述重链具有包含SEQ ID NO: 99的氨基酸残基20-466 的氨基酸序列,且所述轻链具有包含SEQ ID NO: 105的氨基酸残基21-238的氨基酸序列。
但是,源自#32A1抗体的人源化抗体不限于上述的人源化抗体,只要人源化抗体具有#32A1的所有6类CDR序列,且具有抑制破骨细胞形成的活性。此外,#32A1抗体的重链可变区具有:包含由SEQ ID NO: 44表示的氨基酸序列的CDRH1 (DYFMN),包含由SEQ ID NO: 45表示的氨基酸序列的CDRH2 (QIRNKIYTYATFYAESLEG)和由SEQ ID NO: 97表示的CDRH2 (QIRNKIYTYATFYA) 中的任一个,和包含由SEQ ID NO: 46表示的氨基酸序列的CDRH3 (SLTGGDYFDY) 。由SEQ ID NO: 45表示的CDRH2是根据Kabat定义的 (SEQUENCES OF PROTEINS OF IMMUNOLOGICAL INTEREST VOL. I, 第5版 (1991))。与Kabat定义相比,由SEQ ID NO: 97表示的CDRH2在C端处缩短了5个残基。在含有该CDRH2的重链序列中,源自大鼠的CDR序列被制成更短,掺入更多的人框架序列,因此,当将它施用给人时,被识别为异种抗原的可能性更低。此外,#32A1抗体的轻链可变区具有:包含由SEQ ID NO: 47表示的氨基酸序列的CDRL1 (RASQSVTISGYSFIH)、包含由SEQ ID NO: 48表示的氨基酸序列的CDRL2 (RASNLAS)和包含由SEQ ID NO: 49表示的氨基酸序列的CDRL3 (QQSRKSPWT)。
此外,本发明的抗体包括人抗体。人抗-Siglec-15抗体是指只具有源自人染色体的抗体基因序列的人抗体。通过使用生产人抗体的小鼠的方法可以获得人抗-Siglec-15抗体,所述小鼠具有含有人抗体的重链和轻链基因的人染色体片段(参见Tomizuka, K. 等人, Nature Genetics (1997) 16, 第133-143页; Kuroiwa, Y. 等人, Nucl. Acids Res. (1998) 26, 第3447-3448页; Yoshida, H. 等人, Animal Cell Technology: Basic and Applied Aspects vol. 10, 第69-73页 (Kitagawa, Y., Matuda, T.和Iijima, S. 编), Kluwer Academic Publishers, 1999; Tomizuka, K. 等人, Proc. Natl. Acad. Sci. USA (2000) 97, 第722-727页, 等)。
具体地可以如下建立这种生产人抗体的小鼠。通过制备敲除的动物和转基因动物,并使这些动物交配,建立遗传修饰的动物,其中已经破坏了内源免疫球蛋白重链和轻链基因基因座,并替代地,经由酵母人工染色体(YAC)载体等已经导入了人免疫球蛋白重链和轻链基因基因座。
此外,根据基因工程技术,通过使用分别编码这样的人抗体的重链和轻链的cDNA,并优选使用包含该cDNA的载体,转化真核细胞,培养生产重组人单克隆抗体的转化体,由此也可从培养上清液获得该抗体。
在这里,例如,可以使用真核细胞、优选哺乳动物细胞,诸如CHO细胞、淋巴细胞或骨髓瘤细胞作为宿主。
此外,还已知获得从人抗体文库筛选出来的噬菌体展示-衍生的人抗体的方法(参见Wormstone, I. M. 等人, Investigative Ophthalmology & Visual Science. (2002) 43 (7), 第2301-2308页;Carmen, S. 等人, Briefings in Functional Genomics and Proteomics (2002), 1 (2), 第189-203页; Siriwardena, D. 等人, Opthalmology (2002) 109 (3), 第427-431页, 等) 。
例如,可以使用噬菌体展示法,其中在噬菌体表面上表达人抗体的可变区作为单链抗体(scFv),并选择结合抗原的噬菌体(Nature Biotechnology (2005), 23, (9), 第1105-1116页)。
通过分析基于与抗原的结合所选择的噬菌体的基因,可以确定编码结合抗原的人抗体的可变区的DNA序列。
如果确定了结合抗原的scFv的DNA序列,通过制备具有该序列的表达载体,并将该载体导入适当的宿主中以表达它,可以获得人抗体(WO 92/01047,WO 92/20791,WO 93/06213,WO 93/11236,WO 93/19172,WO 95/01438,WO 95/15388,Annu. Rev. Immunol (1994) 12, 第433-455页, Nature Biotechnology (2005) 23 (9), 第1105-1116页)。
通过在实施例25等中所述的方法,可以评价通过上述方法得到的抗体的结合抗原的性质,并可以选择优选的抗体。作为对比抗体的性质时的另一个指标的一个实例,可以例举抗体的稳定性。在实施例33中显示的示差扫描量热法(DSC)是能够快速且准确地测量热变性中点温度(Tm) 的仪器,所述热变性中点温度是蛋白的相对结构稳定性的有利指标。通过使用DSC测量Tm 值并对比该值,可以对比热稳定性的差异。已知,抗体的储存稳定性显示出与抗体的热稳定性的某种关联性 (Lori Burton, 等人, Pharmaceutical Development and Technology (2007) 12, 第265-273页),使用热稳定性作为指标,可以选择出优选的抗体。用于选择抗体的其它指标的实例包括下述:在适当宿主细胞中的产量较高,在水性溶液中的聚集性较低。例如,显示出最高产量的抗体不总是显示出高热稳定性,因此,通过基于上述指标做出综合性评价来选择最适合施用给人的抗体是必要的。
此外,还已知这样的方法,其中使用适当的接头,连接抗体的全长重链和轻链序列,由此得到单链免疫球蛋白(Lee, H-S, 等人, Molecular Immunology (1999) 36, 第61-71页; Shirrmann, T. 等人, mAbs (2010), 2, (1)第1-4页)。通过二聚化这样的单链免疫球蛋白,得到的二聚体可以具有与本身是四聚体的抗体类似的结构和活性。此外,本发明的抗体可以是具有单个重链可变区且不具有轻链序列的抗体。这样的抗体被称作单结构域抗体(sdAb)或纳米体(nanobody),实际上,在骆驼和羊驼中观察到它,且已经报道具有抗原结合亲和力 (Muyldemans S. 等人, Protein Eng. (1994) 7(9), 1129-35, Hamers- Casterman C. 等人, Nature (1993) 363 (6428) 446-8)。可以理解,上述的抗体是根据本发明的抗体的功能片段的类型。
此外,通过控制糖基化(其中聚糖结合到本发明的抗体上),可能增强抗体-依赖性的细胞的细胞毒性活性。关于控制抗体的糖基化的技术,已知WO 99/54342、WO 00/61739、WO 02/31140等。但是,所述技术不限于此。
在首先分离抗体基因、然后将该基因导入适当宿主中来制备抗体的情况下,可以使用适当宿主和适当表达载体的组合。抗体基因的具体实例包括编码在本说明书中描述的抗体的重链序列的基因和编码其轻链序列的基因的组合。当转化宿主细胞时,可能将重链序列基因和轻链序列基因插入相同的表达载体中,且也可能分别插入不同的表达载体中。在使用真核细胞作为宿主的情况下,可使用动物细胞、植物细胞和真核微生物。作为动物细胞,可以例举 (1) 哺乳动物细胞,例如,猿COS细胞(Gluzman, Y., Cell, (1981) 23, 第175-182页, ATCC CRL-1650)的二氢叶酸还原酶-缺陷株 (Urlaub, G.和Chasin, L. A., Proc. Natl. Acad. Sci. USA (1980) 77, 第4126-4220页), 鼠成纤维细胞NIH3T3 (ATCC No. CRL-1658)和中国仓鼠卵巢细胞(CHO细胞; ATCC: CCL-61)。此外,在使用原核细胞的情况下,例如,可以例举大肠杆菌和枯草芽孢杆菌。通过转化,向这些细胞中导入靶抗体基因,并在体外培养这样转化的细胞,可得到该抗体。在上述的培养方法中,产率有时可能随抗体的序列而变化,因此,可能通过使用产量作为指标,在具有相当的结合活性的抗体中选择可以容易地生产作为药物的抗体。
本发明的抗体的同种型没有限制,其实例包括IgG (IgG1、IgG2、IgG3、IgG4)、IgM、IgA (IgA1、IgA2)、IgD和IgE,且其优选的实例包括IgG和IgM,进一步,其更优选的实例包括 IgG1和IgG2。
此外,本发明的抗体可以是具有抗体的抗原结合部位的抗体功能片段或其修饰的片段。通过用诸如木瓜蛋白酶或胃蛋白酶等蛋白酶处理抗体,或根据基因工程技术修饰抗体基因,并在适当的培养细胞中表达修饰的基因,可以得到抗体的片段。在这些抗体片段中,具有全长抗体分子的全部或部分功能的片段可以称为所述抗体的功能片段。作为抗体的功能,通常可以例举抗原结合活性、中和抗原活性的活性、增强抗原活性的活性、抗体-依赖性的细胞毒性活性、补体-依赖性的细胞毒性活性以及补体-依赖性的细胞的细胞毒性活性。根据本发明的抗体功能片段的功能是对Siglec-15的结合活性,优选抑制破骨细胞形成的活性,更优选抑制破骨细胞的细胞融合过程的活性。
抗体片段的实例包括Fab、F(ab’)2、Fv、单链Fv (scFv,其中重链和轻链的Fv 分子通过适当的接头相连)、双特异抗体、线性抗体、以及由抗体片段组成的多特异性抗体。此外,Fab’(通过在还原条件下处理F(ab’)2而得到的抗体可变区中的单价片段)也视作抗体片段。
此外,本发明的抗体可以是对至少两种不同抗原具有特异性的多特异性抗体。通常,这样的分子结合两种抗原(即,双特异性抗体),但是,本文使用的“多特异性抗体”包括具有对两种或更多种(例如,3种)抗原的特异性的抗体。
本发明的多特异性抗体可以是全长抗体或这样的抗体的片段(例如,F(ab’)2双特异性抗体)。通过连接两类抗体的重链和轻链(HL对),可以制备双特异性抗体,或者通过融合生产不同单克隆抗体的杂交瘤,以制备生产双特异性抗体的融合细胞,也可以制备双特异性抗体(Millstein 等人, Nature (1983) 305, 第537-539页)。
本发明的抗体可以是单链抗体(也称为scFv)。通过经由多肽接头连接抗体的重链可变区和轻链可变区,可以获得单链抗体(Pluckthun,The Pharmacology of Monoclonal Antibodies,113( Rosenburg和Moore编,Springer Verlag,New York, 第269-315页(1994),Nature Biotechnology (2005), 23, 第1126-1136页)。此外,通过经由多肽接头连接两个scFv分子生成的BiscFv片段,也可用作双特异性抗体。
制备单链抗体的方法是在本技术领域中已知的(参见,例如,美国专利号4,946,778、5,260,203、5,091,513、5,455,030等)。在该scFv中,通过不形成缀合物的接头,优选通过多肽接头,连接重链可变区和轻链可变区(Huston, J. S. 等人, Proc. Natl. Acad. Sci. USA (1988), 85, 第5879-5883页)。在scFv中,重链可变区和轻链可变区可以源自相同抗体或不同抗体。
作为用于连接可变区的多肽接头,例如使用由12-19个残基组成的给定单链肽。
可如下获得编码scFv的DNA:使用编码全部氨基酸序列或所需的部分氨基酸序列(选自上述抗体的重链或重链可变区及其轻链或轻链可变区)的DNA作为模板,并使用限定其两端的引物对,通过PCR法进行扩增,并通过组合编码多肽接头部分的DNA和限定其两端的引物对,从而将其两端分别连接重链和轻链,进一步进行扩增。
此外,制备编码scFv的DNA以后,根据常规方法,可以获得包含该DNA的表达载体以及用该表达载体转化的宿主。此外,通过使用获得的宿主,根据常规方法,可以获得scFv。通过以与上述相同的方式获得基因并表达该基因,可以在宿主中生产其抗体片段。
可以使本发明的抗体多聚化,以增加对抗原的亲和性。待多聚化的抗体可以是一类抗体,或识别相同抗原的多个表位的多个抗体。作为多聚化抗体的方法,可以例举的是使IgG CH3结构域结合两个scFv分子,结合抗生蛋白链菌素,导入螺旋-转角-螺旋基序等。
本发明的抗体可以是多克隆抗体,它是具有不同氨基酸序列的多类抗-Siglec-15抗体的混合物。作为多克隆抗体的一个实例,可以例举具有不同CDR的多类抗体的混合物。培养生产不同抗体的细胞的混合物,并从得到的培养物纯化抗体可用作这样的多克隆抗体(参见WO 2004/061104)。
也可使用与诸如聚乙二醇 (PEG) 等任意不同类型的分子结合的抗体作为修饰的抗体。
此外,本发明的抗体可以是任一种这样的抗体和另一种药物之间形成的缀合物的形式(免疫缀合物)。这样的抗体的实例包括其中抗体缀合到放射性物质或具有药理作用的化合物上的那些(Nature Biotechnology (2005) 23, 第1137-1146页)。
可以将所得抗体纯化至同质。使用常规的蛋白分离和纯化方法,可以进行抗体的分离和纯化。例如,通过适当地选择和组合柱色谱法、过滤器过滤、超滤、盐析、透析、制备聚丙烯酰胺凝胶电泳、等电聚焦电泳等,可以分离和纯化抗体(Strategies for protein Purification and Charcterization:A Laboratoy Course Manual,Daniel R.Marshak 等人编, Cold Spring Harbor Laboratory Press (1996);Antibodies:A Laboratory Manual. Harlow and David Lane编,Cold Spring Harbor Laboratory(1988)),但是方法不限于此。
这些色谱法的实例包括亲和色谱法、离子交换色谱法、疏水色谱法、凝胶过滤色谱法、反相色谱法和吸附色谱法。
使用液相色谱法,诸如HPLC或FPLC,可以进行这些色谱法。
作为用于亲和色谱法的柱子,可以例举蛋白A柱和蛋白G柱。
例如,作为使用蛋白A柱的柱子,可以例举Hyper D、POROS、琼脂糖 FF (Pharmacia) 等。
此外,通过使用其上固定有抗原的载体,利用抗体对抗原的结合性质,也可纯化抗体。
4. 含有抗-Siglec-15抗体的药物
从在上面的“3. 抗-Siglec-15抗体的制备”一项中所述的方法得到的抗-Siglec-15抗体,可以获得中和Siglec-15的生物活性的抗体。这些中和Siglec-15的生物活性的抗体会在体内抑制Siglec-15的生物活性,即,破骨细胞的分化和/或成熟,因此可用作药物,用作由破骨细胞的异常分化和/或成熟造成的骨代谢异常的治疗剂和/或预防剂。骨代谢异常可以是任何以净骨流失 (骨质减少或骨质溶解)为特征的障碍。一般而言,用抗-Siglec-15抗体的治疗和/或预防应用于需要抑制骨吸收的情况。可用抗-Siglec-15抗体治疗和/或预防的骨代谢异常的实例包括:骨质疏松症 (绝经后骨质疏松症、老年性骨质疏松症、由使用诸如类固醇或免疫抑制剂等治疗剂导致的继发性骨质疏松症、或伴随类风湿性关节炎的骨质疏松症)、伴随类风湿性关节炎的骨破坏、癌性高钙血症、伴随多发性骨髓瘤或癌的骨转移的骨破坏、巨细胞瘤、骨质减少、牙周炎导致的牙缺失、人工关节周围的骨溶解、慢性骨髓炎中的骨破坏、骨佩吉特病、肾性骨营养不良和成骨不全,但是,骨代谢异常不限于此,只要它是伴有破骨细胞造成的净骨流失的疾病即可。作为上述药物使用的抗-Siglec-15抗体的实例包括通过在3. “(3) 其它抗体”一项中所述的方法从#32A1抗体制备的嵌合抗体和人源化抗体。此外,具有与#32A1抗体相同表位的嵌合抗体、人源化抗体和人抗体也可用作药物。通过观察这些抗体是否结合Siglec-15的相同的特定部分肽,可以证实某种抗-Siglec-15抗体是否具有与#32A1抗体相同的表位。此外,如果某种抗-Siglec-15抗体与#32A1抗体竞争结合Siglec-15,则也可判定这些抗体具有相同的表位。
通过例如抑制过表达Siglec-15的细胞分化成破骨细胞的活性,可以测定抗-Siglec-15抗体的中和Siglec-15的生物活性的体外活性。例如,在不同的浓度,向RAW 264.7细胞或RAW 264细胞(小鼠单核细胞-衍生的细胞系)添加抗-Siglec-15抗体,可以测定RANKL或TNF-α的刺激抑制向破骨细胞分化的活性。此外,在不同的浓度,向骨髓-衍生的原代培养细胞添加抗-Siglec-15抗体,可以测定RANKL、TNF-α或活性维生素D3的刺激抑制向破骨细胞分化的活性。此外,在不同的浓度,向正常人破骨细胞前体细胞(正常人天然破骨细胞前体细胞,可从Sanko Junyaku Co., Ltd., 目录号2T-110得到)添加抗-Siglec-15抗体,可以测定RANKL或M-CSF的刺激抑制向破骨细胞分化的活性。通过使用酒石酸盐-抗性的酸性磷酸酶(TRAP)活性的抑制为指标,可以测定这样的对破骨细胞分化的抑制效应。此外,通过使用TRAP-阳性的多核破骨细胞形成的抑制(即破骨细胞的细胞融合的抑制)为指标,也可以测定对破骨细胞分化的抑制效应。此外,在使用股骨-和/或胫骨-衍生的细胞的陷窝测定实验(Takada 等人, Bone和Mineral, (1992) 17, 347-359)中,通过以不同浓度向股骨-和/或胫骨-衍生的细胞添加抗-Siglec-15抗体,并观察牙质切片上的陷窝形成,可以测定抑制破骨细胞的骨吸收的体外活性。作为用于测定抑制破骨细胞导致的骨吸收的体外活性的系统,也可以使用涂有缀合铕的人胶原的板。可以如下确认使用实验动物的抗-Siglec-15抗体对骨代谢异常的体内治疗或预防效应:将抗-Siglec-15抗体施用给骨质疏松症动物模型或过表达Siglec-15的转基因动物,并测量破骨细胞的任何变化。
这样得到的的中和Siglec-15的生物活性的抗体可用作药物,特别是用作治疗或预防骨代谢异常的药物组合物,所述骨代谢异常例如骨质疏松症、伴随类风湿性关节炎的骨破坏、或伴随癌的骨转移的骨破坏,或用作这样的疾病的免疫学诊断的抗体。
在类风湿性关节炎(RA)的治疗中,一个主要的难题是伴随该疾病的发生的骨流失。业已报道在伴随RA的这种骨流失中破骨细胞起主要作用。认为RANKL和TNF-α对于破骨细胞诱导(分化和成熟)和活化而言是最重要的细胞因子,且是RA中骨破坏的原因(Romas E. 等人, Bone 30, 第340-346页, 2002)。作为RANKL的诱饵受体的OCIF/OPG可抑制由RANKL诱导的破骨细胞形成,但是不抑制由TNF-α诱导的破骨细胞形成。另一方面,根据本发明的抗-Siglec-15抗体会有效地抑制由RANKL和TNF-α诱导的破骨细胞形成。因此,预期本发明的抗-Siglec-15抗体可以比RANKL阻滞剂(OCIF/OPG、抗-RANKL抗体等)更强烈地抑制RA等中的TNF-α诱导的骨流失和骨破坏。
作为一个实例,对于骨代谢异常的治疗或预防,抗-Siglec-15抗体可以单独施用或与骨相关疾病的至少一种其它治疗剂组合施用。作为另一个实例,抗-Siglec-15抗体可以与治疗有效量的骨代谢异常的治疗剂组合施用。可与抗-Siglec-15抗体组合施用的治疗剂的实例包括但不限于:二膦酸盐 (例如,阿仑膦酸盐、依替膦酸盐、伊班膦酸盐、伊卡膦酸盐、帕玛二磷酸盐、利塞膦酸盐和唑来膦酸盐), 活性维生素D3、降钙素及其衍生物、激素例如雌二醇、SERM (选择性雌激素受体调节剂)、依普黄酮、维生素K2 (四烯甲萘醌)、钙制剂、PTH (甲状旁腺激素)、非甾体类抗炎药 (例如,塞来考昔和罗非昔布)、可溶性TNF受体 (例如,依那西普)、抗-TNF-α抗体或所述抗体的功能片段(例如,英夫利昔单抗)、抗-PTHrP (甲状旁腺激素相关蛋白)抗体或所述抗体的功能片段、IL-1 受体拮抗剂 (例如,阿那白滞素)、抗-IL-6 受体抗体或所述抗体的功能片段(例如,托珠单抗)、抗-RANKL抗体或所述抗体的功能片段(例如,denosumab)和OCIF (破骨细胞生成抑制因子)。根据骨代谢异常的状态或希望的治疗和/或预防程度,可以施用2或3或更多种药物类型,通过封装在同一制剂中,可以一起供应这些药物。通过将它们封装在同一制剂中,也可以一起供应这些药物和抗-Siglec-15抗体。此外,通过将它们封装为用于治疗和/或预防的试剂盒,也可以一起供应这些药物。此外,可以分别供应这些药物和抗-Siglec-15抗体。在基因治疗中施用的情况下,可以将蛋白性的骨疾病治疗剂的基因和抗-Siglec-15抗体的基因插入到相同启动子区或不同启动子区的下游,且可以导入到相同载体或不同载体中。
通过使骨疾病治疗剂与抗-Siglec-15抗体或其片段缀合,可以制备如在M. C. Garnet “Targeted drug conjugates: principles and progress”, Advanced Drug Delivery Reviews, (2001) 53, 171-216中所述的靶向药物缀合物。为达到该目的,可使用除抗体分子外的任何抗体片段,只要其没有完全失去识别破骨细胞的能力,其实例包括诸如Fab、F(ab’)2和Fv等片段。在本发明中,也可以以相同的方式使用抗体和片段。抗-Siglec-15抗体或其片段与骨疾病治疗剂形成的缀合物可以是在下述文献中所述的多种形式中的任一种:M. C. Garnet “Targeted drug conjugates: principles and progress”, Advanced Drug Delivery Reviews, (2001) 53, 171-216, G. T. Hermanson “Bioconjugate Techniques” Academic Press, California (1996), Putnam和J. Kopecek “Polymer Conjugates with Anticancer Activity” Advances in Polymer Science (1995) 122, 55-123 等。也就是说,可以例举这样的缀合物,其中抗-Siglec-15抗体和骨疾病治疗剂彼此化学地直接缀合,或经由诸如寡肽等间隔物缀合,以及经由适当的药物载体而形成的缀合物。药物载体的实例包括脂质体和水溶性聚合物。经由这种药物载体形成的缀合物的更具体实例包括:这样的缀合物,其中将抗体和骨疾病治疗剂掺入脂质体中,并将脂质体与抗体彼此缀合;和这样的缀合物,其中骨疾病治疗剂与水溶性聚合物(分子量为约1000-100000的化合物)化学地直接缀合,或经由诸如寡肽等间隔物缀合,并将抗体与水溶性聚合物缀合。通过本领域技术人员已知的方法可以实现抗体(或其片段)与骨疾病治疗剂或药物载体(诸如脂质体或水溶性聚合物)的缀合,所述方法例如描述在下述文献中的方法:G. T. Hermanson “Bioconjugate Techniques” Academic Press, California (1996), Putnam和J. Kopecek “Polymer Conjugates with Anticancer Activity” Advances in Polymer Science (1995) 122, 55-123。通过本领域技术人员已知的方法,可以实现骨疾病治疗剂在脂质体中的掺入,所述方法例如描述在下述文献中的方法:D. D. Lasic “Liposomes:From Physics to Applications” Elsevier Science Publishers B. V., Amsterdam (1993)等。通过本领域技术人员已知的方法,可以实现骨疾病治疗剂与水溶性聚合物的缀合,所述方法例如描述在下述文献中的方法:D. Putnam和J. Kopecek “Polymer Conjugates with Anticancer Activity”Advances in Polymer Science(1995)122,55-123。除上述方法外,通过本领域技术人员已知的方法,通过基因工程,可以制备抗体(或其片段)与蛋白性的骨疾病治疗剂(或其片段) 的缀合物。
本发明也提供了药物组合物,其含有治疗和/或预防有效量的抗-Siglec-15抗体和药学上可接受的稀释剂、载体、增溶剂、乳化剂、防腐剂和/或佐剂。
本发明也提供了药物组合物,其含有治疗和/或预防有效量的抗-Siglec-15抗体、治疗和/或预防有效量的至少一种骨疾病治疗剂和药学上可接受的稀释剂、载体、增溶剂、乳化剂、防腐剂和/或佐剂。骨疾病治疗剂的实例包括但不限于:二膦酸盐 (例如,阿屈膦酸盐、依替膦酸盐、伊班膦酸盐、伊卡膦酸盐、帕玛二磷酸盐、利塞膦酸盐和唑来膦酸盐)、活性维生素D3、降钙素及其衍生物、激素例如雌二醇、SERM (选择性雌激素受体调节剂)、依普黄酮、维生素K2 (四烯甲萘醌)、钙制剂、PTH (甲状旁腺激素)、非甾体类抗炎药 (例如,塞来考昔和罗非昔布)、可溶性TNF受体 (例如,依那西普)、抗-TNF-α抗体或所述抗体的功能片段(例如,英夫利昔单抗)、抗-PTHrP (甲状旁腺激素相关蛋白)抗体或所述抗体的功能片段、IL-1 受体拮抗剂 (例如,阿那白滞素)、抗-IL-6 受体抗体或所述抗体的功能片段(例如,托珠单抗)、抗-RANKL抗体或所述抗体的功能片段(例如,denosumab)和OCIF (破骨细胞生成抑制因子)。
在根据本发明的药物组合物中可接受的、用于制剂中的物质优选地在其施用量和施用浓度对施用药物组合物的人没有毒性。
本发明的药物组合物可以包含能改变或维持pH、渗透压、粘度、透明度、颜色、等张性、无菌状态、稳定性、溶解度、释放速率、吸收速率及其渗透性的药用物质。这样的药用物质的实例包括但不限于:氨基酸诸如甘氨酸、丙氨酸、谷氨酰胺、天冬酰胺、精氨酸和赖氨酸;抗微生物剂;抗氧化剂诸如抗坏血酸、硫酸钠和亚硫酸氢钠;缓冲剂诸如磷酸盐、柠檬酸盐、硼酸盐缓冲剂、碳酸氢钠和Tris-HCl溶液;填充剂诸如甘露醇和甘氨酸;螯合剂诸如四乙酸乙二胺(EDTA);络合剂诸如咖啡因、聚乙烯吡咯烷、β-环糊精和羟丙基-β-环糊精;增量剂诸如葡萄糖、甘露糖和糊精;其它碳水化合物诸如单糖类和二糖类;着色剂;矫味剂;稀释剂;乳化剂;亲水聚合物诸如聚乙烯吡咯烷;防腐剂诸如低分子量多肽、成盐抗衡离子、苯扎氯铵、苯甲酸、水杨酸、硫柳汞、苯乙醇、对羟基苯甲酸甲酯、对羟基苯甲酸丙酯、氯己定、山梨酸和过氧化氢;溶剂诸如甘油、丙二醇和聚乙二醇;糖醇诸如甘露醇和山梨醇;助悬剂;表面活性剂诸如脱水山梨糖醇酯、包括聚山梨酯 20和聚山梨酯 80在内的聚山梨酯、三硝基甲苯(Triton)、氨丁三醇、卵磷脂和胆固醇;稳定性增强剂诸如蔗糖和山梨醇;弹性增强剂诸如氯化钠、氯化钾和甘露醇和山梨醇;转运剂;赋形剂;和/或药物佐剂。这些为药用加入的物质的量优选为抗-Siglec-15抗体重量的0.01-100倍,特别优选0.1-10倍。根据组合物应用的疾病、应用的施用途径等,本领域技术人员可以适当地确定制剂中药物组合物的优选组成。
药物组合物中的赋形剂和载体可以是液体或固体形式。适当的赋形剂或载体可以是注射用水、生理盐水、人工脑脊液或肠胃外施用中常用的其它物质。此外,中性生理盐水或含有血清白蛋白的生理盐水也可以用作载体。药物组合物可以含有pH7.0-8.5的Tris 缓冲液、pH4.0-5.5的乙酸盐缓冲液或pH 3.0-6.2的柠檬酸盐缓冲液。此外,这样的缓冲液可以补充山梨醇或另一种化合物。本发明的药物组合物的实例包括含有抗-Siglec-15抗体的药物组合物、以及含有抗-Siglec-15抗体和至少一种骨疾病治疗剂的药物组合物。将本发明的药物组合物制成冻干产品或液体形式,作为具有所选择的组成和所需的纯度的药物。也可以使用适当的赋形剂(如蔗糖),使含有抗-Siglec-15抗体的药物组合物以及含有抗-Siglec-15抗体和至少一种骨代谢异常治疗剂的药物组合物形成冻干产品。
本发明的药物组合物可以制成用于肠胃外施用或用于经口服给药的胃肠道吸收。制剂的组成和浓度可根据施用方法确定。本发明的药物组合物中所含的抗-Siglec-15抗体对Siglec-15的亲和力越高,也就是说,其对Siglec-15的解离常数 (Kd 值)越低,则抗-Siglec-15抗体可以表现出的药物效能越多(甚至在减少给人的剂量时)。因此,基于该考虑,也可以确定本发明的药物组合物对人的剂量。对于剂量,在给人施用人抗-Siglec-15抗体的情况下,可以以约0.1-100 mg/kg(每1~180天一次)的剂量施用抗体。
本发明的药物组合物的剂型的实例包括:包括输注在内的注射剂、栓剂、经鼻剂、舌下剂和经皮吸收剂。
实施例
在下文中,参考实施例更具体地描述本发明,然而,本发明不限于此。应当指出,如无特别说明,在下述实施例中的与基因操作有关的各个操作均根据在“Molecular Cloning”( Sambrook, J., Fritsch, E. F.和Maniatis, T.著, Cold Spring Harbor Laboratory Press,1989年出版)中所述的方法进行,或在使用市售的试剂或试剂盒的情况下,根据附随的说明书使用它们。
实施例1 可溶性小鼠Siglec-15蛋白表达构建体的制备
编码小鼠Siglec-15蛋白的胞外结构域的部分核苷酸序列由序列表中的SEQ ID NO: 5表示,其氨基酸序列由序列表中的SEQ ID NO: 6表示。通过利用这样的部分序列,可以在动物细胞等的培养上清液中生产可溶性小鼠Siglec-15蛋白。
a)通过PCR扩增可溶性小鼠Siglec-15基因
按照常规方法,合成具有序列5’-ggggacaagt ttgtacaaaa aagcaggctt caccATGGAG GGGTCCCTCC AACTC-3’ (mSiglec-15-ECD-F: 序列表中的SEQ ID NO: 7)的寡核苷酸和具有序列5’-ggggaccact ttgtacaaga aagctgggtc TCCGGGGGCG CCGTGGAAGC GGAAC-3’ (mSiglec-15-ECD-R: 序列表中的SEQ ID NO: 8)的寡核苷酸,作为通过PCR扩增小鼠Siglec-15胞外结构域cDNA的引物。此外,将这些引物设计成用于生产gateway进入克隆(entry clone)的扩增引物,使得在mSiglec-15-ECD-F中添加attB1序列,以及在mSiglec-15-ECD-R中添加attB2序列。按照常规方法,使用该引物组合和含有小鼠Siglec-15的开放读码框序列的多核苷酸作为模板,进行PCR。热循环仪的条件设定如下:在94℃加热5分钟后,重复“94℃ 0.5分钟、55℃ 0.5分钟以及68℃ 1.5分钟”的温度循环15次,再在68℃加热5分钟,然后在4℃温育。
b)通过Gateway BP反应制备进入克隆
采用以下方法制备进入克隆,其中通过采用λ噬菌体位点特异性的重组系统的Gateway技术(Invitrogen, Inc.)整合了小鼠Siglec-15胞外结构域cDNA。首先,在两端带有在a)中制备的attB序列的PCR产物和pDNOR221 (由Invitrogen, Inc.生产,它是具有attP序列的供体载体)之间,使用BP Clonase进行BP反应。通过使用该反应液,转化大肠杆菌DH10B,针对耐药的克隆进行菌落PCR,并确认插入片段的大小。然后,对已确认具有正确大小的插入片段的克隆,实施插入片段的总DNA序列的序列分析。结果,得到与编码小鼠Siglec-15蛋白的胞外结构域的靶核苷酸序列(序列表中的SEQ ID NO:5)完全相同的进入克隆。
c)通过Gateway LR反应制备表达克隆
采用以下方法制备表达克隆,其中通过采用λ噬菌体位点特异性的重组系统的Gateway技术(Invitrogen, Inc.)整合了小鼠Siglec-15胞外结构域cDNA。在b)中制备的进入克隆含有在两端具有attL序列的插入片段。在该进入克隆和具有attR序列的两类目标载体之间,使用LR Clonase进行LR反应。此外,作为目标载体,使用两类目标载体:设计成在插入片段的C端添加V5表位标签和6×His标签的pDONM,和设计成在插入片段的C端添加人Fc标签的phIgFc。通过使用由LR反应得到的反应液,转化大肠杆菌DH10B,对所得耐药的克隆进行菌落PCR,并确认插入片段的大小。然后,对已确认具有正确大小的插入片段的克隆,进行从插入片段侧到载体侧的两端的序列分析。
用于序列分析的引物序列:
5’-tgcgtgaagg tgcagggcag-3’ (mSiglec-15-ECD-seq-upstm: 序列表中的SEQ ID NO: 9)
和
5’-cctcgcctgg tcgggtc-3’ (mSiglec-15-ECD-seq-dnstm: 序列表中的SEQ ID NO: 10)。
序列分析的结果是:分别得到其中发生pDONM和phIgFc 的正确重组的表达克隆(可溶性小鼠Siglec-15/pDONM和可溶性小鼠Siglec-15/phIgFc)。通过将可溶性小鼠Siglec-15/pDONM转染进动物细胞等中,具有序列表中的SEQ ID NO:11表示的碱基序列的mRNA被转录,并被翻译成具有序列表中的SEQ ID NO:12表示的氨基酸序列的蛋白(小鼠Siglec-15-His)。此外,通过将可溶性小鼠Siglec-15/phIgFc转染进动物细胞等中,具有序列表中的SEQ ID NO:13表示的碱基序列的mRNA被转录,并被翻译成具有序列表中的SEQ ID NO:14表示的氨基酸序列的蛋白(小鼠Siglec-15-Fc)。
实施例2 使用293-F细胞大规模制备含有可溶性小鼠Siglec-15蛋白的培养液
以约5mg的量,分别制备在实施例1中得到的两类表达质粒(可溶性小鼠Siglec-15/pDONM和可溶性小鼠Siglec-15/phIgFc)。此外,在纯化来自大规模培养的大肠杆菌的质粒时,使用Invitrogen PureLink HiPure Plasmid Gigaprep试剂盒(由Invitrogen, Inc.生产)。将如此制备的质粒与Opti-MEM(由Invitrogen, Inc. 生产)混合,将得到的混合物过滤除菌,向其中加入10 ml转染试剂293fectin (由Invitrogen, Inc. 生产),将得到的混合物在室温温育20分钟。向在锥形瓶中培养的FreeStyle 293-F细胞(由Invitrogen, Inc. 生产)中加入该混合物,使在FreeStyle 293表达培养基(由Invitrogen, Inc. 生产)中的细胞密度达到个1.1×106个细胞/ml×5L(1L/烧瓶,5瓶)。在8.0% CO2浓度、在37℃、振荡(125转/分)培养细胞96小时(4日)后,收集培养液,并离心,以制备培养上清液。认为在这样制备的培养上清液中,分别表达其中在小鼠Siglec-15胞外结构域的C端侧已添加V5表位标签和6×His标签的蛋白(小鼠Siglec-15-His)和其中在小鼠Siglec-15胞外结构域的C端侧已添加人Fc标签的蛋白(小鼠Siglec-15-Fc)。
实施例3 小鼠Siglec-15-His的纯化
a)HisTrap HP柱色谱法
向2L在实施例2中制备的表达小鼠Siglec-15-His的293F细胞的培养液中加入225 ml 10 x 缓冲液(500 mM Tris, 1.5 M NaCl, 200 mM 咪唑, pH 8.0),搅拌均匀得到的混合物,并通过Sterivex-GV过滤器(Millipore Co., Ltd.生产)过滤。以2ml/min的流速,将此培养液加载至串联的3根HisTrap HP 5 ml柱(Amersham Biosciences, Inc.生产),所述柱预先用热原去除剂PyroCLEAN(ALerCHEK, Inc.生产)处理,并用注射用蒸馏水洗涤。以1 ml/min的流速,使用60 ml含有300mM NaCl的50 mM Tris-HCl缓冲液(pH 8.0)洗涤该柱,然后使用50 ml含有300 mM NaCl和500 mM 咪唑的50 mM Tris-HCl缓冲液(pH 8.0),以1ml/min的流速,洗脱吸附于柱上的蛋白。在1 ml/级分,将洗脱液分级(fractionate)到预先加入10 μl 10%吐温20的Mini-sorp管(Nunc, Inc.生产)中。使用离心膜浓缩器Amicon Ultra-15 (Millipore Co., Ltd.生产),将约20ml通过合并级分(级分14-20)得到的、含有洗脱蛋白的溶液浓缩至2.5ml,然后将该浓缩液加载至预先用含有0.01%吐温20的磷酸盐缓冲盐水(T-PBS)平衡的PD-10脱盐柱(Amersham Biosciences, Inc.生产),然后用T-PBS洗脱,由此得到3.5ml样品,其溶剂被替换为T-PBS。
b)Resource Q柱色谱法
向3.5ml通过HisTrap HP柱色谱法纯化的、其溶剂被替换为T-PBS的样品中加入22.5 ml含有0.1% CHAPS的50 mM Tris-HCl缓冲液(pH 7.5),并搅拌得到的混合物。然后,将该混合物在4℃、3,000 rpm离心30分钟,除去沉淀物。通过Millex-GV过滤器(Millipore Co., Ltd.生产)过滤得到的上清液后,将滤液以流速1ml/min加载至用含有0.1% CHAPS的50 mM Tris-HCl缓冲液(pH 7.5)预先平衡的Resource Q 6 ml柱 (Amersham Biosciences, Inc.生产)上。此后,使用该缓冲液以流速1ml/min洗涤柱,收集未吸附于柱上的蛋白级分。以流速1ml/min,用含有0.1% CHAPS和1 M NaCl的50 mM Tris-HCl缓冲液(pH 7.5)洗脱吸附于柱上的蛋白。用离心膜浓缩器Amicon Ultra-15 (Millipore Co., Ltd.生产)将26.5ml未吸附于柱上的级分浓缩至2.0ml,然后将该浓缩液在4℃、3,000 rpm离心10分钟,并除去沉淀物。在-80℃冷冻保存离心后的上清液备用。重复上述纯化操作(HisTrap HP柱色谱法和Resource Q柱色谱法)两次。
c)纯化的小鼠Siglec-15-His的检测和纯度分析
使用通过上述纯化操作(HisTrap HP柱色谱法和Resource Q柱色谱法)制备的样品,进行在还原条件下的SDS-聚丙烯酰胺电泳和银染色。也就是说,向5μl经各纯化步骤纯化的每个样品中加入等量的SDS-处理液,在95℃热处理得到的混合物10分钟。将3μl热处理过的各样品用于SDS-聚丙烯酰胺电泳。使用8-25%聚丙烯酰胺梯度凝胶(Amersham Biosciences, Inc.生产) 作为电泳凝胶,并使用PhastSystem (Amersham Biosciences, Inc.生产)进行电泳。此外,使用彩虹分子量标志物 (Amersham Biosciences, Inc.生产)作为分子量标志物。电泳结束后,使用PhastGel Silver试剂盒(Amersham Biosciences, Inc.生产)和PhastSystem进行银染色。结果如图1所示。经证实,在未吸附于Resource Q柱上的蛋白级分中,分子量为约35 kDa的蛋白(小鼠Siglec-15-His)被有效纯化和浓缩。
除使用ECL DualVue蛋白印迹标志物 (Amersham Biosciences, Inc.生产)作为分子量标志物以外,在相同的条件下进行电泳。使用PhastTransfer 半干转移试剂盒(Amersham Biosciences, Inc.生产)和PhastSystem,将凝胶中的蛋白转移(印迹)到PVDF膜 (Hybond-P, Amersham Biosciences, Inc.生产)上。将该PVDF膜在10ml含有0.1%吐温20的封闭剂(BlockAce, Snow Brand Milk Products, Co., Ltd.生产)中在室温轻轻振荡1小时。向该封闭溶液中加入10 μl S-蛋白HRP (Amersham Biosciences, Inc.生产)和10 μl抗-V5-HRP抗体(Pk-TAG-HRP的单克隆抗体,Acris Antibodies生产),将在溶液中的膜在室温轻轻振荡另外1小时。通过在50ml含有0.01%吐温20的磷酸盐缓冲盐水(PBS)中轻轻振荡5分钟,洗涤PVDF膜4次。洗涤后,按照ECL 检测试剂盒(Amersham Biosciences, Inc.生产)所附的方法,处理PVDF膜,以使蛋白条带显色,并用ECL Mini-Camera (Amersham Biosciences, Inc.生产) 和偏光胶片(Polapan 3200B, Polaroid, Inc.生产)检测显色。结果如图2所示。从该结果也可确认,在未吸附于Resource Q柱的蛋白级分中,分子量为约35kDa并与抗-V5-HRP抗体反应的蛋白(小鼠Siglec-15-His)被有效纯化和浓缩。
d)纯化的小鼠Siglec-15-His的蛋白纯度的测定
对于纯化的小鼠Siglec-15-His(未吸附于Resource Q柱的蛋白级分),使用牛血清白蛋白作为标准品,用DC蛋白测定试剂盒(Bio-Rad Laboratories, Inc.生产)测定蛋白浓度。如表1所示,通过进行2次纯化操作,得到合计1.66mg纯化的小鼠Siglec-15-His蛋白。
表1
。
实施例4小鼠Siglec-15-Fc的纯化
a)HiTrap 蛋白A柱色谱法
通过Sterivex-GV过滤器(Millipore Co., Ltd.生产),过滤1.8 L在实施例2中制备的表达小鼠Siglec-15-Fc的293F细胞的培养液,然后以流速5ml/min,将滤液加载至用Dulbecco氏PBS (D-PBS, Invitrogen, Inc.生产)预先平衡的HiTrap蛋白A 5 ml柱 (Amersham Biosciences, Inc.生产)上。以流速5ml/min使用D-PBS洗涤柱后,使用50 ml 0.1 M柠檬酸钠缓冲液(pH 3.0)以流速5ml/min洗脱吸附于柱上的蛋白。在5 ml/级分,将洗脱液分级到mini-sorp管(Nunc, Inc.生产)中,然后立即加入1.3 ml 1 M Tris,以中和洗脱液。使用离心膜浓缩器Amicon Ultra-15 (Millipore Co., Ltd.生产),将通过合并级分(1和2)得到的溶液(其中检测出洗脱的蛋白)浓缩至2.5ml,然后将该浓缩液加载至预先用含有0.01%吐温20的注射用Otsuka生理盐水 (TO-SS, Otsuka Pharmaceutical Co., Ltd.生产)平衡的PD-10脱盐柱 (Amersham Biosciences, Inc.生产),随后用TO-SS洗脱,由此得到3.5ml样品,其溶剂被替换为TO-SS。在-80℃冷冻保存该样品备用。使用2.9L 293F细胞培养液,再重复同样的纯化操作1次。
b)纯化的小鼠Siglec-15-Fc的检测和纯度测定
使用通过上述纯化方法制备的样品,进行在还原条件下的SDS-聚丙烯酰胺电泳和银染色。也就是说,向5μl经各纯化步骤纯化的每个样品中加入等量的SDS-处理液,在95℃加热得到的混合物10分钟。用半浓度的SDS-处理液稀释每种热处理过的样品至1/300或1/900,然后将0.3 μl样品用于SDS-聚丙烯酰胺电泳。用与实施例3的c)中所述的小鼠Siglec-15-His的纯度测定相同的方式,进行电泳和银染色。结果与小规模检查初步纯化条件的结果(应用的培养液的pH为8.9或7.0)一起显示在图3中。经证实,在从HiTrap蛋白A柱洗脱的蛋白级分中,分子量为约55 kDa的蛋白(小鼠Siglec-15-Fc)被有效纯化和浓缩。
c)纯化的小鼠Siglec-15-Fc的蛋白纯度的测定
对于纯化的小鼠Siglec-15-Fc(从PD-10脱盐柱洗脱的蛋白级分),使用牛血清白蛋白作为标准品,用DC蛋白测定试剂盒(Bio-Rad Laboratories, Inc.生产)测定蛋白浓度。如表2所示,通过2次纯化操作,得到合计92mg纯化的小鼠Siglec-15-Fc蛋白。
表2
。
实施例5 生产大鼠抗-小鼠Siglec-15 单克隆抗体的杂交瘤的建立
a)抗原的制备
以100 μg/0.5 ml制备在实施例3中生产的小鼠Siglec-15-His蛋白,向其中加入等量的佐剂,并使用玻璃注射器制成乳状液。作为佐剂,仅在初次免疫时使用弗氏完全佐剂(FCA, Difco Laboratories, Inc.生产),第二次及后续免疫使用弗氏不完全佐剂(FICA, Difco Laboratories, Inc.生产)。
b)大鼠的免疫
使用4只大鼠(Wistar,雌性,6周龄,购自CLEA Japan, Inc.)作为免疫动物。使用27G注射针,皮下和真皮内注射在a)中得到的乳状液,使抗原的量为50μg/大鼠。第一次免疫后,每7天进行一次免疫,共进行4次。在第4次免疫后7天,从尾静脉少量(200μl)采血,制备抗血清。为了确认抗血清的抗体滴度,进行ELISA,其中使用已经用作抗原的小鼠Siglec-15-His蛋白、在实施例4中制备的小鼠Siglec-15-Fc蛋白或牛血清白蛋白(BSA),它们各自被固相化。结果,在所有4只大鼠(大鼠编号1~4)中,均观察到与小鼠Siglec-15-His蛋白和小鼠Siglec-15-Fc蛋白的反应性。另一方面,未观察到与BSA的反应性。从这些结果可以确认,每个免疫的大鼠的血清中的抗体滴度升高,因此对表现出最高抗体滴度的2号大鼠进行细胞融合操作。
c)细胞融合
按照融合小鼠(大鼠)脾脏细胞和骨髓瘤细胞的普通方法,进行细胞融合。在乙醚麻醉下,从大鼠心脏收集全血,并使大鼠安乐死,然后摘除脾脏。使用聚乙二醇(PEG),对采集的脾细胞和P3X63Ag8.653细胞(ATCC CRL 1580)(小鼠骨髓瘤细胞)进行细胞融合。将得到的细胞接种于96孔板中,并向其中加入含有次黄嘌呤(H)、氨基蝶呤(A)和胸苷(T)的培养基(HAT选择培养基),然后培养细胞7~10天。从确认其中通过细胞融合得到的杂交瘤存活的61个孔中收集培养上清液。然后,使用已经用作抗原的小鼠Siglec-15-His蛋白、在实施例4制备的小鼠Siglec-15-Fc蛋白或BSA(它们各自被固定化),通过ELISA评价抗体滴度,并筛选生产抗-小鼠Siglec-15 单克隆抗体的杂交瘤。根据筛选的结果,选出表现出高抗体滴度的12个孔,对孔中所含的杂交瘤进行克隆操作。
d)杂交瘤的克隆化
通过有限稀释法,对如此选出的杂交瘤进行第一个克隆步骤。有限稀释后,将杂交瘤培养2周,通过使用在实施例4中制备的小鼠Siglec-15-Fc蛋白或BSA(它们各自被固定化)的ELISA,证实培养上清液中的抗体滴度。对于已确认为阳性克隆的11个克隆,进行第二个克隆步骤 (采用与第一个克隆步骤相同的方式),由此最终建立生产抗-小鼠Siglec-15 单克隆抗体的杂交瘤的10个克隆(#1A1、#3A1、#8A1、#24A1、#32A1、#34A1、#39A1、#40A1、#41B1、#61A1)。此外,于2008年8月28日,将杂交瘤#32A1保藏在独立行政法人产业技术综合研究所特许生物寄托中心(International Patent Organism Depositary of the National Institute of Advanced Industrial Science and Technology,位于:Central 6, 1-1-1 Higashi, Tsukuba-shi, Ibaraki-ken, 305-8566, Japan),并已经在抗-Siglec-15 杂交瘤#32A1的名称下获得登录号FERM BP-10999。
实施例6 大鼠抗-小鼠Siglec-15 单克隆抗体的制备
a)裸小鼠腹水的制备
使用含有10% FCS的TIL培养基I (Immuno-biological Laboratories Co., Ltd.生产)培养基,培养在实施例5中建立的杂交瘤。通过下述方法进行细胞的继代培养:其中使用细胞生长至约5 x 105个细胞/ml的时间点作为指标,每2~3天将培养液稀释至约1/4。以1 x 107个细胞/小鼠,给预先腹膜内施用(0.2ml/小鼠)姥鲛烷的裸小鼠腹膜内地植入每种如此培养的杂交瘤。在植入中,10个杂交瘤克隆各使用3只裸小鼠。植入后,在观察到足够的腹水蓄积时,采集腹水,将其与从另外2只植入相同杂交瘤的小鼠采集的腹水合并,测定如此合并的腹水的量,冷冻保存腹水,直至抗体纯化。各杂交瘤的腹水采集量总结于表3中。
表3
。
b)抗体的纯化
使用20ml 蛋白G柱(GE Healthcare, Co., Ltd.生产),对采集的腹水总量进行IgG纯化。通过凝胶过滤分析(Superdex 200柱色谱法),对纯化的IgG进行纯度测定,对一部分抗体进行离心膜浓缩。也就是说,在纯化的抗-小鼠Siglec-15 单克隆抗体中,通过用离心膜浓缩器Amicon Ultra-15 (Millipore Co., Ltd.生产)在3,000 rpm、4℃离心抗体30~60分钟,将除#24A1抗体以外的9类抗体浓缩至原始体积的约1/6-1/8。接着,使用牛血清白蛋白(BSA)作为标准品,对于#24A1抗体和其它浓缩的9类抗体,用DC蛋白测定试剂盒(Bio-Rad Laboratories, Inc.生产)测定蛋白浓度。通过以上操作,制备抗-小鼠Siglec-15 单克隆抗体。
实施例7 评价大鼠抗-小鼠Siglec-15 单克隆抗体结合小鼠Siglec-15蛋白的性质
通过ELISA法,评价大鼠抗-小鼠Siglec-15 单克隆抗体结合小鼠Siglec-15蛋白的性质。用0.1M碳酸钠缓冲液(pH 9.5),将在实施例4中制备的小鼠Siglec-15-Fc蛋白稀释至5 μg/ml,将得到的溶液以100μl/孔加入96孔板(Nalge Nunc International, Inc.生产, 目录号430341)。在室温放置板1小时后,除去溶液,以300μl/孔加入洗涤缓冲液(含有0.05%吐温20的磷酸盐缓冲盐水),再除去。将该洗涤操作再进行一次后,在200μl/孔加入含有25% BlockAce (Dainippon Sumitomo Pharma Co., Ltd.生产)的磷酸盐缓冲盐水,将板在室温放置1小时,由此进行封闭。除去液体,用300μl/孔的洗涤缓冲液洗涤该板2次。然后,使用ELISA缓冲液(含有12.5% BlockAce和0.05%吐温20的磷酸盐缓冲盐水),将在实施例6中制备的每种大鼠抗-小鼠Siglec-15 单克隆抗体或大鼠对照IgG (R&D Systems, Inc.生产) 稀释至1.28~20,000ng/ml的终浓度(5倍稀释系列),并将得到的稀释的抗体溶液以100μl/孔添加至板中。将该板在室温放置1小时后,除去液体,用300μl/孔的洗涤缓冲液洗涤该板3次。接着,以100μl/孔,添加用ELISA缓冲液稀释至1,000倍的HRP (辣根过氧化物酶)-标记的山羊抗-大鼠IgG抗体(Beckman Coulter, Inc.生产),将该板在室温放置1小时。除去溶液,用300μl/孔的洗涤缓冲液洗涤3次,然后使用过氧化物酶用显色试剂盒 (Sumitomo Bakelite Co., Ltd.生产),按照试剂盒所附的说明书,进行显色。显色后,用微量培养板读数器 (Nihon Molecular Devices Corporation生产)测定在492nm的吸光度。结果证实:所有检查的10个大鼠抗-小鼠Siglec-15 单克隆抗体测试样品都以抗体浓度依赖性的方式结合小鼠Siglec-15蛋白(图4)。另一方面,在大鼠对照IgG的情况下,未观察到与小鼠Siglec-15蛋白的结合。
实施例8 小鼠骨髓非贴壁细胞的制备
从5~8周龄的雄性ddY小鼠取出股骨和胫骨,并除去软组织。切除股骨或胫骨的两端,用带有25号注射针的注射器注射D-PBS以推出骨髓细胞,收集在离心管中。在室温、以100g离心5分钟,并去除上清液。向得到的细胞沉淀物中加入1ml 溶血缓冲液 (红细胞溶解缓冲液,Sigma Co., Ltd.生产)以混悬,将得到的悬浮液在室温放置5分钟。向其中加入20ml D-PBS,并在室温以100g离心悬浮液5分钟,去除上清液。向得到的细胞沉淀物中加入10ml含有5 ng/ml M-CSF (R&D Systems, Inc.生产)和10%胎牛血清 (FBS)的MEM-α培养基(由Invitrogen, Inc. 生产)以混悬。然后,使得到的悬浮液通过细胞过滤网(cell strainer )(40μm尼龙,BD Falcon生产)以除去凝集物。将得到的细胞转移至75 cm2 T-培养瓶 (用于贴壁细胞的附着)中,在CO2培养箱中培养过夜。培养过夜后,收集未附着在T-培养瓶上的细胞,用作小鼠骨髓非贴壁细胞。
实施例9基于小鼠破骨细胞形成试验评价大鼠抗-小鼠Siglec-15 单克隆抗体的生物活性
使用在实施例6中制备的所有10个抗-小鼠Siglec-15 单克隆抗体测试样品,检测对小鼠骨髓非贴壁细胞的破骨细胞分化的影响。在含有10% FBS和10 ng/ml M-CSF (R&D Systems, Inc.生产)的α-MEM培养基中,将通过实施例8的方法制备的小鼠骨髓非贴壁细胞制成1.5×105个细胞/ml,并将得到的细胞制品以200μl的量接种在 96孔板的每个孔中,在CO2培养箱中培养细胞2天。除去96孔板中旧的培养液,向每个孔添加100μl MEM-α培养基,所述100μl MEM-α培养基含有 10% FBS,其中已经添加了人RANKL (RANKL, Peprotech, Inc.生产)和M-CSF,终浓度分别为20 ng/ml和10 ng/ml。向该细胞培养液中以32-1,000 ng/ml的浓度添加在实施例6中制备的每种大鼠抗-小鼠Siglec-15 单克隆抗体、从市售的大鼠对照IgG(纯化的大鼠IgG, R&D Systems, Inc.生产)通过除去叠氮化钠所得到的样品、或单独制备的兔抗-小鼠Siglec-15 多克隆抗体(No. 3),在CO2培养箱中培养细胞另外3天。此外,多克隆抗体(No. 3)是已经在该实施例所述的实验系统中证实会抑制破骨细胞形成的抗体。培养结束后,通过下述方法测定形成的破骨细胞的酒石酸盐-抗性的酸性磷酸酶(TRAP)的活性。通过抽吸,去除96-孔板的每个孔中的培养液,向每个孔中加入50 μl 含有1% Triton X-100的50 mM柠檬酸钠缓冲液(pH 6.1)。然后,在板振荡器上振荡板5分钟以裂解细胞。向每个孔中加入50μl底物溶液(50 mM柠檬酸钠缓冲液(pH 6.1),其含有5 mg/ml 对硝基苯基磷酸酯和0.46%酒石酸钠),并将该板在室温温育5分钟。温育后,向96孔板的各个孔中加入50μl 1N氢氧化钠溶液以停止酶反应。酶反应停止后,测定各个孔在405nm的吸光度,得到的测量结果用作TRAP活性的指标。结果如图5和6所示。在市售的大鼠对照IgG的情况下,未观察到TRAP活性的显著抑制。另一方面,在加入32ng/ml至1000ng/ml的范围内的#32A1抗体的情况下,和在加入63 ng/ml至1000 ng/ml的范围内的#8A1抗体和亲和纯化的兔多克隆No. 3抗体的情况下,观察到TRAP活性的显著抑制。还在#3A1抗体、#34A1抗体和#39A1抗体的情况下,在500ng/ml或以上的相对高浓度时,观察到TRAP活性的剂量依赖性的抑制。未观察到其它抗体对小鼠破骨细胞形成的抑制。根据以上结果,在制备的大鼠抗-小鼠Siglec-15 单克隆抗体中,发现了强烈抑制小鼠破骨细胞形成 (破骨细胞分化和成熟)的抗体。此外,作为#3A1抗体、#8A1抗体、#32A1抗体、#34A1抗体和#39A1抗体的共有性质,可以例举在1000ng/ml(即1μg/ml)或以下浓度时抑制破骨细胞形成的活性。
实施例10可溶性人Siglec-15蛋白表达构建体的制备
编码人Siglec-15蛋白的胞外结构域的部分核苷酸序列由序列表中的SEQ ID NO: 15表示,其氨基酸序列由序列表中的SEQ ID NO: 16表示。通过利用这样的部分序列,可以在动物细胞等的培养上清液中生产可溶性人Siglec-15蛋白。
a) 通过PCR扩增可溶性人Siglec-15基因
按照常规方法,合成具有序列5’-ggggacaagt ttgtacaaaa aagcaggctt caccATGGAA AAGTCCATCT GGCTGC-3’ (hSiglec-15-ECD-F: 序列表中的SEQ ID NO: 17)的寡核苷酸和具有序列5’-ggggaccact ttgtacaaga aagctgggtc CCCGCTGGCG CCATGGAAGC GG-3’ (hSiglec-15-ECD-R: 序列表中的SEQ ID NO: 18)的寡核苷酸,作为通过PCR扩增人Siglec-15胞外结构域的引物。此外,将这些引物设计成用于生产gateway进入克隆的扩增引物,使得在hSiglec-15-ECD-F中添加attB1序列,以及在hSiglec-15-ECD-R中添加attB2序列。按照常规方法,使用该引物组合和含有人Siglec-15的开放读码框序列的多核苷酸作为模板,进行PCR。使用PureLink PCR纯化试剂盒(由Invitrogen, Inc. 生产),纯化得到的PCR反应液。
b)通过Gateway BP反应制备进入克隆
采用以下方法制备进入克隆,其中通过采用λ噬菌体位点特异性的重组系统的Gateway技术(Invitrogen, Inc.),在进入克隆中整合了人Siglec-15胞外结构域cDNA。首先,在两端带有在a)中制备的attB序列的PCR产物和pDNOR221 (由Invitrogen, Inc.生产,它是具有attP序列的供体载体)之间,使用BP Clonase进行BP反应。通过使用该反应液,转化大肠杆菌TOP10,针对耐药的克隆进行菌落PCR,并确认插入片段的大小。然后,对已确认具有正确大小的插入片段的克隆,实施插入片段的总DNA序列的序列分析。结果,得到与编码人Siglec-15蛋白的胞外结构域的靶核苷酸序列(序列表中的SEQ ID NO:15)完全相同的进入克隆。
c)通过Gateway LR反应制备表达克隆
采用以下方法制备表达克隆,其中通过采用λ噬菌体位点特异性的重组系统的Gateway技术(Invitrogen, Inc.),在进入克隆中整合了人Siglec-15胞外结构域cDNA。在b)中制备的进入克隆含有在两端具有attL序列的插入片段。在该进入克隆和具有attR序列的两类目标载体之间,使用LR Clonase进行LR反应。此外,作为目标载体,使用两类目标载体:设计成在插入片段的C端添加V5表位标签和6×His标签的pDONM,和设计成在插入片段的C端添加人Fc标签的phIgFc。通过使用由LR反应得到的反应液,转化大肠杆菌TOP10,并对所得耐药的克隆进行序列分析,以确认是否发生正确的重组。
序列分析的结果是:分别得到其中发生pDONM和phIgFc的正确重组的表达克隆(可溶性人Siglec-15/pDONM和可溶性人Siglec-15/phIgFc)。通过将可溶性人Siglec-15/pDONM转染进动物细胞等中,具有序列表中的SEQ ID NO:19表示的碱基序列的mRNA被转录,并被翻译成具有序列表中的SEQ ID NO:20表示的氨基酸序列的蛋白(人Siglec-15-His)。此外,通过将可溶性人Siglec-15/phIgFc转染进动物细胞等中,具有序列表中的SEQ ID NO:21表示的碱基序列的mRNA被转录,并被翻译成具有序列表中的SEQ ID NO:22表示的氨基酸序列的蛋白(人Siglec-15-Fc)。
实施例11使用293-F细胞大规模制备含有可溶性人Siglec-15蛋白的培养液
a) 含有人Siglec-15-His的培养液的制备
以约25mg的量,制备在实施例10中得到的可溶性人Siglec-15/pDONM。此外,在纯化来自大规模培养的大肠杆菌的质粒时,使用Invitrogen PureLink HiPure Plasmid Gigaprep试剂盒(由Invitrogen, Inc.生产)。将如此制备的质粒与Opti-MEM(由Invitrogen, Inc. 生产)混合,向其中加入50 ml转染试剂293fectin(由Invitrogen, Inc. 生产),将得到的混合物在室温温育20分钟。将该混合物添加至FreeStyle 293-F细胞(由Invitrogen, Inc. 生产),所述细胞在含有1% 青霉素-链霉素的FreeStyle 293 表达培养基(由Invitrogen, Inc. 生产)中培养,使得细胞密度达到1.0-3.4 x 106个细胞/ml,其中使用25 L生物过程培养装置 (WAVE生物反应器)。在6-12% CO2浓度、在37℃、振荡(30转/分)培养细胞96小时(4日)后,收集培养液,并离心,以制备培养上清液。观察到在这样制备的培养上清液中,表达其中在人Siglec-15胞外结构域的C端侧已添加V5 表位标签和6 x His标签的蛋白(人Siglec-15-His)。
b) 含有人Siglec-15-Fc的培养液的制备
以约5mg的量,制备在实施例10中得到的可溶性人Siglec-15/phIgFc。此外,在纯化来自大规模培养的大肠杆菌的质粒时,使用Invitrogen PureLink HiPure Plasmid Gigaprep试剂盒(由Invitrogen, Inc.生产)。将如此制备的质粒与Opti-MEM(由Invitrogen, Inc. 生产)混合,然后过滤除菌。然后,向其中加入10 ml转染试剂293fectin(由Invitrogen, Inc. 生产),将得到的混合物在室温温育20分钟。将该混合物加给在锥形瓶中培养的FreeStyle 293-F细胞(由Invitrogen, Inc. 生产),使得在FreeStyle 293 表达培养基(由Invitrogen, Inc. 生产)中的细胞密度达到1.0-3.0 x 106个细胞/ml x 5 L (1 L/培养瓶 x 5瓶)。在8.0% CO2浓度、在37℃、振荡(125转/分)培养细胞96小时(4日)后,收集培养液,并离心,以制备培养上清液。观察到在这样制备的培养上清液中,表达其中在人Siglec-15胞外结构域的C端侧已添加人Fc标签的蛋白(人Siglec-15-Fc)。
实施例12 可溶性人Siglec-15蛋白的纯化
a)可溶性人Siglec-15-His的纯化
a-i) HisTrap HP柱色谱法
向12L在实施例11的a)中制备的表达人Siglec-15-His的293F细胞的培养液中加入1350 ml 10 x缓冲液(500 mM Tris, 1.5 M NaCl, 200 mM 咪唑, pH 8.0),将得到的混合物搅拌均匀,并用MilliPak-60过滤器(Millipore Co., Ltd.生产)过滤。以10ml/min的流速,将该培养液加载至预先用纯水(Milli Q水)洗涤的Ni-琼脂糖 HP (Amersham Biosciences, Inc.生产) 100 ml柱。用400ml含有300mM NaCl的50 mM Tris-HCl缓冲液(pH 8.0)以8mL/min的流速洗涤柱后,用200mL含有300 mM NaCl和500 mM 咪唑的50 mM Tris-HCl缓冲液(pH 8.0)以2.5mL/min的流速洗脱吸附在柱上的蛋白,将洗脱液分级在mini-sorp管(Nunc, Inc.生产)中。为防止蛋白沉淀,向约40mL含有洗脱的蛋白的级分中加入8mL 5M NaCl溶液,随后搅拌,然后使用离心膜浓缩器Amicon Ultra-15 (Millipore Co., Ltd.生产),将得到的混合物浓缩至约20 ml。通过在3000 rpm、在4℃离心30分钟,除去浓缩时产生的不溶物,将2.5mL得到的上清液加载至预先用含有1M NaCl的磷酸盐缓冲盐水(N-PBS)平衡的PD-10脱盐柱 (Amersham Biosciences, Inc.生产),随后用N-PBS洗脱,由此得到溶剂被替换为N-PBS的3.5mL样品。将该操作重复另外7次,得到约28mL部分纯化的人Siglec-15-His溶液。
a-ii) Resource Q柱色谱法
在含有0.1% CHAPS的50 mM Tris-HCl缓冲液(pH 7.5) (1 L, 3次)中,在4℃透析12ml样品过夜,所述样品已经通过Ni-琼脂糖 HP柱色谱法纯化,且其溶剂已经被替换为N-PBS,将得到的透析液在4℃、在3,000rpm离心30分钟,除去沉淀。用Millex-GV过滤器(Millipore Co., Ltd.生产)过滤得到的上清液以后,以1ml/min的流速,将滤液加载至预先用含有0.1% CHAPS的50mM Tris-HCl缓冲液(pH7.5)平衡的Resource Q 6 ml柱 (Amersham Biosciences, Inc.生产)。此后,以1ml/min的流速洗柱,用该缓冲液洗涤柱,并收集未吸附在柱上的蛋白级分。以1ml/min的流速,用含有0.1% CHAPS、1M NaCl的50mM Tris-HCl缓冲液(pH7.5)洗脱吸附在柱上的蛋白。用离心膜浓缩器Amicon Ultra-15 (Millipore Co., Ltd.生产)将26.5ml未吸附在柱上的级分浓缩至3.0ml后,在4℃、3,000rpm离心浓缩液10分钟,并除去沉淀。将2.5mL得到的上清液加载至预先用含有50mM 盐酸精氨酸的磷酸盐缓冲盐水(pH 7.0, A-PBS)平衡的PD-10脱盐柱 (Amersham Biosciences, Inc.生产),然后用A-PBS洗脱,由此得到溶剂被替换为A-PBS的3.5mL样品。为防止可溶性人Siglec-15-His沉淀,在制备的样品溶剂中加入了盐酸精氨酸。将离心后的上清液在-80℃冷冻保存备用。重复以上的纯化操作(Resource Q柱色谱法) 2次。
a-iii)纯化的人Siglec-15-His的检测和纯度测定
使用通过上述纯化操作(Ni-琼脂糖 HP柱色谱法和Resource Q柱色谱法)制备的样品,进行在还原条件下的SDS-聚丙烯酰胺电泳和银染色。也就是说,向5μl通过各自纯化步骤纯化的每个样品中加入等量的SDS-处理液,将得到的混合物在95℃热处理10分钟。将0.3μl热处理后的各样品用于SDS-聚丙烯酰胺电泳。除了使用彩虹分子量标志物(Amersham Biosciences, Inc.生产)作为分子量标志物以外,以与实施例3相同的方式进行电泳操作。电泳结束后,用PhastGel Silver试剂盒(Amersham Biosciences, Inc.生产)和PhastSystem进行银染色。结果如图7所示。显示在未吸附在Resource Q柱上的蛋白级分中,分子量为约35kDa的蛋白(人Siglec-15-His)被有效纯化并浓缩。
a-iv)纯化的人Siglec-15-His的蛋白浓度的测定
对于纯化的人Siglec-15-His(未吸附在Resource Q柱上的蛋白级分),使用牛血清白蛋白作为标准品,用DC蛋白测定试剂盒(Bio-Rad Laboratories, Inc.生产)测定蛋白浓度。通过2次纯化操作,共得到1.66mg纯化的人Siglec-15-His。
b)可溶性人Siglec-15-Fc的纯化
b-i)HiTrap 蛋白A柱色谱法
通过Sterivex-GV过滤器(Millipore Co., Ltd.生产),过滤1.5 L在实施例11的b)中制备的表达人Siglec-15-Fc的293F细胞的培养液,然后以5ml/min的流速,将滤液加载至预先用Dulbecco氏PBS (D-PBS, Invitrogen, Inc.生产)平衡的HiTrap蛋白A 5 ml柱 (Amersham Biosciences, Inc.生产)中。用70ml D-PBS以5ml/min的流速洗涤柱后,用24 ml 0.1 M柠檬酸钠缓冲液(pH 3.0)以1.2ml/min的流速洗脱吸附在柱上的蛋白。以1.2 ml/级分,将洗脱液分级在mini-sorp管(Nunc, Inc.生产)中,然后立即向其中加入0.31ml 1M Tris以中和洗脱液。将通过合并洗脱的蛋白级分(级分5~9)所得到的溶液(约7.5 ml)的2.5 ml 等分试样加载至预先用含有50mM 盐酸精氨酸的磷酸盐缓冲盐水(pH 7.0, A-PBS)平衡的PD-10脱盐柱 (Amersham Biosciences, Inc.生产)中,然后用A-PBS洗脱,由此得到溶剂被替换为A-PBS的3.5ml样品。将该操作重复2次。为了防止可溶性人Siglec-15-Fc沉淀,在溶剂中添加了盐酸精氨酸。将洗脱的蛋白级分(级分5~9)的2.5ml剩余溶液加载至预先用含有1M NaCl的磷酸盐缓冲盐水(pH6.7、N-PBS)平衡的PD-10脱盐柱 (Amersham Biosciences, Inc.生产)中,然后用N-PBS洗脱,由此得到溶剂被替换为N-PBS的3.5ml样品。为了防止可溶性人Siglec-15-Fc沉淀,在制备的样品的溶剂中添加NaCl,而不添加诸如精氨酸等含有氨基的化合物。将通过以上操作所制备的样品在-80℃冷冻保存备用。
b-ii)纯化的人Siglec-15-Fc的检测和纯度测定
使用通过上述纯化操作制备的样品,进行在还原条件下的SDS-聚丙烯酰胺电泳和银染色。也就是说,向5μl通过各自纯化步骤纯化的每个样品中加入等量的SDS-处理液,将得到的混合物在95℃加热10分钟。用半浓度的SDS-处理液稀释每种热处理过的样品至1/100或1/300,将0.3 μl得到的样品用于SDS-聚丙烯酰胺电泳。以与在a-iii)中所述的人Siglec-15-His的纯度测定相同的方法,进行电泳和银染色。结果如图8所示。显示在从HiTrap 蛋白A柱洗脱的蛋白级分中,分子量为约55kDa的蛋白(人Siglec-15-Fc)被有效纯化并浓缩。
b-iii)纯化的人Siglec-15-Fc的蛋白浓度的测定
对于纯化的人Siglec-15-Fc(从PD-10脱盐柱洗脱的蛋白级分),使用牛血清白蛋白作为标准品,用DC蛋白测定试剂盒(Bio-Rad Laboratories, Inc.生产)测定蛋白浓度。如表4所示,通过2次纯化操作,共得到25.2mg纯化的人Siglec-15-Fc。
表4
。
实施例13 大鼠抗-小鼠Siglec-15 单克隆抗体结合人Siglec-15蛋白的性质的评价
通过ELISA法,评价了大鼠抗-小鼠Siglec-15 单克隆抗体结合人Siglec-15蛋白的性质。使用0.1M碳酸钠缓冲液(pH9.5)将在实施例12的b)中制备的人Siglec-15-Fc蛋白(进行A-PBS的替换)稀释至5μg/ml,将得到的溶液以100μl/孔添加于96-孔板 (Nalge Nunc International, Inc.生产, 目录号430341)中。在室温放置该板1小时后,除去溶液,以300μl/孔添加洗涤缓冲液(含有0.05%吐温20的磷酸盐缓冲盐水),然后除去。将该洗涤操作再进行1次后,以200μl/孔添加含有25% BlockAce (Dainippon Sumitomo Pharma Co., Ltd.生产)的磷酸盐缓冲盐水,在室温放置该板1小时,由此进行封闭。除去液体,用300μl/孔的洗涤缓冲液洗涤板2次。然后,使用ELISA缓冲液(含有12.5% BlockAce和0.05%吐温20的磷酸盐缓冲盐水),将在实施例6中制备的每种大鼠抗-小鼠Siglec-15 单克隆抗体或大鼠对照IgG (R&D Systems, Inc.生产)稀释至1.28~20,000ng/ml的终浓度(5倍稀释系列),将得到的稀释的抗体溶液以100μl/孔添加至板中。在室温放置该板1小时后,除去液体,用300μl/孔的洗涤缓冲液洗涤板3次。随后,以100μl/孔,添加用ELISA缓冲液稀释至1,000倍的HRP (辣根过氧化物酶)-标记的山羊抗-大鼠IgG抗体(Beckman Coulter, Inc.生产),在室温放置该板1小时。除去液体,用300μl/孔的洗涤缓冲液洗涤该板3次,然后,通过使用过氧化物酶用显色试剂盒(Sumitomo Bakelite Co., Ltd.生产),按照试剂盒所附说明书进行显色。显色后,用微量培养板读数器 (Nihon Molecular Devices Corporation生产)测定在492nm处的吸光度。结果证实:检测的大鼠抗-小鼠Siglec-15 单克隆抗体的所有10个测试样品都以抗体浓度依赖性的方式结合人Siglec-15蛋白(图9)。具体地,5个测试样品(即#1A1、#3A1、#24A1、#32A1和#61A1)的结合活性较高,3个测试样品(即#8A1、#34A1和#39A1)的结合活性相对较低。另一方面,在大鼠对照IgG的情况下,未观察到与人Siglec-15蛋白的结合。以上结果表明:在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体不仅结合小鼠Siglec-15,而且也结合人Siglec-15,此外,发现有些抗体强烈结合人Siglec-15。
实施例14 添加大鼠抗-小鼠Siglec-15 单克隆抗体对正常人破骨细胞前体细胞的细胞融合和骨吸收活性的影响(体外生物活性评价)
由于在实施例13中证实大鼠抗-小鼠Siglec-15 单克隆抗体也结合人Siglec-15,因此检测这些抗体对人破骨细胞形成和骨吸收活性的影响。
a) 添加大鼠抗-小鼠Siglec-15 单克隆抗体对来自正常人破骨细胞前体细胞的破骨细胞的细胞融合的影响(TRAP染色)
按照细胞所附的说明书,以1×104细胞/孔,将正常人破骨细胞前体细胞(正常人天然破骨细胞前体细胞,购自Sanko Junyaku Co., Ltd., 目录号2T-110)接种于96孔板中。使用添加了OPGM补充套装 (购自Sanko Junyaku Co., Ltd., 目录号PT-9501)的破骨细胞前体细胞用最低必需培养基(OPBM, 购自Sanko Junyaku Co., Ltd., 目录号PT-8201)作为培养基,所述OPGM补充套装含有胎牛血清(终浓度10%)、人RANKL(终浓度66 ng/ml)、人M-CSF(终浓度33 ng/ml)等。向得到的培养上清液中添加在实施例6中制备的每种大鼠抗-小鼠Siglec-15 单克隆抗体或大鼠对照IgG (R&D Systems, Inc.生产)以得到终浓度为30μg/ml,在CO2培养箱中培养细胞4天。培养后,除去上清液,添加10%中性福尔马林以固定细胞。固定细胞后,用蒸馏水洗涤细胞2次,以100μl/孔添加TRAP染色液(0.27mM萘酚AS-MX磷酸盐(Sigma Co., Ltd.生产)、1.6mM 固红紫色LB 盐(Sigma Co., Ltd.生产)、1%二甲基甲酰胺、50mM酒石酸钠、0.1M醋酸钠缓冲液(pH5.0)),在室温反应5分钟。反应后,用蒸馏水洗涤细胞2次,然后通过显微术观察(图10)。结果,通过添加#32A1抗体,几乎完全抑制由高度细胞融合所导致的巨大破骨细胞的形成。此外,在#41B1抗体的情况下,也显著抑制由高度细胞融合所导致的巨大破骨细胞的形成。另一方面,在其它大鼠抗-小鼠Siglec-15 单克隆抗体(#1A1抗体和其它抗体)和大鼠对照IgG的情况下,未观察到这种对破骨细胞细胞融合的显著抑制。以此方式,证实了来自正常人破骨细胞前体细胞的TRAP-阳性的破骨细胞的多核化和细胞融合会被特异性地结合Siglec-15蛋白的单克隆抗体抑制。
b) 添加大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1)对来自正常人破骨细胞前体细胞的破骨细胞的细胞融合的影响(TRAP染色)
按照细胞所附的说明书,以1×104个细胞/孔,将正常人破骨细胞前体细胞(正常人天然破骨细胞前体细胞,购自Sanko Junyaku Co., Ltd., 目录号2T-110)接种于96孔板中。使用添加了OPGM补充套装 (购自Sanko Junyaku Co., Ltd., 目录号PT-9501)的破骨细胞前体细胞用最低必需培养基(OPBM, 购自Sanko Junyaku Co., Ltd., 目录号PT-8201)作为培养基,所述OPGM补充套装含有胎牛血清(终浓度10%)、人RANKL(终浓度68.4 ng/ml)、人M-CSF(终浓度33 ng/ml)等。向得到的培养上清液中添加在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体),得到0.1、0.3、1或3 μg/ml的终浓度,在CO2培养箱中培养细胞3天。培养后,除去上清液,添加10%中性福尔马林以固定细胞。固定细胞后,用蒸馏水洗涤细胞2次,以100μl/孔,添加TRAP染色液(0.27mM萘酚AS-MX磷酸盐(Sigma Co., Ltd.生产)、1.6mM 固红紫色LB 盐(Sigma Co., Ltd.生产)、1%二甲基甲酰胺、50mM酒石酸钠、0.1M醋酸钠缓冲液(pH5.0)),在室温反应5分钟。反应后,用蒸馏水洗涤细胞2次,然后通过显微术观察(图11)。结果,#32A1抗体在0.3μg/ml至3μg/ml的范围内以浓度依赖性的方式抑制TRAP-阳性的多核破骨细胞的形成。
c) 添加大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1)对正常人破骨细胞前体细胞的骨吸收活性的影响(使用胶原-包被的板的评价)
已知破骨细胞会释放诸如组织蛋白酶K等蛋白酶,并降解作为骨组织的构成成分的I型胶原。OsteoLyse测定试剂盒(Lonza, Inc.生产, 目录号PA-1500)提供了涂布有铕-缀合的人胶原的96孔板(96-孔OsteoLyse细胞培养板),通过测定当在板上培养破骨细胞时释放到上清液中的荧光胶原片段的量,可能在体外评价破骨细胞的骨吸收活性。按照细胞所附的说明书,以1×104个细胞/孔,将正常人破骨细胞前体细胞(正常人天然的破骨细胞前体细胞, 购自Sanko Junyaku Co., Ltd., 目录号2T-110)接种于96-孔OsteoLyse细胞培养板中。使用添加了OPGM补充套装 (购自Sanko Junyaku Co., Ltd., 目录号PT-9501)的破骨细胞前体细胞用最低必需培养基(OPBM, 购自Sanko Junyaku Co., Ltd., 目录号PT-8201)作为培养基,所述OPGM补充套装含有胎牛血清(终浓度10%)、人RANKL(终浓度68.4 ng/ml)、人M-CSF(终浓度33 ng/ml)等。向得到的培养上清液中添加在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体),得到0.1、0.3、1或3 μg/ml的终浓度,在CO2培养箱中培养细胞3天。收集培养上清液的10 μl 等分试样,向其中添加200 μl 包含在OsteoLyse测定试剂盒中的荧光团释放试剂,使用荧光板读数器 (ARVO MX, Perkin Elmer Inc.生产)测定荧光强度(激发: 340 nm, 发射: 615 nm),由此测定在培养上清液中释放的游离荧光胶原片段的量 (图12)。结果,#32A1抗体在0.3μg/ml至3μg/ml的范围内,以浓度依赖的方式减少通过添加RANKL所增加的荧光胶原片段的量。该结果表明:人破骨细胞的骨吸收活性被研究的特异性地结合Siglec-15蛋白的单克隆抗体抑制。
实施例15
使用卵巢切除的大鼠评价大鼠抗-小鼠Siglec-15 单克隆抗体的生物活性
a) 动物实验方法
从12周龄的雌性F344大鼠(获自Charles River Laboratories Japan, Inc.)取出双侧卵巢,并将大鼠分成3组:载体施用组;大鼠抗-小鼠Siglec-15 单克隆抗体#8A1施用组;和大鼠抗-小鼠Siglec-15 单克隆抗体#32A1施用组。此外,将一组制成假手术组。在抗体施用组中,从手术次日,以1 mg/kg的剂量,腹膜内地施用在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#8A1或大鼠抗-小鼠Siglec-15 单克隆抗体#32A1,每周3次,重复4周。在载体施用组和假手术组中,腹膜内地施用含有0.01%吐温20的PBS作为载体。在开始施用后4周,在禁食条件下收集尿24小时,并在-80ºC储存尿样品,直到测量。尿收集结束后,将大鼠安乐死,并从每只大鼠切离腰椎。
b) 腰椎骨矿物质密度的测量
去除与切离的腰椎相连的软组织,并提取第4至第6腰椎。将提取的腰椎通过在乙醇中振荡来脱脂并脱水,然后风干,使用骨密度计(DCS-600EX, Aloka Co., Ltd. 生产),测量骨矿物质密度。结果如图13(A) 所示。与假手术组相比,在卵巢切除组中观察到腰椎骨矿物质密度的显著降低,但是,在#8A1和#32A1抗体施用组中,显著抑制了由卵巢切除术导致的骨矿物质密度的降低。
c) 尿脱氧吡啶啉排泄的测量
多种I型胶原交联的代谢物突出地反映了骨代谢更新,尤其是骨吸收。最重要的是,脱氧吡啶啉主要定位于骨胶原中,因此,被认为是骨吸收的非常可靠的指标。
融化冷冻保藏的尿样品,通过离心操作沉淀出不溶物,由此得到上清液。使用Osteolinks “DPD” (DS Pharma Biomedical Co., Ltd. 生产)测量在该上清液中含有的脱氧吡啶啉的量。此外,通过使用肌酸酐Test Wako (Wako Pure Chemical Industries, Ltd. 生产),也测量上清液中的肌酸酐含量,并计算校正过的对于肌酸酐的脱氧吡啶啉的量。结果如图13(B) 所示。与假手术组相比,尿脱氧吡啶啉排泄在卵巢切除组中显著增加,因此,表明在卵巢切除的大鼠中,破骨性骨吸收增加。另一方面,在#8A1和#32A1抗体施用组中,由于卵巢切除术导致的脱氧吡啶啉排泄的增加被抑制,使得脱氧吡啶啉排泄的水平与假手术组的水平相当。从该结果,也在动物模型中证实:研究的特异性地结合Siglec-15的单克隆抗体会抑制破骨性骨吸收,并强烈地指示:由于对骨吸收的抑制作用,抑制了卵巢切除的大鼠中腰椎骨矿物质密度的降低。
实施例16
大鼠抗-小鼠Siglec-15 单克隆抗体的结合位点(表位)的确定
a) 人Siglec-15 V-组结构域的表达和纯化
将编码蛋白的DNA整合进载体 pDEST14 (Invitrogen, Inc., 目录号11801-016)中,在所述蛋白中,His标签和凝血酶识别序列被连接在人Siglec-15 V-组结构域 (一种多肽,其包含具有登录号NP_998767的NCBI蛋白数据库中的氨基酸序列或由序列表中的SEQ ID NO: 2表示的氨基酸序列的氨基酸残基39-165)的N-端侧。使用该质粒,转化大肠杆菌Rosetta-gamiB (DE3) (Novagen, Inc., 目录号71136-4),并在TB培养基 (Invitrogen, Inc., 目录号22711-022)中培养。培养后,通过超声,将细菌细胞匀浆化,将得到的匀浆离心,并使用HisTrap HP柱 (GE Healthcare, Co., Ltd., 目录号17-5247-01),纯化上清液。此后,用凝血酶切割His标签,然后使用Mono S5/50 GL柱 (GE Healthcare, Co., Ltd., 目录号17-5168-01)和Superdex 75 10/300柱 (GE Healthcare, Co., Ltd., 目录号17-5174-01),纯化人Siglec-15 V-组结构域,直到通过电泳得到分子量为14 kDa的单条带。
b) 可溶性人Siglec-15-Fc的纯化
通过在实施例12中所述的方法,纯化可溶性人Siglec-15-Fc。
c) 人V-组结构域和大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的竞争ELISA
在96-孔maxi-sorp 板 (Nunc, Inc.生产, 型号: 442404)中,将100 μl山羊抗-人Fc抗体(Jackson ImmunoResearch, Inc., 型号: 109-005-098) (1.25 μg/ml)加入每个孔中,并在4ºC固定化过夜。用PBS洗涤96-孔maxi-sorp 板2次后,将100 μl可溶性人Siglec-15-Fc (1 μg/ml) 加入每个孔中,并在室温固定化1小时。此后,将300 μl 5% 脱脂乳/PBS溶液加入每个孔中,并在室温封闭孔3小时。与此同时,将2 μg/ml的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1与等量的0、0.032、0.16、0.8、4或20 μg/ml的人Siglec-15 V-组结构域混合,并使反应在室温进行1.5小时。在已经用PBS洗涤96-孔maxi-sorp 板2次后,向其中加入100 μl混合的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1和人Siglec-15 V-组结构域的溶液,并使反应在室温进行1小时。在已经用0.05%吐温20 (Bio-Rad Laboratories, Inc., 目录号170-6531)/PBS溶液 (在下文中称作“PBST溶液”)洗涤96-孔maxi-sorp 板5次以后,向其中加入100 μl HRP-标记的山羊抗-大鼠IgG抗体(Beckman Coulter, Inc., 目录号732664,稀释至2000-倍),并使反应在室温进行1小时。在已经用PBST溶液洗涤板5次以后,向其中加入100 μl ELISA POD底物ABTS试剂盒(Nacalai Tesque Co., Ltd., 目录号14351-80)的显色液,并使反应进行30分钟。然后,向其中加入100 μl反应停止溶液,并测量在405 nm的吸光度。竞争ELISA的结果显示:以人V-组结构域浓度-依赖性的方式抑制了大鼠抗-小鼠Siglec-15 单克隆抗体#32A1与固定化的人Siglec-15的结合。因此,显示了大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的表位是人Siglec-15 V-组结构域 (一种结构域,其包含具有登录号NP_998767的NCBI蛋白数据库中的氨基酸序列或由序列表中的SEQ ID NO: 2表示的氨基酸序列的氨基酸残基39-165) (图14)。
实施例17
编码大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的可变区的cDNA的扩增及其碱基序列分析
a)mRNA的制备
根据实施例6的a),培养杂交瘤#32A1。使用QuickPrep mRNA纯化试剂盒(GE Healthcare, Co., Ltd.),从4 x 107 个杂交瘤#32A1细胞中制备约65 μg mRNA。
b) cDNA (5’-RACE-Ready cDNA)的合成
使用PrimeScript 逆转录酶 (TaKaRa Bio, Inc.)和SMART RACE cDNA扩增试剂盒(Clontech Co., Ltd.),使用0.3 μg 在a) 中制备的mRNA,合成cDNA (5’-RACE-Ready cDNA)。
c) 通过5'-RACE PCR扩增#32A1基因重链可变区的cDNA
使用UPM (通用引物A混合物,SMART RACE cDNA扩增试剂盒所附)和具有序列5’-GGCCGGGTGGGCTACGTTGCAGGTGACGGTCTG-3’ (RG2AR2: 序列表中的SEQ ID NO: 23)的寡核苷酸作为通过PCR扩增#32A1重链基因可变区的cDNA的引物。使用SMART RACE cDNA扩增试剂盒(Clontech Co., Ltd.)所附的UPM作为UPM,从数据库中的大鼠重链(IgG2a)恒定区的序列设计RG2AR2,并按照常规方法合成。
使用该引物组合和在b)中合成的cDNA (5’-RACE-Ready cDNA) 作为模板,通过5'-RACE PCR,扩增#32A1重链基因可变区的cDNA。使用Advantage 2 PCR试剂盒(Clontech Co., Ltd.)进行PCR,并将热循环仪的条件设定如下:在94℃加热1分钟后,重复“94℃ 0.5分钟和72℃ 3分钟”的温度循环5次,然后重复“94℃ 0.5分钟、70℃ 0.5分钟和72℃ 3分钟”的温度循环5次,再重复“94℃ 0.5分钟、68℃ 0.5分钟和72℃ 3分钟”的温度循环20次,然后在4℃温育。
d) 通过5'-RACE PCR扩增#32A1轻链基因可变区cDNA
使用UPM (通用引物A混合物,SMART RACE cDNA扩增试剂盒所附)和具有序列5’-CATGCTGTACGTGCTGTCTTTGCTGTCCTGATCAG-3’ (RKR2: 序列表中的SEQ ID NO: 24)的寡核苷酸作为通过PCR扩增#32A1轻链基因可变区的cDNA的引物。使用SMART RACE cDNA扩增试剂盒(Clontech Co., Ltd.)所附的UPM作为UPM,从数据库中的大鼠轻链(κ链)恒定区的序列设计RKR2,并按照常规方法合成。
使用该引物组合和在b)中合成的cDNA(5’-RACE-Ready cDNA)作为模板,通过5'-RACE PCR扩增#32A1轻链基因可变区的cDNA。使用Advantage 2 PCR试剂盒(Clontech Co., Ltd.)进行PCR,并将热循环仪的条件设定如下:在94℃加热1分钟后,重复“94℃ 0.5分钟和72℃ 3分钟”的温度循环5次,然后重复“94℃ 0.5分钟、70℃ 0.5分钟和72℃ 3分钟”的温度循环5次,再重复“94℃ 0.5分钟、68℃ 0.5分钟和72℃ 3分钟”的温度循环20次,然后在4℃温育。
e) 重链和轻链可变区的cDNA的碱基序列的测定
使用MinElute PCR纯化试剂盒(QIAGEN Inc.),纯化在c)中扩增的重链可变区的cDNA,然后进行DNA序列的序列分析。从数据库中的大鼠重链(IgG2a)恒定区的序列设计具有序列5’-CTCCAGAGTTCCAGGTCACGGTGACTGGC-3’ (RG2AR3: 序列表中的SEQ ID NO: 25)的寡核苷酸作为测序引物,并按照常规方法合成。
使用MinElute PCR纯化试剂盒(QIAGEN Inc.),纯化在d)中扩增的轻链可变区的cDNA,然后进行DNA序列的序列分析。从数据库中的大鼠轻链(κ 链)恒定区的序列,设计具有序列5’-TCCAGTTGCTAACTGTTCCG-3’ (sqRK: 序列表中的SEQ ID NO: 26)的寡核苷酸作为测序引物,并按照常规方法合成。
通过序列分析得到的含有重链可变区的cDNA具有由序列表中的SEQ ID NO: 27表示的碱基序列,且编码由序列表中的SEQ ID NO: 28表示的氨基酸序列。由SEQ ID NO: 28的氨基酸编号1-19表示的氨基酸序列对应着分泌信号,由其氨基酸编号20-140表示的氨基酸序列对应着重链可变区,由其氨基酸编号141-167表示的氨基酸序列对应着重链恒定区 (部分)。上述的重链可变区含有:包含由SEQ ID NO: 44表示的氨基酸序列的CDRH1 (DYFMN),包含由SEQ ID NO: 45表示的氨基酸序列的CDRH2 (QIRNKIYTYATFYAESLEG),和包含由SEQ ID NO: 46表示的氨基酸序列的CDRH3 (SLTGGDYFDY)。此外,含有轻链可变区的cDNA具有由序列表中的SEQ ID NO: 29表示的碱基序列,并编码由序列表中的SEQ ID NO: 30表示的氨基酸序列。由SEQ ID NO: 30的氨基酸编号1-20表示的氨基酸序列对应着分泌信号,由其氨基酸编号21-132表示的氨基酸序列对应着轻链可变区,由其氨基酸编号133-139表示的氨基酸序列对应着轻链恒定区 (部分)。上述的轻链可变区含有:包含由SEQ ID NO: 47表示的氨基酸序列的CDRL1 (RASQSVTISGYSFIH),包含由SEQ ID NO: 48表示的氨基酸序列的CDRL2 (RASNLAS),和包含由SEQ ID NO: 49表示的氨基酸序列的CDRL3 (QQSRKSPWT)。
实施例18
大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体的基因表达构建体的制备
a) 通用表达载体pEF1/FCCU-1和pEF6KCL的制备
a)-i) 人轻链表达载体pEF6KCL的构建
使用质粒 pEF6/V5-HisB (Invitrogen)作为模板,并使用下述引物进行PCR,得到紧邻在BGHpA (2174)至SmaI (2958)下游的DNA片段(含有f1复制起点和SV40启动子和起点的DNA片段,在下文中称作“片段A”):
5’-CCACGCGCCCTGTAGCGGCGCATTAAGC-3’ (引物EFF1: 序列表中的SEQ ID NO: 31)
5’-AAACCCGGGAGCTTTTTGCAAAAGCCTAGG-3’ (引物EFsmaR: SEQ ID NO: 32)。
通过重叠PCR,连接得到的片段A和含有编码人κ链分泌信号、人κ链恒定区和人聚腺苷酸额外信号的DNA序列的DNA片段(SEQ ID NO: 33, 在下文中称作“片段B”)。用限制酶KpnI和SmaI消化如此得到的其中连接了片段A和片段B 的DNA片段 (在下文中称作“片段A+B”),将其连接到用限制酶KpnI和SmaI消化的质粒 pEF6/V5-HisB (Invitrogen) 上,由此构建人轻链表达载体pEF6KCL,其具有信号序列、克隆位点、人κ链恒定区和在EF1启动子下游的人聚腺苷酸额外信号序列。
a)-ii) pEF1/KCL的构建
通过用限制酶KpnI和SmaI切割由上述方法得到的pEF6KCL,得到DNA片段,将其连接到用KpnI和SmaI消化的pEF1/myc-HisB (Invitrogen, Inc.)上,由此构建质粒 pEF1/KCL。
a)-iii) 人重链表达载体pEF1/FCCU-1的构建
使用限制酶NheI和PmeI消化含有编码人IgG1 信号序列和恒定区氨基酸序列的DNA序列的DNA片段(序列表中的SEQ ID NO: 34) ,并连接到用NheI和PmeI消化的质粒 pEF1/KCL上,由此构建人重链表达载体pEF1/FCCU-1,其具有信号序列、克隆位点、人重链恒定区和在EF1启动子下游的人聚腺苷酸额外信号序列。
b)#32A1人嵌合抗体重链表达构建体的制备
按照常规方法,合成具有序列5’-aaagctgagcGAGGTGCAAATTT TGGAGACTGGAGGAGGC-3’ (32A1HF: 序列表中的SEQ ID NO: 35)的寡核苷酸和具有序列5’-aaagctgagctGACTGTGACCATGACTC CTTGGCCCCAG-3’ (32A1HR: 序列表中的SEQ ID NO: 36) 的寡核苷酸,作为通过PCR扩增#32A1重链可变区的cDNA的引物。
此外,为了将PCR产物整合进pEF1/FCCU-1中,消化这些引物,从而添加限制酶BlpI的识别序列。使用该引物组合和在实施例17的e) 中纯化的重链可变区的cDNA作为模板,按照常规方法进行PCR。使用MinElute PCR纯化试剂盒(QIAGEN, Inc.),纯化得到的PCR产物,然后通过用限制酶BlpI (NEW ENGLAND BIOLABS, Inc.)消化PCR产物,得到DNA片段,将其在用限制酶BlpI切割的位点插入pEF1/FCCU-1中,由此构建 #32A1人嵌合抗体重链表达构建体。通过序列分析,证实插入片段的序列。按照常规方法,合成具有序列5’-TAATA CGACTCACTATAGGG-3’ (F11: 序列表中的SEQ ID NO: 37)的寡核苷酸,作为用于测序的引物。将其中正确地插入了插入片段的表达载体命名为“32A1H/pEF1/FCCU”。
c) #32A1人嵌合抗体轻链表达构建体的制备
按照常规方法,合成具有序列5’-aaacatatggcGACATTGTCTTG ACCCAGTCTCCTGCTTTGG-3’ (32A1LF:序列表中的SEQ ID NO: 38)的寡核苷酸和具有序列5’-aaacgtacgTCTCAATTCCAGCTTGGTGCCTCCAGCG-3’ (32A1LR:序列表中的SEQ ID NO: 39) 的寡核苷酸,作为用于PCR扩增#32A1轻链可变区的cDNA的引物。
此外,为了将PCR产物整合进pEF6KCL中,消化这些引物,从而将限制酶NdeI的识别序列添加到32A1LF上,并将限制酶BsiWI的识别序列添加到32A1LR上。使用该引物组合和在实施例17的e) 中纯化的轻链可变区的cDNA作为模板,按照常规方法进行PCR。使用MinElute PCR纯化试剂盒(QIAGEN, Inc.),纯化得到的PCR产物,然后通过用限制酶NdeI (TaKaRa Bio, Inc.)和BsiWI (NEW ENGLAND BIOLABS, Inc.)消化PCR产物,得到DNA片段,将其在用限制酶NdeI (TaKaRa Bio, Inc.)和BsiWI (NEW ENGLAND BIOLABS, Inc.)切割的位点插入pEF6KCL中,由此构建#32A1人嵌合抗体轻链表达构建体。通过序列分析,证实插入片段的序列。使用由序列表中的SEQ ID NO: 37表示的引物F11,作为用于测序的引物。将其中正确地插入了插入片段的表达载体命名为“32A1L/pEF6KCL”。
在b)中制备的#32A1人嵌合抗体重链基因具有由序列表中的SEQ ID NO: 40表示的核苷酸序列,并编码由序列表中的SEQ ID NO: 41表示的氨基酸序列。由SEQ ID NO: 41的氨基酸编号1-19表示的氨基酸序列对应着分泌信号,由其氨基酸编号20-140表示的氨基酸序列对应着重链可变区,由其氨基酸编号141-470表示的氨基酸序列对应着对应着重链恒定区。此外,在c)中制备的#32A1人嵌合抗体轻链基因具有由序列表中的SEQ ID NO: 42表示的核苷酸序列,并编码由序列表中的SEQ ID NO: 43表示的氨基酸序列。由SEQ ID NO: 43的氨基酸编号1-20表示的氨基酸序列对应着分泌信号,由其氨基酸编号21-132表示的氨基酸序列对应着轻链可变区,由其氨基酸编号133-237表示的氨基酸序列对应着轻链恒定区。
实施例19
大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体的制备
a) 人嵌合抗体的制备
将处于对数生长期的293 FreeStyle细胞的3 x 107个细胞接种进30 ml新鲜的FreeStyle 293 表达培养基 (Invitrogen, Inc.)中 (制备4批,其中30-ml培养物作为1批),并在8% CO2 培养箱中在37ºC振荡培养(125 rpm)。将300 μg 聚乙烯亚胺 (Polyscience, Inc. 生产,#24765) 溶解在1 ml Opti-Pro SFM培养基(由Invitrogen, Inc. 生产)中,将得到的溶液在室温放置5分钟。使用PureLink HiPure 质粒试剂盒(Invitrogen, Inc.),制备在实施例18中生产的#32A1人嵌合抗体重链表达载体32A1H/pEF1/FCCU和#32A1人嵌合抗体轻链表达载体32A1L/pEF6KCL。将32A1H/pEF1/FCCU (15 μg)和32A1L/pEF6KCL (45 μg)悬浮于1 ml Opti-Pro SFM培养基 (Invitrogen, Inc.)中,将得到的悬浮液加入1 ml 聚乙烯亚胺/Opti-Pro SFM混合液(已经在室温放置5分钟)中,将得到的混合物在室温放置另外5分钟。随后,将2 ml聚乙烯亚胺/表达载体/Opti-Pro SFM混合液加入每批293 FreeStyle细胞悬浮液中,并继续振荡培养。在8% CO2中在37ºC培养细胞7天后,从每批收集培养上清液。
b) 人嵌合抗体的纯化
通过过滤器(NALGENE, Inc.生产, #295-4545),过滤90 ml上面得到的培养上清液(3批),并将0.5 ml 用PBS平衡的MabSelect SuRe (GE Healthcare Bio-science Co., Ltd. 生产, #17-5438-01) 加入滤液中,在80 rpm和10ºC振荡得到的混合物过夜。次日,收集载体,并用PBS洗涤,此后,用1 M精氨酸溶液 (pH 4.0)洗脱抗体。将洗脱液加载至PD-10柱 (GE Healthcare Bio-science Co., Ltd.生产, #17-0851-01),用PBS替换液体,然后用Amicon Ultra-4 (Millipore Co., Ltd.生产, #UFC805008) 浓缩,由此得到1.2 ml人嵌合抗体(0.98 mg/ml)。使用Hitachi Diode Array BioPhotometer U-0080D,从在280 nm的测量结果计算抗体的浓度。
实施例20
大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体的结合小鼠Siglec-15蛋白的性质的评价
通过下述方法测定大鼠#32A1抗体对在实施例19中制备的纯化的人嵌合抗体的竞争抑制。使用PBS,将在实施例4中纯化的小鼠Siglec-15-Fc 稀释至1 μg/ml,以100 μl/孔,将稀释的溶液分配在免疫板 (Nunc, Inc.生产, #437111)上,并将板在4ºC静置过夜,由此使蛋白吸附在板上。次日,用PBS-T溶液 (PBS, 0.05% (v/v)吐温20)洗涤每个孔2次,以350 μl/孔,分配通过用PBS稀释脱脂乳 (Morinaga Milk Industry Co., Ltd. 生产)至5%所得到的溶液,并将板在室温静置2小时。去除每个孔中的液体,用PBS-T溶液洗涤每个孔3次。此后,以100 μl/孔,分配混合液(含有终浓度为0.5%的脱脂乳的PBS溶液),所述混合液含有0.25 μg/ml的人嵌合抗体和不同浓度(0 μg/ml、0.016 μg/ml、0.05 μg/ml、0.16 μg/ml、0.5 μg/ml、1.66 μg/ml或5 μg/ml)的#32A1抗体或大鼠对照IgG抗体(R&D Systems, Inc.生产, #6-001-A),并将板在室温静置2小时。用PBS-T溶液洗涤每个孔3次后,以100 μl/孔,加入用TBS-T溶液 (TBS, 0.05% (v/v)吐温20)稀释至2500-倍的碱性磷酸酶-缀合的 AffiniPure 山羊抗-人IgG (Jackson ImmunoResearch, Inc. 生产, #109-055-097),并将板在室温静置1 小时。除去每个孔中的液体,用PBS-T溶液洗涤每个孔5次。此后,以100 μl/孔,加入荧光底物溶液 (Roche Co., Ltd. 生产, #11681982001) ,并进行荧光反应。在加入荧光底物溶液后10分钟,使用板读数器,测量荧光强度。结果显示:#32A1抗体以浓度-依赖性的方式抑制人嵌合抗体与小鼠Siglec-15-Fc的结合。此外,当混合相同浓度(1:1)的#32A1抗体和人嵌合抗体时,表现出约40%竞争抑制。因此,认为#32A1抗体和人嵌合抗体具有基本上相同的对小鼠Siglec-15-Fc的亲和力。另一方面,大鼠对照IgG没有表现出竞争抑制 (图15)。
实施例21
基于小鼠破骨细胞形成试验和人破骨细胞形成试验,评价大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体的生物活性
a) 小鼠破骨细胞形成试验
使用在实施例19中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体,检查了对小鼠骨髓非贴壁细胞的破骨细胞分化的影响。在含有10% FBS和10 ng/ml M-CSF (R&D Systems, Inc.生产)的α-MEM培养基中,以1.5 x 105个细胞/ml,制备通过实施例8的方法制备的小鼠骨髓非贴壁细胞,以200 μl的量,将得到的细胞制品接种进96-孔板的每个孔中,并在CO2 培养箱中培养细胞2天。除去96-孔板中的旧培养液,将100 μl MEM-α培养基加入每个孔中,所述100 μl MEM-α培养基含有 10% FBS,其中已经添加了人RANKL (RANKL, Peprotech, Inc.生产)和M-CSF,终浓度分别为20 ng/ml和10 ng/ml。向该细胞培养液中,以3-100 ng/ml的浓度,添加在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1或在实施例19中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体,在CO2培养箱中培养细胞另外3天。培养结束后,通过在实施例9中所述的方法,测量形成的破骨细胞的酒石酸盐-抗性的酸性磷酸酶(TRAP)的活性。酶反应停止后,测定各个孔在405nm的吸光度,得到的测量结果用作TRAP活性的指标。结果如图16所示。在#32A1抗体和大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体的情况下,在50 ng/ml或更高的浓度,观察到TRAP活性的剂量依赖性的抑制。该结果显示:大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体具有基本上与大鼠#32A1抗体相当的抑制破骨细胞形成 (破骨细胞分化和成熟)的活性。
b) 使用正常人破骨细胞前体细胞的评价(TRAP染色)
按照细胞所附的说明书,以1×104个细胞/孔,将正常人破骨细胞前体细胞(正常人天然的破骨细胞前体细胞, 购自Sanko Junyaku Co., Ltd., 目录号2T-110)接种于96孔板中。使用添加了OPGM补充套装 (购自Sanko Junyaku Co., Ltd., 目录号PT-9501)的破骨细胞前体细胞用最低必需培养基(OPBM, 购自Sanko Junyaku Co., Ltd., 目录号PT-8201)作为培养基,所述OPGM补充套装含有胎牛血清(终浓度10%)、人RANKL(终浓度66 ng/ml)、人M-CSF(终浓度33 ng/ml)等。向得到的培养上清液中添加在实施例19中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人嵌合抗体,终浓度为0.3、1、3或10 μg/ml,在CO2培养箱中培养细胞3天。培养后,除去上清液,添加10%中性福尔马林以固定细胞。固定细胞后,用蒸馏水洗涤细胞2次,以100μl/孔,添加TRAP染色液(0.27mM萘酚AS-MX磷酸盐(Sigma Co., Ltd.生产)、1.6mM 固红紫色LB 盐(Sigma Co., Ltd.生产)、1%二甲基甲酰胺、50mM酒石酸钠、0.1M醋酸钠缓冲液(pH5.0)),在室温反应5分钟。反应后,用蒸馏水洗涤细胞2次,然后通过显微术观察(图17)。结果,通过添加在0.3 μg/ml至10 μg/ml范围内的#32A1的人嵌合抗体,抑制了TRAP-阳性的多核破骨细胞的形成。
实施例22
大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体的设计 (1)
a)#32A1的人源化形式的设计
a)-i) #32A1的可变区的分子建模
通过通常称作同源性建模的方法(Methods in Enzymology, 203, 121-153, (1991)),进行#32A1的可变区的分子建模。将在蛋白数据库(Nuc. Acid Res. 35, D301-D303 (2007))中登记的人免疫球蛋白可变区的基本序列(可以得到从X-射线晶体结构衍生出的三维结构)与上面测定的#32A1的可变区相对比。结果,在具有框架中的类似删除的抗体中,选择1LK3作为与#32A1的轻链可变区具有最高序列同源性。此外,选择1AD0作为与#32A1的重链可变区具有最高序列同源性。通过组合与#32A1的轻链和重链相对应的1LK3和1AD0的等同物(coordinate),得到“框架模型”,产生框架区的三维结构。根据Thornton 等人的分类法 (J. Mol. Biol., 263, 800-815, (1996)),分类#32A1的CDR如下:将CDRL1、CDRL2、CDRL3、CDRH1和CDRH2分别分配给簇15A、7A、9A、10A和12B。根据H3-规则 (FEBS letter 399, 1-8 (1996)),将CDRH3分类为k(8)C。然后,将各个CDR的代表性构象整合进框架模型中。
最后,为了在能量方面得到#32A1的可变区的可能分子模型,进行能量计算,以排除不利的原子间接触。使用市售的三维蛋白结构预测程序Prime和等同物搜索程序MacroModel (Schrodinger, LLC),执行上述操作。
a)-ii) 人源化的#32A1的氨基酸序列的设计
根据通常称作CDR移植的方法(Proc. Natl. Acad. Sci. USA 86, 10029-10033 (1989)),构建人源化的#32A1抗体。基于框架区内的氨基酸同源性,以两种方式选择受体抗体。将#32A1的框架区序列与包含抗体氨基酸序列的Kabat 数据库(Nuc. Acid Res. 29, 205-206 (2001))中的所有人框架的序列相对比。结果,基于框架区中的73%的序列同源性,选择M37GO37’CL抗体作为受体。将M37GO37’CL的框架区中的氨基酸残基与#32A1的氨基酸残基相比对,鉴别出其中使用不同氨基酸的位置。使用上面构建的#32A1的三维模型,分析这些残基的位置。然后,根据Queen 等人提供的标准 (Proc. Natl. Acad. Sci. USA 86, 10029-10033 (1989)),选择要移植到受体上的供体残基。
将#32A1的框架区序列与IgBLAST (Nuc. Acid Res.36, D25-D30 (2007))中的所有人框架序列相对比。结果,对于L-链,基于框架区中的79%的序列同源性,选择AAB34430作为受体。对于H-链,基于框架区中的78%的序列同源性,选择CAF31288作为受体。将AAB34430的框架区中的氨基酸残基(关于L-链)和CAF31288的框架区中的氨基酸残基(关于H-链)与#32A1的氨基酸残基相比对,鉴别出其中使用不同氨基酸的位置。使用上面构建的#32A1的三维模型,分析这些残基的位置。然后,根据Queen 等人提供的标准(Proc. Natl. Acad. Sci. USA 86, 10029-10033 (1989)),选择要移植到受体上的供体残基。在这些方法中,通过将一些选择的供体残基转移给受体抗体,构建出如下面的实施例中所述的人源化的#32A1序列。
b) #32A1重链的人源化
b)-i) h#32A1-T1H-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、甘氨酸、苯丙氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、天冬酰胺、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、61 (谷氨酸)、89 (缬氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、105 (丝氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T1H-型重链”。
h#32A1-T1H-型重链的氨基酸序列由序列表中的SEQ ID NO: 51表示。包含SEQ ID NO: 51的氨基酸序列的氨基酸残基1-19的序列、包含其氨基酸残基20-140的序列和包含其氨基酸残基141-470的序列分别对应着信号序列、重链可变区和重链恒定区。编码SEQ ID NO: 51的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 50表示。包含SEQ ID NO: 50的核苷酸序列的核苷酸1-57的序列、包含其核苷酸58-420的序列和包含其核苷酸421-1410的序列分别编码信号序列、重链可变区序列和重链恒定区序列。SEQ ID NO: 50的核苷酸序列和SEQ ID NO: 51的氨基酸序列也显示在图27中。
b)-ii) h#32A1-T2H-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T2H-型重链”。
h#32A1-T2H-型重链的氨基酸序列由序列表中的SEQ ID NO: 53表示。包含SEQ ID NO: 53的氨基酸序列的氨基酸残基1-19的序列、包含其氨基酸残基20-140的序列和包含其氨基酸残基141-470的序列分别对应着信号序列、重链可变区和重链恒定区。编码SEQ ID NO: 53的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 52表示。包含SEQ ID NO: 52的核苷酸序列的核苷酸1-57的序列、包含其核苷酸58-420的序列和包含其核苷酸421-1410的序列分别编码信号序列、重链可变区序列和重链恒定区序列。SEQ ID NO: 52的核苷酸序列和SEQ ID NO: 53的氨基酸序列也显示在图28中。
b)-iii) h#32A1-T3H-型重链:
通过分别用缬氨酸、丝氨酸、谷氨酰胺、甲硫氨酸、缬氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、104 (缬氨酸)、114 (异亮氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T3H-型重链”。
h#32A1-T3H-型重链的氨基酸序列由序列表中的SEQ ID NO: 55表示。包含SEQ ID NO: 55的氨基酸序列的氨基酸残基1-19的序列、包含其氨基酸残基20-140的序列和包含其氨基酸残基141-470的序列分别对应着信号序列、重链可变区和重链恒定区。编码SEQ ID NO: 55的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 54表示。包含SEQ ID NO: 54的核苷酸序列的核苷酸1-57的序列、包含其核苷酸58-420的序列和包含其核苷酸421-1410的序列分别编码信号序列、重链可变区序列和重链恒定区序列。SEQ ID NO: 54的核苷酸序列和SEQ ID NO: 55的氨基酸序列也显示在图29中。
b)-iv) h#32A1-T5H-型重链:
通过分别用缬氨酸、缬氨酸、丝氨酸、谷氨酰胺、甘氨酸、苯丙氨酸、天冬酰胺、赖氨酸、苏氨酸、甲硫氨酸、天冬酰胺、缬氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、61 (谷氨酸)、89 (缬氨酸)、95 (天冬氨酸)、97 (谷氨酸)、99 (丝氨酸)、104 (缬氨酸)、105 (丝氨酸)、114 (异亮氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T5H-型重链”。
h#32A1-T5H-型重链的氨基酸序列由序列表中的SEQ ID NO: 57表示。包含SEQ ID NO: 57的氨基酸序列的氨基酸残基1-19的序列、包含其氨基酸残基20-140的序列和包含其氨基酸残基141-470的序列分别对应着信号序列、重链可变区和重链恒定区。编码SEQ ID NO: 57的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 56表示。包含SEQ ID NO: 56的核苷酸序列的核苷酸1-57的序列、包含其核苷酸58-420的序列和包含其核苷酸421-1410的序列分别编码信号序列、重链可变区序列和重链恒定区序列。SEQ ID NO: 56的核苷酸序列和SEQ ID NO: 57的氨基酸序列也显示在图30中。
b)-v) h#32A1-T6H-型重链:
通过分别用缬氨酸、缬氨酸、丝氨酸、谷氨酰胺、天冬酰胺、赖氨酸、苏氨酸、甲硫氨酸、缬氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、95 (天冬氨酸)、97 (谷氨酸)、99 (丝氨酸)、104 (缬氨酸)、114 (异亮氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T6H-型重链”。
h#32A1-T6H-型重链的氨基酸序列由序列表中的SEQ ID NO: 59表示。包含SEQ ID NO: 59的氨基酸序列的氨基酸残基1-19的序列、包含其氨基酸残基20-140的序列和包含其氨基酸残基141-470的序列分别对应着信号序列、重链可变区和重链恒定区。编码SEQ ID NO: 59的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 58表示。包含SEQ ID NO: 58的核苷酸序列的核苷酸1-57的序列、包含其核苷酸58-420的序列和包含其核苷酸421-1410的序列分别编码信号序列、重链可变区序列和重链恒定区序列。SEQ ID NO: 58的核苷酸序列和SEQ ID NO: 59的氨基酸序列也显示在图31中。
b)-vi) h#32A1-T7H-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、苯丙氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、天冬酰胺、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、89 (缬氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、105 (丝氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T7H-型重链”。
h#32A1-T7H-型重链的氨基酸序列由序列表中的SEQ ID NO: 72表示。包含SEQ ID NO: 72的氨基酸序列的氨基酸残基1-121的序列和包含其氨基酸残基122-451的序列分别对应着重链可变区和重链恒定区。SEQ ID NO: 72的氨基酸序列也显示在图38中。
b)-vii) h#32A1-T8H-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、甘氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、天冬酰胺、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、61 (谷氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、105 (丝氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T8H-型重链”。
h#32A1-T8H-型重链的氨基酸序列由序列表中的SEQ ID NO: 73表示。包含SEQ ID NO: 73的氨基酸序列的氨基酸残基1-121的序列和包含其氨基酸残基122-451的序列分别对应着重链可变区和重链恒定区。SEQ ID NO: 73的氨基酸序列也显示在图38中。
b-viii) h#32A1-T9H-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、甘氨酸、苯丙氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、61 (谷氨酸)、89 (缬氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T9H-型重链”。
h#32A1-T9H-型重链的氨基酸序列由序列表中的SEQ ID NO: 74表示。包含SEQ ID NO: 74的氨基酸序列的氨基酸残基1-121的序列和包含其氨基酸残基122-451的序列分别对应着重链可变区和重链恒定区。SEQ ID NO: 74的氨基酸序列也显示在图38中。
b)-ix) h#32A1-T10H-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、天冬酰胺、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、105 (丝氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T10H-型重链”。
h#32A1-T10H-型重链的氨基酸序列由序列表中的SEQ ID NO: 75表示。包含SEQ ID NO: 75的氨基酸序列的氨基酸残基1-121的序列和包含其氨基酸残基122-451的序列分别对应着重链可变区和重链恒定区。SEQ ID NO: 75的氨基酸序列也显示在图39中。
b)-x) h#32A1-T11H-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、苯丙氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、89 (缬氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T11H-型重链”。
h#32A1-T11H-型重链的氨基酸序列由序列表中的SEQ ID NO: 76表示。包含SEQ ID NO: 76的氨基酸序列的氨基酸残基1-121的序列和包含其氨基酸残基122-451的序列分别对应着重链可变区和重链恒定区。SEQ ID NO: 76的氨基酸序列也显示在图39中。
b)-xi) h#32A1-T12H-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、甘氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的#32A1重链的氨基酸残基23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、61 (谷氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的#32A1重链被命名为“h#32A1-T12H-型重链”。
h#32A1-T12H-型重链的氨基酸序列由序列表中的SEQ ID NO: 77表示。包含SEQ ID NO: 77的氨基酸序列的氨基酸残基1-121的序列和包含其氨基酸残基122-451的序列分别对应着重链可变区和重链恒定区。SEQ ID NO: 77的氨基酸序列也显示在图39中。
c) #32A1轻链的人源化
c)-i) h#32A1-T1L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、赖氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、酪氨酸、甘氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间(即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、68 (精氨酸)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、122 (丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T1L-型轻链”。
h#32A1-T1L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 61表示。包含SEQ ID NO: 61的氨基酸序列的氨基酸残基1-20的序列、包含其氨基酸残基21-133的序列和包含其氨基酸残基134-238的序列分别对应着信号序列、轻链可变区和轻链恒定区。编码SEQ ID NO: 61的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 60表示。包含SEQ ID NO: 60的核苷酸序列的核苷酸1-60的序列、包含其核苷酸61-399的序列和包含其核苷酸400-714的序列分别编码信号序列、轻链可变区序列和轻链恒定区序列。SEQ ID NO: 60的核苷酸序列和SEQ ID NO: 61的氨基酸序列也显示在图32中。
c)-ii) h#32A1-T2L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、脯氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T2L-型轻链”。
h#32A1-T2L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 63表示。包含SEQ ID NO: 63的氨基酸序列的氨基酸残基1-20的序列、包含其氨基酸残基21-133的序列和包含其氨基酸残基134-238的序列分别对应着信号序列、轻链可变区和轻链恒定区。编码SEQ ID NO: 63的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 62表示。包含SEQ ID NO: 62的核苷酸序列的核苷酸1-60的序列、包含其核苷酸61-399的序列和包含其核苷酸400-714的序列分别编码信号序列、轻链可变区序列和轻链恒定区序列。SEQ ID NO: 62的核苷酸序列和SEQ ID NO: 63的氨基酸序列也显示在图33中。
c)-iii) h#32A1-T3L-型轻链:
通过分别用天冬氨酸、丝氨酸、谷氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T3L-型轻链”。
h#32A1-T3L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 65表示。包含SEQ ID NO: 65的氨基酸序列的氨基酸残基1-20的序列、包含其氨基酸残基21-133的序列和包含其氨基酸残基134-238的序列分别对应着信号序列、轻链可变区和轻链恒定区。编码SEQ ID NO: 65的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 64表示。包含SEQ ID NO: 64的核苷酸序列的核苷酸1-60的序列、包含其核苷酸61-399的序列和包含其核苷酸400-714的序列分别编码信号序列、轻链可变区序列和轻链恒定区序列。SEQ ID NO: 64的核苷酸序列和SEQ ID NO: 65的氨基酸序列也显示在图34中。
c)-iv) h#32A1-T4L-型轻链:
通过分别用谷氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号36 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T4L-型轻链”。
h#32A1-T4L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 67表示。包含SEQ ID NO: 67的氨基酸序列的氨基酸残基1-20的序列、包含其氨基酸残基21-132的序列和包含其氨基酸残基133-237的序列分别对应着信号序列、轻链可变区和轻链恒定区。编码SEQ ID NO: 67的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 66表示。包含SEQ ID NO: 66的核苷酸序列的核苷酸1-60的序列、包含其核苷酸61-396的序列和包含其核苷酸397-711的序列分别编码信号序列、轻链可变区序列和轻链恒定区序列。SEQ ID NO: 66的核苷酸序列和SEQ ID NO: 67的氨基酸序列也显示在图35中。
c)-v) h#32A1-T5L-型轻链:
通过分别用天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、赖氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、酪氨酸、甘氨酸、谷氨酰胺、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、68 (精氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、110 (苯丙氨酸)、122 (丙氨酸)、123 (甘氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T5L-型轻链”。
h#32A1-T5L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 69表示。包含SEQ ID NO: 69的氨基酸序列的氨基酸残基1-20的序列、包含其氨基酸残基21-133的序列和包含其氨基酸残基134-238的序列分别对应着信号序列、轻链可变区和轻链恒定区。编码SEQ ID NO: 69的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 68表示。包含SEQ ID NO: 68的核苷酸序列的核苷酸1-60的序列、包含其核苷酸61-399的序列和包含其核苷酸400-714的序列分别编码信号序列、轻链可变区序列和轻链恒定区序列。SEQ ID NO: 68的核苷酸序列和SEQ ID NO: 69的氨基酸序列也显示在图36中。
c)-vi) h#32A1-T6L-型轻链:
通过分别用天冬氨酸、丝氨酸、谷氨酸、脯氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T6L-型轻链”。
h#32A1-T6L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 71表示。包含SEQ ID NO: 71的氨基酸序列的氨基酸残基1-20的序列、包含其氨基酸残基21-133的序列和包含其氨基酸残基134-238的序列分别对应着信号序列、轻链可变区和轻链恒定区。编码SEQ ID NO: 71的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 70表示。包含SEQ ID NO: 70的核苷酸序列的核苷酸1-60的序列、包含其核苷酸61-399的序列和包含其核苷酸400-714的序列分别编码信号序列、轻链可变区序列和轻链恒定区序列。SEQ ID NO: 70的核苷酸序列和SEQ ID NO: 71的氨基酸序列也显示在图37中。
c)-vii) h#32A1-T7L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、脯氨酸、赖氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、酪氨酸、甘氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、68 (精氨酸)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、122 (丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T7L-型轻链”。
h#32A1-T7L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 78表示。包含SEQ ID NO: 78的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 78的氨基酸序列也显示在图40中。
c)-viii) h#32A1-T8L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、酪氨酸、甘氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、122 (丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T8L-型轻链”。
h#32A1-T8L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 79表示。包含SEQ ID NO: 79的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 79的氨基酸序列也显示在图40中。
c)-ix) h#32A1-T9L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、赖氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、甘氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、68 (精氨酸)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、122 (丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T9L-型轻链”。
h#32A1-T9L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 80表示。包含SEQ ID NO: 80的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 80的氨基酸序列也显示在图40中。
c)-x) h#32A1-T10L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、赖氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、酪氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、68 (精氨酸)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T10L-型轻链”。
h#32A1-T10L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 81表示。包含SEQ ID NO: 81的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 81的氨基酸序列也显示在图40中。
c)-xi) h#32A1-T11L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、脯氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、酪氨酸、甘氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、122 (丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T11L-型轻链”。
h#32A1-T11L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 82表示。包含SEQ ID NO: 82的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 82的氨基酸序列也显示在图40中。
c)-xii) h#32A1-T12L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、脯氨酸、赖氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、甘氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、68 (精氨酸)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、122 (丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T12L-型轻链”。
h#32A1-T12L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 83表示。包含SEQ ID NO: 83的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 83的氨基酸序列也显示在图41中。
c)-xiii) h#32A1-T13L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、脯氨酸、赖氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、酪氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、68 (精氨酸)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T13L-型轻链”。
h#32A1-T13L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 84表示。包含SEQ ID NO: 84的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 84的氨基酸序列也显示在图41中。
c)-xiv) h#32A1-T14L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、甘氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、122 (丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T14L-型轻链”。
h#32A1-T14L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 85表示。包含SEQ ID NO: 85的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 85的氨基酸序列也显示在图41中。
c)-xv) h#32A1-T15L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、酪氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T15L-型轻链”。
h#32A1-T15L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 86表示。包含SEQ ID NO: 86的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 86的氨基酸序列也显示在图41中。
c)-xvi) h#32A1-T16L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、赖氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、68 (精氨酸)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T16L-型轻链”。
h#32A1-T16L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 87表示。包含SEQ ID NO: 87的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 87的氨基酸序列也显示在图41中。
c)-xvii) h#32A1-T17L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、脯氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、甘氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、122 (丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T17L-型轻链”。
h#32A1-T17L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 88表示。包含SEQ ID NO: 88的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 88的氨基酸序列也显示在图42中。
c)-xviii) h#32A1-T18L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、脯氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、酪氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T18L-型轻链”。
h#32A1-T18L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 89表示。包含SEQ ID NO: 89的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 89的氨基酸序列也显示在图42中。
c)-xix) h#32A1-T19L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、脯氨酸、赖氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、66 (谷氨酰胺)、68 (精氨酸)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T19L-型轻链”。
h#32A1-T19L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 90表示。包含SEQ ID NO: 90的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 90的氨基酸序列也显示在图42中。
c)-xx) h#32A1-T20L-型轻链:
通过分别用甲硫氨酸、天冬氨酸、丝氨酸、谷氨酸、天冬酰胺、脯氨酸、缬氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、缬氨酸、缬氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29 (丙氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、36 (谷氨酰胺)、41 (丝氨酸)、66 (谷氨酰胺)、81 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T20L-型轻链”。
h#32A1-T20L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 91表示。包含SEQ ID NO: 91的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 91的氨基酸序列也显示在图42中。
c)-xxi) h#32A1-T21L-型轻链:
通过分别用谷氨酸、甲硫氨酸、苏氨酸、丝氨酸、亮氨酸、脯氨酸、谷氨酸、亮氨酸、丙氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、脯氨酸、谷氨酸、苯丙氨酸、亮氨酸、酪氨酸、甘氨酸、谷氨酰胺、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号21 (天冬氨酸)、24 (亮氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、31 (丙氨酸)、32 (缬氨酸)、34 (亮氨酸)、36 (谷氨酰胺)、40 (异亮氨酸)、66 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、102 (谷氨酰胺)、103 (丙氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、122 (丙氨酸)、123 (甘氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T21L-型轻链”。
h#32A1-T21L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 92表示。包含SEQ ID NO: 92的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 92的氨基酸序列也显示在图42中。
c)-xxii) h#32A1-T22L-型轻链:
通过分别用谷氨酸、甲硫氨酸、苏氨酸、丝氨酸、亮氨酸、脯氨酸、谷氨酸、亮氨酸、丙氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、脯氨酸、谷氨酸、苯丙氨酸、亮氨酸、谷氨酰胺、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号21 (天冬氨酸)、24 (亮氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、31 (丙氨酸)、32 (缬氨酸)、34 (亮氨酸)、36 (谷氨酰胺)、40 (异亮氨酸)、66 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、102 (谷氨酰胺)、103 (丙氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、123 (甘氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T22L-型轻链”。
h#32A1-T22L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 93表示。包含SEQ ID NO: 93的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 93的氨基酸序列也显示在图43中。
c)-xxiii) h#32A1-T23L-型轻链:
通过分别用甲硫氨酸、苏氨酸、丝氨酸、亮氨酸、脯氨酸、谷氨酸、亮氨酸、丙氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、脯氨酸、谷氨酸、苯丙氨酸、亮氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号24 (亮氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、31 (丙氨酸)、32 (缬氨酸)、34 (亮氨酸)、36 (谷氨酰胺)、40 (异亮氨酸)、66 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、102 (谷氨酰胺)、103 (丙氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T23L-型轻链”。
h#32A1-T23L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 94表示。包含SEQ ID NO: 94的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 94的氨基酸序列也显示在图43中。
c)-xxiv) h#32A1-T24L-型轻链:
通过分别用谷氨酸、苏氨酸、丝氨酸、亮氨酸、脯氨酸、谷氨酸、亮氨酸、丙氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、脯氨酸、谷氨酸、苯丙氨酸、亮氨酸、酪氨酸、甘氨酸、谷氨酰胺、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号21 (天冬氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、31 (丙氨酸)、32 (缬氨酸)、34 (亮氨酸)、36 (谷氨酰胺)、40 (异亮氨酸)、66 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、102 (谷氨酰胺)、103 (丙氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、122 (丙氨酸)、123 (甘氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T24L-型轻链”。
h#32A1-T24L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 95表示。包含SEQ ID NO: 95的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 95的氨基酸序列也显示在图43中。
c)-xxv) h#32A1-T25L-型轻链:
通过分别用苏氨酸、丝氨酸、亮氨酸、脯氨酸、谷氨酸、亮氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、脯氨酸、谷氨酸、苯丙氨酸、酪氨酸、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的#32A1轻链的氨基酸编号29和30之间 (即插入在大鼠框架中缺失的残基)、31 (丙氨酸)、32 (缬氨酸)、34 (亮氨酸)、36 (谷氨酰胺)、40 (异亮氨酸)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、102 (谷氨酰胺)、103 (丙氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、110 (苯丙氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的#32A1轻链被命名为“h#32A1-T25L-型轻链”。
h#32A1-T25L-型轻链的氨基酸序列由序列表中的SEQ ID NO: 96表示。包含SEQ ID NO: 96的氨基酸序列的氨基酸残基1-113的序列和包含其氨基酸残基114-218的序列分别对应着轻链可变区和轻链恒定区。SEQ ID NO: 96的氨基酸序列也显示在图43中。
实施例23
大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体的基因的制备
a) h#32A1-T1L、h#32A1-T2L、h#32A1-T3L、h#32A1-T4L、h#32A1-T5L和h#32A1-T6L-型轻链表达载体的构建
合成了DNA (Medical & Biological Laboratories Co., Ltd., 人工基因合成服务),所述DNA各自含有编码分别由序列表中的SEQ ID NO: 61的氨基酸编号1-133、SEQ ID NO: 63的氨基酸编号1-133、SEQ ID NO: 65的氨基酸编号1-133、SEQ ID NO: 67的氨基酸编号1-132、SEQ ID NO: 69的氨基酸编号1-133或SEQ ID NO: 71的氨基酸编号1-133表示的与分泌信号融合的h#32A1-T1L、h#32A1-T2L、h#32A1-T3L、h#32A1-T4L、h#32A1-T5L或h#32A1-T6L-型轻链可变区的基因。然后,用限制酶NheI和BsiWI切割合成的DNA,将得到的每个DNA片段在用限制酶NheI和BsiWI切割的位点插入通用的人源化抗体轻链表达载体(pEF6KCL)中,由此构建h#32A1-T1L、h#32A1-T2L、h#32A1-T3L、h#32A1-T4L、h#32A1-T5L和h#32A1-T6L-型轻链表达载体。将如此得到的表达载体分别命名为“pEF6KCL/h#32A1-T1L”、“pEF6KCL/h#32A1-T2L”、“pEF6KCL/h#32A1-T3L”、“pEF6KCL/h#32A1-T4L”、“pEF6KCL/h#32A1-T5L”和“pEF6KCL/h#32A1-T6L”。
b) h#32A1-T1H、h#32A1-T2H、h#32A1-T3H、h#32A1-T5H和h#32A1-T6H重链表达载体的构建
合成了DNA (Medical & Biological Laboratories Co., Ltd., 人工基因合成服务),所述DNA各自含有编码由序列表中的SEQ ID NO: 51、53、55、57或59的氨基酸编号1-140表示的与分泌信号融合的h#32A1-T1H、h#32A1-T2H、h#32A1-T3H、h#32A1-T5H或h#32A1-T6H-型重链可变区的基因。然后,用限制酶BlpI切割合成的DNA,将得到的每个DNA片段在用限制酶BlpI切割的位点插入通用的人源化抗体重链表达载体(pEF1/FCCU-1)中,由此构建h#32A1-T1H、h#32A1-T2H、h#32A1-T3H、h#32A1-T5H和h#32A1-T6H重链表达载体。将如此得到的表达载体分别命名为“pEF1/FCCU/h#32A1-T1H”、“pEF1/FCCU/h#32A1-T2H”、“pEF1/FCCU/h#32A1-T3H”、“pEF1/FCCU/h#32A1-T5H”和“pEF1/FCCU/h#32A1-T6H”。
实施例24
大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体的制备
a)人源化抗体的制备
将处于对数生长期的293 FreeStyle细胞的1.5 x 108个细胞接种进100 ml新鲜的FreeStyle 293 表达培养基 (Invitrogen, Inc.)中,并在8% CO2 培养箱中在37ºC振荡培养(125 rpm)。将1 mg 聚乙烯亚胺 (Polyscience, Inc. 生产#24765)溶解在4 ml Opti-Pro SFM培养基(由Invitrogen, Inc. 生产)中,将得到的溶液在室温放置5分钟。将使用PureLink HiPure 质粒试剂盒(Invitrogen, Inc.)制备的重链表达质粒(0.05mg)和轻链表达质粒(0.15mg)悬浮于4 ml Opti-Pro SFM培养基 (Invitrogen, Inc.)中。然后,将4 ml 得到的表达质粒/Opti-Pro SFM混合液加入已经在室温放置5分钟的4 ml聚乙烯亚胺/Opti-Pro SFM混合液中,将得到的混合物在室温放置另外5分钟。随后,将8 ml 聚乙烯亚胺/表达质粒/Opti-Pro SFM混合液加入293 FreeStyle细胞悬浮液中,并继续振荡培养。在8% CO2中在37ºC培养细胞7天后,收集培养上清液。
b) 人源化抗体的纯化
通过蛋白A 亲和色谱法,纯化在上面a)中得到的培养上清液。将100 ml培养上清液放入500-ml具有塞子的烧瓶中,向其中加入1 ml 用PBS 平衡的MabSelect SuRe (GE Healthcare Bio-science Co., Ltd.生产) 的悬浮液 (50%浆体)。以100 rpm,在10ºC培养箱中搅拌得到的混合物过夜。然后,将293F 培养上清液/MabSelect SuRe 悬浮液加载至 Zeba Spin空柱5 mL (PIERCE, Inc.)。将所有树脂倒入柱中以后,用10 ml 1 M NaCl洗涤柱。随后,将1 ml 1 M 精氨酸溶液 (pH 4.0)加载至柱,收集含有抗体的级分。将该级分加入离心过滤器装置 (Amicon Ultra-4, 分离分子量(fractional molecular weight):50 K, Millipore Co., Ltd.),用柠檬酸盐缓冲液替换液体,并浓缩。将终体积补足至200 μl,将其用作纯化的样品。
由pEF6KCL/h#32A1-T1L和pEF1/FCCU/h#32A1-T1H的组合得到的#32A1的人源化抗体被命名为“h#32A1-T1”;由pEF6KCL/h#32A1-T2L和pEF1/FCCU/h#32A1-T2H的组合得到的#32A1的人源化抗体被命名为“h#32A1-T2”;由pEF6KCL/h#32A1-T3L和pEF1/FCCU/h#32A1-T3H的组合得到的#32A1的人源化抗体被命名为“h#32A1-T3”;由pEF6KCL/h#32A1-T4L和pEF1/FCCU/h#32A1-T3H的组合得到的#32A1的人源化抗体被命名为“h#32A1-T4”;由pEF6KCL/h#32A1-T5L和pEF1/FCCU/h#32A1-T5H的组合得到的#32A1的人源化抗体被命名为“h#32A1-T5”;且由pEF6KCL/h#32A1-T6L和pEF1/FCCU/h#32A1-T6H的组合得到的#32A1的人源化抗体被命名为“h#32A1-T6”。
实施例25
大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体对小鼠Siglec-15蛋白的结合性质的评价
通过下述方法,评价了大鼠抗-小鼠Siglec-15 #32A1 T1-T6的人源化抗体对小鼠Siglec-15-Fc和人Siglec-15-Fc的结合性质。使用PBS,将在实施例4中纯化的小鼠Siglec-15-Fc或在实施例12中纯化的人Siglec-15-Fc稀释至1 μg/ml,以100 μl/孔,将稀释的溶液分配给免疫板 (Nunc, Inc.生产, #437111),在4ºC静置板过夜,由此使每种蛋白吸附到板上。次日,用PBS-T溶液 (PBS, 0.05% (v/v)吐温20)洗涤每个孔5次,并以350 μl/孔,分配用PBS稀释脱脂乳 (Morinaga Milk Industry Co., Ltd. 生产)至5%所得到的溶液,在室温静置板2小时。除去每个孔中的液体,用PBS-T溶液洗涤每个孔5次。此后,使用含有0.5% 脱脂乳的PBS溶液,将在实施例24中制备的每种纯化的大鼠抗-小鼠Siglec-15 #32A1 T1至T6的人源化抗体或在实施例19中制备的人嵌合抗体稀释至1-0.004 μg/ml的终浓度(4-倍稀释系列),以100 μl/孔,分配稀释的溶液,并在室温静置板2小时。在此时,如下测定每种抗体的浓度:通过使用分光光度计 DU-7400 (Beckman Coulter, Inc.生产)在280 nm进行测量,并基于每种抗体的摩尔分子消光系数进行计算。在已经用PBS-T溶液洗涤每个孔5次以后,以100 μl/孔,加入用TBS-T溶液 (TBS, 0.05% (v/v)吐温20)稀释至2500-倍的碱性磷酸酶-缀合的 AffiniPure 山羊抗-人IgG (Jackson ImmunoResearch, Inc. 生产, #109-055-097),并在室温静置板1小时。除去每个孔中的液体,用TBS-T溶液洗涤每个孔5次。此后,以100 μl/孔,加入荧光底物溶液 (Roche Co., Ltd., #11681982001生产) ,并进行荧光反应。在加入荧光底物溶液10分钟以后(在具有与其吸附的小鼠Siglec-15-Fc的板的情况下)和15分钟以后(在具有与其吸附的人Siglec-15-Fc的板的情况下),使用Spectra Max M5 (Molecular Devices, Inc.生产)测量荧光强度。结果证实:所有检查的6种大鼠抗-小鼠Siglec-15的人源化抗体都以抗体浓度依赖性的方式结合小鼠Siglec-15蛋白(图18)和人Siglec-15蛋白(图19)。
实施例26
添加大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的嵌合抗体对小鼠骨髓非贴壁(non-adherent)细胞的破骨细胞分化(由TNFα刺激)的影响
使用在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1和在实施例9中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1 的嵌合抗体,检查了对由TNFα刺激导致的小鼠骨髓非贴壁细胞的破骨细胞分化的影响。用含有10% 胎牛血清 (FBS)、10 ng/ml M-CSF和2 ng/ml TGF-β (R&D Systems、Inc.生产)的α-MEM培养基,将通过实施例8的方法制备的小鼠骨髓非贴壁细胞制成1.5×105个细胞/ml,并将得到的细胞制品以200μl的量接种在 96孔板的每个孔中,在CO2培养箱中培养细胞2天。除去96孔板中旧的培养液,向每个孔添加100μl MEM-α培养基,所述100 μl MEM-α培养基含有 10% FBS,其中已经添加了重组人TNFα (R&D Systems, Inc.生产)和M-CSF,终浓度分别为30 ng/ml和10 ng/ml。向该细胞培养液中,以0.125-4 μg/ml的浓度,添加在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1或在实施例19中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的嵌合抗体,在CO2培养箱中培养细胞另外3天。同时,也准备一个孔,其中以100 ng/ml的浓度,加入通过专利申请公开号WO 96/26217的说明书所述的方法制备的重组人OCIF/OPG进行细胞培养。培养结束后,根据试剂盒所附的说明书,使用白细胞酸性磷酸酶试剂盒(Sigma Co., Ltd.生产)进行TRAP染色,观察到TRAP-阳性的多核破骨细胞的形成。结果,抗-小鼠Siglec-15 多克隆抗体的加入抑制了TRAP-阳性的巨大多核破骨细胞的形成 (图20)。尽管形成了小单核破骨细胞(甚至在加入大鼠抗-小鼠Siglec-15 单克隆抗体#32A1或其嵌合抗体的情况下),由TNFα诱导的大多核破骨细胞的形成几乎完全被抑制。另一方面,甚至当加入足够量的OCIF/OPG(它是一种RANKL阻滞剂)时,大多核破骨细胞的形成仅受到微小抑制。此外,以相同的方式,通过实施例9所述的方法,测量了形成的破骨细胞的酒石酸盐-抗性的酸性磷酸酶(TRAP)的活性。酶反应停止后,测定每个孔在405nm的吸光度,得到的测量结果用作TRAP活性的指标。结果如图21所示。在125 ng/ml至4 μg/ml 的范围内的#32A1抗体的和#32A1的嵌合抗体的情况下,观察到TRAP活性的显著抑制。这些结果显示:大鼠抗-小鼠Siglec-15 单克隆抗体及其嵌合抗体强烈地抑制由TNFα诱导的破骨细胞分化和成熟中的细胞融合过程。
实施例27
基于大鼠破骨细胞形成试验,评价大鼠抗-小鼠Siglec-15 单克隆抗体#8A1和#32A1的生物活性
使用在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#8A1和#32A1,检查了对大鼠骨髓非贴壁细胞的破骨细胞分化的影响。通过改进实施例8的方法,制备了大鼠骨髓非贴壁细胞。也就是说,从12周龄的雌性F344大鼠的双腿切离股骨,并去除软组织。切掉股骨的两端,用带有23号注射针的注射器注射MEM-α培养基以推出骨髓细胞,收集在离心管中。在室温、以100g离心5分钟,去除上清液。向得到的细胞沉淀物中加入1ml 溶血缓冲液 (红细胞溶解缓冲液,Sigma Co., Ltd.生产)以混悬,将得到的悬浮液在室温放置5分钟。向其中加入20 ml D-PBS,并在室温以100g离心悬浮液5分钟,去除上清液。向得到的细胞沉淀物中加入10ml含有5 ng/ml M-CSF (R&D Systems, Inc.生产)和10% 胎牛血清 (FBS)的MEM-α培养基(由Invitrogen, Inc. 生产)以混悬。然后,使得到的悬浮液通过细胞过滤网 (40μm尼龙,BD Falcon生产)以除去凝集物。将得到的细胞转移至75 cm2 T-培养瓶 (用于贴壁细胞的附着)中,在CO2培养箱中培养过夜。培养过夜后,收集未附着在T-培养瓶上的细胞,用作大鼠骨髓非贴壁细胞。
培养通过该方法制备的大鼠骨髓非贴壁细胞,并根据实施例9的方法进行大鼠破骨细胞形成试验。也就是说,在含有10% FBS和10 ng/ml M-CSF (R&D Systems, Inc.生产)的α-MEM培养基中,将这样制备的大鼠骨髓非贴壁细胞制成1.5×105个细胞/ml,并将得到的细胞制品以200μl的量接种在 96孔板的每个孔中,在CO2培养箱中培养细胞2天。除去96孔板中旧的培养液,向每个孔添加100μl MEM-α培养基,所述100 μl MEM-α培养基含有 10% FBS,其中已经添加了人RANKL (RANKL, Peprotech, Inc.生产)和M-CSF,终浓度分别为20 ng/ml和10 ng/ml。向该细胞培养液中,以31-1000 ng/ml的浓度,添加在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体#8A1或#32A1,在CO2培养箱中培养细胞另外3天。培养结束后,通过在实施例9中所述的方法,测定形成的破骨细胞的酒石酸盐-抗性的酸性磷酸酶(TRAP)的活性。酶反应停止后,测定各个孔在405nm的吸光度,得到的测量结果用作TRAP活性的指标。结果如图22所示。在#8A1抗体的情况下,在63 ng/ml或更高的浓度,观察到TRAP活性的剂量依赖性的抑制,且在#32A1抗体的情况下,在31 ng/ml或更高的浓度,观察到TRAP活性的剂量依赖性的抑制。这些结果显示:大鼠抗-小鼠Siglec-15 单克隆抗体#8A1和#32A1以与小鼠破骨细胞形成相同的方式强烈地抑制大鼠破骨细胞形成,且#32A1抗体的抑制大鼠破骨细胞形成的体外活性高于#8A1抗体。
实施例28
大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体的设计 (2)
a)#32A1的人源化形式的设计
a)-i) 人源化的#32A1的氨基酸序列的设计
根据通常称作CDR移植的方法(Proc. Natl. Acad. Sci. USA 86, 10029-10033 (1989)),构建人源化的#32A1抗体。关于CDR区的定义,除了CDRH2以外,全部使用Kabat 定义 (SEQUENCES OF PROTEINS OF IMMUNOLOGICAL INTEREST VOL. I, 第5版 (1991))。关于CDRH2,使用它自己的定义,使其序列比Kabat定义在C端缩短了5个残基。在含有这样定义的CDRH2的重链序列中,源自大鼠的CDR序列被制成更短并掺入更多的人框架序列,因此,当将它施用给人时,被识别为异种抗原的可能性更低。基于框架区内的氨基酸同源性,选择受体抗体。将#32A1的框架区序列与包含抗体氨基酸序列的Kabat 数据库(Nuc. Acid Res. 29, 205-206 (2001))中的所有人框架序列相对比。结果,对于L-链,基于框架区中的71%的序列同源性,选择GF4/1.1’CL作为受体。对于H-链,基于框架区中的73%的序列同源性,选择M37GO37’CL作为受体。将GF4/1.1’CL的框架区中的氨基酸残基(关于L-链)和M37GO37’CL的框架区中的氨基酸残基(关于H-链)与#32A1的氨基酸残基相比对,鉴别出其中使用不同氨基酸的位置。使用在实施例22的a)-i)中构建的#32A1的三维模型,分析这些残基的位置。然后,根据Queen 等人提供的标准(Proc. Natl. Acad. Sci. USA 86, 10029-10033 (1989)),选择要移植到受体上的供体残基。通过将一些选择的供体残基转移给受体抗体,构建出如下面的实施例中所述的人源化的#32A1序列。
a)-ii) #32A1重链的人源化
a)-ii)-i) h#32A1-H1-1-型重链:
通过分别用亮氨酸、缬氨酸、丝氨酸、谷氨酰胺、丙氨酸、甘氨酸、丙氨酸、缬氨酸、赖氨酸、苯丙氨酸、天冬酰胺、丙氨酸、赖氨酸、天冬酰胺、亮氨酸、甲硫氨酸、天冬酰胺、缬氨酸、丙氨酸、苏氨酸、亮氨酸和丝氨酸替换由序列表中的SEQ ID NO: 28表示的32A1的氨基酸编号23 (异亮氨酸)、24 (亮氨酸)、26 (苏氨酸)、32 (赖氨酸)、43 (苏氨酸)、61 (谷氨酸)、83 (谷氨酸)、85 (亮氨酸)、86 (谷氨酸)、89 (缬氨酸)、95 (天冬氨酸)、96 (丝氨酸)、97 (谷氨酸)、98 (丝氨酸)、100 (缬氨酸)、104 (缬氨酸)、105 (丝氨酸)、114 (异亮氨酸)、118 (苏氨酸)、134 (缬氨酸)、135 (甲硫氨酸)和140 (亮氨酸)所设计的人源化的32A1重链被命名为“h#32A1-H1-1-型重链”。
h#32A1-H1-1-型重链的氨基酸序列由序列表中的SEQ ID NO: 99表示。包含SEQ ID NO: 99的氨基酸序列的氨基酸残基1-19的序列、包含其氨基酸残基20-140的序列和包含其氨基酸残基141-466的序列分别对应着信号序列、重链可变区和重链恒定区。编码SEQ ID NO: 99的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 98表示。包含SEQ ID NO: 98的核苷酸序列的核苷酸1-57的序列、包含其核苷酸58-420的序列和包含其核苷酸421-1398的序列分别编码信号序列、重链可变区序列和重链恒定区序列。SEQ ID NO: 98的核苷酸序列和SEQ ID NO: 99的氨基酸序列也显示在图54中。
a)-iii) #32A1轻链的人源化
a)-iii)-i) h#32A1-L2-15-型轻链:
通过分别用谷氨酸、亮氨酸、甲硫氨酸、苏氨酸、丝氨酸、亮氨酸、脯氨酸、谷氨酸、亮氨酸、丙氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、脯氨酸、谷氨酸、苯丙氨酸、亮氨酸、酪氨酸、甘氨酸、谷氨酰胺、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的32A1的氨基酸编号21 (天冬氨酸)、23 (缬氨酸)、24 (亮氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、31 (丙氨酸)、32 (缬氨酸)、34 (亮氨酸)、36 (谷氨酰胺)、40 (异亮氨酸)、66 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、102 (谷氨酰胺)、103 (丙氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、110 (苯丙氨酸)、122 (丙氨酸)、123 (甘氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的32A1轻链被命名为“h#32A1-L2-15-型轻链”。
h#32A1-L2-15-型轻链的氨基酸序列由序列表中的SEQ ID NO: 103表示。包含SEQ ID NO: 103的氨基酸序列的氨基酸残基1-20的序列、包含其氨基酸残基21-133的序列和包含其氨基酸残基134-238的序列分别对应着信号序列、轻链可变区和轻链恒定区。编码SEQ ID NO: 103的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 102表示。包含SEQ ID NO: 102的核苷酸序列的核苷酸1-60的序列、包含其核苷酸61-399的序列和包含其核苷酸400-714的序列分别编码信号序列、轻链可变区序列和轻链恒定区序列。SEQ ID NO: 102的核苷酸序列和SEQ ID NO: 103的氨基酸序列也显示在图56中。
a)-iii)-ii) h#32A1-L2-16-型轻链:
通过分别用谷氨酸、亮氨酸、甲硫氨酸、苏氨酸、丝氨酸、亮氨酸、脯氨酸、谷氨酸、亮氨酸、丙氨酸、丝氨酸、丝氨酸、亮氨酸、谷氨酸、脯氨酸、谷氨酸、苯丙氨酸、亮氨酸、谷氨酰胺、缬氨酸、异亮氨酸、赖氨酸和苏氨酸替换由序列表中的SEQ ID NO: 30表示的32A1的氨基酸编号21 (天冬氨酸)、23 (缬氨酸)、24 (亮氨酸)、29和30之间 (即插入在大鼠框架中缺失的残基)、31 (丙氨酸)、32 (缬氨酸)、34 (亮氨酸)、36 (谷氨酰胺)、40 (异亮氨酸)、66 (谷氨酰胺)、99 (天冬酰胺)、100 (脯氨酸)、101 (缬氨酸)、102 (谷氨酰胺)、103 (丙氨酸)、104 (天冬氨酸)、106 (异亮氨酸)、108 (苏氨酸)、123 (甘氨酸)、127 (亮氨酸)、129 (亮氨酸)、130 (精氨酸)和132 (丙氨酸)所设计的人源化的32A1轻链被命名为“h#32A1-L2-16-型轻链”。
h#32A1-L2-16-型轻链的氨基酸序列由序列表中的SEQ ID NO: 105表示。包含SEQ ID NO: 105的氨基酸序列的氨基酸残基1-20的序列、包含其氨基酸残基21-133的序列和包含其氨基酸残基134-238的序列分别对应着信号序列、轻链可变区和轻链恒定区。编码SEQ ID NO: 105的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 104表示。包含SEQ ID NO: 104的核苷酸序列的核苷酸1-60的序列、包含其核苷酸61-399的序列和包含其核苷酸400-714的序列分别编码信号序列、轻链可变区序列和轻链恒定区序列。SEQ ID NO: 104的核苷酸序列和SEQ ID NO: 105的氨基酸序列也显示在图57中。
b)大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体的基因的制备
b)-i) 通用的人重链表达载体pEG2的构建
合成了DNA片段(Medical & Biological Laboratories Co., Ltd., 人工基因合成服务),所述DNA片段含有由SEQ ID NO: 106表示的DNA序列,其编码信号序列和人IgG2的恒定区的氨基酸,并用限制酶NheI和PmeI消化,由此得到约1100碱基对的DNA片段。将该DNA片段连接到通过用NheI和PmeI消化pEF1/FCCU-1并去除约1100 bp的DNA片段所得到的约6200碱基对的DNA片段上,由此构建通用的人源化抗体(hIgG2型)重链表达载体pEG2。
b)-ii)构建h#32A1-H1-1重链表达载体
通过合成DNA(Invitrogen),所述DNA含有编码由序列表中的SEQ ID NO: 99的氨基酸编号1-140表示的与分泌信号融合的h#32A1-H1-1重链可变区的基因,并用限制酶BlpI切割该DNA,得到DNA片段,将该DNA片段在用限制酶BlpI切割的位点插入通用的人源化抗体(hIgG2型)重链表达载体(pEG2)中,由此构建h#32A1-H1-1重链表达载体。如此得到的表达载体被命名为“pEG2/h#32A1-H1-1”。
b)-iii)h#32A1-H5重链表达载体的构建
其中在实施例22的b)-iv)中设计的h#32A1-T5H-型重链的信号序列和恒定区被改成IgG2型的相应序列的人源化的32A1重链被命名为“h#32A1-H5-型重链”。
h#32A1-H5-型重链的氨基酸序列由序列表中的SEQ ID NO: 101表示。包含SEQ ID NO: 101的氨基酸序列的氨基酸残基1-19的序列、包含其氨基酸残基20-140的序列和包含其氨基酸残基141-466的序列分别对应着信号序列、重链可变区和重链恒定区。编码SEQ ID NO: 101的氨基酸序列的核苷酸序列由序列表中的SEQ ID NO: 100表示。包含SEQ ID NO: 100的核苷酸序列的核苷酸1-57的序列、包含其核苷酸58-420的序列和包含其核苷酸421-1398的序列分别编码信号序列、重链可变区序列和重链恒定区序列。SEQ ID NO: 100的核苷酸序列和SEQ ID NO: 101的氨基酸序列也显示在图55中。
通过用限制酶BlpI切割在实施例23中制备的pEF1/FCCU/ h#32A1-T5H,得到DNA片段,将其在用限制酶BlpI切割的位点插入通用的人源化抗体(hIgG2型)重链表达载体(pEG2)中,由此构建h#32A1-H5重链表达载体。如此得到的表达载体被命名为“pEG2/h#32A1-H5”。
b)-iv) h#32A1-L2-15和h#32A1-L2-16-型轻链表达载体的构建
合成DNA(Invitrogen),所述DNA各自含有编码由序列表中的SEQ ID NO: 103的氨基酸编号1-133或SEQ ID NO: 105的氨基酸编号1-133表示的与分泌信号融合的h#32A1-L2-15或h#32A1-L2-16-型轻链可变区的基因,并以与实施例23相同的方式,构建h#32A1-L2-15和h#32A1-L2-16-型轻链表达载体。如此得到的表达载体分别被命名为“pEF6KCL/h#32A1-L2-15”和“pEF6KCL/h#32A1-L2-16”。
c) 大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体的制备
c)-i) 人源化抗体的制备
将处于对数生长期的FreeStyle 293F细胞(Invitrogen)的1.2 x 109个细胞接种进1.2 L新鲜的FreeStyle 293 表达培养基 (Invitrogen)中,并在8% CO2 培养箱中在90 rpm在37ºC振荡培养1小时。将3.6 mg 聚乙烯亚胺 (Polyscience, #24765)溶解在20 ml Opti-Pro SFM培养基 (Invitrogen)中。随后,将使用PureLink HiPure 质粒试剂盒(Invitrogen, Inc.)制备的H-链表达质粒 (0.4 mg)和L-链表达质粒 (0.8 mg) 悬浮于20 ml Opti-Pro SFM培养基中。然后,将20 ml 得到的表达质粒/Opti-Pro SFM混合液加入20 ml 聚乙烯亚胺/Opti-Pro SFM混合液中,并轻轻搅拌得到的混合物,然后放置5分钟。此后,将该混合物加入FreeStyle 293F细胞中,并在8% CO2 培养箱中在37ºC以90 rpm振荡培养7天。通过一次性胶囊式过滤器(Advantec #CCS-045-E1H)过滤得到的培养上清液。
由pEF6KCL/h#32A1-T5L和pEG2/h#32A1-H5的组合得到的#32A1的人源化抗体被命名为“h#32A1-H5/L5”;由pEF6KCL/h#32A1-T5L和pEG2/h#32A1-H1-1的组合得到的#32A1的人源化抗体被命名为“h#32A1-H1-1/L5”;由pEF6KCL/h#32A1-L2-15和pEG2/h#32A1-H1-1的组合得到的#32A1的人源化抗体被命名为“h#32A1-H1-1/L2-15”;且由pEF6KCL/h#32A1-L2-16和pEG2/h#32A1-H1-1的组合得到的#32A1的人源化抗体被命名为“h#32A1-H1-1/L2-16”。
c)-ii) 人源化抗体的纯化
通过包括rProtein A 亲和色谱法 (4-6ºC)和陶瓷羟基磷灰石 (在室温)的2步法,纯化在上面的c)-i)中得到的培养上清液。在rProtein A 亲和色谱法纯化以后和在陶瓷羟基磷灰石纯化以后,在室温进行缓冲液替换步骤。首先,将1100-1200 ml培养上清液加载至用PBS平衡的MabSelect SuRe (GE Healthcare Bioscience Co., Ltd. 生产, 2 x 1 ml HiTrap柱串联)上。将所有培养液倒入柱中以后,使用15-30 ml PBS洗涤柱。随后,用2 M 盐酸精氨酸溶液 (pH 4.0)进行洗脱,并收集含有抗体的级分。将所述级分加载至脱盐柱 (GE Healthcare Bioscience Co., Ltd.生产, 2 x 5 ml HiTrap脱盐柱串联),并用含有5 mM 磷酸钠、50 mM MES和20 mM NaCl的缓冲液(pH 6.5)替换液体。此外,将经过缓冲液替换的抗体溶液加载至陶瓷羟基磷灰石柱 (Japan Bio-Rad Laboratories, Inc., Bio-Scale CHT2-1 羟基磷灰石柱 (2 ml体积)),所述柱用含有5 mM NaPi、50 mM MES和20 mM NaCl的缓冲液(pH 6.5)平衡。然后,使用氯化钠进行线性浓度梯度洗脱,并收集含有抗体的级分。将所述级分加载至脱盐柱 (GE Healthcare Bioscience Co., Ltd.生产, 2 x 5 ml HiTrap脱盐柱串联),并用CBS (含有140 mM 氯化钠的10 mM 柠檬酸盐缓冲液, pH 6.0)替换液体。最后,使用离心超滤(UF)过滤器装置VIVASPIN 20 (分离分子量: 30 K, Sartorius Co., Ltd., 在4ºC),浓缩得到的溶液,将IgG的浓度调至1.0 mg/ml或更高,将如此得到的溶液用作纯化的样品。各种纯化的样品的体积如下:h#32A1-H1-1/L5:2.2 ml,h#32A1-H5/L5:1.8 ml,h#32A1-H1-1/L2-16:6.0 ml,和h#32A1-H1-1/L2-15:2.2 ml。
实施例29
基于小鼠破骨细胞形成试验,评价大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体的生物活性
通过部分地改进在实施例9中所述的方法,评价了在实施例24和28中得到的人源化抗体所表现出的对小鼠破骨细胞形成的抑制作用。在含有10% FBS和10 ng/ml M-CSF (R&D Systems, Inc.生产)的α-MEM培养基中,以1.5 x 105个细胞/ml,制备通过实施例8的方法制备的小鼠骨髓非贴壁细胞,以200 μl的量,将得到的细胞制品接种进96-孔板的每个孔中,并在CO2 培养箱中培养细胞2天。除去96-孔板中的旧培养液,将100 μl MEM-α培养基加入每个孔中,所述100 μl MEM-α培养基含有 10% FBS,其中已经添加了人RANKL (RANKL, Peprotech, Inc.生产)和M-CSF,终浓度分别为20 ng/ml和10 ng/ml。向该细胞培养液中,以3-100 ng/ml的浓度,添加在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体)或在实施例24中制备的6类人源化的#32A1抗体中的每一种(h#32A1-T1、h#32A1-T2、h#32A1-T3、h#32A1-T4、h#32A1-T5和h#32A1-T6),在CO2培养箱中培养细胞另外3天。培养结束后,通过下述方法测量形成的破骨细胞的酒石酸盐-抗性的酸性磷酸酶(TRAP)的活性。通过抽吸去除96-孔板的每个孔中的培养液,向每个孔中加入50 μl 含有1% Triton X-100的50 mM柠檬酸钠缓冲液(pH 6.1)。然后,在板振荡器上振荡板5分钟,以裂解细胞。向每个孔中加入50μl底物溶液(50 mM柠檬酸钠缓冲液(pH 6.1),其含有5 mg/ml 对硝基苯基磷酸酯和0.46%酒石酸钠),并将该板在室温温育10分钟。温育后,向96孔板的各个孔中加入50μl 1N氢氧化钠溶液以停止酶反应。酶反应停止后,测定各个孔在405nm的吸光度,得到的测量结果用作TRAP活性的指标。结果如图44和45所示。此外,对于在实施例6中制备的大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体)、在实施例19中制备的#32A1抗体的人嵌合抗体、和在实施例28中制备的4类人源化的#32A1抗体(h#32A1-H1-1/L5、h#32A1-H5/L5、h#32A1-H1-1/L2-16和h#32A1-H1-1/L2-15),通过与该试验相同的方法,评价了生物活性,结果如图46和47所示。在评价其活性的10类人源化的#32A1抗体(h#32A1-T1、h#32A1-T2、h#32A1-T3、h#32A1-T4、h#32A1-T5、h#32A1-T6、h#32A1-H1-1/L5、h#32A1-H5/L5、h#32A1-H1-1/L2-16和h#32A1-H1-1/L2-15)的所有情况下,观察到基本上与大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体)或#32A1抗体的人嵌合抗体相当的抑制破骨细胞形成的强活性。
实施例30
添加大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体对正常人破骨细胞前体细胞的骨吸收活性的影响(使用胶原-包被的板进行评价)
按照细胞所附的说明书,以1×104个细胞/孔,将正常人破骨细胞前体细胞(正常人天然的破骨细胞前体细胞, 购自Sanko Junyaku Co., Ltd., 目录号2T-110)接种于96-孔OsteoLyse细胞培养板(购自Sanko Junyaku Co., Ltd., 目录号PA-1500)中。使用添加了OPGM补充套装 (购自Sanko Junyaku Co., Ltd., 目录号PT-9501)的破骨细胞前体细胞用最低必需培养基(OPBM, 购自Sanko Junyaku Co., Ltd., 目录号PT-8201)作为培养基,所述OPGM补充套装含有胎牛血清(终浓度: 10%)、人RANKL (终浓度: 63.8 ng/ml)、人M-CSF (终浓度: 33 ng/ml)等。以4、20、100或500 ng/ml的终浓度,向得到的培养上清液中添加在实施例6中制备的每种大鼠抗-小鼠Siglec-15 单克隆抗体(#32A1抗体)、在实施例19中制备的#32A1抗体的人嵌合抗体和在实施例24和28中制备的10类人源化的#32A1抗体(h#32A1-T1、h#32A1-T2、h#32A1-T3、h#32A1-T4、h#32A1-T5、h#32A1-T6、h#32A1-H1-1/L5、h#32A1-H5/L5、h#32A1-H1-1/L2-16和h#32A1-H1-1/L2-15),在CO2培养箱中培养细胞5天。收集培养上清液的10 μl 等分试样,向其中添加200 μl 包含在OsteoLyse测定试剂盒中的荧光团释放试剂,使用荧光板读数器 (ARVO MX, Perkin Elmer Inc.生产),进行适合使用铕的荧光测量的时间分辨荧光测定 (时间分辨荧光的荧光计) (激发: 340 nm, 发射: 615 nm),由此测定在培养上清液中释放的游离荧光胶原片段的量(图48和49)。结果,大鼠#32A1抗体或人嵌合#32A1抗体的加入以浓度依赖的方式减少了通过添加RANKL所增加的荧光胶原片段的量。以相同的方式,也观察到由检查的所有10类人源化的#32A1抗体进行的浓度-依赖性的抑制。该结果表明:人破骨细胞的骨吸收活性被特异性地结合Siglec-15蛋白的人源化的#32A1抗体抑制。
实施例31
使用卵巢切除的大鼠,对大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体进行生物学评价
通过在实施例15中所述的方法,可以评价在实施例24和28中得到的人源化抗体所表现出的对卵巢切除的大鼠中骨矿物质密度的降低的抑制作用。
实施例32
基于由TNFα刺激的小鼠破骨细胞形成试验,评价大鼠抗-小鼠Siglec-15 单克隆抗体#32A1的人源化抗体的生物活性
通过在实施例26中所述的方法,可以评价在实施例24和28中得到的人源化抗体所表现出的对由TNFα刺激引起的小鼠破骨细胞形成的抑制作用。
实施例33
使用示差扫描量热法(DSC),测定人源化抗体的热稳定性
对于h#32A1-H5/L5、h#32A1-H1-1/L5、h#32A1-H1-1/L2-15和h#32A1-H1-1/L2-16,测定了热稳定性。以0.5 mg/ml的浓度 (在CBS溶液中)制备这些样品中的每一种,并将其400 μl 等分试样用作样品溶液,进行DSC测量。DSC测量的条件设定如下。也就是说,最初温度设定为20ºC,最终温度设定为100ºC,温度增加速率设定为60ºC/小时,过滤时间设定为10 秒。使用CBS作为参照溶液。使用MicroCal Inc. USA生产的VP-毛细管DSC平台作为进行DSC测量的仪器。从为每种样品溶液得到的扫描曲线上减去基线 (通过也在样品池中装填入参照溶液所得到的扫描曲线),由此进行基线校正。图50显示了h#32A1-H5/L5的热分析图,图51 显示了h#32A1-H1-1/L5的热分析图,图52 显示了h#32A1-H1-1/L2-15的热分析图,且图53 显示了h#32A1-H1-1/L2-16的热分析图。当将每个热分析图中的峰顶值取作Fab区中的热变性中点温度(Tm)时,h#32A1-H5/L5的Tm值是81.2ºC,h#32A1-H1-1/L5的Tm 值是84.1ºC,h#32A1-H1-1/L2-15的Tm 值是80.0ºC,且h#32A1-H1-1/L2-16 (IgG2)的Tm 值是77.9ºC。
工业适用性
本发明的嵌合的或人源化的抗-Siglec-15抗体具有抑制破骨细胞分化或骨吸收活性的能力,并且含有所述抗-Siglec-15抗体的药物组合物可以是骨代谢异常疾病的治疗剂或预防剂。
登录号FERM BP-10999。
序列表
<110> DAIICHI SANKYO COMPANY, LIMITED
<120> 抗-Siglec-15抗体
<130> DSPCT-FP1021
<150> JP 2009-94613
<151> 2009-04-09
<160> 106
<170> PatentIn version 3.4
<210> 1
<211> 987
<212> DNA
<213> 智人
<220>
<221> CDS
<222> (1)..(987)
<223> 发明人: Hiruma, Yoshiharu; Tsuda, Eisuke
<223> 发明人: Takizawa, Takeshi; Nakayama, Makiko
<400> 1
atg gaa aag tcc atc tgg ctg ctg gcc tgc ttg gcg tgg gtt ctc ccg 48
Met Glu Lys Ser Ile Trp Leu Leu Ala Cys Leu Ala Trp Val Leu Pro
1 5 10 15
aca ggc tca ttt gtg aga act aaa ata gat act acg gag aac ttg ctc 96
Thr Gly Ser Phe Val Arg Thr Lys Ile Asp Thr Thr Glu Asn Leu Leu
20 25 30
aac aca gag gtg cac agc tcg cca gcg cag cgc tgg tcc atg cag gtg 144
Asn Thr Glu Val His Ser Ser Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
cca ccc gag gtg agc gcg gag gca ggc gac gcg gca gtg ctg ccc tgc 192
Pro Pro Glu Val Ser Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
acc ttc acg cac ccg cac cgc cac tac gac ggg ccg ctg acg gcc atc 240
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
tgg cgc gcg ggc gag ccc tat gcg ggc ccg cag gtg ttc cgc tgc gct 288
Trp Arg Ala Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Ala
85 90 95
gcg gcg cgg ggc agc gag ctc tgc cag acg gcg ctg agc ctg cac ggc 336
Ala Ala Arg Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
cgc ttc cgg ctg ctg ggc aac ccg cgc cgc aac gac ctc tcg ctg cgc 384
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
gtc gag cgc ctc gcc ctg gct gac gac cgc cgc tac ttc tgc cgc gtc 432
Val Glu Arg Leu Ala Leu Ala Asp Asp Arg Arg Tyr Phe Cys Arg Val
130 135 140
gag ttc gcc ggc gac gtc cat gac cgc tac gag agc cgc cac ggc gtc 480
Glu Phe Ala Gly Asp Val His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
cgg ctg cac gtg aca gcc gcg ccg cgg atc gtc aac atc tcg gtg ctg 528
Arg Leu His Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
ccc agt ccg gct cac gcc ttc cgc gcg ctc tgc act gcc gaa ggg gag 576
Pro Ser Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
ccg ccg ccc gcc ctc gcc tgg tcc ggc ccg gcc ctg ggc aac agc ttg 624
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Leu Gly Asn Ser Leu
195 200 205
gca gcc gtg cgg agc ccg cgt gag ggt cac ggc cac cta gtg acc gcc 672
Ala Ala Val Arg Ser Pro Arg Glu Gly His Gly His Leu Val Thr Ala
210 215 220
gaa ctg ccc gca ctg acc cat gac ggc cgc tac acg tgt acg gcc gcc 720
Glu Leu Pro Ala Leu Thr His Asp Gly Arg Tyr Thr Cys Thr Ala Ala
225 230 235 240
aac agc ctg ggc cgc tcc gag gcc agc gtc tac ctg ttc cgc ttc cat 768
Asn Ser Leu Gly Arg Ser Glu Ala Ser Val Tyr Leu Phe Arg Phe His
245 250 255
ggc gcc agc ggg gcc tcg acg gtc gcc ctc ctg ctc ggc gct ctc ggc 816
Gly Ala Ser Gly Ala Ser Thr Val Ala Leu Leu Leu Gly Ala Leu Gly
260 265 270
ttc aag gcg ctg ctg ctg ctc ggg gtc ctg gcc gcc cgc gct gcc cgc 864
Phe Lys Ala Leu Leu Leu Leu Gly Val Leu Ala Ala Arg Ala Ala Arg
275 280 285
cgc cgc cca gag cat ctg gac acc ccg gac acc cca cca cgg tcc cag 912
Arg Arg Pro Glu His Leu Asp Thr Pro Asp Thr Pro Pro Arg Ser Gln
290 295 300
gcc cag gag tcc aat tat gaa aat ttg agc cag atg aac ccc cgg agc 960
Ala Gln Glu Ser Asn Tyr Glu Asn Leu Ser Gln Met Asn Pro Arg Ser
305 310 315 320
cca cca gcc acc atg tgc tca ccg tga 987
Pro Pro Ala Thr Met Cys Ser Pro
325
<210> 2
<211> 328
<212> PRT
<213> 智人
<400> 2
Met Glu Lys Ser Ile Trp Leu Leu Ala Cys Leu Ala Trp Val Leu Pro
1 5 10 15
Thr Gly Ser Phe Val Arg Thr Lys Ile Asp Thr Thr Glu Asn Leu Leu
20 25 30
Asn Thr Glu Val His Ser Ser Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
Pro Pro Glu Val Ser Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
Trp Arg Ala Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Ala
85 90 95
Ala Ala Arg Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
Val Glu Arg Leu Ala Leu Ala Asp Asp Arg Arg Tyr Phe Cys Arg Val
130 135 140
Glu Phe Ala Gly Asp Val His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
Arg Leu His Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
Pro Ser Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Leu Gly Asn Ser Leu
195 200 205
Ala Ala Val Arg Ser Pro Arg Glu Gly His Gly His Leu Val Thr Ala
210 215 220
Glu Leu Pro Ala Leu Thr His Asp Gly Arg Tyr Thr Cys Thr Ala Ala
225 230 235 240
Asn Ser Leu Gly Arg Ser Glu Ala Ser Val Tyr Leu Phe Arg Phe His
245 250 255
Gly Ala Ser Gly Ala Ser Thr Val Ala Leu Leu Leu Gly Ala Leu Gly
260 265 270
Phe Lys Ala Leu Leu Leu Leu Gly Val Leu Ala Ala Arg Ala Ala Arg
275 280 285
Arg Arg Pro Glu His Leu Asp Thr Pro Asp Thr Pro Pro Arg Ser Gln
290 295 300
Ala Gln Glu Ser Asn Tyr Glu Asn Leu Ser Gln Met Asn Pro Arg Ser
305 310 315 320
Pro Pro Ala Thr Met Cys Ser Pro
325
<210> 3
<211> 1026
<212> DNA
<213> 小家鼠
<220>
<221> CDS
<222> (1)..(1026)
<400> 3
atg gag ggg tcc ctc caa ctc ctg gcc tgc ttg gcc tgt gtg ctc cag 48
Met Glu Gly Ser Leu Gln Leu Leu Ala Cys Leu Ala Cys Val Leu Gln
1 5 10 15
atg gga tcc ctt gtg aaa act aga aga gac gct tcg ggg gat ctg ctc 96
Met Gly Ser Leu Val Lys Thr Arg Arg Asp Ala Ser Gly Asp Leu Leu
20 25 30
aac aca gag gcg cac agt gcc ccg gcg cag cgc tgg tcc atg cag gtg 144
Asn Thr Glu Ala His Ser Ala Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
ccc gcg gag gtg aac gcg gag gct ggc gac gcg gcg gtg ctg ccc tgc 192
Pro Ala Glu Val Asn Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
acc ttc acg cac ccg cac cgc cac tac gac ggg ccg ctg acg gcc atc 240
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
tgg cgc tcg ggc gag ccg tac gcg ggc ccg cag gtg ttc cgc tgc acc 288
Trp Arg Ser Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Thr
85 90 95
gcg gcg ccg ggc agc gag ctg tgc cag acg gcg ctg agc ctg cac ggc 336
Ala Ala Pro Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
cgc ttc cgc ctg ctg ggc aac ccg cgc cgc aac gac ctg tcc ctg cgc 384
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
gtc gag cgc ctc gcc ctg gcg gac agc ggc cgc tac ttc tgc cgc gtg 432
Val Glu Arg Leu Ala Leu Ala Asp Ser Gly Arg Tyr Phe Cys Arg Val
130 135 140
gag ttc acc ggc gac gcc cac gat cgc tat gag agt cgc cat ggg gtc 480
Glu Phe Thr Gly Asp Ala His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
cgt ctg cgc gtg act gct gcg ccg cgg atc gtc aac atc tcg gtg ctg 528
Arg Leu Arg Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
ccg ggc ccc gcg cac gcc ttc cgc gcg ctc tgc acc gcc gag ggg gag 576
Pro Gly Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
ccc ccg ccc gcc ctc gcc tgg tcg ggt ccc gcc cca ggc aac agc tcc 624
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Pro Gly Asn Ser Ser
195 200 205
gct gcc ctg cag ggc cag ggt cac ggc tac cag gtg acc gcc gag ttg 672
Ala Ala Leu Gln Gly Gln Gly His Gly Tyr Gln Val Thr Ala Glu Leu
210 215 220
ccc gcg ctg acc cgc gac ggc cgc tac acg tgc acg gcg gcc aat agc 720
Pro Ala Leu Thr Arg Asp Gly Arg Tyr Thr Cys Thr Ala Ala Asn Ser
225 230 235 240
ctg ggc cgc gcc gag gcc agc gtc tac ctg ttc cgc ttc cac ggc gcc 768
Leu Gly Arg Ala Glu Ala Ser Val Tyr Leu Phe Arg Phe His Gly Ala
245 250 255
ccc gga acc tcg acc cta gcg ctc ctg ctg ggc gcg ctg ggc ctc aag 816
Pro Gly Thr Ser Thr Leu Ala Leu Leu Leu Gly Ala Leu Gly Leu Lys
260 265 270
gcc ttg ctg ctg ctt ggc att ctg gga gcg cgt gcc acc cga cgc cga 864
Ala Leu Leu Leu Leu Gly Ile Leu Gly Ala Arg Ala Thr Arg Arg Arg
275 280 285
cta gat cac ctg gtc ccc cag gac acc cct cca cgg tct cag gct cag 912
Leu Asp His Leu Val Pro Gln Asp Thr Pro Pro Arg Ser Gln Ala Gln
290 295 300
gag tcc aat tat gaa aat ttg agc cag atg agt cct cca ggc cac cag 960
Glu Ser Asn Tyr Glu Asn Leu Ser Gln Met Ser Pro Pro Gly His Gln
305 310 315 320
ctg cca cgt gtt tgc tgt gag gaa ctc ctc agc cat cac cat cta gtc 1008
Leu Pro Arg Val Cys Cys Glu Glu Leu Leu Ser His His His Leu Val
325 330 335
att cac cat gag aaa taa 1026
Ile His His Glu Lys
340
<210> 4
<211> 341
<212> PRT
<213> 小家鼠
<400> 4
Met Glu Gly Ser Leu Gln Leu Leu Ala Cys Leu Ala Cys Val Leu Gln
1 5 10 15
Met Gly Ser Leu Val Lys Thr Arg Arg Asp Ala Ser Gly Asp Leu Leu
20 25 30
Asn Thr Glu Ala His Ser Ala Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
Pro Ala Glu Val Asn Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
Trp Arg Ser Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Thr
85 90 95
Ala Ala Pro Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
Val Glu Arg Leu Ala Leu Ala Asp Ser Gly Arg Tyr Phe Cys Arg Val
130 135 140
Glu Phe Thr Gly Asp Ala His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
Arg Leu Arg Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
Pro Gly Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Pro Gly Asn Ser Ser
195 200 205
Ala Ala Leu Gln Gly Gln Gly His Gly Tyr Gln Val Thr Ala Glu Leu
210 215 220
Pro Ala Leu Thr Arg Asp Gly Arg Tyr Thr Cys Thr Ala Ala Asn Ser
225 230 235 240
Leu Gly Arg Ala Glu Ala Ser Val Tyr Leu Phe Arg Phe His Gly Ala
245 250 255
Pro Gly Thr Ser Thr Leu Ala Leu Leu Leu Gly Ala Leu Gly Leu Lys
260 265 270
Ala Leu Leu Leu Leu Gly Ile Leu Gly Ala Arg Ala Thr Arg Arg Arg
275 280 285
Leu Asp His Leu Val Pro Gln Asp Thr Pro Pro Arg Ser Gln Ala Gln
290 295 300
Glu Ser Asn Tyr Glu Asn Leu Ser Gln Met Ser Pro Pro Gly His Gln
305 310 315 320
Leu Pro Arg Val Cys Cys Glu Glu Leu Leu Ser His His His Leu Val
325 330 335
Ile His His Glu Lys
340
<210> 5
<211> 774
<212> DNA
<213> 小家鼠
<220>
<221> CDS
<222> (1)..(774)
<400> 5
atg gag ggg tcc ctc caa ctc ctg gcc tgc ttg gcc tgt gtg ctc cag 48
Met Glu Gly Ser Leu Gln Leu Leu Ala Cys Leu Ala Cys Val Leu Gln
1 5 10 15
atg gga tcc ctt gtg aaa act aga aga gac gct tcg ggg gat ctg ctc 96
Met Gly Ser Leu Val Lys Thr Arg Arg Asp Ala Ser Gly Asp Leu Leu
20 25 30
aac aca gag gcg cac agt gcc ccg gcg cag cgc tgg tcc atg cag gtg 144
Asn Thr Glu Ala His Ser Ala Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
ccc gcg gag gtg aac gcg gag gct ggc gac gcg gcg gtg ctg ccc tgc 192
Pro Ala Glu Val Asn Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
acc ttc acg cac ccg cac cgc cac tac gac ggg ccg ctg acg gcc atc 240
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
tgg cgc tcg ggc gag ccg tac gcg ggc ccg cag gtg ttc cgc tgc acc 288
Trp Arg Ser Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Thr
85 90 95
gcg gcg ccg ggc agc gag ctg tgc cag acg gcg ctg agc ctg cac ggc 336
Ala Ala Pro Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
cgc ttc cgc ctg ctg ggc aac ccg cgc cgc aac gac ctg tcc ctg cgc 384
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
gtc gag cgc ctc gcc ctg gcg gac agc ggc cgc tac ttc tgc cgc gtg 432
Val Glu Arg Leu Ala Leu Ala Asp Ser Gly Arg Tyr Phe Cys Arg Val
130 135 140
gag ttc acc ggc gac gcc cac gat cgc tat gag agt cgc cat ggg gtc 480
Glu Phe Thr Gly Asp Ala His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
cgt ctg cgc gtg act gct gcg ccg cgg atc gtc aac atc tcg gtg ctg 528
Arg Leu Arg Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
ccg ggc ccc gcg cac gcc ttc cgc gcg ctc tgc acc gcc gag ggg gag 576
Pro Gly Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
ccc ccg ccc gcc ctc gcc tgg tcg ggt ccc gcc cca ggc aac agc tcc 624
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Pro Gly Asn Ser Ser
195 200 205
gct gcc ctg cag ggc cag ggt cac ggc tac cag gtg acc gcc gag ttg 672
Ala Ala Leu Gln Gly Gln Gly His Gly Tyr Gln Val Thr Ala Glu Leu
210 215 220
ccc gcg ctg acc cgc gac ggc cgc tac acg tgc acg gcg gcc aat agc 720
Pro Ala Leu Thr Arg Asp Gly Arg Tyr Thr Cys Thr Ala Ala Asn Ser
225 230 235 240
ctg ggc cgc gcc gag gcc agc gtc tac ctg ttc cgc ttc cac ggc gcc 768
Leu Gly Arg Ala Glu Ala Ser Val Tyr Leu Phe Arg Phe His Gly Ala
245 250 255
ccc gga 774
Pro Gly
<210> 6
<211> 258
<212> PRT
<213> 小家鼠
<400> 6
Met Glu Gly Ser Leu Gln Leu Leu Ala Cys Leu Ala Cys Val Leu Gln
1 5 10 15
Met Gly Ser Leu Val Lys Thr Arg Arg Asp Ala Ser Gly Asp Leu Leu
20 25 30
Asn Thr Glu Ala His Ser Ala Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
Pro Ala Glu Val Asn Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
Trp Arg Ser Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Thr
85 90 95
Ala Ala Pro Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
Val Glu Arg Leu Ala Leu Ala Asp Ser Gly Arg Tyr Phe Cys Arg Val
130 135 140
Glu Phe Thr Gly Asp Ala His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
Arg Leu Arg Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
Pro Gly Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Pro Gly Asn Ser Ser
195 200 205
Ala Ala Leu Gln Gly Gln Gly His Gly Tyr Gln Val Thr Ala Glu Leu
210 215 220
Pro Ala Leu Thr Arg Asp Gly Arg Tyr Thr Cys Thr Ala Ala Asn Ser
225 230 235 240
Leu Gly Arg Ala Glu Ala Ser Val Tyr Leu Phe Arg Phe His Gly Ala
245 250 255
Pro Gly
<210> 7
<211> 55
<212> DNA
<213> 人工序列
<220>
<223> 可溶的小鼠Siglec-15 cDNA的PCR引物
<400> 7
ggggacaagt ttgtacaaaa aagcaggctt caccatggag gggtccctcc aactc 55
<210> 8
<211> 55
<212> DNA
<213> 人工序列
<220>
<223> 可溶的小鼠Siglec-15 cDNA的PCR引物
<400> 8
ggggaccact ttgtacaaga aagctgggtc tccgggggcg ccgtggaagc ggaac 55
<210> 9
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 可溶的小鼠Siglec-15 cDNA的测序引物
<400> 9
tgcgtgaagg tgcagggcag 20
<210> 10
<211> 17
<212> DNA
<213> 人工序列
<220>
<223> 可溶的小鼠Siglec-15 cDNA的测序引物
<400> 10
cctcgcctgg tcgggtc 17
<210> 11
<211> 891
<212> DNA
<213> 人工序列
<220>
<223> His-标记的小鼠Siglec-15 cDNA
<220>
<221> CDS
<222> (1)..(891)
<400> 11
atg gag ggg tcc ctc caa ctc ctg gcc tgc ttg gcc tgt gtg ctc cag 48
Met Glu Gly Ser Leu Gln Leu Leu Ala Cys Leu Ala Cys Val Leu Gln
1 5 10 15
atg gga tcc ctt gtg aaa act aga aga gac gct tcg ggg gat ctg ctc 96
Met Gly Ser Leu Val Lys Thr Arg Arg Asp Ala Ser Gly Asp Leu Leu
20 25 30
aac aca gag gcg cac agt gcc ccg gcg cag cgc tgg tcc atg cag gtg 144
Asn Thr Glu Ala His Ser Ala Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
ccc gcg gag gtg aac gcg gag gct ggc gac gcg gcg gtg ctg ccc tgc 192
Pro Ala Glu Val Asn Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
acc ttc acg cac ccg cac cgc cac tac gac ggg ccg ctg acg gcc atc 240
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
tgg cgc tcg ggc gag ccg tac gcg ggc ccg cag gtg ttc cgc tgc acc 288
Trp Arg Ser Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Thr
85 90 95
gcg gcg ccg ggc agc gag ctg tgc cag acg gcg ctg agc ctg cac ggc 336
Ala Ala Pro Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
cgc ttc cgc ctg ctg ggc aac ccg cgc cgc aac gac ctg tcc ctg cgc 384
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
gtc gag cgc ctc gcc ctg gcg gac agc ggc cgc tac ttc tgc cgc gtg 432
Val Glu Arg Leu Ala Leu Ala Asp Ser Gly Arg Tyr Phe Cys Arg Val
130 135 140
gag ttc acc ggc gac gcc cac gat cgc tat gag agt cgc cat ggg gtc 480
Glu Phe Thr Gly Asp Ala His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
cgt ctg cgc gtg act gct gcg ccg cgg atc gtc aac atc tcg gtg ctg 528
Arg Leu Arg Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
ccg ggc ccc gcg cac gcc ttc cgc gcg ctc tgc acc gcc gag ggg gag 576
Pro Gly Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
ccc ccg ccc gcc ctc gcc tgg tcg ggt ccc gcc cca ggc aac agc tcc 624
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Pro Gly Asn Ser Ser
195 200 205
gct gcc ctg cag ggc cag ggt cac ggc tac cag gtg acc gcc gag ttg 672
Ala Ala Leu Gln Gly Gln Gly His Gly Tyr Gln Val Thr Ala Glu Leu
210 215 220
ccc gcg ctg acc cgc gac ggc cgc tac acg tgc acg gcg gcc aat agc 720
Pro Ala Leu Thr Arg Asp Gly Arg Tyr Thr Cys Thr Ala Ala Asn Ser
225 230 235 240
ctg ggc cgc gcc gag gcc agc gtc tac ctg ttc cgc ttc cac ggc gcc 768
Leu Gly Arg Ala Glu Ala Ser Val Tyr Leu Phe Arg Phe His Gly Ala
245 250 255
ccc gga gac cca gct ttc ttg tac aaa gtg gtt gat atc gaa ggt cgt 816
Pro Gly Asp Pro Ala Phe Leu Tyr Lys Val Val Asp Ile Glu Gly Arg
260 265 270
ggg ggt aag cct atc cct aac cct ctc ctc ggt ctc gat tct acg cgt 864
Gly Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser Thr Arg
275 280 285
acc ggt cat cat cac cat cac cat tga 891
Thr Gly His His His His His His
290 295
<210> 12
<211> 296
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 12
Met Glu Gly Ser Leu Gln Leu Leu Ala Cys Leu Ala Cys Val Leu Gln
1 5 10 15
Met Gly Ser Leu Val Lys Thr Arg Arg Asp Ala Ser Gly Asp Leu Leu
20 25 30
Asn Thr Glu Ala His Ser Ala Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
Pro Ala Glu Val Asn Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
Trp Arg Ser Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Thr
85 90 95
Ala Ala Pro Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
Val Glu Arg Leu Ala Leu Ala Asp Ser Gly Arg Tyr Phe Cys Arg Val
130 135 140
Glu Phe Thr Gly Asp Ala His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
Arg Leu Arg Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
Pro Gly Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Pro Gly Asn Ser Ser
195 200 205
Ala Ala Leu Gln Gly Gln Gly His Gly Tyr Gln Val Thr Ala Glu Leu
210 215 220
Pro Ala Leu Thr Arg Asp Gly Arg Tyr Thr Cys Thr Ala Ala Asn Ser
225 230 235 240
Leu Gly Arg Ala Glu Ala Ser Val Tyr Leu Phe Arg Phe His Gly Ala
245 250 255
Pro Gly Asp Pro Ala Phe Leu Tyr Lys Val Val Asp Ile Glu Gly Arg
260 265 270
Gly Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser Thr Arg
275 280 285
Thr Gly His His His His His His
290 295
<210> 13
<211> 1512
<212> DNA
<213> 人工序列
<220>
<223> Fc-融合的小鼠Siglec-15 cDNA
<220>
<221> CDS
<222> (1)..(1512)
<400> 13
atg gag ggg tcc ctc caa ctc ctg gcc tgc ttg gcc tgt gtg ctc cag 48
Met Glu Gly Ser Leu Gln Leu Leu Ala Cys Leu Ala Cys Val Leu Gln
1 5 10 15
atg gga tcc ctt gtg aaa act aga aga gac gct tcg ggg gat ctg ctc 96
Met Gly Ser Leu Val Lys Thr Arg Arg Asp Ala Ser Gly Asp Leu Leu
20 25 30
aac aca gag gcg cac agt gcc ccg gcg cag cgc tgg tcc atg cag gtg 144
Asn Thr Glu Ala His Ser Ala Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
ccc gcg gag gtg aac gcg gag gct ggc gac gcg gcg gtg ctg ccc tgc 192
Pro Ala Glu Val Asn Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
acc ttc acg cac ccg cac cgc cac tac gac ggg ccg ctg acg gcc atc 240
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
tgg cgc tcg ggc gag ccg tac gcg ggc ccg cag gtg ttc cgc tgc acc 288
Trp Arg Ser Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Thr
85 90 95
gcg gcg ccg ggc agc gag ctg tgc cag acg gcg ctg agc ctg cac ggc 336
Ala Ala Pro Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
cgc ttc cgc ctg ctg ggc aac ccg cgc cgc aac gac ctg tcc ctg cgc 384
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
gtc gag cgc ctc gcc ctg gcg gac agc ggc cgc tac ttc tgc cgc gtg 432
Val Glu Arg Leu Ala Leu Ala Asp Ser Gly Arg Tyr Phe Cys Arg Val
130 135 140
gag ttc acc ggc gac gcc cac gat cgc tat gag agt cgc cat ggg gtc 480
Glu Phe Thr Gly Asp Ala His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
cgt ctg cgc gtg act gct gcg ccg cgg atc gtc aac atc tcg gtg ctg 528
Arg Leu Arg Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
ccg ggc ccc gcg cac gcc ttc cgc gcg ctc tgc acc gcc gag ggg gag 576
Pro Gly Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
ccc ccg ccc gcc ctc gcc tgg tcg ggt ccc gcc cca ggc aac agc tcc 624
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Pro Gly Asn Ser Ser
195 200 205
gct gcc ctg cag ggc cag ggt cac ggc tac cag gtg acc gcc gag ttg 672
Ala Ala Leu Gln Gly Gln Gly His Gly Tyr Gln Val Thr Ala Glu Leu
210 215 220
ccc gcg ctg acc cgc gac ggc cgc tac acg tgc acg gcg gcc aat agc 720
Pro Ala Leu Thr Arg Asp Gly Arg Tyr Thr Cys Thr Ala Ala Asn Ser
225 230 235 240
ctg ggc cgc gcc gag gcc agc gtc tac ctg ttc cgc ttc cac ggc gcc 768
Leu Gly Arg Ala Glu Ala Ser Val Tyr Leu Phe Arg Phe His Gly Ala
245 250 255
ccc gga gac cca gct ttc ttg tac aaa gtg gtt gat atc cag gca gag 816
Pro Gly Asp Pro Ala Phe Leu Tyr Lys Val Val Asp Ile Gln Ala Glu
260 265 270
ccc aaa tct tgt gac aaa act cac aca tgc cca ccg tgc cca gca cct 864
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
275 280 285
gaa ctc ctg ggg gga ccg tca gtc ttc ctc ttc ccc cca aaa ccc aag 912
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
290 295 300
gac acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg gtg gtg 960
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
305 310 315 320
gac gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac gtg gac 1008
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
325 330 335
ggc gtg gag gtg cat aat gcc aag aca aag ccg cgg gag gag cag tac 1056
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
340 345 350
aac agc acg tac cgt gtg gtc agc gtc ctc acc gtc ctg cac cag gac 1104
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
355 360 365
tgg ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa gcc ctc 1152
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
370 375 380
cca gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggg cag ccc cga 1200
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
385 390 395 400
gaa cca cag gtg tac acc ctg ccc cca tcc cgg gag gag atg acc aag 1248
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
405 410 415
aac cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc agc gac 1296
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
420 425 430
atc gcc gtg gag tgg gag agc aat ggg cag ccg gag aac aac tac aag 1344
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
435 440 445
acc acg cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc tat agc 1392
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
450 455 460
aag ctc acc gtg gac aag agc agg tgg cag cag ggg aac gtc ttc tca 1440
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
465 470 475 480
tgc tcc gtg atg cat gag gct ctg cac aac cac tac acg cag aag agc 1488
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
485 490 495
ctc tcc ctg tct ccg ggt aaa taa 1512
Leu Ser Leu Ser Pro Gly Lys
500
<210> 14
<211> 503
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 14
Met Glu Gly Ser Leu Gln Leu Leu Ala Cys Leu Ala Cys Val Leu Gln
1 5 10 15
Met Gly Ser Leu Val Lys Thr Arg Arg Asp Ala Ser Gly Asp Leu Leu
20 25 30
Asn Thr Glu Ala His Ser Ala Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
Pro Ala Glu Val Asn Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
Trp Arg Ser Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Thr
85 90 95
Ala Ala Pro Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
Val Glu Arg Leu Ala Leu Ala Asp Ser Gly Arg Tyr Phe Cys Arg Val
130 135 140
Glu Phe Thr Gly Asp Ala His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
Arg Leu Arg Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
Pro Gly Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Pro Gly Asn Ser Ser
195 200 205
Ala Ala Leu Gln Gly Gln Gly His Gly Tyr Gln Val Thr Ala Glu Leu
210 215 220
Pro Ala Leu Thr Arg Asp Gly Arg Tyr Thr Cys Thr Ala Ala Asn Ser
225 230 235 240
Leu Gly Arg Ala Glu Ala Ser Val Tyr Leu Phe Arg Phe His Gly Ala
245 250 255
Pro Gly Asp Pro Ala Phe Leu Tyr Lys Val Val Asp Ile Gln Ala Glu
260 265 270
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
275 280 285
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
290 295 300
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
305 310 315 320
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
325 330 335
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
340 345 350
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
355 360 365
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
370 375 380
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
385 390 395 400
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
405 410 415
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
420 425 430
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
435 440 445
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
450 455 460
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
465 470 475 480
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
485 490 495
Leu Ser Leu Ser Pro Gly Lys
500
<210> 15
<211> 780
<212> DNA
<213> 智人
<220>
<221> CDR
<222> (1)..(780)
<220>
<221> CDS
<222> (1)..(780)
<400> 15
atg gaa aag tcc atc tgg ctg ctg gcc tgc ttg gcg tgg gtt ctc ccg 48
Met Glu Lys Ser Ile Trp Leu Leu Ala Cys Leu Ala Trp Val Leu Pro
1 5 10 15
aca ggc tca ttt gtg aga act aaa ata gat act acg gag aac ttg ctc 96
Thr Gly Ser Phe Val Arg Thr Lys Ile Asp Thr Thr Glu Asn Leu Leu
20 25 30
aac aca gag gtg cac agc tcg cca gcg cag cgc tgg tcc atg cag gtg 144
Asn Thr Glu Val His Ser Ser Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
cca ccc gag gtg agc gcg gag gca ggc gac gcg gca gtg ctg ccc tgc 192
Pro Pro Glu Val Ser Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
acc ttc acg cac ccg cac cgc cac tac gac ggg ccg ctg acg gcc atc 240
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
tgg cgc gcg ggc gag ccc tat gcg ggc ccg cag gtg ttc cgc tgc gct 288
Trp Arg Ala Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Ala
85 90 95
gcg gcg cgg ggc agc gag ctc tgc cag acg gcg ctg agc ctg cac ggc 336
Ala Ala Arg Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
cgc ttc cgg ctg ctg ggc aac ccg cgc cgc aac gac ctc tcg ctg cgc 384
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
gtc gag cgc ctc gcc ctg gct gac gac cgc cgc tac ttc tgc cgc gtc 432
Val Glu Arg Leu Ala Leu Ala Asp Asp Arg Arg Tyr Phe Cys Arg Val
130 135 140
gag ttc gcc ggc gac gtc cat gac cgc tac gag agc cgc cac ggc gtc 480
Glu Phe Ala Gly Asp Val His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
cgg ctg cac gtg aca gcc gcg ccg cgg atc gtc aac atc tcg gtg ctg 528
Arg Leu His Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
ccc agt ccg gct cac gcc ttc cgc gcg ctc tgc act gcc gaa ggg gag 576
Pro Ser Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
ccg ccg ccc gcc ctc gcc tgg tcc ggc ccg gcc ctg ggc aac agc ttg 624
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Leu Gly Asn Ser Leu
195 200 205
gca gcc gtg cgg agc ccg cgt gag ggt cac ggc cac cta gtg acc gcc 672
Ala Ala Val Arg Ser Pro Arg Glu Gly His Gly His Leu Val Thr Ala
210 215 220
gaa ctg ccc gca ctg acc cat gac ggc cgc tac acg tgt acg gcc gcc 720
Glu Leu Pro Ala Leu Thr His Asp Gly Arg Tyr Thr Cys Thr Ala Ala
225 230 235 240
aac agc ctg ggc cgc tcc gag gcc agc gtc tac ctg ttc cgc ttc cat 768
Asn Ser Leu Gly Arg Ser Glu Ala Ser Val Tyr Leu Phe Arg Phe His
245 250 255
ggc gcc agc ggg 780
Gly Ala Ser Gly
260
<210> 16
<211> 260
<212> PRT
<213> 智人
<400> 16
Met Glu Lys Ser Ile Trp Leu Leu Ala Cys Leu Ala Trp Val Leu Pro
1 5 10 15
Thr Gly Ser Phe Val Arg Thr Lys Ile Asp Thr Thr Glu Asn Leu Leu
20 25 30
Asn Thr Glu Val His Ser Ser Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
Pro Pro Glu Val Ser Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
Trp Arg Ala Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Ala
85 90 95
Ala Ala Arg Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
Val Glu Arg Leu Ala Leu Ala Asp Asp Arg Arg Tyr Phe Cys Arg Val
130 135 140
Glu Phe Ala Gly Asp Val His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
Arg Leu His Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
Pro Ser Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Leu Gly Asn Ser Leu
195 200 205
Ala Ala Val Arg Ser Pro Arg Glu Gly His Gly His Leu Val Thr Ala
210 215 220
Glu Leu Pro Ala Leu Thr His Asp Gly Arg Tyr Thr Cys Thr Ala Ala
225 230 235 240
Asn Ser Leu Gly Arg Ser Glu Ala Ser Val Tyr Leu Phe Arg Phe His
245 250 255
Gly Ala Ser Gly
260
<210> 17
<211> 56
<212> DNA
<213> 人工序列
<220>
<223> 可溶的人Siglec-15 cDNA的PCR引物
<400> 17
ggggacaagt ttgtacaaaa aagcaggctt caccatggaa aagtccatct ggctgc 56
<210> 18
<211> 52
<212> DNA
<213> 人工序列
<220>
<223> 可溶的人Siglec-15 cDNA的PCR引物
<400> 18
ggggaccact ttgtacaaga aagctgggtc cccgctggcg ccatggaagc gg 52
<210> 19
<211> 897
<212> DNA
<213> 人工序列
<220>
<223> His-标记的人Siglec-15 cDNA
<220>
<221> CDS
<222> (1)..(897)
<400> 19
atg gaa aag tcc atc tgg ctg ctg gcc tgc ttg gcg tgg gtt ctc ccg 48
Met Glu Lys Ser Ile Trp Leu Leu Ala Cys Leu Ala Trp Val Leu Pro
1 5 10 15
aca ggc tca ttt gtg aga act aaa ata gat act acg gag aac ttg ctc 96
Thr Gly Ser Phe Val Arg Thr Lys Ile Asp Thr Thr Glu Asn Leu Leu
20 25 30
aac aca gag gtg cac agc tcg cca gcg cag cgc tgg tcc atg cag gtg 144
Asn Thr Glu Val His Ser Ser Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
cca ccc gag gtg agc gcg gag gca ggc gac gcg gca gtg ctg ccc tgc 192
Pro Pro Glu Val Ser Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
acc ttc acg cac ccg cac cgc cac tac gac ggg ccg ctg acg gcc atc 240
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
tgg cgc gcg ggc gag ccc tat gcg ggc ccg cag gtg ttc cgc tgc gct 288
Trp Arg Ala Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Ala
85 90 95
gcg gcg cgg ggc agc gag ctc tgc cag acg gcg ctg agc ctg cac ggc 336
Ala Ala Arg Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
cgc ttc cgg ctg ctg ggc aac ccg cgc cgc aac gac ctc tcg ctg cgc 384
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
gtc gag cgc ctc gcc ctg gct gac gac cgc cgc tac ttc tgc cgc gtc 432
Val Glu Arg Leu Ala Leu Ala Asp Asp Arg Arg Tyr Phe Cys Arg Val
130 135 140
gag ttc gcc ggc gac gtc cat gac cgc tac gag agc cgc cac ggc gtc 480
Glu Phe Ala Gly Asp Val His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
cgg ctg cac gtg aca gcc gcg ccg cgg atc gtc aac atc tcg gtg ctg 528
Arg Leu His Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
ccc agt ccg gct cac gcc ttc cgc gcg ctc tgc act gcc gaa ggg gag 576
Pro Ser Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
ccg ccg ccc gcc ctc gcc tgg tcc ggc ccg gcc ctg ggc aac agc ttg 624
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Leu Gly Asn Ser Leu
195 200 205
gca gcc gtg cgg agc ccg cgt gag ggt cac ggc cac cta gtg acc gcc 672
Ala Ala Val Arg Ser Pro Arg Glu Gly His Gly His Leu Val Thr Ala
210 215 220
gaa ctg ccc gca ctg acc cat gac ggc cgc tac acg tgt acg gcc gcc 720
Glu Leu Pro Ala Leu Thr His Asp Gly Arg Tyr Thr Cys Thr Ala Ala
225 230 235 240
aac agc ctg ggc cgc tcc gag gcc agc gtc tac ctg ttc cgc ttc cat 768
Asn Ser Leu Gly Arg Ser Glu Ala Ser Val Tyr Leu Phe Arg Phe His
245 250 255
ggc gcc agc ggg gac cca gct ttc ttg tac aaa gtg gtt gat atc gaa 816
Gly Ala Ser Gly Asp Pro Ala Phe Leu Tyr Lys Val Val Asp Ile Glu
260 265 270
ggt cgt ggg ggt aag cct atc cct aac cct ctc ctc ggt ctc gat tct 864
Gly Arg Gly Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser
275 280 285
acg cgt acc ggt cat cat cac cat cac cat tga 897
Thr Arg Thr Gly His His His His His His
290 295
<210> 20
<211> 298
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 20
Met Glu Lys Ser Ile Trp Leu Leu Ala Cys Leu Ala Trp Val Leu Pro
1 5 10 15
Thr Gly Ser Phe Val Arg Thr Lys Ile Asp Thr Thr Glu Asn Leu Leu
20 25 30
Asn Thr Glu Val His Ser Ser Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
Pro Pro Glu Val Ser Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
Trp Arg Ala Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Ala
85 90 95
Ala Ala Arg Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
Val Glu Arg Leu Ala Leu Ala Asp Asp Arg Arg Tyr Phe Cys Arg Val
130 135 140
Glu Phe Ala Gly Asp Val His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
Arg Leu His Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
Pro Ser Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Leu Gly Asn Ser Leu
195 200 205
Ala Ala Val Arg Ser Pro Arg Glu Gly His Gly His Leu Val Thr Ala
210 215 220
Glu Leu Pro Ala Leu Thr His Asp Gly Arg Tyr Thr Cys Thr Ala Ala
225 230 235 240
Asn Ser Leu Gly Arg Ser Glu Ala Ser Val Tyr Leu Phe Arg Phe His
245 250 255
Gly Ala Ser Gly Asp Pro Ala Phe Leu Tyr Lys Val Val Asp Ile Glu
260 265 270
Gly Arg Gly Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser
275 280 285
Thr Arg Thr Gly His His His His His His
290 295
<210> 21
<211> 1518
<212> DNA
<213> 人工序列
<220>
<223> Fc-融合的人Siglec-15 cDNA
<220>
<221> CDS
<222> (1)..(1518)
<400> 21
atg gaa aag tcc atc tgg ctg ctg gcc tgc ttg gcg tgg gtt ctc ccg 48
Met Glu Lys Ser Ile Trp Leu Leu Ala Cys Leu Ala Trp Val Leu Pro
1 5 10 15
aca ggc tca ttt gtg aga act aaa ata gat act acg gag aac ttg ctc 96
Thr Gly Ser Phe Val Arg Thr Lys Ile Asp Thr Thr Glu Asn Leu Leu
20 25 30
aac aca gag gtg cac agc tcg cca gcg cag cgc tgg tcc atg cag gtg 144
Asn Thr Glu Val His Ser Ser Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
cca ccc gag gtg agc gcg gag gca ggc gac gcg gca gtg ctg ccc tgc 192
Pro Pro Glu Val Ser Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
acc ttc acg cac ccg cac cgc cac tac gac ggg ccg ctg acg gcc atc 240
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
tgg cgc gcg ggc gag ccc tat gcg ggc ccg cag gtg ttc cgc tgc gct 288
Trp Arg Ala Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Ala
85 90 95
gcg gcg cgg ggc agc gag ctc tgc cag acg gcg ctg agc ctg cac ggc 336
Ala Ala Arg Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
cgc ttc cgg ctg ctg ggc aac ccg cgc cgc aac gac ctc tcg ctg cgc 384
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
gtc gag cgc ctc gcc ctg gct gac gac cgc cgc tac ttc tgc cgc gtc 432
Val Glu Arg Leu Ala Leu Ala Asp Asp Arg Arg Tyr Phe Cys Arg Val
130 135 140
gag ttc gcc ggc gac gtc cat gac cgc tac gag agc cgc cac ggc gtc 480
Glu Phe Ala Gly Asp Val His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
cgg ctg cac gtg aca gcc gcg ccg cgg atc gtc aac atc tcg gtg ctg 528
Arg Leu His Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
ccc agt ccg gct cac gcc ttc cgc gcg ctc tgc act gcc gaa ggg gag 576
Pro Ser Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
ccg ccg ccc gcc ctc gcc tgg tcc ggc ccg gcc ctg ggc aac agc ttg 624
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Leu Gly Asn Ser Leu
195 200 205
gca gcc gtg cgg agc ccg cgt gag ggt cac ggc cac cta gtg acc gcc 672
Ala Ala Val Arg Ser Pro Arg Glu Gly His Gly His Leu Val Thr Ala
210 215 220
gaa ctg ccc gca ctg acc cat gac ggc cgc tac acg tgt acg gcc gcc 720
Glu Leu Pro Ala Leu Thr His Asp Gly Arg Tyr Thr Cys Thr Ala Ala
225 230 235 240
aac agc ctg ggc cgc tcc gag gcc agc gtc tac ctg ttc cgc ttc cat 768
Asn Ser Leu Gly Arg Ser Glu Ala Ser Val Tyr Leu Phe Arg Phe His
245 250 255
ggc gcc agc ggg gac cca gct ttc ttg tac aaa gtg gtt gat atc cag 816
Gly Ala Ser Gly Asp Pro Ala Phe Leu Tyr Lys Val Val Asp Ile Gln
260 265 270
gca gag ccc aaa tct tgt gac aaa act cac aca tgc cca ccg tgc cca 864
Ala Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
275 280 285
gca cct gaa ctc ctg ggg gga ccg tca gtc ttc ctc ttc ccc cca aaa 912
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
290 295 300
ccc aag gac acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg 960
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
305 310 315 320
gtg gtg gac gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac 1008
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
325 330 335
gtg gac ggc gtg gag gtg cat aat gcc aag aca aag ccg cgg gag gag 1056
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
340 345 350
cag tac aac agc acg tac cgt gtg gtc agc gtc ctc acc gtc ctg cac 1104
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
355 360 365
cag gac tgg ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa 1152
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
370 375 380
gcc ctc cca gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggg cag 1200
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
385 390 395 400
ccc cga gaa cca cag gtg tac acc ctg ccc cca tcc cgg gag gag atg 1248
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
405 410 415
acc aag aac cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc 1296
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
420 425 430
agc gac atc gcc gtg gag tgg gag agc aat ggg cag ccg gag aac aac 1344
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
435 440 445
tac aag acc acg cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc 1392
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
450 455 460
tat agc aag ctc acc gtg gac aag agc agg tgg cag cag ggg aac gtc 1440
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
465 470 475 480
ttc tca tgc tcc gtg atg cat gag gct ctg cac aac cac tac acg cag 1488
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
485 490 495
aag agc ctc tcc ctg tct ccg ggt aaa taa 1518
Lys Ser Leu Ser Leu Ser Pro Gly Lys
500 505
<210> 22
<211> 505
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 22
Met Glu Lys Ser Ile Trp Leu Leu Ala Cys Leu Ala Trp Val Leu Pro
1 5 10 15
Thr Gly Ser Phe Val Arg Thr Lys Ile Asp Thr Thr Glu Asn Leu Leu
20 25 30
Asn Thr Glu Val His Ser Ser Pro Ala Gln Arg Trp Ser Met Gln Val
35 40 45
Pro Pro Glu Val Ser Ala Glu Ala Gly Asp Ala Ala Val Leu Pro Cys
50 55 60
Thr Phe Thr His Pro His Arg His Tyr Asp Gly Pro Leu Thr Ala Ile
65 70 75 80
Trp Arg Ala Gly Glu Pro Tyr Ala Gly Pro Gln Val Phe Arg Cys Ala
85 90 95
Ala Ala Arg Gly Ser Glu Leu Cys Gln Thr Ala Leu Ser Leu His Gly
100 105 110
Arg Phe Arg Leu Leu Gly Asn Pro Arg Arg Asn Asp Leu Ser Leu Arg
115 120 125
Val Glu Arg Leu Ala Leu Ala Asp Asp Arg Arg Tyr Phe Cys Arg Val
130 135 140
Glu Phe Ala Gly Asp Val His Asp Arg Tyr Glu Ser Arg His Gly Val
145 150 155 160
Arg Leu His Val Thr Ala Ala Pro Arg Ile Val Asn Ile Ser Val Leu
165 170 175
Pro Ser Pro Ala His Ala Phe Arg Ala Leu Cys Thr Ala Glu Gly Glu
180 185 190
Pro Pro Pro Ala Leu Ala Trp Ser Gly Pro Ala Leu Gly Asn Ser Leu
195 200 205
Ala Ala Val Arg Ser Pro Arg Glu Gly His Gly His Leu Val Thr Ala
210 215 220
Glu Leu Pro Ala Leu Thr His Asp Gly Arg Tyr Thr Cys Thr Ala Ala
225 230 235 240
Asn Ser Leu Gly Arg Ser Glu Ala Ser Val Tyr Leu Phe Arg Phe His
245 250 255
Gly Ala Ser Gly Asp Pro Ala Phe Leu Tyr Lys Val Val Asp Ile Gln
260 265 270
Ala Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
275 280 285
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
290 295 300
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
305 310 315 320
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
325 330 335
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
340 345 350
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
355 360 365
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
370 375 380
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
385 390 395 400
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
405 410 415
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
420 425 430
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
435 440 445
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
450 455 460
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
465 470 475 480
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
485 490 495
Lys Ser Leu Ser Leu Ser Pro Gly Lys
500 505
<210> 23
<211> 33
<212> DNA
<213> 人工序列
<220>
<223> 大鼠#32A1抗体的重链cDNA的PCR引物
<400> 23
ggccgggtgg gctacgttgc aggtgacggt ctg 33
<210> 24
<211> 35
<212> DNA
<213> 人工序列
<220>
<223> 大鼠#32A1抗体的轻链cDNA的PCR引物
<400> 24
catgctgtac gtgctgtctt tgctgtcctg atcag 35
<210> 25
<211> 29
<212> DNA
<213> 人工序列
<220>
<223> 大鼠#32A1抗体的重链 cDNA的测序引物
<400> 25
ctccagagtt ccaggtcacg gtgactggc 29
<210> 26
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 大鼠#32A1抗体的轻链cDNA的测序启动子
<400> 26
tccagttgct aactgttccg 20
<210> 27
<211> 501
<212> DNA
<213> 褐家鼠
<220>
<221> CDA
<222> (1)..(501)
<220>
<221> CDS
<222> (1)..(501)
<400> 27
atg aag ttg tgg ttg agc tgg ata ttc ctt gtt gtt ctt ttc aaa ggt 48
Met Lys Leu Trp Leu Ser Trp Ile Phe Leu Val Val Leu Phe Lys Gly
1 5 10 15
gtg agg tgt gag gtg caa att ttg gag act gga gga ggc ttg gtg aag 96
Val Arg Cys Glu Val Gln Ile Leu Glu Thr Gly Gly Gly Leu Val Lys
20 25 30
ccc ggt ggt tcc ctg aga ctg tct tgt gca acg tct gga ttc aat ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
aat gat tat ttc atg aac tgg gtc cgt cag gct cca gaa aag ggg cta 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
gag tgg gtt gct caa ata agg aac aaa att tat act tat gcc aca ttt 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tat gcg gag tct ttg gaa ggc aga gtc aca atc tca cga gac gat tcc 288
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asp Ser
85 90 95
gaa agc agt gtc tac ctg caa gtg agc agt tta aga gct gaa gac act 336
Glu Ser Ser Val Tyr Leu Gln Val Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
gcc att tat tac tgt aca aga tcc cta act ggg gga gac tac ttt gat 384
Ala Ile Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg ggc caa gga gtc atg gtc aca gtc tcc tta gct gaa aca aca 432
Tyr Trp Gly Gln Gly Val Met Val Thr Val Ser Leu Ala Glu Thr Thr
130 135 140
gcc cca tct gtc tat cca ctg gct cct gga act gct ctc aaa agt aac 480
Ala Pro Ser Val Tyr Pro Leu Ala Pro Gly Thr Ala Leu Lys Ser Asn
145 150 155 160
tcc atg gtg acc ctg gga tgc 501
Ser Met Val Thr Leu Gly Cys
165
<210> 28
<211> 167
<212> PRT
<213> 褐家鼠
<400> 28
Met Lys Leu Trp Leu Ser Trp Ile Phe Leu Val Val Leu Phe Lys Gly
1 5 10 15
Val Arg Cys Glu Val Gln Ile Leu Glu Thr Gly Gly Gly Leu Val Lys
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asp Ser
85 90 95
Glu Ser Ser Val Tyr Leu Gln Val Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Ile Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Val Met Val Thr Val Ser Leu Ala Glu Thr Thr
130 135 140
Ala Pro Ser Val Tyr Pro Leu Ala Pro Gly Thr Ala Leu Lys Ser Asn
145 150 155 160
Ser Met Val Thr Leu Gly Cys
165
<210> 29
<211> 417
<212> DNA
<213> 褐家鼠
<220>
<221> CDS
<222> (1)..(417)
<400> 29
atg gag aca gac aga ctc ctg cta tgg gca ctg ctg ctc tgg gtt cca 48
Met Glu Thr Asp Arg Leu Leu Leu Trp Ala Leu Leu Leu Trp Val Pro
1 5 10 15
ggc tcc act ggt gac att gtc ttg acc cag tct cct gct ttg gct gtg 96
Gly Ser Thr Gly Asp Ile Val Leu Thr Gln Ser Pro Ala Leu Ala Val
20 25 30
tct cta ggg cag agg gcc aca atc tcc tgt agg gcc agc caa agt gtc 144
Ser Leu Gly Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val
35 40 45
act att tct gga tat agt ttt ata cac tgg tac caa cag aaa cca gga 192
Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly
50 55 60
cag caa ccc aga ctc ctc atc tat cgt gca tcc aac cta gcc tct ggg 240
Gln Gln Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly
65 70 75 80
atc ccg gcc agg ttc agt ggc agt ggg tct ggg aca gac ttc acc ctc 288
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
85 90 95
acc atc aat cct gtg cag gct gat gat att gca acc tat ttc tgt cag 336
Thr Ile Asn Pro Val Gln Ala Asp Asp Ile Ala Thr Tyr Phe Cys Gln
100 105 110
cag agt agg aaa tct ccg tgg acg ttc gct gga ggc acc aag ctg gaa 384
Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Leu Glu
115 120 125
ttg aga cgg gct gat gct gca cca act gta tct 417
Leu Arg Arg Ala Asp Ala Ala Pro Thr Val Ser
130 135
<210> 30
<211> 139
<212> PRT
<213> 褐家鼠
<400> 30
Met Glu Thr Asp Arg Leu Leu Leu Trp Ala Leu Leu Leu Trp Val Pro
1 5 10 15
Gly Ser Thr Gly Asp Ile Val Leu Thr Gln Ser Pro Ala Leu Ala Val
20 25 30
Ser Leu Gly Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val
35 40 45
Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly
50 55 60
Gln Gln Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly
65 70 75 80
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
85 90 95
Thr Ile Asn Pro Val Gln Ala Asp Asp Ile Ala Thr Tyr Phe Cys Gln
100 105 110
Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Leu Glu
115 120 125
Leu Arg Arg Ala Asp Ala Ala Pro Thr Val Ser
130 135
<210> 31
<211> 28
<212> DNA
<213> 人工序列
<220>
<223> 引物 EFF1
<400> 31
ccacgcgccc tgtagcggcg cattaagc 28
<210> 32
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物 EFsmaR
<400> 32
aaacccggga gctttttgca aaagcctagg 30
<210> 33
<211> 1704
<212> DNA
<213> 人工序列
<220>
<223> 片段B
<400> 33
ggtaccaccc aagctggcta ggtaagcttg ctagcgccac catggtgctg cagacccagg 60
tgttcatctc cctgctgctg tggatctccg gcgcatatgg cgatatcgtg atgattaaac 120
gtacggtggc cgccccctcc gtgttcatct tccccccctc cgacgagcag ctgaagtccg 180
gcaccgcctc cgtggtgtgc ctgctgaata acttctaccc cagagaggcc aaggtgcagt 240
ggaaggtgga caacgccctg cagtccggga actcccagga gagcgtgacc gagcaggaca 300
gcaaggacag cacctacagc ctgagcagca ccctgaccct gagcaaagcc gactacgaga 360
agcacaaggt gtacgcctgc gaggtgaccc accagggcct gagctccccc gtcaccaaga 420
gcttcaacag gggggagtgt taggggcccg tttaaacggg tggcatccct gtgacccctc 480
cccagtgcct ctcctggccc tggaagttgc cactccagtg cccaccagcc ttgtcctaat 540
aaaattaagt tgcatcattt tgtctgacta ggtgtccttc tataatatta tggggtggag 600
gggggtggta tggagcaagg ggcaagttgg gaagacaacc tgtagggcct gcggggtcta 660
ttgggaacca agctggagtg cagtggcaca atcttggctc actgcaatct ccgcctcctg 720
ggttcaagcg attctcctgc ctcagcctcc cgagttgttg ggattccagg catgcatgac 780
caggctcacc taatttttgt ttttttggta gagacggggt ttcaccatat tggccaggct 840
ggtctccaac tcctaatctc aggtgatcta cccaccttgg cctcccaaat tgctgggatt 900
acaggcgtga accactgctc cacgcgccct gtagcggcgc attaagcgcg gcgggtgtgg 960
tggttacgcg cagcgtgacc gctacacttg ccagcgccct agcgcccgct cctttcgctt 1020
tcttcccttc ctttctcgcc acgttcgccg gctttccccg tcaagctcta aatcgggggc 1080
tccctttagg gttccgattt agtgctttac ggcacctcga ccccaaaaaa cttgattagg 1140
gtgatggttc acgtagtggg ccatcgccct gatagacggt ttttcgccct ttgacgttgg 1200
agtccacgtt ctttaatagt ggactcttgt tccaaactgg aacaacactc aaccctatct 1260
cggtctattc ttttgattta taagggattt tgccgatttc ggcctattgg ttaaaaaatg 1320
agctgattta acaaaaattt aacgcgaatt aattctgtgg aatgtgtgtc agttagggtg 1380
tggaaagtcc ccaggctccc cagcaggcag aagtatgcaa agcatgcatc tcaattagtc 1440
agcaaccagg tgtggaaagt ccccaggctc cccagcaggc agaagtatgc aaagcatgca 1500
tctcaattag tcagcaacca tagtcccgcc cctaactccg cccatcccgc ccctaactcc 1560
gcccagttcc gcccattctc cgccccatgg ctgactaatt ttttttattt atgcagaggc 1620
cgaggccgcc tctgcctctg agctattcca gaagtagtga ggaggctttt ttggaggcct 1680
aggcttttgc aaaaagctcc cggg 1704
<210> 34
<211> 1120
<212> DNA
<213> 人工序列
<220>
<223> 人IgG1信号+人IgG1恒定区
<400> 34
tgctagcgcc accatgaaac acctgtggtt cttcctcctg ctggtggcag ctcccagatg 60
ggtgctgagc caggtgcaat tgtgcaggcg gttagctcag cctccaccaa gggcccaagc 120
gtcttccccc tggcaccctc ctccaagagc acctctggcg gcacagccgc cctgggctgc 180
ctggtcaagg actacttccc cgaacccgtg accgtgagct ggaactcagg cgccctgacc 240
agcggcgtgc acaccttccc cgctgtcctg cagtcctcag gactctactc cctcagcagc 300
gtggtgaccg tgccctccag cagcttgggc acccagacct acatctgcaa cgtgaatcac 360
aagcccagca acaccaaggt ggacaagaga gttgagccca aatcttgtga caaaactcac 420
acatgcccac cctgcccagc acctgaactc ctggggggac cctcagtctt cctcttcccc 480
ccaaaaccca aggacaccct catgatctcc cggacccctg aggtcacatg cgtggtggtg 540
gacgtgagcc acgaagaccc tgaggtcaag ttcaactggt acgtggacgg cgtggaggtg 600
cataatgcca agacaaagcc ccgggaggag cagtacaaca gcacgtaccg ggtggtcagc 660
gtcctcaccg tcctgcacca ggactggctg aatggcaagg agtacaagtg caaggtctcc 720
aacaaagccc tcccagcccc catcgagaaa accatctcca aagccaaagg ccagccccgg 780
gaaccacagg tgtacaccct gcccccatcc cgggaggaga tgaccaagaa ccaggtcagc 840
ctgacctgcc tggtcaaagg cttctatccc agcgacatcg ccgtggagtg ggagagcaat 900
ggccagcccg agaacaacta caagaccacc cctcccgtgc tggactccga cggctccttc 960
ttcctctaca gcaagctcac cgtggacaag agcaggtggc agcagggcaa cgtcttctca 1020
tgctccgtga tgcatgaggc tctgcacaac cactacaccc agaagagcct ctccctgtct 1080
cccggcaaat gagatatcgg gcccgtttaa acgggtggca 1120
<210> 35
<211> 40
<212> DNA
<213> 人工序列
<220>
<223> #32A1抗体的重链可变区cDNA的PCR引物
<400> 35
aaagctgagc gaggtgcaaa ttttggagac tggaggaggc 40
<210> 36
<211> 39
<212> DNA
<213> 人工序列
<220>
<223> #32A1抗体的重链可变区cDNA的PCR引物
<400> 36
aaagctgagc tgactgtgac catgactcct tggccccag 39
<210> 37
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> #32A1抗体的重链可变区cDNA的测序引物
<400> 37
taatacgact cactataggg 20
<210> 38
<211> 42
<212> DNA
<213> 人工序列
<220>
<223> #32A1抗体的轻链可变区cDNA的PCR引物
<400> 38
aaacatatgg cgacattgtc ttgacccagt ctcctgcttt gg 42
<210> 39
<211> 37
<212> DNA
<213> 人工序列
<220>
<223> #32A1抗体的轻链可变区cDNA的PCR引物
<400> 39
aaacgtacgt ctcaattcca gcttggtgcc tccagcg 37
<210> 40
<211> 1413
<212> DNA
<213> 人工序列
<220>
<223> #32A1的嵌合重链
<220>
<221> CDS
<222> (1)..(1413)
<400> 40
atg aaa cac ctg tgg ttc ttc ctc ctg ctg gtg gca gct ccc aga tgg 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
gtg ctg agc gag gtg caa att ttg gag act gga gga ggc ttg gtg aag 96
Val Leu Ser Glu Val Gln Ile Leu Glu Thr Gly Gly Gly Leu Val Lys
20 25 30
ccc ggt ggt tcc ctg aga ctg tct tgt gca acg tct gga ttc aat ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
aat gat tat ttc atg aac tgg gtc cgt cag gct cca gaa aag ggg cta 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
gag tgg gtt gct caa ata agg aac aaa att tat act tat gcc aca ttt 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tat gcg gag tct ttg gaa ggc aga gtc aca atc tca cga gac gat tcc 288
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asp Ser
85 90 95
gaa agc agt gtc tac ctg caa gtg agc agt tta aga gct gaa gac act 336
Glu Ser Ser Val Tyr Leu Gln Val Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
gcc att tat tac tgt aca aga tcc cta act ggg gga gac tac ttt gat 384
Ala Ile Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg ggc caa gga gtc atg gtc aca gtc agc tca gcc tcc acc aag 432
Tyr Trp Gly Gln Gly Val Met Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
ggc cca agc gtc ttc ccc ctg gca ccc tcc tcc aag agc acc tct ggc 480
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
ggc aca gcc gcc ctg ggc tgc ctg gtc aag gac tac ttc ccc gaa ccc 528
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
gtg acc gtg agc tgg aac tca ggc gcc ctg acc agc ggc gtg cac acc 576
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
ttc ccc gct gtc ctg cag tcc tca gga ctc tac tcc ctc agc agc gtg 624
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
gtg acc gtg ccc tcc agc agc ttg ggc acc cag acc tac atc tgc aac 672
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
gtg aat cac aag ccc agc aac acc aag gtg gac aag aga gtt gag ccc 720
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
aaa tct tgt gac aaa act cac aca tgc cca ccc tgc cca gca cct gaa 768
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
ctc ctg ggg gga ccc tca gtc ttc ctc ttc ccc cca aaa ccc aag gac 816
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg gtg gtg gac 864
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac gtg gac ggc 912
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
gtg gag gtg cat aat gcc aag aca aag ccc cgg gag gag cag tac aac 960
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
agc acg tac cgg gtg gtc agc gtc ctc acc gtc ctg cac cag gac tgg 1008
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa gcc ctc cca 1056
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggc cag ccc cgg gaa 1104
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
cca cag gtg tac acc ctg ccc cca tcc cgg gag gag atg acc aag aac 1152
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc agc gac atc 1200
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
gcc gtg gag tgg gag agc aat ggc cag ccc gag aac aac tac aag acc 1248
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
acc cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc tac agc aag 1296
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
ctc acc gtg gac aag agc agg tgg cag cag ggc aac gtc ttc tca tgc 1344
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
tcc gtg atg cat gag gct ctg cac aac cac tac acc cag aag agc ctc 1392
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
tcc ctg tct ccc ggc aaa tga 1413
Ser Leu Ser Pro Gly Lys
465 470
<210> 41
<211> 470
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 41
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Ile Leu Glu Thr Gly Gly Gly Leu Val Lys
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asp Ser
85 90 95
Glu Ser Ser Val Tyr Leu Gln Val Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Ile Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Val Met Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys
465 470
<210> 42
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> #32A1的嵌合轻链
<220>
<221> CDS
<222> (1)..(714)
<400> 42
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gac att gtc ttg acc cag tct cct gct ttg gct gtg 96
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Ala Leu Ala Val
20 25 30
tct cta ggg cag agg gcc aca atc tcc tgt agg gcc agc caa agt gtc 144
Ser Leu Gly Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val
35 40 45
act att tct gga tat agt ttt ata cac tgg tac caa cag aaa cca gga 192
Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly
50 55 60
cag caa ccc aga ctc ctc atc tat cgt gca tcc aac cta gcc tct ggg 240
Gln Gln Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly
65 70 75 80
atc ccg gcc agg ttc agt ggc agt ggg tct ggg aca gac ttc acc ctc 288
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
85 90 95
acc atc aat cct gtg cag gct gat gat att gca acc tat ttc tgt cag 336
Thr Ile Asn Pro Val Gln Ala Asp Asp Ile Ala Thr Tyr Phe Cys Gln
100 105 110
cag agt agg aaa tct ccg tgg acg ttc gct gga ggc acc aag ctg gaa 384
Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Leu Glu
115 120 125
ttg aga cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc tcc 432
Leu Arg Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser
130 135 140
gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg aat 480
Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn
145 150 155 160
aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac gcc 528
Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala
165 170 175
ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc aag 576
Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys
180 185 190
gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc gac 624
Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp
195 200 205
tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc ctg 672
Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu
210 215 220
agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt tag 714
Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 43
<211> 237
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 43
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Ala Leu Ala Val
20 25 30
Ser Leu Gly Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val
35 40 45
Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly
50 55 60
Gln Gln Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly
65 70 75 80
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
85 90 95
Thr Ile Asn Pro Val Gln Ala Asp Asp Ile Ala Thr Tyr Phe Cys Gln
100 105 110
Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Leu Glu
115 120 125
Leu Arg Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser
130 135 140
Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn
145 150 155 160
Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala
165 170 175
Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys
180 185 190
Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp
195 200 205
Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu
210 215 220
Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 44
<211> 5
<212> PRT
<213> 褐家鼠
<400> 44
Asp Tyr Phe Met Asn
1 5
<210> 45
<211> 19
<212> PRT
<213> 褐家鼠
<400> 45
Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe Tyr Ala Glu Ser
1 5 10 15
Leu Glu Gly
<210> 46
<211> 10
<212> PRT
<213> 褐家鼠
<400> 46
Ser Leu Thr Gly Gly Asp Tyr Phe Asp Tyr
1 5 10
<210> 47
<211> 15
<212> PRT
<213> 褐家鼠
<400> 47
Arg Ala Ser Gln Ser Val Thr Ile Ser Gly Tyr Ser Phe Ile His
1 5 10 15
<210> 48
<211> 7
<212> PRT
<213> 褐家鼠
<400> 48
Arg Ala Ser Asn Leu Ala Ser
1 5
<210> 49
<211> 9
<212> PRT
<213> 褐家鼠
<400> 49
Gln Gln Ser Arg Lys Ser Pro Trp Thr
1 5
<210> 50
<211> 1410
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T1H
<220>
<221> CDS
<222> (1)..(1410)
<400> 50
atg aaa cac ctg tgg ttc ttc ctc ctg ctg gtg gca gct ccc aga tgg 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
gtg ctg agc gag gtg caa ctg gtg gag agc ggc ggc gga ctt gtg caa 96
Val Leu Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
cct ggc ggc tcc ctg aga ctt agc tgt gcc gcc tcc ggc ttc aac ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe
35 40 45
aac gac tac ttc atg aac tgg gtg aga caa gcc cct ggc aag ggc ctg 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
gag tgg gtg gcc caa atc aga aac aag atc tac acc tac gcc acc ttc 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tac gcc gag agc ctt gag ggc aga ttc acc atc tcc aga gac aac gcc 288
Tyr Ala Glu Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala
85 90 95
aag aac agc ctg tac ctt caa atg aac tcc ctg aga gcc gag gac acc 336
Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
gcc gtg tac tac tgt gcc aga agc ctt acc ggc ggc gac tac ttc gac 384
Ala Val Tyr Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg ggc caa ggc acc ctg gtg acc gtg agc tca gcc tcc acc aag 432
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
ggc cca agc gtc ttc ccc ctg gca ccc tcc tcc aag agc acc tct ggc 480
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
ggc aca gcc gcc ctg ggc tgc ctg gtc aag gac tac ttc ccc gaa ccc 528
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
gtg acc gtg agc tgg aac tca ggc gcc ctg acc agc ggc gtg cac acc 576
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
ttc ccc gct gtc ctg cag tcc tca gga ctc tac tcc ctc agc agc gtg 624
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
gtg acc gtg ccc tcc agc agc ttg ggc acc cag acc tac atc tgc aac 672
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
gtg aat cac aag ccc agc aac acc aag gtg gac aag aga gtt gag ccc 720
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
aaa tct tgt gac aaa act cac aca tgc cca ccc tgc cca gca cct gaa 768
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
ctc ctg ggg gga ccc tca gtc ttc ctc ttc ccc cca aaa ccc aag gac 816
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg gtg gtg gac 864
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac gtg gac ggc 912
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
gtg gag gtg cat aat gcc aag aca aag ccc cgg gag gag cag tac aac 960
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
agc acg tac cgg gtg gtc agc gtc ctc acc gtc ctg cac cag gac tgg 1008
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa gcc ctc cca 1056
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggc cag ccc cgg gaa 1104
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
cca cag gtg tac acc ctg ccc cca tcc cgg gag gag atg acc aag aac 1152
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc agc gac atc 1200
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
gcc gtg gag tgg gag agc aat ggc cag ccc gag aac aac tac aag acc 1248
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
acc cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc tac agc aag 1296
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
ctc acc gtg gac aag agc agg tgg cag cag ggc aac gtc ttc tca tgc 1344
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
tcc gtg atg cat gag gct ctg cac aac cac tac acc cag aag agc ctc 1392
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
tcc ctg tct ccc ggc aaa 1410
Ser Leu Ser Pro Gly Lys
465 470
<210> 51
<211> 470
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 51
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Glu Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala
85 90 95
Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys
465 470
<210> 52
<211> 1410
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T2H
<220>
<221> CDS
<222> (1)..(1410)
<400> 52
atg aaa cac ctg tgg ttc ttc ctc ctg ctg gtg gca gct ccc aga tgg 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
gtg ctg agc gag gtg caa ctg gtg gag agc ggc ggc gga ctg gtg caa 96
Val Leu Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
cct ggc ggc tcc ctg aga ctg agc tgt gcc gcc tcc ggc ttc aac ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe
35 40 45
aac gac tac ttc atg aac tgg gtg aga caa gcc cct gag aag ggc ctg 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
gag tgg gtg gcc caa atc aga aac aag atc tac acc tac gcc acc ttc 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tac gcc gag agc ctg gag ggc aga gtg acc atc tcc aga gac aac gcc 288
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asn Ala
85 90 95
aag aac agc ctg tac ctg caa atg tcc tcc ctg aga gcc gag gac acc 336
Lys Asn Ser Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
gcc gtg tac tac tgt gcc aga agc ctg acc ggc ggc gac tac ttc gac 384
Ala Val Tyr Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg ggc caa ggc acc ctg gtg acc gtg agc tca gcc tcc acc aag 432
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
ggc cca agc gtc ttc ccc ctg gca ccc tcc tcc aag agc acc tct ggc 480
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
ggc aca gcc gcc ctg ggc tgc ctg gtc aag gac tac ttc ccc gaa ccc 528
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
gtg acc gtg agc tgg aac tca ggc gcc ctg acc agc ggc gtg cac acc 576
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
ttc ccc gct gtc ctg cag tcc tca gga ctc tac tcc ctc agc agc gtg 624
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
gtg acc gtg ccc tcc agc agc ttg ggc acc cag acc tac atc tgc aac 672
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
gtg aat cac aag ccc agc aac acc aag gtg gac aag aga gtt gag ccc 720
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
aaa tct tgt gac aaa act cac aca tgc cca ccc tgc cca gca cct gaa 768
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
ctc ctg ggg gga ccc tca gtc ttc ctc ttc ccc cca aaa ccc aag gac 816
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg gtg gtg gac 864
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac gtg gac ggc 912
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
gtg gag gtg cat aat gcc aag aca aag ccc cgg gag gag cag tac aac 960
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
agc acg tac cgg gtg gtc agc gtc ctc acc gtc ctg cac cag gac tgg 1008
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa gcc ctc cca 1056
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggc cag ccc cgg gaa 1104
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
cca cag gtg tac acc ctg ccc cca tcc cgg gag gag atg acc aag aac 1152
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc agc gac atc 1200
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
gcc gtg gag tgg gag agc aat ggc cag ccc gag aac aac tac aag acc 1248
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
acc cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc tac agc aag 1296
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
ctc acc gtg gac aag agc agg tgg cag cag ggc aac gtc ttc tca tgc 1344
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
tcc gtg atg cat gag gct ctg cac aac cac tac acc cag aag agc ctc 1392
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
tcc ctg tct ccc ggc aaa 1410
Ser Leu Ser Pro Gly Lys
465 470
<210> 53
<211> 470
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 53
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asn Ala
85 90 95
Lys Asn Ser Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys
465 470
<210> 54
<211> 1410
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T3H
<220>
<221> CDS
<222> (1)..(1410)
<400> 54
atg aaa cac ctg tgg ttc ttc ctc ctg ctg gtg gca gct ccc aga tgg 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
gtg ctg agc gag gtg cag atc gtg gag agc ggc ggc gga ctt gtg cag 96
Val Leu Ser Glu Val Gln Ile Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
cct ggc ggc tcc ctg aga ctt agc tgt gcc acc tcc ggc ttc aac ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
aac gac tac ttc atg aac tgg gtg aga cag gcc cct gag aag ggc ctg 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
gag tgg gtg gcc cag atc aga aac aag atc tac acc tac gcc acc ttc 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tac gcc gag agc ctt gag ggc aga gtg acc atc tcc aga gac gac agc 288
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asp Ser
85 90 95
gag tcc agc gtg tac ctt cag atg tcc agc ctg aga gcc gag gac acc 336
Glu Ser Ser Val Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
gcc gtg tac tac tgt acc aga agc ctt acc ggc ggc gac tac ttc gac 384
Ala Val Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg ggc cag ggc acc ctg gtg acc gtg agc tca gcc tcc acc aag 432
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
ggc cca agc gtc ttc ccc ctg gca ccc tcc tcc aag agc acc tct ggc 480
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
ggc aca gcc gcc ctg ggc tgc ctg gtc aag gac tac ttc ccc gaa ccc 528
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
gtg acc gtg agc tgg aac tca ggc gcc ctg acc agc ggc gtg cac acc 576
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
ttc ccc gct gtc ctg cag tcc tca gga ctc tac tcc ctc agc agc gtg 624
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
gtg acc gtg ccc tcc agc agc ttg ggc acc cag acc tac atc tgc aac 672
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
gtg aat cac aag ccc agc aac acc aag gtg gac aag aga gtt gag ccc 720
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
aaa tct tgt gac aaa act cac aca tgc cca ccc tgc cca gca cct gaa 768
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
ctc ctg ggg gga ccc tca gtc ttc ctc ttc ccc cca aaa ccc aag gac 816
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg gtg gtg gac 864
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac gtg gac ggc 912
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
gtg gag gtg cat aat gcc aag aca aag ccc cgg gag gag cag tac aac 960
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
agc acg tac cgg gtg gtc agc gtc ctc acc gtc ctg cac cag gac tgg 1008
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa gcc ctc cca 1056
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggc cag ccc cgg gaa 1104
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
cca cag gtg tac acc ctg ccc cca tcc cgg gag gag atg acc aag aac 1152
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc agc gac atc 1200
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
gcc gtg gag tgg gag agc aat ggc cag ccc gag aac aac tac aag acc 1248
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
acc cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc tac agc aag 1296
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
ctc acc gtg gac aag agc agg tgg cag cag ggc aac gtc ttc tca tgc 1344
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
tcc gtg atg cat gag gct ctg cac aac cac tac acc cag aag agc ctc 1392
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
tcc ctg tct ccc ggc aaa 1410
Ser Leu Ser Pro Gly Lys
465 470
<210> 55
<211> 470
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 55
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Ile Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asp Ser
85 90 95
Glu Ser Ser Val Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys
465 470
<210> 56
<211> 1410
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T5H
<220>
<221> CDS
<222> (1)..(1410)
<400> 56
atg aaa cac ctg tgg ttc ttc ctc ctg ctg gtg gca gct ccc aga tgg 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
gtg ctg agc gag gtg cag gtg gtg gag agc ggc ggc gga ctt gtg cag 96
Val Leu Ser Glu Val Gln Val Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
cct ggc ggc tcc ctg aga ctt agc tgt gcc acc tcc ggc ttc aac ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
aac gac tac ttc atg aac tgg gtg aga cag gcc cct ggc aag ggc ctg 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
gag tgg gtg gcc cag atc aga aac aag atc tac acc tac gcc acc ttc 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tac gcc gag agc ctt gag ggc aga ttc acc atc tcc aga gac aac agc 288
Tyr Ala Glu Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
aag tcc acc gtg tac ctt cag atg aac tcc ctg aga gcc gag gac acc 336
Lys Ser Thr Val Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
gcc gtg tac tac tgt acc aga agc ctt acc ggc ggc gac tac ttc gac 384
Ala Val Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg ggc cag ggc acc ctg gtg acc gtg agc tca gcc tcc acc aag 432
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
ggc cca agc gtc ttc ccc ctg gca ccc tcc tcc aag agc acc tct ggc 480
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
ggc aca gcc gcc ctg ggc tgc ctg gtc aag gac tac ttc ccc gaa ccc 528
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
gtg acc gtg agc tgg aac tca ggc gcc ctg acc agc ggc gtg cac acc 576
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
ttc ccc gct gtc ctg cag tcc tca gga ctc tac tcc ctc agc agc gtg 624
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
gtg acc gtg ccc tcc agc agc ttg ggc acc cag acc tac atc tgc aac 672
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
gtg aat cac aag ccc agc aac acc aag gtg gac aag aga gtt gag ccc 720
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
aaa tct tgt gac aaa act cac aca tgc cca ccc tgc cca gca cct gaa 768
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
ctc ctg ggg gga ccc tca gtc ttc ctc ttc ccc cca aaa ccc aag gac 816
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg gtg gtg gac 864
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac gtg gac ggc 912
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
gtg gag gtg cat aat gcc aag aca aag ccc cgg gag gag cag tac aac 960
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
agc acg tac cgg gtg gtc agc gtc ctc acc gtc ctg cac cag gac tgg 1008
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa gcc ctc cca 1056
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggc cag ccc cgg gaa 1104
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
cca cag gtg tac acc ctg ccc cca tcc cgg gag gag atg acc aag aac 1152
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc agc gac atc 1200
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
gcc gtg gag tgg gag agc aat ggc cag ccc gag aac aac tac aag acc 1248
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
acc cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc tac agc aag 1296
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
ctc acc gtg gac aag agc agg tgg cag cag ggc aac gtc ttc tca tgc 1344
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
tcc gtg atg cat gag gct ctg cac aac cac tac acc cag aag agc ctc 1392
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
tcc ctg tct ccc ggc aaa 1410
Ser Leu Ser Pro Gly Lys
465 470
<210> 57
<211> 470
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 57
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Val Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Glu Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Ser Thr Val Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys
465 470
<210> 58
<211> 1410
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T6H
<220>
<221> CDS
<222> (1)..(1410)
<400> 58
atg aaa cac ctg tgg ttc ttc ctc ctg ctg gtg gca gct ccc aga tgg 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
gtg ctg agc gag gtg cag gtg gtg gag agc ggc ggc gga ctt gtg cag 96
Val Leu Ser Glu Val Gln Val Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
cct ggc ggc tcc ctg aga ctt agc tgt gcc acc tcc ggc ttc aac ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
aac gac tac ttc atg aac tgg gtg aga cag gcc cct gag aag ggc ctg 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
gag tgg gtg gcc cag atc aga aac aag atc tac acc tac gcc acc ttc 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tac gcc gag agc ctt gag ggc aga gtg acc atc tcc aga gac aac agc 288
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asn Ser
85 90 95
aag tcc acc gtg tac ctt cag atg tcc agc ctt aga gcc gag gac acc 336
Lys Ser Thr Val Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
gcc gtg tac tac tgt acc aga agc ctt acc ggc ggc gac tac ttc gac 384
Ala Val Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg ggc cag ggc acc ctt gtg aca gtg agc tca gcc tcc acc aag 432
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
ggc cca agc gtc ttc ccc ctg gca ccc tcc tcc aag agc acc tct ggc 480
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
ggc aca gcc gcc ctg ggc tgc ctg gtc aag gac tac ttc ccc gaa ccc 528
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
gtg acc gtg agc tgg aac tca ggc gcc ctg acc agc ggc gtg cac acc 576
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
ttc ccc gct gtc ctg cag tcc tca gga ctc tac tcc ctc agc agc gtg 624
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
gtg acc gtg ccc tcc agc agc ttg ggc acc cag acc tac atc tgc aac 672
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
gtg aat cac aag ccc agc aac acc aag gtg gac aag aga gtt gag ccc 720
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
aaa tct tgt gac aaa act cac aca tgc cca ccc tgc cca gca cct gaa 768
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
ctc ctg ggg gga ccc tca gtc ttc ctc ttc ccc cca aaa ccc aag gac 816
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg gtg gtg gac 864
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac gtg gac ggc 912
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
gtg gag gtg cat aat gcc aag aca aag ccc cgg gag gag cag tac aac 960
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
agc acg tac cgg gtg gtc agc gtc ctc acc gtc ctg cac cag gac tgg 1008
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa gcc ctc cca 1056
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggc cag ccc cgg gaa 1104
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
cca cag gtg tac acc ctg ccc cca tcc cgg gag gag atg acc aag aac 1152
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc agc gac atc 1200
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
gcc gtg gag tgg gag agc aat ggc cag ccc gag aac aac tac aag acc 1248
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
acc cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc tac agc aag 1296
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
ctc acc gtg gac aag agc agg tgg cag cag ggc aac gtc ttc tca tgc 1344
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
tcc gtg atg cat gag gct ctg cac aac cac tac acc cag aag agc ctc 1392
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
tcc ctg tct ccc ggc aaa 1410
Ser Leu Ser Pro Gly Lys
465 470
<210> 59
<211> 470
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 59
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Val Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Glu Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Ser Thr Val Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys
465 470
<210> 60
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T1L
<220>
<221> CDS
<222> (1)..(714)
<400> 60
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gac atc gtg atg acc cag agc cct gac tcc ctt gcc 96
Gly Ala Tyr Gly Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
gtg tcc ctg ggc gag aga gcc acc atc aac tgt aga gcc tcc cag agc 144
Val Ser Leu Gly Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser
35 40 45
gtg acc atc tcc ggc tac agc ttc atc cac tgg tac cag cag aag cct 192
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
ggc cag cct cct aag ctt ctg atc tac aga gcc tcc aac ctt gcc agc 240
Gly Gln Pro Pro Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
ggc gtg cct gcc aga ttc tcc ggc agc ggc tcc ggc acc gac ttc acc 288
Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
ctg acc atc agc tcc ctt cag gcc gag gac gtg gcc gtg tac tac tgt 336
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys
100 105 110
cag cag agc aga aag tcc cct tgg acc ttc ggc ggc ggc acc aag gtg 384
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
gag atc aag cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc 432
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
tcc gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg 480
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
aat aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac 528
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
gcc ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc 576
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
aag gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc 624
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
gac tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc 672
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
ctg agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt 714
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 61
<211> 238
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 61
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
Val Ser Leu Gly Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser
35 40 45
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Pro Pro Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys
100 105 110
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 62
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T2L
<220>
<221> CDS
<222> (1)..(714)
<400> 62
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gac atc gtg atg acc cag agc cct gac tcc ctt gcc 96
Gly Ala Tyr Gly Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
gtg tcc ctg ggc gag aga gcc acc atc agc tgt aga gcc tcc cag agc 144
Val Ser Leu Gly Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser
35 40 45
gtg acc atc tcc ggc tac agc ttc atc cac tgg tac cag cag aag cct 192
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
ggc cag cct cct aga ctt ctg atc tac aga gcc tcc aac ctt gcc agc 240
Gly Gln Pro Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
ggc gtg cct gcc aga ttc tcc ggc agc ggc tcc ggc acc gac ttc acc 288
Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
ctg acc atc agc tcc ctt cag gcc gag gac gtg gcc gtg tac ttc tgt 336
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys
100 105 110
cag cag agc aga aag tcc cct tgg acc ttc gcc ggc ggc acc aag gtg 384
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val
115 120 125
gag atc aag cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc 432
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
tcc gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg 480
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
aat aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac 528
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
gcc ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc 576
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
aag gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc 624
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
gac tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc 672
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
ctg agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt 714
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 63
<211> 238
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 63
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
Val Ser Leu Gly Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser
35 40 45
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Pro Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys
100 105 110
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 64
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T3L
<220>
<221> CDS
<222> (1)..(714)
<400> 64
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gac atc gtg ctt acc cag agc cct gac tcc ctt gcc 96
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
gtg tcc ctg ggc gag aga gcc acc atc agc tgt aga gcc tcc cag agc 144
Val Ser Leu Gly Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser
35 40 45
gtg acc atc tcc ggc tac agc ttc atc cac tgg tac cag cag aag cct 192
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
ggc cag cag cct aga ctt ctg atc tac aga gcc tcc aac ctt gcc agc 240
Gly Gln Gln Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
ggc atc cct gcc aga ttc tcc ggc agc ggc tcc ggc acc gac ttc acc 288
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
ctg acc atc agc tcc ctt cag gcc gag gac gtg gcc acc tac ttc tgt 336
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Thr Tyr Phe Cys
100 105 110
cag cag agc aga aag tcc cct tgg acc ttc gcc ggc ggc acc aag gtg 384
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val
115 120 125
gag atc aag cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc 432
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
tcc gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg 480
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
aat aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac 528
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
gcc ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc 576
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
aag gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc 624
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
gac tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc 672
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
ctg agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt 714
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 65
<211> 238
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 65
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
Val Ser Leu Gly Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser
35 40 45
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Gln Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Thr Tyr Phe Cys
100 105 110
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 66
<211> 711
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T4L
<220>
<221> CDS
<222> (1)..(711)
<400> 66
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gac atc gtg ctt acc cag agc cct gcc ctt gcc gtg 96
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Ala Leu Ala Val
20 25 30
tcc ctg ggc gag aga gcc acc atc agc tgt aga gcc tcc cag agc gtg 144
Ser Leu Gly Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val
35 40 45
acc atc tcc ggc tac agc ttc atc cac tgg tac cag cag aag cct ggc 192
Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly
50 55 60
cag cag cct aga ctt ctg atc tac aga gcc tcc aac ctt gcc agc ggc 240
Gln Gln Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly
65 70 75 80
atc cct gcc aga ttc tcc ggc agc ggc tcc ggc acc gac ttc acc ctg 288
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
85 90 95
acc atc agc tcc ctt cag gcc gag gac gtg gcc acc tac ttc tgt cag 336
Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Thr Tyr Phe Cys Gln
100 105 110
cag agc aga aag tcc cct tgg acc ttc gcc ggc ggc acc aag ctg gag 384
Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Leu Glu
115 120 125
atc aag cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc tcc 432
Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser
130 135 140
gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg aat 480
Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn
145 150 155 160
aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac gcc 528
Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala
165 170 175
ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc aag 576
Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys
180 185 190
gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc gac 624
Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp
195 200 205
tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc ctg 672
Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu
210 215 220
agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt 711
Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 67
<211> 237
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 67
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Ala Leu Ala Val
20 25 30
Ser Leu Gly Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val
35 40 45
Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly
50 55 60
Gln Gln Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly
65 70 75 80
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
85 90 95
Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Thr Tyr Phe Cys Gln
100 105 110
Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Leu Glu
115 120 125
Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser
130 135 140
Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn
145 150 155 160
Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala
165 170 175
Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys
180 185 190
Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp
195 200 205
Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu
210 215 220
Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 68
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T5L
<220>
<221> CDS
<222> (1)..(714)
<400> 68
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gac atc gtg ctt acc cag agc cct gac tcc ctt gcc 96
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
gtg tcc ctg ggc gag aga gcc acc atc aac tgt aga gcc tcc cag agc 144
Val Ser Leu Gly Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser
35 40 45
gtg acc atc tcc ggc tac agc ttc atc cac tgg tac cag cag aag cct 192
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
ggc cag cct cct aag ctt ctg atc tac aga gcc tcc aac ctt gcc agc 240
Gly Gln Pro Pro Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
ggc atc cct gcc aga ttc tcc ggc agc ggc tcc ggc acc gac ttc acc 288
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
ctg acc atc agc tcc ctt cag gcc gag gac gtg gcc acc tac tac tgt 336
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Thr Tyr Tyr Cys
100 105 110
cag cag agc aga aag tcc cct tgg acc ttc ggc cag ggc acc aag gtg 384
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Gly Gln Gly Thr Lys Val
115 120 125
gag atc aag cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc 432
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
tcc gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg 480
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
aat aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac 528
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
gcc ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc 576
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
aag gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc 624
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
gac tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc 672
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
ctg agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt 714
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 69
<211> 238
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 69
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
Val Ser Leu Gly Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser
35 40 45
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Pro Pro Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Thr Tyr Tyr Cys
100 105 110
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Gly Gln Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 70
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-T6L
<220>
<221> CDS
<222> (1)..(714)
<400> 70
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gac atc gtg ctt acc cag agc cct gac tcc ctt gcc 96
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
gtg tcc ctg ggc gag aga gcc acc atc agc tgt aga gcc tcc cag agc 144
Val Ser Leu Gly Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser
35 40 45
gtg acc atc tcc ggc tac agc ttc atc cac tgg tac cag cag aag cct 192
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
ggc cag cct cct aga ctg ctg atc tac aga gcc tcc aac ctt gcc agc 240
Gly Gln Pro Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
ggc atc cct gcc aga ttc tcc ggc agc ggc tcc ggc acc gac ttc acc 288
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
ctg acc atc agc tcc ctt cag gcc gag gac gtg gcc acc tac ttc tgt 336
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Thr Tyr Phe Cys
100 105 110
cag cag agc aga aag tcc cct tgg acc ttc gcc ggc ggc acc aag gtg 384
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val
115 120 125
gag atc aag cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc 432
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
tcc gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg 480
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
aat aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac 528
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
gcc ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc 576
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
aag gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc 624
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
gac tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc 672
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
ctg agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt 714
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 71
<211> 238
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 71
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Asp Ile Val Leu Thr Gln Ser Pro Asp Ser Leu Ala
20 25 30
Val Ser Leu Gly Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser
35 40 45
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Pro Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Thr Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Thr Tyr Phe Cys
100 105 110
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 72
<211> 451
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T7H
<400> 72
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe Asn Asp Tyr
20 25 30
Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu Glu Trp Val
35 40 45
Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe Tyr Ala Glu
50 55 60
Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 73
<211> 451
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T8H
<400> 73
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe Asn Asp Tyr
20 25 30
Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe Tyr Ala Glu
50 55 60
Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 74
<211> 451
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T9H
<400> 74
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe Asn Asp Tyr
20 25 30
Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe Tyr Ala Glu
50 55 60
Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
65 70 75 80
Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 75
<211> 451
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T10H
<400> 75
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe Asn Asp Tyr
20 25 30
Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu Glu Trp Val
35 40 45
Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe Tyr Ala Glu
50 55 60
Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 76
<211> 451
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T11H
<400> 76
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe Asn Asp Tyr
20 25 30
Phe Met Asn Trp Val Arg Gln Ala Pro Glu Lys Gly Leu Glu Trp Val
35 40 45
Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe Tyr Ala Glu
50 55 60
Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
65 70 75 80
Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 77
<211> 451
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T12H
<400> 77
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe Asn Asp Tyr
20 25 30
Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe Tyr Ala Glu
50 55 60
Ser Leu Glu Gly Arg Val Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
65 70 75 80
Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 78
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T7L
<400> 78
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 79
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T8L
<400> 79
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 80
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T9L
<400> 80
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 81
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T10L
<400> 81
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 82
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T11L
<400> 82
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 83
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T12L
<400> 83
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 84
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T13L
<400> 84
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 85
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T14L
<400> 85
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 86
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T15L
<400> 86
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 87
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T16L
<400> 87
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 88
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T17L
<400> 88
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 89
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T18L
<400> 89
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 90
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T19L
<400> 90
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 91
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T20L
<400> 91
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 92
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T21L
<400> 92
Glu Ile Leu Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Leu Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 93
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T22L
<400> 93
Glu Ile Leu Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Leu Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 94
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T23L
<400> 94
Glu Ile Leu Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Leu Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 95
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T24L
<400> 95
Glu Ile Leu Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Leu Tyr Tyr Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 96
<211> 218
<212> PRT
<213> 人工序列
<220>
<223> h#32A1-T25L
<400> 96
Asp Ile Leu Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Gln Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Ser Pro Trp Thr Phe Ala Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 97
<211> 14
<212> PRT
<213> 褐家鼠
<400> 97
Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe Tyr Ala
1 5 10
<210> 98
<211> 1398
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-H1-1
<220>
<221> CDS
<222> (1)..(1398)
<400> 98
atg aaa cac ctg tgg ttc ttc ctc ctg ctg gtg gca gct ccc aga tgg 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
gtg ctg agc gaa gtc cag ctt gtg gaa agc gga ggg gga ctc gtt cag 96
Val Leu Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
cca gga ggc tct ctg cgc ctg tca tgc gct gcc agc gga ttt aat ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe
35 40 45
aat gat tat ttt atg aac tgg gtc agg cag gct ccg gga aaa ggg ctg 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
gaa tgg gtc gcc cag atc aga aac aag atc tat act tac gct aca ttc 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tac gcc gca tct gta aag ggg agg ttt aca att agt cgc gac aat gca 288
Tyr Ala Ala Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala
85 90 95
aaa aat agt ctg tat ctc caa atg aac tcc ctc cgc gca gag gat act 336
Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
gct gtc tac tac tgc gcc agg tcc ttg act ggc ggc gac tat ttt gat 384
Ala Val Tyr Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg gga cag ggc acc ctg gtg acg gtg agc tca gcc agc acc aag 432
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
ggc cct tcc gtg ttc cct ctg gcc cct tgt agc cgt tcc acc agc gag 480
Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu
145 150 155 160
tcc acc gcc gcc ctt ggc tgt ctg gtg aag gac tac ttc cct gag cct 528
Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
gtg acc gtg agc tgg aac tcc gga gcc ctt acc agc ggc gtg cac acc 576
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
ttc cct gcc gtg ctg cag tcc agc ggc ctt tac tcc ctg agc tcc gtg 624
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
gtg acc gtg cct agc tcc aac ttc ggc acc caa acc tac acc tgt aac 672
Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn
210 215 220
gtg gac cac aag cct agc aac acc aag gtg gac aag acc gtg gag cgt 720
Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg
225 230 235 240
aag tgt tgt gtg gag tgt cct cct tgt cct gcc cct cct gtg gcc gga 768
Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly
245 250 255
cct tcc gtg ttc ctt ttc cct cct aag cct aag gac acc ctg atg atc 816
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
agc cgt acc cct gag gtg acc tgt gtg gtg gtg gac gtg tcc cac gag 864
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
gac cct gag gtg cag ttc aac tgg tac gtg gac ggc gtg gag gtg cac 912
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
aac gcc aag acc aag cct cgt gag gag caa ttc aac agc acc ttc cgt 960
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg
305 310 315 320
gtg gtg tcc gtg ctt acc gtg gtg cac caa gac tgg ctg aac ggc aag 1008
Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys
325 330 335
gag tac aag tgt aag gtg agc aac aag gga ctt cct gcc cct atc gag 1056
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu
340 345 350
aag acc atc tcc aag acc aag ggc caa cct cgt gag cct caa gtg tac 1104
Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
acc ctt cct cct agc cgt gag gag atg acc aag aac caa gtg tcc ctt 1152
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
acc tgt ctg gtg aag ggc ttc tac cct agc gac atc gcc gtg gag tgg 1200
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
gag tcc aac gga caa cct gag aac aac tac aag acc acc cct cct atg 1248
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met
405 410 415
ctt gac agc gac ggc tcc ttc ttc ctg tac agc aag ctg acc gtg gac 1296
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
aag tcc cgt tgg caa caa ggc aac gtg ttc agc tgt tcc gtg atg cac 1344
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
gag gcc ctg cac aac cac tac acc caa aag agc ctt tcc ctg agc cct 1392
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
gga aag 1398
Gly Lys
465
<210> 99
<211> 466
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 99
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Ala Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala
85 90 95
Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu
145 150 155 160
Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn
210 215 220
Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg
225 230 235 240
Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
Gly Lys
465
<210> 100
<211> 1398
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-H5
<220>
<221> CDS
<222> (1)..(1398)
<400> 100
atg aaa cac ctg tgg ttc ttc ctc ctg ctg gtg gca gct ccc aga tgg 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
gtg ctg agc gag gtg cag gtg gtg gag agc ggc ggc gga ctt gtg cag 96
Val Leu Ser Glu Val Gln Val Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
cct ggc ggc tcc ctg aga ctt agc tgt gcc acc tcc ggc ttc aac ttc 144
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
aac gac tac ttc atg aac tgg gtg aga cag gcc cct ggc aag ggc ctg 192
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
gag tgg gtg gcc cag atc aga aac aag atc tac acc tac gcc acc ttc 240
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
tac gcc gag agc ctt gag ggc aga ttc acc atc tcc aga gac aac agc 288
Tyr Ala Glu Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
aag tcc acc gtg tac ctt cag atg aac tcc ctg aga gcc gag gac acc 336
Lys Ser Thr Val Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
gcc gtg tac tac tgt acc aga agc ctt acc ggc ggc gac tac ttc gac 384
Ala Val Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
tac tgg ggc cag ggc acc ctg gtg acc gtg agc tca gcc agc acc aag 432
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
ggc cct tcc gtg ttc cct ctg gcc cct tgt agc cgt tcc acc agc gag 480
Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu
145 150 155 160
tcc acc gcc gcc ctt ggc tgt ctg gtg aag gac tac ttc cct gag cct 528
Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
gtg acc gtg agc tgg aac tcc gga gcc ctt acc agc ggc gtg cac acc 576
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
ttc cct gcc gtg ctg cag tcc agc ggc ctt tac tcc ctg agc tcc gtg 624
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
gtg acc gtg cct agc tcc aac ttc ggc acc caa acc tac acc tgt aac 672
Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn
210 215 220
gtg gac cac aag cct agc aac acc aag gtg gac aag acc gtg gag cgt 720
Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg
225 230 235 240
aag tgt tgt gtg gag tgt cct cct tgt cct gcc cct cct gtg gcc gga 768
Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly
245 250 255
cct tcc gtg ttc ctt ttc cct cct aag cct aag gac acc ctg atg atc 816
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
agc cgt acc cct gag gtg acc tgt gtg gtg gtg gac gtg tcc cac gag 864
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
gac cct gag gtg cag ttc aac tgg tac gtg gac ggc gtg gag gtg cac 912
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
aac gcc aag acc aag cct cgt gag gag caa ttc aac agc acc ttc cgt 960
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg
305 310 315 320
gtg gtg tcc gtg ctt acc gtg gtg cac caa gac tgg ctg aac ggc aag 1008
Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys
325 330 335
gag tac aag tgt aag gtg agc aac aag gga ctt cct gcc cct atc gag 1056
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu
340 345 350
aag acc atc tcc aag acc aag ggc caa cct cgt gag cct caa gtg tac 1104
Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
acc ctt cct cct agc cgt gag gag atg acc aag aac caa gtg tcc ctt 1152
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
acc tgt ctg gtg aag ggc ttc tac cct agc gac atc gcc gtg gag tgg 1200
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
gag tcc aac gga caa cct gag aac aac tac aag acc acc cct cct atg 1248
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met
405 410 415
ctt gac agc gac ggc tcc ttc ttc ctg tac agc aag ctg acc gtg gac 1296
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
aag tcc cgt tgg caa caa ggc aac gtg ttc agc tgt tcc gtg atg cac 1344
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
gag gcc ctg cac aac cac tac acc caa aag agc ctt tcc ctg agc cct 1392
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
gga aag 1398
Gly Lys
465
<210> 101
<211> 466
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 101
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Val Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Asn Phe
35 40 45
Asn Asp Tyr Phe Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Gln Ile Arg Asn Lys Ile Tyr Thr Tyr Ala Thr Phe
65 70 75 80
Tyr Ala Glu Ser Leu Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Ser Thr Val Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Ser Leu Thr Gly Gly Asp Tyr Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu
145 150 155 160
Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn
210 215 220
Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg
225 230 235 240
Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
Gly Lys
465
<210> 102
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-L2-15
<220>
<221> CDS
<222> (1)..(714)
<400> 102
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gaa att ctg atg acg cag agt cct gca act ctt agt 96
Gly Ala Tyr Gly Glu Ile Leu Met Thr Gln Ser Pro Ala Thr Leu Ser
20 25 30
ctg tca cct ggc gag aga gcc aca ctc agc tgc cga gcg tcc cag tcc 144
Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser
35 40 45
gtg acc att agc ggc tat tct ttt att cat tgg tat cag caa aag cct 192
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
gga cag gcg cca agg ctg ctc att tac aga gca agc aac ctt gcc tct 240
Gly Gln Ala Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
ggc att cca gca aga ttc agc ggg agc gga tca ggg aca gat ttc acc 288
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
ttg acc atc tcc tcc ctg gag ccg gag gat ttc gcg ttg tat tat tgt 336
Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Leu Tyr Tyr Cys
100 105 110
cag caa tct agg aag agt cca tgg aca ttt ggc cag ggc acc aaa gtg 384
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Gly Gln Gly Thr Lys Val
115 120 125
gag atc aag cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc 432
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
tcc gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg 480
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
aat aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac 528
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
gcc ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc 576
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
aag gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc 624
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
gac tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc 672
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
ctg agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt 714
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 103
<211> 238
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 103
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Glu Ile Leu Met Thr Gln Ser Pro Ala Thr Leu Ser
20 25 30
Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser
35 40 45
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Ala Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Leu Tyr Tyr Cys
100 105 110
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Gly Gln Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 104
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> h#32A1-L2-16
<220>
<221> CDS
<222> (1)..(714)
<400> 104
atg gtg ctg cag acc cag gtg ttc atc tcc ctg ctg ctg tgg atc tcc 48
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
ggc gca tat ggc gaa atc ctg atg aca cag agt cct gcg acc ctc tcc 96
Gly Ala Tyr Gly Glu Ile Leu Met Thr Gln Ser Pro Ala Thr Leu Ser
20 25 30
ctc tca cca ggc gag agg gcc acc ctg tct tgt aga gcc agc cag tca 144
Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser
35 40 45
gtc act att agt gga tac tca ttt atc cat tgg tat caa cag aaa cca 192
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
gga cag gcg cct cgg ctt ctg atc tac cgc gcc tca aac ctt gcc tct 240
Gly Gln Ala Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
ggc atc ccc gcg agg ttc tct ggc tct ggc agc ggt acc gat ttt aca 288
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
ctg act atc tca agc ctc gaa cct gaa gac ttc gca ctg tac ttt tgc 336
Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Leu Tyr Phe Cys
100 105 110
cag cag agc agg aag tcc ccc tgg act ttt gca cag gga acc aaa gtc 384
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gln Gly Thr Lys Val
115 120 125
gaa atc aag cgt acg gtg gcc gcc ccc tcc gtg ttc atc ttc ccc ccc 432
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
tcc gac gag cag ctg aag tcc ggc acc gcc tcc gtg gtg tgc ctg ctg 480
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
aat aac ttc tac ccc aga gag gcc aag gtg cag tgg aag gtg gac aac 528
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
gcc ctg cag tcc ggg aac tcc cag gag agc gtg acc gag cag gac agc 576
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
aag gac agc acc tac agc ctg agc agc acc ctg acc ctg agc aaa gcc 624
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
gac tac gag aag cac aag gtg tac gcc tgc gag gtg acc cac cag ggc 672
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
ctg agc tcc ccc gtc acc aag agc ttc aac agg ggg gag tgt 714
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 105
<211> 238
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 105
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Glu Ile Leu Met Thr Gln Ser Pro Ala Thr Leu Ser
20 25 30
Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser
35 40 45
Val Thr Ile Ser Gly Tyr Ser Phe Ile His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Ala Pro Arg Leu Leu Ile Tyr Arg Ala Ser Asn Leu Ala Ser
65 70 75 80
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Leu Tyr Phe Cys
100 105 110
Gln Gln Ser Arg Lys Ser Pro Trp Thr Phe Ala Gln Gly Thr Lys Val
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 106
<211> 1111
<212> DNA
<213> 人工序列
<220>
<223> 编码信号肽和人IgG2重链的恒定区的核酸序列
<400> 106
aagcttgcta gcgccaccat gaaacacctg tggttcttcc tcctgctggt ggcagctccc 60
agatgggtgc tgagccaggt gcaattgtgc aggcggttag ctcagccagc accaagggcc 120
cttccgtgtt ccctctggcc ccttgtagcc gttccaccag cgagtccacc gccgcccttg 180
gctgtctggt gaaggactac ttccctgagc ctgtgaccgt gagctggaac tccggagccc 240
ttaccagcgg cgtgcacacc ttccctgccg tgctgcagtc cagcggcctt tactccctga 300
gctccgtggt gaccgtgcct agctccaact tcggcaccca aacctacacc tgtaacgtgg 360
accacaagcc tagcaacacc aaggtggaca agaccgtgga gcgtaagtgt tgtgtggagt 420
gtcctccttg tcctgcccct cctgtggccg gaccttccgt gttccttttc cctcctaagc 480
ctaaggacac cctgatgatc agccgtaccc ctgaggtgac ctgtgtggtg gtggacgtgt 540
cccacgagga ccctgaggtg cagttcaact ggtacgtgga cggcgtggag gtgcacaacg 600
ccaagaccaa gcctcgtgag gagcaattca acagcacctt ccgtgtggtg tccgtgctta 660
ccgtggtgca ccaagactgg ctgaacggca aggagtacaa gtgtaaggtg agcaacaagg 720
gacttcctgc ccctatcgag aagaccatct ccaagaccaa gggccaacct cgtgagcctc 780
aagtgtacac ccttcctcct agccgtgagg agatgaccaa gaaccaagtg tcccttacct 840
gtctggtgaa gggcttctac cctagcgaca tcgccgtgga gtgggagtcc aacggacaac 900
ctgagaacaa ctacaagacc acccctccta tgcttgacag cgacggctcc ttcttcctgt 960
acagcaagct gaccgtggac aagtcccgtt ggcaacaagg caacgtgttc agctgttccg 1020
tgatgcacga ggccctgcac aaccactaca cccaaaagag cctttccctg agccctggaa 1080
agtgatatcg ggcccgttta aacgggtggc a 1111
- 还没有人留言评论。精彩留言会获得点赞!