一种鉴别刺槐种质资源亲缘关系的SSR标记引物组及其应用的制作方法
本发明涉及一种鉴别刺槐种质资源亲缘关系的SSR标记引物组及其应用,属于生物技术技术领域。
背景技术:
SSR简称重复序列标记(Simple sequence repeats,SSR),是基于PCR(基因扩增)技术的分子标记,具有高多态性、重复性、共显性、丰富性等优点,对于分析品种亲缘关系和遗传多样性研究具有重要意义,通过确定遗传距离,来分析其亲缘关系,遗传距离为零时,表明植株出自同一无性系。
中国专利文献CN102286613A(申请号201110127982.7)公开了防天麻退化的选育方法,它是采用AFLP或ISSR和SSR及天麻素含量标记同时检测天麻,经软件绘制聚类图并绘制天麻遗传多态性与天麻素含量关系图,从而快速分析天麻种质资源亲缘关系,鉴别天麻自交与杂交体,快速选择确定天麻亲本,快速鉴定天麻杂种纯度等,能使中药天麻优质可持续发展的育种选择方法。
目前,现有技术多采用挖根的方法来调查刺槐是否出于同一无性系,然而由于刺槐连续多代萌生林无性系小株之间的连接根系可能已经死亡,挖根的方法并不能准确判断它们的萌蘖扩散范围和扩散规律。而且挖根的方法耗时费力,在判断刺槐的萌生植株是否来自于同一个基株时存在一定的主观性。如何准确鉴别刺槐的萌生植株是否来自于同一个基株成为研究刺槐扩散格局与过程的关键问题,也是判断是否是同一无性系的主要环节。但由于目前未有采用SSR标记鉴别刺槐种质资源亲缘关系的相关报道,如何利用相关SSR标记进行检测,从而提高检测的准确性和可靠性成为解决上述问题的关键。
技术实现要素:
本发明针对现有技术的不足,提供一种鉴别刺槐种质资源亲缘关系的SSR标记引物组及其应用。
本发明技术方案如下:
一种鉴别刺槐种质资源亲缘关系的SSR标记引物组,SSR标记引物组共四组,每组包括4对引物;第一组的四对SSR标记引物分别为,标记Rops04的上游引物核苷酸序列如SEQ ID NO.1所示,下游引物核苷酸序列如SEQ ID NO.2所示;标记RP106的上游引物核苷酸序列如SEQ ID NO.3所示,下游引物核苷酸序列如SEQ ID NO.4所示;标记Rops09的上游引物核苷酸序列如SEQ ID NO.5所示,下游引物核苷酸序列如SEQ ID NO.6所示;标记RP150的上游引物核苷酸序列如SEQ ID NO.7所示,下游引物核苷酸序列如SEQ ID NO.8所示;
第二组的四对SSR标记引物分别为,标记RP035的上游引物核苷酸序列如SEQ ID NO.9所示,下游引物核苷酸序列如SEQ ID NO.10所示;标记RP109的上游引物核苷酸序列如SEQ ID NO.11所示,下游引物核苷酸序列如SEQ ID NO.12所示;标记RP200的上游引物核苷酸序列如SEQ ID NO.13所示,下游引物核苷酸序列如SEQ ID NO.14所示;标记RP032的上游引物核苷酸序列如SEQ ID NO.15所示,下游引物核苷酸序列如SEQ ID NO.16所示;
第三组的四对SSR标记引物分别为,标记Rops02的上游引物核苷酸序列如SEQ ID NO.17所示,下游引物核苷酸序列如SEQ ID NO.18所示;标记Rops06的上游引物核苷酸序列如SEQ ID NO.19所示,下游引物核苷酸序列如SEQ ID NO.20所示;标记Rops08的上游引物核苷酸序列如SEQ ID NO.21所示,下游引物核苷酸序列如SEQ ID NO.22所示;标记RP206的上游引物核苷酸序列如SEQ ID NO.23所示,下游引物核苷酸序列如SEQ ID NO.24所示;
第四组的四对SSR标记引物分别为,标记Rops05的上游引物核苷酸序列如SEQ ID NO.25所示,下游引物核苷酸序列如SEQ ID NO.26所示;标记Rops10的上游引物核苷酸序列如SEQ ID NO.27所示,下游引物核苷酸序列如SEQ ID NO.28所示;标记RP165的上游引物核苷酸序列如SEQ ID NO.29所示,下游引物核苷酸序列如SEQ ID NO.30所示;标记RP01B的上游引物核苷酸序列如SEQ ID NO.31所示,下游引物核苷酸序列如SEQ ID NO.32所示。
根据本发明优选的,所述标记Rops04的上游引物、标记RP150的上游引物、标记RP200的上游引物、标记RP032的上游引物、标记Rops06的上游引物、标记RP206的上游引物、标记Rops05的上游引物、标记Rops10的上游引物的5’端连接有荧光标签FAM;
所述标记RP106的上游引物、标记RP109的上游引物、标记Rops08的上游引物、标记RP01B的上游引物的上游引物的5’端连接有荧光标签HEX;
所述标记Rops09的上游引物、标记RP035的上游引物、标记Rops02的上游引物、标记RP165的上游引物的5’端连接有荧光标签TAM。
一种利用上述SSR标记引物组鉴别刺槐种质资源亲缘关系的方法,包括如下步骤:
(1)取待鉴别样品,提取总DNA;
(2)以步骤(1)制得的总DNA为模板,分别用上述鉴别刺槐种质资源亲缘关系的四组SSR标记引物进行PCR扩增,制得PCR扩增产物;
(3)对步骤(2)制得的PCR扩增产物进行分析,当扩增出SSR标记时,用1表示,当未扩增出SSR标记时,用0表示,将每对SSR标记扩增后的结果转换为矩阵,用非加权算术平均聚类方法(UPGMA)分析计算遗传距离,得出刺槐种质资源亲缘关系。
根据本发明优选的,所述步骤(1)中,样品为叶片或枝条。
根据本发明优选的,所述步骤(1)中,总DNA的提取方法采用改良的CTAB法。
根据本发明优选的,所述步骤(2)中,PCR扩增体系如下,总体积为10μL:
4μL Takara Multi-plex PCR Master Mix,浓度为10μM的4条上游引物每条各0.2μL,浓度为10μM的4条下游引物每条各0.2μL,10~30ng/μL模板1μL,加3.4μL ddH2O补足至反应总体积;
根据本发明优选的,所述步骤(2)中,PCR扩增程序如下:
94℃预变性15min;94℃变性30s,55℃退火90s,72℃延伸60s,共28个循环;60℃终延伸30min,4℃终止反应。
根据本发明优选的,所述步骤(3)中的分析为:将步骤(2)的PCR扩增产物经电泳后进行测序;然后用软件GeneMarker ver.2.4.0统计每个个体16对SSR标记所有等位基因的长度,并与标准进行对照,当长度在标准范围内时,为有效扩增出SSR标记,当长度在标准范围外时,为未扩增出SSR标记;所述的标准范围如下:
标记Rops04的标准范围为105~110bp;
标记RP106的标准范围为143~154bp;
标记Rops09的标准范围为89~150bp;
标记RP150的标准范围为199~217bp;
标记RP035的标准范围为89~112bp;
标记RP109的标准范围为136~160bp;
标记RP200的标准范围为160~198bp;
标记RP032的标准范围为109~135bp;
标记Rops02的标准范围为107~138bp;
标记Rops06的标准范围为117~144bp;
标记Rops08的标准范围为191~205bp;
标记RP206的标准范围为222~246bp;
标记Rops05的标准范围为120~138bp;
标记Rops10的标准范围为182~187bp;
标记RP165的标准范围为146~159bp;
标记RP01B的标准范围为172~192bp。
根据本发明进一步优选的,所述电泳为采用ABI 3730xl测序仪进行。
根据本发明优选的,所述步骤(3)中用非加权算术平均聚类方法计算遗传距离采用NTSYSpc-2.11F软件。
有益效果
1、本发明首次采用SSR标记引物组鉴别刺槐种质资源亲缘关系,通过筛选16个SSR标记并设计特异引物,从而确保了鉴定的准确性,较现有鉴别方法可以显著缩短鉴别时间;
2、本发明通过在上游引物的5’端连接荧光标签,使每一PCR反应体系中包含4对引物,降低了试验成本及时间;
附图说明
图1是实施例2所述刺槐萌蘖的29个个体的空间分布图;
图中:圆圈表示刺槐个体,圆越大树龄越大,同一颜色为同一基因型。
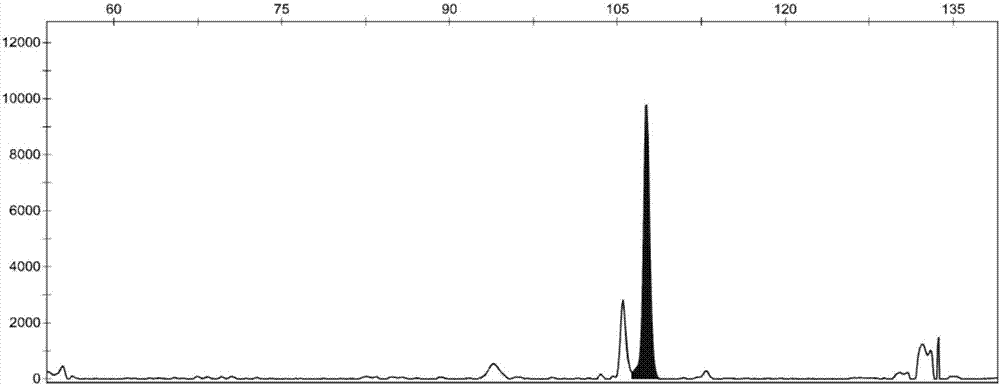
图2是实施例2的W1刺槐个体的PCR产物序列长度结果图;
其中:图2A、带有FAM荧光标签引物(Rops04和RP150)的PCR产物序列长度峰图;图2B、带有HEX荧光标签引物(RP106)的PCR产物序列长度峰图;图2C、带有TAM荧光标签引物(Rops09)的PCR产物序列长度峰图;
图3是实施例2的W1刺槐个体的PCR产物序列长度结果图;
其中:图3A、带有FAM荧光标签引物(RP200和RP032)的PCR产物序列长度峰图;图3B、带有HEX荧光标签引物(RP109)的PCR产物序列长度峰图;图3C、带有TAM荧光标签引物(RP035)的PCR产物序列长度峰图;
图4是实施例2的W1刺槐个体的PCR产物序列长度结果图;
其中:图4A、带有FAM荧光标签引物(Rops06和RP206)的PCR产物序列长度峰图;图4B、带有HEX荧光标签引物(Rops08)的PCR产物序列长度峰图;图4C、带有TAM荧光标签引物(Rops02)的PCR产物序列长度峰图;
图5是实施例2的W1刺槐个体的PCR产物序列长度结果图;
其中:图5A、带有FAM荧光标签引物(Rops04和RP150)的PCR产物序列长度峰图;图5B、带有HEX荧光标签引物(RP106)的PCR产物序列长度峰图;图5C、带有TAM荧光标签引物(Rops09)的PCR产物序列长度峰图;
图6是实施例2所述29株刺槐SSR标记的UPGMA聚类分析图。
具体实施方式
下面结合实施例对本发明的技术方案做进一步阐述,但本发明所保护范围不限于此。
实施例1
参照Lian C et al.(2002,2004)和Mishima K et al.(2009)开发的刺槐的21对SSR引物,选取了16对多态性高且通用性好的引物。
用的16对引物如表1所示:
表1用于刺槐基株、无性系小株鉴定和群体遗传结构分析的SSR引物
实施例2
样品来源
以泰山景区花园小水坝西坡的刺槐纯林为研究对象(东经:117°3′35.96″;北纬:36°16′56.26″;海拔:654.9m)。1958年造林,1976年间伐,1980年皆伐,1984年定株并梯田整地栽植华山松,1986年华山松移走后发展为萌生林。
取样过程
在样地内选取20m×15m范围,将其内的29株个刺槐个体进行SSR标记分析;
选择刺槐纯林内20m×15m的样地,以样地内29株刺槐个体为研究对象,采取29株刺槐个体的叶片或枝条,分别放入乘有硅胶的牛皮纸袋中,并标上样品号。
一种利用SSR标记引物组鉴别刺槐种质资源亲缘关系的方法,包括如下步骤:
(1)取待鉴别样品的叶片或枝条,采用改良的CTAB法提取总DNA;
(2)以步骤(1)制得的总DNA为模板,分别用上述鉴别刺槐种质资源亲缘关系的SSR标记引物组中的特异引物组Multiplex 1、Multiplex 2、Multiplex 3和Multiplex 4(表1所示)进行PCR扩增,制得PCR扩增产物;
PCR扩增体系如下,总体积为10μL:
4μL Takara Multi-plex PCR Master Mix,浓度为10μM的4条上游引物每条各0.2μL,浓度为10μM的4条下游引物每条各0.2μL,10-30ng/μL模板1μL,加3.4μL ddH2O补足至反应总体积;
所述步骤(2)中,PCR扩增程序如下:
94℃预变性15min;94℃变性30s,55℃退火90s,72℃延伸60s,共28个循环;60℃终延伸30min,4℃终止反应。
(3)将步骤(2)的PCR扩增产物经ABI 3730xl测序仪上跑胶(上海生工),条带长度结果以峰图的形式表示;以W1刺槐个体DNA为模板,以Multiplex 1(表1)为引物的PCR产物序列长度示范图如图2所示,其中A、B和C图分别表示带有FAM荧光标签引物(Rops04和RP150)、HEX荧光标签引物(RP106)和TAM荧光标签引物(Rops09)的PCR产物序列长度。
以Multiplex 2(表1)为引物的PCR产物序列长度示范图如图3所示,其中A、B和C图分别表示带有FAM荧光标签引物(RP200和RP032)、HEX荧光标签引物(RP109)和TAM荧光标签引物(RP035)的PCR产物序列长度。
以Multiplex 3(表1)为引物的PCR产物序列长度示范图如图4所示,其中A、B和C图分别表示带有FAM荧光标签引物(Rops06和RP206)、HEX荧光标签引物(Rops08)和TAM荧光标签引物(Rops02)的PCR产物序列长度。
以Multiplex 4(表1)为引物的PCR产物序列长度示范图如图5所示,其中A、B和C图分别表示带有FAM荧光标签引物(Rops04和RP150)、HEX荧光标签引物(RP106)和TAM荧光标签引物(Rops09)的PCR产物序列长度。
然后用软件GeneMarker ver.2.4.0统计,每个个体16对SSR标记所有等位基因的长度和有效性与标准范围进行比较,当扩增出SSR标记时,用1表示,当未扩增出SSR标记时,用0表示,将每对SSR标记扩增后的结果转换为矩阵,采用Popgene version 1.31软件计算Shannon指数(I,Shannon’s information index)、基因多样性指数(H,gene diversity)、有效等位基因数(Ne,effective number of alleles),用NTSYSpc-2.11F软件计算遗传距离和非加权算术平均聚类方法分析(UPGMA),得出刺槐种质资源亲缘关系。
为检查本申请的微卫星分析的可重复性,选择9株刺槐个体进行重复微卫星分析。结果表明,100%的结果相匹配,这表明本申请所述的微卫星分析具有高度可重复性。
将遗传距离为0的株系均定为同一无性系,结果发现同一基因型个体数超过2个的有四组,即W15,W2,W12,W22号刺槐是同一无性系;W16,W19,W11,W17号是同一无性系;W25,W3,W26,W14号是同一无性系;W9和W13号刺槐是同一无性系,如图1所示,以不同颜色表示。
进一步计算它们的空间距离即可得其扩散的范围。对这4组个体分别掘根观察,证实每组个体均为同一无性系。余下15个个体基因型各不相同,可能与样地内未检测的个体以及样地外的其他个体存在相同基因型,也可能是来自种子萌发。该结果证明利用SSR标记技术分析刺槐连续多代萌生林无性系小株的来源,进一步判断刺槐萌蘖扩散规律是可行的。这一尝试的成功,为本研究探索刺槐多代萌生林的株间亲缘关系,揭示刺槐萌蘖扩散范围和扩散规律奠定了坚实基础。
SEQUENCE LISTING
<110> 山东农业大学
<120> 一种鉴别刺槐种质资源亲缘关系的SSR标记引物组及其应用
<160> 32
<170> PatentIn version 3.5
<210> 1
<211> 24
<212> DNA
<213> 人工合成
<400> 1
gtctaatttc acttttctca cgag 24
<210> 2
<211> 21
<212> DNA
<213> 人工合成
<400> 2
ggacaccacc raaattctac c 21
<210> 3
<211> 25
<212> DNA
<213> 人工合成
<400> 3
aaactgaatt atatcccttt acggc 25
<210> 4
<211> 22
<212> DNA
<213> 人工合成
<400> 4
gcatatatcc accagatacc cg 22
<210> 5
<211> 21
<212> DNA
<213> 人工合成
<400> 5
ctccaggtca ctcgattgag g 21
<210> 6
<211> 21
<212> DNA
<213> 人工合成
<400> 6
tttctcattt gatacgaccc c 21
<210> 7
<211> 21
<212> DNA
<213> 人工合成
<400> 7
tcgttggatc aacatgcatg g 21
<210> 8
<211> 20
<212> DNA
<213> 人工合成
<400> 8
acagaaccct aaccctagca 20
<210> 9
<211> 23
<212> DNA
<213> 人工合成
<400> 9
ggagtggaat gcatgctctc atg 23
<210> 10
<211> 26
<212> DNA
<213> 人工合成
<400> 10
tccaaatgga aactcccttg aaacac 26
<210> 11
<211> 22
<212> DNA
<213> 人工合成
<400> 11
gaggaatcac aaaaccgttt gg 22
<210> 12
<211> 23
<212> DNA
<213> 人工合成
<400> 12
tgggatttga gagagtggtg gtg 23
<210> 13
<211> 24
<212> DNA
<213> 人工合成
<400> 13
ggtttctttg ttcacctgct ctgg 24
<210> 14
<211> 20
<212> DNA
<213> 人工合成
<400> 14
acctacgtgt ccacggctct 20
<210> 15
<211> 22
<212> DNA
<213> 人工合成
<400> 15
gcatattgca tatgcgcttg tg 22
<210> 16
<211> 26
<212> DNA
<213> 人工合成
<400> 16
tccctgaagc tcataactgt catgtg 26
<210> 17
<211> 26
<212> DNA
<213> 人工合成
<400> 17
cagaactgtg gagaataatt ctgaac 26
<210> 18
<211> 21
<212> DNA
<213> 人工合成
<400> 18
cgccatctgt tagtttgttg c 21
<210> 19
<211> 20
<212> DNA
<213> 人工合成
<400> 19
ctaaggaggt gctgaccctc 20
<210> 20
<211> 21
<212> DNA
<213> 人工合成
<400> 20
ttaatctgtg atgggacact g 21
<210> 21
<211> 21
<212> DNA
<213> 人工合成
<400> 21
ttctgaggaa gggttccgtg g 21
<210> 22
<211> 21
<212> DNA
<213> 人工合成
<400> 22
gttaaagcaa caggcacatg g 21
<210> 23
<211> 26
<212> DNA
<213> 人工合成
<400> 23
gccaaatccc attagatcac agttga 26
<210> 24
<211> 22
<212> DNA
<213> 人工合成
<400> 24
agaagttaga cttacgtgct gc 22
<210> 25
<211> 21
<212> DNA
<213> 人工合成
<400> 25
tggtgattaa gtcgcaaggt g 21
<210> 26
<211> 21
<212> DNA
<213> 人工合成
<400> 26
gttgtgactt gtacgtaagt c 21
<210> 27
<211> 21
<212> DNA
<213> 人工合成
<400> 27
aactttttcc gtataggggt c 21
<210> 28
<211> 22
<212> DNA
<213> 人工合成
<400> 28
gagttttaca cttggtcaaa cc 22
<210> 29
<211> 22
<212> DNA
<213> 人工合成
<400> 29
ttagatgttg caagtgctga gg 22
<210> 30
<211> 18
<212> DNA
<213> 人工合成
<400> 30
acaatgcctc aatgcagc 18
<210> 31
<211> 20
<212> DNA
<213> 人工合成
<400> 31
accaattagg taacgtcagc 20
<210> 32
<211> 18
<212> DNA
<213> 人工合成
<400> 32
tgttcactga caaagctg 18
- 还没有人留言评论。精彩留言会获得点赞!