一种基于熔解曲线法单管同时检测多种呼吸道病毒的引物及其应用的制作方法
本发明涉及核酸检测
技术领域:
,具体涉及一种基于荧光探针熔解曲线法同时检测多种呼吸道病毒的方法,这7种病毒包括:甲型流感病毒(IFA)、乙型流感病毒(IFB)、呼吸道合胞病毒A型(RSVA)、呼吸道合胞病毒B型(RSVB)、鼻病毒(RV)、柯萨奇病毒B型(CoxB)、腺病毒(AdV)。
背景技术:
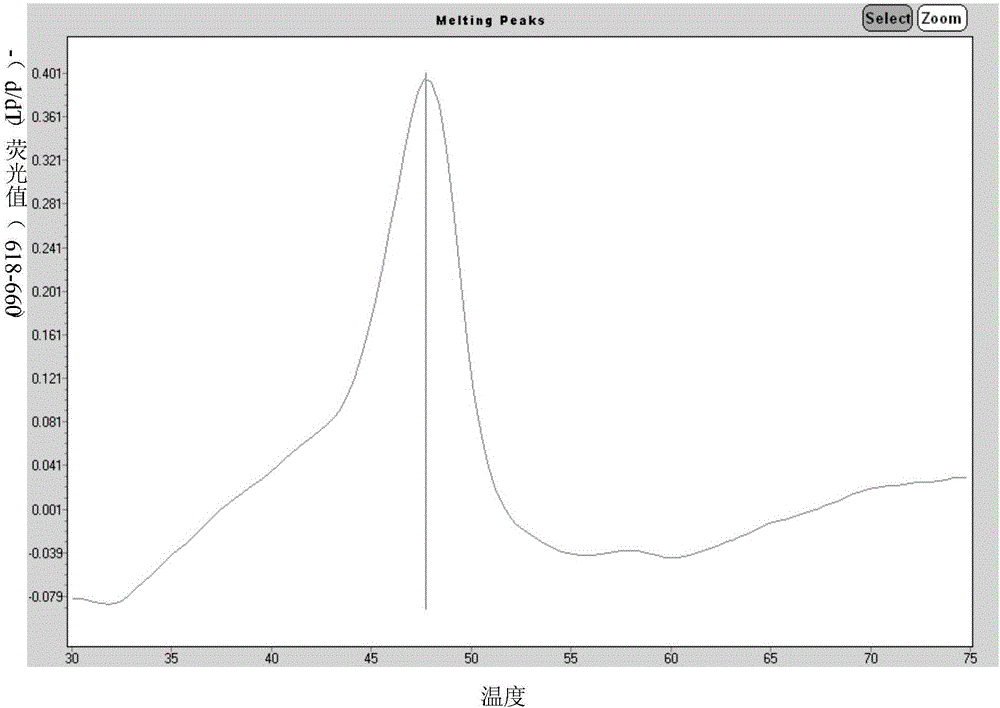
:急性呼吸道感染是临床最常见的疾病之一,可以在任何性别、年龄和地域发生,尤其在老年人和婴幼儿中有较高的发病率和死亡率,严重影响人的身体健康,给社会带来严重的经济损失。仅20世纪,全球就有超过1亿人因感染流感病毒而死亡。急性呼吸道感染可由细菌、病毒和支原体感染引起,呼吸道病毒是其中主要病原体。由于病毒颗粒小,容易通过呼吸道进行传播,且传播速度快,容易造成大流行。另外病毒容易发生突变,产生新的高致病性病原体,感染力增强,混合感染率的概率提高。不同呼吸道病毒所引发的症状和流行特点相似,临床上难以区分从而进行准确诊治。因此,准确鉴定呼吸道病毒的病原体对于制定治疗和用药方案、控制呼吸道病毒的传播具有重要意义。对于呼吸道病毒感染的诊断,其常用的检测方法有以下几种:1、病毒分离培养法,是病毒诊断的“金标准”,缺点:费时费力,且需要有经验操作者;生物安全风险较高。2、免疫学检查,是临床门诊检验常用的方法,该方法操作简单、快速、费用低,但灵敏度特异性不高,病毒抗原发生变异时出现假阴性结果,且一个检测只能针对其中一种病原体。3、分子生物学方法,其优点是:灵敏度高,特异性强,但目前采用的普通荧光PCR检测一般一次只能检测一种病毒,通过重复多次实现对所有病原体的检测,而多通道荧光检测单管最多只能检测3-5个,无法满足一次进行多种病原体的检测。由此可见目前急需一种灵敏、特异、快速、简便的呼吸道病毒的多病原体检测的方法,以便能满足临床诊断、卫生防疫、传染病流行病学研究的需要。Tem-PCR(TargetenrichedmultiplexPCR靶序列富集多重PCR技术)是一种多重PCR技术,能在一次反应中对多种靶序列进行扩增和检测,本发明基于Tem-PCR技术,经荧光分子信标检测通过Tm值分析来实现对多种病原体的同时检测。技术实现要素:本发明的目的是解决多种呼吸道病原体的同时检测,提供一种同时扩增和检测7种呼吸道病毒的引物、探针。为解决上述技术问题,本发明采用如下技术方案:一种基于熔解曲线法单管同时检测多种呼吸道病毒的引物,包括SEQIDNO.1~8所示的反转录引物、SEQIDNO.9~26所示的扩增引物、SEQIDNO:27~28所示的启动扩增的通用引物、SEQIDNO.29~35所示的分子信标探针,所述每条分子信标探针的两端分别带有荧光基团和猝灭基团。所述反转录引物在一次检测中共同使用,其中,腺病毒为DNA病毒,无需反转录。其中,所述的病毒包括:甲型流感病毒、乙型流感病毒、呼吸道合胞病毒A型、呼吸道合胞病毒B型、鼻病毒、柯萨奇病毒B型、腺病毒。其中,所述荧光基团为FAM、HEX或者Cy5,所述淬灭基团为DABCYL。上述基于熔解曲线法单管同时检测多种呼吸道病毒的引物在检测呼吸道病毒中的应用。进一步的,所述通用引物在扩增体系中的终浓度为0.1uM;所述加尾特异性上游引物浓度为0.02uM,加尾特异性下游引物浓度为0.2uM;分子信标探针浓度为0.2uM。进一步的,所述PCR检测程序,具体程序如下:热启动:95℃15min;富集阶段:94℃30s,52℃1min,72℃1min(15cycles);加尾扩增:94℃15s,70℃1.5min(6cycles);PCR扩增:94℃15s,52℃15s,72℃15s(55cycles);延伸阶段:72℃3min;熔解分析:95℃1min,37℃5min,80℃~30℃0.1℃/s。进一步的,所述检测方法,根据熔解曲线的Tm峰值,进行分型。本发明提出的检测7种呼吸道病毒的方法,通过以下技术方案实现:一种可单管同时检测甲型流感病毒(IFA)、乙型流感病毒(IFB)、呼吸道合胞病毒A型(RSVA)、呼吸道合胞病毒B型(RSVB)、鼻病毒(RV,包含RVA、RVB、RVC三型)、柯萨奇病毒B型(CoxB)、腺病毒(AdV)的检测方法,包括用于扩增以上7种呼吸道病毒特异性基因位点的反转录引物、扩增引物对、探针(IFA、IFB、RSVA、RSVB、RV、CoxB、AdV)。所述用于反转录的反转录引物,包含以下序列:反转录甲型流感病毒的反转录引物,其核苷酸序列如SEQIDNO:1所示;反转录乙型流感病毒的反转录引物,其核苷酸序列如SEQIDNO:2所示;反转录呼吸道合胞病毒A型的反转录引物,其核苷酸序列如SEQIDNO:3所示;反转录呼吸道合胞病毒B型的反转录引物,其核苷酸序列如SEQIDNO:4所示;反转录鼻病毒A型的反转录引物,其核苷酸序列如SEQIDNO:5所示;反转录鼻病毒B型的反转录引物,其核苷酸序列如SEQIDNO:6所示;反转录鼻病毒C型的反转录引物,其核苷酸序列如SEQIDNO:7所示;反转录柯萨奇病毒B型的反转录引物,其核苷酸序列如SEQIDNO:8所示;上述反转录引物是各病毒的特异性引物。所述扩增引物对,包含以下序列:扩增甲型流感病毒的引物对,其核苷酸序列如SEQIDNO:9~10所示;扩增乙型流感病毒的引物对,其核苷酸序列如SEQIDNO:11~12所示;扩增呼吸道合胞病毒A型的引物对,其核苷酸序列如SEQIDNO:13~14所示;扩增呼吸道合胞病毒B型的引物对,其核苷酸序列如SEQIDNO:15~16所示;扩增鼻病毒A型的引物对,其核苷酸序列如SEQIDNO:17~18所示;扩增鼻病毒B型的引物对,其核苷酸序列如SEQIDNO:19~20所示;扩增鼻病毒C型的引物对,其核苷酸序列如SEQIDNO:21~22所示;扩增柯萨奇病毒B型的引物对,其核苷酸序列如SEQIDNO:23~24所示;扩增腺病毒的引物对,其核苷酸序列如SEQIDNO:25~26所示;启动扩增的通用引物对,其核酸序列如为SEQIDNO:27~28所示。上述扩增引物是由通用引物和特异性引物组合而成的加尾引物,即在特异性引物的5’端添加一段通用引物序列。上述10对引物对在一次检测中共同使用。所述的分子信标探针序列如下:检测甲型流感病毒的分子信标探针,其核苷酸序列如SEQIDNO:29所示;检测乙型流感病毒的分子信标探针,其核苷酸序列如SEQIDNO:30所示;检测呼吸道合胞病毒A型的分子信标探针,其核苷酸序列如SEQIDNO:31所示;检测呼吸道合胞病毒B型的分子信标探针,其核苷酸序列如SEQIDNO:32所示;检测鼻病毒的分子信标探针,其核苷酸序列如SEQIDNO:33所示;检测柯萨奇病毒B型的分子信标探针,其核苷酸序列如SEQIDNO:34所示;检测腺病毒的分子信标探针,其核苷酸序列如SEQIDNO:35所示;每条分子信标探针的两端分别带有荧光基团和猝灭基团。上述7条探针在一次检测中共同使用。每一条所述分子信标探针的整体为茎环结构,两端为形成发夹结构的互补序列,中间为与检测呼吸道病毒扩增产物的靶特异性序列,每一条分子信标探针的熔解温度范围在38℃~70℃,至少两条所述分子信标探针中若具有相同的荧光基团,则具有相同荧光基团标记的分子信标探针的Tm值设计成梯度,差异在2℃以上。有益效果:采用本发明的技术方案,可以单管同时鉴别7种呼吸道病毒,且操作简便,检测准确、灵敏度高、特异性强、快速,可在4小时内出结果。本发明利用多重Tem-PCR扩增技术与荧光分子信标熔解曲线技术,可同时进行扩增与检测,无需开盖分析,可有效避免产物污染,保证检测的准确性;多重扩增与熔解曲线结合,保证检测的通量和多种病毒的分型,检测成本低,适合临床推广应用。附图说明图1为本发明具体实施方式中的甲型流感病毒的FAM荧光熔解曲线图,Tm值为65℃;图2为本发明具体实施方式中的乙型流感病毒的Cy5荧光熔解曲线图,Tm值为47.5℃;图3为本发明具体实施方式中的呼吸道合胞病毒A型的HEX荧光熔解曲线图,Tm值为38.5℃;图4为本发明具体实施方式中的呼吸道合胞病毒B型的HEX荧光熔解曲线图,Tm值为55℃;图5为本发明具体实施方式中的鼻病毒的Cy5荧光熔解曲线图,Tm值为59.1℃;图6为本发明具体实施方式中的柯萨奇病毒B型的FAM荧光熔解曲线图,Tm值为57.7℃;图7为本发明具体实施方式中的腺病毒的FAM荧光熔解曲线图,Tm值为60.65℃;图8为本发明具体实施方式中以乙型流感病毒为阳性的特异性检测结果;图9位本发明具体实施方式中以乙型流感病毒为例的灵敏度检测结果。具体实施方式下面结合具体实施例详细说明本发明,可以更好的理解本发明。然而,实施例所描述的内容仅用于说明本发明,而不是为了限制本发明的范围和应用。实施例1:同时检测甲型流感病毒、乙型流感病毒、呼吸道合胞病毒A型、呼吸道合胞病毒B型、鼻病毒、柯萨奇病毒B型、腺病毒的引物和探针序列,由英潍捷基(上海)贸易有限公司合成:用于反转录的反转录引物:编号引物名称序列(5’-3’)对应病毒SEQIDNO.1IFA-rtpGGGCACGGTGAGCGTGAA甲型流感病毒SEQIDNO.2IFB-rtpGTTTTCRTATCCTCKGAGAAG乙型流感病毒SEQIDNO.3RSVA-rtpGGTATGTTGGGGTTGTGTT呼吸道合胞病毒A型SEQIDNO.4RSVB-rtpTTTCAGTGTGGYTTTYTATTGTT呼吸道合胞病毒B型SEQIDNO.5RVA-rtpTTGTGTGCACTGGCTGCAGG鼻病毒A型SEQIDNO.6RVB-rtpTTATGCTGCAAGGCTCTGGG鼻病毒B型SEQIDNO.7RVC-rtpGTGTGCGATAGCTGCGGG鼻病毒C型SEQIDNO.8CoxB-rtpCACCCAAAGTASTCGGTTCC柯萨奇病毒B型上述8条反转录引物在一次检测中共同使用,腺病毒为DNA病毒,无需反转录。用于扩增引物和通用引物:上述10对引物对在一次检测中共同使用,序列编号依次为SEQIDNO.9~28。用于检测的分子信标探针:上述7条分子信标探针在一次检测中共同使用,序列编号依次为SEQIDNO.29~35。实施例2:样本核酸提取(1)临床标本采集、保存及运输:适用于咽拭子标本采集。采集时以清晨为宜。用清水漱口,由检查者用压舌板辅助,将咽拭子越过舌根,到达咽峡部的病变处,反复涂抹数次,取出时避免接触舌及口腔粘膜等处,将取样后的棉拭子放入约1mL的生理盐水中充分洗涤,然后贴壁挤干,丢弃棉拭子。待测标本在4℃保存不应超过24小时,-20℃保存不应超过48小时;-80℃保存不超过三个月。标本运送应采用冰壶或加冰的泡沫箱。(2)病毒RNA的提取:按照RneasyMiniKit(Qiagencat74104)说明书进行病毒RNA的提取。实施例3:以提取的核酸为模板进行反转录(1)准备混合反转录引物工作液:将合成好的反转录引物稀释到100uM,各取10uL加入到1.5mL离心管中,补水至500uL,振荡混匀,即为反转录引物工作液。(2)样本病毒RNA反转录:按照RevertAidRTReverseTranscriptionKit(Themo,K1691)对样本中的总RNA进行反转录,反应体系10uL,反转录引物工作液0.5ul,其他成分按照说明书添加,最后补足水至10uL。反应程序:42℃1h;85℃5min;4℃保存。实施例4:Tem-PCR扩增(1)准备Tem-PCR反应工作液:将合成好的通用引物、加尾特异性引物和分子信标探针分别配制成100uM的储存液,分别将上述储存液添加至同一离心管中,其中通用引物终浓度为0.1uM,加尾特异性上游引物终浓度0.02uM、加尾特异性下游引物终浓度0.2uM、分子信标探针终浓度0.2uM;多重Tem-PCR扩增体系使用QiagenMultiplexPCRKit(Qiagen,206143)进行配制;样本反转录产物2uL;补ddH2O至50uL。(2)PCR扩增:使用Roche480荧光定量PCR检测仪,按照下述反应程序进行PCR扩增和熔解曲线分析。热启动:95℃15min;富集阶段:94℃30s,52℃1min,72℃1min(15cycles);加尾扩增:94℃15s,70℃1.5min(6cycles);PCR扩增:94℃15s,52℃15s,72℃15s(55cycles);延伸阶段:72℃3min;熔解分析:95℃1min,37℃5min,80℃—30℃0.1℃/s。实施例5:结果分析根据熔解曲线峰图,确定样本中是否存在待检的7种病毒,根据荧光通道的类型和熔解曲线Tm值,来判断感染的病毒类型,各型病毒对应的熔解峰Tm值如下:实施例6:检测方法特异性分析采用本发明方法对副流感病毒1型、副流感病毒2型、副流感病毒3型、冠状病毒229E型、冠状病毒OC43型、冠状病毒NL63型、冠状病毒HKU1型的T克隆质粒进行Tem-PCR扩增和熔解曲线分析,熔解曲线峰型如图8所示。从图可以看出,本发明检测方法只能特异性检测出阳性对照的熔解峰,而其他7种病毒的T克隆质粒均未有特异性熔解峰出现,表明本发明特异性好。实施例7:检测方法的灵敏度分析将含有各病毒目的片段的T克隆质粒按照10倍梯度稀释,使用Tem-PCR扩增和熔解曲线分析其检测的最低限。熔解峰图以为乙型流感为例,结果见9图。本发明建立的方法的检测下限是10copies/uL,具有较好的灵敏度。实施例8:检测方法的临床样本分析采用本发明方法对有发热症状人群的20份咽拭子临床样本进行检测,检测结果见下表:以上内容是结合具体的实施方式对本发明作进一步的详细说明,本发明也不限于上述举例。本领域技术人员在本发明的实质范围内,作出的变化、改型、添加或替换,也应属于本发明的保护范围。最后所应当说明的是,以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,尽管参照较佳实施例对本发明做了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的实质和范围。SEQUENCELISTING<110>杭州迪安医学检验中心有限公司<120>一种基于熔解曲线法单管同时检测多种呼吸道病毒的引物及其应用<130>SG20161201001<160>35<170>PatentInversion3.5<210>1<211>18<212>DNA<213>ArtificialSequence<220><223>IFA-rtp<400>1gggcacggtgagcgtgaa18<210>2<211>21<212>DNA<213>ArtificialSequence<220><223>IFB-rtp<400>2gttttcrtatcctckgagaag21<210>3<211>19<212>DNA<213>ArtificialSequence<220><223>RSVA-rtp<400>3ggtatgttggggttgtgtt19<210>4<211>23<212>DNA<213>ArtificialSequence<220><223>RSVB-rtp<400>4tttcagtgtggytttytattgtt23<210>5<211>20<212>DNA<213>ArtificialSequence<220><223>RVA-rtp<400>5ttgtgtgcactggctgcagg20<210>6<211>20<212>DNA<213>ArtificialSequence<220><223>RVB-rtp<400>6ttatgctgcaaggctctggg20<210>7<211>18<212>DNA<213>ArtificialSequence<220><223>RVC-rtp<400>7gtgtgcgatagctgcggg18<210>8<211>20<212>DNA<213>ArtificialSequence<220><223>CoxB-rtp<400>8cacccaaagtastcggttcc20<210>9<211>43<212>DNA<213>ArtificialSequence<220><223>IFA-Fi<400>9gcaagcctcagcgaccgaatacaccgatcttgaggctctcatg43<210>10<211>38<212>DNA<213>ArtificialSequence<220><223>IFA-Ri<400>10acgaaccaccagcagaagatgggcacggtgagcgtgaa38<210>11<211>41<212>DNA<213>ArtificialSequence<220><223>IFB-Fi<400>11gcaagcctcagcgaccgaatagcaaragyytcaatactcca41<210>12<211>41<212>DNA<213>ArtificialSequence<220><223>IFB-Ri<400>12acgaaccaccagcagaagatgttttcrtatcctckgagaag41<210>13<211>47<212>DNA<213>ArtificialSequence<220><223>RSVA-Fi<400>13gcaagcctcagcgaccgaataataattgcagccatcatattcatagc47<210>14<211>39<212>DNA<213>ArtificialSequence<220><223>RSVA-Ri<400>14acgaaccaccagcagaagatggtatgttggggttgtgtt39<210>15<211>48<212>DNA<213>ArtificialSequence<220><223>RSVB-Fi<400>15gcaagcctcagcgaccgaataattggcaatgataatctcaacctctct48<210>16<211>43<212>DNA<213>ArtificialSequence<220><223>RSVB-Ri<400>16acgaaccaccagcagaagattttcagtgtggytttytattgtt43<210>17<211>41<212>DNA<213>ArtificialSequence<220><223>RVA-Fi<400>17gcaagcctcagcgaccgaatatgtgaagagccccgtgtgct41<210>18<211>40<212>DNA<213>ArtificialSequence<220><223>RVA-Ri<400>18acgaaccaccagcagaagatttgtgtgcactggctgcagg40<210>19<211>42<212>DNA<213>ArtificialSequence<220><223>RVB-Fi<400>19gcaagcctcagcgaccgaatagtgcgaagacttgcatgtgct42<210>20<211>40<212>DNA<213>ArtificialSequence<220><223>RVB-Ri<400>20acgaaccaccagcagaagatttatgctgcaaggctctggg40<210>21<211>43<212>DNA<213>ArtificialSequence<220><223>RVC-Fi<400>21gcaagcctcagcgaccgaataggtgtgaagagcctagtgtgct43<210>22<211>38<212>DNA<213>ArtificialSequence<220><223>RVC-Ri<400>22acgaaccaccagcagaagatgtgtgcgatagctgcggg38<210>23<211>47<212>DNA<213>ArtificialSequence<220><223>CoxB-Fi<400>23gcaagcctcagcgaccgaatagacatggtgygaagagyctattgagc47<210>24<211>40<212>DNA<213>ArtificialSequence<220><223>CoxB-Ri<400>24acgaaccaccagcagaagatcacccaaagtastcggttcc40<210>25<211>39<212>DNA<213>ArtificialSequence<220><223>Adv-Fi<400>25gcaagcctcagcgaccgaatagccggacaggatgcttcg39<210>26<211>40<212>DNA<213>ArtificialSequence<220><223>AdV-Ri<400>26acgaaccaccagcagaagatgccactgtggggtttctaaa40<210>27<211>21<212>DNA<213>ArtificialSequence<220><223>Fs<400>27gcaagcctcagcgaccgaata21<210>28<211>20<212>DNA<213>ArtificialSequence<220><223>Rs<400>28acgaaccaccagcagaagat20<210>29<211>41<212>DNA<213>ArtificialSequence<220><223>IFA-mb<400>29cgccgtaaacacaagtccaataatgtcacctctgatcggcg41<210>30<211>42<212>DNA<213>ArtificialSequence<220><223>IFB-mb<400>30ggccctgcttaccaataatccacgacagaacaaatatgggcc42<210>31<211>40<212>DNA<213>ArtificialSequence<220><223>RSVA-mb<400>31ctcggaatctcacactaagtactccaatcatactaccgag40<210>32<211>41<212>DNA<213>ArtificialSequence<220><223>RSVB-mb<400>32ccgccttcatcatctctcccaatcacaaagtttcacggcgg41<210>33<211>34<212>DNA<213>ArtificialSequence<220><223>RV-mb<400>33cccgccctccggcccctgaatgaagctaagcggg34<210>34<211>42<212>DNA<213>ArtificialSequence<220><223>CoxB-mb<400>34caccggcttgaatgctgctaatcctaactgtggaacccggtg42<210>35<211>40<212>DNA<213>ArtificialSequence<220><223>AdV-mb<400>35cccgccatttctcccatgcaacagacacctagttcgcggg40当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1