一种质粒载体及利用其建立植物群体的方法与流程
本申请涉及一种质粒载体及利用其建立植物群体的方法。
背景技术:
crispr/cas9基因组定点编辑技术已成为当前的植物基因功能与应用研究的重要手段。其可以实现在生物体内基因组特定位点人为地对遗传物质进行改造的基因打靶技术,其引起的遗传信息改变在世代间能够进行稳定传递和功能呈现。基因组定点编辑的原理在于利用人工核酸酶在生物基因组靶位点处进行切割,产生dna双链断裂(dsb),由此激活细胞的dsb修复机制而达到目的。该技术一般尽可实现一个位点的突变,或少数几个位点突变的同时筛选。
虽然利用crispr/cas9技术已经在植物上进行了大规模突变体群体的创制工作。然而,由于部分质粒载体缺乏负筛选标记(比如自杀基因ccdb)导致载体库构建过程中阳性率不高;而含有负筛选标记ccdb的质粒载体则仅通过t4dna连接酶进行载体库构建,这一过程虽成本低效率高但操作十分繁琐。此外,已有的crispr/cas9建库用质粒载体不含报告基因进行追踪,导致突变体库制备过程中无法直接剔除假阳性材料,使得后期面临比较繁琐、规模庞大的制种过程。因此,无法有效地利用其进行大规模的突变体群体构建和筛选。
另外,以植物中的水稻(oryzasatival.)为例:作为第一个完成基因组测序的作物,水稻研究已经进入功能基因组学时代。挖掘重要经济性状相关功能基因、阐述其作用机制并在生产上加以利用是当下水稻研究的关键。通过分离水稻自然诱发突变体、或通过γ射线、快中子辐射等物理诱变手段、mnu、ems处理等化学诱变手段、t-dna插入、talen、crispr定点编辑技术等生物技术手段人工创制、筛选水稻突变体,目前科研人员结合图位克隆等技术已经分离并鉴定了moc1、ipa1、dsm2、dlt、ltn1等众多控制水稻生长、发育、抗逆、营养吸收相关过程的关键基因,并解析了其中的分子机制。然而,相对于水稻全基因组所携带的数万个功能基因,前期成果仍是杯水车薪。如何全面解析、注释水稻中所有基因的功能,以便最终能因地制宜地进行水稻改良,更好地服务我国农业生产,这是水稻功能基因组研究的重点。这其中,建立水稻突变体群体资源是重中之重。目前,美国、中国、日本等国家多家科研机构均建立了庞大的水稻ems诱导突变体群体,辐射诱变突变体群体、t-dna或转座子(ac/ds系统、tos17系统)插入突变体群体。然而,理化诱变和插入突变构建的突变体群体都存在操作复杂、突变率低、筛选分离耗时长和成本高等问题。如对于理化诱变水稻突变体群体,后期涉及繁琐的杂交、基因图位克隆等过程;对于插入突变体群体,其靶基因较易克隆,但其全基因组编码基因的覆盖率不高。尤其是它们均涉及后期规模庞大的制种过程,耗费大量的人力和物力。
因此,如果能够开发出一种可通过一步克隆方法高效率、高通量地构建质粒库、创制水稻突变群体的技术,通过荧光观察等方法直接剔除掉假阳性的转化材料以免除后续突变体群体材料制种过程中的繁琐工作,做到省事省力、耗资少,那么将大大加快水稻后基因组学时代的基因功能研究、新种质资源创制及品种改良步伐。
技术实现要素:
为了克服现有技术的缺陷,本申请之一提供了一种质粒载体,其包括包含表达cas9蛋白的基因表达盒、第一启动子、包含自杀基因序列的核苷酸序列、grnascaffold元件和终止信号,以及位于所述包含自杀基因序列的核苷酸序列5’端的至少一个第一酶切位点和位于所述包含自杀基因序列的核苷酸序列3’端的至少一个第二酶切位点;并且在所述质粒载体的其他位置中不存在所述第一酶切位点和所述第二酶切位点;其中,所述第一启动子位于所述终止信号的上游,所述包含自杀基因序列的核苷酸序列和所述grnascaffold元件均位于所述第一启动子和所述终止信号之间。
在一个具体实施方式中,所述第一酶切位点和所述第二酶切位点可以相同,也可以不相同。设置所述第一酶切位点和所述第二酶切位点目的在于在使用所述质粒载体时,切除所述包含自杀基因序列的核苷酸序列,同时将所述质粒载体线性化。对于未能成功切除包含自杀基因序列的核苷酸序列的质粒载体,在转化过程中,该自杀基因发挥其特性而使得质粒载体自杀,从而达到提高阳性率的目的。
在一个具体实施方式中,所述质粒载体还包括标记基因表达盒。
在一个具体实施方式中,当含有多个标记基因时,这些标记基因可以独立地使用各自的表达盒,也可以共用一个表达盒,这可以根据本领域的常规技术进行合适的选择。
在一个具体实施方式中,所述标记基因表达盒包括选择基因表达盒和报告基因表达盒。
在一个具体实施方式中,所述选择基因表达盒包括在植物中使用的选择基因表达盒。
在一个具体实施方式中,所述选择基因表达盒为潮霉素抗性基因表达盒或g418抗性基因表达盒。
在一个具体实施方式中,本领域的技术人员可以容易地获得上述抗性基因编码的氨基酸序列或者抗性基因的核苷酸序列,例如,例如,潮霉素抗性基因的核苷酸genebank登陆号为ky420085.1,其编码的氨基酸序列genebank登录号为ask07515.1。
在一个具体实施方式中,所述报告基因选自β半乳糖苷酶基因、荧光素酶基因、荧光蛋白基因和种皮色泽基因中的至少一种。
在一个具体实施方式中,所述报告基因选自荧光蛋白基因中的至少一种。在一个具体实施方式中,荧光蛋白基因选自绿色荧光蛋白基因、红色荧光蛋白基因、青色荧光蛋白基因、蓝色荧光蛋白基因和黄色蛋白基因中的至少一种。
在一个具体实施方式中,绿色荧光蛋白包括各种突变后仍能表现出绿色荧光或者绿色荧光增强的蛋白,例如增强型绿色荧光蛋白。红色荧光蛋白包括各种突变后仍能表现出红色荧光或者红色荧光增强的蛋白,例如增强型红色荧光蛋白。青色荧光蛋白包括各种突变后仍能表现出青色荧光或者青色荧光增强的蛋白,例如增强型青色荧光蛋白。蓝色荧光蛋白包括各种突变后仍能表现出蓝色荧光或者蓝色荧光增强的蛋白,例如增强型蓝色荧光蛋白。黄色荧光蛋白包括各种突变后仍能表现出黄色荧光或者黄色荧光增强的蛋白,例如增强型黄色荧光蛋白。
在一个具体实施方式中,所述报告基因表达盒选自绿色荧光蛋白表达盒。
在一个具体实施方式中,本领域的技术人员可以容易地获得上述报告基因编码的氨基酸序列或报告基因的序列(例如荧光蛋白的氨基酸序列或者荧光蛋白基因的核苷酸序列),例如,可以从genebank中获得。例如绿色荧光蛋白的核苷酸genebank登陆号为ky464890.1,其编码的氨基酸序列genebank登录号为aqt31663.1。
在一个具体实施方式中,所述第一启动子的核苷酸序列选自对待突变植物物种特异的核苷酸序列。
在一个具体实施方式中,所述第一启动子的核苷酸序列选自对禾本科植物特异的核苷酸序列。
在一个具体实施方式中,所述禾本科植物选自水稻、小麦、玉米和高粱中的至少一种。
在一个具体实施方式中,所述第一启动子为rna聚合酶iii型启动子。
在一个具体实施方式中,所述第一启动子的核苷酸序列为如seqidno.1所示的核苷酸序列。
在一个具体实施方式中,所述终止信号的核苷酸序列为如seqidno.2所示的核苷酸序列。
在一个具体实施方式中,所述grnascaffold元件的核苷酸序列如seqidno.3所示。
在一个具体实施方式中,所述包含表达cas9蛋白的基因表达盒包括从5’端到3’端的第二启动子、编码cas9蛋白的基因和第一终止子。
在一个具体实施方式中,所述cas9蛋白可以是本领域常规的cas9蛋白,例如其氨基酸序列可以为如seqidno.4所示的氨基酸序列。
在一个具体实施方式中,所述编码cas9蛋白的基因的核苷酸序列为如seqidno.5所示的核苷酸序列。
在一个具体实施方式中,所述第二启动子为rna聚合酶ii型启动子。
在一个具体实施方式中,所述第二启动子的核苷酸序列如seqidno.6所示。
在一个具体实施方式中,所述第一终止子的核苷酸序列为genebank登陆号为fj362600.1中的第8至第260核苷酸序列。
在一个具体实施方式中,所述选择基因表达盒包括从5’端到3’端的第三启动子、选择基因和第二终止子。
在一个具体实施方式中,所述第三启动子为rna聚合酶ii型启动子。
在一个具体实施方式中,所述第三启动子的核苷酸序列为genebank登陆号为fj362600.1中的第10382至第11162核苷酸序列。
在一个具体实施方式中,所述第二终止子的核苷酸序列为genebank登陆号为fj362600.1中的第8至第260核苷酸序列。
在一个具体实施方式中,所述报告基因表达盒包括从5’端到3’端的第四启动子、报告基因和第三终止子。
在一个具体实施方式中,所述第四启动子为rna聚合酶ii型启动子。
在一个具体实施方式中,所述第四启动子的核苷酸序列为genebank登陆号为fj362600.1中的第10382至第11162核苷酸序列。
在一个具体实施方式中,所述第三终止子的核苷酸序列为genebank登陆号为fj362600.1中的第8至第260核苷酸序列。
在一个具体实施方式中,所述自杀基因为ccdb基因;优选包含自杀基因序列的核苷酸序列如seqidno.7所示。
在一个具体实施方式中,所述选择基因表达盒还包括在细菌中使用的第二选择基因表达盒。
在一个具体实施方式中,所述第二选择基因表达盒选自卡那霉素抗性基因表达盒、青霉素抗性基因表达盒、四环素抗性基因表达盒、链霉素抗性基因表达盒中的至少一种。
在一个具体实施方式中,本领域的技术人员可以容易地获得上述抗性基因编码的氨基酸序列或者抗性基因的核苷酸序列,例如,卡那霉素抗性基因序列为genebank登陆号为kx400856.1中的第9156至第9950核苷酸序列,其编码的氨基酸序列genebank登录号为asn63838.1。
在一个具体实施方式中,所述第二选择基因表达盒包括从5’端到3’端的第五启动子、第二选择基因和第四终止子。
在一个具体实施方式中,优选所述第五启动子为能够在细菌使用的启动子。
在一个具体实施方式中,优选所述第四终止子的核苷酸序列为genebank登陆号为fj362600.1中的第8至第260核苷酸序列。
在一个具体实施方式中,所述质粒载体还包括复制起点,所述复制起点可以在细菌中使用。
在一个具体实施方式中,所述复制起点的核苷酸序列的genebank登陆号为ky420084.1中的第4066至第5066核苷酸序列。
本申请之二提供了一种建立植物突变群体的方法,其通过打靶至少一种植物内源基因获得,包括如下步骤:
步骤一:获得至少一种第i元件,所述第i元件包括依次从5’端到3’端的第i-1核苷酸序列、第i-2核苷酸序列和第i-3核苷酸序列;
其中,所述第i-1核苷酸序列与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第一酶切位点起,向5′端上游20bp以上的序列一致;所述第i-2核苷酸序列为靶核苷酸序列,所述靶核苷酸序列与所述植物内源基因上的部分核苷酸序列一致;所述第i-3核苷酸序列与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第二酶切位点起,向3′端下游20bp以上的序列一致;
步骤二:将所述第i元件通过同源重组分别交换到如本申请之一任意一项的所述质粒载体上,得到打靶载体,在所述打靶载体中,所述第i元件位于所述第一启动子和终止信号之间,并且所述第i-2核苷酸序列和所述grnascaffold能够转录融合;
步骤三:将所述打靶载体导入到植物愈伤组织或植物原生质体中,然后培养获得植物植株;
步骤四:从所述植物植株中筛选获得含有打靶植物内源基因的转基因植株;进一步地,所述含有打靶植物内源基因的转基因植株能够产生含有打靶植物内源基因的转基因植物种子。
在一个具体实施方式中,所述第i-1核苷酸序列与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第一酶切位点起,向5′端上游50-80bp的序列一致。
在一个具体实施方式中,所述第i-3核苷酸序列与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第二酶切位点起,向3′端下游50-80bp的序列一致。
在本申请中,确定所述靶核苷酸序列的原则同属于本领域的常规技术。例如:
在一个具体实施方式中,通过如下方式确定所述靶核苷酸序列:
1)确定植物基因组上需要打靶的至少一种植物内源基因;
2)在所述植物内源基因的编码序列或其反向互补序列中搜索能够被所述cas9蛋白所识别的pam模序,在保证位于所述pam模序5′端上游17至21个核苷酸序列为基因组中的特异性序列(即在基因组中具有很高的特异性,不能为在基因组中有较高序列一致性的那些序列)的情况下,确定所述pam模序5′端上游17至21个核苷酸序列为所述靶核苷酸序列。
在一个具体实施方式中,当所述cas9蛋白为seqidno.4所示的氨基酸序列时,所述识别pam模序为5′-ngg-3′、5′-nga-3′、5′-gagn-3′、5′-aagn-3′中的一种,所述靶核苷酸序列为所述pam模序5′端上游的17至21个核苷酸序列,淘汰含有连续五个t的核苷酸序列;其中,所述n为a、g、c和t中的一种。
一个具体实施方式中,通过如下方式获得至少一种所述第i元件:
i)获得至少一种包括从5’端到3’端依次的ii-1核苷酸序列、ii-2核苷酸序列、ii-3核苷酸序列的寡核苷酸序列;其中,所述ii-1核苷酸序列与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第一酶切位点起,向5′端上游20bp以上的序列一致;所述第ii-2核苷酸序列包括靶核苷酸序列,所述靶核苷酸序列与所述植物内源基因上的部分核苷酸序列一致;所述第ii-3核苷酸序列与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第二酶切位点起,向3′端下游20bp以上的序列一致;
ii)获得上游引物和下游引物;其中所述上游引物与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第一酶切位点起,向5′端上游20bp以上的序列一致;所述下游引物与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第二酶切位点起,向3′端下游20bp以上的序列反向互补;
iii)以步骤i)中的所述寡核苷酸序列为模板,以步骤ii)中的所述上游引物和所述下游引物为引物对进行pcr扩增,从而得到所述第i元件。
在一个具体实施方式中,所述ii-1核苷酸序列与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第一酶切位点起,向5′端上游20-35bp的序列一致。
在一个具体实施方式中,所述第ii-3核苷酸序列与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第二酶切位点起,向3′端下游20-35bp的序列一致。
在一个具体实施方式中,所述上游引物与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第一酶切位点起,向5′端上游50-80bp的序列一致。
在一个具体实施方式中,所述下游引物与从被所述第一酶切位点和所述第二酶切位点线性化后的所述质粒载体的所述第二酶切位点起,向3′端下游50-80bp的序列反向互补。
一个具体实施方式中,当步骤i)中的所述寡核苷酸序列为多种时,在步骤iii)中则以该多种所述寡核苷酸序列的混合物为模板进行pcr扩增,从而得到混合的多种所述第i元件。
一个具体实施方式中,当第i元件为多种时,在多种第i元件中的所述第i-2核苷酸序列两两不同。
一个具体实施方式中,当第i元件为多种时,在多种第i元件中的所述第i-1核苷酸序列可以相同,也可以不同,但是为了操作简便起见,优选所述第i-1核苷酸序列相同。
一个具体实施方式中,当第i元件为多种时,在多种第i元件中的所述第i-3核苷酸序列可以相同,也可以不同,但是为了操作简便起见,优选所述第i-3核苷酸序列相同。
在一个具体实施方式中,所述第i元件与所述质粒载体通过常规分子生物学操作交换至cas9载体中,优选吉布森组装(gibsonassembly)。
在一个具体实施方式中,所述靶核苷酸序列可以为多种靶核苷酸序列,或者可以说,第i元件可以为多种第i元件。如此,将通过例如pcr获得的多种第i元件与本申请之一中任意一项的所述质粒载体混合,通过吉布森组装,将所述多种第i元件与所述质粒载体连接在一起,得到打靶载体库。当将该打靶载体通过常规遗传操作,进行小规模或大规模植物转化,即可创建植物的突变群体。
在本申请中,核苷酸序列以靠近5’端的部分为上游,以靠近3’端的部分为下游,其中的上游和下游均为在核苷酸序列中的相对位置,位于上游的核苷酸序列更靠近序列的5’端。本申请的有益效果在于:
利用本申请提供的质粒载体大大提高了获得突变群体的的阳性率,最高可达100%,降低了植物突变群体中的假阳性植株概率;同时,质粒载体上携带有绿色荧光蛋白基因表达盒,借助荧光蛋白的可视化,可极其方便地对crispr突变体群体中的单株对其后代进行追踪以及后代植株中的t-dna分离情况等,大大降低了田间工作量。
附图说明
图1显示了实施例1构建载体phzlib2的流程示意图。
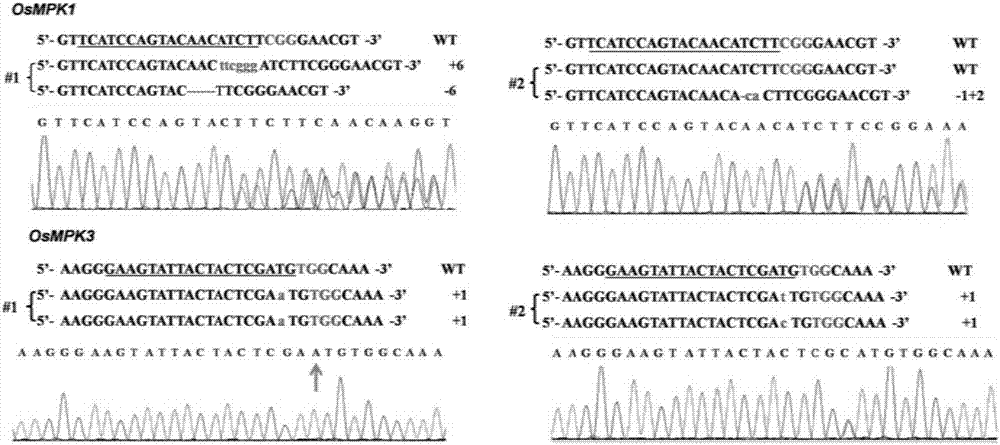
图2显示了利用phzlib2构建的osmpk突变体群体中osmpk1和osmpk3突变体植株典型突变类型。
图3显示了激光共聚焦显微镜观察库转质粒phzlib2-osmpks转化水稻后水稻抗性愈伤中gfp的表达结果图。
图4体视荧光显微镜观察库转质粒phzlib2-osmpks转化水稻后的t0代转基因植株根部的gfp表达结果图。
具体实施方式
下面结合实施例和附图对本发明做以下详细说明。但这些实施例仅是范例性的,并不对本发明的范围构成任何限制。本领域技术人员应该理解的是,在不偏离本发明的精神和范围下可以对本发明技术方案的细节和形式进行修改和替换,但这些修改和替换均落入本发明的保护范围内。
实施例1:重组质粒的构建
构建该载体的技术路线如下:
1、pubi-ccdb-cas9重组质粒构建
对本实验室自有双元载体ph3进行hindiii酶切,回收8.8kb载体骨架,利用t4dnaligase(takara)自连后,命名为ph4。ph4中包括的元件有:camv35s启动子,潮霉素基因,nos终止子,pvs1repa,pvs1复制起点。利用bsai和hindiii对ph4进行双酶切,回收5.5kb大片段,其中包括的主要元件有:camv35s启动子,潮霉素基因,nos终止子。同时以ph4为模板,以h3-f(seqidno.8,引入了限制性内切酶bsai酶切位点)和h3-r(seqidno.9)为引物,利用高保真酶i-5tm2×high-fidelitymastermix(购自克劳宁(北京)生物科技有限公司)从ph4质粒中扩增出3.7kb的小片段(包括的元件为:pvs1repa,pvs1复制起点),与上述5.5kb大片段连接,命名为ph5。ph5进行hindiii酶切,回收线性化载体骨架,包括的元件有:camv35s启动子,潮霉素基因,nos终止子,pvs1repa,pvs1复制起点。
对本实验室自有载体pgemt:attr1r2进行bsmbi酶切,跑胶回收1kb片段(包括attr1、ccdb基因序列、attr2),与上述线性化ph5载体骨架连接,测序鉴定attr1r2为正向插入(即与gfp表达方向一致),命名为ph5-ccdb,使ccdb基因的表达方向与载体上启动子的表达方向一致。将实验室自有载体pgemt:ubip和ph5-ccdb分别用hindiii和saci双酶切,连接后,质粒命名为ph5-ccdb-ubip。将ph5-ccdb-ubip质粒和带有人工合成cas9片段(seqidno.5,其氨基酸序列如seqidno.4所示)的质粒puc57:cas9分别进行bamhi和spei双酶切,回收12kb载体骨架和4.2kb的cas9基因片段,连接,质粒命名为pubi-ccdb-cas9。质粒pubi-ccdb-cas9的构成如下:camv35s启动子,潮霉素基因,nos终止子,pvs1repa,pvs1复制起点,attr1,ccdb基因序列,attr2,ubip启动子,cas9基因,nos终止子。
2、pentr4-grna重组质粒构建
人工合成792bp的gfp基因片段(生工生物工程(上海)股份有限公司),并将其克隆到puc57上,命名为puc57:gfp。利用bamhi和spei对质粒puc57:gfp和载体puc19-3flag进行双酶切,分别回收792bpgfp基因片段和3.9kb的puc19-3flag载体骨架(包括的主要元件有:camv35s启动子,3flag基因,nos终止子等),利用t4dna连接酶(takara)将gfp基因片段连接至puc19-3flag载体,命名为puc19:gfp,经菌落pcr和酶切验证后备用。以35s-f3(seqidno.10,引入了限制性内切酶xhoi酶切位点)和nos-r(seqidno.11,引入了限制性内切酶xhoi酶切位点)为引物,利用i-5tm2×high-fidelitymastermix以载体puc19:gfp为模板进行pcr扩增,获得大小为1.8kb的目的片段(即gfp表达盒:35s-gfp-nos,简写为gfp–cassette,其中35s代表35s启动子,gfp代表gfp基因,nos代表nos终止子),xhoi酶切,回收。同时对实验室自有载体pentr4-grna进行xhoi酶切,回收2.75kb线性化片段(元件为u6启动子,grnascaffold),然后利用fastapthermosensitivealkalinephosphatase(thermoscientific)对线性化pentr4-grna进行去磷酸化处理防止自连,再利用t4dnaligase(takara)将上述1.8kb的gfp片段连接至上述进行了去磷酸化处理的线性化的pentr4-grna片段上,命名为pentr4-grna-gfp,经菌落pcr和酶切验证后测序备用。其中,质粒pentr4-grna-gfp的构成如下:u6启动子,grnascaffold,camv35s启动子,gfp基因,nos终止子。
3、phzlib2载体的构建
用nhei酶切pentr4-grna-gfp,回收约4kb左右的片段(元件为u6启动子,grnascaffold,camv35s启动子,gfp基因,nos终止子),通过gateway技术将pentr4-grna3-gfp中的grna表达盒(包括第一启动子u6启动子(seqidno.1)+终止信号(seqidno.2),此时,该grna表达盒内还不包含grna)和gfp表达盒交换至pubi-ccdb-cas9中,命名为pubi-grna-gfp-cas9(其中的元件包括camv35s启动子,潮霉素基因,nos终止子,pvs1repa,pvs1复制起点,attb1,u6启动子,grnascaffold,终止信号(seqidno.2),camv35s启动子,gfp基因,nos终止子,attb2,ubip启动子,cas9基因,nos终止子),经酶切验证正确后备用。以ccdb-f1(seqidno.12)和ccdb-r1(seqidno.13)为引物,利用i-5tm2×high-fidelitymastermix以载体pgemt-attr1r2为模板进行pcr扩增,获得大小为800bp左右的目的片段(含ccdb基因);用bsai酶切质粒pubi-grna-gfp-cas9并回收10kb左右的线性化条带,再利用trelieftmsosoocloningkit(tsingke)将ccdb基因插入pubi-grna3-gfp-rcas9中,获得最终载体phzlib2,经菌落pcr和酶切验证后备用。其中,质粒phzlib2的构成如下:camv35s启动子(genebank登陆号为fj362600.1,第10382至第11162核苷酸序列),潮霉素基因(genebank登陆号为ky420085.1),nos终止子(genebank登陆号为fj362600.1,第8至第260核苷酸序列),pvs1repa(genebank登陆号为ky420084.1,第5755至第6435核苷酸序列),pvs1复制起点(genebank登陆号为ky420084.1,第4066至第5066核苷酸序列),attb1(seqidno.14),u6启动子(seqidno.1),含有ccdb基因的核酸(seqidno.7),grnascaffold(seqidno.3),终止信号(seqidno.2),camv35s启动子(genebank登陆号为fj362600.1,第10382至第11162核苷酸序列),gfp基因(genebank登陆号为ky464890.1),nos终止子(genebank登陆号为fj362600.1,第8至第260核苷酸序列),attb2(seqidno.15),ubip启动子(seqidno.6),cas9基因(seqidno.5),nos终止子(genebank登陆号为fj362600.1,第8至第260核苷酸序列),kan抗性基因表达盒(genebank登陆号为kx400856.1,其中,卡那抗性基因为位于第9156至第9950核苷酸序列)。
实施例2:利用phzlib2系统构建水稻内源mpk基因家族的质粒库及水稻转化
1、构建phzlib2-osmpks质粒库
合成寡核苷酸序列osmpk1-oligo-1(seqidno.16:cccgcgcgctgtcgcttgtgtgtcatccagtacaacatcttgttttagagctagaaatagcaagttaaaataag,下划线为osmpk1的靶核苷酸序列),osmpk2-oligo-1(seqidno.17:cccgcgcgctgtcgcttgtgtgatggccatcacggtggcatgttttagagctagaaatagcaagttaaaataag,下划线为osmpk2的靶核苷酸序列),osmpk3-oligo-1(seqidno.18:cccgcgcgctgtcgcttgtgtgaagtattactactcgatggttttagagctagaaatagcaagttaaaataag,下划线为osmpk3的靶核苷酸序列),osmpk4-oligo-1(seqidno.19:cccgcgcgctgtcgcttgtgtgctaatggcatgggaaaccagttttagagctagaaatagcaagttaaaataag,下划线为osmpk4的靶核苷酸序列),osmpk5-oligo-1(seqidno.20:cccgcgcgctgtcgcttgtgtgtcaggccgacgatgacgcagttttagagctagaaatagcaagttaaaataag,下划线为osmpk5的靶核苷酸序列),osmpk6-oligo-1(seqidno.21:cccgcgcgctgtcgcttgtgtgtgtacgggaacttcttcggttttagagctagaaatagcaagttaaaataag,下划线为osmpk6的靶核苷酸序列)和osmpk12-oligo-1(seqidno.22:cccgcgcgctgtcgcttgtgtgccaaccagtcgtccaacgcgttttagagctagaaatagcaagttaaaataag,下划线为osmpk12的靶核苷酸序列),以上述七条寡核苷酸等体积等浓度混合后作为osmpks-oligo为模板,array-f1(seqidno.23:cacattgcccagctaactcgaacgcgaccaacttataaacccgcgcgctgtcgcttgtgt)和array-r1(seqidno.24:actcggtgccactttttcaagttgataacggactagccttattttaacttgctatttctagctctaaaac)为引物,利用i-5tm2×high-fidelitymastermix对上述7条寡核苷酸混合扩增,回收pcr产物大约140bp。同时对载体phzlib2用bsai酶切,回收约10kb载体骨架(释放出ccdb基因片段)。再利用trelieftmsosoocloningkit(tsingke)将ccdb基因置换为mpk基因的grna,命名为phzlib2-osmpks。随机挑选10个菌落进行测序,正确率高达100%,并包含所有7种grna。
2、phzlib2-osmpks质粒库转化粳稻kitaake
1)水稻愈伤诱导:
将去壳的成熟的水稻种子用50%的商业化消毒液处理25分钟;无菌水清洗3-5次,然后将种子转移至无菌的培养皿中,吸出多余的水份;将种子放置于msd平板上(4.43g/lms粉;30g/l蔗糖;2ml/l2,4-d;8g/l植物凝胶;ph5.7),于光照培养室培养10天,诱导愈伤组织形成;去除种子的胚和芽,将愈伤组织转移至一个新的msd培养皿上,培养5天,直至能够用于农杆菌的转化。
2)农杆菌转化:
将phzlib2-osmpks质粒库通过电击法转入农杆菌菌株eha105中,构建含有phzlib2-osmpks质粒库的eha105菌株。活化该eha105菌株,在ty培养基中室温过夜培养12小时;离心收集农杆菌,用msd溶液重悬,使其od600=0.2待用。
3)水稻愈伤的农杆菌侵染:
将愈伤组织置于上述农杆菌悬浮液中30分钟;除去农杆菌悬浮液,将愈伤组织转移至无菌的吸水纸上除去多余的农杆菌菌液,再将愈伤组织转移至新的含有100μm乙酰丁香酮的msd培养基上,室温避光培养2-3天。
4)水稻抗性愈伤筛选:
将暗培养后的愈伤组织转移至msd培养基(100mg/l特美汀;50mg/l潮霉素b)上培养2周至2个月,直至愈伤组织的表面出现抗性愈伤;每2周换一次培养基。
5)抗性愈伤分化与生根
将抗性愈伤组织转移至再生培养基上(4.43g/lms粉;30g/l蔗糖;25g/l山梨醇;0.5mg/lnaa;3mg/lba;100mg/l特美汀;50mg/l潮霉素b;12g/l琼脂粉;ph=5.7),直至长成植株幼苗,每7-10天转移一次;转移幼苗至1/2ms培养基(2.21g/lms粉;15g/l蔗糖;8g/l植物凝胶;ph5.7)中生根。
实施例3:osmpks水稻突变体群体的检测
1)基因组dna的提取
剪取t0代植株叶片约0.1g,液氮速冻后利用研磨仪进行研磨;加入600μl2×ctabdna提取液(含1/1000的β-巯基乙醇),震荡混匀后于65℃裂解45min。加入500μl三氯甲烷剧烈摇晃形成乳浊液,14000rpm离心10min。离心后转移上清液于1.5ml离心管,加入等体积异丙醇,颠倒混匀后14000rpm离心10min,弃上清液,加入700μl70%乙醇洗涤白色沉淀,14000rpm离心5min,弃上清液后将离心管置于通风橱风干10min。加入30μlddh2o溶解dna。dna溶液置于-20℃保存备用。
2)突变位点的pcr扩增及测序检测
利用通用引物对u6p-f3(seqidno.25)和g4-r1(seqidno.26)对随机挑选的109株t0代植株幼苗的grna进行检测测序,结果显示其中osmpk1、osmpk4、osmpk5转基因植株各16株,osmpk2转基因植株17株,osmpk3和osmpk12转基因植株各15株,osmpk6转基因植株14株。再根据osmpks基因的靶位点设计特异性的pcr引物:osmpk1-f1(seqidno.27)和osmpk1-r1(seqidno.28);osmpk2-f1(seqidno.29)和osmpk2-r1(seqidno.30);osmpk3-f1(seqidno.31)和osmpk3-r1(seqidno.32);osmpk4-f1(seqidno.33)和osmpk4-r1(seqidno.34);osmpk5-f1(seqidno.35)和osmpk5-r1(seqidno.36);osmpk6-f1(seqidno.37)和osmpk6-r1(seqidno.38);osmpk12-f1(seqidno.39)和osmpk12-r1(seqidno.40);利用i-5tm2×high-fidelitymastermix,以上述引物为模板,分别pcr扩增得osmpk1、osmpk2、osmpk3、osmpk4、osmpk5、osmpk6、osmpk12基因的片段,大小依次为655bp,438bp,399bp,358bp,468bp,530bp,466bp。将pcr产物直接进行sanger测序。sanger测序结果显示:osmpk1、osmpk3和osmpk5突变体各13株,osmpk2和osmpk4突变体各14株,osmpk6突变体12株,osmpk12突变体15株,突变效率为81.25—100%,并且能产生多种类型的突变。例如osmpk1基因,共检测到13株突变体植株,#1号植株突变类型为+6/-6的杂合子,这说明二倍体水稻的osmpk1双等位基因都发生了突变,而且突变类型不同;#2号植株则含有一个野生型的基因,单基因突变为-1+2。又如osmpk3基因,共检测到13株突变体植株,#1号植株突变类型为+a,得到了双等位基因突变的纯合材料;#2号植株突变类型为+t/+c,也得到了双等位基因突变的杂合材料(图2)。
phzlib2-osmpks质粒库构建的t0代转基因植株的突变率高达80%以上,表明该系统具有很高的突变效率。
表2.利用库转质粒phzlib2-osmpks构建osmpks突变体群体的效率
实施例4:转基因抗性愈伤的绿色荧光蛋白gfp观察
分别挑取转基因抗性愈伤和野生型kitaake愈伤,在手持式uv灯下进行观察,同时将愈伤制成临时玻片在激光共聚焦显微镜下进行gfp观察。结果显示在gfp激发光条件下可以观察到细胞水稻愈伤组织显示为绿色(图3左),而在无gfp激发光时,细胞无绿色荧光蛋白表达(图3中),图3右为以上两者的叠加效果,从叠加的效果图可以明显观察到在转基因抗性愈伤中均能观察到明显的绿色荧光(图3右)。同时将水稻t0代植株的根部在体式荧光显微镜的紫外激发下进行观察,在转基因植株的根部均能观察到明显的绿色荧光(图4)。表明该系统中的gfp在水稻细胞中能够正常进行表达,并呈现出绿色荧光,可用于追踪后代分离群体的转基因植株。
序列表
<110>中国农业科学院植物保护研究所
<120>一种质粒载体及利用其建立植物群体的方法
<130>lha1760716
<160>40
<170>siposequencelisting1.0
<210>1
<211>245
<212>dna
<213>人工序列(non)
<400>1
ggatcatgaaccaacggcctggctgtatttggtggttgtgtagggagatggggagaagaa60
aagcccgattctcttcgctgtgatgggctggatgcatgcgggggagcgggaggcccaagt120
acgtgcacggtgagcggcccacagggcgagtgtgagcgcgagaggcgggaggaacagttt180
agtaccacattgcccagctaactcgaacgcgaccaacttataaacccgcgcgctgtcgct240
tgtgt245
<210>2
<211>7
<212>dna
<213>人工序列(non)
<400>2
ttttttt7
<210>3
<211>76
<212>dna
<213>人工序列(non)
<400>3
gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagt60
ggcaccgagtcggtgc76
<210>4
<211>1417
<212>prt
<213>人工序列(non)
<400>4
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalaasplyslystyrserileglyleu
354045
aspileglythrasnservalglytrpalavalilethraspglutyr
505560
lysvalproserlyslysphelysvalleuglyasnthrasparghis
65707580
serilelyslysasnleuileglyalaleuleupheaspserglyglu
859095
thralaglualathrargleulysargthralaargargargtyrthr
100105110
argarglysasnargilecystyrleuglngluilepheserasnglu
115120125
metalalysvalaspaspserphephehisargleuglugluserphe
130135140
leuvalglugluasplyslyshisgluarghisproilepheglyasn
145150155160
ilevalaspgluvalalatyrhisglulystyrprothriletyrhis
165170175
leuarglyslysleuvalaspserthrasplysalaaspleuargleu
180185190
iletyrleualaleualahismetilelyspheargglyhispheleu
195200205
ilegluglyaspleuasnproaspasnseraspvalasplysleuphe
210215220
ileglnleuvalglnthrtyrasnglnleupheglugluasnproile
225230235240
asnalaserglyvalaspalalysalaileleuseralaargleuser
245250255
lysserargargleugluasnleuilealaglnleuproglyglulys
260265270
lysasnglyleupheglyasnleuilealaleuserleuglyleuthr
275280285
proasnphelysserasnpheaspleualagluaspalalysleugln
290295300
leuserlysaspthrtyraspaspaspleuaspasnleuleualagln
305310315320
ileglyaspglntyralaaspleupheleualaalalysasnleuser
325330335
aspalaileleuleuseraspileleuargvalasnthrgluilethr
340345350
lysalaproleuseralasermetilelysargtyraspgluhishis
355360365
glnaspleuthrleuleulysalaleuvalargglnglnleuproglu
370375380
lystyrlysgluilephepheaspglnserlysasnglytyralagly
385390395400
tyrileaspglyglyalaserglnglugluphetyrlyspheilelys
405410415
proileleuglulysmetaspglythrglugluleuleuvallysleu
420425430
asnarggluaspleuleuarglysglnargthrpheaspasnglyser
435440445
ileprohisglnilehisleuglygluleuhisalaileleuargarg
450455460
glngluaspphetyrpropheleulysaspasnargglulysileglu
465470475480
lysileleuthrpheargileprotyrtyrvalglyproleualaarg
485490495
glyasnserargphealatrpmetthrarglysserglugluthrile
500505510
thrprotrpasnpheglugluvalvalasplysglyalaseralagln
515520525
serpheilegluargmetthrasnpheasplysasnleuproasnglu
530535540
lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr
545550555560
asngluleuthrlysvallystyrvalthrgluglymetarglyspro
565570575
alapheleuserglygluglnlyslysalailevalaspleuleuphe
580585590
lysthrasnarglysvalthrvallysglnleulysgluasptyrphe
595600605
lyslysileglucyspheaspservalgluileserglyvalgluasp
610615620
argpheasnalaserleuglythrtyrhisaspleuleulysileile
625630635640
lysasplysasppheleuaspasnglugluasngluaspileleuglu
645650655
aspilevalleuthrleuthrleuphegluaspargglumetileglu
660665670
gluargleulysthrtyralahisleupheaspasplysvalmetlys
675680685
glnleulysargargargtyrthrglytrpglyargleuserarglys
690695700
leuileasnglyileargasplysglnserglylysthrileleuasp
705710715720
pheleulysseraspglyphealaasnargasnphemetglnleuile
725730735
hisaspaspserleuthrphelysgluaspileglnlysalaglnval
740745750
serglyglnglyaspserleuhisgluhisilealaasnleualagly
755760765
serproalailelyslysglyileleuglnthrvallysvalvalasp
770775780
gluleuvallysvalmetglyarghislysprogluasnilevalile
785790795800
glumetalaarggluasnglnthrthrglnlysglyglnlysasnser
805810815
arggluargmetlysargileglugluglyilelysgluleuglyser
820825830
glnileleulysgluhisprovalgluasnthrglnleuglnasnglu
835840845
lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp
850855860
glngluleuaspileasnargleuserasptyraspvalasphisile
865870875880
valproglnserpheleulysaspaspserileaspasnlysvalleu
885890895
thrargserasplysasnargglylysseraspasnvalproserglu
900905910
gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala
915920925
lysleuilethrglnarglyspheaspasnleuthrlysalagluarg
930935940
glyglyleusergluleuasplysalaglypheilelysargglnleu
945950955960
valgluthrargglnilethrlyshisvalalaglnileleuaspser
965970975
argmetasnthrlystyraspgluasnasplysleuilearggluval
980985990
lysvalilethrleulysserlysleuvalseraspphearglysasp
99510001005
pheglnphetyrlysvalarggluileasnasntyrhishisalahis
101010151020
aspalatyrleuasnalavalvalglythralaleuilelyslystyr
1025103010351040
prolysleuglusergluphevaltyrglyasptyrlysvaltyrasp
104510501055
valarglysmetilealalyssergluglngluileglylysalathr
106010651070
alalystyrphephetyrserasnilemetasnphephelysthrglu
107510801085
ilethrleualaasnglygluilearglysargproleuilegluthr
109010951100
asnglygluthrglygluilevaltrpasplysglyargasppheala
1105111011151120
thrvalarglysvalleusermetproglnvalasnilevallyslys
112511301135
thrgluvalglnthrglyglypheserlysgluserileleuprolys
114011451150
argasnserasplysleuilealaarglyslysasptrpaspprolys
115511601165
lystyrglyglypheaspserprothrvalalatyrservalleuval
117011751180
valalalysvalglulysglylysserlyslysleulysservallys
1185119011951200
gluleuleuglyilethrilemetgluargserserpheglulysasn
120512101215
proileasppheleuglualalysglytyrlysgluvallyslysasp
122012251230
leuileilelysleuprolystyrserleuphegluleugluasngly
123512401245
arglysargmetleualaseralaglygluleuglnlysglyasnglu
125012551260
leualaleuproserlystyrvalasnpheleutyrleualaserhis
1265127012751280
tyrglulysleulysglyserprogluaspasngluglnlysglnleu
128512901295
phevalgluglnhislyshistyrleuaspgluileilegluglnile
130013051310
serglupheserlysargvalileleualaaspalaasnleuasplys
131513201325
valleuseralatyrasnlyshisargasplysproileargglugln
133013351340
alagluasnileilehisleuphethrleuthrasnleuglyalapro
1345135013551360
alaalaphelystyrpheaspthrthrileasparglysargtyrthr
136513701375
serthrlysgluvalleuaspalathrleuilehisglnserilethr
138013851390
glyleutyrgluthrargileaspleuserglnleuglyglyasparg
139514001405
prolyslyslysarglysvalglygly
14101415
<210>5
<211>4254
<212>dna
<213>人工序列(non)
<400>5
atggactataaggatcacgatggcgactacaaggatcatgacattgactataaggatgac60
gacgataagatggcacctaagaagaaaaggaaagtcggcattcatggcgttccggcagcc120
gacaaaaagtatagcatcggcctcgatattgggacaaactctgtgggctgggcggtaatt180
accgacgagtacaaggtgcctagtaagaaatttaaagtgctcggaaacactgacaggcac240
tctataaagaagaacctgatcggggcactgcttttcgactccggagagacggcggaggcg300
acgcgtctcaagcgtaccgcgcgccgcaggtacacaagaaggaagaataggatctgctac360
ttgcaggaaatcttcagtaacgagatggcgaaggtcgacgatagtttctttcatcggttg420
gaagaatcgttcctcgtagaggaggacaaaaagcacgagcgtcacccaatattcgggaat480
attgttgacgaggttgcctaccatgagaaatatcctacaatatatcacctccgtaagaag540
cttgtcgattcaactgataaggctgatctcagactcatctatcttgccctcgcacatatg600
attaagtttcgtggccacttcttgattgaaggcgacctcaacccggacaactcagatgtt660
gacaagctttttatacagctcgtccagacatataaccagctgtttgaagagaatcccatc720
aatgcgagtggggttgatgctaaggccattttgtccgccaggttgtccaaatctcgcaga780
ctggaaaacctgatcgcacagcttcccggtgaaaagaaaaacgggctcttcggcaatctc840
atcgcactgtccctcggcctcaccccaaacttcaagtctaacttcgacctggccgaggat900
gcgaagctccagctgtcaaaagatacatacgacgacgatttggacaatctgcttgcgcaa960
ataggcgaccagtatgcggacctgttcctggctgccaaaaatctgtcagatgcaatcctc1020
ctgtccgatatattgcgtgtgaacaccgaaatcacgaaggcaccgcttagcgcatccatg1080
atcaagagatacgacgagcaccatcaggacctcacactcctcaaggcgcttgttcgtcag1140
cagcttcccgagaaatataaggaaatttttttcgatcaaagcaagaatggatatgctggc1200
tatattgacggtggcgcttcgcaggaggagttctataaattcattaagccgattctggag1260
aagatggacggaacggaggagctcctcgtcaagcttaaccgggaagacctgttgcggaag1320
cagaggacttttgataacggctctattccgcaccaaatccatctgggtgagttgcacgca1380
atcttgagaagacaagaggatttctacccgttccttaaggataacagagagaagatagaa1440
aaaatactgaccttcaggataccatactatgtgggcccactggcgcgcggaaatagtcgt1500
ttcgcatggatgactagaaagtccgaagaaacgatcacgccatggaattttgaggaagtg1560
gtcgacaagggcgcctctgcccagagcttcatcgaaaggatgaccaattttgacaaaaat1620
ctgcctaacgaaaaggtgcttccgaagcacagcctgttgtatgaatacttcacagtttat1680
aacgagctcactaaggtcaagtacgtcacggagggcatgcgtaagcctgctttcctgtct1740
ggtgaacaaaaaaaggcgattgtggacctccttttcaagacgaaccgtaaagttactgtg1800
aagcaactgaaagaggattactttaagaaaattgagtgcttcgacagtgtggagatttcc1860
ggtgtcgaggaccggtttaacgccagcctgggtacgtatcatgacctgcttaaaattatc1920
aaggataaagatttcctggataatgaagagaacgaagatatactggaggacattgtgttg1980
actttgaccctcttcgaggacagagagatgattgaggaaagactgaagacctacgcacac2040
ctttttgatgacaaggtcatgaaacaactcaagcgccggcgctatactggctggggccgg2100
ctttctcgcaagctcatcaatgggattcgggataagcaatcaggcaagacaattttggac2160
ttcctcaaatccgacggattcgcaaataggaattttatgcagctgatacatgacgactct2220
ttgacattcaaagaagacatacagaaggctcaggtgtccggccaaggagattctttgcac2280
gagcatatcgctaacttggcaggtagccccgccataaaaaagggcattcttcaaacggta2340
aaagttgttgacgaactcgtgaaggttatgggccgtcataagccggaaaacattgttatt2400
gaaatggctagggaaaatcagacgacccagaagggacagaaaaatagcagggagcggatg2460
aagagaattgaagagggaattaaggagcttggatctcagattcttaaggagcaccctgtg2520
gagaacacccaacttcagaatgaaaagctctacctttactaccttcaaaacggccgggat2580
atgtacgtcgatcaggaacttgacattaaccggttgagcgattatgacgttgaccatatt2640
gtgccccaatctttccttaaagacgactctatcgacaataaagtgctgacgcgcagcgat2700
aaaaatcgcggtaagtcggataatgtcccgtcggaagaggtggttaaaaaaatgaagaac2760
tattggaggcaactcctgaatgccaagctgatcactcagaggaaattcgacaatctcacc2820
aaggcagaaaggggtggacttagcgagctcgacaaggccggttttatcaaaagacagctg2880
gtggagacacgccaaatcaccaaacacgttgcccagatcctggattcgaggatgaacacg2940
aagtatgacgagaacgacaagttgattagggaagtcaaggtcatcactttgaagtccaag3000
ctggtgagcgactttcgcaaagacttccagttttacaaagtcagggaaattaataactac3060
caccacgcccacgacgcctaccttaacgccgtggttggcacagcactcatcaagaaatac3120
cctaagctcgaatctgagttcgtctatggcgactataaggtctacgacgttagaaaaatg3180
atcgcgaaatctgagcaggaaataggcaaggcaactgccaagtacttcttctattccaat3240
atcatgaacttttttaagacggagattaccctggcgaatggtgagatccgcaagcgccct3300
ttgattgagacaaacggagaaacaggagagatcgtatgggacaaagggcgggactttgct3360
actgttaggaaggtgctctctatgccacaagttaacattgtcaaaaaaactgaagtgcag3420
acaggtgggtttagcaaggaatctatcctgccgaagaggaactctgacaagctgatcgcc3480
cgcaagaaagattgggatccgaaaaagtacggaggattcgactcccccacagttgcgtac3540
tccgtgcttgtcgtggccaaagtggagaagggcaagtctaagaagctcaagagcgtcaaa3600
gagttgttggggatcacgattatggagcggtcgtctttcgaaaagaatccgatagatttt3660
ctcgaggccaagggttataaagaagtcaagaaggatcttatcatcaagctccctaagtac3720
tccctctttgagcttgaaaacggacggaaaagaatgctggcttcagcgggtgaacttcag3780
aagggtaatgaactcgctctgccctcaaaatatgtgaatttcctttacctggcatcacac3840
tatgagaagcttaaggggtccccagaggacaacgagcagaagcaactgttcgttgaacaa3900
cacaagcactaccttgacgagattatcgagcaaatcagcgagtttagcaagcgcgttata3960
ctggcagacgcaaatcttgataaggtccttagcgcctacaacaagcatagagacaaaccc4020
atccgggagcaggccgagaacattattcatctcttcaccttgacgaatcttggggccccg4080
gccgcgttcaagtacttcgatactaccatagacagaaagcgctatacatcgacaaaggaa4140
gttcttgacgccacgctgatccaccaaagtataacaggcctctatgagacacgcatcgac4200
ctttcgcagttgggcggtgaccgccccaaaaagaagaggaaagttggcgggtga4254
<210>6
<211>1706
<212>dna
<213>人工序列(non)
<400>6
aaaaaattaccacatattttttttgtcacacttgtttgaagtgcagtttatctatcttta60
tacatatatttaaactttactctacgaataatataatctatagtactacaataatatcag120
tgttttagagaatcatataaatgaacagttagacatggtctaaaggacaattgagtattt180
tgacaacaggactctacagttttatctttttagtgtgcatgtgttctcctttttttttgc240
aaatagcttcacctatataatacttcatccattttattagtacatccatttagggtttag300
ggttaatggtttttatagactaatttttttagtacatctattttattctattttagcctc360
taaattaagaaaactaaaactctattttagtttttttatttaataatttagatataaaat420
agaataaaataaagtgactaaaaattaaacaaataccctttaagaaattaaaaaaactaa480
ggaaacatttttcttgtttcgagtagataatgccagcctgttaaacgccgtcgacgagtc540
taacggacaccaaccagcgaaccagcagcgtcgcgtcgggccaagcgaagcagacggcac600
ggcatctctgtcgctgcctctggacccctctcgagagttccgctccaccgttggacttgc660
tccgctgtcggcatccagaaattgcgtggcggagcggcagacgtgagccggcacggcagg720
cggcctcctcctcctctcacggcacggcagctacgggggattcctttcccaccgctcctt780
cgctttcccttcctcgcccgccgtaataaatagacaccccctccacaccctctttcccca840
acctcgtgttgttcggagcgcacacacacacaaccagatctcccccaaatccacccgtcg900
gcacctccgcttcaaggtacgccgctcgtcctccccccccccccctctctaccttctcta960
gatcggcgttccggtccatggttagggcccggtagttctacttctgttcatgtttgtgtt1020
agatccgtgtttgtgttagatccgtgctgctagcgttcgtacacggatgcgacctgtacg1080
tcagacacgttctgattgctaacttgccagtgtttctctttggggaatcctgggatggct1140
ctagccgttccgcagacgggatcgatttcatgattttttttgtttcgttgcatagggttt1200
ggtttgcccttttcctttatttcaatatatgccgtgcacttgtttgtcgggtcatctttt1260
catgctttttttttgtcttggttgtgatgatgtggtgtggttgggcggtcgttcattcgt1320
tctagatcggagtagaatactgtttcaaactacctggtgtatttattaattttggaactg1380
tatgtgtgtgtcatacatcttcatagttacgagtttaagatggatggaaatatcgatcta1440
ggataggtatacatgttgatgtgggttttactgatgcatatacatgatggcatatgcagc1500
atctattcatatgctctaaccttgagtacctatctattataataaacaagtatgttttat1560
aattattttgatcttgatatacttggatgatggcatatgcagcagctatatgtggatttt1620
tttagccctgccttcatacgctatttatttgcttggtactgtttcttttgtcgatgctca1680
ccctgttgtttggtgttacttctgca1706
<210>7
<211>569
<212>dna
<213>人工序列(non)
<400>7
tcaaaaagaggtatgctatgaagcagcgtattacagtgacagttgacagcgacagctatc60
agttgctcaaggcatatatgatgtcaatatctccggtctggtaagcacaaccatgcagaa120
tgaagcccgtcgtctgcgtgccgaacgctggaaagcggaaaatcaggaagggatggctga180
ggtcgcccggtttattgaaatgaacggctcttttgctgacgagaacaggggctggtgaaa240
tgcagtttaaggtttacacctataaaagagagagccgttatcgtctgtttgtggatgtac300
agagtgatattattgacacgcccgggcgacggatggtgatccccctggccagtgcacgtc360
tgctgtcagataaagtctcccgtgaactttacccggtggtgcatatcggggatgaaagct420
ggcgcatgatgaccaccgatatggccagtgtgccggtctccgttatcggggaagaagtgg480
ctgatctcagccaccgcgaaaatgacatcaaaaacgccattaacctgatgttctggggaa540
tataaatgtcaggctcccttatacacagc569
<210>8
<211>31
<212>dna
<213>人工序列(non)
<400>8
gctggtctcaggaaacagaccttttcgacct31
<210>9
<211>19
<212>dna
<213>人工序列(non)
<400>9
tctagaagcttggcactgg19
<210>10
<211>28
<212>dna
<213>人工序列(non)
<400>10
aggctcgagcaagacgatctacccgagc28
<210>11
<211>33
<212>dna
<213>人工序列(non)
<400>11
gaactcgagccgatctagtaacatagatgacac33
<210>12
<211>47
<212>dna
<213>人工序列(non)
<400>12
acccgcgcgctgtcgcttgtgtagagaccattggcggccgcattagg47
<210>13
<211>48
<212>dna
<213>人工序列(non)
<400>13
cttgctatttctagctctaaaactgagaccgtcgacctgcagactggc48
<210>14
<211>25
<212>dna
<213>人工序列(non)
<400>14
acaagtttgtacaaaaaagcaggct25
<210>15
<211>25
<212>dna
<213>人工序列(non)
<400>15
acccagctttcttgtacaaagtggt25
<210>16
<211>74
<212>dna
<213>人工序列(non)
<400>16
cccgcgcgctgtcgcttgtgtgtcatccagtacaacatcttgttttagagctagaaatag60
caagttaaaataag74
<210>17
<211>74
<212>dna
<213>人工序列(non)
<400>17
cccgcgcgctgtcgcttgtgtgatggccatcacggtggcatgttttagagctagaaatag60
caagttaaaataag74
<210>18
<211>73
<212>dna
<213>人工序列(non)
<400>18
cccgcgcgctgtcgcttgtgtgaagtattactactcgatggttttagagctagaaatagc60
aagttaaaataag73
<210>19
<211>74
<212>dna
<213>人工序列(non)
<400>19
cccgcgcgctgtcgcttgtgtgctaatggcatgggaaaccagttttagagctagaaatag60
caagttaaaataag74
<210>20
<211>74
<212>dna
<213>人工序列(non)
<400>20
cccgcgcgctgtcgcttgtgtgtcaggccgacgatgacgcagttttagagctagaaatag60
caagttaaaataag74
<210>21
<211>73
<212>dna
<213>人工序列(non)
<400>21
cccgcgcgctgtcgcttgtgtgtgtacgggaacttcttcggttttagagctagaaatagc60
aagttaaaataag73
<210>22
<211>74
<212>dna
<213>人工序列(non)
<400>22
cccgcgcgctgtcgcttgtgtgccaaccagtcgtccaacgcgttttagagctagaaatag60
caagttaaaataag74
<210>23
<211>60
<212>dna
<213>人工序列(non)
<400>23
cacattgcccagctaactcgaacgcgaccaacttataaacccgcgcgctgtcgcttgtgt60
<210>24
<211>70
<212>dna
<213>人工序列(non)
<400>24
actcggtgccactttttcaagttgataacggactagccttattttaacttgctatttcta60
gctctaaaac70
<210>25
<211>17
<212>dna
<213>人工序列(non)
<400>25
ctgtgatgggctggatg17
<210>26
<211>18
<212>dna
<213>人工序列(non)
<400>26
ctgcagaattgcccttcg18
<210>27
<211>20
<212>dna
<213>人工序列(non)
<400>27
gccgaaagcgaccaaatctc20
<210>28
<211>20
<212>dna
<213>人工序列(non)
<400>28
cgagctgtcatccaaaccca20
<210>29
<211>19
<212>dna
<213>人工序列(non)
<400>29
ctcctcccaacgccattga19
<210>30
<211>20
<212>dna
<213>人工序列(non)
<400>30
tcacgatcacacgaggttga20
<210>31
<211>20
<212>dna
<213>人工序列(non)
<400>31
cttgaagactgggactgcgt20
<210>32
<211>20
<212>dna
<213>人工序列(non)
<400>32
gctcccgcaaagtccttagt20
<210>33
<211>22
<212>dna
<213>人工序列(non)
<400>33
atgccatgagatcttgtcttgc22
<210>34
<211>21
<212>dna
<213>人工序列(non)
<400>34
ttttcgttggttgcacggttt21
<210>35
<211>20
<212>dna
<213>人工序列(non)
<400>35
gagagagccactagcagcag20
<210>36
<211>20
<212>dna
<213>人工序列(non)
<400>36
ccactcatcgacgcgtatca20
<210>37
<211>20
<212>dna
<213>人工序列(non)
<400>37
ccgagtcaaaaagaggggga20
<210>38
<211>20
<212>dna
<213>人工序列(non)
<400>38
ataacatttttgggccgccg20
<210>39
<211>20
<212>dna
<213>人工序列(non)
<400>39
gtccgagcttggaaggagaa20
<210>40
<211>20
<212>dna
<213>人工序列(non)
<400>40
caggtgccacgaagatctga20
- 还没有人留言评论。精彩留言会获得点赞!