一种用于分析肠道微生物的试剂盒及其应用的制作方法
本发明属于分子生物学领域,涉及一种用于分析肠道微生物的试剂盒及其应用,具体涉及通过两轮pcr构建文库,包括分析分类肠道微生物的引物组合物的试剂盒及应用。
背景技术:
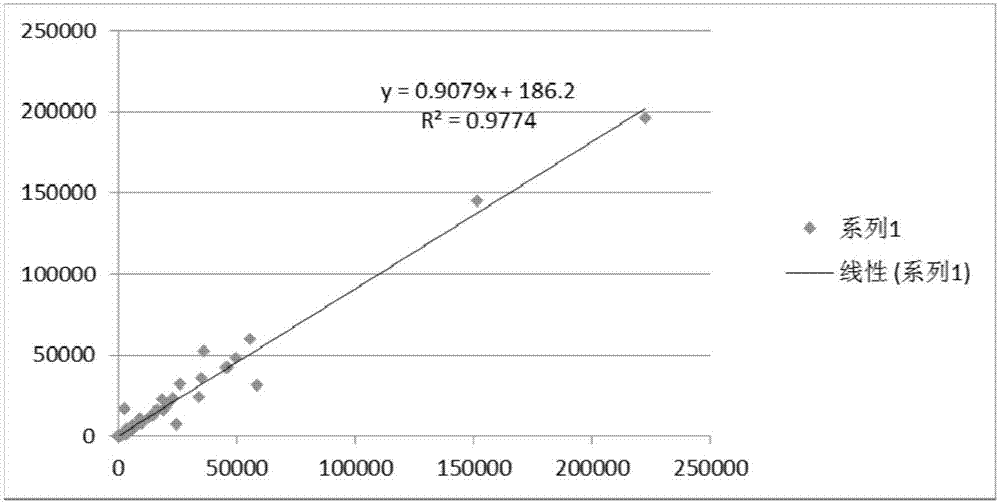
:动物胃肠道内庞大多样的微生物群落与动物的食性、机体的免疫功能、疾病与健康等有着密切联系,越来越引起重视并成为研究热点。然而采用传统方法破环性的采集胃肠道样品对其进行研究受到很大制约,且对于野生保护动物并不可取,因此很多研究都以动物的粪便作为研究样品。科学家们还发现,不同国家人群中的肠道微生物存在很多差异,差异最显著的是微生物的多样性程度。例如:印第安人和马拉维人的肠道微生物组比美国人的肠道微生物具有更大的多样性。有观点认为,微生物多样性程度越高,人体越健康。该研究还发现,尽管来自三种不同地域人群的肠道微生物组存在很多差异,但它们之间也存在惊人的相似性。如,三个不同国家的婴儿微生物组形成过程具有共同的模式,即婴儿需要6-9个月的时间来获得第一组6-700个细菌,然后再经过几年的时间才能获得成人的微生物组。该研究还发现了一个非常有趣的现象,即肠道微生物组的构成会随年龄增长而发生改变,而这一变化恰恰适应了不同年龄段人体的需求。肠道微生物的研究大体经历了培养依赖的方法,非培养依赖的传统分子生物学方法,基于测序的高通量组学方法3个阶段。通过比较3个阶段研究方法的特点与应用,人们可以发现培养依赖的方法在鉴定菌种的同时即可获得相应菌株,方便后续研究,但培养法本身费时费力,肠道微生物以厌氧菌和兼性厌氧菌为主,培养起来更加困难,并且在培养的过程中菌种比例会发生改变,使得其应用存在瓶颈。非培养依赖的传统分子生物学方法可以不经培养直接从样品中提取肠道微生物基因组,利用分子生物学手段进行分离、鉴定和定量,使其结果可以比较准确的反应肠道微生物中高丰度菌种的组成和真实比例。但是传统分子生物学方法存在着通量低的缺陷,靶向方法如实时定量pcr每次只能研究一种或一类肠道微生物,而非靶向方法如最常用的梯度变形凝胶电泳(denaturinggradientgelelectrophoresis,dgge)受限于灵敏度,往往只能研究肠道中高丰度的微生物。肠道微生物群落组成复杂,每种细菌都与其他细菌形成复杂的相互关系网络,低丰度的菌种同样扮演者重要的角色,所以传统分子生物学研究结果往往具有片面性。16srdna因其序列在物种间的高度多样性,成为细菌分类学研究的“分子钟”,具9个可变区和10个保守区间隔排列的特征,可变区因细菌而异,且变异程度与细菌的系统发育密切相关。因此通过分析可变区的序列即可得到各细菌的分类学特征。cn105937053a公开了一种基于高通量基因测序建立粪便菌群基因文库的方法,该方法采用“巢式pcr”的方法对以16srdna进行富集扩增,最大限度减少了宿主和食物残渣基因组污染,同时将v3和v6组合,进行特异扩增并进行大规模并行测序,获得菌群的靶标基因序列库。cn107058490a公开了一种基于新一代高通量测序技术的肠道及口腔菌群多样性及差异性的分析方法,该方法利用illuminamiseq测序系统进行测序,从而分析菌群的组成与功能。但构建的文库覆盖率不够高,检测灵敏度较低。基于上述问题,如何设计引物,能够尽可能全面覆盖所述样本的微生物,提高检测的特异性和灵敏度,就成为亟待解决的问题。技术实现要素:针对现有技术的不足,本发明提供了一种分析肠道微生物的试剂盒及其应用,本发明引物组合物采用两步法扩增获得测序文库,其中包含了1-7bp的随机碱基序列,增加了文库序列的复杂度,降低了phix的比例,保证更多测序质量,提高了文库的覆盖度。第一方面,本发明提供了一种用于分析肠道微生物的试剂盒,其包含用于构建肠道微生物文库的引物组合物,所述引物组合物包括两组引物,具体如下:第一组引物,所述第一组引物包含第一正向引物和第一反向引物,其中,所述第一正向引物从5’到3’端包括依次相连的三个部分:第一测序通用引物、1-7bp的随机碱基序列和肠道微生物16srrnav3-v4区特异性正向引物;所述第一反向引物从5’到3’端包括依次相连的三个部分:第二测序通用引物、1-7bp的随机碱基序列和肠道微生物16srrnav3-v4区特异性反向引物;第二组引物,所述第二组引物包含第二正向引物和第二反向引物,其中,所述第二正向引物从5’到3’端包括依次相连的三个部分:p5端接头序列、第一标签序列和第一测序通用引物的部分序列;所述第二反向引物从5’到3’端包括依次相连的三个部分:p7端接头序列、第二标签序列和第二测序通用引物的部分序列。本发明中,发明人发现,通过采用两步法扩增获得测序文库,所述第一组引物包含v3-v4位点特异性简并引物,提高了扩增片段覆盖率、检测特异性及灵敏度,结合数据分析,可获得更丰富的微生物组信息,此外,所述第一组引物包含随机碱基,增加了文库序列的复杂度,降低了phix的比例,保证更多测序质量;第二组引物包含双index(标签)的设计,可以更大程度地混合更多的样本。根据本发明,所述第一测序通用引物和所述第二测序通用引物独立地选自任意一种二代测序平台的测序通用引物,本发明采用illumina平台的测序通用引物。根据本发明,所述第一测序通用引物的核酸序列如seqidno.1所示,所述第二测序通用引物的核酸序列如seqidno.2所示,所述核酸序列如下:第一测序通用引物(seqidno.1):tcgtcggcagcgtcagatgtgtataagagacag;第二测序通用引物(seqidno.2):gtctcgtgggctcggagatgtgtataagagacag.根据本发明,所述随机碱基序列为1-7个随机碱基,如(n)1-7,优选为3-5个随机碱基,如(n)3-5,其中,n表示为a\t\g\c中的任意一个碱基。发明人发现,通过随机碱基序列可以进一步增加文库的复杂性,提升扩增后的文库的质量,随机碱基不会对所述引物扩增的文库造成影响,选取任意的随机碱基都可以构建文库。根据本发明,所述肠道微生物16srrnav3-v4区特异性正向引物的核酸序列如seqidno.3所示,所述肠道微生物16srrnav3-v4区特异性反向引物的核酸序列如seqidno.4所示,具体核酸序列如下:肠道微生物16srrnav3-v4区特异性正向引物(seqidno.3):cctacggrrbgcascagkvrvgaat;其中,r选自a/g中的任意一个碱基,b选自t/c/g中的任意一个碱基,s选自c/g中的任意一个碱基,k选自t/g中的任意一个碱基,v选自a/c/g中的任意一个碱基;肠道微生物16srrnav3-v4区特异性反向引物(seqidno.4):ggactacnvgggtwtctaatc;其中,n选自a\g\c\t中的任意一个碱基,v选自a/c/g中的任意一个碱基,w选自a/t中的任意一个碱基。本发明中,所述第一正向引物通过seqidno.1所示的第一测序通用引物+1-7bp的随机碱基+seqidno.3所示的肠道微生物16srrnav3-v4区特异性正向引物组合得到,为:5’-tcgtcggcagcgtcagatgtgtataagagacag-(n)1-7-cctacggrrbgcascagkvrvgaat-3',例如可以是5’-tcgtcggcagcgtcagatgtgtataagagacag-atgca-cctacggrrbgcascagkvrvgaat-3';所述第一反向引物通过seqidno.2所示的第二测序通用引物+1-7bp的随机碱基+seqidno.4所示的肠道微生物16srrnav3-v4区特异性反向引物组合得到,为5’-gtctcgtgggctcggagatgtgtataagagacag-tcaga-ggactacnvgggtwtctaatc-3',例如可以是5’-tcgtcggcagcgtcagatgtgtataagagacag-(n)1-7-cctacggrrbgcascagkvrvgaat-3'.根据本发明,所述p5端接头序列的核酸序列如seqidno.5所示,所述p7端接头序列的核酸序列如seqidno.6所示,具体核酸序列如下:p5端接头序列(seqidno.5):aatgatacggcgaccaccgagatctacac;p7端接头序列(seqidno.6):caagcagaagacggcatacgagat.根据本发明,所述第一标签序列的核酸序列如seqidno.7-16所示,所述核酸序列如下:序列接头名称seqidno.7ctctctats502seqidno.8tatcctcts503seqidno.9gtaaggags505seqidno.10actgcatas506seqidno.11aaggagtas507seqidno.12ctaagccts508seqidno.13cgtctaats510seqidno.14tctctccgs511seqidno.15tcgactags513seqidno.16ttctagcts515根据本发明,所述第二标签序列的核酸序列如seqidno.17-26所示,所述核酸序列如下:根据本发明,所述第一测序通用引物的部分序列如seqidno.27所示,所述核酸序列如下:第一测序通用引物的部分序列(seqidno.27):tcgtcggcagcgtc.根据本发明,所述第二测序通用引物的部分序列如seqidno.28所示,所述核酸序列如下:第二测序通用引物的部分序列(seqidno.28):gtctcgtgggctcgg.本发明中,所述第二正向引物通过seqidno.5所示的p5接头+第一标签序列+seqidno.27所示的第一测序通用引物的部分序列组合得到,例如可以是:aatgatacggcgaccaccgagatctacac-ctctctat-tcgtcggcagcgtc;所述第二反向引物通过seqidno.6所示的p7接头+第二标签序列+seqidno.28所示的第二测序通用引物的部分序列组合得到,例如可以是caagcagaagacggcatacgagat-tcgcctta-gtctcgtgggctcgg.第二方面,本发明提供一种肠道微生物的分析方法,采用如第一方面所述的引物组合物,包括如下步骤:(1)样本基因组dna的提取;(2)扩增子文库的构建:进行两轮pcr扩增,第一轮pcr扩增采用第一组引物组,第二轮pcr扩增采用第二组引物组,构建高通量测序文库;(3)数据分析。根据本发明,所述样本来源于粪便、土壤、尿液或唾液中的任意一种或至少两种的组合,优选为来源于粪便。本发明,步骤(1)所述样本基因组dna的提取的方法为常规的dna提取方法,本领域技术人员可以根据需要进行选择,在此不作特殊限定。根据本发明,步骤(2)所述第一轮pcr的条件为:93-96℃预变性1-5min;93-96℃预变性20-40s,50-55℃20-40s,70-75℃20-40s,共进行25-35个循环;70-75℃延伸3-6min。优选地,步骤(2)所述第一轮pcr的条件为:95℃预变性3min;95℃预变性30s,53℃30s,72℃30s,共进行28个循环;72℃延伸5min。根据本发明,步骤(2)所述第二轮pcr的条件为:93-96℃预变性1-5min;93-96℃预变性20-40s,54-58℃20-40s,70-75℃20-40s,共进行6-12个循环;70-75℃延伸3-6min。优选地,步骤(2)所述第二轮pcr的条件为:95℃预变性3min;95℃预变性30s,55.5℃延伸30s,72℃30s,8个循环;72℃5min,1个循环。根据本发明,步骤(2)所述第一轮pcr之后还包括纯化的步骤,优选采用磁珠进行纯化。根据本发明,所述磁珠与扩增产物的体积比为(0.4-0.7):1,例如可以是0.4:1、0.5:1、0.6:1或0.7:1,优选为0.5:1。根据本发明,步骤(2)所述第二轮pcr之后还包括纯化的步骤,优选采用磁珠进行纯化。根据本发明,所述磁珠与扩增产物的体积比为(0.7-1.1):1,例如可以是0.7:1、0.8:1、0.9:1、1:1或1.1:1,优选为0.9:1。根据本发明,步骤(3)所述数据分析为采用illuminamiseq测序仪进行测序分析。根据本发明,所述测序分析具体包括如下步骤:通过illuminamiseq测序仪测序得到按照标签区分的测序读长,利用读长的重叠关系组装得到高可变区v3-v4的全长序列,再对全长序列进行分类分析,得到所述微生物群体的分类。根据本发明,所述全长序列进行分类分析具体包括计算全长序列差异度,根据序列差异度执行操作分类学单元otu的分类,将全长序列分配到otu中,再将每一个otu分类中的全长序列比对到16srrna的v6数据库中,将比对结果根据众数原则对otu进行物种注释。第三方面,本发明提供一种如第一方面所述的试剂盒或如第三方面所述的分析方法用于分析分类肠道微生物。第四方面,本发明提供一种如第三方面所述的分析方法用于免疫组学分析。第五方面,本发明提供一种如第一方面所述的试剂盒用于制备癌症的诊断试剂。优选地,所述癌症选自但不限于肠癌、腺癌或膀胱癌中的任意一种或至少两种的组合。与现有技术相比,本发明具有如下有益效果:(1)本发明中,通过采用两步法扩增获得测序文库,所述试剂盒中的引物组合物可以更大程度地混合更多的样本,提高了扩增片段覆盖率、检测特异性及灵敏度,降低了pcr扩增偏好性,结合数据分析,可获得更丰富的微生物组信息,增加了文库序列的复杂度,降低了phix的比例,保证更多测序质量;(2)本发明提供的数据处理分析方法,实现更多微生物物种在分类学属、种上的鉴别及多样性分析,并得到相关微生物群体的相对丰度值;(3)本发明方法对肠道微生物进行测序及数据分析,实现了更多物种在分类学属甚至种上的鉴别,即获得更多肠道微生物生物学信息,可为免疫组学研究以及临床用药上提供一定指导意义;(4)本发明方法可用于检测癌症,癌症的检出率可达85%以上,尤其针对肠癌的检出率可达96.92%,针对腺瘤的检出率可达85.25%。附图说明图1为本发明实施例中样本a数据分析后相关性分析的结果图;图2为本发明实施例中样本b数据分析后相关性分析的结果图;图3为本发明实施例中样本c数据分析后相关性分析的结果图。具体实施方式下面结合附图和实施例对本发明作进一步的详细说明。可以理解的是,此处所描述的具体实施例仅仅用于解释本发明,而非对本发明的限定。另外还需要说明的是,为了便于描述,附图中仅示出了与本发明相关的部分而非全部结构。材料与试剂:缓冲液a:0.5mtris-cl缓冲液(ph8.0),1.0mnacl;裂解液b:1mtris-cl缓冲液(ph8.0),0.2medta,10%sds,12%tween-40;悬浮液c:30mg/mlrnaasea;去除剂d:1.0mal2(so4)3,250g/lctab,1.5mnacl;结合液e:0.5mtris-cl缓冲液(ph8.0),5m异硫腈酸胍;漂洗液f:5mtris-cl缓冲液(ph8.0),25%(体积比)无水乙醇;洗脱液g:无菌去离子水。实施例1检测试剂盒的制备(1)引物组合物的制备第一组引物:所述第一测序通用引物的核酸序列如seqidno.1所示,所述第二测序通用引物的核酸序列如seqidno.2所示,所述核酸序列如下:第一测序通用引物(seqidno.1):tcgtcggcagcgtcagatgtgtataagagacag;第二测序通用引物(seqidno.2):gtctcgtgggctcggagatgtgtataagagacag;所述随机碱基序列为1-7个随机碱基,如(n)1-7,优选为3-5个随机碱基,如(n)3-5,其中,n表示为a\t\g\c中的任意一个碱基;所述肠道微生物16srrnav3-v4区特异性正向引物的核酸序列如seqidno.3所示,所述肠道微生物16srrnav3-v4区特异性反向引物的核酸序列如seqidno.4所示,具体核酸序列如下:肠道微生物16srrnav3-v4区特异性正向引物(seqidno.3):cctacggrrbgcascagkvrvgaat;肠道微生物16srrnav3-v4区特异性反向引物(seqidno.4):ggactacnvgggtwtctaatc.第二组引物:所述p5端接头序列的核酸序列如seqidno.5所示,所述p7端接头序列的核酸序列如seqidno.6所示,具体核酸序列如下:p5端接头序列(seqidno.5):aatgatacggcgaccaccgagatctacac;p7端接头序列(seqidno.6):caagcagaagacggcatacgagat;所述第一标签序列的核酸序列如seqidno.7-16所示,所述核酸序列如下:序列接头名称seqidno.7ctctctats502seqidno.8tatcctcts503seqidno.9gtaaggags505seqidno.10actgcatas506seqidno.11aaggagtas507seqidno.12ctaagccts508seqidno.13cgtctaats510seqidno.14tctctccgs511seqidno.15tcgactags513seqidno.16ttctagcts515所述第二标签序列的核酸序列如seqidno.17-26所示,所述核酸序列如下:序列接头名称seqidno.17tcgccttan701seqidno.18ctagtacgn702seqidno.19ttctgcctn703seqidno.20gctcaggan704seqidno.21aggagtccn705seqidno.22catgcctan706seqidno.23gtagagagn707seqidno.24cagcctcgn710seqidno.25tgcctcttn711seqidno.26tcctctacn712根据本发明,所述第一测序通用引物的部分序列如seqidno.27所示,所述核酸序列如下:第一测序通用引物的部分序列(seqidno.27):tcgtcggcagcgtc;所述第二测序通用引物的部分序列如seqidno.28所示,所述核酸序列如下:第二测序通用引物的部分序列(seqidno.28):gtctcgtgggctcgg.(2)试剂盒的组装将所述引物组合物和检测探针与试剂盒相关的试剂进行组装,制备成所述检测试剂盒。实施例2样本dna的提取选取3个粪便样本:a、b、c,平均分成2份,标记为:a1/a2,b1/b2,c1/c2,a1、b1、c1三个样本用本专利说明的方法处理并获得数据,a2、b2、c2三个样本直接送去金唯智测序,具体的dna提取方法包括如下步骤:(1)取700~800μl缓冲液a和250mg玻璃珠至2ml离心管中;(2)在上述2ml离心管中加入250mg样本,涡旋混匀10s;(3)向样本中加入70μl裂解液b,涡旋振荡7min混匀样本,12,000rpm(~13,400×g),离心60s。转移上清液(500μl)至新的2ml离心管;(4)加入250μl悬浮液c,涡旋振荡10s,室温放置2min,12,000rpm(~13,400×g),离心60s,沉淀样本颗粒;(5)转移上清至新的2ml离心管,加入300μl去除剂d混匀,4℃放置2~5min;(6)12,000rpm(~13,400×g),离心60s。转移上清液至新的2ml离心管,加入1000μl结合液e颠倒混匀;(7)取上一步所得溶液700μl加入到一个吸附柱中(吸附柱放入收集管中),12,000rpm(~13,400×g),离心30s,倒掉废液,将吸附柱放入收集管中;(8)向吸附柱中,加入700μl漂洗液f,12,000rpm(~13,400×g),离心30s,倒掉废液,将吸附柱放入收集管中;(9)向吸附柱中,加入500μl75%无水乙醇,12,000rpm(~13,400×g),离心30s,倒掉废液,将吸附柱放入收集管中;(10)将吸附柱放回空收集管中,12,000rpm(~13,400×g),离心60s,尽量除去漂洗液,以免漂洗液中残留乙醇抑制下游反应;(11)取出吸附柱,放入一个干净的离心管中,在吸附膜的中间部位加50~100μl洗脱液g(洗脱液可事先在60~70℃水浴中预热),室温放置2min,12,000rpm(~13,400×g),离心60s,将得到的溶液重新加入离心吸附柱中,室温放置2min,12,000rpm(~13,400×g),离心1min;(12)使用分光光度计或qubit对样品进行精确定量,dna可以存放在0~10℃,如果要长时间存放,可以放置在-20℃。将提取后的dna进行纯度测试,od260/od280的比值在1.7~1.9之间,可以直接用于后续的pcr建库。实施例3文库构建(1)在冰上配制以下反应液,除dna提取液和h2o以外,依照如下组份先按反应数+α的量配制扩增预混mix,取配制好的预混mix分装到pcr反应管中,具体引物序列如下:上游引物(seqidno.29):5’-tcgtcggcagcgtcagatgtgtataagagacag-atgca-cctacggagtgcagcagtagcgaat-3';下游引物(seqidno.30):5’-gtctcgtgggctcggagatgtgtataagagacag-tcaga-ggactacacgggtatctaatc-3';反应体系如下:设置一组以水为样本的阴性对照。反应条件如下:(2)第一轮pcr产物的纯化使用agencourtampurexp(beckmancoulter公司)对第一轮pcr扩增产物进行纯化,具体步骤如下:1)4℃冰箱取出,室温放置25min,将agencourtampurexp磁珠充分打散,混匀;2)扩增产物与agencourtampurexp磁珠的结合,所述扩增产物与agencourtampurexp磁珠的体积比为2:1;3)充分混匀,室温孵育15min,瞬离,磁力架上静置5min,去除上清液;4)第一次清洗:反应管放置在磁力架上不动,加入200μl新配的80%的乙醇室温静置(>30s),去除80%的乙醇;5)第二次清洗:重复一次上述4)第一次清洗操作,轻轻离心,去除残留80%的乙醇;6)磁珠的干燥:室温干燥1min;7)洗脱:加入30μl的洗脱液,轻轻地上下吸打,充分混匀,室温孵育15min,瞬离,磁力架上静置5min,回收上清液;(3)片段大小确定琼脂糖电泳确定扩增是否准确,扩增产物长度约550bp。(4)第二轮pcr将步骤(2)中纯化得到的pcr产物为模板,进行pcr导入测序接头,具体步骤如下:1)在冰上配制以下反应液,除第一轮pcr纯化产物以外,依照如下组份先按反应数+α的量配制扩增预混mix,取配制好的预混mix分装到pcr反应管中,具体引物序列如下:上游引物(seqidno.31):aatgatacggcgaccaccgagatctacac-ctctctat-tcgtcggcagcgtc;下游引物(seqidno.32):caagcagaagacggcatacgagat-tcgcctta-gtctcgtgggctcgg;反应体系如下:试剂使用量2×pcr混合液(mg2+,dntps)12.5μl上游引物(1μm)5μl下游引物(1μm)5μldna(第一轮pcr纯化产物)2.5μltotal25μl设置一组以水为样本的阴性对照。反应条件如下:(5)第二轮pcr产物的纯化具体步骤同第一轮pcr产物纯化,即步骤(2),其中pcr产物与agencourtampurexp磁珠的体积比为10:9;(6)片段大小及浓度测定琼脂糖电泳确定扩增是否准确,扩增产物长度约600bp;(7)使用qubitdsdnahsassaykit(thermofisher)等测定pcr产物的浓度;(8)测序建好的文库用illumina公司的miseq测序进行测序,测序侧率为pe300测序。实施例4数据分析(1)对测序得到的原始数据进行质控:通过barcode和引物等信息筛选出高质量的数据,筛选标准如下:含有完整的序列;具有完整前引物序列;去除barcode、引物、接头,序列长度必须大于400bp,不含有模糊碱基;不能存在超过20个的单碱基重复,将得到的序列根据信息分别与各个样品相对应;(2)通过数据库比对工具进行比对,将序列聚类生成out;(3)将得到的otu表,按照a1/a2,b1/b2,c1/c2进行相关性分析,结果如图1-3所示。从图1可以看出,样本a,分成两份后,分别用本专利方法和金唯智公司方法建库测序,得到的数据分析后进行相关性分析发现,两种方法得到的out相关性达到0.9857;从图2可以看出,样本b,分成两份后,分别用本专利方法和金唯智公司方法建库测序,得到的数据分析后进行相关性分析发现,两种方法得到的out相关性达到0.9774;从图3可以看出,样本c,分成两份后,分别用本专利方法和金唯智公司方法建库测序,得到的数据分析后进行相关性分析发现,两种方法得到的out相关性达到0.9809。实施例5分析验证选取65例肠癌患者粪便样本,61例腺瘤患者粪便样本和65例健康患者粪便样本,用本专利所述的方法提取建库测序获得dna序列信息,根据序列信息预测肠癌风险,具体试验方法同实施例2-实施例4,结果如表1所示。表1肠癌腺瘤健康人实际数目656165肠癌检出数目6380腺瘤检出数目2527健康检出数目0158准确率96.92%85.25%89.23%从表1可以看出,本发明检测中,肠癌的检出率为96.92%,腺瘤的检出率为85.25%,健康人的准确率为89.23%,可见,本发明方法用于检测癌症的准确率可达96%以上。综上所述,本发明中,通过采用两步法扩增获得测序文库,所述引物组合物可以更大程度地混合更多的样本,提高了扩增片段覆盖率、检测特异性及灵敏度,结合数据分析,可获得更丰富的微生物组信息,增加了文库序列的复杂度,降低了phix的比例,保证更多测序质量,降低了pcr扩增偏好性;本发明提供的数据处理分析方法,实现更多微生物物种在分类学属、种上的鉴别及多样性分析,并得到相关微生物群体的相对丰度值。注意,上述仅为本发明的较佳实施例及所运用技术原理。本领域技术人员会理解,本发明不限于这里所述的特定实施例,对本领域技术人员来说能够进行各种明显的变化、重新调整和替代而不会脱离本发明的保护范围。因此,虽然通过以上实施例对本发明进行了较为详细的说明,但是本发明不仅仅限于以上实施例,在不脱离本发明构思的情况下,还可以包括更多其他等效实施例,而本发明的范围由所附的权利要求范围决定。序列表<110>苏州普瑞森基因科技有限公司<120>一种用于分析肠道微生物的试剂盒及其应用<130>2017<141>2017-12-29<160>32<170>siposequencelisting1.0<210>1<211>33<212>dna<213>人工合成序列()<400>1tcgtcggcagcgtcagatgtgtataagagacag33<210>2<211>34<212>dna<213>人工合成序列()<400>2gtctcgtgggctcggagatgtgtataagagacag34<210>3<211>25<212>dna<213>人工合成序列()<400>3cctacggrrbgcascagkvrvgaat25<210>4<211>21<212>dna<213>人工合成序列()<220><221>misc_feature<222>(8)..(8)<223>nisa,c,g,ort<400>4ggactacnvgggtwtctaatc21<210>5<211>29<212>dna<213>人工合成序列()<400>5aatgatacggcgaccaccgagatctacac29<210>6<211>24<212>dna<213>人工合成序列()<400>6caagcagaagacggcatacgagat24<210>7<211>8<212>dna<213>人工合成序列()<400>7ctctctat8<210>8<211>8<212>dna<213>人工合成序列()<400>8tatcctct8<210>9<211>8<212>dna<213>人工合成序列()<400>9gtaaggag8<210>10<211>8<212>dna<213>人工合成序列()<400>10actgcata8<210>11<211>8<212>dna<213>人工合成序列()<400>11aaggagta8<210>12<211>8<212>dna<213>人工合成序列()<400>12ctaagcct8<210>13<211>8<212>dna<213>人工合成序列()<400>13cgtctaat8<210>14<211>8<212>dna<213>人工合成序列()<400>14tctctccg8<210>15<211>8<212>dna<213>人工合成序列()<400>15tcgactag8<210>16<211>8<212>dna<213>人工合成序列()<400>16ttctagct8<210>17<211>8<212>dna<213>人工合成序列()<400>17tcgcctta8<210>18<211>8<212>dna<213>人工合成序列()<400>18ctagtacg8<210>19<211>8<212>dna<213>人工合成序列()<400>19ttctgcct8<210>20<211>8<212>dna<213>人工合成序列()<400>20gctcagga8<210>21<211>8<212>dna<213>人工合成序列()<400>21aggagtcc8<210>22<211>8<212>dna<213>人工合成序列()<400>22catgccta8<210>23<211>8<212>dna<213>人工合成序列()<400>23gtagagag8<210>24<211>8<212>dna<213>人工合成序列()<400>24cagcctcg8<210>25<211>8<212>dna<213>人工合成序列()<400>25tgcctctt8<210>26<211>8<212>dna<213>人工合成序列()<400>26tcctctac8<210>27<211>14<212>dna<213>人工合成序列()<400>27tcgtcggcagcgtc14<210>28<211>15<212>dna<213>人工合成序列()<400>28gtctcgtgggctcgg15<210>29<211>63<212>dna<213>人工合成序列()<400>29tcgtcggcagcgtcagatgtgtataagagacagatgcacctacggagtgcagcagtagcg60aat63<210>30<211>60<212>dna<213>人工合成序列()<400>30gtctcgtgggctcggagatgtgtataagagacagtcagaggactacacgggtatctaatc60<210>31<211>51<212>dna<213>人工合成序列()<400>31aatgatacggcgaccaccgagatctacacctctctattcgtcggcagcgtc51<210>32<211>47<212>dna<213>人工合成序列()<400>32caagcagaagacggcatacgagattcgccttagtctcgtgggctcgg47当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1