BCL11A归巢核酸内切酶变体、组合物和使用方法与流程
根据美国法典第35条119(e)款,本申请要求2016年10月28日提交的美国临时申请第62/414,273号,2016年8月16日提交的美国临时申请第62/375,829号,2016年7月27日提交的美国临时申请第62/367,465号,2016年7月25日提交的美国临时申请第62/366,530号,其每一个通过引用整体并入本文。
关于序列表的声明
与本申请相关的序列表以文本格式提供以代替纸质副本,并且通过引用并入本说明书中。包含序列表的文本文件的名称是blbd_071_04wo_st25.txt。该文本文件为141kb,于2017年7月25日创建,并通过efs-web以电子方式提交,与说明书的提交同时进行。
本公开涉及改进的基因组编辑组合物。更具体地,本公开涉及重编程的核酸酶、组合物和使用其编辑b细胞cll/淋巴瘤11a(bcl11a)基因的方法。
背景技术:
血红蛋白病是由血红蛋白的结构和/或合成的变化引起的多种遗传性单基因血液病症。最常见的血红蛋白病是镰状细胞病(scd)、α-地中海贫血和β-地中海贫血。世界上大约5%的人口携带球蛋白基因突变。据世界卫生组织估计,每年有超过30万名婴儿出生时患有严重的血红蛋白病症。血红蛋白病表现出高度不同的临床表现,其范围从轻度低色素性贫血,到中度血液疾病,到多器官受累的重度终身性输血依赖性贫血。
可用于血红蛋白病的唯一潜在治愈性治疗是同种异体造血干细胞移植。然而,据估计,hla兼容的hsc移植物可用于不到20%的受影响个体,并且长期毒性很大。此外,hsc移植物还与患有scd或重度地中海贫血的受试者的显著的死亡率和发病率相关。显著的死亡率和发病率部分归因于hsc前移植输血相关的铁超负荷、移植物抗宿主病(gvhd)、和受试者移植前调理所需的高剂量化疗/放射等等。
血红蛋白病的支持性治疗包含结合铁螯合的终身周期性输血,并在一些情况下需结合脾切除术。scd的其它治疗包含镇痛剂、抗生素、ace抑制剂和羟基脲。然而,与羟基脲治疗相关的副作用包含血细胞减少症、色素沉着过度、体重增加、机会性感染、无精子症、低镁血症和癌症。
充其量,使用现有方法治疗的患者的预计寿命为50到60年。
技术实现要素:
本公开一般涉及包括切割人bcl11a基因中的靶位点的归巢核酸内切酶变体和megatal的组合物及其使用方法。
在各个实施例中,本公开部分地考虑了一种包括归巢核酸内切酶(he)变体的多肽,所述归巢核酸内切酶变体切割人b细胞淋巴瘤/白血病11a(bcl11a)基因中的靶位点。
在特定实施例中,he变体是laglidadg归巢核酸内切酶(lhe)变体。
在一些实施例中,多肽包括he变体的生物活性片段。
在某些实施例中,与相应的野生型he相比,生物活性片段缺少1个、2个、3个、4个、5个、6个、7个或8个n-末端氨基酸。
在另外的实施例中,与相应的野生型he相比,生物活性片段缺少4个n-末端氨基酸。
在某些实施例中,与相应的野生型he相比,生物活性片段缺少8个n-末端氨基酸。
在另外的实施例中,与相应的野生型he相比,生物活性片段缺少1个、2个、3个、4个或5个c-末端氨基酸。
在某些实施例中,与相应的野生型he相比,生物活性片段缺少c-末端氨基酸。
在特定实施例中,与相应的野生型he相比,生物活性片段缺少2个c-末端氨基酸。
在一些实施例中,he变体是选自由以下组成的组的lhe的变体:i-crei和i-scei。
在一些实施例中,he变体是选自由以下组成的组的lhe的变体:i-aabmi、i-aaemi、i-anii、i-apami、i-capiii、i-capiv、i-ckami、i-cpami、i-cpamii、i-cpamiii、i-cpamiv、i-cpamv、i-cpav、i-crami、i-ejemi、i-gpemi、i-gpii、i-gzemi、i-gzemii、i-gzemiii、i-hjemi、i-ltrii、i-ltri、i-ltrwi、i-mpemi、i-mvemi、i-ncrii、i-ncrl、i-ncrmi、i-ohemi、i-onui、i-osomi、i-osomii、i-osomiii、i-osomiv、i-panmi、i-panmii、i-panmiii、i-pnomi、i-scumi、i-smami、i-sscmi和i-vdi141i。
在另外的实施例中,he变体是选自由以下组成的组的lhe的变体:i-cpami、i-hjemi、i-onui、i-panmi和smami。
在特定实施例中,he变体是i-onuilhe变体。
在某些实施例中,he变体包括在dna识别界面中的选自由以下组成的组的氨基酸位置处的一个或多个氨基酸取代:seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的第19位、第24位、第26位、第28位、第30位、第32位、第34位、第35位、第36位、第37位、第38位、第40位、第42位、第44位、第46位、第48位、第68位、第70位、第72位、第75位、第76位、第77位、第78位、第80位、第82位、第168位、第180位、第182位、第184位、第186位、第188位、第189位、第190位、第191位、第192位、第193位、第195位、第197位、第199位、第201位、第203位、第223位、第225位、第227位、第229位、第231位、第232位、第234位、第236位、第238位和第240位。
在一些实施例中,he变体包括在dna识别界面中的选自由以下组成的组的氨基酸位置处的至少5个、至少15个、优选至少25个、更优选至少35个、或甚至更优选至少40个或更多个氨基酸取代:seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的第19位、第24位、第26位、第28位、第30位、第32位、第34位、第35位、第36位、第37位、第38位、第40位、第42位、第44位、第46位、第48位、第68位、第70位、第72位、第75位、第76位、第77位、第78位、第80位、第82位、第168位、第180位、第182位、第184位、第186位、第188位、第189位、第190位、第191位、第192位、第193位、第195位、第197位、第199位、第201位、第203位、第223位、第225位、第227位、第229位、第231位、第232位、第234位、第236位、第238位和第240位。
在特定实施例中,he变体包括选自由以下组成的组的氨基酸位置处的至少5个、至少15个、优选至少25个、更优选至少35个、或甚至更优选至少40个或更多个氨基酸取代:seqidno:1-19所示的i-onuilhe氨基酸序列或其生物活性片段的第26位、第28位、第30位、第32位、第34位、第35位、第36位、第37位、第40位、第41位、第42位、第44位、第48位、第50位、第53位、第68位、第70位、第72位、第76位、第78位、第80位、第82位、第138位、第143位、第159位、第178位、第180位、第184位、第186位、第189位、第190位、第191位、第192位、第193位、第195位、第201位、第203位、第207位、第223位、第225位、第227位、第232位、第236位、第238位和第240位。
在另外的实施例中,he变体包括至少5个、至少15个、优选至少25个、更优选至少35个、或甚至更优选至少40个或更多个以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、l26r、l26y、r28s、r28g、r30q、r30h、n32r、n32s、n32k、n33s、k34d、k34n、s35y、s36a、v37t、s40r、t41i、e42h、e42r、g44t、g44r、t48i、t48g、t48v、h50r、d53e、v68k、v68r、a70n、a70e、a70n、a70q、a70l、a70s、s72a、s72t、s72v、s72m、a76l、a76h、a76r、s78q、k80r、k80v、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44t、v68k、a70n、s72a、a76l、s78q、k80r、t82y、l138m、t143n、s159p、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44t、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在一些实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r30q、n32s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44t、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32k、k34n、s35y、s36a、v37t、s40r、t41i、e42h、g44t、t48i、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42r、g44t、t48i、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在另外的实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28g、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42r、g44t、h50r、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30h、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、v68k、a70n、s72t、a76h、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26r、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、v68k、a70n、s72ta76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26y、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、d53e、v68r、a70e、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在一些实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、d53e、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、t48g、v68k、s72v、a76r、s78q、k80v、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、t48g、v68k、a70q、s72m、a76r、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、t48g、v68k、a70l、s72v、a76h、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,he变体包括以下氨基酸取代:关于seqidno:1-5所示的i-onuilhe氨基酸序列或其生物活性片段的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、t48v、v68k、a70s、s72v、a76h、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,he变体包括与seqidno:6-19中任一个所示的氨基酸序列或其生物活性片段具有至少80%、优选至少85%、更优选至少90%、或甚至更优选至少95%同一性的氨基酸序列。
在特定实施例中,he变体包括seqidno:6所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:7所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:8所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:9所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:10所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:11所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:12所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:13所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:14所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:15所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:16所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:17所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:18所示的氨基酸序列或其生物活性片段。
在一些实施例中,he变体包括seqidno:19所示的氨基酸序列或其生物活性片段。
在一些实施例中,多肽进一步包括dna结合结构域。
在另外的实施例中,dna结合结构域选自由以下组成的组:taledna结合结构域和锌指dna结合结构域。
在另外的实施例中,taledna结合结构域包括约9.5个tale重复单元到约11.5个tale重复单元。
在另外的实施例中,taledna结合结构域包括约9.5个tale重复单元到约12.5个tale重复单元。
在另外的实施例中,taledna结合结构域包括约9.5个tale重复单元到约13.5个tale重复单元。
在另外的实施例中,taledna结合结构域包括约9.5个tale重复单元到约14.5个tale重复单元。
在特定实施例中,taledna结合结构域结合bcl11a基因中的多核苷酸序列。
在特定实施例中,taledna结合结构域结合seqidno:26所示的多核苷酸序列。
在某些实施例中,多肽结合并切割seqidno:27所示的多核苷酸序列。
在某些实施例中,锌指dna结合结构域包括2个、3个、4个、5个、6个、7个或8个锌指基序。
在另外的实施例中,多肽进一步包括肽连接子和末端加工酶或其生物活性片段。
在一些实施例中,多肽进一步包括病毒自切割2a肽和末端加工酶或其生物活性片段。
在特定实施例中,末端加工酶或其生物活性片段具有5'-3'核酸外切酶、5'-3'碱性核酸外切酶、3'-5'核酸外切酶、5'瓣状核酸内切酶、解旋酶、模板依赖性dna聚合酶或模板非依赖性dna聚合酶活性。
在某些实施例中,多肽包括seqidno:20-21中任一个所示的氨基酸序列或其生物活性片段。
在另外的实施例中,多肽包括seqidno:20所示的氨基酸序列或其生物活性片段。
在特定实施例中,多肽包括seqidno:21所示的氨基酸序列或其生物活性片段。
在某些实施例中,末端加工酶包括trex2或其生物活性片段。
在某些实施例中,多肽包括seqidno:22-23中任一个所示的氨基酸序列或其生物活性片段。
在另外的实施例中,多肽包括seqidno:22所示的氨基酸序列或其生物活性片段。
在特定实施例中,多肽包括seqidno:23所示的氨基酸序列或其生物活性片段。
在另外的实施例中,多肽切割seqidno:25或seqidno:27所示的多核苷酸序列处的人bcl11a基因。
在各个实施例中,本公开部分地考虑了一种编码本文考虑的多肽的多核苷酸。
在特定实施例中,本公开部分地考虑了一种编码本文考虑的多肽的mrna。
在特定实施例中,mrna包括seqidno:36-37中任一个所示的序列。
在某些实施例中,本公开部分地考虑了一种编码本文考虑的多肽的cdna。
在另外的实施例中,本公开部分地考虑了一种载体,其包括编码本文考虑的多肽的多核苷酸。
在另外的实施例中,本公开部分地考虑了一种细胞,其包括本文考虑的多肽。
在各个实施例中,本公开部分地考虑了一种细胞,其包括编码本文考虑的多肽的多核苷酸。
在特定实施例中,本公开部分地考虑了一种细胞,其包括本文考虑的载体。
在各个实施例中,本公开部分地考虑了一种细胞,其包括由本文考虑的多肽引入的一个或多个基因组修饰。
在某些实施例中,细胞是造血细胞。
在特定实施例中,细胞是造血干细胞或祖细胞。
在一些实施例中,细胞是cd34+细胞。
在特定实施例中,细胞是cd133+细胞。
在各个实施例中,本公开部分地考虑了一种组合物,其包括本文考虑的基因组编辑的细胞。
在各个实施例中,本公开部分地考虑了一种组合物,其包括本文考虑的基因组编辑的细胞和生理学上可接受的载剂。
在特定实施例中,本公开部分地考虑了一种编辑细胞群中bcl11a基因的方法,其包括:将编码本文考虑的多肽的多核苷酸引入细胞中,其中所述多肽的表达在bcl11a基因中的靶位点处产生双链断裂。
在各个实施例中,本公开部分地考虑了一种编辑细胞群中bcl11a基因的方法,其包括:将编码本文考虑的多肽的多核苷酸引入细胞中,其中所述多肽的表达在bcl11a基因中的靶位点处产生双链断裂,其中通过非同源末端连接(nhej)修复所述断裂。
在特定实施例中,本公开部分地考虑了一种编辑细胞群中bcl11a基因的方法,其包括:将编码本文考虑的多肽的多核苷酸和供体修复模板引入细胞中,其中所述多肽的表达在bcl11a基因中的靶位点处产生双链断裂,并且所述供体修复模板在所述双链断裂(dsb)位点处通过同源定向修复(hdr)掺入bcl11a基因中。
在某些实施例中,细胞是造血细胞。
在另外的实施例中,细胞是造血干细胞或祖细胞。
在一些实施例中,细胞是cd34+细胞。
在特定实施例中,细胞是cd133+细胞。
在另外的实施例中,编码多肽的多核苷酸是mrna。
在特定实施例中,将编码5'-3'核酸外切酶的多核苷酸引入细胞中。
在某些实施例中,将编码trex2的多核苷酸或其生物活性片段引入细胞中。
在另外的实施例中,供体修复模板包括与dsb的bcl11a基因序列5'同源的5'同源臂和与dsb的bcl11a基因序列3'同源的3'同源臂。
在一些实施例中,5'和3'同源臂的长度独立地选自约100bp到约2500bp。
在另外的实施例中,5'和3'同源臂的长度独立地选自约600bp到约1500bp。
在一些实施例中,5'同源臂为约1500bp,并且3'同源臂为约1000bp。
在另外的实施例中,5'同源臂为约600bp,并且3'同源臂为约600bp。
在一些实施例中,使用病毒载体将供体修复模板引入细胞中。
在另外的实施例中,病毒载体是重组腺相关病毒载体(raav)或逆转录病毒。
在特定实施例中,raav具有来自aav2的一个或多个itr。
在另外的实施例中,raav具有选自由以下组成的组的血清型:aav1、aav2、aav3、aav4、aav5、aav6、aav7、aav8、aav9和aav10。
在某些实施例中,raav具有aav2或aav6血清型。
在另外的实施例中,逆转录病毒是慢病毒。
在一些实施例中,慢病毒是整合酶缺陷型慢病毒(idlv)。
在各个实施例中,本公开部分地考虑了一种治疗、预防或改善血红蛋白病或与其相关的病状的至少一种症状的方法,其包括向受试者施用有效量的本文考虑的组合物。
在特定实施例中,受试者具有选自由以下组成的组的β-球蛋白基因型:βe/β0、βc/β0、β0/β0、βe/βe、βc/β+、βe/β+、β0/β+、β+/β+、βc/βc、βe/βs、β0/βs、βc/βs、β+/βs或βs/βs。
在某些实施例中,组合物的量有效地减少受试者中的输血。
在各个实施例中,本公开部分地考虑了一种治疗、预防或改善地中海贫血或与其相关的病状的至少一种症状的方法,其包括向受试者施用有效量的本文考虑的组合物。
在一些实施例中,受试者患有α-地中海贫血或与其相关的病状。
在特定实施例中,受试者患有β-地中海贫血或与其相关的病状。
在某些实施例中,受试者具有选自由以下组成的组的β-球蛋白基因型:βe/β0、βc/β0、β0/β0、βc/βc、βe/βe、βe/β+、βc/βe、βc/β+、β0/β+或β+/β+。
在各个实施例中,本公开部分地考虑了一种治疗、预防或改善镰状细胞病或与其相关的病状的至少一种症状的方法,其包括向受试者施用有效量的本文考虑的组合物。
在特定实施例中,受试者具有选自由以下组成的组的β-球蛋白基因型:βe/βs、β0/βs、βc/βs、β+/βs或βs/βs。
在各个实施例中,本公开部分地考虑了一种增加受试者中γ-球蛋白的量的方法,其包括向受试者施用有效量的本文考虑的组合物。
在各个实施例中,本公开部分地考虑了一种增加受试者中胎儿血红蛋白(hbf)的量的方法,其包括向受试者施用有效量的本文考虑的组合物。
在特定实施例中,受试者患有血红蛋白病。
在一些实施例中,受试者患有α-地中海贫血或与其相关的病状。
在另外的实施例中,受试者患有β-地中海贫血或与其相关的病状。
在特定实施例中,受试者具有选自由以下组成的组的β-球蛋白基因型:βe/β0、βc/β0、β0/β0、βc/βc、βe/βe、βe/β+、βc/βe、βc/β+、β0/β+或β+/β+。
在某些实施例中,受试者患有镰状细胞病或与其相关的病状。
在特定实施例中,受试者具有选自由以下组成的组的β-球蛋白基因型:βe/βs、β0/βs、βc/βs、β+/βs或βs/βs。
附图说明
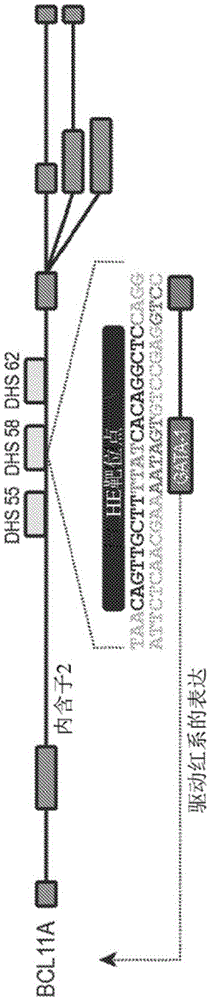
图1示出了人bcl11a基因,其中描绘了可变剪接同种型以及gata-1结合基序(seqidno:77和78)的位置,并且位于脱氧核糖核酸酶高敏位点(dhs)内的重编程的归巢核酸内切酶靶位点位于转录起始位点下游~58kb。
图2a示出了天然归巢核酸内切酶i-smami切割包括ttat作为中心-4序列(seqidno:30)的dna标靶。
图2b示出了i-onui归巢核酸内切酶重编程的标靶ccr5基因能够切割ttat中心-4,同时保留其天然的中心-4切割特异性。
图3示出了通过三轮分选对i-onuin-末端结构域(ntd)和c-末端结构域(ctd)针对嵌合“半位点”重编程,然后融合重编程的结构域以分离切割靶位点的完全重编程的i-onui归巢核酸内切酶。
图4a示出了i-onui衍生的归巢核酸内切酶变体在染色体报告基因测定中对针对bcl11a靶位点的活性的初始筛选。
图4b示出了最初衍生的i-onui衍生的归巢核酸内切酶bcl11a.a4的精制,以获得更具活性的变体bcl11a-b4a3。
图4c示出了bcl11a.a4和bcl11a-b4a3对bcl11a靶序列的催化活性的比较。
图5示出了与野生型i-onui归巢核酸内切酶(seqidno:79)相比,bcl11a.a4(seqidno:80)和bcl11a-b4a3(seqidno:81)归巢核酸内切酶的比对,突出显示了非相同的位置。
图6a示出了bcl11a-b4a3归巢核酸内切酶具有亚纳摩尔亲和性,如使用基于酵母表面展示的底物滴定测定法测量。
图6b示出了每个位置处的靶序列碱基的不同如何影响靶切割特异性。
图7示出了bcl11a-b4a3归巢核酸内切酶的全面的中心-4特异性谱,表明在包含ttat的耐受中心-4序列的轻微移位谱中保留了高度的总体选择性。
图8a示出了靶向bcl11a基因(seqidno:82和83)的bcl11amegatal的示意图。
图8b示出了bcl11a基因中的靶序列在原代人cd34+造血干细胞中的bcl11amegatal编辑的tide分析。
图8c示出了bcl11a基因中的靶序列在编辑原代人cd34+造血干细胞中的bcl11amegatal编辑的基于pcr的分析。
图8d示出了bcl11a基因中的靶序列(seqidno:84-104)在原代人cd34+造血干细胞中的bcl11amegatal编辑的单集落测序分析。
图8e示出了bcl11a基因中的靶序列在原代人cd34+造血干细胞中的bcl11amegatal编辑的额外实验的结果。
图9a示出了供体修复模板的示意图,所述供体修复模板包括位于bcl11a靶序列侧翼的同源臂和嵌入两个同源臂之间的荧光报告基因。
图9b示出了将bcl11amegatal引入cd34+细胞中并用aav6基因组转导细胞,所述aav6基因组包括携带嵌入两个同源臂之间的转基因盒的供体修复模板,导致盒高速率地靶向插入在bcl11a基因中的靶位点处。
图10a示出了将bcl11amegatal引入cd34+细胞中并用aav6基因组转导细胞,所述aav6基因组包括供体修复模板,基本上不改变人cd34+细胞的红系分化能力。
图10b示出了图10a中所示数据的表格表示。
图11a是代表性流式细胞术分析,其示出了当分化为红系细胞时,用bcl11amegatal治疗的原代人cd34+造血干细胞群上调胎儿血红蛋白。
图11b是代表性hplc分析,其示出了当分化为红系细胞时,用bcl11amegatal治疗的原代人cd34+造血干细胞群上调胎儿血红蛋白。
图12示出了在用bcl11amegatal治疗的原代人cd34+造血干细胞群中,集落形成不受影响。
图13示出了在有或没有编码ccr5megatal、ccr5megatal-trex2融合蛋白、bcl11amegatal或bcl11amegatal-trex2融合蛋白的mrna的情况下,电穿孔的人cd34+细胞的编辑速率。
图14示出了在有或没有编码ccr5megatal、ccr5megatal-trex2融合蛋白、bcl11amegatal或bcl11amegatal-trex2融合蛋白的mrna的情况下,电穿孔的人cd34+细胞的hbf产生水平。
图15示出了用bcl11amegatal治疗的原代人cd34+造血干细胞群稳定地植入免疫缺陷小鼠中,其中编辑的细胞的减少最小。
图16示出了人cd34+细胞移植物和具有移植物的移植nsg小鼠的4个月骨髓的hbf产生水平。在有或没有编码ccr5megatal、ccr5megatal-trex2融合蛋白、bcl11amegatal或bcl11amegatal-trex2融合蛋白的mrna的情况下,对人cd34+细胞进行电穿孔。
序列标识符简述
seqidno:1是野生型i-onuilaglidadg归巢核酸内切酶(lhe)的氨基酸序列。
seqidno:2是野生型i-onuilhe的氨基酸序列。
seqidno:3是野生型i-onuilhe的生物活性片段的氨基酸序列。
seqidno:4是野生型i-onuilhe的生物活性片段的氨基酸序列。
seqidno:5是野生型i-onuilhe的生物活性片段的氨基酸序列。
seqidno:6-19是重编程以结合并切割人bcl11a基因中的靶位点的i-onuilhe变体的氨基酸序列。
seqidno:20是结合并切割人bcl11a基因中的靶位点的megatal的氨基酸序列。
seqidno:21是结合并切割人bcl11a基因中的靶位点的megatal的氨基酸序列。
seqidno:22是结合并切割人bcl11a基因中的靶位点的megatal-trex2融合蛋白的氨基酸序列。
seqidno:23是结合并切割人bcl11a基因中的靶位点的megatal-trex2融合蛋白的氨基酸序列。
seqidno:24是包括人bcl11a基因的dna高敏位点58中的gata-1基序的多核苷酸。
seqidno:25是人bcl11a基因中的i-onuilhe变体靶位点。
seqidno:26是人bcl11a基因中的taledna结合结构域靶位点。
seqidno:27是人bcl11a基因中的megatal靶位点。
seqidno:28是i-onuilhe变体n-末端结构域靶位点。
seqidno:29是i-onuilhe变体c-末端结构域靶位点。
seqidno:30是i-smamilhe靶位点。
seqidno:31是人ccr5基因中的i-onuilhe变体靶位点。
seqidno:32是结合并切割人ccr5基因中的靶位点的i-onuilhe变体的i-onuilhe变体表面展示质粒的多核苷酸序列。
seqidno:33是结合并切割人ccr5基因中的靶位点的i-onuilhe变体的中心4阵列的多核苷酸序列。
seqidno:34是结合并切割人bcl11a基因中的靶位点的i-onuilhe变体的i-onuilhe变体表面展示质粒的多核苷酸序列。
seqidno:35是结合并切割人bcl11a基因中的靶位点的i-onuilhe变体的中心4阵列的多核苷酸序列。
seqidno:36是编码切割人bcl11a基因的megatal的mrna序列。
seqidno:37是编码切割人bcl11a基因的megatal-trex2融合体的mrna序列。
seqidno:38是编码鼠trex2的mrna序列。
seqidno:39是编码鼠trex2的氨基酸序列。
seqidno:40-50列出了各种连接子的氨基酸序列。
seqidno:51-75列出了蛋白酶切割位点和自切割多肽切割位点的氨基酸序列。
在前述序列中,x(如果存在的话)是指任何氨基酸或氨基酸的缺失。
具体实施方式
a.概述
本公开一般部分地涉及改进的基因组编辑组合物及其使用方法。不希望受任何特定理论的束缚,本文考虑的基因组编辑组合物用于增加细胞中胎儿血红蛋白的量,以治疗、预防或改善与各种血红蛋白病相关的症状。因此,本文考虑的组合物为患有血红蛋白病的受试者提供了潜在治愈性解决方案。
正常成人血红蛋白包括两种阿尔法-(α)球蛋白和两种贝塔-(β-)球蛋白的四聚复合物。在发育中,胎儿产生胎儿血红蛋白(hbf),其包括两种伽马-(γ)球蛋白而不是两种β-球蛋白。在围产期发育的某个时刻,会出现“球蛋白转换”;红细胞下调γ-球蛋白表达并转向主要产生β-球蛋白。该转换主要由γ-球蛋白基因的转录减少和β-球蛋白基因的转录增加引起。gata结合蛋白-1(gata-1)是影响球蛋白转换的转录因子。gata-1直接反式激活β-球蛋白基因表达,并通过bcl11a表达的反式激活间接阻遏或抑制γ-球蛋白基因表达。对于因β-球蛋白基因突变而患有β-地中海贫血或镰状细胞病的患者而言,该转换的药理学或遗传操作代表了一种有吸引力的治疗策略。
在各个实施例中,考虑了破坏红系细胞中bcl11a基因功能和/或表达的核酸酶变体、基因组编辑组合物、遗传修饰的细胞及其使用方法。红系区室中的bcl11a表达严重依赖于红系增强子,其包括bcl11a基因的第二内含子中的共有gata-1结合基序wgataa(seqidno:24)。不希望受任何特定理论的束缚,预期通过gata-1结合位点的基因组编辑减少或消除红系细胞中的bcl11a表达将导致γ-球蛋白基因表达的再激活或去阻遏和β-球蛋白基因表达的减少,从而增加hbf表达以有效地治疗和/或改善与患有血红蛋白病的受试者相关的一种或多种症状。
在各个实施例中考虑的基因组编辑方法包括核酸酶变体,其被设计成结合并切割b细胞cll/淋巴瘤11a基因(bcl11a)中的转录因子结合位点。特定实施例中考虑的核酸酶变体可以用于将双链断裂引入靶多核苷酸序列中,其可以在不存在多核苷酸模板(例如,供体修复模板)的情况下通过非同源末端连接(nhej)修复,或在存在供体修复模板的情况下通过同源定向修复(hdr)(即,同源重组)修复。在某些实施例中考虑的核酸酶变体也可以被设计成切口酶,这生成单链dna断裂,其可以在存在供体修复模板的情况下使用细胞的碱基切除修复(ber)机制或同源重组来修复。nhej是一个容易出错的过程,经常形成小的插入和缺失,这会破坏基因功能。同源重组需要同源dna作为修复的模板,并且可以利用同源重组产生无限多个修饰,这些修饰通过将在靶位点含有所需序列的供体dna引入以靶位点为侧翼的区域来指定,所述靶位点位于具有同源性的序列的任一侧的侧翼。
在一个优选实施例中,本文考虑的基因组编辑组合物包括靶向人bcl11a基因的归巢核酸内切酶变体或megatal。
在各个实施例中,其中在bcl11a基因的红系特异性增强子中生成dna断裂,切割的基因组序列末端的nhej可能产生bcl11a表达减少的细胞,优选红系细胞,其缺少或基本上缺少功能性bcl11a表达,例如缺少阻遏或抑制γ-球蛋白基因转录的能力并且缺少反式激活β-球蛋白基因转录的能力。
在各个其它实施例中,其中提供了一种用于修复切割的bcl11a基因组序列的供体模板,通过dna断裂位点处的同源重组,用模板的序列修复dsb。在优选实施例中,修复模板包括不同于靶向基因组序列的多核苷酸序列。
在一个优选实施例中,本文考虑的基因组编辑组合物包括核酸酶变体和一种或多种末端加工酶,以增加nhej或hdr效率。
在一个优选实施例中,本文考虑的基因组编辑组合物包括靶向人bcl11a基因的归巢核酸内切酶变体或megatal和末端加工酶,例如trex2。
在各个实施例中,考虑了基因组编辑的细胞。基因组编辑的细胞包括在红系细胞谱系中减少的内源性bcl11a表达。基因组编辑的红系细胞包括增加的γ-球蛋白表达和减少的β-球蛋白表达。
因此,与用于治疗血红蛋白病的现有基因编辑策略相比,本文考虑的方法和组合物代表了重大的改进。
除非特别相反地指出,否则特定实施例的实践将采用本领域技术范围内的化学、生物化学、有机化学、分子生物学、微生物学、重组dna技术、遗传学、免疫学和细胞生物学的常规方法。为了说明的目的,下面描述了这些常规方法中的许多常规方法。这些技术在文献中有充分说明。参见例如sambrook等人,《分子克隆:实验手册(molecularcloning:alaboratorymanual)》(第3版,2001年);sambrook等人,《分子克隆:实验手册(molecularcloning:alaboratorymanual)》(第2版,1989年);maniatis等人,《分子克隆:实验手册(molecularcloning:alaboratorymanual)》(1982年);ausubel等人,《现代分子生物学实验指南(currentprotocolsinmolecularbiology)》(johnwileyandsons,2008年7月更新);《精编分子生物学实验指南:现代分子生物学实验指南的方法纲要(shortprotocolsinmolecularbiology:acompendiumofmethodsfromcurrentprotocolsinmolecularbiology)》,greenepub.associatesandwiley-interscience;glover,《dna克隆:实用方法(dnacloning:apracticalapproach)》,第i卷和第ii卷(irl出版社,牛津,1985年);anand,《复杂基因组分析技术(techniquesfortheanalysisofcomplexgenomes)》(学术出版社,纽约,1992年);《转录和翻译(transcriptionandtranslation)》(b.hames和s.higgins编辑,1984年);perbal,《分子克隆实用指南(apracticalguidetomolecularcloning)》(1984年);harlow和lane,《抗体(antibodies)》,(冷泉港实验室出版社,冷泉港,纽约,1998年),《现代免疫学实验指南(currentprotocolsinimmunology)》,q.e.coligan,a.m.kruisbeek,d.h.margulies,e.m.shevach和w.strober编辑,1991年);《免疫学年鉴(annualreviewofimmunology)》;以及《免疫学进展(advancesinimmunology)》等期刊上的专著。
b.定义
除非另外定义,否则本文使用的所有技术和科学术语具有与本发明所属领域的普通技术人员通常理解的含义相同的含义。尽管与本文描述的那些类似或等同的任何方法和材料可以用于特定实施例的实践或测试,但是本文描述了组合物、方法和材料的优选实施例。出于本公开的目的,以下术语定义如下。
冠词“一个(a/an)”和“所述(the)”在本文中用于指代冠词的一个或多于一个(即,至少一个,或一个或多个)语法对象。举例来说,“一个元件”表示一个元件或一个或多个元件。
选择连词(例如,“或”)的使用应理解为表示选择对象中的一者、两者或其任何组合。
术语“和/或”应理解为表示选择对象中的一者或两者。
如本文使用,术语“约”或“大约”是指相对于参考数量、水平、值、数目、频率、百分比、尺寸、大小、量、重量或长度变化了多达15%、10%、9%、8%、7%、6%、5%、4%、3%、2%或1%的数量、水平、值、数目、频率、百分比、尺寸、大小、量、重量或长度。在一个实施例中,术语“约”或“大约”是指参考数量、水平、值、数目、频率、百分比、尺寸、大小、量、重量或长度±15%、±10%、±9%、±8%、±7%、±6%、±5%、±4%、±3%、±2%或±1%的数量、水平、值、数目、频率、百分比、尺寸、大小、量、重量或长度的范围。
在一个实施例中,范围,例如1到5、约1到5或约1到约5,是指该范围所涵盖的每个数值。例如,在一个非限制性且仅仅是说明性的实施例中,范围“1到5”等同于表达1、2、3、4、5;或1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5或5.0;或1.0、1.1、1.2、1.3、1.4、1.5、1.6、1.7、1.8、1.9、2.0、2.1、2.2、2.3、2.4、2.5、2.6、2.7、2.8、2.9、3.0、3.1、3.2、3.3、3.4、3.5、3.6、3.7、3.8、3.9、4.0、4.1、4.2、4.3、4.4、4.5、4.6、4.7、4.8、4.9或5.0。
如本文使用,术语“基本上”是指为参考数量、水平、值、数目、频率、百分比、尺寸、大小、量、重量或长度的80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的数量、水平、值、数目、频率、百分比、尺寸、大小、量、重量或长度。在一个实施例中,“基本上相同”是指产生与参考数量、水平、值、数目、频率、百分比、尺寸、大小、量、重量或长度大致相同的效应(例如,生理效应)的数量、水平、值、数目、频率、百分比、尺寸、大小、量、重量或长度。
在整个本说明书中,除非上下文另有要求,否则词语“包括(comprise/comprises/comprising)”将被理解为暗示包含所述一个步骤或元件或一组步骤或元件但不排除任何其它步骤或元件或任何其它一组步骤或元件。“由……组成”表示包含并限于短语“由……组成”之后的任何内容。因此,短语“由……组成”表示所列出的元件是必需的或强制性的,并且不存在其它元件。“基本上由……组成”表示包含在该短语之后列出的任何元件,并且限于不干扰或有助于本公开中针对所列元件指定的活性或作用的其它元件。因此,短语“基本上由……组成”表示所列元件是必需的或强制性的,但不存在实质上影响所列元件的活性或作用的其它元件。
在整个本说明书中对“一个实施例(oneembodiment/anembodiment)”、“一个特定实施例”、“一个相关实施例”、“某个实施例”,“一个额外的实施例”或“一个另外的实施例”或其组合的引用表示结合该实施例描述的特定特征、结构或特性包含在至少一个实施例中。因此,在整个本说明书中各处出现的前述短语不一定都指同一实施例。此外,特定特征、结构或特性可以在一个或多个实施例中以任何合适的方式组合。还应理解,在一个实施例中对特征的肯定叙述用作在一个特定实施例中排除该特征的基础。
术语“离体”通常是指在生物体外发生的活动,例如在生物体外的人工环境中在活体组织中或活体组织上进行的实验或测量,优选具有最小的自然条件改变。在特定实施例中,“离体”程序涉及活细胞或活体组织,其取自生物体并在实验室设备中培养或调节,通常在无菌条件下,通常持续数小时或多至约24小时,但包含多至48小时或72小时,视环境而定。在某些实施例中,可以收集并冷冻这些组织或细胞,然后解冻用于离体治疗。使用活细胞或活体组织持续长于几天的组织培养实验或程序通常被认为是“体外”,但是在某些实施例中,该术语可与离体互换使用。
术语“体内”通常是指在生物体内发生的活动。在一个实施例中,在体内改造、编辑或修饰细胞基因组。
“增强”或“促进”或“增加”或“扩增”或“增效”通常是指本文考虑的核酸酶变体、基因组编辑组合物或基因组编辑的细胞产生、引发或引起相较于由媒剂或对照引起的反应更大的反应(即,生理反应)的能力。可测量的反应可以包含γ-球蛋白表达、hbf表达的增加和/或输血独立性的增加,尤其是根据本领域的认识和本文的描述所显而易见的。“增加的”或“增强的”量通常是“统计上显著的”量,并且可以包含由媒剂或对照产生的反应的1.1倍、1.2倍、1.5倍、2倍、3倍、4倍、5倍、6倍、7倍、8倍、9倍、10倍、15倍、20倍、30倍或更多倍(例如,500倍、1000倍)(包含其间的大于1的所有整数和小数点,例如1.5倍、1.6倍、1.7倍、1.8倍等)的增加。
“降低”或“削减”或“减小”或“减少”或“减轻”或“消除”或“抑制”或“减弱”通常是指本文考虑的核酸酶变体、基因组编辑组合物或基因组编辑的细胞产生、引发或引起相较于由媒剂或对照引起的反应更小的反应(即,生理反应)的能力。可测量的反应可以包含内源性β-球蛋白、输血独立性、rbc镰状化等的降低。“降低”或“减少的”量通常是“统计上显著的”量,并且可以包含由载剂或对照产生的反应(参考反应)的1.1倍、1.2倍、1.5倍、2倍、3倍、4倍、5倍、6倍、7倍、8倍、9倍、10倍、15倍、20倍、30倍或更多倍(例如,500倍、1000倍)(包含其间的大于1的所有整数和小数点,例如1.5倍、1.6倍、1.7倍、1.8倍等)的降低。
“维持(maintain/maintenance)”或“保留”或“没有改变”或“没有实质性改变”或“没有实质性降低”通常是指本文考虑的核酸酶变体、基因组编辑组合物或基因组编辑的细胞产生、引发或引起相较于由载剂或对照引起的反应基本上相似或相当的生理反应(即,下游效应)的能力。相当的反应是与参考反应没有显著差异或差异可测量的反应。
本文使用的术语“特异性结合亲和力”或“特异性结合(specificallybinds/specificallybound/specificbinding)”或“特异性靶向”描述了一种分子与另一种分子以比本底结合更大的结合亲和力结合,例如多肽的dna结合结构域结合到dna。如果结合结构域以某一亲和力或ka(即,特定结合相互作用的平衡缔合常数,以1/m为单位)(例如,所述亲和力或ka大于或等于约105m-1)结合到靶位点或与靶位点缔合,则该结合结构域“特异性结合”到该靶位点。在某些实施例中,结合结构域以大于或等于约106m-1、107m-1、108m-1、109m-1、1010m-1、1011m-1、1012m-1或1013m-1的ka结合到靶位点。“高亲和力”结合结构域是指具有至少107m-1、至少108m-1、至少109m-1、至少1010m-1、至少1011m-1、至少1012m-1、至少1013m-1或更大的ka的那些结合结构域。
可替代地,亲和力可以定义为以m为单位的特定结合相互作用的平衡解离常数(kd)(例如,10-5m到10-13m,或更小)。包括用于特定实施例中考虑的dna靶位点的一个或多个dna结合结构域的核酸酶变体的亲和力可以使用常规技术(例如,酵母细胞表面展示),或通过结合缔合,或通过使用标记配体的置换测定容易地确定。
在一个实施例中,特异性结合的亲和力比本底结合大约2倍,比本底结合大约5倍,比本底结合大约10倍,比本底结合大约20倍,比本底结合大约50倍,比本底结合大约100倍,或比本底结合大约1000倍或更多。
术语“选择性地结合(selectivelybinds/selectivelybound/selectivelybinding)”或“选择性地靶向”描述了在存在多个脱靶分子的情况下的一个分子与靶分子的优先结合(靶上结合)。在特定实施例中,he或megatal选择性地结合靶上dna结合位点比he或megatal结合脱靶dna靶结合位点多约5次、10次、15次、20次、25次、50次、100次或1000次或更频繁的次数。
“靶上”是指靶位点序列。
“脱靶”是指与靶位点序列相似但不相同的序列。
“靶位点”或“靶序列”是染色体或染色体外核酸序列,如果存在足够的结合和/或切割条件,其定义了结合分子将结合到和/或切割的核酸的一部分。当提及的是参考靶位点或靶序列的仅一条链的多核苷酸序列或seqidno.时,应理解,由核酸酶变体结合和/或切割的靶位点或靶序列是双链的并且包括参考序列及其互补序列。在一个优选实施例中,靶位点是人bcl11a基因中的序列。
“重组”是指在两个多核苷酸之间交换遗传信息的过程,包含但不限于通过非同源末端连接(nhej)和同源重组的供体捕获。出于本公开的目的,“同源重组(hr)”是指这种交换的专化形式,此类交换例如发生在通过同源定向修复(hdr)机制修复细胞中的双链断裂期间。该过程需要核苷酸序列同源性,使用“供体”分子作为模板来修复“靶”分子(即,经历双链断裂的分子),并且被不同地称为“非交换基因转换”或“短序列(tract)基因转换”,因为它导致遗传信息从供体转移到靶标。不希望受任何特定理论的束缚,此类转移可能涉及在断裂的靶标和供体之间形成的异源双链dna的错配校正和/或“合成依赖性链退火”,其中供体用于重新合成遗传信息,这将成为靶标和/或相关过程的一部分。此类专化hr通常导致靶分子序列的改变,使得供体多核苷酸序列的部分或全部掺入靶多核苷酸中。
“nhej”或“非同源末端连接”是指在不存在供体修复模板或同源序列的情况下的双链断裂的消除。nhej可能导致断裂位点处的插入和缺失。nhej由几种子途径介导,每种途径都有明显的突变后果。经典nhej途径(cnhej)需要ku/dna-pkcs/lig4/xrcc4复合物,以最少的处理将末端连接回一起,并且经常导致断裂的精确修复。替代nhej途径(altnhej)也在消除dsdna断裂方面具有活性,但是这些途径具有相当大的诱变性,并且通常导致对由插入和缺失标记的断裂的不精确修复。尽管不希望受任何特定理论的束缚,但是预期通过末端加工酶(诸如例如,核酸外切酶,例如trex2)对dsdna断裂的修饰可能使修复偏向于altnhej途径。
“切割”是指dna分子的共价骨架的断裂。切割可以通过多种方法引发,包含但不限于磷酸二酯键的酶促或化学水解。单链切割和双链切割都是可能的。由于两个不同的单链切割事件,可能发生双链切割。dna切割可能导致平末端或交错末端的产生。在某些实施例中,本文考虑的多肽和核酸酶变体(例如,归巢核酸内切酶变体、megatal等)用于靶向双链dna切割。核酸内切酶切割识别位点可位于任一dna链上。
“外源”分子是通常不存在于细胞中但通过一种或多种遗传、生物化学或其它方法引入细胞的分子。示例性外源分子包含但不限于小有机分子、蛋白质、核酸、碳水化合物、脂质、糖蛋白、脂蛋白、多糖、上述分子的任何修饰衍生物、或包括上述分子中一种或多种的任何复合物。将外源分子引入细胞的方法是本领域技术人员已知的,包含但不限于脂质介导的转移(即,脂质体,包含中性和阳离子脂质)、电穿孔、直接注射、细胞融合、粒子轰击、生物聚合物纳米粒子、磷酸钙共沉淀、deae-葡聚糖介导的转移和病毒载体介导的转移。
“内源”分子是在特定环境条件下在特定发育阶段通常存在于特定细胞中的分子。另外的内源分子可以包含蛋白质,例如内源性球蛋白。
“基因”是指编码基因产物的dna区,以及调节基因产物产生的所有dna区,无论这些调节序列是否与编码和/或转录序列相邻。基因包含但不限于启动子序列、增强子、沉默子、绝缘子、边界元件、终止子、多聚腺苷酸化序列、转录后反应元件、翻译调节序列(例如,核糖体结合位点和内部核糖体进入位点)、复制起点、基质附着位点和基因座控制区。
“基因表达”是指基因中含有的信息转化为基因产物。基因产物可以是基因的直接转录产物(例如,mrna、trna、rrna、反义rna、核酶、结构rna或任何其它类型的rna)或通过mrna翻译产生的蛋白质。基因产物还包含通过过程(例如,加帽、多聚腺苷酸化、甲基化和编辑)修饰的rna,以及通过例如甲基化、乙酰化、磷酸化、泛素化、adp-核糖基化、十四酰化和糖基化修饰的蛋白质。
如本文使用,术语“遗传改造的”或“遗传修饰的”是指额外的遗传物质以dna或rna的形式染色体或染色体外加入到细胞中的总遗传物质。遗传修饰可以靶向或非靶向细胞基因组中的特定位点。在一个实施例中,遗传修饰是位点特异性的。在一个实施例中,遗传修饰不是位点特异性的。
如本文使用,术语“基因组编辑”是指遗传物质在细胞基因组中的靶位点的取代、缺失和/或引入,其恢复、校正、破坏和/或修饰基因或基因产物的表达。特定实施例中考虑的基因组编辑包括将一种或多种核酸酶变体引入细胞中以在细胞基因组中的靶位点处或邻近靶位点生成dna损伤,任选地在存在供体修复模板的情况下。
如本文使用,术语“基因疗法”是指将额外的遗传物质引入细胞中的总遗传物质中,其恢复、校正或修饰基因或基因产物的表达,或用于表达治疗性多肽的目的。在特定实施例中,通过基因组编辑将遗传物质引入细胞的基因组中来恢复、校正、破坏或修饰基因或基因产物的表达或用于表达治疗性多肽的目的被认为是基因疗法。
c.核酸酶变体
本文特定实施例中考虑的核酸酶变体适合于基因组编辑bcl11a基因中的靶位点,并且包括一个或多个dna结合结构域和一个或多个dna切割结构域(例如,一个或多个核酸内切酶和/或核酸外切酶结构域),以及任选的本文考虑的一个或多个连接子。术语“重编程的核酸酶”、“改造的核酸酶”或“核酸酶变体”可互换使用,并且是指包括一个或多个dna结合结构域和一个或多个dna切割结构域的核酸酶,其中核酸酶已从亲本核酸酶或天然存在的核酸酶设计和/或修饰,以结合并切割bcl11a基因中,优选bcl11a基因中的gata-1结合位点中,更优选bcl11a基因的第二内含子中的共有gata-1结合位点中,甚至更优选seqidno:25(其互补序列包含共有gata-1基序wgatar)所示的靶位点中的双链dna靶序列。可以从天然存在的核酸酶或从先前的核酸酶变体设计和/或修饰核酸酶变体。特定实施例中考虑的核酸酶变体可以进一步包括一个或多个另外的功能结构域,例如,显示5'-3'核酸外切酶、5'-3'碱性核酸外切酶、3'-5'核酸外切酶(例如,trex2)、5'瓣状核酸内切酶、解旋酶、模板依赖性dna聚合酶或模板非依赖性dna聚合酶活性的末端加工酶的末端加工酶结构域。
结合并切割bcl11a基因中的靶序列的核酸酶变体的说明性实例包含但不限于归巢核酸内切酶变体(大范围核酸酶变体)和megatal。
1.归巢核酸内切酶(大范围核酸酶)变体
在各个实施例中,归巢核酸内切酶或大范围核酸酶被重编程以将双链断裂(dsb)引入bcl11a基因中的红系特异性增强子中,优选bcl11a基因中的gata-1结合位点中,更优选bcl11a基因的第二内含子中的共有gata-1结合位点中,甚至更优选seqidno:25(其互补序列包含共有gata-1基序wgatar)所示的靶位点中。“归巢核酸内切酶”和“大范围核酸酶”可互换使用,是指识别12到45个碱基对切割位点的天然存在的核酸酶,并且其通常基于序列和结构基序分为五个家族:laglidadg、giy-yig、hnh、his-cys盒和pd-(d/e)xk。
“参考归巢核酸内切酶”或“参考大范围核酸酶”是指野生型归巢核酸内切酶或在自然界中存在的归巢核酸内切酶。在一个实施例中,“参考归巢核酸内切酶”是指经过修饰以增加基础活性的野生型归巢核酸内切酶。
“改造的归巢核酸内切酶”、“重编程的归巢核酸内切酶”、“归巢核酸内切酶变体”、“改造的大范围核酸酶”、“重编程的大范围核酸酶”或“大范围核酸酶变体”是指包括一个或多个dna结合结构域和一个或多个dna切割结构域的归巢核酸内切酶,其中归巢核酸内切酶已经从亲本归巢核酸内切酶或天然存在的归巢核酸内切酶设计和/或修饰,以结合并切割bcl11a基因中的dna靶序列。可以从天然存在的归巢核酸内切酶或从另一种归巢核酸内切酶变体设计和/或修饰归巢核酸内切酶变体。特定实施例中考虑的归巢核酸内切酶变体可以进一步包括一个或多个另外的功能结构域,例如,显示5'-3'核酸外切酶、5'-3'碱性核酸外切酶、3'-5'核酸外切酶(例如,trex2)、5'瓣状核酸内切酶、解旋酶、模板依赖性dna聚合酶或模板非依赖性dna聚合酶活性的末端加工酶的末端加工酶结构域。
归巢核酸内切酶(he)变体在自然界中不存在,并且可以通过重组dna技术或通过随机诱变获得。he变体可以通过在天然存在的he或he变体中进行一个或多个氨基酸改变(例如,突变、取代、加入或缺失一个或多个氨基酸)来获得。在特定实施例中,he变体包括dna识别界面的一个或多个氨基酸改变。
特定实施例中考虑的he变体可以进一步包括一个或多个连接子和/或另外的功能结构域,例如,显示5'-3'核酸外切酶、5'-3'碱性核酸外切酶、3'-5'核酸外切酶(例如,trex2)、5'瓣状核酸内切酶、解旋酶、模板依赖性dna聚合酶或模板非依赖性dna聚合酶活性的末端加工酶的末端加工酶结构域。在特定实施例中,将he变体引入具有显示5'-3'核酸外切酶、5'-3'碱性核酸外切酶、3'-5'核酸外切酶(例如,trex2)、5'瓣状核酸内切酶、解旋酶、模板依赖性dna聚合酶或模板非依赖性dna聚合酶活性的末端加工酶的t细胞中。he变体和3'加工酶可以分开引入,例如,在不同的载体或分开的mrna中,或者一起引入,例如作为融合蛋白,或在由病毒自切割肽或ires元件分开的多顺反子构建体中引入。
“dna识别界面”是指与核酸靶碱基相互作用的he氨基酸残基以及相邻的那些残基。对于每个he,dna识别界面包括侧链到侧链和侧链到dna接触的广泛网络,其中大多数必须是识别特定核酸靶序列所独有的。因此,对应于特定核酸序列的dna识别界面的氨基酸序列显著不同,并且是任何天然he或he变体的特征。作为非限制性实例,可以通过构建he变体文库来衍生特定实施例中考虑的he变体,其中位于天然he(或先前生成的he变体)的dna识别界面中的一个或多个氨基酸残基是各不相同的。可以使用切割测定针对相对于每个预测的bcl11a靶位点的靶切割活性来筛选文库(参见例如jarjour等人,2009年,《核酸研究(nuc.acidsres.)》,37(20):6871-6880)。
laglidadg归巢核酸内切酶(lhe)是研究最深入的大范围核酸酶家族,主要编码于古细菌和绿藻和真菌中的细胞器dna中,并显示出最高的整体dna识别特异性。lhe包括每条蛋白质链的一个或两个laglidadg催化基序,并分别起同二聚体或单链单体的作用。laglidadg蛋白的结构研究标识了高度保守的核心结构(stoddard,2005年),其特征在于αββαββα折叠,其中laglidadg基序属于该折叠的第一螺旋。lhe的高效和特异性切割代表了蛋白质支架,以衍生新的高度特异性的核酸内切酶。然而,改造lhe以结合并切割非天然或非规范靶位点需要在靶位点中多达三分之二的碱基对位置处选择合适的lhe支架,检查靶基因座,选择推定的靶位点,并广泛改变lhe以改变其dna接触点和切割特异性。
在一个实施例中,可以设计重编程的lhe或lhe变体的lhe包含但不限于i-crei和i-scei。
可以设计重编程的lhe或lhe变体的lhe的说明性实例包含但不限于i-aabmi、i-aaemi、i-anii、i-apami、i-capiii、i-capiv、i-ckami、i-cpami、i-cpamii、i-cpamiii、i-cpamiv、i-cpamv、i-cpav、i-crami、i-ejemi、i-gpemi、i-gpii、i-gzemi、i-gzemii、i-gzemiii、i-hjemi、i-ltrii、i-ltri、i-ltrwi、i-mpemi、i-mvemi、i-ncrii、i-ncrl、i-ncrmi、i-ohemi、i-onui、i-osomi、i-osomii、i-osomiii、i-osomiv、i-panmi、i-panmii、i-panmiii、i-pnomi、i-scumi、i-smami、i-sscmi和i-vdi141i。
在一个实施例中,重编程的lhe或lhe变体选自由以下组成的组:i-cpami变体、i-hjemi变体、i-onui变体、i-panmi变体和i-smami变体。
在一个实施例中,重编程的lhe或lhe变体是i-onui变体。参见例如seqidno:6-19。
在一个实施例中,靶向bcl11a基因的重编程的i-onuilhe或i-onui变体由天然i-onui或其生物活性片段(seqidno:1-5)生成。在一个优选实施例中,由现有的i-onui变体生成靶向人bcl11a基因的重编程的i-onuilhe或i-onui变体。在一个实施例中,针对seqidno:25所示的人bcl11a基因靶位点生成重编程的i-onuilhe。
在一个特定实施例中,结合并切割人bcl11a基因的重编程的i-onuilhe或i-onui变体在dna识别界面中包括一个或多个氨基酸取代。在特定实施例中,结合并切割人bcl11a基因的i-onuilhe包括与i-onui(taekuchi等人,2011年,《美国国家科学院学报(procnatlacadsciu.s.a.)》,2011年8月9日;108(32):13077-13082)或seqidno:6-19所示的i-onuilhe变体或其另外的变体的dna识别界面至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的序列同一性。
在一个实施例中,结合并切割人bcl11a基因的i-onuilhe包括与i-onui(taekuchi等人,2011年,《美国国家科学院学报(procnatlacadsciu.s.a.)》,2011年8月9日;108(32):13077-13082)或seqidno:6-19所示的i-onuilhe变体或其另外的变体的dna识别界面至少70%、更优选至少80%、更优选至少85%、更优选至少90%、更优选至少95%、更优选至少97%、更优选至少99%的序列同一性。
在一个特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括seqidno:1-19中任一项所示的i-onui的dna识别界面中的一个或多个氨基酸取代或修饰。
在一个特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括在dna识别界面中的一个或多个氨基酸取代或修饰,特别是在位于i-onui(seqidno:1-5)、seqidno:6-19所示的i-onui变体或其另外的变体的第24位到第50位、第68位到第82位、第180位到第203位以及第223位到第240位的亚结构域中。
在一个特定实施例中,结合并切割人bcl11a基因的i-onuilhe包括在dna识别界面中的选自由以下组成的组的氨基酸位置处的一个或多个氨基酸取代或修饰:i-onui(seqidno:1-5)、seqidno:6-19所示的i-onui变体或其另外的变体的第19位、第24位、第26位、第28位、第30位、第32位、第34位、第35位、第36位、第37位、第38位、第40位、第42位、第44位、第46位、第48位、第68位、第70位、第72位、第75位、第76位、第77位、第78位、第80位、第82位、第168位、第180位、第182位、第184位、第186位、第188位、第189位、第190位、第191位、第192位、第193位、第195位、第197位、第199位、第201位、第203位、第223位、第225位、第227位、第229位、第231位、第232位、第234位、第236位、第238位和第240位。
在一个特定实施例中,结合并切割人bcl11a基因的i-onuilhe包括在dna识别界面中的5个、10个、15个、20个、25个、30个、35个或40个或更多个氨基酸取代或修饰,特别是在位于i-onui(seqidno:1-5)、seqidno:6-19所示的i-onui变体或其另外的变体的第24位到第50位、第68位到第82位、第180位到第203位以及第223位到第240位的亚结构域中。
在一个特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括在dna识别界面中的选自由以下组成的组的氨基酸位置处的5个、10个、15个、20个、25个、30个、35个或40个或更多个氨基酸取代或修饰:i-onui(seqidno:1-5)、seqidno:6-19所示的i-onui变体或其另外的变体的第19位、第24位、第26位、第28位、第30位、第32位、第34位、第35位、第36位、第37位、第38位、第40位、第42位、第44位、第46位、第48位、第68位、第70位、第72位、第75位、第76位、第77位、第78位、第80位、第82位、第168位、第180位、第182位、第184位、第186位、第188位、第189位、第190位、第191位、第192位、第193位、第195位、第197位、第199位、第201位、第203位、第223位、第225位、第227位、第229位、第231位、第232位、第234位、第236位、第238位和第240位。
在一个实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括在位于整个i-onui序列内任何位置的其它位置处的一个或多个氨基酸取代或修饰。可以被取代和/或修饰的残基包含但不限于与核酸靶标接触或与核酸骨架或与核苷酸碱基直接或通过水分子相互作用的氨基酸。在一个非限制性实例中,本文考虑的结合并切割人bcl11a基因的i-onuilhe变体包括至少一个位置处的一个或多个取代和/或修饰,优选至少5个、优选至少10个、优选至少15个、优选至少20个、更优选至少25个、更优选至少30个、甚至更优选至少35个、或甚至更优选至少40个取代和/或修饰,所述至少一个位置选自由以下位置组成的位置组:关于seqidno:1-19中任一个的第26位、第28位、第30位、第32位、第34位、第35位、第36位、第37位、第40位、第41位、第42位、第44位、第68位、第70位、第72位、第76位、第78位、第80位、第82位、第138位、第143位、第159位、第178位、第180位、第184位、第186位、第189位、第190位、第191位、第192位、第193位、第195位、第201位、第203位、第207位、第223位、第225位、第227位、第232位、第236位、第238位和第240位。
在特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括选自由以下组成的组的氨基酸位置处的至少5个、至少15个、优选至少25个、更优选至少35个、或甚至更优选至少40个或更多个氨基酸取代:seqidno:1-19所示的i-onuilhe氨基酸序列或其生物活性片段的第26位、第28位、第30位、第32位、第34位、第35位、第36位、第37位、第40位、第41位、第42位、第44位、第48位、第50位、第53位、第68位、第70位、第72位、第76位、第78位、第80位、第82位、第138位、第143位、第159位、第178位、第180位、第184位、第186位、第189位、第190位、第191位、第192位、第193位、第195位、第201位、第203位、第207位、第223位、第225位、第227位、第232位、第236位、第238位和第240位。
在另外的实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括至少5个、至少15个、优选至少25个、更优选至少35个、或甚至更优选至少40个或更多个以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、l26r、l26y、r28s、r28g、r30q、r30h、n32r、n32s、n32k、n33s、k34d、k34n、s35y、s36a、v37t、s40r、t41i、e42h、e42r、g44t、g44r、t48i、t48g、t48v、h50r、d53e、v68k、v68r、a70n、a70e、a70n、a70q、a70l、a70s、s72a、s72t、s72v、s72m、a76l、a76h、a76r、s78q、k80r、k80v、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44t、v68k、a70n、s72a、a76l、s78q、k80r、t82y、l138m、t143n、s159p、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44t、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在一些实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r30q、n32s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44t、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32k、k34n、s35y、s36a、v37t、s40r、t41i、e42h、g44t、t48i、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42r、g44t、t48i、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在另外的实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28g、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42r、g44t、h50r、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30h、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、v68k、a70n、s72t、a76h、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26r、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、v68k、a70n、s72ta76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26y、r28s、r30q、n32r、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、d53e、v68r、a70e、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在一些实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、d53e、v68k、a70n、s72t、a76l、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、t48g、v68k、s72v、a76r、s78q、k80v、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在某些实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、t48g、v68k、a70q、s72m、a76r、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、t48g、v68k、a70l、s72v、a76h、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括以下氨基酸取代:i-onui(seqidno:1-5)或seqidno:6-19中任一个所示的i-onui变体、其生物活性片段和/或其另外的变体的l26v、r28s、r30q、n32r、n33s、k34d、s35y、s36a、v37t、s40r、t41i、e42h、g44r、t48v、v68k、a70s、s72v、a76h、s78q、k80r、t82y、l138m、t143n、s159p、e178d、c180s、n184r、i186r、k189n、s190v、k191n、l192a、g193r、q195r、s201e、t203s、k207r、y223h、k225y、k227g、f232r、d236q、v238r和t240e。
在特定实施例中,结合并切割人bcl11a基因的i-onuilhe变体包括与seqidno:6-19中任一个所示的氨基酸序列或其生物活性片段具有至少80%、优选至少85%、更优选至少90%、或甚至更优选至少95%同一性的氨基酸序列。
在特定实施例中,i-onuilhe变体包括seqidno:6-19中任一个所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:6所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:7所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:8所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:9所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:10所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:11所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:12所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:13所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:14所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:15所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:16所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:17所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:18所示的氨基酸序列或其生物活性片段。
在特定实施例中,i-onuilhe变体包括seqidno:19所示的氨基酸序列或其生物活性片段。
2.megatal
在各个实施例中,将包括归巢核酸内切酶变体的megatal重编程以将双链断裂(dsb)引入bcl11a基因中的红系特异性增强子中,优选bcl11a基因中的gata-1结合位点中,更优选bcl11a基因的第二内含子中的共有gata-1结合位点中,甚至更优选seqidno:25(其互补序列包含共有gata-1基序wgatar)所示的靶位点中。“megatal”是指包括taledna结合结构域和归巢核酸内切酶变体的多肽,其结合并切割bcl11a基因中的dna靶序列,并且任选地包括一个或多个连接子和/或另外的功能结构域,例如,显示5'-3'核酸外切酶、5'-3'碱性核酸外切酶、3'-5'核酸外切酶(例如,trex2)、5'瓣状核酸内切酶、解旋酶或模板非依赖性dna聚合酶活性的末端加工酶的末端加工酶结构域。
在特定实施例中,可以将megatal与显示5'-3'核酸外切酶、5'-3'碱性核酸外切酶、3'-5'核酸外切酶(例如,trex2)、5'瓣状核酸内切酶、解旋酶、模板依赖性dna聚合酶或模板非依赖性dna聚合酶活性的末端加工酶一起引入细胞中。megatal和3'加工酶可以分开引入,例如,在不同的载体或分开的mrna中,或者一起引入,例如作为融合蛋白,或在由病毒自切割肽或ires元件分开的多顺反子构建体中引入。
“taledna结合结构域”是类转录激活因子效应子(tale或tal效应子)的dna结合部分,其模拟植物转录激活因子以操纵植物转录组(参见例如kay等人,2007年,《科学(science)》,318:648-651)。特定实施例中考虑的taledna结合结构域是从头改造的或从天然存在的tale改造的,所述天然存在的tale例如来自野油菜黄单胞菌疮痂致病变体(xanthomonascampestrispv.vesicatoria)、加德纳黄单胞菌(xanthomonasgardneri)、半透明黄单胞菌(xanthomonastranslucens)、地毯草黄单胞菌(xanthomonasaxonopodis)、穿孔黄单胞菌(xanthomonasperforans)、苜蓿黄单胞菌(xanthomonasalfalfa)、柑橘黄单胞菌(xanthomonascitri)、易疮痂黄单胞菌(xanthomonaseuvesicatoria)和水稻黄单胞菌(xanthomonasoryzae)的avrbs3以及来自青枯雷尔氏菌(ralstoniasolanacearum)的brg11和hpx17。用于衍生和设计dna结合结构域的tale蛋白的说明性实例公开在美国专利第9,017,967号和其中引用的参考文献中,所有这些文献都通过引用整体并入本文。
在特定实施例中,megatal包括taledna结合结构域,其包括一个或多个重复单元,所述重复单元参与taledna结合结构域与其相应的靶dna序列的结合。单个“重复单元”(也称为“重复”)的长度通常为33到35个氨基酸。每个taledna结合结构域重复单元包含构成重复可变二残基(rvd)的1个或2个dna结合残基,通常在重复的第12位和/或第13位。已经确定了用于这些taledna结合结构域的dna识别的天然(规范)密码,使得第12位和第13位的hd序列会结合到胞嘧啶(c),ng结合到t,ni结合到a,nn结合到g或a,并且ng结合到t。在某些实施例中,考虑了非规范(非典型)rvd。
适用于特定实施例中考虑的特定megatal的非规范rvd的说明性实例包含但不限于用于识别鸟嘌呤(g)的hh、kh、nh、nk、nq、rh、rn、ss、nn、sn、kn;用于识别腺嘌呤(a)的ni、ki、ri、hi、si;用于识别胸腺嘧啶(t)的ng、hg、kg、rg;用于识别胞嘧啶(c)的rd、sd、hd、nd、kd、yg;用于识别a或g的nv、hn;以及用于识别a或t或g或c的h*、ha、ka、n*、na、nc、ns、ra、s*,其中(*)表示不存在第13位的氨基酸。适用于特定实施例中考虑的特定megatal的rvd的另外的说明性实例进一步包含美国专利第8,614,092号中公开的那些,其通过引用整体并入本文。
在特定实施例中,本文考虑的megatal包括taledna结合结构域,其包括3到30个重复单元。在某些实施例中,megatal包括3个、4个、5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个或30个taledna结合结构域重复单元。在一个优选实施例中,本文考虑的megatal包括taledna结合结构域,其包括5到15个重复单元,更优选7到15个重复单元,更优选9到15个重复单元,更优选9个、10个、11个、12个、13个、14个或15个重复单位。
在特定实施例中,本文考虑的megatal包括taledna结合结构域和另外的单截短tale重复单元,所述taledna结合结构域包括3到30个重复单元,所述另外的单截短tale重复单元包括位于一组tale重复单元的c-末端的20个氨基酸,即其为另外的c-末端半taledna结合结构域重复单元(本文其它地方公开的c-帽的氨基酸-20到-1,见下文)。因此,在特定实施例中,本文考虑的megatal包括taledna结合结构域,其包括3.5到30.5个重复单元。在某些实施例中,megatal包括3.5个、4.5个、5.5个、6.5个、7.5个、8.5个、9.5个、10.5个、11.5个、12.5个、13.5个、14.5个、15.5个、16.5个、17.5个、18.5个、19.5个、20.5个、21.5个、22.5个、23.5个、24.5个、25.5个、26.5个、27.5个、28.5个、29.5个或30.5个taledna结合结构域重复单元。在一个优选实施例中,本文考虑的megatal包括taledna结合结构域,其包括5.5到15.5个重复单元,更优选7.5到15.5个重复单元,更优选9.5到15.5个重复单元,更优选9.5个、10.5个、11.5个、12.5个、13.5个、14.5个或15.5个重复单位。
在特定实施例中,megatal包括tal效应子构造,其包括“n-末端结构域(ntd)”多肽、一个或多个tale重复结构域/单元、“c-末端结构域(ctd)”多肽和归巢核酸内切酶变体。在一些实施例中,ntd、tale重复和/或ctd结构域来自相同物种。在其它实施例中,ntd、tale重复和/或ctd结构域中的一个或多个来自不同物种。
如本文使用,术语“n-末端结构域(ntd)”多肽是指天然存在的taledna结合结构域的n-末端部分或片段侧翼的序列。如果存在,ntd序列可以是任何长度,只要taledna结合结构域重复单元保留结合dna的能力即可。在特定实施例中,ntd多肽包括在taledna结合结构域的n-末端的至少120到至少140个或更多个氨基酸(0是最n-末端重复单元的氨基酸1)。在特定实施例中,ntd多肽包括在taledna结合结构域的n-末端的至少约120个、121个、122个、123个、124个、125个、126个、127个、128个、129个、130个、131个、132个、133个、134个、135个、136个、137个、138个、139个或至少140个氨基酸。在一个实施例中,本文考虑的megatal包括ntd多肽,其为黄单胞菌tale蛋白的至少约氨基酸+1到+122到至少约+1到+137(0是最n-末端重复单元的氨基酸1)。在特定实施例中,ntd多肽包括在黄单胞菌tale蛋白的taledna结合结构域的n-末端的至少约122个、123个、124个、125个、126个、127个、128个、129个、130个、131个、132个、133个、134个、135个、136个或137个氨基酸。在一个实施例中,本文考虑的megatal包括ntd多肽,其为雷尔氏菌tale蛋白的至少氨基酸+1到+121(0是最n-末端重复单元的氨基酸1)。在特定实施例中,ntd多肽包括在雷尔氏菌tale蛋白的taledna结合结构域的n-末端的至少约121个、122个、123个、124个、125个、126个、127个、128个、129个、130个、131个、132个、133个、134个、135个、136个或137个氨基酸。
如本文使用,术语“c-末端结构域(ctd)”多肽是指天然存在的taledna结合结构域的c-末端部分或片段侧翼的序列。如果存在,ctd序列可以是任何长度,只要taledna结合结构域重复单元保留结合dna的能力即可。在特定实施例中,ctd多肽包括在taledna结合结构域的最后一个完整重复的c-末端的至少20到至少85个或更多个氨基酸(前20个氨基酸是在最后一个c-末端完整重复单元的c-末端的半重复单元)。在特定实施例中,ctd多肽包括在taledna结合结构域的最后一个完整重复的c-末端的至少约20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、31个、32个、33个、34个、35个、36个、37个、38个、39个、40个、41个、42个、443个、44个、45个、46个、47个、48个、49个、50个、51个、52个、53个、54个、55个、56个、57个、58个、59个、60个、61个、62个、63个、64个、65个、66个、67个、68个、69个、70个、71个、72个、73个、74个、75个、76个、77个、78个、79个、80个、81个、82个、83个、84个或至少85个氨基酸。在一个实施例中,本文考虑的megatal包括ctd多肽,其为黄单胞菌tale蛋白的至少约氨基酸-20到-1(-20是最后一个c-末端完全重复单元的c-末端的半重复单元的氨基酸1)。在特定实施例中,ctd多肽包括在黄单胞菌tale蛋白的taledna结合结构域的最后一个完全重复的c-末端的至少约20个、19个、18个、17个、16个、15个、14个、13个、12个、11个、10个、9个、8个、7个、6个、5个、4个、3个、2个或1个氨基酸。在一个实施例中,本文考虑的megatal包括ctd多肽,其为雷尔氏菌tale蛋白的至少约氨基酸-20到-1(-20是最后一个c-末端完全重复单元的c-末端的半重复单元的氨基酸1)。在特定实施例中,ctd多肽包括在雷尔氏菌tale蛋白的taledna结合结构域的最后一个完全重复的c-末端的至少约20个、19个、18个、17个、16个、15个、14个、13个、12个、11个、10个、9个、8个、7个、6个、5个、4个、3个、2个或1个氨基酸。
在特定实施例中,本文考虑的megatal包括融合多肽,其包括被改造以结合靶序列的taledna结合结构域、被重编程以结合并切割靶序列的归巢核酸内切酶和任选的ntd和/或ctd多肽,所述taledna结合结构域、归巢核酸内切酶和任选的ntd和/或ctd多肽任选地通过一个或多个本文其它地方考虑的连接子多肽彼此连接。不希望受任何特定理论的束缚,预期包括taledna结合结构域和任选的ntd和/或ctd多肽的megatal与连接子多肽融合,其进一步与归巢核酸内切酶变体融合。因此,taledna结合结构域结合距离由归巢核酸内切酶变体的dna结合结构域结合的靶序列约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、11个、12个、13个、14个或15个核苷酸之内的dna靶序列。以这种方式,本文考虑的megatal增加了基因组编辑的特异性和效率。
在一个实施例中,megatal包括归巢核酸内切酶变体和taledna结合结构域,所述taledna结合结构域结合在重编程的归巢核酸内切酶的结合位点上游约4个、5个或6个核苷酸(优选6个核苷酸)内的核苷酸序列。
在一个实施例中,megatal包括归巢核酸内切酶变体和taledna结合结构域,所述taledna结合结构域结合seqidno:26所示的核苷酸序列,其在由归巢核酸内切酶变体(seqidno:25)结合并切割的核苷酸序列的上游6个核苷酸处。在优选实施例中,megatal靶序列是seqidno:27。
在特定实施例中,本文考虑的megatal包括一个或多个taledna结合重复单元和从lhe设计或重编程的lhe变体,所述lhe变体选自由以下组成的组:i-aabmi、i-aaemi、i-anii、i-apami、i-capiii、i-capiv、i-ckami、i-cpami、i-cpamii、i-cpamiii、i-cpamiv、i-cpamv、i-cpav、i-crami、i-ejemi、i-gpemi、i-gpii、i-gzemi、i-gzemii、i-gzemiii、i-hjemi、i-ltrii、i-ltri、i-ltrwi、i-mpemi、i-mvemi、i-ncrii、i-ncrl、i-ncrmi、i-ohemi、i-onui、i-osomi、i-osomii、i-osomiii、i-osomiv、i-panmi、i-panmii、i-panmiii、i-pnomi、i-scumi、i-smami、i-sscmi、i-vdi141i及其变体,或优选i-cpami、i-hjemi、i-onui、i-panmi、smami及其变体,或更优选i-onui及其变体。
在特定实施例中,本文考虑的megatal包括ntd、一个或多个taledna结合重复单元、ctd和lhe变体,所述lhe变体选自由以下组成的组:i-aabmi、i-aaemi、i-anii、i-apami、i-capiii、i-capiv、i-ckami、i-cpami、i-cpamii、i-cpamiii、i-cpamiv、i-cpamv、i-cpav、i-crami、i-ejemi、i-gpemi、i-gpii、i-gzemi、i-gzemii、i-gzemiii、i-hjemi、i-ltrii、i-ltri、i-ltrwi、i-mpemi、i-mvemi、i-ncrii、i-ncrl、i-ncrmi、i-ohemi、i-onui、i-osomi、i-osomii、i-osomiii、i-osomiv、i-panmi、i-panmii、i-panmiii、i-pnomi、i-scumi、i-smami、i-sscmi、i-vdi141i及其变体,或优选i-cpami、i-hjemi、i-onui、i-panmi、smami及其变体,或更优选i-onui及其变体。
在特定实施例中,本文考虑的megatal包括ntd、约9.5到约15.5个taledna结合重复单元和lhe变体,所述lhe变体选自由以下组成的组:i-aabmi、i-aaemi、i-anii、i-apami、i-capiii、i-capiv、i-ckami、i-cpami、i-cpamii、i-cpamiii、i-cpamiv、i-cpamv、i-cpav、i-crami、i-ejemi、i-gpemi、i-gpii、i-gzemi、i-gzemii、i-gzemiii、i-hjemi、i-ltrii、i-ltri、i-ltrwi、i-mpemi、i-mvemi、i-ncrii、i-ncrl、i-ncrmi、i-ohemi、i-onui、i-osomi、i-osomii、i-osomiii、i-osomiv、i-panmi、i-panmii、i-panmiii、i-pnomi、i-scumi、i-smami、i-sscmi、i-vdi141i及其变体,或优选i-cpami、i-hjemi、i-onui、i-panmi、smami及其变体,或更优选i-onui及其变体。
在特定实施例中,本文考虑的megatal包括约122个氨基酸到137个氨基酸的ntd、约9.5个、约10.5个、约11.5个、约12.5个、约13.5个、约14.5个或约15.5个结合重复单元、约20个氨基酸到约85个氨基酸的ctd和i-onuilhe变体。在特定实施例中,ntd、dna结合结构域和ctd中的任何一个、两个或全部可以从相同的物种或不同的物种以任何合适的组合设计。
在特定实施例中,本文考虑的megatal包括seqidno:20或21中任一个所示的氨基酸序列。
在特定实施例中,本文考虑的megatal-trex2融合蛋白包括seqidno:22或23所示的氨基酸序列。
在某些实施例中,megatal包括taledna结合结构域,并且i-onuilhe变体结合并切割seqidno:27所示的核苷酸序列。
3.末端加工酶
特定实施例中考虑的基因组编辑组合物和方法包括使用核酸酶变体和末端加工酶编辑细胞基因组。在特定实施例中,单个多核苷酸编码归巢核酸内切酶变体和末端加工酶,其由连接子、自切割肽序列(例如,2a序列)分开,或由ires序列分开。在特定实施例中,基因组编辑组合物包括编码核酸酶变体的多核苷酸和分开的编码末端加工酶的多核苷酸。
术语“末端加工酶”是指修饰多核苷酸链的暴露末端的酶。多核苷酸可以是双链dna(dsdna)、单链dna(ssdna)、rna、dna和rna的双链杂交体和合成dna(例如,含有除a、c、g和t以外的碱基)。末端加工酶可以通过加入一个或多个核苷酸,去除一个或多个核苷酸,去除或修饰磷酸根基团和/或去除或修饰羟基基团来修饰暴露的多核苷酸链末端。末端加工酶可以修饰核酸内切酶切割位点处的末端或通过其它化学或机械手段生成的末端,所述其它化学或机械手段例如剪切(例如,通过细隔距针、加热、超声处理、迷你珠滚磨和雾化)、电离辐射、紫外线辐射、氧自由基、化学水解和化疗剂。
在特定实施例中,特定实施例中考虑的基因组编辑组合物和方法包括使用归巢核酸内切酶变体或megatal和dna末端加工酶编辑细胞基因组。
术语“dna末端加工酶”是指修饰dna的暴露末端的酶。dna末端加工酶可以修饰平末端或交错末端(具有5'或3'突出端的末端)。dna末端加工酶可以修饰单链或双链dna。dna末端加工酶可以修饰核酸内切酶切割位点处的末端或通过其它化学或机械手段生成的末端,所述其它化学或机械手段例如剪切(例如,通过细隔距针、加热、超声处理、迷你珠滚磨和雾化)、电离辐射、紫外线辐射、氧自由基、化学水解和化疗剂。dna末端加工酶可以通过加入一个或多个核苷酸,去除一个或多个核苷酸,去除或修饰磷酸根基团和/或去除或修饰羟基基团来修饰暴露的dna末端。
适用于本文考虑的特定实施例的dna末端加工酶的说明性实例包含但不限于:5'-3'核酸外切酶、5'-3'碱性核酸外切酶、3'-5'核酸外切酶、5'瓣状核酸内切酶、解旋酶、磷酸酶、水解酶和模板非依赖性dna聚合酶。
适用于本文考虑的特定实施例的dna末端加工酶的另外的说明性实例包含但不限于trex2、trex1、不含跨膜结构域的trex1、apollo、artemis、dna2、exo1、exot、exoiii、fen1、fan1、mreii、rad2、rad9、tdt(末端脱氧核苷酸转移酶)、pnkp、rece、recj、recq、λ核酸外切酶、sox、牛痘dna聚合酶、核酸外切酶i、核酸外切酶iii、核酸外切酶vii、ndk1、ndk5、ndk7、ndk8、wrn、t7-核酸外切酶基因6、禽类成髓细胞瘤病毒整合蛋白(in)、bloom、热敏磷酸酶、碱性磷酸酶、多核苷酸激酶(pnk)、apei、绿豆核酸酶、hex1、ttrap(tdp2)、sgs1、sae2、cup、polμ、polλ、mus81、eme1、eme2、slx1、slx4和ul-12。
在特定实施例中,用于编辑本文考虑的细胞基因组的基因组编辑组合物和方法包括多肽,其包括归巢核酸内切酶变体或megatal和核酸外切酶。术语“核酸外切酶”是指通过断裂在3'或5'末端的磷酸二酯键的水解反应在多核苷酸链的末端处切割磷酸二酯键的酶。
适用于本文考虑的特定实施例的核酸外切酶的说明性实例包含但不限于:hexoi、酵母exoi、大肠杆菌exoi、htrex2、小鼠trex2、大鼠trex2、htrex1、小鼠trex1、大鼠trex1和大鼠trex1。
在特定实施例中,dna末端加工酶是3'或5'核酸外切酶,优选trex1或trex2,更优选trex2,甚至更优选人或小鼠trex2。
d.靶位点
与天然存在的核酸酶相比,特定实施例中考虑的核酸酶变体可以设计为结合任何合适的靶序列并且可以具有新的结合特异性。在特定实施例中,靶位点是基因的调节区,包含但不限于启动子、增强子、阻遏子元件等。在特定实施例中,靶位点是基因或剪接位点的编码区。在某些实施例中,设计核酸酶变体以下调或降低基因的表达。在特定实施例中,可以设计核酸酶变体和供体修复模板以删除期望的靶序列。
在各个实施例中,核酸酶变体结合并切割b细胞cll/淋巴瘤11a(bcl11a)基因中的靶序列。bcl11a基因编码类似于小鼠bcl11a/evi9蛋白的c2h2型锌指转录因子。bcl11a是一种转录阻遏子,其在调节球蛋白基因表达中起作用。在胎儿发育中,不表达全长形式的bcl11a,并且红系细胞产生γ-球蛋白,其与α-球蛋白复合以形成胎儿血红蛋白(hbf)。在出生时,bcl11a表达在红系细胞中增加,与γ-球蛋白启动子中的转录元件结合并抑制或阻遏γ-球蛋白表达,这与β-球蛋白表达增加有关。以γ-球蛋白为代价的β-球蛋白表达的增加导致从hbf到hba(两种β-球蛋白/两种α-球蛋白)的“球蛋白转换”。然而,在具有导致血红蛋白病的β-球蛋白基因中的一个或多个突变的受试者中,重新转换γ-球蛋白基因表达并且以突变的β-球蛋白基因表达为代价将潜在地治疗血红蛋白病。一种解决方案是降低bcl11a表达以去阻遏γ-球蛋白基因表达并减少突变的β-球蛋白基因表达。
在特定实施例中,归巢核酸内切酶变体或megatal将双链断裂(dsb)引入bcl11a基因中的红系特异性增强子中,优选bcl11a基因中的gata-1结合位点中,更优选bcl11a基因的第二内含子中的共有gata-1结合位点中,甚至更优选seqidno:25(其互补序列包含共有gata-1基序wgatar)所示的靶位点中。在特定实施例中,重编程的核酸酶或megatal包括i-onuilhe变体,其通过切割与共有gata-1结合基序(wgataa)互补的链上的序列“ttat”来将双链断裂引入在bcl11a基因的第二内含子中的gata-1位点处。
在一个优选实施例中,归巢核酸内切酶变体或megatal切割双链dna并将dsb引入seqidno:25或27所示的多核苷酸序列中。
在一个优选实施例中,bcl11a基因是人bcl11a基因。
e.供体修复模板
核酸酶变体可以用于将dsb引入靶序列中;可以在存在一个或多个供体修复模板的情况下,通过同源定向修复(hdr)机制修复dsb。在特定实施例中,供体修复模板用于将序列插入基因组中。在特定优选实施例中,供体修复模板用于删除或修复基因组中的基因组序列。
在各个实施例中,通过用包括供体修复模板的腺相关病毒(aav)、逆转录病毒(例如,慢病毒、idlv等)、单纯疱疹病毒、腺病毒或牛痘病毒载体转导细胞,将供体修复模板引入造血细胞中,例如造血干细胞或祖细胞或cd34+细胞中。
在特定实施例中,供体修复模板包括位于dsb位点侧翼的一个或多个同源臂。
如本文使用,术语“同源臂”是指供体修复模板中与以由核酸酶在靶位点处引入的dna断裂为侧翼的dna序列相同或几乎相同的核酸序列。在一个实施例中,供体修复模板包括5'同源臂,其包括与dna断裂位点的dna序列5'相同或几乎相同的核酸序列。在一个实施例中,供体修复模板包括3'同源臂,其包括与dna断裂位点的dna序列3'相同或几乎相同的核酸序列。在一个优选实施例中,供体修复模板包括5'同源臂和3'同源臂。供体修复模板可以包括与dsb位点紧邻的基因组序列的同源性,或与来自dsb位点的任何数目碱基对内的基因组序列的同源性。在一个实施例中,供体修复模板包括与基因组序列同源约5bp、约10bp、约25bp、约50bp、约100bp、约250bp、约500bp、约1000bp、约2500bp、约5000bp、约10000bp或更多的核酸序列,包含任何中间长度的同源序列。
可以独立地选择特定实施例中考虑的同源臂的合适长度的说明性实例,包含但不限于:约100bp、约200bp、约300bp、约400bp、约500bp、约600bp、约700bp、约800bp、约900bp、约1000bp、约1100bp、约1200bp、约1300bp、约1400bp、约1500bp、约1600bp、约1700bp、约1800bp、约1900bp、约2000bp、约2100bp、约2200bp、约2300bp、约2400bp、约2500bp、约2600bp、约2700bp、约2800bp、约2900bp或约3000bp或更长的同源臂,包含所有中间长度的同源臂。
合适的同源臂长度的另外的说明性实例包含但不限于:约100bp到约3000bp、约200bp到约3000bp、约300bp到约3000bp、约400bp到约3000bp、约500bp到约3000bp、约500bp到约2500bp、约500bp到约2000bp、约750bp到约2000bp、约750bp到约1500bp、或约1000bp到约1500bp,包含所有中间长度的同源臂。
在一个特定实施例中,5'和3'同源臂的长度独立地选自约500bp到约1500bp。在一个实施例中,5'同源臂为约1500bp,3'同源臂为约1000bp。在一个实施例中,5'同源臂在约200bp到约600bp之间,3'同源臂在约200bp到约600bp之间。在一个实施例中,5'同源臂为约200bp,3'同源臂为约200bp。在一个实施例中,5'同源臂为约300bp,3'同源臂为约300bp。在一个实施例中,5'同源臂为约400bp,3'同源臂为约400bp。在一个实施例中,5'同源臂约为500bp,3'同源臂约为500bp。在一个实施例中,5'同源臂为约600bp,3'同源臂为约600bp。
f.多肽
本文考虑了各种多肽,包含但不限于归巢核酸内切酶变体、megatal和融合多肽。在优选实施例中,多肽包括seqidno:1-23和39所示的氨基酸序列。除非另有相反的说明,否则“多肽”、“多肽片段”、“肽”和“蛋白质”可互换使用,并且根据常规含义,即用作为氨基酸的序列。在一个实施例中,“多肽”包含融合多肽和其它变体。可以使用各种熟知的重组和/或合成技术中的任何一种制备多肽。多肽不限于具体长度,例如,它们可以包括全长蛋白质序列、全长蛋白质片段或融合蛋白,并且可以包含多肽的翻译后修饰(例如,糖基化、乙酰化、磷酸化等)以及本领域已知的其它修饰(包括天然存在的和非天然存在的)。
如本文使用,“分离的蛋白质”、“分离的肽”或“分离的多肽”等是指从细胞环境以及从与细胞的其它组分的缔合体外合成、分离和/或纯化肽或多肽分子,即它与体内物质没有显著关联。
特定实施例中考虑的多肽的说明性实例包含但不限于归巢核酸内切酶变体、megatal、末端加工核酸酶、融合多肽及其变体。
多肽包含“多肽变体”。多肽变体与天然存在的多肽的不同之处可以是一个或多个氨基酸取代、缺失、加入和/或插入。这些变体可以是天然存在的或可以通过合成生成,例如,通过修饰上述多肽序列的一个或多个氨基酸。例如,在特定实施例中,可能需要通过将一个或多个取代、缺失、加入和/或插入引入多肽中来改善结合并切割人bcl11a基因中的靶位点的归巢核酸内切酶、megatal等的生物性质。在特定实施例中,多肽包含与本文考虑的任何参考序列具有至少约65%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸同一性的多肽,通常其中变体保持参考序列的至少一种生物活性。
多肽变体包含生物活性“多肽片段”。生物活性多肽片段的说明性实例包含dna结合结构域、核酸酶结构域等。如本文使用,术语“生物活性片段”或“最小生物活性片段”是指保留天然存在的多肽活性的至少100%、至少90%、至少80%、至少70%、至少60%、至少50%、至少40%、至少30%、至少20%、至少10%或至少5%的多肽片段。在优选实施例中,生物活性是针对靶序列的结合亲和力和/或切割活性。在某些实施例中,多肽片段可以包括长度为至少5到约1700个氨基酸的氨基酸链。应当理解,在某些实施例中,片段的长度为至少5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、31个、32个、33个、34个、35个、36个、37个、38个、39个、40个、41个、42个、43个、44个、45个、46个、47个、48个、49个、50个、55个、60个、65个、70个、75个、80个、85个、90个、95个、100个、110个、150个、200个、250个、300个、350个、400个、450个、500个、550个、600个、650个、700个、750个、800个、850个、900个、950个、1000个、1100个、1200个、1300个、1400个、1500个、1600个、1700个或更多个氨基酸。在特定实施例中,多肽包括归巢核酸内切酶变体的生物活性片段。在特定实施例中,本文所述的多肽可以包括一个或多个表示为“x”的氨基酸。如果存在于氨基酸中seqidno,则“x”表示任何氨基酸。一个或多个“x”残基可以存在于本文考虑的特定seqidno所示的氨基酸序列的n-末端和c-末端。如果不存在“x”氨基酸,则余下的seqidno所示的氨基酸序列可以被认为是生物活性片段。
在特定实施例中,多肽包括归巢核酸内切酶变体的生物活性片段,例如seqidno:3-19或megatal(seqidno:20-21)。生物活性片段可以包括n-末端截短和/或c-末端截短。在一个特定实施例中,生物活性片段与相应的野生型归巢核酸内切酶相比缺少归巢核酸内切酶变体的1个、2个、3个、4个、5个、6个、7个或8个n-末端氨基酸或包括其缺失,更优选与相应的野生型归巢核酸内切酶序列相比包括归巢核酸内切酶变体的4个n-末端氨基酸的缺失。在一个特定实施例中,生物活性片段与相应的野生型归巢核酸内切酶序列相比缺少归巢核酸内切酶变体的1个、2个、3个、4个或5个c-末端氨基酸或包括其缺失,更优选与相应的野生型归巢核酸内切酶序列相比包括归巢核酸内切酶变体的2个c-末端氨基酸的缺失。在一个特定优选实施例中,生物活性片段与相应的野生型归巢核酸内切酶序列相比缺少归巢核酸内切酶变体的4个n-末端氨基酸和2个c-末端氨基酸或包括其缺失。
在一个特定实施例中,i-onui变体包括以下n-末端氨基酸中的1个、2个、3个、4个、5个、6个、7个或8个的缺失:m、a、y、m、s、r、r、e;和/或以下1个、2个、3个、4个或5个c-末端氨基酸的缺失:r、g、s、f、v。
在一个特定实施例中,i-onui变体包括以下n-末端氨基酸中的1个、2个、3个、4个、5个、6个、7个或8个的缺失或取代:m、a、y、m、s、r、r、e;以下1个、2个、3个、4个或5个c-末端氨基酸的缺失或取代:r、g、s、f、v。
在一个特定实施例中,i-onui变体包括以下n-末端氨基酸中的1个、2个、3个、4个、5个、6个、7个或8个的缺失:m、a、y、m、s、r、r、e;和/或以下1个或2个c-末端氨基酸的缺失:f、v。
在一个特定实施例中,i-onui变体包括以下n-末端氨基酸中的1个、2个、3个、4个、5个、6个、7个或8个的缺失或取代:m、a、y、m、s、r、r、e;和/或以下1个或2个c-末端氨基酸的缺失或取代:f、v。
如上所述,可以以各种方式改变多肽,包含氨基酸取代、缺失、截短和插入。此类操作的方法通常是本领域已知的。例如,参考多肽的氨基酸序列变体可以通过dna中的突变来制备。诱变和核苷酸序列改变的方法是本领域熟知的。参见例如kunkel(1985年,《美国科学院院报(proc.natl.acad.sci.usa.)》,82:488-492),kunkel等人,(1987年,《酶学方法(methodsinenzymol)》,154:367-382),美国专利第4,873,192号,watson,j.d.等人,(《基因分子生物学(molecularbiologyofthegene)》,第四版,benjamin/cummings,门洛帕克,加利福尼亚,1987年)和其中引用的参考文献。关于不影响目标蛋白质的生物活性的适当氨基酸取代的指导可以在dayhoff等人的模型中找到,(1978年)《蛋白质序列和结构地图集(atlasofproteinsequenceandstructure)》(国家生物医学研究基金会,华盛顿特区)。
在某些实施例中,变体将含有一个或多个保守取代。“保守取代”是其中一种氨基酸取代具有相似性质的另一种氨基酸,使得肽化学领域的技术人员将预期多肽的二级结构和亲/疏水性质(hydropathicnature)基本上不变。可以在特定实施例中考虑的多核苷酸和多肽的结构中进行修饰,多肽包含具有至少约的多肽并且仍然获得编码具有所需特性的变体或衍生多肽的功能分子。当需要改变多肽的氨基酸序列以产生等同的或甚至改进的变体多肽时,本领域技术人员例如可以改变编码dna序列的一个或多个密码子,例如根据表1。
表1-氨基酸密码子
可以使用本领域熟知的计算机程序,例如dnastar、dnastrider、geneious、macvector或vectornti软件,找到确定哪些氨基酸残基可以在不消除生物活性的情况下被取代、插入或缺失的指导。优选地,本文公开的蛋白质变体中的氨基酸变化是保守氨基酸变化,即相似带电荷或不带电荷氨基酸的取代。保守氨基酸改变涉及在其侧链中相关的氨基酸家族之一的取代。天然存在的氨基酸通常分为四个家族:酸性(天冬氨酸盐、谷氨酸盐)、碱性(赖氨酸、精氨酸、组氨酸)、非极性(丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、蛋氨酸、色氨酸)、不带电荷极性(甘氨酸、天冬酰胺、谷氨酰胺、半胱氨酸、丝氨酸、苏氨酸、酪氨酸)氨基酸。苯丙氨酸、色氨酸和酪氨酸有时被主要分类为芳香族氨基酸。在肽或蛋白质中,合适的氨基酸保守取代是本领域技术人员已知的,并且通常可以在不改变所得分子的生物活性的情况下进行。本领域技术人员认识到,通常,多肽的非必需区中的单个氨基酸取代基本上不改变生物活性(参见例如watson等人,《基因分子生物学(molecularbiologyofthegene)》,第4版,1987年,thebenjamin/cummingspub.co.,第224页)。
在一个实施例中,当需要表达两种或更多种多肽时,编码它们的多核苷酸序列可以通过ires序列分开,如本文其它地方所公开。
特定实施例中考虑的多肽包含融合多肽。在特定实施例中,提供了融合多肽和编码融合多肽的多核苷酸。融合多肽和融合蛋白是指具有至少两个、三个、四个、五个、六个、七个、八个、九个或十个多肽片段的多肽。
在另一个实施例中,两种或更多种多肽可以表达为包括一个或多个本文其它地方公开的自切割多肽序列的融合蛋白。
在一个实施例中,本文考虑的融合蛋白包括一个或多个dna结合结构域和一个或多个核酸酶,以及一个或多个连接子和/或自切割多肽。
在一个实施例中,本文考虑的融合蛋白包括核酸酶变体;连接子或自切割肽;和末端加工酶,其包含但不限于5'-3'核酸外切酶、5'-3'碱性核酸外切酶和3'-5'核酸外切酶(例如,trex2)。
融合多肽可以包括一个或多个多肽结构域或片段,包含但不限于信号肽、细胞渗透肽结构域(cpp)、dna结合结构域、核酸酶结构域等、表位标签(例如,麦芽糖结合蛋白(“mbp”)、谷胱甘肽s转移酶(gst)、his6、myc、flag、v5、vsv-g和ha)、多肽连接子和多肽切割信号。融合多肽通常是c-末端到n-末端连接的,但是它们也可以是c-末端到c-末端、n-末端到n-末端或n-末端到c-末端连接的。在特定实施例中,融合蛋白的多肽可以是任何顺序。融合多肽或融合蛋白还可以包含保守修饰的变体、多态变体、等位基因、突变体、亚序列和种间同源物,只要保留了融合多肽的所需活性即可。融合多肽可以通过化学合成方法或通过两个部分之间的化学连接产生,或者通常可以使用其它标准技术制备。包括融合多肽的连接dna序列与合适的转录或翻译控制元件可操作地连接,如本文其它地方所公开。
融合多肽可以任选地包括可以用于连接多肽内的一个或多个多肽或结构域的连接子。肽连接子序列可以用于将任何两个或更多个多肽组分分开足够的距离,以确保每个多肽折叠成其合适的二级和三级结构,从而使多肽结构域发挥其所需功能。使用本领域的标准技术将此类肽连接子序列掺入融合多肽中。可以基于以下因素选择合适的肽连接子序列:(1)它们能够采用柔性延伸构象;(2)它们不能采用可以与第一和第二多肽上的功能性表位相互作用的二级结构;(3)缺少可能与多肽功能性表位反应的疏水或带电残基。优选的肽连接子序列含有gly、asn和ser残基。其它近中性氨基酸,例如thr和ala,也可以用于连接子序列。可以用作连接子的氨基酸序列包含maratea等人,《基因(gene)》,40:39-46,1985;murphy等人,《美国国家科学院学报(proc.natl.acad.sci.usa)》,83:8258-8262,1986;美国专利第4,935,233号和美国专利第4,751,180号中公开的那些。当特定融合多肽片段含有可以用于分开功能结构域并防止空间干扰的非必需n-末端氨基酸区时,不需要连接子序列。优选的连接子通常是柔性氨基酸子序列,其作为重组融合蛋白的一部分合成。连接子多肽的长度可以是1到200个氨基酸、1到100个氨基酸,或者1到50个氨基酸,包含其间的所有整数值。
示例性连接子包含但不限于以下氨基酸序列:甘氨酸聚合物(g)n;甘氨酸-丝氨酸聚合物(g1-5s1-5)n,其中n是至少一、二、三、四或五的整数;甘氨酸-丙氨酸聚合物;丙氨酸-丝氨酸聚合物;ggg(seqidno:40);dgggs(seqidno:41);tgekp(seqidno:42)(参见例如liu等人,《美国科学院院报(pnas)》,5525-5530(1997));ggrr(seqidno:43)(pomerantz等人,1995年,同上);(ggggs)n,其中n=1、2、3、4或5(seqidno:44)(kim等人,《美国科学院院报(pnas)》,93,1156-1160(1996.);egkssgsgseskvd(seqidno:45)(chaudhary等人,1990年,《美国科学院院报(proc.natl.acad.sci.u.s.a.)》,87:1066-1070);kesgsvsseqlaqfrsld(seqidno46)(bird等人,1988年,《科学(science)》,242:423-426),ggrrgggs(seqidno:47);lrqrdgerp(seqidno:48);lrqkdgggserp(seqidno:49);lrqkd(gggs)2erp(seqidno:50)。可替代地,柔性连接子可以使用能够对dna结合位点和肽本身进行建模的计算机程序(desjarlais和berg,《美国科学院院报(pnas)》,90:2256-2260(1993),《美国科学院院报(pnas)》,91:11099-11103(1994))或通过噬菌体展示方法来合理设计。
融合多肽可以进一步包括在本文描述的每个多肽结构域之间或在内源性开放阅读框和由供体修复模板编码的多肽之间的多肽切割信号。另外,可以将多肽切割位点置于任何连接子肽序列中。示例性多肽切割信号包含多肽切割识别位点,例如蛋白酶切割位点、核酸酶切割位点(例如,稀有限制酶识别位点、自切割核酶识别位点)和自切割病毒寡肽(参见defelipe和ryan,2004年,《运输(traffic)》,5(8);616-26)。
合适的蛋白酶切割位点和自切割肽是本领域技术人员已知的(参见例如ryan等人,1997年,《普通病毒学杂志(j.gener.virol.)》,78,699-722;scymczak等人(2004年),《自然生物技术(naturebiotech.)》,5,589-594)。示例性蛋白酶切割位点包含但不限于马铃薯y病毒nia蛋白酶(例如,烟草蚀刻病毒蛋白酶)、马铃薯y病毒hc蛋白酶、马铃薯y病毒p1(p35)蛋白酶、大麦花叶病毒(byovirus)nia蛋白酶、大麦花叶病毒rna-2编码的蛋白酶、口疮病毒l蛋白酶、肠病毒2a蛋白酶、鼻病毒2a蛋白酶、小rna病毒3c蛋白酶、豇豆花叶病毒24k蛋白酶、线虫传多面体病毒24k蛋白酶、rtsv(水稻东格鲁球状病毒)3c样蛋白酶、pyvf(欧防风黄点病毒)3c样蛋白酶、肝素、凝血酶、因子xa和肠激酶的切割位点。由于其高切割严格性,在一个实施例中优选tev(烟草蚀刻病毒)蛋白酶切割位点,例如exxyxq(g/s)(seqidno:51),例如enlyfqg(seqidno:52)和enlyfqs(seqidno:53),其中x代表任何氨基酸(由tev引起的发生在q和g或q和s之间的切割)。
在某些实施例中,自切割多肽位点包括2a或2a样位点、序列或结构域(donnelly等人,2001年,《普通病毒学杂志(j.gen.virol.)》,82:1027-1041)。在一个特定实施例中,病毒2a肽是口疮病毒2a肽、马铃薯y病毒2a肽或心病毒2a肽。
在一个实施例中,病毒2a肽选自由以下组成的组:口蹄疫病毒(fmdv)2a肽、马鼻炎a病毒(erav)2a肽、明脉扁刺蛾β四体病毒(tav)2a肽、猪捷申病毒-1(ptv-1)2a肽、泰勒病毒2a肽和脑心肌炎病毒2a肽。
表2中提供了2a位点的说明性实例。
表2:示例性2a位点包含以下序列:
g.多核苷酸
在特定实施例中,提供了编码本文考虑的一种或多种归巢核酸内切酶变体、megatal、末端加工酶和融合多肽的多核苷酸。如本文使用,术语“多核苷酸”或“核酸”是指脱氧核糖核酸(dna)、核糖核酸(rna)和dna/rna杂合体。多核苷酸可以是单链或双链的,并且是重组的、合成的或分离的。多核苷酸包含但不限于:前信使rna(前mrna)、信使rna(mrna)、rna、短干扰rna(sirna)、短发夹rna(shrna)、微rna(mirna)、核酶、合成rna、基因组rna(grna)、正链rna(rna(+))、负链rna(rna(-))、tracrrna、crrna、单向导rna(sgrna)、合成rna、合成mrna、基因组dna(gdna)、pcr扩增dna、互补dna(cdna)、合成dna或重组dna。多核苷酸是指长度为至少5个、至少10个、至少15个、至少20个、至少25个、至少30个、至少40个、至少50个、至少100个、至少200个、至少300个、至少400个、至少500个、至少1000个、至少5000个、至少10000个或至少15000个或更多个核苷酸的核苷酸聚合形式,或是核糖核苷酸,或是脱氧核糖核苷酸,或是任一类型核苷酸的修饰形式,以及所有中间长度。容易理解的是,在这种背景下,“中间长度”表示引用值之间的任何长度,例如6个、7个、8个、9个核苷酸等,101个、102个、103个核苷酸等;151个、152个、153个核苷酸等;201个、202个、203个核苷酸等。在特定实施例中,多核苷酸或变体与参考序列具有至少或约50%、55%、60%、65%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性。
在特定实施例中,多核苷酸可以是密码子优化的。如本文使用,术语“密码子优化的”是指取代编码多肽的多核苷酸中的密码子以增加多肽的表达、稳定性和/或活性。影响密码子优化的因素包含但不限于以下一种或多种:(i)两种或多种生物体或基因或合成构建的偏好表之间的密码子偏好的变化,(ii)生物体、基因或基因组内密码子偏好程度的变化,(iii)密码子(包含环境)的系统性变化,(iv)根据其解码trna的密码子的变化,(v)根据gc%的密码子的变化,无论是整体还是在三联体的一个位置,(vi)与参考序列(例如,天然存在的序列)的相似程度的变化,(vii)密码子频率截止的变化,(viii)从dna序列转录的mrna的结构性质,(ix)关于密码子取代组的设计所基于的dna序列的功能的先前知识,和/或(x)每个氨基酸的密码子组的系统性变化,和/或(xi)假翻译起始位点的分离去除。
如本文使用,术语“核苷酸”是指n-糖苷键中的杂环含氮碱基与磷酸化糖。核苷酸应被理解为包含天然碱基和多种本领域公认的修饰碱基。这些碱基通常位于核苷酸糖部分的1'位置。核苷酸通常包括碱基、糖和磷酸酯基团。在核糖核酸(rna)中,糖是核糖,并且在脱氧核糖核酸(dna)中,糖是脱氧核糖,即缺少核糖中存在的羟基基团的糖。示例性天然含氮碱基包含嘌呤、腺苷(a)和胍(g)、以及嘧啶、胞苷(c)和胸苷(t)(或在rna背景下,尿嘧啶(u))。脱氧核糖的c-1原子与嘧啶的n-1或嘌呤的n-9键合。核苷酸通常是单磷酸酯、二磷酸酯或三磷酸酯。核苷酸可以在糖、磷酸酯和/或碱基部分处被或不被修饰(也可互换地称为核苷酸类似物、核苷酸衍生物、修饰的核苷酸、非天然核苷酸和非标准核苷酸;参见例如,wo92/07065和wo93/15187)。limbach等人(1994年,《核酸研究(nucleicacidsres.)》,22,2183-2196)总结了修饰的核酸碱基的实例。
核苷酸也可以被认为是核苷的磷酸酯,其中酯化发生在与糖的c-5连接的羟基基团上。如本文使用,术语“核苷”是指n-糖苷键中的杂环含氮碱基与糖。核苷在本领域中被认为包含天然碱基,并且还包含众所周知的修饰碱基。这些碱基通常位于核苷糖部分的1'位置。核苷通常包括碱和糖基团。核苷可以在糖和/或碱基部分处被或不被修饰(也可互换地称为核苷类似物、核苷衍生物、修饰的核苷、非天然核苷或非标准核苷)。同样如上所述,limbach等人(1994年,《核酸研究(nucleicacidsres.)》,22,2183-2196)总结了修饰的核酸碱基的实例。
多核苷酸的说明性实例包含但不限于编码seqidno:1-19和39的多核苷酸和seqidno:20-38所示的多核苷酸序列。
在各个说明性实施例中,本文考虑的多核苷酸包含但不限于编码归巢核酸内切酶变体、megatal、末端加工酶、融合多肽的多核苷酸,和包括本文考虑的多核苷酸的表达载体、病毒载体和转移质粒。
如本文使用,术语“多核苷酸变体”和“变体”等是指表现出与参考多核苷酸序列的基本序列同一性的多核苷酸或在下文定义的严格条件下与参考序列杂交的多核苷酸。这些术语还涵盖通过加入、缺失、取代或修饰至少一个核苷酸而区别于参考多核苷酸的多核苷酸。因此,术语“多核苷酸变体”和“变体”包含其中已加入或缺失或修饰一个或多个核苷酸或已用不同核苷酸取代一个或多个核苷酸的多核苷酸。在这一点,本领域充分理解,可以对参考多核苷酸进行某些改变,包含突变、加入、缺失和取代,由此改变的多核苷酸保留参考多核苷酸的生物功能或活性。
在一个实施例中,多核苷酸包括在严格条件下与靶核酸序列杂交的核苷酸序列。在“严格条件”下杂交描述了杂交方案,其中彼此具有至少60%同一性的核苷酸序列保持杂交。通常,严格条件选择为在确定的离子强度和ph下比具体序列的热熔点(tm)低约5℃。tm是温度(在确定的离子强度、ph和核酸浓度下),在此温度下,50%与靶序列互补的探针在平衡时与靶序列杂交。由于靶序列通常过量存在,所以在tm下,50%的探针在平衡时被占用。
如本文使用,叙述“序列同一性”或例如包括“与……具有50%同一性的序列”是指序列在比较窗口上在逐个核苷酸的基础上或在逐个氨基酸的基础上具有同一性的程度。因此,“序列同一性百分比”可以通过以下计算:在比较窗口上比较两个最佳比对序列,确定两个序列中存在相同的核酸碱基(例如,a、t、c、g、i)或相同的氨基酸残基(例如,ala、pro、ser、thr、gly、val、leu、ile、phe、tyr、trp、lys、arg、his、asp、glu、asn、gln、cys和met)的位置的数量以得到匹配位置的数量,用匹配位置的数量除以比较窗口中的位置总数(即,窗口大小),并将结果乘以100以得到序列同一性百分比。包含与本文所述的任何参考序列具有至少约50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%的序列同一性的核苷酸和多肽,通常其中多肽变体保持参考多肽的至少一种生物活性。
用于描述两个或更多个多核苷酸或多肽之间的序列关系的术语包含“参考序列”、“比较窗口”、“序列同一性”、“序列同一性百分比”和“基本同一性”。“参考序列”的长度为至少12个单体单元,但通常为15到18个单体单元,在大多数情况下为至少25个单体单元,所述单体单元包含核苷酸和氨基酸残基。因为两个多核苷酸可以各自包括(1)两个多核苷酸之间相似的序列(即,仅完整多核苷酸序列的一部分),和(2)两个多核苷酸之间不同的序列,通常通过在“比较窗口”上比较两个多核苷酸的序列来进行两个(或更多个)多核苷酸之间的序列比较,以标识和比较序列相似性的局部区域。“比较窗口”是指至少6个连续位置,通常约50到约100个连续位置,更通常约100到约150个连续位置的概念性片段,其中将序列与连续位置数量相同的参考序列进行比较,然后对两个序列进行最佳比对。对于两个序列的最佳比对,比较窗口可以包括与参考序列(其不包括加入或缺失)相比约20%或更少的加入或缺失(即,缺口)。用于比对比较窗口的序列的最佳比对可以通过算法的计算机化实现(威斯康星遗传学软件包7.0版本中的gap、bestfit、fasta和tfasta,geneticscomputergroup,575sciencedrivemadison,wi,usa)进行,或者通过选择的各个方法中的任一个生成的检查和最佳比对(即,导致比较窗口上的同源性百分比最高)。还可以参考blast程序家族,例如altschul等人,1997年,《核酸研究(nucl.acidsres.)》,25:3389。序列分析的详细讨论可以在ausubel等人,《现代分子生物学实验指南(currentprotocolsinmolecularbiology)》,johnwiley&sonsinc.,1994-1998年,第15章的单元19.3中找到。
如本文使用,“分离的多核苷酸”是指从天然存在状态侧翼的序列纯化的多核苷酸,例如已从通常与片段相邻的序列中去除的dna片段。在特定实施例中,“分离的多核苷酸”是指互补dna(cdna)、重组多核苷酸、合成多核苷酸或自然界中不存在并且已由人工制造的其它多核苷酸。
在各个实施例中,多核苷酸包括编码本文考虑的多肽的mrna,所述多肽包含但不限于归巢核酸内切酶变体、megatal和末端加工酶。在某些实施例中,mrna包括帽、一个或多个核苷酸和聚(a)尾。
如本文使用,术语“5'帽”或“5'帽结构”或“5'帽部分”是指化学修饰,其已掺入mrna的5'末端。5'帽参与核输出、mrna稳定性和翻译。
在特定实施例中,本文考虑的mrna包括5'帽,其包括mrna分子的末端鸟苷帽残基与5'-末端转录的正义核苷酸之间的5'-ppp-5'-三磷酸酯键。然后,可以将该5'-鸟苷酸帽甲基化以生成n7-甲基-鸟苷酸残基。
适用于本文考虑的mrna多核苷酸的特定实施例的5'帽的说明性实例包含但不限于:未甲基化的5'帽类似物,例如g(5')ppp(5')g、g(5')ppp(5')c、g(5')ppp(5')a;甲基化的5'帽类似物,例如m7g(5')ppp(5')g、m7g(5')ppp(5')c和m7g(5')ppp(5')a;二甲基化的5'帽类似物,例如m2,7g(5')ppp(5')g、m2,7g(5')ppp(5')c和m2,7g(5')ppp(5')a;三甲基化的5'帽类似物,例如m2,2,7g(5')ppp(5')g、m2,2,7g(5')ppp(5')c和m2,2,7g(5')ppp(5')a;二甲基化的对称5'帽类似物,例如m7g(5')pppm7(5')g、m7g(5')pppm7(5')c和m7g(5')pppm7(5')a;和抗反向5'帽类似物,例如抗反向帽类似物(arca)帽、指定3'o-me-m7g(5')ppp(5')g、2'o-me-m7g(5')ppp(5')g、2'o-me-m7g(5')ppp(5')c、2'o-me-m7g(5')ppp(5')a、m72'd(5')ppp(5')g、m72'd(5')ppp(5')c、m72'd(5')ppp(5')a、3'o-me-m7g(5')ppp(5')c、3'o-me-m7g(5')ppp(5')a、m73'd(5')ppp(5')g、m73'd(5')ppp(5')c、m73'd(5')ppp(5')a及其四磷酸酯衍生物)(参见例如jemielity等人,《核糖核酸(rna)》,9:1108-1122(2003))。
在特定实施例中,mrna包括5'帽,其为7-甲基鸟苷酸(“m7g”),其通过三磷酸酯桥连接到第一转录核苷酸的5'-末端,产生m7g(5')ppp(5')n,其中n是任何核苷。
在一些实施例中,mrna包括5'帽,其中帽是cap0结构(cap0结构缺少与碱基1和2连接的核糖的2'-o-甲基残基)、cap1结构(cap1结构具有碱基2处的2'-o-甲基残基或cap2结构(cap2结构具有与碱基2和3连接的2'-o-甲基残基)。
在一个实施例中,mrna包括m7g(5')ppp(5')g帽。
在一个实施例中,mrna包括arca帽。
在特定实施例中,本文考虑的mrna包括一种或多种修饰的核苷。
在一个实施例中,mrna包括一种或多种修饰的核苷,其选自由以下组成的组:假尿苷、吡啶-4-酮核糖核苷、5-氮杂-尿苷、2-硫代-5-氮杂-尿苷、2-硫尿苷、4-硫代-假尿苷、2-硫代-假尿苷、5-羟基尿苷、3-甲基尿苷、5-羧甲基-尿苷、1-羧甲基-假尿苷、5-丙炔基-尿苷、1-丙炔基-假尿苷、5-牛磺酸甲基尿苷、1-牛磺酸甲基-假尿苷、5-牛磺酸甲基-2-硫代-尿苷、1-牛磺酸甲基-4-硫代-尿苷、5-甲基-尿苷、1-甲基-假尿苷、4-硫代-1-甲基-假尿苷、2-硫代-1-甲基-假尿苷、1-甲基-1-脱氮-假尿苷、2-硫代-1-甲基-1-脱氮-假尿苷、二氢尿苷、二氢假尿苷、2-硫代-二氢尿苷、2-硫代-二氢假尿苷、2-甲氧基尿苷、2-甲氧基-4-硫代-尿苷、4-甲氧基-假尿苷、4-甲氧基-2-硫代-假尿苷、5-氮杂-胞苷、假异胞苷、3-甲基-胞苷、n4-乙酰基胞苷、5-甲酰基胞苷、n4-甲基胞苷、5-羟甲基胞苷、1-甲基-假异胞苷、吡咯-胞苷、吡咯-假异胞苷、2-硫代-胞苷、2-硫代-5-甲基-胞苷、4-硫代-假异胞苷、4-硫代-1-甲基-假异胞苷、4-硫代-1-甲基-1-脱氮-假异胞苷、1-甲基-1-脱氮-假异胞苷、泽布莱林(zebularine)、5-氮杂-泽布莱林、5-甲基-泽布莱林、5-氮杂-2-硫代-泽布莱林、2-硫代-泽布莱林、2-甲氧基-胞苷、2-甲氧基-5-甲基-胞苷、4-甲氧基-假异胞苷、4-甲氧基-1-甲基-假异胞苷、2-氨基嘌呤、2,6-二氨基嘌呤、7-脱氮-腺嘌呤、7-脱氮-8-氮杂-腺嘌呤、7-脱氮-2-氨基嘌呤、7-脱氮-8-氮杂-2-氨基嘌呤、7-脱氮-2,6-二氨基嘌呤、7-脱氮-8-氮杂-2,6-二氨基嘌呤、1-甲基腺苷、n6-甲基腺苷、n6-异戊烯基腺苷、n6-(顺-羟基异戊烯基)腺苷、2-甲硫基-n6-(顺-羟基异戊烯基)腺苷、n6-甘氨酰基氨基甲酰基腺苷、n6-苏氨酰基氨基甲酰基腺苷、2-甲硫基-n6-苏氨酰氨基甲酰腺苷、n6,n6-二甲基腺苷、7-甲基腺嘌呤、2-甲硫基-腺嘌呤、2-甲氧基-腺嘌呤、肌苷、1-甲基-肌苷、怀俄苷、怀丁苷、7-脱氮-鸟苷、7-脱氮-8-氮杂-鸟苷、6-硫代-鸟苷、6-硫代-7-脱氮-鸟苷、6-硫代-7-脱氮-8-氮杂-鸟苷、7-甲基-鸟苷、6-硫代-7-甲基-鸟苷、7-甲基肌苷、6-甲氧基-鸟苷、1-甲基鸟苷、n2-甲基鸟苷、n2,n2-二甲基鸟苷、8-氧代-鸟苷、7-甲基-8-氧代-鸟苷、1-甲基-6-硫代-鸟苷、n2-甲基-6-硫代-鸟苷和n2,n2-二甲基-6-硫代-鸟苷。
在一个实施例中,mrna包括一种或多种修饰的核苷,其选自由以下组成的组:假尿苷、吡啶-4-酮核糖核苷、5-氮杂-尿苷、2-硫代-5-氮杂-尿苷、2-硫尿苷、4-硫代-假尿苷、2-硫代-假尿苷、5-羟基尿苷、3-甲基尿苷、5-羧甲基-尿苷、1-羧甲基-假尿苷、5-丙炔基-尿苷、1-丙炔基-假尿苷、5-牛磺酸甲基尿苷、1-牛磺酸甲基-假尿苷、5-牛磺酸甲基-2-硫代-尿苷、1-牛磺酸甲基-4-硫代-尿苷、5-甲基-尿苷、1-甲基-假尿苷、4-硫代-1-甲基-假尿苷、2-硫代-1-甲基-假尿苷、1-甲基-1-脱氮-假尿苷、2-硫代-1-甲基-1-脱氮-假尿苷、二氢尿苷、二氢假尿苷、2-硫代-二氢尿苷、2-硫代-二氢假尿苷、2-甲氧基尿苷、2-甲氧基-4-硫代-尿苷、4-甲氧基-假尿苷和4-甲氧基-2-硫代-假尿苷。
在一个实施例中,mrna包括一种或多种修饰的核苷,其选自由以下组成的组:5-氮杂-胞苷、假异胞苷、3-甲基-胞苷、n4-乙酰基胞苷、5-甲酰基胞苷、n4-甲基胞苷、5-羟甲基胞苷、1-甲基-假异胞苷、吡咯-胞苷、吡咯-假异胞苷、2-硫代-胞苷、2-硫代-5-甲基-胞苷、4-硫代-假异胞苷、4-硫代-1-甲基-假异胞苷、4-硫代-1-甲基-1-脱氮-假异胞苷、1-甲基-1-脱氮-假异胞苷、泽布莱林、5-氮杂-泽布莱林、5-甲基-泽布莱林、5-氮杂-2-硫代-泽布莱林、2-硫代-泽布莱林、2-甲氧基-胞苷、2-甲氧基-5-甲基-胞苷、4-甲氧基-假异胞苷和4-甲氧基-1-甲基-假异胞苷。
在一个实施例中,mrna包括一种或多种修饰的核苷,其选自由以下组成的组:2-氨基嘌呤、2,6-二氨基嘌呤、7-脱氮-腺嘌呤、7-脱氮-8-氮杂-腺嘌呤、7-脱氮-2-氨基嘌呤、7-脱氮-8-氮杂-2-氨基嘌呤、7-脱氮-2,6-二氨基嘌呤、7-脱氮-8-氮杂-2,6-二氨基嘌呤、1-甲基腺苷、n6-甲基腺苷、n6-异戊烯基腺苷、n6-(顺-羟基异戊烯基)腺苷、2-甲硫基-n6-(顺-羟基异戊烯基)腺苷、n6-甘氨酰基氨基甲酰基腺苷、n6-苏氨酰基氨基甲酰基腺苷、2-甲硫基-n6-苏氨酰氨基甲酰腺苷、n6,n6-二甲基腺苷、7-甲基腺嘌呤、2-甲硫基-腺嘌呤和2-甲氧基-腺嘌呤。
在一个实施例中,mrna包括一种或多种修饰的核苷,其选自由以下组成的组:肌苷、1-甲基-肌苷、怀俄苷、怀丁苷、7-脱氮-鸟苷、7-脱氮-8-氮杂-鸟苷、6-硫代-鸟苷、6-硫代-7-脱氮-鸟苷、6-硫代-7-脱氮-8-氮杂-鸟苷、7-甲基-鸟苷、6-硫代-7-甲基-鸟苷、7-甲基肌苷、6-甲氧基-鸟苷、1-甲基鸟苷、n2-甲基鸟苷、n2,n2-二甲基鸟苷、8-氧代-鸟苷、7-甲基-8-氧代-鸟苷、1-甲基-6-硫代-鸟苷、n2-甲基-6-硫代-鸟苷和n2,n2-二甲基-6-硫代-鸟苷。
在一个实施例中,mrna包括一个或多个假尿苷、一个或多个5-甲基-胞嘧啶和/或一个或多个5-甲基-胞苷。
在一个实施例中,mrna包括一个或多个假尿苷。
在一个实施例中,mrna包括一个或多个5-甲基-胞苷。
在一个实施例中,mrna包括一个或多个5-甲基-胞嘧啶。
在特定实施例中,本文考虑的mrna包括聚(a)尾,以帮助保护mrna免于核酸外切酶降解,稳定mrna并促进翻译。在某些实施例中,mrna包括3'聚(a)尾结构。
在特定实施例中,聚(a)尾的长度为至少约10个、25个、50个、75个、100个、150个、200个、250个、300个、350个、400个、450个或至少约500个或更多个腺嘌呤核苷酸或任何中间数量的腺嘌呤核苷酸。在特定实施例中,聚(a)尾的长度为至少约125个、126个、127个、128个、129个、130个、131个、132个、133个、134个、135个、136个、137个、138个、139个、140个、141个、142个、143个、144个、145个、146个、147个、148个、149个、150个、151个、152个、153个、154个、155个、156个、157个、158个、159个、160个、161个、162个、163个、164个、165个、166个、167个、168个、169个、170个、171个、172个、173个、174个、175个、176个、177个、178个、179个、180个、181个、182个、183个、184个、185个、186个、187个、188个、189个、190个、191个、192个、193个、194个、195个、196个、197个、198个、199个、200个、201个、202个、202个、203个、205个、206个、207个、208个、209个、210个、211个、212个、213个、214个、215个、216个、217个、218个、219个、220个、221个、222个、223个、224个、225个、226个、227个、228个、229个、230个、231个、232个、233个、234个、235个、236个、237个、238个、239个、240个、241个、242个、243个、244个、245个、246个、247个、248个、249个、250个、251个、252个、253个、254个、255个、256个、257个、258个、259个、260个、261个、262个、263个、264个、265个、266个、267个、268个、269个、270个、271个、272个、273个、274个或275个或更多个腺嘌呤核苷酸。
在特定实施例中,聚(a)尾的长度为约10到约500个腺嘌呤核苷酸、约50到约500个腺嘌呤核苷酸、约100到约500个腺嘌呤核苷酸、约150到约500个腺嘌呤核苷酸、约200到约500个腺嘌呤核苷酸、约250到约500个腺嘌呤核苷酸、约300到约500个腺嘌呤核苷酸、约50到约450个腺嘌呤核苷酸、约50到约400个腺嘌呤核苷酸、约50到约350个腺嘌呤核苷酸、约100到约500个腺嘌呤核苷酸、约100到约450个腺嘌呤核苷酸、约100到约400个腺嘌呤核苷酸、约100到约350个腺嘌呤核苷酸、约100到约300个腺嘌呤核苷酸、约150到约500个腺嘌呤核苷酸、约150到约450个腺嘌呤核苷酸、约150到约400个腺嘌呤核苷酸、约150到约350个腺嘌呤核苷酸、约150到约300个腺嘌呤核苷酸、约150到约250个腺嘌呤核苷酸、约150到约200个腺嘌呤核苷酸、约200到约500个腺嘌呤核苷酸、约200到约450个腺嘌呤核苷酸、约200到约400个腺嘌呤核苷酸、约200到约350个腺嘌呤核苷酸、约200到约300个腺嘌呤核苷酸、约250到约500个腺嘌呤核苷酸、约250到约450个腺嘌呤核苷酸、约250到约400个腺嘌呤核苷酸、约250到约350个腺嘌呤核苷酸、或约250到约300个腺嘌呤核苷酸或任何中间范围的腺嘌呤核苷酸。
描述多核苷酸方向的术语包含:5'(通常是具有游离磷酸根基团的多核苷酸的末端)和3'(通常是具有游离羟基(oh)基团的多核苷酸的末端)。多核苷酸序列可以以5'到3'方向或3'到5'方向注释。对于dna和mrna,5'到3'链被称为“正义”、“正”或“编码”链,因为其序列与前信使(前mrna)的序列相同[rna中的尿嘧啶(u)除外,而不是dna中的胸腺嘧啶(t)]。对于dna和mrna,互补3'到5'链,其是由rna聚合酶转录的链,被称为“模板”、“反义”、“负”或“非编码”链。如本文使用,术语“反向”是指以3'到5'方向写入的5'到3'序列或以5'到3'方向写入的3'到5'序列。
术语“互补的”和“互补性”是指通过碱基配对规则相关的多核苷酸(即,核苷酸序列)。例如,dna序列5'agtcatg3'的互补链是3'tcagtac5'。后一序列通常写为反向互补,其中5'末端在左,3'末端在右,5'catgact3'。与其反向互补相等的序列称为回文序列。互补性可以是“部分的”,其中根据碱基配对规则仅匹配一些核酸碱基。或者,核酸之间可以存在“完全”或“全”互补性。
如本文使用,术语“核酸盒”或“表达盒”是指载体内可以表达rna并随后表达多肽的遗传序列。在一个实施例中,核酸盒含有目标基因,例如目标多核苷酸。在另一个实施例中,核酸盒含有一个或多个表达控制序列,例如启动子、增强子、聚(a)序列和目标基因(例如,目标多核苷酸)。载体可以包括1个、2个、3个、4个、5个、6个、7个、8个、9个或10个或更多个核酸盒。核酸盒在载体内定位和顺序定向,使得盒中的核酸可以转录成rna,并且在必要时翻译成蛋白质或多肽,经历转化的细胞中活性所需的适当翻译后修饰,通过靶向合适的细胞内区室或分泌到细胞外区室中移位到生物活性所需的适当区室。优选地,盒具有适于容易插入载体的3'末端和5'末端,例如,其在每个末端具有限制性核酸内切酶位点。在一个优选实施例中,核酸盒含有用于治疗、预防或改善遗传病症的治疗基因的序列。可以去除盒并将其作为单个单元插入质粒或病毒载体中。
多核苷酸包含目标多核苷酸。如本文使用,术语“目标多核苷酸”是指编码多肽或融合多肽的多核苷酸或用作抑制性多核苷酸转录的模板的多核苷酸,如本文所考虑。
此外,本领域普通技术人员将理解,由于遗传密码的简并性,存在许多核苷酸序列,其可以编码如本文考虑的多肽或其变体的片段。这些多核苷酸中的一些与任何天然基因的核苷酸序列具有最小的同源性。尽管如此,在特定实施例中特别考虑了由于密码子使用的差异而不同的多核苷酸,例如针对人和/或灵长类动物密码子选择而优化的多核苷酸。在一个实施例中,提供了包括特定等位基因序列的多核苷酸。等位基因是内源多核苷酸序列,其由于一个或多个突变而改变,例如核苷酸的缺失、加入和/或取代。
在某个实施例中,目标多核苷酸包括供体修复模板。
在某个实施例中,目标多核苷酸包括抑制性多核苷酸,其包含但不限于sirna、mirna、shrna、核酶或另一种抑制性rna。
在一个实施例中,包括抑制性rna的供体修复模板包括一个或多个调节序列,诸如例如强组成型poliii,例如人或小鼠u6snrna启动子、人和小鼠h1rna启动子、或人trna-val启动子、或强组成型polii启动子,如本文其它地方所述。
无论编码序列本身的长度,特定实施例中考虑的多核苷酸可以与其它dna序列组合,例如启动子和/或增强子、非翻译区(utr)、kozak序列、多聚腺苷酸化信号、其它限制酶位点、多克隆位点、内部核糖体进入位点(ires)、重组酶识别位点(例如,loxp、frt和att位点)、终止密码子、转录终止信号、转录后反应元件、和编码自切割多肽的多核苷酸、表位标签,如本文其它地方所公开或本领域已知,使得它们的总长度可以相当大地不同。因此在特定实施例中考虑,可以使用几乎任何长度的多核苷酸片段,其总长度优选受在预期的重组dna方案中的制备和使用的容易性的限制。
可以使用本领域已知和可获得的各种成熟技术中的任何一种来制备、操作、表达和/或递送多核苷酸。为了表达所需多肽,可以将编码多肽的核苷酸序列插入合适的载体中。还可以通过将编码多肽的mrna递送到细胞中来表达所需多肽。
载体的说明性实例包含但不限于质粒、自主复制序列和转座因子,例如sleepingbeauty、piggybac。
另外的载体的说明性实例包含但不限于质粒、噬菌粒、粘粒、人工染色体(例如,酵母人工染色体(yac)、细菌人工染色体(bac)或p1衍生的人工染色体(pac))、噬菌体(例如,λ噬菌体或m13噬菌体)和动物病毒。
可用作载体的病毒的说明性实例包含但不限于逆转录病毒(包含慢病毒)、腺病毒、腺相关病毒、疱疹病毒(例如,单纯疱疹病毒)、痘病毒、杆状病毒、乳头瘤病毒和乳多空病毒(例如,sv40)。
表达载体的说明性实例包含但不限于用于在哺乳动物细胞中的表达的pclneo载体(promega);用于慢病毒介导的基因转移和在哺乳动物细胞中的表达的plenti4/v5-desttm、plenti6/v5-desttm和plenti6.2/v5-gw/lacz(invitrogen)。在特定实施例中,本文公开的多肽的编码序列可以连接到这些用于在哺乳动物细胞中表达多肽的表达载体中。
在特定实施例中,载体是附加型载体或在染色体外维持的载体。如本文使用,术语“附加型的”是指能够复制而不整合到宿主的染色体dna中且不会从分裂的宿主细胞中逐渐丧失的载体,这也表示所述载体在染色体外或附加地复制。
表达载体中存在的“表达控制序列”、“控制元件”或“调节序列”是载体的非翻译区——复制起点、选择盒、启动子、增强子、翻译起始信号(shinedalgarno序列或kozak序列)内含子、转录后调节元件、多聚腺苷酸化序列、5'和3'非翻译区——其与宿主细胞蛋白相互作用以进行转录和翻译。这些元件的强度和特异性可能不同。取决于利用的载体系统和宿主,可以使用任何数量的合适的转录和翻译元件,包含普遍存在的启动子和可诱导启动子。
在特定实施例中,多核苷酸包括载体,其包含但不限于表达载体和病毒载体。载体可以包括一个或多个外源、内源或异源控制序列,例如启动子和/或增强子。“内源控制序列”是与基因组中的给定基因天然连接的序列。“外源控制序列”是通过遗传操作(即,分子生物学技术)与基因并置放置的序列,使得该基因的转录由连接的增强子/启动子指导。“异源控制序列”是来自与遗传操作的细胞不同的物种的外源序列。“合成的”控制序列可以包括一个或多个内源和/或外源序列的元件,和/或体外或计算机中确定的序列,其为特定疗法提供最佳启动子和/或增强子活性。
本文使用的术语“启动子”是指rna聚合酶结合的多核苷酸(dna或rna)的识别位点。rna聚合酶启动并转录与启动子可操作地连接的多核苷酸。在特定实施例中,在哺乳动物细胞中起作用的启动子包括位于转录起始位点上游约25到30个碱基的富含at的区域和/或在转录起始上游70到80个碱基处发现的另一个序列cncaat区域,其中n可以是任何核苷酸。
术语“增强子”是指dna片段,其含有能够提供增强的转录的序列,并且在一些情况下可以独立于其相对于另一个控制序列的方向而起作用。增强子可以与启动子和/或其它增强子元件协同或相加地起作用。术语“启动子/增强子”是指dna片段,其含有能够提供启动子和增强子功能的序列。
术语“可操作地连接”是指并置,其中所述组分处于允许它们以其预期方式起作用的关系中。在一个实施例中,该术语是指核酸表达控制序列(例如,启动子和/或增强子)与第二多核苷酸序列(例如,目标多核苷酸)之间的功能性连接,其中表达控制序列指导对应于第二序列的核酸的转录。
如本文使用,术语“组成型表达控制序列”是指持续地或连续地允许可操作地连接的序列的转录的启动子、增强子或启动子/增强子。组成型表达控制序列可以分别是允许在多种细胞和组织类型中的表达的“普遍存在的”启动子、增强子或启动子/增强子,或允许在受限制的多种细胞和组织类型中的表达的“细胞特异性”、“细胞类型特异性”、“细胞谱系特异性”或“组织特异性”启动子、增强子或启动子/增强子。
适用于特定实施例的示例性普遍存在的表达控制序列包含但不限于巨细胞病毒(cmv)立即早期启动子、病毒猿猴病毒40(sv40)(例如,早期或晚期)、莫洛尼小鼠白血病病毒(momlv)ltr启动子、劳氏肉瘤病毒(rsv)ltr、单纯疱疹病毒(hsv)(胸苷激酶)启动子、来自牛痘病毒的h5、p7.5和p11启动子、短延长因子1-α(ef1a-短)启动子、长延长因子1-α(ef1a-长)启动子、早期生长反应1(egr1)、铁蛋白h(ferh)、铁蛋白l(ferl)、甘油醛3-磷酸脱氢酶(gapdh)、真核翻译起始因子4a1(eif4a1)、热休克70kda蛋白5(hspa5)、热休克蛋白90kdaβ成员1(hsp90b1)、热休克蛋白70kda(hsp70)、β-驱动蛋白(β-kin)、人rosa26基因座(irions等人,《自然生物技术(naturebiotechnology)》,25,1477-1482(2007))、泛素c启动子(ubc)、磷酸甘油酸激酶-1(pgk)启动子、巨细胞病毒增强子/鸡β-肌动蛋白(cag)启动子、β-肌动蛋白启动子和骨髓增生性肉瘤病毒增强子、阴性对照区缺失的dl587rev引物结合位点取代(mnd)启动子(challita等人,《病毒学杂志(jvirol.)》,69(2):748-55(1995))。
在一个特定实施例中,可能需要使用细胞、细胞类型、细胞谱系或组织特异性表达控制序列来实现所需多核苷酸序列的细胞类型特异性、谱系特异性或组织特异性表达(例如,以表达仅在细胞类型、细胞谱系或组织的子集中或在特定发育阶段期间编码多肽的特定核酸。
如本文使用,“条件表达”可以是指任何类型的条件表达,包含但不限于可诱导的表达;可阻遏的表达;在具有特定生理、生物或疾病状态等的细胞或组织中的表达。该定义不旨在排除细胞类型或组织特异性表达。某些实施例提供了目标多核苷酸的条件表达,例如,通过使细胞、组织、生物等经受治疗,或受到导致多核苷酸表达的条件,或受到引起由目标多核苷酸编码的多核苷酸的表达的增加或减少的条件来控制表达。
可诱导启动子/系统的说明性实例包含但不限于类固醇可诱导启动子,例如编码糖皮质激素或雌激素受体的基因的启动子(可通过用相应的激素治疗诱导)、金属硫蛋白启动子(可通过用各种重金属治疗诱导)、mx-1启动子(可通过干扰素诱导)、“基因开关”米非司酮可调系统(sirin等人,2003年,《基因(gene)》,323:67)、异丙基苯甲酸(cumate)可诱导基因开关(wo2002/088346)、四环素依赖性调节系统等。
也可以通过使用位点特异性dna重组酶实现条件表达。根据某些实施例,多核苷酸包括至少一个(通常两个)位点,用于由位点特异性重组酶介导的重组。如本文使用,术语“重组酶”或“位点特异性重组酶”包含涉及一个或多个重组位点(例如,两个、三个、四个、五个、六个、七个、八个、九个、十个或更多个)的重组反应中涉及的切除或整合蛋白、酶、辅因子或相关蛋白,其可能是野生型蛋白质(参见landy,《生物技术发展现状(currentopinioninbiotechnology)》,3:699-707(1993)),或其突变体、衍生物(例如,含有重组蛋白序列或其片段的融合蛋白)、片段及其变体。适用于特定实施例的重组酶的说明性实例包含但不限于:cre、int、ihf、xis、flp、fis、hin、gin、φc31、cin、tn3解离酶、tndx、xerc、xerd、tnpx、hjc、gin、spcce1和para。
多核苷酸可以包括多种位点特异性重组酶中的任何一种的一个或多个重组位点。应当理解,位点特异性重组酶的靶位点是整合载体(例如,逆转录病毒载体或慢病毒载体)所需的任何位点的补充。如本文使用,术语“重组序列”、“重组位点”或“位点特异性重组位点”是指重组酶识别并结合的特定核酸序列。
例如,cre重组酶的一个重组位点是loxp,其是34碱基对序列,其包括位于8碱基对核心序列侧翼的两个13碱基对反向重复(用作重组酶结合位点)(参见sauer,b.,《生物技术发展现状(currentopinioninbiotechnology)》,5:521-527(1994)的图1)。其它示例性loxp位点包含但不限于:lox511(hoess等人,1996年;bethke和sauer,1997年)、lox5171(lee和saito,1998年)、lox2272(lee和saito,1998年)、m2(langer等人,2002年)、lox71(albert等人,1995年)和lox66(albert等人,1995年)。
flp重组酶的合适识别位点包含但不限于:frt(mcleod等人,1996年)、f1、f2、f3(schlake和bode,1994年)、f4、f5(schlake和bode,1994年)、frt(le)(senecoff等人,1988年)、frt(re)(senecoff等人,1988年)。
识别序列的其它实例是attb、attp、attl和attr序列,其被重组酶λ整合酶识别,例如
在一个实施例中,本文考虑的多核苷酸包括侧翼为一对重组酶识别位点的修复模板多核苷酸。在特定实施例中,修复模板多核苷酸侧翼为loxp位点、frt位点或att位点。
在特定实施例中,本文考虑的多核苷酸包含编码一个或多个多肽的一个或多个目标多核苷酸。在特定实施例中,为了实现多个多肽中的每一个的有效翻译,可以通过一种或多种ires序列或编码自切割多肽的多核苷酸序列分开多核苷酸序列。
如本文使用,“内部核糖体进入位点”或“ires”是指促进直接内部核糖体进入顺反子(蛋白质编码区)的起始密码子(例如,atg)的元件,从而导致基因的帽非依赖性翻译。参见例如jackson等人,1990年,《生物化学的发展趋势(trendsbiochemsci)》,15(12):477-83)和jackson和kaminski,1995年,《核糖核酸(rna)》,1(10):985-1000。本领域技术人员通常采用的ires的实例包含美国专利第6,692,736号中所述的那些。本领域已知的“ires”的另外的实例包含但不限于可从小核糖核酸病毒获得的ires(jackson等人,1990年)和可从病毒或细胞mrna来源获得的ires,诸如例如免疫球蛋白重链结合蛋白(bip)、血管内皮生长因子(vegf)(huez等人,1998年,《分子细胞生物学(mol.cell.biol.)》,18(11):6178-6190)、成纤维细胞生长因子2(fgf-2)和胰岛素样生长因子(igfii)、翻译起始因子eif4g和酵母转录因子tfiid和hap4、可从novagen商购获得的脑心肌炎病毒(emcv)(duke等人,1992年,《病毒学杂志(j.virol)》,66(3):1602-9)和vegfires(huez等人,1998年,《分子细胞生物学(molcellbiol)》,18(11):6178-90)。在小核糖核酸病毒科、二顺反子病毒科和黄病毒科种的病毒基因组中以及hcv、弗里德小鼠白血病病毒(frmlv)和莫洛尼小鼠白血病病毒(momlv)中也报道了ires。
在一个实施例中,本文考虑的多核苷酸中使用的ires是emcvires。
在特定实施例中,多核苷酸包括具有共有kozak序列并编码所需多肽的多核苷酸。如本文使用,术语“kozak序列”是指短核苷酸序列,其极大地促进mrna与核糖体的小亚单位的初始结合并增加翻译。共有kozak序列是(gcc)rccatgg(seqidno:76),其中r是嘌呤(a或g)(kozak,1986年,《细胞(cell)》,44(2):283-92,和kozak,1987年,《核酸研究(nucleicacidsres.)》,15(20):8125-48)。
指导异源核酸转录物的有效终止和多聚腺苷酸化的元件增加了异源基因表达。通常在多聚腺苷酸化信号的下游发现转录终止信号。在特定实施例中,载体包括编码待表达多肽的多核苷酸的多聚腺苷酸化序列3'。本文使用的术语“聚a位点”、“聚a序列”、“聚(a)位点”或“聚(a)序列”表示通过rna聚合酶ii指导新生rna转录物的终止和多聚腺苷酸化的dna序列。多聚腺苷酸化序列可以通过向编码序列的3'末端加入聚(a)尾来促进mrna稳定性,因此有助于提高翻译效率。重组转录物的有效多聚腺苷酸化是理想的,因为缺少聚(a)尾的转录物是不稳定的并且快速降解。可以用于载体的聚(a)信号的说明性实例包含理想的聚(a)序列(例如,aataaa、attaaa、agtaaa)、牛生长激素聚(a)序列(bghpa)、兔β-球蛋白聚(a)序列(rβgpa)、或本领域已知的另一种合适的异源或内源聚(a)序列。
在一些实施例中,多核苷酸或携带多核苷酸的细胞利用自杀基因,包含可诱导自杀基因,以降低直接毒性和/或不受控制的增殖的风险。在具体实施例中,自杀基因对携带多核苷酸或细胞的宿主没有免疫原性。可以使用的自杀基因的某个实例是半胱天冬酶-9或半胱天冬酶-8或胞嘧啶脱氨酶。半胱天冬酶-9可以使用特异性二聚化学诱导剂(cid)激活。
在某些实施例中,多核苷酸包括基因片段,其导致本文考虑的遗传修饰的细胞易于在体内进行阴性选择。“阴性选择”是指由于个体体内条件的改变而可以消除的输注细胞。阴性可选择表型可以通过赋予施用试剂(例如,化合物)敏感性的基因的插入而产生。阴性选择基因是本领域已知的,包含但不限于:赋予更昔洛韦敏感性的单纯疱疹病毒i型胸苷激酶(hsv-itk)基因;细胞次黄嘌呤磷酸核糖转移酶(hprt)基因、细胞腺嘌呤磷酸核糖转移酶(aprt)基因和细菌胞嘧啶脱氨酶。
在一些实施例中,遗传修饰的细胞包括进一步包括阳性标志物的多核苷酸,所述阳性标志物使得能够在体外选择阴性可选择表型的细胞。阳性可选择标志物可以是基因,其在被引入宿主细胞后表达显性表型,允许携带该基因的细胞的阳性选择。这种类型的基因是本领域已知的,包含但不限于赋予潮霉素b耐药性的潮霉素-b磷酸转移酶基因(hph)、来自编码抗生素g418耐药性的tn5的氨基糖苷磷酸转移酶基因(neo或aph)、二氢叶酸还原酶(dhfr)基因、腺苷脱氨酶基因(ada)和多药耐药性(mdr)基因。
在一个实施例中,阳性可选择标志物和阴性可选择元件连接,使得阴性可选择元件的损失也必然伴随着阳性可选择标志物的损失。在一个特定实施例中,阳性和阴性可选择标志物被融合,使得一种必需标志物的损失导致另一种标志物的损失。作为表达产物产生的融合多核苷酸的实例是赋予上述所需的阳性和阴性选择特征的多肽,其是潮霉素磷酸转移酶胸苷激酶融合基因(hytk)。该基因的表达产生了赋予用于体外阳性选择的潮霉素b耐药性以及用于体内阴性选择的更昔洛韦敏感性的多肽。还参见s.d.lupton的pctus91/08442和pct/us94/05601的出版物,其描述了通过使显性阳性可选择标志物与阴性可选择标志物融合而得到的双功能可选择融合基因的使用。
优选的阳性可选择标志物衍生自基因,所述基因选自由以下组成的组:hph、nco和gpt,优选的阴性可选择标志物衍生自基因,所述基因选自由以下组成的组:胞嘧啶脱氨酶、hsv-itk、vzvtk、hprt、aprt和gpt。特定实施例中考虑的示例性双功能可选择融合基因包含但不限于其中阳性可选择标志物衍生自hph或neo,阴性可选择标志物衍生自胞嘧啶脱氨酶或tk基因或可选择标志物的基因。
在特定实施例中,可以通过非病毒和病毒方法将编码一种或多种归巢核酸内切酶变体、megatal、末端加工酶或融合多肽的多核苷酸引入造血细胞,例如cd34+细胞。在特定实施例中,可以通过相同的方法或通过不同的方法,和/或通过相同的载体或通过不同的载体提供一种或多种编码核酸酶和/或供体修复模板的多核苷酸的递送。
术语“载体”在本文中用于指能够转移或转运另一种核酸分子的核酸分子。转移的核酸通常与载体核酸分子连接,例如插入载体核酸分子中。载体可以包含指导细胞中自主复制的序列,或者可以包含足以允许整合到宿主细胞dna中的序列。在特定实施例中,非病毒载体用于将本文考虑的一个或多个多核苷酸递送到cd34+细胞。
非病毒载体的说明性实例包含但不限于质粒(例如,dna质粒或rna质粒)、转座子、粘粒和细菌人工染色体。
特定实施例中考虑的非病毒递送多核苷酸的说明性方法包含但不限于:电穿孔、声致穿孔、脂质转染、显微注射、基因枪(biolistics)、病毒体、脂质体、免疫脂质体、纳米粒子、聚阳离子或脂质:核酸缀合物、裸dna、人工病毒粒子、deae-葡聚糖介导的转移、基因枪和热休克。
适用于特定实施例中考虑的特定实施例的多核苷酸递送系统的说明性实例包含但不限于由amaxabiosystems、maxcyte,inc.、btxmoleculardeliverysystems和copernicustherapeuticsinc.提供的那些。脂质转染试剂是市售的(例如,transfectamtm和lipofectintm)。适用于多核苷酸的有效受体识别脂质转染的阳离子和中性脂质已在文献中描述。参见例如liu等人(2003年),《基因疗法(genetherapy)》,10:180–187;和balazs等人(2011年),《药物递送杂志(journalofdrugdelivery)》,2011:1-12。在特定实施例中还考虑了抗体靶向的、细菌衍生的、基于非生物纳米细胞的递送。
包括特定实施例中考虑的多核苷酸的病毒载体可以通过向个体患者施用而体内递送,通常通过全身给药(例如,静脉内、腹膜内、肌肉内、皮下或颅内输注)或局部给予,如下所描述。可替代地,可以将载体离体递送到细胞,例如从个体患者(例如,动员的外周血、淋巴细胞、骨髓穿刺液、组织活检等)或通用供体造血干细胞移出的细胞,然后将细胞重新植入患者体内。
在一个实施例中,将包括核酸酶变体和/或供体修复模板的病毒载体直接施用于生物体以在体内转导细胞。可替代地,可以施用裸dna或mrna。施用是通过通常用于引入分子与血液或组织细胞最终接触的任何途径,包含但不限于注射、输注、局部给予和电穿孔。施用此类核酸的合适方法是本领域技术人员可获得的并且是公知的,并且尽管可以使用多于一种途径来施用特定组合物,但是特定途径通常可以提供比另一途径更直接且更有效的反应。
适用于本文考虑的特定实施例的病毒载体系统的说明性实例包含但不限于腺相关病毒(aav)、逆转录病毒、单纯疱疹病毒、腺病毒和牛痘病毒载体。
在各个实施例中,通过用包括一个或多个多核苷酸的重组腺相关病毒(raav)转导细胞,将编码核酸酶变体和/或供体修复模板的一个或多个多核苷酸引入造血细胞中,例如造血干细胞或祖细胞中,或cd34+细胞中。
aav是一种小的(~26nm)复制缺陷型(主要是附加型)无包膜病毒。aav可以感染分裂细胞和非分裂细胞,并且可以将其基因组掺入宿主细胞的基因组中。重组aav(raav)通常至少由转基因及其调节序列和5'和3'aav反向末端重复(itr)构成。itr序列的长度为约145bp。在特定实施例中,raav包括与aav1、aav2、aav3、aav4、aav5、aav6、aav7、aav8、aav9或aav10分离的itr和衣壳序列。
在一些实施例中,使用嵌合raav,itr序列从一种aav血清型分离,并且衣壳序列从不同的aav血清型分离。例如,具有衍生自aav2的itr序列和衍生自aav6的衣壳序列的raav称为aav2/aav6。在特定实施例中,raav载体可以包括来自aav2的itr,和来自aav1、aav2、aav3、aav4、aav5、aav6、aav7、aav8、aav9或aav10中任一种的衣壳蛋白。在一个优选实施例中,raav包括衍生自aav2的itr序列和衍生自aav6的衣壳序列。在一个优选实施例中,raav包括衍生自aav2的itr序列和衍生自aav2的衣壳序列。
在一些实施例中,可以将改造和选择方法应用于aav衣壳,以使它们更可能转导目标细胞。
raav载体的构建及其生产和纯化已公开于例如美国专利第9,169,494号;第9,169,492号;第9,012,224号;第8,889,641号;第8,809,058号;和第8,784,799号,其每一个通过引用整体并入本文。
在各个实施例中,通过用包括一个或多个多核苷酸的逆转录病毒(例如,慢病毒)转导细胞,将编码核酸酶变体和/或供体修复模板的一个或多个多核苷酸引入造血细胞中,例如造血干细胞或祖细胞中,或cd34+细胞中。在一个实施例中,通过用整合酶缺陷型慢病毒转导细胞,将核酸酶变体和/或供体修复模板引入造血细胞中,例如造血干细胞或祖细胞中,或cd34+细胞中。
如本文使用,术语“逆转录病毒”是指rna病毒,其将其基因组rna逆转录为线性双链dna拷贝,随后将其基因组dna共价整合到宿主基因组中。适用于特定实施例的说明性逆转录病毒包含但不限于:莫洛尼小鼠白血病病毒(m-mulv)、莫洛尼小鼠肉瘤病毒(momsv)、哈维小鼠肉瘤病毒(hamusv)、小鼠乳腺肿瘤病毒(mumtv)、长臂猿白血病病毒(galv)、猫白血病病毒(flv)、泡沫病毒、弗里德小鼠白血病病毒、小鼠干细胞病毒(mscv)和劳氏肉瘤病毒(rsv)和慢病毒。
如本文使用,术语“慢病毒”是指复杂逆转录病毒的组(或属)。说明性慢病毒包含但不限于:hiv(人免疫缺陷病毒;包含hiv1型和hiv2型);维斯纳-梅依迪病毒(vmv)病毒;山羊关节炎-脑炎病毒(caev);马传染性贫血病毒(eiav);猫免疫缺陷病毒(fiv);牛免疫缺陷病毒(biv);和猿猴免疫缺陷病毒(siv)。在一个实施例中,优选基于hiv的载体骨架(即,hiv顺式作用序列元件)。
在各个实施例中,本文考虑的慢病毒载体包括一种或多种ltr,以及以下辅助元件中的一种或多种或全部:cppt/flap、psi(ψ)包装信号、输出元件、聚(a)序列,并且可以任选地包括wpre或hpre、绝缘子元件、可选择标志物和细胞自杀基因,如本文其它地方所讨论。
在特定实施例中,本文考虑的慢病毒载体可以是整合型慢病毒或非整合型慢病毒或整合缺陷型慢病毒。如本文使用,术语“整合缺陷型慢病毒”或“idlv”是指具有整合酶的慢病毒,所述整合酶缺少将病毒基因组整合到宿主细胞基因组中的能力。在专利申请wo2006/010834中已描述了不能整合的病毒载体,该专利申请通过引用整体并入本文。
适于降低整合酶活性的hiv-1pol基因中的说明性突变包含但不限于:h12n、h12c、h16c、h16v、s81r、d41a、k42a、h51a、q53c、d55v、d64e、d64v、e69a、k71a、e85a、e87a、d116n、d1161、d116a、n120g、n1201、n120e、e152g、e152a、d35e、k156e、k156a、e157a、k159e、k159a、k160a、r166a、d167a、e170a、h171a、k173a、k186q、k186t、k188t、e198a、r199c、r199t、r199a、d202a、k211a、q214l、q216l、q221l、w235f、w235e、k236s、k236a、k246a、g247w、d253a、r262a、r263a和k264h。
在一个实施例中,hiv-1整合酶缺陷型pol基因包括d64v、d116i、d116a、e152g或e152a突变;d64v、d116i和e152g突变;或d64v、d116a和e152a突变。
在一个实施例中,hiv-1整合酶缺陷型pol基因包括d64v突变。
术语“长末端重复(ltr)”是指位于逆转录病毒dna末端的碱基对的结构域,其在其天然序列环境中是直接重复并含有u3、r和u5区。
如本文使用,术语“flap元件”或“cppt/flap”是指核酸,其序列包含逆转录病毒(例如,hiv-1或hiv-2)的中心多嘌呤管道和中心终止序列(cppt和cts)。合适的flap元件描述于美国专利第6,682,907号和zennou等人,2000年,《细胞(cell)》,101:173。在另一个实施例中,慢病毒载体含有在cppt和/或cts元件中具有一个或多个突变的flap元件。在又一个实施例中,慢病毒载体包括cppt或cts元件。在又一个实施例中,慢病毒载体不包括cppt或cts元件。
如本文使用,术语“包装信号”或“包装序列”是指位于逆转录病毒基因组内的psi[ψ]序列,其是将病毒rna插入病毒衣壳或粒子所需的,参见例如clever等人,1995年,《病毒学杂志(j.ofvirology)》,第69卷,第4期;第2101-2109页。
术语“输出元件”是指顺式作用的转录后调节元件,其调节rna转录物从细胞核到细胞质的转运。rna输出元件的实例包含但不限于人免疫缺陷病毒(hiv)rev反应元件(rre)(参见例如cullen等人,1991年,《病毒学杂志(j.virol.)》,65:1053;和cullen等人,1991年,《细胞(cell)》,58:423),和乙型肝炎病毒转录后调节元件(hpre)。
在特定实施例中,通过将转录后调节元件、有效的多聚腺苷酸化位点和任选的转录终止信号掺入载体中来增加病毒载体中异源序列的表达。多种转录后调节元件可以增加异源核酸在蛋白质处的表达,例如土拨鼠肝炎病毒转录后调节元件(wpre;zufferey等人,1999年,《病毒学杂志(j.virol.)》,73:2886);乙型肝炎病毒中存在的转录后调节元件(hpre)(huang等人,《分子细胞生物学(mol.cell.biol.)》,5:3864);等(liu等人,1995年,《基因发展(genesdev.)》,9:1766)。
由于修饰ltr,慢病毒载体优选含有几种安全性增强作用。“自失活”(sin)载体是指复制缺陷型载体,例如,其中右(3')ltr增强子-启动子区(称为u3区)已被修饰(例如,通过缺失或取代)以防止病毒转录超过第一轮病毒复制。通过用异源启动子替换5'ltr的u3区来提供额外的安全性增强,以在病毒粒子的产生过程中驱动病毒基因组的转录。可以使用的异源启动子的实例包含例如病毒猿猴病毒40(sv40)(例如,早期或晚期)、巨细胞病毒(cmv)(例如,立即早期)、莫洛尼小鼠白血病病毒(momlv)、劳氏肉瘤病毒(rsv)和单纯疱疹病毒(hsv)(胸苷激酶)启动子。
本文使用的术语“假型”或“假型分型”是指其病毒包膜蛋白已被具有优选特性的另一种病毒的病毒包膜蛋白取代的病毒。例如,hiv可以用水疱性口炎病毒g蛋白(vsv-g)包膜蛋白假型化,这允许hiv感染更广泛的细胞,因为hiv包膜蛋白(由env基因编码)通常将病毒靶向cd4+呈递细胞。
在某些实施例中,根据已知方法生产慢病毒载体。参见例如kutner等人,《bmc生物技术(bmcbiotechnol.)》,2009;9:10.doi:10.1186/1472-6750-9-10;kutner等人,《自然实验手册(nat.protoc.)》,2009;4(4):495–505.doi:10.1038/nprot.2009.22。
根据本文考虑的某些具体实施例,大多数或所有病毒载体骨架序列衍生自慢病毒,例如hiv-1。然而,应当理解,可以使用或组合许多不同的逆转录病毒和/或慢病毒序列来源,并且可以适应某些慢病毒序列中的许多取代和改变而不损害转移载体执行本文所述功能的能力。此外,本领域已知多种慢病毒载体,参见naldini等人,(1996年a、1996年b和1998年);zufferey等人,(1997年);dull等人,1998年,美国专利第6,013,516号;和第5,994,136号,其中许多可以适于生产本文考虑的病毒载体或转移质粒。
在各个实施例中,通过用包括一个或多个多核苷酸的腺病毒转导细胞,将编码核酸酶变体和/或供体修复模板的一个或多个多核苷酸引入造血细胞中,例如造血干细胞或祖细胞中,或cd34+细胞中。
基于腺病毒的载体在许多细胞类型中具有非常高的转导效率,并且不需要细胞分裂。使用此类载体,已经获得了高滴度和高水平的表达。该载体可以在相对简单的系统中大量生产。大多数腺病毒载体被改造,使得转基因取代ade1a、e1b和/或e3基因;随后,复制缺陷型载体在人293细胞中繁殖,该细胞反式提供缺失的基因功能。ad载体可以在体内转导多种类型的组织,包含非分裂的分化细胞,例如在肝、肾和肌肉中发现的那些。常规ad载体具有较大承载能力。
当前的复制缺陷型腺病毒载体的生成和繁殖可以利用命名为293的独特辅助细胞系,其通过ad5dna片段从人胚胎肾细胞转化并组成型表达e1蛋白(graham等人,1977年)。由于e3区是腺病毒基因组中非必需的(jones和shenk,1978年),当前的腺病毒载体,在293细胞的辅助下,在e1区、d3区或两个区承载外源dna(graham和prevec,1991年)。腺病毒载体已用于真核基因表达(levrero等人,1991年;gomez-foix等人,1992年)和疫苗开发(grunhaus和horwitz,1992年;graham和prevec,1992年)。向不同组织施用重组腺病毒的研究包含气管滴注(rosenfeld等人,1991年;rosenfeld等人,1992年)、肌肉注射(ragot等人,1993年)、外周静脉注射(herz和gerard,1993年)和立体定向接种到大脑(legallasalle等人,1993年)。在临床试验中使用ad载体的实例涉及用于肌肉内注射的抗肿瘤免疫的多核苷酸疗法(sterman等人,《人类基因疗法(hum.genether.)》,7:1083-9(1998))。
在各个实施例中,通过用包括一个或多个多核苷酸的单纯疱疹病毒(例如,hsv-1、hsv-2)转导细胞,将编码核酸酶变体和/或供体修复模板的一个或多个多核苷酸引入造血细胞中,例如造血干细胞或祖细胞中,或cd34+细胞中。
成熟的hsv病毒粒子由包膜的二十面体衣壳组成,其中病毒基因组由152kb的线性双链dna分子组成。在一个实施例中,基于hsv的病毒载体缺少一种或多种必需或非必需的hsv基因。在一个实施例中,基于hsv的病毒载体是复制缺陷型的。大多数复制缺陷型hsv载体含有缺失以去除一个或多个中早期、早期或晚期hsv基因以防止复制。例如,hsv载体可能缺少立即早期基因,所述立即早期基因选自由以下组成的组:icp4、icp22、icp27、icp47及其组合。hsv载体的优点是其进入潜伏期的能力,其可以导致长期dna表达,并且其较大的病毒dna基因组可以容纳高达25kb的外源dna插入物。基于hsv的载体描述于例如美国专利第5,837,532号、第5,846,782号和第5,804,413号,以及国际专利申请wo91/02788、wo96/04394、wo98/15637和wo99/06583,它们各自通过引用整体并入本文。
h.基因组编辑的细胞
通过特定实施例中考虑的方法制备的基因组编辑的细胞提供用于治疗血红蛋白病的改进的基于细胞的治疗剂。不希望受任何特定理论的束缚,据信本文考虑的组合物和方法共同选择胎儿球蛋白转换机制,以提供更强健的基因组编辑的细胞组合物,其可以用于治疗(并且在一些实施例中可能治愈)血红蛋白病。
特定实施例中考虑的基因组编辑的细胞可以是自体的(autologous/autogeneic)(“自身的”)或非自体的(“非自身的”,例如同种异体的、同源的或异种的)。如本文使用,“自体的”是指来自相同受试者的细胞。如本文使用,“同种异体的”是指相同物种的与比较细胞在遗传学上不同的的细胞。如本文使用,“同源的”是指不同受试者的与比较细胞在遗传学相同的细胞。如本文使用,“异种的”是指与比较细胞不同的物种的细胞。在优选的实施例中,细胞获自哺乳动物受试者。在一个更优选的实施例中,细胞获自灵长类动物受试者,任选地获自非人灵长类动物。在最优选的实施例中,细胞获自人类受试者。
“分离的细胞”是指非天然存在的细胞,例如自然界中不存在的细胞、修饰的细胞、改造的细胞等,其已经从体内组织或器官获得并且基本上不含细胞外基质。
可以使用本文考虑的组合物和方法编辑其基因组的细胞类型的说明性实例包含但不限于细胞系、原代细胞、干细胞、祖细胞和分化细胞。
术语“干细胞”是指一种细胞,其是未分化细胞,能够(1)长期自我更新,或能够生成原始细胞的至少一个相同拷贝,(2)在单细胞水平上分化成多个,在一些情况下分化成仅一个特化细胞类型和(3)组织的体内功能性再生。根据干细胞的发育潜能将干细胞细分为全能、多能、专能和寡能/单能。“自我更新”是指具有产生未改变的子细胞并生成特化细胞类型(效力)的独特能力的细胞。自我更新可以通过两种方式实现。不对称细胞分裂产生一个与亲代细胞相同的子细胞和一个与亲代细胞不同且是祖细胞或分化细胞的子细胞。对称细胞分裂产生两个相同的子细胞。细胞的“增殖”或“扩增”是指对称分裂的细胞。
如本文使用,术语“祖细胞(progenitor/progenitorcell)”是指具有自我更新和分化成更成熟细胞的能力的细胞。许多祖细胞沿着单一谱系分化,但是可能具有相当广泛的增殖能力。
在特定实施例中,细胞是原代细胞。本文使用的术语“原代细胞”在本领域中是已知的,是指已从组织中分离并已被建立用于体外或离体生长的细胞。相应的细胞经历了非常少的群体倍增(如果有的话),因此与连续细胞系相比更能代表它们所衍生自的组织的主要功能组分,因此代表了体内状态的更具代表性的模型。从各种组织获得样品的方法和建立原代细胞系的方法是本领域公知的(参见例如jones和wise,《分子生物学方法(methodsmolbiol.)》,1997年)。用于本文考虑的方法的原代细胞衍生自脐带血、胎盘血、动员的外周血和骨髓。在一个实施例中,原代细胞是造血干细胞或祖细胞。
在一个实施例中,基因组编辑的细胞是胚胎干细胞。
在一个实施例中,基因组编辑的细胞是成体干细胞或祖细胞。
在一个实施例中,基因组编辑的细胞是原代细胞。
在一个优选实施例中,基因组编辑的细胞是造血细胞,例如造血干细胞、造血祖细胞、红系细胞或包括造血细胞的细胞群。
如本文使用,术语“细胞群”是指多个细胞,其可以由任何数量和/或组合的同质或异质细胞类型构成,如本文其它地方所述。例如,为了转导造血干细胞或祖细胞,可以从脐带血、胎盘血、骨髓或动员的外周血分离或获得细胞群。细胞群可以占待编辑的靶细胞类型的约10%、约20%、约30%、约40%、约50%、约60%、约70%、约80%、约90%或约100%。在某些实施例中,可以使用本领域已知的方法从异质细胞群分离或纯化造血干细胞或祖细胞。
获得造血细胞的示例性来源把汗但不限于:脐带血、骨髓或动员的外周血。
造血干细胞(hsc)产生定型的造血祖细胞(hpc),其能够在生物体的整个寿命期间生成整个成熟血细胞库。术语“造血干细胞”或“hsc”是指专能干细胞,其产生生物体的所有血细胞类型,包含骨髓(例如,单核细胞和巨噬细胞、中性粒细胞、嗜碱性粒细胞、嗜酸性粒细胞、红细胞、巨核细胞/血小板、树突细胞)和淋巴谱系(例如,t细胞、b细胞、nk细胞)和本领域已知的其它细胞(参见fei,r.等人,美国专利第5,635,387号;mcglave等人,美国专利第5,460,964号;simmons,p.等人,美国专利第5,677,136号;tsukamoto等人,美国专利第5,750,397号;schwartz等人,美国专利第5,759,793号;diguisto等人,美国专利第5,681,599号;tsukamoto等人,美国专利第5,716,827号)。当移植到致命辐射的动物或人类中时,造血干细胞和祖细胞可以再植红系、中性粒细胞-巨噬细胞、巨核细胞和淋巴造血细胞库。
适用于本文考虑的方法和组合物的造血干细胞或祖细胞的另外的说明性实例包含为cd34+cd38locd90+cd45ra-的造血细胞,为cd34+、cd59+、thy1/cd90+、cd38lo/-、c-kit/cd117+和lin(-)的造血细胞,和为cd133+的造血细胞。
在一个优选实施例中,造血细胞是cd133+cd90+。
在一个优选实施例中,造血细胞是cd133+cd34+。
在一个优选实施例中,造血细胞是cd133+cd90+cd34+。
存在各种方法来表征造血层级结构。一种表征方法是slam代码。slam(信号淋巴细胞激活分子)家族是一组>10个分子,其基因大部分串联位于染色体1(小鼠)的单个基因座中,都属于免疫球蛋白基因超家族的一个子集,最初被认为参与了t细胞刺激。该家族包含cd48、cd150、cd244等,cd150是创始成员,因此也称为slamf1,即slam家族成员1。造血层级结构的签名slam代码是造血干细胞(hsc)-cd150+cd48-cd244-;专能祖细胞(mpp)-cd150-cd48-cd244+;谱系限制性祖细胞(lrp)-cd150-cd48+cd244+;常见的髓系祖细胞(cmp)-lin-sca-1-c-kit+cd34+cd16/32mid;粒细胞-巨噬细胞祖细胞(gmp)-lin-sca-1-c-kit+cd34+cd16/32hi;和巨核细胞-红系祖细胞(mep)-lin-sca-1-c-kit+cd34-cd16/32low。
用本文考虑的组合物和方法编辑的优选靶细胞类型包含造血细胞,优选人造血细胞,更优选人造血干细胞和祖细胞,甚至更优选cd34+人造血干细胞。如本文使用,术语“cd34+细胞”是指在其细胞表面上表达cd34蛋白的细胞。如本文使用,“cd34”是指细胞表面糖蛋白(例如,唾液黏蛋白),其通常充当细胞-细胞粘附因子。cd34+是造血干细胞和祖细胞的细胞表面标志物。
在一个实施例中,基因组编辑的造血细胞是cd150+cd48-cd244-细胞。
在一个实施例中,基因组编辑的造血细胞是cd34+cd133+细胞。
在一个实施例中,基因组编辑的造血细胞是cd133+细胞。
在一个实施例中,基因组编辑的造血细胞是cd34+细胞。
在特定实施例中,包括造血干细胞和祖细胞(hspc)的造血细胞群包括编辑的bcl11a基因,其中编辑是由nhej修复的dsb。编辑可以在bcl11a基因的红系特异性增强子中,优选在bcl11a基因的gata-1结合位点中,更优选在bcl11a基因的第二内含子中的共有gata-1结合位点中。
在特定实施例中,包括造血干细胞和祖细胞(hspc)的造血细胞群包括编辑的bcl11a基因,其包括在bcl11a基因中的红系特异性增强子中,优选在bcl11a基因中的gata-1结合位点中,更优选在bcl11a基因的第二内含子中的共有gata-1结合位点中,甚至更优选在seqidno:25(其互补序列包含共有gata-1基序wgatar)所示的靶位点中的约1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个或更多个核苷酸的插入或缺失(indel);从而降低、减少或消除bcl11a表达。
在一个实施例中,编辑是在bcl11a基因中的红系特异性增强子中,优选在bcl11a基因中的gata-1结合位点中,更优选在bcl11a基因的第二内含子中的共有gata-1结合位点中,甚至更优选在seqidno:25(其互补序列包含共有gata-1基序wgatar)所示的靶位点中的1个核苷酸的插入或约1个、2个、3个或4个核苷酸的缺失;从而降低、减少或消除bcl11a表达。
在特定实施例中,基因组编辑的细胞包括红系细胞。
在特定实施例中,基因组编辑的细胞包括β-球蛋白基因中的一个或多个突变。在一个实施例中,受试者的β-球蛋白等位基因选自由以下组成的组:βe/β0、βc/β0、β0/β0、βe/βe、βc/β+、βe/β+、β0/β+、β+/β+、βc/βc、βe/βs、β0/βs、βc/βs、β+/βs或βs/βs。
在特定实施例中,基因组编辑的细胞包括导致地中海贫血的β-球蛋白基因中的一个或多个突变。在一个实施例中,地中海贫血是α-地中海贫血。在一个实施例中,地中海贫血是β-地中海贫血。在一个实施例中,受试者的β-球蛋白等位基因选自由以下组成的组:βe/β0、βc/β0、β0/β0、βc/βc、βe/βe、βe/β+、βc/βe、βc/β+、β0/β+或β+/β+。
在特定实施例中,基因组编辑的细胞包括导致镰状细胞病的β-球蛋白基因中的一个或多个突变。在一个实施例中,受试者的β-球蛋白βe/βs、β0/βs、βc/βs、β+/βs或βs/βs。
i.组合物和制剂
特定实施例中考虑的组合物可以包括一个或多个多肽、多核苷酸、包括其的载体以及基因组编辑组合物和基因组编辑的细胞组合物,如本文所考虑。特定实施例中考虑的基因组编辑组合物和方法可用于编辑细胞或细胞群中人bcl11a基因中的靶位点。在优选实施例中,基因组编辑组合物用于编辑造血细胞(例如,造血干细胞或祖细胞,或cd34+细胞)中的bcl11a基因。
在各个实施例中,本文考虑的组合物包括核酸酶变体,和任选的末端加工酶,例如3'-5'核酸外切酶(trex2)。核酸酶变体可以是mrna的形式,其通过上文公开的多核苷酸递送方法多(例如,电穿孔、脂质纳米颗粒等)引入细胞中。在一个实施例中,包括编码归巢核酸内切酶变体或megatal和任选的3'-5'核酸外切酶的mrna的组合物通过上文公开的多核苷酸递送方法引入细胞中。该组合物可以用于通过容易出错的nhej生成基因组编辑的细胞或基因组编辑的细胞群。
在特定实施例中,本文考虑的组合物包括细胞群、核酸酶变体和任选的供体修复模板。在特定实施例中,本文考虑的组合物包括细胞群、核酸酶变体、末端加工酶和任选的供体修复模板。核酸酶变体和/或末端加工酶可以是mrna的形式,其通过上文公开的多核苷酸递送方法引入细胞中。
在特定实施例中,本文考虑的组合物包括细胞群、归巢核酸内切酶变体或megatal和任选的供体修复模板。在特定实施例中,本文考虑的组合物包括细胞群、归巢核酸内切酶变体或megatal、3'-5'核酸外切酶和任选的供体修复模板。归巢核酸内切酶变体、megatal和/或3'-5'核酸外切酶可以是mrna的形式,其通过上文公开的多核苷酸递送方法引入细胞中。
在特定实施例中,细胞群包括遗传修饰的造血细胞,包含但不限于造血干细胞、造血祖细胞、cd133+细胞和cd34+细胞。
组合物包含但不限于药物组合物。“药物组合物”是指药学上可接受的或生理学上可接受的溶液中配制的组合物,其单独或与一种或多种其它治疗方式组合施用于细胞或动物。还应理解,如果需要,组合物也可以与其它试剂联合施用,例如细胞因子、生长因子、激素、小分子、化学治疗剂、前药、药物、抗体或其它各种药物活性剂。对组合物中也可能包含的其它组分实际上没有限制,条件是另外的试剂不会不利地影响组合物。
短语“药学上可接受的”在本文中用于指在合理的医学判断范围内适合用于与人类和动物的组织接触而不引起过量毒性、刺激、过敏反应或其它问题或并发症,与合理的利益/风险比相称的那些化合物、材料、组合物和/或剂型。
术语“药学上可接受的载剂”是指与治疗细胞一起施用的稀释剂、佐剂、赋形剂或媒剂。药物载剂的说明性实例可以是无菌液体,例如细胞培养基、水和油,所述油包含石油、动物、植物或合成来源的那些,例如花生油、大豆油、矿物油、芝麻油等。盐水溶液和葡萄糖水溶液和甘油溶液也可以用作液体载剂,特别是用于可注射溶液。在特定实施例中,合适的药物赋形剂包含淀粉、葡萄糖、乳糖、蔗糖、明胶、麦芽、大米、面粉、白垩、硅胶、硬脂酸钠、单硬脂酸甘油酯、滑石、氯化钠、脱脂奶粉、甘油、丙烯、乙二醇、水、乙醇等。除非任何常规培养基或试剂与活性成分不相容,否则考虑其在治疗组合物中的使用。补充的活性成分也可以掺入组合物中。
在一个实施例中,包括药学上可接受的载剂的组合物适于施用于受试者。在特定实施例中,包括载剂的组合物适于肠胃外施用,例如血管内(静脉内或动脉内),腹膜内或肌肉内施用。在特定实施例中,包括药学上可接受的载剂的组合物适用于心室内、脊柱内或鞘内施用。药学上可接受的载剂包括无菌水溶液、细胞培养基或分散体。这些培养基和试剂用于药物活性物质的用途是本领域熟知的。除非任何常规培养基或试剂与转导细胞不相容,否则考虑其在药物组合物中的使用。
在特定实施例中,本文考虑的组合物包括遗传修饰的造血干细胞和/或祖细胞和药学上可接受的载剂。包括本文考虑的基于细胞的组合物的组合物可以通过肠内或肠胃外施用方法单独施用或与其它合适的化合物组合施用以实现期望的治疗目标。
药学上可接受的载剂必须具有足够高的纯度和足够低的毒性,以使其适合于向待治疗的人类受试者施用。它还应该保持或增加组合物的稳定性。药学上可接受的载剂可以是液体或固体,并且考虑到计划的施用方式,当与组合物的其它组分组合时,选择药学上可接受的载剂以提供所需的体积、稠度等。例如,药学上可接受的载剂可以是但不限于粘合剂(例如,预胶化的玉米淀粉、聚乙烯吡咯烷酮或羟丙基甲基纤维素等)、填充剂(例如,乳糖和其它糖、微晶纤维素、果胶、明胶、硫酸钙、乙基纤维素、聚丙烯酸酯、磷酸氢钙等)、润滑剂(例如,硬脂酸镁、滑石、二氧化硅、胶体二氧化硅、硬脂酸、金属硬脂酸盐、氢化植物油、玉米淀粉、聚乙二醇、苯甲酸钠、乙酸钠等)、崩解剂(例如,淀粉、羟基乙酸淀粉钠等)、或润湿剂(例如,月桂基硫酸钠等)。用于本文考虑的组合物的其它合适的药学上可接受的载剂包含但不限于水、盐溶液、醇、聚乙二醇、明胶、直链淀粉、硬脂酸镁、滑石、硅酸、粘性石蜡、羟甲基纤维素、聚乙烯吡咯烷酮等。
这些载剂溶液还可以含有缓冲剂、稀释剂和其它合适的添加剂。本文使用的术语“缓冲剂”是指其化学组成中和酸或碱而没有显著ph变化的溶液或液体。本文考虑的缓冲剂的实例包含但不限于杜尔贝科磷酸盐缓冲盐水(pbs)、林格氏溶液、5%葡萄糖水溶液(d5w)、正常/生理盐水(0.9%nacl)。
药学上可接受的载剂可以以足以维持组合物的ph为约7的量存在。可替代地,组合物的ph在约6.8到约7.4的范围内,例如为6.8、6.9、7.0、7.1、7.2、7.3和7.4。在再一个实施例中,组合物的ph为约7.4。
本文考虑的组合物可以包括无毒的药学上可接受的培养基。该组合物可以是混悬液。本文使用的术语“混悬”是指其中细胞不附着于固体支持物的非粘附条件。例如,可以对保持为混悬液的细胞搅动或搅拌,并且不将其粘附到支持物上,例如培养皿。
在特定实施例中,本文考虑的组合物配制在混悬液中,其中基因组编辑的造血干细胞和/或祖细胞分散在可接受的液体培养基或溶液(例如,盐水或无血清培养基)、静脉输液(iv)袋等等中。可接受的稀释剂包含但不限于水、勃脉力、林格氏溶液、等渗氯化钠(盐水)溶液、无血清细胞培养基和适于低温储存的培养基(例如,
在某些实施例中,药学上可接受的载剂基本上不含人类或动物来源的天然蛋白质,并且适合于储存包括基因组编辑的细胞群(例如,造血干细胞和祖细胞)的组合物。治疗组合物旨在施用于人类患者,因此基本上不含细胞培养组分,例如牛血清白蛋白、马血清和胎牛血清。
在一些实施例中,将组合物配制在药学上可接受的细胞培养基中。此类组合物适合于向人类受试者施用。在特定实施例中,药学上可接受的细胞培养基是无血清培养基。
与含有血清的培养基相比,无血清培养基具有几个优点,包含简化并更好地定义了组合物,降低了污染程度,消除了传染源的潜在来源并降低了成本。在各个实施例中,无血清培养基是无动物的,并且可以任选地不含蛋白质。任选地,培养基可以含有生物药学上可接受的重组蛋白。“无动物”培养基是指其中组分来源于非动物来源的培养基。重组蛋白质在无动物培养基中代替天然动物蛋白质,并且营养物质从合成、植物或微生物来源获得。相反,“无蛋白”培养基定义为基本上不含蛋白质。
用于特定组合物的无血清培养基的说明性实例包含但不限于qbsf-60(qualitybiological,inc.)、stempro-34(lifetechnologies)和x-vivo10。
在优选实施例中,将包括基因组编辑的造血干细胞和/或祖细胞的组合物配制在勃脉力中。
在各个实施例中,将包括造血干细胞和/或祖细胞的组合物配制在低温保存培养基中。例如,具有低温保存剂的低温保存培养基可以用于在解冻后维持高细胞活力结果。用于特定组合物的低温保存培养基的说明性实例包含但不限于cryostorcs10、cryostorcs5和cryostorcs2。
在一个实施例中,将组合物配制在包括50:50的勃脉力a与cryostorcs10的溶液中。
在特定实施例中,该组合物基本上不含支原体、内毒素和微生物污染。相对于内毒素而言,“基本上不含”是指每细胞剂量的内毒素含量少于fda针对生物剂所允许的含量,其为每天5eu/kg体重的总内毒素,即对于平均70公斤的人而言,每总细胞剂量为350eu。在特定实施例中,包括用本文考虑的逆转录病毒载体转导的造血干细胞或祖细胞的组合物含有约0.5eu/ml到约5.0eu/ml,或约0.5eu/ml、1.0eu/ml、1.5eu/ml、2.0eu/ml、2.5eu/ml、3.0eu/ml、3.5eu/ml、4.0eu/ml、4.5eu/ml或5.0eu/ml。
在某些实施例中,考虑了适合于递送多核苷酸的组合物和制剂,包含但不限于编码一种或多种重编程的核酸酶和任选的末端加工酶的一种或多种mrna。
用于离体递送的示例性制剂还可以包含使用本领域已知的各种转染剂,例如磷酸钙、电穿孔、热休克和各种脂质体制剂(即脂质介导的转染)。如下面更详细描述,脂质体是包裹一部分水性流体的脂质双层。dna自发地缔合到阳离子脂质体的外表面(凭借其电荷),并且这些脂质体将与细胞膜相互作用。
在特定实施例中,药学上可接受的载剂溶液的制剂是本领域技术人员公知的,例如开发合适的给药和治疗方案以将本文所述的特定组合物用于各种治疗方案中,包含例如肠内和肠胃外,例如血管内、静脉内、动脉内、骨内、心室内、脑内、颅内、脊柱内、鞘内和髓内施用和配制。本领域技术人员将理解,本文考虑的特定实施例可以包括其它制剂,例如制药领域熟知的制剂,其描述于例如《雷明顿:药学的科学与实践(remington:thescienceandpracticeofpharmacy)》,第i卷和第ii卷,第22版,loydv.allenjr.编辑,pa:医药出版社;2012年中,其全部内容通过引用并入本文。
j.基因组编辑的细胞疗法
通过特定实施例中考虑的方法制备的基因组编辑的细胞提供了改进的药物产品,用于预防、治疗和改善血红蛋白病或用于预防、治疗或改善与血红蛋白病或具有β-球蛋白基因中的血红蛋白病变异的受试者相关的至少一种症状。如本文使用,术语“药物产品”是指使用本文考虑的组合物和方法生产的遗传修饰的细胞。在特定实施例中,药物产品包括遗传修饰的造血干细胞或祖细胞,例如cd34+细胞。遗传修饰的造血干细胞或祖细胞产生具有增加的γ-球蛋白基因表达的成体红系细胞,并且允许治疗在体内没有γ-球蛋白基因表达或具有最小γ-球蛋白基因表达的受试者,从而显著扩大了将基因组编辑的细胞疗法用于受试者的机会,此类型的治疗以前并不是受试者的可行治疗选项。
在特定实施例中,基因组编辑的造血干细胞或祖细胞包括bcl11a基因中的非功能性或破坏的、消除的或缺失的红系特异性增强子,从而减少或消除红系细胞中的功能性bcl11a表达,例如bcl11a表达不足以阻遏或抑制γ-球蛋白基因转录并反式激活β-球蛋白基因转录,从而增加红系细胞中γ-球蛋白基因表达。
在特定实施例中,基因组编辑的造血干细胞或祖细胞包括在bcl11a基因中,优选在bcl11a基因中的gata-1结合位点中,更优选在bcl11a基因的第二内含子中的共有gata-1结合位点中,甚至更优选在seqidno:25(其互补序列包含共有gata-1基序wgatar)所示的靶位点中的非功能性或破坏的、消除的或缺失的gata-1结合位点,从而减少或消除在红系细胞中的功能性bcl11a表达,导致红系细胞中γ-球蛋白基因表达的增加。
在特定实施例中,基因组编辑的造血干细胞或祖细胞为被诊断患有或疑似患有单基因疾病、病症或病状或者造血系统的疾病、病症或病状(例如,血红蛋白病)的受试者提供治愈、预防或改善疗法。
如本文使用,“造血作用”是指来自祖细胞的血细胞的形成和发育以及来自干细胞的祖细胞的形成。血细胞包含但不限于红细胞或红血细胞(rbc)、网织红细胞、单核细胞、嗜中性粒细胞、巨核细胞、嗜酸性粒细胞、嗜碱性粒细胞、b细胞、巨噬细胞、粒细胞、肥大细胞、凝血细胞和白细胞。
如本文使用,术语“血红蛋白病”或“血红蛋白病病状”是指多种遗传性血液病症,其涉及由血红蛋白的结构和/或合成的改变引起的异常血红蛋白分子的存在。通常,血红蛋白由四个蛋白质亚单位组成:两个β-球蛋白亚单位和两个α-球蛋白亚单位。这些蛋白质亚单位中的每一个都与称为血红素的含铁分子连接(结合);每个血红素在其中心含有一个铁分子,其可以与一个氧分子结合。红血细胞内的血红蛋白与肺中的氧分子结合。然后,这些细胞经过血流并将氧递送到整个身体的组织。
血红蛋白a(hba)是出生之后存在的正常血红蛋白的名称。血红蛋白a是具有两条α链和两条β链(α2β2)的四聚体。血红蛋白a2是出生之后红细胞中发现的血红蛋白的次要组分,其由两条α链和两条δ链(α2δ2)组成。血红蛋白a2通常占总红细胞血红蛋白的不到3%。血红蛋白f(hbf)是胎儿发育期间的主要血红蛋白。该分子是两条α链和两条γ链(α2γ2)的四聚体。在优选实施例中,向受试者施用基因组编辑的造血干细胞或祖细胞,其产生具有增加的γ-球蛋白基因表达和/或降低的血红蛋白病β-球蛋白基因表达的红系细胞,从而增加受试者中的hbf的量。
最常见的血红蛋白病包含镰状细胞病、β-地中海贫血和α-地中海贫血。
在特定实施例中,本文考虑的组合物和方法为患有镰状细胞病的受试者提供基因组编辑的细胞疗法。术语“镰状细胞贫血”或“镰状细胞病”在本文中定义为包含由红血细胞的镰状化引起的任何症状性贫血病状。镰状细胞贫血βs/βs是一种常见的镰状细胞病(scd),由血红蛋白s(hbs)引起。通过在β-球蛋白中的第6位处用缬氨酸(v)取代谷氨酸(e)生成hbs,记为glu6val或e6v。用缬氨酸取代谷氨酸会导致异常的hbs亚单位粘在一起,形成长而刚性的分子,其将红血细胞弯曲成镰状(新月形)。镰状细胞过早死亡,可能导致红血细胞缺少(贫血)。此外,镰状细胞是刚性的,可以阻塞小血管,导致严重的疼痛和器官损伤。
β-球蛋白基因中的另外的突变也可以引起β-球蛋白的其它异常,导致其它类型的镰状细胞病。这些异常形式的β-球蛋白通常用字母表中的字母或有时用名称表示。在这些其它类型的镰状细胞病中,一个β-球蛋白亚单位被hbs取代,而另一个β-球蛋白亚单位被不同的异常变体取代,例如血红蛋白c(hbc;记为βc的β-球蛋白等位基因)或血红蛋白e(hbe;记为βe的β-球蛋白等位基因)。
在血红蛋白sc(hbsc)病中,β-球蛋白亚单位被hbs和hbc取代。hbc是由β-球蛋白基因中的突变引起的,是患有hbc病(α2βc2)的人中发现的主要血红蛋白。当氨基酸赖氨酸在β-球蛋白中的第6位处取代氨基酸谷氨酸(记为glu6lys或e6k)时,产生hbc。hbc病相对良性,产生轻度溶血性贫血和脾肿大。hbsc病的严重程度是可变的,但是它可能与镰状细胞贫血一样严重。
当氨基酸谷氨酸在β-球蛋白中的第26位处被氨基酸赖氨酸取代(记为glu26lys或e26k)时,引起hbe。患有hbe病的人患有轻度溶血性贫血和轻度脾肿大。hbe在东南亚极为常见,并且在一些地区中与血红蛋白a频率相当。在一些情况下,hbe突变与hbs一起存在。在这些情况下,一个人可能具有更严重的与镰状细胞贫血相关的体征和症状,例如疼痛发作、贫血和脾脏功能异常。
当产生血红蛋白s和β-地中海贫血的突变一起发生时,引起称为血红蛋白镰状-β-地中海贫血(hbsbetathal)的其它病状。结合了镰状细胞病和β-零(β0;防止β-球蛋白产生的基因突变)地中海贫血的突变导致严重疾病,而与β-正(β+;降低β-球蛋白产生的基因突变)地中海贫血相结合的镰状细胞病较轻微。
如本文使用,“地中海贫血”是指以缺陷型血红蛋白产生为特征的遗传性病症。地中海贫血的实例包含α-和β-地中海贫血。
在特定实施例中,本文考虑的组合物和方法为患有β-地中海贫血的受试者提供基因组编辑的细胞疗法。β-地中海贫血是由β-球蛋白链中的突变引起的,并且可以以主要或次要形式存在。已发现β-球蛋白基因中近400个突变引起β-地中海贫血。大多数突变涉及β-球蛋白基因内或附近的单个dna构件(核苷酸)的变化。其它突变插入或缺失β-球蛋白基因中的少量核苷酸。如上所述,降低β-球蛋白产生的β-球蛋白基因突变导致一种称为β-正(β+)地中海贫血的病状。阻止细胞产生任何β-球蛋白的突变导致β-零(β0)地中海贫血。在β-地中海贫血的主要形式中,儿童在出生时是正常的,但是在出生后的第一年就会出现贫血。β-地中海贫血的次要形式产生小的红血细胞。如果仅从一位亲本那里收到有缺陷的基因,就会发生轻型地中海贫血。患有该形式的病症的人是该疾病的携带者并且通常没有症状。
hbe/β-地中海贫血是由hbe和β-地中海贫血(βe/β0,βe/β+)的组合引起的,并且产生比hbe性状或β-地中海贫血性状可见更严重的病状。该病症表现为中度严重的地中海贫血,属于中间型地中海贫血。hbe/β-地中海贫血在东南亚背景人群中最常见。
在特定实施例中,本文考虑的组合物和方法为患有α-地中海贫血的受试者提供基因组编辑的细胞疗法。α-地中海贫血是世界范围内相当常见的血液病症。每年都有数以千计的患有hb巴特综合征和hbh病的婴儿出生,特别是在东南亚。地中海国家、北非、中东、印度和中亚的人们经常发生α-地中海贫血。α-地中海贫血通常由涉及hba1和hba2基因的缺失引起。这两种基因都提供了制造称为α-球蛋白的蛋白质的指令,所述α-蛋白质是血红蛋白的组分(亚单位)。人们在每个细胞中都有hba1基因的两个拷贝和hba2基因的两个拷贝。不同类型的α-地中海贫血是由于部分或全部hba1和hba2等位基因的损失所致。
hb巴特综合征是α-地中海贫血的最严重形式,是由于所有四个α-球蛋白等位基因的损失所致。hbh病是由四个α-球蛋白等位基因中的三个的损失引起的。在这两种病状下,α-球蛋白的短缺会阻止细胞制造正常的血红蛋白。相反,细胞产生异常形式的血红蛋白,称为血红蛋白巴特(hb巴特)或血红蛋白h(hbh)。这些异常的血红蛋白分子不能有效地将氧运送到人体组织。将hb巴特或hbh替换为正常血红蛋白会导致贫血和其它与α-地中海贫血相关的严重健康问题。
另外两种α-地中海贫血变体与减少量的α-球蛋白有关。因为细胞仍然产生一些正常的血红蛋白,这些变体往往很少导致或不导致健康问题。四个α-球蛋白等位基因中的两个的损失导致α-地中海贫血性状。具有α-地中海贫血性状的人可能具有异常小的淡红血细胞并患有轻度贫血。在α-地中海贫血隐性携带者中发现一个α-球蛋白等位基因的损失。这些个体通常没有与地中海贫血相关的体征或症状。
在一个优选实施例中,本文考虑的基因组编辑的细胞疗法用于治疗、预防或改善血红蛋白病,其选自由以下组成的组:血红蛋白c病、血红蛋白e病、镰状细胞贫血、镰状细胞病(scd)、地中海贫血、β-地中海贫血、重型地中海贫血、中间型地中海贫血、α-地中海贫血、血红蛋白巴特综合征和血红蛋白h病。
在各个实施例中,基因组编辑组合物通过向需要基因治疗的受试者的细胞、组织或器官(例如,骨髓)体内直接注射来施用。在各个其它实施例中,细胞用本文考虑的重编程的核酸酶体外或离体编辑,并任选地离体扩增。然后,将基因组编辑的细胞施用于需要治疗的受试者。
用于本文考虑的基因组编辑方法的优选细胞包含自体的(“自身的”)细胞,优选造血细胞,更优选造血干细胞或祖细胞,甚至更优选cd34+细胞。
如本文使用,术语“个体”和“受试者”通常可互换使用,并且是指表现出可以用本文其它地方考虑的重编程的核酸酶、基因组编辑组合物、基因治疗载体、基因组编辑载体、基因组编辑的细胞和方法治疗的血红蛋白病的症状的任何动物。合适的受试者(例如,患者)包含实验动物(例如,小鼠、大鼠、兔或豚鼠)、农场动物和家养动物或宠物(例如,猫或狗)。包含非人灵长类动物和优选的人类患者。典型的受试者包含患有、已经被诊断患有或有风险患有血红蛋白病的人类患者。
如本文使用,术语“患者”是指已经被诊断患有血红蛋白病的受试者,所述血红蛋白病可以用本文其它地方考虑的重编程的核酸酶、基因组编辑组合物、基因治疗载体、基因组编辑载体、基因组编辑的细胞和方法治疗。
如本文使用,“治疗(treatment/treating)”包含对血红蛋白病或血红蛋白病病状的症状或病理学的任何有益或期望的作用,并且可以包含血红蛋白病或血红蛋白病病状的一种或多种可测量标志物的甚至最小量的减少。治疗可以任选地涉及延迟血红蛋白病或血红蛋白病病状的进展。“治疗”不一定表示完全根除或治愈血红蛋白病或血红蛋白病病状或其相关症状。
如本文使用,“预防(prevent)”和相似词语(例如,prevention/prevented/preventing等)表示预防、抑制或降低血红蛋白病或血红蛋白病病状发生或复发的可能性的方法。它还指延迟血红蛋白病或血红蛋白病病状的发作或复发或延迟血红蛋白病或血红蛋白病病状的症状的发生或复发。如本文使用,“预防”和相似词语还包含在血红蛋白病或血红蛋白病病状发作或复发之前降低血红蛋白病或血红蛋白病病状的强度、作用、症状和/或负荷。
如本文使用,短语“改善至少一种症状”是指降低受试者所治疗的血红蛋白病或血红蛋白病病状(例如,地中海贫血、镰状细胞病等)的一种或多种症状。在特定实施例中,所治疗的血红蛋白病或血红蛋白病病状是β-地中海贫血,其中改善的一种或多种症状包含但不限于虚弱、疲劳、面色苍白、黄疸、面部骨骼畸形、生长缓慢、腹部肿胀、尿色深、缺铁(在没有输血的情况下)、需要频繁输血。在特定实施例中,所治疗的血红蛋白病或血红蛋白病病状是镰状细胞病(scd),其中改善的一种或多种症状包含但不限于贫血;不明原因的疼痛发作,例如腹部、胸部、骨骼或关节疼痛;手部或脚部肿胀;腹部肿胀;发热;经常感染;皮肤或甲床苍白;黄疸;生长延迟;视力问题;中风的体征或症状;缺铁(在没有输血的情况下)、需要频繁输血。
如本文使用,术语“量”是指核酸酶变体、基因组编辑组合物或基因组编辑的细胞足以实现有益或所需的预防或治疗结果(包含临床结果)的“有效量(anamounteffective/aneffectiveamount)”。
“预防有效量”是指核酸酶变体、基因组编辑组合物或基因组编辑的细胞足以有效实现所需预防结果的量。通常但非必要地,由于预防剂量是在疾病之前或疾病的早期阶段在受试者中使用,所以预防有效量小于治疗有效量。
核酸酶变体、基因组编辑组合物或基因组编辑的细胞的“治疗有效量”可以根据诸如个体的疾病状态、年龄、性别和体重以及在引发个体中的期望反应中的能力等因素而不同。治疗有效量还是其中治疗有益效果超过任何毒性或有害作用的量。术语“治疗有效量”包含有效“治疗”受试者(例如,患者)的量。当指示治疗量时,医生可以根据说明书并考虑年龄、体重、肿瘤大小、感染或转移程度以及患者(受试者)病状的个体差异来确定待施用的在特定实施例中考虑的组合物的精确量。
基因组编辑的细胞可以在已经历或未经历骨髓消融治疗的个体中作为骨髓或脐带血移植物的一部分施用。在一个实施例中,本文考虑的基因组编辑的细胞在骨髓移植物中施用于已经历化学消融或放射性消融骨髓治疗的个体。
在一个实施例中,将一定剂量的基因组编辑的细胞静脉内递送到受试者。在优选实施例中,将基因组编辑的造血干细胞静脉内施用于受试者。
在一个示例性实施例中,提供给受试者的基因组编辑细胞的有效量为至少2x106个细胞/kg、至少3x106个细胞/kg、至少4x106个细胞/kg、至少5x106个细胞/kg、至少6x106个细胞/kg、至少7x106个细胞/kg、至少8x106个细胞/kg、至少9x106个细胞/kg、或至少10x106个细胞/kg、或更多个细胞/kg,包含所有中间剂量的细胞。
在另一个说明性实施例中,提供给受试者的基因组编辑的细胞的有效量为约2x106个细胞/kg、约3x106个细胞/kg、约4x106个细胞/kg、约5x106个细胞/kg、约6x106个细胞/kg、约7x106个细胞/kg、约8x106个细胞/kg、约9x106个细胞/kg、或约10x106个细胞/kg、或更多个细胞/kg,包含所有中间剂量的细胞。
在另一个说明性实施例中,提供给受试者的基因组编辑的细胞的有效量为约2x106个细胞/kg到约10x106个细胞/kg、约3x106个细胞/kg到约10x106个细胞/kg、约4x106个细胞/kg到约10x106个细胞/kg、约5x106个细胞/kg到约10x106个细胞/kg、2x106个细胞/kg到约6x106个细胞/kg、2x106个细胞/kg到约7x106个细胞/kg、2x106个细胞/kg到约8x106个细胞/kg、3x106个细胞/kg到约6x106个细胞/kg、3x106个细胞/kg到约7x106个细胞/kg、3x106个细胞/kg到约8x106个细胞/kg、4x106个细胞/kg到约6x106个细胞/kg、4x106个细胞/kg到约7x106个细胞/kg、4x106个细胞/kg到约8x106个细胞/kg、5x106个细胞/kg到约6x106个细胞/kg、5x106个细胞/kg到约7x106个细胞/kg、5x106个细胞/kg到约8x106个细胞/kg、或6x106个细胞/kg到约8x106个细胞/kg,包含所有中间剂量的细胞。
取决于所治疗的受试者的病状,必然会发生剂量的一些变化。在任何情况下,负责施用的人员将确定个体受试者的适当剂量。
在特定实施例中,基因组编辑的细胞疗法用于治疗、预防或改善血红蛋白病或与其相关的病状,包括向具有β-球蛋白基因型的受试者施用治疗有效量的本文考虑的基因组编辑的细胞,所述β-球蛋白基因型选自由以下组成的组:βe/β0、βc/β0、β0/β0、βe/βe、βc/β+、βe/β+、β0/β+、β+/β+、βc/βc、βe/βs、β0/βs、βc/βs、β+/βs或βs/βs。在一个实施例中,基因组编辑的细胞疗法缺少在红系细胞中的功能性bcl11a表达,例如缺少足够bcl11a表达来阻遏或抑制γ-球蛋白基因转录和反式激活β-球蛋白基因转录的能力。在一个实施例中,基因组编辑的细胞具有引入bcl11a基因中的gata-1结合位点中的突变。在一个实施例中,基因组编辑的细胞具有引入bcl11a基因的第二内含子中的共有gata-1结合位点(seqidno.24)中的突变。
在特定实施例中,本文考虑的基因组编辑的细胞疗法用于治疗、预防或改善地中海贫血或与其相关的病状。可用本文考虑的基因组编辑的细胞治疗的地中海贫血包含但不限于α-地中海贫血和β-地中海贫血。在特定实施例中,基因组编辑的细胞疗法用于治疗、预防或改善β-地中海贫血或与其相关的病状,包括向具有β-球蛋白基因型的受试者施用治疗有效量的本文考虑的基因组编辑的细胞,所述β-球蛋白基因型选自由以下组成的组:βe/β0、βc/β0、β0/β0、βc/βc、βe/βe、βe/β+、βc/βe、βc/β+、β0/β+或β+/β+。在一个实施例中,基因组编辑的细胞疗法缺少在红系细胞中的功能性bcl11a表达,例如缺少足够bcl11a表达来阻遏或抑制γ-球蛋白基因转录和反式激活β-球蛋白基因转录的能力。在一个实施例中,基因组编辑的细胞具有引入bcl11a基因中的gata-1结合位点中的突变。在一个实施例中,基因组编辑的细胞具有引入bcl11a基因的第二内含子中的共有gata-1结合位点(seqidno.24)中的突变。
在特定实施例中,本文考虑的基因组编辑的细胞疗法用于治疗、预防或改善镰状细胞病或与其相关的病状。在特定实施例中,基因组编辑的细胞疗法用于治疗、预防或改善镰状细胞病或与其相关的病状,包括向具有β-球蛋白基因型的受试者施用治疗有效量的本文考虑的基因组编辑的细胞,所述β-球蛋白基因型选自由以下组成的组:βe/βs、β0/βs、βc/βs、β+/βs或βs/βs。在一个实施例中,基因组编辑的细胞疗法缺少在红系细胞中的功能性bcl11a表达,例如缺少足够bcl11a表达来阻遏或抑制γ-球蛋白基因转录和反式激活β-球蛋白基因转录的能力。在一个实施例中,基因组编辑的细胞具有引入bcl11a基因中的gata-1结合位点中的突变。在一个实施例中,基因组编辑的细胞具有引入bcl11a基因的第二内含子中的共有gata-1结合位点(seqidno.24)中的突变。
在各个实施例中,向受试者施用一定量的包括bcl11a基因中的红系特异性增强子中的突变的基因组编辑的细胞,有效增加受试者中γ-球蛋白的表达。在特定实施例中,未经历基因组编辑的细胞中的γ-球蛋白基因表达相比,包括bcl11a基因中的红系特异性增强子中的突变的基因组编辑的细胞中的γ-球蛋白基因表达的量增加至少约10%、至少约20%、至少约30%、至少约40%、至少约50%、至少约60%、至少约70%、至少约80%、至少约90%、至少约100%、至少约2倍、至少约5倍、至少约10倍、至少约50倍、至少约100倍、至少约200倍、至少约300倍、至少约400倍、至少约500倍、或至少约1000倍、或更多倍。
在各个实施例中,向受试者施用一定量的包括bcl11a基因中的红系特异性增强子中的突变的基因组编辑的细胞,有效增加受试者中的hbf水平。在特定实施例中,与未经历基因组编辑的细胞中的hbf量相比,包括bcl11a基因中的红系特异性增强子中的突变的基因组编辑的细胞中的hbf量增加至少约10%、至少约20%、至少约30%、至少约40%、至少约50%、至少约60%、至少约70%、至少约80%、至少约90%、至少约100%、至少约2倍、至少约5倍、至少约10倍、至少约50倍、至少约100倍、至少约200倍、至少约300倍、至少约400倍、至少约500倍、或至少约1000倍、或更多倍。
本领域普通技术人员将能够使用常规方法来确定合适的施用途径和有效量的包括本文考虑的基因组编辑的细胞的组合物的正确剂量。本领域普通技术人员还应当知道,在某些疗法中,可能需要多次施用本文考虑的药物组合物来实现治疗。
用于治疗适合用基因组编辑的造血干细胞和祖细胞疗法治疗的受试者的主要方法之一是输血。因此,本文考虑的组合物和方法的主要目标之一是减少输血的次数或消除对输血的需要。
在特定实施例中,药物产品施用一次。
在某些实施例中,药物产品在1年、2年、5年、10年或更多年的范围内施用1次、2次、3次、4次、5次、6次、7次、8次、9次或10次或更多次。
本说明书中引用的所有出版物、专利申请和授权专利均通过引用并入本文中,如同每个单独的出版物、专利申请或授权专利被具体地和单独地指出以通过引用并入。
尽管为了清楚理解的目的已经通过说明和实例详细地描述了前述实施例,但是根据本文考虑的教导,本领域普通技术人员将容易明白,可以在不脱离所附权利要求的精神或范围的情况下做出某些改变和修改。以下实例仅以说明的方式提供,而不是作为限制。本领域技术人员将容易地认识到可以改变或修改以产生基本相似结果的各种非关键参数。
实例
实例1
bcl11a基因中的红系增强子中的非规范i-onui归巢核酸内切酶靶位点的标识
存在于bcl11a基因中的核心gata-1基序(ctgnnnnnnnwgatar;参见seqidno:24;图1)不含有规范i-onui“中心-4”切割基序:attc、tttc、atac、atat、ttac和attt。
令人惊讶的是,本发明人发现i-onui是用于开发靶向gata-1基序的归巢核酸内切酶变体或megatal的合适起始支架。选择靶位点“ttat”(参见seqidno:25),因为其反向互补序列“ataa”存在于bcl11a基因中的核心gata-1基序(参见seqidno:24)中。尽管不是规范i-onui切割位点,但是“ttat”是野生型i-smamilhe(与i-onui具有~45%的同一性)的中心-4序列(seqidno:30)。图2a。
此外,使用高通量酵母表面展示体外核酸内切酶测定谱分析(profile)了靶向ccr5基因的i-onui变体he(seqidno:31)的中心4特异性(jarjour,west-foyle等人,2009年)。将编码靶向he的ccr5的质粒(seqidno:32)转化到酿酒酵母中用于表面展示,然后测试针对pcr生成的双链dna底物的切割活性,所述双链dna底物包括含有256个可能的中心-4序列(seqidno:33)中的每一个的ccr5靶位点dna序列,包含“ttat”。特异性谱示出了重编程的i-onui能够切割包括非规范“ttat”中心-4序列的靶位点。图2b。
选择i-onui作为用于开发靶向bcl11a中的gata-1基序的归巢核酸内切酶变体或megatal的起始支架。
实例2
重编程i-onui以靶向bcl11a基因中的gata-1基序
通过在dna识别界面中构建含有可变氨基酸残基的模块文库,对i-onui重编程以靶向bcll11a基因中的gata-1基序。为了构建变体,使用寡核苷酸将简并密码子掺入i-onuidna结合结构域。编码简并密码子的寡核苷酸用作pcr模板,以通过酵母菌株酿酒酵母中的缺口重组生成变体文库。每个变体文库跨越n-或c-末端i-onuidna识别结构域并且含有~107到108个独特的转化体。针对包括相应结构域“半位点”的靶位点(seqidno:28-29)的切割活性,通过流式细胞术对所得的表面展示文库进行筛选。图3。
纯化表现出n-和c-末端结构域重编程的i-onuihe的酵母,并提取质粒dna。进行pcr反应以扩增重编程的结构域,随后将其转化到酿酒酵母中以产生重编程的结构域组合的文库。从该文库中标识识别存在于bcl11a基因中的gata-1基序中的完整靶位点(seqidno:25)的完全重编程的i-onui变体,并进行纯化。
实例3
有效地靶向bcl11a基因中的gata-1基序的重编程的i-onui归巢核酸内切酶
使用染色体整合的荧光报告基因系统测量靶向bcl11a基因中的gata-1基序的重编程的i-onuihe的活性(certo等人,2011年)。将结合并切割bcl11a靶序列的完全重编程的i-onuihe克隆到哺乳动物表达质粒中,然后单独转染到hek293t成纤维细胞系中,该细胞系被重编程以含有编码荧光mcherry蛋白的框外基因上游的bcl11a靶序列。通过非同源末端连接(nhej)途径进行dna修复引起的he对嵌入的靶位点的切割以及随后的小插入或缺失的累积,导致了大约三分之一的修复基因座将荧光报告基因重新放回“框内”。因此,mcherry荧光是染色体嵌入的靶序列处的核酸内切酶活性的读数。结合并切割bcl11a靶位点的完全重编程的i-onuihe在细胞染色体环境中表现出中等效率的mcherry表达。图4a。
通过进行随机诱变生成第二i-onui变体文库,靶向bcl11a靶位点的重编程的i-onuihe中的一个在初始荧光报告筛选中标识(bcl11.a.b4,seqidno:6)。此外,在更严格的分割条件(ph调节到7.2)下进行基于展示的流式分选,以尽力分离具有改善的催化效率的变体。图4b。该过程标识了i-onui变体bcl11a.b4.a3(seqidno:7),其相对于亲本i-onui变体在dna识别界面中含有两个氨基酸突变,并且具有高于亲本i-onui变体大约3倍的mcherry表达细胞的速率。图4c。图5示出了代表性i-onui的相对比对以及包括dna识别界面的残基的位置信息。
通过进行随机诱变生成第三i-onui变体文库,靶向bcl11a靶位点的重编程的i-onuihe中的一个在二级筛选中标识(bcl11a.b4.a3(seqidno:7)。此外,在更严格的亲和力条件(50pm)下进行基于展示的流式分选,以分离具有改善的结合特征的变体。该过程标识了i-onui变体:bcl11a.b4.a3.c7(seqidno:8)、bcl11a.b4.a3.e3(seqidno:9)、bcl11a.b4.a3.b6(seqidno:10)、bcl11a.b4.a3.h4(seqidno:11)、bcl11a.b4.a3.b12(seqidno:12)、bcl11a.b4.a3.a7(seqidno:13)、bcl11a.b4.a3.c2(seqidno:14)、bcl11a.b4.a3.g8(seqidno:15)、bcl11a.b4.a3.a1(seqidno:16)、bcl11a.b4.a3.a5(seqidno:17)、bcl11a.b4.a3.b6.2(seqidno:18)和bcl11a.b4.a3.b7(seqidno:19)。
实例4
有效地靶向bcl11a基因中的gata-1基序的重编程的i-onui归巢核酸内切酶的亲和力和特异性
表征了i-onui变体bcl11a.b4.a3的dna结合亲和力和切割特异性。将编码在重编程期间标识的bcl11a.b4.a3变体的质粒(seqidno:34)转化到酿酒酵母中用于表面展示。通过平衡结合滴定确定i-onui变体bcl11a.b4.a3的亲和力,平衡解离常数估计为~500pm,其在i-onui亚家族中的几种其它野生型he的范围内(图6a)。
连续取代分析用于确定切割特异性。在一组dna底物上评估切割活性,其中每个靶位点位置(seqidno:25)突变为3个备选碱基对中的每一个。图6b。ctd示出了比ntd更高程度的切割特异性。
还评估了bcl11a.b4.a3的靶特异性,因为它是第一个被重编程以靶向在其靶位点含有非天然中心-4序列的序列的归巢核酸内切酶。生成了包括bcl11a靶位点内的所有256个可能的中心-4序列的dna底物(seqidno:35)。针对在酵母表面上展示的i-onui变体bcl11a.b4.a3测定每个底物(图7)。与图2b中呈现的数据类似,i-onui变体bcl11a.b4.a3示出了包含ttat基序的中心-4谱,但是其保留了天然i-onui中心-4特异性。
实例5
bcl11a基因中的gata-1基序的有效破坏
通过使用boissel等人,2013年中描述的方法在bcl11a靶位点(seqidno:26)上游附加对应于11碱基对tal阵列靶位点的n-末端10.5tal阵列(例如,seqidno:21和36)将i-onui变体bcl11a.b4.a3格式化为megatal。图8a。另一种形式的megatal包括trex2的c-末端融合(例如,seqidno:23和37)。
通过在细胞因子补充的培养基中预刺激细胞48-72小时,然后用编码bcl11amegatal的体外转录的mrna(例如,seqidno:36)和任选地格式化为trex2融合蛋白的megatal(例如,seqidno:37)电穿孔细胞,在原代人cd34+细胞中评估bcl11amegatal编辑效率。电穿孔后,将细胞在细胞因子补充的培养基中培养1-4天,在此期间取出等分试样用于基因组dna分离,然后进行跨bcl11a靶位点的pcr扩增。
使用通过分解跟踪插入缺失(tide,参见brinkman等人,2014年)、体外切割测定和集落测序来测量小插入/缺失(indel)事件的频率。图8b示出了扩增子插入缺失的代表性tide分析,并说明了bcl11amegatal的靶位点处的+1、-1、-2、-3或-4插入缺失的优势。通过测试跨bcl11a靶位点的pcr扩增子是否能够被重组bcl11a归巢核酸内切酶重新切割来确认megatal编辑速率。用编码bcl11amegatal或bcl11amegatal-trex2融合蛋白的mrna治疗细胞导致显著部分的扩增子被修饰至不再被重组bcl11amegatal识别和切割的程度。图8c。插入缺失的谱还通过对单个集落的pcr扩增子克隆并测序来表征。bcl11amegatal靶位点处的插入缺失谱如图8d所示。图8e总结了具有不同的原代cd34+供体细胞、不同的预刺激窗口、细胞浓度和mrna产生批次的多个实验的插入缺失分析。
dna测序研究表明,i-onui变体在治疗的细胞的显著部分中破坏了gata-1共有基序。通过与trex2融合,提高了bcl11amegatal的编辑效率。
实例6
bcl11a基因中的gata-1基序处的高效hdr
将bcl11amegatalmrna电穿孔到原代人cd34+细胞中,以评估bcl11a基因中的gata-1靶序列处的aav-递送的转基因的同源定向修复。使用标准方法制备aav2/6载体,其包括驱动bfp表达的组成型启动子,所述bfp位于与bcl11amegatal靶位点侧翼的5'和3'区域dna同源的序列之间。图9a。在细胞因子补充的培养基中预刺激原代人cd34+细胞,然后在存在或不存在编码bcl11amegatal的mrna(例如,seqidno:36)的情况下进行洗涤和电穿孔。在电穿孔之前或在电穿孔后恢复步骤期间用aav转导细胞。将细胞在细胞因子补充的培养基中培养2-10天,在此期间取出等分试样用于bfp表达的流式细胞术分析以测量同源定向修复。
相对于单一试剂对照样品,在megatal加aav样品中观察到相当频率的bfp+细胞。图9b。数据示出了用含bfp的转基因同源定向修复bcl11a靶序列带来的稳定bfp表达,因为来自瞬时附加型aav基因组的bfp表达在转导后2-4天的培养期间消失。
进行甲基纤维素测定以确定基于megatal的nhej或hdr是否改变了原代cd34+细胞的谱系特性。如本实例的前述段落中所述治疗原代人cd34+细胞,只是在电穿孔后恢复步骤之后,对细胞进行计数并将其接种到甲基纤维素培养基中14天。培养14天后,对集落的频率和形态进行评分。bcl11amegatal治疗的样品示出了相对于对照样品相当的成熟集落表型频率,并且未示出与bcl11a基因座的内含子2中的gata-1位点处的基因组编辑相关的明显谱系倾斜的证据。图10a。
此外,bcl11amegatal加aav治疗的样品在一式两份培养物中示出了30%和29.8%bfp+细胞,而暴露于ccr5megatal或无核酸酶的细胞产生<1%bfp+细胞。图10b。这些结果与bcl11amegatal在原始造血干细胞和祖细胞中介导的显著同源定向修复一致。
实例7
用靶向megatal的bcl11a编辑的cd34+细胞上调hbf水平
有效地破坏原代人cd34+细胞中的bcl11a基因中的gata-1序列的megatal增加了编辑的细胞中的hbf水平。在细胞因子补充的培养基中预刺激原代人cd34+细胞,然后在存在或不存在bcl11amegataltrex2融合物(例如,seqidno:37)的情况下进行洗涤和电穿孔。电穿孔后,将细胞在含有血清、rhscf、rhil-3和rhepo的基于imdm的培养基中培养5-7天,其促进了培养的cd34+细胞中的红系分化。通过使用直接缀合的抗hbf抗体进行染色和流式细胞术,或通过球蛋白链的hplc分析,在分化的红系细胞中分析hbf水平。
与对照培养的细胞相比,用编码bcl11amegatal-trex2融合体的mrna电穿孔的细胞中增加了通过流式细胞术的hbf+细胞的频率。图11a。与对照培养的细胞相比,在用编码bcl11amegatal-trex2融合体的mrna电穿孔的细胞中还观察到通过hplc的hbf+细胞的显著增加。图11b。这些数据表明,靶向bcl11a基因中的gata-1位点的bcl11amegatal去阻遏γ-球蛋白基因表达,导致γ-球蛋白与β-球蛋白表达基因比率的增加,从而增加编辑的红系细胞中的hbf水平。
实例8
异种移植模型中的人原代长期nsg-再植细胞的持久基因组编辑
介绍
用megatal电穿孔人原代cd34+细胞并移植到nsg小鼠中,以确定长期再植造血干细胞中基因组编辑的持久性,这有助于移植后造血谱系的长期重建。
方法
将新鲜的人动员外周血(mpb)cd34+细胞在标准的湿润组织培养箱(5%co2)中于含有细胞因子的培养基(scf、tpo、flt3-l)中预刺激48小时。预刺激后,收获细胞并统计数量。将细胞分成六组25x106个细胞,并重悬于400μl电穿孔缓冲液中。使用maxcyte电穿孔装置和具有媒剂或具有编码bcl11amegatal、bcl11amegatal-trex2、ccr5megatal和ccr5megatal-trex2的mrna的oc400比色皿以100μg/ml的浓度电穿孔细胞。电穿孔后,将细胞转移到烧瓶中并用含细胞因子的培养基(scf、tpo、flt3-l、il-3)稀释到2x106个细胞/ml,并在30℃温育大约20小时。电穿孔后第二天,在移植前低温保存细胞。
将细胞解冻、洗涤、并分成两等份,并重悬于2mlscgm+细胞因子或红系分化培养基中,并转移到标准的12孔非贴壁组织培养板。将在scgm+细胞因子中培养的细胞在标准的湿润组织培养箱(5%co2)中维持另外最多6天,并在培养过程中统计细胞的数量以建立生长曲线。另外,在培养5天后,收集一部分细胞用于分析插入缺失频率,详述如下。在红系分化培养基中培养的细胞培养长达三周或直至至少30%的细胞是血型糖蛋白a+和cd71+,其为红系分化的标志物。一旦确定了足够的红系分化水平,将细胞洗涤并重悬于水中并在干冰上快速冷冻。然后通过离子交换高效液相色谱(ie-hplc)分析提取的蛋白质的血红蛋白含量。
将洗涤的细胞重悬于200μlscgm中,然后转移到3ml等份的细胞因子补充的甲基纤维素(例如,methocultm4434classic)。然后使用平端16号针将1.1ml转移到平行的35-mm组织培养皿中。将培养皿保持在标准的湿润组织培养箱中14-16天,并对集落的大小、形态和细胞组成进行评分。
从细胞中提取基因组dna并进行pcr扩增,以扩增目标区域。在pcr纯化后,扩增子适于miseq分析,并通过针对插入和缺失事件的靶向扩增子重测序进行分析。
为了评估基因编辑对人长期造血干细胞的影响,将对照和megatal治疗的细胞解冻并洗涤,然后移植到亚骨髓清除的成年nsg小鼠的尾静脉中。根据标准iacuc动物护理指南将小鼠圈养在无病原体的环境中。分别收获在移植后2个月和4个月时外周血(pb)和骨髓(bm)并分析插入缺失频率,通过用抗hcd45抗体(bd#561864)染色植入人细胞,然后进行流式细胞术分析,并在红系分化后进行hbf诱导。
为了评估用megatal治疗的hbf诱导,使用美天旎小规模柱对bm进行cd34+富集。然后将cd34+细胞置于红系分化培养物中长达三周或直至至少30%的细胞为cd71+和gpa+。然后通过ie-hplc分析细胞的血红蛋白含量。
结果
megatal电穿孔不影响cfc的形成
将低温保存的对照和megatal治疗的小规模药物产品解冻并统计数量。将来自每个治疗组的500个细胞转移到methocult(h4434)并开始半固体培养。培养两周后,使用stemvision(stemcelltechnologies)对含有造血集落的板成像并统计数量。用megatalmrna电穿孔的细胞未示出集落形成、每组的集落总数或髓系、红系和干细胞样表型的倾斜的差异。图12。
megatal-trex2融合蛋白提高编辑速率
将低温保存的对照和megatal治疗的小规模药物产品解冻并统计数量。然后在插入缺失频率分析之前,将细胞在含细胞因子的培养基中培养五天。hcd34+细胞megatal针对ccr5或bcl11a的治疗生成约10%插入缺失。ccr5或bcl11amegatal-trex2融合蛋白将编辑速率分别提高2.9倍和4.1倍到大约30-35%插入缺失。本底编辑速率小于1%。图13。
bcl11amegatal-trex2融合蛋白诱导胎儿血红蛋白(hbf)
将低温保存的对照和megatal治疗的小规模药物产品解冻,统计数量并置于红系分化培养物中。培养~3周后,收获红系分化标志物、细胞,进行洗涤并在水中溶解。通过ie-hplc分析蛋白质的血红蛋白含量。该细胞批次中hbf的本底水平为~18%。在有或没有编码ccr5megatal、ccr5megatal-trex2megatal融合蛋白或bcl11amegatal的mrna的情况下,电穿孔的细胞没有显著地改变hbf水平。然而,与未治疗的细胞相比,用bcl11amegatal-trex2融合蛋白电穿孔的细胞使hbf增加64%,以达到~28%的hbf。
长期再植细胞的编辑频率
在移植物(前)、移植后2个月的pb分析(2个月pbl)和4个月bm编辑分析(4个月bm)之间比较插入缺失的编辑速率或频率。跨megatal靶位点进行pcr扩增,并使用下一代测序对扩增子进行测序。在用bcl11a-trex2megatal电穿孔的cd34+细胞中,在4个月时间点的基因组编辑率保持在20%以上。图15。
bcl11amegatal-trex2融合蛋白增加长期再植细胞中的hbf
通过ie-hplc分析来自nsgbm的红系分化的人cd34+富集细胞。所得的hbf水平反映了移植物的水平。这些培养物中的本底hbf水平为大约11%。在有或没有编码ccr5megatal、ccr5megatal-trex2megatal融合蛋白或bcl11amegatal的mrna的情况下电穿孔的细胞没有显著地改变hbf水平。然而,用bcl11a-trex2megatal治疗使hbf产量增加了~18%。与对照细胞相比,这增加了>50%。
结论
bcl11amegatal生成了高基因组编辑速率,其与编辑的移植细胞cd34+群中长期再植造血干细胞群的持久基因组编辑一致。
通常,在以下权利要求中,使用的术语不应被解释为将权利要求限制于说明书和权利要求中公开的具体实施例,而是应该被解释为包含所有可能的实施例以及这些权利要求的等同物的全部范围。因此,权利要求不受本公开的限制。
序列表
<110>蓝鸟生物公司(bluebirdbio,inc.)
乔丹·贾儒尔(jarjour,jordan)
贾斯迪普·曼恩(mann,jasdeep)
<120>bcl11a归巢核酸内切酶变体、组合物及其使用方法
<130>blbd-071/04wo315698-2598
<150>us62/414,273
<151>2016-10-28
<150>us62/375,829
<151>2016-08-16
<150>us62/367,465
<151>2016-07-27
<150>us62/366,530
<151>2016-07-25
<160>104
<170>patentin版本3.5
<210>1
<211>303
<212>prt
<213>榆树枯萎菌美洲亚种(线粒体)(ophiostomanovo-ulmisubsp.americana(mitochondrion))
<400>1
metalatyrmetserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserpheleuleuargileargasnasn
202530
asnlysserservalglytyrserthrgluleuglypheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglyvalilealaasnserglyaspasnalavalserleulys
65707580
valthrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrmetleuphelysgln
100105110
alaphecysvalmetgluasnlysgluhisleulysileasnglyile
115120125
lysgluleuvalargilelysalalysleuasntrpglyleuthrasp
130135140
gluleulyslysalapheprogluileileserlysgluargserleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glygluglycysphephevalasnleuilelysserlysserlysleu
180185190
glyvalglnvalglnleuvalpheserilethrglnhisilelysasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglytyrile
210215220
lysglulysasnlyssergluphesertrpleuaspphevalvalthr
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalphe
290295300
<210>2
<211>303
<212>prt
<213>榆树枯萎菌美洲亚种(线粒体)(ophiostomanovo-ulmisubsp.americana(mitochondrion))
<400>2
metalatyrmetserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserpheleuleuargileargasnasn
202530
asnlysserservalglytyrserthrgluleuglypheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglyvalilealaasnserglyaspasnalavalserleulys
65707580
valthrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysleuasntrpglyleuthrasp
130135140
gluleulyslysalapheprogluasnileserlysgluargserleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glygluglycysphephevalasnleuilelysserlysserlysleu
180185190
glyvalglnvalglnleuvalpheserilethrglnhisilelysasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglytyrile
210215220
lysglulysasnlyssergluphesertrpleuaspphevalvalthr
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalphe
290295300
<210>3
<211>303
<212>prt
<213>榆树枯萎菌美洲亚种(线粒体)(ophiostomanovo-ulmisubsp.americana(mitochondrion))
<220>
<221>mod_res
<222>(1)..(3)
<223>任何氨基酸或不存在
<400>3
xaaxaaxaametserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserpheleuleuargileargasnasn
202530
asnlysserservalglytyrserthrgluleuglypheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglyvalilealaasnserglyaspasnalavalserleulys
65707580
valthrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysleuasntrpglyleuthrasp
130135140
gluleulyslysalapheprogluasnileserlysgluargserleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glygluglycysphephevalasnleuilelysserlysserlysleu
180185190
glyvalglnvalglnleuvalpheserilethrglnhisilelysasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglytyrile
210215220
lysglulysasnlyssergluphesertrpleuaspphevalvalthr
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalphe
290295300
<210>4
<211>303
<212>prt
<213>榆树枯萎菌美洲亚种(线粒体)(ophiostomanovo-ulmisubsp.americana(mitochondrion))
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(302)..(303)
<223>任何氨基酸或不存在
<400>4
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserpheleuleuargileargasnasn
202530
asnlysserservalglytyrserthrgluleuglypheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglyvalilealaasnserglyaspasnalavalserleulys
65707580
valthrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysleuasntrpglyleuthrasp
130135140
gluleulyslysalapheprogluasnileserlysgluargserleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glygluglycysphephevalasnleuilelysserlysserlysleu
180185190
glyvalglnvalglnleuvalpheserilethrglnhisilelysasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglytyrile
210215220
lysglulysasnlyssergluphesertrpleuaspphevalvalthr
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargxaaxaa
290295300
<210>5
<211>303
<212>prt
<213>榆树枯萎菌美洲亚种(线粒体)(ophiostomanovo-ulmisubsp.americana(mitochondrion))
<220>
<221>mod_res
<222>(1)..(8)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(302)..(303)
<223>任何氨基酸或不存在
<400>5
xaaxaaxaaxaaxaaxaaxaaxaaserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserpheleuleuargileargasnasn
202530
asnlysserservalglytyrserthrgluleuglypheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglyvalilealaasnserglyaspasnalavalserleulys
65707580
valthrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysleuasntrpglyleuthrasp
130135140
gluleulyslysalapheprogluasnileserlysgluargserleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glygluglycysphephevalasnleuilelysserlysserlysleu
180185190
glyvalglnvalglnleuvalpheserilethrglnhisilelysasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglytyrile
210215220
lysglulysasnlyssergluphesertrpleuaspphevalvalthr
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargxaaxaa
290295300
<210>6
<211>303
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(302)..(303)
<223>任何氨基酸或不存在
<400>6
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnarg
202530
asnasptyralathrglytyrargilehisleuthrpheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnalaglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glygluglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargxaaxaa
290295300
<210>7
<211>303
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(302)..(303)
<223>任何氨基酸或不存在
<400>7
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnarg
202530
asnasptyralathrglytyrargilehisleuthrpheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnthrglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargxaaxaa
290295300
<210>8
<211>306
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<400>8
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuargileglnasnser
202530
asnasptyralathrglytyrargilehisleuthrpheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnthrglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyarg
305
<210>9
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>9
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnlys
202530
asnasntyralathrglytyrargilehisleuthrpheglnileile
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnthrglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>10
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>10
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnarg
202530
asnasptyralathrglytyrargileargleuthrpheglnileile
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnthrglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>11
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>11
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuglyileglnasnarg
202530
asnasptyralathrglytyrargileargleuthrpheglnilethr
354045
leuargasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnthrglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>12
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>12
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserilehisasnarg
202530
asnasptyralathrglytyrargilehisleuthrpheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnthrglyaspasnhisvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>13
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>13
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserpheargleuserileglnasnarg
202530
asnasptyralathrglytyrargilehisleuargpheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnthrglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>14
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>14
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphetyrleuserileglnasnarg
202530
asnasptyralathrglytyrargilehisleuargpheglnilethr
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglyargilegluasnthrglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>15
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>15
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnarg
202530
serasptyralathrglytyrargilehisleuargpheglnilethr
354045
leuhisasnlysglulysserileleugluasnileglnserthrtrp
505560
lysvalglylysileasnasnthrglyaspasnleuvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>16
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>16
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnarg
202530
asnasptyralathrglytyrargilehisleuargpheglnilegly
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysilealaasnvalglyaspasnargvalglnleuval
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>17
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>17
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnarg
202530
asnasptyralathrglytyrargilehisleuargpheglnilegly
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileglnasnmetglyaspasnargvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>18
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>18
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnarg
202530
asnasptyralathrglytyrargilehisleuargpheglnilegly
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileleuasnvalglyaspasnhisvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>19
<211>308
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<220>
<221>mod_res
<222>(1)..(4)
<223>任何氨基酸或不存在
<220>
<221>mod_res
<222>(307)..(308)
<223>任何氨基酸或不存在
<400>19
xaaxaaxaaxaaserargarggluserileasnprotrpileleuthr
151015
glyphealaaspalagluglyserphevalleuserileglnasnarg
202530
asnasptyralathrglytyrargilehisleuargpheglnileval
354045
leuhisasnlysasplysserileleugluasnileglnserthrtrp
505560
lysvalglylysileserasnvalglyaspasnhisvalglnleuarg
65707580
valtyrargphegluaspleulysvalileileasphispheglulys
859095
tyrproleuilethrglnlysleuglyasptyrlysleuphelysgln
100105110
alapheservalmetgluasnlysgluhisleulysgluasnglyile
115120125
lysgluleuvalargilelysalalysmetasntrpglyleuasnasp
130135140
gluleulyslysalapheprogluasnileserlysgluargproleu
145150155160
ileasnlysasnileproasnphelystrpleualaglyphethrser
165170175
glyaspglyserphephevalargleuarglysserasnvalasnala
180185190
argvalargvalglnleuvalphegluileserglnhisileargasp
195200205
lysasnleumetasnserleuilethrtyrleuglycysglyhisile
210215220
tyrgluglyasnlyssergluargsertrpleuglnpheargvalglu
225230235240
lyspheseraspileasnasplysileileprovalpheglngluasn
245250255
thrleuileglyvallysleugluaspphegluasptrpcyslysval
260265270
alalysleuilegluglulyslyshisleuthrgluserglyleuasp
275280285
gluilelyslysilelysleuasnmetasnlysglyargvalpheser
290295300
glyargxaaxaa
305
<210>20
<211>875
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成megatal氨基酸序列
<400>20
metglyseralaproprolyslyslysarglysvalvalaspleuarg
151015
thrleuglytyrserglnglnglnglnglulysilelysprolysval
202530
argserthrvalalaglnhishisglualaleuvalglyhisglyphe
354045
thrhisalahisilevalalaleuserglnhisproalaalaleugly
505560
thrvalalavalthrtyrglnhisileilethralaleuprogluala
65707580
thrhisgluaspilevalglyvalglylysglntrpserglyalaarg
859095
alaleuglualaleuleuthraspalaglygluleuargglypropro
100105110
leuglnleuaspthrglyglnleuvallysilealalysargglygly
115120125
valthralametglualavalhisalaserargasnalaleuthrgly
130135140
alaproleuasnleuthrproaspglnvalvalalailealaserasn
145150155160
ileglyglylysglnalaleugluthrvalglnargleuleuproval
165170175
leucysglnasphisglyleuthrproaspglnvalvalalaileala
180185190
serasnasnglyglylysglnalaleugluthrvalglnargleuleu
195200205
provalleucysglnasphisglyleuthrproaspglnvalvalala
210215220
ilealaserasnglyglyglylysglnalaleugluthrvalglnarg
225230235240
leuleuprovalleucysglnasphisglyleuthrproaspglnval
245250255
valalailealaserhisaspglyglylysglnalaleugluthrval
260265270
glnargleuleuprovalleucysglnasphisglyleuthrproasp
275280285
glnvalvalalailealaserasnglyglyglylysglnalaleuglu
290295300
thrvalglnargleuleuprovalleucysglnasphisglyleuthr
305310315320
proaspglnvalvalalailealaserasnileglyglylysglnala
325330335
leugluthrvalglnargleuleuprovalleucysglnasphisgly
340345350
leuthrproaspglnvalvalalailealaserasnasnglyglylys
355360365
glnalaleugluthrvalglnargleuleuprovalleucysglnasp
370375380
hisglyleuthrproaspglnvalvalalailealaserasnglygly
385390395400
glylysglnalaleugluthrvalglnargleuleuprovalleucys
405410415
glnasphisglyleuthrproaspglnvalvalalailealaserasn
420425430
asnglyglylysglnalaleugluthrvalglnargleuleuproval
435440445
leucysglnasphisglyleuthrproaspglnvalvalalaileala
450455460
serhisaspglyglylysglnalaleugluthrvalglnargleuleu
465470475480
provalleucysglnasphisglyleuthrproaspglnvalvalala
485490495
ilealaserasnileglyglylysglnalaleugluserilevalala
500505510
glnleuserargproaspproalaleualaalaleuthrasnasphis
515520525
leuvalalaleualacysleuglyglyargproalametaspalaval
530535540
lyslysglyleuprohisalaprogluleuileargargvalasnarg
545550555560
argileglygluargthrserhisargvalalaileserargvalgly
565570575
glyserserargarggluserileasnprotrpileleuthrglyphe
580585590
alaaspalagluglyserphevalleuserileglnasnargasnasp
595600605
tyralathrglytyrargilehisleuthrpheglnilethrleuhis
610615620
asnlysasplysserileleugluasnileglnserthrtrplysval
625630635640
glylysileasnasnalaglyaspasnleuvalglnleuargvaltyr
645650655
argphegluaspleulysvalileileasphispheglulystyrpro
660665670
leuilethrglnlysleuglyasptyrlysleuphelysglnalaphe
675680685
servalmetgluasnlysgluhisleulysgluasnglyilelysglu
690695700
leuvalargilelysalalysmetasntrpglyleuasnaspgluleu
705710715720
lyslysalapheprogluasnileserlysgluargproleuileasn
725730735
lysasnileproasnphelystrpleualaglyphethrserglyglu
740745750
glyserphephevalargleuarglysserasnvalasnalaargval
755760765
argvalglnleuvalphegluileserglnhisileargasplysasn
770775780
leumetasnserleuilethrtyrleuglycysglyhisiletyrglu
785790795800
glyasnlyssergluargsertrpleuglnpheargvalglulysphe
805810815
seraspileasnasplysileileprovalpheglngluasnthrleu
820825830
ileglyvallysleugluaspphegluasptrpcyslysvalalalys
835840845
leuilegluglulyslyshisleuthrgluserglyleuaspgluile
850855860
lyslysilelysleuasnmetasnlysglyarg
865870875
<210>21
<211>875
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成megatal氨基酸序列
<400>21
metglyseralaproprolyslyslysarglysvalvalaspleuarg
151015
thrleuglytyrserglnglnglnglnglulysilelysprolysval
202530
argserthrvalalaglnhishisglualaleuvalglyhisglyphe
354045
thrhisalahisilevalalaleuserglnhisproalaalaleugly
505560
thrvalalavalthrtyrglnhisileilethralaleuprogluala
65707580
thrhisgluaspilevalglyvalglylysglntrpserglyalaarg
859095
alaleuglualaleuleuthraspalaglygluleuargglypropro
100105110
leuglnleuaspthrglyglnleuvallysilealalysargglygly
115120125
valthralametglualavalhisalaserargasnalaleuthrgly
130135140
alaproleuasnleuthrproaspglnvalvalalailealaserasn
145150155160
ileglyglylysglnalaleugluthrvalglnargleuleuproval
165170175
leucysglnasphisglyleuthrproaspglnvalvalalaileala
180185190
serasnasnglyglylysglnalaleugluthrvalglnargleuleu
195200205
provalleucysglnasphisglyleuthrproaspglnvalvalala
210215220
ilealaserasnglyglyglylysglnalaleugluthrvalglnarg
225230235240
leuleuprovalleucysglnasphisglyleuthrproaspglnval
245250255
valalailealaserhisaspglyglylysglnalaleugluthrval
260265270
glnargleuleuprovalleucysglnasphisglyleuthrproasp
275280285
glnvalvalalailealaserasnglyglyglylysglnalaleuglu
290295300
thrvalglnargleuleuprovalleucysglnasphisglyleuthr
305310315320
proaspglnvalvalalailealaserasnileglyglylysglnala
325330335
leugluthrvalglnargleuleuprovalleucysglnasphisgly
340345350
leuthrproaspglnvalvalalailealaserasnasnglyglylys
355360365
glnalaleugluthrvalglnargleuleuprovalleucysglnasp
370375380
hisglyleuthrproaspglnvalvalalailealaserasnglygly
385390395400
glylysglnalaleugluthrvalglnargleuleuprovalleucys
405410415
glnasphisglyleuthrproaspglnvalvalalailealaserasn
420425430
asnglyglylysglnalaleugluthrvalglnargleuleuproval
435440445
leucysglnasphisglyleuthrproaspglnvalvalalaileala
450455460
serhisaspglyglylysglnalaleugluthrvalglnargleuleu
465470475480
provalleucysglnasphisglyleuthrproaspglnvalvalala
485490495
ilealaserasnileglyglylysglnalaleugluserilevalala
500505510
glnleuserargproaspproalaleualaalaleuthrasnasphis
515520525
leuvalalaleualacysleuglyglyargproalametaspalaval
530535540
lyslysglyleuprohisalaprogluleuileargargvalasnarg
545550555560
argileglygluargthrserhisargvalalaileserargvalgly
565570575
glyserserargarggluserileasnprotrpileleuthrglyphe
580585590
alaaspalagluglyserphevalleuserileglnasnargasnasp
595600605
tyralathrglytyrargilehisleuthrpheglnilethrleuhis
610615620
asnlysasplysserileleugluasnileglnserthrtrplysval
625630635640
glylysileasnasnthrglyaspasnleuvalglnleuargvaltyr
645650655
argphegluaspleulysvalileileasphispheglulystyrpro
660665670
leuilethrglnlysleuglyasptyrlysleuphelysglnalaphe
675680685
servalmetgluasnlysgluhisleulysgluasnglyilelysglu
690695700
leuvalargilelysalalysmetasntrpglyleuasnaspgluleu
705710715720
lyslysalapheprogluasnileserlysgluargproleuileasn
725730735
lysasnileproasnphelystrpleualaglyphethrserglyasp
740745750
glyserphephevalargleuarglysserasnvalasnalaargval
755760765
argvalglnleuvalphegluileserglnhisileargasplysasn
770775780
leumetasnserleuilethrtyrleuglycysglyhisiletyrglu
785790795800
glyasnlyssergluargsertrpleuglnpheargvalglulysphe
805810815
seraspileasnasplysileileprovalpheglngluasnthrleu
820825830
ileglyvallysleugluaspphegluasptrpcyslysvalalalys
835840845
leuilegluglulyslyshisleuthrgluserglyleuaspgluile
850855860
lyslysilelysleuasnmetasnlysglyarg
865870875
<210>22
<211>1116
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成megatal氨基酸序列
<400>22
metglyseralaproprolyslyslysarglysvalvalaspleuarg
151015
thrleuglytyrserglnglnglnglnglulysilelysprolysval
202530
argserthrvalalaglnhishisglualaleuvalglyhisglyphe
354045
thrhisalahisilevalalaleuserglnhisproalaalaleugly
505560
thrvalalavalthrtyrglnhisileilethralaleuprogluala
65707580
thrhisgluaspilevalglyvalglylysglntrpserglyalaarg
859095
alaleuglualaleuleuthraspalaglygluleuargglypropro
100105110
leuglnleuaspthrglyglnleuvallysilealalysargglygly
115120125
valthralametglualavalhisalaserargasnalaleuthrgly
130135140
alaproleuasnleuthrproaspglnvalvalalailealaserasn
145150155160
ileglyglylysglnalaleugluthrvalglnargleuleuproval
165170175
leucysglnasphisglyleuthrproaspglnvalvalalaileala
180185190
serasnasnglyglylysglnalaleugluthrvalglnargleuleu
195200205
provalleucysglnasphisglyleuthrproaspglnvalvalala
210215220
ilealaserasnglyglyglylysglnalaleugluthrvalglnarg
225230235240
leuleuprovalleucysglnasphisglyleuthrproaspglnval
245250255
valalailealaserhisaspglyglylysglnalaleugluthrval
260265270
glnargleuleuprovalleucysglnasphisglyleuthrproasp
275280285
glnvalvalalailealaserasnglyglyglylysglnalaleuglu
290295300
thrvalglnargleuleuprovalleucysglnasphisglyleuthr
305310315320
proaspglnvalvalalailealaserasnileglyglylysglnala
325330335
leugluthrvalglnargleuleuprovalleucysglnasphisgly
340345350
leuthrproaspglnvalvalalailealaserasnasnglyglylys
355360365
glnalaleugluthrvalglnargleuleuprovalleucysglnasp
370375380
hisglyleuthrproaspglnvalvalalailealaserasnglygly
385390395400
glylysglnalaleugluthrvalglnargleuleuprovalleucys
405410415
glnasphisglyleuthrproaspglnvalvalalailealaserasn
420425430
asnglyglylysglnalaleugluthrvalglnargleuleuproval
435440445
leucysglnasphisglyleuthrproaspglnvalvalalaileala
450455460
serhisaspglyglylysglnalaleugluthrvalglnargleuleu
465470475480
provalleucysglnasphisglyleuthrproaspglnvalvalala
485490495
ilealaserasnileglyglylysglnalaleugluserilevalala
500505510
glnleuserargproaspproalaleualaalaleuthrasnasphis
515520525
leuvalalaleualacysleuglyglyargproalametaspalaval
530535540
lyslysglyleuprohisalaprogluleuileargargvalasnarg
545550555560
argileglygluargthrserhisargvalalaileserargvalgly
565570575
glyserserargarggluserileasnprotrpileleuthrglyphe
580585590
alaaspalagluglyserphevalleuserileglnasnargasnasp
595600605
tyralathrglytyrargilehisleuthrpheglnilethrleuhis
610615620
asnlysasplysserileleugluasnileglnserthrtrplysval
625630635640
glylysileasnasnalaglyaspasnleuvalglnleuargvaltyr
645650655
argphegluaspleulysvalileileasphispheglulystyrpro
660665670
leuilethrglnlysleuglyasptyrlysleuphelysglnalaphe
675680685
servalmetgluasnlysgluhisleulysgluasnglyilelysglu
690695700
leuvalargilelysalalysmetasntrpglyleuasnaspgluleu
705710715720
lyslysalapheprogluasnileserlysgluargproleuileasn
725730735
lysasnileproasnphelystrpleualaglyphethrserglyglu
740745750
glyserphephevalargleuarglysserasnvalasnalaargval
755760765
argvalglnleuvalphegluileserglnhisileargasplysasn
770775780
leumetasnserleuilethrtyrleuglycysglyhisiletyrglu
785790795800
glyasnlyssergluargsertrpleuglnpheargvalglulysphe
805810815
seraspileasnasplysileileprovalpheglngluasnthrleu
820825830
ileglyvallysleugluaspphegluasptrpcyslysvalalalys
835840845
leuilegluglulyslyshisleuthrgluserglyleuaspgluile
850855860
lyslysilelysleuasnmetasnlysglyargvalphealaserthr
865870875880
glysergluproproargalagluthrphevalpheleuaspleuglu
885890895
alathrglyleuproasnmetaspprogluilealagluileserleu
900905910
phealavalhisargserserleugluasnprogluargaspaspser
915920925
glyserleuvalleuproargvalleuasplysleuthrleucysmet
930935940
cysprogluargprophethralalysalasergluilethrglyleu
945950955960
sersergluserleumethiscysglylysalaglypheasnglyala
965970975
valvalargthrleuglnglypheleuserargglngluglyproile
980985990
cysleuvalalahisasnglypheasptyrasppheproleuleucys
99510001005
thrglyleuglnargleuglyalahisleuproglnaspthrval
101010151020
cysleuaspthrleuproalaleuargglyleuaspargalahis
102510301035
serhisglythrargalaglnglyarglyssertyrserleuala
104010451050
serleuphehisargtyrpheglnalagluproseralaalahis
105510601065
seralagluglyaspvalhisthrleuleuleuilepheleuhis
107010751080
argalaprogluleuleualatrpalaaspgluglnalaargser
108510901095
trpalahisileglupromettyrvalproproaspglyproser
110011051110
leugluala
1115
<210>23
<211>1116
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成megatal氨基酸序列
<400>23
metglyseralaproprolyslyslysarglysvalvalaspleuarg
151015
thrleuglytyrserglnglnglnglnglulysilelysprolysval
202530
argserthrvalalaglnhishisglualaleuvalglyhisglyphe
354045
thrhisalahisilevalalaleuserglnhisproalaalaleugly
505560
thrvalalavalthrtyrglnhisileilethralaleuprogluala
65707580
thrhisgluaspilevalglyvalglylysglntrpserglyalaarg
859095
alaleuglualaleuleuthraspalaglygluleuargglypropro
100105110
leuglnleuaspthrglyglnleuvallysilealalysargglygly
115120125
valthralametglualavalhisalaserargasnalaleuthrgly
130135140
alaproleuasnleuthrproaspglnvalvalalailealaserasn
145150155160
ileglyglylysglnalaleugluthrvalglnargleuleuproval
165170175
leucysglnasphisglyleuthrproaspglnvalvalalaileala
180185190
serasnasnglyglylysglnalaleugluthrvalglnargleuleu
195200205
provalleucysglnasphisglyleuthrproaspglnvalvalala
210215220
ilealaserasnglyglyglylysglnalaleugluthrvalglnarg
225230235240
leuleuprovalleucysglnasphisglyleuthrproaspglnval
245250255
valalailealaserhisaspglyglylysglnalaleugluthrval
260265270
glnargleuleuprovalleucysglnasphisglyleuthrproasp
275280285
glnvalvalalailealaserasnglyglyglylysglnalaleuglu
290295300
thrvalglnargleuleuprovalleucysglnasphisglyleuthr
305310315320
proaspglnvalvalalailealaserasnileglyglylysglnala
325330335
leugluthrvalglnargleuleuprovalleucysglnasphisgly
340345350
leuthrproaspglnvalvalalailealaserasnasnglyglylys
355360365
glnalaleugluthrvalglnargleuleuprovalleucysglnasp
370375380
hisglyleuthrproaspglnvalvalalailealaserasnglygly
385390395400
glylysglnalaleugluthrvalglnargleuleuprovalleucys
405410415
glnasphisglyleuthrproaspglnvalvalalailealaserasn
420425430
asnglyglylysglnalaleugluthrvalglnargleuleuproval
435440445
leucysglnasphisglyleuthrproaspglnvalvalalaileala
450455460
serhisaspglyglylysglnalaleugluthrvalglnargleuleu
465470475480
provalleucysglnasphisglyleuthrproaspglnvalvalala
485490495
ilealaserasnileglyglylysglnalaleugluserilevalala
500505510
glnleuserargproaspproalaleualaalaleuthrasnasphis
515520525
leuvalalaleualacysleuglyglyargproalametaspalaval
530535540
lyslysglyleuprohisalaprogluleuileargargvalasnarg
545550555560
argileglygluargthrserhisargvalalaileserargvalgly
565570575
glyserserargarggluserileasnprotrpileleuthrglyphe
580585590
alaaspalagluglyserphevalleuserileglnasnargasnasp
595600605
tyralathrglytyrargilehisleuthrpheglnilethrleuhis
610615620
asnlysasplysserileleugluasnileglnserthrtrplysval
625630635640
glylysileasnasnthrglyaspasnleuvalglnleuargvaltyr
645650655
argphegluaspleulysvalileileasphispheglulystyrpro
660665670
leuilethrglnlysleuglyasptyrlysleuphelysglnalaphe
675680685
servalmetgluasnlysgluhisleulysgluasnglyilelysglu
690695700
leuvalargilelysalalysmetasntrpglyleuasnaspgluleu
705710715720
lyslysalapheprogluasnileserlysgluargproleuileasn
725730735
lysasnileproasnphelystrpleualaglyphethrserglyasp
740745750
glyserphephevalargleuarglysserasnvalasnalaargval
755760765
argvalglnleuvalphegluileserglnhisileargasplysasn
770775780
leumetasnserleuilethrtyrleuglycysglyhisiletyrglu
785790795800
glyasnlyssergluargsertrpleuglnpheargvalglulysphe
805810815
seraspileasnasplysileileprovalpheglngluasnthrleu
820825830
ileglyvallysleugluaspphegluasptrpcyslysvalalalys
835840845
leuilegluglulyslyshisleuthrgluserglyleuaspgluile
850855860
lyslysilelysleuasnmetasnlysglyargvalphealaserthr
865870875880
glysergluproproargalagluthrphevalpheleuaspleuglu
885890895
alathrglyleuproasnmetaspprogluilealagluileserleu
900905910
phealavalhisargserserleugluasnprogluargaspaspser
915920925
glyserleuvalleuproargvalleuasplysleuthrleucysmet
930935940
cysprogluargprophethralalysalasergluilethrglyleu
945950955960
sersergluserleumethiscysglylysalaglypheasnglyala
965970975
valvalargthrleuglnglypheleuserargglngluglyproile
980985990
cysleuvalalahisasnglypheasptyrasppheproleuleucys
99510001005
thrglyleuglnargleuglyalahisleuproglnaspthrval
101010151020
cysleuaspthrleuproalaleuargglyleuaspargalahis
102510301035
serhisglythrargalaglnglyarglyssertyrserleuala
104010451050
serleuphehisargtyrpheglnalagluproseralaalahis
105510601065
seralagluglyaspvalhisthrleuleuleuilepheleuhis
107010751080
argalaprogluleuleualatrpalaaspgluglnalaargser
108510901095
trpalahisileglupromettyrvalproproaspglyproser
110011051110
leugluala
1115
<210>24
<211>29
<212>dna
<213>智人(homosapiens)
<400>24
cctggagcctgtgataaaagcaactgtta29
<210>25
<211>22
<212>dna
<213>智人(homosapiens)
<400>25
cagttgcttttatcacaggctc22
<210>26
<211>11
<212>dna
<213>智人(homosapiens)
<400>26
agtctagtgca11
<210>27
<211>39
<212>dna
<213>智人(homosapiens)
<400>27
agtctagtgcaagcttacagttgcttttatcacaggctc39
<210>28
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体靶位点
<400>28
cagttgcttttataacctttta22
<210>29
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体ctd靶位点
<400>29
tttccacttttatcacaggctc22
<210>30
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-smami靶位点
<400>30
tatcctccattatcaggtgtac22
<210>31
<211>22
<212>dna
<213>智人(homosapiens)
<400>31
cttccaggaattctttggcctg22
<210>32
<211>7078
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体表面展示质粒
<400>32
gacgaaagggcctcgtgatacgcctatttttataggttaatgtcatgataataatggttt60
cttaggacggatcgcttgcctgtaacttacacgcgcctcgtatcttttaatgatggaata120
atttgggaatttactctgtgtttatttatttttatgttttgtatttggattttagaaagt180
aaataaagaaggtagaagagttacggaatgaagaaaaaaaaataaacaaaggtttaaaaa240
atttcaacaaaaagcgtactttacatatatatttattagacaagaaaagcagattaaata300
gatatacattcgattaacgataagtaaaatgtaaaatcacaggattttcgtgtgtggtct360
tctacacagacaagatgaaacaattcggcattaatacctgagagcaggaagagcaagata420
aaaggtagtatttgttggcgatccccctagagtcttttacatcttcggaaaacaaaaact480
attttttctttaatttctttttttactttctatttttaatttatatatttatattaaaaa540
atttaaattataattatttttatagcacgtgatgaaaaggacccaggtggcacttttcgg600
ggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccg660
ctcatgagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagt720
attcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgttttt780
gctcacccagaaacgctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtg840
ggttacatcgaactggatctcaacagcggtaagatccttgagagttttcgccccgaagaa900
cgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtatt960
gacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgag1020
tactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagt1080
gctgccataaccatgagtgataacactgcggccaacttacttctgacaacgatcggagga1140
ccgaaggagctaaccgctttttttcacaacatgggggatcatgtaactcgccttgatcgt1200
tgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgta1260
gcaatggcaacaacgttgcgcaaactattaactggcgaactacttactctagcttcccgg1320
caacaattaatagactggatggaggcggataaagttgcaggaccacttctgcgctcggcc1380
cttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggt1440
atcattgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacg1500
ggcagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactg1560
attaagcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaa1620
cttcatttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaa1680
atcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaagga1740
tcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccg1800
ctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaact1860
ggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccac1920
cacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtg1980
gctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccg2040
gataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcga2100
acgacctacaccgaactgagatacctacagcgtgagcattgagaaagcgccacgcttccc2160
gaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacg2220
agggagcttccaggggggaacgcctggtatctttatagtcctgtcgggtttcgccacctc2280
tgacttgagcgtcgatttttgtgatgctcgtcaggggggccgagcctatggaaaaacgcc2340
agcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttcttt2400
cctgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgatacc2460
gctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgc2520
ccaatacgcaaaccgcctctccccgcgcgttggccgattcattaatgcagctggcacgac2580
aggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatgtgagttacctcact2640
cattaggcaccccaggctttacactttatgcttccggctcctatgttgtgtggaattgtg2700
agcggataacaatttcacacaggaaacagctatgaccatgattacgccaagctcggaatt2760
aaccctcactaaagggaacaaaagctgggtacccgacaggttatcagcaacaacacagtc2820
atatccattctcaattagctctaccacagtgtgtgaaccaatgtatccagcaccacctgt2880
aaccaaaacaattttagaagtactttcactttgtaactgagctgtcatttatattgaatt2940
ttcaaaaattcttactttttttttggatggacgcaaagaagtttaataatcatattacat3000
ggcattaccaccatatacatatccatatacatatccatatctaatcttacttatatgttg3060
tggaaatgtaaagagccccattatcttagcctaaaaaaaccttctctttggaactttcag3120
taatacgcttaactgctcattgctatattgaagtacggattagaagccgccgagcgggtg3180
acagccctccgaaggaagactctcctccgtgcgtcctcgtcttcaccggtcgcgttcctg3240
aaacgcagatgtgcctcgcgccgcactgctccgaacaataaagattctacaatactagct3300
tttatggttatgaagaggaaaaattggcagtaacctggccccacaaaccttcaaatgaac3360
gaatcaaattaacaaccataggatgataatgcgattagttttttagccttatttctgggg3420
taattaatcagcgaagcgatgatttttgatctattaacagatatataaatgcaaaaactg3480
cataaccactttaactaatactttcaacattttcggtttgtattacttcttattcaaatg3540
taataaaagatcgaatcctacttcatacattttcaattaagatgcagttacttcgctgtt3600
tttcaatattttctgttattgcttcagttttagcacaggaactgacaactatatgcgagc3660
aaatcccctcaccaactttagaatcgacgccgtactctttgtcaacgactactattttgg3720
ccaacgggaaggcaatgcaaggagtttttgaatattacaaatcagtaacgtttgtcagta3780
attgcggttctcacccctcaacaactagcaaaggcagccccataaacacacagtatgttt3840
ttaaggacaatagctcgacgattgaaggtagatacccatacgacgttccagactacgctc3900
tgcaggctagtggtggaggaggctctggtggaggcggtagcggaggcggagggtcggcta3960
gctccatcaacccatggattctgactggtttcactgatgccgaaggatcattcatgctaa4020
gaatccgtaacacgaacaaccggtcagtagggtactacacttcactggtattcgaaatca4080
ctctgcacaacaaggacaaatcgattcttgagaatatccagtcgacttggaaggtcggca4140
caatcaacaaccgaggcgacggcaccgccagactgagcgtcactcgtttcgaagatttga4200
aagtgattatcgaccacttcgagaaatatccgctgattacccagaaattgggcgattaca4260
agttgtttaaacaggcattcagcgtcatggagaacaaagaacatcttaaggagaatggga4320
ttaaggagctcgtacgaatcaaagctaagatgaattggggtctcaatgacgaattgaaaa4380
aagcatttccagagaacatcagcaaagagcgcccccttatcaataagaacattccgaatc4440
tcaaatggctggctggattcacatctggtgaaggcacattctacgtgcacctagcaaagt4500
ctgaagctagcggcaaggtatacgtgcgactgaggttcataatcggccagcacatcagag4560
acaagaacctgatgaattcattgataacatacctaggctgtggtacgatccaggagaaga4620
acaggtctaagggcagtatgctccacttcatagtaactaaattcagcgatatcaacgaca4680
agatcattccggtattccaggaaaatactctgattggcgtcaaactcgaggactttgaag4740
attggtgcaaggttgccaaattgatcgaagagaagaaacacctgaccgaatccggtttgg4800
atgagattaagaaaatcaagctgaacatgaacaaaggtcgttctagagaacaaaagttaa4860
tttctgaagaggacttgtaagatctgataacaacagtgtagatgtaacaaaatcgacttt4920
gttcccactgtacttttagctcgtacaaaatacaatatacttttcatttctccgtaaaca4980
acatgttttcccatgtaatatccttttctatttttcgttccgttaccaactttacacata5040
ctttatatagctattcacttctatacactaaaaaactaagacaattttaattttgctgcc5100
tgccatatttcaatttgttataaattcctataatttatcctattagtagctaaaaaaaga5160
tgaatgtgaatcgaatcctaagagaattgagctccaattcgccctatagtgagtcgtatt5220
acaattcactggccgtcgttttacaacgtcgtgactgggaaaaccctggcgttacccaac5280
ttaatcgccttgcagcacatccccctttcgccagctggcgtaatagcgaagaggcccgca5340
ccgatcgcctttcccaacagttgcgcagcctgaatggcgaatggacgcgccctgtagcgg5400
cgcattaagcgcggcgggtgtggtggttacgcgcagcgtgaccgctacacttgccagcgc5460
cctagcgcccgctcctttcgctttcttcccttcctttctcgccacgttcgccggctttcc5520
ccgtcaagctctaaatcgggggctccctttagggttccgatttagtgctttacggcacct5580
cgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctgatagac5640
ggtttttcgccctttgacgttggagtccacgttctttaatagtggactcttgttccaaac5700
tggaacaacactcaaccctatctcggtctattcttttgatttataagggattttgccgat5760
ttcggcctattggttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaa5820
aatattaacgtttacaatttcctgatgcggtattttctccttacgcatctgtgcggtatt5880
tcacaccgcaggcaagtgcacaaacaatacttaaataaatactactcagtaataacctat5940
ttcttagcatttttgacgaaatttgctattttgttagagtcttttacaccatttgtctcc6000
acacctccgcttacatcaacaccaataacgccatttaatctaagcgcatcaccaacattt6060
tctggcgtcagtccaccagctaacataaaatgtaagctttcggggctctcttgccttcca6120
acccagtcagaaatcgagttccaatccaaaagttcacctgtcccacctgcttctgaatca6180
aacaagggaataaacgaatgaggtttctgtgaagctgcactgagtagtatgttgcagtct6240
tttggaaatacgagtcttttaataactggcaaaccgaggaactcttggtattcttgccac6300
gactcatctccatgcagttggacgatatcaatgccgtaatcattgaccagagccaaaaca6360
tcctccttaggttgattacgaaacacgccaaccaagtatttcggagtgcctgaactattt6420
ttatatgcttttacaagacttgaaattttccttgcaataaccgggtcaattgttctcttt6480
ctattgggcacacatataatacccagcaagtcagcatcggaatcaagagcacattctgcg6540
gcctctgtgctctgcaagccgcaaactttcaccaatggaccagaactacctgtgaaatta6600
ataacagacatactccaagctgcctttgtgtgcttaatcacgtatactcacgtgctcaat6660
agtcaccaatgccctccctcttggccctctccttttcttttttcgaccgaattaattctt6720
aatcggcaaaaaaagaaaagctccggatcaagattgtacgtaaggtgacaagctattttt6780
caataaagaatatcttccactactgccatctggcgtcataactgcaaagtacacatatat6840
tacgatgctgtctattaaatgcttcctatattatatatatagtaatgtcgtttatggtgc6900
actctcagtacaatctgctctgatgccgcatagttaagccagccccgacacccgccaaca6960
cccgctgacgcgccctgacgggcttgtctgctcccggcatccgcttacagacaagctgtg7020
accgtctccgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcga7078
<210>33
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>i-onuilhe变体的中心4阵列的合成多核苷酸序列
<220>
<221>misc_feature
<222>(10)..(13)
<223>nisa,c,g,ort
<400>33
cttccaggannnntttggcctg22
<210>34
<211>7243
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>i-onuilhe变体表面展示质粒的合成多核苷酸序列
<400>34
gacgaaagggcctcgtgatacgcctatttttataggttaatgtcatgataataatggttt60
cttaggacggatcgcttgcctgtaacttacacgcgcctcgtatcttttaatgatggaata120
atttgggaatttactctgtgtttatttatttttatgttttgtatttggattttagaaagt180
aaataaagaaggtagaagagttacggaatgaagaaaaaaaaataaacaaaggtttaaaaa240
atttcaacaaaaagcgtactttacatatatatttattagacaagaaaagcagattaaata300
gatatacattcgattaacgataagtaaaatgtaaaatcacaggattttcgtgtgtggtct360
tctacacagacaagatgaaacaattcggcattaatacctgagagcaggaagagcaagata420
aaaggtagtatttgttggcgatccccctagagtcttttacatcttcggaaaacaaaaact480
attttttctttaatttctttttttactttctatttttaatttatatatttatattaaaaa540
atttaaattataattatttttatagcacgtgatgaaaaggacccaggtggcacttttcgg600
ggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccg660
ctcatgagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagt720
attcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgttttt780
gctcacccagaaacgctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtg840
ggttacatcgaactggatctcaacagcggtaagatccttgagagttttcgccccgaagaa900
cgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtatt960
gacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgag1020
tactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagt1080
gctgccataaccatgagtgataacactgcggccaacttacttctgacaacgatcggagga1140
ccgaaggagctaaccgctttttttcacaacatgggggatcatgtaactcgccttgatcgt1200
tgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgta1260
gcaatggcaacaacgttgcgcaaactattaactggcgaactacttactctagcttcccgg1320
caacaattaatagactggatggaggcggataaagttgcaggaccacttctgcgctcggcc1380
cttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggt1440
atcattgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacg1500
ggcagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactg1560
attaagcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaa1620
cttcatttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaa1680
atcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaagga1740
tcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccg1800
ctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaact1860
ggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccac1920
cacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtg1980
gctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccg2040
gataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcga2100
acgacctacaccgaactgagatacctacagcgtgagcattgagaaagcgccacgcttccc2160
gaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacg2220
agggagcttccaggggggaacgcctggtatctttatagtcctgtcgggtttcgccacctc2280
tgacttgagcgtcgatttttgtgatgctcgtcaggggggccgagcctatggaaaaacgcc2340
agcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttcttt2400
cctgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgatacc2460
gctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgc2520
ccaatacgcaaaccgcctctccccgcgcgttggccgattcattaatgcagctggcacgac2580
aggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatgtgagttacctcact2640
cattaggcaccccaggctttacactttatgcttccggctcctatgttgtgtggaattgtg2700
agcggataacaatttcacacaggaaacagctatgaccatgattacgccaagctcggaatt2760
aaccctcactaaagggaacaaaagctgggtacccgacaggttatcagcaacaacacagtc2820
atatccattctcaattagctctaccacagtgtgtgaaccaatgtatccagcaccacctgt2880
aaccaaaacaattttagaagtactttcactttgtaactgagctgtcatttatattgaatt2940
ttcaaaaattcttactttttttttggatggacgcaaagaagtttaataatcatattacat3000
ggcattaccaccatatacatatccatatacatatccatatctaatcttacttatatgttg3060
tggaaatgtaaagagccccattatcttagcctaaaaaaaccttctctttggaactttcag3120
taatacgcttaactgctcattgctatattgaagtacggattagaagccgccgagcgggtg3180
acagccctccgaaggaagactctcctccgtgcgtcctcgtcttcaccggtcgcgttcctg3240
aaacgcagatgtgcctcgcgccgcactgctccgaacaataaagattctacaatactagct3300
tttatggttatgaagaggaaaaattggcagtaacctggccccacaaaccttcaaatgaac3360
gaatcaaattaacaaccataggatgataatgcgattagttttttagccttatttctgggg3420
taattaatcagcgaagcgatgatttttgatctattaacagatatataaatgcaaaaactg3480
cataaccactttaactaatactttcaacattttcggtttgtattacttcttattcaaatg3540
taataaaagatcgaatcctacttcatacattttcaattaagatgcagttacttcgctgtt3600
tttcaatattttctgttattgcttcagttttagcacaggaactgacaactatatgcgagc3660
aaatcccctcaccaactttagaatcgacgccgtactctttgtcaacgactactattttgg3720
ccaacgggaaggcaatgcaaggagtttttgaatattacaaatcagtaacgtttgtcagta3780
attgcggttctcacccctcaacaactagcaaaggcagccccataaacacacagtatgttt3840
ttaaggacaatagctcgacgattgaaggtagatacccatacgacgttccagactacgctc3900
tgcaggctagtggtggaggaggctctggtggaggcggtagcggaggcggagggtcggcta3960
gctccatcaacccatggattctgactggtttcgctgatgccgaaggaagcttcgtgctaa4020
gtatccaaaacagaaacgattatgctactggttacagaattcacctgacattccaaatca4080
ccctgcacaacaaggacaaatcgattctggagaatatccagtcgacttggaaggtcggca4140
aaatcaataacacgggcgacaacctcgtccaactgagagtctaccgtttcgaagatttga4200
aagtgattatcgaccacttcgagaaatatccgctgataacacagaaattgggcgattaca4260
agttgtttaaacaggcattcagcgtcatggagaacaaagaacatcttaaggagaatggga4320
ttaaggagctcgtacgaatcaaagctaagatgaattggggtctcaatgacgaattgaaaa4380
aagcatttccagagaacattagcaaagagcgcccccttatcaataagaacattccgaatt4440
tcaaatggctggctggattcacatctggtgatggctccttcttcgtgcgcctaagaaagt4500
ctaatgttaatgctagagtacgtgtgcaactggtattcgagatctcacagcacatcagag4560
acaagaacctgatgaattcattgataacatacctaggctgtggtcacatctacgagggaa4620
acaaatctgagcgcagttggctccaattcagagtagaaaaattcagcgatatcaacgaca4680
agatcattccggtattccaggaaaatactctgattggcgtcaaactcgaggactttgaag4740
attggtgcaaggttgccaaattgatcgaagagaagaaacacctgaccgaatccggtttgg4800
atgagattaagaaaatcaagctgaacatgaacaaaggtcgtgtcttctctagaggcggtt4860
ccagaagcggatctggtactggcgaacagaaactcataagcgaagaagaccttagcggga4920
ctggagagcaaaagttgatttctgaggaggatttgtcgggaaccggggagcagaagttaa4980
tcagtgaagaggatctcagtggaacgggcgaacaaaagttgatctcggaggaagacttat5040
aatgagatctgataacaacagtgtagatgtaacaaaatcgactttgttcccactgtactt5100
ttagctcgtacaaaatacaatatacttttcatttctccgtaaacaacatgttttcccatg5160
taatatccttttctatttttcgttccgttaccaactttacacatactttatatagctatt5220
cacttctatacactaaaaaactaagacaattttaattttgctgcctgccatatttcaatt5280
tgttataaattcctataatttatcctattagtagctaaaaaaagatgaatgtgaatcgaa5340
tcctaagagaattgagctccaattcgccctatagtgagtcgtattacaattcactggccg5400
tcgttttacaacgtcgtgactgggaaaaccctggcgttacccaacttaatcgccttgcag5460
cacatccccctttcgccagctggcgtaatagcgaagaggcccgcaccgatcgcctttccc5520
aacagttgcgcagcctgaatggcgaatggacgcgccctgtagcggcgcattaagcgcggc5580
gggtgtggtggttacgcgcagcgtgaccgctacacttgccagcgccctagcgcccgctcc5640
tttcgctttcttcccttcctttctcgccacgttcgccggctttccccgtcaagctctaaa5700
tcgggggctccctttagggttccgatttagtgctttacggcacctcgaccccaaaaaact5760
tgattagggtgatggttcacgtagtgggccatcgccctgatagacggtttttcgcccttt5820
gacgttggagtccacgttctttaatagtggactcttgttccaaactggaacaacactcaa5880
ccctatctcggtctattcttttgatttataagggattttgccgatttcggcctattggtt5940
aaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaatattaacgtttac6000
aatttcctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcaggcaa6060
gtgcacaaacaatacttaaataaatactactcagtaataacctatttcttagcatttttg6120
acgaaatttgctattttgttagagtcttttacaccatttgtctccacacctccgcttaca6180
tcaacaccaataacgccatttaatctaagcgcatcaccaacattttctggcgtcagtcca6240
ccagctaacataaaatgtaagctttcggggctctcttgccttccaacccagtcagaaatc6300
gagttccaatccaaaagttcacctgtcccacctgcttctgaatcaaacaagggaataaac6360
gaatgaggtttctgtgaagctgcactgagtagtatgttgcagtcttttggaaatacgagt6420
cttttaataactggcaaaccgaggaactcttggtattcttgccacgactcatctccatgc6480
agttggacgacatcaatgccgtaatcattgaccagagccaaaacatcctccttaggttga6540
ttacgaaacacgccaaccaagtatttcggagtgcctgaactatttttatatgcttttaca6600
agacttgaaattttccttgcaataaccgggtcaattgttctctttctattgggcacacat6660
ataatacccagcaagtcagcatcggaatcaagagcacattctgcggcctctgtgctctgc6720
aagccgcaaactttcaccaatggaccagaactacctgtgaaattaataacagacatactc6780
caagctgcctttgtgtgcttaatcacgtatactcacgtgctcaatagtcaccaatgccct6840
ccctcttggccctctccttttcttttttcgaccgaattaattcttaatcggcaaaaaaag6900
aaaagctccggatcaagattgtacgtaaggtgacaagctatttttcaataaagaatatct6960
tccactactgccatctggcgtcataactgcaaagtacacatatattacgatgctgtctat7020
taaatgcttcctatattatatatatagtaatgtcgtttatggtgcactctcagtacaatc7080
tgctctgatgccgcatagttaagccagccccgacacccgccaacacccgctgacgcgccc7140
tgacgggcttgtctgctcccggcatccgcttacagacaagctgtgaccgtctccgggagc7200
tgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcga7243
<210>35
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>i-onuilhe变体的中心4阵列的合成多核苷酸序列
<220>
<221>misc_feature
<222>(10)..(13)
<223>nisa,c,g,ort
<400>35
cagttgcttnnnncacaggctc22
<210>36
<211>2634
<212>rna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>编码megatal的合成mrna
<400>36
augggauccgcgccaccuaagaagaaacgcaaagucguggaucuacgcacgcucggcuac60
agucagcagcagcaagagaagaucaaaccgaaggugcguucgacaguggcgcagcaccac120
gaggcacuggugggccauggguuuacacacgcgcacaucguugcgcucagccaacacccg180
gcagcguuagggaccgucgcugucacguaucagcacauaaucacggcguugccagaggcg240
acacacgaagacaucguuggcgucggcaaacagugguccggcgcacgcgcccuggaggcc300
uugcucacggaugcgggggaguugagagguccgccguuacaguuggacacaggccaacuu360
gugaagauugcaaaacguggcggcgugaccgcaauggaggcagugcaugcaucgcgcaau420
gcacugacgggugccccccugaacuuaacacccgaucaagugguagcgauagcgucaaau480
aucggggguaaacaggcuuuggagacgguacagcgguuauugccgguacucugccaggac540
cacggauugacaccggaccaagugguggcgauugcguccaauaacggaggcaagcaggca600
cuagagaccguccaacggcuucuucccguucuuugucaggaucaugggcuaaccccugau660
cagguagucgcuauagcuucaaauggcgggggcaagcaagcacuggagaccguucaacga720
cuccugccagugcucugccaagaccacggacuuacgccagaucaggugguugcuauugcc780
ucccacgauggcgggaaacaagcguuggaaacugugcagagacuguuaccugucuugugu840
caagaccacggccucacgccagaucaggugguagccauagcgucgaauggaggugguaag900
caagcccuugaaacgguccagcgucuucugccgguguugugccaggaccacggacuaacg960
ccggaucaggucguagccauugcuucaaauaucggcggcaaacaggcgcuagagacaguc1020
cagcgccucuugccuguguuaugccaggaucacggcuuaaccccagaccaaguuguggcu1080
auugcaucuaacaaugguggcaaacaagccuuggagacagugcaacgauuacugccuguc1140
uuaugucaggaucauggccugacgcccgaucagguaguggcaaucgcaucuaacggcgga1200
gguaagcaagcacuggagacuguccagagauuguuacccguacuaugucaagaucauggu1260
uugacgccugaucagguuguugcgauagccagcaacaacggagggaaacaggcucuugaa1320
accguacagcgacuucucccagucuugugccaagaucacgggcuuacuccugaucaaguc1380
guagcuaucgccagccacgacggugggaaacaggcccuggaaaccguacaacgucuccuc1440
ccaguacuuugucaagaccacggguugacuccggaucaagucgucgcgaucgcgagcaau1500
auaggggggaagcaggcgcuggaaagcauuguggcccagcugagccggccugauccggcg1560
uuggccgcguugaccaacgaccaccucgucgccuuggccugccucggcggacguccugcc1620
auggaugcagugaaaaagggauugccgcacgcgccggaauugaucagaagagucaaucgc1680
cguauuggcgaacgcacgucccaucgcguugcgauaucuagagugggaggaagcucucgc1740
agagaguccaucaacccauggauucugacugguuucgcugaugccgaaggaagcuucgug1800
cuaaguauccaaaacagaaacgauuaugcuacugguuacagaauucaccugacauuccaa1860
aucacccugcacaacaaggacaaaucgauucuggagaauauccagucgacuuggaagguc1920
ggcaaaaucaauaacacgggcgacaaccucguccaacugagagucuaccguuucgaagau1980
uugaaagugauuaucgaccacuucgagaaauauccgcugauaacacagaaauugggcgau2040
uacaaguuguuuaaacaggcauucagcgucauggagaacaaagaacaucuuaaggagaau2100
gggauuaaggagcucguacgaaucaaagcuaagaugaauuggggucucaaugacgaauug2160
aaaaaagcauuuccagagaacauuagcaaagagcgcccccuuaucaauaagaacauuccg2220
aauuucaaauggcuggcuggauucacaucuggugauggcuccuucuucgugcgccuaaga2280
aagucuaauguuaaugcuagaguacgugugcaacugguauucgagaucucacagcacauc2340
agagacaagaaccugaugaauucauugauaacauaccuaggcuguggucacaucuacgag2400
ggaaacaaaucugagcgcaguuggcuccaauucagaguagaaaaauucagcgauaucaac2460
gacaagaucauuccgguauuccaggaaaauacucugauuggcgucaaacucgaggacuuu2520
gaagauuggugcaagguugccaaauugaucgaagagaagaaacaccugaccgaauccggu2580
uuggaugagauuaagaaaaucaagcugaacaugaacaaaggucgugucuucuaa2634
<210>37
<211>3351
<212>rna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>编码megatal的合成mrna
<400>37
augggauccgcgccaccuaagaagaaacgcaaagucguggaucuacgcacgcucggcuac60
agucagcagcagcaagagaagaucaaaccgaaggugcguucgacaguggcgcagcaccac120
gaggcacuggugggccauggguuuacacacgcgcacaucguugcgcucagccaacacccg180
gcagcguuagggaccgucgcugucacguaucagcacauaaucacggcguugccagaggcg240
acacacgaagacaucguuggcgucggcaaacagugguccggcgcacgcgcccuggaggcc300
uugcucacggaugcgggggaguugagagguccgccguuacaguuggacacaggccaacuu360
gugaagauugcaaaacguggcggcgugaccgcaauggaggcagugcaugcaucgcgcaau420
gcacugacgggugccccccugaacuuaacacccgaucaagugguagcgauagcgucaaau480
aucggggguaaacaggcuuuggagacgguacagcgguuauugccgguacucugccaggac540
cacggauugacaccggaccaagugguggcgauugcguccaauaacggaggcaagcaggca600
cuagagaccguccaacggcuucuucccguucuuugucaggaucaugggcuaaccccugau660
cagguagucgcuauagcuucaaauggcgggggcaagcaagcacuggagaccguucaacga720
cuccugccagugcucugccaagaccacggacuuacgccagaucaggugguugcuauugcc780
ucccacgauggcgggaaacaagcguuggaaacugugcagagacuguuaccugucuugugu840
caagaccacggccucacgccagaucaggugguagccauagcgucgaauggaggugguaag900
caagcccuugaaacgguccagcgucuucugccgguguugugccaggaccacggacuaacg960
ccggaucaggucguagccauugcuucaaauaucggcggcaaacaggcgcuagagacaguc1020
cagcgccucuugccuguguuaugccaggaucacggcuuaaccccagaccaaguuguggcu1080
auugcaucuaacaaugguggcaaacaagccuuggagacagugcaacgauuacugccuguc1140
uuaugucaggaucauggccugacgcccgaucagguaguggcaaucgcaucuaacggcgga1200
gguaagcaagcacuggagacuguccagagauuguuacccguacuaugucaagaucauggu1260
uugacgccugaucagguuguugcgauagccagcaacaacggagggaaacaggcucuugaa1320
accguacagcgacuucucccagucuugugccaagaucacgggcuuacuccugaucaaguc1380
guagcuaucgccagccacgacggugggaaacaggcccuggaaaccguacaacgucuccuc1440
ccaguacuuugucaagaccacggguugacuccggaucaagucgucgcgaucgcgagcaau1500
auaggggggaagcaggcgcuggaaagcauuguggcccagcugagccggccugauccggcg1560
uuggccgcguugaccaacgaccaccucgucgccuuggccugccucggcggacguccugcc1620
auggaugcagugaaaaagggauugccgcacgcgccggaauugaucagaagagucaaucgc1680
cguauuggcgaacgcacgucccaucgcguugcgauaucuagagugggaggaagcucucgc1740
agagaguccaucaacccauggauucugacugguuucgcugaugccgaaggaagcuucgug1800
cuaaguauccaaaacagaaacgauuaugcuacugguuacagaauucaccugacauuccaa1860
aucacccugcacaacaaggacaaaucgauucuggagaauauccagucgacuuggaagguc1920
ggcaaaaucaauaacacgggcgacaaccucguccaacugagagucuaccguuucgaagau1980
uugaaagugauuaucgaccacuucgagaaauauccgcugauaacacagaaauugggcgau2040
uacaaguuguuuaaacaggcauucagcgucauggagaacaaagaacaucuuaaggagaau2100
gggauuaaggagcucguacgaaucaaagcuaagaugaauuggggucucaaugacgaauug2160
aaaaaagcauuuccagagaacauuagcaaagagcgcccccuuaucaauaagaacauuccg2220
aauuucaaauggcuggcuggauucacaucuggugauggcuccuucuucgugcgccuaaga2280
aagucuaauguuaaugcuagaguacgugugcaacugguauucgagaucucacagcacauc2340
agagacaagaaccugaugaauucauugauaacauaccuaggcuguggucacaucuacgag2400
ggaaacaaaucugagcgcaguuggcuccaauucagaguagaaaaauucagcgauaucaac2460
gacaagaucauuccgguauuccaggaaaauacucugauuggcgucaaacucgaggacuuu2520
gaagauuggugcaagguugccaaauugaucgaagagaagaaacaccugaccgaauccggu2580
uuggaugagauuaagaaaaucaagcugaacaugaacaaaggucgugucuucgcuagcacc2640
gguucugagccaccucgggcugagaccuuuguauuccuggaccuagaagccacugggcuc2700
ccaaacauggacccugagauugcagagauaucccuuuuugcuguucaccgcucuucccug2760
gagaacccagaacgggaugauucugguuccuuggugcugccccguguucuggacaagcuc2820
acacugugcaugugcccggagcgccccuuuacugccaaggccagugagauuacugguuug2880
agcagcgaaagccugaugcacugcgggaaggcugguuucaauggcgcugugguaaggaca2940
cugcagggcuuccuaagccgccaggagggccccaucugccuuguggcccacaauggcuuc3000
gauuaugacuucccacugcugugcacggggcuacaacgucugggugcccaucugccccaa3060
gacacugucugccuggacacacugccugcauugcggggccuggaccgugcucacagccac3120
ggcaccagggcucaaggccgcaaaagcuacagccuggccagucucuuccaccgcuacuuc3180
caggcugaacccagugcugcccauucagcagaaggugaugugcacacccugcuucugauc3240
uuccugcaucgugcuccugagcugcucgccugggcagaugagcaggcccgcagcugggcu3300
cauauugagcccauguacgugccaccugaugguccaagccucgaagccuga3351
<210>38
<211>711
<212>rna
<213>小家鼠(musmusculus)
<400>38
augucugagccaccucgggcugagaccuuuguauuccuggaccuagaagccacugggcuc60
ccaaacauggacccugagauugcagagauaucccuuuuugcuguucaccgcucuucccug120
gagaacccagaacgggaugauucugguuccuuggugcugccccguguucuggacaagcuc180
acacugugcaugugcccggagcgccccuuuacugccaaggccagugagauuacugguuug240
agcagcgaaagccugaugcacugcgggaaggcugguuucaauggcgcugugguaaggaca300
cugcagggcuuccuaagccgccaggagggccccaucugccuuguggcccacaauggcuuc360
gauuaugacuucccacugcugugcacggggcuacaacgucugggugcccaucugccccaa420
gacacugucugccuggacacacugccugcauugcggggccuggaccgugcucacagccac480
ggcaccagggcucaaggccgcaaaagcuacagccuggccagucucuuccaccgcuacuuc540
caggcugaacccagugcugcccauucagcagaaggugaugugcacacccugcuucugauc600
uuccugcaucgugcuccugagcugcucgccugggcagaugagcaggcccgcagcugggcu660
cauauugagcccauguacgugccaccugaugguccaagccucgaagccuga711
<210>39
<211>236
<212>prt
<213>小家鼠(musmusculus)
<400>39
metsergluproproargalagluthrphevalpheleuaspleuglu
151015
alathrglyleuproasnmetaspprogluilealagluileserleu
202530
phealavalhisargserserleugluasnprogluargaspaspser
354045
glyserleuvalleuproargvalleuasplysleuthrleucysmet
505560
cysprogluargprophethralalysalasergluilethrglyleu
65707580
sersergluserleumethiscysglylysalaglypheasnglyala
859095
valvalargthrleuglnglypheleuserargglngluglyproile
100105110
cysleuvalalahisasnglypheasptyrasppheproleuleucys
115120125
thrglyleuglnargleuglyalahisleuproglnaspthrvalcys
130135140
leuaspthrleuproalaleuargglyleuaspargalahisserhis
145150155160
glythrargalaglnglyarglyssertyrserleualaserleuphe
165170175
hisargtyrpheglnalagluproseralaalahisseralaglugly
180185190
aspvalhisthrleuleuleuilepheleuhisargalaprogluleu
195200205
leualatrpalaaspgluglnalaargsertrpalahisileglupro
210215220
mettyrvalproproaspglyproserleugluala
225230235
<210>40
<211>3
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>40
glyglygly
1
<210>41
<211>5
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>41
aspglyglyglyser
15
<210>42
<211>5
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>42
thrglyglulyspro
15
<210>43
<211>4
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>43
glyglyargarg
1
<210>44
<211>5
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>44
glyglyglyglyser
15
<210>45
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>45
gluglylysserserglyserglysergluserlysvalasp
1510
<210>46
<211>18
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>46
lysgluserglyservalsersergluglnleualaglnpheargser
151015
leuasp
<210>47
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>47
glyglyargargglyglyglyser
15
<210>48
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>48
leuargglnargaspglygluargpro
15
<210>49
<211>12
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>49
leuargglnlysaspglyglyglysergluargpro
1510
<210>50
<211>16
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>示例性连接子序列
<400>50
leuargglnlysaspglyglyglyserglyglyglysergluargpro
151015
<210>51
<211>7
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>tev蛋白酶的切割序列
<220>
<221>site
<222>(2)..(3)
<223>xaa为任何氨基酸
<220>
<221>site
<222>(5)..(5)
<223>xaa为任何氨基酸
<220>
<221>site
<222>(7)..(7)
<223>xaa=gly或ser
<400>51
gluxaaxaatyrxaaglnxaa
15
<210>52
<211>7
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>tev蛋白酶的切割序列
<400>52
gluasnleutyrpheglngly
15
<210>53
<211>7
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>tev蛋白酶的切割序列
<400>53
gluasnleutyrpheglnser
15
<210>54
<211>22
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>54
glyserglyalathrasnpheserleuleulysglnalaglyaspval
151015
glugluasnproglypro
20
<210>55
<211>19
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>55
alathrasnpheserleuleulysglnalaglyaspvalglugluasn
151015
proglypro
<210>56
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>56
leuleulysglnalaglyaspvalglugluasnproglypro
1510
<210>57
<211>21
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>57
glyserglygluglyargglyserleuleuthrcysglyaspvalglu
151015
gluasnproglypro
20
<210>58
<211>18
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>58
gluglyargglyserleuleuthrcysglyaspvalglugluasnpro
151015
glypro
<210>59
<211>13
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>59
leuleuthrcysglyaspvalglugluasnproglypro
1510
<210>60
<211>23
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>60
glyserglyglncysthrasntyralaleuleulysleualaglyasp
151015
valgluserasnproglypro
20
<210>61
<211>20
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>61
glncysthrasntyralaleuleulysleualaglyaspvalgluser
151015
asnproglypro
20
<210>62
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>62
leuleulysleualaglyaspvalgluserasnproglypro
1510
<210>63
<211>25
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>63
glyserglyvallysglnthrleuasnpheaspleuleulysleuala
151015
glyaspvalgluserasnproglypro
2025
<210>64
<211>22
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>64
vallysglnthrleuasnpheaspleuleulysleualaglyaspval
151015
gluserasnproglypro
20
<210>65
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>65
leuleulysleualaglyaspvalgluserasnproglypro
1510
<210>66
<211>19
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>66
leuleuasnpheaspleuleulysleualaglyaspvalgluserasn
151015
proglypro
<210>67
<211>19
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>67
thrleuasnpheaspleuleulysleualaglyaspvalgluserasn
151015
proglypro
<210>68
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>68
leuleulysleualaglyaspvalgluserasnproglypro
1510
<210>69
<211>17
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>69
asnpheaspleuleulysleualaglyaspvalgluserasnprogly
151015
pro
<210>70
<211>20
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>70
glnleuleuasnpheaspleuleulysleualaglyaspvalgluser
151015
asnproglypro
20
<210>71
<211>24
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>71
alaprovallysglnthrleuasnpheaspleuleulysleualagly
151015
aspvalgluserasnproglypro
20
<210>72
<211>40
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>72
valthrgluleuleutyrargmetlysargalagluthrtyrcyspro
151015
argproleuleualailehisprothrglualaarghislysglnlys
202530
ilevalalaprovallysglnthr
3540
<210>73
<211>18
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>73
leuasnpheaspleuleulysleualaglyaspvalgluserasnpro
151015
glypro
<210>74
<211>40
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>74
leuleualailehisprothrglualaarghislysglnlysileval
151015
alaprovallysglnthrleuasnpheaspleuleulysleualagly
202530
aspvalgluserasnproglypro
3540
<210>75
<211>33
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>包括2a位点的自切割多肽
<400>75
glualaarghislysglnlysilevalalaprovallysglnthrleu
151015
asnpheaspleuleulysleualaglyaspvalgluserasnprogly
202530
pro
<210>76
<211>10
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>共有kozak序列
<400>76
gccrccatgg10
<210>77
<211>29
<212>dna
<213>智人(homosapiens)
<400>77
taacagttgcttttatcacaggctccagg29
<210>78
<211>29
<212>dna
<213>智人(homosapiens)
<400>78
attctcaacgaaaatagtgtccgaggtcc29
<210>79
<211>293
<212>prt
<213>榆树枯萎菌美洲亚种(线粒体)
<400>79
serileasnprotrpileleuthrglyphealaaspalagluglyser
151015
pheleuleuargileargasnasnasnlysserservalglytyrser
202530
thrgluleuglypheglnilethrleuhisasnlysasplysserile
354045
leugluasnileglnserthrtrplysvalglyvalilealaasnser
505560
glyaspasnalavalserleulysvalthrargphegluaspleulys
65707580
valileileasphispheglulystyrproleuilethrglnlysleu
859095
glyasptyrlysleuphelysglnalapheservalmetgluasnlys
100105110
gluhisleulysgluasnglyilelysgluleuvalargilelysala
115120125
lysleuasntrpglyleuthraspgluleulyslysalapheproglu
130135140
asnileserlysgluargserleuileasnlysasnileproasnphe
145150155160
lystrpleualaglyphethrserglygluglycysphephevalasn
165170175
leuilelysserlysserlysleuglyvalglnvalglnleuvalphe
180185190
serilethrglnhisilelysasplysasnleumetasnserleuile
195200205
thrtyrleuglycysglytyrilelysglulysasnlyssergluphe
210215220
sertrpleuaspphevalvalthrlyspheseraspileasnasplys
225230235240
ileileprovalpheglngluasnthrleuileglyvallysleuglu
245250255
aspphegluasptrpcyslysvalalalysleuilegluglulyslys
260265270
hisleuthrgluserglyleuaspgluilelyslysilelysleuasn
275280285
metasnlysglyarg
290
<210>80
<211>293
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<400>80
serileasnprotrpileleuthrglyphealaaspalagluglyser
151015
phevalleuserileglnasnargasnasptyralathrglytyrarg
202530
ilehisleuthrpheglnilethrleuhisasnlysasplysserile
354045
leugluasnileglnserthrtrplysvalglylysileasnasnala
505560
glyaspasnleuvalglnleuargvaltyrargphegluaspleulys
65707580
valileileasphispheglulystyrproleuilethrglnlysleu
859095
glyasptyrlysleuphelysglnalapheservalmetgluasnlys
100105110
gluhisleulysgluasnglyilelysgluleuvalargilelysala
115120125
lysmetasntrpglyleuasnaspgluleulyslysalapheproglu
130135140
asnileserlysgluargproleuileasnlysasnileproasnphe
145150155160
lystrpleualaglyphethrserglygluglyserphephevalarg
165170175
leuarglysserasnvalasnalaargvalargvalglnleuvalphe
180185190
gluileserglnhisileargasplysasnleumetasnserleuile
195200205
thrtyrleuglycysglyhisiletyrgluglyasnlyssergluarg
210215220
sertrpleuglnpheargvalglulyspheseraspileasnasplys
225230235240
ileileprovalpheglngluasnthrleuileglyvallysleuglu
245250255
aspphegluasptrpcyslysvalalalysleuilegluglulyslys
260265270
hisleuthrgluserglyleuaspgluilelyslysilelysleuasn
275280285
metasnlysglyarg
290
<210>81
<211>293
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>
<222>
<223>合成i-onuilhe变体
<400>81
serileasnprotrpileleuthrglyphealaaspalagluglyser
151015
phevalleuserileglnasnargasnasptyralathrglytyrarg
202530
ilehisleuthrpheglnilethrleuhisasnlysasplysserile
354045
leugluasnileglnserthrtrplysvalglylysileasnasnthr
505560
glyaspasnleuvalglnleuargvaltyrargphegluaspleulys
65707580
valileileasphispheglulystyrproleuilethrglnlysleu
859095
glyasptyrlysleuphelysglnalapheservalmetgluasnlys
100105110
gluhisleulysgluasnglyilelysgluleuvalargilelysala
115120125
lysmetasntrpglyleuasnaspgluleulyslysalapheproglu
130135140
asnileserlysgluargproleuileasnlysasnileproasnphe
145150155160
lystrpleualaglyphethrserglyaspglyserphephevalarg
165170175
leuarglysserasnvalasnalaargvalargvalglnleuvalphe
180185190
gluileserglnhisileargasplysasnleumetasnserleuile
195200205
thrtyrleuglycysglyhisiletyrgluglyasnlyssergluarg
210215220
sertrpleuglnpheargvalglulyspheseraspileasnasplys
225230235240
ileileprovalpheglngluasnthrleuileglyvallysleuglu
245250255
aspphegluasptrpcyslysvalalalysleuilegluglulyslys
260265270
hisleuthrgluserglyleuaspgluilelyslysilelysleuasn
275280285
metasnlysglyarg
290
<210>82
<211>47
<212>dna
<213>智人(homosapiens)
<400>82
agctagtctagtgcaagctaacagttgcttttatcacaggctccagg47
<210>83
<211>47
<212>dna
<213>智人(homosapiens)
<400>83
tcgatcagatcacgttcgattctcaacgaaaatagtgtccgaggtcc47
<210>84
<211>21
<212>dna
<213>智人(homosapiens)
<400>84
caaaccctcctggagcctgtg21
<210>85
<211>28
<212>dna
<213>智人(homosapiens)
<400>85
caaaccctcctggagcctgtggggataa28
<210>86
<211>21
<212>dna
<213>智人(homosapiens)
<400>86
caaaccctcctggggcctgtg21
<210>87
<211>21
<212>dna
<213>智人(homosapiens)
<400>87
caaaccctcctggagcccgtg21
<210>88
<211>18
<212>dna
<213>智人(homosapiens)
<400>88
caaaccctcctggagccc18
<210>89
<211>12
<212>dna
<213>智人(homosapiens)
<400>89
caaaccctcctg12
<210>90
<211>20
<212>dna
<213>智人(homosapiens)
<400>90
caaaccctcctggagccggt20
<210>91
<211>20
<212>dna
<213>智人(homosapiens)
<400>91
caaaccctcctggagcctgg20
<210>92
<211>19
<212>dna
<213>智人(homosapiens)
<400>92
caaaccctcctggagcctg19
<210>93
<211>22
<212>dna
<213>智人(homosapiens)
<400>93
caaaccctcctggagcctgtgg22
<210>94
<211>25
<212>dna
<213>智人(homosapiens)
<400>94
aagcaactgttagcttgcactagac25
<210>95
<211>25
<212>dna
<213>智人(homosapiens)
<400>95
aagcgactgttagcttgcactagac25
<210>96
<211>31
<212>dna
<213>智人(homosapiens)
<400>96
ataaggaagcaactgttagcttgcactagac31
<210>97
<211>26
<212>dna
<213>智人(homosapiens)
<400>97
aaagcaactgttagcttgcactagac26
<210>98
<211>16
<212>dna
<213>智人(homosapiens)
<400>98
ttagcttgcactagac16
<210>99
<211>15
<212>dna
<213>智人(homosapiens)
<400>99
tagcttgcactagac15
<210>100
<211>18
<212>dna
<213>智人(homosapiens)
<400>100
tgttagcttgcactagac18
<210>101
<211>22
<212>dna
<213>智人(homosapiens)
<400>101
caactgttagcttgcactagac22
<210>102
<211>14
<212>dna
<213>智人(homosapiens)
<400>102
agcttgcactagac14
<210>103
<211>20
<212>dna
<213>智人(homosapiens)
<400>103
actgttagcttgcactagac20
<210>104
<211>25
<212>dna
<213>智人(homosapiens)
<400>104
aagcaactgttagcttgcactggac25
- 还没有人留言评论。精彩留言会获得点赞!
- 改进的广谱核酸内切酶及其工业生产方法
- 一种依赖内切酶结合侧向层析的核酸等温检测技术和试剂的制作方法
- 松墨天牛中性肽内切酶2基因orf全长序列及其克隆方法
- 用于高效且特异性靶向包含高度重复基序的dna序列的稀有切割核酸内切酶的设计的制作方法
- 一种Cas9核酸内切酶分泌型表达载体及其构建方法和应用
- CRISPR-Cas9引导序列及其引物、转基因表达载体及其构建方法
- 一种稳定表达Cas9蛋白的猪肠道上皮细胞系的制作方法
- 一种用于猪基因编辑的高效特异性sgRNA识别位点引导序列及其筛选方法
- CRISPR-Cas9特异性敲除人PCSK9基因的方法以及用于特异性靶向PCSK9基因的sgRNA的制作方法
- 用于拟无枝菌酸菌的穿梭载体和表达载体的制作方法