一种带有黄色或红色荧光蛋白标签的非洲爪蟾卵母细胞表达载体及应用的制作方法
本发明涉及分子生物学及基因工程领域。具体来说,涉及带有黄色或红色荧光标签的非洲爪蟾卵母细胞表达载体及应用。
背景技术
非洲爪蟾卵母细胞是一种功能强大、适用范围广的异源性表达体系,被广泛应用于外源性钙离子通道、氯离子通道、钾离子通道、钠离子通道、γ-氨基丁酸受体、谷氨酸受体、乙酰胆碱受体等异源蛋白的表达及其生理学和药理学特性的研究。它在探讨化合物的作用机制、评价和筛选靶标化合物的药效和安全性等方面发挥着重要作用,是生物学、医药学、神经学、农药学等领域研究中不可或缺的异源型表达系统。
黄色和红色荧光蛋白是指能在绿色光(500-565nm)激发下会发出黄色和红色荧光的蛋白质,已经作为新型报告基因在生物学的许多研究领域中得到应用。黄色荧光蛋白的突变体—增强型黄色荧光蛋白(enhancedyellowfluorescentprotein,eyfp)和单体红色荧光蛋白(monomericredfluorescentprotein,mrfp)因其发光稳定、亮度和区分度高,因此被广泛应用于基因表达、细胞分化及蛋白质的体内定位和转运等研究。
pgh19质粒是一种含有多种限制性内切酶位点且应用广泛的蛋白质表达载体,因其启动子和终止子附近分别含有非洲爪蟾β-球珠蛋白的5’utr和3’utr碱基序列,能够在非洲爪蟾卵母细胞中成功地表达异源性蛋白质,所以被广泛应用于非洲爪蟾卵母细胞的异源表达研究。
利用pgh19质粒在非洲爪蟾卵母细胞中表达的蛋白质可通过电生理实验检测其生理学和药理学特性,但是不能在显微镜下观察目标蛋白表达与否及时空表达动态,无法进行有效的亚细胞定位和目的蛋白的纯化及鉴定,而针对目的蛋白特异性抗体的获取也需要消耗大量的时间和经济成本。
技术实现要素:
本发明的目的在于提供带有黄色或红色荧光蛋白标签的非洲爪蟾卵母细胞表达载体,该载体既可以在非洲爪蟾卵母细胞中成功表达有活性的目的蛋白,又带有可直接镜检的黄色或红色荧光,能够对蛋白的表达进行实时监测和亚细胞定位,并可利用通用型抗体anti-eyfp和mrfp对表达蛋白进行纯化和检测不同亚基的结合模式,从而免去制备特异性抗体的时间和经济成本。
为达到上述目的,本发明采用如下技术方案:
带有黄色或红色荧光蛋白标签的非洲爪蟾卵母细胞表达载体,其特征在于该载体为带有修饰序列的eyfp或带有修饰序列的mrfp基因连接到pgh19质粒上获得;带有修饰序列的eyfp或带有修饰序列的mrfp基因具体为eyfp基因或mrfp基因的5’端加入限制性内切酶a的识别碱基序列和一段人工设计的酶切位点碱基序列,及3’端自身携带的限制性内切酶b的识别碱基序列。
所述人工设计的酶切位点碱基序列如seqidno.3所示,通过添加该序列的酶切位点,可以解决pgh19质粒插入eyfp和mrfp基因后无法插入目的基因的问题,且可以确保成功插入目的基因后,不会破坏eyfp和mrfp基因活性和pgh19质粒原有功能,即在保障pgh19质粒原有功能的前提下,又可使目的蛋白带有黄色或红色荧光标签。
本发明所述限制性内切酶a可以为ecori限制性内切酶、xmai限制性内切酶、smai限制性内切酶、bamhi限制性内切酶、bstbi限制性内切酶、xhoi限制性内切酶或ecorv限制性内切酶。
本发明所述限制性内切酶a的碱基识别序列可以为ecori限制性内切酶碱基识别序列(gaattc,如seqidno.4所示)、xmai限制性内切酶碱基识别序列(cccggg,如seqidno.5所示)、smai限制性内切酶碱基识别序列(cccggg,如seqidno.6所示)、bamhi限制性内切酶碱基识别序列(ggatcc,如seqidno.7所示)、bstbi限制性内切酶碱基识别序列(ttcgaa,如seqidno.8所示)、xhoi限制性内切酶碱基识别序列(ctcgag,如seqidno.9所示)或ecorv限制性内切酶碱基识别序列(gatatc,如seqidno.10所示)。
本发明所述限制性内切酶b可以为xbai限制性内切酶、bsrgi限制性内切酶、pspomi限制性内切酶、apai限制性内切酶、bmgbi限制性内切酶或bstpi限制性内切酶。
本发明所述限制性内切酶b的碱基识别序列可以为xbai限制性内切酶碱基识别序列(tctaga,如seqidno.11所示)、bsrgi限制性内切酶碱基识别序列(tgtaca,如seqidno.12所示)、pspomi限制性内切酶碱基识别序列(gggccc,如seqidno.13所示)、apai限制性内切酶碱基识别序列(gggccc,如seqidno.14所示)、bmgbi限制性内切酶碱基识别序列(cacgtc,如seqidno.15所示)或bstpi限制性内切酶碱基识别序列(ggtnacc,如seqidno.16所示)。
具体的,本发明提供带有黄色荧光标签的非洲爪蟾卵母细胞表达载体pgh19-eyfp,该载体的碱基序列如seqidno.1所示。
具体的,本发明还提供带有红色荧光标签的非洲爪蟾卵母细胞表达载体pgh19-mrfp,该载体的碱基序列如seqidno.2所示。
本发明还提供本发明所述载体在生物检测中的应用,特别是在实时观察蛋白表达动态或蛋白纯化或检测中的应用。
本发明还提供本发明所述载体中插入目的基因形成的基因表达载体。
本发明还提供本发明所述基因表达载体在生物检测中的应用,特别是在实时观察蛋白表达动态或蛋白纯化或检测中的应用。
本发明还提供一种seqidno.1或seqidno.2所示序列的载体pgh19-eyfp和pgh19-mrfp的构建方法为:
1)从ncbi网站下载eyfp和mrfp碱基序列,并设计引物,对正向引物和反向引物进行修饰,分别在它们的5’端加入内切酶ecori和xbai的酶切位点和保护性碱基,并在正向引物的5’端ecori酶切位点后加入一段人工设计的酶切位点碱基序列。引物序列为:
forwardprimer(5’-3’):
aaggaattcgaagcttctcgagatatcatggtgagcaagggcgaggagc,
reverseprimer(5’-3’):ttgtctagattacttgtacagctcgtccatgccg.
其中,小写字母代表保护性碱基,斜体字母代表ecori和xbai限制性内切酶识别碱基序列,下划线字母代表人工设计插入的酶切位点碱基序列。
2)通过pcr扩增和dna回收操作,得到带有修饰序列的eyfp或mrfp基因产物;
3)对pgh19质粒进行限制性内切酶ecori和xbai的双酶切。将酶切后的质粒及带有修饰序列的eyfp或mrfp基因通过t4连接酶进行连接,即得到带有黄色或红色荧光标签的非洲爪蟾卵母细胞表达载体—pgh19-eyfp或pgh19-mrfp。
本发明中,步骤3)中的ecori限制性内切酶可由xmai限制性内切酶、smai限制性内切酶、bamhi限制性内切酶、bstbi限制性内切酶、xhoi限制性内切酶、ecorv限制性内切酶或含有上述酶切位点的任意限制性内切酶替换。
本发明步骤3)中的xbai限制性内切酶可由bsrgi限制性内切酶、pspomi限制性内切酶、apai限制性内切酶、bmgbi限制性内切酶或bstpi限制性内切酶或含有上述酶切位点的任意限制性内切酶替换。
本发明通过将在非洲爪蟾卵母细胞表达体系中广泛应用的pgh19载体进行改造,将pgh19载体与eyfp或mrfp基因连接,并在其5’端插入一段人工设计的酶切位点碱基序列以保证目的基因的正常插入和表达。当在该载体插入目的基因时,只需对pgh19-eyfp或pgh19-mrfp载体进行酶切,然后与带有相同酶切位点的目的基因进行同源重组或t4连接,即可成功构建既能表达目的基因蛋白,又带有黄色或红色荧光标签,且可应用于非洲爪蟾卵母细胞表达体系的表达载体。这种载体既可以进行正常的电生理实验检测,又可以通过荧光显微镜检测目的蛋白在非洲爪蟾卵母细胞的时空表达动态,且便于进行亚细胞定位及目的蛋白的提取和纯化,为生物学实验提供了一种直观、便捷的工具。
本发明通过将pgh19质粒与eyfp或mrfp基因进行融合改造,在不影响pgh19质粒原有功能的前提下,又可使目的蛋白带有黄色或红色荧光标签;可在不影响靶蛋白活性和电生理检测的同时,又可用于表达蛋白状态的检测。通过本载体表达的靶标蛋白,可以实时观察蛋白表达动态;后期检测时,无需特异性抗体,只需使用通用型抗体即可对靶标蛋白进行纯化并检测靶标蛋白的表达情况,从而大大地简化了实验流程,节省了实验时间和成本。
附图说明
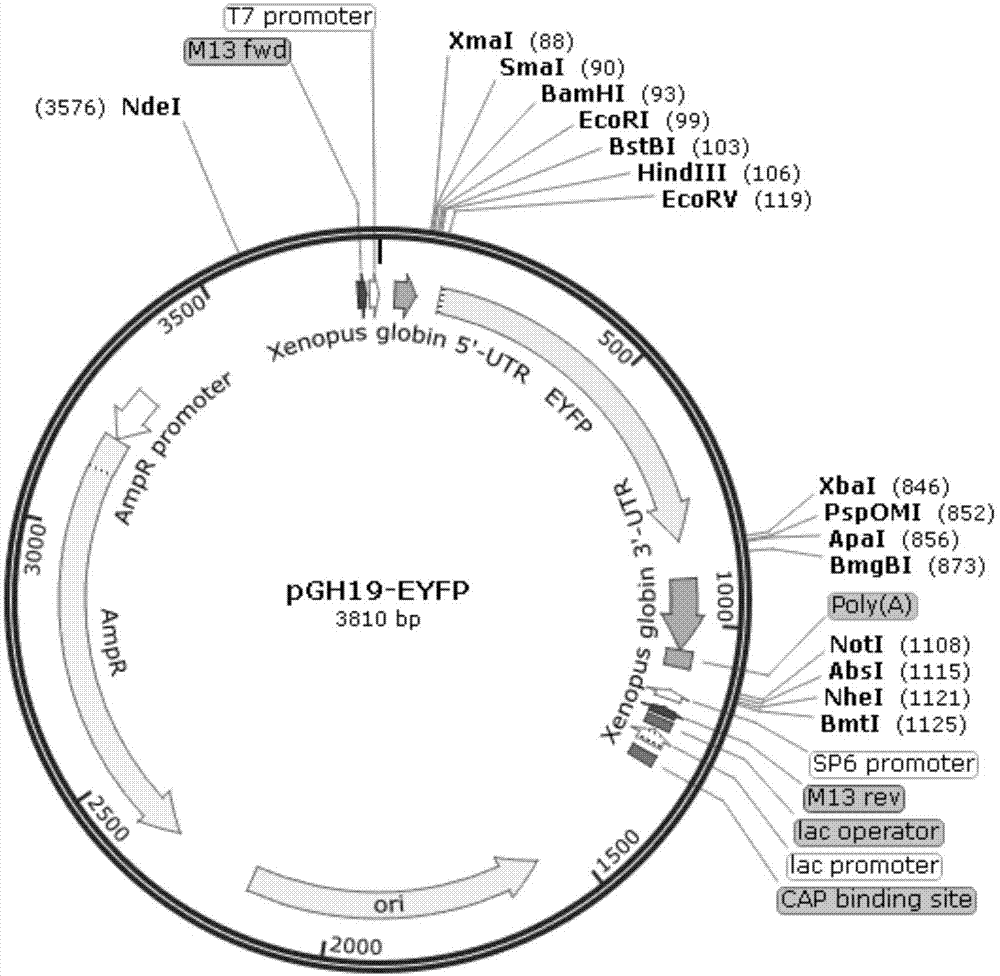
图1本发明提供的带有荧光标签的非洲爪蟾卵母细胞表达载体pgh19-eyfp的质粒图谱;
图2本发明提供的带有荧光标签的非洲爪蟾卵母细胞表达载体pgh19-mrfp的质粒图谱;
图3egfp和mrfp基因的pcr扩增电泳图;
图4pgh19载体的酶切电泳图;
图5amrdl基因碱基序列的pcr扩增电泳图;
图6pgh19-eyfp和pgh19-mrfp载体的酶切电泳图;
图7本发明提供的带有荧光标签的能够表达amrdl基因的非洲爪蟾卵母细胞表达载体pgh19-eyfp-amrdl的质粒图谱;
图8本发明提供的带有荧光标签的能够表达amrdl基因的非洲爪蟾卵母细胞表达载体pgh19-mrfp-amrdl的质粒图谱;
图9pgh19-amrdl及pgh19-eyfp-amrdl和pgh19-mrfp-amrdl表达载体的电生理检测;
图10pgh19-eyfp-amrdl基因在非洲爪蟾卵母细胞上表达的荧光检测;
图11pgh19-mrfp-amrdl基因在非洲爪蟾卵母细胞上表达的荧光检测。
具体实施方式
为便于本领域技术人员对本发明的技术方案和有益效果进行理解,特结合附图对具体实施方式进行如下描述。应该指出,以下具体说明都是例示性的,旨在对本发明提供进一步的说明。除非另有说明,本发明使用的所有科学和专业术语具有与本发明所属技术领域人员通常理解的相同含义。
实施例1pgh19-eyfp和pgh19-mrfp重组载体的构建
1.实验材料
2.实验步骤
2.1eyfp和mrfp序列的pcr扩增及回收
1)设计eyfp和mrfp序列的扩增引物,由南京金斯瑞生物科技有限公司合成,纯化方式为rpc。
2)采用takara公司的
3)根据
4)将pcr扩增产物进行琼脂糖凝胶电泳,在紫外凝胶成像系统中检测目的条带(附图3,其中泳道1为扩增的eyfp基因片段;泳道2为扩增的mrfp基因片段)。将目的条带切胶,并进行目的片段的回收。
2.2pgh19质粒载体的酶切及回收
1)采用限制性内切酶ecori和xbai将pgh19质粒进行双酶切。分别在200μl的pcr管中加入以下组分,配置成20μl反应体系:
将以上反应体系混匀,然后在pcr仪中37℃保温2h,80℃加热5min,即完成pgh19质粒的线性化;
2)将线性化的pgh19质粒进行琼脂糖凝胶电泳检测,在紫外凝胶成像系统中检测酶切后的目的条带(附图4,其中泳道1为未消化的pgh19质粒,作为阴性对照;泳道2为消化的pgh19质粒)。将目的条带切胶并进行目的片段的回收。
2.3重组pg19-eyfp和mrfp载体的连接转化
1)将回收得到的eyfp和mrfp产物与pgh19载体产物通过thermo公司的t4dnaligase试剂盒进行连接。在200μl的pcr管中分别加入以下组分,配置成20μl反应体系:
将以上反应体系混匀,然后在pcr仪中22℃保温30min,即完成重组pgh19-eyfp和pgh19-mrfp载体的连接。
2)将重组pgh19-eyfp和pgh19-mrfp载体转化到trans-t1大肠杆菌感受态细胞中,过夜培养并挑取单克隆菌株测序。
3.实验结果
测序结果显示,重组pgh19-eyfp和pgh19-mrfp载体分别含有完整的eyfp和mrfp基因序列和人工设计的酶切位点碱基序列,载体构建成功,带有荧光标签的非洲爪蟾卵母细胞表达载体pgh19-eyfp和pgh19-mrfp的质粒图谱分别如图1和2。带有黄色荧光标签的非洲爪蟾卵母细胞表达载体pgh19-eyfp的碱基序列如seqidno.1所示;带有红色荧光标签的非洲爪蟾卵母细胞表达载体pgh19-mrfp的碱基序列如seqidno.2所示。
实施例2蜜蜂(apismellifera)rdl基因表达载体pgh19-eyfp-amrdl和pgh19-mrfp-amrdl的构建
1.实验材料
2.实验步骤
2.1amrdl基因编码区碱基序列的克隆
1)从ncbi网站下载蜜蜂rdl基因(amrdl)的核苷酸序列,设计扩增其完整编码区序列的引物,由南京金斯瑞生物科技有限公司合成,纯化方式为rpc。
2)采用takara公司的
3)根据
4)将pcr扩增产物进行琼脂糖凝胶电泳,在紫外凝胶成像系统中检测目的条带(附图5)。将目的条带切胶并进行目的片段的回收。
2.2pgh19-eyfp和pgh19-mrfp载体的酶切及回收
1)采用thermo公司的hindiii限制性内切酶试剂盒进行pgh19-eyfp或pgh19-mrfp质粒载体的酶切。在200μl的pcr管分别中加入以下组分,配置成20μl反应体系:
将以上反应体系混匀,然后在pcr仪中37℃保温2h,80℃加热5min,即完成pgh19-eyfp或pgh19-mrfp载体的线性化;
2)将线性化的pgh19-eyfp或pgh19-mrfp载体进行琼脂糖凝胶电泳检测,在紫外凝胶成像系统中检测酶切后的目的条带(附图6,其中泳道1为酶切线性化的pgh19-eyfp质粒;泳道2为未酶切的pgh19-eyfp质粒,作为阴性对照;泳道3为酶切线性化的pgh19mrfp质粒;泳道4为未酶切的pgh19-mrfp质粒,作为阴性对照)。将目的条带切胶并进行目的片段的回收。
2.3重组pgh19-eyfp-amrdl或pgh19-mrfp-amrdl载体的连接转化
1)将回收得到的pgh19-eyfp或pgh19-mrfp载体产物和amrdl胶回收产物通过clonexpressiionestepcloningkit试剂盒进行连接。在200μl的pcr管中分别加入以下组分,配置成10μl反应体系:
将以上反应体系混匀,然后在pcr仪中37℃保温30min,然后置于4℃冰箱保存,即完成重组pgh19-eyfp-amrdl和pgh19-mrfp-amrdl载体的连接。
2)将重组pgh19-eyfp-amrdl和pgh19-mrfp-amrdl载体转化到trans-t1大肠杆菌感受态细胞中,过夜培养并挑取单克隆菌株送往南京金斯瑞生物科技有限公司测序。
3.实验结果
测序结果显示,重组pgh19-eyfp-amrdl或pgh19-mrfp-amrdl载体含有完整的eyfp和mrfp基因序列和amrdl基因序列,载体构建成功(附图7和8)。
实施例3amrdl带有egfp或mrfp基因和不带荧光标签基因在非洲爪蟾卵母细胞中的表达及检测
1.实验材料
noti限制性内切酶和mmessagemmachinetmt7试剂盒购自thermo公司,酚氯仿(苯酚:氯仿:异戊醇=25:24:1,ph>7.8)购自北京索莱宝科技有限公司,非洲爪蟾由中国科学院上海生命科学研究所提供。
2.实验步骤
2.1pgh19-amrdl、pgh19-eyfp-amrdl和pgh19-mrfp-amrdl载体的线性化
1)采用thermo公司的noti限制性内切酶试剂盒进行pgh19-amrdl、pgh19-eyfp-amrdl和pgh19-mrfp-amrdl载体的酶切。分别在200μl的pcr管中加入以下组分,配置成150μl反应体系:
将以上反应体系混匀,然后在pcr仪中37℃酶切7.5h,65℃加热15min使酶失活,然后置于4℃冰箱保存,即完成pgh19-amrdl、pgh19-eyfp-amrdl和pgh19-mrfp-amrdl载体的线性化。
2.2线性化产物的纯化
1)向线性化产物中加ddh2o至400μl,再加入200μl酚氯仿,13000g/min离心10min;
2)吸取上清液400μl,再加入200μl酚氯仿,13000g/min离心10min;
3)吸取上清液300μl,加入800μl无水乙醇和30μl醋酸钠溶液,静置7-8h;
4)13000g/min离心15min,弃上清,加入1ml预冷的75%乙醇,洗涤沉淀,再用13000g/min离心10min,弃上清;
5)将沉淀在超净工作台内吹干,加入10μlddh2o溶解,测定浓度,保存备用。
2.3体外合成crna
采用thermo公司的mmessagemmachinetmt7试剂盒体外合成crna,并置于-80℃超低温冰箱保存备用。
2.4非洲爪蟾卵母细胞的获取及crna的注射
解剖成熟的雌性非洲爪蟾并从腹部取出适量卵母细胞,然后将取出的卵母细胞用typeia型胶原酶处理去掉卵母细胞外围的膜,洗涤备用。将crna用显微注射器注入新准备好的非洲爪蟾卵母细胞中,每颗卵母细胞注入23nl体积的crna。
2.5非洲爪蟾卵母细胞的电生理实验和荧光检测
将注射过crna的卵母细胞放在16℃恒温箱中培养48h后,开始进行电生理实验和荧光检测。
3.实验结果
通过双电极电压钳实验检测融合了荧光标签的amrdl基因与未融合荧光标签的amrdl基因,结果显示荧光标签的存在对检测结果无影响(附图9)。
通过荧光显微镜实时监测amrdl蛋白在非洲爪蟾卵母细胞的时空表达动态,且可以对amrdl蛋白进行初步的亚细胞定位(附图10和图11),这将有利于下一步amrdl蛋白的提取和纯化及更深层次的功能研究。
对本领域的技术人员来说,可根据以上描述的技术方案以及构思,做出其它各种相应的改变以及形变,而所有的这些改变以及形变都属于本发明权利要求的保护范围之内。
序列表
<110>南京农业大学
<120>一种带有黄色或红色荧光蛋白标签的非洲爪蟾卵母细胞表达载体及应用
<160>16
<170>siposequencelisting1.0
<210>1
<211>3810
<212>dna
<213>人工序列(artificialsequence)
<400>1
gggcgaattaattcgagctcggtacccagcttgcttgttctttttgcagaagctcagaat60
aaacgctcaactttggcagatcaattccccggggatccgaattcgaagcttctcgagata120
tcatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctgg180
acggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacct240
acggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggccca300
ccctcgtgaccaccttcggctacggcctacagtgcttcgcccgctaccccgaccacatga360
agcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatct420
tcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccc480
tggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggc540
acaagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaaga600
acggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcg660
ccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctacccgacaacc720
actacctgagctaccagtccgccctgagcaaaggccccaacgagaagcgcgatcacatgg780
tcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacagat840
cttaatctagagggcccgatcgccgagcatcacgtcgcgcacggcggatgtctgagccag900
gagcttgatctggttaccactaaaccagcctcaagaacacccgaatggagtctctaagct960
acataataccaacttacactttacaaaatgttgtcccccaaaatgtagccattcgtatct1020
gctcctaataaaaagaaagtttcttcacattctaaaaaaaaaaaaaaaaaaaaaaaaaaa1080
aaaaaaccccccccccccccctgcaggcggccgcctcgaggctagcttgagtattctata1140
gtgtcacctaaatagcttggcgtaatcatggtcatagctgtttcctgtgtgaaattgtta1200
tccgctcacaattccacacaacatacgagccggaagcataaagtgtaaagcctggggtgc1260
ctaatgagtgagctaactcacattaattgcgttgcgctcactgcccgctttccagtcggg1320
aaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcg1380
tattgggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcg1440
gcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataa1500
cgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgc1560
gttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcgacgctc1620
aagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctggaag1680
ctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgcctttct1740
cccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctcagttcggtgta1800
ggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgc1860
cttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggc1920
agcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttctt1980
gaagtggtggcctaactacggctacactagaagaacagtatttggtatctgcgctctgct2040
gaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaaccaccgc2100
tggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaaggatctca2160
agaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaactcacgtta2220
agggattttggtcatgagattatcaaaaaggatcttcacctagatccttttaaattaaaa2280
atgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagttaccaatg2340
cttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctg2400
actccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgc2460
aatgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccagc2520
cggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatccagtctattaa2580
ttgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaacgttgttgc2640
cattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattcagctccgg2700
ttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctc2760
cttcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactcatggttat2820
ggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttctgtgactgg2880
tgagtactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttgctcttgccc2940
ggcgtcaatacgggataataccgcgccacatagcagaactttaaaagtgctcatcattgg3000
aaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatccagttcgat3060
gtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagcgtttctgg3120
gtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatg3180
ttgaatactcatactcttcctttttcaatattattgaagcatttatcagggttattgtct3240
catgagcggatacatatttgaatgtatttagaaaaataaacaaataggggttccgcgcac3300
atttccccgaaaagtgccacctgacgtctaagaaaccattattatcatgacattaaccta3360
taaaaataggcgtatcacgaggccctttcgtctcgcgcgtttcggtgatgacggtgaaaa3420
cctctgacacatgcagctcccggagacggtcacagcttgtctgtaagcggatgccgggag3480
cagacaagcccgtcagggcgcgtcagcgggtgttggcgggtgtcggggctggcttaacta3540
tgcggcatcagagcagattgtactgagagtgcaccatatgcggtgtgaaataccgcacag3600
atgcgtaaggagaaaataccgcatcaggcgccattcgccattcaggctgcgcaactgttg3660
ggaagggcgatcggtgcgggcctcttcgctattacgccagctggcgaaagggggatgtgc3720
tgcaaggcgattaagttgggtaacgccagggttttcccagtcacgacgttgtaaaacgac3780
ggccagtgaattgtaatacgactcactata3810
<210>2
<211>3765
<212>dna
<213>人工序列(artificialsequence)
<400>2
gggcgaattaattcgagctcggtacccagcttgcttgttctttttgcagaagctcagaat60
aaacgctcaactttggcagatcaattccccggggatccgaattcgaagcttctcgagata120
tcatggcctcctccgaggacgtcatcaaggagttcatgcgcttcaaggtgcgcatggagg180
gctccgtgaacggccacgagttcgagatcgagggcgagggcgagggccgcccctacgagg240
gcacccagaccgccaagctgaaggtgaccaagggcggccccctgcccttcgcctgggaca300
tcctgtcccctcagttccagtacggctccaaggcctacgtgaagcaccccgccgacatcc360
ccgactacttgaagctgtccttccccgagggcttcaagtgggagcgcgtgatgaacttcg420
aggacggcggcgtggtgaccgtgacccaggactcctccctgcaggacggcgagttcatct480
acaaggtgaagctgcgcggcaccaacttcccctccgacggccccgtaatgcagaagaaga540
ccatgggctgggaggcctccaccgagcggatgtaccccgaggacggcgccctgaagggcg600
agatcaagatgaggctgaagctgaaggacggcggccactacgacgccgaggtcaagacca660
cctacatggccaagaagcccgtgcagctgcccggcgcctacaagaccgacatcaagctgg720
acatcacctcccacaacgaggactacaccatcgtggaacagtacgagcgcgccgagggcc780
gccactccaccggcgcctaatctagagggcccgatcgccgagcatcacgtcgcgcacggc840
ggatgtctgagccaggagcttgatctggttaccactaaaccagcctcaagaacacccgaa900
tggagtctctaagctacataataccaacttacactttacaaaatgttgtcccccaaaatg960
tagccattcgtatctgctcctaataaaaagaaagtttcttcacattctaaaaaaaaaaaa1020
aaaaaaaaaaaaaaaaaaaaaccccccccccccccctgcaggcggccgcctcgaggctag1080
cttgagtattctatagtgtcacctaaatagcttggcgtaatcatggtcatagctgtttcc1140
tgtgtgaaattgttatccgctcacaattccacacaacatacgagccggaagcataaagtg1200
taaagcctggggtgcctaatgagtgagctaactcacattaattgcgttgcgctcactgcc1260
cgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggg1320
gagaggcggtttgcgtattgggcgctcttccgcttcctcgctcactgactcgctgcgctc1380
ggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccac1440
agaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaa1500
ccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatca1560
caaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggc1620
gtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggata1680
cctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggta1740
tctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttca1800
gcccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacga1860
cttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcgg1920
tgctacagagttcttgaagtggtggcctaactacggctacactagaagaacagtatttgg1980
tatctgcgctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccgg2040
caaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcag2100
aaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaa2160
cgaaaactcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagat2220
ccttttaaattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggtc2280
tgacagttaccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttcgttc2340
atccatagttgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatc2400
tggccccagtgctgcaatgataccgcgagacccacgctcaccggctccagatttatcagc2460
aataaaccagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgcctc2520
catccagtctattaattgttgccgggaagctagagtaagtagttcgccagttaatagttt2580
gcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggc2640
ttcattcagctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaa2700
aaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcagtgtt2760
atcactcatggttatggcagcactgcataattctcttactgtcatgccatccgtaagatg2820
cttttctgtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgacc2880
gagttgctcttgcccggcgtcaatacgggataataccgcgccacatagcagaactttaaa2940
agtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgtt3000
gagatccagttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttacttt3060
caccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataag3120
ggcgacacggaaatgttgaatactcatactcttcctttttcaatattattgaagcattta3180
tcagggttattgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaat3240
aggggttccgcgcacatttccccgaaaagtgccacctgacgtctaagaaaccattattat3300
catgacattaacctataaaaataggcgtatcacgaggccctttcgtctcgcgcgtttcgg3360
tgatgacggtgaaaacctctgacacatgcagctcccggagacggtcacagcttgtctgta3420
agcggatgccgggagcagacaagcccgtcagggcgcgtcagcgggtgttggcgggtgtcg3480
gggctggcttaactatgcggcatcagagcagattgtactgagagtgcaccatatgcggtg3540
tgaaataccgcacagatgcgtaaggagaaaataccgcatcaggcgccattcgccattcag3600
gctgcgcaactgttgggaagggcgatcggtgcgggcctcttcgctattacgccagctggc3660
gaaagggggatgtgctgcaaggcgattaagttgggtaacgccagggttttcccagtcacg3720
acgttgtaaaacgacggccagtgaattgtaatacgactcactata3765
<210>3
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>3
gaagcttctcgagatatc18
<210>4
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>4
gaattc6
<210>5
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>5
cccggg6
<210>6
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>6
cccggg6
<210>7
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>7
ggatcc6
<210>8
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>8
ttcgaa6
<210>9
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>9
ctcgag6
<210>10
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>10
gatatc6
<210>11
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>11
tctaga6
<210>12
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>12
tgtaca6
<210>13
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>13
gggccc6
<210>14
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>14
gggccc6
<210>15
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>15
gacgtc6
<210>16
<211>7
<212>dna
<213>人工序列(artificialsequence)
<400>16
ggtnacc7
- 还没有人留言评论。精彩留言会获得点赞!
- 用于粘接剂活化的使用光的加热装置及其在预成型装置中的集成的制造方法
- 全程C‑反应蛋白荧光免疫层析用活化荧光乳胶微球的制造方法与工艺
- 一种不依赖激发光强的上转换荧光强度比测温方法与制造工艺
- 一种PEP‑1肽介导绿色荧光蛋白转导水牛胚胎的方法与制造工艺
- 一种利用级联放大效应高敏监测水体PAHs的转荧光蛋白基因斑马鱼的研制方法与制造工艺
- 一株表达绿色荧光蛋白(EGFP)基因的人肠道病毒EV71型重组病毒及其应用的制造方法与工艺
- 一种HPV16型E7蛋白的POCT荧光定量检测试剂盒及其应用的制造方法与工艺
- 一种基于双荧光发色团的力和光双重响应型自组装体及其制备方法与制造工艺
- 基于机器视觉的禽类受精蛋检测方法与制造工艺
- 一种荧光蛋白质染色剂及其制备方法和应用与制造工艺