基因表达的调控的制作方法
本发明涉及gdp-l-半乳糖磷酸化酶(ggp)表达的控制和操纵以及抗坏血酸的制备。本发明也涉及控制所述表达和制备的序列元件,及其使用。
背景技术:
抗坏血酸是植物中最丰富的可溶性抗氧化剂,并且对于人和一些其它动物也是必需的营养素。抗坏血酸显著地促进人类膳食中“游离自由基清除剂”或“抗氧化代谢产物”的全面摄入。令人信服的证据现在表明,这种代谢产物单独或者联合地作为抗癌症形成剂和预防冠心病而有利于健康和身体。
人类摄入的几乎全部膳食抗坏血酸都来源于植物产品。然而,植物组织的抗坏血酸含量显著不同。虽然在草本和木本植物中,叶子的抗坏血酸含量通常很高并且相对一致,但是在非绿色的可食用植物组织中,发现抗坏血酸含量有巨大的无法解释的差异。例如,在果实中,其水平在mirciariadubia的卡姆果(camucamu)中高达30mggfw-1asa,而在mespilusgermanica的欧楂果(medlar)中低于3μggfw-1asa(rodriguez等,1992,jchromatogrsci,30:433-437)。据报道猕猴桃中抗坏血酸值的变化很大(ferguson,a.r.,botanicalnominclature:actinidiachinensis,actinidiadeliciosa,andactinidiasetosa.kiwifruit:scienceandmanagement,ed.i.j.warrington和g.c.weston.1990,palmerstonnorth;newzealand:newzealandsocietyforhorticulturalscience.576.beever,d.j.和g.hopkirk,fruitdevelopmentandfruitphysiology.kiwifruit:scienceandmanagement,ed.i.j.warringtonandg.c.weston.1990,palmerstonnorth;newzealand:newzealandsocietyforhorticulturalscience.576.)。不同的藤本植物水果的抗坏血酸含量,对于美味猕猴桃(a.deliciosa)范围是30-400mg/100g(ferguson,a.r.,1991actahort.290:p.603-656,spano,d.等,1997actahort.,.444:p.501-506.),而对于栽培品种“hayward”报道的范围在80-120mg/100g(beever,d.j.和g.hopkirk,fruitdevelopmentandfruitphysiology.kiwifruit:scienceandmanagement,ed.i.j.warrington和g.c.weston.1990,palmerstonnorth;newzealand:newzealandsocietyforhorticulturalscience.576.)。据报道,软枣猕猴桃(a.arguta)、中华猕猴桃(a.chinensis)(muggleston,s.等,orchardist,1998.71(8):p.38-40,chen,q.和q.chen,cropgeneticresources,1998(2):p.3,coggiatti,s.,1971italagr,oct,.108(10):p.935-941)、金花猕猴桃(a.chrysantha)和葛枣猕猴桃(a.polygama)的果实中含有较高浓度的抗坏血酸,在毛花猕猴桃(a.eriantha)和阔叶猕猴桃(a.latifolia)中水平非常高(>1%鲜重)(ferguson1991actahort.290:p.603-656.和a.kolomikta(kola,j.andj.pavelka,1988nahrung,.32(5):p.513-515)。
已经提出植物中生物合成抗坏血酸的三种途径,一种是通过l-半乳糖(l-gal)(wheeler等,1998,nature393,365-369),另一种是由肌醇(myo-inositol)合成(loewus和kelly,1961,arch.biochem.biophys.95,483-493;lorence等,(2004)plantphysiol.134,1200-1205),以及第三种途径是通过半乳糖醛酸(agius等,2003,natbiotechnol21,177-81)。l-gal途径通过l-半乳糖生产半乳糖酸-1,4-内酯(galactono-1,4-lactone),并由此形成抗坏血酸(wheeler等,1998,nature393,365-369)。
对于l-半乳糖途径,已经鉴定并且至少部分表征出来编码这些酶的所有基因和它们相关的酶活性。
被表征的基因和酶活性包括gdp-d-甘露糖焦磷酸化酶(conklin,1998,trendsplantsci3:329-330.;conklin等,1999procnatlacadsciusa96:4198-4203.;keller等,1999plantj19:131-141.)、gdp-d-甘露糖3',5'-茶香异构酶(woluckaetal.,2001,analbiochem294:161-168;woluckaandvanmontagu,2003,j.biol.chem.278:47483-47490;watanabeetal.,2006phytochemistry67:338-346.)、l-半乳糖-1-p磷酸酶(laing等,2004,proceedingsofthenationalacademyofsciences(usa)101:16976-16981.;conklin等,2006,j.biol.chem.281:15662-15670.)、l-半乳糖脱氢酶(wheeler等,1998,nature393:365-369.;gatzek等,2002,plantj.30,541(2002;laing等,2004proceedingsofthenationalacademyofsciences(usa)101:16976-16981)、l-半乳糖酸-1,4-内酯脱氢酶(imai等,1998plantandcellphysiology39:1350-1358.;bartoli等,2005,plant,cellandenvironment28:1073-1081.)和gdp-l半乳糖磷酸化酶(ggp)(laing等,2007,proceedingsofthenationalacademyofsciences(usa)104:9534-9)。申请人之前已经示出了gdp-l半乳糖磷酸化酶在决定抗坏血酸的产生中是重要的(bulleys等,2012plantbiotechnolj2012,10:390-397)。
抗坏血酸的浓度是根据需求调控的。当在高光强度下对高抗坏血酸的需求最高时,叶子中抗坏血酸浓度升高(bartoli等,j.exp.bot.57,1621(2006);gatzek,等,plantj.30,541(2002))。然而,对植物中抗坏血酸的生物合成的调控机制知之甚少(bulley等,plantbiotechnolj10,390(2012);bulley等,j.exp.bot.60,765(2009))。了解抗坏血酸的生物合成如何调控可以为在植物中操纵生物合成提供工具。了解基因表达的调控、以及控制所述表达的因子/元件也能为基因操纵提供有价值的工具。
本发明的一个目的在于提供用于调节ggp(也称为gdp-l-半乳糖磷酸化酶)活性;和/或植物中的抗坏血酸含量的改进组合物和方法;和/或提供可用于遗传操纵的改进工具;或至少为公众提供有用的选择。
技术实现要素:
第一方面,本发明提供了一种分离的多核苷酸,包含编码具有以下氨基酸序列的多肽或其变体或片段的序列,所述氨基酸序列选自:seqidno:1至20、以及132至134(uorf肽)。
在一个实施方案中,所述变体或片段包含与选自seqidno:21至40、以及135至137的氨基酸序列(uorf肽的保守区域)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含选自seqidno:21至40、以及135至137的氨基酸序列(uorf肽的保守区域)。
在一个实施方案中,所述变体或片段包含与选自氨基酸序列seqidno:21至30、33至37以及135至137(双子叶植物uorf肽的保守区域)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含选自seqidno:21至30、33至37以及135至137的氨基酸序列(双子叶植物uorf肽的保守区域)。
在一个实施方案中,所述变体或片段包含与氨基酸序列seqidno:108(uorf肽共有基序)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含氨基酸序列seqidno:108(uorf肽共有基序)。
在另一个实施方案中,所述变体包含与选自seqidno:1至20、以及132至134的氨基酸序列(uorf肽)有至少70%同一性的序列。
在另一个实施方案中,所述变体包含选自seqidno:1至20、以及132至134的氨基酸序列(uorf肽)。
在另一个实施方案中,所述变体包含与选自seqidno:1至10、13至17以及132至134的氨基酸序列(双子叶植物uorf肽)有至少70%同一性的序列。
在另一个实施方案中,所述变体包含选自seqidno:1至10、13至17以及132至134的氨基酸序列(双子叶植物uorf肽)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:61至80、以及138至140的序列(uorfdna序列保守区域)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:61至80、以及138至140的序列(uorfdna序列保守区域)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:61至70、73至77以及138至140的序列(双子叶植物uorfdna序列保守区域)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:61至70、73至77以及138至140的序列(双子叶植物uorfdna序列保守区域)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:41至60、以及129至131的序列(uorfdna序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:41至60、以及129至131的序列(uorfdna序列)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:41至50、53至57以及129至131的序列(双子叶植物uorfdna序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:41至50、53至57以及129至131的序列(双子叶植物uorfdna序列)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:111至125的序列(5’-utr子序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:111至125的序列(5’-utr子序列)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:81至100、以及126至128的序列(完整5’-utr序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:81至100、以及126至128的序列(完整5’-utr序列)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:81至90、93至97以及125至128的序列(完整双子叶植物5’-utr序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:81至90、93至97以及125至128的序列(完整双子叶植物5’-utr序列)。
在另一个方面,本发明提供了一种分离的多核苷酸,包含选自于seqidno:41至60、以及129至131(uorfdna序列)、或其变体或片段的序列。
在一个实施方案中,所述变体或片段包含与选自seqidno:61至80、以及138至140的序列(uorfdna序列保守区域)有至少70%同一性的序列。
在一个实施方案中,所述变体或片段包含选自seqidno:61至80、以及138至140的序列(uorfdna序列保守区域)。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:41至60、以及129至131的序列(uorfdna序列)。
在一个实施方案中,所述变体包含与选自seqidno:41至60、以及129至131的序列(uorfdna序列)有至少70%同一性的序列。
在一个实施方案中,所述变体包含与选自seqidno:41至50、53至57以及129至131的序列(双子叶植物uorfdna序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:111至125的序列(5’-utr子序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:111至125的序列(5’-utr子序列)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:81至100、以及126至128的序列(完整5’-utr序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:81至100、以及126至128的序列(完整5’-utr序列)。
在另一个实施方案中,所述分离的多核苷酸包含与选自seqidno:81至90、93至97以及126至128的序列(完整双子叶植物5’-utr序列)有至少70%同一性的序列。
在另一个实施方案中,所述分离的多核苷酸包含选自seqidno:81至90、93至97以及126至128的序列(完整双子叶植物5’-utr序列)。
在另一个方面,本发明提供了一种分离的多核苷酸,具有选自seqidno:81至100、以及126至128(完整5’-utr序列)、或其变体或片段的序列。
在一个实施方案中,所述变体与选自seqidno:81至100、以及126至128(完整5’-utr序列)有至少70%同一性的序列。
在另一个方面,本发明提供了一种分离的多核苷酸,具有选自seqidno:111至125(5’-utr子序列)、或其变体或片段的序列。
在一个实施方案中,所述变体与选自seqidno:111至125(5’-utr子序列)的序列有至少70%同一性。
在另一个实施方案中,所述变体或片段包含与选自seqidno:41至60、以及129至131(uorfdna序列)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含选自seqidno:41至60、以及129至131的序列(uorfdna序列)。
在另一个实施方案中,所述变体或片段包含与选自seqidno:41至50、53至57以及129至131(双子叶植物uorfdna序列)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含选自seqidno:41至50、53至57以及129至131的序列(双子叶植物uorfdna序列)。
在另一个实施方案中,所述变体或片段包含与选自seqidno:61至80、以及138至140的序列(uorfdna序列保守区域)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含选自seqidno:61至80、以及138至140的序列(uorfdna序列保守区域)的序列。
在另一个实施方案中,所述变体或片段包含与选自seqidno:61至70、73至77以及138至140的序列(双子叶植物uorfdna序列保守区域)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含选自seqidno:61至70、73至77以及138至140的序列(双子叶植物uorfdna序列保守区域)。
在另一个实施方案中,所述变体编码与seqidno:21至40、以及135至137(uorf肽的保守区域)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体编码选自seqidno:21至40、以及135至137(uorf肽的保守区域)中任一者的序列。
在另一个实施方案中,所述变体编码与seqidno:21至30、33至37以及135至137(双子叶植物uorf肽的保守区域)中至少一者有至少70%同一性的序列。
在另一个实施方案中,所述变体编码选自seqidno:21至30、33至37以及135至137(双子叶植物uorf肽的保守区域)中的至少一者的序列。
在一个实施方案中,所述变体或片段包含与氨基酸序列seqidno:108(uorf肽共有基序)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含氨基酸序列seqidno:108(uorf肽共有基序)。
在另一个实施方案中,所述变体编码与seqidno:1至20、以及132至134(uorf肽)中的至少一者有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段编码选自seqidno:1至20、以及132至134(uorf肽)中至少一者的序列。
在另一个实施方案中,所述变体编码与seqidno:1至10、13至17以及132至134(双子叶植物uorf肽)中至少一者有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段编码选自seqidno:1至10、13至17以及132至134(双子叶植物uorf肽)中至少一者的序列。
在一个实施方案中,所述分离的多核苷酸是修饰的。
在一个实施方案中,所述修饰为在编码5'-utr的序列中至少一个核苷酸的缺失、添加或置换的至少一者。
在一个实施方案中,所述修饰减少、干扰或阻止uorf多肽或其变体的翻译,所述uorf多肽具有seqidno:1和20以及132至134(uorf肽)中任一者的序列。
在另一个实施方案中,所述修饰减少、干扰或破坏uorf多肽或其变体的活性,所述uorf多肽具有seqidno:1和20以及132至134(uorf肽)中任一者的序列。
在一个实施方案中,所述变体包含与seqidno:1和20以及132至134(uorf肽)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含与seqidno:1至10、13至17以及132至134(双子叶植物uorf肽)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含与seqidno:21至40、以及135至137(uorf肽保守区域)中至少一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含具有seqidno:21至40、以及135至137(uorf肽保守区域)中至少一者的序列。
在另一个实施方案中,所述变体包含与seqidno:1至30、33至37以及135至137(双子叶植物uorf肽保守区域)中至少一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含具有seqidno:1至30、33至37以及135至137(双子叶植物uorf肽保守区域)中至少一者的序列。
在一个实施方案中,所述变体或片段包含与氨基酸序列seqidno:108(uorf肽共有基序)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含氨基酸序列seqidno:108(uorf肽共有基序)。
在一个实施方案中,所述多核苷酸、或变体或片段可操作地连接至目标核酸序列。
在另一个实施方案中,所述目标核酸序列编码目标蛋白。
在一个实施方案中,所述多核苷酸和核酸序列在自然界中通常不相连。
当如上所述修饰多核苷酸时,为了干扰uorf多肽的表达或活性,可操作地连接的序列可以为ggp序列。在该实施方案中,所述修饰除去通过抗坏血酸对uorf的阻遏。在经修饰的多核苷酸的控制下表达ggp可以有利地保留与天然ggp启动子和5'-utr的控制类似的空间和/或时间表达,但是停止了抗坏血酸经由uorf多肽的负向调控表达。在该实施方案中,所述多核苷酸与目标核酸序列在自然界可以正常地相连,但是所述多核苷酸处于如上所述的修饰形式。
多肽
在另一个方面,本发明提供了一种分离的多肽,其包含选自seqidno:1至20以及132至134(uorf肽)、或其变体或片段中任一者的序列。
在一个实施方案中,所述变体或片段包含与选自seqidno:21至40、以及135至137(uorf肽的保守区域)的序列有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含选自seqidno:21至40、以及135至137(uorf肽的保守区域)的序列。
在一个实施方案中,所述变体或片段包含与选自seqidno:21至30、33至37以及135至137的序列(双子叶植物uorf肽的保守区域)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含选自seqidno:21至30、33至37以及135至137的序列(双子叶植物uorf肽的保守区域)。
在一个实施方案中,所述变体或片段包含与氨基酸序列seqidno:108(uorf肽保守基序)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含氨基酸序列seqidno:108(uorf肽保守基序)。
在另一个实施方案中,所述变体包含与选自seqidno:1至20、以及132至134的序列(uorf肽)有至少70%同一性的序列。
在另一个实施方案中,所述变体包含选自seqidno:1至10、13至17以及132至134的序列(双子叶植物uorf肽)有至少70%同一性的序列。
构建体
在另一个实施方案中,本发明提供了一种构建体,包含本发明的多核苷酸。
在一个实施方案中,所述多核苷酸可操作地连接至目标核酸序列。
在另一个实施方案中,所述多核苷酸与核酸序列在自然界中通常不相连。
在另一个实施方案中,所述目标核酸序列编码目标蛋白。
多核苷酸的活性
在一个实施方案中,本发明的多核苷酸受到化合物的调控。
在该实施方案中,可操作地连接至本发明的多核苷酸的任意核酸序列的表达受到所述化合物的调控。
在一个优选的实施方案中,调控为转录后的。
优选地,由可操作地连接的核酸编码的多肽的表达受到化合物的调控。
在一个实施方案中,所述可操作地连接的核酸的表达是由所述化合物与所述uorf肽之间的相互作用调控的,所述uorf肽由本发明的多核苷酸表达。在一个实施方案中,相互作用是直接的。在另一个实施方案中,相互作用是间接的。在另一个实施方案中,该间接相互作用是通过另一种蛋白进行的。
在一个实施方案中,所述化合物为抗坏血酸、或相关代谢物。在一个优选的实施方案中,所述化合物为抗坏血酸。
在如上所述修饰时,所述多核苷酸可能不再受到所述化合物的调控。该实施方案的应用可以在经修饰的多核苷酸序列的控制下表达ggp编码序列。在该实施方案中,所述修饰导致该化合物阻遏的减轻或消除。当该化合物为抗坏血酸时,这导致对ggp翻译阻遏的降低,因此导致了升高的ggp产生和升高的抗坏血酸积聚。在该实施方案中,uorf或uorf编码序列的修饰可能处于启动子和5'-utr序列中。完整ggp启动子和5'-utr序列的例子参见seqidno:101至107或其变体。在启动子和5'-utr序列中,使用经修饰的uorf可保留天然ggp序列的一些时空表达,而不会导致抗坏血酸经由uorf多肽对ggp翻译的阻遏。
细胞
在另一个实施方案中,本发明提供了一种细胞,包含本发明的多核苷酸或本发明的构建体。
优选地,所述细胞或其前体细胞已经经过遗传修饰从而包含了本发明的多核苷酸或本发明的构建体。
优选地,所述细胞或其前体细胞已经经过转化从而包含了本发明的多核苷酸或本发明的构建体。
植物细胞和植物
在另一个实施方案中,本发明提供了一种植物细胞或植物,其包含本发明的多核苷酸或本发明的构建体。
优选地,所述植物细胞或植物、或其前体植物细胞或植物已经经过遗传修饰从而包含了本发明的多核苷酸或本发明的构建体。
优选地,所述植物细胞或植物、或其前体植物细胞或植物已经经过转化从而包含了本发明的多核苷酸或本发明的构建体。
还表达差向异构酶
在一个实施方案中,所述细胞或植物还经过遗传修饰或经过转化从而表达编码gdp-d-甘露糖差向异构酶的多核苷酸。
优选地,所述细胞或植物还包含本发明的表达构建体,所述表达构建体能够表达gdp-d-甘露糖差向异构酶。
植物部分或繁殖体
在另一个实施方案中,本发明提供了一种植物部分或繁殖体,其包含本发明的多核苷酸或本发明的构建体。
优选地,所述植物部分或繁殖体、或其前体植物细胞或植物已经经过遗传修饰从而包含了本发明的多核苷酸或本发明的构建体。
优选地,所述植物部分或繁殖体、或其前体植物细胞或植物已经经过转化从而包含了本发明的多核苷酸或本发明的构建体。
在另一个方面,本发明提供了一种在细胞中控制或调节至少一种核酸序列的表达的方法,包括用本发明的多核苷酸或构建体转化所述细胞。
在另一个方面,本发明提供了一种在植物细胞或植物中控制或调节至少一种核酸序列的表达的方法,包括用本发明的多核苷酸或构建体转化植物细胞或植物。
在另一个方面,本发明提供了一种制备具有修饰基因表达的细胞的方法,所述方法包括用本发明的多核苷酸或构建体转化所述细胞。
在另一个方面,本发明提供了一种制备具有修饰基因表达的植物细胞或植物的方法,所述方法包括用本发明的多核苷酸或构建体转化植物细胞或植物。
在另一个方面,本发明提供了一种改变植物表型的方法,所述方法包括将本发明的多核苷酸或构建体稳定整合进所述植物的基因组中。
本领域技术人员能够理解,将本发明的多核苷酸引入细胞、植物细胞或植物中可能导致在这些序列引入之前,对与核酸序列可操作地连接的序列的调节或控制。在这样的实施方案中,本发明的多核苷酸和可操作地连接的目标核酸将一起引入到例如本发明的构建体中。
在可选的实施方案中,本发明的多核苷酸可插入基因组,并控制或调节插入位点附近的核酸序列(如编码蛋白质的核酸序列)的表达。
在一个优选的实施方案中,所述细胞、植物细胞或植物产生这样的化合物,所述化合物通过引入的本发明的多核苷酸、或是通过由引入的本发明的多核苷酸编码的uorf多肽调控或控制表达。
可选地,所述化合物可施用于细胞、植物细胞或植物。
在另一个方面,本发明提供了由本发明的方法制备的植物细胞或植物。
在一个方面,本发明提供了一种制备具有以下至少一者的植物细胞或植物的方法:
a)增多的ggp翻译,
b)增多的ggp产生,
c)增强的ggp活性,以及
d)增多的抗坏血酸产生,
所述方法包括在所述植物细胞或植物中ggp基因的5’-utr的修饰。
在一个实施方案中,所述5'-utr位于选自seqidno:101至107(具有启动子5'-utr的ggp基因组序列)中任一者或其变体的多核苷酸序列中。
优选地,所述变体与seqidno:101至107(具有启动子5'-utr的ggp基因组序列)中任一者有至少70%同一性。
在另一个实施方案中,所述5'-utr具有选自seqidno:81至100、以及126至128(完整5'-utr序列)中任一者或其变体的多核苷酸序列。
优选地,所述变体与seqidno:81至100、以及126至128(完整5'-utr序列)中任一者有至少70%同一性。
在一个优选的实施方案中,所述修饰位于5'-utr中的uorf序列中。
在一个优选的实施方案中,所述uorf具有选自seqidno:41至60、以及129至131(uorfdna序列)中任一者或其变体的序列。
在一个优选的实施方案中,所述变体与seqidno:41至60、以及129至131(uorfdna序列)中任一者有至少70%同一性。
在一个优选的实施方案中,所述变体与seqidno:41至50、53至57、以及129至131(双子叶植物uorfdna序列)中任一者有至少70%同一性。
在另一个实施方案中,所述变体包含与选自seqidno:61至80、以及138至140(uorfdna序列保守区域)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含选自seqidno:61至80、以及138至140(uorfdna序列保守区域)中任一者的序列。
在另一个实施方案中,所述变体包含与选自seqidno:61至70、73至77、以及138至140(双子叶植物uorfdna序列保守区域)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含seqidno:61至70、73至77、以及138至140(双子叶植物uorfdna序列保守区域)中任一者的序列。
在另一个实施方案中,所述uorf具有选自seqidno:1至20、以及132至134(uorf多肽序列)中任一者或其变体的序列。
在另一个实施方案中,所述变体包含与选自seqidno:1至20、以及132至134(uorf多肽序列)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含选自seqidno:1至20、以及132至134(uorf多肽序列)中任一者的序列。
在另一个实施方案中,所述变体包含与选自seqidno:1至10、13至17以及132至134(双子叶植物uorf多肽序列)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含选自seqidno:1至10、13至17以及132至134(双子叶植物uorf多肽序列)中任一者的序列。
在另一个实施方案中,所述变体包含与选自seqidno:21至40、以及135至137(uorf多肽序列的保守区域)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含seqidno:21至40、以及135至137(uorf多肽序列的保守区域)中任一者的序列。
在另一个实施方案中,所述变体包含与选自seqidno:21至30、33至37以及135至137(双子叶植物uorf多肽序列的保守区域)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含seqidno:21至30、33至37以及135至137(双子叶植物uorf多肽序列的保守区域)中任一者的序列。
在一个实施方案中,所述变体或片段包含与氨基酸序列seqidno:108(uorf肽共有基序)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含氨基酸序列seqidno:108(uorf肽共有基序)。
修饰
在一个实施方案中,所述修饰为在编码5'-utr的序列中至少一个核苷酸的缺失、添加或置换。
在一个实施方案中,所述修饰减少、干扰或阻止uorf多肽的翻译,所述uorf多肽具有与seqidno:1和20、以及132至134(uorf肽)中任一者或其变体的序列。
在另一个实施方案中,所述修饰减少、干扰或破坏uorf多肽的活性,所述uorf多肽具有seqidno:1和20、以及132至134(uorf肽)中任一者或其变体的序列。
在一个实施方案中,所述变体包含与seqidno:1和20、以及132至134(uorf肽)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含与seqidno:1至10、13至17以及132至134(双子叶植物uorf肽)中任一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含与seqidno:21至40、以及135至137(uorf肽保守区域)中至少一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含seqidno:21至40、以及135至137(uorf肽保守区域)中至少一者的序列。
在另一个实施方案中,所述变体包含与seqidno:21至30、33至37以及135至137(双子叶植物uorf肽保守区域)中至少一者有至少70%同一性的序列。
在另一个实施方案中,所述变体包含seqidno:21至30、33至37以及135至137(双子叶植物uorf肽保守区域)中至少一者的序列。
在一个实施方案中,所述变体或片段包含与氨基酸序列seqidno:108(uorf肽共有基序)有至少70%同一性的序列。
在另一个实施方案中,所述变体或片段包含氨基酸序列seqidno:108(uorf肽共有基序)。
在另一个方面,本发明提供了一种筛选具有以下至少一者的植物的方法:
a)增多的ggp翻译,
b)增多的ggp产生,
c)增强的ggp活性,以及
d)增多的抗坏血酸产生,
所述方法包括在检测植物是否存在本发明的多核苷酸的第一多态性,或与所述第一多态性相关的另一多态性。
在一个实施方案中,第一多态性的存在、或与所述第一多态性相关的另一多态性的存在表明具有a)至d)中任一者。
在另一个实施方案中,另一多态性与第一多态性有连锁不平衡(ld)。
在另一个实施方案中,所述方法包括从一种或多种未经筛选的植物中分离选定的植物。
在另一个方面,本发明提供了由本发明的方法选定的植物。
在另一个方面,本发明提供了一种由本发明的方法选定的一组植物。优选地,该组包含至少2种、更优选至少3种、更优选至少4种、更优选至少5种、更优选至少6种、更优选至少7种、更优选至少8种、更优选至少9种、更优选至少10种、更优选至少11种、更优选至少12种、更优选至少13种、更优选至少14种、更优选至少15种、更优选至少16种、更优选至少17种、更优选至少18种、更优选至少19种、更优选至少20种植物。
在另一个方面,本发明提供了一种制备抗坏血酸的方法,所述方法包括从本发明的植物细胞或植物中提取抗坏血酸。
在另一个方面,本发明提供了一种针对本发明的多核苷酸的抗体。在另一个方面,本发明提供了一种对本发明的多核苷酸有特异性的抗体。
本发明的多核苷酸、多肽、变体或片段可以来源于任意物种。所述多核苷酸、多肽、变体或片段可以是自然存在的或非自然存在的。多核苷酸、多肽、变体或片段可以由重组制备,也可以是“基因改组”方法的产物。
在一个实施方案中,所述多核苷酸、多肽、变体或片段来源于任意植物物种。在本发明的方法中用于转化或修饰的植物可以来源于任意植物物种。在本发明的方法中用于转化或修饰的细胞可以来源于任意植物物种。
在另一个实施方案中,所述植物来源于裸子植物物种。
在另一个实施方案中,所述植物来源于被子植物物种。
在另一个实施方案中,所述植物来源于双子叶植物物种。
在另一个实施方案中,所述植物来源于选自下列但不限于下列属的水果物种:猕猴桃属(actinidia)、苹果属(malus)、柑橘属(citrus)、草莓属(fragaria)或越桔属(vaccinium)。
特别优选的水果植物物种是:美味猕猴桃(actidiniadeliciosa)、中华猕猴桃(a.chinensis)、毛花猕猴桃(a.eriantha)、软枣猕猴桃(a.arguta)、四种猕猴桃物种的杂交品种、苹果(malusdomestica)和三叶海棠(malussieboldii)。
在另一个实施方案中,所述植物选自由下列物种构成的组:毛花猕猴桃(actinidiaeriantha)、黄瓜(cucumissativus)、大豆(glycinemax)、番茄(solanumlycopersicum)、葡萄(vitisvinifera)、拟南芥(arabidopsisthaliana)、苹果(malusxdomesticus)、蒺藜苜蓿(medicagotruncatula)、毛果杨(populustrichocarpa)、软枣猕猴桃(actinidiaarguta)、中华猕猴桃(actinidiachinensis)、草莓(fragariavulgaris)、马铃薯(solanumtuberosum)和玉米(zeamays)。
在另一个实施方案中,所述植物选自来源于下列但不限于下列属的蔬菜物种:芸苔属(brassica)、番茄属(lycopersicon)和茄属(solanum)。
特别优选的蔬菜植物物种是:番茄和马铃薯。
在另一个实施方案中,所述植物来源于单子叶植物物种。
在另一个实施方案中,所述植物选自来源于下列但不限于下列属的农作物物种:大豆属(glycine)、玉蜀黍属(zea)、大麦属(hordeum)和稻属(oryza)。
特别优选的农作物物种是:水稻(oryzasativa)、大豆和玉米。
在另一个实施方案中,所述植物选自由下列物种构成的组:毛花猕猴桃、黄瓜、大豆、番茄、葡萄、拟南芥、苹果、蒺藜苜蓿、毛果杨、软枣猕猴桃、中华猕猴桃、草莓、马铃薯和玉米。
发明详述
在本说明书中引用专利说明书、其他外部文件或其他来源的信息处,通常是为讨论本发明的特征提供的背景。除非另外特别说明,否则对这类外部文献的参考不应解释为以任何权限承认这类文献、或这类信息来源是现有技术、或形成本领域的一般公知常识的一部分。
本说明书中所使用的术语“包含”意味着“至少由...部分组成”。当在本说明书中解释每个包括术语“包含”的陈述时,其他特征或者那些以该术语开始的特征也可存在。相关术语如“包括”和“含有”以相同方式解释。
在某些实施方案中,术语“包含”及其相关术语可以用“由...构成”及其相关术语替代。
多核苷酸和片段
本文中所用术语“多核苷酸”是指,任何长度但优选至少15个核苷酸的单链或双链脱氧核糖核苷酸或核糖核苷酸聚合物,包括以下非限制性实例:基因的编码及非编码序列、有义及反义互补序列、外显子、内含子、基因组dna、cdna、pre-mrna、mrna、rrna、sirna、mirna、trna、核酸酶、重组多肽、分离和纯化的天然存在的dna或rna序列、合成的rna和dna序列、核酸探针、引物和片段。
优选地,术语“多核苷酸”包括指定序列及其互补序列。
本文提供的多核苷酸序列的“片段”是连续核苷酸的子序列,例如至少长度为15个核苷酸的序列。本发明所述的片段包含本发明所述的多核苷酸的连续核苷酸中的15个核苷酸,优选至少20个核苷酸,更优选至少30个核苷酸,更优选至少50个核苷酸,更优选至少60个核苷酸。
术语“引物”是指通常有自由的3’oh的短的多核苷酸,其与模板杂交并用于引发与靶体互补的多核苷酸的聚合。
多肽和片段
本文中所用术语“多肽”包括任何长度但优选至少5个氨基酸的氨基酸链,包括全长蛋白,其中氨基酸残基通过共价肽键相连。本发明的多肽可为纯化的天然产物,或可使用重组或合成技术部分地或整体地制备。该术语可指多肽、多肽的聚体(如二聚体或其他多聚体)、融合多肽、多肽片段、多肽变体或其衍生物。
多肽的“片段”是多肽的子序列。优选地,所述片段执行对于该多肽的生物学活性所需的功能和/或提供该多肽的三维结构。
本发明用于多核苷酸或多肽序列的术语“分离的”是指从它们的天然细胞环境中提取出来的序列。在一个实施方案中,所述序列从其自然界发现的侧翼序列中分离出来。分离的分子可通过任何方法或方法的组合获得,包括生物化学的、重组的及合成的技术。
术语“重组”是指在其天然环境中从其周围的序列中提取出来的多核苷酸序列,和/或与其在天然环境中不存在的序列重组的多核苷酸序列。
通过从“重组的”多核苷酸序列翻译,制备“重组的”多肽序列。
就源自特定属或种的本发明的多核苷酸或多肽而言,术语“源自”是指,所述多核苷酸或多肽具有与在该属或种中天然发现的多核苷酸或多肽相同的序列。源自特定属或种的多核苷酸或多肽因此可以合成地或重组地产生。
变体
本文中所用术语“变体”是指,不同于具体鉴别出的序列的多核苷酸或多肽序列,其中缺失、置换或添加了一个或多个核苷酸或氨基酸残基。变体可为天然存在的等位基因变体或非天然存在的变体。变体可来自同一物种或来自其它物种,且可包括同系物、旁系同源物及直系同源物。在某些实施方案中,本发明多肽的变体具有与本发明多肽相同或类似的生物活性。就多肽而言,术语“变体”包括所有形式的多肽和如本文所定义的多肽。
多核苷酸变体
变体多核苷酸序列优选地与本发明的序列表现出至少50%、更优选至少51%、更优选至少52%、更优选至少53%、更优选至少54%、更优选至少55%、更优选至少56%、更优选至少57%、更优选至少58%、更优选至少59%、更优选至少60%、更优选至少61%、更优选至少62%、更优选至少63%、更优选至少64%、更优选至少65%、更优选至少66%、更优选至少67%、更优选至少68%、更优选至少69%、更优选至少70%、更优选至少71%、更优选至少72%、更优选至少73%、更优选至少74%、更优选至少75%、更优选至少76%、更优选至少77%、更优选至少78%、更优选至少79%、更优选至少80%、更优选至少81%、更优选至少82%、更优选至少83%、更优选至少84%、更优选至少85%、更优选至少86%、更优选至少87%、更优选至少88%、更优选至少89%、更优选至少90%、更优选至少91%、更优选至少92%、更优选至少93%、更优选至少94%、更优选至少95%、更优选至少96%、更优选至少97%、更优选至少98%和最优选至少99%的同一性。在本发明的核苷酸的至少20个核苷酸位置、优选至少50个核苷酸位置、更优选至少100个核苷酸位置且最优选本发明多核苷酸的全长范围的比较窗口上,存在同一性。
可以如下方式测定多核苷酸序列同一性。在bl2seq(tatianaa.tatusova,thomasl.madden(1999),“blast2sequences-anewtoolforcomparingproteinandnucleotidesequences”,femsmicrobiollett.174:247-250)中,使用blastn(来自blast程序套件,2.2.5版[2002年11月]),将主题多核苷酸序列与候选多核苷酸序列进行比较,所述bl2seq可从ncbi(ftp://ftp.ncbi.nih.gov/blast/)公开获得。除应关闭对低复杂性部分的过滤以外,利用bl2seq的预设参数。
可使用以下unix命令行参数检验多核苷酸序列的同一性:
bl2seq–inucleotideseq1–jnucleotideseq2–ff–pblastn
参数-ff关闭对低复杂性区段的过滤。参数-p为序列对选出适当算法。bl2seq程序在“identities=”行中将序列同一性报告为相同核苷酸的数量及百分比。
使用总体序列比对程序(例如needleman,s.b.和wunsch,c.d.(1970)j.mol.biol.48,443-453),也可以在候选序列与主题多核苷酸序列之间的重叠部分的整个长度上计算多核苷酸序列同一性。needleman-wunsch总体比对算法的一个完整实现,参见emboss套件(rice,p.longden,i.andbleasby,a.emboss:theeuropeanmolecularbiologyopensoftwaresuite,trendsingeneticsjune2000,第16卷,第6期,第276-277页)中的needle程序。该emboss套件可从http://www.hgmp.mrc.ac.uk/software/emboss/得到。europeanbioinformaticsinstitute服务器也在http:/www.ebi.ac.uk/emboss/align/上在线提供执行两个序列之间的emboss-needle总体比对的工具。
或者,可使用gap程序,其计算两个序列在无处罚末端空隙的情况下的最佳总体比对。gap描述在以下论文中:huang,x.(1994)onglobalsequencealignment.computerapplicationsinthebiosciences10,227-235。
计算多核苷酸%序列同一性的优选方法是基于使用clustalx(jeanmougin等人,1998,trendsbiochem.sci.23,403-5.)对比待比较的序列。
本发明的多核苷酸变体也涵盖这样的变体:所述变体展现与可能保留那些序列的功能等效性的一个或多个特别鉴别的序列的相似性,且不能恰当地预期随机发生。使用从来从ncbi(ftp://ftp.ncbi.nih.gov/blast/)的blast程序套件(2.2.5版[2002年11月])可公开获得的bl2seq程序,可以测定有关多肽的这种序列相似性。
可使用以下unix命令行参数检验多核苷酸序列的相似性:
bl2seq–inucleotideseq1–jnucleotideseq2–ff–ptblastx
参数-ff关闭对低复杂性区段的过滤。参数-p为序列对选出适当算法。该程序发现序列之间的相似性区域,且为每个这样的区域报导一个“e值”,所述e值为预期在含有随机序列的固定参考尺寸的数据库中找到该偶然匹配的预期次数。此数据库的尺寸是由bl2seq程序中的默认值设定。对于远小于1的小e值而言,e值大致为这样的随机匹配的机率。
当与任一个特别鉴别的序列相比较时,变体多核苷酸序列优选地表现出小于1x10-6、更优选地小于1x10-9、更优选地小于1x10-12、更优选地小于1x10-15、更优选地小于1x10-18、更优选地小于1x10-21、更优选地小于1x10-30、更优选地小于1x10-40、更优选地小于1x10-50、更优选地小于1x10-60、更优选地小于1x10-70、更优选地小于1x10-80、更优选地小于1x10-90和最优选地小于1x10-100的e值。
或者,本发明的变体多核苷酸在严谨条件下与指定的多核苷酸序列或其互补序列杂交。
术语“在严谨条件下杂交”及其语法等效描述是指,多核苷酸分子在限定的温度及盐浓度条件下与靶多核苷酸分子(诸如固定于dna或rna印迹(诸如dna印迹或rna印迹)上的靶多核苷酸分子)杂交的能力。通过最初在更低严谨性条件下杂交,随后将严谨性增加至希望的严谨性,可以测定在严谨杂交条件下杂交的能力。
关于长度大于约100个碱基的多核苷酸分子,典型的严谨杂交条件为,比天然双链体的解链温度(tm)低不超过25至30℃(例如10℃)(一般参见,sambrook等人编,1987,molecularcloning,alaboratorymanual,第2版.coldspringharborpress;ausubel等人,1987,currentprotocolsinmolecularbiology,greenepublishing)。大于约100个碱基的多核苷酸分子的tm可通过下式来计算:tm=81.5+0.41%(g+c-log(na+).(sambrook等人,eds,1987,molecularcloning,alaboratorymanual,第2版.coldspringharborpress;boltonandmccarthy,1962,pnas84:1390)。长度大于100个碱基的多核苷酸的典型严谨条件是这样的杂交条件,诸如在6×ssc、0.2%sds的溶液中预洗涤;在65℃,在6×ssc、0.2%sds中杂交过夜;随后在1×ssc、0.1%sds中在65℃进行两次各30分钟的洗涤,并在0.2×ssc、0.1%sds中在65℃进行两次各30分钟的洗涤。
关于长度小于100个碱基的多核苷酸分子,示例性的严谨杂交条件是,比tm低5至10℃。平均而言,长度小于100碱基对的多核苷酸分子的tm降低大约(500/寡核苷酸长度)℃。
关于称为肽核酸(pna)的dna模拟物(nielsen等人,science.1991年12月6日;254(5037):1497-500),tm值高于dna-dna或dna-rna杂交物的tm值,且可使用giesen等人,nucleicacidsres.1998年11月1日;26(21):5004-6中所述的公式来计算。长度小于100个碱基的dna-pna杂交物的示例性的严谨杂交条件为,比tm低5至10℃。
本发明的变体多核苷酸也涵盖这样的多核苷酸:其不同于本发明的序列,但因遗传密码的简并性而编码具有与由本发明的多核苷酸所编码的多肽相似的活性的多肽。不改变多肽的氨基酸序列的序列变化是“沉默修饰”。除atg(甲硫氨酸)和tgg(色氨酸)以外,同一氨基酸的其它密码子可通过本领域认可的技术发生改变,例如,以优化在特定宿主有机体中的密码子表达。
引起编码的多肽序列中的一个或若干个氨基酸的保守置换但不显著改变其生物活性的多核苷酸序列变化,也包括于本发明中。技术人员知晓制造表型沉默的氨基酸置换的方法(参见,例如,bowie等人,1990,science247,1306)。
使用从ncbi(ftp://ftp.ncbi.nih.gov/blast/)公开获得的来自blast程序套件(2.2.5版[2002年11月])的bl2seq程序,通过先前所述的tblastx算法,可以测定由于编码的多肽序列中的沉默修饰及保守置换而产生的变体多核苷酸。
本发明所述的变体多核苷酸作为ggp的功能可通过(例如)如在实施例的部分所述的通过这种序列在细菌中的表达,并检测其编码的蛋白质的活性而评价。变体的功能还可通过其改变植物中的ggp活性或抗坏血酸含量的能力而检测,该方法在本文的实施例的部分也做了描述。
本发明所述的变体多核苷酸作为gdp-d-甘露糖差向异构酶的功能可通过(例如)如在实施例的部分所述的通过这种序列在细菌中的表达,并检测其编码的蛋白质的活性而评价。变体的功能还可通过其改变植物中的gdp-d-甘露糖差向异构酶活性或抗坏血酸含量的能力而检测,该方法在本文的实施例的部分也做了描述。
多肽变体
就多肽而言,术语“变体”涵盖天然存在的、重组地和合成地生产的多肽。变体多肽序列优选地与本发明的序列表现出至少50%、更优选至少51%、更优选至少52%、更优选至少53%、更优选至少54%、更优选至少55%、更优选至少56%、更优选至少57%、更优选至少58%、更优选至少59%、更优选至少60%、更优选至少61%、更优选至少62%、更优选至少63%、更优选至少64%、更优选至少65%、更优选至少66%、更优选至少67%、更优选至少68%、更优选至少69%、更优选至少70%、更优选至少71%、更优选至少72%、更优选至少73%、更优选至少74%、更优选至少75%、更优选至少76%、更优选至少77%、更优选至少78%、更优选至少79%、更优选至少80%、更优选至少81%、更优选至少82%、更优选至少83%、更优选至少84%、更优选至少85%、更优选至少86%、更优选至少87%、更优选至少88%、更优选至少89%、更优选至少90%、更优选至少91%、更优选至少92%、更优选至少93%、更优选至少94%、更优选至少95%、更优选至少96%、更优选至少97%、更优选至少98%和最优选至少99%同一性。在本发明的多肽的至少20个氨基酸位置、优选至少50个氨基酸位置、更优选至少100个氨基酸位置且最优选整个长度的比较窗上,存在同一性。
多肽序列同一性可以如下方式测定。在bl2seq中使用blastp(来自blast程序套件,2.2.5版[2002年11月])将主题多肽序列与候选多肽序列进行比较,bl2seq可从ncbi(ftp://ftp.ncbi.nih.gov/blast/)公开获得。除应关闭对低复杂性区域的过滤以外,利用bl2seq的默认参数。
使用总体序列比对程序,也可在候选序列与主题多核苷酸序列之间的重叠部分的整个长度上计算多肽序列同一性。如上文所论述的emboss-needle(可获自http:/www.ebi.ac.uk/emboss/align/)及gap(huang.x.(1994)onglobalsequencealignment.computerapplicationsinthebiosciences10,227-235.),也是适用于计算多肽序列同一性的总体序列比对程序。
用于计算多肽%序列同一性的优选方法,是基于使用clustalx(jeanmougin等人,1998,trendsbiochem.sci.23,403-5)比对待比较的序列。
本发明的多肽变体也涵盖这样的多肽变体:其展现与可能保留序列的功能等效性的一个或多个特别鉴别的序列的相似性,且不能恰当预期随机发生。使用从ncbi(ftp://ftp.ncbi.nih.gov/blast/)公开获得的来自blast程序套件(2.2.5版[2002年11月])的bl2seq程序,可以测定有关多肽的这种序列相似性。可使用以下unix命令行参数检验多肽序列的相似性:
bl2seq–ipeptideseq1–jpeptideseq2-ff–pblastp
当与任一个特别鉴别的序列相比较时,变体多肽序列优选地表现出小于1x10-6、更优选地小于1x10-9、更优选地小于1x10-12、更优选地小于1x10-15、更优选地小于1x10-18、更优选地小于1x10-21、更优选地小于1x10-30、更优选地小于1x10-40、更优选地小于1x10-50、更优选地小于1x10-60、更优选地小于1x10-70、更优选地小于1x10-80、更优选地小于1x10-90和最优选地小于1x10-100的e值。
参数-ff关闭对低复杂性区段的过滤。参数-p为序列对选出适当算法。该程序发现序列之间的相似性区域,且为每一个这样的区域报导一个“e值”,所述e值为预期在含有随机序列的固定参考尺寸的数据库中找到该偶然匹配的预期次数。对于远小于1的小e值而言,e值大致为该随机匹配的机率。
所述多肽序列的一个或若干个氨基酸的保守置换(不显著改变其生物活性)也包括于本发明中。技术人员知晓制造表型沉默的氨基酸置换的方法(参见,例如,bowie等人,1990,science247,1306)。
变体多肽包括这样的多肽,其中氨基酸序列与本文的多肽存在一个或多个保守的氨基酸置换、缺失、添加或插入而不影响所述多肽的生物学活性。保守置换通常包括用另一个具有类似性质的另一个氨基酸置换一个氨基酸,例如在下列组内的置换:缬氨酸、甘氨酸;甘氨酸、丙氨酸;缬氨酸、异亮氨酸、亮氨酸;天冬氨酸、谷氨酸酸;天冬酰胺、谷氨酰胺;丝氨酸、苏氨酸;赖氨酸,精氨酸;以及苯丙氨酸、酪氨酸。
非保守置换将引起这些组中一个组中的成员变为另一组中的成员。
对进化生物学序列的分析示出并非所有序列改变的可能性相等,这至少部分反应了在生物学水平上保守置换与非保守置换之间的差异。例如,某些氨基酸置换可能频繁发生,而其它氨基酸置换却很少发生。氨基酸残基的进化修饰或置换可由记分矩阵(也称为置换矩阵)作为模型。这些矩阵用于生物统计学分析仪鉴定序列之间的关系,一个例子为如下示出的blosum62矩阵(表a)。
表a:含有所有可能置换分数的blosum62矩阵[henikoff和henikoff,1992]。
所示的blosum62矩阵用于在对应列和行交叉处的每个比对的氨基酸对生成分数。例如,谷氨酸残基(e)置换为天冬氨酸残基(d)的置换分数为2。对角线示出了未改变的氨基酸的分数。大部分置换的改变分数为负。该矩阵仅包含整数。
据信本领域技术人员熟知如何确定合适的记分矩阵为指定组的序列产生最佳的比对。尽管未做限定,表1所示的blosum62矩阵也用作blast检索中的缺省矩阵。
其它变体包括具有能影响肽稳定性的修饰的肽。这样的类似物可包含例如肽序列中的一个或多个非肽键(其替换肽键)。也包括含有天然存在的l-氨基酸之外的残基(如d-氨基酸)、或非天然存在的合成氨基酸(如β或γ氨基酸和环形类似物)的类似物,
多肽变体作为ggp的功能可由本文中实施例的部分所述的方法来评价。
多肽变体作为gdp-d-甘露糖差向异构酶的功能可由本文中实施例的部分所述的方法来评价。
构建体、载体及其组分
术语“遗传构建体”是指多核苷酸分子,通常为双链dna,其中可能已插入另一个多核苷酸分子(插入多核苷酸分子),例如,但不限于,cdna分子。遗传构建体可含有允许转录所述插入多核苷酸分子且任选地将转录物翻译为多肽的必需元件。所述插入多核苷酸分子可源自宿主细胞,或可源自不同细胞或有机体,和/或可为重组多核苷酸。一旦在宿主细胞内,遗传构建体则可整合进宿主染色体dna中。所述遗传构建体可连接至载体上。
术语“载体”是指多核苷酸分子,通常为双链dna,其用于将遗传构建体转运至宿主细胞中。所述载体可能能够在至少一个额外宿主系统(诸如大肠杆菌)中复制。
术语“表达构建体”是指这样的遗传构建体:其包括允许转录所述插入多核苷酸分子且任选地将转录物翻译为多肽的必需元件。表达构建体通常在5’至3’方向包含:
a)在宿主细胞(构建体将转化进其中)中具有功能的启动子,
b)待表达的多核苷酸,和
c)在宿主细胞(构建体将转化进其中)中具有功能的终止子。
术语“编码区”或“开放读码框”(orf)是指,能够在适当调控序列控制下生产转录产物和/或多肽的基因组dna序列或cdna序列的有义链。在某些情况下,编码序列可通过5’翻译起始密码子和3’翻译终止密码子的存在来鉴别。当插入遗传构建体中时,“编码序列”在可操作地连接至启动子及终止子序列的情况下能够被表达。
“可操作地连接”是指,将目的序列,如待表达的序列放置在与含有调控元件的另一序列的控制下,并且通常与所述的另一序列连接,所述调控元件可包括启动子、组织特异性调控元件、临时调控元件、增强子、抑制子及终止子、5'-utr序列、包括uorf的5'-utr序列以及uorf。
在一个优选的实施方案中,所述调控元件包括本发明的多核苷酸序列。
优选地,本发明的序列包含5'-utr序列。优选地所述5'-utr序列包含uorf。
术语“非编码区”是指,在翻译起始位点的上游并在翻译终止位点的下游的非翻译序列。这些序列也分别称为5’utr和3’utr。这些区域包括转录起始及终止及调节翻译效率所需的元件。
5'-utr序列位于转录起始位点以及翻译起始位点之间。
5'-utr序列是由基因组dna编码的mrna序列。然而,如本文所用,术语5'-utr序列包括编码5'-utr序列的基因组序列、以及该基因组序列的互补序列、以及5'-utrmrna序列。
终止子为终止转录的序列,且见于翻译序列的下游基因的3’不翻译端。终止子为mrna稳定性的重要决定因素,且在有些情况下,已发现具有空间调节功能。
术语“uorf”或“上游开放读码框”为这样的mrna元件,其在5'-utr由起始密码子(任意三个碱基对密码子,其中至少两个碱基为如下顺序:aug)限定,其中框终止密码子(uaa、uag、uga)主要编码序列的上游(即以5’方向)并且与其不重叠。
术语“启动子”是指,在编码区上游的调节基因转录的顺式调控元件。启动子包含指定转录起始位点的顺式引发元件及保守盒(诸如tata盒),及被转录因子结合的基序。
“转基因”是这样的多核苷酸:其取自一种有机体,且通过转化引入不同的有机体中。转基因可源自与引入该转基因的有机体物种相同的物种或不同的物种。
“转基因植物”是指,含有经遗传操纵或转化而得到的新遗传物质的植物。所述新遗传物质可源自与所得转基因植物相同的物种或不同物种的植物。
“反向重复序列”是存在重复的序列,其中重复序列的另一半是在互补链中,例如
(5’)gatcta…….tagatc(3’)
(3’)ctagat…….atctag(5’)
通读转录将产生这样的转录物:其经历互补碱基配对,以形成发夹结构,条件是,在重复区之间存在3-5碱基对间隔物。
涉及本发明的多核苷酸或多肽的术语“改变...的表达”和“表达改变”旨在包含这种情况:本发明的多核苷酸对应的基因组dna经修饰,从而改变本发明的多核苷酸或多肽的表达。基因组dna的修饰可通过遗传转化或本领域已知引入突变的其它方法。“表达改变”能与信使rna和/或产生的多肽的量的增加或减少相关,且还可由于多核苷酸和所产生多肽的序列改变而导致多肽活性改变。
分离或制备多核苷酸的方法
使用本领域普通技术人员已知的多种技术,可以分离本发明的多核苷酸分子。作为实例,通过使用在mullis等人编,1994版,thepolymerasechainreaction,birkhauser(通过引用并入本文)中所述的聚合酶链式反应(pcr),可以分离这样的多肽。使用源自本发明的多核苷酸序列的如本文所定义的引物,可以扩增本发明的多肽。
用于分离本发明的多核苷酸的其它方法包括:使用具有本文所述的序列的所有或部分多肽作为杂交探针。使标记的多核苷酸探针与固定在固体支持物(诸如硝化纤维素滤膜或尼龙膜)上的多核苷酸杂交的技术,可以用于筛选基因组或cdna文库。示例性的杂交及洗涤条件为:在65℃,在5.0xssc、0.5%十二烷基硫酸钠、1x登哈特溶液中杂交20小时;在1.0xssc、1%(w/v)十二烷基硫酸钠中洗涤(在55℃进行三次各20分钟的洗涤),和任选地在60℃、在0.5×ssc、1%(w/v)十二烷基硫酸钠中洗涤一次(20分钟)。可选的进一步洗涤(20分钟)可在60℃、在0.1×ssc、1%(w/v)十二烷基硫酸钠的条件下进行。
通过本领域中熟知的技术,诸如限制性核酸内切酶消化、寡核苷酸合成及pcr扩增,可以生产本发明的多核苷酸片段。
可在本领域熟知的方法中使用部分多核苷酸序列来鉴别相应的全长多核苷酸序列。这样的方法包括基于pcr的方法、5’race(frohmanma,1993,methodsenzymol.218:340-56)及基于杂交的方法、基于计算机/数据库的方法。此外,作为实例,反向pcr允许获取未知序列,所述序列侧接本文中所公开的多核苷酸序列,从基于已知区域的引物起始(triglia等人,1998,nucleicacidsres16,8186,通过引用并入本文)。该方法使用若干限制酶,以产生在基因的已知区域中的合适片段。随后通过分子内连接环化该片段,且将其用作pcr模板。从已知区域设计不同引物。为了以物理方式装配全长克隆,可利用标准分子生物学方法(sambrook等,molecularcloning:alaboratorymanual,第2版,coldspringharborpress,1987)。
当从特定物种生产转基因植物时,有益地用源自该物种的一个或多个序列转化这样的植物。所述益处可以是,减少公众对于生产转基因有机体中的跨物种转化的关注。另外,当基因减量调节是希望的结果时,可能必须利用与需要减少其表达的植物中的序列相同(或至少高度相似)的序列。尤其出于这些原因,希望能够在若干不同植物物种中鉴别及分离特定基因的直系同源物。
变体(包括直系同源物)可通过所述方法来鉴别。
鉴别变体的方法
物理方法
变体多肽可使用基于pcr的方法来鉴别(mullis等人编,1994thepolymerasechainreaction,birkhauser)。通常,引物的多核苷酸序列用于通过pcr扩增本发明的多核苷酸分子的变体,所述引物的多核苷酸序列可以是基于编码相应氨基酸序列的保守区的序列。
或者,可使用本领域技术人员熟知的文库筛选法(sambrook等人,molecularcloning:alaboratorymanual,第2版,coldspringharborpress,1987)。当鉴别探针序列的变体时,通常相对地降低杂交和/或洗涤严谨性,直至找到确切序列匹配。
也可通过物理方法鉴别多肽变体,例如使用针对本发明多肽产生的抗体筛选表达文库(sambrook等人,molecularcloning:alaboratorymanual,第2版,coldspringharborpress,1987),或借助于这样的抗体鉴别来自天然来源的多肽。
基于计算机的方法
通过本领域技术人员熟知的基于计算机的方法,使用公共域序列比对算法及用于搜寻序列数据库的序列相似性搜寻工具(公共域数据库包括genbank、embl、swiss-prot、pir和其它),也可以鉴别本发明的变体序列(包括多核苷酸及多肽变体)。关于在线资源的实例,参见,例如,nucleicacidsres.29:1-10和11-16,2001。相似性搜寻会检索及比对目标序列,以供与待分析的序列(即,查询序列)进行比较。序列比较算法使用计分矩阵来为每一比对指派总分。
可用于鉴别序列数据库中的变体的程序的一个示例性家族为blast程序套件(2.2.5版[2002年11月]),包括blastn、blastp、blastx、tblastn和tblastx,它们可从(ftp://ftp.ncbi.nih.gov/blast/)或国家生物技术信息中心(nationalcenterforbiotechnologyinformation,ncbi)、国立医学图书馆(nationallibraryofmedicine,building38a,room8n805,bethesda,md20894usa)公开获得。ncbi服务器也提供了使用程序来筛选许多可公开获得的序列数据库的设施。blastn对照核苷酸序列数据库比较核苷酸查询序列。blastp对照蛋白序列数据库比较氨基酸查询序列。blastx对照蛋白序列数据库比较在所有阅读框架中翻译的核苷酸查询序列。tblastn对照核苷酸序列数据库比较在所有阅读框架中动态翻译的蛋白查询序列。tblastx对照核苷酸序列数据库的六-框架翻译物比较核苷酸查询序列的六-框架翻译物。blast程序可以预设参数来使用,或可视需要改变参数以改进筛选。
算法的blast家族(包括blastn、blastp及blastx)的应用,描述于altschul等人,nucleicacidsres.25:3389-3402,1997的出版物中。
通过blastn、blastp、blastx、tblastn、tblastx或相似算法产生的查询序列对一个或多个数据库序列的“命中”,会比对及鉴别序列的相似部分。以相似性程度及序列重叠部分的长度的顺序,排列命中。命中一个数据库序列,一般表示仅在查询序列的一小部分序列长度上具有重叠。
blastn、blastp、blastx、tblastn及tblastx算法也产生比对的“预期”值。预期值(e)指示,当搜寻含有随机邻接序列的相同尺寸的数据库时,可“预期”偶然见到的命中数目。预期值系用作判定命中数据库是否表明真正相似性的有效阈值。例如,指派给多核苷酸命中的0.1的e值解释为是指,在所筛选数据库的尺寸的数据库中,可能预期在具有相似分数的序列的比对部分上仅偶然见到0.1匹配。对于在比对及匹配部分上具有0.01或小于0.01的e值的序列而言,使用blastn、blastp、blastx、tblastn或tblastx算法发现数据库中偶然匹配的机率为1%或1%以下。
可以用clustalw(thompson,j.d.,higgins,d.g.andgibson,t.j.(1994)clustalw:improvingthesensitivityofprogressivemultiplesequencealignmentthroughsequenceweighting,positions-specificgappenaltiesandweightmatrixchoice.nucleicacidsresearch,22:4673-4680,http://www-igbmc.u-strasbg.fr/bioinfo/clustalw/top.html)或t-coffee(cedricnotredame,desmondg.higgins,jaapheringa,t-coffee:anovelmethodforfastandaccuratemultiplesequencealignment,j.mol.biol.(2000)302:205-217)),或使用渐进成对比对的pileup(feng和doolittle,1987,j.mol.evol.25,351),进行一组相关序列的多重序列比对。
可利用图样识别软件应用来找到基序或标签序列。例如,meme(用于基序引出的多个em)在一组序列中找到基序及标签序列,且mast(基序比对及搜寻工具)使用这些基序在查询序列中鉴别相似或相同基序。提供mast结果作为与适当统计资料及所找到基序的目视全览的一系列比对。在圣地亚哥的加利福尼亚大学(universityofcalifornia,sandiego)开发出meme和mast。
prosite(bairoch和bucher,1994,nucleicacidsres.22,3583;hofmann等人,1999,nucleicacidsres.27,215)是鉴别从基因组或cdna序列翻译的未表征蛋白的功能的方法。prosite数据库(www.expasy.org/prosite)含有生物学显著图样及特性,且被设计成使得其可与适当计算工具一起使用,以向已知蛋白家族指派新序列或判定哪个已知域存在于该序列中(falquet等人,2002,nucleicacidsres.30,235)。prosearch是可以用既定序列图样或标签搜寻swiss-prot及embl数据库的工具。
分离多肽的方法
本发明的多肽(包括变体多肽)可使用本领域中熟知的肽合成方法来制备,诸如使用固相技术进行直接肽合成(例如stewart等人,1969,solid-phasepeptidesynthesis,whfreemanco,sanfranciscocalifornia),或(例如)使用appliedbiosystems431a肽合成仪(fostercity,california)进行自动合成。多肽的突变形式也可在所述合成期间产生。
本发明的多肽及变体多肽也可使用本领域中熟知的多种技术(例如deutscher编,1990,methodsinenzymology,第182卷,guidetoproteinpurification)从天然来源纯化。
或者,本发明的多肽及变体多肽可在合适的宿主细胞中重组表达,并与细胞分离,如下文所论述。
修饰序列的方法
对蛋白质序列、或编码所述蛋白质序列的多核苷酸序列进行修饰的方法是本领域技术人员熟知的。可以通过改变/修饰编码蛋白质的序列并表达经修饰的蛋白质来方便地修饰蛋白质序列。诸如定点突变等方法也可以用于修饰现有的多核苷酸序列。或者,可使用限制性内切酶来切割现有序列的部分。改变的多核苷酸序列也可以方便地以修饰的形式表达。
制备构建体及载体的方法
本发明的遗传构建体包含一个或多个本发明的多核苷酸序列和/或编码本发明的多肽的多核苷酸,且可用于转化例如细菌、真菌、昆虫、哺乳动物或植物有机体。本发明的遗传构建体意欲包括如本文中所定义的表达构建体。
生产及使用遗传构建体及载体的方法是本领域中熟知的,且一般描述于:sambrook等人,molecularcloning:alaboratorymanual,第2版,coldspringharborpress,1987;ausube1等人,currentprot
生产包含多核苷酸、构建体或载体的宿主细胞的方法
本发明提供了一种宿主细胞,其包含本发明的遗传构建体或载体。宿主细胞可以来源于例如细菌、真菌、昆虫、哺乳动物或植物有机体。
包含本发明的遗传构建体(诸如表达构建体)的宿主细胞可用于本领域熟知的方法中(例如sambrook等人,molecularcloning:alaboratorymanual,第2版coldspringharborpress,1987;ausubel等人,currentprotocolsinmolecularbiology,greenepublishing,1987),用于重组生产本发明的多肽。这样的方法可能包括,在适用于或有助于表达本发明的多肽的条件下,在适当介质中培养宿主细胞。随后,通过本领域熟知的方法(例如deutscher编,1990,methodsinenzymology,第182卷,guidetoproteinpurification),可以将表达的重组多肽(其可以任选地分泌至培养物中)与介质、宿主细胞或培养基分离。
生产包含构建体及载体的植物细胞及植物的方法
本发明另外提供了包含本发明的遗传构建体的植物细胞,及经修饰以改变本发明多核苷酸或多肽的表达的植物细胞。包含这样的细胞的植物也形成本发明的一个方面。
用多肽转化植物细胞、植物及其部分的方法,参见:draper等人,1988,plantgenetic转化andgeneexpression.alaboratorymanual.blackwellsci.pub.oxford,第365页;potrykusandspangenburg,1995,genetransfertoplants.springer-verlag,berlin.;和gelvin等人,1993,plantmolecularbiol.manual.kluweracad.pub.dordrecht。对转基因植物(包括转化技术)的综述,参见:galun和breiman,1997,transgenicplants.imperialcollegepress,london。
植物的遗传操纵方法
可利用许多植物转化策略(例如,birch,1997,annrevplantphysplantmolbiol,48,297,hellensrp,等人(2000)plantmolbiol42:819-32,hellensr等人plantmeth1:13)。例如,策略可设计成增加多核苷酸/多肽在通常表达所述多核苷酸/多肽的植物细胞、器官中和/或在特定发育阶段的表达,或在通常不表达所述多核苷酸/多肽的细胞、组织、器官中和/或在特定发育阶段异位表达所述多核苷酸/多肽。表达的多核苷酸/多肽可源自待转化的植物物种,或可源自不同植物物种。
转化策略可设计成降低多核苷酸/多肽在通常表达所述多核苷酸/多肽的植物细胞、组织、器官中或在特定发育阶段的表达。这样的策略称为基因沉默策略。
用于在转基因植物中表达基因的遗传构建体通常包括:用于驱动一个或多个克隆的多核苷酸的表达的启动子,终止子,及用于检测遗传构建体在转化植物中的存在的选择标记序列。
适用于本发明的构建体中的启动子在单子叶植物或双子叶植物的细胞、组织或器官中具有功能,且包括细胞特异性的、组织特异性的及器官特异性的启动子、细胞周期特异性的启动子、时间启动子、诱导型启动子、在大多数植物组织中具有活性的组成型启动子、及重组启动子。在必要时,启动子的选择将取决于克隆的多核苷酸的时间及空间表达。启动子可为通常与目标转基因有关的启动子,或源自其它植物、病毒及植物病原性细菌及真菌的基因的启动子。本领域技术人员无需过多试验就能够选出适用于使用包含本发明的多核苷酸序列的遗传构建体改良及调节植物特性的启动子。组成型植物启动子的实例包括:camv35s启动子、胆脂碱合酶启动子及章鱼碱合酶启动子、及来自玉蜀黍的ubi1启动子。在特定组织中具有活性的植物启动子,会对内部发育信号或外部非生物的或生物的应激做出响应,这描述于科学文献中。示例性的启动子描述于例如wo02/00894中,该文献通过引用并入本文中。
常用于植物转化遗传构建体中的示例性的终止子包括,例如,花椰菜花叶病毒(camv)35s终止子、根瘤土壤杆菌(agrobacteriumtumefaciens)胆脂碱合酶或章鱼碱合酶终止子、玉蜀黍zein基因终止子、水稻(oryzasativa)adp-葡萄糖焦磷酸化酶终止子及马铃薯pi-ii终止子。
常用于植物转化中的选择标记包括:赋予卡那霉素抗性的新霉素磷酸转移酶ii基因(nptii)、赋予壮观霉素及链霉素抗性的aada基因、赋予ignite(agrevo)及basta(hoechst)抗性的草胺膦乙酰转移酶(bar基因)及赋予潮霉素抗性的潮霉素磷酸转移酶基因(hpt)。
也预见到遗传构建体的应用,所述遗传构建体包含可用于植物及植物组织中的启动子表达分析的报告基因(表达对于宿主而言外来的活性、通常为酶活性和/或可见信号(例如荧光素酶、gus、gfp)的编码序列)。报告基因文献论述于herrera-estrella等人,1993,nature303,209,和schrott,1995,见:genetransfertoplants(potrykus,t.,spangenberg编)springerverlag.berline,第325-336页。
关于基因沉默策略,人们关注基因本身或影响编码多肽表达的调节元件。本文所用的“调节元件”具有最广泛的可能的意义,并且包括与目标基因相互作用的其它基因。
设计减少或沉默本发明所述的多核苷酸/多肽表达的遗传构建体可包括本发明所述多核苷酸的反义拷贝。在这种构建体内,多核苷酸被置于与启动子和终止子反义的方向上。
“反义”多核苷酸通过使多核苷酸或多核苷酸的片段倒置,使得产生的转录本与该基因的mrna转录本互补而获得,例如
5’gatcta3’(编码链)3’ctagat5’(反义链)
3’cuagau5’mrna5’gaucucg3’反义rna
设计用于基因沉默的遗传构建体还包括倒置重复序列。“倒置重复序列”是其中重复的第二半部分在互补链上的重复序列,例如:
5’-gatcta………tagatc-3’
3’-ctagat………atctag-5’
形成的转录本可进行互补性碱基配对以形成发卡结构。通常,需要重复区域间至少3-5bp的间隔物来形成发卡。
另一种沉默方法包括使用靶向等同于mirna的转录本的小反义rna(llave等,2002,science297,2053)。使用这种与本发明的多核苷酸对应的小反义rna是明确包含的。
如本文所用的术语遗传构建体也包括小反义rna和影响基因沉默的其它这样的多肽。
以本文所定义的表达构建体进行转化也可通过称之为正义抑制的过程导致基因沉默(例如,napoli等,1990,plantcell2,279;decarvalhoniebel等,1995,plantcell,7,347)。在某些情况下,正义抑制可包括完整或部分编码序列过表达,还可包括基因的非编码区的表达,基因的非编码区例如为内含子或5’或3’非翻译区(utr)。嵌合的部分正义构建体能够协同用于使多个基因沉默(abbott等,2002,plantphysiol.128(3):844-53;jones等,1998,planta204:499-505)。使用这种正义抑制策略来使本发明的多核苷酸的表达沉默也是包含在本文中。
插在设计用于基因沉默的遗传构建体中的多核苷酸可对应于编码序列和/或非编码序列,如启动子和/或内含子和/或5’或3’-utr序列、或对应的基因。
其它基因沉默的策略包括显性抑制(dominantnegative)方法和使用核酸酶构建体(mcintyre,1996,transgenicres,5,257)。
转录前沉默可通过基因本身或其调节元件的突变而产生。这种突变可包括点突变、移码、插入、缺失或置换。
下面是公开了可用于遗传转化以下植物物种的遗传转化方案的代表性出版物:稻米(alam等,1999,plantcellrep.18,572);苹果(yao等,1995,plantcellreports14,407-412);玉米(美国专利第5,177,010号及第5,981,840号);小麦(ortiz等,1996,plantcellrep.15,1996,877);西红柿(美国专利第5,159,135号)、马铃薯(kumar等人,1996plantj.9,:821);木薯(li等,1996nat.biotechnology14,736);莴苣(michelmore等,1987,plantcellrep.6,439);烟草(horsch等,1985,science227,1229);棉花(美国专利第5,846,797号及第5,004,863号)、草类(美国专利第5,187,073号及第6.020,539号);胡椒薄荷(niu等人,1998,plantcellrep.17,165);橘类植物(pena等,1995,plantsci.104,183);香菜(krens等,1997,plantcellrep,17,39);香蕉(美国专利第5,792,935号);大豆(美国专利第5,416,011号、第5,569,834号、第5,824,877号、第5,563,04455号及第5,968,830);菠萝(美国专利第5,952,543号);杨树(美国专利第4,795,855号);单子叶统称(美国专利第5,591,616号及第6,037,522号);芸苔属(美国专利第5,188,958号、第5,463,174号及第5,750,871号);谷类(美国专利第6,074,877号);梨(matsuda等,2005,plantcellrep.24(1):45-51);李属(ramesh等,2006plantcellrep.25(8):821-8;songandsink2005plantcellrep.2006;25(2):117-23;gonzalezpadilla等,2003plantcellrep.22(1):38-45);草莓(oosumi等,2006planta.223(6):1219-30;folta等,2006plantaapr14;pmid:16614818)、玫瑰(li等,2003)、树莓(graham等,1995methodsmolbiol.1995;44:129-33)、西红柿(dan等,2006,plantcellreportsv25:432-441)、苹果(yao等,1995,plantcellrep.14,407–412)及毛花猕猴桃(wang等,2006,plantcellrep.25,5:425-31)。本发明也涵盖其它物种的转化。合适的方法及方案可获自科学文献中。
可使用本领域已知的几种其它方法来改变本发明的核苷酸和/或多肽的表达。这些方法包括但不限于tilling(till等,2003,methodsmolbiol,2%,205)所谓的“deletagene”技术(li等,2001,plantjournal27(3),235)、以及使用人工转录因子如合成锌指转录因子(例如,jouvenot等,2003,genetherapy10,513)。另外,靶向特定多肽的抗体或其片段也可在植物中表达来调节那种多肽的活性(jobling等,2003,nat.biotechnol.,21(1),35)。还可使用转座子示踪法。此外,与本发明的多肽相互作用的肽可通过如相显示(phase-display)(dyax公司)等技术来鉴定。这种相互作用的肽可在植物中表达或应用于植物以影响本发明的多肽的活性。改变本发明的核苷酸和/或多肽表达的上述方法中每一种方法的使用也特别地包含在本文中。
在植物中修饰内源dna序列的方法
在植物中修饰内源基因组dna序列的方法是本领域人员已知的。这些方法可包括使用序列特异性核酸酶,其在目标基因中产生靶向双链dna断裂。用于植物的这些方法可包括:锌指核酸酶(curtin等,2011.plantphysiol.156:466–473.;sander,等,2011.nat.methods8:67–69.)、转录激活样效应因子核酸酶或“talens”(cermak等,2011,nucleicacidsres.39:e82;mahfouz等,2011proc.natl.acad.sci.usa108:2623–2628;li等,2012nat.biotechnol.30:390–392)、以及laglidadg归位内切酶,也称为“巨核酶”(tzfira等,2012.plantbiotechnol.j.10:373–389)。
在本发明的一些实施方案中,这些技术中的一者(如talen或锌指核酸酶)可用于改性uorf中的一个或多个碱基对以使其失去能力,从而不能翻译。
在一个实施方案中,acg起始密码子的第一个碱基对变为tcg以实现该过程。这将使抗坏血酸对ggp翻译的反馈调控失去活性,并允许植物中抗坏血酸浓度升高。
或者,uorf中高度保守的氨基酸的密码子可改变以在抗坏血酸浓度高时停止uorf下调ggp翻译。例如,在uorf的保守区域中的组氨酸残基可以变为亮氨酸。
在另一个实施方案中,将uorf的早期碱基对改变以引入终止密码子,从而使uorf在早期终止,从而停止抗坏血酸对ggp翻译的反馈调控。
因此,本领域技术人员能够理解存在众多方式来干扰uorf以除去抗坏血酸引起的负向调控以及提高抗坏血酸的产生。任意这样的方法包括在本发明的范围中。
植物
术语“植物”意欲包括整个植物、植物的任何部分、植物的繁殖体及子代。
术语“繁殖体”是指植物中可用于繁殖或增殖的任何部分,其为有性或无性的,包括种子及插枝。
可培养本发明的植物,并自交或与不同植物品系杂交,且由两代或更多代得到的后代也构成了本发明的一个方面,条件是它们保留本发明的转基因或修饰。
从植物中提取和测定抗坏血酸的方法
还提供了通过从本发明的植物中提取抗坏血酸,以生产抗坏血酸的方法。可按照如下方式从植物中提取抗坏血酸:
将冰冻组织样品在cryomill中在液氮温度下研碎成精细的粉末。然后将约200mg冰冻粉末组织用5倍体积的含2mmtcep(pierce公司)的7%偏磷酸中悬浮,振荡混合20秒,并在40℃在加热块中孵育2小时。在提取溶液中使用tcep是因为其在酸性条件下是比dtt更有效的还原试剂,确保所有维生素c是抗坏血酸的还原形式。提取物在4℃离心,且将20μl上清液注射入rocket柱内,并用两种溶剂a(0.28%正磷酸,0.1mmedta和0.25%甲醇)和b(乙腈)洗脱。使用5分钟梯度(直至90%b)洗脱抗坏血酸及其它组分。每批或处理20个样品后运行标准试样。根据在240nm处洗脱1分钟的吸收曲线下方的面积计算抗坏血酸。
该方法可使用本领域人员熟知的方法为大规模抗坏血酸提取扩大规模。
本发明还可以宽泛得解释为包括本申请说明书中分别或一起提及或所提示的各部分、元素和特征,和任意两个或更多个所述部分、元素或特征的任一种或所有组合,并且在本文中提及特定整体的情况下,所述整体具有本发明所涉及领域内的已知等同物,这种已知的等同物被认为是引入本文中,好像其被独立地阐述那样。
附图简要说明
参考下列附图可更好地理解本发明,其中:
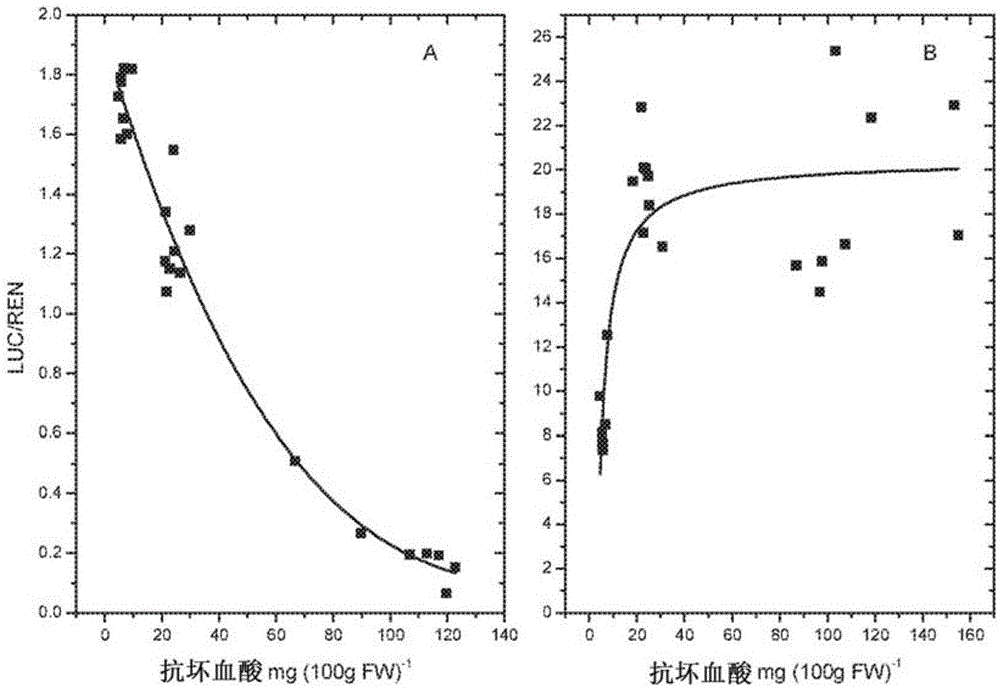
图1示出了抗坏血酸对由ggp启动子或对照启动子驱动的报告基因活性的影响。a:对于ggp启动子,luc/ren比作为叶抗坏血酸浓度的函数;b:对于tt8启动子,luc/ren比作为叶抗坏血酸浓度的函数。▲低抗坏血酸(ko)叶,■对照叶,○高抗坏血酸(ggp)叶。
图2示出了在ggp基因的5'-utr中存在的acguorf的各种改变形式下,抗坏血酸对ggp启动子强度的影响(图s7b)。a:luc/ren比;b:相同处理中抗坏血酸的浓度。vtc2是野生型ggp5'-utr和启动子。acg1是acg1的启动acg变为tcg,不再为起始密码子。acg2是在uorf的高保守区域(图s5、s7b)中第一个组氨酸his变为亮氨酸leu。加入ggp以操纵抗坏血酸浓度。在a中,vtc2+ggp处理比所有其它处理(差异不显著)具有显著更低的luc/ren值(p=0.001)。在b中,向叶中加入ggp显著提高了抗坏血酸(p=0.001),但是高或低抗坏血酸的处理之间的差异不显著。棒为标准误差,n=4。
图3示出了检测ggp5'-utr的非典型uorf是以顺式还是反式的方式作用的结果。vtc2wt是指抗坏血酸阻遏启动子和5'-utr,acg1指uorf的突变acg密码子(无响应),uorf是指添加了由35s驱动子驱动的uorf并且ggp是指添加了也由35s驱动的ggp编码序列以提高抗坏血酸。
图4示出了对于来自猕猴桃和拟南芥的ggp全长启动子构建体,报告基因对抗坏血酸的反应的比较。ggp用于如图1所示提高抗坏血酸的水平。■拟南芥ggp(启动子和5'-utr),□标准猕猴桃ggp(启动子和5'-utr)。其他的细节可见方法部分。
图5示出了在瞬时转化的烟叶中用抗luc抗体测量(由ggp启动子或对照tt8启动子驱动),高抗坏血酸浓度对luc蛋白的量的影响。如图1所示的那样进行试验。在319998(ggp)泳道中抗坏血酸的浓度为55至63mg/100gfw,而没有ggp的泳道中抗坏血酸浓度为21mg/100gfw。其他的细节可见方法部分。
图6示出了独立于提高叶抗坏血酸的ggp酶,在不同的ggp水平下通过使用进一步提高抗坏血酸的gme来操纵抗坏血酸浓度的效果。a:ggp和gme的不同组合对luc/ren比的影响;b:ggp和gme的不同组合对抗坏血酸浓度的影响。在a和b中,具有相同的字母的柱在5%水平无显著差异。c:单个叶片luc/ren和叶抗坏血酸之间的关系。其他的细节可见方法部分。在c中,■,对照;●,gme;◆,0.33xggp;▲,1xggp;□,0.33xggp+1xgme;▼,1xggp+gme。1和0.33指注入叶中的ggp和gme的相对量。
图7示出了对来自一系列双子叶物种的5'-utr序列的比对。使用clustalx完成比对,用vectornti执行。后缀字母表示5'-utr序列来自的物种:aa为软枣猕猴桃,ae为毛花猕猴桃并且ac为中华猕猴桃,mxd为苹果,fv为野草莓,sl为番茄,st为马铃薯,vv为葡萄,gm为大豆,mt为蒺藜苜蓿,pt为毛果杨,cs为黄瓜。各名之前的前缀为genbank登陆号。两种基因组序列来自公开的来源(t.p.g.s.consortium,2011nature475,189);velasco等,2010natgenet42,833)。以atg起始的短uorf为粗体,而以acg起始的高度保守的非典型uorf用下划线粗体(acg1)示出。各序列括号内的数字表示其5'-utr的总长度。下方的图示出了5'-utr的高保守区域以及用于检测它们功能的三个缺失。着色区域为高保守区域,其单独删除(从左至右缺失2和3)或是完整删除(缺失1)。黄色基序为以atg起始的小的保守uorf(atg1),而橙色基序为以acg起始的两个非典型uorf(acg1底部、acg2顶部)。所有3个uorf在物种间保守,而acg2(家族间同一性为~40%至~60%)在蛋白质水平方面比acg1(~60%至~80%)保守性差。
图8示出了ggp基因5'-utr区域的缺失对抗坏血酸下调启动子的影响。缺失如图s4标记。底部图示出了测量luc/ren比时相同叶中对应的抗坏血酸浓度。棒表示标准误差。
图9示出了ggpcds前方5'-utr存在或缺失对叶抗坏血酸的影响的时间过程。所有构建体由35s启动子驱动。其他的细节可见方法部分。■,对照(仅p19);●,-5'-utrggp;▲,+5'-utrggp;▼,-5'-utrggp+gme;◆,+5'-utrggp;○,gme。
图10示出了对来自一大系列物种的ggp的5'-utr中的acguorf的预测肽序列的比对。用clustalx完成比对,用vectornti执行(thompson等,nucleicacidsres25,4876(1997).)。后缀字母如图7所示,还增加了双子叶植物植物之外的植物。cr,莱茵衣藻(chlamydomonasrheinhardtii);pp,小立碗藓(physcomitrellapatens);ps,北美云杉(piceasitchensis);sm,江南卷柏(selaginellamoellendorffi);zm,玉米。各名之前的前缀为genbank登陆号。还示出了完整uorf的共有序列。此外标记下划线的为高度保守的共有基序npsphggrgalpseggspsdllflaggg(seqidno:108)。
图11示出了小atguorf对报告基因的活性对抗坏血酸浓度反应的影响,报告基因的活性由ggp启动子和5'-utr驱动。5'-utr中小的30bporf的起始密码子通过将起始的atg变为ttg而失活。□,失活uorf;■,uorf起始密码子存在。在该试验以及重复试验中,受到不具有起始密码子的5'-utr控制的报告基因的活性在低和高的抗坏血酸浓度下的表达都高于uorf完整的基因。实心线为对数据的多项拟合。其他的细节可见方法部分。
图12示出了抗坏血酸的还原状态作为叶中的总抗坏血酸的函数。这些数据来自三个独立的试验。使用ggp操纵抗坏血酸,用还原剂和不用还原剂测量。当抗坏血酸浓度分为低(27±0.7mg/100gfw)、中(59±2.7)和高(179±17.5)(平均值±标准误差)时,氧化还原电势值显著不同并且随抗坏血酸的升高而下降:74.8±1.0、70.1±1.2和65.6±0.8(p=0.05)。实线是还原%数据的双曲线拟合,而虚线是对氧化还原电势的线性拟合。
图13示出了抗坏血酸对报告基因活性的影响,报告基因的活性受到ggp启动子或对照启动子驱动。a,对ggp启动子,luc活性对比ren活性;b,对tt8启动子,luc活性对比ren活性。在a中的三个斜率差异显著(p<0.001),而在b中只有低抗坏血酸斜率明显(p<0.001)比斜率差异不显著的其他两条线的斜率更低。
图14示出了基于pgreen0800的报告基因构建体的位置的图,所述构建体被设计用于检测来源于不同物种的ggp的5’utr。
图15示出了来源于马铃薯、番茄和苹果的ggp基因的5’utr序列。uorf用粗体示出。
图16示出了对番茄5’utr而言,高(+319998)和低抗坏血酸下luc值对ren的图。
图17示出了对马铃薯5’utr而言,高(+319998)和低抗坏血酸下luc值对ren的图。
图18示出了对苹果5’utr而言,高(+319998)和低抗坏血酸下luc值对ren的图。
图19示出了对于对照启动子-5’utr-luc构建体而言,高(+319998)和低抗坏血酸下luc值对ren的图。
图20示出了对于野生型猕猴桃ggp启动子-5’utr-luc构建体而言,高(+319998)和低抗坏血酸下luc值对ren的图。
例子
现在参考下列非限制性例子来说明本发明。
不能认为本发明的范围仅限于上述的例子。本领域技术人员能够理解,在不脱离本发明的范围的情况下可对本发明进行多种变形。
实施例1:对ggp的表达控制的阐明
概要
抗坏血酸(维生素c)是植物和动物体内必需的抗氧化剂和辅酶因子。在植物中抗坏血酸浓度受到密切调节,部分地对应激反应。申请人已经示出了通过gdp-l-半乳糖磷酸化酶(ggp)的转录后阻遏来控制抗坏血酸浓度,ggp是抗坏血酸生物合成途径中的限速酶。该调节要求顺式作用的uorf(上游开放读码框)的翻译,其由非典型的起始密码子启动并在高抗坏血酸浓度下阻遏下游ggporf的翻译。除去该uorf允许植物产生高浓度的抗坏血酸。更高级和低级的植物中ggp基因中uorf的存在表明其是控制抗坏血酸的一种古老的机制。
抗坏血酸(维生素c)是在大部分生物体中发现的必需生物化学品,其主要作用在于控制细胞的氧化还原电势(asensi-fabado等,2010,trendsplantsci.15,582;foyer等,plantphysiol.155,2(2011))以及作为酶辅因子(mandl等,2009,br.j.pharmacol.157,1097)。根据需要调节抗坏血酸的浓度;例如,在高光强度下对抗坏血酸的需要最高时,叶抗坏血酸浓度升高(bartoli等,2006,j.exp.bot.57,1621;gatzek等,2002,plantj.30,541)。然而,如何调节抗坏血酸生物合成的机制是未知的。申请人之前已经示出了酶gdpl-半乳糖磷酸化酶(ggp)对决定植物中抗坏血酸是重要的(bulley等,2012,plantbiotechnolj10,390;bulley等,2009,j.exp.bot.60,765),表明其可能具有调节作用。
结果和讨论
为了研究ggp基因是否受到抗坏血酸水平的调节,申请人将猕猴桃ggp启动子及其5'-utr(seqidno:101)与荧光素酶(luc)报告基因融合,并在本氏烟(nicotianabenthamiana)叶中瞬时表达该构建体(hellens等,2005,plantmethods1,13.)。申请人也在强的组成型启动子下仅表达ggp编码序列来操纵抗坏血酸。抗坏血酸浓度从2mm(20mg/100gfw)翻倍到4mm足以将相对luc活性降低50%,并且当抗坏血酸升高至接近10mm时,>90%的luc活性被破坏(图1)。类似地,拟南芥ggp(vtc2;at4g26850)启动子和5'-utr(seqidno:102)也对luc报告基因赋予抗坏血酸依赖性的阻遏(图4)。
相反,使用与抗坏血酸代谢不相关的基因的对照启动子(tt8:控制多酚生物合成的拟南芥bhlh转录因子),高抗坏血酸对相对luc活性无影响(图1)。其它的对照示出该调控对ggp序列是特异的,与转基因的表达水平无关,并且由luc蛋白的改变体现(表1-3,图5)。
表1:luc或ren的绝对值不显著影响luc/ren比。luc和ren之间的关系在超过200倍的范围内是线性的。下标p是指整个启动子,包括得自该基因的任意5'-utr基因。其他的细节可见方法部分。
表2:抗坏血酸对一系列对照基因的效果比较。+ggp是指为了提高抗坏血酸,共转化处于35s启动子控制下的来源于猕猴桃的ggpcds(genbank登录号fg528585)。lucren斜率是luc值相对于ren值的绘图强制通过原点的斜率,以作为与luc/ren比的比较。n是独立luc/ren比的测量次数。下标p是指整个启动子,包括得自该基因的任意5'-utr基因。在一组两行内,具有相同字母的luc/ren值没有显著差异p<0.01。其他的细节可见方法部分。
3.对用来操纵抗坏血酸浓度的基因添加对照基因的影响。为了检测额外基因(ggp)的表达增加抗坏血酸并不直接影响luc/ren比,我们用另一个对抗坏血酸浓度无直接影响的对照基因进行置换(genbank登录号fg429343),猕猴桃甲基转移酶。下标p是指整个启动子。在一组三行内,具有相同字母的luc/ren值没有显著差异p<0.01。其他的细节可见方法部分。
在所述的试验中,通过ggp编码序列的表达变化操纵叶抗坏血酸。为了将抗坏血酸的影响与ggp蛋白的可能影响分开(
为了检测抗坏血酸的影响是否是通过非转录的启动子或5’-utr介导,申请人进行了两个试验。首先,申请人在ggptt8启动子之间交换5'-utr。我们在叶中瞬时表达这些物质并测量相对luc活性。仅在ggp的5'-utr存在时,升高的抗坏血酸降低luc活性(表4)。其次,申请人删除了5'-utr中物种间在dna水平高度保守的两个区域。第一个是从387到432bp,并且第二个是从514到597bp(图7),而ggp启动子的其余部分不变,并且使用报告分析对它们进行检测。所有的缺失引起抗坏血酸对报告基因表达下调的能力的损失(图8)。这些试验表明,5’-utr是抗坏血酸实现下调所必要的和充分地。
表4:抗坏血酸下调ggp启动子通过该基因的5'-utr区进行表达。下标utr指基因的5'-utr,并且下标p’是指相应基因的非转录启动子。ggp是指共转化了35s启动子控制下的ggpcds从而提高抗坏血酸水平,而fg429343为甲基转移酶对照基因。lucren斜率是luc值相对于ren值的绘图强制通过原点的斜率。n是独立luc/ren比的测量次数。在一组三行内,具有相同字母的luc/ren值没有显著差异p<0.01。其他的细节可见方法部分。
为了研究抗坏血酸控制是在转录水平还是在转录后水平,申请人测量了报告基因构建体的转录水平。我们的数据示出抗坏血酸对lucmrna水平的影响不大(表5),表明抗坏血酸直接地或间接地通过5'-utr控制ggp的翻译起作用。
表5:抗坏血酸水平对lucrna水平的影响(通过ggp启动子或tt8对照基因驱动)。在相同的rna制备品中,相对于ren的表达测量基因表达。所得值为三个生物学重复的平均值,每个包括三个合并的叶。标准误差在括号中表示。在每个启动子对内,对于tt8启动子,基因表达或luc活性没有显著差异。对于ggp启动子,luc活性的改变是显著的(p<0.001),如同两种启动子的抗坏血酸改变(p<0.003)那样。
为了验证5'-utr直接起作用以影响叶抗坏血酸浓度,我们构建了35s启动子驱动的ggp编码序列,在该编码序列前方具有或不具有ggp5'-utr。两种构建体都能在瞬时体系中增强叶抗坏血酸,但是不含5'-utr的构建体比具有5'-utr的构建体多约30%的抗坏血酸(图9)。此外,将gme共渗透入叶中从而使抗坏血酸比仅由ggp还高(bulley等,2009,j.exp.bot.60,765.),这使得在不含5'-utr的构建体的情况下抗坏血酸浓度是含5'-utr的构建体的两倍。因此,在高抗坏血酸情况下,ggp5'-utr同时限制了ggp的产生和抗坏血酸的合成。除去该调控提供了一种制备具有高抗坏血酸水平的植物的方式。
考虑到抗坏血酸的影响是通过ggp的5'-utr区介导的,我们研究了5'-utr的性能。在具有严格保守元件的许多物种中,ggp是不同寻常的,具有超过500bp的长5'-utr(图7)。比较来自不同物种(包括藻类和苔藓)的ggp5'-utr,示出了存在高度保守的uorf,其可能编码60-65个氨基酸的肽(图10)。有趣的是,为了制备该肽,需要在非典型acg起始密码子启动翻译。已经描述了多个非典型翻译起始的例子(ivanov等,2008,proc.natl.acad.sci.usa105,10079)。高效的翻译需要kozak序列,对于该orf也是这种情况(图8)。为了检测这种uorf是否是ggp基因的抗坏血酸依赖性调控所需要的,申请人将可能的acg起始密码子突变为tcg。来自突变构建体的luc活性仍然很高,即使在高抗坏血酸的存在下也是如此(图2)。为了进一步检测对uorf的需要,申请人将残基36处的高度保守的his(cgg密码子)突变为leu(ctg)。再次,这破坏了抗坏血酸依赖性调控(图2)。突变内部atg密码子(可能编码10个氨基酸的短uorf)将相对luc活性提高了超过两倍,这可能是因为它移除了竞争的起始密码子,但并没有改变该启动子对抗坏血酸浓度的相对灵敏度(图11)。
申请人然后用对抗坏血酸没有反应的acguorf突变体检测预计的uorf是以顺式还是反式构型起作用。申请人检测了独立表达acguorf是否能在该突变载体中恢复抗坏血酸对luc活性的阻遏。在图3中,申请人示出了acguorf的存在对任何处理无影响,并且不能补偿acguorf的突变形式。这与ggpcds情况下uorf以顺式构象起作用是一致的。
在该工作中,申请人提供的证据表明,抗坏血酸或抗坏血酸的前体通过中间物与由ggp5'-utr中的非典型uorf产生的肽(抗坏血酸生物合成的关键调控基因)直接或间接相互作用,导致对ggp酶的翻译的抑制。在真核细胞中通过生物合成途径的产物控制蛋白表达的报导是罕见的。通常,基因表达是通过借助于单独受体到转录因子的信号级联或通过目标蛋白的翻译后修饰来控制的(smeekens等,2010,curr.opin.plantbiol.13,273)。虽然已经报导5'-utr序列在控制蛋白表达中是重要的(hulzink等,2003,plantphysiol.132,75),但是在真核生物中报告利用小分子通过mrna的5'-utr控制基因表达是罕见的(rahmani等,2009,plantphysiol.150,1356),并且通过非典型起始密码子uorf进行控制是极其罕见的。
一个简单的作用模型可能是acguorf被翻译,但在高抗坏血酸存在时,核糖体停留在uorf。在低抗坏血酸时,翻译在终止密码子终止,并且在ggp的起始atg下游立即重新启动。没有明显的kozak序列与ggp主要起始密码子相关,但有相当强的kozak序列与acg1相关。这有效地引起在高抗坏血酸时,准备进行翻译的ggpmrna水平的核糖体快速应对应激条件下抗坏血酸的任意减少。
似乎该反馈环的作用需要另一个因素。这是因为来源于毛花猕猴桃(具有很高抗坏血酸的猕猴桃物种)的ggp5'-utr(bulley等,2009,j.exp.bot.60,765.)的抗坏血酸调控,在本氏烟中起作用。猕猴桃有高抗坏血酸表明毛花猕猴桃中发生突变干扰了抗坏血酸对ggp翻译的反馈。然而,抗坏血酸对毛花猕猴桃ggp的控制在本氏烟中起作用表明在抗坏血酸和acg1uorf之间介导的因素在本氏烟中起作用。该因素可能是蛋白质。
有两种类型的uorf(tran等,2008.bmcgenomics9,361):序列独立性uorf,其中uorf翻译影响下游orf的重新启动的效率,从而影响整体翻译(calvo等,2009.proc.natl.acad.sci.usa106,7507),但uorf编码的肽序列是不重要的(ggp5'-utr中短atg10氨基酸uorf似乎符合该类(图7)),以及序列依赖的orf,其中新生uorf肽在翻译延伸和终止期间引起核糖体停止。事实上,ggpuorf在很宽范围的植物分类学中编码高度保守的肽,并且通过uorf中的单个氨基酸突变破坏抗坏血酸阻遏,这表明是后一种类型的uorf。植物中的两个例子,多胺和蔗糖调节(rahmani等,2009,plantphysiol.150,1356;gongandpua,2005,plantphysiol.138,276)涉及序列依赖uorf。我们的新的例子的不同之处在于其由高度保守的非典型密码子启动翻译。
总之,我们已经示出,叶片中抗坏血酸的水平可以通过抗坏血酸生物合成的控制基因ggp的长5'-utr中的非典型uorf介导的抗坏血酸反馈来控制。我们的证据表明,这种反馈作用是通过控制ggp酶的水平通过转录后控制起作用的。我们认为这是一个重要的机制,即在抗坏血酸生物合成的l-半乳糖途径控制抗坏血酸浓度。
材料和方法:
植物材料和化学分析
如之前记载,本氏烟叶瞬时报告基因体系使用荧光素酶(luc)作为启动子特异报告因子,以及海肾(ren)作为转化报告因子(hellens等,2005,plantmethods1,13)。通过以下方法操纵叶中抗坏血酸浓度:共注射在pgreen(hellens等,2000,plantmol.biol.42,819)中的中华猕猴桃ggp的编码序列,pgreen受到35s启动子的控制,并且被转化入土壤农杆菌(agrobacteriumtumifaciens)(bulley等,2009,j.exp.bot.60,765.);或是共注射使用来源于本氏烟的ggp序列(由如(snowden等,theplantcell17,746)所述从genbank中的七个est构建)作为模板构建的ko载体。另外,之前所述(bulley等,2009,j.exp.bot.60,765.)的毛花猕猴桃gme(genbank登录号fg424114)的cds版本用于与ggp一起协同增强抗坏血酸。还如前所述,通过基于hplc的分析法测定相同叶子的提取物中的抗坏血酸(rassam等,2005,j.agric.foodchem.53,2322)。通过在hplc之前还原提取物测定抗坏血酸作为总抗坏血酸(rassam等,2005,j.agric.foodchem.53,2322)。对抗坏血酸氧化还原状态的测量发现,随着抗坏血酸浓度的增加,氧化还原电势显著降低(图12)。这就提出了一个可能性,抗坏血酸的影响可能是通过抗坏血酸本身或通过抗坏血酸升高时氧化还原电势降低而进行的。
图13示出了其中每个点(两到三个测量的平均值)表示本氏烟的不同叶的典型试验,其示出了抗坏血酸如何影响luc活性但对ren活性影响不大。这个数据也绘制在图1,其中每个叶子的平均luc/ren比值相对于抗坏血酸浓度进行作图。
为了检测抗坏血酸的影响是否是通过启动子或5'-utr介导的,我们构建了两个载体,其中在ggp启动子和tt8启动子之间交换5'-utr区域。由此得到的构建体包括tt8核心启动子(tt8p’)之后是ggp5'-utr(ggputr),反之亦然。我们在叶中瞬时表达这些构建体,并测量相对luc活性。
为了检测位于ggp编码序列前方的5'-utr对抗坏血酸浓度的影响,使用了不同的ggp基因(genbank登录号fg460629)替代在其它试验中的ggp。在蛋白质水平,其与标准ggp有96%一致,并且在5'-utr缺乏时,升高的抗坏血酸浓度达到通常ggp所见的类似水平。具有5'-utr的版本具有全长5'-utr,而不具有utr的标记版本使用xhoi限制酶将起始atg上游除37bp之外的部分完整删除。在5'-utr的3’端的该区域,ggp之间几乎没有同源性(图8)。两个版本均连接到part277载体中(gleave,a.,1992,plantmol.biol.20,1203(1992)。
通过western印迹使用luc抗体(promega公司)测定luc蛋白量,每个泳道采用来自在本氏烟叶中瞬时表达的各种构建体的50μg可溶细胞内蛋白进行(提取到40mm磷酸盐缓冲液中,ph值7.4,150mmnacl)。用sypro橘蛋白凝胶染色的rubisco大亚基作为加载对照。
为了将抗坏血酸和ggp蛋白的影响分开,我们初步尝试用抗坏血酸或其前体直接用注射器注入叶。但是我们不可能得到叶中抗坏血酸持久的变化。我们也尝试让离体叶片或之前注射了农杆菌luc/ren构建体的叶片吸收抗坏血酸。虽然这些叶子中抗坏血酸非常显著增加,但是在luc/ren值上升到能够测量之前叶片变质。然后我们试图通过敲除参与抗坏血酸生物合成途径中的两个基因(编码半乳糖脱氢酶和gdp甘露糖差向异构酶)来降低抗坏血酸而不降低ggp浓度。然而,我们也没有看到叶中抗坏血酸显著变化,表明其表达的酶可能比较稳定或七天的试验范围中是过量的。
试验至少重复两次得到类似的结果,虽然在某些情况下,高抗坏血酸降低ren的表达以及luc的表达,这并没有改变抗坏血酸对降低ggp启动子(而非tt8)关系的斜率(即luc/ren比)的影响。
基因克隆和质粒
通过基因组步移法克隆来自毛花猕猴桃的ggp启动子(seqidno:101),其已经以登录号jx486682记录在genbank中。使用下列七种平末端限制性内切酶对毛花猕猴桃gdna(2.0μg)进行酶切:drai、eclii136、ecorv、hpai、msci、scai、sspi和stui。纯化酶切产物并用pcr清洁和离心浓缩柱(zymogen公司)用10μl洗脱。使用t4快速连接酶(roche公司),在16℃下将含有巢式pcr引物位点的双链接头序列(clontech公司)与酶切片段过夜连接。连接产物用柱纯化第二次,并用30μl洗脱。使用1ul各酶切产物以及引物319998nrwlk1、rph-149和extaq聚合酶(takara公司),通过以下两步循环条件进行第一轮pcr。对于第一高严谨步骤,一个循环是在94℃下变性2分钟,随后七个循环是在94℃下变性25秒,并在72℃下延伸/退火3分钟。第二步由32个循环构成,每个循环中在94℃下25秒以及67℃下3分钟,最后67℃延伸3分钟。第一轮的产物在1%琼脂糖凝胶中电泳,并使用1μl1:50的稀释物作为第二轮pcr的模板,其中第二轮pcr加入319998nrwlk2和rph-150。第二轮pcr也是两步pcr,最初在94℃下变性2分钟,之后5个循环是在94℃下25秒以及72℃下3分钟。之后是另外20个循环:94℃下25秒以及67℃下3分钟,然后67℃延伸3分钟。根据制造商说明,使用凝胶电泳鉴定大小在500-2kb之间的pcr产物,将其克隆进pgemteasy载体(promega公司)。对克隆进行dna测序证实为与已知的5'-utr重叠。设计的第二组嵌套引物在第一个启动子的步移末端,以使已知的毛花猕猴桃启动子序列延伸至2kb。然后使用猕猴桃gdnapcr5’和319998nrwlk2引物由从毛花猕猴桃gdna对该2kb的启动子序列进行pcr扩增,并使用ecorv和ncoi限制性酶将其亚克隆入pgreen0800-5’_luc(hellens等,2000,plantmol.biol.42,819)。最终构建体称为ggp-启动子-pgreenii0800-5luc载体。
通过pcr克隆来源于拟南芥的ggp启动子(at4g26850)(seqidno:102)以及来源于各种来源的对照启动子。对照启动子为tt8(at4g09820)、ef1α(at1g07940)、act2p(at3g18780)和act7p(at5g09810)。
起始密码子失活或5'-utr的uorf中的其他缺失以及突变的产生是由突变和对照序列的化学合成(genscript,www.genscript.com)实现的。在失活版本中,uorf中的atg或acg起始密码子变为ttg。其他改变是通过点特异性突变完成的。在5'-utr中utr28bp的5’侧有stui位点,其用作合成片段的5’边界。我们在合成基因的3’端添加了额外的cc以产生ncoi位点(ccatgg),并用stui和ncoi酶切移去相当于来自ggp-启动子-pgreenii0800-5luc的合成片段的序列。然后将合成片段分别克隆入载体以得到含有和不含uorf的两个版本。
rna分离和cdna合成:
使用rneasyplantmini试剂盒,从100mg的叶组织中提取总rna,使用nanodrop1000分光光度计(thermofisherscientific公司)测量浓度。然后由1μg总rna合成互补dna,并且使用blueprintreagent试剂盒根据制造商的说明用总体积10μl的随机引物进行实时pcr(takarabio公司)。cdna合成后,将制备物稀释75倍以用于实时定量pcr。
定量pcr
使用
所用体系的对照检测
由于对照基因启动子tt8的表达比ggt启动子高约10倍,因此我们考虑了tt8启动子可能使烟草细胞表达转录本或翻译luc的能力饱和,因此无法观测到任何抑制的可能性。为了检验这一点,我们在200倍的范围内滴定了含有tt8-luc构建体的农杆菌。luc/ren比以及luc和ren之间关系的斜率不受到注入农杆菌的量的影响(表1),表明几乎没有报告基因的表达发生饱和的迹象。tt8启动子驱动的luc值与使用ggp启动子表达的luc值重叠。我们还测试了几种不同的可供选择的启动子是否被抗坏血酸抑制。这些包括ef1α、act2p和act7p。这些启动子都没有受到抗坏血酸负面影响(表2),但是在该试验中,tt8启动子的强度实际上被抗坏血酸增强。在第三个测试中,通过检测来自拟南芥ggp的相同启动子(at4g26850),我们证实了抗坏血酸对ggp启动子强度的影响不限于猕猴桃ggp基因启动子。如图4所示,对于来自不同物种的两种启动子而言,luc/ren比对抗坏血酸的反应基本上是相同的。在最后的测试中,我们检查是否表达基因以提高抗坏血酸(猕猴桃ggp)本身可能影响结果。因此,我们增加了额外的对照基因,其为甲基转移酶的形式。在该试验中,虽然增加的基因的luc/ren比少量降低(表3),但是这没有改变以下结论,即升高的抗坏血酸降低ggp启动子强度但对其它对照启动子的影响很小。
实施例2:在luc/ren报告检验中检测抗坏血酸对其它5’utr序列的影响。
方法
35s驱动的-luc构建体来源于pgreen0800luc(hellens等,2000,plantmol.biol.42,819),其中不含5’utr的35s启动子的第二拷贝克隆入位于luc编码序列前方的多克隆位点,并且在该35s启动子和luc编码序列起始点之间插入来源于苹果、马铃薯和番茄的5’utr(分别为seqidno:126、127和128)以替代35s5’utr(图14)。5’utr序列如图15所示。
按照如上实施例1中对其它ggp启动子构建体所述的那样检测这些构建体。
结果
对于由不同的ggp5’utr插入物驱动的每种luc基因,其均被抗坏血酸下调(图16、17和18,表6)。在每种情况下,luc/ren比以及luc和ren之间关系的斜率被抗坏血酸降低。在这些试验中,抗坏血酸的升高小于实施例1所示(表6),这可能是由于不同的生长条件(更少的光照和更高的温度)所致,而升高的抗坏血酸仍然显著降低了luc值。还示出了典型的非反应性启动子-5’utr(ef1alpha:图19)以及标准ggp启动子-5’utr(图20)的对应数据。其它的变体5'utr序列当然可以以同样的方式进行测试。
表6:对来源于三个不同物种的ggp的各种5’utr的luc/ren比以及luc相对于ren作图的斜率进行表格化。我们也包括了对照启动子及其本身的5’utr;以及之前所用的初始ggp启动子及5’utr作为对照。p值为各构建体高和低抗坏血酸之间差异的统计学显著性。
结论
对来源于三个不同物种的ggp5'utr构建体的检测均示出了由升高的抗坏血酸所实现的显著下调。连同来自拟南芥和猕猴桃的ggp的5’utr(实施例1),这共检测了5种不同的ggp5’utr,它们均示出了由升高的抗坏血酸下调,强烈暗示这是普遍的现象。
序列总结
序列表
<110>新西兰植物和食品研究院有限公司
<120>基因表达的调控
<130>685038hcf/tab
<150>us61/740,751
<151>2012-12-21
<160>140
<170>patentinversion3.5
<210>1
<211>60
<212>prt
<213>毛花猕猴桃(a.eriantha)
<400>1
thralailepheglyvalserargalaleuvalhisvalargserval
151015
argarglysglycysvalvalgluserasnproserprohisglygly
202530
argglyalaleuprosergluglyglyserproseraspleuleuphe
354045
leualaglyglyglyhisphealapheservaltyr
505560
<210>2
<211>62
<212>prt
<213>黄瓜(cucumissativus)
<400>2
thralailehisvalvalserargserphephehisvalargalaval
151015
argarglysglycysilethrprothrasnproserprohisglygly
202530
argglyalaleuprosergluglyglyserproseraspleuleuphe
354045
leualaglyglyglyphesercysphephesersersertyr
505560
<210>3
<211>58
<212>prt
<213>大豆(glycinemax)
<400>3
thralaileleuargvalserargserleuilehisvalprothrval
151015
argargargthrglycysvalthralathrasnproserprohisgly
202530
glyargglyalaleuprosergluglyglyserproseraspleuleu
354045
pheleualaglyglyglyseralavalphe
5055
<210>4
<211>61
<212>prt
<213>番茄(solanumlycopersicum)
<400>4
thralailehislysvalasnargargproleuleuhisvalproala
151015
valargarglysglycysvalthralathrasnproalaprohisgly
202530
glyargglyalaleuprosergluglyglyserproseraspleuleu
354045
pheleualaglyglyglyserpheleuserphesertyr
505560
<210>5
<211>59
<212>prt
<213>葡萄(vitisvinifera)
<400>5
thralaileglnargileproproproleuilehisvalargalaval
151015
argarglysglycysvalilegluserasnproserprohisglygly
202530
argglyalaleuprosergluglyglyserproseraspleuleuphe
354045
leualaglyglyglyserasnalapheleucys
5055
<210>6
<211>64
<212>prt
<213>拟南芥(arabidopsisthaliana)
<400>6
thralailehisglyileserargglyvalserserhisvalhisile
151015
valargglnlysglycysleuilegluthrasnproleuprohisgly
202530
glyargglyalaleuprosergluglyglyserproseraspleuleu
354045
pheleualaglyglyglyserserpheasnphepheserpheargphe
505560
<210>7
<211>57
<212>prt
<213>拟南芥(arabidopsisthaliana)
<400>7
thralailetyrglyilelysproargproleuserphehisvalgln
151015
arglysglycysleuileilethrasnproleuprohisglyglyarg
202530
glyalaleuprosergluglyglyserproseraspleuleupheleu
354045
alaglyglyglyserthrasnserhis
5055
<210>8
<211>64
<212>prt
<213>苹果(malusxdomesticus)
<400>8
thralaileproargalaproargproleuvalhisvalargglyval
151015
glyarglysglycysvalilegluserasnproserprohisglygly
202530
argglyalaleuprosergluglyglyserproseraspleuleuphe
354045
leualaglyglyglyserproserservalpheleuphecysphetyr
505560
<210>9
<211>61
<212>prt
<213>蒺藜苜蓿(medicagotruncatula)
<400>9
thralapheleuargvaltyrargserleuserhisalaargthrval
151015
argarglysglycyscysleuthrprothrasnproserprohisgly
202530
glyargglyalaleuprosergluglyglyserproseraspleuleu
354045
pheleualaglyglyglyvalphealaproserserphe
505560
<210>10
<211>63
<212>prt
<213>毛果杨(populustrichocarpa)
<400>10
thralailehisglyvalthrargserleuilehisvalargalaval
151015
argarglysglycysvalilegluserserasnproserprohisgly
202530
glyargglyalaleuprosergluglyglyserproseraspleuleu
354045
pheleualaglyglyglyphephephephecysleuvalvalval
505560
<210>11
<211>80
<212>prt
<213>北美云杉(piceasitchensis)
<400>11
thralailelysargileleuargpheglnprohisaspargargarg
151015
ileleuargserargasnalaglycysalaleugluserthrproser
202530
prohisglyglyargglyalaleuprosergluglyglyserproser
354045
aspleuleupheleualaglyglyglycysaspargalaleuphecys
505560
leualahispropheleuleuserargglygluileiletrpgluarg
65707580
<210>12
<211>57
<212>prt
<213>小立碗藓(physcomitrellapatens)
<400>12
metserlysaspphetyrargalaglyileglnleuserargserleu
151015
serserserleuserleuhisglyglyargglyalaalaproserglu
202530
glyglyargproseraspleuseralaleualaglyglyglypheleu
354045
serasnphehishisglyalaasppro
5055
<210>13
<211>60
<212>prt
<213>软枣猕猴桃(a.arguta)
<400>13
thralaileleuglyvalserargproleuilehisvalargserval
151015
argarglysglycysvalvalgluserasnproserprohisglygly
202530
argglyalaleuprosergluglyglyserproseraspleuleuphe
354045
leualaglyglyglyhisphealapheservaltyr
505560
<210>14
<211>60
<212>prt
<213>毛花猕猴桃(a.eriantha)
<400>14
thralaileleuglyvalserargproleuilehisvalargserval
151015
argarglysglycysvalvalgluserasnproserprohisglygly
202530
argglyalaleuprosergluglyglyserproseraspleuleuphe
354045
leualaglyglyglyhisphealapheservaltyr
505560
<210>15
<211>60
<212>prt
<213>中华猕猴桃(a.chinensis)
<400>15
thralaileleuglyvalserargproleuilehisvalargserval
151015
argarglysglycysvalvalgluserasnproserprohisglygly
202530
argglyalaleuprosergluglyglyserproseraspleuleuphe
354045
leualaglyglyglyhisphealapheservaltyr
505560
<210>16
<211>64
<212>prt
<213>草莓(fragaria)
<400>16
thralailehisargvalserargproleuilehisvalargargthr
151015
valargarglysglycysvalilegluserasnproserprohisgly
202530
glyargglyalaleuprosergluglyglyserproseraspleuleu
354045
pheleualaglyglyglyvalhisvalseraspleuargphephephe
505560
<210>17
<211>61
<212>prt
<213>马铃薯(solanumtuberosum)
<400>17
thralailehislysvalasnargargproleuleuhisvalproala
151015
valargarglysglycysvalthralathrasnproalaprohisgly
202530
glyargglyalaleuprosergluglyglyserproseraspleuleu
354045
pheleualaglyglyglyserpheleuserphesertyr
505560
<210>18
<211>55
<212>prt
<213>莱茵衣藻(chlamydomonasrheinhardtii)
<400>18
methisleuarggluprovallysthralapheserglualaalaarg
151015
valglnseralaalaserglnproalathralaasnargcyssergly
202530
glyargglyalaalaprosercysglyglylysprolysaspalaleu
354045
serlysalaglyglyglygln
5055
<210>19
<211>58
<212>prt
<213>玉米(zeamays)
<220>
<221>misc_feature
<222>(56)..(56)
<223>xaa可以为任意天然存在的氨基酸。
<400>19
thralaservalalaalaproargargglyproalaalaalaglnval
151015
gluprothrglythrilealaseralavalalaserserproalapro
202530
hisglyglyargglyalaleuproseralaglyglyserproserasp
354045
leuleupheleualaglyglyxaaargleu
5055
<210>20
<211>61
<212>prt
<213>江南卷柏(selaginellamoellendorffi)
<400>20
thralaserilegluleuleuserarghisproileilecystyrgly
151015
hisservalsersercyslysthrleuleuserserleusercyshis
202530
glyglyargglyalaserprosergluglyglyhisproseraspleu
354045
thrpheleualaglyglyglyleuleuleuglyalapro
505560
<210>21
<211>28
<212>prt
<213>毛花猕猴桃(a.eriantha)
<400>21
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>22
<211>28
<212>prt
<213>黄瓜(cucumissativus)
<400>22
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>23
<211>28
<212>prt
<213>大豆(glycinemax)
<400>23
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>24
<211>28
<212>prt
<213>番茄(solanumlycopersicum)
<400>24
asnproalaprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>25
<211>28
<212>prt
<213>葡萄(vitisvinifera)
<400>25
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>26
<211>28
<212>prt
<213>拟南芥(arabidopsisthaliana)
<400>26
asnproleuprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>27
<211>28
<212>prt
<213>拟南芥(arabidopsisthaliana)
<400>27
asnproleuprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>28
<211>28
<212>prt
<213>苹果(malusxdomesticus)
<400>28
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>29
<211>28
<212>prt
<213>蒺藜苜蓿(medicagotruncatula)
<400>29
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>30
<211>28
<212>prt
<213>毛果杨(populustrichocarpa)
<400>30
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>31
<211>28
<212>prt
<213>北美云杉(piceasitchensis)
<400>31
thrproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>32
<211>28
<212>prt
<213>小立碗藓(physcomitrellapatens)
<400>32
serleuserleuhisglyglyargglyalaalaprosergluglygly
151015
argproseraspleuseralaleualaglyglygly
2025
<210>33
<211>28
<212>prt
<213>软枣猕猴桃(a.arguta)
<400>33
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>34
<211>28
<212>prt
<213>毛花猕猴桃(a.eriantha)
<400>34
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>35
<211>28
<212>prt
<213>中华猕猴桃(a.chinensis)
<400>35
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>36
<211>28
<212>prt
<213>草莓(fragaria)
<400>36
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>37
<211>28
<212>prt
<213>马铃薯(solanumtuberosum)
<400>37
asnproalaprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>38
<211>28
<212>prt
<213>莱茵衣藻(chlamydomonasrheinhardtii)
<400>38
alaasnargcysserglyglyargglyalaalaprosercysglygly
151015
lysprolysaspalaleuserlysalaglyglygly
2025
<210>39
<211>26
<212>prt
<213>玉米(zeamays)
<400>39
proalaprohisglyglyargglyalaleuproseralaglyglyser
151015
proseraspleuleupheleualaglygly
2025
<210>40
<211>28
<212>prt
<213>江南卷柏(selaginellamoellendorffi)
<400>40
serleusercyshisglyglyargglyalaserprosergluglygly
151015
hisproseraspleuthrpheleualaglyglygly
2025
<210>41
<211>180
<212>dna
<213>毛花猕猴桃(a.eriantha)
<400>41
acggctatactcggagtttctcggccgctcatacatgtccggtctgtacgacgcaagggt60
tgtgtagtcgagagcaacccttcgccgcacggaggacgtggcgccttgccgtccgaaggc120
ggtagcccctccgacctcctcttcctcgccggcggcggtcacttcgctttctccgtctac180
<210>42
<211>186
<212>dna
<213>黄瓜(cucumissativus)
<400>42
acggctatacacgtagtttcccggtcgttctttcatgtcagggctgtacgacggaagggt60
tgtataactccgacaaacccttcgccgcacggcggacgtggtgctttgccttccgaaggt120
ggtagtccttctgatcttctttttctcgccggcggtggtttctcttgcttcttctcttct180
tcgtat186
<210>43
<211>174
<212>dna
<213>大豆(glycinemax)
<400>43
acggctattctaagagtgtctcgctctctcattcatgtcccaactgtacgacggaggaca60
ggttgcgtaactgccaccaacccttcgccgcacggtgggcgtggtgctttgccttctgaa120
ggtggtagcccttcagacttgctcttcttagctggtggtggttctgctgtcttc174
<210>44
<211>183
<212>dna
<213>番茄(solanumlycopersicum)
<400>44
acggctatacacaaagtaaaccgccgaccacttttacatgttccagcagtacgtcgtaag60
ggttgtgtaacagctactaaccctgcgccgcacggtggacgtggcgctttgccttctgaa120
ggtggtagtccttccgacctcctcttccttgccggcggcggttctttcctctccttctcc180
tac183
<210>45
<211>177
<212>dna
<213>葡萄(vitisvinifera)
<400>45
acggctatacaaagaattccgccgcctctcatacatgtccgggcggtacgacgcaagggy60
tgtgtaattgagagcaacccttcgccgcacggcgggcgtggcgctttgccttcagaaggc120
ggtagtccctctgatctgctcttcctcgccggcggtggttccaacgctttcctctgc177
<210>46
<211>192
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>46
acggctatacacgggatatcacggggtgttagctcacatgtccatattgtccgacagaag60
ggttgtttaatcgaaactaatcctttgccgcacggaggacgtggagctctgccgtctgaa120
ggcggcagcccttccgatctcctctttctcgccggtggcggttccagctttaacttcttt180
tcctttaggttt192
<210>47
<211>183
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>47
acggcttttcttagagtttatcggtcactttcacatgcccgaactgtacgacgtaagggt60
tgttgcttaactccgactaacccttcgccgcacggtgggcgtggtgctctgccttctgaa120
ggtggtagcccctccgatcttctcttcctcgccggtggtggtgtctttgctccttcttcc180
ttc183
<210>48
<211>192
<212>dna
<213>苹果(malusxdomesticus)
<400>48
acggctatacccagagctcctcggccgctcgttcatgtccggggtgtcggacgaaagggt60
tgtgtaattgagagcaacccttcgccgcacggcgggcgtggtgctttgccttccgaaggc120
ggtagcccgtctgacctcctcttcctcgccggtggcggctccccttcctctgtttttctc180
ttctgcttttat192
<210>49
<211>183
<212>dna
<213>蒺藜苜蓿(medicagotruncatula)
<400>49
acggcttttcttagagtttatcggtcactttcacatgcccgaactgtacgacgtaagggt60
tgttgcttaactccgactaacccttcgccgcacggtgggcgtggtgctctgccttctgaa120
ggtggtagcccctccgatcttctcttcctcgccggtggtggtgtctttgctccttcttcc180
ttc183
<210>50
<211>189
<212>dna
<213>毛果杨(populustrichocarpa)
<400>50
acggctatacacggagtaactcggtccctaattcatgtccgggctgttcgacgtaagggc60
tgtgtaatagagagcagcaacccttcgccgcacggtggacgtggtgctttaccctcggaa120
ggcggtagcccttctgatctcctctttctagctggtggcggtttcttcttcttctgttta180
gtagtagtt189
<210>51
<211>240
<212>dna
<213>北美云杉(piceasitchensis)
<400>51
acggctattaaacgtattttgcggttccagccccatgatcgcagaagaattctgcggagc60
aggaacgctggttgtgcactagagagtaccccgtcgccgcacggtgggagaggagccttg120
ccctcggaaggcgggagcccctctgatctcctctttctcgcaggaggcggttgcgaccga180
gcccttttctgcctcgcccatccatttcttctgagcagaggagaaataatttgggagagg240
<210>52
<211>171
<212>dna
<213>小立碗藓(physcomitrellapatens)
<400>52
atgagcaaagacttctaccgtgcaggaatccaactgtcccgatctctctcttcctctctt60
tccttgcacggtggtcggggggccgcccccagtgaaggcggtcgtccctctgacctatct120
gcacttgctgggggaggttttctctcaaactttcaccacggtgctgatcct171
<210>53
<211>180
<212>dna
<213>软枣猕猴桃(a.arguta)
<400>53
acggctatactcggagtttctcggccgctcatacatgtccggtctgtacgacgcaagggt60
tgtgtagtcgagagcaacccttcgccgcacggcggacgtggcgccttgccgtccgaaggc120
ggtagcccctccgacctcctcttcctcgccggtggcggtcacttcgctttctccgtctac180
<210>54
<211>180
<212>dna
<213>毛花猕猴桃(a.eriantha)
<400>54
acggctatactcggagtttctcggccgctcatacatgtccggtctgtacgacgcaagggt60
tgtgtagtcgagagcaacccttcgccgcacggaggacgtggcgccttgccgtccgaaggc120
ggtagcccctccgacctcctcttcctcgccggcggcggtcacttcgctttctccgtctac180
<210>55
<211>180
<212>dna
<213>中华猕猴桃(a.chinensis)
<400>55
acggctatactcggagtttctcggccgctcatacatgtccggtctgtacgacgcaagggt60
tgtgtagtcgagagcaacccttcgccgcacggcggacgtggcgccttgccgtccgaaggc120
ggtagcccctccgacctcctcttcctcgccggcggcggtcacttcgctttctccgtctac180
<210>56
<211>192
<212>dna
<213>草莓(fragaria)
<400>56
acggctatacacagagtttcacggccactcattcatgtccgccggactgtccgacgtaag60
ggttgtgtaattgagagcaacccttcgccgcacggcgggcgtggtgctttgccgtccgaa120
ggaggaagtccttccgacctccttttcctcgccggcggtggtgtccacgtttccgacttg180
cgtttctttttc192
<210>57
<211>183
<212>dna
<213>马铃薯(solanumtuberosum)
<400>57
acggctatacacaaagtaaaccgccgaccacttttacatgttccagcagtacgtcgtaag60
ggttgtgtaacagctactaaccctgcgccgcacggtggacgtggcgctttgccctctgaa120
ggtggcagtccttccgacctcctcttccttgccggcggcggttctttcctctccttctcc180
tac183
<210>58
<211>165
<212>dna
<213>莱茵衣藻(chlamydomonasrheinhardtii)
<400>58
atgcatttgcgtgagccggtaaaaacggcgttctctgaggcagcccgcgttcagtcggct60
gcgtcgcagcctgcgaccgccaatcggtgctccggtggccgtggtgcagccccctcgtgc120
ggtggcaagcccaaggacgctctgagcaaggcgggcggtggccag165
<210>59
<211>174
<212>dna
<213>玉米(zeamays)
<400>59
acggctagcgttgcagccccccggcgcggccccgcagctgcgcaggtggagcccacgggc60
accatcgcctccgctgtcgcgtccagccccgctccgcacggcggccgcggggcgctgccc120
tcggccggaggaagcccgtccgatctcctcttcctcgccggcggsgktcgcctc174
<210>60
<211>183
<212>dna
<213>江南卷柏(selaginellamoellendorffi)
<400>60
acggcttcaattgagcttttgagtcgacaccccatcatttgttatggacacagtgtttcc60
agttgtaagactctgctgagttccctctcttgccacggtggtcgaggggctagcccctcg120
gaaggaggacacccttcagatcttacctttcttgctggtggtggccttcttctcggagca180
cca183
<210>61
<211>105
<212>dna
<213>毛花猕猴桃(a.eriantha)
<400>61
aagggttgtgtagtcgagagcaacccttcgccgcacggaggacgtggcgccttgccgtcc60
gaaggcggtagcccctccgacctcctcttcctcgccggcggcggt105
<210>62
<211>105
<212>dna
<213>黄瓜(cucumissativus)
<400>62
aagggttgtataactccgacaaacccttcgccgcacggcggacgtggtgctttgccttcc60
gaaggtggtagtccttctgatcttctttttctcgccggcggtggt105
<210>63
<211>108
<212>dna
<213>大豆(glycinemax)
<400>63
aggacaggttgcgtaactgccaccaacccttcgccgcacggtgggcgtggtgctttgcct60
tctgaaggtggtagcccttcagacttgctcttcttagctggtggtggt108
<210>64
<211>105
<212>dna
<213>番茄(solanumlycopersicum)
<400>64
aagggttgtgtaacagctactaaccctgcgccgcacggtggacgtggcgctttgccttct60
gaaggtggtagtccttccgacctcctcttccttgccggcggcggt105
<210>65
<211>105
<212>dna
<213>葡萄(vitisvinifera)
<400>65
aagggytgtgtaattgagagcaacccttcgccgcacggcgggcgtggcgctttgccttca60
gaaggcggtagtccctctgatctgctcttcctcgccggcggtggt105
<210>66
<211>105
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>66
aagggttgtttaatcgaaactaatcctttgccgcacggaggacgtggagctctgccgtct60
gaaggcggcagcccttccgatctcctctttctcgccggtggcggt105
<210>67
<211>105
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>67
aagggttgtttaatcataactaatcctttgcctcacggaggacgtggagctctgccgtct60
gaaggcggcagtccctccgatctcctcttcctcgccggaggcggt105
<210>68
<211>105
<212>dna
<213>苹果(malusxdomesticus)
<400>68
aagggttgtgtaattgagagcaacccttcgccgcacggcgggcgtggtgctttgccttcc60
gaaggcggtagcccgtctgacctcctcttcctcgccggtggcggc105
<210>69
<211>107
<212>dna
<213>蒺藜苜蓿(medicagotruncatula)
<400>69
aagggttgttgcttaactccgactaacccttcgccgcacggtgggcgtggtgctctgcct60
tctgaaggtggtagcccctccgatcttctcttcctcgccggtggtgg107
<210>70
<211>108
<212>dna
<213>毛果杨(populustrichocarpa)
<400>70
aagggctgtgtaatagagagcagcaacccttcgccgcacggtggacgtggtgctttaccc60
tcggaaggcggtagcccttctgatctcctctttctagctggtggcggt108
<210>71
<211>102
<212>dna
<213>北美云杉(piceasitchensis)
<400>71
ggttgtgcactagagagtaccccgtcgccgcacggtgggagaggagccttgccctcggaa60
ggcgggagcccctctgatctcctctttctcgcaggaggcggt102
<210>72
<211>78
<212>dna
<213>小立碗藓(physcomitrellapatens)
<400>72
tccttgcacggtggtcggggggccgcccccagtgaaggcggtcgtccctctgacctatct60
gcacttgctgggggaggt78
<210>73
<211>105
<212>dna
<213>软枣猕猴桃(a.arguta)
<400>73
aagggttgtgtagtcgagagcaacccttcgccgcacggcggacgtggcgccttgccgtcc60
gaaggcggtagcccctccgacctcctcttcctcgccggtggcggt105
<210>74
<211>105
<212>dna
<213>毛花猕猴桃(a.eriantha)
<400>74
aagggttgtgtagtcgagagcaacccttcgccgcacggaggacgtggcgccttgccgtcc60
gaaggcggtagcccctccgacctcctcttcctcgccggcggcggt105
<210>75
<211>105
<212>dna
<213>中华猕猴桃(a.chinensis)
<400>75
aagggttgtgtagtcgagagcaacccttcgccgcacggcggacgtggcgccttgccgtcc60
gaaggcggtagcccctccgacctcctcttcctcgccggcggcggt105
<210>76
<211>105
<212>dna
<213>草莓(fragaria)
<400>76
aagggttgtgtaattgagagcaacccttcgccgcacggcgggcgtggtgctttgccgtcc60
gaaggaggaagtccttccgacctccttttcctcgccggcggtggt105
<210>77
<211>105
<212>dna
<213>马铃薯(solanumtuberosum)
<400>77
aagggttgtgtaacagctactaaccctgcgccgcacggtggacgtggcgctttgccctct60
gaaggtggcagtccttccgacctcctcttccttgccggcggcggt105
<210>78
<211>69
<212>dna
<213>莱茵衣藻(chlamydomonasrheinhardtii)
<400>78
ggtggccgtggtgcagccccctcgtgcggtggcaagcccaaggacgctctgagcaaggcg60
ggcggtggc69
<210>79
<211>81
<212>dna
<213>玉米(zeamays)
<400>79
cccgctccgcacggcggccgcggggcgctgccctcggccggaggaagcccgtccgatctc60
ctcttcctcgccggcggsgkt81
<210>80
<211>72
<212>dna
<213>江南卷柏(selaginellamoellendorffi)
<400>80
cacggtggtcgaggggctagcccctcggaaggaggacacccttcagatcttacctttctt60
gctggtggtggc72
<210>81
<211>655
<212>dna
<213>毛花猕猴桃(a.eriantha)
<220>
<221>misc_feature
<222>(219)..(219)
<223>n为a、c、g或t。
<220>
<221>misc_feature
<222>(356)..(356)
<223>n为a、c、g或t。
<220>
<221>misc_feature
<222>(364)..(364)
<223>n为a、c、g或t。
<400>81
tctctccgtccctctcaatagttgtctccattcgcagtaaaatcactaaggccgctcgtc60
cctcagtgcacaccacggcccctccacagccgcattcacctctctctctctctctctgct120
ctatctatatatccccccaatctggcctctcttcacctcacccccaaaatctacacaaaa180
tcaatccttcatcttccacatcggcctccaaaacccacnctcttctccacaatccagaca240
caccttgagcggctggcgttgagcgaatagatagagatagagagagagattttctgcttc300
gatcgggggtaaaacccggtgtttgacaagttgtagacatcacggctatactcggnagtt360
tctncggccgctcatacatgtccggtctgtacgacgcaagggttgtgtagtcgagagcaa420
cccttcgccgcacggcggacgtggcgccttgccgtccgaaggcggtagcccctccgacct480
cctcttcctcgccggcggcggtcacttcgctttctccgtctactagcttattaggtttat540
tcttacttagtgagtaattcgtcctattatagttcgtaagttcatcaaagatctgttact600
tgattcgtctttcgttgctcgagtcttggtgttttttgcgttttctgagttcgag655
<210>82
<211>600
<212>dna
<213>黄瓜(cucumissativus)
<400>82
tccctttctatataattgcttcattccccacctttccatgttcgtgcagcccattcaatc60
ccctcattttaacccacttcctctttttctttttctccttcctcttccagttcccttttc120
cccatctgggttctcttgatttctcttaaaatccaacaacccatgttcgactttgaggaa180
ttggtgcgttggaattgagttttcggagaagatttttcgttttttatcacaacccatcta240
ctccaggtaaggggtaaaacccgggttcgtcaggctgtagacatcacggctatacacgta300
gtttcccggtcgttctttcatgtcagggctgtacgacggaagggttgtataactccgaca360
aacccttcgccgcacggcggacgtggtgctttgccttccgaaggtggtagtccttctgat420
cttctttttctcgccggcggtggtttctcttgcttcttctcttcttcgtattagctttcc480
gtttgtgttttagctctaccggtttaggatttgacatcagcaagtttctgtttcgcgttt540
atttcttttgggtgggggagattttggtgttcggtttggtttgaattagaagcagacgat600
<210>83
<211>429
<212>dna
<213>大豆(glycinemax)
<400>83
gacaaactcacaccacatccatctttgccttctgcatgttggttctctgtaaacagatac60
tgcaaaagaagaataatattgaatatttggttgggttgaatccctgggttgaagcgttgc120
agacatcacggctattctaagagtgtctcgctctctcattcatgtcccaactgtacgacg180
gaggacaggttgcgtaactgccaccaacccttcgccgcacggtgggcgtggtgctttgcc240
ttctgaaggtggtagcccttcagacttgctcttcttagctggtggtggttctgctgtctt300
ctagcttcttcttaactcttttttctttttactacttttaagctaccttgttttcaaaat360
aacaaaaacaaacactttcttacttcataagatcaccttttcttcatcttctacttctcc420
ttcactaaa429
<210>84
<211>680
<212>dna
<213>番茄(solanumlycopersicum)
<400>84
atttgttcggtatactgtaaccccctgtttgcgattggccttgtagccccgttttacatc60
ttccagagactccatttgtatcggttcacatacagtagcaaagcgccattatcttactct120
accccattggcaaacccacagccacaattttccaatcctccattatcccttctacaattt180
tctatataaatacccacatctctctgctctactcccttattatcaacaacaaccaccaaa240
tttcttcttttttttcttcgatagtagcaatctatcaacaaaaacagagaccccatcaca300
agaatcttggaattttagtgttgggtttaagaggaaaaggggttattgtattttgcagtt360
ttgagggtaaagcccagtttaacaagttgtagacatcacggctatacacaaagtaaaccg420
ccgaccacttttacatgttccagcagtacgtcgtaagggttgtgtaacagctactaaccc480
tgcgccgcacggtggacgtggcgctttgccttctgaaggtggtagtccttccgacctcct540
cttccttgccggcggcggttctttcctctccttctcctactagatatagttatacttact600
atagatctctagcttattacgtacagttgtatctagtattctattgattattcgaagaaa660
acacacaaaaagaagtaaag680
<210>85
<211>600
<212>dna
<213>葡萄(vitisvinifera)
<400>85
acacaaaagcctctcaagtccaacagataggccgccttcttcctcatttcactgcagcta60
gcttcctatttttcttcttccaaaatccagggatttcaagagaagagaaagagaaataag120
ggttctgcgggttgtgggtcggtgtgttttgtggttttctatatagggtttctaaggagg180
gagggattttgatttgggttgtgacggtggtggggtaaaacccttgttcgacgagttgta240
gacatcacggctatacaaagaattccgccgcctctcatacatgtccgggcggtacgacgc300
aagggytgtgtaattgagagcaacccttcgccgcacggcgggcgtggcgctttgccttca360
gaaggcggtagtccctctgatctgctcttcctcgccggcggtggttccaacgctttcctc420
tgctagtttaggcttatattctgcataatatagctactgtctttaggattagatcaacca480
atccgtatcgaacactcgatctctcgctttagccatttctttagatcaatcaatccgtat540
cgaacaatcgatctctcgctttagccatttctttaggttggtgtttgagttcttgaaaaa600
<210>86
<211>503
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>86
gtatcatcaaaaaacacctcaaagaattattcattcaggcatcttctcaaatttttgttt60
gtgaaaaaaacccacatcaaaagatctctcatttattcgtttcgtttctgctgttttgag120
tgtcgggttcgttttagctgtaatctttttttccggcgttcgatttgaaaaaatccgggg180
aacaggtgatcggaatcacggctatacacgggatatcacggggtgttagctcacatgtcc240
atattgtccgacagaagggttgtttaatcgaaactaatcctttgccgcacggaggacgtg300
gagctctgccgtctgaaggcggcagcccttccgatctcctctttctcgccggtggcggtt360
ccagctttaacttcttttcctttaggttttaggagttagggtttgttagtgttttttcct420
tcttctttttttggtgctcttgaatcgcttttttcttgggggaagttttttcttttgctc480
ttcgaaatttgtcttttttgaga503
<210>87
<211>420
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>87
caaaatctcacacatcaaacacgtgaatttgctctctttctggttcactcacctaccatt60
actataagtctgaaagagtgattgaaacccacctcgaaaaatctatccttttttttgttt120
tccttctccggcgaatccccggggagattggtaatcggtaatcacggctatttacgggat180
aaagccacggcctttgagctttcatgtccaacgaaagggttgtttaatcataactaatcc240
tttgcctcacggaggacgtggagctctgccgtctgaaggcggcagtccctccgatctcct300
cttcctcgccggaggcggttccacaaatagccactaaccctaaccctttttctaattagg360
tttttagttcttagagtcctgtattaatctgttatttcgagattataatatttgtgagca420
<210>88
<211>554
<212>dna
<213>苹果(malusxdomesticus)
<400>88
ccccacctgctcccccaattccccagtacccatttgtgtttttcgattcagttcgaaacc60
aggcggtcttcacctttctggttgttttcctatctcggttttaaggaggaagaagaaagg120
aaggcgttttgatcattttcttttcgaatttcttttggggtaagacccaggttcgacgag180
ttgtagacatcacggctatacccagagctcctcggccgctcgttcatgtccggggtgtcg240
gacgaaagggttgtgtaattgagagcaacccttcgccgcacggcgggcgtggtgctttgc300
cttccgaaggcggtagcccgtctgacctcctcttcctcgccggtggcggctccccttcct360
ctgtttttctcttctgcttttattagttttatttttatagagtttcttgcttagattttt420
agagattttttgttctataaagcgctcgagtagatcgtatttttgttttcgggggttttt480
tttttttgtggtgtttgatttttactgagaaatcgagaaaaaaagggagagagagagaga540
gaaagaaggcgagt554
<210>89
<211>600
<212>dna
<213>蒺藜苜蓿(medicagotruncatula)
<400>89
aacaaaacaccctcctcgtgcttgattctagtacaagtataccaaactcattcacaacca60
catttccttaatctcttttcattttctcttatctatataacactttacaatctccacctc120
attttcttcatcaaaaacaattcatctttcttctttgctatcatcactaaattttcttga180
gaattcagaaaataaaagagagatttgaatgtttgggttgattccctgtattgacacgtt240
gtagacatcacggcttttcttagagtttatcggtcactttcacatgcccgaactgtacga300
cgtaagggttgttgcttaactccgactaacccttcgccgcacggtgggcgtggtgctctg360
ccttctgaaggtggtagcccctccgatcttctcttcctcgccggtggtggtgtctttgct420
ccttcttccttctaatttcttgtttttagtttaactttctttagatttttacgactcaaa480
actataagctaaaactagactttgtaagtaagatttatttgaaggtgcttcttcttcttc540
ttctttcttcttcttgttgttaaaaaaaaaagtatttttgtgtttggtttggtgaaaatg600
<210>90
<211>621
<212>dna
<213>毛果杨(populustrichocarpa)
<400>90
gaaccattagtgtgtaccctcctcaccaaaaaagaataaaaactctcacaaccaaacccc60
aattcacagcaaaaccataaccacaattcccacttctctcttatctatacaatatcccat120
ctcctttttcttcttttatctcttgctctcatcaaaatcccagcacctctcctctcgctg180
ctaataaacttcaactcccatttctgcggcggcggctgcagcccagtggttcttgttttt240
tctttatttattgtgatctgtaaaaaaaaatctaaagagtacaagaacaagggttaaaat300
cccagggtagacaagttgtagacatcacggctatacacggagtaactcggtccctaattc360
atgtccgggctgttcgacgtaagggctgtgtaatagagagcagcaacccttcgccgcacg420
gtggacgtggtgctttaccctcggaaggcggtagcccttctgatctcctctttctagctg480
gtggcggtttcttcttcttctgtttagtagtagtttagtgctattgttgttgttaattat540
tattattattattattaaatctaaaataaaaggccagtctgcaaataagtaaaagtatta600
gatctcgaataagcgaacaag621
<210>91
<211>640
<212>dna
<213>北美云杉(piceasitchensis)
<400>91
cggtgaagaagtggtgagaagtgggtttgtggttaacgtttgttcagccgtcctctgata60
tctcttcagccttacacgtattttctggtataaacgtattgtcatctgaatctgaattta120
ttagctgcgtctttatgaggttcgaaagcgggatctcctcagcttcagagtttactttct180
ggacggaaagaaatctgtattcatagtttgtgaaggcagggaggggattcgatccggtgc240
agttgtggaggaaattcggagggtaacatcacggggactggtgtcgggagacgaaaataa300
ggccattcaagaagggaaagaaaggtctggcaagttggggcaacacggctattaaacgta360
ttttgcggttccagccccatgatcgcagaagaattctgcggagcaggaacgctggttgtg420
cactagagagtaccccgtcgccgcacggtgggagaggagccttgccctcggaaggcggga480
gcccctctgatctcctctttctcgcaggaggcggttgcgaccgagcccttttctgcctcg540
cccatccatttcttctgagcagaggagaaataatttgggagaggtagaggtggtttagcc600
taatttaggcgtcaatcaatcgctcatattaaccaccacc640
<210>92
<211>253
<212>dna
<213>小立碗藓(physcomitrellapatens)
<400>92
cgcgtccatatgagcaaagacttctaccgtgcaggaatccaactgtcccgatctctctct60
tcctctctttccttgcacggtggtcggggggccgcccccagtgaaggcggtcgtccctct120
gacctatctgcacttgctgggggaggttttctctcaaactttcaccacggtgctgatcct180
taggggggggtgctagggagaattttctgctcaggtaacaggaagtgtctaggagactat240
tcaacctgcaaaa253
<210>93
<211>477
<212>dna
<213>软枣猕猴桃(a.arguta)
<400>93
gcaaaatcaacccttcatcttccacatcggcctcccaaacccacctcttctccacaatcc60
aggcacaccttgagcggctggcgttgagggaatagagagagagagagagagagattttct120
gcttcgatcgggggtaaaacccggtgtttgacaagttgtagacatcacggctatactcgg180
agtttctcggccgctcatacatgtccggtctgtacgacgcaagggttgtgtagtcgagag240
caacccttcgccgcacggcggacgtggcgccttgccgtccgaaggcggtagcccctccga300
cctcctcttcctcgccggtggcggtcacttcgctttctccgtctactagcttattaggtt360
tattcttacttagtgagtaatttgtcctattatagttcgtaagttcgtcgaagatctgtt420
gcttgattcgtctttcgttgctcgagtcttggtgtttttgcgttttctgagttcgag477
<210>94
<211>655
<212>dna
<213>毛花猕猴桃(a.eriantha)
<220>
<221>misc_feature
<222>(219)..(219)
<223>n为a、c、g或t。
<220>
<221>misc_feature
<222>(356)..(356)
<223>n为a、c、g或t。
<220>
<221>misc_feature
<222>(364)..(364)
<223>n为a、c、g或t。
<400>94
tctctccgtccctctcaatagttgtctccattcgcagtaaaatcactaaggccgctcgtc60
cctcagtgcacaccacggcccctccacagccgcattcacctctctctctctctctctgct120
ctatctatatatccccccaatctggcctctcttcacctcacccccaaaatctacacaaaa180
tcaatccttcatcttccacatcggcctccaaaacccacnctcttctccacaatccagaca240
caccttgagcggctggcgttgagcgaatagatagagatagagagagagattttctgcttc300
gatcgggggtaaaacccggtgtttgacaagttgtagacatcacggctatactcggnagtt360
tctncggccgctcatacatgtccggtctgtacgacgcaagggttgtgtagtcgagagcaa420
cccttcgccgcacggcggacgtggcgccttgccgtccgaaggcggtagcccctccgacct480
cctcttcctcgccggcggcggtcacttcgctttctccgtctactagcttattaggtttat540
tcttacttagtgagtaattcgtcctattatagttcgtaagttcatcaaagatctgttact600
tgattcgtctttcgttgctcgagtcttggtgttttttgcgttttctgagttcgag655
<210>95
<211>473
<212>dna
<213>中华猕猴桃(a.chinensis)
<400>95
cacaaaatcaatccttcatcttccacatcggcctccaaaacccacctcttctccacaatc60
cagacacaccttgagctgctggcgttgagcgaatagatagagagagagattttctgcttc120
gatcgggggtaaaacccggtgtttgacaagttgtagacatcacggctatactcggagttt180
ctcggccgctcatacatgtccggtctgtacgacgcaagggttgtgtagtcgagagcaacc240
cttcgccgcacggcggacgtggcgccttgccgtccgaaggcggtagcccctccgacctcc300
tcttcctcgccggcggcggtcacttcgctttctccgtctactagcttattaggtttattc360
ttacttagtgagtaattcgtcctattatagttcgtaagttcatcaaagatctgttacttg420
attcgtctttcgttgctcgagtcttggtgttttttgcgttttctgagttcgag473
<210>96
<211>444
<212>dna
<213>草莓(fragaria)
<400>96
aatacaccaccacataaccaaaacccactgccccattttctcgattctcattcgcatctg60
actaggaaggagagattttctgcgttgggtttgattccggtttggggtaaaacccgggtc120
gacgagttgtagacatcacggctatacacagagtttcacggccactcattcatgtccgcc180
ggactgtccgacgtaagggttgtgtaattgagagcaacccttcgccgcacggcgggcgtg240
gtgctttgccgtccgaaggaggaagtccttccgacctccttttcctcgccggcggtggtg300
tccacgtttccgacttgcgtttctttttctagctttttgtagattcgggtttagctcaaa360
gattattgtttcgcaagtagatcgtgtttgctgtcgttgctgtgctttgatttttctttt420
gagaagaacaagaacataacaaaa444
<210>97
<211>600
<212>dna
<213>马铃薯(solanumtuberosum)
<400>97
ccatttgtatcggttcacacacagtaagcaagcgccattatccatctctacccccattgg60
caaacccacagccacaattttcctatcctccattatcccttctccaatttattatataaa120
tacccacatctccctgctctttctcccttatcatcaacaacaacaaccaaatttcttctt180
tttttcttccacagtagcaatctatcaacaaaacagagaccccattacaagaatcttgga240
attttagttttgggtttaagaggaaggggttattgtatttgcagttttgagggtaaagcc300
cagtttaacaaggtgtagacatcacggctatacacaaagtaaaccgccgaccacttttac360
atgttccagcagtacgtcgtaagggttgtgtaacagctactaaccctgcgccgcacggtg420
gacgtggcgctttgccctctgaaggtggcagtccttccgacctcctcttccttgccggcg480
gcggttctttcctctccttctcctactagatatagttatacttaccgtagatctctagct540
tattacgtacagttctatctagtattctcttgattattcgaagaaaaacacaaaaagatg600
<210>98
<211>499
<212>dna
<213>莱茵衣藻(chlamydomonasrheinhardtii)
<400>98
cagcttccaaactttgtctgtattatctgccacgcttgctcgagctgcattacctgagag60
tgcgacaaagcgacctcgcgtaaattgaatggcttgtgcggctactttctcccaacttct120
tcatgcatttgcgtgagccggtaaaaacggcgttctctgaggcagcccgcgttcagtcgg180
ctgcgtcgcagcctgcgaccgccaatcggtgctccggtggccgtggtgcagccccctcgt240
gcggtggcaagcccaaggacgctctgagcaaggcgggcggtggccagtaaccttcctcgc300
gcaacacaccgcgcgagcgcctgcggctgttggacagccagcagcgtgtgtcgaccccgc360
gccaggacaccggcagcgacgtcgacggctagtatcatctagcctttagcaactctagcc420
tagaaacttagtattcgctcacgaaacttttaggagcttttcgtcgatcaacatcgcctc480
gctcgtcgccgcggacaca499
<210>99
<211>480
<212>dna
<213>玉米(zeamays)
<400>99
aaaccaaacccagctcaggcacaaccccaacgcaccgccgccgctcgcctggcctcacct60
cgccggagaagagttataagacaggagaaccccgtccccggggcgcaggcctcggcagct120
tccgtgaccaccgcccaatcctgccacagctgccctccctcctcccctctgggcgtggcc180
gagttgtaggcatcacggctagcgttgcagccccccggcgcggccccgcagctgcgcagg240
tggagcccacgggcaccatcgcctccgctgtcgcgtccagccccgctccgcacggcggcc300
gcggggcgctgccctcggccggaggaagcccgtccgatctcctcttcctcgccggcggsg360
ktcgcctctgagccgttcctgtctctcaccttcttcctttcctagcaattagtcccttct420
gctaacccccccggaccccgacgagttctcaccccgagagtaactgaccgacctaccggc480
<210>100
<211>337
<212>dna
<213>江南卷柏(selaginellamoellendorffi)
<400>100
gcttttgattcaggtatggttgctccccaattcagcacatttcctttctttttccttgca60
gggtttattcttatgcactgactagcctggaagaaggctagtccgtgccaaaattttgac120
aaccacggcttcaattgagcttttgagtcgacaccccatcatttgttatggacacagtgt180
ttccagttgtaagactctgctgagttccctctcttgccacggtggtcgaggggctagccc240
ctcggaaggaggacacccttcagatcttacctttcttgctggtggtggccttcttctcgg300
agcaccatagtgggggtttcaatcttattggctaaag337
<210>101
<211>2130
<212>dna
<213>毛花猕猴桃(a.eriantha)
<220>
<221>misc_feature
<222>(556)..(556)
<223>n为a、c、g或t。
<220>
<221>misc_feature
<222>(1261)..(1261)
<223>n为a、c、g或t。
<220>
<221>misc_feature
<222>(1750)..(1750)
<223>n为a、c、g或t。
<220>
<221>misc_feature
<222>(1754)..(1755)
<223>n为a、c、g或t。
<400>101
atcccaaaatatttgttcacttagaaaattaactgataaaataatgcaaactctcctttt60
tgttctcctcttttgaattgacgtgacacatttatctttttaattttagataatttcgaa120
ttattgaaaaaaaattaaactgttttccaaataataattttttagaaataatgcaaataa180
tagttttttaaactattttccaaatattttttttcaaaaataatgcattattaagaataa240
tattaaaaaatatcttcaaatattaaaaaatatatttttgataaaatttaataatatata300
aaaataaatgaatgtttttgtttgcataaacaatttctaataaaatattttgcgaaaatg360
tttttgtttgcataaacaatttctaataaaatattttgcgaaaatgtttttgttcgcata420
aacaatttctgataaaattttttgcaaaactaaacctaacacaaatgggtagcatttttg480
cttctttaaaatcttggattccctaaattagacaaataaattgggacggatcaacattta540
ttttcttcttaattwntttctctaacactacaacaaaataattaaatagataagaaaaga600
gaaaaaggaacttgagaacccacccaacttttaaacattgcagttgggtccttccgtacg660
ttgcagtggtcctccacaacgtccacatgaaccacatgggcgtggttaatacaacgcacc720
ccactctctctctctctctctctctctcgataattgtctccattcgcagtaaaattacca780
aggccactcgtcccacagtgcacaccacggccgatccacagccacactcaccaatcacct840
ctctctctctctctctctctagaatttatttgttgctcttggagcaacacgtcacttttt900
gacacgtggtggtcggatccaatcatctcacgccatccaagcactcagtttcatgtgttt960
gccacgtcaccacaacaattccaccacaaacccaggtaaacacaagactaacagaacctc1020
actccgttaatgccatcttcctgtcgctgactcgcatgaaataccaccacttttggaaac1080
caaacgccagaaaagattactctcaccaatattctctatgaacaaagaaattgggttatt1140
atttattatttacaagaaataaatggcaccaaccaaatttaaaaagacgtctctgcagcg1200
attttcacctcattttattttttgagcttttaggtgtctcgtccgaaaccgacgccttct1260
ntattatgcaatttttcactcttctttgccttctcagtcccgaaatgactattttcaggc1320
aacatcatagggtgattgggttgtttagctatgtaggtacgaaatctaaaaatttgaatt1380
tgtaaagtttatgaatatttcatcgcatcgagtactggcggaatgttcacggggttaaca1440
ggatttgaactccgttatttctttcttgagtaaacggacgtggctgaatacacggacaac1500
caattaaatggtgtatgatatttcgtgtggagcaccacgcgtagaaagtgaggtgttgcg1560
tcaagagcatcaaataatttctcctctctctctctccctcttctctatctatatatcccc1620
caatctggcctctcctcacctcacccccaaagtctacacagaatcaacccttcatctccc1680
gcataggcctcccaaacccacctcttctccacaatccagacacacctcgagtggccggag1740
tttagagagnaganngagagagagattttctgcttcgatcggggggtaaaacccggtgtt1800
tgacaagttgtagacatcacggctataatcggagtttctcggccgctcatacatgtccgg1860
tctgtacgacgcaagggttgtgtagtcgagagcaacccttcgccgcacggcggacgtggc1920
gccttgccgtccgaaggcggtagcccctccgacctcctcttcctcgccggcggcggtcac1980
ttcgctttctccgtctactagcttattaggtttattcttacttagtgagtaattcgtcct2040
attatagttcgtaagttcatcaaagatctgttacttgattcgtctttcgttgctcgagtc2100
ttggtgttttttgcgttttctgagttcgag2130
<210>102
<211>1959
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>102
ttttgatacttgtcctctcgaaattgatgtttactgacgcacgattattgggaatttggg60
acttagtggtgtaccaatagcatctcagctttcatggttacaaagaggccagtgaaagag120
gcttggtcaaccgcaattatgatccgttacttgcactgactaccaccgatccaccgccgt180
aactctttaaatcttaatgcaatactaattcgaattacatgtcgaggtcttaaaacatga240
taaatggttgtttaaagtaaaaaaagaaaagaaacaataaataaaacaaacggtgatgga300
aggagggacgtacgatcgtagtcgtagagactgccaaataaataatggaccactattatg360
tggttcaagaactatatccttttcttagattccgaatcttaacttgtgtttcttgccttg420
tttggttttttttttgtttattttgtagttttgtctcataaaataacgaacaaaaagcaa480
tggtctaaatatttctcaatatatgtatttaaaaattatatcatagtttcgcaaaaaaac540
gaccgaacgaacgatggtagtcatttgcccaaattgagcctcgtagtagttgctacgacc600
acacccccgtcccatgaataaacacgaccatgtaaatattatctataaagacaacataaa660
tttacaaattaatgttgaacagaaaaagtcaaaaagaaaatttcgttaatataataataa720
tattctatgataatataaaacgtggcataacacatgacttcacatgacatcataagaaga780
catatgccacatgaactcttcatcgcctccatccttttagtctcgtttacatgcagcaaa840
ctacgatctacgattatatacaatgaaattcaaattcataatcaattggcattaaacata900
cgtatatcataattcataaggttaactaggtttagcaaatgttattctctttggataaac960
gtttagcaattgtttatctcaaatttaattgaaaatacttgtaagacacagttacaatta1020
gttctcaatttccaatctacacaagatttaaatctttctttagttcattctgatctattt1080
ttctctcaaagaaaatgtagtgctccaacttttctcccaagactaataaaaattttctta1140
attgcatgcaaaaatattttttccaactttaatcttatcaaacttagatctcattttatc1200
ttccgaataagatttttgttttggtcctttgcctaatcaaactaactaattagattagaa1260
gaaaaatacaattagacaaaataatatcgatgaaaaaataaaatccacaagaaaggacct1320
aagaaatttcacgtccgaatcacaaccacagaaaaaaagagagaaatattagtatataat1380
taaaattttgtcgtctgtcttctctggttcactcatctcctatctatttaaagcccattc1440
gatatcctaaaacactgtatcatcaaaaaacacctcaaagaattattcattcaggcatct1500
tctcaaatttttgtttgtgaaaaaaacccacatcaaaagatctctcatttattcgtttcg1560
tttctgctgttttgagtgtcgggttcgttttagctgtaatctttttttccggcgttcgat1620
ttgaaaaaatccggggaacaggtgatcggaatcacggctatacacgggatatcacggggt1680
gttagctcacatgtccatattgtccgacagaagggttgtttaatcgaaactaatcctttg1740
ccgcacggaggacgtggagctctgccgtctgaaggcggcagcccttccgatctcctcttt1800
ctcgccggtggcggttccagctttaacttcttttcctttaggttttaggagttagggttt1860
gttagtgttttttccttcttctttttttggtgctcttgaatcgcttttttcttgggggaa1920
gttttttcttttgctcttcgaaatttgtcttttttgaga1959
<210>103
<211>1161
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>103
acgcgtgatgaatcaatcattcattcacgccaacgtaaaaaaaaaaaaaaaaaaaaaatt60
taactaaaattatacggatgatttatttacatggacgctgattaaaaaaggtcggtccaa120
ttaattatttacttaataattaattaaagtgttgtgtgtatttttggattcccataatgt180
tttgaggggtatataaaggagattattttttctattttcaatgtctacgaataaaatgga240
acatcctcgttccaatatatactgctgttttattactaaggtttataattggagtgtata300
aaaacactccaaaataatatcatgaataatgtttttatacgtataatacataaaataaaa360
tctctttcctcatagctggtcctctgattactatcaaacatatgattcaattgcaaattt420
gcaattattaatgcaagaagaatgagtggaacattaaagttagatcaaaaactcttcatt480
acaaacataaataaattcattttggtgctttctaaattaatttattgatcattttttggt540
atgtagctaggggttaactcatcaggaagaacattacctagatgatgtcattgtcaaagt600
caataggttgacttatccgatggatactaatccaacgggtcaaaaagtgccaaatcgggt660
caaaatcaaaaacctcaaagatgacccgtgaacacttgtttttatcttcctcacaaaatc720
cacacgtccaaatcacaacctcaaaatctcacacatcaaacacgtgaatttgctctcttt780
ctggttcactcacctaccattactataagtctgaaagagtgattgaaacccacctcgaaa840
aatctatccttttttttgttttccttctccggcgaatccccggggagattggtaatcggt900
aatcacggctatttacgggataaagccacggcctttgagctttcatgtccaacgaaaggg960
ttgtttaatcataactaatcctttgcctcacggaggacgtggagctctgccgtctgaagg1020
cggcagtccctccgatctcctcttcctcgccggaggcggttccacaaatagccactaacc1080
ctaaccctttttctaattaggtttttagttcttagagtcctgtattaatctgttatttcg1140
agattataatatttgtgagca1161
<210>104
<211>2003
<212>dna
<213>苹果(malusxdomesticus)
<400>104
gacgagatcagcacgatgttggaggagttagaggcctgacagaaaaaaaaaayattaaaa60
caggcgtctaacgtcagatccacctcaggctctcgggccggcgagaatcgacgggcctgg120
ggctcggggttgctcaggctcccttgccagccaacgctggacttcctacctcgggcaacc180
aagctcggggttgttcaacatctttctctctctccctctttcacatattcccatcaattt240
tttctctcttctttcacctcacccatccaaaactacaatattcggttttattttttttat300
ggaaaataaatttaktaaagagaaaatcatctcgaacaacatgttccaactccmaattca360
attaaagttcaaagcatctaactccgaagcawggaggcaataattctaaaagtggggatt420
ggacaccaacaccccaacgtaagcaagcttatcaaccacaaaattgattttattatgttt480
ccagaagaagtcgattgcaacacttggaaaataaaccaacagctacggaaaatcaaaaca540
aacacaacagaatttctatataaaaatgcttgtacacattgccataccctaattttcacc600
atgttatgaaattttcaatctgaattttagcaattgtaaatttccgaaatcttgaacaat660
tttcttcatttcaacaaaagagaaatgttgtaaacgggaattgacatatttttaatccga720
taaacyctttctctktctcaattaattcaagagaactttaacgaaaaactytcgttactg780
ttcaytttaataaaaaatcacatttttaaactaaaaagtcaatcmtkgtactatwcactt840
taccctttattttgtccttatcattaaaacttttcragtttttttcattagtttcytttt900
aattcaaarcsgtttaagtattatatatttcaaccctcattttcttgcatataaagtcaa960
gtcttttttccctcsatccgtacacaaagtggtccatatacggcaatcataagtaccagt1020
gaacctcaatctctcacacattttttttctcaattattgtcccaaaaaaggacccggctt1080
ttcttcgttctcactttctatacactttcaactcttcttaatttatttatgcarttacgs1140
ttaagttatgttaatattttatattaatttttttttataaarataataaaacaaaaaaya1200
ataataatataaaatattaatrtaayttaaccgtaatygcayaaataaaaaaaataasaa1260
tgtattaaarrtgggaggacaaagaagtcaggtccctagaaaatacaccatcaaaccaac1320
caaggccacgaaaccccccaaaatacaaacacaaaccccacggcacatcctctaccacaa1380
atccacttttcctccccatgcctataaatccaccccctcatctcctctccacacccccac1440
aaccacaaaccccacctgctcccccaattccccagtacccatttgtgtttttcgattcag1500
ttcgaaaccaggcggtcttcacctttctggttgttttcctatctgggttttaaggaggaa1560
gaagaaaggaaggcgttttgatcattttcttttcgaatttcttttggggtaagacccagg1620
ttcgacgagttgtagacatcacggctatacccagagctcctcggccgctcgttcatgtcc1680
ggggtgtcggacgaaagggwtgtgtaattgagagcaacccttcgccgcacggcgggcgtg1740
gtgctttgccttccgaaggcggtagcccgtctgacctcctcttcctcgccggtggcggct1800
ccccttcctctgtttttctcttctgcttttattagttttatttttatagagtttcttgct1860
tagatttttagagattttttgttctataaagcgctcgagtagatcgtatttttgttttcg1920
ggggttttttttttttgtggtgtttgatttttactgagaaatcgagaaaaaaagrgagag1980
agagagagagagagaaagaaggc2003
<210>105
<211>2000
<212>dna
<213>苹果(malusxdomesticus)
<400>105
tgcccttctcaataacaataacatagaatgatactttgcakaactcaaayagactagggc60
tcatctggactrcttttaaaatgattaaaasygtttttkgtgaaaatattttaatatcaa120
tcyttagtaaaaattcaagtggaycctcaaaaacayttggagtgcttcctataagaatca180
tattggtgcttcttccaacaaacaattgaagtgctttttgaactcaaaattcatttcacc240
aaaaacactttcaaatattactatgcttcctcttttgtagttaagttcaaatcatctatc300
gctatgtccaaaaacaaaacattaaaaaaatgtgagatcatattaaaaaaaagaaaattg360
gaactttgtcttcttgaactaaaatatcatgagtactcttgaacttgtcaaatatagaac420
aatggccatctagataacttgttaggacttacataacttatttgctcctttaattcttta480
ttttttatgaaagttctaactgtrttatctaaagaggttattgctccacattacgacaag540
ctcaaaaaattcatcaaaattaattaggtataaaatctcttattgttggttacatgttca600
atttactaaacttcagcgttggataccaatatatatgtgtttaggtacaagctttagaaa660
caaggataatgcagccatstagtgaaactggcatgaagaccccatgtttttatggtcacc720
gcccacacccatgtgagcaagacatggacaatggataaggtgattgtccttggatattmt780
ggggcatgtcccttaattttaaggacgacgactgctactagtctactaccaccacttttt840
gctttgcatagtactttgggccctgtgcgtccttgaatgtyctggtgcatttactataac900
tttaaggacgacgacttagactactatttatttattttaattaactgttattaagagaaa960
aagatgggagttcgaactattacaaaaaataaaagagmgggtttagaattcataatgcat1020
agataaaaatttaacattctattcattaagatattgaactacatgcactactaatacttc1080
tttagttgggragttgcaggcaaaacaactcgtgcatgtgaacacattgtgatttggttt1140
tctctcgagtacaacacactatcattttggttttctctagagttttagcgagttgtcatg1200
aaattagtttttatttttatttgttattcaacgacatrgttacaccaagtgtagatgcat1260
aatttgggaaaaccayataataagctaactataacaatttaatatcaaatgcrtcatcta1320
tgaagttgagccttgagctcctacttatgtacaaatgaaaatgaataccgtartactaaa1380
tgactaacttagacatcaagaaattgataaaataaaawttaaaaaaaaaaactacaagca1440
tgatgatgaggaatgaggatcctatctgaatcctctttatgaggactctaaagattctcc1500
aatcacgtccgtttattatagatcgtacgacttgtttttgttagatgttatttatattca1560
atttaaaaaaaattacaataatatataatcacatgatatacaataaaatagatataatta1620
aaagattcctataattaaagaatcctcacaaagagatttccgaacgctctttcgaccatg1680
agggtagttaggctcctaaatacttgaaaggtacagtacatcatcatggtaggtttgaag1740
ttataagtccacctcatccaaaacacagcatatagaatgattccctcatggatcccgaat1800
cctaaatttttatttcaattaaaatttgtgtgcttgctttgtggaatgcggaccgtcttc1860
ttgtaaatttcgtaggaagactcggctccaattttttawgattaaaatccgctgccatgg1920
tcaattcactgtcgtctaattatcaacaggcagccttccaacccacgtgtttgtttgcac1980
gcattagaatattcactgtc2000
<210>106
<211>2018
<212>dna
<213>苹果(malusxdomesticus)
<400>106
ccgaggacctcagaagcgagggggcaatgtttgggcccaaaataacagtttgggccgagt60
gtagggtcattctcggcccggaaggccttacgacaagaatactatggatcgttcagtacg120
tgggcatccaaacctaggtcagctaggtcataacgcaagaggtcgaatcctagtgcaata180
aggagtctcggcaggacccggaagtgaatccggctcagttagggactaggttcacagtcc240
tagtgaaggtaggactggtcgagttgatgctgatcaggggaagaaaacctagtccgagta300
ggtttttaactcgaccttggggggagccattgctataaatagaaaaggttgcacatcctt360
caatgacccctgcaaatcaatacaaaattgccctgcgcaaattctcacaacttgtgattt420
ttctttttccttttttcgctgacacatcttccgttggcatcaacagcactgtggaagcaa480
ccggtgatatcttaagtcggcatagatagctctgtcaccgtagagttagtcggtctcgca540
gtatcttccgttggcatcaacagcactgcggcgagaayggttgattacctatccaagtct600
cggtcragaagggtttctaaatccttattggtcgaggtcatctcatcagccttctcggcg660
ragtgaggtgttacagttattacattcggcacattgaaagccgaatttgatattgaactt720
cktaagaatagtaaccttgtcttcaggttcgagagcccaagaggycgagacgtgttcctt780
tctcggccgcaatcgcaagacgcagaagtcagtagcgcgacccaacgcaacatcaayaaa840
tttactcmtcggccgagctcggcckacgagttggcacgccccgcattcaccgaaggacgt900
agttagctcattaattactcggcctgcgcgccacgtaggctttgtagtttctagggtcaa960
caaggtgaaattcttgaatttatgaaagacaaacaattacaaaaatgtttgctaatgatg1020
ttttcattaattaagaatgaaaattggaaatccgaagatgatcacataccaatgggcttt1080
gtgatgtcatttatgttttagaccgatgcccctctcgaatgaggatcctctctggatcct1140
ctctgtgagaatcatctccggatcctctttttaagaatctcgagaattctcaaatcgtgt1200
tcgtttatcgtacatcatgcgatcaagtttcttcagttattgttcatgtttaattttaaa1260
taaaaatatttaaaatgatttttgatcgaacaatgtacgataaaagaacacgatttaaga1320
attttcagaattatagtaaagaatatccgaagaggatcctcattcgccccctctataccc1380
aaaaaaccaataaatagatccgaccgaatatccaaaatccaaaaacagttgcgtaatatc1440
tttacacatgaacggttgggatcggtcaaaggacggaaaaccagaacacacgtgtccgtc1500
tccgttcgtcccagtagcagaccgtagaccggaactctgtatcgcgaaatcaaaagacgt1560
cacgatatagaagaagaagagaaagagaaacggagatagatatttgatacagattttccg1620
atcgcacaatcatcgacattctcgaccaatttgaagactcggaatcgctggtccggcgag1680
ggctggcttcgtagatgcaatcacggctctaatcaaagcaatcactagtattaagatttc1740
gagaggcagcagagaatgcatgagagccttccctaattccctgcttgcatgcccttcgtt1800
tcacggcggtcgcggtgccctaccctccgccggtgggcatccatccgatctcacctacct1860
ggccggcggcggttgcttctgaaaacgacgaccgcctgtgtttccccttctgggttttga1920
gtgtacttcgttttttattaagcagtaatcaatggttacggttaagcagcttcaagatgg1980
taactttacgtcgcaattccctccattttctctgcaag2018
<210>107
<211>2000
<212>dna
<213>中华猕猴桃(a.chinensis)
<400>107
ttggtgtataaagatgagtaaatagggatcccggcaaatggtctccacaaaagtgtagtg60
tcttaggggcactatagagattccaaatctccctagatattttgttccacagtatatgat120
ccacaatgagccatgtattttatataattttatttatttttttataattactcataggtt180
atgtttgaatgctagtaatcacaaggagaatagcaatatgatttcaaatctttggaattt240
taccatttggataaagtaggaataagaatataatatttattgatattttaaattagattc300
ataaagtaacgaggaatctgattctgcaaagatcctaattaatttcaaatttctccgtca360
tttttttataaatttgataacaaatataaatccttttttaaaatattatttcgtgattaa420
gattctgaacaaaggcaagtacttgaataagattaactctttcattaaattattaaaaat480
tatattattaataacgaaaacgatcatccaaacacagcataggggaatggatatattccg540
tggtatcaatgaaatttgcaaatgtgcgtgagaacccaactttattcatggtgaaaataa600
gcgaataggaagggcaaatttcgccaatggtctctgtagtttaacaaaagtttcactttg660
gttcttataattttaatataaacaattaaaattttgaacttttaattttatttttatata720
gttgttctatctataatcgttaagaatttgaatggaaagtgagaacatagagctcacgta780
ggccctttttagaggataattttactactatttttgcttataatttggttaaaattttac840
tttagtccttataaataatcacgagttttcacatgctttttctccatttatgttattaat900
ttttattgacagagggaagctacattgaaatccaattgaaactttagagatataactgtc960
caaattgaaaataaaggaactaaagtgaacttttaatcaaattacaaagacaagatgaaa1020
ttttcccgaataacaacttagcacattggcatattcgtcgctctcttgtgtatagacaac1080
actgccctcaactattcactgttactacaaaattgccacacattcaagctaaagcatcaa1140
cccactatgatactatgatgctgcaacacagccttgaatattaggtatttattgtagttg1200
agtttatatttaaattgaatttttattaaaaattaaatatttaaaaatagaagttaaaaa1260
atattaaaataaacttttttaaaatttaactttttaattttttagaagttatttatagtc1320
cataagtagaaaaactataataaacacagactttaaggcaaaataaatcaaaagtttcta1380
ctaataagtaacaacaaaacatgggtgtttgatagagtgaacttacactataattgccaa1440
tttaaaaataaaatttaaaaaattttaaaataaagattttaagaatcagctatttttcaa1500
ctttcgtagtttgttttggcccataagtagaagtagacgcaaaaactgtgctaaacactt1560
tggcgcaacttttaagacaaaaatggctataagttgtatgccaaacatgcaatattcgct1620
ctaaaattgccaagtatgtcaccattggtcattccgattaaacccaggggtacttttgtc1680
tttaaatttctataatttatgtactttttcattttttttttgctttttgttataattata1740
aattaatgatgtccactctgttcttccatgtaccacctgaaagatcaagaagcaagatat1800
atatattgttattctaccattctctctctctctctctctctctcatcagacaattctctt1860
tcaaaaaacaaaggaatggccaaaaacggtacggccccattaatctctcttctgtgtgtg1920
tgtgtgtgtgtgtgtgtgtgtaaatgtgtatattgtgtacaatgtatttgttatctgtgt1980
ttcgatggatttgttgtgtg2000
<210>108
<211>28
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>构建体
<400>108
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>109
<211>20
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>109
thralathrcyscysglycysthrglyglyalaalaglyalathrgly
151015
glyalaalacys
20
<210>110
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>110
tccacctcgatatgtgcatc20
<210>111
<211>246
<212>dna
<213>黄瓜(cucumissativus)
<400>111
gggtaaaacccgggttcgtcaggctgtagacatcacggctatacacgtagtttcccggtc60
gttctttcatgtcagggctgtacgacggaagggttgtataactccgacaaacccttcgcc120
gcacggcggacgtggtgctttgccttccgaaggtggtagtccttctgatcttctttttct180
cgccggcggtggtttctcttgcttcttctcttcttcgtattagctttccgtttgtgtttt240
agctct246
<210>112
<211>254
<212>dna
<213>毛花猕猴桃(a.eriantha)
<400>112
gggtaaaacccggtgtttgacaagttgtagacatcacggctataatcggagtttctcggc60
cgctcatacatgtccggtctgtacgacgcaagggttgtgtagtcgagagcaacccttcgc120
cgcacggcggacgtggcgccttgccgtccgaaggcggtagcccctccgacctcctcttcc180
tcgccggcggcggtcacttcgctttctccgtctactagcttattaggtttattcttactt240
agtgagtaattcgt254
<210>113
<211>253
<212>dna
<213>软枣猕猴桃(a.arguta)
<400>113
ggtaaaacccggtgtttgacaagttgtagacatcacggctatactcggagtttctcggcc60
gctcatacatgtccggtctgtacgacgcaagggttgtgtagtcgagagcaacccttcgcc120
gcacggcggacgtggcgccttgccgtccgaaggcggtagcccctccgacctcctcttcct180
cgccggtggcggtcacttcgctttctccgtctactagcttattaggtttattcttactta240
gtgagtaatttgt253
<210>114
<211>255
<212>dna
<213>毛花猕猴桃(a.eriantha)
<220>
<221>misc_feature
<222>(49)..(49)
<223>n为a、c、g或t。
<220>
<221>misc_feature
<222>(57)..(57)
<223>n为a、c、g或t。
<400>114
ggtaaaacccggtgtttgacaagttgtagacatcacggctatactcggnagtttctncgg60
ccgctcatacatgtccggtctgtacgacgcaagggttgtgtagtcgagagcaacccttcg120
ccgcacggcggacgtggcgccttgccgtccgaaggcggtagcccctccgacctcctcttc180
ctcgccggcggcggtcacttcgctttctccgtctactagcttattaggtttattcttact240
tagtgagtaattcgt255
<210>115
<211>253
<212>dna
<213>中华猕猴桃(a.chinensis)
<400>115
ggtaaaacccggtgtttgacaagttgtagacatcacggctatactcggagtttctcggcc60
gctcatacatgtccggtctgtacgacgcaagggttgtgtagtcgagagcaacccttcgcc120
gcacggcggacgtggcgccttgccgtccgaaggcggtagcccctccgacctcctcttcct180
cgccggcggcggtcacttcgctttctccgtctactagcttattaggtttattcttactta240
gtgagtaattcgt253
<210>116
<211>254
<212>dna
<213>苹果(malusxdomesticus)
<400>116
ggtaagacccaggttcgacgagttgtagacatcacggctatacccagagctcctcggccg60
ctcgttcatgtccggggtgtcggacgaaagggttgtgtaattgagagcaacccttcgccg120
cacggcgggcgtggtgctttgccttccgaaggcggtagcccgtctgacctcctcttcctc180
gccggtggcggctccccttcctctgtttttctcttctgcttttattagttttatttttat240
agagtttcttgctt254
<210>117
<211>247
<212>dna
<213>草莓(fragaria)
<400>117
ggtaaaacccgggtcgacgagttgtagacatcacggctatacacagagtttcacggccac60
tcattcatgtccgccggactgtccgacgtaagggttgtgtaattgagagcaacccttcgc120
cgcacggcgggcgtggtgctttgccgtccgaaggaggaagtccttccgacctccttttcc180
tcgccggcggtggtgtccacgtttccgacttgcgtttctttttctagctttttgtagatt240
cgggttt247
<210>118
<211>256
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>118
tgaaaaaatccggggaacaggtgatcggaatcacggctatacacgggatatcacggggtg60
ttagctcacatgtccatattgtccgacagaagggttgtttaatcgaaactaatcctttgc120
cgcacggaggacgtggagctctgccgtctgaaggcggcagcccttccgatctcctctttc180
tcgccggtggcggttccagctttaacttcttttcctttaggttttaggagttagggtttg240
ttagtgttttttcctt256
<210>119
<211>245
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>119
aatccccggggagattggtaatcggtaatcacggctatttacgggataaagccacggcct60
ttgagctttcatgtccaacgaaagggttgtttaatcataactaatcctttgcctcacgga120
ggacgtggagctctgccgtctgaaggcggcagtccctccgatctcctcttcctcgccgga180
ggcggttccacaaatagccactaaccctaaccctttttctaattaggtttttagttctta240
gagtc245
<210>120
<211>256
<212>dna
<213>毛果杨(populustrichocarpa)
<400>120
ggttaaaatcccagggtagacaagttgtagacatcacggctatacacggagtaactcggt60
ccctaattcatgtccgggctgttcgacgtaagggctgtgtaatagagagcagcaaccctt120
cgccgcacggtggacgtggtgctttaccctcggaaggcggtagcccttctgatctcctct180
ttctagctggtggcggtttcttcttcttctgtttagtagtagtttagtgctattgttgtt240
gttaattattattatt256
<210>121
<211>252
<212>dna
<213>大豆(glycinemax)
<400>121
ggttgaatccctgggttgaagcgttgcagacatcacggctattctaagagtgtctcgctc60
tctcattcatgtcccaactgtacgacggaggacaggttgcgtaactgccaccaacccttc120
gccgcacggtgggcgtggtgctttgccttctgaaggtggtagcccttcagacttgctctt180
cttagctggtggtggttctgctgtcttctagcttcttcttaactcttttttctttttact240
acttttaagcta252
<210>122
<211>253
<212>dna
<213>蒺藜苜蓿(medicagotruncatula)
<400>122
ggttgattccctgtattgacacgttgtagacatcacggcttttcttagagtttatcggtc60
actttcacatgcccgaactgtacgacgtaagggttgttgcttaactccgactaacccttc120
gccgcacggtgggcgtggtgctctgccttctgaaggtggtagcccctccgatcttctctt180
cctcgccggtggtggtgtctttgctccttcttccttctaatttcttgtttttagtttaac240
tttctttagattt253
<210>123
<211>252
<212>dna
<213>番茄(solanumlycopersicum)
<400>123
gggtaaagcccagtttaacaagttgtagacatcacggctatacacaaagtaaaccgccga60
ccacttttacatgttccagcagtacgtcgtaagggttgtgtaacagctactaaccctgcg120
ccgcacggtggacgtggcgctttgccttctgaaggtggtagtccttccgacctcctcttc180
cttgccggcggcggttctttcctctccttctcctactagatatagttatacttactatag240
atctctagctta252
<210>124
<211>252
<212>dna
<213>马铃薯(solanumtuberosum)
<400>124
gggtaaagcccagtttaacaaggtgtagacatcacggctatacacaaagtaaaccgccga60
ccacttttacatgttccagcagtacgtcgtaagggttgtgtaacagctactaaccctgcg120
ccgcacggtggacgtggcgctttgccctctgaaggtggcagtccttccgacctcctcttc180
cttgccggcggcggttctttcctctccttctcctactagatatagttatacttaccgtag240
atctctagctta252
<210>125
<211>258
<212>dna
<213>葡萄(vitisvinifera)
<400>125
gggtaaaacccttgttcgacgagttgtagacatcacggctatacaaagaattccgccgcc60
tctcatacatgtccgggcggtacgacgcaagggytgtgtaattgagagcaacccttcgcc120
gcacggcgggcgtggcgctttgccttcagaaggcggtagtccctctgatctgctcttcct180
cgccggcggtggttccaacgctttcctctgctagtttaggcttatattctgcataatata240
gctactgtctttaggatt258
<210>126
<211>682
<212>dna
<213>番茄(solanumlycopersicum)
<400>126
atttgttcggtatactgtaaccccctgtttgcgattggccttgtagccccgttttacatc60
ttccagagactccatttgtatcggttcacatacagtagcaaagcgccattatcttactct120
accccattggcaaacccacagccacaattttccaatcctccattatcccttctacaattt180
tctatataaatacccacatctctctgctctactcccttattatcaacaacaaccaccaaa240
tttcttcttttttttcttcgatagtagcaatctatcaacaaaaacagagaccccatcaca300
agaatcttggaattttagtgttgggtttaagaggaaaaggggttattgtattttgcagtt360
ttgagggtaaagcccagtttaacaagttgtagacatcacggctatacacaaagtaaaccg420
ccgaccacttttacatgttccagcagtacgtcgtaagggttgtgtaacagctactaaccc480
tgcgccgcacggtggacgtggcgctttgccttctgaaggtggtagtccttccgacctcct540
cttccttgccggcggcggttctttcctctccttctcctactagatatagttatacttact600
atagatctctagcttattacgtacagttgtatctagtattctattgattattcgaagaaa660
acacacaaaaagaagtaaagcc682
<210>127
<211>472
<212>dna
<213>马铃薯(solanumtuberosum)
<400>127
taagggggtgcttatataaagttggggagtctaccaatgagacgaactcattgaccaaat60
acgtctgcaggagaaagaccaccggagcaccaaacgccacccaacaaccacccattaaat120
tcttccagaaaaaaacatcttcctcaaaattatcgatgaaggatcgttccttagtagttg180
ttcgttgatcctacaaattcaatcacggctcttcttggatctttcgtttgtattctcaca240
attcatcatcaccgcaaagtgttgacccttaatccaactcttctggtggacgataagcac300
cggaccccttcccctcacggaggtaggggtgcctcacccgctgaaggcggttgcccctcc360
gatctcctcttcctcgccggcggcggtccaattcttcctttctctttctccttctcctaa420
tttttcgtgtaagaattgtatttttgattatccatccaagaacaggaccgcc472
<210>128
<211>607
<212>dna
<213>苹果(malusxdomesticus)
<400>128
ccacggtacaccctcagccacgaacaccccttcttctccccacacctataaatccacccc60
ctcatctcctccccacacccccactcacttcagttcgaaacaggcgatcctcgcctttct120
gggttgtttcctattttatctgagggagaagaaaggaaggtgtttgatcaattttttggt180
atatttttaggggtaagacccaggttcgacgagttgtagacatcacggctatacacggag240
ctcctcggccgctcattcatgtccgggctgtccgacgaaagggttgtgtaattgagagca300
acccttcgccgcacggcgggcgtggcgctttgccttccgaaggcggtagcccctccgacc360
tgctcttcctcgctggtggcggttctgcatcctctgtttttctcttctgcttatattagc420
ttttttagactttcttggttagattcttaggagattttagagattttttttcttctataa480
agcgcacgagtagatcgtattgttgttttcggggggttttgggtttggtggtgtttgatt540
ttactgagaattaagaaaaaataaaaggaaaaaaaagagagagagaaagaaggggaggga600
gcatgcc607
<210>129
<211>183
<212>dna
<213>番茄(solanumlycopersicum)
<400>129
acggctatacacaaagtaaaccgccgaccacttttacatgttccagcagtacgtcgtaag60
ggttgtgtaacagctactaaccctgcgccgcacggtggacgtggcgctttgccttctgaa120
ggtggtagtccttccgacctcctcttccttgccggcggcggttctttcctctccttctcc180
tac183
<210>130
<211>213
<212>dna
<213>马铃薯(solanumtuberosum)
<400>130
acggctcttcttggatctttcgtttgtattctcacaattcatcatcaccgcaaagtgttg60
acccttaatccaactcttctggtggacgataagcaccggaccccttcccctcacggaggt120
aggggtgcctcacccgctgaaggcggttgcccctccgatctcctcttcctcgccggcggc180
ggtccaattcttcctttctctttctccttctcc213
<210>131
<211>192
<212>dna
<213>苹果(malusxdomesticus)
<400>131
acggctatacacggagctcctcggccgctcattcatgtccgggctgtccgacgaaagggt60
tgtgtaattgagagcaacccttcgccgcacggcgggcgtggcgctttgccttccgaaggc120
ggtagcccctccgacctgctcttcctcgctggtggcggttctgcatcctctgtttttctc180
ttctgcttatat192
<210>132
<211>61
<212>prt
<213>番茄(solanumlycopersicum)
<400>132
thralailehislysvalasnargargproleuleuhisvalproala
151015
valargarglysglycysvalthralathrasnproalaprohisgly
202530
glyargglyalaleuprosergluglyglyserproseraspleuleu
354045
pheleualaglyglyglyserpheleuserphesertyr
505560
<210>133
<211>71
<212>prt
<213>马铃薯(solanumtuberosum)
<400>133
thralaleuleuglyserphevalcysileleuthrilehishishis
151015
arglysvalleuthrleuasnprothrleuleuvalaspasplyshis
202530
argthrproserprohisglyglyargglyalaserproalaglugly
354045
glycysproseraspleuleupheleualaglyglyglyproileleu
505560
propheserpheserpheser
6570
<210>134
<211>64
<212>prt
<213>苹果(malusxdomesticus)
<400>134
thralailehisglyalaproargproleuilehisvalargalaval
151015
argarglysglycysvalilegluserasnproserprohisglygly
202530
argglyalaleuprosergluglyglyserproseraspleuleuphe
354045
leualaglyglyglyseralaserservalpheleuphecysleutyr
505560
<210>135
<211>28
<212>prt
<213>番茄(solanumlycopersicum)
<400>135
asnproalaprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>136
<211>28
<212>prt
<213>马铃薯(solanumtuberosum)
<400>136
thrproserprohisglyglyargglyalaserproalagluglygly
151015
cysproseraspleuleupheleualaglyglygly
2025
<210>137
<211>28
<212>prt
<213>苹果(malusxdomesticus)
<400>137
asnproserprohisglyglyargglyalaleuprosergluglygly
151015
serproseraspleuleupheleualaglyglygly
2025
<210>138
<211>84
<212>dna
<213>番茄(solanumlycopersicum)
<400>138
aaccctgcgccgcacggtggacgtggcgctttgccttctgaaggtggtagtccttccgac60
ctcctcttccttgccggcggcggt84
<210>139
<211>84
<212>dna
<213>马铃薯(solanumtuberosum)
<400>139
accccttcccctcacggaggtaggggtgcctcacccgctgaaggcggttgcccctccgat60
ctcctcttcctcgccggcggcggt84
<210>140
<211>84
<212>dna
<213>苹果(malusxdomesticus)
<400>140
aacccttcgccgcacggcgggcgtggcgctttgccttccgaaggcggtagcccctccgac60
ctgctcttcctcgctggtggcggt84
- 还没有人留言评论。精彩留言会获得点赞!