一种无标签用于基因编辑的试剂组合及其应用的制作方法
本发明涉及生物技术领域,具体地,涉及一种无标签用于基因编辑的试剂组合及其应用。
背景技术:
基因编辑技术近年来发展迅速,其最终目的是能在基因组的任何位置产生稳定且可遗传的基因序列改变,包括指定片段或碱基的定点插入、删除或替换。
目前,锌指核酸酶技术(zincfingernuclease,zfn)、转录激活因子样效应物核酸酶技术(transcriptionactivator-likeeffectornucleases,talen)和crispr/cas9技术(clusteredregularlyinterspacedshortpalindromicrepeats/crispr-associatedprotein9)已成功地应用于多种生物,包括大肠杆菌、酵母、水稻、拟南芥、果蝇、小鼠和人类细胞等的基因编辑中。这三种技术都能在生物体基因组指定位点特异性地切割dna,产生双链断裂,从而利用生物自身的dna修复机制,进行定点编辑。细胞dna自我修复方式有两种,非同源末端连接(non-homologousend-joining,nhej)和同源重组(homology-directed,hdr)。nhej倾向于在基因组中随机删除、插入或替换一段序列或单个碱基,在修复时发生概率较高;hdr可实现精准修复,但需要有同源序列为模板,且发生概率较低。因此现有的利用hdr的基因编辑技术,为提高阳性率,需要在引入的外源同源序列中加上筛选标签。
当下,在植物基因组中实现指定序列或碱基的定点插入或替换仍是一项难题。zfn和talen技术需要特异性的蛋白引导,但蛋白元件的构建相对复杂,成本较高,且编辑效率较低。crispr/cas9技术以rna引导,体外构建简单,成本较低,同时结合hdr,可实现精准基因编辑。但需要在引入的外源序列中加上筛选标签,否则编辑效率非常低,难以有效地应用于植物研究和育种。
综上所述,为了植物科学研究和育种生产的需要,本领域迫切需要开发一种高效的无需筛选标签的基因靶向敲入或替换技术。
技术实现要素:
本发明的目的在于提供一种高效的无需筛选标签的基因靶向敲入或替换技术。
本发明第1a方面提供了一种无标签用于基因编辑的试剂组合,包括:
(i)第一核酸构建物,或含有所述第一核酸构建物的第一载体,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织特异性启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;和
(ii)第二核酸构建物,或含有所述第二核酸构建物的第二载体,所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna的编码序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
在另一优选例中,所述核苷酸连接序列为1-60nt。
在另一优选例中,所述核苷酸连接序列不影响各元件的正常转录和翻译。
在另一优选例中,所述组织特异性启动子包括生殖细胞特异表达的启动子。
在另一优选例中,所述生殖细胞特异表达的启动子选自下组:胚珠特异表达的启动子、花粉特异表达的启动子、胚胎发育早期特异表达的启动子、或其组合。
在另一优选例中,所述胚珠特异表达的启动子包括dd45启动子。
在另一优选例中,所述花粉特异表达的启动子包括lat52启动子。
在另一优选例中,所述胚胎发育早期特异表达的启动子包括dd45启动子。
在另一优选例中,所述cas9蛋白选自下组:cas9、cas12a(cpf1)、cas12b、或其组合。
在另一优选例中,所述cas9蛋白的来源选自下组:酿脓链球菌(streptococcuspyogenes)、葡萄球菌(staphylococcusaureus)、或其组合。
在另一优选例中,所述cas12a蛋白的来源选自下组:氨基酸球菌属(acidaminococcus)、毛螺科菌(lachnospiraceaebacterium)、或其组合。
在另一优选例中,所述cas12b蛋白的来源包括噬酸耐热菌(alicyclobacillusacidoterrestris)。
在另一优选例中,所述终止子选自下组:nos终止子、ubq终止子、或其组合。
在另一优选例中,所述供体dna的长度为600-3000bp,较佳地,700-2800bp。
在另一优选例中,所述polyt序列选自下组:ttttttt。
在另一优选例中,所述第一核酸构建物、和/或第二核酸构建物还包括增强子元件。
在另一优选例中,所述增强子元件选自下组:atubi10基因的第一个内含子(intron)序列、tmvomega序列、或其组合。
在另一优选例中,所述第一载体和所述第二载体为不同的载体。
在另一优选例中,所述第一核酸构建物和所述第二核酸构建物位于不同的载体上。
在另一优选例中,所述第一载体和所述第二载体为相同的载体。
在另一优选例中,所述第一核酸构建物和所述第二核酸构建物位于相同的载体上。
在另一优选例中,所述供体dna上不带有筛选标签。
在另一优选例中,所述的载体为可转染或转化植物细胞的双元表达载体。
在另一优选例中,所述的载体为植物表达载体。
在另一优选例中,所述的载体为pcambia载体。
在另一优选例中,所述的植物表达载体选自下组:pcambia1300、pcambia3301、pcambia2300、或其组合。
在另一优选例中,所述的载体为农杆菌ti载体。
在另一优选例中,所述载体是环状的或者是线性的。
在另一优选例中,所述的植物选自下组:十字花科植物、禾本科植物、豆科植物、茄科、或其组合。
在另一优选例中,所述的植物选自下组:拟南芥、水稻、白菜、大豆、番茄、玉米、烟草、小麦、高粱、大麦、燕麦、粟、花生、或其组合。
本发明第1b方面提供了一种无标签用于基因编辑的试剂组合,包括:
(i)第一核酸构建物,或含有所述第一核酸构建物的第一载体,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织被转化时期高表达启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;和
(ii)第二核酸构建物,或含有所述第二核酸构建物的第二载体,所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna的编码序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
在另一优选例中,所述组织被转化时期高表达启动子包括组织特异性启动子、和/或组成型启动子。
在另一优选例中,所述组织被转化时期高表达启动子包括农杆菌侵染期高表达启动子。
在另一优选例中,所述组织被转化时期高表达启动子选自下组:maizeubiquitin1(简称ubi1)、rubisco、actin启动子、或其组合。
在另一优选例中,所述组织被转化时期高表达启动子(px)包括满足以下条件的组织被转化时期高表达启动子:
浸花法转化时,px/p1的比值≥80%,更佳地,≥90%;
px/p1的比值≥80%,更佳地,≥90%;和/或
愈伤组织转化时,px/p2的比值≥80%,更佳地,≥90%;
px/p2的比值≥80%,更佳地,≥90%。
其中,px为组织被转化时期高表达启动子的启动强度;p1为拟南芥dd45启动子的启动强度;p2为ubi1启动子的启动强度。
在另一优选例中,所述组成型启动子选自下组:ubi1、actin、rubisco、ribosomal启动子、或其组合。
在另一优选例中,所述组织特异性启动子包括生殖细胞特异表达的启动子和/或愈伤组织高表达的启动子。
在另一优选例中,所述生殖细胞特异表达的启动子选自下组:胚珠特异表达的启动子、花粉特异表达的启动子、胚胎发育早期特异表达的启动子、或其组合。
在另一优选例中,所述胚珠特异表达的启动子包括dd45启动子。
在另一优选例中,所述花粉特异表达的启动子包括lat52启动子。
在另一优选例中,所述胚胎发育早期特异表达的启动子包括dd45启动子。
在另一优选例中,所述愈伤组织高表达的启动子包括thaumatin启动子、ribulosebisphosphatecarboxylase启动子、slahrd启动子、slrbcsc启动子、elongationfactor启动子。
在另一优选例中,所述thaumatin启动子包括选自下组的一种或多种植物来源的启动子:水稻、玉米、竹芋、或其组合。
在另一优选例中,所述ribulosebisphosphatecarboxylase启动子包括选自下组的一种或多种植物来源的启动子:番茄、甘薯、烟草、水稻、或其组合。
在另一优选例中,所述slahrd启动子包括选自下组的一种或多种植物来源的启动子:番茄、甘薯、木薯、或其组合。
在另一优选例中,slrbcsc启动子包括选自下组的一种或多种植物来源的启动子:番茄、甘薯、木薯、或其组合。
在另一优选例中,所述组成型启动子选自下组:ubi1、actin、rubisco、ribosome启动子、或其组合。
在另一优选例中,所述第一核酸构建物、和/或第二核酸构建物还包括增强子元件。
在另一优选例中,所述增强子元件选自下组:atubi10基因的第一个内含子(intron)序列、tmvomega序列、或其组合。
在另一优选例中,所述第一载体和所述第二载体为相同或不同的载体。
在另一优选例中,所述第一核酸构建物和所述第二核酸构建物位于相同或不同的载体上。
在另一优选例中,所述第一核酸构建物和所述第二核酸构建物位于同一载体。
在另一优选例中,所述供体dna上不带有筛选标签。
本发明第二方面提供了一种试剂盒,所述试剂盒含有本发明第1a方面或本发明第2a方面所述的试剂组合。
在另一优选例中,所述试剂盒还含有标签或说明书。
本发明第3a方面提供了一种对植物进行基因编辑的方法,包括步骤:
(i)提供待编辑植物,作为亲代植物;
(ii)将第一核酸构建物或含所述第一核酸构建物的第一载体导入所述待编辑植物的植物细胞,从而获得导入所述待编辑植物的植物细胞;
其中所述植物细胞选自下组:
(a1)来自所述植物的离体细胞;
(a2)所述植物的离体细胞形成的愈伤组织的细胞;
(a3)位于所述植株上的来自繁殖器官的细胞;
(iii)获得来源于所述导入所述待编辑植物的植物细胞的子代植物的植株;
(iv)将第二核酸构建物或含有所述第二核酸构建物的第二载体导入所述子代植株的植物细胞,
其中所述植物细胞选自下组:
(b1)来自所述子代植物的离体细胞;
(b2)所述子代植物的离体细胞形成的愈伤组织的细胞;
(b3)位于所述子代植物的植株上的来自繁殖器官的细胞;
其中,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织特异性启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;
所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna的编码序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
在另一优选例中,所述的繁殖器官包括花。
在另一优选例中,所述的细胞包括:花粉细胞。
在另一优选例中,步骤(ii)中,植物细胞为(a3);和/或步骤(iv)中,植物细胞为(b3)。
在另一优选例中,所述方法包括:
(1)提供待编辑植物;
(2)将第一核酸构建物或含所述第一核酸构建物的第一载体导入所述待编辑植物的植物细胞;和
(3)经过t1时间后,将第二核酸构建物或含有所述第二核酸构建物的第二载体导入所述待编辑植物的植物细胞;
其中,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织特异性启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;
所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna的编码序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
在另一优选例中,所述t1为10天-500天,较佳地,15天-200天。
在另一优选例中,所述植物细胞来自花、愈伤组织、或其组合。
在另一优选例中,所述的愈伤组织用选自下组的植物细胞诱导形成:根、茎、叶、花、和/或种子的细胞。
在另一优选例中,当步骤(ii)中植物细胞为(a3)位于所述植株上的来自繁殖器官的细胞(如采用拟南芥的花的细胞)时,则所述t1为42天-70天,较佳地,49天-63天。
在另一优选例中,当步骤(ii)中植物细胞为(a2)所述植物的离体细胞形成的愈伤组织的细胞(如采用水稻愈伤组织的细胞)时,则所述t1为15天-40天,较佳地,25天-30天。
在另一优选例中,所述步骤(ii)和步骤(iv)中的植物细胞来自同一部位。
在另一优选例中,所述步骤(2)和步骤(3)中的植物细胞来自同一部位。
在另一优选例中,所述导入为通过农杆菌导入。
在另一优选例中,所述导入为通过基因枪导入。
在另一优选例中,所述的基因编辑为定点敲入或替换。
本发明第3b方面提供了一种对植物进行基因编辑的方法,包括步骤:
(i)提供待编辑植物,作为亲代植物;
(ii)将第一核酸构建物或含所述第一核酸构建物的第一载体导入所述待编辑植物的植物细胞,从而获得导入所述待编辑植物的植物细胞;
其中所述植物细胞选自下组:
(a1)来自所述植物的离体细胞;
(a2)所述植物的离体细胞形成的愈伤组织的细胞;
(a3)位于所述植株上的来自繁殖器官的细胞;
(iii)获得来源于所述导入所述待编辑植物的植物细胞的子代植物的植株;
(iv)将第二核酸构建物或含有所述第二核酸构建物的第二载体导入所述子代植株的植物细胞,
其中所述植物细胞选自下组:
(b1)来自所述子代植物的离体细胞;
(b2)所述子代植物的离体细胞形成的愈伤组织的细胞;
(b3)位于所述子代植物的植株上的来自繁殖器官的细胞;
其中,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织被转化时期高表达启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;
所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna的编码序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
本发明第3c方面提供了一种对植物进行基因编辑的方法,包括步骤:
(i)提供待编辑植物,作为亲代植物;
(ii)将第一核酸构建物或含所述第一核酸构建物的第一载体、和第二核酸构建物或含所述第二核酸构建物的第二载体导入所述待编辑植物的植物细胞,从而获得导入所述待编辑植物的植物细胞;
其中所述植物细胞选自下组:
(a1)来自所述植物的离体细胞;
(a2)所述植物的离体细胞形成的愈伤组织的细胞;
(a3)位于所述植株上的来自繁殖器官的细胞;
(iii)获得来源于所述导入所述待编辑植物的植物细胞的植株;其中,
其中,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织特异性启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;
所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna的编码序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
在另一优选例中,所述第一载体、第二载体为相同或不同的载体。
在另一优选例中,所述第一核酸构建物和第二核酸构建物位于相同或不同的载体上。
本发明第3d方面提供了一种对植物进行基因编辑的方法,包括步骤:
(i)提供待编辑植物,作为亲代植物;
(ii)将第一核酸构建物或含所述第一核酸构建物的第一载体、和第二核酸构建物或含所述第二核酸构建物的第二载体导入所述待编辑植物的植物细胞,从而获得导入所述待编辑植物的植物细胞;
其中所述植物细胞选自下组:
(a1)来自所述植物的离体细胞;
(a2)所述植物的离体细胞形成的愈伤组织的细胞;
(a3)位于所述植株上的来自繁殖器官的细胞;
(iii)获得来源于所述导入所述待编辑植物的植物细胞的植株;其中,
其中,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织被转化时期高表达启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;
所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna的编码序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
在另一优选例中,所述第一载体、第二载体为相同或不同的载体。
在另一优选例中,所述第一核酸构建物和第二核酸构建物位于相同或不同的载体上。
本发明第四方面提供了一种制备转基因植物细胞的方法,包括步骤:
(i)将本发明第1a方面或本发明第1b方面所述的试剂组合转染植物细胞,使得所述试剂组合中的所述构建物与所述植物细胞中的染色体发生定点敲入和/或替换,从而制得所述转基因植物细胞。
在另一优选例中,所述的转染采用农杆菌转化法或基因枪轰击法。
本发明第五方面提供了一种制备转基因植物细胞的方法,包括步骤:
(i)将本发明第1a方面或本发明第1b方面所述的试剂组合转染植物细胞,使得所述植物细胞含有所述试剂组合中的所述构建物,从而制得所述转基因植物细胞。
本发明第六方面提供了一种制备转基因植物的方法,包括步骤:
将本发明第四方面或本发明第五方面所述方法制备的所述转基因植物细胞再生为植物体,从而获得所述转基因植物。
本发明第七方面提供了一种转基因植物,所述的植物是用本发明第六方面所述的方法制备的。
应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
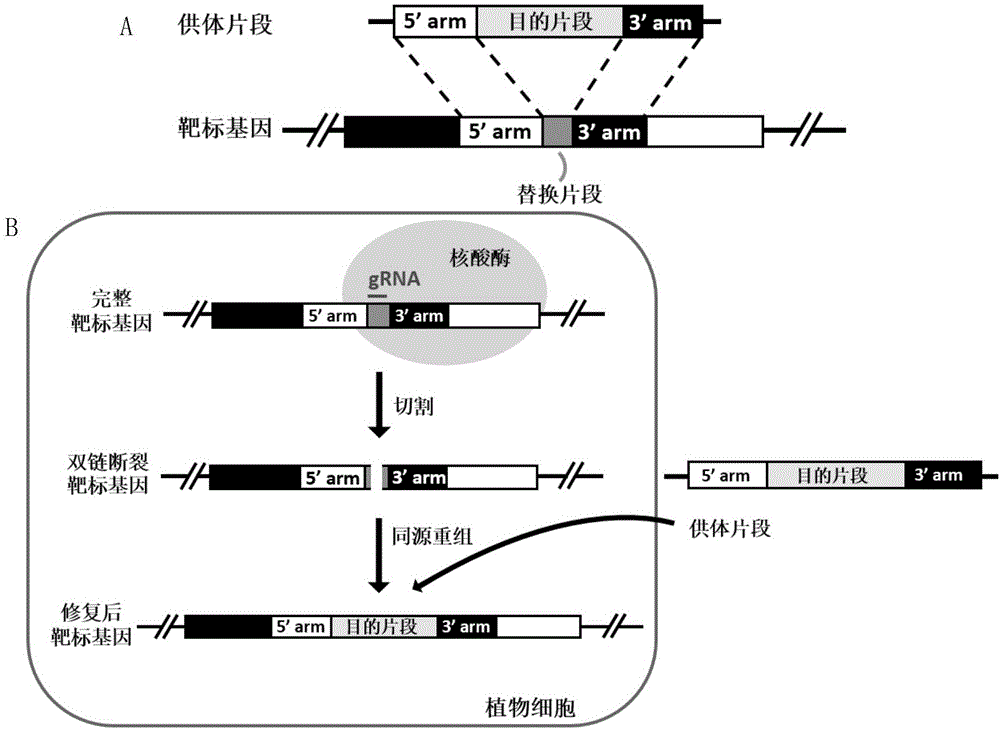
图1为靶向敲入或替换示意图。
(a)外源供体dna片段制备示意图。
(b)利用不含筛选标签的供体dna通过hdr机制进行植物靶向敲入示意图。
图2为在拟南芥ros1基因3’端靶向敲入gfp片段示意图。
(a)dd45::cas9载体构建示意图。35s::hpt是双元载体pcambia1300原有组件。
(b)gfp供体片段和表达grna的载体构建示意图。35s::bar是双元载体pcambia3301原有组件。
(c)gfp供体片段、ros121st外显子序列、grna序列、5’arm、3’arm和pcr检测引物设计示意图。
图3为在拟南芥ros1基因3’端靶向敲入gfp片段检测结果。
(a)目的植株t1代pcr筛选结果。
(b)目的植株t2代pcr基因型鉴定结果。
(c)gfp靶向敲入dna测序结果。
图4为拟南芥ros1-gfp表达水平和功能检测结果。
(a)荧光定量pcr检测gfp纯合、杂合和未敲入的拟南芥转基因植株中ros1-gfpmrna的表达水平。野生型col-0为对照。
(b)激光共聚焦荧光显微镜检测gfp纯合植株根尖细胞中gfp信号。野生型col-0为阴性对照。
(c)定量chop-pcr检测gfp纯合、杂合和未敲入的拟南芥转基因植株中ros1-gfp蛋白去甲基化功能。at1g26400和at1g03890是已知的在col-0中的低甲基化、在ros1-4功能缺失突变体中的高甲基化位点。
图5为在拟南芥dme基因3’端靶向敲入gfp片段示意图。
(a)gfp供体片段和表达grna的载体的构建示意图。35s::bar是双元载体pcambia3301原有组件。
(b)gfp供体片段、dme20th外显子序列、grna序列、5’arm、3’arm和pcr检测引物设计示意图。
在上述各图中,“arm”指同源臂。
图6为在水稻osros1a基因3’端靶向敲入gfp基因示意图。
(a)ubi1::cas9载体构建示意图。35s::hpt是双元载体pcambia1300原有组件。
(b)gfp供体片段和表达grna的载体构建示意图。35s::bar是双元载体pcambia3301原有组件。
(c)gfp供体片段、osros118th外显子序列、grna序列、5’arm、3’arm和pcr检测引物设计示意图。
图7为在水稻osros1a基因3’端靶向敲入gfp片段检测结果。
(a)t0阳性单株pcr筛选结果。
(b)t1单株pcr基因型鉴定结果。
(c)gfp靶向敲入dna测序结果。
图8为使用一步法在拟南芥ros1基因3’端靶向敲入gfp片段示意图及检测结果;
(a)一步法载体构建示意图。其上包括dd45::cas9组件,gfp供体片段,和表达grna的组件。35s::hpt是双元载体pcambia1300原有组件。
(b)t1单株pcr基因型鉴定结果。
图9为在一步法基础上使用增强子在拟南芥ros1基因3’端靶向敲入gfp片段示意图及检测结果;
(a)一步法载体构建示意图。其上包括dd45::增强子-cas9组件,gfp供体片段,和表达grna的组件。35s::hpt是双元载体pcambia1300原有组件。
(b)t1单株pcr基因型鉴定结果。
具体实施方式
本发明人经过广泛而深入地研究,通过大量筛选,筛选出特定的组织被转化时期高表达启动子和/或农杆菌侵染期高表达启动子(包括组织特异性启动子、和/或组成型启动子),通过采用特定结构的核酸构建物,本发明在植物中成功实现了高效的rna引导的基因编辑(碱基定点敲入和/或替换),并且在本发明中,敲入和/或替换效率可高达5~10%或更高,并且本发明的供体dna不带有筛选标签,首次实现了目的基因的无标签无累赘、精确无缝、稳定可遗传的编辑。此外,本发明首次发现,本发明的方法为通用方法,一步法或两步法均可达到非常高的敲除和/或替换效率。在此基础上,本发明人完成了本发明。
术语
如本文所用,术语“植物启动子”指能够在植物细胞中启动核酸转录的核酸序列。该植物启动子可以是来源于植物、微生物(如细菌、病毒)或动物等,或者是人工合成或改造过的启动子。
如本文所用,术语“cas蛋白”指一种核酸酶。一种优选的cas蛋白是cas9蛋白。典型的cas9蛋白包括(但并不限于):来源于酿脓链球菌(streptococcuspyogenes)、葡萄球菌(staphylococcusaureus)的cas9。如本文所用,术语“cas蛋白的编码序列”指编码具有切割活性的cas蛋白的核苷酸序列。在插入的多聚核苷酸序列被转录和翻译从而产生功能性cas蛋白的情况下,技术人员会认识到,因为密码子的简并性,有大量多聚核苷酸序列可以编码相同的多肽。另外,技术人员也会认识到不同物种对于密码子具有一定的偏好性,可能会根据在不同物种中表达的需要,会对cas蛋白的密码子进行优化,这些变异体都被术语“cas蛋白的编码序列”所具体涵盖。此外,术语特定地包括了全长的、与cas基因序列基本相同的序列,以及编码出保留cas蛋白功能的蛋白质的序列。
如本文所用,术语“植物”包括全植株、植物器官(如叶、茎、根等)、种子和植物细胞以及它们的子代。可用于本发明方法的植物的种类没有特别限制,一般包括任何可进行转化技术的高等植物类型,包括单子叶、双子叶植物和裸子植物。
如本文所用,术语“碱基敲入”指大片段的置换,尤其是当置换上的是和原基因完全不同的序列时。
如本文所用,术语“碱基替换”指小片段、几个氨基酸、几个碱基的置换。
如本文所用,术语“表达盒”是指含有待表达基因以及表达所需元件的序列组件的一段多聚核苷酸序列。表达所需的组件包括启动子和聚腺苷酸化信号序列。此外,本发明的表达盒还可以含有或不含有其他序列,包括(但并不限于):增强子、分泌信号肽序列等。
无标签用于基因编辑的试剂组合
本发明提供了一种无标签用于基因编辑的试剂组合,所述试剂组合包括第一核酸构建物或含有所述第一构建物的第一载体;和第二构建物或含有所述第二构建物的第二载体,其中,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织特异性启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;
所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna转录序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
在本发明中,还提供了一种无标签用于基因编辑的试剂组合,包括:
(i)第一核酸构建物,或含有所述第一核酸构建物的第一载体,所述第一核酸构建物具有从5’-3’的式i结构:
p1-z1-z2(i)
其中,p1为第一启动子,所述第一启动子为组织被转化时期高表达启动子;
z1为编码cas9蛋白编码序列;
z2为终止子;
并且,“-”为键或核苷酸连接序列;和
(ii)第二核酸构建物,或含有所述第二核酸构建物的第二载体,所述第二核酸构建物具有从5’-3’的式ii所示的结构:
p2-z3-z4-z5(ii)
其中,p2为第二启动子,所述第二启动子选自下组:u6、u3、7sl、或其组合;
z3为grna的编码序列;
z4为polyt序列;
z5为供体dna序列;
并且,“-”为键或核苷酸连接序列。
在本发明中,所述组织被转化时期高表达启动子是指在植物组织(如花蕾、愈伤组织、子叶等)的生长发育阶段高表达的强启动子,具体地,所述组织被转化时期高表达启动子包括满足以下条件的组织被转化时期高表达启动子:
px/p1的比值≥80%,更佳地,≥90%;和/或
px/p2的比值≥80%,更佳地,≥90%;
其中,px为组织被转化时期高表达启动子的启动强度;p1为拟南芥dd45启动子的启动强度;p2为maizeubi1启动子的启动强度。
在本发明中,组织被转化时期包括转染和侵染。当适用于农杆菌侵染时,使用农杆菌侵染期高表达启动子。在本发明中,农杆菌侵染期高表达启动子指在农杆菌转染阶段可高表达的强启动子。
本发明的构建物中所用的各种元件可以用常规方法,如pcr方法、全人工化学合成法、酶切方法获得,然后通过熟知的dna连接技术连接在一起,就形成了本发明的构建物。
将本发明的载体转化植物细胞从而介导本发明的载体对植物细胞染色体进行整合,制得转基因植物细胞。
将本发明的转基因植物细胞再生为植物体,从而获得转基因植物。
将本发明构建好的上述核酸构建物,通过常规的植物重组技术(例如农杆菌转化技术),可以导入植物细胞,从而获得携带所述核酸构建物(或带有所述核酸构建物的载体)的植物细胞,或获得基因组中整合有所述核酸构建物的植物细胞。
基因编辑(geneediting)
如本文所述,“基因编辑”是指对基因进行人为有目标的改变,就是在生物基因组的特定位点上产生dna的删除、插入或替换。
“基因打靶(genetargeting)”或“基因靶向”是指基于同源重组原理(一种细胞内固有的dna损伤修复机制)对基因组进行的改造,主要是基因敲入、替换。
在本发明中,基因编辑包括基因打靶。
载体构建
该载体的主要特征是利用植物组织被转化时期高表达启动子(农杆菌浸花法中如dd45,农杆菌愈伤组织转化法中如ubi1)驱动crispr/cas系统中的cas蛋白在被转化的植物组织中大量表达,并由guiderna引导至基因组中的靶点位置,由cas蛋白切割靶点,并通过hdr机制进行植物靶向敲入或替换。
一般的,为了增加蛋白的活性,蛋白间一般通过一些柔性短肽连接,即linker(连接肽序列)。优选的,该linker可以选用attb。
为了增加敲入和/或替换效率,本发明选择特定的适用于植物细胞的启动子,比如dd45启动子、u6启动子、lat52启动子、ubi1启动子等。选择适用于植物细胞的guiderna的表达框,并将其与上述蛋白的开放表达框(orf)构建在不同的载体。
在本发明中,所述载体没有特别限制,任何双元载体都可以,不限于pcambia载体,也不限于这两种抗性,只要满足如下要求的载体都可以用在本发明中:(1)能通过农杆菌介导,转化进入植物中;(2)让rna正常转录;(3)让植物获得新的抗性。
在一优选实施方式中,所述载体选自下组:pcambia1300、pcambia3301、pcambia2300、或其组合。
遗传转化
将上述载体通过合适的方法导入到植物受体中。导入方法包括但不局限于:农杆菌转染法、基因枪法、显微注射法、电击法、超声波法和聚乙二醇(peg)介导法等。受体植物包括但不限于拟南芥、水稻、大豆、番茄、玉米、烟草、小麦、高粱等。上述dna载体或片段导入植物细胞后,使转化的植物细胞中的dna表达该蛋白和guiderna。cas蛋白在其guiderna的引导下,对靶点位置进行基因编辑(敲入和/或替换)。
对于用本发明方法进行植物基因组定点替换后的植物细胞或组织或器官,可以用常规方法再生获得相应的转基因植株。例如,通过农杆菌浸花法获得基因编辑后的植株。
应用
本发明可以用于植物基因工程领域,用于植物研究和育种,尤其是具有经济价值的农作物、林业作物或园艺植物的遗传改良。
本发明的主要优点包括:
(1)本发明首次提供了一种适用于植物的高效的指定片段或碱基定点敲入或替换方法,实现了目的基因的无标签无累赘、精确无缝、稳定可遗传的编辑,其效率可高达5~10%或更高,可以广泛地应用于植物科学研究和育种生产。
(2)本发明的供体dna不需含有筛选标签。
(3)本发明首次使用组织被转化时期高表达启动子(包括组织特异性启动子(如生殖细胞特异表达的启动子dd45)和/或愈伤组织高表达的启动子ubi1)驱动cas9核酸酶的表达,从而高效的实现指定片段或碱基定点敲入或替换,编辑效率可达3%~10%或更高。
(4)本发明首次使用农杆菌“两步转化法”获得目的转化子植株,即首次采用先导入cas核酸酶、再导入供体dna和guiderna的方法,实现了高效的编辑。
(5)本发明仅对期望编辑的基因位点进行基因编辑(敲入和/或替换),该基因的其他序列,尤其是作为同源臂的5’arm和3’arm序列并没有发生碱基或片段的改变。
(6)本发明的植物基因编辑方法简便,容易推广应用。
(7)本发明也可用“一步法”获得目的转化子植株,因此本发明的方法具有普适性。
(8)本发明中的“一步法”在进行元件优化后,包括在cas9核酸酶的表达元件中添加增强子等组分(如atubi10基因的第一个intron序列、tmvomega序列等),编辑效率可达4%或更高。
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。除非另外说明,否则百分比和份数按重量计算。本发明中所涉及的实验材料和试剂如无特殊说明均可从市售渠道获得。
实验方法
①拟南芥“两步转化”方法
野生型拟南芥,正常培养至盛花期(一般4周左右),用携带cas9质粒的农杆菌,按照传统方法,浸染花序,此为第一步侵染。
浸染的操作一小时内能完成,只要载体构建好,从准备农杆菌到浸染完成,只需要5天。
浸花结束后,再等3周左右,花序结实,种子完全成熟,收获。
培养种子,用潮霉素筛选出具有抗性的幼苗,比如获得36株,再鉴定cas9的存在和活性,挑出多株(如2株)cas9活性最高的(此为母株),正常培养至盛花期,再用携带donor质粒的农杆菌,浸染花序,此为第二步转染,再等3周,种子成熟收获,鉴定筛选。
cas9活性最好的植株,保留好没有被农杆菌浸染过花序所结的种子,即为含有cas9的母株,后续实验可备用。
②拟南芥“一步转化”方法
野生型拟南芥,正常培养至盛花期(一般4周左右),用同时携带cas9和donor的质粒的农杆菌,按照传统方法,侵染花序,再等3周,种子成熟收获,就可以鉴定筛选。
③作物的转化方法
如水稻、番茄、玉米、木薯等不适合用浸花法转化的作物,一般是利用农杆菌浸染愈伤组织来转化。区别只有转化材料不同,农杆菌菌种不同,其他的载体设计、植株鉴定筛选方法等都一样。
“两步转化”策略中,愈伤组织可以几小时内连续转化两次,再用2种抗性同时做阳性筛选;也可以先转化cas9载体,经过一次抗性筛选培养数天至数周,选出阳性转化子之后,再转化donor载体,再做第二次抗性筛选。
“一步转化”策略中,只有一个载体,因此只需要转化一次,抗性筛选一次。
实施例1“两步转化”法在拟南芥ros1基因3’端敲入gfp基因
利用农杆菌浸花法,将表达dd45::cas9的载体和含有供体dna片段gfp绿色荧光蛋白基因并表达guiderna的载体先后转化植物,最终将体外合成的gfp基因插入到拟南芥ros1基因的3’端区域。pcr、dna测序、定量pcr、定量chop-pcr和激光共聚焦荧光显微镜检测表明,该片段能够高效地整合至目标位置,正常发挥作用,且不影响原基因正常功能。具体操作流程如下:
1.1cas9载体制备和转基因母株(第一转基因植株)的获得
体外将拟南芥dd45启动子介导的cas9序列整合到双元载体pcambia1300(潮霉素抗性)上(图2a)。可参考毛妍斐等人的方法(maoetal.,2013)。
将此cas9质粒以热击法转化到感受态农杆菌gv3101中,再以农杆菌浸花法转化拟南芥col-0花序,收获成熟种子。利用潮霉素作为筛选剂,经常规幼苗培养筛选出具抗性的植株,即表达cas9蛋白的拟南芥母株,称为第一转基因植株。
1.2供体片段载体制备和目的植株(第二转基因植株)筛选库的获得
针对拟南芥ros1基因的最后一个外显子,即21stexon的3’端设计grna,启动子是拟南芥atu6。可参考毛妍斐等人的方法(maoetal.,2013)。
以ros121stexon的终止密码子taa为界限,向上游取801bp作为5’同源臂(5’arm),以taatccgtt为内源基因上需要替换掉的片段,向下游取325bp作为3’同源臂(3’arm)。体外将5’arm、目的片段720bpgfp基因与3’arm依次连接起来,作为供体片段(donor)(图2c)。
最后将atu6::grna组件和donor组件共同整合到双元载体pcambia3301(除草剂basta抗性)上(图2b)。
将此载体以热击法转化到农杆菌gv3101中,再以浸花法转化之前拿到的拟南芥cas9母株(即第一转基因植株)花序,收获成熟种子。
利用除草剂basta作为筛选剂,经常规幼苗培养筛选出具抗性的植株(即t1代),作为目的植株的筛选库。
拟南芥ros1基因21stexon及其上下游序列如下:
其中,单下划线所示为grna靶序列(正向),加粗黑色碱基cc之间为cas9切割位点,黑框所示为将会被替换掉的序列,斜体部分序列为5’arm,加粗斜体部分序列为3’arm。
gfp序列如下:
其中,双下划线所示为防止基因沉默的连接序列。
1.3目的植株单株的获得和靶向敲入效率检测
对筛选库中所有t1植株依次编号,单株取成熟花序提取基因组dna。
采用以下3对特异性设计引物进行pcr扩增检测(图2c):
ros1-gfp5’-f:gcagttggaaaagagagaacctgatgatcc(seqidno.:3)
ros1-gfp5’-r:ctgaacttgtggccgttcacgtc(seqidno.:4)
ros1-gfpfull-f:acctgatgatccatgttcttatttg(seqidno.:5)
ros1-gfpfull-r:ccttgtacaactctaggactgtt(seqidno.:6)
ros1-gfp3’-f:acaaccactacctgagcacc(seqidno.:7)
ros1-gfp3’-r:tgaagatcggagctggttcc(seqidno.:8)
拟南芥为二倍体植物,gfp供体dna成功靶向插入的植株中,纯合和杂合植株ros1-gfp5’-f/r的扩增子为1011bp,ros1-gfp3’-f/r的扩增子为559bp;对于ros1-gfpfull-f/r,纯合植株只有一个1758bp的扩增子,杂合还有一个1018bp的扩增子。pcr扩增后电泳检测结果如图3a所示,来自dd45-#58母株的#1是杂合子,#2、#5是纯合子,#3、#4、#6是野生型;来自dd45-#70母株的#9是杂合子,#11是纯合子,#7、#8、#10、#12是野生型。
取杂合单株#1的种子继续培养,单株pcr检测结果如图3b所示,即杂合t1的t2后代发生了基因分离,符合孟德尔遗传定律,说明gfp的替换插入是可遗传的、稳定的。
进一步的测序结果显示,gfp供体dna正确替换掉了内源基因taatccgtt序列,无缝插入了ros13’端,ros121stexon的其余序列均未产生突变(图3c)。
统计来自2个不同cas9母株背景的筛选结果,如表1所示,不含筛选标签的外源gfp的靶向敲入效率分别达到7.7%和8.3%。
表1.拟南芥ros1基因gfp靶向敲入效率
1.4敲入片段功能和靶标基因功能检测
取21天大的拟南芥真叶提总rna,反转录成cdna,荧光定量pcr检测表明,gfp稳定遗传的t3纯合和杂合植株,ros1基因的表达水平与野生型col-0相当(图4a)。取7天大的gfp纯合拟南芥幼苗根尖细胞,激光共聚焦荧光显微镜检测表明,ros1-gfp蛋白在拟南芥根尖细胞中成功表达(图4b)。取bstui酶切后的dna作为模板,定量chop-pcr检测表明,ros1-gfp蛋白仍能执行内源ros1原有去甲基化酶的正常功能(图4c)。
荧光定量pcr引物如下:
ros1-qrtpcr-f:accaaacgaagggaacagaga(seqidno.:9)
ros1-qrtpcr-r:acagtcctagagttgtacaaggt(seqidno.:10)
荧光定量chop-pcr引物如下:
at1g26400-bstui-qchop-f:tgacctgcataggctataacaca(seqidno.:11)
at1g26400-bstui-qchop-r:attggaatcaatccgagtgg(seqidno.:12)
at1g03890-bstui-qchop-f:cgtgcattattttggcagtaaca(seqidno.:13)
at1g03890-bstui-qchop-r:atgcgtccggatttcagtat(seqidno.:14)
实施例2
“两步转化”法在拟南芥ros1基因3’端敲入luc基因
利用农杆菌浸花法,将表达dd45::cas9的载体和含有供体dna片段luc荧光素酶基因并表达guiderna的载体,先后转化植物,最终将外源片段luc插入到拟南芥ros1基因的3’端区域。
2.1cas9载体制备和第一转基因植株的获得
载体制备和植株获得方法同实施例1。
2.2供体片段载体制备和第二转基因植株的获得
载体制备和植株获得方法同实施例1。不同在于,体外合成的供体dna片段变为1653bp的luc基因。
统计来自一个cas9母株背景的筛选结果,如表2所示,不含筛选标签的外源luc的在ros1基因的靶向敲入效率可达6.3%。
表2.拟南芥ros1基因luc靶向敲入效率
实施例3
“两步转化”法在拟南芥dme基因3’端敲入gfp基因
利用农杆菌浸花法,将表达dd45::cas9的载体和含有供体dna片段gfp基因并表达guiderna的载体,先后转化植物,最终将外源片段gfp插入到拟南芥dme基因的3’端区域。
3.1cas9载体制备和第一转基因植株的获得
载体制备和植株获得方法同实施例1。
3.2供体片段载体制备和第二转基因植株的获得
载体制备和植株获得方法同实施例1。不同在于:①是针对拟南芥dme基因的最后一个外显子,即20thexon的3’端设计grna。②同源臂序列取自dme基因20thexon的终止密码子的上下游(图5a、5b)。
统计来自一个cas9母株背景的筛选结果,如表3所示,不含筛选标签的外源gfp的在dme基因3’端的靶向敲入效率可达9.1%。
表3.拟南芥dme基因3’端gfp靶向敲入效率
实施例4
“两步转化”法在拟南芥dme基因5’端敲入gfp基因
利用农杆菌浸花法,将表达dd45::cas9的载体和含有供体dna片段gfp基因并表达guiderna的载体,先后转化植物,最终将外源片段gfp插入到拟南芥dme基因的5’端区域。
4.1cas9载体制备和第一转基因植株的获得
载体制备和植株获得方法同实施例1。
4.2供体片段载体制备和第二转基因植株的获得
载体制备和植株获得方法同实施例1。不同在于:①是针对拟南芥dme基因的第二个外显子,即2rdexon的5’端设计grna。②同源臂序列取自dme基因2rdexon起始密码子的上下游。
统计来自2个不同cas9母株背景的筛选结果,如表4所示,不含筛选标签的外源gfp的在dme基因5’端的靶向敲入效率可达8.3%。
表4.拟南芥dme基因5’端gfp靶向敲入效率
实施例5
“两步转化”法将拟南芥dme蛋白1633位点的脯氨酸p替换成丙氨酸a
利用农杆菌浸花法,将表达dd45::cas9的载体和含有供体dna片段gfp基因并表达guiderna的载体,先后转化植物,最终将拟南芥dme基因1633位点的脯氨酸p序列替换成丙氨酸a序列。
5.1cas9载体制备和第一转基因植株的获得
载体制备和植株获得方法同实施例1。
5.2供体片段载体制备和第二转基因植株的获得
载体制备和植株获得方法同实施例1。不同在于:①是针对拟南芥dme蛋白的1633位点设计grna。②同源臂序列取自dme蛋白1633位点的上下游基因。
统计来自一个cas9母株背景的筛选结果,如表5所示,拟南芥dme基因定点替换效率为5.3%。
表5.拟南芥dme基因定点替换效率
综上所述,利用“两步转化”法,将表达dd45::cas9的载体和含有供体dna并表达guiderna的载体先后转化植物,可以实现长度至1653bp大小的目的片段无标签、无突变、可遗传地靶向敲入或替换并正常发挥功能,编辑效率可以高达9.1%。
实施例6
“两步转化”法在水稻osros1a基因3’端敲入gfp基因
利用农杆菌愈伤组织侵染法,将表达ubi1::cas9的载体和含有供体dna片段gfp基因并表达guiderna的载体先后转化植物,最终将体外合成的外源片段gfp替换插入到水稻osros1a基因的3’端区域。pcr和dna测序表明,该片段能够整合至目标位置。具体操作流程如下:
6.1cas9载体制备和转基因母株(第一转基因植株)的获得
体外将玉米ubi1启动子介导的cas9序列整合到双元载体pcambia1300(潮霉素抗性)上(图6a)。可参考张辉等人的方法(zhangetal.,2014)。
将此cas9质粒以热击法转化到感受态农杆菌eh105中,再以农杆菌侵染法转化水稻日本晴愈伤组织。利用潮霉素作为筛选剂,经筛选后在分化培养基上培养后获得阳性的植株,即表达cas9蛋白的水稻母株,称为第一转基因植株。收集该母株的种子,诱导表达cas9蛋白的水稻愈伤组织备用。
6.2供体片段载体制备和目的植株(第二转基因植株)的获得
针对水稻osros1a基因的最后一个外显子,即18thexon的3’端设计grna,启动子是osu6。可参考张辉等人的方法(zhangetal.,2014)。
以osros1a18thexon的终止密码子tag为界限,向上游取1000bp作为5’同源臂(5’arm),以tag为内源基因上需要替换掉的片段,向下游取1000bp作为3’同源臂(3’arm)。体外将5’arm、目的片段720bpgfp基因与3’arm依次连接起来,作为供体片段(donor)(图6c)。
最后将osu6::grna组件和donor组件共同整合到双元载体pcambia3301(除草剂basta抗性)上(图6b)。
将此载体以热击法转化到农杆菌eh105中,再以农杆菌侵染法转化之前拿到的表达cas9蛋白的水稻愈伤组织(即由第一转基因植株种子诱导的愈伤组织),得到第二转基因愈伤组织库。利用除草剂basta作为筛选剂,经常规愈伤组织培养筛选后获得t0阳性的愈伤组织,后经诱导分化为水稻t0代植株,经pcr和测序检测后可得目的植株(第二转基因植株)。
水稻osros1a基因18thexon及其上下游序列如下:
其中,单下划线所示为grna靶序列(正向),加粗双下划线黑色碱基ca之间为cas9切割位点,黑框所示为将会被替换掉的序列,斜体部分序列为5’arm,加粗部分序列为3’arm。
gfp序列如前所述:
其中,双下划线所示为防止基因沉默的连接序列。
6.3靶向敲入效率检测
①阳性植株的筛选与获得
对不同t0植株依次编号,单株取叶片提取基因组dna。采用2对特异性进行pcr扩增检测。
osros1a-gfp5’-f:tcgcaggtttgacaactgaag(seqidno.:17)
osros1a-gfp5’-r:ccggtggtgcagatgaactt(seqidno.:18)
osros1a-gfpfull-f:gcagaggagcatgtctctattct(seqidno.:19)
osros1a-gfpfull-r:tggtcagcgatccatttcagg(seqidno.:20)
水稻为二倍体植物,gfp基因成功敲入的目的植株(即第二转基因植株)中,纯合和杂合植物osros1a-gfp5’-f/r的扩增子为1383bp;对于osros1a-gfpfull-f/r,杂合植株的扩增子为1857bp和1137bp。pcr产物琼脂糖凝胶电泳检测结果如图7a所示,其中#95植株(t0-95)的扩增条带明亮且大小正确,为杂合子。
进一步的测序结果显示,gfp基因正确替换掉了内源基因tag序列,无缝插入了osros1a3’端,osros1a18thexon的其余序列均未产生突变(图7c)。
取杂合植株#95的种子(t1-95)继续培养,pcr检测结果如图7b所示,即杂合t0的t1后代发生了基因分离,符合孟德尔遗传定律,说明gfp的敲入是可遗传的、稳定的。
统计显示,以供体片段载体转化水稻第一愈伤组织后,诱导分化得到33份水稻第二愈伤组织,继续培养即得33株t0单株,其中,基因打靶成功植株1株。因此计算水稻基因打靶效率为3.0%(表6)。
表6.水稻osros1a基因3’端gfp靶向敲入效率
因此,利用农杆菌二次转化法,将表达ubi1::cas9的载体和含有供体dna并表达guiderna的载体先后转化水稻愈伤组织,可以实现长至720bp大小的目的片段无标签、无突变、可遗传地靶向敲入/替换,编辑效率可以达到3.0%。
此外,采用实施例6的方法,区别在于,采用水稻愈伤组织超高表达启动子(如thaumatin启动子)介导的cas9序列,结果表明,编辑效率可以达到10%到20%。
综上所述,双子叶和单子叶的模式植物上,运用本发明的方法(“两步转化”法)都可以实现高效率的基因打靶。而且拟南芥的转化方式是农杆菌浸花法,水稻的转化方式是农杆菌愈伤组织转化,那么适合这两类转化方式的植物都可以运用此方法进行基因打靶,适用范围大大扩展。
实施例7
“一步转化”法在拟南芥ros1基因3’端敲入gfp基因
采用实施例1的方法,将外源片段gfp基因靶向敲入拟南芥ros1基因的3’端,区别在于,如图8a所示,把cas9核酸酶表达元件、grna表达元件、供体dna装到一个质粒上(pcambia1300),以哥伦比亚col-0生态型拟南芥为背景,只利用农杆菌转化一次,得到293个t1抗性植株(潮霉素抗性),作为筛选库。pcr检测结果如图8b所示。结果表明,t1植株中能筛选到目的单株,效率为0.68%(表7)。
表7.拟南芥ros1基因gfp单载体靶向敲入编辑效率
实施例8
农杆菌侵染期高表达启动子启动强度的评价方法
①拟南芥的转基因通常采用的是农杆菌浸花法,因此转化组织是盛花期的花序。当待检启动子是拟南芥内源基因的启动子时,直接提取拟南芥盛花期花序rna,然后用qrt-pcr方法检测该基因表达量(以actin7为内参基因),与dd45启动子的表达量比较。
若待检基因表达量≥80%dd45表达量,就可视为农杆菌侵染期(更广泛地说是,组织被转化时期)高表达启动子。
当待检启动子是植物外源基因的启动子时,构建载体(用该启动子驱动cas9)转化进入植物,然后同样提取转化组织的rna,qrt-pcr法检测cas9基因表达量,与dd45驱动的cas9基因表达量比较。
例如花椰菜花叶病毒camv35s启动子的评价。将35s::cas9和dd45::cas9分别转化进入拟南芥中,拿到纯合子后,检测拟南芥花序中cas9基因的表达量,结果表明,dd45的是35s的10倍。因此35s不是农杆菌侵染期高表达启动子。
②水稻等作物的转基因通常采用的是农杆菌愈伤组织转化法,因此将检测材料换成愈伤组织,其他类似,并改为与maizeubi1驱动的cas9表达量比较。
若待检基因表达量≥80%ubi1的cas9表达量,就可视为农杆菌侵染期(更广泛地说是,组织被转化时期)高表达启动子。
实施例9
添加增强子元件的“一步转化”法在拟南芥ros1基因3’端敲入gfp基因
采用实施例7的方法,将外源片段gfp基因靶向敲入拟南芥ros1基因的3’端,区别在于,如图9a所示,在cas9基因之前添加翻译增强子元件(如来自烟草花叶病毒tmv的蛋白翻译增强子omega序列),把这个优化了的cas9表达元件,再与grna表达元件、供体dna装到一个质粒上(pcambia1300),以哥伦比亚col-0生态型拟南芥为背景,只利用农杆菌转化一次,得到125个t1抗性植株(潮霉素抗性),作为筛选库。pcr检测结果如图9b所示。结果表明,t1植株中能筛选到目的单株,效率为2.4%(表8)。
同样的方法,区别在于,在cas9基因之前添加转录增强子元件(如来自拟南芥atubiquitin10基因的1stintron序列),效率为1.7%。
同样的方法,区别在于,在cas9基因之前同时添加转录和翻译增强子元件(atubi10和omega),效率为3.8%。
表8.添加增强子的拟南芥ros1基因gfp单载体靶向敲入编辑效率
对比例1
采用实施例1的方法,区别在于,cas9核酸酶的启动子用35s(强启动子,也是组成型启动子,但是在农杆菌侵染的拟南芥花序组织中表达量较低)、cdc45或yao(茎尖分生组织特异启动子,但不是花序组织这个被侵染部位的高表达启动子),统计来自7个t2种子库,结果如表9所示。结果表明,无论是利用35s::cas9将gfp片段靶向敲入拟南芥ros1基因3’端,还是利用cdc45::cas9或yao::cas9将gfp片段靶向敲入dme基因5’端,t2单株中筛选不到阳性植株,所以这3种启动子的编辑效率很低,筛选不到成功编辑的植株。
表9.由其他启动子调控cas9时对拟南芥基因编辑效率
参考文献
1.mao,y.,zhang,h.,xu,n.,zhang,b.,gou,f.,andzhu,j.k.(2013).applicationofthecrispr-cassystemforefficientgenomeengineeringinplants.molecularplant.2.zhang,h.,zhang,j.,wei,p.,zhang,b.,gou,f.,feng,z.,mao,y.,yang,l.,zhang,h.,xu,n.,andzhu,j.k.(2014).thecrispr/cas9systemproducesspecificandhomozygoustargetedgeneeditinginriceinonegeneration.plantbiotechnol.
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
序列表
<110>中国科学院上海生命科学研究院
<120>一种无标签用于基因编辑的试剂组合及其应用
<130>p2018-2526
<150>cn201810130658.2
<151>2018-02-08
<160>20
<170>siposequencelisting1.0
<210>1
<211>1242
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>1
gtaatttaatgatttgcatatcttttaatcatgaaatcaaatcggtatctttaatcgatg60
tgttattcaacaggtgagacggctgattctattcaaccgtctgttagtacgtgcatattc120
caagcaaatggtatgctttgtgacgaggagacttgtttctcctgcaacagcatcaaggag180
actagatctcaaattgtgagagggacaattttggtaagtagttgcatcaatctccttatg240
cggcttactacacttatgttttctacagattaatatgttgtaaattgtacagattccttg300
tagaacagcgatgaggggtagttttcctctaaatggaacgtactttcaagtaaatgaggt360
aagacttgaactccaataactccaactctttcattttaattatttgcattgaaacttgtg420
atgtattctattgatacaggtgtttgcggatcatgcatccagcctaaacccaatcaatgt480
cccaagggaattgatatgggaattacctcgaagaacggtctattttggtacctctgttcc540
tacgatattcaaaggtataagattttctttattggattgtgatgcgattgtttatataaa600
agaaagttgttctaaccacttgagctctatttttctcaggtttatcaactgagaagatac660
aggcttgcttttggaaaggtacattttgtattaagcagtttcttcttgttctgaaacata720
acttatcttttgatattatgtagggtacgtatgtgtacgtggatttgatcgaaagacgag780
gggaccgaagcctttgattgcaagattgcacttcccggcgagcaaactgaagggacaaca840
agctaacctcgcctaatccgttggcaagcaaacaaatacaagcttatggttaagagtgag900
agagcacactgttccaatctagttaatgtaagaaagtgaaaacgtaaagttaacagtcct960
agagttgtacaaggtttctaaatcccattttagtttcgtcttaaatttgtatcaaacact1020
tgtcacaaaaaacagacccgtagctgtgtaaactctctgttcccttcgtttggtttatat1080
ctgaatttacggttagcttttggttacctgaatcaagatctggttcgatatgtggaggat1140
ctcacttaacttggtttaaatcagatagttcaaccggaccaaagaattctattgttgtca1200
aatattagatgatgggcctattatacatggaatgtgggtcat1242
<210>2
<211>740
<212>dna
<213>人工序列(artificialsequence)
<400>2
acaagtttgtatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggt60
cgagctggacggcgacgtgaacggccacaagttcagcgtgtccggcgagggcgagggcga120
tgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgcc180
ctggcccaccctcgtgaccaccttcacctacggcgtgcagtgcttcagccgctaccccga240
ccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcg300
caccatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgaggg360
cgacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacat420
cctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaa480
gcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgt540
gcagctcgccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcc600
cgacaaccactacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcga660
tcacatggtcctgctggagttcgtgaccgccgccgggatcactcacggcatggacgagct720
gtacaagtaatacaaagtgg740
<210>3
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>3
gcagttggaaaagagagaacctgatgatcc30
<210>4
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>4
ctgaacttgtggccgttcacgtc23
<210>5
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>5
acctgatgatccatgttcttatttg25
<210>6
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>6
ccttgtacaactctaggactgtt23
<210>7
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>7
acaaccactacctgagcacc20
<210>8
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>8
tgaagatcggagctggttcc20
<210>9
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>9
accaaacgaagggaacagaga21
<210>10
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>10
acagtcctagagttgtacaaggt23
<210>11
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>11
tgacctgcataggctataacaca23
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
attggaatcaatccgagtgg20
<210>13
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>13
cgtgcattattttggcagtaaca23
<210>14
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>14
atgcgtccggatttcagtat20
<210>15
<211>2003
<212>dna
<213>水稻(oryzasativa)
<400>15
tatccttttttttgccatttcagctgatagctactctccagtcaaaatatttgccatctc60
tattgaacttttcattgtcttctgaatgtatcttactcttggatcattaatatttcattt120
tgtcacgatatagtggtataggacaataaaatcatgggaagtatttattttcatcaccaa180
tctactcatataattttcaaatgacaattataaatatcttaaaaatatattgttagttgt240
cctgtataaaataattgtcacaccctagtccacagcgacaagaatttgtgtctacaggct300
agagtgagtactctagaagtatcttcataggaatcggaataaaatgccaatgtgaatgaa360
caaggatatcaagtataccctcaaaatctctagagaggattgcgtaaatatgtaggtgta420
attaaacaattgtttcatatggagggttttcttaaaggaggtacaagacttatcaatatg480
ggtaaagtagtttttatccataggcattgttggcagaaagctgcttagggtagaatgcta540
ctccctccgtcccacaatataagagattttgagtttttgcttgcaacgtttgaccactcg600
gcttattcaaaaatttttgaaattattatttattttatttgtgacttactttattattca660
cagtactttaagtacaacttttcgttttttatatttgcaaaaaaaattgtataagacgag720
tggtcaaacgttgtacgcaaaaactcaaaatcccttatattgtgggacggagggagtact780
tatggatgccttttttgtccaagatgtcagtaacattttctttcagggatgtggattttt840
acttcttttttccctaactttttcaggatttgtgtgcgtgagaggctttgataggacatc900
aagagcacccagaccactgtatgcaagactccactttccagcaagcaaaattaccaggaa960
taaaaaatctgcaggttctgctccaggaagagatgatgaataggccatctggaaaaccag1020
aaaggaaataaagaggaggtacatatgatctgccagaagatcactgacctgaaatggatc1080
gctgaccaataagttgccgtaggcaattcaattatttctggccatatacatctgctgaaa1140
gttatgaactccagccactgacgaattcgtggtgctggtattcttcggcaacatgatcca1200
tcatacagattctatgcttggttgttgcaagcaattcttatgcggtgacagttgctgctg1260
atagggagaaaaggcatgtccggcggctcagcggctctaactgtactttcatatgagtgg1320
aaccgattgttgtacatgtgaaaagtttgccattcaaaatggtcattcatgttgttaggt1380
cattcatgtagtcgatgtcaaattaatcatcaattatttgatttgattcattcacaagtt1440
taattggctctagatacttgtgagctgcaggccagcaatgtcataatgtatgtcgggaaa1500
aggtagatataaatcgtcagcatttacctgaaatacccttcctctgtccataaatacaag1560
ggattttgggttgttaagcgtattttaagttcgatatgaaaattactttgatgttccgaa1620
gggcttattttgtcttgggttgtttagcatgtcataaggttgatattaaattactttcat1680
gcccttaggagttttggaaagttgacgcttaacagatgcgactagcagtaagcaataaat1740
gtctagatggattgagaaagtagtatttcttccgtctcaaaatattgctataagctctgt1800
ttgttagggttctaacttctaaattactttagaagttaggtatatagtgaagttgtggaa1860
ctatttaaatatagttatatctctctagtttattttttattttatgagagcacttcaatt1920
cattccactctttttttttttggaattgaaatcatttggttgtgcttcagttctagaaaa1980
tgtggagctagagttgaagacgt2003
<210>16
<211>740
<212>dna
<213>人工序列(artificialsequence)
<400>16
acaagtttgtatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggt60
cgagctggacggcgacgtgaacggccacaagttcagcgtgtccggcgagggcgagggcga120
tgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgcc180
ctggcccaccctcgtgaccaccttcacctacggcgtgcagtgcttcagccgctaccccga240
ccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcg300
caccatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgaggg360
cgacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacat420
cctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaa480
gcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgt540
gcagctcgccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcc600
cgacaaccactacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcga660
tcacatggtcctgctggagttcgtgaccgccgccgggatcactcacggcatggacgagct720
gtacaagtaatacaaagtgg740
<210>17
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>17
tcgcaggtttgacaactgaag21
<210>18
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>18
ccggtggtgcagatgaactt20
<210>19
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>19
gcagaggagcatgtctctattct23
<210>20
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>20
tggtcagcgatccatttcagg21
- 还没有人留言评论。精彩留言会获得点赞!