一种快速、无损鉴别转基因大豆方法与流程
本发明涉及到食品品质检测领域,特别是常见转基因大豆的鉴别方法,具体为一种结合PLS-DA判别法快速、无损鉴别转基因大豆方法。
背景技术:
我国为大豆的原产国也是大豆的主产国之一,但是近年来,国外的转基因大豆大量涌入中国,中国国产大豆失去了价格优势,很多企业选择低成本的转基因大豆作为原料。中国是个饮食文化上很依赖大豆的国家,各种大豆制品充斥着我们的餐桌,但是由于转基因大豆可能存在安全性问题,很多消费者选择不食用转基因食品,但是有调查显示我国存在很多大豆制品中是使用转基因大豆制作的,但是都没有进行标示,所以在食品安全监测方面,亟待一种有效的检测产品中是为转基因的方法。
目前针对于转基因大豆的定性鉴别方法有许多仍未感官评定为主,感官评定的方法的检测结果因主观因素影响较大,不适合大批量样本的检测。目前针对于转基因大豆的检测的方法主要有,酶联免疫吸附检测法,试条法,PCR技术,基因芯片等方法,但是存在实验过程繁杂,操作复杂,需要添加化学试剂等缺点。因此,有必要寻找一种更加简单、快速的转基因大豆鉴别方法。近红外光谱分析技术是今年来一种新兴的光谱分析技术,无需样品预处理,不用破坏样品,有望成为一种简单快捷的转基因大豆鉴别方法。近红外光谱仪(Near Infrared Spectrum Instrument,NIRS)是介于可见光(Vis)和中红外(MIR)之间的电磁辐射波,近红外光谱区定义为780-2526nm的区域,是人们在吸收光谱中发现的第一个非可见光区。
近红外光谱区与有机分子中含氢基团(O-H、N-H、C-H)振动的合频和各级倍频的吸收区一致,通过扫描样品的近红外光谱,可以得到样品中有机分子含氢基团的特征信息,而且利用近红外光谱技术分析样品具有方便、快速、高效、准确和成本较低,不破坏样品,不消耗化学试剂,不污染环境,仪器便携等优点,因此该技术受到越来越多人的青睐。
在实际工作中,只需要知道样品的类别和质量等级,并不需要知道样品中含有的组分数和其含量的问题,这时候需要模式识别法。近红外光谱的定性依据主要是光谱本身,因为光谱反映了真实样品的组成和结构信息,相同或者近似的样品有着相同或相近的光谱。通过应用含糊的数学光谱匹配方式进行实际的定性分析。近红外光谱常用模式识别方法主要有聚类分析、主成分分析结合马氏距离的判别、主成分分析结合最小二乘支持向量机的方式等。而偏最小二乘判别分析是一种常用的化学模式识别方法。
技术实现要素:
综上,当前亟需一种快速、无损的转基因大豆的鉴别方法,解决现有技术中转基因大豆检测技术,实验过程繁殖,操作复杂,并且往往需要破坏样品等问题。本发明拟借助近红外光谱技术构建一种快速、准确、无损的方法用于鉴别转基因大豆。
为上述目的,本发明采用以下技术方案实现:一种快速、无损鉴别转基因大豆方法,其包括以下:步骤S1:收集不同种类的转基因大豆和非转基因的大豆样品;步骤S2:将样本进行近红外光谱扫描,采集其近红外光谱,并剔除异常的样本数据;步骤S3:选取1100~2500 nm特征波段下的光谱信息,运用多元散射校正预处理方法进行对步骤S2中的光谱信息进行处理;并对处理后的光谱信息进行散射校正;步骤S4:采用留一法交叉验证,求PLS-DA最佳的因子数;具体步骤为:假设有N个样本,将每一个样本作为测试样本,其它N-1个样本作为训练样本,这样得到N个分类器,N个测试结果,用这N个结果的平均值来衡量模型的性能,从而找到求PLS-DA最佳的因子数;N为大于1的自然数;步骤S5:采用步骤S4的最佳的因子数,进行PLS-DA建模;步骤S6:对于未知的样品,利用扫描器获取其近红外光谱,再利用建立好的PLS-DA模型,预测其所属类别。
进一步的,所述步骤S3包括以下步骤:首先建立一个待测样品的理想光谱,所述理想光谱的变化与样品中成分的含量满足直接的线性关系,以该光谱为标准要求对所有其他样品的近红外光谱进行修正,其中包括基线平移和偏移校正。
本发明的上述技术方案相比现有的技术具有以下的优点:本发明所采取的主成分分析法结合Fisher判别引入到鉴别常见的致病菌,不仅能够快速鉴别出致病菌的种类,并且具有高精度、操作简单等优点,在后续的临床菌种鉴别工作中有重大贡献,具有良好的应用前景。
附图说明
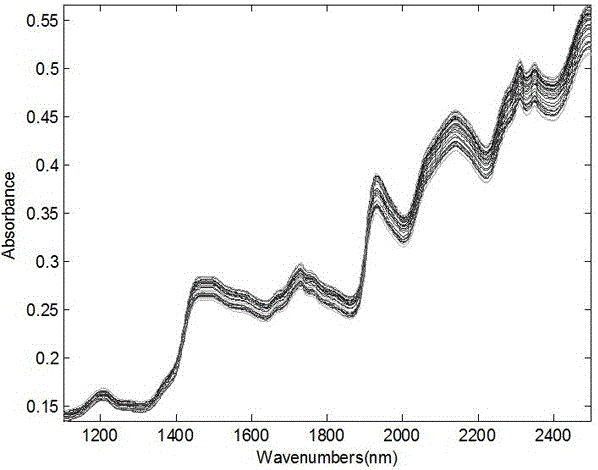
图1实施所述转基因大豆的近红外原始吸收光谱图。
图2实施所述非转基因大豆的近红外原始吸收光谱图。
图3经过多元散射校正预处理后的转基因大豆的近红外吸收光谱图。
图4经过多元散射校正预处理后的非转基因大豆的近红外吸收光谱图。
图5 利用留一法交叉验证取得最佳主成分因子数变化图。
具体实施方式
下面结合附图和具体实施例对本发明做进一步解释说明。
为了更好的理解本发明,下面结合实例对本发明进一步的详细说明。
本发明提供一种快速、无损的转基因大豆的鉴别方法,其包括以下:步骤S1:收集不同种类的转基因大豆和非转基因的大豆样品;步骤S2:将样本进行近红外光谱扫描,采集其近红外光谱,并剔除异常的样本数据;步骤S3:选取1100~2500 nm特征波段下的光谱信息,运用多元散射校正预处理方法进行对步骤S2中的光谱信息进行处理;并对处理后的光谱信息进行散射校正;步骤S4:采用留一法交叉验证,求PLS-DA最佳的因子数;具体步骤为:假设有N个样本,将每一个样本作为测试样本,其它N-1个样本作为训练样本,这样得到N个分类器,N个测试结果,用这N个结果的平均值来衡量模型的性能,从而找到求PLS-DA最佳的因子数;N为大于1的自然数;步骤S5:采用步骤S4的最佳的因子数,进行PLS-DA建模;步骤S6:对于未知的样品,利用扫描器获取其近红外光谱,再利用建立好的PLS-DA模型,预测其所属类别。
一个交叉验证将样本数据集分成两个互补的子集,一个子集用于训练(分类器或模型)称为训练集(training set);另一个子集用于验证(分类器或模型的)分析的有效性称为测试集(testing set)。利用测试集来测试训练得到的分类器或模型,以此作为分类器或模型的性能指标。得到高度预测精确度和低的预测误差,是研究的期望。为了减少交叉验证结果的可变性,对一个样本数据集进行多次不同的划分,得到不同的互补子集,进行多次交叉验证。取多次验证的平均值作为验证结果。
在给定的建模样本中,拿出大部分样本进行建模型,留小部分样本用刚建立的模型进行预报,并求这小部分样本的预报误差,记录它们的平方加和。这个过程一直进行,直到所有的样本都被预报了一次而且仅被预报一次。把每个样本的预报误差平方加和,称为PRESS(predicted Error Sum of Squares)。
假设分类器或模型有一个或多个未知的参数,并且设这个训练器(模型)与已有样本数据集(训练数据集)匹配。训练的过程是指优化模型的参数,以使得分类器或模型能够尽可能的与训练数据集匹配。我们在同一数据集总体中,取一个独立的测试数据集。
进一步的,所述步骤S3包括以下步骤:首先建立一个待测样品的理想光谱,所述理想光谱的变化与样品中成分的含量满足直接的线性关系,以该光谱为标准要求对所有其他样品的近红外光谱进行修正,其中包括基线平移和偏移校正。
具体实施例:
(1)收集转基因大豆与非转基因大豆样本各44个样本,在温度为25℃时,湿度为30 %左右条件下,利用Nicolet 6700傅里叶变换近红外光仪(赛默飞世尔科技公司,Thermo Fisher)采集其漫反射光谱图,扫描范围1100~2500 nm,如图1和图2。
(2)对于异常的样本进行剔除,并运用多元散射校正预处理方法进行对光谱信息进行处理。经过散射校正后得到的光谱数据可以有效地消除散射影响,增强了与成分含量相关的光谱吸收信息。该方法的使用首先要求建立一个待测样品的“理想光谱”,即光谱的变化与样品中成分的含量满足直接的线性关系,以该光谱为标准要求对所有其他样品的近红外光谱进行修正,其中包括基线平移和偏移校正等,如图3和图4。
(3)将共88个样本利用K-S算法分别对转基因大豆与非转基因划分校正集与预测集,校正集66个样本,预测集22个样本。
(4)采用留一法交叉验证求的PLS-DA最佳的因子数,如图5。
(5)采用最佳的因子数4,进行PLS-DA建模。对于未知的样品,扫描器近红外光谱,就可以利用建立好的PLS-DA模型,预测其归属,准确率如表格1,预测成功率在校正集与预测集均为100%。
表1
以上是本发明的较佳实施例,凡依本发明技术方案所作的改变,所产生的功能作用未超出本发明技术方案的范围时,均属于本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!