基于决策模板预测药物‑靶蛋白相互作用关系的方法和系统与流程
本发明涉及生物及信息技术领域,具体涉及一种基于决策模板预测药物-靶蛋白相互作用关系的方法和系统。
背景技术:
研制一种新药大致需要花费数十亿美元,耗时9-12年,而且还存在高失败率和高召回率的风险,据统计,自1950年以来,新授权的药物数量几乎为零(参考文献:Scannell J W,Blanckley A,Boldon H,et al.Diagnosing the decline in pharmaceutical R&D efficiency[J].Nature reviews Drug discovery,2012,11(3):191-200.)。然而随着测序技术和生物技术的发展,产生了大量药物及生物组学数据。研究已存在药物的新作用靶蛋白可用于发现已存在药物的新用途,同时也可发现该药物的偏靶。前者称为药物功能重定位,是当前药物研发的主要方向;后者可用于寻找药物副作用产生的原因。因此,识别新的药物-靶蛋白作用关系是药物设计的第一步,其在学术领域和药物制造工业领域都受到广泛关注。
生物实验方法识别新的药物-靶蛋白作用关系虽然可靠,但实验过程费时,费力,费钱,而且实验缺少重复性(参考文献:Whitebread,S.;Hamon,J.;Bojanic,D.;Urban,L.(2005),Keynote review:in vitro safety pharmacology profiling:an essential tool for successful drug development.Drug discovery today,10(21),1421-1433.Haggarty,S.J.;Koeller,K.M.;Wong,J.C.;Butcher,R.A.;Schreiber,S.L.(2003)Multidimensional chemical genetic analysis of diversity-oriented synthesis-derived deacetylase inhibitors using cell-based assays.Chemistry&biology,10(5),383-396.)。因此研究人员提出大量计算方法预测新的药物-靶蛋白作用关系,其不仅可以指导实验科学家更好地设计实验方案,也可以减少候选样本集的数量,以解决实验方法耗时、耗钱的问题(参考文献:X.-Y.Yan,S.-W.Zhang,S.-Y.Zhang,(2016)Prediction of drug –target interaction by label propagation with mutual interaction information derived from heterogeneous network,Molecular BioSystems.Z.Mousavian,A.Masoudi-Nejad,Drug-target interaction prediction via chemogenomic space:learning-based methods,Expert opinion on drug metabolism&toxicology,10(2014)1273-1287.)。
传统的基于配体方法和基于靶蛋白的方法需要靶蛋白的三维结构已知,且只能对有已知作用药物的靶蛋白进行预测。此外,现存的大多数方法只是基于药物的结构相似性和靶蛋白的序列相似性进行预测,其不能反映具有不同结构的药物可能会与同一个靶蛋白作用的情况以及具有不同序列相似性的靶蛋白可能会与同一个药物作用的情况,而且对提出的多种相似性度量值,其组合方式多数采用求平均,求最大值等方式进行,太过简单,不能充分利用所提出的多种相似性度量(参考文献:Y.Yamanishi,M.Kotera,Y.Moriya,R.Sawada,M.Kanehisa,S.Goto,(2014)DINIES:drug–target interaction network inference engine based on supervised analysis,Nucleic acids research,42W39-W45.J.-Y.Shi,S.-M.Yiu,Y.Li,H.C.Leung,F.Y.Chin,(2015)Predicting drug–target interaction for new drugs using enhanced similarity measures and super-target clustering,Methods,83 98-104.)。
药物的治疗效果是通过绑定到一些靶蛋白上,影响靶蛋白的化学、物理结构及活动,进而影响靶蛋白对应的基因功能,最终实现治疗目的的。而靶蛋白化学及物理结构功能的改变当且仅当其参与的代谢通路发生激活或抑制,因此共同参与的代谢通路是描述靶蛋白的重要特征之一。(参考文献:Li,Z.,Wang,R.S.,&Zhang,X.S.(2011).Two-stage flux balance analysis of metabolic networks for drug target identification.BMC systems biology,5(Suppl 1),S11)。GO基因本体论从生物过程,分子功能和细胞组件方面对基因及基因产物进行了描述,因此共同具有的GO术语是描述靶蛋白的重要特征之一。
技术实现要素:
为了克服上述现有技术的不足,本发明提出了一种基于决策模板预测药物-靶蛋白相互作用关系的方法和系统,通过提出两种新的靶蛋白相似性度量策略,即基于GO本体注释和基于pathway通路功能映射的相似性度量,结合已有的药物化合物分子结构相似性,药物ATC注释相似性,以及靶蛋白序列相似性和功能相似性,可构成多种相似性组合,同时基于相似药物较容易与相似靶蛋白作用的假定,分别采用KNN分类算法预测药物-靶蛋白作用关系;更重要的是,本发明提出基于决策模板融合的策略,将基于多种相似性度量的分类器预测结果进行决策级融合,同时结合靶蛋白组和药物组的概念,有效解决了已知的药物和靶蛋白作用关系比较稀疏(即正样本数目较少)的问题,本发明提出的算法模型提高了预测精度,可用于实现新药物的靶蛋白预测和新靶蛋白的药物预测。
为了达到上述目的,本发明所采用的技术方案:基于决策模板预测药物-靶蛋白相互作用关系的方法,包括如下步骤:
步骤1)收集药物-靶蛋白作用数据集,构建二部图描述药物和靶蛋白对的相互作用;
步骤2)收集药物和靶蛋白的多种不同特征描述数据,并采用有效的数学方法将其转换为特征向量形式,其中,药物描述数据包括:药物的化合物分子结构,药物的ATC注释;靶蛋白的描述数据包括:靶蛋白的序列信息,靶蛋白的FC功能注释,靶蛋白参与的代谢通路信息,靶蛋白的GO功能注释;
步骤3)计算药物-药物相似性和靶蛋白-靶蛋白相似性
根据步骤2)中得到的药物和靶蛋白的不同描述信息,分别构建相应的相似矩阵,其中,药物的相似性通过其共同拥有的化合物分子结构,ATC注释得到,分别为和靶蛋白对的相似性通过计算序列相似性,共同拥有的FC功能注释项数目,共同拥有的GO功能注释项及参与的代谢通路数目得到,分别为和
步骤4)基于K近邻(KNN)分类算法预测药物与靶蛋白相互作用
对步骤3)中得到的2种药物相似性和4种靶蛋白相似性度量方法进行两两组合,形成8个分类特征组合,在不同的相似性特征组合下采用K近邻(KNN)分类算法预测药物与靶蛋白相互作用关系的得分,即:
药物的相似性:
靶蛋白的相似性:
组合形式:
当用于对新药物的靶蛋白预测(di,tj)时,采用靶蛋白特异的分类器,分两个阶段进行,第一阶段预测该新药di与一组相似靶蛋白组(包含候选靶蛋白tj)作用的可能性,第二阶段直接预测该新药di与该候选靶蛋白tj之间作用的可能性,并对两阶段的预测结果进行组合,得到在本组特征组合下药物和靶蛋白对(di,tj)的预测得分,对8个不同的特征相似性组合,样本集中的每个药物-靶蛋白作用关系对(di,tj)均会得到8个预测结果;
或者,当用于对新靶蛋白的药物预测(tj,di)时,采用药物特异的分类器,分两个阶段进行,第一阶段预测该新靶蛋白与一组相似药物组(包含候选药物di)作用的可能性,第二阶段直接预测该新靶蛋白tj与该候选药物di之间作用的可能性,并对两阶段的预测结果进行组合,得到在本组特征组合下药物和靶蛋白对(tj,di)的预测得分,对8个不同的特征相似性组合,样本集中的每个药物-靶蛋白作用关系对(tj,di)(即训练样本)均会得到8个预测结果;
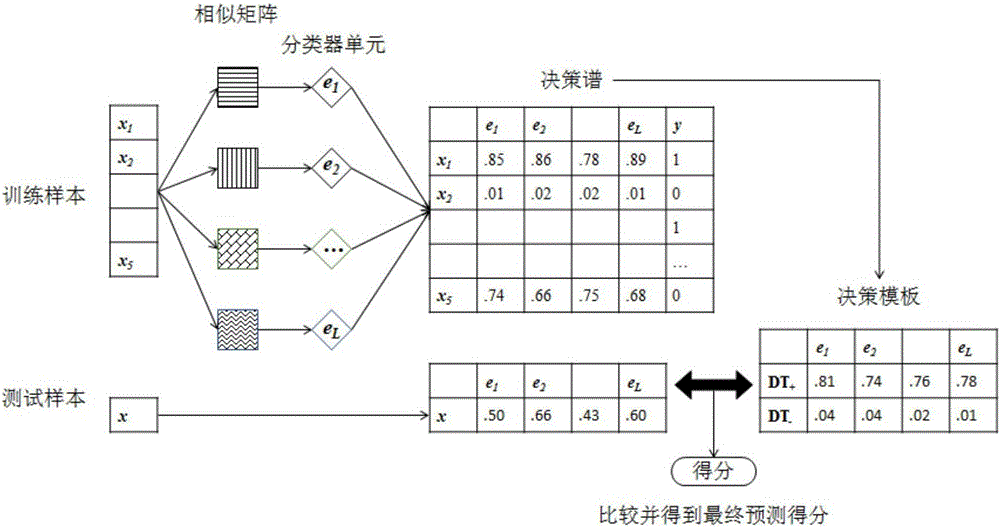
步骤5)对步骤4)中得到的8个预测结果,采用决策模板进行决策级融合,其中训练样本的8个预测结果用于构建决策模板,由此得到药物与靶蛋白作用关系的预测模型;
步骤6)对新药物和候选靶蛋白对逐个进行独立集测试,预测该新药可能存在的相互作用靶蛋白;同样地,对新靶蛋白和候选药物逐个进行独立集测试,预测该新靶蛋白可能存在的相互作用药物。
步骤4)中,新药物指在当前数据库中没有任何靶蛋白与该药物存在已知的作用关系,新靶蛋白指在当前数据库中没有任何药物与该靶蛋白存在已知的作用关系。
步骤4)中,包含候选靶蛋白tj的相似靶蛋白组是由靶蛋白相似性矩阵(或网络),经过凝聚聚类算法得到,以药物相似性作为分类特征,采用K近邻(KNN)分类算法预测新药与候选靶蛋白tj所在的靶蛋白组以及候选靶蛋白tj之间的作用关系得分;
步骤4)中,构建包含候选药物di的相似药物组时,是由药物相似性网络,经过凝聚聚类算法得到的,以靶蛋白相似性为分类特征,采用K近邻(KNN)分类算法预测新靶蛋白与候选药物di所在组以及候选药物di之间的作用关系得分。
所述步骤5)中,根据训练样本中每个药物-靶蛋白作用关系对(即样本)在8种相似性组合下得到的8个预测结果,组成决策谱DP(xi,yi)={dp1(xi),dp2(xi),…,dpL(xi)},i=1,...,N
其中,N为训练样本数目,L为分类器数目,L=8;
根据样本的标签值将所有训练样本的DP决策谱分成两组,并计算决策模板
其中,N+和N-为训练样本中的正、负样本数目;
所述步骤6)中独立集测试方法如下,对测试集中的样本(x,y),其决策谱为DP(x,y)={p1(x),p2(x),…,pL(x)},判断该样本为正样本的得分为其中,μΔ(x)(Δ∈{+,-})是DP(x,y)和DTΔ之间的相似性得分,
还包括如下步骤,步骤7)通过数据库和文献检索对预测结果进行验证,即通过最新的药物靶蛋白作用关系数据库和文献检索验证预测得到的新作用对,为进一步的生物实验提供有力的依据。
基于决策模板预测药物-靶蛋白相互作用关系的系统,包括:
构建数据集模块,用于收集及整理已知的药物和靶蛋白相关作用关系数据集
获取数据模块,用于获取描述药物和靶蛋白特征的数据;
数学建模模块,用于利用药物和靶蛋白的多种特征描述信息,分别构建多个药物相似性和靶蛋白相似性矩阵;由药物相似性矩阵构建相似药物组,由靶蛋白相似性矩阵构建相似靶蛋白组,对新药物的靶蛋白预测或新靶蛋白的药物预测均采用K近邻(KNN)分类算法,分两个阶段进行预测,并对两个阶段的预测结果进行组合,得到该药物和靶蛋白对在当前相似性特征组合下分类器的预测结果;多个分类器的预测结果采用决策模型进行决策级融合,建立药物-靶蛋白相互作用预测的模型;
模型测试模块,用于对新药物和靶蛋白之间相互作用进行独立集测试,预测该新药可能存在的相互作用靶蛋白,或用于对新靶蛋白和药物之间相互作用进行独立集测试,预测该新靶蛋白可能存在的相互作用药物,对模型新预测出的作用关系对,通过最新数据库和文献资料检索进行验证。
预测新药物的相互作用靶蛋白,如预测(di,tj)的相互作用得分,可分两个阶段进行,第一阶段预测该新药di与一组相似靶蛋白组(包含候选靶蛋白tj)作用的可能性,由此得到新的药物-靶蛋白作用关系矩阵,并以此新矩阵为标签,药物相似性为分类特征,采用K近邻(KNN)分类算法预测该新药与候选靶蛋白tj的作用关系得分,实质为预测该新药与靶蛋白组的作用关系得分;
第二阶段直接预测该新药di与该候选靶蛋白tj之间作用的可能性,即以原始药物-靶蛋白作用关系矩阵为标签,药物di与所有药物的相似性为分类特征,采用K近邻分类算法预测di与候选靶蛋白tj之间的作用关系。
靶蛋白组是由靶蛋白相似性矩阵(或网络),经过凝聚聚类算法得到。
新靶蛋白的药物预测,如预测(tj,di)的相互作用得分,分两个阶段进行,第一阶段预测该新靶蛋白与一组相似药物组(包含候选药物di)作用的可能性,第二阶段直接预测该新靶蛋白tj与该候选药物di之间作用的可能性。
构建包含候选药物di的相似药物组时,是由药物相似性网络,经过凝聚聚类算法得到的。
与现有技术相比,本发明至少具有以下有益效果,本发明提出了一种基于决策模板预测药物-靶蛋白相互作用关系的系统,通过提出两种新的靶蛋白相似性度量策略,即基于GO本体注释和基于pathway通路功能映射的相似性度量,结合已有的药物化合物分子结构相似性,药物ATC注释相似性,以及靶蛋白序列相似性和功能相似性,构成多种相似性组合,同时基于相似药物较容易与相似靶蛋白作用的假定,分别采用KNN分类算法预测药物-靶蛋白作用关系;更重要的是,本发明提出基于决策模板融合的策略,将基于多种相似性度量的分类器预测结果进行决策级融合,同时结合靶蛋白组和药物组的概念,有效解决了已知的药物和靶蛋白作用关系比较稀疏(即正样本数目较少)的问题;本发明提出了一种全新的基于决策模板融合方法预测药物-靶蛋白作用关系的计算方法,不同于以往的特征融合方法,本发明提出根据不同特征组合分别构建分类器,之后对多分类器结果采用决策模板进行决策级融合的模型策略,考虑药物治疗作用是通过激活或抑制代谢通路上蛋白的代谢反应实现的,而GO基因本体论术语对基因及基因产物从生物过程,分子功能和细胞组件方面进行了描述,本发明提出了基于pathway代谢通路和GO功能注释的靶蛋白相似性的新的计算方法,本发明提出的算法模型提高了预测精度,可用于实现新药物的靶蛋白预测和新靶蛋白的药物预测。
附图说明
图1是本发明的系统框图;
图2是基于决策模板融合的预测模型框图;
图3是靶蛋白组构建解释图,其中,(a)为已知的药物和靶蛋白之间的作用关系,其中椭圆形表示药物,矩形表示靶蛋白,虚框表示两个靶蛋白组(tg1,tg2);图(b)表示由原始的药物和靶蛋白作用关系,即由图(a),得到的药物-靶蛋白二分图矩阵表示,行为药物,列为靶蛋白;图(c)表示对靶蛋白组的药物进行并操作,得到的新的药物与靶蛋白组之间的作用关系矩阵。
具体实施方式
为使本发明的上述目的,特征和优点能够更加明显易懂,下面通过具体实施例和附图,对本发明做进一步详细说明。
图1所示为本发明的融合多种相似性度量预测药物与靶蛋白相互作用系统框图。该系统包括四个模块,数据集收集模块,描述数据获取模块,数学模型模块和模型测试模块。
1)药物和靶蛋白相互作用数据集构建模块
通过收集人类蛋白质和药物相互作用数据库,构建药物-靶蛋白相互作用数据集。
2)描述数据获取模块(即特征提取)
获取药物和靶蛋白的相关描述信息,采用有效数学方法将其转换为特征向量形式。具体包括:
I、描述药物的化合物分子结构特征,药物的Anatomical Therapeutic Chemical(ATC)注释信息;
II、描述靶蛋白的序列信息特征,靶蛋白的功能分类FC特征,新提出了通过靶蛋白参与的代谢通路Pathway信息以及靶蛋白对应基因的基因本体论注释GO信息描述靶蛋白的方法。
3)数学建模模块
从机器学习方法论的角度入手探求药物-靶蛋白相互作用预测模型和算法,提出了基于决策模板融合的多相似性整合模型。具体地,首先利用药物-靶蛋白相互作用数据集建立二部图,用以表征药物和靶蛋白之间的作用关系;其次利用药物和靶蛋白的多种描述数据分别计算并构建药物相似性矩阵和靶蛋白相似性矩阵,其中,药物的相似性通过其化合物分子结构,ATC注释描述;靶蛋白的相似性通过其序列信息,功能分类FC信息,并新提出了通过靶蛋白参与的代谢通路Pathway和靶蛋白对应基因的基因本体论注释GO信息。将药物的两种相似性信息和靶蛋白的四种相似性信息两两组合,形成8种相似性特征组合方式,对每组组合特征,分别采用机器学习算法(如使用K-近邻分类算法(KNN))预测药物-靶蛋白相互作用。最后对8个预测结果采用决策模板进行决策级融合,得到最终的预测得分。
具体而言,分为四个步骤:
1、构建药物-靶蛋白相互作用关系的二部图网络;
2、计算并构建药物-药物相似性矩阵,靶蛋白-靶蛋白相似性矩阵;
3、基于机器学习算法例如K近邻分类算法,分别采用各组相似性特征组合,分两阶段预测新药物与靶蛋白(或新靶蛋白与药物)的相互作用得分;
4、采用决策模板,对所有药物-靶蛋白对的8个预测结果进行决策级融合,建立药物-靶蛋白相互作用关系的预测模型。
4)模型测试模块
对新药物和所有候选靶蛋白对进行独立集测试,预测该新药物可能存在的相互作用靶蛋白(或对新靶蛋白和所有候选药物对进行独立集测试,预测该新靶蛋白可能存在的相互作用药物);通过数据库和文献检索验证新预测的结果。
下面结合图1,针对数据集构建,数据特征获取,数学建模和模型测试四部分进行详细阐述。
(1)数据集构建
通过收集人类蛋白质和药物相互作用数据库,构建药物-靶蛋白相互作用数据集。具体而言,首先从KEGG BRITE和DrugBank数据库收集人类蛋白质和药物相互作用对,或者从已有文献提供的数据集中获取药物-靶蛋白相互作用对,本发明的测试数据集,由Yamanishi等人收集整理,并已广泛地用于药物-靶蛋白作用关系预测方法的研究,该数据集涉及四类蛋白质:酶,离子通道,G蛋白偶联受体和核受体(分别简写为:EN,IC,GPCR和NR),下载网址:http://web.kuicr.kyoto-u.ac.jp/supp/yoshi/drugtarget/。
(2)药物和靶蛋白描述数据获取
描述药物的数据包括:
A.药物化合物分子数据:DrugBank
B.药物ATC注释:KEGG Brite
描述靶蛋白的数据包括:
A.靶蛋白的序列信息:UniProt
B.靶蛋白的功能分类FC:HGNC
C.靶蛋白的代谢通路Pathway信息:MSigDB3.0
D.靶蛋白对应基因的基因本体论注释GO信息:UniProt
(3)药物-靶蛋白相互作用预测的数学模型构建
如图2所示,建模过程包括四部分:由收集的药物-靶蛋白作用关系数据集中提取正负样本集;根据药物和靶蛋白的不同描述信息计算药物-药物相似性矩阵,靶蛋白-靶蛋白相似性矩阵;利用各组药物相似性矩阵和靶蛋白相似性矩阵组合作为特征,分别采用KNN进行分类预测;采用决策模板对多组预测结果进行决策级融合,得到该样本的预测得分。
当前的药物-靶蛋白作用关系数据集中,已知有作用关系的药物-靶蛋白对为正样本,其它未知的药物-靶蛋白对为负样本。
3.1)采用多种度量方式分别计算药物的相似性
药物结构相似性:
表示药物di的子结构,∪和∩分别表示集合的“并”和“交”运算,表示集合中元素的数目。
基于ATC的药物相似性计算:其中f表示药物di和dj共同拥有的ATC编码中子码的数目,N为药物ATC编码中子码的数目,N=5
3.2)采用多种度量方式分别计算靶蛋白的相似性
靶蛋白的序列相似性采用公式其中align(ti,tj)表示归一化的靶蛋白ti和tj的Smith-Waterman得分。
靶蛋白的FC功能相似性:靶蛋白的功能注释信息可从HGNC获取,也可以从文献(J.-Y.Shi,S.-M.Yiu,Y.Li,H.C.Leung,F.Y.Chin,(2015)Predicting drug–target interaction for new drugs using enhanced similarity measures and super-target clustering,Methods,83 98-104)中下载,相似性计算与药物基于ATC相似性的计算相似,采用公式其中,N表示靶蛋白FC功能注释中子域的数目,f表示FC功能注释中靶蛋白ti和tj共同拥有的子域的数目。
基于GO功能注释的靶蛋白相似性
其中符号Gi表示靶蛋白ti的GO注释项目,∪和∩分别表示集合的“并”和“交”运算,|G|表示集合G中的元素数目。
基于pathway通路注释的靶蛋白相似性
其中符号Pi表示靶蛋白ti参与的通路项目,∪和∩分别表示集合的“并”和“交”运算,|P|表示集合P中的元素数目。
3.3)对2种药物相似性和4种靶蛋白相似性度量方法,两两组合,形成8个分类特征组合,分别采用KNN分类器进行药物-靶蛋白作用关系对的分类预测
药物的两种相似性:
靶蛋白的四种相似性:
药物和靶蛋白相似性的不同组合形式:
具体来说,本发明提出的基于决策模板预测药物-靶蛋白相互作用关系的方法可适用于预测新药物的靶蛋白和新蛋白的作用药物,这里“新药物”指对该药物,当前没有任何靶蛋白与该药物存在作用关系;“新靶蛋白”指对该靶蛋白,当前没有任何药物与该靶蛋白存在作用关系。
当对新药物的靶蛋白预测进行预测时,如预测(di,tj)的作用关系,采用靶蛋白特异的分类器,分两个阶段进行:第一阶段预测该新药di与一组相似靶蛋白组(包含候选靶蛋白tj)作用的可能性。这里,包含候选靶蛋白tj的相似靶蛋白组是由靶蛋白相似性矩阵(或网络),如Stseq,经过凝聚聚类算法得到,其基本思想是,如果一个药物与一个靶蛋白组中其中一个有相互作用,则认为该药物与该靶蛋白组中所有靶蛋白都有相互作用;因此是其作用谱集合的并集,由此得到新的药物-靶蛋白作用关系矩阵,参见图3,以此新矩阵为标签,以药物相似性为分类特征,采用K近邻(即,KNN)分类算法预测新药与候选靶蛋白tj的作用关系,实质为预测该新药与靶蛋白组的作用关系;
第二阶段直接预测该新药di与该候选靶蛋白tj之间作用的可能性,以原始药物-靶蛋白作用关系矩阵为标签,参见图3,药物di与所有药物的相似性为分类特征,采用K近邻(即,KNN)分类算法预测di与候选靶蛋白tj之间的作用关系;
将两个阶段的预测结果相乘即可得到在一种相似性特征组合下,该样本的预测得分结果,因此,8种不同的组合方式,会得到8个预测结果。
与之相似,当对新靶蛋白tj的药物di进行预测时(tj,di),采用药物特异的分类器,同样分两个阶段进行:第一阶段预测该新靶蛋白与一组相似药物组(包含候选药物di)作用的可能性,第二阶段直接预测该新靶蛋白tj与该候选药物di之间作用的可能性;构建包含候选药物di的相似药物组时,是由药物相似性网络,经过凝聚聚类算法得到的,分别以新构建的药物-靶蛋白作用关系矩阵和原始的作用关系矩阵为标签,以靶蛋白相似性为分类特征,采用K近邻(即,KNN)分类算法,预测新靶蛋白tj与候选药物di所在组以及候选药物di之间的作用关系。
对每个样本,经过上述步骤后得到8个预测结果。
采用KNN分类算法,预测新药物di与候选靶蛋白tj之间的作用关系得分。
首先根据药物相似性网络,寻找样本x=(di,tj)中,对应于新药di的K个最近邻居,记为N(x,K),n(x,K)表示在K个邻居中,与所有候选靶蛋白有作用关系的邻居的数目,药物di与靶蛋白tj关系对样本的预测得分见公式(1),本发明中K=3,
其中,Pr[y=b],b=0/1,是正/负样本的先验概率,其计算公式为Pr[y=1]≈(1+k)/(m+2),这里m是当前已知的药物数目,k是药物中已知与给定靶点有作用关系的药物数目。
Pr[n(x,K)=c|y=b]是在已知当前正/负标签的前提下,样本x有c个正样本邻居的概率,其计算公式为这里Ind[S]是一个指示函数,表示状态S正确与否。
(4)对步骤(3)得到的8个预测结果,采用决策模板进行决策级融合,其中训练样本的8个预测结果用于构建决策模板,由此得到药物与靶蛋白作用关系的预测模型,具体如下:
将所有训练样本的8个预测结果,组成决策谱DP(xi,yi)={dp1(xi),dp2(xi),…,dpL(xi)},i=1,...,N,其中,N为样本数目,L为分类器数目,L=8;
根据样本的标签值将所有训练样本的DP决策谱分成两组,并计算决策模板其中,N+和N-为训练样本中的正、负样本数目。
对新药物和候选靶蛋白对逐个进行独立集测试,预测该新药可能存在的相互作用靶蛋白;同样地,对新靶蛋白和候选药物逐个进行独立集测试,预测该新靶蛋白可能存在的相互作用药物,具体如下:对测试集中的样本(x,y),其决策谱为DP(x,y)={p1(x),p2(x),…,pL(x)},判断该样本为正样本的得分为
其中,μΔ(x)(Δ∈{+,-})是DP(x,y)和DTΔ之间的相似性得分:模型测试过程:
ROC(Receriver Operating Characteristic)曲线(参考文献Gribskov,M.and Robinson,N.L.(1996).Use of receiveroperating characteristic(roc)analysis to evaluate sequence matching.Computers and Chemistry,20,25–33.)以及ROC曲线下面积(AUC),PR(Precision-Recall)曲线及PR曲线下面积(AUPR)常被用于评价预测模型的性能,对于正负样本不均衡的情况,即正样本数量明显少于负样本的情况,PR曲线及AUPR更能表征算法性能(J.Davis,M.Goadrich,The relationship between Precision-Recall and ROC curves,Proceedings of the 23rd international conference on Machine learning,ACM,2006,pp.233-240.)。
测试1
为了测试引入基于GO注释的相似性度量与基于通路Pathway的相似性度量是否会提高药物-靶点作用关系预测的性能,分别采用两组不同的策略产生靶点的组合相似性,采用KNN作为分类算法预测DTI。其中药物相似性采用和的平均,即
第一组靶点相似性采用
第二组靶点相似性采用
采用5折叠交叉验证方法进行新药物(记为,S2)和新靶点(记为,S3)的作用关系预测,结果见表1;
如表1所示,引入新的相似性测量方法和提高了算法性能。其中对新靶蛋白的药物预测(即S3)性能提升较大(对EN,GPCR和NR数据集的AUPR提高了2.9~3.7%),对新药物的靶蛋白预测(即S2)的性能提升一般,其原因是,靶蛋白相似性在S2预测中主要用于构造靶蛋白组,而对于S3预测,靶蛋白相似性是KNN分类器的主要特征。
测试2
为了测试所提出的采用决策模板对多个相似性组合的预测结果进行决策级融合,是否会提高药物-靶蛋白作用关系预测的性能。比较了对药物相似性和靶蛋白相似性的8种相似性组合方式,采用DT决策模板融合策略的预测结果(DT-all)与直接对药物相似性和靶蛋白相似性进行特征级组合,采用KNN的预测结果(Average)进行比较,如表2所示,DT决策模板融合策略大大提高了预测性能。
表1.组合不同的相似性的KNN算法预测性能比较
表2 特征级融合策略与DT决策级融合策略结果比较
本发明提出多种新的靶蛋白相似性度量策略,并采用决策模板对多个分类器的预测结果进行决策级融合,充分地利用了药物和靶蛋白的多种相似性特征,同时,在分类器算法实施过程中引入了靶蛋白组和药物组的概念,进一步解决了已知正样本数量不足的问题。提出的算法策略首次引入决策模板融合8个分类器的预测结果,可用于预测新药物的靶蛋白和新靶蛋白的药物,并进一步提高了预测精度。
- 还没有人留言评论。精彩留言会获得点赞!