一种特异性结合肿瘤内皮标记物8的单克隆抗体及其应用的制作方法
本发明属于基因工程抗体技术领域,具体涉及一种特异性结合肿瘤内皮标记物8的单克隆抗体及其应用。
背景技术:
肿瘤内皮标记物(TEM8)是肿瘤内皮标记物(tumor endothelial markers,TEMs)中的一种,最初分别在肿瘤血管形成和体外正常血管形成过程中的内皮细胞中被发现表达上调。TEM8是一种由564个氨基酸残基组成的具有完整跨膜螺旋的I型跨膜蛋白,胞外有大约300个氨基酸,包括一个约200个氨基酸组成的vWA结构域和近膜的一个功能未知的Ig样结构域。其中v WA结构域主要负责蛋白质相互作用,TEM8的vWA结构域具有典型的Ⅰ结构域特征:一个NAD结合的Rossmann折叠,这一结构是由个α﹣螺旋围绕着中央的一个六层的β﹣折叠组成,在α﹣螺旋和β﹣折叠之间创造了一个疏水核心。目前对跨膜结构域的了解相当有限,只知道其由23个氨基酸残基组成,比一般的跨膜蛋白的跨膜区多两个残基,此外TEM8还有一个非常特殊的胞质尾。
在研究肿瘤血管形成相关基因的表达时,发现TEM8在鼠和人的各种肿瘤血管细胞表面或者肿瘤细胞表面的表达量均有上调。起初发现TEM8表达于结肠癌内皮细胞中,而在周围正常组织中表达微乎其微,随后在结肠癌组织的血管中也检测到TEM8蛋白,后来TEM8还被发现在许多不同类型的人类肿瘤(包括膀胱癌、结肠癌、食管癌和肺癌等)的血管内皮细胞中表达增加,而在正常组织的血管中没有表达或表达很少。这些研究都表明TEM8可以成为治疗肿瘤的新靶点。
肿瘤是一类严重危害人类生命健康的疾病,除了早期的化疗外,其余主要依靠抗体类的药物,目前大多数药物针对的靶点是血管内皮生长因子和血管内皮生长因子受体,除了价格昂贵,疗效也很难达到预期。针对靶点TEM8的抗体将会是一个新的治疗途径。
技术实现要素:
本发明的目的是提供一种特异性结合TEM8的单克隆抗体,以及含有该抗体的药物组合物或试剂、试剂盒或芯片,在检测肿瘤发生,发展及转移中的用途。
本发明的特异性结合TEM8的单克隆抗体对TEM8具有特异性结合。
经检测,本发明的特异性结合TEM8的单克隆抗体与TEM8重组蛋白结的结合活性的EC50值为32.42ng/ml,与TEM8过表达细胞结合,EC50值为133ng/ml。
本发明的特异性结合TEM8的单克隆抗体,其氨基酸序列区由重链和轻链中的高度可变区CDR1、CDR2和CDR3,以及连接序列和框架区组成,其中重链可变区含有SEQ ID NO.1、SEQ ID N0.2和SEQ ID NO.3所示的氨基酸序列,和/或其轻链可变区含有SEQ ID NO.4、SEQ ID NO.5和SEQ ID NO.6所示的氨基酸序列,重链和轻链中的高度可变区CDR1、CDR2和CDR3是与TEM8结合的关键部位,框架区可在结合所需的三维结构不受影响的条件下被其他序列置换。
在某些优选例中,一个单克隆抗体包含如下内容的可变区:一个包含了如SEQ ID NO.12的重链可变区,其含有CDR1(SEQ ID NO.1)、CDR2(SEQ ID NO.2)和CDR3(SEQ ID NO.3),和一个包含了如SEQ ID NO.13的轻链可变区,其含有CDR1(SEQ ID NO.4)、CDR2(SEQ ID NO.5),CDR3(SEQ ID NO.6)。
可变区CDR的变体与上述CDR区除了有至多6个氨基酸取代的不同外基本上是一致的(例如,1、2、3、4或5个氨基酸取代),在该单克隆抗体的CDR区具有与TEM8结合活性。
在其它实施方案中,抗体可包含:a)一个重链可变区,其氨基酸序列与SEQ ID NO.12相比有至多6个氨基酸序列取代的不同,例如1、2、3、4、5、或6个取代;和b)一个轻链可变区,其氨基酸序列与SEQ ID NO.8相比有至多6个氨基酸序列取代的不同,例如1、2、3、4、5、或6个氨基酸取代。目标抗体可能包含这些取代中的任何一个或其组合。
拥有这些取代位置中任一个的抗体和拥有所有取代位置的抗体同样具有结合TEM8的活性。氨基酸取代可同时存在于框架区和CDR区,或单独出现在框架区或CDR区。因此,在某些优选例中,重链可变区的框架区的氨基酸序列与SEQ ID NO.13相比可能有最多6个氨基酸序列取代的不同,例如1、2、3、4、5、或6个取代,和轻链可变区的框架区的氨基酸序列与SEQ ID NO.8相比可能有最多6个氨基酸序列取代的不同,例如1、2、3、4、5、或6个取代。
在一些抗体中,氨基酸取代可能分布在多个CDR区。因此,重链可变区的多个CDR区的氨基酸序列与SEQ ID NO.12相比可能有至多6个氨基酸序列取代的不同,例如1、2、3、4、5、或6个取代,和轻链可变区的多个CDR区的氨基酸序列与SEQ ID NO.13相比可能有至多6个氨基酸序列取代的不同,例如1、2、3、4、5、或6个取代。
在特殊的优选例中,抗体可能包含a)一个重链可变区,其氨基酸序列与SEQ ID NO.12一致和b)一个轻链可变区,其氨基酸序列与SEQ ID NO.13一致。
在特殊的优选例中,抗体可能包含a)一个重链可变区,其氨基酸序列与SEQ ID NO.12至少有95%的一致性和b)一个轻链可变区,其氨基酸序列与SEQ ID NO.13至少有95%的一致性。因此,目标抗体可能包含a)一个重链可变区,其氨基酸序列与SEQ ID NO.12至少有约95%、96%、97%、98%、99%或100%的一致性和b)一个轻链可变区,其氨基酸序列与SEQ ID NO.13至少有约95%、96%、97%、98%、99%或100%的一致性。
在特殊的优选例中,抗体可能包含a)一个重链,其氨基酸序列与SEQ ID NO.16一致和b)一个轻链,其氨基酸序列与SEQ ID NO.17一致。
在特殊的优选例中,抗体可能包含a)一个重链,其氨基酸序列与SEQ ID NO.9至少有95%的一致性和b)一个轻链,其氨基酸序列与SEQ ID NO.10至少有95%的一致性。因此,目标抗体可能包含a)一个重链,其氨基酸序列与SEQ ID NO.16至少有约95%、96%、97%、98%、99%或100%的一致性和b)一个轻链,其氨基酸序列与SEQ ID NO.17至少有约95%、96%、97%、98%、99%或100%的一致性。除了上面所描述的氨基酸取代,目标抗体可能在重链或轻链的两端有附加的氨基酸。例如,目标抗体在重链和/或轻链的C或N末端分别可能包含至少1、2、3、4、5或6或更多的附加氨基酸。在某些实施例中,目标抗体可能比这里所描述的示范性氨基酸短,其主要区别为在重链和轻链的两端分别比示范性氨基酸少1、2、3、4、5或6个氨基酸。
本发明所述特异性结合TEM8的单克隆抗体包括具有所需特异性的结合结构域的任意特异性结合因子,是单价的或是单链抗体、双链抗体、嵌合抗体以及上述抗体的衍生物、功能等同物和同源物,也包括抗体片段和含有抗原结合结构域的任何多肽;如免疫球蛋白亚型:IgG,IgE,IgM,IgD和IgA及其亚型亚类,如包含抗原结合结构域的片段:Fab、scFv、Fv、dAb、Fd。
本发明的特异性结合TEM8的单克隆抗体的制备方法是杂交瘤方法,根据本发明公开的氨基酸序列人工合成或用PCR法扩增,或者采用重组DNA方法,将本发明公开的氨基酸序列连入合适的表达载体中。
本发明的特异性结合TEM8的单克隆抗体的纯化采用本领域已知的纯化免疫球蛋白分子的任何方法对其进行纯化,如:色谱法,包括离子交换色谱、亲和色谱、特别是通过蛋白A对特异性抗原的亲和色谱和其它柱色谱,离心,利用溶解度差异,或通过任何其它纯化蛋白质的标准技术。
本发明的特异性结合TEM8的单克隆抗体可通过多种方式修饰,如:用DNA重组技术产生保留原来抗体特异性的其它抗体或嵌合分子,包括将编码抗体的免疫球蛋白可变区或互补决定区(CDRs)的DNA引入不同免疫球蛋白的恒定区或恒定区加框架区(参见,EP.A-184187,GB 2188638A或EP.A-239400),或者对杂交瘤细胞或产生抗体的其它细胞进行遗传突变或其它改变,改变或者不改变所产生抗体的结合特异性。
本发明还提供所述的特异性结合TEM8的单克隆抗体在制备备用于治疗TEM8相关性疾病药物中的用途。所述的TEM8相关性疾病包括膀胱癌、结肠癌、食管癌和胰腺癌等TEM8过表达肿瘤。
本发明还提供含有特异性结合TEM8的单克隆抗体的药物组合物或试剂、试剂盒或芯片。
本发明的优点及有益效果是:
本发明的特异性结合TEM8的单克隆抗体,与TEM8重组蛋白和TEM8过表达细胞中的TEM8抗原都具有极强的特异性和敏感性,经实验验证,本发明的特异性结合TEM8的单克隆抗体可用于与TEM8相关性疾病药物中,如膀胱癌、结肠癌、食管癌和胰腺癌等,以及包含TEM8的单克隆抗体的药物组合物或试剂、试剂盒或芯片,采用含有该抗体的试剂盒进行TEM8相关诊断与检测,具有很高的应用价值。
此外,本发明的特异性结合TEM8的单克隆抗体还可以用于与TEM8相关的科学研究,如发育生物学、细胞生物学、代谢、结构生物学、功能基因组学等多个领域的科学研究、或肿瘤等医学和药学的应用研究,用于临床中检测肿瘤的发生、发展、转移等实验中。
附图说明
图1是实施例1中重组表达的抗人TEM8的嵌合抗体SDS-PAGE的还原电泳图;
图2是实施例3中单克隆抗体的噬菌体与TEM8过表达细胞的结合活性即吸光值的检测结果;
图3是实施例4的单克隆抗体NMT802和NMT803与TEM8蛋白的结合活性测试结果;
图4是实施例4的单克隆抗体NMT802和NMT803与TEM8过表达细胞的结合活性测试结果;
图5是实施例4的流式细胞仪检测NMT802抗体与细胞膜TEM8结合活性测试结果。
具体实施方式
实施例1
本实施例通过杂交瘤细胞技术制备鼠源特异性结合TEM8单克隆抗体,有关实验方案参见文献(Ed Harlow,David Lane.Antibody:A laboratory manual.1988),具体按照以下步骤进行:
首先通过基因合成技术合成TEM8单克隆抗体序列的TEM8全长基因(SEQ ID NO.20),将TEM8全长基因通过HindIII和XbaI两个酶切位点的克隆入PCDNA3.1(Invitrogen),转染该质粒进入CHO-K1(ATCC No.CCL-60)细胞株,经过G418(GIBCO)加压筛选,获得TEM8稳定过表达细胞株;
用1×107个TEM8稳定表达细胞,对BALB/c小鼠进行腹腔注射,首次免疫后,分别在第14d和第35d,用1×107个细胞腹腔注射对小鼠进行加强免疫,第56d用1×106个对BALB/c小鼠静脉注射进行最终免疫,4天后取脾脏进行融合;
将鼠脾细胞与SP2/0细胞(ATCC No.CRL-1581)按数量比5:1比例融合,在96孔板(Corning)中以HAT(GBICO)培养基培养,然后进行杂交瘤细胞筛选,所得细胞克隆进行鉴定筛选;
鉴定筛选过程分为两个步骤:
①将重组TEM8蛋白(来自北京义翘神州生物技术有限公司)固定化于96孔酶联免疫吸附板(Costar)上,加入克隆表达上清孵育1h后,用PBST洗涤3次,使用羊抗鼠IgG抗体-HRP(Jackson Immuno)鉴定具有TEM8结合活性细胞克隆上清,从而获得可与TEM8直接结合的阳性克隆;
②随后将步骤①中的阳性克隆转移入24孔板(Corning)培养,以获得更多的表达产物,将TEM8稳定过表达细胞株接种于96孔板(Corning)上过夜培养,加入阳性克隆表达上清共同孵育1h,再用PBS洗涤3次,使用EU标记的羊抗鼠IgG(Perkin Elmer)进行检测,从而鉴别出能够结合细胞表面TEM8的阳性克隆;
将所筛选的阳性克隆的杂交瘤细胞裂解,提取mRNA后逆转录得cDNA,以此cDNA为模板,采用PCR方法分别扩增出鼠IgG抗体的轻链和重链可变区核酸序列,对重链可变区、轻链可变区进行分析,其编码的重链可变区含有SEQ ID NO.1的亲本序列(TSGMGVG)、SEQ ID NO.2的亲本序列(WWDDVKRYYPALKS)和SEQ ID NO.3的亲本序列(IGYDWFPY)、轻链可变区含有SEQ ID NO.4的亲本序列(KASQNVGIAVA)、SEQ ID NO.5的亲本序列(SASNRYT)和SEQ ID NO.6的亲本序列(QQYRSYPLT);
然后,通过序列末端互补的方法,拼接重链可变区或轻链可变区与其对应的人源抗体的恒定区序列,将全长的嵌合抗体的重链和轻链片段,均包含信号肽,分别克隆入PCDNA3.1(Invitrogen)质粒,共转染轻、重链质粒至293C18细胞株(ATCC No.CRL-10852),培养7天后,用Protein A(GE)纯化上清,最终获得重组表达的抗人TEM8的嵌合抗体,其氨基酸序列如SEQ ID NO.7所示的重链和SEQ ID NO.8所示的轻链,SDS-PAGE还原蛋白电泳分析抗体如图1所示,其中:重链分子量约55KD,轻链分子量约30KD。
实施例2
本实施例是将实施例1中制备的鼠源特异性结合TEM8单克隆抗体人源化,具体按照以下步骤进行:
分析鼠源抗体序列,与IMGT的人胚系(germ line)基因比对,最终确定IGKV1-39*02,IGKV1-13*02和IGKV1-8*01分别为轻链FR1,FR2和FR3的人源化框架序列,确定IGHV2-70*06,IGHV2-5*02和IGHV2-26*01为重链FR1,FR2和FR3的人源化框架序列,通过CDR-grafting,将重链和轻链的CDR并置到构架序列,构建人源化抗体,称为NMT801,其重链可变区氨基酸序列如SEQ ID NO.9所示,轻链可变区如SEQ ID NO.10所示。
实施例3
本实施例是对实施例2中的NMT801抗体进行亲和力筛选,基因合成NMT801抗体的scfv片段,如SEQ ID NO.11所示,并将scfv连接到PCOMB3噬菌体质粒中,分析鼠源抗体CDR的DNA序列,确定可变区CDR中的突变热点,设计引物序列,将突变热点位点所在的引物位置设计为NNS,使之编码任意的氨基酸,以NMT801抗体scfv为模板,PCR扩增scfv抗体库,将scfv的抗体库,通过sfiI酶切位点,构建到PCOMB3噬菌体质粒中,构建二级抗体库,然后通过噬菌体展示进行高亲和力抗体筛选,具体步骤如下:
a.通过电转化,将含scfv的抗体库的噬菌体质粒转化到TG1中,经过37度,220rpm,1h的恢复后,将helper phage加入到剩余的菌液中,另加入Ampicillin,37度,220rpm,1h,2500rpm x5min离心去上清,用2x YT-AK吹悬菌泥,37度,220rpm过夜培养;
b.包被抗原:用包被缓冲液稀释TEM8蛋白,混匀加入到免疫管中,4度包被过夜;
c.重组噬菌体收集:上述过夜培养菌液,2500rpm x5min离心,收集上清10ml,加入2ml PEG/NaCl,混匀放置冰上30-60min,10000g×20min离心,去上清,用2x YT溶解噬菌体库;
d.封闭:免疫管用PBS洗两次,加入封闭液,室温1h;另外,取等体积封闭液与噬菌体库混合,室温封闭10-15min;
e.孵育噬菌体库:免疫管用PBS洗2次,加入封闭好的噬菌体库,37度培养箱2-3h;
f.洗脱:取100TG1菌液(前一天接种)到10ml2x YT中,37度,220rpm培养到A600值0.4-0.5。用PBST洗涤免疫管8次,再用PBS洗2次,加入5ml对数期生长的菌液,37度,220rpm,1h;
g.OUTPUT:稀释上述菌液10-1,10-2,分别取100ul涂平板。
h.INPUT:取稀释10-2,10-4,10-6的重组噬菌体库各1ul,加入到100ul对数期生长的菌液中,37度,220rpm,1h,然后将感染的菌液涂平板;
i.下-轮筛选:取200ul helper phage加入到5ml洗脱后的菌液中,同时加入5ul Ampicillin,37度,220rpm,1h,2500rpm x5min离心去上清,用10ML 2x YT-AK吹悬菌泥,37度,220rpm过夜培养,重复步骤b-h。
经过三轮筛选,挑选单克隆抗体,通过噬菌体ELISA方法,检测phage活性,具体如下:
a.包被TEM8蛋白,0.5ug/ml,4度过夜;
b.PBST洗两次,加入50ul phage上清和50ul 1%BSA,25℃,1h;
c.PBST洗三次,加入100ul 1:10000稀释的anti-M13-Ab-HRP,25℃,1h。;
d.PBST洗三次,加入100ul预热的TMB,25℃,10min;
e.加入50ul 1M H2SO4中止反应,OD450检测吸光值。
挑选阳性单克隆抗体,通过噬菌体与TEM8过表达细胞结合实验,检测单克隆抗体的细胞结合活性,具体如下:
a.用胰酶消化TEM8过表达细胞,200gx5min;
b.弃上清,用1%BSA重悬细胞,25℃,封闭20min;
c.50ul phage上清和50ul 1%BSA,25℃,1h;
d..细胞铺板,2500rpmx5min;
e.弃上清,用封闭好的phage重悬细胞,100ul/well,rt,1h;
f.PBS洗两次,加入100ul 1:10000稀释的anti-M13-Ab-HRP,rt,1h;
g.PBST洗两次,加入100ul预热的TMB,25℃,10min;
h.加入50ul 1M H2SO4中止反应,OD450检测吸光值。
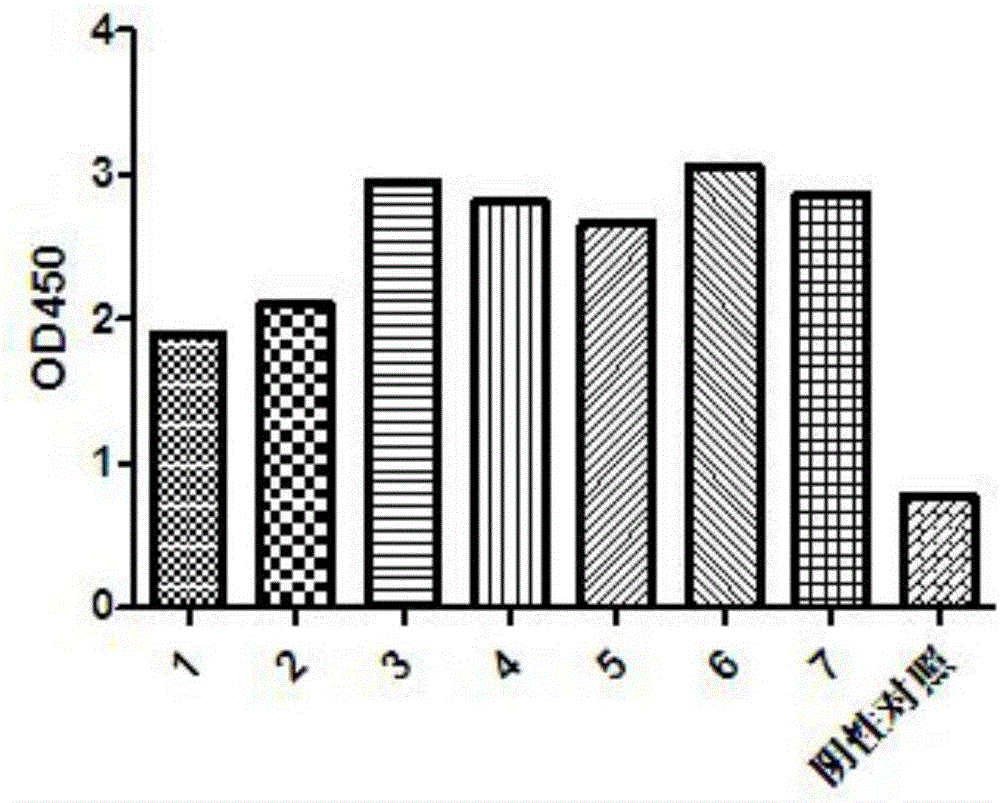
结果如图2所示,显示出通过细胞结合实验,挑选五个细胞结合克隆体,分别是clone 3,clone 4,clone 5,clone 6,clone 7,并进行测序,共鉴定出2株序列不同的亲和力成熟单克隆抗体,来自clone 3的抗体命名为NMT802,另外,clone 4,clone 5,clone 6,clone 7序列一致,抗体命名为NMT803。其中,NMT802的重链可变区氨基酸序列如SEQ ID NO.12所示,轻链可变区如SEQ ID NO.13所示,NMT803的重链可变区氨基酸序列如SEQ ID NO.14所示,轻链可变区如SEQ ID NO.15所示。另外,构建NMT802和NMT803的全长抗体,NMT802的重链氨基酸序列和轻链氨基酸序列如SEQ ID NO.16和SEQ ID NO.17所示,NMT802的重链氨基酸序列和轻链氨基酸序列如SEQ ID NO.18和SEQ ID NO.19所示。将NMT802和NMT803表达质粒瞬转293C18细胞株(ATCC No.CRL-10852),用Protein A(GE)纯化上清,获取纯化抗体。
实施例4
本实施例是对单克隆抗体的活性进行测定,具体按照以下步骤进行:
A:与重组TEM8蛋白结合活性测定
将TEM8蛋白包被于96孔酶联免疫吸附板(Costar)上,然后将1:4系列稀释实施例3中的纯化抗体加入包被TEM8蛋白板的孔中,加入鼠抗人IgG抗体-HRP(Jackson Immuno),显色进行检测。结果显示如图3,所筛选的单克隆抗体NMT802和NMT803与TEM8蛋白的直接结合EC50分别为32.42ng/ml和5.441ng/ml。
B:与TEM8过表达细胞结合活性测定
将PD1过表达细胞铺板于黑色96孔板,培养过夜,1%BSA封闭,37度,孵育1小时,抗体5ug/ml起始,3倍稀释,8浓度,100ul/孔,25度孵育1小时,PBS洗一次,然后,加入anti-human-EU,50ng/ml,每孔100ul,25度反应0.5小时,PBS洗一次,最后,加入荧光增强液,激发光337nm/发射光620nm读数。结果显示如图4所示,所筛选的单克隆抗体NMT802和NMT803与TEM8过表达细胞结合EC50分别为133ng/ml和764.7ng/ml。
C:采用流式细胞仪检测抗体与细胞膜TEM8的结合活性
取1×106个细胞,PBS洗一次,加入1%BSA封闭,冰上1小时,加入TEM8抗体,冰上孵育1小时,PBS洗两次,加入FITC-鼠抗人IgG二抗,冰上孵育1小时。PBS洗两次,用500ul PBS吹悬,采用BD流式细胞仪检测抗体与细胞膜TEM8结合水平,结果如图5显示,NMT802抗体能够特异结合细胞膜TEM8,空白CHO-K1作为阴性对照,NMT802抗体不与CHO-K1结合。
实施例5
在实施例1~4所披露的特异性结合TEM8的单克隆抗体的基础上,所得到的特异性结合TEM8的单克隆抗体的药物组合物或试剂、试剂盒或芯片,在用于治疗或检测TEM8相关性疾病药物中的用途。
SEQUENCE LISTING
<110> 江苏诺迈博生物医药科技有限公司
<120> 一种特异性结合肿瘤内皮标记物8的单克隆抗体及其应用
<130> 0
<160> 20
<170> PatentIn version 3.5
<210> 1
<211> 7
<212> PRT
<213> Homo sapiens
<400> 1
Thr Ser Gly Met Gly Val Gly
1 5
<210> 2
<211> 14
<212> PRT
<213> Homo sapiens
<400> 2
Trp Trp Asp Asp Val Lys Arg Tyr Tyr Pro Ala Leu Lys Ser
1 5 10
<210> 3
<211> 8
<212> PRT
<213> Homo sapiens
<400> 3
Ile Gly Tyr Asp Trp Phe Pro Tyr
1 5
<210> 4
<211> 11
<212> PRT
<213> Homo sapiens
<400> 4
Lys Ala Ser Gln Asn Val Gly Ile Ala Val Ala
1 5 10
<210> 5
<211> 7
<212> PRT
<213> Homo sapiens
<400> 5
Ser Ala Ser Asn Arg Tyr Thr
1 5
<210> 6
<211> 9
<212> PRT
<213> Homo sapiens
<400> 6
Gln Gln Tyr Arg Ser Tyr Pro Leu Thr
1 5
<210> 7
<211> 448
<212> PRT
<213> Homo sapiens
<400> 7
Gln Val Thr Leu Lys Glu Ser Gly Pro Gly Ile Leu Gln Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Met Gly Val Gly Trp Ile Arg Gln Pro Ser Gly Arg Gly Leu Glu
35 40 45
Trp Leu Ala His Ile Trp Trp Asp Asp Val Lys Arg Tyr Tyr Pro Ala
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Ser Ser Gln Val
65 70 75 80
Phe Leu Lys Ile Ala Ser Val Asp Thr Ala Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Ile Gly Tyr Asp Trp Phe Pro Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 8
<211> 214
<212> PRT
<213> Homo sapiens
<400> 8
Asp Ile Val Met Thr Gln Ser Gln Lys Phe Met Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Ser Ile Ala Cys Lys Ala Ser Gln Asn Val Gly Ile Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly His Ser Pro Lys Leu Leu Ile
35 40 45
His Ser Ala Ser Asn Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Tyr Gly Thr Asp Phe Thr Leu Thr Ile Ser Asn Met Gln Ser
65 70 75 80
Glu Asp Leu Ala Asp Tyr Phe Cys Gln Gln Tyr Arg Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Val Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 9
<211> 118
<212> PRT
<213> Homo sapiens
<400> 9
Glu Val Thr Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Met Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Trp Trp Asp Asp Val Lys Arg Tyr Tyr Pro Ala
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Ile Gly Tyr Asp Trp Phe Pro Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 10
<211> 107
<212> PRT
<213> Homo sapiens
<400> 10
Asp Ile Gln Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asn Val Gly Ile Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Asn Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Cys Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Arg Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 11
<211> 241
<212> PRT
<213> Homo sapiens
<400> 11
Glu Val Thr Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Met Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Trp Trp Asp Asp Val Lys Arg Tyr Tyr Pro Ala
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Ile Gly Tyr Asp Trp Phe Pro Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Phe Leu
130 135 140
Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln
145 150 155 160
Asn Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala
165 170 175
Pro Lys Leu Leu Ile Tyr Ser Ala Ser Asn Arg Tyr Thr Gly Val Pro
180 185 190
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
195 200 205
Ser Cys Leu Gln Ser Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr
210 215 220
Arg Ser Tyr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
225 230 235 240
Arg
<210> 12
<211> 118
<212> PRT
<213> Homo sapiens
<400> 12
Glu Val Thr Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Met Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Trp Trp Asp Asp Val Lys Arg Tyr Tyr Pro Ala
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Ala Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Ile Gly Tyr Asp Trp Phe Pro Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 13
<211> 107
<212> PRT
<213> Homo sapiens
<400> 13
Asp Ile Gln Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asn Val Gly Ile Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Asn Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Tyr Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Arg Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 14
<211> 118
<212> PRT
<213> Homo sapiens
<400> 14
Glu Val Thr Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Met Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Trp Trp Asp Asp Val Lys Gln Tyr Tyr Pro Ala
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Ile Gly Tyr Asp Trp Phe Pro Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 15
<211> 107
<212> PRT
<213> Homo sapiens
<400> 15
Asp Ile Gln Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asn Val Gly Ile Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Asn Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Cys Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Arg Phe Lys Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 16
<211> 448
<212> PRT
<213> Homo sapiens
<400> 16
Glu Val Thr Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Met Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Trp Trp Asp Asp Val Lys Arg Tyr Tyr Pro Ala
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Ala Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Ile Gly Tyr Asp Trp Phe Pro Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 17
<211> 214
<212> PRT
<213> Homo sapiens
<400> 17
Asp Ile Gln Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asn Val Gly Ile Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Asn Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Tyr Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Arg Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 18
<211> 448
<212> PRT
<213> Homo sapiens
<400> 18
Glu Val Thr Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Met Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Trp Trp Asp Asp Val Lys Gln Tyr Tyr Pro Ala
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Ile Gly Tyr Asp Trp Phe Pro Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 19
<211> 214
<212> PRT
<213> Homo sapiens
<400> 19
Asp Ile Gln Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asn Val Gly Ile Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Asn Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Cys Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Arg Phe Lys Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 20
<211> 1695
<212> DNA
<213> Homo sapiens
<400> 20
atggctaccg ccgaaagaag ggctctgggc attggattcc agtggctcag cctcgccaca 60
ctcgtgctga tttgcgctgg ccaaggcgga agaagagaag acggcggccc cgcttgctat 120
ggaggattcg acctgtactt catcctggat aagagcggct ccgtgctcca ccactggaac 180
gagatttact acttcgtcga gcagctggcc cacaaattta tcagccccca gctgaggatg 240
tccttcatcg tcttcagcac caggggcacc acactgatga agctgacaga ggatagggag 300
caaattaggc agggcctcga agagctgcaa aaggtcctgc ccggcggcga cacctacatg 360
cacgaaggct tcgagagggc tagcgagcag atctattacg aaaataggca aggctacaga 420
accgccagcg tcatcatcgc cctcaccgat ggagaactgc acgaggacct cttcttctac 480
agcgaaaggg aggccaatag gtccagggac ctgggagcca ttgtgtactg cgtgggagtc 540
aaggacttca acgaaaccca gctcgccaga atcgccgaca gcaaggacca cgtgttccct 600
gtgaatgacg gcttccaggc cctccaggga atcatccaca gcattctgaa gaagagctgc 660
attgagatcc tggccgccga acccagcaca atttgcgccg gcgagagctt ccaggtcgtg 720
gtcaggggaa atggcttcag acatgccaga aacgtcgaca gagtgctgtg cagcttcaag 780
atcaacgaca gcgtcaccct gaacgagaag cccttttccg tcgaggatac ctacctgctg 840
tgtcctgccc ctatcctgaa ggaggtgggc atgaaagccg ccctccaggt gagcatgaac 900
gacggactga gcttcatcag cagctccgtc atcatcacca ccacacactg tagcgatggc 960
agcatcctcg ccatcgctct gctgatcctc tttctgctgc tcgccctggc cctcctctgg 1020
tggttctggc ctctctgctg caccgtgatc atcaaggaag tgcctcctcc ccccgccgag 1080
gaaagcgaag aggaggatga cgacggcctc cctaagaaga agtggcctac agtggacgct 1140
agctactacg gcggaagggg cgtcggcgga atcaagagga tggaggtgag gtggggagag 1200
aaaggctcca cagaggaggg cgctaagctg gagaaggcca agaatgccag agtgaagatg 1260
cccgagcagg agtacgagtt ccccgagcct agaaacctga acaacaatat gaggaggccc 1320
tccagcccta ggaaatggta ctcccccatc aaaggaaagc tcgacgccct gtgggtgctc 1380
ctgagaaagg gctacgacag ggtgagcgtg atgagacctc aacccggaga caccggcagg 1440
tgtatcaact tcacaagggt caagaacaat cagcccgcta aatatcccct caacaacgcc 1500
tatcacacca gctcccctcc tcctgccccc atctacaccc ctcctcctcc tgctcctcat 1560
tgccctcccc ctcctcctag cgctcccaca ccccctattc ctagccctcc ttccaccctg 1620
cctccccctc ctcaagctcc tcctcccaac agagcccctc ctcctagcag gcctcctccc 1680
agaccttccg tgtag 1695
- 还没有人留言评论。精彩留言会获得点赞!