提高基因组编辑效率的方法与流程
本发明提供一种提高基因组编辑效率的方法,特别是涉及一种新的能够显著提高同源重组概率的增效因子,将所述增效因子与基因组编辑系统结合可提高基因组编辑效率。
背景技术:
在CRISPR/Cas9基因编辑系统中,设计不同的sgRNA足以指导Cas9内切酶完成对DNA的定点切割,形成DNA双链断裂(DSB),进而启动细胞自身的两种修复机制——非同源末端连接(NHEJ)和同源重组(HR),实现错误修复或者引入改变,造成DNA序列改变,从而实现基因的定向修饰操作。
DNA断裂产生后,当细胞内不存在同源片段时,细胞会启动非同源末端连接修复机制,在切口附近随机缺失或插入碱基,连接断裂的DNA双链。当细胞内存在同源片段时,细胞会发生极低概率的同源重组;如单链退火(SSA)是细胞内同源重组的一种常见方式。
CRISPR/Cas9系统是通过同源重组修复机制实现目标基因中不同类型的修饰,包括基因的删除、添加和替换等。当CRISPR/Cas9系统应用于植物时,即使存在外源引入的同源片段,植物体内发生同源重组的概率依然很低,这成为限制基因组编辑效率的瓶颈,所以提高同源重组概率对于基因组编辑系统的优化具有重要的意义。
目前,提高同源重组概率主要有以下途径:1)抑制非同源末端连接;实验表明,若抑制非同源末端连接途径中的相关蛋白如Ku70、Ku80、Lig4等的表达,会提高同源重组的概率。2)引入增效因子;已报道在拟南芥中引入酵母的Rad54基因(ScRad54),能够使同源重组概率提高约27倍,但目前没有其他有效的增效因子。
技术实现要素:
本发明的目的是提供一种提高基因组编辑效率的方法,有效克服了现有技术在植物体内发生同源重组概率低进而影响精确编辑的技术缺陷。
为实现上述目的,本发明提供一种提高同源重组概率的方法,包括向宿主细胞中引入增效因子,所述增效因子具有SEQ ID NO:1所示的氨基酸序列组成的蛋白质。
进一步地,所述增效因子包含a)或b):
a)编码SEQ ID NO:1所示的氨基酸序列组成的蛋白质的多核苷酸序列;
b)SEQ ID NO:2所示的多核苷酸序列。
进一步地,编码所述增效因子的多核苷酸序列有效连接于第一调控序列。
更进一步地,所述第一调控序列包括Ⅱ型启动子和/或定位结构域。
优选地,所述Ⅱ型启动子包括花椰菜花叶病毒35S启动子或泛素启动子。
在上述技术方案的基础上,所述方法还包括向宿主细胞中引入序列操纵系统。
具体地,所述序列操纵系统为CRISPR/Cas系统。
进一步地,编码所述基因组编辑系统的多核苷酸序列有效连接于第二调控序列。
更进一步地,所述第二调控序列包括Ⅲ型启动子。
优选地,所述Ⅲ型启动子包括U6启动子或U3启动子。
在上述技术方案的基础上,所述宿主细胞为植物细胞。
为实现上述目的,本发明还提供一种基因组编辑系统,包括包含a)和b):
a)编码增效因子的多核苷酸序列;
b)编码序列操纵系统的多核苷酸序列;
其中,所述增效因子具有SEQ ID NO:1所示的氨基酸序列组成的蛋白质。
可选地,所述增效因子与所述序列操纵系统由不同载体表达。
可选地,所述增效因子与所述序列操纵系统由同一载体表达。
进一步地,所述增效因子包含a)或b):
a)编码SEQ ID NO:1所示的氨基酸序列组成的蛋白质的多核苷酸序列;
b)SEQ ID NO:2所示的多核苷酸序列。
进一步地,编码所述增效因子的多核苷酸序列有效连接于第一调控序列。
更进一步地,所述第一调控序列包括Ⅱ型启动子和/或定位结构域。
优选地,所述Ⅱ型启动子包括花椰菜花叶病毒35S启动子或泛素启动子。
在上述技术方案的基础上,所述序列操纵系统为CRISPR/Cas系统。
进一步地,编码所述CRISPR/Cas系统的多核苷酸序列有效连接于第二调控序列。
更进一步地,所述第二调控序列包括Ⅲ型启动子。
优选地,所述Ⅲ型启动子包括U6启动子或U3启动子。
为实现上述目的,本发明还提供一种提高基因组编辑效率的方法,包括向宿主细胞中引入所述基因组编辑系统。
进一步地,所述宿主细胞为植物细胞。
为实现上述目的,本发明还提供一种实现高效基因组编辑的方法,包括在生物体中表达所述基因组编辑系统。
为实现上述目的,本发明还提供一种高效编辑靶位点序列的方法,包括将所述基因组编辑系统导入生物体中。
为实现上述目的,本发明还提供一种产生基因组编辑的植物的方法,包括向植物基因组中引入编码所述基因组编辑系统的核苷酸序列。
为实现上述目的,本发明还提供一种产生基因组编辑植物种子的方法,包括将所述方法产生的基因组编辑的植物自交,从而获得具有基因组编辑植物种子。
为实现上述目的,本发明还提供一种培育基因组编辑植物的方法,包括:
种植至少一粒所述方法产生的所述基因组编辑植物种子;
使所述种子长成植株。
为实现上述目的,本发明还提供一种增效因子在提高同源重组概率中的用途,其中,所述增效因子具有SEQ ID NO:1所示的氨基酸序列组成的蛋白质。
为实现上述目的,本发明还提供一种所述基因组编辑系统在提高同源重组概率和/或提高基因组编辑效率中的用途。
本发明中所述的CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats),是指成簇的规律间隔的短回文重复序列,含有多个短同向重复的基因座,其被发现存在于约40%测序细菌的基因组中和90%测序古细菌的基因组中。CRISPR作为原核的免疫系统发挥功能,其赋予对外来遗传元件例如质粒和噬菌体的抵抗性。CRISPR系统提供了一种获得性免疫形式。外源DNA的短片段(称为间隔区)整合在CRISPR重复序列之间的基因组中,然后CRISPR间隔区以类似于真核生物中RNAi的方式用于识别和沉默外来遗传元件。
本发明所述Cas蛋白的非限制性实例包括:Cas1、Cas1B、Cas2、Cas3、Cas4、Cas5、Cas6、Cas7、Cas8,Cas9(也称为Csn1和Csx12)、Cas10、Csy1、Csy2、Csy3、Cse1、Cse2、Csc1、Csc2、Csa5、Csn2、Csm2、Csm3、Csm4、Csm5、Csm6、Cmr1、Cmr3、Cmr4、Cmr5、Cmr6、Csb1、Csb2、Csb3、Csx17、Csx14、Csx10、Csx16、CsaX、Csx3、Csx1、Csx15、Csf1、Csf2、Csf3、Csf4、其同系物、或其修饰形式。这些酶是已知的;例如,化脓链球菌Cas9蛋白的氨基酸序列可见于SwissProt数据库登录号Q99ZW2下。在一些实施例中,未修饰的CRISPR酶如Cas9具有DNA切割活性。在一些实施例中,该CRISPR酶是Cas9,并且可以是来自化脓链球菌或肺炎链球菌的Cas9。
本发明中所述的Cas9,Ⅱ型CRISPR/Cas系统中一种重要的蛋白质成分,可以从生物体如链球菌属物种(Streptococcus sp.),优选酿脓链球菌(Streptococcus pyogenes)中分离得到。当Cas9与称为CRISPR RNA(crRNA)和反式激活crRNA(TracrRNA)的两个RNA复合时,形成活性核酸内切酶,从而切断入侵噬菌体或质粒中的外源遗传元件,以保护宿主细胞。crRNA从宿主基因组中的CRISPR元件转录,其中该CRISPR元件之前自外源入侵物捕获。研究表明通过融合crRNA和tracrRNA的必要部分产生的单链嵌合RNA可以取代Cas9/RNA复合体中的两个RNA以形成功能性核酸内切酶。Cas9蛋白质的变体可以是Cas9的突变体形式,其中催化性天冬氨酸残基改变为任何其它氨基酸。优选地,所述其它氨基酸可以是丙氨酸。
本发明中所述的向导RNA或引导RNA(guide RNA,gRNA),也称为小向导RNA(small guide RNA,sgRNA),作用于动质体(kinetoplastid)体内一种称为RNA编辑(RNA editing)的后转录修饰过程中,也是一种小型非编码RNA。可与pre-mRNA配对,并在其中插入一些尿嘧啶(U),产生具有作用的mRNA。向导RNA编辑的RNA分子,长度大约是60-80个核苷酸,是由单独的基因转录的,在gRNA的5’末端有一段锚定区,以特殊的G-U配对方式与非编辑的pre-mRNA序列互补,锚定序列促进gRNA与pre-mRNA中的编辑区互补,特意结合;在gRNA分子的中间部位有一个编辑区负责在被编辑的pre-mRNA分子中插入U的位置,其与被编辑mRNA精确互补;在gRNA分子的3’末端,有一段转录后加入的由大约15个非编码的PolyU序列,功能为把gRNA链接到pre-mRNA的编辑区的5’上游富含嘌呤碱基的核苷酸序列上。在编辑时,形成一个编辑体(editosome),以gRNA内部的序列作为模板进行转录物的校正,同时产生编辑的mRNA。
有三种类型CRISPR/Cas系统,其中涉及Cas9蛋白质和crRNA、tracrRNA的Ⅱ型CRISPR/Cas系统是代表性的。Cas9蛋白质通过人工修饰的向导RNA的导向作用可以打靶DNA序列的5’-N20-NGG-3’(N代表任何脱氧核苷酸碱基),N20是与gRNA的5’序列相同的20个碱基,NGG是PAM区(原型间隔子邻近基序,Protospacer-adjacent motif)。Cas9剪切的位点就是PAM附近的区域。相对于锌指和转录激活因子样效应物DNA结合蛋白提供了优势-因为在核苷酸结合CRISPR-Cas蛋白中的位点特异性由RNA分子调控而不是DNA结合蛋白调控。
本发明中所述重组,当用于例如细胞、核酸、蛋白质或载体时,表示该细胞、核酸、蛋白质或载体已通过引入异源核酸或蛋白质、或改变天然核酸或蛋白质而被修饰,或该细胞源自此修饰的细胞。
本发明中向导RNA可以以编码该向导RNA的RNA或DNA的形式转移到细胞或生物体中。向导RNA可以是分离的RNA、并入病毒载体的RNA的形式、或者在载体中编码。优选地,载体可以是病毒载体、质粒载体、或农杆菌载体。
编码向导RNA的DNA可以是包含编码向导RNA序列的载体。例如,可以通过用分离的向导RNA或包含编码向导RNA的序列和启动子的质粒DNA转染细胞或生物体,将向导RNA转染到细胞或生物体。
本发明中所述切割或剪切是指核苷酸分子共价骨架的断裂。向导RNA可以制备为特异于任何待切割的靶,通过向导RNA的靶特异性部分切割任何靶DNA。
本发明中所述非同源末端连接(Non-homologous end joining,NHEJ)指完全不需要任何模版的帮助,修复蛋白可以直接将DNA断裂的末端彼此拉近,再借由DNA连接酶的帮助将断裂的末端重新接合的修复机制。
本发明中所述同源重组(Homologous Recombination,HR)是指发生在非姐妹染色单体之间或同一染色体上含有同源序列的DNA分子之间或分子之内的重新组合。同源重组需要一系列的蛋白质催化,如原核生物细胞内的RecA、RecBCD、RecF、RecO、RecR等;以及真核生物细胞内的Rad51、Mre11-Rad50等等。同源重组反应通常根据交叉分子或Holliday结构的形成和拆分分为三个阶段,即前联会体阶段、联会体形成和Holliday结构的拆分。同源重组反应依赖于DNA分子之间的同源性,100%同源性的DNA分子之间的重组常见于非姐妹染色体之间的同源重组,称为Homologous Recombination,而小于100%同源性的DNA分子之间或分子之内的重组,则被称为Hemologus Recombination。后者可被负责碱基错配对的蛋白如原核细胞内的MutS或真核生物细胞内的MSH2-3等蛋白质“编辑”。同源重组可以双向交换DNA分子,也可以单向转移DNA分子,后者又被称为基因转换(Gene Conversion)。
本发明中,单链退火(SSA)模型是由Lin等于1984年提出的。在SSA模型中,重组在DNA双链断裂处开始,在单链特异性核酸外切酶的作用下,断裂点两侧逐渐形成DNA单链区域,这一过程持续到两个断点处出现互补DNA单链。互补DNA单链退火,非互补末端被切除,单链缺口修复连接,完成DNA重组。SSA模型没有其它模型所需的双链DNA的识别配对过程,不形成Holliday结构作为重组的中间过渡形式。因此,重组产生DNA双链交换,并丢失非退火区域单链DNA顺序。
本发明中所述的植物、植物组织或植物细胞的基因组,是指植物、植物组织或植物细胞内的任何遗传物质,且包括细胞核和质体和线粒体基因组。
本发明中所述的多核苷酸和/或核苷酸形成完整“基因”,在所需宿主细胞中编码蛋白质或多肽。本领域技术人员很容易认识到,可以将本发明的多核苷酸和/或核苷酸置于目的宿主中的调控序列控制下。
如本申请,包括权利要求中使用的,除非上下文中清楚地另有说明,否则单数和单数形式的术语,例如“一”、“一个”和“该”,包括复数指代物。因此,例如“植物”、“该植物”或“一个植物”也指示多个植物。并且根据上下文,使用术语“植物”还可指示该植物在遗传上相似或相同的后代。类似地,术语“核酸”可以指核酸分子的许多拷贝。类似地,术语“探针”可以指代相同或相似的探针分子。
数字范围包括限定该范围的数字,并明确地包括所限定的范围内的每个整数和非整数分数。除非另外指出,否则本文所用的全部技术和科学术语具有与本领域普通技术人员普遍理解的相同的含义。
本发明中,术语“核酸”、“核苷酸”、“核苷酸序列”、“寡核苷酸”和“多核苷酸”可互换使用,它们是指具有任何长度的核苷酸的聚合形式,根据上下文含义,可指代DNA或RNA、或其类似物。其中DNA包括但不限于cDNA、基因组DNA、合成DNA(例如人工合成)和含核酸类似物的DNA(或RNA)。多核苷酸可具有任何三维结构,并且可以执行已知或未知的任何功能。核酸可为双链或单链(既有义链或反义单链)。多核苷酸的非限制性示例包括基因、基因片段、外显子、内含子、信使RNA(mRNA)、转移RNA、核糖体RNA、核酶、cDNA、重组多核苷酸、分支多核苷酸、质粒、载体、任何序列的分离DNA、任何序列的分离RNA、核酸探针和引物、以及核酸类似物。
本发明中“非天然存在的”表明人工的参与。当指核酸分子或多肽时,表示该核酸分子或多肽至少基本上从它们在自然界中或如发现于自然界中的与其结合的至少另一种组分游离出来。
本发明中“表达”指感兴趣的序列转录产生对应mRNA并且该mRNA翻译产生对应产物,即肽、多肽或蛋白。调节元件控制或调整感兴趣的序列表达,所述调节元件包括5’调节元件如启动子。
本发明中“多肽”、“肽”和“蛋白质”可互换地使用,是指具有任何长度的氨基酸聚合物。该聚合物可以是直链或支链的,它可以包含修饰的氨基酸,并且它可以被非氨基酸中断。这些术语还涵盖已经被修饰的氨基酸聚合物;这些修饰例如二硫键形成、糖基化、脂化、乙酰化、磷酸化、或任何其他操作,如与标记组分的结合。术语“氨基酸”包括天然的和/或非天然的或者合成的氨基酸,包括甘氨酸以及D和L旋光异构体、以及氨基酸类似物和肽模拟物。
本发明中术语“载体”是指能够在宿主细胞内复制的DNA分子。质粒和粘粒是示例性载体。此外,术语“载体”和“媒介”可互换使用以指将DNA片段从一种细胞转移至另一种细胞的核酸分子,因此细胞不必要属于相同的生物(例如将DNA片段从农杆菌细胞转移至植物细胞)。
本发明中术语“表达载体”是指含有所需要的编码序列和在特定宿主生物中表达有效连接的编码序列所需要的适宜核酸序列的重组DNA分子。
本发明中术语“重组表达载体”指来自任何来源、能够整合入基因组或自主复制的任何因子如质粒、粘粒、病毒、BAC(细菌人工染色体)、自主复制型序列、噬菌体、或者线性或环状单链或双链DNA或RNA核苷酸序列,包括其中一种或多种DNA序列使用众所周知的重组DNA技术以功能性可操作方式连接的DNA分子。
本发明中,真核生物有3类RNA聚合酶,负责转录3类不同的启动子。由RNA聚合酶I负责转录的rRNA基因,启动子(I型)比较单一,由转录起始位点附近的两部分序列构成:第一部分是核心启动子(core promoter),由-45—+20位核苷酸组成,单独存在时就足以起始转录;另一部分由-170—-107位序列组成,称为上游调控元件,能有效地增强转录效率。
由RNA聚合酶Ⅲ负责转录的是5S rRNA、tRNA和某些核内小分子RNA(snRNA),其启动子(Ⅲ型)组成较复杂,又可被分为两个亚类:一类属于结构基因内启动子,一类属于结构基因外启动子。内启动子的有效启动依赖于基因内部包含的两个不连续的DNA片段,这两个DNA片段包括一些不同的连续DNA序列A、B或C区,两个区之间被隔开。根据两个区的不同组合,可以分为I类和II类两种:I类包括A和C区,目前发现其仅存在于5S rRNA的基因中;II类包括A和B区,存在于tRNA的基因、7SLRNA的基因、腺病毒VAI与VAII的RNA中。A、B或A、C内部DNA序列是转录因子TFⅢA和TFⅢC转录启动结合部位。在多数情况下,5’端还会有其它调节或关键元件,这些元件对RNA的高效转录是必需的。这些序列的存在与否之间影响着转录效率。这些序列呈现复杂的多样性,不过大多数启动子5’端-30到-20处存在着TATA盒样序列,与外启动子相似。外启动子缺乏相应的内部序列,仅在5’端有顺式作用元件,在基因的末端有一组由4个或更多胸腺嘧啶组成的终止信号,如脊椎动物U6核小RNA和7SK RNA启动子,这些启动子都高度相似或完全相同,其位置和碱基序列高度保守,其结构与pol II启动子也有一定的相似性。它们的5’端顺式作用元件包括几个控制元件,在其上游约-30处存在一个TATA样序列,近-60处有一个snRNA PSE(snRNA近似序列)和一个或多个被称为OCT的修饰序列5’-ATGCAAAT-3’。TATA样序列是pol III对snRNA基因进行转录特异的。TATA样元件和PSE元件共同决定了转录起始位点的选择和转录效率。TATA样元件和PSE元件之间的距离决定了RNA聚合酶转录的特异性,但似乎TATA样元件更重要,因为在PSE缺失的U6 RNA和7SK RNA基因转录中,只有转录效率的下降。而且,PSE元件可能和B盒(boxB)相关,B盒在一定程度上可以替代PSE元件。这些序列对下游基因的转录具有决定性的作用,而且它们与起始位点相距较远,往往超过150bp,而对pol III内启动子来说,一般在80bp以内;与pol III外启动子相比,pol II启动子5’端各顺式作用元件的作用刚好相反,如果TATA样元件缺失,PSE则可以履行TATA样元件功能,决定转录的起始部位。在PSE上游,外启动子还有一个远端控制序列,其结构与pol II增强子OCT骨架相似,但较其复杂。而在-223处还有一个CACC序列与OCT骨架相连。这些远端控制序列的存在可大大提高U6 RNA和7SK RNA的表达效率。
由RNA聚合酶II负责转录的II型基因包括所有蛋白质编码基因和部分snRNA基因,后者的启动子结构与III型基因启动子中的第三亚类相似,编码蛋白质的II型基因启动子在结构上有共同的保守序列。转录起始位点没有广泛的序列同源性,但第一个碱基为腺嘌呤,而两侧是嘧啶碱基。这个区域被称为起始子(initiator,Inr),序列可表示为Py2CAPy5。Inr元件位于-3—+5。仅由Inr元件组成的启动子是具有可被RNA聚合酶II识别的最简单启动子形式。多数II型启动子有一个被称为TATA盒的共有序列,通常处于-30区,相对于转录起始位点的位置比较固定。TATA盒存在于所有真核生物中,TATA盒是一个保守的七碱基对,也有一些II型启动子不含有TATA盒,这样的启动子称为无TATA盒启动子。
本发明中所述“有效连接”或“可操作地连接”表示核酸序列的联结,所述联结使得一条序列可提供对相连序列来说需要的功能。在本发明中所述“有效连接”可以为将启动子与感兴趣的序列相连,使得该感兴趣的序列的转录受到该启动子控制和调控。当感兴趣的序列编码蛋白并且想要获得该蛋白的表达时“有效连接”表示:启动子与所述序列相连,相连的方式使得得到的转录物高效翻译。如果启动子与编码序列的连接是转录物融合并且想要实现编码的蛋白的表达时,制造这样的连接,使得得到的转录物中第一翻译起始密码子是编码序列的起始密码子。备选地,如果启动子与编码序列的连接是翻译融合并且想要实现编码的蛋白的表达时,制造这样的连接,使得5’非翻译序列中含有的第一翻译起始密码子与启动子相连结,并且连接方式使得得到的翻译产物与编码想要的蛋白的翻译开放读码框的关系是符合读码框的。可以“有效连接”的核酸序列包括但不限于:提供基因表达功能的序列(即基因表达元件,例如启动子、5’非翻译区域、内含子、蛋白编码区域、3’非翻译区域、聚腺苷化位点和/或转录终止子)、提供DNA转移和/或整合功能的序列(即T-DNA边界序列、位点特异性重组酶识别位点、整合酶识别位点)、提供选择性功能的序列(即抗生素抗性标记物、生物合成基因)、提供可计分标记物功能的序列、体外或体内协助序列操作的序列(即多接头序列、位点特异性重组序列)和提供复制功能的序列(即细菌的复制起点、自主复制序列、着丝粒序列)。
本发明中,所述调控序列包括但不限于启动子、转运肽、终止子,增强子,前导序列,内含子以及其它可操作地连接到CRISPR系统的一个或多个元件,从而驱动该CRISPR系统的所述一个或多个元件的表达。一般而言,CRISPR(规律间隔成簇短回文重复),也称为SPIDR(Spacer间隔开的同向重复),构成通常对于特定细菌物种而言特异性的DNA基因座的家族。该CRISPR座位包含在大肠杆菌中被识别的间隔开的短序列重复(SSR)的一个不同类、以及相关基因。类似的间隔开的SSR已经鉴定于地中海富盐菌、化脓链球菌、鱼腥草属和结核分枝杆菌中。这些CRISPR座位典型地不同于其它SSR的重复结构,这些重复已被称为规律间隔的短重复(SRSR)。一般而言,这些重复是以簇存在的短元件,其被具有基本上恒定长度的独特间插序列规律地间隔开。虽然重复序列在菌株之间是高度保守的,许多间隔开的重复和这些间隔区的序列一般在菌株与菌株之间不同,已经在40种以上的原核生物中鉴定出CRISPR座位。
本发明中,“靶序列”或“靶位点序列”是待被作用的任何期望的预定的核酸序列,包括但不限于编码或非编码序列、基因、外显子或内含子、调节序列、基因间序列、合成序列和细胞内寄生物序列。在一些实施方案中,靶序列存在于靶细胞、组织、器官或生物体内。
术语“引物”是一段分离的核酸分子,其通过核酸杂交,退火结合到互补的目标DNA链上,在引物和目标DNA链之间形成杂合体,然后在聚合酶(例如DNA聚合酶)的作用下,沿目标DNA链延伸。本发明的引物对涉及其在目标核酸序列扩增中的应用,例如,通过聚合酶链式反应(PCR)或其他常规的核酸扩增方法。
引物的长度一般是11个多核苷酸或更多,优选的是18个多核苷酸或更多,更优选的是24个多核苷酸或更多,最优选的是30个多核苷酸或更多。这种引物在高度严格杂交条件下与目标序列特异性地杂交。尽管不同于目标DNA序列且对目标DNA序列保持杂交能力的引物是可以通过常规方法设计出来的,但是,优选的,本发明中的引物与目标序列的连续核酸具有完全的DNA序列同一性。
本发明中氨基酸序列的取代、缺失或添加是本领域的常规技术,优选这种氨基酸变化为:小的特性改变,即不显著影响蛋白的折叠和/或活性的保守氨基酸取代;小的缺失,通常约1-30个氨基酸的缺失;小的氨基或羧基端延伸,例如氨基端延伸一个甲硫氨酸残基;小的连接肽,例如约20-25个残基长。
保守取代的实例是在下列氨基酸组内发生的取代:碱性氨基酸(如精氨酸、赖氨酸和组氨酸)、酸性氨基酸(如谷氨酸和天冬氨酸)、极性氨基酸(如谷氨酰胺、天冬酰胺)、疏水性氨基酸(如亮氨酸、异亮氨酸和缬氨酸)、芳香氨基酸(如苯丙氨酸、色氨酸和酪氨酸),以及小分子氨基酸(如甘氨酸、丙氨酸、丝氨酸、苏氨酸和甲硫氨酸)。通常不改变特定活性的那些氨基酸取代在本领域内是众所周知的,并且已由,例如,N.Neurath和R.L.Hill在1979年纽约学术出版社(Academic Press)出版的《Protein》中进行了描述。最常见的互换有Ala/Ser,Val/Ile,Asp/Glu,Thu/Ser,Ala/Thr,Ser/Asn,Ala/Val,Ser/Gly,Tyr/Phe,Ala/Pro,Lys/Arg,Asp/Asn,Leu/Ile,Leu/Val,Ala/Glu和Asp/Gly,以及它们相反的互换。
对于本领域的技术人员而言显而易见地,这种取代可以在对分子功能起重要作用的区域之外发生,而且仍产生活性多肽。对于由本发明的多肽,其活性必需的并因此选择不被取代的氨基酸残基,可以根据本领域已知的方法,如定点诱变或丙氨酸扫描诱变进行鉴定(如参见,Cunningham和Wells,1989,Science244:1081-1085)。后一技术是在分子中每一个带正电荷的残基处引入突变,检测所得突变分子的抗虫活性,从而确定对该分子活性而言重要的氨基酸残基。底物-酶相互作用位点也可以通过其三维结构的分析来测定,这种三维结构可由核磁共振分析、结晶学或光亲和标记等技术测定(参见,如de Vos等,1992,Science 255:306-312;Smith等,1992,J.Mol.Biol 224:899-904;Wlodaver等,1992,FEBS Letters 309:59-64)。
本发明的引物在严格条件下与目标DNA序列杂交。核酸分子或其片段在一定情况下能够与其他核酸分子进行特异性杂交。如本发明使用的,如果两个核酸分子能形成反平行的双链核酸结构,就可以说这两个核酸分子彼此间能够进行特异性杂交。如果两个核酸分子显示出完全的互补性,则称其中一个核酸分子是另一个核酸分子的“互补物”。如本发明使用的,当一个核酸分子的每一个核苷酸都与另一个核酸分子的对应核苷酸互补时,则称这两个核酸分子显示出“完全互补性”。如果两个核酸分子能够以足够的稳定性相互杂交从而使它们在至少常规的“低度严格”条件下退火且彼此结合,则称这两个核酸分子为“最低程度互补”。类似地,如果两个核酸分子能够以足够的稳定性相互杂交从而使它们在常规的“高度严格”条件下退火且彼此结合,则称这两个核酸分子具有“互补性”。从完全互补性中偏离是可以允许的,只要这种偏离不完全阻止两个分子形成双链结构。为了使一个核酸分子能够作为引物或探针,仅需保证其在序列上具有充分的互补性,以使得在所采用的特定溶剂和盐浓度下能形成稳定的双链结构。
如本发明使用的,基本同源的序列是一段核酸分子,该核酸分子在高度严格条件下能够和相匹配的另一段核酸分子的互补链发生特异性杂交。促进DNA杂交的适合的严格条件,例如,大约在45℃条件下用6.0×氯化钠/柠檬酸钠(SSC)处理,然后在50℃条件下用2.0×SSC洗涤,这些条件对本领域技术人员是公知的。例如,在洗涤步骤中的盐浓度可以选自低度严格条件的约2.0×SSC、50℃到高度严格条件的约0.2×SSC、50℃。此外,洗涤步骤中的温度条件可以从低度严格条件的室温约22℃,升高到高度严格条件的约65℃。温度条件和盐浓度可以都发生改变,也可以其中一个保持不变而另一个变量发生改变。优选地,本发明的一个核酸分子可以在中度严格条件下,例如在约2.0×SSC和约65℃下与SEQ ID NO:2或其互补序列,或者上述序列的任一片段发生特异性杂交。更优选地,本发明的一个核酸分子在高度严格条件下与SEQ ID NO:2或其互补序列,或者上述序列的任一片段发生特异性杂交。本发明中,优选的标记物核酸分子具有SEQ ID NO:2或其互补序列,或者上述序列的任一片段。本发明另一优选的标记物核酸分子与SEQ ID NO:2或其互补序列,或者上述序列的任一片段具有80%到100%或90%到100%的序列同一性。SEQ ID NO:2可以用作植物育种方法中的标记物以鉴定遗传杂交的后代。探针与目标DNA分子的杂交可以通过任何一种为本领域技术人员所熟知的方法进行检测,这些方法包括但不限于,荧光标记、放射性标记、抗体类标记和化学发光标记。
关于使用特定的扩增引物对目标核酸序列进行的扩增(例如,通过PCR),“严格条件”指的是在DNA热扩增反应中仅允许引物对目标核酸序列发生杂交的条件,具有与目标核酸序列相应的野生型序列(或其互补序列)的引物,能够与所述目标核酸序列结合,并且优选产生唯一的扩增产物,扩增产物即扩增子。
术语“特异性结合(目标序列)”是指在严格杂交条件下引物仅与包含目标序列的样品中的目标序列发生杂交。
本发明中,“试剂盒”可包括本发明描述的基因组修饰系统与以下的任一种或全部:测定试剂、缓冲液、探针和/或引物、和无菌盐水或另一种药学上可接受的乳剂和悬液基底。此外,试剂盒可包括含有用于实践本发明描述的方法的用法说明(例如,操作方案)的说明性材料。
转化方案以及将核苷酸序列导入植物的方案依定向转化的植物或植物细胞类型而异,即单子叶植物或双子叶植物。将核苷酸序列导入植物细胞并随后插入植物基因组中的适合方法包括但不限于,农杆菌介导的转化、微量发射轰击、直接将DNA摄入原生质体、电穿孔或晶须硅介导的DNA导入。
已经转化的细胞可按照常规的方式生长成植物。这些植物被培育,用相同的转化株或不同的转化株授粉,得到的杂交体表达所需的被鉴定的表现型特征。可培育二代或多代以保证稳定地保持和遗传所需表现型特征的表达,然后收获可保证得到所需表现型特征表达的种子。
本发明提供了一种提高基因组编辑效率的方法,具有以下优点:
1、本发明提供了一些新的增效因子,能够显著提高细胞内同源重组概率,促进同源重组的发生,为提高植物体中同源重组概率提供了新的途径;
2、本发明提供了一种更高效的基因组编辑系统,当本发明所述增效因子在与分子剪刀共同使用时,可以显著提升分子剪刀的切割效率,进而提升基因组编辑的效率。
下面通过附图和实施例,对本发明的技术方案做进一步的详细描述。
附图说明
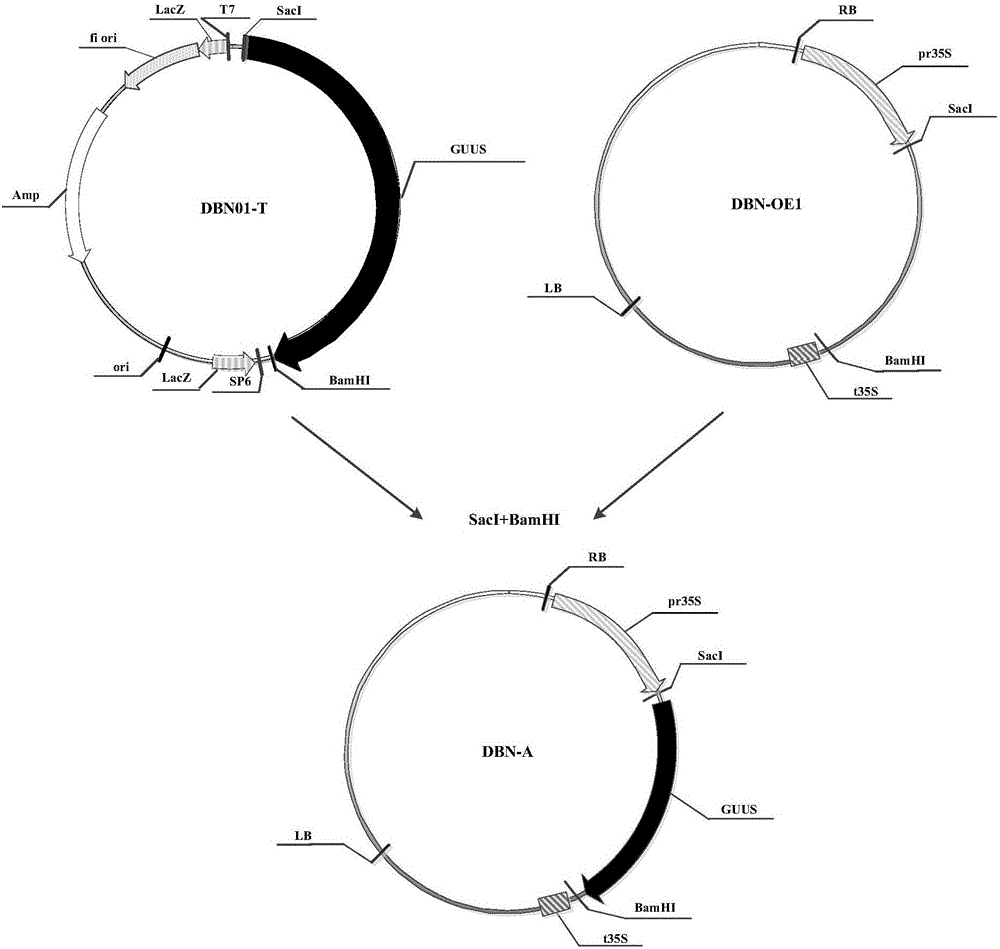
图1为本发明提高基因组编辑效率的方法中重组克隆载体DBN01-T构建流程图;
图2为本发明提高基因组编辑效率的方法中重组表达载体DBN-A构建流程图;
图3为本发明提高基因组编辑效率的方法中重组表达载体DBN-B01载体结构图;
图4为本发明提高基因组编辑效率的方法中剪刀载体DBN-C载体结构图;
图5为本发明提高基因组编辑效率的方法中不同增效因子对于报告载体同源重组概率的增效倍数示意图;
图6为本发明提高基因组编辑效率的方法中不同增效因子对于剪刀载体切割效率的增效倍数示意图。
具体实施方式
下面通过具体实施例进一步说明本发明提高基因组编辑效率的方法的技术方案。
第一实施例、报告载体DBN-A的构建
1、获得GUUS核苷酸序列
编码相应于所述GUUS核苷酸序列(3086bp),如序列表SEQ ID NO:6所示,所述GUUS核苷酸序列(如序列表中SEQ ID NO:6所示)由南京金斯瑞生物科技有限公司合成。合成的所述GUUS核苷酸序列(如序列表中SEQ ID NO:6所示)的5’端还连接有SacI酶切位点;合成的所述GUUS核苷酸序列(如序列表中SEQ ID NO:6所示)的3’端还连接有BamHI酶切位点。
2、重组克隆载体的构建
将合成的GUUS核苷酸序列连入克隆载体pGEM-T(Promega,Madison,USA,CAT:A3600)上,操作步骤按Promega公司产品pGEM-T载体说明书进行,得到重组克隆载体DBN01-T,其构建流程如图1所示(其中,Amp表示氨苄青霉素抗性基因;f1表示噬菌体f1的复制起点;LacZ为LacZ起始密码子;SP6为SP6RNA聚合酶启动子;T7为T7RNA聚合酶启动子;GUUS为GUUS核苷酸序列(GUUS核苷酸序列如SEQ ID NO:6所示);MCS为多克隆位点)。
然后将重组克隆载体DBN01-T用热激方法转化大肠杆菌T1感受态细胞(Transgen,Beijing,China,CAT:CD501),其热激条件为:50μl大肠杆菌T1感受态细胞、10μl质粒DNA(重组克隆载体DBN01-T),42℃水浴30秒;37℃振荡培养1小时(100rpm转速下摇床摇动),在表面涂有IPTG(异丙基硫代-β-D-半乳糖苷)和X-gal(5-溴-4-氯-3-吲哚-β-D-半乳糖苷)的氨苄青霉素(100mg/L)的LB平板(胰蛋白胨10g/L,酵母提取物5g/L,NaCl 10g/L,琼脂15g/L,用NaOH调pH至7.5)上生长过夜。挑取白色菌落,在LB液体培养基(胰蛋白胨10g/L,酵母提取物5g/L,NaCl 10g/L,氨苄青霉素100mg/L,用NaOH调pH至7.5)中于温度37℃条件下培养过夜。碱法提取其质粒:将菌液在12000rpm转速下离心1min,去上清液,沉淀菌体用100μl冰预冷的溶液I(25mM Tris-HCl,10mM EDTA(乙二胺四乙酸),50mM葡萄糖,pH8.0)悬浮;加入200μl新配制的溶液II(0.2M NaOH,1%SDS(十二烷基硫酸钠)),将管子颠倒4次,混合,置冰上3-5min;加入150μl冰冷的溶液III(3M醋酸钾,5M醋酸),立即充分混匀,冰上放置5-10min;于温度4℃、转速12000rpm条件下离心5min,在上清液中加入2倍体积无水乙醇,混匀后室温放置5min;于温度4℃、转速12000rpm条件下离心5min,弃上清液,沉淀用浓度(V/V)为70%的乙醇洗涤后晾干;加入30μl含RNase(20μg/ml)的TE(10mM Tris-HCl,1mM EDTA,pH8.0)溶解沉淀;于温度37℃下水浴30min,消化RNA;于温度-20℃保存备用。
提取的质粒经SacI和BamHI酶切鉴定后,对阳性克隆进行测序验证,结果表明重组克隆载体DBN01-T中插入的所述GUUS核苷酸序列为序列表中SEQ ID NO:6所示的核苷酸序列,即GUUS核苷酸序列正确插入。
3、重组表达载体的构建
将pCAMBIA2300(CAMBIA机构可以提供)载体进行改造,利用常规的酶切方法构建载体是本领域技术人员所熟知的,通过点突变去掉pCAMBIA2300载体上的BsaI位点,同时将卡那霉素表达盒去掉,得到pDBN骨架载体。向所述pDBN骨架载体引入花椰菜花叶病毒35S启动子和终止子,得到表达载体DBN-OE1,用于下述载体构建。
用限制性内切酶SacI和BamHI酶切重组克隆载体DBN01-T和表达载体DBN-OE1,将切下的GUUS核苷酸序列插入表达载体DBN-OE1,利用常规的酶切方法构建载体是本领域技术人员所熟知的,构建成重组表达载体DBN-A,其构建流程如图2所示(Kan:卡那霉素基因;RB:右边界;pr35S:花椰菜花叶病毒35S启动子(SEQ ID NO:7);GUUS:GUUS核苷酸序列(SEQ ID NO:6);t35S:花椰菜花叶病毒35S终止子(SEQ ID NO:8);LB:左边界)。
第二实施例、测试载体的构建
1、序列合成
拟南芥RecQ4增效因子(AtRecQ4)的氨基酸序列(1188个氨基酸),如序列表中SEQ ID NO:1所示;编码相应于AtRecQ4氨基酸序列的AtRecQ4核苷酸序列(3567个核苷酸),如序列表中SEQ ID NO:2所示。
炭疽菌DNTT增效因子(CoDNTT)的氨基酸序列(291个氨基酸),如序列表中SEQ ID NO:3所示;依据水稻密码子优化后编码相应于CoDNTT氨基酸序列的CoDNTT核苷酸序列(876个核苷酸),如序列表中SEQ ID NO:4所示。
依据水稻密码子优化后编码相应于酵母Rad54增效因子(ScRad54)的氨基酸序列的ScRad54核苷酸序列(2697个核苷酸),如序列表中SEQ ID NO:5所示。
所述AtRecQ4核苷酸序列(如序列表中SEQ ID NO:2所示)、所述CoDNTT核苷酸序列(如序列表SEQ ID NO:4)和所述ScRad54核苷酸序列(如序列表中SEQ ID NO:5所示)由南京金斯瑞生物科技有限公司合成。合成的所述AtRecQ4核苷酸序列(SEQ ID NO:2)、所述CoDNTT核苷酸序列(SEQ ID NO:4)和所述ScRad54核苷酸序列(SEQ ID NO:5)的5’端和3’端分别均连接有BsaI酶切位点。
2、重组表达载体的构建
按照上述第一实施例2中的方法,将所述AtRecQ4核苷酸序列连入克隆载体pGEM-T上,得到重组克隆载体DBN02-T,其中AtRecQ4为拟南芥RecQ4增效因子的核苷酸序列(SEQ ID NO:2)。酶切和测序验证重组克隆载体DBN02-T中所述AtRecQ4核苷酸序列正确插入。
按照上述第一实施例2中的方法,将所述CoDNTT核苷酸序列连入克隆载体pGEM-T上,得到重组克隆载体DBN03-T,其中CoDNTT为依据水稻密码子优化的炭疽菌DNTT增效因子的核苷酸序列(SEQ ID NO:4)。酶切和测序验证重组克隆载体DBN03-T中所述CoDNTT核苷酸序列正确插入。
按照上述第一实施例2中的方法,将所述ScRad54核苷酸序列连入克隆载体pGEM-T上,得到重组克隆载体DBN04-T,其中ScRad54为依据水稻密码子优化的酵母Rad54增效因子的核苷酸序列(SEQ ID NO:5)。酶切和测序验证重组克隆载体DBN04-T中所述ScRad54核苷酸序列正确插入。
按照第一实施例3中所述方法,向所述表达载体DBN-OE1引入prZmUbi启动子和tNos终止子,中间引入BsaI酶切位点,得到表达载体DBN-OE2,用于下述载体构建。
用BsaI酶切重组克隆载体DBN02-T和表达载体DBN-OE2,将切下的AtRecQ4核苷酸序列插入表达载体DBN-OE2,利用常规的酶切方法构建载体是本领域技术人员所熟知的,构建成重组表达载体DBN-B01,其载体结构如图3所示(Kan:卡那霉素基因;RB:右边界;pr35S:花椰菜花叶病毒35S启动子(SEQ ID NO:7);GUUS:GUUS核苷酸序列(SEQ ID NO:6);t35S:花椰菜花叶病毒35S终止子(SEQ ID NO:8);prZmUbi:玉米泛素(Ubiquitin)基因启动子(SEQ ID NO:9);AtRecQ4:拟南芥RecQ4增效因子的核苷酸序列(SEQ ID NO:2);tNos:胭脂碱合成酶基因的终止子(SEQ ID NO:10);LB:左边界)。
按照上述构建重组表达载体DBN-B01方法,用BsaI酶切重组克隆载体DBN03-T切下的所述CoDNTT核苷酸序列插入表达载体DBN-OE2,得到重组表达载体DBN-B02,酶切和测序验证重组表达载体DBN-B02中的核苷酸序列含有为序列表中SEQ ID NO:4所示核苷酸序列,即依据水稻密码子优化的CoDNTT核苷酸序列,所述依据水稻密码子优化的CoDNTT核苷酸序列可以连接所述prZmUbi启动子和tNos终止子。
按照上述构建重组表达载体DBN-B01方法,用BsaI酶切重组克隆载体DBN04-T切下的所述ScRad54核苷酸序列插入表达载体DBN-OE2,得到重组表达载体DBN-B03,酶切和测序验证重组表达载体DBN-B03中的核苷酸序列含有为序列表中SEQ ID NO:5所示核苷酸序列,即依据水稻密码子优化的ScRad54核苷酸序列,所述依据水稻密码子优化的ScRad54核苷酸序列可以连接所述prZmUbi启动子和tNos终止子。
第三实施例、剪刀载体的构建
根据Cas9蛋白识别PAM序列(NGG/NGGNG)的原则,在CRISPRDirect网站(http://crispr.dbcls.jp/)筛选到来源于玉米的一个靶位点序列(20bp),即45CS1靶位点序列,如SEQ ID NO:11所示。
45CS1正向引物:aggcaCCCAAGAGCGGGCGAGGTAG,如SEQ ID NO:12所示;
45CS1反向引物:gaaacCTACCTCGCCCGCTCTTGGG,如SEQ ID NO:13所示;
其中,上述引物5’端的粗体小写字母为保护碱基,斜体小写字母为BsaI酶切位点,带下划线的小写字母为BsaI酶切位点的粘性末端,正常字体大写字母为45CS1靶位点序列或其反向互补序列。
所述45CS1正向引物和所述45CS1反向引物分别包括45CS1靶位点序列,二者反向互补,互为模板,用Pfu酶(NEB)进行PCR扩增,PCR体系如下:
PCR反应条件为:98℃预变性30s,然后进入下列循环:98℃变性10s,56-60℃退火30s,72℃延伸30s/kb,共30-32个循环,最后72℃延伸5-10min;于4℃保存。
使用过柱纯化试剂盒(购自北京全式金生物技术有限公司)对上述PCR产物过柱纯化,具体方法参考其产品说明书;用BsaI酶切PCR产物和市售表达载体pRGEB32(购自Addgene),切胶回收对应酶切产物后,将酶切后的表达载体pRGEB32产物和PCR产物按照1:10的比例用T4连接酶于温度16℃下连接30min,利用常规的酶切方法构建载体是本领域技术人员所熟知的,获得引入了45CS1靶位点的剪刀载体DBN-C,其载体结构如图4所示(Kan:卡那霉素基因;LB:右边界;Hpt:潮霉素磷酸转移酶基因;pr35S:花椰菜花叶病毒35S启动子;prU3:水稻U3启动子;45CS1:45CS1靶位点序列(SEQ ID NO:11);sgRNA:sgRNA序列;prZmUbi:玉米泛素(Ubiquitin)基因启动子;Cas9:Cas9核苷酸序列;Ter:可以为花椰菜花叶病毒35S终止子;RB:右边界),按照第一实施例2中的方法,将剪刀载体DBN-C用热激方法转化大肠杆菌T1感受态细胞,碱法提取其质粒,提取的质粒进行测序验证,结果表明剪刀载体DBN-C构建正确。
第四实施例、重组表达载体转化农杆菌
对己经构建正确的重组表达载体DBN-A、DBN-B01、DBN-B02、DBN-B03和DBN-C用液氮法转化到农杆菌LBA4404(Invitrgen,Chicago,USA,CAT:18313-015)中,其转化条件为:100μl农杆菌LBA4404、3μl质粒DNA(重组表达载体);置于液氮中5min,37℃温水浴5min;将转化后的农杆菌LBA4404接种于LB试管中于温度28℃、转速为200rpm条件下培养2小时,涂于含25mg/L的利福平(Rifampicin)和50mg/L的卡那霉素的LB平板上直至长出阳性单克隆,挑取单克隆培养并提取其质粒,用限制性内切酶对重组表达载体DBN-A、DBN-B01、DBN-B02、DBN-B03和DBN-C酶切后进行酶切验证,结果表明重组表达载体DBN-A、DBN-B01、DBN-B02、DBN-B03和DBN-C结构完全正确。
第五实施例、增效因子在报告载体中的功能验证
1、瞬时转化的烟草植株的获得
活化菌液:用包含重组表达载体DBN-A、DBN-B01、DBN-B02和DBN-B03的LBA4404菌种划线,按照1:50的比例将温度-80℃冻存菌液(LBA4404)接种至1ml含25mg/L利福平和50mg/L卡那霉素的YEP培养基(牛肉浸膏10g/L、酵母提取液10g/L、NaCl 5g/L,pH7.0)中;约20h后,按照1:100的比例,将上述培养菌液增加至4ml,并于温度28℃下过夜培养16-18h(OD600值约为2),而后在转速为8000rpm条件下离心4min,弃上清,用10mM MgCl2将弃上清后的菌液浓度调至OD600=1;加入终浓度为10mM的2-吗啉乙磺酸(MES,pH5.6),然后加入终浓度为200μM的乙酰丁香酮(AS),室温静止放置3小时后获得侵染液。
用不带针头的注射器将侵染液注射进烟草叶片(6-8周),设5组处理,每组处理为在同一片叶片左边注射含有DBN-A的侵染液,右边分别注射含有DBN-B01、DBN-B02和DBN-B03的侵染液,并黑暗处理48-72h。获得转入DBN-A-B01的烟草叶片、转入DBN-A-B02的烟草叶片和转入DBN-A-B03的烟草叶片,每组处理设3次重复。
2、瞬时转化烟草植株的GUS定量测定
分别取瞬时转化获得的转入DBN-A-B01的烟草叶片、转入DBN-A-B02的烟草叶片和转入DBN-A-B03的烟草叶片作为样品,参照Jefferson等(Jefferson,R.A.,Kavanagh,T.A.and Bevan,M.W.GUS fusions:beta-glucuronidase as a sensitive and versatile gene fusion marker in higher plants.EMBO J.,1987,3901-3907)的方法,用4-甲基伞形酮(4-MU)通过荧光法测定GUS活性,即增效因子有助于使GUUS恢复GUS染色。实验设3次重复,以注射含有DBN-A的侵染液的部分作为对照,取平均值。测定GUS活性的具体方法如下:
步骤1、分别称取新鲜的转入DBN-A-B01的烟草叶片、转入DBN-A-B02的烟草叶片和转入DBN-A-B03的烟草叶片各20mg,用植物总蛋白提取试剂盒(采购自北京康为世纪生物科技有限公司)提取上述样品的叶片总蛋白,具体方法参考其产品说明书;
步骤2、在温度4℃、转速13000rpm下离心10min后,收集上清液,并置4℃下保存;
步骤3、在250μl预热(37℃、至少30min)的反应缓冲液(50mM Na3PO4(pH7.0)、1mM二硫苏糖醇(DTT)、10mM EDTA、0.1%十二烷基肌氨酸钠(Sarcosyl)、0.1%聚乙二醇辛基苯基醚(Triton X-100)、1mM 4-甲基伞形酮酰-β-葡萄糖苷酸(MUG))中加入50μl所述上清液,混匀,于温度37℃下反应15min后,取出30μl加入到270μl反应终止液(2%(m/v)碳酸钠)中终止反应;
步骤4、制作4-MU的标准曲线(0、50、100、250、500、1000、1500和2000pmol 4-MU稀释于2%碳酸钠),用SpectraMax M2测定各样品的Ex365/Em455值。
3、增效因子在报告载体中的功能验证
分别测得转入DBN-A-B01的烟草叶片、转入DBN-A-B02的烟草叶片和转入DBN-A-B03的烟草叶片的GUS活性平均值(pmol 4-MU/mg蛋白/min)为34.35/12863.76、29.50/502.86和32.16/501.71。
根据GUS活性值分别计算增效因子对GUUS的增效倍数,增效因子的增效倍数=测试载体恢复GUS表达的活性值/报告载体恢复GUS表达的活性值。如图5所示,上述实验结果表明,所述3个增效因子均能使GUUS恢复GUS染色,即当存在同源片段时,增效因子可以提高同源重组概率;所述AtRecQ4对同源重组概率的增效倍数均显著高于所述ScRad54(依据水稻密码子优化),为374.49,所述ScRad54(依据水稻密码子优化)对同源重组概率的增效倍数仅为15.60。
第六实施例、增效因子在剪刀载体中的增效功能验证
1、瞬时转化的烟草植株的获得
活化菌液:按照第五实施例1中所述方法,由包含重组表达载体DBN-A、DBN-C、DBN-B01、DBN-B02和DBN-B03的LBA4404菌种分别获得DBN-A、DBN-C、DBN-B01、DBN-B02和DBN-B03的菌液。按照以下组合将菌液等体积混合:DBN-C和DBN-A菌液(对照处理)、DBN-C和DBN-B01菌液(处理1)、DBN-C和DBN-B02菌液(处理2)、DBN-C和DBN-B03菌液(处理3),室温静置3h,获得对应处理的侵染液。
用不带针头的注射器将侵染液注射进烟草叶片(6-8周),上述3组处理中每组处理为在同一片叶片左边注射含有对照处理的侵染液,右边分别注射含有处理1、处理2和处理3的侵染液,并黑暗处理48-72h。获得转入DBN-AC-B01C的烟草叶片、转入DBN-AC-B02C的烟草叶片和转入DBN-AC-B03C的烟草叶片,每组处理设3次重复。
2、增效因子在剪刀载体中的增效功能验证
按照第五实施例2中的方法,分别测得转入DBN-AC-B01C的烟草叶片、转入DBN-AC-B02C的烟草叶片和转入DBN-AC-B03C的烟草叶片的GUS活性值(pmol 4-MU/mg蛋白/min)如表1所示。
表1、3个处理的GUS活性值(pmol 4-MU/mg蛋白/min)和增效倍数
根据GUS活性值分别计算增效因子对剪刀的增效倍数,增效因子的增效倍数=测试载体与剪刀载体共转化后恢复GUS表达的活性值/报告载体与剪刀载体共转化后恢复GUS表达的活性值。由图6和表1可得,所述3个增效因子通过有效切割可以提高CRISPR/Cas9系统的剪切效率,使GUUS恢复GUS染色,进而可以提高基因组编辑效率;所述AtRecQ4对CRISPR/Cas9系统剪切效率的提升效果(增效倍数和GUS活性值)明显优于所述ScRad54(依据水稻密码子优化),进而也使基因组编辑效率得到显著提高。
综上所述,本发明首次提供了一些新的增效因子,特别是所述AtRecQ4增效因子,能够显著提高细胞内同源重组概率,促进同源重组的发生,为提高植物体中同源重组概率提供了新的途径;当本发明所述增效因子在与分子剪刀共同使用时,可以显著提升分子剪刀的切割效率,进而提升基因组编辑的效率。
最后所应说明的是,以上实施例仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的精神和范围。
SEQUENCE LISTING
<110> 北京大北农生物技术有限公司
<120> 提高基因组编辑效率的方法
<130> DBN117
<160> 13
<170> PatentIn version 3.5
<210> 1
<211> 1188
<212> PRT
<213> Arabidopsis thaliana
<400> 1
Met Ile Asn Ser Asn Gln Met Ser Arg Ser His Leu Pro Glu Val Gln
1 5 10 15
Lys Pro Arg Gly Pro Gln Thr Asn Trp Ser Glu His Ala Lys Ala Leu
20 25 30
Glu Ser Ser Ser Ser Val Thr Lys Phe Leu Ser Ser Asn Val Leu Tyr
35 40 45
Ala Leu Glu Ser Gln Lys Pro Arg Asp Met Ala Ala Arg Ser Ile Ala
50 55 60
Phe Pro Ser Val Asn Val His Thr Leu Ala His Pro Gln Ile Ser Lys
65 70 75 80
Ala Trp Arg Ala Leu Ser Ser Leu Ser Val Asn Asn Thr Tyr Leu Arg
85 90 95
Pro Gly Val Thr Pro Pro Ile Asp Val Gly Thr Asn Asp Ser Tyr Ser
100 105 110
Ala Arg Glu Arg Ser Thr Ala Lys Val Ile Ser Ser Thr Gly Gly Ser
115 120 125
Val Tyr Ser Ser Thr Arg Pro Asn Leu Ser Ala Met Asn Val Ser Gly
130 135 140
Thr Gly Arg Ser Phe His Ser Phe Pro Ser Ser Val Pro Gly Asp Asp
145 150 155 160
Lys Ile Val Ala Glu Lys Phe Pro Arg Gly Asn Asn Glu Ile Arg Glu
165 170 175
Ser Glu Pro Ser Cys Thr His Leu Asn Gly Val Glu Lys Ser Phe Gly
180 185 190
Asn Ser Ala Phe Pro Ala Glu Gln Phe Glu Ser Arg Lys Ala Cys Leu
195 200 205
Asp Asp Met Asp Asp Asp Asp Ile Leu Glu Asn Ile Asp Val Asp Gln
210 215 220
Ile Val Met Glu His Tyr His Ser Thr Ser Thr Pro Gln Pro Ser Val
225 230 235 240
Ser Asn Phe Ser Leu Arg Thr Pro Pro Val Asp Arg Ser Ala Ser Arg
245 250 255
Leu Glu Glu Glu Cys Asn Leu Pro Pro Glu Leu Cys Ser Asn Cys Ser
260 265 270
His Gly Ile Lys Leu Gly Leu Cys Pro Glu Ala Ser Thr His Val Glu
275 280 285
Gln Met Lys Asp Val Leu Leu Ala Ile Ser Asn Glu Leu Leu Asp Asp
290 295 300
Ala Thr Asp Leu Ser Pro Asp Arg Val Gly Gln Leu Arg Gln Glu Arg
305 310 315 320
Leu Arg Leu Lys Lys Gln Ile Gln Gln Leu Glu Asn His Ile Arg Asp
325 330 335
Lys Glu Ser Gln Lys Ser Gln Phe Leu Ser Ser Thr Ala Thr Arg Ile
340 345 350
Phe Gln Tyr Glu Thr Pro Lys Ser Thr Asn Tyr Lys Met Asp Gln Pro
355 360 365
Gln Thr Asp Phe Arg Ala His Val Ser Asp Gln Gly Arg Tyr Ala Cys
370 375 380
Asp Ser Trp Asn Thr Pro Arg Asp Ser Ser Phe Ser Val Asp Arg Tyr
385 390 395 400
Gly Leu Ser Ser Ala Pro Val Glu Arg Glu Gln Tyr Val Pro Lys Ile
405 410 415
Ile Asp Val Thr Tyr Thr Glu Gly Ser Asn Asp Lys Lys Trp Ser Ser
420 425 430
Arg Glu Phe Pro Trp Thr Arg Lys Leu Glu Val Asn Asn Lys Lys Val
435 440 445
Phe Gly Asn His Ser Phe Arg Pro Asn Gln Arg Glu Ile Ile Asn Ala
450 455 460
Thr Met Ser Gly Ser Asp Val Phe Val Leu Met Pro Thr Gly Gly Gly
465 470 475 480
Lys Ser Leu Thr Tyr Gln Leu Pro Ala Leu Ile Cys Gly Gly Ile Thr
485 490 495
Leu Val Ile Ser Pro Leu Val Ser Leu Ile Gln Asp Gln Ile Met Asn
500 505 510
Leu Leu Gln Ala Asn Ile Pro Ala Ala Ser Leu Ser Ala Gly Met Glu
515 520 525
Trp Ala Glu Gln Leu Lys Ile Phe Gln Glu Leu Asn Ser Glu His Ser
530 535 540
Lys Tyr Lys Leu Leu Tyr Val Thr Pro Glu Lys Val Ala Lys Ser Asp
545 550 555 560
Ser Leu Leu Arg His Leu Glu Asn Leu Asn Ser Arg Gly Leu Leu Ala
565 570 575
Arg Phe Val Ile Asp Glu Ala His Cys Val Ser Gln Trp Gly His Asp
580 585 590
Phe Arg Pro Asp Tyr Gln Ser Leu Gly Ile Leu Lys Gln Lys Phe Pro
595 600 605
Asn Ile Pro Val Leu Ala Leu Thr Ala Thr Ala Thr Ala Ser Val Lys
610 615 620
Glu Asp Val Val Gln Ala Leu Gly Leu Val Asn Cys Val Val Phe Arg
625 630 635 640
Gln Ser Phe Asn Arg Pro Asn Leu Trp Tyr Ser Val Val Pro Lys Thr
645 650 655
Lys Lys Cys Leu Glu Asp Ile Asp Lys Phe Ile Lys Glu Asn His Phe
660 665 670
Asp Glu Cys Gly Ile Ile Tyr Cys Leu Ser Arg Met Asp Cys Glu Lys
675 680 685
Val Ser Glu Arg Leu Gln Glu Phe Gly His Lys Ala Ala Phe Tyr His
690 695 700
Gly Ser Met Glu Pro Glu Gln Arg Ala Phe Ile Gln Thr Gln Trp Ser
705 710 715 720
Lys Asp Glu Ile Asn Ile Ile Cys Ala Thr Val Ala Phe Gly Met Gly
725 730 735
Ile Asn Lys Pro Asp Val Arg Phe Val Ile His His Ser Leu Pro Lys
740 745 750
Ser Ile Glu Gly Tyr His Gln Glu Cys Gly Arg Ala Gly Arg Asp Gly
755 760 765
Gln Arg Ser Ser Cys Val Leu Tyr Tyr Gly Tyr Gly Asp Tyr Ile Arg
770 775 780
Val Lys His Met Ile Ser Gln Gly Gly Val Asp Gln Ser Pro Met Ala
785 790 795 800
Thr Gly Tyr Asn Arg Val Ala Ser Ser Gly Arg Leu Leu Glu Thr Asn
805 810 815
Thr Glu Asn Leu Leu Arg Met Val Arg Tyr Cys Glu Asn Glu Val Glu
820 825 830
Cys Arg Arg Phe Leu Gln Leu Val His Leu Gly Glu Lys Phe Asp Ser
835 840 845
Thr Asn Cys Lys Lys Thr Cys Asp Asn Cys Cys Ser Ser Gln Ser Leu
850 855 860
Ile Asp Lys Asp Val Thr Leu Ile Thr Arg Gln Leu Val Glu Leu Val
865 870 875 880
Lys Gln Thr Gly Glu Arg Phe Ser Ser Ala His Ile Leu Glu Val Tyr
885 890 895
Arg Gly Ser Leu Asn Gln Met Val Lys Lys His Arg His Glu Thr Leu
900 905 910
Gln Phe His Gly Ala Gly Lys His Leu Ser Lys Ile Glu Val Ser Arg
915 920 925
Ile Leu His Tyr Leu Val Thr Glu Asp Ile Leu Val Glu Asp Val Arg
930 935 940
Lys Ser Asp Met Tyr Gly Ser Val Ser Ser Leu Leu Gln Val Asn Asn
945 950 955 960
Ala Lys Ala Thr Ile Leu Phe Ser Gly Ser Gln Thr Ile Val Met Lys
965 970 975
Phe Pro Ser Ser Val Lys Val Leu Lys Pro Ser Lys Gln Gly Ala Thr
980 985 990
Ala Ala Lys Gly Pro Leu Thr Ser Glu Lys Gln Ser Thr Leu Pro Leu
995 1000 1005
Thr Thr Glu Asp Ala Pro Pro Lys Asp Val Asn Leu Ser Ala Asn
1010 1015 1020
Met Tyr Thr Ala Leu Arg Lys Leu Arg Thr Ala Leu Val Lys Glu
1025 1030 1035
Ala Pro Asp Gly Val Met Ala Tyr His Ile Phe Ile Asn Ser Thr
1040 1045 1050
Leu Gln Gln Ile Ser Arg Arg Ile Pro Arg Thr Lys Glu Glu Leu
1055 1060 1065
Leu Glu Ile Asn Gly Leu Gly Lys Ala Lys Val Ser Lys Tyr Gly
1070 1075 1080
Asp Gln Leu Leu Glu Thr Ile Glu Thr Thr Val Asn Glu Tyr Tyr
1085 1090 1095
Gly Thr Asn Lys Lys Asp Ser Ile Ile Ser Asn Asp Ser Pro Asp
1100 1105 1110
Ser Gly Lys Arg Arg Arg Asp Glu Asn Ile Ser Pro Asn Val Ala
1115 1120 1125
Glu Asp Asp Asp Phe Glu Val Ser Pro Ser Gln Ser Cys Lys Lys
1130 1135 1140
Thr Val Arg Asn Lys Ser Asn Glu Val Leu His Gly Glu Cys Ile
1145 1150 1155
Asp Gly Asp Arg Arg Gly Met Glu Leu Asp Phe Asp Phe Lys Asp
1160 1165 1170
Glu Asp Gly Ser Glu Ile Arg Pro Glu Gly Arg Val Leu Pro Trp
1175 1180 1185
<210> 2
<211> 3567
<212> DNA
<213> Arabidopsis thaliana
<400> 2
atgattaatt cgaatcaaat gagtaggagt cacctccccg aagttcagaa acctagaggt 60
cctcaaacga attggtcaga acatgcaaag gcgctcgaga gttcatcatc ggtgactaag 120
tttctgagct ccaatgtcct ttacgcccta gaaagccaaa agcctagaga tatggctgca 180
aggtcaattg cgtttcccag tgtgaatgtg catactttgg cgcacccgca aatttcaaag 240
gcttggcgtg ctctttcaag tctttcagta aataatacct acctaagacc tggcgttact 300
ccccccattg atgttggcac caatgactct tattctgcaa gagagagatc taccgcaaaa 360
gttatatcta gcactggtgg ttctgtttat tcgagcactc gaccaaacct aagtgcaatg 420
aatgtttcag gaactggaag atcctttcat agtttccctt cctctgtccc tggtgatgat 480
aaaatagttg ctgaaaagtt tccacgggga aacaatgaga tcagagaatc agaacccagt 540
tgcacgcatt taaatggtgt ggagaagtca ttcgggaatt ctgcattccc agctgagcag 600
ttcgaatcta gaaaagcatg tcttgatgat atggatgatg atgacatact tgagaatatt 660
gacgtcgacc aaatagtgat ggaacactac cattcaacaa gtaccccaca gccatcagtg 720
tctaattttt cgttgagaac tccacctgta gatagaagtg cttccagatt agaagaagag 780
tgcaatctgc ctcctgaatt atgttccaac tgtagtcatg gaataaagtt ggggctttgt 840
ccagaagctt caacccatgt ggagcagatg aaggacgtgt tgcttgcaat atcaaatgaa 900
ctccttgacg atgctactga cctgagtcct gatcgtgttg gacagctccg tcaagagagg 960
ttacggctga agaaacaaat tcagcagctc gagaaccata tccgagataa agaaagccaa 1020
aaatctcagt ttttgtcatc gacagctact cgtattttcc agtatgaaac acctaaatca 1080
acaaattata aaatggatca accacaaact gattttcgag cacatgtaag tgaccaaggc 1140
agatatgcct gtgatagctg gaacacgcca agagattctt ccttttctgt agatagatat 1200
ggtctttcat ctgctcctgt agagagggaa caatatgttc caaagattat tgacgtaacc 1260
tacactgaag gttcaaacga caaaaaatgg agcagtcgtg aatttccctg gacaaggaaa 1320
ttggaggtta ataacaaaaa agtgtttgga aatcattcgt ttcgacctaa ccagagagaa 1380
attatcaatg caacaatgag tggctctgat gtttttgttt tgatgccaac tggaggaggg 1440
aagagtttga catatcagct ccccgctctg atctgtggag gaataacatt agtaatttct 1500
ccacttgtgt ctcttattca agaccagata atgaacctat tgcaggccaa tataccagct 1560
gcttccctaa gtgccgggat ggaatgggct gaacaactaa agatctttca ggagctgaat 1620
tccgagcatt ctaagtacaa gttgctatat gtcacacctg aaaaagtagc aaaaagcgat 1680
tctcttttaa ggcatctgga aaacttaaat tctcgtggtt tgcttgctcg gtttgttatt 1740
gatgaagctc attgtgtgag ccaatgggga catgactttc gaccagacta ccagagtctt 1800
ggtattctga agcagaaatt tcccaacatt cccgtgttag ctttaacagc tacagcaaca 1860
gccagtgtga aagaagatgt ggtacaagcc cttggccttg ttaactgtgt cgtgttccgg 1920
caaagtttta atcgtccaaa tctatggtat tctgttgttc ccaagacaaa aaagtgtctg 1980
gaagacatcg acaaatttat taaggaaaac cattttgatg agtgtggaat tatatattgt 2040
ctttcaagaa tggactgcga aaaagtctct gaacggttgc aggagtttgg tcataaagca 2100
gcattctacc atggcagtat ggagcctgag caacgtgcct ttatacagac acaatggagc 2160
aaggacgaaa tcaatataat atgtgctact gtggcatttg gaatgggaat caacaagcct 2220
gatgtccgct ttgttattca tcactcgctc ccaaagtcta tagaaggcta tcatcaggaa 2280
tgcggtcgtg ctggcaggga tggccagcgg tcatcttgtg ttttatatta cggttatggc 2340
gactatatac gagttaagca tatgattagc caaggaggag ttgaccaaag ccccatggca 2400
actgggtata atcgtgtagc aagttcaggg aggttacttg aaacaaatac cgaaaacctt 2460
ctccgcatgg ttagatactg cgaaaatgag gtggaatgcc gacgtttcct acaactagtg 2520
catttggggg aaaaattcga ttccacaaac tgcaaaaaaa cgtgtgataa ttgttgcagt 2580
agtcagagtt tgattgacaa agatgtcacc ttgatcacaa ggcaactggt tgaactggtg 2640
aagcaaacag gagaaaggtt ttcttcagct catattcttg aagtctacag aggttcctta 2700
aaccaaatgg tcaagaaaca cagacatgag actttgcagt tccatggagc tggaaaacat 2760
ttatcgaaaa ttgaagtctc tcgtatcttg cattatctcg taacagagga tattcttgtg 2820
gaagatgtta ggaaaagtga tatgtatgga tctgtgtcat cgttgctcca ggtgaataac 2880
gcaaaggcta ccatactatt ttctggaagc cagactatag tgatgaagtt cccttcttct 2940
gtaaaagtat taaagccaag taaacagggg gcaaccgcag caaaaggtcc gctaacatca 3000
gagaagcaaa gtactcttcc acttacaaca gaagatgctc ctccaaaaga tgtgaatctc 3060
tctgccaaca tgtacacagc cttgaggaag cttcgtacag ctcttgtcaa agaagcccct 3120
gatggtgtca tggcatatca tatattcatc aattcaacgc ttcagcagat aagcagacgg 3180
atacccagaa ccaaagagga actccttgaa atcaatggcc ttgggaaggc caaagtaagc 3240
aagtacggtg atcagttgtt ggaaacaata gaaacaacgg tgaacgagta ctatggaacc 3300
aataagaaag actccatcat cagcaacgat agtcccgatt cagggaagag aagaagagac 3360
gagaacatta gtccaaatgt agccgaggat gatgatttcg aagtaagccc aagccagtcc 3420
tgcaaaaaaa cagtgagaaa caaaagcaat gaggttcttc atggtgaatg tatagatgga 3480
gatagaagag gcatggagct tgattttgat ttcaaagacg aagatgggtc agagatacga 3540
ccggagggaa gagtattacc gtggtga 3567
<210> 3
<211> 291
<212> PRT
<213> Colletotrichum orbiculare
<400> 3
Met Asp Pro Glu Ile Thr Arg Leu Pro Gly Cys Gly Ser Lys Val Ala
1 5 10 15
Glu Leu Trp His Glu Trp Lys Glu Ser Gly Arg Leu Arg Glu Val Asp
20 25 30
Glu Ala His Gly Asp Pro Lys Leu Ser Val Leu Gln Ala Phe Tyr Asp
35 40 45
Ile Trp Gly Val Gly Asp Ala Thr Ala Arg Asp Phe Tyr Asn Lys Asp
50 55 60
Leu Asp Asp Val Val Asp Phe Gly Trp Gln Ser Leu Ser Arg Ala Gln
65 70 75 80
Gln Ile Gly Val Lys Phe Tyr Asp Glu Phe Lys Leu Lys Ile Gln Arg
85 90 95
Asp Glu Val Glu Ala Ile Ala Glu Asp Ile Leu Lys His Ala Arg Asn
100 105 110
Phe Ser Pro Asp Phe Gln Met Val Ile Val Gly Gly Tyr Arg Arg Gly
115 120 125
Lys Gln Asp Ser Gly Asp Val Asp Val Ile Ile Ser His Pro Asp Glu
130 135 140
Ser Ala Thr Leu Asn Phe Val Asp Lys Leu Val Leu Ser Leu Glu Lys
145 150 155 160
Ser Arg Arg Val Thr His Thr Leu Ser Leu Ser Thr His Asn Ser Gln
165 170 175
Arg Gly Gln Arg Pro Val Ser Trp Arg Gly Asn Glu Arg Arg Gly Ser
180 185 190
Gly Phe Asp Thr Leu Asp Lys Ala Leu Val Val Trp Gln Glu Pro Leu
195 200 205
Arg Asp Ala Asp Asp Arg Ile Ser Leu Glu Lys Gly Arg Ala Cys Gln
210 215 220
Ala Pro His Arg Arg Val Asp Ile Ile Lys Glu Arg Gly Leu Arg Phe
225 230 235 240
Asp Ser Ser Gly Ile Arg Ser Arg Leu Asp Gly Thr Trp Val Asn Phe
245 250 255
Glu Gly Gly Asp Gly Gly Thr Ala Gln Asp Met Leu Thr Ala Glu Lys
260 265 270
Arg Val Phe Asp Gly Leu Gly Leu Arg Phe Val Asp Pro Tyr Asn Arg
275 280 285
Cys Thr Gly
290
<210> 4
<211> 876
<212> DNA
<213> Artificial Sequence
<220>
<223> CoDNTT核苷酸序列
<400> 4
atggaccctg agataacacg gcttcctggc tgtgggagta aagtcgctga actgtggcac 60
gagtggaaag aatcggggcg gttgcgtgag gtggatgaag ctcacgggga ccctaagctc 120
tcggttctgc aggccttcta cgatatctgg ggtgtcgggg atgctacagc ccgggacttc 180
tataacaagg atttggatga cgtggttgac tttggatggc agagcctctc aagagctcag 240
caaatcggtg tgaaattcta cgatgagttt aagttgaaga tacaaaggga cgaggttgaa 300
gcgattgcag aagatatact caagcacgcc cgcaacttct ctccggactt tcagatggtt 360
attgtcggcg gatatagaag gggtaaacaa gattccgggg atgtggacgt tatcattagc 420
catcccgacg agtcagcgac gcttaatttc gtcgataagc tcgtgctttc tctggaaaaa 480
tcccgccggg tcactcacac cttgtcgctc agtactcata actcgcagag agggcaaagg 540
ccggtgagtt ggagaggcaa tgagcgtaga ggcagcggat ttgatacctt ggacaaggcg 600
ctcgtcgtgt ggcaggagcc tcttcgtgac gcagatgaca gaatatcact ggaaaagggt 660
agggcttgcc aagccccaca taggcgcgtt gatataatca aagagcgcgg ccttcggttc 720
gactcttccg gaattcggag ccgtctggat ggtacatggg tcaattttga gggtggggac 780
ggcggaacag cgcaagatat gctcacggca gaaaagcgtg tgtttgatgg gctcgggctg 840
cggtttgttg acccctataa tcgttgcact ggttga 876
<210> 5
<211> 2697
<212> DNA
<213> Artificial Sequence
<220>
<223> ScRad54核苷酸序列
<400> 5
atggcacggc gtaggcttcc cgataggccc ccgaacggta ttggagcagg cgaaagacct 60
cgtctggtcc cgcgtccgat aaatgtccag gattcagtta atcgtctgac taagccattc 120
agagtcccgt acaaaaacac ccatatccct ccagctgccg gacggattgc aacaggttct 180
gacaacatag tgggcggacg tagcctcaga aagaggtctg ctacggtttg ctactctggc 240
ctcgatatca acgcagacga agctgagtat aactcccagg atatctcttt ctcccaactc 300
actaagagaa ggaaagatgc cctttctgca cagcggctgg ccaaggaccc tacccgtctt 360
tcgcacattc aatacacact gcgccggagt tttacggtcc ccataaaagg gtatgtgcag 420
aggcattccc tccctcttac tctgggcatg aagaaaaaga ttaccccaga gccccgccct 480
ctgcacgatc caacagacga attcgctatc gttttgtatg atccgtcggt cgacggagag 540
atgattgtgc atgatacttc tatggataat aaggaggaag agtcgaaaaa gatgattaag 600
agtacccaag agaaggataa cataaataag gagaagaact ctcaggaaga gaggcctact 660
caacgtatcg ggagacaccc tgcactcatg accaatggcg tgcgcaacaa gccacttcgg 720
gagctcctcg gcgattctga aaattccgcc gagaacaaaa agaaattcgc gtctgttcca 780
gtggttatcg atccgaagct cgcgaaaatt ctgaggccac accaggtgga gggagttcgg 840
ttcctttacc gttgtgtgac aggtttggtt atgaaggact atctcgaagc tgaggccttt 900
aacacttctt ctgaagatcc gttgaagtct gacgaaaaag cactcacaga gtcccaaaag 960
actgagcaaa acaatagagg ggcatacggc tgcattatgg ctgatgagat gggcctcggc 1020
aagactctcc agtgtatcgc tctcatgtgg acgctgttga gacagggacc acaaggcaag 1080
agactcatcg acaaatgcat cattgtttgt ccgagctcat tggtcaacaa ttgggcaaac 1140
gagcttatca agtggctggg gcccaacact ttgacccctc tcgctgtcga tggcaagaaa 1200
tcgagtatgg gtgggggcaa tactaccgtg tcgcaggcca tccatgcgtg ggcacaggct 1260
caaggtagaa atattgtcaa gccagtgctt ataatcagct acgagactct gcgtagaaac 1320
gtcgatcaat tgaagaactg caatgtgggc ttgatgctcg ccgacgaagg acacaggctc 1380
aaaaacggcg atagcttgac attcacggcg ctcgacagca tttcatgtcc aaggcgcgtt 1440
attctgtctg gcaccccgat acagaatgat ttgtccgagt atttcgccct cctttcgttt 1500
agtaacccgg ggctgttggg ctcaagagcg gagttcagga agaactttga aaatcctatt 1560
cttcgcgggc gggatgccga cgcgactgat aaggagatca ccaaaggcga agcccagctt 1620
caaaagctga gcacaatagt gtcaaaattc attatacggc gtacgaatga catcttggcg 1680
aagtacctcc cctgcaaata tgagcatgtt atattcgtca acttgaagcc tctccagaat 1740
gagctttaca acaagctgat caaaagccgc gaagtgaaga aagtcgtgaa gggagttgga 1800
ggttcacaac ccctcagagc catcggtatt cttaagaaac tgtgtaacca cccaaatctc 1860
cttaacttcg aagatgagtt tgatgacgaa gatgaccttg agctgccaga tgactacaat 1920
atgccgggct ctaaggcacg cgacgtccag acaaagtatt cggctaaatt cagtatattg 1980
gagcggtttc tccataagat caaaacggaa agcgatgaca agatagtgct catctcaaac 2040
tacactcaaa ccttggatct cattgagaag atgtgccgct acaaacacta tagcgctgtt 2100
cggctcgacg ggaccatgtc aattaataag aggcagaaac ttgtcgatcg cttcaacgac 2160
ccggaaggac aagagttcat ctttctgctc tcttctaagg ccgggggctg tgggattaat 2220
cttataggcg ctaacaggct tatcctcatg gaccctgact ggaatcctgc ggcagatcag 2280
caagcactgg ctagggtttg gcgcgatgga cagaagaaag actgcttcat ctatagattc 2340
atcagcactg gtacgataga agagaagatc tttcagaggc aatctatgaa aatgtccctg 2400
agctcatgtg ttgtcgatgc caaggaggat gtggagcgtt tgttctcgag tgataatttg 2460
agacagctct ttcaaaagaa cgagaatacc atctgcgaaa ctcacgagac ttaccattgc 2520
aaaaggtgta acgcgcaggg aaagcaactc aaacgcgccc ctgcgatgct ttatggtgac 2580
gcaacaacgt ggaatcacct taaccatgat gctctggaaa agactaacga ccatctcctc 2640
aaaaacgagc accactacaa cgacatctcc ttcgcctttc aatacataag ccactaa 2697
<210> 6
<211> 3086
<212> DNA
<213> Artificial Sequence
<220>
<223> GUUS核苷酸序列
<400> 6
atggtagatc tgagggtaaa tttctagttt ttctccttca ttttcttggt taggaccctt 60
ttctcttttt atttttttga gctttgatct ttctttaaac tgatctattt tttaattgat 120
tggttatggt gtaaatatta catagcttta actgataatc tgattacttt atttcgtgtg 180
tctatgatga tgatgatagt tacagaaccg acgaacttct ctgtacccga tcaacaccga 240
aacccgtggc gtcttcgacc tcaatggcgt ctggaacttc aagctggact acgggaaagg 300
actggaagag aagtggtacg aaagcaagct gaccgacact attagtatgg ccgtcccaag 360
cagttacaat gacattggcg tgaccaagga aatccgcaac catatcggat atgtctggta 420
cgaacgtgag ttcacggtgc cggcctatct gaaggatcag cgtatcgtgc tccgcttcgg 480
ctctgcaact cacaaagcaa ttgtctatgt caatggtgag ctggtcgtgg agcacaaggg 540
cggattcctg ccattcgaag cggaaatcaa caactcgctg cgtgatggca tgaatcgcgt 600
caccgtcgcc gtggacaaca tcctcgacga tagcaccctc ccggtggggc tgtacagcga 660
gcgccacgaa gagggcctcg gaaaagtcat tcgtaacaag ccgaacttcg acttcttcaa 720
ctatgcaggc ctgcaccgtc cggtgaaaat ctacacgacc ccgtttacgt acgtcgagga 780
catctcggtt gtgaccgact tcaatggccc aaccgggact gtgacctata cggtggactt 840
tcaaggcaaa gccgaaaaca ggataatgag gtactggctg gaaggcccaa gagcgggcga 900
ggtagaggtg ttcgcgaacc tgccgggctt ccccgacaac gtgcgctcca acggcagggg 960
ccagttctgg gtggcgatcg actgctgccg gacgccggcg caggaggtgt tcgccaagag 1020
gccgtggctc cggaccctat acttcaagtt cccgctgtcg ctcaaggtgc tcacttggaa 1080
ggccgccagg aggatgcaca cggtgctcgc gctcctcgac ggcgaagggc gcgtcgtgga 1140
ggtgctcgag gaccggggcc acgaggtgat gaagctggtg agtgaggtgc gggaggtggg 1200
cagcaagctg tggatcggaa ccgtggcgca caaccacatc gccaccatcc cctacccttt 1260
agaggactaa ttttacccgt ggcgtcttcg acctcaatgg cgtctggaac ttcaagctgg 1320
actacgggaa aggactggaa gagaagtggt acgaaagcaa gctgaccgac actattagta 1380
tggccgtccc aagcagttac aatgacattg gcgtgaccaa ggaaatccgc aaccatatcg 1440
gatatgtctg gtacgaacgt gagttcacgg tgccggccta tctgaaggat cagcgtatcg 1500
tgctccgctt cggctctgca actcacaaag caattgtcta tgtcaatggt gagctggtcg 1560
tggagcacaa gggcggattc ctgccattcg aagcggaaat caacaactcg ctgcgtgatg 1620
gcatgaatcg cgtcaccgtc gccgtggaca acatcctcga cgatagcacc ctcccggtgg 1680
ggctgtacag cgagcgccac gaagagggcc tcggaaaagt cattcgtaac aagccgaact 1740
tcgacttctt caactatgca ggcctgcacc gtccggtgaa aatctacacg accccgttta 1800
cgtacgtcga ggacatctcg gttgtgaccg acttcaatgg cccaaccggg actgtgacct 1860
atacggtgga ctttcaaggc aaagccgaaa ccgtgaaagt gtcggtcgtg gatgaggaag 1920
gcaaagtggt cgcaagcacc gagggcctga gcggtaacgt ggagattccg aatgtcatcc 1980
tctgggaacc actgaacacg tatctctacc agatcaaagt ggaactggtg aacgacggac 2040
tgaccatcga tgtctatgaa gagccgttcg gcgtgcggac cgtggaagtc aacgacggca 2100
agttcctcat caacaacaaa ccgttctact tcaagggctt tggcaaacat gaggacactc 2160
ctatcaacgg ccgtggcttt aacgaagcga gcaatgtgat ggatttcaat atcctcaaat 2220
ggatcggtgc caacagcttc cggaccgcac actatccgta ctctgaagag ttgatgcgtc 2280
ttgcggatcg cgagggtctg gtcgtgatcg acgagactcc ggcagttggc gtgcacctca 2340
acttcatggc caccacggga ctcggcgaag gcagcgagcg cgtcagtacc tgggagaaga 2400
ttcggacgtt tgagcaccat caagacgttc tccgtgaact ggtgtctcgt gacaagaacc 2460
atccaagcgt cgtgatgtgg agcatcgcca acgaggcggc gactgaggaa gagggcgcgt 2520
acgagtactt caagccgttg gtggagctga ccaaggaact cgacccacag aagcgtccgg 2580
tcacgatcgt gctgtttgtg atggctaccc cggagacgga caaagtcgcc gaactgattg 2640
acgtcatcgc gctcaatcgc tataacggat ggtacttcga tggcggtgat ctcgaagcgg 2700
ccaaagtcca tctccgccag gaatttcacg cgtggaacaa gcgttgccca ggaaagccga 2760
tcatgatcac tgagtacggc gcagacaccg ttgcgggctt tcacgacatt gatccagtga 2820
tgttcaccga ggaatatcaa gtcgagtact accaggcgaa ccacgtcgtg ttcgatgagt 2880
ttgagaactt cgtgggtgag caagcgtgga acttcgcgga cttcgcgacc tctcagggcg 2940
tgatgcgcgt ccaaggaaac aagaagggcg tgttcactcg tgaccgcaag ccgaagctcg 3000
ccgcgcacgt ctttcgcgag cgctggacca acattccaga tttcggctac aagaacgcta 3060
gccatcacca tcaccatcac gtgtga 3086
<210> 7
<211> 328
<212> DNA
<213> Cauliflower mosaic virus
<400> 7
ccattgccca gctatctgtc actttattgt gaagatagtg gaaaaggaag gtggctccta 60
caaatgccat cattgcgata aaggaaaggc catcgttgaa gatgcctctg ccgacagtgg 120
tcccaaagat ggacccccac ccacgaggag catcgtggaa aaagaagacg ttccaaccac 180
gtcttcaaag caagtggatt gatgtgatat ctccactgac gtaagggatg acgcacaatc 240
ccactatcct tcgcaagacc cttcctctat ataaggaagt tcatttcatt tggagaggac 300
acgctgacaa gctgactcta gcagatct 328
<210> 8
<211> 195
<212> DNA
<213> Cauliflower mosaic virus
<400> 8
ctgaaatcac cagtctctct ctacaaatct atctctctct ataataatgt gtgagtagtt 60
cccagataag ggaattaggg ttcttatagg gtttcgctca tgtgttgagc atataagaaa 120
cccttagtat gtatttgtat ttgtaaaata cttctatcaa taaaatttct aattcctaaa 180
accaaaatcc agtgg 195
<210> 9
<211> 1992
<212> DNA
<213> Zea mays
<400> 9
ctgcagtgca gcgtgacccg gtcgtgcccc tctctagaga taatgagcat tgcatgtcta 60
agttataaaa aattaccaca tatttttttt gtcacacttg tttgaagtgc agtttatcta 120
tctttataca tatatttaaa ctttactcta cgaataatat aatctatagt actacaataa 180
tatcagtgtt ttagagaatc atataaatga acagttagac atggtctaaa ggacaattga 240
gtattttgac aacaggactc tacagtttta tctttttagt gtgcatgtgt tctccttttt 300
ttttgcaaat agcttcacct atataatact tcatccattt tattagtaca tccatttagg 360
gtttagggtt aatggttttt atagactaat ttttttagta catctatttt attctatttt 420
agcctctaaa ttaagaaaac taaaactcta ttttagtttt tttatttaat aatttagata 480
taaaatagaa taaaataaag tgactaaaaa ttaaacaaat accctttaag aaattaaaaa 540
aactaaggaa acatttttct tgtttcgagt agataatgcc agcctgttaa acgccgtcga 600
cgagtctaac ggacaccaac cagcgaacca gcagcgtcgc gtcgggccaa gcgaagcaga 660
cggcacggca tctctgtcgc tgcctctgga cccctctcga gagttccgct ccaccgttgg 720
acttgctccg ctgtcggcat ccagaaattg cgtggcggag cggcagacgt gagccggcac 780
ggcaggcggc ctcctcctcc tctcacggca cggcagctac gggggattcc tttcccaccg 840
ctccttcgct ttcccttcct cgcccgccgt aataaataga caccccctcc acaccctctt 900
tccccaacct cgtgttgttc ggagcgcaca cacacacaac cagatctccc ccaaatccac 960
ccgtcggcac ctccgcttca aggtacgccg ctcgtcctcc cccccccccc ctctctacct 1020
tctctagatc ggcgttccgg tccatggtta gggcccggta gttctacttc tgttcatgtt 1080
tgtgttagat ccgtgtttgt gttagatccg tgctgctagc gttcgtacac ggatgcgacc 1140
tgtacgtcag acacgttctg attgctaact tgccagtgtt tctctttggg gaatcctggg 1200
atggctctag ccgttccgca gacgggatcg atttcatgat tttttttgtt tcgttgcata 1260
gggtttggtt tgcccttttc ctttatttca atatatgccg tgcacttgtt tgtcgggtca 1320
tcttttcatg cttttttttg tcttggttgt gatgatgtgg tctggttggg cggtcgttct 1380
agatcggagt agaattctgt ttcaaactac ctggtggatt tattaatttt ggatctgtat 1440
gtgtgtgcca tacatattca tagttacgaa ttgaagatga tggatggaaa tatcgatcta 1500
ggataggtat acatgttgat gcgggtttta ctgatgcata tacagagatg ctttttgttc 1560
gcttggttgt gatgatgtgg tgtggttggg cggtcgttca ttcgttctag atcggagtag 1620
aatactgttt caaactacct ggtgtattta ttaattttgg aactgtatgt gtgtgtcata 1680
catcttcata gttacgagtt taagatggat ggaaatatcg atctaggata ggtatacatg 1740
ttgatgtggg ttttactgat gcatatacat gatggcatat gcagcatcta ttcatatgct 1800
ctaaccttga gtacctatct attataataa acaagtatgt tttataatta ttttgatctt 1860
gatatacttg gatgatggca tatgcagcag ctatatgtgg atttttttag ccctgccttc 1920
atacgctatt tatttgcttg gtactgtttc ttttgtcgat gctcaccctg ttgtttggtg 1980
ttacttctgc ag 1992
<210> 10
<211> 253
<212> DNA
<213> Agrobacterium tumefaciens
<400> 10
gatcgttcaa acatttggca ataaagtttc ttaagattga atcctgttgc cggtcttgcg 60
atgattatca tataatttct gttgaattac gttaagcatg taataattaa catgtaatgc 120
atgacgttat ttatgagatg ggtttttatg attagagtcc cgcaattata catttaatac 180
gcgatagaaa acaaaatata gcgcgcaaac taggataaat tatcgcgcgc ggtgtcatct 240
atgttactag atc 253
<210> 11
<211> 20
<212> DNA
<213> Zea mays
<400> 11
cccaagagcg ggcgaggtag 20
<210> 12
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> 45CS1正向引物
<400> 12
tacccaggtc tcaggcaccc aagagcgggc gaggtag 37
<210> 13
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> 45CS1反向引物
<400> 13
taagttggtc tcgaaaccta cctcgcccgc tcttggg 37
- 还没有人留言评论。精彩留言会获得点赞!