一种光合作用调控蛋白LPE1及其应用的制作方法
本发明属于植物基因工程技术领域,更具体地,涉及一种光合作用调控蛋白LPE1及其应用
背景技术:
随着全球人口增长,耕地面积减少,气候变化导致的异常天气及生物能源的消耗,使全球粮食危机日益严峻。植物和细菌通过吸收光能,利用二氧化碳和水等无机物合成糖类等有机物质同时释放出氧的过程称为光合作用。光合作用被认为是地球上最重要的化学反应,是生物界赖以生存的基础,90%~95%的植物干重来自光合作用,光合作用效率的高低直接影响作物的产量。因此,提高光合作用效率已成为增加作物产量的重要途径,为解决粮食短缺问题找到了新出路。
目前,提高光合作用效率的方法有:①植物光合作用促进剂,一般利用促进剂中营养物促进植物光合作用。如专利CN101913951A公开了的一种植物光合作用促进剂,能够在弱光、低温、干热等逆境条件下显著提高植物光合作用效率,保证植株正常生长。②通过基因改造的技术,调控光合作用系统中的关键基因。叶绿体是绿色植物和藻类进行光合作用的主要场所,其上的基质和基粒片层结构是光合作用发生的主要位点,在这类片层膜上集聚了进行光合作用电子传递的四大复合体,即光系统II,光系统I,细胞色素b6f和ATP酶复合体,通过调控这些参与影响光合作用电子传递的因子对提高光合作用具有重要的意义。例如Miyagawa等通过过表达光合作用暗反应SBPase酶,有效增加了光合作用暗反应速率;再如Lin等通过基因工程方法向烟草中转入外源蓝细菌Rubisco,从而使得光合作用碳固定更加高效。
尽管如此,到目前为止,有效提高光合作用效率的方法还非常有限,因此寻找一种光合作用调控因子,应用于高光效植物的培育和筛选,具有现实意义。
技术实现要素:
本发明的目的在于提供一种光合作用调控蛋白LPE1及其应用。
本发明所采取的技术方案是:
光合作用调控蛋白LPE1,其氨基酸序列如SEQ ID NO:1所示。
编码光合作用调控蛋白LPE1的基因,其核苷酸序列如SEQ ID NO:2所示或为其同源保守序列。
一种提高植物光合作用效率的方法,包括将植物的光合作用调控基因LPE1上调或使光合作用调控蛋白LPE1过表达。
其中,所述的植物为禾本科作物、豆科作物、茄科作物、蔷薇科作物。
一种快速筛查高光合作用效率植物的方法,包括检测植物的光合作用调控基因LPE1的表达情况,光合作用调控基因LPE1上调则判定植物的光合作用效率更高。
其中,所述的植物为禾本科作物、豆科作物、茄科作物、蔷薇科作物。
一种获得高光合作用效率植物的转基因方法,包括将植物的光合作用调控基因LPE1上调。
其中,所述的植物为禾本科作物、豆科作物、茄科作物、蔷薇科作物。
本发明的有益效果是:
发明人利用正向遗传学方法从拟南芥突变体库中筛选到一个光合作用调控蛋白突变体lpe1,通过分子鉴定,克隆到了一个编码含PPR结构域叶绿体蛋白的基因LPE1(AT3G46610),并证实了其编码基因LPE1参与拟南芥光效调控,是一种重要的调控植物光合效率的功能基因,利用正向遗传学方法,获得两个LPE1的干涉株系(lpe1-1、lpe1-2)及一个纯合缺失突变体株系(lpe1-3),发现LPE1基因的功能缺失导致植株发育迟缓,最大光合效率降低。这为利用基因工程手段调控LPE1以增加光合速率打下基础。
结合生物信息学分析,发现LPE1蛋白在其他作物中具有序列保守性,提示LPE1在其他作物中的功能可能存在保守性。可以预见,通过对LPE1基因进行上调或蛋白LPE1过表达,有望获得高光效的植物,
附图说明
图1:拟南芥LPE1突变体LPE1基因表达、光合作用效率和植株生长情况图;
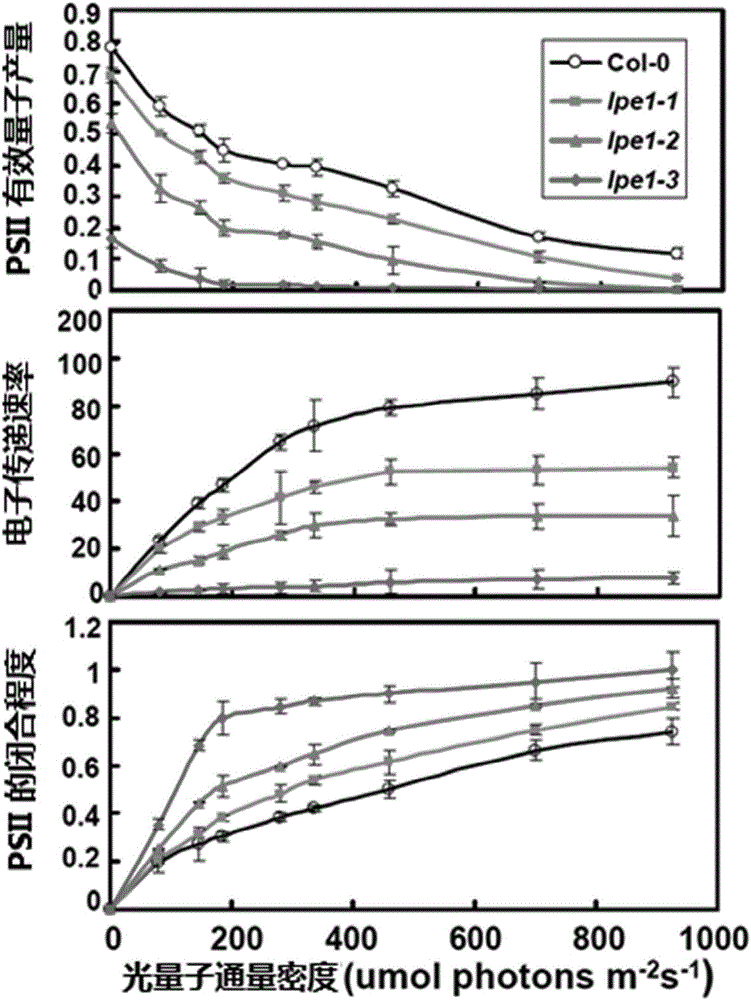
图2:拟南芥LPE1突变体叶绿素荧光参数图;
图3:LPE1蛋白特异性定位于叶绿体图;
图4:LPE1蛋白序列在作物中的保守性分析图。
具体实施方式
发明人通过实验发现一种光合作用调控蛋白LPE1,其氨基酸序列如SEQ ID NO:1所示,存在有叶绿体信号肽(Chloroplast Transit Peptide,CTP)结构域和PPR结构域(pentatricopeptide repeat,PPR)(如图3所示)。
一种编码光合作用调控蛋白LPE1的基因(如SEQ ID NO:2所示),位于拟南芥3号染色体上,基因座位号为LOC_AT3G46610(TAIR登录号)。其全长基因组序列约为2471bp,包括1个外显子。其cDNA(如SEQ ID NO:3所示)全长2471bp,编码665个氨基酸。
发明人通过实验证实,光合作用调控蛋白LPE1定位于叶绿体,其编码基因LPE1参与拟南芥光效调控,是一种重要的调控植物光合效率的功能基因。利用正向遗传学方法,获得三个LPE1的纯合缺失突变体株系,发现LPE1基因的功能缺失会导致植株发育迟缓,最大光合效率降低。
鉴于LPE1基因在植物中具有较高的保守性,可以预见在其他陆生植物中同样具有相同或相近的功能,例如杨树,葡萄,黄瓜,蓖麻,大豆,草莓,茄子,玉米,水稻,大麦,苔藓等,但不仅限于此。因此,通过对LPE1基因进行上调或蛋白LPE1过表达,有望获得高光效的植物,
下面结合试验,进一步说明本发明,其中未详细说明的方法为本领域公知的常规方法。
一.拟南芥LPE1突变体的纯合性鉴定
1.拟南芥总DNA的提取:
称取0.5~1.0g的三周龄拟南芥野生型(Col-0)和LPE1(lpe1-1,lpe1-2,lpe1-3)突变体嫩叶,液氮速冻后研磨,加入600μL 2×CTAB提取液,植物组织破碎仪上破碎20s,重复两次,将破碎后的植物材料,放入65℃金属浴中温浴30min,每隔十分钟翻转一次;室温冷却5min后,加入等体积的预冷的氯仿/异戊醇(24:1),振荡混匀后室温静置10min;8000g/min离心10min;离心后液面分上下两层,小心取出离心管,轻轻吸取上清400μL转至新的1.5mL EP管中,注意不要吸到沉淀;加入预冷的300μL异丙醇,轻轻上下摇动离心管,缓慢混合上清与异丙醇,放置-20℃,20min;随后12000g/min,离心10min;弃上清,加入1mL无水乙醇洗涤沉淀,轻微振荡,12 000g/min离心去上清,重复此步骤一次;DNA沉淀于室温晾干,在见到无色胶状物附在管壁时,加入30μL无菌蒸馏水溶解沉淀的DNA,存放于-20℃备用。其中所述2×CTAB提取液(500mL)的配方为:10g CTAB、6.05g Tris、3.722g EDTA-Na2、40.95g NaCl(1.4M)。
2.拟南芥LPE1突变体纯合性鉴定
利用三引物法鉴定LPE1突变体,设计如下三对引物(引物作用位置如图1A):
SALK_059367-L:5′-TGGACTACTTTGCACACGATG-3′(SEQ ID NO:4);
SALK_059367-R:5′-ACTACCGAAGCAAGCCTGTTC-3′(SEQ ID NO:5);
SALK_030882-L:5′-TGGACTACTTTGCACACGATG-3′(SEQ ID NO:6);
SALK_030882-R:5′-ATCCTACCCCAAACGATGATC-3′(SEQ ID NO:7);
SALK_110539-F:5′-ATCCTACCCCAAACGATGATC-3′(SEQ ID NO:8);
SALK_110539-R:5′-ATCTTGCTGAGTGCAGCTAGC-3′(SEQ ID NO:9);
中间引物LBa1:5′-TGGTTCACGTAGTGGGCCATCG-3′(SEQ ID NO:10)。
PCR反应体系为:DNA模板1μL、引物L 1μL、引物R 1μL、引物LBa1 1μL、2×PCR Mix 12.5μL、H2O 8.5μL。
扩增条件为:94℃变性5min;94℃30s,60℃30s,72℃60s,30个循环;72℃延伸5min,PCR产物在1%琼脂糖胶中分离并拍照。
结果如图1B所示,突变体的扩增片段大小明显小于野生型拟南芥植株,说明突变体有T-DNA插入,且突变体没有也野生型大小一致的条带,说明这些突变体为是纯合突变体。
二.拟南芥LPE1突变体LPE1基因表达分析
1.拟南芥总RNA的提取
取三周龄拟南芥野生型(Col-0)和LPE1(lpe1-1,lpe1-2,lpe1-3)突变体嫩叶,置于1.5mL EP管中液氮速冻后研磨成粉状,按照每0.1g材料加入1mL提取试剂的比例加入Trizol(Invitrogen公司产RNA专用提取试剂),充分振荡混匀后,室温放置5min;再按照每0.1g材料加入200μL氯仿的比例加入氯仿,快速振荡后静置分层,12 000g,4℃离心10min;将上层水相转至新的1.5mL EP管中,加入600μL异丙醇充分混匀,室温静置10min;12 000g,4℃离心10min,收集沉淀,加入1mL 75%的乙醇(DEPC处理)洗涤沉淀;12 000g,4℃离心5min,沉淀于超净工作台中吹干,用30~50μL无RNase水(DEPC处理)溶解RNA,检测RNA的完整性及纯度符合要求后,于-80℃超低温冰箱保存备用。
2.LPE1基因的RT-PCR表达分析:
(1)cDNA第一链的合成
RNA需要进行去除基因组的处理,体系为5×gDNA Eraser Buffer 2.0μL,gDNA 1μL,RNA 2μg,RNase Free dH2O补足到10μL,42℃2min进行DNA去除试验并得到反应液1。再采用20μL体系进行反转录反应,体系为反应液1 10μL,PrimeScript RT Enzyme Mix I 1.0μL,Primer Mix 1.0μL,5×PrimeScript Buffer 2 4.0μL,RNase Free dH2O 4.0μL。按以下程序进行反转录反应:37℃15min;85℃5s,生成的cDNA第一链保存于-20℃备用。
(2)cDNA为模板的qPCR
设计如下一对引物:
LPE1-F:5′-GTATTGAACCGAGCGTTGTGAC-3′(SEQ ID NO:11);
LPE1-R:5′-AGCCTCAATCAACATCTCGTAAGTT-3′(SEQ ID NO:12);
Real-time PCR反应体系为15μL:SYBR Premix Ex TaqTM II 7.5μL、引物LPE1-F 0.5μL、引物LPE1-R 0.5μL、cDNA模板1μL,dH2O 5.5μL。
扩增条件为:95℃预变性30s;95℃变性5s,60℃退火30s,72℃延伸20s,39个循环,添加熔解曲线;每个组织样品添加3个重复。
采用Pfaffl method,以野生型中基因表达量为对照,计算LPE1基因在其他各突变株系的相对表达量,同时应用SPASS 18.0软件计算重复样品之间Ct均值以及标准偏差,最后使用Excel制图功能绘制出基因在各突变株系中的相对表达柱状图。
结果如图1C所示,突变体的LPE1基因的相对表达量显著低于野生型拟南芥植株。
三.拟南芥LPE1突变体叶绿素荧光参数及含量分析
植株暗适应20min后,利用M系列叶绿素荧光仪IMAGING-PAM MAXI版(Heinz-Walz),测定响应的叶绿素荧光参数。
(1)最小荧光(F0)、最大荧光(Fm)以及最大光合作用效率(Fv/Fm)的测定:
打开叶绿素荧光仪IMAGING-PAM饱和脉冲(2800μmol photons m-2s-1)测定F0、Fm以及Fv/Fm。
结果如图1D所示,突变体的最大光合作用效率Fv/Fm显著低于野生型拟南芥植株。
(2)光响应曲线测定:
F0和Fm测定后,设置光强参数以及时间间隔,测定实际光合作用效率(ФPSII)和电子传递速率(ETR)和1-qP的光响应曲线。光强设置为0,81,145,186,281,335,461,701,以及926μmol photons m-2s-1。每隔3min打开饱和脉冲(2800μmol photons m-2s-1)记录一次数值。
结果如图2所示,突变体的实际光合作用效率和电子传递速率均低于野生型拟南芥植株,而1-qP的光响应曲线均高于野生型拟南芥植株。
(3)动力学曲线测定
F0和Fm测定后,打开光化光(531μmol photons m-2s-1),测定715s,每隔20s记录一次数值,共测定36个饱和脉冲。光化光以后,Fm’的恢复测定如“Non-Photochemical Quenching.A Response to Excess Light Energy.Muller,Plant Physiology,2001”所述,测定16个饱和脉冲。根据Fm′=Fm′36,Fmr=Fm′52,qE=Fm/fm′–fm/Fmr,qI=(fm-fmr)/fmr分别算出qE和qI,并记录相应的qN及Y(NO)(结果见表1)。
(4)叶绿素含量测定
利用分光光度法分别对野生型(Col-0)及突变体(lpe1-1,lpe1-2,lpe1-3)中叶绿素a(Chl a)和叶绿素b(Chl b)的含量进行检测(结果见表1)。具体步骤如下:
叶片用80%丙酮4℃抽提过夜,10000g,离心5min,弃沉淀。测定OD645及OD663值,Chl a和Chl b含量按照如下公式计算,单位mg/g FW:
Chl a=(12.7×A663–2.69×A645)×稀释倍数/1000
Chl b=(22.9×A645–4.48×A663)×稀释倍数/1000
表1野生型(Col-0)和lpe1突变体叶绿素含量和叶绿素荧光参数
从表1可以看出,突变体的Chl a、Chl b和qE均显著降低。叶绿素是植物进行光合作用的主要色素,叶绿素含量降低提示光系统可能发育异常。F0、Fm、qN、Y(NO)、qI均显著提高。qI代表光抑制依赖的非光化学淬灭,lpe1突变体中qI明显增加,提示植株受到了明显的光抑制。F0代表最小荧光值,Fm代表最大荧光值,Y(NO)代表除了热扩散以外的荧光淬灭部分,qN代表非光化学猝灭,这些参数升高,提示实际参与光合作用的效率降低。
综上,LPE1基因的缺失会降低拟南芥植株的光合作用效率和叶绿体功能。
四.拟南芥LPE1突变体莲座叶长度及鲜重测定
对野生型(Col-0)及突变体(lpe1-1,lpe1-2,lpe1-3)中莲座叶的长度及鲜重进行检测。取生长4周龄的拟南芥野生型(Col-0)及突变体(lpe1-1,lpe1-2,lpe1-3)植株,用游标卡尺测量莲座叶的长度并用电子天平测定除根以外的整株植株的鲜重。各实验至少重复三次。
结果如图1E所示,突变体的莲座叶的长度和除根以外的整株植株的鲜重均明显小于野生型拟南芥植株,说明LPE1基因的缺失会导致植株发育迟缓。
五.LPE1蛋白亚细胞定位分析
1.LPE1基因的克隆
设计如下两条引物:
LPE1-F:5′-GTATTGAACCGAGCGTTGTGAC-3′(SEQ ID NO:11);
LPE1-R:5′-CAGGTACCAGGTCTATTCTTTTTGTCTGGT-3(SEQ ID NO:12)′。
PCR反应体系为:cDNA模板1μL、引物LPE1-F 1μL、引物LPE1-R 1μL、2×PCR Mix 12.5μL、H2O 9.5μL。
扩增条件为:94℃变性5min;94℃30s,60℃30s,72℃120s,30个循环;72℃延伸5min,PCR产物在1%琼脂糖胶中分离并拍照。
2.LPE1-GFP融合蛋白表达载体的构建
以Columbia-0(Col-0)cDNA为模板,PCR得到的DNA片段先连接至pMD 19-T载体中,挑取单克隆进行鉴定,鉴定的阳性克隆送至英潍捷基(上海)贸易有限公司测序。将经测序正确的pMD19-LPE1载体用XbaI和Kpn I双酶切;原生质体瞬时表达载体pUC-GFP分别用XbaI和Kpn I双酶切,回收并纯化。将回收得到的目的片段用T4DNA连接酶连接入原生质体瞬时表达载体pUC-GFP的相应位点中,连接产物转化大肠杆菌DH5α感受态细胞,挑选阳性克隆提取质粒DNA,进行酶切鉴定,最后得到在35S启动子驱动下的LPE1-GFP融合蛋白表达载体。
3.利用拟南芥原生质体系统瞬时表达LPE1-GFP融合蛋白
(1)试剂配置
1)酶解液
总体系20mL:蔗糖0.5mol/L、0.2M的MES(pH 5.7)1mL、1M的CaCl2 0.4mL、2M的KCl 0.4mL、1%Cellose(R10)0.2g、1%Macerozyme(R10)0.2g、H2O 18.4mL。
5~10mL酶解液能酶解10~20片叶子,最终能分离出0.5~1×106个原生质体,每个样品需要1~2×104个原生质体。
2)TVL溶液
总体系15mL:山梨醇0.3mol/L、1M的CaCl2 0.75mL、H2O 14.25mL。
3)W5溶液
总体系300mL:葡萄糖0.3g、KCl 0.24g、NaCl 2.7g、CaCl2 5.52g、0.2M的MES 3mL、H2O补齐至300mL
4)Mmg溶液
总体系20mL:0.2M的MES 0.4mL、0.8M的Mannitol 10mL、1M的MgCl2 0.3mL、H2O 9.3mL。
5)PEG/Ca2+溶液
总体系20mL:PEG4000 8g、0.8M的Mannitol 5mL、1M的CaCl2 2mL、H2O 7mL。
(2)原生质体分离
1)用新的刀片在含有15mL过滤的无菌TVL溶液中切割2g培养14d的拟南芥幼苗(注意勤换刀片),切成0.5~1mm的细条,均匀分散在无菌的培养皿中;
2)切割完成后,将含TVL溶液的拟南芥细条转移到200mL烧杯中,再添加20mL过滤的无菌酶解液,使组织和酶解液充分混合,用封口膜和铝箔封闭;
3)在黑暗的室温条件下,35rpm摇晃培养植物组织16~18h;
4)用8层纱布收集释放的原生质体至50mL尖底离心管中,纱布事先用W5溶液湿润;
5)用W5溶液清洗纱布上所残留的原生质体,清洗的次数根据所需要的原生质体的含量来确定;
6)用W5溶液稀释收集的原生质体至100mL,随后100g离心7min(注意一定要用水平的离心机);
7)去上清,用W5溶液洗涤原生质体,随后60g离心5min,重复2~3次;
8)去上清,用W5溶液重悬原生质体,用血细胞计数板镜检计数;
9)将重悬的原生质体其置于冰上30min,随后60g离心5min;
10)去上清,再MMG溶液重悬原生质体,定量至2×105个/mL。
(3)PEG转化
1)在一个2mL的圆底EP管加入10μL DNA(5kb大小的质粒DNA 10~20ug),如果是双转,则每种质粒都需要加入10μL;
2)加入100μL原生质体(2×104个原生质体),混匀;
3)加入110μL PEG/Ca溶液,混匀;
4)从第一个开始计时,黑暗放置15min;
5)用440μL W5溶液稀释,并轻轻混匀;
6)800rmp离心2min,去上清;
7)加入500μL的W5溶液,分散原生质体,平铺于6/12/24孔板中培养;
8)过夜培养8-~12h。
4.激光共聚焦观察LPE1-GFP融合蛋白
荧光蛋白标记的目的蛋白在原生质体细胞中表达后,利用激光扫描共聚焦显微镜进行观察。GFP使用433nm激发,并使用500~530nm波长收集GFP信号;叶绿素自发荧光用488或514nm波长的激光激发,并用650-750nm收集。
结果如图3所示,LPE1-GFP融合蛋白荧光特异性和叶绿素自发荧光重合,证实LPE1在叶绿体中的特异性定位。
六.LPE1的生物信息学分析
利用ClustalX进行拟南芥LPE1蛋白在其他陆生植物中的同源性分析(www.clustal.org),包括杨树,葡萄,黄瓜,蓖麻,大豆,草莓,茄子,玉米,水稻,大麦,山羊草,苔藓等。
结果如图4所示,LPE1蛋白序列在其他陆生植物中有较高的保守性,可以预见,LPE1基因在其他陆生植物中极有可能存在相同的作用,通过调控LPE1基因,可以获得相应的高光效植物。
SEQUENCE LISTING
<110> 中山大学
<120> 一种光合作用调控蛋白LPE1及其应用
<130>
<160> 12
<170> PatentIn version 3.5
<210> 1
<211> 665
<212> PRT
<213> Arabidopsis thaliana
<400> 1
Met Gln Ala Leu Ser Ile Leu Pro Leu Lys Ser Gly Leu Leu Val Gly
1 5 10 15
Ser Arg Leu Glu Phe Glu Leu Asp Cys Ser Cys Phe Val Val Ser Pro
20 25 30
Lys Thr Thr Arg Lys Arg Leu Cys Phe Leu Glu Gln Ala Cys Phe Gly
35 40 45
Ser Ser Ser Ser Ile Ser Ser Phe Ile Phe Val Ser Ser Asn Arg Lys
50 55 60
Val Leu Phe Leu Cys Glu Pro Lys Arg Ser Leu Leu Gly Ser Ser Phe
65 70 75 80
Gly Val Gly Trp Ala Thr Glu Gln Arg Glu Leu Glu Leu Gly Glu Glu
85 90 95
Glu Val Ser Thr Glu Asp Leu Ser Ser Ala Asn Gly Gly Glu Lys Asn
100 105 110
Asn Leu Arg Val Asp Val Arg Glu Leu Ala Phe Ser Leu Arg Ala Ala
115 120 125
Lys Thr Ala Asp Asp Val Asp Ala Val Leu Lys Asp Lys Gly Glu Leu
130 135 140
Pro Leu Gln Val Phe Cys Ala Met Ile Lys Gly Phe Gly Lys Asp Lys
145 150 155 160
Arg Leu Lys Pro Ala Val Ala Val Val Asp Trp Leu Lys Arg Lys Lys
165 170 175
Ser Glu Ser Gly Gly Val Ile Gly Pro Asn Leu Phe Ile Tyr Asn Ser
180 185 190
Leu Leu Gly Ala Met Arg Gly Phe Gly Glu Ala Glu Lys Ile Leu Lys
195 200 205
Asp Met Glu Glu Glu Gly Ile Val Pro Asn Ile Val Thr Tyr Asn Thr
210 215 220
Leu Met Val Ile Tyr Met Glu Glu Gly Glu Phe Leu Lys Ala Leu Gly
225 230 235 240
Ile Leu Asp Leu Thr Lys Glu Lys Gly Phe Glu Pro Asn Pro Ile Thr
245 250 255
Tyr Ser Thr Ala Leu Leu Val Tyr Arg Arg Met Glu Asp Gly Met Gly
260 265 270
Ala Leu Glu Phe Phe Val Glu Leu Arg Glu Lys Tyr Ala Lys Arg Glu
275 280 285
Ile Gly Asn Asp Val Gly Tyr Asp Trp Glu Phe Glu Phe Val Lys Leu
290 295 300
Glu Asn Phe Ile Gly Arg Ile Cys Tyr Gln Val Met Arg Arg Trp Leu
305 310 315 320
Val Lys Asp Asp Asn Trp Thr Thr Arg Val Leu Lys Leu Leu Asn Ala
325 330 335
Met Asp Ser Ala Gly Val Arg Pro Ser Arg Glu Glu His Glu Arg Leu
340 345 350
Ile Trp Ala Cys Thr Arg Glu Glu His Tyr Ile Val Gly Lys Glu Leu
355 360 365
Tyr Lys Arg Ile Arg Glu Arg Phe Ser Glu Ile Ser Leu Ser Val Cys
370 375 380
Asn His Leu Ile Trp Leu Met Gly Lys Ala Lys Lys Trp Trp Ala Ala
385 390 395 400
Leu Glu Ile Tyr Glu Asp Leu Leu Asp Glu Gly Pro Glu Pro Asn Asn
405 410 415
Leu Ser Tyr Glu Leu Val Val Ser His Phe Asn Ile Leu Leu Ser Ala
420 425 430
Ala Ser Lys Arg Gly Ile Trp Arg Trp Gly Val Arg Leu Leu Asn Lys
435 440 445
Met Glu Asp Lys Gly Leu Lys Pro Gln Arg Arg His Trp Asn Ala Val
450 455 460
Leu Val Ala Cys Ser Lys Ala Ser Glu Thr Thr Ala Ala Ile Gln Ile
465 470 475 480
Phe Lys Ala Met Val Asp Asn Gly Glu Lys Pro Thr Val Ile Ser Tyr
485 490 495
Gly Ala Leu Leu Ser Ala Leu Glu Lys Gly Lys Leu Tyr Asp Glu Ala
500 505 510
Phe Arg Val Trp Asn His Met Ile Lys Val Gly Ile Glu Pro Asn Leu
515 520 525
Tyr Ala Tyr Thr Thr Met Ala Ser Val Leu Thr Gly Gln Gln Lys Phe
530 535 540
Asn Leu Leu Asp Thr Leu Leu Lys Glu Met Ala Ser Lys Gly Ile Glu
545 550 555 560
Pro Ser Val Val Thr Phe Asn Ala Val Ile Ser Gly Cys Ala Arg Asn
565 570 575
Gly Leu Ser Gly Val Ala Tyr Glu Trp Phe His Arg Met Lys Ser Glu
580 585 590
Asn Val Glu Pro Asn Glu Ile Thr Tyr Glu Met Leu Ile Glu Ala Leu
595 600 605
Ala Asn Asp Ala Lys Pro Arg Leu Ala Tyr Glu Leu His Val Lys Ala
610 615 620
Gln Asn Glu Gly Leu Lys Leu Ser Ser Lys Pro Tyr Asp Ala Val Val
625 630 635 640
Lys Ser Ala Glu Thr Tyr Gly Ala Thr Ile Asp Leu Asn Leu Leu Gly
645 650 655
Pro Arg Pro Asp Lys Lys Asn Arg Pro
660 665
<210> 2
<211> 2471
<212> DNA
<213> Arabidopsis thaliana
<400> 2
caaaaattct atcttcttct tcgcggctct gaatccgcca ttgaagttat tttcaagccc 60
tcctcaattt tcgtttttgt tcaatattaa tttgtaagag atctttacgc ttcagtttct 120
gaaagaactt tgcgcttctg attttgcaaa attggaagag agtgcttccg ttgcgctttt 180
gaagcgtgaa atcgtgtgtc ttttgacgct tccatgctac ataacgctag ttgtgagatt 240
gtgtttggaa aaagatgcaa gctttaagca ttttgccatt aaagtctggt cttttagttg 300
gatcaagatt ggaatttgag ttggattgtt cttgttttgt agttagccct aagactacta 360
gaaagagact ttgctttctt gaacaggctt gcttcggtag tagtagtagt atctctagct 420
tcatcttcgt ttcgagtaac aggaaagttc tgtttttgtg tgagccaaag agaagtttgt 480
tgggatcatc gtttggggta ggatgggcca ctgagcaacg agagctcgag ctcggggaag 540
aagaagtttc cacggaagat ttaagttcag caaatggagg agaaaagaac aatttaagag 600
ttgatgttag agaattagcg tttagtttac gagctgctaa gacagctgat gatgtggatg 660
ctgttctcaa ggacaaggga gagttgcctc tgcaagtctt ttgtgctatg ataaagggtt 720
ttggcaagga caagagattg aagcctgcgg tagctgtagt agattggctt aagaggaaga 780
agagtgaatc aggcggtgtc ataggtccaa atctttttat atataacagc cttttgggtg 840
ctatgagagg gtttggggaa gcagagaaga tactgaaaga tatggaggaa gaagggattg 900
ttccaaacat tgttacttac aatactttga tggttattta catggaggaa ggtgagtttc 960
tcaaggctct tgggattctt gatttaacca aggaaaaggg ttttgagcct aacccgatta 1020
catattcaac ggctttgctt gtttacagaa gaatggaaga cggtatggga gctttagagt 1080
tctttgttga attgagagag aaatacgcaa agagggaaat agggaatgat gttggttacg 1140
attgggaatt tgagtttgtt aagcttgaga actttattgg tagaatatgc taccaagtga 1200
tgaggcggtg gttggtgaaa gatgataatt ggactactag agtgttgaag cttttgaatg 1260
caatggacag tgcaggggtt agaccgagta gggaagagca cgagaggctt atatgggcat 1320
gtactcgtga agagcattat attgttggta aagagttgta taaaagaatc agagagaggt 1380
tttctgagat aagtctgtct gtttgcaacc atttgatttg gctgatgggt aaggctaaga 1440
aatggtgggc agcgttggag atctacgagg atcttcttga cgaaggacct gaacctaaca 1500
atttgtctta tgagttagtg gtctctcatt tcaatatctt gctgagtgca gctagcaaga 1560
gagggatatg gagatgggga gttaggttac ttaacaaaat ggaagataaa ggattgaaac 1620
cgcagaggag acattggaac gcggttcttg tagcatgttc aaaagcctct gagaccacag 1680
ctgcgatcca aatctttaaa gcaatggttg acaatggtga aaagccaacg gtcatctcat 1740
acggtgcact actcagtgct cttgagaaag ggaagctata tgatgaggcg tttcgggttt 1800
ggaaccatat gattaaggta gggattgagc caaatcttta cgcgtacaca acaatggctt 1860
cagttttgac aggacagcaa aagttcaatc ttttggatac ccttctcaag gaaatggctt 1920
caaagggtat tgaaccgagc gttgtgactt tcaacgcggt tataagtggt tgcgcgagga 1980
acgggttaag tggtgtggct tatgagtggt ttcatagaat gaaaagtgag aatgttgagc 2040
ctaacgagat aacttacgag atgttgattg aggctcttgc aaatgatgca aagccaaggc 2100
ttgcttatga gttacatgtg aaggctcaaa acgaagggct taaactctct tctaagccat 2160
atgatgcggt tgttaaatca gctgagactt atggagccac cattgatctt aatcttttgg 2220
gtcctcgacc agacaaaaag aatagacctt agtctgacga aaacacattg tacaaggtta 2280
ccgtggggta aggcgcatcc aaggaccctt aaaggcaaga aaattgccat tacagggaag 2340
aagaaagttt ccaagtaatc actttcttgt tctttaatac tctttttagt ttgtctagtc 2400
tctctgttta tattgtgaat ttttatcttc gtacaacatc tgtattatca tcagacacat 2460
tgatatgcaa a 2471
<210> 3
<211> 2471
<212> DNA
<213> Arabidopsis thaliana
<400> 3
caaaaattct atcttcttct tcgcggctct gaatccgcca ttgaagttat tttcaagccc 60
tcctcaattt tcgtttttgt tcaatattaa tttgtaagag atctttacgc ttcagtttct 120
gaaagaactt tgcgcttctg attttgcaaa attggaagag agtgcttccg ttgcgctttt 180
gaagcgtgaa atcgtgtgtc ttttgacgct tccatgctac ataacgctag ttgtgagatt 240
gtgtttggaa aaagatgcaa gctttaagca ttttgccatt aaagtctggt cttttagttg 300
gatcaagatt ggaatttgag ttggattgtt cttgttttgt agttagccct aagactacta 360
gaaagagact ttgctttctt gaacaggctt gcttcggtag tagtagtagt atctctagct 420
tcatcttcgt ttcgagtaac aggaaagttc tgtttttgtg tgagccaaag agaagtttgt 480
tgggatcatc gtttggggta ggatgggcca ctgagcaacg agagctcgag ctcggggaag 540
aagaagtttc cacggaagat ttaagttcag caaatggagg agaaaagaac aatttaagag 600
ttgatgttag agaattagcg tttagtttac gagctgctaa gacagctgat gatgtggatg 660
ctgttctcaa ggacaaggga gagttgcctc tgcaagtctt ttgtgctatg ataaagggtt 720
ttggcaagga caagagattg aagcctgcgg tagctgtagt agattggctt aagaggaaga 780
agagtgaatc aggcggtgtc ataggtccaa atctttttat atataacagc cttttgggtg 840
ctatgagagg gtttggggaa gcagagaaga tactgaaaga tatggaggaa gaagggattg 900
ttccaaacat tgttacttac aatactttga tggttattta catggaggaa ggtgagtttc 960
tcaaggctct tgggattctt gatttaacca aggaaaaggg ttttgagcct aacccgatta 1020
catattcaac ggctttgctt gtttacagaa gaatggaaga cggtatggga gctttagagt 1080
tctttgttga attgagagag aaatacgcaa agagggaaat agggaatgat gttggttacg 1140
attgggaatt tgagtttgtt aagcttgaga actttattgg tagaatatgc taccaagtga 1200
tgaggcggtg gttggtgaaa gatgataatt ggactactag agtgttgaag cttttgaatg 1260
caatggacag tgcaggggtt agaccgagta gggaagagca cgagaggctt atatgggcat 1320
gtactcgtga agagcattat attgttggta aagagttgta taaaagaatc agagagaggt 1380
tttctgagat aagtctgtct gtttgcaacc atttgatttg gctgatgggt aaggctaaga 1440
aatggtgggc agcgttggag atctacgagg atcttcttga cgaaggacct gaacctaaca 1500
atttgtctta tgagttagtg gtctctcatt tcaatatctt gctgagtgca gctagcaaga 1560
gagggatatg gagatgggga gttaggttac ttaacaaaat ggaagataaa ggattgaaac 1620
cgcagaggag acattggaac gcggttcttg tagcatgttc aaaagcctct gagaccacag 1680
ctgcgatcca aatctttaaa gcaatggttg acaatggtga aaagccaacg gtcatctcat 1740
acggtgcact actcagtgct cttgagaaag ggaagctata tgatgaggcg tttcgggttt 1800
ggaaccatat gattaaggta gggattgagc caaatcttta cgcgtacaca acaatggctt 1860
cagttttgac aggacagcaa aagttcaatc ttttggatac ccttctcaag gaaatggctt 1920
caaagggtat tgaaccgagc gttgtgactt tcaacgcggt tataagtggt tgcgcgagga 1980
acgggttaag tggtgtggct tatgagtggt ttcatagaat gaaaagtgag aatgttgagc 2040
ctaacgagat aacttacgag atgttgattg aggctcttgc aaatgatgca aagccaaggc 2100
ttgcttatga gttacatgtg aaggctcaaa acgaagggct taaactctct tctaagccat 2160
atgatgcggt tgttaaatca gctgagactt atggagccac cattgatctt aatcttttgg 2220
gtcctcgacc agacaaaaag aatagacctt agtctgacga aaacacattg tacaaggtta 2280
ccgtggggta aggcgcatcc aaggaccctt aaaggcaaga aaattgccat tacagggaag 2340
aagaaagttt ccaagtaatc actttcttgt tctttaatac tctttttagt ttgtctagtc 2400
tctctgttta tattgtgaat ttttatcttc gtacaacatc tgtattatca tcagacacat 2460
tgatatgcaa a 2471
<210> 4
<211> 21
<212> DNA
<213> 人工序列
<400> 4
tggactactt tgcacacgat g 21
<210> 5
<211> 21
<212> DNA
<213> 人工序列
<400> 5
actaccgaag caagcctgtt c 21
<210> 6
<211> 21
<212> DNA
<213> 人工序列
<400> 6
tggactactt tgcacacgat g 21
<210> 7
<211> 21
<212> DNA
<213> 人工序列
<400> 7
atcctacccc aaacgatgat c 21
<210> 8
<211> 21
<212> DNA
<213> 人工序列
<400> 8
atcctacccc aaacgatgat c 21
<210> 9
<211> 21
<212> DNA
<213> 人工序列
<400> 9
atcttgctga gtgcagctag c 21
<210> 10
<211> 22
<212> DNA
<213> 人工序列
<400> 10
tggttcacgt agtgggccat cg 22
<210> 11
<211> 22
<212> DNA
<213> 人工序列
<400> 11
gtattgaacc gagcgttgtg ac 22
<210> 12
<211> 25
<212> DNA
<213> 人工序列
<400> 12
agcctcaatc aacatctcgt aagtt 25
- 还没有人留言评论。精彩留言会获得点赞!